Método de detección asistida por RPA-CRISPR-Cas13a del virus de la gastroenteritis transmisible
 Haoyu Wang1†
Haoyu Wang1† Zhimeng Cheng2,3†
Zhimeng Cheng2,3† Ran Luo1†
Ran Luo1† Qiyue Yang1
Qiyue Yang1 Yongping Zeng1
Yongping Zeng1 Yijun Yang4
Yijun Yang4 Yuankun Chen4
Yuankun Chen4 Wenting Li4,5*
Wenting Li4,5* Xiao Liu1,6*
Xiao Liu1,6*- 1Universidad del Suroeste, Facultad de Medicina Veterinaria, Chongqing, China
- número arábigoDivisión de Cirugía del Tracto Biliar, Departamento de Cirugía General, Hospital de China Occidental, Universidad de Sichuan, Chengdu, Sichuan, China
- 3Centro de Investigación de Enfermedades Biliares, Hospital de China Occidental, Universidad de Sichuan, Chengdu, Sichuan, China
- 4Departamento de Enfermedades Infecciosas y Tropicales, Segundo Hospital Afiliado de la Universidad Médica de Hainan, Haikou, China
- 5Departamento de Enfermedades Infecciosas, el primer hospital afiliado de la Universidad Médica de Anhui, Hefei, China
- 6Laboratorio Estatal Clave de Biología del Genoma del Gusano de Seda, Chongqing, China
Antecedentes y objetivo: El virus de la gastroenteritis transmisible (TGEV, por sus siglas en inglés) es un virus gastrointestinal altamente contagioso que causa diarrea, vómitos, anorexia, deshidratación y pérdida de peso en lechones. En la práctica clínica, a menudo ocurre en infecciones mixtas con otros patógenos y, por lo tanto, es difícil de diagnosticar y prevenir. Perjudica principalmente a los lechones de aproximadamente 2 semanas de edad, causando enormes pérdidas en las granjas. La confirmación clínica del TGEV suele requerir un diagnóstico de laboratorio, pero los ensayos tradicionales de PCR e inmunofluorescencia tienen algunas limitaciones. Además, la mayoría de las granjas en China están mal equipadas para diagnosticar con precisión la enfermedad. Por lo tanto, se requiere un nuevo método de detección con alta sensibilidad y especificidad y menos dependencia de la instrumentación.
Métodos: Utilizamos la amplificación de la polimerasa recombinasa (RPA), combinada con las características de la nucleasa de la proteína Cas13a activada para establecer un método de detección visual asistido por CRISPR-Cas13a para TGEV mediante la adición de un ARN reportero con fracciones fluorescentes y de extinción al sistema.
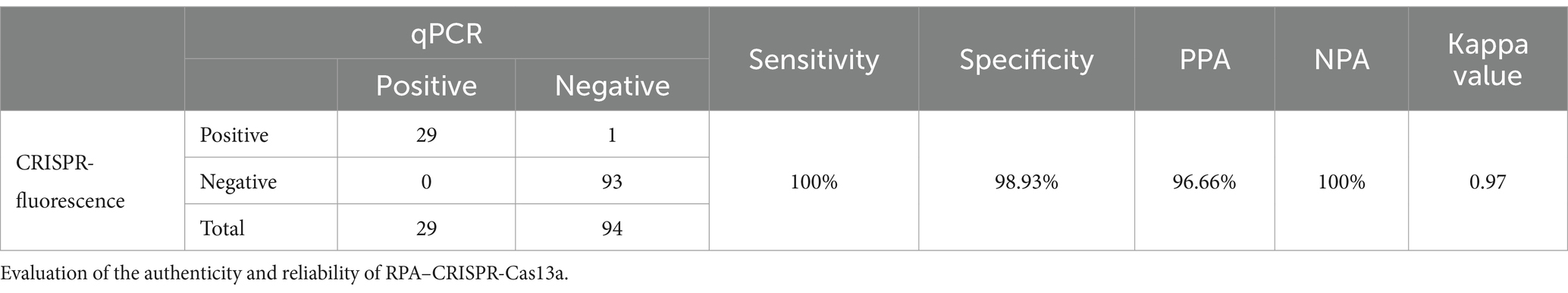
Resultado: Seleccionamos el cebador RPA óptimo y el mejor ARN CRISPR (crRNA). Se optimizó el sistema de reacción y se verificó su repetibilidad, especificidad y sensibilidad. El sistema de detección de TGEV no tuvo reacción cruzada con otros virus de diarrea comunes, y su límite de detección fue de 101 copias, lo que es similar a la sensibilidad de la qPCR. Establecimos con éxito un método de detección asistida por RPA-CRISPR-Cas13a y utilizamos este sistema de detección para analizar 123 muestras de sangre de cerdo. Se utilizó la qPCR como método de referencia. La sensibilidad, la especificidad, la tasa de coincidencia positiva y la tasa de coincidencia negativa del nuevo método fueron de 100, 98,93, 96,66 y 100%, respectivamente.
1 Introducción
El virus de la gastroenteritis transmisible (TGEV) es un miembro del género Coronavirus de la familia Coronaviridae, y es un coronavirus de tipo α (1). El TGEV causa una enfermedad intestinal altamente contagiosa en los cerdos y es una de las causas importantes de muerte de los lechones. Por lo tanto, ha traído enormes pérdidas a la industria porcina de China (2, 3). El TGEV ha sido prevalente en muchas regiones de China desde el primer brote en 1956. En la actualidad, la vacuna inactivada contra el TGEV no induce componentes inmunes en la leche de las cerdas gestantes, por lo que no confiere ninguna protección contra la infección a los lechones. Sin embargo, la vacuna atenuada a menudo presenta un riesgo de mayor toxicidad y requiere múltiples rondas de inmunización. Por lo tanto, el diagnóstico rápido y preciso de la infección por TGEV es extremadamente importante para la prevención y el tratamiento de la enfermedad (4, 5).
Debido a que el aislamiento del TGEV es complicado en la práctica clínica y a menudo se presenta como coinfecciones con otros virus, como el virus de la diarrea epidémica porcina (PEDV) (6), no se puede detectar ni diagnosticar rápidamente. Además, la transcripción inversa diagnóstica (RT)-PCR requiere la instrumentación pertinente y personal técnicamente capacitado en la práctica clínica. Los requisitos de equipo son altos, pero las condiciones en la mayoría de las granjas en China son limitadas, por lo que el diagnóstico de la infección por TGEV es difícil. La amplificación de la polimerasa recombinasa (RPA) es una nueva tecnología que puede sustituir a la detección por PCR (7). Utiliza recombinasa, una enzima de desplazamiento de hebra y proteína de unión monocatenaria, para lograr la unión específica de cebadores y plantillas a temperatura ambiente. La RPA puede amplificar la secuencia diana a 37 °C, tardando solo 5-20 minutos, lo que reduce en gran medida el tiempo necesario para el diagnóstico. También requiere poco instrumentación, es eficiente en el tiempo, tiene una alta especificidad y sensibilidad, y se puede utilizar en entornos en los que la PCR no puede (8).
La técnica de repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) fue desarrollada por primera vez en Japón en 1987 por los científicos Ishino et al., y nombrada por Mojica, Jansen (9-11). Las proteínas asociadas a CRISPR forman parte del sistema inmunitario de los procariotas, que capta las secuencias extrañas integradas y guía la función inmunitaria mediante la interferencia del ARN tras la identificación de secuencias foráneas (12). En los últimos años, CRISPR se ha utilizado como un nuevo tipo de tecnología de detección de ácidos nucleicos, con alta sensibilidad y especificidad, atrayendo cada vez más la atención de los investigadores y con amplias aplicaciones en muchas disciplinas. Entre estas proteínas, Cas13a actúa sobre el ARN en el sistema CRISPR. A diferencia de Cas9 y Cas12a, tiene una función de ARNasa bajo la guía del ARN CRISPR (ARNcr). Leptotrichia wadei Cas13a (LwaCas13a) no tiene ninguna restricción en su sitio de flanqueo protoespaciador (PFS) (13), lo que lo hace extremadamente flexible cuando se utiliza como herramienta de detección de ácidos nucleicos. Después de la activación, la proteína Cas13a escinde de forma inespecífica cualquier ARN monocatenario circundante (ssRNA) (14). Sobre la base de esta característica, se pueden diseñar sondas indicadoras fluorescentes de ARN específicas para permitir la detección de ácido nucleico asistida por CRISPR-Cas13a. Por lo tanto, la combinación de RPA y CRISPR-Cas13a proporciona una nueva opción más conveniente y menos dependiente del instrumento para el diagnóstico de TGEV.
2 Materiales y métodos
2.1 Construcción de plásmido positivo
Seleccionamos el fragmento de secuencia de la cepa TGEV-CHN-SC (GenBank ID: ON016092.1) del suroeste de China del Centro Nacional de Información Biotecnológica (NCBI), y lo insertamos en el plásmido pUC57, utilizando la secuencia de la región N (Sitio: 26888 a 28,036) (1,149 pb) de TGEV como plantilla sobre la cual construir el plásmido positivo pUC57–TGEV. El plásmido positivo para pUC57-TGEV de 1ul (1 ng) se transformó en Escherichia coli competente para DH5α (Shanghai Weidi Biotechnology Co., Ltd.), luego se seleccionó y cultivó Escherichia coli competente en agar Luria-Bertani (LB) que contenía ampicilina. Las colonias se seleccionaron e inocularon en LB líquido, y se cultivaron durante la noche a 37°C y 180 rpm. Centrifugar el caldo a 8000 rpm durante 10 min. Utilizamos el mini kit de plásmidos (Tiangen Biotechnology Co., Ltd.) para extraer el plásmido de las células y lo almacenamos a -80 °C.
2.2 Síntesis de cebadores, crRNA y reporteros
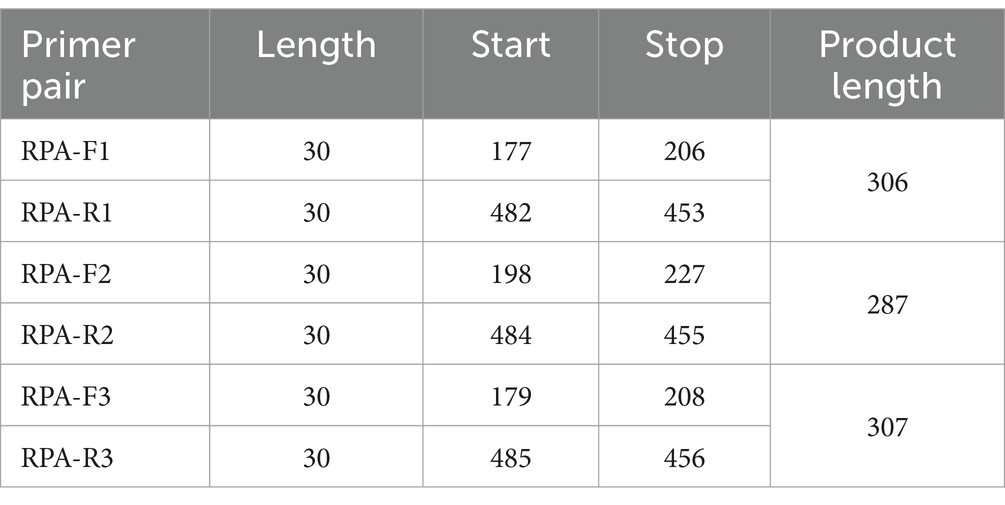
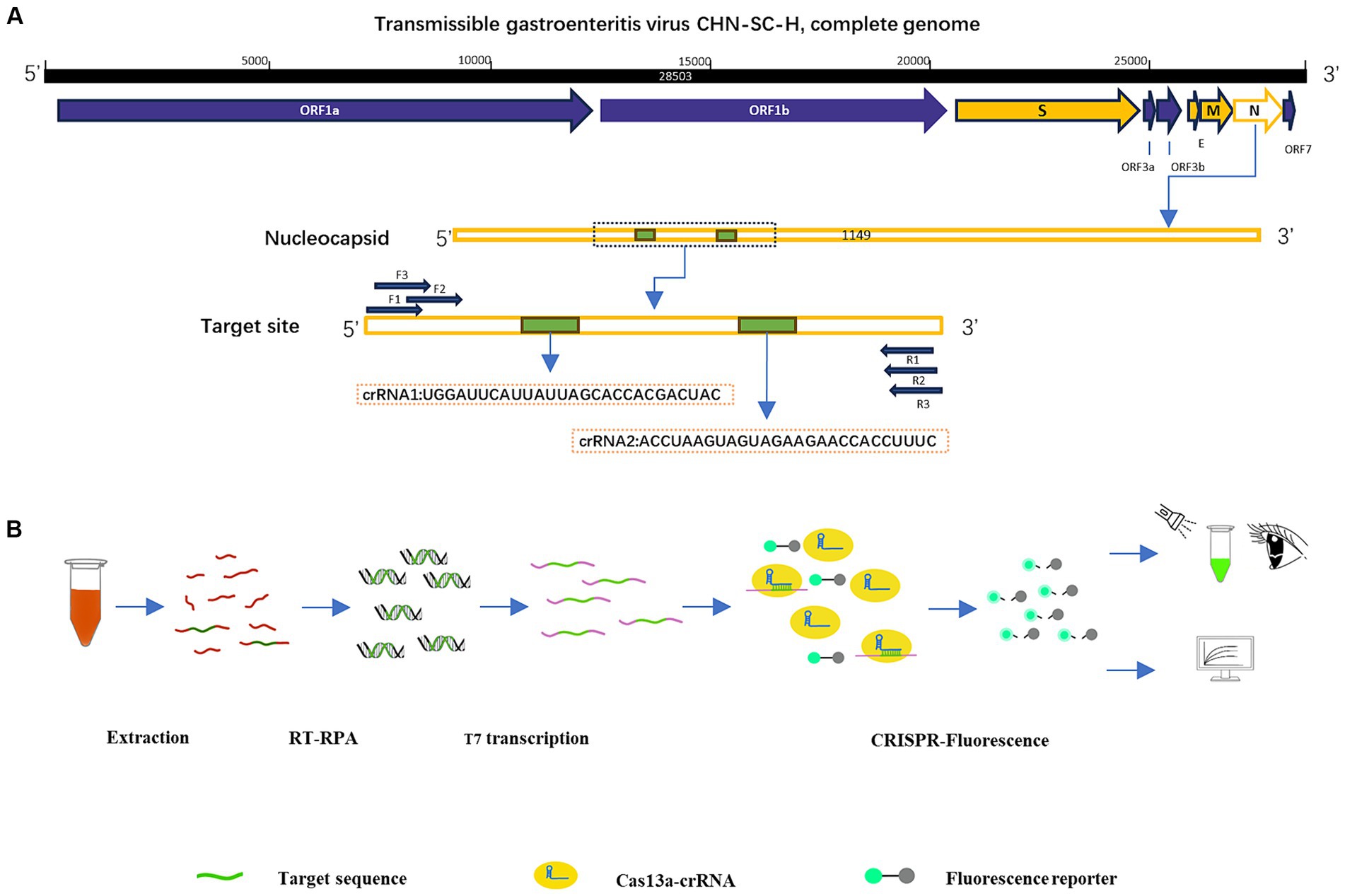
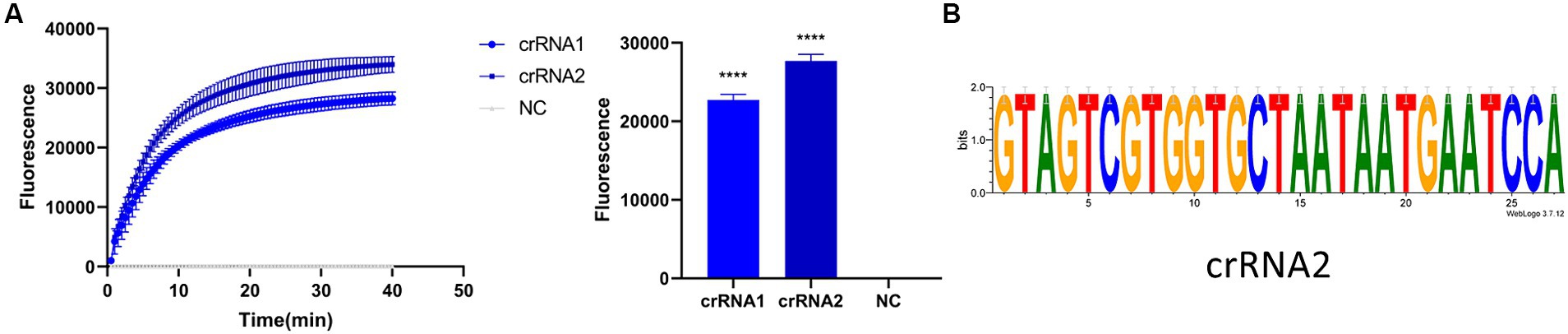
De acuerdo con la secuencia de plásmidos positivos insertada, utilizamos la herramienta Primer-BLAST del NCBI para diseñar tres conjuntos de cebadores de RPA, cada uno de los cuales tenía una longitud de 30 nt, y utilizamos Weblogo1 para analizar la conservación de los cebadores de RPA. La ubicación de los cebadores en la secuencia N se muestra en la Tabla 1. El promotor T7 (20 nt) se añadió al cebador aguas arriba de RPA para facilitar la transcripción posterior del producto. Los principios de diseño del ARNcr son proporcionados por Changzhou Amplification Future Biotechnology Co., Ltd. Weblogo se utilizó para analizar la conservación del ARNcr. Consiste en una secuencia complementaria al gen diana (28 nt) y una secuencia de repetición relacionada con CRISPR (36 nt), con una longitud total de 64 nt. El reportero fue un ARN corto (Figura 1A) con un grupo fluorescente y un grupo de extinción agregados en cada extremo, 5′ modificado como FAM, 3′ modificado como BHQ1, y la secuencia fue sintetizada por Shenzhen BGI Gene Co., Ltd. La secuencia diseñada para el experimento se muestra en la Tabla 2.
2.3 Amplificación de la secuencia diana
El plásmido positivo se utilizó como plantilla a partir de la cual amplificar la secuencia de ácido nucleico objetivo con un kit de amplificación rápida de ADN (Changzhou Amplification Future Biotechnology Co., Ltd.). Se añadió tampón A (29,4 μL) a cada tubo de reacción que contenía polvo seco, y luego se añadieron 2 μL de cebador (10 μM), 2 μL de plantilla de ácido nucleico, 2,5 μL de tampón B y 12,1 μL de agua, se mezclaron y se incubaron a 37 °C durante 30 min. Después de la reacción, se agregaron fenol tris-saturado (25 μL), cloroformo (24 μL) y alcohol isoamílico (1 μL), se centrifugaron a 12000 rpm durante 5 min a temperatura ambiente y luego se extrajo el sobrenadante para obtener el producto de amplificación purificado. El producto RPA se almacenó temporalmente a -20 °C.
Para verificar la amplificación de la secuencia diana de RPA, se preparó agarosa al 1% para electroforesis en gel de agarosa. El primer carril contenía 5 μL de marcador de ADN de 100-600 pb (Tiangen Biotechnology Co., Ltd.), y los carriles siguientes contenían 6 μL de producto de amplificación. La electroforesis se realizó a 100 V. Cuando las muestras alcanzaron 3/4 de la longitud del gel, el gel se retiró y se analizó con un generador de imágenes de quimioluminiscencia.
2.4 Detección de fluorescencia de ácido nucleico CRISPR-Cas13a
La detección de fluorescencia incluyó tres procesos. En primer lugar, se transcribió el producto de la amplificación del cebador en el sistema de reacción T7. El kit T7 fue comprado a New England Biolabs Inc. El sistema de reacción T7 contenía 8,5 μL de ddH2O, 1,5 μL de tampón de reacción de 10 ×, 6 μL de NTP (7,5 mM), 2 μL de ADN molde y 2 μL de ARN polimerasa T7 (10 mM), y se incubó a 37 °C durante 4 h o toda la noche. El producto se añadió a 1 μL de inhibidor de la RNasa (Shanghai Beyotime Biotechnology Co., Ltd.). La concentración de ácido nucleico se determinó utilizando un espectrofotómetro (Implen P330 NanoPhotometer), y posteriormente el ARN se almacenó a -80 °C. La proteína Cas13a se mezcló con ARNcr, después de que el ARNcr se diluyera con agua tratada con pirocarbonato de dietilo (DEPC) hasta una concentración final de 8,0 ng/μl. El sistema de reacción incluyó 1 μL de tampón de reacción de 10 ×, 2 μL de crRNA, 2 μL de nucleasa Cas13a (100 ng) y 5 μL de agua tratada con DEPC, y se incubó a 37 °C durante 10 min.
Finalmente, se construyó el método de detección CRISPR-Cas13a. El sistema de reacción incluyó 4 μL de tampón de reacción, 2 μL de ssRNA, 29 μL de agua tratada con DEPC, 5 μL de reportero (2 pmol/μl) y 10 μL de solución mixta Cas13a-crRNA, y se incubó a 37 °C durante 30 min. Los resultados se analizaron con el Sistema de Detección Cuantitativa de PCR de Fluorescencia (FQD-96A, Hangzhou Bori Technology Co., Ltd.).
2.5 Optimización de la condición
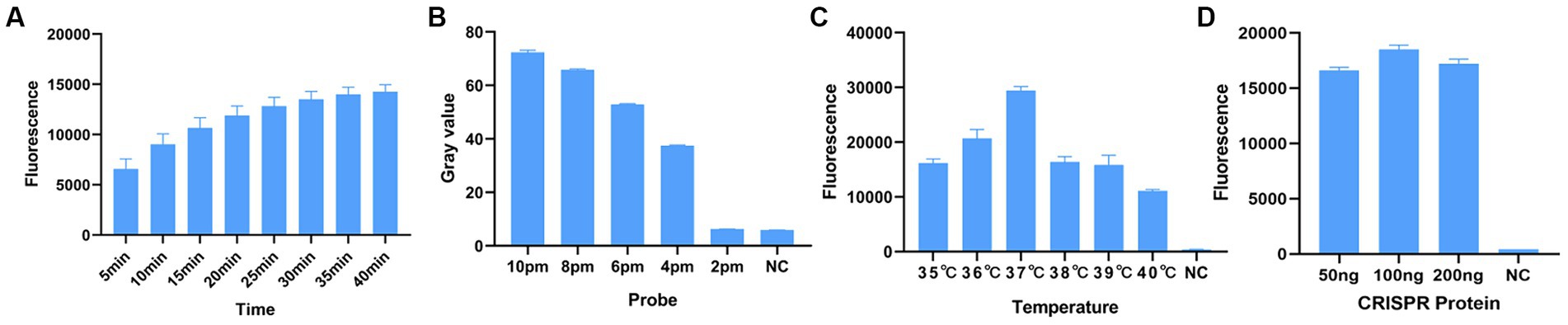
Identificamos las condiciones óptimas de reacción probando secuencialmente diferentes niveles de variables individuales y determinando la intensidad de fluorescencia final del reportero Cas13a. Las variables fueron: temperatura (35, 36, 37, 38, 39 y 40°C), tiempo de reacción (5, 10, 15, 20, 25, 30, 35 y 40 min), cantidad de ARN reportero (10, 8, 6, 4 y 2 pmol) y cantidad de proteína Cas13a (50, 100 y 200 ng).
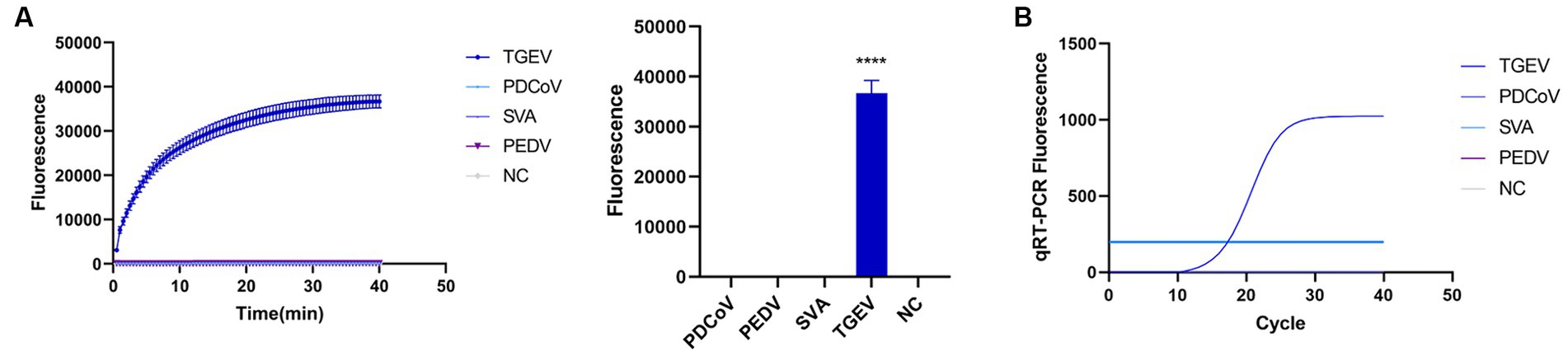
2.6 Evaluación de la sensibilidad y la especificidad
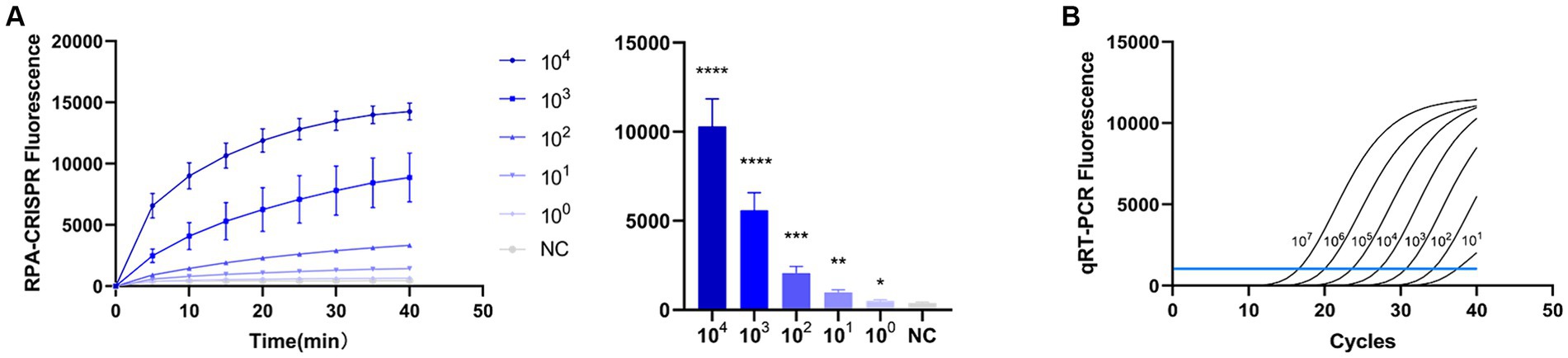
En este estudio, utilizamos virus clínicos comunes de la diarrea porcina para probar la especificidad del método de detección, incluidos el deltacoronavirus porcino (PDCoV), el Senecavirus A (SVA), PEDV y TGEV (las muestras procedían del laboratorio Xiao Liu, Southwest University, Facultad de Medicina Veterinaria, Chongqing, China), amplificándolas con RPA y detectándolas con el método de detección CRISPR-Cas13a. Para facilitar la recogida y el análisis de los datos, se utilizó un instrumento de qPCR para la detección de muestras. Para evaluar la sensibilidad del sistema de detección, diluimos las muestras estándar positivas a un gradiente (104, 103, 102, 101 y 100 copias/ul) y los resultados de los datos se analizaron mediante el instrumento qPCR. Al mismo tiempo, se utilizó un método de qPCR para detectar el mismo gradiente de concentración de estas muestras estándar positivas, y diferentes virus clínicos de la diarrea porcina, para comparar la especificidad y sensibilidad de los dos métodos. Los resultados de la qPCR se analizaron con el Sistema de Detección de PCR Cuantitativa de Fluorescencia y se dibujó la curva estándar.
2.7 Análisis de muestras clínicas
Para confirmar la viabilidad del método de detección optimizado establecido, se adquirieron muestras clínicas de sangre de cerdos (principalmente lechones de 30 días) en varias áreas de Sichuan, China. El ARN se extrajo de las muestras con el reactivo Trizol y se purificó con cloroformo e isopropanol. Después de la transcripción inversa del ARN de la muestra con un kit de síntesis de primera cadena de ADNc (Shanghai Biyuntian Biotechnology Co., Ltd.), el ADNc se combinó con RPA-CRISPR-Cas13a. El crRNA unido a la proteína Cas13a se utilizó para identificar la secuencia de ARN en la región conservada del virus, y se activó la actividad de escisión de ácido nucleico inespecífico de Cas13a. El reportero se cortó para emitir fluorescencia, y la fluorescencia se detectó mediante observación visual bajo una fuente de luz ultravioleta. Para el análisis de los valores de fluorescencia se utilizó el Sistema de Detección Cuantitativa por PCR de Fluorescencia (Figura 1B).
3 Resultados
3.1 Verificación positiva de plásmidos y cribado de cebadores RPA
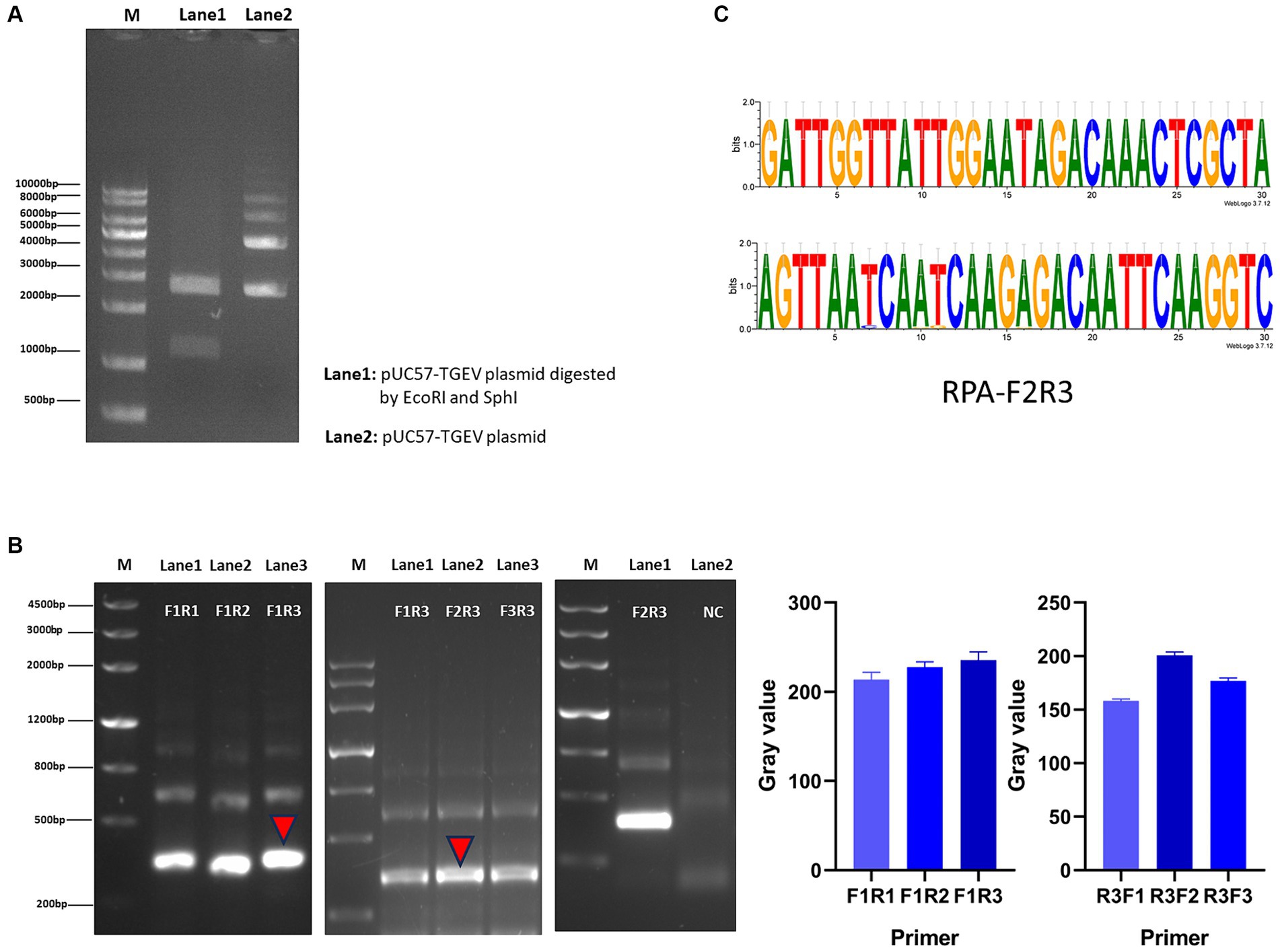
Cribamos los transformadores mediante el cultivo de las células en medio LB que contenía ampicilina. Después de extraer el plásmido, se digierió dos veces y los productos se resolvieron con electroforesis en gel de agarosa. Se detectaron dos bandas de aproximadamente 1.100 pb y 2.700 pb, lo que confirma que el plásmido positivo se construyó con éxito (Figura 2A). Utilizando el plásmido positivo como plantilla de ADN, se probaron tres pares de cebadores de RPA para la amplificación de RPA. Después de la electroforesis en gel de agarosa, las imágenes se obtuvieron con un generador de imágenes químicas y se analizaron con el software ImageJ. Finalmente, se identificó que el par de cebadores F2R3 proporciona la mayor eficiencia de amplificación (Figura 2B). Después de realizar un análisis con WebLogo, se confirmó que los cebadores F2R3 exhiben un alto conservadurismo (Figura 2C).
3.2 Optimización de las condiciones de detección de fluorescencia RPA-CRISPR-Cas13a
Antes de optimizar las condiciones, verificamos el sistema de reacción RPA-CRISPR-Cas13a. Los resultados mostraron que el grupo de control positivo emitió fluorescencia bajo luz azul-violeta, y el control negativo no emitió fluorescencia (Figura 3A). A continuación, optimizamos el ARNcr, el tiempo de reacción, la temperatura, la cantidad de reportero y la cantidad de proteína Cas13a. La amplificación de RPA se realizó con los cebadores F2R3. Después de la transcripción T7 combinada con CRISPR-Cas13a, los resultados se analizaron con el instrumento qPCR para identificar el crRNA óptimo, crRNA2 (Figura 4A). Se utilizó el análisis WebLogo para comparar múltiples cepas, y los resultados mostraron que la secuencia de ARNcr estaba altamente conservada (Figura 4B). La intensidad de fluorescencia del sistema de reacción fue alta y el cambio en la fluorescencia fue pequeño cuando la reacción se realizó durante aproximadamente 30 min, por lo que se seleccionó 30 min como el mejor tiempo de reacción (Figura 5A). Cuando se utilizó diferente cantidad de reportero, los resultados de 10, 8 y 6 pmol mostraron una alta intensidad de fluorescencia. Con el fin de ahorrar consumibles, se consideró que 6 pmol era la cantidad óptima de reportero (Figura 5B; Figura complementaria S1). La temperatura y la cantidad optimizadas de Cas13a fueron de 37 °C (Figura 5C) y 100 ng (Figura 5D), respectivamente.

3.3 Capacidad de detección de RPA-CRISPR-Cas13a
Para evaluar la capacidad de detección del método de detección asociado a CRISPR-Cas13a, probamos su especificidad para TGEV, SVA, PDCoV y PEDV. El resultado de la detección mostró que solo el grupo TGEV dio positivo (Figura 6A), lo que es consistente con el método qPCR (Figura 6B). Esto indica que el método de detección asociado a CRISPR-Cas13a exhibe una buena especificidad. Las pruebas repetidas confirmaron que la especificidad del ensayo para TGEV era del 100%. A continuación, verificamos la sensibilidad del sistema de detección y lo comparamos con el método qPCR. Se utilizaron diluciones en gradiente de las muestras positivas como plantillas de detección. Dado que los resultados de la prueba se analizaron en función de la intensidad final de la señal de fluorescencia, una intensidad de fluorescencia superior a 1447,56 se consideró indicativa de un resultado positivo. Los resultados mostraron que el método de detección establecido asociado a CRISPR-Cas13a generó una señal fluorescente a un mínimo de 7,7 × 101 copias del virus (Figura 7A). Además, se empleó un método de qPCR para detectar el mismo gradiente de concentración de estas muestras, clasificándose como positivas todas las muestras que presentaban valores de Ct ≤35. La qPCR mostró una sensibilidad de 7,7 × 102 copias (Figura 7B), lo que confirma que nuestro método de detección es altamente sensible.
Para probar la repetibilidad del ensayo, dividimos la misma muestra en tres concentraciones (105, 104 y 103 copias/ul) y repetimos la prueba en 3 días. Los coeficientes de variación fueron de 1,06, 4,13 y 16,65% (Tabla Suplementaria S1), respectivamente. El grupo de control negativo no emitió fluorescencia. Estos resultados indican que la repetibilidad del ensayo fue buena.
3.4 Detección de muestras clínicas en zonas de Sichuan, China
Utilizando el sistema de detección establecido, analizamos muestras de campo de diferentes granjas, incluidas Mingxing (MX), KA y Yuanda (YD), en diferentes regiones de Chengdu, Leshan y Guangyuan (provincia de Sichuan, China). Seleccionamos al azar 123 muestras de toda la colección y utilizamos el método de detección asistida por CRISPR-Cas13a para analizar estas muestras. Los resultados del sistema de detección asistida por CRISPR-Cas13a se compararon con los resultados de la qPCR. La sensibilidad, la especificidad, la tasa de coincidencia positiva, la tasa de coincidencia negativa y el valor kappa del método de detección asistida por CRISPR-Cas13a fueron 100, 98,93, 96,66, 100% y 0,97 (Tabla 3), respectivamente. Estos resultados fueron muy cercanos a los resultados de la qPCR, lo que confirma que el sistema de detección asistida por CRISPR-Cas13a es una tecnología de detección muy fiable.
4 Discusión
Desde que se descubrió el TGEV en los Estados Unidos en 1946 (15), se ha extendido por todo el mundo, provocando enormes pérdidas económicas a la industria mundial de la cría de cerdos. La prevalencia del TGEV en China no es particularmente grave (16) y la tasa de positividad en cerdos es baja. Sin embargo, cabe destacar que China es un gran país de cría de cerdos. Como virus de ARN, el TGEV es altamente mutable. Por ejemplo, la aparición de su mutante respiratorio, el coronavirus respiratorio porcino (PRCV), demuestra el riesgo que potencialmente representa. El TGEV tiene una tasa de mortalidad del 100% en lechones. En consecuencia, una mutación del virus o un brote repentino e impredecible de una epidemia de TGEV aumenta la presión sobre la industria porcina de China para que introduzca medidas de prevención y control. Cuando los lechones se infectan con TGEV, es probable que mueran, y aún no existe un tratamiento farmacológico. La mayoría de los cerdos tienen un efecto inmunológico deficiente después de la vacunación (4, 5), por lo que el diagnóstico oportuno de la infección por TGEV es extremadamente importante.
En la actualidad, el diagnóstico de laboratorio de TGEV se realiza principalmente utilizando la tecnología tradicional de amplificación por PCR, que depende en gran medida de la instrumentación, y este requisito no puede cumplirse en muchas regiones de los países en desarrollo. En 2006, Piepenburg et al. desarrollaron una tecnología RPA para reemplazar la PCR, y esta reacción es sensible y rápida. Es importante destacar que la reacción se realiza a una temperatura baja constante y no requiere los engorrosos pasos cíclicos de la tecnología de PCR (17). En comparación con la tecnología de PCR, la aparición de la tecnología RPA ofrece una nueva oportunidad para escenarios de detección en tiempo real e in situ (18). La tecnología RPA reduce la dependencia de los instrumentos y disminuye la necesidad de un control estricto de la temperatura. Esta tecnología ayuda a agilizar los procesos y a reducir la dependencia de condiciones ambientales precisas (17). Por lo tanto, la tecnología RPA está recibiendo cada vez más atención, con continuas innovaciones y mejoras, incluida la RPA combinada con tiras de flujo lateral, RPA multiplex, método de detección molecular isotérmica dobleplex relacionado con RPA, etc. (19-21). La RPA también se utiliza en la detección de bacterias, hongos, virus, parásitos, alimentos modificados genéticamente y genes de bacterias resistentes a los medicamentos (22-24). Su función se ha afirmado en diversos campos.
CRISPR-Cas es un sistema inmunitario adaptativo de procariotas. Las proteínas Cas reconocen y cortan secuencias de ácidos nucleicos extraños y las almacenan en regiones espaciadoras CRISPR (12). Con el estudio en profundidad del sistema CRISPR, el descubrimiento de Cas9 condujo al descubrimiento de su utilidad potencial en la edición del ADN (25). La caracterización de la proteína Cas13 identificó sus características de empalme de ARN, que ahora se utilizan en la edición de genes relacionados con el ARN. Además, utilizando la actividad nucleasa de la proteína Cas13, los investigadores han añadido de forma innovadora ARN reporteros con un grupo fluorescente FAM al sistema, y han construido un método de diagnóstico para ácidos nucleicos in vitro (26-28). En 2017, el laboratorio Zhang Feng construyó un método de detección de ácidos nucleicos basado en CRISPR-Cas13a (27). Recientemente, esta tecnología se ha utilizado en la detección de virus.
Dadas las deficiencias de la tecnología actual de diagnóstico clínico para TGEV, establecimos un nuevo método de detección de TGEV basado en RPA y CRISPR-Cas13a. Sus requisitos de equipamiento son bajos. En comparación con qPCR, solo requiere un dispositivo de calentamiento a temperatura constante y un destello azul-violeta para completar la detección de la muestra, y puede completar la detección de TGEV a baja temperatura y la interpretación de los resultados es relativamente simple. Como excelente método de detección comprobado, la qPCR se utiliza ampliamente (29). Por lo tanto, comparamos el método de detección asistida por CRISPR-Cas13a con el método de detección por qPCR para evaluar la sensibilidad y especificidad del método de detección asistida por CRISPR-Cas13a. En el estudio, solo TGEV mostró resultados positivos tanto en qPCR como en el método de detección asistida por CRISPR-Cas13a. En la prueba de sensibilidad, los límites de detección de los dos métodos fueron de 101 copias y 102 copias, respectivamente. En este estudio, la qPCR solo se utilizó como método de detección de referencia, el sistema de reacción y los cebadores no se optimizaron, los resultados de la qPCR pueden verse afectados por los cebadores, pero aún así puede reflejar su especificidad y sensibilidad. A continuación, verificamos la repetibilidad del método de detección RPA-CRISPR-Cas13a establecido. El coeficiente de variación de las muestras a10 3 copias fue mayor que 105 y 104 copias. Es posible que las muestras de copias bajas sean más susceptibles a los efectos del almacenamiento a largo plazo y de los ciclos repetidos de congelación y descongelación, lo que podría dar lugar a una mayor variabilidad en los resultados. Optimizamos varias de las condiciones de reacción, incluido el tiempo de reacción, la cantidad de proteína Cas13a utilizada y la cantidad de reportero utilizado, para que sea más factible y económico dentro de un cierto rango y para evitar el desperdicio de reactivos. El método de detección asistida por RPA-CRISPR-Cas13a proporciona una opción más conveniente y altamente sensible para la detección de TGEV en países en desarrollo, en pequeñas empresas o en granjas privadas. Aunque el sistema de detección todavía tiene algunos inconvenientes, como la necesidad de evitar la contaminación de las muestras, la incapacidad de eliminar por completo la dependencia de los instrumentos, los altos costos de RPA y los múltiples pasos operativos, creemos que los defectos de este nuevo método de detección no oscurecen sus virtudes. Durante el experimento, observamos que la transcripción de CRISPR-Cas13a y T7 se puede realizar a 37 °C, por lo que estos pasos se pueden integrar en un solo paso. Además, podemos intentar integrar la mayoría de los reactivos en un sistema de reacción. Cuando se usa en el sitio, puede reducir los pasos de operación y los requisitos de capacitación para el personal, y ahorrar tiempo. En resumen, todo el método de detección muestra una buena especificidad, sensibilidad y repetibilidad y facilitará la detección del TGEV y ayudará en la prevención y el control de la enfermedad asociada al TGEV.
Declaración de disponibilidad de datos
En este estudio se analizaron conjuntos de datos disponibles públicamente. Estos datos se pueden encontrar en: https://www.ncbi.nlm.nih.gov/nuccore/2428737012.
Declaración ética
El estudio en animales fue aprobado por el Centro de Animales de Laboratorio de la Universidad del Suroeste. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
HW: Redacción – borrador original, curación de datos, software, metodología. ZC: Escritura – borrador original. LR: Escritura – borrador original. QY: Escritura – borrador original. YZ: Escritura – borrador original. YY: Escritura – borrador original. YC: Escritura – borrador original. WL: Obtención de fondos, redacción, revisión y edición. XL: Obtención de fondos, Metodología, Redacción, revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este estudio contó con el apoyo del Proyecto de Investigación Básica y Tecnología de Frontera de Chongqing (cstc2020jcyj-msxm1458) (XL), la Fundación Nacional de Ciencias de China (No. 82260125) (WL), la Fundación Provincial de Ciencias Naturales de Hainan de China (No. 822MS181 y 823RC591) (WL) y el tema abierto del Laboratorio Estatal Clave de Biología del Genoma del Gusano de Seda (SKLSGB-ORP202104) (XL).
Reconocimientos
Agradecemos a Liwen Bianji (Edanz) (www.liwenbianji.cn) por editar el texto en inglés de un borrador de este manuscrito.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1428591/full#supplementary-material
Notas
1. ^https://weblogo.threeplusone.com
Referencias
1. Wagner, JE, Beamer, PD y Ristic, M. Microscopía electrónica de células epiteliales intestinales de lechones infectados con un virus de gastroenteritis transmisible. Can J Comp Med. (1973) 37:177–88.
2. Wesley, RD, Woods, RD, y Cheung, AK. Bases genéticas de la patogénesis del virus transmisible de la gastroenteritis. J Virol. (1990) 64:4761–6. doi: 10.1128/jvi.64.10.4761-4766.1990
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Sirinarumitr, T, Kluge, JP y Paul, PS. Apoptosis inducida por el virus de la gastroenteritis transmisible en cultivos celulares de testículos porcinos. Arch Virol. (1998) 143:2471–85. doi: 10.1007/s007050050477
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Register, KB y Wesley, RD. Caracterización molecular de cepas vacunales atenuadas del virus transmisible de la gastroenteritis. J Vet Diagn Invest. (1994) 6:16–22. doi: 10.1177/104063879400600104
5. Park, S, Sestak, K, Hodgins, DC, Shoup, DI, Ward, LA, Jackwood, DJ, et al. Respuesta inmunitaria de cerdas vacunadas con vacunas atenuadas contra el virus de la gastroenteritis transmisible atenuada (Tgev) y la proteína de la espícula Tgev recombinante y protección de sus lechones frente a la exposición virulenta al desafío con Tgev. Am J Vet Res. (1998) 59:1002–8. doi: 10.2460/ajvr.1998.59.08.1002
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Zhang, Q, Hu, R, Tang, X, Wu, C, He, Q, Zhao, Z, et al. Ocurrencia e investigación de infecciones virales entéricas en cerdos con diarrea en China. Arch Virol. (2013) 158:1631–6. doi: 10.1007/s00705-013-1659-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Jirawannaporn, S, Limothai, U, Tachaboon, S, Dinhuzen, J, Kiatamornrak, P, Chaisuriyong, W, et al. Detección rápida y sensible de Leptospira en el punto de atención mediante Rpa-Crispr/Cas12a dirigida a Lipl32. PLoS negl trop dis. (2022) 16:E0010112. doi: 10.1371/journal.pntd.0010112
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. James, A, y Macdonald, J. Amplificación de la polimerasa recombinasa: emergencia como una tecnología molecular crítica para diagnósticos rápidos y de bajos recursos. Experto Rev Mol Diagn. (2015) 15:1475–89. doi: 10.1586/14737159.2015.1090877
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Jansen, R, Embden, JD, Gaastra, W, y Schouls, LM. Identificación de genes que se asocian con repeticiones de ADN en procariotas. Mol Microbiol. (2002) 43:1565–75. doi: 10.1046/j.1365-2958.2002.02839.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Mojica, FJ, Díez-Villaseñor, C, Soria, E, y Juez, G. Significado biológico de una familia de repeticiones regularmente espaciadas en los genomas de arqueas, bacterias y mitocondrias. Mol Microbiol. (2000) 36:244–6. doi: 10.1046/j.1365-2958.2000.01838.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Ishino, Y, Shinagawa, H, Makino, K, Amemura, M y Nakata, A. Secuencia de nucleótidos del gen Iap, responsable de la conversión de isoenzimas de fosfatasa alcalina en Escherichia Coli e identificación del producto génico. J Bacteriol. (1987) 169:5429–33. doi: 10.1128/jb.169.12.5429-5433.1987
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Koonin, EV, y Makarova, KS. CRISPR-Cas: un sistema de inmunidad adaptativo en procariotas. F1000 Biol Rep. (2009) 1:95. DOI: 10.3410/B1-95
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Abudayyeh, OO, Gootenberg, JS, Essletzbichler, P, Han, S, Joung, J, Belanto, JJ, et al. Segmentación de ARN con Crispr-Cas13. Naturaleza. (2017) 550:280–4. doi: 10.1038/nature24049
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Gootenberg, JS, Abudayyeh, OO, Lee, JW, Essletzbichler, P, Dy, AJ, Joung, J, et al. Detección de ácidos nucleicos con Crispr-Cas13a/C2c2. Science (Nueva York, NY). (2017) 356:438–42. doi: 10.1126/science.aam9321
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Doyle, LP, y Hutchings, LM. Una gastroenteritis transmisible en cerdos. J Am Vet Med Assoc. (1946) 108:257–9.
16. Zhang, F, Luo, S, Gu, J, Li, Z, Li, K, Yuan, W, et al. Prevalencia y análisis filogenético de los virus asociados a la diarrea porcina en el sur de China de 2012 a 2018. BMC Vet Res. (2019) 15:470. doi: 10.1186/s12917-019-2212-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Piepenburg, O, Williams, CH, Stemple, DL y Armes, NA. Detección de ADN mediante proteínas de recombinación. PLoS Biol. (2006) 4:E204. doi: 10.1371/journal.pbio.0040204
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Munawar, MA. Información crítica sobre la tecnología de amplificación de la polimerasa recombinasa. Experto Rev Mol Diagn. (2022) 22:725–37. doi: 10.1080/14737159.2022.2109964
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Lei, R, Kong, J, Qiu, Y, Chen, N, Zhu, S, Wang, X, et al. Detección rápida de los hongos patógenos causantes de la pata negra de Brassica Napus utilizando un detector de fluorescencia portátil en tiempo real. Químico de los alimentos. (2019) 288:57–67. doi: 10.1016/j.foodchem.2019.02.089
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Tran, DH, Tran, HT, Pham, TNM y Phung, HTT. Amplificación directa de la polimerasa recombinasa multiplex para la detección rápida de S Taphylococcus aureus y P Seudomonas aeruginosa en alimentos. Mol Biol Res Commun. (2022) 11:1–10. doi: 10.22099/mbrc.2021.41503.1664
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Wang, F, Wang, L, Chen, H, Li, N, Wang, Y, Li, Y, et al. Detección rápida de carbapenemasas Bla(Kpc), Bla(Ndm), Bla(Oxa-48-like) y Bla(imp) Carbapenemasas en Enterobacterales utilizando amplificación de polimerasa recombinasa combinada con tira de flujo lateral. Las células frontales infectan el microbiol. (2021) 11:772966. doi: 10.3389/fcimb.2021.772966
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Wang, X, Xie, S, Chen, X, Peng, C, Xu, X, Wei, W, et al. Un método rápido y conveniente para la detección in situ de maíz Mon863 mediante amplificación de la polimerasa recombinasa por fluorescencia en tiempo real. Químico de los alimentos. (2020) 324:126821. doi: 10.1016/j.foodchem.2020.126821
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Liu, S, Huang, G, Gong, Y, Jin, X, Meng, Y, Peng, Y, et al. Detección rápida y precisa del gen de resistencia a carbapenémicos mediante amplificación isotérmica en Acinetobacter Baumannii. Traumatismo por quemaduras. (2020) 8:tkaa026. doi: 10.1093/burnst/tkaa026
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Kersting, S, Rausch, V, Bier, FF y von Nickisch-Rosenegk, M. Detección rápida de plasmodium falciparum con amplificación isotérmica de polimerasa recombinasa y análisis de flujo lateral. Malar J. (2014) 13:99. doi: 10.1186/1475-2875-13-99
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Jinek, M, Chylinski, K, Fonfara, I, Hauer, M, Doudna, JA, y Charpentier, E. Una endonucleasa de ADN guiada por ARN dual programable en la inmunidad bacteriana adaptativa. Science (Nueva York, NY). (2012) 337:816–21. doi: 10.1126/science.1225829
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Eisenstein, M. Siete tecnologías a tener en cuenta en 2022. Naturaleza. (2022) 601:658–61. doi: 10.1038/d41586-022-00163-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Cox, DBT, Gootenberg, JS, Abudayyeh, OO, Franklin, B, Kellner, MJ, Joung, J, et al. Edición de ARN con Crispr-Cas13. Science (Nueva York, NY). (2017) 358:1019–27. doi: 10.1126/science.aaq0180
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Abudayyeh, OO, Gootenberg, JS, Konermann, S, Joung, J, Slaymaker, IM, Cox, DB, et al. C2c2 es un efector Crispr programable de un solo componente guiado por ARN dirigido por ARN. Ciencia. (2016) 353:AAF5573. doi: 10.1126/science.aaf5573
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Taylor, SC, Nadeau, K, Abbasi, M, Lachance, C, Nguyen, M, y Fenrich, J. El experimento definitivo de Qpcr: producir datos reproducibles y de calidad de publicación a la primera. Tendencias Biotechnol. (2019) 37:761–74. doi: 10.1016/j.tibtech.2018.12.002
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: virus de la gastroenteritis transmisible porcina (TGEV), amplificación de la polimerasa recombinasa (RPA), CRISPR-Cas13a, detección, amplificación isotérmica
Cita: Wang H, Cheng Z, Luo R, Yang Q, Zeng Y, Yang Y, Chen Y, Li W y Liu X (2024) Método de detección asistida por RPA-CRISPR-Cas13a del virus de la gastroenteritis transmisible. Frente. Vet. Sci. 11:1428591. doi: 10.3389/fvets.2024.1428591
Recibido: 07 de mayo de 2024; Aceptado: 21 de junio de 2024;
Publicado: 02 julio 2024.
Editado por:
Fanfan Zhang, Academia de Ciencias Agrícolas de Jiangxi (CAAS), China
Revisado por:
Yonghou Jiang, Universidad de Ciencia y Tecnología de Zhejiang, China
Gaurav Kumar Sharma, Instituto Indio de Investigación Veterinaria (IVRI), India
Derechos de autor © 2024 Wang, Cheng, Luo, Yang, Zeng, Yang, Chen, Li y Liu. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Xiao Liu, Drliuxiao@126.com; Wenting Li, wtl9911002@163.com
†Estos autores han contribuido igualmente a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía