Un objetivo alternativo de la vacuna para la anaplasmosis bovina
Un objetivo alternativo de la vacuna para la anaplasmosis bovina basada en enolasa, una proteína de luz lunar
 Rosa Estela Quiroz-Castañeda*†
Rosa Estela Quiroz-Castañeda*†  Hugo Aguilar-Díaz†
Hugo Aguilar-Díaz†  Itzel Amaro-Estrada
Itzel Amaro-Estrada- Centro Nacional de Investigación Disciplinaria en Salud Animal e Inocuidad, INIFAP. Carretera Federal Cuernavaca-Cuautla, Col. Progreso, Jiutepec, Morelos, México
El descubrimiento de nuevos objetivos para prevenir la anaplasmosis bovina se ha alejado de centrarse en proteínas que ya se han estudiado ampliamente en Anaplasma marginal, incluidas las proteínas principales de superficie, las proteínas de la membrana externa y las proteínas del sistema de secreción tipo IV. Una alternativa son las proteínas multifuncionales o moonlighting, capaces de realizar diversas funciones biológicas dentro de varios compartimentos celulares. Hay varios informes sobre el papel de las proteínas moonlighting como factores de virulencia en diversos microorganismos. Además, se sabe que alrededor del 25% de toda la luz de la luna está involucrada en la virulencia de los patógenos. En este trabajo, por primera vez, presentamos la identificación de tres proteínas enolasas (AmEno01, AmEno15 y AmEno31) en el genoma de cepas mexicanas de A. marginale. Usando herramientas bioinformáticas, predijimos los dominios catalíticos, la firma de enolasa y los aminoácidos que se unen al ion magnesio del dominio catalítico y realizamos una reconstrucción filogenética. Además, mediante análisis de acoplamiento molecular, encontramos que AmEno01 se uniría a las proteínas eritrocitarias espectrina, anquirina y estomatina. Esta función de adhesión ha sido reportada para enolasas de otros patógenos. Se considera una diana prometedora ya que el bloqueo de esta función impediría el proceso de adhesión fundamental que facilita la infección de los eritrocitos. Además, el acoplamiento molecular predice que AmEno01 podría unirse a la proteína de la matriz extracelular fibronectina, lo que sería significativo si consideramos que algunas proteínas con dominios de fibronectina se localizan en las células intestinales de las garrapatas y se utilizan como una estrategia de adhesión para recolectar bacterias antes de viajar a las glándulas salivales. Derivado del análisis de acoplamiento molecular de AmEno01, planteamos la hipótesis de que las enolasas podrían ser proteínas impulsadas por el patógeno y redirigidas a expensas de las necesidades del patógeno.
1. Introducción
El desempeño de más de una función (moonlighting) por una sola proteína ha sido reconocido como un fenómeno común con implicaciones significativas en los procesos metabólicos y otras funciones en bacterias, plantas, levaduras, hongos, parásitos y vertebrados (1-8). Las proteínas moonlighting se describieron a fines de la década de 1980 como proteínas estructurales en el cristalino del ojo (cristalinas) con una segunda e incluso una tercera función (9-11). La mayoría de las funciones de las proteínas de luz lunar están relacionadas con funciones bioquímicas o biofísicas fisiológicamente relevantes (12-14). Las funciones de repertorio de las proteínas moonlighting reportadas en los últimos años incluyen su participación como enzimas del ciclo TCA (aconitasa, Homo sapiens, Saccharomyces cerevisiae, Mycobacterium tuberculosis) (15, 16); metabolismo de la glucosa (aldolasa, Arabidopsis thaliana, hexoquinasa; Plasmodium vivax) (17, 18); chaperonas (GroEL, S. cerevisiae, HSP60; Enterobacter aerogenes) proteínas antioxidantes (tiorredoxina, Escherichia coli) (19); funciones asociadas a virulencia (factor de elongación Tu y enolasa, Mycoplasma pneumoniae, Pseudomonas aeruginosa, Streptococcus pneumoniae, Plasmodium spp.) (20, 21); entre otras funciones en diferentes organismos que han sido ampliamente reportadas (6, 22-24) En los últimos años, debido a las tecnologías de secuenciación y los avances metagenómicos, se están descubriendo más proteínas de luz lunar y sus funciones en diversos organismos (25).
Actualmente, se han propuesto dos grandes grupos para clasificar las proteínas de luz lunar: 1) «enzimas desencadenantes» y 2) proteínas intracelulares/secretadas de luz lunar (4, 26). El primer subconjunto comprende enzimas que regulan la transcripción o la traducción uniéndose directamente al ADN o ARN o uniéndose a otros factores de traducción o transcripción proteicos (27). El segundo subconjunto incluye las proteínas de luz lunar más extensas conocidas, con actividades como enzimas de limpieza, chaperonas, factores de traducción, adhesión, proteínas de unión al ADN y muchas otras que se secretan y residen unidas a la superficie celular, actuando como receptores para proteínas solubles o moléculas pequeñas, o funcionan en la fase fluida, a menudo para la señalización intracelular (4). En este sentido, como las proteínas moonlighting realizan sus funciones canónicas y lunares en compartimentos celulares separados (citoplasma y superficie celular), esta localización celular dual de la proteína sugiere fuertemente una actividad multifuncional (28).
La presencia de proteínas moonlighting es relevante en bacterias, y su estudio se centra en dilucidar sus funciones alternativas, ya que están presentes tanto en bacterias patógenas como comensales (23).
En este sentido, Anaplasma marginale es un patógeno intracelular gramnegativo conocido como el agente causal de la Anaplasmosis bovina, una enfermedad infecciosa no contagiosa caracterizada por anemia hemolítica progresiva, abortos, pérdida de condición, producción de leche e incluso la muerte (6, 29, 30). Hasta ahora, siete genomas (~1.2 Mbp) de cepas mexicanas de A. marginale han sido reportados y anotados (31-33). Debido a su tamaño reducido del genoma, las proteínas de luz lunar en este patógeno podrían ser una estrategia para maximizar eficientemente el uso de sus proteínas (31, 34).
Actualmente, nuestro interés se centra en las proteínas moonlighting de este patógeno transmitido por vectores, ya que podrían participar en su patogenicidad o evasión del sistema inmune del huésped, como se ha informado para muchos patógenos, que emplean proteínas moonlighting/multitarea como factores de virulencia para interferir con múltiples procesos celulares, en diferentes compartimentos en diferentes momentos durante la infección, aumentando su virulencia (6). Por lo tanto, realizamos un análisis genómico profundo y agudo que nos permitió identificar posibles proteínas de luz lunar en A. marginale, incluida la enolasa (AmEno), que no se han reportado antes en este patógeno. La enolasa (2-fosfo-D-glicerato hidrolasa, EC 4.2.1.11) es una proteína de luz lunar intensamente estudiada que convierte el 2-fosfoglicerato en fosfoenolpiruvato en glucólisis. Además de participar en esta vía metabólica, la enolasa facilita la unión a las células huésped, como se informa en Anaplasma phagocytophilum, en el que la enolasa se une al plasminógeno del huésped. En 2018, Gao et al. (35) demostraron que la enolasa recombinante de A. phagocytophilum puede unirse y activar el plasminógeno y promover la conversión en plasmina, por lo que es crucial para la infección por patógenos. Además, esta enolasa se consideró un objetivo potencial para controlar la infección por anaplasmosis. En Borrelia burgdorferi, la enolasa expresada en la superficie juega un papel esencial durante la invasión de patógenos al unirse al plasminógeno de mamíferos (36, 37). Recientemente, Xie et al., (38) confirmaron que Mycoplasma hyopneumoniae enolasa se localiza en su superficie y es capaz de adherirse a las células epiteliales traqueales porcinas.
En este trabajo, realizamos análisis bioinformáticos, modelado tridimensional (3D) y acoplamiento de la enolasa de A. marginale, AmEno. Este estudio tuvo como objetivo identificar in silico su potencial para interactuar con diferentes proteínas de la matriz extracelular (ECM), la membrana de eritrocitos (EM) y el plasminógeno zimógeno que circula en la sangre de los mamíferos. Además, podría guiar el desarrollo de una estrategia racional y aguda para comprender la interacción y las funciones de la enolasa y algunos ligandos, que son esenciales para el éxito del establecimiento del patógeno.
2. Materiales y métodos
2.1. Identificación de enolasas lunares en genomas marginales de Anaplasma
Los siete borradores de genomas de A. marginale reportados (MEX-01-001-01, MEX-14-010-01, MEX-15-099-01, MEX-17-017-01, MEX-30-184-02, MEX-30-193-01 y MEX-31-096-01) habían sido previamente anotados automáticamente utilizando el servidor RAST (versión 2.0) (39). Derivado de esta anotación, identificamos un gen de enolasa en cada uno de los siete genomas. Además, se recuperaron secuencias de enolasas de diferentes organismos de una búsqueda de Blastp en NCBI. Además, una búsqueda en AlphaFold Protein Structure Database (40) y MoonProt 2.0 (41) permitió comparar la enolasa de A. marginale con las reportadas como proteínas de virulencia patógena. Las secuencias seleccionadas fueron utilizadas en la reconstrucción filogenética.
2.2. Reconstrucción filogenética
Las secuencias de enolasa fueron seleccionadas de los dominios Eukarya y Bacteria (Tabla S1). Todas las alineaciones múltiples se realizaron con Clustal Omega (42) y se visualizaron con Jalview (43). Se utilizó un método de unión vecina para reconstruir una filogenia utilizando el software Mega 11 (44) con un modelo de sustitución de Poisson y un valor de arranque de 1.000 réplicas.
2.3. Análisis bioinformáticos de Anaplasma marginale enolasas
El magnesio (Mg2+) los sitios de unión, que son esenciales para la actividad catalítica de la enolasa y los dominios conservados, se predijeron en la base de datos de dominios conservados (CDD-NCBI) (44) y ScanProsite (45). Las regiones transmembrana se predijeron con DeepTMHMM (46). La localización subcelular de las proteínas se predijo en PSORTb 3.0 (46), y la estructura y función secundarias se predijeron en PSIPRED Workbench (47). La topología de las proteínas se predijo en CATH (48).
2.4. Modelado tridimensional (3D)
SwissModel es un servidor de modelado de homología de estructura de proteínas ampliamente utilizado para predecir la estructura 3D de proteínas (49). Utilizamos este servidor para predecir las estructuras 3D de las enolasas de A. marginale de las cepas MEX-01-001-01 (AmEno01), MEX-15-099-01 (AmEno15) y MEX-31-096-01 (AmEno31). El resto de las enolasas de las cepas MEX-30-193-01, MEX-30-184-02, MEX-14-010-01 y MEX-17-017-01 son esencialmente idénticas a MEX-01-001-01; En consecuencia, estas estructuras no fueron modeladas.
El modelado de homología es actualmente un método preciso para generar modelos confiables de estructura de proteínas 3D, utilizando estructuras de proteínas experimentales de PDB («plantillas») para construir modelos para proteínas relacionadas con la evolución («objetivos»). Todos los modelos generados en SwissModel se basan en la calidad del modelo GMQE (Global Model Quality Estimate) y QMEAN.
2.5. Acoplamiento molecular y análisis de interacción
El acoplamiento del AmEno01 modelado y cinco posibles ligandos se realizó en ClusPro (50) para analizar su afinidad de unión. Los números de identificación PDB de los ligandos son plasminógeno (4DUR), fibronectina (3M7P), espectrina (3LBX), anquirina (4RLV) y estomatina (4FVF).
ClusPro es un servidor que utiliza un método de transformada rápida de Fourier (FFT) llamado Piper, donde una de las proteínas se coloca en el origen del sistema de coordenadas en una cuadrícula fija, la segunda proteína se coloca en una cuadrícula móvil y la energía de interacción se escribe como una suma de unas pocas funciones de correlación. El algoritmo gira el ligando con 70.000 rotaciones. Se eligen las 1.000 combinaciones de rotaciones/traslación de las 70.000 rotaciones con la puntuación más baja, y estas 1.000 posiciones de ligando se agrupan con un radio RMSD de 9 Å C-alfa (51). En ClusPro, generamos modelos de acoplamiento utilizando AmEno01 y los cinco ligandos potenciales. Para cada acoplamiento molecular, se generaron diez modelos que se descargaron en formato PDB y se visualizaron en UCSF ChimeraX (52). El modelo con la puntuación más alta de los diez modelos de acoplamiento fue seleccionado para visualizar el modelo de superficie de contacto en HDOCK (53).
El análisis de las interacciones entre aminoácidos de los cinco complejos ligando AmEno01 y la visualización se realizaron en PDBsum (54). Para esto, primero usamos la opción PDBsum Generate para cargar cada modelo de acoplamiento y generar un código PDB. Una vez que recuperamos el código PDB de los cinco modelos de acoplamiento, lo usamos como una entrada en PDBsum para analizar las interacciones del ligando AmEno01.
3. Resultados
3.1. Identificación de enolasas moonlighting en genomas marginales de Anaplasma mexicanos
Identificamos una enolasa por genoma en las siete cepas mexicanas de A. marginale. Las enolasas de las cepas MEX-30-184-02 (GenBank KAB0450913.1), MEX-17-017-01 (KAB0451331.1), MEX-30-193-01 (KAB0450361.1), MEX-14-010-01 (TZF77690.1) y MEX-01-001-01 (RCL19410.1) tenían 425 aminoácidos de longitud. La enolasa de la cepa MEX-15-099-01 (KAA8472002.1) tenía 450 aminoácidos, incluyendo 26 aminoácidos adicionales (MLYLSLLCLLFRKDCLFCPPLGVRAV) en el extremo N-terminal, y finalmente, la enolasa de la cepa MEX-31-096-01 (KAA8473352.1) tenía 431 aminoácidos, considerando seis aminoácidos adicionales (MGVRAV) en el extremo N-terminal. Sólo se observaron once diferencias en las secuencias de aminoácidos en las siete secuencias de enolasa (Figura 1). Se realizaron predicciones bioinformáticas a todas las enolasas de los tres Grupos; sin embargo, los análisis de modelado y acoplamiento que presentamos aquí se realizaron solo con la cepa MEX-01-001-01, secuencia AmEno01. Este modelo era representativo de las tres enolasas de A. marginale. Sin embargo, para confirmar que el modelado 3D y el acoplamiento molecular de las enolasas de los Grupos 2 y 3 no variaron sustancialmente debido a las diferencias a nivel de secuencia, también realizamos un acoplamiento de estas proteínas con los ligandos. Sin embargo, no se encontró variación significativa a nivel de estructura o interacción (datos no mostrados).
 Figura 1. Alineaciones múltiples de cepas mexicanas de A. marginale enolasas usando Clustal Omega. Las enolasas se clasifican en tres grupos. El grupo 1 comprende las cepas MEX-01-001-01, MEX-14-010-01, MEX-17-017-01, MEX-30-184-02 y MEX-30-193-01. Estas secuencias comparten la misma longitud de 425 aa. El grupo 2 comprende la cepa MEX-15-099-01 con 450 aa. Observe los 26 aminoácidos adicionales en el extremo N-terminal. El Grupo 3 comprende MEX-31-096-01 con 431 aa, incluyendo seis aminoácidos adicionales en el extremo N-terminal. El porcentaje de identidad oscila entre el 97,41 y el 99,76%.
Figura 1. Alineaciones múltiples de cepas mexicanas de A. marginale enolasas usando Clustal Omega. Las enolasas se clasifican en tres grupos. El grupo 1 comprende las cepas MEX-01-001-01, MEX-14-010-01, MEX-17-017-01, MEX-30-184-02 y MEX-30-193-01. Estas secuencias comparten la misma longitud de 425 aa. El grupo 2 comprende la cepa MEX-15-099-01 con 450 aa. Observe los 26 aminoácidos adicionales en el extremo N-terminal. El Grupo 3 comprende MEX-31-096-01 con 431 aa, incluyendo seis aminoácidos adicionales en el extremo N-terminal. El porcentaje de identidad oscila entre el 97,41 y el 99,76%.
3.2. Reconstrucción filogenética
Se reconstruyó un árbol filogenético para determinar la relación filogenética de AmEno01 y otras enolasas reportadas. Se sabe que las enolasas están bien definidas en grupos alfa, beta y gamma en mamíferos. En la reconstrucción filogenética, encontramos esta clasificación en Chordata. Las enolasas de las garrapatas Rhipicephalus spp. e Ixodes scapularis se agruparon en un clado perteneciente a Arthropoda, y las de organismos protistas se separaron de Animalia. En Bacterias, las enolasas MEX-01-001-01 (AmEno01), MEX-14-010-01 (AmEno14), MEX-17-017-01 (AmEno17), MEX-30-184-02 [(AmEno30-02) y MEX-30-193-01 (AmEno30-01)] se agruparon en un clado único que llamamos Grupo 1. La enolasa MEX-15-099-01 (AmEno15) y las cepas brasileñas Jaboticabal y Palmeira se agruparon en el Grupo 2. Finalmente, la enolasa MEX-31-096-01 (AmEno31) se agrupó con la cepa de referencia A. marginale St. Maries en el Grupo 3 (Figura 2). Además, la identidad secuencial de las cepas mexicanas varió de 97.65 a 99.53% en el Grupo 1; la identidad de secuencia entre AmEno15 y las cepas brasileñas fue del 100%; y la identidad entre AmEno31 y la cepa de referencia St. Maries fue del 98,38%.
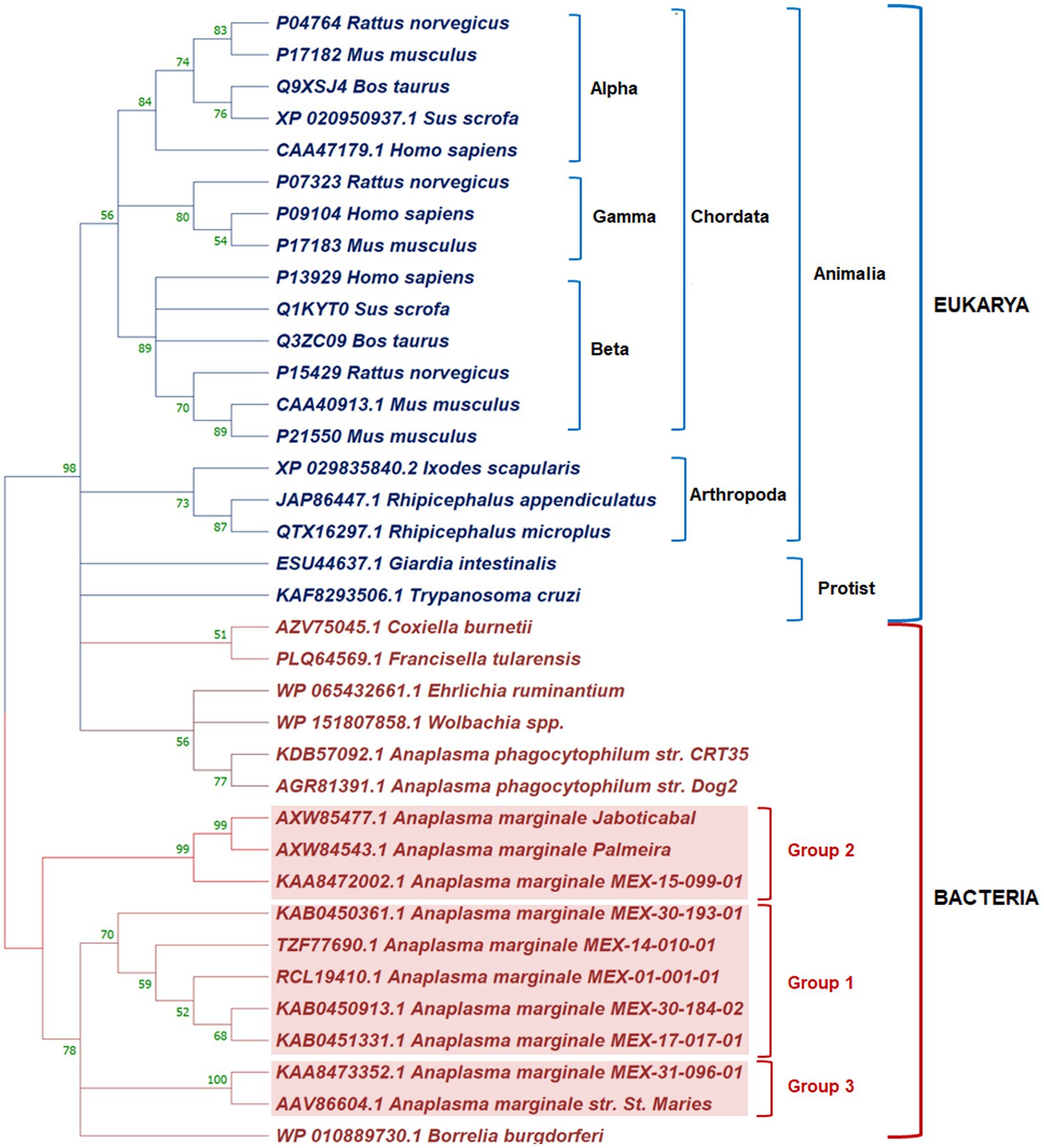 Figura 2. Reconstrucción filogenética de enolasas de los dominios Eukarya y Bacteria en Mega 11. En Eukarya, las enolasas de Chordata están organizadas en grupos alfa, beta y gamma. Las enolasas de garrapatas forman un clado separado de Animalia y Protist. En Bacterias, las cepas mexicanas de A. marginale se agruparon con cepas brasileñas y cepas norteamericanas (cajas rojas).
Figura 2. Reconstrucción filogenética de enolasas de los dominios Eukarya y Bacteria en Mega 11. En Eukarya, las enolasas de Chordata están organizadas en grupos alfa, beta y gamma. Las enolasas de garrapatas forman un clado separado de Animalia y Protist. En Bacterias, las cepas mexicanas de A. marginale se agruparon con cepas brasileñas y cepas norteamericanas (cajas rojas).
3.3. Análisis bioinformáticos de Anaplasma marginale enolasas
El análisis en la base de datos CDD mostró cuatro Mg2+ sitios de unión identificados en AmEno01, AmEno14, AmEno17, AmEno30-02, AmEno30-01, AmEno15 y AmEno31, que fueron S, D, E y D, variando en posición a lo largo de las secuencias (Tabla 1). Estos residuos son significativos debido a su papel en la actividad catalítica de la enolasa. La firma de enolasa se identificó en el análisis de ScanProsite. Además, DeepTMHMM predijo una localización dentro de la célula para todas las enolasas, ya que no se identificaron regiones transmembrana y no se predijo ningún péptido señal con PSORT. También se identificaron hebras beta, hélices alfa y bobinas (Figura Suplementaria S1). Para identificar y comparar las secuencias de firma de enolasa, los aminoácidos del sitio catalítico y los bucles del sitio activo, contrastamos las enolasas AmEno01, AmEno14, AmEno17, AmEno30-02, AmEno30-01, AmEno15 y AmEno31 con información previamente reportada para enolasas de buena fe de H. sapiens, T. cruzi, S. pneumoniae, A. phagocytophilum y, R. microplus (Tabla 1). Encontramos que los aminoácidos con un papel significativo en el sitio catalítico de las enolasas eran esencialmente H, E, E, E, D, K, R, S y K, que variaban en la posición de la secuencia. Además, encontramos variantes de la firma de enolasa y el sitio de unión al plasminógeno en cepas mexicanas (Tabla 1 y Figura 3).
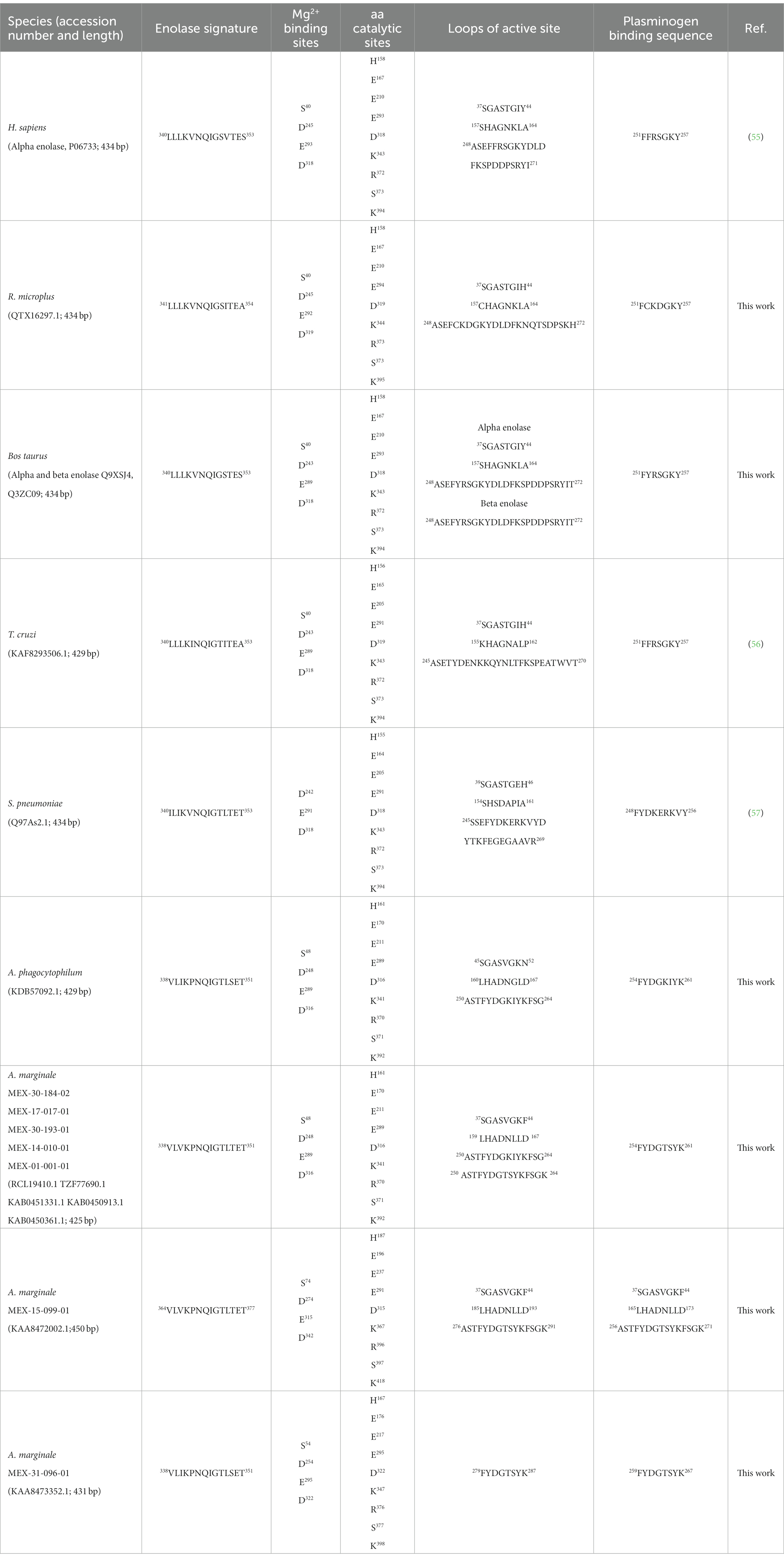 Tabla 1. Características de algunas enolasas reportadas para animales, protistas y bacterias, incluyendo enolasas de A. marginale.
Tabla 1. Características de algunas enolasas reportadas para animales, protistas y bacterias, incluyendo enolasas de A. marginale.
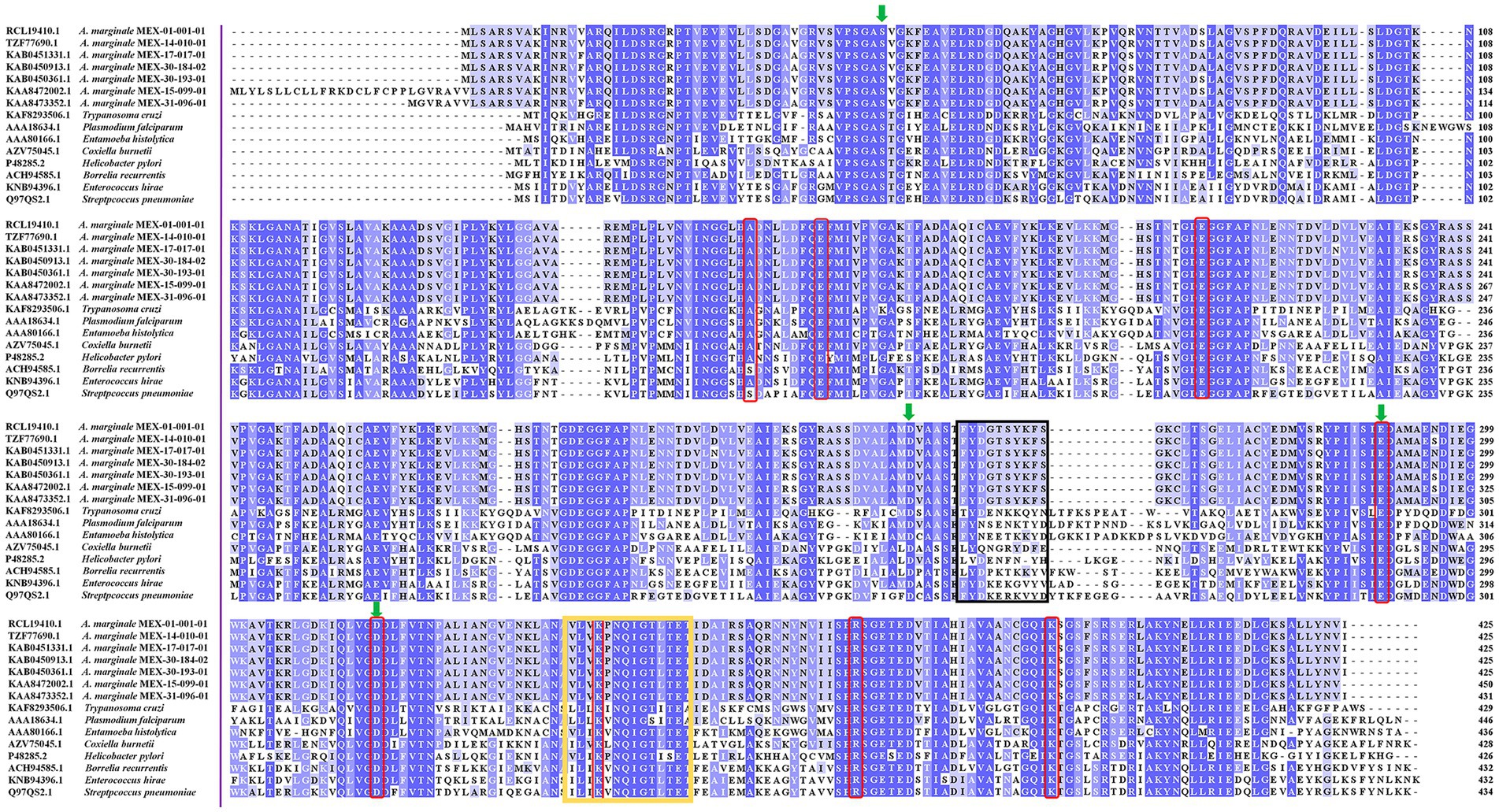 Figura 3. Enolasas de parásitos y bacterias alineadas con Clustal Omega. La secuencia de unión al plasminógeno se muestra en la caja negra. La secuencia FYDGTSYKFS en cepas de A. marginale varía en un 70% de la reportada en S. pneumoniae, que se une al plasminógeno por la secuencia FYDKERKVYD. La firma de enolasa en las cepas de A. marginale tiene siete cambios de aminoácidos en comparación con las secuencias que se muestran en el recuadro amarillo. Los aminoácidos que se unen a los átomos de magnesio se muestran en flechas verdes y los aminoácidos del sitio catalítico se muestran en cuadros rojos.
Figura 3. Enolasas de parásitos y bacterias alineadas con Clustal Omega. La secuencia de unión al plasminógeno se muestra en la caja negra. La secuencia FYDGTSYKFS en cepas de A. marginale varía en un 70% de la reportada en S. pneumoniae, que se une al plasminógeno por la secuencia FYDKERKVYD. La firma de enolasa en las cepas de A. marginale tiene siete cambios de aminoácidos en comparación con las secuencias que se muestran en el recuadro amarillo. Los aminoácidos que se unen a los átomos de magnesio se muestran en flechas verdes y los aminoácidos del sitio catalítico se muestran en cuadros rojos.
3.4. Modelado tridimensional (3D) y acoplamiento molecular
Seleccionamos las tres enolasas representativas AmEno01, AmEno15 y AmEno31, como secuencias de destino de entrada en el servidor SwissModel. Los resultados en SwissModel para plantillas que coincidían con la secuencia objetivo se ordenaron por mayor valor GMQE, y la enolasa de Enterococcus hirae (PDB 1IYX) con un valor de GMQE de 0,88 se seleccionó como plantilla para modelar las tres enolasas. Los porcentajes de identidad y cobertura entre la plantilla 1IYX y el modelo AmEno01 fueron 56,83 y 98%, respectivamente; para el modelo AmEno15, fueron 56,49 y 92%, respectivamente; y para el modelo AmEno31 fueron 56.83 y 97%, respectivamente (Figuras 4A-C).
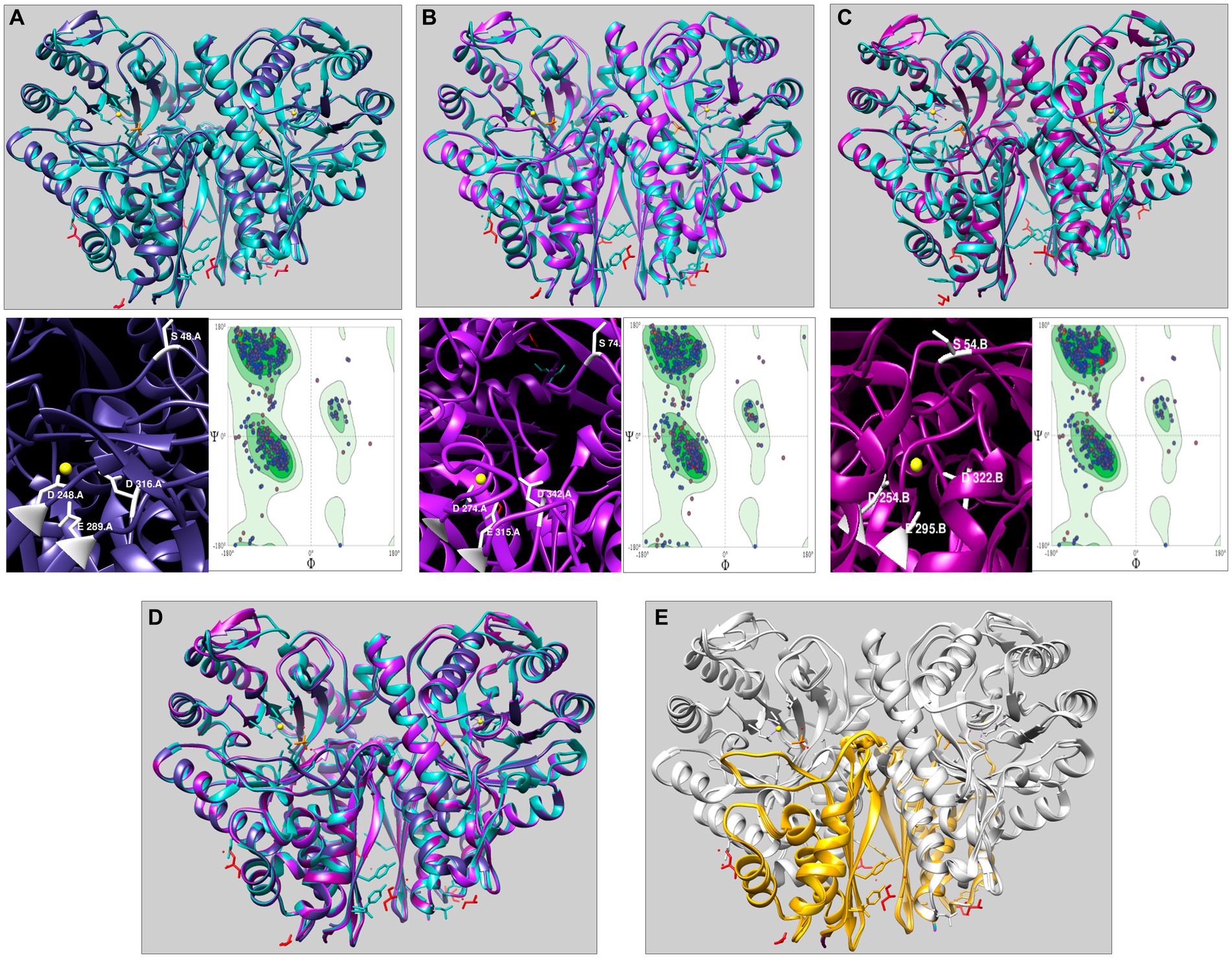 Figura 4. Modelado tridimensional de enolasas de cepas mexicanas de A. marginale utilizando Enterococcus hirae enolasa como plantilla (PDB 1IYX, cian). En las tres enolasas modeladas obtenidas en el modelo suizo (A) AmEno01 (púrpura), (B) AmEno15 (magenta) y (C) AmEno31, se muestran los átomos de unión de magnesio S, D, E y D (que varían en posición). Los modelos se consideraron factibles de acuerdo con las parcelas de Ramachandran. (D) Superposición de AmEno01, AmEno15, AmEno31 y 1IYX. (E) Las enolasas tienen la topología de un sándwich de dos capas en el extremo N-terminal (dorado) y un barril alfa-beta (barril TIM) en el extremo C-terminal (plata). Para todos los modelos, los átomos de magnesio se muestran en círculos amarillos; La estructura de las moléculas de glicerol está en rojo y los sulfatos en naranja. Todos los modelos 3D fueron visualizados y coloreados en ChimeraX.
Figura 4. Modelado tridimensional de enolasas de cepas mexicanas de A. marginale utilizando Enterococcus hirae enolasa como plantilla (PDB 1IYX, cian). En las tres enolasas modeladas obtenidas en el modelo suizo (A) AmEno01 (púrpura), (B) AmEno15 (magenta) y (C) AmEno31, se muestran los átomos de unión de magnesio S, D, E y D (que varían en posición). Los modelos se consideraron factibles de acuerdo con las parcelas de Ramachandran. (D) Superposición de AmEno01, AmEno15, AmEno31 y 1IYX. (E) Las enolasas tienen la topología de un sándwich de dos capas en el extremo N-terminal (dorado) y un barril alfa-beta (barril TIM) en el extremo C-terminal (plata). Para todos los modelos, los átomos de magnesio se muestran en círculos amarillos; La estructura de las moléculas de glicerol está en rojo y los sulfatos en naranja. Todos los modelos 3D fueron visualizados y coloreados en ChimeraX.
El valor GMQE para las enolasas modeladas AmEno01, AmEno15 y AmEno31 fue de 0,82, 0,80 y 0,82, respectivamente, que es un valor de precisión significativo; Por lo tanto, estos eran modelos confiables. El valor QMEAN para los modelos AmEno01, AmEno15 y AmEno31 fue de 0,82 ± 0,05.
Dado que AmEno01, AmEno15 y AmEno31 presentaron una estructura 3D muy similar (Figura 4D), con una topología de un barril alfa-beta (barril TIM) en el extremo C-terminal y un sándwich de dos capas en el extremo N-terminal (Figura 4E), realizamos el acoplamiento con sólo AmEno01 como modelo representativo de las cepas mexicanas enolasas.
Por lo tanto, el modelo de AmEno01 se descargó en formato PDB para ser utilizado en acoplamiento molecular en ClusPro. Se realizaron cinco acoplamientos proteína-proteína en ClusPro, donde AmEno01 se consideró como receptor, y las proteínas plasminógeno (4DUR), fibronectina (3M7P), espectrina (3LBX), anquirina (4RLV) y estomatina (4FVF), como ligandos.
Los diez modelos de acoplamiento generados por ClusPro se recuperaron como «modelos con coeficientes equilibrados ya que no sabemos qué fuerzas dominan el complejo proteína-proteína», como se recomienda en el manual de ClusPro. Uno de los diez modelos de acoplamiento para cada ligando AmEno01 fue visualizado por UCSF ChimeraX. Los acoplamientos moleculares se muestran en la Figura 5, y los modelos de superficie de contacto para las cinco interacciones de los ligandos AmEno01 se muestran en la Figura 2 Suplementaria.
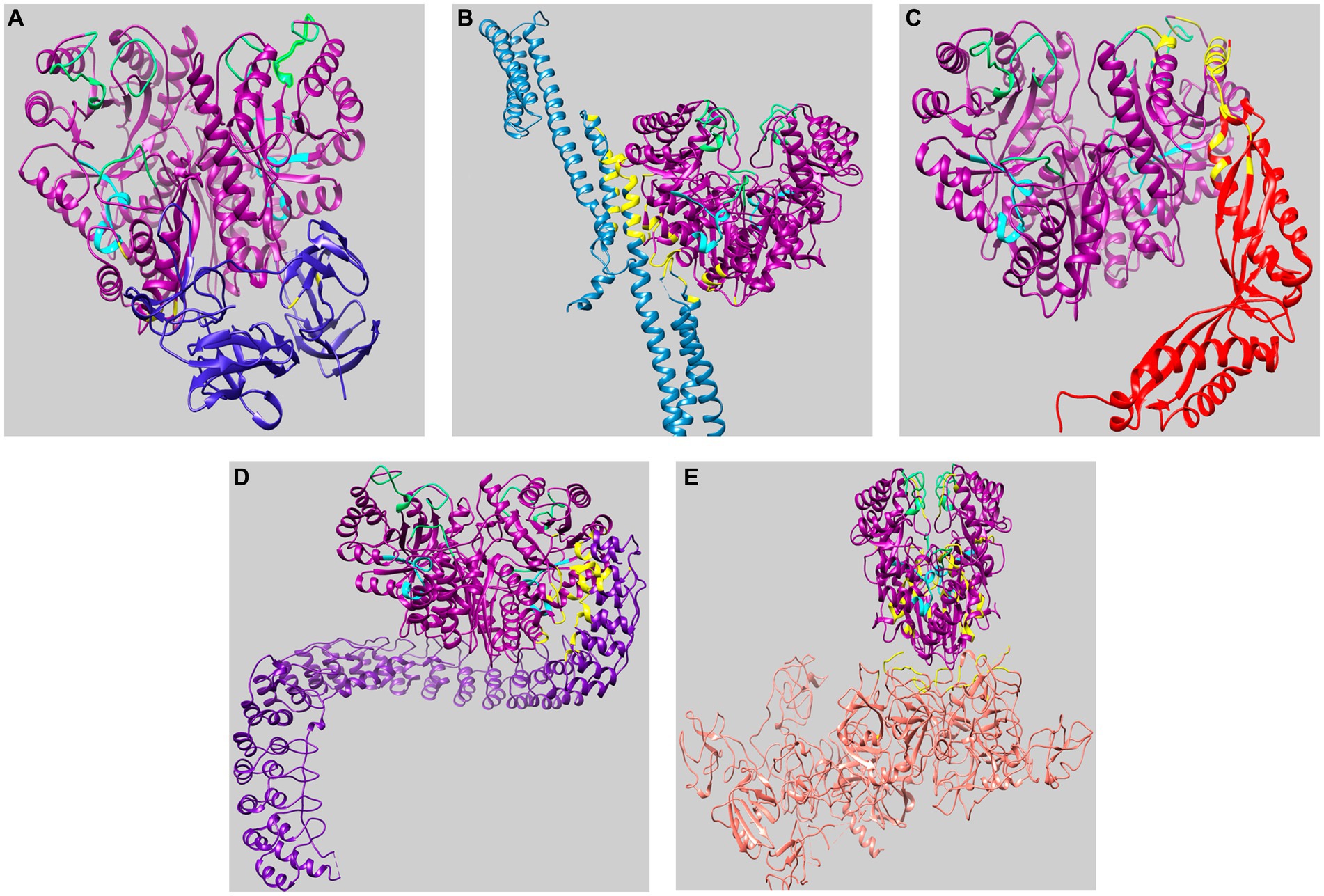
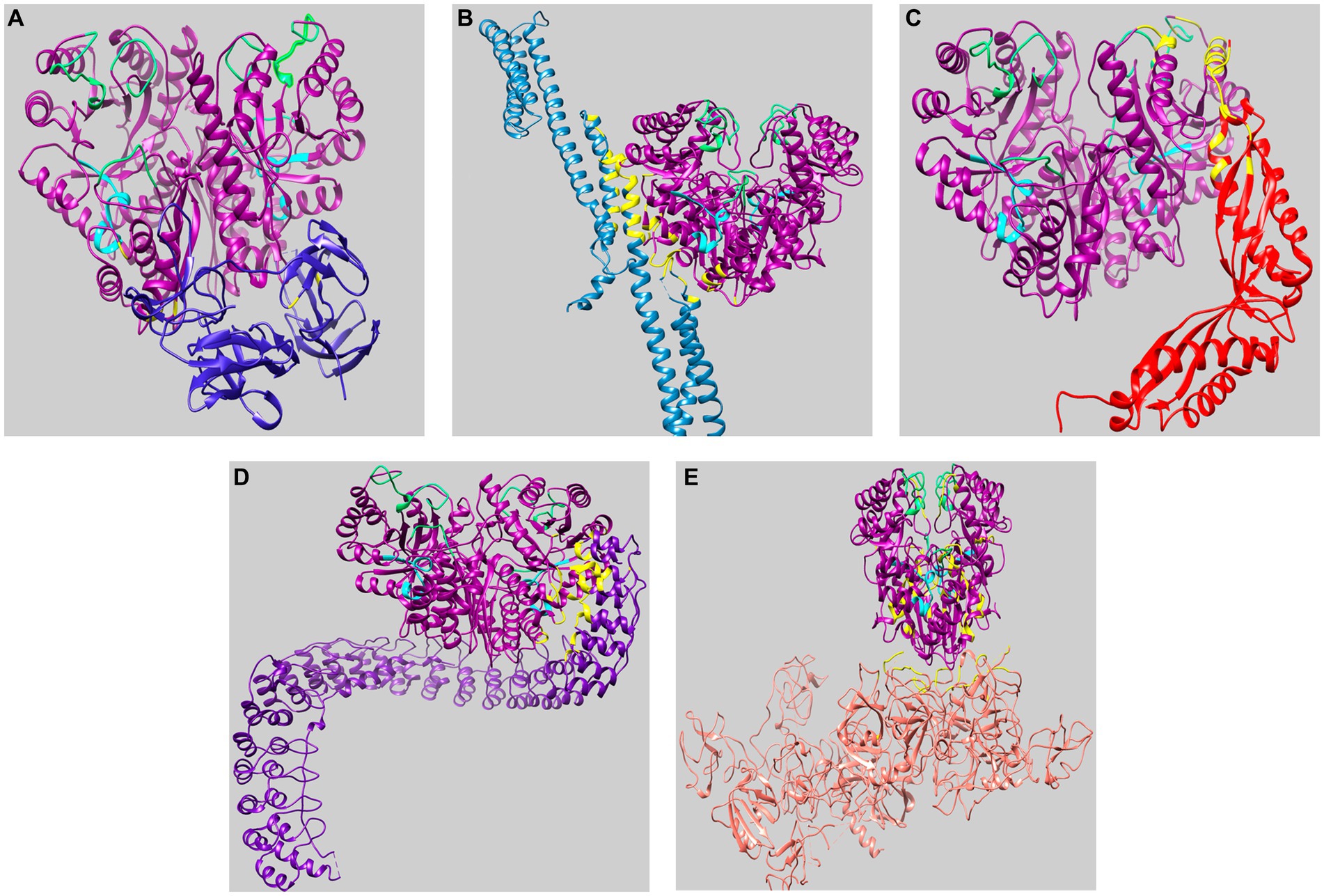 Figura 5. Visualización del acoplamiento molecular de AmEno01 con ligandos recuperados en ClusPro. (A) fibronectina (3M7P); (B) espectrina (3LBX); (C) estomatina (4FVF); (D) anquirina (4RLV); y (E) plasminógeno (4DUR). Los aminoácidos predichos que participan en la interacción entre AmEno01 y cada ligando se muestran en amarillo, la firma de la enolasa se muestra en cian y los bucles del sitio activo se muestran en verde. Las estructuras acopladas fueron visualizadas y coloreadas en ChimeraX.
Figura 5. Visualización del acoplamiento molecular de AmEno01 con ligandos recuperados en ClusPro. (A) fibronectina (3M7P); (B) espectrina (3LBX); (C) estomatina (4FVF); (D) anquirina (4RLV); y (E) plasminógeno (4DUR). Los aminoácidos predichos que participan en la interacción entre AmEno01 y cada ligando se muestran en amarillo, la firma de la enolasa se muestra en cian y los bucles del sitio activo se muestran en verde. Las estructuras acopladas fueron visualizadas y coloreadas en ChimeraX.
Además, los cinco acoplamientos moleculares se evaluaron utilizando las parcelas de Ramachandran para validar la calidad del acoplamiento mediante PDBsum. De acuerdo con esto, los valores obtenidos para cada acoplamiento fueron 86.6% (AmEno01-3M7P), 87.3% (AmEno01-3LBX), 86.6% (AmEno01-4FVF), 83.9% (AmEno01-4RLV) y 76.3% (AmEno01-4DUR).
Por último, el análisis de proteína-proteína (AmEno01-ligando) realizado en PDBsum para los cinco modelos de acoplamiento permitió la identificación de las interacciones de residuos a través de la interfaz. Por lo tanto, se identificaron los puentes salinos, los enlaces disulfuro, los enlaces de hidrógeno y los contactos no unidos para cada modelo de acoplamiento (Figura complementaria 3).
4. Discusión
Las funciones reportadas para las enolasas como proteínas de luz lunar en los últimos años amplían sus aplicaciones potenciales en el área poco explorada de enfermedades veterinarias, como la Anaplasmosis bovina. A. marginale es el agente causal de esta enfermedad, y hasta ahora se han reportado siete genomas de cepas mexicanas sin ninguna proteína de luz lunar reportada en este patógeno (5, 55, 57). Sin embargo, su presencia en genomas de cepas mexicanas de A. marginale sugiere que podrían tener funciones similares reportadas en otros lugares para este tipo de proteína. La A. marginale enolasa (AmEno), es de gran interés ya que esta proteína podría tener un papel significativo durante la invasión globular del patógeno, como se ha informado para Mycoplasma suis donde la enolasa participaría en la adhesión del patógeno a los eritrocitos porcinos en las primeras etapas del proceso de invasión (58).
El uso de enfoques bioinformáticos nos permitió identificar solo una secuencia de enolasa por genoma en cepas mexicanas de A. marginale, siete secuencias de enolasa. Como mostró la reconstrucción filogenética, estas secuencias se agruparon con otras enolasas en tres subclados diferentes: Grupo 1 (cinco cepas mexicanas enolasas de 425 aa), Grupo 2 (una cepa mexicana enolasa de 450 aa y cepas brasileñas enolasas Palmeira y Jaboticabal), y Grupo 3 (una cepa mexicana enolasa de 431 aa y cepa norteamericana St. Maries). Esto sugirió que algunas enolasas de cepas mexicanas de A. marginale podrían estar relacionadas con cepas brasileñas y norteamericanas, mientras que otras podrían ser exclusivas de México.
Aunque la principal diferencia entre las enolasas de los grupos 1 a 3 son aminoácidos adicionales en el extremo N-terminal, tienen estructuras secundarias similares de hebras beta, hélice alfa y estructuras de bobina. Además, estas diferencias a nivel de secuencia y longitud observadas en las enolasas mexicanas de A. marginale no causaron modificaciones significativas en las disposiciones estructurales 3D, como dilucidamos cuando realizamos el modelado 3D de miembros representativos de cada grupo de enolasas: AmEno01, AmEno15 y AmEno31. Después de predecir estructuras 3D para estas tres enolasas y darnos cuenta de que su estructura era prácticamente idéntica, seleccionamos AmEno01 para un análisis más detallado.
Con respecto al modelado 01D de AmEno3, nuestros resultados revelaron una estructura dimérica de la proteína, ya que se ha observado en otras estructuras de enolasa, incluidas Helicobacter pylori (59), Aeromonas hydrophyla (60), Candida albicans (61) y Mycoplasma pneumoniae (62). Además, se ha reportado una estructura octamérica de enolasa en Bacillus subtilis, Streptococcus spp. y Thermotoga maritima (63-65), y también se ha identificado una estructura monomérica en condiciones in vitro (66).
Funcionalmente, en la estructura 01D AmEno3, identificamos las secuencias de los bucles del sitio activo (37SGASVGKF44, 159LHADNLLD167, 250ASTFYDGKIYKFSG264y 250ASTFYDGTSYKFSGK 264) y la firma enolasa (338VLVKPNQIGTLTET351), que eran similares a las secuencias del resto de las cepas de las enolasas mexicanas.
Por otro lado, se ha reportado que dos Mg2+ iones se unen a los aminoácidos S, D, E y D en el sitio activo de la enolasa clásica, facilitando su reacción catalítica (67). En AmEno01, encontramos que el aminoácido S48 tiene una ubicación espacial diferente a la observada en otras enolasas; sin embargo, no podemos descartar su posible unión al Mg2+ ion. Además, en los modelos 3D predichos de AmEno01, AmEno15 y AmEno31, los gráficos de Ramachandran, que predicen la posible conformación de una proteína, revelaron que estos modelos con una ubicación espacial diferente de la S48 eran factibles.
Posteriormente, para analizar el potencial de AmEno01 para unirse a ligandos, exploramos sus interacciones con proteínas de la matriz extracelular o ECM (fibronectina), membrana de eritrocitos (espectrina, anquirina y estomatina) y plasminógeno mediante enfoques in silico.
En este sentido, nuestro análisis de la interacción AmEno01-fibronectina mostró que la gráfica de Ramachandran contiene 86,6% de residuos en las regiones más favorecidas [A, B, L], lo que sugiere la posibilidad de ocurrencia de esta interacción. La importancia de esta interacción se basa en la capacidad de unirse a la fibronectina como una característica reportada para muchos patógenos en los primeros pasos de la infección de los tejidos del huésped (68). Curiosamente, como A. marginale es un patógeno que no infecta tejidos, propusimos que el potencial de AmEno01 para unirse a la fibronectina es muy relevante para infectar garrapatas si consideramos la posibilidad de adherirse a las células intestinales de las garrapatas. Como en el espiroqueto B. burgdorferi, cuyo extracto de proteína de membrana interactúa con una proteína con dominios de fibronectina III (Ixofin3D) identificada en el intestino de Ixodes spp., facilitando la congregación de espiroquetas al intestino y proporcionando una salida molecular a las glándulas salivales antes de la transmisión al huésped humano (69, 70).
Por otro lado, decidimos realizar un análisis de acoplamiento para dilucidar si AmEno01 podría adherirse a las proteínas eritrocitarias espectrina y anquirina como parte del proceso de invasión inicial. De acuerdo con la gráfica de Ramachandran con 87.3 y 83.9% de residuos, respectivamente, en las regiones más favorecidas, nuestros resultados de acoplamiento sugirieron la unión de AmEno01 con espectrina y anquirina. Por lo tanto, planteamos la hipótesis de que AmEno01 se adhiere a estas proteínas y probablemente medie el proceso inicial de invasión de eritrocitos. Debemos destacar que en A. marginale, el proceso de entrada al eritrocito no es bien conocido; Sin embargo, la capacidad de adherirse a las células huésped es esencial para la infección (71).
Se han descrito algunos ejemplos de enolasa como molécula de adhesión a los eritrocitos. Al igual que M. suis enolase, que podría actuar como un factor de adhesión a los eritrocitos porcinos. Schreiner et al. (58) encontraron que la enolasa recombinante M. suis unida a lisados eritrocitos de una manera dependiente de la dosis, e incluso los transformadores E. coli adquirieron la capacidad de unirse a los eritrocitos debido a la expresión de la enolasa en su superficie.
Además de la espectrina y la anquirina, otra proteína eritrocitaria es la estomatina, una proteína integral que desempeña un papel como proteína de andamiaje unida a la membrana que modula la proteína de transporte (72). De acuerdo con esto, los resultados de acoplamiento de AmEno01 con estomatina mostraron 86.6% de residuos en las regiones más favorecidas en la parcela de Ramachandran. Por lo tanto, propusimos que AmEno01 podría reconocer la estomatina en la membrana de los eritrocitos como un paso esencial para la adhesión y una mayor internalización. En resumen, la espectrina, la anquirina y la estomatina podrían ser objetivos potenciales a ser evaluados experimentalmente para evitar la invasión de A. marginale a los eritrocitos.
Como se sabe, A. marginale infecta los eritrocitos pero no los tejidos. Por lo tanto, se debe analizar su unión al plasminógeno, un zimógeno que facilita la migración e invasión de patógenos a los tejidos del huésped cuando se convierte en plasmina por su actividad proteolítica. Curiosamente, encontramos que el 76.3% de los residuos en el acoplamiento AmEno01-plasminógeno se encuentran en regiones favorecidas de la parcela de Ramachandran, un porcentaje por debajo de los valores esperados para ser considerado un modelo de buena calidad. Además, la secuencia de unión al plasminógeno identificada en cepas mexicanas de A. marginale es diferente a la secuencia de enolasas bien conocidas que se unen al plasminógeno (73, 74).
En este sentido, teorizamos que AmEno01 podría estar uniéndose a proteínas de membrana de eritrocitos en lugar de unirse al plasminógeno; esta última es una estrategia que utilizan los parásitos para invadir tejidos como Leishmania mexicana (cysticercus), Taenia solium y las bacterias S. pneumoniae, Bacillus antracis y B. burgdorferi, entre otros (37, 73, 75-77).
Finalmente, el papel de las enolasas en muchos organismos aún está bajo estudio; Sin embargo, se han logrado muchos avances en sus funciones como proteínas moonlighting (78). Por lo tanto, el estudio de las actividades lunares atribuidas a enolasas en bacterias patógenas como A. marginale es relevante ya que podría ser un candidato para controlar la Anaplasmosis bovina. Esta propuesta se basa en los enfoques realizados con proteínas moonlighting como posibles candidatos a vacuna contra varias enfermedades animales. Por ejemplo, se observó una respuesta inmune robusta en ratones y lechones cuando se usó una subunidad enolasa de M. suis como antígeno (20), o la respuesta inmune protectora obtenida cuando la enolasa recombinante de P. vivax se expresó en E. coli y se usó como antígeno contra la malaria (79).
En este sentido, dilucidar las funciones de las enolasas de cepas mexicanas de A. marginale podría ser la base para desarrollar estrategias como un anticuerpo antienolasa que interfiera, evite la invasión de eritrocitos o bloquee algunos otros procesos vitales para la patogénesis. Además, no excluimos que las enolasas puedan ser proteínas impulsadas por el patógeno según sus necesidades ni descartamos su posible interacción con proteínas de garrapata que contribuyen a la supervivencia del patógeno dentro del vector.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio / repositorios y el (los) número (s) de acceso se pueden encontrar en el artículo / Material complementario.
Contribuciones del autor
HA-D y RQ-C realizaron los experimentos. HA-D, RQ-C e IA-E analizaron los datos. RQ-C y HA-D imaginaron y diseñaron el estudio, y RQ-C y HA-D escribieron el manuscrito. RQ-C, IA-E y HA-D editaron el manuscrito. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Esta investigación fue financiada por los proyectos INIFAP-Fiscal SIGI números 112353134456, 13232936047 y 1515235065; y CONAHCYT CF-2023-I-2600.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1225873/full#supplementary-material
Figura complementaria 1 | (A) fibronectina (3M7P); (B) espectrina (3LBX); (C) estomatina (4FVF) (D) anquirina (4RLV) y (E) plasminógeno (4DUR).
Figura complementaria 2 | (1) fibonectina (3M7P); (2) espectrina (3LBX); (3) estomatina (4FVF) (4) anquirina (4RLV) y (5) plasminógeno (4DUR).
Referencias
1. Krantz, M, y Klipp, E. Proteínas de luz lunar – un enfoque para sistematizar el concepto. In Silico Biol. (2020) 14:71–83. doi: 10.3233/ISB-190473
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Copley, SD. La iluminación lunar es la corriente principal: se requiere un ajuste de paradigma. BioEnsayos. (2012) 34:578–88. doi: 10.1002/bies.201100191
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Arvizu-Rubio, VJ, García-Carnero, LC, y Mora-Montes, HM. Proteínas de luz lunar en hongos médicamente relevantes. PeerJ. (2022) 10:E14001. doi: 10.7717/peerj.14001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Satala, D, Karkowska-Kuleta, J, Zelazna, A, Rapala-Kozik, M, and Kozik, A. Moonlighting proteins at the candidal cell surface. Microorganismos. (2020) 8:1–25. doi: 10.3390/microorganismos8071046
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Hemmadi, V, y Biswas, M. Una visión general de las proteínas moonlighting en la infección por Staphylococcus aureus. Arco Microbiol. (2021) 203:481–98. doi: 10.1007/s00203-020-02071-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Matos, AL, Curto, P, y Simões, I. Moonlighting in Rickettsiales: expanding virulence landscape. Trop Med Infect Dis. (2022) 7:1–19. doi: 10.3390/tropicalmed7020032
7. Schmid, L-M, Ohler, L, Möhlmann, T, Brachmann, A, Muiño, JM, Leister, D, et al. PUMPKIN, la única quinasa UMP plastidio, se asocia con intrones del grupo II y altera su metabolismo. Planta Physiol. (2019) 179:248–64. doi: 10.1104/pp.18.00687
8. Collingridge, PW, Brown, RWB, and Ginger, ML. Moonlighting enzymes in parasitic protozoa. Parasitología. (2010) 137:1467–75. doi: 10.1017/S0031182010000259
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Piatigorsky, J, y Wistow, GJ. Enzimas/cristalinas: el intercambio de genes como estrategia evolutiva. Células. (1989) 57:197–9. doi: 10.1016/0092-8674(89)90956-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Wistow, G, y Piatigorsky, J. Reclutamiento de enzimas como proteínas estructurales de la lente. Ciencia. (1987) 236:1554–6. doi: 10.1126/science.3589669
11. Ligabue-Braun, R, y Carlini, CR. Moonlighting toxins: ureases and beyond. Toxinas vegetales. (2015):1–21. DOI: 10.1007/978-94-007-6728-7_10-1
12. Jeffery, CJ. Proteínas moonlighting: proteínas viejas aprendiendo nuevos trucos. Tendencias Genet. (2003) 19:415–7. doi: 10.1016/S0168-9525(03)00167-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Jeffery, CJ. Protein moonlighting: ¿qué es y por qué es importante? Trad. R. Soc. (2017) B373:20160523. DOI: 10.1098/RSTB.2016.0523
14. Jeffery, CJ. Proteínas de luz lunar. Tendencias Biochem Sci. (1999) 24:8–11. DOI: 10.1016/S0968-0004(98)01335-8
15. Philpott, CC, Klausner, RD, y Rouault, TA. La proteína bifuncional de unión al elemento sensible al hierro / aconitasa citosólica: el papel de los residuos del sitio activo en la unión y regulación del ligando. Proc Natl Acad Sci U S A. (1994) 91:7321–5. DOI: 10.1073/PNAS.91.15.7321
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Banerjee, S, Nandyala, AK, Raviprasad, P, Ahmed, N, and Hasnain, SE. Iron-dependent RNA-binding activity of Mycobacterium tuberculosis aconitase. J Bacteriol. (2007) 189:4046–52. doi: 10.1128/JB.00026-07
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Moore, B, Zhou, L, Rolland, F, Hall, Q, Cheng, W-H, Liu, Y-X, et al. Papel del sensor de glucosa de Arabidopsis HXK1 en la señalización de nutrientes, luz y hormonales. Procedimientos resumidos para el período desde y un directorio de miembros a partir de. (2003) 300:332–6. doi: 10.1126/science.1080585
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Bosch, J, Buscaglia, CA, Krumm, B, Ingason, BP, Lucas, R, Roach, C, et al. La aldolasa proporciona un sitio de unión inusual para la proteína anónima relacionada con la trombospondina en la maquinaria de invasión del parásito de la malaria. Proc Natl Acad Sci U S A. (2007) 104:7015–20. DOI: 10.1073/PNAS.0605301104
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Bedford, E, Tabor, S, y Richardson, CC. El dominio de unión a tiorredoxina de la ADN polimerasa del bacteriófago T7 confiere procesividad a la ADN polimerasa I. Proc Natl Acad Sci. (1997) 94:479–84. DOI: 10.1073/PNAS.94.2.479
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Xue, S, Seo, K, Yang, M, Cui, C, Yang, M, Xiang, S, et al. La vacuna de subunidad Mycoplasma suis alfa-enolasa induce una respuesta inmune en animales de experimentación. Vacuna. (2021) 9:18–20. doi: 10.3390/vaccines9121506
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Hassan, M, Baig, AA, Attique, SA, Abbas, S, Khan, F, Zahid, S, et al. Acoplamiento molecular de alfa-enolasa para dilucidar los candidatos prometedores contra la infección por Streptococcus pneumoniae. Daru. (2021) 29, 73–84.
22. Ebner, P, y Götz, F. Excreción bacteriana de proteínas citoplasmáticas (ECP): ocurrencia, mecanismo y función. Tendencias Microbiol. (2019) 27:176–87. doi: 10.1016/j.tim.2018.10.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Wang, G, Xia, Y, Cui, J, Gu, Z, Song, Y, Chen, YQ, et al. Los roles de las proteínas moonlighting en bacterias. Curr emite mol biol. (2014) 16:15–22. doi: 10.21775/CIMB.016.015
24. Franco-Serrano, L, Sánchez-Redondo, D, Nájar-García, A, Hernández, S, Amela, I, Perez-Pons, JA, et al. Proteínas de luz lunar patógenas: desde enzimas metabólicas clave ancestrales hasta factores de virulencia. Microorganismos. (2021) 9:1300. doi: 10.3390/microorganismos9061300
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Ko, K-C, Lee, JH, Han, Y, Choi, JH, y Song, JJ. Una nueva enzima celulolítica multifuncional examinada a partir de recursos metagenómicos que representan bacterias ruminales. Biochem Biophys Res Commun. (2013) 441:567–72. doi: 10.1016/j.bbrc.2013.10.120
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Jeffery, CJ. Proteínas intracelulares/de superficie que ayudan en la unión de la microbiota intestinal al huésped. AIMS Microbiol. (2019) 5:77–86. doi: 10.3934/microbiol.2019.1.77
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Commichau, FM, y Stülke, J. Trigger enzymes: coordination of metabolism and virulence gene expression. Microbiol Spectr. (2015) 3:105–127. DOI: 10.1128/microbiolspec. MBP-0010-2014
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Kainulainen, V, y Korhonen, TK. Bailando con otra melodía: proteínas adhesivas de luz lunar en bacterias. Biología (Basilea). (2014) 3:178–204. doi: 10.3390/biology3010178
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Theiler, A. Investigación adicional sobre la anaplasmosis del ganado sudafricano En: A Theiler, editor. Primer informe del director de investigación veterinaria. Departamento de Agricultura de la Unión de Sudáfrica : Sudáfrica (1911). 7–47.
30. Aubry, P, y Geale, DW. Una revisión de la anaplasmosis bovina. Transbound Emerg Dis. (2011) 58:1–30. doi: 10.1111/j.1865-1682.2010.01173.x
31. Quiroz-Castañeda, RE, Amaro Estrada, I, Martínez Ocampo, F, Rodríguez Camarillo, S, Dantán González, E, Cobaxin Cárdenas, M, et al. Proyecto de secuencia genómica de Anaplasma marginale cepa MEX-01-001-01, una cepa mexicana que causa anaplasmosis bovina. Microbiol Resour Announc. (2018) 7:E01101–18. doi: 10.1128/MRA.01101-18
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Martínez-Ocampo, F, Quiroz-Castañeda, RE, Amaro-Estrada, I, Cobaxin Cárdenas, M, Dantán-González, E, and Rodríguez-Camarillo, S. Draft genome sequences of Anaplasma marginale strains MEX-15-099-01 and MEX-31-096-01, two Mexican isolates with different degrees of virulence. Microbiol Resour Announc. (2019) 8. doi: 10.1128/MRA.01184-19
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Martínez-Ocampo, F, Quiroz-Castañeda, RE, Amaro-Estrada, I, Dantán-González, E, De La Torre, JFP, and Rodríguez-Camarillo, S. Whole-genome sequencing of Mexican strains of Anaplasma marginale: an approach to the causal agent of bovine Anaplasmosis. Int J Genómica. (2020) 2020:1–7. DOI: 10.1155/2020/5902029
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Rodríguez-Camarillo, SD, Quiroz-Castañeda, RE, Aguilar-Díaz, H, Vara-Pastrana, JE, Pescador-Pérez, D, Amaro-Estrada, I, et al. Análisis inmunoinformático para identificar proteínas a utilizar como dianas potenciales para el control de la Anaplasmosis bovina. Int J Microbiol. (2020) 2020:8882031–8. doi: 10.1155/2020/8882031
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Gao, X, Zheng, C, and Liu, X. Expression, purification, and biological characterization of Anaplasma phagocytophilum enolase. Tendencias Biosci. (2018) 11:651–7. doi: 10.5582/bst.2017.01195
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Nogueira, SV, Smith, AA, Qin, J-H, y Pal, U. Una enolasa de superficie participa en la interacción Borrelia burgdorferi-plasminógeno y contribuye a la supervivencia del patógeno dentro de las garrapatas que se alimentan. Infectar inmune. (2012) 80:82–90. doi: 10.1128/IAI.05671-11
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Floden, AM, Watt, JA, y Brissette, CA. Borrelia burgdorferi enolasa es una proteína de unión al plasminógeno expuesta a la superficie. PLoS One. (2011) 6:E27502. doi: 10.1371/journal.pone.0027502
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Xie, Q, Xing, H, Wen, X, Liu, B, Wei, Y, Yu, Y, et al. Identificación de las múltiples funciones de la enolasa como receptor de plasminógeno y adhesina en Mycoplasma hyopneumoniae. Microb Pathog. (2023) 174:105934. doi: 10.1016/j.micpath.2022.105934
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Aziz, RK, Bartels, D, Best, AA, DeJongh, M, Disz, T, Edwards, RA, et al. El servidor RAST: anotaciones rápidas utilizando tecnología de subsistemas. BMC Genómica. (2008) 9:75. doi: 10.1186/1471-2164-9-75
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Jumper, J, Evans, R, Pritzel, A, Green, T, Figurnov, M, Ronneberger, O, et al. Predicción de la estructura de proteínas de alta precisión con AlphaFold. Naturaleza. (2021) 596:583–9. DOI: 10.1038/S41586-021-03819-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Chen, C, Zabad, S, Liu, H, Wang, W, and Jeffery, C. MoonProt 2.0: an expansion and update of the moonlighting proteins database. Ácidos nucleicos res. (2018) 46:D640–4. doi: 10.1093/nar/gkx1043
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Sievers, F, Wilm, A, Dineen, D, Gibson, TJ, Karplus, K, Li, W, et al. Generación rápida y escalable de alineamientos de secuencias múltiples de proteínas de alta calidad utilizando Clustal omega. Mol Syst Biol. (2011) 7:539. DOI: 10.1038/MSB.2011.75
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Waterhouse, AM, Procter, JB, Martin, DMA, Clamp, M, y Barton, GJ. Jalview versión 2: un editor de alineación de secuencias múltiples y un banco de trabajo de análisis. Bioinformática. (2009) 25:1189–91. DOI: 10.1093/bioinformática/BTP033
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Tamura, K, Stecher, G, y Kumar, S. MEGA11: molecular evolutionary genetics analysis version 11. Mol Biol Evol. (2021) 38:3022–7. DOI: 10.1093/molbev/msab120
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Sigrist, CJA, de Castro, E, Cerutti, L, Cuche, BA, Hulo, N, Bridge, A, et al. Nuevos y continuos desarrollos en PROSITE. Ácidos nucleicos res. (2013) 41:D344–7. doi: 10.1093/nar/gks1067
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Yu, NY, Wagner, JR, Laird, MR, Melli, G, Rey, S, Lo, R, et al. PSORTb 3.0: predicción mejorada de localización subcelular de proteínas con subcategorías de localización refinadas y capacidades predictivas para todos los procariotas. Bioinformática. (2010) 26:1608–15. DOI: 10.1093/bioinformatics/btq249
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Buchan, DWA, y Jones, DT. El banco de trabajo de análisis de proteínas PSIPRED: 20 años después. Ácidos nucleicos res. (2019) 47:W402–7. doi: 10.1093/nar/gkz297
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Sillitoe, I, Bordin, N, Dawson, N, Waman, VP, Ashford, P, Scholes, HM, et al. CATH: aumento de la cobertura estructural del espacio funcional. Ácidos nucleicos res. (2020) 49:D266–73. doi: 10.1093/nar/gkaa1079
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Waterhouse, A, Bertoni, M, Bienert, S, Studer, G, Tauriello, G, Gumienny, R, et al. SWISS-MODEL: homology modelling of protein structures and complexes. Ácidos nucleicos res. (2018) 46:W296–303. doi: 10.1093/nar/gky427
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Kozakov, D, Hall, DR, Xia, B, Porter, KA, Padhorny, D, Yueh, C, et al. El servidor web ClusPro para el acoplamiento proteína-proteína. Nat Protoc. (2017) 12:255–78. doi: 10.1038/nprot.2016.169
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Desta, IT, Porter, KA, Xia, B, Kozakov, D, and Vajda, S. Performance and its limits in rigid body protein-protein docking. Estructura. (2020) 28:1071–1081.e3. doi: 10.1016/j.str.2020.06.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Goddard, TD, Huang, CC, Meng, EC, Pettersen, EF, Couch, GS, Morris, JH, et al. UCSF ChimeraX: meeting modern challenges in visualization and analysis. Proteína Sci. (2018) 27:14–25. doi: 10.1002/pro.3235
Resumen de PubMed | Texto completo de CrossRef | Google Académico
53. Yan, Y, Tao, H, He, J, y Huang, S-Y. El servidor HDOCK para el acoplamiento proteína-proteína integrado. Nat Protoc. (2020) 15:1829–52. DOI: 10.1038/S41596-020-0312-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
54. Laskowski, RA, Jabłońska, J, Pravda, L, Vařeková, RS, y Thornton, JM. PDBsum: resúmenes estructurales de las entradas del AP. Proteína Sci. (2018) 27:129–34. DOI: 10.1002/PRO.3289
Resumen de PubMed | Texto completo de CrossRef | Google Académico
55. Kang, HJ, Jung, S-K, Kim, SJ, y Chung, SJ. Estructura de la {$α$}-enolasa humana (hENO1), una enzima glucolítica multifuncional. Acta Crystallogr Secta D. (2008) 64:651–7. doi: 10.1107/S0907444908008561
Resumen de PubMed | Texto completo de CrossRef | Google Académico
56. Diaz-Hernandez, A, Gonzalez-Vazquez, MC, Arce-Fonseca, M, Rodríguez-Morales, O, Cedillo-Ramirez, ML, and Carabarin-Lima, A. Consensus enolase of Trypanosoma Cruzi: Evaluation of their immunogenic properties using a bioinformatics approach. Life (Basilea, Suiza). (2022) 12:12050746. doi: 10.3390/life12050746
Resumen de PubMed | Texto completo de CrossRef | Google Académico
57. Ehinger, S, Schubert, W-D, Bergmann, S, Hammerschmidt, S, y Heinz, DW. Plasmin(ogen)-binding α-enolase from Streptococcus pneumoniae: crystal structure and evaluation of plasmin(ogen)-binding sites. J Mol Biol. (2004) 343:997–1005. doi: 10.1016/j.jmb.2004.08.088
58. Schreiner, SA, Sokoli, A, Felder, KM, Wittenbrink, MM, Schwarzenbach, S, Guhl, B, et al. La α-enolasa localizada en superficie de Mycoplasma suis es una proteína de adhesión. Veterinario Microbiol. (2012) 156:88–95. doi: 10.1016/j.vetmic.2011.10.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
59. López-López, MJ, Rodríguez-Luna, IC, Lara-Ramírez, EE, López-Hidalgo, M, Benítez-Cardoza, CG, and Guo, X. Biochemical and biophysical characterization of the enolase from Helicobacter pylori. Biomed Res Int. (2018) 2018:9538193. doi: 10.1155/2018/9538193
Resumen de PubMed | Texto completo de CrossRef | Google Académico
60. Sha, J, Erova, TE, Alyea, RA, Wang, S, Olano, JP, Pancholi, V, et al. La enolasa expresada en superficie contribuye a la patogénesis de la SSU aislada clínica de Aeromonas hydrophila. J Bacteriol. (2009) 191:3095–107. doi: 10.1128/JB.00005-09
Resumen de PubMed | Texto completo de CrossRef | Google Académico
61. Satala, D, Satala, G, Karkowska-Kuleta, J, Bukowski, M, Kluza, A, Rapala-Kozik, M, et al. Conocimientos estructurales sobre las interacciones de la enolasa candidal con la vitronectina humana, fibronectina y plasminógeno. Int J Mol Sci. (2020) 21. DOI: 10.3390/IJMS21217843
Resumen de PubMed | Texto completo de CrossRef | Google Académico
62. Chen, R, Zhao, L, Gan, R, Feng, Z, Cui, C, Xie, X, et al. Evidencia de la evolución rápida y divergente de micoplasmas: análisis estructural y filogenético de enolasas. Frente Mol Biosci. (2022) 8:811106. doi: 10.3389/fmolb.2021.811106
Resumen de PubMed | Texto completo de CrossRef | Google Académico
63. Newman, JA, Hewitt, L, Rodrigues, C, Solovyova, AS, Harwood, CR y Lewis, RJ. Disección de la red de interacciones que vincula el procesamiento del ARN con la glucólisis en el Bacillus subtilis Degradosome. J Mol Biol. (2012) 416:121–36. doi: 10.1016/j.jmb.2011.12.024
Resumen de PubMed | Texto completo de CrossRef | Google Académico
64. Lu, Q, Lu, H, Qi, J, Lu, G y Gao, GF. Un octámero de enolasa de Streptococcus suis. Célula proteica. (2012) 3:769–80. DOI: 10.1007/S13238-012-2040-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
65. Schurig, H, Rutkat, K, Jaenicke, R, and Rachel, R. Octameric enolase from the hyperthermophilic bacterium Thermotoga maritima: purification, characterization, and image processing. Proteína Sci. (1995) 4:228–36. doi: 10.1002/pro.5560040209
Resumen de PubMed | Texto completo de CrossRef | Google Académico
66. Mirasol-Meléndez, E, Lima, E, Lara, V, Brieba, LG, Lara-González, S, y Benitez-Cardoza, CG. Autoasociación de Enolasa de Tricomonas vaginalis. Monómeros, dímeros y octámeros coexisten en solución. ACS. Omega. (2018) 3:17871–80. doi: 10.1021/acsomega.8b02197
67. Schreier, B, y Höcker, B. Engineering the enolase magnesium II binding site: implications for its evolution. Bioquímica. (2010) 49:7582–9. DOI: 10.1021/BI100954F
Resumen de PubMed | Texto completo de CrossRef | Google Académico
68. Joh, D, Wann, ER, Kreikemeyer, B, Speziale, P, and Höök, M. Role of fibronectin-binding MSCRAMMs in bacterial adherence and entry into mammalian cells. Matriz Biol. (1999) 18:211–23. doi: 10.1016/s0945-053x(99)00025-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
69. Narasimhan, S, Coumou, J, Schuijt, TJ, Boder, E, Hovius, JW y Fikrig, E. Una proteína intestinal de garrapata con dominios de fibronectina III ayuda a la congregación de Borrelia burgdorferi al intestino durante la transmisión. PLoS Pathog. (2014) 10:E1004278–8. doi: 10.1371/journal.ppat.1004278
Resumen de PubMed | Texto completo de CrossRef | Google Académico
70. Kurokawa, C, Lynn, GE, Pedra, JHF, Pal, U, Narasimhan, S, and Fikrig, E. Interactions between Borrelia burgdorferi and ticks. Nat Rev Microbiol. (2020) 18:587–600. DOI: 10.1038/S41579-020-0400-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
71. Rikihisa, Y. Mecanismos de infección intracelular obligatoria con Anaplasma phagocytophilum. Clin Microbiol Rev. (2011) 24:469–89. doi: 10.1128/CMR.00064-10
Resumen de PubMed | Texto completo de CrossRef | Google Académico
72. Genetet, S, Desrames, A, Chouali, Y, Ripoche, P, Lopez, C, y Mouro-Chanteloup, I. Stomatin modula la actividad del intercambiador de aniones 1 (AE1, SLC4A1). Sci Rep. (2017) 7:46170. DOI: 10.1038/srep46170
Resumen de PubMed | Texto completo de CrossRef | Google Académico
73. Vanegas, G, Quiñones, W, Carrasco-López, C, Concepción, JL, Albericio, F, and Avilán, L. Enolase as a plasminogen binding protein in Leishmania mexicana. Parasitol Res. (2007) 101:1511–6. DOI: 10.1007/S00436-007-0668-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
74. Pancholi, V, y Fischetti, VA. Α-enolasa, una nueva proteína de unión a plasmina fuerte (Ogen) en la superficie de estreptococos patógenos. J Biol Chem. (1998) 273:14503–15. DOI: 10.1074/JBC.273.23.14503
Resumen de PubMed | Texto completo de CrossRef | Google Académico
75. Ayón-Núñez, DA, Fragoso, G, Espitia, C, García-Varela, M, Soberón, X, Rosas, G, et al. Identificación y caracterización de la enolasa Taenia solium como proteína de unión al plasminógeno. Acta Trop. (2018) 182:69–79. doi: 10.1016/j.actatropica.2018.02.020
Resumen de PubMed | Texto completo de CrossRef | Google Académico
76. Bergmann, S, Schoenen, H, y Hammerschmidt, S. La interacción entre la enolasa bacteriana y el plasminógeno promueve la adherencia de Streptococcus pneumoniae a las células epiteliales y endoteliales. Int J Med Microbiol. (2013) 303:452–62. doi: 10.1016/j.ijmm.2013.06.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
77. Agarwal, S, Kulshreshtha, P, Bambah Mukku, D, y Bhatnagar, R. Alpha-enolase se une al plasminógeno humano en la superficie de Bacillus anthracis. Biochim Biophys Acta. (2008) 1784:986–94. doi: 10.1016/j.bbapap.2008.03.017
Resumen de PubMed | Texto completo de CrossRef | Google Académico
78. Didiasova, M, Schaefer, L, y Wygrecka, M. Cuando el lugar importa: transporte de enolasa-1 a través de compartimentos celulares. Celda frontal. Dev Biol. (2019) 7:61. DOI: 10.3389/fcell.2019.00061
Resumen de PubMed | Texto completo de CrossRef | Google Académico
79. Zhang, C, Gu, Y, Tang, J, Lu, F, Cao, Y, Zhou, H, et al. Producción de plasmodium vivax enolasa en Escherichia coli y sus propiedades protectoras. Hum Vaccin Immunother. (2016) 12:2855–61. doi: 10.1080/21645515.2016.1208328
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: proteínas multifuncionales, enolasa, eritrocitos, garrapatas, matriz extracelular, enfermedades veterinarias
Cita: Quiroz-Castañeda RE, Aguilar-Díaz H y Amaro-Estrada I (2023) Un objetivo de vacuna alternativo para la anaplasmosis bovina basada en enolasa, una proteína de luz lunar. Frente. Vet. Sci. 10:1225873. doi: 10.3389/fvets.2023.1225873
Recibido: 20 de mayo de 2023; Aprobado: 11 de septiembre de 2023;
Publicado: 22 septiembre 2023.
Editado por:
Yadong Zheng, Universidad de Agricultura y Silvicultura de Zhejiang, China
Revisado por:
Panat Anuracpreeda, Universidad de Mahidol, Tailandia
Eliane Esteves, Universidad del Sur de Alabama, Estados Unidos
Copyright © 2023 Quiroz-Castañeda, Aguilar-Díaz y Amaro-Estrada. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Rosa Estela Quiroz-Castañeda, requiroz79@yahoo.com.mx; quiroz.rosa@inifap.gob.mx
†Estos autores han contribuido igualmente a este trabajo
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía
