Identificación de yeguas equinas como hospederos reservorio de especies patógenas de Leptospira
Identificación de yeguas equinas como hospederos reservorio de especies patógenas de Leptospira
 Camila Hamond1,2
Camila Hamond1,2  Emma N. Adam3
Emma N. Adam3  Nathan E. Piedra4
Nathan E. Piedra4  Karen LeCount1,2
Karen LeCount1,2  Tammy Anderson1,2
Tammy Anderson1,2  Ellie J. Putz5
Ellie J. Putz5  Campamento de Patricio1
Campamento de Patricio1  Jessica Hicks1
Jessica Hicks1  Tod Stuber1
Tod Stuber1  Hans van der Linden6
Hans van der Linden6  Darrell O. Bayles5
Darrell O. Bayles5  Jason W. Sahl4
Jason W. Sahl4  Linda K. Schlater1,2
Linda K. Schlater1,2  David M. Wagner4
David M. Wagner4  Jarlath E. Nally2,5*
Jarlath E. Nally2,5*- 1Laboratorios Nacionales de Servicios Veterinarios, Servicio de Inspección de Sanidad Animal y Vegetal, Departamento de Agricultura de los Estados Unidos, Ames, IA, Estados Unidos
- 2Grupo de Trabajo de Leptospira de los Centros Nacionales de Salud Animal, Departamento de Agricultura de EE. UU., Ames, IA, Estados Unidos
- 3Departamento de Ciencias Veterinarias, Universidad de Kentucky, Centro de Investigación Equina Maxwell H. Gluck, Lexington, KY, Estados Unidos
- 4Instituto de Patógenos y Microbioma, Universidad del Norte de Arizona, Flagstaff, Arizona, Estados Unidos
- 5Unidad de Investigación de Enfermedades Bacterianas Infecciosas, Servicio de Investigación Agrícola, Departamento de Agricultura de los Estados Unidos, Ames, IA, Estados Unidos
- 6Departamento de Microbiología Médica y Prevención de Infecciones, Organización Mundial de Sanidad Animal (OMSA) y Centro Nacional Colaborador para la Referencia e Investigación sobre Leptospirosis, Centro Médico de la Universidad de Ámsterdam, Universidad de Ámsterdam, Ámsterdam, Países Bajos
La leptospirosis equina puede provocar aborto, muerte fetal, muerte neonatal, placentitis y uveítis. Los caballos también pueden actuar como reservorios subclínicos de la infección, que se caracterizan por ser portadores asintomáticos que excretan leptospiras de forma persistente y transmiten enfermedades. En este estudio, se utilizó PCR y cultivo para evaluar la excreción urinaria de Leptospira patógena de 37 yeguas asintomáticas. Tres yeguas asintomáticas, designadas como H2, H8 y H9, dieron positivo para lipL32, un gen específico de especies patógenas de Leptospira. Una yegua asintomática, H9, fue positiva para cultivo, y el aislado recuperado se clasificó como L. kirschneri serogrupo Australis serovar Rushan. La captura de ADN y el enriquecimiento del ADN genómico de Leptospira a partir de muestras positivas para PCR y cultivo negativo determinaron que la yegua asintomática H8 también estaba excretando el serogrupo Australis de L. kirschneri, mientras que la yegua asintomática H2 estaba excretando el serogrupo Icterohaemorrhagiae de L. interrogans. Los sueros de todas las yeguas asintomáticas se analizaron mediante la prueba de aglutinación microscópica (MAT) y 35 de 37 (94,6%) fueron seropositivos con títulos que oscilaron entre 1:100 y 1:3200. A diferencia de las yeguas asintomáticas, la yegua H44 presentó aborto espontáneo agudo y un título sérico de MAT de 1:102.400 al serogrupo de L. interrogans Pomona serovar Pomona. La comparación de la cepa H9 del serogrupo Australis de L. kirschneri con la del serogrupo Pomona H44 de L. interrogans en el modelo de hámster de leptospirosis corroboró las diferencias en la virulencia de las cepas. Dado que el lipopolisacárido (LPS) es un antígeno protector en las vacunas bacterinicas, se comparó el LPS de la cepa H9 (asociada con portación subclínica) con la cepa H44 (asociada con aborto espontáneo). Esto reveló diferentes perfiles de LPS e inmunorreactividad con antisueros de referencia. Es fundamental conocer qué especies y serovares de Leptospira están circulando en las poblaciones equinas para diseñar vacunas y pruebas diagnósticas eficaces. Nuestros resultados demuestran que los caballos en los EE. UU. pueden actuar como huéspedes reservorios de leptospirosis y eliminar diversas especies patógenas de Leptospira a través de la orina. Este informe también detalla la detección del serogrupo de L. kirschneri Australis serovar Rushan, una especie y serotipo de Leptospira, no reportada previamente en los EE. UU.
1 Introducción
La leptospirosis es una enfermedad bacteriana, zoonótica y muy desatendida que causa una importante morbilidad y mortalidad en los animales domésticos. Los agentes causales, especies patógenas del género Leptospira, se excretan a través de la orina o se encuentran en el tracto genital del ganado doméstico y pueden sobrevivir en condiciones ambientales húmedas adecuadas para facilitar la transmisión adicional de enfermedades (1, 2). Hasta la fecha, se han descrito 41 especies patógenas de Leptospira que comprenden cientos de serovares (2-6). Se estima que la leptospirosis humana causa 1,03 millones de casos y 58.900 muertes cada año (7).
La leptospirosis equina puede provocar aborto, muerte fetal, muerte neonatal, placentitis y uveítis (1, 8, 9). Los procedimientos diagnósticos para la leptospirosis se dividen en dos grupos: (1) detección de anticuerpos y (2) demostración directa de la presencia de leptospiras. La prueba de aglutinación microscópica (MAT) es el ensayo de diagnóstico de anticuerpos de elección. Un aumento del título de anticuerpos en sueros emparejados agudos y convalecientes concurrentes con signos clínicos de enfermedad aguda es diagnóstico (1). La presencia de anticuerpos en el suero fetal es diagnóstica de infección fetal. La detección directa de leptospiras se facilita mediante ensayos moleculares y/o cultivos. El cultivo es definitivo y proporciona un aislado que puede caracterizarse exhaustivamente mediante secuenciación del genoma y serotipificación. Se cultivó el serogrupo Pomona serovar Pomona tipo kennewicki de Pomona a partir de tejidos y placenta asociados con fetos equinos abortados en los Estados Unidos (10-14).
En Europa, el serogrupo Pomona ha sido identificado como causa de aborto equino, al igual que los serogrupos Australis, Hebdomadis e Icterohaemorrhagiae (15). En Europa, la uveítis recurrente equina se asocia con los serogrupos Grippotyphosa, Australis, Sejroe, Pomona y Javanica (16, 17). Los caballos en Europa también han sido identificados como huéspedes reservorios de Leptospira, portadores asintomáticos que liberan persistentemente Leptospira patógena viva a través de la orina en el medio ambiente para mantener la transmisión de la enfermedad (18). En Irlanda del Norte, se plantea la hipótesis de que los caballos actúan como reservorio del serogrupo Australis serovar Bratislava de L. interrogans, que se ha cultivado a partir de riñones equinos (19). Los estudios de seroprevalencia en caballos de todo el mundo demuestran altos niveles de reactividad con el serogrupo Australis serovar Bratislava, que se utiliza para respaldar aún más el papel de los caballos que sirven como huéspedes reservorio del serogrupo Australis serovar Bratislava de L. interrogans a nivel mundial (20-25).
El ensayo serológico MAT no puede diagnosticar caballos que actúan como huéspedes reservorio de la infección, ya que los estudios de seroprevalencia en poblaciones equinas normales demuestran exposición, no infección clínica activa (1, 21, 26, 27). Se requiere la detección directa de leptospiras mediante cultivo o métodos moleculares para identificar huéspedes reservorio asintomáticos de infección (1, 28, 29). Sin embargo, el cultivo no se realiza de forma rutinaria debido a los exigentes requisitos de crecimiento de Leptospira, la duración y los altos niveles de experiencia requeridos (30, 31). Para diseñar estrategias eficaces de vacunación y diagnóstico de la leptospirosis equina, es esencial identificar a los animales infectados y determinar tanto el genotipo (especie) como el fenotipo (serogrupo/serovar) de las especies de Leptospira asociadas a infecciones equinas. El cultivo exitoso proporciona aislados que pueden caracterizarse de manera integral y usarse en vacunas bacterinológicas. Dado que no hay informes publicados que confirmen que los caballos en los EE. UU. actúen como huéspedes reservorios de leptospirosis, se examinó una población de yeguas pura sangre asintomáticas mediante ensayos moleculares para determinar si alguna estaba excretando Leptospira a través de la orina. Las muestras de yeguas positivas se examinaron más a fondo mediante técnicas de cultivo y enriquecimiento del genoma de Leptospira para identificar la especie y el serotipo de Leptospira asociados con el porte subclínico y para compararlos con una cepa de Leptospira comúnmente asociada con una infección clínica manifiesta en caballos.
2 Materiales y métodos
2.1 Muestras
Se muestrearon un total de 37 yeguas pura sangre asintomáticas de la granja #1 en el centro de Kentucky sin antecedentes de leptospirosis desde diciembre de 2022 hasta febrero de 2023. La sangre se recogió por venopunción yugular en tubos separadores de suero (Vacutainer, BD Diagnostics, Franklin Lakes, NJ, Estados Unidos). La orina para PCR se recolectó por captura libre y se enfrió en hielo. En resumen, se envolvió la cola y se usó agua estéril para eliminar todos los desechos visibles y se limpió a fondo la vulva y el área circundante. La orina intermedia se recogió en recipientes estériles y se envió en bolsas de hielo para su procesamiento por PCR en un plazo de 24 h. Para recolectar orina para cultivo, si una yegua era positiva para PCR, se envolvía la cola y se retiraba del área perineal. Se utilizó agua estéril para eliminar todos los residuos visibles y limpiar a fondo la vulva y el área circundante. La región vulvar se limpió ligeramente con alcohol etílico al 70% y se secó con suaves palmaditas. El proceso se repetía si se producía alguna contaminación del sitio antes de orinar. Se administró un diurético por inyección intravenosa (furosemida inyectable al 5%, 1 mg/kg según el peso estimado de la yegua) y se recogió la orina media de los sucesivos vacíos en recipientes estériles. La orina se pasó a un asistente con guantes limpios y se utilizó una pipeta estéril para transferir 1 mL de orina de cada muestra miccionada a tubos cónicos separados que contenían 9 mL de medio HAN que contenía 5-fluorouracilo (100 μg/μL) (30). La orina restante de esta segunda recolección se transportó en bolsas de hielo para una PCR repetida dentro de las 24 h.®
Se recolectaron muestras de la granja #2 en el centro de Kentucky, una granja con un historial muy reciente de aborto por Leptospira diagnosticado por el Laboratorio de Diagnóstico Veterinario de la Universidad de Kentucky, de una sola yegua que sufrió un aborto en el último trimestre de gestación (16 de enero de 2023). La orina, la membrana corioalantoidea, el amnios y el líquido alantoideo se enviaron inmediatamente en bolsas de hielo para su procesamiento en un plazo de 24 horas. Un segundo conjunto de muestras recogidas 2 días después (18 de enero de 2023), que incluía orina, exudado y un hisopo uterino, se inoculó directamente en el medio HAN y se envió inmediatamente para su procesamiento en un plazo de 24 horas.
2.2 Ensayo de aglutinación microscópica
El MAT se realizó utilizando un panel de 18 antígenos representativos de 15 serogrupos (Tabla Suplementaria S1), como se describió anteriormente (32, 33). Un título se consideró positivo a ≥1:100. También se probaron los seros para determinar su reactividad en el MAT con la cepa H9, un aislado recuperado de una yegua en este estudio.
2.3 Detección molecular de Leptospira patógena
El gen que codifica la principal proteína de la membrana externa, LipL32, discrimina las especies patógenas de Leptospira de las saprófitas. Antes de analizar muestras de orina equina mediante rtPCR lipL32 (34, 35), se realizaron estudios preliminares con orina equina enriquecida para optimizar los parámetros de almacenamiento y preprocesamiento de la orina equina. En resumen, 1 mL de L. borgpetersenii serovar Tarassovi cepa Perepelitsin, a una densidad de 108 leptospiras/mL, se inoculó en 9 mL de orina equina recién recolectada. Una alícuota de 1 mL de esto se diluyó en serie 10 veces para preparar muestras enriquecidas que contenían 107 Leptospira/mL a 100 Leptospira/mL de orina. Las leptospiras móviles intactas se enumeraron mediante microscopía de campo oscuro como se describió anteriormente (36). En total, se procesó 1 ml de cada dilución para la extracción de ADN después del almacenamiento en varias condiciones/puntos de tiempo, que incluyen: (A) ese mismo día, (B) ese mismo día después de la centrifugación a 900 × g durante 10 minutos para eliminar el «lodo», (C) después del almacenamiento durante 24 h en un recipiente de espuma de poliestireno con bolsas de hielo, (D) después del almacenamiento durante 24 h en un recipiente de espuma de poliestireno con bolsas de hielo seguido de centrifugación para eliminar «lodo, E) después del almacenamiento durante 48 h en un recipiente de espuma de poliestireno con bolsas de hielo, F) después del almacenamiento durante 48 h en un recipiente de espuma de poliestireno con bolsas de hielo, seguido de centrifugación para eliminar el «lodo», G) después de la congelación a -20 °C durante 24 h, y (H) después de que las muestras se hayan centrifugado a 900 × g para eliminar el «lodo» y se hayan almacenado a -20 °C durante 24 h. Los tratamientos de almacenamiento de la A a la H se correlacionan con las etiquetas y se muestran en la Figura Suplementaria S1. Posteriormente, la orina se centrifugó a 12.000 × g durante 30 min. Se retiró el sobrenadante y los gránulos se lavaron dos veces resuspendiéndolos en solución salina tamponada con fosfato (PBS) de 1 mL y centrifugando a 12.000 × g durante 15 min, dejando los gránulos finales en ~100 μL. El ADN se extrajo del gránulo urinario utilizando el kit de patógenos de purificación Maxwell RSC Purefood (Promega Corporation, Madison, Wisconsin, Estados Unidos), siguiendo las instrucciones del fabricante, excepto utilizando una incubación de 1 h con 200 μL de tampón de lisis A y un volumen de elución de 100 μL (37). La rtPCR lipL32 se realizó con 10 μL de Perfect taq qPCR ToughMix low ROX™ (Quantabio, Beverly, MA, Estados Unidos), 400 nmol/L de cada cebador, 132,5 nmol/L de sonda, TaqMan™ Reactivos de control positivo interno exógeno: 2 μL de mezcla 10X Exo IPC, 0,4 μL de ADN 50X Exo IPC y 5 μL de extracto de ADN de cultivo o muestra. El ciclo de rtPCR se realizó en un QuantStudio™ 7 (Thermo Fisher Scientific, Estados Unidos) comenzando con una desnaturalización inicial de 3 min a 95 °C para la activación de la polimerasa Taq, seguida de 40 ciclos de desnaturalización a 95 °C durante 15 s y recocido y extensión del cebador a 60 °C durante 1 min. La PCR de las muestras se realizó por triplicado y se consideró positiva por rtPCR lipL32 cuando el duplicado o los triplicados fueron positivos con valores de Ct <40, como se describió anteriormente (34, 35). Se utilizó ADN de la cepa Perepelitsin de L. borgpetersenii serovar Tarassovi para preparar una curva estándar como se describió anteriormente (38).
Se centrifugó una alícuota de orina de 45 mL de cada yegua asintomática a 900 × g durante 10 min a 4 °C, y el sobrenadante se transfirió a un tubo cónico limpio de 50 mL. A continuación, la orina se centrifugó a 12.000 × g durante 30 min a 4 °C. Se retiró el sobrenadante y el pellet se lavó dos veces resuspendiendo en 1 mL de PBS y centrifugando a 12.000 × g durante 10 min a 4°C. Se extrajo el ADN como se describió anteriormente, y se realizó rtPCR con lipL32 como se describió anteriormente (34, 35).
2.4 Cultura
Se inoculó inmediatamente una alícuota de 1 mL de orina miccional recién recolectada en 9 mL de medio HAN (30) y se transportó al Centro Nacional de Enfermedades Animales (NADC), Ames, para el cultivo de Leptospira, como se describió anteriormente (37). Se realizaron dos diluciones seriadas de 10 veces a partir del inóculo inicial (es decir, 500 μL) en 5 mL de medio HAN líquido y se incubaron a 37 °C en CO al 5%2. Las muestras de exudado vulvar y corioalantois se sometieron a vórtice con 9 mL de medio HAN y se realizaron dos diluciones seriadas de 10 veces (es decir, 500 μL) en 5 mL de HAN líquido a 37 °C en CO al 5%2. Los tubos inoculados se examinaron diariamente mediante microscopía de campo oscuro durante las primeras 2 semanas y luego periódicamente durante 6 meses.
2.5 Tipificación molecular de aislados de Leptospira
El ADN se extrajo de un cultivo de 5 mL de las cepas aisladas utilizando el kit de patógenos de purificación Maxwell RSC Purefood (Promega Corporation, Madison, WI), siguiendo las instrucciones del fabricante. La concentración de ADN genómico se determinó mediante Qubit (Qubit dsDNA Broad Range Assay Assay Kit, Qubit 3.0 fluorometer, Invitrogen, Carlsbad, CA, Estados Unidos). Se obtuvo la secuencia del genoma completo (WGS) de Illumina (Nextera XT DNA Library Preparation Kit and the MiSeq Sequencer, 2 × 250 v2 paired-end chemistry, Illumina, San Diego, CA, Estados Unidos), de acuerdo con las instrucciones del fabricante. Las lecturas de Illumina WGS se identificaron taxonómicamente utilizando Kraken 2 versión 2.1 (39). Las lecturas se ensamblaron con SPAdes 3.13 (40) y se verificaron comparando el tamaño esperado del genoma con el tamaño real del ensamblaje y verificando los contigs como Leptospira por BLASTN (41) contra la base de datos de nucleótidos (NT) del NCBI.
2.6 Serotipificación de aislados de Leptospira
El serogrupo de las cepas H9 y H44 se determinó por el método MAT utilizando un panel de antisueros policlonales de referencia de conejo que representan 13 serogrupos (Tabla suplementaria S2). El serovar de las cepas H9 y H44 se determinó mediante la realización de MAT con paneles de anticuerpos monoclonales (mAbs) que aglutinan característicamente serovares de los serogrupos Australis y Pomona, respectivamente, como se describió anteriormente (42).
2.7 Captura y enriquecimiento de ADN
El ADN de dos muestras de orina positivas para cultivo de Leptospira con PCR negativo (designadas como H2 y H8) se sometió a captura y enriquecimiento de ADN panpatogénico de Leptospira, como se describió anteriormente (43). Las dos muestras se procesaron de manera diferente porque el ADN de la muestra H8 estaba muy fragmentado y mostraba bajas concentraciones de ácidos nucleicos (~0,5 ng/μL), mientras que la muestra H2 tenía ADN más intacto (tamaño medio del fragmento >6.000 pb) y concentrado (~4 ng/μL). Para la muestra H2, el ADN se diluyó a ~2 ng/μL en un volumen de 40 μL y se sonicó a un tamaño medio de 228 pb utilizando un sonicador Q800R2 (QSonica, Newtown, CT, Estados Unidos). Para la muestra H8, 40 μL de ADN sin diluir se sometieron a una breve sonicación, y el tamaño medio final del fragmento fue de 103 pb. Las bibliotecas de próxima generación de lectura corta se prepararon por separado utilizando la metodología Agilent Sure-Select. A continuación, las bibliotecas se agruparon en cantidades equimolares y se sometieron a una ronda de captura y enriquecimiento de ADN y luego se secuenciaron en un instrumento Illumina MiSeq utilizando un kit de 600 ciclos MiSeq v3 (lecturas de 2 × 300 pb).
Para estimar el porcentaje de lecturas de Leptospira en las secuencias enriquecidas, las lecturas se compararon con la base de datos estándar de Kraken con Kraken v2.1.2 (39). Las lecturas asignadas como Leptospira se extrajeron y ensamblaron utilizando SPAdes v3.13.0 (40) con la configuración predeterminada; también se generaron ensamblajes para lecturas que se generaron a partir de aislados para las muestras H9 y H44. Los ensamblajes de H2, H8, H9 y H44 se colocaron en un dendrograma de género que contenía 66 genomas de referencia de Leptospira con Mashtree v1.2.046 (44) para confirmar la identificación de la especie. Los números de acceso de GenBank para cada genoma se presentan en figuras.
2.8 Mapeo de lectura y filogenómica
Se identificaron polimorfismos de un solo nucleótido (SNP) entre dos genomas enriquecidos (H2 y H8), dos genomas de aislados cultivados (H9 y H44) y 103 genomas de Leptospira disponibles públicamente (los números de acceso del GenBank para cada genoma se presentan en figuras) mediante la alineación de lecturas con los genomas de referencia Leptospira interrogans serogrupo Canicola serovar Cepa de Canicola LJ178 (GCA_008831445.1), Leptospira interrogans serogrupo Icterohaemorrhagiae serovar Copenhageni cepa Fiocruz_L1-130 (GCA_000007685.1), o Leptospira kirschneri serogrupo Grippotyphosa serovar cepa Grippotyphosa RedPanda1 (GCA_027563495.1) utilizando minimap2 v2.22 (45) y llamando a SNPs desde el archivo BAM con GATK v4.2.2 (46) utilizando una profundidad de cobertura ≥3× y una proporción de lectura de 0,9. Sobre la base de una autoalineación de referencia con NUCmer v3.1 (47), los SNP que se encontraban dentro de regiones duplicadas se filtraron de los análisis posteriores. Todos estos métodos fueron envueltos por NASP v1.2.1 (48). A continuación, se dedujeron filogenias de máxima verosimilitud en las alineaciones SNP concatenadas utilizando IQ-TREE v2.2.0.3 con parámetros predeterminados (49), 1.000 réplicas de bootstrap y el método ModelFinder integrado (50); las filogenias se enraizaron con Leptospira interrogans serovar cepa Canicola LJ178, Leptospira interrogans serovar cepa Copenhageni Fiocruz_L1-130, o Leptospira kirschneri serovar cepa Grippotyphosa RedPanda1, según corresponda. Para determinar la amplitud de la cobertura de los genomas enriquecidos, las lecturas se alinearon con Leptospira interrogans serovar Copenhageni cepa Fiocruz_L1-130 para la muestra H2 y Leptospira kirschneri serovar Grippotyphosa cepa RedPanda1 para la muestra H8 con minimap2, y la profundidad de cobertura por base se calculó con Samtools v1.6 (51).
2.9 Evaluación de la virulencia
Toda la experimentación con animales se llevó a cabo de acuerdo con los protocolos revisados y aprobados por el Comité de Cuidado y Uso de Animales de la NADC y las pautas institucionales del USDA. Las cepas H9 y H44 se propagaron en medio HAN líquido a 29 °C y se evaluó su virulencia mediante inyección intraperitoneal de 10 °C8 leptospiras en 500 μL en grupos (n = 4 por grupo) de hámsters sirios dorados (Mesocricetus auratus). Un grupo control negativo (n = 4) recibió el medio HAN solo. Después de 3 semanas de inoculación, los animales fueron sacrificados y desangrados por punción cardíaca, y se evaluaron frotis de sangre total. Los portaobjetos secos se enviaron al laboratorio de Servicios de Microscopía del Centro Nacional de Enfermedades Animales para la tinción de Giemsa como se informó anteriormente (52), y los portaobjetos teñidos con Giemsa se evaluaron contando los primeros 100 glóbulos blancos para identificar la porción de macrófagos espumosos. Los recuentos diferenciales de células se evaluaron en R (53), donde se utilizó un desafío de ajuste del modelo de regresión lineal como efecto fijo para generar medias de mínimos cuadrados y errores estándar. Se recolectaron tejidos renales y hepáticos para su cultivo. La qPCR para lipL32 se realizó en muestras de hígado y riñón, como se describió anteriormente (54), excepto utilizando ADN de L. interrogans serovar cepa de Canicola Hond Utrecht IV para una curva estándar. Se recolectaron sueros para MAT, que se realizó de acuerdo con las directrices de la Organización Mundial de Sanidad Animal (OMSA) (32), utilizando un panel de 18 antígenos representativos de 15 serogrupos (Tabla suplementaria S1), así como las cepas H9 y H44. Un título se consideró positivo a ≥ 1:100.
2.10 Electroforesis e inmunotransferencias
Además de las cepas H9 y H44, el serogrupo de L. interrogans Icterohemorrhagiae serovar Copenhageni cepa Fiocruz_L1-130 (55, 56) y la cepa PigK151 de Bratislava del serogrupo de L. interrogans Australis serovar (57) se propagaron en medio HAN hasta mediados de la fase logarítmica tardía y se cosecharon por centrifugación (10.000 × g, 4 °C, 30 min), se lavaron dos veces con PBS y se procesaron para electroforesis en gel de poliacrilamida dodecililsulfato de sodio unidimensional (1-D) (SDS-PAGE) en geles de acrilamida al 12% (Bio-Rad, Hercules, CA, Estados Unidos), de acuerdo con las directrices del fabricante. Las proteínas se visualizaron mediante tinción con SYPRO Ruby (Invitrogen), y LPS se visualizó mediante tinción con Pro-Q Emerald 300 (Invitrogen), de acuerdo con las pautas del fabricante. Para el inmunotransferencia, las muestras se transfirieron por transferencia semiseca (Amersham TE77 PWR) a una membrana de transferencia Immobilon-P (Millipore, 220 Bedford, MA, Estados Unidos) y se bloquearon durante la noche a 4 °C con tampón de bloqueo Starting Block (PBS) (Thermo Fisher). Las membranas se incubaron individualmente con antisueros indicados [anti-LipL32, anti-Loa22 o anti-LipL21 (58, 59)] o antisueros de referencia [anti-Pomona, anti-Bratislava, anti-Copenhageni o anti-Ramisi (NVSL, USDA, Ames, IA)] diluidos en tampón de bloqueo seguido de incubación con conjugado de rábano picante-peroxidasa anti-inmunoglobulina G de conejo (Sigma, St. Louis, MO, Estados Unidos) diluido 1:4.000 en tampón de bloqueo. Los conjugados unidos se detectaron utilizando el sustrato Clarity Western ECL (Bio-Rad) y las imágenes se adquirieron utilizando un sistema de imágenes Bio-Rad ChemiDoc MP.
3 Resultados
3.1 Detección de Leptospira patógena
La PCR de muestras de orina equina centrifugadas a 900 × g durante 10 min para eliminar el «lodo» y procesadas el mismo día o dentro de las 24 h posteriores al almacenamiento a 4 °C proporcionó los límites más bajos de detección a 10 Leptospira/mL de orina (Figura Suplementaria S1). El almacenamiento de muestras a 4 °C durante 48 h, la congelación o la no eliminación de «lodos» por centrifugación a 900 × g durante 10 min aumentaron el límite de detección a 102 Leptospira/ml de orina. La congelación de muestras sin eliminar el «lodo» aumentó el límite de detección a 103 Leptospira/ml de orina.
La orina de 37 yeguas asintomáticas, designadas como H1 a H37, se analizó mediante rtPCR para lipL32 utilizando protocolos optimizados como se describió anteriormente. Tres muestras de orina (8%), designadas como H2, H8 y H9 de tres yeguas gestantes diferentes, fueron positivas para PCR con valores de Ct de 37, 38,2 y 36,9, respectivamente (Tabla 1). La repetición de la prueba de orina recolectada 4 días después fue positiva para PCR para las yeguas H8 y H9, pero negativa para la yegua H2. La orina de la yegua H9 fue positiva para Leptospira. Los datos completos de todas las muestras equinas se muestran en la Tabla Suplementaria S3.
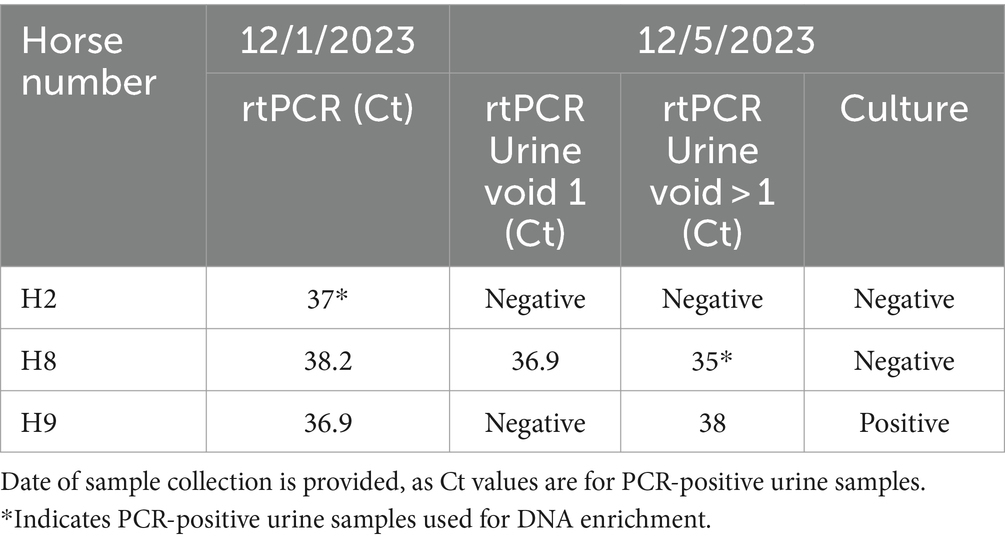 Tabla 1. Detección de Leptospira en orina de yeguas asintomáticas mediante PCR en tiempo real y cultivo.
Tabla 1. Detección de Leptospira en orina de yeguas asintomáticas mediante PCR en tiempo real y cultivo.
La orina, la membrana corioalantoidea, el amnios y el líquido alantoideo recolectados de una yegua (designada como H44) que mostraba enfermedad clínica, y en proceso de aborto en el momento de la recolección de la muestra, fueron positivos por rtPCR para lipL32 con valores de Ct de 26, 31.8, 29.9 y 25.4, respectivamente (Tabla Suplementaria S3). La membrana corioalantoidea fue positiva para Leptospira. Las muestras recolectadas de la yegua H44 después de 2 días después del aborto, incluyendo orina, exudado vulvar e hisopos uterinos, fueron todas positivas por rtPCR para lipL32, pero todos los cultivos estaban muy contaminados (Tabla Suplementaria S3).
3.2 Genotipado y serotipado de aislados de Leptospira
Un aislado de Leptospira cultivado a partir de orina de yegua asintomática H9, designada como cepa H9, fue genotipado como L. kirschneri (Figura 1; Figura complementaria S2). La serotipificación de la cepa H9 con antisueros de referencia identificó el aislado como perteneciente al serogrupo Australis (Tabla Suplementaria S3). La serotipificación adicional con anticuerpos monoclonales para identificar el serovar confirmó que la cepa H9 era más similar a la de serovar Rushan debido a patrones de reactividad similares con la cepa de referencia de Rushan dentro del serogrupo Australis (Figura suplementaria S3). La cepa H9 está clasificada como L. kirschneri serogrupo Australis serovar Rushan.
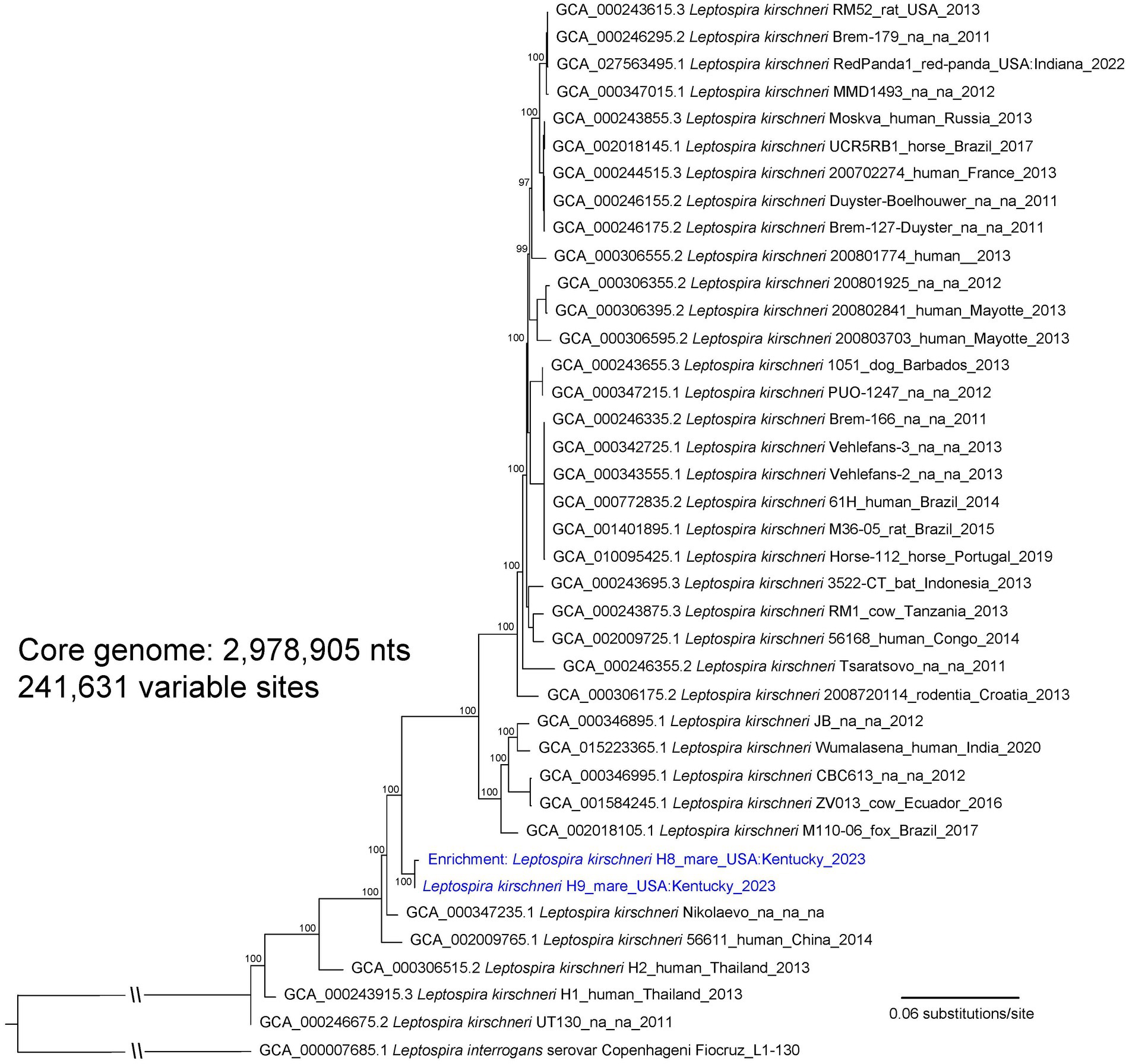 Figura 1. Una filogenia de máxima verosimilitud de 36 genomas de referencia de L. kirschneri junto con ADN genómico de Leptospira obtenido de las yeguas H8 y H9 (resaltado con texto azul). El árbol fue inferido por IQ-TREE a partir de una alineación de SNP concatenada de 241.631 posiciones de un tamaño de genoma central de 2.978.905 nts. La Leptospira de estos dos animales se agrupa en un clado distinto de L. kirschneri. Los números de acceso de GenBank para los genomas de referencia se incluyen en las anotaciones junto con el nombre de la cepa, el hospedador, la ubicación geográfica y el año de recolección cuando están disponibles. La filogenia está enraizada con la cepa Fiocruz_L1-130 de L. interrogans, y los valores de arranque basados en 1.000 réplicas se indican en los nodos principales.
Figura 1. Una filogenia de máxima verosimilitud de 36 genomas de referencia de L. kirschneri junto con ADN genómico de Leptospira obtenido de las yeguas H8 y H9 (resaltado con texto azul). El árbol fue inferido por IQ-TREE a partir de una alineación de SNP concatenada de 241.631 posiciones de un tamaño de genoma central de 2.978.905 nts. La Leptospira de estos dos animales se agrupa en un clado distinto de L. kirschneri. Los números de acceso de GenBank para los genomas de referencia se incluyen en las anotaciones junto con el nombre de la cepa, el hospedador, la ubicación geográfica y el año de recolección cuando están disponibles. La filogenia está enraizada con la cepa Fiocruz_L1-130 de L. interrogans, y los valores de arranque basados en 1.000 réplicas se indican en los nodos principales.
Un aislado de Leptospira cultivado a partir de corioalantois de yegua H44 que presentó aborto fue genotipado como L. interrogans (Figura 2; Figura complementaria S2). La serotipificación de la cepa H44 con antisueros de referencia identificó el aislado como perteneciente al serogrupo Pomona (Tabla suplementaria S3). La serotipificación adicional con anticuerpos monoclonales para identificar el serovar confirmó que la cepa H44 era más similar a la de Serovar Pomona debido a patrones de reactividad similares con la cepa de referencia de Pomona dentro del serogrupo Pomona (Figura suplementaria S4). La cepa H44 está clasificada como L. interrogans serogrupo Pomona serovar Pomona.
 Figura 2. Una filogenia de máxima verosimilitud de 16 L. interrogans hace referencia a genomas que caen dentro del clado «serovar Pomona» junto con la cepa H44 de Leptospira (resaltada con texto azul). El árbol fue inferido por IQ-TREE a partir de una alineación SNP concatenada de 10.732 posiciones de un tamaño de genoma central de 4.165.981 nts. Leptospira de este grupo de animales entre otros cuatro genomas de Pomona obtenidos de dos leones marinos, un zorro y un humano del GenBank de EE. UU. Los números de acceso para los genomas de referencia se incluyen en las anotaciones junto con el nombre de la cepa, el huésped, la ubicación geográfica y el año de recolección cuando estén disponibles. La filogenia está enraizada con L. interrogans serovar Canicola cepa LJ178, y los valores de bootstrap basados en 1.000 réplicas se indican en los nodos principales.
Figura 2. Una filogenia de máxima verosimilitud de 16 L. interrogans hace referencia a genomas que caen dentro del clado «serovar Pomona» junto con la cepa H44 de Leptospira (resaltada con texto azul). El árbol fue inferido por IQ-TREE a partir de una alineación SNP concatenada de 10.732 posiciones de un tamaño de genoma central de 4.165.981 nts. Leptospira de este grupo de animales entre otros cuatro genomas de Pomona obtenidos de dos leones marinos, un zorro y un humano del GenBank de EE. UU. Los números de acceso para los genomas de referencia se incluyen en las anotaciones junto con el nombre de la cepa, el huésped, la ubicación geográfica y el año de recolección cuando estén disponibles. La filogenia está enraizada con L. interrogans serovar Canicola cepa LJ178, y los valores de bootstrap basados en 1.000 réplicas se indican en los nodos principales.
3.3 Captura y enriquecimiento de ADN
Dados los exigentes requisitos de crecimiento de Leptospira patógena y las dificultades inherentes a su cultivo a partir de orina de especies animales grandes, las muestras de orina que eran positivas para la PCR para lipL32 pero negativas para el cultivo para Leptospira se procesaron utilizando un sistema de captura y enriquecimiento de ADN independiente del cultivo para obtener Leptospira información genómica directamente de las muestras de orina H2 y H8. Las muestras enriquecidas H2 y H8 tuvieron proporciones muy altas y moderadas, respectivamente, de lecturas de secuenciación que las asignadas a Leptospira: 98,36% para H2 (1.529.676 de 1.555.190 lecturas) y 60,08% para H8 (562.885 de 936.958 lecturas totales). La muestra H2 reveló una amplitud de cobertura del 99,95% con una profundidad promedio de 77,5× (72-78×) contra el genoma de referencia L. interrogans serogrupo Icterohemorrhagiae serovar Copenhageni cepa Fiocruz_L1-130. La muestra H8 mostró una amplitud y profundidad de cobertura de 92,72% y 32,5× (3-176×) cuando se alineó con el serogrupo de Leptospira kirschneri Grippotyphosa serovar cepa RedPanda1. El dendrograma del genoma completo de Leptospira colocó genomas enriquecidos y aislados para las cepas H8 y H9 en el clado de L. kirschneri, mientras que los genomas enriquecidos y aislados para las cepas H2 y H44 agrupadas con L. interrogans (Figura suplementaria S2). Para obtener una mayor resolución dentro de L. interrogans, se construyó una filogenia del genoma central que contiene el genoma enriquecido H2 y el genoma aislado H44 más 51 genomas diversos de L. interrogans (Figura suplementaria S5). Este análisis ubicó a H44 dentro del clado «serovar Pomona» y a H2 dentro del clado «serovar Copenhageni».
Se construyeron tres filogenias separadas basadas en los genomas principales de: (1) genoma enriquecido H8, genoma aislado H9 y 36 genomas de referencia de L. kirschneri (Figura 1); (2) aislar el genoma H44 y 16 L. interrogans hacen referencia a los genomas que se asignan al clado «serovar Pomona» (Figura 2); y (3) genoma enriquecido H2 y 9 L. interrogans genomas de referencia que caen dentro del clado «serovar Copenhageni» (Figura 3). Los genomas de las cepas H8 y H9 se agruparon en un clado dentro de otros genomas de L. kirschneri, pero distintos de ellos (Figura 1). Genoma H44 agrupado con otros genomas de L. interrogans serovar Pomona a partir de aislados estadounidenses de Leptospira derivados de un zorro, leones marinos y un humano (Figura 2). Finalmente, el genoma enriquecido H2 era más parecido a otros genomas de L. interrogans serovar Copenhageni (Figura suplementaria S5), pero aún así se distinguía al mostrar 376 diferencias de SNP para separarlo del clado «serovar Copenhageni» (Figura 3).
 Figura 3. Una filogenia de máxima verosimilitud de nueve genomas de referencia de L. interrogans que caen dentro del clado «serovar Copenhageni» junto con ADN genómico enriquecido de Leptospira de la yegua H2 (resaltado con texto azul). El árbol fue inferido por IQ-TREE a partir de una alineación SNP concatenada de 37.071 posiciones de un tamaño de genoma central de 4.247.835 nts. La Leptospira de este animal es en su mayoría similar a otros genomas del clado «serovar Copenhageni», pero es distinta. Los números de acceso de GenBank para los genomas de referencia se incluyen en las anotaciones junto con el nombre de la cepa, el hospedador, la ubicación geográfica y el año de recolección cuando están disponibles. La filogenia está enraizada con L. interrogans serovar Canicola cepa LJ178, y los valores de bootstrap basados en 1.000 réplicas se indican en los nodos principales.
Figura 3. Una filogenia de máxima verosimilitud de nueve genomas de referencia de L. interrogans que caen dentro del clado «serovar Copenhageni» junto con ADN genómico enriquecido de Leptospira de la yegua H2 (resaltado con texto azul). El árbol fue inferido por IQ-TREE a partir de una alineación SNP concatenada de 37.071 posiciones de un tamaño de genoma central de 4.247.835 nts. La Leptospira de este animal es en su mayoría similar a otros genomas del clado «serovar Copenhageni», pero es distinta. Los números de acceso de GenBank para los genomas de referencia se incluyen en las anotaciones junto con el nombre de la cepa, el hospedador, la ubicación geográfica y el año de recolección cuando están disponibles. La filogenia está enraizada con L. interrogans serovar Canicola cepa LJ178, y los valores de bootstrap basados en 1.000 réplicas se indican en los nodos principales.
3.4 Ensayo de aglutinación microscópica
Los sueros de 37 yeguas asintomáticas fueron analizados por MAT. De estos, 35 (94,6%) fueron seropositivos (título ≥1:100). Se observaron títulos equivalentes más altos para más de un serogrupo en 10 muestras (Tabla Suplementaria S3). El título de MAT de mayor reacción más frecuente fue con el serogrupo Australis (31,4%), seguido de los serogrupos Pomona (20%), Djasiman (5,7%), Grippotyphosa (5,7%) e Icterohaemorrhagiae (2,8%). Todos los sueros equinos fueron seronegativos cuando fueron analizados por el MAT utilizando la cepa H9.
El suero de la yegua clínicamente infectada (H44) que se presentó con aborto tuvo un título alto de MAT al serogrupo Pomona (1:102,400).
3.5 Evaluación de la virulencia
Los hámsteres inoculados experimentalmente no mostraron ningún signo clínico de infección o pérdida de peso después de la inoculación intraperitoneal con L. kirschneri serogrupo Australis cepa H9. De manera similar, los hámsters inoculados con la cepa H44 del serogrupo Pomona de L. interrogans no mostraron signos clínicos de la enfermedad, excepto un hámster que estaba perdiendo peso y fue sacrificado al día 14 después de la inoculación. Después de 3 semanas de infección, todos los hámsters restantes fueron sacrificados. Todas las muestras renales de cada grupo resultaron positivas mediante qPCR lipL32 (Tabla 2 y Figura 4). Los hígados de hámsters inoculados con la cepa H44 también fueron positivos para la PCR, pero los hígados de hámsteres inoculados con la cepa H9 fueron negativos para la PCR. Los riñones y el hígado de hámsteres inoculados con la cepa H44 fueron positivos para el cultivo, pero solo dos de los cuatro hámsteres inoculados con la cepa H9 fueron positivos para el cultivo renal (Tabla 2). Los hámsters inoculados con L. kirschneri serogrupo Australis cepa H9 tuvieron títulos positivos de MAT frente a la cepa H9 y la cepa de referencia L. interrogans serogrupo Australis serovar Bratislava cepa Jez Bratislava, pero fueron seronegativos cuando se realizaron las pruebas frente a otros antígenos de referencia (Tabla 2). Los hámsters inoculados con L. interrogans serogrupo Pomona cepa H44 tuvieron títulos positivos de MAT frente a la cepa H44 y la cepa de referencia L. interrogans serogrupo Pomona serovar Pomona cepa Pomona, pero fueron seronegativos cuando se analizaron frente a otros antígenos de referencia (Tabla 2). Los diferenciales de hemograma manual determinaron que todos los hámsteres desafiados con H9 y H44 produjeron macrófagos espumosos circulantes (1,50 ± 0,66 y 1,55 ± 0,66, respectivamente, macrófagos espumosos por cada 100 glóbulos blancos evaluados), un marcador asociado con la virulencia y la gravedad de la enfermedad en el modelo de hámster de leptospirosis (52, 60).
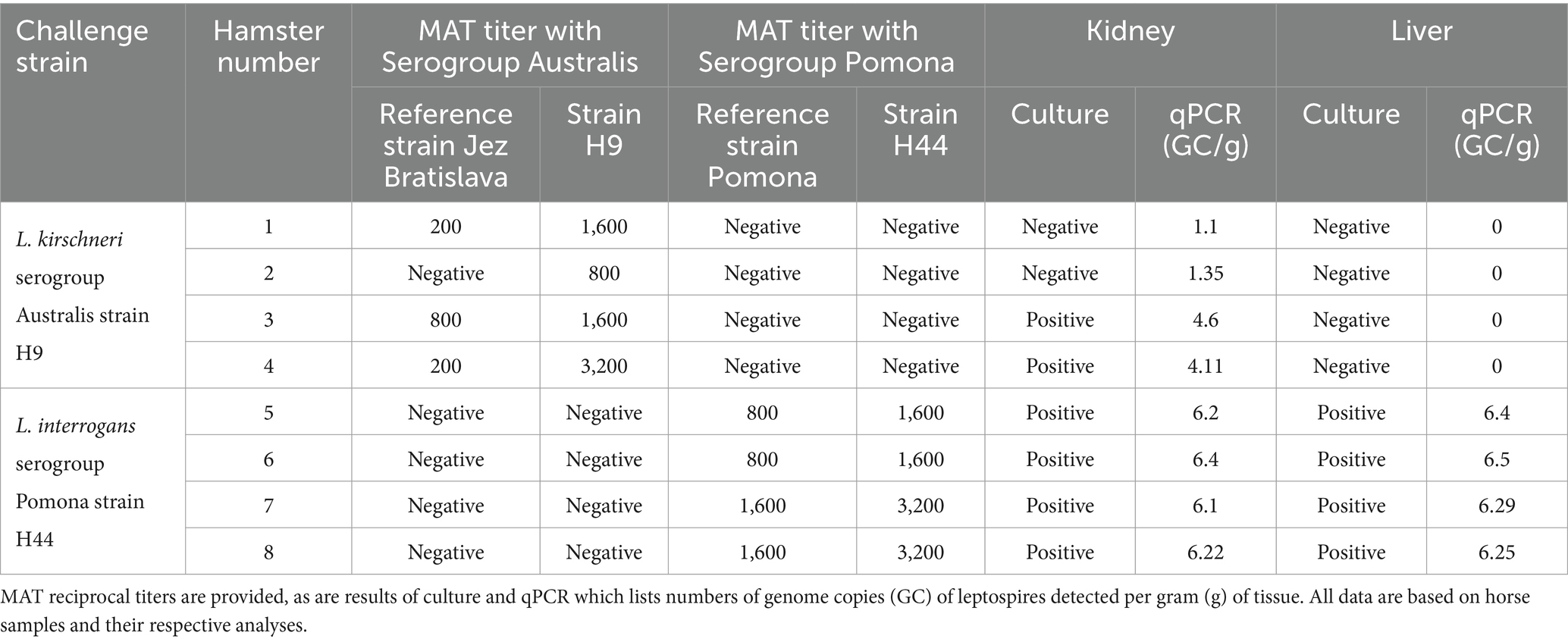 Tabla 2. Desafío de hámster con L. kirschneri serogrupo Australis cepa H9 y L. interrogans serogrupo Pomona cepa H44.
Tabla 2. Desafío de hámster con L. kirschneri serogrupo Australis cepa H9 y L. interrogans serogrupo Pomona cepa H44.
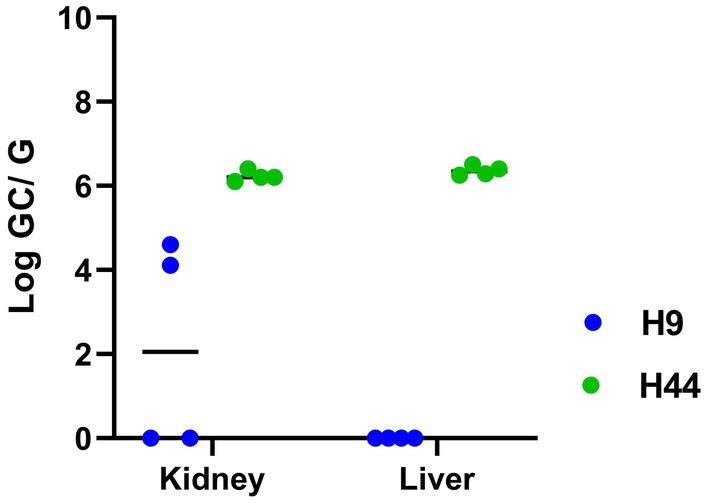 Figura 4. Detección de Leptospira en el riñón y el hígado de hámsters después de la inoculación con L. kirschneri serogrupo Australis cepa H9 (asociada a porte asintomático en una yegua) y L. interrogans serogrupo Pomona cepa H44 (asociada a aborto equino espontáneo) mediante la detección por qPCR de lipL32. El número de copias del genoma (GC) de leptospiras por gramo (G) de tejido se muestra en la Tabla 2.
Figura 4. Detección de Leptospira en el riñón y el hígado de hámsters después de la inoculación con L. kirschneri serogrupo Australis cepa H9 (asociada a porte asintomático en una yegua) y L. interrogans serogrupo Pomona cepa H44 (asociada a aborto equino espontáneo) mediante la detección por qPCR de lipL32. El número de copias del genoma (GC) de leptospiras por gramo (G) de tejido se muestra en la Tabla 2.
3.6 Perfiles proteicos y lipopolisacáridos de Leptospira
Se compararon los perfiles proteicos totales del serogrupo Australis H9 de L. kirschneri y de la cepa H44 del serogrupo Pomona de L. interrogans con los del serogrupo Icterohemorrhagiae de L. interrogans serovar Copenhageni cepa Fiocruz_L1-130 y del serogrupo de L. interrogans Australis serovar Bratislava cepa PigK151 (Figura 5A). Como se esperaba para Leptospira patógena, se detectaron perfiles proteicos similares en diferentes especies y serovares, y se confirmó que todas las cepas expresaban la proteína asociada al patógeno LipL32 y los factores de virulencia conocidos Loa22 y LipL21 (Figuras 5B, C).
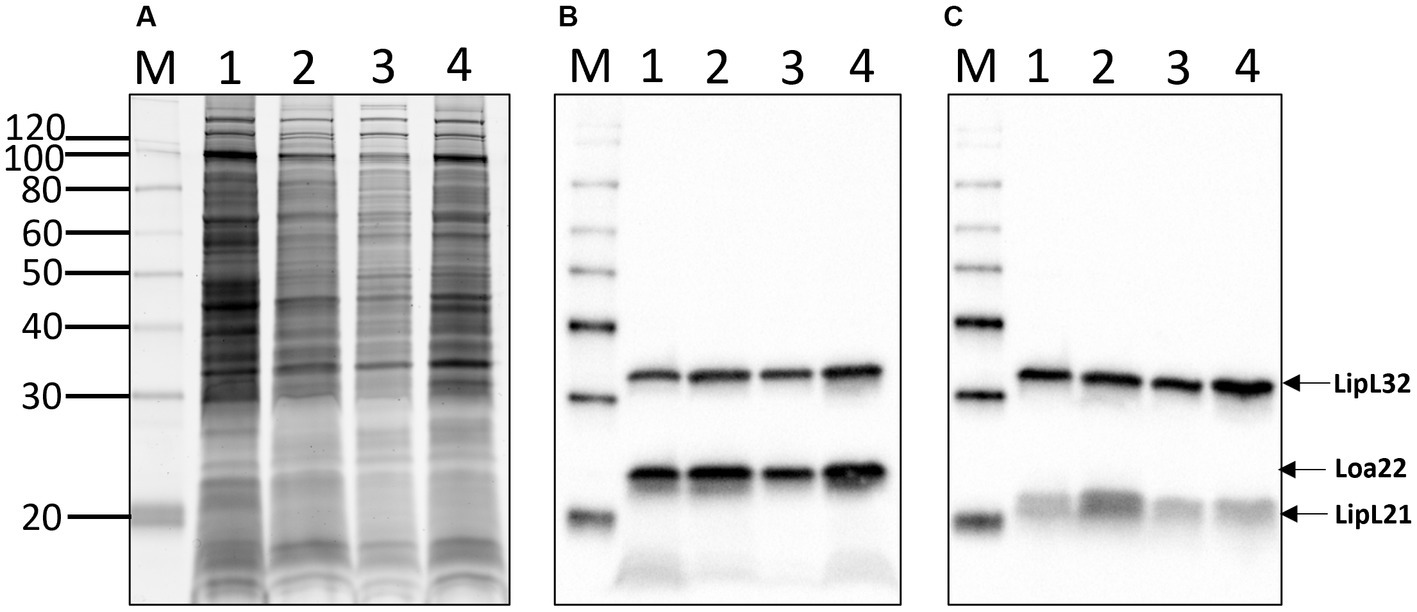 Figura 5. Perfiles proteicos totales (A) de 1: L. kirschneri serogrupo Australis cepa H9, 2: L. interrogans serogrupo Pomona cepa H44, 3: L. interrogans serogrupo Icterohaemorrhagiae serovar Copenhageni cepa Fiocruz_L1-130, y 4: L. interrogans serogrupo Australis serovar Bratislava cepa PigK151 e inmunotransferencia con (B) anti-LipL32/Loa22 o (C) anti-LipL32/LipL21. Se indican marcadores de masa molecular.
Figura 5. Perfiles proteicos totales (A) de 1: L. kirschneri serogrupo Australis cepa H9, 2: L. interrogans serogrupo Pomona cepa H44, 3: L. interrogans serogrupo Icterohaemorrhagiae serovar Copenhageni cepa Fiocruz_L1-130, y 4: L. interrogans serogrupo Australis serovar Bratislava cepa PigK151 e inmunotransferencia con (B) anti-LipL32/Loa22 o (C) anti-LipL32/LipL21. Se indican marcadores de masa molecular.
También se compararon los perfiles de lipopolisacáridos totales (LPS) de cada cepa (Figura 6A). Los resultados confirman el perfil inusual y atípico de LPS de Leptospira patógena en comparación con el de E. coli y confirman diferentes perfiles de LPS entre serovares de Leptospira de diferentes serogrupos. Las diferencias antigénicas de LPS entre serogrupos y diferentes serovares dentro del mismo serogrupo se confirmaron mediante inmunotransferencia con antisueros de referencia específicos para (1) el serogrupo Australis serovar Ramisi, (2) el serogrupo Pomona serovar Pomona, (3) el serogrupo Icterohemorrhagiae serovar Copenhageni y (4) el serogrupo Australis serovar Bratislava (Figuras 6B-E).
 Figura 6. Perfiles de lipopolisacáridos de leptospiras: perfiles totales de LPS (A) de 1: L. kirschneri serogrupo Australis cepa H9, 2: L. interrogans serogrupo Pomona cepa H44, 3: L. interrogans serogrupo Icterohaemorrhagiae serovar Copenhageni cepa Fiocruz_L1-130, 4: L. interrogans serogrupo Australis serovar Bratislava cepa PigK151, y control positivo de LPS (+) que comprende 10 μg de LPS de E. coli serotipo 055:B5. Inmunotransferencia con antisueros de referencia contra (B) el serogrupo Australis serovar Ramisi, (C) el serogrupo Pomona serovar Pomona, (D) el serogrupo Icterohaemorrhagiae serovar Copenhageni y (E) el serogrupo Australis serovar Bratislava. Se indican marcadores de masa molecular.
Figura 6. Perfiles de lipopolisacáridos de leptospiras: perfiles totales de LPS (A) de 1: L. kirschneri serogrupo Australis cepa H9, 2: L. interrogans serogrupo Pomona cepa H44, 3: L. interrogans serogrupo Icterohaemorrhagiae serovar Copenhageni cepa Fiocruz_L1-130, 4: L. interrogans serogrupo Australis serovar Bratislava cepa PigK151, y control positivo de LPS (+) que comprende 10 μg de LPS de E. coli serotipo 055:B5. Inmunotransferencia con antisueros de referencia contra (B) el serogrupo Australis serovar Ramisi, (C) el serogrupo Pomona serovar Pomona, (D) el serogrupo Icterohaemorrhagiae serovar Copenhageni y (E) el serogrupo Australis serovar Bratislava. Se indican marcadores de masa molecular.
3.7 Disponibilidad de los datos
Los datos de secuenciación y los números de acceso están disponibles en el NCBI bajo el ID de BioProject PRJNA994138.
4 Discusión
El objetivo principal de este estudio fue determinar si las yeguas asintomáticas pueden actuar como huéspedes reservorios de la infección y, de ser así, qué especies y serovares de Leptospira están involucrados. Nuestro enfoque se limitó a la buena voluntad y cooperación de la gerencia y el personal de una sola granja comercial de cría de pura sangre que facilitó la recolección de muestras de un número limitado de yeguas disponibles, con un historial de vacunación incompleto, en una sola granja, en un período de tiempo relativamente corto. Nuestros resultados demuestran que las yeguas asintomáticas pueden eliminar diferentes especies y serovares de Leptospira a través de la orina. Inesperadamente, un solo aislado recuperado de una yegua asintomática se clasifica como perteneciente al serogrupo Australis serovar Rushan de L. kirschneri, que no se ha reportado previamente en los EE. UU. ni en caballos. Estos hallazgos justifican estudios más exhaustivos en caballos en todo Estados Unidos para determinar con precisión la prevalencia de la leptospirosis equina, y las especies y serovares de Leptospira se asocian con la muda asintomática.
La literatura está repleta de evidencia serológica de que los caballos están expuestos a serovares dentro del serogrupo Australis y junto con el aislamiento del serogrupo Australis serovar Bratislava de L. interrogans de caballos en Irlanda y Portugal (18, 19); se plantea la hipótesis de que los caballos actúan como reservorios de Bratislava. Sin embargo, la serología por MAT sola no detecta ni refleja la diseminación de Leptospira en los huéspedes reservorios de leptospirosis (1). Por lo tanto, las muestras de orina de yeguas asintomáticas se analizaron inicialmente mediante rtPCR dirigida a lipL32, un gen presente en especies patógenas de Leptospira que discrimina de las especies saprófitas. La detección óptima de lipL32 en orina equina requiere que se procesen para PCR dentro de las 24 horas y se envíen en bolsas de hielo (Figura suplementaria S1). Se identificaron tres yeguas positivas para PCR (designadas como H2, H8 y H9) y se seleccionaron para cultivo y repetición de rtPCR, de las cuales dos (H8 y H9) siguieron siendo positivas para PCR y una (H9) fue positiva para cultivo, lo que validó los resultados iniciales. El excreción intermitente de leptospiras en la orina de los huéspedes reservorio no es infrecuente, lo que puede explicar la rtPCR negativa en la yegua H2 (38, 61). Debido a que el veterinario clínico de la granja inició la terapia antimicrobiana después de que se dispusiera de resultados positivos para la PCR, no se realizaron rtPCR ni cultivos adicionales. El cultivo de Leptospira a partir de orina de animales domésticos grandes es notoriamente difícil (1), por lo que se utilizó un enfoque independiente del cultivo para caracterizar el genoma de Leptospira en muestras positivas para PCR para caracterizar genómicamente las especies asociadas con la infección subclínica en yeguas H2 y H8 (43). Se determinó que la yegua H2 estaba desprendiendo L. interrogans, cuyo genoma se alineaba más estrechamente con el serogrupo Icterohemorrhagiae serovar Copenhageni, pero aún bastante distinto, con 376 SNP que lo separan de otros genomas en este clado (Figura 3). El serogrupo Icterohemorrhagiae es una de las principales causas de leptospirosis aguda en pacientes humanos, y su huésped reservorio clásico es la rata, no los animales domésticos. Sin embargo, este serogrupo se ha aislado de riñón equino y fetos abortados anteriormente en Europa (15, 19) y más recientemente en orina de una vaca lechera estadounidense en el momento del sacrificio (Hamond y Nally, datos no publicados). Se determinó que la yegua H8 estaba excretando L. kirschneri, cuyo genoma se alineaba más estrechamente con el del aislado cultivado de L. kirschneri serogrupo Australis serovar Rushan cepa H9 (Figura 1). Cabe destacar que los genomas de H8 y H9 diferían en solo 16 SNP cuando se comparaban 4.565.109 posiciones de nucleótidos compartidos.
El serogrupo Australis contiene al menos 14 serovares, incluidos los serovares Australis y Bratislava, que se utilizan comúnmente como serovares representativos en los paneles de antígenos MAT y ambos pertenecen a L. interrogans (Tabla suplementaria S1). Los paneles de anticuerpos monoclonales de referencia tipificaron la cepa H9 como serovar Rushan. La cepa de referencia para el serovar Rushan se aisló originalmente de un sapo (Bombina orientalis) en China y se clasificó como L. noguchii (62, 63). La identidad promedio de nucleótidos (ANI) la clasificó posteriormente como L. alstonii (64). Solo un serovar dentro del serogrupo Australis se asigna a L. kirschneri, la cepa de referencia para el serovar Ramisi, que se aisló originalmente en un paciente humano en Kenia, alrededor de 1971 (65, 66). La eficacia de las vacunas contra bacterinas en animales depende de la inclusión de serovares relevantes asociados con la infección animal. El estado de Serovar se define utilizando antisueros aglutinantes de referencia o anticuerpos monoclonales, que se dirigen a LPS, un antígeno protector y específico de serovar. El LPS de Leptospira tiene una estructura atípica de la de otras bacterias gramnegativas y es mucho menos tóxico, probablemente debido a los lípidos A modificados (67). Se comparó el perfil LPS de la cepa H9 del serogrupo Australis de L. kirschneri (una cepa asociada aquí con portación subclínica) con el del serogrupo H44 de L. interrogans Pomona (cepa asociada con el aborto equino espontáneo), el serogrupo de L. interrogans Icterohemorrhagiae serovar, la cepa Copenhageni Fiocruz_L1-130 (una cepa estrechamente relacionada con el genoma enriquecido H2) y L. interrogans serogrupo Australis serovar Bratislava cepa PigK151 [una cepa utilizada recientemente en un estudio de provocación equina (68)] para resaltar las diferencias en los perfiles de LPS (Figura 6A). Y lo que es más importante, el uso de antisueros policlonales de referencia en inmunoblots confirma las diferencias antigénicas en la expresión del antígeno protector LPS (Figuras 6B-E) entre los serovares, lo que demuestra su estatus individual y la necesidad de su inclusión en las bacterinas como antígenos protectores o en los paneles MAT como antígenos diagnósticos. La reactividad de LPS derivada de la cepa H9 del serogrupo Australis de L. kirschneri con antisuero de referencia específico para el serogrupo Australis serovar Ramisi de L. kirschneri confirma suficientes diferencias antigénicas con el serogrupo Australis serovar Bratislava de L. interrogans, demostrando una reactividad cruzada limitada en la vacuna bacterina o en los antígenos de diagnóstico MAT (Figura 6E), como lo confirman los títulos de MAT en hámsteres infectados experimentalmente (Tabla 2). A diferencia del LPS, las proteínas están altamente conservadas entre las especies patógenas y los serovares (Figura 5A). La expresión de los factores de virulencia conocidos Loa22 y LipL21 se confirmó en todas las cepas (Figuras 5B, C) (59, 69-71).
Se plantea la hipótesis de que los caballos actúan como reservorio de serovar Bratislava en los EE. UU., según múltiples estudios de seroprevalencia (9). Sin embargo, en un panel de TMA se utiliza un serovar como serovar representativo del serogrupo, de modo que una reacción positiva es indicativa de exposición a un serogrupo, no específicamente al serovar utilizado en el panel (2). Todavía no se han aportado pruebas directas de que el serovar Bratislava infecte de forma persistente a los caballos estadounidenses (72). Estudios recientes han intentado forzar esta hipótesis mediante la infección experimental de caballos con un aislado de serovar Bratislava que se recuperó de un cerdo hace unas décadas (57), pero no se informó evidencia de infección establecida (68). En los EE.UU., el serogrupo Australis serovar Bratislava solo se ha aislado de los riñones o del tracto genital de los cerdos (73, 74) y se ha informado universalmente que es una causa importante de pérdida reproductiva porcina (1). Las vacas actúan como reservorios del serogrupo Sejroe serovar Hardjo, que también puede colonizar los riñones y/o el tracto genital. La evidencia de exposición bovina al serogrupo Sejroe se asocia con un aumento en el tiempo medio desde el parto hasta la concepción, así como con el doble de probabilidades de no concebir (75). En Brasil, Leptospira se ha detectado por PCR en biopsias endometriales o fluidos vaginales de yeguas con problemas reproductivos que incluyen muerte embrionaria temprana y endometritis (76); por lo tanto, sería beneficioso examinar los caballos estadounidenses para detectar la colonización del tracto genital por Leptospira, ya sea por L. kirschneri serogrupo Australis u otras cepas.
La leptospirosis animal se presenta con una serie de signos clínicos que dependen, en parte, tanto del serovar de Leptospira como de especies animales hospedadoras específicas (77). Los mecanismos patogénicos de infección no se comprenden claramente ni la razón por la que una especie huésped animal actúa como huésped reservorio para un serovar, pero como huésped incidental para otros. En este estudio, más del 90% de los caballos fueron seropositivos. Sin embargo, no se detectó reactividad cuando se incluyó la cepa H9 del serogrupo Australis serovar Rushan de L. kirschneri en el panel de antígenos MAT, a pesar de que aquí se reportan dos caballos (H8 y H9) como excrementos activos. Del mismo modo, el H2 de caballo no tuvo título reactivo contra el serogrupo Icterohemorragiae, aunque se detectó en orina. La identificación de hospederos reservorios seronegativos no es infrecuente cuando los sueros se analizan a 1:100 y, por lo tanto, los estudios con animales subclínicos pueden analizar sueros a diluciones iniciales inferiores a 1:100 (28, 78, 79). La infección incidental se caracteriza por la diseminación aguda de Leptospira con excreción limitada en comparación con los huéspedes reservorio, que tienen un porte subclínico y una excreción prolongada (80). La cepa H9 del serogrupo Australis serovar Rushan de L. kirschneri no se detectó en el hígado después de la infección experimental en hámsteres, pero se detectó en niveles bajos en el riñón, mientras que la cepa H44 de Pomona serovar del serogrupo de L. interrogans se detectó en niveles más altos en todos los hígados y riñones (Tabla 2). Serovar Pomona se asocia con enfermedad aguda en muchos huéspedes animales, y la cepa H44 está estrechamente relacionada con otras cepas de Pomona aisladas de múltiples animales distribuidos geográficamente en los EE. UU. (81). Un total de 195 SNPs diferencian el genoma de la cepa H44 de la cepa aislada de león marino de California CSL4002 cuando se comparan 4.524.948 posiciones de nucleótidos compartidos (Figura 2). Se ha informado que genotipos únicos de Pomona están asociados con el aborto equino (11), y el advenimiento de la secuenciación del genoma de alta resolución brinda oportunidades para explorar más a fondo estas asociaciones. Del mismo modo, los análisis genómicos comparativos de las cepas asociadas con la enfermedad aguda en comparación con las cepas asociadas con el porte subclínico pueden proporcionar información sobre el mecanismo patogénico de la infección y la adaptación del huésped.
Estudios previos sobre la leptospirosis equina se han basado en los medios EMJH para aislar Leptospira de caballos abortados y nacidos muertos (8, 82). Sin embargo, las Leptospira patógenas comprenden un género diverso de bacterias altamente exigentes que requieren formulaciones de medios alternativos, ya que muchos serovares no crecerán en medios comerciales de EMJH (2, 30). La dependencia de EMJH para cultivar leptospiras de mamíferos hospederos es selectiva solo para aquellas leptospiras patógenas capaces de sobrevivir o adaptarse a los medios de crecimiento y las condiciones utilizadas, por lo que se recomiendan formulaciones de medios alternativos como HAN. El serogrupo Australis serovar Bratislava se aisló de cerdas estadounidenses utilizando medio T80/40/LH, no EMJH (74, 83). Históricamente, el aislamiento de Leptospira patógena de huéspedes mamíferos se ha limitado a cultivos incubados a 28-30 °C (31). Sin embargo, el uso de formulaciones de medios más nuevos, como HAN, permite el aislamiento de huéspedes mamíferos a 28-30 °C y 37 °C, una temperatura que emula más de cerca la encontrada durante la infección del huésped (30). La temperatura es una señal ambiental importante utilizada por Leptospira para regular la expresión génica y proteica y los antígenos expresados por Leptospira que reaccionan con sueros equinos (84-86). La leptospira patógena regula la expresión génica y proteica durante la infección aguda y crónica (59, 69, 87, 88), por lo que se plantea la hipótesis de que las bacterinas producidas a temperaturas más similares a las de un huésped expresarán niveles más altos de antígenos proteicos cruzados y factores de virulencia (89).
La leptospirosis es una enfermedad desatendida a nivel mundial y paradigma de una sola salud (90). Se siguen identificando nuevas especies y serovares (91, 92), lo que pone de manifiesto su compleja epidemiología y la necesidad de determinar las especies y serovares que circulan dentro de las poblaciones de animales domésticos. El uso de nuevas formulaciones de medios para Leptospira ha facilitado la caracterización de una amplia gama de nuevas especies y serovares que circulan en el medio ambiente (4) y en el ganado estadounidense (37, 38). De igual forma, se reporta la identificación de una nueva especie y serovar asociada a porte asintomático y excreción en orina de caballos, L. kirschneri serogrupo Australis serovar Rushan. En ausencia de cultivo, la PCR en tiempo real de la orina identifica a los portadores asintomáticos, mientras que la captura y el enriquecimiento del ADN proporcionan información genómica completa sobre Leptospira. Para la PCR en tiempo real, la orina equina debe enfriarse tan pronto como se recoja y procesarse dentro de las 24 horas para una sensibilidad óptima. La identificación y caracterización de las cepas asociadas a la leptospirosis equina es esencial para diseñar estrategias eficaces de diagnóstico, vacunación, prevención y tratamiento.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/genbank/, BioProject ID PRJNA994138.
Declaración ética
Los estudios en animales fueron aprobados por el Comité de Cuidado y Uso de Animales de la NADC y las directrices institucionales del USDA. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. No se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio debido a que el agente de los animales proporcionó el consentimiento verbal.
Contribuciones de los autores
CH: Curación de datos, Análisis formal, Investigación, Metodología, Supervisión, Validación, Visualización, Redacción – borrador original, Redacción – revisión y edición. EA: Conceptualización, Curación de datos, Adquisición de fondos, Investigación, Metodología, Administración de proyectos, Recursos, Redacción, revisión y edición. NS: Curación de datos, Análisis formal, Investigación, Metodología, Software, Visualización, Escritura – revisión y edición. KL: Curación de datos, investigación, metodología, redacción, revisión y edición. TA: Análisis formal, Investigación, Metodología, Redacción – revisión y edición. EP: Curación de datos, Análisis formal, Investigación, Metodología, Redacción – revisión y edición. PC: Curación de datos, Análisis formal, Investigación, Metodología, Redacción – revisión y edición. JH: Curación de datos, Análisis formal, Investigación, Metodología, Redacción – revisión y edición. TS: Curación de datos, Análisis formal, Investigación, Metodología, Redacción – revisión y edición. NS: Curación de datos, Análisis formal, Investigación, Metodología, Redacción, revisión y edición. DB: Curación de datos, Análisis formal, Investigación, Metodología, Redacción – revisión y edición. JS: Curación de datos, Análisis formal, Investigación, Metodología, Supervisión, Redacción, revisión y edición. LS: Adquisición de fondos, Recursos, Redacción – revisión y edición. DW: Curación de datos, Análisis formal, Investigación, Metodología, Recursos, Supervisión, Redacción, revisión y edición. JN: Conceptualización, Curación de datos, Análisis formal, Obtención de fondos, Investigación, Metodología, Administración de proyectos, Recursos, Supervisión, Validación, Visualización, Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Esta investigación fue apoyada por el Departamento de Agricultura de los Estados Unidos (USDA, por sus siglas en inglés) y, en parte, por un nombramiento para el Programa de Participación en la Investigación del Servicio de Inspección de Sanidad Agropecuaria (APHIS, por sus siglas en inglés) administrado por el Instituto Oak Ridge para la Ciencia y la Educación (ORISE, por sus siglas en inglés) a través de un acuerdo interinstitucional entre el Departamento de Energía de los Estados Unidos (DOE, por sus siglas en inglés) y el Departamento de Agricultura de los Estados Unidos (USDA, por sus siglas en inglés). ORISE es administrada por ORAU bajo el número de contrato del DOE DE-SC0014664. El Departamento de Agricultura de los Estados Unidos (USDA, por sus siglas en inglés) es un proveedor y empleador que ofrece igualdad de oportunidades. La mención de nombres comerciales o productos comerciales en esta publicación es únicamente con el propósito de proporcionar información específica y no implica una recomendación o respaldo por parte del Departamento de Agricultura de los Estados Unidos.
Reconocimientos
Los autores agradecen al médico de la granja, a la gerencia y al personal por su compromiso, la ayuda experta y el tiempo requerido para proporcionar muestras de diagnóstico para estos estudios.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
El/los autor/es declararon, en el momento de la presentación, ser miembro del consejo editorial de Frontiers. Esto no tuvo ningún impacto en el proceso de revisión por pares ni en la decisión final.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1346713/full#supplementary-material
FIGURA COMPLEMENTARIA S1 | Límites inferiores de detección de Leptospira por qPCR lipL32 en orina equina procesada fresca el día de la recolección (A,B), después del almacenamiento durante 24 h en bolsas de hielo (C,D), después del almacenamiento durante 48 h en bolsas de hielo (E,F) o después de ser congelada a -20 hoC (G,H). Las muestras B, D, F, H se centrifugaron a 900 × g durante 10 min para eliminar el «lodo» antes del procesamiento.
FIGURA COMPLEMENTARIA S2 | Dendrograma del genoma completo basado en distancias MASH por pares de genomas de Leptospira, que muestra las secuencias de yeguas H2, H8, H9 y H44 dentro del clado P1 de Leptospira (resaltado en azul) (3). Los genomas enriquecidos y aislados de las cepas H8 y H9 se colocaron en el clado de L. kirschneri, mientras que las cepas H2 y H44 se agruparon con L. interrogans. El dendrograma incluye genomas de 66 cepas de Leptospira que representan todas las especies conocidas de los clados P1, P2, S1 y S2.
FIGURA COMPLEMENTARIA S3 | Serotipificación de la cepa H9 con anticuerpos monoclonales (mAb) que aglutinan característicamente serovares del serogrupo Australis. Se proporcionan títulos de reactividad para cada AcM para L. Cepa H9 del serogrupo Australis de Kirschneri y cepa de referencia L. noguchii serogrupo Australis serovar Rushan cepa 507. Los títulos recíprocos se muestran en el eje y; El número de anticuerpos monoclonales se muestra en el eje x.
FIGURA COMPLEMENTARIA S4 | Serotipificación de la cepa H44 con anticuerpos monoclonales (mAb) que aglutinan característicamente serovares del serogrupo Pomona. Se proporcionan títulos de reactividad para cada AcM para L. interrogans serogrupo Pomona cepa H44 y cepa de referencia L. interrogans serogrupo Pomona serovar Pomona cepa Pomona. Los títulos recíprocos se muestran en el eje y; El número de anticuerpos monoclonales se muestra en el eje x.
FIGURA COMPLEMENTARIA S5 | Una filogenia de máxima verosimilitud de 51 genomas de referencia de L. interrogans junto con ADN genómico de Leptospira de las cepas H2 y H44 (resaltado con texto azul). El árbol fue inferido por IQ-TREE a partir de una alineación SNP concatenada de 248.698 posiciones de un tamaño de genoma central de 2.688.796 nts. Leptospira de la cepa H2 cae entre otros genomas de L. interrogans serovar Copenhageni, mientras que la cepa H44 se agrupa entre otros L. interrogans serovar genomas de Pomona. Los números de acceso al GenBank para los genomas de referencia se incluyen en las anotaciones. La filogenia está enraizada con la cepa RedPanda1 de L. kirschneri.
Referencias
1. Ellis, WA. Leptospirosis animal. Curr Top Microbiol Immunol. (2015) 387:99–137. doi: 10.1007/978-3-662-45059-8_6
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Faine, S, Adler, B, Bolin, C, y Perolat, P. Leptospira y leptospirosis. 2ª ed. Melbourne, Australia: MediSci (1999).
3. Vincent, AT, Schiettekatte, O, Goarant, C, Neela, VK, Bernet, E, Thibeaux, R, et al. Revisando la taxonomía y evolución de la patogenicidad del género leptospira a través del prisma de la genómica. PLoS negl trop dis. (2019) 13:e0007270. doi: 10.1371/journal.pntd.0007270
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Fernandes, LGV, Stone, NE, Roe, CC, Goris, MGA, van der Linden, H, Sahl, JW, et al. Nov., una especie patógena del género leptospira aislada del suelo en Puerto Rico. Int J Syst Evol Microbiol. (2022) 72:5560. doi: 10.1099/ijsem.0.005560
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Casanovas-Massana, A, Vincent, AT, Bourhy, P, Neela, VK, Veyrier, FJ, Picardeau, M, et al. Leptospira dzianensis y Leptospira putramalaysiae son sinónimos heterotípicos posteriores de Leptospira yasudae y Leptospira stimsonii. Int J Syst Evol Microbiol. (2021) 71:4713. doi: 10.1099/ijsem.0.004713
6. Korba, AA, Lounici, H, Kainiu, M, Vincent, AT, Mariet, JF, Veyrier, FJ, et al. Leptospira abararensis sp. nov. y Leptospira chreensis sp. nov., cuatro nuevas especies aisladas de fuentes de agua en Argelia. Int J Syst Evol Microbiol. (2021) 71:5148. doi: 10.1099/ijsem.0.005148
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Costa, F, Hagan, JE, Calcagno, J, Kane, M, Torgerson, P, Martinez-Silveira, MS, et al. Morbilidad y mortalidad global de la leptospirosis: una revisión sistemática. PLoS negl trop dis. (2015) 9:E0003898. doi: 10.1371/journal.pntd.0003898
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Donahue, JM, Smith, BJ, Redmon, KJ, y Donahue, JK. Diagnóstico y prevalencia de la infección por Leptospira en caballos abortados y nacidos muertos. J Vet Diagn Invest. (1991) 3:148–51. doi: 10.1177/104063879100300208
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Buzos, TJ, Chang, YF, Irby, NL, Smith, JL y Carter, CN. Leptospirosis: una importante enfermedad infecciosa en caballos norteamericanos. Veterinario Equino J. (2019) 51:287–92. doi: 10.1111/evj.13069
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Polle, F, Storey, E, Eades, S, Alt, D, Hornsby, R, Zuerner, R, et al. Papel de las infecciones intraoculares por Leptospira en la patogénesis de la uveítis recurrente equina en el sur de los Estados Unidos. J Veterinario Equino. (2014) 34:1300–6. doi: 10.1016/j.jevs.2014.09.010
11. Timoney, JF, Kalimuthusamy, N, Velineni, S, Donahue, JM, Artiushin, SC, y Fettinger, M. Un genotipo único de Leptospira interrogans serovar Pomona tipo kennewicki se asocia con el aborto equino. Microbiol veterinario. (2011) 150:349–53. doi: 10.1016/j.vetmic.2011.02.049
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Donahue, JM, Smith, BJ, Poonacha, KB, Donahoe, JK y Rigsby, CL. Prevalencia y serovares de leptospira involucrados en abortos equinos en el centro de Kentucky durante las temporadas de parto de 1991-1993. J Vet Diagn Invest. (1995) 7:87–91. doi: 10.1177/104063879500700114
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Poonacha, KB, Donahue, JM, Giles, RC, Hong, CB, Petrites-Murphy, MB, Smith, BJ, et al. Leptospirosis en fetos equinos, potros nacidos muertos y placentas. Veterinario Pathol. (1993) 30:362–9. doi: 10.1177/030098589303000405
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Donahue, JM, Smith, BJ, Donahoe, JK, Rigsby, CL, Tramontin, RR, Poonacha, KB, et al. Prevalencia y serovares de leptospira involucrados en abortos equinos en Kentucky Central durante la temporada de partos de 1990. J Vet Diagn Invest. (1992) 4:279–84. doi: 10.1177/104063879200400309
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Ellis, W, Bryson, D, O’brien, J y Neill, S. Infección por leptospiral en fetos equinos abortados. Veterinario Equino J. (1983) 15:321–4. doi: 10.1111/j.2042-3306.1983.tb01811.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Brem, S, Gerhards, H, Wollanke, B, Meyer, P, y Kopp, H. Demostración de leptospira intraocular en 4 caballos que padecen uveítis recurrente equina (URE). Berliner und Münchener Tierarztliche Wochenschrift. (1998) 111:415–7.
17. Hartskeerl, R, Goris, M, Brem, S, Meyer, P, Kopp, H, Gerhards, H, et al. Clasificación de la leptospira a partir de los ojos de caballos que sufren de uveítis recurrente. J Veterinary Med Ser B. (2004) 51:110–5. doi: 10.1111/j.1439-0450.2004.00740.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Rocha, T, Ellis, W, Montgomery, J, Gilmore, C, Regalla, J y Brem, S. Estudio microbiológico y serológico de la leptospirosis en caballos en el momento del sacrificio: primeros aislamientos. Res Vet Sci. (2004) 76:199–202. doi: 10.1016/j.rvsc.2003.12.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Ellis, WA, O’Brien, JJ, Cassells, JA y Montgomery, J. Infección por leptoespiral en caballos en Irlanda del Norte: hallazgos serológicos y microbiológicos. Veterinario Equino J. (1983) 15:317–20. doi: 10.1111/j.2042-3306.1983.tb01809.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Lees, VW, y Gale, SP. Títulos de especies de Leptospira en caballos en Alberta. ¿Puede Vet J. (1994) 35:636–40.
21. Trimble, AC, Blevins, CA, Beard, LA, Deforno, AR y Davis, EG. Seroprevalencia, frecuencia de leptospiuria y factores de riesgo asociados en caballos en Kansas, Missouri y Nebraska de 2016 a 2017. PLoS Uno. (2018) 13:e0206639. doi: 10.1371/journal.pone.0206639
22. Benvin, I, Perko, VM, Maljković, MM, Habuš, J, Štritof, Z, Hađina, S, et al. Vigilancia serológica de la leptospirosis equina en Croacia en el período de 2012 a 2022: una visión clave de la evolución de la epizootiología. J Veterinario Equino. (2023) 127:104844. doi: 10.1016/j.jevs.2023.104844
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Odontsetseg, N, Boldbaatar, D, Mweene, AS, y Kida, H. Prevalencia serológica de Leptospira interrogans serovar Bratislava en caballos en Mongolia. Rec. Veterinario (2005) 157:518–9. doi: 10.1136/vr.157.17.518
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Simbizi, V, Saulez, MN, Potts, A, Lötter, C, y Gummow, B. Un estudio de la leptospirosis en caballos sudafricanos y los factores de riesgo asociados. Prev Vet Med. (2016) 134:6–15. doi: 10.1016/j.prevetmed.2016.09.019
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Båverud, V, Gunnarsson, A, Engvall, EO, Franzén, P, y Egenvall, A. Seroprevalencia de leptospira y asociaciones entre seropositividad, enfermedad clínica y factores del huésped en caballos. Acta Vet Scand. (2009) 51:1–10. doi: 10.1186/1751-0147-51-15
26. Williams, D, Smith, B, Donahue, J y Poonacha, K. Hallazgos serológicos y microbiológicos en 3 granjas con abortos equinos leptoespirales. Veterinario Equino J. (1994) 26:105–8. doi: 10.1111/j.2042-3306.1994.tb04345.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Fagre, AC, Mayo, CE, Pabilonia, KL, y Landolt, GA. Seroprevalencia de Leptospira spp. en équidos de Colorado y asociación con enfermedad clínica. J Vet Diagn Invest. (2020) 32:718–21. doi: 10.1177/1040638720943155
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Nally, JE, Hornsby, RL, Alt, DP, Bayles, D, Wilson-Welder, JH, Palmquist, DE, et al. Aislamiento y caracterización de leptospiras patógenas asociadas al ganado bovino. Microbiol veterinario. (2018) 218:25–30. doi: 10.1016/j.vetmic.2018.03.023
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Nally, JE, Ahmed, AA, Putz, EJ, Palmquist, DE, y Goris, MG. Comparación de PCR en tiempo real, cultivo bacteriológico y prueba de anticuerpos fluorescentes para la detección de Leptospira borgpetersenii en orina de bovinos infectados de forma natural. Veterinaria Sci. (2020) 7:66. doi: 10.3390/vetsci7020066
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Hornsby, RL, Alt, DP y Nally, JE. Aislamiento y propagación de leptospiras a 37 grados C directamente del huésped mamífero. Sci Rep. (2020) 10:9620. doi: 10.1038/s41598-020-66526-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Cole, JR, Sulzer, CR y Pursell, AR. Microtécnica mejorada para la prueba de aglutinación microscópica leptospiral. Appl Microbiol. (1973) 25:976–80. doi: 10.1128/am.25.6.976-980.1973
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Goris, MG, y Hartskeerl, RA. Serodiagnóstico de leptospirosis mediante la prueba de aglutinación microscópica. Curr Protoc Microbiol. (2014) 32:5.1-E. doi: 10.1002/9780471729259.mc12e05s32
34. Galloway, RL y Hoffmaster, AR. Optimización del ensayo de PCR LipL32 para aumentar la sensibilidad en el diagnóstico de la leptospirosis. Diagn Microbiol Infect Dis. (2015) 82:199–200. doi: 10.1016/j.diagmicrobio.2015.03.024
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Stoddard, RA, Gee, JE, Wilkins, PP, McCaustland, K y Hoffmaster, AR. Detección de Leptospira spp. patógena a través de la reacción en cadena de la polimerasa TaqMan dirigida al gen LipL32. Diagn Microbiol Infect Dis. (2009) 64:247–55. doi: 10.1016/j.diagmicrobio.2009.03.014
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Miller, JN. Espiroquetas en fluidos corporales y tejidos. Springfield, Illinois, Estados Unidos: Charles C. Thomas (1971).
37. Hamond, C, Dirsmith, KL, LeCount, K, Soltero, FV, Rivera-García, S, Camp, P, et al. Leptospira borgpetersenii serovar Hardjo y Leptospira santarosai serogrupo Pyrogenes aislados de rebaños lecheros bovinos en Puerto Rico. Fronteras en veterinaria. Ciencia. (2022) 9:1795. doi: 10.3389/fvets.2022.1025282
38. Hamond, C, LeCount, K, Putz, EJ, Bayles, DO, Camp, P, Goris, MGA, et al. Leptospirosis bovina por portación renal persistente de Leptospira borgpetersenii Serovar Tarassovi. Front Vet Sci. (2022) 9:848664. doi: 10.3389/fvets.2022.848664
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Wood, DE, Lu, J y Langmead, B. Análisis metagenómico mejorado con kraken 2. Genoma Biol. (2019) 20:1–13. doi: 10.1186/s13059-019-1891-0
40. Bankevich, A, Nurk, S, Antipov, D, Gurevich, AA, Dvorkin, M, Kulikov, AS, et al. SPAdes: un nuevo algoritmo de ensamblaje del genoma y sus aplicaciones a la secuenciación de una sola célula. J Comput Biol. (2012) 19:455–77. doi: 10.1089/cmb.2012.0021
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Altschul, SF, Gish, W, Miller, W, Myers, EW y Lipman, DJ. Herramienta básica de búsqueda de alineaciones locales. J Mol Biol. (1990) 215:403–10. doi: 10.1016/S0022-2836(05)80360-2
42. Hartskeerl, R, Smits, H, Korver, H, Goris, M, y Terpstra, W. Manual curso internacional sobre métodos de laboratorio para el diagnóstico de la leptospirosis. Ámsterdam, Países Bajos: KIT (2006).
43. Stone, NE, McDonough, RF, Hamond, C, LeCount, K, Busch, JD, Dirsmith, KL, et al. Captura y enriquecimiento de ADN: un enfoque independiente del cultivo para caracterizar la diversidad genómica de especies patógenas de Leptospira. Microorganismos. (2023) 11:51282. doi: 10.3390/microorganismos11051282
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Katz, LS, Griswold, T, Morrison, SS, Caravas, JA, Zhang, S, den Bakker, HC, et al. Mashtree: una comparación rápida de archivos de secuencia de genoma completo. J Software de código abierto. (2019) 4:1762. doi: 10.21105/joss.01762
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. Li, H. Minimapa2: alineación por pares para secuencias de nucleótidos. Bioinformática. (2018) 34:3094–100. doi: 10.1093/bioinformatics/bty191
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. McKenna, A, Hanna, M, Banks, E, Sivachenko, A, Cibulskis, K, Kernytsky, A, et al. El kit de herramientas de análisis del genoma: un marco de MapReduce para analizar datos de secuenciación de ADN de próxima generación. Genoma Res. (2010) 20:1297–303. doi: 10.1101/gr.107524.110
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Delcher, AL, Salzberg, SL, y Phillippy, AM. Uso de MUMmer para identificar regiones similares en grandes conjuntos de secuencias. Curr Protoc Bioinformática. (2003) 10:s00. doi: 10.1002/0471250953.bi1003s00
48. Sahl, JW, Lemmer, D, Travis, J, Schupp, JM, Gillece, JD, Aziz, M, et al. NASP: un método preciso y rápido para la identificación de SNP en conjuntos de datos WGS que admite formatos flexibles de entrada y salida. Genoma Microb. (2016) 2:E000074. doi: 10.1099/mgen.0.000074
Resumen de PubMed | Texto completo de Crossref | Google Académico
49. Nguyen, LT, Schmidt, HA, von Haeseler, A, y Minh, BQ. IQ-TREE: un algoritmo estocástico rápido y eficaz para estimar filogenias de máxima verosimilitud. Mol Biol Evol. (2015) 32:268–74. DOI: 10.1093/Molbev/MSU300
Resumen de PubMed | Texto completo de Crossref | Google Académico
50. Kalyaanamoorthy, S, Minh, BQ, Wong, TKF, von Haeseler, A, y Jermiin, LS. ModelFinder: selección rápida de modelos para estimaciones filogenéticas precisas. Métodos Nat. (2017) 14:587–9. doi: 10.1038/nmeth.4285
Resumen de PubMed | Texto completo de Crossref | Google Académico
51. Li, H, Handsaker, B, Wysoker, A, Fennell, T, Ruan, J, Homer, N, et al. El formato de alineación/mapa de secuencia y SAMtools. Bioinformática. (2009) 25:2078–9. doi: 10.1093/bioinformatics/BTP352
Resumen de PubMed | Texto completo de Crossref | Google Académico
52. Putz, EJ, Andreasen, CB, Stasko, JA, Fernandes, LG, Palmer, MV, Rauh, MJ, et al. Macrófagos espumosos circulantes en el modelo de leptospirosis del hámster sirio dorado (Mesocricetus auratus). J Comp Pathol. (2021) 189:98–109. doi: 10.1016/j.jcpa.2021.10.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
53. R. Equipo central. R: Un lenguaje y un entorno para la computación estadística. R Foundation for Statistical Computing, Viena, Austria. Disponible en: https://www.R-project.org/. (2021)
54. Hamond, C, Browne, AS, de Wilde, LH, Hornsby, RL, LeCount, K, Anderson, T, et al. Evaluación de roedores como portadores de especies patógenas de Leptospira en las Islas Vírgenes de EE. UU. y su riesgo para la salud animal y pública. Sci Rep. (2022) 12:4846. doi: 10.1038/s41598-022-04846-3
55. Nascimento, AL, Ko, AI, Martins, EA, Monteiro-Vitorello, CB, Ho, PL, Haake, DA, et al. La genómica comparativa de dos serovares de Leptospira interrogans revela nuevos conocimientos sobre fisiología y patogénesis. J Bacteriol. (2004) 186:2164–72. doi: 10.1128/JB.186.7.2164-2172.2004
Resumen de PubMed | Texto completo de Crossref | Google Académico
56. Nascimento, AL, Verjovski-Almeida, S, Van Sluys, MA, Monteiro-Vitorello, CB, Camargo, LE, Digiampietri, LA, et al. Características del genoma de Leptospira interrogans serovar Copenhageni. Braz J Med Biol Res. (2004) 37:459–77. doi: 10.1590/S0100-879X2004000400003
Resumen de PubMed | Texto completo de Crossref | Google Académico
57. Alt, DP, Wilson-Welder, JH, Bayles, DO, Cameron, C, Adler, B, Bulach, DM, et al. Secuencia completa del genoma de Leptospira interrogans Serovar Bratislava, cepa PigK151. Anuncio del genoma. (2015) 3:e15. doi: 10.1128/genomaA.00678-15
Resumen de PubMed | Texto completo de Crossref | Google Académico
58. Nally, JE, Wilson-Welder, JH, Hornsby, RL, Palmer, MV y Alt, DP. Ratas endogámicas como modelo para estudiar la leptospirosis renal persistente y la respuesta inmune celular asociada. Las células frontales infectan el microbiol. (2018) 8:66. doi: 10.3389/fcimb.2018.00066
Resumen de PubMed | Texto completo de Crossref | Google Académico
59. Nally, JE, Whitelegge, JP, Bassilian, S, Blanco, DR, y Lovett, MA. Caracterización del proteoma de la membrana externa de Leptospira interrogans expresado durante la infección letal aguda. Infectar a Immun. (2007) 75:766–73. doi: 10.1128/IAI.00741-06
Resumen de PubMed | Texto completo de Crossref | Google Académico
60. Fernandes, LG, Putz, EJ, Stasko, J, Lippolis, JD, Nascimento, AL, y Nally, JE. Evaluación de mutantes knockdown de LipL32 y LigA/LigB en Leptospira interrogans Serovar Copenhageni: impactos en el proteoma y virulencia. Microbiol frontal. (2022) 12:799012. doi: 10.3389/fmicb.2021.799012
61. Monti, G, Montes, V, Tortosa, P, Tejeda, C, y Salgado, M. Patrones de excreción de orina de Leptospira spp. patógena en vacas lecheras. Res. Veterinaria. (2023) 54:1190. DOI: 10.1186/s13567-023-01190-w
62. Stallman, Dakota del Norte. Subcomité del Comité Internacional de Bacteriología Sistemática sobre la taxonomía de Leptospira: actas de la reunión, 5 y 6 de septiembre de 1986, Manchester, Inglaterra. Int J Syst Evol Microbiol. (1987) 37:472–3. doi: 10.1099/00207713-37-4-472
63. Brenner, DJ, Kaufmann, AF, Sulzer, KR, Steigerwalt, AG, Rogers, FC, y Weyant, RS. Determinación adicional de la relación del ADN entre serogrupos y serovares de la familia Leptospiraceae con una propuesta para Leptospira alexanderi sp. nov. y cuatro nuevas genoespecies de Leptospira. Int J Syst Evol Microbiol. (1999) 49:839–58. doi: 10.1099/00207713-49-2-839
Resumen de PubMed | Texto completo de Crossref | Google Académico
64. Guglielmini, J, Bourhy, P, Schiettekatte, O, Zinini, F, Brisse, S y Picardeau, M. Tipificación de secuencias multilocus del genoma central de Leptospira en todo el género para la taxonomía de cepas y la vigilancia global. PLoS negl trop dis. (2019) 13:e0007374. doi: 10.1371/journal.pntd.0007374
Resumen de PubMed | Texto completo de Crossref | Google Académico
66. Dikken, H, Kmety, E, De Geus, A, y Timmer, V. Un nuevo serovar leptospiral en el serogrupo Australis. Trop Geogr Med. (1979) 31:263–8.
67. Que-Gewirth, NL, Ribeiro, AA, Kalb, SR, Cotter, RJ, Bulach, DM, Adler, B, et al. Un grupo fosfato metilado y cuatro cadenas de acilo unidas a amida en el lípido a. de Leptospira interrogans, el anclaje de membrana de un lipopolisacárido inusual que activa TLR2. J Biol Chem. (2004) 279:25420–9. doi: 10.1074/jbc. M400598200
Resumen de PubMed | Texto completo de Crossref | Google Académico
68. Zilch, TJ, Lee, JJ, Saleem, MZ, Zhang, H, Cortese, V, Voris, N, et al. Leptospirosis equina: el desafío experimental de Leptospira interrogans serovar Bratislava no logra establecer la infección en caballos naïf. Veterinario Equino J. (2021) 53:845–54. doi: 10.1111/evj.13442
Resumen de PubMed | Texto completo de Crossref | Google Académico
69. Nally, JE, Monahan, AM, Miller, IS, Bonilla-Santiago, R, Souda, P, y Whitelegge, JP. Análisis proteómico comparativo de proteínas expresadas diferencialmente en orina de hospederos reservorio de leptospirosis. PLoS Uno. (2011) 6:e26046. doi: 10.1371/journal.pone.0026046
Resumen de PubMed | Texto completo de Crossref | Google Académico
70. Ristow, P, Bourhy, P, da Cruz McBride, FW, Figueira, CP, Huerre, M, Ave, P, et al. La proteína Loa22, similar a OmpA, es esencial para la virulencia de la leptoespiral. PLoS Pathog. (2007) 3:E97. doi: 10.1371/journal.ppat.0030097
Resumen de PubMed | Texto completo de Crossref | Google Académico
71. Fernandes, LGV, Teixeira, AF y Nascimento, A. Evaluación de mutantes knockdown de Leptospira interrogans para las proteínas LipL32, LipL41, LipL21 y OmpL1. Microbiol frontal. (2023) 14:1199660. doi: 10.3389/fmicb.2023.1199660
Resumen de PubMed | Texto completo de Crossref | Google Académico
72. Chang, YF. El síndrome reproductivo en la leptospirosis equina. Veterinario Equino J. (2021) 53:856. doi: 10.1111/evj.13447
Resumen de PubMed | Texto completo de Crossref | Google Académico
73. Ellis, W, y Thiermann, A. Aislamiento de Leptospira interrogans serovar Bratislava de cerdas en Iowa. Am J Vet Res. (1986) 47:1458–60.
74. Bolin, CA, y Cassells, JA. Aislamiento de Leptospira interrogans, serovares, Bratislava y hardjo de cerdos en el momento del sacrificio. J Vet Diagn Invest. (1992) 4:87–9. doi: 10.1177/104063879200400121
Resumen de PubMed | Texto completo de Crossref | Google Académico
75. Guitian, J, Thurmond, MC y Hietala, SK. Infertilidad y aborto entre vacas lecheras de primera lactancia seropositivas o seronegativas para Leptospira interrogans serovar Hardjo. J Am Vet Med Assoc. (1999) 215:515–8. doi: 10.2460/javma.1999.215.04.515
Resumen de PubMed | Texto completo de Crossref | Google Académico
76. Hamond, C, Pestana, CP, Rocha-de-Souza, CM, Cunha, LE, Brandão, FZ, Medeiros, MA, et al. Presencia de leptospiras en el tracto genital de yeguas con problemas reproductivos. Microbiol veterinario. (2015) 179:264–9. doi: 10.1016/j.vetmic.2015.06.014
Resumen de PubMed | Texto completo de Crossref | Google Académico
77. Putz, EJ, y Nally, JE. Investigando el equilibrio inmunológico y biológico de los huéspedes reservorio y la Leptospira patógena: ¿equilibrando la solución a un problema agudo? Microbiol frontal. (2020) 11:2005. doi: 10.3389/fmicb.2020.02005
Resumen de PubMed | Texto completo de Crossref | Google Académico
78. Steven, EW, Rogers, GM, Ramachandran, S, Engelken, TJ, Epperson, WB, Larson, RL, et al. Prevalencia del rebaño y factores de riesgo de infección por Leptospira en operaciones de vacas y terneros de carne en los Estados Unidos. El. Practicante bovino. (2007) 41:15–23. doi: 10.21423/bovine-vol41no1p15-23
79. Ellis, WA, O’Brien, JJ y Cassells, J. Papel del ganado en el mantenimiento de la infección por Herjo por el serotipo de Leptospira interrogans en Irlanda del Norte. Rec. Veterinario (1981) 108:555–7. doi: 10.1136/vr.108.26.555
Resumen de PubMed | Texto completo de Crossref | Google Académico
80. Babudieri, B. Reservorios animales de leptospiras. Ann N Y Acad Sci. (1958) 70:393–413. doi: 10.1111/j.1749-6632.1958.tb35398.x
81. Helman, SK, Tokuyama, AFN, Mummah, RO, Stone, NE, Gamble, MW, Snedden, CE, et al. La leptospira patógena está muy extendida en la vida silvestre urbana del sur de California. Sci Rep. (2023) 13:14368. doi: 10.1038/s41598-023-40322-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
82. Johnson, RC, y Harris, VG. Diferenciación de leptospiras patógenas y saprófitas I. Crecimiento a bajas temperaturas. J Bacteriol. (1967) 94:27–31. doi: 10.1128/jb.94.1.27-31.1967
Resumen de PubMed | Texto completo de Crossref | Google Académico
83. Ellis, W, Montgomery, J y Cassells, J. Tratamiento con dihidroestreptomicina de bovinos portadores de Leptospira interrogans serovar Hardjo. Res Vet Sci. (1985) 39:292–5. doi: 10.1016/S0034-5288(18)31716-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
84. Nally, JE, Artiushin, S, y Timoney, JF. Caracterización molecular de las proteínas inmunogénicas termoinducidas Qlp42 y Hsp15 de Leptospira interrogans. Infectar a Immun. (2001) 69:7616–24. doi: 10.1128/IAI.69.12.7616-7624.2001
Resumen de PubMed | Texto completo de Crossref | Google Académico
85. Nally, JE, Timoney, JF y Stevenson, B. Síntesis de proteínas reguladas por temperatura por Leptospira interrogans. Infectar a Immun. (2001) 69:400–4. doi: 10.1128/IAI.69.1.400-404.2001
Resumen de PubMed | Texto completo de Crossref | Google Académico
86. Putz, EJ, Sivasankaran, SK, Fernandes, LGV, Brunelle, B, Lippolis, JD, Alt, DP, et al. Perfiles transcripcionales distintos de Leptospira borgpetersenii serovar Hardjo cepas JB197 y HB203 cultivadas a diferentes temperaturas. PLoS negl trop dis. (2021) 15:E0009320. doi: 10.1371/journal.pntd.0009320
Resumen de PubMed | Texto completo de Crossref | Google Académico
87. Monahan, AM, Callanan, JJ, y Nally, JE. Análisis proteómico de Leptospira interrogans excretada en orina de huéspedes con infección crónica. Infectar a Immun. (2008) 76:4952–8. doi: 10.1128/IAI.00511-08
Resumen de PubMed | Texto completo de Crossref | Google Académico
88. Nally, JE, Grassmann, AA, Planchon, S, Sergeant, K, Renaut, J, Seshu, J, et al. Las leptospiras patógenas modulan la expresión de proteínas y las modificaciones postraduccionales en respuesta a las señales del huésped de mamíferos. Las células frontales infectan el microbiol. (2017) 7:362. doi: 10.3389/fcimb.2017.00362
Resumen de PubMed | Texto completo de Crossref | Google Académico
89. Putz, EJ, Fernandes, LG, Sivasankaran, SK, Bayles, DO, Alt, DP, Lippolis, JD, et al. A algunos les gusta caliente, a otros les gusta frío; Comparación del proteoma de cepas de Leptospira borgpetersenii serovar Hardjo propagadas a diferentes temperaturas. J Proteoma. (2022) 262:104602. doi: 10.1016/j.jprot.2022.104602
90. Sykes, JE, Haake, DA, Gamage, CD, Mills, WZ y Nally, JE. Una perspectiva global de «una sola salud» sobre la leptospirosis en humanos y animales. J Am Vet Med Assoc. (2022) 260:1589–96. doi: 10.2460/javma.22.06.0258
91. Hamond, C, LeCount, K, Anderson, T, Putz, EJ, Stuber, T, Hicks, J, et al. Aislamiento y caracterización de cepas saprófitas y patógenas de Leptospira de fuentes de agua en el Medio Oeste de los Estados Unidos. Fronteras en el agua. (2024) 6:8088. doi: 10.3389/frwa.2024.1278088
92. Nally, JE, aren’t, Z, Bayles, DO, Hornsby, RL, Gilmore, C, Regan, S, et al. Implicaciones de las especies de mamíferos invasores en las enfermedades infecciosas emergentes: la musaraña de dientes blancos (Crocidura russula) se asocia con un nuevo Serovar de Leptospira patógena en Irlanda. PLoS negl trop dis. (2016) 10:E0005174. doi: 10.1371/journal.pntd.0005174
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: Leptospira, leptospirosis, equino, Australis, kirschneri
Cita: Hamond C, Adam EN, Stone NE, LeCount K, Anderson T, Putz EJ, Camp P, Hicks J, Stuber T, van der Linden H, Bayles DO, Sahl JW, Schlater LK, Wagner DM y Nally JE (2024) Identificación de yeguas equinas como huéspedes reservorio de especies patógenas de Leptospira. Frente. Vet. Sci. 11:1346713. doi: 10.3389/fvets.2024.1346713
Recibido: 29 de noviembre de 2023; Aceptado: 24 de abril de 2024;
Publicado: 09 Mayo 2024.
Editado por:
Valentina Virginia Ebani, Universidad de Pisa, Italia
Revisado por:
Balamurugan Vinayagamurthy, Instituto Nacional de Epidemiología Veterinaria e Informática de las Enfermedades (ICAR), India
Dhani Prakoso, Fundación Profesor Nidom, Indonesia
Derechos de autor © 2024 Hamond, Adam, Stone, LeCount, Anderson, Putz, Camp, Hicks, Stuber, van der Linden, Bayles, Sahl, Schlater, Wagner y Nally. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Jarlath E. Nally, jarlath.nally@usda.gov
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

