Identificación de dos nuevos epítopos lineales en la proteína E165R del virus de la peste porcina africana
 Jian He1,2†
Jian He1,2† Jieqiong Li1†
Jieqiong Li1† Mingzhan Luo1
Mingzhan Luo1 Yangkun Liu1
Yangkun Liu1 Jingchen Sol2*
Jingchen Sol2* Lunguang Yao1*
Lunguang Yao1*- 1Centro Provincial de Ingeniería y Tecnología de Henan de Productos Sanitarios para el Ganado y las Aves de Corral, Estación de Investigación y Observación de Campo de Henan del Ecosistema de Humedales de Cabecera de la Ruta Central del Proyecto de Desvío de Agua de Sur a Norte, Escuela de Ciencias de la Vida e Ingeniería Agrícola, Universidad Normal de Nanyang, Nanyang, China
- número arábigoLaboratorio Provincial Clave de Guangdong de Genómica Agroanimal y Mejoramiento Molecular, Facultad de Ciencias Animales, Universidad Agrícola del Sur de China, Guangzhou, China
La peste porcina africana (PPA) es una enfermedad infecciosa muy mortal en los cerdos, causada por el virus de la peste porcina africana (PPA). Se caracteriza por una corta duración de la enfermedad y una alta morbimortalidad. En agosto de 2018, la PPA se notificó por primera vez en China y, posteriormente, se extendió rápidamente por todo el país, causando graves pérdidas económicas para la industria porcina china. La detección precoz desempeña un papel fundamental en la prevención y el control de la PPA, ya que en la actualidad no existe una vacuna eficaz ni medicamentos terapéuticos dirigidos. Además, la identificación de epítopos antigénicos protectores conservados del virus de la PPA es esencial para el desarrollo de reactivos de diagnóstico. La proteína E165R, que se expresa en gran medida en las primeras etapas de la infección por el virus de la peste porcina africana, puede servir como un indicador importante para la detección temprana. En este estudio, obtuvimos con éxito la expresión procariota soluble de alta pureza de la proteína E165R. A continuación, utilizamos la proteína E165R recombinante purificada para la inmunización en ratones con el fin de preparar anticuerpos monoclonales (mAb) mediante la técnica de fusión de hibridomas. Después de tres cribados subclonales, obtuvimos con éxito tres mAbs contra la proteína E165R del virus de la PPA en células denominadas 1B7, 1B8 y 10B8. A través del ensayo de inmunofluorescencia (IFA) y Western blot, confirmamos que los mAbs preparados reconocen específicamente la proteína E165R expresada por baculovirus. Mediante el uso de la proteína E165R truncada superpuesta y el análisis de barrido de péptidos superpuestos, identificamos tentativamente dos nuevos epítopos lineales de células B (13EAEAYYPPSV22 y 55VACEHMGKKC64) que están altamente conservados en los genotipos I y II de la PPA. Por lo tanto, como anticuerpo de detección, tiene la capacidad de detectar el virus de la PPA en una amplia gama de genotipos, proporcionando información valiosa para el desarrollo de reactivos de inmunodiagnóstico relacionados.
1 Introducción
La peste porcina africana (PPA) es una enfermedad infecciosa grave y aguda de los cerdos causada por el virus de la peste porcina africana (PPA) (1). La fase aguda de la PPA puede causar una mortalidad del 100% en los cerdos, y los síntomas incluyen letargo, diarrea sanguinolenta, hipertermia y cambios vasculares (2). La transmisión del virus de la peste porcina africana se produce principalmente a través del contacto directo entre cerdos infectados y sanos. La PPA puede transmitirse por vía oral y nasal, pero también a través de picaduras blandas de garrapatas, arañazos o inyecciones (3, 4). Debido a la compleja estructura y a la diversa funcionalidad del virus de la PPA, el control de la propagación del virus presenta desafíos. Ante la ausencia de una vacuna comercial disponible, es necesario tomar medidas de sacrificio para contener la epidemia (5). Por lo tanto, el desarrollo de métodos de diagnóstico altamente sensibles y específicos se vuelve crucial para detectar y aislar rápidamente a los cerdos infectados por el virus de la PPA.
La peste porcina africana ha tenido consecuencias económicas y ecológicas devastadoras en todo el mundo y se considera una de las enfermedades más destructivas (6). Surgió por primera vez en Kenia en la década de 1920 y resurgió en Georgia en 2007, extendiéndose rápidamente por África subsahariana, el Caribe y Europa del Este, hasta que llegó a China en 2018. Desde entonces, ha continuado extendiéndose a otros países asiáticos (6-8). Una de las características que distinguen al virus de la PPA de diferentes cepas o generaciones es la duplicación y deleción de secuencias específicas en su genoma. El extremo C-terminal de la proteína p72 codificada por el gen B646L ha sido sometido a un análisis filogenético, revelando que el virus de la PPA se clasifica actualmente en 24 genotipos (genotipos I a XXIV). En particular, la cepa de genotipo II, altamente virulenta, es la principal cepa que se transmite actualmente en China (1, 9).
El virus de la PPA es un virus de ADN, de 260-300 nm de diámetro, con un genoma de 170-190 kb de longitud. Codifica más de 170 proteínas, incluyendo enzimas, proteínas estructurales y proteínas de andamiaje (10-12). La replicación del virus de la peste porcina africana se produce principalmente en macrófagos y monocitos. La infección viral experimenta adhesión, internalización, proliferación del genoma, ensamblaje viral y liberación (13, 14). En última instancia, el virus se libera de las células como una excrecencia (15). Las proteínas dUTPasa (E165R), K196R y A240L codificadas por los genes E165R, K196R y A240L son las principales enzimas metabolizadoras de nucleótidos responsables de proporcionar energía durante la replicación viral (16). Se ha demostrado que la dUTPasa está asociada con la inmunidad innata (17, 18) y puede activar la vía de señalización del factor nuclear kappa B (NF-κB) (19). El gen E165R del virus de la peste porcina africana codifica una desoxiuridina trifosfato nucleotidohidrolasa (20). Al igual que otras enzimas dUTPasa, la dUTPasa del virus de la PPA tiene una estructura trimérica y se expresa a lo largo de la infección, desempeñando un papel crucial en el mantenimiento de la integridad de la replicación viral. Se ha descubierto que el ASFV codifica tanto un sistema de reparación de escisión de base (BER) como una dUTPasa. El sistema BER repara el daño de la base del ADN causado por hidrólisis u oxidación, mientras que la dUTPasa degrada la 2′-desoxiuridina 5′-trifosfato (dUTPasa) en el citoplasma, reduciendo la probabilidad de incorporación de uracilo en los errores de replicación del ADN viral y contribuyendo a la replicación del genoma viral de alta fidelidad (21). La deleción del gen E165R inhibe fuertemente la replicación del virus de la PPA in vitro, lo que indica su correlación con la virulencia y la eficiencia de la replicación (21). Sin embargo, estudios recientes han demostrado que la deleción del gen dUTPasa no afecta la replicación y virulencia del virus de la PPA (22). Los anticuerpos monoclonales (mAb) que se dirigen a la región altamente conservada de E165R pueden inhibir específicamente la actividad dUTPasa de E165R (23). Además, la dUTPasa de la PPA exhibe una notable tolerancia a altas temperaturas, de hasta 83 °C, lo que la convierte en una herramienta enzimática prometedora (24). Los epítopos, que son las unidades estructurales y funcionales básicas de las proteínas antigénicas, desempeñan un papel crucial en el diagnóstico de enfermedades, el diseño de vacunas y otras aplicaciones que requieren respuestas inmunitarias específicas.
En este estudio, se eligió E165R como proteína diana, y se generaron con éxito tres anticuerpos monoclonales (mAb) contra la expresión procariota de la proteína E165R. Mediante la superposición de análisis de barrido de proteínas y péptidos E165R truncados, se identificaron dos nuevos epítopos conservados dirigidos por los mAbs. En conclusión, estos hallazgos sientan las bases para el desarrollo de nuevos reactivos de diagnóstico.
2 Materiales y métodos
2.1 Genes, células y animales
El plásmido recombinante pET32a-E165R de la cepa ASFV Pig/HLJ/2018 (Genbank: MK333180.1), fue sintetizado por Sangon Bioengineering Co., Ltd. (Shanghái, China). En nuestro laboratorio se almacenaron células Sf9 y el baculovirus recombinante Ac MutliBac-E165R (el gen E165R impulsado por el promotor de poliedrín). Las células SP2/0 fueron donadas por el Laboratorio Clave de Inmunología Animal de la Academia de Ciencias Agrícolas de Henan. Se obtuvieron ratones hembra BALB/c de seis semanas de edad de Autobio Diagnostics Co., Ltd. (Zhengzhou, China).
2.2 Preparación e identificación de la proteína recombinante E165R
El plásmido recombinante pET32a-E165R se transformó en células competentes para E. coli Bl21 (DE3) (Weidi, Shanghai, China). Después de la detección de resistencia a la ampicilina, se seleccionaron bacterias monoclonales positivas para la inducción de la expresión. En concreto, se añadió un inductor de isopropil-β-D-tiogalactopiranósido (IPTG) a una concentración final de 0,5 mmoL/L cuando el valor de OD600 nm de la solución bacteriana alcanzó aproximadamente 0,4-0,6. La inducción se llevó a cabo a 37°C durante 6 h. Los productos de la solución bacteriana se recogieron por centrifugación a 12.000 g y luego se lavaron tres veces con 1 × PBS (Solarbio, Beijing, China). Tras la sonicación y la lisis de las bacterias, se aislaron los sobrenadantes y se analizó su solubilidad mediante SDS-PAGE. La proteína se purificó utilizando una columna a base de resina Ni-NTA (Solarbio, Beijing, China) de acuerdo con las instrucciones. La elución de la proteína diana se logró variando la concentración de imidazol en la solución de elución. La muestra purificada de la proteína E165R se analizó mediante SDS-PAGE y Western blot utilizando His-tag mAb (Proteintech, Wuhan, China).
2.3 Experimentación con animales y preparación de anticuerpos monoclonales E165R
Todos los protocolos de animales se llevaron a cabo siguiendo las directrices del comité de ética de la Universidad Normal de Nanyang. Ratones BALB/C de seis semanas de edad fueron inmunizados con proteína E165R purificada. Para la inmunización primaria, se mezcló una dosis de 20 μg de la proteína con una cantidad igual de adyuvante de Freund completo. Posteriormente, se administraron vacunas de refuerzo los días 14 y 28. Siete días después de la inmunización final, se recogieron muestras de sangre de las venas de la cola para determinar los títulos de anticuerpos séricos mediante ELISA indirecto. Se seleccionó el ratón con el título sérico más alto para las siguientes inmunizaciones de refuerzo intraperitoneal. Se recogieron muestras de suero positivas de la sangre del ojo después de tres días, y se aislaron esplenocitos de ratones con alta potencia sérica. A continuación, estos esplenocitos se fusionaron con células de mieloma SP2/0 en crecimiento logarítmico mediante el tratamiento con PEG 1500. Las células se examinaron, se cultivaron en un medio que contenía HAT y se cultivaron en placas de 96 pocillos. Se seleccionaron células de hibridoma positivas 10 días después de la fusión, y se realizaron tres exámenes de detección subclonales utilizando el método de dilución limitada para obtener anticuerpos monoclonales cultivados de manera estable. A continuación, las líneas celulares monoclonales se amplificaron mediante inducción in vivo de ascitis.
2.4 ELISA indirecto
Se utilizó el método ELISA indirecto para detectar el título de ascitis mAb anti-E165R. La proteína E165R se diluyó a una concentración de 1 μg/mL utilizando 1 × tampón de recubrimiento ELISA (Solarbio, Pekín, China). A continuación, se añadieron 100 μL de la solución diluida por pocillo en la placa ELISA. La placa ELISA se recubrió con un 5% de leche desnatada en polvo y se incubó durante la noche. Los mAb ascitis se diluyeron en una dilución de doble proporción de 1:400 a 1:819.200 como anticuerpo primario. A continuación, se añadió el anticuerpo secundario, IgG (H + L) anti-ratón de cabra conjugado con HRP (diluido 1:5.000, Proteintech, Wuhan, China), y se incubó en los pocillos durante 1 h a 37 °C. A continuación, se añadieron 100 μL de TMB a la reacción y se incubaron durante 10 min. A continuación, la reacción se detuvo añadiendo 2 moL/L H2SO4, y la densidad óptica a 450 nm se midió mediante la lectura de la placa.
De acuerdo con las instrucciones proporcionadas por el kit de identificación de isotipos de anticuerpos monoclonales de ratón (Proteintech, Wuhan, China), los isotipos de mAbs se identificaron mediante la lectura del valor de OD450 nm.
2.5 Ensayo de inmunofluorescencia
La especificidad del mAb se confirmó aún más mediante el método IFA. Las células Sf9 se infectaron utilizando el baculovirus recombinante Ac MutliBac-E165R, mientras que las no infectadas sirvieron como control negativo (NC). 72 h después de la infección, se retiró el sobrenadante y las células se fijaron en paraformaldehído al 4% a temperatura ambiente (RT) durante 20 min. Luego, las células se permeabilizaron con Triton X-100 al 0,1%, se enjuagaron tres veces con PBST y se bloquearon con leche en polvo descremada al 5% a 37 °C durante 1 h. Posteriormente, las células se incubaron con la IgG H&L anti-ratón de cabra (FITC) (diluida 1:100, Abcam, UK) a 37°C durante 1 h. Después del lavado, los núcleos celulares se tiñeron con DAPI (Solarbio, Beijing, China) y las células se observaron bajo un microscopio de fluorescencia invertida.
2.6 Western blot
Las células Sf9 se infectaron con baculovirus recombinante Ac MutliBac-E165R, mientras que las células no infectadas se establecieron como control negativo. Después de 48 h de infección, se recogieron las muestras celulares y se utilizaron muestras de 50 μg para luego analizarlas mediante Western blot con mAbs E165R como anticuerpos primarios. Las membranas de PVDF se recubrieron con leche desnatada en polvo al 5% a 37 °C durante 1 h. Posteriormente, las membranas de PVDF se incubaron con el mAbs anti-PPA E165R (diluido 1:2.000) a RT durante 1 h. Después de tres lavados con PBST, luego se incubó con la IgG anti-ratón de cabra conjugada con HRP (diluida 1:5.000) a RT durante 1 h. Las membranas se lavaron tres veces y luego se sometieron a un desarrollo de color de quimioluminiscencia (ECL) mejorado en la oscuridad.
2.7 Diseño y síntesis de polipéptidos
Determinar los epítopos reconocidos por el mAb. La proteína E165R se truncó en cuatro segmentos que contenían regiones de superposición y se ligó al vector de expresión procariota pET32a (+) para la expresión inducida, denominada A, B, C y D, respectivamente (cebador utilizado como se muestra en la Tabla 1). Posteriormente, los epítopos reconocidos por mAb se identificaron mediante análisis de Western blot. Sobre la base de los resultados del Western blot, se sintetizaron 12 fragmentos de péptidos que contenían seis aminoácidos superpuestos. Para facilitar el acoplamiento a la proteína BSA, se añadió una C-cisteína (C) en el extremo N-terminal del fragmento peptídico para asegurar la presencia de al menos un residuo de cisteína. Todos los péptidos fueron sintetizados por GenScript (Nanjing, China) (pureza >95%).
2.8 Identificación y cribado de polipéptidos
Los epítopos antigénicos reconocidos por los mAbs se identificaron por el método dot-blot. El péptido se conjugó con la proteína transportadora BSA utilizando Sulfo-SMCC y el péptido acoplado se detectó en una membrana de filtro de nitrocelulosa (NC). Se incluyó un control negativo (proteína BSA) y un control positivo (proteína E165R). Los anticuerpos monoclonales preparados se utilizaron como anticuerpo primario y la IgG (H + L) anti-ratón de cabra conjugada con HRP como anticuerpo secundario. La reactividad de los mAbs con el péptido se detectó utilizando un reactivo ECL.
Ensayo ELISA de péptidos para la localización de mAbs. Los péptidos acoplados a BSA se alcanzaron a una concentración de 5 μg/mL utilizando tampón de recubrimiento ELISA, seguidos de 100 μL/pocillo recubiertos en placas ELISA con control negativo de BSA y control positivo de proteína E165R. Las placas se incubaron a 4°C durante la noche. Se realizaron tres repeticiones para cada grupo experimental. Después de la incubación, se añadieron 100 μL de mAbs diluidos (diluidos 1:1.000) como anticuerpo primario, seguido de la adición de IgG anti-ratón de cabra conjugada con HRP (diluida 1:3.000) como anticuerpo secundario. Finalmente, la reacción de desarrollo del color se llevó a cabo utilizando 100 μL de TMB por pocillo, seguido de la adición de 50 μL de 2 M H2SO4 para terminar la reacción.
2.9 Análisis de homología
El análisis de homología del epítopo de reconocimiento de mAb E165R en este estudio se realizó utilizando MEGA 11 (Mega Limited, Auckland, Nueva Zelanda). Se compararon individualmente las secuencias E165R de 35 cepas de PPA de diferentes regiones. La estructura de ASFV E165R (PDB: 6LIS) se simuló utilizando PyMOL y se marcaron los epítopos identificados.
3 Resultados
3.1 Expresión y purificación de la proteína E165
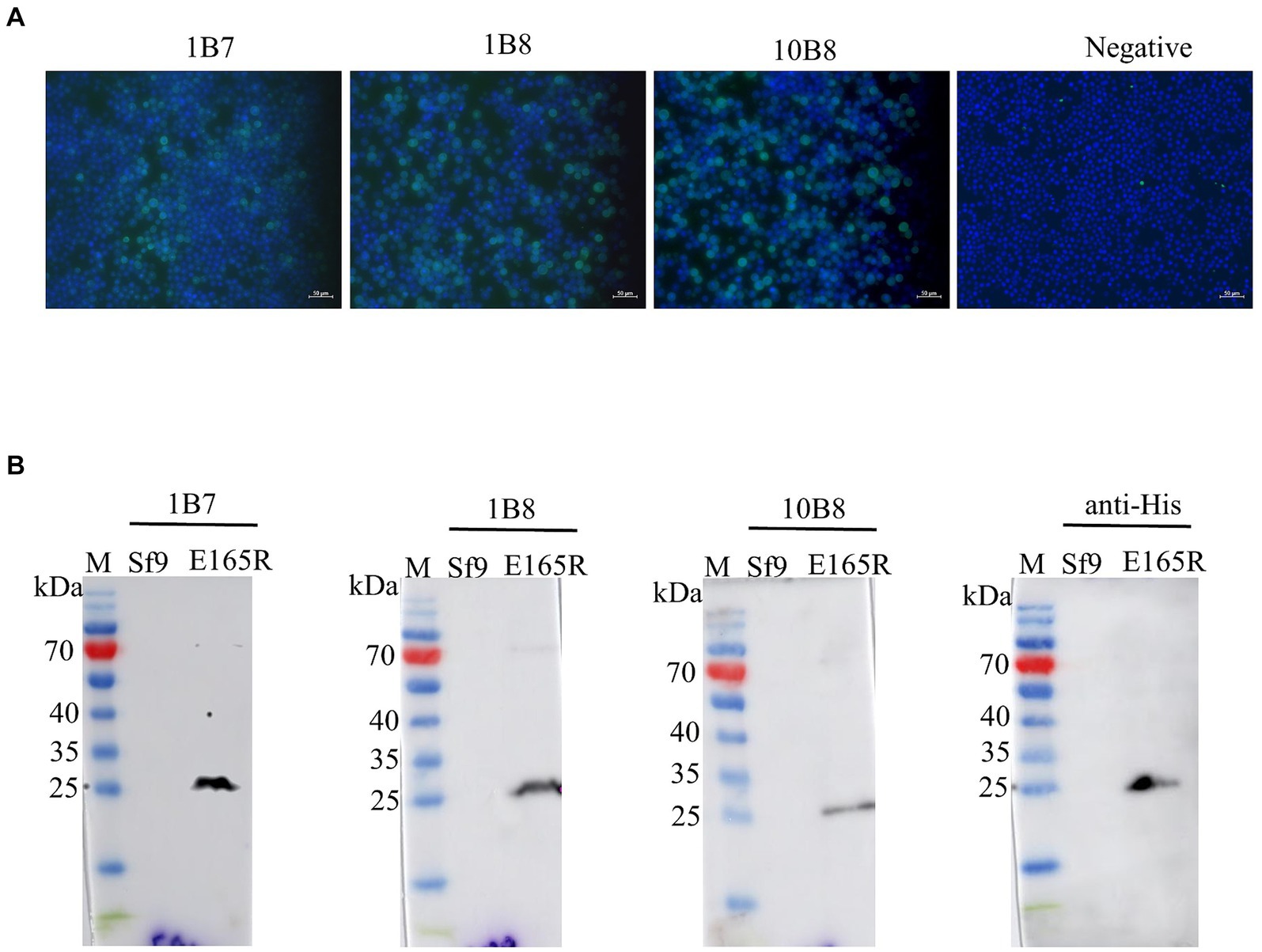
El plásmido recombinante pET32a-E165R se transformó en E. coli BL21(DE3) para su expresión mediante inducción con IPTG. Los resultados mostraron que los resultados mostraron un alto nivel de expresión de la proteína E165R predominantemente en forma soluble, con una masa molecular de aproximadamente 38 kDa (Figura 1A). Después de la expresión, la proteína recombinante E165R se purificó utilizando Ni Sepharose 6FF, lo que resultó en una pureza superior al 90% (Figura 1B). Posteriormente, se confirmó la identidad de la proteína recombinante E165R mediante Western blot, que demostró claramente su interacción específica con los mAbs de la marca His (Figura 1C).
3.2 Preparación de mAbs frente a la proteína E165R
Los ratones BALB/c fueron inmunizados con la proteína E165R purificada. Después del procedimiento de inmunización (Figura 2A), se recolectó sangre de las venas de cola al 7º día después de la tercera inmunización. La potencia de los anticuerpos séricos se midió mediante ELISA, los resultados mostraron que el título de anticuerpos del suero de ratón fue de 1:5,12 × 105, lo que demuestra la producción exitosa de anticuerpos específicos contra la proteína E165R del virus de la PPA (Figura 2B). Se obtuvieron tres líneas celulares de hibridoma mAb de proteína E165R estables dirigidas al crecimiento, denominadas 1B7, 1B8 y 10B8, mediante métodos de subclonación de dilución limitada y ELISA.
3.3 Tres mAbs mostraron una fuerte reactividad con la proteína recombinante E165R
La especificidad de los mAbs se evaluó infectando células Sf9 con el baculovirus recombinante Ac MultiBac-E165R mediante análisis de Western blot e IFA. Los resultados de IFA demostraron fluorescencia verde específica, lo que indica que los mAbs generados reconocieron específicamente la proteína E165R expresada en células Sf9 (Figura 3A). Además, los resultados de la Figura 3B confirmaron que los tres mAbs eran capaces de reconocer las proteínas E165R.
3.4 Identificación de la potencia y el subtipo de tres mAbs
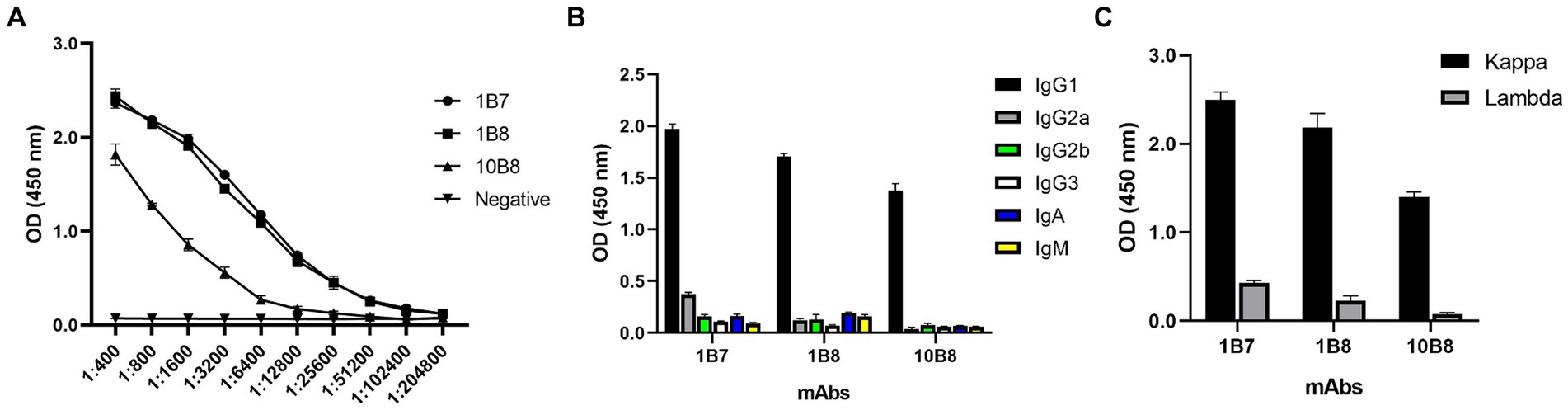
Para revelar aún más las características de los tres mAbs, la potencia de los mAbs se determinó mediante ELISA indirecto. Se preparó un gran número de anticuerpos monoclonales por inducción de ascitis y el título se midió de la siguiente manera: 1B7 (1:1,024 × 105), 1B8 (1:1,024 × 105) y 10B8 (1:1,28 ×10 4) (Figura 4A). Además, se utilizó un kit comercial para confirmar que todos los mAbs preparados pertenecían al subtipo IgG1 (Figura 4B) y contenían la cadena ligera kappa (Figura 4C).
3.5 Mapeo de epítopos de los tres mAbs
El mapeo de epítopos de tres mAbs se determinó mediante Western blot, dot-blot y peptide-ELISA. En primer lugar, diseñamos y expresamos cuatro péptidos E165R truncados superpuestos (Figura 5A). Para garantizar la detección, los fragmentos truncados se expresaron utilizando el vector pET32a, que incorpora una secuencia de etiquetas trxA de 11,8 kD en el extremo N. El análisis de Western blot demostró que las proteínas A, B y C fueron reconocidas por 1B7 y 1B8, mientras que las proteínas A, B, C y D fueron reconocidas por 10B8. Estos hallazgos indicaron que las regiones reconocidas por 1B7, 1B8 y estaban entre 1-58 aa, mientras que la región reconocida por 10B8 estaba entre 58-65 aa (Figura 5B).
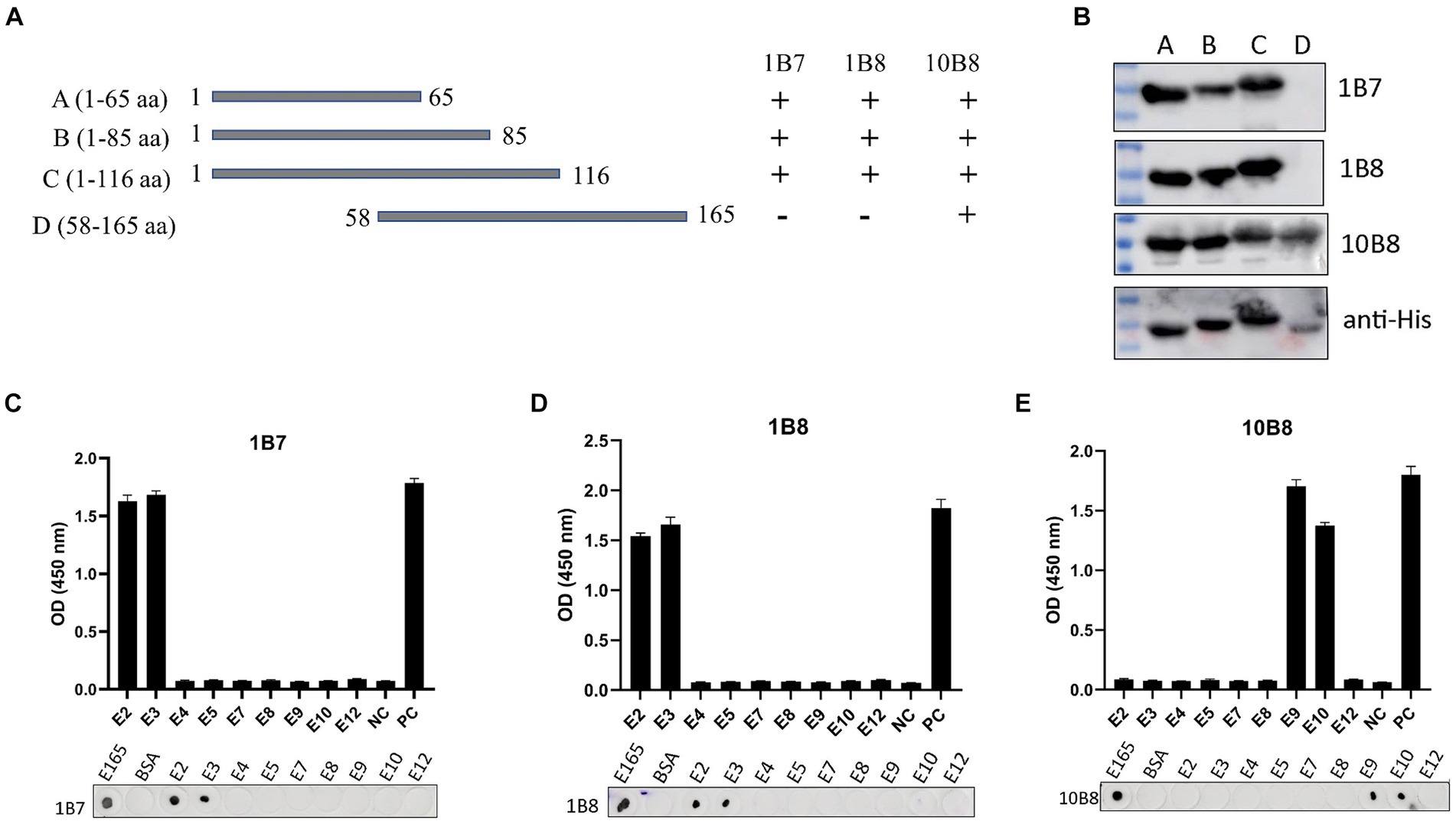
Posteriormente, diseñamos doce péptidos cortos, cada uno de los cuales contenía 6 aminoácidos superpuestos (Tabla 2). Sin embargo, solo nueve de estos péptidos se sintetizaron con éxito debido a sus propiedades hidrofóbicas. Se validó aún más mediante dot-blot y peptide-ELISA. En particular, los péptidos E2 y E3 fueron reconocidos por 1B7 y 1B8 (Figuras 5C,D), y los péptidos E9 y E10 fueron reconocidos por 10B8 (Figura 5E). Como resultado, se determinó que los sitios de reconocimiento de 1B7 y 1B8 fueron 13EAEAYYPPSV22, y 10B8 fue 55VACEHMGKKC64.
3.6 Análisis conservador de los epítopos de mAb
Se realizó un análisis de alineamiento para evaluar la conservación de los epítopos identificados entre 35 aislados de PPA pertenecientes a seis genotipos. Los resultados del alineamiento de aminoácidos que se muestran en la Figura 6A, los epítopos de 13EAEAYYPPSV22 estaban altamente conservados en los genotipos I, II y XXII del virus de la PPA. Por el contrario, los epítopos 55VACEHMGKKC64 exhibieron una alta conservación en los genotipos I, II, VIII y XXII. El análisis del árbol filogenético confirmó que la secuencia E165R utilizada en este estudio tiene una gran similitud con la cepa epidémica de genotipo I y II (Figura 6B). Dado que los genotipos I y II son las principales cepas epidémicas de PPA en Asia y Europa, los epítopos conservados revelados en este estudio ofrecen una estrategia práctica y una opción para desarrollar métodos de detección del virus de la PPA.
Se utilizó el software PyMOL para simular la estructura de la proteína E165R y observar la distribución espacial de los epítopos identificados por los mAbs desarrollados en este estudio (Figura 6C).
4 Discusión
La PPA aguda tiene una tasa de letalidad de hasta el 100% y tiene un impacto devastador en la industria porcina. China, como principal país de cría de cerdos en el mundo, ha causado enormes pérdidas económicas debido a la PPA (25). Sin embargo, la falta de conocimiento suficiente sobre los mecanismos de las respuestas inmunitarias del huésped y del virus de la PPA ha obstaculizado el desarrollo de vacunas (26, 27). La complejidad del propio virus de la peste porcina africana y la limitada comprensión de los factores de virulencia de las diferentes cepas, así como de sus genes protectores asociados, han creado nuevos retos en los procesos de tratamiento y desarrollo de vacunas. Como resultado, aún no se han identificado vacunas preventivas seguras y eficaces ni agentes terapéuticos dirigidos (28). La aparición de nuevas características en las epidemias de PPA, incluidas cepas naturalmente atenuadas y muy virulentas, junto con el largo período de incubación de los cerdos infectados, ha dificultado aún más la detección del virus de la PPA. Por lo tanto, existe una necesidad urgente de desarrollar un reactivo de diagnóstico rápido y sensible para la PPA.
La dUTPasa es una hidrolasa esencial que se encuentra en una amplia gama de organismos, incluidos procariotas, eucariotas, virus de ADN y virus de ARN (29, 30). La investigación actual sobre la dUTPasa se centra principalmente en los virus. Estos estudios confirman que la dUTPasa desempeña un papel crucial en la facilitación de la replicación viral eficiente dentro del organismo huésped. Además, la dUTPasa ha sido identificada como un factor importante en la determinación de la virulencia viral (31, 32). Además, ciertas dUTPasas monoméricas tienen implicaciones más amplias que van más allá de la síntesis de nucleótidos y el metabolismo. Por ejemplo, se ha observado que la dUTPasa codificada por el virus de Epstein-Barr (VEB) tiene un impacto en el sistema inmunitario del huésped al promover la producción de citocinas proinflamatorias y ejercer efectos reguladores no enzimáticos (33, 34).
El gen E165R del virus de la peste porcina africana codifica una enzima dUTPasa que funciona principalmente para prevenir la incorporación incorrecta de uracilos en el ADN viral, asegurando así la fidelidad de la replicación del genoma (16, 35, 36). Esta enzima cataliza la hidrólisis del trifosfato de desoxiuridina (dUTP) en dUMP y pirofosfato, eliminando eficazmente el dUTP de la síntesis de ADN (24). Tanto la dTTP como la dUTP pueden ser incorporadas al ADN con la misma eficiencia por la ADN polimerasa durante la síntesis de ADN. Sin embargo, los altos niveles de dUTP conducen a la mala incorporación de uracilo (37), que es crucial para la viabilidad celular de todos los organismos (30, 37). Al igual que la mayoría de las dUTPasas, la dUTPasa de la PPAA exhibe una estructura trimérica con un centro enzimático activo compuesto por distintas subunidades. Se cree que la expresión de esta enzima durante las fases temprana y tardía de la infección desempeña un papel crucial en el mantenimiento de la integridad de la replicación viral (38).
El alto entorno oxidativo de los macrófagos, junto con el porcentaje significativamente mayor de dUTP en relación con dTTP, hace que el virus de la PPA dependa en gran medida de su dUTPasa codificada (24). La enzima dUTPasa desempeña un papel crucial en el mantenimiento de la fidelidad e integridad del genoma viral del virus de la PPA. Oliveros et al. (38) demostraron que la dUTPasa influye en la replicación eficiente de la PPA en macrófagos porcinos mediante la generación de mutantes de deleción de la dUTPasa de la PPA. Además, la dUTPasa del VEB regula al alza la expresión de TNF-α e IL-6 a través de la vía de señalización NF-κB (17, 18). Se ha demostrado que el tratamiento de las células cancerosas con el inhibidor de 2-desoxi-5 TAS-114, en combinación con 2-desoxi-5-fluorouracilo (FDURD), aumenta los niveles de FDUTP y dUTP, así como la incorporación incorrecta de 5-FU y uracilo, lo que resulta en un aumento de la mortalidad de las células cancerosas (39-41). Por lo tanto, la inhibición de la dUTPasa aumenta la actividad antitumoral de las fluoropirimidinas. Además, la dUTPasa está fuertemente correlacionada con la virulencia de la PPA y su alto nivel de eficiencia de replicación (19, 42). Aunque las proteínas dUTPasa de diferentes orígenes de especies tienen diferentes pesos moleculares, todas contienen secuencias de aminoácidos de cinco motivos típicamente conservados (motivo I-V), con el motivo III generalmente considerado el centro catalítico de la actividad enzimática de la dUTPasa (43).
La proteína recombinante E165R, producida en este estudio, exhibe altos niveles de expresión y solubilidad, lo que facilita la preservación de su estructura natural. Además, los mAbs exhiben una fuerte reactividad hacia la proteína E165R expresada heterólogamente. Posteriormente, los epítopos de mAb se reconocieron utilizando proteínas E165R truncadas superpuestas en combinación con análisis de escaneo de péptidos. Desafortunadamente, los mAbs no logran una actividad viral de neutralización completa. En este estudio, se examinaron dos nuevos epítopos antigénicos conservados: 13EAEAYYPPSV22 y 55VACEHMGKKC64. El epítopo 55VACEHMGKKC64 se encuentra en la región conservada del Motivo I de la enzima dUTPasa. Dado que el E165R se expresa en gran medida en las primeras etapas de la infección por el virus de la PPA, los anticuerpos dirigidos a esta región conservada son importantes para la detección temprana del virus. Analizando la conservación de la secuencia de esta región, el epítopo 13EAEAYYPPSV22, reconocido por los mAbs 1B7 y 1B8, está altamente conservado en los genotipos I, II y XXII del virus de la PPA, lo que lo convierte en un valioso anticuerpo de detección con amplia aplicabilidad. De manera similar, la región 55VACEHMGKKC64 reconocida por mAb 10B8 está altamente conservada en los genotipos I, II, VIII y XXIII, y con una diferencia de bases de 1-2 en los genotipos IX y X. En consecuencia, la información de epítopos obtenida en este estudio tiene implicaciones significativas para el diagnóstico futuro de la PPA.
En conclusión, obtuvimos altos niveles de expresión y alta solubilidad de la proteína E165R utilizando el sistema de expresión procariota de E. coli. Además, examinamos tres cepas de mAbs que se dirigen específicamente al virus de la peste porcina africana E165R, lo que dio como resultado la identificación de dos nuevos epítopos conservados. Estos hallazgos ofrecen una valiosa base teórica para el desarrollo futuro de reactivos de diagnóstico.
Declaración de disponibilidad de datos
Los datos brutos que respaldan las conclusiones de este artículo serán puestos a disposición por los autores, sin reservas indebidas.
Declaración de ética
El estudio en animales fue aprobado por el Comité de Cuidado Animal de la Universidad Normal de Nanyang, China. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
JH: Conceptualización, Curación de datos, Análisis formal, Metodología, Redacción – borrador original, Redacción – revisión y edición. JL: Conceptualización, Curación de datos, Análisis formal, Metodología, Redacción – borrador original. ML: Análisis formal, Metodología, Redacción – borrador original. YL: Curación de datos, Supervisión, Redacción – borrador original. JS: Conceptualización, Supervisión, Redacción – revisión y edición. LY: Conceptualización, Obtención de fondos, Recursos, Supervisión, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Esta investigación fue financiada por el Proyecto de Talento Líder en Innovación Científica y Tecnología de las llanuras centrales de China (244200510040), el Proyecto de Investigación y Práctica sobre la Reforma de la Enseñanza basada en la Investigación en las Universidades de Pregrado de la Provincia de Henan (2022SYJXLX079) y el Proyecto Principal de Ciencia y Tecnología de la Provincia de Henan seleccionado por «Licitación abierta para seleccionar a los mejores candidatos» (211110110700).
Reconocimientos
Agradecemos al Laboratorio Clave de Inmunología Animal de la Academia de Ciencias Agrícolas de Henan por el regalo de las células SP2/0.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Urbano, AC, y Ferreira, F. Control y prevención de la peste porcina africana: una actualización sobre el desarrollo de vacunas. Los microbios emergentes infectan. (2022) 11:2021–33. doi: 10.1080/22221751.2022.2108342
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Mebus, CA. Peste porcina africana. adv virus res. (1988) 35:251–69. doi: 10.1016/s0065-3527(08)60714-9
3. Penrith, ML y Vosloo, W. Revisión de la peste porcina africana: transmisión, propagación y control. J S Afr Vet Assoc. (2009) 80:58–62. doi: 10.4102/jsava.v80i2.172
4. Zhou, X, Li, N, Luo, Y, Liu, Y, Miao, F, Chen, T, et al. Aparición de la peste porcina africana en China, 2018. Transbound emerg dis. (2018) 65:1482–4. doi: 10.1111/tbed.12989
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Dixon, LK, Stahl, K, Jori, F, Vial, L y Pfeiffer, DU. Epidemiología y control de la peste porcina africana. Annu Rev Anim Biosci. (2020) 8:221–46. doi: 10.1146/annurev-animal-021419-083741
6. Rojo, G, García-Beato, R, Viñuela, E, Salas, ML y Salas, J. Replicación del ADN del virus de la peste porcina africana en células infectadas. Virología. (1999) 257:524–36. doi: 10.1006/viro.1999.9704
7. Salas, ML, y Andrés, G. Morfogénesis del virus de la peste porcina africana. Res. de Virus. (2013) 173:29–41. doi: 10.1016/j.virusres.2012.09.016
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Gaudreault, NN, Madden, DW, Wilson, WC, Trujillo, JD y Richt, JA. Virus de la peste porcina africana: un arbovirus de ADN emergente. Veterinario delantero Sci. (2020) 7:215. doi: 10.3389/fvets.2020.00215
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Qu, H, Ge, S, Zhang, Y, Wu, X y Wang, Z. Una revisión sistemática de los genotipos y serogrupos del virus de la peste porcina africana. Genes de virus. (2022) 58:77–87. doi: 10.1007/s11262-021-01879-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Bao, J, Wang, Q, Lin, P, Liu, C, Li, L, Wu, X, et al. Comparación del genoma de la cepa del virus de la peste porcina africana China/2018/AnhuiXCGQ y las cepas europeas relacionadas del genotipo II p72. Transbound emerg dis. (2019) 66:1167–76. doi: 10.1111/tbed.13124
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Zhang, Y, Chen, Y, Zhou, J, Wang, X, Ma, L, Li, J, et al. Virus de la diarrea epidémica porcina: una visión actualizada de la epidemiología del virus, los patrones de variación de la virulencia y las interacciones virus-huésped. Virus. (2022) 14:2434. doi: 10.3390/v14112434
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Liu, S, Luo, Y, Wang, Y, Li, S, Zhao, Z, Bi, Y, et al. Estructura crio-EM del virus de la peste porcina africana. Microbio huésped celular. (2019) 26:836–843.e3. doi: 10.1016/j.chom.2019.11.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Sánchez, EG, Pérez-Núñez, D, y Revilla, Y. Mecanismos de entrada y vía endosomal del virus de la peste porcina africana. Vacunas (Basilea). (2017) 5:42. doi: 10.3390/vaccines5040042
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Andrés, G. El virus de la peste porcina africana se desnuda: nuevos conocimientos sobre la vía de entrada. J Virol. (2017) 91:E01906–16. doi: 10.1128/jvi.01906-16
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Jouvenet, N, Monaghan, P, Way, M y Wileman, T. El transporte del virus de la peste porcina africana desde los sitios de ensamblaje hasta la membrana plasmática depende de los microtúbulos y la kinesina convencional. J Virol. (2004) 78:7990–8001. doi: 10.1128/JVI.78.15.7990-8001.2004
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Li, G, Wang, C, Yang, M, Cao, L, Fu, D, Liu, X, et al. El conocimiento estructural de la dUTPasa del virus de la peste porcina africana revela un nuevo patrón de plegamiento en la familia de las dUTPasa. J Virol. (2020) 94:E01698–19. doi: 10.1128/JVI.01698-19
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Waldman, WJ, Williams, MV Jr, Lemeshow, S, Binkley, P, Guttridge, D, Kiecolt-Glaser, JK, et al. La dUTPasa codificada por el virus de Epstein-Barr aumenta la producción de citocinas proinflamatorias por parte de los macrófagos en contacto con las células endoteliales: evidencia de riesgo aterosclerótico inducido por depresión. El cerebro se comporta inmune. (2008) 22:215–23. doi: 10.1016/j.bbi.2007.07.007
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Ariza, ME, Glaser, R, y Williams, MV. DUTPasas codificadas por herpesvirus humanos: familia de proteínas que modulan la función de las células dendríticas y la inmunidad innata. Microbiol frontal. (2014) 5:504. doi: 10.3389/fmicb.2014.00504
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Ariza, ME, Glaser, R, Kaumaya, PT, Jones, C, y Williams, MV. La dUTPasa codificada por EBV activa NF-kappa B a través de la vía de señalización dependiente de TLR2 y MyD88. J Immunol. (2009) 182:851–9. doi: 10.4049/jimmunol.182.2.851
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Alejo, A, Matamoros, T, Guerra, M, y Andrés, G. Un atlas proteómico de la partícula del virus de la peste porcina africana. J Virol. (2018) 92:e01293–18. doi: 10.1128/jvi.01293-18
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Oliveros, M, Yáñez, RJ, Salas, ML, Salas, J, Viñuela, E y Blanco, L. Caracterización de una ADN polimerasa de 20 kDa del virus de la peste porcina africana involucrada en la reparación del ADN. J Biol Chem. (1997) 272:30899–910. doi: 10.1074/jbc.272.49.30899
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Vuono, EA, Ramírez-Medina, E, Pruitt, S, Rai, A, Espinoza, N, Silva, E, et al. La deleción del gen E165R de la dUTPasa del virus de la PPA Peste Porcina Africana altamente virulento Georgia 2010 no afecta a la replicación ni a la virulencia del virus en cerdos domésticos. Virus. (2022) 14:1409. doi: 10.3390/v14071409
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Zhang, S, Wang, R, Zhu, X, Jin, J, Lu, W, Zhao, X, et al. Identificación y caracterización de un nuevo epítopo de dUTPasa codificada por PPA mediante anticuerpos monoclonales. Virus. (2021) 13:2175. doi: 10.3390/v13112175
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Liang, R, Wang, G, Zhang, D, Ye, G, Li, M, Shi, Y, et al. Las comparaciones estructurales de las dUTPasas del huésped y del virus de la peste porcina africana revelan nuevas pistas para el desarrollo de inhibidores. J Biol Chem. (2021) 296:100015. DOI: 10.1074/jbc. RA120.014005
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Li, Z, Chen, W, Qiu, Z, Li, Y, Fan, J, Wu, K, et al. Virus de la peste porcina africana: una revisión. Vida (Basilea). (2022) 12:1255. doi: 10.3390/life12081255
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Teklue, T, Sun, Y, Abid, M, Luo, Y y Qiu, HJ. Situación actual y evolución de los enfoques para el desarrollo de vacunas contra la peste porcina africana. Transbound emerg dis. (2020) 67:529–42. doi: 10.1111/tbed.13364
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Zhu, JJ. Vacunología de la peste porcina africana: los desafíos biológicos desde perspectivas inmunológicas. Virus. (2022) 14:2021. doi: 10.3390/v14092021
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Blome, S, Franzke, K, y Beer, M. Peste porcina africana: una revisión del conocimiento actual. Res. de Virus. (2020) 287:198099. doi: 10.1016/j.virusres.2020.198099
29. Nyíri, K, Mertens, HDT, Tihanyi, B, Nagy, GN, Kőhegyi, B, Matejka, J, et al. Modelo estructural de dUTPasa humana en complejo con un nuevo inhibidor proteico. Sci Rep. (2018) 8:4326. doi: 10.1038/s41598-018-22145-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Kumar, H, Kehrer, J, Singer, M, Reinig, M, Santos, JM, Mair, GR, et al. Evaluación genética funcional de las enzimas de limpieza del ADN en el parásito de la malaria: la dUTPasa y la Ap4AH son esenciales en el plasmodium berghei, pero la ITPasa y la NDH son prescindibles. Expertos en la óptica de otros objetivos. (2019) 23:251–61. doi: 10.1080/14728222.2019.1575810
31. Ladner, RD, McNulty, DE, Carr, SA, Roberts, GD, y Caradonna, SJ. Caracterización de distintas formas nucleares y mitocondriales de la desoxiuridina trifosfato nucleotidohidrolasa humana. J Biol Chem. (1996) 271:7745–51. doi: 10.1074/jbc.271.13.7745
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Saltarelli, M, Querat, G, Konings, DA, Vigne, R, y Clements, JE. Secuencia de nucleótidos y análisis transcripcional de clones moleculares de CAEV que generan virus infecciosos. Virología. (1990) 179:347–64. doi: 10.1016/0042-6822(90)90303-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Ariza, ME, Rivailler, P, Glaser, R, Chen, M, y Williams, MV. Los exosomas que contienen dUTPasa codificada por el virus de Epstein-Barr modulan las respuestas inmunitarias innatas y adaptativas en células dendríticas humanas y células mononucleares de sangre periférica. PLoS Uno. (2013) 8:E69827. doi: 10.1371/journal.pone.0069827
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Williams Ph, DM, Cox, B, Lafuse Ph, DW y Ariza, ME. La dUTPasa del virus de Epstein-Barr induce mediadores neuroinflamatorios: implicaciones para la encefalomielitis miálgica/síndrome de fatiga crónica. Clin Ther. (2019) 41:848–63. doi: 10.1016/j.clinthera.2019.04.009
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Li, C, Chai, Y, Song, H, Weng, C, Qi, J, Sun, Y, et al. La estructura cristalina de la dUTPasa del virus de la peste porcina africana revela un posible objetivo farmacológico. MBio. (2019) 10:e02483–19. doi: 10.1128/mBio.02483-19
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Payne, SL, y Elder, JH. El papel de las dUTPasas retrovirales en la replicación y virulencia. Curr Protein Pept Sci. (2001) 2:381–8. doi: 10.2174/1389203013381008
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Vodenkova, S, Buchler, T, Cervena, K, Veskrnova, V, Vodicka, P y Vymetalkova, V. 5-fluorouracilo y otras fluoropirimidinas en el cáncer colorrectal: pasado, presente y futuro. Pharmacol Ther. (2020) 206:107447. doi: 10.1016/j.pharmthera.2019.107447
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Oliveros, M, García-Escudero, R, Alejo, A, Viñuela, E, Salas, ML y Salas, J. La dUTPasa del virus de la peste porcina africana es una enzima altamente específica necesaria para una replicación eficiente en macrófagos porcinos. J Virol. (1999) 73:8934–43. doi: 10.1128/JVI.73.11.8934-8943.1999
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Diasio, RB, y Harris, BE. Farmacología clínica del 5-fluorouracilo. Clin Farmacokinet. (1989) 16:215–37. doi: 10.2165/00003088-198916040-00002
40. Yamamoto, N, Hayashi, H, Planchard, D, Morán, T, Gregorc, V, Dowell, J, et al. Estudio aleatorizado de fase 2 del inhibidor trifosfatasa de la desoxiuridina, TAS-114, en combinación con S-1 versus S-1 solo en pacientes con cáncer de pulmón de células no pequeñas avanzado. Invertir en nuevos fármacos. (2020) 38:1588–97. doi: 10.1007/s10637-020-00930-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Yokogawa, T, Yano, W, Tsukioka, S, Osada, A, Wakasa, T, Ueno, H, et al. La inhibición de la dUTPasa confiere susceptibilidad a un inhibidor de la timidilato sintasa en células cancerosas humanas defectuosas en la reparación del ADN. Cáncer Sci. (2021) 112:422–32. doi: 10.1111/cas.14718
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Leang, RS, Wu, TT, Hwang, S, Liang, LT, Tong, L, Truong, JT, et al. La actividad antiinterferón de la dUTPasa viral ORF54 conservada es esencial para una infección efectiva por MHV-68. PLoS Pathog. (2011) 7:E1002292. doi: 10.1371/journal.ppat.1002292
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. McGeoch, DJ. Las comparaciones de secuencias de proteínas muestran que las «pseudoproteasas» codificadas por los poxvirus y ciertos retrovirus pertenecen a la familia de las desoxiuridina trifosfatasas. Ácidos nucleicos Res. (1990) 18:4105–10. doi: 10.1093/nar/18.14.4105
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: Peste porcina africana, virus de la peste porcina africana, proteína E165R, anticuerpo monoclonal, epítopo conservado
Cita: He J, Li J, Luo M, Liu Y, Sun J y Yao L (2024) Identificación de dos nuevos epítopos lineales en la proteína E165R del virus de la peste porcina africana reconocidos por anticuerpos monoclonales. Frente. Vet. Sci. 11:1392350. doi: 10.3389/fvets.2024.1392350
Recibido: 27 de febrero de 2024; Aceptado: 24 de julio de 2024;
Publicado: 06 de agosto de 2024.
Editado por:
Francisco José Pallarés, Universidad de Córdoba, España
Revisado por:
Verónica Martín García, Directora del Departamento de Anatomía Patológica, Inmunología y Control de Enfermedades Infecciosas (CISA-INIA-CSIC), España
Tong-Qing An, Academia China de Ciencias Agrícolas, China
Derechos de autor © 2024 Él, Li, Luo, Liu, Sun y Yao. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Jingchen Sun, cyfz@scau.edu.cn; Lunguang Yao, lunguangyao@163.com
†Estos autores han contribuido igualmente a este trabajo
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía