La especificidad de la escisión de protamina del patógeno aviar Escherichia coli OmpT revela dos sitios de unión al sustrato relacionados con la virulencia
 Juanhua Liu1,2
Juanhua Liu1,2 Luyao Jiang1,2
Luyao Jiang1,2 Cuelga a Wang1,2
Cuelga a Wang1,2 Jiayan Wu1,2
Jiayan Wu1,2 Qingqing Gao1,2
Qingqing Gao1,2 Changchao Huan1,2
Changchao Huan1,2 Song Gao1,2*
Song Gao1,2*- 1Centro de Co-Innovación de Jiangsu para la Prevención y el Control de Enfermedades Infecciosas Animales Importantes y Zoonosis, Facultad de Medicina Veterinaria, Universidad de Yangzhou, Yangzhou, China
- número arábigoLaboratorio Conjunto Internacional para la Cooperación en Agricultura e Inocuidad de Productos Agrícolas, Ministerio de Educación, Yangzhou (China)
La naturaleza patógena de las bacterias se puede aumentar mediante la escisión de péptidos antimicrobianos mediante omptinas, para evitar o contrarrestar las defensas inmunitarias naturales del huésped. El OmpT codificado por plásmidos (pOmpT o ArlC) en la Escherichia coli patógena aviar (APEC), al igual que el OmpT codificado por cromosomas (cOmpT), pertenece a la familia de las omptinas y ambos exhiben secuencias y estructuras muy similares. A través de la alineación de secuencias y exámenes fisiológicos, se ha identificado pOmpT como un factor de virulencia, distinto de cOmpT en términos de especificidad del sustrato. Cuando se compara pOmpT con cOmpT en cuanto a sus actividades proteolíticas y sustratos objetivo, se encuentra que Asp267 y Ser276 en el bucle 5 de cOmpT son sitios de unión que facilitan el anclaje del sustrato y mejoran la escisión del sustrato (protamina o péptido sintético) por el centro catalítico. Por el contrario, las características de los residuos en las posiciones 267 y 276 del bucle 5 de pOmpT inhiben la escisión de la protamina, pero permiten la escisión específica del péptido antimicrobiano humano RNasa 7, que desempeña un papel en la defensa del huésped. Este hallazgo sugiere una relación entre estos dos sitios de unión y la especificidad del sustrato. Además, se determina que los sitios de unión al sustrato (residuos 267 y 276, en particular el residuo 267) de cOmpT y pOmpT son críticos en la virulencia de APEC. En resumen, los residuos 267 y 276 de pOmpT son cruciales para la patogenicidad de APEC y ofrecen nuevos conocimientos sobre los determinantes de la virulencia de APEC y el desarrollo de fármacos antimicrobianos.
1 Introducción
Escherichia coli (E. coli) es una bacteria común que se encuentra en el tracto gastrointestinal de mamíferos y aves, pero también puede causar una variedad de enfermedades graves en humanos y animales debido a su naturaleza patógena multifuncional. En las aves de corral, la E. coli patógena aviar (APEC), una E. coli patógena extraintestinal (ExPEC), que actúa como agente primario o secundario, puede infectar a todo tipo de aves a todas las edades, causando infecciones localizadas y sistémicas, a menudo denominadas colibacilosis aviar. En la actualidad, la colibacilosis aviar es una de las principales causas de mortalidad y morbilidad asociadas a pérdidas económicas en la industria avícola en todo el mundo. El desarrollo de resistencia bacteriana a los antibióticos y la ineficacia de las vacunas plantean riesgos significativos para la salud pública y la industria avícola. En consecuencia, existe una necesidad urgente de estrategias de prevención y tratamiento más eficaces.
Los péptidos antimicrobianos (AMP) representan uno de los principales desafíos a los que se enfrentan los patógenos bacterianos al infectar a un huésped, desempeñando un papel crucial en el sistema inmunitario innato (1). Estos péptidos, de tamaño pequeño (20-50 aminoácidos), catiónicos y anfifílicos, son secretados principalmente por las células epiteliales y los neutrófilos del huésped (2-4). Su capacidad para adherirse a las membranas bacterianas aniónicas, crear poros y, en consecuencia, causar la muerte bacteriana a través de la lisis celular está bien documentada (5-7). Más allá de sus capacidades bactericidas, los AMP también tienen la capacidad de atraer las células inmunitarias del huésped a los sitios de infección, induciendo así un amplio espectro de actividades inmunomoduladoras para controlar las infecciones bacterianas (8).
A pesar del papel fundamental de los AMP en el mecanismo de defensa del huésped, las bacterias han desarrollado numerosas estrategias para contrarrestar los efectos de los AMP, como la utilización de modificaciones de LPS, bombas de eflujo, cápsulas y proteasas (1). Las omptinas, un grupo distinto de proteasas de membrana externa (MO) con capacidades proteolíticas, han atraído una atención significativa. Estas proteasas, que se encuentran en varias bacterias gramnegativas de la familia Enterobacteriaceae, influyen en la virulencia bacteriana a través de la modificación o degradación de numerosas proteínas bacterianas y del huésped (9-19). Las omptinas identificadas actualmente incluyen OmpT, OmpP y ArlC en E. coli, Pla en Yersinia pestis, PgtE en Salmonella enteritidis, IcsP en Shigella flexneri y CroP en Citrobacter rodentium. Comparten un alto grado de similitud en la secuencia de aminoácidos (45-80%) con sitios activos altamente conservados (11, 19-24). OmpT, la primera en ser caracterizada y codificada por el cromosoma E. coli, es una proteína de 37 kDa que forma una estructura hueca de β barril con sus sitios activos expuestos al medio externo (24, 25). Los estudios inhibitorios sobre omptina han demostrado que elementos como el Zn2+, el Cu2+ y la benzamidina pueden dificultar la actividad de OmpT (26-28), mientras que los inhibidores de la serina proteasa aprotinina y ulinastatina también pueden alterar la funcionalidad de OmpT (29, 30). Además, la estructura altamente conservada de OmpT y la naturaleza de sus residuos de sitio activo (residuos Asp83, Asp85, Asp210 y His212) son cruciales para su especificidad de escisión del sustrato, a menudo prefiriendo motivos dibásicos (24, 31-33). Se ha observado que OmpT en E. coli patógena urinaria (UPEC) ayuda a la supervivencia bacteriana dentro del huésped al escindir los péptidos antimicrobianos secretados como la protamina P1 y la catelicidina LL-37 de las células epiteliales uretrales humanas (13, 34-36). Además, la OmpT presente en E. coli enterohemorrágica (EHEC) y E. coli enteropatógena (EPEC) puede escindir e inactivar LL-37 en diversos grados (12, 37). Se ha demostrado que la proteasa OmpP codificada por plásmidos, que se encuentra en aislados de infecciones del tracto urinario (ITU), escinde la protamina AMP, y ArlC está relacionada con la resistencia a AMP (p. ej., ARNasa 7 de AMP humana); por lo tanto, ayuda a la supervivencia bacteriana (23, 38, 39).
Las omptinas, como factores de virulencia clave que desempeñan un papel central en la interfaz huésped, son objetivos potenciales para el desarrollo de antimicrobianos y vacunas, por lo que tienen potencial de investigación. De hecho, para la mayoría de las E. coli, OmpT solo está codificada por el gen ompT en el cromosoma. Los estudios previos se han centrado principalmente en la actividad proteolítica y la patogenicidad de OmpT localizada en el cromosoma (24, 25, 32, 40). Encontramos que la cepa E058 patógena aviar (APEC) tiene tanto el gen OmpT (cOmpT) (compT) en su cromosoma como el gen OmpT (pOmpT) codificado por el plásmido ColV (pompT), los cuales exhiben secuencias de aminoácidos altamente homólogas de hasta aproximadamente el 76 por ciento y están involucrados en la patogenicidad de APEC en el huésped (40, 41). Aunque la cOmpT ha sido ampliamente estudiada, la investigación sobre la pOmpT es relativamente escasa. Aquí, nuestro objetivo fue explorar el papel especial de la pOmpT en la fisiología en comparación con la de la cOmpT y aclarar el efecto de la pOmpT en la patogenicidad de APEC tanto in vitro como in vivo. El estudio en profundidad de proteasas como OmpT es propicio para el diseño de nuevos péptidos antimicrobianos resistentes a las enzimas y ofrece la posibilidad de explorar nuevos enfoques antiinfecciosos y el cribado de nuevas dianas farmacológicas.
2 Materiales y métodos
2.1 Cepas bacterianas, plásmidos, anticuerpos y condiciones de crecimiento
Las cepas bacterianas y plásmidos utilizados en este estudio se enumeran en la Tabla Suplementaria S1. Los cebadores de oligonucleótidos utilizados se enumeran en la Tabla Suplementaria S2. La cepa E058 de APEC se aisló de un pollo con los síntomas clínicos típicos de colibacilosis en China (42). El anticuerpo monoclonal OmpT de origen mratón fue desarrollado por nuestro laboratorio (43). Los demás detalles relacionados con las cepas son los siguientes. Las cepas se cultivaron en caldo Luria-Bertani (LB), en placas de agar LB o en medio N-mínimo (44) ajustado a pH 7.5 y suplementado con 0.2% de glucosa y 1 mM MgCl2. Se utilizaron antibióticos como 50 μg/mL de kanamicina, 50 μg/mL de espectinomicina, 30 μg/mL de cloranfenicol o 50 μg/mL de tetraciclina para la selección donde fue necesario. Todos los cultivos se cultivaron a 37°C o 30°C en condiciones aeróbicas.
2.2 Generación de bacterias mutantes, revertentes y de expresión recombinante
Las cepas bacterianas utilizadas en este estudio se enumeran en la Tabla Suplementaria S1. Las deleciones de genes simples/dobles de los genes compT y pompT de la cepa APEC E058 se realizaron utilizando el sistema de recombinasa roja lambda (41). Simultáneamente, los genes nativos compT y pompT, junto con sus promotores putativos, fueron amplificados, clonados e insertados en el plásmido pACYC184. Tras la PCR y la secuenciación del ADN, los plásmidos p184-ccompT y p184-ppompT se transformaron en las cepas de deleción de un solo gen compT/pompT para crear cepas de complementación mediante electroporación.
Para minimizar la interferencia de proteínas de membrana externa distintas de la proteína cOmpT/pOmpT en la cepa E058, se clonó todo el marco de lectura abierto (ORF) de los genes compT y pompT y se insertó en el plásmido de expresión pET-28a. Posteriormente, los plásmidos correctos se transformaron en E. coli BL21(DE3), que naturalmente carece del gen ompT, después de la PCR y la secuenciación del ADN. Además, las secuencias de aminoácidos de las proteínas cOmpT y pOmpT se dividieron en cinco bucles basados en la estructura topológica de la proteína cOmpT. Empleando la tecnología de PCR por fusión, utilizamos el mismo método de clonación de genes mencionado anteriormente para construir una serie de bacterias recombinantes con los correspondientes intercambios de bucle y mutaciones dirigidas al sitio en el bucle 5 (L5). Para determinar aún más la importancia de L5 y el aminoácido en las posiciones 83/85/210/212/267/276 de cOmpT/pOmpT en la cepa de tipo salvaje E058, se debe excluir la interferencia mutua entre cOmpT y pOmpT. Por lo tanto, primero insertamos fragmentos de ADN mutantes quiméricos o dirigidos al sitio a través de la deleción génica y luego eliminamos o insertamos otro gen sobre la base de la cepa de deleción de un solo gen compT/pompT utilizando una estrategia de edición continua del genoma basada en el sistema CRISPR-Cas9 (45). La secuencia guía (secuencia N20), que se dirige a la secuencia FRT de la cepa mutante de un solo gen compT/pompT y a las secuencias de genes compT y pompT, se utilizó para construir plásmidos de la serie pTarget. El ADN del donante se amplificó en consecuencia utilizando el ADN genómico de la cepa de deleción de un solo gen compT/pompT y los plásmidos de la serie pET correctos anteriormente con intercambio de 5 bucles y mutación/intercambio de sitios del gen compT/pompT como plantillas.
La plantilla de ADN de la RNasa 7 se derivó de la muestra de hisopo nasal del experimentador. El gen de la rnasa 7 sin un péptido señal y con una etiqueta His y mutaciones dirigidas al sitio se amplificó mediante PCR superpuesta y posteriormente se clonó e insertó en el plásmido de expresión pDEST17 después de la digestión. A continuación, el plásmido correcto pDEST17-RNasa 7 se transformó en células BL21(AI) competentes (46, 47).
2.3 Aislamiento de ARN bacteriano, RT-PCR/qPCR, secuenciación y análisis de alineación
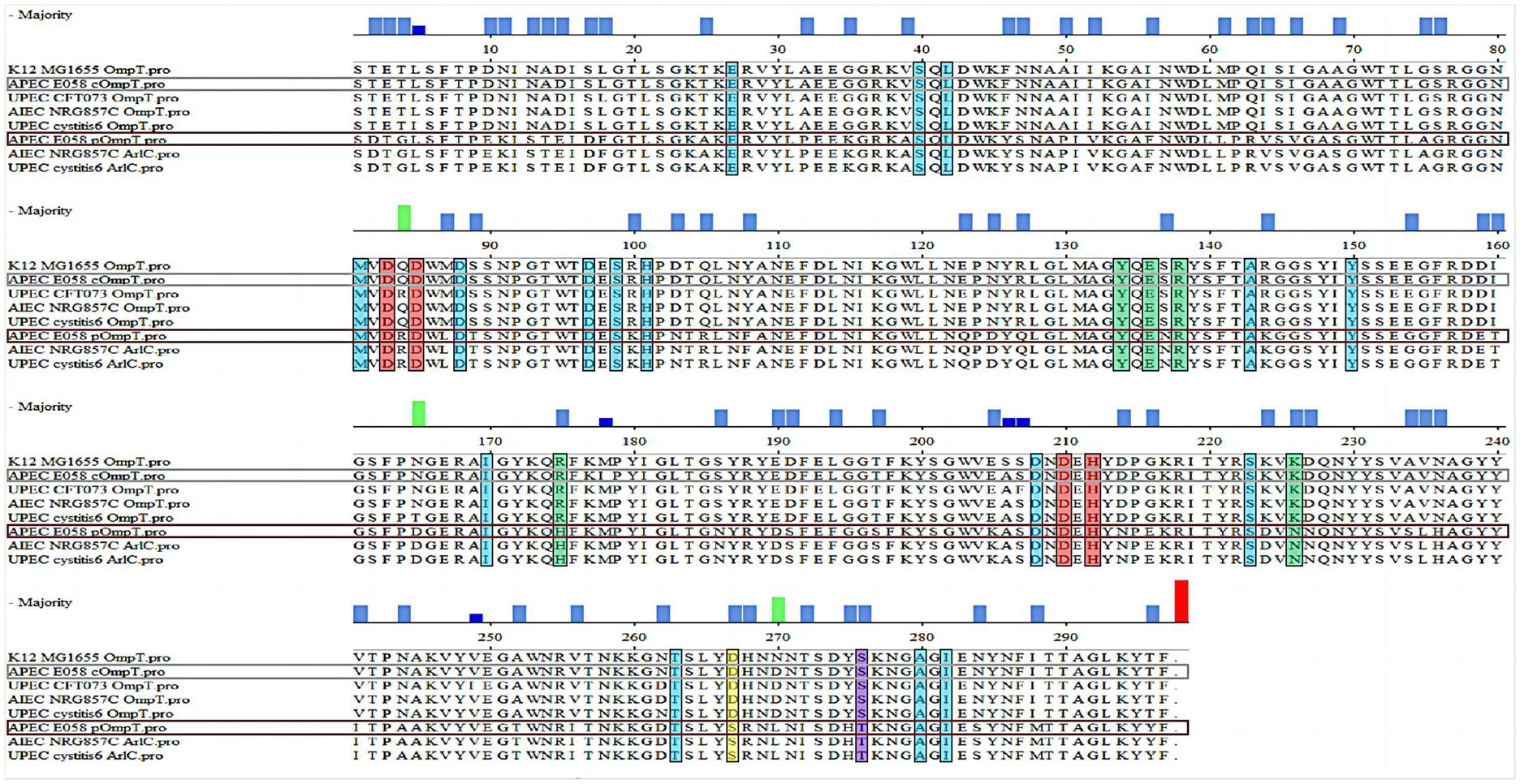
El ARN total se extrajo de la cepa E058 de APEC y se transcribió de forma inversa en ADNc utilizando el kit de reactivos PrimeScript RT (TaKaRa, China) de acuerdo con el protocolo del fabricante. Los conjuntos de cebadores para la amplificación por PCR de los genes diana compT y pompT en muestras de ADNc se detallan en la Tabla Suplementaria S2. Al mismo tiempo, las PCR se llevaron a cabo utilizando ADN de la cepa E058 como controles positivos y muestras de ADNc sin activación de transcripción inversa (RT) como controles negativos. Los productos de PCR se separaron en geles de agarosa al 0,8%. A continuación, se extrajeron los fragmentos correspondientes a los genes compT y pompT amplificados por PCR de los geles de agarosa utilizando un kit de extracción en gel de ADN de Axygen (Corning, China) y se sometieron a verificación de secuenciación. El sistema de qPCR fue seguido con ChamQ SYBR qPCR Master Mix (Vazyme, China). Las condiciones térmicas fueron las siguientes: 95 °C durante 30 s, 40 ciclos de 95 °C durante 10 s y 60 °C durante 30 s. El gen gapA se utilizó como control interno para la normalización. Los cebadores utilizados para la qRT-PCR de los genes compT y pompT se enumeran en la Tabla Suplementaria S2. Se realizó el alineamiento secuencial entre las secuencias de aminoácidos de los genes compT y pompT de APEC E058, la secuencia de aminoácidos del gen ompT localizada en el cromosoma de la cepa K12 de E. coli subcepa MG1655 (NC_000913.3), UPEC CFT073 (CP051263.1), AIEC NRG857C (CP001855.1) y UPEC cistitis aislada 6 (CP041302.1) y la secuencia de aminoácidos de la arlC gen localizado en los plásmidos AIEC NRG857C (CP001856.1) y UPEC aislado de cistitis 6 (CP041301.1) publicados en el NCBI.
2.4 Extracción de proteínas de la membrana externa
Las bacterias se cultivaron durante la noche en un medio mínimo de 200 mL de N. El aislamiento de las fracciones de la membrana externa se realizó de la siguiente manera (48): las células bacterianas se centrifugaron a 6.000 rpm durante 10 min a 4 °C, y los gránulos se resuspendieron en tampón HEPES de 7 mL (10 mM HEPES, pH 7,4) y se sonicaron. Una alícuota de 1 mL del lisado se reservó como proteína bacteriana completa para su uso posterior. A continuación, las muestras restantes se centrifugaron a 6.000 rpm durante 10 min a 4 °C. Los sobrenadantes se recolectaron y se suplementaron con 48 mL de tampón sarcosil (2% de sarcosyl), seguido de una incubación de 30 min a 4°C. Después de la centrifugación durante 1 h a 35.000 rpm, el pellet que contenía la proteína de la membrana externa se resuspendió en 1 mL de tampón A (20 mM de Tris-HCl pH 7,5, 10% glicerol) para su posterior análisis.
2.5 Western blot
Se utilizó un kit de determinación de la concentración de proteína BCA (Beyotime, China) para determinar la concentración de la proteína bacteriana total y la proteína total de la membrana externa extraídas, que posteriormente se normalizaron. Las muestras se resolvieron en un gel SDS-PAGE al 12% y se transfirieron a una membrana de fluoruro de polivinilideno. Las membranas se bloquearon durante la noche en tampón PBST [solución salina tamponada con fosfato de 10 mM (PBS, pH 7,4), 0,05% Tween-20] suplementada con leche descremada al 5% a 4 °C. A continuación, las membranas se incubaron con el anticuerpo monoclonal OmpT de origen ratón para su detección y luego con anticuerpos secundarios [IgG anti-ratón de cabra conjugada con peroxidasa de rábano picante (Beyotime, China)]. Finalmente, las membranas se expusieron a un sustrato HRP quimioluminiscente (Sharebio, China).
2.6 Expresión proteica y purificación de la RNasa 7
La RNasa 7 se expresó en células BL21(AI) de E. coli que albergan el plásmido de expresión pDEST17-RNasa 7. Los cultivos se indujeron con L-arabinosa (concentración final 2%) en medio caldo LB durante 3 h. El pellet celular se recogió por centrifugación y se resuspendió en 20 mL de tampón D (20 mM de Tris-Cl, pH 7,5). Tras la lisis celular por sonicación, el sobrenadante se recogió por centrifugación y se purificó a través de una columna de Ni-NTA (eluyente: 50 mM Na2HPO4, 0,3 M de NaCl y 250 mM de imidazol, pH 8,0) y una columna de Superdex-200 (tampón E: 20 mM de Tris-HCl, 150 mM de NaCl y 10% de glicerol, pH 8,0). La RNasa 7 se almacenó en el tampón E (20 mM de Tris-HCl, 150 mM de NaCl, 10% de glicerol) a 4 °C. La concentración de proteína, determinada por el método BCA, fue de aproximadamente 0,15 mg/mL.
2.7 Cinética de crecimiento de bacterias co-incubadas con protamina
Para estudiar la cinética de crecimiento de las bacterias incubadas con protamina, cultivamos bacterias en medios N-mínimos para evitar la posible interferencia de algunos componentes de medios complejos en la función de OmpT. Se prepararon y centrifugaron cultivos bacterianos durante la noche en medio N-mínimo. Los cultivos se resuspendieron en 10 mL de medio N-mínimo fresco y se normalizaron a un OD600 de 0,35 (una concentración final de 3,5 × 108 unidades formadoras de colonias [UFC]/mL), y luego se agregó protamina a la suspensión bacteriana a una concentración final de 100 μg/mL. La mezcla se agitó a 37 °C y se midió el diámetro exteriorde 600 cada 2 h. Los resultados se introdujeron en la curva de crecimiento de las bacterias y se utilizó el software GraphPad Prim 7 para el análisis diferencial.
2.8 Escisión proteolítica de los AMP
Las células bacterianas cultivadas en un medio N-mínimo a un diámetro exterior de600 nm de 0,6-0,8 se lavaron, se peletizaron por centrifugación, se resuspendieron en PBS (pH 7,4) y se normalizaron a una densidad bacteriana de 3 × 1010 UFC/mL. Las bacterias se combinaron en una proporción de 1:4 (v/v) con 2,5 μg/μL de protamina o en una proporción de 1:12 (v/v) con 0,15 μg/μL de ARNasa 7 para facilitar la visualización de los productos de degradación y se incubaron a 37 °C durante varios puntos de tiempo. Las bacterias se separaron de los productos de escisión de péptidos por centrifugación, y los sobrenadantes se mezclaron con tampón de muestra de tricina 2× (Beyotime, China) o tampón de carga de proteínas SDS-PAGE 5× (Yeasen Biotechnology, China), luego se hirvieron y almacenaron a -20 °C. Los productos de escisión de péptidos se calentaron a 96 °C durante 10 min y se separaron con un 16,5% de Tris-Tricine SDS-PAGE (Beyotime, China) o un 13% de SDS-PAGE. Después de la fijación durante 30 min en glutaraldehído al 5% y el posterior lavado durante 30 min con agua desionizada, los péptidos se tiñeron durante 1 h con azul de Coomassie G-250.
2.9 Ensayo de actividad de transferencia de energía por resonancia fluorescente (FRET)
El sustrato sintético FRET que contiene ácido orto-aminobenzoico (Abz) como fluoróforo y grupo 2, 4-nitrofenilo (Dnp) como extintor y un motivo dibásico (RK) en su centro (2Abz-SLGRKIQI-K(Dnp)-NH2) se compró a GL Biochem Ltd. (China)1 (25, 49). Para realizar el ensayo, las bacterias se cultivaron en un medio N-mínimo hasta la fase exponencial media y se normalizaron a un OD600 de 0,6-0,8. Las células bacterianas se centrifugaron, se resuspendieron en PBS (pH 7,4) y se normalizaron a 3 × 108 UFC/mL. Las bacterias (~2,25×10 7 UFC en 75 μL) se mezclaron en una placa de 96 pocillos con 75 μL de sustrato FRET (concentración final 40 μM). La emisión de fluorescencia se monitorizó durante 360 minutos a 25 °C utilizando un lector de placas BioTek Synergy 2 con una longitud de onda de excitación de 325 nm y una longitud de onda de emisión de 430 nm. Las mediciones iniciales de fondo se restaron de los valores finales de la muestra de reacción. Los parámetros cinéticos (Km, Kcat y Kcat/Km) se calcularon midiendo la actividad de OmpT a 0-240 μM de sustrato y ajustando la ecuación de Michaelis-Menten resultante.
2.10 Ensayo colorimétrico
Las muestras de las fracciones de membrana se diluyeron en el tampón A a concentraciones apropiadas antes de las mediciones de la actividad de OmpT. La actividad de OmpT se evaluó en un ensayo espectrofotométrico acoplado utilizando el sustrato cromogénico IAA-Arg-Arg-pNA comprado a GL Biochem Ltd. (China) (49). En un sistema de reacción de 200 μL, el ensayo incluyó 100 μg de proteína total de membrana externa, 0,5 mM de IAA-Arg-Arg-pNA, 1 mM de Tween 20, 20 mM de Mes (pH 7,0) y 0,5 U·mL-1 de aminopeptidasa M. La escisión específica de OmpT entre las dos argininas da como resultado la liberación de Arg-pNA, que posteriormente es escindida por la aminopeptidasa M (Sigma, EE. UU.), presente en exceso para garantizar que su actividad no limite la velocidad. Este proceso libera pNA, detectado espectrofotométricamente a 405 nm durante 12 h a 37 °C utilizando un lector de placas BioTek Synergy 2. Las mediciones iniciales de fondo se restaron de los valores finales de la muestra de reacción. Los datos de estos experimentos se trazaron en una curva cinética y se analizaron utilizando el software GraphPad Prism 7.
2.11 Inhibición de la actividad proteolítica
Para investigar la inhibición de la actividad proteolítica, se utilizaron tres inhibidores de la serina proteasa [PMSF (Beyotime, China), leupeptina (Beyotime, China) y aprotinina (todos de Beyotime, China)] para estudiar las funciones de Asp267 (ácido aspártico, D) y Ser276 (serina, S) en cOmpT a través de un ensayo de actividad FRET. Las bacterias cultivadas en la fase N-mínima de medio a medio exponencial se normalizaron a un ODde 600 nm de 0,6-0,8. Las células bacterianas se peletizaron por centrifugación, se resuspendieron en PBS (pH 7,4) y se ajustaron a una densidad bacteriana de 3 × 108 UFC/mL. En un sistema de reacción de 175 μL dentro de una placa de 96 pocillos, el ensayo incluyó bacterias (~2,25 × 107 UFC), 40 μM de sustrato FRET y 0,8 mM de PMSF, 0,8 mM de leupeptina o 1 mM de aprotinina. La fluorescencia (con una excitación de 325 nm y una emisión de 430 nm) se monitorizó durante 360 min a 25 °C utilizando un lector de placas BioTek Synergy 2. Las mediciones iniciales de fondo se restaron de los valores finales de la muestra de reacción. Los datos se utilizaron para construir curvas cinéticas, y el software GraphPad Prism 7 se utilizó para el análisis de expresión diferencial.
2.12 Acoplamiento molecular
El método de acoplamiento molecular se utilizó para simular las interacciones entre la protamina y la cOmpT/pOmpT y sus mutantes mediante el uso del software Autodock (50). La estructura cristalina de cOmpT (PDB ID: 1I78) se descargó de la PDB. 2 Las estructuras de los mutantes pOmpT, cOmpTD267S/S276T y pOmpTS267D/T276S se obtuvieron mediante modelado en línea utilizando un modelo suizo basado en la estructura cristalina de cOmpT. 3 Protamine (P69015) se basa en la estructura predicha de la base de datos de estructuras de proteínas Alpha Fold. 4 Se utilizó el software Autodock Tools para eliminar agua, agregar hidrógenos, calcular las cargas de Gasteiger y asignarles átomos de tipo AD4. La caja de la cuadrícula era 126 × 126 × 126 con un espaciado entre puntos de la cuadrícula de 0,7 Å y centrada en 25,946, 58,190 y 13,075 (x, y, z). Se utilizó AutoDock para acoplar la protamina con cOmpT, pOmpT y sus mutantes dobles, y las posibles conformaciones de unión se identificaron mediante un algoritmo genético. Los parámetros del algoritmo genético incluían un tamaño de población de 150, un número máximo de medios de 2.500.000, un número máximo de generaciones de 27.000, un modo de cruce de GA de dos puntos y un número de series de GA de 10. Se utilizó una función de puntuación para evaluar la energía libre de enlace calculada, y el agrupamiento se llevó a cabo utilizando el error cuadrático medio (RMSD) entre los modos de enlace. Se eligió la conformación menos energética como el modo de unión óptimo para OmpT y protamina.
2.13 Experimento con animales
Este estudio recibió la aprobación del Comité Institucional de Cuidado y Uso de Animales (IACUC) de la Universidad de Yangzhou (Pollo: SCXK(Su)2021–0027) y se llevó a cabo de acuerdo con los Procedimientos y Directrices de Ética Animal de la República Popular China.
El ensayo LD50 se llevó a cabo en pollos libres de patógenos específicos (SPF) de 3 días de edad (White Leghorn; Jinan SPAFAS Poultry Co., Ltd., Jinan, China) para evaluar la patogenicidad de la cepa de tipo salvaje E058 y sus cepas mutantes en los genes compT y pompT. Los cultivos de la cepa de tipo silvestre y sus derivados mutantes se cultivaron hasta la fase logarítmica a 37 °C. Las bacterias de cada cepa se recolectaron, se lavaron dos veces y se suspendieron en PBS estéril (que contenía 10% de glicerol) antes de diluirse a concentraciones adecuadas (1010 o 109 UFC/mL) y luego se diluyeron aún más a 109, 108, 107, 106, 105, 104 y 103 UFC/mL, respectivamente. Seis aves de cada grupo fueron desafiadas a través del saco de aire con 0.1 mL de cada suspensión de cultivo. Al grupo simulado se le inyectó PBS estéril. Los pollos fueron monitoreados durante 7 días hasta que las tasas de supervivencia se estabilizaron. Los resultados de LD50 se estimaron utilizando el método Reed-Muench y el software estadístico IBM SPSS. La significación estadística se evaluó con la prueba t y las diferencias con valores de p <0,05 se consideraron estadísticamente significativas.
3 Resultados
3.1 pOmpT es un derivado homólogo de cOmpT
El alineamiento de la secuencia reveló que APEC E058 pOmpT compartía una secuencia de aminoácidos altamente homóloga con las de cOmpT de UPEC CFT073 y cistitis 6, E. coli adherente invasiva (AIEC) NRG857C y APEC E058 (≈ 76%) (Figura 1). En particular, el pOmpT de E058 exhibió 226 de las 297 identidades de secuencia de aminoácidos con las de cOmpT, mientras que compartió una identidad de secuencia idéntica (297/297) con las del ArlC codificado por plásmidos tanto en la cistitis UPEC 6 como en la AIEC NRG857C (Figura 1), lo que indica un origen ancestral común para estos OmpT codificados por plásmidos. La alineación sugirió que E058 cOmpT es una proteasa de membrana externa típica, similar a OmpT de las cepas E. coli K12 MG1655, UPEC CFT073, AIEC NRG857C y UPEC cistitis 6. A pesar de que pOmpT compartió solo alrededor del 76% de la identidad de secuencia con cOmpT, los residuos críticos de la proteasa OmpT entre E058 pOmpT y E058 cOmpT, incluidos los sitios catalíticos y activos, son idénticos. No obstante, dos residuos en los sitios de unión a LPS de E058 pOmpT diferían de los de las cepas de cOmpT. Además, se utilizó AlphaFold 3.0 para predecir la estructura de pOmpT en función de la secuencia de aminoácidos de E058 pOmpT, lo que indica que tanto cOmpT como E058 pOmpT exhibieron una estructura de β barril.
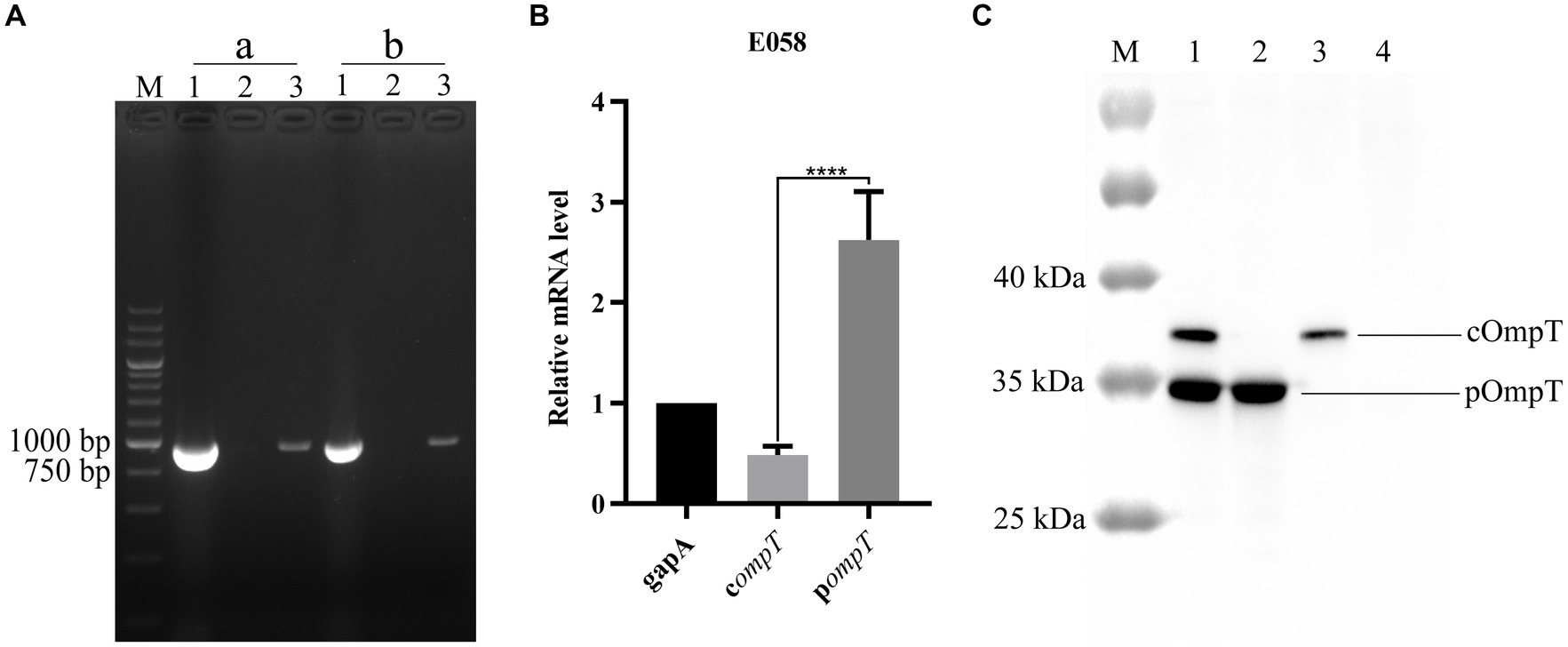
La PCR de transcripción inversa (RT-PCR) reveló que tanto los genes compT como pompT se pueden transcribir constitutivamente y tienen una longitud de 954 pb en la cepa APEC E058 (Figura 2A). Los resultados de la PCR cuantitativa mostraron que el nivel de transcripción del gen pompT fue significativamente mayor que el del gen compT (Figura 2B). No está claro si pOmpT es lo mismo que cOmpT y se encuentra en la membrana externa, aunque comparte el mismo número de nucleótidos y una secuencia de aminoácidos muy similar a la cOmpT hasta aproximadamente el 76 por ciento y también se transcribe en E058. Para determinar la ubicación de pOmpT, se extrajeron las proteínas de la membrana externa de la cepa de tipo salvaje E058 y los mutantes de deleción de genes simple/dobles compT/pompT y se detectaron mediante Western blot utilizando un anticuerpo monoclonal específico de OmpT. Los resultados mostraron que se podía detectar una sola banda en los mutantes de deleción de un solo gen E058 compT/pompT, y los tamaños de las bandas diferían entre las dos cepas, lo que sugiere que se expresaron dos proteínas diferentes en estos dos mutantes de deleción (Figura 2C). Además, se detectaron dos bandas en la cepa de tipo salvaje E058, y las posiciones de las bandas fueron consistentes con las observadas en los mutantes de deleción de un solo gen E058 compT/pompT (Figura 2C). Los resultados anteriores indicaron que tanto pOmpT como cOmpT se expresan en la membrana externa de E058. Además, el peso molecular proteico estimado de pOmpT (≈36 kDa) fue menor que el de cOmpT (≈37 kDa) en función de sus secuencias de aminoácidos. Además, se encontró que el nivel de expresión de pOmpT en la membrana era mayor que el de cOmpT, lo que coincide con los resultados de la qPCR.
3.2 La pOmpT no puede resistir a la protamina
La escisión de la protamina es una de las funciones importantes de la cOmpT en la resistencia del huésped (13, 34-36). Para comprender el papel de pOmpT en la resistencia al huésped, se evaluó la cinética de crecimiento de la cepa de tipo salvaje E058, las cepas de deleción de genes simples/dobles compT/pompT y las cepas de complementación incubadas con protamina. En comparación con el de la cepa de tipo salvaje, el crecimiento de las cepas E058ΔpompT y ReE058ΔcompT-cc ompT no difirió significativamente cuando cOmpT estaba presente en las cepas (p > 0,05), mientras que la capacidad de crecimiento de la cepa se redujo significativamente cuando solo pOmpT estaba presente en la cepa de deleción E058ΔcompT y la cepa de complementación ReE058ΔcompT-pp ompT (p < 0,01) (Figura 3A). El mismo fenómeno se verificó en bacterias recombinantes que expresan el gen compT/pompT (Figura 3B), lo que indica que la cOmpT puede resistir a la protamina pero que la pOmpT no.
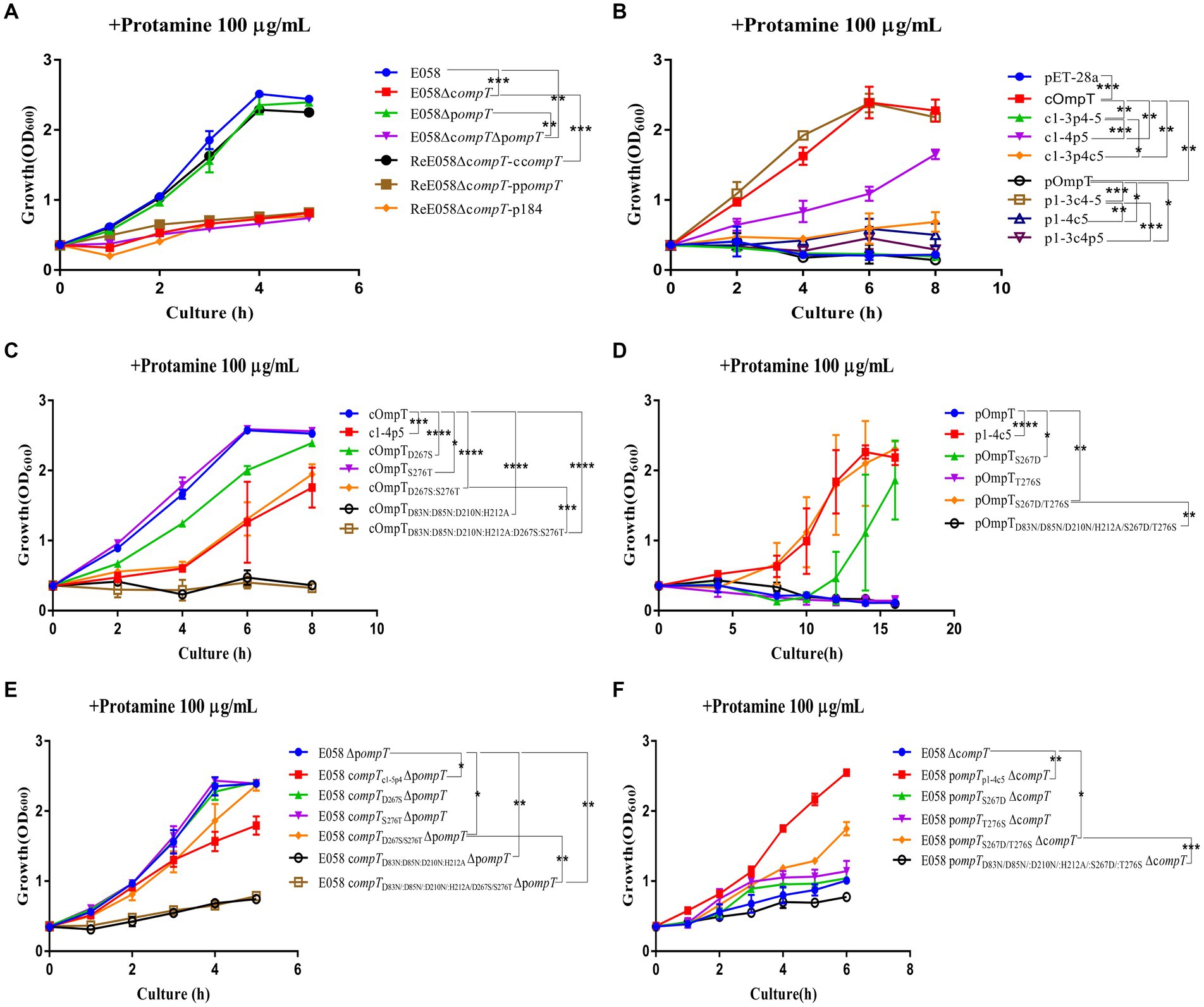
Figura 3. La cinética del crecimiento bacteriano bajo tratamiento con protamina. (A) Perfiles de crecimiento de la cepa de tipo salvaje E058 de APEC, cepas de deleción de genes simples/dobles deompT/p ompT y sus cepas de complementación bajo tratamiento con protaminas. (B) Perfiles de crecimiento de las cepas de E. coli BL21(DE3) que expresan cOmpT, pOmpT y la proteína quimérica con los diferentes bucles intercambiados entre cOmpT y pOmpT bajo tratamiento con protamina. (C) Perfiles de crecimiento de cepas de E. coli BL21(DE3) que expresan mutagénesis dirigida al sitio de cOmpT bajo tratamiento con protamina. (D) Perfiles de crecimiento de las cepas de E. coli BL21(DE3) que expresan mutagénesis de pOmpT dirigida al sitio bajo tratamiento con protaminas. (E) Perfiles de crecimiento de cepas de deleción del gen pompT que expresan mutagénesis dirigida al sitio de cOmpT de APEC E058 bajo tratamiento con protamina. (F) Perfiles de crecimiento de cepas de deleción del gen compT que expresan mutagénesis dirigida al sitio de pOmpT de APEC E058 bajo tratamiento con protaminas. Las cepas bacterianas utilizadas aquí se enumeran en la Tabla Suplementaria S1. Todos los experimentos se repitieron tres veces. La significación estadística se determinó mediante el ANOVA de dos vías. Las diferencias con valores de p <0,05 se consideraron estadísticamente significativas. *p < 0,05; **p < 0,01; p < 0,001; p < 0,0001.
3.3 La diferencia en el tipo de residuos en las posiciones 267 y 276 discrimina la especificidad de la escisión de la protamina por cOmpT y pOmpT
Para explorar cuál de los cinco bucles de pOmpT desempeña un papel fundamental en la desactivación de la protamina, se diseñaron mutantes de pOmpT y cOmpT intercambiando los bucles entre los dos. Se evaluó la sensibilidad de estos constructos modificados a la protamina. Los hallazgos revelaron una disminución significativa en la resistencia a la protamina para la variante cOmpT que contiene el segmento pOmpT L5 (Figura 3B). Por el contrario, la sustitución de la región L5 de cOmpT por la de pOmpT en el constructo pOmpT aumentó sustancialmente su resistencia a la protamina (Figura 3B). La experimentación adicional con mutantes que involucran otros intercambios de bucles entre cOmpT y pOmpT no indicó un impacto significativo en la resistencia a la protamina, lo que subraya el papel fundamental de la región L5 en la mediación de las diferencias en la resistencia a la protamina entre cOmpT y pOmpT. Con el objetivo de identificar residuos cruciales en la L5 de la cOmpT que contribuyen a la resistencia a la protamina, se llevaron a cabo intercambios de residuos L5 simples o múltiples entre la cOmpT y la pOmpT. Los ensayos de resistencia a la protamina demostraron que la sustitución de Ser276 en cOmpT por Thr276 de pOmpT redujo significativamente la resistencia a la protamina (p < 0,05) (Figura 3C). La sustitución de Asp267 en cOmpT por Ser267 de pOmpT, o el intercambio simultáneo de Asp267 y Ser276 en cOmpT por Ser267 y Thr276 de pOmpT, dio lugar a una marcada disminución de la resistencia a la protamina (p < 0,001) (Figura 3C). En comparación con pOmpT, una forma mutantepOmpT S267D mostró un crecimiento notablemente mayor en presencia de resistencia a la protamina (p < 0,05), y el mutante pOmpTS267D/T276S mostró un aumento significativo de la resistencia a la protamina (p < 0,01) (Figura 3D). La cinética de crecimiento de la resistencia bacteriana a la protamina afirmó aún más la contribución de los residuos 267 y 276 en cOmpT a la resistencia a la protamina en APEC E058 (Figuras 3E, F).
También analizamos la eficiencia de la escisión de protaminas en E. coli que expresa cOmpT, pOmpT y sus diversos mutantes. La eficiencia de escisión de la protamina por el mutante simple D267S y el doble mutante D267S/S276T de cOmpT fue significativamente menor que la de cOmpT (p < 0,001) (Figuras suplementarias S1A,C). Por el contrario, la eficiencia de escisión de la protamina por el mutante simple T276S y el mutante doble S267D/T276S de pOmpT aumentó significativamente (p < 0,01) (Figuras suplementarias S1B,D). En particular, la eficiencia de escisión del mutante único S267D de pOmpT fue incluso mayor que la de los otros mutantes (p < 0,001) (Figuras suplementarias S1B,D). Los resultados anteriores indicaron que L5 de cOmpT es el bucle clave que contribuye a las diferencias en la resistencia a la protamina entre cOmpT y pOmpT, y los residuos clave son los residuos 267 y 276.
Para investigar la posible interacción entre OmpT y la protamina, la protamina se acopló a cOmpT, pOmpT y sus mutantes utilizando el software AutoDock (AutoDock 4.0, Scripps Research Institute, EE. UU.)5 (50). La configuración con la energía de enlace más baja se sometió a un análisis visual (51). Dado que cOmpT y pOmpT son proteínas de membrana externa, se excluyeron los modelos de simulación que representan la región transmembrana (50). Los resultados de la simulación de acoplamiento indicaron que la protamina no podía acoplarse en el bolsillo de unión de pOmpT, mientras que se acoplaba fácilmente en el de cOmpT (Figuras suplementarias S2A,B). En el modelo de cOmpT, se encontró que seis residuos (D159, I160, K216, D267, S273 y D274) formaban enlaces de hidrógeno con los residuos correspondientes de protamina, lo que destaca su importancia para la unión de la protamina a cOmpT (Figura suplementaria S2B). Sin embargo, el complejo cOmpTD267S/S276T-protamina reveló solo tres residuos de anclaje (E34, D270 y N269) para la unión de protamina (Figuras suplementarias S2C,D), alineándose con la disminución de la capacidad de escisión del mutante cOmpTD267S/S276T hacia la protamina. Además, la sustitución de los residuos 267 y 276 de pOmpT por los de cOmpT permitió el acoplamiento de la protamina en su sitio de unión, formando enlaces de hidrógeno en tres residuos, incluido el D267 (Figuras suplementarias S2E,F). Esto sugiere que D267 en cOmpT es un residuo clave para el anclaje de la protamina.
3.4 Los residuos 267 y 276 de cOmpT y pOmpT están implicados en la afinidad del sustrato
OmpT escinde específicamente sustratos en motivos dibásicos (RR, RK y KK) utilizando péptidos sintéticos como sustratos (31, 32). La protamina, debido a su abundancia de motivos dibásicos en la estructura primaria, es escindida por cOmpT, lo que explica por qué los productos de escisión de protamina en geles no son una sola banda. Los péptidos sintéticos que contienen motivos dibásicos, como el sustrato sintético FRET [2Abz-SLGRKIQI-K(Dnp)-NH2] y el sustrato cromogénico [IAA-Arg-Arg-pNA], se utilizan comúnmente para detectar la actividad de la enzima OmpT (25, 49). Examinamos si el cOmpT y el pOmpT comparten características similares y si las diferencias en los residuos 267 y 276 afectan su actividad hacia el sustrato sintético. Los resultados de la digestión mostraron que tanto la pOmpT como la cOmpT en cantidades iguales de E. coli BL21(DE3) podían escindir el sustrato sintético (Figuras 4A-C). Aunque la eficiencia de escisión de la cOmpT sigue siendo mayor que la de la pOmpT, esto difiere de la actividad de escisión hacia el sustrato protamina, que es digerido exclusivamente por la cOmpT. Las mutaciones de reemplazo simple y doble de cOmpT (S276T y D267S/S276T) disminuyeron significativamente la actividad de cOmpT en cantidades iguales de E. coli BL21(DE3) (p < 0,01). Por el contrario, la mutación de reemplazo de pOmpT aumentó significativamente la eficiencia de escisión en comparación con pOmpT (p < 0,001) en cantidades iguales de E. coli BL21(DE3) (Figura 4A). La notable eficiencia en el corte de sustratos por parte de la variante pOmpTS276T subraya el papel crítico del residuo 276. Esta observación apoya aún más la contribución de los residuos 267 y 276 a las diferencias de escisión observadas entre cOmpT y pOmpT.
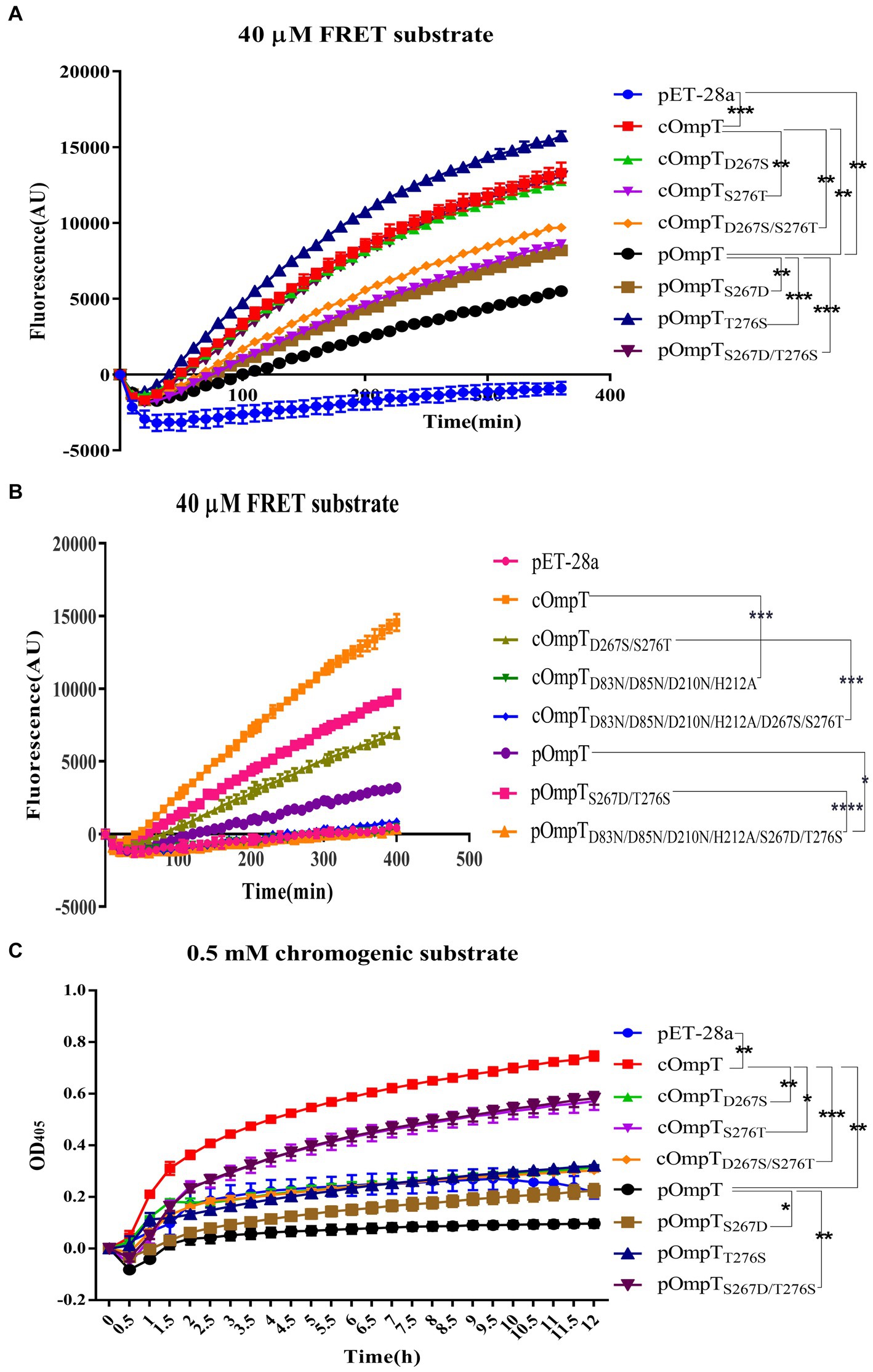
Figura 4. Determinación de la actividad enzimática de cOmpT, pOmpT y sus mutantes dirigidos al sitio. (A,B) Actividad enzimática de cOmpT, pOmpT y sus variantes expresadas en E. coli BL21(DE3) utilizando un sustrato FRET (2Abz-SLGRKIQI-K(Dnp)-NH2). (C) Actividad enzimática de cOmpT, pOmpT y sus variantes expresadas en E. coli BL21(DE3) mediante la utilización de un sustrato cromogénico (IAA-Arg-Arg-pNA). La significación estadística se determinó mediante el ANOVA de dos vías. Las diferencias con valores de p <0,05 se consideraron estadísticamente significativas. *p < 0,05; **p < 0,01; p < 0,001; p < 0,0001.
Se ha documentado que la cOmpT puede ser inhibida por los inhibidores de la serina proteasa (27, 52). Se llevaron a cabo experimentos para determinar si los inhibidores de la serina proteasa afectan a la pOmpT de manera similar a la cOmpT. Los resultados demostraron que la pOmpT en E. coli BL21(DE3) fue inhibida por la aprotinina (Figura 5) y la leupeptina (Figura suplementaria S3), junto con mutantes de los residuos 267 y 276 en las variantes cOmpT y pOmpT (Figura 5; Figura complementaria S3). Esto indica que la diferencia entre pOmpT y cOmpT no influye en su inhibición por los inhibidores de la serina proteasa. Los residuos Asp83, Asp85, Asp210 y His212 han sido propuestos como sitios catalíticos para cOmpT (24, 25). Las mutaciones en estos sitios, junto con las mutaciones en los residuos 267 y 276 tanto en cOmpT como en pOmpT, dieron lugar a una pérdida de actividad hacia los sustratos sintéticos, tanto con como sin presencia de inhibidores. Esto sugiere que estos residuos también son esenciales para la actividad de pOmpT (Figuras 4B; 5C,D; Figuras suplementarias S3C,D; S4C,D). Además, implica que los residuos 267 y 276 pueden no actuar como sitios catalíticos en ninguna de las variantes de la proteína. Para dilucidar aún más las funciones de los residuos 267 y 276, se determinaron los valores de Km de cOmpT, pOmpT y sus respectivos mutantes. Se encontró que los valores de Km para los mutantes de cOmpT eran más altos que los del tipo salvaje (1,56-4,44 veces) (Tabla 1), lo que indica una disminución en la afinidad de unión al sustrato. Por el contrario, el valor de Km de pOmpT fue mayor que el de sus mutantes (2,37-4,24 veces) (Tabla 1), lo que sugiere que los residuos 267 y 276 están involucrados en la unión al sustrato más que en la catálisis. Los valores de Kcat/Km para cOmpT y pOmpT y sus mutantes revelaron además que la eficiencia catalítica de cOmpT es significativamente mayor que la de pOmpT. Las mutaciones en los residuos 267 y 276 mejoraron la eficiencia catalítica de pOmpT, mientras que disminuyeron drásticamente la de cOmpT (Tabla 1), lo que indica que los cambios en la afinidad de unión debidos a estos residuos también afectan la eficiencia catalítica.
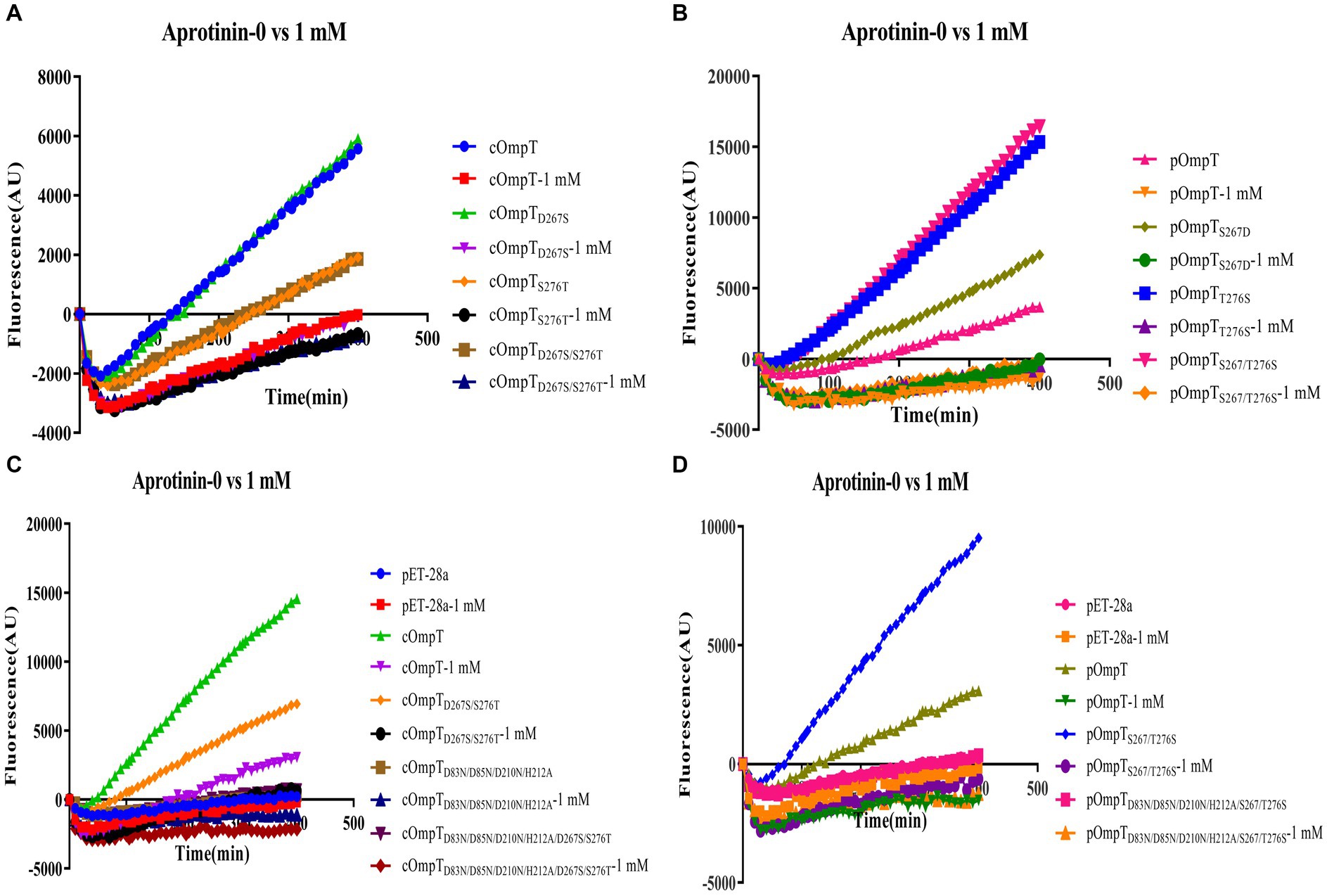
Figura 5. Inhibición de la actividad enzimática de cOmpT, pOmpT y sus mutantes dirigidos al sitio expresados en E. coli BL21 por aprotinina. (De la A a la D) Los ensayos FRET se realizaron con cOmpT, pOmpT y sus mutantes dirigidos al sitio en PBS o en presencia de aprotinina (1 mM).
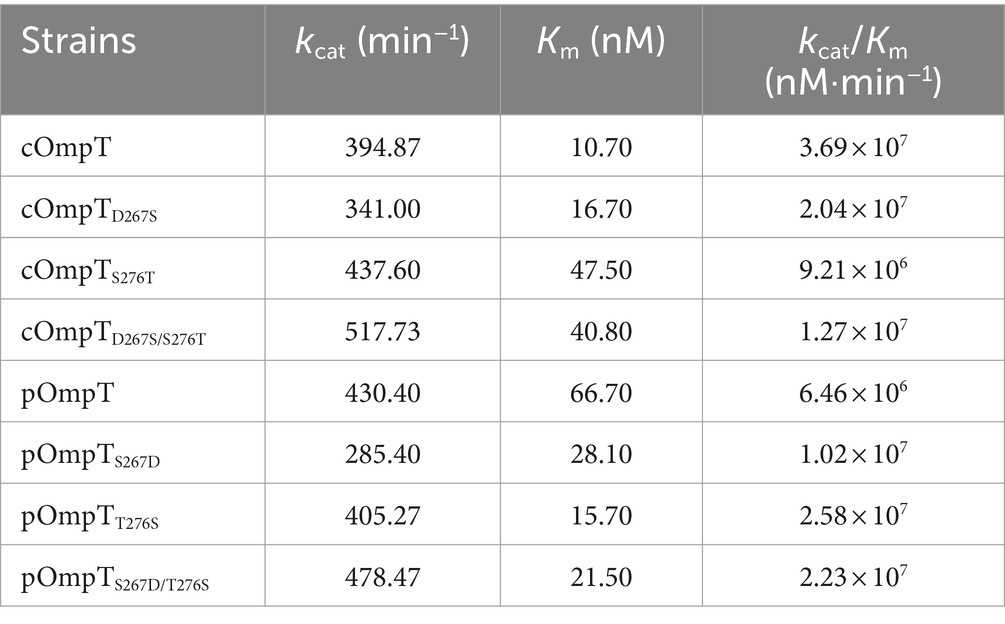
Tabla 1. Parámetros cinéticos de (c/p)OmpT y sus mutantes dirigidos al sitio expresados en E. coli BL21 utilizando péptido fluorimétrico sintético como sustrato.
3.5 Los residuos 267 y 276 de pOmpT contribuyen a la escisión de la RNasa 7 humana
El ArlC en UPEC, que comparte el 100% de identidad con el pOmpT, puede promover la resistencia bacteriana al huésped mediante la escisión de la gran molécula AMP ARNasa humana 7 (23). Como se esperaba, la RNasa 7 se escindió con una banda de producto de escisión clara restante (<17,7 kDa) cuando se incubó con la cepa que expresa pOmpT (como las cepas E058 y E058ΔcompT). Sin embargo, la cepa E058ΔcompTΔpompT sin c/pOmpT no escindió la RNasa 7 con una banda clara de proteína RNasa 7 restante (~17,7 kDa), lo que indica que pOmpT también puede escindir la RNasa7 (Figura 6). Curiosamente, la RNasa 7 también fue escindida por cOmpT (cepa E058ΔpompT), que demostró no tener ningún efecto sobre la RNasa 7 en la cistitis aislada 6 de UPEC (23). La digestión del sustrato sintético mostró que los residuos 267 y 276 de c/pOmpT están involucrados en el reconocimiento del sustrato (Figura 6; Tabla 1), por lo que también comprobamos la influencia de los cambios en los residuos 267 y 276 sobre la actividad proteolítica de c/pOmpT sobre la RNasa 7. Los resultados mostraron que la sustitución de los residuos 267 y 276 de cOmpT por la de pOmpT disminuyó drásticamente la actividad de la cOmpT sobre la ARNasa 7, y se observó una banda clara de ARNasa 7 intacta en comparación con la de la cOmpT (Figura 6).
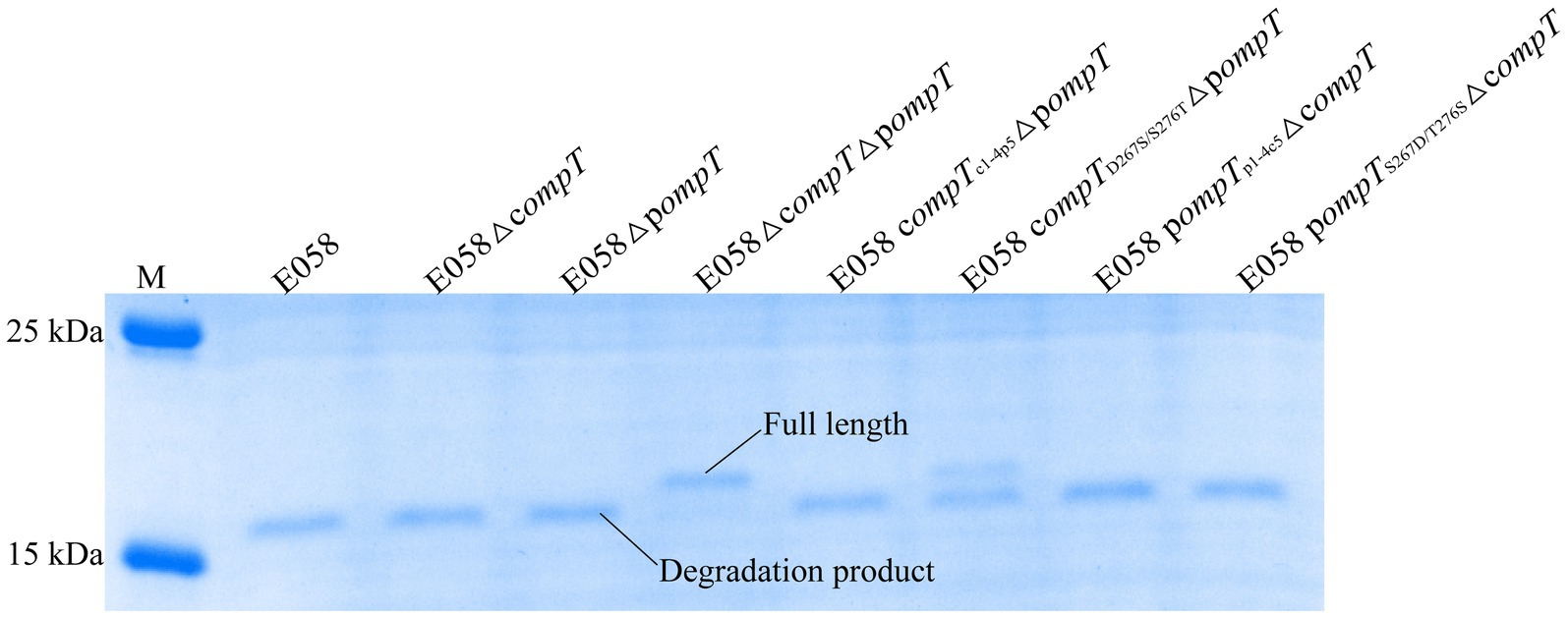
Figura 6. Los cambios de residuos en las posiciones 267 y 276 de pOmpT también pueden cambiar su capacidad para escindir la RNasa 7 humana. La RNasa 7 se incubó con APEC E058 expresando cOmpT, pOmpT o sus mutantes durante 15 min. Se utilizó un marcador de proteína preteñido de 180 kDa (Vazyme, China) como estándar de tamaño molecular de la proteína (carril M).
3.6 Los residuos 267 y 276 de cOmpT y pOmpT están estrechamente relacionados con la patogenicidad de APEC
La dosis letal del 50% (LD50) en un modelo de infección por pollo se utilizó para evaluar la patogenicidad de cOmpT, pOmpT y sus mutantes dirigidos al sitio en APEC E058. La LD50 para todas las cepas se presenta en la Tabla 2 y la Figura 7. La patogenicidad de las cepas de deleción de uno o dos genes de cOmpT y pOmpT en APEC E058 se redujo significativamente en comparación con la cepa de tipo salvaje E058, en casi 5 (103.569/102.833, p < 0.05) (deleción simple de cOmpT), 112 (104.883/102.833, p < 0.01) (deleción simple de pOmpT) y 463 (105.499/102.833, p < 0,001) (cOmpT y pOmpT doble deleción), respectivamente. Esto confirma el papel de cOmpT y pOmpT como factores de virulencia en la patogenicidad de APEC E058. Curiosamente, la LD50 de la cepa mutante E058 compTD267SΔpompT fue significativamente menor, casi 80 veces menor que la cepa de deleción de un solo gen E058ΔpompT (106.784/104.883, p < 0.01), lo que indica que el residuo 267 de cOmpT contribuye más a la patogenicidad de APEC que el residuo 276. Por el contrario, la sustitución de los residuos 267 y 276 de pOmpT por los de cOmpT en las cepas de deleción del gen compT dio como resultado una LD50 superior a la de la cepa de deleción de un solo gen E058ΔcompT. La doble sustitución de los residuos 267 y 276 (102.741) presentó la mayor virulencia, casi equivalente a la cepa silvestre E058 (102.883/102.741, p > 0.05). A continuación, se produjo la sustitución del residuo 267 (102.910) y del residuo 276 (103.193), respectivamente. Esta observación subraya un fenómeno significativo: tras la deleción de cOmpT en E058, la sustitución de sólo 2 residuos de pOmpT dio lugar a que su virulencia coincidiera con la de la cepa parental. Dicho de otro modo, la sustitución de dos residuos no solo preservó la patogenicidad del propio pOmpT, sino que también compensó plenamente la virulencia del cOmpT. Estos hallazgos sugieren que los residuos 267 y 276 de pOmpT también juegan un papel esencial en la virulencia de APEC.
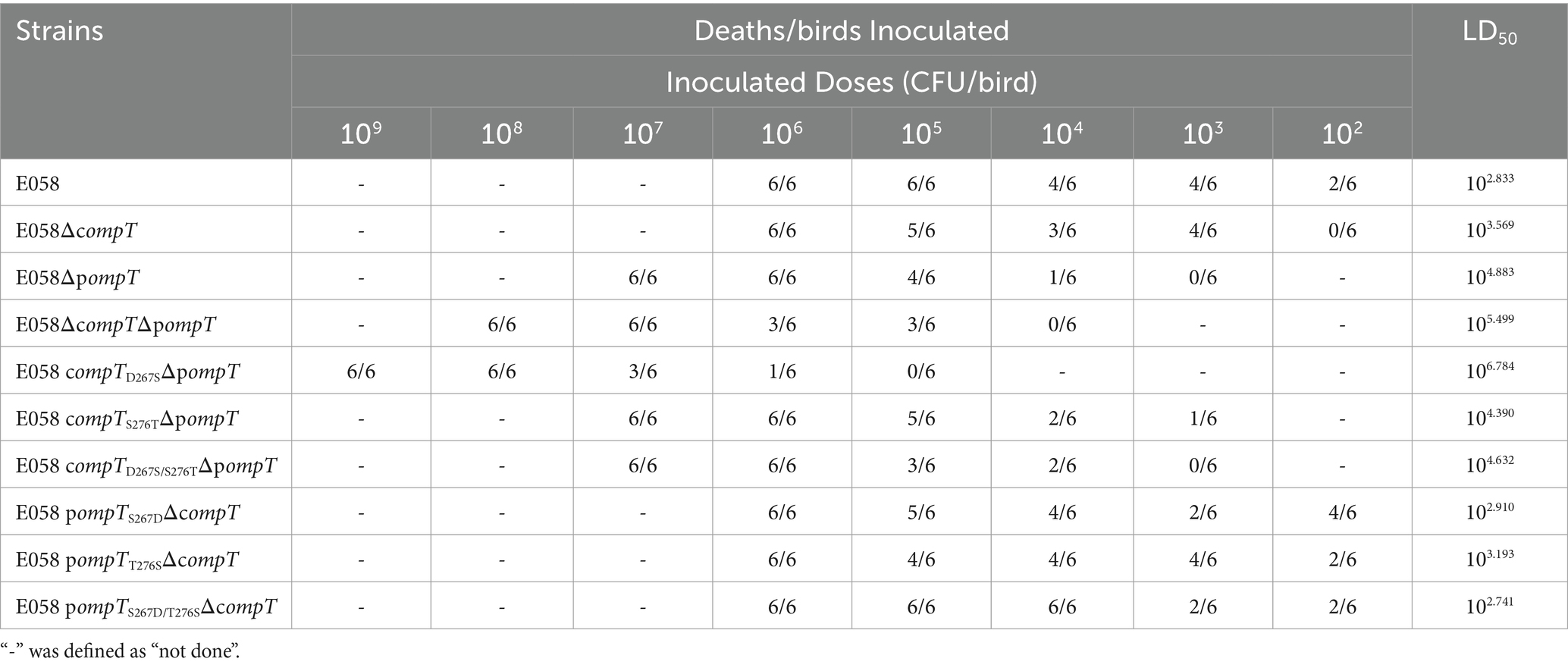
Tabla 2. LD50 de cepa de tipo salvaje y sus cepas mutantes.
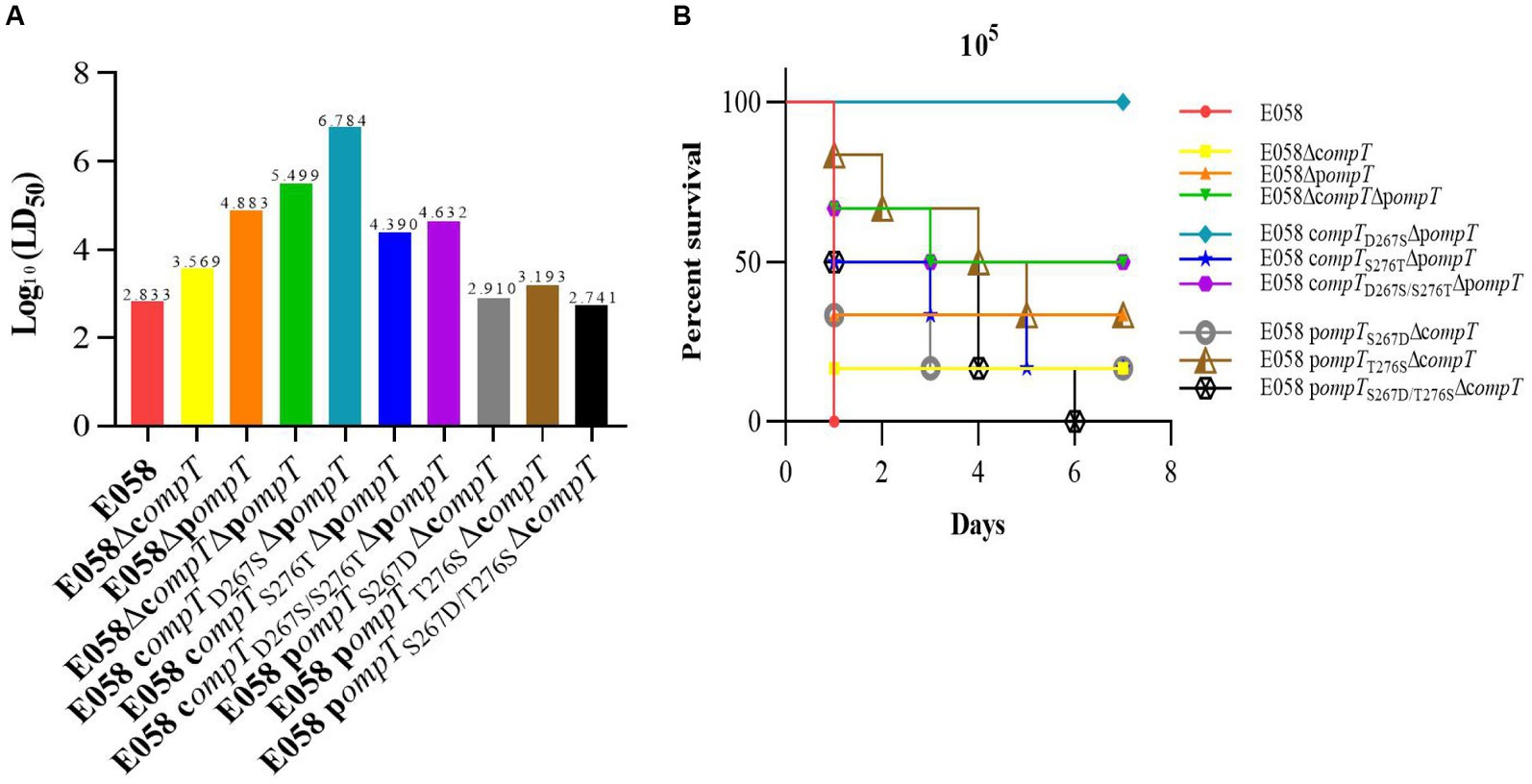
Figura 7. Los residuos 267 y 276 de cOmpT y pOmpT están estrechamente relacionados con la patogenicidad de APEC. (A) Se utilizó LD50 en un modelo de infección de pollo para evaluar el efecto de cOmpT, pOmpT y sus mutantes dirigidos al sitio sobre la patogenicidad de APEC E058. Los datos de LD50 para todas las cepas se presentaron en la Tabla 2. (B) La supervivencia de las aves desafiadas con cada cepa a una dosis de 105 UFC/ave se monitoreó durante 7 días después del desafío en base a las curvas de supervivencia de Kaplan Meyer.
4 Discusión
Las omptinas son factores de virulencia clave involucrados en varios patógenos bacterianos gramnegativos (14, 15, 17, 39). Numerosos estudios han sugerido que las proteínas similares a OmpT están presentes en cepas de E. coli patógenas intestinales o extraintestinales que infectan a humanos y animales y que aumentan la virulencia bacteriana al participar en la escisión de los AMP del huésped (12, 13, 23, 41). Su contribución a la virulencia de E. coli depende principalmente de su propia actividad proteolítica y especificidad del sustrato (53). Las omptinas en E. coli incluyen principalmente OmpT, que se codifica en los cromosomas, y OmpP/ArlC, que se codifican en plásmidos episomales (23, 38, 39). La mayoría de las cepas patógenas de E. coli (por ejemplo, EPEC, EHEC, UPEC) solo albergan OmpT en sus cromosomas. Sin embargo, descubrimos que la cepa APEC E058 (serotipo O2), miembro de las ExPEC, posee el pompT en el plásmido ColV junto con el compT en el cromosoma. Sorprendentemente, a pesar de tener solo un 76% de identidad de secuencia de aminoácidos, ambos homólogos se expresan en la membrana externa de APEC E058. Además, los sitios activos caracterizados (incluidos los residuos catalíticos) y los sitios de unión a LPS de cOmpT se conservan en pOmpT, con la excepción de los dos sitios de unión a LPS. De acuerdo con este estudio, APEC cOmpT y UPEC OmpT (de UPEC CFT073 y UPEC cistitis aislado 6) descendieron del mismo ancestro, mientras que pOmpT en APEC y ArlC en UPEC cistitis aislada 6 fueron homólogos. OmpT puede escindir la protamina (35), lo que fue confirmado por APEC E058. ArlC se descubrió en la isla de patogenicidad en el plásmido de la cepa AIEC NRG 857c y está involucrado en la resistencia a las HDP humanas HD5, HBD2 y LL-37 (23). Sin embargo, aún no está claro si pOmpT o ArlC pueden resistir a la protamina. Aquí, los hallazgos de que pOmpT incapaz de escindir la protamina llenan el vacío.
Las proteasas similares a OmpT exhiben variabilidad en la especificidad del sustrato, lo que indica diferencias estructurales significativas en los surcos enriquecidos con el sitio activo proteolítico entre cOmpT y pOmpT a pesar de su alta similitud en la secuencia de aminoácidos (54). La única estructura previamente resuelta de una subfamilia de proteasas similar a E. coli OmpT fue la de cOmpT, codificada en el cromosoma. Esta estructura presentaba un β-barril antiparalelo de 10 cadenas, con su asa extracelular sobresaliendo por encima de la bicapa lipídica (24). Dentro de esta configuración estructural se identificaron sitios de unión putativos para LPS y los sitios activos (incluidos los sitios catalíticos), ubicados en el surco en la parte superior extracelular del barril de β en forma de jarrón (24). Aunque los resultados de la simulación de software indican que la estructura general de pOmpT se parece mucho a la de cOmpT, la verdadera estructura de pOmpT sigue sin descubrirse. Se ha encontrado que la actividad proteolítica y la especificidad del sustrato de las omptinas dependen de los residuos dentro de los bucles 1-5 (L1-L5), y las mutaciones en estos bucles alteran su actividad (55). El análisis de la estructura cristalina de cOmpT confirma que los sitios activos están situados en la región de la bolsa de unión al sustrato en la parte superior del bucle extracelular (24). Para investigar la resistencia de la protamina a la pOmpT, utilizamos los cinco bucles extracelulares de OmpT como puntos de entrada para examinar la actividad proteolítica y la especificidad del sustrato de la pOmpT. La cinética de la escisión de protamina por mutantes de intercambio de bucles indicó que L5 de cOmpT contribuye a la actividad de escisión de protamina. Además, se identificaron Asp267 y Ser276 en L5 de cOmpT como los residuos clave que escinden la protamina. Aunque se han caracterizado los sitios activos y los sitios putativos de unión a LPS de cOmpT, ni el residuo 267 ni el 276 están involucrados en estos sitios activos. Este resultado sugiere que, debido a las características de L5 e incluso a sus residuos 267 y 276 de pOmpT, pierde la capacidad de resistir a la protamina. Por lo tanto, nuestros hallazgos son particularmente importantes para complementar la información crítica.
Escherichia coli OmpT se clasificó previamente como una serina proteasa, con los residuos Ser99 y His212 identificados como sitios activos típicos (56). Estudios posteriores también clasificaron a OmpT como una proteasa aspártico (24), proponiendo un nuevo mecanismo proteolítico que involucra una díada His212-Asp 210 y un par Asp83-Asp 85 que activa una supuesta molécula de agua nucleofílica, basada en la estructura cristalina de E. coli OmpT (24). Estos sitios activos están completamente conservados en toda la familia de las omptinas (24). Este estudio investiga si el pOmpT opera a través del mismo mecanismo proteolítico que el cOmpT y examina el impacto de los residuos 267 y 276 en la actividad proteolítica. El análisis de alineamiento de secuencias confirmó la conservación de estos residuos catalíticos entre los dos homólogos. La mutación de estos cuatro sitios catalíticos (Asp83, Asp85, Asp210 y His212) resultó en la abolición completa de la actividad proteolítica de los mutantes, demostrando que ni el residuo 267 ni el intercambio 276 entre cOmpT y pOmpT pudieron restaurar la capacidad de escindir sustratos, lo que indica que estos residuos no son sitios catalíticos de OmpT. Un estudio previo apoyó la hipótesis de que OmpT es un miembro de la familia de las serina proteasas (57), ya que su actividad proteolítica podría ser inhibida significativamente por inhibidores de la serina proteasa como el diisopropilfluorofosfato (DFP) (27), el fluoruro de fenilmetanosulfonilo (PMSF) (27) y la aprotinina (49). De acuerdo con las observaciones en cOmpT, la actividad proteolítica tanto de la pOmpT de tipo salvaje como de su variante S267D/T276S fue inhibida significativamente por los inhibidores de la serina proteasa (aprotinina, PMSF y leupeptina), lo que sugiere un mecanismo catalítico compartido entre pOmpT y cOmpT. El análisis posterior de las actividades enzimáticas de cOmpT de tipo salvaje, pOmpT, y sus sustituciones en los residuos 267 y 276 revelaron una actividad proteolítica significativamente menor para pOmpT en comparación con cOmpT, alineándose con la incapacidad de pOmpT para escindir la protamina. La sustitución con S276T o D267S/S276T afectó significativamente la actividad proteolítica de cOmpT de tipo salvaje; por el contrario, la sustitución con S267D, T276S o S267D/T276S mejoró significativamente la actividad proteolítica de pOmpT de tipo salvaje. Estos resultados demostraron plenamente que los residuos 267 y 276 de cOmpT y pOmpT son sitios activos que contribuyen a la actividad proteolítica tanto de cOmpT como de pOmpT, lo que conduce a diferentes especificidades de escisión del sustrato reconocidas por estas dos omptinas. Para dilucidar el papel de los residuos 267 y 276, se examinó la cinética proteolítica de cOmpT, pOmpT y sus mutantes que involucran estos residuos utilizando péptidos fluorescentes sintéticos. En comparación con el cOmpT de tipo salvaje, los mutantes mostraron valores de constante catalítica (Kcat) prácticamente inalterados, pero los valores de Km aumentaron significativamente: aproximadamente un 56% para la sustitución de Asp267 a Ser [(16,7-10,7)/10,7], un 344% para la sustitución de Ser276 a Thr [(47,5-10,7)/10,7] y un 281% para las sustituciones de Asp267 y Ser276 [(40.8–10.7)/10.7]. Por el contrario, para los mutantes de pOmpT en relación con el pOmpT de tipo salvaje, los valores de Km se redujeron en aproximadamente un 58% (sustitución de Ser267 a Asp, [(66.7–28.1)/66,7]), 76% (sustitución de Thr276 a Ser, [(66,7–15,7)/66,7]) y 68% (sustituciones de Ser267 y Thr276, [(66,7–21,5)/66,7]). Esto sugiere que los residuos 267 y 276 tanto en cOmpT como en pOmpT están involucrados en la unión al sustrato en lugar de en la catálisis, similar a Tyr248 en la carboxipeptidasa A, que está involucrada en la unión del sustrato como lo indica una sustitución de Tyr248 a Phe que mantuvo el mismo valor de Kcat mientras aumentaba su valor de Km seis veces en comparación con el tipo silvestre (57). Por lo tanto, los residuos 267 y 276 en cOmpT y pOmpT probablemente sirven como sitios de unión al sustrato, facilitando y promoviendo la escisión del sustrato por el centro catalítico.
La protamina es un sustrato específico para la cOmpT pero no para la pOmpT; por lo tanto, buscamos un sustrato específico para pOmpT e investigamos el mecanismo subyacente a la especificidad de la escisión del sustrato. Se informó que ArlC en aislados clínicos de UPEC escinde específicamente la ARNasa 7 de AMP humana (23). Sin embargo, se observó en este estudio que tanto la cOmpT como la pOmpT de la cepa APEC E058 podían escindir la ARNasa 7 humana. Las sustituciones de los residuos 267 y 276 entre cOmpT y pOmpT también influyeron en su capacidad para escindir la RNasa 7, destacando una característica única de la pOmpT de la cepa APEC E058: no escinde la protamina, pero sí la ARNasa 7, a pesar de que ambas son AMP humanas. La proteasa OmpT exhibe una especificidad de escisión estrecha, prefiriendo sustratos en motivos dibásicos (RR, RK, KK) (31, 32), una especificidad determinada por los residuos conservados Glu27 y Asp208 en el fondo de la bolsa S1 profunda y Asp97 en la bolsa S1′ menos profunda (24). A pesar de la alta conservación de estos residuos en pOmpT y cOmpT, el análisis de acoplamiento molecular sugiere que los residuos 267 y 276 de cOmpT juegan un papel crucial en la unión al sustrato a través de la interacción con los residuos de arginina en la protamina. Esta variación de especificidad entre diferentes omptinas, impulsada por la variabilidad de la secuencia en el bucle externo (55), revela diferencias en el reconocimiento del sustrato objetivo por el bucle 5 y sus residuos 267 y 276 de pOmpT, lo que sugiere una especificidad estructural de pOmpT que evita la interacción con los residuos de arginina en la protamina, lo que permite una unión y escisión efectivas de la RNasa 7 en su lugar.
La evasión de APEC de los efectos mortales del AMP huésped sigue siendo una preocupación central. OmpT, como factor de virulencia, permite a APEC protegerse contra el huésped. Es vital comprender si los sitios clave que determinan la especificidad del sustrato influyen en la virulencia de APEC. Este estudio confirma que los cambios en los sitios de unión al sustrato de cOmpT y pOmpT afectan la infectividad de APEC, validando la hipótesis de que la especificidad del sustrato de las proteasas similares a OmpT contribuye a la virulencia de E. coli. Sin embargo, hubo un resultado inexplicable de que la sustitución del residuo 267 en cOmpT por el de pOmpT en un mutante pOmpT (cepa E058 compTD267SΔpompT, 106.784) fue menos patogénico que el doble mutante (cepa E058ΔcompTΔpompT, 105.499), aunque se había descartado in vitro la posibilidad potencial de defecto de crecimiento de este mutante. Es posible que haya algunos efectos potenciales del residuo 267 en cOmpT sobre otros residuos, incluso sobre otros factores de virulencia (a saber, sideróforos), lo que conduce a una virulencia más atenuada en el mutante. Los hallazgos no solo fueron interesantes, sino que también valía la pena estudiarlos más a fondo en el futuro.
Teniendo en cuenta los informes de la OMS de 2020 y 2021 que ponen de manifiesto la falta de tratamientos eficaces o de prevención de las infecciones bacterianas debidas a la resistencia a los antibióticos, las omptinas podrían representar nuevos objetivos para el desarrollo de fármacos y vacunas. La inhibición o neutralización de estas opptinas es crucial para prevenir las infecciones bacterianas septicémicas (52, 53). Los péptidos antimicrobianos, con su actividad antibacteriana de amplio espectro, su baja propensión al desarrollo de resistencia a los fármacos y su papel en el sistema inmunitario innato, han suscitado un gran interés (2, 58, 59). Sin embargo, la composición natural de la mayoría de los péptidos antimicrobianos los hace susceptibles a la degradación por proteasas como la tripsina y la pepsina en fluidos fisiológicos ricos en proteasa (60). Por lo tanto, los péptidos antimicrobianos requieren modificaciones para resistir la hidrólisis enzimática, lo que mejora su eficacia bactericida para su aplicación clínica. Además, los péptidos antimicrobianos también necesitan escapar de la escisión por parte de las opptinas bacterianas. Por ejemplo, basándonos en nuestros hallazgos, podemos reemplazar la arginina en la posición 15 (R15) en la protamina, que se une fácilmente a D267 en cOmpT, con el aminoácido no natural D-arginina, lo que lleva a escapar del reconocimiento y la unión de péptidos antimicrobianos modificados por proteasas similares a OmpT. Dado que los aminoácidos no naturales no tienen características de unión al reconocimiento de proteasas, su introducción en péptidos antimicrobianos naturales puede reducir significativamente e incluso bloquear la degradación de la proteasa, mejorando en última instancia la actividad antimicrobiana (61).
En resumen, caracterizamos la fisiología de pOmpT, una proteasa de la subfamilia OmpT recientemente descubierta en E. coli patógena que causa infecciones intestinales y extraintestinales tanto en humanos como en aves de corral, y revelamos su mecanismo molecular involucrado en la escisión del AMP humano. Los residuos 267 y 276 se caracterizaron por primera vez como sitios de unión al sustrato de las opptinas y desempeñan un papel crítico en la eficiencia de la escisión del AMP (62) y la patogenicidad de cOmpT y pOmpT. Es importante destacar que tanto el cOmpT como el pOmpT de APEC, que se investigaron en este estudio, son homólogos del ExPEC humano; en particular, el cOmpT y el pOmpT de origen aviar confieren a APEC la capacidad de escindir AMP humanos, lo que sugiere que APEC es un patógeno humano potencial (63). Nuestros hallazgos proporcionan nuevos conocimientos para el desarrollo de fármacos antibacterianos que inhiban la actividad de la omptina.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en el artículo/Material complementario.
Declaración ética
El estudio en animales fue aprobado por el Comité Institucional de Cuidado y Uso de Animales (IACUC) de la Universidad de Yangzhou (Pollo: SCXK(Su)2021-0027). El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
JL: Conceptualización, Análisis formal, Investigación, Metodología, Administración de proyectos, Validación, Visualización, Redacción – borrador original, Redacción – revisión y edición. LJ: Análisis formal, Investigación, Metodología, Recursos, Validación, Visualización, Redacción – borrador original, Redacción – revisión y edición. HW: Metodología, Recursos, Redacción – borrador original, Redacción – revisión y edición. JW: Metodología, Recursos, Redacción – borrador original, Redacción – revisión y edición. QG: Metodología, Recursos, Supervisión, Redacción – borrador original, Redacción – revisión y edición. CH: Metodología, Recursos, Supervisión, Redacción – borrador original, Redacción – revisión y edición. SG: Conceptualización, Obtención de fondos, Administración de proyectos, Recursos, Supervisión, Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este estudio contó con el apoyo de subvenciones del Programa Nacional de Investigación y Desarrollo Clave de China (2021YFD1800404, 2017YFD0500203-2, 2017YFD0500705), el Proyecto 111 (D18007), la Fundación Nacional de Ciencias Naturales de China (32373011, 31672553, 31972711), el Programa Nacional de Investigación y Desarrollo de Alta Tecnología en China (2003 AA 222141), el Fondo Especial para la Investigación Agrocientífica en el Interés Público (201303044) a SG, y el Proyecto Financiado por el Desarrollo del Programa Académico Prioritario de las Instituciones de Educación Superior de Jiangsu (PAPD). Los financiadores no tuvieron ningún papel en el diseño del estudio, la recopilación de datos y la interpretación, ni en la decisión de enviar el trabajo para su publicación.
Reconocimientos
Muchas gracias bioRxiv por nuestro estudio proporcionando un preprint, puedes acceder al preprint utilizando el siguiente enlace: https://www.biorxiv.org/content/10.1101/2022.06.17.496525v1.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1410113/full#supplementary-material
Notas
1. ^http://www.glschina.com/en/profile.htm
2. ^https://www.rcsb.org
3. ^https://swissmodel.expasy.org/
4. ^https://alphafold.ebi.ac.uk
5. ^http://autodock.scripps.edu
Referencias
1. Samantha, G, y Le, MH. Resistencia a péptidos antimicrobianos en bacterias gramnegativas. FEMS Microbiol Lett. (2012) 330:81–9. doi: 10.1111/j.1574-6968.2012.02528.x
2. Zasloff, M . Péptidos antimicrobianos de organismos pluricelulares. Naturaleza. (2002) 415:389–95. doi: 10.1038/415389a
3. Hancock, R, y Sahl, HG. Péptidos antimicrobianos y de defensa del huésped como nuevas estrategias terapéuticas antiinfecciosas. Nat Biotechnol. (2006) 24:1551–7. DOI: 10.1038/NBT1267
4. Gallo, RL, y Hooper, LV. Defensa antimicrobiana epitelial de la piel y el intestino. Nat Rev Immunol. (2012) 12:503–16. doi: 10.1038/nri3228
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Piers, KL, y Hancock, RE. La interacción de un péptido híbrido recombinante de cecropina/melitina con la membrana externa de Pseudomonas aeruginosa. Mol Microbiol. (1994) 12:951–8. doi: 10.1111/j.1365-2958.1994.tb01083.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Zhang, L, Rozek, A, y Hancock, RE. Interacción de péptidos antimicrobianos catiónicos con membranas modelo. J Biol Chem. (2001) 276:35714–22. DOI: 10.1074/JBC. M104925200
7. Brogden, KA . Péptidos antimicrobianos: ¿formadores de poros o inhibidores metabólicos en bacterias? Nat Rev Microbiol. (2005) 3:238–50. doi: 10.1038/nrmicro1098
8. Hilchie, AL, Wuerth, K, y Hancock, RE. Modulación inmunitaria por péptidos multifacéticos de defensa catiónica del huésped (antimicrobianos). Nat Chem Biol. (2013) 9:761–8. doi: 10.1038/nchembio.1393
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Haiko, J, Suomalainen, M, Ojala, T, Lahteenmaki, K, y Korhonen, TK. Revisión invitada: rompiendo barreras: ataque a las defensas inmunitarias innatas por las proteasas de superficie de Omptin de patógenos enterobacterianos. Inmunidad innata. (2009) 15:67–80. doi: 10.1177/1753425909102559
10. Kukkonen, M, y Korhonen, TK. La familia Omptin de proteasas de superficie/adhesinas enterobacterianas: desde la limpieza en Escherichia coli hasta la propagación sistémica de Yersinia pestis. Int J Med Microbiol. (2004) 294:7–14. doi: 10.1016/j.ijmm.2004.01.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Le Sage, V, Zhu, L, Lepage, C, Portt, A, Viau, C, Daigle, F, et al. Una proteasa de membrana externa de la familia Omptin impide la activación del sistema de dos componentes PhoPQ de Citrobacter rodentium por péptidos antimicrobianos. Mol Microbiol. (2009) 74:98–111. doi: 10.1111/j.1365-2958.2009.06854.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Thomassin, JL, Brannon, JR, Gibbs, BF, Gruenheid, S, y Le Moual, H. Las proteasas de membrana externa OmpT de Escherichia coli enterohemorrágica e enteropatógena contribuyen de manera diferente a la degradación de Ll-37 humano. Infectar a Immun. (2012) 80:483–92. doi: 10.1128/IAI.05674-11
13. Brannon, JR, Thomassin, JL, Desloges, I, Gruenheid, S, y Le Moual, H. Papel de la Escherichia coli OmpT uropatógena en la resistencia contra la catelicidina humana Ll-37. FEMS Microbiol Lett. (2013) 345:64–71. doi: 10.1111/1574-6968.12185
14. Sodeinde, OA, Subrahmanyam, YV, Stark, K, Quan, T, Bao, Y, y Goguen, JD. Una proteasa superficial y el carácter invasivo de la peste. Ciencia. (1992) 258:1004–7. doi: 10.1126/ciencia.1439793
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Lathem, WW, Price, PA, Miller, VL y Goldman, WE. Una proteasa activadora de plasminógenos controla específicamente el desarrollo de la peste neumónica primaria. Ciencia. (2007) 315:509–13. doi: 10.1126/ciencia.1137195
16. Korhonen, TK, Haiko, J, Laakkonen, L, Jarvinen, HM, y Westerlund-Wikstrom, B. Actividades fibrinolíticas y coagulativas de Yersinia pestis. La célula frontal infecta el microbiol. (2013) 3:35. doi: 10.3389/fcimb.2013.00035
17. Caulfield, AJ, Walker, ME, Gielda, LM y Lathem, WW. La proteasa Pla de Yersinia pestis degrada el ligando Fas para manipular la muerte y la inflamación de la célula huésped. Microbio huésped celular. (2014) 15:424–34. doi: 10.1016/j.chom.2014.03.005
18. Franco, AA, Kothary, MH, Gopinath, G, Jarvis, KG, Grim, CJ, Hu, L, et al. El Cpa, la proteasa de la membrana externa de Cronobacter sakazakii, activa el plasminógeno y media la resistencia a la actividad bactericida sérica. Infectar a Immun. (2011) 79:1578–87. doi: 10.1128/IAI.01165-10
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Grodberg, J y Dunn, JJ. OmpT codifica la proteasa de la membrana externa de Escherichia coli que escinde la ARN polimerasa T7 durante la purificación. J Bacteriol. (1988) 170:1245–53. DOI: 10.1128/jb.170.3.1245-1253.1988
20. Sodeinde, OA, y Goguen, JD. Secuencia de nucleótidos del gen activador del plasminógeno de Yersinia pestis: relación con OmpT de Escherichia coli y el gen E de Salmonella typhimurium. Infectar a Immun. (1989) 57:1517–23. doi: 10.1128/IAI.57.5.1517-1523.1989
21. Grodberg, J y Dunn, JJ. Comparación de la proteasa de membrana externa OmpT de Escherichia Coli K-12 y la proteína E de Salmonella typhimurium. J Bacteriol. (1989) 171:2903–5. DOI: 10.1128/jb.171.5.2903-2905.1989
22. Egile, C, D’Hauteville, H, Parsot, C, y Sansonetti, PJ. SopA, la proteasa de membrana externa responsable de la localización polar de IcsA en Shigella flexneri. Mol Microbiol. (2010) 23:1063–73. doi: 10.1046/j.1365-2958.1997.2871652.x
23. Desloges, I, Taylor, JA, Leclerc, JM, Brannon, JR, Portt, A, Spencer, JD, et al. Identificación y caracterización de proteasas tipo OmpT en aislados clínicos de Escherichia coli uropatógena. Microbiología. (2019) 8:E915. DOI: 10.1002/MBO3.915
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Vandeputte-Rutten, L, Kramer, RA, Kroon, J, Dekker, N, Egmond, MR y Gros, P. La estructura cristalina de la proteasa de membrana externa OmpT de Escherichia coli sugiere un nuevo sitio catalítico. EMBO J. (2001) 20:5033–9. doi: 10.1093/emboj/20.18.5033
25. Kramer, RA, Vandeputte-Rutten, L, Roon, G, Gros, P, Dekker, N y Egmond, MR. La identificación de residuos ácidos esenciales de la proteasa de membrana externa OmpT apoya un nuevo sitio activo. FEBS Lett. (2001) 505:426–30. doi: 10.1016/s0014-5793(01)02863-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Sugimura, K, y Higashi, N. Una nueva proteasa asociada a la membrana externa en Escherichia coli. J Bacteriol. (1988) 170:3650–4. doi: 10.1128/jb.170.8.3650-3654.1988
27. Sugimura, K, y Nishihara, T. Purificación, caracterización y estructura primaria de la proteasa VII de Escherichia coli con especificidad para residuos básicos emparejados: identidad de la proteasa VII y OmpT. J Bacteriol. (1988) 170:5625. doi: 10.1128/jb.170.12.5625-5632.1988
28. Yam, CH, Siu, WY y Kaganovich, D. Escisión de la ciclina a en R70/R71 por la proteasa bacteriana OmpT. Proc Natl Acad Sci USA. (2001) 98:497–501. doi: 10.1073/pnas.98.2.497
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Gill, RT, Delisa, MP, Shiloach, M, Holoman, TR, y Bentley, WE. La expresión y actividad de OmpT aumentan en respuesta a la sobreexpresión de cloranfenicol acetiltransferasa recombinante y al choque térmico en E. coli. J Mol Microbiol Biotechnol. (2000) 66:1304. doi: 10.1001/archgenpsychiatry.2009.156
30. Hui, CY, Guo, Y, He, QS, Liang, P, Wu, SC, Hong, C, et al. La proteasa de membrana externa de Escherichia coli OmpT confiere resistencia a los péptidos catiónicos urinarios. Microbiol Immunol. (2010) 54:452–9. doi: 10.1111/j.1348-0421.2010.00238.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Dekker, N, Cox, RC, Kramer, RA, y Egmond, MR. Especificidad del sustrato de la proteasa de membrana integral OmpT determinada por bibliotecas de péptidos abordadas espacialmente. Bioquímica EE.UU. (2001) 40:1694–701. doi: 10.1021/bi0014195
32. McCarter, JD, Stephens, D, Shoemaker, K, Rosenberg, S, Kirsch, JF y Georgiou, G. Especificidad del sustrato de la proteasa de membrana externa de Escherichia coli OmpT. J Bacteriol. (2004) 186:5919–25. doi: 10.1128/JB.186.17.5919-5925.2004
33. Varadarajan, N, Gam, J, Olsen, MJ, Georgiou, G e Iverson, BL. Ingeniería de variantes de proteasa que exhiben alta actividad catalítica y exquisita selectividad de sustrato. Proc Natl Acad Sci USA. (2005) 102:6855–60. doi: 10.1073/pnas.0500063102
34. Stathopoulos, C . Características estructurales, roles fisiológicos y aplicaciones biotecnológicas de las proteasas de membrana de la familia de endopeptidasas bacterianas OmpT: una microrevisión. Membr Cell Biol. (1998) 12:1–8.
35. Stumpe, S, Schmid, R, Stephens, DL, Georgiou, G y Bakker, EP. Identificación de OmpT como la proteasa que hidroliza el péptido antimicrobiano protamina antes de que ingrese a las células en crecimiento de Escherichia coli. J Bacteriol. (1998) 180:4002–6. doi: 10.1128/JB.180.15.4002-4006.1998
36. Él, XL, Wang, Q, Peng, L, Qu, YR, Puthiyakunnon, S, Liu, XL, et al. Papel de la proteína T de la membrana externa de Escherichia coli uropatógena en la patogénesis de la infección del tracto urinario. Pathog Dis. (2015) 73:FTV006. doi: 10.1093/femspd/ftv006
37. Thomassin, JL, Brannon, JR, Kaiser, J, Gruenheid, S y Le Moual, H. La Escherichia coli enterohemorrágica y enteropatógena evolucionó diferentes estrategias para resistir a los péptidos antimicrobianos. Microbios intestinales. (2012) 3:556–61. doi: 10.4161/gmic.21656
38. Hwang, BY, Varadarajan, N, Li, H, Rodríguez, S, Iverson, BL y Georgiou, G. Especificidad del sustrato de la proteasa de membrana externa de Escherichia coli OmpP. J Bacteriol. (2007) 189:522–30. doi: 10.1128/JB.01493-06
39. Mcphee, JB, Small, CL, Reid-Yu, SA, Brannon, JR, Moual, HL y Coombes, BK. La resistencia a los péptidos de defensa del huésped contribuye a la colonización y a la patología intestinal máxima por Escherichia coli adherente invasiva asociada a la enfermedad de Crohn. Infectar a Immun. (2014) 82:3383–93. doi: 10.1128/IAI.01888-14
40. Chen, J . Construcción de mutantes knockout del gen compT, pompT de E. coli patógena aviar E058 y evaluación de su patogenicidad. Yangzhou: Universidad de Yangzhou (2016).
41. Liu, J, Mu, X, Wang, X, Huan, H, Gao, Q, Chen, J, et al. Un transcriptoma inesperado pompT‘ contribuye al aumento de la patogenicidad de un mutante pompT de Escherichia coli patógena aviar. Microbiol veterinario. (2019) 228:61–8. doi: 10.1016/j.vetmic.2018.11.011
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Gao, S, Liu, X, Zhang, R, Jiao, X, Wen, Q, Wu, C, et al. El aislamiento e identificación de aislados patógenos de Escherichia coli de origen policio de algunas regiones de China. Acta Veterinaria et Zootechnica Sinica. (1999) 30:164–71. doi: 10.3321/j.issn:0366-6964.1999.02.015
43. Xue, F, Yang, Z, Xu, Y, Wang, Y, Yu, T, Fan, M, et al. Desarrollo de anticuerpos moloclonales contra la proteína cOmpT de Escherichia coli patógena aviar e identificación de epítopos reconocidos por anticuerpos moloclonales. Acta Veterinaria et Zootechnica Sinica. (2020) 51:2270–80. doi: 10.11843/j.issn.0366-6964.2020.12.021
44. Nelson, DL y Kennedy, EP. Transporte de magnesio en Escherichia coli: inhibición por ion cobaltous. J Biol Chem. (1971) 246:3042–9. doi: 10.1016/S0021-9258(18)62288-4
45. Jiang, Y, Chen, B, Duan, C, Sun, B, Yang, J y Yang, S. Edición multigénica en el genoma de Escherichia coli a través del sistema Crispr-Cas9. Appl Environ Microbiol. (2015) 81:2506–14. doi: 10.1128/AEM.04023-14
46. Koten, B, Simanski, M, Glaser, R, Podschun, R, Schroder, JM y Harder, J. Rnase 7 contribuye a la defensa cutánea contra Enterococcus faecium. PLoS Uno. (2009) 4:E6424. doi: 10.1371/journal.pone.0006424
47. Wang, H, Schwaderer, AL, Kline, J, Spencer, JD, Kline, D, y Hains, DS. Contribución de los dominios estructurales a la actividad de la ribonucleasa 7 frente a bacterias uropatógenas. Agentes antimicrobianos quimioterápicos. (2013) 57:766–74. doi: 10.1128/AAC.01378-12
48. Gao, S, Liu, X y Zhang, R. Un método mejorado para el aislamiento rápido de las proteínas de la membrana externa de los aislados de Escherichia coli de Chichén Origen. Microbiología China. (1996) 23:122–4.
49. Kramer, RA, Zandwijken, D, Egmond, MR y Dekker, N. Plegamiento, purificación y caracterización in vitro de la proteasa de membrana externa OmpT de Escherichia coli. Eur J Biochem. (2000) 267:885–93. doi: 10.1046/j.1432-1327.2000.01073.x
50. Morris, GM, Huey, R, Lindstrom, W, Sanner, MF, Belew, RK, Goodsell, DS, et al. Autodock4 y Autodocktools4: acoplamiento automatizado con flexibilidad de receptor selectivo. J Comput Chem. (2009) 30:2785–91. doi: 10.1002/jcc.21256
Resumen de PubMed | Texto completo de Crossref | Google Académico
51. Zhang, S, Wang, Y, Han, L, Fu, X, Wang, S, Li, W, et al. Targeting N-terminal human maltasa-Glucoamylase para desentrañar posibles inhibidores utilizando acoplamiento molecular, simulaciones de dinámica molecular y simulaciones de dinámica molecular dirigida adaptativa. Frente Químico. (2021) 9:711242. doi: 10.3389/fchem.2021.711242
Resumen de PubMed | Texto completo de Crossref | Google Académico
52. Brannon John, R, Burk David, L, Leclerc, J-M, Thomassin, J-L, Portt, A, Berghuis, Albert, M, et al. Inhibición de las proteasas de la membrana externa de la familia Omptin por Aprotinina. Infectar a Immun. (2015) 83:2300–11. doi: 10.1128/iai.00136-15
Resumen de PubMed | Texto completo de Crossref | Google Académico
53. Hritonenko, V, y Stathopoulos, C. Proteínas de omptina: una familia en expansión de proteasas de membrana externa en enterobacterias gramnegativas (revisión). Mol Membr Biol. (2007) 24:395–406. doi: 10.1080/09687680701443822
54. Brannon John, R, Thomassin, J-L, Gruenheid, S y Le Moual, H. Conformación del péptido antimicrobiano como determinante estructural de la especificidad de la proteasa de Omptina. J Bacteriol. (2015) 197:3583–91. doi: 10.1128/jb.00469-15
Resumen de PubMed | Texto completo de Crossref | Google Académico
55. Kukkonen, M, Lähteenmäki, K, Suomalainen, M, Kalkkinen, N, Emödy, L, Lång, H, et al. Regiones proteicas importantes para la activación e inactivación del plasminógeno Α2-Antiplasmina en la proteasa de superficie Pla de Yersinia pestis. Mol Microbiol. (2001) 40:1097–111. doi: 10.1046/j.1365-2958.2001.02451.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
56. Kramer, RA, Dekker, N y Egmond, MR. Identificación de residuos de serina e histidina del sitio activo en la proteasa de membrana externa OmpT de Escherichia coli. FEBS Lett. (2000) 468:220–4. doi: 10.1016/S0014-5793(00)01231-X
Resumen de PubMed | Texto completo de Crossref | Google Académico
57. Gardell, SJ, Craik, CS, Hilvert, D, Urdea, MS, y Rutter, WJ. La mutagénesis dirigida al sitio muestra que la tirosina 248 de la carboxipeptidasa a no desempeña un papel crucial en la catálisis. Naturaleza. (1985) 317:551–5. doi: 10.1038/317551a0
58. Lee, MO, Jang, H-J, Rengaraj, D, Yang, S-Y, Han, JY, Lamont, SJ, et al. Expresión tisular y actividad antibacteriana de péptidos de defensa del huésped en pollo. BMC Vet Res. (2016) 12:231. doi: 10.1186/s12917-016-0866-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
59. Hancock, REW, Nijnik, A, y Philpott, DJ. Modulación de la inmunidad como terapia para infecciones bacterianas. Nat Rev Microbiol. (2012) 10:243–54. doi: 10.1038/nrmicro2745
60. Ong, ZY, Wiradharma, N y Yang, YY. Estrategias empleadas en el diseño y optimización de péptidos antimicrobianos sintéticos anfifilos con potencial terapéutico mejorado. Adv Droga Deliv Rev. (2014) 78:28–45. doi: 10.1016/j.addr.2014.10.013
61. Ong, ZY, Cheng, J, Huang, Y, Xu, K, Ji, Z, Fan, W, et al. Efecto de la estereoquímica, la longitud de la cadena y el patrón de secuencia sobre las propiedades antimicrobianas de los anfifílos formadores de láminas Β sintéticas cortas. Biomateriales. (2014) 35:1315–25. doi: 10.1016/j.biomaterials.2013.10.053
62. Liu, JH, Ran, TT, Jiang, LY, Gao, QQ, Huan, CC, Wang, H, et al. La fisiología y la estructura de la pOmpT patógena de Escherichia coli revelan dos sitios de unión al sustrato. bioRxiv. (2022). DOI: 10.1101/2022.06.17.496525
63. Johnson, TJ, Wannemuehler, Y, Johnson, SJ, Stell, AL, Doetkott, C, Johnson, JR, et al. La comparación de cepas de Escherichia coli patógenas extraintestinales de fuentes humanas y aviares revela un subconjunto mixto que representa posibles patógenos zoonóticos. Appl Environ Microbiol. (2008) 74:7043–50. doi: 10.1128/AEM.01395-08
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: Escherichia coli patógena aviar, actividad proteolítica, especificidad de escisión, afinidad por el sustrato, sitio de unión al sustrato, virulencia
Cita: Liu J, Jiang L, Wang H, Wu J, Gao Q, Huan C y Gao S (2024) La especificidad de la escisión de la protamina del patógeno aviar Escherichia coli OmpT revela dos sitios de unión al sustrato relacionados con la virulencia. Frente. Vet. Sci. 11:1410113. doi: 10.3389/fvets.2024.1410113
Recibido: 31 de marzo de 2024; Aceptado: 26 de agosto de 2024;
Publicado: 05 de septiembre de 2024.
Editado por:
Dirk Werling, Real Colegio Veterinario (RVC), Reino Unido
Revisado por:
Timothy James Wells, Universidad de Queensland, Australia
Kezong Qi, Universidad Agrícola de Anhui, China
Derechos de autor © 2024 Liu, Jiang, Wang, Wu, Gao, Huan y Gao. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Atribución (CC BY).
*Correspondencia: Song Gao, gsong@yzu.edu.cn
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

