Un análisis comparativo basado en variantes compartidas de la secuencia de amplicones
 Yutaro ItoUn †
Yutaro ItoUn † Miho Nagasawa2†
Miho Nagasawa2† Kahori Koyama2
Kahori Koyama2 Kohei Ito1*
Kohei Ito1* Takefumi Kikusui2*
Takefumi Kikusui2*- 1BIOTA Inc., Tokio, Japón
- número arábigoLaboratorio de Interacción y Reciprocidad Humano-Animal, Departamento de Ciencia y Biotecnología Animal, Universidad de Azabu, Kanagawa, Japón
Introducción: El concepto de One Health es una comprensión integral de la interacción entre los seres humanos, los animales y el medio ambiente. La convivencia de humanos y mascotas afecta positivamente su bienestar físico, mental y social. Se reconoce como un factor esencial desde la perspectiva de Una Salud. Además, un equilibrio saludable en el microbioma intestinal es esencial para una buena salud, y los cambios en el microbioma intestinal asociados con la cohabitación entre humanos y mascotas podrían afectar varios aspectos de la salud de ambos huéspedes. Por lo tanto, dilucidar el intercambio de bacterias intestinales entre humanos y mascotas asociado con la cohabitación es importante para comprender One Health. Sin embargo, la mayoría de los estudios han examinado el intercambio a nivel taxonómico, y no está claro si las mismas bacterias se transfieren entre humanos y mascotas, y si se influyen mutuamente.
Métodos: En este caso, se realizó un análisis del microbioma y un análisis de la variante de secuencia de amplicón (ASV) del gen 16S rRNA compartido antes del inicio de la convivencia entre humanos y perros, así como a las 2 semanas, 1 mes y 3 meses después de la convivencia.
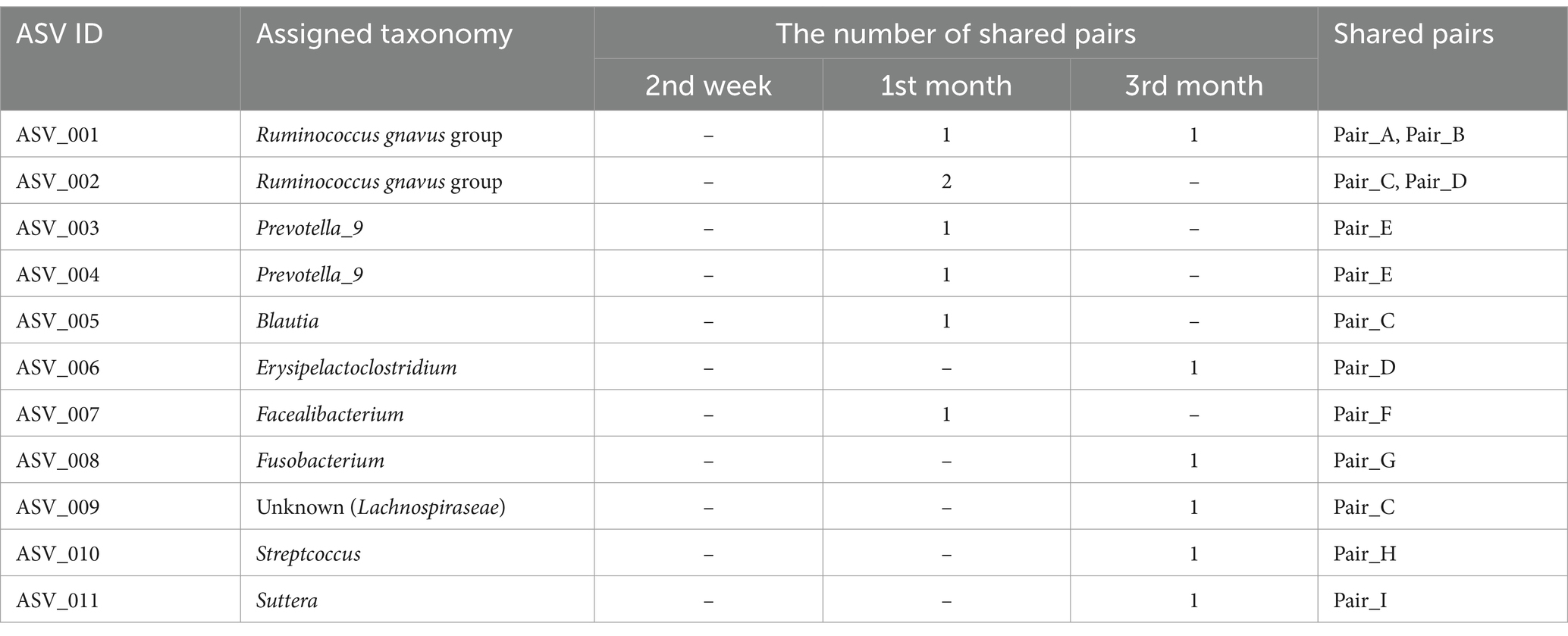
Resultados: El análisis de los ASV del gen 16S rRNA indicó que los microbios intestinales se han transferido entre humanos y perros. La estructura general de la microbiota intestinal en parejas humano-perro se mantuvo sin cambios después de 3 meses de adaptación. Sin embargo, se compartieron 11ASV entre parejas de humanos y perros. Muchos ASV compartidos fueron muy abundantes dentro de cada huésped, y esta alta abundancia puede considerarse un factor que influye en la transferencia bacteriana entre huéspedes.
Discusión: Nuestros resultados proporcionan información importante sobre el potencial de la transferencia de bacterias intestinales entre humanos y perros. Estos hallazgos se consideran cruciales para comprender el impacto de la cohabitación entre humanos y perros en varios aspectos de la salud.
1 Introducción
El concepto de «Una sola salud» se basa en la comprensión integral de que los seres humanos, los animales y el medio ambiente que los rodea están interconectados. Es un enfoque interdisciplinario para resolver problemas a través de la colaboración entre personas involucradas en la salud humana, animal y ambiental. Por lo tanto, se están llevando a cabo discusiones para promover la salud integral de las mascotas, que pasan un tiempo considerable en el mismo entorno que los humanos (1, 2). Uno de los temas centrales es el intercambio de microbios y enfermedades infecciosas; Las cuestiones relacionadas con las zoonosis, en particular, han recibido siempre una gran atención.
Los microbiomas residenciales humanos coexisten en varios sitios del cuerpo, como el intestino, la piel, los pulmones y la cavidad oral. Se estima que el número total de bacterias en el «hombre de referencia» de 70 kg es de 38 billones de células (3). El microbioma intestinal es el principal factor que mantiene la salud. Un desequilibrio debido a cambios externos puede conducir al desarrollo de enfermedades cardiovasculares, cáncer, enfermedades respiratorias, diabetes, enfermedad inflamatoria intestinal (EII), enfermedades cerebrales, enfermedades renales crónicas y enfermedades hepáticas (4, 5).
Las bacterias residenciales humanas se ven sustancialmente afectadas por múltiples factores en el entorno externo, incluidos los espacios habitables (6, 7). Se ha informado que las mascotas que comparten entornos de vida con humanos son un factor considerable que influye en la composición taxonómica y la diversidad filogenética de los microbiomas intestinales y cutáneos humanos a través de la transferencia microbiana directa o indirecta (8-14). El contacto entre humanos y mascotas altera la composición de las bacterias intestinales y potencialmente reduce el riesgo de enfermedades alérgicas en los bebés (9, 15, 16) y síndromes metabólicos (17). El perro es considerado como el primer animal domesticado (18). Los perros domésticos están en contacto diario con sus dueños y comparten sus entornos de vida. En cuanto a la salud mental, algunos estudios han demostrado que tener un perro tiene un impacto en la mejora del bienestar humano a través de cambios en las funciones fisiológicas, como la regulación endocrina (19-21). Otro estudio informó que la modificación de la microbiota canina por probióticos específicos se reflejaba en el microbioma intestinal de los niños (22). Por lo tanto, debemos comprender el impacto de las interacciones ecológicas en las estructuras microbianas y cómo afectan a la salud humana y canina.
Aunque el efecto de los perros sobre el microbioma humano se considera sustancial, la mayoría de los estudios lo han discutido a nivel taxonómico. La transferencia directa de microbios intestinales de los perros o el intercambio coincidente de los mismos taxones entre humanos y perros no está clara. El microbioma intestinal del perro era similar al microbioma intestinal humano, con un mapeo del 63% con el catálogo de genes humanos (23), lo que sugiere una posible interacción. En este estudio, planteamos la hipótesis de que pasar tiempo con los dueños conduce al intercambio microbiano entre humanos y perros, lo que resulta en microbiomas intestinales similares. Para probar esta hipótesis, analizamos el intercambio microbiano a nivel de secuencia de amplicones.
2 Materiales y métodos
2.1 Diseño del estudio
Examinamos 28 familias individuales con perros y participantes humanos entre las edades de 20 y 72 años (48,5 ± 15,7; 13 machos y 15 hembras), y perros entre las edades de 1 y 10 años (4,4 ± 2,6; 18 machos y 10 hembras; 5 raza pura, 23 mestizos; 16 callejeros, 8 reproductores, 4 entregados). Los perros se obtuvieron originalmente de refugios y criadores y fueron adoptados por la Universidad de Azabu. Estos perros se mantuvieron en un centro de entrenamiento canino de la universidad durante 6 meses y luego se adoptaron en nuevas familias como parte de un programa educativo de la Universidad de Azabu que se desarrolló de 2015 a 2022 (24). Todos los perros se mantuvieron en el interior, libres de enfermedades, sin medicación, y fueron alimentados con comida comercial para perros (incluidas pequeñas cantidades de golosinas). Las características detalladas del estudio se resumen en la Tabla 1.
2.2 Recogida de muestras
Se recogieron muestras fecales tanto de humanos como de perros. Se recogieron muestras fecales de los perros en el centro donde se mantuvieron durante 2-3 meses antes de la adopción y de los propietarios 1 semana antes de la adopción, así como de los propietarios y los perros a las 2 semanas, 1 mes y 3 meses después de la adopción. En cuanto a las muestras humanas, los dueños de perros defecaron en una hoja de inspección fecal (Nagasale 0-9761-01, AS ONE Co. Ltd., Osaka, Japón) colocada en el inodoro. Una porción de la muestra fecal se extrajo sin contacto con el agua utilizando palillos desechables, se colocó en un tubo (tubo de centrífuga de la tapa del filtro del reactor CELL, 227245, Greiner Bio-one, Tokio, Japón) y se cubrió con una tapa. Las muestras de los perros se recogieron en interiores o exteriores utilizando el mismo tipo de sábana cuando el perro defecaba. Cuando los perros defecaban, como durante un paseo, se recogía una porción no contaminada de heces sin tierra ni arena con palillos desechables y se colocaba en un tubo en las mismas condiciones anaeróbicas que se utilizan para los humanos. El tubo que contenía la muestra fecal se colocó en una bolsa (A-58, Mitsubishi Chemical Corporation, Tokio, Japón) junto con AnaeroPack-Anaero™ (A-03, Mitsubishi Gas Chemical Corporation, Tokio, Japón) y se hizo anaeróbico. Después de la recolección, las muestras se colocaron en una nevera con refrigerante congelado, se sellaron y se refrigeraron hasta el día siguiente. Las muestras de tres de los 28 pares se almacenaron en soluciones de conservación (RNAlater™ Stabilization Solution, AM7022, Invitrogen, Thermo Fisher Scientific Inc.) debido a cambios en los métodos de transporte. Este método es comparable a la congelación inmediata (25). Inmediatamente después de la defecación, se extrajo una pequeña cantidad de heces con una microespátula desechable (1-9404-02, AS ONE Co. Ltd., Osaka, Japón), se colocó en un tubo de 1,5 mL que contenía ARNlater y se selló. Estas muestras se almacenaron en un congelador a -80 °C en el laboratorio hasta su análisis.
2.3 Ética
El protocolo del estudio fue aprobado por el Comité de Ética Animal de la Universidad de Azabu (#210325-12) y el Comité de Ética para la Investigación Médica y de Salud con Sujetos Humanos de la Universidad de Azabu (#097). Todos los procedimientos se llevaron a cabo de acuerdo con las directrices y reglamentos del Comité de Ética. Se obtuvo el consentimiento informado de todos los participantes, a quienes se les proporcionó información detallada sobre los objetivos del estudio, los procedimientos, los riesgos potenciales y su derecho a retirarse en cualquier momento sin penalización. Para proteger la privacidad de los participantes, se eliminaron todos los identificadores personales y los datos se codificaron para mantener la confidencialidad y el anonimato.
2.4 Extracción total de ADN y secuenciación de alto rendimiento
Las muestras se trataron con Lysis Solution F (NIPPON GENE Co., Ltd., Tokio, Japón) y se homogeneizaron durante 2 min a 1.500 rpm utilizando un Shake Master Neo (Biomedical Science, Japón). La suspensión se sometió a un tratamiento térmico a 65 °C durante 10 minutos y se centrifugó durante 2 minutos a 12.000 × g. El ADN se extrajo del sobrenadante separado utilizando un kit de extracción de ADN Lab-Aid824s (Zeesan Biotech Co., China) de acuerdo con el protocolo del fabricante. Además, se llevaron a cabo reacciones de PCR con los cebadores universales bacterianos 1st-341f_MIX (5′-ACACTCTTTCCCTACACGACGCTCTTCCGATCT-NNNNN-CCTACGGGNGGCWGCAG-3′) y 1st-805r_MIX (5′-GTGACTGGAGTAGACGTGTGCTCTTCCGATCT-NNNNN-GACTACHVGGGTATCTAATCC-3′), para amplificar el V3-V4 del gen 16S rRNA. Las condiciones térmicas fueron de 94 °C durante 2 min, seguidas de 98 °C durante 10 s, 55 °C durante 30 s y 68 °C durante 30 s, con una extensión final a 68 °C durante 7 min. Las muestras de ADN, la preparación de la biblioteca y la secuenciación de amplicones se realizaron utilizando secuenciación de extremos emparejados de 300 pb en el kit de reactivos MiSeq v3 (Illumina Inc., San Diego, CA, EE.UU.) y la plataforma Illumina MiSeq (Illumina Inc., San Diego, CA, EE.UU.) en el Bioengineering Lab. Co., Ltd. (Kanagawa, Japón).
2.5 Análisis del microbioma
El análisis del microbioma se realizó como se describió anteriormente (26). En resumen, los datos brutos de FASTQ se importaron a la plataforma QIIME2 (versión 2023.5) como archivos qza (27). Las secuencias de eliminación de ruido y el control de calidad se llevaron a cabo utilizando QIIME dada2, que demostró identificar la variación real a las escalas más finas en los datos de secuencias de amplicones mientras arrojaba pocos falsos positivos sin construir Unidades Taxonómicas Operativas (OTU) (28). A continuación, las secuencias se convirtieron en variantes de secuencia de amplicones (ASV). Los ASV se asignaron a la base de datos SILVA SSU 138.1 utilizando el paquete de clasificación scikit-learn del clasificador de características QIIME (29, 30). Los análisis posteriores excluyeron los ASV clasificados como mitocondriales, cloroplastos o no asignados. Para evaluar el efecto de los recuentos de lecturas de secuencias en la diversidad del microbioma, trazamos los cambios en el índice de diversidad de Shannon, una medida de la diversidad alfa que tiene en cuenta tanto la riqueza como la uniformidad de las especies, en un rango de recuentos de lecturas de 0 a 10.000 utilizando curvas de rarefacción. En las curvas de rarefacción, el número de ASV se estabilizó cuando el número de lecturas alcanzó aproximadamente 4.000 (Figura suplementaria S1). Los índices de diversidad beta ponderados por las distancias de UniFrac, que evalúan las diferencias en la estructura de la comunidad microbiana en función de las distancias, se visualizaron mediante análisis de coordenadas principales (PCoA). Los datos se visualizaron utilizando ggplot2 (versión 3.4.4) (31) y ggprism (versión 1.0.4)1 (Creadores: Charlotte Dawson1, Mostrar afiliaciones 1. Universidad de Cambridge, sin fecha; Wickham, sin fecha).
2.6 Cálculo de los ASV compartidos
Se realizaron análisis de ASV compartidos como se describió anteriormente (32). En el análisis de ASV compartido, para excluir los ASV ruidosos, nos enfocamos en ASV con una abundancia de más del 1% y definimos los ASV compartidos como aquellos que se compartieron entre parejas de humanos y perros al mismo tiempo. El cálculo se llevó a cabo utilizando código Python personalizado 2 con 0,01 para p-porcentaje.
2.7 Análisis estadístico
Se utilizaron las pruebas U de Mann-Whitney para comparar el índice de diversidad de Shannon y las distancias UniFrac por pares para tener en cuenta las comparaciones entre los cursos de tiempo de muestreo. Todas las correcciones de pruebas múltiples se realizaron mediante el cálculo de la tasa de falsos descubrimientos mediante el método de Benjamini-Hochberg, y los valores Q (valores p ajustados) < 0,05 se consideraron estadísticamente significativos. Las pruebas estadísticas se realizaron utilizando SciPy (versión 1.9.3) (33) y Scikit-bio (versión 0.5.9). 3 Para comparar las diferencias en la diversidad beta (distancia UniFrac ponderada) entre especies, para todos los análisis de PERMANOVA, se realizaron 5.000 ensayos para evaluar la significación estadística. Para validar la abundancia de géneros en humanos y perros, utilizamos el análisis de composición de microbiomas (ANCOM) (34).
3 Resultados
3.1 Composición taxonómica de los microbiomas intestinales en humanos y perros
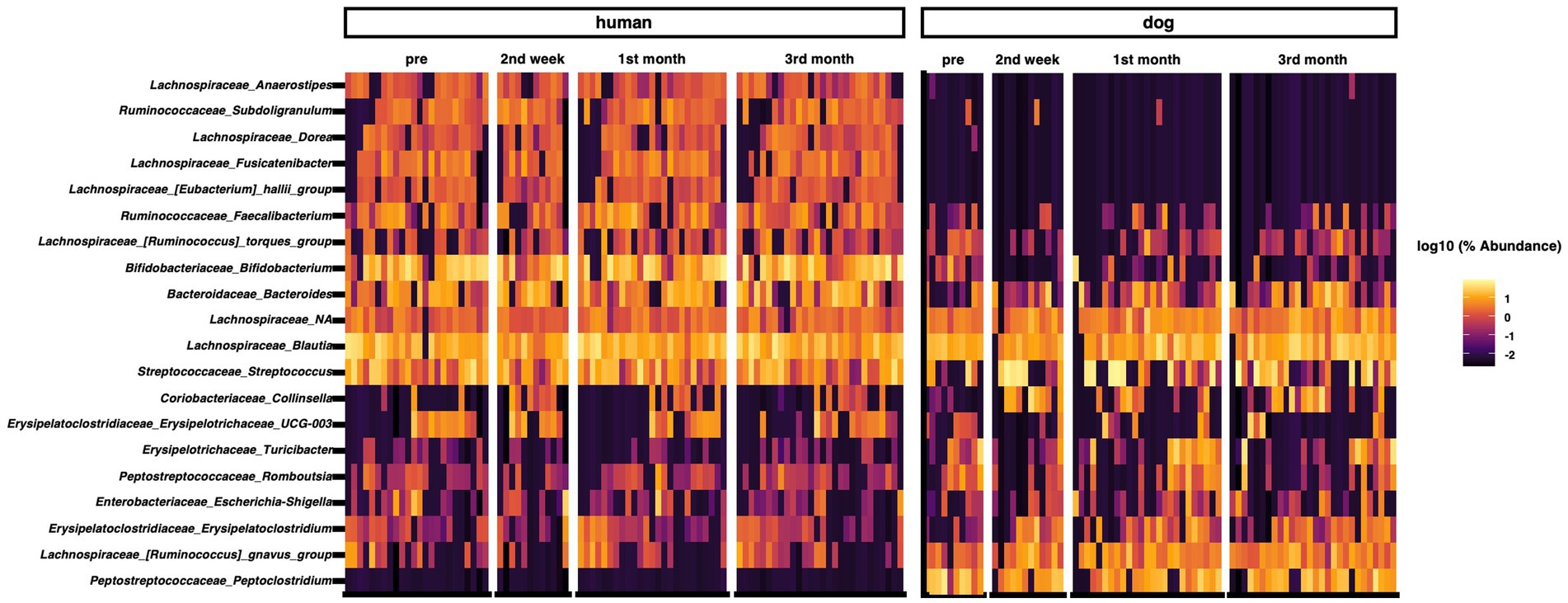
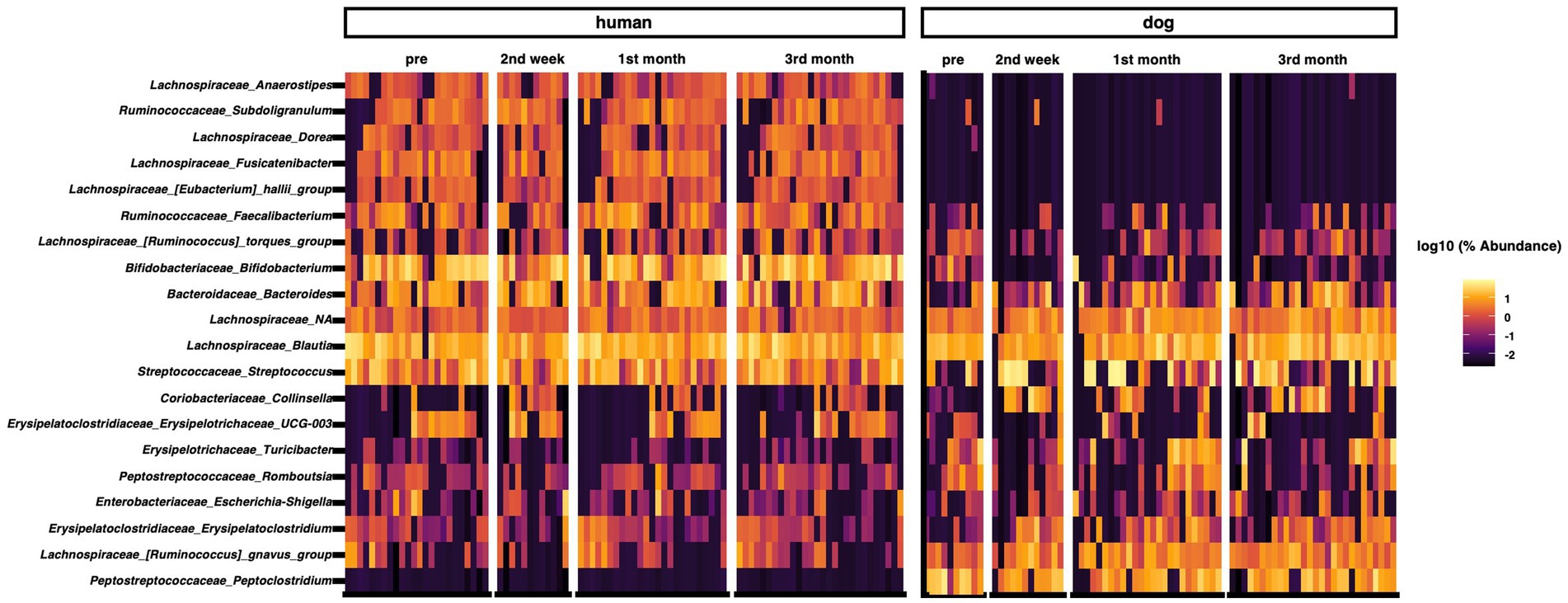
Analizamos la composición de la microbiota intestinal en humanos y perros. Los géneros más abundantes en el intestino humano fueron Bifidobacterium, Blautia, Streptococcus, Bacteroides y Faecalibacterium (Figura 1). Fusicatenibacter fue significativamente abundante solo en humanos, según lo determinado por ANCOM (Tabla Suplementaria S1). Los géneros más abundantes en el intestino de los perros fueron Streptococcus, Blautia, Peptoclostridium, Fusobacterium y Ruminococcus gnavus (Figura 1). Peptocrostridium y Blautia fueron significativamente más abundantes en los perros, en comparación con los humanos, utilizando ANCOM (Tabla Suplementaria S1). Blautia y Streptococcus eran abundantes en humanos y perros. Los cinco géneros dominantes principales en cada huésped representaron colectivamente el 51,6% (rango intercuartílico [RIC] 42,0-63,7) en el intestino humano y el 46,2% (IQR 33,0-63,7) en el intestino del perro, según la abundancia relativa mediana (Figura suplementaria S2A). El índice de diversidad de Shannon, el índice más utilizado para medir la diversidad alfa del microbioma intestinal (35), no cambió a lo largo del período de convivencia de tres meses (Figura suplementaria S2B).
3.2 Los cambios en la diversidad y las estructuras microbianas a través de la cohabitación de humanos y perros
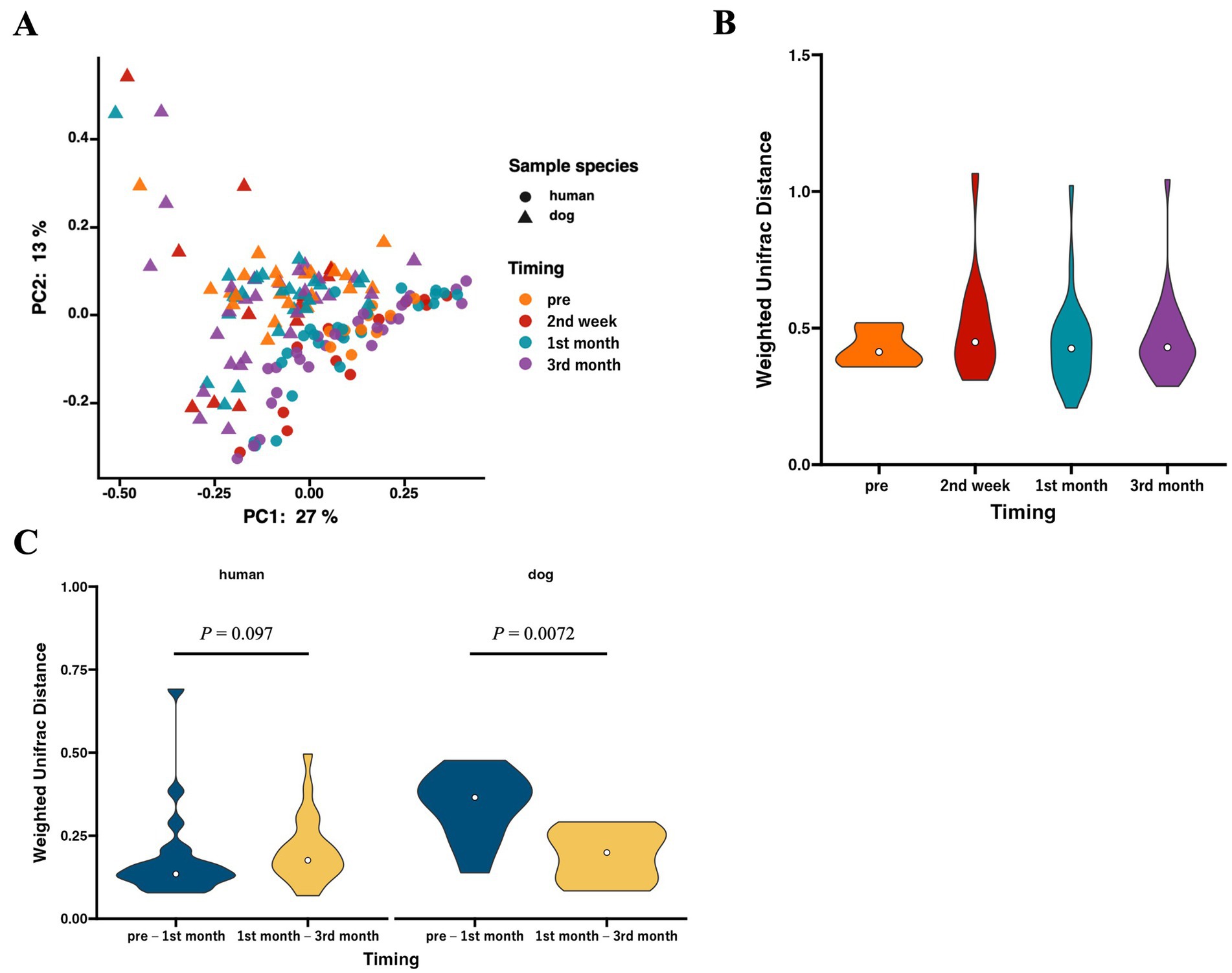
Analizamos la diversidad beta para investigar la influencia de la cohabitación entre humanos y perros en las comunidades bacterianas. De acuerdo con el PCoA utilizando distancias UniFrac ponderadas, se formaron dos conglomerados distintos basados en la especie huésped (p = 0,00020 basado en PERMANOVA), mientras que no se formaron conglomerados dentro de las especies en función de la duración de la cohabitación (Figura 2A). Se calcularon y compararon las distancias ponderadas de UniFrac para cada pareja de convivencia humano-perro a lo largo de diferentes períodos de convivencia, y no se observaron variaciones en función de la duración de la convivencia (Figura 2B). Los cálculos de los cambios en las distancias ponderadas de UniFrac durante el período de cohabitación para los mismos individuos revelaron que, si bien el microbioma intestinal humano no cambió con el tiempo después de la cohabitación, el microbioma intestinal del perro mostró alteraciones significativas durante el primer mes de cohabitación (Figura 2C).
3.3 Cambios en las series temporales de los ASV compartidos de la microbiota intestinal entre humanos y perros
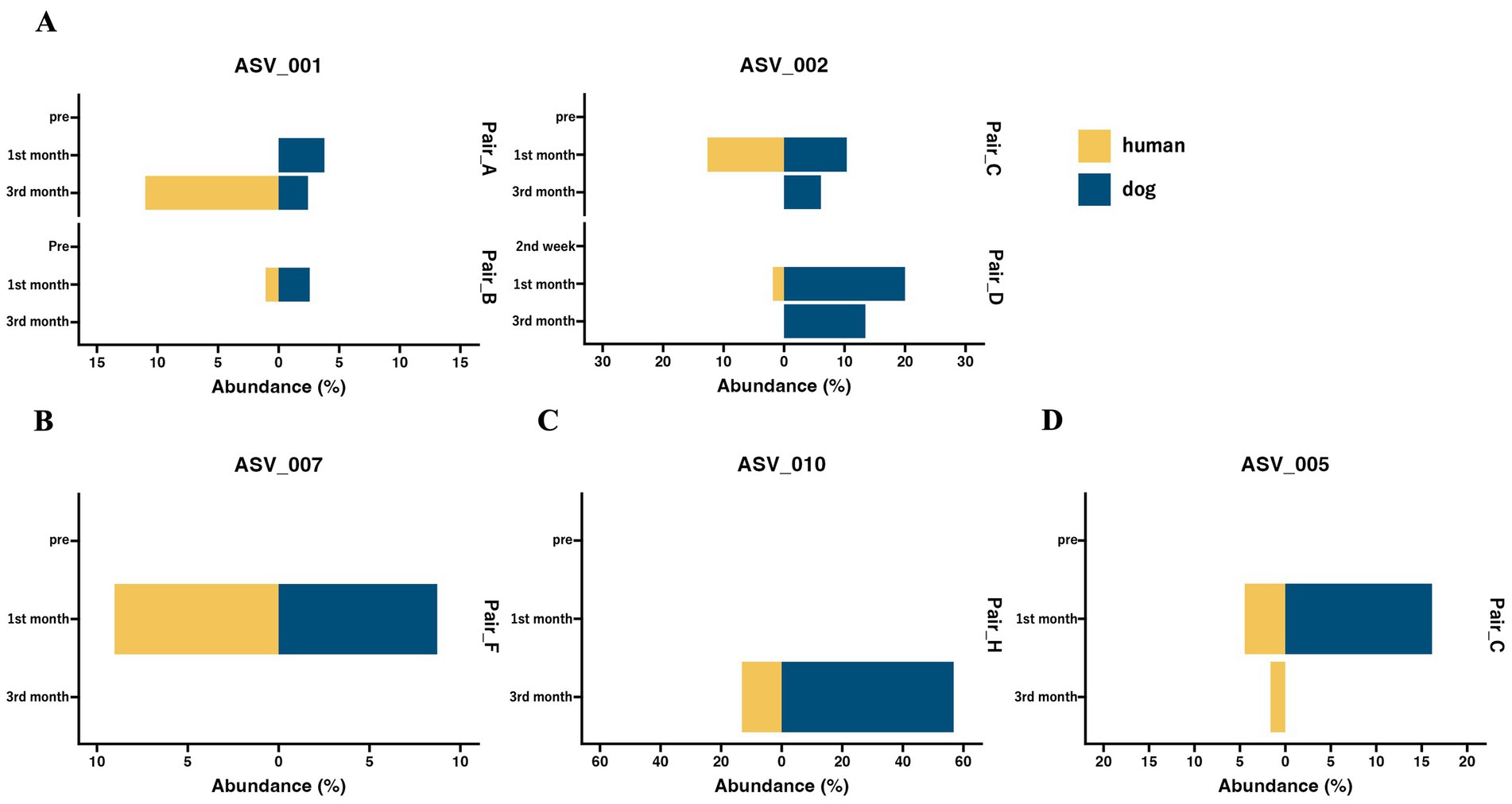
Aunque los microbiomas intestinales generales de las parejas humano-perro no se vieron influidos por las condiciones de vida compartidas, se consideró la posibilidad de compartir el mismo taxón a nivel de ASV dentro de cada pareja. De todas las muestras se obtuvieron un total de 5.709 ASVs. El análisis del ASV compartido reveló que solo se compartieron 11 ASV dentro de parejas humano-perro durante el 1er y 3er mes de cohabitación, pero no durante la 2da semana de cohabitación (Tabla 2). Se compartieron múltiples ASV en algunos pares, como Pair_C, D y E. ASV001 y ASV002 se asignaron al grupo R. gnavas, el principal género bacteriano en los intestinos de los perros, y se compartieron entre múltiples pares (Figura 3A). En una pareja, estos ASV se identificaron exclusivamente en una muestra de perro en el primer mes y luego se compartieron entre humanos y perros en el tercer mes. Las otras parejas compartieron ASV en el mismo momento. ASV007, asignado a Faecalibacterium, se compartió durante el primer mes (Figura 3B). El ASV no se detectó en ningún otro punto temporal de ninguna de las cepas hospedadoras. El ASV010, asignado a Streptococcus, se detectó solo en el tercer mes, con una abundancia relativa del 56,8% en la muestra de perro y se compartió en este momento (Figura 3C). El ASV005, asignado a Blautia, apareció simultáneamente y se compartió durante el primer mes (Figura 3D). Los ASV asignados a Prevotella_9, Erysipelactoclostridium, Fusobacterium, Lachnospiraceae y Sutterella también se compartieron entre varias parejas que cohabitaban (Tabla suplementaria S2).
4 Discusión
En el presente estudio, demostramos que los cinco géneros dominantes en cada huésped constituían colectivamente aproximadamente la mitad de la abundancia relativa en sus respectivos huéspedes. Los cinco géneros dominantes en el microbioma intestinal humano, Bifidobacterium, Blautia, Streptococcus, Bacteroides y Faecalibacterium, han sido reportados como componentes principales del microbioma intestinal humano japonés (36). Los géneros abundantes en el microbioma intestinal de los perros, incluidos Streptococcus, Blautia, Peptoclostridium y Fusobacterium, también se han identificado como componentes principales del microbioma intestinal del perro (37). R. gnavas ha sido reportada como la especie más abundante en el intestino del perro (23), que también se identificó en este estudio. Se consideró que la microbiota intestinal humana y canina de este estudio no presentaba diferencias significativas en comparación con las de informes anteriores.
Evaluamos el índice de diversidad de Shannon para investigar el impacto de la cohabitación entre humanos y perros en la diversidad de la comunidad. Sin embargo, no se observó variación en la duración de la convivencia. Estudios anteriores han informado que las diversidades alfa en el microbioma intestinal humano no muestran diferencias sustanciales (10, 38), lo que coincide con los resultados actuales. La diversidad beta basada en las distancias ponderadas de UniFrac se comparó a lo largo del tiempo, sin revelar cambios sustanciales en las estructuras microbianas generales entre parejas dentro del período de cohabitación de tres meses. Al igual que en estudios anteriores, en nuestro estudio, los huéspedes (humanos o perros) fueron los principales factores que explicaron las diferencias en la microbiota intestinal, y la cohabitación no pareció ser uno de los principales factores que afectaran a la composición general del microbioma intestinal (12, 22). Finalmente, analizamos las variaciones temporales en la diversidad beta para dilucidar los cambios en las estructuras microbianas dentro de las especies debido a la cohabitación. La distancia ponderada de UniFrac de los microbiomas intestinales de los perros entre el período previo a la prueba y el primer mes fue significativamente diferente de la distancia entre el primer y el tercer mes. Los cambios en las condiciones de vida, como la dieta y el lugar de residencia, cuando los perros rescatados comienzan a convivir con los humanos provocan alteraciones significativas en su microbiota intestinal durante las primeras etapas de la convivencia (39, 40). Sin embargo, no se observaron cambios temporales en la microbiota intestinal humana debido a la cohabitación, lo que sugiere que, en comparación con los perros, el impacto de los cambios ambientales causados por la cohabitación es limitado.
Aunque no se observó una correlación entre las parejas de humanos y perros al considerar todo el microbioma, consideramos la posibilidad de bacterias individuales compartidas entre humanos y perros. Estudios previos han comparado los cambios en la microbiota intestinal debidos a la cohabitación a nivel de géneros bacterianos u OTU (10, 22, 38). Sin embargo, estos análisis no determinaron si las mismas bacterias se transfirieron o se compartieron. Para evaluar con precisión el reparto de la microbiota intestinal entre humanos y perros, llevamos a cabo un análisis de ASV compartido, que es un enfoque para inferir la proporción de reparto del microbioma a nivel de ASV entre muestras y que se ha utilizado anteriormente (32, 41, 42). Si bien el intercambio de ASVs no fue detectable después de 2 semanas de convivencia, se observó en el 1er y 3er mes. Estos resultados sugieren que un período de convivencia de al menos 1 mes puede ser importante para el intercambio microbiano. Por otro lado, el número de ASV compartidos fue similar a 1 mes y 3 meses de convivencia. Esto sugiere que, más allá del primer mes, el número de ASV compartidos no sigue aumentando con el tiempo, sino que se estabiliza en un cierto nivel. Entre las nueve parejas en las que se confirmó el uso compartido de ASV, tres parejas compartían múltiples ASV, lo que sugiere la posibilidad de que existan condiciones que faciliten el intercambio de ASV. La presencia o ausencia de la mayoría de los ASV compartidos fluctuó a lo largo del tiempo. Se sabe que las bacterias intestinales fluctúan en diversas condiciones, como el entorno externo, la dieta y el estado de salud, y se considera necesario establecer líneas de base individuales mediante la observación a largo plazo para un análisis detallado.
Entre los 11 ASV, seis se clasificaron como los géneros bacterianos dominantes en los cinco primeros de cada huésped. ASV001 y ASV002, asignados al grupo R. gnavas, los géneros bacterianos comunes en el intestino del perro, estuvieron presentes en las muestras de perro de las parejas compartidas en más puntos de tiempo y se detectaron abundantemente en las muestras de perro (Tabla suplementaria S3). Estos resultados sugirieron que los dos ASV se transfirieron de perros a humanos. Estudios previos han demostrado que el Ruminococcus grupo 2 es más abundante en los intestinos de los niños con perros, lo que sugiere que el Ruminococcus se transfiere fácilmente de los perros a los humanos (22). El grupo R. gnavus, que se encuentra en abundancia en las heces de pacientes humanos con EII, produce polisacáridos y desencadena la secreción de TNF-α de las células dendríticas (43). Se especula que la transferencia del grupo R. gnavus de los perros a los humanos afecta negativamente a la salud humana. ASV007, asignado a Faecalibacterium, el principal género bacteriano en el intestino humano, se compartió en un par y se detectó principalmente en muestras humanas (Tabla suplementaria S3). Estos resultados implican que los ASV son compartidos de humanos a perros. Faecalibacterium es conocida como una bacteria beneficiosa en el intestino humano, donde se reduce en varias enfermedades, incluida la enfermedad inflamatoria intestinal (EII) (44). Puede tener efectos beneficiosos cuando se transfiere a los perros. El ASV010, asignado a Streptococcus, existió principalmente en muestras de perros (Tabla Suplementaria S3) y se detectó en el tercer mes, con una abundancia relativa del 56,8% en la muestra de perros. Se han reportado efectos negativos, como un aumento en la proporción de estreptococos en el intestino debido a la enteropatía inflamatoria crónica en perros, y esta puede ser una de las razones por las que ASV010 se volvió dominante (45). Es posible que este aumento temporal de la proporción en los perros estuviera asociado a su transmisión a los humanos, donde también podría causar efectos negativos. ASV005, asignado a Blautia, se detectó significativamente en el intestino del perro según ANCOM, mientras que Blautia es un género abundantemente presente en ambos huéspedes. Estos resultados sugieren que este ASV es compartido entre perros y humanos. Cabe destacar que este ASV se detectó exclusivamente en el intestino humano 2 meses después de compartirlo. Blautia es el segundo género más abundante en los humanos, y es posible que Blautia se haya transferido de los perros a colonizar fácilmente a los humanos. Las OTU clasificadas como Blautia se vuelven más abundantes en el intestino humano debido a la cohabitación con mascotas, lo que apoya esta hipótesis (38). Blautia es reconocida por su potencial función probiótica en el intestino humano y se especula que la transferencia de Blautia de los perros a los humanos puede tener efectos beneficiosos (46). Algunas bacterias anaeróbicas, como Ruminococcus y Blautia, se transfieren de los perros a los humanos. Estos resultados sugieren que la transferencia de bacterias puede ser desencadenada por la limpieza de heces que contienen una alta abundancia de bacterias anaeróbicas, similar a las infecciones zoonóticas (47). En conclusión, el patrón de este ASV compartido sugiere que el intercambio mutuo de bacterias entre humanos y perros y la alta abundancia son factores críticos para la transferencia microbiana entre huéspedes.
En este estudio, nuestro objetivo era analizar las interacciones entre la microbiota intestinal de humanos adultos y perros. En consecuencia, eran muy pocos (solo 11) los ASV compartidos. Los lactantes expuestos a perros a una edad temprana tienen una microbiota intestinal alterada, lo que respalda un posible mecanismo que explica la reducción del riesgo de atopia y asma (48). El efecto de la transferencia microbiana puede depender de la edad del huésped. Además, la tenencia de un perro aumenta la similitud de la microbiota cutánea entre humanos y perros, en lugar de la microbiota intestinal (12). La naturaleza cerrada del ambiente intestinal puede reducir la probabilidad de transferencia bacteriana entre huéspedes. En el futuro, puede ser importante evaluar los ASV compartidos en varios grupos de edad y ubicaciones para comprender mejor las interacciones entre humanos y perros.
Este estudio tiene tres limitaciones. En primer lugar, utilizamos secuencias de amplicones de la región V3-V4 del gen 16S rRNA, lo que limitaba la identificación de las bacterias al nivel del género (30). El sesgo de amplificación por PCR y las diferencias en los métodos de extracción de ADN afectan a la precisión de la composición relativa del microbioma intestinal (49, 50). El análisis compartido de ASV es una forma conveniente de rastrear las fuentes microbianas, y el gen 16S rRNA de longitud completa se puede usar para predecir el intercambio microbiano entre muestras de manera más rigurosa. Los linajes de cepas bacterianas entre los dos grupos se pueden comparar utilizando genomas ensamblados con metagenoma, que muestran más precisión en el seguimiento a nivel de genoma completo, no solo en las regiones hipervariables específicas restringidas del gen 16S rRNA. La segunda limitación es un desequilibrio en el tamaño de la muestra en cada punto de tiempo. A diferencia del primer y tercer mes, donde se recogieron 25 y 28 muestras, respectivamente, el análisis se realizó utilizando solo 10 muestras del pretest del perro y 12 muestras recogidas durante la 2ª semana. No se puede descartar que la falta de ASVs compartidos en las muestras de la 2ª semana pueda atribuirse al pequeño tamaño de la muestra. Por último, todavía tenemos que demostrar el impacto de los ASV compartidos en cada host. El análisis de los ASVs compartidos reveló la posibilidad de transferencia bacteriana. No se pudo aclarar la influencia del intercambio de bacterias en la salud de cada huésped. Creemos que son necesarios experimentos adicionales, como los experimentos con animales con bacterias aisladas demostrados en este estudio.
En conclusión, este estudio combinó el amplicón del gen ARNr 16S y análisis compartidos de ASV para proporcionar evidencia de alta resolución de la transferencia del microbioma intestinal durante la cohabitación entre humanos y perros. Los ASV compartidos en el intestino exhibieron una alta abundancia relativa en cada huésped, lo que sugiere que es más probable que el intercambio de ASV ocurra en el taxón dominante. Muchos ASV que se confirmó que eran compartidos fueron los taxones dominantes en cada hospedador. Se necesita un tamaño de muestra más grande en estudios futuros para diferenciar los efectos de los diferentes entornos de vida, las razas de perros, el sexo del huésped, la edad del huésped y el tiempo que se pasa con los perros. Se requieren más análisis para determinar la relevancia de los ASV específicamente compartidos por cada individuo para los riesgos posteriores para la salud.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/, DRR542122–DRR542285.
Declaración ética
Los estudios con seres humanos fueron aprobados por el Comité de Ética para la Investigación Médica y de la Salud con Seres Humanos de la Universidad de Azabu. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. El consentimiento informado por escrito para participar en este estudio fue proporcionado por los tutores legales o familiares más cercanos de los participantes. Los estudios en animales fueron aprobados por el Comité de Ética Animal de la Universidad de Azabu. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones de los autores
YI: Curación de datos, Análisis formal, Validación, Visualización, Escritura – borrador original, Escritura – revisión y edición. MN: Conceptualización, Obtención de fondos, Metodología, Supervisión, Validación, Visualización, Redacción – revisión y edición. KK: Curación de datos, Investigación, Redacción, revisión y edición. KI: Curación de datos, Análisis formal, Investigación, Metodología, Redacción – borrador original, Redacción – revisión y edición. TK: Conceptualización, Curación de datos, Análisis formal, Obtención de fondos, Investigación, Metodología, Supervisión, Validación, Redacción, revisión y edición.
Financiación
El/los autor/es declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo contó con el apoyo de JSPS KAKENHI (números de subvención 21H05173 y 21H03333), del cual MN fue el investigador principal, y JSPS KAKENHI (número de subvención 23H05472) y JST (número de subvenciónJPMJMI21J3), del cual TK es el investigador principal.
Reconocimientos
Todos los autores agradecen a Morgenrot Inc. por proporcionar el entorno computacional para el análisis. Nos gustaría agradecer a Editage (www.editage.jp) por la edición en inglés.
Conflicto de intereses
KI es miembro de la junta directiva de BIOTA Inc., Tokio, Japón. YI es empleado de BIOTA Inc. como desarrollador a tiempo parcial.
El resto de los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un potencial conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1417461/full#supplementary-material
Notas
1. ^https://csdaw.github.io/ggprism/
2. ^Q2-shared_asv, https://github.com/biota-inc/q2-shared_asv.
3. ^http://scikit-bio.org
Referencias
1. Overgaauw, PAM, Vinke, CM, van Hagen, MAE y Lipman, LJA. Una perspectiva de One Health sobre la relación humano-animal de compañía con énfasis en los aspectos zoonóticos. Int J Environ Res Salud Pública. (2020) 17:3789. doi: 10.3390/ijerph17113789
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Zeigler, MK, y Vander Wyst, KB. Asociaciones y transferencias microbianas a través de la tríada One Health, efectos sobre la adiposidad y el temperamento humanos y animales: un protocolo para un estudio piloto observacional. Frente Salud Pública. (2023) 11:1225188. doi: 10.3389/fpubh.2023.1225188
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Sender, R, Fuchs, S y Milo, R. Estimaciones revisadas para el número de células humanas y bacterianas en el cuerpo. PLoS Biol. (2016) 14:E1002533. doi: 10.1371/journal.pbio.1002533
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Shreiner, AB, Kao, JY y Young, VB. El microbioma intestinal en la salud y en la enfermedad. Curr Opin Gastroenterol. (2015) 31:69–75. doi: 10.1097/MOG.00000000000000139
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Hou, K, Wu, Z-X, Chen, X-Y, Wang, J-Q, Zhang, D, Xiao, C, et al. La microbiota en la salud y las enfermedades. Objetivo de transducción de señal. (2022) 7:135. doi: 10.1038/s41392-022-00974-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Ito, K. Comprender las características de los microbiomas del entorno construido y sus efectos en el cuerpo humano. Ambiente interior. (2023) 26:29–42. doi: 10.7879/siej.26.29
7. Gilbert, JA, y Stephens, B. Microbiología del entorno construido. Nat Rev Microbiol. (2018) 16:661–70. doi: 10.1038/s41579-018-0065-5
8. Sitarik, AR, Havstad, S, Levin, AM, Lynch, SV, Fujimura, KE, Ownby, DR, et al. La introducción de perros altera la microbiota del polvo doméstico. Aire interior. (2018) 28:539–47. doi: 10.1111/ina.12456
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Tun, HM, Konya, T, Takaro, TK, Brook, JR, Chari, R, Field, CJ, et al. La exposición a mascotas peludas domésticas influye en la microbiota intestinal de los lactantes a los 3-4 meses después de varios escenarios de parto. Microbioma. (2017) 5:40. doi: 10.1186/s40168-017-0254-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Jiang, C, Cui, Z, Fan, P y Du, G. Efectos de la tenencia de un perro en la microbiota intestinal de los propietarios de edad avanzada. PLoS Uno. (2022) 17:e0278105. doi: 10.1371/journal.pone.0278105
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Torres, S, Clayton, JB, Danzeisen, JL, Ward, T, Huang, H, Knights, D, et al. Existen diversas comunidades bacterianas en la piel canina y se ven afectadas por la convivencia y el tiempo. ParesJ. (2017) 5:E3075. doi: 10.7717/peerj.3075
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Song, SJ, Lauber, C, Costello, EK, Lozupone, CA, Humphrey, G, Berg-Lyons, D, et al. Los miembros de la familia que cohabitan comparten la microbiota entre sí y con sus perros. eVida. (2013) 2:E00458. doi: 10.7554/eLife.00458
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Wetzels, SU, Strachan, CR, Conrady, B, Wagner, M, Burgener, IA, Virányi, Z, et al. Los lobos, los perros y los humanos, en contacto regular, pueden afectar mutuamente a la microbiota cutánea de los demás. Sci Rep. (2021) 11:17106. doi: 10.1038/s41598-021-96160-7
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Kuthyar, S, y Reese, AT. Variación en la exposición microbiana en la interfaz humano-animal y las implicaciones para los resultados de salud mediados por el microbioma. mSystems. (2021) 6:E0056721. doi: 10.1128/mSystems.00567-21
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Azad, MB, Konya, T, Maughan, H, Guttman, DS, Field, CJ, Sears, MR, et al. La microbiota intestinal infantil y la hipótesis higiénica de las enfermedades alérgicas: impacto de los animales domésticos y los hermanos en la composición y diversidad de la microbiota. Alergia Asma Clin Immunol. (2013) 9:15. doi: 10.1186/1710-1492-9-15
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Nermes, M, Endo, A, Aarnio, J, Salminen, S e Isolauri, E. Las mascotas peludas modulan la composición de la microbiota intestinal en lactantes con riesgo de enfermedad alérgica. J Alergia Clin Immunol. (2015) 136:1688–90.e1. doi: 10.1016/j.jaci.2015.07.029
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Arenas-Montes, J, Pérez-Martínez, P, Vals-Delgado, C, Romero-Cabrera, JL, Cardelo, MP, León-Acuña, A, et al. Tener una mascota se asocia con cambios en la composición de la microbiota intestinal y podría influir en el riesgo de trastornos metabólicos en los seres humanos. Animales (Basilea). (2021) 11:2347. doi: 10.3390/ani11082347
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Wang, Dios, Zhai, W, Yang, H-C, Wang, L, Zhong, L, Liu, Y-H, et al. Del sur de Asia Oriental: la historia natural de los perros domésticos en todo el mundo. Res. de celda. (2016) 26:21–33. DOI: 10.1038/CR.2015.147
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Koyasu, H, Ogasawara, S, Kikusui, T y Nagasawa, M. La propiedad de perros y gatos conduce a niveles más altos de bienestar y confianza general a través de la participación familiar en la adolescencia tardía. Frente Vet Sci. (2023) 10:1220265. doi: 10.3389/fvets.2023.1220265
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Nagasawa, M, Kikusui, T, Onaka, T y Ohta, M. La mirada del perro a su dueño aumenta la oxitocina urinaria del dueño durante la interacción social. Horm Compórtate. (2009) 55:434–41. doi: 10.1016/j.yhbeh.2008.12.002
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Endo, K, Yamasaki, S, Ando, S, Kikusui, T, Mogi, K, Nagasawa, M, et al. La tenencia de perros y gatos predice el bienestar mental de los adolescentes: un estudio longitudinal basado en la población. Int J Environ Res Salud Pública. (2020) 17:884. doi: 10.3390/ijerph17030884
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Gómez-Gallego, C, Forsgren, M, Selma-Royo, M, Nermes, M, Collado, MC, Salminen, S, et al. La composición y la diversidad de la microbiota intestinal de los niños son modificables por los perros domésticos: el impacto de un probiótico específico para perros. Microorganismos. (2021) 9:557. doi: 10.3390/microorganismos9030557
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Coelho, LP, Kultima, JR, Costea, PI, Fournier, C, Pan, Y, Czarnecki-Maulden, G, et al. Similitud de los microbiomas intestinales del perro y del ser humano en el contenido de genes y la respuesta a la dieta. Microbioma. (2018) 6:72. doi: 10.1186/s40168-018-0450-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Nagasawa, M, Mogi, K y Kikusui, T. Angustia continua entre los perros abandonados en Fukushima. Sci Rep. (2012) 2:724. doi: 10.1038/srep00724
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Wang, Z, Zolnik, CP, Qiu, Y, Usyk, M, Wang, T, Strickler, HD, et al. Comparación de métodos de recolección fecal para estudios de microbioma y metabolómica. La célula frontal infecta el microbiol. (2018) 8:301. doi: 10.3389/fcimb.2018.00301
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Ito, K, Niwa, R, Yamagishi, Y, Kobayashi, K, Tsuchida, Y, Hoshino, G, et al. Un caso único en el que la fermentación al estilo Kimoto se completó con leuconostoc como género dominante sin pasar a lactobacillus. J Biosci Bioeng. (2023) 135:451–7. doi: 10.1016/j.jbiosc.2023.03.007
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Bolyen, E, Rideout, JR, Dillon, MR, Bokulich, NA, Abnet, CC, Al-Ghalith, GA, et al. Ciencia de datos de microbioma reproducible, interactiva, escalable y extensible utilizando QIIME 2. Nat Biotechnol. (2019) 37:852–7. doi: 10.1038/s41587-019-0209-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Callahan, BJ, McMurdie, PJ, Rosen, MJ, Han, AW, Johnson, AJA y Holmes, SP. DADA2: inferencia de muestras de alta resolución a partir de datos de amplicones de Illumina. Métodos Nat. (2016) 13:581–3. doi: 10.1038/nmeth.3869
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Quast, C, Pruesse, E, Yilmaz, P, Gerken, J, Schweer, T, Yarza, P, et al. El proyecto SILVA ribosomal RNA gene database: mejora del procesamiento de datos y herramientas basadas en la web. Ácidos nucleicos Res. (2013) 41:D590-6. doi: 10.1093/nar/gks1219
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Bokulich, NA, Kaehler, BD, Rideout, JR, Dillon, M, Bolyen, E, Knight, R, et al. Optimización de la clasificación taxonómica de secuencias de amplicones de genes marcadores con el complemento q2-feature-classifier de QIIME 2. Microbioma. (2018) 6:90. doi: 10.1186/s40168-018-0470-z
31. Villanueva, RAM, y Chen, ZJ. ggplot2: Gráficos elegantes para el análisis de datos (2ª ed.). Medición. (2019) 17:160–7. doi: 10.1080/15366367.2019.1565254
32. Niwa, R, Chen, D, Seong, YA, Jo, K e Ito, K. El contacto directo de los lechos de salvado de arroz fermentado promueve la transmisión de bacterias del ácido láctico de los alimentos a las manos. FEMS Microbiol Lett. (2023) 370:fnad120. doi: 10.1093/femsle/fnad120
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Virtanen, P, Gommers, R, Oliphant, TE, Haberland, M, Reddy, T, Cournapeau, D, et al. SciPy 1.0: algoritmos fundamentales para la computación científica en Python. Métodos Nat. (2020) 17:261–72. doi: 10.1038/s41592-019-0686-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Mandal, S, Van Treuren, W, White, RA, Eggesbø, M, Knight, R, y Peddada, SD. Análisis de la composición de los microbiomas: un método novedoso para estudiar la composición microbiana. Microb Ecol Health Dis. (2015) 26:27663. doi: 10.3402/mehd.v26.27663
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Samuthpongtorn, C, Nopsopon, T y Pongpirul, K. Medidas de la diversidad del microbioma intestinal para afecciones metabólicas: una revisión sistemática del alcance. bioRxiv. (2021). doi: 10.1101/2021.06.25.21259549
36. Takagi, T, Inoue, R, Oshima, A, Sakazume, H, Ogawa, K, Tominaga, T, et al. Tipificación de la comunidad de microbiota intestinal en sujetos japoneses. Microorganismos. (2022) 10:664. DOI: 10.3390/Microorganismos10030664
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Kubinyi, E, Bel Rhali, S, Sándor, S, Szabó, A y Felföldi, T. La composición del microbioma intestinal se asocia con la edad y el rendimiento de la memoria en perros domésticos. Animales (Basilea). (2020) 10:1488. doi: 10.3390/ani10091488
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Kates, AE, Jarrett, O, Skarlupka, JH, Sethi, A, Duster, M, Watson, L, et al. La tenencia de mascotas domésticas y la diversidad microbiana de la microbiota intestinal humana. La célula frontal infecta el microbiol. (2020) 10:73. doi: 10.3389/fcimb.2020.00073
39. Kim, J, An, J-U, Kim, W, Lee, S y Cho, S. Diferencias en la microbiota intestinal de perros (Canis lupus familiaris) alimentados con una dieta natural o un alimento comercial reveladas por la plataforma Illumina MiSeq. Pathog de tripas. (2017) 9:68. doi: 10.1186/s13099-017-0218-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Pilla, R, y Suchodolski, JS. El microbioma intestinal de perros y gatos, y la influencia de la dieta. Vet Clin North Am Pequeño Anim Pract. (2021) 51:605–21. DOI: 10.1016/j.cvsm.2021.01.002
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Kageyama, S, Furuta, M, Takeshita, T, Ma, J, Asakawa, M y Yamashita, Y. Adquisición de alto nivel de bacterias orales maternas en la microbiota oral infantil alimentada con fórmula. MBio. (2022) 13:E0345221. doi: 10.1128/mbio.03452-21
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Ito, K, Niwa, R, Kobayashi, K, Nakagawa, T, Hoshino, G, y Tsuchida, Y. Una materia oscura en la elaboración del sake: el origen de los microbios que producen un iniciador de fermentación al estilo Kimoto. Microbiol frontal. (2023) 14:1112638. doi: 10.3389/fmicb.2023.1112638
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Henke, MT, Kenny, DJ, Cassilly, CD, Vlamakis, H, Xavier, RJ y Clardy, J. Ruminococcus gnavus, un miembro del microbioma intestinal humano asociado con la enfermedad de Crohn, produce un polisacárido inflamatorio. Proc Natl Acad Sci USA. (2019) 116:12672–7. doi: 10.1073/pnas.1904099116
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Martín, R, Ríos-Covian, D, Huillet, E, Auger, S, Khazaal, S, Bermúdez-Humarán, LG, et al. Faecalibacterium: un género bacteriano con aplicaciones prometedoras para la salud humana. FEMS Microbiol Rev. (2023) 47:fuad039. doi: 10.1093/femsre/fuad039
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. AlShawaqfeh, MK, Wajid, B, Minamoto, Y, Markel, M, Lidbury, JA, Steiner, JM, et al. Un índice de disbiosis para evaluar los cambios microbianos en muestras fecales de perros con enteropatía inflamatoria crónica. FEMS Microbiol Ecol. (2017) 93. doi: 10.1093/femsec/fix136
46. Liu, X, Mao, B, Gu, J, Wu, J, Cui, S, Wang, G, et al. Blautia: ¿un nuevo género funcional con posibles propiedades probióticas? Microbios intestinales. (2021) 13:1–21. doi: 10.1080/19490976.2021.1875796
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Ghasemzadeh, I, y Namazi, SH. Revisión de infecciones zoonóticas bacterianas y virales transmitidas por perros. J Med Vida. (2015) 8:1–5.
48. Panzer, AR, Sitarik, AR, Fadrosh, D, Havstad, SL, Jones, K, Davidson, B, et al. El impacto de la tenencia prenatal de perros en el desarrollo de la microbiota intestinal infantil. Clin Exp Alergia. (2023) 53:833–45. doi: 10.1111/cea.14303
Resumen de PubMed | Texto completo de Crossref | Google Académico
49. Bonk, F, Popp, D, Harms, H y Centler, F. Cuantificación basada en PCR de abundancias específicas de taxones en comunidades microbianas: cuantificación y prevención de errores comunes. J Métodos de Microbiol. (2018) 153:139–47. doi: 10.1016/j.mimet.2018.09.015
Resumen de PubMed | Texto completo de Crossref | Google Académico
Keywords: dogs, gut microbiome, human-pet microbial interaction, shared amplicon sequence variants, One Health
Citation: Ito Y, Nagasawa M, Koyama K, Ito K and Kikusui T (2024) Comparative analysis based on shared amplicon sequence variants reveals that cohabitation influences gut microbiota sharing between humans and dogs. Front. Vet. Sci. 11:1417461. doi: 10.3389/fvets.2024.1417461
Received: 15 April 2024; Accepted: 25 September 2024;
Published: 07 October 2024.
Edited by:
Kate Worthing, The University of Sydney, Australia
Reviewed by:
Stephanie Salyer, Centers for Disease Control and Prevention (CDC), United States
George Golovko, University of Texas Medical Branch at Galveston, United States
Copyright © 2024 Ito, Nagasawa, Koyama, Ito and Kikusui. This is an open-access article distributed under the terms of the Creative Commons Attribution License (CC BY).
*Correspondencia: Takefumi Kikusui, takkiku@carazabu.com; Kohei Ito, kohei@biota.ne.jp
†Estos autores han contribuido por igual a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía