Cribado e identificación de los antígenos dominantes del virus de la peste porcina africana
 Zhaoyang Xu1,2†
Zhaoyang Xu1,2† Yifan Hu1†
Yifan Hu1† Junbo Li1,3
Junbo Li1,3 Ancheng Wang1
Ancheng Wang1 Xin Meng1,3
Xin Meng1,3 Lingchao Chen1
Lingchao Chen1 Jianchao Wei1
Jianchao Wei1 Wu Tong1
Wu Tong1 Ning Kong1
Ning Kong1 Lingxue Yu1
Lingxue Yu1 Hai Yu1
Hai Yu1 Tongling Shan1
Tongling Shan1 Guangzhi Tong1
Guangzhi Tong1 Guihua Wag2*
Guihua Wag2* Hao Zheng1,4*
Hao Zheng1,4*- 1Instituto de Investigación Veterinaria de Shanghái de la Academia China de Ciencias Agrícolas, Shanghái, China
- número arábigoFacultad de Medicina Veterinaria de la Universidad Agrícola de Shandong, Tai’an, China
- 3Facultad de Vida Silvestre y Área Protegida, Universidad Forestal del Noreste, Harbin, China
- 4Centro de Co-Innovación de Jiangsu para la Prevención y el Control de Enfermedades Infecciosas Animales Importantes y Zoonosis, Universidad de Yangzhou, Yangzhou, China
La peste porcina africana es una enfermedad contagiosa altamente letal de los cerdos para la que no existe vacuna. Su agente causal, el virus de la peste porcina africana (PPA), es un virus de ADN con envoltura muy complejo que codifica más de 150 marcos de lectura abiertos. La antigenicidad del virus de la PPA aún no está clara en la actualidad. En este estudio, Escherichia coli expresó 35 proteínas del virus de la PPA, y se desarrolló ELISA para la detección de anticuerpos contra estas proteínas. p30, p54 y p22 se presentaron como los antígenos principales del virus de la peste porcina africana, reaccionando positivamente con los cinco sueros clínicos de cerdos positivos para el virus de la peste porcina africana y 10 sueros de cerdos infectados experimentalmente por el virus de la peste porcina. Cinco proteínas (pB475L, pC129R, pE199L, pE184L y pK145R) reaccionaron bien con sueros positivos para el virus de la peste porcina africana. El p30 indujo una respuesta inmunitaria rápida y fuerte de anticuerpos durante la infección por PPA. Estos resultados promoverán el desarrollo de vacunas de subunidades y métodos de diagnóstico sérico contra el virus de la peste porcina africana.
1. Introducción
La peste porcina africana, causada por el virus de la peste porcina africana (PPA), es una enfermedad contagiosa altamente letal en cerdos domésticos y jabalíes. Afecta a cerdos de todas las razas y edades, dando lugar a una variedad de signos clínicos y lesiones, desde fiebre hemorrágica aguda con alta mortalidad hasta infección crónica con úlceras cutáneas e hinchazón articular, y causa graves consecuencias económicas en la industria porcina (1). Por lo tanto, está incluida en la lista de enfermedades obligadas a declarar a la Organización Mundial de Sanidad Animal (OIE).
La peste porcina africana (PPA) se describió por primera vez en 1921 en Kenia y pronto se convirtió en enzoótica en todo el África subsahariana. Cruzó continentes por primera vez hasta Portugal en 1957 y desde allí a otros países europeos y latinoamericanos, desde donde la enfermedad fue erradicada con éxito a mediados de la década de 1990, a excepción de Cerdeña (2). Una nueva era de transmisión comenzó cuando la PPA se introdujo en Georgia desde el sudeste africano en 2007. Posteriormente, se extendió progresivamente por el continente euroasiático (2). En la actualidad, la mayoría de los países de Europa Central y Oriental y Oriental y Sudeste Asiático han notificado casos de PPA, e incluso ha aparecido en varios países de Europa Occidental (3, 4). La PPA se notificó por primera vez en China en agosto del 2018 (5). En 6 meses, la PPA aguda con casi el 100% de mortalidad se había extendido por la mayoría de las provincias de China y había causado devastación en miles de granjas porcinas chinas (4, 6). Después de 2 años de grandes esfuerzos, la PPA aguda se ha controlado en gran medida. Sin embargo, a principios de 2021 se encontró un virus de la PPA con baja virulencia en algunas granjas porcinas chinas (7). Luego, los aislados poco virulentos se volvieron predominantes en la epidemia de PPA en China. En la actualidad, el virus de la peste porcina africana está clasificado como un patógeno infeccioso de clase I, y la peste porcina africana se ha convertido en la enfermedad porcina número uno en China (6).
El virus de la peste porcina africana (PPA) es el único miembro caracterizado de Asfarviridae y tiene un genoma lineal de ADN bicatenario de 170-194 kbp que codifica entre 150 y 170 marcos de lectura abiertos (ORF) predichos (3). Las diferencias en la longitud del genoma viral y el número de genes se originan predominantemente en la pérdida o ganancia de genes en familias multigénicas en el extremo del genoma. Un total de 94 proteínas virales se expresaron en líneas celulares infectadas con PPA (8). Sin embargo, los perfiles de expresión de las proteínas del virus de la PPA en diferentes líneas celulares variaron notablemente. Se han identificado un total de 68 proteínas virales en viriones maduros (9). Muchas proteínas virales, especialmente las que contienen dominios transmembrana, tienen una función desconocida. La caracterización de los antígenos virales es importante para el desarrollo de vacunas contra la PPA y métodos de serodiagnóstico contra el virus de la PPA. Varias proteínas virales, como p30, p54, p72 y CD2v, son los principales antígenos del virus de la PPA, y las vacunas de subunidades elaboradas con estas proteínas proporcionan una protección parcial contra el desafío virulento del virus de la PPA (10). Los cerdos inmunizados con vacunas de ADN mixto o virus vaccinia recombinante que expresan estas proteínas están parcialmente protegidos contra el virulento virus de la PPA (11, 12). La inmunización con una biblioteca de expresión de ADN que contenía >4.000 plásmidos clonados con fragmentos del genoma de la PPA mejoró claramente la protección contra el virus de la PPA (13). Estos resultados sugirieron que muchos antígenos protectores estaban inexplorados.
En este estudio, se prepararon 35 proteínas estructurales y altamente expresadas del virus de la PPA mediante un sistema de expresión procariota. Para evaluar la antigenicidad de estas proteínas en la infección por PPA, se detectaron los niveles de anticuerpos específicos contra estas proteínas en sueros porcinos positivos para PPA mediante ELISA.
2. Materiales y métodos
2.1. Sueros
Un total de 26 sueros inactivados de cerdos infectados experimentalmente con el candidato a vacuna contra el virus de la PPA CD2v y con deleción genética del Reino Unido (ASFV-SY18-ΔCD2v/UK) fueron amablemente proporcionados por el Dr. Sun en el Instituto de Investigación Veterinaria de Harbin, Harbin, China. Entre estos, 10 sueros de cerdos infectados con PPA-SY18-ΔCD2v/UK a los 49 días después de la inoculación (dpi) se sirvieron como sueros positivos para PPA (14). Se recogieron un total de 16 sueros de cuatro cerdos infectados con ASFV-SY18-ΔCD2v/UK a 6, 10, 15 y 21 dpi. En total, se obtuvieron cinco sueros clínicos inactivados de cerdos de granja que se habían recuperado de la infección por el virus de la peste porcina africana y que también sirvieron como sueros positivos para el virus de la peste porcina africana. En 2017 se recogieron un total de cinco sueros como testigos negativos de cerdos sanos. Los sueros positivos o negativos para el virus de la PPA se analizaron con el kit de detección de anticuerpos ID Vet PPAV p30 y el kit ELISA Ingenasa ASFV p72.
2.2. Construcción de plásmidos de expresión de genes del virus de la peste porcina africana
Se utilizó como secuencia de referencia el genoma del genotipo II cepa de PPA Pig/HLJ/2018 (MK333180). Un total de 38 genes de PPA, incluyendo 32 proteínas estructurales principales y siete proteínas altamente expresadas en las células, fueron analizados con Protean en el LaserGene (DNAStar, Madison, Wisconsin, EE.UU.) y el software en línea de ProtScale (URL), SignalP y TMHMM. El péptido señal y las secuencias transmembrana se eliminaron de las proteínas transmembrana. Cuando una proteína tenía >500 aminoácidos de longitud, se utilizó el fragmento antigénico principal con 200-300 aminoácidos para su expresión. Después de la optimización con el codón de Escherichia coli, estas secuencias fueron sintetizadas por General Biol (Anhui, China) y clonadas en el vector de expresión procariota pCold I con las enzimas de restricción NdeI/XbaI, respectivamente. Todos los constructos se verificaron mediante secuenciación de ADN y se transformaron en la cepa competente de E. coli BL21 (TaKaRa Biotechnology (Dalian), Dalian, China).
2.3. Purificación de proteínas
Las células BL21 transformadas se cultivaron durante la noche en medio líquido Luria-Bertani (LB) con ampicilina (100 μg/ml). El cultivo se diluyó 1:100 en medio LB fresco y se cultivó durante 2,5 h. La expresión de proteínas recombinantes se indujo con 1 mM de isopropilo β-d-1-tiogalactopiranósido (IPTG) durante 16 h a 16°C, y las células se cosecharon por centrifugación a 5.000 rpm en un rotor Eppendorf F-34-6-38 durante 10 min. Las proteínas solubles se purificaron utilizando WorkBeads 40 Ni-NTA (Bio-Works, Upasala, Suecia). Las proteínas insolubles se purificaron después de la solubilización con urea. Los gránulos celulares se resuspendieron en 10 ml 50 mM de Tris/HCl y se añadió lisozima (1,25 mg). Las células se lisaron mediante cinco ciclos de sonicación y luego se centrifugaron. El pellet se lavó tres veces en 5 ml 50 mM Tris/HCl con 1% Triton X-100. A continuación, se añadieron 8 M de urea (2,5 ml) y la mezcla se sonicó hasta que el pellet se disolvió. La suspensión se centrifugaba a 6.000 rpm en un rotor Eppendorf F-34-6-38 durante 10 min. La proteína desnaturalizada se purificó con WorkBeads 40 Ni-NTA y luego se repliegó mediante diálisis en PBS.
2.4. ELISA
Las placas ELISA (Corning Inc., Corning, Nueva York, EE. UU.) se recubrieron con proteínas recombinantes del virus de la PPA (100 μl por pocillo) diluidas a las concentraciones adecuadas (1 μg/ml) en tampón de recubrimiento (50 mM de carbonato de sodio/tampón bicarbonato, pH 9,6) y se incubaron durante la noche a 4 °C. Los pocillos se lavaron tres veces con PBS más 0,05% Tween 20 y se bloquearon con PBS más leche al 5% (200 μl por pocillo) a 37°C durante 1 h. Después del bloqueo, las placas se lavaron cinco veces como se mencionó anteriormente y se incubaron durante 1 h a 37 °C con sueros de cerdo diluidos 1:100 en PBS más leche al 5% (100 μl por pocillo). Las placas se lavaron de nuevo cinco veces y se incubaron con anticuerpo IgG anti-cerdo conjugado con peroxidasa de rábano picante (Sigma-Aldrich, Saint Louis, Missouri, EE. UU.) diluido 1:4.000 (100 μl por pocillo) durante 1 h a 37 °C. Finalmente, las placas se lavaron de nuevo y se revelaron con TMB en la oscuridad a temperatura ambiente durante 15 min. Después de detener la reacción con 2M H2SO4 (50 μl por pocillo), el A450 se leyó en un lector de microplacas Bio-Rad (Bio-Rad, Hercules, California, EE. UU.).
2.5. Análisis estadístico
Los datos se analizaron con el software GraphPad Prism 6.0. Todos los datos se presentan como media ± desviación estándar (DE). Un valor de p de <0,05 se consideró estadísticamente significativo.
3. Resultados
3.1. Expresión y purificación de 35 proteínas del virus de la PPA
El genoma del virus de la peste porcina africana contiene más de 150 ORF, y el virión consta de casi 70 proteínas. Para explorar la antigenicidad del virus de la peste porcina africana en cerdos infectados, E. coli expresó 38 genes virales (Tabla 1). Entre estos genes, O61R (gen p12), D117L (gen p17) y K78R (gen p10) se fusionaron con secuencias enlazadoras, e I73R se fusionó con B169L. Las secuencias de aminoácidos de las proteínas expresadas por el virus de la PPA se presentan en el material suplementario S1. Después de la inducción con IPTG, cuatro proteínas (p54, pA104R, p11. 5 y pI73R-pB169L) se expresaron en forma soluble, y las otras 31 proteínas (p30, pH171R, pCP312R, pE199L, pC257L, pE120R, p72, p22, pI177L, pK196R, pE146L, pF317L, pC717R, pB475L, pB602L, pEP152R, pE248R, pE184L, pH240R, pM1249L, pB125R, pC129R, pK421R, pEP153R, pK205R, pK145R, p150, CD2v, p15, p34 y p12-p17-p10) se expresaron en forma de cuerpos de inclusión. Como se muestra en el material suplementario S2, 35 proteínas se purificaron con Ni-NTA y se utilizaron para recubrir placas de ELISA.
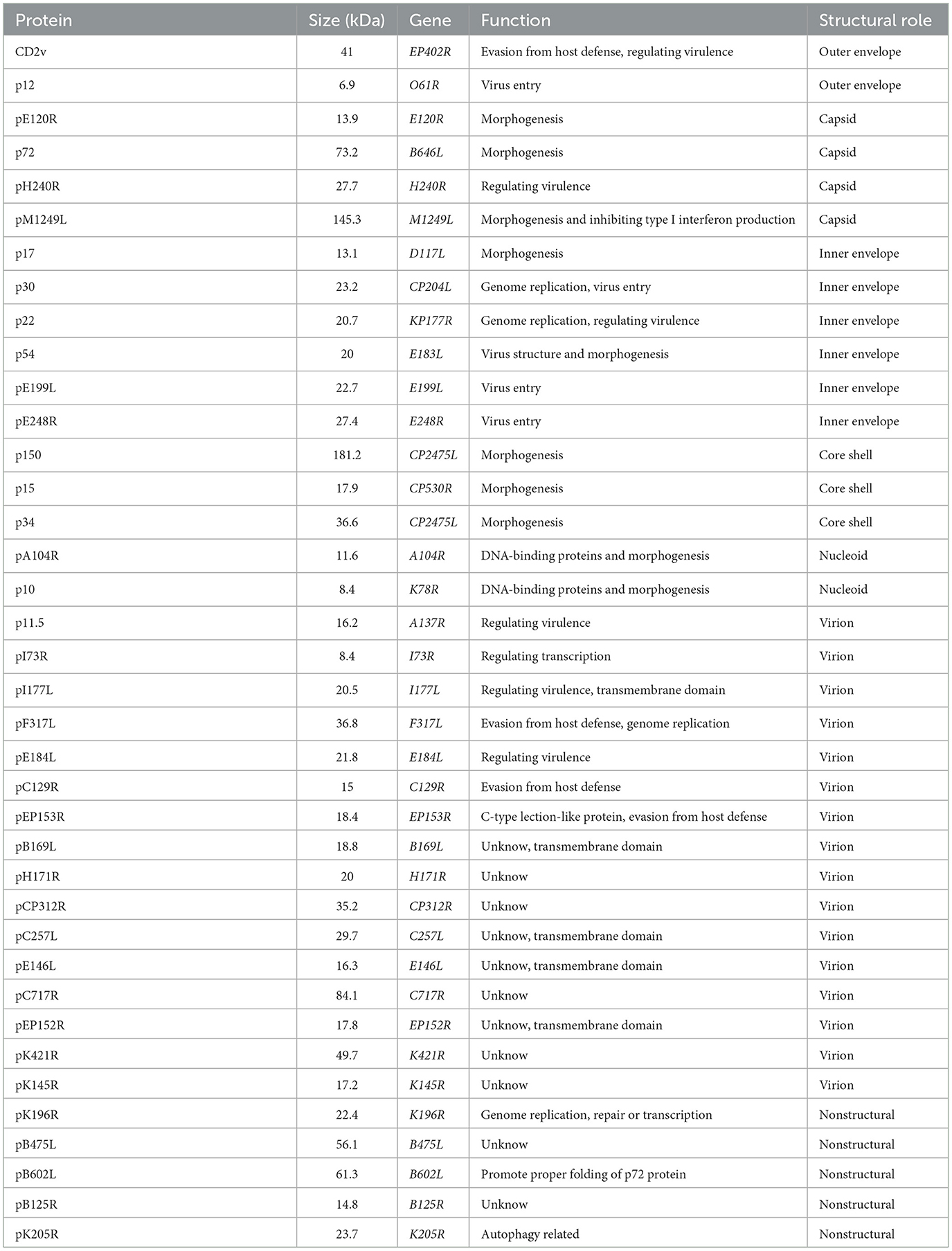
3.2. Antígenos dominantes del virus de la peste porcina africana
Se detectaron un total de 15 sueros porcinos positivos para anticuerpos anti-PPA (10 de cerdos infectados experimentalmente con PPA atenuada y cinco de muestras clínicas de campo) y cinco sueros porcinos libres de PPA con placas ELISA recubiertas con proteínas PPA. Como se muestra en la Figura 1, 24 proteínas reaccionaron con los sueros del virus de la PPA en diversos grados. p30, p54 y p22 mostraron una reacción positiva con todos los sueros del virus de la PPA (el valor de muestra/negativo ≥3), lo que sugiere que eran los antígenos dominantes del virus de la PPA. pA104R, pB475L, pE120R, pC129R, pE199L, pCP312R, pE184L, pB602L, pK205R, pH171R y la proteína de fusión p12-p17-p10 mostraron una reacción positiva con los cinco sueros clínicos del virus de la PPA y solo con algunos de los sueros infectados experimentalmente con el virus de la PPA. p34, p11.5, pB125R, pK145R, CD2v, pK421R, p15, pI177L, p72 y la proteína de fusión pI73R-pB169L mostraron una reacción positiva con algunos de los sueros infectados clínica y experimentalmente por el virus de la PPA. Las otras 11 proteínas mostraron una reacción negativa con todos los sueros del virus de la PPA (material suplementario S3). Estos resultados indicaron que algunas proteínas del virus de la PPA, como p30, p54 y p22, tienen una excelente antigenicidad y estimulan fuertes respuestas inmunitarias. Sin embargo, algunas proteínas virales no indujeron ninguna respuesta inmunitaria de anticuerpos en la infección por PPA.
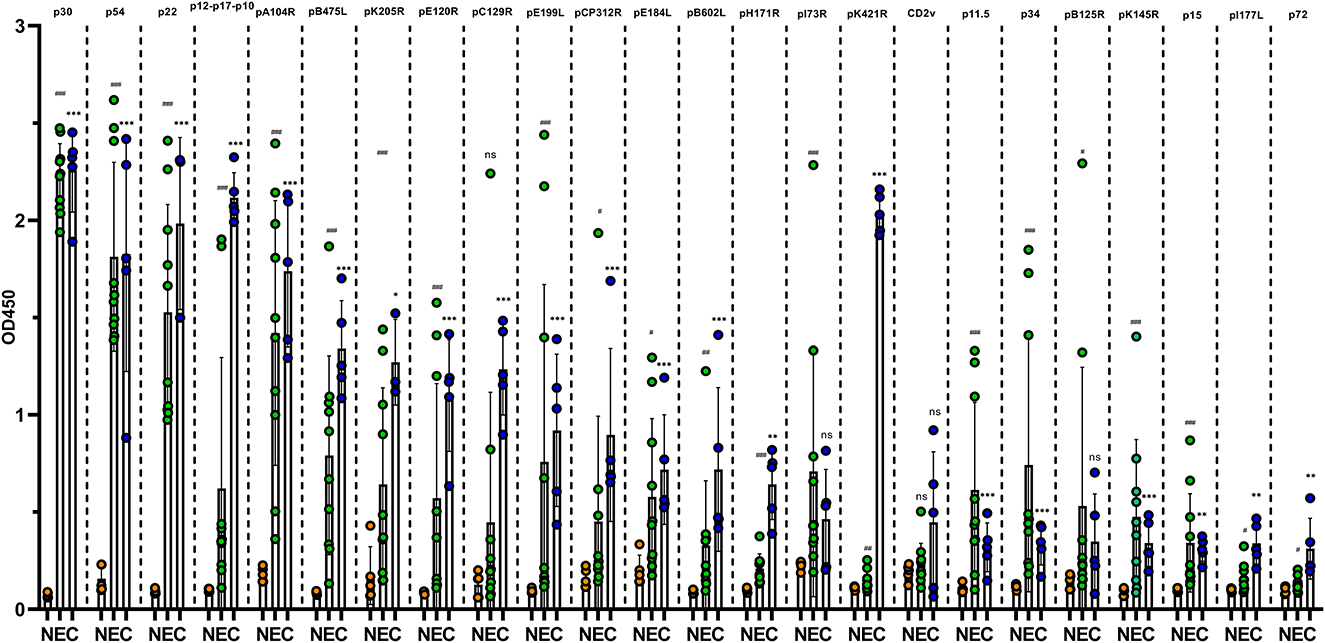
3.3. El virus de la peste porcina africana indujo una respuesta rápida y fuerte del anticuerpo p30 en cerdos infectados
Algunas proteínas del virus de la peste porcina africana fueron capaces de inducir inmunidad contra los anticuerpos del huésped. Para explorar más a fondo las propiedades de la generación de anticuerpos durante la infección por PPA, se recolectaron sueros de cerdo a los 6, 10, 15 y 21 días después de la infección atenuada por PPA, y se detectaron niveles de anticuerpos contra 15 proteínas de alta antigenicidad de PPA mediante ELISA. Como se muestra en la Figura 2, tres de los cuatro cerdos mostraron una conversión positiva de anticuerpos p30 (S/N ≥3) a 10 dpi. Los cuatro sueros de cerdo fueron positivos para anticuerpos p30, y la relación S/N para los anticuerpos p30 fue de >10 a los 15 dpi. Un total de tres cerdos dieron positivo para p22, p34 y p54 a los 15 dpi y para pB475L y pK145R a los 21 dpi, pero el nivel de anticuerpos contra estas proteínas siguió siendo negativo en el resto de un cerdo. En total, uno o dos cerdos desarrollaron anticuerpos contra pA104R, pE184L, pC129R, pE199L, pE120R y pCP312R y la proteína de fusión pI73R-pB169L. Estos resultados mostraron que la infección atenuada por el virus de la peste porcina africana indujo una respuesta rápida y fuerte del anticuerpo p30.
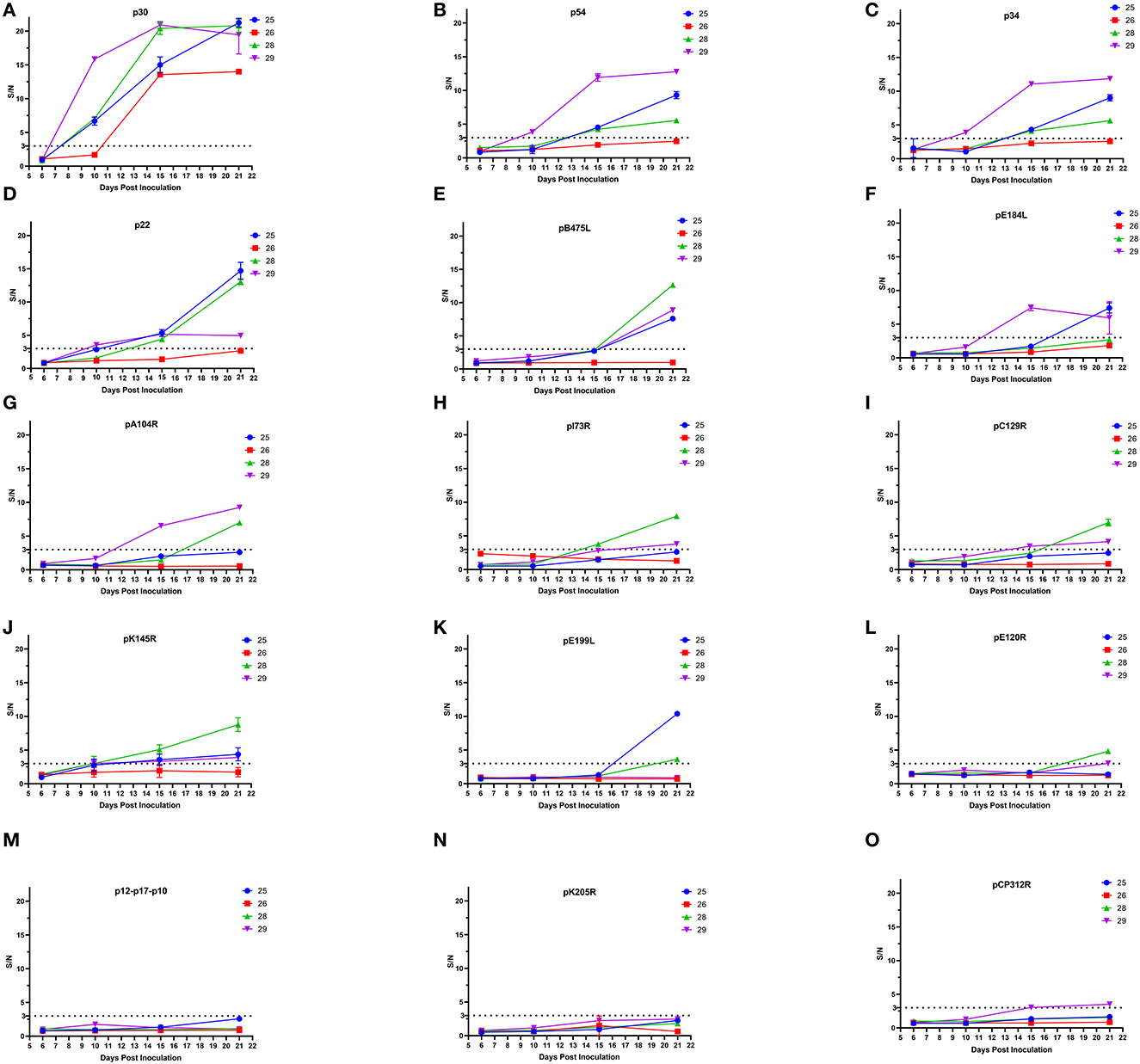
Figura 2. Desarrollo de anticuerpos específicos contra el virus de la peste porcina africana en cerdos infectados con PPA de baja virulencia. Métodos ELISA basados en 15 proteínas recombinantes del virus de la PPA, entre las que se incluyen p30 (A), p54 (B), p34 (C), p22 (D), pB475L (E), pE184L (F), pA104R (G), pI73R-pB169L (H), pC129R (I), pK145R (J), pE199L (K), pE120R (L), p12-p17-p10 (M), pK205R (N) y pCP312R (O) se utilizaron para analizar los sueros de cuatro cerdos (los números 25, 26, 28 y 29) a 6, 10, 15 y 21 dpi con PPA de baja virulencia. Un total de cinco sueros porcinos negativos para el virus de la peste porcina africana sirvieron como controles negativos. Si S/N≥3 (valor de corte), la muestra fue positiva para anticuerpos contra la proteína del virus de la PPA.
4. Discusión
El virus de la peste porcina africana (PPA) es un virus de ADN con envoltura muy complejo y codifica >150 ORF (3). En la actualidad, se ha confirmado que ~100 proteínas son expresadas por el virus de la PPA, y 63 proteínas forman el virión (8, 9). Sin embargo, las funciones de muchas proteínas estructurales no están caracterizadas. Aclarar la antigenicidad de las proteínas del virus de la PPA es importante para desarrollar métodos de diagnóstico serológico y vacunas contra el virus de la PPA. En este estudio, E. coli expresó 33 genes de proteínas estructurales con alta abundancia en el virión y cinco genes de proteínas no estructurales con altos niveles de expresión en las células huésped para detectar los antígenos dominantes del virus de la PPA (8, 9). Entre las 33 proteínas de estructura, muchas proteínas son componentes importantes del virión y desempeñan un papel importante en la entrada y morfogénesis del virus, y las otras no están caracterizadas (Tabla 1). Se establecieron ELISA indirectos basados en estas proteínas para detectar los niveles de anticuerpos en los sueros de cerdos infectados por PPA. Se confirmó que la infección por el virus de la peste porcina africana indujo fuertes respuestas de anticuerpos contra p30, p54 y p22. pA104R, pB475L, p34, pE120R, pC129R, pE199L, pE184L, pCP312R, pK145R y pB602L también indujeron anticuerpos distintos.
En este estudio, las proteínas del virus de la PPA se prepararon utilizando un sistema de expresión de E. coli. Para mejorar los niveles de expresión de las proteínas del virus de la PPA en E. coli, se optimizaron las secuencias génicas. Se eliminaron los péptidos señal y los dominios transmembrana de las proteínas. En el caso de varias proteínas grandes del virus de la PPA, como pM1249L, pB602L, pCP717R y p72, se expresaron las regiones dominantes del antígeno predichas. El truncamiento de proteínas puede reducir la detección de anticuerpos especiales contra proteínas naturales. Reis et al. mostraron fuertes respuestas de anticuerpos contra pB602L (15). En nuestro estudio, la mayoría de los sueros de cerdos infectados con PPA atenuado mostraron una reacción débil con la proteína pB602L truncada. El genotipo del virus de la peste porcina africana puede afectar a la generación de anticuerpos especiales. Los sueros de las muestras clínicas de nuestro estudio mostraron una reacción más fuerte con la proteína pB602L truncada que los sueros de los cerdos infectados con PPA atenuada. La proteína p72 es la principal proteína de la cápside y una de las proteínas más inmunogénicas del virus de la PPA, que es un objetivo importante para el desarrollo de pruebas y vacunas (16-19). En nuestro estudio, sin embargo, la p72 expresada mostró una pobre reactividad con los sueros positivos para el virus de la PPA, lo que puede deberse a que la proteína p72 no formó la estructura correcta. Se ha demostrado que el p72 correctamente plegado necesitó la ayuda del pB602L (20). Por lo tanto, la proteína p72 que se expresó sola en E. coli puede no ser un antígeno diagnóstico adecuado.
Entre las 35 proteínas del virus de la peste porcina africana expresadas en este estudio, p30, p54 y p22 fueron los antígenos dominantes durante la infección por el virus de la peste porcina, lo que concuerda con estudios previos (15, 17). Para mejorar la sensibilidad y la especificidad de los ELISA basados en p30 y p54, la mayoría de p30 y p54 se produjeron en células de insectos utilizando un sistema de expresión de baculovirus (21). Aunque la proteína p30 se expresó en cuerpos de inclusión en E. coli en nuestro estudio, la proteína p30 replegada mostró una fuerte reacción con todos los sueros positivos para el virus de la peste porcina africana. El anticuerpo p30 se desarrolló precozmente durante la infección atenuada por el virus de la peste porcina africana y fue la diana más sensible detectada por ELISA. A los 10 dpi, los anticuerpos p30 se volvieron positivos en la mayoría de los cerdos. Incluso en un cerdo que resultó negativo para anticuerpos contra otras proteínas del virus de la PPA, los anticuerpos p30 se volvieron positivos a los 15 dpi. La prueba de inmunoperoxidasa indirecta es la mejor para el diagnóstico serológico de la PPA debido a su mayor sensibilidad. Los anticuerpos contra la PPA detectados con inmunoperoxidasa se volvieron positivos a los 8-10 dpi en la infección subaguda o crónica, lo que es consistente con el tiempo de seroconversión del anticuerpo p30 en nuestro estudio (22, 23). Por lo tanto, el anticuerpo p30 fue un excelente objetivo para el diagnóstico serológico de la PPA. La proteína p30 expresada en E. coli también fue adecuada como antígeno diagnóstico para ELISA.
En la actualidad, no existe una vacuna segura y eficaz contra el virus de la peste porcina africana (24). Varias vacunas de subunidades, incluidas las vacunas basadas en ADN y péptidos basadas en algunos antígenos principales, como p30, p54, p72, CD2v y/o pEP153R, brindan protección parcial contra el desafío virulento del virus de la PPA (25, 26). Además de p54, p30 y hemaglutinina, Lacasta et al. sugirieron que algunos determinantes protectores no identificados dentro del genoma del virus de la PPA podrían desempeñar un papel importante en la prevención de la infección por el virus de la PPA (13). La combinación de los resultados de la reacción de las proteínas del virus de la PPA con sueros positivos para el virus de la PPA y el desarrollo de anticuerpos contra el virus de la PPA en este estudio, pB475L, pC129R, pE199L, pE184L y pK145R indujeron un rápido desarrollo de anticuerpos en la infección por el virus de la PPA, además de p30, p54 y p22. pB475L es una proteína no estructural, mientras que pC129R, pE199L, pE184L y pK145R son proteínas estructurales. E199L puede mediar la entrada del virus, pero las otras cuatro proteínas no están caracterizadas (27). No está claro si estas cinco proteínas pueden inducir inmunidad protectora contra el virus de la PPA.
5. Conclusión
En este estudio, se identificaron p30, p54 y p22 como los antígenos dominantes del virus de la PPA. En particular, el desarrollo del anticuerpo p30 fue rápido y fuerte durante la infección por el virus de la PPA. Por lo tanto, fue un objetivo importante del serodiagnóstico. pB475L, pC129R, pE199L, pE184L y pK145R también indujeron el desarrollo de anticuerpos en la infección por el virus de la PPA. Estos resultados promovieron el desarrollo de vacunas de subunidades y métodos de serodiagnóstico contra el virus de la PPA.
Declaración de disponibilidad de datos
Los datos brutos que respaldan las conclusiones de este artículo serán puestos a disposición por los autores, sin reservas indebidas.
Contribuciones de los autores
ZX y YH: metodología, investigación y validación. JL, AW y XM: análisis formal y validación. LC: investigación. JW, WT, LY, NK y HY: metodología y recursos. TS: revisión y edición. GT: administración de proyectos. GW: supervisión y redacción, revisión y edición. HZ: conceptualización, metodología, redacción-borrador original, supervisión y captación de fondos. Todos los autores han leído y están de acuerdo con la versión publicada del manuscrito.
Financiación
Este estudio contó con el apoyo del Programa Nacional de Investigación y Desarrollo Clave de China (Subvención Nº 2021YFD1801300), el Plan de Acción de Innovación Científica y Tecnológica de Shanghái (Subvención Nº 20392002500) y la Fundación Municipal de Ciencias Naturales de Shanghái (Nº 20ZR1469500).
Reconocimientos
Los autores están agradecidos al Dr. Yuan Sun del Instituto de Investigación Veterinaria de Harbin, CAAS, quien amablemente proporcionó sueros de cerdos infectados experimentalmente con PPA atenuado.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1175701/full#supplementary-material
Referencias
1. Pikalo J, Zani L, Hühr J, Beer M, Blome S. Patogénesis de la peste porcina africana en cerdos domésticos y jabalíes europeos: lecciones aprendidas de ensayos animales recientes. Res. de Virus (2019) 271:197614. doi: 10.1016/j.virusres.2019.04.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Galindo I y Alonso C. Virus de la peste porcina africana: una revisión. Virus. (2017) 9:103–13. doi: 10.3390/v9050103
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Dixon LK, Sun H, Roberts H. Peste porcina africana. Antiviral Res. (2019) 165:34–41. doi: 10.1016/j.antiviral.2019.02.018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Gavier-Widen D, Stahl K, Dixon L. No hay soluciones apresuradas para la peste porcina africana. Ciencia. (2020) 367:622–4. doi: 10.1126/ciencia.aaz8590
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Zhou X, Li N, Luo Y, Liu Y, Miao F, Chen T, et al. Aparición de la peste porcina africana en China, 2018. Transbound emerg Dis. (2018) 65:1482–4. doi: 10.1111/tbed.12989
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Wu K, Liu J, Wang L, Fan S, Li Z, Li Y, et al. Estado actual del desarrollo mundial de vacunas contra la peste porcina africana bajo la prevalencia y transmisión de la PPA en China. Vacunas. (2020) 8:531–58. doi: 10.3390/vaccines8030531
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Sun E, Zhang Z, Wang Z, He X, Zhang X, Wang L, et al. Aparición y prevalencia de virus de la peste porcina africana de baja virulencia natural en cerdos domésticos en China en 2020. Sci China Life Sci. (2021) 64:752–65. doi: 10.1007/s11427-021-1904-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Keßler C, Forth JH, Keil GM, Mettenleiter TC, Blome S, Karger A. El proteoma intracelular del virus de la peste porcina africana. Sci Rep. (2018) 8:14714–23. doi: 10.1038/s41598-018-32985-z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Alejo A, Matamoros T, Guerra M, Andrés G. Atlas proteómico de la partícula del virus de la peste porcina africana. J Virol. (2018) 92:e01293–18. doi: 10.1128/JVI.01293-18
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Sánchez EG, Pérez-Núñez D, Revilla Y. Desarrollo de vacunas contra el virus de la peste porcina africana. Res. de Virus (2019) 265:150–5. doi: 10.1016/j.virusres.2019.03.022
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Argilaguet JM, Pérez-Martín E, Nofrarías M, Gallardo C, Accensi F, Lacasta A, et al. La vacunación con ADN protege parcialmente contra el desafío letal del virus de la peste porcina africana en ausencia de anticuerpos. PLoS UNO. (2012) 7:E40942. doi: 10.1371/journal.pone.0040942
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Jancovich JK, Chapman D, Hansen DT, Robida MD, Loskutov A, Craciunescu F, et al. Inmunización de cerdos mediante DNA prime y refuerzo recombinante del virus Vaccinia para identificar y clasificar las proteínas inmunogénicas y protectoras del virus de la peste porcina africana. J Virol. (2018) 92:E02219–17. doi: 10.1128/JVI.02219-17
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Lacasta A, Ballester M, Monteagudo PL, Rodríguez JM, Salas ML, Accensi F, et al. La inmunización puede conferir protección contra el desafío letal con el virus de la peste porcina africana. J Virol. (2014) 88:13322–32. doi: 10.1128/JVI.01893-14
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Teklue T, Wang T, Luo Y, Hu R, Sun Y, Qiu H-J. Generación y evaluación de un mutante del virus de la peste porcina africana con deleción de los genes CD2v y UK. Vacunas. (2020) 8:763–80. doi: 10.3390/vaccines8040763
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Reis AL, Parkhouse RME, Penedos AR, Martins C, Leitão A. Análisis sistemático de las respuestas serológicas longitudinales de cerdos infectados experimentalmente con el virus de la peste porcina africana. J Gen Virol. (2007) 88:2426–34. doi: 10.1099/vir.0.82857-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Sanz A, García-Barreno B, Nogal ML, Viñuela E, Enjuanes L. Anticuerpos monoclonales específicos para proteínas del virus de la peste porcina africana. J Virol. (1985) 54:199–206. doi: 10.1128/jvi.54.1.199-206.1985
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Kollnberger SD, Gutiérrez-Castañeda B, Foster-Cuevas M, Corteyn A, Parkhouse RME. Identificación de los principales inmunodeterminantes serológicos del virus de la peste porcina africana mediante el cribado de una biblioteca de ADNc del virus con anticuerpos. J Gen Virol. (2002) 83:1331–42. doi: 10.1099/0022-1317-83-6-1331
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Liu S, Luo Y, Wang Y, Li S, Zhao Z, Bi Y, et al. Estructura crio-EM del virus de la peste porcina africana. Microbio huésped celular. (2019) 26:836–43. doi: 10.1016/j.chom.2019.11.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Cubillos C, Gómez-Sebastian S, Moreno N, Nuñez MC, Mulumba-Mfumu LK, Quembo CJ, et al. Serodiagnóstico del virus de la peste porcina africana: una revisión general centrada en el análisis de muestras de suero africano. Res. de Virus (2013) 173:159–67. doi: 10.1016/j.virusres.2012.10.021
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Liu Q, Ma B, Qian N, Zhang F, Tan X, Lei J, et al. Estructura de la proteína p72 de la cápside principal del virus de la peste porcina africana. Cell Res. (2019) 29:953–5. doi: 10.1038/s41422-019-0232-x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Oviedo JM, Rodríguez F, Gómez-Puertas P, Brun A, Gómez N, Alonso C, et al. Expresión de alto nivel de las principales proteínas antigénicas p54 y p30 del virus de la peste porcina africana en baculovirus y su posible uso como reactivos de diagnóstico. J Métodos de Virol. (1997) 64:27–35. doi: 10.1016/S0166-0934(96)02140-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Gallardo C, Fernández-Piñero J, Arias M. Diagnóstico de la peste porcina africana (PPA), una herramienta esencial en la investigación epidemiológica. Res. de Virus (2019) 271:197676. doi: 10.1016/j.virusres.2019.197676
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Gallardo C, Nieto R, Soler A, Pelayo V, Fernández-Piñero J, Markowska-Daniel I, et al. Evaluación de las técnicas de diagnóstico de la peste porcina africana como respuesta a los brotes epidémicos en los países de la Unión Europea del Este: Cómo mejorar los programas de vigilancia y control. J Clin Microbiol. (2015) 53:2555–65. doi: 10.1128/JCM.00857-15
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Urbano AC y Ferreira F. Control y prevención de la peste porcina africana: una actualización sobre el desarrollo de vacunas. Los microbios emergentes infectan. (2022) 11:2021–33. doi: 10.1080/22221751.2022.2108342
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Sang H, Miller G, Lokhandwala S, Sangewar N, Waghela SD, Bishop RP, et al. Avances hacia el desarrollo de vacunas eficaces y seguras contra el virus de la peste porcina africana. Frente Vet Sci. (2020) 7:84. doi: 10.3389/fvets.2020.00084
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Revilla Y, Pérez-Núñez D, Richt JA. Biología del virus de la peste porcina africana y enfoques vacunales. adv virus res. (2018) 100:41–74. doi: 10.1016/bs.aivir.2017.10.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Matamoros T, Alejo A, Rodríguez JM, Hernáez B, Guerra M, Fraile-Ramos A, et al. La proteína pE199L del virus de la peste porcina africana media la entrada del virus al permitir la fusión de la membrana y la penetración en el núcleo. MBio. (2020) 11:E00789–20. doi: 10.1128/mBio.00789-20
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: Virus de la peste porcina africana, antígeno dominante, proteína p30, proteína p54, proteína p22
Cita: Xu Z, Hu Y, Li J, Wang A, Meng X, Chen L, Wei J, Tong W, Kong N, Yu L, Yu H, Shan T, Tong G, Wang G y Zheng H (2023) Cribado e identificación de los antígenos dominantes del virus de la peste porcina africana. Frente. Vet. Sci. 10:1175701. doi: 10.3389/fvets.2023.1175701
Recibido: 28 de febrero de 2023; Aceptado: 27 de marzo de 2023;
Publicado: 04 mayo 2023.
Editado por:
Shixing Yang, Universidad de Jiangsu, China
Revisado por:
Fernando Costa Ferreira, Universidad de Lisboa, Portugal
Tao Wang, Academia China de Ciencias Agrícolas (CAAS), China
Yanhua Li, Universidad de Yangzhou, China
Chao Ye, Universidad del Suroeste, China
Derechos de autor © 2023 Xu, Hu, Li, Wang, Meng, Chen, Wei, Tong, Kong, Yu, Yu, Shan, Tong, Wang y Zheng. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Guihua Wang, de wguihua1126@163.com; Hao Zheng, haozheng@shvri.ac.cn
†Estos autores han contribuido igualmente a este trabajo
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

