Alteraciones del microbioma intestinal en la enfermedad renal crónica canina
 Un estudio piloto de alteraciones del microbioma intestinal en la enfermedad renal crónica canina
Un estudio piloto de alteraciones del microbioma intestinal en la enfermedad renal crónica canina
 Kyung-Ryung Kim1
Kyung-Ryung Kim1  Seon-Myung Kim2
Seon-Myung Kim2  Jung-Hyun Kim1*
Jung-Hyun Kim1*- 1Departamento de Medicina Interna Veterinaria, Facultad de Medicina Veterinaria, Universidad Konkuk, Seúl, República de Corea
- 2KR Lab Bio Incorporation, Seúl, República de Corea
Introducción: La disbiosis intestinal se ha observado en humanos y animales con enfermedad renal crónica (ERC). Sin embargo, se sabe poco sobre el microbioma intestinal en pacientes caninos con ERC. Este estudio tuvo como objetivo analizar y comparar los perfiles del microbioma intestinal de perros sanos y ERC, incluidas las diferencias en el microbioma intestinal entre cada etapa de ERC.
Métodos: El estudio se realizó en 29 perros propiedad del cliente que se sometieron a un examen físico, conteo sanguíneo completo (CSC), bioquímica sérica y análisis de orina. El perfil del microbioma intestinal de perros sanos (n = 10) y perros con ERC (n = 19) se analizó empleando secuenciación de ARNr 16S.
Resultados: Se observaron diferencias significativas en la composición del microbioma intestinal, con un aumento de las unidades taxonómicas operativas del filo Proteobacteria (p = 0,035), la familia Enterobacteriaceae (p < 0,001) y el género Enterococcus (p = 0,002) en perros con ERC, y una disminución en el género Ruminococcus (p = 0,007). Además, se observó un aumento tanto en la progresión de la ERC como en la abundancia del género Klebsiella (valor estadístico de la prueba de Jonckheere-Terpstra (JT) = 2,852, p = 0,004) y Clostridium (JT = 2,018, p = 0,044).
Discusión: Nuestro estudio demostró que en perros con ERC, la composición del microbioma intestinal varió dependiendo de la etapa de la ERC. Las alteraciones en la composición del microbioma intestinal observadas en pacientes con ERC se caracterizan por un aumento de las bacterias proteolíticas y una disminución de las bacterias sacarolíticas. Estos hallazgos sugieren que la microbiota intestinal específica podría ser el objetivo del tratamiento clínico de perros urémicos con ERC.
1. Introducción
En los perros, la enfermedad renal crónica (ERC) es la enfermedad renal más prevalente, representando hasta el 7% de los casos (1). Los perros con ERC experimentan manifestaciones similares a las humanas, ya que ambos se caracterizan por cambios funcionales o estructurales progresivos en uno o ambos riñones (2). Iniciada por numerosos factores, incluidas las propiedades familiares, congénitas y adquiridas, la ERC da como resultado una tasa de filtración glomerular renal total reducida y, en última instancia, puede conducir a crisis urémicas y muerte (3).
La relación íntima entre las enfermedades urémicas, incluida la ERC y los perfiles del microbioma intestinal, es un tema emergente de investigación en el campo de la medicina humana y veterinaria (4, 5). En medicina humana, se han descrito vínculos patogénicos entre el microbioma intestinal y la progresión de la ERC, incluido el aumento de la permeabilidad intestinal y la producción excesiva de nefrotoxina generada por la microbiota (6, 7). Además, en gatos y pacientes humanos con ERC, se ha reportado una composición anormal del microbioma intestinal (8-10). El microbioma intestinal disbiótico y la ERC se promueven mutuamente a través de diversos mecanismos fisiopatológicos (11). En medicina humana, los pacientes con ERC pueden ser identificados por una desigualdad en los tipos de microbiomas, con una mayor prevalencia de microbiomas proteolíticos sobre los sacarolíticos (12). Este desequilibrio acelera la progresión de la ERC, ya que las bacterias sacarolíticas proporcionan protección contra la inflamación asociada a la ERC y ayudan a prevenir la progresión de la enfermedad (12, 13).
Recientemente, el concepto de «diálisis entérica» en pacientes con ERC ha atraído mucha atención, especialmente, con respecto a la necesidad de considerar la salud intestinal al diseñar estrategias de tratamiento para la ERC (11). Para diseñar nuevos enfoques terapéuticos dirigidos a las comunidades microbianas intestinales para aliviar la progresión de la ERC, el análisis del microbioma intestinal de perros sanos y de aquellos con ERC es esencial. Sin embargo, las características del microbioma de los perros con ERC aún no se han estudiado bien. En este estudio, buscamos caracterizar la microbiota intestinal en perros con ERC en comparación con perros sanos y explorar posibles relaciones entre la composición de la microbiota intestinal y la ERC en diferentes etapas.
2. Materiales y métodos
2.1. Diseño del estudio y selección de animales
Este estudio prospectivo se realizó en el Hospital Docente de Medicina Veterinaria de la Universidad de Konkuk entre marzo y diciembre de 2022. Todos los protocolos de este estudio fueron revisados y aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de Konkuk (número de aprobación: KU22036-1). Cada propietario dio su consentimiento informado para que su perro participara en el estudio. Los perros sanos se inscribieron en función de su historial, examen físico, hemograma completo y bioquímica sérica. Los perros fueron excluidos del grupo sano si fueron diagnosticados con enfermedad gastrointestinal, cáncer, enfermedad hepática, ERC o infección del tracto urinario.
Se consideró que los perros tenían ERC si se cumplía cualquiera de los siguientes criterios: (1) aumento de la concentración sérica (Cr ≥1.6 mg / dL) en al menos dos ocasiones o (2) aumento de la dimetilarginina simétrica sérica (SDMA >14 μg / dL) en al menos dos ocasiones. La ERC se estadificó de acuerdo con las guías de la Sociedad Internacional de Interés Renal (IRIS). Los criterios de exclusión fueron la administración de antibióticos o probióticos dentro de las 4 semanas anteriores a la recolección de la muestra, hembras embarazadas y perros con otros hallazgos clínicos anormales, incluidos tumores malignos, infecciones del tracto urinario o enfermedades gastrointestinales. Todos los perros que participaron en el estudio fueron alimentados con dietas comerciales o recetadas sin regulación de la composición de los ingredientes.
En total, diecinueve perros con ERC (IRIS etapa 1, n = 5; etapa 2, n = 6; etapa 3, n = 4; y etapa 4, n = 4) y diez perros control sanos se incluyeron en este estudio. El microbioma intestinal se analizó para todos los perros para examinar y comparar su estructura en los grupos de ERC y sanos.
3. Recolección de muestras de microbioma
Se recogieron muestras de hisopos rectales de perros sanos y con ERC utilizando un aplicador de hisopo de cultivo estéril (REST™ NBgene-GUT, Noble Bio, Hwaseong, Corea). Los hisopos se almacenaron a 4 ° C hasta un análisis adicional.
3.1. Extracción de ADN y secuenciación del ARNr 16S
El ADN genómico total bacteriano se extrajo utilizando un kit de purificación de ADN del microbioma PureLink™ (Invitrogen, Waltham, MA, Estados Unidos) de acuerdo con las instrucciones del fabricante. Cada muestra de ADN se eluyó en 50 μL de tampón de elución S6. La concentración y pureza del ADN eluyente se evaluaron utilizando un espectrofotómetro NanoDrop ND-1000 (Thermo Fisher Scientific, Wilmington, DE, Estados Unidos) y Qubit (Thermo Fisher Scientific).
El ADN se extrajo de muestras de heces y se utilizó como plantilla para la amplificación por PCR de la región variable V3-V4 del gen bacteriano 16S rRNA utilizando cebadores con código de barras que contienen adaptadores para el sistema de secuenciación Ion S5™ (Thermo Fisher Scientific). Cada mezcla de reacción contenía Platinum PCR SuperMix High Fidelity (23 μL), 10 μM F Primer (1 μL), 10 μM R Primer (1 μL) y 2,5 ng/μL plantilla de ADN genómico (2 μL). Las condiciones de termociclado para la amplificación por PCR fueron las siguientes: 3 min a 94°C, seguidos de 25 ciclos de 30 s a 94°C, 30 s a 50°C y 30 s a 72°C, una extensión final de 5 min a 72°C y mantener a 4°C. Los productos de PCR se purificaron utilizando el reactivo AMPure™ XP de acuerdo con las instrucciones del fabricante. Los amplicones se cuantificaron utilizando un kit de ensayo Qubit dsDNA HS (Invitrogen, CA, Estados Unidos, Q32854). Después de la cuantificación, cada amplicón se diluyó a una concentración de 100 pM. Se recolectaron cantidades iguales de amplicones diluidos en tubos de 1,5 ml para hacer un volumen final de 50 μL.
3.2. Análisis bioinformático de las secuencias
Los datos de secuenciación se analizaron utilizando Quantitative Insights into Microbial Ecology (QIIME) en el software Thermo Fisher Ion Reporter v5.18.4.0. Las opciones predeterminadas incluían un filtro de longitud de lectura ≥150, una cobertura de alineación mínima ≤90,0, un límite de género ≤97,0, un límite de especies ≤99,0 y un porcentaje de notificación de ID de barra diagonal ≤ 0,2. El filtro de abundancia de lectura se estableció en ≤5, que no es el valor predeterminado. Las secuencias se agruparon a nivel de unidad taxonómica operacional (OTU) utilizando Greengenes v13.5, la biblioteca de referencia microseq(R) 16S curada v2013.1 y QIIME en las dos bases de datos.
Para evaluar la diversidad de especies bacterianas en muestras fecales, se generaron curvas de rarefacción alfa utilizando las OTU observadas y los índices de diversidad Chao1, Shannon y Simpson, y se identificaron métricas OTU. Luego comparamos las comunidades bacterianas en perros sanos y perros con etapas de ERC 1 a 4 agrupándolas. Utilizamos el Análisis de similitudes (ANOSIM) en el paquete scikit-bio (v0.5.8) en Python (v3.9.7) para evaluar la diversidad beta utilizando la matriz de distancia UniFrac y lo visualizamos utilizando un gráfico de Análisis de coordenadas principales (PCoA). UniFrac es una métrica de diversidad beta basada en la filogenia que calcula la similitud en las comunidades microbianas en función de la distancia evolutiva entre los linajes microbianos. Se utilizaron UniFrac ponderados y no ponderados, considerando este último solo la presencia de OTU en los dos grupos comparados, y el primero también considerando la abundancia de taxones dentro de los grupos. La abundancia de OTU se refiere al número de lecturas o secuencias asignadas a una OTU particular en la muestra de la comunidad microbiana.
Además, analizamos la composición de los microbiomas (ANCOM) utilizando el paquete scikit-bio en Python para identificar características diferencialmente enriquecidas en los datos del microbioma. ANCOM utiliza la estadística W para probar diferencias significativas en la abundancia relativa de características entre grupos de muestras, como perros de control sanos y perros experimentales con ERC. ANOSIM genera un valor R entre −1 y 1, con valores cercanos a 1 que indican una gran diferencia entre los grupos y valores cercanos a 0 que indican que no hay diferencias significativas en la composición entre los grupos. La estadística W se utilizó para probar las diferencias en la abundancia relativa de características entre los diferentes grupos de muestra, y se obtuvieron varias características significativamente diferentes después de corregir las comparaciones múltiples dentro del nivel de significación.
3.3. Análisis estadístico
El análisis estadístico se realizó utilizando SPSS (IBM Corporation, Armonk, NY). La normalidad de los datos se evaluó mediante la prueba de Shapiro-Wilk. Normalmente, los datos distribuidos se presentan como medias (± s.d.), mientras que los datos que no cumplían con los supuestos de distribución normal se representan como medianas e intervalos. Las variables continuas de los grupos sanos y de ERC se analizaron mediante la prueba de Mann-Whitney y la prueba t de muestra independiente para dos grupos, y la prueba de Kruskal-Wallis se utilizó para comparar más de dos grupos. Se realizó una corrección post-hoc de Bonferroni para determinar las diferencias en los taxones bacterianos entre los grupos. Se utilizó la prueba de Jonckheere-Terpstra para evaluar la importancia de las tendencias en el análisis de taxones de cada grupo ERC y control. GraphPad Prism 7 (GraphPad Software, San Diego, CA, Estados Unidos) se utilizó para producir gráficos. Para todos los análisis, la significación estadística se estableció en p < 0,05.
4. Resultados
4.1. Animales
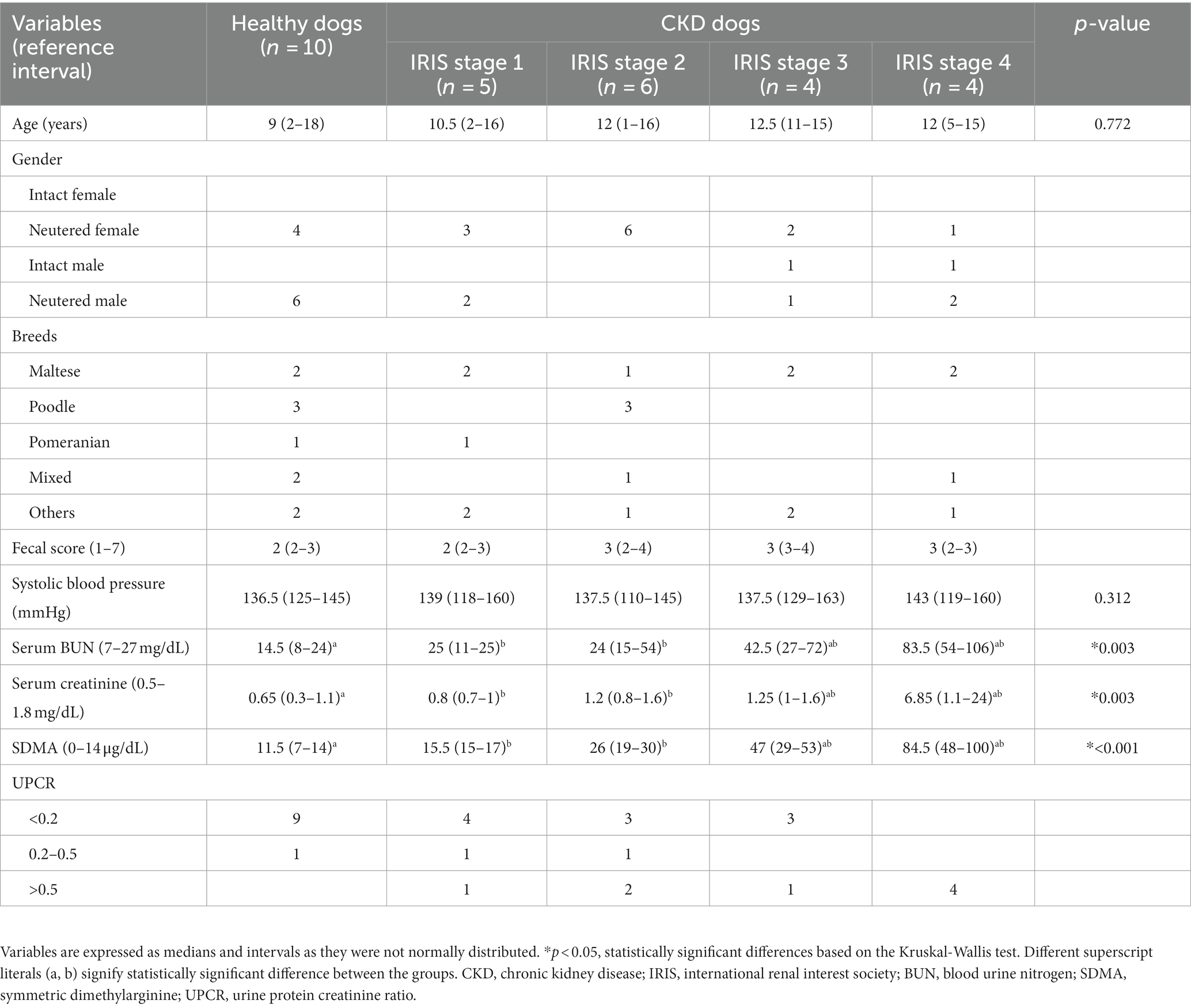
Los datos de referencia, que incluyen edad, sexo, razas, puntuaciones clínicas, presión arterial sistólica y propiedades hematológicas y bioquímicas, se presentan en la Tabla 1. Como era de esperar, los perros con ERC habían aumentado significativamente las concentraciones séricas de nitrógeno ureico en sangre (BUN; p = 0,003), creatinina (p = 0,003) y SDMA (p < 0,001). Además, los perros con ERC tenían un cociente de proteína de creatinina en orina (UPCR) significativamente mayor que los perros sanos (p = 0,009). No se encontraron diferencias significativas en edad, sexo, puntuación fecal o presión arterial sistólica entre los perros sanos y los de ERC.
Tabla 1. Demografía, química sérica y analitos de orina en perros sanos (n = 10) y ERC (n = 19).
4.2. Análisis del microbioma intestinal
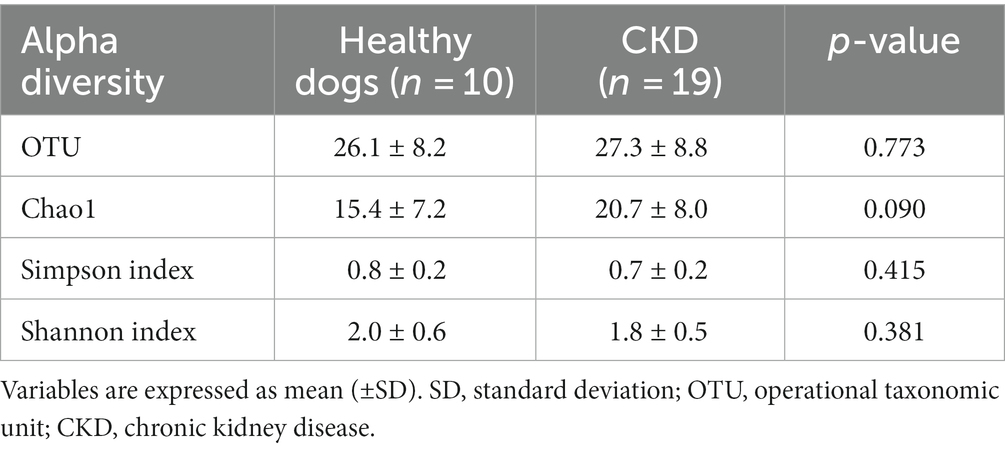
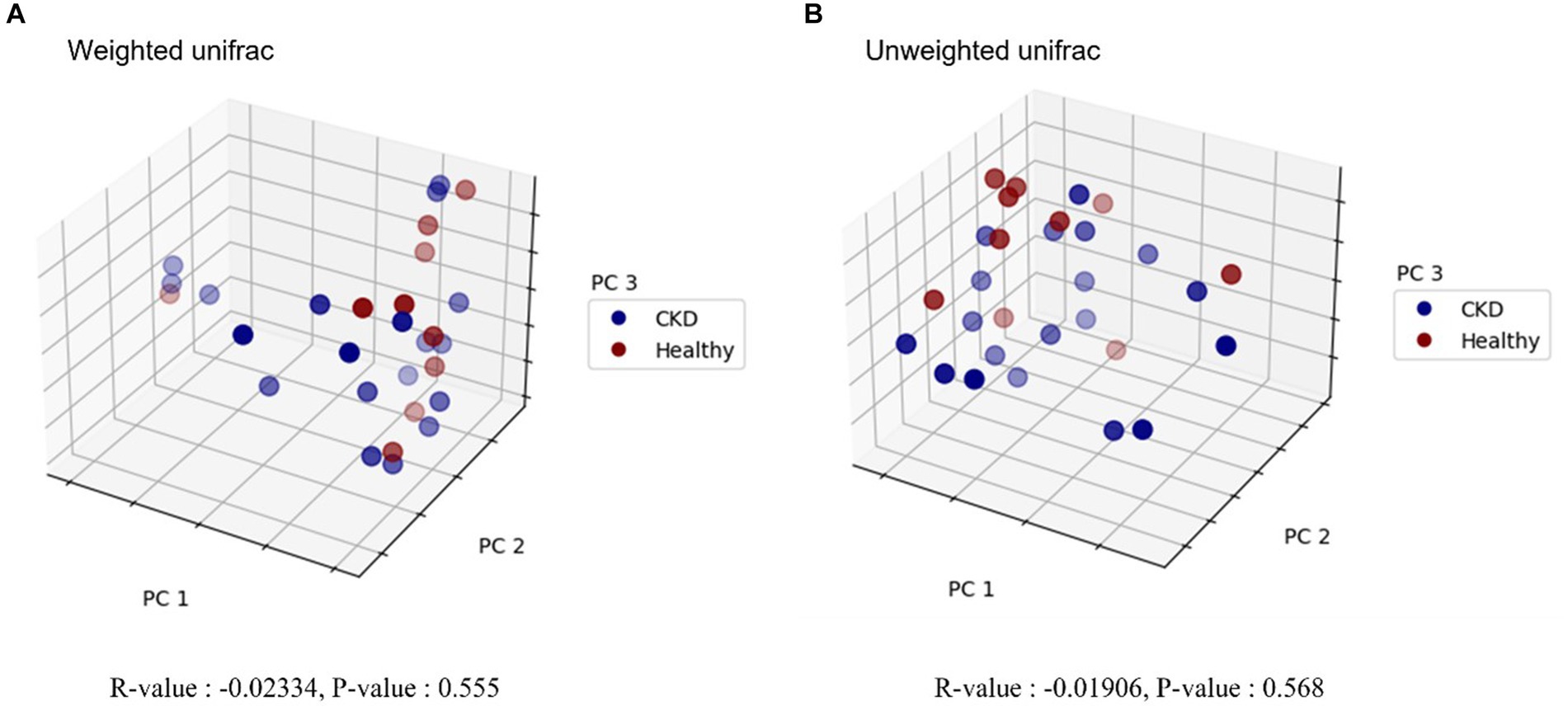
En general, se encontraron 779 OTU en todas las muestras analizadas (n = 29). El análisis de secuencia generó 12.738.717 lecturas de 29 muestras. Para la evaluación de la diversidad alfa, se compararon los índices Chao1, Shannon y Simpson entre perros sanos y perros con ERC (Tabla 2). No se observaron diferencias claras en la diversidad alfa entre los grupos sanos y los de ERC utilizando los índices de diversidad OTU, Chao1, Shannon o Simpson. La diversidad beta, que se presentó con gráficos de PCoA (Figura 1), no mostró diferencias significativas en el microbioma intestinal entre perros sanos y ERC basados en ANOSIM de distancias UniFrac ponderadas (R = −0.02) y no ponderadas (R = −0.02). Para el análisis adicional de la abundancia diferencial de OTU entre los grupos de control experimentales, se emplearon los métodos ANCOM y ALDEx2; sin embargo, ninguno de los análisis mostró una diferencia significativa en la diversidad entre los dos grupos.
Figura 1. Gráfico de análisis de coordenadas principales (PCoA) en el microbioma de perros sanos y perros con enfermedad renal crónica. Se muestra el análisis de diversidad beta utilizando distancias UniFrac ponderadas (A) y no ponderadas (B), respectivamente. Los círculos azules y rojos representan muestras de microbioma intestinal de ERC y perros sanos. Cada eje representa la coordenada principal que explica el mayor cambio de datos, y la distancia espacial de los respectivos puntos de muestra indica la similitud entre las muestras. Los valores R y p del análisis de similitudes (ANOSIM) se proporcionan debajo de cada gráfico. El valor R menor que cero indica que la variabilidad dentro del grupo es mayor que la variabilidad entre grupos. No se detectaron diferencias estadísticamente significativas entre los perros sanos y los de ERC.
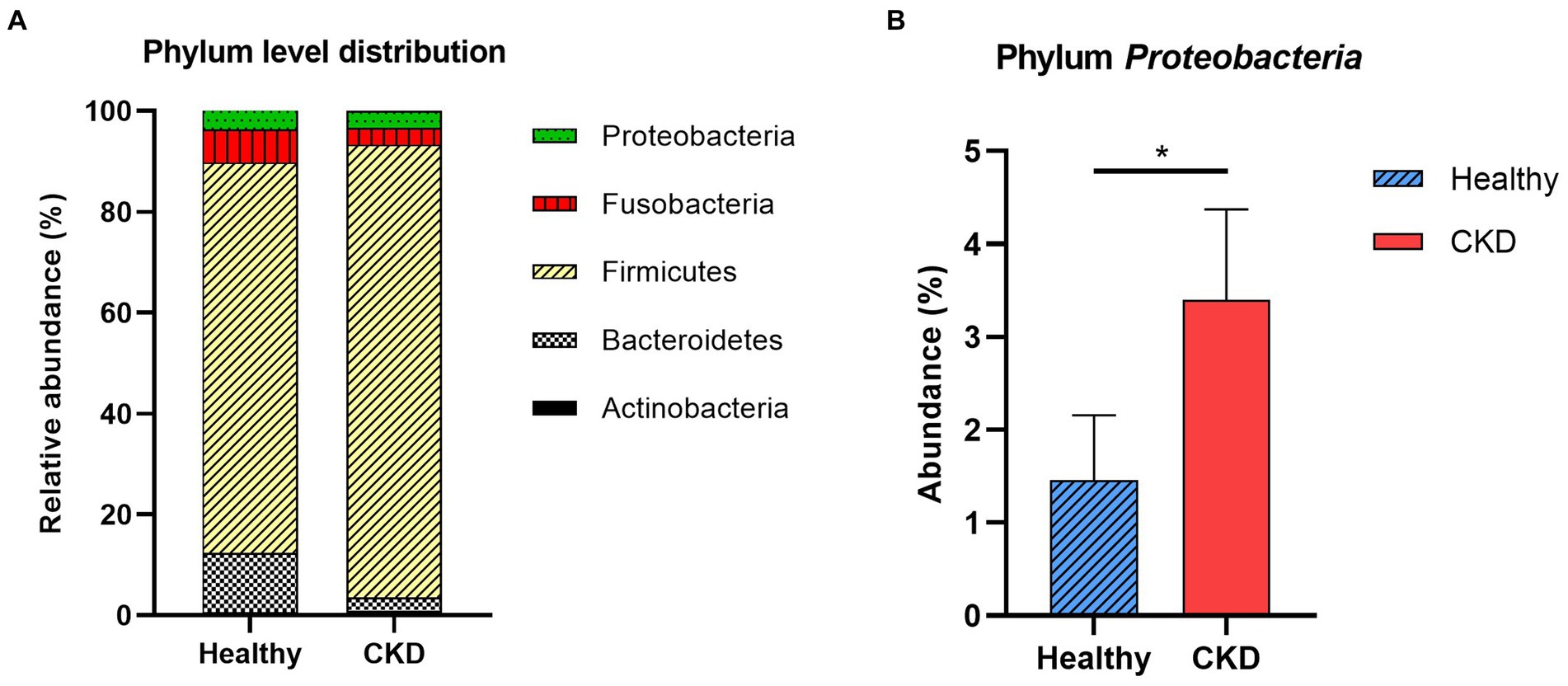
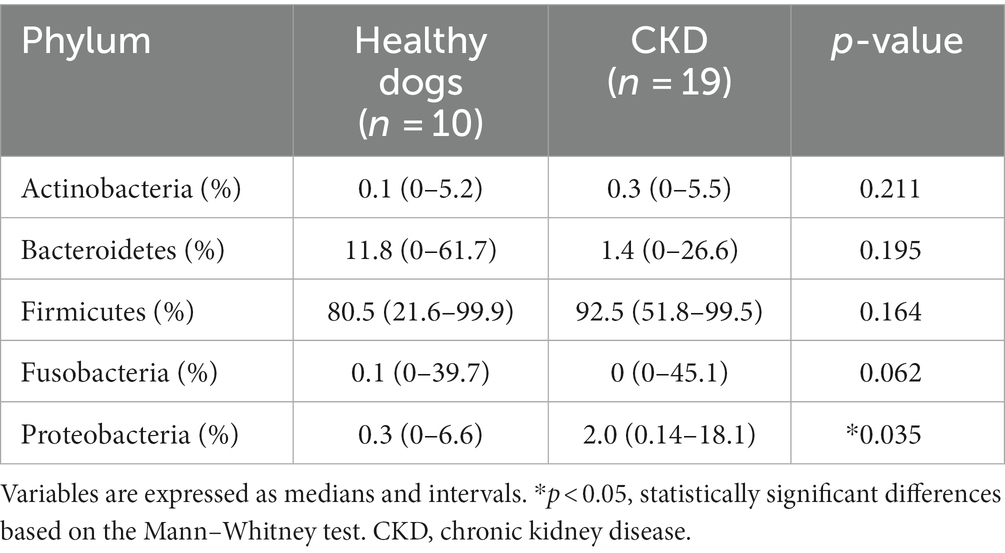
A nivel taxonómico, se observaron los siguientes cinco filos principales: Actinobacteria, Bacteroidetes, Firmicutes, Fusobacteria y Proteobacteria (Figura 2). Entre estos, el filo más predominante en perros sanos y con ERC fue Firmicutes, que representó más del 85% del microbioma total. La abundancia relativa de proteobacterias fue significativamente mayor en perros con ERC que en el grupo control sano (p = 0,035; Figura 2; Tabla 3).
Tabla 3. Análisis de taxones de perros sanos y ERC a nivel de filo.
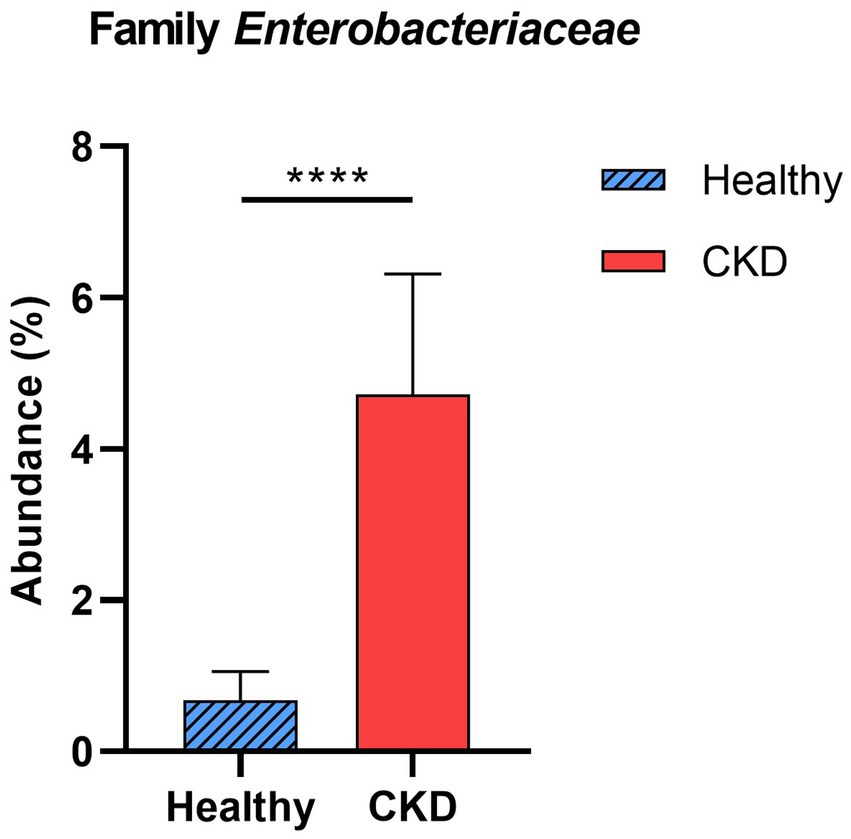
Enterobacteriaceae, una familia bacteriana, mostró un aumento significativo en los perros ERC en comparación con el de los perros control sanos (p < 0,001; Figura 3). Otras especies bacterianas productoras de toxinas urémicas, incluyendo Bacteroidaceae, Pseudomonadaceae, Clostridiaceae, fueron más abundantes en perros con ERC que en perros sanos (sin significación estadística). No se encontró correlación en los taxones entre las comunidades microbianas y la progresión de la ERC a nivel de filo y familia.
Figura 3. Comparación del análisis de taxones de perros sanos y con enfermedad renal crónica (ERC) a nivel familiar. La abundancia relativa media de la familia Enterobacteriaceae se mostró como gráfico de barras con ± error estándar de la media (SEM). Se observó un aumento estadísticamente significativo en la abundancia relativa de enterobacterias de la familia en pacientes con ERC en comparación con perros sanos. *p < 0,05; **p < 0,01; p < 0,005; p < 0,001.
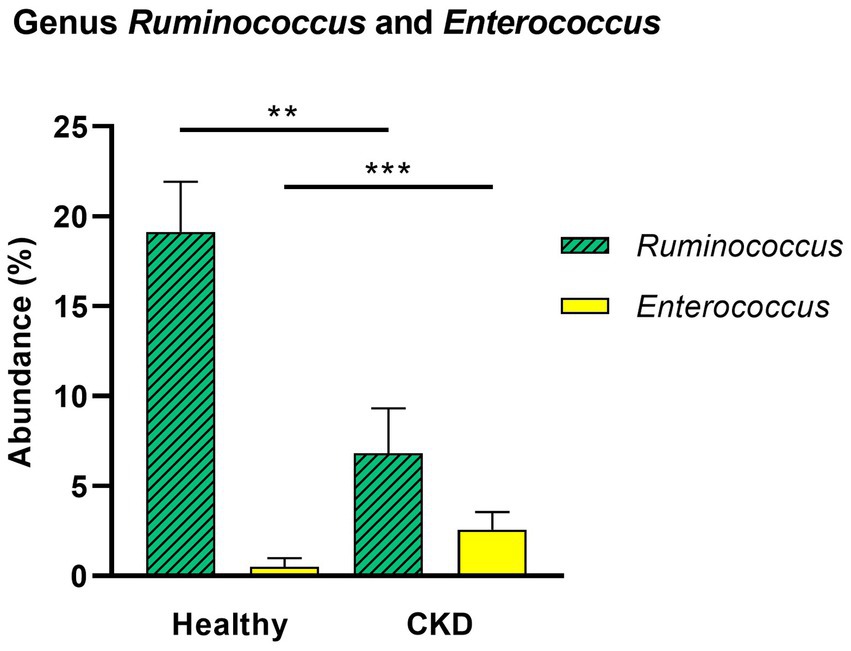
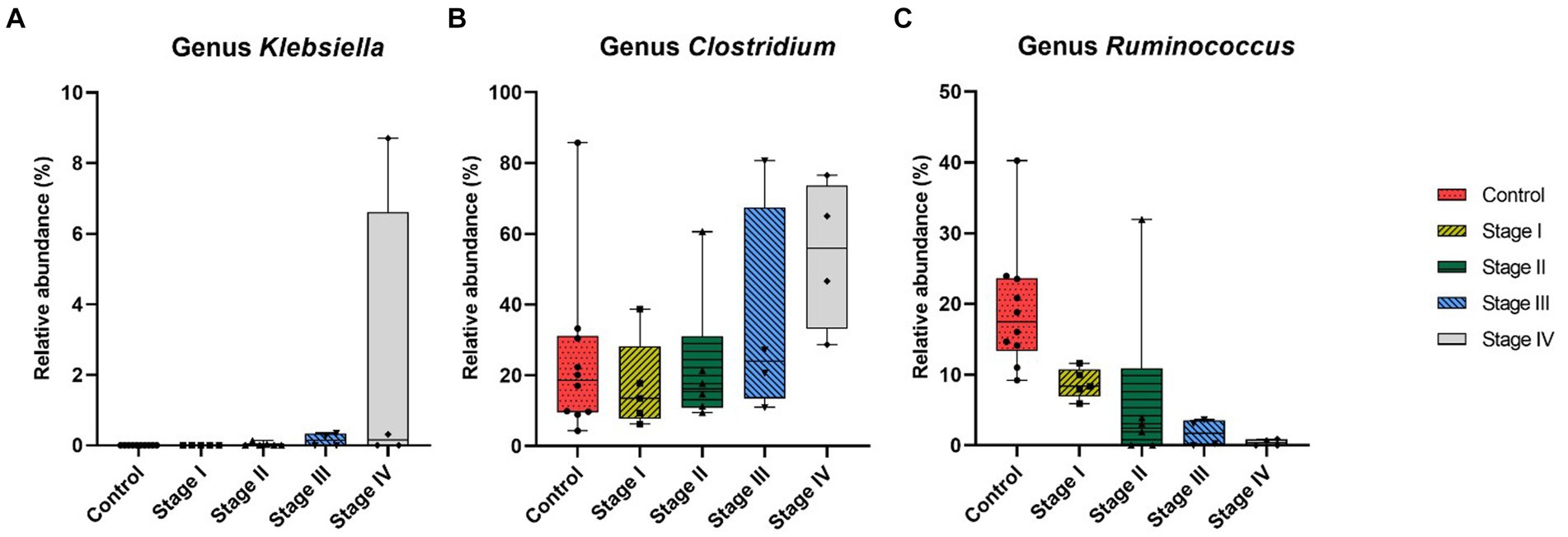
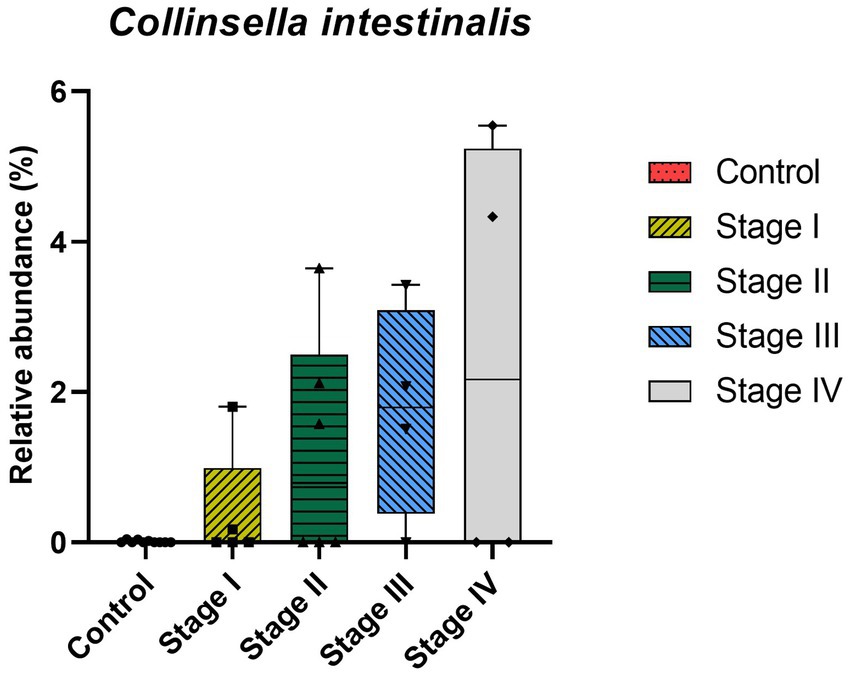
A nivel de género, los perros ERC tendían a tener una abundancia relativa significativamente mayor de Enterococcus (p = 0,002) y una menor abundancia relativa de Ruminococcus (p = 0,007; Figura 4). Además, se observaron tendencias crecientes de progresión de la ERC y abundancia relativa de los géneros Klebsiella (valor estadístico de la prueba de Jonckheere-Terpstra (JT) = 2,026, p = 0,043) y Clostridium (JT = 2,018, p = 0,044). En contraste, la abundancia relativa de Ruminococcus se relacionó inversamente con la progresión de la ERC (JT = −4,311, p < 0,001; Figura 5). No se encontraron diferencias significativas o correlación a nivel de especie entre perros con ERC y perros sanos, excepto que la abundancia relativa de Collinsella intestinalis aumentó con el progreso de la ERC (JT = 2,302, p = 0,021; Figura 6).
Figura 6. Análisis de correlación a nivel de especie de perros sanos y perros con enfermedad renal crónica (ERC) mediante el test de Jonckheere-Terpstra. Los límites inferior y superior de la casilla indican los valores mínimo y máximo, respectivamente; La línea dentro del cuadro representa los valores medianos. Cada cinco colores indica el grupo de control y 4 etapas de la ERC. Se observó una tendencia positiva estadísticamente significativa en la abundancia relativa de Collinsella intestinalis a medida que avanzaba la ERC.
5. Discusión
La evidencia reciente sugiere que la disbiosis de la microbiota intestinal desempeña un papel crítico en pacientes humanos con ERC (14). Sin embargo, la relación entre los perfiles alterados del microbioma intestinal y la ERC sigue sin estar clara en los perros. Aquí, analizamos los perfiles del microbioma intestinal de perros sanos y perros con ERC en diferentes etapas.
En este estudio, los filos bacterianos más predominantes en perros sanos y con ERC fueron Actinobacteria, Bacteroidetes, Firmicutes, Fusobacteria y Proteobacteria, que son consistentes con los hallazgos en la medicina humana y veterinaria (15, 16). Especialmente se observó un crecimiento excesivo del filo Proteobacteria en perros con ERC en comparación con el de los perros sanos, lo que está de acuerdo con estudios previos (17, 18). La proteobacteria, que es el filo bacteriano más grande, es un marcador diagnóstico potencial de disbiosis. En los seres humanos, una mayor prevalencia de proteobacterias se asocia con numerosas enfermedades crónicas e inflamatorias, incluyendo ERC, enfermedad inflamatoria intestinal, enfermedades cardiovasculares y enfermedades pulmonares crónicas (14, 19, 20). Aunque no se puede dar una explicación precisa para estos fenómenos, generalmente se sabe que los pacientes con ERC experimentan un aumento de las respuestas inflamatorias inmunes sistémicas y la susceptibilidad intestinal.
La hiperproliferación de bacterias formadoras de lipopolisacáridos (LPS), género Klebsiella, fue prominente en pacientes con ERC en este estudio. La disbiosis del microbioma intestinal en pacientes con ERC puede causar un aumento de la inflamación sistémica debido a la presencia de LPS derivado del microbioma intestinal (21). Además, en este estudio se observó un aumento gradual de Collinsella intestinalis en perros con ERC; esta especie pertenece a la familia Coriobacteriaceae y es un patobiológico (22). Algunos estudios han demostrado que la administración de Collinsella induce el aflojamiento de la integración intestinal mediante la regulación a la baja de la expresión de proteínas de unión estrecha de enterocitos y está fuertemente relacionada con el aumento de la producción de citoquinas proinflamatorias en modelos de ratón. La desintegración de la barrera intestinal es causada por los productos resultantes de la disbiosis del microbioma intestinal, lo que lleva a la translocación de productos bacterianos (por ejemplo, LPS, toxinas urémicas y citoquinas) del intestino a la circulación sistémica (9, 23). Como se discutió anteriormente, la correlación entre la ERC y la disbiosis intestinal, el deterioro de la barrera intestinal, la translocación bacteriana intestinal y las moléculas inflamatorias derivadas del intestino sugiere un mecanismo fisiopatológico en el riñón y el intestino. No se ha demostrado evidencia clara de inflamación sistémica y deterioro de la barrera intestinal causado por disbiosis en perros; sin embargo, se han observado hallazgos similares de disbiosis en pacientes con ERC y perros, lo que sugiere un mecanismo renal-intestinal subyacente cercano.
También observamos que la abundancia de las familias Enterobacteriaceae, Clostridiaceae, Pseudomonadaceae y Bacteroidaceae fue mayor en perros con ERC que en el grupo control. Aunque no se encontró significación estadística, a excepción de la familia Enterobacteriaceae, estas especies bacterianas son bien conocidas por producir toxinas urémicas (24). A nivel de género, Enterococcus fue significativamente más abundante en el microbioma intestinal de los perros con ERC en comparación con el de los perros sanos. Comparando cada estadio de la ERC y el grupo sano, algunas bacterias proteolíticas, los géneros Klebsiella y Clostridium, mostraron un aumento gradual de la abundancia a medida que avanzaba la ERC; Como se sabe que ambos géneros exhiben actividad proteolítica, se espera una relación íntima con el aumento de los niveles de toxinas urémicas. Mientras que los individuos con función renal normal pueden excretar metabolitos urémicos, se retienen en individuos con ERC, afectando negativamente sus funciones biológicas (25). La descomposición de los aminoácidos aromáticos por las especies proteolíticas debido a un desequilibrio en el microbioma intestinal ha sido reconocida como un contribuyente a la producción de toxinas urémicas unidas a proteínas, como el sulfato de indoxilo y el sulfato de p-cresol (26). En humanos, la acumulación de metabolitos de aminoácidos tóxicos acelera la progresión de la ERC a través de diversos mecanismos fisiopatológicos, incluida la exacerbación del estrés oxidativo en las células tubulares renales, la inducción de la esclerosis glomerular y la acumulación de toxinas intracelulares (27). Estos resultados sugieren que las toxinas urémicas derivadas del intestino pueden contribuir al desarrollo y progresión de la ERC en perros mediante la síntesis de moléculas de toxina urémica relacionadas con el deterioro de la ERC. Sin embargo, el mecanismo exacto subyacente a este fenómeno requiere una mayor clarificación.
Nuestro estudio también reveló una disminución significativa de la abundancia del género Ruminococcus en perros con ERC, que fue responsable de producir ácidos grasos de cadena corta (AGCC). Además, observamos una posible asociación entre la abundancia de Ruminococcus y la progresión de la ERC en perros. Ruminococcus spp. son bacterias anaerobias grampositivas que pertenecen al filo Firmicutes. Este género es una bacteria sacarolítica bien conocida que produce butirato que está inversamente asociado con las concentraciones séricas de LPS en humanos (28). El butirato, un AGCC, se produce principalmente por la fermentación bacteriana sacarolítica de la fibra dietética en el intestino (29). Este ácido graso no solo es una fuente de energía esencial para las células del tracto intestinal inferior, sino que también es un modulador inmune y regulador de la presión arterial en humanos (30). Los AGCC disminuyen la inflamación y suprimen la lesión oxidativa y la fibrosis del riñón a través de numerosos mecanismos biológicos, incluidos los receptores acoplados a proteínas G, la regulación epigenética y la acetilación de histonas (29). En medicina humana, una reducción en las especies de bacterias productoras de butirato se asocia con la progresión de la ERC (9). Aunque la función bioquímica del butirato en perros no se conoce bien, dada la disminución significativa de las especies bacterianas productoras de butirato en perros con ERC, los AGCC podrían considerarse como una diana terapéutica para frenar la progresión de la ERC en perros. Nuestros resultados muestran que la disbiosis en perros con ERC puede promover el crecimiento excesivo de especies proteolíticas al tiempo que reduce la población de especies sacarolíticas, lo que puede contribuir a la acumulación de toxinas urémicas y una disminución en la producción de AGCC. También conduce a un estado de inflamación crónica mediante la translocación de metabolitos bacterianos, citoquinas y moléculas inflamatorias, que son objetivos terapéuticos clave en la ERC.
A diferencia del estudio en humanos, nuestro estudio no reveló diferencias significativas en los índices de diversidad alfa y beta entre perros sanos y ERC. Esta discrepancia podría deberse a diferencias en las especies de comunidades microbianas. Debido a que muchos otros estudios realizados en especies distintas de los humanos (por ejemplo, ratas, perros) no encontraron diferencias significativas en la diversidad del microbioma intestinal entre pacientes sanos y con ERC (16, 31). Se requiere más investigación con respecto a las diferencias entre especies para evaluar los cambios en la diversidad del microbioma intestinal en perros con ERC.
Existen algunas limitaciones para este estudio, que comprende un tamaño de muestra pequeño de perros ERC, particularmente aquellos con enfermedad en etapa avanzada. En este estudio, la inscripción de perros con ERC en etapa avanzada cumple con los estrictos criterios de exclusión que prohíben la terapia con antibióticos o probióticos dentro de las 4 semanas fue difícil, ya que a los pacientes con ERC a menudo se les recetan probióticos en las primeras etapas de la enfermedad en los hospitales de animales locales. Otra limitación de este estudio es la dieta y el entorno descontrolados de los perros propiedad del cliente. Para minimizar las limitaciones en el estudio, restringimos a los perros involucrados en el experimento de consumir alimentos de mesa y solo les proporcionamos dietas recetadas o comerciales. Además, también se restringió el uso de antibióticos sistémicos y suplementos probióticos, que podrían tener un impacto en la composición y diversidad de la microbiota intestinal.
6. Conclusion
Our study demonstrates that the gut microbiome composition in dogs with CKD undergoes a transition from symbiosis to dysbiosis, which can be explained by increased abundance of proteolytic bacteria and decreased abundance of saccharolytic bacteria. Additionally, a few bacterial species known to be associated with systemic inflammatory response in human CKD patients were found to be overgrown in dogs in this study, indicating that a renal–gut axis like that in humans may also exist in dogs. These findings aid in enhancing comprehension of the relationship between CKD and the intestinal microbiome in dogs. Further studies are needed to evaluate the correlation between the gut microbiome composition and microbiome-derived metabolites in dogs with CKD to understand the mechanisms of gut–kidney interactions.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio / repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/, PRJNA944392.
Declaración ética
Los estudios en animales fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de Konkuk. Los estudios se realizaron de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones del autor
K-RK: conceptualización, análisis formal, investigación, recurso, preparación de borradores originales, revisión y edición de escritura, y visualización. S-MK: análisis formal, curación de datos y redacción-revisión y edición. J-HK: conceptualización, preparación de borradores originales, revisión y edición, supervisión, administración de proyectos y adquisición de fondos. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Conflicto de intereses
S-MK fue empleado por KR Lab Bio Incorporation.
Los autores restantes declaran que la investigación se realizó en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Dunaevich, A, Chen, H, Musseri, D, Kuzi, S, Mazaki-Tovi, M, Aroch, I, et al. Agudo en la enfermedad renal crónica en perros: etiología, hallazgos clínicos y clínicos-patológicos, marcadores pronósticos y supervivencia. J Vet Pasante Med. (2020) 34:2507–15. DOI: 10.1111/JVIM.15931
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Polzin, DJ. Enfermedad renal crónica en animales pequeños. Veterinario Clin North am Small Anim Pract. (2011) 41:15–30. doi: 10.1016/J.CVSM.2010.09.004
3. O’Neill, DG, Elliott, J, Church, DB, McGreevy, PD, Thomson, PC y Brodbelt, DC. Enfermedad renal crónica en perros en las prácticas veterinarias del Reino Unido: prevalencia, factores de riesgo y supervivencia. J Vet Pasante Med. (2013) 27:814–21. doi: 10.1111/JVIM.12090
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Lohia, S, Vlahou, A, y Zoidakis, J. Microbioma en la enfermedad renal crónica (ERC): una perspectiva ómica. Toxinas. (2022) 14:14. DOI: 10.3390/TOXINS14030176
5. Suchodolski, JS. Análisis del microbioma intestinal en perros y gatos. Veterinario Clin Pathol. (2022) 50:6–17. doi: 10.1111/VCP.13031
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Castillo-Rodriguez, E, Fernandez-Prado, R, Esteras, R, Perez-Gomez, MV, Gracia-Iguacel, C, Fernandez-Fernandez, B, et al. Impacto de la microbiota intestinal alterada en la progresión de la enfermedad renal crónica. Toxinas. (2018) 10:10. DOI: 10.3390/TOXINS10070300
7. Fernandez-Prado, R, Esteras, R, Perez-Gomez, MV, Gracia-Iguacel, C, Gonzalez-Parra, E, Sanz, AB, et al. Nutrientes convertidos en toxinas: modulación de la microbiota de las propiedades nutritivas en la enfermedad renal crónica. Nutrientes. (2017) 9:9. DOI: 10.3390/NU9050489
8. Hall, JA, Jackson, MI, Jewell, DE, y Ephraim, E. La enfermedad renal crónica en gatos altera la respuesta del metaboloma plasmático y el microbioma fecal a la fibra dietética. PLoS One. (2020) 15:E0235480. doi: 10.1371/journal.pone.0235480
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Jazani, NH, Savoj, J, Lustgarten, M, Lau, WL y Vaziri, ND. Impacto de la disbiosis intestinal en las vías neurohormonales en la enfermedad renal crónica. Enfermedades. (2019) 7:21. doi: 10.3390/enfermedades7010021
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Sircana, A, De Michieli, F, Parente, R, Framarin, L, Leone, N, Berrutti, M, et al. Microbiota intestinal, hipertensión y enfermedad renal crónica: avances recientes. Farmacol Res. (2019) 144:390–408. doi: 10.1016/j.phrs.2018.01.013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Cosola, C, Rocchetti, MT, Sabatino, A, Fiaccadori, E, Di Iorio, BR, and Gesualdo, L. Microbiota issue in CKD: how promising are gut-targeted approaches? J Nefrol. (2019) 32:27–37. DOI: 10.1007/S40620-018-0516-0
12. Yang, T, Richards, EM, Pepine, CJ, and Raizada, MK. The gut microbiota and the brain-gut-kidney axis in hypertension and chronic kidney disease. Nat Rev Nephrol. (2018) 14:442–56. doi: 10.1038/s41581-018-0018-2
13. Jiang, S, Xie, S, Lv, D, Zhang, Y, Deng, J, Zeng, L, et al. A reduction in the butyrate producing species Roseburia spp. and Faecalibacterium prausnitzii is associated with chronic kidney disease progression. Antonie Leeuwenhoek. (2016) 109:1389–96. doi: 10.1007/s10482-016-0737-y
14. Hu, X, Ouyang, S, Xie, Y, Gong, Z, and Du, J. Characterizing the gut microbiota in patients with chronic kidney disease. Postgrad Med. (2020) 132:495–505. doi: 10.1080/00325481.2020.1744335
15. Rinninella, E, Raoul, P, Cintoni, M, Franceschi, F, Miggiano, GAD, Gasbarrini, A, et al. What is the healthy gut microbiota composition? A changing ecosystem across age, environment, diet, and diseases. Microorganisms. (2019) 7:14. doi: 10.3390/MICROORGANISMS7010014
16. Seo, J, Matthewman, L, Xia, D, Wilshaw, J, Chang, YM, and Connolly, DJ. The gut microbiome in dogs with congestive heart failure: a pilot study. Sci Rep. (2020) 10:13777. doi: 10.1038/S41598-020-70826-0
17. Vaziri, ND, Wong, J, Pahl, M, Piceno, YM, Yuan, J, Desantis, TZ, et al. Chronic kidney disease alters intestinal microbial flora. Kidney Int. (2013) 83:308–15. doi: 10.1038/KI.2012.345
18. Wilkins, LJ, Monga, M, and Miller, AW. Defining dysbiosis for a cluster of chronic diseases. Sci Rep. (2019) 9:12918. doi: 10.1038/S41598-019-49452-Y
19. Amar, J, Lange, C, Payros, G, Garret, C, Chabo, C, Lantieri, O, et al. Blood microbiota dysbiosis is associated with the onset of cardiovascular events in a large general population: the D.E.S.I.R. study. PLoS One. (2013) 8:e54461. doi: 10.1371/JOURNAL.PONE.0054461
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Rizzatti, G, Lopetuso, LR, Gibiino, G, Binda, C, y Gasbarrini, A. Proteobacteria: un factor común en enfermedades humanas. Biomed Res Int. (2017) 2017:9351507. DOI: 10.1155/2017/9351507
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Pan, W, and Kang, Y. Gut microbiota and chronic kidney disease: Implications for novel mechanistic insights and therapeutic strategies. Int Urol Nefrol. (2018) 50:289–99. DOI: 10.1007/S11255-017-1689-5
22. Gomez-Arango, LF, Barrett, HL, Wilkinson, SA, Callaway, LK, McIntyre, HD, Morrison, M, et al. La baja ingesta de fibra dietética aumenta la abundancia de Collinsella en la microbiota intestinal de mujeres embarazadas con sobrepeso y obesas. Microbios intestinales. (2018) 9:189–201. doi: 10.1080/19490976.2017.1406584
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Yang, J, Lim, SY, Ko, YS, Lee, HY, Oh, SW, Kim, MG, et al. La ruptura de la barrera intestinal y la inmunidad desregulada de la mucosa contribuyen a la fibrosis renal en la enfermedad renal crónica. Trasplante de dial de nefol. (2019) 34:419–28. doi: 10.1093/ndt/gfy172
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Onal, EM, Afsar, B, Covic, A, Vaziri, ND, and Kanbay, M. Gut microbiota and inflammation in chronic kidney disease and their roles in the development of cardiovascular disease. Hypertens Res. (2019) 42:123–40. doi: 10.1038/S41440-018-0144-Z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Kikuchi, M, Ueno, M, Itoh, Y, Suda, W, and Hattori, M. Uremic toxin-producing gut microbiota in rats with chronic kidney disease. Nefrona. (2017) 135:51–60. doi: 10.1159/000450619
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Ren, Z, Fan, Y, Li, A, Shen, Q, Wu, J, Ren, L, et al. Alteraciones del microbioma intestinal humano en la enfermedad renal crónica. Adv Sci (Weinh). (2020) 7:2001936. doi: 10.1002/ADVS.202001936
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Lim, YJ, Sidor, NA, Tonial, NC, Che, A, and Urquhart, BL. Toxinas urémicas en la progresión de la enfermedad renal crónica y la enfermedad cardiovascular: mecanismos y dianas terapéuticas. Toxinas. (2021) 13:13. doi: 10.3390/TOXINAS13020142
28. Biruete, A, Allen, JM, Kistler, BM, Jeong, JH, Fitschen, PJ, Swanson, KS, et al. Microbiota intestinal y factores de riesgo cardiometabólicos en pacientes en hemodiálisis: un estudio piloto. Arriba Clin Nutr. (2019) 34:153–60. doi: 10.1097/TIN.0000000000000170
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Cheng, X, Zhou, T, He, Y, Xie, Y, Xu, Y, y Huang, W. El papel y el mecanismo del butirato en la prevención y el tratamiento de la enfermedad renal diabética. Microbiol frontal. (2022) 13:961536. DOI: 10.3389/fmicb.2022.961536
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Rukavina Mikusic, NL, Kouyoumdzian, NM, and Choi, MR. Gut microbiota and chronic kidney disease: evidences and mechanisms that mediate a new communication in the gastrointestinal-renal axis. Arco de Pflugers. (2020) 472:303–20. DOI: 10.1007/S00424-020-02352-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Chen, L, Chen, DQ, Liu, JR, Zhang, J, Vaziri, ND, Zhuang, S, et al. La obstrucción ureteral unilateral causa disbiosis microbiana intestinal y trastornos del metaboloma que contribuyen a la fibrosis tubulointersticial. Exp Mol Med. (2019) 51:1–18. doi: 10.1038/S12276-019-0234-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: perro, enfermedad renal crónica, microbioma intestinal, disbiosis, bacterias proteolíticas, bacterias sacarolíticas
Cita: Kim K-R, Kim S-M y Kim J-H (2023) Un estudio piloto de alteraciones del microbioma intestinal en la enfermedad renal crónica canina. Frente. Vet. Sci. 10:1241215. doi: 10.3389/fvets.2023.1241215
Recibido: 16 de junio de 2023; Aprobado: 11 de agosto de 2023;
Publicado: 24 agosto 2023.
Editado por:
Isaac Karimi, Universidad Razi, Irán
Revisado por:
Guiyan Yang, Universidad Agrícola de China, China
Rashmi Hogarehalli Mallappa, Instituto Nacional de Investigación Láctea (ICAR), India
Derechos de autor © 2023 Kim, Kim y Kim. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Jung-Hyun Kim, junghyun@konkuk.ac.kr
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía