Análisis comparativo de conjuntos de cebadores para la evaluación de la clonalidad en linfomas felinos
Análisis comparativo de conjuntos de cebadores para la evaluación de la clonalidad en linfomas felinos
 Angelika Weyrich
Angelika Weyrich  Werner Hecht
Werner Hecht  Kernt Köhler
Kernt Köhler  Christiane Herden
Christiane Herden  Manfred Henrich*
Manfred Henrich*- Institut für Veterinär-Pathologie, Justus-Liebig-Universität Giessen, Giessen, Alemania
Introducción: Los linfomas se encuentran entre los tumores malignos más importantes y comunes en los gatos. Diferenciar los linfomas de las proliferaciones linfoides reactivas puede ser un desafío, por lo que las herramientas adicionales, como la evaluación de la clonalidad por PCR, son importantes para encontrar el diagnóstico. Se han desarrollado varios ensayos de PCR para evaluar la clonalidad en linfomas felinos. En el caso de los linfomas de células T, los genes TRG (receptor gamma de células T) son el objetivo preferido, mientras que en el caso de los linfomas de células B, la mayoría de los conjuntos de cebadores se dirigen a los genes de la cadena pesada de inmunoglobulinas (IGH). Aquí comparamos los conjuntos de cebadores diagnósticos más utilizados para la evaluación de la clonalidad en linfomas felinos en condiciones controladas (es decir, conjunto de muestras idéntico, configuración de PCR, sistema de detección de amplicones).
Métodos: Se analizaron muestras fijadas en formol e incluidas en parafina de 31 linfomas felinos de células T, 29 linfomas de células B y 11 controles no neoplásicos mediante PCR combinada con electroforesis capilar.
Resultados y discusión: Mostramos que la combinación de los conjuntos de cebadores publicados por Weiss et al. y Mochizuki et al. proporcionó los mejores resultados para la clonalidad de las células T, es decir, asigna correctamente la mayoría de las poblaciones como clonales o policlonales. Para la clonalidad de las células B, la combinación de los conjuntos de cebadores de Mochizuki et al. y Rout et al. dio los mejores resultados al omitir el reordenamiento del gen Kde debido a su baja especificidad. Este estudio evaluó rigurosamente varios conjuntos de cebadores en condiciones experimentales uniformes para mejorar la precisión del diagnóstico del linfoma y proporciona una recomendación para lograr la mayor precisión diagnóstica en el análisis de la clonalidad del linfoma.
1 Introducción
Los gatos son animales de compañía populares en todo el mundo. Entre las neoplasias malignas en gatos, los linfomas son una de las más importantes (1). La mayoría de los linfomas surgen de los dos linajes principales de linfocitos, los linfocitos B y T, por lo que existen muchos subtipos diferentes con diferente comportamiento biológico (2). Tanto la inflamación crónica como el linfoma causan proliferación de linfocitos. Las dos entidades pueden ser tan similares histológica y citológicamente que la diferenciación puede ser extremadamente difícil. Un ejemplo clásico de este dilema diagnóstico es la diferenciación, a menudo imposible, de la enteritis crónica del linfoma alimentario (3). Si bien las células de un linfoma y una inflamación pueden parecer muy similares morfológicamente, existen diferencias significativas y útiles para el diagnóstico a nivel genético. Las células de un linfoma generalmente se derivan de una sola célula transformada y son genéticamente uniformes (clonales) en sus genes receptores de antígenos, mientras que las células de una inflamación se reclutan a partir de diferentes células y, por lo tanto, difieren en sus genes receptores de antígenos (policlonales) (4-6). Se ha demostrado que la evaluación de esta clonalidad mediante PCR ayuda a encontrar un diagnóstico preciso (7, 8). Los genes receptores de antígenos se reorganizan durante el desarrollo de los linfocitos de modo que cada linfocito contiene una secuencia génica única en esa región del genoma (9). Los linfocitos T organizan directamente los genes variables (V) del gen del receptor gamma (TRG) de los linfocitos T con los genes de conexión (J) en múltiples casetes (10–12). En las cadenas ligeras de inmunoglobulina (IG), es decir, IG kappa (IGK) e IG lambda (IGL), los genes V también se reorganizan directamente con los genes J, mientras que en las cadenas pesadas de IG (IGH), la diversidad (D) y los genes J se reorganizan en un primer paso. A continuación, los genes D-J reordenados se fusionan con un gen V (9). En el caso de las cadenas ligeras de inmunoglobulinas, primero se reorganizan los genes IGK. Si esto no da lugar a una proteína funcional, se inicia el reordenamiento de los genes IGL y el alelo IGK se inactiva mediante el reordenamiento del llamado elemento de eliminación kappa (Kde) (10, 13, 14). Debido a que los reordenamientos implican la inserción y deleción aleatoria de nucleótidos, los genes reordenados difieren en longitud entre los diferentes linfocitos. Este principio se puede aplicar en ensayos de PCR para distinguir clones de linfocitos en función del tamaño del gen. La comparación de genes receptores de antígenos de diferentes linfocitos revela regiones con diversos grados de variabilidad dentro de estos genes. Las regiones marco (FR) muestran una baja variabilidad entre los diferentes linfocitos, mientras que las regiones determinantes de la complementariedad (CDR) muestran una alta variabilidad. Aquí, CDR3, que cubre las regiones de unión entre genes reordenados, exhibe el mayor grado de variabilidad (15). La amplificación de CDR3 con cebadores flanqueantes en las regiones marco y la posterior separación de los productos por longitud revelan un producto uniforme en una población neoplásica clonal y una gama de productos diferentes en poblaciones policlonales reactivas de linfocitos (7, 8).
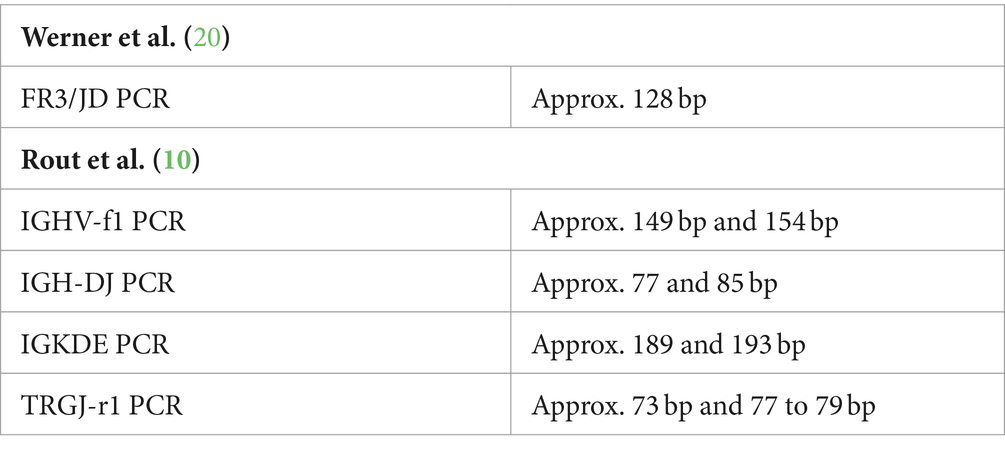
Se han desarrollado varios juegos de cebadores para evaluar la clonalidad en gatos (6, 10, 16–21). En los linfomas de células T, los genes TRG son el objetivo preferido. Este es el primer locus que se reordena y, por lo tanto, se reorganiza no solo en las células T gamma-delta, sino también en las células T alfa-beta (22, 23). El locus TRG felino contiene 12 genes TRGV (incluidos los pseudogenes) que pertenecen a 6 subconjuntos, 12 genes TRGJ (incluidos los pseudogenes y los marcos de lectura abiertos) y 6 genes TRGC (incluidos los pseudogenes) dispuestos en 5 casetes V-J-(J)-C completos y 1 incompleto (24). En los linfomas de células B, la mayoría de los conjuntos de cebadores se dirigen a los genes de la cadena pesada de inmunoglobulinas (IGH). Hay tres subconjuntos de genes IGHV en gatos (25). Es más probable que el subgrupo IGHV3 participe en los reordenamientos (18-20), mientras que el subgrupo IGHV1 tiene menos probabilidades de participar en los reordenamientos (18, 19). Los genes IGHJ pueden dividirse en dos variantes que se reordenan con frecuencia y varias variantes menos frecuentes (18-20). Rout et al. ampliaron la gama de objetivos potenciales para evaluar la clonalidad en poblaciones de células B en gatos al agregar un ensayo que incluye adicionalmente el reordenamiento incompleto de los genes IGH (reordenamiento IGH-DJ), el reordenamiento de los genes IGL y el reordenamiento de los genes Kde (10). En 2021, se publicó un ensayo adicional para el análisis de clonalidad de linfomas felinos de células T que incorporó loci de receptores de células T beta, delta y gamma (21).
La comparación directa de los conjuntos de cebadores para analizar la clonalidad en gatos es un desafío debido a las diferencias en los enfoques metodológicos, los conjuntos de muestras y las condiciones de laboratorio utilizadas en su diseño experimental original publicado (Tabla 1). Los primeros estudios utilizaron la electroforesis en gel de poliacrilamida (PAGE) para separar los productos de PCR, mientras que los estudios más recientes separan los productos mediante electroforesis capilar, que actualmente se considera el estándar de oro para el análisis de clonalidad. Algunos conjuntos de cebadores publicados no cubren todos los subgrupos de genes descritos, y el método de fijación también varía (tejido fresco o células frente a muestras FFPE).
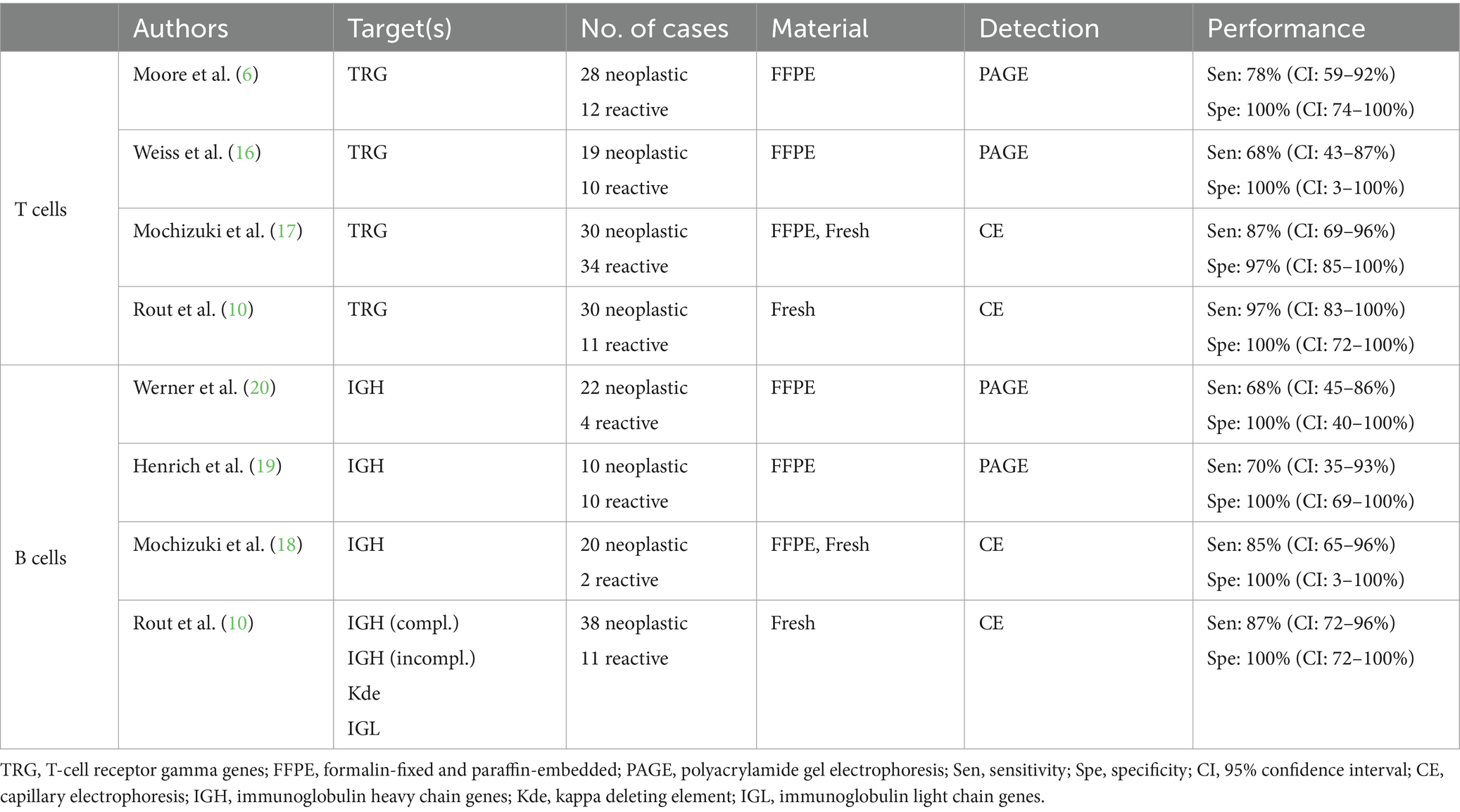 Tabla 1. Comparación de los conjuntos de cebadores publicados que se utilizan actualmente para el análisis de la clonalidad de los linfomas felinos.
Tabla 1. Comparación de los conjuntos de cebadores publicados que se utilizan actualmente para el análisis de la clonalidad de los linfomas felinos.
Por lo tanto, es importante realizar una comparación sistemática y controlada. En este trabajo presentamos un análisis comparativo de los sets de cebadores de PCR para la detección de clonalidad en linfomas felinos. Hasta la fecha, se compararon diferentes conjuntos de cebadores comúnmente utilizados en el diagnóstico para evaluar mejor la clonalidad en linfomas felinos en condiciones estandarizadas de laboratorio y muestras. Se identificaron combinaciones de conjuntos de cebadores para el diagnóstico de clonalidad del linfoma de células T y del linfoma de células B con una precisión diagnóstica significativamente mayor que los conjuntos de cebadores individuales utilizados hasta la fecha.
2 Materiales y métodos
2.1 Comparación de los conjuntos de cebadores por alineación
Para los conjuntos de cebadores que se unen a las mismas dianas, las secuencias de los cebadores de los diferentes ensayos se alinearon con las secuencias correspondientes de los genes receptores de antígenos felinos para evaluar y visualizar la relación espacial de los cebadores. El alineamiento se realizó utilizando BioEdit versión 7.2.0 (26), y las secuencias de los genes receptores de antígenos felinos se tomaron de estudios previos (19, 27).
2.2 Muestras de pacientes
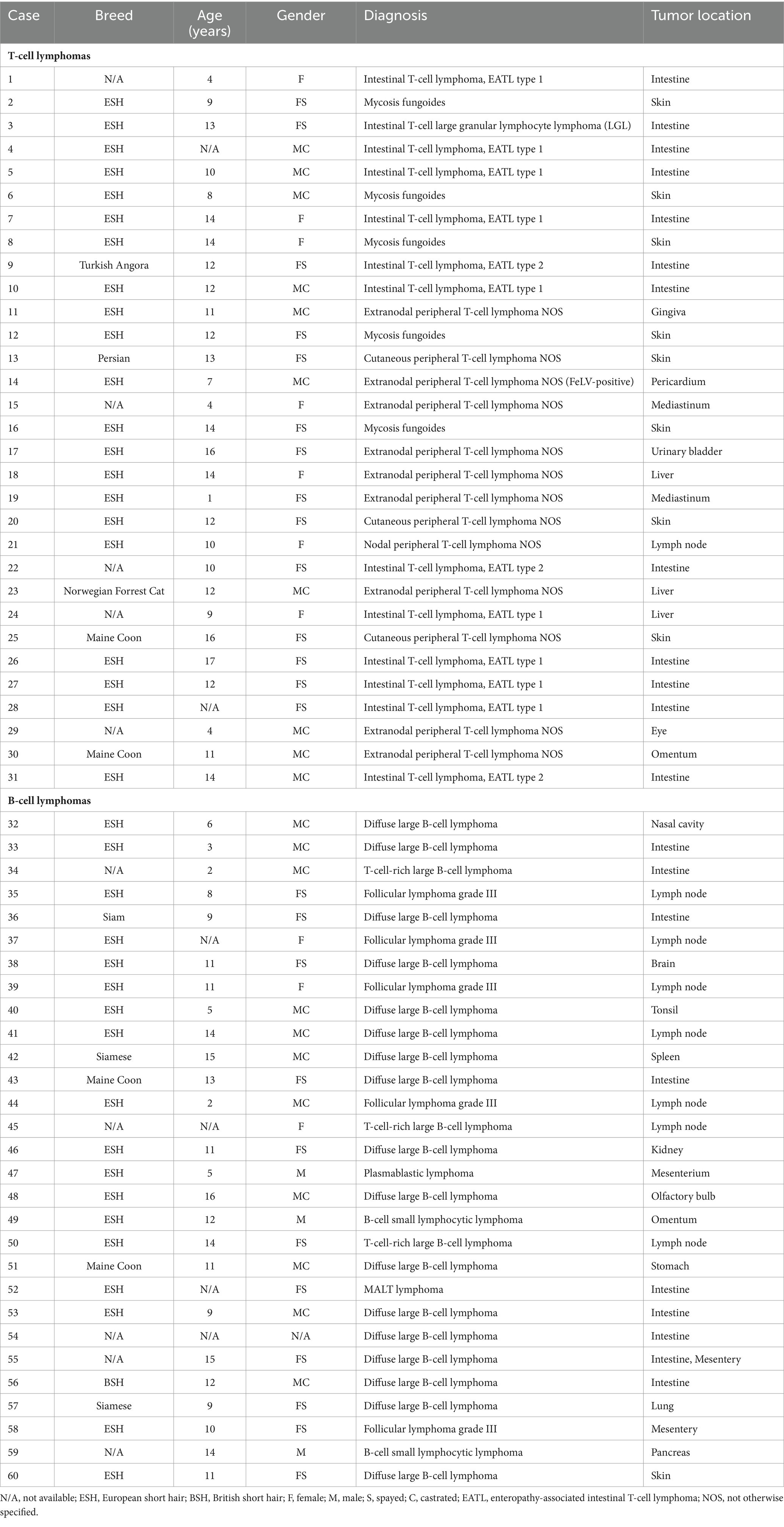
En el estudio se incluyeron muestras de FFPE de 31 gatos con linfomas de células T, 29 gatos con linfomas de células B (Tabla 2) y 11 gatos con proliferaciones linfoides reactivas (Tabla 3). El diagnóstico de linfoma y lesiones linfoides reactivas se realizó mediante histología [según criterios de la OMS (2)] e inmunohistoquímica frente al marcador de células T CD3 (policlonal conejo anti-humano CD3, Dako, Hamburgo, Alemania) y el marcador CD45R (rata anti-ratón CD45R, clon B220/RA3-6B2 [Ly 5], Cedarlane, Burlington, Ontario, Canadá) para identificar células B (28-31) según protocolos de rutina (32). Todos los casos fueron revisados por dos patólogos senior (KK, MH), y solo se incluyeron los casos con un diagnóstico inequívoco.
 Tabla 3. Características del paciente, diagnóstico y localización tumoral de gatos con proliferaciones linfocíticas reactivas.
Tabla 3. Características del paciente, diagnóstico y localización tumoral de gatos con proliferaciones linfocíticas reactivas.
2.3 Controles clonales
Para los reordenamientos de TRG, se utilizó ADN de una línea de células T (S87) con reordenamiento conocido de TRG (33). Para los reordenamientos de GH, el ADN de un linfoma difuso de células B grandes con patrón de electroforesis clonal confirmado (19) sirvió como control clonal. Para los conjuntos de cebadores que no lograron amplificar este linfoma, se utilizaron plásmidos que contenían las variantes correspondientes de los genes IGHV y los genes IGHJ (19).
2.4 Extracción y calidad del ADN
El ADN de las muestras de FFPE se aisló de los bloques de parafina utilizando el kit de tejido QIAamp DNA FFPE (QIAGEN GmbH, Hilden, Alemania) de acuerdo con las instrucciones del fabricante con las siguientes modificaciones: En los casos con áreas pequeñas de células tumorales, el material de la región de interés se raspó manualmente de los bloques utilizando una cuchilla de micrótomo estéril para minimizar la cantidad de ADN de las células no neoplásicas. El ADN del material de cultivo celular (línea celular S87) se aisló de acuerdo con un protocolo modificado de Miller et al. (34). Brevemente, 1 mL de suspensión celular (1 × 10®6 células/ml) (15.000 × g, 5 s). El gránulo celular se incubó con 300 μL de tampón de lisis celular (10 mM de Tris-HCl, 400 mM de NaCl y 2 mM de Na2EDTA, pH 8,2) a 37°C hasta que sea homogéneo. El ARN se eliminó mediante la adición de RNasa A e incubación a 37 °C durante 30 min. Después de enfriar a temperatura ambiente, la proteína en la solución se precipitó agregando 100 μL de solución de precipitación de proteína (NaCl saturado, aproximadamente 6 M) y se peletizó después de vórtice vigorosamente. El ADN del sobrenadante se precipitó en 300 μL de isopropanol y después del vórtice y la centrifugación, el gránulo se lavó en 300 μL de etanol al 70%. El ADN se resuspendió en una solución de hidratación de ADN (10 mM de Tris-HCl, 0,2 mM de Na2EDTA, pH 7,5). Todos los productos químicos y enzimas se compraron a Carl Roth GmbH & Co. KG, Karlsruhe, Alemania. La concentración de ADN se determinó fotométricamente (NanoDrop 2000; Thermo Scientific, Waltham, MA, EE. UU.) después de la homogeneización a 63 °C. Todas las muestras se almacenaron a -20 °C. La calidad del ADN se evaluó amplificando fragmentos de aproximadamente 300 pb (19), aproximadamente 188 pb (18) y aproximadamente 150 pb (19) de las porciones no reordenadas del genoma felino.
2.5 Protocolos de PCR para el análisis de reordenamientos de IG y TRG
Para cada caso, todos los juegos de cebadores utilizados (Tabla Suplementaria 1) se probaron en el mismo aislamiento de ADN para evitar sesgos debidos a la variabilidad en el ADN aislado. Para facilitar la detección de la pseudoclonalidad, todas las reacciones se ejecutaron por duplicado (linfomas de células T) o por triplicado (linfomas de células B) a 50 ng de ADN por reacción de PCR de 25 μL, según lo recomendado por Keller et al. (7). El ADN se amplificó utilizando Bioline MyTaq™ HS Mix (Bioline GmbH, Luckenwalde, Alemania) y las respectivas imprimaciones (adquiridas a Biomers.net GmbH, Ulm, Alemania). Se utilizaron cebadores en las combinaciones y concentraciones indicadas (Tabla Suplementaria 1). Para su uso en electroforesis capilar, los juegos de cebadores se marcaron con colorantes fluorescentes de modo que cada amplicón llevara una molécula de colorante fluorescente (Tabla suplementaria 1). Las condiciones de ciclismo fueron las indicadas para los juegos de cebadores (Tabla suplementaria 2) con las siguientes modificaciones: Si no se incluyen en las condiciones de ciclismo originales, un paso de activación inicial (5 min, 95 °C) de Bioline MyTaq™ Se añadió polimerasa HS (hot start). Además, se agregó un paso de elongación terminal (10 min, 72 °C) para asegurar los voladizos de 3′-adenina y, por lo tanto, minimizar los artefactos «plus A» en la electroforesis capilar. En los protocolos de Mochizuki et al. (17, 18), el paso de elongación terminal ya estaba incluido. Se utilizó un control sin plantilla para todas las reacciones.
2.6 Electroforesis capilar
Los amplicones se separaron en un analizador genético ABI Prism® 310 (Applied Biosystems® by life technologies, Darmstadt, Alemania). Los productos de PCR se analizaron sin diluir o diluidos en agua después de la PCR. La dilución estándar fue de 1:5 o 1:10 (dependiendo del juego de cebadores). El producto de PCR (diluido) se desnaturalizó en formamida (Hi-Di™ Formamide, Applied Biosystems® by life technologies, Darmstadt, Alemania) mezclada con el estándar de tamaño LIZ (GeneScan™ 500 LIZ™ dye Size Standard, Applied Biosystems® by life technologies, Darmstadt, Alemania) a 95 °C durante 5 min, seguido de 10 min de incubación en hielo. La inyección se realizó a 15.000 V durante 5 s. La electroforesis se realizó a 60°C, 15.000 V y 10 mA de corriente láser. Si el resultado de la electroforesis era inadecuado, se ajustaba el tiempo de dilución y/o inyección: si las señales alcanzaban el máximo de detección, se aumentaba la dilución. Si los picos eran demasiado pequeños o estaban ausentes, el tiempo de inyección se aumentaba a 10 s. Si esto no mejoraba los resultados, se repetía la electroforesis con producto de PCR menos diluido o sin diluir.
2.7 Análisis de resultados
La representación gráfica y el análisis de tamaño se realizaron utilizando el software Peak Scanner™ versión 1.0 (Applied Biosystems® by life technologies, Darmstadt, Alemania). Para el análisis estadístico de los datos se utilizó Microsoft Excel 2019. Los resultados de la electroforesis se interpretaron/describieron de acuerdo con las guías publicadas (7, 8). Para objetivar la comparación de los diferentes conjuntos de cebadores, se definieron algunos criterios de la siguiente manera:
De acuerdo con la definición comúnmente utilizada de un intervalo de referencia, el rango de tamaño esperado para cada conjunto de cebadores se definió como el 95% central de la distribución de referencia (distribución de tamaño de todos los amplicones de los controles policlonales del conjunto respectivo). Por lo tanto, el rango de tamaño esperado se calculó como μ ± 2σ del tamaño de los amplicones de los controles policlonales (con μ = media y σ = desviación estándar).
Los picos específicos se definieron como picos dentro del rango de tamaño esperado [± 10 pb, según Rothberg et al. (17)]. Todos los picos fuera del rango de tamaño esperado se consideraron picos no específicos. Esta última categoría también incluyó picos que fueron reproducibles en muestras no relacionadas y/o controles no relacionados (los llamados «picos constantes»), independientemente de si estos picos ocurrieron dentro o fuera del rango de tamaño esperado (7, 8).
Los picos reproducibles se consideraron como tales si los picos del mismo tamaño (en bases) ocurrieron en reacciones duplicadas/triplicadas. Dado que el software analítico calcula el tamaño del pico en función del estándar de tamaño agregado, el tamaño de un pico se redondea a dos decimales. Los picos se consideraron iguales en tamaño si el tamaño del pico no difería de la media de los dos tamaños de pico en más de ±0,49 bases.
Los picos múltiples se han definido como ≥3 picos (8). En este estudio, los picos múltiples se definieron como 3 a 5 picos distintos sin fondo (6, 20). Las señales de menos de 50 unidades de fluorescencia relativa (RFU) se consideraron ruido de fondo, independientemente de si eran estructuras «en forma de pico» que se destacaban de la señal de fluorescencia de fondo circundante.
Los picos pequeños/altura de pico baja se definieron como picos entre 50 RFU y el «límite de cuantificación» (LOQ) del analizador genético ABI Prism® 310. El LOQ es el umbral inferior del intervalo de intensidad de la señal de fluorescencia con una relación lineal entre la intensidad de la señal y el número de fragmentos de ADN. Para el analizador genético ABI Prism® 310, el LOQ es de 150 RFU.
Todos los patrones de electroforesis se asignaron a una de las siguientes categorías:
No se definieron picos específicos como la ausencia de picos específicos como se definió anteriormente. Si la calidad del ADN era adecuada, la muestra se interpretaba como no amplificable con el conjunto de cebadores correspondiente. Si la calidad del ADN era deficiente, la ausencia de picos específicos podría deberse a un conjunto de cebadores no vinculantes y/o a un ADN generalmente no amplificable. Por lo tanto, estas muestras fueron clasificadas como no evaluables.
Uno o dos picos específicos y reproducibles se consideraron indicativos de una población clonal (véase la Figura 1A). En los casos con dos picos, se asumió una población biclonal o un reordenamiento bialélico. Para la interpretación de la clonalidad, la reproducibilidad fue obligatoria para al menos uno de los picos. Los picos en esta categoría con baja altura máxima (50 a 150 RFU) se interpretaron con precaución y solo como indicativos de una población clonal. El modificador «con antecedentes» se aplicó cuando los picos reproducibles se acompañaron de un patrón indicativo de una población policlonal/oligoclonal en el mismo tejido (ver Figura 1B). Para distinguir los picos clonales del fondo, los picos clonales en todas las reacciones duplicadas/triplicadas tenían que ser al menos dos veces más altos que los picos adyacentes (relación 2:1). Esto corresponde a un 5% de células clonales en la muestra (35). Las muestras que tenían picos reproducibles con un fondo por debajo de la proporción 2:1 se interpretaron como «policlonales con clones menores de significado incierto».
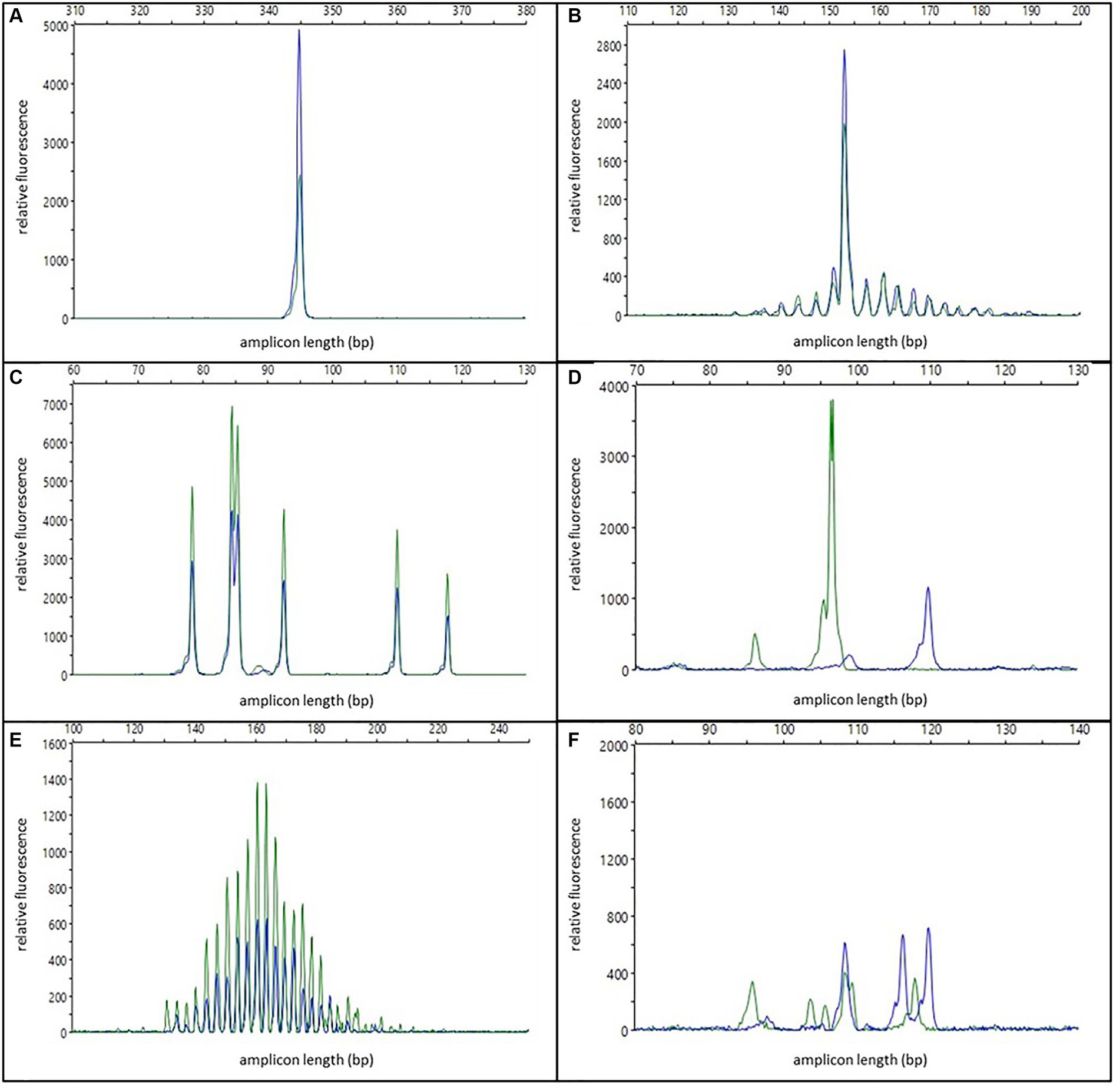 Figura 1. Ejemplos de diferentes patrones de electroforesis; los colores azul y verde representan reacciones duplicadas; (A) un pico/clonal específico reproducible; (B) un pico específico reproducible con fondo/clonal con fondo policlonal; (C) múltiples picos específicos reproducibles/oligoclonales; (D) dos picos no reproducibles/pseudoclonales; (E) Curva gaussiana/policlonal; (F) múltiples picos específicos no reproducibles/irregularmente policlonales.
Figura 1. Ejemplos de diferentes patrones de electroforesis; los colores azul y verde representan reacciones duplicadas; (A) un pico/clonal específico reproducible; (B) un pico específico reproducible con fondo/clonal con fondo policlonal; (C) múltiples picos específicos reproducibles/oligoclonales; (D) dos picos no reproducibles/pseudoclonales; (E) Curva gaussiana/policlonal; (F) múltiples picos específicos no reproducibles/irregularmente policlonales.
Los picos múltiples específicos y reproducibles (≤5) se interpretaron como el patrón típico de una población oligoclonal (véase la Figura 1C). A diferencia de otras publicaciones (10), estos casos se consideraron no clonales y, por lo tanto, se excluyeron del cálculo de la sensibilidad.
Uno o dos picos no reproducibles se consideraron como resultados pseudoclonales debido a la amplificación aleatoria de diferentes clones individuales de una población oligoclonal o policlonal en reacciones repetidas de PCR de muestras con mala calidad de ADN (ver Figura 1D).
Los patrones de electroforesis con múltiples picos (>5) dispuestos en una curva gaussiana más o menos «en forma de campana», se interpretaron como poblaciones policlonales (ver Figura 1E). Esta interpretación podría modificarse: las muestras con curvas de forma irregular, las muestras con curvas regulares en una reacción pero solo de tres a cinco picos individuales en las reacciones replicadas, o las muestras con picos discernibles no reproducibles que no tenían una relación de 2:1 con los picos adyacentes en la reacción replicada se designaron como irregularmente policlonales. Las curvas gaussianas con baja altura (50 a 150 RFU) se interpretaron como «débilmente policlonales».
Los picos múltiples específicos y no reproducibles también se interpretaron como irregularmente policlonales (véase la Figura 1F).
Muestras que no encajaban en una de las categorías antes mencionadas, por ejemplo, muestras con picos solo dentro del ruido de fondo, muestras sin picos específicos en una reacción y al menos tres picos o una curva gaussiana en las réplicas, o muestras con uno o dos picos específicos sin fondo en una muestra, pero una curva gaussiana o múltiples picos no reproducibles en las réplicas, se consideraban no evaluables.
3 Resultados
3.1 Comparación de los juegos de cebadores por alineación
En la Figura 2 se muestra la relación espacial de los conjuntos de cebadores dirigidos a los genes TRG y a los genes IGH, respectivamente. Aunque existe una clara diferencia en los conjuntos de objetivos individuales, también existe una superposición significativa.
 Figura 2. Comparación de los sitios de unión de cebadores de los diferentes conjuntos de prueba en los genes del receptor gamma de células T (TRG) y la cadena pesada de inmunoglobulinas (IGH). TRGV1, 2 y 3, subgrupos de genes de región variable del receptor gamma de células T; TRGJ: genes de la región de unión gamma del receptor de células T; CDR3, región determinante de la complementariedad; IGHV1 y 3, subgrupos de regiones variables de cadena pesada de inmunoglobulinas; IGHJ: genes de la región de unión de cadenas pesadas de inmunoglobulinas. Resaltado en rojo: Combinaciones de cebadores que, en conjunto, clasificaron correctamente la mayoría de las poblaciones como clonales o policlonales en el estudio.
Figura 2. Comparación de los sitios de unión de cebadores de los diferentes conjuntos de prueba en los genes del receptor gamma de células T (TRG) y la cadena pesada de inmunoglobulinas (IGH). TRGV1, 2 y 3, subgrupos de genes de región variable del receptor gamma de células T; TRGJ: genes de la región de unión gamma del receptor de células T; CDR3, región determinante de la complementariedad; IGHV1 y 3, subgrupos de regiones variables de cadena pesada de inmunoglobulinas; IGHJ: genes de la región de unión de cadenas pesadas de inmunoglobulinas. Resaltado en rojo: Combinaciones de cebadores que, en conjunto, clasificaron correctamente la mayoría de las poblaciones como clonales o policlonales en el estudio.
3.2 Comparación experimental de conjuntos de cebadores
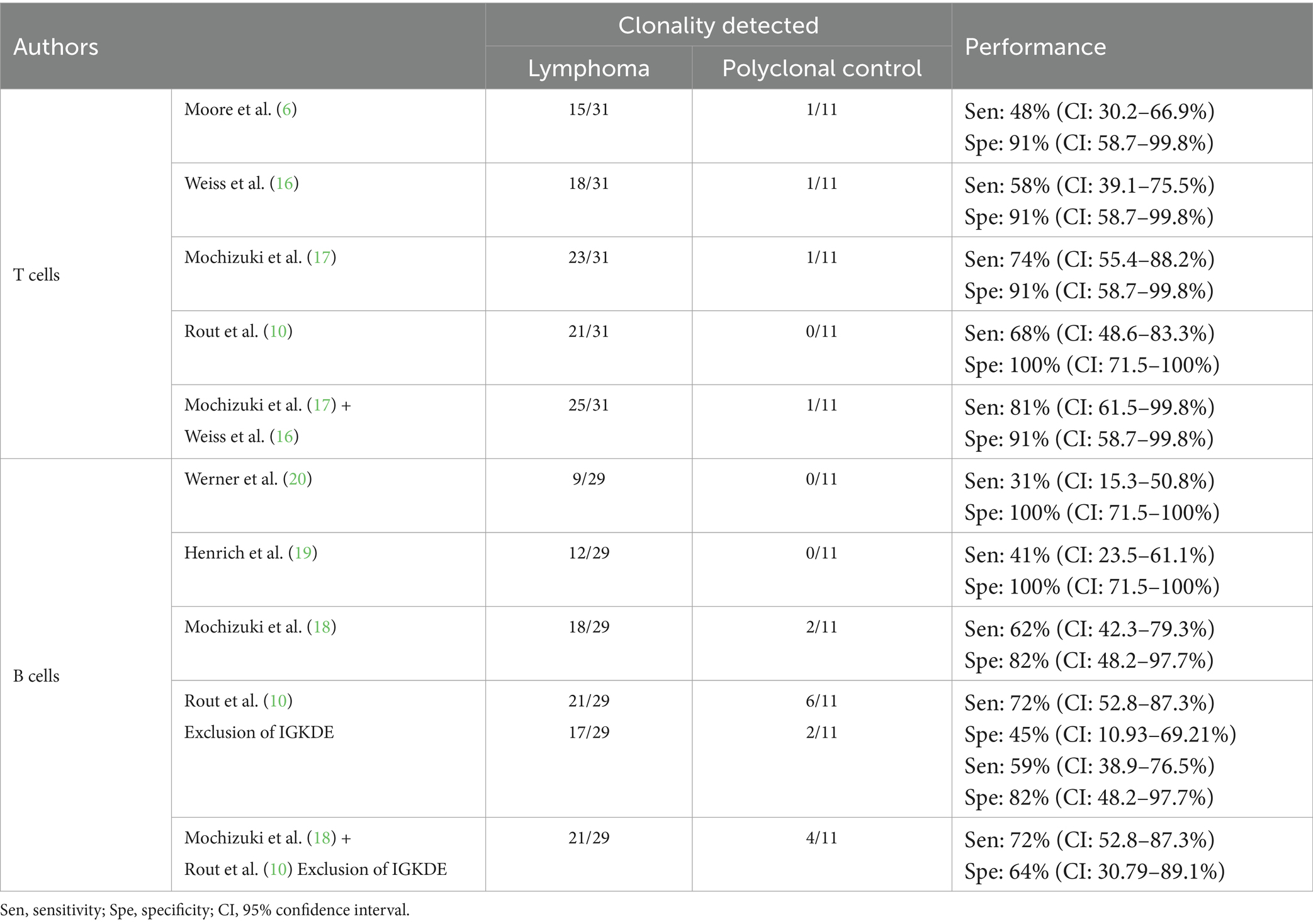
La comparación de los resultados y rendimientos de los diferentes conjuntos de cebadores se muestra en la Tabla 4. Además de los ratios de los conjuntos de cebadores individuales, también se enumeran los valores correspondientes de la combinación de juegos de cebadores que juntos lograron los mejores resultados de ensayo (para obtener resultados detallados de las muestras individuales, véanse las tablas complementarias 3 y 4).
En el caso de los linfocitos T, los mejores resultados se obtuvieron combinando los conjuntos de cebadores de Mochizuki et al. (17) y Weiss et al. (16). Los cebadores de Weiss et al. (16), detectaron con éxito la clonalidad en dos muestras donde los cebadores de Mochizuki et al. (17) indicaron policlonalidad. Por el contrario, los cebadores de Mochizuki et al. (17) detectaron clonalidad en siete muestras, mientras que los cebadores de Weiss et al. (16) mostraron policlonalidad, sin reordenamiento o resultados no evaluables. La combinación de los conjuntos de Mochizuki et al. (17) y Weiss et al. (16) incluyó todos los resultados clonales obtenidos con los conjuntos de cebadores de Moore et al. (6) y Rout et al. (10), cada uno de los cuales arrojó menos resultados clonales.
Weiss et al. (16) concluyeron en su estudio que mediante el uso de los cebadores inversos FTGFR4r2 y FTGFR4r3 la clonalidad solo podía detectarse en el 58% de los linfomas y se producían resultados clonales falsos en el 86% del grupo control policlonal. Por lo tanto, no recomendaron el uso de estas imprimaciones. Sin embargo, en el presente estudio, la sensibilidad de las reacciones de PCR con FTGFR4r2 fue del 45% y la especificidad del 91%. El resultado clonal de la muestra de control 69 como causa de la menor especificidad también podría reproducirse con varios otros conjuntos de cebadores. Además, en cuatro muestras, la clonalidad solo fue detectable con FTGFR4r2. Por lo tanto, los resultados del presente estudio sugieren que el uso de esta imprimación puede ser valioso.
El conjunto de cebadores de Moore et al. (6), que cubre solo los genes del subgrupo 1 de TRGV, mostró clonalidad en 15/31 linfomas, lo que sugiere que el subgrupo 1 de TRGV se reorganizó con mayor frecuencia en este estudio. En 6/31 muestras la detección de clonalidad fue exitosa con los conjuntos de cebadores de Weiss et al. (16) y/o Mochizuki et al. (17) (que cubren los tres subgrupos de TRGV) y adicionalmente con el conjunto de Rout et al. (10) (que cubre los subgrupos 1 y 2 de TRGV), pero no con el de Moore et al. (6). Esta distribución sugiere que estos clones neoplásicos utilizaron genes del subgrupo 2 del TRGV. En 4/31 muestras, la clonalidad se detectó con los conjuntos de cebadores de Weiss et al. (16) y/o Mochizuki et al. (17), pero no con los de Moore et al. (6) y Rout et al. (10). Esto indica el uso de genes del subgrupo 3 de TRGV.
En seis linfomas de células T, no se pudo demostrar la clonalidad con ningún sistema. Todos los juegos de cebadores, excepto el de Rout et al. (10), mostraron clonalidad en una muestra de control policlonal (nº 69, enteritis crónica). Sin embargo, con la excepción de la PCR FR2f1 de Weiss et al. (16), se detectó una población policlonal de fondo en todas las PCR. Las combinaciones de cebadores con las que no se pudo detectar la clonalidad dieron resultados policlonales claros. Una posible explicación para esta aparente detección de clonalidad falsa positiva podría ser un número limitado de linfocitos en la biopsia del intestino delgado. Por otro lado, se ha descrito la progresión de la enteritis crónica a linfoma intestinal (6, 36). Por lo tanto, no se puede excluir que esta muestra contenga una población neoplásica de linfocitos.
Para las células B, los mejores resultados se obtuvieron combinando los conjuntos de cebadores de Mochizuki et al. (18) y Rout et al. (10), excluyendo la PCR para el reordenamiento de los genes Kde (IGKDE-PCR). La IGKDE-PCR alcanzó una sensibilidad del 38%. Sin embargo, se detectaron seis resultados clonales en el grupo control policlonal, lo que resultó en una especificidad de solo el 45%. Esto se debió a la ocurrencia de picos reproducibles en torno a 189 y 193 pb (véase el Cuadro 5). La mayoría de las muestras en el grupo de linfoma evaluado como clonal también mostraron estos picos. No está claro si se trata de productos específicos de PCR o de «picos constantes» inespecíficos que deben excluirse de la evaluación. Por lo tanto, este conjunto no se incluyó en los cálculos posteriores. Sin embargo, con la combinación de los conjuntos de cebadores de Mochizuki et al. (18) y Rout et al. (10) modificados de esta manera, se detectó clonalidad en tres muestras con solo uno de estos dos conjuntos.
Todos los resultados clonales obtenidos con el conjunto de cebadores de Werner et al. (20) y Henrich et al. (19) también se obtuvieron con el conjunto de Mochizuki et al. (18).
La muestra 55 fue el único linfoma de células B en el que la detección de clonalidad con cebadores específicos de IGHV1 fue exitosa, lo que apoya la hipótesis de que el reordenamiento de este subgrupo es raro en la población de gatos en su conjunto (18, 19, 37).
En ocho linfomas de células B, no fue posible la detección de la clonalidad.
Cuatro muestras de control policlonal mostraron un resultado clonal. La muestra 71 era tejido tímico y la muestra 69 era enteritis crónica del intestino delgado con un posible bajo número de células B en las muestras. Las muestras 65 y 67 mostraron una población policlonal además de los picos clonales. Estos picos tenían longitudes de fragmento en el medio de la curva gaussiana, es decir, en el rango de longitud de fragmentos donde estadísticamente se esperarían los reordenamientos más frecuentes. Por lo tanto, estos picos podrían representar múltiples clones de la misma longitud que no se pueden distinguir por electroforesis capilar.
Además de los resultados de la comparación de cebadores, se obtuvo información adicional con respecto al rendimiento técnico del ensayo.
La calidad del ADN de todas las muestras de FFPE fue suficiente para la amplificación de un fragmento de 150 pb de largo. La amplificación del fragmento largo de 300 pb falló en una muestra de control policlonal, seis linfomas de células B y seis linfomas de células T. Sin embargo, el fragmento de 188 pb pudo amplificarse en todas estas muestras. Según lo propuesto por Lenze (38), la pureza del ADN se comprobó fotométricamente (Tabla suplementaria 5). Los cocientes subóptimos A260/A280 y A260/A230 estuvieron presentes en el 28% de los linfomas de células B (8/29) y el 35% de los linfomas de células T (11/31), respectivamente. Sin embargo, el efecto sobre la sensibilidad del ensayo pareció ser insignificante, ya que la detección de clonalidad falló en el 20% de las muestras con pureza subóptima y en el 27% de las muestras con proporciones óptimas. Con la excepción de una muestra con una pureza subóptima, se podrían generar amplicones de al menos 188 pb en todas las muestras.
Al realizar la electroforesis capilar, es importante mantener la intensidad de la señal (altura del pico en RFU) en el rango evaluable, es decir, en el rango con una relación lineal entre el número de fragmentos de ADN de cierta longitud y la intensidad de la señal (39). En el caso de muchas muestras, este no fue el caso cuando se utilizó el producto de PCR sin diluir. La altura del pico superó el llamado «límite de linealidad» y las imágenes de electroforesis no fueron interpretables. En estos casos, se requirió la predilución del producto de PCR. Ni la concentración del ADN aislado ni la pureza del ADN permitieron predecir la dilución requerida. Sin embargo, las muestras clonales generalmente requirieron una dilución más alta. La única publicación sobre el diagnóstico de clonalidad por PCR por electroforesis capilar que menciona la necesidad de predilución (10) especifica una dilución estándar de diez veces para cada muestra. Sin embargo, en nuestro estudio, se requirió una dilución de hasta 1:100 cuando se utilizó el mismo sistema de prueba. Parece haber una correlación con la eficiencia de la amplificación y muy probablemente también con los parámetros técnicos del instrumento analítico. Por lo tanto, podría ser necesario un examen de la dilución óptima para cada juego de cebadores, que tendría que ajustarse adicionalmente en función de la evaluabilidad de los resultados de la electroforesis para muestras individuales.
Para la mayoría de los ensayos, el cálculo del rango de tamaño esperado de los conjuntos de cebadores dio como resultado rangos más grandes que en las respectivas publicaciones originales (Tabla suplementaria 6).
En algunas reacciones de PCR, se generaron amplicones reproducibles que estaban más de 10 pb fuera del rango de tamaño esperado. Es necesario realizar un análisis de la secuencia para determinar si se trata de reordenamientos específicos (40).
Además, se detectaron picos constantes en algunas reacciones de PCR (ver Tabla 5).
4 Discusión
En los últimos años, se ha desarrollado una variedad de ensayos diagnósticos con múltiples conjuntos de cebadores para evaluar la clonalidad en los linfomas felinos. Cada conjunto de cebadores ha sido validado en las publicaciones originales y estudios posteriores. Sin embargo, la comparación precisa de estos conjuntos de cebadores se complica por los diferentes enfoques y materiales utilizados. En este estudio, empleamos un enfoque estandarizado para comparar varios conjuntos de cebadores, lo que en última instancia ofrece una recomendación concluyente para lograr una alta precisión diagnóstica en el análisis de clonalidad del linfoma felino.
En los linfomas de células T, la mayoría de las poblaciones podrían asignarse correctamente como clonales o policlonales mediante la combinación de los conjuntos de cebadores de Weiss et al. (16) y Mochizuki et al. (17). Esto no es sorprendente, ya que estos dos pueden detectar una amplia gama de posibles variantes genéticas, incluidos objetivos en todas las regiones del marco. Sin embargo, el uso del juego de cebadores de Weiss et al. (16) requiere relativamente tiempo y material debido a las numerosas reacciones de PCR. Por lo tanto, para su uso en diagnósticos rutinarios, particularmente en tejidos FFPE, se debe considerar el uso de los cebadores de Mochizuki et al. (17) como primer paso, que también tienen la ventaja de amplicones más cortos. Si no se puede demostrar la clonalidad y todavía se sospecha fuertemente de linfoma, se podría utilizar posteriormente el conjunto de cebadores de Weiss et al. (16).
En seis linfomas de células T, la clonalidad no se pudo detectar con ningún sistema. La falta de productos de amplificación específicos puede tener numerosas causas además de la mala calidad del ADN, lo que tiene un impacto negativo, especialmente en sistemas con longitudes de fragmentos más largas. La cobertura insuficiente de las diferentes variantes genéticas por parte de los cebadores disponibles, la mutación de los sitios de unión de los cebadores, que sin embargo desempeña un papel particularmente en los linfocitos B debido a la hipermutación somática, y las aberraciones cromosómicas son posibles causas (7). De acuerdo con Wang et al. (41), la ausencia de amplificación de diana combinada con una buena amplificación de los genes de control también puede indicar una falta de ADN linfocitario en la muestra. Finalmente, en ausencia de evidencia de clonalidad por parte de todos los conjuntos de cebadores, el diagnóstico histológico también debe ser cuestionado de manera autocrítica. Aunque los casos se clasificaron como linfomas según los criterios histológicos de la OMS (2) y por la determinación inmunohistoquímica del inmunofenotipo de los linfocitos, no se puede excluir completamente que se trate de lesiones reactivas, es decir, no neoplásicas.
En el caso de los linfomas de células B, el mejor resultado se obtuvo combinando los conjuntos de Mochizuki et al. (18) y Rout et al. (10), pero sin utilizar IGKDE-PCR. Incluso sin el análisis de los reordenamientos de Kde, la combinación de estos dos conjuntos de cebadores cubre la gama más amplia de variantes genéticas, lo que explica el buen resultado general. Se excluyó el análisis de reordenamiento de Kde debido a la baja especificidad del 45% basada en los picos constantes en el grupo control. Es concebible que la variabilidad de longitud generalmente baja en este locus conduzca a la formación de productos de PCR de longitud similar, pero con secuencia diferente. La electroforesis capilar podría entonces fallar en distinguir estos productos.
En ocho linfomas de células B no fue posible la detección de clonalidad. Como se mencionó anteriormente, el fenómeno de la hipermutación somática en los linfomas de células B, probablemente tenga un gran impacto en la integridad de los sitios de unión del cebador, especialmente en FR3. Esto también puede asumirse como la causa de la menor sensibilidad de los ensayos de clonalidad de células B en comparación con los ensayos de clonalidad de células T en el estudio presentado (18, 20).
En general, el presente trabajo no detectó ninguna influencia del tipo de linfoma en la sensibilidad de ciertos conjuntos de cebadores o en el reordenamiento preferencial de ciertas dianas. Sin embargo, el número de cada tipo de linfoma en el material de la muestra fue bajo. Además, hubo una clara disparidad en la frecuencia de los diferentes tipos: aproximadamente el 42% de los linfomas de células T eran formas intestinales (13/31) y aproximadamente el 59% de los linfomas de células B eran linfomas difusos de células B grandes (DLBCL) (17/29).
Los llamados «picos constantes» (véase el Cuadro 3), es decir, los picos que son reproducibles en muchas o incluso en todas las muestras, pueden tener diferentes causas (7). La amplificación inespecífica ocurre principalmente cuando solo están presentes unos pocos sitios específicos de unión de cebadores, es decir, en muestras con un bajo número de linfocitos B o T. Este efecto es particularmente común en las reacciones de PCR múltiplex debido a la mayor competencia por el ADN molde. En el caso de los ensayos de clonalidad en medicina humana, existen numerosas descripciones de estos productos de amplificación inespecíficos, que deben tenerse en cuenta en la evaluación (8). En los sistemas de pruebas veterinarias, estas descripciones son raras. Para evitar interpretaciones erróneas, sería útil disponer de información más detallada sobre este fenómeno. Keller et al. (7) mencionan una baja diversidad de CDR3 en ciertas recombinaciones V-J en células T caninas. Este fenómeno también se ha descrito en humanos y se conoce como «reordenamientos canónicos» (42, 43). Esto también podría explicar la aparición de «picos constantes» en las muestras de linfocitos felinos.
El desarrollo ulterior de los diagnósticos de clonalidad basados en la PCR en la medicina veterinaria en general y en el gato en particular requiere un estudio prospectivo a mayor escala utilizando material de muestra relevante para la práctica, es decir, casos dudosos en los que se sospecha claramente un linfoma pero no es posible un diagnóstico definitivo sobre la base de exámenes preliminares. De forma análoga a la situación del estudio en perros (44), debe realizarse una comparación de diferentes materiales de muestra (tejido FFPE, tejido fresco, aspirados con aguja fina, gránulos de células). Para evaluar la relevancia de los resultados del análisis de clonalidad de la PCR para la progresión de la enfermedad, la respuesta a la quimioterapia y el pronóstico de los diferentes tipos de linfoma, se requeriría un seguimiento clínico detallado.
Los avances en curso en el análisis de genes de receptores de antígenos felinos deben incorporarse a las pruebas de clonalidad. Tras la finalización de nuestro estudio, Radtanakatikanon et al. presentaron nuevos hallazgos sobre el receptor de células T felinas utilizando un ensayo recientemente desarrollado (21). Esto incluye los genes delta, beta y gamma del receptor de células T, a lo largo de la estructura multicasete del locus TRG felino. La incorporación directa de este novedoso conjunto de cebadores en nuestra comparación actual habría comprometido la estricta comparabilidad de las muestras iniciales utilizadas y, por lo tanto, socavado la solidez de nuestro estudio. Si bien esta es una limitación de nuestro estudio, la realización de un análisis comparativo separado del nuevo ensayo junto con los conjuntos de cebadores de Weiss et al. (16) y Mochizuki et al. (17) debería permitir una evaluación del rendimiento de este nuevo ensayo.
De acuerdo con Keller et al. (7), la adopción de métricas de rendimiento y criterios de evaluación estandarizados como en la medicina humana (45) sería beneficiosa para garantizar la comparabilidad de los resultados de las pruebas entre laboratorios.
En resumen, mediante la comparación sistemática de diferentes conjuntos de cebadores, este estudio reveló combinaciones útiles de ensayos [Weiss et al. (16) y Mochizuki et al. (17) para linfocitos T y Mochizuki et al. (18) y Rout et al. (10) para linfocitos B] para el análisis de clonalidad de linfomas felinos.
Además, los resultados aclararon algunas definiciones que pueden ser útiles para interpretar los resultados del análisis de clonalidad en linfomas felinos.
Declaración de disponibilidad de datos
Los datos brutos que respaldan las conclusiones de este artículo serán puestos a disposición por los autores, sin reservas indebidas.
Declaración ética
No se requirió aprobación ética para el estudio con animales de acuerdo con la legislación local y los requisitos institucionales, ya que el estudio se realizó en material de archivo fijo.
Contribuciones de los autores
AW: Metodología, Redacción – revisión y edición, Redacción – borrador original, Investigación, Obtención de fondos, Conceptualización. PM: Investigación, Redacción – revisión y edición, Validación, Metodología. KK: Escritura – revisión y edición, Validación, Investigación. CH: Redacción – revisión y edición, Validación, Supervisión. MH: Redacción – revisión y edición, Validación, Supervisión, Metodología, Investigación, Conceptualización.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. El apoyo financiero para la realización de la investigación fue proporcionado por la fundación Ewald e Hilde Berge (Stiftung der Eheleute Ewald und Hilde Berge). El patrocinador no tuvo ningún papel en el diseño del estudio, en la recopilación, análisis e interpretación de los datos, en la redacción del manuscrito y en la decisión de enviar el artículo para su publicación.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
El/los autor/es declararon, en el momento de la presentación, ser miembro del consejo editorial de Frontiers. Esto no tuvo ningún impacto en el proceso de revisión por pares ni en la decisión final.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1356330/full#supplementary-material
Referencias
1. Graf, R, Grüntzig, K, Boo, G, Hässig, M, Axhausen, KW, Fabrikant, S, et al. Registro suizo de cáncer felino 1965-2008: la influencia del sexo, la raza y la edad en los tipos y localizaciones de los tumores. J Comp Pathol. (2016) 154:195–210. doi: 10.1016/j.jcpa.2016.01.008
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Valli, VE. Clasificación histológica de los tumores hematopoyéticos de animales domésticos. Washington, DC: Instituto de Patología de las Fuerzas Armadas, en cooperación con el Registro Americano de Patología y el Centro Colaborador de la Organización Mundial de la Salud para la Referencia Mundial en Oncología Comparativa (2002).
3. Briscoe, KA, Krockenberger, M, Beatty, JA, Crowley, A, Dennis, MM, Canfield, PJ, et al. Evaluación histopatológica e inmunohistoquímica de 53 casos de enteritis linfoplasmocítica felina y linfoma alimentario de bajo grado. J Comp Pathol. (2011) 145:187–98. doi: 10.1016/j.jcpa.2010.12.011
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Kiupel, M, Smedley, RC, Pfent, C, Xie, Y, Xue, Y, Wise, AG, et al. Algoritmo diagnóstico para diferenciar el linfoma de la inflamación en muestras de biopsia de intestino delgado felino. Veterinario Pathol. (2011) 48:212–22. doi: 10.1177/0300985810389479
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Mooney, SC, Patnaik, AK, Hayes, AA, y MacEwen, EG. Linfadenopatía generalizada parecida al linfoma en gatos: seis casos (1972-1976). J Am Vet Med Assoc. (1987) 190:897–900.
6. Moore, PF, Woo, JC, Vernau, W, Kosten, S y Graham, PS. Caracterización de los genes de la región variable del receptor gamma de células T felinas (TCRG) para el diagnóstico molecular del linfoma intestinal felino de células T. Veterinario Inmunol Inmunopatíaco. (2005) 106:167–78. doi: 10.1016/j.vetimm.2005.02.014
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Keller, SM, Vernau, W, y Moore, PF. Pruebas de clonalidad en medicina veterinaria: una revisión con pautas diagnósticas. Veterinario Pathol. (2016) 53:711–25. doi: 10.1177/0300985815626576
8. Langerak, AW, Groenen, PJ, Brüggemann, M, Beldjord, K, Bellan, C, Bonello, L, et al. Directrices de EuroClonality/BIOMED-2 para la interpretación y notificación de las pruebas de clonalidad de Ig/TCR en sospechas de linfoproliferación. Leucemia. (2012) 26:2159–71. doi: 10.1038/leu.2012.246
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Tonegawa, S. Generación somática de diversidad de anticuerpos. Naturaleza. (1983) 302:575–81. doi: 10.1038/302575a0
10. Rout, ED, Burnett, RC, Yoshimoto, JA, Avery, PR y Avery, AC. Evaluación de las pruebas de clonalidad de la cadena pesada de inmunoglobulinas, cadenas ligeras de inmunoglobulinas y receptores de células T en el diagnóstico de la neoplasia linfoide felina. Veterinario Clin Pathol. (2019) 48:45–58. doi: 10.1111/vcp.12767
11. Keller, SM y Moore, PF. Patrones de reordenamiento del locus TCRγ canino en un grupo distinto de linfomas de células T. Veterinario Inmunol Inmunopatíaco. (2012) 145:350–61. doi: 10.1016/j.vetimm.2011.12.008
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Krangel, MS. Mecánica del reordenamiento génico del receptor de células T. Curr Opin Immunol. (2009) 21:133–9. doi: 10.1016/j.coi.2009.03.009
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Alt, FW, Enea, V, Bothwell, AL, y Baltimore, D. Actividad de múltiples genes de cadena ligera en células de mieloma murino que producen una sola cadena ligera funcional. Célula. (1980) 21:1–12. doi: 10.1016/0092-8674(80)90109-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Siminovitch, KA, Bakhshi, A, Goldman, P, y Korsmeyer, SJ. Un elemento de eliminación uniforme media la pérdida de genes kappa en las células B humanas. Naturaleza. (1985) 316:260–2. doi: 10.1038/316260a0
15. Kabat, EA, Wu, TT y Bilofsky, H. Evidencia que respalda el ensamblaje somático de los segmentos de ADN (minigenes), la codificación del marco y los segmentos determinantes de la complementariedad de las regiones variables de inmunoglobulina. J Exp Med. (1979) 149:1299–313. doi: 10.1084/jem.149.6.1299
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Weiss, AT, Klopfleisch, R, y Gruber, AD. Los genes variables de cadena γ del receptor de células T y de la región de unión del subgrupo 1 se reordenan clonalmente en el linfoma felino de células B y T. J Comp Pathol. (2011) 144:123–34. doi: 10.1016/j.jcpa.2010.08.001
17. Mochizuki, H, Nakamura, K, Sato, H, Goto-Koshino, Y, Sato, M, Takahashi, M, et al. Análisis GeneScan para detectar la clonalidad del reordenamiento del gen gamma del receptor de células T en neoplasias linfoides felinas. Veterinario Inmunol Inmunopatíaco. (2012) 145:402–9. doi: 10.1016/j.vetimm.2011.12.015
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Mochizuki, H, Nakamura, K, Sato, H, Goto-Koshino, Y, Sato, M, Takahashi, M, et al. Análisis de PCR múltiple y Genescan para detectar el reordenamiento de genes de cadena pesada de inmunoglobulinas en neoplasias de células B felinas. Veterinario Inmunol Inmunopatíaco. (2011) 143:38–45. doi: 10.1016/j.vetimm.2011.05.030
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Henrich, M, Hecht, W, Weiss, AT, y Reinacher, M. Un nuevo subgrupo de genes de región variable de cadena pesada de inmunoglobulinas para la evaluación de la clonalidad en linfomas felinos de células B. Veterinario Inmunol Inmunopatíaco. (2009) 130:59–69. doi: 10.1016/j.vetimm.2009.01.006
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Werner, JA, Woo, JC, Vernau, W, Graham, PS, Grahn, RA, Lyons, LA, et al. Caracterización de genes de región variable de cadena pesada de inmunoglobulinas felinas para el diagnóstico molecular de neoplasias de células B. Veterinario Pathol. (2005) 42:596–607. doi: 10.1354/vp.42-5-596
21. Radtanakatikanon, A, Moore, PF, Keller, SM y Vernau, W. Nuevos ensayos de clonalidad para el linfoma de células T en gatos dirigidos al receptor de células T beta, el receptor de células T delta y los loci gamma del receptor de células T. J Veterinario Pasante Med. (2021) 35:2865–75. doi: 10.1111/jvim.16288
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Joachims, ML, Chain, JL, Hooker, SW, Knott-Craig, CJ y Thompson, LF. Desarrollo de timocitos alfa beta y gamma delta humanos: reordenamientos del gen TCR, expresión intracelular de TCR beta y potencial de desarrollo gamma delta: diferencias entre hombres y ratones. J Immunol. (2006) 176:1543–52. doi: 10.4049/jimmunol.176.3.1543
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Thompson, SD, Manzo, AR, Pelkonen, J, Larche, M, y Hurwitz, JL. Reordenamientos de genes del receptor de linfocitos T en desarrollo: relación del precursor de linfocitos T alfa/beta y gamma/delta. Eur J Immunol. (1991) 21:1939–50. doi: 10.1002/eji.1830210824
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Radtanakatikanon, A, Keller, SM, Darzentas, N, Moore, PF, Folch, G, Nguefack Ngoune, V, et al. Topología y repertorio expresado de los loci receptores de células T de Felis catus. BMC Genómica. (2020) 21:20. doi: 10.1186/s12864-019-6431-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Lu, Z, Tallmadge, RL, Callaway, HM, Felippe, MJ y Parker, JS. Análisis de secuencia de ARNm de inmunoglobulinas felinas y desarrollo de un anticuerpo monoclonal felinizado específico para el virus de la panleucopenia felina. Sci Rep. (2017) 7:12713. doi: 10.1038/s41598-017-12725-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Salón, TA. BioEdit: un editor de alineación de secuencias biológicas fácil de usar y un programa de análisis para Windows 95/98/NT. (1999) 41:95–8.
27. Weiss, AT, Hecht, W y Reinacher, M. Secuencias de la región V y J del receptor gamma de células T felinas recuperadas del archivo de trazas y del análisis del transcriptoma de gatos. Vet Med Int. (2010) 2010:953272. doi: 10.4061/2010/953272
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Jackson, ML, Wood, SL, Misra, V y Haines, DM. Identificación inmunohistoquímica de linfocitos B y T en linfosarcomas felinos fijados en formol e incluidos en parafina: relación con el estado del virus de la leucemia felina, el sitio del tumor y la edad del paciente. Can J Vet Res. (1996) 60:199–204.
29. Monteith, CE, Chelack, BJ, Davis, WC y Haines, DM. Identificación de anticuerpos monoclonales para la tinción inmunohistoquímica de linfocitos B felinos en tejidos congelados y fijados en formol incluidos en parafina. Can J Vet Res. (1996) 60:193–8.
30. Treggiari, E, Ressel, L, Polton, GA, Benoit, J, Desmas, I y Blackwood, L. Resultado clínico, expresión de PDGFRβ y KIT en trastornos histiocíticos felinos: un estudio multicéntrico. Veterinario Comp Oncol. (2017) 15:65–77. doi: 10.1111/vco.12142
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Willett, BJ, Cannon, CA, y Hosie, MJ. Expresión de CXCR4 en células mononucleares de sangre periférica felina: efecto de la infección por el virus de la inmunodeficiencia felina. J Virol. (2003) 77:709–12. doi: 10.1128/jvi.77.1.709-712.2003
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Kipar, A, Bellmann, S, Kremendahl, J, Köhler, K, y Reinacher, M. Composición celular, expresión de antígenos de coronavirus y producción de anticuerpos específicos en lesiones en peritonitis infecciosa felina. Veterinario Inmunol Inmunopatíaco. (1998) 65:243–57. doi: 10.1016/s0165-2427(98)00158-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Rydzewski, L, Scheffold, S, Hecht, W, Burkhardt, E, Kerner, K, Klymiuk, MC, et al. Identificación de una nueva línea celular de linfoma granular felino (S87) como línea de células T citotóxicas no restringidas por MHC y evaluación de su inestabilidad genética. Veterinario Inmunol Inmunopatíaco. (2016) 177:24–34. doi: 10.1016/j.vetimm.2016.05.012
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Miller, S.A., Dykes, DD, y Polesky, HF. Un procedimiento simple de salado para extraer ADN de células nucleadas humanas. Ácidos nucleicos Res. (1988) 16:1215. doi: 10.1093/nar/16.3.1215
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Meier, VS, Rufle, A y Gudat, F. Evaluación simultánea de la clonalidad de las células T y B, t (11,14) y t (14,18), en una sola reacción mediante un ensayo de reacción en cadena de polimerasa multiplex de cuatro colores y análisis automatizado de fragmentos de alta resolución. Am J Pathol. (2001) 159:2031–43. doi: 10.1016/s0002-9440(10)63055-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Barrs, VR, y Beatty, JA. Linfoma alimentario felino: 2. Diagnóstico, tratamiento y pronóstico adicionales. J Feline Med Surg. (2012) 14:191–201. doi: 10.1177/1098612X12439266
37. Steiniger, SC, Glanville, J, Harris, DW, Wilson, TL, Ippolito, GC, y Dunham, SA. Análisis comparativo del repertorio de inmunoglobulinas felinas. Biológicos. (2017) 46:81–7. doi: 10.1016/j.biologicals.2017.01.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Lenze, D, Müller, H-H y Hummel, M. Consideraciones para el uso de muestras de tejido fijadas en formol e incluidas en parafina para el análisis de clonalidad. J Hematop. (2012) 5:27–34. doi: 10.1007/s12308-012-0138-8
39. Mayordomo, JM. Temas avanzados en tipificación forense de ADN: interpretación. Burlington, MA: Elsevier Academic Press (2014). 609 págs.
40. Rothberg, PG, Langerak, AW, Verhaaf, B, van Dongen, JJ, Burack, WR, Johnson, MD, et al. Productos de PCR del gen del receptor de antígeno clonal fuera del rango de tamaño esperado. J Hematop. (2012) 5:57–67. doi: 10.1007/s12308-011-0135-3
41. Wang, H-W y Raffeld, M. Evaluación molecular de la clonalidad en neoplasias linfoides. Semin Hematol. (2019) 56:37–45. doi: 10.1053/j.seminhematol.2018.05.008
42. Breit, TM, Wolvers-Tettero, IL y van Dongen, JJ. Determinante de selección único en las regiones de unión policlonal V delta 2-J delta 1 de los linfocitos T gamma delta periféricos humanos. J Immunol. (1994) 152:2860–4. doi: 10.4049/jimmunol.152.6.2860
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Delfau, MH, Hance, AJ, Lecossier, D, Vilmer, E y Grandchamp, B. Diversidad restringida de reordenamientos de V gamma 9-JP en linfocitos T humanos gamma/delta no estimulados. Eur J Immunol. (1992) 22:2437–43. doi: 10.1002/eji.1830220937
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Ehrhart, EJ, Wong, S, Richter, K, Zismann, V, Grimes, C, Hendricks, W, et al. Reacción en cadena de la polimerasa para el reordenamiento del receptor de antígenos: evaluación comparativa del rendimiento de un ensayo de clonalidad linfoide en diversos tipos de muestras caninas. J Veterinario Pasante Med. (2019) 33:1392–402. doi: 10.1111/jvim.15485
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. van Dongen, JJ, Langerak, AW, Brüggemann, M, Evans, PA, Hummel, M, Lavender, FL, et al. Diseño y estandarización de cebadores y protocolos de PCR para la detección de inmunoglobulinas clonales y recombinaciones génicas de receptores de células T en sospechas de linfoproliferaciones: informe de la acción concertada de BIOMED-2 BMH4-CT98-3936. Leucemia. (2003) 17:2257–317. doi: 10.1038/sj.leu.2403202
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: PCR, reordenamientos de receptores de antígenos, clonalidad, Felis catus, linfoma, gato, PARR
Cita: Weyrich A, Hecht W, Köhler K, Herden C y Henrich M (2024) Análisis comparativo de conjuntos de cebadores para la evaluación de la clonalidad en linfomas felinos. Frente. Vet. Sci. 11:1356330. doi: 10.3389/fvets.2024.1356330
Recibido: 15 de diciembre de 2023; Aceptado: 22 de abril de 2024;
Publicado: 07 Mayo 2024.
Editado por:
Irina Amorim, Universidad de Oporto, Portugal
Revisado por:
Aleksandra Pawlak, Universidad de Ciencias Ambientales y de la Vida de Wrocław, Polonia
Sabine E. Hammer, Universidad de Medicina Veterinaria de Viena, Austria
Derechos de autor © 2024 Weyrich, Hecht, Köhler, Herden y Henrich. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Manfred Henrich, Manfred.henrich@vetmed.uni-giessen.de
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía