Análisis transcriptómico y metabolómico exhaustivo de las células epiteliales intestinales porcinas después de la infección por PDCoV
Análisis transcriptómico y metabolómico exhaustivo de las células epiteliales intestinales porcinas después de la infección por PDCoV
 Guangzheng Wang1
Guangzheng Wang1  Yanan Cao1
Yanan Cao1  Chao Xu1
Chao Xu1  Shuoshuo Zhang1
Shuoshuo Zhang1  Yanjie Huang1
Yanjie Huang1  Shuai Zhang1*
Shuai Zhang1*  Wenbin Bao1,2*
Wenbin Bao1,2*- 1Laboratorio Clave de Genética Animal, Cría, Reproducción y Diseño Molecular, Facultad de Ciencia y Tecnología Animal, Universidad de Yangzhou, Yangzhou, China
- 2Laboratorio Internacional Conjunto de Investigación de Agricultura e Inocuidad de Productos Agropecuarios, Universidad de Yangzhou, Yangzhou (China)
Introducción: El deltacoronavirus porcino (PDCoV), un coronavirus enteropatógeno porcino emergente con distribución mundial, infecta principalmente a los lechones recién nacidos con diarrea severa, vómitos, deshidratación e incluso la muerte, causando enormes pérdidas económicas a la industria porcina. Sin embargo, los mecanismos patogénicos subyacentes de la infección por PDCoV y los efectos de la infección por PDCoV en los transcritos y metabolitos del huésped siguen sin comprenderse completamente.
Métodos: Este estudio investigó un análisis transcriptómico y metabolómico combinado de células epiteliales intestinales porcinas (IPEC-J2) después de la infección por PDCoV mediante técnicas de LC/MS y RNA-seq. Se detectaron un total de 1.401 genes expresados diferencialmente y 254 metabolitos acumulados diferencialmente en el grupo de comparación de infectados por PDCoV frente a infectados simulados.
Resultados y discusión: Descubrimos que la infección por PDCoV regula los conjuntos de genes asociados con múltiples vías de señalización, incluida la interacción neuroactiva ligando-receptor, la interacción citocina-receptor de citocinas, la vía de señalización MAPK, la vía de señalización de quimiocinas, la vía de señalización ras, etc. Además, los resultados metabolómicos mostraron que la biosíntesis de cofactores, el metabolismo de nucleótidos, la digestión y absorción de proteínas y la biosíntesis de aminoácidos estaban involucrados en la infección por PDCoV. Además, los análisis transcriptómicos y metabolómicos integrados revelaron la participación de la ferroptosis en la infección por PDCoV, y la adición exógena del activador de la ferroptosis erastina inhibió significativamente la replicación de PDCoV. En general, estas características únicas de reprogramación transcripcional y metabólica pueden proporcionar una mejor comprensión de las células IPEC-J2 infectadas con PDCoV y de las posibles dianas para el tratamiento antiviral.
1 Introducción
El deltacoronavirus porcino (PDCoV) es un coronavirus enteropatógeno porcino emergente. El primer PDCoV notificado se describió en hisopos rectales porcinos de Hong Kong en 2012 (1), y el primer brote de PDCoV en lechones y cerdas se anunció en Ohio, EE. UU. en 2014, seguido de una rápida propagación a otros estados vecinos de Ohio con una tasa de mortalidad de lechones de hasta el 30 % al 40 % (2). Desde entonces, se ha informado de la aparición de PDCoV en muchos países, entre ellos China, Canadá, Japón, Corea, Tailandia, República Democrática Popular Lao y Vietnam (3-6). El genoma de PDCoV, de aproximadamente 25,4 kb, codifica 15 proteínas no estructurales maduras (nsp2-16), cuatro proteínas estructurales (proteína S, proteína E, proteína M y proteína N) y tres proteínas accesorias (NS6, NS7 y NS7a) (7, 8). La infección por PDCoV causa principalmente enfermedad intestinal en lechones recién nacidos caracterizada por diarrea aguda, vómitos y deshidratación (9-11). Además, estudios recientes han reportado que los terneros, pavos y pollos también son susceptibles a PDCoV (12, 13). Es importante destacar que el PDCoV tiene el potencial de infectar a los seres humanos, lo que representa una amenaza significativa para la salud humana y animal (14). Por lo tanto, el estudio del mecanismo de infección de PDCoV es de gran importancia para prevenir la epidemia de PDCoV.
Los virus dependen de los mecanismos de la célula huésped para reproducirse, generar macromoléculas necesarias para su propia replicación, como aminoácidos o nucleótidos. Los virus se ensamblan alterando el ciclo celular del huésped y promoviendo el anabolismo del huésped. La infección por PDCoV causa cambios en el transcriptoma de las células huésped y activa múltiples vías asociadas con la inmunidad innata de la célula huésped (15). La infección por PDCoV obstaculizó drásticamente la expresión del interferón tipo III (IFN-λ) (16), y la infección por PDCoV también inhibió la producción de IFN-β mediada por RIG-I (17). Las múltiples proteínas codificadas por PDCoV secuestran el sistema inmunitario innato del huésped para beneficiarse de la replicación. Además, aunque se cree que la apoptosis es parte del sistema inmune innato del huésped, varios virus promueven la apoptosis para facilitar la liberación y diseminación de la progenie viral (18, 19). La apoptosis inducida por PDCoV se acompañó con la activación de cascadas de caspasas y promovió una replicación viral efectiva in vitro (20). La infección por PDCoV induce apoptosis dependiente de caspasas a través de la vía mitocondrial intrínseca (20). Por otro lado, los virus podrían regular la ferroptosis, una forma de muerte celular dependiente del hierro caracterizada por la peroxidación lipídica acumulada. Se ha reportado que la ferroptosis está involucrada en procesos de infección por virus divergentes (21). Se ha confirmado que la activación de la ferroptosis inhibe la replicación del PEDV (22). Por el contrario, la infección por el virus Infuenza A (IAV) provoca una generación excesiva de ROS en las células A549, lo que provoca estrés oxidativo y peroxidación lipídica, lo que en última instancia induce ferroptosis, lo que mejora la replicación del virus (23). Por lo tanto, la exploración de la muerte celular reguladora inducida por PDCoV es beneficiosa para investigar más a fondo los mecanismos de la infección por PDCoV.
En este estudio, escaneamos los genes y metabolitos expresados diferencialmente en células IPEC-J2 infectadas con PDCoV a las 24 h utilizando enfoques transcriptómicos y metabolómicos. El análisis transcriptómico y metabolómico combinado mostró que múltiples vías de señalización están involucradas en la infección por PDCoV, como la vía de señalización PI3K/AKT y la ferroptosis. Cabe destacar que la ferroptosis inducida por erastina impidió la replicación de PDCoV. En general, la infección por PDCoV altera la transcripción y el metabolismo de las células huésped para promover su replicación.
2 Materiales y métodos
2.1 Cultivo celular
Las células IPEC-J2 utilizadas en este estudio se mantuvieron en nuestro laboratorio. Las células IPEC-J2 se cultivaron en medio DMEM suplementado con 10% de suero fetal bovino (FBS, GIBCO, Australia) y 1% de Pen-Strep [penicilina (100 U/ml), estreptomicina (0,1 mg/ml); Solarbio, Pekín, China]. Las células se cultivaron a 37 °C en una incubadora humidificada ajustada al 5% de CO2.
2.2 Infección por PDCoV
Las células IPEC-J2 se sembraron en placas de cultivo celular de 100 mm y se cultivaron durante la noche en una incubadora a 37 °C con 5% de CO2. Se utilizó PDCoV a una multiplicidad de infección (MOI) para la adsorción en monocapa de células IPEC-J2 durante 1 h a 37 °C con 5% de CO2, luego se retiró el inóculo y se lavó tres veces con solución salina tamponada con fosfato (PBS), posteriormente se agregó DMEM suplementado con FBS al 2% y pancreatina al 1%, y se continuó la incubación a 37 °C con CO al 5%2 durante 24 h.
2.3 Recogida de muestras
Las células se recolectaron y se lavaron con PBS helado tres veces. Para el análisis metabolómico, se añadieron 500 μl de metanol-agua helada (4:1, V/V, MACKLIN, Shanghái, China) para raspar las células, luego se transfirieron las células recogidas a un tubo de centrífuga de 1,5 ml, y se añadieron otros 500 μl de metanol-agua helada (4:1, V/V) y se almacenaron a -80 °C; Para el análisis transcriptómico, se recolectaron células y el ARN total se extrajo mediante RNAiso Plus (TaKaRa, Dalian, China) de acuerdo con las instrucciones del fabricante y el ARN extraído se almacenó a -80 ° C.
2.4 Secuenciación y análisis del transcriptoma
La secuenciación del transcriptoma y el análisis de la expresión diferencial fueron proporcionados por Novogene (Beijing, China). Los genes con ajuste de P ≤ 0.05 y |log2FC| ≥ 1 se consideraron como genes de expresión diferencial (DEGs). La clasificación de funciones y el análisis de enriquecimiento de los DEG se realizaron utilizando el software GOseq basado en la base de datos GO (http://geneontology.org/page/go-enrichment-analysis). Sobre la base de las anotaciones de los DEG, las transcripciones se clasificaron en tres categorías, incluyendo el proceso biológico (BP), el componente celular (CC) y la función molecular (MF). El enriquecimiento de la vía se analizó con base en la base de datos KEGG (https://www.kegg.jp/kegg/kegg1.html) y se llevó a cabo utilizando el servidor web KOBAS 3.0 (con un P ≤ 0,05) en cada nivel de comparación para diferentes genotipos y tratamientos.
2.5 Análisis de metabolitos
La extracción de metabolitos y el análisis de LC-MS/MS fueron proporcionados por Novogene (Pekín, China). Los metabolitos diferenciales identificados se combinaron mediante el valor VIP del modelo PLS-DA (≥1), el valor P de la prueba t de student (≤0,05) y |log2FC| (≥0,5). Se consideró que los metabolitos elegibles habían cambiado significativamente. Se utilizó la base de datos KEGG para anotar y mostrar los metabolitos diferenciales. Otros análisis incluyeron el análisis discriminante de mínimos cuadrados parciales (PLS-DA) y el enriquecimiento de vías.
2.6 PCR cuantitativa en tiempo real
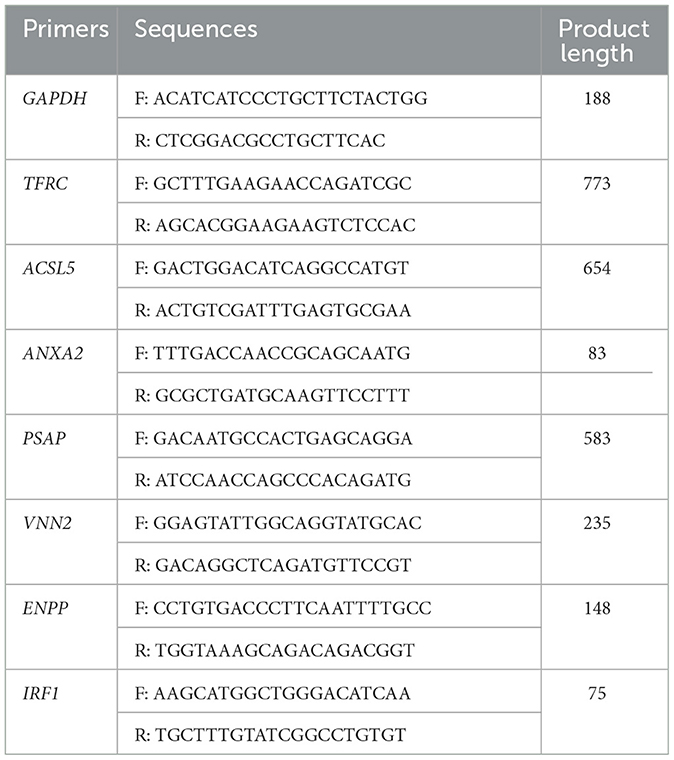
Los niveles de expresión relativa de genes seleccionados se examinaron mediante RT-qPCR utilizando AceQ Universal SYBR qPCR Master Mix (Vazyme, Nanjing, China). Los niveles relativos de expresión génica de los 8 genes se normalizaron a la expresión de GAPDH utilizando los 2−ΔΔCT método. Los cebadores utilizados en la RT-qPCR se enumeraron en la Tabla 1. Las reacciones de RT-qPCR se realizaron en el sistema de PCR en tiempo real StepOnePlus (Thermo Fisher Scientific, Massachusetts, EE. UU.).
2.7 Ensayos de Western blot
Las células se lavaron tres veces con PBS y se lisaron con tampón de lisis celular RIPA, y luego se sometieron cantidades iguales de proteína a electroforesis en gel de poliacrilamida de dodecil sulfato de sodio (SDS-PAGE). Las muestras se transfirieron a membranas de PVDF, y las membranas se bloquearon con leche desnatada en polvo al 5% disuelta en TBST (TBS que contenía 2% de Tween 20) durante 2 h a temperatura ambiente. A continuación, las membranas se incubaron con anticuerpos primarios específicos durante 12 h, se lavaron tres veces con TBST y se incubaron con anticuerpos secundarios durante 2 h. Las bandas fueron fotografiadas utilizando un sistema de imágenes quimioluminiscentes Tanon-5200 (Tanon Science & Technology Co., Ltd.).
2.8 Análisis estadístico
El análisis estadístico se realizó mediante el Programa Estadístico de Ciencias Sociales (SPSS 16.0, SPSS Inc., Chicago, IL, USA). Los datos se presentaron como la media ± la desviación estándar (DE), y las diferencias entre los grupos control y experimental emplearon la prueba t de Student y el análisis de varianza de una vía (ANOVA). Para todos los análisis, los valores de p de < 0,05 se consideraron estadísticamente significativos (P < 0,05; P < 0,01).***
3 Resultados
3.1 La infección por PDCoV induce los cambios en el perfil del transcriptoma en las células IPEC-J2
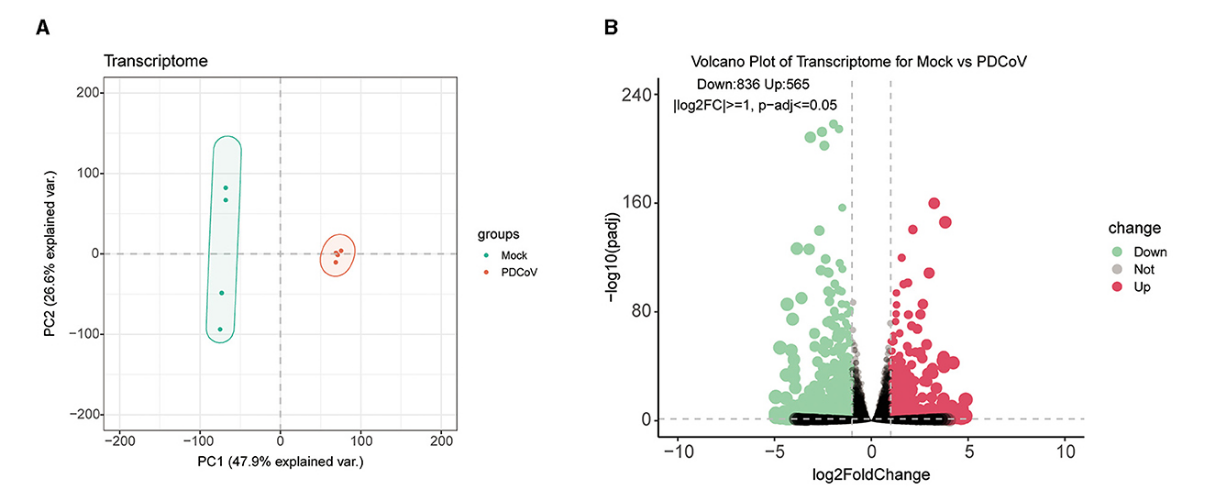
Para determinar los cambios en el transcriptoma en el pico de replicación de PDCoV, se realizó la secuenciación del ARN en células IPEC-J2 infectadas con PDCoV a las 24 h. El análisis de componentes principales (PCA) mostró que las muestras del grupo infectado por PDCoV y del grupo simulado estaban bien separadas (Figura 1A), lo que indica que el patrón de expresión génica de las células infectadas por PDCoV era diferente al de las células infectadas por el simulacro.
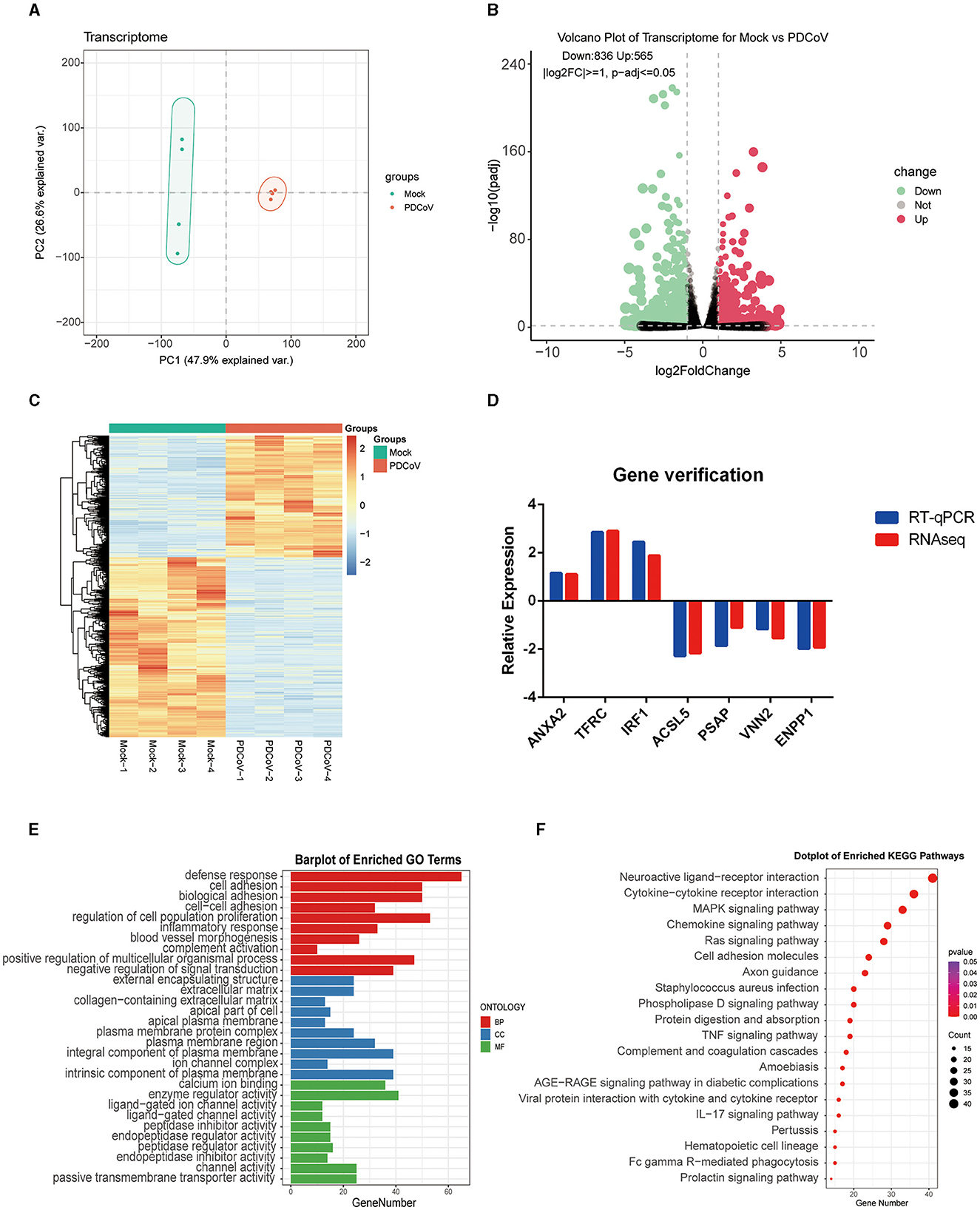 Figura 1. Análisis de los cambios en el transcriptoma en las células IPEC-J2 durante la infección por PDCoV. (A) Análisis de componentes principales (ACP). (B) Diagrama volcánico de genes expresados diferencialmente entre PDCoV y grupo Mock. Cada parcela representa un gen identificado. (C) Mapa de calor de genes expresados diferencialmente. D) Verificación genética. Los niveles de genes expresados diferencialmente se detectaron mediante RT-qPCR. Los niveles de genes seleccionados se normalizaron con GAPDH. (E) Los 10 términos de GO significativamente enriquecidos de cada categoría, incluidos el proceso biológico (BP), el componente celular (CC) y la función molecular (MF). (F) Clasificación de la vía KEGG de genes expresados diferencialmente en células infectadas con PDCoV (MOI = 1).
Figura 1. Análisis de los cambios en el transcriptoma en las células IPEC-J2 durante la infección por PDCoV. (A) Análisis de componentes principales (ACP). (B) Diagrama volcánico de genes expresados diferencialmente entre PDCoV y grupo Mock. Cada parcela representa un gen identificado. (C) Mapa de calor de genes expresados diferencialmente. D) Verificación genética. Los niveles de genes expresados diferencialmente se detectaron mediante RT-qPCR. Los niveles de genes seleccionados se normalizaron con GAPDH. (E) Los 10 términos de GO significativamente enriquecidos de cada categoría, incluidos el proceso biológico (BP), el componente celular (CC) y la función molecular (MF). (F) Clasificación de la vía KEGG de genes expresados diferencialmente en células infectadas con PDCoV (MOI = 1).
Se detectaron un total de 1.401 DEG, de los cuales 565 DEG estaban regulados al alza y 836 DEG estaban regulados a la baja. Todos los DEG identificados fueron mapeados en el mapa del volcán (Figura 1B). Los perfiles de expresión génica con diferencia significativa se presentaron en el mapa de calor (Figura 1C). Para validar aún más la precisión de los datos del transcriptoma, seleccionamos aleatoriamente ANXA2, TFRC y TIRF1 de DEG regulados al alza, PSAP, ACSL5C3, VNN2 y ENPP a partir de DEG regulados a la baja para el análisis de RT-qPCR. De acuerdo con los datos de secuenciación de ARN, los niveles de ARNm de ANXA2, TFRC y TIRF1 aumentaron significativamente y los niveles de ARNm de PSAP, ACSL5C3, VNN2 y ENPP disminuyeron significativamente, lo que indica la confiabilidad de los resultados de secuenciación de ARN (Figura 1D).
Todos los DEG se anotaron en la base de datos Gene Ontology (GO) (http://geneontology.org) y se enriquecieron a 528 vías de señalización. Se seleccionaron las 10 vías principales con el mayor enriquecimiento en cada categoría para su visualización (Figura 1E). El análisis de GO en la PA se relacionó principalmente con la respuesta de defensa, la adhesión celular y la adhesión biológica; El CC se enriqueció principalmente en estructura encapsulante externa, matriz extracelular y matriz extracelular que contiene colágeno; La MF se enriqueció principalmente en la unión de iones de calcio, la actividad reguladora de enzimas y la actividad de los canales iónicos activados por ligandos. Además, se anotaron 1.401 DEG en la base de datos de la Enciclopedia de Genes y Genomas de Kioto (KEGG) (http://www.kegg.jp/kegg/pathway.html), y se asignaron a 68 vías KEGG diferentes. Los diagramas de burbujas de KEGG muestran las 20 principales vías de señalización enriquecidas (Figura 1F), relacionadas con la interacción ligando-receptor neuroactivo, la interacción receptor citoquina-citocina, la vía de señalización MAPK, la vía de señalización de quimiocinas y la vía de señalización ras.
3.2 Cambios metabolómicos en células IPEC-J2 infectadas con PDCoV
Para investigar más a fondo los diferentes metabolitos implicados en las respuestas celulares a la infección por PDCoV, se llevaron a cabo cambios metabolómicos en las células IPEC-J2 infectadas con PDCoV por LC/MS. Los grupos infectados con PDCoV y los simulados se distinguieron fácilmente, y el modelo PLS-DA fue confiable con una gran brecha y un buen agrupamiento intragrupo dentro de los grupos, lo que indica que la infección por PDCoV causó los cambios en los metabolitos (Figura 2A). Todos los metabolitos identificados se trazaron como mapas volcánicos (Figura 2B), y se identificaron 254 metabolitos diferenciales en el grupo infectado por PDCoV en comparación con el grupo simulado, en el que los puntos rojos representaban 112 metabolitos regulados al alza y los puntos azules representaban 142 metabolitos regulados a la baja. Los 10 metabolitos más regulados al alza tras la infección por PDCoV fueron metionina, pidotimod, PG (22:5/22:5), L-valina, DL-α-ácido aminocaprílico, 5-aminopentanoato, cuminaldehído, ácido N-acetil-aspártico, L-(+)-citrulina, ácido N-acetilaspártico. Mientras tanto, los 10 principales metabolitos regulados a la baja fueron LPG 20:1, N1-metil-5-metoxi-2-({2-[(metilamino)carbonil]fenil}tio) benzamida, prostaglandina H2, penicilina G, DL-carnitina, 6-metil-4-(morfolinometil)-2H-cromeno-2-ona, N1-pirazina-2-il-4clorobenzamida, L-glutatión (reducido), D-manosa 6-fosfato, DL-arginina. Además, se mostró el agrupamiento jerárquico para mostrar los patrones de expresión de los metabolitos diferenciales entre y dentro del PDCoV y el grupo simulado (Figura 2C).
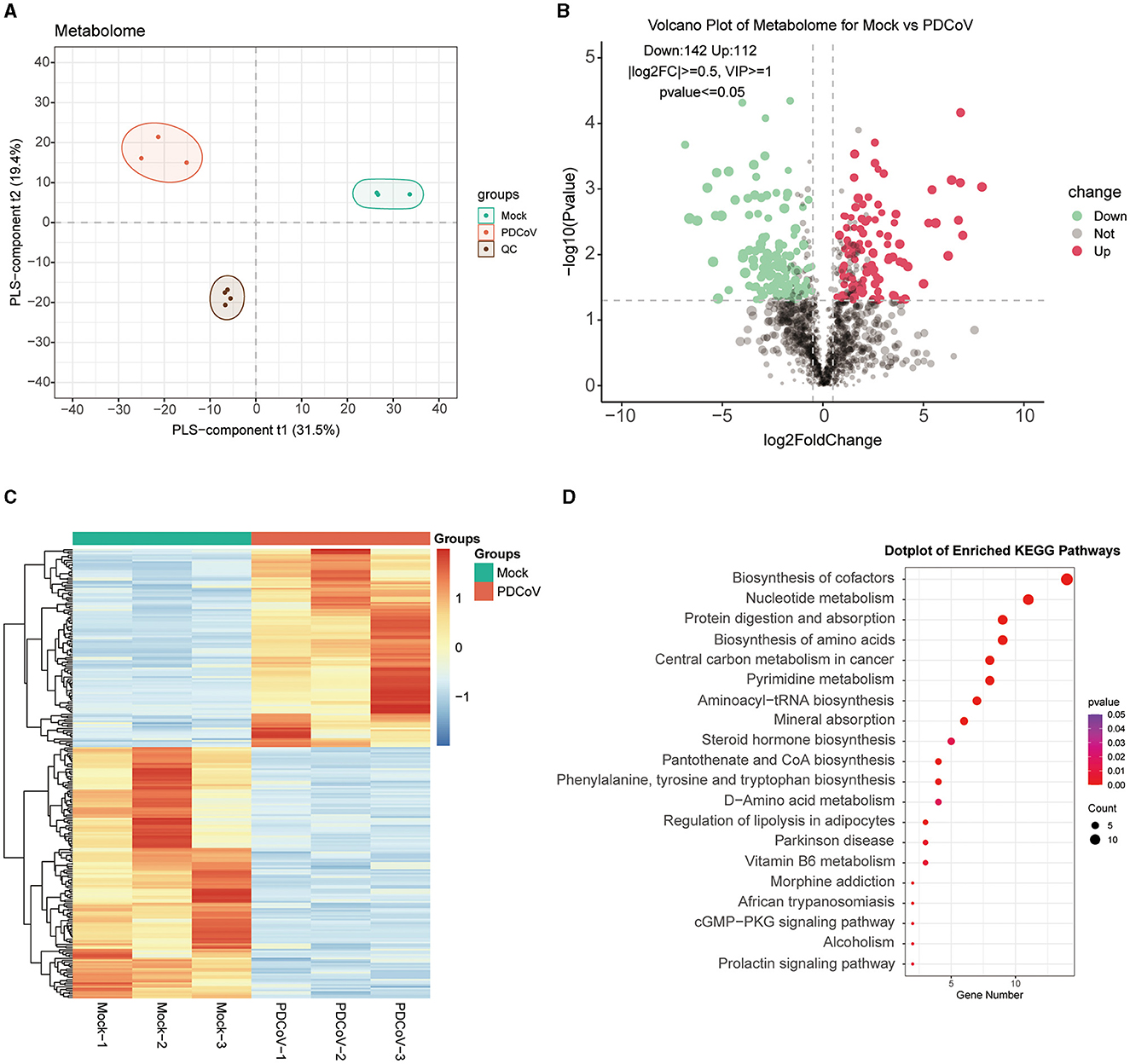 Figura 2. Identificación diferencial de metabolitos y análisis de enriquecimiento de vías. (A) Modelo PLS-DA. (B) Diagrama volcánico de metabolitos expresados diferencialmente entre PDCoV y grupo simulado. Cada parcela representa un metabolito identificado. (C) Análisis de mapas de calor de metabolitos significativamente modificados entre PDCoV y el grupo simulado. (D) Clasificación de la vía KEGG de metabolitos expresados diferencialmente tras la infección por PDCoV (MOI = 1).
Figura 2. Identificación diferencial de metabolitos y análisis de enriquecimiento de vías. (A) Modelo PLS-DA. (B) Diagrama volcánico de metabolitos expresados diferencialmente entre PDCoV y grupo simulado. Cada parcela representa un metabolito identificado. (C) Análisis de mapas de calor de metabolitos significativamente modificados entre PDCoV y el grupo simulado. (D) Clasificación de la vía KEGG de metabolitos expresados diferencialmente tras la infección por PDCoV (MOI = 1).
Posteriormente, los metabolitos expresados diferenciales se enriquecieron en base a la base de datos KEGG (http://www.kegg.jp/kegg/pathway.html), el mapa de burbujas enumera las 20 vías KEGG más enriquecidas (Figura 2D), que contenían específicamente Biosíntesis de cofactores, Metabolismo de nucleótidos, Digestión y absorción de proteínas, Biosíntesis de aminoácidos y Metabolismo central del carbono en el cáncer. En particular, la expresión de varios aminoácidos cambió notablemente. Una regulación positiva significativa de la L-glutamina y una regulación negativa significativa de la gamma-glutamilcisteína indicaron que la infección por PDCoV dio lugar a la acumulación de glutamina. Además, también encontramos que la infección por PDCoV alteró el metabolismo de nucleótidos de las células huésped, con cambios significativos en la expresión de varios nucleótidos esenciales para la proliferación celular y la replicación viral. Los nucleótidos pueden ser consumidos por PDCoV para su propia replicación. Además, la expresión del ácido 5′-adenilico se reguló significativamente a la baja, lo que también sugirió el consumo de nucleótidos de las células huésped tras la infección por PDCoV. En general, estos resultados sugirieron que PDCoV utiliza numerosos componentes del huésped como aminoácidos y nucleótidos para completar su ciclo de vida.
3.3 Análisis integrado de los datos del transcriptoma y del metaboloma
Para revelar las moléculas y las vías de señalización asociadas con la respuesta a la infección por PDCoV, combinamos el análisis del transcriptoma y el metaboloma entre los grupos simulado y PDCoV. Los DEG y los metabolitos acumulados diferencialmente (DAM) causados por la infección por PDCoV se mapearon en el mapa de vías KEGG. Los DEG y los DAM se enriquecieron en la vía de señalización PI3K-Akt (P < 0,01) y en la vía de señalización de la ferroptosis (P < 0,01; Figura 3). En concreto, se asignaron 32 genes clave y un metabolito (ácido 5′-adenilico) a la vía de señalización PI3K/Akt. Además, siete genes clave expresados diferencialmente (ACSL5, TFRC, SLC39A8, LPCAT3, SLC7A11, SAT2 y CP) y dos DAM (ácido mevalónico, gamma-glutamilcisteína) participaron en la vía de señalización de la ferroptosis (Figura 4).
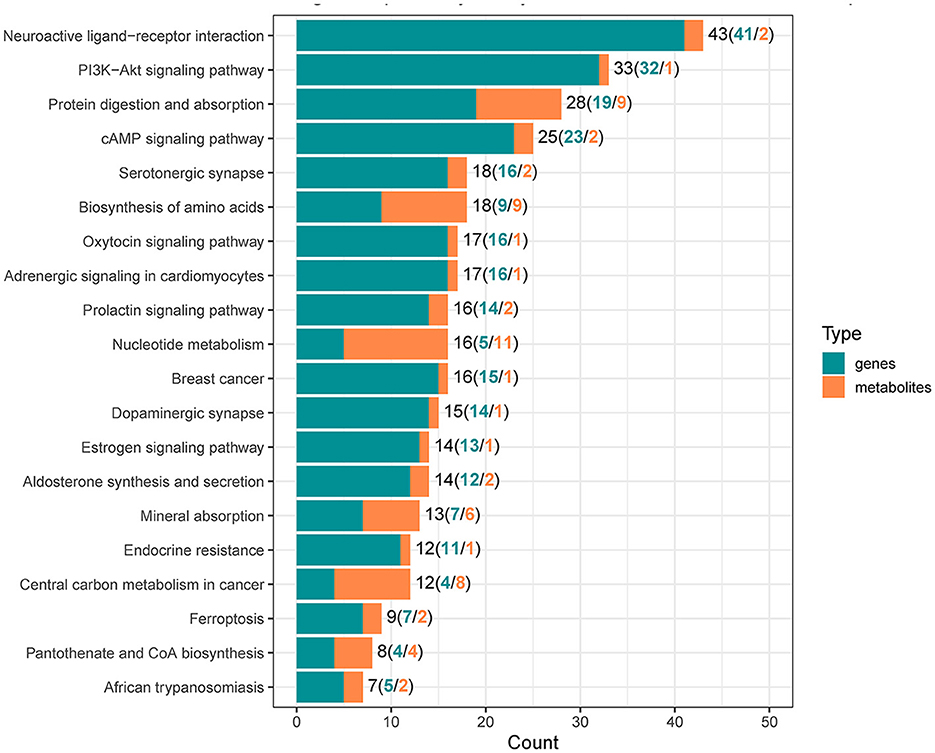 Figura 3. Análisis integrado de los genes expresados diferencialmente y de los metabolitos acumulados diferencialmente.
Figura 3. Análisis integrado de los genes expresados diferencialmente y de los metabolitos acumulados diferencialmente.
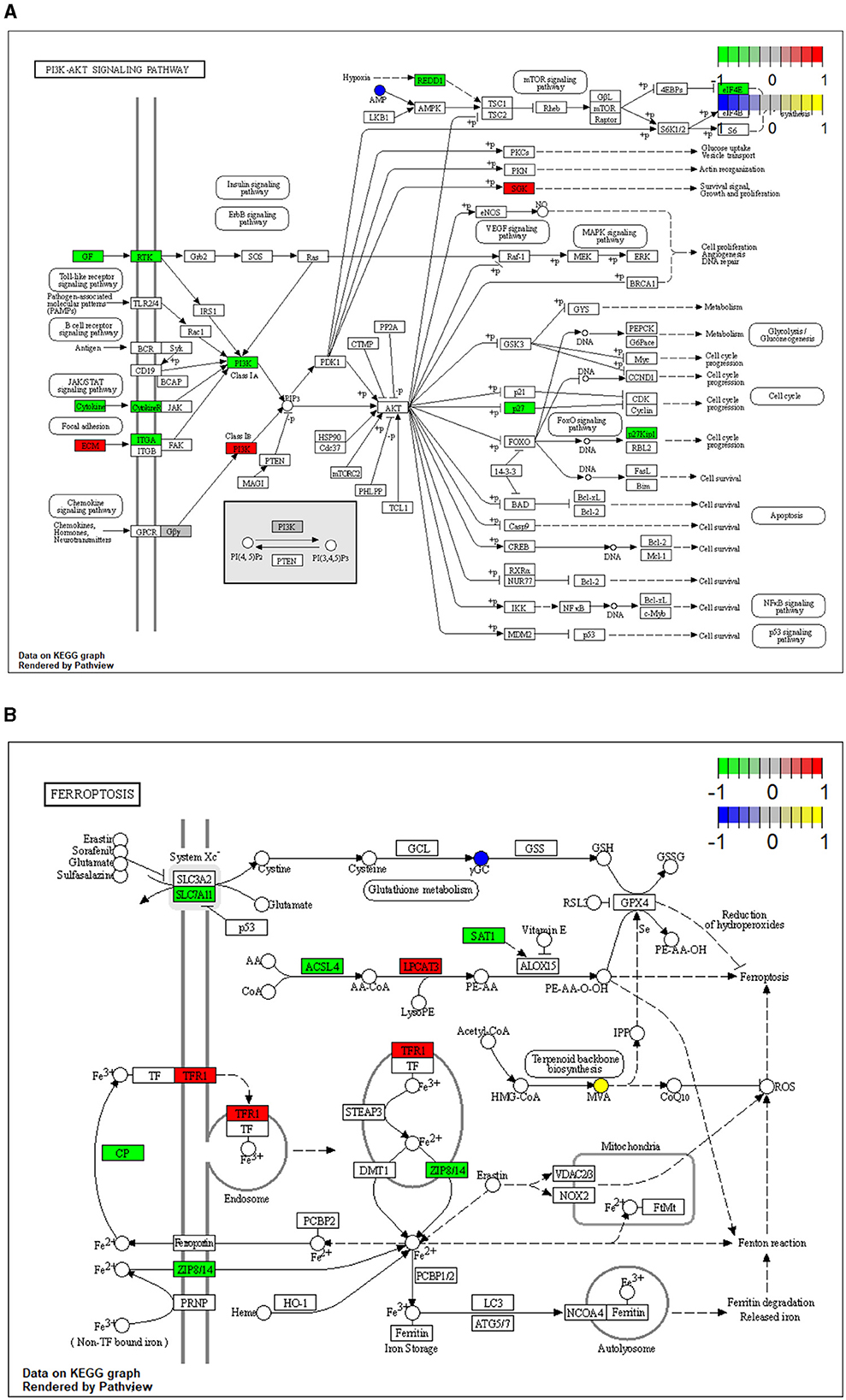 Figura 4. Mapeo de genes expresados diferencialmente (DEGs) y metabolitos diferencialmente acumulados (DAMs) a las vías KEGG. (A) Vía de señalización PI3K/Akt (ssc04151). (B) Ferroptosis (ssc04216). Los cuadrados representan los genes y los círculos representan los metabolitos. El verde indica la regulación negativa de los genes, el rojo indica la regulación positiva de los genes, el azul representa la regulación negativa de los metabolitos y el amarillo representa la regulación positiva de los metabolitos.
Figura 4. Mapeo de genes expresados diferencialmente (DEGs) y metabolitos diferencialmente acumulados (DAMs) a las vías KEGG. (A) Vía de señalización PI3K/Akt (ssc04151). (B) Ferroptosis (ssc04216). Los cuadrados representan los genes y los círculos representan los metabolitos. El verde indica la regulación negativa de los genes, el rojo indica la regulación positiva de los genes, el azul representa la regulación negativa de los metabolitos y el amarillo representa la regulación positiva de los metabolitos.
3.4 El activador de la vía de señalización de la ferroptosis inhibe la infección por PDCoV
La ferroptosis es una importante forma celular no apoptótica de muerte celular regulada que desempeña un papel en varias infecciones virales (23, 24). Para explorar más a fondo el papel específico que desempeña la vía de la ferroptosis en la infección por PDCoV, utilizamos erastina, un activador clásico de la ferroptosis (22), para pretratarlo con células IPEC-J2, y luego infectarlo con PDCoV a un MOI de 1. Los resultados de la RT-qPCR (Figuras 5A, B) y de la Western blot (Figura 5C) indicaron que el tratamiento con erastina dificultó la transcripción de genes virales y la expresión de proteínas virales.
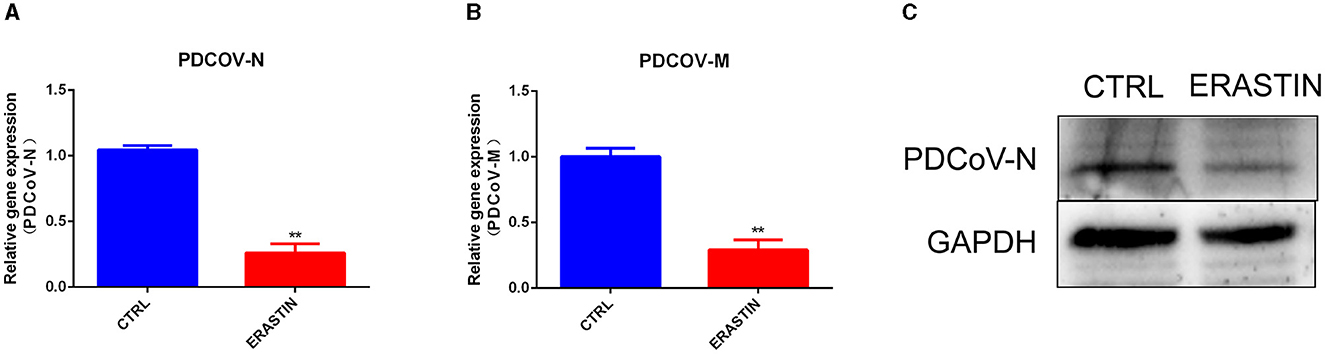 Figura 5. La erastina inhibe la replicación de PDCoV en células IPEC-J2. El nivel de ARNm de PDCoV-N (A) y el nivel de ARNm de PDCoV-M (B) se detectaron mediante RT-qPCR (MOI = 1). (C) El nivel de la proteína PDCoV-N se detectó mediante Western blot (MOI = 1). **P < 0,01.
Figura 5. La erastina inhibe la replicación de PDCoV en células IPEC-J2. El nivel de ARNm de PDCoV-N (A) y el nivel de ARNm de PDCoV-M (B) se detectaron mediante RT-qPCR (MOI = 1). (C) El nivel de la proteína PDCoV-N se detectó mediante Western blot (MOI = 1). **P < 0,01.
4 Discusión
Desde que el PDCoV se identificó por primera vez en 2012 y provocó un brote de diarrea porcina en los EE. UU. en 2014, se había convertido rápidamente en una nueva amenaza para la industria porcina. Hasta la fecha, la transcriptómica se ha utilizado ampliamente para desentrañar los mecanismos de la infección viral y para encontrar los genes clave y las vías de señalización implicadas en la infección viral (25). Además, la metabolómica también se ha utilizado para investigar los efectos de la infección viral en el metabolismo del huésped y para detectar posibles dianas terapéuticas en las redes metabólicas (26). En este estudio, investigamos los efectos de la infección por PDCoV en las transcripciones y metabolitos del huésped a partir de un análisis transcriptómico y metabolómico combinado. A nivel metabólico, la infección por PDCoV alteró principalmente el metabolismo de nucleótidos, proteínas y aminoácidos. A nivel transcripcional, encontramos que la infección por PDCoV activó la vía de señalización PI3K/Akt y la vía de señalización de la ferroptosis, alteró múltiples genes relacionados, incluidos TFRC, ACSL5, SLC39A8, LPCAT3, SLC7A11, SAT2 y CP, y la activación de la vía de señalización de la ferroptosis inhibió significativamente la replicación de PDCoV.
Los aminoácidos son las sustancias más importantes de los organismos vivos. No solo se utilizan en la síntesis de proteínas y otras biomoléculas importantes, sino que también proporcionan metabolitos intermedios para el ciclo del ácido tricarboxílico y la gluconeogénesis. El metabolismo de los aminoácidos está involucrado en el proceso de infección viral y en la inmunidad innata (27). En el presente estudio, la infección por PDCoV dio lugar a cambios en los patrones de expresión de una variedad de aminoácidos y nucleótidos. La L-glutamina, la metionina, el L-triptófano, la fenilalanina, la L-tirosina, el L-aspartato, la L-valina, la citrulina y la L-quinurenina estaban obviamente reguladas al alza, mientras que la L-cisteína y sus derivados mostraron una importante regulación a la baja. Como parásitos intracelulares, los virus responden completamente a la energía celular y a la maquinaria molecular para entrar, proliferar y construir una infección exitosa. En general, los virus son capaces de remodelar la red metabólica del huésped durante su proceso de infección (28, 29). Se ha reportado que el virus de la peste porcina africana secuestra la energía del huésped y el metabolismo de los ácidos animogénicos para promover la infección viral (30). El KSHV induce alteraciones en la vía glucolítica al mejorar la captación de glutamina, que proporciona intermediarios para el ciclo del ácido tricarboxílico (31, 32). Por lo tanto, planteamos la hipótesis de que, de acuerdo con KSHV, PDCoV regula al alza la expresión de varios aminoácidos, especialmente la glutamina, aumenta la reserva de aminoácidos intracelulares y regula el ciclo del ácido tricarboxílico en la célula huésped para proporcionar aminoácidos y energía para su propia replicación. Los virus satisfacen las demandas de síntesis de ácidos nucleicos virales mediante la regulación del anabolismo de nucleótidos (33). Niveles elevados de UDP, CMP, citosina, uracilo, UMP y GMP durante la infección por el VEN. Las alteraciones en el metabolismo de los nucleótidos pueden desempeñar un papel importante en la promoción de la rápida replicación del genoma del virus del VEN. Las células PK-15 infectadas con el virus de la peste porcina clásica mostraron una disminución del contenido de inosina, ácido inosínico y guanina, lo que sugiere que la infección por el virus de la peste porcina clásica promueve el agotamiento de las nucleobases por la actividad de síntesis de ARN viral (34). En nuestro estudio, la producción de uracilo, adenina, timina, citosina y la mayoría de sus derivados o materias primas se redujo significativamente, lo que puede deberse al alto consumo de nucleótidos durante la rápida replicación de PDCoV en las primeras etapas. Estos resultados revelan que las PDCoV regulan el metabolito primario (es decir, la biosíntesis de aminoácidos y el metabolismo de nucleótidos) para promover su replicación.
La ferroptosis es una forma no apoptótica dependiente del hierro de muerte celular regulada (RCD, por sus siglas en inglés). Está mediada principalmente por la peroxidación lipídica dependiente del hierro y se caracteriza por niveles intracelulares elevados de peróxidos de hierro y fosfolípidos (35), con un exceso de hierro que promueve la acumulación anormal de ROS y acelera la peroxidación lipídica (36). En nuestro estudio, los resultados transcriptómicos mostraron que la expresión de varios genes diferencialmente significativos se asoció con la vía de la ferroptosis. En concreto. La activación de la vía de señalización de la ferroptosis por la erastina impidió significativamente la infección por PDCoV. Además, se encontró que el virus del VIS reguló al alza la expresión de TFRC durante el proceso de infección, lo que provocó que las células tomaran grandes cantidades de hierro antes de que se iniciaran los mecanismos de defensa, lo que finalmente indujo la ferroptosis en las células (23). Además, los resultados metabolómicos también mostraron que el contenido de γ-GC estaba significativamente regulado a la baja. γ-GC funciona como sintetizador y regulador al alza de GSH, que es un sustrato reducido para GPX4, un importante regulador negativo de la ferroptosis. Se demostró que el γ-GC inhibía significativamente la ferroptosis en las células neuronales (37). Nuestros resultados sugieren que la infección por PDCoV activa la vía de señalización de la ferroptosis en las células IPEC-J2, y el huésped utiliza la activación de la ferropotosis para inhibir la replicación del virus.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están a disposición del público. Estos datos se pueden encontrar en: https://www.ncbi.nlm.nih.gov/bioproject/, PRJNA1078001; https://www.ebi.ac.uk/metabolights/, MTBLS9591.
Contribuciones de los autores
GW: Redacción – borrador original, Curación de datos, Análisis formal, Investigación, Metodología, Validación. YC: Investigación, Software, Escritura – Revisión y Edición. CX: Curación de datos, Metodología, Software, Redacción – revisión y edición. ShuoZ: Metodología, Visualización, Escritura – revisión y edición. YH: Análisis formal, Software, Redacción – revisión y edición. ShuaZ: Conceptualización, Obtención de Fondos, Administración de Proyectos, Recursos, Supervisión, Validación, Redacción, Revisión y Edición. WB: Redacción, revisión y edición, obtención de fondos, administración de proyectos, recursos, validación.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo fue financiado por la Fundación de Ciencias Naturales de la Provincia de Jiangsu (BK20220581), el fondo destinado al Sistema de Tecnología de la Industria Agrícola de Jiangsu (JATS [2023] 450), la Fundación de Investigación Postdoctoral de China (2021M692712) y el Desarrollo de Programas Académicos Prioritarios de las Instituciones de Educación Superior de Jiangsu.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
El revisor ZZ declaró una afiliación compartida con el/los autor/es al editor encargado en el momento de la revisión.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Woo PCY, Lau SKP, Lam CSF, Lau CCY, Tsang AKL, Lau JHN, et al. El descubrimiento de siete nuevos coronavirus de mamíferos y aves del género deltacoronavirus respalda a los coronavirus de murciélagos como fuente genética de alfacoronavirus y betacoronavirus y a los coronavirus aviares como fuente genética de gammacoronavirus y deltacoronavirus. J Virol. (2012) 86:3995–4008. doi: 10.1128/JVI.06540-11
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Wang L, Byrum B, Zhang Y. Detección y caracterización genética de deltacoronavirus en cerdos, Ohio, EE. UU., 2014. Emerg Infect Dis. (2014) 20:140296. doi: 10.3201/eid2007.140296
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Wang Y-W, Yue H, Fang W, Huang Y-W. Secuencia completa del genoma de la cepa porcina de deltacoronavirus CH/Sichuan/S27/2012 de China continental. Anuncio del genoma. (2015) 3:E00945-15. doi: 10.1128/genomaA.00945-15
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Ajayi T, Dara R, Misener M, Pasma T, Moser L, Poljak Z. Prevalencia e incidencia a nivel de rebaño del virus de la diarrea epidémica porcina (PEDV) y deltacoronavirus porcino (PDCoV) en piaras porcinas en Ontario, Canadá. Transbound Emerg Dis. (2018) 65:1197–207. doi: 10.1111/tbed.12858
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Lee S, Lee C. Caracterización completa del genoma de la cepa coreana porcina deltacoronavirus KOR/KNU14-04/2014. Anuncio del genoma. (2014) 2:E01191-14. doi: 10.1128/genomaA.01191-14
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Stramer SL, Moritz ED, Foster GA, Ong E, Linnen JM, Hogema BM, et al. Virus de la hepatitis E: seroprevalencia y frecuencia de detección de ARN viral entre donantes de sangre de EE. UU. Transfusión. (2016) 56:481–8. doi: 10.1111/trf.13355
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Fang P, Fang L, Liu X, Hong Y, Wang Y, Dong N, et al. Identificación y localización subcelular de la proteína accesoria NS6 del deltacoronavirus porcino. Virología. (2016) 499:170–7. doi: 10.1016/j.virol.2016.09.015
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Fang P, Fang L, Hong Y, Liu X, Dong N, Ma P, et al. Descubrimiento de una nueva proteína accesoria NS7a codificada por deltacoronavirus porcino. J Gen Virol. (2017) 98:173–8. doi: 10.1099/jgv.0.000690
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Chen Q, Gauger P, Stafne M, Thomas J, Arruda P, Burrough E, et al. Patogenicidad y patogenia de un aislado de células de deltacoronavirus porcino de los Estados Unidos en lechones neonatos de 5 días de edad. Virología. (2015) 482:51–9. doi: 10.1016/j.virol.2015.03.024
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Jung K, Hu H, Saif LJ. Infección porcina por deltacoronavirus: etiología, cultivo celular para aislamiento y propagación del virus, epidemiología molecular y patogenia. Virus Res. (2016) 226:50–9. doi: 10.1016/j.virusres.2016.04.009
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Zhang J. Deltacoronavirus porcino: descripción general de la dinámica de la infección, métodos de diagnóstico, prevalencia y evolución genética. Virus Res. (2016) 226:71–84. doi: 10.1016/j.virusres.2016.05.028
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Boley PA, Alhamo MA, Lossie G, Yadav KK, Vásquez-Lee M, Saif LJ, et al. Infección y transmisión porcina por deltacoronavirus en aves de corral, Estados Unidos1. Emerg Infect Dis. (2020) 26:255–65. doi: 10.3201/eid2602.190346
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Jung K, Hu H, Saif LJ. Los terneros son susceptibles a la infección por el deltacoronavirus porcino recién aparecido, pero no por el alfacoronavirus entérico porcino, el virus de la diarrea epidémica porcina. Arch Virol. (2017) 162:2357–62. doi: 10.1007/s00705-017-3351-z
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Li W, Hulswit RJG, Kenney SP, Widjaja I, Jung K, Alhamo MA, et al. La amplia participación de los receptores de un coronavirus global emergente puede potenciar su diversa transmisibilidad entre especies. Proc Natl Acad Sci USA. (2018) 115:E5135–43. doi: 10.1073/pnas.1802879115
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Gladue D, Jiang S, Li F, Li X, Wang L, Zhang L, et al. Análisis del transcriptoma de las células PK-15 en la respuesta inmune innata a la infección porcina por deltacoronavirus. Plos ONE. (2019) 14:e0223177. doi: 10.1371/journal.pone.0223177
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Liu S, Fang P, Ke W, Wang J, Wang X, Xiao S, et al. La infección porcina por deltacoronavirus (PDCoV) antagoniza la producción de interferón-λ1. Microbiol veterinario. (2020) 247:108785. doi: 10.1016/j.vetmic.2020.108785
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Luo J, Fang L, Dong N, Fang P, Ding Z, Wang D, et al. La infección porcina por deltacoronavirus (PDCoV) suprime la producción de interferón-β mediada por RIG-I. Virología. (2016) 495:10–7. doi: 10.1016/j.virol.2016.04.025
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Zhang H, Li F, Pan Z, Wu Z, Wang Y, Cui Y. La activación de la vía PI3K/Akt limita la apoptosis mediada por JNK durante la infección por EV71. Virus Res. (2014) 192:74–84. doi: 10.1016/j.virusres.2014.07.026
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Ren Z, Yu Y, Zhang X, Wang Q, Deng J, Chen C, et al. Exploración de la apoptosis inducida por PDCoV a través del desequilibrio de la dinámica mitocondrial y el efecto antagónico de las SeNPs. Frente Immunol. (2022) 13:972499. doi: 10.3389/fimmu.2022.972499
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Lee YJ, Lee C. El deltacoronavirus porcino induce apoptosis dependiente de caspasas a través de la activación de la vía mitocondrial intrínseca mediada por el citocromo c. Virus Res. (2018) 253:112–23. doi: 10.1016/j.virusres.2018.06.008
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Gao J, Wang Q, Tang Y-D, Zhai J, Hu W, Zheng C. Cuando la ferroptosis se encuentra con infecciones patógenas. Tendencias Microbiol. (2023) 31:468–79. doi: 10.1016/j.tim.2022.11.006
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Zhang H, Li Y, Yang R, Xiao L, Dong S, Lin J, et al. La erastina inhibe la replicación del virus de la diarrea epidémica porcina en las células Vero. Las células frontales infectan el microbiol. (2023) 13:1142173. doi: 10.3389/fcimb.2023.1142173
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Cheng J, Tao J, Li B, Shi Y, Liu H. El virus de la influenza porcina desencadena la ferroptosis en las células A549 para mejorar la replicación del virus. Virol J. (2022) 19:104. doi: 10.1186/s12985-022-01825-y
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Li Y, Bao Y, Li Y, Duan X, Dong S, Lin J, et al. RSL3 inhibe la replicación del virus de la diarrea epidémica porcina mediante la activación de la ferroptosis. Virus. (2023) 15:2080. doi: 10.3390/v15102080
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Liu X, Zhu L, Liao S, Xu Z, Zhou Y. Respuesta del transcriptoma del microARN porcino a la infección por el virus de la gastroenteritis transmisible. PLoS UNO. (2015) 10:e0120377. doi: 10.1371/journal.pone.0120377
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Manchester M, Anand A. Metabolómica: estrategias para definir el papel del metabolismo en la infección y patogénesis del virus. Adv Virus Res. (2017) 98:57–81. doi: 10.1016/bs.aivir.2017.02.001
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Tomo D. Metabolismo de aminoácidos y vías de señalización: objetivos potenciales en el control de la infección y la inmunidad. Eur J Clin Nutr. (2021) 75:1319–27. doi: 10.1038/s41430-021-00943-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Moreno-Altamirano MMB, Kolstoe SE, Sánchez-García FJ. Control del metabolismo celular por parte de los virus para la replicación y evasión de las respuestas inmunitarias del huésped. Las células frontales infectan el microbiol. (2019) 9:95. doi: 10.3389/fcimb.2019.00095
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Rosenwasser S, Ziv C, Creveld SG, Vardi A. Metabolismo de Virocell: innovaciones metabólicas durante las interacciones huésped-virus en el océano. Tendencias Microbiol. (2016) 24:821–32. doi: 10.1016/j.tim.2016.06.006
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Xue Q, Liu H, Zhu Z, Yang F, Song Y, Li Z, et al. El virus de la peste porcina africana regula la energía del huésped y el metabolismo de los aminoácidos para promover la replicación viral. J Virol. (2022) 96:e0191921. doi: 10.1128/JVI.01919-21
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Delgado T, Sánchez EL, Camarda R, Lagunoff M. Perfil metabólico global de la infección por un virus oncogénico: el KSHV induce y requiere lipogénesis para la supervivencia de la infección latente. PLoS Pathog. (2012) 8:e1002866. doi: 10.1371/journal.ppat.1002866
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Sanchez EL, Carroll PA, Thalhofer AB, Lagunoff M. Las células endoteliales latentes infectadas con KSHV son adictas a la glutamina y requieren glutaminólisis para sobrevivir. PLoS Pathog. (2015) 11:e1005052. doi: 10.1371/journal.ppat.1005052
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Thai M, Graham NA, Braas D, Nehil M, Komisopoulou E, Kurdistani SK, et al. La activación de MYC inducida por adenovirus E4ORF1 promueve el metabolismo anabólico de la glucosa de la célula huésped y la replicación del virus. Célula Metab. (2014) 19:694–701. doi: 10.1016/j.cmet.2014.03.009
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Gou H, Zhao M, Yuan J, Xu H, Ding H, Chen J. Perfiles metabólicos en líneas celulares infectadas con el virus de la peste porcina clásica. Microbiol frontal. (2017) 8:691. doi: 10.3389/fmicb.2017.00691
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Dixon SJ, Lemberg KM, Lamprecht MR, Skouta R, Zaitsev EM, Gleason CE, et al. Ferroptosis: forma de muerte celular no apoptótica dependiente del hierro. Celda. (2012) 149:1060–72. doi: 10.1016/j.cell.2012.03.042
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Xu T, Ding W, Ji X, Ao X, Liu Y, Yu W, et al. Mecanismos moleculares de la ferroptosis y su papel en la terapia del cáncer. J Cell Mol Med. (2019) 23:4900–12. doi: 10.1111/jcmm.14511
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Zhang R, Lei J, Chen L, Wang Y, Yang G, Yin Z, et al. La gamma-glutamilcisteína ejerce efectos neuroprotectores contra la lesión por isquemia/reperfusión cerebral mediante la inhibición de la peroxidación lipídica y la ferroptosis. Antioxidantes. (2022) 11:1653. doi: 10.3390/antiox11091653
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: PDCoV, células IPEC-J2, transcriptoma, metaboloma, ferroptosis
Cita: Wang G, Cao Y, Xu C, Zhang S, Huang Y, Zhang S y Bao W (2024) Análisis transcriptómico y metabolómico exhaustivo de las células epiteliales intestinales porcinas después de la infección por PDCoV. Frente. Vet. Sci. 11:1359547. doi: 10.3389/fvets.2024.1359547
Recibido: 21 de diciembre de 2023; Aceptado: 07 de mayo de 2024;
Publicado: 24 mayo 2024.
Editado por:
Michael Kogut, Departamento de Agricultura de los Estados Unidos, Estados Unidos
Revisado por:
Zhenbang Zhu, Universidad de Yangzhou, China
Hongxu Du, Universidad del Suroeste, China
Derechos de autor © 2024 Wang, Cao, Xu, Zhang, Huang, Zhang y Bao. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Wenbin Bao, de wbbao@yzu.edu.cn; Shuai Zhang, shuai_zhang1990@163.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía