Caracterización metagenómica en cerdos alimentados con Boehmeria nivea fermentada
Caracterización metagenómica de la comunidad de microbiota cecal y funciones en cerdos de engorde alimentados con Boehmeria nivea fermentada
- 1Facultad de Ciencia y Tecnología Animal, Universidad Agrícola de Henan, Zhengzhou, China
- 2Laboratorio de Ingeniería Aditiva de Alimentación Funcional de Jiangxi, Instituto de Recursos Biológicos, Academia de Ciencias de Jiangxi, Nanchang, China
- 3CAS Key Laboratory of Agro-Ecological Processes in Subtropical Region, Institute of Subtropical Agriculture, Chinese Academy of Sciences, National Engineering Laboratory for Pollution Control and Waste Utilization in Livestock and Poultry Production, Changsha, China
- 4Facultad de Ciencia y Tecnología Animal, Universidad Agrícola de Hunan, Changsha, China
- 5Departamento de Ciencia y Tecnología Animal, Universidad Konkuk, Seúl, República de Corea
- 6Instituto de Biotecnología Industrial de Tianjin, Academia China de Ciencias, Tianjin, China
El ramio (Boehmeria nivea, BN) se utiliza como forraje para el ganado a través de una fermentación de ensilaje adecuada debido a su valor nutricional. Hasta la fecha, relativamente pocos estudios han investigado los efectos del BN fermentado dietético (FBN) en la salud intestinal en cerdos de engorde. El objetivo del presente estudio fue investigar los efectos de la suplementación dietética con FBN al 20% sobre la morfología intestinal, la expresión génica y la respuesta funcional de la microbiota intestinal en cerdos de engorde. Encontramos que FBN no afectó significativamente las actividades de las enzimas antioxidantes séricas, la morfología ileal o la expresión de genes que codifican enzimas antioxidantes, citoquinas inflamatorias o proteínas de unión estrecha en el hígado de los cerdos. Sin embargo, los niveles de expresión génica del receptor de hidrocarburos de arilo (AHR) y la interleucina 6 (IL6) se regularon significativamente a la baja en el íleon. Un análisis metagenómico demostró que, en comparación con el observado en el grupo control, la microbiota cecal de los cerdos en el grupo de tratamiento con FBN estaba más estrechamente agrupada y contenía un mayor número de microbios únicos. Las bacterias fueron el reino predominante en la microbiota cecal, mientras que Firmicutes, Bacteroidetes y Proteobacteria fueron los filos dominantes, y Streptococcus, Lactobacillus y Prevotella fueron los géneros dominantes. El FBN dietético aumentó significativamente la abundancia de la bacteria probiótica Roseburia inulinivorans (p < 0,05). El análisis funcional de la microbiota cecal mostró que los niveles del transportador ABC y las funciones asociadas a la glucólisis/gluconeogénesis estaban disminuidas en cerdos alimentados con FBN. Mientras tanto, el análisis de CAZyme reveló que el FBN dietético redujo significativamente el contenido de las enzimas activas en carbohidratos, como GT2, GH1, GH25 y GH13_31. Además, el análisis del citocromo P450 reveló que la abundancia de CYP51 y CYP512 disminuyó con el tratamiento con FBN. Una evaluación de la resistencia a los antibióticos basada en la anotación de la Base de Datos Integral de Resistencia a los Antibióticos (CARD) indicó que los microbios cecales de cerdos en el grupo de tratamiento con FBN habían aumentado la resistencia a la lincosamida, la estreptogramina y el cloranfenicol y una resistencia reducida a la amikacina, isepamicina, neomicina, lividomicina, gentamicina, paromomicina, ribostamicina y butirosina. Finalmente, el análisis relacionado con el factor de virulencia mostró que las funciones putativas asociadas a la hemolisina disminuyeron, mientras que las funciones asociadas a la proteína de unión a fibronectina, los flagelos y el alginato aumentaron. Tomados en conjunto, nuestros datos mostraron que la suplementación con FBN ejerció solo efectos menores sobre la morfología intestinal y la composición de la comunidad microbiana, lo que sugiere que es potencialmente seguro para su uso como suplemento en las dietas de cerdos de engorde. Sin embargo, se necesitan más estudios para validar su funcionalidad.
1. Introducción
En las últimas décadas, China ha importado una enorme cantidad de harina de soja (SBM) debido a la rápida ampliación de la industria porcina moderna y al limitado suministro interno de SBM. Los investigadores y los fabricantes de piensos se han centrado cada vez más en identificar alternativas nativas rentables a la SBM para reducir la dependencia de los SBM importados y mantener el desarrollo sostenible de la industria porcina china. El ramio (Boehmeria nivea) es un antiguo cultivo de fibra textil perenne de rápido crecimiento ampliamente cultivado en el sur de China (1). Las hojas y las partes superiores tiernas del ramio son bajas en fibra pero altas en proteínas (20% de contenido de proteína cruda sobre una base de materia seca) y ricas en aminoácidos (especialmente lisina), minerales, flavonoides, polifenoles y vitaminas (1). El ramio se ha aplicado en piensos para cabras negras como sustituto de la alfalfa (2) y no ha afectado la salud, la calidad de la carne o la diversidad de la microbiota intestinal de las cabras Boer, incluso a una tasa de inclusión de hasta el 40% (3). Se ha demostrado que el ensilaje mixto dietético de ramio y paja de arroz aumenta el porcentaje de grasa láctea, el contenido de sólidos lácteos y la digestibilidad de la materia seca en vacas lecheras (4). El ramio tiene buena palatabilidad y valor nutricional como suplemento en la alimentación de rumiantes, pero sigue siendo infrautilizado. El ramio también se ha utilizado en las dietas de cerdos de engorde para mejorar los rasgos de la canal, la composición química muscular (5), las actividades enzimáticas antioxidantes séricas y la composición de ácidos grasos de cerdo (6). Se ha informado que un bajo nivel de inclusión dietética de ramio aumenta la ganancia diaria promedio y la relación alimento-ganancia en cerdos, aunque los niveles de inclusión más altos afectaron negativamente el rendimiento de crecimiento de los animales (6).
El ensilaje es uno de los mejores métodos para preservar los recursos forrajeros en regiones con fuertes lluvias porque contiene altos niveles de bacterias del ácido láctico y ácidos orgánicos y bajos niveles de fibra no digerible (7). En general, el ensilaje se produce a través de la fermentación natural mediada por bacterias, con algunos aditivos que se incluyen para mejorar la calidad del ensilaje. Aspergillus niger se usa comúnmente como inoculante para tratar una amplia gama de residuos agrícolas y domésticos para la producción de productos de valor agregado debido a sus actividades multienzimáticas (mananasa, xilanasa, celulasa, glucanasa y lipasa) (8, 9). A. niger también se utiliza para la producción de ácidos orgánicos y la hidrólisis de polifenoles unidos utilizando materiales de bajo valor (10, 11).
La microbiota intestinal y los ingredientes del alimento pueden afectar mutuamente la nutrición, el metabolismo y la salud general del huésped (12-14). Sin embargo, no es posible predecir cómo la introducción de un nuevo ingrediente alimenticio en la dieta de los cerdos afectará el desarrollo de la microbiota intestinal de los animales. Los avances en la tecnología de secuenciación de alto rendimiento han permitido un análisis más completo de la microbiota intestinal compleja (15). Los estudios han empleado tradicionalmente la secuenciación del gen 16S rRNA de muestras fecales para analizar la diversidad y composición de la microbiota intestinal (16); Sin embargo, este método no puede lograr una comprensión profunda de los efectos de la dieta en la funcionalidad de los microorganismos intestinales. En este estudio, preparamos ensilaje de ramio utilizando una cepa autóctona de A. niger previamente aislada de ramio y exploramos sus efectos en cerdos de engorde. Realizamos un análisis metagenómico de secuenciación de escopeta del contenido cecal de los animales para dilucidar exhaustivamente cómo un alto nivel de inclusión de ensilaje de ramio afecta a su microbiota intestinal. También se evaluaron los efectos del ensilaje de ramio dietético sobre la morfología ileal y la expresión de genes antioxidantes y antiinflamatorios.
2. Materiales y métodos
2.1. Animales y dietas experimentales
Todos los procedimientos experimentales con animales fueron aprobados (2015-8A) por el Comité de Cuidado y Uso de Animales del Instituto de Agricultura Subtropical de la Academia China de Ciencias. En el estudio se utilizaron un total de 12 cerdos finales DLY (Duroc × Landrace × Yorkshire) con un peso corporal inicial promedio de 60,11 ± 0,09 kg. Se formuló una dieta basal que contenía maíz, SBM y salvado de arroz sobre la base de las recomendaciones del Consejo Nacional de Investigación (Tabla Suplementaria S1). Los cerdos fueron asignados aleatoriamente a dos grupos de tratamiento, con seis réplicas por grupo. Los cerdos en el grupo de control (Con) fueron alimentados con una dieta basal, y los del grupo fermentado Boehmeria nivea (FBN) fueron alimentados con una dieta basal en la que la mezcla de maíz, SBM y salvado de arroz fue reemplazada por la inclusión isocalórica e isonitrogenada de 20% FBN. El ramio fermentado fue proporcionado por la Universidad Agrícola de Hunan. El ensilaje de ramio se preparó mediante la inoculación de 100 mL de 1 × 106 UFC/ml de A. niger en 1 kg de ramio en polvo. Después del envasado y fermentación a 30 ° C durante 60 días, el FBN fue formulado y peletizado. Los cerdos recibían alimento dos veces al día y tenían acceso gratuito a sus dietas. El ensayo de alimentación duró 60 días después de un período de adaptación de 7 días.
2.2. Eutanasia y recogida de muestras
Al final del experimento de alimentación, los cerdos fueron sacrificados de acuerdo con los procedimientos comerciales estándar. Se recolectaron muestras de sangre, hígado, íleon y contenido de colon de cada cerdo. La sangre se recogió de la vena yugular como se describió anteriormente (17). Las muestras de suero se prepararon por centrifugación de la sangre a 3.000 × g durante 10 min a 4 °C y se almacenaron a -20 °C para el análisis de la actividad enzimática antioxidante. Las muestras de tejido hepático e ileal y el contenido cecal (exprimido del ciego) se congelaron a presión en nitrógeno líquido y se almacenaron a -80 ° C para el análisis de la expresión génica y la secuenciación metagenómica de escopeta.
2.3. Parámetros antioxidantes séricos
La capacidad antioxidante total sérica (TAC) y las actividades de superóxido dismutasa (SOD), catalasa (CAT) y glutatión peroxidasa (GSH-Px) se determinaron utilizando kits disponibles comercialmente obtenidos de Suzhou Comin Biotechnology Co., Ltd. (Suzhou, Jiangsu, China) siguiendo las instrucciones del fabricante.
2.4. Análisis histológico de la morfología ileal
Las secciones de tejido ileal incrustadas en parafina (5 μm) se tiñeron con hematoxilina y eosina (H&E). La integridad del tejido ileal se evaluó midiendo la longitud de las vellosidades (VL) y la profundidad de la cripta (CD) y determinando la relación VL/CD bajo un microscopio óptico (Olympus, Japón) equipado con una cámara digital.
2.5. Análisis cuantitativo por PCR en tiempo real (qPCR) de la expresión génica en el íleon y el hígado
La extracción total de ARN, la síntesis de ADNc y la qPCR se realizaron utilizando métodos descritos previamente (5). Brevemente, el ARN total se extrajo de 0,1 g de tejido hepático o ileal utilizando kits de extracción de ARN de columna (Magen, Guangzhou, China). La concentración y pureza de ARN se determinaron utilizando un espectrofotómetro NanoDrop 2000 (Thermo Fisher Scientific, Waltham, MA, EUA) y electroforesis en gel de agarosa al 1%, respectivamente. Aproximadamente 1 μg del ARN aislado se transcribió inversamente en ADNc utilizando un kit de síntesis de ADNc (CWBIO, Jiangsu, China) siguiendo las instrucciones del fabricante. La qPCR se realizó en un sistema de PCR en tiempo real ABI 7900HT (Applied Biosystems, Branchburg, NJ, EUA). Los cebadores utilizados en el estudio se diseñaron con Primer3 y BLAST utilizando parámetros predeterminados (Premier Biosoft International, Palo Alto, CA, EUA) y se describen en la Tabla Suplementaria S2. Las condiciones de ciclo de PCR fueron las siguientes: 95 ° C durante 10 min, seguido de 40 ciclos de 95 ° C durante 15 s y 60 ° C durante 60 s, y luego 1 ciclo de 72 ° C durante 30 s. Los niveles relativos de expresión génica se calcularon utilizando el 2–ΔΔCt después de la normalización a GAPDH o β-actina (18).
2.6. Extracción de ADN microbiano y secuenciación metagenómica
El ADN genómico se extrajo del contenido cecal utilizando un kit de ADN del suelo E.Z.N.A. (Omega Bio-Tek, Norcross, GA, EUA) de acuerdo con el protocolo del fabricante. La concentración y la calidad del ADN se evaluaron mediante un espectrofotómetro NanoDrop 2000 (Thermo Fisher Scientific, Waltham, MA, EUA) y electroforesis en gel de agarosa al 1%, respectivamente. El ADN metagenómico se secuenció en el extremo pareado (fragmentos de 400 pb) en la plataforma NovaSeq / HiSeq X 10 (Illumina Inc., San Diego, CA, EUA) en Majorbio Bio-Pharm Technology Co., Ltd. (Shanghai, China) utilizando kits de reactivos NovaSeq / HiSeq X 10.
2.7. Análisis de los datos de secuenciación
Los datos de secuenciación se analizaron en línea en la plataforma Majorbio Cloud.1 Las lecturas de baja calidad se eliminaron usando fastp2 (19). Las lecturas se alinearon utilizando el alineador Burrows-Wheeler (BWA)3 (20). Se seleccionaron contigs de al menos 300 pb y se utilizaron para una mayor predicción y anotación de genes. Los marcos de lectura abiertos (ORF) de cada contig ensamblado se predijeron utilizando MetaGene4 (21). Los ORFs predichos (longitud ≥ 100 pb) fueron recuperados y traducidos en secuencias de aminoácidos basadas en la tabla de traducción NCBI.5 Se construyó un catálogo de genes no redundantes utilizando CD-HIT6 (22) con 90% de identidad de secuencia y 90% de cobertura. Después del control de calidad, las lecturas se asignaron al catálogo de genes no redundantes con 95% de identidad utilizando SOAPaligner7 (23), y se calculó la abundancia de genes en cada muestra.
2.8. Anotación funcional
Las secuencias representativas del catálogo de genes no redundantes se alinearon con la base de datos no redundante del NCBI para la anotación taxonómica y el análisis de proteínas del Grupo de Grupos Ortólogos (COG). Anotación funcional de las secuencias representativas contra la Enciclopedia de Genes y Genomas de Kioto (KEGG)8 se realizó utilizando Diamond9 (24). Además, la anotación funcional de los genes representativos se analizó más a fondo en la base de datos Carbohydrate-Active enZYme (CAZy) (v07312018)10, la Base de Datos Integral de Resistencia a los Antibióticos (CARD)11y la base de datos de factores de virulencia (VFDB)12 usando Diamante (24).
2.9. Análisis estadístico
Las diferencias entre los tratamientos Con y FBN se evaluaron mediante la prueba t en SPSS 24.0 (SPSS IBM, NY, USA). Los resultados se expresaron como media ± error estándar de la media (SEM), y un valor de p <0,05 se consideró significativo.
3. Resultados
3.1. Parámetros antioxidantes séricos
Los efectos de la FBN dietética sobre las actividades de las enzimas antioxidantes séricas en cerdos de engorde se muestran en la Tabla 1. Las actividades séricas de TAC, SOD, CAT y GSH-Px no se vieron afectadas significativamente por la suplementación con 20% de FBN en la dieta.
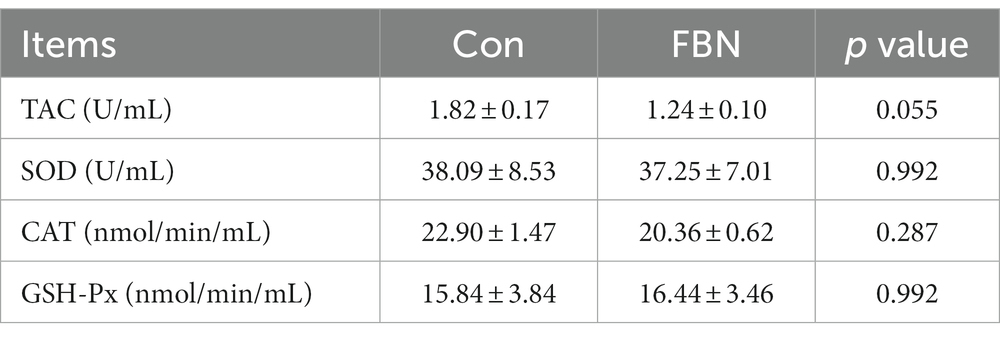 Tabla 1. Efecto dietético de las hojas fermentadas de Boechmeria nivea sobre las enzimas antioxidantes séricas.
Tabla 1. Efecto dietético de las hojas fermentadas de Boechmeria nivea sobre las enzimas antioxidantes séricas.
3.2. Morfología ileal
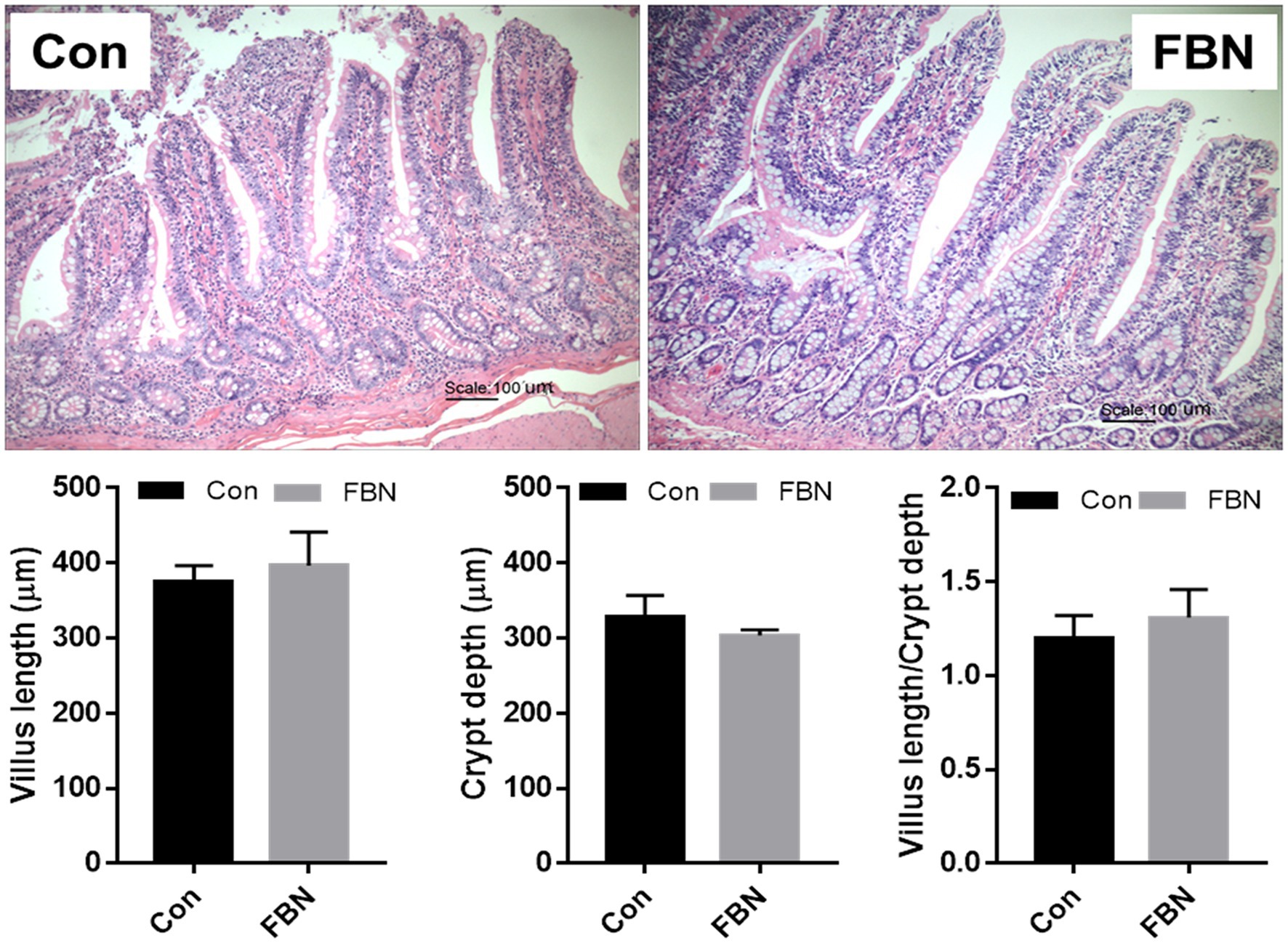
Los efectos de FBN dietético sobre la histomorfología ileal se muestran en la Figura 1. No se encontraron diferencias significativas en la relación LV, EC o VL/CD entre los dos grupos de tratamiento (p > 0,05).
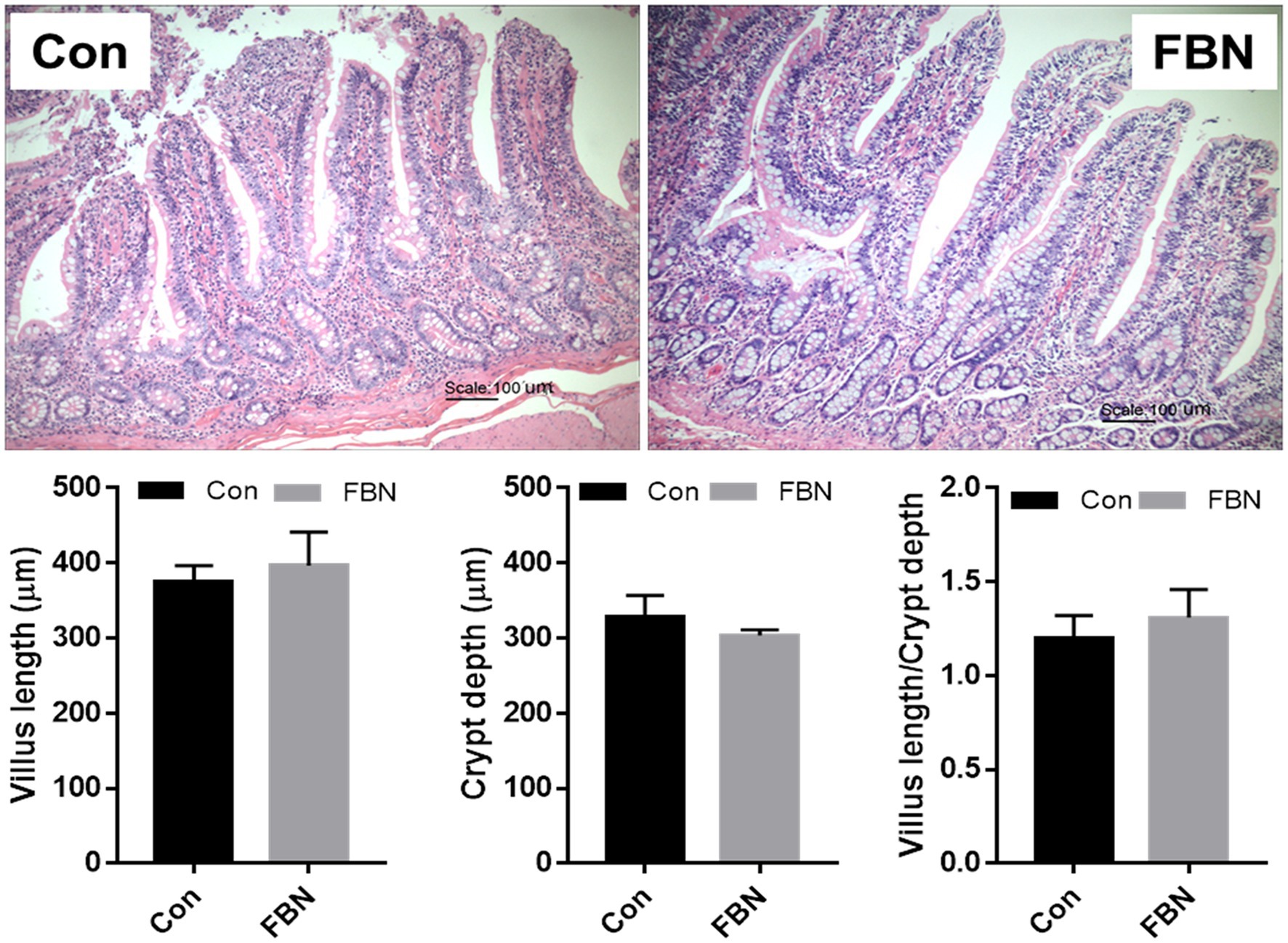 Figura 1. Efecto dietético de Boechmeria nivea fermentada sobre la morfología ileal de cerdos de engorde.
Figura 1. Efecto dietético de Boechmeria nivea fermentada sobre la morfología ileal de cerdos de engorde.
3.3. Expresión génica en el hígado y el íleon
En comparación con el tratamiento Con, no se detectaron cambios significativos en los niveles de expresión relativos de los genes que codifican antioxidantes, citoquinas inflamatorias y proteínas de unión estrecha en los hígados de animales administrados con FBN (Figura 2A); sin embargo, la expresión de los genes AHR e IL6 en el íleon fue significativamente regulada a la baja por la suplementación dietética de FBN (p < 0,05) (Figura 2B).
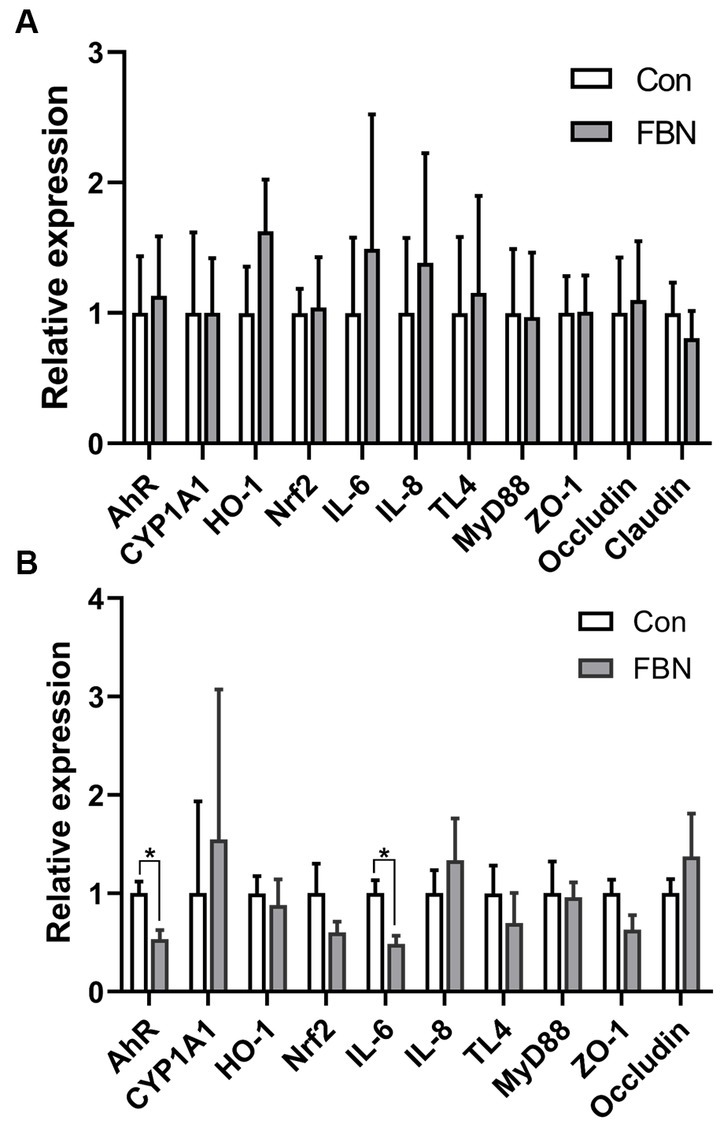 Figura 2. Efectos dietéticos de Boechmeria nivea fermentada sobre la expresión génica relativa en hígado (A) e íleon (B) de cerdos de engorde.
Figura 2. Efectos dietéticos de Boechmeria nivea fermentada sobre la expresión génica relativa en hígado (A) e íleon (B) de cerdos de engorde.
3.4. Composición taxonómica de los microbiomas cecales de los dos grupos
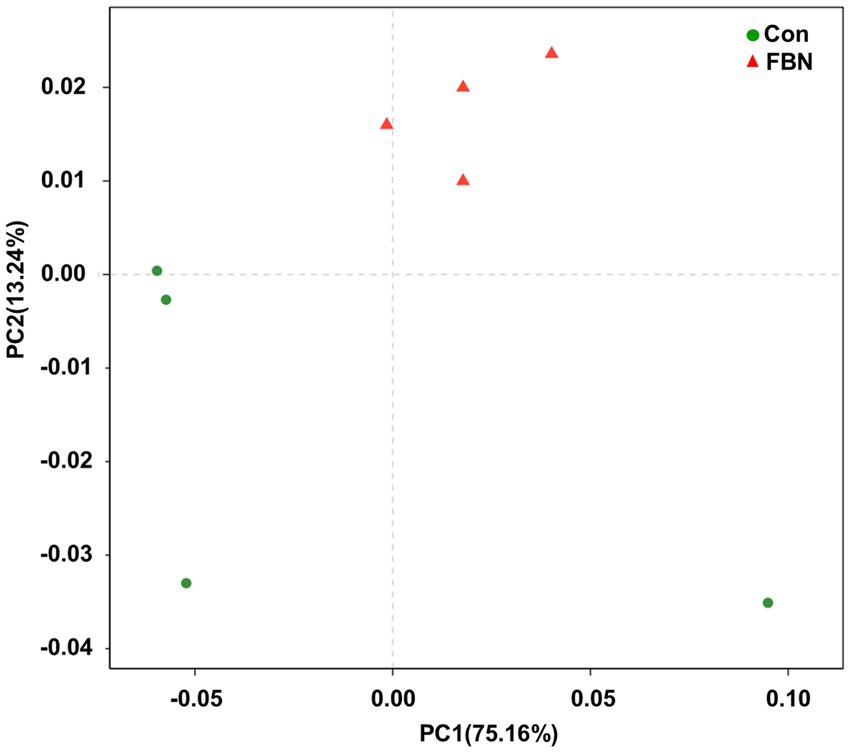
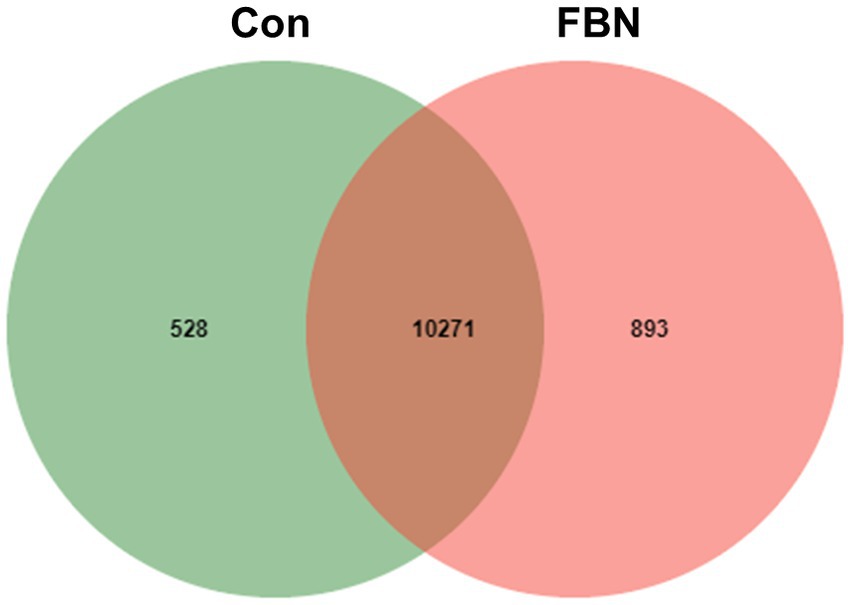
El análisis de componentes principales (ACP) indicó que los microorganismos cecales estaban más estrechamente agrupados en el grupo de tratamiento con FBN que en el grupo de tratamiento Con, y hubo una clara separación entre la microbiota de los dos grupos (Figura 3). Se identificaron un total de 11.692 especies mediante secuenciación metagenómica de escopeta, de las cuales 10.271 se compartieron entre los dos grupos y 528 y 893 fueron exclusivas de los grupos Con y FBN, respectivamente (Figura 4). La composición taxonómica del microbioma cecal se determinó a nivel de dominio, reino, filo y género (Figura 5). Las bacterias fueron los organismos más abundantes a nivel de dominio y reino, y otros organismos constituyeron solo una proporción muy pequeña del total (Figuras 5A, B). A nivel de filo, Firmicutes, Bacteroidetes y Proteobacteria predominaron en ese orden de abundancia (Figura 5C). A nivel de género, se identificaron 15 géneros dominantes en el ciego de los cerdos de engorde. La suplementación dietética de FBN redujo significativamente la abundancia relativa de Streptococcus y Ruminococcus, y aumentó la de Clostridium, unclassified_f_Lachnospiraceae, Eubacterium y unclassified_o_Clostridiales en el contenido cecal de los animales (Figura 5D).
Figura 4. Diagrama de Venn que muestra las especies únicas y compartidas en la microbiota cecal.
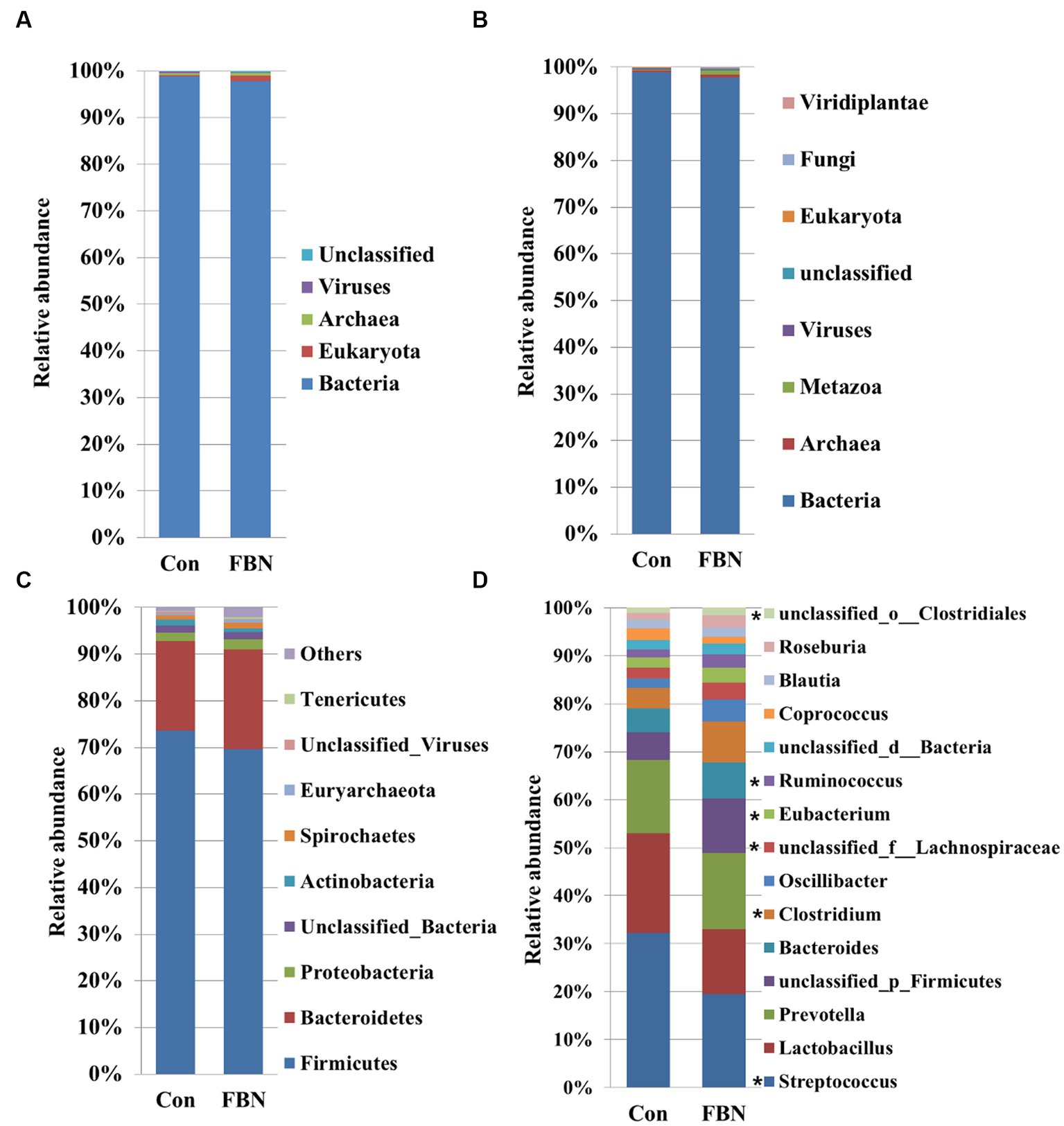 Figura 5. El microbioma cecal dominante en los niveles de dominio (A), reino (B), filo (C) y género (D). *indica diferencias significativas en el nivel P < 0,05.
Figura 5. El microbioma cecal dominante en los niveles de dominio (A), reino (B), filo (C) y género (D). *indica diferencias significativas en el nivel P < 0,05.
3.5. Anotación funcional del microbioma
Los cambios en los perfiles funcionales de las comunidades microbianas cecales se investigaron utilizando el software Tax4FUN basado en los conjuntos de datos de genes 16S rRNA. Para analizar la funcionalidad putativa del microbioma, los genes se anotaron en base a seis bases de datos de anotaciones funcionales. Las secuencias en el catálogo final de genes se alinearon y analizaron en la base de datos de probióticos. Se identificaron un total de 15 cepas probióticas dominantes entre las secuencias del microbioma cecal de cerdos de engorde, con Lactobacillus reuteri exhibiendo la mayor abundancia. Además, la abundancia de Roseburia inulinivorans mejoró significativamente con el tratamiento con FBN (Figura 6). El análisis de correlación mostró que la abundancia de R. inulinivorans se correlacionó negativamente con la expresión génica de AHR e IL6 en el íleon (Figura 7). El catálogo de genes se anotó funcionalmente en la base de datos KEGG y se identificaron las 15 categorías funcionales principales (en el nivel 3). Los genes involucrados en las vías que se predice que están relacionadas con los transportadores ABC y la glucólisis/gluconeogénesis estuvieron significativamente subrepresentados, mientras que los asociados con la reparación de desajustes se enriquecieron con el tratamiento dietético de FBN (Figura 8). Los catálogos de genes se alinearon aún más con sus secuencias de proteínas y se analizaron en la base de datos CAZy. Las secuencias se clasificaron principalmente en tres grupos de enzimas, a saber, glicosil transferasas (GT), glucósidos hidrolasas (GH) y esterasas de carbohidratos (CE). Entre ellos, GT2, GH1, GH25 y GH13_31 se redujeron significativamente con el tratamiento dietético de FBN, junto con una disminución en la abundancia de CE1 (Figura 9). Además, las secuencias de genes se analizaron en la base de datos de ingeniería del citocromo P450. Los resultados mostraron que CYP51, CYP505 y CYP125 eran las familias predominantes de CYP450. Entre estas familias de P450, la abundancia de CYP51 y CYP512 disminuyó significativamente con la suplementación dietética de FBN (Figura 10). También analizamos el catálogo de genes en el CARD para identificar y comparar la abundancia de genes de resistencia a antibióticos (ARG) (Figura 11). Los resultados mostraron que el FBN dietético redujo la abundancia de ARG y aumentó la de genes sensibles a los antibióticos. Finalmente, analizamos los factores de virulencia en el microbioma cecal en la VFDB. Encontramos que la abundancia de genes relacionados con la hemolisina disminuyó, mientras que la de genes relacionados con la proteína de unión a fibronectina, flagelos y alginato aumentó significativamente en el grupo de suplementación con FBN (Figura 12).
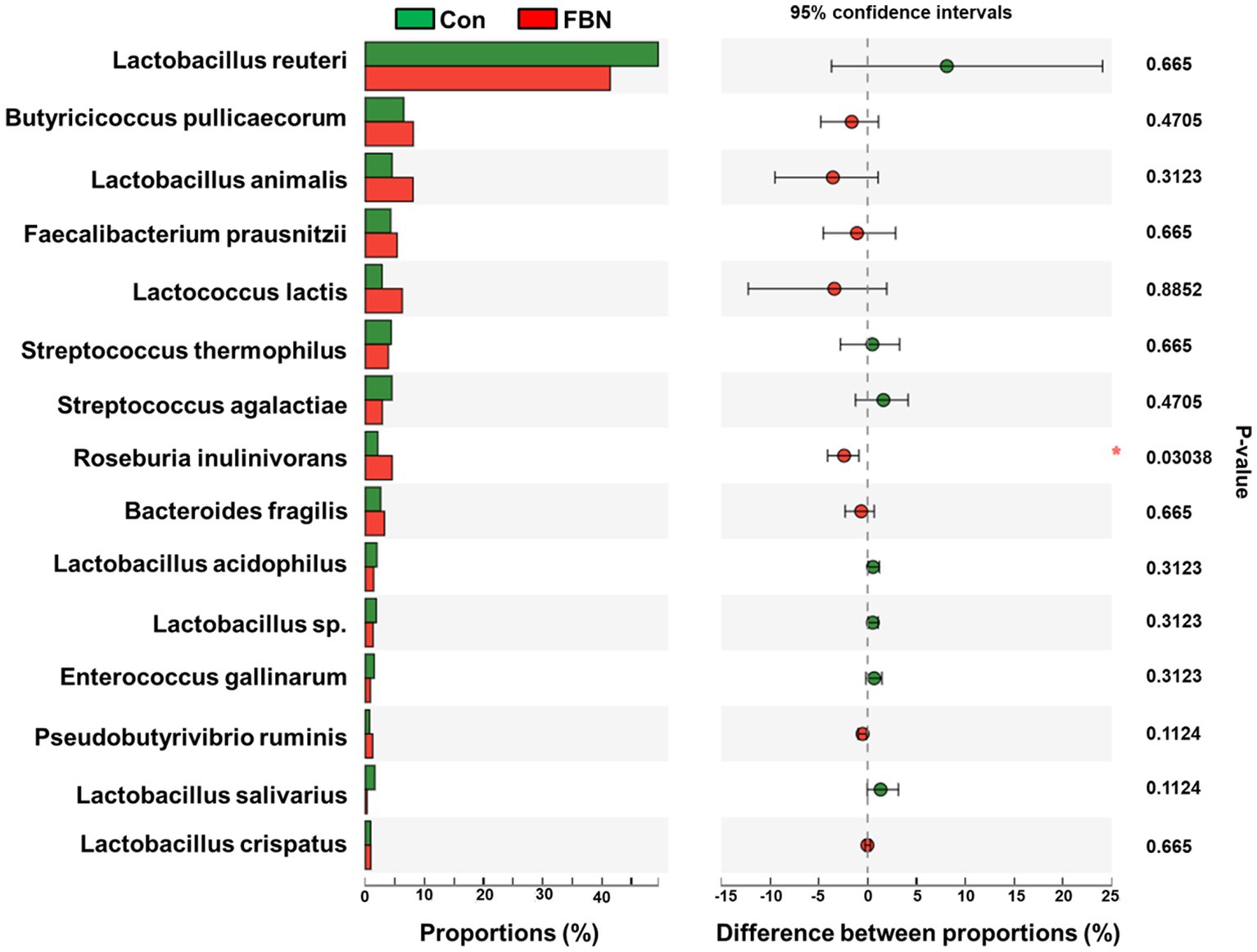 Figura 6. Supuesta abundancia de probióticos en la microbiota cecal anotada por la base de datos de probióticos.
Figura 6. Supuesta abundancia de probióticos en la microbiota cecal anotada por la base de datos de probióticos.
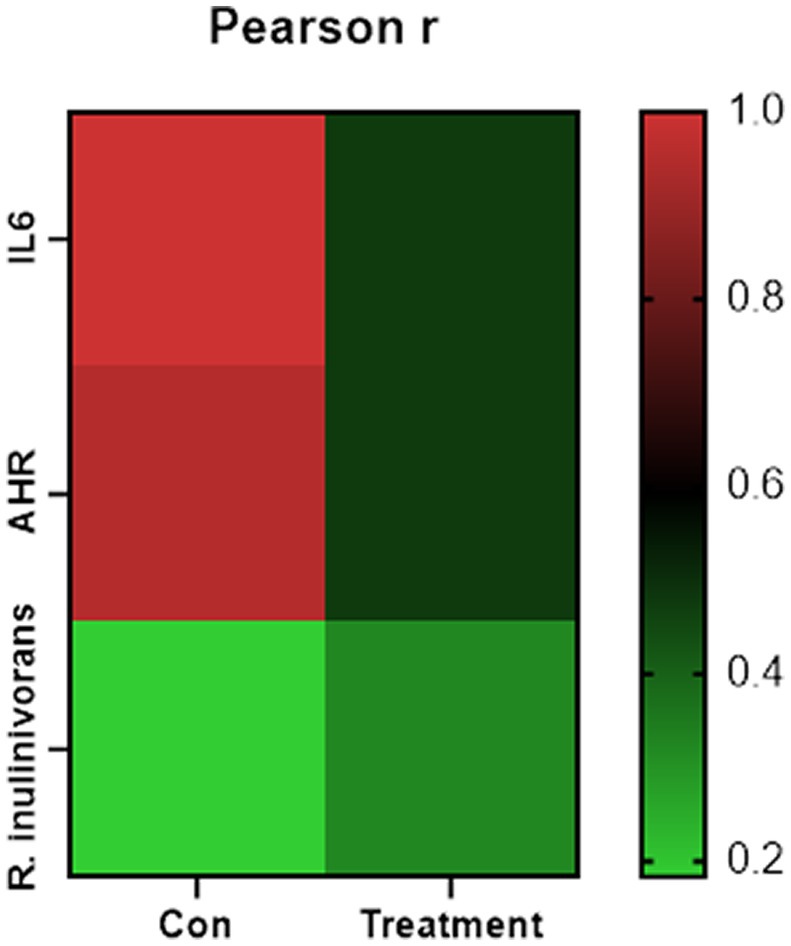 Figura 7. Análisis de correlación entre las especies alteradas diferencialmente y la expresión génica.
Figura 7. Análisis de correlación entre las especies alteradas diferencialmente y la expresión génica.
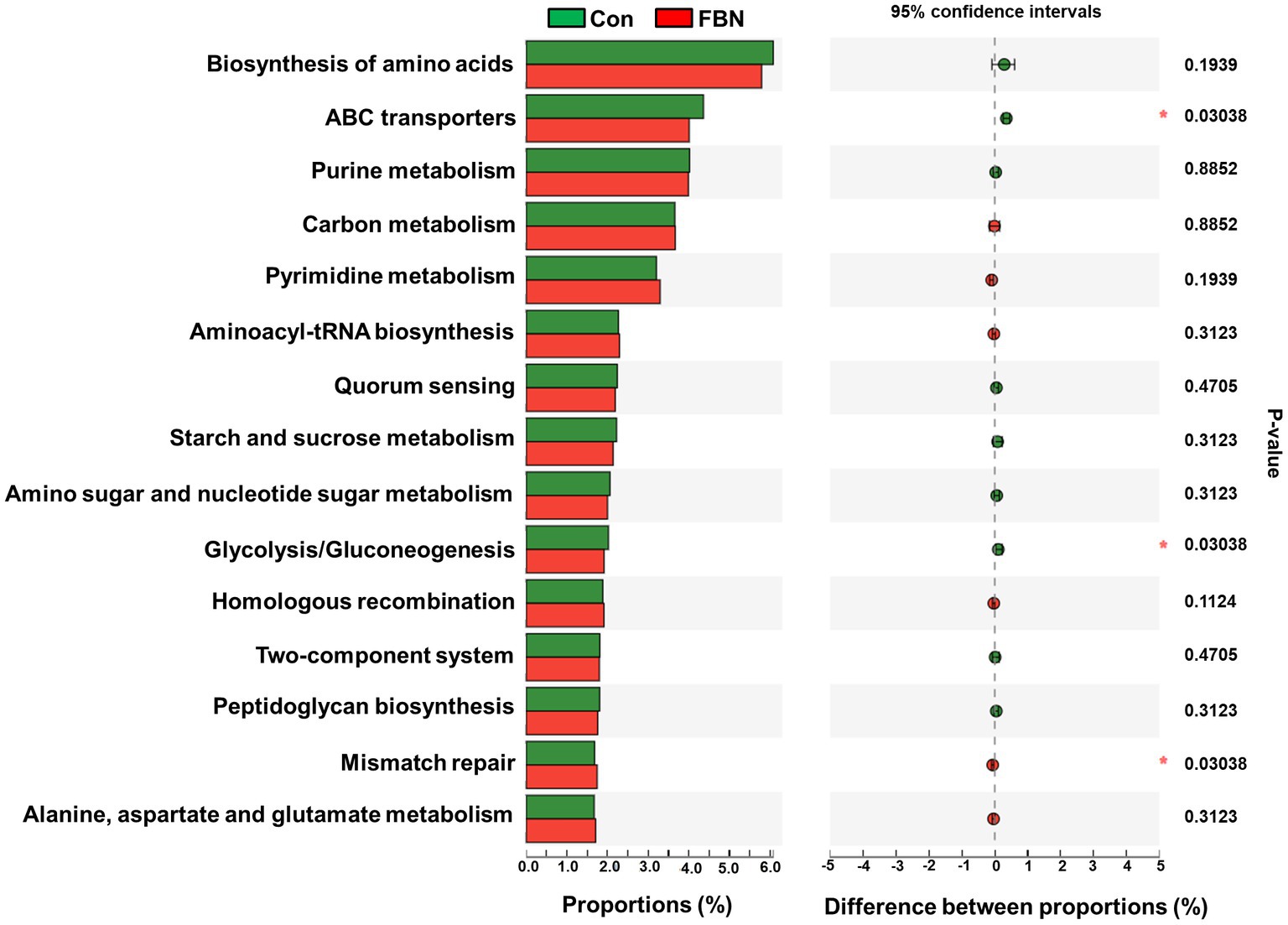 Figura 8. Funciones putativas del microbioma cecal anotadas por la Enciclopedia de Genes y Genomas de Kioto (KEGG) en el nivel 3.
Figura 8. Funciones putativas del microbioma cecal anotadas por la Enciclopedia de Genes y Genomas de Kioto (KEGG) en el nivel 3.
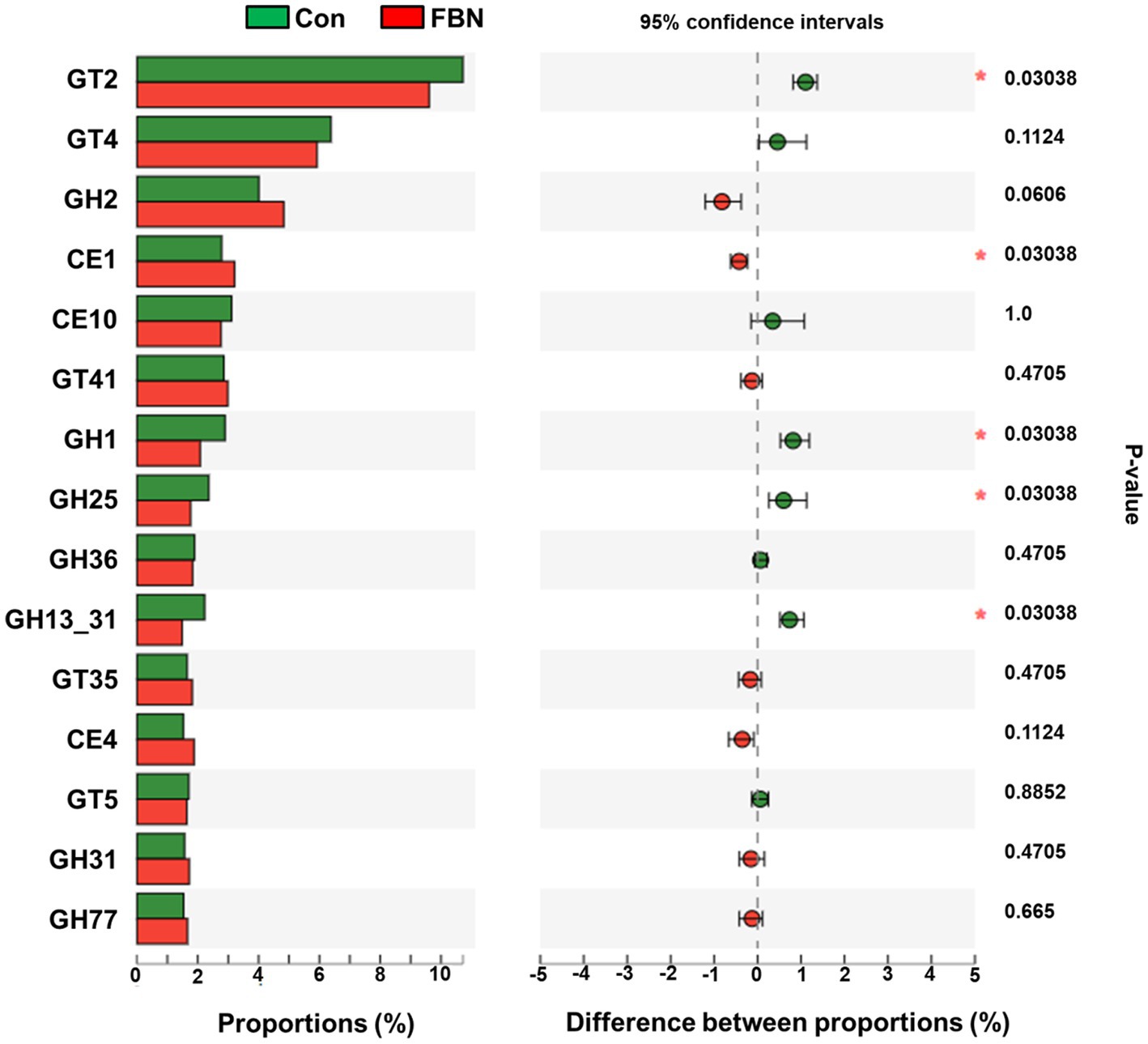 Figura 9. Sumativas de microbios cecales de la enzima carbohidrato activa (CAZyme) a nivel familiar, anotadas utilizando la base de datos CAZy. GT: glicosiltransferasas; GH: glucósidos hidrolasas; CE: esterasas de carbohidratos.
Figura 9. Sumativas de microbios cecales de la enzima carbohidrato activa (CAZyme) a nivel familiar, anotadas utilizando la base de datos CAZy. GT: glicosiltransferasas; GH: glucósidos hidrolasas; CE: esterasas de carbohidratos.
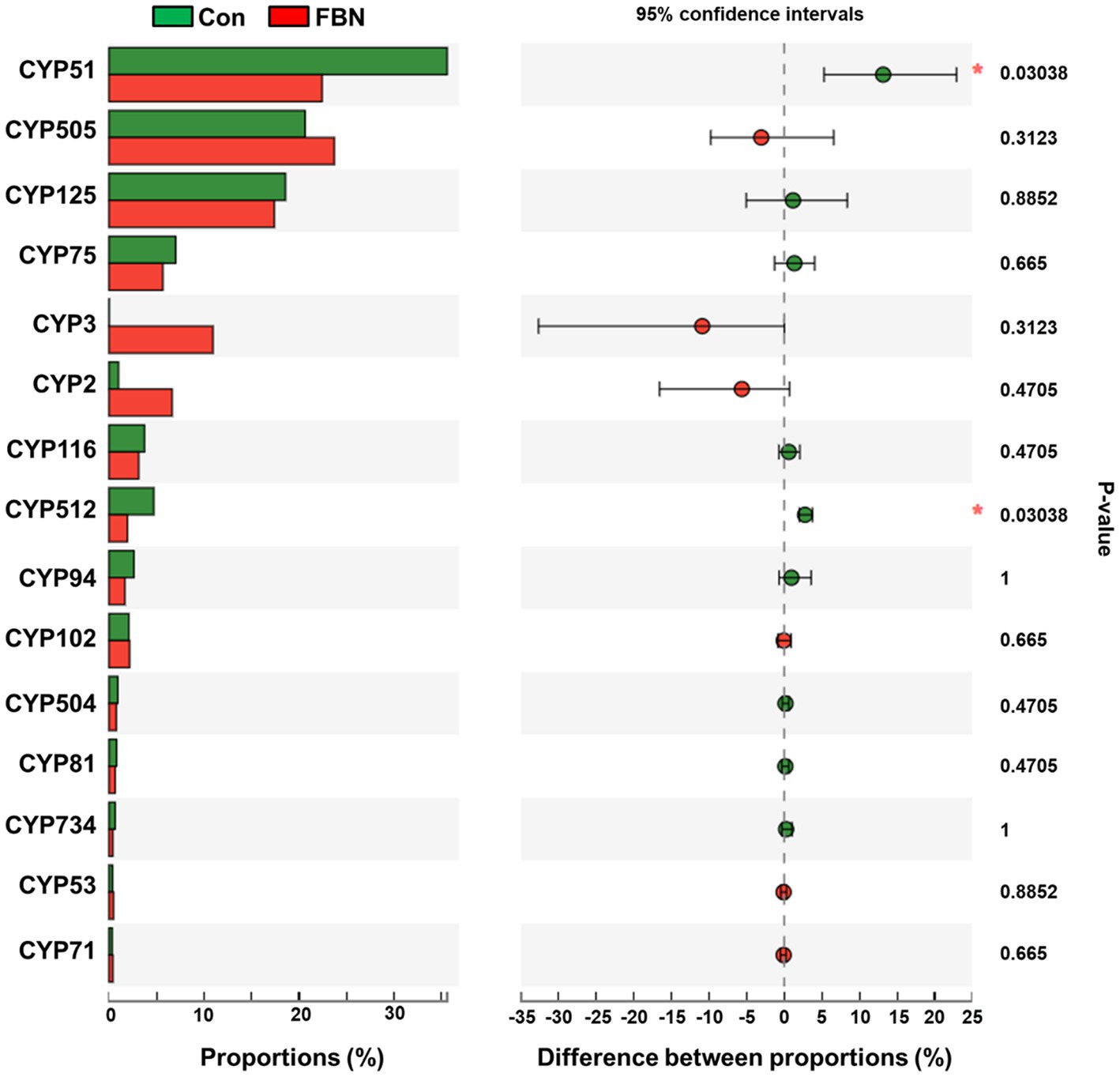 Figura 10. Familia putativa CYP450 de microbios cecales anotados utilizando la base de datos Cytochrome P450 Engineering.
Figura 10. Familia putativa CYP450 de microbios cecales anotados utilizando la base de datos Cytochrome P450 Engineering.
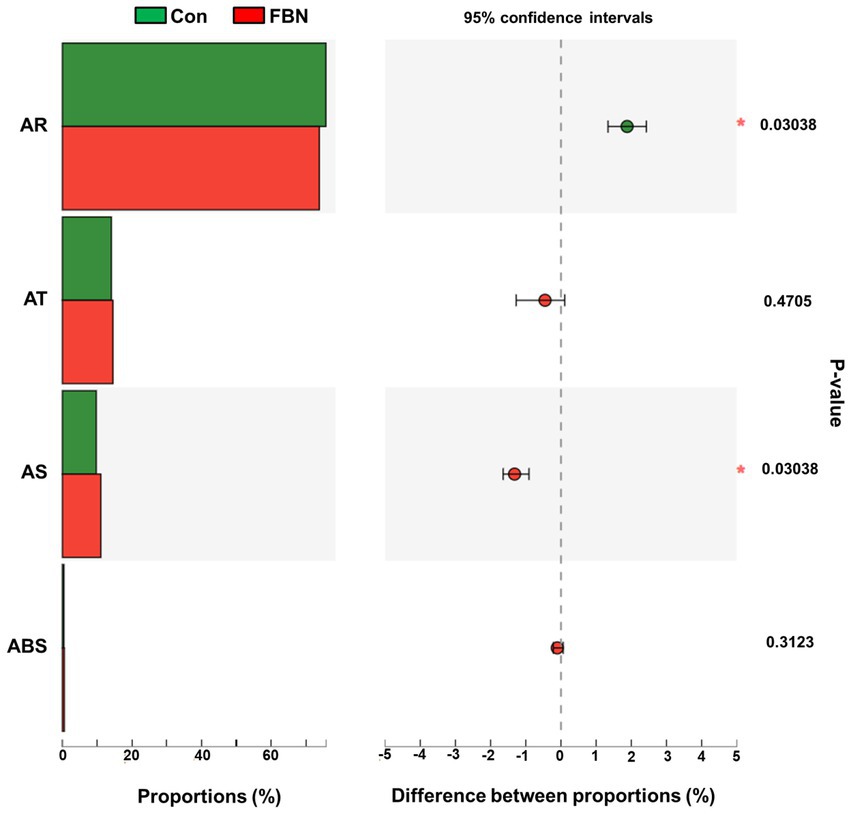 Figura 11. Supuesta resistencia a los antibióticos de microbios cecales a nivel de clase anotada utilizando la base de datos CARD (Comprehensive Antibiotic Resistance Database). AR: Resistencia a los antibióticos; AT: Diana Antibiótico; AS: sensible a los antibióticos; ABS: Biosíntesis de antibióticos.
Figura 11. Supuesta resistencia a los antibióticos de microbios cecales a nivel de clase anotada utilizando la base de datos CARD (Comprehensive Antibiotic Resistance Database). AR: Resistencia a los antibióticos; AT: Diana Antibiótico; AS: sensible a los antibióticos; ABS: Biosíntesis de antibióticos.
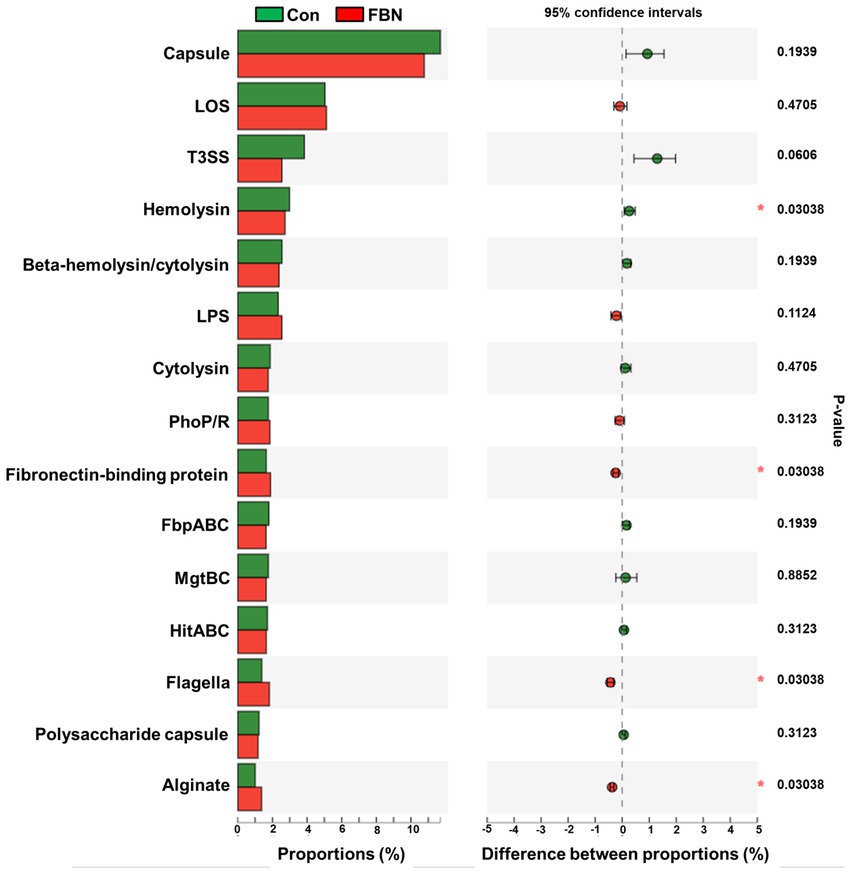 Figura 12. Factores de virulencia putativos de microbios cecales anotados por la Virulence Factor Database (VFDB).
Figura 12. Factores de virulencia putativos de microbios cecales anotados por la Virulence Factor Database (VFDB).
4. Discusión
El uso del ramio como fuente de forraje no convencional en la dieta de los cerdos se ha explorado en algunas áreas del sur de China con el objetivo de reducir la dependencia de la región de la SBM importada (5, 6). Se ha informado que la inclusión de ramio en polvo a menos del 9% en la dieta de cerdos de engorde mejoró parcialmente los rasgos de la canal y la capacidad antioxidante sérica sin afectar el crecimiento (5, 6). Del mismo modo, en este estudio, no observamos ningún efecto perjudicial de FBN sobre las actividades de las enzimas antioxidantes en suero cuando se suplementó al 20% en las dietas de cerdos de engorde. Las enzimas antioxidantes como SOD, CAT y GSH-Px juegan un papel importante en la eliminación de ROS y el mantenimiento de la homeostasis redox (6). El hecho de que el contenido sérico de enzimas antioxidantes no se alterara sugiere que incluso los altos niveles de inclusión de FBN no inducen estrés oxidativo en cerdos de engorde.
En cabras, se ha informado que los altos niveles de inclusión dietética de ramio (hasta el 40%) mejoran las concentraciones de butirato en el rumen sin alterar la diversidad de la microbiota ruminal (3). En comparación con los animales monogástricos, los rumiantes son mejores para digerir el forraje de ramio. Durante la fermentación del ensilaje, el contenido de fibra generalmente se reduce, lo que mejora la digestibilidad del forraje resultante. Esto implica que incluso un alto nivel de inclusión de ramio fermentado puede no ser perjudicial para los cerdos. Sin embargo, cuando se introduce un nuevo ingrediente en la alimentación animal, los efectos sobre la salud intestinal deben evaluarse cuidadosamente. En este estudio, no se observaron cambios en la arquitectura intestinal o la morfología en el grupo de tratamiento con FBN, lo que indica que FBN puede incluirse de manera segura en las dietas de los cerdos de engorde. Dadas sus funciones desintoxicantes, también es importante evaluar el estado del hígado cuando se introduce un nuevo ingrediente alimenticio. AhR ha sido ampliamente estudiado por su papel en la mediación del metabolismo xenobiótico; Sin embargo, también se sabe que es un regulador crítico de la inmunidad y la inflamación (25, 26). Un estudio informó que un metabolito microbiano de polifenoles mejoró la función de barrera intestinal a través de la activación de las vías dependientes del factor eritroide 2 relacionado con AhR/factor nuclear 2 (Nrf2) (27). Nrf2 es un objetivo aguas abajo del AhR y juega un papel vital en el sistema de defensa antioxidante (28). Evaluamos la expresión de genes implicados en la vía AhR-Nrf2, como los implicados en las funciones antioxidantes, inmunes y de barrera, tanto en el hígado como en el íleon. Encontramos que la expresión de los genes AHR e IL6 estaba regulada negativamente en el íleon, lo que puede deberse a la presencia de fitoquímicos desconocidos en el FBN. La IL-6 es una citoquina proinflamatoria y también está involucrada en las respuestas inmunes (29). La disminución observada en la expresión del gen IL6 sugiere que FBN tiene potencial antiinflamatorio.
La microbiota intestinal desempeña un papel fundamental en la digestión y adsorción de nutrientes, la inmunidad gastrointestinal y la renovación de las células epiteliales (30). Muchos factores, tanto intrínsecos como extrínsecos, pueden influir en la diversidad microbiana intestinal, la composición, la función y las actividades metabólicas. Hasta la fecha, ningún estudio ha investigado los efectos de la FBN dietética en la microbiota intestinal de cerdos de engorde. El conocimiento de las alteraciones que ocurren en la composición y las funciones relevantes de la microflora intestinal en respuesta a los cambios en la dieta ayudará en el desarrollo de nuevos forrajes prácticos para cerdos. En este estudio, los cambios inducidos por el tratamiento FBN en la composición de la microbiota cecal se caracterizaron por un mayor número de especies únicas en comparación con el tratamiento control. La FBN dietética no afectó significativamente la composición microbiana a nivel de dominio, reino y filo, lo que sugiere que incluso los altos niveles de inclusión dietética de FBN no alteran la estabilidad microbiana intestinal. A nivel de género, la abundancia de Streptococcus se redujo, mientras que la de Clostridium, Unclassified_f_Lachnospiraceae, Eubacterium, Ruminococcus, Unclassified_o_Clostridiales y el probiótico Roseburia inulinivorans aumentó cuando FBN se incluyó en la dieta de los cerdos. Las especies de Streptococcus, Lactobacillus y Clostridium son prevalentes en especies específicas de cerdos, como lo revela un metanálisis de secuencias de ARNr 16S y metagenómica (31). Las especies de estreptococos como S. alactolyticus son prevalentes en cerdos de China, pero no en cerdos de Francia o Dinamarca, lo que puede atribuirse al uso generalizado de antibióticos en China (31). Se han encontrado varios ARG que confieren resistencia a aminoglucósidos, macrólidos y fluoroquinolonas en el genoma de Streptococcus suis aislados de cerdos enfermos (32). S. suis es un importante patógeno productor de hemolisina de los cerdos (33). La disminución en la abundancia de Streptococcus puede explicar la reducción de la resistencia a los antibióticos y los niveles de hemolisina en el microbioma intestinal de los cerdos alimentados con FBN. Wylensek et al. identificaron varias especies de Clostridium en la microbiota cecal de cerdos, como C. innocuum, C. beijerinckii, C. perfringens, C. cochlearium, C. cadaveris, C. porci, C. celerecrescens y C. scindens (31). Entre estos, C. porci era una especie recientemente identificada, y se encontró un gen codificador de fucosiltransferasa en su genoma (31). Eubacterium y Ruminococcus son géneros intestinales comunes y se han detectado en cerdas tanto en períodos prenatal como postnatal (34). Yang et al. (35) informaron que la abundancia de Eubacterium y Ruminococcus disminuyó en lechones diarreicos, lo que sugiere que un mayor número de los dos géneros puede indicar una microbiota intestinal más saludable en cerdos. Un análisis metagenómico mostró que Roseburia inulinivorans estaba enriquecida en la microbiota intestinal de cerdos alimentados con FBN. Se sabe que R. inulinivorans produce butirato a partir de polisacáridos dietéticos a través de la secreción de fructofuranosidasa, α-amilasa y α-glucanotransferasas (36). Predicción de funciones basada en tLa base de datos KEGG indicó que la función del transportador ABC disminuyó con la suplementación con FBN, lo que es consistente con un informe anterior que muestra que la medicina tradicional china disminuyó significativamente la abundancia de transportadores ABC en ratones (37). Las actividades enzimáticas de los microbios intestinales son muy importantes para la digestión y absorción de nutrientes. Los conocimientos funcionales sobre el microbioma basados en la base de datos CAZy mostraron que las actividades de GT2, GH1, GH13 y GT2 disminuyeron con el tratamiento con FBN. Entre estos, se informó que GT2 es la enzima más abundante (nivel familiar) en el íleon, el ciego y el colon de los cerdos (16). La disminución de las actividades de estas familias de enzimas puede atribuirse a los efectos inhibitorios de algunos fitoquímicos en FBN sobre microbios capaces de producir glucósidos transferasa y enzimas glucósidos hidrolasas, que están involucradas en la glucólisis/gluconeogénesis. El citocromo P450 juega un papel importante en la activación y desintoxicación de drogas, tóxicos ambientales e ingredientes dietéticos. El microbioma intestinal está involucrado en la biotransformación de los xenobióticos en el huésped, ya sea directamente a través de la producción de enzimas que metabolizan los xenobióticos o indirectamente al influir en los receptores del huésped y las vías de señalización a través de la producción de metabolitos (38). La familia CYP51 está ampliamente distribuida en hongos (39). Inesperadamente, la suplementación con FBN en la dieta redujo la abundancia de CYP51 en el microbioma intestinal de los cerdos de engorde. Esto puede deberse a la baja abundancia de hongos en el microbioma intestinal de los animales. Además, AhR puede estar asociado con la abundancia reducida de proteínas de la familia CYP dado que actúa aguas arriba de los genes CYP (27).
5. Conclusión
En conclusión, este estudio fue el primero en explorar los efectos de los altos niveles de inclusión dietética (20%, p/p) de Boehmeria nivea fermentada con Aspergillus niger sobre la composición, estructura y funciones putativas de la microbiota cecal de cerdos de engorde. Los altos niveles de suplementación con FBN en la dieta no afectaron la capacidad antioxidante sérica, la morfología intestinal o la expresión de genes que codifican proteínas antioxidantes, inmunes y relacionadas con la unión estrecha en el hígado, excepto por una disminución observada en los niveles de expresión génica AHR e IL6 en el íleon. El tratamiento con FBN alteró la composición de la microbiota intestinal a nivel de género, como lo demuestra la reducción de la abundancia de Streptococcus y el enriquecimiento de Clostridium, Eubacterium, Ruminococcus y Roseburia inulinivorans. Además, la inclusión de FBN en la dieta redujo las funciones predichas de las glucósidos hidrolasas (GH1, GH13, GH25), que pueden estar asociadas con vías involucradas en la glucólisis/gluconeogénesis. Las funciones génicas relacionadas con la resistencia a los antibióticos y la virulencia basada en hemolisina en la microbiota cecal se atenuaron con el tratamiento con FBN. Estos resultados indican que los altos niveles de inclusión dietética de FBN pueden no afectar la salud intestinal en cerdos de engorde. Sin embargo, los efectos de FBN en el rendimiento del crecimiento, la calidad de la carne y los costos de alimentación requieren más investigación.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio / repositorios y el número (s) de acceso se pueden encontrar en el artículo / material complementario.
Declaración ética
El estudio en animales fue aprobado por todos los procedimientos experimentales que involucran animales fueron aprobados (2015-8A) por el Comité de Cuidado y Uso de Animales del Instituto de Agricultura Subtropical de la Academia China de Ciencias. El estudio se realizó de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones del autor
XL: Curación de datos, Análisis formal, Redacción – borrador original, Metodología, Software. ZZ: Curación de datos, Análisis formal, Redacción – borrador original. FR: Análisis formal, investigación, redacción – borrador original. YJ: Conceptualización, Escritura – revisión y edición, Recursos. S-KK: Conceptualización, Escritura – revisión y edición. K-MN: Redacción – revisión y edición, Curación de datos, Análisis formal, Adquisición de fondos, Redacción – borrador original. XW: Redacción – revisión y edición, adquisición de fondos, conceptualización, administración de proyectos.
Financiación
Este trabajo fue apoyado financieramente por el proyecto piloto de demostración para el Sistema General de Racionamiento de la Academia de Ciencias de Jiangxi (Subvención Nos. 2022YSBG22002 y 2022YJC2002), el proyecto de cooperación de la Academia China de Ciencias y gobiernos locales (Subvención No. 2022-03JXZT-02), el Programa de Investigación Colaborativa Clave de la Alianza de Organizaciones Científicas Internacionales (Subvención No. ANSO-CR-KP-2021-10), y el Fondo de Desarrollo de Ciencia y Tecnología Local de Guías del Gobierno Central de China (Subvención No. 20231zdf03076).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1253778/full#supplementary-material
Notas
1. ^www.majorbio.com
2. ^https://github.com/OpenGene/fastp, versión 0.20.0
3. ^http://bio-bwa.sourceforge.net, versión 0.7.9a
4. ^http://metagene.cb.k.u-tokyo.ac.jp/
5. ^http://www.ncbi.nlm.nih.gov/Taxonomy/taxonomyhome.html/index.cgi?chapter=tgencodes#SG1
6. ^http://www.bioinformatics.org/cd-hit/, versión 4.6.1
7. ^http://soap.genomics.org.cn/, versión 2.21
8. ^http://www.genome.jp/kegg/
9. ^http://www.diamondsearch.org/index.php, versión 0.8.35
10. ^http://www.cazy.org/
11. ^https://card.mcmaster.ca/home
12. ^http://www.mgc.ac.cn/VFs/
Referencias
1. Rehman, M, Gang, D, Liu, QQ, Chen, YL, Wang, B, Peng, DX, et al. Ramie, un cultivo polivalente: aplicaciones potenciales, limitaciones y estrategias de mejora. Ind Crop Prod. (2019) 137:300–7. doi: 10.1016/j.indcrop.2019.05.029
2. Tang, SX, He, Y, Zhang, PH, Jiao, JZ, Han, XF, Yan, QX, et al. La digestión de nutrientes, la fermentación ruminal y el rendimiento como ramio (Boehmeria nivea) se incrementa en las dietas de las cabras. Anim Feed Sci Technol. (2019) 247:15–22. doi: 10.1016/j.anifeedsci.2018.10.013
3. Du, E, Guo, W, Chen, F, Fan, Q, Zhao, N, Zhang, W, et al. Efectos del ramio a diversos niveles sobre la fermentación ruminal y la microbiota ruminal de cabras. Comida Sci Nutr. (2020) 8:1628–35. DOI: 10.1002/FSN3.1449
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Gao, S, Liao, Y, Li, Z, Hou, Z, Zhong, R, and Wu, D. Including ramie (Boehmeria nivea L. gaud) in the diet of dairy cows: effects on production performance, milk composition, rumen fermentation, and nutrient digestion. Ital J Anim Sci. (2020) 19:240–4. doi: 10.1080/1828051X.2020.1726831
5. Li, YH, Liu, YY, Li, FN, Lin, Q, Dai, QZ, Sun, JB, et al. Efectos del polvo de ramio dietético en varios niveles sobre los rasgos de la canal y la calidad de la carne en cerdos de engorde. Carne Sci. (2018) 143:52–9. doi: 10.1016/j.meatsci.2018.04.019
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Li, YH, Liu, YY, Li, FN, Sun, A, Lin, Q, Huang, XG, et al. Efectos del ramio en polvo dietético en varios niveles sobre el rendimiento de crecimiento, la capacidad antioxidante y el perfil de ácidos grasos de cerdos de engorde. J Anim Physiol Anim Nutr. (2019) 103:564–73. doi: 10.1111/jpn.13031
7. Coblentz, WK, and Akins, MS. Silage review: recent advances and future technologies for embaled silages. J Dairy Sci. (2018) 101:4075–92. DOI: 10.3168/JDS.2017-13708
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Bansal, N, Tewari, R, Soni, R, y Soni, SK. Producción de celulasas de aspergillus niger NS-2 en fermentación en estado sólido en residuos de residuos agrícolas y de cocina. Gestión de residuos. (2012) 32:1341–6. doi: 10.1016/j.wasman.2012.03.006
9. Li, C, Zhou, JW, Du, GC, Chen, J, Takahashi, S, and Liu, S. Developing Aspergillus niger as a cell factory for food enzyme production. Biotechnol Adv. (2020) 44:107630. doi: 10.1016/j.biotechadv.2020.107630
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Ma, X, Gao, M, Wang, N, Liu, S, Wang, Q, and Sun, X. Producción de ácido láctico a partir de la cofermentación de residuos de alimentos y sustancia de hongos gastada con celulasa de Aspergillus niger. Bioresour Technol. (2021) 337:125365. doi: 10.1016/j.biortech.2021.125365
11. Xue, PY, Liao, W, Chen, Y, Xie, JH, Chang, XX, Peng, GY, et al. Libera la característica y el mecanismo de los polifenoles unidos de la fibra dietética insoluble de la cáscara de naranja navel a través de la fermentación mixta en estado sólido con Trichoderma reesei y Aspergillus niger. LWT. (2022) 161:113387. doi: 10.1016/j.lwt.2022.113387
12. Lynch, SV, y Pedersen, O. El microbioma intestinal humano en la salud y la enfermedad. N Engl J Med. (2016) 375:2369–79. doi: 10.1056/NEJMra1600266
13. Gresse, R, Chaucheyras-Durand, F, Fleury, MA, Van de Wiele, T, Forano, E, and Blanquet-Diot, S. Gut microbiota dysbiosis in postweaning piglets: understanding the keys to health. Tendencias Microbiol. (2017) 25:851–73. doi: 10.1016/j.tim.2017.05.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Niu, KM, Lee, BJ, Kothari, D, Lee, WD, Hur, SW, Lim, SG, et al. Efecto dietético del alimento acuícola con bajo contenido de harina de pescado sobre la microbiota intestinal en platija de olivo (Paralichthys olivaceus) en diferentes etapas de crecimiento. Microbiología. (2020) 9:e992. DOI: 10.1002/MBO3.992
15. Jovel, J, Patterson, J, Wang, W, Hotte, N, O’Keefe, S, Mitchel, T, et al. Caracterización del microbioma intestinal mediante 16S o metagenómica de escopeta. Microbiol frontal. (2016) 7:459. doi: 10.3389/fmicb.2016.00459
16. Quan, JP, Cai, GY, Yang, M, Zang, ZH, Ding, RR, Wang, XW, et al. Explorar la composición microbiana fecal y las capacidades funcionales metagenómicas asociadas con la eficiencia alimenticia en cerdos comerciales DLY. Microbiol frontal. (2019) 10:52. DOI: 10.3389/fmicb.2019.00052
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Wu, X, Xie, CY, Long, CM, Li, J, Zhou, XH, Fan, ZY, et al. Efectos de un patrón diario de tres comidas con diferentes contenidos de proteínas dietéticas en el rendimiento del crecimiento porcino, los rasgos de calidad de la canal y el músculo. J Sci Food Agric. (2018) 98:415–21. DOI: 10.1002/JSFA.8467
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Wu, X, Gao, LM, Liu, YL, Xie, CY, Cai, LC, Xu, K, et al. La suplementación con uridina en la dieta materna reduce la incidencia de diarrea en lechones al regular la barrera mucosa intestinal y los perfiles de citoquinas. J Sci Food Agric. (2020) 100:3709–18. doi: 10.1002/jsfa.10410
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Chen, SF, Zhou, YQ, Chen, YR y Gu, J. Fastp: un preprocesador FASTQ todo en uno ultrarrápido. Bioinformática. (2018) 34:I884–90. DOI: 10.1093/Bioinformatics/BTY560
20. Li, H, and Durbin, R. Fast and accurate short read alignment with burrows-wheeler transform. Bioinformática. (2009) 25:1754–60. DOI: 10.1093/bioinformatics/btp324
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Noguchi, H, Park, J, and Takagi, T. MetaGene: prokaryotic gene finding from environmental genome shotgun sequences. Ácidos nucleicos res. (2006) 34:5623–30. doi: 10.1093/nar/gkl723
22. Fu, LM, Niu, BF, Zhu, ZW, Wu, ST y Li, WZ. CD-HIT: acelerado para agrupar los datos de secuenciación de próxima generación. Bioinformática. (2012) 28:3150–2. DOI: 10.1093/Bioinformatics/BTS565
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Li, RQ, Li, YR, Kristiansen, K, and Wang, J. SOAP: short oligonucleotide alignment program. Bioinformática. (2008) 24:713–4. DOI: 10.1093/Bioinformatics/BTN025
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Buchfink, B, Xie, C, y Huson, DH. Alineación rápida y sensible de proteínas utilizando DIAMOND. Métodos Nat. (2015) 12:59–60. DOI: 10.1038/NMETH.3176
25. Hooper, LV. AhR lo que comes: vincular dieta e inmunidad. Células. (2011) 147:489–91. doi: 10.1016/j.cell.2011.10.004
26. Zhou, L. AHR function in lymphocytes: emerging concepts. Tendencias Immunol. (2016) 37:17–31. doi: 10.1016/j.it.2015.11.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Singh, R, Chandrashekharappa, S, Bodduluri, SR, Baby, BV, Hegde, B, Kotla, NG, et al. Mejora de la integridad de la barrera intestinal por un metabolito microbiano a través de la vía Nrf2. Nat Commun. (2019) 10:89. DOI: 10.1038/S41467-018-07859-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Kohle, C, y Bock, KW. Activación del receptor de ah acoplado y las baterías de genes Nrf2 por fitoquímicos dietéticos en relación con la quimioprevención. Biochem Pharmacol. (2006) 72:795–805. doi: 10.1016/j.bcp.2006.04.017
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Hirano, T. IL-6 en inflamación, autoinmunidad y cáncer. Int Immunol. (2021) 33:127–48. doi: 10.1093/intimm/dxaa078
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Wang, AR, Ran, C, Ringo, E, y Zhou, ZG. Avances en la investigación de la microbiota gastrointestinal de peces. Rev Aquac. (2018) 10:626–40. doi: 10.1111/raq.12191
31. Wylensek, D, Hitch, TCA, Riedel, T, Afrizal, A, Kumar, N, Wortmann, E, et al. Una colección de aislados bacterianos del intestino del cerdo revela diversidad funcional y taxonómica. Nat Commun. (2020) 11:6389. DOI: 10.1038/S41467-020-19929-W
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Yongkiettrakul, S, Wongsurawat, T, Jenjaroenpun, P, Acheampong, DA, Srimanote, P, Maneerat, K, et al. Secuencias genómicas de cepas de Streptococcus suis resistentes a los antibióticos aisladas de pacientes humanos y cerdos enfermos y asintomáticos en Tailandia. Infectar Genet Evol. (2021) 87:104674. doi: 10.1016/j.meegid.2020.104674
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Jacobs, AAC, Vandenberg, AJG, Baars, JC, Nielsen, B, y Johannsen, LW. Producción de suilisina, la hemolisina activada por tiol de Streptococcus-Suis, por aislados de campo de cerdos enfermos. Veterinario Rec. (1995) 137:295–6. DOI: 10.1136/VR.137.12.295
34. Sun, L, Zhang, Y, Chen, W, Lan, T, Wang, Y, Wu, YB, et al. Los cambios dinámicos de la microbiota intestinal durante el período perinatal en cerdas. Animales. (2020) 10:2254. doi: 10.3390/ani10122254
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Yang, QL, Huang, XY, Zhao, SG, Sun, WY, Yan, ZQ, Wang, PF, et al. Estructura y función de la microbiota fecal en lechones neonatales diarreicos. Microbiol frontal. (2017) 8:502. DOI: 10.3389/fmicb.2017.00502
36. Scott, KP, Martin, JC, Chassard, C, Clerget, M, Potrykus, J, Campbell, G, et al. Substrate-driven gene expression in Roseburia inulinivorans: importance of inducible enzymes in the utilization of inulin and stmid. Proc Natl Acad Sci U S A. (2011) 108:4672–9. DOI: 10.1073/PNAS.1000091107
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Zhang, XP, Yang, YN, Zhang, F, Yu, JQ, Sun, WY, Wang, RQ, et al. Las medicinas tradicionales chinas modulan diferencialmente la microbiota intestinal en función de su naturaleza (Yao-Xing). Fitomedicina. (2021) 85:153496. doi: 10.1016/j.phymed.2021.153496
38. Dempsey, JL, y Cui, JY. El microbioma es un modificador funcional del metabolismo del fármaco P450. Curr Pharmacol Rep. (2019) 5:481–90. DOI: 10.1007/S40495-019-00200-W
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Chen, WP, Lee, MK, Jefcoate, C, Kim, SC, Chen, FS y Yu, JH. Monooxigenasas del citocromo P450 fúngico: su distribución, estructura, funciones, expansión familiar y origen evolutivo. Genoma Biol Evol. (2014) 6:1620–34. doi: 10.1093/gbe/evu132
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: cerdos, metagenómica, Boehmeria nivea , fermentación, microbiota cecal
Cita: Liang X, Zhai Z, Ren F, Jie Y, Kim S-K, Niu K-M y Wu X (2023) Caracterización metagenómica de la comunidad de microbiota cecal y funciones en cerdos de engorde alimentados con Boehmeria nivea fermentada. Frente. Vet. Sci. 10:1253778. doi: 10.3389/fvets.2023.1253778
Recibido: 06 Julio 2023; Aprobado: 05 Septiembre 2023;
Publicado: 28 septiembre 2023.
Editado por:
Tao Wang, Universidad Agrícola de Jilin, China
Revisado por:
Shad Mahfuz, Universidad Agrícola de Sylhet, Bangladesh
Jian Ying Zhang, Universidad de Linyi, China Cheng-long Jin, Academia de Ciencias Agrícolas de Guangdong (GDAAS), China
Derechos de autor © 2023 Liang, Zhai, Ren, Jie, Kim, Niu y Wu. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Kai-Min Niu, niulele88@126.com; Xin Wu, wuxin@isa.ac.cn
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía


 Zhenya Zhai2,3
Zhenya Zhai2,3 Fengyun Ren4
Fengyun Ren4 Yucheng Jie4
Yucheng Jie4 Soo-Ki Kim5
Soo-Ki Kim5 Kai-Min Niu2,3*
Kai-Min Niu2,3* Xin Wu2,3,6*
Xin Wu2,3,6*