Desarrollo de un ensayo de detección visual rápida basado en RPA-CRISPR/Cas12a para el parvovirus porcino 7
 Shubo Wen1,2,3
Shubo Wen1,2,3 Lemuge Ella1,2,3
Lemuge Ella1,2,3 Sheng Dang2,3
Sheng Dang2,3 Ao Liao4
Ao Liao4 Xiaorui Li1
Xiaorui Li1 Shuai Zhang2,3
Shuai Zhang2,3 Canción Yang1,2,3*
Canción Yang1,2,3* Xiangyang Li1,2,3*
Xiangyang Li1,2,3* Jingbo Zhai1,2,3*
Jingbo Zhai1,2,3*- 1Facultad de Ciencia y Tecnología Animal, Universidad Minzu de Mongolia Interior, Tongliao, China
- número arábigoCentro de Investigación Tecnológica para la Prevención y el Tratamiento de la Brucelosis, Tongliao, China
- 3Laboratorio clave de prevención y control de zoonosis en las universidades de la Región Autónoma de Mongolia Interior, Tongliao, China
- 4Guangzhou Yitun Pig Industry Co. Ltd., Guangzhou, China
Introducción: El parvovirus porcino (PPV) es un patógeno importante en la industria porcina, con ocho genotipos, incluido el PPV7, identificados desde su aparición en 2016. Las coinfecciones con virus como el Circovirus Porcino 2 (PCV2) y el Virus del Síndrome Respiratorio y Reproductivo Porcino (PRRSV) plantean graves riesgos para la salud porcina. Por lo tanto, existe una necesidad urgente de métodos de detección rápidos, sensibles y específicos adecuados para su uso en entornos de campo o laboratorios con recursos limitados.
Métodos: Desarrollamos un ensayo basado en CRISPR/Cas12a combinado con amplificación de polimerasa recombinasa (RPA) para la detección rápida de PPV7. Se diseñaron cebadores específicos de RPA y cinco ARN CRISPR (ARNcr) para dirigirse a una región altamente conservada dentro del gen NS1 de PPV7. Se optimizaron las concentraciones de crRNA y ADN monocatenario (ssDNA) para mejorar el rendimiento del ensayo.
Resultados: La optimización del ARNcr identificó al crRNA-05 como el candidato óptimo para la detección de PPV7 basada en Cas12a, ya que todos los ARNcr sintetizados demostraron un rendimiento similar. Se determinó que la concentración óptima de crRNA era de 200 nM, lo que arrojó resultados consistentes en todas las concentraciones probadas. Para la optimización del ssDNA, la señal de fluorescencia más fuerte se logró con 500 nM del receptor de ssDNA FAM-BHQ. El ensayo mostró un límite mínimo de detección de 100 copias/μl para PPV7, confirmado mediante métodos de detección de fluorescencia y flujo lateral. Las pruebas de especificidad indicaron que solo las muestras de ADN PPV7 arrojaron resultados positivos, lo que confirma la precisión del ensayo. En las pruebas de 50 muestras de tejido pulmonar de cerdos enfermos, el ensayo RPA-Cas12a identificó 29 muestras positivas (58%), superando las 22 muestras positivas (44%) detectadas por PCR convencional. Esto pone de manifiesto la capacidad de detección mejorada del método RPA-Cas12a y su utilidad potencial en la vigilancia clínica y el tratamiento de PPV7 en poblaciones porcinas.
Discusión: El ensayo RPA-Cas12a detecta eficazmente el PPV7 en muestras clínicas, lo que mejora la vigilancia y el control de la enfermedad en los cerdos. Su adaptabilidad a entornos con recursos limitados mejora significativamente las estrategias de manejo y prevención de PPV7, apoyando así la salud general y el desarrollo de la industria porcina.
1 Introducción
Los parvovirus porcinos (PPV), miembros de la familia Parvovirinae, son virus de ADN monocatenario pequeños, sin envoltura con un tamaño del genoma que oscila entre aproximadamente 4 y 6,3 kb. Hasta la fecha, se han caracterizado un total de 8 subtipos de VPP (PPV1-PPV8). Taxonómicamente, PPV1 se clasifica dentro del género Protoparvovirus, mientras que los genotipos identificados más recientemente (PPV2-PPV7) se han clasificado en tres géneros distintos: Tetraparvovirus (PPV2-PPV3), Copiparvovirus (PPV4-PPV6) y Chapparvovirus (PPV7), en función de la similitud de la secuencia de aminoácidos de la proteína NS1 (1, 2). En 2022 se descubrió una nueva especie, el PPV8, que actualmente se considera que pertenece al mismo género que el PPV1 original (Protoparvovirus). Sin embargo, su clasificación taxonómica aún no ha sido determinada oficialmente (3). El PPV1, detectado por primera vez en Alemania en 1965 (4, 5), fue el único genotipo de PPV que se ha determinado que está asociado con el fracaso reproductivo en los cerdos. Como PPV recientemente descubierto, el PPV7 se identificó por primera vez en los EE. UU. en 2016 a partir de muestras de hisopo rectal de cerdos sanos utilizando secuenciación de próxima generación (2). Desde entonces, se ha informado de PPV7 en varios países, entre ellos Suecia (6), Polonia (7, 8), Corea (9-11), China (12-17), Brasil (18) y Colombia (19), lo que subraya su distribución mundial y su importancia en el contexto de la salud porcina.
Si bien las manifestaciones clínicas resultantes de las infecciones por PPV2 a PPV8 aún no se comprenden completamente, el PPV7 se ha relacionado con ciertas interacciones con otros virus porcinos. Los estudios han indicado que PPV7 puede provocar un aumento de la viremia de PCV2 en cerdos individuales (8) y elevar los niveles de ácido nucleico viral de PCV3 en el suero de animales infectados (16). Esto sugiere que la PPV7 puede exacerbar los síntomas clínicos asociados con PCV2 o infecciones por PCV3 (9). Además, un estudio realizado en Corea ha informado de una asociación entre el PPV7 y la infección por PRRSV (11). Estos hallazgos ponen de manifiesto el impacto potencial de PPV7 en la gravedad y los resultados de las coinfecciones con otros virus porcinos. Dada la falta de vacunas efectivas o tratamientos específicos para el PPV7, existe una necesidad apremiante de desarrollar métodos de diagnóstico precisos y en tiempo real para facilitar el control y la gestión de la propagación del PPV7 y sus posibles interacciones con otros patógenos porcinos. Estas herramientas de diagnóstico serían cruciales para la intervención oportuna y el manejo de la enfermedad en las poblaciones porcinas.
Los sistemas de proteínas palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) y asociados a CRISPR (Cas) han sido reconocidos por su capacidad para dirigirse y escindir secuencias específicas de ácidos nucleicos guiadas por ARN CRISPR (crRNA) en un proceso conocido como cis-clivage (20, 21). Además de esta escisión dirigida, ciertas proteínas Cas como Cas12a, Cas12b, Cas13a y Cas14 han demostrado la capacidad de transescisión inespecífica de secuencias no diana tras el reconocimiento de una plantilla de ADN diana (22-25). Esta propiedad ha llevado a la aplicación de métodos de detección basados en CRISPR en la identificación de diversos ácidos nucleicos, en particular los de microorganismos patógenos.
Sin embargo, se supone que el sistema CRISPR/Cas12a o Cas13a por sí solo no es lo suficientemente sensible como para reconocer niveles bajos de ácidos nucleicos, por lo que se han realizado esfuerzos para mejorar sus capacidades de detección. En los últimos años se ha perseguido la integración de CRISPR/Cas12a o Cas13a con la reacción en cadena de la polimerasa (PCR) o técnicas de amplificación isotérmica como la amplificación de la polimerasa recombinasa (RPA), la amplificación isotérmica mediada por bucle (LAMP) y la amplificación de cebado cruzado (23, 26-28). El RPA, como método fiable de detección de ácidos nucleicos, podría permitir una amplificación sensible, específica y rápida del ADN isotérmico a una temperatura que oscila entre 37 y 42 °C (29). Los sistemas combinados Cas12a o Cas13a con RPA y una lectura de fluorescencia o tira de flujo lateral con altas sensibilidades, especificidades y convicciones han demostrado ser aplicables a la detección de patógenos (30, 31).
En este estudio, nos propusimos establecer un método de detección visual factible, específico, sensible y confiable basado en RPA-CRISPR/Cas12a para la detección rápida de PPV7.
2 Materiales y métodos
2.1 Muestras clínicas y preparación nucleica
El ADN genómico viral de PPV2-PPV6, PCV2, PCV3, PRV, ADNc de PRRSV se almacenó en nuestro laboratorio. Las muestras de tejido pulmonar de cerdos enfermos se recibieron de diferentes granjas porcinas en el este de la Región Autónoma de Mongolia Interior y se almacenaron en nuestro laboratorio.
2.2 Diseño de objetivos y cebadores PPV7
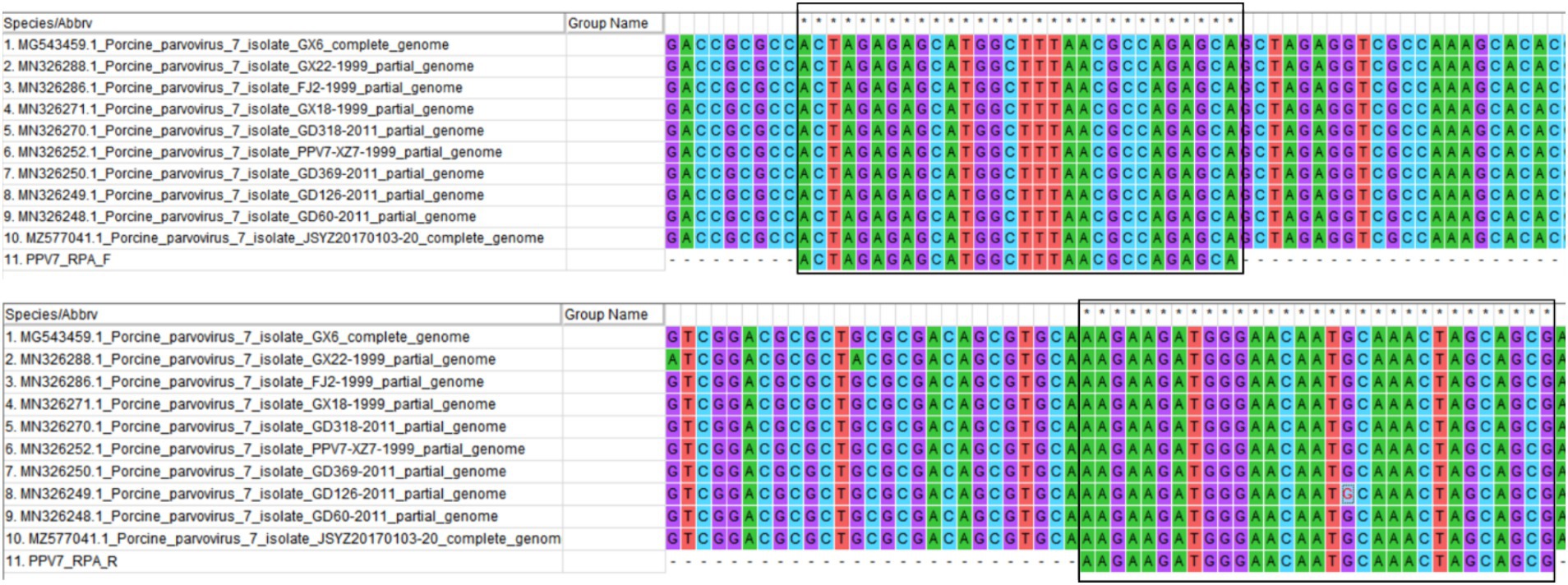
Las secuencias del gen NS1 de las cepas de PPV7 identificadas en diferentes países disponibles en la base de datos del GenBank están altamente conservadas. Se eligió una región conservadora dentro del gen PPV7 NS1 como secuencia objetivo, que luego se sintetizó y clonó en un plásmido pUC57, designado como plásmido pUC57-NS1. Los cebadores de RPA (RPA-F y RPA-R) se diseñaron utilizando Primer Premier 5.0 para dirigirse al plásmido pUC57-NS1, siguiendo los principios del diseño de cebadores de RPA para la amplificación (32). La conservación de los cebadores se evaluó alineándolos con las secuencias NS1 de diez cepas PPV7 seleccionadas aleatoriamente de GenBank (Figura 1). Además, se evaluó la especificidad de los cebadores RPA utilizando Primer-BLAST del NCBI. Los crRNAs fueron diseñados para reconocer regiones específicas dentro del gen PPV7 NS1 presente en el amplicón RPA. Cada crRNA se une a una secuencia diana de 20 pb adyacente a un sitio PAM (TTTN o NAAA). Se diseñaron cinco crRNAs basados en la secuencia del gen PPV7 NS1. Además, se diseñaron un par de cebadores dirigidos a la región NS1 conservada de PPV7 para la detección tradicional de PPV7 por PCR en muestras clínicas. Todos los cebadores y crRNAs enumerados en la Tabla 1 fueron sintetizados por Sangon Biotech (Shanghai, China).

2.3 Ensayo basado en RPA-Cas12a
La plataforma de detección de ácidos nucleicos basada en RPA-CRISPR/Cas12a funciona de la siguiente manera (Figura 2). Después de la amplificación, el producto RPA se emplea para el ensayo de detección mediante CRISPR/Cas12a. Se introduce un ssDNA marcado con una molécula fluorescente verde apagada con fines de detección visual. Cuando la molécula objetivo está presente en el sistema, Cas12a escinde el ssDNA, desencadenando una señal de fluorescencia verde visible. En ausencia de dsDNA derivado del virus, el ssDNA permanece sin escarniar, lo que resulta en la no generación de señales de fluorescencia. Los resultados de la detección se pueden observar directamente a simple vista bajo luz azul o luz ultravioleta después de un período de incubación de aproximadamente 15 minutos.
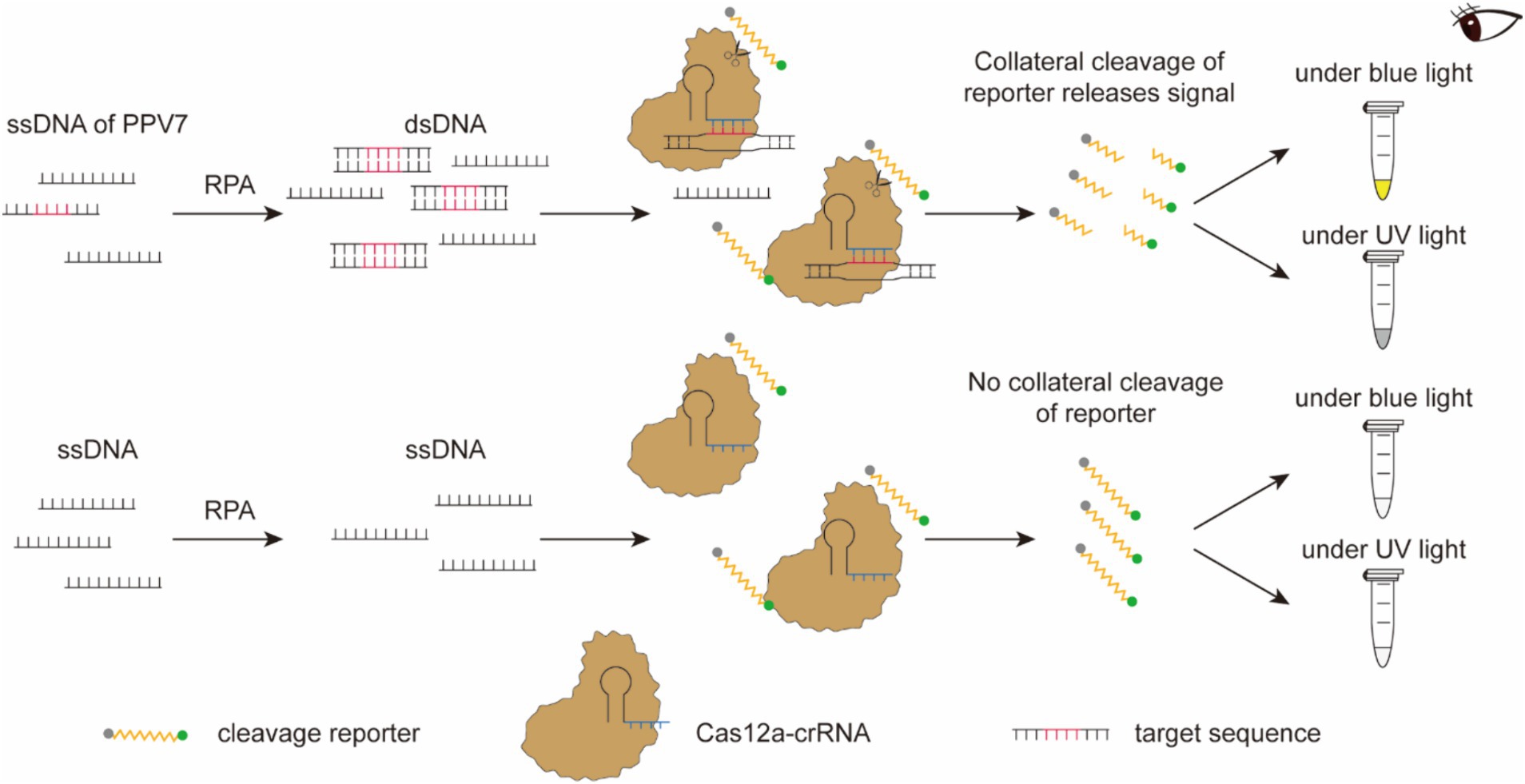
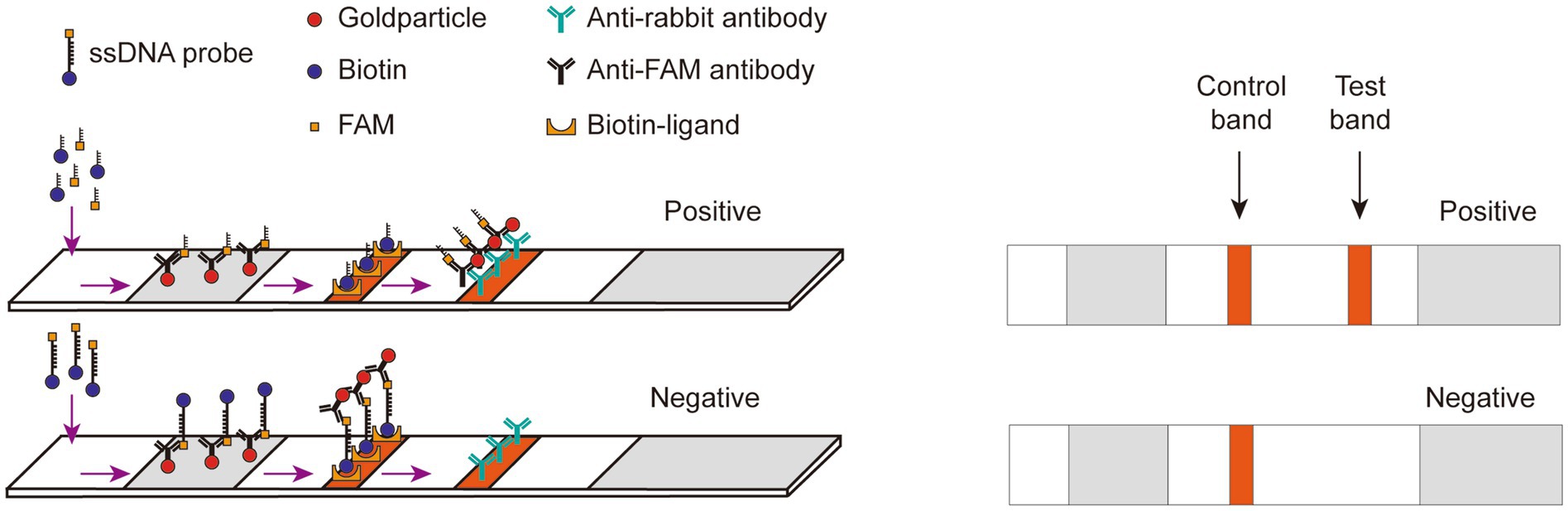
La técnica de detección de flujo lateral podría describirse de la siguiente manera (Figura 3): se colocaron nanopartículas de oro, conjugadas con un anticuerpo anti-FAM, en la almohadilla de unión. La estreptavidina y la IgG se fijaron en la membrana NC, sirviendo como línea de control para la unión específica de biotina y como línea de prueba para la unión específica a un anticuerpo anti-FAM, respectivamente. El reportero de ssDNA de FAM-Biotina se adhirió selectivamente a las nanopartículas de oro para crear un complejo debido al reconocimiento de FAM por el anticuerpo anti-FAM en las nanopartículas de oro. Cuando el Cas12a no degradó el ssDNA, este complejo se unió a la estreptavidina en la línea de control. Por el contrario, cuando el ssDNA se degradó, lo que llevó a la liberación de biotina del complejo, el complejo pudo pasar por la línea de control y unirse al anticuerpo IgG en la línea de prueba.
La reacción de RPA se llevó a cabo utilizando el kit (TwistDx, Reino Unido), con el pUC57-NS1 pLasmid como plantilla. En pocas palabras, la mezcla de reacción de amplificación de RPA consistió en 2,4 μL de cada cebador de RPA (10 μM), 29,5 μL de tampón de rehidratación, 2,5 μL de MgOAc (208 mM), 12,2 μL de ddH2O y 1 μl de ADN. El volumen total de la mezcla fue de 50 μL, que luego se incubó a 39°C durante 20 min. Posteriormente, se añadió una mezcla 1:1 de 50 μL de fenol/cloroformo, y la mezcla se mezcló completamente mediante agitación. Después de la centrifugación a 12000 rpm durante 5 min, se analizaron 5-8 μL del sobrenadante mediante electroforesis en gel de agarosa para su verificación.
En cada ensayo de detección de fluorescencia basado en CRISPR/Cas12a, se utilizó el receptor de ssDNA FAM-BHQ (5′-FAM-TTATT-BHQ-3′). La mezcla de reacción consistió en 200 nM de LbCas12a (Bio-lifesci, Guangzhou, China), 100 nM de crRNA, 0,5 μL de inhibidor de RNasa, 3 μL de tampón 10 × LbCas12a, 200 nM de ssDNA reportero marcado con FAM-BHQ1 y 3 μL de producto RPA. Después de una mezcla completa en un tubo de 1,5 mL utilizando un vórtice, la mezcla con un volumen total de 25 μL se incubó a 45 °C durante 20 minutos. Posteriormente, la reacción se fotografió con un teléfono inteligente bajo luz azul o se fotografió con un sistema de imágenes en gel (Bio-Rad).
En el ensayo de tira de flujo lateral basado en CRISPR/Cas12a para la detección de PPV7, se utilizó la sonda FAM-biotina ssDNA marcada con FAM en el extremo 5′ y biotina en el extremo 3′ (5′-FAM-TTATT-Biotin-3′). Cuando el ADN PPV7 está presente en la reacción, tanto la línea C como la línea T en la tira de flujo lateral serán visibles. Por el contrario, si la muestra es muy positiva, solo será visible la línea T. La reacción se llevó a cabo utilizando plásmidos pUC57-NS1 como plantilla, junto con un reactivo de mezcla basado en Cas-12a que comprendía 200 nM de Lba Cas12a, 100 nM de crRNA, 200 nM de FAM-Biotin ssDNA reporter, 0,5 μL de inhibidor de RNasa, 3 μL de tampón LbCas12a, 3 μL de producto RPA y 0,5 μL de inhibidor de RNasa, con un volumen total de hasta 50 μL utilizando agua sin ARN. La reacción se llevó a cabo a 45°C durante 20 min. Después de la reacción, se insertó la tira de flujo lateral (Bio-lifesci, Guangzhou, China) en la mezcla, y los resultados se observaron visualmente a simple vista en 10 minutos.
2.4 Selección de crRNA y optimización de la concentración
Para seleccionar el crRNA más óptimo para el ensayo basado en RPA-Cas12a, se llevaron a cabo las reacciones descritas anteriormente utilizando cada uno de los 5 crRNAs. El número de copias del plásmido diana de dsDNA se probó a 105 copias/μl. Para optimizar aún más la concentración de crRNA en el ensayo basado en RPA-Cas12a, se llevaron a cabo reacciones adicionales con la misma configuración, pero con diferentes concentraciones de crRNA. En concreto, las concentraciones de crRNA se ajustaron a 200 nM, 400 nM, 1,2 μM, 2 μM y 4 μM, respectivamente.
2.5 Optimización de la concentración del reportero de ssDNA
Para optimizar la concentración reportera de ssDNA, las reacciones se llevaron a cabo como se indicó anteriormente con las siguientes modificaciones: el crRNA se estableció en la concentración optimizada anteriormente. Para cada ensayo de detección de fluorescencia basado en CRISPR/Cas12a, la concentración del receptor de ssDNA FAM-BHQ varió a 25 nM, 50 nM, 100 nM, 200 nM, 250 nM y 500 nM, respectivamente. Del mismo modo, para cada ensayo de tira de flujo lateral basado en CRISPR/Cas12a, la concentración del reportero de ssDNA FAM-Biotina se ajustó a 50 nM, 100 nM, 200 nM, 500 nM y 1 μM, respectivamente.
2.6 Evaluación de la sensibilidad y especificidad de los ensayos basados en RPA-Cas12a
Para evaluar la sensibilidad del ensayo desarrollado basado en RPA-Cas12a, se utilizó un gradiente de concentración de plásmidos pUC57-NS1 que oscilaba entre 108 y 100 copias/μl como plantillas para las reacciones. El límite de detección del ensayo se determinó como el número mínimo de copias de plásmidos objetivo necesarias para producir una señal de fluorescencia o una banda de prueba visible. Para validar la especificidad de los ensayos desarrollados basados en RPA-Cas12a, se analizaron muestras de ácido nucleico de cinco virus porcinos diferentes. Estos incluyeron la genómica viral de PCV2, PCV3, PRV, ADNc de PRRSV y una mezcla de ADN genómico viral de PPV2-PPV6. Estas muestras de ácido nucleico se prepararon y almacenaron en nuestro laboratorio con fines de prueba. Además, se evaluó la sensibilidad y especificidad del método tradicional de detección por PCR utilizando el mismo gradiente de concentración de los plásmidos pUC57-NS1 y las muestras de ácido nucleico empleadas en la evaluación de la sensibilidad y especificidad de los ensayos basados en RPA-Cas12a.
2.7 Detección de ADN genómico de muestras clínicas mediante ensayo basado en RPA-Cas12a
Un total de 50 muestras de tejido pulmonar obtenidas de cerdos enfermos en varias granjas porcinas comerciales en el este de Mongolia Interior fueron homogeneizadas para extraer ADN. Todos los cerdos presentaban diferentes síntomas clínicos, principalmente enfermedades gastrointestinales o respiratorias. El ADN genómico total de las muestras se extrajo utilizando el kit de ADN del virus TIANamp (TIANGEN BIOTECH, Beijing, China) como se describió anteriormente (33). Posteriormente, todas las muestras de ADN se analizaron para detectar la presencia de PPV7 utilizando el ensayo de PCR tradicional o los ensayos basados en RPA-Cas12a desarrollados en este estudio.
2.8 Análisis estadístico
Los datos se analizaron con el software GraphPad Prism, versión 10.1.2. Se utilizó la prueba t de Student de dos colas para analizar la diferencia de fluorescencia entre las diferentes reacciones detectadas mediante el ensayo basado en CRISPR-Cas12a. p < 0,05 se consideró estadísticamente significativo. Además, el análisis estadístico de los resultados de la detección clínica se realizó utilizando el software IBM SPSS Statistics 22 a través de pruebas χ2 y análisis de consistencia Kappa, donde un valor de p de <0,05 indicó una diferencia estadísticamente significativa.
3 Resultados
3.1 Selección y optimización de la concentración de crRNAs para la detección de PPV7 basada en Cas12a
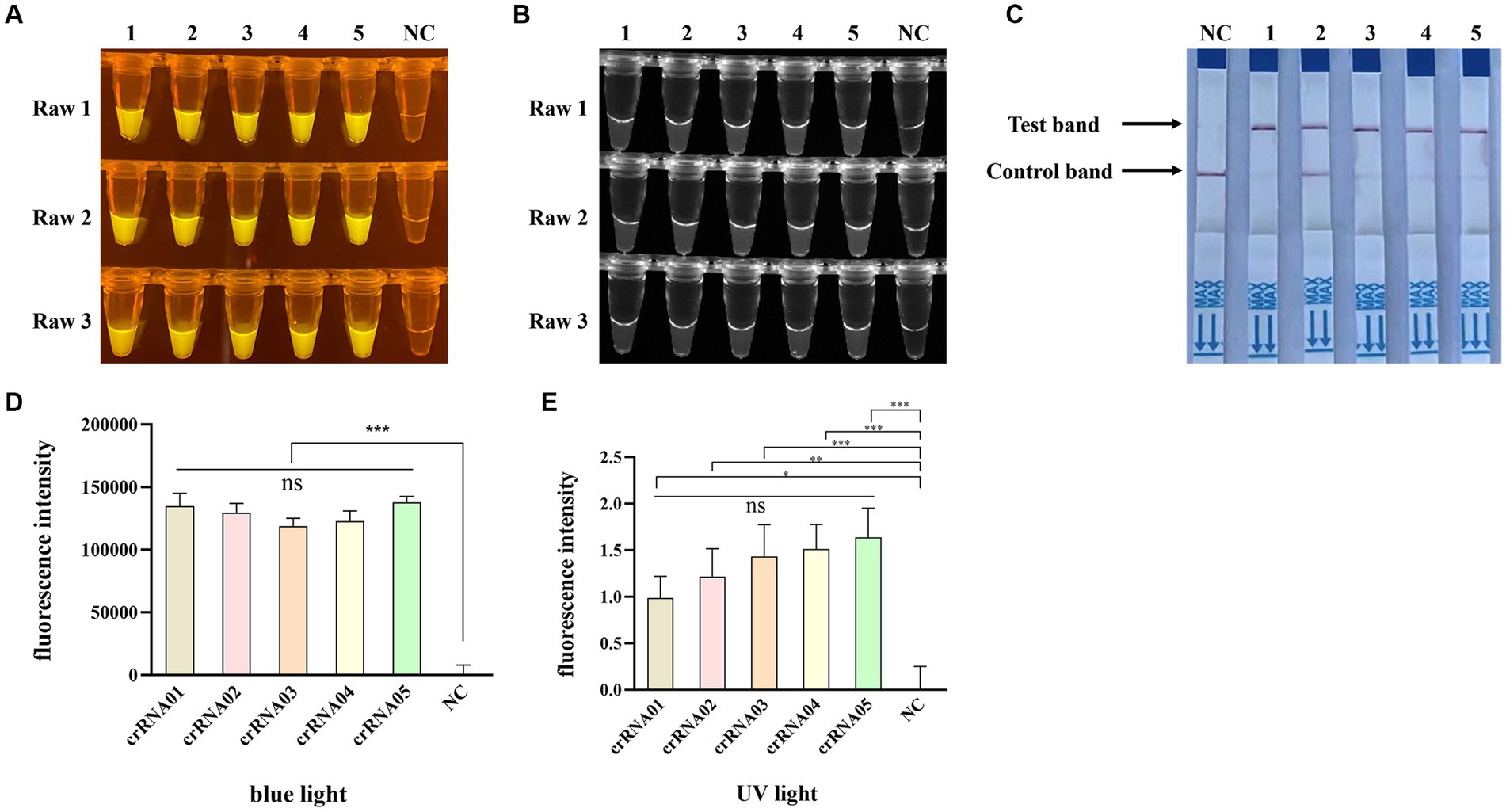
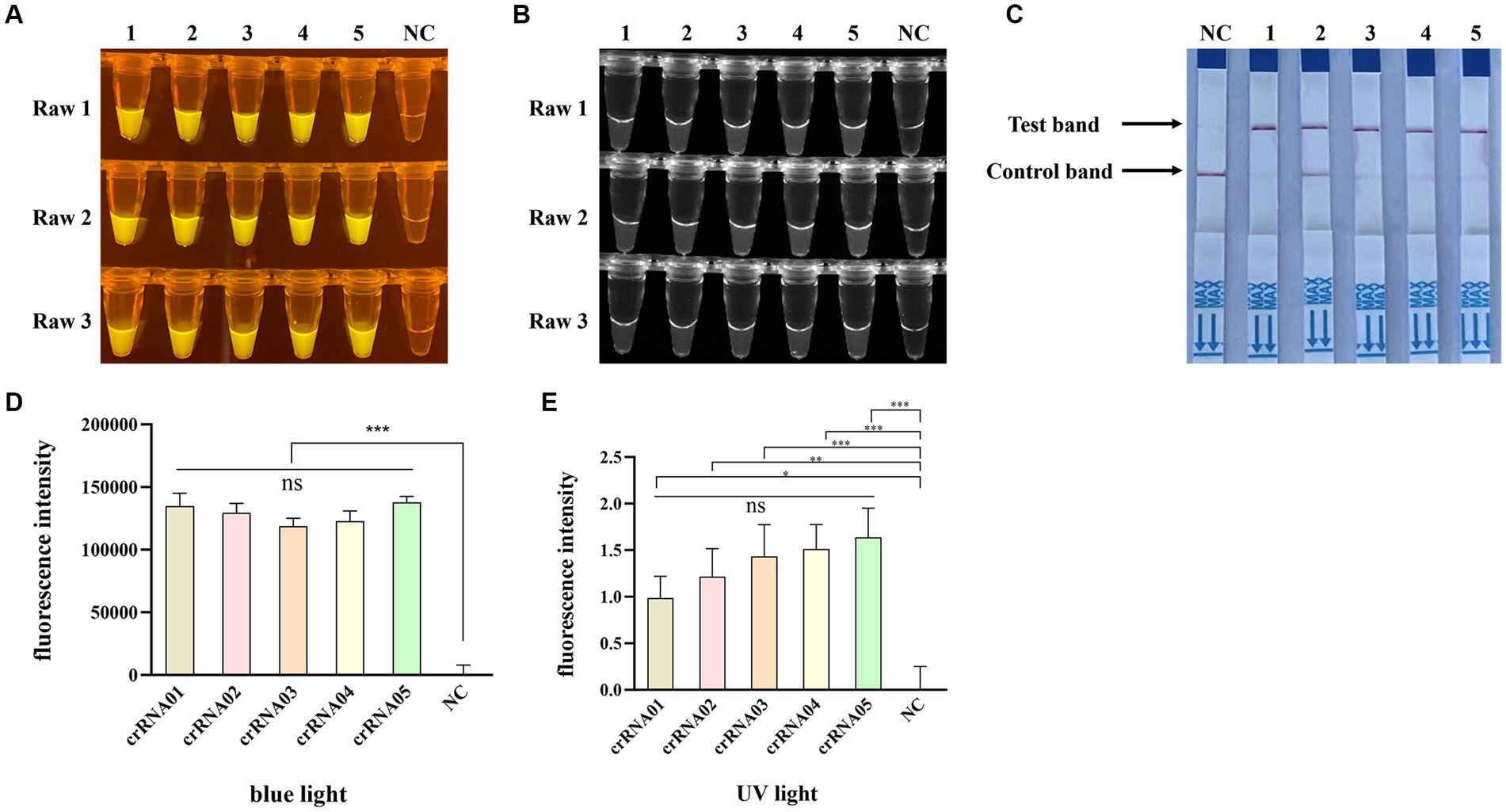
Para obtener un crRNA óptimo para la detección de PPV7 basada en Cas12a, se sintetizaron y probaron un total de 5 crRNAs (designados como crRNA-1 a crRNA-5) utilizando el plásmido pUC57-NS1 que contiene la región diana del gen PPV7 NS1 como plantilla. Cada crRNA se empleó individualmente en ensayos de detección basados en Cas12a para PPV7. Los resultados indicaron que todos los crRNAs producían señales de fluorescencia observables bajo luz azul (Figura 4A) y luz UV (Figura 4B), y mostraban bandas de prueba específicas (Figura 4C). Además, la intensidad fluorescente de las reacciones utilizando crRNA1-5 bajo luz azul (Figura 4D) o luz UV (Figura 4E) calculada con el software Image J fue significativamente mayor que la del control negativo. Sin embargo, no hubo diferencias significativas en la intensidad de fluorescencia o la intensidad de la banda de prueba entre las reacciones que utilizaron los diferentes crRNAs. Sobre la base de estos hallazgos, se eligió arbitrariamente crRNA-05 para experimentos posteriores.
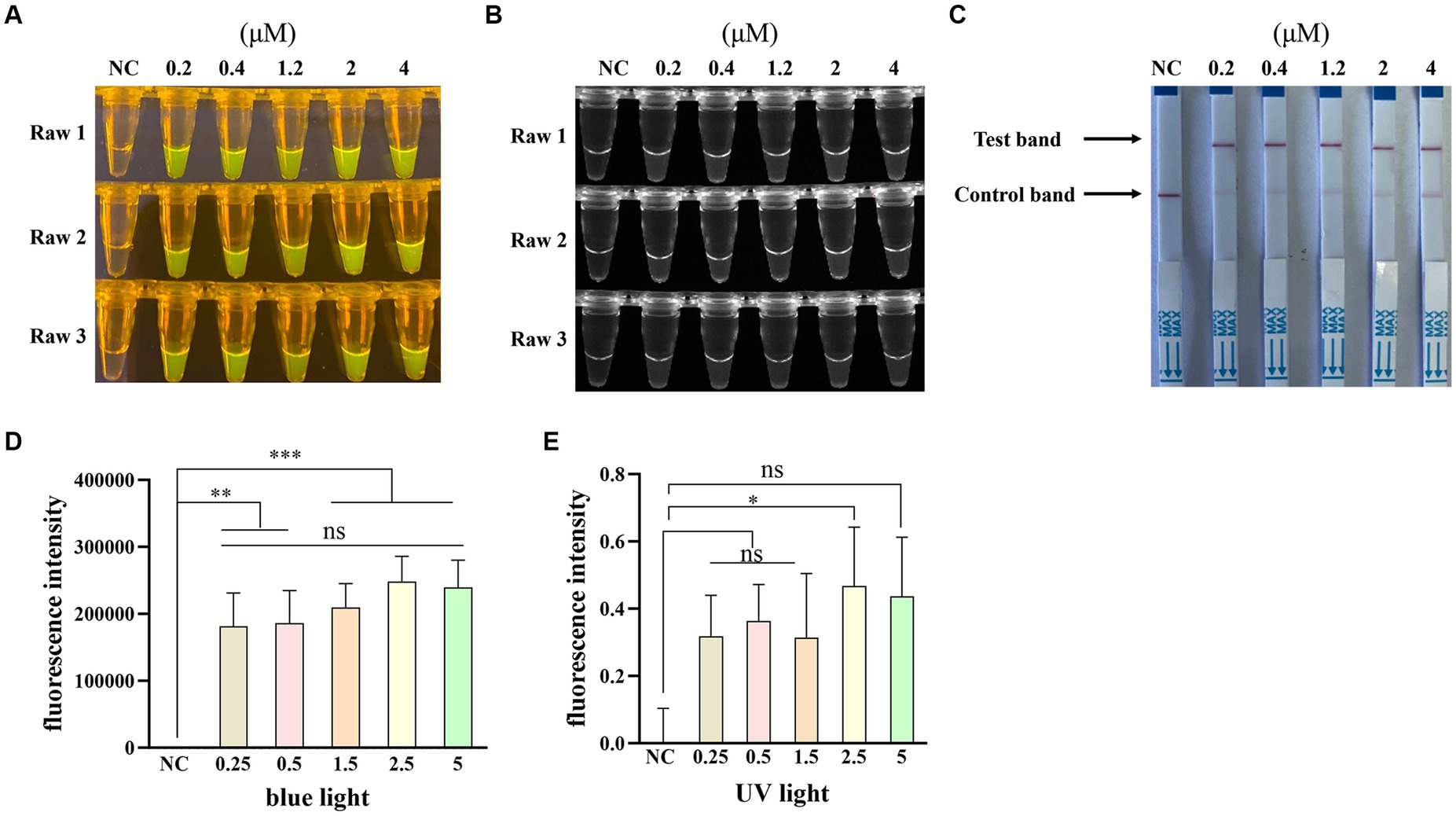
Además, para optimizar la concentración de crRNA para la detección de PPV7 basada en Cas12a, se probaron cinco concentraciones diferentes (0,2 μM, 0,4 μM, 1,2 μM, 2 μM y 4 μM) de crRNA en las reacciones. Los resultados mostraron que todas las concentraciones condujeron a la observación de señales de fluorescencia (Figuras 5A, B) y bandas de prueba específicas (Figura 5C) en las reacciones. Sin embargo, al igual que en las observaciones anteriores con diferentes crRNAs, no hubo diferencias significativas en la intensidad de fluorescencia o la intensidad de la banda de prueba entre las reacciones que utilizaron diferentes concentraciones de crRNA (Figuras 5D,E). Dada la falta de diferencias sustanciales en el rendimiento en función de la concentración de ARNcr, se seleccionó la concentración más baja de 200 nM para experimentos posteriores.
3.2 Optimización de la concentración de ssDNA para la detección de PPV7 basada en Cas12a
Para optimizar la concentración de ADN monocatenario (ssDNA), probamos seis concentraciones diferentes (25 nM, 50 nM, 100 nM, 200 nM, 250 nM y 500 nM) del receptor de ssDNA FAM-BHQ y cinco concentraciones diferentes (50 nM, 100 nM, 200 nM, 250 nM y 500 nM) del reportero de ssDNA FAM-biotina para el ensayo RAA-Cas12a. Las figuras 6A, B demuestran que la reacción utilizando 500 nM del receptor de ssDNA FAM-BHQ exhibió la fluorescencia más fuerte. Además, la intensidad de fluorescencia bajo luz azul (Figura 6D) y luz ultravioleta (Figura 6E) de la reacción utilizando un reportero de ssDNA de 500 nM fue significativamente mayor que la de las otras reacciones, según lo calculado por el software Image J. Sin embargo, la banda de prueba más prominente se observó en reacciones que utilizaron 200 nM, 250 nM y 500 nM del reportero de ssDNA de FAM-biotina (Figura 6C). Por lo tanto, seleccionamos una concentración de 500 nM para el receptor de ssDNA FAM-BHQ y de 200 nM para el reportero de ssDNA de FAM-biotina para experimentos posteriores.
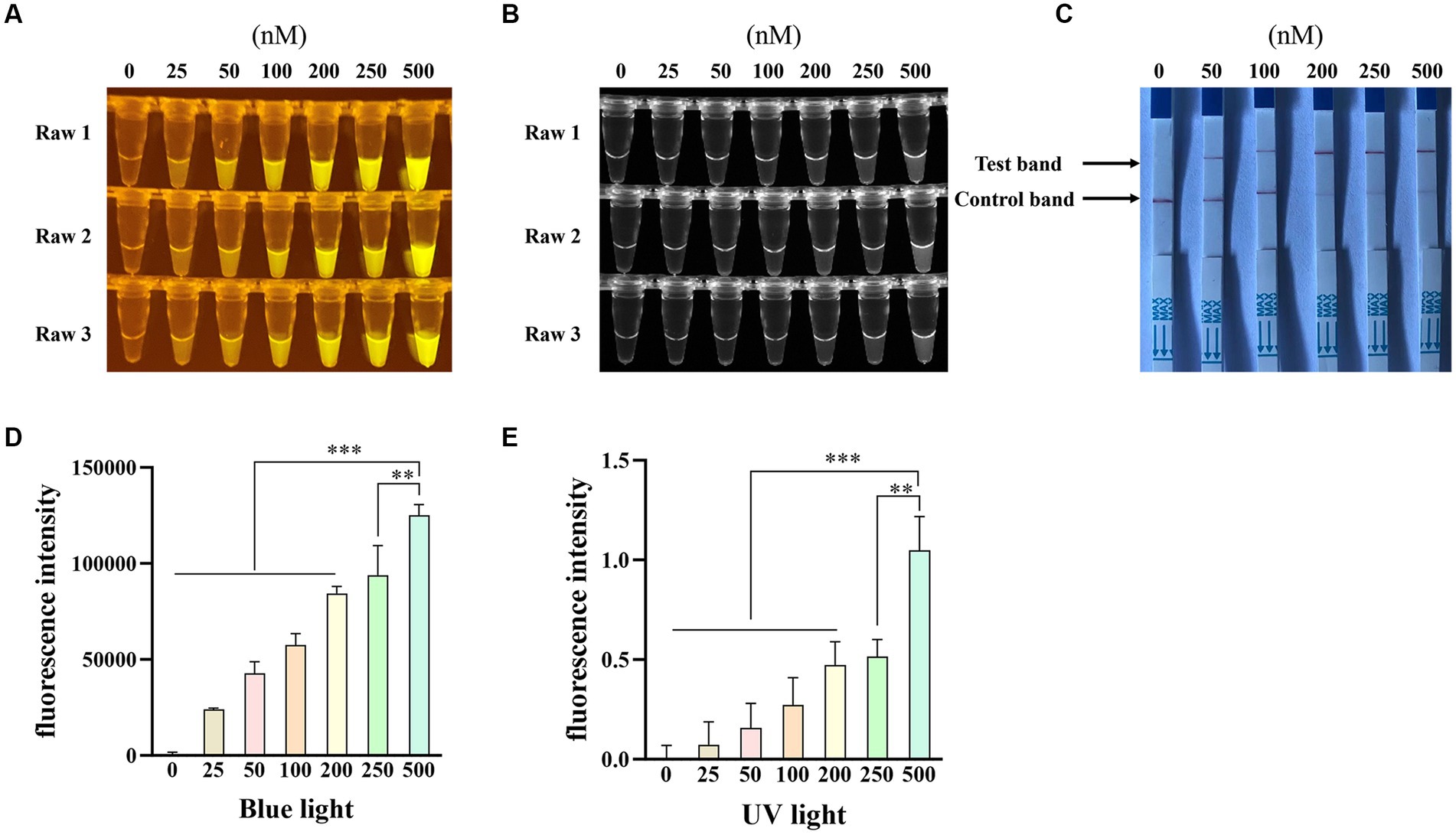
Figura 6. Optimización de la concentración de reporteros de extinción fluorescente de ADN monocatenario (ssDNA-FQ) y biotina para la detección de parvovirus porcino 7 (PPV7) mediante un ensayo basado en CRISPR/Cas12a. El ensayo de detección de fluorescencia basado en CRISPR/Cas12a con concentraciones variables de reporteros ssDNA-FQ bajo luz azul (A) y luz UV (B). Ensayo de tira de flujo lateral realizado con diferentes concentraciones de ssDNA-biotina (C). NC: control negativo (ddH2O). Todos los experimentos se realizaron tres veces. Los valores se presentan como medias ± s.d. (barras de error) (n = 3 repeticiones; * p < 0,05, ** p < 0,01, *** p < 0,001 entre muestras, prueba t de dos muestras). La intensidad fluorescente se calculó mediante el software Image J (D y E).
3.3 Evaluación de la sensibilidad y especificidad de los ensayos basados en RPA-Cas12a
Para evaluar la sensibilidad de detección de la amplificación de la polimerasa recombinasa (RPA) combinada con el ensayo basado en CRISPR/Cas12a, el ADN plasmídico pUC57-NS1 se diluyó en serie diez veces de 1 × 108 a 1 × 100 copias/μl y se utilizó como plantilla. Los resultados indicaron que la sensibilidad del RPA combinada con la detección de fluorescencia basada en CRISPR/Cas12a (Figura 7A) y la detección de tiras de flujo lateral (Figura 7B) para el parvovirus porcino 7 (PPV7) alcanzó un nivel tan bajo como 1 × 102 copias/μl, que es 10 veces más sensible que el método tradicional de detección por PCR (1 × 103 copias/μl, Figura 7C). En el análisis de especificidad de los ensayos PPV7 basados en RPA-Cas12a, solo la muestra de ADN viral PPV7 mostró resultados positivos (Figuras 8A,B). Del mismo modo, en el análisis de especificidad del ensayo de detección de PCR tradicional, solo la muestra de ADN viral PPV7 arrojó resultados positivos (Figura 8C).
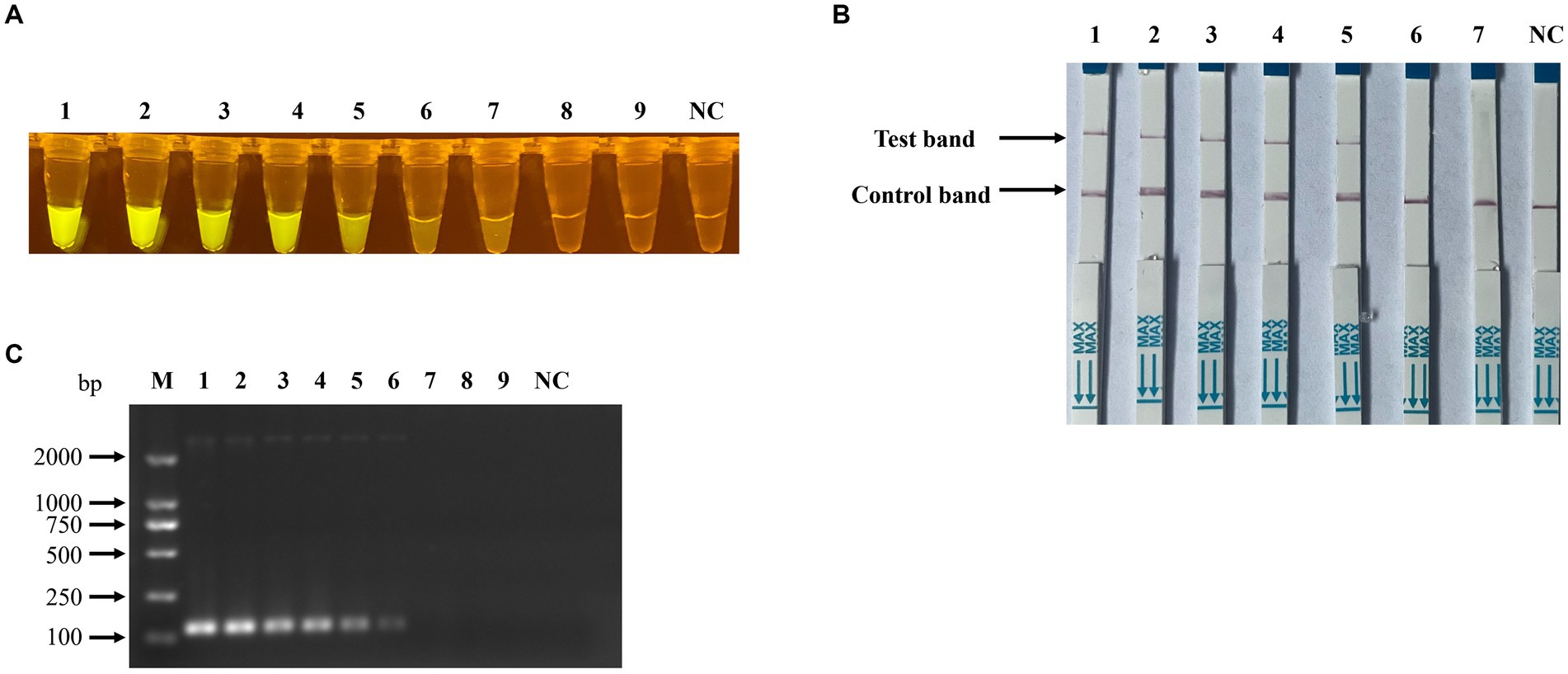
Figura 7. Sensibilidad de la detección basada en RPA-CRISPR/Cas12a para PPV7. Se detectaron diluciones seriadas de 10 veces de plásmidos PPV7 pUC57-NS1 (10 8-100 copias) utilizando el ensayo de detección de fluorescencia basado en RPA-CRISPR/Cas12a (A), o (10 6-10 0 copias) utilizando el ensayo de tira de flujo lateral (B). Además, se evaluó la sensibilidad del método tradicional de detección por PCR utilizado en este estudio utilizando el mismo gradiente de concentración de pUC57-NS1 (C). El n.º 1-9 representa plásmidos con concentraciones de 108-10 0 copias/μl en (A,C), y el n.º 1-7 representa plásmidos con concentraciones de 106-10 0 copias/μl en (B). NC: control negativo (ddH2O).
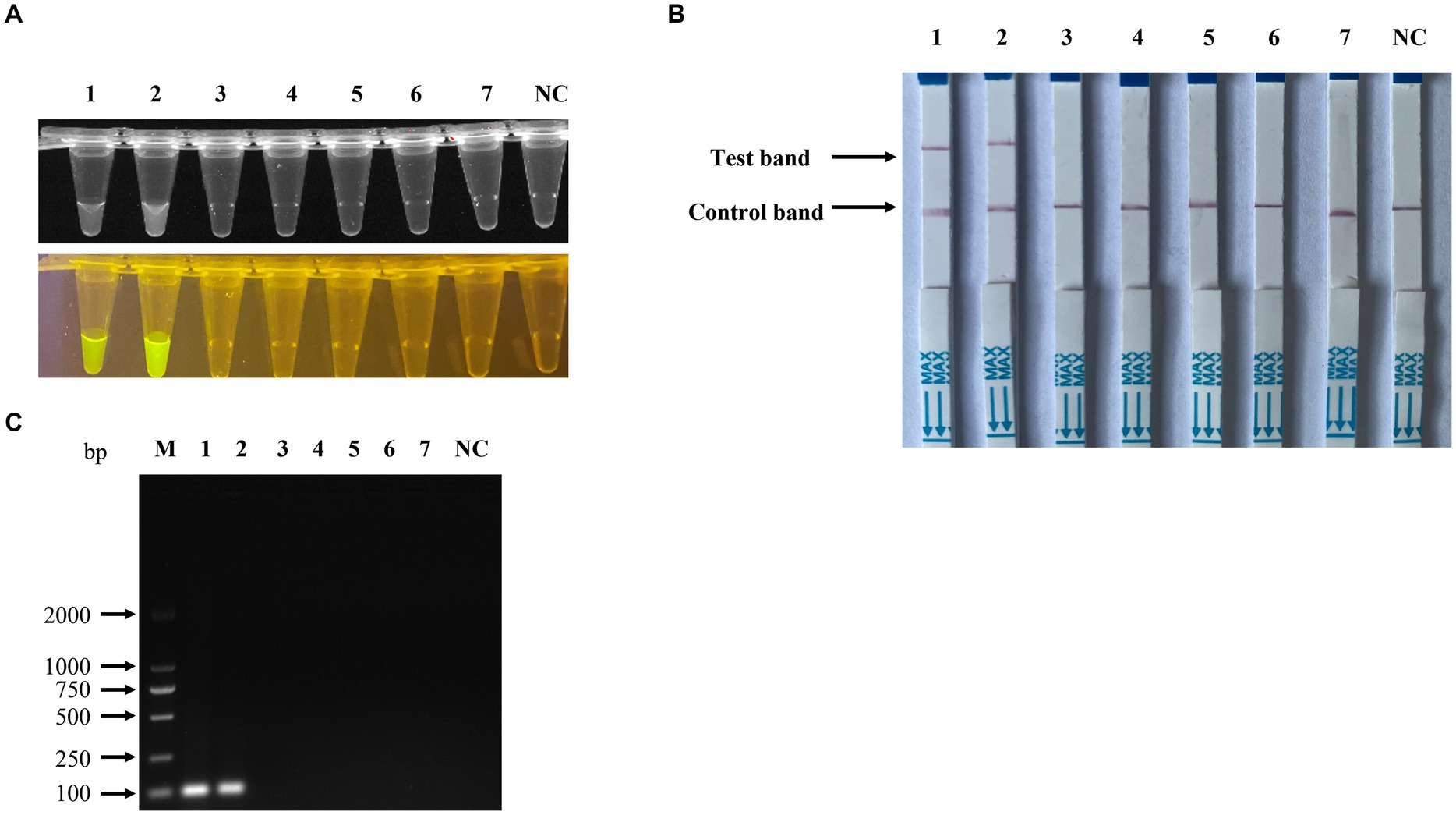
Figura 8. Especificidad de la detección basada en RPA-CRISPR/Cas12a para PPV7. El análisis de especificidad del ensayo de detección de fluorescencia basado en RPA-CRISPR/Cas12a se llevó a cabo utilizando el ADN genómico viral de una mezcla de PPV2-PPV6, PRV, PCV2, PCV3 y ADN complementario (ADNc) de PRRSV en el ensayo de detección de fluorescencia (A), en el ensayo de tira de flujo lateral (B) y en el método tradicional de detección de PCR (C). Los números 1-2 representan los plásmidos PPV7 pUC57-NS1, mientras que los números 3-7 representan el ADN genómico viral de la mezcla PPV2-PPV6, PRV, PCV2, PCV3 y ADNc del PRRSV. NC: control negativo (ddH2O).
3.4 Detección de PPV7 en muestras clínicas mediante ensayos basados en RPA-Cas12a
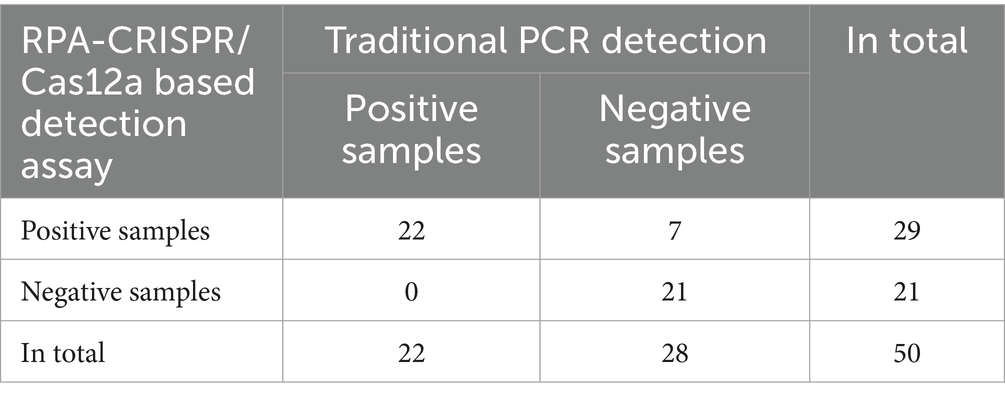
Para validar la precisión de la RPA combinada con los ensayos Cas12a para la detección de muestras clínicas, se utilizaron como muestras de prueba un total de 50 muestras de tejido pulmonar obtenidas de cerdos enfermos. El ADN genómico extraído de estas muestras se cribó y confirmó inicialmente mediante ensayos de PCR convencionales, seguidos de la detección mediante ensayos basados en RPA-CRISPR/Cas12a. Los resultados revelaron que 22 de 50 (44%) muestras dieron positivo con PCR tradicional, mientras que 29 de 50 (58%) muestras dieron positivo con ensayos basados en RPA-CRISPR/Cas12a.
Los resultados de detección de los dos métodos se analizaron utilizando una tabla de contingencia para la prueba χ2 (Tabla 2) y un análisis de consistencia Kappa. Los resultados indicaron una diferencia estadísticamente significativa entre los dos métodos de detección (χ2 = 7, p = 0,016). El análisis de consistencia Kappa arrojó p = 0,000 y un valor de Kappa de 0,725, lo que indica un buen nivel de concordancia entre los dos métodos.
4 Discusión
El parvovirus porcino 7 (PPV7) se identificó por primera vez en los EE. UU. en 2016 como una cepa nueva e infecciosa de PPV, lo que generó preocupaciones sobre su posible impacto en la industria porcina. El virus se ha detectado en varias muestras, incluyendo suero, hisopos fecales, hisopos nasales y tejidos pulmonares, lo que sugiere varios modos de transmisión. Una investigación reciente llevada a cabo en el este de Mongolia Interior por nuestro equipo reveló una notable prevalencia de PPV7 en tejidos pulmonares de cerdos enfermos en múltiples granjas porcinas comerciales, lo que pone de relieve su amplia distribución en la región (17). Debido a su asociación con infecciones exacerbadas como PCV2, PCV3 y PRRSV, el PPV7 representa una amenaza significativa para el bienestar de la industria porcina, lo que pone de manifiesto la necesidad de métodos de detección eficaces para frenar su transmisión.
El ADN genómico monocatenario de PPV7 codifica dos proteínas principales: la proteína no estructural 1 (NS1), crucial para la replicación viral, y la proteína Cap, un componente antigénico clave de PPV7. Si bien los genes NS1 y Cap se han utilizado como objetivos para la detección de PPV7 en varios estudios (7, 10, 12, 17, 34, 35), el gen NS1 generalmente se ve favorecido debido a su mayor conservación en comparación con la secuencia del gen Cap. En diversos estudios se han descrito varios métodos para detectar los ácidos nucleicos PPV7. Kim et al. desarrollaron una técnica de PCR multiplex capaz de detectar PPV1-PPV7, con una sensibilidad de 3 × 103 copias virales/μl específicamente para PPV7 (35). Li et al. introdujeron un ensayo de PCR en tiempo real basado en SYBR Green I que podía detectar tan solo 35,6 copias/μl de molde de ADN (36). Estos métodos de diagnóstico basados en ácidos nucleicos, como la PCR y la RT-PCR, muestran una alta especificidad y sensibilidad en la identificación de PPV7. Sin embargo, su dependencia de equipos costosos los hace inadecuados para laboratorios de campo con recursos limitados, lo que pone de manifiesto la necesidad urgente de un ensayo basado en ácidos nucleicos para la detección de PPV7 que sea fácilmente implementable con una instrumentación mínima.
El sistema CRISPR, que sirve como mecanismo de defensa bacteriana contra ácidos nucleicos extraños, alberga varias proteínas Cas con actividad nucleasa. En particular, Cas12a se destaca por su función de nucleasa dirigida al ADN, lo que la convierte en una herramienta valiosa para la edición de genes a través de las capacidades de escisión y escisión colateral (37). Cas12a se ha mostrado prometedor en la detección de microorganismos patógenos (38), como se ha demostrado en estudios que utilizan una combinación de amplificación de polimerasa recombinasa (RPA) y ensayos de Cas12a para la detección del virus de la viruela del mono y el SARS-CoV-2 (39, 40). A diferencia de otras proteínas Cas como Cas13a y Cas13b, que se centran en secuencias de ARN, la especificidad de Cas12a radica en el reconocimiento y la escisión del ADN, ahorrando así tiempo al eliminar los pasos de transcripción en la construcción de sistemas de detección de ácidos nucleicos (41). Además, la ausencia de reactivos de transcripción en el sistema de reacción Cas12a simplifica los componentes, lo que podría mejorar la precisión. Sin embargo, en comparación con otros patógenos, actualmente existe una brecha en el desarrollo de métodos de diagnóstico basados en CRISPR para los PPV.
En este estudio, se diseñaron cebadores de RPA basados en las secuencias NS1 de aislados de PPV7 disponibles en GenBank, específicamente dirigidos a las regiones más conservadas. Además de los cebadores RPA, el diseño de crRNAs es esencial para el sistema de detección basado en Cas12a. Las proteínas Cas12a requieren crRNAs para unirse e iniciar la trans-escisión del ssDNA no específico, reconociendo las secuencias adyacentes a la secuencia del motivo adyacente del protoespaciador (PAM) (TTTN o AAAN) dentro de la plantilla de ADN diana (23, 26, 42, 43). Hay cinco sitios PAM (TTTN o AAAN) en los amplicones RPA de PPV7, y las secuencias correspondientes que flanquean estos sitios PAM se seleccionaron como crRNAs. La especificidad de estas secuencias de crRNA se validó mediante el análisis Blast frente a GenBank, lo que confirma su especificidad para el gen de la proteína PPV7 NS1.
Nuestro estudio presentó un método de detección rápido y fácil de implementar que requiere un equipo mínimo para monitorear PPV7 en granjas porcinas. Optimizamos meticulosamente las condiciones de reacción, y el ensayo basado en Cas12a demostró una alta sensibilidad con un límite de detección de 100 copias/μl, comparable al ensayo de PCR en tiempo real basado en SYBR Green I realizado por Li et al. (36). Además, la evaluación de la especificidad y las pruebas de muestras clínicas revelaron que el enfoque diagnóstico basado en Cas12a no mostró reactividad cruzada con otros patógenos porcinos. En el ensayo de detección de muestras clínicas, 7 de las 50 muestras mostraron resultados de detección inconsistentes, que se atribuyeron a cargas virales bajas que los métodos tradicionales de PCR no podían detectar. Los ensayos basados en RPA-CRISPR/Cas12a demostraron una tasa de detección mejorada para estas muestras de baja concentración. En general, los resultados sugieren que el ensayo basado en RPA-CRISPR/Cas12a para detectar PPV7 es más sensible que los métodos tradicionales de PCR, lo que lo convierte en una opción fiable para detectar muestras recogidas de varias granjas porcinas.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo, las consultas posteriores pueden dirigirse a los autores correspondientes.
Declaración ética
El estudio en animales fue aprobado por el Comité Institucional de Ética y Cuidado de Animales de la Universidad Minzu de Mongolia Interior. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
SW: Metodología, Software, Redacción – borrador original, Redacción – revisión y edición, Análisis formal. LS: Investigación, Metodología, Redacción – borrador original. SD: Investigación, Metodología, Redacción – borrador original. AL: Investigación, Validación, Redacción – revisión y edición. XiaoL: Investigación, Metodología, Redacción – borrador original. SZ: Investigación, Metodología, Redacción – borrador original. YS: Análisis formal, Obtención de fondos, Validación, Redacción – revisión y edición. XianL: Adquisición de fondos, Recursos, Redacción – revisión y edición. JZ: Adquisición de fondos, Administración de proyectos, Supervisión, Redacción, revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo contó con el apoyo de los principales proyectos de ciencia y tecnología de Mongolia Interior de China (2019ZD006), el proyecto de laboratorio clave para las universidades superiores de la Región Autónoma de Mongolia Interior, el Proyecto de Investigación Científica Básica para las Universidades de la Región Autónoma de Mongolia Interior y el Proyecto de Desarrollo de Jóvenes Talentos Científicos y Tecnológicos 2023 (Equipo de Innovación) para el Proyecto de Investigación de Tecnología Clave de Prevención y Control de Zoonosis (NMGIRT2315). Los jóvenes talentos científicos y tecnológicos de Mongolia Interior (núms. NJYT23095, NJYT22053).
Conflicto de intereses
AL era empleada de Guangzhou Yitun Pig Industry Co., Ltd.
El resto de los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un potencial conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Streck, AF, Canal, CW y Truyen, U. Epidemiología molecular y evolución de los parvovirus porcinos. Infectar Genet Evol. (2015) 36:300–6. doi: 10.1016/j.meegid.2015.10.007
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Palinski, RM, Mitra, N, y Hause, BM. Descubrimiento de un nuevo virus Parvovirinae, parvovirus porcino 7, mediante secuenciación metagenómica de hisopos rectales porcinos. Genes de virus. (2016) 52:564–7. doi: 10.1007/s11262-016-1322-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Guo, Y, Yan, G, Chen, S, Han, H, Li, J, Zhang, H, et al. Identificación y caracterización genómica de un nuevo parvovirus porcino en China. Frente Vet Sci. (2022) 9:1009103. doi: 10.3389/fvets.2022.1009103
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Opriessnig, T, y Halbur, PG. Las infecciones concurrentes son importantes para la expresión de la enfermedad asociada al circovirus porcino. Virus Res. (2012) 164:20–32. doi: 10.1016/j.virusres.2011.09.014
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Allan, GM, Kennedy, S, McNeilly, F, Foster, JC, Ellis, JA, Krakowka, SJ, et al. Reproducción experimental de la enfermedad de emaciación severa por coinfección de cerdos con circovirus porcino y parvovirus porcino. J Comp Pathol. (1999) 121:1–11. doi: 10.1053/jcpa.1998.0295
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Blomström, AL, Ye, X, Fossum, C, Wallgren, P y Berg, M. Caracterización del Virom de las amígdalas de cerdos convencionales y de cerdos libres de patógenos específicos. Virus. (2018) 10:382. doi: 10.3390/v10070382
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Miłek, D, Woźniak, A, y Stadejek, T. Detección y diversidad genética del nuevo parvovirus porcino 7 (PPV7) en granjas porcinas polacas. Res Vet Sci. (2018) 120:28–32. doi: 10.1016/j.rvsc.2018.08.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Miłek, D, Woźniak, A, Podgórska, K, y Stadejek, T. ¿Los parvovirus porcinos 1 a 7 (PPV1-PPV7) tienen un impacto en la viremia del circovirus porcino tipo 2 (PCV2) en cerdos? Microbiol veterinario. (2020) 242:108613. doi: 10.1016/j.vetmic.2020.108613
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Ouh, IO, Park, S, Lee, JY, Song, JY, Cho, IS, Kim, HR, et al. Primera detección y caracterización genética del parvovirus porcino 7 en granjas de cerdos domésticos coreanos. J Vet Sci. (2018) 19:855–7. doi: 10.4142/jvs.2018.19.6.855
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Chung, HC, Nguyen, VG, Huynh, TM, Park, YH, Park, KT y Park, BK. Detección y caracterización genética de parvovirus porcinos basados en PCR en Corea del Sur en 2018. BMC Vet Res. (2020) 16:113. doi: 10.1186/s12917-020-02329-z
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Kim, SC, Kim, JH, Kim, JY, Park, GS, Jeong, CG y Kim, WI. Prevalencia de parvovirus porcino 1 a 7 (PPV1-PPV7) y asociación cofactorial con PCV2 y PRRSV en Corea. BMC Vet Res. (2022) 18:133. doi: 10.1186/s12917-022-03236-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Xing, X, Zhou, H, Tong, L, Chen, Y, Sun, Y, Wang, H, et al. Primera identificación del parvovirus porcino 7 en China. Arch Virol. (2018) 163:209–13. doi: 10.1007/s00705-017-3585-9
13. Wang, W, Cao, L, Sun, W, Xin, J, Zheng, M, Tian, M, et al. Secuencia y análisis filogenético de nuevos aislados de parvovirus porcino 7 de cerdos en Guangxi, China. PLoS Uno. (2019) 14:E0219560. doi: 10.1371/journal.pone.0219560
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Wang, Y, Yang, KK, Wang, J, Wang, XP, Zhao, L, Sun, P, et al. Detección y caracterización molecular del nuevo parvovirus porcino 7 en la provincia de Anhui desde el centro-este de China. Infectar Genet Evol. (2019) 71:31–5. doi: 10.1016/j.meegid.2019.03.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Li, J, Xiao, Y, Qiu, M, Li, X, Li, S, Lin, H, et al. Una investigación sistemática revela un alto estado de coinfección de los tipos 1 a 7 de parvovirus porcino en China entre 2016 y 2020. Microbiol Spectr. (2021) 9:E0129421. doi: 10.1128/Spectrum.01294-21
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Mai, J, Wang, D, Zou, Y, Zhang, S, Meng, C, Wang, A, et al. Alto estado de coinfección del nuevo parvovirus porcino 7 con el circovirus porcino 3 en cerdas que experimentaron fallos reproductivos. Frente Vet Sci. (2021) 8:695553. doi: 10.3389/fvets.2021.695553
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Wen, S, Song, Y, Lv, X, Meng, X, Liu, K, Yang, J, et al. Detección y caracterización molecular del parvovirus porcino 7 en la región autónoma oriental de Mongolia Interior. China Frente Vet Sci. (2022) 9:930123. doi: 10.3389/fvets.2022.930123
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Da Silva, MS, Budaszewski, RF, Weber, MN, Cibulski, SP, Paim, WP, Mósena, ACS, et al. El viroma hepático de cerdos sanos revela diversos genomas virales pequeños de ssDNA. Infectar Genet Evol. (2020) 81:104203. doi: 10.1016/j.meegid.2020.104203
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Vargas-Bermúdez, DS, Rendón-Marín, S, Ruiz-Sáenz, J, Mogollón, D, y Jaime, J. El primer reporte de parvovirus porcino 7 (PPV7) en Colombia demuestra la presencia de variantes asociadas a modificaciones a nivel de la proteína VP2-cápside. PLoS Uno. (2021) 16:e0258311. doi: 10.1371/journal.pone.0258311
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Cong, L, Ran, FA, Cox, D, Lin, S, Barretto, R, Habib, N, et al. Ingeniería genómica multiplexada mediante sistemas CRISPR/Cas. Ciencia. (2013) 339:819–23. doi: 10.1126/ciencia.1231143
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Jinek, M, Chylinski, K, Fonfara, I, Hauer, M, Doudna, JA, y Charpentier, E. Una endonucleasa de ADN guiada por ARN dual programable en inmunidad bacteriana adaptativa. Ciencia. (2012) 337:816–21. doi: 10.1126/ciencia.1225829
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Abudayyeh, OO, Gootenberg, JS, Essletzbichler, P, Han, S, Joung, J, Belanto, JJ, et al. Segmentación de ARN con CRISPR-Cas13. Naturaleza. (2017) 550:280–4. doi: 10.1038/nature24049
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Chen, JS, Ma, E, Harrington, LB, Da Costa, M, Tian, X, Palefsky, JM, et al. La unión a dianas CRISPR-Cas12a desencadena una actividad indiscriminada de DNasa monocatenaria. Ciencia. (2018) 360:436–9. doi: 10.1126/ciencia.aar6245
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Harrington, LB, Burstein, D, Chen, JS, Paez-Espino, D, Ma, E, Witte, IP, et al. Destrucción programada de ADN por enzimas CRISPR-Cas14 en miniatura. Ciencia. (2018) 362:839–42. doi: 10.1126/ciencia.aav4294
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Yang, H, Gao, P, Rajashankar, KR y Patel, DJ. Reconocimiento y escisión del ADN diana dependiente de PAM por la endonucleasa C2c1 CRISPR-Cas. Célula. (2016) 167:1814-28.e12. doi: 10.1016/j.cell.2016.11.053
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Gootenberg, JS, Abudayyeh, OO, Kellner, MJ, Joung, J, Collins, JJ y Zhang, F. Plataforma de detección de ácidos nucleicos multiplexada y portátil con Cas13, Cas12a y Csm6. Ciencia. (2018) 360:439–44. doi: 10.1126/ciencia.aaq0179
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Myhrvold, C, Freije, CA, Gootenberg, JS, Abudayyeh, OO, Metsky, HC, Durbin, AF, et al. Diagnóstico viral desplegable en campo utilizando CRISPR-Cas13. Ciencia. (2018) 360:444–8. doi: 10.1126/ciencia.aas8836
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Shan, Y, Zhou, X, Huang, R y Xing, D. Alta fidelidad y cuantificación rápida de miRNA que combina la programabilidad del crRNA y la actividad trans-escisión de CRISPR / Cas13a. Químico Anal. (2019) 91:5278–85. doi: 10.1021/acs.analchem.9b00073
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Kanitchinda, S, Srisala, J, Suebsing, R, Prachumwat, A, y Chaijarasphong, T. Ensayo de escisión fluorescente CRISPR-Cas junto con amplificación de polimerasa recombinasa para la detección sensible y específica de Enterocytozoon hepatopenaei. Biotechnol Rep (Amst). (2020) 27:E00485. doi: 10.1016/j.btre.2020.e00485
30. Lu, S, Li, F, Chen, Q, Wu, J, Duan, J, Lei, X, et al. Detección rápida del virus de la peste porcina africana mediante diagnósticos portátiles en papel basados en Cas12a. Celular Discov. (2020) 6:18. doi: 10.1038/s41421-020-0151-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Li, F, Ye, Q, Chen, M, Zhou, B, Zhang, J, Pang, R, et al. Un biosensor electroquímico ultrasensible basado en CRISPR/Cas12a para la detección de Listeria monocytogenes. Bioelectrón Biosens. (2021) 179:113073. doi: 10.1016/j.bios.2021.113073
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Piepenburg, O, Williams, CH, Stemple, DL, y Armes, NA. Detección de ADN mediante proteínas de recombinación. PLoS Biol. (2006) 4:E204. doi: 10.1371/journal.pbio.0040204
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Wen, S, Sun, W, Li, Z, Zhuang, X, Zhao, G, Xie, C, et al. Detección de circovirus porcino 3 en Guangxi, China. Transbound emerg dis. (2018) 65:27–31. doi: 10.1111/tbed.12754
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Wang, Y, Zhang, D, Bai, C, Li, M, Liu, X, Peng, M, et al. Uso de la amplificación isotérmica mediada por bucle para la detección visual del parvovirus porcino 7. 3. Biotecnología. (2021) 11:111. doi: 10.1007/s13205-020-02623-5
35. Kim, SC, Jeong, CG, Nazki, S, Lee, SI, Baek, YC, Jung, YJ, et al. Evaluación de un método de PCR múltiplex para la detección de parvovirus porcino tipos 1 a 7 utilizando varias muestras de campo. PLoS Uno. (2021) 16:e0245699. doi: 10.1371/journal.pone.0245699
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Li, YD, Yu, ZD, Bai, CX, Zhang, D, Sun, P, Peng, ML, et al. Desarrollo de un ensayo de PCR en tiempo real SYBR green I para la detección del nuevo parvovirus porcino 7. Pol J Vet Sci. (2021) 24:43–9. doi: 10.24425/pjvs.2021.136791
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Nguyen, LT, Smith, BM y Jain, PK. La mejora de la actividad de transescisión de Cas12a con crRNA diseñado permite la detección de ácidos nucleicos amplificados. Nat Commun. (2020) 11:4906. doi: 10.1038/s41467-020-18615-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Wang, R, Qian, C, Pang, Y, Li, M, Yang, Y, Ma, H, et al. opvCRISPR: plataforma RT-LAMP-CRISPR visual de un solo recipiente para la detección del SARS-cov-2. Bioelectrón Biosens. (2021) 172:112766. doi: 10.1016/j.bios.2020.112766
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Yu, T, Rong, Z, Gu, Z, Wei, H, Wang, Y, Song, R, et al. Detección del virus de la viruela del mono basada en una estrategia RPA-CRISPR/Cas12a conveniente y sensible de un solo paso. RSC Adv. (2024) 14:14775–83. doi: 10.1039/D4RA02049A
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Zeng, Q, Zhou, M, Deng, W, Gao, Q, Li, Z, Wu, L, et al. Detección sensible y visual del SARS-CoV-2 mediante el ensayo de un solo paso RPA-Cas12a con crRNA modificado con ssDNA. Chim Acta Anal. (2024) 1309:342693. doi: 10.1016/j.aca.2024.342693
41. Yin, K, Ding, X, Li, Z, Zhao, H, Cooper, K y Liu, C. Sistema dinámico de reacción acuosa multifásica para el diagnóstico molecular cuantitativo y ultrasensible basado en CRISPR-Cas12a en un solo recipiente. Químico Anal. (2020) 92:8561–8. doi: 10.1021/acs.analchem.0c01459
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Yan, WX, Hunnewell, P, Alfonse, LE, Carte, JM, Keston-Smith, E, Sothiselvam, S, et al. Sistemas CRISPR-Cas de tipo V funcionalmente diversos. Ciencia. (2019) 363:88–91. doi: 10.1126/ciencia.aav7271
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: CRISPR/Cas12a, Parvovirus Porcino 7, detección, visual, PCR
Cita: Wen S, She L, Dang S, Liao A, Li X, Zhang S, Song Y, Li X y Zhai J (2024) Desarrollo de un ensayo de detección visual rápida basado en RPA-CRISPR/Cas12a para el parvovirus porcino 7. Frente. Vet. Sci. 11:1440769. doi: 10.3389/fvets.2024.1440769
Recibido: 30 de mayo de 2024; Aceptado: 20 de agosto de 2024;
Publicado: 09 de septiembre de 2024.
Editado por:
Juan Manuel Sanhueza, Universidad Católica de Temuco, Chile
Revisado por:
Jinyang Zhang, Universidad de Ciencia y Tecnología de Kunming, China
Kai Zhao, Universidad de Taizhou, China
Derechos de autor © 2024 Wen, ella, Dang, Liao, Li, Zhang, Song, Li y Zhai. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Atribución (CC BY).
*Correspondencia: Yang Song, yangyang2130@126.com; Xiangyang Li, 187101414@qq.com; Jingbo Zhai, jbzhai@imun.edu.cn
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía