Detección rápida del virus de la leucemia aviar mediante tira reactiva de flujo lateral basada en CRISPR/Cas13a
 Jing Li1†
Jing Li1† Zichuang Zhang2†
Zichuang Zhang2† Zongshu Zhang1
Zongshu Zhang1 Xi Chen1
Xi Chen1 Chunguang Wang1
Chunguang Wang1 Xianghe Zhai1
Xianghe Zhai1 Corbata Zhang1*
Corbata Zhang1*- 1Facultad de Medicina Veterinaria, Universidad Agrícola de Hebei, Baoding, China
- número arábigoInstituto de Ciencias Animales y Vegetales Especiales, Academia China de Ciencias Agrícolas, Changchun, China
El virus de la leucemia aviar (ALV) es uno de los principales patógenos de las enfermedades tumorales de las aves de corral y ha causado importantes pérdidas económicas a la industria avícola desde su descubrimiento. Por lo tanto, establecer un método de detección rápida es esencial para prevenir y controlar eficazmente la propagación del ALV. En este estudio, se diseñaron cebadores específicos de ARN CRISPR (crRNA) y amplificación asistida por recombinasa (RAA) con promotor T7 basados en la secuencia relativamente conservada del virus de la leucemia aviar. Cuando el crRNA reconoció la secuencia objetivo, se activó la proteína Cas13a para cortar las sondas de informe y, a continuación, los resultados de la detección se leyeron mediante el uso de una tira reactiva de flujo lateral (LFD). Se construyó el sistema de reacción RAA-CRISPR/Cas13a-LFD. El tiempo de amplificación de RAA, la concentración de proteína Cas13a, la concentración de crRNA y el tiempo de reacción CRISPR se optimizaron para evaluar la especificidad, sensibilidad y reproducibilidad del sistema. Finalmente, se comparó el método RAA-CRISPR/Cas13a-LFD con el método de electroforesis de reacción en cadena de la polimerasa (PCR)-Agarosa y el método qPCR en la detección de muestras clínicas, y se evaluó la fiabilidad del método RAA-CRISPR/Cas13a-LFD. Los resultados mostraron que el método RAA-CRISPR/Cas13a-LFD podía amplificar eficazmente el gen objetivo a 37 °C durante 40 minutos, y los resultados de las pruebas podían determinarse mediante la observación visual de LFD. El método tuvo buena especificidad y no tuvo reacción cruzada con el virus de la enfermedad de Marek (MDV), el adenovirus aviar (FAdV), el virus de la bursitis infecciosa (IBDV), el virus de la enfermedad de Newcastle (NDV), el virus de la laringotraqueítis infecciosa (ILTV) y el virus de la bronquitis infecciosa (IBV). El límite mínimo de detección del método fue de 100 copias/μL, y tuvo buena repetibilidad y estabilidad. La tasa de coincidencia de detección clínica alcanzó el 97,69% y el 99,23%. En resumen, este estudio estableció un método de detección simple, eficiente, preciso y visualizado de ALV, que puede ser utilizado para la prevención y el diagnóstico clínico rápido de la leucosis aviar (AL).
Introducción
La leucosis aviar (AL) es una enfermedad inmunosupresora causada por la infección por el virus de la leucemia aviar (ALV) (1). Las principales manifestaciones de la enfermedad son tumores multiorgánicos, inmunosupresión e infecciones fáciles o secundarias de otros patógenos. Conduce a pérdidas económicas directas después de la disminución del rendimiento de la producción, lo que afecta gravemente el desarrollo saludable de la industria avícola (2). Entre los 11 subgrupos de ALV conocidos, los pollos más infectados en China fueron el subgrupo J, seguido por el subgrupo A y el subgrupo B (3). El ALV es un retrovirus aviar común y natural. La transmisión del ALV se produce principalmente a través de la vía vertical desde el embrión infectado hasta la descendencia, y también puede transmitirse horizontalmente tras el contacto directo o indirecto con los pollos infectados o los contaminantes contaminados por el virus (4), lo que tiene una velocidad de transmisión rápida y una fuerte infectividad. No solo eso, sino que el ALV también puede transmitirse a través de vacunas vivas comerciales contaminadas (5). Las hembras infectadas con ALV-J a través de la inseminación artificial pueden transmitir el virus verticalmente a su descendencia a través de sus huevos (6), por lo que la única forma de aislar la transmisión del ALV es eliminar los pollos positivos mediante pruebas para detectar el patógeno (7). En respuesta, se han realizado estudios sobre el desarrollo de una vacuna contra el ALV (8), pero la vacuna no proporciona una protección adecuada para las aves de corral. Por lo tanto, el cribado precoz del virus y la detección rápida son esenciales para la prevención y el control de los virus antivirus.
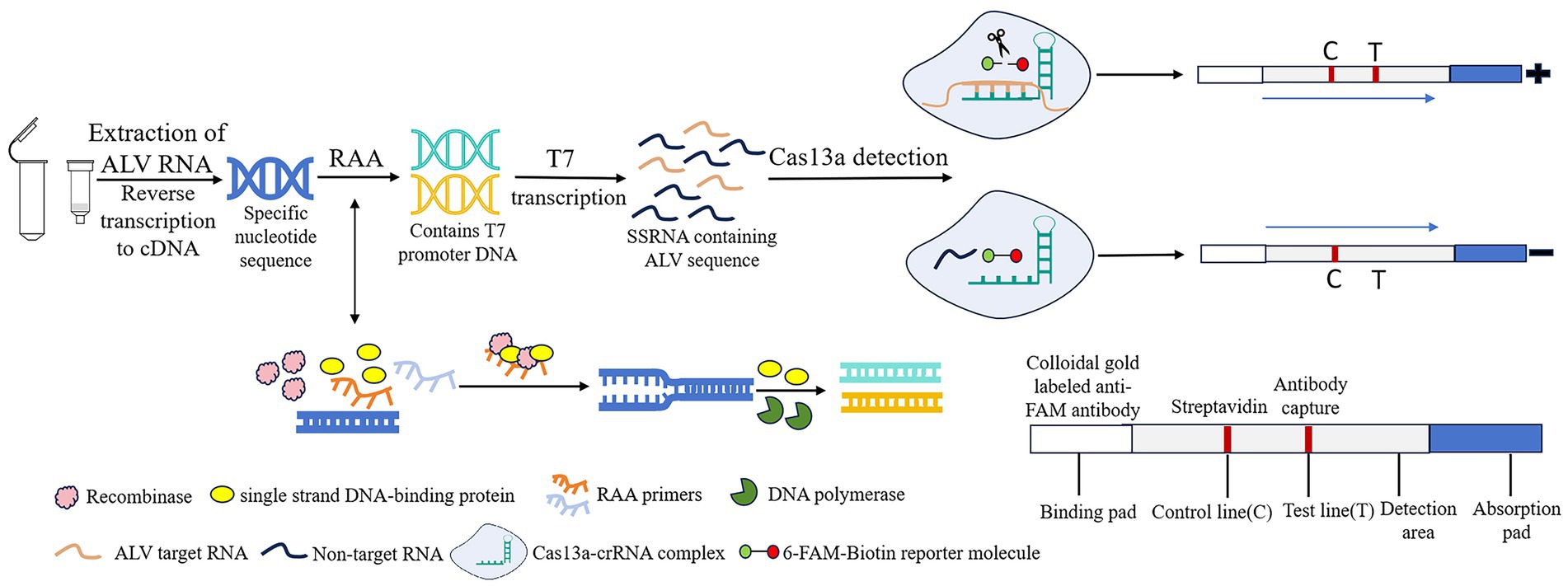
La amplificación por adición de recombinasa (RAA) es un nuevo tipo de tecnología de amplificación rápida de ácidos nucleicos que permite una rápida expansión de ADN o ARN a baja temperatura (generalmente 37 °C), con el producto de amplificación obtenido en 30 min (9, 10). Las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) y sus proteínas relacionadas (Cas) constituyen el sistema CRISPR/Cas. Su capacidad de corte transversal permite la identificación y el corte precisos de objetivos específicos de ácidos nucleicos. El sistema se utiliza como una herramienta útil para la edición del genoma y el diagnóstico de ácidos nucleicos debido a su alta sensibilidad y especificidad (11, 12). Entre ellos, Cas13a tiene la capacidad de reconocer objetivos de ARN a través de ARN CRISPR (crRNA) y actividad de corte colateral (13). Basado en la proteína Cas13a, el sistema SHERLOCK combina Cas13a con la amplificación de la polimerasa recombinasa (RPA) para lograr el diagnóstico molecular mediante la utilización de la actividad de escisión colateral (14). En este experimento, se utilizó el método RAA-CRISPR/Cas13a-tira reactiva de flujo lateral (LFD) para detectar el ALV, y el principio de reacción se mostró en la Figura 1. Después de la extracción de ácido nucleico del patógeno que se va a probar, se realiza la reacción de amplificación de RAA. El complejo formado por la recombinasa, la proteína de unión monocatenaria y el cebador RAA escanea el ADN bicatenario y el ADN bicatenario no rotado en la secuencia homóloga al cebador. La proteína de unión monocatenaria evita que el ADN monocatenario se repliegue, y luego la ADN polimerasa completa la extensión de la cadena. Después de eso, el ADN que contiene el promotor T7 es transcrito por T7 in vitro para formar ARN monocatenario que contiene un gran número de secuencias diana. Cas13a se une al crRNA para reconocer la secuencia diana. Si el complejo reconoce la secuencia diana, se activa la actividad de escisión colateral de la nucleasa Cas13a. La 6-FAM-Biotina informó que la molécula fue cortada y unida al complejo de anticuerpos anti-FAM marcado con oro coloidal, y la línea de detección (T) fue positiva. Si el complejo no reconoce la secuencia diana, la nucleasa Cas13a no se activa, y el complejo de la molécula reportera con anticuerpos anti-FAM marcados con oro coloidal es capturado por estreptavidina, y solo la línea de control (C) es negativa. En la actualidad, la tecnología RAA-CRISPR/Cas13a se ha utilizado ampliamente en la detección del nuevo coronavirus (SARS-CoV-2) (15), virus de plantas (16), virus de la peste porcina africana (PPA) (17) y otros campos.
En este estudio, se combinaron RAA, CRISPR/Cas13a y LFD para diseñar el cebador RAA y el crRNA de acuerdo con la secuencia conservada del gen ALV env. Después de establecer y optimizar el sistema de reacción RAA-CRISPR/Cas13a-LFD, se evaluó su especificidad, sensibilidad, repetibilidad y fiabilidad clínica, con el fin de establecer un método de detección de ALV rápido, sensible, específico y visualizado en resultados, con el objetivo de proporcionar una herramienta poderosa para la detección, prevención y control rápidos de ALV en el campo.
Materiales y métodos
Extracción de ácidos nucleicos
ALV, FAdV, IBDV, NDV y MDV fueron cepas almacenadas en el laboratorio de enfermedades infecciosas animales de la Universidad Agrícola de Hebei. El IBV (AV1511) y el ILTV (AV195) se compraron a la Administración de Medicamentos Veterinarios de China. El ADN de FAdV, ILTV, MDV y el ARN de ALV, IBDV, NDV e IBV se extrajeron mediante TIANamp Virus DNA/RNA Kit (Tiangen Biochemical Technology Co., Ltd.), y el ARN se transcribió inversamente en ADNc y se almacenó a -20 °C para su uso futuro.
Diseño de cebadores y crRNAs y cribado de cebadores
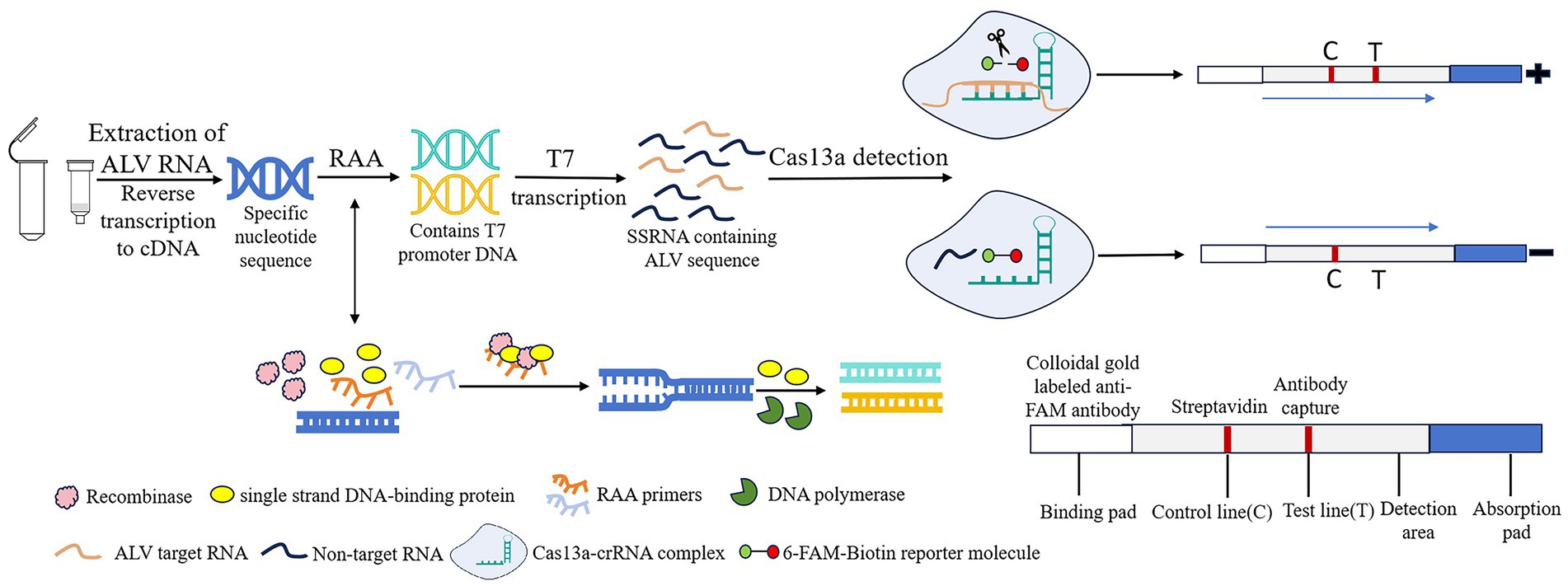
De acuerdo con la secuencia del gen env de ALV registrada en GenBank (número de entrada: MF926336.1, OP837418.1, ON840108.1, HQ260975.1, MG700538.1, MG700535.1, MG700534.1, MG700537.1, MG700533.1, HM235664.1, KC841153.1), se utilizaron el software SnapGene y DNAMAN para el análisis comparativo para determinar la secuencia conservada en la región gp37 del gen env (Figura 2A), y se diseñaron tres pares de cebadores específicos de RAA (Tabla 1). Añadir en el cebador delantero secuencias promotoras de ARN polimerasa T7 del extremo 5′ (T7; GAAATTAAT ACGACTCACTATAGGG; Figura 2B). Utilizando el ADNc de ALV como plantilla, se realizó una reacción de amplificación de RAA. El sistema de reacción fue de 50 μL, incluyendo tampón 25 μL, agua purificada 13,8 μL, cebador directo 2,1 μL (10 μM), cebador inverso 2,1 μL (10 μM), molde de ADN 2 μL y 5 μL de acetato de magnesio. La amplificación se realizó a 37°C durante 30 min, y luego se purificaron los productos amplificados. Los resultados se observaron mediante electroforesis de agarosa al 2% y se seleccionaron un par de cebadores óptimos. Además, se diseñó crRNA en la secuencia conservada del gen env, crRNA por LwaCas13a estructura de anillo de tallo fijo (GTTTTAGTCCCCTTCGTTTGGGGTAGTCTAA ATCCCCTAGTGTGAGTCATTTC) y la longitud de la secuencia diana de 28 nt (Tabla 1), que se incluyó en el área de amplificación del cebador. En este experimento se utilizó PCR-F/R para verificar la sensibilidad de la electroforesis de PCR-agarosa. Tanto el cebador como el crRNA fueron sintetizados por la empresa Sangon Biotech (Shanghai).

Construir plásmido estándar
El ADNc de ALV se utilizó como molde para la amplificación por PCR. El sistema de reacción de PCR fue de 25 μL, incluyendo 2 × Taq Mix 12,5 μL, cebador directo (10 μM) 0,5 μL, cebador inverso (10 μM) 0,5 μL, plantilla 1 μL, ddH2O 10,5 μL. El procedimiento de reacción fue el siguiente: 94 °C 5 min, 94 °C 30 s, 53 °C 30 s, 72 °C 30 s, 35 ciclos, 72 °C extendido durante 5 min. Los productos de PCR se verificaron mediante electroforesis en gel de agarosa al 2%. Se utilizó el kit de extracción de gel de ADN de columna SanPrep (Sangon Bioengineering Co., Ltd., Shanghai, China) para la recuperación de gel. El ADN recuperado se conectó al vector T-Vector pMD 19 (Takara Biomedical Technology Co., Ltd., Beijing, China) y se introdujo en células competentes DH5α. Después del cultivo nocturno, se examinaron las colonias azules y blancas, se seleccionaron las colonias blancas y se extrajeron los plásmidos con el kit de extracción de plásmidos (Tiangen Biochemical Technology Co., Ltd.). El plásmido estándar fue secuenciado por Sangon Biotech.
Según la fórmula, se calcula el número de copias de ADN contenidas en un plásmido por unidad de volumen:
El plásmido estándar construido se diluyó a 109–100 copias/μL mediante el método de dilución de 10 veces y se almacenó a -20 °C para su uso posterior.
Establecimiento del método de detección RAA-CRISPR/Cas13a-LFD
La reacción RAA se realizó de acuerdo con las instrucciones del kit de amplificación de ácido nucleico RAA (Anhui Microanaly Genetech Co., Ltd., Hefei, China). El sistema de reacción RAA fue de 50 μL, incluyendo tampón 25 μL, agua purificada 13,8 μL, cebador directo 2,1 μL (10 μM), cebador inverso 2,1 μL (10 μM), molde de ADNc 2 μL y 5 μL de acetato de magnesio. En primer lugar, los reactivos, excepto el acetato de magnesio, se añadieron sucesivamente al tubo de expansión que contenía la enzima de liofilización, y después de mezclar suavemente a mano, se añadieron 5 μL de acetato de magnesio a la tapa, que luego se amplificó en un dispositivo de amplificación a temperatura constante a 37 °C durante 30 min.
El sistema de reacción RAA-CRISPR/Cas13a-LFD es de 50 μL, incluyendo 4 μL de mezcla de tampón NTP (New England Biolabs; 25 mmol/L), 2 μL de inhibidor de la RNasa (Takara Biomedical Technology Co., Ltd., Pekín, China; 40 U/μL), 4 μL Cas13a (Magigen China; 80 nmol/L), 2 μL de crRNA (80 ng/uL), 2 μL de sonda de doble marcado de ARN (Bio-Lifesci, China; 100 μmol/L), 1 μL de mezcla de ARN polimerasa T7 (New England Biolabs), 0,5 μL de MgCl2 (Coolaber, China), 1 μL de tampón HEPES (Solarbio, China), 28,5 μL de agua estéril sin enzimas y 5 μL de producto de amplificación RAA. Los reactivos se mezclaron completamente y se incubaron a 37 °C durante 25 min. Posteriormente, se añadieron 50 μL de productos de reacción a la LFD y los resultados se observaron en un plazo de 3 a 5 minutos.
Optimización del sistema de reacción RAA-CRISPR/Cas13a-LFD
Los factores que afectan la eficiencia de la amplificación, incluido el tiempo de amplificación de RAA, la concentración de Cas13a, la concentración de crRNA y el tiempo de reacción CRISPR, se optimizaron mediante el uso del sistema de reacción anterior. El tiempo de amplificación de RAA está configurado para comenzar a partir de 10 minutos y aumentar el gradiente cada 5 minutos. Si aparecen líneas de detección claras, este tiempo se selecciona como el tiempo óptimo de amplificación de RAA. La concentración de Cas13a se fijó en 40 nmol/L y aumentó en un gradiente de cada 20 nmol/L, mientras que otros factores permanecieron sin cambios. Cuando apareció una línea de detección clara, esta concentración se seleccionó como la concentración óptima de Cas13a. Bajo la concentración óptima de Cas13a, la concentración de crRNA se estableció en 10 ng/μL, y el aumento del gradiente fue cada 10 ng/μL, y otros factores permanecieron sin cambios. Si aparecía una línea de detección clara, la concentración se seleccionaba como la concentración óptima de ARNcr. Bajo la concentración óptima de Cas13a y crRNA, el tiempo de reacción de CRISPR se estableció para comenzar a partir de 10 minutos y aumentar el gradiente cada 5 minutos hasta que apareciera una línea de detección clara.
Detección específica
El ADN de FAdV, ILTV y MDV y el ADNc de ALV, IBDV, NDV e IBV se utilizaron como plantillas, respectivamente, y ddH2O se estableció como control negativo para evaluar la especificidad del ensayo RAA-CRISPR/Cas13a-LFD.
Detección de sensibilidad
Se utilizó el plásmido estándar ALV diluido (107-10 0 copias/μL) como plantilla, y ddH2O como control negativo. Para la detección se utilizó el método PCR-Agar-gel, electroforesis, qPCR y RAA-CRISPR/Cas13a-LFD, y cada método se repitió tres veces para evaluar su sensibilidad.
El sistema de reacción de PCR fue de 25 μL, compuesto por 2 × Taq Mix de 12,5 μL, cebador directo (10 μM) 0,5 μL, cebador inverso (10 μM) 0,5 μL, molde (107-10 0 copias/μL) 1 μL, ddH2O 10,5 μL. El procedimiento de reacción fue el siguiente: 94 °C 5 min, 94 °C 30 s, 53 °C 30 s, 72 °C 30 s, 35 ciclos, 72 °C extendido durante 5 min. Los productos de PCR se sometieron a electroforesis en gel de agarosa para determinar el límite mínimo de detección.
El sistema de reacción de qPCR fue de 20 μL, incluyendo TB Green Premix Ex TaqTMII (TliRNaseH Plus; 2×; Takara Biomedical Technology Co., Ltd., Pekín, China) 12,5 μL, cebadores directos e inversos PCR (10 μM) 1,0 μL cada uno, plantillas (106–100 copias/μL) 1,0 μL, ddH2O 9,5 μL, Procedimiento de reacción: 95 °C 30 s; Los resultados se observaron después de 40 ciclos a 95 °C durante 5 s y a 60 °C durante 30 s.
El ensayo RAA-CRISPR/Cas13a-LFD utilizó 105-10 0 copias/plásmido estándar μL como plantilla y se llevó a cabo de acuerdo con el sistema de reacción optimizado para determinar el límite mínimo de detección del método.
Detección de repetibilidad
Se utilizaron tres plásmidos estándar de ALV con diferente concentración después de la dilución (106 copias/μL, 103 copias/μL y 100 copias/μL) como plantilla, y la prueba se repitió 3 veces para cada concentración, y ddH2O se estableció como control negativo. Evaluar la repetibilidad del método RAA-CRISPR/Cas13a-LFD.
Pruebas de muestras clínicas
Las muestras de hígado y riñón de 130 pollos enfermos del norte de China con diagnóstico clínico preliminar de AL se detectaron mediante PCR-Agar-electroforesis, RAA-CRISPR/Cas13a-LFD y qPCR (las muestras se recogieron en 7 granjas de pollos). Se calculó la tasa de coincidencia del método RAA-CRISPR/Cas13a-LFD y los dos métodos de detección, y se evaluó la factibilidad de la aplicación del método en muestras clínicas.
Resultados
Cribado de cebadores RAA
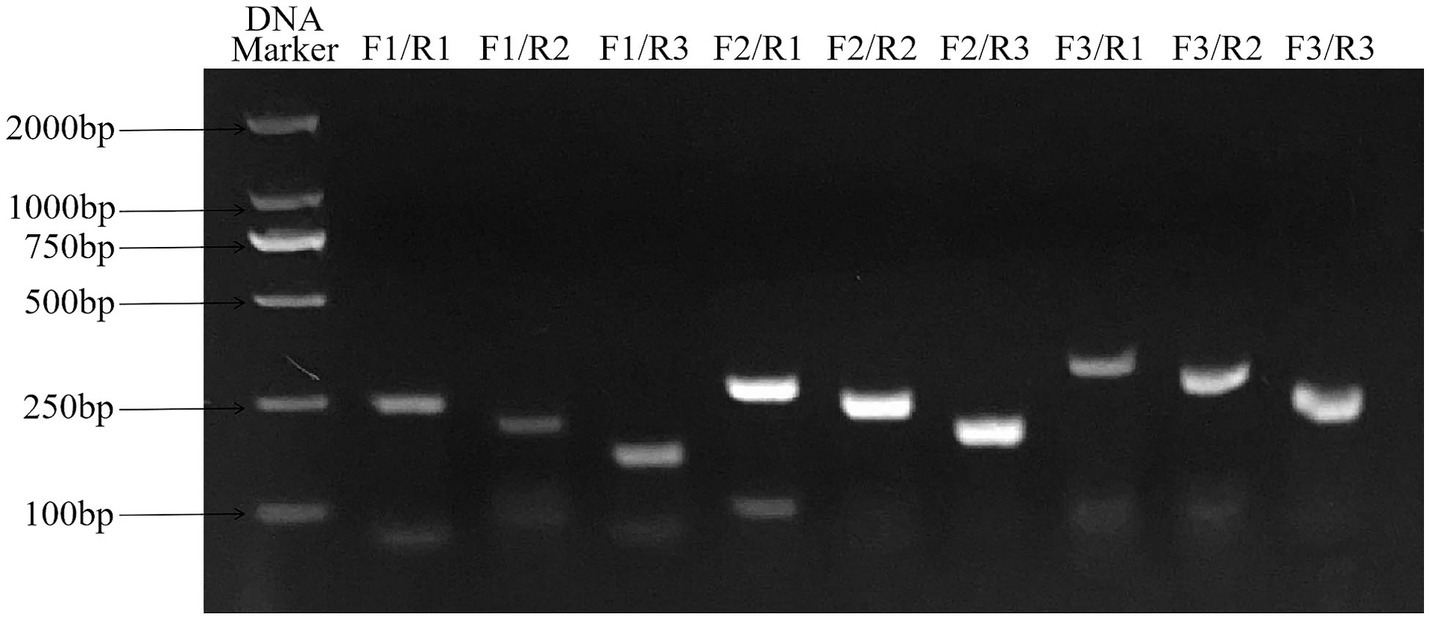
Los resultados del cribado de cebadores mostraron que la eficiencia de amplificación de las combinaciones de cebadores RAA-F2/R1, F2/R2 y F2/R3 fue mayor que la de otras combinaciones de cebadores, entre las cuales la combinación de cebadores F2/R3 no mostró amplificación inespecífica, por lo que se seleccionó la combinación RAA-F2/R3 como el mejor cebador. El resultado se muestra en la Figura 3.
Optimización del sistema de reacción RAA-CRISPR/Cas13a-LFD
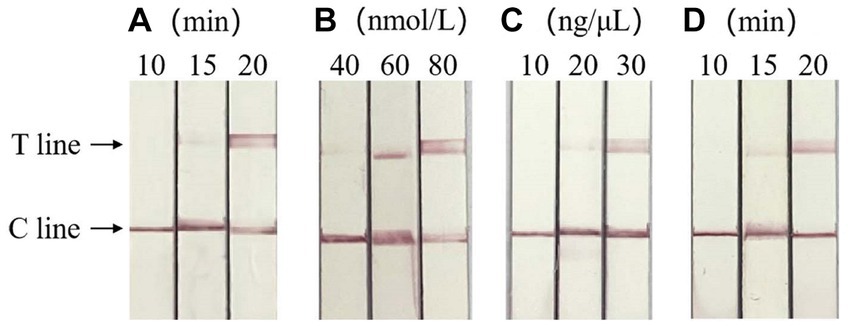
Sobre la base de la línea de detección clara de LFD, se verificaron las condiciones óptimas de reacción de RAA-CRISPR/Cas13a-LFD como un tiempo de amplificación de RAA de 20 min, una concentración de crRNA de 30 ng/μL y un tiempo de reacción CRISPR de 20 min, respectivamente. Cuando la concentración de Cas13a es de 60 y 80 nmol/L, aparecen líneas de detección claras en ambos casos, por lo que la concentración óptima de Cas13a es de 60 nmol/L. El resultado se muestra en la Figura 4.
Detección específica
La prueba de especificidad mostró que solo el ALV fue positivo y no se encontró una línea de prueba entre otros virus y los controles negativos, lo que indica que no hubo reacción cruzada entre el ALV y el MDV, FAdV, IBDV, NDV, ILTV e IBV, y el método tuvo buena especificidad. El resultado se muestra en la figura 5.

Detección de sensibilidad
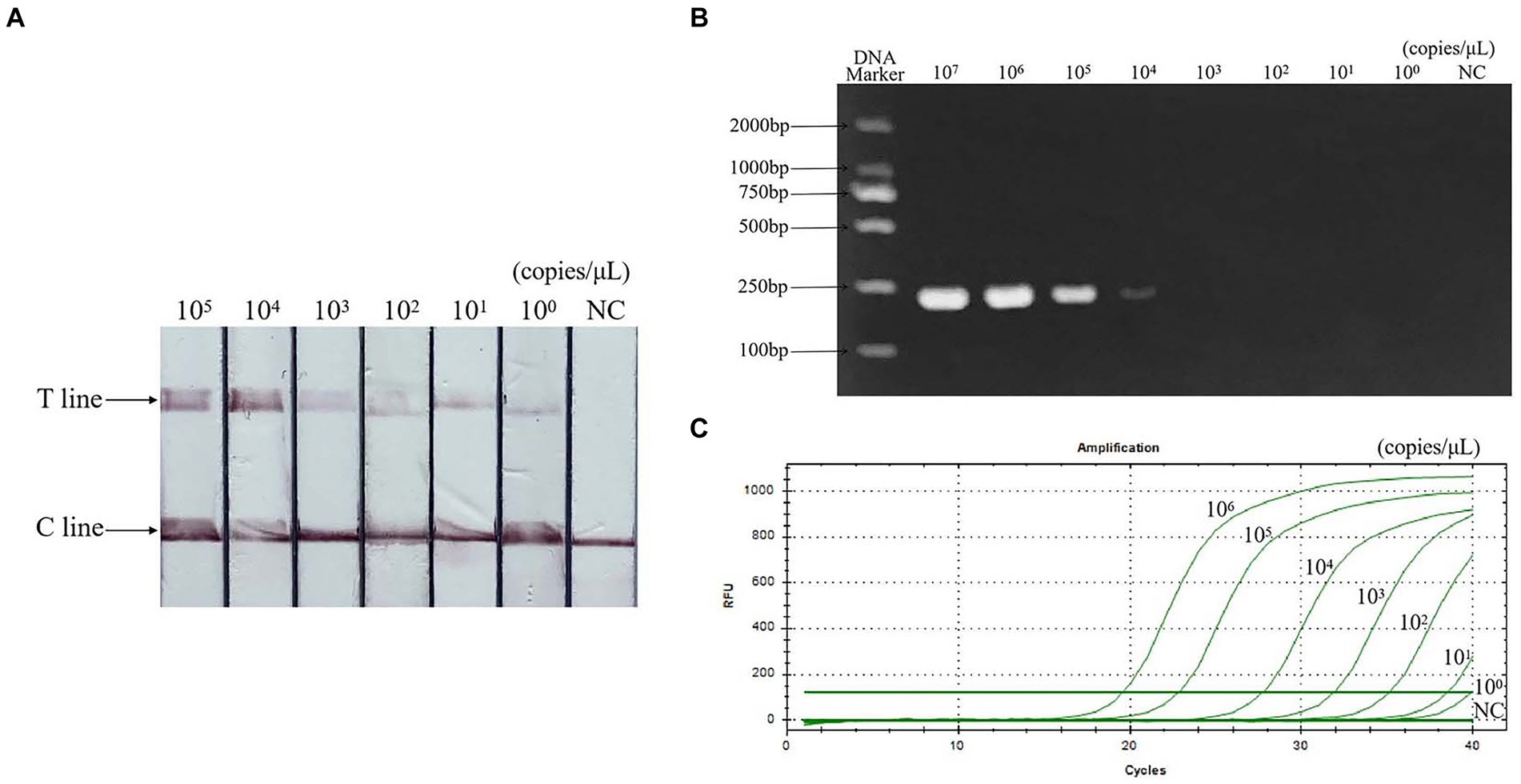
Los resultados de la evaluación de sensibilidad mostraron que el límite mínimo de detección de PCR fue de 104 copias/μL, qPCR fue de 101 copias/μL y RAA-CRISPR/Cas13a-LFD fue de 100 copias/μL. La sensibilidad es 10 veces mayor que la de la qPCR y 10.000 veces mayor que la de la PCR. El resultado se muestra en la Figura 6.
Detección de repetibilidad
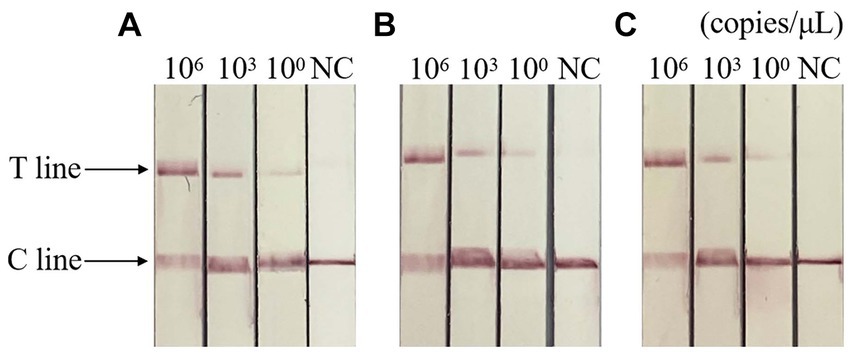
Los resultados de la prueba de repetibilidad mostraron que los plásmidos estándar de diferentes concentraciones fueron positivos y el grupo control negativo, lo que indica que el método de detección tuvo buena repetibilidad y estabilidad. El resultado se muestra en la Figura 7.
Pruebas de muestras clínicas
Los resultados de las pruebas de muestras clínicas de ALV mostraron (Tabla 2) que se detectaron 42 muestras positivas mediante RAA-CRISPR/Cas13a-LFD, y la tasa de coincidencia con la electroforesis de PCR-Agarosa fue del 97,69% y del 99,23% con el método qPCR, lo que indica que el método RAA-CRISPR/Cas13a-LFD tiene una capacidad de detección precisa y puede utilizarse para la detección clínica.
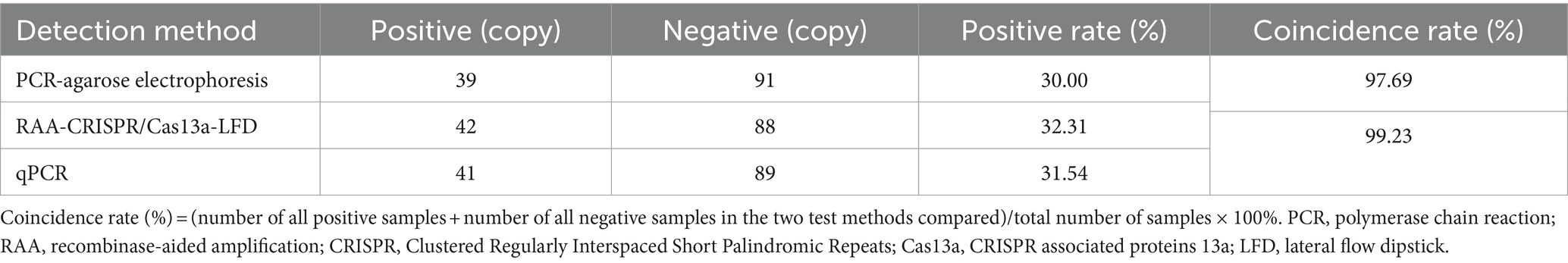
Tabla 2. Resultados de las pruebas clínicas de los dos métodos.
Discusión
Desde su primer descubrimiento en China en 1999, el ALV-J se ha extendido rápidamente por todo el país. En su día fue una enfermedad importante que ponía en peligro a la industria avícola de China, con una tasa de mortalidad y morbilidad de hasta el 50%, lo que supuso una gran amenaza para el suministro de productos de pollo ponedor durante mucho tiempo y provocó graves pérdidas económicas a la industria avícola (18).
En la actualidad, los métodos comúnmente utilizados para detectar el ALV en la clínica incluyen el aislamiento del virus, el ensayo de inmunoabsorción ligado a enzimas (ELISA), la PCR, la PCR cuantitativa de fluorescencia en tiempo real (qPCR) y la amplificación isotérmica mediada por bucle (LAMP). El aislamiento y la identificación de virus es el estándar de oro para la detección de ALV, pero requiere cultivo celular, lo que requiere mucho tiempo y laboriosidad. El método de detección LAMP tiene una alta sensibilidad, pero su diseño de cebador es complejo y fácil de contaminar (19, 20), lo que no es adecuado para una detección rápida a nivel de base. El método ELISA también debe combinarse con el aislamiento del virus y la PCR, y la operación es complicada (21). Aunque el método de detección de qPCR tiene una alta sensibilidad y especificidad (22) y puede juzgarse con precisión, requiere instrumentos complejos y costosos y experiencia del operador, y no es adecuado para un diagnóstico rápido in situ. Por lo tanto, establecer un método de detección rápido, preciso y conveniente es particularmente importante para el diagnóstico, la prevención y el control rápidos de la AL. El sistema CRISPR/Cas es un sistema de diagnóstico desarrollado por primera vez para ácidos nucleicos moleculares en 2016 (13). El RAA puede amplificar ácidos nucleicos virales a una temperatura de 37 °C a 42 °C sin instrumentos especiales de temperatura variable, y puede amplificar la señal inicial, que puede utilizarse para mejorar la sensibilidad del sistema CRISPR/Cas (12). Con la rápida evolución del diagnóstico molecular, la necesidad de pruebas rápidas ha aumentado, y los sistemas CRISPR/Cas, combinados con diversas tecnologías de amplificación isotérmica, se han utilizado para detectar varios tipos de patógenos, incluidas enfermedades virales, bacterianas y parasitarias. Zhang et al. (23) combinaron RAA con el sistema CRISPR-Cas13a para detectar el virus de la gripe aviar y utilizaron la amplificación específica de fragmentos de genes diana para proporcionar un nuevo método de detección para la detección del virus de la gripe aviar. An et al. (24) combinaron RPA con el sistema CRISPR-Cas13a para proporcionar un método de detección simple, rápido y específico para Salmonella. Zhao et al. (25) combinaron RAA con CRISPR/Cas13a para establecer un método de detección visual rápida y sensible para Toxoplasma gondii.
En este estudio, se desarrolló un método RAA-CRISPR/Cas13a-LFD para la detección rápida de la leucemia aviar, y los resultados se determinaron mediante el uso de tiras de flujo transversal. El método RAA-CRISPR/Cas13a-LFD puede completar la detección de genes objetivo en solo 40 minutos, y la capacidad de corte dirigido de la unión de Cas13a con crRNA hace que este método tenga una fuerte especificidad. La combinación de la tecnología RAA y el sistema CRISPR/Cas mejora la sensibilidad de detección. El límite mínimo de detección fue de 100 copias/μL, que fue 10 veces más sensible que la qPCR y 10.000 veces más sensible que la PCR. Miao et al. (26) utilizaron RAA-CRISPR/Cas13a-LFD para detectar el virus Nipah con un límite de detección de 103 copias/μL, que fue ligeramente inferior a la sensibilidad de este estudio. Dou et al. (27) establecieron un método de PCR cuantitativa multiplex para la detección de leucemia aviar en los subgrupos A, B, J y K, con un límite de detección de 101 copias/μL por respuesta, pero su funcionamiento es complejo y el instrumento es costoso. Xiang et al. (28) establecieron un método CPA para la detección del subgrupo J de leucemia aviar con un límite de detección de 101 copias/μL, pero su tiempo de respuesta fue largo y la sensibilidad fue solo 10 veces mayor que la del método de PCR. El método RAA-CRISPR / Cas13a-LFD tiene buena repetibilidad y estabilidad, y una alta tasa de coincidencia con el método de electroforesis PCR-Agarosa y el método qPCR para la detección del mismo lote de muestras, que es adecuado para la detección clínica.
En este estudio, se estableció el método RAA-CRISPR/Cas13a-LFD para la detección de campo de ALV, que tiene las ventajas de ser rápido, sensible y conveniente, y proporciona una tecnología confiable para el diagnóstico, prevención y control de AL.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio o repositorios y el número de acceso se pueden encontrar en el artículo/material complementario.
Declaración de ética
Los estudios con animales fueron aprobados por la Facultad de Medicina Veterinaria de la Universidad Agrícola de Hebei, Baoding, China. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones de los autores
JL: Redacción – revisión y edición, Redacción – borrador original, Metodología, Análisis formal, Curación de datos, Conceptualización. ZiZ: Escritura – revisión y edición, validación. ZoZ: Escritura – revisión y edición, Investigación. XC: Escritura – revisión y edición, Investigación. CW: Redacción – revisión y edición, supervisión. XZ: Redacción – revisión y edición, supervisión. TZ: Redacción – revisión y edición, supervisión.
Financiación
El/los autor/es declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo fue apoyado por el Programa de Ciencia y Tecnología de Hebei (no. 22326608D).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Payne, LN, y Nair, V. Visión a largo plazo: 40 años de investigación sobre la leucosis aviar. Pathol aviar. (2012) 41:11–9. doi: 10.1080/03079457.2011.646237
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Zheng, LP, Teng, M, Li, GX, Zhang, WK, Wang, WD, Liu, JL, et al. Epidemiología actual y coinfecciones de enfermedades neoplásicas e inmunosupresoras aviares en parvadas de pollos en China Central. Virus. (2022) 14:2599. doi: 10.3390/v14122599
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Mo, GD, Wei, P, Hu, BW, Nie, QH y Zhang, XQ. Avances en estudios genéticos y genómicos de la resistencia a ALV. J Anim Sci Biotechnol. (2022) 13:123. doi: 10.1186/s40104-022-00769-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Tang, S, Li, J, Chang, YF y Lin, WC. Interacción virus-huésped de la leucosis aviar: la participación de los factores del huésped en la replicación viral. Frente Immunol. (2022) 13:907287. doi: 10.3389/fimmu.2022.907287
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Wang, PK, Li, M, Li, HJ, Bi, YY, Lin, LL, Shi, MY, et al. Las vacunas vivas comerciales contaminadas con ALV-J indujeron patogenicidad en pollos de tres amarillos: una de las rutas de transmisión de ALV-J a pollos comerciales. Sci. (2021) 100:101027. doi: 10.1016/j.psj.2021.101027
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Li, Y, Cui, S, Li, WH, Wang, YX, Cui, ZZ, Zhao, P, et al. Transmisión vertical del virus de la leucosis aviar subgrupo J (ALV-J) de gallinas infectadas por inseminación artificial con semen infectado con ALV-J. BMC Vet Res. (2017) 13:204. doi: 10.1186/s12917-017-1122-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Feng, M y Zhang, XQ. Inmunidad frente al virus de la leucosis aviar: ¿dónde estamos ahora y qué debemos hacer? Frente Immunol. (2016) 7:624. doi: 10.3389/fimmu.2016.00624
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Jing, WF, Zhou, JR, Wang, CY, Qiu, JH, Guo, HJ y Li, HM. Preparación de la proteína secretora recombinante ALV-J gp85 utilizando Pichia pastoris y su inmunoprotección como antígeno vacunal combinando con el adyuvante CpG-ODN. Inmunología Viral. (2018) 31:407–16. doi: 10.1089/vim.2017.0170
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Wu, XH, Chu, FS, Zhang, LX, Chen, S, Gao, LG, Zhang, H, et al. Nueva detección rápida mediante el uso de un método de temperatura constante para los virus de la leucosis aviar. Microbiol frontal. (2022) 13:968559. doi: 10.3389/fmicb.2022.968559
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Wu, XH, Liu, YJ, Gao, LG, Yan, ZQ, Zhao, QQ, Chen, F, et al. Desarrollo y aplicación de un ensayo de amplificación asistida por recombinasa con transcripción inversa para el virus de la diarrea epidémica porcina. Virus. (2022) 14:591. doi: 10.3390/v14030591
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Li, XP, Zhu, SY, Zhang, XL, Ren, YL, He, J, Zhou, JW, et al. Avances en la aplicación de la amplificación asistida por recombinasa combinada con la tecnología CRISPR-Cas en la detección rápida de microbios patógenos. Frente Bioeng Biotechnol. (2023) 11:1215466. doi: 10.3389/fbioe.2023.1215466
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Li, Y, Shang, JJ, Luo, J, Zhang, FY, Meng, G, Feng, YJ, et al. Detección rápida del virus de la gripe aviar del subtipo H5 mediante tira reactiva de flujo lateral basada en CRISPR Cas13a. Microbiol frontal. (2023) 14:1283210. doi: 10.3389/fmicb.2023.1283210
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Abudayyeh, OO, Gootenberg, JS, Konermann, S, Joung, J, Slaymaker, IM, Cox, DB, et al. C2c2 es un efector CRISPR dirigido por ARN guiado por ARN programable de un solo componente. Ciencia. (2016) 353:AAF5573. doi: 10.1126/ciencia.aaf5573
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Kellner, MJ, Koob, JG, Gootenberg, JS, Abudayyeh, OO y Zhang, F. SHERLOCK: detección de ácidos nucleicos con nucleasas CRISPR. Nat Protoc. (2019) 14:2986–3012. doi: 10.1038/s41596-019-0210-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Rahman, MR, Hossain, MA, Mozibullah, M, Mujib, FA, Afrose, A, Shahed-Al-Mahmud, M, et al. CRISPR es una herramienta biológica útil para detectar el ácido nucleico del SARS-CoV-2 en muestras clínicas humanas. Farmacia Biomédica. (2021) 140:111772. doi: 10.1016/j.biopha.2021.111772
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Robertson, G, Burger, J y Campa, M. Herramientas basadas en CRISPR/Cas para el control dirigido de virus de plantas. Mol Plant Pathol. (2022) 23:1701–18. doi: 10.1111/mpp.13252
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Wei, N, Zheng, BH, Niu, JJ, Chen, T, Ye, J, Si, YH, et al. Detección rápida del virus de la peste porcina africana de genotipo II mediante tira de flujo lateral basada en CRISPR Cas13a. Virus. (2022) 14:179. doi: 10.3390/v14020179
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Gao, YL, Qin, LT, Pan, W, Wang, YQ, Qi, XL, Gao, HL, et al. Virus de la leucosis aviar subgrupo J en pollos ponedoras, China. Emerg Infect Dis. (2010) 16:1637–8. doi: 10.3201/eid1610.100780
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Peng, H, Qin, LL, Bi, YY, Wang, PK, Zou, GZ, Li, J, et al. Detección rápida de los subgrupos comunes del virus de la leucosis aviar mediante amplificación isotérmica mediada por bucle en tiempo real. Virol J. (2015) 12:195. doi: 10.1186/s12985-015-0430-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Zhang, X, Liao, M, Jiao, P, Luo, K, Zhang, H, Ren, T, et al. Desarrollo de un ensayo de amplificación isotérmica mediado por bucle para la detección rápida del virus de la leucosis aviar del subgrupo J. J Clin Microbiol. (2010) 48:2116–21. doi: 10.1128/JCM.02530-09
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Zhou, XY, Wang, L, Shen, AN, Shen, X, Xu, MR, Qian, K, et al. Detección de ALV p27 en hisopos cloacales y medio de aislamiento viral por sELISA. BMC Vet Res. (2019) 15:383. doi: 10.1186/s12917-019-2150-z
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Wu, M, Zhi, BH, Sheng, R, Zhou, ET, Zhou, Q, Li, JM, et al. Establecimiento de un método de PCR cuantitativo en tiempo real basado en TaqMan para la detección de adenovirus aviares exógenos tipo I, tipo III y virus de la leucosis aviar en vacunas vivas atenuadas contra la influenza adaptadas al frío humano. Heliyon. (2023) 9:e23033. doi: 10.1016/j.heliyon.2023.e23033
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Zhang, ZS, Wang, CG, Chen, X, Zhang, ZC, Shi, GQ, Zhai, XH, et al. Basado en el sistema CRISPR-Cas13a, para establecer un método de detección visual rápida de los virus de la gripe aviar. Veterinario delantero Sci. (2024) 10:1272612. doi: 10.3389/fvets.2023.1272612
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. An, BL, Zhang, HB, Su, X, Guo, Y, Wu, T, Ge, YY, et al. Detección rápida y sensible de Salmonella spp. mediante CRISPR-Cas13a combinado con amplificación de polimerasa recombinasa. Microbiol frontal. (2021) 12:732426. doi: 10.3389/fmicb.2021.732426
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Zhao, JH, Li, YY, Xue, QQ, Zhu, ZW, Zou, MH y Fang, F. Un nuevo ensayo de detección visual rápida para toxoplasma gondii que combina amplificación asistida por recombinasa y tira reactiva de flujo lateral junto con fluorescencia CRISPR-Cas13a (RAA-Cas13a-LFD). Parásito. (2022) 29:21. doi: 10.1051/parásito/2022021
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Miao, J, Zuo, LL, He, DM, Fang, ZX, Berthet, N, Yu, C, et al. Detección rápida del virus Nipah mediante el ensayo RPA-CRISPR/Cas13a de un solo recipiente. Res. de Virus. (2023) 332:199130. doi: 10.1016/j.virusres.2023.199130
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Dou, JF, Wang, Z, Li, L, Lu, Q, Jin, XX, Ling, XC, et al. Reacción en cadena de la polimerasa cuantitativa múltiple para la detección diferencial rápida de los virus de la leucosis aviar de los subgrupos a, B, J y K. Virus. (2023) 15:1789. doi: 10.3390/v15091789
28. Xiang, Y, Li, LZ, Liu, P, Yan, L, Jiang, Z, Yu, Y, et al. Detección rápida del subgrupo J del virus de la leucosis aviar mediante amplificación de cebado cruzado. Sci Rep. (2021) 11:10946. doi: 10.1038/s41598-021-90479-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: virus de la leucemia aviar, amplificación asistida por recombinasa, CRISPR/Cas13a, tira reactiva de flujo lateral, detección rápida
Cita: Li J, Zhang Z, Zhang Z, Chen X, Wang C, Zhai X y Zhang T (2024) Detección rápida del virus de la leucemia aviar mediante tira reactiva de flujo lateral basada en CRISPR/Cas13a. Frente. Vet. Sci. 11:1424238. doi: 10.3389/fvets.2024.1424238
Recibido: 27 de abril de 2024; Aceptado: 06 de agosto de 2024;
Publicado: 16 de agosto de 2024.
Editado por:
Perumal Arumugam Desingu, Instituto Indio de Ciencias (IISc), India
Revisado por:
Naidong Wang, Universidad Agrícola de Hunan, China
Xueyang Chen, Universidad de Yangtze, China
Peikun Wang, Universidad de Linyi, China
Derechos de autor © 2024 Li, Zhang, Zhang, Chen, Wang, Zhai y Zhang. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Tie Zhang, zhangtie1998@163.com
†Estos autores han contribuido a partes iguales a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía