Dinámica global del perfil proteico en cerdos durante el ciclo estral

El análisis proteómico cuantitativo y la verificación identifican la dinámica global del perfil proteico en cerdos durante el ciclo estral
 Haiyun Xin1,2
Haiyun Xin1,2  Baohong Li1
Baohong Li1  Fanming Meng1,2
Fanming Meng1,2  Bin Hu1,2
Bin Hu1,2  Sutian Wang1,2
Sutian Wang1,2  Ying Wang3
Ying Wang3  Jianhao Li1,2*
Jianhao Li1,2*- 1Laboratorio Estatal Clave de la Industria de Cría de Porcinos y Aves de Corral, Laboratorio Clave de Cría y Nutrición Animal de Guangdong, Instituto de Ciencia Animal, Academia de Ciencias Agrícolas de Guangdong, Guangzhou, China
- 2Rama de Maoming, Laboratorio de Guangdong para la Agricultura Moderna de Lingnan, Maoming, China
- 3Centro Técnico de Aduanas de Guangzhou, Guangzhou, China
El método actual de detección de estro generalmente requiere mucho tiempo y tiene baja precisión. Como tal, una comprensión más profunda de los procesos fisiológicos durante el ciclo estral acelera el desarrollo de la eficiencia y precisión de la detección del estro. En este estudio, se utilizó la espectrometría de masas de adquisición sin etiqueta para explorar los perfiles de proteoma salival durante el ciclo estral (día -3, día 0, día 3 y día 8) en cerdos, y se aplicó el monitoreo de reacción paralela (PRM) para verificar los perfiles relativos de expresión de proteínas. Se identificaron un total de 1.155 proteínas en el análisis libre de etiquetado, de las cuales 115 se identificaron como proteínas expresadas diferencialmente (DEP) entre diferentes grupos (p ≤ 0,05). La anotación funcional reveló que los DEP se agruparon en la unión de iones de calcio, el citoesqueleto de actina y la actividad de la liasa. PRM verificó los perfiles relativos de expresión de proteínas, en los que se confirmó que la proteína que contiene el dominio PHB, la proteína 2 unida al receptor del factor de crecimiento, el factor de elongación Tu, la carboxipeptidasa D, la anhidrasa carbónica y el factor trébol 3 eran consistentes tanto en los enfoques libres de etiqueta como en los PRM. Los ensayos proteómicos comparativos sobre la saliva aumentarían nuestro conocimiento del ciclo estral en cerdas y proporcionarían métodos potenciales para la detección del estro.
Introducción
Como parte indispensable del manejo reproductivo de las cerdas, la detección rápida y precisa del estro determina directamente la productividad general de la utilización de las cerdas al afectar el momento óptimo de inseminación, reducir la alimentación ineficaz y eliminar oportunamente las cerdas con bajo rendimiento reproductivo (1). Se han aplicado varios métodos para detectar el estro en cerdas, por ejemplo, la observación del comportamiento, la prueba del jabalí y la estimación de esteroides (2). El método más común es el reconocimiento manual basado en la hinchazón de la vulva y los signos de comportamiento a una prueba de contrapresión o jabalí. El método de observación requiere mucho tiempo y mano de obra, mientras que la precisión depende en gran medida de la experiencia del técnico (2). Pocos esteroides están presentes en la saliva del jabalí y se considera que contribuyen en gran medida a promover los comportamientos de estro en las cerdas (3). En este contexto, se sintetizó una mezcla análoga de saliva de jabalí de androstenona, androstanol y quinolina y se demostró eficaz para la detección del estro (4, 5). Como feromona esteroide, el análogo protege a las cerdas de la exposición al jabalí y la probable infección por enfermedad por jabalí teaser (6, 7).
Con el desarrollo de la inteligencia artificial, muchos investigadores se han centrado en la identificación automática del estro de cerda mediante la detección de cambios regulares de temperatura (8, 9), actividad (10) o la frecuencia y duración de la visita de la cerda a un jabalí (11) después de recopilar imágenes o videos masivos (12, 13). Estas identificaciones automáticas mostraron una precisión relativamente menor o equivalente en comparación con la observación manual (9, 11). Un estudio realizado por Lei (11) combinó por primera vez la visión artificial con un modelo de jabalí biónico para identificar cerdas en celo. A pesar del alto costo de las máquinas, el reconocimiento tiene una alta precisión del 98,25% y otras ventajas, como una mayor inteligencia y bioseguridad, pero menos estrés que los métodos de detección manual. También se han investigado estimaciones basadas en parámetros físicos o bioquímicos del moco vaginal (14), la concentración hormonal o las formas cristalinas en los fluidos corporales (15, 16), pero no se utilizaron ampliamente debido al alto costo de los kits de ensayo o sus características inestables. Sigue siendo una preocupación vital para la industria de la cría explorar métodos de diagnóstico de estro oportunos, eficientes y sensibles. Mientras tanto, se deben abordar las brechas de conocimiento con respecto a la fisiología de los ciclos estrales.
Como una secreción corporal importante, la saliva es rica en hormonas esteroides, ácidos nucleicos, proteínas y polipéptidos, porciones de las cuales pueden difundirse pasivamente o transportarse activamente desde la sangre, revelando una gran similitud con la sangre (17-19). Recientemente, la saliva se ha convertido en un material eficaz, no invasivo, fácilmente disponible y conveniente para detectar el estado fisiológico de los animales. La estimación de la saliva tiene grandes perspectivas de aplicación para la detección del estro (2). Aquí, comparamos la proteómica salival en cerdas durante el ciclo estral y verificamos la expresión relativa de varias proteínas para complementar nuestra comprensión de la fisiología del estro y proporcionar una referencia para el desarrollo del reconocimiento del estro.
Materiales y métodos
Recolección de animales y saliva
Para este estudio se seleccionaron ocho cerdas híbridas primíparas sanas (Duroc × cerdo cruzado tibetano) que mostraban estro normal. El bienestar animal en este estudio fue salvaguardado y aprobado por el Comité de Cuidado y Uso de Animales de la Academia de Ciencias Agrícolas de Guangdong (Protocolo número: 2018001). Las cerdas experimentales se observaron diariamente por la mañana y por la noche para evaluar su estado de celo. La identificación del estro se realizó mediante observación externa y la reacción de contrapresión. Una cerda parada y aceptando un jabalí arrastrándose con una vulva hinchada se consideraba estro, y el día del estro se definía como el día 0. La saliva, que representa cuatro etapas del ciclo estral, se recolectó todas las mañanas antes de la alimentación, incluyendo ED-3 (proestro, n = 3): 3 días antes del estro; ED0 (estro, n = 3): el día del estro; ED3 (metestro, n = 3): 3 días después del estro; y ED8 (diestro, n = 3): 8 días después del estro. El número de colecciones de saliva para cada punto de tiempo fue de tres. Los cerdos tenían curiosidad y masticaban las bolsas de algodón colgantes envueltas con bolas de algodón absorbentes. La saliva infiltrada en la bola de algodón absorbente fue exprimida, liberada en la bolsa de muestras y luego transferida a tubos limpios. Posteriormente, las muestras se centrifugaron a 4000 × g durante 5 min a 4 °C y el sobrenadante se mezcló con un cóctel inhibidor de la proteasa (10 μL/mL, Sigma) antes del almacenamiento a -80 °C hasta el ensayo de proteómica cuantitativa.
Extracción de proteínas, prueba de calidad, digestión y desalinización
La extracción de proteínas y el análisis de LS-MS fueron realizados por Novogene Co., Ltd. La muestra se lisó con tampón de lisis DB (8 M Urea, 100 mM TEAB, pH 8.5, Sigma) en un tubo de centrífuga de 1.5 mL, y el lisado se centrifugó a 12000 × g durante 15 min a 4°C. El sobrenadante se redujo con 10 mM de TDT (Sigma) a 56°C durante 1 h y se alquiló con suficiente IAM (Sigma) durante 1 h a temperatura ambiente en la oscuridad. La concentración total de proteína se determinó utilizando el kit cuantitativo de proteínas de Bradford (Beyotime). Luego, se cargaron 20 μg de proteína en cada pocillo para la electroforesis en gel para evaluar la calidad de la proteína. A continuación, se mezclaron 120 μg de muestras de proteínas con tampón de lisis DB (8 M Urea, 100 mM TEAB, pH 8.5, Sigma), tripsina (Promega) y tampón TEAB de 100 mM para hacer que el volumen final sea de hasta 100 μL. La mezcla se incubó durante 4 h a 37°C. Posteriormente, tripsina y CaCl2 (Sinopharm chemical reagent Co., Ltd) se añadieron, y las muestras se incubaron durante la noche. Se añadió ácido fórmico (Thermo Fisher Scientific) a las muestras digeridas, se ajustó a pH < 3 y se centrifugó a 12000 × g durante 5 min a temperatura ambiente. El sobrenadante se cargó en una columna de desalinización C18 (Thermo Fisher Scientific), se lavó tres veces con tampón de lavado (0,1% de ácido fórmico, 3% de acetonitrilo, Thermo Fisher), se eluió con tampón de elución (0,1% de ácido fórmico, 70% de acetonitrilo) y los eluyentes de cada muestra se recolectaron y liofilizaron.
Análisis LC-MS
Las proteínas liofilizadas se disolvieron en 10 μL de fase móvil A (0,1% de ácido fórmico en agua destilada desionizada), centrifugadas a 14.000 × g durante 20 min a 4 °C, y 1 μL de sobrenadante se cargó en una columna casera C18 Nano-Trap (4,5 cm × 75 μm, 3 μm). Luego, las proteínas se fraccionaron linealmente mediante una columna analítica casera (15 cm × 150 μm, 1,9 μm) con fases móviles A y B (80% acetonitrilo, 0,1% de ácido fórmico) y se analizaron utilizando un espectrómetro de masas Q Exactive™ HF-X (Thermo Fisher Scientific). La fuente de iones de Nanospray Flex™ (ESI) se escaneó completamente, variando de 350 a 1.500 m / z, en la que los 40 precursores principales de la mayor abundancia se fragmentaron por disociación de colisión de mayor energía (HCD) y se analizaron por MS / MS utilizando los siguientes parámetros: la resolución fue de 15,000 (a m / z 200), el valor objetivo de control automático de ganancia (20) fue de 1 × 105, el tiempo máximo de inyección de iones fue de 45 ms, la energía de colisión normalizada se estableció en 27%, el umbral de intensidad fue de 2.2 × 104, y el parámetro de exclusión dinámica fue de 20 s.
Identificación y cuantificación de proteínas
Los espectros fuera de línea se buscaron utilizando Proteome Discoverer 2.2 (PD 2.2, Thermo Fisher Scientific), con un máximo de dos sitios de escisión perdidos permitidos. Los péptidos con credibilidad superior al 99% se consideraron coincidencias de espectro peptídico (PSM). Una proteína que contiene al menos un péptido único se definió como confiable. Estos PSM preservados y proteínas confiables se sometieron a un análisis de tasa de descubrimiento falso (FDR) para eliminar datos mayores de 0.01. El carácter de distribución general de todas las proteínas identificadas se analizó con el programa en línea Jvenn.1 Las diferencias cuantitativas relativas se analizaron mediante la prueba t. Las proteínas con un cambio de pliegue ≥1,5 o ≤ 0,67 y p ≤ 0,05 se definieron como DEP.
Análisis bioinformático
Tanto las proteínas identificadas como los DEP se sometieron a análisis bioinformáticos. La predicción funcional de Gene Ontology (GO) e InterPro (21) se realizó utilizando un programa interproscan (versión 5.22-61.0) contra las bases de datos de proteínas no redundantes, incluidas Pfam, PRINTS, ProDom, SMART, ProSite y PANTHER (22). Las predicciones de la familia de proteínas y las vías se analizaron utilizando las bases de datos Clusters of Orthologous Groups (23) y Kyoto Encyclopedia of Genes and Genomes (KEGG). Las posibles interacciones proteína-proteína se analizaron utilizando el servidor STRING-db (24).2
Validación del nivel de expresión relativo por PRM
Para verificar los niveles de expresión relativos de DEP identificados en el análisis proteómico cuantitativo sin etiquetado, las muestras de saliva recolectadas en días −3, 0,3, 8 para cada día (n = 3) durante el ciclo estral fueron confirmadas por PRM con un espectro adquirido de MS / MS. Las proteínas extraídas de la saliva fueron probadas en cantidad y calidad, hidrolizadas enzimáticamente, desalinizadas y liofilizadas, como se describió anteriormente. Para el análisis LC-MS/MS previo al experimento, se eluyó 1 μg de la mezcla como un método «sin etiqueta» utilizando el sistema UHPLC EASY-nLCTM1200 (Thermo Fisher Scientific) junto con un espectrómetro de masas de la serie Q Exactive (Thermo Fisher Scientific). Los datos brutos se buscaron mediante el software PD2.2 con modo de escaneo completo y el patrón PRM secuencialmente, y los péptidos seleccionados se determinaron utilizando el software Skyline basado en la reproducibilidad y estabilidad. En los experimentos formales LC-MS/MS, péptidos equivalentes pretratados con tripsina fueron enriquecidos con la misma cantidad de péptido marcado con isótopos que un estándar interno. Las muestras se analizaron mediante escaneo completo y patrones PRM como se describió anteriormente. Para el análisis de datos fuera de línea, el área pico de cada proteína diana se corrigió de acuerdo con el péptido estándar interno para que estuviera disponible para la evaluación posterior de la abundancia relativa.
Análisis de datos
Los niveles relativos de expresión de proteínas fueron representados por cambios en el pliegue comparando la abundancia de proteínas durante el ciclo estral. El análisis estadístico fue realizado por GraphPad Prism, versión 8. Las comparaciones de datos entre dos grupos se analizaron mediante la prueba t. Las comparaciones de datos entre tres o más grupos se realizaron mediante ANOVA unidireccional, seguido de la prueba de rango múltiple de Tukey. La significación estadística se fijó en p < 0,05.
Resultados
Identificación de proteínas salivales durante el ciclo estral
Se identificaron un total de 8.091 péptidos y 1.155 proteínas en muestras salivales, incluidas 885, 923, 942 y 908 proteínas en los grupos ED-3, ED0, ED3 y ED8, respectivamente. El carácter de distribución general de todas las proteínas identificadas se analizó con el programa en línea Jvenn, como se muestra en la Figura 1. El número de proteínas compartidas en cuatro grupos fue de 791, y el número de proteínas específicas para ED-3, ED0, ED3 y ED8 fue de 6, 17, 9 y 4, respectivamente (Figura 1).
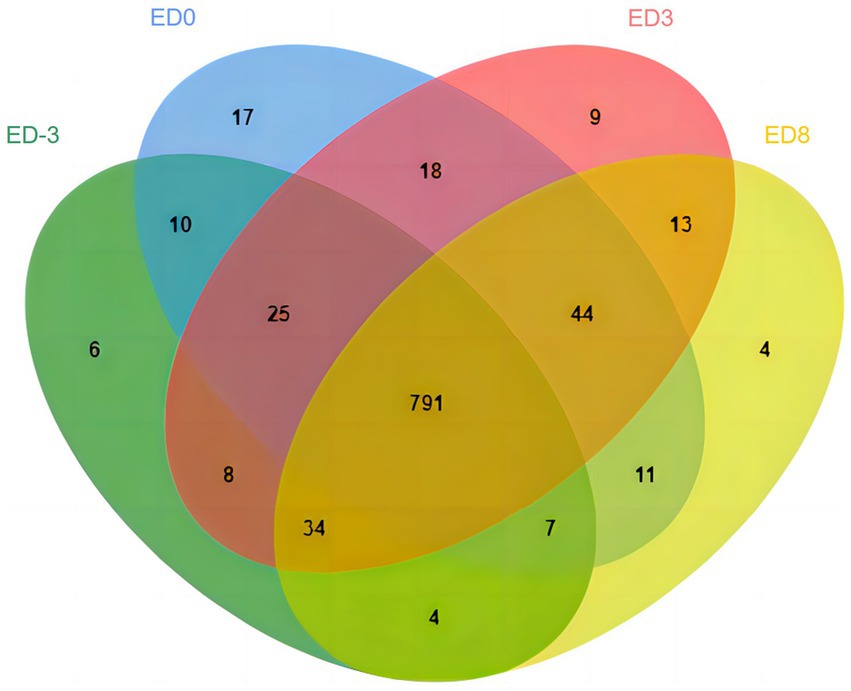 Figura 1. Diagrama de Venn de proteínas identificadas en las etapas de proestro (ED-3), estro (ED0), metestro (ED3) y diestro (ED8) en saliva de cerdo.
Figura 1. Diagrama de Venn de proteínas identificadas en las etapas de proestro (ED-3), estro (ED0), metestro (ED3) y diestro (ED8) en saliva de cerdo.
Predicción funcional de proteínas totales
La predicción funcional y la clasificación de las proteínas totales se realizaron utilizando el programa interproscan, como se muestra en la Figura 2. Los resultados del enriquecimiento de GO indicaron que todas las proteínas se clasificaron en tres dominios: 666 proteínas en función molecular, 399 proteínas en procesos biológicos y 202 proteínas en componente celular (Figura 2A). La función molecular tuvo el mayor número de ítems, y los tres ítems principales fueron unión a proteínas (n = 96), unión a iones de calcio (n = 44) y actividad endopeptidasa de tipo serina (n = 36). Los resultados del proceso biológico indicaron que las proteínas estaban involucradas en la proteólisis (n = 57), el proceso de oxidación-reducción (n = 47) y el proceso metabólico de carbohidratos (n = 23). De acuerdo con los resultados de los componentes celulares, la mayoría de las proteínas están involucradas en la región extracelular (n = 43), la membrana (n = 23), el filamento intermedio (n = 21) y el espacio extracelular (n = 20). Para el análisis COG (Figura 2B), las tres clases funcionales principales se clasificaron como O (modificación postraduccional, recambio de proteínas, chaperonas), R (predicción de función general solamente) y G (transporte y metabolismo de carbohidratos) con 159, 75 y 42, respectivamente. El análisis KEGG (Figura 2C) reveló que la mayoría de las proteínas estaban involucradas en el metabolismo (n = 362), los procesos organísmicos (n = 300) y celulares (n = 208), mientras que el procesamiento de información ambiental (n = 117) y la transducción de señales (n = 94) enriquecieron menos proteínas. En la predicción de IPR (Figura 2D), el dominio V-set de inmunoglobulina (n = 50) contenía la mayoría de las proteínas.
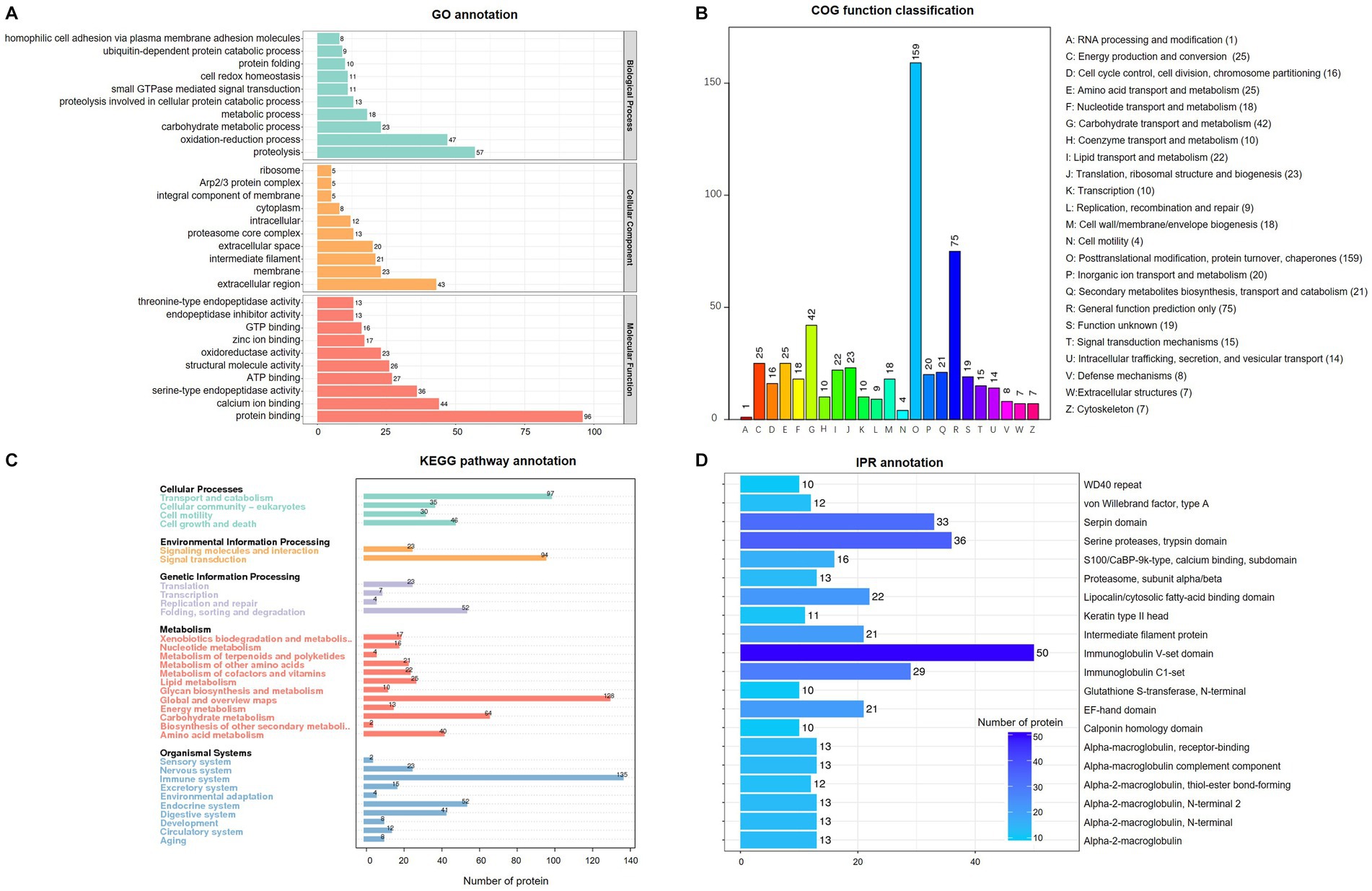 Figura 2. Análisis de enriquecimiento de proteínas totales identificadas en saliva de cerdo durante el ciclo estral. (A) Análisis de ontología génica (GO); b) grupos de grupos ortólogos de enriquecimiento de proteínas; (C) Anotación de la vía de la Enciclopedia de Genes y Genomas de Kioto (KEGG). D) Análisis funcional de InterPro (DPI).
Figura 2. Análisis de enriquecimiento de proteínas totales identificadas en saliva de cerdo durante el ciclo estral. (A) Análisis de ontología génica (GO); b) grupos de grupos ortólogos de enriquecimiento de proteínas; (C) Anotación de la vía de la Enciclopedia de Genes y Genomas de Kioto (KEGG). D) Análisis funcional de InterPro (DPI).
Anotación funcional y predicción de DEPs
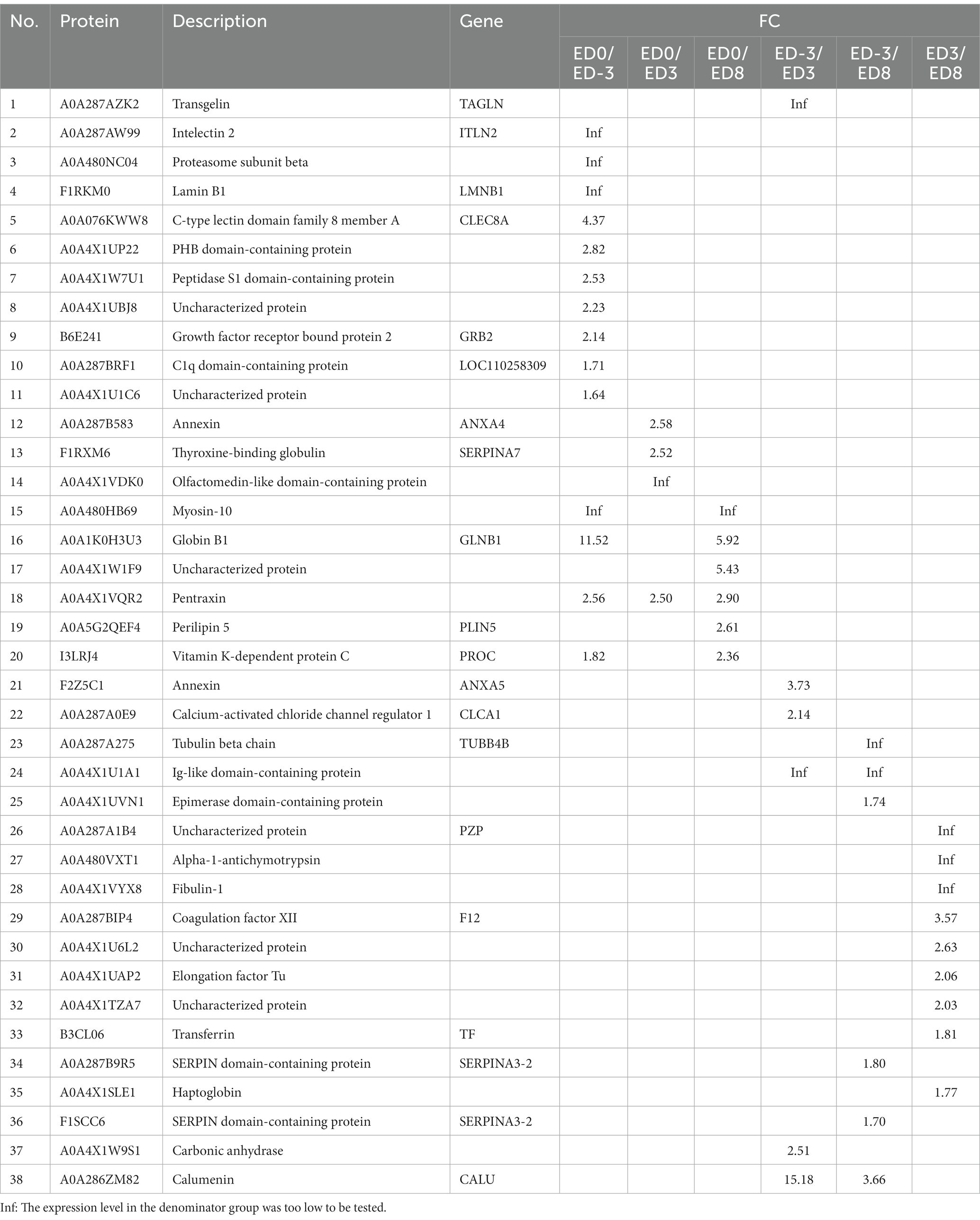
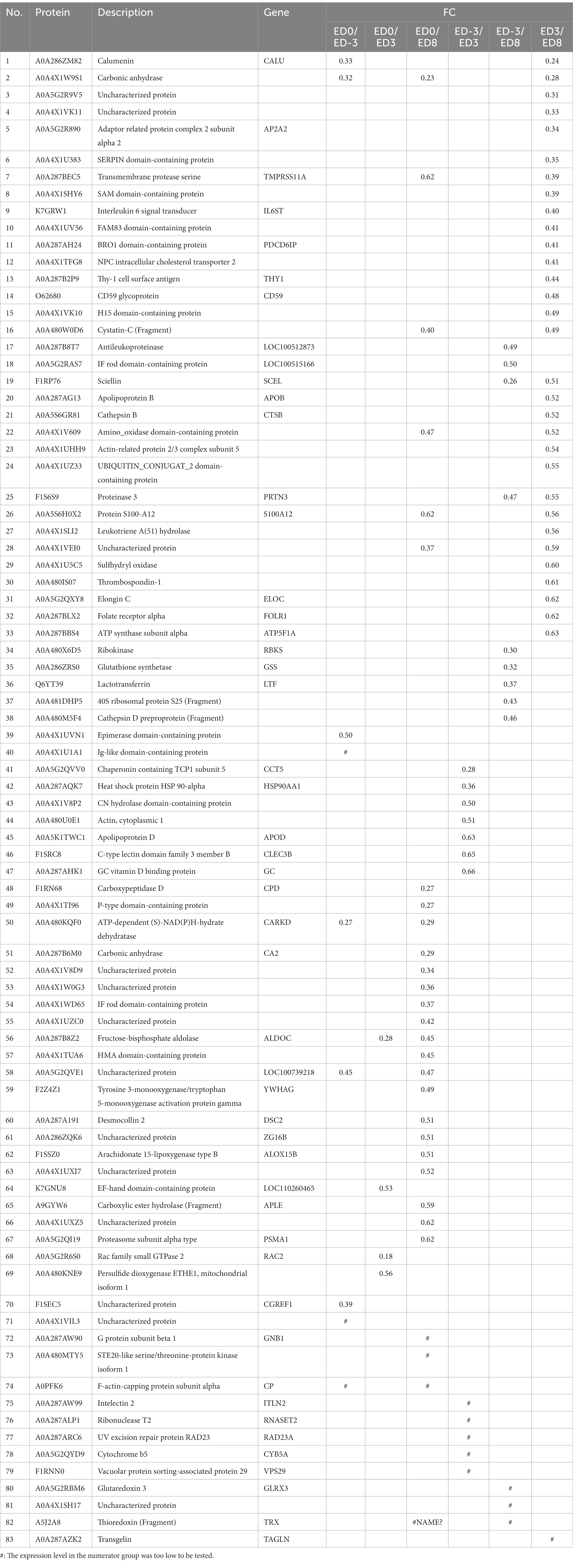
La cantidad de proteína se comparó entre los diferentes grupos para aislar DEPs (FC ≥ 1,5, p ≤ 0,05). En total, se identificaron 93 proteínas como DEP, de las cuales la expresión de 38 proteínas se reforzó al alza y 83 se regularon a la baja. Los cambios relativos en el pliegue se muestran en las Tablas 1, 2. La comparación ED3 vs. ED8 y ED0 vs. ED8 mostró el mayor número de proteínas con expresiones reguladas a la baja, con 30 y 34, respectivamente. La mayoría de las proteínas con expresiones reguladas al alza provenían de los grupos ED0 vs. ED-3 y ED3 vs. ED8 (15 y 11, respectivamente). En ED0 vs. ED-3 y ED0 vs. ED8, las expresiones de 20 proteínas fueron reguladas al alza, y las de 12 fueron reguladas a la baja con FC > 2. Todos los DEP se dividieron en seis conjuntos de acuerdo con los patrones de expresión de series temporales, en los que se agruparon 21, 14, 10, 36, 17 y 17 proteínas (Figura 3).
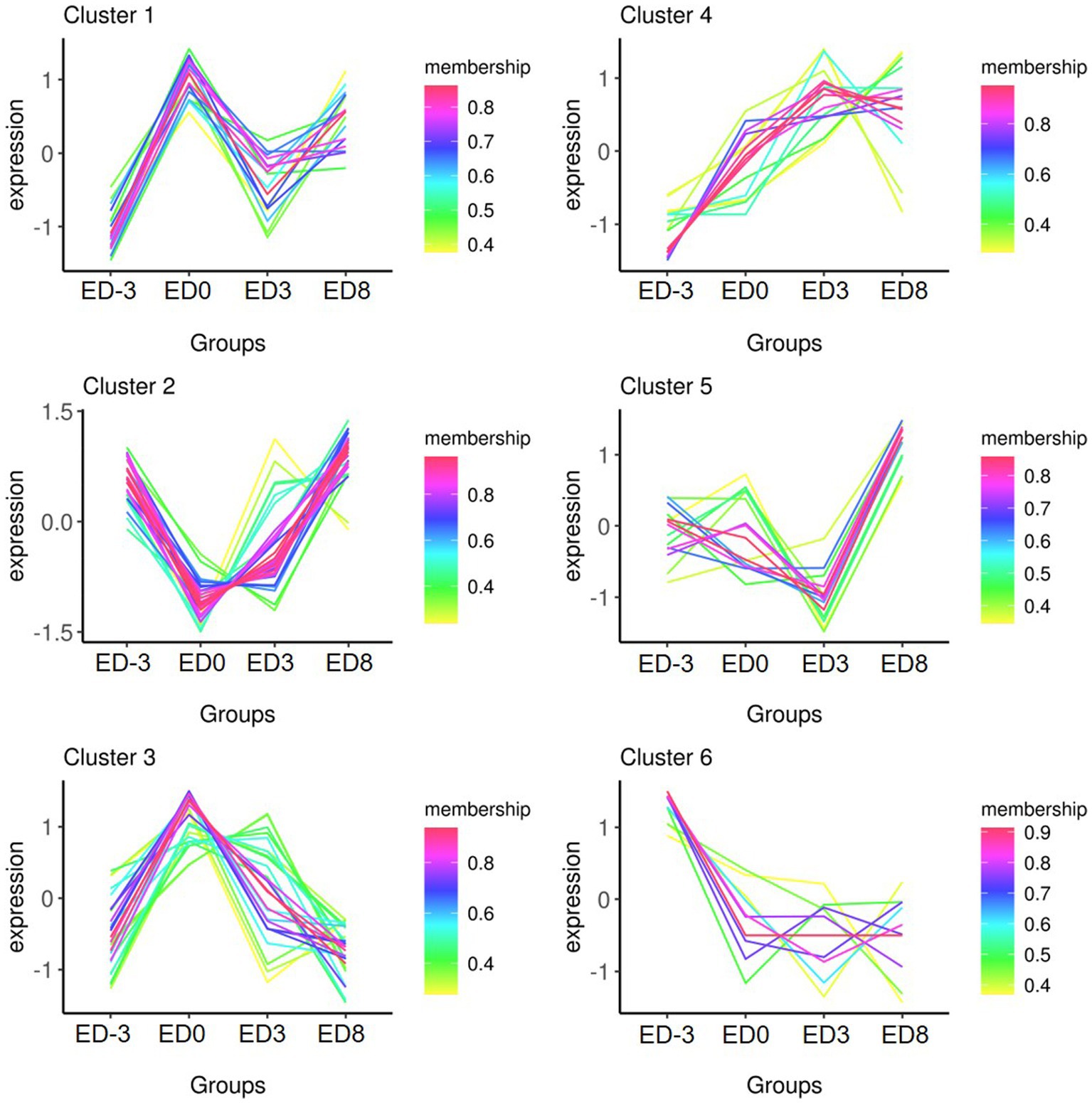 Figura 3. Tendencias de expresión de series temporales de seis grupos de DEP en saliva de cerdo durante el ciclo estral.
Figura 3. Tendencias de expresión de series temporales de seis grupos de DEP en saliva de cerdo durante el ciclo estral.
La anotación funcional se realizó mediante análisis GO y KEGG. De acuerdo con el análisis de enriquecimiento (Tabla 3), los ítems significativos de GO generados entre las diferentes comparaciones con ED0 se clasificaron principalmente por función molecular, incluyendo citoesqueleto de actina, unión a iones de calcio, actividad de la deshidratasa de hidratos de hidrato NAD(P)H-dependiente de ADP, proceso glucolítico, actividad fructosa-bisfosfato aldolasa, unión fosfolípida dependiente de calcio y actividad liasa. En el análisis de enriquecimiento de KEGG (Tabla 4), las vías significativas se relacionaron principalmente con la vía de señalización de los receptores activados por proliferadores de peroxisomas (PPAR), la resistencia al inhibidor de la tirosina quinasa del receptor del factor de crecimiento epidérmico (EGFR), la vía de señalización de la prolactina, la vía de señalización del factor de crecimiento endotelial vascular (VEGF), la vía de señalización Wnt, la vía de señalización cAMP, el metabolismo del nitrógeno y la sinapsis serotoninérgica.
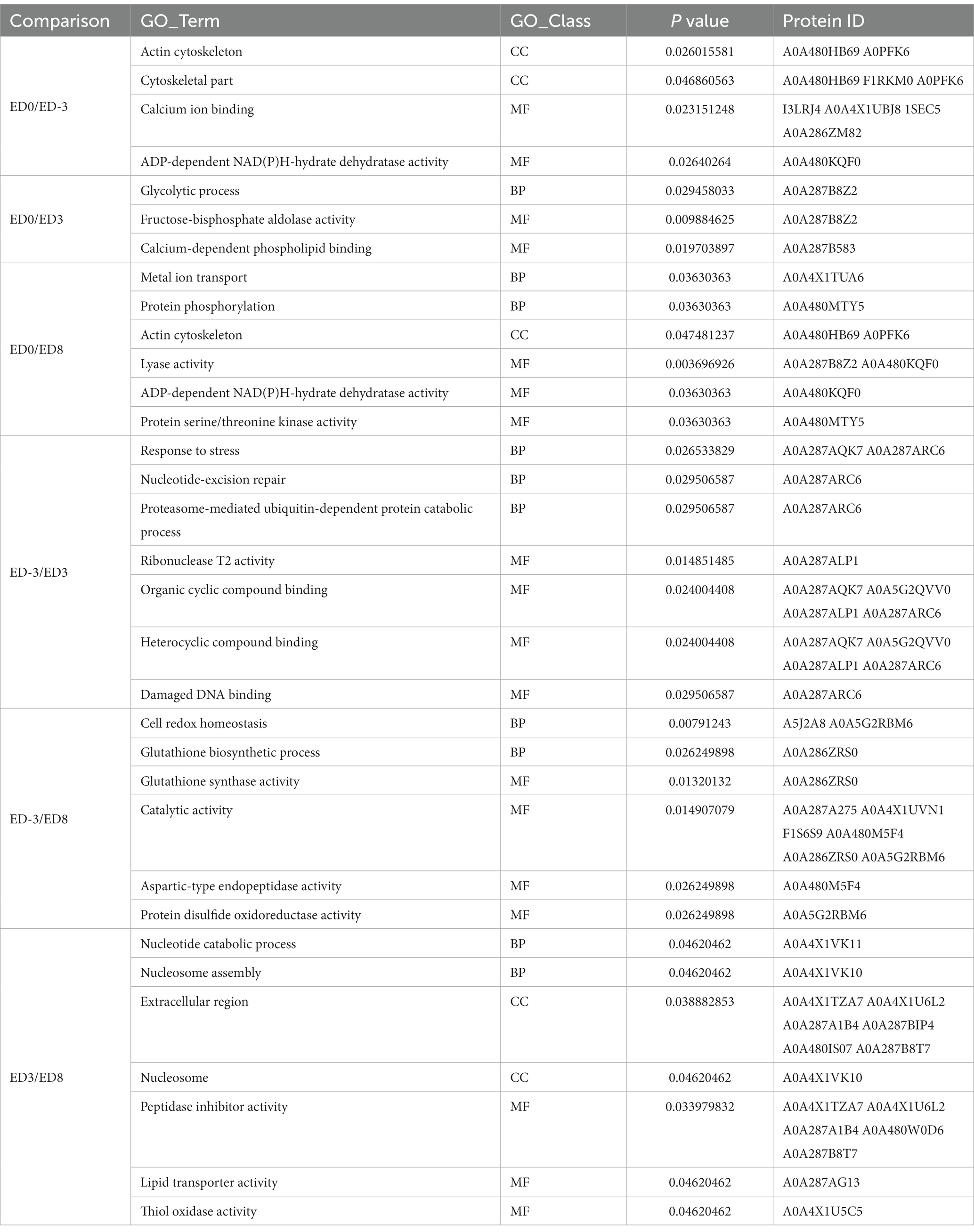 Tabla 3. Los elementos GO se enriquecieron de manera significativamente diferente entre las diferentes comparaciones.
Tabla 3. Los elementos GO se enriquecieron de manera significativamente diferente entre las diferentes comparaciones.
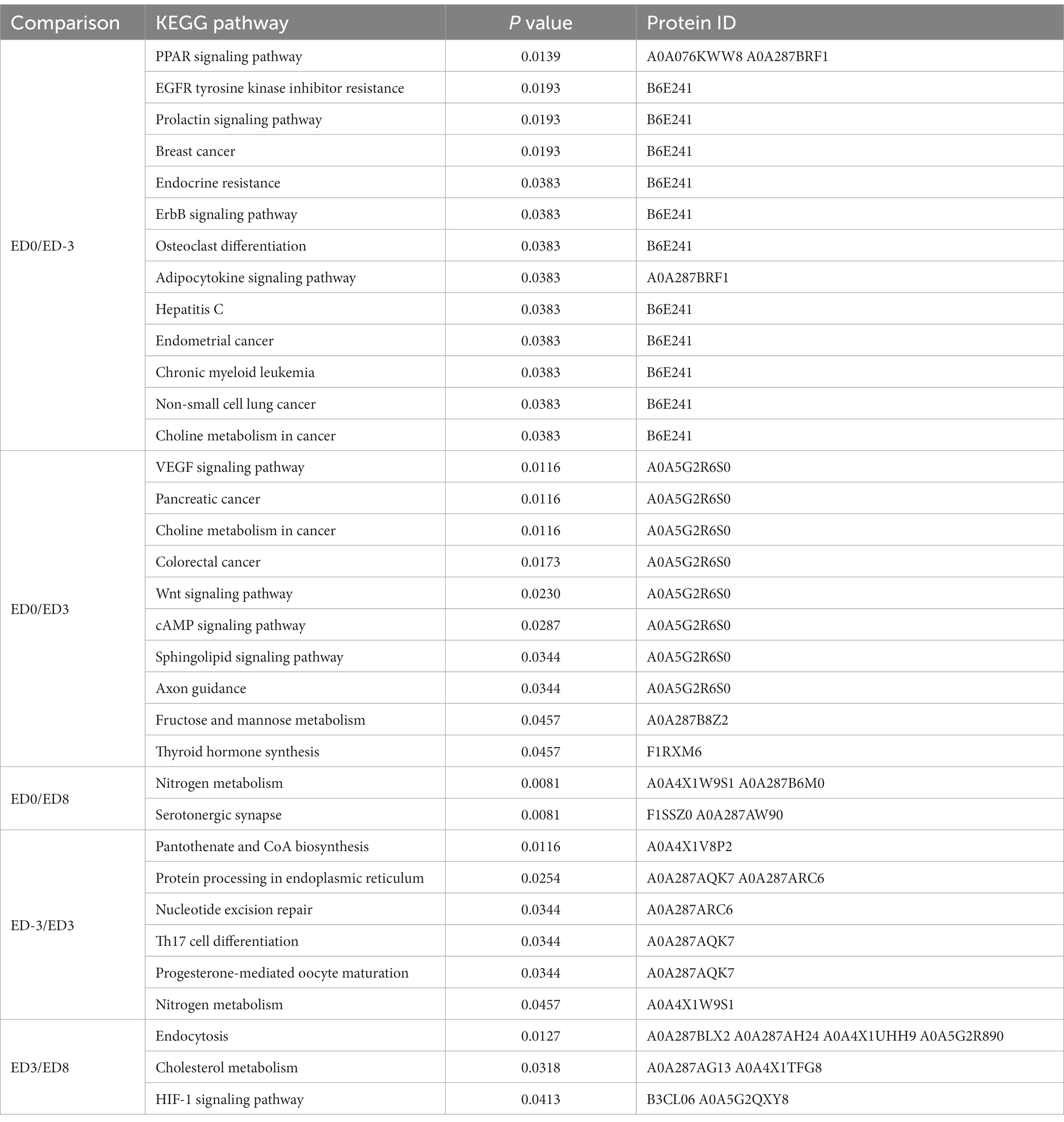 Tabla 4. Lista de vías enriquecidas de manera significativamente diferente entre diferentes comparaciones.
Tabla 4. Lista de vías enriquecidas de manera significativamente diferente entre diferentes comparaciones.
Verificación de expresiones relativas por PRM
La cohorte de 12 muestras (3 réplicas) derivada de cuatro puntos de tiempo representativos del ciclo estral se envió para el ensayo PRM para verificar la expresión relativa. El análisis cuantitativo PRM de los DEP mostró un alto acuerdo con los resultados sin etiqueta (Figura 4). Las tendencias relativas de expresión de seis proteínas, incluida la proteína que contiene el dominio de estomatina (STOM, A0A4X1UP22), la proteína 2 unida al receptor del factor de crecimiento (Grb2, B6E241), el factor de elongación Tu (EFTU, A0A4X1UAP2), la carboxipeptidasa D (CPD, F1RN68), la anhidrasa carbónica (CA, A0A4X1W9S1) y el factor trébol 3 (TFF3, A0A4X1TI96), se validaron utilizando el método PRM y mostraron tendencias de expresión similares como libres de etiquetado. Las tres primeras proteínas mostraron una actualización, seguida de una tendencia descendente, con un nivel máximo de expresión de 1,34 × 107, 1,48 × 106, 1,30 × 107, respectivamente, para el análisis sin etiqueta, y 0,0017, 0,00044, 0,00063, respectivamente, para el análisis PRM, en celo. En cambio, las otras tres proteínas mostraron una tendencia significativa de ‘V’, con el nivel más bajo de 6.6 × 105, 1,91 × 109, 1,10 × 107, respectivamente, para el análisis sin etiqueta, y 0,00043, 0,15, 0,0027, respectivamente, para el análisis PRM, que aparece en celo.
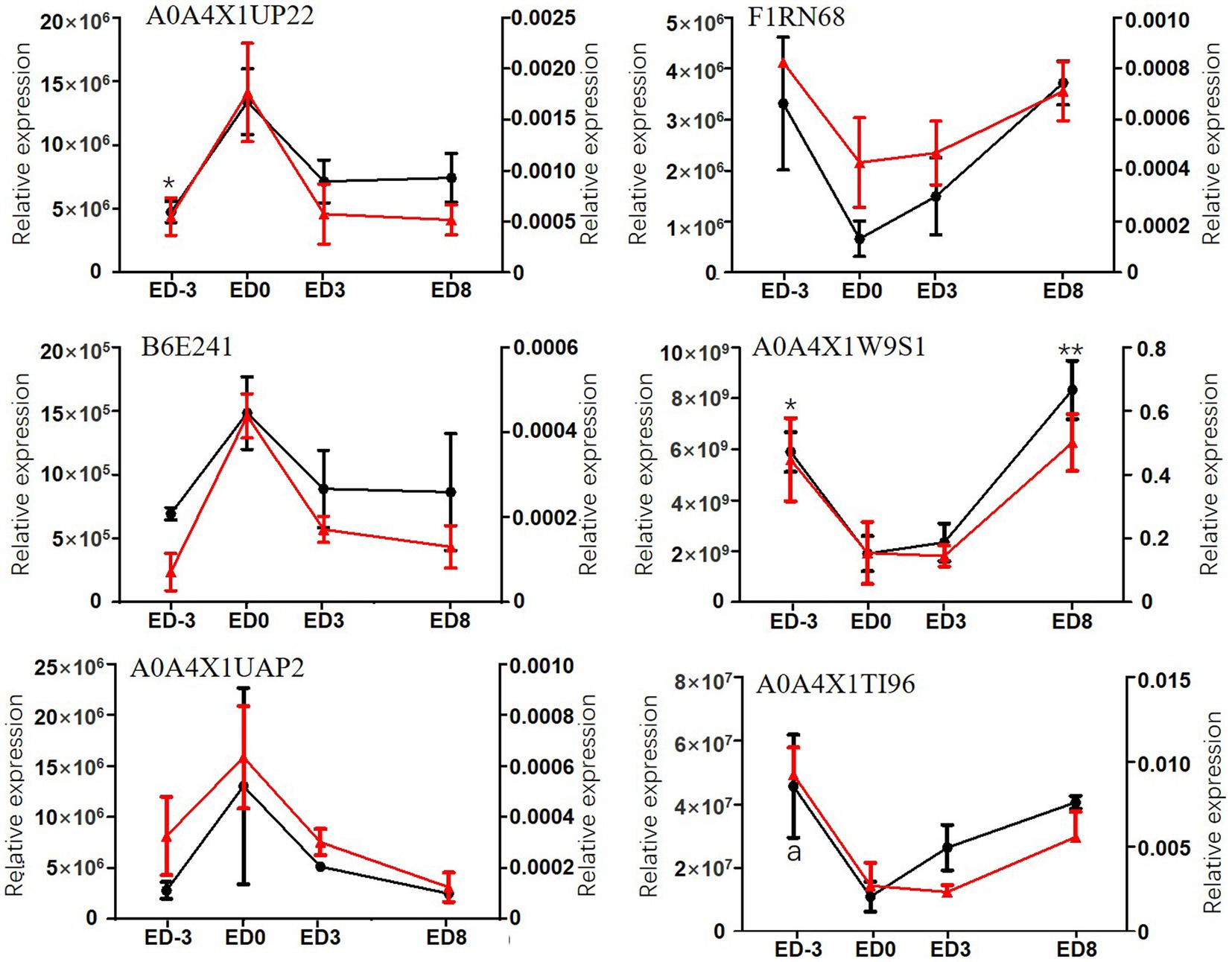 Figura 4. Niveles relativos de expresión de seis DEPs ensayados por método sin etiqueta y PRM en saliva de cerdo durante el ciclo estral. La coordenada principal representa el ensayo sin etiqueta, izquierda, líneas rojas; * y ** indican diferencias significativas en comparación con ED0 , p < 0,05, p < 0,01, respectivamente. La coordenada secundaria representa el ensayo PRM, derecha, líneas negras; a representa la diferencia significativa en comparación con ED0.
Figura 4. Niveles relativos de expresión de seis DEPs ensayados por método sin etiqueta y PRM en saliva de cerdo durante el ciclo estral. La coordenada principal representa el ensayo sin etiqueta, izquierda, líneas rojas; * y ** indican diferencias significativas en comparación con ED0 , p < 0,05, p < 0,01, respectivamente. La coordenada secundaria representa el ensayo PRM, derecha, líneas negras; a representa la diferencia significativa en comparación con ED0.
Discusión
El presente estudio se centró en caracterizar la proteómica general durante el ciclo estral en saliva de cerdo y buscó proteínas relevantes o específicas del estro. Dadas las evidentes ventajas de la saliva en la diversidad de especies proteicas, la alta abundancia y la inmensa popularidad en el diagnóstico biológico, es razonable plantear la hipótesis de que la exploración de perfiles de proteínas y proteínas específicas del estro en la saliva puede ayudar a desarrollar nuevos métodos de diagnóstico del estro no invasivos. Datos recientes mostraron que se encontraron 1.155 proteínas totales en cuatro grupos representativos, y hasta 115 proteínas se expresaron diferencialmente entre los diferentes grupos. Tanto el número total como los DEP fueron mayores que los obtenidos por Li (2), lo que puede ser causado por diferentes razas y comparaciones. La cantidad de PRM validó los patrones de expresión de las seis proteínas, incluida la proteína que contiene el dominio de estomatina, la proteína unida al receptor del factor de crecimiento, el factor de elongación Tu, la carboxipeptidasa, las anhidras carbónicas y el factor de trébol 3, mostrando tendencias de expresión consistentes con el método sin etiquetado.
Los niveles de proteína cambiaron con el tiempo durante el ciclo estral. En este estudio, muchos DEP fueron regulados al alza en el estro, como el dominio de lectina tipo C miembro 8 A, STOM, Grb2, anexina A4, globulina fijadora de tiroxina, globina B1, pentraxina, perilipina 5 (Plin5), proteína C dependiente de vitamina K. La expresión de pentraxina en las células del cúmulo se correlaciona positivamente con la fertilización in vivo al afectar la expansión del cúmulo oophorus, la calidad de los ovocitos correspondientes y la ovulación (25). Plin5 regula bidireccionalmente el equilibrio dinámico del metabolismo lipídico en los tejidos oxidativos mediante el control de la actividad de la triglicéridos adiposa lipasa. Plin5 inhibe la lipólisis en el estado básico, mientras que el proceso de lipólisis suprimida se aceleraría una vez que se produjera el estrés (26). Sin embargo, hay pocos estudios sobre la función de las otras proteínas reguladas al alza en el estro.
En particular, las expresiones de muchas proteínas fueron reguladas a la baja durante el diestro. La anhidrasa carbónica (AC), como enzima que contiene zinc, está ampliamente distribuida en los tejidos de mamíferos, incluido el tracto reproductivo (27). La AC desempeña un papel clave en el equilibrio de los fluidos corporales al influir en el transporte metabólico de dióxido de carbono (28). LTF es un componente importante de la defensa de los fluidos de la mucosa debido a las propiedades antimicrobianas e inmunomoduladoras. Es evidente que la distribución bimodal de LTF en los componentes inmunes cervicales está determinada por el ciclo menstrual (29). Además, LTF se expresa correlacionado con el nivel circulante de 17β-estradiol (E2), y sintetizados selectivamente en el epitelio uterino dependiente del receptor de estrógeno α (ERα) (30). Para varias otras proteínas reguladas a la baja durante el diestro, por ejemplo, calumenina, proteína que contiene el dominio SERPIN, sciellin, riboquinasa, glutatión sintetasa, hay pocos informes disponibles para su papel en la reproducción.
La clasificación de la función GO de los DEP estableció su estrecha asociación con el proceso biológico, los componentes celulares y la función molecular. La unión a iones de calcio se enriqueció en el mayor número de proteínas en el grupo ED0 vs. ED-3. Como segundo mensajero intracelular, los iones de calcio son cruciales en los procesos fisiológicos celulares y son esenciales para el crecimiento de ovocitos de mamíferos, la meiosis, la producción de hormonas, la secreción del factor de crecimiento y la maduración meiótica (31). En esta categoría, las cuatro proteínas enriquecidas, I3LRJ4, A0A4X1UBJ8, 1SEC5 y A0A286ZM82, sugirieron su participación en la maduración de los ovocitos. El citoesqueleto de actina contribuye a la maduración meiótica de los ovocitos al mediar el ensamblaje del huso, la longitud y la segregación cromosómica (32). La superposición del citoesqueleto de actina en los grupos ED0 vs. ED8 y ED0 vs. ED-3 enfatizó su importancia durante la etapa de estro.
Se realizó un análisis de enriquecimiento KEGG de DEP para identificar las vías funcionales durante el ciclo reproductivo. En comparación con ED0, las vías más enriquecidas incluyeron la vía de señalización PPAR, la resistencia al inhibidor de la tirosina cinasa EGFR, la vía de señalización de la prolactina, la vía de señalización del VEGF, la vía de señalización Wnt, la vía de señalización cAMP, el metabolismo del nitrógeno y la sinapsis serotoninérgica. Se demostró que la vía PPAR es fundamental en la gametogénesis canina, la ovulación o la regresión de CL durante el ciclo estral (33). La señalización del VEGF es una vía clave para la proliferación de células de la granulosa, la síntesis de hormonas esteroides, la inhibición de la apoptosis y la angiogénesis ovárica durante el desarrollo folicular y la ovulación (34). La vía Wnt es necesaria para el desarrollo ovárico embrionario y adulto. Wnt-4 estimula la diferenciación de las células de la granulosa y el desarrollo sexual femenino durante la etapa embrionaria. En contraste, la función ovárica adulta y la fertilidad dependen de las acciones sincronizadas de los genes y hormonas de la familia Wnt (35). Los efectos inhibitorios del óxido nítrico (NO), un producto representativo del metabolismo del nitrógeno, sobre la esteroidogénesis se han reportado en varios modelos animales. Los efectos controvertidos del NO durante la foliculogénesis, que protege o induce la apoptosis folicular, fueron dependientes de la concentración. Además, la vía del NO ejerce una influencia central en la maduración meiótica de los ovocitos de mamíferos entre las numerosas moléculas mediadoras (36). La fuerte relación entre estas vías y la fisiología reproductiva fortalece la confiabilidad de nuestros resultados de enriquecimiento.
Tanto los grupos sin etiqueta como los PRM revelaron consistentemente los niveles máximos de STOM, Grb2 y EFTU durante el estro. Hay cinco miembros en la familia de proteínas que contienen dominio estomatína: STOM, STOML1, STOML2, STOML3 y podocina (37). La proliferación e invasión de trofoblastos humanos puede ser mediada por STOML2 a través de la modulación de la función mitocondrial (38). Un aumento significativo en la expresión de STOML2 podría promover la proliferación y decidualización de las células del estroma endometrial en ratones y humanos (39). Grb2 induce varios eventos celulares como una proteína adaptadora en las vías de transducción de señales (40). El complejo Grb2/Sos activa Ras en respuesta a factores de crecimiento celular. Grb2 está implicado en el desarrollo vulvar y la migración de mioblastos sexuales en Caenorhabditis. elegans (40). En los ovocitos de Xenopus, Grb2 indujo el reinicio de la meiosis (40). Un estudio identificó el Grb2 como un gen candidato clave en la producción de progesterona durante la ovulación a través del análisis bioinformático y verificó su función en las vías progestogénicas en AOC de rata (41). En el estudio actual, Grb2 mostró máxima expresión durante el estro, lo que podría estar relacionado con la ovulación. EFTU aumentó del día 12 al 16 en el cuerpo lúteo de las ovejas cíclicas (42), lo que puede ayudar a la regresión del cuerpo lúteo durante el estro. En contraste, EFTU fue más abundante en el día del estro en nuestro estudio, lo que indica sus efectos potenciales sobre el desarrollo folicular, la ovulación y la síntesis de LH. Esta tendencia distinta puede atribuirse a diferentes organismos o tejidos.
Simultáneamente, se observó una expresión mínima de CPD, CA y TFF3 en los productos libres de etiqueta y PRM. CPD es un miembro de la familia de las carboxipeptidasas (CP) caracterizado por un dominio metalocarboxipeptidasa conservado. La familia de CP en mamíferos incluye CPH, CPM, CPE y otras nueve proteínas (43). Las señales de CPD son significativamente altas en los folículos bovinos dominantes y se consideran un marcador confiable para los folículos dominantes (44). La DPC tiene grandes similitudes funcionales y estructurales con otras PC (45); sin embargo, hay estudios limitados disponibles sobre la función de CPD en la reproducción (44, 46). En nuestro estudio, CPD mostró el nivel más bajo de saliva en el día del estro, lo que sugiere que CPD puede regular la fisiología del estro mediante la modulación de los procesos precursores de hormonas y neuropéptidos. Un estudio previo exploró la localización de AC en los genitales de cerdos hembras. La AC fue positiva en el endotelio de los vasos sanguíneos y ausente en el parénquima ovárico. En el oviducto, se observó actividad conspicua de AC en el epitelio superficial de la ampolla tubárica, donde ocurrió la fertilización y el desarrollo temprano del embrión. Además, la AC se tiñó en el istmo trompa de la unión uterotubárica, que actúa como reservorio de espermatozoides. Tanto la intensidad como la localización de la AC se mantuvieron constantes durante el ciclo estral. La localización de AC puede indicar su papel crucial en el mantenimiento de la homeostasis ácido-base en el fluido luminal (47). Asimismo, la implicación de la AC en la secreción de bicarbonato durante el ciclo estral también se observó en ratones y ratas uterinas (48, 49) ya que la anhidrasa carbónica 2 (CAR2, CA2) tuvo la máxima expresión en el estro durante el ciclo estral cuando el pH de la superficie uterina en reposo fue significativamente mayor que en el diestro; sin embargo, el pH alto podría reducirse significativamente con el inhibidor de CA (48). Sorprendentemente, el estrógeno indujo un aumento paralelo en la expresión de CAR2 y el pH de la superficie endometrial. Nuestros datos mostraron el resultado opuesto; La AC tuvo la expresión más baja en el estro. Por lo tanto, el patrón de expresión de AC puede cambiar con las especies. En humanos, la expresión de TFF3 está regulada al alza en el día 4 frente al día 2 de la menstruación y está implicada en la regeneración y reparación endometrial durante la menstruación (50). La presencia de TFF3 en la saliva puede estar asociada con el endometrio, mientras que la función subyacente y los mecanismos del estro aún no se conocen y deben resolverse.
Conclusión
El presente estudio comparó los perfiles globales de proteoma de saliva durante el ciclo estral basado en la proteómica cuantitativa libre de etiquetas en un cerdo cruzado. La identificación y el análisis de enriquecimiento de las proteínas expresadas diferencialmente indicaron la participación de muchas proteínas en las funciones fisiológicas del estro porcino. Las tendencias de expresión de las seis proteínas se confirmaron utilizando PRM. Este estudio proporcionó nuevos conocimientos sobre la fisiología del ciclo reproductivo y sirvió como referencia para el desarrollo de kits o tiras de detección de estro. Sin embargo, se necesitan más investigaciones para descubrir la confusión con respecto a la generación de DEP, la secreción, los mecanismos de función y el potencial práctico en la detección del celo.
Declaración de disponibilidad de datos
Los datos presentados en el estudio están depositados en el repositorio del Consorcio ProteomeXchange, número de acceso PXD043141.
Contribuciones del autor
HX concibió el estudio, diseñó y realizó los experimentos, realizó análisis de datos, preparó figuras y tablas, y escribió el manuscrito. BL, BH y YW prepararon materiales y recolectaron muestras. SW y FM modificaron el manuscrito. JL supervisó el estudio. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Este estudio fue apoyado por los siguientes proyectos: Programa Provincial Clave de Investigación y Desarrollo de Guangdong (2022B0202090003); Equipo de innovación porcina del sistema de tecnología industrial agrícola moderna de Guangdong (2022KJ126); Proyecto de formación de equipos científicos de la industria agrícola superior de la Academia de Ciencias Agrícolas de Guangdong (202118TD); Proyecto de revitalización de la industria de siembra de fondos especiales de la estrategia de revitalización rural de la provincia de Guangdong (2021-44000-24010202-8887); Parque de innovación de la industria de semillas de cerdo Guangdong Xiaoerhua (2022-4408X1-43010402-0019).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Notas
Referencias
1. Endo, N. Posibles causas y estrategias de tratamiento para el estro y los trastornos de la ovulación en vacas lecheras. J Reprod Dev. (2022) 68:85–9. DOI: 10.1262/JRD.2021-125
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Li, C, Song, C, Qi, K, Liu, Y, Dou, Y, Li, X, et al. Identificación del estro en cerdas basada en la proteómica salival. Animales (Basilea). (2022) 12:1656. doi: 10.3390/ani12131656
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Sankarganesh, D, Kirkwood, RN, Nagnan-Le Meillour, P, Angayarkanni, J, Achiraman, S, and Archunan, G. Pheromones, binding proteins, and olfactory systems in the pig (Sus scrofa): an updated review. Front Vet Sci. (2022) 9:989409. DOI: 10.3389/FVETS.2022.989409
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. McGlone, JJ, Garcia, A, and Rakhshandeh, A. Multi-farm analysis indicate a novel jabai pheromone improve sow reproductive performance. Animales (Basilea). (2019) 9:37. DOI: 10.3390/ANI9020037
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Mcglone, JJ, Devaraj, S, y Garcia, A. Una nueva mezcla de feromonas de jabalí induce comportamientos de celo de cerda y éxito reproductivo. Appl Anim Behav Sci. (2019) 219:104832. doi: 10.1016/j.applanim.2019.104832
6. Sankarganesh, D, Kirkwood, R, Angayarkanni, J, Achiraman, S, and Archunan, G. Pig pheromones and behaviors: a review. Teriogenología. (2021) 175:1–6. doi: 10.1016/j.theriogenology.2021.08.032
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Van den Berghe, F, Paris, MCJ, Sarnyai, Z, Vlamings, B, Millar, RP, Ganswindt, A, et al. La feromona apaciguadora del perro previene el aumento de andrógenos y puede reducir el dominio del contacto y la sumisión activa después de intervenciones estresantes en perros salvajes africanos (Lycaon pictus). PLoS One. (2019) 14:E0212551. doi: 10.1371/journal.pone.0212551
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Simões, VG, Lyazrhi, F, Picard-Hagen, N, Gayrard, V, Martineau, GP, y Waret-Szkuta, A. Variaciones en la temperatura vulvar de las cerdas durante el proestro y el estro según lo determinado por termografía infrarroja y su relación con la ovulación. Teriogenología. (2014) 82:1080–5. doi: 10.1016/j.theriogenology.2014.07.017
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Sykes, DJ, Couvillion, JS, Cromiak, A, Bowers, S, Schenck, E, Crenshaw, M, et al. El uso de imágenes térmicas infrarrojas digitales para detectar estros en primerizas. Teriogenología. (2012) 78:147–52. doi: 10.1016/j.theriogenology.2012.01.030
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Freson, L, Godrie, S, Bos, N, Jourquin, J, y Geers, R. Validación de un sensor infrarrojo para la detección del celo de cerdas alojadas individualmente. Comput Electron Agric. (1998) 20:21–9. doi: 10.1016/S0168-1699(98)00005-2
11. Lei, K, Zong, C, Du, X, Teng, G, and Feng, F. Oestrus analysis of sows based on bionic baars and machine vision technology. Animales (Basilea). (2021) 11:1485. doi: 10.3390/ani11061485
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Chen, C, Zhu, W, Ma, C, Guo, Y, Huang, W, and Ruan, C. Image motion feature extraction for recognition of aggressive behaviors among group-alojed pigs. Comput Electron Agric. (2017) 142:380–7. doi: 10.1016/j.compag.2017.09.013
13. Kim, J, Chung, Y, Choi, Y, Sa, J, Kim, H, Chung, Y, et al. Detección basada en la profundidad de cerdos de pie en entornos de ruido en movimiento. Sensores (Basilea). (2017) 17:2757. doi: 10.3390/s17122757
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Agbugba, LC, Oyewunmi, AO, Ogundumade, TP, y Leigh, OO. Investigación de parámetros del moco vaginal: desarrollo de modelos para la estadificación del ciclo estral de la vaca Bunaji. Reprod Domest Anim. (2020) 55:1044–53. DOI: 10.1111/RDA.13694
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Talukder, MRI, Hasan, M, Rosy, TA, Bari, FY y Juyena, NS. Monitoreo de la resistencia eléctrica vaginal, las ondas foliculares y el perfil hormonal durante el ciclo estral en el período de transición en ovejas de Bangladesh. J Vet Res. (2018) 62:571–9. DOI: 10.2478/JVETRES-2018-0080
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Chavan, NB, Kumaresan, A, Chhillar, S, Nayak, S, Prakash, MA, Lathika, S, et al. Patrón de cristalización salival: una posible herramienta no convencional para el momento de la inseminación y el diagnóstico temprano del embarazo en vacas cebú. J Dairy Res. (2023) 90:21–5. doi: 10.1017/s0022029922000887
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Chojnowska, S, Baran, T, Wilińska, I, Sienicka, P, Cabaj-Wiater, I, and Knaś, M. Human saliva as a diagnostic material. Adv Med Sci. (2018) 63:185–91. doi: 10.1016/j.advms.2017.11.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Lindsay, A, y Costello, JT. Realising the potential of urine and saliva as diagnostic tools in sport and exercise medicine. Medicina Deportiva. (2017) 47:11–31. doi: 10.1007/s40279-016-0558-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Michalke, B, Rossbach, B, Göen, T, Schäferhenrich, A, y Scherer, G. Saliva as a matrix for human biomonitoring in occupational and environmental medicine. Int Arch Occup Environ Health. (2015) 88:1–44. DOI: 10.1007/S00420-014-0938-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Green, JA, Kim, JG, Whitworth, KM, Agca, C, y Prather, RS. El uso de microarrays para definir genes funcionalmente relacionados que se expresan diferencialmente en el útero ciclista del cerdo. Soc Reprod Fertil Suppl. (2006) 62:163–76.
21. Rodrigues, AD, Cooke, RF, Cipriano, RS, Silva, LGT, Cerri, RLA, Cruppe, LH, et al. Impactos de la expresión e intensidad del estro durante un protocolo de IA cronometrada sobre variables asociadas con la fertilidad y el éxito del embarazo en vacas de carne influenciadas por Bos indicus. J Anim Sci. (2018) 96:236–49. doi: 10.1093/jas/skx043
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Jones, P, Binns, D, Chang, HY, Fraser, M, Li, W, McAnulla, C, et al. Inter ProScan 5: clasificación de la función de la proteína a escala del genoma. Bioinformática. (2014) 30:1236–40. doi: 10.1093/bioinformática/BTU031
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Fedorka, CE, Scoggin, KE, Boakari, YL, Hoppe, NE, Squires, EL, Ball, BA, et al. El efecto antiinflamatorio de la lactoferrina exógena en la endometritis inducida por la reproducción cuando se administra después de la reproducción en yeguas susceptibles. Teriogenología. (2018) 114:63–9. doi: 10.1016/j.theriogenology.2018.03.017
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Hermawan, A, y Putri, H. Los estudios de bioinformática proporcionan información sobre posibles objetivos y mecanismos de acción de Nobiletin contra las células madre cancerosas. Asian Pac J Cancer Prev. (2020) 21:611–20. doi: 10.31557/apjcp.2020.21.3.611
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Zheng, X, Zheng, Y, Qin, D, Yao, Y, Zhang, X, Zhao, Y, et al. Papel regulador e importancia potencial de GDF-8 en la actividad reproductiva ovárica. Frente Endocrinol (Lausana). (2022) 13:878069. doi: 10.3389/fendo.2022.878069
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Zhang, X, Xu, W, Xu, R, Wang, Z, Zhang, X, Wang, P, et al. Plin5 Regula bidireccionalmente el metabolismo de los lípidos en los tejidos oxidativos. Oxidativo Med Cell Longev. (2022) 2022:4594956. doi: 10.1155/2022/4594956
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Karahan, SC, Guven, S, Mentese, A, Bacak, A, Kopuz, M, and Ozeren, M. Serum anti-carbonic anhydrase I and II antibodies and idiopathic recurrent pregnancy loss. Reprod Biomed Online. (2009) 19:859–63. doi: 10.1016/j.rbmo.2009.09.030
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Göktuğ Kadıoğlu, B, Nalçakan, A, y Dilek, E. Relationship between zinc content and carbic anhydrase activity in blood of anemic pregnant women in Turkey. J Obstet Gynaecol Res. (2020) 46:2612–7. doi: 10.1111/jog.14506
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Santacroce, L, Palmirotta, R, Bottalico, L, Charitos, IA, Colella, M, Topi, S, et al. La diafonía entre la microbiota residente y las células inmunes regula la salud del tracto genital femenino. Vida (Basilea). (2023) 13:1531. doi: 10.3390/life13071531
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Winuthayanon, W, Hewitt, SC, Orvis, GD, Behringer, RR y Korach, KS. El receptor epitelial uterino de estrógeno α es prescindible para la proliferación, pero esencial para respuestas biológicas y bioquímicas completas. Proc Natl Acad Sci U S A. (2010) 107:19272–7. doi: 10.1073/PNAS.1013226107
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Dua, D, Nagoorvali, D, Chauhan, MS, Palta, P, Mathur, P, y Singh, MK. Calcium ionophore enhanced developmental competence and apoptotic dynamics of goat parthenogenetic embryos produced in vitro. In Vitro Cell Dev Biol Anim. (2019) 55:159–68. DOI: 10.1007/S11626-019-00322-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Li, Z, Guo, Y, Ndandala, CB, Chen, H, Huang, C, Zhao, G, et al. Análisis de perfiles de expresión de circ RNA y miRNA en maduración ovárica inducida por IGF3 en excrementos manchados (Scatophagus argus). Frente Endocrinol (Lausana). (2022) 13:998207. doi: 10.3389/fendo.2022.998207
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Socha, BM, Socha, P, Szóstek-Mioduchowska, AZ, Nowak, T, y Skarżyński, DJ. Expresión del receptor activado por proliferador de peroxisomas en el endometrio canino con complejo hiperplasia endometrial-piometra quística. Reprod Domest Anim. (2022) 57:771–83. DOI: 10.1111/RDA.14121
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Xiao, L, Wang, Z, Lu, N, He, Y, Qiao, L, Sheng, X, et al. GPER media la vía IL6/JAK2/STAT3 implicada en la expresión de VEGF en GCs de ovario porcino. J Mol Endocrinol. (2021) 68:23–33. DOI: 10.1530/JME-21-0125
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Zhang, G, Michener, CM, and Yang, B. Low-grade ovarian stromal tumors with genetic alterations of the Wnt/β-catenin pathway that is crucial in ovarian follicle development and regulation. Cánceres (Basilea). (2022) 14:5622. doi: 10.3390/cánceres14225622
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Budani, MC, y Tiboni, GM. Novel insights on the role of nitric oxide in the ovary: a review of the literature. Int J Medio Ambiente Salud Pública. (2021) 18:18. DOI: 10.3390/IJERPH18030980
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Gonzalez-Velandia, KY, Hernandez-Clavijo, A, Menini, A, Dibattista, M, and Pifferi, S. Expression pattern of Stomatin-domain proteins in the peripheral olfactory system. Sci Rep. (2022) 12:11447. DOI: 10.1038/S41598-022-15572-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Zhang, X, Li, BY, Fu, LJ, Adu-Gyamfi, EA, Xu, BR, Liu, TH, et al. La proteína similar a la estomatina 2 (SLP2) regula la proliferación e invasión de las células trofoblásticas mediante la modulación de las funciones mitocondriales. Placenta. (2020) 100:13–23. doi: 10.1016/j.placenta.2020.07.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Feng, Q, Hu, ZY, Liu, XQ, Zhang, X, Lan, X, Geng, YQ, et al. La proteína 2 similar a la estomatina está involucrada en la proliferación y diferenciación de las células del estroma endometrial durante la decidualización en ratones y humanos. Reprod Biomed Online. (2017) 34:191–202. doi: 10.1016/j.rbmo.2016.11.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Bai, X, Lee, JY, Kim, TI, Dai, F, Lee, TJ y Hong, SJ. Clonación molecular y caracterización de la proteína unida al receptor del factor de crecimiento en Clonorchis sinensis. PLoS One. (2014) 9:e85577. doi: 10.1371/journal.pone.0085577
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Yang, F, Wang, M, Zhang, B, Xiang, W, Zhang, K, Chu, M, et al. Identificación de nuevas redes asociadas a progestágenos en la ovulación de mamíferos mediante bioinformática. BMC Syst Biol. (2018) 12:36. DOI: 10.1186/S12918-018-0577-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Arianmanesh, M, McIntosh, RH, Lea, RG, Fowler, PA, y Al-Gubory, KH. Las proteínas del cuerpo lúteo ovino, con funciones que incluyen el estrés oxidativo y el metabolismo de los lípidos, muestran alteraciones complejas durante la implantación. J Endocrinol. (2011) 210:47–58. doi: 10.1530/joe-10-0336
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Reznik, SE, y Fricker, LD. Carboxypeptidases from a to z: implications in embryonic development and Wnt binding. Cell Mol Life Sci. (2001) 58:1790–804. DOI: 10.1007/PL00000819
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Sisco, B, Hagemann, LJ, Shelling, AN, y Pfeffer, PL. Isolation of genes differentially expressed in dominant and subordinate bovine follicles. Endocrinología. (2003) 144:3904–13. doi: 10.1210/es.2003-0485
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Cawley, NX, Wetsel, WC, Murthy, SR, Park, JJ, Pacak, K, y Loh, YP. Nuevas funciones de la carboxipeptidasa E en la función endocrina y neural y el cáncer. Endocr Rev. (2012) 33:216–53. DOI: 10.1210/ER.2011-1039
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Singh, U, Yu, Y, Kalinina, E, Konno, T, Sun, T, Ohta, H, et al. Carboxipeptidasa E en la placenta del ratón. Diferenciación. (2006) 74:648–60. doi: 10.1111/j.1432-0436.2006.00093.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Rodriguez-Martinez, H, Ekstedt, E, and Ridderstråle, Y. Histochemical localization of carbic anhydrase in the female genitalia of pigs during the oestrous cycle. Acta Anat (Basilea). (1991) 140:41–7. DOI: 10.1159/000147036
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. He, Q, Chen, H, Wong, CH, Tsang, LL, y Chan, HC. Mecanismo regulador subyacente a los cambios cíclicos en la secreción de bicarbonato uterino de ratón: papel del estrógeno. Reproducción. (2010) 140:903–10. DOI: 10.1530/REP-10-0178
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Gholami, K, Muniandy, S, and Salleh, N. Estudio funcional in vivo sobre la participación de CFTR, SLC26A6, NHE-1 y CA isoenzimas II y XII en el pH del líquido uterino, el volumen y la regulación electrolítica en ratas bajo diferente influencia de esteroides sexuales. Int J Med Sci. (2013) 10:1121–34. DOI: 10.7150/IJMS.5918
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Paiva, P, Lockhart, MG, Girling, JE, Olshansky, M, Woodrow, N, Marino, JL, et al. Identificación de genes expresados diferencialmente en la descomposición y reparación menstrual. Mol Hum Reprod. (2016) 22:898–912. doi: 10.1093/molehr/gaw060
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: cerda, detección de celo, saliva, proteómica cuantitativa, proteínas expresadas diferencialmente, eficiencia reproductiva
Cita: Xin H, Li B, Meng F, Hu B, Wang S, Wang Y y Li J (2023) El análisis proteómico cuantitativo y la verificación identifican la dinámica global del perfil de proteínas en cerdos durante el ciclo estral. Frente. Vet. Sci. 10:1247561. doi: 10.3389/fvets.2023.1247561
Recibido: 26 de junio de 2023; Aprobado: 11 de septiembre de 2023;
Publicado: 28 septiembre 2023.
Editado por:
Regiane R. Santos, Schothorst Feed Research, Países Bajos
Revisado por:
Danuza Leão, Instituto de Desenvolvimento Sustentável Mamirauá, Brasil
Dagan Mao, Universidad Agrícola de Nanjing, China
Derechos de autor © 2023 Xin, Li, Meng, Hu, Wang, Wang y Li. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Jianhao Li, 3562047763@qq.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía