Diseño racional asistido por inmunoinformática de una vacuna multiepítopo dirigida al virus de la peritonitis infecciosa felina
Diseño racional asistido por inmunoinformática de una vacuna multiepítopo dirigida al virus de la peritonitis infecciosa felina
 Mohit Chawla1
Mohit Chawla1  Andrés Felipe Cuspoca2,3
Andrés Felipe Cuspoca2,3  Nahid Akthar4
Nahid Akthar4  Jorge Samuel León Magdaleno1
Jorge Samuel León Magdaleno1  Siriluk Rattanabunyong5
Siriluk Rattanabunyong5  Chonticha Suwattanasophon5
Chonticha Suwattanasophon5  Nathjanan Jongkon6
Nathjanan Jongkon6  Kiattawee Choowongkomon5*
Kiattawee Choowongkomon5*  Abdul Rajjak Shaikh4*
Abdul Rajjak Shaikh4*  Tabarak Malik7*
Tabarak Malik7*  Luigi Cavallo1*
Luigi Cavallo1*- 1División de Ciencias Físicas e Ingeniería, Centro de Catálisis Kaust, Universidad de Ciencia y Tecnología Rey Abdullah (KAUST), Thuwal, Arabia Saudita
- 2Grupo de Investigación en Epidemiología Clínica de Colombia (GRECO), Universidad Pedagógica y Tecnológica de Colombia, Tunja, Colombia
- 3Centro de Atención e Investigación Médica–CAIMED, Chía, Colombia
- 4Departamento de Investigación e Innovación, STEMskills Research and Education Lab Private Limited, Faridabad, Haryana, India
- 5Departamento de Bioquímica, Facultad de Ciencias, Universidad de Kasetsart, Bangkok, Tailandia
- 6Departamento de Ciencias Sociales y Aplicadas, Facultad de Tecnología Industrial, Universidad Tecnológica Rey Mongkut del Norte de Bangkok, Bangkok (Tailandia)
- 7Departamento de Ciencias Biomédicas, Instituto de Salud, Universidad de Jimma, Jimma, Etiopía
La peritonitis infecciosa felina (PIF) es una dolencia grave y frecuentemente letal provocada por el coronavirus felino (FCoV) en especies felinas silvestres y domésticas. La proteína de la espícula (S) del FCoV asume una función crítica en la entrada e infección viral, lo que presenta una vía prometedora para el desarrollo de una vacuna. En esta investigación, se empleó un enfoque inmunoinformático para determinar los epítopos inmunogénicos dentro de la proteína S de la FIP y formular una vacuna candidata innovadora. Al someter la secuencia de aminoácidos de la proteína S FIP a un escrutinio computacional, se predijeron epítopos de células T de unión al MHC-I, que posteriormente se evaluaron para determinar su antigenicidad, toxicidad y alergenicidad a través de herramientas in silico. Nuestros análisis permitieron identificar 11 epítopos potenciales capaces de provocar una respuesta inmune robusta contra el FIPV. Además, el análisis de acoplamiento molecular demostró la capacidad de estos epítopos para unirse a moléculas de MHC felino de clase I. A través de la utilización de enlazadores adecuados, estos epítopos, junto con los adyuvantes, se integraron para diseñar una vacuna candidata de múltiples epítopos. Además, se estableció la estabilidad de la interacción entre la vacuna candidata y el receptor felino tipo Toll 4 (TLR4) mediante análisis de acoplamiento molecular y simulación de dinámica molecular. Esto sugiere buenas perspectivas para futuras validaciones experimentales para determinar la eficacia de nuestra vacuna candidata en la inducción de una respuesta inmunitaria protectora contra la PIF .
1 Introducción
El coronavirus felino (FCoV) es el agente subyacente responsable de la peritonitis infecciosa felina (PIF), una dolencia generalizada que afecta tanto a los felinos domésticos como a los salvajes. Esta enfermedad es particularmente prevalente entre los gatos que viven en entornos densamente poblados y muestra un alcance global (1). La prevalencia de FCoV oscila entre el 70 y el 86%, observándose una mayor susceptibilidad en gatitos menores de 2 años (2-4). Clasificados dentro de la familia Coronaviridae y del género α coronavirus, los FCoV son virus de ARN monocatenario con envoltura, de sentido positivo y que se distinguen por sus proteínas de pico distintivamente similares a un garrote (5). Existen dos patotipos discernibles de FCoV: el coronavirus entérico felino (FECV) y el virus de la peritonitis infecciosa felina (FIPV). Si bien el FECV es benigno, marcadamente contagioso y, a menudo, se manifiesta de forma asintomática, hay casos en los que conduce a diarrea leve o incluso enteritis grave. Su modo de transmisión gira principalmente en torno a la vía fecal-oral (6, 7). Por el contrario, la FIPV puede ser letal, no se transmite por vía fecal-oral y surge de mutaciones (sustituciones de aminoácidos en proteínas de pico) en cepas virulentas de FECV (6, 8). Las mutaciones provocan un cambio de tropismo celular de la FIPV del epitelio gastrointestinal a monocitos/macrófagos, lo que conduce a la infección sistémica del virus (5). Entre los gatos infectados con FCoV en entornos con varios gatos, como refugios para gatos, centros de rescate o criaderos de gatos, una pequeña población (7-14%) está afectada por FIPV (5). Los gatos que padecen PIF muestran signos clínicos como pérdida de peso, ictericia, anemia, letargo, anorexia, fiebre, palidez de las mucosas, agrandamiento de los ganglios linfáticos, distensión abdominal, disnea y algunos trastornos neurológicos y oculares (5). Además, el FIPV causa piogranulomas, microcitosis, derrame seroso en cavidades corporales, flebitis, linfopenia y serositis en los huéspedes infectados (6, 8). En particular, la PIF es responsable del 0,3-1,4% de las muertes felinas en las clínicas veterinarias (5).
El genoma de FCoV abarca un repertorio de elementos genéticos, que incluyen 16 proteínas no estructurales (NSP), cinco proteínas accesorias (3a, 3b, 3c, 7a y 7b) y cuatro proteínas estructurales: espiga, envoltura, membrana y proteína de la nucleocápside (8-10). Las NSP son necesarias para la síntesis de ARN viral, mientras que las proteínas de la envoltura y la membrana ayudan en el ensamblaje viral, la maduración y la interacción de la célula huésped (10). El papel de las proteínas accesorias FCoV no se ha determinado bien, pero la proteína accesoria 3c se ha asociado con la replicación intestinal tanto de FECV como de FIPV (10, 11). La proteína de la espícula, una proteína transmembrana de tipo I, en el coronavirus es vital para la especificidad del tipo de célula huésped (tropismo de macrófagos), la unión viral y la fusión de las membranas celulares y virales del huésped (8, 12). Además, la proteína de la espícula también es importante para la inducción de la inmunidad mediada por células y la respuesta de anticuerpos en los félidos infectados por FCoV (10). Además, la proteína de la espícula de FCoV es un determinante significativo de la virulencia, la patogénesis y el cambio de un FECV virulento a un FIPV virulento (13). Estudios anteriores han utilizado un péptido de repetición heptada 2 (57 aminoácidos) de la proteína de pico FECV para desarrollar una vacuna oral recombinante candidata (13).
En la actualidad, faltan vacunas eficaces o terapias clínicamente aprobadas para el tratamiento del coronavirus felino (FCoV) (14). La PIF puede ser mortal para los felinos, por lo que es importante desarrollar vacunas candidatas que puedan generar inmunidad protectora contra el FcoV en los gatos.
Dado el papel fundamental de la proteína de la espícula FcoV en la inducción inmunitaria, tanto en términos de respuestas humorales como mediadas por células, así como su implicación en la patogénesis y virulencia de la FcoV, este estudio se centra en la predicción de epítopos dentro de la proteína de la espícula para el desarrollo de una vacuna candidata contra la FIPV utilizando inmunoinformática. El potencial antigénico, los atributos alergénicos y las propiedades tóxicas de estos epítopos se sometieron a una evaluación exhaustiva empleando diversas herramientas computacionales. Además, el escrutinio se extendió a las simulaciones de acoplamiento molecular y dinámica molecular, lo que facilitó el análisis de las interacciones entre la vacuna candidata ideada y las células inmunitarias específicas de los felinos.
2 Materiales y métodos
2.1 Recuperación y análisis de la glicoproteína de superficie de la espícula (proteína S) para FIPV
La secuencia de aminoácidos de la proteína de la espícula (S) del virus de la peritonitis infecciosa felina (FIPV), que consta de 1.452 aminoácidos, se obtuvo de UniProt (número de acceso: P10033). Posteriormente, se evaluó la antigenicidad de la proteína S de la FIPV utilizando Vaxijen 2.0, un servidor en línea (15).
2.2 Creación de un contendiente de vacunas: predicción de epítopos dentro de la proteína S a través de NetMHCpan 4.1
Los epítopos citotóxicos (Tc) de células T derivados de la proteína de la espícula (S) del virus de la peritonitis infecciosa felina (FIPV) se predijeron utilizando el servidor web NetMHCpan 4.1 (16). La secuencia de la proteína S FIPV se empleó como entrada en formato FASTA dentro del servidor web NetMHCpan 4.1, y los epítopos con una longitud de 9 aminoácidos (9-mers) se predijeron utilizando parámetros predeterminados. Como la proteína S de FIPV comparte el 100% de identidad de secuencia con la proteína de pico del coronavirus canino, se seleccionaron los alelos DLA (DLA-8803401, DLA-8850101, DLA-8850801) para la predicción de epítopos. Los epítopos de Tc que exhiben una fuerte afinidad de unión se seleccionaron posteriormente para análisis posteriores.
2.3 Elucidación de epítopos a partir de la proteína S para FIPV y acoplamiento molecular de epítopos identificados con MHC-I felino
La antigenicidad, el potencial alergénico y la toxicidad del epítopo se predijeron utilizando los servidores web Vaxijen 2.0, AllergenFP y ToxinPred, respectivamente (15, 17, 18). Los parámetros predeterminados se emplearon mientras se utilizaban estos servidores web, y las secuencias de epítopos se ingresaron en formato de código de una sola letra. Para Vaxijen 2.0, el organismo objetivo se estableció como el virus. Se eligieron epítopos que se predijo que poseían propiedades antigénicas, no alergénicas y no tóxicas para su inclusión en el diseño final de la vacuna.
Los péptidos seleccionados se modelaron utilizando el programa AmberTools (19). Posteriormente, se realizó un cribado molecular de estos péptidos utilizando el software AutoDock Vina (20). Durante el proceso de selección, se permitió que los péptidos exhibieran flexibilidad, mientras que la proteína permaneció en una conformación fija. Para el estudio se eligió la cadena A del PDB ID: 5XMF (21), que representa la estructura cristalina del complejo mayor de histocompatibilidad (MHC) felino clase I. El sitio de unión se definió utilizando la proteína gag cocristalizada. La cuadrícula utilizada para los cálculos de acoplamiento se colocó en las coordenadas (32, 22, 21,5) con un tamaño de 30 Å en cada dimensión (X, Y y Z). Se consideró que el péptido que demostraba la energía de enlace más negativa poseía la mayor afinidad de unión.
2.4 Diseño de la construcción final de la vacuna
En la formulación de la vacuna, incorporamos la región no tóxica de la toxina del cólera (CtxB), que exhibe una mayor afinidad por el gangliósido «prototipo» (GM1) cuando se coloca en el N-terminal de la vacuna (22). GM1 se manifiesta en la superficie celular de las células presentadoras de antígenos (APC) especializadas, como las células dendríticas y las células B. Esta presencia desencadena un aumento en la expresión del complejo mayor de histocompatibilidad clase II (MHC-II) y favorece la diferenciación en inmunoglobulinas (Ig) (23). Esto da como resultado una mejor absorción de antígenos, lo que aumenta la disponibilidad de antígenos y facilita una mejor interacción con las células TCD4. Para mejorar aún más esta estimulación inmunitaria, incluimos un epítopo combinado de tétanos y toxoide diftérico (TpD), que se ha identificado como un adyuvante universal para las células TCD4, lo que podría superar la eficacia de «PADRE», un péptido conocido por unirse promiscuamente a varias moléculas de antígeno leucocitario humano-DR (HLA-DR) (24). Los estudios in vitro han demostrado la capacidad de la TpD para inducir la producción de anticuerpos neutralizantes (25) y conferir protección a las membranas mucosas (26). Estos resultados persisten en diferentes especies de mamíferos y están marcados por la creación de células de memoria central CD4 T duraderas, la síntesis de anticuerpos neutralizantes y la liberación de citocinas de interferón-gamma (IFN-γ) y TNF-α. Este patrón significa una respuesta inmune dominante de células T auxiliares (Th1) (27). Hacia el extremo C-terminal de la vacuna, incorporamos la subunidad final de la fimbria tipo 1 de Escherichia coli (FimH), que se ha encontrado que interactúa con el receptor tipo Toll 4 (TLR4) de manera dependiente. Esta interacción promueve la maduración, activación y proliferación de células dendríticas locales y células periféricas y exhibe una regulación más favorable y segura de las moléculas MHC de clase I y clase II en comparación con el lipopolisacárido (LPS). Al igual que la TpD, la FimH también estimula la producción de IFN-γ y TNF-α (28, 29). En particular, el uso de FimH ha sido reconocido por su eficacia en la inmunidad de las mucosas (28).+
Para provocar una respuesta celular y humoral dirigida al tiempo que se minimiza la interacción entre la proteína de entrada y el huésped, utilizamos un fragmento específico de la proteína Spike conocido por su interacción con las células huésped. Este fragmento se empleó para inducir una respuesta adyuvante de células que involucraba tanto a las células T como a las células B. Estudios previos han demostrado la utilidad de utilizar fragmentos de la proteína diana que se unen al receptor del huésped en el diseño de vacunas, lo que resulta en la generación de anticuerpos neutralizantes y una respuesta celular robusta (30). Se incorporaron enlazadores apropiados, como «GGGGS» y «EAAAK», para formar configuraciones de proteínas rígidas o flexibles basadas en la actividad biológica deseada. El aumento de la flexibilidad permite una mayor variación en la distancia entre los extremos N y C de la proteína de fusión, lo que afecta la presentación antigénica al separar eficazmente los dominios de interés incluidos (31, 32). La inclusión de lisina (K) en las construcciones de la vacuna mejora la solubilidad al evitar los epítopos de las células B y ofrece una mejor respuesta inmunitaria, al tiempo que proporciona un sitio de escisión favorable para las proteasas dentro de los lisosomas, un paso esencial en la presentación de las células T CD4 (33, 34).
2.5 Modelización molecular y estudio de acoplamiento
La predicción de la estructura tridimensional (3D) de la vacuna multiepítopo FIPV (FIPV-MEV) se llevó a cabo utilizando AlphaFold v2 (35). Sin embargo, la estructura del receptor tipo Toll 4 (TLR4) de Felis catus (UniProt ID: P58727) se obtuvo directamente de la base de datos AlphaFold (36). En el caso de la estructura TLR4, solo se conservó el dominio extracelular que abarca los aminoácidos 24-632, mientras que se excluyeron otras regiones. Las puntuaciones de la prueba de diferencia de distancia local predicha (pLDDT) fueron accesibles a través de Alphafoldv2.0, mientras que los gráficos de Ramachandran y las puntuaciones Z se produjeron (37) empleando el servidor web ProCheck y ProSA para evaluar la calidad estructural (38). Para el acoplamiento de FIPV-MEV con TLR4, se activó el servidor HADDOCK (39), adhiriéndose a la configuración predeterminada. Es importante señalar que la secuencia de FimH, integrada en la estructura FIPV-MEV (que abarca los residuos 586-610) y reconocida por su capacidad para activar TLR4 (28, 40), se marcó claramente como «residuos activos» dentro de la arquitectura FIPV-MEV. Por el contrario, para TLR4, el dominio central que abarca los segmentos LRR8 y LRR9 humanos (residuos 225-245) se especificó como «Residuos activos» durante la implementación del programa HADDOCK (41).
2.6 Estudio de simulaciones de dinámica molecular
Las simulaciones de Dinámica Molecular (MD) se realizaron utilizando el software de simulación GROMACS 2022 (42). La caja de simulación cúbica contenía la vacuna multiepítopo complejada con TLR-4, inmersa en un entorno de solvatación que consistía en una combinación de modelos de agua atomística TIP3P y WatFour de grano grueso (WT4) (43). Se incluyeron iones de grano grueso NaW y ClW. Los parámetros proteicos, el modelo de agua TIP3P y el campo de fuerza SIRAH (44) se utilizaron para el modelo de agua de grano grueso WT4 y los iones, mientras que para las proteínas se aplicó el campo de fuerza CHARMM-36 (45). Para garantizar la neutralidad general y replicar las condiciones fisiológicas, se añadieron iones Na+ y Cl-, lo que dio como resultado una fuerza iónica aparente de 0,15 M. La caja de simulación estaba compuesta por 1.048 NaW, 1.068 ClW, 23.990 moléculas de agua TIP3P y 51.000 moléculas de agua WT4, con un total de 142.215 átomos. Se adoptó un enfoque de minimización de dos pasos: primero, una minimización del descenso más pronunciado con 500.000 pasos, seguido de una minimización del gradiente conjugado de 50.000 pasos. El equilibrio del sistema se logró con 100 ps de equilibrio NVT y 100 ps de equilibrio NPT, ambos realizados sin restricciones. Las simulaciones de producción posteriores abarcaron 50 ns, empleando el conjunto NPT. La temperatura se mantuvo a 310 K (temperatura fisiológica) utilizando un enfoque de reescalado de velocidad con un tiempo de acoplamiento de 0,1 ps. La presión se controló a 1 atm durante las simulaciones de NPT utilizando el barostato Parrinello-Rahman (46) con un tiempo de acoplamiento de 2 ps. La integración de las ecuaciones de movimiento se llevó a cabo utilizando el algoritmo leapfrog con un paso de tiempo de 2.0 fs. Las interacciones electrostáticas se calcularon utilizando el método de suma de Ewald de malla de partículas (PME) (47), con los puntos de corte de Coulomb y van der Waals establecidos en 1,0 nm. Se aplicaron condiciones de contorno periódicas a lo largo de los ejes x, y y z para emular el comportamiento masivo. Las longitudes de enlace que involucran hidrógeno se restringieron utilizando el algoritmo LINCS (48). Las coordenadas de simulación se registraron en archivos de trayectoria cada 20 ps. Se emplearon herramientas GROMACS para el procesamiento de trayectorias y diversos análisis. La visualización y los gráficos moleculares se generaron con el software PyMOL (49), mientras que las ilustraciones complejas se prepararon con VMD (50). Para generar el mapa de interacción proteína-proteína se utilizó ProLIF (51). Las parcelas se generaron utilizando Matplotlib (52).
3 Resultados
3.1 Recuperación y análisis de la secuencia de la proteína S de FIPV
La proteína S de la FIPV fue identificada por el UniProt ID P10033 y consta de 1.452 aminoácidos y exhibe una puntuación de anotación de 4/5. Está asociado a la cepa de coronavirus felino FIPV WSU-79/1146. La antigenicidad potencial de la proteína FIPV Spike se evaluó utilizando el servidor web Vaxijen, lo que dio como resultado una puntuación Vaxijen de 0,5234. Esta puntuación corrobora las características antigénicas de la proteína.
3.2 Predicción de epítopos a partir de la proteína S para el diseño de vacunas candidatas y su acoplamiento molecular con la proteína FLA-E*01801
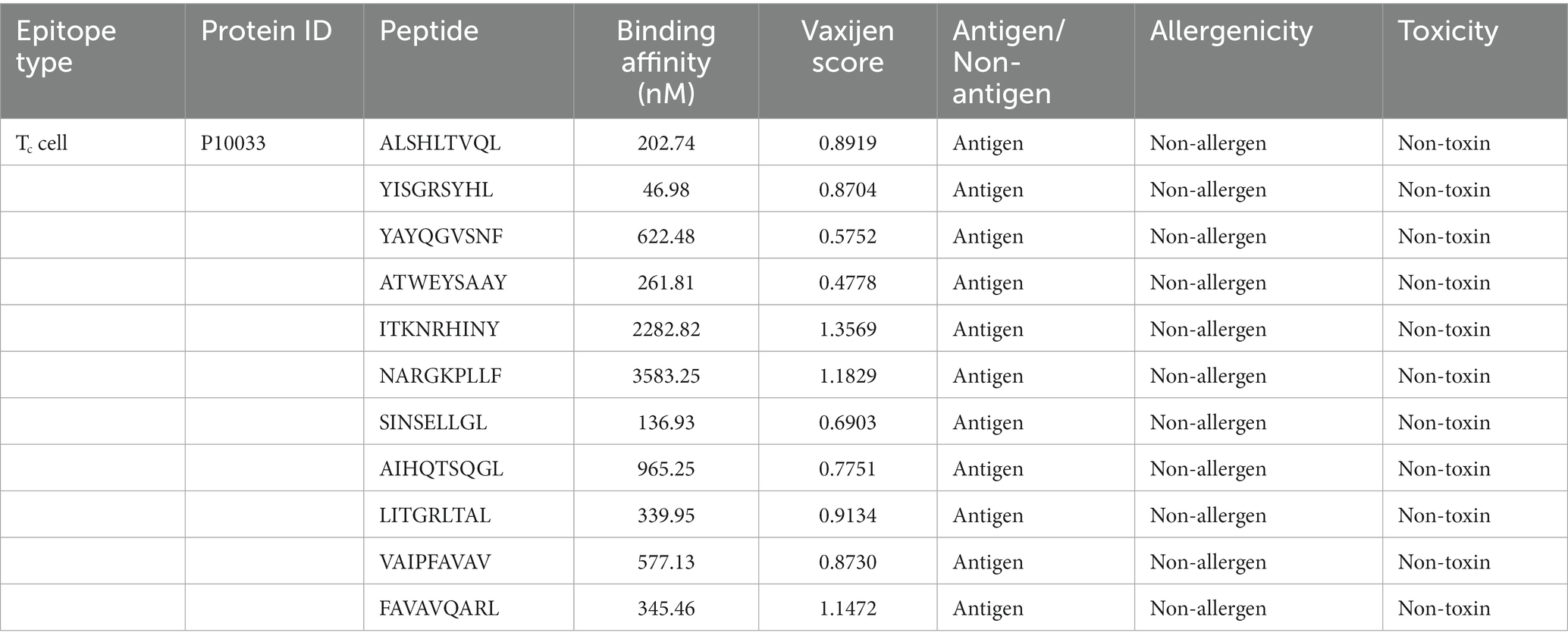
Se pronosticó un número completo de epítopos de 4.332 Tc a partir de la proteína FIPV-S empleando el servidor web MetMHCpan 4.1. Entre estos epítopos predichos, 89 fueron identificados como aglutinantes fuertes y seleccionados para su posterior análisis (Tabla Suplementaria S1). Después de una evaluación cuidadosa, se eligieron 11 epítopos de Tc para el diseño de la vacuna en función de su antigenicidad, no alergenicidad y no toxicidad predichas (Tabla 1). Además, se llevó a cabo un análisis de acoplamiento molecular en los 11 péptidos seleccionados con FLA-E*01801-MHC-I, lo que dio como resultado la unión de estos péptidos a los mismos sitios de unión que el péptido gag cocristalizado de referencia en la proteína FLA-E*01801, ver Figura 1. Los valores de afinidad de unión para todos los péptidos examinados fueron comparables al péptido gag de referencia unido a la proteína FLA-E*01801, lo que indica una fuerte afinidad de los péptidos seleccionados por la proteína FLA-E*01801.
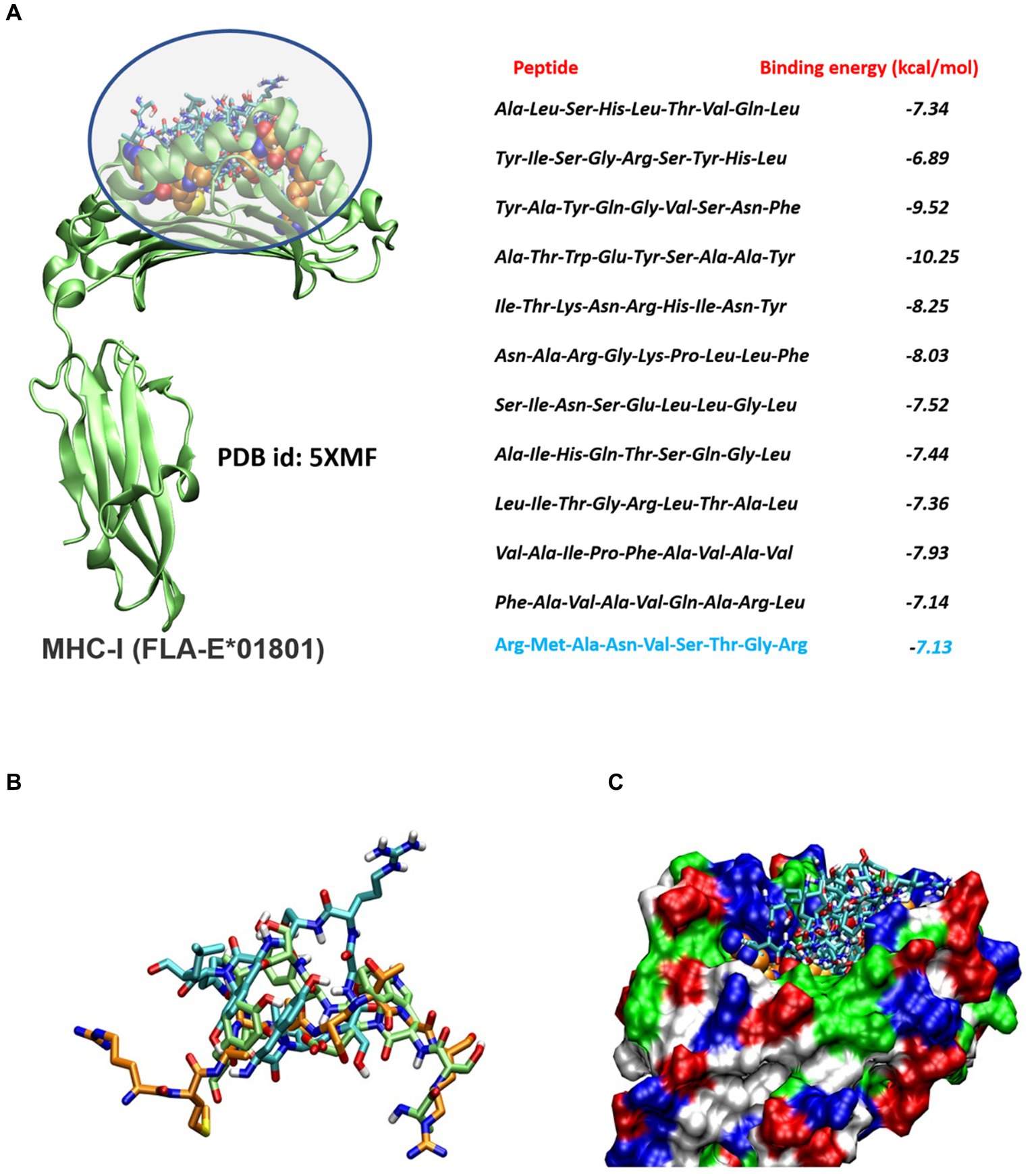 Figura 1. (A) Análisis de acoplamiento molecular de los péptidos cribados y del complejo mayor de histocompatibilidad FLA-E*01801 clase I (MHC-I). Sitio de unión al péptido dentro de la estructura MHC-I de FLA-E*01801, representado resaltando la región. Los péptidos cribados se representan como modelos en barra. El péptido de referencia (péptido gag) cocristalizado con el antígeno FLA se muestra como un modelo esférico. El panel derecho muestra los valores de afinidad de unión correspondientes, con la afinidad de unión del péptido gag de referencia resaltada en color azul. (B) Análisis de acoplamiento molecular de los péptidos cribados y del complejo mayor de histocompatibilidad FLA-E*01801 clase I (MHC-I). Los péptidos cribados compuesto 2 (cian), compuesto 4 (cal) y la proteína gag (naranja) se representan como modelos de barra. (C) La proteína se muestra como una superficie molecular y toda la conformación acoplada se muestra como un modelo de barra.
Figura 1. (A) Análisis de acoplamiento molecular de los péptidos cribados y del complejo mayor de histocompatibilidad FLA-E*01801 clase I (MHC-I). Sitio de unión al péptido dentro de la estructura MHC-I de FLA-E*01801, representado resaltando la región. Los péptidos cribados se representan como modelos en barra. El péptido de referencia (péptido gag) cocristalizado con el antígeno FLA se muestra como un modelo esférico. El panel derecho muestra los valores de afinidad de unión correspondientes, con la afinidad de unión del péptido gag de referencia resaltada en color azul. (B) Análisis de acoplamiento molecular de los péptidos cribados y del complejo mayor de histocompatibilidad FLA-E*01801 clase I (MHC-I). Los péptidos cribados compuesto 2 (cian), compuesto 4 (cal) y la proteína gag (naranja) se representan como modelos de barra. (C) La proteína se muestra como una superficie molecular y toda la conformación acoplada se muestra como un modelo de barra.
3.3 Diseño de la construcción final de la vacuna FIPV
En resumen, se diseñó una construcción de vacuna basada en péptidos de múltiples epítopos dirigida a los epítopos de Tc, un dominio de unión al receptor y los adyuvantes relevantes (como se enumeran a continuación) mediante la unión de 11 epítopos utilizando varios enlazadores. El objetivo era crear una vacuna estable, antigénica y no alergénica específica para la FIPV. La construcción de la vacuna resultante consta de un total de 744 aminoácidos, y su secuencia de aminoácidos se muestra en la Figura 2. Además, la Figura 2 presenta los atributos fisicoquímicos de la construcción final de la vacuna, que abarcan características como el punto isoeléctrico, el recuento de átomos, el índice alifático y otras propiedades pertinentes.
 Figura 2. Ilustración que describe el diseño esquemático de la construcción FIPV-MEV proyectada, acompañada de las secuencias de aminoácidos correspondientes para las construcciones empleadas, junto con una descripción general de los atributos fisicoquímicos de la construcción FIPV-MEV anticipada.
Figura 2. Ilustración que describe el diseño esquemático de la construcción FIPV-MEV proyectada, acompañada de las secuencias de aminoácidos correspondientes para las construcciones empleadas, junto con una descripción general de los atributos fisicoquímicos de la construcción FIPV-MEV anticipada.
3.4 Estimulación del sistema inmunitario mediante componentes de construcción de vacunas
La construcción de la vacuna propuesta demuestra una afinidad específica por el receptor TLR4 a través de su interacción con el extremo fimbrial (FimH) de la fimbria tipo 1 de E. coli. Esta interacción se dirige específicamente a los receptores TLR4 presentes en las células dendríticas derivadas de la médula ósea, donde se expresan moderadamente en los felinos (53). Al unirse, inicia una interacción canónica que desencadena varios procesos celulares, incluyendo la maduración, la transcripción, la autofagia, la endocitosis, la fagocitosis y los estallidos oxidativos (54, 55). La vía de señalización que sigue a la unión de la construcción FIPV-MEV al receptor TLR4 posiblemente implica la dimerización citoplasmática del receptor Toll IL-1 (TIR), véase la Figura 3. Esta dimerización facilita la formación de un nuevo sitio de ensamblaje, lo que permite el reclutamiento de proteínas adaptadoras que inician dos mecanismos de señalización intracelular distintos (56, 57).
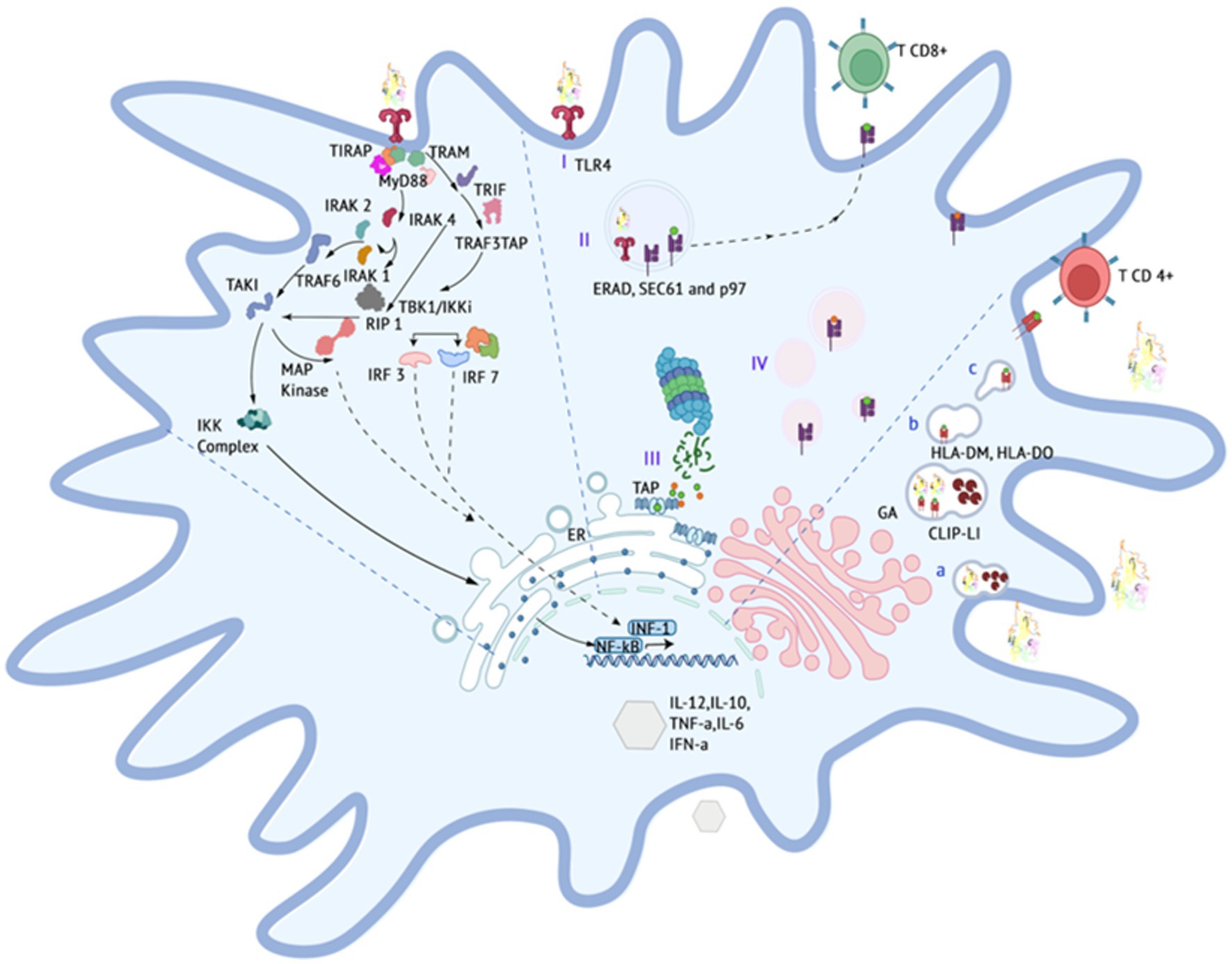 Figura 3. Eventos esperados que ocurren en el sistema inmunitario, en particular la estimulación del sistema inmunitario por los componentes de la construcción de la vacuna.
Figura 3. Eventos esperados que ocurren en el sistema inmunitario, en particular la estimulación del sistema inmunitario por los componentes de la construcción de la vacuna.
El mecanismo inicial implica la interacción entre la proteína adaptadora que contiene el dominio del receptor Toll/Interleucina-1 (TIRAP), que luego recluta la proteína de respuesta primaria de diferenciación mieloide 88 (MyD88). Este reclutamiento desencadena la activación de las proteínas quinasas asociadas al receptor de interleucina-1 (IRAK), iniciando posteriormente la cascada de activación del factor 6 asociado al receptor del factor de necrosis tumoral (TNFR) (TRAF6) (58). La vía progresa con la participación del factor de crecimiento transformante (TAK1) y el complejo inhibidor de la quinasa NF-kappa-B (IKK), culminando en la translocación de las subunidades NFkB al núcleo celular. En consecuencia, esta translocación provoca la regulación transcripcional positiva de citoquinas y quimiocinas proinflamatorias, incluyendo en particular el factor de necrosis tumoral (59).
El segundo mecanismo, distinto de la participación de MyD88, se centra en la molécula adaptadora relacionada con TRIF (TRAM), que instiga la activación del interferón inductor del adaptador (TRIF) que contiene el dominio TIR. Esta activación, a su vez, desencadena la activación de la proteína que interactúa con el receptor (RIP1) y TRAF6, iniciando así la cascada de activación de las vías NFkB y MAPK. Además, TRIF interactúa con TRAF3, lo que conduce a la activación de la quinasa de unión al activador NF-kappa-B asociada a miembros de la familia TRAF (TBK1) / I-kappa-B quinasa i (IKKi). Esta activación, posteriormente, activa los factores reguladores del interferón (IRF3) e (IRF7). Estos factores sufren translocación nuclear y facilitan la transcripción de interferones tipo 1 (IFN tipo 1).
En conjunto, estas vías establecen una respuesta inmunitaria innata proinflamatoria, que conduce a la producción de citoquinas y quimiocinas, a la regulación positiva de las moléculas implicadas en la respuesta inmunitaria adaptativa, a la activación de las células T hacia un fenotipo efector y a la coestimulación de otras células presentadoras de antígenos (55).
TLR-4 es capaz de internalizar la construcción FIPV-MEV, facilitando la presentación cruzada y la estimulación de las células TCD-8. Este proceso comienza con el engullimiento del constructo en el fagosoma (I) y la posterior desnaturalización a través de mecanismos dependientes del pH o transporte al citoplasma a través de maquinaria de degradación asociada al retículo endoplásmico (ERAD), como SEC61 o la AAA hexámera (ATPasa) p97 (60). La escisión proteasómica genera múltiples fragmentos peptídicos (II), que luego se transportan al RE a través de proteínas transportadoras (TAP) y se ensamblan con moléculas MHC de clase I. El complejo ensamblado sufre tráfico desde el aparato de Golgi (III) hasta la membrana celular (61).
El esquema propuesto incluye la posibilidad de presentación del MHC-II, que tiene lugar dentro de los compartimentos lisosomales. En este contexto, la desnaturalización de la construcción de la vacuna dentro del ambiente lisosomal da como resultado la generación de péptidos cortos en condiciones de pH bajo y agentes reductores. Estas condiciones son propicias para la maduración adecuada de las proteasas lisosomales (a). La formación de un complejo inmune estable entre los péptidos cortos y el MHC-II requiere la eliminación de la proteína de cadena invariante (LI) y su fragmento CLIP del marco nativo del MHC-II. Las moléculas homólogas en los felinos, como el antígeno leucocitario humano DM (HLA-DM) y el complejo de histocompatibilidad leucocitaria humana DO (HLA-DO), participan en la estabilización y localización de los péptidos. En última instancia, este complejo migra a la membrana plasmática, donde activa las células T CD4.
En cuanto a la estimulación de las células B, la construcción de la vacuna incorpora un sitio de predicción de unión al huésped para mejorar su interacción con las células B. Esta interacción tiene como objetivo inducir la generación de anticuerpos neutralizantes que puedan evitar la internalización del virus.
3.5 Modelado y acoplamiento de la construcción TLR4 y FIPV-MEV
Dada la falta de disponibilidad de estructuras experimentales de TLR4 para Felis catus, la estructura de TLR4 (Uniprot ID: P58727) se obtuvo de la base de datos AlphaFold. La estructura obtenida mostró valores robustos de pLDDT, notablemente superiores a 90 para la mayoría de los residuos, lo que refleja la naturaleza de alta confianza de la predicción (Figura suplementaria S1). Posteriormente, se proyectó la estructura 3D del constructo FIPV-MEV utilizando Alphafoldv2.0, arrojando valores elevados de pLDDT por encima de 90 para las regiones pertinentes al constructo de la vacuna, incluidas las secciones donde se insertaron el FimH y la unión del receptor a la secuencia del huésped. Por el contrario, las regiones que albergan adyuvantes/enlazadores y secuencias de epítopos (aminoácidos 309-461) exhibieron valores de pLDDT más bajos por debajo de 50, lo que implica un estado estructuralmente desordenado para este segmento (Figura suplementaria S1).
Para validar aún más la calidad estructural, se emplearon gráficos de Ramachandran y puntuaciones Z del servidor web ProSA. Para el receptor TLR4, aproximadamente el 81,6% de los aminoácidos residían dentro de la región central aceptable. Aproximadamente el 18% ocupaba las regiones permitidas y generosamente permitidas, mientras que solo el 0,4% pertenecía a la región no permitida (Figura 4A). En cuanto al constructo FIPV-MEV, el 66,9% de los residuos se ubicaron dentro de las regiones favorecidas, y el 24,5% de los residuos se ubicaron dentro de las regiones permitidas y generosamente permitidas. Sin embargo, el 8,6% de los residuos se localizaron en regiones no permitidas (Figura Suplementaria S2). Dado que una alta proporción de residuos se encuentran dentro de la región no permitida, realizamos el refinamiento del modelo FIPV-MEV mediante la realización de simulaciones de dinámica molecular de 50 ns para la construcción FIPV-MEV y recalculamos el gráfico de Ramachandran. Nuestros resultados mostraron una mejora significativa en la proporción de residuos en las regiones favorecidas y solo un pequeño porcentaje (0,3%) en las regiones no permitidas, ver Figura 4A, lo que indica un modelo refinado. La superposición estructural del constructo FIPV-MEV antes y después de la ejecución de la simulación se ilustra en la Figura Suplementaria S2. El modelo refinado FIPV-MEV se utilizó para estudios de acoplamiento posteriores.
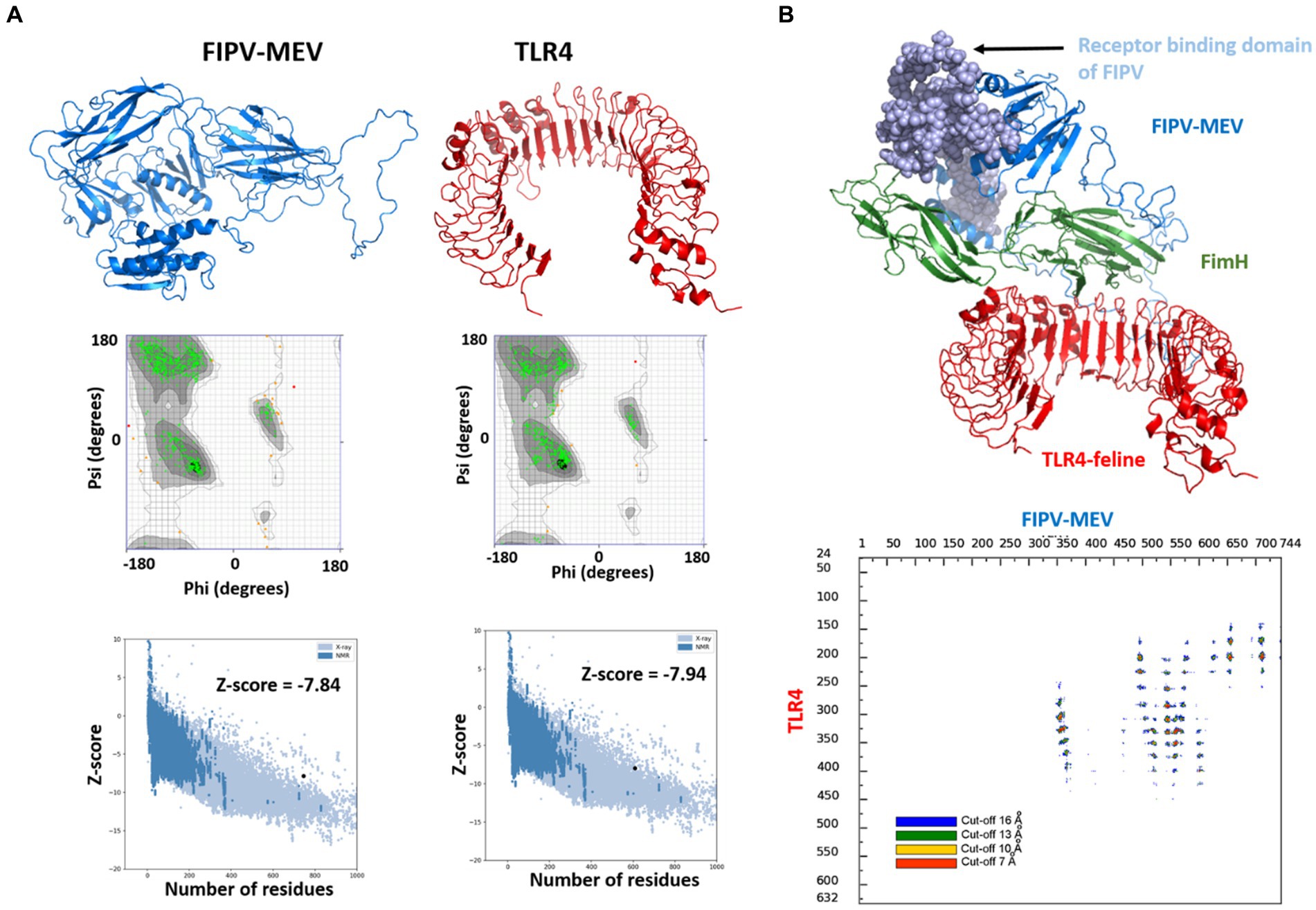 Figura 4. Estructuras 3D modeladas de (A) construcción FIPV-MEV y TLR4 y sus respectivos gráficos de Ramachandran y puntuaciones Z calculadas por el servidor web Pro-SA. (B) Acoplamiento molecular entre FIPV-MEV y TLR4 con complejo acoplado (construcción de vacuna coloreada con diferentes componentes y receptor TLR4 en rojo). El mapa de interacción de la construcción TLR4 y FIPV-MEV con residuos que interactúan con una distancia umbral dada se presenta en diferentes colores.
Figura 4. Estructuras 3D modeladas de (A) construcción FIPV-MEV y TLR4 y sus respectivos gráficos de Ramachandran y puntuaciones Z calculadas por el servidor web Pro-SA. (B) Acoplamiento molecular entre FIPV-MEV y TLR4 con complejo acoplado (construcción de vacuna coloreada con diferentes componentes y receptor TLR4 en rojo). El mapa de interacción de la construcción TLR4 y FIPV-MEV con residuos que interactúan con una distancia umbral dada se presenta en diferentes colores.
Para profundizar en el mecanismo de interacción entre la construcción MEV y TLR4, que es fundamental para orquestar las respuestas inmunitarias, realizamos un acoplamiento molecular entre FIPV-MEV y TLR4 utilizando el servidor web HADDOCK 2.4. Se emplearon parámetros predeterminados para este proceso de acoplamiento; se describen detalles adicionales en la sección Métodos. La estructura final del complejo FIPV-MEV y TLR4 se seleccionó del grupo mejor clasificado con la puntuación HADDOCK más baja. Esta estructura compleja, que representa el resultado del acoplamiento molecular, se representa en la Figura 4B. Además, para obtener más información, generamos «mapas de rango de distancia» para el complejo acoplado utilizando la herramienta COCOMAPS (Figura 4B). Las instancias de contacto se determinaron considerando una distancia de corte de 5 Å entre dos átomos. Específicamente, identificamos 79 contactos entre residuos hidrófilos, 39 contactos que involucran residuos hidrófilos e hidrofóbicos, y 14 contactos adicionales entre dos residuos hidrofóbicos.
3.6 Estabilidad del complejo receptor TLR4 de la construcción de la vacuna
La simulación de dinámica molecular ha surgido como un método valioso para investigar la estabilidad y el análisis de sistemas biológicos (62-67). En este estudio, empleamos el software GROMACS para evaluar la estabilidad de una vacuna multiepítopo complejada con TLR-4. Se llevaron a cabo tres simulaciones independientes de dinámica molecular, cada una con una duración de 100 ns, para el FIPV-MEV complejado con TLR4 utilizando diferentes velocidades iniciales, obteniendo resultados muy comparables. En la Figura 5 se presentan los parámetros calculados para las tres réplicas, demostrando resultados consistentes. Para mantener la claridad, presentaremos los resultados únicamente para la réplica inicial.
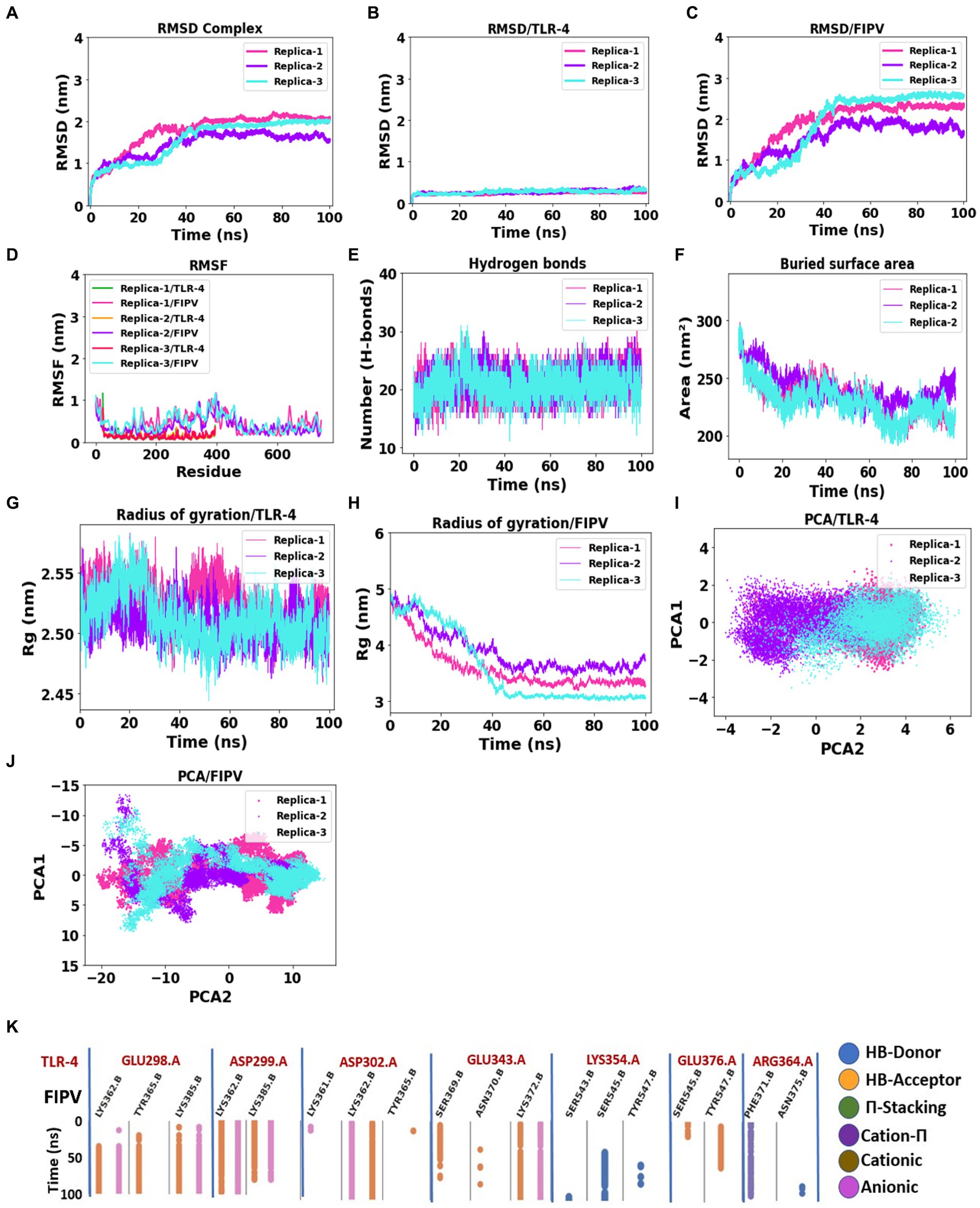 Figura 5. Evaluación de la estabilidad del complejo: construcción FIPV-MEV unida al receptor TLR4. (De la A a la C) Progresión temporal de la RMSD de la red troncal para todo el complejo, FIPV-MEV y TLR4 individualmente, durante el período de simulación de MD. (D) Diagramas que representan las fluctuaciones de la red troncal RMSF. (E) Evolución de los números de enlaces de hidrógeno entre TLR4 y FIPV-MEV a lo largo de la simulación. (F) Tendencias alteradas de la superficie enterrada en las simulaciones de MD para las tres réplicas. (G,H) Fluctuaciones en el radio de giro para TLR4 y FIPV-MEV. (I,J) Resultados del análisis de componentes principales que dilucidan el comportamiento de TLR4 y FIPV-MEV. (K) Análisis de huellas dactilares que proporciona información sobre la estabilidad duradera de las interacciones intermoleculares dentro de la red FIPV-MEV y TLR4 a lo largo de la duración de la simulación.
Figura 5. Evaluación de la estabilidad del complejo: construcción FIPV-MEV unida al receptor TLR4. (De la A a la C) Progresión temporal de la RMSD de la red troncal para todo el complejo, FIPV-MEV y TLR4 individualmente, durante el período de simulación de MD. (D) Diagramas que representan las fluctuaciones de la red troncal RMSF. (E) Evolución de los números de enlaces de hidrógeno entre TLR4 y FIPV-MEV a lo largo de la simulación. (F) Tendencias alteradas de la superficie enterrada en las simulaciones de MD para las tres réplicas. (G,H) Fluctuaciones en el radio de giro para TLR4 y FIPV-MEV. (I,J) Resultados del análisis de componentes principales que dilucidan el comportamiento de TLR4 y FIPV-MEV. (K) Análisis de huellas dactilares que proporciona información sobre la estabilidad duradera de las interacciones intermoleculares dentro de la red FIPV-MEV y TLR4 a lo largo de la duración de la simulación.
Para evaluar la estabilidad y los posibles cambios conformacionales dentro del complejo, se calculó la desviación cuadrática media (RMSD) de la columna vertebral con respecto a su configuración inicial. En particular, el complejo exhibió un RMSD sustancial, con una medición promedio de 1,78 ± 0,42 nm (Figura 5A). En particular, el complejo alcanzó un estado estable después de aproximadamente 40 ns de simulación, como se muestra en la Figura 5A, y mantuvo la estabilidad durante toda la duración de la simulación en las tres réplicas. El receptor TLR-4 mostró una estabilidad notable, con un valor RMSD promedio de 0,24 ± 0,02 nm (Figura 5B). Por el contrario, la construcción de la vacuna multiepítopo, caracterizada por largos bucles que acomodan epítopos y enlazadores, mostró una flexibilidad considerable, lo que dio lugar a una RMSD media de 1,94 ± 0,54 nm (Figura 5C).
La exploración posterior implicó el examen de la fluctuación cuadrática media (RMSF) individualmente tanto para TLR-4 como para la construcción de la vacuna. Cabe destacar que el análisis de la RMSF reveló residuos específicos, localizados principalmente en regiones periféricas, que exhibieron fluctuaciones pronunciadas, registrando una medición promedio de 0,48 ± 0,30 nm (Figura 5D). En marcado contraste, TLR-4 demostró una flexibilidad limitada, evidenciada por sus valores RMSF promedio notablemente más bajos de 0,13 ± 0,08 nm (Figura 5D).
Además, examinamos el número de enlaces de hidrógeno intermoleculares dentro del complejo MEV-TLR4 en el transcurso de 20 ns, revelando un recuento constante durante todo el período de simulación (Figura 5E). Además, el área de superficie enterrada en la interfaz de interacción de la construcción de la vacuna y TLR-4 mantuvo la estabilidad en las tres réplicas durante toda la duración de la simulación (Figura 5F). Estos análisis exhaustivos subrayan colectivamente la solidez y la coherencia de las interacciones de interfaz entre TLR-4 y la construcción de la vacuna.
El análisis de giro demostró que tanto la proteína FIPV como el receptor TLR-4 mantuvieron su compacidad y forma durante la simulación, destacando las interacciones equilibradas y las fuerzas que gobiernan sus estructuras, evitando así cambios conformacionales significativos o el despliegue, ver Figuras 5G, H.
Para investigar los movimientos significativos durante la simulación de MEV y TLR-4, se empleó el análisis de componentes principales (ACP). Las Figuras 5I, J muestran los muestreos conformacionales de las réplicas MEV-TLR-4 en el subespacio esencial, ilustrando los movimientos globales a lo largo de PC1 y PC2 proyectados por el átomo de Cα. Todos los complejos exhibieron estabilidad y ocuparon un espacio de fase confinado en la proyección bidimensional, lo que indica un complejo estable en las tres réplicas. La proyección 2D de PCA reveló pequeños subespacios conformacionales y patrones de movimiento similares, lo que confirma aún más la formación de un complejo estable en los tres análisis de réplicas.
La Figura 5K ilustra las interacciones moleculares dentro de la interfaz de interacción entre MEV y TLR-4, como se observa en los resultados de la simulación de dinámica molecular. Para centrarse en las interacciones relevantes, se aplicó un umbral de corte basado en la ocupación de las interacciones a lo largo de la simulación de 100 ns. El umbral elegido de 0,3 indica que estas interacciones estuvieron presentes durante al menos el 30% de toda la duración de la simulación. En particular, las cadenas laterales de TLR-4 involucradas en estas interacciones son las siguientes: Glu298, que forma enlaces de hidrógeno e interacciones aniónicas con las cadenas laterales de Lys362, Tyr365 y Lys385 de MEV; Asp299, que establece interacciones aniónicas y de enlace de hidrógeno con las cadenas laterales de Lys362 y Lys385. Además, el Asp302 de TLR-4 exhibe principalmente interacciones aniónicas y de hidrógeno con Lys362. Además, el TLR-4 Glu343 forma interacciones como un enlace aceptor de hidrógeno e interacciones aniónicas con Lys372 de MEV. Por el contrario, la cadena lateral de TLR-4 Lys354 forma una interacción donante de enlace H con la cadena lateral Ser545 de MEV, y Arg364 de TLR-4 forma una interacción de catión π con Phe371 de MEV. Este análisis de huellas dactilares destaca la importancia de estas interacciones con alta ocupación a lo largo de toda la simulación de dinámica molecular, enfatizando su papel en la promoción de la estabilidad dentro de la interfaz FIPV-MEV y TLR4.
La robustez de la integridad estructural se reafirmó a través de la superposición del complejo completo que involucra la construcción de la vacuna y el receptor TLR-4. Este análisis mostró una alineación favorable entre las estructuras, como se muestra en la Figura 6. Los altos valores de la desviación cuadrática media (RMSD) observados en las estructuras representadas en la Figura 6 pueden atribuirse a la presencia de regiones flexibles no alineadas dentro de la construcción de la vacuna, en particular los bucles largos y flexibles. A pesar de estas discrepancias estructurales, el patrón de interacción entre los residuos involucrados en la interacción entre FIPV-MEV y TLR-4 mostró una consistencia constante. Para profundizar en la interfaz que conecta TLR-4 y la construcción de la vacuna acoplada, se sometieron instantáneas específicas a un análisis de interfaz utilizando la herramienta COCOMAPS (68) (Figura 6). Este análisis generó mapas de contacto, ofreciendo una representación visual de las distancias por pares entre los residuos pertenecientes a la construcción de la vacuna y el TLR-4. Dentro de estos mapas de contacto, los puntos están codificados por colores en rojo, amarillo, verde y azul, denotando distancias inferiores a 7, 10, 13 y 16 Å, respectivamente. La Figura 6 demuestra notablemente la estabilidad inquebrantable de la interfaz en las instantáneas muestreadas, como lo demuestran los contactos entre residuos.
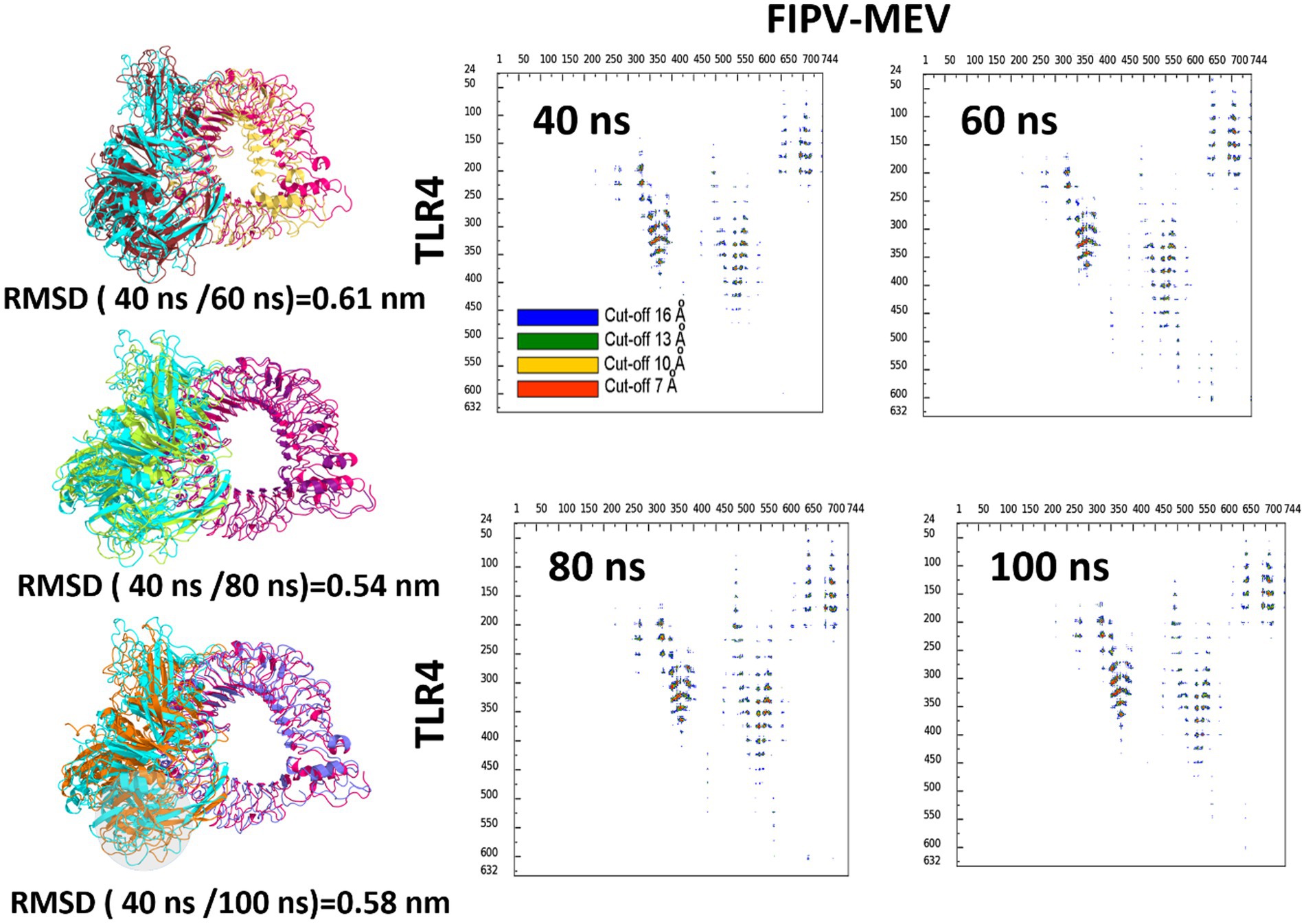 Figura 6. Superposición de instantáneas seleccionadas de la construcción TLR4 y FIPV-MEV y sus respectivos valores RMSD para las primeras simulaciones replicadas. Mapas de contacto que ilustran interacciones: Representación de contactos intermoleculares basada en la distancia para instantáneas idénticas. Los puntos en la unión de dos residuos están codificados por colores rojo, amarillo, verde y azul, lo que indica la proximidad de cualquier par de átomos dentro de 7, 10, 13 y 16 Å, respectivamente.
Figura 6. Superposición de instantáneas seleccionadas de la construcción TLR4 y FIPV-MEV y sus respectivos valores RMSD para las primeras simulaciones replicadas. Mapas de contacto que ilustran interacciones: Representación de contactos intermoleculares basada en la distancia para instantáneas idénticas. Los puntos en la unión de dos residuos están codificados por colores rojo, amarillo, verde y azul, lo que indica la proximidad de cualquier par de átomos dentro de 7, 10, 13 y 16 Å, respectivamente.
4 Discusión
Dependiendo de los biotipos, los FCoV pueden causar infecciones benignas o sistémicas y letales tanto en felinos domésticos como silvestres (1, 6). Existe una falta de una vacuna eficaz y de fármacos clínicamente aprobados para el tratamiento del FCoV (14). Sin embargo, diferentes estudios han identificado posibles fármacos antivirales que podrían ser eficaces para el tratamiento de la PIF (69-72). Por ejemplo, un estudio in vitro demostró que ERDRP-0519, un inhibidor no nucleósido dirigido a la ARN polimerasa, inhibe eficazmente la replicación de FIPV de una manera dependiente de la dosis (69). De manera similar, la difilina, un bloqueador vacuolar de la ATPasa, y su nanoformulación inhibieron la replicación de FIPV (71). El nelfinavir, un inhibidor de la proteasa del VIH-1, en combinación con la aglutinina de Galanthus nivalis, ha demostrado un efecto sinérgico en la inhibición de la replicación del FCoV (73). Otro estudio examinó 90 compuestos e identificó la actividad anti-FIPV en 26 compuestos diferentes (74). Además, los interferones felinos recombinantes, omega-a y omega-b, también han mostrado actividad antiviral contra FCoV (75).
Además de los compuestos antivirales, se han realizado numerosos esfuerzos para desarrollar vacunas contra la FCOV (13, 76, 77). Administración oral de una vacuna recombinante basada en la técnica de visualización de superficie de Bacillus subtilis que expresa el péptido de dominio 2 de repetición heptada FECV protegido contra la infección por FECV y mejoró la patología del tracto digestivo en modelos de ratones (13). Una vacuna FIP viva atenuada fabricada utilizando la deleción de ORF 3abc protegió a los gatos de la provocación letal de FIPV (78). La vacunación con la proteína de nucleocápside de FIPV expresada por baculovirus recombinante no indujo anticuerpos neutralizantes del virus, pero aumentó la tasa de supervivencia de los gatos desafiados con FIPV heteróloga en comparación con el grupo de control (79). Sin embargo, una vacuna de ADN que contenía plásmidos que codificaban la nucleocápside y la membrana de la FIPV no protegió a los gatitos de la PIF (80).
En ausencia de terapias efectivas contra la PIF es imperativo investigar estrategias novedosas, seguras y efectivas, como el desarrollo de vacunas, inmunoterapias y medicamentos antivirales, para tratar y proteger a los gatos de las infecciones por FIV. Una posible vía es la inmunoinformática, que podría ser un método barato y rápido para diseñar nuevas vacunas candidatas para la protección de los felinos frente a la infección mortal por FIPV. Este es el primer estudio que tiene como objetivo desarrollar un candidato vacunal multiepítopo contra FIPV utilizando inmunoinformática. Sin embargo, en un estudio anterior, una vacuna basada en péptidos que consistía en dos epítopos de células T-helper-1 (GQRKELPERWFFYFLGTGPH y EPLRFDGKIPPQFQLEVNRS) derivada de la proteína de la nucleocápside de FIPV en conjugación con el adyuvante felino CpG-oligodesoxinucleótidos evitó que los gatos contrajeran FIPV (81). Un estudio similar también identificó dos epítopos, NNYLTFNKFCLSLSPVGANC (de la proteína de la espícula) y QYGRPQFSWLVYGIKMLIMW (de la proteína de membrana), de FIPV que indujeron la actividad del T-helper 1 en gatos específicos libres de patógenos cuando se administraron junto con el adyuvante felino de oligodesoxinucleótidos CpG (82). Anteriormente, se ha utilizado un enfoque inmunoinformático para el desarrollo de vacunas candidatas contra diversos virus animales, como el circovirus canino, el virus de la gripe aviar, el virus de la dermatosis nodular contagiosa y el virus de la peste porcina africana (66, 83-86).
En el presente estudio, utilizamos métodos computacionales para predecir 11 epítopos antigénicos derivados de la proteína S de la FIPV. Se encontró que estos epítopos no eran tóxicos, no eran alérgicos y eran capaces de interactuar con la molécula MHC-I felina. Para diseñar una vacuna eficaz contra la FIPV, estos epítopos se combinaron con un dominio de unión al receptor de la FIPV y los adyuvantes relevantes (CtxB, TpD y FimH) utilizando diferentes enlazadores. La construcción resultante de la vacuna, que comprende 744 aminoácidos, demuestra una sólida estabilidad, antigenicidad y atributos no alergénicos. Para profundizar nuestra comprensión de la interacción entre el contendiente de la vacuna y el receptor felino tipo Toll 4 (TLR4), realizamos simulaciones de acoplamiento molecular y dinámica molecular, afirmando efectivamente la firmeza de esta interacción. Estas evaluaciones computacionales proporcionan una valiosa corroboración del potencial inmunogénico de la vacuna candidata FIPV. Sin embargo, sigue siendo imperativo llevar a cabo más investigaciones in vivo para validar la seguridad y eficacia de los epítopos identificados y de la vacuna candidata propuesta. Los futuros esfuerzos de investigación podrían abarcar la síntesis de los epítopos inmunogénicos identificados en este documento, seguida de una evaluación de su capacidad para desencadenar respuestas de anticuerpos protectores en sujetos felinos. Además, la vacuna candidata propuesta podría clonarse y expresarse como una proteína recombinante, lo que facilitaría su administración a gatos infectados por FIPV para medir su inmunogenicidad y su potencial para conferir protección contra la infección por FIPV.
A pesar del potencial del uso de la inmunoinformática para el diseño de vacunas multiepítopos FIPV, también existen algunos desafíos. La complejidad, las diversas cepas y los mutantes de FIPV plantean dificultades para predecir con precisión los epítopos. Las herramientas bioinformáticas pueden tener dificultades para predecir con precisión la inmunogenicidad in vivo. La variabilidad en las respuestas inmunitarias individuales, los diversos perfiles de MHC y una comprensión en evolución de las interacciones huésped-virus añaden más complejidad. La precisión de la predicción de los epítopos estimulantes de células T a través de la especificidad de unión del complejo mayor de histocompatibilidad (MHC) varía, lo que requiere una validación experimental frecuente. Es posible que la presentación y el procesamiento efectivos de los epítopos predichos por las células presentadoras de antígenos no sean completamente capturados por la bioinformática. La incertidumbre con respecto a la durabilidad a largo plazo de la respuesta inmunitaria y el potencial de la FIPV para generar mutantes de escape plantean desafíos para el éxito de la vacuna. Además, el proceso de aprobación regulatoria para las vacunas desarrolladas mediante inmunoinformática puede verse obstaculizado por la necesidad de una validación experimental sólida y las preocupaciones sobre la seguridad y la eficacia. A pesar de estas limitaciones, la combinación de enfoques computacionales y experimentales sigue siendo crucial para mejorar la eficacia de las vacunas multiepítopo de la FIPV.
5 Conclusion
La fuerza impulsora de esta investigación fue la utilización de metodologías bioinformáticas para identificar un posible contendiente de vacuna contra la FIPV, dirigida a la proteína S. Se diseñó meticulosamente una vacuna candidata innovadora para la FIPV, que contaba con estabilidad proyectada, características no alergénicas y antigenicidad anticipada. Sin embargo, la trayectoria que tenemos por delante exige análisis in vivo en profundidad para determinar meticulosamente la seguridad y eficacia de la vacuna candidata FIPV. La posibilidad de una vacuna sugerida surge como un trampolín auspicioso, que sienta las bases para futuras exploraciones experimentales que buscan cultivar una vacuna eficaz contra la FIPV.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo/Material complementario, las consultas adicionales pueden dirigirse a los autores correspondientes.
Contribuciones de los autores
MC: Conceptualización, Metodología, Redacción – borrador original, Redacción – revisión y edición, Curación de datos, Supervisión, Validación, Visualización. AC: Curación de datos, Investigación, Redacción – borrador original. NA: Curación de datos, Metodología, Software, Redacción – borrador original. JM: Metodología, Software, Redacción – borrador original. SR: Curación de datos, Redacción – borrador original. CS: Curación de datos, Investigación, Redacción – borrador original. NJ: Curación de datos, Software, Escritura – borrador original. KC: Conceptualización, Obtención de Fondos, Redacción – Revisión y Edición. AS: Análisis formal, Metodología, Software, Redacción – revisión y edición. TM: Administración de proyectos, Redacción – borrador original. LC: Administración de proyectos, Recursos, Supervisión, Redacción, revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. La investigación fue financiada por la Universidad de Ciencia y Tecnología Rey Abdullah (KAUST, por sus siglas en inglés). Esta investigación utilizó el Laboratorio de Supercomputación de la Universidad de Ciencia y Tecnología Rey Abdullah (KAUST) en Thuwal, Arabia Saudita, para el tiempo de computadora. Los miembros del equipo de STEMskills Research and Education Lab Private Limited son reconocidos por la lectura crítica del manuscrito y el apoyo computacional. Además, este trabajo contó con el apoyo financiero de la Secretaría de Educación Superior, Ciencia, Investigación e Innovación; y el Programa de Investigación e Innovación Científica de Tailandia a través del Programa de Reinvención de la Universidad de Kasetsart 2022. Instituto de Investigación y Desarrollo de la Universidad de Kasetsart (KURDI) Bangkok, Tailandia, KURDI (FF(KU)16.66) también es reconocido.
Conflicto de intereses
NA y AS fueron empleados por la empresa STEMskills Research and Education Lab Private Limited, India.
El resto de los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un potencial conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1280273/full#supplementary-material
Referencias
1. Kennedy, MA. Feline infectious peritonitis: update on pathogenesis, diagnostics, and treatment. Vet Clin North Am Small Anim Pract. (2020) 50:1001–11. doi: 10.1016/j.cvsm.2020.05.002
2. Klein-Richers, U, Hartmann, K, Hofmann-Lehmann, R, Unterer, S, Bergmann, M, Rieger, A, et al. Prevalence of feline coronavirus shedding in German catteries and associated risk factors. Viruses. (2020) 12:1000. doi: 10.3390/v12091000
3. Sharif, S, Arshad, SS, Hair-Bejo, M, Omar, AR, Zeenathul, NA, and Hafidz, MA. Prevalence of feline coronavirus in two cat populations in Malaysia. J Feline Med Surg. (2009) 11:1031–4. doi: 10.1016/j.jfms.2009.08.005
4. Herrewegh, AA, Mähler, M, Hedrich, HJ, Haagmans, BL, Egberink, HF, Horzinek, MC, et al. Persistence and evolution of feline coronavirus in a closed cat-breeding Colony. Virology. (1997) 234:349–63. doi: 10.1006/viro.1997.8663
5. Thayer, V, Gogolski, S, Felten, S, Hartmann, K, Kennedy, M, and Olah, GA. 2022 AAFP/EveryCat feline infectious peritonitis diagnosis guidelines. J Feline Med Surg. (2022) 24:905–33. doi: 10.1177/1098612X221118761
6. Felten, S, and Hartmann, K. Diagnosis of feline infectious peritonitis: a review of the current literature. Viruses. (2019) 11:1068. doi: 10.3390/v11111068
7. Vogel, L, Van der Lubben, M, Te Lintelo, EG, Bekker, CPJ, Geerts, T, Schuijff, LS, et al. Pathogenic characteristics of persistent feline enteric coronavirus infection in cats. Vet Res. (2010) 41:71. doi: 10.1051/vetres/2010043
8. Tekes, G, and Thiel, H-J. Feline coronaviruses. Adv Virus Res. (2016) 96:193–218. doi: 10.1016/bs.aivir.2016.08.002
9. Tekes, G, Hofmann-Lehmann, R, Stallkamp, I, Thiel, V, and Thiel, H-J. Genome organization and reverse genetic analysis of a type I feline coronavirus. J Virol. (2008) 82:1851–9. doi: 10.1128/JVI.02339-07
10. Kipar, A, and Meli, ML. Feline infectious peritonitis: still an enigma? Vet Pathol. (2014) 51:505–26. doi: 10.1177/0300985814522077
11. Pedersen, NC, Liu, H, Scarlett, J, Leutenegger, CM, Golovko, L, Kennedy, H, et al. Feline infectious peritonitis: role of the feline coronavirus 3c gene in intestinal tropism and pathogenicity based upon isolates from resident and adopted shelter cats. Virus Res. (2012) 165:17–28. doi: 10.1016/j.virusres.2011.12.020
12. Rottier, PJM, Nakamura, K, Schellen, P, Volders, H, and Haijema, BJ. Acquisition of macrophage tropism during the pathogenesis of feline infectious peritonitis is determined by mutations in the feline coronavirus spike protein. J Virol. (2005) 79:14122–30. doi: 10.1128/JVI.79.22.14122-14130.2005
13. Chen, C, Li, Y-L, Lv, F-L, Xu, L-D, and Huang, Y-W. Surface display of peptides corresponding to the heptad repeat 2 domain of the feline enteric coronavirus spike protein on Bacillus Subtilis spores elicits protective immune responses against homologous infection in a feline aminopeptidase-N-transduced mouse model. Front Immunol. (2022) 13:925922. doi: 10.3389/fimmu.2022.925922
14. Wang, G, Hu, G, Liang, R, Shi, J, Qiu, X, Yang, Y, et al. Establishment of full-length CDNA clones and an efficient Oral infection model for feline coronavirus in cats. J Virol. (2021) 95:e00745-21. doi: 10.1128/JVI.00745-21
15. Doytchinova, IA, and Flower, DR. VaxiJen: a server for prediction of protective antigens, tumour antigens and subunit vaccines. BMC Bioinformatics. (2007) 8:4. doi: 10.1186/1471-2105-8-4
16. Reynisson, B, Alvarez, B, Paul, S, Peters, B, and Nielsen, M. NetMHCpan-4.1 and NetMHCIIpan-4.0: improved predictions of MHC antigen presentation by concurrent motif deconvolution and integration of MS MHC eluted ligand data. Nucleic Acids Res. (2020) 48:W449–54. doi: 10.1093/nar/gkaa379
17. Dimitrov, I, Naneva, L, Doytchinova, I, and Bangov, I. AllergenFP: allergenicity prediction by descriptor fingerprints. Bioinformatics. (2014) 30:846–51. doi: 10.1093/bioinformatics/btt619
18. Gupta, S, Kapoor, P, Chaudhary, K, Gautam, A, Kumar, R, Consortium, OSDD, et al. In silico approach for predicting toxicity of peptides and proteins. PLoS One. (2013) 8:e73957. doi: 10.1371/journal.pone.0073957
19. Case, DA, Cheatham, TE III, Darden, T, Gohlke, H, Luo, R, Merz, KM Jr, et al. The Amber biomolecular simulation programs. J Comput Chem. (2005) 26:1668–88. doi: 10.1002/jcc.20290
20. Eberhardt, J, Santos-Martins, D, Tillack, AF, and Forli, S. AutoDock Vina 1.2.0: new docking methods, expanded force field, and python bindings. J Chem Inf Model. (2021) 61:3891–8. doi: 10.1021/acs.jcim.1c00203
21. Liang, R, Sun, Y, Liu, Y, Wang, J, Wu, Y, Li, Z, et al. Major histocompatibility complex class I (FLA-E*01801) molecular structure in domestic cats demonstrates species-specific characteristics in presenting viral antigen peptides. J Virol. (2018) 92:e01631-17. doi: 10.1128/jvi.01631-17
22. Liljeqvist, S, Ståhl, S, Andréoni, C, Binz, H, Uhlén, M, and Murby, M. Fusions to the cholera toxin B subunit: influence on pentamerization and GM1 binding. J Immunol Methods. (1997) 210:125–35. doi: 10.1016/s0022-1759(97)00170-1
23. Stratmann, T. Cholera toxin subunit B as adjuvant–an accelerator in protective immunity and a break in autoimmunity. Vaccines (Basel). (2015) 3:579–96. doi: 10.3390/vaccines3030579
24. Alexander, J, Sidney, J, Southwood, S, Ruppert, J, Oseroff, C, Maewal, A, et al. Development of high potency universal DR-restricted helper epitopes by modification of high affinity DR-blocking peptides. Immunity. (1994) 1:751–61. doi: 10.1016/s1074-7613(94)80017-0
25. Chan, Y, Jazayeri, SD, Ramanathan, B, and Poh, CL. Enhancement of tetravalent immune responses to highly conserved epitopes of a dengue peptide vaccine conjugated to polystyrene nanoparticles. Vaccines (Basel). (2020) 8:417. doi: 10.3390/vaccines8030417
26. Li, X, Zhang, S, Lei, J, Zhu, Y, Zhou, X, Xiao, J, et al. Prophylactic herpes simplex virus type 2 vaccine Adjuvanted with a universal CD4 T cell helper peptide induces long-term protective immunity against lethal challenge in mice. Int Immunopharmacol. (2018) 61:100–8. doi: 10.1016/j.intimp.2018.05.024
27. Fraser, CC, Altreuter, DH, Ilyinskii, P, Pittet, L, LaMothe, RA, Keegan, M, et al. Generation of a universal CD4 memory T cell recall peptide effective in humans, mice and non-human Primates. Vaccine. (2014) 32:2896–903. doi: 10.1016/j.vaccine.2014.02.024
28. Zhang, W, Xu, L, Park, H-B, Hwang, J, Kwak, M, Lee, PCW, et al. Escherichia Coli adhesion portion FimH functions as an adjuvant for cancer immunotherapy. Nat Commun. (2020) 11:1187. doi: 10.1038/s41467-020-15030-4
29. Zhang, Y, Huo, F, Cao, Q, Jia, R, Huang, Q, Wang, ZA, et al. FimH confers mannose-targeting ability to Bacillus Calmette-Guerin for improved immunotherapy in bladder cancer. J Immunother Cancer. (2022) 10:e003939. doi: 10.1136/jitc-2021-003939
30. Law, JLM, Logan, M, Joyce, MA, Landi, A, Hockman, D, Crawford, K, et al. SARS-COV-2 recombinant receptor-binding-domain (RBD) induces neutralizing antibodies against variant strains of SARS-CoV-2 and SARS-CoV-1. Vaccine. (2021) 39:5769–79. doi: 10.1016/j.vaccine.2021.08.081
31. Li, G, Huang, Z, Zhang, C, Dong, B-J, Guo, R-H, Yue, H-W, et al. Construction of a linker library with widely controllable flexibility for fusion protein design. Appl Microbiol Biotechnol. (2016) 100:215–25. doi: 10.1007/s00253-015-6985-3
32. Arai, R, Ueda, H, Kitayama, A, Kamiya, N, and Nagamune, T. Design of the linkers which effectively separate domains of a bifunctional fusion protein. Protein Eng Des Sel. (2001) 14:529–32. doi: 10.1093/protein/14.8.529
33. Gu, Y, Sun, X, Li, B, Huang, J, Zhan, B, and Zhu, X. Vaccination with a Paramyosin-based multi-epitope vaccine elicits significant protective immunity against Trichinella spiralis infection in mice. Front Microbiol. (2017) 8:1475. doi: 10.3389/fmicb.2017.01475
34. Chen, X, Zaro, JL, and Shen, W-C. Fusion protein linkers: property, design and functionality. Adv Drug Deliv Rev. (2013) 65:1357–69. doi: 10.1016/j.addr.2012.09.039
35. Jumper, J, Evans, R, Pritzel, A, Green, T, Figurnov, M, Ronneberger, O, et al. Highly accurate protein structure prediction with AlphaFold. Nature. (2021) 596:583–9. doi: 10.1038/s41586-021-03819-2
36. Varadi, M, Anyango, S, Deshpande, M, Nair, S, Natassia, C, Yordanova, G, et al. AlphaFold protein structure database: massively expanding the structural coverage of protein-sequence space with high-accuracy models. Nucleic Acids Res. (2022) 50:D439–44. doi: 10.1093/nar/gkab1061
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Lovell, SC, Davis, IW, Arendall, WB, de Bakker, PIW, Word, JM, Prisant, MG, et al. Validación de la estructura mediante geometría Calpha: phi,psi y desviación Cbeta. Proteínas. (2003) 50:437–50. doi: 10.1002/prot.10286
38. Wiederstein, M, y Sippl, MJ. ProSA-web: servicio web interactivo para el reconocimiento de errores en estructuras tridimensionales de proteínas. Ácidos nucleicos Res. (2007) 35:W407-10. doi: 10.1093/nar/gkm290
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. van Zundert, GCP, Rodrigues, JPGLM, Trellet, M, Schmitz, C, Kastritis, PL, Karaca, E, et al. El servidor web HADDOCK2.2: modelado integrador fácil de usar de complejos biomoleculares. J Mol Biol. (2016) 428:720–5. doi: 10.1016/j.jmb.2015.09.014
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Mossman, KL, Mian, MF, Lauzon, NM, Gyles, CL, Lichty, B, Mackenzie, R, et al. Filo de corte: FimH La adhesina de las fimbrias tipo 1 es un nuevo ligando TLR4. J Immunol. (2008) 181:6702–6. doi: 10.4049/jimmunol.181.10.6702
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Kim, HM, Park, BS, Kim, J-I, Kim, SE, Lee, J, Oh, SC, et al. Estructura cristalina del complejo TLR4-MD-2 con el antagonista de la endotoxina unida Eritoran. Células. (2007) 130:906–17. doi: 10.1016/j.cell.2007.08.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Abraham, MJ, Murtola, T, Schulz, R, Páll, S, Smith, JC, Hess, B, et al. GROMACS: simulaciones moleculares de alto rendimiento a través del paralelismo multinivel de computadoras portátiles a supercomputadoras. SoftwareX. (2015) 1–2:19–25. doi: 10.1016/j.softx.2015.06.001
43. González, HC, Darré, L, y Pantano, S. Mezcla transferible de modelos de agua atomística y de grano grueso. J Phys Chem B. (2013) 117:14438–48. doi: 10.1021/jp4079579
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Machado, MR, Barrera, EE, Klein, F, Sóñora, M, Silva, S, y Pantano, S. El campo de fuerza SIRAH 2.0: altius, fortius, citius. J Cálculo de la teoría química. (2019) 15:2719–33. doi: 10.1021/acs.jctc.9b00006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Huang, J, Rauscher, S, Nawrocki, G, Ran, T, Feig, M, de Groot, BL, et al. CHARMM36m: un campo de fuerza mejorado para proteínas plegadas e intrínsecamente desordenadas. Métodos Nat. (2017) 14:71–3. doi: 10.1038/nmeth.4067
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Parrinello, M, y Rahman, A. Transiciones polimórficas en monocristales: un nuevo método de dinámica molecular. J Appl Phys. (1981) 52:7182–90. doi: 10.1063/1.328693
47. Darden, T, York, D, y Pedersen, L. Malla de partículas Ewald: un método N·log(N) para sumas de Ewald en grandes sistemas. J Chem Phys. (1993) 98:10089–92. doi: 10.1063/1.464397
48. Hess, B, Bekker, H, Berendsen, HJC, y Fraaije, JGEM. LINCS: un solucionador de restricciones lineales para simulaciones moleculares. J Comput Chem. (1997) 18:1463–72. doi: 10.1002/(SICI)1096-987X(199709)18:12<1463::AID-JCC4>3.0.CO; 2-H
49. Schrödinger, LLC, (2015). El sistema de gráficos moleculares PyMOL, versión 2.0 Schrödinger, LLC.
50. Humphrey, W, Dalke, A y Schulten, K. VMD: dinámica molecular visual. J Mol Gráfico. (1996) 14:33–8. doi: 10.1016/0263-7855(96)00018-5
51. Bouysset, C, y Fiorucci, S. ProLIF: una biblioteca para codificar interacciones moleculares como huellas dactilares. J Chem. (2021) 13:72. doi: 10.1186/s13321-021-00548-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Cazador, JD. Matplotlib: un entorno gráfico 2D. Comput Sci Eng. (2007) 9:90–5. doi: 10.1109/MCSE.2007.55
53. Lehman, TL, O’Halloran, KP, Fallon, SA, Habermann, LM, Campbell, JA, Nordone, S, et al. Alteración de la producción de citocinas de células dendríticas de la médula ósea al receptor tipo Toll y a la ligadura de CD40 durante la infección crónica por el virus de la inmunodeficiencia felina. Inmunología. (2009) 126:405–12. doi: 10.1111/j.1365-2567.2008.02907.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
54. Kagan, JC. Detectar endotoxinas desde dentro. Ciencia. (2013) 341:1184–5. doi: 10.1126/science.1243939
Resumen de PubMed | Texto completo de CrossRef | Google Académico
55. Liu, G, Zhang, L y Zhao, Y. Modulación de las respuestas inmunitarias a través de la activación directa de receptores tipo Toll a las células T. Clin Exp Immunol. (2010) 160:168–75. doi: 10.1111/j.1365-2249.2010.04091.x
56. Vaure, C, y Liu, Y. Una revisión comparativa de la expresión y funcionalidad del receptor tipo toll 4 en diferentes especies animales. Frente Immunol. (2014) 5:316. doi: 10.3389/fimmu.2014.00316
57. Molteni, M, Gemma, S, y Rossetti, C. El papel del receptor tipo toll 4 en la inflamación infecciosa y no infecciosa. Mediat Inflamm. (2016) 2016:6978936–9. doi: 10.1155/2016/6978936
Resumen de PubMed | Texto completo de CrossRef | Google Académico
58. Walsh, MC, Lee, J y Choi, Y. Regulación del desarrollo, la función y la homeostasis del sistema inmunitario del factor asociado al receptor del factor de necrosis tumoral (TRAF6). Immunol Rev. (2015) 266:72–92. doi: 10.1111/imr.12302
Resumen de PubMed | Texto completo de CrossRef | Google Académico
59. Jursza, E, Kowalewski, MP, Boos, A, Skarzynski, DJ, Socha, P, y Siemieniuch, MJ. El papel de los receptores tipo toll 2 y 4 en la patogénesis de la piometra felina. Teriogenología. (2015) 83:596–603. doi: 10.1016/j.theriogenology.2014.10.023
60. Embgenbroich, M, and Burgdorf, S. Current concepts of antigen cross-presentation. Front Immunol. (2018) 9:1643. doi: 10.3389/fimmu.2018.01643
61. Joffre, OP, Segura, E, Savina, A, and Amigorena, S. Cross-presentation by dendritic cells. Nat Rev Immunol. (2012) 12:557–69. doi: 10.1038/nri3254
62. Karplus, M, and Kuriyan, J. Molecular dynamics and protein function. Proc Natl Acad Sci U S A. (2005) 102:6679–85. doi: 10.1073/pnas.0408930102
63. Akhtar, N, Kaushik, V, Grewal, RK, Wani, AK, Suwattanasophon, C, Choowongkomon, K, et al. Immunoinformatics-aided design of a peptide based multiepitope vaccine targeting glycoproteins and membrane proteins against monkeypox virus. Viruses. (2022) 14:2374. doi: 10.3390/v14112374
64. Akhtar, N, Magdaleno, JSL, Ranjan, S, Wani, AK, Grewal, RK, Oliva, R, et al. Secreted aspartyl proteinases targeted multi-epitope vaccine design for Candida dubliniensis using immunoinformatics. Vaccine. (2023) 11:364. doi: 10.3390/vaccines11020364
65. Kaushik, V, G, S.K, Gupta, LR, Kalra, U, Shaikh, AR, Cavallo, L, et al. Immunoinformatics aided design and in-vivo validation of a cross-reactive peptide based multi-epitope vaccine targeting multiple serotypes of dengue virus. Front Immunol. (2022) 13:865180. doi: 10.3389/fimmu.2022.865180
66. Kaushik, V, Jain, P, Akhtar, N, Joshi, A, Gupta, LR, Grewal, RK, et al. Immunoinformatics-aided design and in vivo validation of a peptide-based multiepitope vaccine targeting canine circovirus. ACS Pharmacol Transl Sci. (2022) 5:679–91. doi: 10.1021/acsptsci.2c00130
67. Chawla, M, Gorle, S, Shaikh, AR, Oliva, R, y Cavallo, L. Sustitución de la timina por una quinta base fuertemente emparejada: un estudio combinado de mecánica cuántica y dinámica molecular. Comput Struct Biotechnol J. (2021) 19:1312–24. doi: 10.1016/j.csbj.2021.02.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
68. Vangone, A, Spinelli, R, Scarano, V, Cavallo, L, y Oliva, R. COCOMAPS: una aplicación web para analizar y visualizar contactos en la interfaz de complejos biomoleculares. Bioinformática. (2011) 27:2915–6. doi: 10.1093/bioinformatics/btr484
Resumen de PubMed | Texto completo de CrossRef | Google Académico
69. Camero, M, Lanave, G, Catella, C, Lucente, MS, Sposato, A, Mari, V, et al. ERDRP-0519 inhibe el coronavirus felino in vitro. BMC Vet Res. (2022) 18:55. doi: 10.1186/s12917-022-03153-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
70. Tanaka, Y, Sato, Y y Sasaki, T. La replicación del coronavirus felino se ve afectada tanto por la ciclofilina A como por la ciclofilina B. J Gen Virol. (2017) 98:190–200. doi: 10.1099/jgv.0.000663
Resumen de PubMed | Texto completo de CrossRef | Google Académico
71. Hu, C-MJ, Chang, W-S, Fang, Z-S, Chen, Y-T, Wang, W-L, Tsai, H-H, et al. El bloqueador de la ATPasa vacuolar nanopartícula exhibe una potente actividad antiviral dirigida al huésped contra el coronavirus felino. Sci Rep. (2017) 7:13043. doi: 10.1038/s41598-017-13316-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
72. Perera, KD, Galasiti Kankanamalage, AC, Rathnayake, AD, Honeyfield, A, Groutas, W, Chang, K-O, et al. Inhibidores de la proteasa ampliamente eficaces contra los coronavirus felinos, hurones y visones. Antiviral Res. (2018) 160:79–86. doi: 10.1016/j.antiviral.2018.10.015
Resumen de PubMed | Texto completo de CrossRef | Google Académico
73. Hsieh, L-E, Lin, C-N, Su, B-L, Jan, T-R, Chen, C-M, Wang, C-H, et al. Efecto antiviral sinérgico de la aglutinina de Galanthus Nivalis y nelfinavir contra el coronavirus felino. Antivir Res. (2010) 88:25–30. doi: 10.1016/j.antiviral.2010.06.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
74. Cook, SE, Vogel, H, Castillo, D, Olsen, M, Pedersen, N, y Murphy, BG. Investigación de monoterapia y terapias anticoronavirales combinadas contra el coronavirus felino serotipo II in vitro. J Feline Med Surg. (2022) 24:943–53. doi: 10.1177/1098612X211048647
Resumen de PubMed | Texto completo de CrossRef | Google Académico
75. Wang, X, Li, F, Han, M, Jia, S, Wang, L, Qiao, X, et al. Clonación, expresión soluble procariota y análisis de la actividad antiviral de dos nuevas proteínas IFN-ω felinas. Virus. (2020) 12:335. doi: 10.3390/v12030335
Resumen de PubMed | Texto completo de CrossRef | Google Académico
76. Bálint, Á, Farsang, A, Szeredi, L, Zádori, Z y Belák, S. Los coronavirus felinos recombinantes como candidatos a vacunas confieren protección en SPF pero no en gatos convencionales. Microbiol veterinario. (2014) 169:154–62. doi: 10.1016/j.vetmic.2013.10.015
Resumen de PubMed | Texto completo de CrossRef | Google Académico
77. Hebben, M, Duquesne, V, Cronier, J, Rossi, B, y Aubert, A. Virus vaccinia modificado de Ankara como vacuna contra el coronavirus felino: inmunogenicidad y eficacia. J Feline Med Surg. (2004) 6:111–8. doi: 10.1016/j.jfms.2003.12.011
78. Haijema, BJ, Volders, H, and Rottier, PJM. Live, attenuated coronavirus vaccines through the directed deletion of group-specific genes provide protection against feline infectious peritonitis. J Virol. (2004) 78:3863–71. doi: 10.1128/JVI.78.8.3863-3871.2004
79. Hohdatsu, T, Yamato, H, Ohkawa, T, Kaneko, M, Motokawa, K, Kusuhara, H, et al. Vaccine efficacy of a cell lysate with recombinant Baculovirus-expressed feline infectious peritonitis (FIP) virus nucleocapsid protein against progression of FIP. Vet Microbiol. (2003) 97:31–44. doi: 10.1016/j.vetmic.2003.09.016
80. Glansbeek, HL, Haagmans, BL, te Lintelo, EG, Egberink, HF, Duquesne, V, Aubert, A, et al. Adverse effects of feline IL-12 during DNA vaccination against feline infectious peritonitis virus. J Gen Virol. (2002) 83:1–10. doi: 10.1099/0022-1317-83-1-1
81. Takano, T, Tomizawa, K, Morioka, H, Doki, T y Hohdatsu, T. Evaluación de la eficacia protectora de la vacuna peptídica sintética que contiene el epítopo T-helper 1 con Cpg Oligodesoxinucleótido contra la infección por el virus de la peritonitis infecciosa felina en gatos. Antivir Ther. (2014) 19:645–50. doi: 10.3851/IMP2735
Resumen de PubMed | Texto completo de CrossRef | Google Académico
82. Takano, T, Morioka, H, Gomi, K, Tomizawa, K, Doki, T y Hohdatsu, T. Cribado e identificación del ayudante T 1 y epítopos lineales de unión a anticuerpos inmunodominantes en el dominio de la espícula 1 y la proteína de membrana del virus de la peritonitis infecciosa felina. Vacuna. (1834) 32:1834–40. doi: 10.1016/j.vaccine.2014.01.074
Resumen de PubMed | Texto completo de CrossRef | Google Académico
83. Jain, P, Joshi, A, Akhtar, N, Krishnan, S, y Kaushik, V. Un estudio inmunoinformático: diseño de una vacuna multivalente de epítopos de células T contra circovirus caninos. J Genet Eng Biotechnol. (2021) 19:121. doi: 10.1186/s43141-021-00220-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
84. Mia, MM, Hasan, M, Ahmed, S, y Rahman, MN. Información sobre la primera vacuna de subunidades peptídicas basadas en epítopos múltiples contra el virus de la influenza aviar A (H5N6): un enfoque inmunoinformático. Infectar Genet Evol. (2022) 104:105355. doi: 10.1016/j.meegid.2022.105355
Resumen de PubMed | Texto completo de CrossRef | Google Académico
85. Kar, PP, Araveti, PB, Kuriakose, A, y Srivastava, A. Diseño de una proteína multiepítopo como vacuna de subunidad contra la dermatosis nodular contagiosa utilizando un enfoque inmunoinformático. Sci Rep. (2022) 12:19411. doi: 10.1038/s41598-022-23272-z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
86. Nguyen, TL, Samuel Leon Magdaleno, J, Rajjak Shaikh, A, Choowongkomon, K, Li, V, Lee, Y, et al. Diseño de una vacuna candidata multiepítopo mediante el empleo de enfoques inmunoinformáticos para controlar la propagación de la peste porcina africana. J Biomol Struct Dyn. (2022) 41:10214–29. doi: 10.1080/07391102.2022.2153922
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: coronavirus felino, peritonitis infecciosa felina, vacuna, inmunoinformática, vacunología inversa, proteína de la espícula
Cita: Chawla M, Cuspoca AF, Akthar N, Magdaleno JSL, Rattanabunyong S, Suwattanasophon C, Jongkon N, Choowongkomon K, Shaikh AR, Malik T y Cavallo L (2023) Diseño racional asistido por inmunoinformática de una vacuna multiepítopo dirigida al virus de la peritonitis infecciosa felina. Frente. Vet. Sci. 10:1280273. doi: 10.3389/fvets.2023.1280273
Recibido: 19 de agosto de 2023; Aceptado: 27 de noviembre de 2023;
Publicado: 13 diciembre 2023.
Editado por:
Xiaoyu Niu, Universidad Estatal Técnica y Agrícola de Carolina del Norte, Estados Unidos
Revisado por:
Mamoon Rashid, Centro Internacional de Investigación Médica Rey Abdullah (KAIMRC), Arabia
Saudita Albert Poater, Universidad de Girona, España
Suresh Gorle, PharmCADD, República de Corea
Derechos de autor © 2023 Chawla, Cuspoca, Akthar, Magdaleno, Rattanabunyong, Suwattanasophon, Jongkon, Choowongkomon, Shaikh, Malik y Cavallo. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Kiattawee Choowongkomon, de fsciktc@ku.ac.th; Abdul Rajjak Shaikh, de ab_rajjak@yahoo.co.in; Tabarak Malik, tabarak.malik@ju.edu.et; Luigi Cavallo, luigi.cavallo@kaust.edu.sa
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía