Ensayo digital de reacción en cadena de la polimerasa de gotas múltiples para enterovirus, coronavirus y rotavirus bovino

Establecimiento y aplicación del ensayo digital de reacción en cadena de la polimerasa de gotas múltiples para enterovirus bovino, coronavirus bovino y rotavirus bovino
 Junzhen Chen1†
Junzhen Chen1† Dan Li1,2†
Dan Li1,2† Yafang Xu1
Yafang Xu1 Zeyu Li1
Zeyu Li1 Siqi Ma1
Siqi Ma1 Xinyi Liu1
Xinyi Liu1 Yuanyuan Yuan1
Yuanyuan Yuan1 Chengyuan Zhang1
Chengyuan Zhang1 Qiang Fu1*
Qiang Fu1* Huijun Shi1*
Huijun Shi1*- 1Facultad de Medicina Veterinaria, Universidad Agrícola de Xinjiang, Ürümqi, China
- 2Tecon Biology Co., Ltd., Ürümqi, China
El enterovirus bovino (BEV), el coronavirus bovino (BCoV) y el rotavirus bovino (BRV) siguen siendo las principales preocupaciones mundiales en el cuidado de la salud del ganado, causando graves pérdidas económicas en la industria ganadera. Es urgente establecer métodos específicos y sensibles para detectar virus para el control temprano de enfermedades. La PCR digital de gotitas (ddPCR) se ha propuesto para detectar eficazmente partículas virales, y no implica valores de Ct o curvas estándar. En este estudio, diseñamos cebadores y sondas específicos, basados en regiones conservadas de genomas virales, para optimizar los protocolos para un ensayo dual ddPCR para detectar BCoV y BRV y un ensayo ddPCR multiplex para BEV, BCoV y BRV. Los ensayos de sensibilidad revelaron que el límite inferior de detección para qPCR fue de 1.000 copias/μL y para ddPCR para BEV, BCoV y BRV, 2,7 copias/μL, 1 copia/μL y 2,4 copias/μL, respectivamente. Al estudiar 82 muestras recolectadas de terneros diarreicos en una granja, nuestro método dual ddPCR detectó BCoV, BRV y coinfección a tasas de 18.29%, 14.63% y 6.1%, respectivamente. En contraste, los métodos convencionales de qPCR detectaron BCoV, BRV y coinfección a tasas de 10.98%, 12.2% y 3.66%, respectivamente. Por otro lado, estudiando 68 muestras de otra granja, la qPCR detectó BCoV, BRV, BEV y coinfección de BCoV y BEV a tasas de 14.49%, 1.45%, 5.80% y 1.45%, respectivamente. Nuestro método ddPCR multiplex detectó BCoV, BRV, BEV, coinfección de BCoV y BEV, y coinfección de BRV y BEV. a tasas de 14,49%, 2,9%, 8,7%, 2,9% y 1,45%, respectivamente. Al estudiar 93 muestras de otra granja, la qPCR detectó BCoV, BRV, BEV y se detectó coinfección de BCoV y BEV a tasas de 5.38%, 1.08%, 18.28% y 1.08%, respectivamente. La coinfección de BCoV, BRV, BEV, BCoV y BEV, y la coinfección de BRV y BEV, se detectaron mediante métodos de ddPCR multiplex a tasas de 5,38%, 2,15%, 20,45%, 1,08% y 1,08%, respectivamente. Estos resultados indicaron que nuestros métodos optimizados de ddPCR dual y múltiplex fueron más efectivos que los ensayos convencionales de qPCR para detectar estas infecciones virales.
1. Introducción
El enterovirus bovino (BEV) y el coronavirus bovino (BCoV) son virus de ARN monocatenario de cadena positiva (1, 2). La infección por BEV en el ganado causa diarrea, heces con sangre, dificultad respiratoria, disminución de la producción de leche e infertilidad (3, 4). BCoV causa diarrea en terneros y diarrea invernal y enfermedades respiratorias en ganado adulto (5). El rotavirus bovino (BRV) es un virus de ARN bicatenario (6). BRV infecta a los terneros para producir diarrea acuosa, depresión y pérdida de apetito, lo que lleva a una tasa de morbilidad del 90% al 100% y una tasa de mortalidad del 50% (7, 8). BCoV y BRV son capaces de infectar a un animal. BCoV y BRV también pueden infectar a los humanos (2, 9). Como una preocupación seria, BEV, BCoV y BRV se han extendido a la mayoría de las áreas de China (10-12). Por lo tanto, es urgente establecer un método confiable, específico y cuantitativo para detectar y diferenciar eficazmente estos virus para el control temprano de las infecciones virales y la prevención oportuna de su propagación.
En 1985, Mullis y Faloona (13) informaron de la primera generación del método de reacción en cadena de la polimerasa (PCR). En 1992, Higuchi et al. (14) propusieron la segunda generación de PCR cuantitativa en tiempo real (qPCR), que se convirtió en un método mejorado para detectar patógenos cuantitativamente, específicamente, sensiblemente y reproduciblemente para el diagnóstico clínico de enfermedades animales/humanas y problemas de calidad de los alimentos. Sin embargo, el método qPCR implica la construcción de curvas estándar y valores de Ct (15). En 1999, Vogelstein y Kinzler (16) propusieron la PCR digital de tercera generación (dPCR) capaz de detectar cantidades bajas de múltiples objetivos de manera efectiva sin depender de la curva estándar y los valores de Ct (17). Recientemente, el grupo de Nyaruaba (18) informó sobre el ensayo ddPCR multiplex para la detección del SARS-CoV-2. Este nuevo método ayuda a desarrollar un ensayo de diagnóstico avanzado para el SARS-CoV-2 con resultados reducidos falsos positivos y falsos negativos. También ayuda a la investigación de laboratorio a maximizar el número de objetivos que deben detectarse dentro de un pequeño volumen de muestra (19). Sin embargo, la técnica de ddPCR multiplex no se ha aplicado a la detección simultánea de BEV, BCoV y BRV. Por lo tanto, en esta investigación, perseguimos el desarrollo de ensayos híbridos de ddPCR simples, rápidos, específicos y sensibles para detectar y diferenciar infecciones por BEV, BCoV y BRV.
2. Materiales y métodos
2.1. Muestras de tejido clínico y principales reactivos
Se obtuvieron muestras de 82 terneros diarreicos de una granja ganadera en Ürümqi, Xinjiang, China; Se obtuvieron 68 submuestras anales de ganado diarreico de una granja ganadera en Bole, Xinjiang, China; y se recolectaron 93 fórmulas anales de ganado en granjas ganaderas en el condado de Ürümqi, Xinjiang, China.
TRIzol fue comprado a Invitrogeng; 2× Taq PCR Mix, Common Agarose Gel DNA Recovery Kit (DP209), DH5α receptor cells, Rapid Plplasmid Small Extraction Kit y SuperReal Fluorescence Quantitative Premix Reagent (TaqMan) fueron adquiridos a Tiangen Biochemical Technology Co., Ltd. (Beijing, China); RT EasyTM II Reverse Transcription Kit fue comprado a FIC Biotechnology Co., Ltd. (Chengdu, China); El kit vectorial T-Vector pMD19 fue comprado a TaKaRa; 2× ddPCR SuperMix for Probes fue comprado a Bio-Rad.
2.2. Construcción de plásmidos estándar BEV, BCoV y BRV
Según la base de datos GenBank, combinada con las secuencias publicadas de BEV-E, BCoV Npro, y el gen BRV-VP6, las secuencias se enviaron a Sangon Bioengineering (Shanghai) Co., Ltd. BEV, BCoV y BRV PCR se clonaron en vectores pMD18-T, pGEM-T Easy Vector y pUC18 (comprados a ADDGENG), secuenciados para validación y mapeados (Figura suplementaria S1). El sistema específico de reacción por PCR se muestra en la Tabla Suplementaria S1. Las condiciones de reacción fueron 95°C, 5 min; 95°C, 30 s; recocido, 30 s; 72°C, 1 min; 34 ciclos; y 72°C, 10 min.
2.3. Diseño de cebadores y sondas de DDPCR BEV, BCoV y BRV
Los plásmidos recombinantes pMD18-T-BEV-E, pGEM-T-BCoV-N y pUC18-BRV-VP6 positivos correctamente secuenciados se utilizaron como plantillas de acuerdo con la base de datos GenBank. El software Primer Premier 5.0 (Primer Canada Inc.) se utilizó para diseñar cebadores y sondas específicos para las regiones conservadas de BEV E (GenBank No. MN607031.1), BCoV Npro (GenBank No. LC494174.1) y BRV-VP6 (GenBank No. AB573082.1) genes para reacciones qPCR y ddPCR (Tabla suplementaria S2). La secuencia de la sonda fue enviada a Suzhou GENEWIZ Biotechnology Co., Ltd. para su síntesis.
2.4. Establecimiento del método de PCR cuantitativa en tiempo real TaqMan
De acuerdo con las instrucciones del kit SuperReal Pre Mix (TaqMan), los estándares plásmidos de BEV, BCoV y BRV a una concentración de 1 × 106 copias/μL fueron seleccionadas como plantillas, y otras condiciones fueron constantes controladas. Las reacciones de qPCR se realizaron en las concentraciones de cebador aguas arriba y aguas abajo, seleccionadas como gradientes de concentración de 200, 300, 400, 500, 600, 700, 800 y 900 nM, para detectar la mejor concentración de cebadores a utilizar. Las reacciones de qPCR se realizaron cuando las concentraciones finales de las sondas se ajustaron a 150, 200, 250, 300, 350, 400 y 450 nM, respectivamente, para determinar las concentraciones óptimas de la sonda para BEV, BCoV y BRV. Las concentraciones iniciales de 1 × 1010 copias/μL de patrones de plásmidos BEV, BCoV y BRV se diluyeron en un gradiente de 10 veces (1 × 109–1 × 102 copias/μL), se realizaron reacciones de qPCR, se construyeron curvas estándar y se realizaron pruebas de sensibilidad, especificidad y reproducibilidad. El programa de PCR fue de 94 ° C durante 3 min, 94 ° C durante 7 s, 60 ° C durante 30 s y 40 ciclos.
2.5. Detección de sensibilidad
BEV, BCoV y BRV se diluyeron en un gradiente de 10 veces (1 × 109–1 × 102 copias/μL), y se estableció un control negativo; Cada concentración se repitió tres veces. Las reacciones de qPCR se realizaron utilizando las condiciones de reacción optimizadas para determinar la sensibilidad del método.
2.6. Detección de especificidad
La especificidad del método de qPCR se evaluó mediante el método qPCR establecido para la detección de plásmidos positivos de BEV, BCoV y BRV, respectivamente, con un solo plásmido estándar como control positivo, agua como control negativo y el resto de plásmidos estándar como grupo de detección para la prueba.
2.7. Detección de la repetibilidad
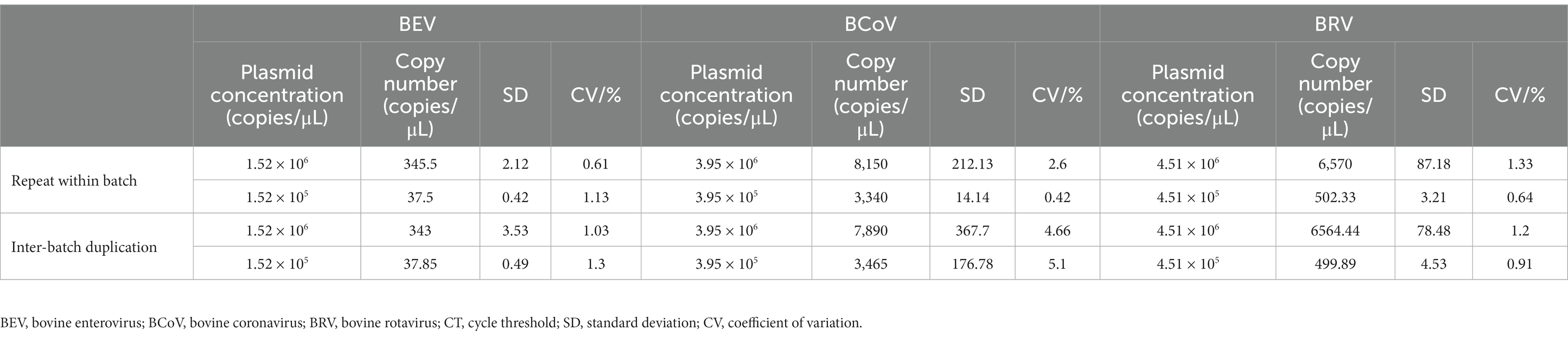
El método optimizado se utilizó para realizar reacciones de qPCR utilizando 1 × 107 copias/μL, 1 × 106 copias/μl y 1 × 105 copias/μL de patrones de plásmidos como concentraciones de plantilla, que se repitieron tres veces. A intervalos, se realizaron tres experimentos de replicación utilizando las condiciones anteriores para detectar la variación de lote a lote en el método. El coeficiente de variación (CV) de los valores de Ct en experimentos de replicación intra e interlotes se calculó utilizando la fórmula CV = desviación estándar / media, y el CV se utilizó para responder a la reproducibilidad.
2.8. Establecimiento del método único de detección de ddPCR BEV, BCoV y BRV
La reacción ddPCR se realizó utilizando el sistema QX200 ddPCR de la empresa Bio-Rad, el programa fue de 94 ° C durante 10 min, 94 ° C durante 30 s, 58 ° C durante 30 s, 72 ° C durante 30 s, 40 ciclos, 98 ° C durante 10 min y 4 ° C para el almacenamiento. El sistema de reacción se configuró de acuerdo con el kit ddPCR SuperMix for Probes (Bio-Rad) para garantizar una mezcla completa y sin burbujas de aire. Las gotas se colocaron en el lector para lecturas de señal baja. Se llevaron a cabo experimentos de sensibilidad, especificidad y repetibilidad.
2.9. Establecimiento de reacciones de detección de ddPCR dual BCoV y BRV
De acuerdo con el flujo de trabajo y el método ddPCR, se utilizaron cebadores y sondas para BCoV y BRV. El canal FAM detectó el BCoV de destino y el canal HEX detectó el BRV de destino. El sistema de reacción dúplex ddPCR fue: 2× ddPCR SuperMix para sondas de 10 μL, dos pares de concentraciones superiores de cebador aguas abajo fueron de 450 nM cada una, las concentraciones de sonda fueron de 250 nM cada una, la plantilla fue de 2 μL, ddH2O se complementó a 20 μL, y las condiciones de reacción fueron las mismas que las anteriores.
2.10. Optimización de las condiciones de reacción de ddPCR multiplex para BEV, BCoV y BRV
La temperatura de recocido se estableció en 58 ° C, los cebadores fueron todos de 500 nM y se establecieron cuatro grupos de diferentes concentraciones de sonda. Se optimizaron las condiciones de las reacciones. Las secuencias y concentraciones específicas de la sonda se muestran en la Tabla Suplementaria S3.
2.11. Extracción de ARN y transcripción inversa
Las muestras de heces se agregaron tres veces el volumen de solución salina tamponada con fosfato esterilizada (PBS), vórtice y mezclada, y centrifugadas a 10,000 g durante 10 min a 4 ° C. Se recogió el sobrenadante, se añadió líquido de penicilina-estreptomicina y se dejó durante 4 h en nevera a 4°C, y la solución de tratamiento del virus se obtuvo por filtración utilizando un filtro de 0,22 μm. El ARN total de las muestras se extrajo utilizando TRIzol (Invitrogen), se determinó la concentración de ARN total y el ARN total se transcribió inversamente a ADNc utilizando un kit de transcripción inversa.
2.12. Detección de muestras clínicas
Las 82 muestras frescas recolectadas de terneros diarreicos de la granja ganadera en Ürümqi se agregaron al PBS esterilizado, vórtice y mezclado. El ARN extraído se transcribió inversamente en ADNc. Estas muestras se analizaron utilizando los métodos construidos de qPCR y ddPCR dúplex. Las 68 muestras frescas recolectadas de terneros diarreicos de la granja ganadera en Bole se trataron con PBS esterilizado, vórtice y mixto, y se extrajo ARN y se transcribió inversamente en ADNc. Estas muestras se examinaron utilizando métodos establecidos de qPCR y ddPCR múltiple.
3. Resultados
3.1. Establecimiento de métodos de qPCR BEV, BCoV y BRV
Se utilizaron estándares plásmidos de BEV, BCoV y BRV como plantillas, y se establecieron concentraciones de gradiente de cebadores aguas arriba y aguas abajo para las reacciones de qPCR. Se encontró que las concentraciones óptimas de cebador para BEV, BCoV y BRV eran 200 nM, 300 nM y 300 nM, respectivamente, de acuerdo con el análisis basado en el principio de producir el valor de Ct más pequeño y la dosis más económica (Tabla Suplementaria S4). Las concentraciones óptimas de la sonda fueron 400 nM para BEV, 150 nM para BCoV y 500 nM para BRV (Tabla Suplementaria S5). Las curvas estándar se construyeron con las condiciones anteriores y mostraron variaciones regulares entre cada rango de concentración y los valores de Ct, todas con excelente linealidad. La ecuación de regresión de la curva estándar para BEV fue y = −0,3295x + 42,281; el coeficiente de correlación (R2) de la curva estándar fue de 0,9996, y la eficiencia de amplificación fue del 99,68% (Figura 1A). La ecuación de regresión de la curva estándar para BCoV fue y = −3,4792x + 43,435, R2 = 0,9993, y la eficiencia de amplificación fue del 93,83% (Figura 1B). La ecuación de regresión de la curva estándar de BRV fue y = −3.369x + 44.292, R2 = 0,9997, y la eficiencia de amplificación fue del 98,07% (Figura 1C).
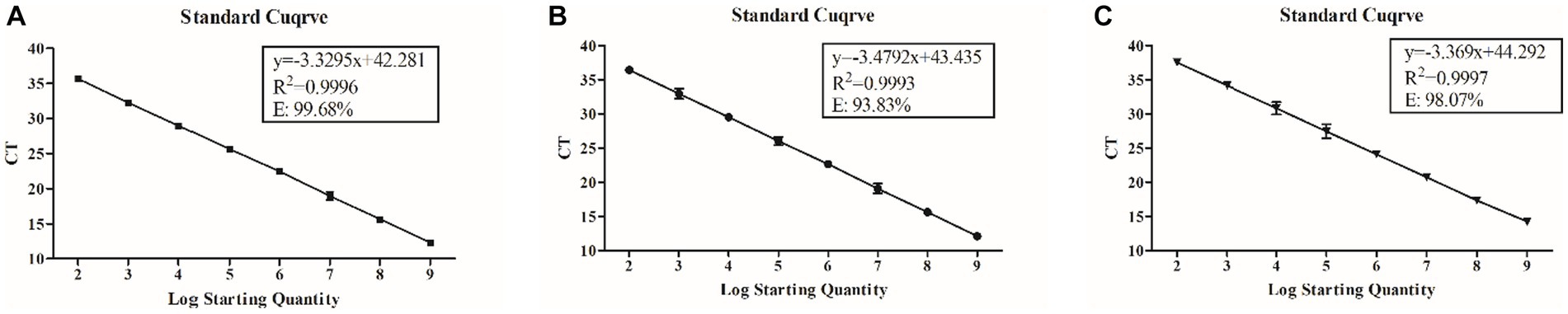 Figura 1. Ecuación de regresión, coeficiente de correlación (R2), y la eficiencia de amplificación de curvas estándar para ensayos de qPCR BEV, BCoV y BRV. (A) Curva estándar de BEV con una concentración de cebador de 200 nM y una concentración de sonda de 400 nM. (B) Curva estándar de BCoV con una concentración de cebador de 300 nM y una concentración de sonda de 150 nM. (C) Curva estándar de BRV con una concentración de cebador de 300 nM y una concentración de sonda de 500 nM. Coordenada horizontal: número de copias (A,B) o logaritmo del número de copias (C); coordenada vertical: umbral del ciclo (A,B); ecuación: ecuación de curva estándar; R2:coeficiente de correlación; E: eficiencia de amplificación.
Figura 1. Ecuación de regresión, coeficiente de correlación (R2), y la eficiencia de amplificación de curvas estándar para ensayos de qPCR BEV, BCoV y BRV. (A) Curva estándar de BEV con una concentración de cebador de 200 nM y una concentración de sonda de 400 nM. (B) Curva estándar de BCoV con una concentración de cebador de 300 nM y una concentración de sonda de 150 nM. (C) Curva estándar de BRV con una concentración de cebador de 300 nM y una concentración de sonda de 500 nM. Coordenada horizontal: número de copias (A,B) o logaritmo del número de copias (C); coordenada vertical: umbral del ciclo (A,B); ecuación: ecuación de curva estándar; R2:coeficiente de correlación; E: eficiencia de amplificación.
3.2. Detección de sensibilidad
Los patrones plásmidos de BEV, BCoV y BRV se diluyeron a 1 × 109–1 × 102 copias/μL utilizando el método de dilución de 10 veces y se realizaron experimentos de sensibilidad a qPCR y ddPCR. Los resultados del ensayo de qPCR mostraron que los valores de Ct de las concentraciones de plásmidos BEV, BCoV y BRV fueron inferiores a 35 a 1.000 copias/μL, por lo que el límite inferior de detección del método fue de 1.000 copias/μL (Tabla suplementaria S6). Los resultados del ensayo de ddPCR mostraron que las concentraciones de detección más bajas de los ensayos de ddPCR establecidos para BEV, BCoV y BRV fueron 2,7 copias/μL (Figura 2A), 1 copia/μL (Figura 2B) y 2,4 copias/μL (Figura 2C), respectivamente. La sensibilidad de los ensayos de ddPCR establecidos para BEV, BCoV y BRV demostró ser 370,37, 1.000 y 416,67 veces mayor que la de los ensayos de qPCR correspondientes, lo que indica que la ddPCR es significativamente más sensible que la qPCR y es capaz de detectar niveles más bajos de virus y reducir los falsos negativos.
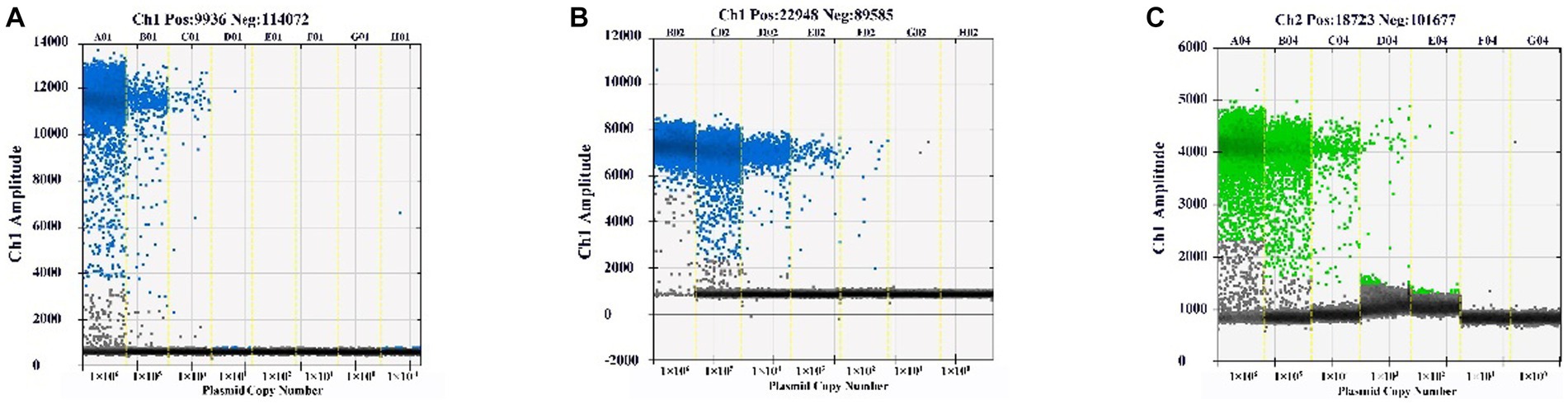 Figura 2. Sensibilidad del ensayo ddPCR. Los estándares de BEV, BCoV y BRV se diluyeron a 1 × 109–1 × 102 copias/μL en un gradiente de 10 veces y ensayadas por el método ddPCR construido. (A) Ensayo de sensibilidad del método BEV ddPCR. (B) Ensayo de sensibilidad del método BCoV ddPCR. (C) Ensayo de sensibilidad del método BRV ddPCR. Coordenada horizontal: número de copia plásmida; Coordenadas verticales: amplitud de fluorescencia. Amplitud Ch1: FAM, respectivamente.
Figura 2. Sensibilidad del ensayo ddPCR. Los estándares de BEV, BCoV y BRV se diluyeron a 1 × 109–1 × 102 copias/μL en un gradiente de 10 veces y ensayadas por el método ddPCR construido. (A) Ensayo de sensibilidad del método BEV ddPCR. (B) Ensayo de sensibilidad del método BCoV ddPCR. (C) Ensayo de sensibilidad del método BRV ddPCR. Coordenada horizontal: número de copia plásmida; Coordenadas verticales: amplitud de fluorescencia. Amplitud Ch1: FAM, respectivamente.
3.3. Detección de especificidad
La especificidad de los ensayos de qPCR y ddPCR se evaluó con los plásmidos de BEV, BCoV, BRV, BVDV y H2O como plantillas. Los resultados de la qPCR mostraron que solo el control positivo del virus correspondiente tenía una curva de amplificación, mientras que los otros virus no lo tenían (Figuras suplementarias S2A-C). Los resultados de ddPCR mostraron que solo el virus correspondiente produjo gotitas positivas, mientras que los otros virus no lo hicieron (Figuras 3A-C), lo que indica que ambos ensayos tuvieron buena especificidad.
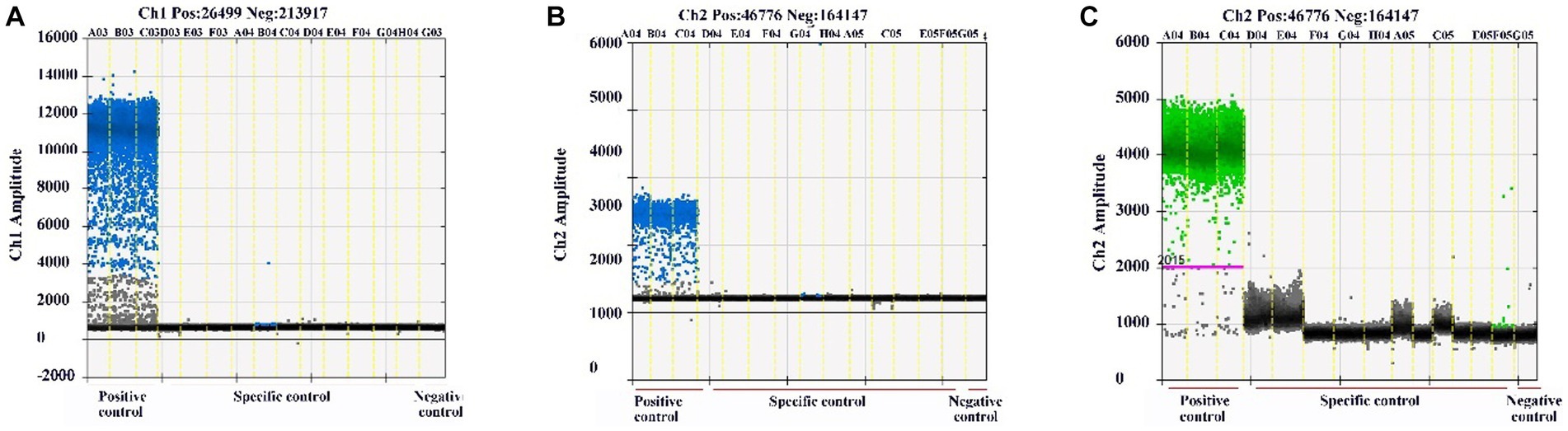 Figura 3. Resultados de detección de especificidad de ensayos de ddPCR. Detección de especificidad de BEV, BCoV, BRV y H2O utilizando ensayos de ddPCR establecidos. (A) Detección de especificidad de BEV mediante ddPCR. (B) Detección de especificidad de BCoV mediante ddPCR. (C) Detección de especificidad para BRV ddPCR. Coordenadas horizontales: conjunto grupo experimental; Coordenadas verticales: amplitud de fluorescencia. Amplitud Ch1: FAM, respectivamente.
Figura 3. Resultados de detección de especificidad de ensayos de ddPCR. Detección de especificidad de BEV, BCoV, BRV y H2O utilizando ensayos de ddPCR establecidos. (A) Detección de especificidad de BEV mediante ddPCR. (B) Detección de especificidad de BCoV mediante ddPCR. (C) Detección de especificidad para BRV ddPCR. Coordenadas horizontales: conjunto grupo experimental; Coordenadas verticales: amplitud de fluorescencia. Amplitud Ch1: FAM, respectivamente.
3.4. Detección de la repetibilidad
Se realizaron pruebas de replicación intra e interlotes del ensayo qPCR en plásmidos estándar de 1 × 107 a 1 × 105 copias/μL. Los resultados mostraron que los CV de las pruebas replicadas intra e interlotes para BEV, BCoV y BRV (Tabla Suplementaria S7) fueron inferiores al 3%. Las pruebas de reproducibilidad intra e interlotes BEV se realizaron diluyendo los patrones de plásmidos BEV a 1,52 × 105 y 1,52 × 106 copias/μL. Los CV intra e interlotes fueron inferiores al 1,3%, 0,5 veces mayor que el de la qPCR. Las pruebas de reproducibilidad de BCoV se realizaron diluyendo los patrones de plásmidos BCoV a 3,95 × 105 y 3,95 × 106 copias/μL. Los CV intra e interlotes fueron inferiores al 5,1%, 2,13 veces superiores a los de la qPCR. Las pruebas de reproducibilidad de BRV se realizaron diluyendo los patrones de plásmidos BRV a 4,51 × 105 y 4,51 × 106 copias/μL. Los CV intra e interlotes fueron inferiores al 1,33% (Tabla 1), 0,48 veces mayor que el de la qPCR. Esto indicó que los ensayos establecidos de BEV, BCoV y BRV qPCR y ddPCR tenían buena reproducibilidad y estabilidad, siendo los ensayos de ddPCR para BEV y BRV los más estables.
3.5. Establecimiento del sistema de reacción dual ddPCR BCoV y BRV
Los resultados de las reacciones duales de ddPCR para la detección de BCoV y BRV (Figura 4; Cifras complementarias S3A,B) mostró que las gotas azules indican un positivo para BCoV, las gotas verdes indican un positivo para BRV y las gotas naranjas indican un doble positivo para BCoV y BRV. Los resultados mostraron que el sistema de reacción dual ddPCR para BCoV y BRV establecido en este experimento podría detectar BCoV, BRV o BCoV y BRV simultáneamente.
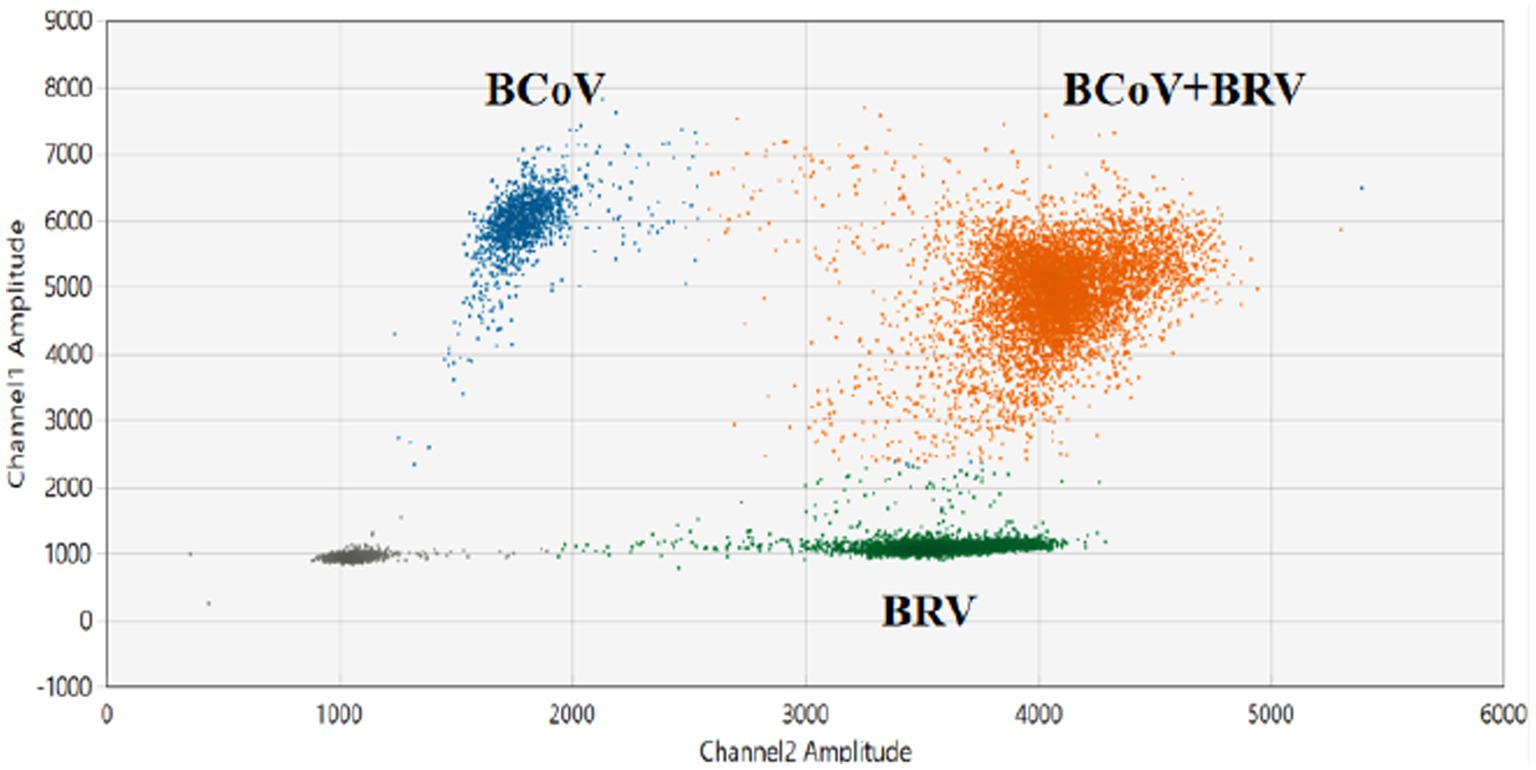 Figura 4. Establecimiento de los sistemas duales de detección de ddPCR BCoV y BRV. Gráfica 2D de los resultados de la ddPCR dual BCoV y BRV; azul: gotitas positivas para BCoV; verde: gotitas positivas para BRV; naranja: gotitas BCoV y BRV doblemente positivas; Gris: negativo. Las coordenadas horizontales y verticales son las amplitudes de fluorescencia de HEX y FAM, respectivamente. Amplitud del canal 1: FAM, respectivamente; amplitud del canal 2: HEX, respectivamente.
Figura 4. Establecimiento de los sistemas duales de detección de ddPCR BCoV y BRV. Gráfica 2D de los resultados de la ddPCR dual BCoV y BRV; azul: gotitas positivas para BCoV; verde: gotitas positivas para BRV; naranja: gotitas BCoV y BRV doblemente positivas; Gris: negativo. Las coordenadas horizontales y verticales son las amplitudes de fluorescencia de HEX y FAM, respectivamente. Amplitud del canal 1: FAM, respectivamente; amplitud del canal 2: HEX, respectivamente.
3.6. Establecimiento y optimización de ensayos multiplex ddPCR para BEV, BCoV y BRV
La temperatura de recocido de la reacción se estableció en 58 °C, la concentración del cebador fue de 500 nM y se realizaron diferentes concentraciones de sonda en el ensayo (Tabla suplementaria S3). Los resultados mostraron que había gotitas simples positivas, gotitas doblemente positivas y gotitas triple positivas, y las gotitas aparecieron en las concentraciones de la sonda de BCoV FAM 200 nM, BRV FAM 100 nM, HEX 100 nM y BEV HEX 200 nM. Los límites de gotas eran obvios y concentrados (Figura 5), por lo que esta concentración de sonda, temperatura de recocido y concentración de cebador se seleccionaron como las mejores condiciones de reacción. Otros resultados de concentración de la sonda se representan en las figuras suplementarias S4A-C.
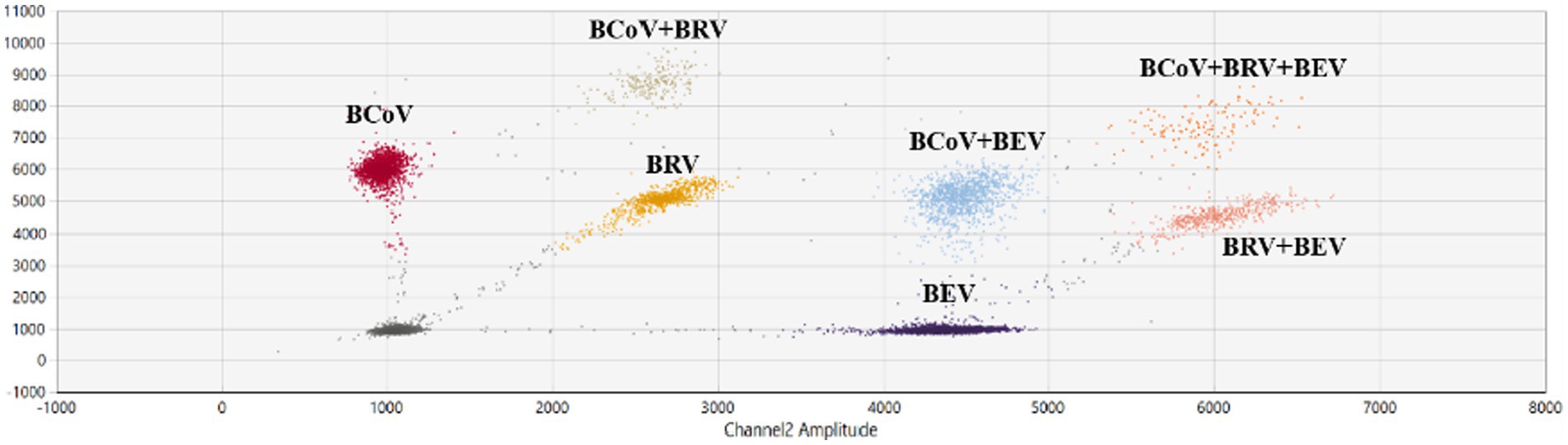 Figura 5. Establecimiento y optimización de resultados de ensayos multiplex ddPCR para BEV, BCoV y BRV. Resultados de ddPCR multiplex para concentraciones de sonda: BCoV FAM 200 nM, BRV FAM 100 nM, HEX 100 nM y BEV HEX 200 nM; a una temperatura de recocido de 58°C y una concentración de cebador de 500 nM. Rojo: gotitas positivas para BCoV; amarillo: gotitas positivas para el BRV; púrpura: gotitas positivas para BEV; rojo anaranjado: gotas dobles positivas BRV y BEV; marrón: gotitas BCoV y BRV doblemente positivas; azul: gotas de BCoV y BEV doblemente positivas; naranja: BEV, BCoV y BRV gotas triple positivas; Gris: negativo. Las coordenadas horizontales y verticales son las amplitudes de fluorescencia de HEX y FAM, respectivamente. Amplitud del canal 1: FAM, respectivamente; amplitud del canal 2: HEX, respectivamente.
Figura 5. Establecimiento y optimización de resultados de ensayos multiplex ddPCR para BEV, BCoV y BRV. Resultados de ddPCR multiplex para concentraciones de sonda: BCoV FAM 200 nM, BRV FAM 100 nM, HEX 100 nM y BEV HEX 200 nM; a una temperatura de recocido de 58°C y una concentración de cebador de 500 nM. Rojo: gotitas positivas para BCoV; amarillo: gotitas positivas para el BRV; púrpura: gotitas positivas para BEV; rojo anaranjado: gotas dobles positivas BRV y BEV; marrón: gotitas BCoV y BRV doblemente positivas; azul: gotas de BCoV y BEV doblemente positivas; naranja: BEV, BCoV y BRV gotas triple positivas; Gris: negativo. Las coordenadas horizontales y verticales son las amplitudes de fluorescencia de HEX y FAM, respectivamente. Amplitud del canal 1: FAM, respectivamente; amplitud del canal 2: HEX, respectivamente.
3.7. Detección de muestras clínicas
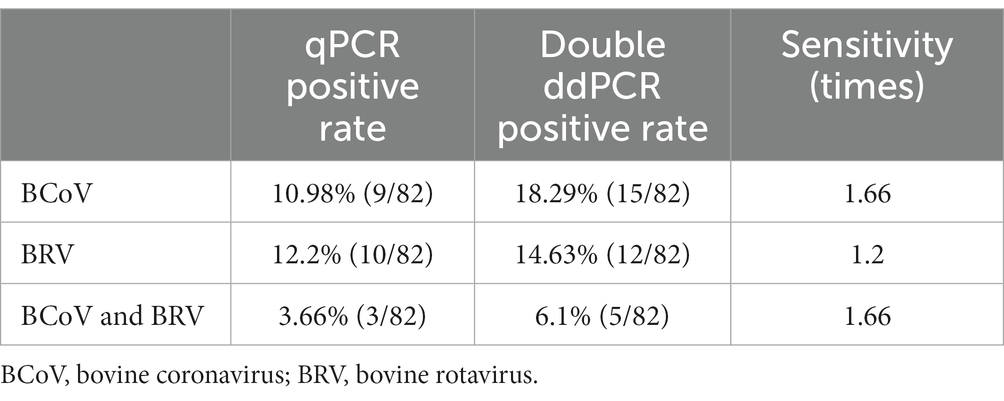
Ochenta y dos hisopos anales recolectados de terneros diarreicos se probaron utilizando los métodos establecidos de qPCR y ddPCR dúplex. Los resultados mostraron que las tasas de positividad de la qPCR para BCoV y BRV fueron 10,98% y 12,2%, respectivamente, con una tasa de coinfección de 3,66%. Las tasas de positividad de BCoV y BRV por ddPCR dúplex fueron de 18,29% y 14,63%, respectivamente, con una tasa de coinfección de 6,1% (Figura 6A). La sensibilidad fue 1,66 veces mayor que la de la qPCR para la detección de infecciones mixtas (Tabla 2; Cuadro suplementario S8). En comparación con los dos métodos, el ensayo dual ddPCR tuvo una mejor eficiencia de detección, lo que indica además que el ensayo de ddPCR fue más sensible y confiable que la qPCR.
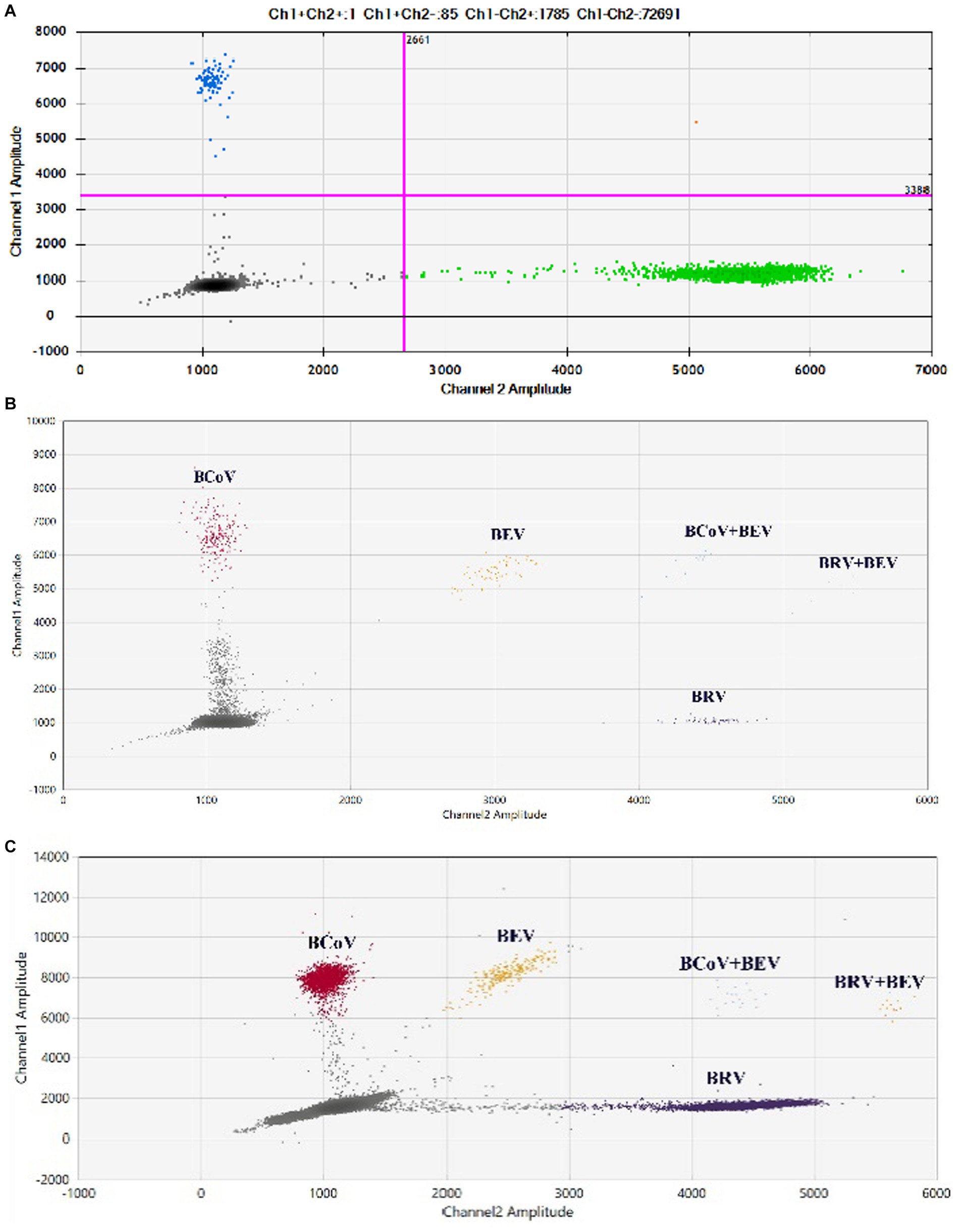 Figura 6. Resultados de muestras clínicas analizadas mediante ddPCR dúplex y múltiple. (A) Resultados de la coinfección en muestras clínicas probadas por ddPCR dúplex para BCoV y BRV; azul: gotitas positivas para el BRV; verde: gotitas positivas para BCoV; rojo: gotas dobles positivas BRV y BCoV. (B,C) Resultados de pruebas clínicas mediante ddPCR multiplex para BCoV, BEV y BRV. (B) Resultados de pruebas de 68 muestras recolectadas en Bole, Xinjiang (China). (C) Resultados de pruebas de 93 muestras recogidas en el condado de Ürümqi, Xinjiang (China). Rojo: gotitas positivas para BCoV; amarillo: gotitas positivas para el BRV; púrpura: gotitas positivas para BEV; naranja: gotitas dobles positivas BRV y BEV; azul: gotas de BCoV y BEV doblemente positivas; Gris: negativo. Las coordenadas horizontales y verticales son las amplitudes de fluorescencia de HEX y FAM, respectivamente. Amplitud del canal 1: FAM, respectivamente; amplitud del canal 2: HEX, respectivamente.
Figura 6. Resultados de muestras clínicas analizadas mediante ddPCR dúplex y múltiple. (A) Resultados de la coinfección en muestras clínicas probadas por ddPCR dúplex para BCoV y BRV; azul: gotitas positivas para el BRV; verde: gotitas positivas para BCoV; rojo: gotas dobles positivas BRV y BCoV. (B,C) Resultados de pruebas clínicas mediante ddPCR multiplex para BCoV, BEV y BRV. (B) Resultados de pruebas de 68 muestras recolectadas en Bole, Xinjiang (China). (C) Resultados de pruebas de 93 muestras recogidas en el condado de Ürümqi, Xinjiang (China). Rojo: gotitas positivas para BCoV; amarillo: gotitas positivas para el BRV; púrpura: gotitas positivas para BEV; naranja: gotitas dobles positivas BRV y BEV; azul: gotas de BCoV y BEV doblemente positivas; Gris: negativo. Las coordenadas horizontales y verticales son las amplitudes de fluorescencia de HEX y FAM, respectivamente. Amplitud del canal 1: FAM, respectivamente; amplitud del canal 2: HEX, respectivamente.
Tabla 2. Resultados comparativos de muestras clínicas de qPCR y doble ddPCR.
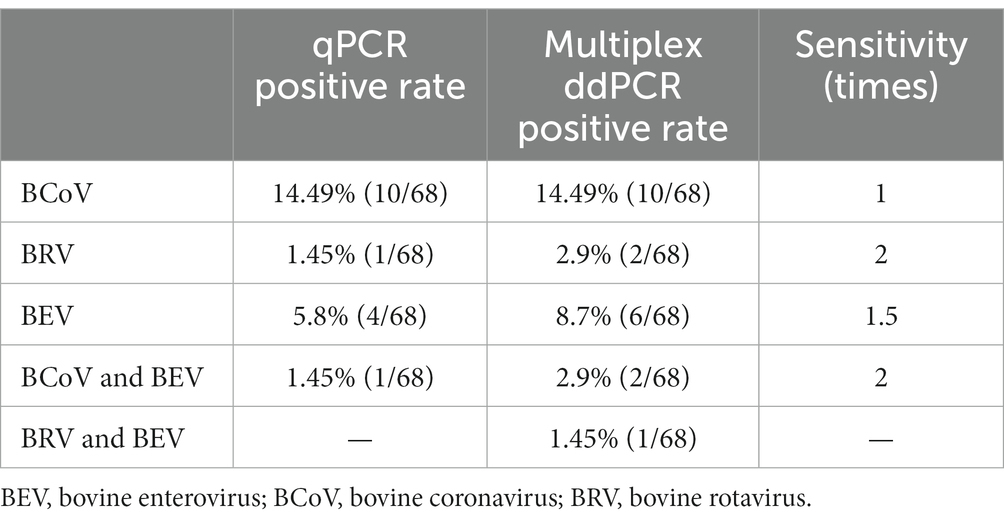
Los métodos establecidos de qPCR y ddPCR múltiple se utilizaron para analizar 68 hisopos anales recolectados de terneros diarreicos. Los resultados mostraron que las tasas de positividad de qPCR para BCoV, BRV y BEV fueron 14,49%, 1,45% y 5,80%, respectivamente, con una tasa de coinfección de 1,45% para BCoV y BEV. Las tasas de positividad de ddPCR multiplex para BCoV, BRV y BEV fueron 14,49%, 2,9% y 8,70%, respectivamente, con una tasa de coinfección de 2,9% para BCoV y BEV y 1,45% para BRV y BEV (Figura 6B). La ddPCR múltiple fue dos veces más sensible que la qPCR para la detección de BRV, 1,5 veces más sensible que la qPCR para detectar BEV y dos veces más sensible que qPCR para detectar coinfecciones con BCoV y BEV; La qPCR no detectó muestras con infecciones mixtas por BRV y BEV, mientras que la ddPCR múltiple detectó un caso (Tabla 3; Cuadro suplementario S9).
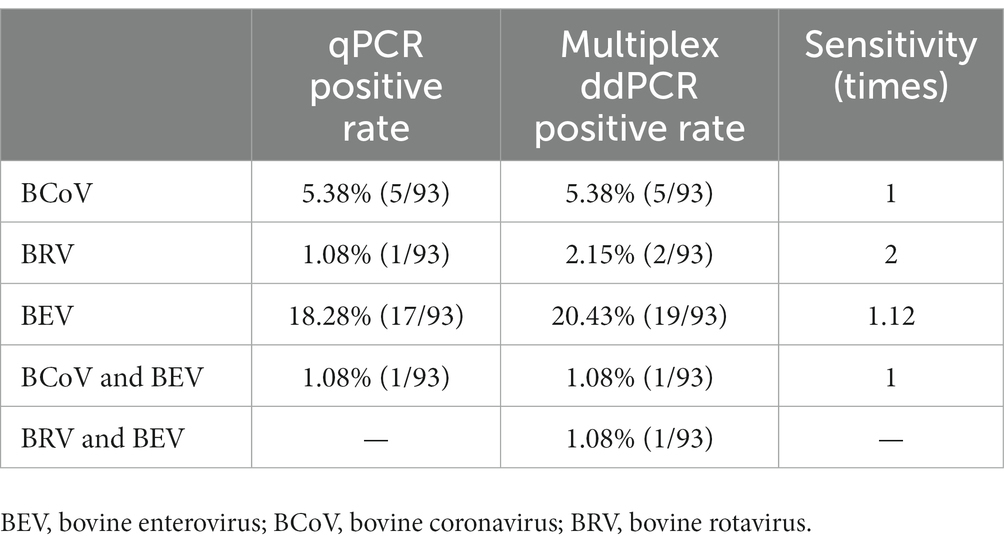
Se analizaron noventa y tres hisopos anales recolectados en el condado de Ürümqi. Los resultados mostraron que las tasas positivas de qPCR para BCoV, BRV y BEV fueron 5,38%, 1,08% y 18,28%, respectivamente; entre ellos, la tasa de coinfección de BCoV y BEV fue de 1,08%. Las tasas positivas de ddPCR multiplex para BCoV, BRV y BEV fueron 5,38%, 2,15% y 20,45%, respectivamente; la tasa de coinfección para BCoV y BEV fue de 1,08% y para BRV y BEV fue de 1,08% (Figura 6C). La ddPCR múltiple fue dos veces más sensible que la qPCR para detectar BRV y 1,12 veces más sensible que qPCR para detectar BEV. La qPCR no detectó muestras con infecciones mixtas por BRV y BEV, mientras que la ddPCR múltiple detectó un caso (Tabla 4; Cuadro suplementario S10).
4. Discusión
La diarrea de terneros es una enfermedad común y frecuente en la cría de ganado y se puede dividir en dos categorías principales según las causas: enfermedades infecciosas, como infecciones virales, bacterianas y parasitarias, y enfermedades no infecciosas, relacionadas, por ejemplo, con malestar ambiental, nutrición o manejo (20, 21). Entre estas causas, la diarrea causada por virus es la más peligrosa y difícil de prevenir. Los patógenos más comunes que causan diarrea viral en China son BEV, BCoV, BRV y el virus de la diarrea viral bovina (BVDV) (22, 23). Estas infecciones causan diarrea y una variedad de complicaciones en el cuerpo, lo que lleva a la muerte de terneros en casos graves (24, 25). Las infecciones por BEV, BCoV y BRV tienen un alto grado de similitud clínica, por lo que el diagnóstico de epidemias de diarrea viral no puede confirmarse solo con diagnóstico clínico (26), lo que requiere pruebas de laboratorio para determinar la etiología. El método más común para las pruebas de laboratorio de BEV, BCV y BEV es la PCR, pero la PCR tiene baja sensibilidad y poca especificidad, lo que no es propicio para la detección temprana y el tratamiento de la enfermedad. Por lo tanto, existe una necesidad urgente de un método de detección altamente sensible, específico, rápido y efectivo.
Uno de los métodos más comunes en el diagnóstico de biología molecular es el ensayo de PCR (27). Ji et al. (28) establecieron un ensayo cuádruple de RT-PCR de un solo paso para BVDV, BEV, BRV y BCoV, que es económico y eficiente, pero el análisis de los resultados de la prueba requiere el uso de gel, que es propenso a la contaminación con falsos positivos, tiene baja sensibilidad y no se puede cuantificar. Liu et al. (29) establecieron un ensayo cuádruple de fluorescencia qPCR en tiempo real para BAstV, BVDV-1, BCoV y BRV, que es rápido, eficiente y puede cuantificarse, pero se basa en valores de Ct, que se ven fácilmente afectados por la eficiencia de amplificación, y la construcción de la curva estándar debe calcularse en función del valor de Ct, lo que conduce a una disminución de la precisión (26, 30–32). El rango efectivo de valores de Ct está entre 15-35. Cuando la concentración de la plantilla es demasiado alta, el valor de Ct es inferior a 15, lo que hace que la amplificación no alcance el umbral de fluorescencia dentro del período de referencia, y cuando la concentración de la plantilla es demasiado baja, el valor de Ct es mayor que 35, lo que se considera sin sentido. Mediante el uso del ensayo qPCR establecido con optimización del gradiente de la temperatura de recocido y la concentración del cebador, el ensayo ddPCR para BEV, BCoV y BRV se construyó utilizando grupos fluorescentes FAM y HEX involucrados en la reacción. Debido a la ausencia de una cepa BRV, se utilizaron los plásmidos estándar construidos para el ensayo de especificidad. Los resultados mostraron que sólo los plásmidos positivos pueden producir señales fluorescentes, lo que indica una buena especificidad. en términos de reproducibilidad, los CV intra e interlotes para los ensayos de qPCR BEV, BCoV y BRV fueron inferiores al 3%; los CV para la ddPCR BEV fueron inferiores al 1,3%, 0,5 veces mayores que para la qPCR; los CV para BCoV ddPCR fueron inferiores al 5,1%, 2,13 veces mayores que para la qPCR; y el CV de BRV fue inferior a 1,33%, 2,13 veces mayor que el de qPCR. Esto demostró que los ensayos de qPCR y ddPCR establecidos para BEV, BCoV y BRV tenían buena reproducibilidad y estabilidad, siendo los ensayos de ddPCR para BEV y BRV los más estables. En términos de sensibilidad, los límites inferiores de los ensayos de qPCR establecidos fueron todos de 1.000 copias/μL, mientras que las concentraciones más bajas en los ensayos de ddPCR establecidos para BEV, BCoV y BRV fueron 2,7 copias/μL, 1 copia/μL y 2,4 copias/μL, respectivamente; estos valores son 370,37, 1.000 y 416,67 veces superiores a los de los ensayos de qPCR correspondientes. La prueba detectó una menor carga viral y redujo los falsos negativos. ddPCR también mostró una señal fluorescente negativa cuando la concentración de plásmidos era demasiado alta, lo que indica que ddPCR tiene un límite superior de detección. Una revisión de datos relacionados reveló (33) que los principios de ddPCR se basan en la distribución de Poisson, lo que significa que las concentraciones de plásmidos que exceden el límite superior de detección no pueden ser corregidas por el sistema. Esto resulta en la incapacidad de lograr una cuantificación precisa de ddPCR, por lo que una gota negativa debe estar presente en el sistema de reacción ddPCR. Las concentraciones en nuestras pruebas de plásmidos estuvieron entre 1 × 106–1 × 10−1 copias/μL, que no superaron el límite superior de detección.
Un estudio de Chang et al. (34) reveló que la coinfección con BRV y BCoV es más grave, por lo que era imperativo un método que pueda detectar BCoV y BRV simultáneamente. Se estableció un ensayo dual de ddPCR para BCoV y BRV basado en la ddPCR anterior. Las interacciones entre cebadores y sondas en ddPCR dual son más complejas en comparación con el sistema de detección de ddPCR simple, y la calidad del diseño del cebador y la sonda es la clave para la implementación exitosa de ddPCR dúplex. En referencia al ensayo de ddPCR multiplex del SARS-CoV-2 establecido por Nyaruaba et al. (18), la relación de sondas FAM y HEX en la ddPCR dúplex fue de 1:1. Los resultados de la prueba mostraron la presencia de gotitas dobles positivas, lo que indica el establecimiento exitoso del método. El método de qPCR establecido y el método de ddPCR dúplex se utilizaron para analizar el lote de 82 muestras de diarrea de terneros, y la qPCR mostró que la tasa de detección positiva fue del 10,98% para BCoV y del 12,2% para BRV. La ddPCR dúplex mostró que la tasa de positividad fue del 18,29% para BCoV, 7,31% mayor que la qPCR y 14,63% para BRV, 2,43% mayor que qPCR, con una tasa de coinfección del 6,1%, 2,44% más alta que la qPCR. Esto demostró que el ensayo de ddPCR dúplex no solo fue capaz de detectar muestras positivas, sino que también fue más sensible que la qPCR.
El ensayo multiplex ddPCR para BEV, BCoV y BRV se desarrolló y optimizó aún más para lograr una temperatura de recocido de 58 ° C, utilizando una concentración de cebador de 500 nM y concentraciones de sonda de 200 nM para BCoV FAM, 100 nM para BRV FAM, 100 nM para HEX y 200 nM para BEV HEX. Las 68 muestras restantes se analizaron mediante qPCR y la ddPCR multiplex establecida. La qPCR mostró tasas de positividad de 14,49% para BCoV, 1,45% para BRV y 5,80% para BEV, con una tasa de coinfección de 1,45% entre BCoV y BEV. La ddPCR múltiple mostró tasas positivas de 14,49% para BCoV, 1,45% para BRV y 8,70% para BEV, con una tasa de coinfección de 2,9% para BCoV y BEV y 1,45% para BRV y BEV; La qPCR no detectó muestras con coinfección por BRV y BEV, mientras que la ddPCR multiplex mostró una tasa de positividad del 1,45%. En otro campo, los ensayos de qPCR mostraron tasas positivas de 5,38%, 1,08% y 18,28% para BCoV, BRV y BEV, respectivamente; con una tasa de coinfección del 1,08% para BCoV y BEV. La ddPCR múltiple mostró tasas positivas de 5,38%, 2,15% y 20,45% para BCoV, BRV y BEV, respectivamente; la tasa de coinfección fue del 1,08% para BCoV y del 1,08% para BRV y BEV, donde qPCR no detectó muestras con infección mixta de BRV y BEV, mientras que la ddPCR multiplex detectó un caso.
5. Conclusión
Este estudio muestra cómo desarrollar una prueba de ddPCR multiplex optimizando las condiciones de detección para un solo sistema ddPCR. Se determinó que este era un método específico, sensible y conveniente para la detección simultánea de BEV, BCoV y BRV. Fue capaz de reducir el número de resultados falsos positivos y falsos negativos durante las pruebas y contribuyó al desarrollo de mejores métodos de diagnóstico.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio se incluyen en el artículo / material complementario, las consultas adicionales pueden dirigirse al autor correspondiente.
Declaración ética
Las muestras para este estudio se recolectaron con el consentimiento de los dueños de los animales, y no se manejaron animales vivos en el estudio, por lo que no se requirió aprobación ética para este estudio.
Contribuciones del autor
QF, DL y CZ contribuyeron para la concepción y el diseño del estudio. DL y YX organizaron la base de datos. YY y CZ realizaron el muestreo y procesamiento de los materiales estudiados. SM, XL y ZL realizaron el análisis estadístico. JC escribió el primer borrador del manuscrito. HS es el tutor principal del estudio y revisó los resultados obtenidos y los manuscritos. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Este estudio ha contado con el apoyo del Proyecto Fondo Juventud Sobresaliente de la Comunidad Autónoma (número de subvención: 2022D01E15); Gran Proyecto de Ciencia y Tecnología de la Región Autónoma (número de subvención: 2020A01001-2); Fundación Nacional de Ciencias Naturales de China (número de subvención: 32260881); Proyecto Nacional de Innovación Estudiantil (número de subvención: 202110758005), Proyecto de Innovación de Postgrado de la Comunidad Autónoma (número de subvención: XJ2022G131).
Reconocimientos
Un agradecimiento especial a Gang Yao y Yawei Sun por su ayuda en el muestreo.
Conflicto de intereses
DL fue empleado por Tecon Biology Co., Ltd.
Los autores restantes declaran que la investigación se realizó en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1157900/full#supplementary-material
Referencias
1. Beato, MS, Marcacci, M, Schiavon, E, Bertocchi, L, Di Domenico, M, Peserico, A, et al. Identificación y caracterización genética de enterovirus bovino mediante combinación de dos plataformas de secuenciación de última generación. J Virol Métodos. (2018) 260:21–5. doi: 10.1016/j.jviromet.2018.07.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Suzuki, T, Otake, Y, Uchimoto, S, Hasebe, A, and Goto, Y. Caracterización genómica y clasificación filogenética de coronavirus bovinos mediante análisis de secuencia del genoma completo. Virus. (2020) 12:183. DOI: 10.3390/v12020183
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Liu, YJ, Li, X, Zhang, Q, Zhao, YN, Jia, YQ, Liu, HB, et al. Variación genética de aislados de BEV en diferentes áreas de la provincia de Jilin. Chin J Vet Sci. (2018) 38:59–63. doi: 10.16303/j.cnki.1005-4545.2018.01.08
4. Zhu, L, Xing, Z, Gai, X, Li, S, San, Z, y Wang, X. Identificación de un nuevo enterovirus E aislado de HY12 de ganado con enfermedades respiratorias y entéricas graves. PLoS One. (2014) 9:E97730. doi: 10.1371/journal.pone.0097730
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Kapil, S, Pomeroy, KA, Goyal, SM y Trent, AM. Infección experimental con un aislado neumoentérico virulento del coronavirus bovino. J Vet Diagn Investig. (1991) 3:88–9. DOI: 10.1177/104063879100300123
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Donato, CM, y Bines, JE. Rotavirus y vacunas contra rotavirus. Patógenos. (2021) 10:959. doi: 10.3390/patógenos10080959
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Bai, XQ, and Wei, SC. Establishment of bovine rotavirus animal model and development of corresponding vaccine. J North Minzu Univ. (2020) 41:32–7. doi: 10.14084/j.cnki.cn62-1188/n.2020.04.007
8. Cao, C, Yu, DB, Xu, XJ, Yu, L, and Chang, JT. Isolation, identification and complete gene sequence analysis of a bovine rotavirus G6P[5] strain. Chin J Prev Vet Med. (2021) 43:939–45. doi: 10.3969/j.issn.1008-0589.202010011
9. Kumar, N, Malik, YS, Sharma, K, Dhama, K, Ghosh, S, Bányai, K, et al. Caracterización molecular de cepas inusuales de rotavirus bovino a que tienen alta relación genética con el rotavirus humano: evidencia de transmisión zooantroponótica. J Vet Med B. (2018) 65:431–42. doi: 10.1111/zph.12452
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Ji, C, Zhang, Y, Sun, R, Pan, Z, Ma, J y Yao, H. Aislamiento e identificación de dos cepas clínicas del nuevo genotipo enterovirus E5 en China. Microbiol Spectr. (2022) 10:E02662–21. doi: 10.1128/spectrum.02662-21
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Keha, A, Xue, L, Yan, S, Yue, H, and Tang, C. Prevalence of a novel bovine coronavirus strain with a recombinant hemagglutininin/esterase gene in dairy terners in China. Transbound Emerg Dis. (2019) 66:1971–81. doi: 10.1111/tbed.13228
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Qin, YF, Gong, QL, Zhang, M, Sun, ZY, Wang, W, Wei, XY, et al. Prevalencia del rotavirus bovino entre los bóvidos en China durante 1984-2021: una revisión sistemática y metanálisis. Microb Pathog. (2022) 169:105661. doi: 10.1016/j.micpath.2022.105661
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Mullis, KB, y Faloona, FA. Síntesis específica de ADN in vitro a través de una reacción en cadena catalizada por polimerasa. Métodos Enzymol. (1987) 155:335–50. DOI: 10.1016/0076-6879(87)55023-6
14. Higuchi, R, Fockler, C, Dollinger, G, and Watson, R. Kinetic PCR analysis: real-time monitoring of DNA amplification reactions. Biotecnología. (1993) 11:1026–30. DOI: 10.1038/NBT0993-1026
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Taylor, S, Wakem, M, Dijkman, G, Alsarraj, M, y Nguyen, M. Un enfoque práctico para la RT-qPCR: publicar datos que se ajusten a las directrices de MIQE. Métodos. (2010) 50:S1–5. doi: 10.1016/j.ymeth.2010.01.005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Vogelstein, B, y Kinzler, KW. PCR digital. Proc Natl Acad Sci U S A. (1999) 96:9236–41. DOI: 10.1073/PNAS.96.16.9236
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Guo, HW, Zhao, XY, Li, HW, Zhang, Z, Ma, H, and Zheng, M. Progress on application of digital PCR in animal disease diagnosis. Prog Vet Med. (2021) 42:102–6. doi: 10.16437/j.cnki.1007-5038.2021.02.019
18. Nyaruaba, R, Li, C, Mwaliko, C, Mwau, M, Odiwuor, N, Muturi, E, et al. Desarrollo de ensayos ddPCR multiplex para la detección de SARS-CoV-2 basados en mezcla de sondas y multiplexación basada en amplitud. Experto Rev Mol Diagn. (2021) 21:119–29. doi: 10.1080/14737159.2021.1865807
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Alcaide, M, Cheung, M, Hillman, J, Rassekh, S, Deyell, RJ, Batist, G, et al. Evaluación de la cantidad, calidad y distribución de tamaño del ADN libre de células mediante PCR digital de gotas múltiples. Sci Rep. (2020) 10:12564–10. DOI: 10.1038/S41598-020-69432-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Cho, YI, y Yoon, KJ. Una visión general de la diarrea de ternera: etiología, diagnóstico e intervención infecciosa. J Vet Sci. (2014) 15:1–17. doi: 10.4142/jvs.2014.15.1.1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Wilson, DJ, Habing, G, Winder, CB y Renaud, DL. Una revisión de alcance de las definiciones de casos de diarrea neonatal de terneros. Prev Vet Med. (2022) 211:105818. doi: 10.1016/j.prevetmed.2022.105818
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Cho, YI, Han, JI, Wang, C, Cooper, V, Schwartz, K, Engelken, T, et al. Estudio de casos y controles de etiología microbiológica asociada a diarrea de terneros. Veterinario Microbiol. (2013) 166:375–85. doi: 10.1016/j.vetmic.2013.07.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Sobhy, NM, Mor, SK, Mohammed, MEM, Bastawecy, IM, Fakhry, HM, Youssef, CRB, et al. Aislamiento y caracterización molecular de enterovirus bovinos en Egipto. Veterinario J. (2015) 206:317–21. doi: 10.1016/j.tvjl.2015.10.011
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Baker, JC. Las manifestaciones clínicas de la infección por diarrea viral bovina. Veterinario Clin N am Food Anim Pract. (1995) 11:425–45. doi: 10.1016/s0749-0720(15)30460-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Smith, DR. Investigación diagnóstica de la enfermedad de campo de la diarrea neonatal de terneros. Veterinario Clin North Am Food Anim Pract. (2012) 28:465–81. doi: 10.1016/j.cvfa.2012.07.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Schroeder, ME, Bounpheng, MA, Rodgers, S, Baker, RJ, Black, W, Naikare, H, et al. Desarrollo y evaluación del rendimiento del flujo de trabajo de purificación y detección de ácidos nucleicos patógenos de diarrea de terneros. J Vet Diagn Investig. (2012) 24:945–53. doi: 10.1177/1040638712456976
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Hou, QL, Wang, HM, y He, HB. Establecimiento y aplicación de una RT-PCR múltiple para la detección de BVDV, BCoV y BEV. Chin J Vet Sci. (2016) 36:1672–5. doi: 10.16303/j.cnki.1005-4545.2016.10.04
28. Ji, B, Peng, YN, Ye, Z, Wang, SS, y Wang, ZY. Establecimiento y aplicación del método cuádruple de RT-PCR de un paso para la identificación del virus de la diarrea viral bovina, el enterovirus bovino, el rotavirus bovino y el coronavirus bovino. Chin J Anim Infect Dis. (2021):1–8. doi: 10.19958/j.cnki.cn31-2031/s.20210525.001
29. Liu, MY, Wang, ZH, Wu, H, Gu, Y y Wu, WX. El desarrollo de la RT-PCR cuádruple en tiempo real para los asztrovirus bovinos, el virus de la diarrea del virus bovino 1, el coronavirus bovino y el rotavirus bovino. Acta Vet Zootech Sin. (2021) 52:1942–52. doi: 10.11843/j.issn.0366-6964.2021.07.016
30. Adams, G. Una guía para principiantes sobre RT-PCR, qPCR y RT-qPCR. Bioquímico. (2020) 42:48–53. doi: 10.1042/BIO20200034
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Barua, SR, Islam, S, Siddiki, АZ, Masuduzzaman, M, Hossain, MA, and Chowdhury, S. Comparison of diagnostic tests for detection of bovine rotavirus a in ternf feces. Maced Vet Rev. (2021) 44:37–45. DOI: 10.2478/Macvetrev-2020-0033
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Wang, C, Ding, Q, Plant, P, Basheer, M, Yang, C, Tawedrous, E, et al. La PCR digital de gotitas mejora la detección de miARN exosomal urinario en comparación con la PCR en tiempo real. Clin Biochem. (2019) 67:54–9. doi: 10.1016/j.clinbiochem.2019.03.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Whale, AS, Huggett, JF, and Tzonev, S. Fundamentals of multiplexing with digital PCR. Biomol Detectar Quantif. (2016) 10:15–23. doi: 10.1016/j.bdq.2016.05.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: enterovirus bovino, coronavirus bovino, rotavirus bovino, PCR digital de gotitas, PCR cuantitativa en tiempo real
Cita: Chen J, Li D, Xu Y, Li Z, Ma S, Liu X, Yuan Y, Zhang C, Fu Q y Shi H (2023) Establecimiento y aplicación del ensayo de reacción en cadena de la polimerasa digital de gotas múltiples para enterovirus bovino, coronavirus bovino y rotavirus bovino. Frente. Vet. Sci. 10:1157900. doi: 10.3389/fvets.2023.1157900
Recibido: 03 Febrero 2023; Aprobado: 30 de agosto de 2023;
Publicado: 12 septiembre 2023.
Editado por:
Inmaculada Martín-Burriel, Universidad de Zaragoza, España
Revisado por:
John Dustin Loy, Universidad de Nebraska-Lincoln, Estados Unidos
Minakshi Prasad, Lala Lajpat Rai Universidad de Ciencias Veterinarias y Animales, India
Derechos de autor © 2023 Chen, Li, Xu, Li, Ma, Liu, Yuan, Zhang, Fu y Shi. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Qiang Fu, 466183013@qq.com; Huijun Shi, shihuijunmm@163.com
†Estos autores comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía