Infección por Brucella suis biovar 1 en un perro con orquitis
Infección por Brucella suis biovar 1 en un perro con orquitis en Alemania
 Sofía Aurich1*†
Sofía Aurich1*†  Juliane Schneider2†
Juliane Schneider2†  Hanka Brangsch3
Hanka Brangsch3  Ad Kots4
Ad Kots4  Falk Melzer3
Falk Melzer3  Christa Ewers1
Christa Ewers1  Ellen Prenger-Berninghoff1
Ellen Prenger-Berninghoff1- 1Departamento de Ciencias Veterinarias, Instituto de Higiene y Enfermedades Infecciosas de los Animales, Universidad Justus Liebig, Giessen, Alemania
- 2Tierärztliche Klinik für Kleintiere am Kaiserberg, Duisburg, Alemania
- 3Instituto de Infecciones Bacterianas y Zoonosis, Instituto Friedrich-Loeffler, Instituto Federal de Investigación de Sanidad Animal, Jena, Alemania
- 4Departamento de Bacteriología, Interacción huésped-patógeno y diagnóstico, Investigación Bioveterinaria de Wageningen, Lelystad, Países Bajos
En 2021, un caso de brucelosis canina diagnosticada en un perro con orquitis se presentó a una práctica veterinaria en Alemania. Las pruebas serológicas excluyeron Brucella (B.) canis como agente causal, pero el análisis molecular reveló la presencia de B. suis biovar 1. Dado que biovar 1 no es endémico en Europa y el perro no tenía antecedentes de viajes a áreas endémicas, se realizó una investigación epidemiológica exhaustiva utilizando datos de secuencia del genoma completo para determinar la fuente de infección. Describimos el progreso clínico del animal y la posible infección de un empleado de una clínica veterinaria. Los hallazgos resaltan la importancia de considerar especies menos comunes de Brucella como posibles causas de brucelosis canina. Los datos también enfatizan que es bastante difícil identificar especies de Brucella en un laboratorio de diagnóstico de rutina y realizar investigaciones epidemiológicas para revelar posibles rutas de transmisión.
1. Introducción
La brucelosis se manifiesta principalmente en el sistema reproductivo de varios mamíferos terrestres y acuáticos. Se ha asociado con el aborto y otros trastornos reproductivos y crónicos, lo que ha causado pérdidas económicas significativas y representa una amenaza para la salud humana debido a su potencial zoonótico (1). Es causada por el género Brucella, un cocoide, gramnegativo, aerobio a microaerofílico varilla que es de crecimiento lento y requiere nutrientes complejos para el cultivo (2). De las seis especies clásicas de Brucella, a saber, B. melitensis (ovejas y cabras), B. abortus (bovino), B. suis (cerdos, liebres y renos), B. canis (perros), B. ovis (ovejas) y B. neotomae (roedores), solo las cuatro primeras son patógenos zoonóticos terrestres relevantes bien conocidos (3-5). Para los humanos, B. melitensis se considera la especie más patógena, seguida de B. suis, B. abortus y B. canis (5-10).
La brucelosis canina es más comúnmente causada por B. canis y se nota principalmente por infertilidad y fracaso reproductivo en perreras de cría de perros (11, 12). Tiene una alta afinidad por los testículos, los epidídimos, la próstata y el útero, pero también coloniza los órganos linfáticos, los ojos y la columna vertebral. Los signos clínicos típicos en las mujeres son infertilidad en forma de fracaso de la concepción, reabsorción embrionaria, aborto tardío y flujo vaginal después del aborto o el parto; En los hombres, se puede notar epididimitis, orquitis o prostatitis. La uveítis, la discospondilitis y la meningitis pueden presentarse como síntomas independientes del tracto reproductivo o como efectos a largo plazo (12-15). La principal vía de infección es la transmisión sexual (11, 16). Un estudio sobre la incidencia de B. canis en Europa entre 2011 y 2016 detectó un 5,4% de animales serológicamente positivos. Sin embargo, solo se analizaron muestras de perros con sospecha de infecciones por B. canis. Por lo tanto, la incidencia real de brucelosis canina debido a B. canis en Europa sigue siendo desconocida (17).
Además de B. canis, los agentes causales de la brucelosis canina también se han identificado esporádicamente como B. suis, B. abortus y B. melitensis (18). En cuanto a los cinco biovares de B. suis, los biovars (bv) 1-3 son la causa predominante de brucelosis porcina y se manifiestan en los tejidos reproductivos, causando trastornos reproductivos e infertilidad (19). Bv 1 exhibe el mayor potencial zoonótico y virulencia con respecto a humanos y cerdos (8, 20): En regiones donde B. suis bv 1 es endémica, como Australia o los Estados Unidos, se ha documentado la infección de humanos y perros asociada con la caza o alimentación de carne cruda de cerdos salvajes / cerdos salvajes (21-27). Mientras que B. suis bv 1 es más prevalente en el sur de Asia, América del Norte y del Sur, y Oceanía, B. suis bv 2 está presente en las poblaciones europeas de liebre marrón en Europa y se encuentra con frecuencia en jabalíes y cerdos domésticos (28, 29). A diferencia de bv 1, bv 2 rara vez infecta a los humanos (30).1
En Europa, hay pocos informes de B. infecciones por suis bv 1 en humanos (20, 31–34); en perros, se notificó infección con B. suis bv 1 en Berlín, Alemania, en 1978 y en los Países Bajos en 2016, ambas presumiblemente causadas por el consumo de carne cruda (32, 33).
El objetivo de este informe es proporcionar una descripción exhaustiva de la presentación clínica, las características microbiológicas y el análisis molecular de un caso de brucelosis canina causada por B. suis bv 1 en Alemania, incluidas las investigaciones de la fuente por tipificación molecular.
2. Métodos
2.1. Historia clínica
En julio de 2021, un macho de 2 años intacto de Rhodesian Ridgeback fue presentado a una práctica veterinaria de atención primaria en Renania del Norte-Westfalia por apatía e inapetencia. El perro mostró cojera, renuencia a acostarse y testículos hinchados dolorosos bilaterales. Sospechando una orquitis infecciosa, el perro fue tratado con amoxicilina/ácido clavulánico (12,5 mg/kg dos veces al día). Después de 2 días de terapia con antibióticos, el perro fue castrado porque los signos clínicos no mejoraron. En la cirugía, se observó exudación purulenta de los testículos y un cordón espermático engrosado. El tratamiento antibiótico se modificó mediante la administración de amoxicilina/ácido clavulánico (20 mg/kg dos veces al día) y marbofloxacino (2 mg/kg al día).
Cuatro días después de que el perro fue castrado, fue remitido a una clínica veterinaria debido a fiebre, vómitos y pérdida de peso. La ecografía abdominal reveló ascitis y una próstata agrandada y no homogénea. El análisis de sangre resultó en leucocitosis (28/μl), neutrofilia (21,56/μl), monocitosis (4,38/μl), hipoalbuminemia (2,2 g/dl), hipocloremia (105 mg/dl) y un alto nivel de proteína C reactiva (0,85 mg/dl). El análisis de orina mostró altos niveles de leucocitos, eritrocitos, proteínas (30 mg / dl), alta gravedad específica (1050) y presumiblemente la presencia de bacterias cocoides. Se reveló que el líquido ascítico era un exudado séptico con una gravedad específica de 1.026, proteína total de 30 g/dl, recuento celular de 107.200 células/μl, presencia de bacterias cocoides intracelulares fagocitadas, células mesoteliales activadas y neutrófilos degenerados. Durante la siguiente laparotomía diagnóstica al día siguiente, se recogieron 2 L de exudado pardusco, se observó peritonitis en toda la cavidad abdominal y se encontraron múltiples abscesos en el cordón espermático izquierdo. Posteriormente, se resecó el cordón espermático izquierdo y se realizó un lavado abdominal. Después de la intervención quirúrgica, se estableció un diagnóstico tentativo de brucelosis y el perro fue colocado en la sala de aislamiento. El tratamiento antibiótico se ajustó a trimetoprima/sulfametoxazol (5 mg/kg al día) y doxiciclina (10 mg/kg al día). Los síntomas clínicos mejoraron y el perro tuvo una recuperación completa en 7 días.
2.2. Muestreo
Durante la castración en la práctica veterinaria de atención primaria, se recolectó exudado purulento de los testículos y el cordón espermático para su examen microbiológico. Esta muestra fue enviada al Instituto de Higiene y Enfermedades Infecciosas de los Animales de la Universidad Justus Liebig en Giessen, Alemania (IHIT). Además, durante una laparotomía posterior realizada en la clínica veterinaria, se recogió líquido ascítico y material de absceso. Estas muestras fueron enviadas a un laboratorio de diagnóstico externo para su análisis microbiológico. Las muestras de sangre tomadas durante la laparotomía se enviaron al IHIT para realizar pruebas serológicas de B. canis.
Para determinar la fuente de infección, el departamento veterinario local obtuvo muestras de la alimentación de los perros proporcionada por los propietarios, una dieta de carne cruda disponible comercialmente que consiste en carne de res, caballo, salmón (origen desconocido) y canguro procedente de granjas australianas. En el momento de la investigación sólo se disponía de carne de salmón y canguro y posteriormente se sometieron a pruebas para detectar especies de Brucella en el Instituto de Análisis Químico y Veterinario correspondiente.
2.3. Investigación microbiológica
Para el análisis microbiológico inicial, la muestra se rayó en agar nutriente estándar (Oxoid, Wesel, Alemania) que contenía un 5% de sangre de oveja defibrinada (agar sangre) y en agar lactosa amarillo metacromo azul agua, según Gassner (Sifin Diagnostics GmbH, Berlín, Alemania). Las placas se incubaron a 37°C durante 48 h en aire ambiente. Para el enriquecimiento bacteriano, la muestra se cultivó durante 24 h a 37 °C en caldo nutriente estándar I (E. Merck KG, Darmstadt, Alemania) y se rayó en agar sangre de oveja al 5% y agar Gassner. Para la detección de bacterias microaerofílicas, la muestra se rayó adicionalmente en agar de infusión cerebro-corazón (Oxoid) y se incubó durante 5 días en 10% de CO2 a 37°C. El agar Schaedler (Becton Dickinson GmbH, Heidelberg, Alemania) y el agar Zeissler (E. Merck KG) se incubaron durante 72 h a 37 °C en condiciones anaeróbicas en un frasco utilizando los sobres de gas AnaeroGen™ (Oxoid). Después de 48 h, el crecimiento abundante (>200) de colonias pequeñas, no hemolíticas y brillantes fue evidente en el agar sangre. También se observaron colonias similares en agar de infusión cerebro-corazón. Se realizó la identificación con espectrometría de masas de tiempo de vuelo de desorción láser asistida por matriz (MALDI-TOF-MS, Bruker Daltonics, Bremen, Alemania) utilizando la biblioteca de referencia MBT Compass estándar (versión 10.0.0.0) y la biblioteca relevante para la seguridad (SR).
Para su posterior identificación, el aislado se envió al laboratorio de referencia de la Organización Mundial de Sanidad Animal (WOAH) y al Laboratorio Nacional de Referencia (NRL) para la brucelosis en animales, el Instituto de Infecciones Bacterianas y Zoonosis del Friedrich-Loeffler-Institut, Jena, Alemania. La identificación de patógenos y la tipificación de biovares se realizaron mediante la llamada reacción en cadena de la polimerasa (PCR) de Bruce-Ladder y New Bruce-Ladder (35, 36).
La alimentación de los perros se analizó utilizando métodos de detección de cultivos y PCR múltiple en tiempo real de acuerdo con los protocolos publicados para la detección de brucelosis en bovinos, porcinos, ovinos y caprinos de la NRL (37).
2.4. Investigación serológica
Las pruebas serológicas para B. canis se realizaron con antígenos específicos de B. canis (antígeno interno) utilizando la prueba de aglutinación en tubo (TAT) en el IHIT. Además, se realizaron pruebas serológicas [prueba de aglutinación de diapositivas (SAT), prueba de fijación del complemento y prueba de Rosa de Bengala] con antígenos de B. abortus (IDEXX Montpellier SAS, Montpellier, Francia) de reacción cruzada para B. abortus, B. suis y B. melitensis en el NRL para brucelosis en animales.
2.5. Caracterización genómica
2.5.1. Aislamiento del ADN y secuenciación del genoma completo
El ADN se extrajo de cultivos puros utilizando el kit de preparación de plantillas de PCR de alta pureza (Roche Molecular Systems, Pleasanton, CA, Estados Unidos). Para la secuenciación de lectura corta mediante la tecnología Illumina, se preparó una biblioteca genómica utilizando el kit NexteraXT (Illumina Inc., San Diego, CA, Estados Unidos), que se secuenció en un MiSeq (Illumina Inc., San Diego, CA, Estados Unidos) en modo de extremo pareado. Para obtener un genoma cerrado, el ADN se secuenció adicionalmente mediante la tecnología Nanopore en un dispositivo MinION Mk1B (Oxford Nanopore Technologies Ltd., Oxford, Inglaterra). La biblioteca genómica correspondiente se preparó con el kit de secuenciación de ligadura (SQK-LSK109) y se le dio un código de barras utilizando el kit EXP-NBD 104 (Oxford Nanopore Technologies Ltd., Oxford, Inglaterra). Esta biblioteca se ejecutó en una celda de flujo R9.4.1 durante 24 h.
2.5.2. Montaje y anotación a novo
Se utilizaron lecturas de las tecnologías Illumina y Nanopore para el ensamblaje combinado del genoma de novo utilizando microPIPE (38) con basecalling en modo de súper precisión. Las estadísticas de calidad del ensamblaje se evaluaron utilizando QUAST versión 5.0.2 (39), y la anotación se realizó utilizando Bakta versión 1.6.1 con base de datos versión 4.0 (40).
2.5.3. Comparación del genoma y genotipado
Para determinar el origen del aislado, se examinaron las bases de datos del genoma Sequence Read Archive (SRA) y RefSeq del NCBI (a las que se accedió en enero de 2023) en busca de secuencias de B. suis bv 1 (Tabla suplementaria 1). Además, se buscó en MLVAbank [https://microbesgenotyping.i2bc.paris-saclay.fr/; consultado el 9 de febrero de 2023 (41)] para obtener múltiples perfiles de análisis de repeticiones en tándem (MLVA) con similitud con el aislado investigado que muestra un máximo de tres alelos de diferencia.
La identidad nucleótida promedio del genoma recién ensamblado en comparación con otras cepas de B. suis bv 1 depositadas en la base de datos NCBI RefSeq se evaluó utilizando fastANI versión 1.1 (42). Para excluir que el aislado fuera idéntico a la cepa S1 de la vacuna B. suis bv 2, se realizó una PCR in silico utilizando un script de Egon A. Ozer (versión 0.5.1) (https://github.com/egonozer/in_silico_pcr) con cebadores IclRP1 (5′-TGGCAAGAGCGGTTTCAG-3′) e IclRP2 (5′-TCCAAGGTCGGCTACGAA-3′) (43). In silico La AMML se llevó a cabo utilizando el MISTReSS (https://github.com/Papos92/MISTReSS) descrito por Sacchini et al. (44). Sobre la base de las diferencias en los alelos, se calculó un árbol de expansión mínimo utilizando GrapeTree versión 1.0 (45) con el algoritmo MSTreeV2 implementado. Además, se realizó la tipificación de secuencias multilocus del genoma central (cgMLST) utilizando Ridom Seqsphere+ versión 7.7 (46) y el esquema de Abdel-Glil et al. (47). Las cepas extranjeras para las que se han depositado exclusivamente lecturas crudas en NCBI se ensamblaron utilizando Shovill versión 1.0.4 (https://github.com/tseemann/shovill) con el ensamblador SPAdes y la opción «- – trim». Se utilizaron distancias alélicas CgMLST para el cálculo de un árbol de expansión mínimo como se implementó en Ridom Seqsphere+. La tipificación basada en polimorfismos de nucleótido único del genoma central (cgSNPs) se realizó utilizando Snippy versión 4.6.0 (https://github.com/tseemann/snippy) utilizando B. suis bv 1 cepa 1330 (GCF_000223195.1) como referencia. En este análisis, se incluyeron datos de SRA. La alineación cgSNP se utilizó para el cálculo de un árbol filogenético de máxima verosimilitud utilizando RAxML versión 8.2.12 (48) con el modelo GTRGAMMA. El árbol fue visualizado usando FigTree versión 1.4.3 (http://tree.bio.ed.ac.uk/soft-ware/figtree/).
3. Resultados
3.1. Análisis microbiológico
La identificación inicial del aislado, denominado 21RB23181, con MALDI-TOF-MS reveló Brucella melitensis, arrojando una puntuación de 2,39. Aunque la identificación de especies debe considerarse cuestionable debido al hecho de que la base de datos comercial solo proporciona espectros de referencia para B. melitensis, debe suponerse que la identificación del género es correcta. Por lo tanto, después de la diferenciación como Brucella sp., los resultados se enviaron inmediatamente al departamento veterinario local y la muestra se manejó de acuerdo con las medidas de protección oficiales (49, 50). El aislado fue probado por el NRL para la brucelosis en animales por PCR, que identificó 21RB23181 como una cepa de B. suis bv 1.
La muestra de líquido ascítico y material de absceso enviada a un laboratorio de diagnóstico externo no resultó en la detección de Brucella sp. Del mismo modo, las muestras restantes de carne de salmón y canguro obtenidas por el departamento veterinario local dieron negativo para Brucella sp.
3.2. Análisis serológico
La prueba serológica de una muestra de sangre para B. canis utilizando el ensayo TAT arrojó un resultado negativo (<40 UI/ml). Sin embargo, cuando se probó con antígenos específicos para el grupo B. melitensis, B. abortus y B. suis, la muestra mostró un resultado positivo (SAT 841 UI / ml, prueba de fijación del complemento 1,189 SensE / ml y positivo en la prueba de Rosa de Bengala).
3.3. Análisis genómico
3.3.1. Caracterización y similitud del genoma
Usando un enfoque de ensamblaje combinado de lecturas de Illumina y Nanopore, el genoma de 21RB23181 podría ensamblarse hasta completarse con una cobertura media de 271×. El genoma consistió en dos contigs circulares de 2.107.952 y 1.207.151 pb con un contenido promedio de GC del 57,25% y 3.113 secuencias codificantes predichas.
Los valores más altos de ANI se observaron para las cepas B. suis bv 1 Human/AR/US/1981 (99.9958% ANI) y VBI22 (99.9949% ANI), ambas aisladas en los Estados Unidos, y la cepa de la vacuna S2 (99.9946% ANI) aislada en China. In silico La PCR arrojó un resultado negativo para el par de cebadores específicos de S2, descartando así la posibilidad de que 21RB23181 fuera la cepa de la vacuna.
3.3.2. Genotipado mediante métodos basados en alelos
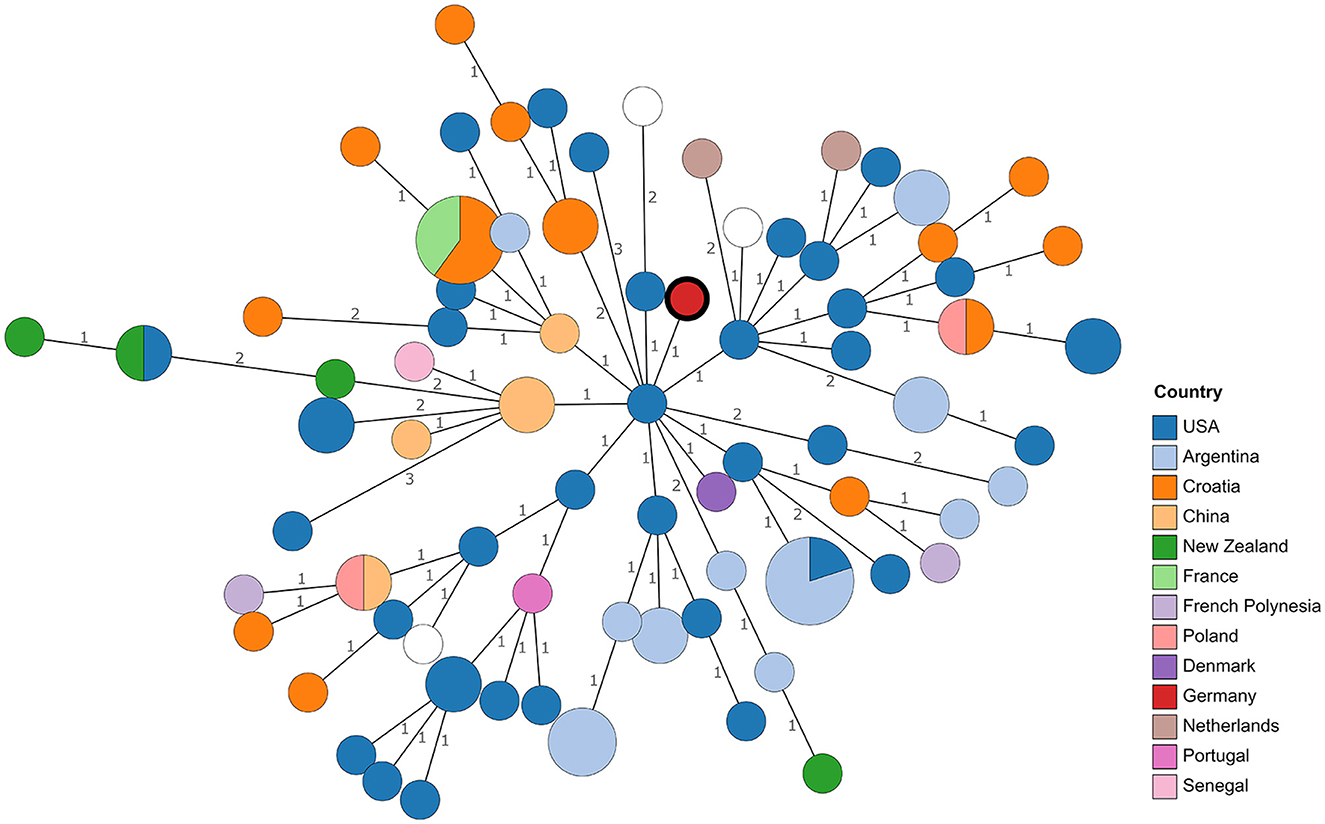
Aunque WGS ofrece la posibilidad de comparar cepas a nivel de nucleótidos, la falta de secuencias genómicas de aislados reportados requiere recurrir a la comparación de perfiles MLVA, a pesar de la menor resolución. Se incluyeron perfiles publicados en MLVAbank y en la literatura (ver Tabla Suplementaria 2). Sorprendentemente, en el árbol de expansión mínimo resultante (Figura 1), no se pudo observar ningún agrupamiento de cepas según su origen. Especialmente cepas de Croacia, Estados Unidos y Argentina se dispersaron en el árbol. Con la excepción de un alelo, el perfil MLVA de 21RB23181 coincidió con el de una cepa aislada en Dinamarca en 1987 (BCCN#87-85) y la cepa B93-0078, que se originó en el ganado bovino en los Estados Unidos en 1993. A pesar de la ubicación geográfica más cercana, la distancia a Brucella aislados de un perro (WBVR_2016) y una liebre en los Países Bajos (WBVR_2017) mostró mayores diferencias con cuatro alelos diferentes cada uno.
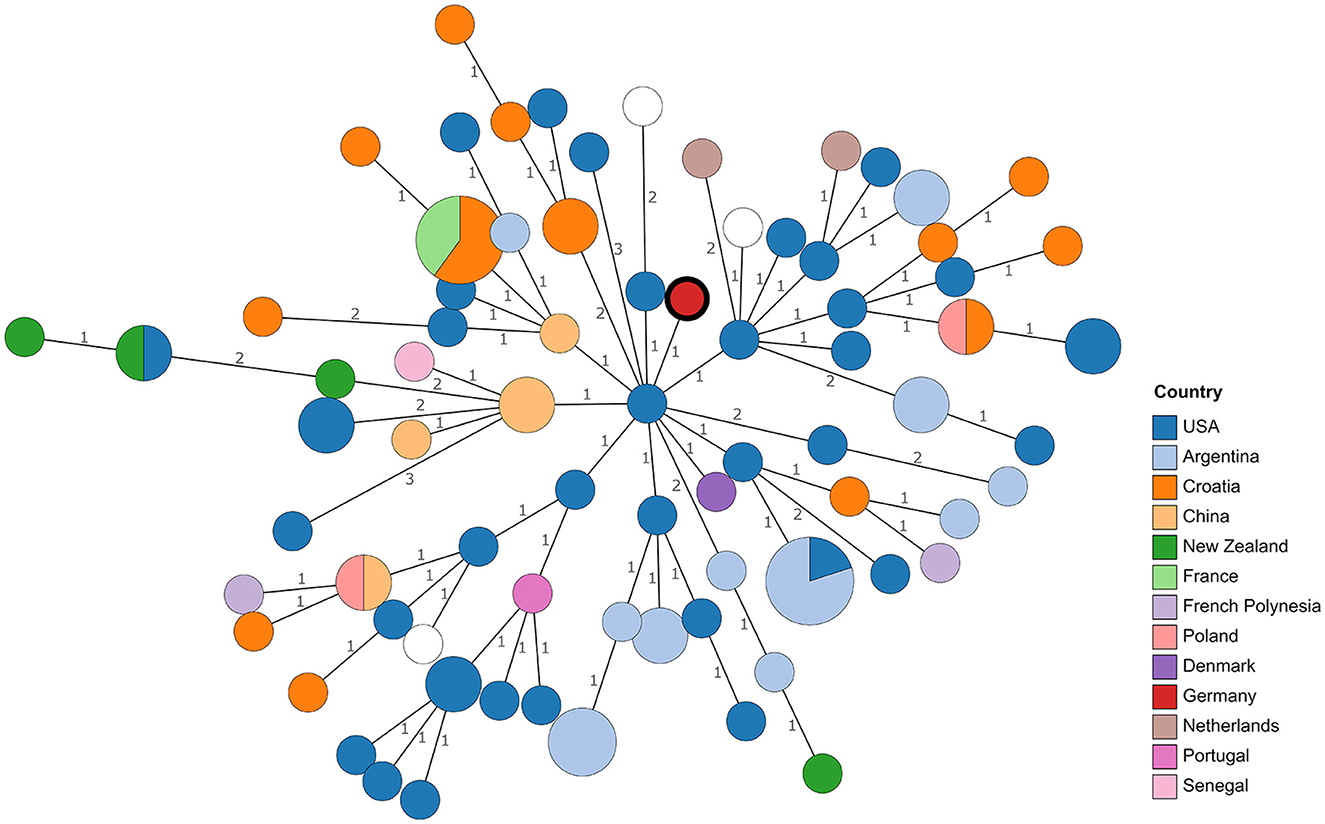 Figura 1. Árbol de expansión mínimo basado en las diferencias de perfil MLVA. Los números en las ramas indican diferencias alélicas. Las hojas están coloreadas según el origen de las cepas. La hoja que representa el aislado 21RB23181 tiene un margen en negrita. Para las hojas vacías, el origen es desconocido.
Figura 1. Árbol de expansión mínimo basado en las diferencias de perfil MLVA. Los números en las ramas indican diferencias alélicas. Las hojas están coloreadas según el origen de las cepas. La hoja que representa el aislado 21RB23181 tiene un margen en negrita. Para las hojas vacías, el origen es desconocido.
En el análisis cgMLST, 21RB23181 exhibió al menos 31 diferencias alélicas en comparación con otras cepas de B. suis bv 1 (Figura 2). La cepa VBI22 aislada del ganado en los Estados Unidos mostró el mayor grado de similitud. En este análisis, también se incluyó un aislado de B. suis bv 1 de un caso de brucelosis humana en Alemania en 2018 (Bw_180660). Sin embargo, mostró 45 diferencias alélicas con el aislado de perro alemán, por lo que no se pudo inferir ninguna conexión entre estos dos casos.
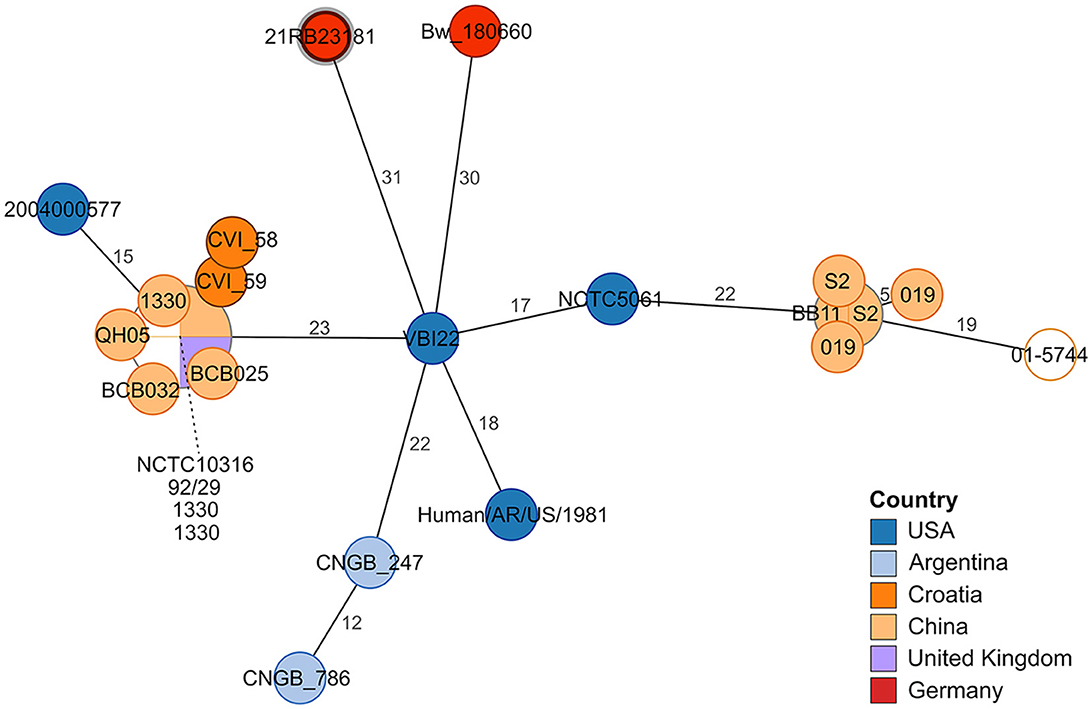 Figura 2. Árbol de expansión mínimo basado en distancias alélicas determinadas por análisis cgMLST. Los números en las ramas indican distancias alélicas. Las hojas están coloreadas según el origen de la cepa. La hoja que representa el aislado 21RB23181 tiene un margen en negrita. Para una mejor legibilidad, los nombres de las hojas agrupadas están conectados a las hojas por una línea discontinua. Para las hojas vacías, el origen es desconocido.
Figura 2. Árbol de expansión mínimo basado en distancias alélicas determinadas por análisis cgMLST. Los números en las ramas indican distancias alélicas. Las hojas están coloreadas según el origen de la cepa. La hoja que representa el aislado 21RB23181 tiene un margen en negrita. Para una mejor legibilidad, los nombres de las hojas agrupadas están conectados a las hojas por una línea discontinua. Para las hojas vacías, el origen es desconocido.
3.3.3. Genotipado mediante SNP
En un enfoque cgSNP (Figura 3), 21RB23181 se comparó con aislados de B. suis bv 1 para los cuales se disponía de datos de lectura sin procesar (Tabla suplementaria 1). En total, se llamaron 476 SNP del genoma central. El aislado 21RB23181 fue más similar a las cepas originarias de Tonga, con la identidad SNP más alta a una cepa aislada en 1979 de un humano en Tonga (69 SNP). Sin embargo, las diferencias entre las cepas de los Estados Unidos y Argentina fueron solo ligeramente mayores, oscilando entre 77 y 96 SNP. Sobre la base de este resultado, los genomas de las cepas de Tonga se ensamblaron utilizando los datos de secuenciación sin procesar, y se repitió el análisis cgMLST (Figura complementaria 1). Los perfiles alélicos de dos cepas de Tonga mostraron una mayor concordancia con 21RB23181 que la cepa estadounidense VBI22, difiriendo en 27 alelos, pero aún así, esta diferencia no es mucho menor que la de la cepa estadounidense estadounidense. Estas cepas de Tonga también están parcialmente representadas en el árbol MLVA; sin embargo, en la base de datos MLVAbank, su origen se da como Nueva Zelanda (B13-0234, B13-0236, B13-0237 y B13-0239). En el MLVA, 21RB23181 difiere en tres alelos de las cepas originarias de Tonga.
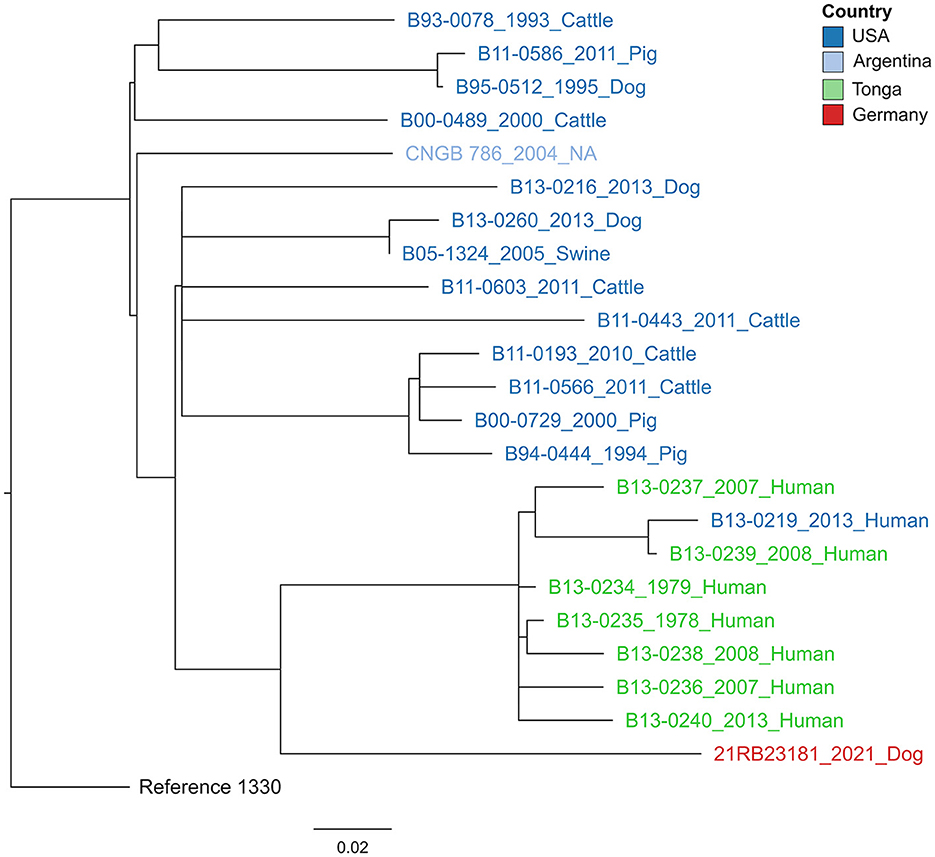 Figura 3. Árbol de máxima verosimilitud basado en la alineación del SNP del genoma central. El árbol está enraizado en la cepa de referencia B. suis bv 1 cepa 1330. Los colores de las etiquetas dan el origen de los aislados. El año de aislamiento y el huésped se dan después de los nombres de las cepas. La barra indica el número de sustituciones de base por sitio.
Figura 3. Árbol de máxima verosimilitud basado en la alineación del SNP del genoma central. El árbol está enraizado en la cepa de referencia B. suis bv 1 cepa 1330. Los colores de las etiquetas dan el origen de los aislados. El año de aislamiento y el huésped se dan después de los nombres de las cepas. La barra indica el número de sustituciones de base por sitio.
3.4. Medidas viables relativas a la posible transmisión
Inmediatamente después de la detección de Brucella sp., se evaluó el riesgo zoonótico para las prácticas veterinarias involucradas y el laboratorio microbiológico. Los empleados con mayor riesgo debido al contacto cercano con el animal o la manipulación de la muestra microbiológica y el cultivo puro antes de la identificación del aislado fueron informados y sometidos a control serológico. Un miembro del personal de la práctica veterinaria de atención primaria que había estado en contacto cercano con el animal mostró un valor de anticuerpos IgM ligeramente aumentado de 21 U/ml (rango de referencia, <15 U/ml) utilizando el ensayo inmunoabsorbente ligado a enzimas (ELISA) de Brucella IgM e IgG (Virion/Serion, Würzburg, Alemania) ~14 días después del primer contacto con el perro. Los valores de las pruebas de anticuerpos IgG y de inmunocaptura-aglutinación (bestbion dx GmbH, Colonia, Alemania) se mantuvieron dentro del rango normal. El empleado afectado también reportó síntomas como fatiga y sudores nocturnos que duraron aproximadamente una semana. Los síntomas mejoraron rápidamente mientras recibía doxiciclina (100 mg dos veces al día durante 3 meses). La serología de seguimiento 6 semanas después todavía mostró valores de IgM ligeramente elevados de 19 U/ml, pero ningún aumento en los valores de anticuerpos IgG. La prueba de inmunocaptura-aglutinación de Brucella resultó negativa nuevamente. Sin embargo, no se hicieron intentos de aislar la bacteria mediante cultivo directo. Hasta la fecha, 1,5 años después, el empleado sigue sin síntomas.
4. Discusión
Este informe ilustra la primera infección notificada por B. suis en un perro en Alemania desde 1978. El individuo infectado era un macho intacto de Rhodesian Ridgeback que sufría de orquitis con síntomas que incluían fiebre, hinchazón testicular y dolor, cordón espermático con absceso, líquido abdominal libre debido a peritonitis, anorexia y debilidad. El análisis microbiológico de un absceso testicular después de la castración reveló la presencia de Brucella sp., identificada por PCR como B. suis bv 1.
En áreas donde B. suis bv 1 es endémico en la vida silvestre, la infección canina con el patógeno no es infrecuente. Se ha informado que la incidencia de infección en perros aumenta en ciertas regiones de los Estados Unidos y Australia (51-53). En la mayoría de estos casos, la exposición a cerdos salvajes a través de la caza o el consumo de carne cruda de cerdo salvaje se asoció con la infección. En Europa, hasta la fecha solo se han notificado dos casos de infección por B. suis en perros: en 1978, se detectó B. suis bv 1 en dos perros machos en Berlín, Alemania, mostrando fiebre y orquitis / epididimitis. Dado que se mantuvieron solo en entornos urbanos y bv 1 no es endémico en Alemania, se sospechó que el alimento para perros (carne cruda de países de Europa del Este) era la fuente de infección (33). El segundo caso reportado fue un perro infectado con B. suis bv 1 en los Países Bajos en 2016. No tenía antecedentes de caza o viajes a B. suis bv 1 países endémicos, pero fue alimentado con carne de liebre cruda importada de Argentina. Mediante PCR, se detectó B. suis bv 1 en estas canales de liebre y se caracterizó in silico por MLVA y MLST. Tanto las muestras del perro como la carne mostraron una alta similitud en MLVA (32).
Desafortunadamente, en el caso presentado aquí, no fue posible determinar la fuente de infección. La paciente no era ni un perro de caza ni un perro de cría, aunque no se podía excluir el contacto con el fluido vaginal infectado o la orina de las perras. El perro, criado en Alemania, tenía un historial de viajes a los Países Bajos, pero nunca había estado en las regiones endémicas de B. suis bv 1. Dado que los casos de infección por B. suis bv 1 se asociaron con el consumo de carne cruda, esta vía de transmisión es probable pero incierta, especialmente porque Brucella sp. no pudo detectarse en la carne analizada. Este resultado está limitado por la incapacidad de analizar todos los tipos de carne y la incertidumbre con respecto a si la carne analizada era del mismo lote consumido durante el tiempo de la infección. Dado el período de incubación variable de la infección por Brucella, que puede variar de 2 semanas a varios meses, puede haber transcurrido algún tiempo desde la infección hasta la aparición de los síntomas (11).
La transmisión de B. suis de perros a humanos aún no se ha demostrado claramente. Un solo informe de los Estados Unidos sugiere una infección por B. suis en una mujer como resultado de manipular fetos caninos abortados sin guantes (54). Sin embargo, en el caso aquí informado, los resultados serológicos del funcionario no prueban en última instancia la infección. Un aumento en los anticuerpos IgM por sí solo puede indicar una reacción no específica y no necesariamente una infección. Los síntomas presentados por el empleado afectado eran característicos de una infección por Brucella, pero no eran específicos y, por lo tanto, no concluyentes. Desafortunadamente, no se ha obtenido evidencia cultural o directa por PCR, dejando la posibilidad de infección humana como especulativa pero notable, especialmente porque también se ha reportado un caso de infección por B. suis bv 1 en un humano sin ninguna respuesta serológica (55). En Europa, solo se describieron dos casos humanos de infección por B. suis bv 1 según el conocimiento de los autores: un trabajador español de una planta de tratamiento de residuos médicos se infectó con B. suis bv 1 en 2014, probablemente después de una punción accidental con una aguja contaminada (34). Presumiblemente como consecuencia del procesamiento privado de carne, un alemán se infectó con B. suis bv 1 en 2018 (20).
Las pruebas serológicas estándar para la sospecha de brucelosis canina solo detectarán B. canis, ya que no hay reacción cruzada serológica entre B. canis y B. suis ya que tienen diferentes características antigénicas. B. canis y B. ovis llevan un lipopolisacárido rugoso (LPS) sin el antígeno más externo, el O-polisacárido, mientras que todas las demás especies de Brucella son cepas lisas de Brucella con el O-polisacárido presente en el LPS (2, 56). Esto puede dar lugar a resultados falsos negativos para la infección con especies de Brucella distintas de B. canis, por lo que se prefiere el cultivo directo. Aún así, la identificación precisa de los aislamientos utilizando el método estándar de MALDI-TOF MS es un desafío, ya que solo los espectros de referencia para B. melitensis están disponibles en la base de datos. Dado que este organismo es altamente clonal y demanda métodos con alto potencial discriminatorio, la secuenciación del genoma completo se emplea ampliamente para la identificación y diferenciación de Brucella sp. (47, 57). Tres años antes del caso presentado, también se diagnosticó brucelosis en un perro en los Países Bajos, y se aisló B. suis bv 1 (32). Sin embargo, sobre la base de los resultados de MLVA, no se pudo establecer una conexión epidemiológica entre ambos casos. Del mismo modo, el aislamiento de un caso de brucelosis humana en Alemania en 2018 (20) difirió notablemente de 21RB23181. La determinación del origen geográfico de la fuente de infección en el caso presentado se ve obstaculizada por la falta de datos completos de secuenciación. En cuanto a Europa, solo se dispone de un número limitado de datos de B. suis bv 1 WGS, de los cuales 21RB23181 no mostraron una similitud notable. Con respecto a la distribución conocida de las cepas de B. suis bv 1, cabe esperar que la cepa no sea de origen endémico sino importada, tal vez de la región del Pacífico (por ejemplo, Polinesia), ya que la tipificación de SNP reveló una mayor similitud con las cepas de Tonga. El hecho de que MLVA pondría 21RB23181 más cerca de los aislados estadounidenses ya que la distancia alélica a las cepas de Tonga era comparativamente alta puede ser ignorado. Se demostró para B. melitensis que los resultados de MLVA pueden llevar a conclusiones falsas con respecto al origen de la cepa y que los métodos basados en WGS son más apropiados (57).
A pesar de su recuperación, el perro fue sacrificado después de ser diagnosticado con B. suis bv 1, dado el alto riesgo de transmisión zoonótica. Brucella se disemina a través del contacto directo, la ingestión o la aerosolización de fluidos corporales. Puede eliminarse intermitentemente hasta por 60 semanas y permanece persistente durante al menos 2 años después de la inoculación (12). Debido a su naturaleza intracelular y bacteriemia periódica, el tratamiento con antibióticos a menudo no tiene éxito. La terapia más prometedora implica una combinación de tetraciclinas (dosis altas de doxiciclina o minociclina durante 1-2 meses) y aminoglucósidos (estreptomicina o gentamicina durante las primeras 2 semanas). Sin embargo, las recaídas pueden ocurrir poco después de la interrupción del antibiótico. Dado el alto riesgo de transmisión zoonótica, la excreción prolongada y las malas opciones de tratamiento, se recomienda la eutanasia de los perros afectados (12).
A continuación, informamos de la detección del tercer caso de brucelosis canina causada por B. suis bv 1 en Europa. Se utilizó la secuenciación del genoma completo para determinar la relación filogenética del aislado con otras cepas, con el objetivo de rastrear el origen de la infección e identificar posibles rutas de transmisión. Sin embargo, debido a la limitada disponibilidad de datos de secuencia relevantes, no fue posible determinar claramente el origen de los aislados, pero se pudieron excluir las conexiones con otros casos europeos. Aunque la transmisión de perros a humanos no se ha demostrado claramente, B. suis bv 1 es un linaje altamente virulento que con frecuencia infecta a los humanos con síntomas leves a severos, lo que representa una amenaza para los dueños de perros y el personal veterinario. Los laboratorios deben ser conscientes de las dificultades en el cultivo y las pruebas serológicas que pueden resultar en un diagnóstico insuficiente de esta enfermedad.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio / repositorios y los números de acceso se pueden encontrar a continuación: https://www.ebi.ac.uk/ena, PRJEB60627.
Contribuciones del autor
EP-B, CE y FM supervisaron el proyecto. EP-B, CE y SA realizaron análisis microbiológicos. HB realizó la secuenciación del genoma y escribió secciones del manuscrito. AK contribuyó con datos del genoma de B. suis. HB y FM analizaron los datos de secuenciación. JS y SA redactaron la primera versión del manuscrito. Todos los autores contribuyeron a la revisión del manuscrito, leyeron y aprobaron la versión presentada.
Reconocimientos
Nos gustaría agradecer a los dueños de perros por la información detallada sobre la historia del perro; el personal de la consulta veterinaria para obtener información detallada sobre el progreso clínico del perro y para proporcionar datos de las pruebas serológicas; El Dr. Thomas Mönig, jefe del departamento veterinario local, y el Instituto Analítico Químico y Veterinario de Westfalia por la cooperación en las investigaciones de la alimentación para perros.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1233118/full#supplementary-material
Notas
1. ^https://www.efsa.europa.eu/de/efsajournal/pub/1144
Referencias
1. Figueiredo P de, Ficht TA, Rice-Ficht A, Rossetti CA, Adams LG. Patogénesis e inmunobiología de la brucelosis: revisión de las interacciones Brucella-huésped. Soy J Pathol. (2015) 185:1505–17. doi: 10.1016/j.ajpath.2015.03.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Corbel MJ, Menachem B. Género I. Brucella Meyer y Shaw 1920. En: Garrity GM, Bell JA, Lilburn T, editores. Clase I. Alphaproteobacteria class. nov: En: Brenner, D.J., Krieg, N.R., Staley, J.T. (eds) Bergey’s Manual of Systematic Bacteriology®. 2ª ed. Boston, MA: Springer (2005). págs. 370–86.
3. Glynn MK, Lynn TV. Brucelosis. J Am Vet Med Assoc. (2008) 233:900–8. doi: 10.2460/javma.233.6.900
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Yagupsky P, Morata P, Colmenero JD. Diagnóstico de laboratorio de la brucelosis humana. Clin Microbiol Rev. (2019) 33:3. doi: 10.1128/CMR.00073-19
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Young EJ, Corbel MJ. Brucelosis: aspectos clínicos y de laboratorio. Boca Raton, FL: Taylor & Francis Group (1989). Disponible en línea en: https://ebookcentral.proquest.com/lib/kxp/detail.action?docID=6269747 (consultado el 14 de abril de 2023).
6. Seleem MN, Boyle SM, Sriranganathan N. Brucelosis: una zoonosis reemergente. Veterinario Microbiol. (2010) 140:392–8. doi: 10.1016/j.vetmic.2009.06.021
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Dentinger CM, Jacob K, Lee LV, Mendez HA, Chotikanatis K, McDonough PL, et al. Infección humana por Brucella canis y exposiciones de laboratorio posteriores asociadas con un cachorro, Ciudad de Nueva York, 2012. Zoonosis Salud Pública. (2015) 62:407–14. doi: 10.1111/zph.12163
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Lucero NE, Ayala SM, Escobar GI, Jacob NR. Brucella aislada en humanos y animales en América Latina desde 1968 hasta 2006. Epidemiol Infect. (2008) 136:496–503. doi: 10.1017/S0950268807008795
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Lucero NE, Corazza R, Almuzara MN, Reynes E, Escobar GI, Boeri E, et al. Brote de Brucella canis humano relacionado con la infección en perros. Epidemiol Infect. (2010) 138:280–5. doi: 10.1017/S0950268809990525
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Lucero NE, Escobar GI, Ayala SM, Jacob N. Diagnosis of human brucellosis caused by Brucella canis. J Med Microbiol. (2005) 54 (Pt 5):457–61. DOI: 10.1099/JMM.0.45927-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Hollett RB. Brucelosis canina: brotes y cumplimiento. Teriogenología. (2006) 66:575–87. doi: 10.1016/j.theriogenology.2006.04.011
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Davidson PA, Skyes JE. Brucelosis canina. En: Sykes JE, editor. Enfermedades infecciosas de Greene del perro y el gato. 5ª ed. St. Louis: Elsevier Saunders (2022). págs. 876–92.
13. Makloski CL. Manejo de la brucelosis canina. Veterinario Clin N am Small Anim Pract. (2011) 41:1209–19. doi: 10.1016/j.cvsm.2011.08.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Wanke MM. Brucelosis canina. Anim Reprod Sci. (2004) 82–3:195–207. doi: 10.1016/j.anireprosci.2004.05.005
15. Hohmann M. Canine Brucellose – Ein Globalisierungsproblem? Deutsches Tierärzteblatt (2012). p. 1066–70. Available online at: https://www.bund estieraerztekammer.de/btk/dtbl/archiv/2012/artikel/DTBl_08_2012_Brucellose.pdf (accessed April 25, 2023).
16. Holst BS, Löfqvist K, Ernholm L, Eld K, Cedersmyg M, Hallgren G. The first case of Brucella canis in Sweden: background, case report and recommendations from a northern European perspective. Acta Vet Scand. (2012) 54:18. doi: 10.1186/1751-0147-54-18
17. Buhmann G, Paul F, Herbst W, Melzer F, Wolf G, Hartmann K, et al. Canine brucellosis: insights into the epidemiologic situation in Europe. Front Vet Sci. (2019) 6:151. doi: 10.3389/fvets.2019.00151
18. Woldemeskel M. Zoonosis due to Brucella suis with special reference to infection in dogs (carnivores): a brief review. OJVM. (2013) 3:213–21. doi: 10.4236/ojvm.2013.33034
19. Olsen SC, Boggiatto P, Nol P, Samartino L. Brucellosis. In: Zimmerman JJ, Karriker LA, Ramirez A, Schwartz KJ, Stevenson GW, Jianqiang Z, editors. Diseases of Swine. 11th ed. Hoboken, NJ: Wiley-Blackwell/American Association of Swine Veterinarians (2019). p. 778–91.
20. Zange S, Schneider K, Georgi E, Scholz HC, Antwerpen MH, Walter MC, et al. A headache with surprising outcome: first case of brucellosis caused by Brucella suis biovar 1 in Germany. Infection. (2019) 47:863–8. doi: 10.1007/s15010-019-01312-7
21. Irwin MJ, Massey PD, Walker B, Durrheim DN. Feral pig hunting: a risk factor for human brucellosis in north-west NSW? N S W Public Health Bull. (2009) 20:192–4. doi: 10.1071/NB09023
22. Eales KM, Norton RE, Ketheesan N. Brucellosis in northern Australia. Am J Trop Med Hyg. (2010) 83:876–8. doi: 10.4269/ajtmh.2010.10-0237
23. Centers for Disease Control and Prevention. Brucella suis infection associated with feral swine hunting in three states, 2007-2008. Morbid Mortal Wkly Rep. (2009) 58:618–21.
24. Mason RJ, Fleming PJ. Serological survey for Brucella antibodies in feral pigs from eastern Australia. Aust Vet J. (1999) 77:331–2. doi: 10.1111/j.1751-0813.1999.tb10276.x
25. Crichton R, Medveczky NE. The identity, distribution and epizootiological significance of Brucella isolates in Australia, 1981 to 1985. Aust Vet J. (1987) 64:48–52. doi: 10.1111/j.1751-0813.1987.tb16128.x
26. Norton TH, Thomas AD. Letter: Brucella suis in feral pigs. Aust Vet J. (1976) 52:293–4. doi: 10.1111/j.1751-0813.1976.tb00122.x
27. Aldrick SJ. Typing of Brucella strains from Australia and Papua-New Guinea received by the regional W.H.O. Brucellosis Centre. Aust Vet J. (1968) 44:130–3. doi: 10.1111/j.1751-0813.1968.tb09054.x
28. Massis F de, Zilli K, Di Donato G, Nuvoloni R, Pelini S, Sacchini L, et al. Distribution of Brucella field strains isolated from livestock, wildlife populations, and humans in Italy from 2007 to 2015. PLoS ONE. (2019) 14:e0213689. doi: 10.1371/journal.pone.0213689
29. Grégoire F, Mousset B, Hanrez D, Michaux C, Walravens K, Linden A, et al. serological and bacteriological survey of brucellosis in wild boar (Sus scrofa) in Belgium. BMC Vet Res. (2012) 8:80. doi: 10.1186/1746-6148-8-80
30. EFSA. Porcine brucellosis (Brucella suis). EFSA J. (2009) 7:1–112. doi: 10.2903/j.efsa.2009.1144
31. Kutlu M, Cevahir N, Erdenlig-Gürbilek S, Akalin S, Uçar M, diciendo-Kutlu S. El primer informe de aislamiento de Brucella suis biovar 1 en humanos en Turquía. J Infectar la salud pública. (2016) 9:675–8. doi: 10.1016/j.jiph.2016.01.011
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. van Dijk MAM, Engelsma MY, Visser VXN, Spierenburg MAH, Holtslag ME, Willemsen PTJ, et al. Infección por Brucella suis en carne cruda alimentada con perros, Países Bajos. Emerg infectar dis. (2018) 24:1127–9. DOI: 10.3201/EID2406.171887
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Hellmann E, Sprenger HU. Infecciones por Brucella suis en el perro. Berl Munch Tierarztl Wochenschr. (1978) 91:385–7.
34. Compés Dea C, Guimbao Bescós J, Alonso Pérez de Ágreda JP, Muñoz Álvaro PM, Blasco Martínez JM, Villuendas Usón MC. Investigación epidemiológica del primer caso de brucelosis humana en España debido a Brucella suis biovar 1 cepa 1330. Enferm Infecc Microbiol Clin. (2017) 35:179–81. doi: 10.1016/j.eimc.2016.06.005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. García-Yoldi D, Marín CM, Miguel MJ de, Muñoz PM, Vizmanos JL, López-Goñi I. Multiplex PCR assay for the identification and differentiation of all Brucella species and the vaccine strains Brucella abortus S19 and RB51 and Brucella melitensis Rev1. Clin Chem. (2006) 52:779–81. DOI: 10.1373/clinchem.2005.062596
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. López-Goñi I, García-Yoldi D, Marín CM, Miguel MJ de, Barquero-Calvo E, Guzmán-Verri C, et al. Nuevo ensayo de PCR multiplex Bruce-ladder para la tipificación biovar de Brucella suis y la discriminación de Brucella suis y Brucella canis. Veterinario Microbiol. (2011) 154:152–5. doi: 10.1016/j.vetmic.2011.06.035
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Friedrich-Loeffler-Institut. Brucellose der Rinder, Schweine, Schafe und Ziegen: Amtliche Methode und Falldefinition: Amtliche Methode und Falldefinition. Friedrich-Loeffler-Institut & Friedrich-Loeffler-Institut, Amtliche Methodensammlung und Falldefinitionen: Anzeigepflichtige Tierseuchen, Greifswald – Insel Riems, Alemania. (2021).
38. Murigneux V, Roberts LW, Forde BM, Phan M-D, Nhu NTK, Irwin AD, et al. MicroPIPE: validación de un flujo de trabajo de extremo a extremo para la construcción completa del genoma bacteriano de alta calidad. BMC Genom. (2021) 22:474. DOI: 10.1186/S12864-021-07767-Z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Gurevich A, Saveliev V, Vyahhi N, Tesler G. QUAST quality assessment tool for genome assemblies. Bioinformática. (2013) 29:1072–5. doi: 10.1093/bioinformática/BTT086
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Schwengers O, Jelonek L, Dieckmann MA, Beyvers S, Blom J, Goesmann A. Bakta: anotación rápida y estandarizada de genomas bacterianos a través de la identificación de secuencias libres de alineación. Género Microb. (2021) 7. DOI: 10.1099/mgen.0.000685
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Grissa I, Bouchon P, Pourcel C, Vergnaud G. Recursos en línea para estudios de microevolución bacteriana utilizando la tipificación MLVA o CRISPR. Biochimie. (2008) 90:660–8. doi: 10.1016/j.biochi.2007.07.014
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Jain C, Rodriguez-R LM, Phillippy AM, Konstantinidis KT, Aluru S. El análisis ANI de alto rendimiento de 90K genomas procariotas revela límites claros de especies. Nat Commun. (2018) 9:5114. DOI: 10.1038/S41467-018-07641-9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Nan W, Tan P, Wang Y, Xu Z, Mao K, Peng D, et al. PCR dúplex para la diferenciación de la cepa vacunal Brucella suis S2 y B. suis biovar 1 de otras cepas de Brucella spp. Vet J. (2014) 201:427–8. doi: 10.1016/j.tvjl.2014.05.033
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Sacchini L, Wahab T, Di Giannatale E, Zilli K, Abass A, Garofolo G, et al. Secuenciación del genoma completo para rastrear el origen geográfico de casos importados de brucelosis humana en Suecia. Microorganismos. (2019) 7:3. doi: 10.3390/microorganismos7100398
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Zhou Z, Alikhan N-F, Sergeant MJ, Luhmann N, Vaz C, Francisco AP, et al. GrapeTree: visualización de las relaciones genómicas centrales entre 100.000 patógenos bacterianos. Genoma Res. (2018) 28:1395–404. doi: 10.1101/gr.232397.117
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Jünemann S, Sedlazeck FJ, Prior K, Albersmeier A, John U, Kalinowski J, et al. Actualización de la comparación de rendimiento de secuenciación de sobremesa. Nat Biotechnol. (2013) 31:294–6. DOI: 10.1038/NBT.2522
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Abdel-Glil MY, Thomas P, Brandt C, Melzer F, Subbaiyan A, Chaudhuri P, et al. Esquema de tipificación de secuencias multilocus del genoma central para mejorar la caracterización y la vigilancia epidemiológica de Brucella patógena. J Clin Microbiol. (2022) 60:E0031122. DOI: 10.1128/JCM.00311-22
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Stamatakis A. RAxML versión 8: una herramienta para el análisis filogenético y el post-análisis de filogenias grandes. Bioinformática. (2014) 30:1312–3. doi: 10.1093/bioinformática/btu033
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. TRBA 466 Clasificación de procariotas (bacterias y arqueas) en grupos de riesgo: norma técnica para agentes biológicos. GMBI (2010). págs. 1428–667.
50. TRBA 100 Medidas de protección para actividades con agentes biológicos en laboratorios: norma técnica para agentes biológicos. GMBI (2013). págs. 1010–42.
51. Landis M, Rogovskyy AS. Cerrando el breve caso: infección por Brucella suis en un hogar de perros. J Clin Microbiol. (2022) 60:E0098521. DOI: 10.1128/JCM.00985-21
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Mor SM, Wiethoelter AK, Lee A, Moloney B, James DR, Malik R. Emergence of Brucella suis in dogs in New South Wales, Australia: clinical findings and implications for zoonotic transmission. BMC Vet Res. (2016) 12:199. DOI: 10.1186/S12917-016-0835-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
53. Ramamoorthy S, Woldemeskel M, Ligett A, Snider R, Cobb R, Rajeev S. Brucella suis infection in dogs, Georgia, USA. Emerg infectar dis. (2011) 17:2386–7. doi: 10.3201/eid1712.111127
Resumen de PubMed | Texto completo de CrossRef | Google Académico
54. Nicoletti PL, Quinn BR, Minor PW. Transmisión canina a humana de la brucelosis. N Y Estado J Med. (1967) 67:2886–7.
55. Naha K, Dasari S, Pandit V, Seshadri S. Un caso raro de infección comprobada por cultivo seronegativo con Brucella suis. Aust Med J. (2012) 5:340–3. doi: 10.4066/AMJ.2012.1177
Resumen de PubMed | Texto completo de CrossRef | Google Académico
56. Cosford KL. Brucella canis: an update on research and clinical management. Can Vet J. (2018) 59:74–81.
57. Schaeffer J, Revilla-Fernández S, Hofer E, Posch R, Stoeger A, Leth C, et al. Seguimiento del origen de los casos de brucelosis humana austriaca utilizando la secuenciación del genoma completo. Front Med. (2021) 8:635547. DOI: 10.3389/FMED.2021.635547
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: brucelosis canina, zoonosis, Brucella suis biovar 1, epididimitis, enfermedad reproductiva, secuenciación del genoma completo, genotipado, dieta de carne cruda
Cita: Aurich S, Schneider J, Brangsch H, Koets A, Melzer F, Ewers C y Prenger-Berninghoff E (2023) Infección por Brucella suis biovar 1 en un perro con orquitis en Alemania. Frente. Vet. Sci. 10:1233118. doi: 10.3389/fvets.2023.1233118
Recibido: 01 Junio 2023; Aprobado: 10 de julio de 2023;
Publicado: 03 agosto 2023.
Editado por:
Yogesh Chander, Varigen Biosciences Corporation, Estados Unidos
Revisado por:
Giuseppe Marruchella, Universidad de Teramo, Italia
Hosny El-Adawy, Friedrich Loeffler Institut, Alemania
Derechos de autor © 2023 Aurich, Schneider, Brangsch, Koets, Melzer, Ewers y Prenger-Berninghoff. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Sophie Aurich, Sophie.Aurich@vetmed.uni-giessen.de
†Estos autores comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

