Influencia del sistema de cría en el microbioma, el metaboloma y el transcriptoma del íleon en conejos de carne
 Zhoulin Wu1
Zhoulin Wu1 Xiaoyu Li1
Xiaoyu Li1 Maoqin Xu1
Maoqin Xu1 Contenedor Wen2
Contenedor Wen2 Xiangchao Fu2
Xiangchao Fu2 Zhonghua Tang3
Zhonghua Tang3 Xueqin Liu3
Xueqin Liu3 Jiamin Zhang1*
Jiamin Zhang1*- 1Laboratorio clave de procesamiento de carne de la provincia de Sichuan, Facultad de Ingeniería Biológica y de Alimentos, Universidad de Chengdu, Chengdu, China
- número arábigoAcademia de Ciencias de los Pastizales de Sichuan, Chengdu, China
- 3Sichuan Aichi Rabbit Food Co., Ltd., Bazhong, China
El sistema de cría del ganado desempeña un papel vital en la producción animal, la calidad de la carne y el bienestar general. El objetivo de este estudio fue evaluar la influencia del sistema de cría en jaulas y del sistema de cría forestal en la microbiota del íleon, el metaboloma y el transcriptoma de la mucosa ileal en conejos de carne. Además, la secuenciación del ADNr 16S reveló diferencias significativas en la composición del microbioma ileal: los conejos enjaulados exhibieron una mayor abundancia de los géneros Erysipelotrichaceae y Delftia no cultivados, mientras que los niveles de Muribaculaceae, Burkholderiales no clasificados y Eubacteriaceae no cultivados fueron menores en comparación con los conejos criados en el bosque. El análisis del metaboloma identificó 372 metabolitos acumulados diferencialmente en el contenido de íleon, que se mapearon predominantemente con el metabolismo de aminoácidos, el metabolismo de nucleótidos y las vías del metabolismo energético. Se encontró que el sistema de cría en jaulas se correlaciona positivamente con la utilización eficiente de las fuentes de nutrientes. Además, el análisis del transcriptoma de la mucosa ileal reveló 984 genes expresados diferencialmente, predominantemente involucrados en vías metabólicas, vías de transducción de señales y procesos de respuesta inmune. A través del análisis de correlación de Pearson, pudimos dilucidar la ruta metabólica, las respuestas inmunitarias y los mecanismos de resistencia a las enfermedades que se vieron afectados por el sistema de cría. En general, los hallazgos sugirieron que la adaptación metabólica, la utilización de nutrientes y la respuesta inmunitaria desempeñan un papel crucial en la forma en que los conejos se adaptan a los diferentes sistemas de cría. Si bien el sistema de jaulas puede mejorar la eficiencia de los nutrientes, parece suprimir la función inmunológica y la resistencia a las enfermedades.
1 Introducción
El consumo y la producción de carne de conejo no están generalizados a nivel mundial, pero se han convertido en una industria ganadera altamente especializada en algunos países asiáticos y en la mayoría de los países mediterráneos, particularmente en China, Italia, Francia y España (1). Entre estos países, China por sí sola representa aproximadamente el 60% de la producción total mundial, siendo Europa el segundo mayor productor (1, 2). Los conejos se consideran ideales para la producción de carne debido a sus muchas cualidades ventajosas, como un ciclo vital y un período de gestación cortos, un aumento de peso diario significativo y una alta fertilidad. Como herbívoros monogástricos altamente especializados, los conejos poseen un sistema digestivo bien adaptado a una dieta alta en fibra, lo que les permite tener una notable eficiencia de conversión alimenticia.
La carne de conejo también es magra, rica en aminoácidos esenciales, y contiene grasas altamente insaturadas (3). Aporta niveles moderados de energía y bajo contenido en colesterol (1). A pesar de estas cualidades beneficiosas, el consumo de carne de conejo está disminuyendo en los países occidentales, en gran parte debido a las preocupaciones sobre el bienestar animal y las preferencias de los consumidores (4).
En los últimos años, ha habido un creciente interés de los consumidores por el bienestar animal, la agricultura ecológica, la nutrición de la carne y la salud humana. Muchos consumidores prefieren comprar productos cárnicos de sistemas de cría al aire libre debido a sus cualidades de sensor superiores en comparación con las de los sistemas de alojamiento convencionales (5, 6). En el caso de los conejos, el sistema de cría es uno de los factores que afecta moderadamente el rendimiento del crecimiento, el comportamiento, la inmunidad, el estrés oxidativo y la calidad de la canal y la carne (7-9). Por ejemplo, los sistemas de cría al aire libre se asocian con la expresión de comportamientos más naturales y una menor incidencia de trastornos digestivos (10). Los trastornos digestivos son una de las principales causas de deterioro del bienestar, con una alta tasa de incidencia en los sistemas de alojamiento convencionales.
El íleon, la parte terminal del intestino delgado, alberga billones de microbios que interactúan íntimamente con el huésped (11). La composición del microbioma residente está influenciada por la condición fisiológica del huésped y, a su vez, afecta la salud general (12). Algunos estudios han demostrado que la microbiota intestinal puede interactuar directamente con las células epiteliales intestinales y modular aún más el sistema inmunitario intestinal (13), la diferenciación del epitelio (14) y la protección de la mucosa mediada por el sistema inmunitario (15).
Actualmente, los efectos de los sistemas de cría sobre la histomorfología intestinal y la composición microbiana intestinal se han explorado ampliamente en cerdos (18), pollos (19) y gansos (20). De manera similar, estudios previos realizados en conejos han concluido que los diferentes sistemas de cría afectan significativamente el rendimiento del crecimiento, el rendimiento del sacrificio y la composición de la carne (21), al tiempo que reducen la incidencia de trastornos digestivos (10). Es importante destacar que el análisis integrador del microbioma, el transcriptoma y el metaboloma proporciona nuevos conocimientos sobre cómo las interacciones entre el huésped y la microbiota afectan al rendimiento de los animales y a su bienestar general (16, 17). Hasta la fecha, las complejas interacciones entre la microbiota intestinal y las respuestas genéticas del huésped en conejos de carne criados bajo diferentes sistemas de cría siguen siendo en gran medida inexploradas.
Por lo tanto, para abordar esta brecha de conocimiento, se empleó un enfoque multiómico para explorar la composición bacteriana ileal, el metaboloma y la expresión génica del huésped en conejos de carne criados en jaulas o en un entorno forestal. Este enfoque tiene como objetivo identificar la microbiota clave, descubrir las vías metabólicas reguladoras y aclarar los mecanismos moleculares que subyacen a las respuestas fisiológicas. En última instancia, se espera que estos resultados identifiquen las asociaciones huésped-microbio y proporcionen una visión integral de los sistemas biológicos involucrados, ofreciendo información valiosa sobre cómo los sistemas de cría contribuyen al bienestar de la cunicultura.
2 Materiales y métodos
2.1 Diseño de experimentos y tratamiento animal
En este estudio se utilizaron un total de 30 conejos machos sanos de Nueva Zelanda de una línea de raza pura. De los 18 a los 20 días de edad, los animales jóvenes fueron introducidos gradualmente a la alimentación sólida, junto con la leche materna, para ayudarlos a adaptarse a los alimentos peletizados. A los 30 días de edad, todos los conejos fueron destetados y se les proporcionó alimento comercial granulado (los ingredientes de la dieta se muestran en la Tabla Suplementaria 1), alimentados ad libitum tres veces al día a las 8:00, 13:00 y 18:00, respectivamente.
A los 40 días de edad, los conejos fueron asignados aleatoriamente a dos grupos en función de sus sistemas de cría: sistema de cría en jaulas (RC) y sistema de cría forestal (RF). El grupo RC se alojó individualmente en jaulas de acero inoxidable con una densidad de 0,2m2/cabeza en condiciones estándar con temperaturas entre 15 °C y 23 °C. En el sistema de RF, un área de bosque de aproximadamente 200m2 estaba rodeada por una cerca metálica de 3 m de altura, con una densidad de población calculada de 13 m2/cabeza. Ambos grupos fueron alimentados con el mismo alimento comercial granulado tres veces al día ad libitum, y el agua estuvo disponible gratuitamente a través de autobebederos con válvula durante todo el período experimental de 50 días.
2.2 Preparación de la muestra
A los 90 días de edad, se seleccionaron aleatoriamente seis conejos con pesos corporales similares (2.180,5 ± 102,5 g) de cada grupo. Los animales seleccionados fueron sometidos a aturdimiento eléctrico, seguido de procedimientos de exanguinación, desollado y evisceración. Inmediatamente después del sacrificio, las muestras de contenido de íleon se recogieron asépticamente, se congelaron en nitrógeno líquido y se almacenaron a -80 °C para su posterior análisis microbiano y metabolómico. Al mismo tiempo, se recolectaron secciones del íleon y la digesta se lavó del revestimiento epitelial utilizando solución salina estéril tamponada con fosfato (PBS) helada. Posteriormente, la mucosa del íleon se raspó suavemente después de lavarla tres veces con PBS y luego se almacenó rápidamente en nitrógeno líquido para la secuenciación del ARN.
2.3 Análisis de la microbiota asociada al contenido de íleon mediante secuenciación del gen ARNr 16S
La extracción, amplificación, construcción de bibliotecas y secuenciación del ADN bacteriano se llevó a cabo como se describió anteriormente (22). En resumen, las muestras de contenido de íleon congelado se sometieron a la extracción de ADN genómico microbiano utilizando el QIAamp DNA Stool Mini Kit (Qiagen, Shanghai, China) de acuerdo con el protocolo del fabricante. La concentración y pureza del ADN se evaluaron utilizando un espectrofotómetro NanoDrop ND-1000 (NanoDrop Technologies, Montchanin, DE, EE. UU.). La región hipervariable V3-V4 del gen de ARNr 16S bacteriano se amplificó mediante PCR utilizando los cebadores específicos 338F y 806R. Después de la amplificación por PCR, todos los amplicones calificados se sometieron a la construcción de bibliotecas y, posteriormente, se secuenciaron en la plataforma Illumina HiSeq 2500, generando lecturas de extremos emparejados de 250 pb.
Las lecturas brutas se filtraron y analizaron utilizando el software QIIME2 (23). Las etiquetas se agruparon en unidades taxonómicas operativas (OTU) y, a continuación, la asignación taxonómica se realizó utilizando la base de datos SILVA v138 (silva-138-99-nb-classifier.qza) con el algoritmo classify-sklearn. Las diversidades alfa y beta se calcularon mediante la prueba de Kruskal-Wallis y el método PERMANOVA, respectivamente (23, 24). Los análisis estadísticos se realizaron utilizando el software R (v4.1.3) (https://www.r-project.org/). El criterio de significancia se determinó con un valor de p de <0,05 y los valores se presentaron como medias. Finalmente, se utilizó PICRUST2 (v1.7.3) (25) para predecir los perfiles funcionales de los datos del gen ARNr 16S, y las vías se predijeron utilizando la base de datos KEGG.
2.4 Análisis de la metabolómica asociada al contenido de íleon por LC-MS
Se utilizó la tecnología de cromatografía líquida-espectrometría de masas (LC-MS/MS) para analizar el perfil metabólico del contenido intestinal, siguiendo un método descrito anteriormente (26). Brevemente, los metabolitos se extrajeron utilizando una solución de metanol de 400 μL (4:1, v/v) con 0,02 mg/mL de L-2-clorofenilalanina como patrón interno. La mezcla se sonicó a 40 kHz durante 30 min a 5 °C, seguida de una precipitación de proteínas a -20 °C durante 30 min. Posteriormente, se obtuvo el sobrenadante por centrifugación a 13.000 rpm durante 10 min a 4°C. Finalmente, el sobrenadante se evaporó hasta la sequedad bajo una corriente suave de nitrógeno para el análisis de LC-MS, y se preparó una muestra de control de calidad (QC) agrupada mezclando cantidades iguales de metabolitos de cada muestra para garantizar la consistencia de los datos.
El análisis de LC-MS se llevó a cabo utilizando el sistema UHPLC-Q Exactive HF-X de Thermo Fisher Scientific, siguiendo las condiciones descritas en nuestro estudio anterior (27). Para la separación por cromatografía líquida, se mantuvo un caudal de 0,25 mL min-1 y la temperatura de la columna se ajustó a 40 °C. Cada muestra se equilibró y se inyectó un volumen de 2 μL para su análisis. Los datos espectrales de masas se adquirieron utilizando voltajes de pulverización de 3,8 kV para el modo de iones positivos (ESI+) y de −2,5 kV para el modo de iones negativos (ESI−).
Finalmente, los datos brutos de LC-MS se procesaron utilizando el software Progenesis QI (Waters Inc., Milford, MA, EE. UU.). La identificación de metabolitos se llevó a cabo mediante la búsqueda en las bibliotecas espectrales estándar de referencia de MS/MS o bases de datos como la base de datos HumanMetabolome Database (HMDB, http://www.hmdb.ca), Metlin (http://metlin.scripps.edu) y mzCloud (https://www.mzcloud.org). Los metabolitos acumulados diferencialmente (DAM) se identificaron a partir de una variable de importancia en el umbral de proyección (VIP) >1.0 en el modelo OPLS-DA y un valor p de <0.05 en la prueba t de un estudiante. El análisis de enriquecimiento funcional de los DAM se llevó a cabo utilizando la base de datos de la Enciclopedia de Genes y Genomas de Kioto (KEGG).
2.5 Análisis del perfil del transcriptoma de la mucosa ileal mediante secuenciación de ARN
El ARN total se extrajo de la mucosa ileal utilizando el reactivo TRIzol (TaKaRa, Dalian, China), y el ADN se extrajo utilizando el RNeasy Midi Kit (Qiagen, Valencia, CA, USA) según los protocolos del fabricante. La calidad del ARN se evaluó con un Bioanalizador 5.300 (Agilent Technologies, EE.UU.) y se cuantificó utilizando el espectrofotómetro NanoDrop ND-2000 (NanoDrop Technologies).
La biblioteca de transcriptomas RNA-seq se construyó utilizando el kit de preparación de bibliotecas de ARN NEBNextUltra TM de Illumina (NEB, EE. UU.) de acuerdo con las instrucciones del fabricante. La secuenciación de la biblioteca se llevó a cabo en la plataforma Illumina NovaSeq™ X Plus, generando lecturas de 150 pb en el extremo emparejado.
Las lecturas en bruto se sometieron a la retirada del adaptador y al control de calidad, y las lecturas de baja calidad se filtraron como se describe en nuestro informe anterior (28). A continuación, los datos limpios se asignaron al último genoma de referencia del conejo (OryCun2.0.110 en Ensembl) utilizando el software HISAT2 (v2.2.1) con parámetros predeterminados (29). La expresión génica se cuantificó utilizando featureCounts (v2.0.1) (30), que contó el número de lecturas mapeadas para cada gen. Los genes expresados diferencialmente (DEGs) entre los dos grupos se analizaron utilizando el paquete DESeq2 R (v3.2.3) (31), con DEGs significativos definidos por un valor P ajustado (Padj) de <0.05 y un |log2 (FoldChange)| de >1.
Finalmente, se utilizó el software DAVID (v6.8) (32) para analizar el enriquecimiento estadístico de los términos de Gene Ontology (GO) y de las vías de la Enciclopedia de Genes y Genomas de Kyoto (KEGG), con un umbral de FDR de <0,05 que indica significación.
2.6 Análisis estadístico
Todos los resultados de seis réplicas por grupo se presentan como valor medio ± desviación estándar (DE). Se utilizó una prueba t de Student no pareada para comparar los dos grupos utilizando el programa SPSS 21.0 (IBM Corp., Nueva York, EE.UU.), con significación estadística definida a un valor de p de <0,05. El coeficiente de correlación de Spearman y el análisis de conglomerados se realizaron utilizando el paquete R (v4.2.0), y todos los resultados se visualizaron usando ggplot2 (v3.3.6) en el paquete R (33). Se utilizó el coeficiente de correlación de Pearson para identificar correlaciones significativas en los datos multiómicos, con valores de P de <0,05 considerados estadísticamente significativos.
3 Resultados
3.1 El sistema de cría indujo un cambio en la composición de la microbiota del contenido de íleon
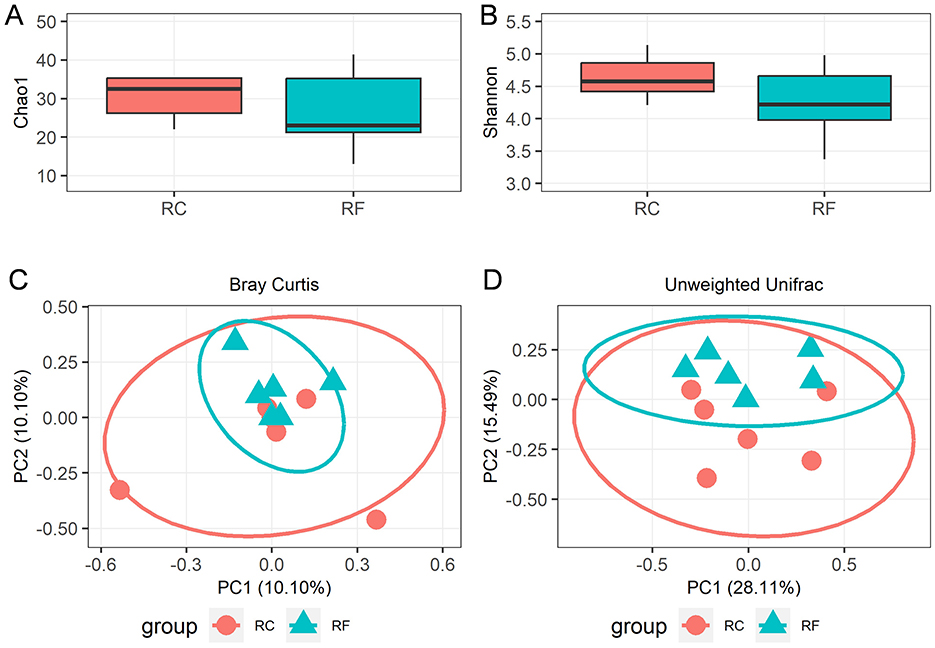
Un total de 1.119.688 secuencias bacterianas con una longitud promedio de 448 pb fueron retenidas y categorizadas en 396 unidades taxonómicas operativas (OTUs) utilizando el programa DEBLUR. No hubo diferencias significativas en los índices de diversidad alfa entre los dos grupos de conejos, como lo indica la riqueza de Chao1 (Figura 1A) y el índice de diversidad de Shannon (Figura 1B) (P > 0.05). Sin embargo, se observó un cambio notable en las diversidades beta a través del análisis de coordenadas principales (PCoA) basado en los métodos de Bray-Curtis y unifrac no ponderado. Los gráficos de PCoA revelaron que la microbiota con contenido de íleon de los animales con RF se agrupaba y estaba claramente separada de la de los conejos RC (Figuras 1C, D), lo que indica que las comunidades bacterianas se correlacionaban positivamente con el sistema de cría.
A nivel de filo, la microbiota con contenido de íleon estaba compuesta predominantemente por cuatro filos principales en ambos grupos de conejos: Proteobacteria, Firmicutes, Bacteroidota y Actinobacteriota, que en conjunto representaron el 89,24% de las OTU (Figura 2A).
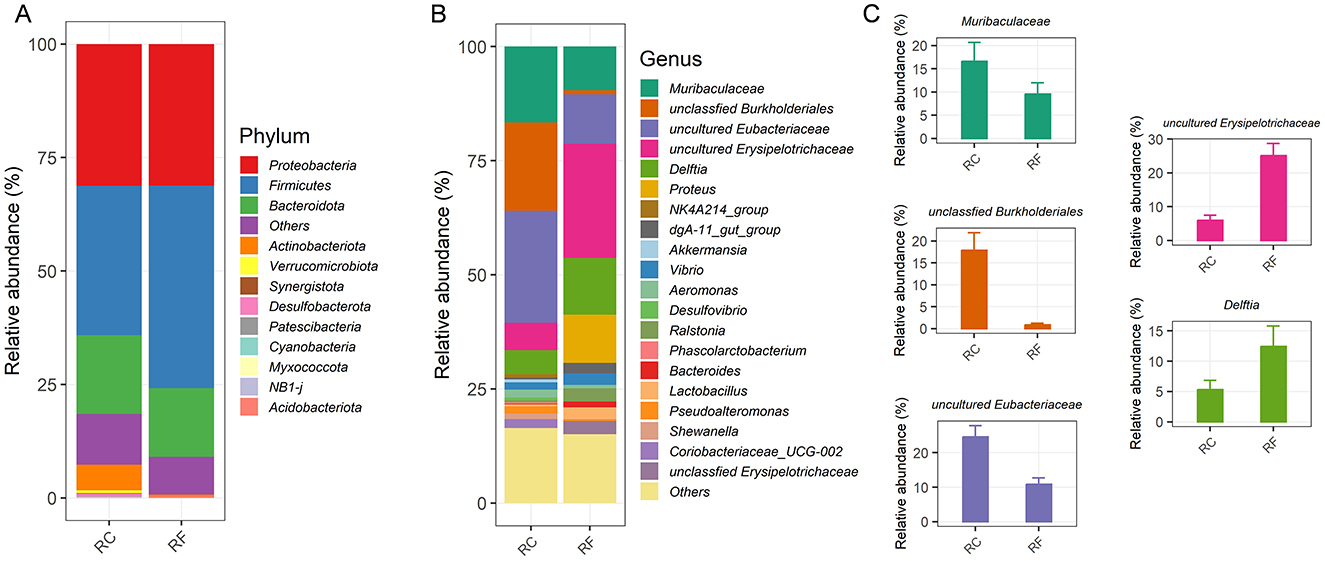
Figura 2. Diferentes sistemas de cría alteraron las composiciones bacterianas ileales específicas de los conejos. Abundancia relativa del filo ileal, nivel taxonómico (A). Abundancia relativa de las 20 principales bacterias a nivel taxonómico del género (B). Géneros de bacterias más modificados (C). RF: sistema de cría forestal; RC, sistema de cría en jaulas.
Se observaron diferencias significativas entre los grupos en la abundancia relativa de Firmicutes y Actinobacteriota, siendo Firmicutes más abundante en los conejos RF y Actinobacteriota más prevalente en los conejos RC (Figura 2A). Las composiciones promedio de la comunidad bacteriana de los 20 géneros principales se muestran en la Figura 2B. Además, se observaron diferencias significativas en la abundancia de los cinco géneros más prevalentes entre los conejos RF y RC. Los conejos RF exhibieron una mayor colonización de varios géneros, incluidos Erysipelotrichaceae, Delftia y Proteus no cultivados. Sin embargo, los conejos RC presentaron mayor abundancia de Muribaculaceae, Burkholderiales no clasificados y Eubacteriaceae no cultivados en su contenido de íleon (Figura 2C).
3.2 Funciones previstas de la microbiota con contenido de íleon
A partir de PICRUST2 análisis, se dedujo el perfil funcional de la microbiota con contenido de íleon en función del sistema de cría, revelando 28 vías KEGG significativamente enriquecidas entre los dos grupos. En los conejos criados en el sistema forestal, las vías relacionadas con el metabolismo de los lípidos, como la biosíntesis de ácidos grasos insaturados, el metabolismo de la riboflavina y la degradación del limoneno, estuvieron altamente representadas (Figura 3).
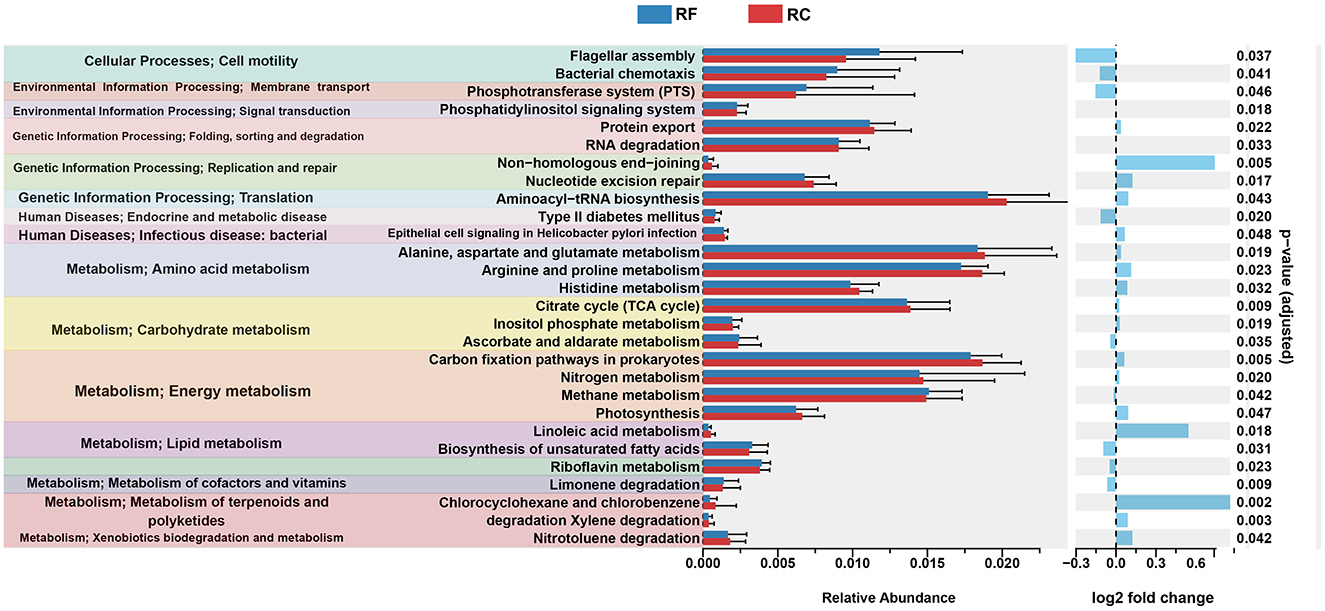
Además, las vías asociadas con los procesos celulares, incluido el ensamblaje flagelar y la quimiotaxis bacteriana, así como las vías involucradas en el sistema fosfotransferasa, la diabetes mellitus tipo II y el metabolismo del ascorbato y el aldarato, fueron más prominentes en los conejos con FR (Figura 3).
Por el contrario, los conejos en el sistema de cría en jaulas mostraron una mayor expresión de vías relacionadas con el metabolismo de aminoácidos, como el metabolismo de la histidina, la alanina, el aspartato, el glutamato, la arginina y la prolina. Las vías del metabolismo energético, como la fijación de carbono en procariotas, el metabolismo del nitrógeno y la fotosíntesis, también fueron más pronunciadas en los conejos RC en comparación con el grupo RF (Figura 3).
3.3 El sistema de cría indujo un cambio en el perfil metabolómico del contenido de íleon
Se detectaron un total de 1.174 metabolitos en el contenido de íleon, de los cuales 627 se identificaron en modo de iones positivos y 547 en modo de iones negativos. El análisis OPLS-DA indicó una clara separación entre los dos grupos de conejos (Figuras 4A, B), lo que indica distintos perfiles metabólicos entre ellos con modelos estables y confiables. Sobre la base de los umbrales de un VIP de > 1,00 y un valor P de <0,05, se identificaron 372 DAMs, de los cuales 181 estaban regulados al alza y 191 a la baja (Tabla complementaria 2).
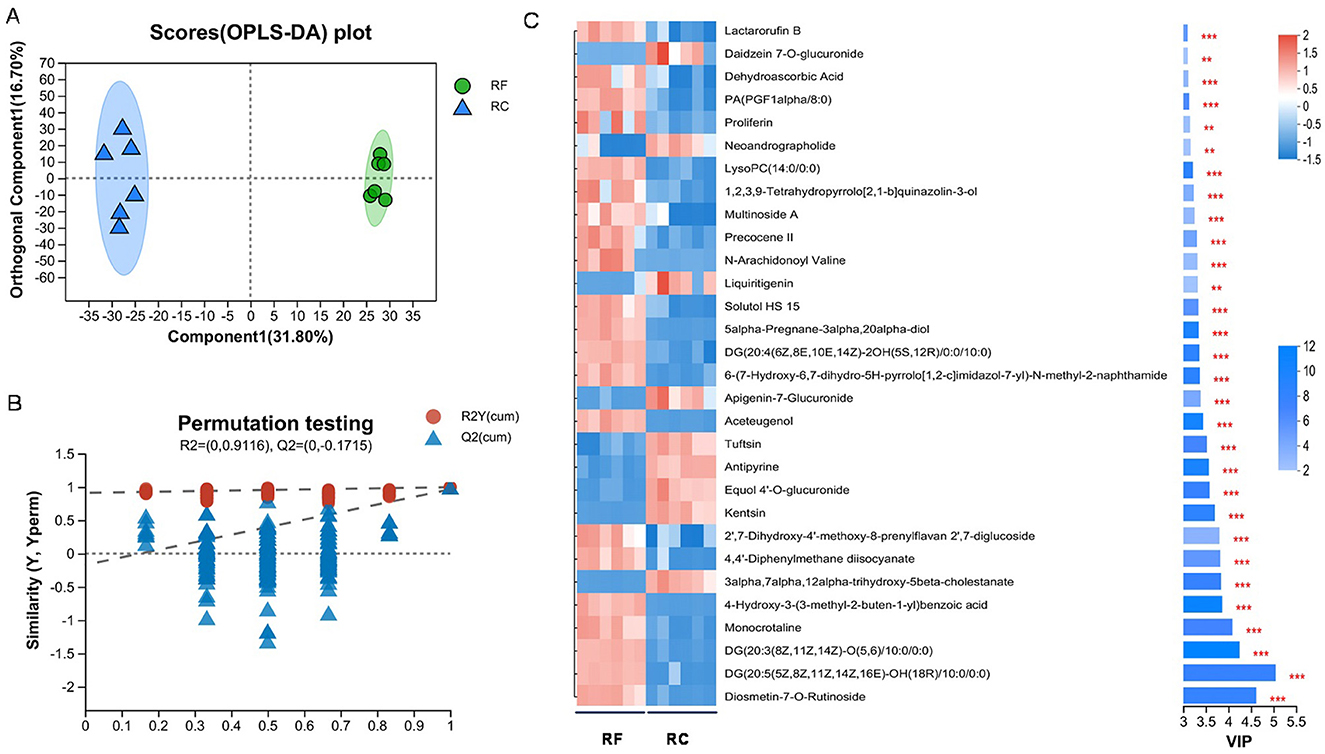
Figura 4. Análisis del metaboloma de muestras de contenido de íleon de diferentes sistemas de cría. Se obtuvieron gráficos de puntuación (A) y pruebas de permutación de análisis discriminante de mínimos cuadrados parciales ortogonales (OPLS-DA) para los grupos RF y RC (B). Los 30 metabolitos principales acumulados diferencialmente en el contenido de íleon de conejos identificados por OPLS-DA (C) (*0,01 < P ≤ 0,05; **0,001 < P ≤ 0,01; ***P ≤ 0,001). RF: sistema de cría forestal; RC, sistema de cría en jaulas.
Estos DAM se clasificaron en 13 categorías, como 118 lípidos y moléculas similares a los lípidos, 59 ácidos orgánicos y derivados, 47 compuestos organoheterocíclicos, 44 fenilpropanoides y policétidos, 27 compuestos orgánicos de oxígeno y 22 bencenoides, entre otros. En particular, los lípidos y moléculas similares a los lípidos, los ácidos orgánicos y derivados, los compuestos organoheterocíclicos y los fenilpropanoides y policétidos representaron el 33,62%, 16,81%, 13,39% y 12,54% de los DAM, respectivamente (Figura suplementaria 1). Luego, un análisis de mapa de calor de conglomerados de los 30 metabolitos principales confirmó aún más que los conejos RF y RC podían separarse claramente en función de sus perfiles metabolómicos (Figura 4C).
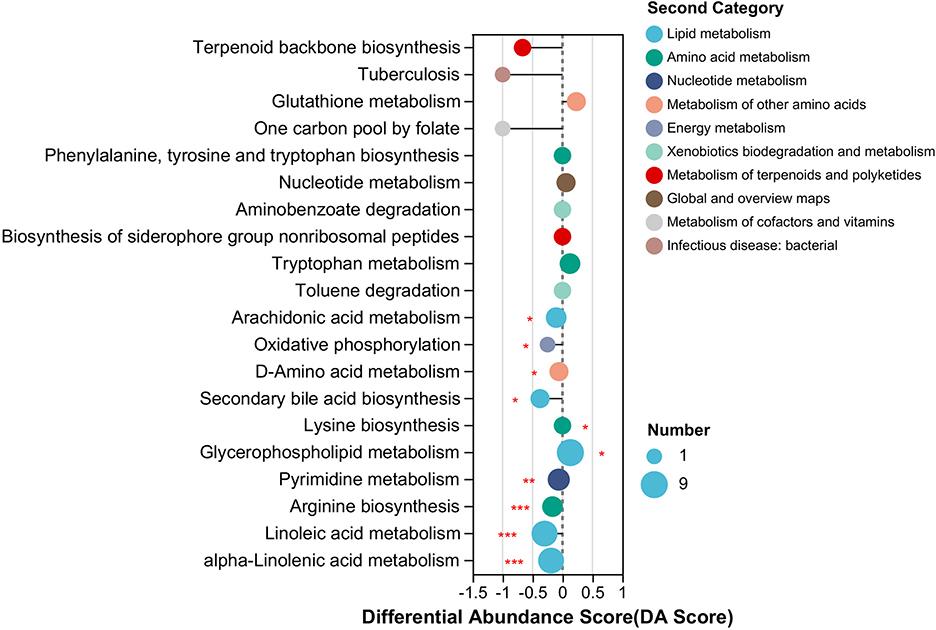
El análisis de enriquecimiento de la vía KEGG de los DAM de conejos RF y RC mostró que los DAMs estaban principalmente enriquecidos en 10 vías KEGG significativas, entre las cuales ocho vías estaban reguladas a la baja y dos vías estaban reguladas al alza en conejos RF en comparación con los conejos RC. Las vías enriquecidas incluyeron el metabolismo de los lípidos, el metabolismo de los aminoácidos, el metabolismo energético, el metabolismo de los nucleótidos y otras vías del metabolismo de los aminoácidos (Figura 5). En particular, la mayoría de los metabolitos alterados mostraron una mayor abundancia en los conejos RC, y la mayoría estuvo involucrada en las vías del metabolismo de los lípidos, como el metabolismo del ácido alfa-linolénico, el metabolismo del ácido linoleico, la biosíntesis secundaria de ácidos biliares y el metabolismo del ácido araquidónico (Figura 5).
3.4 Perfil de expresión génica de la mucosa ileal bajo diferentes sistemas de cría
Se generaron un total de 567.304.736 lecturas brutas a partir de 12 muestras epiteliales de íleon. Después de filtrar las secuencias de adaptadores y las lecturas de baja calidad, se conservaron un total de 562.212.578 lecturas limpias de 150 pares de bases. Más del 82,72% de estas lecturas limpias se mapearon con éxito en el genoma del conejo utilizando el software HISAT2. Después de filtrar genes con no más de 10 lecturas de conteo bruto en un máximo de dos muestras, se identificaron un total de 9.734 genes anotados, lo que representa el 46,05% del conjunto de 21.140 genes. En general, se detectaron 984 DEG en el epitelio del íleon, con 648 DEG regulados al alza y 336 DEG a la baja en conejos con FR en comparación con el grupo RC (Figura 6A). Estos DEG se definieron utilizando un umbral de |log2(FoldChange)| > 1 y Padj de <0.05.
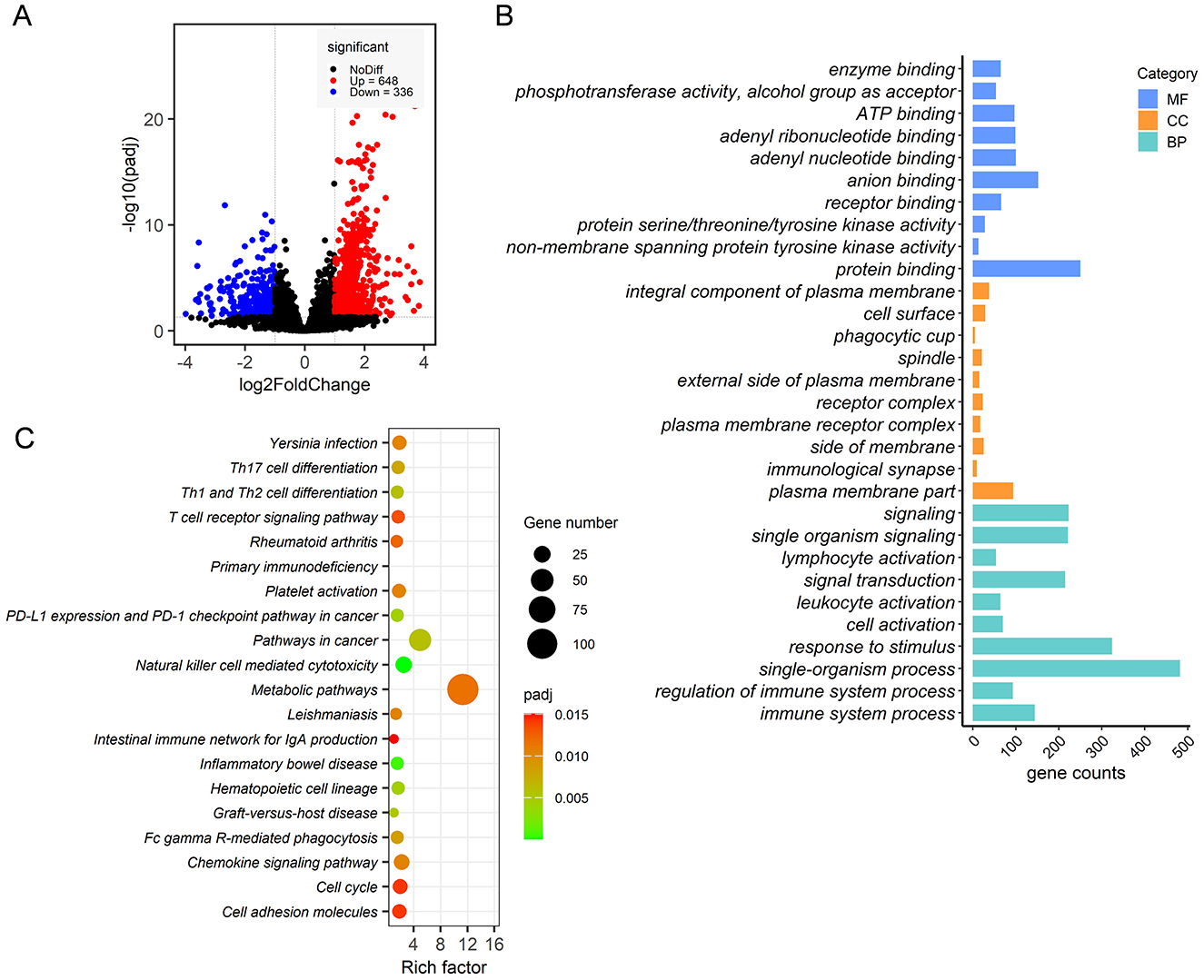
Se llevaron a cabo las vías de enriquecimiento de las vías GO y KEGG para evaluar los procesos biológicos y las vías asociadas con estos DEG. El análisis de enriquecimiento de GO reveló que los DEG se enriquecieron en 335 términos de GO, con los 10 términos más significativos resumidos para cada categoría.
En la categoría de procesos biológicos (BP), los DEG se asociaron principalmente con la activación celular, los procesos del sistema inmunitario y su regulación. En la categoría de función molecular (MF), los DEG se asociaron principalmente con la actividad y la unión de la quinasa. Para la categoría de componentes celulares (CC), los DEG se enriquecieron principalmente en procesos relacionados con la señalización en la sinapsis inmunológica, los complejos de receptores, la formación de copas fagocíticas y la membrana plasmática (Figura 6B). El análisis de la vía KEGG mostró que los DEG estaban significativamente enriquecidos en las vías metabólicas, las vías de transducción de señales, incluida la vía de señalización de quimiocinas y la vía de señalización del receptor de células T, y los procesos de respuesta inmunitaria, como la enfermedad de injerto contra huésped, la diferenciación de células Th1 y Th2, la diferenciación de células Th17, la fagocitosis mediada por Fc gamma R e inmunodeficiencia primaria (Figura 6C).
3.5 Análisis de correlación de bacterias de íleon, metabolitos y expresión génica del huésped
Se exploró la influencia del sistema de cría en las tres ómicas: microflora, metaboloma y expresión génica del huésped. Para las características del metaboloma, se incluyeron 78 metabolitos acumulados diferencialmente (DAM) anotados enriquecidos en metabolismo de lípidos, aminoácidos y energía.
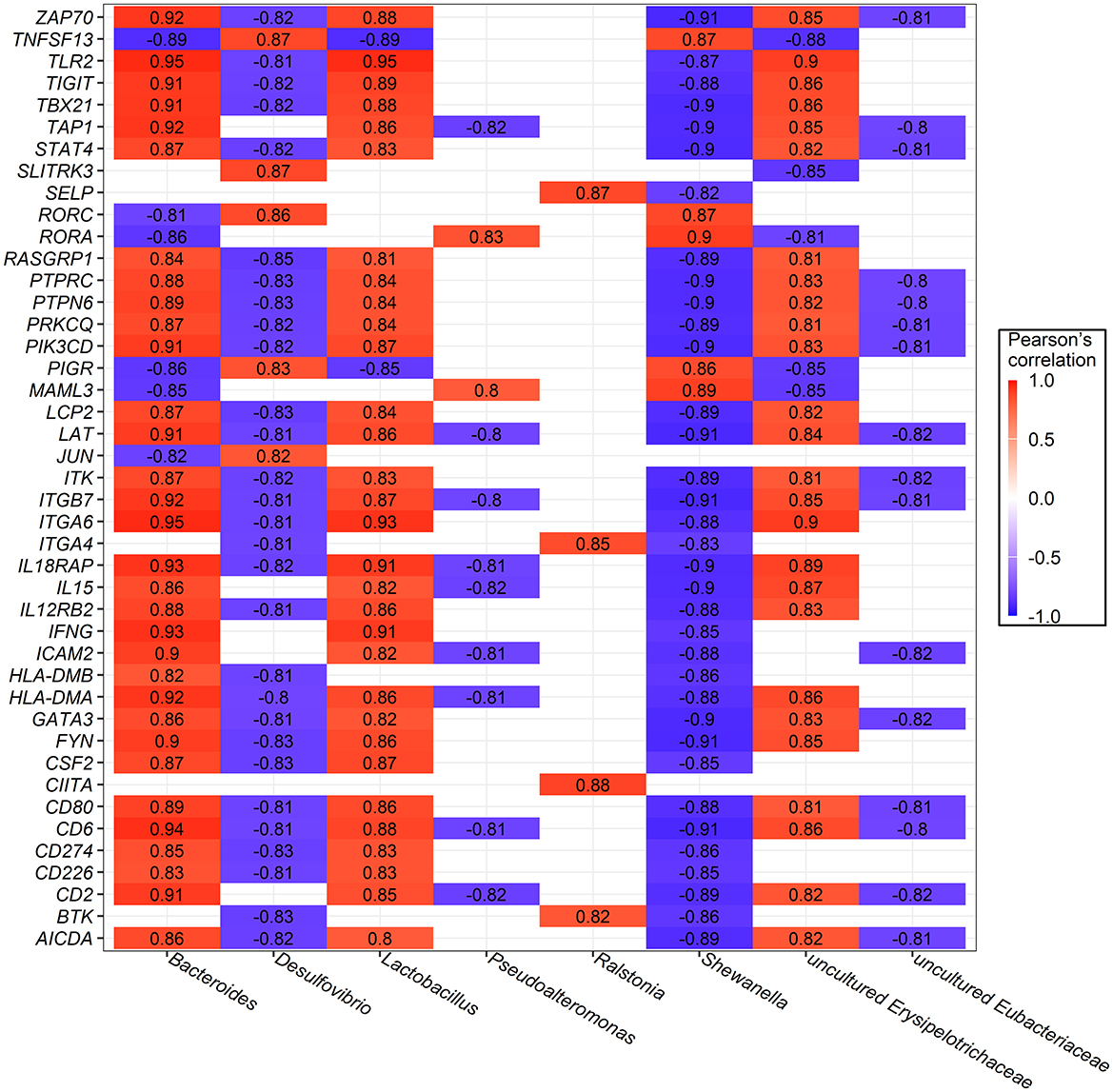
Además, se seleccionaron los 10 géneros bacterianos más abundantes y un subconjunto de 43 DEG enriquecidos en los procesos de respuesta inmunitaria y las vías metabólicas para el análisis de correlación de Pearson. Solo los coeficientes con |r| > 0,8 y los valores de P de <0,05 se muestran en la Figura 7. Se encontraron correlaciones significativas entre la expresión génica de las bacterias y el huésped, con Bacteroides correlación fuerte y positivamente con 32 genes, Lactobacillus con 31 genes y Erysipelotrichaceae sin cultivo con 26 genes. Por el contrario, las Burkholderiales no clasificadas se correlacionaron negativamente con 36 genes, mientras que Shewanella y Desulfovibrio se correlacionaron negativamente con 35 y 29 genes, respectivamente.
Además, las relaciones entre las comunidades bacterianas y los metabolitos proporcionaron información sobre las funciones de la microbiota y los metabolitos clave en diferentes sistemas de cría. Correlaciones con coeficientes |r| > 0,8 y los valores de P de <0,05 se muestran en la Figura complementaria 2. Entre ellos, Bacteroides mostró una amplia gama de correlaciones fuertes y significativas, con 25 correlaciones positivas y 28 negativas con los metabolitos. Además, las Erysipelotrichaceae no cultivadas se correlacionaron positivamente con 12 metabolitos y negativamente con 8 metabolitos. Curiosamente, también observamos que el género Lactobacillus se correlacionó positivamente con 14 metabolitos, mientras que Eubacteriaceae no cultivado se correlacionó positivamente con 12 metabolitos.
Tomados en conjunto, estos resultados indican que bacterias específicas, específicamente Bacteroides, Erysipelotrichaceae no cultivadas y Lactobacillus, podrían desempeñar un papel importante en la interacción con numerosos metabolitos y genes del huésped, influyendo en la adaptación del sistema de cría mediante la modulación de las respuestas inmunitarias y las vías metabólicas del huésped.
4 Discusión
Para satisfacer la creciente demanda de los consumidores de opciones de compra sostenibles, estudios anteriores han examinado los efectos de los sistemas de cría en el rendimiento del crecimiento y el bienestar del ganado. En particular, el microbioma intestinal es un determinante crucial de la salud y la productividad de los animales, y es bien sabido que su composición está asociada con el entorno de cría. Por ejemplo, Lin et al. (34) demostraron que los gansos criados en interiores exhibían una mayor abundancia de géneros patógenos y niveles más bajos de géneros comensales en comparación con los criados al aire libre.
De manera similar, para los pollos de engorde, las aves criadas en el suelo mostraron una abundancia relativamente mayor de bacterias potencialmente patógenas y asociadas a la cama (35), lo que podría deberse a una mayor exposición a los microbios ambientales. Esta exposición puede mejorar la función inmunológica y la resistencia a las enfermedades (35, 36). A pesar de estos conocimientos sobre las aves de corral, sigue habiendo una laguna en la comprensión del impacto de los sistemas de cría en la microbiota intestinal de los conejos y los posibles vínculos entre los géneros bacterianos, los patrones de expresión génica del huésped y el metaboloma. Los resultados de la secuenciación del ADNr 16S revelaron que los filos dominantes tanto en los conejos criados en bosques (RF) como en los criados en jaulas (RC) eran Proteobacteria, Firmicutes, Bacteroidota y Actinobacteriota. Aunque las abundancias relativas difieren, estos filos dominantes se alinean con estudios previos sobre la microbiota cecal de conejos blancos de Nueva Zelanda (37).
Si bien las comunidades bacterianas en general fueron en su mayoría similares, las diferencias clave de los taxones pueden reflejar adaptaciones a diferentes entornos de cría. Muribaculaceae, Burkholderiales no clasificadas y Eubacteriaceae no cultivadas se redujeron, mientras que Erysipelotrichaceae y Delftia no cultivadas aumentaron significativamente en los conejos RF en comparación con los conejos RC. Muribaculaceae se considera beneficiosa y está vinculada a vías que involucran citocinas y ácidos grasos de cadena corta (15), y su abundancia varía estacionalmente en conejos (38). Las burkholderiales incluyen bacterias con diversas funciones metabólicas (39), mientras que las Eubacterium no cultivadas, un miembro de Eubacteriaceae, pueden producir butirato, que desempeña un papel fundamental en la homeostasis energética, la motilidad colónica, la inmunomodulación y la supresión de la inflamación en el intestino (40, 41).
Los taxones bacterianos Erysipelotrichaceae se han asociado recurrentemente con fenotipos dislipidémicos en huéspedes, incluidos ratones y humanos, particularmente en el contexto de la obesidad, el síndrome metabólico y la hipercolesterolemia (42). Además, PICRUST2 análisis de estos taxones clave indicó que las vías relacionadas con el metabolismo de los lípidos y la resistencia a las enfermedades, como el sistema fosfotransferasa y la diabetes mellitus tipo II, eran más prominentes en los conejos criados en bosques (FR). Por el contrario, las funciones asociadas con el metabolismo de aminoácidos y energía fueron más prevalentes en los conejos criados en jaulas (RC). Estos hallazgos sugieren que los cambios en la composición de la microbiota intestinal pueden haber provocado cambios sustanciales en el metabolismo del huésped y en la resistencia a las enfermedades, lo que podría explicar por qué los conejos criados en jaulas mostraron un rendimiento de crecimiento favorable (21). Esto también es consistente con hallazgos previos en conejos (20, 43) y pollos de engorde (35) criados en sistemas de jaulas.
El intestino no solo es el órgano digestivo primario, sino también un órgano inmunológico importante en los animales. Desempeña un papel importante en la digestión y absorción de nutrientes de los alimentos ingeridos, mientras que la mucosa intestinal funciona como un componente clave de las barreras físicas y químicas, ya que puede reconocer y combatir las infecciones por patógenos, manteniendo la homeostasis entre el huésped y la microflora intestinal comensal (44). Numerosos estudios han demostrado la influencia de la composición de la microbiota intestinal en la expresión génica del epitelio intestinal del huésped y en la función inmunitaria de la mucosa intestinal (45, 46).
En el presente estudio, exploramos los efectos de diferentes sistemas de cría sobre la expresión génica en la mucosa ileal mediante análisis de RNA-seq. Se identificaron un total de 984 DEGs entre los conejos criados en el grupo FR y los del grupo RC. Se descubrió que estos genes estaban implicados en múltiples procesos y vías biológicas, y muchos de ellos estaban implicados en los procesos del sistema inmunitario y su regulación. Además, el análisis de enriquecimiento de KEGG reveló que estos DEG estaban significativamente enriquecidos en las vías metabólicas, las vías de transducción de señales y los procesos de respuesta inmunitaria, como la diferenciación de las células Th1 y Th2, la diferenciación de las células Th17, la fagocitosis mediada por Fc gamma R y la inmunodeficiencia primaria. Estudios previos han informado que, en comparación con los pollos enjaulados, las aves criadas en la planta baja exhibieron niveles más altos de IL-1β e IFN-γ ARNm en el íleon (47).
Del mismo modo, los resultados del transcriptoma y del metagenoma indicaron que los conejos criados en el bosque exhibieron una función inmunitaria de la mucosa intestinal más fuerte. En particular, los genes relacionados con el sistema inmunológico como IL9, IL15, IL2RG, IL12RB2, IL1RN, IL18RA, IF2A, IRF1, IFNG, TLR2 y TLR8 se regularon significativamente al alza en los conejos con RF en comparación con los criados en jaulas. En línea con nuestros hallazgos, Inman et al. (48) informaron que los lechones criados en un aislador habían aumentado significativamente los niveles de IL-2 producidos por las células T de la mucosa y habían reducido significativamente los niveles de IL-4 en comparación con los lechones criados al aire libre, lo que respalda aún más la idea de que las condiciones de cría pueden afectar la respuesta inmunitaria. Estos resultados sugieren que los cambios en las condiciones de cría pueden conducir a respuestas inmunitarias mejoradas en la mucosa ileal del conejo a nivel del transcriptoma.
El contenido intestinal sirve como un valioso indicador de la actividad microbiana intestinal y del metabolismo del huésped. Para investigar la respuesta metabólica a los cambios en el sistema de cría, comparamos el metaboloma del contenido ileal entre conejos RF y RC. Los metabolitos acumulados diferencialmente entre los dos grupos se asociaron principalmente con el metabolismo de lípidos, el metabolismo de aminoácidos, el metabolismo energético y el metabolismo de nucleótidos (Figura 5). En particular, estas vías fueron más abundantes en los conejos criados en jaulas, lo que sugiere una correlación positiva entre el sistema de cría en jaulas y la utilización eficiente de las fuentes de nutrientes y el rendimiento del crecimiento. Este hallazgo se alinea con estudios anteriores que demuestran que los animales criados en condiciones de suelo tienden a tener una mayor eficiencia alimenticia y un rendimiento de crecimiento superior en varias especies de ganado, incluidos patos (49), pollos (47) y cerdos (50).
Además, un análisis exhaustivo de correlación entre metagenómica, metabolómica y transcriptómica reveló que se encontró que géneros bacterianos específicos, como Bacteroides, Lactobacillus y Erysipelotrichaceae no cultivados, estaban significativamente asociados positivamente con múltiples metabolitos involucrados en el metabolismo nutricional, así como genes asociados con la respuesta inmune y las vías metabólicas. Estos hallazgos sugieren que estas bacterias pueden desempeñar un papel esencial en la interacción con los metabolitos ileales y los genes de la mucosa, influyendo así en la adaptación del huésped a diferentes sistemas de cría mediante la modulación de las respuestas inmunitarias y los procesos metabólicos.
5 Conclusión
El presente estudio demostró que el sistema de cría tiene un impacto significativo en la composición microbiana, la metabolómica del contenido ileal y la transcriptómica del huésped. Los conejos criados en el bosque exhibieron el microbioma intestinal con una menor abundancia relativa de Muribaculaceae, Burkholderiales no clasificados y Eubacteriaceae no cultivados, pero una mayor abundancia relativa de Erysipelotrichaceae y Delftia no cultivados en comparación con los criados en jaulas.
Además, el perfil metabolómico del contenido ileal difirió significativamente entre los grupos, con cambios principalmente en las vías relacionadas con el metabolismo de los aminoácidos, el metabolismo de los nucleótidos y el metabolismo energético. En particular, el sistema de cría en jaulas se asoció positivamente con una mejor utilización de nutrientes. Sin embargo, también se observaron cambios transcripcionales significativos en la mucosa ileal, particularmente en las vías metabólicas, la transducción de señales y los procesos de respuesta inmune. En general, si bien el sistema de cría en jaulas mejora la utilización de nutrientes, parece estar asociado con una respuesta inmunitaria deprimida y una menor resistencia a las enfermedades.
Declaración de disponibilidad de datos
Los datos de RNA-seq generados en este proyecto se depositan en el repositorio Sequence Read Archive (SRA), número de acceso PRJNA1105390. Los datos de secuenciación del gen ARNr 16S se depositan en el repositorio SRA, número de acceso PRJNA1105432.
Declaración ética
El Comité de Cuidado y Uso de Animales de la Universidad de Chengdu aprobó los procedimientos experimentales y de cuidado animal involucrados en este estudio y se adhirió a las directrices de la universidad para la investigación con animales (SSXY600008). Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones de los autores
ZW: Conceptualización, Curación de datos, Análisis formal, Investigación, Metodología, Recursos, Software, Supervisión, Validación, Visualización, Redacción – borrador original, Redacción – revisión y edición. MX: Curación de datos, Análisis formal, Metodología, Validación, Redacción – borrador original. XLi: Investigación, Metodología, Validación, Redacción – borrador original. BW: Curación de datos, Metodología, Validación, Redacción – borrador original. XF: Curación de datos, Investigación, Escritura – borrador original. ZT: Curación de datos, Redacción – borrador original. XLiu: Curación de datos, Redacción – borrador original. JZ: Curación de datos, Investigación, Metodología, Recursos, Visualización, Redacción – borrador original.
Financiación
El (los) autor(es) declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este estudio contó con el apoyo del Fondo Asignado para el Sistema de Investigación de la Industria Agrícola de China (CARS-43), la Fundación de Apertura del Laboratorio Clave de Procesamiento de Carne de la Provincia de Sichuan (23-R-19) y el Proyecto del Equipo de Innovación en Tecnología Forestal y de Pastizales de Sichuan «Investigación y Utilización de Tecnologías Clave en la Industria de Pastizales y Ganadería».
Conflicto de intereses
ZT y XL fueron empleados por Sichuan Aichi Rabbit Food Co., Ltd.
El resto de los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un potencial conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1456790/full#supplementary-material
Referencias
1. Cullere M, Dalle Zotte A. Producción y consumo de carne de conejo: estado del conocimiento y perspectivas futuras. Ciencia de la carne. (2018) 143:137–46. doi: 10.1016/j.meatsci.2018.04.029
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Zhang J, Wu Z-l, Wang W, Hou B, Bai T, Wen Y, et al. Mejora de la calidad del producto de carne de conejo curado (Chan Si Tu) utilizando Staphylococcus xylosus como cultivo iniciador. Mundo Conejo Sci. (2023) 31:191–200. doi: 10.4995/wrs.2023.19015
3. Lazzaroni C, Biagini D, Lussiana C. Composición de ácidos grasos de la carne y la grasa perirrenal en conejos de dos sistemas de cría diferentes. Ciencia de la carne. (2009) 83:135–9. doi: 10.1016/j.meatsci.2009.04.011
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Mancini S, Paci G. Probióticos en la cunicultura: rendimiento del crecimiento, estado sanitario y calidad de la carne. Animales. (2021) 11:3388. doi: 10.3390/ani11123388
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Barnett, J. L. Efectos del confinamiento y las necesidades de investigación para apuntalar los estándares de bienestar. J Veterinario Compórtate. (2007) 2:213–8. doi: 10.1016/j.jveb.2007.08.006
6. Szendro K, Szabó-Szentgróti E, Szigeti O. Actitud de los consumidores hacia el consumo de carne de conejo en ocho países según el método de producción y su forma de compra. Alimentos (Basilea). (2020) 9:654. doi: 10.3390/foods9050654
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Tufarelli V, Tateo A, Schiavitto M, Mazzei D, Calzaretti G, Laudadio V. Evaluación del rendimiento productivo, la calidad de la carne y los productos de oxidación de conejos de raza blanca italiana bajo sistema de cría en jaulas y en libertad. Anim Biosci. (2022) 35:884–91. doi: 10.5713/ab.21.0327
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Trocino A, Menegon F, Zomeño C, Pasqualin D, Cunial G, Xiccato G, et al. Un estudio piloto sobre la evaluación de la salud y el bienestar en la granja en conejos mantenidos en diferentes sistemas de alojamiento. Frente Vet Sci. (2022) 9:936643. doi: 10.3389/fvets.2022.936643
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Krunt O, Zita L, Kraus A, Bureš D, Needham T, Volek Z. El efecto del sistema de alojamiento en el rendimiento del crecimiento del conejo, las características de la canal y la calidad de la carne de diferentes músculos. Ciencia de la carne. (2022) 193:108953. doi: 10.1016/j.meatsci.2022.108953
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Fetiveau M, Savietto D, Fillon V, Bannelier C, Pujol S, Fortun-Lamothe L. Efecto del tiempo de acceso al área de pastoreo al aire libre y el enriquecimiento en el uso del espacio y el pasto, el comportamiento, la salud y los rasgos de crecimiento de los conejos destetados. Animal. (2023) 17:100724. doi: 10.1016/j.animal.2023.100724
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Krautkramer K A, Fan J, Bäckhed F. Metabolitos microbianos intestinales como intermediarios de múltiples reinos. Nat Rev Microbiol. (2021) 19:77–94. doi: 10.1038/s41579-020-0438-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Gilbert J A, Blaser M J, Caporaso J G, Jansson J K, Lynch S V, Knight R. Comprensión actual del microbioma humano. Nat Med. (2018) 24:392–400. doi: 10.1038/nm.4517
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Mahapatro M, Erkert L, Becker C. Diafonía mediada por citocinas entre las células inmunitarias y las células epiteliales en el intestino. Células. (2021) 10:111. doi: 10.3390/cells10010111
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Sommer F, Bäckhed F. La microbiota intestinal: maestros del desarrollo y la fisiología del huésped. Nat Rev Microbiol. (2013) 11:227–38. doi: 10.1038/nrmicro2974
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Li Z, He H, Ni M, Wang Z, Guo C, Niu Y, et al. Análisis del microbioma-metaboloma del microambiente inmune del contenido cecal, heces blandas y heces duras de conejos hyplus. Oxid Med Cell Longev. (2022) 2022:5725442. DOI: 10.1155/2022/5725442
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Tous N, Marcos S, Goodarzi Boroojeni F, Pérez De Rozas A, Zentek J, Estonba A, et al. Estrategias novedosas para mejorar el rendimiento y el bienestar de los pollos mediante la revelación de las interacciones entre el huésped y la microbiota a través de la hologenómica. Fisiola delantera. (2022) 13:884925. doi: 10.3389/fphys.2022.884925
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Saladrigas-García M, D’angelo M, Ko HL, Nolis P, Ramayo-Caldas Y, Folch JM, et al. Comprender las interacciones entre el huésped y la microbiota en el lechón comercial alrededor del destete. Sci Rep. (2021) 11:23488. doi: 10.1038/s41598-021-02754-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Holman D B, Gzyl K E, Kommadath A. El microbioma intestinal y el resistoma de los cerdos criados convencionalmente frente a los criados en pastos. Genoma Microb. (2023) 9:001061. doi: 10.1099/mgen.0.001061
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Chen S, Xiang H, Zhang H, Zhu X, Wang D, Wang J, et al. El sistema de cría provoca cambios en el comportamiento, el microbioma y la expresión génica de los pollos. Poult Sci. (2019) 98:3365–76. doi: 10.3382/ps/pez140
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Li X, Hu S, Wang W, Tang B, Zheng C, Hu J, et al. Efectos del sistema de cría en jaula frente al sistema de cría en el suelo sobre la histomorfología intestinal y la composición microbiana cecal. Poult Sci. (2022) 101:101931. doi: 10.1016/j.psj.2022.101931
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. D’agata M, Preziuso G, Russo C, Zotte AD, Mourvaki E, Paci G. Efecto de un sistema de cría al aire libre en el bienestar, el rendimiento del crecimiento, la calidad de la canal y la carne de una población de conejos de crecimiento lento. Ciencia de la carne. (2009) 83:691–696. doi: 10.1016/j.meatsci.2009.08.005
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Wu Z L, Yang X, Zhang J, Wang W, Liu D, Hou B, et al. Efectos del tipo de forraje sobre la microbiota ruminal, el rendimiento del crecimiento, las características de la canal y la calidad de la carne en cabras de engorde. Frente Vet Sci. (2023) 10:1147685. doi: 10.3389/fvets.2023.1147685
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Bolyen E, Rideout JR, Dillon M R, Bokulich NA, Abnet CC, Al-Ghalith GA, et al. Ciencia de datos de microbioma reproducible, interactiva, escalable y extensible utilizando QIIME 2. Nat Biotechnol. (2019) 37:852–7. doi: 10.1038/s41587-019-0252-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Lima J, Manning T, Rutherford KM, Baima ET, Dewhurst RJ, Walsh P, et al. La anotación taxonómica de secuencias de ARNr 16S de muestras intestinales de cerdos utilizando MG-RAST y QIIME2 generó diferentes composiciones de microbiota. J Métodos de Microbiol. (2021) 186:106235. doi: 10.1016/j.mimet.2021.106235
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Douglas GM, Maffei VJ, Zaneveld JR, Yurgel SN, Brown JR, Taylor CM, et al. PICRUSt2 para la predicción de las funciones del metagenoma. Nat Biotechnol. (2020) 38:685–8. doi: 10.1038/s41587-020-0548-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Wang S, Zuo Z, Ye B, Zhang L, Cheng Y, Xie S, et al. El análisis metabolómico revela efectos beneficiosos del almidón resistente a las algas marinas en la dieta sobre las funciones intestinales de las cabezas de serpiente híbridas (Channa maculata ♀ × Channa argus ♂ ). Antioxidantes. (2023) 12:1631. doi: 10.3390/antiox12081631
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Wu ZL, Chen SY, Hu S, Jia X, Wang J, Lai SJ. Perfiles metabolómicos y proteómicos asociados a la cetosis en vacas lecheras. Delantero Genet. (2020) 11:551587. doi: 10.3389/fgene.2020.551587
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Wu Z-L, Chen S-Y, Qin C, Jia X, Deng F, Wang J, et al. Alteración clínica de la expresión génica asociada a la cetosis en vacas Holstein. Genes. (2020) 11:219. doi: 10.3390/genes11020219
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Kim D, Langmead B, Salzberg SL. HISAT un alineador de empalme rápido con bajos requisitos de memoria. Métodos Nat. (2015) 12:357–60. doi: 10.1038/nmeth.3317
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Liao Y, Smyth GK, Shi W. featureCounts: un programa eficiente de propósito general para asignar lecturas de secuencias a características genómicas. Bioinformática. (2014) 30:923–30. doi: 10.1093/bioinformática/BTT656
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Love MI, Huber W, Anders S. Estimación moderada del cambio de pliegue y la dispersión para datos de RNA-seq con DESeq2. Genoma Biol. (2014) 15:550. doi: 10.1186/s13059-014-0550-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Huang Da W, Sherman BT, Lempicki RA. Análisis sistemático e integrador de grandes listas de genes utilizando los recursos bioinformáticos de DAVID. Nat Protoc. (2009) 4:44–57. doi: 10.1038/nprot.2008.211
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Gómez-Rubio V. ggplot2-gráficos elegantes para el análisis de datos. J Stat Softw. (2017) 77:1–3. doi: 10.18637/jss.v077.b02
34. Lin YY, Chang PE, Shen SY, Wang SD. Efectos del sistema de cría en interiores y exteriores sobre los parámetros bioquímicos de los gansos y la composición microbiana cecal. Poult Sci. (2023) 102:102731. doi: 10.1016/j.psj.2023.102731
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Song B, Yan S, Li P, Li G, Gao M, Yan L, et al. Análisis comparativo y de correlación de la función inmunitaria y la microbiota intestinal de pollos de engorde criados en jaulas de doble capa y corrales de suelo de cama. Microbiol Spectr. (2022) 10:E0004522. doi: 10.1128/spectrum.00045-22
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Jones DR, Cox NA, Guard J, Fedorka-Cray PJ, Buhr RJ, Gast RK, et al. Impacto microbiológico de tres sistemas comerciales de alojamiento de gallinas ponedoras. Poult Sci. (2015) 94:544–51. doi: 10.3382/ps/peu010
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Chen S-Y, Deng F, Jia X, Liu H, Zhang G-W, Lai S-J. Perfil de la microbiota intestinal con tolerancia diferencial frente al nivel reducido de fibra dietética en conejo. Sci Rep. (2019) 9:288. doi: 10.1038/s41598-018-36534-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Ye D, Ding X, Pang S, Gan Y, Li Z, Gan Q, et al. Variaciones estacionales en el rendimiento de la producción, el estado sanitario y la microbiota intestinal de conejos de carne criados en condiciones semiconfinadas. Animales. (2024) 14:113. doi: 10.3390/ani14010113
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Eggers S, Safdar N, Sethi AK, Suen G, Peppard PE, Kates AE, et al. Concentración urinaria de plomo y composición de la microbiota intestinal adulta en una muestra transversal poblacional. Environ Int. (2019) 133:105122. doi: 10.1016/j.envint.2019.105122
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Colombino E, Biasato I, Michetti A, Rubino MG, Franciosa I, Giribaldi M, et al. Efectos de la suplementación dietética de lactobacillus acidophilus sobre los parámetros sanguíneos y la salud intestinal de los conejos. Animales. (2022) 12:3543. doi: 10.3390/ani12243543
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Mukherjee A, Lordan C, Ross RP, Cotter PD. Microbios intestinales del género Filogenéticamente diverso Eubacterium y sus diversas contribuciones a la salud intestinal. Microbios intestinales. (2020) 12:1802866. doi: 10.1080/19490976.2020.1802866
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Martínez I, Perdicaro DJ, Brown AW, Hammons S, Carden TJ, Carr TP, et al. Es probable que las alteraciones del metabolismo del colesterol del huésped inducidas por la dieta afecten a la composición de la microbiota intestinal de los hámsters. Appl Environ Microbiol. (2013) 79:516–24. doi: 10.1128/AEM.03046-12
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Lazzaroni C, Biagini D, Lussiana C. Diferentes sistemas de cría para conejos de engorde: rendimiento y características de la canal. Ciencia de la carne. (2009) 82:200–4. doi: 10.1016/j.meatsci.2009.01.011
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Yu Y, Sitaraman S, Gewirtz AT. Regulación de la inflamación de la mucosa por las células epiteliales intestinales. Immunol Res. (2004) 29:55–68. doi: 10.1385/IR:29:1-3:055
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. Yang C, Qiu M, Zhang Z, Song X, Yang L, Xiong X, et al. Los galactooligosacáridos y xilooligosacáridos afectan el sabor de la carne al alterar el microbioma cecal, el metaboloma y el transcriptoma de los pollos. Poult Sci. (2022) 101:102122. doi: 10.1016/j.psj.2022.102122
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. Pan W-H, Sommer F, Falk-Paulsen M, Ulas T, Best P, Fazio A, et al. La exposición a la microbiota intestinal provoca distintos cambios en el metiloma y el transcriptoma en las células epiteliales intestinales durante el desarrollo postnatal. Genoma Med. (2018) 10:27. doi: 10.1186/s13073-018-0534-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Yan L, Lv ZZ, An S, Xing K, Wang ZG, Lv MB, et al. Efectos del sistema de cría y la narasina en el rendimiento del crecimiento, el desarrollo gastrointestinal y la microbiota intestinal de los pollos de engorde. Poult Sci. (2021) 100:100840. doi: 10.1016/j.psj.2020.10.073
Resumen de PubMed | Texto completo de Crossref | Google Académico
48. Inman CF, Haverson K, Konstantinov SR, Jones PH, Harris C, Smidt H, et al. El entorno de cría afecta el desarrollo del sistema inmunitario en los recién nacidos. Clin Exp Immunol. (2010) 160:431–9. doi: 10.1111/j.1365-2249.2010.04090.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
49. Zhang C, Ah Kan Razafindrabe RH, Chen K, Zhao X, Yang L, Wang L, et al. Efectos de diferentes sistemas de cría sobre el rendimiento del crecimiento, las características de la canal, la calidad de la carne y los parámetros bioquímicos séricos de los patos Chaohu. Anim Sci J. (2018) 89:672–8. doi: 10.1111/asj.12976
Resumen de PubMed | Texto completo de Crossref | Google Académico
50. Sirtori F, Crovetti A, Zilio DM, Pugliese C, Acciaioli A, Campodoni G, et al. Efecto de la raza paterna y el sistema de cría sobre el crecimiento, la composición de la canal y las características de la carne de los cerdos cruzados con Cinta Senese. Ital J Anim Sci. (2011) 10:E47. doi: 10.4081/ijas.2011.e47
Palabras clave: conejo, sistema de cría, microbioma ileal, metaboloma, transcriptoma de la mucosa ileal
Cita: Wu Z, Li X, Xu M, Wen B, Fu X, Tang Z, Liu X y Zhang J (2024) Influencia del sistema de cría en el microbioma, el metaboloma y el transcriptoma del íleon en conejos de carne. Frente. Vet. Sci. 11:1456790. doi: 10.3389/fvets.2024.1456790
Recibido: 29 de junio de 2024; Aceptado: 24 de septiembre de 2024;
Publicado: 08 noviembre 2024.
Editado por:
Shuai Liu, Universidad Agrícola de China, China
Revisado por:
Lianbin Li, Universidad de Hainan, China
Long Jin, Universidad Agrícola de Sichuan, China
Derechos de autor © 2024 Wu, Li, Xu, Wen, Fu, Tang, Liu y Zhang. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Jiamin Zhang, cdusp@qq.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

