La proteómica comparativa de los ovarios en ovejas

La proteómica comparativa de los ovarios elucidó las dianas potenciales relacionadas con la prolificación ovina
 Chunyan Li1,2†
Chunyan Li1,2†  Mei Zhou1,3†
Mei Zhou1,3†  Xiaoyun He1
Xiaoyun He1  Ran Di1
Ran Di1  Zijun Zhang4
Zijun Zhang4  Chunhuan Ren4
Chunhuan Ren4  Qiuyue Liu1*
Qiuyue Liu1*  Mingxing Chu1*
Mingxing Chu1*- 1Laboratorio Clave de Genética Animal, Cría y Reproducción del Ministerio de Agricultura y Asuntos Rurales, Instituto de Ciencia Animal, Academia China de Ciencias Agrícolas, Beijing, China
- 2Laboratorio Provincial de Ingeniería de Yunnan de Conservación de Recursos Genéticos Animales y Mejora de Germoplasma, Academia de Ganadería y Ciencias Veterinarias de Yunnan, Kunming, China
- 3Laboratorio clave de genética cuantitativa molecular porcina de la Academia de Ciencias Agrícolas de Anhui, Laboratorio clave provincial de Anhui de Ingeniería de Seguridad de Productos Ganaderos y Avícolas, Instituto de Ganadería y Medicina Veterinaria, Academia de Ciencias Agrícolas de Anhui, Hefei, China
- 4Facultad de Ciencia y Tecnología Animal, Universidad Agrícola de Anhui, Hefei, China
Las ovejas Small Tail Han (STH), una raza china única, son reconocidas por su madurez temprana, celo durante todo el año y prolificidad. Sin embargo, el mecanismo molecular de su alta prolificidad no ha sido completamente dilucidado. El enfoque proteómico es factible y eficaz para revelar las proteínas involucradas en los complejos procesos fisiológicos de cualquier organismo. Ante esto, realizamos el perfil de expresión de proteínas de tejidos ováricos durante la fase lútea utilizando ovejas STH politocosas (tamaño de camada ≥2, tres partos consecutivos) y ovejas STH monotocosas (tamaño de camada = 1, tres partos consecutivos) (PL vs. ML), y la fase folicular utilizando ovejas STH politocosas (tamaño de camada ≥2, tres partos consecutivos) y ovejas STH monotocosas (tamaño de camada = 1, tres partos consecutivos) (PF vs. MF), respectivamente. Se realizó un monitoreo de reacción paralela (PRM) para validar las proteínas diferencialmente abundantes (DAP). Los resultados proteómicos cuantitativos de la etiqueta de masa en tándem (TMT) mostraron que se identificaron un total de 5.237 proteínas, de las cuales 49 y 44 mostraron abundancia diferencial en los grupos PL vs. ML y PF vs. MF, respectivamente. Los análisis de enriquecimiento indicaron que los DAP que incluyen la proteína 1 de unión a ARN asociada a gránulos citotóxicos TIA1 (TIAL1), la nicotinamida fosforribosiltransferasa (NAMPT) y la proteína 1 de unión al ácido retinoico celular (CRABP1) se enriquecieron en la fase lútea, mientras que TIAL1, inhibina beta-a-subunidad (A2ICA4) y W5PG55 se enriquecieron en la fase folicular, mediando potencialmente los procesos reproductivos en ovejas politócas. Además, seis DAP fueron verificados utilizando PRM, confirmando la precisión de los datos de TMT adquiridos en este estudio. Juntos, nuestro trabajo amplió la base de datos de razas ovinas autóctonas y proporcionó nuevas dianas moleculares candidatas ováricas, que ayudarán en el estudio de los mecanismos genéticos de la prolificidad ovina.
Introducción
El tamaño de la camada es un indicador importante de reproducción en la cría de ovejas; Un aumento en el tamaño de la camada tiene un impacto efectivo en la rentabilidad de la industria del cordero, promoviendo el suministro de carne, leche y lana para la vida diaria en la agricultura moderna. Sin embargo, el tamaño de la camada tiene una heredabilidad baja (aproximadamente 0.1) (1), que está influenciada por muchos factores, incluido el crecimiento ovárico, el estro, la tasa de ovulación y la receptividad uterina (2, 3). Los múltiples mecanismos moleculares para el tamaño de la camada aún no se han dilucidado completamente. La mayoría de los estudios de los mecanismos genéticos subyacentes a la prolificidad en ovejas hasta ahora se han centrado en genes candidatos y sus reguladores, como lncRNA, circRNA y múltiples vías reguladas, que probablemente estén interrelacionadas, para facilitar el rendimiento reproductivo ovino (4, 5). Las ovejas prolíficas y no prolíficas se caracterizan por diferencias en el ciclo estral. Los genes PRLR (6), BMP15 (7, 8), GDF9 (9, 10), FecB (11, 12), BMPR1B (13), NTRK2 (14) y LHβ (15), así como vías moleculares cruciales como BMP/Smad (16, 17) y TGF-beta vías de señalización (18) están involucrados en el desarrollo folicular y el aumento de la tasa de ovulación y se ha demostrado que están estrechamente asociados con el rasgo de prolificidad de las ovejas. Además, los péptidos intraováricos y los factores de crecimiento también participan en la regulación de la actividad ovárica a través de vías paracrinas o autocrinas, por lo que la actividad ovárica, a su vez, determina el ciclo del estro (19).
La proteómica es una biotecnología factible y efectiva que puede proporcionar información global de proteínas que incluye estructura, abundancia, función, localización, interacción y modificación. El avance en la técnica proteómica ha mejorado la sensibilidad y precisión de la identificación y cuantificación de proteínas o péptidos en una célula, tejido u organismo. El etiquetado de etiquetas de masa en tándem (TMT) recientemente desarrollado combinado con espectrometría de masas (MS) se ha utilizado como un enfoque proteómico particularmente robusto en diferentes tipos de campos de investigación, detectando información cuantitativa de proteínas en múltiples muestras biológicas (20). Hasta la fecha, se han ejecutado reactivos TMT para analizar biomarcadores de proteínas en plasma (21) para evaluar proteínas expresadas diferencialmente entre adenoma hipofisario no funcional positivo y negativo para la hormona foliculoestimulante (FSH) en pacientes (22) e identificar biomarcadores potenciales en mastitis subclínica en vacas (23). Además, previamente demostramos algunas proteínas potenciales involucradas en los rasgos de tamaño de la camada ovina como STAR, HSD3B1 y CYP11A1 utilizando la técnica basada en TMT (24-26). Además, el monitoreo de reacciones paralelas (PRM) se ha convertido en un poderoso enfoque proteómico dirigido que se ha verificado para la cuantificación de proteínas debido a su mayor sensibilidad, resolución, precisión y reproducibilidad (27).
Small Tail Han (STH) es una notable raza ovina materna politócica en el norte de China conocida por su estro durante todo el año y su alta prolificidad, particularmente con una tasa de parto de hasta el 267.1% (26). Esta raza se ha convertido en un excelente modelo prolífico para la investigación de la alta fecundidad ovina. En este estudio, se aplicó una técnica proteómica cuantitativa basada en TMT para mapear el perfil del proteoma de los ovarios en ovejas STH con dos rasgos de fecundidad para caracterizar la abundancia de las proteínas y buscar proteínas candidatas relacionadas con la fecundidad. Además, también se realizó el ensayo PRM para verificar los resultados del TMT. La identificación de proteínas importantes combinadas con el análisis bioinformático relevante repondrá biomarcadores potenciales para revelar el mecanismo subyacente de prolificidad en ovejas.
Materiales y métodos
Declaración ética
Todos los procedimientos experimentales se realizaron siguiendo las directrices y regulaciones pertinentes establecidas por el Ministerio de Agricultura de la República Popular China. La aprobación ética sobre la supervivencia animal fue dada por el comité de ética animal del Instituto de Ciencia Animal de la Academia China de Ciencias Agrícolas (No. NIC 2019-49).
Diseño experimental y flujo de trabajo
Se utilizó la tecnología TMT para identificar y caracterizar las proteínas diferencialmente abundantes (DAPs) en los ovarios de ovejas politócicas y monotócolas en las fases lútea y folicular, respectivamente. Cada grupo de muestras biológicas consistió en tejidos ováricos aislados de tres ovejas STH. Se realizó la extracción de proteínas, el etiquetado TMT y la espectrometría de masas para la identificación de proteínas, seguido de análisis bioinformático (Figura suplementaria S1).
Agrupación de animales y recogida de muestras
Se seleccionaron ovejas STH experimentales de la granja de cría de ovejas Chenglian STH en la región suroeste de la provincia de Shandong, República Popular China. Fueron alojados en las mismas condiciones, con acceso gratuito a agua y alimentos. En primer lugar, se recogió sangre de vena yugular de ovejas sanas no preñadas (n = 890) que se utilizó para la identificación de los genotipos FecB mediante el ensayo TaqMan (28), por lo que se obtuvieron tres genotipos. Las ovejas elegidas tenían aproximadamente entre 2 y 4 años y pesaban entre 65 y 75 kg. Un total de 12 ovejas (seis politócanos con FecBBB genotipo y seis monotocosos con genotipo FecB) con índices similares de edad y peso fueron seleccionados para nuestro estudio. La sincronización del estro se realizó en todas las ovejas seleccionadas, donde se colocó un dispositivo interno controlado de liberación de fármaco (CIDR, progesterona 300 mg, Zoetis Australia Pty. Ltd., NSW, Australia) en la vagina de estas ovejas durante 12 días, y se inyectaron 5 ml de vitamina AD por vía intramuscular para proteger el epitelio vaginal. La tasa de ovulación (el número de cuerpo lúteo) se detectó mediante el procedimiento de laparoscopia después de 7 días de la extracción del CIDR (fase lútea), que es un índice clave que determina la prolificidad de las ovejas. Luego, la sincronización del estro mencionada anteriormente se realizó nuevamente después de 45 h de la extracción del CIDR, y se obtuvieron ovarios con diámetro folicular ≥ 3.5 mm (fase folicular) (12, 29). Las 12 ovejas se dividieron en cuatro grupos, incluyendo ovejas politócicas en la fase folicular (PF, n = 3), ovejas politócicas en la fase lútea (PL, n = 3), ovejas monotocous en la fase folicular (MF, n = 3) y ovejas monotocous en la fase lútea (ML, n = 3). Se recolectaron tejidos ováricos enteros frescos de 12 ovejas, se congelaron inmediatamente en nitrógeno líquido y se almacenaron a -80 ° C para experimentos proteómicos posteriores.++
Extracción de proteínas, digestión y etiquetado TMT
En primer lugar, según el método dado por Tang et al. (25), la proteína se extrajo por lisis con lisado SDT (incluyendo 4% p/v SDS, 150 mM Tris/HCl (pH8) y 100 mM DTT). Además, se agregaron 30 mg de cada tejido ovárico a 900 μL SDT; el lisado se sonilizó (80 W, 10 ciclos de 10 s con intervalos de 15 s) y luego se hirvió durante 15 min. Después de la centrifugación a 14.000 × g durante 40 min, el sobrenadante final se cuantificó mediante un ensayo de BCA (Bio-Rad, CA, Estados Unidos) y se detectó mediante SDS-PAGE (Figura suplementaria S2). La proteína se trató con el método de preparación de muestras asistida por filtro (FASP) para hidrólisis enzimática (30), y se recolectó el filtrado. La fracción peptídica se calificó a 280 nm, OD280. Después de la digestión de tripsina, las muestras de péptidos se secaron por centrifugación al vacío, luego se resuspendieron 100 μg de péptidos de cada muestra en 0,5 M TEAB. El kit de etiquetado TMT (Thermo Fisher Scientific, Waltham, MA, Estados Unidos) con las etiquetas isobáricas 126, 127, 128 y 129 se utilizó para etiquetar las muestras PF, PL, MF y ML, respectivamente.
Fraccionamiento peptídico con columna de espín de fase inversa de pH alto
La mezcla de péptidos se fraccionó utilizando el kit de fraccionamiento de fase inversa de pH alto (Thermo Fisher Scientific, Waltham, MA, Estados Unidos) de acuerdo con las instrucciones del fabricante.
Análisis LC–MS/MS
Las muestras liofilizadas se disolvieron en disolvente A (0,1% de ácido fórmico, v/v) y se cargaron en una precolumna Acclaim PepMap 100 (fase inversa C18, Thermo Fisher) a un caudal de 300 nL/min y luego se separaron en una columna analítica Acclaim pepMap RSLC (fase inversa C18, Thermo Fisher). El gradiente consistió en un aumento del disolvente B (84% de acetonitrilo con 0,1% de ácido fórmico) de la siguiente manera: 0-35% durante 50 min, 35-100% durante 5 min y 100% durante 5 min. El análisis LC-MS/MS se realizó en un espectrómetro de masas Q Exactive que se acopló a un sistema Easy-nLC 1.000 UPLC (Thermo Fisher Scientific, Waltham, MA, Estados Unidos) durante 60 min. Se detectaron iones positivos, y los parámetros del escaneo de iones precursores variaron de 300 a 1.800 m/z. La resolución primaria de MS fue de 70.000 a 200 m/z, el control automático de ganancia (AGC) fue de 3e6, el tiempo máximo de inyección fue de 10 ms y la duración de la exclusión dinámica fue de 40,0 s. La resolución secundaria de MS fue de 17.500 a 200 m/z, el tiempo máximo de inyección fue de 1,60 ms y la energía de colisión normalizada fue de 30 eV, con una relación de llenado insuficiente del 0,1%.
Búsqueda en bases de datos de EM y detección de DAP
Los datos sin procesar de MS se generaron en formato RAW y se buscaron utilizando la base de datos (uniprot_Ovis_aries_27500_20170904.fasta), que se convirtió al formato de archivo de formato genérico de mascota (MGF) utilizando Proteome Discoverer 1.4 (Thermo Fisher Scientific, 2012) y se recuperó a través del servidor Mascot 2.21 Identificar y cuantificar proteínas. Los parámetros relacionados fueron los siguientes: máximo de escisiones perdidas = 2; modificaciones fijas = carbamidometilo (C), TMT-6 plex (N-término) y TMT-6 plex (K); modificaciones variables = Oxidación (M) y TMT-4 plex (Y); tolerancia a la masa de iones peptídicos = ± 20 ppm; tolerancia de masa de iones de fragmento = 0,1 Da; base de datos = uniprot_Ovis_aries_27500_20170904.fasta, patrón de base de datos = Señuelo; y la tasa de falso descubrimiento de péptidos (FDR) ≤0,01 como punto de corte para obtener resultados cualitativos altamente confiables. Las proporciones de proteínas se calcularon como la mediana de solo péptidos únicos de la proteína, todas las proporciones de péptidos se normalizaron por la proporción mediana de proteínas y la proporción mediana de proteínas debe ser una después de la normalización. Los valores de p de proteínas de confianza calculados con la prueba t de Student mostraron diferencias significativas (cambio de pliegue >1,2 o < 0,833; valor p ajustado <0,05) entre los grupos.
Verificación de la monitorización de reacciones paralelas
Para validar los datos proteómicos obtenidos por el análisis TMT, se utilizó el método PRM (una técnica basada en el análisis de EM) para determinar los niveles de abundancia de DAP seleccionados relacionados con la función ovárica (péptidos únicos ≥2, cambio de pliegue (FC) >1.2 o < 0.833) (31). Después de la hidrólisis enzimática, los péptidos fueron desalados, liofilizados y redisueltos en ácido fórmico al 0,1%. La concentración de los péptidos fue determinada por OD280. La información de los péptidos adecuada para el análisis PRM se importó al software Xcalibur™ (Thermo Fisher Scientific) para la configuración PRM. Cada muestra contenía péptidos de 1 μg, y se añadieron 200 péptidos estándar de fmol (PRTC: GILFVGSGVSGGEEGAR) para la separación cromatográfica utilizando el sistema HPLC. Se utilizó Q-Exactive HF MS (Thermo Fisher Scientific) para el análisis PRM/MS durante 60 min y se detectaron iones positivos. Todas las exploraciones PRM se realizaron de acuerdo con la lista de inclusión después de cada exploración de espectro de masa completo de primer orden. Los datos brutos de LC-PRM/MS se analizaron utilizando el software Skyline (MacCoss Lab, Universidad de Washington) (32).
Análisis bioinformático
Enriquecimiento GO2 el análisis para identificar la proteína se realizó utilizando el software Blast2GO, herramienta BLAST3 como base de datos de secuencias de proteínas, mapeo, anotación y aumento de anotaciones. Se utilizó el software KAAS para anotar la vía KEGG,4 mapeo de DAP a la base de datos de genes KEGG; las proteínas mapeadas se clasificaron según la ortología KEGG. Las vías GO y KEGG se ejecutaron utilizando la prueba exacta de Fisher (20). Además, las redes de interacción proteína-proteína (PPI) de DAP fueron realizadas por STRING5 combinado con el software Cytoscape para intentar capturar la información de interacción de proteínas subyacente.
Resultados
Análisis de datos de EM
Los tejidos ováricos de dos grupos de comparación se aislaron y agruparon para la extracción de proteínas, el etiquetado de TMT y el análisis de EM. Un total de 167.020 números de coincidencia del espectro peptídico y 4.749 péptidos únicos se obtuvieron en este estudio (Tabla Suplementaria S1), que se mapearon a 5.237 proteínas, en las que 732 no estaban caracterizadas y 4.505 eran funciones conocidas. Una lista completa de las proteínas y péptidos detectados en el ovario, con el número de acceso UniProt, el nombre de la proteína y el número de puntuación de péptidos y aminoácidos únicos, el peso molecular y la cobertura de la secuencia se muestran en la Tabla Suplementaria S2. Entre estos resultados, la mayoría de las distribuciones de los pesos moleculares de las proteínas (MW) variaron de 10 a 200 kDa (Figura 1A), y los valores del punto isoeléctrico (pI) variaron de 4 a 10 (Figura 1B). Más de la mitad de la cobertura de la secuencia de proteínas fue del ≤15% (Figura 1C). La mayoría de las proteínas identificadas contenían más de uno o incluso hasta 201 péptidos únicos, en los que el 29,4% de las proteínas tenían un solo péptido (Figura 1D).
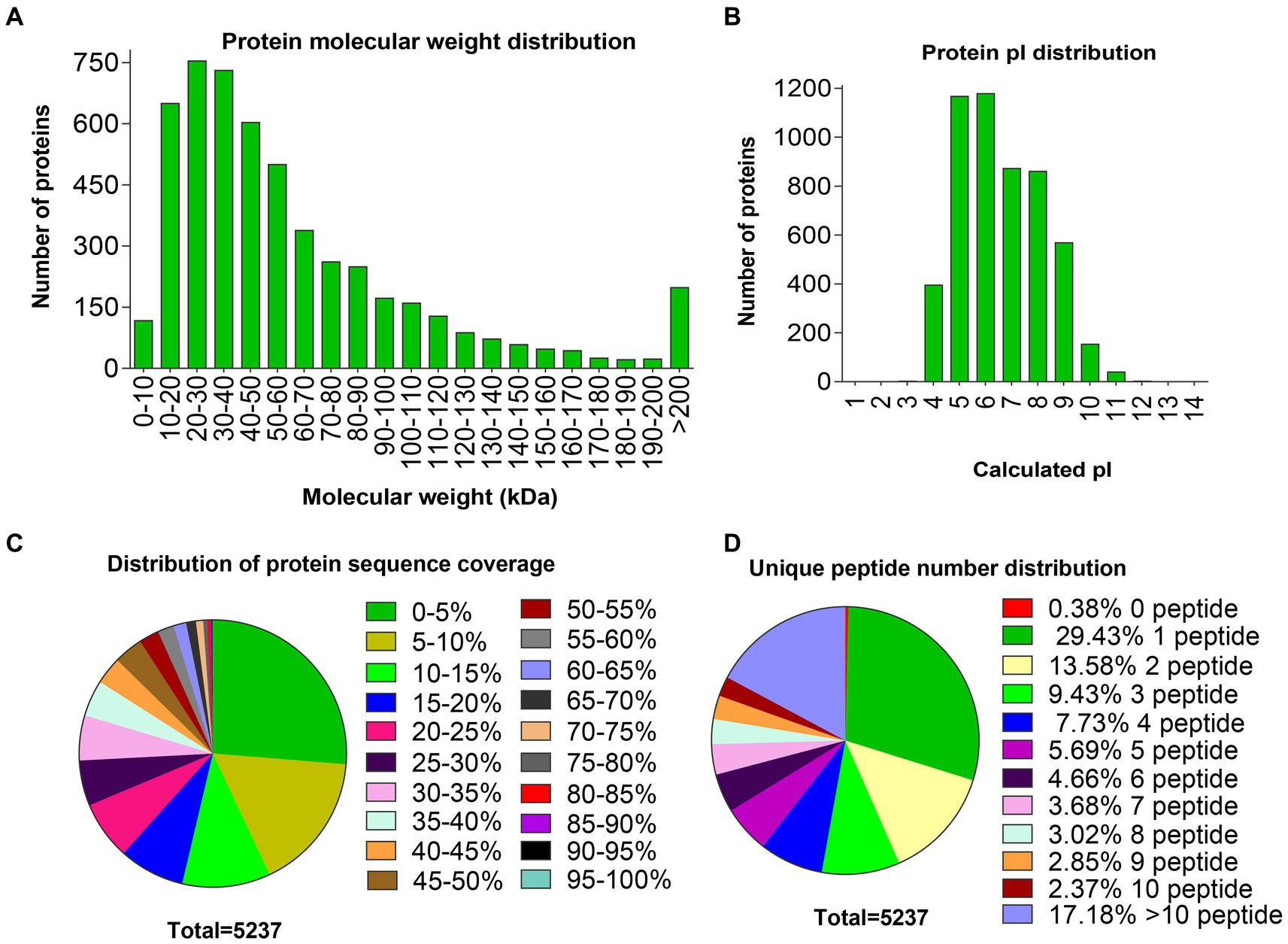 Figura 1. Distribuciones de peso molecular, pI, cobertura de secuencia y número de péptidos únicos de proteínas identificadas en los ovarios. (A) Distribución de pesos moleculares de proteínas. (B) Distribución de los valores de pI de la proteína. (C) Distribución de la cobertura de la secuencia de proteínas. (D) Distribución de números únicos de péptidos.
Figura 1. Distribuciones de peso molecular, pI, cobertura de secuencia y número de péptidos únicos de proteínas identificadas en los ovarios. (A) Distribución de pesos moleculares de proteínas. (B) Distribución de los valores de pI de la proteína. (C) Distribución de la cobertura de la secuencia de proteínas. (D) Distribución de números únicos de péptidos.
Identificación de proteínas diferencialmente abundantes
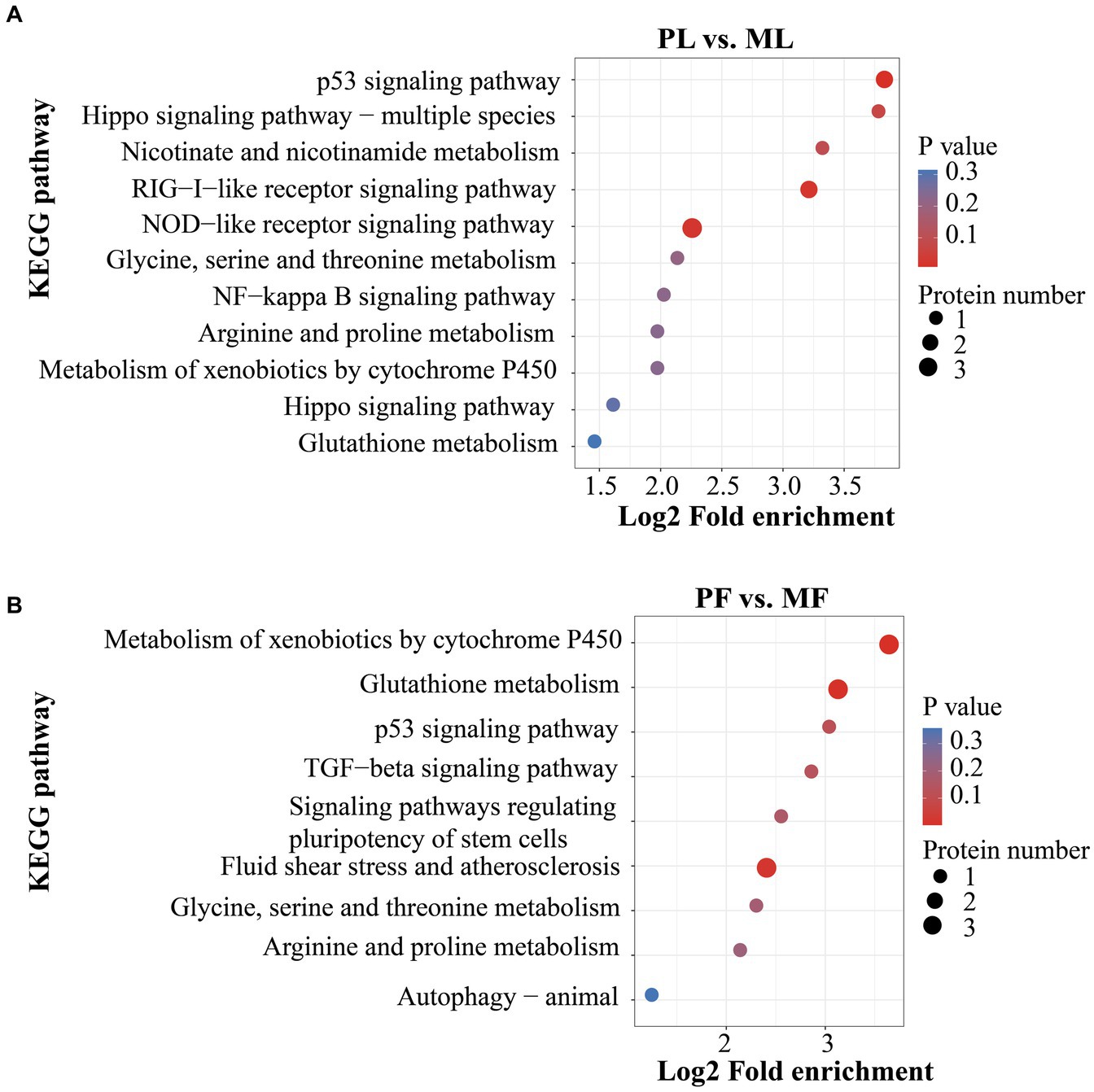
Los DAP entre los dos grupos de comparación (PL vs. ML y PF vs. MF) se clasificaron según proteínas con FC > 1,20 y p < 0,05. Hubo 49 DAP (9 regulados al alza y 40 regulados a la baja) y 44 DAP (18 regulados al alza y 26 regulados a la baja) en los grupos de comparación de PL vs. ML y PF vs. MF, respectivamente (Figura 2B; Cuadro suplementario S3). En la fase lútea, las proteínas diferencialmente abundantes, como la proteína 1 de unión a ARN asociada a gránulos citotóxicos TIA1 (TIAL1), la nicotinamida fosforribosiltransferasa (NAMPT) y la proteína 1 de unión al ácido retinoico celular (CRABP1) fueron mayores en PL que en ML, mientras que la proteína de unión al componente 4 del complemento alfa (C4BPA), el gen 17 estimulado por interferón (ISG17), la proteína 1 asociada a sí (YAP1), la globulina fijadora de hormonas sexuales (SHBG), y la proteína no caracterizada (W5P9M5) fueron menores en PL que en ML. En la fase folicular, las proteínas diferencialmente abundantes, incluyendo TIAL1, proteína no caracterizada (W5PG55), inhibina beta-A-subunidad (A2ICA4) y mu cristalina (CRYM) fueron mayores en PF que en MF, mientras que la proteína de membrana lisosomal asociada 1 (LAMP1), la cadena alfa 1 de colágeno tipo XII (COL12A1) y la molécula CD74 (CD74) fueron menores en PF que en MF. El análisis de agrupamiento jerárquico de los DAP se muestra en la Figura 3; la similitud de los patrones de abundancia de proteínas dentro de cada grupo fue relativamente alta, mientras que fue relativamente baja entre los grupos politocosos y monotocosos, y los grupos estaban claramente divididos, lo que sugirió la racionalidad de los DAP en este experimento.
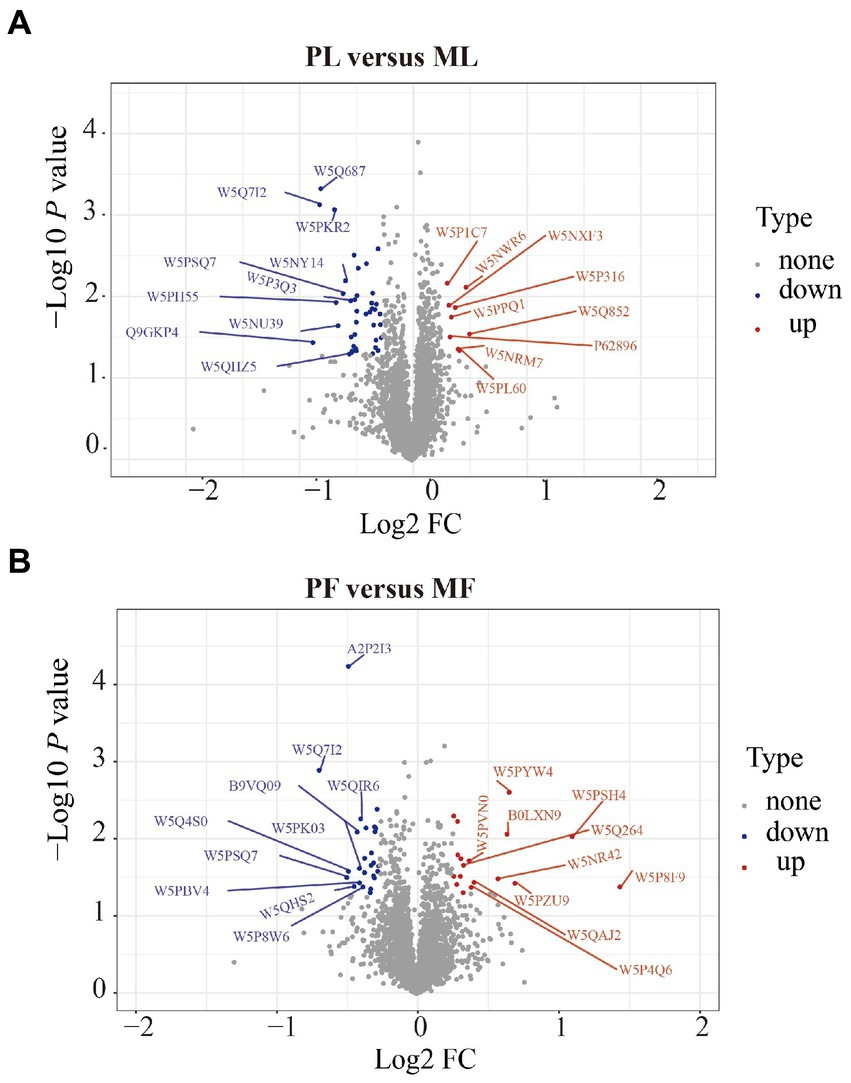 Figura 2. Diagramas volcánicos de DAPs en PL vs. ML (A), y en PF vs. MF (B). Donde los círculos rojos en el lado positivo representan proteínas reguladas al alza y los del lado negativo representan proteínas reguladas a la baja.
Figura 2. Diagramas volcánicos de DAPs en PL vs. ML (A), y en PF vs. MF (B). Donde los círculos rojos en el lado positivo representan proteínas reguladas al alza y los del lado negativo representan proteínas reguladas a la baja.
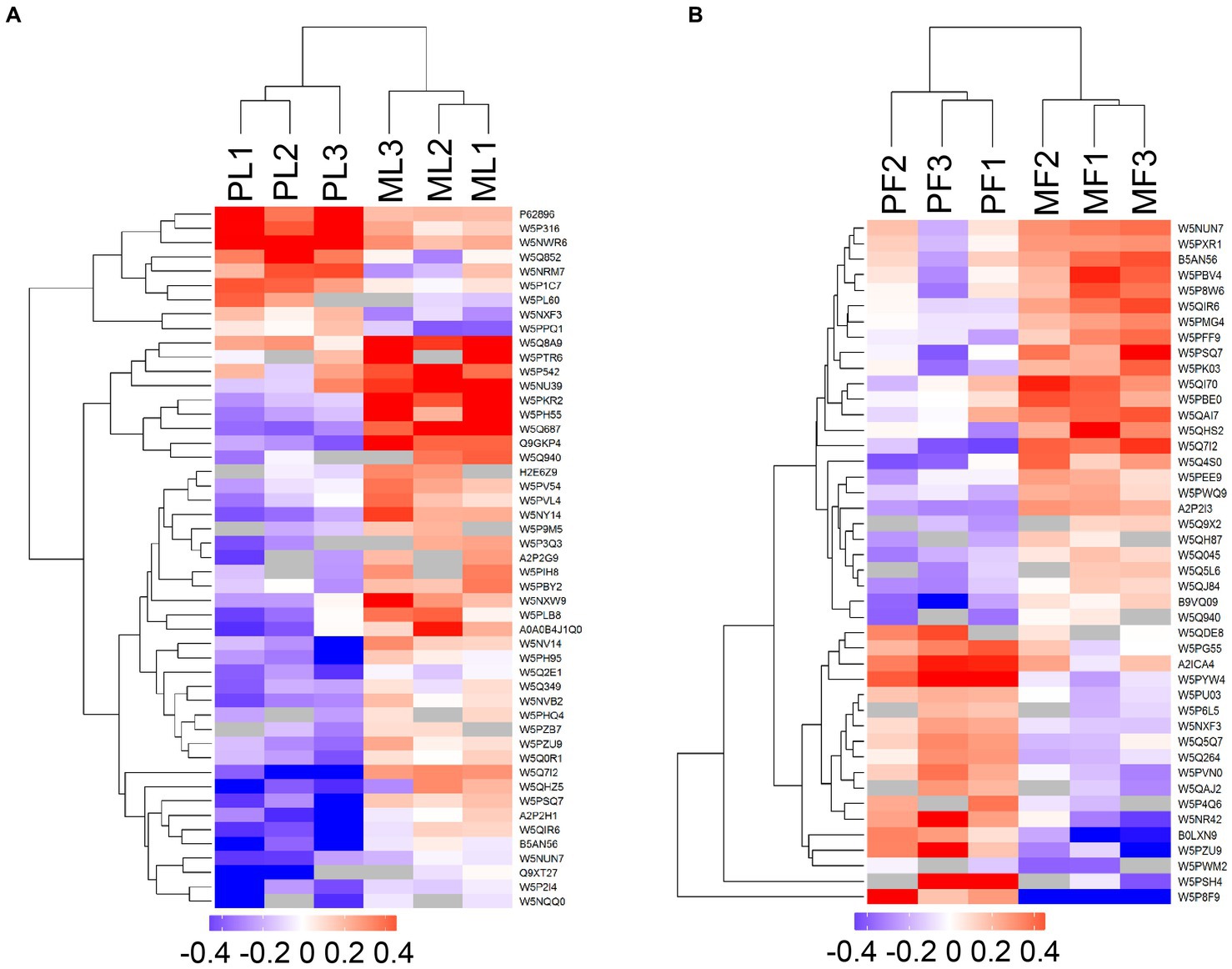 Figura 3. Mapas de calor de DAPs en PL vs. ML (A), y PF vs. MF (B). Donde los tonos rojo y azul representan proteínas significativamente reguladas al alza y a la baja, respectivamente, y las áreas grises indican que no hay información cuantitativa sobre las proteínas.
Figura 3. Mapas de calor de DAPs en PL vs. ML (A), y PF vs. MF (B). Donde los tonos rojo y azul representan proteínas significativamente reguladas al alza y a la baja, respectivamente, y las áreas grises indican que no hay información cuantitativa sobre las proteínas.
Análisis de enriquecimiento funcional de DAPs
Para identificar las posibles funciones de los DAP en los dos grupos de comparación, se realizaron análisis de enriquecimiento de los términos GO y las vías KEGG. En general, el análisis GO contiene tres ontologías significativas de funciones biológicas: proceso biológico (BP), componente celular (CC) y función molecular (MF). Entre ellos, a cada DAP se le asignó más de un término; los 20 términos GO enriquecidos relacionados con la reproducción se muestran en la Figura 4. En el grupo PL vs. ML, los términos PA incluyeron regulación positiva de la señalización, proceso reproductivo y reproducción sexual (Figura 4A; Tabla Suplementaria S4), así como proteínas predominantemente enriquecidas incluyendo TIAL1, NAMPT, CRABP1, C4BPA, ISG17, W5P9M5 y proteínas SHBG, mientras que la generación de gametos, el desarrollo embrionario, el proceso reproductivo y el proceso celular involucrados en la reproducción en organismos multicelulares se enriquecieron en PF vs. MF (Figura 4B), incluidas las proteínas de TIAL1, W5PG55, LAMP1 y A2ICA4.
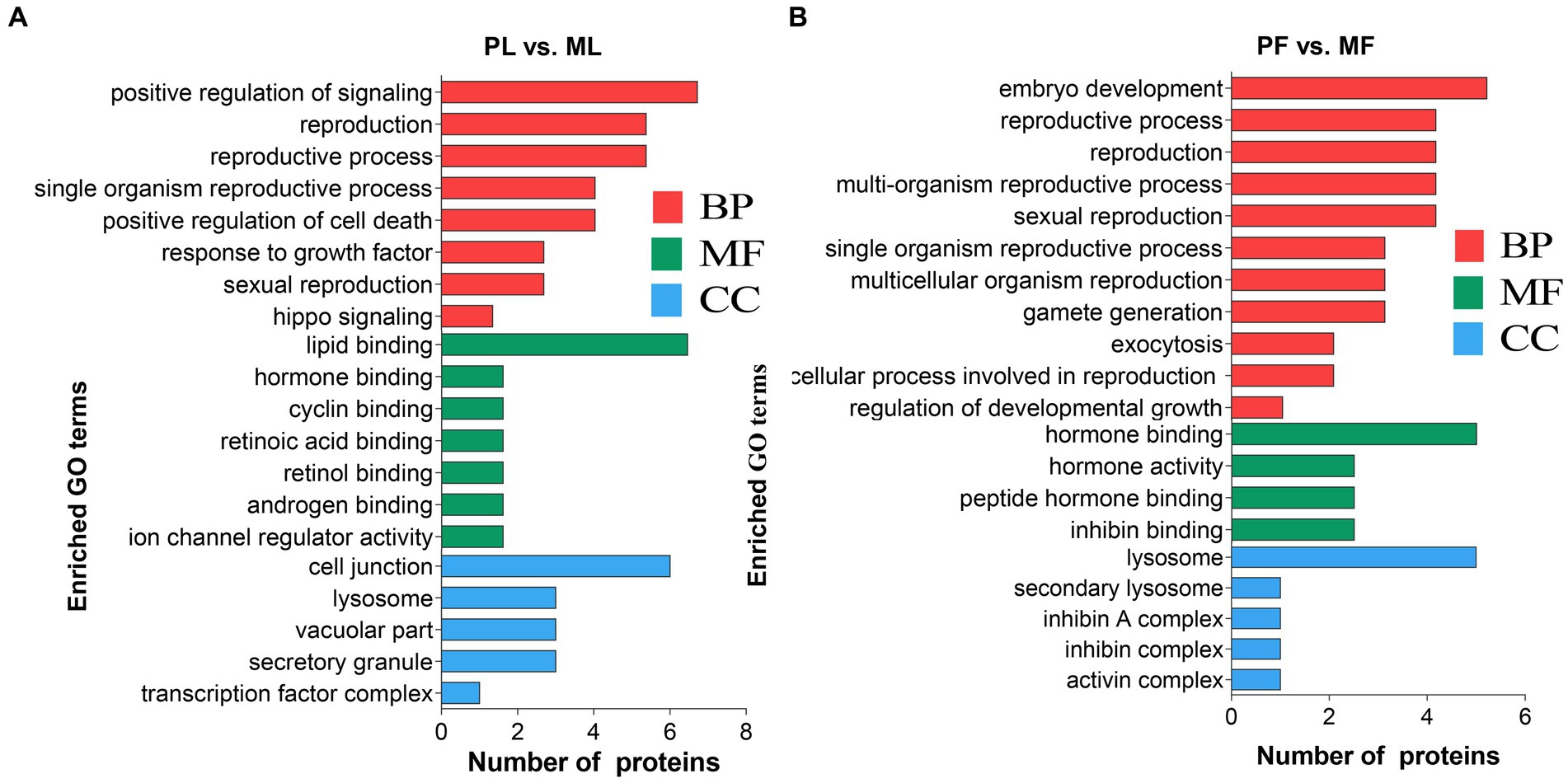 Figura 4. Los 20 términos enriquecidos de GO relacionados con la reproducción concluyen el proceso biológico (BP), la función molecular (MF) y el componente celular (CC) en PL vs. ML (A) y PF vs. MF (B).
Figura 4. Los 20 términos enriquecidos de GO relacionados con la reproducción concluyen el proceso biológico (BP), la función molecular (MF) y el componente celular (CC) en PL vs. ML (A) y PF vs. MF (B).
Además, los DAP en los grupos de comparación PL vs. ML y PF vs. MF se enriquecieron en 64 y 42 vías, respectivamente (Tabla Suplementaria S5). p < 0,05 se estableció como punto de corte para un análisis adicional del enriquecimiento. En la fase lútea, varias vías relacionadas con la función ovárica se enriquecieron significativamente, como el metabolismo del nicotinato y la nicotinamida (NAMPT), la señalización del hipopótamo (W5P9M5), la señalización del receptor similar a NOD (NAMPT, W5NY14 y W5PH55) y las vías de señalización del receptor similar a RIG-I (ISG17 y DDX58) (Figura 5A). En la fase folicular, las vías relacionadas con el desarrollo folicular se enriquecieron significativamente, incluyendo la señalización de TGF-beta y las vías de señalización que regulan la pluripotencia de las células madre (A2ICA4) (Figura 5B). En general, los análisis de enriquecimiento de GO y KEGG proporcionaron pistas cruciales sobre los mecanismos moleculares subyacentes a la prolificidad de las ovejas.
Análisis de red PPI de DAPs
Utilizando la base de datos STRING y el software Cytoscape (v.3.9.1), construimos una red PPI para los DAP. Los datos de STRING se muestran en la Tabla Suplementaria S6, y la intermediación se seleccionó durante el análisis de CytoNCA. Nuestros resultados mostraron que algunos DAP estaban involucrados en el IBP, y se encontraron proteínas de centro. Es importante destacar que YAP1 y NAMPT interactuaron con otros DAP en PL vs. ML (Figura 6A), mientras que COL12A1, LAMP1 y CD74 interactuaron con otros DAP en PF vs. MF (Figura 6B), respectivamente.
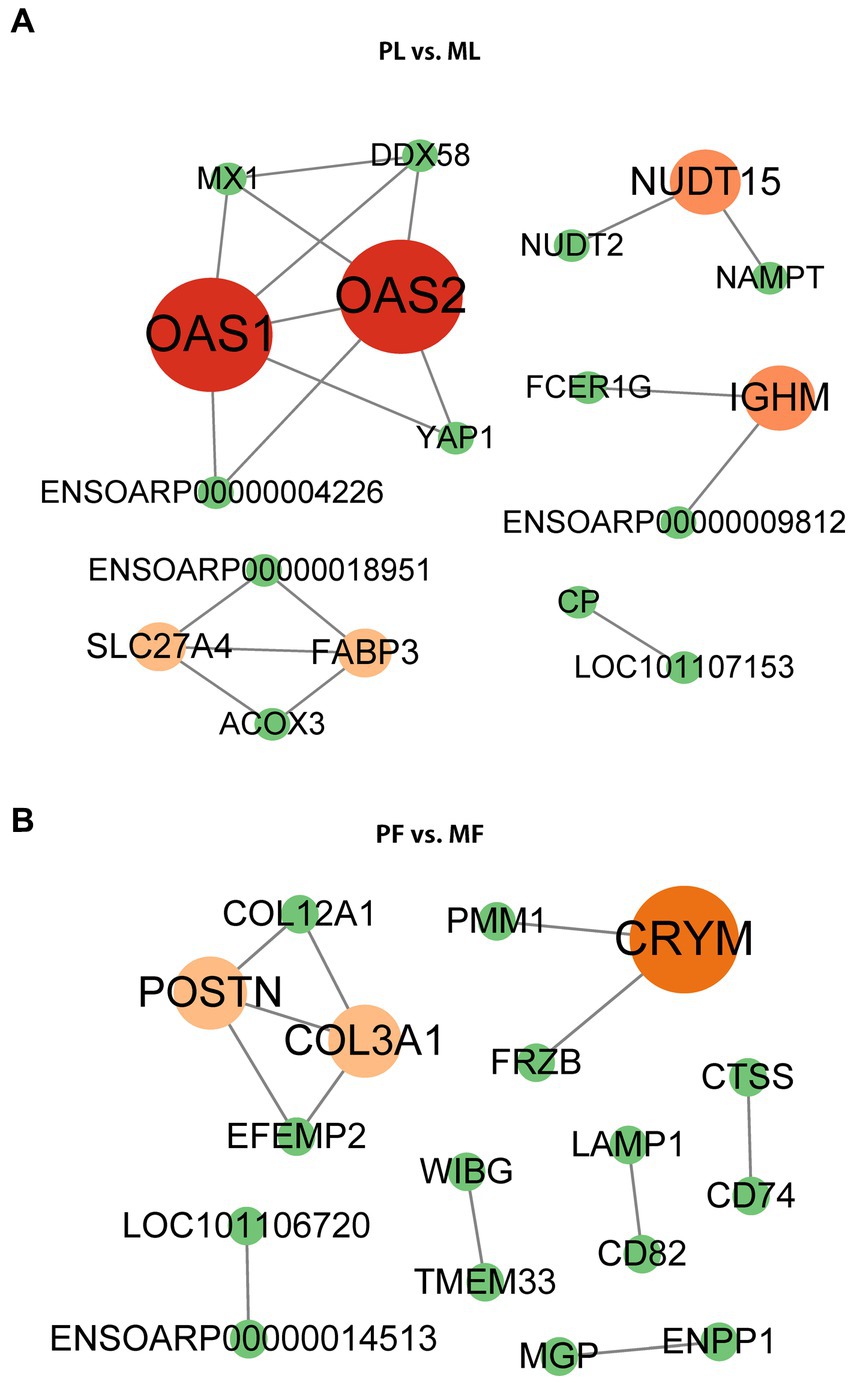 Figura 6. Red PPI de DAPs en PL vs. ML (A) y PF vs. MF (B). Diferentes colores representan los valores de puntuación de intermediación de las proteínas en el proceso de análisis utilizando el cistoscopio (v.3.9.1).
Figura 6. Red PPI de DAPs en PL vs. ML (A) y PF vs. MF (B). Diferentes colores representan los valores de puntuación de intermediación de las proteínas en el proceso de análisis utilizando el cistoscopio (v.3.9.1).
Validación PRM de resultados basados en TMT
Se seleccionaron seis DAP (NAMPT, CRABP1, TIAL1 e ISG17 en PL vs. ML, y W5PG55, TIAL1 y LAMP1 en PF vs. MF) que contienen al menos dos péptidos únicos para su verificación mediante análisis cuantitativo PRM. Entre los DAP, FC de NAMPT, CRABP1, TIAL1 y W5PG55 estuvieron por encima de 1,2, mientras que ISG17 y LAMP1 estuvieron por debajo de 0,833. La tendencia de abundancia de PRM fue consistente con la de los resultados basados en TMT, lo que indicó la credibilidad de nuestros datos proteómicos (Figura 7; Cuadro suplementario S7).
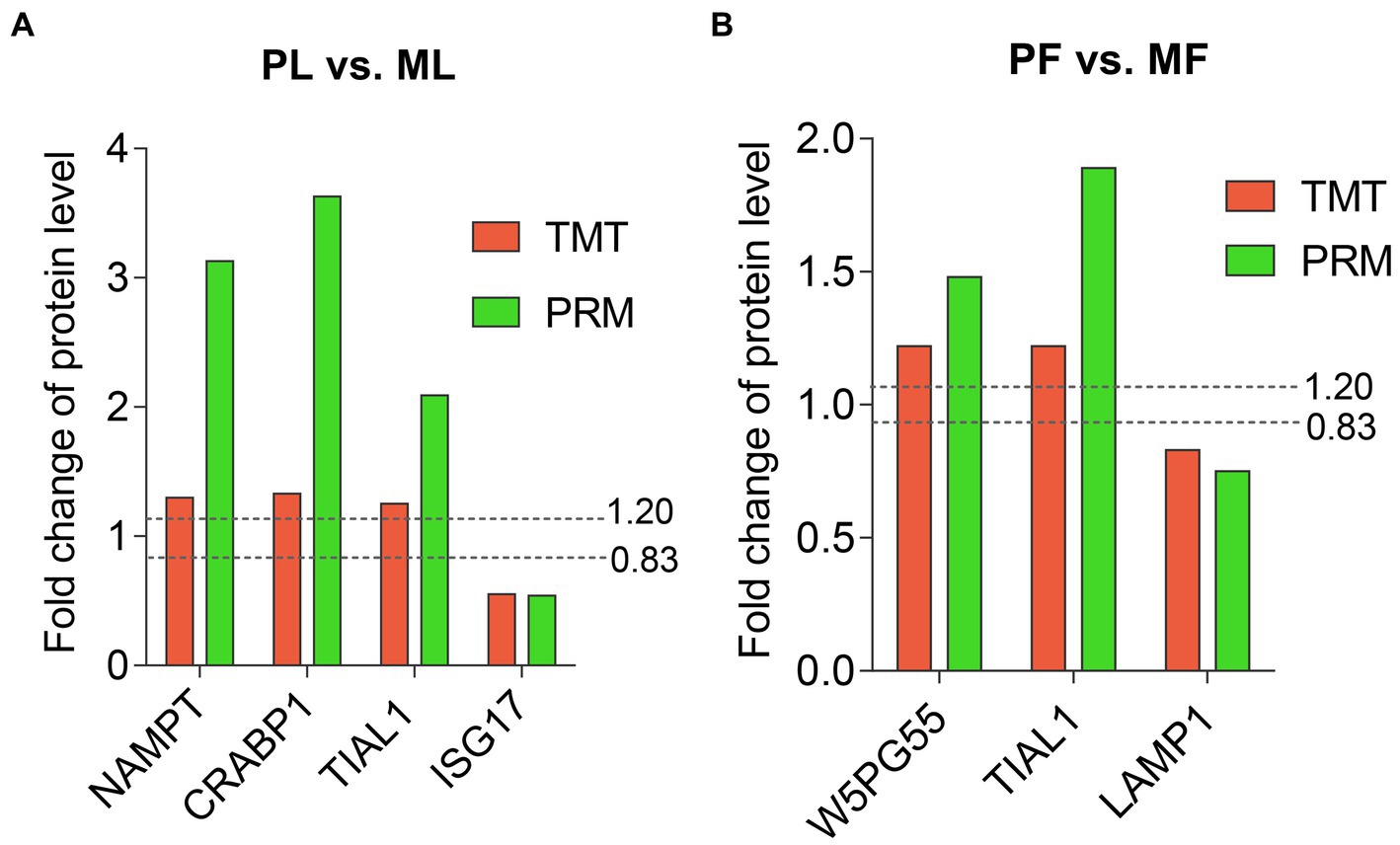 Figura 7. Patrones abundantes de seis DAP utilizando análisis TMT y validación PRM en PL vs. ML (A), y en PF vs. MF (B). Dos líneas punteadas representan FC = 1.2 (regulación ascendente) y FC = 0.833 (regulación descendente).
Figura 7. Patrones abundantes de seis DAP utilizando análisis TMT y validación PRM en PL vs. ML (A), y en PF vs. MF (B). Dos líneas punteadas representan FC = 1.2 (regulación ascendente) y FC = 0.833 (regulación descendente).
Discusión
La reproducción es un proceso extraordinariamente complejo con una regulación coordinada en múltiples órganos reproductivos y niveles moleculares. En particular, el mecanismo molecular preciso de la reproducción de las ovejas no está completamente aclarado. Como uno de los tejidos reproductivos directos, el ovario apoya el desarrollo de folículos y ovocitos (33). La investigación de este tejido de ovejas en diferentes fases de celo podría proporcionar información potencial sobre una mejora adicional del tamaño de la camada (34). En este estudio, la secuenciación de alto rendimiento del proteoma en ovarios involucrados en los rasgos de prolificidad de las ovejas STH se realizó en diferentes fases de estro. La adquisición de los DAP ováricos entre grupos politócicos y monotocosos podría participar en la regulación molecular de la prolificidad ovina.
En la comparación de PL vs. ML, el análisis de enriquecimiento funcional de proteínas diferenciales importantes reveló que la nicotinamida fosforribosil transferasa (NAMPT), el gen 17 estimulado por interferón (ISG17) y W5P9M5 se asociaron con el rasgo de prolificidad. Como hormona citoquina y enzima limitante de la velocidad, NAMPT está implicada en la mediación del metabolismo energético, la apoptosis y la biosíntesis de nicotinamida adenina dinucleótido celular (NAD), que es una coenzima crucial para las deshidrogenasas en el metabolismo celular (35). En un mamífero, NAMPT generalmente coopera con SIRT1 para regular los niveles intracelulares de NAD y, por lo tanto, pone a disposición la energía celular, lo cual es importante para la supervivencia de las células placentarias y un embarazo exitoso (36). Además, NAMPT aumenta la esteroidogénesis inducida por el factor de crecimiento similar a la insulina 1 en las células primarias de la granulosa del ovario humano (37) y mejora la calidad de los ovocitos y la fertilidad durante la superovulación de ratas (38). La proteína ISG17 en el útero ovino responde al interferón-tau (IFNtau), que es una señal importante para el reconocimiento materno del embarazo (MRP). MRP podría facilitar la transición de la ciclicidad al embarazo (39, 40). Una alta abundancia de ISG17 en el ovario de ovejas monotocous sugirió que el ISG17 puede regular la función del útero con IFNtau como un efector negativo aguas abajo. Además, encontramos que W5P9M5 estaba codificado por el gen YAP1 y significativamente enriquecido en la vía de señalización del hipopótamo. La investigación ha demostrado que la vía del hipopótamo se conserva e implica en la correlación espacio-temporal con el tamaño de la reserva de folículos primordiales (41), así como en el control del crecimiento folicular ovárico y la maduración de los ovocitos (42). YAP1 es uno de los componentes de la vía del hipopótamo, y su sobreexpresión puede mejorar el síndrome de ovario poliquístico (SOP), que es una etiología de la oligoovulación.++
Informes anteriores han demostrado que TIA1 cytotoxic granule-associated RNA binding protein-like 1 (TIAL1) se expresó en células germinales primordiales y desempeña un papel en el desarrollo de espermatogonias u oogonia (43), así como un marcador de gránulos de estrés para responder a condiciones de estrés como la infección por virus del síndrome respiratorio y reproductivo porcino, protegiendo la reproducción de la cerda y la salud de los jóvenes (44). Nuestro estudio reveló que TIAL1 era muy abundante en los ovarios de ovejas politócicas y se asociaba con gránulos de estrés citoplasmático y la regulación de los procesos celulares. La proteína 1 de unión al ácido retinoico celular (CRABP1) podría inhibir directamente el Ca endógeno2+/proteína quinasa II dependiente de calmodulina (CaMKII) compitiendo con la calmodulina (45). Si bien CaMKII es un regulador clave de los eventos de activación del óvulo, como la finalización de la meiosis y la progresión a la interfase embrionaria, su actividad contribuye al bloqueo de la membrana para prevenir la fertilización por polispermia (46). La regulación positiva de CRABP1 en ovejas politócicas implicó además su efecto inhibitorio sobre CaMKII. Además, el enriquecimiento de A2ICA4 en los ovarios PF estuvo involucrado principalmente en la vía de señalización que regula la pluripotencia de las células madre y la regulación positiva de la secreción de la hormona foliculoestimulante. A2ICA4 también estimula la producción de SMAD2/SMAD3, que es la señalización SMAD aguas abajo de TGF-β1 (47) y participa en el desarrollo gonadal, la diferenciación embrionaria y la formación de placenta. La proteína de membrana asociada a lisosomas 1 (LAMP1) se expresa en tejidos reproductivos como los ovarios y el útero en humanos y está involucrada en la estabilidad lisosomal y la autofagia (48). Una mayor abundancia y enriquecimiento funcional de LAMP1 en los ovarios MF de ovejas confirmó aún más el papel de esta proteína en la mediación de la respuesta de supervivencia de las células reproductivas, como la autofagia durante la fase folicular. Curiosamente, nuestro estudio también reveló que W5PG55 se asoció con el desarrollo, crecimiento y reproducción de la estructura anatómica durante la fase folicular, que está codificada por el gen SELENOP. El SELENOP tiene un dominio funcional de tiorredoxina que contiene selenocisteína N-terminal, que es indicativo de su potencial función redox (49). La investigación ha demostrado que las proteínas importantes que controlan la condición oxidativa en el líquido folicular pueden regular el crecimiento del folículo y la maduración de los ovocitos (50). Estos sugieren el potencial efecto antioxidante de la región extracelular o proteína secretada W5PG55.
Conclusión
En general, los cambios en los componentes proteicos de los ovarios ovinos en dos grupos de comparación se investigaron mediante proteómica cuantitativa basada en TMT, y los DAP se caracterizaron aún más mediante análisis bioinformáticos. Los niveles de abundancia de proteínas en el ovario están asociados con su estado fisiológico. En total, se identificaron 49 y 44 DAP en PL vs. ML y PF vs. MF, respectivamente. La regulación positiva de TIAL1, NAMPT y CRABP1 en PL vs. ML, y TIAL1, A2ICA4 y W5PG55 en PF vs. MF podría modular positivamente la reproducción en ovejas politócas. Estas proteínas son de importancia sustancial para el estudio en profundidad del mecanismo genético de la prolificidad ovina.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio / repositorios y el número (s) de acceso se pueden encontrar en el artículo / material complementario.
Declaración ética
El estudio en animales fue revisado y aprobado por el Departamento de Investigación Científica (a cargo de los temas de bienestar animal) del Instituto de Ciencia Animal de la Academia China de Ciencias Agrícolas (IAS-CAAS).
Contribuciones del autor
CL analizó los datos, escribió y revisó el manuscrito. MZ diseñó y realizó el experimento. XH y RD prepararon las muestras. ZZ y CR investigaron los recursos. QL diseñó el experimento y revisó el manuscrito. MC administró el proyecto y revisó el manuscrito final. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Este trabajo fue apoyado por la Fundación Nacional de Ciencias Naturales de China (32172704), el Programa de Innovación en Ciencia y Tecnología Agrícola de China (CAAS-ZDRW202106 y ASTIP-IAS13), el Fondo Asignado para el Sistema de Investigación Agrícola de China (CARS-38) y el Fondo de Investigación Basal de la Institución Científica Central de Interés Público (Y2017JC24).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
El revisor YaL declaró una afiliación compartida con los autores ZZ y CR al editor de manejo en el momento de la revisión.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1096762/full#supplementary-material
Notas
1. ^http://www.matrixscience.com/
2. ^http://www.ebi.ac.uk/interpro/
3. ^https://blast.ncbi.nlm.nih.gov/
4. ^http://www.genome.jp/kaas-bin/kaasmain
5. ^https://cn.string-db.org/
Referencias
1. Notter, DR. Aspectos genéticos de la reproducción en ovejas. Reprod Domest Anim. (2010) 43:122–8. DOI: 10.1111/J.1439-0531
2. Li, XY, Li, CY, Xu, YR, Yao, R, Li, HX, Ni, W, et al. El análisis de la transcriptómica hipofisaria indica que los lncRNAs están involucrados en la regulación del estro ovino. Genómica de Funct Integr. (2020) 20:563–73. DOI: 10.1007/S10142-020-00735-Y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Gao, XX, Yao, XL, Li, XD, Liang, YX, Liu, ZF, Wang, ZB, et al. Funciones de WNT6 en la progresión del ciclo celular epitelial endometrial de ovejas y organogénesis de las glándulas uterinas. Veterinario Sci. (2021) 8:316. DOI: 10.3390/VETSci8120316
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Chang, C, He, XY, Di, R, Wang, XY, Han, MC, Liang, C, et al. El perfil transcriptómico tiroideo revela la regulación diferencial de la fase folicular de lncRNA y ARNm relacionada con la prolificidad en ovejas Han de cola pequeña con dos genotipos FecB. Genes (Basilea). (2022) 13:849. doi: 10.3390/genes13050849
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Sadeghi, M, Bahrami, A, Hasankhani, A, Kioumarsi, H, Nouralizadeh, R, Abdulkareem, SA, et al. Red LncRNA-miRNA-Mrna ceRNA implicada en la prolificidad ovina: un enfoque integrado. Genes (Basilea). (2022) 13:1295–360. doi: 10.3390/genes13081295
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Chu, MX, Mu, YL, Fang, L, Ye, SC, and Sun, SH. Receptor de prolactina como gen candidato para la prolificidad de ovejas Han de cola pequeña. Anim Biotechnol. (2007) 18:65–73. DOI: 10.1080/10495390601090950
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Chu, MX, Liu, ZH, Jiao, CJ, He, YQ, Fang, L, Ye, SC, et al. Las mutaciones en los genes BMPR-IB y BMP-15 están asociadas con el tamaño de la camada en ovejas Han de cola pequeña (Ovis aries). J Anim Sci. (2007) 85:598–603. DOI: 10.2527/JAS.2006-324
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Di, R, Wang, FY, Yu, P, Wang, XY, He, XY, Mwacharo, JM, et al. Detección de nuevas variaciones relacionadas con el tamaño de la camada en el gen BMP15 de ovejas cordero luzhong (Ovis aries). Animales (Basilea). (2021) 11:3528. DOI: 10.3390/ani11123528
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Chu, MX, Yang, J, Feng, T, Cao, GL, Fang, L, Di, R, et al. GDF9 como gen candidato para la prolificidad de ovejas Han de cola pequeña. Mol Biol Rep. (2011) 38:5199–204. DOI: 10.1007/S11033-010-0670-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Vague, DI, Husdal, M, Kent, MP, Klemetsdal, G y Boman, IA. Una mutación sin sentido en el factor de diferenciación de crecimiento 9 (GDF9) está fuertemente asociada con el tamaño de la camada en ovejas. BMC Genet. (2013) 14:1–8. DOI: 10.1186/1471-2156-14-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Miao, XY, Luo, QM, Zhao, HJ y Qin, XY. El estudio proteómico ovárico revela el posible mecanismo molecular para la hiperprolificidad de las ovejas Han de cola pequeña. Sci Rep. (2016) 6:27606–37. DOI: 10.1038/SEP27606
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Guo, XF, Wang, XY, Di, R, Liu, QY, Hu, WP, He, XY, et al. Efectos metabólicos del gen FecB sobre el líquido folicular y el suero de las venas ováricas en ovejas (Ovis aries). Int J Mol Sci. (2018) 19:539. doi: 10.3390/ijms19020539
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Tang, JS, Hu, WP, Di, R, Liu, QY, Wang, XY, Zhang, XS, et al. Análisis de expresión de los genes candidatos prolíficos, BMPR1B, BMP15 y GDF9 en ovejas Han de cola pequeña con tres genotipos de fecundidad (gen FecB). Animales (Basilea). (2018) 8:166. doi: 10.3390/ani8100166
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Esmaeili-Fard, SM, Gholizadeh, M, Hafezian, SH, and Abdollahi-Arpanahi, R. Genome-wide association study and pathway analysis identify NTRK2 as a novel candidate gene for litter size in sheep. PLoS One. (2021) 16:E0244408. doi: 10.1371/journal.pone.0244408
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Wang, WM, La, YF, Li, F, Liu, SJ, Pan, XY, Li, C, et al. Caracterización molecular y perfiles de expresión del gen LHβ ovino y su asociación con el tamaño de la camada en ovejas Han de cola pequeña indígenas chinas. Animales (Basilea). (2020) 10:460. DOI: 10.3390/ANI10030460
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Ruoss, C, Tadros, A, Oshea, T, Mc-Farlane, J, and Almahbobi, G. Ovarian follicle development in Booroola sheep exhibiting impaired bone morphogenetic protein signalling pathway. Reproducción. (2009) 138:689–96. doi: 10.1530/REP-09-0190
17. Xu, YF, Li, EL, Han, YD, Chen, L, and Xie, Z. Differential expression of mRNAs encoding BMP/Smad pathway molecules in antral follicles of high- and low-fecundity Hu sheep. Anim Reprod Sci. (2010) 120:47–55. doi: 10.1016/j.anireprosci.2010.02.009
18. Bodin, L, Pasquale, ED, Fabre, S, Bontoux, M, Monget, P, Persani, L, et al. Una nueva mutación en el gen de la proteína morfogenética ósea 15 que causa la secreción defectuosa de proteínas se asocia tanto con una mayor tasa de ovulación como con esterilidad en las ovejas Lacacune. Endocrinología. (2007) 148:393–400. doi: 10.1210/es.2006-0764
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Jones, ASK, y Shikanov, A. Follicle development as an orchestrated signaling network in a 3D organoid. J Biol Ing. (2019) 13:2–13. DOI: 10.1186/S13036-018-0134-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Zhang, ZB, Tang, JS, Di, R, Liu, QY, Wang, XY, Gan, SQ, et al. Identificación de proteínas expresadas diferencialmente relacionadas con la prolificidad del hipotálamo de ovejas (Ovis aries) mediante proteómica comparativa. Proteómica. (2019) 19:E1900118. doi: 10.1002/pmic.201900118
21. Robert, M, Santosh, DB, David, RG, and Riitta, L. Analysis of the plasma proteome using iTRAQ and TMT-based isobaric labeling. Espectro de masas Rev. (2018) 37:583–606. DOI: 10.1002/MAS.21550
22. Wang, Y, Cheng, TT, Lu, ML, Mu, Y, Li, B, Li, XJ, et al. La proteómica cuantitativa basada en TMT reveló caracterizaciones moleculares relacionadas con la hormona foliculoestimulante (FSH) para la evaluación potencialmente pronóstica y el tratamiento personalizado de adenomas hipofisarios no funcionales positivos para FSH. EPMA J. (2019) 10:395–414. DOI: 10.1007/S13167-019-00187-W
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Shveta, B, Anil, S, Sudarshan, K, Shivam, KD, Smaranika, P, Preeti, R, et al. La proteómica cuantitativa basada en la etiqueta de masa en tándem (TMT) revela objetivos potenciales asociados con la aparición de mastitis subclínica en vacas. Sci Rep. (2020) 10:3921–55. DOI: 10.1038/S41598-020-66211-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Tang, JS, Hu, WP, Chen, S, Di, R, Liu, QY, Wang, XY, et al. El mecanismo genético de alta prolificidad en ovejas Han de cola pequeña por proteómica comparativa de ovarios en las etapas folicular y lútea. J Proteoma. (2019) 204:103394. doi: 10.1016/j.jprot.2019.103394
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. La, YF, Tang, JS, Guo, XF, Zhang, LP, Gan, SQ, Zhang, XS, et al. El análisis proteómico del útero ovino revela su papel en la prolificidad. J Proteoma. (2020) 210:103526. doi: 10.1016/j.jprot.2019.103526
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Hu, WP, Liu, MQ, Tian, ZL, Liu, QY, Zhang, ZB, Tang, JS, et al. Análisis de polimorfismo, expresión y estructura de genes clave en la vía de la esteroidogénesis ovárica en ovejas (Ovis aries). Vet Med Sci. (2021) 7:1303–15. DOI: 10.1002/VMS3.485
27. Vidova, V, y Spacil, Z. Una revisión sobre la proteómica cuantitativa basada en espectrometría de masas: adquisición dirigida e independiente de los datos. Anal Chim Acta. (2017) 964:7–23. doi: 10.1016/j.aca.2017.01.059
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Zhang, ZB, Tang, JS, Di, R, Liu, QY, Wang, XY, Gan, SQ, et al. El perfil integrado del transcriptoma hipotalámico revela las funciones reproductivas de los ARNm y los miARN en las ovejas. Genet frontal. (2020) 10:1296. doi: 10.3389/fgene.2019.01296
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Li, CY, He, XY, Zhang, ZJ, Ren, CH y Chu, MX. El perfil transcriptómico de la glándula pineal revela la regulación diferencial de lncRNA y mRNA relacionada con la prolificidad en ovejas STH con dos genotipos FecB. Datos de BMC Genom. (2021) 22:9. DOI: 10.1186/S12863-020-00957-W
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Wisniewski, JR, Zougman, A, Nagaraj, N, y Mann, M. Universal sample preparation method for proteome analysis. Métodos Nat. (2009) 6:359–62. DOI: 10.1038/NMETH.1322
31. Xue, T, Ping, L, Yong, Z, Liu, K, Li, Y, Moritz, RL, et al. El microambiente fenotípico «agudo» inducido por interleucina-6 promueve la inmunidad antitumoral Th1 en la terapia criotérmica revelada por la proteómica de monitoreo de reacción paralela y escopeta. Teranósticos. (2016) 6:773–94. doi: 10.7150/thno.14394
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Maclean, B, Tomazela, DM, Shulman, N, Chambers, M, Finney, GL, Frewen, B, et al. Skyline: un editor de documentos de código abierto para crear y analizar experimentos proteómicos específicos. Bioinformática. (2010) 26:966–8. DOI: 10.1093/Bioinformatics/BTQ054
33. Briley, SM, Jasti, S, Mccracken, JM, Hornick, JE, Fegley, B, Pritchard, MT, et al. Fibrosis asociada a la edad reproductiva en el estroma del ovario de mamífero. Reproducción. (2016) 152:245–60. doi: 10.1530/REP-16-0129
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Liu, YF, Zhou, ZY, He, XY, Jiang, YT, Ouyang, YN, Hong, QH, et al. Firmas de perfil de ARN circular expresadas diferencialmente identificadas en el rasgo de prolificidad del ovario de cabra negra de Yunshang en el ciclo de celo. Frente Physiol. (2022) 13:820459. DOI: 10.3389/FPHYS.2022.820459
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Ocon-Grove, OM, Krzysik-Walker, SM, Maddineni, SR, Hendricks, GL, and Ramachandran, R. NAMPT (visfatin) in the chicken testis: influence of sexual maturation on cellular localization, plasma levels and gene and protein expression. Reproducción. (2010) 139:217–26. doi: 10.1530/REP-08-0377
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Broady, AJ, Loichinger, MH, Ahn, HJ, Davy, PMC, Allsopp, RC, y Bryant-Greenwood, GD. Proteínas protectoras y longitud de los telómeros en placentas de pacientes con preeclampsia en el último trimestre de gestación. Placenta. (2017) 50:44–52. doi: 10.1016/j.placenta.2016.12.018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Reverchon, M, Cornuau, M, Cloix, L, Rame, C, Guerif, F, Royere, D, et al. La visfatina se expresa en las células de la granulosa humana: regulación por metformina a través de las vías AMPK / SIRT1 y su papel en la esteroidogénesis. Mol Hum Reprod. (2013) 19:313–26. DOI: 10.1093/molehr/gat002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Choi, KH, Joo, BS, Sun, ST, Park, MJ, Son, JB, Joo, JK, et al. La administración de visfatina durante la superovulación mejora la capacidad de desarrollo de los ovocitos y el potencial de fertilidad en ratones hembra de edad avanzada. Esterilidad fertilizante. (2012) 97:1234–1241.e3. doi: 10.1016/j.fertnstert.2012.02.032
39. Pru, JK, Austin, KJ, Perry, DJ, Nighswonger, AM, y Hansen, TR. Production, purification, and carboxy-terminal sequencing of bioactive recombinant bovine interferon-stimulated gene product 17. Biol Reprod. (2000) 63:619–28. doi: 10.1095/biolreprod63.2.619
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Der-Weijden, VAV, Puntar, B, Vegas, AR, Milojevic, V, Schanzenbach, CI, Kowalewski, MP, et al. Las células epiteliales luminales endometriales detectan el alargamiento embrionario en el corzo independiente del interferón-tau. Biol Reprod. (2019) 101:882–92. doi: 10.1093/biolre/ioz129
41. Xiang, C, Li, J, Hu, LL, Huang, J, Luo, T, Zhong, ZS, et al. La vía de señalización del hipopótamo revela una correlación espacio-temporal con el tamaño del folículo primordial en ratones. Fisioquímica Celular. (2015) 35:957–68. DOI: 10.1159/000369752
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Kawamura, K, Cheng, Y, Suzuki, N, Deguchi, M, Sato, Y, Takae, S, et al. Interrupción de la señalización del hipopótamo y estimulación Akt de los folículos ováricos para el tratamiento de la infertilidad. Proc Natl Acad Sci U S A. (2013) 110:17474–9. doi: 10.1073/PNAS.1312830110
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Beck, AR, Miller, IJ, Anderson, P, y Streuli, M. La proteína de unión al ARN TIAR es esencial para el desarrollo de las células germinales primordiales. Proc Natl Acad Sci U S A. (1998) 95:2331–6. DOI: 10.1073/PNAS.95.5.2331
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Catanzaro, N, y Meng, XJ. Los gránulos de estrés inducidos por el virus del síndrome reproductivo y respiratorio porcino (PRRSV) se asocian con complejos de replicación viral y supresión de la traducción del huésped. Virus Res. (2019) 265:47–56. doi: 10.1016/j.virusres.2019.02.016
45. Park, SW, Persaud, SD, Ogokeh, S, Meyers, TA, Townsend, DW y Wei, LN. CRABP1 protege el corazón de la remodelación aguda y crónica inducida por isoproterenol. J Endocrinol. (2018) 236:151–65. doi: 10.1530/JOE-17-0613
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Gardner, AJ, Knott, JG, Jones, KT, y Evans, JP. CaMKII puede participar pero no es suficiente para el establecimiento del bloqueo de membrana a la polispermia en óvulos de ratón. J Cell Physiol. (2007) 212:275–80. doi: 10.1002/jcp.21046
47. Zhang, ZH, Li, S, Deng, J, Yang, SR, Xiang, ZW, Guo, HY, et al. La aspirina inhibe la fibrosis endometrial al suprimir la vía TGF-β1-Smad2/Smad3 en las adherencias intrauterinas. Int J Mol Med. (2020) 45:1351–60. doi: 10.3892/ijmm.2020.4506
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Ma, Y, Liu, HH, Du, XD, Petlulu, P, Chen, XH, Wang, R, et al. IRE1 y CaMKKβ pathways to reveal the mechanism involved in microcystin-LR-induced autophagy in mouse ovarian cells. Alimentos Químicos Toxicol. (2021) 147:111911. doi: 10.1016/j.fct.2020.111911
49. Qazi, IH, Angel, C, Yang, HX, Zoidis, E, Pan, B, Wu, ZZ, et al. Papel del selenio y las selenoproteínas en la función reproductiva masculina: una revisión de evidencias pasadas y presentes. Antioxidantes (Basilea). (2019) 8:268–301. DOI: 10.3390/antiox8080268
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Freitas, C, Neto, AC, Matos, L, Silva, E, Ribeiro, A, Silva-Carvalho, JL, et al. Afectación redox del líquido folicular para el crecimiento del folículo ovárico. J Ovarian Res. (2017) 10:44–53. DOI: 10.1186/S13048-017-0342-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: ovejas, prolificidad, ovarios, etiqueta de masa en tándem, proteína
Cita: Li C, Zhou M, He X, Di R, Zhang Z, Ren C, Liu Q y Chu M (2023) La proteómica comparativa de los ovarios elucidó los objetivos potenciales relacionados con la prolificación ovina. Frente. Vet. Sci. 10:1096762. doi: 10.3389/fvets.2023.1096762
Recibido: 12 de noviembre de 2022; Aprobado: 02 Agosto 2023;
Publicado: 22 agosto 2023.
Editado por:
Kangfeng Jiang, Universidad Agrícola de Yunnan, China
Revisado por:
Ya Liu, Universidad Agrícola de Anhui, China Yong Liu, Universidad Normal de Fuyang, China Hui Li, Universidad de Guangxi, China
Derechos de autor © 2023 Li, Zhou, He, Di, Zhang, Ren, Liu y Chu. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Mingxing Chu, mxchu@263.net; Qiuyue Liu, liuqiuyue@caas.cn
†Estos autores han contribuido igualmente a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía