Micobacterias no tuberculosas: presencia en reactores bovinos de prueba cutánea de rebaños oficiales libres de tuberculosis
 Micobacterias no tuberculosas: presencia en reactores bovinos de prueba cutánea de rebaños oficiales libres de tuberculosis
Micobacterias no tuberculosas: presencia en reactores bovinos de prueba cutánea de rebaños oficiales libres de tuberculosis
 Alberto Gómez-Buendía1,2
Alberto Gómez-Buendía1,2  Julio Álvarez1,2
Julio Álvarez1,2  Javier bezos1,2
Javier bezos1,2  Jorge Mourelo3
Jorge Mourelo3  Javier Amado4
Javier Amado4  José Luis Sáez5
José Luis Sáez5  Lucía de Juan1,2
Lucía de Juan1,2  Beatriz Romero1,2*
Beatriz Romero1,2*- 1Centro de Vigilancia Sanitaria VISAVET, Universidad Complutense de Madrid, Madrid, España
- 2Departamento de Sanidad Animal, Facultad de Veterinaria, Universidad Complutense de Madrid, Madrid, España
- 3Servicio de Sanidad Animal, Xunta de Galicia, Consellería de Medio Rural, Santiago de Compostela, España
- 4Servicio de Laboratorio de Sanidad Animal y Vegetal, Dirección General de Ganadería y Sanidad Agraria, Consejería de Medio Rural y Política Agraria, Principado de Asturias, España
- 5Subdirección General de Sanidad e Higiene Animal y Trazabilidad, Dirección General de la Producción Agraria, Ministerio de Agricultura, Pesca y Alimentación, Madrid, España
Las micobacterias no tuberculosas (MNT) se consideran una causa relevante de reacciones inespecíficas a la prueba de tuberculosis bovina (TBb) más aplicada, la prueba de tuberculina intradérmica. Con el fin de establecer qué especies de MNT podrían actuar como fuente potencial de dicha interferencia diagnóstica, se identificó a nivel de especie una colección de 373 aislamientos obtenidos de vacas positivas en pruebas cutáneas de 359 rebaños oficialmente libres de tuberculosis (OTF), sacrificados en el marco de la campaña de erradicación de la tuberculosis bovina en España, mediante PCR y secuenciación Sanger del ADNr 16S. Genes hsp65 y rpoB. De los 308 aislamientos para los que se logró una identificación confiable, se identificaron 32 especies micobacterianas diferentes, siendo algunas las especies más representadas: entre los miembros del complejo M. avium (n = 142, 46,1%), M. avium subsp. hominissuis (98; 69,0%) fue el más abundante, seguido de M. avium subsp. avium (33, 23,2%) y M. intracellulare (7, 4,9%). Entre los miembros no MAC (n = 166, 53,9%), las especies predominantes fueron M. nonchromogenicum (85; 27,6%) y M. bourgelatii (11; 5,6%). Además, se obtuvieron resultados mixtos en 53 aislamientos que presentaban hasta 30 genotipos diferentes, lo que podría ser indicativo de nuevas especies de micobacterias. Nuestros resultados representan un primer paso hacia la caracterización de la diversidad de especies de MNT que podrían interferir con las pruebas diagnósticas oficiales para la erradicación de la tuberculosis bovina en España.
1 Introducción
El género Mycobacterium es muy grande, abarcando 196 taxones hijos diferentes con nombres válidamente publicados y descritos correctamente (1). Las micobacterias pueden asumir diferentes funciones: hay patógenos animales y humanos de gran relevancia, como M. bovis y M. caprae, miembros del complejo Mycobacterium tuberculosis (MTC) y los agentes causales más comunes de la tuberculosis bovina (TBB), mientras que otros, como las micobacterias no tuberculosas (MNT), suelen ser microorganismos de vida libre ampliamente distribuidos en el medio ambiente. Estas MNT pueden encontrarse en el suelo, el agua, el polvo, etc. (2), pero en ciertos casos, generalmente ligados a procesos inmunosupresores, pueden infectar a humanos y animales y actuar como patógenos oportunistas (3). En bovinos, la infección por MNT no se ha asociado tradicionalmente con la presencia de signos clínicos [con la notable excepción de M. paratuberculosis (Map), el agente causal de la paratuberculosis], aunque se ha descrito la presencia de lesiones granulomatosas como consecuencia de la infección (4-7).
Desde el inicio del establecimiento de programas de erradicación de la tuberculosis bovina en diferentes regiones del mundo a principios de 1900, varios países han tenido éxito en la erradicación de la enfermedad (8). En otros, sin embargo, a pesar de todos los esfuerzos realizados, la enfermedad sigue siendo endémica (9). El fracaso de estos programas se ha atribuido en parte a las limitaciones en la sensibilidad y especificidad de las pruebas diagnósticas existentes. Entre los factores que comprometen el desempeño de estas pruebas, las MNA se han relacionado repetidamente con la ocurrencia de reacciones inespecíficas en las pruebas de tuberculina intradérmica única y comparativa (SIT y CIT), las principales herramientas diagnósticas utilizadas como base de los programas de erradicación a nivel mundial (10).
La mayoría de los estudios que evalúan el papel de la MNT en los problemas diagnósticos de TBb se han centrado en los producidos por la PAM, mostrando que puede afectar tanto a la especificidad como a la sensibilidad (11-14). Sin embargo, las MNT distintas de la PMA, también conocidas como atípicas o ambientales, también pueden afectar a la fiabilidad de las pruebas diagnósticas de tuberculosis bovina debido a la aparición de reacciones cruzadas en animales libres de tuberculosis (15, 16), mientras que su impacto en la sensibilidad de las pruebas aún no se ha cuantificado. Varios estudios han identificado qué especies pueden asociarse más comúnmente con estas reacciones cruzadas en las pruebas diagnósticas de tuberculosis bovina en diferentes entornos, con M. Las subespecies avium y M. nonchromogenicum son las micobacterias más comúnmente recuperadas de muestras bovinas (15, 17-19). Sin embargo, no todas las muestras evaluadas en estos estudios procedían de animales con resultados positivos en las pruebas o de granjas oficialmente libres de tuberculosis (OTF). Por ejemplo, en España, un estudio reciente que describe la presencia de MNT en animales incluyó solo tres bovinos de reactor de rebaños OTF, con otros aislados procedentes de rebaños en los que la enfermedad estaba presente, por lo que el papel de estas especies bacterianas en la aparición de reacciones cruzadas no está claro (19).
Por lo tanto, el objetivo de este estudio fue identificar las especies asociadas a reacciones inespecíficas detectadas en el marco de la campaña de erradicación de la TBb en España en una gran cohorte (n = 373) de bovinos de rebaños de OTF ubicados en áreas con diferente prevalencia de la enfermedad. Esta información puede ser útil para comprender mejor las especies primarias que afectan a la especificidad de las pruebas cutáneas de tuberculosis bovina y para evaluar la variabilidad geográfica en su aparición.
2 Materiales y métodos
Se evaluaron todos los aislamientos de MNT disponibles en el Centro de Vigilancia Sanitaria VISAVET recuperados entre 2011 y 2020 de bovinos positivos en pruebas cutáneas localizados en 359 rebaños de OTF detectados en el marco del programa español de erradicación. Los aislamientos se cultivaron directamente en VISAVET a partir de muestras de ganado recogidas en el matadero y analizadas en el laboratorio de dicho matadero o se enviaron a laboratorios veterinarios oficiales ubicados en dos regiones de OTF (Asturias y Galicia, 72,1% de los rebaños), y el resto de los animales muestreados procedían de diferentes puntos del país (Figura 1). Los laboratorios veterinarios enviaron los aislamientos a VISAVET para su identificación una vez que se confirmó que no pertenecían al complejo M. tuberculosis, excepto en la región de OTF de Asturias, que en su mayoría presentó aislamientos ya identificados como pertenecientes al complejo M. avium (MAC).
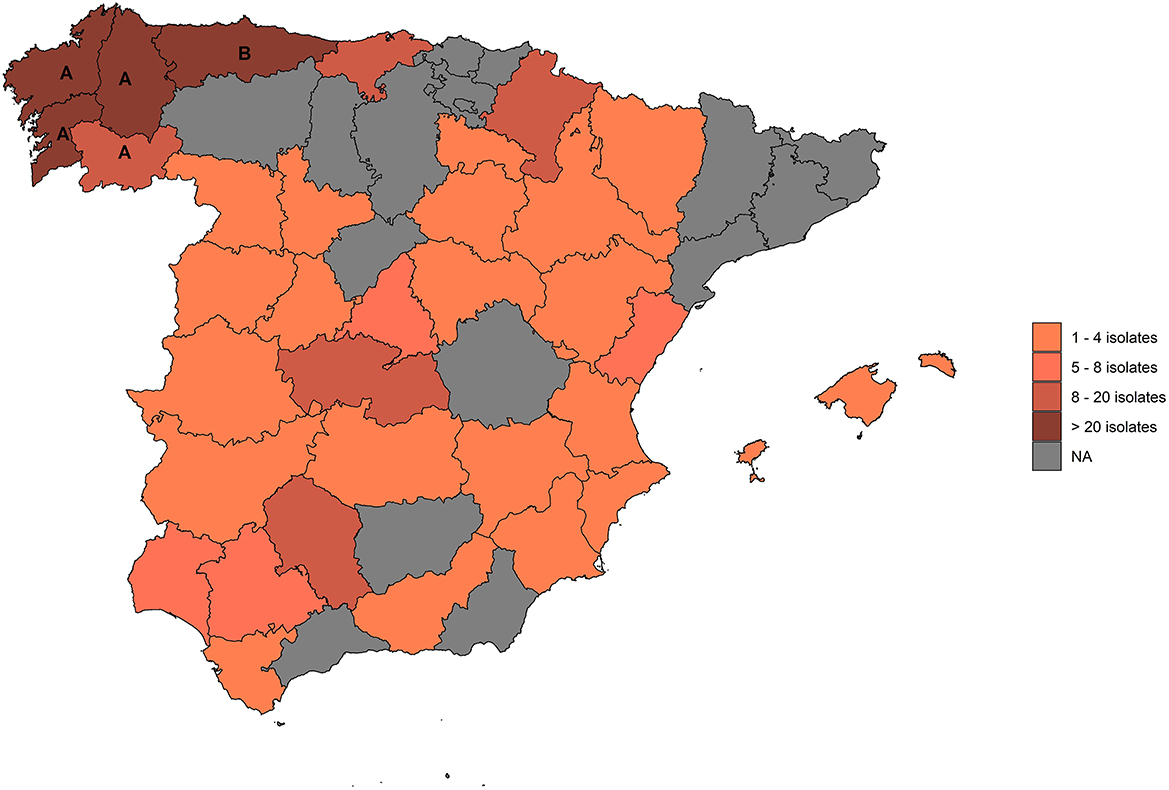 Figura 1. Provincias de las que procedían los aislamientos incluidos en este estudio. (A): Galicia; (B): Asturias.
Figura 1. Provincias de las que procedían los aislamientos incluidos en este estudio. (A): Galicia; (B): Asturias.
De acuerdo con el programa de erradicación, cuando se identifica un animal positivo en una prueba cutánea en un rebaño de OTF, el animal debe ser sacrificado en un plazo máximo de 15 días y se recogen muestras de tejido en el matadero y se envían a un laboratorio autorizado para aislar o detectar en las muestras la presencia de miembros del MTC mediante bacteriología (20) y, más recientemente, PCR directa (21). El análisis bacteriológico de estas muestras puede conducir ocasionalmente a la identificación de especies de MNT, que se identifican como tales en el laboratorio de acuerdo con el siguiente protocolo (Figura 2): una vez que se observa el crecimiento, se extrae el ADN y se somete a PCR multiplex para la detección de un fragmento de ADN específico de Mycobacterium (secuencia de ADNr 16S) (22) y de una diana específica de MTC (gen mpb70) (22). Si no se detecta la presencia de un miembro del CMT, se realiza otra PCR dirigida a un ADNr 16S específico de MAC (22), seguida en caso de resultado positivo de dos PCR dirigidas a las secuencias IS901 e IS1245 asociadas a M. avium subsp. avium (Maa) (23) y M. avium subsp. hominissuis (Mah) (24). La colección de cepas NTM disponibles incluyó todos los aislados identificados como Mycobacterium spp. sobre la base de la detección del fragmento de Mycobacterium de ADNr 16S pero que no pertenecía al MTC sobre la base de la ausencia del gen mpb70 que se había cultivado inicialmente en VISAVET o que se cultivaron en otros laboratorios autorizados y luego se enviaron a VISAVET para su posterior identificación. Los aislados no pertenecientes a MAC según la metodología explicada anteriormente se sometieron a amplificación por PCR seguida de secuenciación Sanger de varias dianas: en primer lugar, se generaron secuencias de fragmentos parciales de los genes 16S rDNA (22) y hsp65 (fragmento Telenti) (25, 26). Las secuencias directas e inversas se seleccionaron utilizando el software BioEdit (27) y se combinaron para producir una secuencia de consenso que luego se examinó utilizando la Herramienta de Búsqueda de Alineación Local Básica (BLAST) del NCBI para identificar las especies bacterianas. Se consideró que la identificación era fiable si la similitud de la secuencia y la cobertura con una diana eran >99 y 100%, respectivamente (28). Si el análisis de ADNr/hsp65 16S arrojó una identificación no confiable (debido a la baja similitud/cobertura y/o desacuerdo en las especies identificadas a través de cada diana), se realizó la amplificación y secuenciación de fragmentos del gen rpoB (29) y luego de la región 3′ del gen hsp65 (30) como se describió anteriormente. El flujo de trabajo completo se muestra en la Figura 2. Previo al cultivo bacteriológico, las muestras fueron evaluadas para detectar la presencia de lesiones macroscópicas compatibles con TBBb (31).
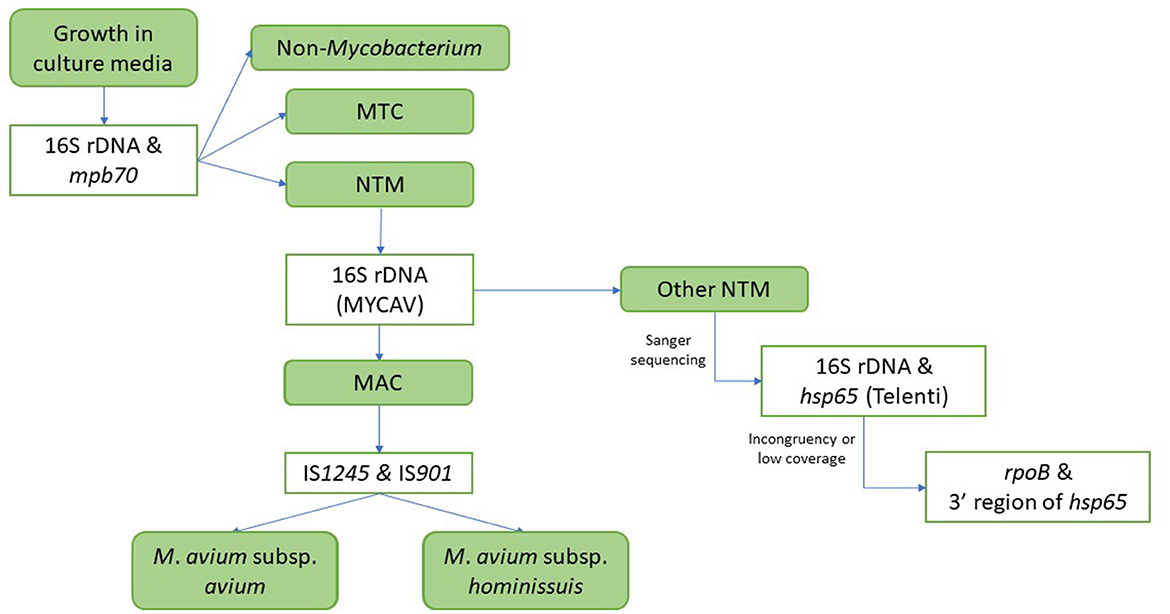 Figura 2. Flujo de trabajo para la identificación de aislados. MTC: complejo Mycobacterium tuberculosis; MNT: micobacterias no tuberculosas; MAC, complejo Mycobacterium avium; MYCAV, PCR de ADNr 16S específica para MAC.
Figura 2. Flujo de trabajo para la identificación de aislados. MTC: complejo Mycobacterium tuberculosis; MNT: micobacterias no tuberculosas; MAC, complejo Mycobacterium avium; MYCAV, PCR de ADNr 16S específica para MAC.
Todas las imprimaciones utilizadas se muestran en la Tabla Suplementaria 1. La secuenciación fue realizada por STABvida (Lisboa, Portugal).
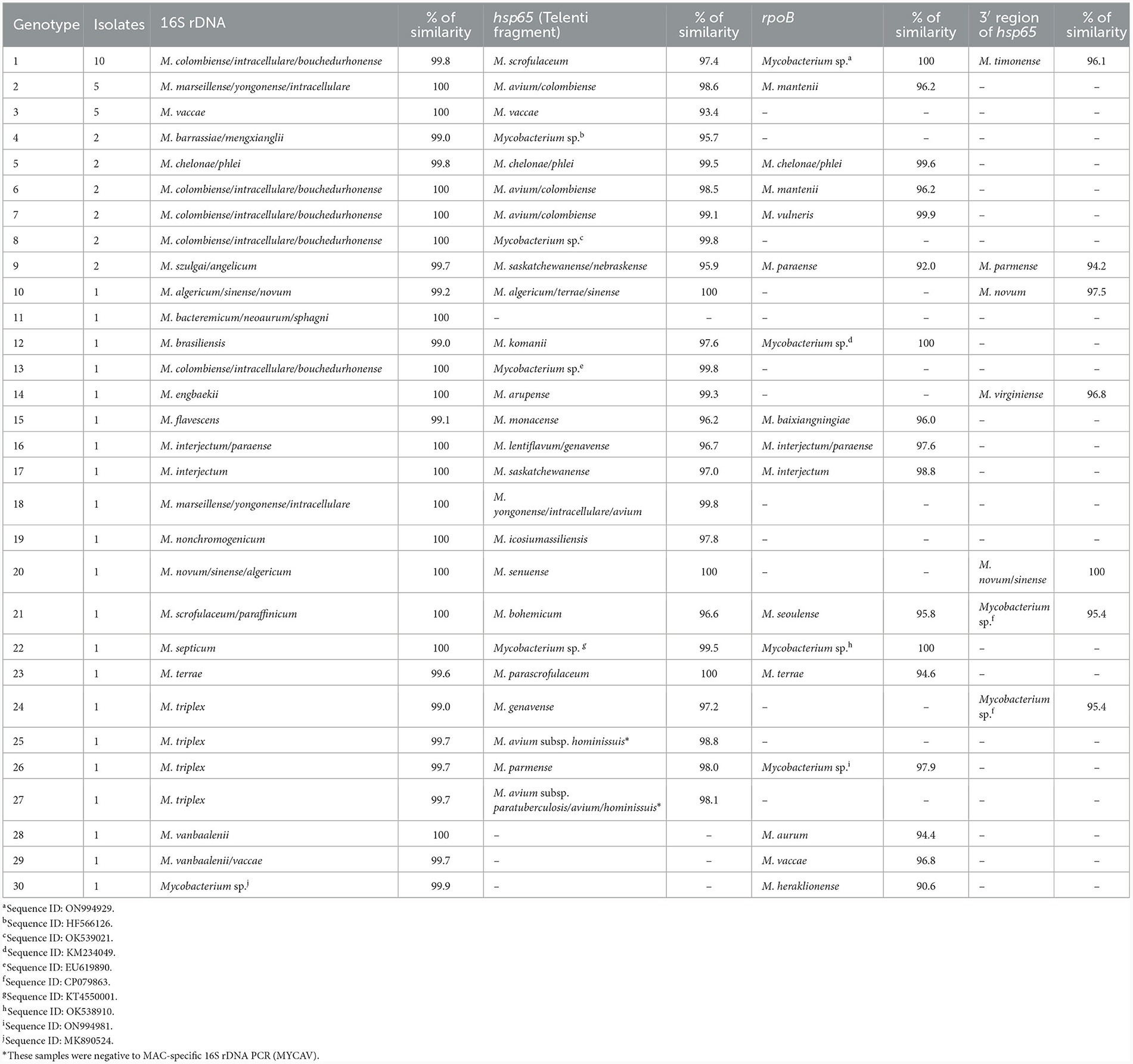
3 Resultados
En total, se incluyeron en el estudio 373 aislamientos recuperados de bovinos positivos ubicados en 359 rebaños de OTF en 34 provincias (15 Comunidades Autónomas) (Figura 1). De estos, 194 (54,0%) eran rebaños de carne, 150 (41,8%) eran lecherías, ocho (2,2%) eran unidades de engorde y siete (1,9%) eran ganaderías de lidia. De los 373 aislamientos, se logró una identificación confiable en 308 (82,6%). De estas, 166 (53,9%) fueron clasificadas como MNT no MAC y 142 (46,1%) como especies MAC. En los 65 aislamientos restantes (17,4% del total de muestras) no se logró una identificación fiable. De ellas, 30 (46,2%) fueron clasificadas como NTM no MAC, 23 (35,4%) como MAC, y la secuencia de ADNr 16S reveló una bacteria no perteneciente al género Mycobacterium para los 12 (18,5%) aislamientos restantes.
Dentro de los 166 aislamientos de MNT no MAC, se identificaron de manera confiable 27 especies (Tabla 1), siendo la especie más frecuente M. nonchromogenicum (n = 85; 51,2%), seguida de M. bourgelatii (n = 11; 6,6%), M. shimoidei (8; 4,8%), M. kansasii (7; 4,2%) y M. intermedium (7; 4,2%). No se logró una identificación confiable en 30 aislamientos (15,3%), de los cuales 8 y 16 no arrojaron resultado positivo para la región rpoB y 3′ de los genes hsp65, respectivamente (Tabla 2). Entre estos aislados, identificamos 23 «genotipos» presentes en 1-5 aislamientos. Un genotipo se define como el ADNr parcial distinto de 16S y hsp65 (fragmento de Telenti) y, cuando está disponible, la región rpoB y 3′ de las secuencias de hsp65. La mayor parte de las MNA no MAC se recuperaron de Galicia (141/196). Sin embargo, la diversidad de genotipos fue casi la misma que en las regiones no OTF (29 y 28 genotipos, respectivamente).
 Tabla 1. Especies de MNT con una identificación fiable desde el bovino positivo hasta la prueba cutánea.
Tabla 1. Especies de MNT con una identificación fiable desde el bovino positivo hasta la prueba cutánea.
Para los 142 aislados de MAC se identificaron cinco especies (Mah, Maa, M. intracellulare, M. yongonense y M. colombiense). De estos, la subespecie de M. avium representó el 92,3% (n = 131) de los aislados recuperados, siendo Mah (98; 69,0%) más representada que Maa (33; 23,2%). La mayoría de los aislamientos de MAC (63/165) procedían de Asturias, ya que esta región presentó principalmente cultivos identificados preliminarmente como MAC, pero también se incluyeron 56 y 46 aislamientos de MAC procedentes de Galicia y regiones no OTF, respectivamente. En cuanto a las especies predominantes de MAC, Mah se recuperó con mayor frecuencia que Maa en todas las regiones, y en el caso de Galicia, M. intracellulare (4) se identificó incluso con mayor frecuencia que Maa (1).
De los 23 aislamientos con identificación no confiable, no obtuvimos un amplicón de PCR para la región rpoB y 3′ de los genes hsp65 en 11 y 20 aislamientos, respectivamente. Entre estos 23 aislados, se identificaron siete genotipos, como se definió anteriormente, en entre 1 y 10 aislamientos cada uno. El genotipo más común se encontró en 10 aislamientos de cinco provincias en los que se obtuvo una similitud del 99,8% con la secuencia de ADNr 16S de M. colombiense, M. intracellulare y M. bouchedurhonense junto con una similitud del 97,4% con la secuencia del gen hsp65 de M. scrofulaceum. En la Tabla 2 se muestran todos los genotipos de aislados con una identificación no fiable.
Finalmente, la secuencia de ADNr 16S reveló que 12 aislamientos no pertenecían al género Mycobacterium sino a las siguientes especies: Prauserella rugosa (n = 3), Streptomyces hydrogenans (3), actinobacteria no cultivada (3), Corynebacterium pseudotuberculosis (2) y Brevibacillus brevis (1).
En general, se observaron lesiones macroscópicas granulomatosas similares a la tuberculosis bovina en muestras de ganglios linfáticos de 20 animales (5,3%). A partir de estas muestras, se identificaron 11 especies diferentes: Mah (n = 6), genotipo 2 (2) (Tabla 2), M. nonchromogenicum (2), M. shimoidei (2), Maa (1), M. intracellulare (1), M. xenopi (1), Corynebacterium pseudotuberculosis (1), M. interjectum (1), M. yongonense y genotipo 7 (1) (Tabla 2).
4 Discusión
Las micobacterias no tuberculosas son uno de los factores limitantes que comprometen el rendimiento diagnóstico de las pruebas de tuberculosis bovina (15, 32). Dado que la carga de la tuberculosis bovina está disminuyendo en la mayoría de los países en los que se han aplicado sistemáticamente campañas de erradicación, la necesidad de mantener una alta especificidad es clave para mantener el valor predictivo positivo lo más alto posible, pero sin comprometer la sensibilidad general del sistema de vigilancia. La caracterización de los factores que afectan a la especificidad diagnóstica, como la MNT más prevalente asociada a reacciones inespecíficas a las pruebas cutáneas de TBBb, puede ser útil para diseñar estrategias destinadas a minimizar su impacto. En este trabajo se presenta una caracterización exhaustiva de un amplio panel de aislados de MNT recuperados de bovinos de rebaños de OTF para evaluar su diversidad en España.
Un gran porcentaje de los aislados incluidos en el estudio pertenecían a MAC (específicamente a las especies Mah, Maa, M. intracellulare, M. colombiense y M. yongonense, de las cuales las dos primeras fueron, con mucho, las más comunes), en parte debido a que la segunda región de la que se disponía de un mayor número de aislamientos (Asturias) proporcionaba mayoritariamente MNT identificada preliminarmente como MAC (Tabla 1) (33). Sin embargo, la alta frecuencia de aislamiento de los miembros de MAC del ganado bovino aquí concuerda con estudios previos en los que ciertos miembros de MAC (Mah, Maa, Map, M. arosiense, M. bouchedurhonense, M. chimaera, M. colombiense, M. intracellulare y M. vulneris) también fueron aislados de muestras bovinas e identificados como una posible causa de reacciones inespecíficas (15, 19, 34). La posibilidad de provocar una respuesta inmunitaria de reacción cruzada en las pruebas diagnósticas de tuberculosis bovina por parte de los miembros del MAC (y, en cierta medida, también por otras MNT) debe reducirse mediante el uso de la CIT, que también tiene en cuenta la respuesta provocada por el derivado purificado de proteínas aviares (PPD-A) obtenido a partir de un aislado de Maa (35, 36), por lo que la interferencia diagnóstica debe limitarse en las zonas en las que se utiliza esta prueba (33). Esto, sin embargo, puede conducir a una disminución de la sensibilidad diagnóstica de entre 14 y 44 puntos porcentuales según algunas estimaciones (37), por lo que se deben utilizar pruebas comparativas cuando el riesgo de tuberculosis se considera bajo. En nuestro estudio, la mayoría de los animales (353/373) eran reactores en el SIT (basados solo en el PPD bovino), y de hecho, de los únicos 20 animales que eran reactores en el CIT (todos provenientes de un área actual de OTF), se aisló M. nonchromogenicum de 13 de ellos y solo se aisló una especie de MAC (M. colombiense). lo que sugiere que el CIT podría verse comprometido en mayor medida por ciertas especies micobacterianas.
M. nonchromogenicum fue la MNT no MAC aislada con mayor frecuencia. Esta especie fue la MNT más común recuperada en muestras de ganado bovino en Irlanda del Norte (23 de 48 animales) (18) y Hungría (30 de 104) (17), y la segunda más común en Francia (81 de 310) (15), aunque en estos estudios no todas las muestras procedían de animales con piel positiva o rebaños de OTF. M. nonchromogenicum también se ha descrito en muestras de ganado bovino con lesiones recogidas en mataderos de Suiza (6), Sudáfrica (34) y Etiopía (7, 38), así como en muestras de leche de vacas positivas a la CIT en Brasil (39) y en la mucosa nasal del ganado (40). En otro estudio llevado a cabo en España (19), que incluyó muestras de tejido de bovinos y jabalíes del País Vasco (del que no se incluyeron muestras aquí), se aisló M. nonchromogenicum en tres de 21 bovinos y nueve jabalíes positivos a SIT. Estos resultados, junto con nuestros hallazgos, demuestran que M. nonchromogenicum se puede encontrar en bovinos de reactores de las regiones del norte de España, de las que se recuperaron la mayoría de los aislamientos, y podría ser un contribuyente relevante para el desarrollo de reacciones cruzadas en la prueba cutánea. La capacidad de M. nonchromogenicum para provocar reacciones después de la inoculación de ciertos antígenos micobacterianos (incluidos PPD-A y PPD-B) se exploró en un modelo experimental de infección en cobayas, pero las reacciones inducidas observadas fueron limitadas (16). Del mismo modo, aún no se ha demostrado la capacidad de esta especie para causar reacciones falsas positivas en pruebas cutáneas en bovinos, ya que los estudios experimentales para evaluar el potencial de reacción cruzada de la MNT solo se han limitado a M. kansasii (4, 41) y M. fortuitum (42).
En cuanto a la distribución geográfica de las MNT no MAC, el 93% de los 85 aislamientos de M. nonchromogenicum identificados aquí se recuperaron de bovinos de Galicia, lo que representa el 56% de todas las MNT no MAC disponibles en esta región en particular. Por el contrario, ninguna MNT no MAC representó más del 10% de los aislamientos recuperados de regiones no OTF, lo que sugiere una distribución más heterogénea de las especies de MNT. Al considerar los aislamientos de MAC, Mah fue la especie más comúnmente encontrada, pero su frecuencia relativa en comparación con otras especies de MAC varió entre regiones: en Galicia el 85,4% de todos los aislamientos de MAC con una identificación confiable (41/48) fueron Mah y solo se recuperó un aislado de Maa. Por el contrario, en Asturias se identificaron 34 aislamientos de Mah y 25 de Maa, lo que representa el 57,6 y el 42,4% de los 59 aislamientos de MAC con identificación fiable, respectivamente, mientras que en las regiones no OTF Mah y Maa representaron el 65,7 y el 20,0% de los 46 aislamientos de MAC disponibles en estas regiones, respectivamente. Aunque estas diferencias pueden estar señalando diferencias en la epidemiología de las reacciones cruzadas debidas a MNT en bovinos entre las regiones OTF y las que no lo son, los esfuerzos de muestreo altamente heterogéneos pueden haber dado lugar a sesgos que contribuyen a estos resultados y, por lo tanto, estos hallazgos deben interpretarse con cuidado. Las diferencias en las especies de MNT más prevalentes asociadas a reacciones cruzadas no estuvieron influenciadas por el tipo de producción, siendo M. nonchromogenicum la más abundante tanto en rebaños de carne como de leche en Galicia (42,3 y 38,4% de todos los aislados en cada tipo, respectivamente). Del mismo modo, la distribución de los miembros de MAC en los diferentes tipos de rebaños de producción en Asturias también fue similar, estando aislado Mah en el 52,5 y 56,5% de los rebaños de carne y leche, respectivamente, y Maa en el 40,0 y 34,8% de ellos. Esto sugiere que la prevalencia de la MNA puede estar más influenciada por factores regionales que vinculada al tipo de producción.
Curiosamente, 53 aislamientos en nuestro estudio no pudieron ser asignados de manera confiable a una especie específica. Entre ellos, 10 aislamientos provenientes de rebaños de carne exhibieron secuencias idénticas en los cuatro genes secuenciados (genotipo 1, Tabla 2), lo que constituiría la quinta «especie» más común en nuestro estudio. Estos aislamientos tuvieron una similitud del 99,8% con M. colombiense/intracellulare/bouchedurhonense (miembros MAC) basados en el ADNr 16S y del 100% con una secuencia previamente clasificada como perteneciente a MAC basada en el gen rpoB (19). Sin embargo, la coincidencia más cercana a su secuencia corta hsp65 fue M. scrofulaceum (97,4% de similitud), mientras que la mayor similitud con una secuencia hsp65 de un miembro MAC fue solo del 95,7%, por debajo del umbral del 97,3% sugerido anteriormente para los miembros MAC para este gen (33). Este resultado resalta la importancia del uso de múltiples marcadores genéticos para lograr una identificación completa de las especies involucradas (28, 43). Se necesitarían más estudios para concluir sobre la naturaleza de estos aislados.
Nuestros resultados demuestran que múltiples especies de MNT podrían estar asociadas con la aparición de reacciones inespecíficas en la prueba cutánea en bovinos en España. Sin embargo, la importancia relativa de los miembros de MAC (especialmente Mah y Maa) y M. nonchromogenicum, que constituyeron hasta el 69% de todos los aislamientos considerados en el estudio, demuestra que ciertas especies tienen más probabilidades de conducir a interferencias diagnósticas en rebaños de OTF en áreas de baja prevalencia/OTF del país, de acuerdo con lo descrito en otros países europeos (6, 15, 17-19) y países no europeos (34, 38, 39).
Al incluir solo aislamientos recuperados de animales positivos en rebaños de OTF (que mantuvieron este estado durante al menos 3 años después de que se encontró el reactor), intentamos minimizar la posibilidad de que los animales también pudieran haber sido infectados con un miembro del MTC. Se llevarán a cabo otros estudios basados en modelos experimentales de cobayas y bovinos para confirmar la capacidad de las cepas caracterizadas para inducir reacciones inespecíficas en bovinos no infectados por tuberculosis bovina, y para explorar la capacidad de diferentes antígenos para discriminar la respuesta inmunitaria inducida en pruebas cutáneas y ensayos de interferón gamma. En conjunto, esto contribuirá a limitar la interferencia en los resultados de las pruebas diagnósticas rutinarias, que pueden comprometer la fiabilidad del estado del rebaño obtenido y las medidas de gestión de riesgos indeseables que es necesario implementar en el contexto de un programa de erradicación.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. A continuación se indican los nombres de los repositorios y los números de acceso: https://www.ncbi.nlm.nih.gov/genbank/, OR642807—OR642833, OR648408—OR648468, OR671981—OR672046.
Declaración ética
No se requirió aprobación ética para el estudio con animales de acuerdo con la legislación local y los requisitos institucionales porque no era necesario. Todos los animales fueron sacrificados por los resultados positivos a la prueba cutánea oficial del Programa de Erradicación de la Tuberculosis Bovina en España.
Contribuciones de los autores
AG-B: Curación de datos, Análisis formal, Redacción: borrador original, Redacción: revisión y edición. JAl: Curación de datos, Adquisición de fondos, Recursos, Supervisión, Redacción, revisión y edición. JB: Obtención de fondos, redacción, revisión y edición. JM: Conceptualización, Supervisión, Redacción, revisión y edición. JAm: Curación de datos, recursos, redacción, revisión y edición. JS: Investigación, Recursos, Redacción, revisión y edición. LdJ: Curación de datos, adquisición de fondos, redacción, revisión y edición. BR: Curación de datos, metodología, supervisión, redacción, revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. AG-B es miembro de una beca de doctorado (referencia CT58/21-CT59/21) de la Universidad Complutense de Madrid y del Banco Santander. Este trabajo ha sido financiado por el Laboratorio de Referencia de la Unión Europea para la Tuberculosis Bovina del Ministerio de Agricultura, Pesca y Alimentación de España, y es una contribución al proyecto Estrategias Integradas para el Control y la Erradicación de la Tuberculosis en España (ERATUB) (RTI2018-096010-BC22, Ministerio de Ciencia, Innovación y Universidades, MICINN).
Reconocimientos
Los autores agradecen la colaboración y el apoyo de la EU-RL para la Tuberculosis Bovina, del Ministerio de Agricultura, Pesca y Alimentación, de la Consellería do Medio Rural de la Xunta de Galicia, y de la Consejería de Medio Rural y Política Agraria del Principado de Asturias. Asimismo, queremos agradecer a Francisco Lozano, Nuria Moya, Tatiana Alende, Alexandra Gutiérrez, Cristina Viñolo, Daniela de la Cruz y Laura Sánchez Martín de la Unidad de Micobacterias del Centro de Vigilancia Sanitaria VISAVET por su apoyo técnico.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial de interés.
El/los autor/es declararon, en el momento de la presentación, ser miembro del consejo editorial de Frontiers. Esto no tuvo ningún impacto en el proceso de revisión por pares ni en la decisión final.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1361788/full#supplementary-material
Referencias
1. Parte AC, Sardà Carbasse J, Meier-Kolthoff JP, Reimer LC, Göker M. La lista de nombres procariotas con nomenclatura permanente (LPSN) se traslada a la DSMZ. Int J Syst Evol Microbiol. (2020) 70:5607–12. doi: 10.1099/ijsem.0.004332
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Honda JR, Virdi R, Chan ED. Micobacterias no tuberculosas ambientales globales y sus nichos naturales y artificiales contemporáneos. Microbiol frontal. (2018) 9:e02029. doi: 10.3389/fmicb.2018.02029
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Primm TP, Falkinham JO. Micobacterias no tuberculosas. En: Módulo de Referencia en Ciencias Biomédicas. Elsevier (2014).
4. Waters WR, Whelan AO, Lyashchenko KP, Greenwald R, Palmer M V, Harris BN, et al. Respuestas inmunitarias en bovinos inoculados con Mycobacterium bovis, Mycobacterium tuberculosis o Mycobacterium kansasii. Clin Vacc Immunol. (2010) 17:247–52. doi: 10.1128/CVI.00442-09
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Hernández-Jarguín AM, Martínez-Burnes J, Molina-Salinas GM, de la Cruz-Hernández NI, Palomares-Rangel JL, Mayagoitia AL, et al. Aislamiento y cambios histopatológicos asociados a micobacterias no tuberculosas en ganglios linfáticos condenados en un matadero bovino. Veterinaria Sci. (2020) 7:1–10. doi: 10.3390/vetsci7040172
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Ghielmetti G, Friedel U, Scherrer S, Sarno E, Landolt P, Dietz O, et al. Micobacterias no tuberculosas aisladas de ganglios linfáticos y muestras fecales de ganado sano sacrificado y el entorno del matadero. Transbound Emerg Dis. (2018) 65:711–8. doi: 10.1111/tbed.12793
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Nuru A, Zewude A, Mohammed T, Wondale B, Teshome L, Getahun M, et al. Las micobacterias no tuberculosas son las principales causas de lesiones similares a la tuberculosis en el ganado sacrificado en el matadero de Bahir Dar, en el noroeste de Etiopía. BMC Vet Res. (2017) 13:237. doi: 10.1186/s12917-017-1168-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Más SJ, Radunz B, Glanville RJ. Lecciones aprendidas durante la erradicación exitosa de la tuberculosis bovina de Australia. Rec. Veterinario (2015) 177:224–32. doi: 10.1136/vr.103163
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Autoridad Europea de Seguridad Alimentaria (EFSA). Informe de zoonosis 2021 de la Unión Europea «Una sola salud». EFSA J. (2022) 20:e8442. doi: 10.2903/j.efsa.2023.8442
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. WOAH. Tuberculosis de mamíferos (infección por el complejo Mycobacterium tuberculosis). En: Manual de Pruebas Diagnósticas y Vacunas para Animales Terrestres. OMSA (2023).
11. Alvarez J, de Juan L, Bezos J, Romero B, Sáez JL, Reviriego Gordejo FJ, et al. Interferencia de la paratuberculosis con el diagnóstico de tuberculosis en un rebaño caprino con una infección mixta natural. Microbiol veterinario. (2008) 128:72–80. doi: 10.1016/j.vetmic.2007.08.034
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Roupie V, Alonso-Velasco E, Van Der Heyden S, Holbert S, Duytschaever L, Berthon P, et al. Evaluación de las respuestas de interferón gamma y anticuerpos específicos de micobacterias antes y después de una única prueba cutánea intradérmica en bovinos expuestos naturalmente a M. avium subsp paratuberculosis e infectados experimentalmente con M. bovis. Veterinario Inmunol Inmunopatíaco. (2018) 196:35–47. doi: 10.1016/j.vetimm.2017.12.007
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Álvarez J, de Juan L, Bezos J, Romero B, Sáez JL, Marqués S, et al. Efecto de la paratuberculosis en el diagnóstico de tuberculosis bovina en un hato bovino con una infección mixta mediante ensayo de detección de interferón-gamma. Microbiol veterinario. (2009) 135:389–93. doi: 10.1016/j.vetmic.2008.09.060
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Dunn JR, Kaneene JB, Grooms DL, Bolin SR, Bolin CA, Bruning-Fann CS. Efectos de los resultados positivos para Mycobacterium avium subsp paratuberculosis determinados por cultivo microbiano de heces o ELISA de anticuerpos sobre los resultados de la prueba de tuberculina del pliegue caudal y el ensayo de γ de interferón para la tuberculosis en bovinos. J Am Vet Med Assoc. (2005) 226:429–35. doi: 10.2460/javma.2005.226.429
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Biet F, Boschiroli ML. Infecciones micobacterianas no tuberculosas de relevancia veterinaria. Res Vet Sci. (2014) 97:S69–77. doi: 10.1016/j.rvsc.2014.08.007
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Fernández-Veiga L, Fuertes M, Geijo MV, Pérez de Val B, Vidal E, Michelet L, et al. Diferencias en las reacciones cutáneas a antígenos oficiales y definidos en cobayas expuestas a bacterias tuberculosas y no tuberculosas. Sci Rep. (2023) 13:2936. doi: 10.1038/s41598-023-30147-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Rónai Z, Eszterbauer E, Csivincsik, Guti CF, Dencso L, Jánosi S, et al. Detección de una amplia diversidad genética y de varias cepas nuevas entre micobacterias no aviar no tuberculosas aisladas de animales de granja y salvajes en Hungría. J Appl Microbiol. (2016) 121:41–54. doi: 10.1111/jam.13152
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Hughes MS, Ball NW, McCarroll J, Erskine M, Taylor MJ, Pollock JM, et al. Análisis molecular de micobacterias distintas del complejo M. tuberculosis aisladas de bovinos de Irlanda del Norte. Microbiol veterinario. (2005) 108:101–12. doi: 10.1016/j.vetmic.2005.03.001
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Varela-Castro L, Barral M, Arnal MC, Fernández de Luco D, Gortázar C, Garrido JM, et al. Más allá de la tuberculosis: diversidad e implicaciones de las micobacterias no tuberculosas en la interfaz entre la fauna silvestre y el ganado. Transbound Emerg Dis. (2022) 69:E2978–93. doi: 10.1111/tbed.14649
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Ministerio de Agricultura, Pesca y Alimentación. Programa Nacional de Erradicación de Tuberculosis Bovina 2023 (Infección por el complejo Mycobacterium tuberculosis). España (2023).
21. Lorente-Leal V, Liandris E, Pacciarini M, Botelho A, Kenny K, Loyo B, et al. PCR directa en muestras de tejido para detectar el complejo Mycobacterium tuberculosis: una alternativa al cultivo bacteriológico. J Clin Microbiol. (2021) 59:E01404-20. doi: 10.1128/JCM.01404-20
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Wilton S, Cousins D. Detección e identificación de múltiples patógenos micobacterianos mediante amplificación de ADN en un solo tubo. Genoma Res. (1992) 1:269–73. doi: 10.1101/gr.1.4.269
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Kunze ZM, Portaels F, McFadden JJ. Los subtipos biológicamente distintos de Mycobacterium avium difieren en la posesión de la secuencia de inserción IS901. J Clin Microbiol. (1992) 30:2366–72. doi: 10.1128/jcm.30.9.2366-2372.1992
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Guerrero C, Bernasconi C, Burki D, Bodmer T, Telenti A. Un nuevo elemento de inserción de Mycobacterium avium, IS1245, es una diana específica para el análisis de la relación de cepas. J Clin Microbiol. (1995) 33:304–7. doi: 10.1128/jcm.33.2.304-307.1995
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Swanson DS, Kapur V, Stockbauer K, Pan X, Fronthingham R, Musser JM. Diferenciación subespecífica de cepas del complejo Mycobacterium avium mediante secuenciación automatizada de una región del gen (hsp65) que codifica una proteína de choque térmico de 65 kilodalton. Int J Syst Bacteriol. (1997) 47:414–9. doi: 10.1099/00207713-47-2-414
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Telenti A, Marchesi F, Balz M, Bally F, Böttger EC, Bodmer T. Identificación rápida de micobacterias a nivel de especie mediante reacción en cadena de la polimerasa y análisis de enzimas de restricción. J Clin Microbiol. (1993) 31:175–8. doi: 10.1128/jcm.31.2.175-178.1993
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Sala TA. BioEdit: un editor de alineación de secuencias biológicas fácil de usar y un programa de análisis para Windows 95/98/NTBioEdit: un editor de alineación de secuencias biológicas fácil de usar y un programa de análisis para Windows 95/98/NT. Nucleic Acids Symp Ser. (1999) 41:95–8.
28. Tortoli E. Procedimiento operativo estándar para la identificación óptima de micobacterias mediante secuencias de genes de ARNr 16S. Stand Genomic Sci. (2010) 3:145–52. doi: 10.4056/sigs.932152
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Adékambi T, Drancourt M. Disección de las relaciones filogenéticas entre 19 especies de Mycobacterium de rápido crecimiento mediante la secuenciación de genes 16S rRNA, hsp65, sodA, recA y rpoB. Int J Syst Evol Microbiol. (2004) 54:2095–105. doi: 10.1099/ijs.0.63094-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Turenne CY, Semret M, Cousins D V, Collins DM, Behr MA. La secuenciación de hsp65 distingue entre subconjuntos del complejo Mycobacterium avium. J Clin Microbiol. (2006) 44:433–40. doi: 10.1128/JCM.44.2.433-440.2006
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Gómez-Buendía A, Romero B, Bezos J, Lozano F, de Juan L, Álvarez J. Riesgo específico de espoligotipo de encontrar lesiones en tejidos de bovinos infectados por Mycobacterium bovis. BMC Vet Res. (2021) 17:148. doi: 10.1186/s12917-021-02848-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Jenkins AO, Gormley E, Gcebe N, Fosgate GT, Conan A, Aagaard C, et al. Respuestas inmunitarias de reacción cruzada en bovinos derivadas de la exposición a Mycobacterium bovis y micobacterias no tuberculosas. Prev Vet Med. (2018) 152:16–22. doi: 10.1016/j.prevetmed.2018.02.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. van Ingen J, Turenne CY, Tortoli E, Wallace Jr RJ, Brown-Elliott BA. Una definición del complejo Mycobacterium avium con fines taxonómicos y clínicos, una revisión. Int J Syst Evol Microbiol. (2018) 68:3666–77. doi: 10.1099/ijsem.0.003026
34. Gcebe N, Rutten V, Gey van Pittius NC, Michel A. Prevalencia y distribución de micobacterias no tuberculosas (MNT) en bovinos, búfalos africanos (syncerus caffer) y sus entornos en Sudáfrica. Transbound Emerg Dis. (2013) 60:74–84. doi: 10.1111/tbed.12133
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Gcebe N, Michel A, Gey van Pittius NC, Rutten V. Genómica comparativa y análisis proteómico de cuatro especies de Mycobacterium no tuberculosas y complejo Mycobacterium tuberculosis: ocurrencia de proteínas inmunogénicas compartidas. Microbiol frontal. (2016) 7:795. doi: 10.3389/fmicb.2016.00795
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Infantes-Lorenzo JA, Moreno I, Risalde MDLÁ, Roy Á, Villar M, Romero B, et al. Caracterización proteómica de derivados proteicos purificados bovinos y aviares e identificación de antígenos específicos para el serodiagnóstico de tuberculosis bovina. Clin Proteom. (2017) 17:36. doi: 10.1186/s12014-017-9171-z
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Núñez-García J, Downs SH, Parry JE, Abernethy DA, Broughan JM, Cameron AR, et al. Metaanálisis de la sensibilidad y especificidad de las pruebas diagnósticas ante-mortem y post-mortem para la tuberculosis bovina en el Reino Unido e Irlanda. Prev Vet Med. (2018) 153:94–107. doi: 10.1016/j.prevetmed.2017.02.017
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Berg S, Firdessa R, Habtamu M, Gadisa E, Mengistu A, Yamuah L, et al. La carga de la enfermedad micobacteriana en el ganado etíope: implicaciones para la salud pública. PLoS UNO. (2009) 4:E5068. doi: 10.1371/journal.pone.0005068
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Bolaños CAD, de Paula CL, Guerra ST, Franco MMJ, Ribeiro MG. Diagnóstico de micobacterias en leche bovina: una visión general. Rev Inst Med Trop São Paulo. (2017) 59:1–13. doi: 10.1590/s1678-9946201759040
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. McCorry TP, McCormick CM, Hughes MS, Pollock JM, Neill SD. Mycobacterium nonchromogenicum en moco nasal de ganado en un rebaño infectado con tuberculosis bovina. Microbiol veterinario. (2004) 99:281–5. doi: 10.1016/j.vetmic.2003.12.006
41. Waters WR, Palmer M V, Thacker TC, Payeur JB, Harris NB, Minion FC, et al. Respuestas inmunitarias a antígenos definidos de Mycobacterium bovis en bovinos infectados experimentalmente con Mycobacterium kansasii. Clin Vacc Immunol. (2006) 13:611–9. doi: 10.1128/CVI.00054-06
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Michel AL. Interferencia de la infección por Mycobacterium fortuitum con el diagnóstico de Mycobacterium bovis: casos de infección natural y una infección experimental piloto. J Vet Diagn Investig. (2008) 20:501–3. doi: 10.1177/104063870802000415
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Fedrizzi T, Meehan CJ, Grottola A, Giacobazzi E, Fregni Serpini G, Tagliazucchi S, et al. Caracterización genómica de micobacterias no tuberculosas. Sci Rep. (2017) 7:45258. doi: 10.1038/srep45258
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: bovinos, diagnóstico, rebaños oficialmente libres de tuberculosis (OTF), interferencia, micobacterias no tuberculosas, tuberculosis, pruebas cutáneas
Cita: Gómez-Buendía A, Álvarez J, Bezos J, Mourelo J, Amado J, Sáez JL, de Juan L y Romero B (2024) Micobacterias no tuberculosas: ocurrencia en reactores de bovinos de prueba cutánea de rebaños oficiales libres de tuberculosis. Frente. Vet. Sci. 11:1361788. doi: 10.3389/fvets.2024.1361788
Recibido: 26 de diciembre de 2023; Aceptado: 15 de enero de 2024;
Publicado: 31 enero 2024.
Editado por:
Cinzia Marianelli, Instituto Nacional de Salud (ISS), Italia
Revisado por:
Marta Alonso-Hearn, Basque Research and Technology Alliance (BRTA), España
Carly Kanipe, Departamento de Agricultura de los Estados Unidos (USDA), Estados Unidos
Copyright © 2024 Gómez-Buendía, Álvarez, Bezos, Mourelo, Amado, Sáez, de Juan y Romero. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Beatriz Romero, bromerom@visavet.ucm.es
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía