Pequeños mamíferos de compañía no tradicionales en España como reservorios de estafilococos resistentes a los antimicrobianos
 Ana Marco-Fuertes1
Ana Marco-Fuertes1 Clara Marín1
Clara Marín1 José Villora-González2
José Villora-González2 Concepción Gimeno-Cardona3,4
Concepción Gimeno-Cardona3,4 Violeta Artal-Muñoz3
Violeta Artal-Muñoz3 Santiago Vega1*
Santiago Vega1* Laura Montoro-Dasi1*
Laura Montoro-Dasi1*- 1Facultad de Veterinaria, Instituto de Ciencias Biomédicas, Universidad Cardenal Herrera-CEU, Universidades CEU, Valencia, España
- número arábigoClínica Veterinaria Selvätica, Valencia, España
- 3Servicio de Microbiología, Consorcio Hospital General Universitario de Valencia, Valencia, España
- 4Universidad de Valencia, Facultad de Medicina, Valencia, España
Introducción: La creciente prevalencia de la resistencia a los antimicrobianos (RAM) y la resistencia a múltiples fármacos (MDR) en los microorganismos plantea una preocupación importante tanto en la medicina humana como en la veterinaria. Los animales de compañía no tradicionales, especialmente populares entre los hogares con niños, desempeñan un papel crucial en la epidemiología de la resistencia a los antimicrobianos debido a su creciente población. De hecho, se sabe que algunos de estos animales pueden actuar como reservorios de patógenos zoonóticos y, por lo tanto, ser capaces de propagarlos y transmitirlos a los miembros de la familia, junto con su resistencia a los antimicrobianos, a través de su entorno compartido. Por lo tanto, es imperativo abordar esta preocupación con la participación de profesionales de la salud humana, animal y ambiental. Este estudio piloto tuvo como objetivo evaluar la prevalencia y los patrones de resistencia a los antimicrobianos de las cepas de Staphylococcus spp. obtenidas de muestras de infección comensal de la mucosa y de la piel en pequeños mamíferos NTC, centrándose en cepas como el Staphylococcus spp. resistente a la meticilina (MRS) que son críticas para la salud pública.
Métodos: Para ello, se tomaron muestras de 81 animales de diferentes especies de pequeños mamíferos, evaluando la susceptibilidad antimicrobiana a 27 agentes antimicrobianos (AMA) relevantes en la salud humana mediante ensayos de concentración mínima inhibitoria, e interpretándolos de acuerdo con las directrices EUCAST y CLSI. Las cepas aisladas de Staphylococci fueron identificadas por MALDI-TOF, siendo las especies predominantes Mammalicoccus sciuri y Staphylococcus aureus.
Resultados y discusión: Incluyendo todas las cepas aisladas, se observó RAM contra los 27 AMA, incluidos seis AMA de último recurso en medicina humana. Además, más del 85% de las cepas exhibieron MDR. Estos hallazgos subrayan la necesidad de monitorear las tendencias de RAM y MDR en animales de compañía y enfatizan el papel potencial de los NTCA en la propagación de la resistencia a humanos, otros animales y su entorno compartido, lo que exige un enfoque integral de «Una sola salud».
1 Introducción
Los animales de compañía no tradicionales, incluidos los pequeños mamíferos (como conejos o hurones), serpientes, lagartijas o aves exóticas, representan actualmente casi el 30 % de todos los animales de compañía en Europa. En particular, se ha producido un notable aumento en el número de pequeños mamíferos, hasta alcanzar los 29 millones en los hogares europeos en la actualidad (1).
Los mamíferos pequeños, como conejos, conejillos de indias o roedores, se consideran animales de compañía ideales para los niños debido a su tamaño manejable, su mantenimiento relativamente fácil y su bajo riesgo de lesiones. Son socialmente interactivos y pueden vincularse con los niños, brindando oportunidades para aprender sobre la responsabilidad y el comportamiento animal. Estos animales se adaptan fácilmente a espacios pequeños, lo que facilita su cuidado en entornos domésticos, y su presencia ofrece a los niños la oportunidad de aprender sobre la naturaleza y las necesidades básicas de los seres vivos (2). Por este motivo, esta tendencia creciente a la tenencia de NTCA favorece su estrecho contacto con sus propietarios, cobrando especial importancia en los hogares compartidos con poblaciones en riesgo (3). Además, se ha observado que estos animales pueden albergar diferentes microorganismos, como bacterias comensales y patógenas, y transmitirlos junto con su resistencia a los antimicrobianos (RAM) (4).
La resistencia a los antimicrobianos se caracteriza por la capacidad de los microorganismos de evolucionar con el tiempo y volverse resistentes a los fármacos utilizados para combatir las infecciones que inducen (5, 6). Esto es especialmente crítico debido a la aparición de cepas multirresistentes (MDR), que son cepas de bacterias que han desarrollado resistencia a varias clases de agentes antimicrobianos (AMA) (5) y, por lo tanto, actualmente se dispone de recursos limitados para una intervención eficaz (7). De hecho, debido a los desafíos que plantean la resistencia a los antimicrobianos y la resistencia a los antimicrobianos tanto en medicina humana como veterinaria, la Organización Mundial de la Salud (OMS) las ha declarado como una de las principales amenazas para la salud pública en la actualidad (6), ya que estas resistencia a los antimicrobianos no son exclusivas de una sola especie y pueden propagarse a través del entorno compartido entre humanos y animales. subrayando la necesidad de abordar este problema a través de una estrategia de «Una sola salud» (4, 8).
Tradicionalmente, se ha estudiado la importancia de la resistencia a los antimicrobianos en el ganado, junto con su asociación con los ganaderos (9), pero se han realizado pocos estudios en animales domésticos a pesar de la importancia de su impacto en los propietarios, que a menudo son niños. De hecho, en la Unión Europea (UE) se han puesto en marcha diferentes programas de seguimiento y vigilancia de la resistencia a los antimicrobianos para las bacterias zoonóticas y comensales en animales destinados a la producción de alimentos por parte de la Autoridad Europea de Seguridad Alimentaria (EFSA) (10), y en medicina humana por el Centro Europeo para la Prevención y el Control de las Enfermedades (ECDC) (11). Sin embargo, especialmente en la UE, cada Estado miembro ha puesto en marcha además sus propios programas, por ejemplo, el Plan Nacional de Resistencia Antibióticos (PRAN) en España (12). En la actualidad, es necesario homogeneizar todos estos programas para comparar los datos disponibles y establecer la situación epidemiológica actual de la resistencia a los antimicrobianos, incluso en animales destinados a la producción de alimentos y de compañía. Por este motivo, la UE tiene la intención de crear la Red Europea de Vigilancia de la Resistencia a los Antimicrobianos en Medicina Veterinaria (EARS-Vet) (13, 14), pero solo se incluyen en este proyecto los animales de compañía tradicionales, como perros y gatos, dejando de lado las NTCA.
En los estudios epidemiológicos de la resistencia a los antimicrobianos, las especies de la familia Staphylococceae son de especial relevancia, ya que forman parte de la microbiota comensal de la piel y la mucosa de animales y humanos, pero también se consideran patógenos oportunistas que pueden causar infecciones tanto en humanos como en animales (15). Esta familia se divide en estafilococos coagulasa positivos (CoPS) y estafilococos coagulasa negativos (CoNS). Ambos grupos han sido identificados como bacterias patógenas con un potencial significativo para causar infecciones graves tanto en medicina humana como veterinaria (16). En particular, una de las principales bacterias monitoreadas a nivel mundial es Staphylococcus aureus (CoPS), que es una de las especies de Staphylococcus más ampliamente distribuidas, ya que está ampliamente presente tanto en humanos como en animales y también ha sido designada por la OMS como una de las bacterias de alta prioridad para la investigación y desarrollo de nuevos AMAs debido a su alta resistencia (17). Esto es de especial importancia debido a la aparición de cepas de Staphylococcus aureus resistentes a la meticilina (SARM), uno de los patógenos de alta prioridad listados por la OMS (17). Sin embargo, debe destacarse la necesidad de monitorizar todas las cepas de estafilococos resistentes a la meticilina (MRS) debido a su importancia para la salud pública (18). Además, es importante no olvidar las cepas de CoNS, ya que también se ha informado que muchas de ellas son resistentes a la meticilina, lo que podría conducir a fracasos terapéuticos en el tratamiento de infecciones causadas por estas cepas desafiantes (19, 20).
Sin embargo, a pesar de toda esta información, no existen programas disponibles centrados en las NTCA, a pesar de que se consideran portadoras de Staphylococcus spp. y pueden transmitirlas a sus propietarios. Además, en Europa se han llevado a cabo pocos estudios sobre NTCAs, y los pocos que se han realizado se han centrado principalmente en conejos o roedores, los pequeños mamíferos NTC más populares (21-24). Por lo tanto, se necesitan más estudios para lograr una visión global de la RAM y la MDR en las diferentes especies animales incluidas en este grupo bacteriano. Por lo tanto, para obtener una visión general inicial completa, el objetivo de este estudio piloto fue evaluar la prevalencia y los patrones de resistencia a los antimicrobianos de cepas de Staphylococcus spp. aisladas de muestras de mucosa comensal y muestras de infección cutánea tomadas de pequeños mamíferos NTC. Además, el estudio también tuvo como objetivo investigar la presencia de MDR y MRS en estas cepas.
2 Materiales y métodos
2.1 Diseño experimental
El Comité de Ética Animal de la Universidad UCH-CEU (número de investigación CEEA 22/04) revisó y aprobó el presente estudio con animales realizado en la Comunitat Valenciana.
Con este fin, se invitó a participar de forma voluntaria a un importante centro veterinario (CV), que se ocupa exclusivamente de los exóticos y los NTCA. Este centro atiende a casi el 70% de la población de animales exóticos de la Comunitat Valenciana, ya que recibe animales procedentes de varias clínicas y hospitales de la Comunidad Valenciana, lo que lo convierte en un sitio de muestreo exhaustivo y representativo para el estudio.
2.2 Recopilación de datos epidemiológicos
Con el objetivo de tomar las muestras y recopilar toda la información epidemiológica de estos animales muestreados, primero se solicitó el consentimiento informado de todos los propietarios de animales. En primer lugar, los veterinarios de la consulta rellenaron un cuestionario epidemiológico que contenía detalles sobre el origen de los animales. En la segunda parte se proporcionaron datos generales sobre los animales, como su sexo, edad, si compartían el hogar con otros animales y si tenían acceso al exterior. Por último, la tercera y última sección del cuestionario se centró en los datos clínicos relacionados con los animales. Incluía información sobre si el animal tenía alguna enfermedad crónica, si estaba tomando actualmente algún medicamento diario, detalles sobre su tratamiento más reciente con AMAs y un registro de AMAs específicos administrados a lo largo de su vida. Además, para estudiar el impacto que tienen los AMAs en el desarrollo de la RAM y la MDR, se realizaron cuatro grupos para clasificar a los animales en función de cuándo fueron tratados por última vez: (I) Nunca; (II) En los últimos 6 meses: (III) En el último mes; (IV) En tratamiento en el momento de la toma de muestras. El cuestionario está disponible en el material complementario.
2.3 Recogida de muestras
Para estudiar la prevalencia de Staphylococcus spp., sus patrones de resistencia a los antimicrobianos y la resistencia a múltiples fármacos de pequeños mamíferos NTC, se recogieron muestras entre enero y junio de 2023, de todos los animales que acudieron a la CV. Se tomaron dos tipos de muestras: para la primera, se introdujo un hisopo (hisopos de transporte estéril de Cary-Blair, DELTALAB, Barcelona, España) en la cavidad nasal y luego en la auricular, de pequeños mamíferos sanos y asintomáticos, basándose en estudios previos (25-27). Para verificar el estado de salud de los animales, los veterinarios realizaron un examen clínico, evaluando los signos vitales, como la temperatura corporal (Tª), y el estado cardíaco, respiratorio y corporal (28), para asegurarse de que se encontraban dentro de los rangos normales, de modo que pudieran clasificarse como animales sanos asintomáticos. La segunda muestra se tomó para aislar Staphylococcus spp. Para ello, se tomó un hisopo (hisopos de transporte estéril Cary-Blair, DELTALAB, Barcelona, España) de animales con infecciones cutáneas activas, que se introdujo en heridas aparentemente infectadas por la piel.
Para su posterior análisis, todas las muestras se transportaron al laboratorio de microbiología de la Facultad de Ciencias Veterinarias de la Universidad CEU Cardenal Herrera, se conservaron en medio de transporte Cary-Blair y se refrigeraron a ≤4°C dentro de las 24 h posteriores a la recolección.
2.4 Aislamiento e identificación de Staphylococcus spp.
Los hisopos de muestra se sometieron a un enriquecimiento previo en agua de peptona tamponada (BPW; Scharlau, Barcelona, España) en una proporción de 1:10 vol/vol y luego se incubó a 37 ± 1°C durante 24 h. Luego, la suspensión se sembró en agar inespecífico, agar Columbia CNA con 5% de sangre de oveja, Mejorado II (BD, Becton Dickinson, Madrid, España), y se incubó a 37 ± 1°C durante 24 a 48 h. La observación de las placas se produjo tanto a las 24 como a las 48 h. Las colonias sospechosas que mostraban una morfología típica de Staphylococcus spp. en agar sangre, junto con un resultado positivo en la prueba de catalasa, fueron identificadas por MALDI-TOF MS Biotyper System (Bruker Daltonics, Madrid, España) en el Servicio de Microbiología del Consorcio Hospital General Universitario de Valencia. Para interpretar los resultados obtenidos se utilizó el criterio estándar de Bruker, que osciló entre 0,00 y 3,00 (29). Estas puntuaciones se clasifican en tres grupos: el rango de 2,00 a 3,00 significa una identificación de alta confianza por especie; los rangos entre 1,70 y < 2,00 proporcionaron una identificación de baja confianza por especie (solo confiable a nivel de género); Y, por último, los rangos <1,70 no proporcionan una identificación fiable. Solo se incluyeron en este estudio puntuaciones superiores a 2,00.
2.5 Pruebas de susceptibilidad a los antimicrobianos
Las pruebas de susceptibilidad a los antimicrobianos, que incluyeron AMAs importantes para la salud pública, se realizaron siguiendo el protocolo descrito en estudios previos (30). Además, el MDR se definió como la resistencia adquirida a al menos un agente en tres o más clases de antimicrobianos (5).
Sin embargo, dado que se sabe poco sobre el estado epidemiológico de los pequeños mamíferos NTC en relación con su resistencia a los antimicrobianos por Staphylococcus spp., y que no existe un programa específico de seguimiento y vigilancia para su resistencia a los antimicrobianos, se realizaron dos paneles de AMA. El primer panel, realizado con la placa GPALL1F Gram-Positive Sensititre (Thermo Scientific™ Sensititre™, Madrid, España) (Tabla 1), incluyó 20 AMAs de relevancia para la salud pública y AMAs clínicamente importantes para la medicina humana e incluidos en el programa EARS-Vet (32). Además, la placa tenía dos pocillos de prueba D, que combinaban clindamicina (CLI) y eritromicina (ERY). Estos pozos indicaron si la cepa probada tenía resistencia inducible a CLI en presencia de ERY, lo que podría conducir a un fracaso terapéutico. La interpretación de los resultados se realizó siguiendo las directrices de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) (33). El segundo panel, que se realizó con la placa EU Surveillance Staphylococcus EUST2 Sensititre (Thermo Scientific™ Sensititre™, Madrid, España) (Tabla 1), incluyó los AMAs con relevancia en salud pública establecidos en la Decisión (UE) 2023/1017 en lo que respecta al seguimiento del SARM en cerdos de engorde (34), la única legislación disponible actualmente sobre esta bacteria en la UE.
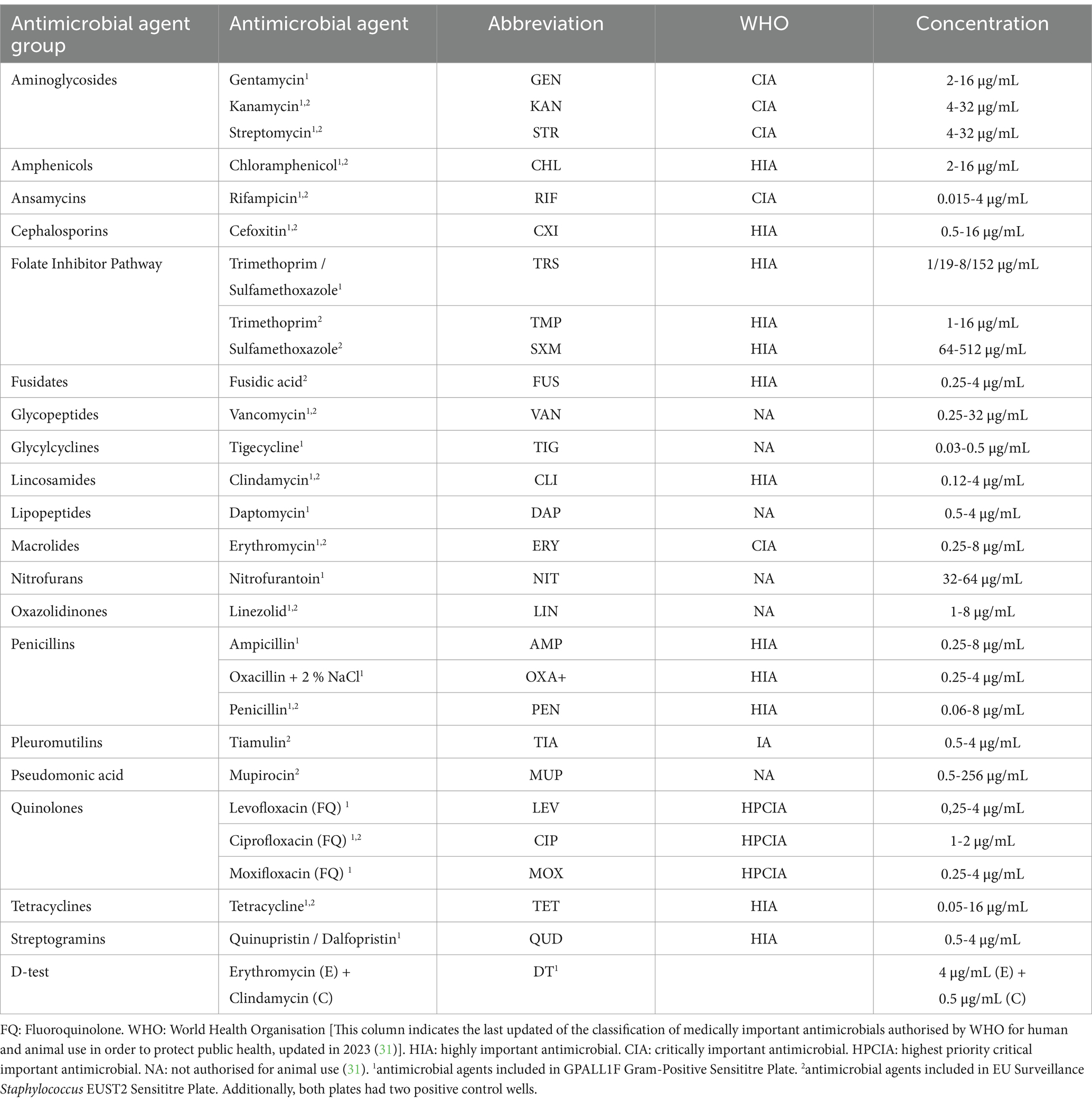
Tabla 1. Agentes antimicrobianos, la última clasificación antimicrobiana de la OMS y sus concentraciones estudiadas incluidas en GPALL1F placa de sensibilización Gram-Positive y en la placa de sensibilización EUST2 de Staphylococcus de vigilancia de la UE (ambas Thermo Scientific™ Sensititre™, Madrid, España).
Con este fin, se realizaron análisis de acuerdo con las instrucciones del fabricante (ThermoFisher Scientific™, Madrid, España) (35). La lectura manual de las placas se realizó utilizando un Sensititre Vizion (Thermo Scientific™ Sensititre™ Vizion™ Digital MIC Viewing System, ThermoFisher Scientific, Madrid, España).
Todos los resultados se interpretaron sobre la base de las directrices del Comité Europeo de Pruebas de Susceptibilidad a los Antimicrobianos (EUCAST) en su último informe (14.ª ed., 2024) (36). Las cepas de MRS se examinaron evaluando la resistencia a los antimicrobianos frente a la cefoxitina (el antibiótico utilizado para el cribado de SARM y las cepas de estafilococos coagulasa negativos resistentes a la meticilina (MR-CoNS)), y frente a la oxacilina + NaCl al 2%, el antibiótico utilizado para el cribado del Staphylococcus pseudintermedius resistente a la meticilina (MRSP). Sin embargo, dado que algunos valores de CMI de estos antibióticos para el cribado de MR-CoNS y MRSP no están disponibles actualmente en EUCAST, se siguieron las recomendaciones del Clinical and Laboratory Standards Institute (CLSI), especificadas en M100 (37) y VET01 (38), en esos casos.
2.6 Análisis estadístico
Una vez finalizados los análisis y obtenidos todos los datos del estudio, se procedió a su análisis mediante un modelo lineal generalizado (GLM) con función de enlace probit, asumiendo una distribución binomial. Esto se hizo para examinar la influencia de los factores epidemiológicos intrínsecos y externos de cada animal en la aparición de patrones de RAM y MDR en pequeños mamíferos Staphylococcus spp. El objetivo de este análisis fue determinar asociaciones con variables categóricas, incluyendo origen animal, sexo, convivencia con otros animales, relación con animales fuera del hogar e información clínica sobre enfermedades crónicas, medicación diaria y tratamientos antibióticos previos. Se consideró un nivel de significancia de p ≤0,05 indicativo de una diferencia estadísticamente significativa. El análisis estadístico se realizó utilizando los paquetes del software R (versión 4.3.1) EMMs (39), car (40) y multicompView (41).
3 Resultados
3.1 Resultados epidemiológicos
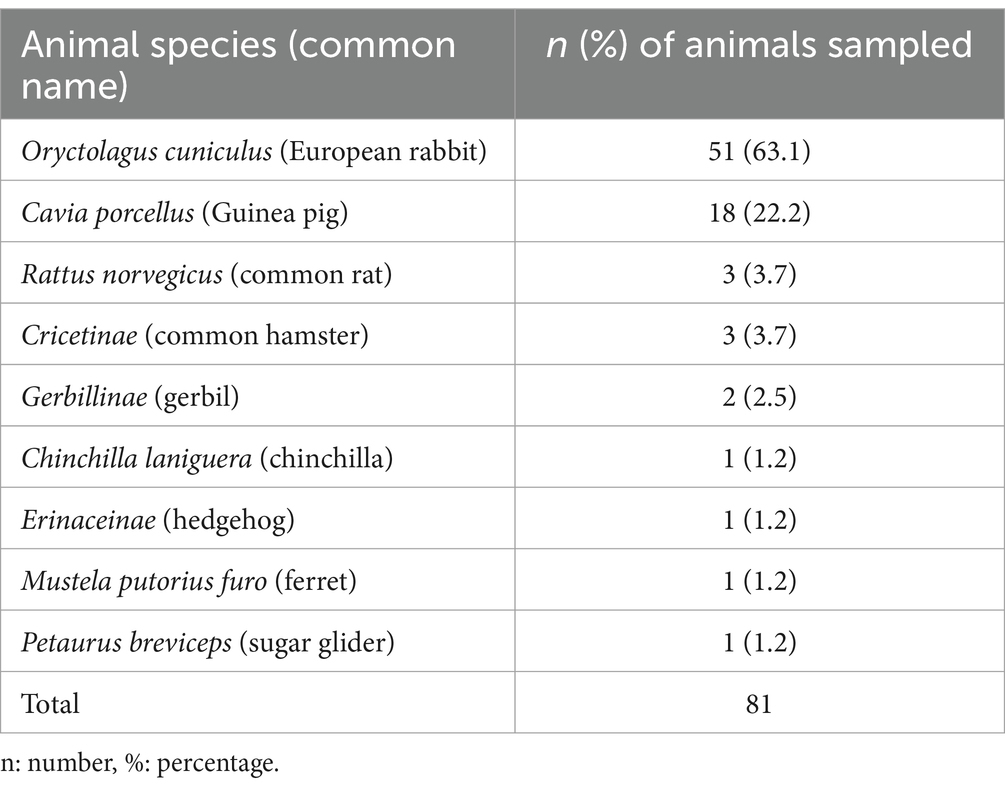
En el presente estudio se muestrearon 81 pequeños mamíferos de nueve especies diferentes. Todas ellas y el número de muestras tomadas por cada especie animal se encuentran en la Tabla 2.
En primer lugar, se recopiló información epidemiológica, incluyendo sexo y edad, de todos los animales de este estudio. Sin embargo, debido a la naturaleza diversa de la población de estudio, que incluye varias especies animales de diferentes familias, los datos no son directamente comparables. En cuanto a su estilo de vida, el 61,7% (50/81) de los animales convivían en el mismo hogar con otros animales, pero ninguno de ellos salía de su casa. En segundo lugar, de acuerdo con la información clínica recopilada, el 68% (55/81) de los animales presentaba una enfermedad crónica y el 14,8% (12/81) tomaba medicación diaria. Por último, del total de animales, el 70,4% (57/81) había sido tratado previamente con AMAs en algún momento de su vida. Los datos presentados en la Figura 1 muestran el historial de tratamiento con AMAs de la población estudiada, detallando el grupo específico de AMAs y la fecha del último tratamiento.
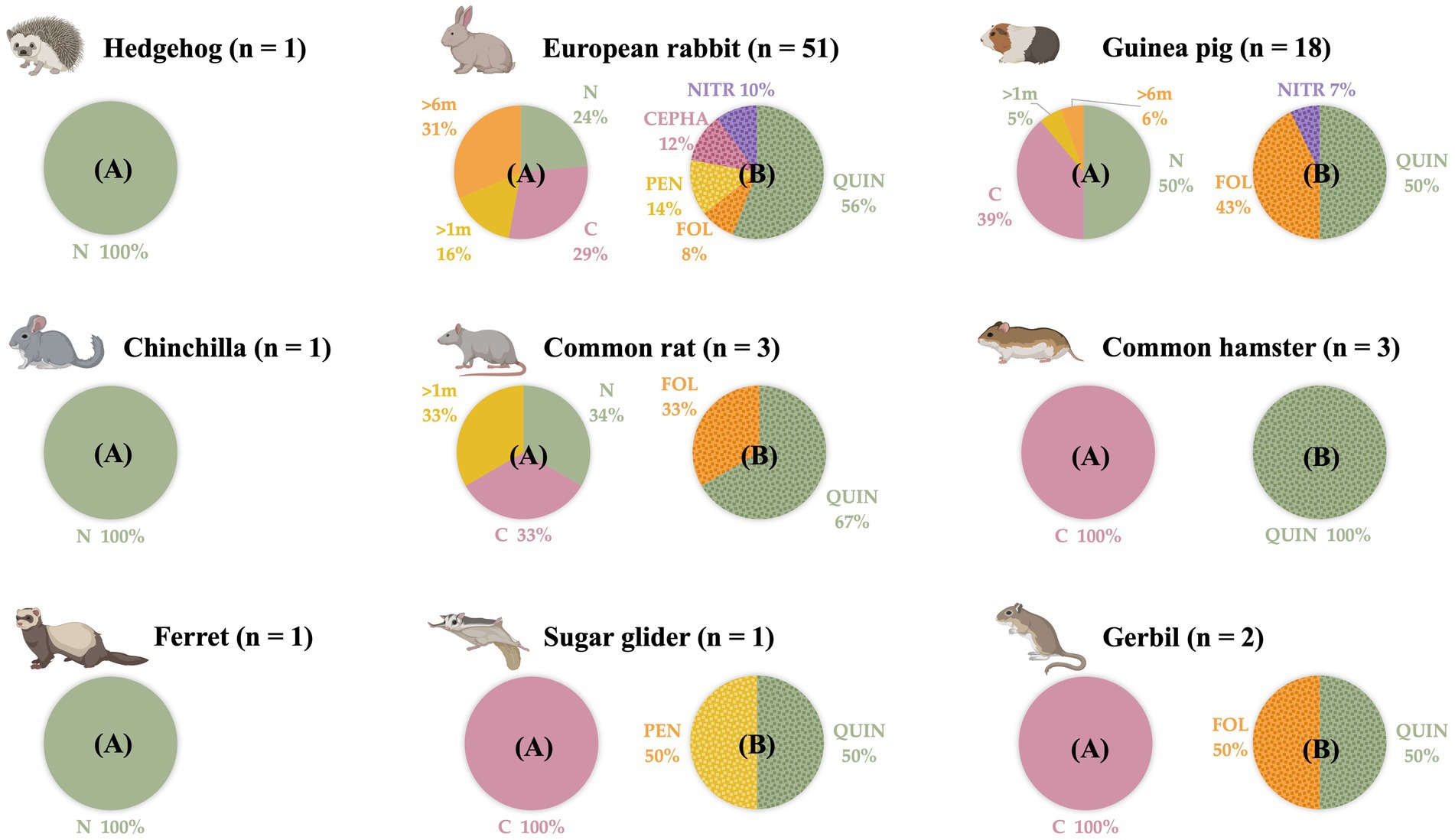
Figura 1. Distribución por especies animales de la población de pequeños mamíferos estudiada, según cuándo fueron tratados por última vez con agentes antimicrobianos y con qué se agrupan los agentes antimicrobianos. n: número de animales muestreados. A) Momento de la última administración de agentes antimicrobianos. N: Nunca. C: actualmente. >1 m: en el último mes. >6 m: en los últimos 6 meses. (B) Grupos de agentes antimicrobianos administrados en la población del estudio en algún momento de su vida. QUIN: quinolonas. FOL: vía inhibidora del folato. CEPHA: cefalosporinas. PEN: penicilinas. NITR: Nitroimidazoles. (Creado por Biorender).
3.2 Prevalencia de Staphylococcus spp.
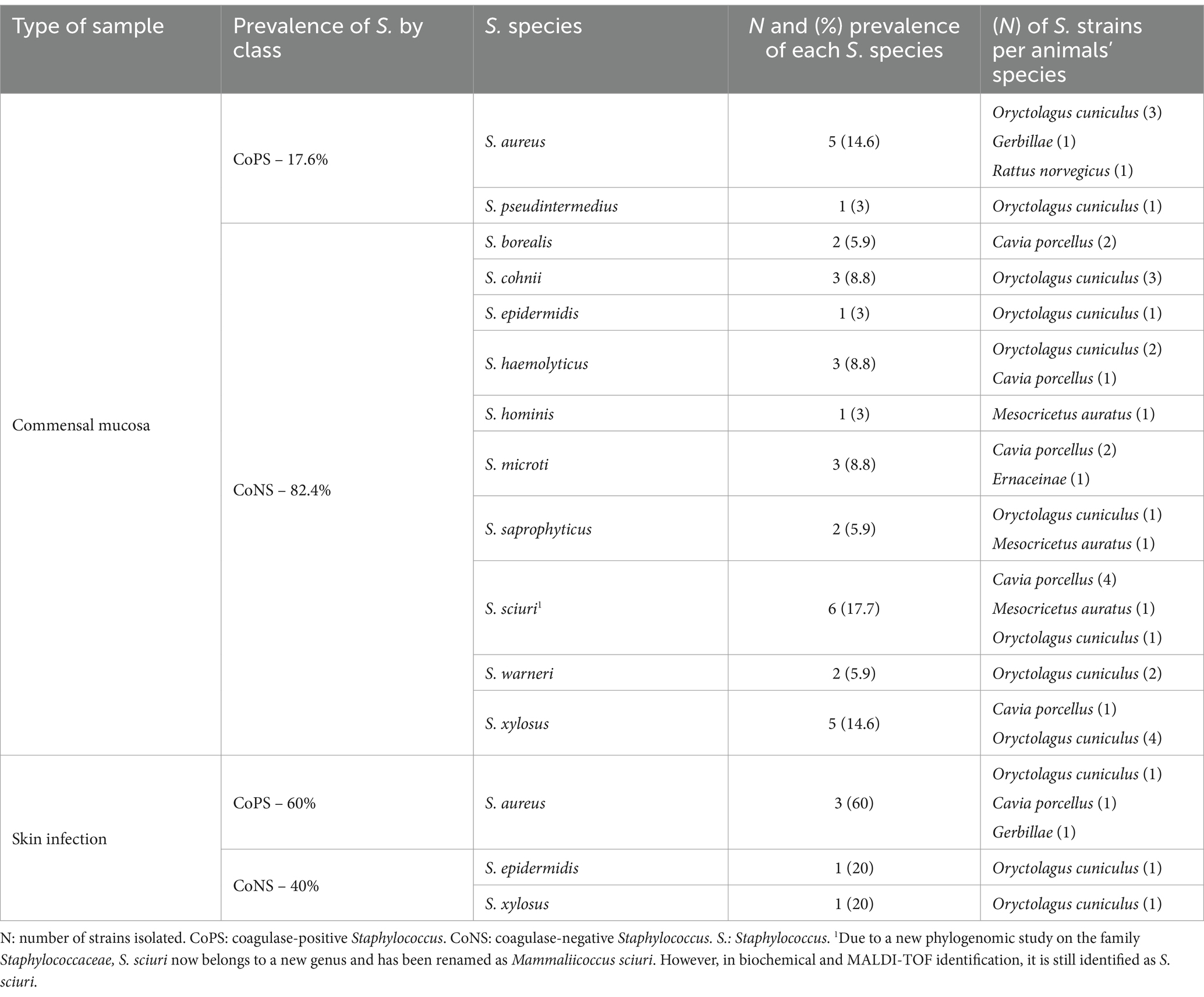
De los 81 especímenes muestreados, 72 eran animales asintomáticos y 9 presentaban una infección cutánea. De todos ellos, la prevalencia total de Staphylococcus spp. fue del 48,2% (39/81), de los cuales el 42% (34/81) y el 6,2% (5/81) fueron comensales y causantes de infecciones por Staphylococcus spp., respectivamente. Todos los Staphylococcus spp. aislados de cada uno de los pequeños mamíferos, junto con el tipo de muestra de la que se derivaron, se enumeran en la Tabla 3.
3.3 Susceptibilidad antimicrobiana en cepas de Staphylococcus spp.
3.3.1 Cepas resistentes a la meticilina
En el presente estudio, todas las cepas de MRS provinieron de los aislados de bacterias comensales, y ninguna de las cepas aisladas de infecciones cutáneas activas mostró resistencia a la meticilina. Las cepas de MRS representaron un 14,7% (5/34), perteneciendo cada una a una especie diferente: Mammaliicoccus sciuri, S. aureus, S. xylosus, S. haemolyticus y S. epidermidis.
3.3.2 Perfil de resistencia a los antimicrobianos
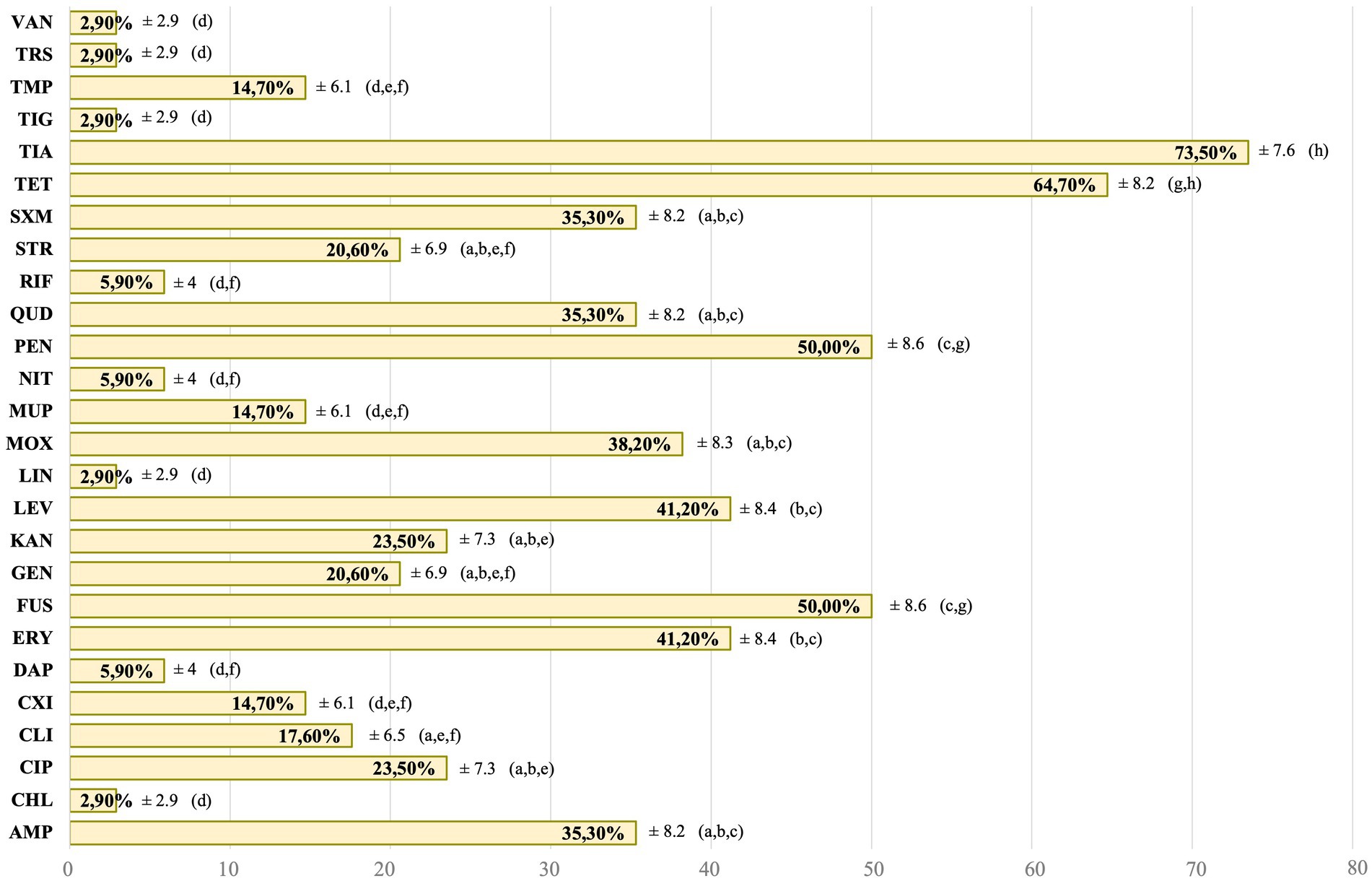
Con respecto a las cepas de Staphylococcus spp. y Mammaliicoccus spp. aisladas de muestras comensales en el presente estudio, todas las cepas (34/34) mostraron RAM en al menos uno de los 27 AMAs estudiados, y el 85,3% (29/34) fueron MDR. Del total de cepas comensales, el 17,6% (6/34) fueron positivas a la prueba D realizada. Los valores de AMR para los grupos de AMA, donde se estudió más de un AMA, fueron del 34,3% para las quinolonas, del 29,4% para las penicilinas y del 17,6% para la vía de los inhibidores del folato. Para el resto de los grupos de AMA, solo se estudió un AMA de cada grupo, por lo que la Figura 2 muestra la RAM para cada AMA individualmente, con la excepción de la oxacilina, que se probó contra una sola cepa de S. pseudintermedius y se encontró que era susceptible. La RAM observada para cada uno de los aislados, se detalla en la Tabla Suplementaria 1.
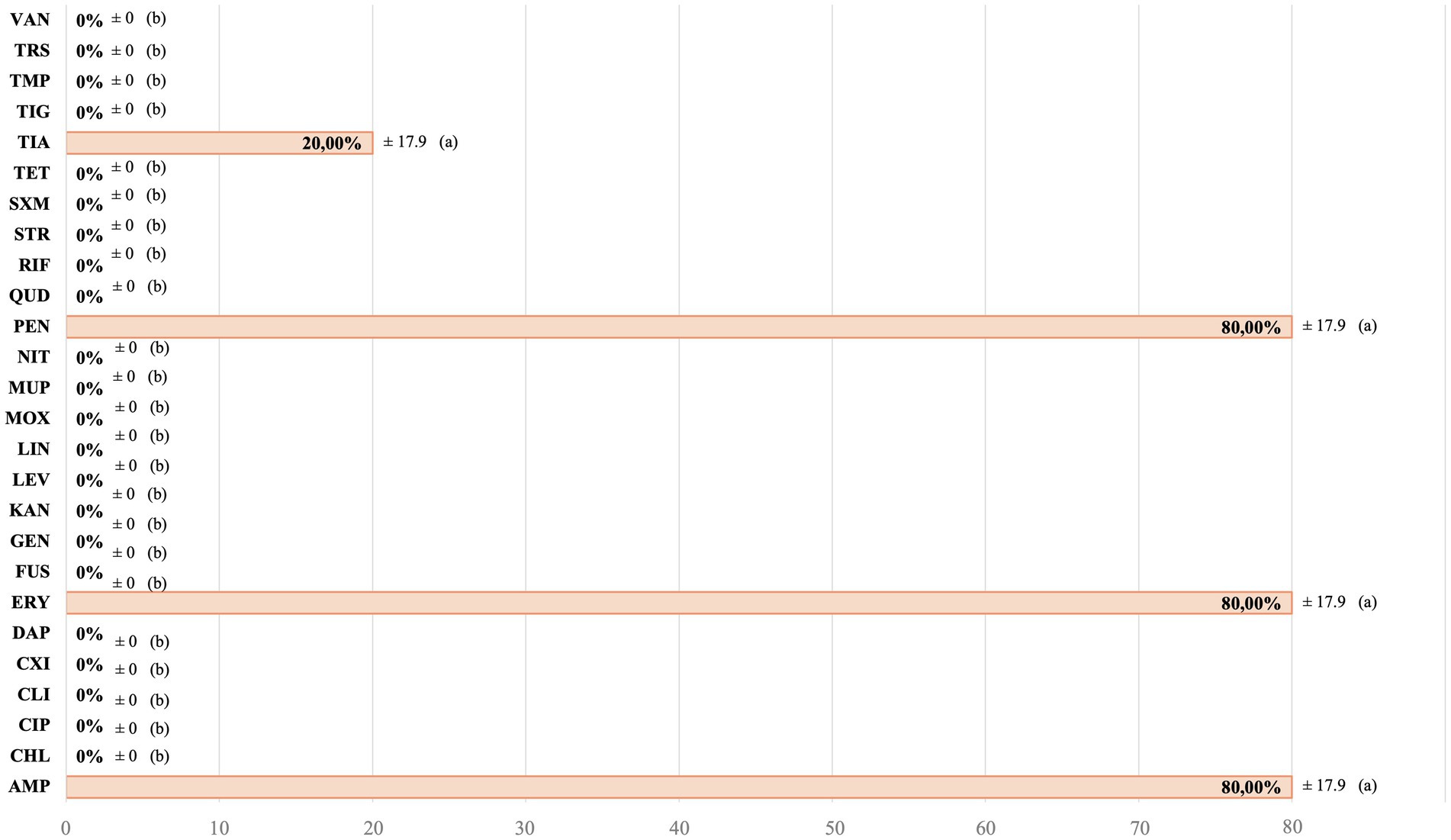
Para todos los Staphylococcus spp. causantes de infección aislados de animales con infecciones cutáneas activas, todos ellos (5/5) fueron resistentes a al menos uno de los AMAs estudiados, y el 40% (2/5) fueron MDR. Además, la prueba D realizada en estas cepas fue positiva en el 80% (4/5) de ellas. Para los grupos de AMA con más de un AMA estudiado, las tasas de AMR fueron del 60 % para las penicilinas y del 20 % para las quinolonas. En cuanto a la vía de los inhibidores del folato, no se observaron RAM. En la Figura 3 se muestra la RAM para cada AMA individual estudiado. En cuanto a la RAM observada en cada uno de los aislados, se detallan en la Tabla Suplementaria 1.
Además, no se observó relación entre los datos epidemiológicos y clínicos recogidos en el cuestionario y la aparición de RAM y MDR, ni en las cepas comensales ni en las cepas de Staphylococcus spp. causantes de infección (valor de p >0,05).
Por último, en este estudio no se observó un patrón discernible en las tendencias generales de la resistencia a los antimicrobianos. Entre los 39 aislados de Staphylococcus spp. y Mammaliicoccus spp., se identificaron 37 patrones distintos de RAM, lo que indica una amplia gama de perfiles de RAM. Solo se duplicaron dos patrones, uno para las vías inhibidoras del folato junto con pleuromutilinas y quinolonas, y el segundo para fusidatos, pleuromutilinas y tetraciclinas, ambos en dos aislados comensales de Staphylococcus spp. (2/39). La lista de patrones de resistencia a los antimicrobianos se puede encontrar en la Tabla Suplementaria 1.
4 Discusión
La familia Staphylococcaceae es una de las bacterias más comunes en general, y en particular entre las bacterias grampositivas, ya que este microorganismo forma parte de la microbiota normal de la piel y las membranas mucosas de los seres humanos y de la mayoría de los animales. Recientemente, se han llevado a cabo nuevos estudios filogenómicos de esta familia, reubicando algunos Staphylococcus spp. en otros géneros, como el antiguo S. sciuri, ahora llamado Mammaliicoccus sciuri. Sin embargo, la importancia de esta cepa sigue siendo la misma, ya que se considera el reservorio evolutivo del gen mecA, que codifica la resistencia a la meticilina. Abarcando todas las especies bacterianas encontradas en este estudio, la prevalencia observada es consistente con la encontrada en otros estudios tanto de NTC como de pequeños mamíferos de vida libre (21, 42, 43), siendo la mayoría de los aislados CoNS. En el presente estudio, M. sciuri fue la bacteria más prevalente en muestras comensales, seguida de S. aureus. En los aislados de infección cutánea, S. aureus fue la especie más prevalente. Aunque se encontró una alta variabilidad, ya que se han observado 10 especies bacterianas adicionales, como se ha reportado en otros estudios realizados en pequeños mamíferos (44). Una de las hipótesis para esta alta diversidad de estafilococos en los pequeños mamíferos NTC podría ser su entorno doméstico. Lo comparten con humanos de todas las edades, y más del 60% de los animales lo comparten con otros animales de compañía, de la misma especie o de otras especies como los perros, que salen a la calle a diario, enriqueciendo el microbioma ambiental de los hogares y favoreciendo así a diferentes especies bacterianas que colonizan la mucosa de NTCAs. Otra posible razón podría ser la alta tasa (más del 70%) de tratamiento antimicrobiano previo en la población estudiada, lo que ejerce presión sobre las comunidades bacterianas y puede favorecer el crecimiento de especies bacterianas seleccionadas.
Además, el aumento de la resistencia a los antimicrobianos y la resistencia multirresistente en las cepas de Staphylococcus spp. en medicina veterinaria plantea un desafío para la salud pública mundial. Las investigaciones indican que estas cepas resistentes pueden persistir en el medio ambiente y transferirse entre animales y humanos (45, 46). Esto subraya la necesidad de evaluar la prevalencia de tales resistencias tanto en bacterias comensales como en bacterias patógenas, lo que requiere un enfoque integral de «Una sola salud». Abordar este problema es vital no solo para evitar fracasos terapéuticos en medicina veterinaria, sino también para salvaguardar la salud humana, especialmente con los resultados observados en este estudio, donde todas las cepas fueron resistentes a al menos uno de los AMAs estudiados, y más del 85% de ellas eran MDR, con una diversa gama de perfiles de RAM, no seguir ningún patrón discernible. Se han observado resultados similares en otros NTCA (21, 47) y animales de compañía tradicionales (48), lo que pone de manifiesto este problema mundial. Además de la MDR, la vigilancia de las cepas de MRS es crucial, principalmente debido a su resistencia a los AMA comunes, lo que complica la selección de tratamientos efectivos. Además, las cepas de MRS son conocidas por su capacidad de propagarse rápidamente en los centros de atención médica. En este estudio, se han observado cepas de MRS (14,7%) tanto en CoPS como en CoNS, tal y como se ha informado en estudios realizados en otros países, como Austria (47) o Turquía (49), en perros, gatos y NTCAs, lo que subraya la necesidad global de monitorizar estas cepas.
En cuanto a cada AMA, destaca la RAM observada frente a la tiamulina (AIT) por encima de las demás con un 73,5%. El AIT es un AMA utilizado exclusivamente en medicina veterinaria, en particular para los animales destinados a la producción de alimentos, especialmente cerdos y aves de corral, para los que se han observado tasas similares de RAM (50). Sin embargo, también está aprobado para su uso, aunque en menor medida, en conejos productores de carne (51), lo que puede contribuir a su uso en conejos mantenidos como animales de compañía y no con fines de producción. Los siguientes AMAs con mayor RAM fueron la tetraciclina (TET; 64,7%) y el ácido fusídico (FUS; 50%), ambos AMAs pertenecientes a la categoría de antimicrobianos de gran importancia (HIA) en la última categorización de la OMS (31). Por lo tanto, cabe esperar que se observen porcentajes más altos de resistencia a los antimicrobianos frente a estos AMA (52) y no frente a los que pertenecen a categorías superiores. Otros AMAs de esta categoría, que son uno de los tratamientos de primera línea, son las vías inhibidoras del folato, como el trimetoprim (TMP), el sulfametoxazol (SMX) o la combinación de ambos (trimetoprim-sulfametoxazol, TRS). Aunque estos AMA pueden administrarse por separado, se observaron tasas más altas de resistencia a la RAM individualmente (TMP, 14,7%; SMX, 35,5%) que las observadas a TRS (2,9%) en combinación, lo que pone de manifiesto la importancia del uso de esta combinación en medicina veterinaria, hasta agotar esta opción terapéutica (53). Sin embargo, se ha observado una mayor resistencia a los antimicrobianos en esta combinación en animales de compañía tradicionales, alcanzando casi el 50% (30, 54).
De los AMAs estudiados, la eritromicina (ERY) representa uno de los primeros tratamientos de elección para las infecciones estafilocócicas, especialmente en pacientes con alergias a la penicilina (55). Las altas tasas de RAM encontradas en este estudio para ERY se alinean con las encontradas en otros estudios en pequeños mamíferos (42), perros y gatos en España (48) y Canadá (56), o en perros y sus dueños en Italia (27), lo que indica que las primeras opciones terapéuticas para tratar estas infecciones pueden comenzar a fallar. Por esta razón, es importante explorar la resistencia a los antimicrobianos con otras opciones terapéuticas, como la clindamicina (CLI), un AMA de categoría HIA, pero utilizada para tratar infecciones cutáneas adquiridas en la comunidad probablemente debidas a MRS (55). Sin embargo, para evaluar si este AMA puede utilizarse en la práctica o no, se debe realizar la prueba D, para confirmar si existe o no un fenotipo de resistencia a CLI inducible (57). En el presente estudio, el 17,6 y el 80% de las cepas comensales y causantes de infección, respectivamente, fueron positivas a la prueba D. Este resultado indica que, aunque la CLI por sí sola puede parecer eficaz, las bacterias pueden desarrollar resistencia durante el tratamiento, lo que puede tener graves implicaciones para el tratamiento de la infección, ya que la resistencia a los antimicrobianos puede comprometer el tratamiento (58). Además, la resistencia inducible a la CLI confirma el fenotipo de resistencia del grupo de macrólidos, lincosamidas, estreptogramina ß y pleuromutilina (MLS_B-P), ya que los genes de resistencia que inducen resistencia a la CLI también pueden inducir resistencia a MLS_B-P, que son antibióticos comúnmente utilizados para el tratamiento del SARM (59). Esta puede ser una de las razones por las que se observaron altas tasas de RAM a estos AMA en este estudio.
Cuando todas las demás opciones terapéuticas fallan, los últimos AMAs que se pueden utilizar en medicina veterinaria son los antimicrobianos críticos importantes (HPCIAs) de mayor prioridad, incluyendo las quinolonas (31), aunque en la población estudiada, las quinolonas fueron el grupo de AMAs más administrado. En este estudio, las tres quinolonas evaluadas: levofloxacino (LEV), ciprofloxacino (CIP) y moxifloxacina (MOX), son AMAs solo aprobados para su uso en humanos, pero no en medicina veterinaria en la UE (31). Por lo tanto, la alta RAM observada (34,3%) en este grupo, similar a la observada en otro estudio en conejos (21), es preocupante debido a los fracasos terapéuticos que podría plantear, y la posibilidad de transmisión de estas RAM a otras bacterias patógenas (60).
Por último, la última categoría disponible cuando todas las demás han fracasado está reservada para la medicina humana y no está autorizada para la medicina veterinaria, siendo más comúnmente conocida como AMA de último recurso (31). Los AMAs de esta categoría estudiados fueron vancomicina (VAN), tigeciclina (TIG), linezolid (LIN), daptomicina (DAP), nitrofurantoína (NIT) y mupirocina (MUP). Estos AMA generalmente se reservan para infecciones graves o potencialmente mortales que no responden a las terapias estándar de AMA que utilizan las categorías anteriores. En cuanto a la RAM observada, se encontró una baja prevalencia de casi todos los AMA, en línea con otros estudios realizados en pequeños mamíferos en la República Checa (43) y en perros, gatos y conejos en Lituania (61), pero no para el MUP. Este AMA es solo para uso tópico, utilizado para infecciones cutáneas complicadas, incluidas las causadas por MRS, y para descolonizar portadores nasales de S. aureus. Aunque dada la importancia de esta AMA debería observarse un menor porcentaje de RAM, la prevalencia reportada en este estudio (14,7%) se encuentra dentro de rangos normales, considerando que en España la RAM para esta AMA en aislados de CoNS es de alrededor del 40% y para S. aureus entre el 8 y el 10%, en medicina humana (62).
El presente estudio se centra en evaluar la prevalencia y los patrones de resistencia a los antimicrobianos de cepas de Staphylococcaceae aisladas de muestras de mucosa e infecciones cutáneas en pequeños mamíferos. Los resultados ponen de manifiesto la alta prevalencia de la resistencia a los antimicrobianos y la resistencia a los microrrusos en los pequeños mamíferos, lo que subraya la necesidad de un enfoque integral de «Una sola salud» para abordar este problema, ya que estos animales comparten el entorno doméstico con los seres humanos y otros animales. Además, la diversidad de especies bacterianas y la alta tasa de tratamientos antimicrobianos previos sugieren una presión selectiva significativa, lo que puede favorecer la aparición de RAM. Esta investigación es un paso inicial para futuras iniciativas destinadas a controlar y prevenir la proliferación de AMR y MDR en las NTCA. Sin embargo, es esencial realizar más investigaciones para validar nuestros resultados en una población de estudio más grande y representativa.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo/Material complementario, las consultas posteriores pueden dirigirse a los autores correspondientes.
Declaración de ética
Los estudios en animales fueron aprobados por los Comités de Ética Animal de la Universidad Cardenal Herrera-CEU. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones de los autores
AM-F: Curación de datos, Análisis formal, Investigación, Metodología, Recursos, Software, Visualización, Redacción – borrador original, Redacción – revisión y edición. CM: Conceptualización, Obtención de fondos, Investigación, Metodología, Administración de proyectos, Recursos, Supervisión, Validación, Visualización, Redacción – revisión y edición. JV-G: Metodología, Recursos, Supervisión, Validación, Visualización, Redacción – revisión y edición. CG-C: Investigación, Metodología, Recursos, Supervisión, Validación, Redacción – Revisión y Edición. VA-M: Investigación, Metodología, Supervisión, Validación, Redacción – Revisión y Edición. SV: Conceptualización, Obtención de fondos, Investigación, Administración de proyectos, Recursos, Supervisión, Validación, Visualización, Redacción – revisión y edición. LM-D: Conceptualización, Análisis formal, Investigación, Metodología, Administración de proyectos, Recursos, Supervisión, Validación, Visualización, Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. Esta investigación ha sido financiada por la Universidad Cardenal Herrera-CEU, subvención número INDI23/39.
Reconocimientos
Los autores queremos agradecer a los propietarios de los animales de compañía y a los veterinarios, que aceptaron voluntariamente participar en el estudio, por permitirnos llevar a cabo este trabajo en la Clínica Veterinaria Selvätica. Los autores también quieren expresar su agradecimiento al Servicio de Microbiología del Consorcio Hospital General de Valencia por su inestimable colaboración y apoyo técnico. Por último, los autores quieren agradecer al grupo de investigación «Mejora de la Seguridad Alimentaria y de los Productos Finales en el Sistema Productivo» (Facultad de Veterinaria, Universidad Cardenal Herrera-CEU) su apoyo técnico y financiero. La versión en inglés fue revisada por N. Macowan English Language Service.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda hacer su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en:https://www.frontiersin.org/articles/10.3389/fvets.2024.1378346/full#supplementary-material
Referencias
1. FEDIAF. La industria europea de animales de compañía y alimentos. Informe anual 2023. (2023). Disponible en: https://europeanpetfood.org/about/annual-report/. Consultado el 7 de julio de 2023
2. Walsh, F. Vínculos humano-animal II: el papel de las mascotas en los sistemas familiares y la terapia familiar. Proceso de Fam. (2009) 48:481–99. doi: 10.1111/J.1545-5300.2009.01297.X
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Marín, C, Lorenzo-Rebenaque, L, Laso, O, Villora-González, J, y Vega, S. Reptiles domésticos: una fuente potencial de transmisión de Salmonella multirresistente. Frente. Vet. Sci. (2021) 7:1157. doi: 10.3389/FVETS.2020.613718/BIBTEX
4. Overgaauw, PAM, Vinke, CM, van Hagen, MAE, y Lipman, LJA. Una perspectiva de una sola salud en la relación humano-animal de compañía con énfasis en los aspectos zoonóticos. Int J Environ Res Salud Pública. (2020) 17:3789. doi: 10.3390/IJERPH17113789
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Magiorakos, AP, Srinivasan, A, Carey, RB, Carmeli, Y, Falagas, ME, Giske, CG, et al. Bacterias multirresistentes, extensivamente resistentes a los fármacos y panrresistentes: una propuesta de expertos internacionales para definiciones estándar provisionales de resistencia adquirida. Clin Microbiol Infectar. (2012) 18:268–81. doi: 10.1111/j.1469-0691.2011.03570.X
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Organización Mundial de la Salud. Sistema mundial de vigilancia de la resistencia y el uso de antimicrobianos (GLASS). Memoria: 2022. Ginebra: Organización Mundial de la Salud. (2022) 2003–5 p.
7. Zhu, Y, Huang, WE y Yang, Q. Perspectiva clínica de la resistencia a los antimicrobianos en bacterias. Infectar la resistencia a los medicamentos. (2022) 15:735–46. doi: 10.2147/IDR. S345574
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Pomba, C, Rantala, M, Greko, C, Baptiste, KE, Catry, B, van Duijkeren, E, et al. Riesgo para la salud pública de la transferencia de resistencia a los antimicrobianos de los animales de compañía. J Quimiomadre antimicrobiana. (2017) 72:DKW481–968. doi: 10.1093/JAC/DKW481
9. Anjum, MF, Marco-Jimenez, F, Duncan, D, Marín, C, Smith, RP, and Evans, SJ. Livestock-associated methicillin-resistant Staphylococcus aureus from animals and animal products in the UK. Front Microbiol. (2019) 10:442883. doi: 10.3389/FMICB.2019.02136/BIBTEX
10. European Food and Safety Authority. The European Union summary report on antimicrobial resistance in zoonotic and indicator bacteria from humans, animals and food in 2020/2021. EFSA J. (2023) 21:e07867. doi: 10.2903/J.EFSA.2023.7867
11. European Centre for Disease Prevention and Control. European Antimicrobial Resistance Surveillance Network (EARS-Net). (2023). Available at: https://www.ecdc.europa.eu/en/about-us/networks/disease-networks-and-laboratory-networks/ears-net-data. Accessed July 19, 2023
12. Plan Nacional de Resistencias a los Antibióticos. ¿Quiénes somos? (2023). Available at: https://www.resistenciaantibioticos.es/es/quienes-somos Accessed July 7, 2023
13. Marco-Fuertes, A, Marin, C, Lorenzo-Rebenaque, L, Vega, S, and Montoro-Dasi, L. Antimicrobial resistance in companion animals: a new challenge for the one health approach in the European Union. Vet Sci. (2022) 9:208. doi: 10.3390/VETSCI9050208
14. Mader, R, Damborg, P, Amat, JP, Bengtsson, B, Bourély, C, Broens, EM, et al. Building the European antimicrobial resistance surveillance network in veterinary medicine (EARS-vet). Eur Secur. (2021) 26:2001359. doi: 10.2807/1560-7917.ES.2021.26.4.2001359/CITE/PLAINTEXT
15. Misic, AM, Davis, MF, Tyldsley, AS, Hodkinson, BP, Tolomeo, P, Hu, B, et al. La microbiota compartida de humanos y animales de compañía evaluada a partir de sitios de transporte de Staphylococcus. Microbioma. (2015) 3:1–19. doi: 10.1186/S40168-014-0052-7/FIGURES/4
16. Khairullah, AR, Kurniawan, SC, Sudjarwo, SA, Effendi, MH, Afnani, DA, Silaen, OSM, et al. Detección de Staphylococcus aureus multirresistente (MDR) y estafilococos coagulasa negativos (CoNS) en leche de vaca y manos de agricultores en Java Oriental, Indonesia. Biodiversitas. (2023) 24:658–64. doi: 10.13057/BIODIV/D240174
17. Organización Mundial de la Salud. Lista de bacterias para las que se necesitan urgentemente nuevos antibióticos. (2017). Disponible en: https://www.who.int/news/item/27-02-2017-who-publishes-list-of-bacteria-for-which-new-antibiotics-are-urgently-needed. Consultado el 10 de diciembre de 2023
18. Lord, J, Millis, N, Jones, RD, Johnson, B, Kania, SA, y Odoi, A. Estudio epidemiológico de los predictores de multirresistencia y resistencia a meticilina entre Staphylococcus spp. aislados de especímenes caninos enviados a un laboratorio de diagnóstico en Tennessee, EE. UU. ParesJ. (2023) 11:E15012. doi: 10.7717/PEERJ.15012/SUPP-1
19. França, A, Gaio, V, Lopes, N y Melo, LDR. Factores de virulencia en los estafilococos coagulasa negativos. Patógenos. (2021) 10:170. doi: 10.3390/PATHOGENS10020170
20. González, J, Hernández, L, Tabera, A, Bustamante, AV, y Sanso, AM. Staphylococcus aureus resistente a meticilina y Staphylococcus coagulasa negativo de comedores escolares de Argentina. Foodborne Pathog Dis. (2023) 21:44–51. doi: 10.1089/FPD.2023.0071
21. Fernández, M, Garcías, B, Durán, I, Molina-López, RA, y Darwich, L. Situación actual de las infecciones bacterianas y perfiles de resistencia a los antimicrobianos en conejos de compañía en España. Veterinario Sci. (2023) 10:352. doi: 10.3390/VETSCI10050352/S1
22. Ju, LIMJ, Hyeok, KIMD, Ju, LEEJ, Geun, KIMD, Hun, KIMS, Min, W, et al. Prevalencia de Lawsonia intracellularis, Salmonella spp. y Eimeria spp. en conejos sanos y diarreicos. J Vet Med Sci. (2012) 74:263–5. doi: 10.1292/jvms.11-0389
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Russo, TP, Borrelli, L, Minichino, A, Fioretti, A, y Dipineto, L. Ocurrencia y resistencia antimicrobiana de Staphylococcus aureus aislado de conejos domésticos sanos. Vector Transmitido Zoonótico Dis. (2024) 24:135–40. doi: 10.1089/VBZ.2023.0066
24. Cardoso, S, Le, L’h A, Marques, I, Almeida, A, Sousa, S, Saavedra, MJ, et al. Desvelar la aparición de patógenos multirresistentes en animales de compañía exóticos de Francia: un estudio exhaustivo (2017-2019). Implemento de una sola salud Res. (2023) 3:161–76. doi: 10.20517/OHIR.2023.30
25. Marco-Fuertes, A, Marín, C, Gimeno-Cardona, C, Artal-Muñoz, V, Vega, S, y Montoro-Dasi, L. Staphylococcus spp. comensal multirresistente y causante de infecciones aislado de animales de compañía en la Comunidad Valenciana. Veterinario Sci. (2024) 11:54. doi: 10.3390/VETSCI11020054/S1
26. Frosini, SM, Bond, R, King, RH y Loeffler, A. El olfato no es suficiente: el muestreo multicéntrico es el mejor para la detección de MRSP en perros y hogares. Veterinario Dermatol. (2022) 33:576–80. doi: 10.1111/VDE.13118
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Nocera, FP, Pizzano, F, Masullo, A, Cortese, L, y De Martino, L. Colonización de especies de Staphylococcus resistentes a los antimicrobianos en perros, sus propietarios y personal veterinario del Hospital de enseñanza veterinaria de Nápoles, Italia. Patógenos. (2023) 12:1016. DOI: 10.3390/PATHOGENS12081016
28. DeCubellis, J. Emergencias comunes en conejos, conejillos de indias y chinchillas. Vet Clin North Am Exot Anim Pract. (2016) 19:411–29. doi: 10.1016/j.cvex.2016.01.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Biotipor® MALDI para la investigación microbiana | Bruker. Disponible en: https://www.bruker.com/en/applications/microbiology-and-diagnostics/microbiological-research/maldi-biotyper-for-microbial-research.html. Consultado el 14 de julio de 2024
30. Marco-Fuertes, A, Jordá, J, Marín, C, Lorenzo-Rebenaque, L, Montoro-Dasi, L, y Vega, S. Cepas de Escherichia coli multirresistentes a antibióticos humanos de último recurso aislados de animales de compañía sanos en la región de Valencia. Antibióticos. (2023) 12:1638. DOI: 10.3390/ANTIBIOTICS12111638
31. QUIÉN. Lista de Antimicrobianos de Importancia Médica de la OMS. Una herramienta de gestión de riesgos para mitigar la resistencia a los antimicrobianos debido al uso no humano, vol. 2023.
32. Mader, R, Demay, C, Jouvin-Marche, E, Ploy, MC, Barraud, O, Bernard, S, et al. Definición del ámbito de aplicación de la Red Europea de Vigilancia de la Resistencia a los Antimicrobianos en Medicina Veterinaria (EARS-vet): un enfoque ascendente y de una sola salud. J Quimiomadre antimicrobiana. (2022) 77:816–26. doi: 10.1093/JAC/DKAB462
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Ardanuy, C, María, EC, Morosini, I, y Torres, C. Detección fenotípica de mecanismos de resistencia en grampositivos. Recomendaciones de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC): Procedimientos en Microbiología Clínica (2011), Oxford University Press, en nombre de la Sociedad Británica de Quimioterapia Antimicrobiana.
34. Comisión Europea. EUR-Lex – 32023D1017 – ES – EUR-Lex. Disponible en: https://eur-lex.europa.eu/eli/dec_impl/2023/1017/oj Consultado el 21 de diciembre de 2023
35. ThermoFisher Scientific. Guía de placas Thermo Scientific Sensititre para pruebas de susceptibilidad a los antimicrobianos. Disponible en: https://assets.thermofisher.com/TFS-Assets/MBD/brochures/Sensititre-Plate-Guide-Booklet-EN.pdf. Consultado el 21 de diciembre de 2023
36. Comité Europeo de Pruebas de Susceptibilidad a los Antimicrobianos. Tablas de puntos de corte para la interpretación de MIC y diámetros de zona. Versión 14.0, 2024. (2024). Disponible en: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_14.0_Breakpoint_Tables.pdf. Consultado el 4 de enero de 2024
37. Instituto de Normas Clínicas y de Laboratorio. Normas de rendimiento para las pruebas de susceptibilidad a los antimicrobianos. 33ª ed. Suplemento CLSI M100. EE.UU.: Las nuevas directrices del Comité Europeo de Pruebas de Susceptibilidad a los Antimicrobianos (2023).
38. Instituto de Normas Clínicas y de Laboratorio. Normas de rendimiento para discos antimicrobianos y pruebas de susceptibilidad a la dilución para bacterias aisladas de animales. 6ª ed. Suplemento CLSI VET01S. EE. UU.: Las nuevas directrices del Clinical and Laboratory Standards Institute (2023).
39. Searle, SR, Speed, FM y Milliken, GA. Medias marginales de la población en el modelo lineal: una alternativa a las medias de mínimos cuadrados. Am Stat. (1980) 34:216–21. doi: 10.1080/00031305.1980.10483031
40. Fox, J y Weisberg, S. Un compañero de R para la regresión aplicada. Tercero. Thousand Oaks CA: Sabio (2019).
41. Graves, S, Piepho, H, Selzer, L y Dorai-Raj, S. Multcompview: Visualizaciones de comparaciones pareadas. Paquete de R versión 0.1–9. (2023). Disponible en: https://cran.r-project.org/web/packages/multcompView/index.html. Consultado el 27 de julio de 2023
42. Muñoz-Ibarra, E, Molina-López, RA, Durán, I, Garcias, B, Martín, M, y Darwich, L. Resistencia antimicrobiana en bacterias aisladas de animales de compañía exóticos: la situación en la Península Ibérica. Animales. (2022) 12:1912. doi: 10.3390/ANI12151912
43. Santana, JA, Colombo, SA, Silva, BA, Diniz, AN, de Almeida, LR, Oliveira Junior, CA, et al. Clostridioides difficile y estafilococos multirresistentes en roedores y marsupiales de vida libre en parques de Belo Horizonte, Brasil. Braz J Microbiol. (2022) 53:401–10. doi: 10.1007/s42770-021-00640-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Kmeť, V, Čuvalová, A, y Stanko, M. Pequeños mamíferos como centinelas de los estafilococos resistentes a los antimicrobianos. Folia Microbiol. (2018) 63:665–8. doi: 10.1007/s12223-018-0594-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. Fletcher, S. Comprender la contribución de los factores ambientales en la propagación de la resistencia a los antimicrobianos. Environ Health Prev Med. (2015) 20:243–52. doi: 10.1007/S12199-015-0468-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. Graham, DW, Bergeron, G, Bourassa, MW, Dickson, J, Gomes, F, Howe, A, et al. Complejidades en la comprensión de la resistencia a los antimicrobianos en los sistemas animales domésticos, humanos y ambientales. Ann n y Acad Sci. (2019) 1441:17–30. doi: 10.1111/NYAS.14036
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Loncaric, I, Tichy, A, Handler, S, Szostak, MP, Tickert, M, Diab-Elschahawi, M, et al. Prevalencia de Staphylococcus sp. (MRS) resistente a meticilina en diferentes animales de compañía y determinación de factores de riesgo para la colonización con antibióticos MRS. (2019) 8:17–30. doi: 10.3390/ANTIBIOTICS8020036
Resumen de PubMed | Texto completo de Crossref | Google Académico
48. Li, Y, Fernández, R, Durán, I, Molina-López, RA, y Darwich, L. Resistencia antimicrobiana en bacterias aisladas de perros y gatos de la Península Ibérica. Microbiol frontal. (2021) 11:621597. doi: 10.3389/fmicb.2020.621597
Resumen de PubMed | Texto completo de Crossref | Google Académico
49. Cengiz, S, Okur, S, Oz, C, Turgut, F, Gumurcinler, B, Sevuk, NS, et al. Prevalencia y diversidad clonal de Staphylococcus aureus resistente a meticilina y Staphylococcus pseudintermedius resistente a meticilina aislados de perros y gatos con secreción ocular. Acta Microbiol Immunol colgado. (2023) 70:134–41. doi: 10.1556/030.2023.01899
50. Li, B, Wendlandt, S, Yao, J, Liu, Y, Zhang, Q, Shi, Z, et al. Detección y nuevo entorno genético del gen de resistencia lsa(E) a pleuromutilina-lincosamida-estreptogramina en Staphylococcus aureus resistente a meticilina de origen porcino. J Quimadre antimicrobiana. (2013) 68:1251–5. doi: 10.1093/JAC/DKT015
Resumen de PubMed | Texto completo de Crossref | Google Académico
51. van Duijkeren, E, Greko, C, Pringle, M, Baptiste, KE, Catry, B, Jukes, H, et al. Pleuromutilinas: uso en animales productores de alimentos en la Unión Europea, desarrollo de resistencias e impacto en la salud humana y animal. J Quimiomadre antimicrobiana. (2014) 69:2022–31. doi: 10.1093/JAC/DKU123
Resumen de PubMed | Texto completo de Crossref | Google Académico
52. Schwarz, S, Feßler, AT, Loncaric, I, Wu, C, Kadlec, K, Wang, Y, et al. Resistencia antimicrobiana entre estafilococos de origen animal. Microbiol Spectr. (2018) 6:1–29. DOI: 10.1128/microbiolspec. ARBA-0010-2017
53. Broens, EM, y van Geijlswijk, IM. Uso prudente de los antimicrobianos en la medicina de animales exóticos. Vet Clin North Am Exot Anim Pract. (2018) 21:341–53. doi: 10.1016/j.cvex.2018.01.014
Resumen de PubMed | Texto completo de Crossref | Google Académico
54. Teng, L, Feng, M, Liao, S, Zheng, Z, Jia, C, Zhou, X, et al. Un estudio transversal de Escherichia coli multirresistente derivada de animales de compañía en Hangzhou, China. Microbiol Spectr. (2023) 11:e0211322. doi: 10.1128/spectrum.02113-22
Resumen de PubMed | Texto completo de Crossref | Google Académico
55. Manual MSD Edición Profesional. Infecciones estafilocócicas – Enfermedades infecciosas. Disponible en: https://www.msdmanuals.com/professional/infectious-diseases/gram-positive-cocci/staphylococcal-infections. Consultado el 12 de enero de 2024
56. Awosile, BB, Mcclure, JT, Saab, ME y Heider, LC. Resistencia a los antimicrobianos en bacterias aisladas de gatos y perros de las provincias atlánticas, Canadá, entre 1994 y 2013. ¿Puede Vet J. (2018) 59:885.
57. Hamzah, AMC, Yeo, CC, Puah, SM, Chua, KH, Rahman, NIA, Abdullah, FH, et al. Resistencia a tigeciclina y clindamicina inducible en aislados clínicos de Staphylococcus aureus resistente a meticilina de Terengganu, Malasia. J Med Microbiol. (2019) 68:1299–305. doi: 10.1099/jmm.0.000993
Resumen de PubMed | Texto completo de Crossref | Google Académico
58. Shrestha, B. D test: una prueba simple con grandes implicaciones para el patrón de resistencia de Staphylococcus aureus macrólido-Lincosamida-Estreptograma B. Revisa el artículo Nepal Med Coll J. (2014) 16:88–94.
59. Thapa, D, Pyakurel, S, Thapa, S, Lamsal, S, Chaudhari, M, Adhikari, N, et al. Staphylococcus aureus con resistencia inducible a clindamicina y resistencia a meticilina en un hospital terciario en Nepal. Trop Med Salud. (2021) 49:1–7. doi: 10.1186/S41182-021-00392-2/TABLES/3
60. Belas, A, Menezes, J, Gama, LT y Pomba, C. Intercambio de genes de resistencia a los antimicrobianos clínicamente importantes por parte de animales de compañía y sus miembros humanos del hogar (2020) 26: 1174–85. doi: 10.1089/MDR.2019.0380,
61. Ruzauskas, M, Couto, N, Kerziene, S, Siugzdiniene, R, Klimiene, I, Virgailis, M, et al. Prevalencia, distribución de especies y patrones de resistencia a los antimicrobianos de estafilococos resistentes a la meticilina en animales de compañía lituanos. Acta Vet Scand. (2015) 57:1–7. doi: 10.1186/S13028-015-0117-Z/TABLES/3
Palabras clave: resistencia a antimicrobianos, resistencia a meticilina, animales de compañía no tradicionales, pequeños mamíferos, Staphylococcus spp.
Cita: Marco-Fuertes A, Marín C, Villora-González J, Gimeno-Cardona C, Artal-Muñoz V, Vega S y Montoro-Dasi L (2024) Pequeños mamíferos de compañía no tradicionales en España como reservorios de Estafilococos resistentes a los antimicrobianos. Frente. Vet. Sci. 11:1378346. doi: 10.3389/fvets.2024.1378346
Recibido: 29 de enero de 2024; Aceptado: 26 de julio de 2024;
Publicado: 09 de agosto de 2024.
Editado por:
Ioannis Magouras, Universidad de Berna, Suiza
Revisado por:
Francesca Paola Nocera, Universidad de Nápoles Federico II, Italia
Madubuike Umunna Anyanwu, Universidad de Nigeria, Nigeria
Copyright © 2024 Marco-Fuertes, Marín, Villora-González, Gimeno-Cardona, Artal-Muñoz, Vega y Montoro-Dasi. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Laura Montoro-Dasi, de laura.montoro@uchceu.es; Santiago Vega, svega@uchceu.es
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

