Un SNP intrónico afecta el desarrollo del músculo esquelético regulando la expresión de TP63
Un SNP intrónico afecta el desarrollo del músculo esquelético regulando la expresión de TP63
 Yufen Chen1,2
Yufen Chen1,2  Zhen Wang1
Zhen Wang1  Xiaolu Qu1
Xiaolu Qu1  Canción de Bangmin1
Canción de Bangmin1  Yueting Tang1
Yueting Tang1  Bugao Li2
Bugao Li2  Guoqing Cao2*
Guoqing Cao2*  Guoqiang Yi1,3,4*
Guoqiang Yi1,3,4*- 1Laboratorio de Guangdong de Agricultura Moderna de Lingnan, Laboratorio Clave de Multiómica Ganadera y Avícola de MARA, Instituto de Genómica Agrícola de Shenzhen, Academia China de Ciencias Agrícolas, Shenzhen, China
- 2Facultad de Ciencia Animal, Universidad Agrícola de Shanxi, Jinzhong, China
- 3Instituto Kunpeng de Agricultura Moderna en Foshan, Academia China de Ciencias Agrícolas, Foshan, China
- 4Instituto de Investigación para la Revitalización Rural del Condado Autónomo de Bama Yao, Bama, China
Fondo: El desarrollo del músculo esquelético porcino es fundamental para mejorar la producción de carne. El TP63, un factor de transcripción, regula los procesos celulares vitales, pero su papel en la proliferación del músculo esquelético no está claro.
Métodos: Los efectos de TP63 sobre la viabilidad y proliferación de las células del músculo esquelético se investigaron utilizando mioblastos del músculo esquelético de ratón y porcino. El análisis de barrido selectivo en cerdos occidentales identificó a TP63 como un gen candidato potencial para el desarrollo del músculo esquelético. La correlación entre la sobreexpresión de TP63 y la proliferación celular se evaluó mediante PCR cuantitativa en tiempo real (RT-qPCR) y 5-etinil-2′-desoxiuridina (EDU).
Resultados: El estudio reveló una correlación positiva entre la sobreexpresión de TP63 y la proliferación de células del músculo esquelético. El análisis bioinformático predijo una interacción entre MEF2A, otro factor de transcripción, y el sitio de mutación de TP63. La validación experimental a través de ensayos de luciferasa dual confirmó que un SNP potenciador candidato podría influir en la unión a MEF2A, regulando posteriormente la expresión de TP63 y promoviendo la proliferación de células musculares esqueléticas.
Conclusión: Estos hallazgos ofrecen evidencia experimental para una mayor exploración de los mecanismos de desarrollo del músculo esquelético y el avance de estrategias de mejoramiento genético destinadas a mejorar los rasgos de producción de carne.
1 Introducción
La capacidad de producción de carne de los cerdos está estrechamente relacionada con el desarrollo del músculo esquelético, que sirve como fuente primaria de proteína animal (1, 2). La masa muscular esquelética representa una proporción significativa del peso corporal, lo que enfatiza la importancia de la diferenciación y proliferación de las células musculares en la mejora de la tasa de crecimiento de los cerdos (3-5). La miogénesis es un proceso coordinado que involucra varias etapas, incluida la identificación de células mesodérmicas multipotenciales, la proliferación y migración de mioblastos adultos, la fusión de mioblastos en fibras musculares multinucleadas y la maduración de las fibras musculares (6-8). Por lo tanto, el estudio de los mecanismos moleculares que subyacen al crecimiento y desarrollo del músculo esquelético porcino es esencial para mejorar la tasa de crecimiento y el rendimiento de la carne de cerdo (9, 10).
La formación del músculo esquelético y los rasgos de producción de carne están regulados por intrincados mecanismos moleculares que involucran la interacción de varios genes y vías (11, 12). Se han llevado a cabo extensas investigaciones en todo el mundo, lo que ha dado como resultado un progreso significativo y la identificación de varios genes y marcadores moleculares asociados con los rasgos de producción de carne (13, 14). Por ejemplo, muchos estudios relacionados con QTL encontraron que los genes seleccionados de cerdos locales en China estaban relacionados principalmente con la grasa y los rasgos de reproducción, mientras que los genes seleccionados de razas de cerdo magro estaban enriquecidos en peso corporal, tasa de crecimiento y rasgos de la canal (15-18). Con la implementación de técnicas de genotipado de alto rendimiento, se ha hecho posible la identificación de barridos selectivos a nivel del genoma (19). Rubin et al. identificaron tres genes (NR6A1, PLAG1 y LCORL) que contribuyen a la longitud corporal de los cerdos domésticos europeos (20). Ma et al. revelaron fuertes firmas de selección en Duroc que pueden afectar la masa muscular magra (21, 22). Sin embargo, es necesaria una comprensión más profunda de los mecanismos moleculares subyacentes para optimizar las estrategias de cría y mejorar la calidad de la carne.
TP63 y TP73, su contraparte, abarcan múltiples isoformas que regulan de forma independiente varios genes o interactúan con otras familias de factores de transcripción (23). Por ejemplo, se ha demostrado que TAp63α promueve la proliferación en la epidermis del ratón (24, 25). TP63 es un factor de transcripción conservado con múltiples sitios de unión distribuidos por todo el genoma (26). Al unirse directamente a los promotores de genes, TP63 ejerce control sobre la expresión génica aguas abajo (27). En particular, TP63 ha sido implicado en la miogénesis y la contracción muscular, lo que resulta en alteraciones significativas en la expresión de genes de diferenciación miogénica como MYH9, MYH10 y CDKN1A (28). Sin embargo, el papel preciso de TP63 en el desarrollo del músculo esquelético sigue siendo difícil de alcanzar (29).
Este estudio tuvo como objetivo investigar los mecanismos moleculares por los cuales TP63 regula el desarrollo del músculo esquelético. En primer lugar, caracterizamos los patrones de expresión de TP63 a través del transcriptoma del músculo esquelético en 27 puntos de tiempo de desarrollo, e investigamos la función de TP63 en el desarrollo del músculo esquelético a través del knockdown y la sobreexpresión. Se utilizó la base de datos JASPAR para predecir los motivos de los factores de transcripción unidos por los sitios de mutación de TP63, y se exploró el mecanismo molecular de los factores de transcripción de unión a SNP MEF2A que regulan el desarrollo del músculo esquelético. Este estudio proporciona una base teórica para mejorar el rendimiento de la producción de carne y mejorar la genética en cerdos.
2 Materiales y métodos
2.1 Cultivo celular
Las células del músculo esquelético porcino (BIOSPECIES-0017a) se compraron a Guangzhou Suyan Biotechnology Co., Ltd., Guangdong, China. Los mioblastos C2C12 y las células T 293 se adquirieron en la American Type Culture Collection (ATCC). El medio de cultivo fue el Medio de Águila Modificado de Dulbecco (DMEM, Corning, China). contenía un 10% de suero fetal bovino (FBS, Gibco, California, EE. UU.) y un 1% de penicilina-estreptomicina (PS, Thermo Scientific, Massachusetts, EE. UU.). A continuación, las células se colocaron en una incubadora celular a 37 °C que contenía un 5% de oxígeno y un 95% de dióxido de carbono.
2.2 Colección de músculo esquelético
Se recolectaron meticulosamente muestras de músculo Longissimus dorsi de cerdos Landrace y Tongcheng en un espectro completo de 27 puntos de tiempo de desarrollo. A estos animales se les concedió acceso irrestricto a alimentos y agua, y se les alojó de manera uniforme en condiciones controladas para minimizar las variaciones ambientales. En particular, en cada momento, se recolectaron meticulosamente muestras de tejido de tres cerdos distintos para garantizar una replicación biológica robusta. Después de la recolección, todos los especímenes se congelaron rápidamente en nitrógeno líquido y se conservaron hasta los análisis posteriores de secuenciación de ARN (GSE157045 y PRJNA754250) (18, 30).
2.3 Interferencia y sobreexpresión de ARN
Para la interferencia de ARN, se compraron siRNA de control negativo (siRNA-NC) y siRNA-TP63 de ratón a Gemma Pharmaceutical Technology (Shanghai, China). Las secuencias de los siRNAs dirigidos a TP63 se enumeran en la Tabla Suplementaria S1. El vector de expresión de TP63 (pcDNA3.1-TP63) fue sintetizado por Gene Create (Wuhan, China). El plásmido de control pcDNA3.1 se obtuvo de nuestro laboratorio. Las secuencias codificantes (CDS) del gen TP63 porcino se insertaron en el vector PLV3 (Invitrogen). Las células del músculo esquelético porcino y las células C2C12 se inocularon en placas de 6 o 12 pocillos 12 h antes del tratamiento y luego se transfectaron con siRNA o plásmido utilizando el reactivo de transfección Attractene (Qiagen) de acuerdo con sus instrucciones. El ARN interferente pequeño (siRNA) y el plásmido se transfectaron a una concentración final de 50 nM. Esta concentración precisa se seleccionó después de exhaustivos experimentos de optimización, alineándose con el rango efectivo ampliamente reportado documentado en la literatura existente para la eliminación de genes competente.
2.4 Extracción de ARN y PCR cuantitativa en tiempo real (qPCR)
Las células o tejidos se lisaron con Triol (Invitrogen, Shanghai, China), cloroformo y se desnaturalizaron con isopropanol para precipitar ARN, luego se lavaron con etanol al 75 y 100%, respectivamente, y finalmente se solubilizaron en agua DPC. La calidad del ARN se determinó mediante NanoDrop 2000 (Thermo Fisher Scientific, Massachusetts, EE. UU.). Los ARN identificados estaban disponibles para estudios posteriores. Se utilizó el kit de síntesis de ADNc de primera cadena HiScript III (+limpiador de ADNg) (R312-01, Vazyme, Nanjing, China) para la síntesis de transcripción inversa de ADNc de ARNm, respectivamente, de acuerdo con las instrucciones. Además, se utilizó Taq Pro Universal SYBR qPCR Master Mix (Vazyme, Nanjing, China) en la reacción total para la qPCR de ARN mensajero en un volumen de 20 μL, incluidos 10 μL 2× SYBR Master Mix, 0,4 μL de cebador directo de PCR, 0,4 μL de cebador inverso de reacción en cadena de polimerasa, 2 μL de ADNc y 7,2 μL de agua estéril libre de enzimas. Las condiciones de reacción fueron de 95 °C durante 30 s, seguidas de 95 °C durante 10 s y 65 °C durante 30 s durante 40 ciclos; este gen de referencia era Gapdh. Los niveles relativos de expresión del ácido ribonucleico mensajero (ARN) se analizaron mediante el método 2-∆∆CT. La información de la secuencia de los cebadores utilizados para la transcripción inversa y la cuantificación (Sangon Biotech, Shanghai, China) se muestra en las Tablas Suplementarias S2 y S3.
2.5 Ensayo de 5-etinil-2′-desoxiuridina (EdU)
El ensayo de EdU se realizó utilizando el kit de ensayo EdU (Beyotime, China) y la capacidad de proliferación celular se determinó utilizando el kit de ensayo de proliferación celular EdU-488 (Beyotime, China). Las células se extendieron en placas de 6 pocillos, que se interrumpieron y sobreexpresaron, y la concentración final de EdU se ajustó al 10% μm, se agregó a cada pocillo y se continuó la incubación durante 1-2 h en la incubadora celular. Las células se lavaron tres veces con PBS (Thermo Fisher) y luego se fijaron con fijador de formaldehído al 4% durante 15 min a temperatura ambiente. Se retiró el fijador y se lavaron las células tres veces con detergente. Se añadió 1 mL de Triton X-100 al 0,3% diluido en PBS y se incubó a temperatura ambiente durante 15 min para aumentar la permeabilidad de la membrana celular. Además, se añadieron 500 μL de la solución de reacción de clic preparada a cada pocillo, y las células se incubaron durante 30 min a temperatura ambiente y se protegieron de la luz para observar y cuantificar el número de células teñidas con EdU, y los núcleos se tiñeron con DAPI (1:1000 PBS). Se seleccionaron aleatoriamente tres campos para el análisis estadístico.
2.6 Ensayo de proliferación del kit de recuento celular-8
Los mioblastos adultos y los mioblastos esqueléticos C2C12 se inocularon en placas de 96 pocillos y se cosecharon a las 0 h, 24 h, 36 h, 48 h y 72 h después de la transfección, respectivamente. La proliferación de miocitos adultos se midió utilizando el Cell Counting Kit-8 (CCK-8) (Beyotime C0038, Beijing, China). Se añadió una mezcla 1:9 de reactivo CCK-8 y medio de cultivo completo a placas de 96 pocillos y se continuó a 37 °C durante 1 h. Las células se contaron con un lector de microplacas. La densidad óptica (DO) a 450 nm de cada muestra se midió con un lector de microplacas y se trazaron curvas de crecimiento.
2.7 Ensayo del ciclo celular
Las células del músculo esquelético se extendieron en placas de 6 pocillos, se interrumpieron y sobreexpresaron, y se recolectaron después de 48 h. Las células se digierieron con tripsina, se centrifugaron a 1000 g durante 3-5 min, se precipitaron y se prepararon suspensiones unicelulares. Las células se lavaron con PBS preenfriado y se centrifugaron de nuevo. Fije las células, vuelva a suspenderlas con 1 ml de etanol al 70% preenfriado y vuelva a suspenderlas a 4 °C durante 2 h o durante la noche. Se preparó una cantidad adecuada de solución de tinción de yoduro de propidio (PI) de acuerdo con el kit de ensayo del ciclo celular (#C1052, Beyotime Biotechnology, Shanghai, China). Las células se resuspendieron con 500 μL de solución de tinción de PI por tubo, se incubaron a 37 °C durante 30 min, se protegieron de la luz y se sometieron a citometría de flujo (CytoFLEX, BD Biosciences, NY, Estados Unidos) tras la excitación.
2.8 Ensayos de genes indicadores duales de luciferasa
Se empleó la amplificación por PCR para aislar un fragmento de ADN de 400 pb ubicado aguas arriba y aguas abajo del locus SNP rs327571319. Posteriormente, este fragmento se clonó en un vector PGK para la validación de la actividad potenciadora. A continuación, el plásmido construido se transfectó en 293 células T utilizando un reactivo de transfección (Qiagen). Al mismo tiempo, la co-transfección involucró a un plásmido que albergaba Renilla luciferasa (PRL-TK) para servir como control interno. Después de 24 h de transfección, la transducción de la señal de luciferasa se cuantificó utilizando el kit de ensayo reportero de luciferasa dual (Vazyme).
2.9 Análisis de motivos
Para encontrar los factores de transcripción unidos, se analizaron los motivos de los factores de transcripción (TF) unidos a rs327571319 utilizando TF de la base de datos pública JASPAR.
2.10 Análisis estadístico
Los niveles de expresión de ARNm se calcularon con el método 2-ΔΔCt y se mostraron como media ± desviación estándar. Para calcular el valor p se utilizó la prueba t de Student de dos colas no apareadas. Se adoptó la prueba t para detectar diferencias entre los grupos en cuanto a significación estadística. Todos los datos analíticos se obtuvieron de tres experimentos independientes y cada experimento se realizó por triplicado.
3 Resultados
3.1 TP63 sirve como candidato regulador del desarrollo del músculo esquelético
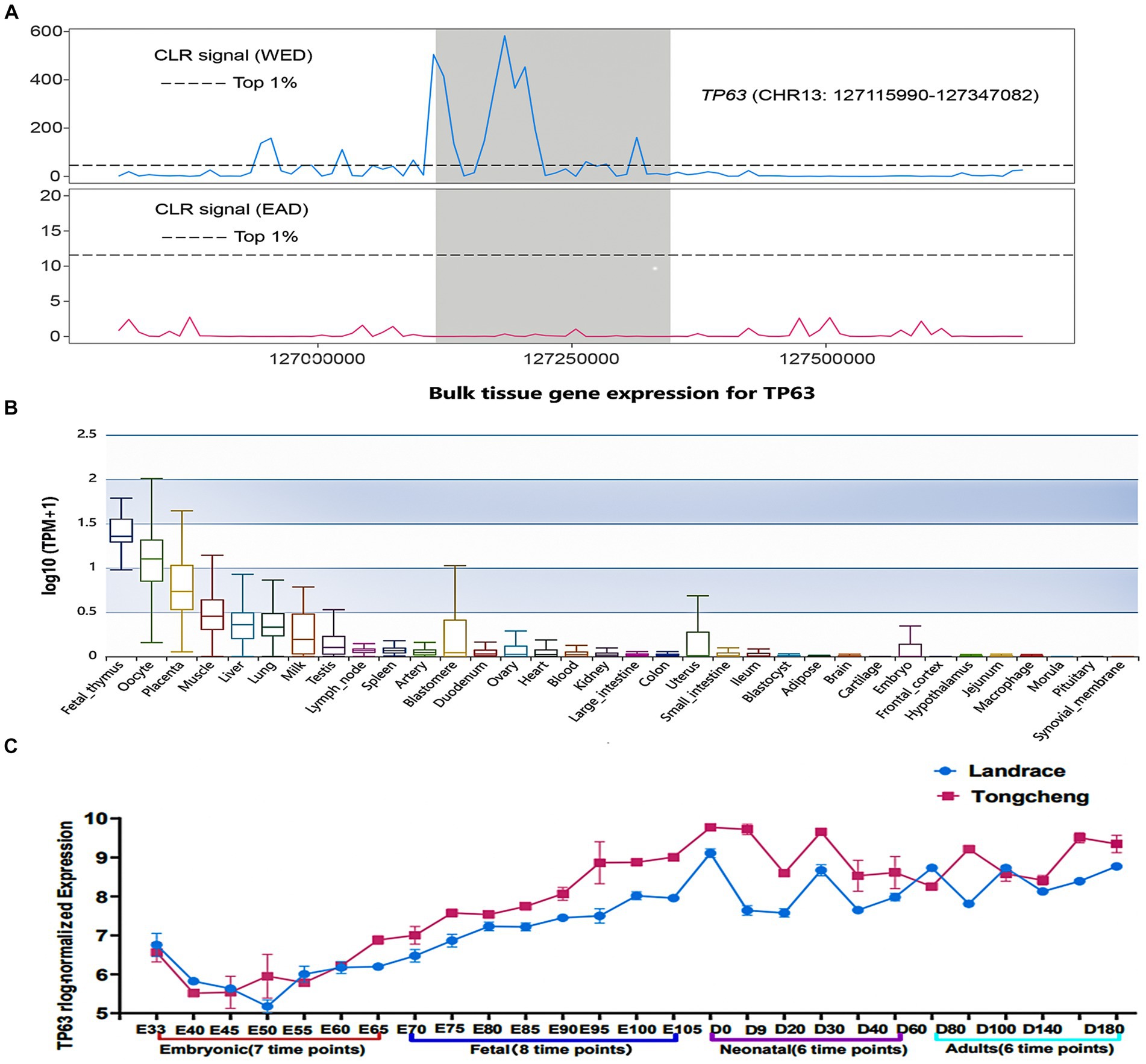
Los intrincados mecanismos moleculares que gobiernan el desarrollo del músculo esquelético han atraído una atención significativa debido a su impacto en el desarrollo muscular y las estrategias terapéuticas para los trastornos musculares. Los análisis de barrido selectivo demostraron que TP63 es un gen altamente seleccionado en las razas de cerdos magros occidentales (WED) en comparación con los cerdos orientales (EAD) (Figura 1A), lo que indica su posible participación en el desarrollo del músculo esquelético. Para validar su expresión específica de tejido, aprovechamos la base de datos Pig GTEx Atlas, revelando una expresión robusta de TP63 en el tejido muscular en relación con otros tejidos (Figura 1B). Además, basándonos en nuestros datos previos de RNA-seq del músculo esquelético en diferentes etapas de desarrollo, examinamos los perfiles de expresión de TP63 en cerdos Tongcheng y Landrace durante varias etapas de desarrollo del músculo esquelético. Observamos una disparidad significativa en los niveles de expresión de TP63 entre estas dos razas de cerdos, con cerdos Tongcheng exhibiendo una expresión notablemente mayor de TP63 en comparación con los cerdos Landrace en las etapas fetal, neonatal y adulta del desarrollo del músculo esquelético (Figura 1C). Los hallazgos anteriores destacan a TP63 como un gen candidato prometedor implicado en la regulación del desarrollo del músculo esquelético.
 Figura 1. TP63 es un candidato a regulador del desarrollo del músculo esquelético. (A) Análisis de barrido selectivo que indica una fuerte selección artificial que actúa sobre TP63 en razas de cerdos occidentales (WED) en comparación con cerdos orientales (EAD). (B) Expresión tisular específica de TP63 en cerdos, destacando su expresión enriquecida en tejidos musculares en relación con otros tejidos. (C) Patrones de expresión diferencial de TP63 en diferentes etapas de desarrollo del músculo esquelético.
Figura 1. TP63 es un candidato a regulador del desarrollo del músculo esquelético. (A) Análisis de barrido selectivo que indica una fuerte selección artificial que actúa sobre TP63 en razas de cerdos occidentales (WED) en comparación con cerdos orientales (EAD). (B) Expresión tisular específica de TP63 en cerdos, destacando su expresión enriquecida en tejidos musculares en relación con otros tejidos. (C) Patrones de expresión diferencial de TP63 en diferentes etapas de desarrollo del músculo esquelético.
3.2 TP63 promueve la proliferación de mioblastos de ratón
Para investigar el efecto de TP63 en la proliferación de mioblastos, primero utilizamos mioblastos adultos C2C12 de ratón y realizamos experimentos funcionales que involucraron la transfección con vectores de sobreexpresión de TP63 y siRNA-TP63 diseñados para atacar TP63. Nuestros resultados demostraron que la eliminación de TP63 atenuó efectivamente el nivel de ARNm de TP63 en mioblastos C2C12 de ratón, esta tasa de eliminación es de aproximadamente el 60% en comparación con el grupo de control (Figura 2A), así como varios genes marcadores de proliferación, incluidos CYCLINE, CYCLIND y KI67 (Figura 2B). Los resultados del ensayo EdU revelaron además una disminución de la actividad de proliferación celular tras la eliminación de TP63 (Figura 2C). El ensayo CCK8 reveló una disminución en la viabilidad de la proliferación celular tras la eliminación de TP63, pero no un cambio significativo. (Figura 2D). Además, los experimentos de RT-qPCR mostraron que la sobreexpresión de TP63, esta eficiencia de sobreexpresión aumentó aproximadamente 100 veces en comparación con el control (Figura 2E) y aumentó significativamente la expresión de genes marcadores de proliferación (Figura 2F). Los ensayos de EdU mostraron un aumento en el número de células positivas para EdU después de la sobreexpresión de TP63 (Figura 2G), lo que sugiere una mayor proliferación. Además, los ensayos de viabilidad celular demostraron un aumento significativo de la viabilidad celular tras la sobreexpresión de TP63 (Figura 2H). Los hallazgos indican que TP63 mejora la proliferación de mioblastos murinos y contribuye potencialmente al proceso de miogénesis.
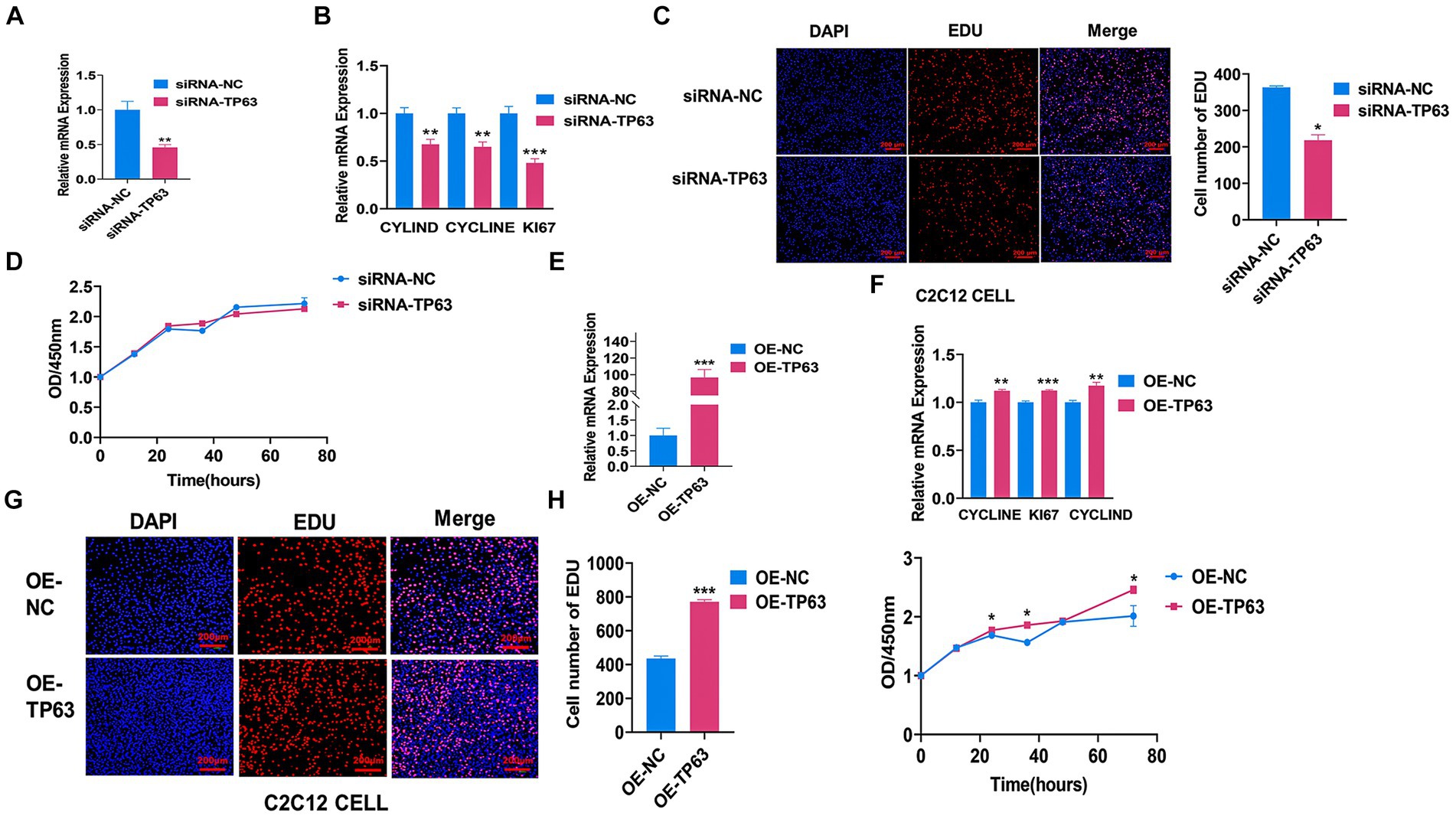 Figura 2. TP63 promueve la proliferación de células C2C12. (A) Se evaluó la eficiencia de la eliminación de TP63 en celdas C2C12. (B) Se realizó un análisis de RT-qPCR para determinar los niveles de expresión de ARNm de genes marcadores de proliferación inhibidos por siRNA-TP63. (C) La tinción de EdU se utilizó para detectar la capacidad proliferativa de las células C2C12 después de la inhibición de TP63. Se cuantificó el número de células positivas teñidas con EdU (rojo), células replicantes de ADN y núcleos teñidos con Hoechst (azul). Barra de escala = 200 μm. (D) La proliferación celular se evaluó mediante el ensayo CCK-8 después de la transfección de siRNA-TP63 en células C2C12. (E) Se evaluó la eficiencia de la sobreexpresión de TP63 en células C2C12. (F) Se realizó el ensayo RT-qPCR para determinar el nivel de expresión de ARNm del gen marcador de proliferación aumentado por la sobreexpresión de TP63. (G) La tinción de EdU se utilizó para detectar la capacidad de proliferación de las células C2C12 después de la sobreexpresión de TP63. Barra de escala = 200 μm. (H) La proliferación celular se evaluó mediante el ensayo CCK-8. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (t-test). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
Figura 2. TP63 promueve la proliferación de células C2C12. (A) Se evaluó la eficiencia de la eliminación de TP63 en celdas C2C12. (B) Se realizó un análisis de RT-qPCR para determinar los niveles de expresión de ARNm de genes marcadores de proliferación inhibidos por siRNA-TP63. (C) La tinción de EdU se utilizó para detectar la capacidad proliferativa de las células C2C12 después de la inhibición de TP63. Se cuantificó el número de células positivas teñidas con EdU (rojo), células replicantes de ADN y núcleos teñidos con Hoechst (azul). Barra de escala = 200 μm. (D) La proliferación celular se evaluó mediante el ensayo CCK-8 después de la transfección de siRNA-TP63 en células C2C12. (E) Se evaluó la eficiencia de la sobreexpresión de TP63 en células C2C12. (F) Se realizó el ensayo RT-qPCR para determinar el nivel de expresión de ARNm del gen marcador de proliferación aumentado por la sobreexpresión de TP63. (G) La tinción de EdU se utilizó para detectar la capacidad de proliferación de las células C2C12 después de la sobreexpresión de TP63. Barra de escala = 200 μm. (H) La proliferación celular se evaluó mediante el ensayo CCK-8. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (t-test). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
3.3 TP63 promueve la proliferación de células musculares esqueléticas porcinas
Para validar aún más la función de TP63 en la proliferación celular, realizamos múltiples experimentos en las células del músculo esquelético porcino. Nuestros hallazgos demostraron que la reducción de TP63 mediada por siRNA, la eficiencia de eliminación alcanzó aproximadamente el 80% en comparación con el grupo de control, lo que subraya una reducción significativa en la expresión génica diana como consecuencia de la intervención experimental (p < 0,01; Figura 3A) y redujo eficientemente los niveles de expresión de ARNm de genes marcadores de proliferación críticos, a saber, KI67, PCNA y CYCLINA (Figura 3B). Consistentemente, la depleción de TP63 resultó en una disminución en el número de mioblastos positivos para EdU, lo que refleja una disminución de la capacidad proliferativa (p < 0,05; Figura 3C). Además, el ensayo CCK8 reveló una disminución significativa en la viabilidad de la proliferación celular tras la eliminación de TP63 (Figura 3D). Sorprendentemente, el análisis del ciclo celular reveló una distribución alterada de las células que progresan a través del ciclo celular, con una mayor proporción de células detenidas en la fase G0/G1 y un número reducido de células que pasan a la fase S (Figuras 3E, F). Por el contrario, la sobreexpresión de TP63 en células basales esqueléticas porcinas. La eficiencia de sobreexpresión aumentó aproximadamente 130 veces en relación con el control (p < 0,001; Figura 3G), lo que conduce a un aumento notable en los niveles de expresión de ARNm de TP63 y genes marcadores de proliferación clave (Figura 3H). Curiosamente, tanto los ensayos de EdU como los de CCK-8 confirmaron que la sobreexpresión de TP63 mejoró significativamente la actividad proliferativa celular (Figuras 3I, J). Además, el análisis del ciclo celular demostró un aumento en el número de células que progresan a la fase S tras la regulación positiva de TP63 (Figuras 3K, L). Estos hallazgos arrojan luz sobre la función esencial de TP63 en la orquestación de la proliferación de células musculares esqueléticas porcinas, lo que podría contribuir a nuestra comprensión del desarrollo muscular y los mecanismos de regeneración.
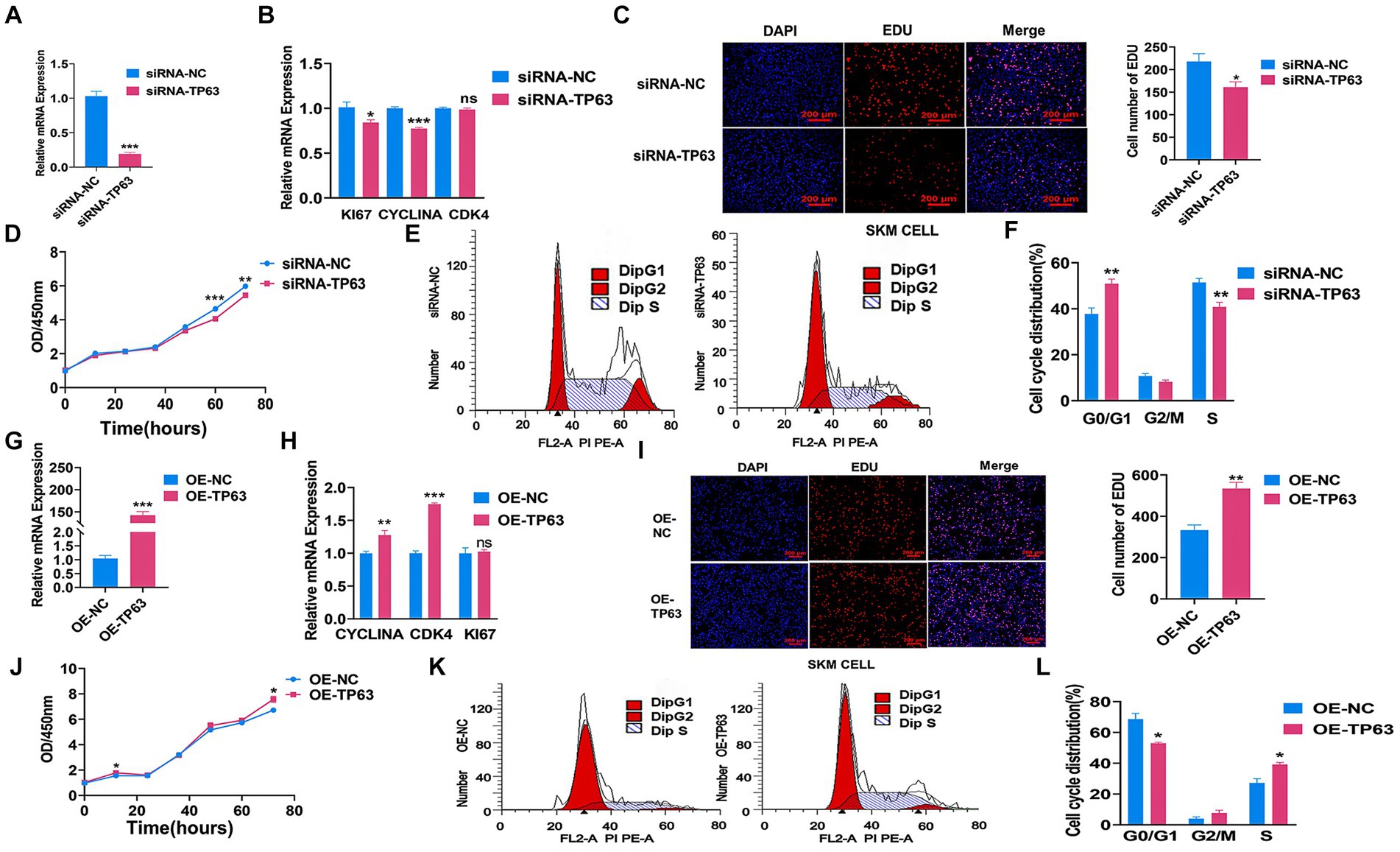 Figura 3. TP63 regula la proliferación de células musculares esqueléticas porcinas. (A) Se evaluó la eficiencia de la eliminación de TP63 en células basales esqueléticas porcinas. (B) Se realizó un análisis de RT-qPCR para determinar el efecto de derribo de TP63 en los niveles de expresión de ARNm de los genes marcadores de proliferación celular. (C) Se realizó la tinción de EdU para detectar la capacidad proliferativa de las células del músculo esquelético porcino después de la inhibición de TP63. Barra de escala = 200 μm. (D) La proliferación celular se evaluó mediante el ensayo CCK-8 después de la transfección de siRNA-TP63 en células basales esqueléticas porcinas. (E,F) El análisis del ciclo celular se realizó mediante citometría de flujo después de la transfección de siRNA-TP63 en células proliferantes del músculo esquelético porcino y su control negativo. (G) Se evaluó la eficiencia de la sobreexpresión de TP63 en células basales esqueléticas porcinas. (H) La sobreexpresión de TP63 aumentó el nivel de expresión de ARNm de los genes marcadores de proliferación celular. (I) Se realizó tinción con EdU para detectar la capacidad proliferativa de las células del músculo esquelético porcino después de la sobreexpresión de TP63. Barra de escala = 200 μm. (J) La proliferación celular se evaluó mediante el ensayo CCK-8 después de la transfección de OE-TP63 en células basales esqueléticas porcinas. (K,L) El análisis del ciclo celular se realizó mediante citometría de flujo después de la transfección de OE-TP63 en células musculares esqueléticas porcinas proliferantes y su control negativo. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (t-test). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
Figura 3. TP63 regula la proliferación de células musculares esqueléticas porcinas. (A) Se evaluó la eficiencia de la eliminación de TP63 en células basales esqueléticas porcinas. (B) Se realizó un análisis de RT-qPCR para determinar el efecto de derribo de TP63 en los niveles de expresión de ARNm de los genes marcadores de proliferación celular. (C) Se realizó la tinción de EdU para detectar la capacidad proliferativa de las células del músculo esquelético porcino después de la inhibición de TP63. Barra de escala = 200 μm. (D) La proliferación celular se evaluó mediante el ensayo CCK-8 después de la transfección de siRNA-TP63 en células basales esqueléticas porcinas. (E,F) El análisis del ciclo celular se realizó mediante citometría de flujo después de la transfección de siRNA-TP63 en células proliferantes del músculo esquelético porcino y su control negativo. (G) Se evaluó la eficiencia de la sobreexpresión de TP63 en células basales esqueléticas porcinas. (H) La sobreexpresión de TP63 aumentó el nivel de expresión de ARNm de los genes marcadores de proliferación celular. (I) Se realizó tinción con EdU para detectar la capacidad proliferativa de las células del músculo esquelético porcino después de la sobreexpresión de TP63. Barra de escala = 200 μm. (J) La proliferación celular se evaluó mediante el ensayo CCK-8 después de la transfección de OE-TP63 en células basales esqueléticas porcinas. (K,L) El análisis del ciclo celular se realizó mediante citometría de flujo después de la transfección de OE-TP63 en células musculares esqueléticas porcinas proliferantes y su control negativo. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (t-test). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
3.4 rs327571319 priorizado como un posible SNP funcional
Para explorar las variantes genéticas funcionales que regulan TP63 en la miogénesis, realizamos análisis sistemáticos basados en conjuntos de datos públicos. Utilizamos archivos de genotipos que comprenden 570 muestras de la población oriental (EAD) y 296 muestras de cerdos occidentales (WED) para identificar SNP en la región promotora o intrón de TP63 que exhibían una diferencia de frecuencia génica superior a 0,9 entre las dos comunidades de nuestro estudio anterior (18). Un SNP candidato, chr13: 127266445 (rs327571319 T/C) localizado en el quinto intrón de TP63, se destacó por mostrar una frecuencia del alelo T de 0,972 en cerdos occidentales y solo de 0,051 en cerdos orientales (Figura 4A). Los datos del estado de la cromatina mostraron que la mutación se localizaba en la región potenciadora de TP63 (Figura 4B). Investigaciones posteriores revelaron que este SNP estaba asociado con el área del músculo del lomo a 100 kg (LMA) (Figura 4C) (31). Para determinar su impacto funcional, realizamos un ensayo de luciferasa dual utilizando vectores que contenían la secuencia de tipo salvaje (PGL4.23-rs327571319-T) o la secuencia mutada (PGL4.23-rs327571319-C). Los resultados demostraron que la mutación T-to-C alteró significativamente la actividad potenciadora (p < 0,001; Figura 4D). Además, en 293 linfocitos T cotransfectados con sobreexpresión de TP63 (TP63-OE) junto con rs327571319T/C y plásmido PRL-TK, la actividad de luciferasa fue significativamente mayor en presencia de OE-TP63-rs327571319-C + TK en comparación con OE-TP63-rs327571319-T + TK, lo que confirma la relación regulatoria entre TP63 y rs327571319 (p < 0,001; Figura 4E). Estos hallazgos sugieren que rs327571319 podría ser potencialmente un SNP candidato funcional involucrado en la regulación de TP63 y la miogénesis.
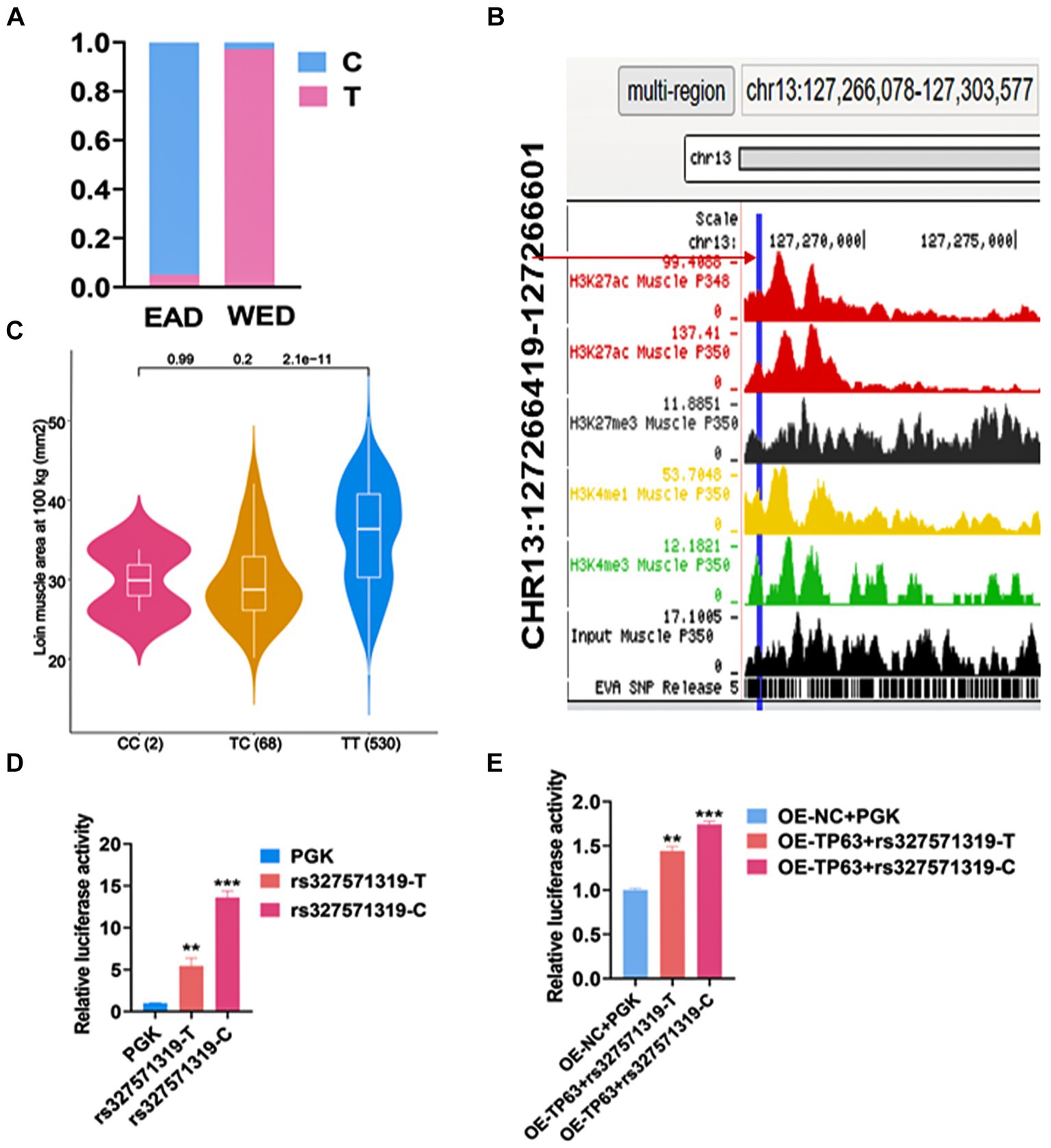 Figura 4. rs327571319 priorizado como un posible SNP funcional. (A) La frecuencia génica del locus rs327571319 indica una diferencia significativa en las frecuencias alélicas entre los cerdos occidentales y orientales. (B) Se presentan datos sobre el estado de la cromatina para el locus rs327571319, lo que sugiere que puede tener importancia regulatoria. (C) Este SNP se asoció con el área del músculo del lomo a los 100 kg (LMA). (D) Los ensayos de luciferasa reportera realizados en células HEK293T comparan la actividad potenciadora entre los dos alelos de rs327571319, lo que indica una diferencia significativa en la actividad. (E) En 293 linfocitos T cotransfectados con un plásmido reportero de luciferasa que contenía el locus SNP y un plásmido de sobreexpresión de TP63, se ensayó la actividad relativa de la luciferasa. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (prueba t). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
Figura 4. rs327571319 priorizado como un posible SNP funcional. (A) La frecuencia génica del locus rs327571319 indica una diferencia significativa en las frecuencias alélicas entre los cerdos occidentales y orientales. (B) Se presentan datos sobre el estado de la cromatina para el locus rs327571319, lo que sugiere que puede tener importancia regulatoria. (C) Este SNP se asoció con el área del músculo del lomo a los 100 kg (LMA). (D) Los ensayos de luciferasa reportera realizados en células HEK293T comparan la actividad potenciadora entre los dos alelos de rs327571319, lo que indica una diferencia significativa en la actividad. (E) En 293 linfocitos T cotransfectados con un plásmido reportero de luciferasa que contenía el locus SNP y un plásmido de sobreexpresión de TP63, se ensayó la actividad relativa de la luciferasa. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (prueba t). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
3.5 MEF2A se une preferentemente a rs327571319(C) para regular la expresión de TP63
El desarrollo del músculo esquelético es un proceso muy complejo gobernado por las acciones coordinadas de numerosos factores de transcripción (TF) y variantes genéticas. Aquí, nos esforzamos por investigar el impacto de un polimorfismo funcional putativo de un solo nucleótido (SNP), rs327571319, ubicado dentro de la región intrónica de TP63, en el desarrollo del músculo esquelético. A través del análisis de motivos utilizando JASPAR, MEF2A llamó nuestra atención debido a su promoción reportada de la diferenciación de mioblastos (Figura 5A) (32). Para dilucidar el papel regulador específico de MEF2A en relación con el alelo rs327571319 (T/C), realizamos experimentos de co-transfección mediante la sobreexpresión de MEF2A junto con plásmidos que contienen el alelo rs327571319-C o rs327571319-T en 293 células T. Los experimentos de co-transfección revelaron un aumento significativo en la actividad potenciadora asociada con el alelo rs327571319-C, lo que sugiere la mayor capacidad de unión de MEF2A en el alelo C (p < 0.01; Figura 5B). Además, la sobreexpresión de MEF2A en células musculares esqueléticas porcinas (p < 0,001; Figura 5C) resultó en una regulación positiva sustancial de la expresión de TP63 (p < 0,001; Figura 5D). De acuerdo con estos hallazgos, el ensayo EdU demostró un aumento de la actividad proliferativa celular (p < 0,01; Figuras 5E, F). Nuestro estudio proporciona evidencia de que el alelo rs327571319-C dentro de la región intrónica TP63 interactúa con MEF2A, lo que conduce a un aumento de la expresión de TP63 y la actividad potenciadora (Figura 5G). Estos cambios reguladores promueven la proliferación celular y contribuyen a la regulación del desarrollo del músculo esquelético.
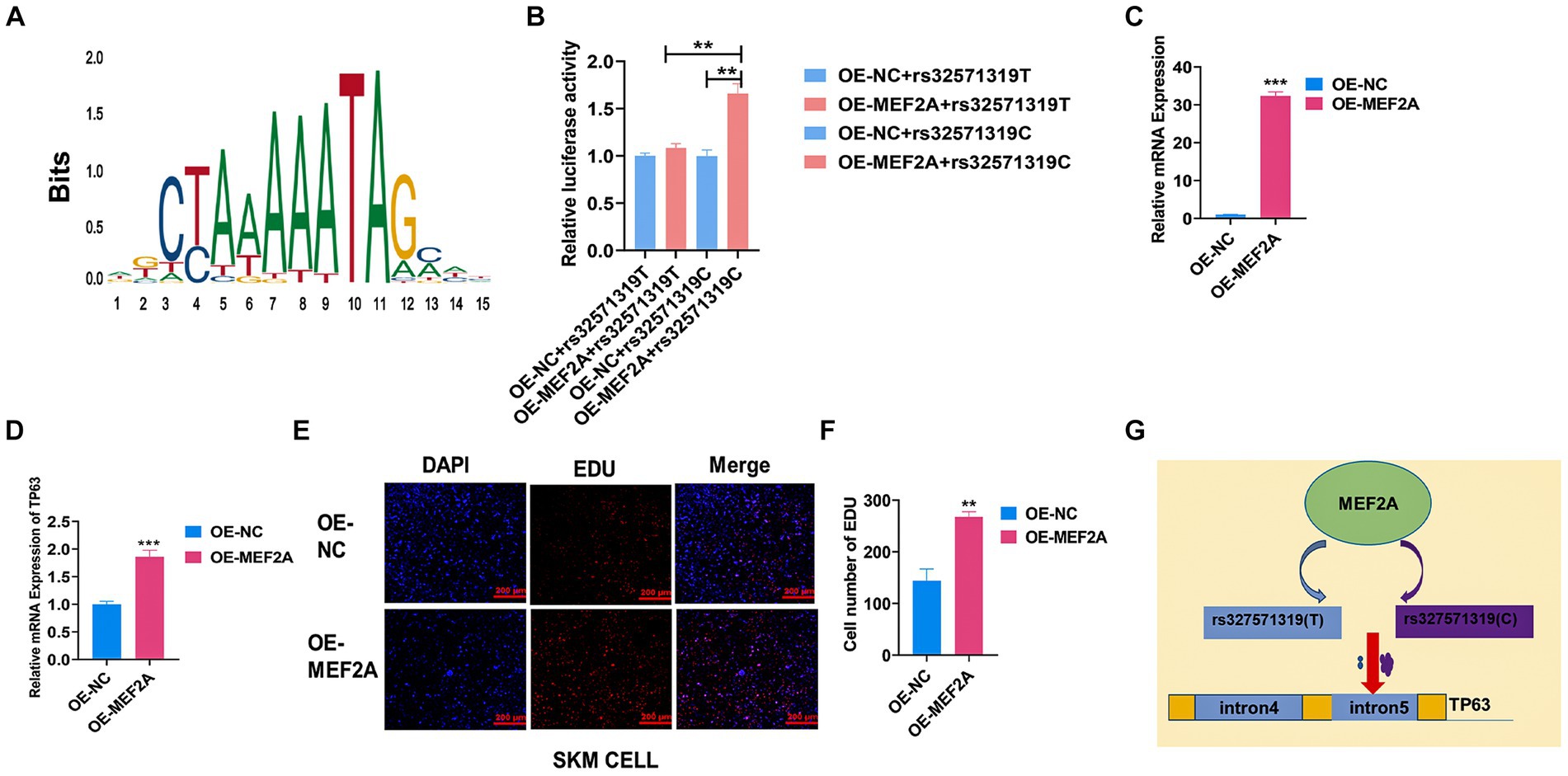 Figura 5. MEF2A se une preferentemente a rs327571319(C) para regular la expresión de TP63. (A) Predicción de secuencias de motivos para la unión de sitios mutantes utilizando la base de datos JASPAR. (B) Se realizaron experimentos de co-transfección en 293 células T, con plásmidos de luciferasa que contenían el sitio SNP y plásmidos de sobreexpresión de MEF2A, para evaluar la actividad relativa de la luciferasa. (C) Ensayo de eficiencia de la sobreexpresión de MEF2A en células basales esqueléticas porcinas. (D) La sobreexpresión de MEF2A aumentó significativamente el nivel de expresión de ARNm de TP63. (E,F) Se realizó un ensayo de tinción de EdU para evaluar la actividad proliferativa de las células del músculo esquelético porcino que sobreexpresan MEF2A. Barra de escala = 200 μm. (G) Representación esquemática que ilustra cómo el locus rs327571319 afecta a la expresión de TP63. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (prueba t). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
Figura 5. MEF2A se une preferentemente a rs327571319(C) para regular la expresión de TP63. (A) Predicción de secuencias de motivos para la unión de sitios mutantes utilizando la base de datos JASPAR. (B) Se realizaron experimentos de co-transfección en 293 células T, con plásmidos de luciferasa que contenían el sitio SNP y plásmidos de sobreexpresión de MEF2A, para evaluar la actividad relativa de la luciferasa. (C) Ensayo de eficiencia de la sobreexpresión de MEF2A en células basales esqueléticas porcinas. (D) La sobreexpresión de MEF2A aumentó significativamente el nivel de expresión de ARNm de TP63. (E,F) Se realizó un ensayo de tinción de EdU para evaluar la actividad proliferativa de las células del músculo esquelético porcino que sobreexpresan MEF2A. Barra de escala = 200 μm. (G) Representación esquemática que ilustra cómo el locus rs327571319 afecta a la expresión de TP63. Todos los experimentos se llevaron a cabo con al menos tres réplicas biológicas y los datos fueron normalizados por GAPDH. Los resultados se presentan como media ± SEM (prueba t). La significación estadística se determinó de la siguiente manera: *p < 0,05, **p < 0,01, ***p < 0,001, p ≥ 0,05: ns (no estadísticamente significativo).
4 Discusión
El objetivo de este estudio fue explorar el mecanismo regulador de TP63 en células musculares esqueléticas porcinas. El desarrollo del músculo esquelético es un proceso crucial en el crecimiento y desarrollo animal, en el que la proliferación, diferenciación y fusión de células satélite juegan un papel fundamental en este proceso (33). Los análisis de barrido selectivo han identificado a TP63 como un gen altamente seleccionado en las razas de cerdos magros occidentales en comparación con los cerdos orientales, lo que llama nuestra atención sobre su papel potencial en el desarrollo del músculo esquelético. Su papel en el desarrollo del músculo esquelético porcino sigue siendo en gran medida inexplorado. Nuestro estudio proporciona nuevos conocimientos sobre los mecanismos reguladores que subyacen al desarrollo del músculo esquelético porcino.
Estudios anteriores han demostrado que TP63 desempeña diversas funciones en diferentes tipos de células, incluida la promoción de la proliferación celular y la regulación de las últimas etapas de la diferenciación miogénica (34). Por ejemplo, se ha demostrado que TP63 promueve la proliferación de células de carcinoma de células escamosas de esófago (ESCC) al unirse al potenciador en el motivo LINC01503 (35, 36). Además, se ha informado que TP63 funciona en una etapa tardía de diferenciación miogénica (37), afectando la expresión de genes asociados con la miogénesis y la contractilidad del músculo esquelético tras la eliminación de TAp63γ (38). Sin embargo, Li et al. informaron que la inhibición de la expresión de TP63 promueve las células ciliadas (39). Nuestros resultados mostraron que TP63 se correlacionó positivamente con la proliferación de células del músculo esquelético. Estos hallazgos mejoran nuestra comprensión de los factores que influyen en el desarrollo muscular y arrojan luz sobre la participación de TP63 en la proliferación de células musculares esqueléticas.
En el contexto de la eucromatina activa, los factores de transcripción (TF) exhiben la capacidad de unirse a regiones abiertas de cromatina que albergan elementos reguladores del ADN, modulando así la expresión génica (40). Investigaciones recientes han revelado el papel regulador fundamental de TP63 en la orquestación de la accesibilidad de la cromatina y la reprogramación del potenciador en las células formadoras de queratina (41, 42). La importancia funcional de los potenciadores de unión a p63 se subraya por su correlación con la dinámica de la expresión génica, lo que indica la participación crucial de TP63 en la regulación génica a través de la activación del potenciador (42). Nuestra investigación ha delineado la implicación funcional de la unión de MEF2A a un SNP funcional, rs327571319-C, que sirve como modulador crítico de la actividad potenciadora y la expresión de TP63. Este eje regulador ejerce influencia sobre la proliferación celular, arrojando luz sobre los intrincados mecanismos reguladores que gobiernan la expresión de TP63 y sus efectos posteriores en los procesos celulares relevantes para los rasgos de producción de carne. En particular, el análisis previo de datos de RNA-seq ha revelado patrones de expresión diferenciales de TP63 en cerdos Landrace de tipo magro en comparación con cerdos Tongcheng de tipo obeso, alineándose con la distribución de frecuencia observada del alelo rs327571319-T en las poblaciones de cerdos occidentales. Esta observación subraya la influencia potencial de TP63 en los rasgos de producción de carne y lo posiciona como un candidato prometedor para su utilización como marcador de mejoramiento, ofreciendo vías para el mejoramiento genético en el contexto de la producción de carne.
El factor potenciador de miocitos 2 (MEF2) es un miembro de la superfamilia MADS de factores de transcripción que desempeñan un papel crucial en la regulación de la expresión génica específica del músculo (43). Estudios previos han demostrado la participación de MEF2A en la promoción de la proliferación de cardiomiocitos y la proliferación de miocitos adultos (44, 45). Nuestros hallazgos indican que MEF2A también promueve la proliferación de células musculares esqueléticas, ampliando aún más nuestra comprensión de sus funciones reguladoras en el desarrollo muscular. Sin embargo, el estudio actual está limitado por la ausencia de experimentos de validación funcional in vivo, así como por la falta de investigación sobre la regeneración muscular y los efectos de la eliminación de TP63 y MEF2A en ratones. Se llevarán a cabo estudios futuros para abordar estas brechas y proporcionar una comprensión más completa de los mecanismos subyacentes a la miogénesis.
5 Conclusión
En resumen, nuestro estudio proporciona información valiosa sobre los factores reguladores implicados en el desarrollo del músculo esquelético y destaca la implicación de TP63 y MEF2A en la proliferación y diferenciación de las células del músculo esquelético. Estos hallazgos proporcionan una base para una mayor exploración del papel de TP63 en la configuración de rasgos fenotípicos relevantes para la producción de carne y ofrecen vías potenciales para la mejora genética en la industria porcina.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo/Material complementario, las consultas adicionales pueden dirigirse a los autores correspondientes.
Declaración ética
No se requirió aprobación ética para los estudios en animales de acuerdo con la legislación local y los requisitos institucionales, ya que solo se utilizaron líneas celulares establecidas disponibles comercialmente.
Contribuciones de los autores
YC: Curación de datos, Validación, Redacción – borrador original, Redacción – revisión y edición. ZW: Análisis formal, Validación, Redacción – borrador original. XQ: Curación de datos, Análisis formal, Redacción – borrador original. BS: Curación de datos, Software, Validación, Escritura – borrador original. YT: Curación de datos, Software, Validación, Escritura – borrador original. BL: Escritura – revisión y edición. GC: Redacción – revisión y edición. GY: Conceptualización, Obtención de fondos, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. El Programa Nacional de Investigación y Desarrollo Clave de China (2021YFF1000600), la Fundación Nacional de Ciencias Naturales de China (32002150), el Programa de Innovación en Ciencia y Tecnología Agrícola de la Academia China de Ciencias Agrícolas (CAAS-NBSCA-202301), la Fundación de Investigación Básica y Básica Aplicada de la Provincia de Guangdong (2020B1515120053), la Comisión de Innovación Científica y Tecnológica de Shenzhen (JCYJ20190813114401691) y los Fondos de Orientación del Gobierno Central para la Ciencia Local y Desarrollo Tecnológico de China (He-Ke ZY220603).
Reconocimientos
Agradecemos al Dr. Yuchun Pan y al Dr. Zhe Zhang de la Facultad de Ciencias Animales de la Universidad de Zhejiang por proporcionar los datos de genotipo y fenotipo para nuestro trabajo.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1396766/full#supplementary-material
Referencias
1. Frontera, WR, y Ochala, J. Músculo esquelético: una breve revisión de la estructura y la función. Calcif Tissue Int. (2015) 96:183–95. doi: 10.1007/s00223-014-9915-y
2. Daniel, JE, y David, C. Músculo esquelético: anatomía funcional y fisiopatología. Semin Musculoskelet Radiol. (2010) 14:97–105. doi: 10.1055/s-0030-1253154
3. Hongmei, L, Wei, L, Qian, T, Jianjun, J, Zaiyan, X y Bo, Z. ARN funcional no codificante durante la miogénesis embrionaria y el desarrollo muscular postnatal y la enfermedad. Front Cell Dev Biol. (2021) 9:628339. doi: 10.3389/fcell.2021.628339
4. Le Grand, F, y Rudnicki, MA. Células satélite del músculo esquelético y miogénesis adulta. Curr Opin Cell Biol. (2007) 19:628–33. doi: 10.1016/j.ceb.2007.09.012
5. Jinryong, P, Lee, J, Song, KD, Kim, SJ, Kim, DC y Lee, SC. Los factores de crecimiento mejoran la proliferación de las células musculares del cerdo negro de Jeju mediante la regulación de la diferenciación miogénica 1 y los genes relacionados con el crecimiento. Anim Biosci. (2021) 34:1392–402. doi: 10.5713/ab.20.0585
6. Grefte, S, Kuijpers-Jagtman, AM, Torensma, R, y Von den Hoff, JW. Desarrollo y regeneración del músculo esquelético. Desarrollo de Células Madre. (2007) 16:857–68. doi: 10.1089/scd.2007.0058
7. Raquel, EW, Anne-Sophie, B, Viola, FG, y Peter, SZ. Dinámica del crecimiento de las fibras musculares durante el desarrollo postnatal del ratón. BMC Dev Biol. (2010) 10:21. doi: 10.1186/1471-213X-10-21
8. Bismuto, K, y Relaix, F. Regulación genética del desarrollo del músculo esquelético. Exp Cell Res. (2010) 316:3081–6. doi: 10.1016/j.yexcr.2010.08.018
9. Bertol, TM, de Campos, RM, Ludke, JV, Terra, NN, de Figueiredo, EA y Coldebella, A. Efectos del genotipo y la suplementación con aceite dietético sobre el rendimiento, las características de la canal, la calidad de la carne de cerdo y la composición de ácidos grasos de la grasa dorsal y la grasa intramuscular. Ciencia de la carne. (2013) 93:507–16. doi: 10.1016/j.meatsci.2012.11.012
10. Jong-Hyun, J, Kwan Seob, S, Chong-Sam, N y Ho-Sung, C. Estudios sobre el porcentaje de grasa intramuscular en cerdos vivos utilizando ultrasonido en tiempo real para determinar la calidad de la carne de cerdo. Australas Asiáticas J Anim Sci. (2015) 28:318–22. doi: 10.5713/ajas.14.0927
11. Xinyun, L, Liangliang, F, Hui-Ling, C y Shuhong, Z. Avances en el microARN en la regulación del desarrollo del músculo esquelético de los mamíferos. Yi chuan. (2017) 39:1046–53. doi: 10.16288/j.yczz.17-112
12. Hong, R, Li, Y, Tang, Z, Yang, S, Mu, Y y Cui, W. Estructura genómica, localización cromosómica y perfil de expresión de un ARN porcino largo no codificante aislado de bibliotecas largas de SAGE. Anim Genet. (2009) 40:499–508. doi: 10.1111/j.1365-2052.2009.01868.x
13. He, Z, Qing, C, Cheng-Lin, T y Zhongkui, Z. Efecto del ejercicio y masaje temprano en la cinta de correr sobre los factores activadores de las células satélite musculares después de una lesión aguda del músculo esquelético en ratas. Chin J Rehabil Teoría Práctica. (2020) 29:49–54. doi: 10.3969/j.issn.1006-9771.2020.01.008
14. Cristina, O, Oliver, A, Noguera, JL, Clop, A, Barragán, C, y Varona, L. Prueba de genes candidatos posicionales para la composición corporal en el cromosoma 6 de cerdo. Genet Sel Evol. (2002) 34:465–79. doi: 10.1186/1297-9686-34-4-465
15. Yuwen, L, Fu, Y, Yang, Y, Yi, G, Lian, J, Xie, B, et al. La integración de datos multiómicos revela variantes cis-reguladoras que están asociadas con la diferenciación fenotípica de los cerdos orientales de los occidentales. Genet Sel Evol. (2022) 54:62. doi: 10.1186/s12711-022-00754-2
16. Cassady, JP, Johnson, RK, Pomp, D, Rohrer, GA, van Vleck, LD, Spiegel, EK, et al. Identificación de loci de rasgos cuantitativos que afectan a la reproducción en cerdos. J Anim Sci. (2001) 79:623–33. doi: 10.2527/2001.793623x
17. Xianwei, L, Zheng, H, Wu, W, Liu, H, Wang, J, Jia, Y, et al. Mapeo de QTL y análisis de genes candidatos para la tolerancia a los álcalis en arroz japónica en la etapa de yema basado en el mapeo de ligamiento y el estudio de asociación de todo el genoma. Arroz. (2020) 13:48. doi: 10.1186/s12284-020-00412-5
18. Guoqiang, Y, Lei, L, Yilong, Y, Yuwen, L, Jiang, L, Yalan, Y, et al. El análisis multiómico revela firmas de selección y loci asociadas con rasgos complejos en cerdos. Nueva York: Laboratorio Cold Spring Harbor (2023).
19. Sharon, RG, Shlyakhter, I, Karlsson, EK, Byrne, EH, Morales, S, y Frieden, G. Un compuesto de múltiples señales distingue variantes causales en regiones de selección positiva. Ciencia. (2010) 327:883–6. doi: 10.1126/science.1183863
20. Carl-Johan, R, Hendrik-Jan, M, y Álvaro Martínez, B. Fuertes firmas de selección en el genoma del cerdo doméstico. Proc Natl Acad Sci USA. (2012) 109:19529–36. doi: 10.1073/pnas.1217149109
21. Yunlong, M, Zhang, S, Zhang, K, Fang, C, Xie, S, Du, X, et al. Análisis genómico para identificar firmas de selección artificial y loci asociados con rasgos económicos importantes en cerdos Duroc. G3. (2018) 8:3617–25. doi: 10.1534/g3.118.200665
22. Zifeng, G, Yang, Q, Huang, F, Zheng, H, Sang, Z, Xu, Y, et al. Desarrollo de arrays multi-SNP de alta resolución para análisis genéticos y mejoramiento molecular mediante genotipado por secuenciación diana y chip líquido. Planta Comunal. (2021) 2:100230. doi: 10.1016/j.xplc.2021.100230
23. Christine, AJ, María, CM y William, GK. Fe de erratas: p73 es una proteína humana relacionada con p53 que puede inducir apoptosis. Naturaleza. (1999) 399:817
24. Maranke, IK, Shi-Long, L, Lisa, DW, Xiaojing, W, y Dennis, RR. La reactivación de las isoformas de p63 expresadas en el desarrollo predispone al desarrollo y progresión tumoral. Cáncer Res. (2006) 66:3981–6. doi: 10.1158/0008-5472.CAN-06-0027
25. Guojun, W, Nomoto, S, Hoque, MO, Dracheva, T, Osada, M, Lee, CC, et al. DeltaNp63alfa y TAp63alfa regulan la transcripción de genes con distintas funciones biológicas en el cáncer y el desarrollo. Cáncer Res. (2003) 63:2351–7.
26. Simon, SM, Patel, D, Moran, M, Campbell, J, Fenwick, K y Kozarewa, I. La caracterización de todo el genoma revela una interacción compleja entre TP53 y TP63 en respuesta al estrés genotóxico. Ácidos nucleicos Res. (2014) 42:6270–85. doi: 10.1093/nar/gku299
27. Simon, SM, Henry, AE, Pivato, GP, Kozarewa, I, Mitsopoulos, C, Fenwick, K, et al. El análisis de todo el genoma de los sitios de unión de p63 identifica los factores AP-2 como co-reguladores de la diferenciación epidérmica. Ácidos nucleicos Res. (2012) 40:7190–206. doi: 10.1093/nar/gks389
28. Wen, L, Ren, X, Chen, J, Li, L, Lu, S, Chen, T, et al. Las transcripciones de TP63 desempeñan papeles opuestos en la diferenciación del músculo esquelético de pollo. Fisiola delantera. (2018) 6:1298. doi: 10.3389/fphys.2018.01298
29. Yang, X, Lu, H, Yan, B, Romano, RA, Bian, Y, Friedman, J, et al. ΔNp63 regula de manera versátil un amplio programa de genes NF-κB y promueve la proliferación, migración e inflamación epitelial escamosa. Cáncer Res. (2011) 71:3688–700. doi: 10.1158/0008-5472.CAN-10-3445
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Yalan, Y, Fan, X, Yan, J, Chen, M, Zhu, M, Tang, Y, et al. Un completo atlas del epigenoma revela la metilación del ADN que regula el desarrollo del músculo esquelético. Ácidos nucleicos Res. (2021) 49:1313–29. doi: 10.1093/nar/gkaa1203
31. Wei, Z, Zhang, Z, Ma, P, Wang, Z, Wang, Q y Pan, Y. El efecto de los datos genotípicos de alta densidad y diferentes métodos en la predicción genómica conjunta: un estudio de caso en cerdos blancos grandes. Anim Genet. (2022) 54:45–54. doi: 10.1111/age.13275
32. Xinyi, G, Wang, S, Li, D, Jin, B, Qi, Z, Deng, J, et al. El microARN-142a-3p regula la atrofia del músculo esquelético neurogénico dirigiéndose a Mef2a. Mol otros ácidos nucleicos. (2023) 33:191–204. doi: 10.1016/j.omtn.2023.05.023
33. Lianjie, H, Xu, J, Li, H, Ou, J, Jiao, Y, Hu, C, et al. MiR-34c reprime el desarrollo muscular formando un bucle regulador con Notch1. Sci Rep. (2017) 7:9346. doi: 10.1038/s41598-017-09688-y
34. Anderson, T, Renuka, MP, Chaerin, L, y Eek-hoon, J. Perspectivas pasadas, presentes y futuras del factor de transcripción EB (TFEB): mecanismos de regulación y asociación con la enfermedad. La muerte celular difiere. (2022) 29:1433–49. doi: 10.1038/s41418-022-01028-6
35. Jian Jun, X, Jiang, YY, Jiang, Y, Li, CQ, Lim, MC, An, O, et al. Super-enhancer-driven Long non-coding RNA LINC01503, regulado por TP63, está sobreexpresado y es oncogénico en el carcinoma de células escamosas. Gastroenterología. (2018) 154:2137–2151.e1. doi: 10.1053/j.gastro.2018.02.018
36. Yan-Yi, J, Jiang, Y, Li, CQ, Zhang, Y, Dakle, P, Kaur, H, et al. TP63, SOX2 y KLF5 establecen un circuito regulador central que controla los patrones epigenéticos y de transcripción en líneas celulares de carcinoma de células escamosas de esófago. Gastroenterología. (2020) 159:1311–1327.e19. doi: 10.1053/j.gastro.2020.06.050
37. Bénard, J, Douc-Rasy, S, y Ahomadegbe, J-C. Los miembros de la familia TP53 y los cánceres humanos. Hum Mutat. (2003) 21:182–91. doi: 10.1002/humu.10172
38. Cefalù, S, Lena, AM, Vojtesek, B, Musarò, A, Rossi, A, Melino, G, et al. TAp63gamma es necesario para las últimas etapas de la miogénesis. Ciclo celular. (2015) 14:894–901. doi: 10.4161/15384101.2014.988021
39. Qiuling, L, Jiao, J, Heng, Y, Lu, Q, Zheng, Y, Li, H, et al. Prmt5 promueve la especificación celular ciliada de los progenitores epiteliales de las vías respiratorias a través de la inhibición transcripcional de Tp63. J Biol Chem. (2023) 299:104964. doi: 10.1016/j.jbc.2023.104964
40. Sai, M, Zhang, B, LM, LF, Earl, AS, Chiang, Z, Hu, Y, et al. Potencial de cromatina identificado mediante el perfil unicelular compartido de ARN y cromatina. Célula. (2020) 183:1103–1116.e20. doi: 10.1016/j.cell.2020.09.056
41. Xiaomin, B, Rubin, AJ, Qu, K, Zhang, J, Giresi, PG, Chang, HY, et al. Un nuevo enfoque ATAC-seq revela el refuerzo específico del linaje del paisaje de la cromatina abierta a través de la cooperación entre BAF y p63. Genoma Biol. (2015) 16:284. doi: 10.1186/s13059-015-0840-9
42. Evelyn, NK, Oti, M, Niehues, H, van Heeringen, SJ y Schalkwijk, J. El factor de transcripción p63 marca y regula los potenciadores dinámicos durante la diferenciación epidérmica. Rep. EMBO (2015) 16:863–78. doi: 10.15252/embr.201439941
43. Timothy, AM, Chun Li, Z y Eric, NO. MEF2: un regulador dependiente del calcio de la división, diferenciación y muerte celular. Tendencias Biochem Sci. (2002) 20:40–7. DOI: 10.1016/s0968-0004(01)02031-x
44. Amanda, C, y Francisco, JN. Los microARN en el locus de ARN no codificante Gtl2-Dio3 regulado por el factor potenciador de miocitos 2 (MEF2) promueven la proliferación de cardiomiocitos al dirigirse al coactivador transcripcional Cited2. J Biol Chem. (2015) 290:23162–72. doi: 10.1074/jbc. M115.672659
Palabras clave: TP63, mioblastos, proliferación, variación alélica, MEF2A
Cita: Chen Y, Wang Z, Qu X, Song B, Tang Y, Li B, Cao G y Yi G (2024) Un SNP intrónico afecta el desarrollo del músculo esquelético mediante la regulación de la expresión de TP63. Frente. Vet. Sci. 11:1396766. doi: 10.3389/fvets.2024.1396766
Recibido: 16 de marzo de 2024; Aceptado: 28 de mayo de 2024;
Publicado: 12 de junio de 2024.
Editado por:
Ning Song, Universidad Agrícola de Anhui, China
Revisado por:
Jiao Wu, Universidad Agrícola de Yunnan, China
Ling Lian, Universidad Agrícola de China, China
Derechos de autor © 2024 Chen, Wang, Qu, Song, Tang, Li, Cao y Yi. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Guoqiang Yi, yiguoqiang@caas.cn; Guoqing Cao, anniecao710502@aliyun.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía