Uso del aprendizaje profundo para la clasificación de ganglios linfáticos hiperplásicos y subtipos comunes de linfomas caninos
Uso del aprendizaje profundo para la clasificación de ganglios linfáticos hiperplásicos y subtipos comunes de linfomas caninos: un estudio preliminar
 Magdalena Hubbard-Pérez
Magdalena Hubbard-Pérez  Andreea Luchian
Andreea Luchian  Charles Milford
Charles Milford  Lorenzo Ressel*
Lorenzo Ressel*- DiMoLab, Instituto de Infecciones Veterinarias y Ciencias Ecológicas, Departamento de Anatomía Veterinaria, Fisiología y Patología, Universidad de Liverpool, Liverpool, Reino Unido
La Inteligencia Artificial ha observado un crecimiento significativo en su capacidad para clasificar diferentes tipos de tumores en humanos debido a los avances en la tecnología de patología digital. Entre estos tumores, los linfomas son bastante comunes en perros, a pesar de que los estudios sobre la aplicación de la IA en especies domésticas son escasos. Esta investigación tiene como objetivo emplear el aprendizaje profundo (DL) a través de redes neuronales convolucionales (CNN) para distinguir entre los ganglios linfáticos normales y 3 subtipos comunes de linfomas caninos de la OMS. Para entrenar y validar la CNN, se utilizaron 1.530 imágenes microscópicas de alta resolución derivadas de escaneos de portaobjetos completos (WSI, por sus siglas en inglés), incluidas las de áreas de fondo, ganglios linfáticos hiperplásicos (n = 4) y tres subtipos diferentes de linfoma: linfoma difuso de células B grandes (DLBCL; n = 5), linfoblásticas (LBL; n = 5) y el linfoma de la zona marginal (MZL; n = 3). La CNN fue capaz de identificar correctamente 456 imágenes de los 457 equipos de prueba posibles, logrando una precisión máxima del 99,34%. Los resultados de este estudio han demostrado la viabilidad de utilizar el aprendizaje profundo para diferenciar entre ganglios linfáticos hiperplásicos y linfomas, así como para clasificar los subtipos comunes de la OMS. Se requiere más investigación para explorar las implicaciones de estos hallazgos y validar la capacidad de la red para clasificar una gama más amplia de linfomas.
Introducción
Los linfomas son un tipo común de neoplasia que se encuentra en los caninos (1). Por lo general, se derivan de los tejidos linfoides, incluidos los ganglios linfáticos, la médula ósea y el bazo, aunque pueden desarrollarse dentro de cualquier tejido del cuerpo (2). Los linfomas pueden surgir tanto de linfocitos B como de células T, y el origen del linfoma a menudo determina su forma (2). Los linfomas de células B son los más comunes en los caninos, con aproximadamente el 65-75% de células B y el 25-35% de células T (3). Existen múltiples formas de linfomas, siendo el más común el multicéntrico, con una prevalencia del 84% en perros (2). Otras formas son la alimentaria, la mediastínica y la cutánea, que son mucho menos frecuentes (2). Los linfomas caninos comparten muchas características con los linfomas no Hodgkin humanos (LNH) (4). Debido a la similitud entre los LNH humanos y los linfomas caninos, el sistema de la Organización Mundial de la Salud (OMS) para los LNH humanos se ha adaptado para clasificar los tipos de linfomas caninos (4). A pesar de ser considerada obsoleta por algunos autores, la clasificación actualizada de Kiel es también una clasificación apropiada y comúnmente utilizada para los linfomas caninos (5). Según la clasificación de la OMS, tres de los linfomas más diagnosticados son el linfoma difuso de células B grandes (DLBCL), el linfoma de la zona marginal (MZL) y el linfoma linfoblástico (LBL). El linfoma difuso de células B grandes (DLCBL, por sus siglas en inglés) es la forma más común de linfoma que se encuentra en perros, y el 79% de los sujetos son diagnosticados con este subtipo (3). Desde el punto de vista histológico, se definen como de patrón difuso con núcleos uniformemente grandes (6). Estos núcleos son generalmente redondos y con poca frecuencia hendidos o hendidos, con una tasa mitótica variable (4). Los perros con este tipo de linfoma suelen presentar linfadenopatía generalizada y, por lo general, se clasifican en estadios III a V según el sistema de la OMS (7). No está claro si este tipo de linfoma es más común en razas específicas. Sin embargo, algunos estudios indican que los Golden Retrievers, los Labradores Retriever, los Boyeros de Berna y los Pastores Alemanes se ven afectados con mayor frecuencia (6). Los linfomas de la zona marginal (LZM) son el segundo linfoma más frecuente en perros, con un 17% de los casos clasificados como LZM (3). Es un linfoma de células B que generalmente se encuentra en los ganglios linfáticos o en el bazo (4). Se caracterizan por tener un patrón nodular, con células de tamaño intermedio con nucléolo central y abundante citoplasma ligeramente teñido en comparación con otros linfomas (6). Las mitosis son prominentes solo en casos avanzados (4). Este tipo de linfoma se caracteriza morfológicamente por sus «centros germinales desvanecidos», lo que hace que se asemeje a la zona marginal de un folículo ganglionar típico (3). Además de esto, los MZL en etapa tardía mantienen sus características celulares, pero pierden su patrón nodular distintivo (6). Esto hace que sea difícil para los patólogos diferenciar entre este subtipo y DLBCL (6). Los perros con este tipo de linfoma generalmente acuden a la clínica con un ganglio linfático submandibular o cervical agrandado que permanece móvil debajo de la piel. La mayoría de las veces, los MZL están presentes en perros de razas grandes (4). Los linfomas linfoblásticos pueden originarse a partir de células B o T, aunque el fenotipo T se encuentra con mucha más frecuencia para este tipo (8). Es un linfoma menos frecuenteun tipo si se compara con DLBCL o MZL (9). Histológicamente, se caracterizan por tener núcleos uniformemente de tamaño intermedio con cromatina uniformemente distribuida, lo que oculta el detalle nuclear y una alta tasa mitótica (4). Es el subtipo más agresivo que se encuentra con frecuencia en las consultas con perros que presentan uno o más ganglios linfáticos periféricos agrandados (4). El estudio de Comazzi y Riondato (9) también sugiere que este tipo de linfoma se encuentra con mayor frecuencia en boxeadores. Debido a sus diferentes características morfológicas, estos tres tipos comunes de linfoma representan un buen objetivo para explorar la capacidad de la Inteligencia Artificial para discriminar entre ellos y distinguir una proliferación neoplásica linfoide (para la que ofrecen ejemplos comunes) del tejido linfoide hiperplásico. De hecho, la hiperplasia ganglionar reactiva representa el principal diagnóstico diferencial de un ganglio linfático agrandado. El enfoque más utilizado para la clasificación de imágenes es el aprendizaje profundo (DL) implementado con redes neuronales convolucionales (CNN) (10). El aprendizaje profundo es una forma de aprendizaje automático en la que el diseño de una red neuronal está influenciado por la estructura del cerebro humano (11) y aprende con el ejemplo directamente de los datos dados. Algunos ejemplos de tipos de datos son las imágenes, el sonido y el texto. Esta forma de aprendizaje automático es conocida por lograr precisiones muy altas en la clasificación de imágenes. Estas redes pueden alcanzar altas precisiones si se entrenan con una gran cantidad de datos y una red profunda con múltiples capas que permiten aprender millones de características diferentes (11). El aprendizaje por transferencia es un método común utilizado en la enseñanza a distancia. En esta forma de aprendizaje, se selecciona una red previamente entrenada y se utiliza como punto de partida para entrenar una red para aprender una nueva tarea. Se agregan o intercambian características específicas para alterar el proceso antes de que ocurra el entrenamiento. Esto permite que la red construya a partir de sus conocimientos previos y logre el objetivo con más éxito (12). Este es uno de los métodos de entrenamiento más utilizados, ya que es más fácil de implementar y, por lo general, es más rápido, ya que solo implica una puesta a punto (13). El uso del aprendizaje automático y la enseñanza a distancia, en particular para el procesamiento y análisis de imágenes digitales en patología, se ha popularizado en los últimos años. Esto se debe al aumento en el uso de escáneres de portaobjetos digitales completos y a un mayor interés en la patología digital, lo que permite abordar el análisis de imágenes con mayor libertad (14). Este proyecto tiene como objetivo investigar la viabilidad de una CNN para distinguir el ganglio linfático hiperplásico del linfoma y clasificar tres tipos comunes de linfoma canino. Esto se aborda utilizando una estrategia de aprendizaje por transferencia.
Materiales y métodos
Se seleccionaron diferentes casos de linfoma y láminas de ganglios linfáticos hiperplásicos reactivos del archivo de láminas del Departamento de Anatomía Veterinaria, Fisiología y Patología, Sección de Patología Veterinaria de la Universidad de Liverpool. En total, se seleccionaron 4 casos de ganglio hiperplásico, 5 casos por cada DLBCL y LBL, y 3 casos de MZL como ejemplos clásicos de cada categoría, siguiendo los criterios presentes en la clasificación de la OMS (4). Todos los portaobjetos se tiñeron previamente con tinción de hematoxilina y eosina (HE) y fueron diagnosticados por patólogos certificados por la junta con el uso de inmunohistoquímica para la determinación del fenotipo. Los portaobjetos se escanearon con un escáner de portaobjetos microscópico Aperio CS2 (Leica Biosystems, Wetzlar, Alemania) utilizando un aumento nativo de 40X. En la vista previa de las diapositivas, se recortó la región de interés (ROI) y se generó una imagen final de diapositiva completa (WSI). Se utilizó el software de análisis de imágenes QuPath (15) para convertir todos los WSI en los mosaicos que luego se seleccionarían para entrenar la red neuronal. Para cada portaobjetos de linfoma, el área del linfoma se anotó manualmente como «tumor», mientras que para el ganglio linfático reactivo, todas las áreas de los ganglios linfáticos (corteza y médula) se anotaron como «ganglio linfático». Se seleccionó la pestaña automatizar, seguida del editor de scripts, y se utilizó un script personalizado, que permitió la creación de mosaicos no superpuestos y de 512x512x3 píxeles de tamaño. Esto se repitió para cada tipo de linfoma, las áreas de fondo del portaobjetos y los portaobjetos de ganglios linfáticos hiperplásicos. Se seleccionaron ejemplos de cada clasificación entre el gran número de teselas generadas con este proceso (rango: 2000-18.000 dependiendo del tamaño de la muestra). Para cada linfoma, un patólogo veterinario certificado (LR) seleccionó manualmente 75 mosaicos de los mejores ejemplos de cada DLBCL (Figura 1A), LBL (Figura 1B), MZL (Figura 1C) o ganglio linfático hiperplásico (Figura 1D) y los cotejó, asegurándose de que representaran los criterios morfológicos mencionados en la clasificación de la OMS. Para el fondo, también se seleccionaron 15 mosaicos de cada diapositiva de áreas sin tejido. Estos mosaicos de cada diapositiva se agruparon en sus clases y se colocaron en carpetas, lo que dio como resultado 5 carpetas de mosaicos, con un máximo de 375 mosaicos por clase para ingresar en la red neuronal. La formación se llevó a cabo en MATLAB (R2022a, Natick, Massachusetts: The MathWorks Inc.) con Deep Network Toolbox instalado, y el método de aprendizaje fue el aprendizaje por transferencia. GoogLeNet fue elegida entre las posibles CNN como una red rápida, conservando una alta profundidad y precisión (19). La última capa totalmente conectada se eliminó y se reemplazó por una nueva capa totalmente conectada no entrenada, mientras que el tamaño de salida se cambió a cinco clases (DLBCL, LBL, MZL, Hyperplastic y Background). Los valores de WeightLearnRateFactor y BiasLearnRateFactor también se aumentaron a 2 para que la tasa de aprendizaje general sea más rápida. Todas las teselas clasificadas se importaron a la red. Entre 1.530 mosaicos, cada clase se dividió aleatoriamente entre el conjunto de entrenamiento (70 %) y el conjunto de prueba (30 %), y se aplicó una estrategia de validación cruzada de exclusión, ya que el conjunto de prueba se consideró una buena representación estadística de todo el conjunto de datos. La red cambió automáticamente el tamaño de las imágenes para que se ajustaran al formato aceptado por la capa de entrada de red previamente entrenada de 512 x 512 x 3 a un tamaño de entrada de 224 x 224 x 3 (reducción de muestreo). Las teselas se aleatorizaron entre tlloviendo y probando cada vez que se ejecutó la red. En la pestaña de opciones de entrenamiento, se seleccionó uno de los tres solucionadores (sgdm, adam o rmsprop) (un solucionador es el algoritmo de descenso de gradiente estocástico, que evalúa el gradiente de aprendizaje y actualiza los parámetros de la red utilizando una parte de los datos de entrenamiento durante el aprendizaje de una red neuronal para reducir la pérdida), así como una tasa de aprendizaje inicial. Una vez que se completó el proceso de entrenamiento de aprendizaje profundo, se registraron los resultados de la ejecución. Después de revisar y registrar los resultados, la red se volvió a ejecutar con una combinación de diferentes solucionadores y/o valores iniciales de la tasa de aprendizaje hasta que se logró una combinación con alta precisión y bajas pérdidas. Una vez que este experimento se ejecutó varias veces y se identificó el mejor solucionador, los datos se ejecutaron en un experimento de refinamiento utilizando la optimización bayesiana para encontrar la mejor tasa de aprendizaje y obtener la mayor precisión. En esta fase de optimización, se utilizó el solucionador sgdm, y dado que una tasa de aprendizaje inicial de 0,0075 tenía una alta precisión, el rango de tasa de aprendizaje se estableció entre 0,00075 y 0,075. Se seleccionó de antemano un límite máximo de 30 ensayos. Se llevaron a cabo los 30 ensayos. Una vez completada la optimización, los hiperparámetros finales se muestran en la Tabla Suplementaria S1. A partir de estos datos, se generaron gráficos que muestran las precisiones y pérdidas de entrenamiento y validación durante el proceso de aprendizaje. Cuando se completó el entrenamiento, la red produjo una matriz de confusión para mostrar qué tan bien aprendió los datos y las áreas de errores (rendimiento) en las imágenes de prueba (30% de las teselas generadas). Se calcularon parámetros como Falsos Positivos, Sensibilidad, Falsos Negativos y Especificidad para cada clase. Todos los pasos experimentales se ejecutaron en una PC computacionalmente eficiente equipada con una CPU AMD Ryzen Threadripper PRO 3995WX, 6 GPU NVIDIA RTX 3090, 1,00 TB de RAM y Windows 10 Professional. En la Figura 2 se presenta un resumen gráfico del flujo de trabajo de la CNN para el entrenamiento y las pruebas.
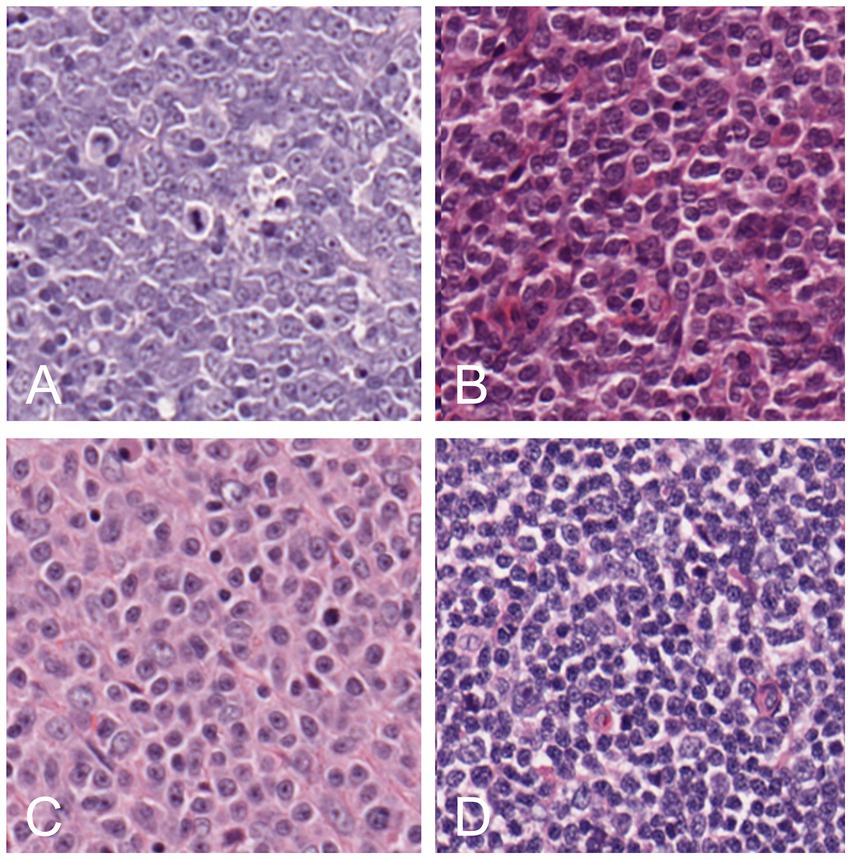 Figura 1. Ejemplos de mosaicos de diferentes clases: DLBCL (A), LBL (B), MZL (C) y ganglio linfático hiperplásico (D).
Figura 1. Ejemplos de mosaicos de diferentes clases: DLBCL (A), LBL (B), MZL (C) y ganglio linfático hiperplásico (D).
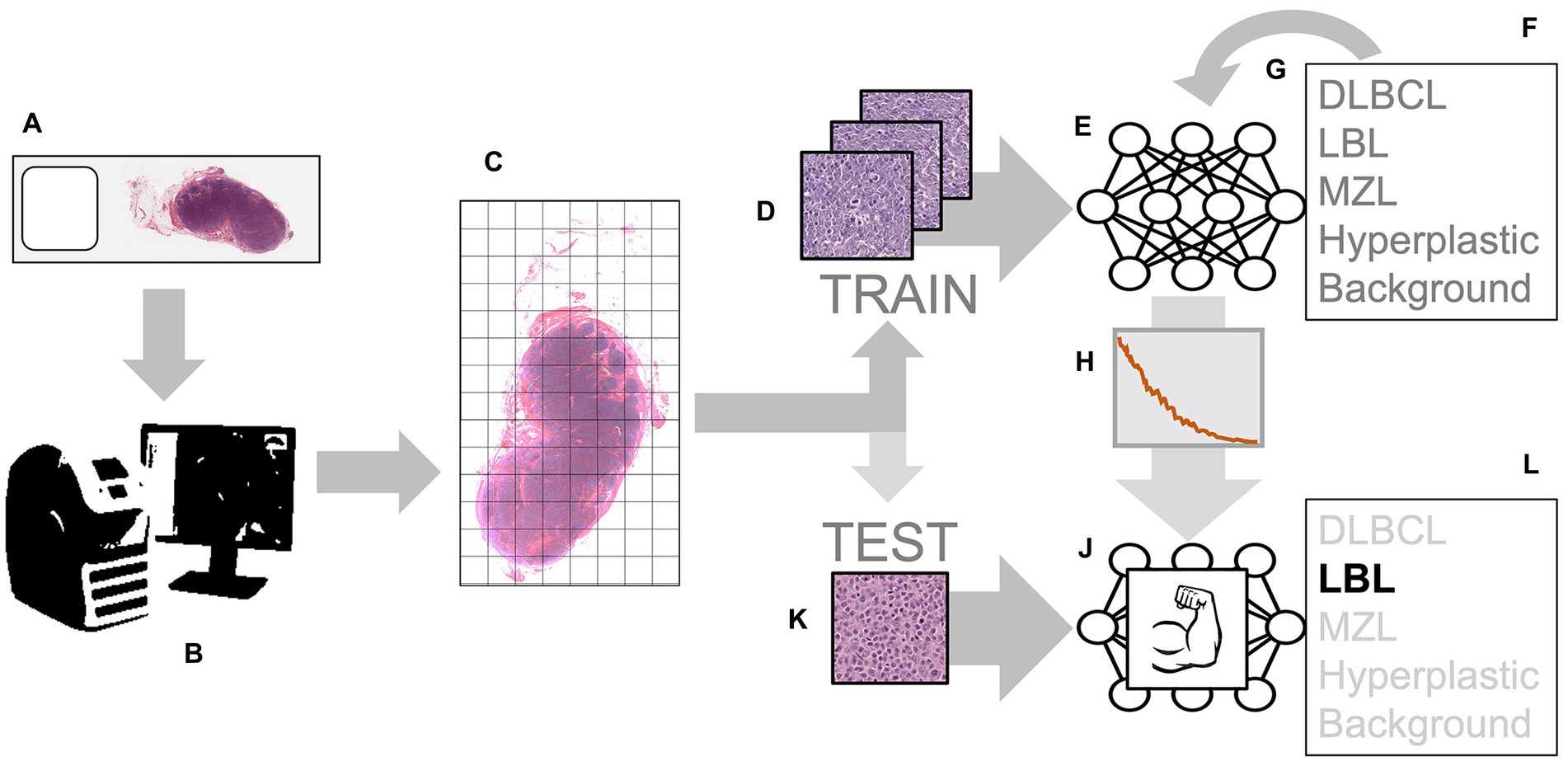 Figura 2. Flujo de trabajo para entrenar y probar la red neuronal convolucional (CNN): el proceso comienza con diapositivas histológicas que representan varias clases, como DLBCL, LBL, MZL y ganglios linfáticos hiperplásicos (A). Estas diapositivas se escanean con un escáner digital de diapositivas, creando una imagen de diapositiva completa (WSI) (B). El WSI se divide en fichas más pequeñas (C). Estas teselas de diferentes clases se utilizan como ejemplos para entrenar la capa de entrada de la CNN (D). La CNN procesa los mosaicos de entrada (E) y predice la probabilidad de cada clase (F). El error en estas predicciones se utiliza para ajustar los pesos de las neuronas a través de la propagación hacia atrás (G). Este proceso continúa hasta que el error se minimiza a un valor óptimo (H). El resultado es una red «entrenada» (J). Esta red entrenada ahora se puede usar para analizar teselas nunca antes vistas obtenidas a través del mismo proceso de ordenamiento en teselas (K). Cuando se alimenta con estas nuevas teselas, la red predice con precisión la clase (L).
Figura 2. Flujo de trabajo para entrenar y probar la red neuronal convolucional (CNN): el proceso comienza con diapositivas histológicas que representan varias clases, como DLBCL, LBL, MZL y ganglios linfáticos hiperplásicos (A). Estas diapositivas se escanean con un escáner digital de diapositivas, creando una imagen de diapositiva completa (WSI) (B). El WSI se divide en fichas más pequeñas (C). Estas teselas de diferentes clases se utilizan como ejemplos para entrenar la capa de entrada de la CNN (D). La CNN procesa los mosaicos de entrada (E) y predice la probabilidad de cada clase (F). El error en estas predicciones se utiliza para ajustar los pesos de las neuronas a través de la propagación hacia atrás (G). Este proceso continúa hasta que el error se minimiza a un valor óptimo (H). El resultado es una red «entrenada» (J). Esta red entrenada ahora se puede usar para analizar teselas nunca antes vistas obtenidas a través del mismo proceso de ordenamiento en teselas (K). Cuando se alimenta con estas nuevas teselas, la red predice con precisión la clase (L).
Resultados
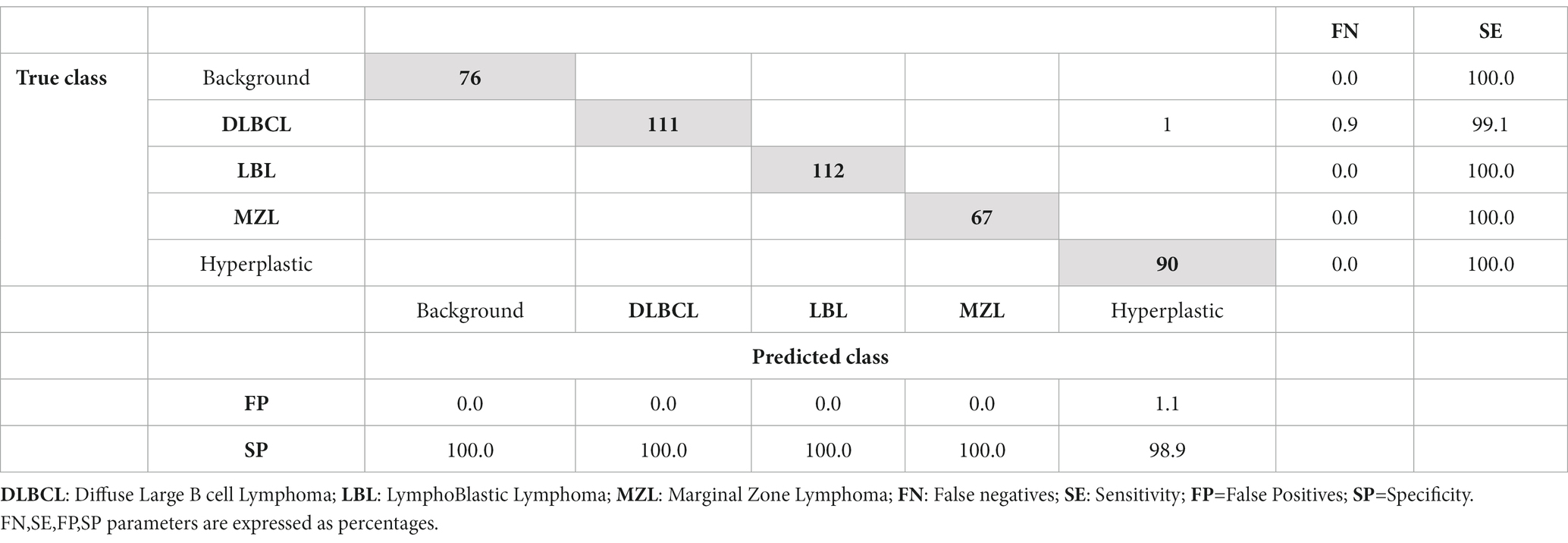
La red se ejecutó tres veces para cada solucionador, cada una con una tasa de aprendizaje diferente, para calcular un rango aproximado de qué tasa de aprendizaje tendría una mayor precisión de validación. A partir de estos experimentos, el solucionador sgdm pudo ejecutarse con una tasa de aprendizaje más alta (0,005) y, al mismo tiempo, tener una alta precisión de validación del 99,34 %. La precisión media de tres experimentos diferentes, con una división aleatoria de mosaicos de entrenamiento y prueba utilizando el solucionador sgdm con diferentes tasas de aprendizaje, fue del 97,01% (Tabla complementaria S2). Después de la optimización bayesiana, 18 de los 30 ensayos fueron identificados como los mejores, que tuvieron una tasa de aprendizaje de 0,0057 (Figura Suplementaria S1). Los diagramas de entrenamiento mostraron que la red convergió aproximadamente 50 iteraciones, alcanzando una precisión cercana al 100% para los conjuntos de entrenamiento y prueba antes de la 250ª iteración (Figura suplementaria S2). Los resultados del conjunto de prueba (ejemplos de DLBCL, LBL, MZL, ganglio linfático normal y antecedentes) presentados en una matriz de confusión (Tabla 1) mostraron que la CNN pudo identificar correctamente 456 imágenes de las 457 posibles del conjunto de prueba. Todos los LBL, MZL, hiperplásicos y mosaicos de fondo se predijeron correctamente, mientras que 90 ejemplos de DLBCL de 91 (98,9) se identificaron correctamente, con un ejemplo de DLBCL clasificado erróneamente como ganglio linfático hiperplásico. Teniendo en cuenta el rendimiento de distinguir los ganglios linfáticos hiperplásicos del linfoma (DLBCL+LBL + MZL), todas las predicciones para el linfoma fueron correctas, mientras que una sola predicción para el ganglio linfático hiperplásico de 90 se clasificó erróneamente como linfoma.
Discusión
El presente trabajo preliminar demostró que el aprendizaje por transferencia aplicado al problema de la diferenciación entre ganglios linfáticos hiperplásicos y neoplásicos y de clasificación de las neoplasias linfoides en perros puede ser exitoso. También demostramos que nuestra estrategia metodológica para la optimización de hiperparámetros de aprendizaje en red es prometedora. Nuestro enfoque utilizó un enfoque de validación cruzada de retención, ya que el conjunto de prueba se consideró una buena representación estadística de todo el conjunto de datos. Dada la precisión media del 97,01 % en 3 experimentos diferentes con diferentes tasas de aprendizaje y aleatorización de los mosaicos de entrenamiento y prueba, la red parece funcionar de forma consistente. A pesar de que el estudio es preliminar y se centra principalmente en la prueba de principio de aplicación de la técnica, en la actualidad, no hay estudios publicados que apliquen las CNN a los linfomas caninos, mientras que el aprendizaje automático se ha aplicado solo marginalmente a los datos clínicos en linfomas caninos (16), sin explorar el potencial de las CNN. La precisión de la red era muy alta. En la única imagen en la que la clasificación de la red era incorrecta, se clasificó erróneamente un mosaico DLBCL como un mosaico de ganglios linfáticos hiperplásicos. Esto es alentador, ya que la red aún pudo clasificar correctamente el 99,1% de las teselas DLBCL que se le presentaron, y se puede argumentar que una vista de muy alta potencia de un centro germinal en un nodo hiperplásico puede superponerse con un área pequeña de DLBCL. Considerado dentro de un escenario en el que el resultado de varias teselas debería lograr el diagnóstico final, este error puede considerarse casi irrelevante. Es probable que siga siendo un desafío establecer, a nivel de mosaico, lo que debe considerarse como ganglio linfático normal, debido a la heterogeneidad de la estructura ganglionar linfática, y probablemente un enfoque de segmentación a menor aumento, similar al que abordamos recientemente en una estructura anatómica igualmente compleja (riñón) (17) podría resultar una estrategia alternativa efectiva. Sin embargo, es interesante observar que la red parece capturar características de diferentes clases sin la necesidad de una estructura submicroscópica del ganglio linfático, probablemente basándose en características citológicas. Una limitación de este estudio preliminar es, inherentemente, el bajo número de portaobjetos utilizados en los experimentos, que pueden no simular completamente la complejidad de un experimento a mayor escala utilizando un mayor número de casos. Sin embargo, la sugerencia de que las características se aprenden a esta tasa de precisión utilizando un número razonablemente bajo de ejemplos (cientos por clase) en el contexto de un enfoque de aprendizaje por transferencia es prometedora para futuros experimentos más grandes, que deberían incluir un conjunto más grande y diverso de conjuntos de datos de entrenamiento y prueba. En un escenario ideal, el desarrollo de una CNN integral para facilitar el diagnóstico asistido por IA a gran escala también obtendría ventajas significativas mediante la incorporación de diversos escáneres de portaobjetos y protocolos de tinción procedentes de varios laboratorios. Es importante señalar que, en la actualidad, este aspecto sigue siendo una de las principales limitaciones en el contexto de los abordajes histológicos de la CNN (18). Los resultados de este experimento han demostrado que la red GoogLeNet es una red altamente confiable para usar para el aprendizaje profundo cuando se aplica a imágenes histológicas de linfoma utilizando un enfoque de aprendizaje por transferencia. Esto sugiere fuertemente que los patrones básicos aprendidos en las primeras capas durante el entrenamiento original en 1,2 millones de imágenes y 1.000 clases diferentes, representande objetos comunes y seres vivos (19), representan un sólido punto de partida para afinar las características de las imágenes histológicas de los linfomas caninos. Se utilizó un número menor de baldosas para una de las categorías de neoplasias (MZL). Sin embargo, a pesar de esto, no hubo ningún efecto sobre el rendimiento, lo que sugiere que los linfomas son, a los «ojos» de la CNN, un tipo de tumor razonablemente homogéneo, donde un número relativamente bajo de ejemplos puede resultar en un entrenamiento exitoso. Hay muchos estudios que analizan las similitudes entre los linfomas caninos y humanos y cómo los modelos caninos pueden contribuir a la investigación del linfoma. Debido a las similitudes y al uso de un sistema de clasificación derivado, este resultado preliminar parece probablemente traslacional a la medicina humana. Los linfomas DLBCL tanto en humanos como en caninos son especialmente similares, ya que se parecen entre sí en muchas características, incluido que son el subtipo más común de linfoma de células B tanto en perros como en humanos (6). El flujo de trabajo propuesto utiliza software de fácil acceso: MATLAB y QuPath son software de código abierto u ofrecen versiones gratuitas, lo que los hace accesibles a investigadores con presupuestos limitados. Además, este software se usa comúnmente en entornos académicos y tiene una amplia base de usuarios, lo que promueve la transparencia, la reproducibilidad y la colaboración dentro de la comunidad científica. En conclusión, este estudio ha demostrado con éxito que se puede utilizar una red neuronal convolucional para diferenciar entre ganglios linfáticos hiperplásicos y neoplásicos, así como para clasificar subtipos específicos de linfomas caninos. La integración del diagnóstico asistido por IA en la histología del linfoma canino, entre otras áreas, promete revolucionar el diagnóstico veterinario, pero aún no se han desentrañado los posibles beneficios y desafíos que podríamos encontrar a medida que esta tecnología se vuelve más frecuente; por lo tanto, se anima a realizar más estudios. Estos hallazgos no solo demuestran el potencial del aprendizaje profundo para distinguir los linfomas caninos de los ganglios linfáticos normales, sino que también sientan las bases para estudios a mayor escala que abarquen diferentes especies y una gama más amplia de subtipos de linfoma.
Declaración de disponibilidad de datos
Los datos brutos que respaldan las conclusiones de este artículo pueden ser puestos a disposición por los autores, sin reservas indebidas previa solicitud.
Declaración ética
El estudio en animales fue aprobado por el Comité de Ética de la Facultad de Ciencias Veterinarias de la Universidad de Liverpool. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales (RETH000942).
Contribuciones de los autores
MH-P: Conceptualización, Curación de datos, Investigación, Visualización, Redacción – borrador original, Redacción – revisión y edición. AL: Conceptualización, Investigación, Metodología, Escritura – Revisión y Edición. CM: Conceptualización, Investigación, Escritura – Revisión y Edición. LR: Conceptualización, Administración de Proyectos, Recursos, Supervisión, Visualización, Redacción, Revisión y Edición.
Financiación
El/los autor/es declaran/n que no se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo.
Reconocimientos
Los autores agradecen a NVIDIA por el apoyo de las GPU y al Laboratorio de Histología de la Universidad de Liverpool por el corte y tinción de los portaobjetos histológicos.®
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1309877/full#supplementary-material
Figura complementaria S1 | Captura de pantalla del resultado de la optimización bayesana, con el experimento elegido 18 resaltado.
Figura complementaria S2 | Curvas de precisión (azul) y pérdida (rojo) para la ejecución 18: la red alcanzó un entrenamiento óptimo debido a la alta precisión y la muy baja pérdida.
CUADRO S1 COMPLEMENTARIO | Hiperparámetros después de la optimización.
CUADRO S2 COMPLEMENTARIO | Precisión de validación ejecutando 3 experimentos con 3 solucionadores diferentes.
Referencias
1. Greenlee, PG, Filippa, DA, Quimby, FW, Patnaik, AK, Calvano, SE, Matus, RE, et al. Linfomas en perros: un estudio morfológico, inmunológico y clínico. Cáncer. (1990) 66:480–90. doi: 10.1002/1097-0142(19900801)66:3<480::aid-cncr2820660314>3.0.co; 2-x
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Vail, DM, Pinkerton, ME, y Young, KM. Tumores hematopoyéticos En: SJ Withrow, R Page y DM Vail, editores. Oncología clínica de pequeños animales de Withrow y MacEwen. 5ª ed. Saint Louis: Elsevier (2013). 608–78.
3. Richards, KL, y Suter, SE. El mejor amigo del hombre: ¿qué pueden enseñarnos los perros sobre el linfoma no Hodgkin?’. Immunol Rev. (2014) 263:173–91. doi: 10.1111/imr.12238
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Valli, VE, San Myint, M, Barthel, A, Bienzle, D, Caswell, J, Colbatzky, F, et al. Clasificación de los linfomas malignos caninos según los criterios de la Organización Mundial de la Salud. Veterinario Pathol. (2010) 48:198–211. doi: 10.1177/0300985810379428
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Sayag, D, Fournel-Fleury, C y Ponce, F. Significado pronóstico de los morfotipos en los linfomas caninos: una revisión sistemática de la literatura. Veterinario Comp Oncol. (2017) 19:12–9. doi: 10.1111/vco.12320
6. Avery, A.C. Bases genéticas y moleculares de modelos caninos de leucemia y linfoma humanos. Frente Oncol. (2020) 10:32. doi: 10.3389/fonc.2020.00023
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Assumpção, ALFV, Jark, PC, Hong, CC, Lu, Z, Ruetten, HM, Heaton, CM, et al. La expresión y la actividad de STAT3 se regeneran al alza en el linfoma de células B grandes de perros. J Veterinario Pasante Med. (2017) 32:361–9. doi: 10.1111/jvim.14860
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Holm, F, Hardon, T, Clasen-Linde, E, Hjorth Mikkelsen, L, y Heegaard, S. Linfoma linfoblástico de células B de la membrana nictitante como primer signo de presentación en un springer spaniel de 2 años. Clin Case Rep. (2018) 6:2246–51. doi: 10.1002/ccr3.1862
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Comazzi, S, y Riondato, F. Citometría de flujo en el diagnóstico del linfoma canino de células T. Front Vet Sci. (2021) 8:600963. doi: 10.3389/fvets.2021.600963
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Krizhevsky, A, Sutskever, I, y Hinton, GE. Clasificación de redes de imágenes con redes neuronales convolucionales profundas. Commun ACM. (2012) 60:84–90. doi: 10.1145/3065386
11. LeCun, Y, Bengio, Y y Hinton, G. Aprendizaje profundo. Naturaleza. (2015) 521:436–44. doi: 10.1038/nature14539
12. Janowczyk, A, y Madabhushi, A. Aprendizaje profundo para el análisis de imágenes de patología digital: un tutorial completo con casos de uso seleccionados. J Pathol Informar. (2016) 7:29. doi: 10.4103/2153-3539.186902
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Pan, SJ y Yang, Q. Un estudio sobre el aprendizaje por transferencia. IEEE Trans Knowl Data Eng. (2010) 22:1345–59. doi: 10.1109/TKDE.2009.191
14. Madabhushi, A, y Lee, G. Análisis de imágenes y aprendizaje automático en patología digital: desafíos y oportunidades. Med Image Anal. (2016) 33:170–5. doi: 10.1016/j.media.2016.06.037
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Bankhead, P, Loughrey, MB, Fernández, JA, Dombrowski, Y, McArt, DG, Dunne, PG, et al. QuPath: software de código abierto para el análisis digital de imágenes de patología. Sci Rep. (2017) 7:16878. doi: 10.1038/s41598-017-17204-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Koo, J, Choi, K, Lee, P, Polley, A, Pudupakam, RS, Tsang, J, et al. Predicción de resultados clínicos dinámicos de la quimioterapia para pacientes con linfoma canino mediante un modelo de aprendizaje automático. Veterinaria Sci. (2021) 8:301. doi: 10.3390/vetsci8120301
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Luchian, A, Cepeda, KT, Harwood, R, Murray, P, Wilm, B, Kenny, S, et al. Cuantificación de la lesión renal aguda en un modelo de ratón con lesión por isquemia-reperfusión mediante segmentación semántica basada en el aprendizaje profundo en histología. Biol Abierto. (2023) 12:bio059988. doi: 10.1242/bio.059988
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Litjens, G, Kooi, T, Bejnordi, BE, Setio, AAA, Ciompi, F, Ghafoorian, M, et al. Una encuesta sobre el aprendizaje profundo en el análisis de imágenes médicas. Med Image Anal. (2017) 42:60–88. doi: 10.1016/j.media.2017.07.005
Palabras clave: linfoma, canino, red neuronal convolucional, Inteligencia Artificial, aprendizaje por transferencia
Cita: Hubbard-Perez M, Luchian A, Milford C y Ressel L (2024) Uso del aprendizaje profundo para la clasificación de ganglios linfáticos hiperplásicos y subtipos comunes de linfomas caninos: un estudio preliminar. Frente. Vet. Sci. 10:1309877. doi: 10.3389/fvets.2023.1309877
Recibido: 08 de octubre de 2023; Aceptado: 04 de diciembre de 2023;
Publicado: 12 enero 2024.
Editado por:
Vittoria Castiglioni, Laboratorios IDEXX (Alemania), Alemania
Revisado por:
Jorge Del Pozo, Universidad de Edimburgo, Reino Unido
Diana Giannuzzi, Universidad de Padua, Italia
Derechos de autor © 2024 Hubbard-Pérez, Luchian, Milford y Ressel. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Lorenzo Ressel, ressel@liverpool.ac.uk
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía