Utilidad limitada de la PCR de aterrizaje IS6110 en sangre para la prueba cutánea de la tuberculina en bovinos
Utilidad limitada de la PCR de aterrizaje IS6110 en sangre para la prueba cutánea de la tuberculina en bovinos falsos negativos con respuesta serológica a Mycobacterium bovis
 Micaela Encinas1
Micaela Encinas1  Ximena Ferrara Muñiz1
Ximena Ferrara Muñiz1  Romina Ayelén Sammarruco2
Romina Ayelén Sammarruco2  Victori Ruiz Menna2
Victori Ruiz Menna2  Carlos Javier Garro2
Carlos Javier Garro2  Fernando Delgado2
Fernando Delgado2  Analía Macías3
Analía Macías3  Gabriel Magnano3
Gabriel Magnano3  Martín José Zumárraga1*
Martín José Zumárraga1*  Sergio Gabriel Garbaccio2†
Sergio Gabriel Garbaccio2†  María Emili Eirin1†
María Emili Eirin1†- 1Instituto de Agrobiotecnología y Biología Molecular (IABiMo) UEDD CONICET-INTA, Centro de Investigación en Ciencias Veterinarias y Agronómicas (CICVyA)-CNIA, Hurlingham, Argentina
- 2Instituto de Patobiología Veterinaria (IPVET), UEDD CONICET-INTA, Instituto Nacional de Tecnología Agropecuaria (INTA), INTA-CONICET, Hurlingham, Argentina
- 3Departamento de Patología Animal, Facultad de Agronomía y Veterinaria, Universidad Nacional de Río Cuarto, Río Cuarto, Argentina
El diagnóstico ante mortem de la tuberculosis bovina (TBBb) se basa principalmente en la prueba cutánea de la tuberculina (TST) y el ensayo de liberación de ɣ-IFN (IGRA). Algunos animales infectados escapan a las pruebas de detección, por lo que limitan el saneamiento del rebaño. Informes anteriores han sugerido un patrón predominante de lesiones multiorgánicas atribuibles a la bacteriemia por Mycobacterium bovis (el agente causal de la tuberculosis bovina). Se realizó un estudio de casos y controles para investigar la PCR en sangre como una herramienta alternativa para mejorar la detección ante-mortem de bovinos falsos negativos de TST. Los casos comprendieron 70 bovinos falsos negativos a la TST (casos), que fueron serológicamente positivos, y los controles incluyeron 81 bovinos positivos a la TST; todos ellos confirmados como infectados por M. bovis. Se realizó la detección de la diana IS6110 mediante PCR en sangre (IS6110 TD-PCR). La positividad de la PCR en sangre fue del 27,2% en el grupo control. Este desempeño fue similar al 15% obtenido entre los casos (p = 0,134). La mayoría de los casos identificados por la TD-PCR IS6110 presentaron lesiones focalizadas (p = 0,002). Los resultados demostraron que la PCR en sangre podía detectar bovinos falsos negativos para TST, incluso si son negativos para IGRA. Teniendo en cuenta que los casos exhibieron respuesta humoral a M. bovis, estudios adicionales realizados en una etapa pre-serológica podrían proporcionar evidencia sobre la contribución real de la técnica en rebaños.
Introducción
La tuberculosis bovina (TBB), una enfermedad crónica diseminada en todo el mundo, afecta a los países agrícolas. Su agente etiológico, Mycobacterium bovis, infecta a una amplia gama de mamíferos, incluidos los humanos. Los programas de control y erradicación de esta enfermedad consisten en una política de «prueba y sacrificio» mediante el uso de la prueba cutánea de la tuberculina in vivo (TST) como herramienta primaria de cribado (1). La TST es muy eficaz, pero tiene limitaciones. El diagnóstico puede verse comprometido por las altas tasas de exposición a micobacterias no tuberculosas (MNT) (2) y la existencia de no reactores a las pruebas de cribado. Informes previos mostraron la presencia de animales falsos negativos a las pruebas de cribado en rebaños endémicos (3-10).
Los bovinos falsos negativos de TST constituyen un desafío para la sanidad del rebaño, ya que permanecen en el rebaño como fuente de infección. En Argentina, Garbaccio et al. (5) reportaron que el 76% de los bovinos lecheros falsos negativos de TST también fueron negativos para el IGRA, lo que sugiere que las pruebas basadas en la respuesta inmune mediada por células detectan parcialmente la infección entre estos animales. Además, la respuesta humoral detecta falsos negativos de TST en bovinos de rebaños endémicos, lo que probablemente se asocie a etapas tardías de la enfermedad (4, 5, 10). Sin embargo, algunos animales con falsos negativos de TST e IGRA podrían estar en una etapa pre-serológica y, por lo tanto, no serían detectados en los rebaños.
Los bovinos falsos negativos para la PT presentaron lesiones macroscópicas exacerbadas con un compromiso multiorgánico predominante (5), que puede ocurrir debido a la diseminación hematógena de los bacilos desde el área de infección primaria. Este hecho sugiere que las micobacterias viables podrían ser detectables en muestras de sangre, como se ha confirmado previamente en bovinos infectados de forma natural (11). En cuanto a la detección de M. bovis en sangre, estudios previos desarrollaron diferentes PCRs específicas en sangre, dirigidas a secuencias de M. bovis (mpb70, gen esxB, región RD4, IS6110), con resultados variables (12-16).
Zumárraga et al. (17) desarrollaron un ciclo de aterrizaje para PCR, con el elemento IS6110 como diana (IS6110 TD-PCR), que es altamente específico para los miembros del complejo Mycobacterium tuberculosis (MTC) (17). La TD-PCR IS6110 mostró una sensibilidad mejorada en comparación con la IS6110-PCR estándar y detectó ADN de M. bovis en diferentes muestras biológicas (tejidos, leche, hisopos nasales) y cultivos de colonias (PCR de colonias), para su diagnóstico directo en bovinos infectados de M. bovis (17-20). Sin embargo, la utilidad de la TD-PCR IS6110 en muestras de sangre sigue sin explorarse.
El objetivo del estudio fue evaluar el rendimiento de la TD-PCR IS6110 en bovinos falsos negativos TST de rebaños endémicos de tuberculosis bovina. Los resultados confirman la presencia de ADN de M. bovis en sangre, y constituyen evidencia para futuros estudios que evalúen la utilidad de la técnica en una etapa pre-serológica de la infección.
Materiales y métodos
Diseño del estudio
Se realizó un estudio de casos y controles para evaluar el rendimiento de la PCR IS6110 TD en sangre de bovinos Holstein frisones (n = 151), predominantemente mayores de 2 años, con TBB confirmada. Los animales provenían de 31 hatos lecheros diferentes bajo programas de saneamiento ubicados en la región lechera productiva central de Argentina.
El grupo control (n = 81) incluyó bovinos positivos a la PT de acuerdo con los lineamientos oficiales argentinos. La selección de casos (n = 70) se realizó entre los no reactores a la PT, considerando aquellos animales positivos para respuesta humoral contra M. bovis mediante Enzyme-Linked Immunosorbent Assay (ELISA) (5). La sangre de los casos y controles se analizó mediante PCR.
Los casos también fueron analizados por IGRA, como una prueba auxiliar a la TST que también evidencia una respuesta inmune mediada por células.
El patrón patológico se evaluó en la inspección del matadero para caracterizar la presentación de la enfermedad entre los casos. Se registró como un órgano con lesiones o más de un órgano con lesiones (lesiones multiorgánicas).
Se confirmó la presencia de tuberculosis bovina en los animales que dieron positivo en al menos una de las siguientes pruebas: bacteriología, histopatología-tinción de ZN y PCR de tejidos.
Prueba cutánea de la tuberculina
Los veterinarios acreditados realizaron la TST del pliegue caudal (CF-TST) de acuerdo con las directrices oficiales del Servicio Nacional de Sanidad y Calidad Agroalimentaria (Ley 128/12)1. Debido a que la CF-TST se aplicó en rebaños en saneamiento, un animal que presentó un aumento del grosor de los pliegues cutáneos ≥3 mm fue positivo (prueba de interpretación severa).
Ensayo de liberación de interferón gamma (IGRA)
Todos los casos fueron analizados por el IGRA. La sangre heparinizada (200 μL) de la vena yugular se estimuló con 25 μL de los siguientes antígenos: Tuberculina aviar PPD 2500 (250 UI/mL), Tuberculina bovina PPD 3000 (300 UI/mL), solución salina tamponada con fosfato estéril (PBS) 1X y BOVIGAM® Pokeweed Mitogen (5 μg/mL) en una placa de cultivo celular de 96 pocillos. El plasma recolectado se almacenó a -20 °C para la medición de γ-IFN mediante ELISA utilizando un ELISA comercial de microplacas de γ IFN bovino. La estimulación sanguínea, el ELISA y la interpretación de los datos (interpretación estándar, punto de corte = 0,1) se realizaron de acuerdo con las instrucciones del fabricante (kit de tuberculosis BOVIGAM, Thermofisher).
Detección de la respuesta humoral mediante ELISA
Sueros humorales probados con ELISA de animales TST no reactores. Se obtuvo sangre (10 ml) de la vena yugular entre 15 y 20 días después de la prueba de fibrosis quística. El suero (100 μL, 1.100 en solución PBS 1X), se centrifugó a 2500 g y luego se utilizó para probar anticuerpos contra el derivado purificado de proteína bovina (PPDB). El ELISA consistió en una etapa de unión (12 μL de PPDB por pocillo, 1 μg/mL) realizada durante la noche a 4 °C, una etapa de lavado (cinco veces con una solución de PBS 1X-Tween 0,5, 1% de leche desnatada) y una etapa de incubación con sueros (100 μL, diluido 1.100 en solución de PBS 1X) 1 h a 37 °C. La placa se lavó con PBS 1X y se adicionaron 100 μL de anticuerpo conjugado anti-IgG-Peroxidasa bovina (Sigma, dilución 1:7000) para incubar las muestras con este anticuerpo secundario durante 1 h a 37°C. Los platos se lavaron cinco veces más. Finalmente, 100 μL del sustrato revelador (Tampón Citrato + ABTS (Sigma) + H2O2) se añadieron y se incubaron durante 10 min en la oscuridad. La lectura de la reacción se realizó a 405 nm. Las densidades ópticas (DO) ≥ 0,42 clasificaron a los animales como positivos (5).
PCR en sangre
Se obtuvo sangre (10 mL) anticoagulada con EDTA (0,5 mg/mL) de la vena yugular. La extracción de ADN se realizó con un kit comercial (kit PuriPrep-S, InbioHighway), siguiendo las instrucciones del fabricante. Como la PCR en sangre no es un protocolo rutinario que se realiza en nuestro laboratorio, se realizaron algunas evaluaciones antes de comprobar la presencia de ADN de M. bovis en las muestras. La calidad (A260nm/A280nm) y la concentración (A260) se evaluaron mediante espectrofotometría (NanodropTM, Thermo Fisher Scientist). La integridad del ADN también se analizó mediante electroforesis en un gel de agarosa al 0,8% (tampón TAE 1X, 80 voltios durante 60 min.) teñido con 5 μg/mL de bromuro de etidio (Promega, EE.UU.). También se realizó la amplificación de un fragmento de 450 pb del gen endógeno de ARN ribosómico mitocondrial 16S (16SRNArmt) (21) mediante el uso de la ADN polimerasa GoTaq® G2 (Promega, EE. UU.). Los productos de amplificación se visualizaron por electroforesis en un gel de agarosa al 1,2% (tampón TAE 1X, 90 voltios durante 45 min) teñido con bromuro de etidio, como se ha descrito anteriormente, y con un marcador de peso molecular de 100 pb (100 pb Plus DNA Ladder, Trans, China).
La presencia de ADN de M. bovis en sangre se evaluó utilizando un fragmento específico de MCT IS6110 (IS6110 TD-PCR) utilizando el kit HotStarTaq® Master Mix (Qiagen, EE. UU.), como se describió anteriormente (17). Los productos se visualizaron por electroforesis en un gel de agarosa al 2% (tampón TAE 1X, 80 voltios durante 45 min) teñido con bromuro de etidio, como se ha descrito anteriormente, y con un marcador de peso molecular de 100 pb (100 pb Plus DNA Ladder, Trans, China). El producto de amplificación esperado era de 245 pb.
Inspección post mortem y toma de muestras
Los veterinarios oficiales realizaron una inspección detallada en busca de lesiones compatibles con la tuberculosis bovina, de acuerdo con los procedimientos oficiales del matadero. Se tomaron muestras de porciones representativas de los ganglios linfáticos (LN) retrofaríngeos y submandibulares, respiratorios (LN traqueobronquiales y mediastínicos, y pulmón), digestivos (LN mesentéricos y hepáticos, e hígado) y mamarios (ubre y supramamarios) en contenedores individuales para realizar PCR de tejidos y bacteriología. Además, otras piezas fueron dispuestas en recipientes con formalina tamponada al 10% para la fijación de tejidos para histopatología. En cuanto a los casos, después de realizar la inspección patológica, los animales se clasificaron como aquellos que presentaban un órgano con lesiones macroscópicas o aquellos con lesiones multiorgánicas.
Bacteriología e histopatología
Para bacteriología, 30 g de cada tejido se cortaron en trozos pequeños con unas tijeras y se colocaron en una bolsa estéril con 20 mL de agua estéril de doble destilación. Se realizó una maceración de 3 min. (Basic Masticator, IUL Instruments tipo 470, España) y el homogeneizado se descontaminó por el método de Petroff (22). Se inoculó una alícuota de 2 mL de cada muestra descontaminada en medios sólidos Stonebrink a base de huevo a 37 °C con observación quincenal, durante al menos 8 semanas. Esta inoculación se realizó por triplicado. Las colonias sospechosas de M. bovis se tiñeron con Ziehl-Neelsen (ZN) para identificar bacterias acidorresistentes (23). Para la histopatología, las muestras fijas se deshidrataron con diferentes soluciones alcohólicas de concentración creciente (50, 70, 80, 95% y etanol absoluto), posteriormente se clarificaron con xileno y, finalmente, se incluyeron en parafina. Los tapones de parafina se cortaron en secciones de 5 μm de espesor (Leica RM2125 RTS, Biosystems), se desparafinaron, se hidrataron y se tiñeron con hematoxilina-eosina y tinción con ZN (23).
PCR de tejidos
Se utilizaron indistintamente dos PCR diferentes para comprobar la presencia de ADN de M. bovis en los tejidos recogidos: la TD-PCR IS6110 (17), con un producto esperado de 245 pb, y una PCR de aterrizaje adaptada a partir de una PCR anidada Rv2807 (TD-PCR Rv2807) previamente descrita (24). El ADN de los tejidos se obtuvo utilizando un kit de extracción comercial (ADN PuriPrep-T Kit, InbioHighway, Argentina) de acuerdo con las instrucciones del fabricante. Se utilizó la ADN polimerasa GoTaq® G2 (Promega, EE. UU.) de acuerdo con las instrucciones del fabricante. Brevemente, la etapa de adaptación de la TD-PCR Rv2807 consistió en una desnaturalización inicial a 96 °C durante 3 min, con ocho ciclos de 96 °C durante 1 min, con una temperatura de recocido que se redujo gradualmente de 72 °C a 64 °C, y una etapa de extensión a 72 °C durante 1 min. El procedimiento fue seguido por 30 ciclos que incluyeron una etapa de desnaturalización a 96 °C durante 1 min, recocido a 66 °C durante 1 min, extensión a 72 °C durante 1 min 45 s y una extensión final a 72 °C durante 8 min. El producto de amplificación esperado fue de 443 pb. Los productos de PCR se visualizaron en un gel de agarosa al 2% (tampón TAE 1X), 80 voltios durante 45 min, teñido con 5 μg/mL de bromuro de etidio (Promega, EE. UU.) y comparado con un marcador de peso molecular de 100 pb (100 pb Plus DNA Ladder, Trans, China). La digitalización de las imágenes se realizó en un analizador genético Geldoc (Bio-Rad Laboratories).
Análisis estadístico
La comparación estadística de proporciones se realizó con el software EpiDat versión 3.0 (Xunta de Galicia, OPS-OMS). Los valores de p calculados iguales o inferiores a 0,05 se consideraron estadísticamente significativos. Se analizó la concentración y calidad del ADN para identificar valores atípicos significativos (p > 0,05) mediante la prueba de Grubbs (25). La distribución de los datos se probó mediante la prueba de Shapiro Wilk con mediana y rango intercuartílico de 25-75 (RIQ25–75) como estadística descriptiva. Las comparaciones de medias se realizaron mediante la prueba t de Student no apareada (GraphPad Software, Inc.). Se utilizó el Test exacto de Fisher para evaluar la asociación entre los resultados de la TD-PCR IS6110 en sangre y el perfil patológico.
Resultados
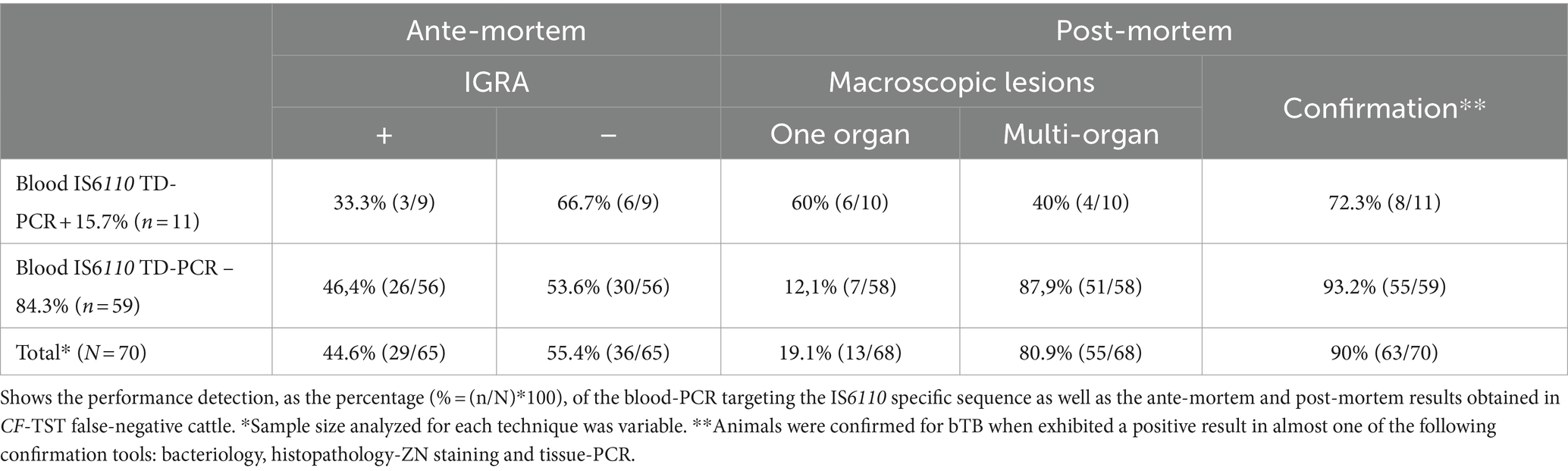
La prueba IGRA in vitro, que representa una herramienta adicional que mide la respuesta inmune mediada por células, registró un 55,4% de negatividad entre los casos. Este resultado muestra que los animales, positivos o negativos a esta prueba de tamizaje auxiliar, estuvieron representados de manera similar dentro de los bovinos falsos negativos CF-TST (p > 0.05) (Tabla 1).
En la inspección del matadero, los casos presentaron 97,1% de lesiones macroscópicas compatibles con TBB. De estos animales, el 80,9% desarrollaron lesiones multiorgánicas y, en menor proporción, el 19,1% de ellos granulomas limitados a un órgano. Dos animales carecían de lesiones macroscópicas, aunque con resultado positivo de tinción histopatológica-ZN (Tabla 1).
Se realizó extracción de ADN de sangre en todos los bovinos estudiados (n = 151). La mediana de la concentración genómica de ADN de los casos fue de 5,9 ± 3,6; 11,2 ng/μL, significativamente inferior a la obtenida en el grupo control, en el que la mediana de concentración fue de 27,0 ± 19,0; 56,3 ng/μL (p < 0,0001). A pesar de esta diferencia, la calidad de las plantillas según A260nm/Un280nm y los resultados positivos para la PCR 16SRNArmt sugirieron buenas condiciones del ADN extraído (Figura suplementaria 1). Por lo tanto, la plantilla fue útil para otras determinaciones posteriores, como la TD-PCR IS6110.
La PCR en sangre realizada en casos y controles (n = 151), arrojó un 22% de positividad para la TD-PCR IS6110. La estratificación de los datos reveló que el 27,2% de los controles fueron positivos para la TD-PCR IS6110, mientras que una proporción menor (15,7%) de los casos fue positiva para esta prueba; sin embargo, las diferencias no fueron significativas (p = 0,134).
Por otro lado, la TD-PCR IS6110 arrojó resultados positivos significativamente mayores en animales con lesiones focalizadas (46,2%), en comparación con animales con lesiones multiorgánicas (7,3%) (p = 0,002). Además, el 66,7% de las muestras positivas para TD-PCR IS6110 fueron IGRA negativas (Tabla 1).
Discusión
La tuberculosis bovina, un problema de salud para los animales y los seres humanos, es una enfermedad milenaria distribuida en todo el mundo, que se encuentra entre las principales amenazas zoonóticas (26). En la actualidad, los bovinos falsos negativos de TST constituyen un reto para el diagnóstico, ya que estos animales permanecen en los rebaños que propagan la enfermedad. Algunos de estos bovinos tampoco reaccionan a las herramientas de diagnóstico auxiliares como el IGRA (3, 5).
En Argentina, nuestro grupo reportó casos de estos bovinos falsos negativos para TST con resultados negativos repetidos para CF-TST y una patología multiorgánica predominante en hatos en saneamiento, la mayoría de estos animales fueron negativos para IGRA (5). En España, los bovinos doble negativo TST e IGRA fueron identificados como positivos para bacteriología. Los investigadores informaron que estos animales produjeron una proporción significativamente mayor de cultivo positivo en el ganado vacuno de carne en comparación con el ganado lechero (3).
En las infecciones experimentales, M. bovis promueve un predominio de la inmunidad mediada por células, pero con anticuerpos bajos o ausentes durante las etapas iniciales. La respuesta humoral aumenta a medida que avanza la infección, lo que se asocia a la exacerbación de la patología de la enfermedad (27, 28). Los bovinos falsos negativos de TST exhiben una respuesta humoral, y estudios previos demostraron que la detección de anticuerpos contra M. bovis podría contribuir a identificar a estos animales en rebaños naturalmente endémicos (5, 6, 10). Sin embargo, la asociación de anticuerpos con estadios más tardíos de la enfermedad podría limitar el rendimiento de las herramientas basadas en la serología para detectar aquellos bovinos falsos negativos que aún no han alcanzado este estadio humoral.
El presente estudio constituye una evidencia valiosa para comprender cómo tratar este fenotipo en rebaños endémicos de tuberculosis bovina. Sin embargo, es importante destacar que los resultados aquí obtenidos están sesgados dados dados los criterios de selección para identificar bovinos falsos negativos CF-TST en hatos endémicos. Esta selección se basó en la presencia de respuesta humoral. Algunas preguntas siguen abiertas: ¿Se pueden identificar los bovinos falsos negativos de doble TST e IGRA antes de la etapa humoral? ¿Hay animales falsos negativos con lesiones pero sin respuesta humoral, a pesar de un supuesto estadio avanzado de la infección?
La detección del genoma de M. bovis en muestras de sangre constituye un enfoque poco desarrollado entre los reactores positivos en la literatura, y sin antecedentes entre el fenotipo falso negativo de CF-TST. En Etiopía, la PCR dirigida al gen mpb70 de M. bovis en muestras de sangre detectó una proporción significativamente mayor (68%) de animales positivos (12) que la obtenida en la presente investigación (p < 0,0001). Estos animales pertenecían a bovinos infectados crónicamente, según lo examinado por el TST cervical. En nuestro estudio, los bovinos positivos a CF-TST procedían de rebaños en saneamiento, donde no se espera que los bovinos altamente infectados estén presentes durante un período prolongado. Estas diferencias podrían limitar la sensibilidad de la TD-PCR IS6110 en muestras de sangre. Por el contrario, un estudio indio anterior no informó de muestras de sangre positivas para IS6110-PCR de bovinos positivos para TST cervical con infección confirmada (14). Los investigadores utilizaron el método de fenol-cloroformo-alcohol isoamílico para extraer el ADN, en contraste con el presente estudio y la investigación de Elsohaby et al., que utilizaron kits comerciales optimizados. Thakur et al. no realizaron controles de la cantidad y la calidad del ADN extraído, que son esenciales para garantizar la utilidad de una muestra de ADN para aplicaciones posteriores, incluida la PCR (29).
Mediante la realización de la TD-PCR IS6110 en leche bovina, Zumárraga et al. (20) demostraron la presencia de M. bovis en hatos libres de tuberculosis bovina de un área productiva lechera con focos de infección en la provincia de Santa Fe, Argentina. Con base en este estudio, las autoridades incorporaron la detección de M. bovis en hatos lecheros como herramienta complementaria de vigilancia en el Plan Regional para el control y erradicación de la tuberculosis bovina en la provincia de Santa Fe (Ley 949/12, Ministerio de la Producción de la provincia de Santa Fe, Argentina). Sin embargo, los resultados del presente estudio sugieren que el uso de PCR para muestras de sangre podría ser útil, no solo para el ganado lechero, sino también para el ganado vacuno de carne, y en todos los períodos del ciclo productivo.
A pesar de las lesiones multiorgánicas predominantes observadas entre los bovinos falsos negativos para CF-TST, este patrón patológico no necesariamente reflejaría una mayor presencia de ADN micobacteriano en sangre.
Una posible explicación sería que el ADN desnudo circulante, bacilos viables asociados a bacteriemia o ambos, podrían estar presentes en la sangre pero de forma intermitente.
Los bovinos falsos negativos de CF-TST limitan el saneamiento de los rebaños infectados por la tuberculosis bovina basándose en la aplicación de pruebas de cribado ante mortem, lo que apoya la búsqueda de mejores herramientas de diagnóstico. Los resultados aquí presentados, aunque preliminares, confirman la presencia de ADN de M. bovis en muestras de sangre bovina de bovinos falsos negativos CF-TST, lo que sugiere la utilidad de esta técnica a considerar. Sin embargo, otros estudios realizados en una etapa pre-serológica podrían proporcionar más evidencia sobre la contribución real de la técnica en rebaños.
Declaración de disponibilidad de datos
Los datos brutos que respaldan las conclusiones de este artículo serán puestos a disposición por los autores, sin reservas indebidas.
Declaración ética
Los estudios en animales fueron aprobados por el CICUAE Instituto Nacional de Tecnología Agropecuaria, CICVyA. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. No se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio debido a que el estudio se realizó en rebaños endémicos. Los animales detectados como infectados por M. bovis deben ser enviados al matadero debido a que la infección en Argentina es una enfermedad de declaración obligatoria.
Contribuciones de los autores
MEn: Curación de datos, Análisis formal, Investigación, Metodología, Redacción – borrador original, Redacción – revisión y edición. XF: Curación de datos, metodología, redacción, revisión y edición. RS: Curación de datos, Investigación, Metodología, Redacción, revisión y edición. VR: Curación de datos, metodología, redacción, revisión y edición. CG: Curación de datos, metodología, redacción, revisión y edición. FD: Curación de datos, Investigación, Metodología, Redacción – revisión y edición. AM: Investigación, Metodología, Redacción – revisión y edición. GM: Investigación, Metodología, Redacción – Revisión y Edición. MZ: Conceptualización, Obtención de Fondos, Recursos, Supervisión, Redacción – Revisión y Edición. SG: Conceptualización, Curación de datos, Análisis formal, Obtención de fondos, Administración de proyectos, Recursos, Supervisión, Redacción, revisión y edición. MEi: Conceptualización, Curación de datos, Análisis formal, Obtención de fondos, Metodología, Administración de proyectos, Recursos, Supervisión, Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declaran/n que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. El presente estudio contó con el apoyo de las siguientes fuentes de financiamiento: Instituto Nacional de Tecnología Agropecuaria PNSA-PDi-113, PICT 2019 04085, PICT Start Up 2019 00038 y PIP 2021-2023 11220200101912CO.
Reconocimientos
Los autores agradecen a Julia Sabio y García por la revisión inglesa del manuscrito. MEi y MZ son investigadores del Consejo Nacional de Investigaciones Científicas de Argentina (CONICET).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1359205/full#supplementary-material
FIGURA S1 | Características de las plantillas de ADN y de la TD-PCR IS6110 en bovinos falsos negativos para CF-TST. (A) Concentración y calidad del ADN. El diagrama de dispersión de la concentración (ng/μL) y la calidad (proporción A260/A280) del ADN de las muestras de sangre de bovinos falsos negativos para CF-TST (n = 70) se determinó mediante espectrofotometría. Los valores atípicos, previamente identificados por la prueba de Grubbs (GraphPad), se excluyeron del análisis estadístico. B) Plantillas de integridad del ADN obtenidas a partir de muestras de sangre bovina. Electroforesis en gel de agarosa al 0,8% teñido con bromuro de etidio para evaluar la integridad del ADN y la contaminación por ARN de las muestras (muestra de carga: 5 μL). S1-S9 se refiere a nueve ADN genómicos diferentes extraídos de muestras de sangre pertenecientes a bovinos falsos negativos de CF-TST. El «alto peso molecular» representa la región del gel que se espera que muestre ADN no alterado, como una banda densa de ADN genómico de alto peso molecular. «Degradación» muestra la región del gel en la que se visualiza la degradación del ADN genómico, observada como una banda continua y difusa, y finalmente, la «contaminación por ARN» indica el área del gel en la que se espera detectar el ARN contaminante que ha coeluido con el ADN genómico durante el proceso de extracción. (C) Amplificación de IS6110 TD-PCR en muestras de sangre bovina. Electroforesis en gel de agarosa al 2% para visualizar la amplificación de la diana IS6110 en muestras de sangre de bovinos falsos negativos para CF-TST. La flecha roja indica una banda de 245 pb correspondiente a la secuencia de amplificación IS6110. S1-S9 se refiere al ADN extraído de diferentes animales. PC, control positivo (ADN de M. bovis, cepa AN5). CC, control de la contaminación (agua libre de DNasa y RNasa); MWM: marcador de peso molecular.
Referencias
1. Reis, AC, Ramos, B, Pereira, AC, y Cunha, MV. Tendencias mundiales de la investigación epidemiológica en tuberculosis bovina durante las últimas cuatro décadas. Transbound Emerg Dis. (2021) 68:333–46. doi: 10.1111/tbed.13763
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Jenkins, AO, Gormley, E, Gcebe, N, Fosgate, GT, Conan, A, Aagaard, C, et al. Respuestas inmunitarias de reacción cruzada en bovinos derivadas de la exposición a Mycobacterium bovis y micobacterias no tuberculosas. Prev Vet Med. (2018) 152:16–22. doi: 10.1016/j.prevetmed.2018.02.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Álvarez, J, Pérez, A, Marqués, S, Bezos, J, Grau, A, de la Cruz, ML, et al. Factores de riesgo asociados a resultados diagnósticos in vivo negativos en bovinos infectados por tuberculosis bovina en España. BMC Vet Res. (2014) 10:14. doi: 10.1186/1746-6148-10-14
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Casal, C, Díez-Guerrier, A, Álvarez, J, Rodríguez-Campos, S, Mateos, A, Linscott, R, et al. Uso estratégico de la serología para el diagnóstico de tuberculosis bovina tras pruebas cutáneas intradérmicas. Microbiol veterinario. (2014) 170:342–51. doi: 10.1016/j.vetmic.2014.02.036
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Garbaccio, SG, Garro, CJ, Delgado, F, Tejada, GA, Eirin, ME, Huertas, PS, et al. Ensayo de inmunoadsorción enzimática como complemento de la prueba cutánea intradérmica para la detección de la infección por Mycobacterium bovis en bovinos. Tuberculosis. (2019) 117:56–61. doi: 10.1016/j.tube.2019.05.006
6. Griffa, N, Moyano, RD, Canal, AM, Travería, GE, Santangelo, MP, Alonso, N, et al. Desarrollo y validación diagnóstica de un ELISA basado en una mezcla antigénica para la detección de tuberculosis bovina. Veterinario J. (2020) 256:105426. doi: 10.1016/j.tvjl.2020.105426
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Houlihan, MG, Dixon, FW y Page, NA. Brote de tuberculosis bovina con anergia cutánea, lesiones de ubre y enfermedades transmitidas por la leche en terneros jóvenes. Rec. Veterinario (2008) 163:357–61. doi: 10.1136/vr.163.12.357
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Raffo, E, Steuer, P, Tomckowiack, C, Tejeda, C, Collado, B, y Salgado, M. Más información sobre el efecto interferente de la infección por Mycobacterium avium subsp. paratuberculosis (MAP) en la detección de Mycobacterium bovis (M. bovis) en el ganado lechero. Trop Anim Salud Prod. (2020) 52:1479–85. doi: 10.1007/s11250-019-02151-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Waters, WR, Buddle, BM, Vordermeier, HM, Gormley, E, Palmer, MV, Thacker, TC, et al. Desarrollo y evaluación de un ensayo de inmunoadsorción enzimática para su uso en la detección de tuberculosis bovina en bovinos. Clin Vacc Immunol. (2011) 18:1882–8. doi: 10.1128/CVI.05343-11
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Waters, WR, Vordermeier, HM, Rhodes, S, Khatri, B, Palmer, MV, Maggioli, MF, et al. Potencial de detección rápida de anticuerpos para identificar bovinos tuberculosos con resultados de pruebas cutáneas de tuberculina no reactiva. BMC Vet Res. (2017) 13:164. doi: 10.1186/s12917-017-1085-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Maggioli, MF . Una prueba contundente: ¿es frecuente la bacteriemia por Mycobacterium bovis en el ganado? Virulencia. (2016) 7:748–50. doi: 10.1080/21505594.2016.1213477
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Elsohaby, I, Mahmmod, YS, Mweu, MM, Ahmed, HA, El-Diasty, MM, Elgedawy, AA, et al. a. Precisión de los ensayos de PCR, cultivo de micobacterias y γ de interferón para la detección de Mycobacterium bovis en muestras de sangre y leche de vacas lecheras egipcias mediante modelización bayesiana. Prev Vet Med. (2020) 181:105054. doi: 10.1016/j.prevetmed.2020.105054
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Elsohaby, I, Ahmed, HA, El-Diasty, MM, Elgedawy, AA, Mahrous, E, y El Hofy, FI. Evidencia serológica y molecular de Mycobacterium bovis en ganado lechero y trabajadores de granjas lecheras bajo el sistema de producción lechera intensiva en Egipto. J Appl Microbiol. (2020) 129:1207–19. doi: 10.1111/jam.14734
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Thakur, MK, Sinha, DK y Singh, BR. Evaluación de herramientas diagnósticas complementarias para la detección de la tuberculosis bovina en rebaños lecheros de la India. Mundo Veterinario. (2016) 9:862–8. doi: 10.14202/vetworld.2016.862-868
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Brahma, D, Narang, D, Chandra, M y Singh, ST. Comparación de PCR múltiple y ordinaria para el diagnóstico de paratuberculosis y tuberculosis en muestras de sangre (capa leucocitaria) de bovinos y búfalos. Revista iraní de investigación veterinaria. (2020) 21, 52–56.
16. Cezar, RD, Lucena-Silva, N, Filho, AF, Borges, J, de, M, y de Oliveira, PR. Detección molecular de Mycobacterium bovis en rebaños bovinos del estado de Pernambuco. BMC investigación veterinaria. (2016) 12, 31. doi: 10.1186/s12917-016-0656-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Zumárraga, MJ, Meikle, V, Bernardelli, A, Abdala, A, Tarabla, H, Romano, MI, et al. Uso de la reacción en cadena de la polimerasa de aterrizaje para mejorar la sensibilidad de la detección de Mycobacterium bovis. J Vet Diagnostic Investig. (2005) 17:232–8. doi: 10.1177/104063870501700303
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Barandiaran, S, Pérez Aguirreburualde, MS, Marfil, MJ, Martínez Vivot, M, Aznar, N, Zumárraga, M, et al. Evaluación bayesiana de la precisión de una prueba de diagnóstico rápido basada en PCR para la tuberculosis bovina en cerdos. Front Vet Sci. (2019) 6:204. doi: 10.3389/fvets.2019.00204
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Marfil, MJ, Huertas, PS, Garbaccio, SG, Barandiaran, S, Martínez Vivot, M, Garro, C, et al. Detección de Mycobacterium bovis viable en pulmones e hígados vendidos en carnicerías de Buenos Aires, Argentina. Foodborne Pathog Dis. (2018) 15:758–62. doi: 10.1089/fpd.2018.2467
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Zumárraga, MJ, Soutullo, A, García, MI, Marini, R, Abdala, A, Tarabla, H, et al. Detección de rebaños lecheros infectados por Mycobacterium bovis mediante PCR en muestras de leche de tanque a granel. Foodborne Pathog Dis. (2012) 9:132–7. doi: 10.1089/fpd.2011.0963
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Roellig, DM, Gómez-Puerta, LA, Mead, DG, Pinto, J, Ancca-Juárez, J, Calderón, M, et al. Metodologías de PCR y RFLP hemianidadas para la identificación de la sangre del vector de la enfermedad de Chagas, Triatoma infestans. PLoS Uno. (2013) 8:e74713. doi: 10.1371/journal.pone.0074713
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. de Kantor, IN . Bacteriología de la Tuberculosis humana y animal. CEPANZO OPS/OMS Serie de Monografías. (1989) 11:63.
23. AAVLD. Manual de Diagnóstico de Micobacterias de Importancia en Medicina Veterinaria. Comisión Científica de Micobacterias. (2005):20–8.
24. Araújo, CP, Osório, AL, Jorge, KS, Ramos, CA, Souza Filho, AF, Vidal, CE, et al. Detección directa del complejo Mycobacterium tuberculosis en tejidos bovinos y bubalinos mediante PCR anidada. Braz J Microbiol. (2014) 45:633–40. doi: 10.1590/s1517-83822014000200035
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Software GraphPad de prueba de Grubbs, Inc. (s.f.). Disponible en: https://www.graphpad.com/quickcalcs/Grubbs1.cfm (consultado el 28 de noviembre de 2023).
26. Kock, R, Michel, AL, Yeboah-Manu, D, Azhar, EI, Torrelles, JB, Cadmus, SI, et al. Tuberculosis zoonótica: el panorama cambiante. Int J Dis. Infecciosa. (2021) 113:S68–72. doi: 10.1016/j.ijid.2021.02.091
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Pollock, JM, McNair, J, Welsh, MD, Girvin, RM, Kennedy, HE, Mackie, DP, et al. Respuestas inmunitarias en tuberculosis bovina. Tuberculosis. (2001) 81:103–7. doi: 10.1054/tube.2000.0258
28. Welsh, MD, Cunningham, RT, Corbett, DM, Girvin, RM, McNair, J, Skuce, RA, et al. Influencia de la progresión patológica en el equilibrio entre las respuestas inmunes celulares y humorales en tuberculosis bovina. Inmunología. (2005) 114:101–11. doi: 10.1111/j.1365-2567.2004.02003.x
29. Lucena-Aguilar, G, Sánchez-López, AM, Barberán-Aceituno, C, Carrillo-Ávila, JA, López-Guerrero, JA, y Aguilar-Quesada, R. Selección de fuentes de ADN para aplicaciones posteriores basadas en el análisis de indicadores de calidad del ADN. Biopreservación, Biobancos. (2016) 14:264–70. doi: 10.1089/bio.2015.0064
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Garbaccio, S, Barandiaran, S, Fernández, A, Macías, A, Magnano, G, Vivot, M, et al. Ensayo interlaboratorio: aislamiento de Mycobacterium bovis a partir de lesiones granulomatosas en bovinos. Revista Argentina de Microbiología. (2016) 48:161–5. doi: 10.1016/j.ram.2016.03.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Menin, Á, Fleith, R, Reck, C, Marlow, M, Fernandes, P, Pilati, C, et al. Los bovinos asintomáticos infectados naturalmente con Mycobacterium bovis presentan patología tisular exacerbada y diseminación bacteriana. PLoS Uno. (2013) 8:e53884. doi: 10.1371/journal.pone.0053884
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: tuberculosis bovina, anergia, diagnóstico, IS6110 touchdown-PCR, sangre, falso negativo, prueba cutánea de tuberculina
Cita: Encinas M, Ferrara Muñiz X, Sammarruco RA, Ruiz Menna V, Garro CJ, Delgado F, Macías A, Magnano G, Zumárraga MJ, Garbaccio SG y Eirin ME (2024) Utilidad limitada de la IS6110 touchdown-PCR en sangre para bovinos falsos negativos de prueba cutánea de tuberculina con respuesta serológica a Mycobacterium bovis. Frente. Vet. Sci. 11:1359205. doi: 10.3389/fvets.2024.1359205
Recibido: 21 de diciembre de 2023; Aceptado: 23 de abril de 2024;
Publicado: 20 de mayo de 2024.
Editado por:
Francisco Javier Salguero, Agencia de Seguridad Sanitaria del Reino Unido (UKHSA), Reino Unido
Revisado por:
Gobena Ameni, Universidad de los Emiratos Árabes Unidos, Emiratos Árabes
Unidos Aman Ullah Khan, Universidad de Ciencias Veterinarias y Animales, Pakistán
Copyright © 2024 Encinas, Ferrara Muñiz, Sammarruco, Ruiz Menna, Garro, Delgado, Macías, Magnano, Zumárraga, Garbaccio y Eirin. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Martín José Zumárraga, zumarraga.martin@inta.gob.ar
†Estos autores han contribuido por igual a este trabajo y comparten la última autoría
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.