Detección molecular de los genes mcr-1 y mcr-5 en Enterobacteriaceae de aves de corral

La aparición y detección molecular de los genes mcr-1 y mcr-5 en Enterobacteriaceae aislados de aves de corral y carnes de aves de corral en Malasia
 Md Rezaul Karim1,2
Md Rezaul Karim1,2  Zunita Zakaria1,3*
Zunita Zakaria1,3*  Latiffah Hassan4
Latiffah Hassan4  Nik Mohd Faiz5
Nik Mohd Faiz5  Nur Indah Ahmad1
Nur Indah Ahmad1- 1Departamento de Patología Veterinaria y Microbiología, Facultad de Medicina Veterinaria, Universiti Putra Malaysia, UPM, Serdang, Selangor, Malasia
- 2Instituto de Investigación Ganadera de Bangladesh, Dhaka, Bangladesh
- 3Instituto de Biociencias, Universiti Putra Malaysia, UPM, Serdang, Selangor, Malasia
- 4Departamento de Diagnóstico de Laboratorio Veterinario, Facultad de Medicina Veterinaria, Universiti Putra Malaysia, UPM, Serdang, Selangor, Malasia
- 5Departamento de Estudios Clínicos Veterinarios, Facultad de Medicina Veterinaria, Universiti Putra Malaysia, UPM, Serdang, Selangor, Malasia
El advenimiento de los antimicrobianos resistentes (RAM), incluidas las bacterias resistentes a la colistina, plantea un desafío significativo para la salud animal y humana, la inocuidad de los alimentos, el crecimiento socioeconómico y el medio ambiente mundial. Este estudio tuvo como objetivo determinar la prevalencia de resistencia a la colistina y los mecanismos moleculares de la resistencia a la colistina en Enterobacteriaceae. La resistencia a la colistina se determinó mediante ensayo de microdilución en caldo, PCR; y secuenciación de Sanger de genes mcr responsables de la resistencia a la colistina en Enterobacteriaceae (n = 627), incluyendo Escherichia coli (436), Salmonella spp. (n = 140) y Klebsiella pneumoniae (n = 51), obtenida de pollo y carnes de pollo. De 627 Enterobacteriaceae, el 8,6% de los aislados exhibieron resistencia a la colistina fenotípicamente. Entre estos aislados resistentes a la colistina, el 9,3% (n = 37) se aisló de la carne de pollo, el 7,2% (n = 11) del hisopo cloacal de pollo y el 7,9% (n = 6) de las muestras de camada. En general, el 12,96% de los aislados resistentes a la colistina fueron positivos con los genes mcr, en los que los genes mcr-1 y mcr-5 se determinaron en el 11,11% y el 1,85% de los aislados resistentes a la colistina, respectivamente. Los aislados de E. coli obtenidos de carnes de pollo, hisopos cloacales y muestras de cama se encontraron positivos para mcr-1, y Salmonella spp. originada a partir de la muestra de carne de pollo se observó con mcr-5, mientras que no se observaron genes mcr en cepas de K. pneumoniae aisladas de ninguna de las muestras recolectadas. Los otros genes de resistencia a la colistina, incluidos mcr-2, mcr-3, mcr-4, mcr-6, mcr-7, mcr-8, mcr-9 y mcr-10, no se detectaron en las muestras estudiadas. Los genes mcr-1 y mcr-5 fueron secuenciados y se encontró que eran 100% idénticos a las secuencias de genes mcr-1 y mcr-5 disponibles en la base de datos del NCBI. Este es el primer informe de resistencia a la colistina mcr-5 gen en Malasia que podría presagiar la aparición de mcr-5 que albergan cepas bacterianas para la infección. Se necesitan más estudios para caracterizar la bacteria mr-5 que alberga para la determinación del plásmido asociado con el gen mcr-5.
Introducción
Los agentes antimicrobianos son medicamentos esenciales en animales y humanos para frenar las infecciones. Debido al uso excesivo y el abuso de agentes antimicrobianos, el mundo se enfrenta a la rápida proliferación de microbios resistentes. Actualmente, el advenimiento de bacterias resistentes a los antimicrobianos (AMR) plantea un desafío significativo para la salud animal y humana, la seguridad alimentaria, el crecimiento socioeconómico y el medio ambiente global (Theuretzbacher, 2017). La proliferación de cepas bacterianas gramnegativas resistentes a múltiples fármacos, y la ausencia de nuevos fármacos para combatir dichos microbios, reintrodujeron la colistina como terapia de última línea (Ezadi et al., 2019).
La resistencia a la colistina es un problema crucial que debe abordarse hoy en día. Numerosos estudios han demostrado que esta resistencia a la colistina estaba presente en varias cepas bacterianas de todo el mundo. Los genes mcr mediados por plásmidos son responsables de una resistencia excepcional a la colistina, ya que es un conducto que se propaga a través de la transmisión horizontal de una cepa bacteriana a otra y a través de la cadena alimentaria o el contacto directo con humanos, animales y entornos (Gharaibeh y Shatnawi, 2019). Antes de 2015, toda la resistencia a la colistina documentada estaba regulada cromosómicamente, lo que implica la modificación de una estructura reguladora de dos componentes, phoPQ y pmrAB con el regulador negativo, la alteración del gen mgrB (Cannatelli et al., 2013; Liu et al., 2016). En 2016, las cepas de Escherichia coli aisladas de humanos, carne de pollo y cerdo al por menor, y cepas de Klebsiella pneumoniae aisladas de humanos se informaron con el gen mcr-1 codificado por plásmidos en China (Liu et al., 2016). Los nuevos genes mcr mediados por plásmidos han surgido rápidamente. La efectividad terapéutica de la colistina se ha visto comprometida por el advenimiento de los genes mcr codificados por plásmidos, incluidos mcr-1 a mcr-10, que se informaron durante los últimos cuatro años (Ling et al., 2020). Los aislados de E. coli recuperados del hígado de pollo y el alimento para pollos en el comedero (Yu et al., 2016), y la carne de aves de corral en Malasia resultaron positivos para el gen mcr-1 (Aklilu y Raman, 2020).
La carne de aves de corral es una fuente importante de proteínas para los seres humanos, también podría ser un conducto importante para la propagación de especies bacterianas resistentes a múltiples fármacos de animales productores de alimentos a los seres humanos. Estudios anteriores han demostrado que la carne de pollo al por menor desempeña un papel en la diseminación de múltiples cepas resistentes a los antibióticos entre los humanos y su entorno, lo que representa una grave amenaza para la salud ambiental y la seguridad alimentaria (Aidara-Kane et al., 2013). El gen productor de resistencia a la colistina, mcr-1, estaba presente en el 52.1 por ciento de los aislados de E. coli de la carne cruda de pollo (Aklilu y Raman, 2020). Se encontró resistencia a la colistina en más del 36,4% de las bacterias del pollo de aves de corral y en el 20% de las cepas aisladas de pollo nativo en Bangladesh (Islam et al., 2020). En Nepal, se informó que 27 (22,8%) de E. coli resistentes a la colistina en granjas de pollos de engorde portaban el gen mcr-1 (Joshi et al., 2019). Aparte de eso, se cree que la transmisión horizontal es el principal mecanismo para la propagación de genes mcr de resistencia a la colistina en Enterobacteriaceae en todo el mundo (Gharaibeh y Shatnawi, 2019).
Con potentes tasas de transferencia in vitro y frecuentemente albergadas junto con otros determinantes de resistencia como β -lactamasas, se han identificado genes mcr en una variedad de plásmidos conjugativos (IncI2, IncHI2, IncX4 y pHNSHP45) (EMA (European Medicine Agencey), 2016). Dado que los genes movilizados de resistencia a la colistina (mcr) impulsados por plásmidos están surgiendo rápidamente, se requiere información adecuada sobre la resistencia a la colistina, incluida la incidencia y los estudios epidemiológicos de los casos positivos para MCR, a fin de aplicar medidas para prevenir y gestionar su diseminación. Por lo tanto, el objetivo del estudio fue determinar la prevalencia y los determinantes moleculares subyacentes a la resistencia a la colistina en aislados de Enterobacteriaceae (E. coli, Salmonella spp. y Klebsiella pneumoniae) recuperados de aves de corral y carnes de aves de corral en Malasia.
Materiales y métodos
Aprobación ética
El comité de ética de la Universiti Putra Malaysia (UPM), Institutional Animal Care and Use Committee (IACUC) aprobó el protocolo de estudio de investigación para recolectar hisopos cloacales de aves de corral vivas (UPM / IACUC / AUP-R091 / 2019).
Diseño del estudio y muestras
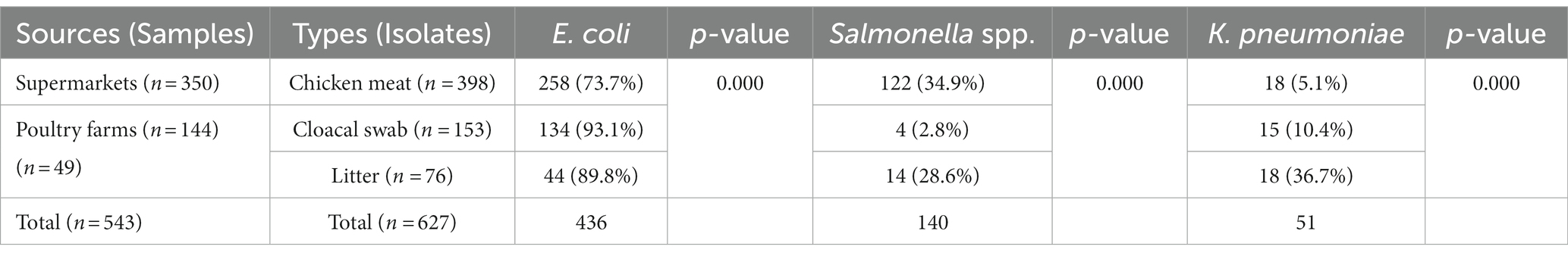
El estudio de investigación se realizó en el que se recolectaron muestras de carne de pollo de supermercados e hisopos cloacales, y muestras de basura de granjas de pollos en Selangor, Malasia (Figura 1) desde julio de 2019 hasta febrero de 2021. Selangor es el área densamente poblada en Malasia y la mayoría de las granjas avícolas se encuentran en este estado. Se utilizaron bolsas de plástico estériles para recoger las muestras de carne. Las muestras de hisopo cloacal se recolectaron asépticamente del pollo sano y se mantuvieron en medios de transporte estériles, medios Stuart. Las muestras de basura se recogieron de los pisos de las granjas con una cuchara estéril y se colocaron en una bolsa de plástico estéril. En total, se recolectaron 543 muestras, incluidas 350 carnes de pollo, 144 hisopos cloacales y 49 muestras de basura (Tabla 1), de supermercados y granjas avícolas en diferentes áreas de Selangor en Malasia (Figura 1). Todas las muestras recolectadas se transportaron inmediatamente en una nevera sellada al Laboratorio de Bacteriología, Facultad de Medicina Veterinaria de la Universidad Putra Malasia (UPM), Serdang, Selangor, Malasia.
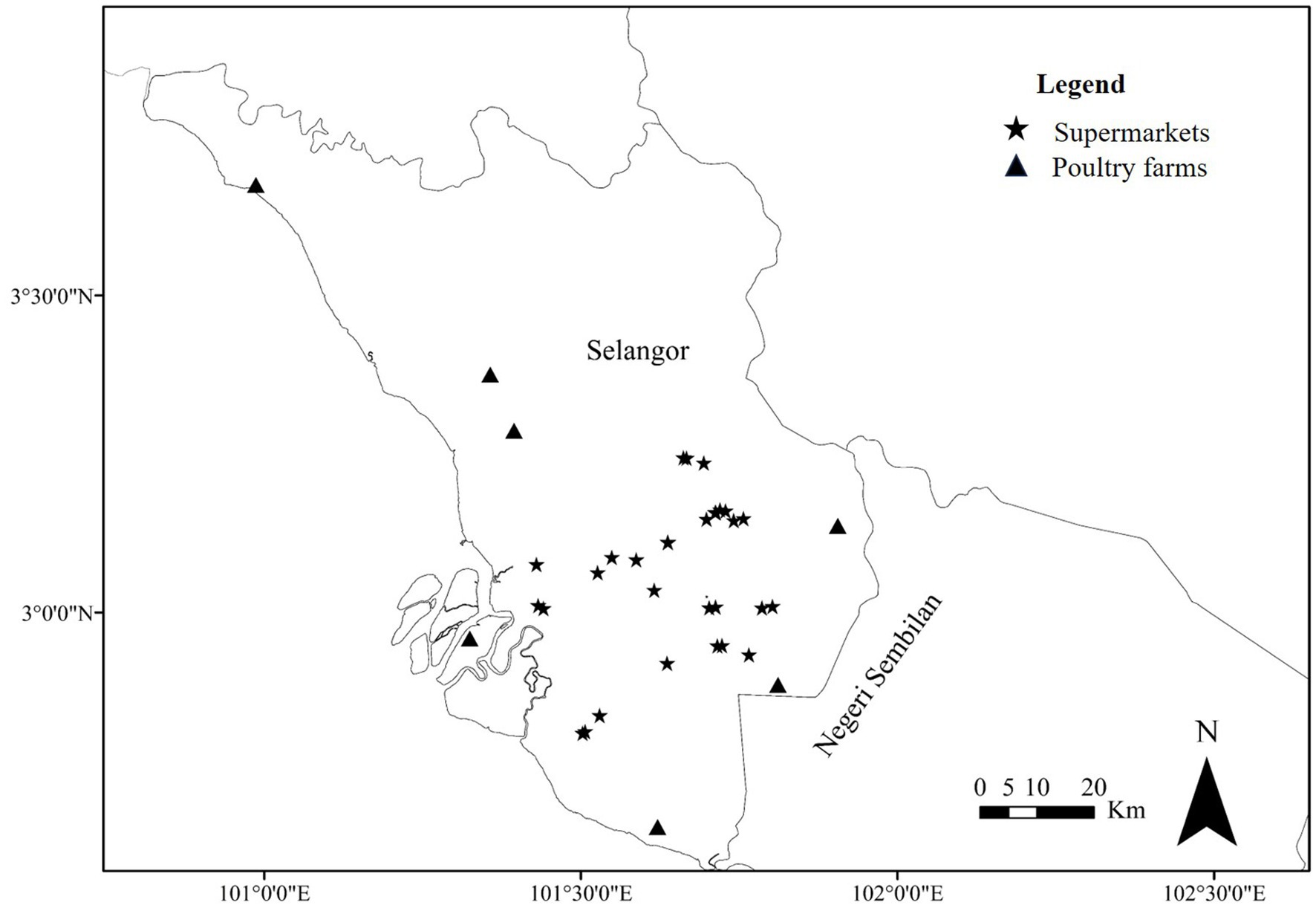 Figura 1. Mapa que muestra las áreas de muestreo de Selangor, Malasia. Marcas de estrella (*) y triángulo (▲) que indican supermercados y granjas avícolas, respectivamente. Se utilizó el software de información geográfica, ArcGIS v.10.4.1 for Windows para generar el punto de muestreo en el mapa.
Figura 1. Mapa que muestra las áreas de muestreo de Selangor, Malasia. Marcas de estrella (*) y triángulo (▲) que indican supermercados y granjas avícolas, respectivamente. Se utilizó el software de información geográfica, ArcGIS v.10.4.1 for Windows para generar el punto de muestreo en el mapa.
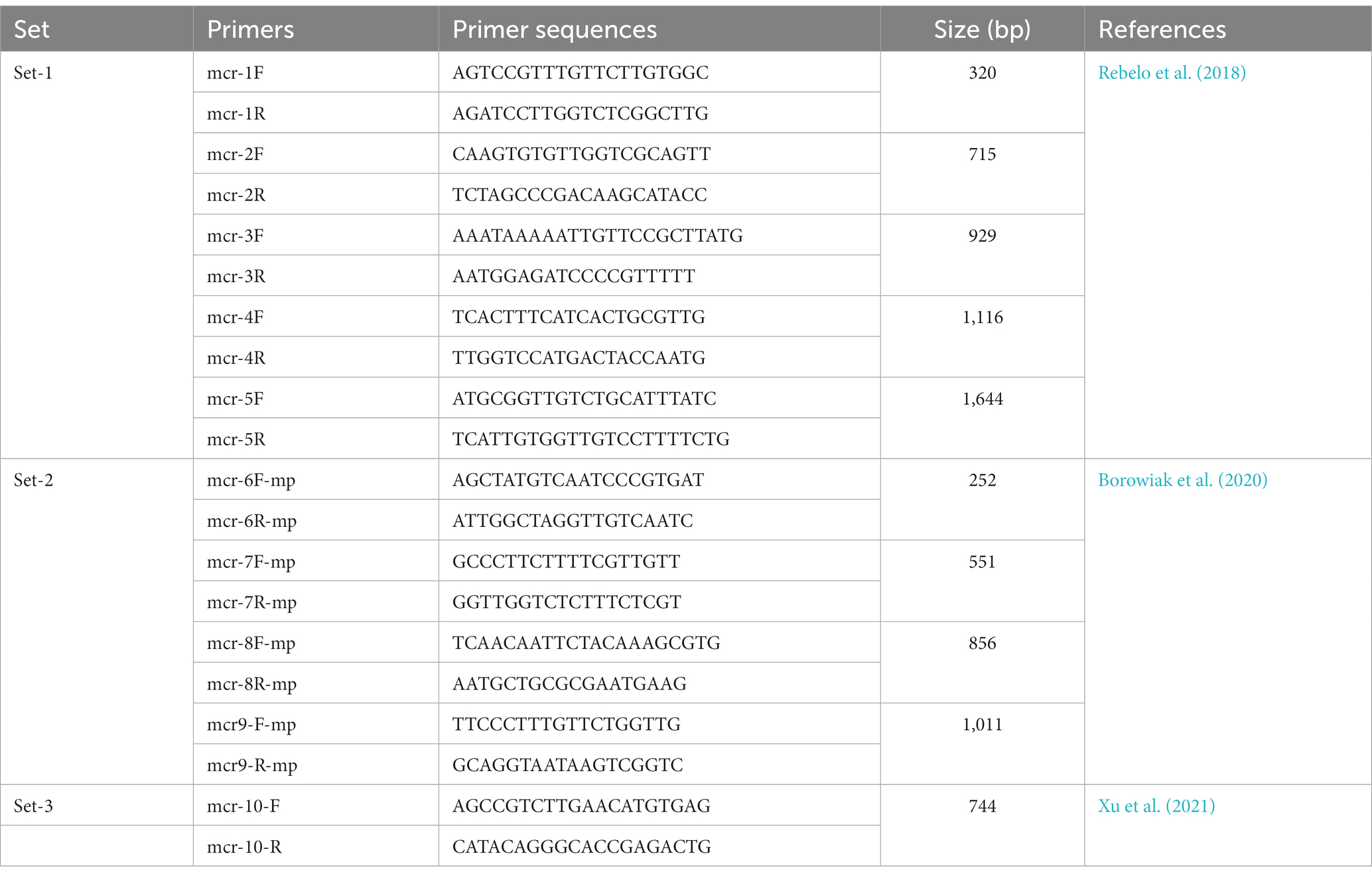
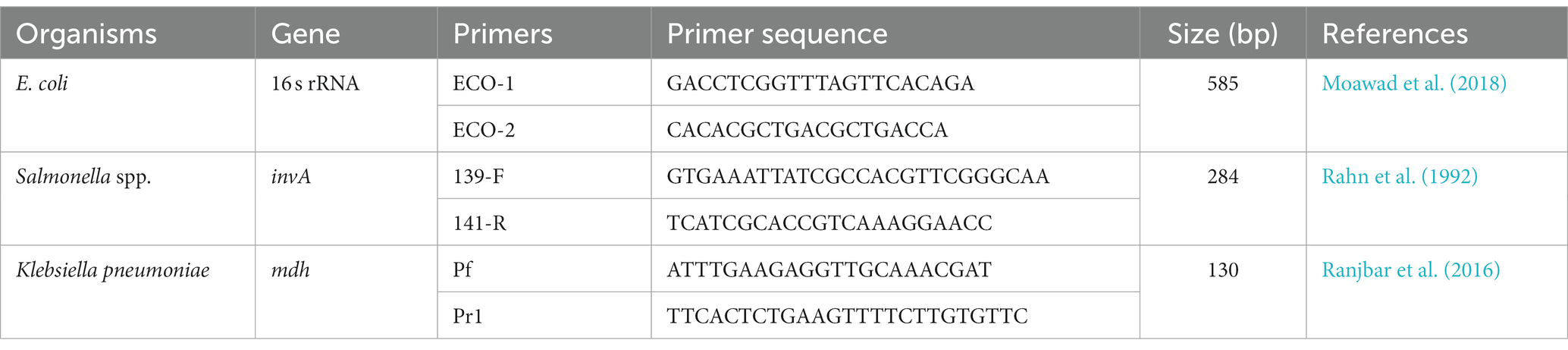 Tabla 1. Lista de los cebadores y secuencias para la confirmación de E. coli, Salmonella spp., y Klebsiella pneumoniae.
Tabla 1. Lista de los cebadores y secuencias para la confirmación de E. coli, Salmonella spp., y Klebsiella pneumoniae.
Aislamiento e identificación de cepas bacterianas
En la familia Enterobacteriaceae, Salmonella spp., E. coli y K. pneumoniae tienen importancia para la salud pública y son más prevalentes con genes mcr de resistencia a la colistina (Elbediwi et al., 2019). Por esta razón, Salmonella spp., E. coli y K. pneumoniae, se aislaron e identificaron fenotípicamente utilizando el protocolo estándar de pruebas culturales y bioquímicas tradicionales (Ghafur et al., 2019; Sharma et al., 2019). En la etapa previa al enriquecimiento, las muestras se cultivaron en agua tamponada de peptona (BPW, Oxoid, Reino Unido), incluyendo 25 g de carne en 225 mL, 10 g de cama en 90 mL e hisopos cloacales en medio BPW de 10 mL, las muestras se incubaron a 37°C durante 24 h. Las muestras de carne con BPW se homogeneizaron durante 2 min en un estómago. Para el aislamiento de Salmonella spp., se mezclaron 100 μL de BPW homogeneizado con 10 mL de Rappaport-Vassiliadis (RVS; Oxoid, Reino Unido). Luego las mezclas de RVS se incubaron a 42°C durante 24 h. Un RVS lleno de bucle de cada muestra se subcultivó en agar xilosa lisina desoxicolato (XLD; Oxoid, Reino Unido) y mantenido a 37°C durante 24 h para incubación. En XLD, típicamente, las colonias de Salmonella eran rojas con un centro negro. Una colonia pura de XLD se cultivó en agar nutriente (NA, Oxoid, Reino Unido). Para la identificación presuntiva, se realizaron pruebas bioquímicas (TSI, ureasa, citrato y SIM) con cultivos puros cultivados en NA. La confirmación serológica de Salmonella spp. se realizó mediante una prueba de aglutinación en portaobjetos utilizando antisueros Poly ‘O’ y ‘H’ (Remel, Reino Unido). Para el aislamiento de E. coli y K. pneumoniae, se inoculó un bucle de suspensión de BPW en MacConkey (Oxoid, Reino Unido). Una presunta colonia de E. coli y K. pneumoniae se subcultivó en NA y se sometió a una prueba bioquímica estándar para su presunta confirmación.
Confirmación molecular de Escherichia coli, Salmonella spp. y Klebsiella pneumoniae
El ADN genómico se extrajo del cultivo puro de los aislados cultivados en agar nutriente utilizando el método de ebullición y enfriamiento rápido (Pui et al., 2011). Las especies fenotípicamente positivas de E. coli, Salmonella spp. y Klebsiella pneumoniae se confirmaron con PCR convencional con cebadores genéticos específicos de la especie (Tabla 1), en los que los controles positivos incluyeron E. coli ATCC 25922, Salmonella ATCC 14028 y Klebsiella pneumoniae ATCC 700603 (Rahn et al., 1992; Ranjbar et al., 2016; Moawad et al., 2018).
Evaluación de la susceptibilidad a la colistina y la concentración inhibitoria mínima (CMI)
La susceptibilidad a la colistina y la CMI en aislados se evaluaron mediante la técnica de microdilución en caldo (DMO) estándar ISO-20776 recomendada conjuntamente por EUCAST (2016a) con pocas modificaciones. En resumen, se preparó una dilución doble (0.125-128 μg / ml) de la sal de sulfato de colistina (Sigma-Aldrich) en una placa de microtitulación de 96 pocillos. El inóculo bacteriano se inoculó en cada pocillo con una concentración final de 5 × 105 UFC/ml. A continuación, la placa de microtitulación se incubó a 37°C durante 16-20 h. Después del período de incubación, se agregaron 30 μL de solución de resazurina al 0,015% a cada pocillo de la placa de microtitulación y se incubaron nuevamente a 37C durante 1 h, durante la cual la placa se verificó rutinariamente cada 15 min. Las CMI de colistina se interpretaron a simple vista y se registraron observando el cambio de color de la resazurina (la decoloración de azul a rosa o púrpura indica resistencia a la colistina, mientras que la susceptibilidad se deduce cuando no cambia de color: color azul). La prueba se realizó por triplicados. Durante las pruebas de susceptibilidad a la colistina, se utilizaron E. coli ATCC25922 susceptible a la colistina y E. coli resistente a la colistina (ColR) NCTC 13846 como controles negativos y positivos, respectivamente.
Detección de los determinantes de resistencia a la colistina, genes mcr
Se identificaron aislados de Enterobacteriaceae resistentes a la colistina con valores de CMI superiores a 2 μg/ml de colistina (EUCAST, 2016b). El ADN genómico de los aislados resistentes a la colistina (col-R) se evaluó con PCR convencional para detectar variantes del gen de resistencia a la colistina (mcr-1 a mcr-10). Según estudios previos, la detección de mcr-1 a mcr-5 (Rebelo et al., 2018) y mcr-6 a mcr-9 (Borowiak et al., 2020) se realizó con PCR múltiple. La PCR uniplex se realizó para mcr-10 (Xu et al., 2021) mediante los cebadores de oligonucleótidos previamente diseñados y el protocolo (Tabla 2).
Análisis de resultados de PCR
Los productos de PCR amplificada se examinaron en agarosa al 1,5% (Conda, Madrid, España) preparada en 100 mL de tampón TBE de 0,5 × teñido con 4 μL de tinción de ácido nucleico (ETB «out» Nucleic Acid, Cat. No. FYD007-200P, Yestern Biotech Co. ltd, Taiwán) para el gen mcr. Las bandas esperadas para mcr-1 a mcr-10 (Tabla 2) se visualizaron y fotografiaron bajo luz UV utilizando AlphaImager 2.200 (AlphaImager, Estados Unidos).
Confirmación del gen mcr (mcr-1 y mcr-5) mediante secuenciación
Los productos de PCR amplificados para el gen mcr-1 de las cepas de E. coli, E. coli E172 y el gen mcr-5 de una cepa S283 de Salmonella spp. se enviaron a una empresa comercial para la secuenciación de ADN con los mismos cebadores para la secuenciación de Sanger (Tabla 2) y se compararon con las secuencias mcr-1 y mcr-5 previamente informadas en la base de datos NCBI. Las secuencias de consenso de mcr-1 y mcr-5 se obtuvieron en base a la alineación de las secuencias hacia adelante y hacia atrás utilizando el programa BioEdit v. 7.2 (Hall, 1999).
Análisis filogenético
Las secuencias del gen mcr se utilizaron en la construcción del árbol filogenético, y el análisis se llevó a cabo de acuerdo con Li et al. (2019). Las secuencias de este estudio y las de GenBank para mcr-1 y mcr-5 se alinearon por separado utilizando el software MEGA X (Kumar et al., 2018) para comparar sus similitudes. Hay cuatro tipos diferentes de variantes del gen mcr-5 como mcr-5.1, mcr-5.2, mcr-5.3 y mcr-5.4 que se ha identificado en el mundo. Hemos seleccionado secuencias parciales de estas cuatro variantes mcr-5 de GenBank en la base de datos NCBI y analizado con nuestros datos de secuencia mcr-5. El árbol filogenético para mcr-1 y mcr-5 se construyó con secuencias alineadas por el método de unión vecina utilizando el modelo de 2 parámetros de Kimura, y los valores de Bootstrap se calcularon utilizando 500 réplicas.
Análisis estadístico
Se utilizaron hojas de Microsoft Excel (MS-2019) para ingresar datos, que luego se cargaron en el programa SPSS v. 25.0 (IBM, Armonk, NY, Estados Unidos). Se utilizó el análisis descriptivo para cuantificar la prevalencia, en el que se evaluó el nivel de significancia mediante el χ2 prueba. Para la significación estadística, se tomaron en consideración valores de p inferiores a 0,05 (p < 0,05).
Resultados
Frecuencia de aislados de Enterobacteriaceae
En total, se aislaron e identificaron 627 cepas de Enterobacteriaceae (E. coli, Salmonella spp. y K. pneumoniae), incluidos 398 aislados de muestras de carne de supermercados, 153 aislados de hisopos cloacales y 76 aislados de muestras de basura de granjas avícolas (Tabla 3). Los 398 aislamientos de muestras de carne de supermercados se clasificaron como E. coli (n = 258, 73,7%), Salmonella spp. (n = 122, 34,9%) y K. pneumoniae (n = 18, 5,1%). Los hisopos cloacales de aves de corral produjeron E. coli (n = 134, 93,1%), Salmonella spp. (n = 4, 2,8%) y K. pneumoniae (n = 15, 10,4%), y las muestras de hojarasca de granjas avícolas generaron E. coli (n = 44, 89,8%), Salmonella spp. (n = 14, 28,6%) y K. pneumoniae (n = 18, 36,7%). Salmonella spp. fue altamente prevalente en muestras de carne de pollo recolectadas (p = 0.000), E. coli en hisopo cloacal (p = 0.000) y K. pneumoniae fue en muestras de cama (p = 0.000) (Tabla 3). Los aislados de E. coli, Salmonella spp. y K. pneumoniae se confirmaron mediante PCR mostrando un tamaño de banda de 585 pb, 284 pb y 130 pb, respectivamente.
Determinación de la susceptibilidad fenotípica a la colistina
De los 627 aislados de Enterobacteriaceae, el 8,6% (n = 54) de los aislados mostraron resistencia a la colistina utilizando el ensayo de microdilución en caldo. Entre estos, 9,3% (n = 37) fueron aislados de carne de pollo, 7,2% (n = 11) del hisopo cloacal de pollo y 7,9% (n = 6) de las muestras de camada (Tabla 4). En general, la resistencia fenotípica a la colistina de los aislados de carne de pollo, hisopos cloacales y muestras de cama fue indiferente (Tablas 4, p = 0,712). Por otro lado, 54 aislados de Enterobacteriaceae resistentes a colistina estaban compuestos por E. coli (n = 32, 7,34%), Salmonella spp. (n = 16, 11,4%) y K. pneumoniae (n = 6, 11,76%) (Figura 2).
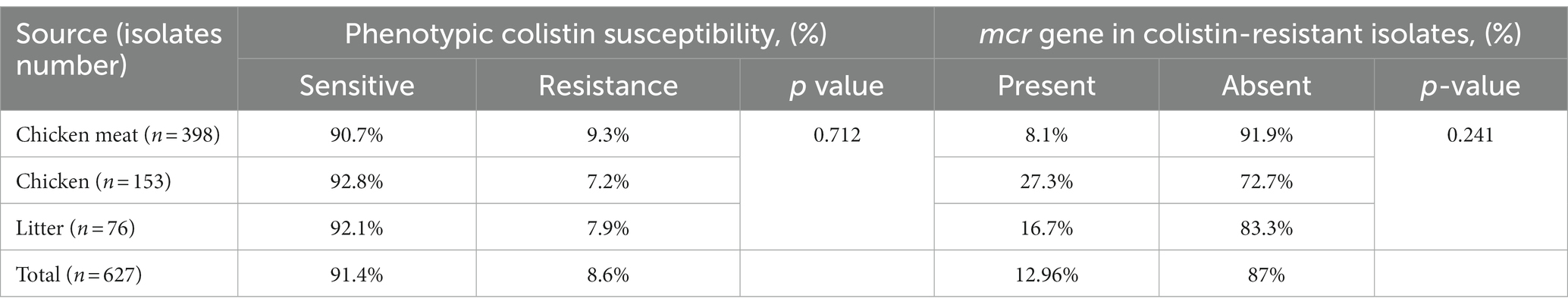 Tabla 4. Resistencia fenotípica a la colistina y mecanismo de resistencia a la colistina con genes mcr en aislados de carne de pollo, pollo y cama.
Tabla 4. Resistencia fenotípica a la colistina y mecanismo de resistencia a la colistina con genes mcr en aislados de carne de pollo, pollo y cama.
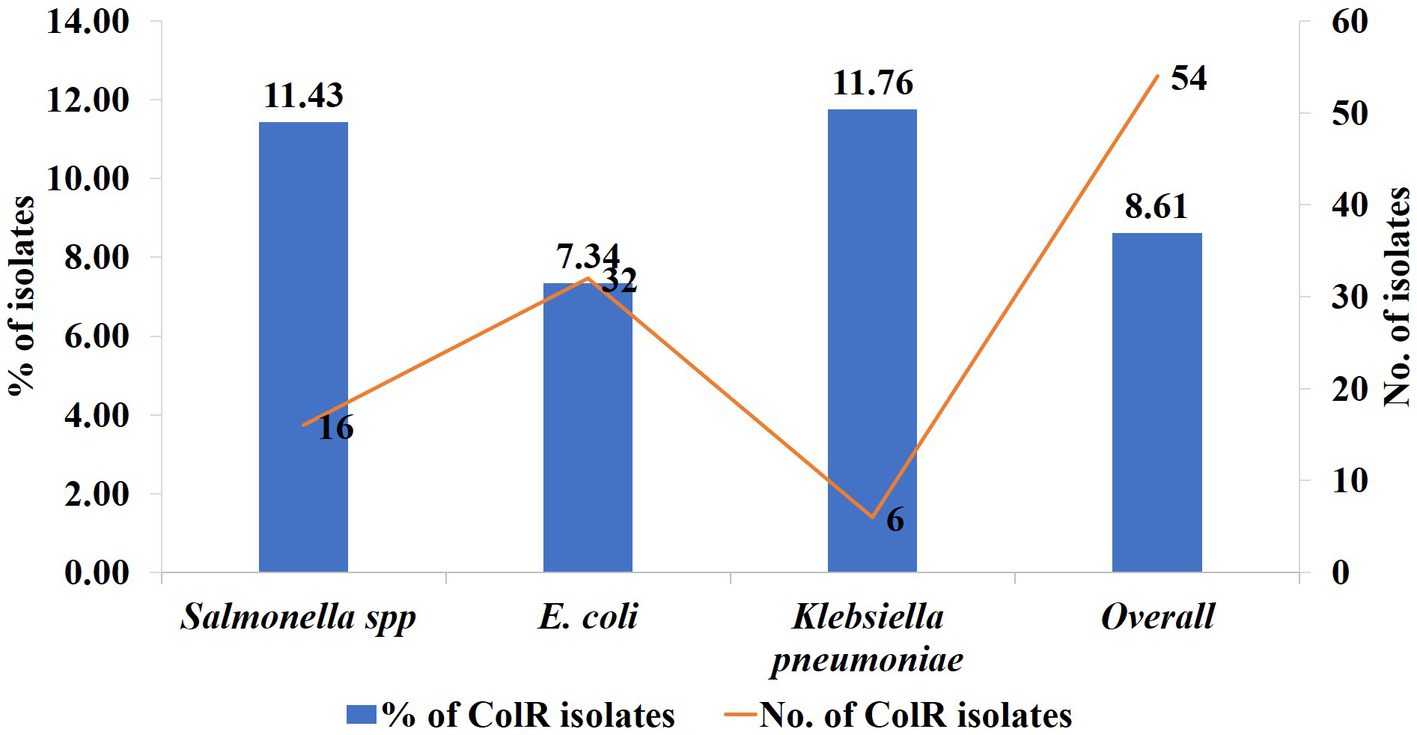 Figura 2. La tendencia de la tasa y el número de aislados de Enterobacteriaceae fenotípicamente resistentes a la colistina, E. coli, Salmonella spp. y K. pneumoniae. La columna azul = tendencia de la tasa y la línea roja = número de aislados, eje y izquierdo = % de aislados colR, eje y derecho = número de aislados colR.
Figura 2. La tendencia de la tasa y el número de aislados de Enterobacteriaceae fenotípicamente resistentes a la colistina, E. coli, Salmonella spp. y K. pneumoniae. La columna azul = tendencia de la tasa y la línea roja = número de aislados, eje y izquierdo = % de aislados colR, eje y derecho = número de aislados colR.
Detección de determinantes de resistencia a colistina, variantes del gen mcr
Se analizaron las 54 Enterobacteriaceae aisladas resistentes a la colistina (Col-R) para observar la presencia de mcr-1 a mcr-10. En general, se encontró que el 12,96% (n = 7) de los aislados de Enterobacteriaceae resistentes a la colistina poseían genes mcr de resistencia a la colistina que comprenden el 8,1% (n = 3), el 27,3% (n = 3) y el 16,67% (n = 1) de los aislados de Col-R de las muestras de carne de pollo, pollo y cama, respectivamente. Las variaciones de la resistencia a la colistina de diferentes fuentes no fueron estadísticamente significativas (valor de p > 0,05) como se muestra en la Tabla 4. De siete mcr que albergaban aislados de Col-R, 11,11% (n = 6) y 1,85% (n = 1) se encontraron con mcr-1 y mcr-5, respectivamente. Los aislados de E. coli obtenidos de carnes de pollo, hisopos cloacales y muestras de cama resultaron positivos para mcr-1, y Salmonella spp. originada a partir de la muestra de carne de pollo se observó con mcr-5, mientras que no se observaron genes mcr en cepas de K. pneumoniae aisladas de ninguna de las muestras recolectadas (Figura 3). Los otros genes de resistencia a la colistina, incluidos mcr-2, mcr-3, mcr-4, mcr-6, mcr-7, mcr-8, mcr-9 y mcr-10, no se detectaron en las muestras estudiadas.
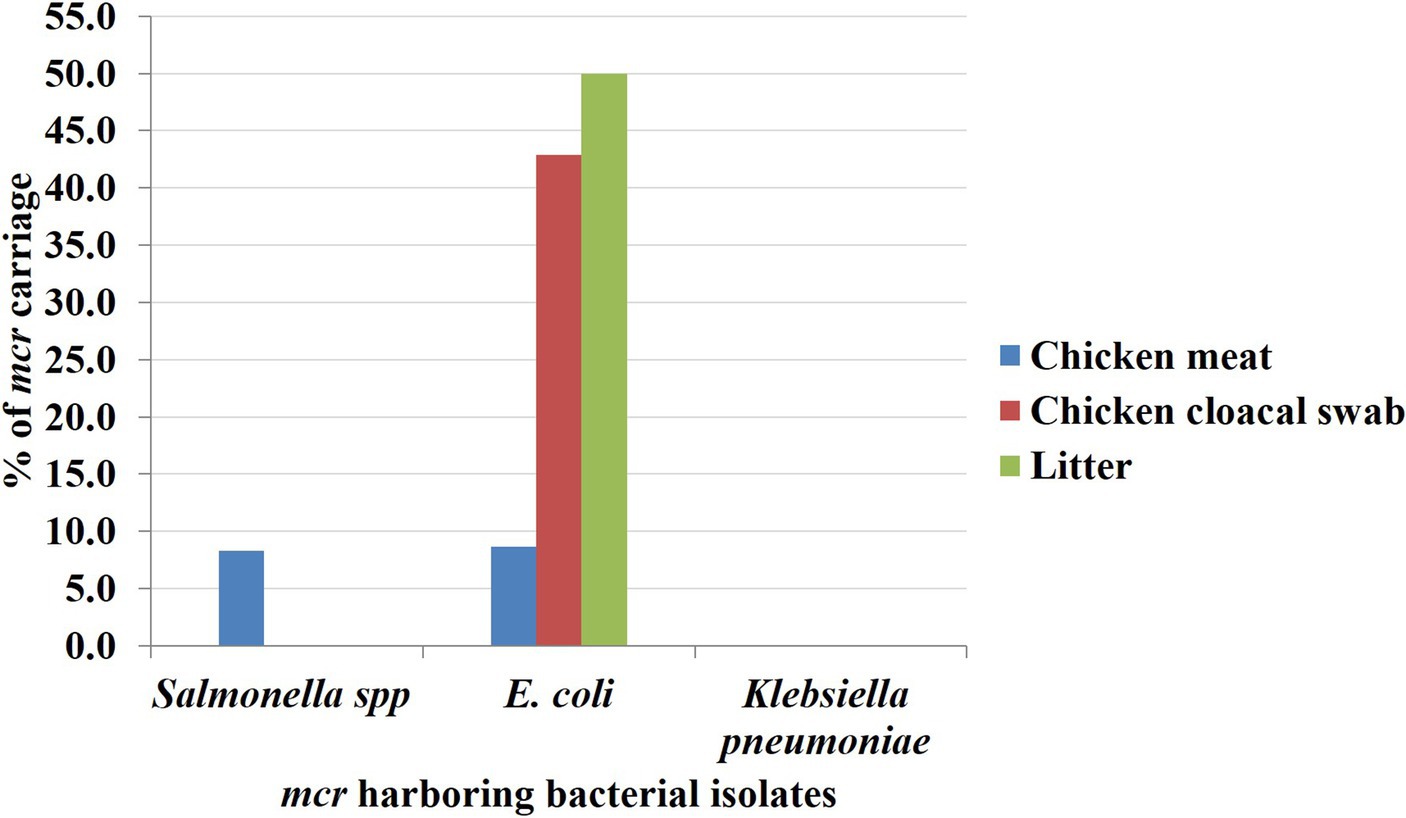 Figura 3. La tasa de Enterobacteriaceae aislados que albergan el gen mcr basado en fuentes de muestra. La tasa de genes mcr se caracterizó por barras azules, rojas y verdes en el eje Y para muestras de carne de pollo, hisopo cloacal de pollo y basura, respectivamente.
Figura 3. La tasa de Enterobacteriaceae aislados que albergan el gen mcr basado en fuentes de muestra. La tasa de genes mcr se caracterizó por barras azules, rojas y verdes en el eje Y para muestras de carne de pollo, hisopo cloacal de pollo y basura, respectivamente.
Confirmación de variantes del gen mcr
En el análisis BLAST, se encontró que nuestra secuencia ¬mcr-1 estudiada era 100% idéntica a la secuencia mcr-1 (Genbank: NG_050417.1) en la base de datos NCBI con una cobertura de consulta del 99%. Por otro lado, se encontró que nuestra secuencia ¬mcr-5 estudiada era 100% idéntica a la secuencia mcr-5 (Genbank: NG_055658) en la base de datos NCBI con una cobertura de consulta del 98%.
Determinación de la concentración inhibitoria mínima
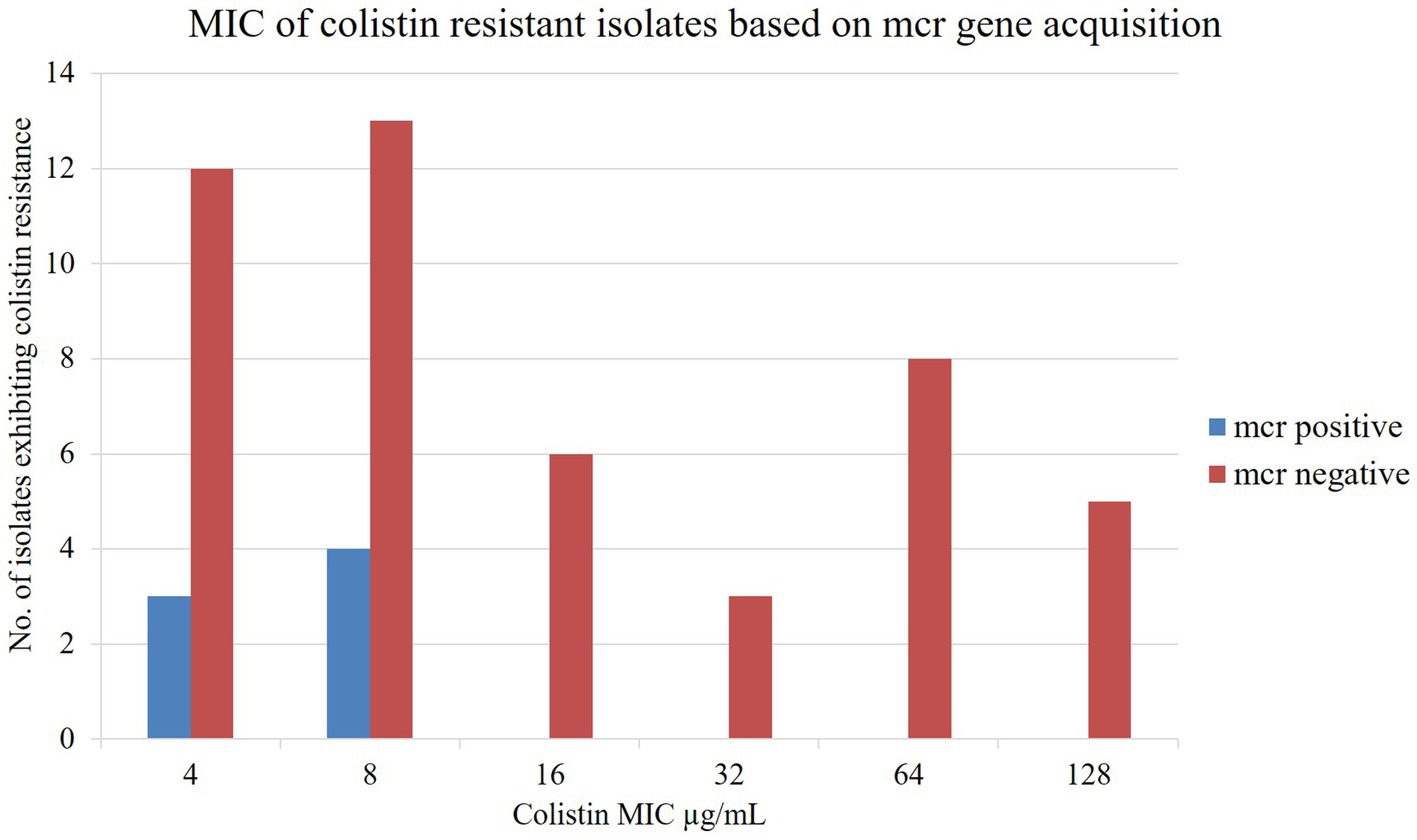
La prueba de microdilución en caldo se realizó para evaluar el valor de CMI de todos los aislados siguiendo las directrices EUCAST, con el valor de corte epidemiológico >2 μg/ml para la resistencia a la colistina. Las cepas control, aislados resistentes a la colistina Escherichia coli NTCC 13846 y aislados susceptibles de Escherichia coli ATCC 25922, mostraron un crecimiento de hasta 4 μg/mL y 0,5 μg/mL de concentración de colistina, respectivamente. El valor de CMI de bacterias aisladas resistentes a la colistina exhibió de 4 a 128 μg/ml de colistina. Los aislados portadores de mcr se observaron con valores de CMI de 4 y 8 μg/ml de colistina (Figura 4). En contraste, los aislados resistentes a la colistina mcr negativos tenían niveles extremadamente altos de CMI 128 μg/ml de colistina (Figura 4). La mayoría de los aislados de Col-R de tres fuentes exhibieron valores de CMI de 4 a 8 μg/ml de colistina (Figura suplementaria S1).
Comparación filogenética
En el análisis de filogenia, se observa que los genes mcr-1 en los aislados obtenidos se dividieron en dos clados, llamados clado A, incluyendo subclado I y subclado II, y clado B. La secuencia del gen mcr-1 de resistencia a la colistina de las cepas E48, E13, E278, E297, E331 recuperada de hisopos cloacales y muestras de carne de pollo se agrupó en el subclado I. Estos aislamientos estaban estrechamente relacionados con el subclado II, agrupado con cepas de E. coli y Salmonella spp. recuperadas de humanos y animales obtenidas de la base de datos GenBank del NCBI. La secuencia de nucleótidos del gen mcr-1 de E. coli E172 obtenida de muestras de cama de pollo fue muy cercana al aislado previamente identificado en Malasia y al aislado humano en China. Por otro lado, las secuencias mcr-1 de aislados de E. coli recuperados de carnes de pollo y muestras de hisopos cloacales de pollo se agruparon en un grupo y se encontraron cerca de aislados de China y Brasil (Figura 5).
 Figura 5. Árbol filogenético del gen mcr-1 que muestra la relación de las regiones codificantes parciales de las secuencias del gen mcr-1. Las secuencias estudiadas fueron marcadas por cajas rojas. MEGA X se utilizó para crear el árbol filogenético.
Figura 5. Árbol filogenético del gen mcr-1 que muestra la relación de las regiones codificantes parciales de las secuencias del gen mcr-1. Las secuencias estudiadas fueron marcadas por cajas rojas. MEGA X se utilizó para crear el árbol filogenético.
En el análisis de filogenia del gen mcr-5, se reveló que la secuencia de nucleótidos del gen mcr-5 de Salmonella spp. obtenida de carne de pollo tiene una estrecha relación con el gen mcr-5.3 en el aislado de E. coli obtenido del caballo en Brasil (Figura 6).
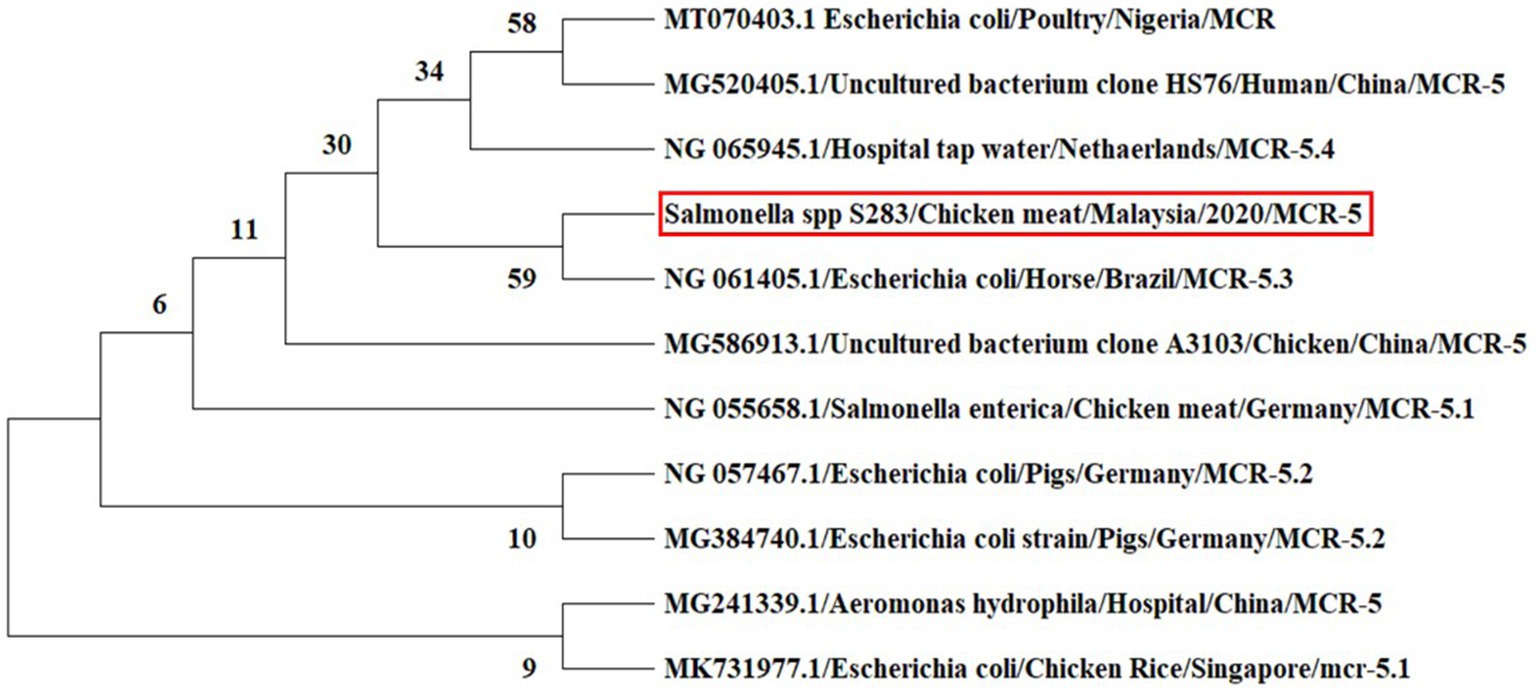 Figura 6. Árbol filogenético inferido de las secuencias de nucleótidos del gen mcr-5 utilizando el método de máxima verosimilitud para cálculos de distancia utilizando el software MEGA X. Los porcentajes de arranque recuperados en 500 replicaciones se muestran en los nodos. La secuencia estudiada está marcada por el recuadro rojo.
Figura 6. Árbol filogenético inferido de las secuencias de nucleótidos del gen mcr-5 utilizando el método de máxima verosimilitud para cálculos de distancia utilizando el software MEGA X. Los porcentajes de arranque recuperados en 500 replicaciones se muestran en los nodos. La secuencia estudiada está marcada por el recuadro rojo.
Discusión
Las bacterias más comunes asociadas con infecciones bacterianas en pollos son E. coli, Salmonella spp. y K. pneumoniae. Se sabe que estos microorganismos causan graves problemas de salud, lo que puede resultar en un aumento de la mortalidad, disminución de la productividad y mayores costos tanto para la prevención como para el tratamiento de enfermedades (Ibrahim et al., 2021). Los aislados de Escherichia coli fueron los más prevalentes en las muestras recolectadas, seguidos por los aislados de Salmonella spp. y K. pneumoniae. Los aislados de Escherichia coli fueron de alta prevalencia en hisopos cloacales de pollo (93,1%), Salmonella spp. en carnes de pollo (34,9%) y aislados de K. pneumoniae en muestras de cama (36,7%). Estudios anteriores también han informado que el 83 y el 53,04% de los hisopos cloacales dieron positivo para E. coli en Malasia y China, respectivamente (Suryadevara et al., 2020; Li et al., 2022). En Indonesia, el 13,75% de los hisopos cloacales obtenidos de granjas de pollos de engorde dieron positivo para K. pneumoniae (Permatasari et al., 2020). Otro estudio en Malasia mostró que la prevalencia de Salmonella spp. y E. coli de hisopos cloacales obtenidos de pollos de engorde fue de 6.5 y 51.8%, respectivamente (Ibrahim et al., 2021). Los pollos de traspatio en Malasia se documentaron con 2.5% de Salmonella spp. (Jajere et al., 2019). En contraste, se observó una prevalencia mucho mayor de Salmonella spp. (48%) en muestras de carne de pollo en Bangladesh (Islam et al., 2016). En Nepal, el 33,33% de las muestras de carne de pollo fueron positivas para E. coli (Joshi et al., 2019), que es inferior a nuestros hallazgos, lo que indica que la frecuencia de E. coli en la carne de pollo podría variar ampliamente. Los diferentes niveles de prácticas higiénicas en diversas regiones geográficas y factores ambientales, como la exposición a un saneamiento deficiente, podrían ser la fuente de las variaciones en la prevalencia (Dawadi et al., 2021).
La colistina se prescribe con fines terapéuticos de infección bacteriana en humanos que son resistentes a múltiples medicamentos a pesar de sus efectos secundarios. La introducción del gen de resistencia a la colistina (mcr) en cepas bacterianas que ya son resistentes a muchos antibióticos, crea la ineficacia de la colistina, el fármaco de último recurso. En este estudio, los aislados de Enterobacteriaceae recuperados de diversas fuentes, que comprenden carne de pollo de supermercados e hisopos cloacales y muestras de basura de granjas avícolas, prevalecieron con resistencia a la colistina y genes mcr. El valor de CMI de los aislados resistentes a la colistina varió de 4 μg/ml a 128 μg/ml de colistina, sin embargo, se encontraron valores bajos de CMI en los aislados mcr positivos. En estudios anteriores, se había documentado el mcr-1 de aislados de Klebsiella pneumoniae y E. coli en Malasia (Yu et al., 2016; Mobasseri et al., 2019; Aklilu y Raman, 2020). El gen mcr-1 de resistencia a la colistina también se observa en aislados de E. coli de aves de corral en muchos países de Asia (Trung et al., 2017; Joshi et al., 2019; Amin et al., 2020). Un mayor porcentaje de mcr-1 (>94%) se representó en cepas bacterianas de heces de pavo y pollos de engorde en Alemania (Irrgang et al., 2016). El gen de resistencia a la colistina (mcr-1) se encontró en el 25,8% de las carnes de aves de corral (pavo y pollo) de Italia (5 muestras) y Alemania (28 muestras), pero no en ninguna muestra (carnes de pavo y pollo) de Suiza, Dinamarca, Austria o Hungría, según un estudio suizo (Zurfluh et al., 2016). En Brasil, el 19,5% de las muestras de carne de pollo e hígado fueron positivas para E. coli que albergaba mcr-1 (Monte et al., 2017). Todas las E. coli resistentes a la colistina obtenidas de carne cruda y productos de carne de res en Egipto resultaron positivas para mcr-1 (Sabala et al., 2021). En comparación con estudios previos, se encontró una baja prevalencia del gen mcr-1 en aislados resistentes a la colistina en la investigación actual. Se desconoce la explicación de la baja incidencia en los aislados de Malasia, aunque podrían estar implicados otros mecanismos de resistencia, como la alteración cromosómica y la modulación en el gen mgrB (Chen et al., 2017).
El gen de resistencia a la colistina, mcr-5 se detectó en Salmonella spp. por primera vez en Malasia. No hubo informes sobre este gen en ninguna cepa bacteriana de animales o humanos en Malasia hasta donde sabemos. En este estudio, el gen mcr-5 en Salmonella spp. se encontró a una tasa muy baja. Este hallazgo es consistente con un informe anterior en el que el 2,5% (8/315) de Salmonella spp. resistente a la colistina se originó en cerdos y carnes (cerdo) en Alemania (Borowiak et al., 2019), y el 0,7% de las muestras de hisopos vaginales humanos en Yangzhou en China (Zhang et al., 2018) resultaron positivas para el gen mcr-5. El gen mcr-5 también se observó en cepas de E. coli resistentes a la colistina obtenidas de hisopos cloacales de aves de corral en Nigeria (Ngbede et al., 2020), ternera en Bélgica (Timmermans et al., 2021), muestras de carne de cerdo en Camboya (Pungpian et al., 2021) y muestras de aves y cerdos en España y China (García-Meniño et al., 2019)). La situación es preocupante porque estos patógenos resistentes podrían propagarse a los humanos a través de la cadena alimentaria o el contacto cercano con animales (Dawadi et al., 2021).
El gen mcr-1 se encontró entre los aislados que mostraban valores de CMI de 4 μg/ml y 8 μg/ml de colistina en este estudio, lo que es consistente con los estudios previos en Bangladesh y China, que mostraron que los aislados resistentes a la colistina con mcr-1 tenían un valor de CMI de 4 μg/ml, y de 8 a 16 μg/ml de colistina. respectivamente (Islam et al., 2017; Amin et al., 2020).
En el nucleótido BLAST, se encontró que las secuencias del gen mcr-1 aislado de E. coli y el gen mcr-5 obtenido de Salmonella spp. eran 100% idénticas a las secuencias de genes mcr-1 y mcr-5 previamente informadas en la base de datos NCBI. En el análisis filogenético, las secuencias del gen mcr-1 estaban estrechamente relacionadas con informes anteriores, la secuencia del gen mcr-1 de aislados de E. coli recuperados de pollo en Malasia (Yu et al., 2016) y la cepa de E. coli aislada de humanos en China (Genbank_MZ450868). Esto sugiere que el gen mcr-1 en E. coli ha estado circulando en Malasia, lo que es una amenaza para los animales y la salud pública. El gen mcr-5 de resistencia a la colistina se detectó por primera vez en el aislado de Salmonella spp. en Malasia. Los diversos tipos de variantes del gen mcr-5, incluidos mcr-5.1 a mcr-5.4, se identificaron en el mundo (Fleres et al., 2019; Ling et al., 2020). En la filogenia, la secuencia del gen mcr-5 del presente estudio estaba estrechamente relacionada con el gen mcr-5.3 recuperado de aislados de E. coli obtenidos del caballo en Brasil (base de datos GenBank, NG_061405).
Conclusión
La introducción y diseminación de la resistencia a la colistina con genes mcr en Enterobacteriaceae es un problema importante en todo el mundo. En el presente estudio se observaron enterobacterias resistentes a la colistina en carnes de aves de corral y granjas avícolas. Los genes mcr-1 y mcr-5 se encontraron en E. coli resistente a la colistina y Salmonella spp., respectivamente. El mcr-5 se ha identificado en Malasia por primera vez, lo que podría indicar el advenimiento de mcr-5 que alberga cepas bacterianas para la infección. La existencia de enterobacterias positivas para mcr en aves de corral y carne de aves de corral en Malasia enfatiza la importancia de la eliminación adecuada de los desechos de aves de corral y las buenas prácticas de higiene entre las personas que están expuestas a aves de corral y carnes de aves de corral.
Declaración de disponibilidad de datos
Las secuencias de nucleótidos de mcr-1 y mcr-5 se han presentado al GenBank del NCBI (acceso No. OR333822, OR333835). Los datos brutos que respaldan los hallazgos de este artículo serán puestos a disposición por los autores, sin reservas indebidas.
Declaración ética
El estudio en animales fue revisado y aprobado por el consejo ético de la Universiti Putra Malaysia (UPM), el Comité Institucional de Cuidado y Uso de Animales (IACUC) aprobó el protocolo del estudio de investigación para recolectar hisopos cloacales de aves de corral vivas (UPM / IACUC / AUP-R091 / 2019).
Contribuciones del autor
MK y ZZ: conceptualización, software, investigación, curación de datos, redacción – preparación de borradores originales, visualización y adquisición. MK, ZZ, LH, NF y NA: metodología, validación y redacción – revisión y edición. RK: análisis formal. ZZ: recursos, supervisión y administración de proyectos. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Esta investigación fue financiada por el fondo fiduciario de la UPM (número de subvención: 6282525) en colaboración con el Consejo de Investigación Agrícola de Bangladesh (BARC), Dhaka, Bangladesh.
Reconocimientos
Los autores desean agradecer a las autoridades del Consejo de Investigación Agrícola de Bangladesh, Bangladesh, y la Universidad Putra Malasia por su apoyo financiero durante esta investigación.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fmicb.2023.1208314/full#supplementary-material
Referencias
Aidara-Kane, A., Andremont, A. y Collignon, P. (2013). Resistencia a los antimicrobianos en la cadena alimentaria y la iniciativa AGISAR. J. Infectar. Salud Pública 6, 162–165. doi: 10.1016/j.jiph.2013.04.001
Aklilu, E. y Raman, K. (2020). El gen MCR-1 codifica Escherichia coli resistente a la colistina en carne cruda de pollo y brotes de frijol en Malasia. Int. J. Microbiol. 2020:8853582. doi: 10.1155/2020/8853582
Amin, M. B., Sraboni, A. S., Hossain, M. I., Roy, S., Mozmader, T. A. U., Unicomb, L., et al. (2020). Aparición y características genéticas de E. coli resistente a la colistina mcr-1-positiva de ambientes avícolas en Bangladesh. J. Glob. Antimicrobiano. Resistir. 22, 546–552. doi: 10.1016/j.jgar.2020.03.028
Borowiak, M., Baumann, B., Fischer, J., Thomas, K., Deneke, C., Hammerl, J. A., et al. (2020). Desarrollo de una nueva PCR múltiple de mcr-6 a mcr-9 y evaluación de la presencia de mcr-1 a mcr-9 en aislados de Salmonella enterica resistentes a la colistina del medio ambiente, los piensos, los animales y los alimentos (2011-2018) en Alemania. Frente. Microbiol. 11:80. DOI: 10.3389/fmicb.2020.00080
Borowiak, M., Hammerl, J. A., Deneke, C., Fischer, J., Szabo, I. y Malorny, B. (2019). Caracterización de aislados de Enterica Serovar typhimurium de origen animal y alimentario en Alemania. Antimicrobiano. Agentes Chemother. 5:E63-00063. doi: 19.10/AAC.1128-00063
Cannatelli, A., D’Andrea, M. M., Giani, T., Di Pilato, V., Arena, F., Ambretti, S., et al. (2013). Aparición in vivo de resistencia a la colistina en Klebsiella pneumoniae productora de carbapenemasas de tipo KPC mediada por la inactivación insercional del regulador PhoQ/PhoP mgrB. Antimicrobiano. Agentes Chemother. 57, 5521–5526. doi: 10.1128/AAC.01480-13
Chen, Y., Luo, Y., Pettengill, J., Timme, R., Melka, D., Doyle, M., et al. (2017). Secuencia singleton tipo 382, un grupo clonal emergente de Listeria monocytogenes asociado con tres brotes multiestatales relacionados con frutas de hueso contaminadas, manzanas de caramelo y ensalada verde de hoja verde. J. Clin. Microbiol. 55, 931–941. doi: 10.1128/JCM.02140-16
Dawadi, P., Bista, S. y Bista, S. (2021). Prevalencia de Escherichia coli resistente a la colistina de aves de corral en países en desarrollo del sur de Asia. Vet. Med. Int. 2021, 1–5. DOI: 10.1155/2021/6398838
Elbediwi, M., Li, Y., Paudyal, N., Pan, H., Li, X., Xie, S., et al. (2019). Carga global de bacterias resistentes a colistina: estudio movilizado de genes de resistencia a colistina. Microorganismos 7, 1–18. doi: 10.3390/microorganismos7100461
EMA (Agencia Europea del Medicamento) (2016). Asesoramiento actualizado sobre el uso de productos de colistina en animales dentro de la Unión Europea: desarrollo de resistencia y posible impacto en la salud humana y animal. Agencia Europea de Medicamentos, Comité de Medicamentos de Uso Veterinario (CVMP); Londres, Reino Unido.
EUCAST (2016a) Recomendaciones para la determinación de la colistina (polimixina E) por CMI según lo recomendado por el Grupo de trabajo conjunto CLSI-EUCAST sobre puntos de interrupción de polimixina. Disponible en: https://Www.Eucast.Org
EUCAST (2016b). Tablas de puntos de interrupción EUCAST para la interpretación de CMI y diámetros de zona, versión 1.0. Basilea: Comité Europeo de Pruebas de Susceptibilidad a los Antimicrobianos. Versión 6.
Ezadi, F., Ardebili, A. y Mirnejad, R. (2019). Pruebas de susceptibilidad antimicrobiana para polimixinas: desafíos, problemas y recomendaciones. J. Clin. Microbiol. 57, 1–20. doi: 10.1128/JCM.01390-18
Fleres, G., Couto, N., Schuele, L., Chlebowicz, M. A., Mendes, C. I., Van Der Sluis, L. W. M., et al. (2019). Detección de una nueva variante del gen mcr-5.4 en el agua del grifo del hospital mediante secuenciación metagenómica de escopeta. J. Antimicrobiano. Chemother. 74, 3626–3628. DOI: 10.1093/JAC/DKZ363
García-Meniño, I., Díaz-Jiménez, D., García, V., de Toro, M., Flament-Simon, S. C., Blanco, J., et al. (2019). Caracterización genómica de la prevalencia de mcr-1, mcr-4 y mcr-5 Escherichia coli dentro de la colibacilosis entérica porcina en España. Frente. Microbiol. 10:2469. doi: 10.3389/fmicb.2019.02469
Ghafur, A., Shankar, C., GnanaSoundari, P., Venkatesan, M., Mani, D., Thirunarayanan, M. A., et al. (2019). Detección de mecanismos cromosómicos y mediados por plásmidos de resistencia a la colistina en Escherichia coli y Klebsiella pneumoniae a partir de muestras de alimentos indios. J. Glob. Antimicrobiano. Resistir. 16, 48–52. doi: 10.1016/j.jgar.2018.09.005
Gharaibeh, M. H. y Shatnawi, S. Q. (2019). Una visión general de la resistencia a la colistina, la diseminación movilizada de genes de resistencia a la colistina, las respuestas globales y las alternativas a la colistina: una revisión. Mundo Veterinario 12, 1735–1746. doi: 10.14202/vetworld.2019.1735-1746
Hall, T. A. (1999). BioEdit: un editor de alineación de secuencias biológicas fácil de usar y un programa de análisis para Windows 95/98/NT. Ácidos. Symp. Ser. 41, 95–98.
Ibrahim, S., Wei Hoong, L., Lai Siong, Y., Mustapha, Z., CS, C. W. Z., Aklilu, E., et al. (2021). Prevalencia de resistencia a los antimicrobianos (RAM) salmonella spp. y Escherichia coli aisladas de pollos de engorde en la costa este de Malasia peninsular. Antibióticos 10. doi: 10.3390/antibióticos10050579
Irrgang, A., Roschanski, N., Tenhagen, B. A., Grobbel, M., Skladnikiewicz-Ziemer, T., Thomas, K., et al. (2016). Prevalencia de mcr-1 en E. coli de ganado y alimentos en Alemania, 2010-2015. PLoS One 11:e0159863. doi: 10.1371/journal.pone.0159863
Islam, M. J., Mahbub-E-Elahi, A. T. M., Ahmed, T. y Hasan, M. K. (2016). Aislamiento e identificación de salmonella spp. de pollos de engorde y su estudio de antibiograma en Sylhet, Bangladesh. J. Appl. Biol. Biotechnol. 4, 046–051. doi: 10.7324/jabb.2016.40308
Islam, A., Rahman, Z., Monira, S., Rahman, M. A., Camilli, A., George, C. M., et al. (2017). Escherichia coli resistente a la colistina portadora de mcr-1 en muestras de lodos urbanos: Dhaka, Bangladesh. Gut Pathog. 9:77. doi: 10.1186/s13099-017-0227-4
Islam, S., Urmi, U. L., Rana, M., Sultana, F., Jahan, N., Hossain, B., et al. (2020). Alta abundancia del gen de resistencia a la colistina mcr-1 en bacterias intestinales de pollo en Bangladesh. 10:17292. DOI: 10.1038/S41598-020-74402-4
Jajere, S. M., Hassan, L., Abdul Aziz, S., Zakaria, Z., Abu, J., Nordin, F., et al. (2019). Salmonella en pollos nativos de «aldea» (Gallus domesticus): prevalencia y factores de riesgo de granjas en el centro-sur de Malasia peninsular. Poult. Sci. 98, 5961–5970. doi: 10.3382/ps/pez392
Joshi, P. R., Thummeepak, R., Paudel, S., Acharya, M., Pradhan, S., Banjara, M. R., et al. (2019). Caracterización molecular de Escherichia coli resistente a la colistina aislada de pollos: primer informe de Nepal. Microbio. Resistencia a las drogas. 25, 846–854. DOI: 10.1089/MDR.2018.0326
Kumar, S., Stecher, G., Li, M., Knyaz, C. y Tamura, K. (2018). MEGA X: análisis de genética evolutiva molecular a través de plataformas informáticas. Mol. Biol. Evol. 35, 1547–1549. doi: 10.1093/molbev/msy096
Li, J., Liu, S., Fu, J., Yin, J., Zhao, J., Zhong, C., et al. (2019). Co-ocurrencia de determinantes de resistencia a Colistin y Meropenem en una cepa de Stenotrophomonas aislada de aguas residuales. Microbio. Resistencia a las drogas. 25, 317–325. DOI: 10.1089/MDR.2018.0418
Li, Z., Xin, L., Peng, C., Liu, C., Wang, P., Yu, L., et al. (2022). Perfiles de prevalencia y susceptibilidad antimicrobiana de Klebsiella pneumoniae productora de BLEE de granjas de pollos de engorde en la provincia de Shandong, China. Poult. Sci. 101:102002. doi: 10.1016/j.psj.2022.102002
Ling, Z., Yin, W., Shen, Z., Wang, Y., Shen, J. y Walsh, T. R. (2020). Epidemiología de los genes móviles de resistencia a la colistina mcr-1 a mcr-9. J. Antimicrobiano. Chemother. 75, 3087–3095. doi: 10.1093/jac/dkaa205
Liu, Y.-Y., Ang, Y., Walsh, T. R., Yi, L.-X., Zhang, R., Spencer, J., et al. (2016). Aparición del mecanismo de resistencia a la colistina MCR-1 mediado por plásmidos en animales y seres humanos en China: un estudio microbiológico y biológico molecular. Lancet Infect. Dis. 16, 161–168. doi: 10.1016/S1473-3099(15)00424-7
Moawad, A. A., Hotzel, H., Neubauer, H., Ehricht, R., Monecke, S., Tomaso, H., et al. (2018). Resistencia a los antimicrobianos en Enterobacteriaceae de pollos de engorde sanos en Egipto: aparición de Escherichia coli resistente a la colistina y de espectro extendido productora de β-lactamasa. Gut Pathog. 10, 1–12. DOI: 10.1186/S13099-018-0266-5
Mobasseri, G., Teh, C. S. J., Ooi, P. T. y Thong, K. L. (2019). La aparición de cepas de Klebsiella pneumoniae resistentes a la colistina de cerdos en Malasia. J. Glob. Antimicrobiano. Resistir. 17, 227–232. doi: 10.1016/j.jgar.2018.12.015
Monte, D. F., Mem, A., Fernandes, M. R., Cerdeira, L., Esposito, F., Galvão, J. A., et al. (2017). Carne de pollo como reservorio de cepas de Escherichia coli resistentes a la colistina portadoras de genes mcr-1 en América del Sur. Antimicrobiano. Agentes Chemother. 61, 2–5. doi: 10.1128/AAC.02718-16
Ngbede, E. O., Poudel, A., Kalalah, A., Yang, Y., Adekanmbi, F., Adikwu, A. A., et al. (2020). Identificación de genes móviles de resistencia a la colistina (mcr-1.1, mcr-5 y mcr-8.1) en Enterobacteriaceae y Alcaligenes faecalis de origen humano y animal, Nigeria. Int. J. Antimicrobiano. Agentes 56:106108. doi: 10.1016/j.ijantimicag.2020.106108
Permatasari, D. A., Witaningrum, A. M., Wibisono, F. J. y Effendi, M. H. (2020). Detección y prevalencia de cepas de Klebsiella pneumoniae multirresistentes aisladas de granjas avícolas en Blitar, Indonesia. Biodiversitas 21, 4642–4647. doi: 10.13057/biodiv/d211024
Pui, C. F., Wong, W. C., Chai, L. C., Nillian, E., Ghazali, F. M., Cheah, Y. K., et al. (2011). Salmonella Typhi y Salmonella Typhimurium en lonchas de frutas mediante PCR múltiple. Food Control 22, 337–342. doi: 10.1016/j.foodcont.2010.05.021
Pungpian, C., Lee, S., Trongjit, S., Sinwat, N., Angkititrakul, S., Prathan, R., et al. (2021). Resistencia a la colistina y genes MCR mediados por plásmidos en Escherichia coli y salmonella aisladas de cerdos, canales de cerdos y carne de cerdo en las provincias fronterizas de Tailandia, la República Democrática Popular Lao y Camboya. J. Vet. Sci. 22, 1–15. DOI: 10.4142/JVS.2021.22.E68
Rahn, K., De Grandis, S. A. A., Clarke, R. C. C., McEwen, S. A. A., Galán, J. E. E., Ginocchio, C., et al. (1992). Amplificación de una secuencia del gen invA de Salmonella typhimurium mediante reacción en cadena de la polimerasa como método específico de detección de salmonela. Mol. Sondas 6, 271–279. doi: 10.1016/0890-8508(92)90002-F
Ranjbar, R., Izadi, M., Hafshejani, T. T. y Khamesipour, F. (2016). Detección molecular y resistencia antimicrobiana de Klebsiella pneumoniae de moscas domésticas (Musca domestica) en cocinas, granjas, hospitales y mataderos. J. Infectar. Salud Pública 9, 499–505. doi: 10.1016/j.jiph.2015.12.012
Rebelo, A. R., Bortolaia, V., Kjeldgaard, J. S., Pedersen, S. K., Leekitcharoenphon, P., Hansen, I. M., et al. (2018). PCR multiplex para la detección de determinantes de resistencia a la colistina mediados por plásmidos, mcr-1, mcr-2, mcr-3, mcr-4 y mcr-5 con fines de vigilancia. Eur. Secur. 23:17-00672. doi: 10.2807/1560-7917.ES.2018.23.6.17-00672
Sabala, R. F., Usui, M., Tamura, Y., Abd-Elghany, S. M., Sallam, K. I. y Elgazzar, M. M. (2021). Prevalencia de Escherichia coli resistente a la colistina que alberga mcr-1 en carne cruda y productos de carne de vacuno listos para el consumo en Egipto. Control de alimentos 119:107436. doi: 10.1016/j.foodcont.2020.107436
Sharma, J., Kumar, D., Hussain, S., Pathak, A., Shukla, M., Kumar, V. P., et al. (2019). Prevalencia, resistencia antimicrobiana y caracterización de genes de virulencia de salmonela no tifoidea aislada de tiendas minoristas de carne de pollo en el norte de la India. Control de alimentos 102, 104–111. doi: 10.1016/j.foodcont.2019.01.021
Suryadevara, N., Yong, K. B., Ganapathy, B., Subramonie, S., Ragavan, N. D., Ramachandiran, M., et al. (2020). Caracterización molecular de Escherichia coli de pollos en granjas avícolas de Malasia. Res. J. Biotechnol. 15, 1–10.
Theuretzbacher, U. (2017). Resistencia antimicrobiana global en patógenos gramnegativos y necesidad clínica. Curr. Opinar. Microbiol. 39, 106–112. doi: 10.1016/j.mib.2017.10.028
Timmermans, M., Wattiau, P., Denis, O. y Boland, C. (2021). Genes de resistencia a la colistina mcr-1 a mcr-5, incluido un caso de triple aparición (mcr-1, -3 y -5), en aislados de Escherichia coli de heces de cerdos, bovinos y aves sanos en Bélgica, 2012-2016. Int. J. Antimicrobiano. Agentes 57:106350. doi: 10.1016/j.ijantimicag.2021.106350
Trung, N. V., Matamoros, S., Carrique-Mas, J. J., Nghia, N. H., Nhung, N. T., Chieu, T. T. B., et al. (2017). Transmisión zoonótica del gen de resistencia a la colistina mcr-1 de granjas avícolas a pequeña escala, Vietnam. Emerg. Infecta. Dis. 23, 529–532. DOI: 10.3201/EID2303.161553
Xu, T., Zhang, C., Ji, Y., Song, J., Liu, Y., Guo, Y., et al. (2021). Identificación de mcr-10 transportado por plásmidos autotransmisibles y cromosomas en cepas de Enterobacter roggenkampii aisladas de aguas residuales hospitalarias. Entorno. Contaminante. 268:115706. doi: 10.1016/j.envpol.2020.115706
Yu, C. Y., Ang, G. Y., Chin, P. S., Ngeow, Y. F., Yin, W.-F., y Chan, K.-G. (2016). Aparición de resistencia a la colistina mediada por mcr-1 en Escherichia coli en Malasia. Int. J. Antimicrobiano. Agentes 47, 504–505. doi: 10.1016/j.ijantimicag.2016.04.004
Zhang, J., Chen, L., Wang, J., Butaye, P., Huang, K., Qiu, H., et al. (2018). Detección molecular de genes de resistencia a la colistina (mcr-1 a mcr-5) en hisopos vaginales humanos. BMC. Res. Notes 11:143. DOI: 10.1186/S13104-018-3255-3
Palabras clave: Enterobacteriaceae, MCR, resistencia a la colistina, aves de corral, Malasia
Cita: Karim MR, Zakaria Z, Hassan L, Faiz NM y Ahmad NI (2023) La aparición y detección molecular de los genes mcr-1 y mcr-5 en Enterobacteriaceae aislados de aves de corral y carnes de aves de corral en Malasia. Frente. Microbiol. 14:1208314. doi: 10.3389/fmicb.2023.1208314
Recibido: 18 de abril de 2023; Aprobado: 12 de julio de 2023;
Publicado: 03 agosto 2023.
Editado por:
Zuowei Wu, Universidad Estatal de Iowa, Estados Unidos
Revisado por:
Nikolaos Strepis, Centro Médico Erasmus, Países Bajos
Hazem Ramadan, Universidad de Mansoura, Egipto
Derechos de autor © 2023 Karim, Zakaria, Hassan, Faiz y Ahmad. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Zunita Zakaria, zunita@upm.edu.my
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía