Distribución de detección de CNVs de SNX29 en tres razas caprinas

Distribución de detección de CNVs de SNX29 en tres razas caprinas y sus asociaciones con rasgos de crecimiento
 Qian Wang1†
Qian Wang1†  Canción Xiaoyue2,3†
Canción Xiaoyue2,3†  Yi Bi1
Yi Bi1  Haijing Zhu2,3
Haijing Zhu2,3  Xianfeng Wu4
Xianfeng Wu4  Zhengang Guo5
Zhengang Guo5  Mei Liu6*
Mei Liu6*  Chuanying Pan1*
Chuanying Pan1*- 1Facultad de Ciencia y Tecnología Animal, Laboratorio Clave de Genética Animal, Cría y Reproducción de la Provincia de Shaanxi, Universidad Northwest A&F, Yangling, Shaanxi, China
- 2Centro Provincial de Investigación de Ingeniería y Tecnología de Shaanxi de Cabras de Cachemira, Universidad de Yulin, Yulin, Shaanxi, China
- 3Centro de Investigación de Ciencias de la Vida, Universidad de Yulin, Yulin, Shaanxi, China
- 4Instituto de Ganadería y Veterinaria, Academia de Ciencias Agrícolas de Fujian, Fuzhou, Fujian, China
- 5Instituto de Ganadería y Ciencias Veterinarias de la ciudad de Bijie, Bijie, Guizhou, China
- 6Facultad de Ciencia y Tecnología Animal, Universidad Agrícola de Hunan, Changsha, Hunan, China
Como miembro de la familia SNX, la nexina 29 clasificadora de cabras (SNX29) se identifica inicialmente como un gen de miogénesis. Por lo tanto, este estudio tuvo como objetivo examinar el polimorfismo en el gen SNX29 y su asociación con los rasgos de crecimiento. En este estudio, utilizamos una plataforma en línea para predecir las estructuras de la proteína SNX29 y utilizamos PCR cuantitativa en tiempo real para detectar la posible variación del número de copias (CNV) en cabras de cachemira blanca Shaanbei (SBWC) (n = 541), cabras negras de Guizhou (GB) (n = 48) y cabras de Nubia (NB) (n = 39). Los resultados mostraron que la proteína SNX29 de cabra pertenecía a la proteína no secretora. Luego, se detectaron cinco CNV y se analizó su asociación con rasgos de crecimiento. En cabras SBWC, CNV1, CNV3, CNV4 y CNV5 se asociaron con el ancho del pecho y la longitud del cuerpo (P < 0,05). Entre ellos, los individuos CNV1 con genotipos de ganancia y pérdida fueron superiores a aquellos individuos con un genotipo mediano, pero CNV4 y CNV5 de individuos con genotipo mediano fueron superiores a aquellos con los genotipos de pérdida y ganancia. Además, los individuos con el genotipo de ganancia tenían rasgos de crecimiento superiores en CNV3. En resumen, este estudio sugiere que el CNV de SNX29 se puede utilizar como un marcador molecular en la cría de cabras.
Introducción
Los miembros de la familia SNX se encuentran en el citoplasma de unión a la membrana y pueden unirse al fosfatidilinositol a través del dominio PX e interactuar con complejos de proteínas asociadas a la membrana, que desempeñan un papel importante en la regulación de la endocitosis y el transporte de proteínas a través de los compartimentos de la membrana celular (1, 2). Hasta la fecha, se han identificado 32 miembros, y se dividen en cinco subgrupos basados en el dominio de la proteína. Entre ellos, el gen SNX29 pertenece a la subfamilia SNX-PX (3), que se ha informado que está involucrada en la enfermedad, el desarrollo del sistema nervioso y el crecimiento animal. Los estudios han relacionado el gen SNX29 con la esquizofrenia (SCZ), el autismo y otros trastornos psiquiátricos (4, 5). La deleción del intrón 29 de SNX14 puede conducir a linfoma testicular primario (6). Zhu et al. encontraron que la regulación a la baja del gen SNX29 se asoció con células de carcinoma epitelial de ovario (7). Además, Sparks et al. mostraron una fuerte asociación entre los niveles de IgA y la región entre 6,89 y 14,95 Mb en el cromosoma 24 de las ovejas, que corresponde al gen SNX29 (8). Un circRNA del gen SNX29 regula la proliferación y diferenciación de las células musculares (9). Los estudios han demostrado que el gen SNX29 desempeña un papel clave en la deposición de grasa subcutánea en cabras negras de Xiangdong (XDB), y la CNV SNX29 se asocia significativamente con la circunferencia torácica y abdominal de cabras XDB (P < 0.01) (10). Con base en lo anterior, el gen SNX29 fue seleccionado para ser estudiado en este estudio.
La variación del número de copias (NVC) existe ampliamente en los genomas de los organismos, y se considera una fuente importante de diferencias genéticas entre individuos (11, 12). En los últimos años, algunos estudios informaron que la NVC se correlacionó significativamente con los rasgos económicos del ganado, como el tamaño de la camada (13), la calidad de la carne (14), la producción de leche (15), la tasa de aumento de peso (16) y la tasa de conversión alimenticia (17). Las ventajas de la diversidad, simplicidad y eficiencia de la población que promueve la CNV fueron descubiertas por más personas (18). Como marcador molecular aplicable, CNV puede hacer que la selección asistida por marcadores (MAS) juegue mejor las ventajas de conveniencia, simplicidad, etc. En resumen, proporciona nuevas ideas y métodos para el trabajo de cría.
Las cabras de cachemira blanca Shaanbei (SBWC) fueron criadas a partir de la cabra de cachemira blanca de Liaoning y la cabra negra Ziwuling (19), que tiene un alto valor de cachemira y valor de carne (20). Las cabras negras de Guizhou (GB) son una excelente raza local con buena calidad de carne y tolerancia a la alimentación gruesa (21). Las cabras de Nubia (NB) tienen un buen valor para la carne y la leche y tienen un mayor contenido de carne que otras cabras de doble propósito (22). Sin embargo, su rendimiento de crecimiento no logra los resultados esperados, por lo que es útil aumentar el valor económico de las cabras mejorando sus rasgos de crecimiento a través de MAS.
Actualmente, las CNV del gen SNX29 y su asociación con rasgos de crecimiento en cabras SBWC no han sido reportadas. Por lo tanto, este estudio se caracteriza en función de los aspectos de la estructura de la proteína, las propiedades fisicoquímicas y la variación del ADN. A continuación, exploramos cinco CNV potenciales, que se detectaron en cabras SBWC, cabras GB y cabras NB mediante PCR cuantitativa en tiempo real (qRT-PCR). Se realizó un análisis de asociación entre el gen SNX29 y los rasgos de crecimiento de las cabras. Estos resultados tendrán una comprensión más profunda de la variación genética y los rasgos de crecimiento del ganado, con el fin de sentar una base teórica para la cría de cabras MAS.
Materiales y métodos
Explicación del bienestar animal
Las muestras utilizadas en este experimento cumplen con el Reglamento sobre la Administración de Animales de Experimentación de la Universidad Northwest A&F (NWAFU-314020038).
Predicción de las propiedades fisicoquímicas y la estructura de la proteína SNX29
Utilizando secuencias de proteínas SNX29 buscadas por el NCBI, se calculó el número de aminoácidos de la proteína SNX29, el peso molecular y el punto isoeléctrico de las cabras utilizando la plataforma en línea Expasy, y la aplicación ProtScale y ProtParam se utilizaron para predecir la hidrofobicidad de la proteína. La proteína SNX29 del péptido señal transmembrana se predijo utilizando la base de datos TMHMM y SignalP 4.1. Se utilizaron las plataformas en línea AlphaFold y SOPMA para predecir la estructura avanzada de la proteína SNX29 (23) (Tabla suplementaria S1).
Recolección de muestras y extracción de ADN genómico
En las mismas condiciones de alimentación, se seleccionaron tejidos auditivos de 541 cabras SBWC, cabras de 48 GB y 39 cabras NB de la granja de cabras Yulin en la provincia de Shaanxi, la granja de cabras Bijie en la provincia de Guizhou y la cooperativa de cría de cabras de Nubia de Zhangzhou en la provincia de Fujian. Todos los individuos eran cabras hembras (2-3 años) y no estaban relacionados entre sí. El ADN genómico se extrajo del tejido de la oreja de cabra utilizando el método de extracción con alto contenido de sal (24) y se almacenó al 70% de alcohol a -80 °C (25). Se utilizó un espectrofotómetro NanoDrop™2000 (Thermo Scientific, Waltham, MA, EUA) para medir el OD260/280 y una relación entre 1,8 y 2,0 significa que la concentración de ácido nucleico está calificada (26). Luego, el ADN extraído se colocó a -40 ° C.
Diseño de imprimaciones
Se realizaron búsquedas en la base de datos Animal Omics (27) (Tabla Suplementaria S1) y se encontraron cinco loci CNV del gen SNX29 en cabras. Cinco pares de cebadores amplificados fueron referenciados en un artículo anterior (28).
Detección de genotipado CNV del gen SNX29
Para garantizar que los cebadores puedan amplificar el fragmento objetivo, los cebadores se detectan a través del grupo mixto (CNV1 = 137 pb, CNV2 = 138 pb, CNV3 = 104 pb, CNV4 = 151 pb y CNV5 = 109 pb). A continuación, se utilizaron 541 muestras de cabra SBWC, muestras de cabra de 48 GB y 39 muestras de cabra NB para detectar los loci de CNV. Los sistemas y procedimientos de amplificación qRT-PCR se refieren a artículos de laboratorio anteriores (29, 30). El resultado se procesó utilizando el método 2 2(−δCt) (31).*
Análisis estadísticos
La asociación entre las variantes y los rasgos de crecimiento se exploró utilizando el análisis de varianza (ANOVA) y la prueba t de muestra independiente en SPSS 26.0 (IBM, EUA), y el chi-cuadrado (χ2) se utilizó para analizar la significación entre las tres razas (32). Y el modelo lineal fue utilizado como referencia por Liu et al. (33). Donde YIJK = αYo + βj + eIJK + u actúa como modelo de análisis, YIJK es la evaluación de los rasgos de crecimiento en el nivel I de la edad de factor fijo (αYo) y el nivel j del genotipo del factor fijo (βj), u es la media global, y eIJK es el error aleatorio.
Resultados
Predicción de las propiedades fisicoquímicas y la estructura de la proteína SNX29
Para caracterizar las funciones del gen SNX29, se predijo la estructura de la proteína y las propiedades fisicoquímicas. Los resultados mostraron que la proteína contenía 817 aminoácidos, el peso molecular era de 9.143,14 y el punto isoeléctrico era de 5,90 por la plataforma en línea Expasy. El software en línea ProtScale predijo la hidrofobicidad de la proteína, y los resultados mostraron que había más residuos hidrófilos en la proteína SNX29 de cabra, lo que indicaba que esta proteína era hidrófila (Figura 1A). Los resultados fueron consistentes con las predicciones del software en línea ProtParam. Los resultados de la predicción de TMHMM mostraron que la proteína codificada por el gen SNX29 no tenía hélice transmembrana (Figura 1B). Los resultados de la predicción de SignalP 4.1 mostraron que el valor crítico D del péptido señal y el péptido no señal de esta proteína fue 0.450, y el valor crítico D de la proteína SNX29 fue 0.155 (Figura 1C). De acuerdo con la hipótesis del péptido señal, la proteína SNX29 no tenía péptido señal y pertenecía a la proteína no secretora. La plataforma en línea SOPMA predijo la información detallada sobre la estructura secundaria de la proteína SNX29, y los resultados mostraron que la hélice alfa representó el 47,98%, la hebra extendida representó el 12,24%, la β vuelta representó el 4,04% y la bobina aleatoria representó el 35,74% (Figura 1E). El software en línea AlphaFold predijo la estructura tridimensional de la proteína SNX29 (Figura 1D).
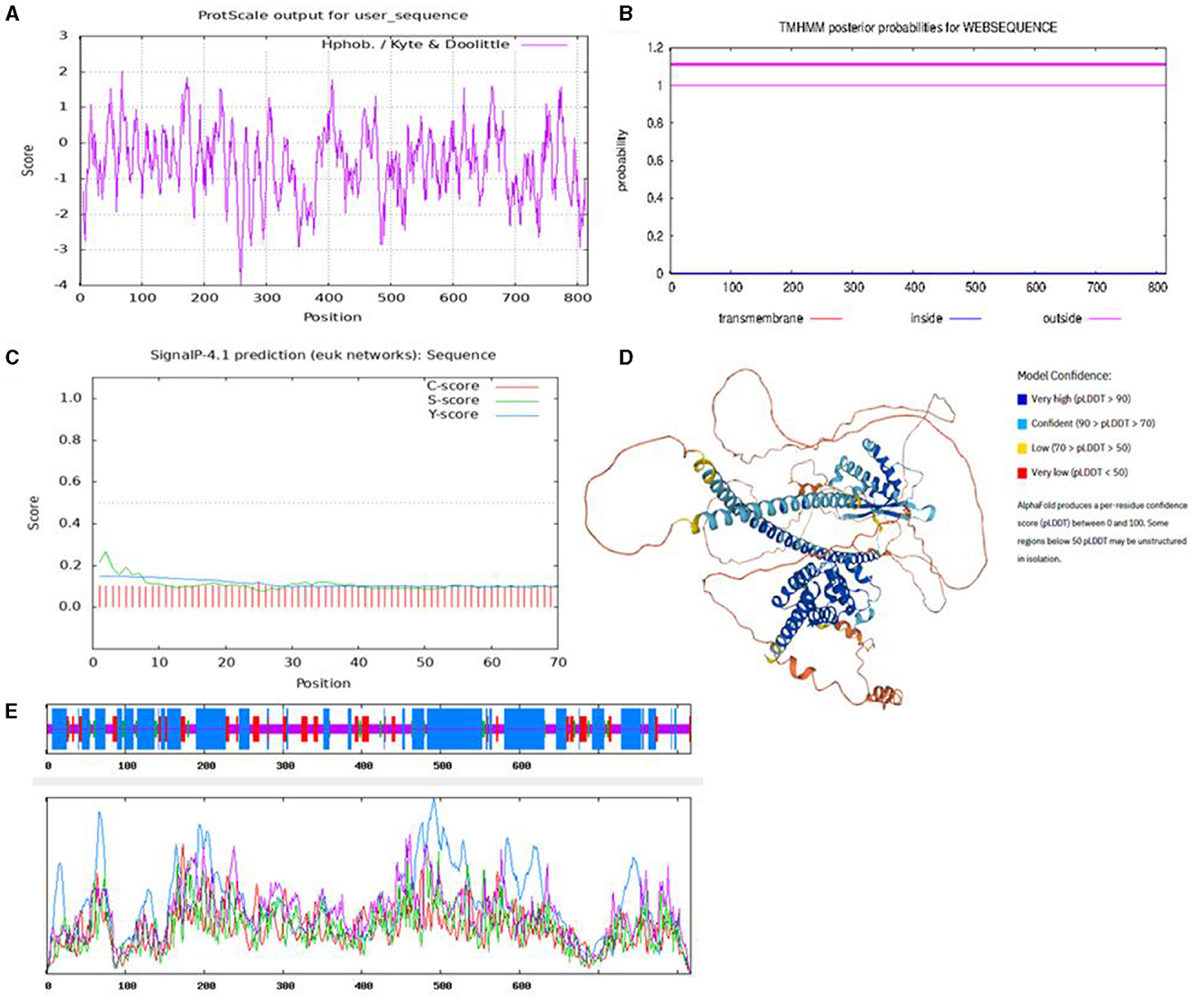 Figura 1. Predicción de las propiedades fisicoquímicas y estructura de la proteína SNX29. (A) Hidrofobicidad de la proteína SNX29 de cabra. (B) Péptido de señal transmembrana de proteína SNX29 de cabra. (C) Predicción del péptido de señal de señal de la proteína SNX29 de cabra. El eje de abscisas representa el número de secuencia de residuos de aminoácidos correspondiente a la secuencia de proteínas presentada; el valor del eje de ordenadas es el valor de probabilidad de cada aminoácido ubicado en el interior, exterior y TMhelix en el eje de abscisas. (D) Modelo tridimensional de la estructura terciaria de la proteína SNX29 de cabra. (E) Parámetros estructurales secundarios de la proteína SNX29 de cabra. Azul significa una hélice, rojo significa columna vertebral extendida, verde significa plegado de β y amarillo significa engarzado aleatorio.
Figura 1. Predicción de las propiedades fisicoquímicas y estructura de la proteína SNX29. (A) Hidrofobicidad de la proteína SNX29 de cabra. (B) Péptido de señal transmembrana de proteína SNX29 de cabra. (C) Predicción del péptido de señal de señal de la proteína SNX29 de cabra. El eje de abscisas representa el número de secuencia de residuos de aminoácidos correspondiente a la secuencia de proteínas presentada; el valor del eje de ordenadas es el valor de probabilidad de cada aminoácido ubicado en el interior, exterior y TMhelix en el eje de abscisas. (D) Modelo tridimensional de la estructura terciaria de la proteína SNX29 de cabra. (E) Parámetros estructurales secundarios de la proteína SNX29 de cabra. Azul significa una hélice, rojo significa columna vertebral extendida, verde significa plegado de β y amarillo significa engarzado aleatorio.
Frecuencia de genotipos CNV en cabras
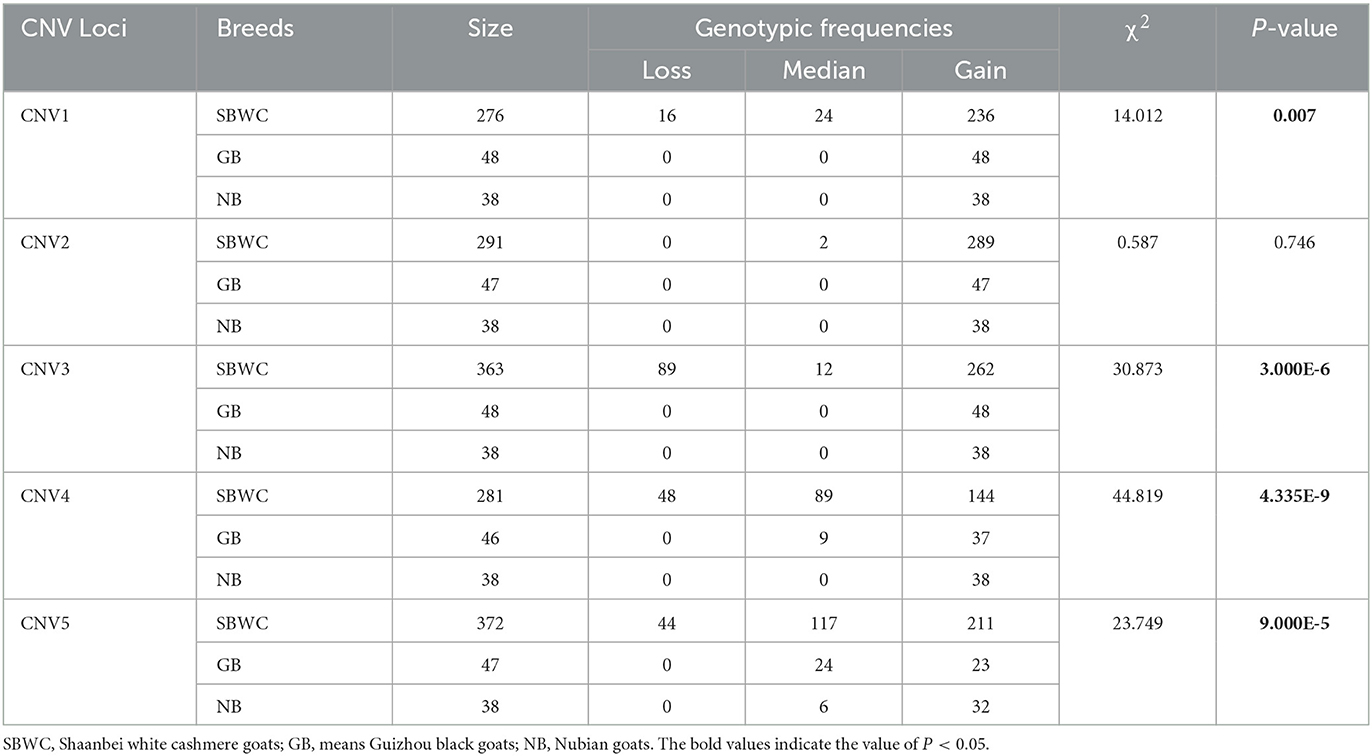
Después de la detección de grupos mixtos, se encontró que los cinco CNV eran consistentes con la banda objetivo (Figura 2). Luego, al ampliar el tamaño de la muestra para las pruebas, se obtuvieron los siguientes resultados: En CNV1, la proporción de genotipo de ganancia fue mayor que la de genotipos de mediana y pérdida en cabras. Hubo 85,61% de individuos con genotipos de ganancia en las cabras SBWC; sin embargo, y todos los individuos en las cabras GB y cabras NB fueron genotipos ganadores; en CNV2, las tres razas caprinas fueron genotipo ganador; en CNV3, hubo 72,18% de individuos de genotipo de ganancia, 3,31% de individuos de genotipo mediano y 24,52% de individuos de genotipo de pérdida en cabras SBWC, y todas las cabras GB y cabras NB fueron genotipo de ganancia; en CNV4, hubo 51,25% de individuos de genotipo de ganancia, 31,67% de individuos de genotipo mediano y 17,08% de individuos de genotipo de pérdida en cabras SBWC, hubo 80,43% de individuos de genotipo de ganancia, 19,57% de individuos de genotipo mediano en cabras GB, y cabras NB fueron todos genotipo de ganancia; y en CNV5, hubo 56,72% de individuos de genotipo de ganancia, 31,45% de individuos de genotipo mediano y 11,83% de individuos de genotipo de pérdida en cabras SBWC, hubo 48,94% de individuos de genotipo de ganancia, 51,06% de individuos de genotipo mediano en cabras GB, hubo 84,21% de individuos de genotipo de ganancia y 15,79% de individuos de genotipo mediano en cabras NB (Figura 3).
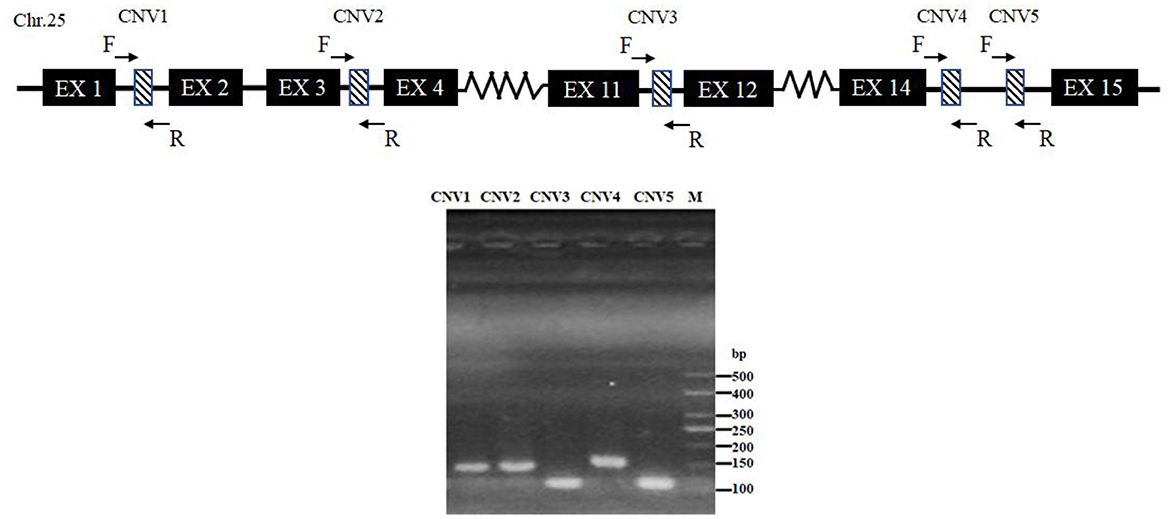 Figura 2. Diagrama esquemático del ensayo de PCR para CNVs del gen SNX29 de cabra. Chr: cromosoma; EX: exón; F: imprimación hacia adelante; R: imprimación inversa; M, significa marcador de ADN.
Figura 2. Diagrama esquemático del ensayo de PCR para CNVs del gen SNX29 de cabra. Chr: cromosoma; EX: exón; F: imprimación hacia adelante; R: imprimación inversa; M, significa marcador de ADN.
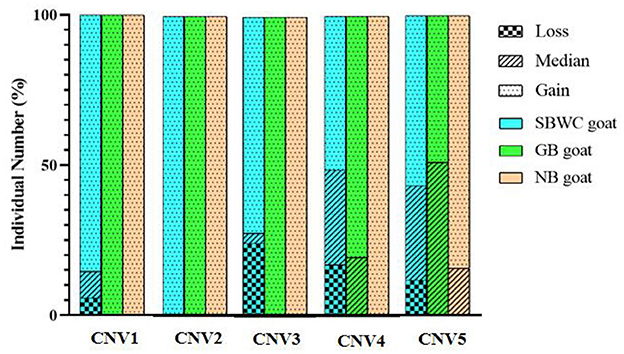 Figura 3. Proporción de genotipado de CNVs en cabra SBWC, cabra GB y cabra NB. En CNV1: el número total individual de cabras SBWC fue de 278, el de cabras GB fue de 48 y el de cabras NB fue de 38; en CNV2: el número total individual de cabras SBWC fue de 290, el de cabras GB fue de 48 y el de cabras NB fue de 39; en CNV3: el número total individual de cabras SBWC fue de 363, el de cabras GB fue de 48, y las cabras NB fueron 281, las cabras GB fueron 46 y las cabras NB fueron 38; en CNV5: el número total individual de cabras SBWC fue de 372, el de cabras GB fue de 48 y el de cabras NB fue de 39.
Figura 3. Proporción de genotipado de CNVs en cabra SBWC, cabra GB y cabra NB. En CNV1: el número total individual de cabras SBWC fue de 278, el de cabras GB fue de 48 y el de cabras NB fue de 38; en CNV2: el número total individual de cabras SBWC fue de 290, el de cabras GB fue de 48 y el de cabras NB fue de 39; en CNV3: el número total individual de cabras SBWC fue de 363, el de cabras GB fue de 48, y las cabras NB fueron 281, las cabras GB fueron 46 y las cabras NB fueron 38; en CNV5: el número total individual de cabras SBWC fue de 372, el de cabras GB fue de 48 y el de cabras NB fue de 39.
Análisis de asociación entre CNVs y el gen SNX29 de cabra
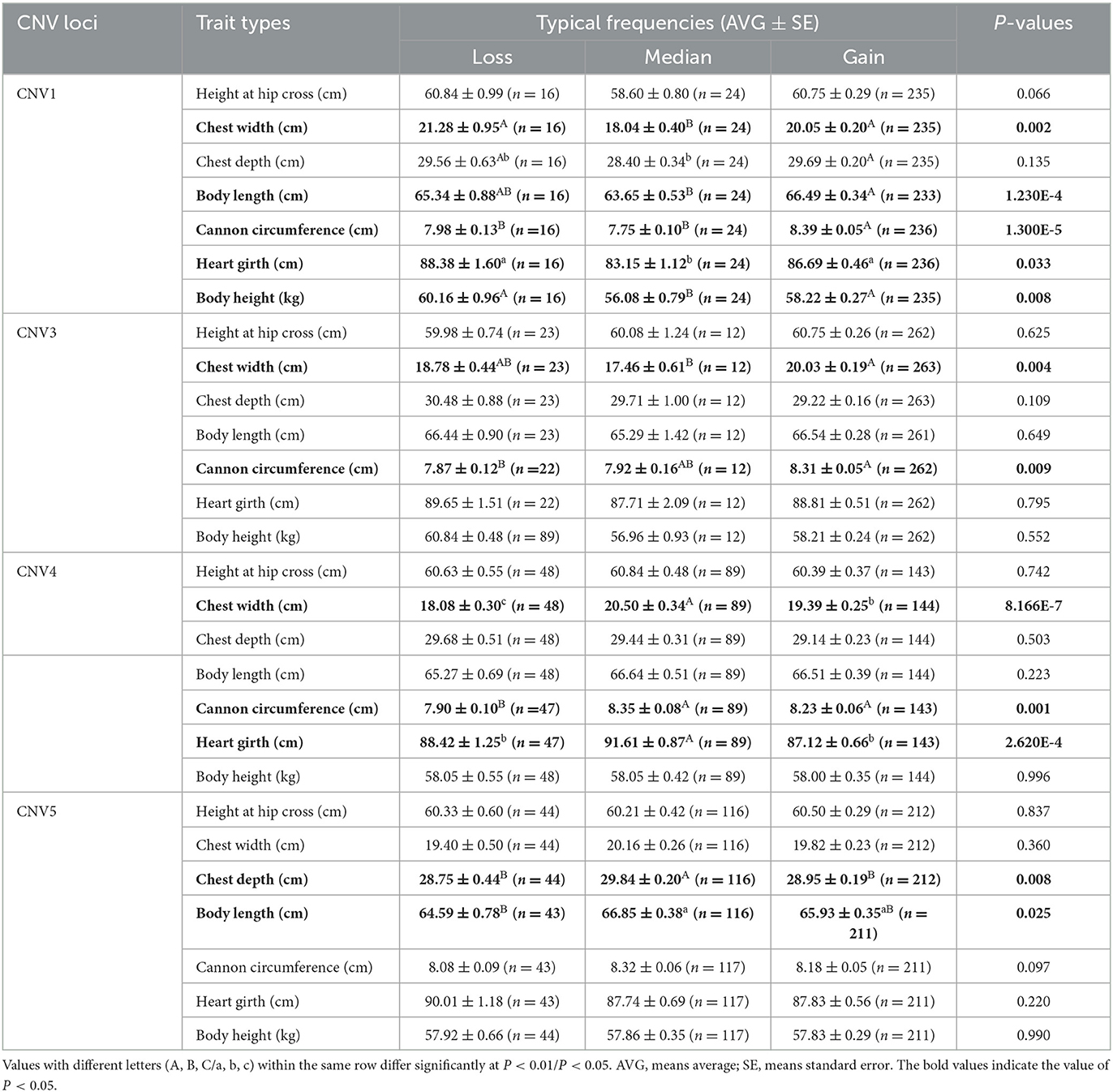
Los resultados del análisis de asociación mostraron que cuatro CNV estaban relacionados con rasgos de crecimiento en cabras SBWC. La CNV1 se asoció significativamente con el ancho del pecho (P = 0,002), la longitud del cuerpo (P = 1,230E-4), la altura del cuerpo (P = 0,008), la circunferencia del cañón (P = 1,300E-5) y la circunferencia del corazón (P = 0,033). La CNV3 se asoció significativamente con el ancho del pecho (P = 0,004) y la circunferencia del cañón (P = 0,009). La CNV4 se asoció significativamente con el ancho del pecho (P = 8.166E-7), la circunferencia del corazón (P = 2.620E-4) y la circunferencia del cañón (P = 0.001). La CNV5 se asoció significativamente con la profundidad torácica (P = 0,008) y la longitud corporal (P = 0,025). Además, en el análisis de asociación entre los rasgos de crecimiento de cabras SBWC y CNV, encontramos que en individuos CNV1, los genotipos de ganancia y pérdida fueron superiores a aquellos con genotipo mediano en el aspecto de los rasgos de crecimiento, pero en individuos CNV4 y CNV5, los genotipos medianos fueron superiores a la pérdida y ganancia. Además, en la CNV3, el genotipo de ganancia realizó mejores rasgos de crecimiento (Tabla 1). El χ2 test results showed that except for CNV2, the remaining CNV loci were significantly associated among the SBWC goats, GB goats, and NB goats (P < 0.01) (Table 2).
Discusión
Estudios relevantes han demostrado que los genes SNX7 (34, 35), SNX8 (36), SNX9 (37), SNX10 (38) y SNX19 (39) se asociaron con rasgos de crecimiento animal. Como miembro de la misma familia, especulamos que los CNV SNX29 pueden tener una influencia notable en los rasgos de crecimiento. Para explorar preliminarmente la función del gen SNX29, se predijo la estructura de la proteína SNX29 de cabra utilizando una plataforma en línea. Los resultados mostraron que la proteína SNX29 era hidrófila y no tenía hélice transmembrana ni péptido señal, y es una proteína no secretora y realizó una función relevante en el citoplasma, lo que fue consistente con la descripción anterior (40).
Para explorar más a fondo la relación entre este gen y los rasgos de crecimiento, realizamos la validación de la población. Se recuperaron cinco CNV de la base de datos. Después de la detección de la distribución de la población, se encontró que los genotipos de cabras de las tres razas eran diferentes en diferentes loci. Esto se debe a que las variaciones genéticas varían de una raza a otra (41). En las tres razas caprinas, más individuos realizaron ganancia de genotipo. Esto puede deberse a que el genotipo de ganancia mostró una mejor eficiencia económica y se mantuvo en la selección artificial. En particular, el análisis de asociación mostró que cuatro CNV se asociaron observablemente con el ancho del pecho, la longitud del cuerpo, la altura del cuerpo, la circunferencia del cañón y la circunferencia del pecho (P < 0.05) en cabras SBWC, lo que apoya nuestra conjetura. Además, encontramos que en individuos CNV1, los genotipos de ganancia y pérdida fueron superiores a aquellos con los genotipos medianos en términos de rasgos de crecimiento, pero en individuos CNV4 y CNV5, los genotipos medianos fueron mejores que los genotipos de pérdida y ganancia. Además, en la CNV3, el genotipo de ganancia realizó mejores rasgos de crecimiento, lo que podría deberse a la mutación, selección, recombinación génica y migración de deriva genética (42). Estos resultados sugieren que el genotipo de ganancia/pérdida de CNV1, el genotipo de ganancia de CNV3 y el genotipo mediano de CNV4 y CNV5 tienen un efecto positivo sobre los rasgos de crecimiento (43).
En este estudio, encontramos que las CNV de SNX29 se asociaron con los rasgos de crecimiento de las cabras, lo que es consistente con la función de SNX29 en estudios previos asociados con el crecimiento. Un escaneo de todo el genoma identificó los marcadores SNP relacionados con el crecimiento de SNX29 en ganado Wagyu chino (44). El análisis de asociación de todo el genoma mostró que CNV27 del gen SNX29 se asoció con rasgos de crecimiento de cabras africanas (45), y también dos InDels dentro de este gen se correlacionan significativamente con el ancho del pecho, el ancho de la cadera y otros rasgos de crecimiento en cabras (46). Además, este gen ha mostrado funciones relacionadas con el crecimiento en diferentes especies. En cerdos de York, el análisis de asociación de todo el genoma de cinco rasgos de calidad de la carne encontró que 12 SNP intrón del gen SNX29 estaban asociados con el contenido de grasa intramuscular (47). Por lo tanto, el SNX29 ha sido identificado como un gen candidato asociado con rasgos de crecimiento, cuyas CNV también pueden actuar como una influencia en los rasgos de crecimiento del ganado. Continuaremos explorando el mecanismo molecular entre este gen y los rasgos de crecimiento en estudios posteriores
Conclusión
En este estudio, el efecto de crecimiento del gen SNX29 se dilucidó a partir de los aspectos de la estructura de la proteína, las propiedades fisicoquímicas y la variación del ADN. La proteína codificada por SNX29 fue una proteína no secretada, cuyas cinco CNV se identificaron en cabras SBWC, cabras GB y cabras NB. Además, se encontró que las CNV estaban asociadas con rasgos de crecimiento en cabras SBWC. El CNV1, CNV3, CNV4 y CNV5 se asociaron significativamente con las cabras SBWC, cabras GB y cabras NB (P < 0,01). Por lo tanto, el gen SNX29 puede ser un gen candidato funcional esencial para los rasgos de crecimiento.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio se incluyen en el artículo / Material complementario, las consultas adicionales pueden dirigirse a los autores correspondientes.
Declaración ética
El estudio en animales fue revisado y aprobado por la Universidad Northwest A&F. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones del autor
XS, HZ, XW, ZG y CP: recolección de muestras. QW y YB: operación experimental. QW, YB, HZ, XW y ZG: recopilación y análisis de datos. QW: redacción de artículos. QW, YB y CP: revisión y edición de manuscritos. ML y CP: gestión de proyectos. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Este estudio fue apoyado por la Fundación Nacional de Ciencias Naturales de China (No.32002166).
Reconocimientos
Los autores agradecen sinceramente a la granja de cabras Shaanxi Yulin, la granja de cabras Guizhou Bijie y la cooperativa de cabras Fujian ZhangZhou por proporcionarles muestras. Los autores también desean agradecer a Lei Qu, Hailong Yan y Jinwang Liu de la Universidad de Yulin por su ayuda en la recolección de muestras, y a XW por su ayuda en el Instituto de Ganadería y Veterinaria de la Academia de Ciencias Agrícolas de Fujian.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1132833/full#supplementary-material
Referencias
1. Hao X, Wang Y, Ren F, Zhu S, Ren Y, Jia B, et al. SNX25 regula la señalización de TGF-β al mejorar la degradación del receptor. Señal celular. (2011) 23:935–46. doi: 10.1016/j.cellsig.2011.01.022
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Lin YJ, Chang JS, Liu X, Lin TH, Huang SM, Liao CC, et al. La clasificación de la variación genética de la nexina 24 se asocia con la gravedad del aneurisma de la arteria coronaria en pacientes con enfermedad de Kawasaki. Biosci celular. (2013) 3:44. doi: 10.1186/2045-3701-3-44
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Amatya B, Lee H, Asico LD, Konkalmatt P, Armando I, Felder RA, et al. Subfamilia de SNX en la regulación de la señalización mediada por receptores y el tráfico de membrana. Int J Mol Sci. (2021) 22:2319. DOI: 10.3390/IJMS22052319
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Xia L, Ou J, Li K, Guo H, Hu Z, Bai T, et al. El análisis de asociación de todo el genoma del autismo identificó múltiples loci que se han reportado como señales fuertes para trastornos neuropsiquiátricos. Autismo Res. (2020) 13:382–96. doi: 10.1002/aur.2229
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Chen JH, Zhao Y, Khan RAW, Li ZQ, Zhou J, Shen JW, et al. SNX29, un nuevo gen de susceptibilidad compartido con los principales trastornos mentales en la población china Han. World J Biol Psiquiatría. (2021) 22:526–34. doi: 10.1080/15622975.2020.1845793
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Twa DD, Mottok A, Chan FC, Ben-Neriah S, Woolcock BW, Tan KL, et al. Reordenamientos genómicos recurrentes en el linfoma testicular primario. J Pathol. (2015) 236:136–41. doi: 10.1002/path.4522
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Zhu L, Hu Z, Liu J, Gao J, Lin B. El análisis del perfil de expresión génica identifica metástasis y genes asociados a quimiorresistencia en células de carcinoma epitelial de ovario. Med Oncol. (2015) 32:426. DOI: 10.1007/S12032-014-0426-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Sparks AM, Watt K, Sinclair R, Pilkington JG, Pemberton JM, McNeilly TN, et al. La arquitectura genética de las respuestas inmunes específicas de helmintos en una población salvaje de ovejas Soay (Ovis aries). PLoS Genet. (2019) 15:E1008461. doi: 10.1371/journal.pgen.1008461
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Peng S, Song C, Li H, Cao X, Ma Y, Wang X, et al. El ARN circular SNX29 esponja miR-744 para regular la proliferación y diferenciación de mioblastos mediante la activación de la vía de señalización Wnt5a/Ca2+. Mol Terapia Nucl Ácidos. (2019) 16:481–93. doi: 10.1016/j.omtn.2019.03.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Chen Y, Yang L, Lin X, Peng P, Shen W, Tang S, et al. Efectos de la variación genética del gen de la nexina 29 (SNX29) en los rasgos de crecimiento de la cabra negra de xiangdong. Animales. (2022) 12:3461. doi: 10.3390/ani12243461
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Wright D, Boije H, Meadows JR, Bed’hom B, Gourichon D, Vieaud A, et al. (2009). La variación del número de copias en el intrón 1 de SOX5 causa el fenotipo Pea-comb en pollos. iPLoS Geneti. 5, e1000512. doi: 10.1371/journal.pgen.1000512
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Henkel J, Saif R, Jagannathan V, Schmocker C, Zeindler F, Bangerter E, et al. Las firmas de selección en cabras revelan variantes de número de copias subyacentes a los fenotipos de color de pelaje que definen la raza. PLoS Genet. (2019) 15:E1008536. doi: 10.1371/journal.pgen.1008536
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Zhang RQ, Wang JJ, Zhang T, Zhai HL, Shen W. Copy-number variation in goat genome sequence: a comparative analysis of the different litter size trait groups. Gen. (2019) 696:40–6. doi: 10.1016/j.gene.2019.02.027
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Wang, L, Xu L., Liu X., Zhang T., Li N., Hay el H., et al. El estudio de asociación de genoma amplio basado en la variación del número de copias revela variantes adicionales que contribuyen a la calidad de la carne en cerdos. Sci Rep. (2015) 5:12535. DOI: 10.1038/SEP12535
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Kang X, Li M, Liu M, Liu S, Pan MG, Wiggans GR, et al. El análisis de variación del número de copias revela variantes asociadas con los rasgos de producción de leche en cabras lecheras. Genómica. (2020) 112:4934–7. doi: 10.1016/j.ygeno.2020.09.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Fernandes AC, da Silva VH, Goes CP, Moreira GC, Godoy TF, Ibelli AM, et al. Detección de CNV en todo el genoma y su asociación con rasgos de rendimiento en pollos de engorde. BMC Genom. (2021) 22:354. DOI: 10.1186/S12864-021-07676-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Strillacci MG, Gorla E, Ríos-Utrera A, Vega-Murillo VE, Montaño-Bermudez M, Garcia-Ruiz A, et al. Copy number variation mapping and genomic variation of autochthonous and commercial turkey populations. Front Genet. (2019) 10:982. doi: 10.3389/fgene.2019.00982
18. Pös O, Radvanszky J, Buglyó G, Pös Z, Rusnakova D, Nagy B, et al. Copy number variation: main characteristics, evolutionary significance, and pathological aspects. Biomed J. (2021) 44:548–59. doi: 10.1016/j.bj.2021.02.003
19. Bi Y, Feng B, Wang Z, Zhu H, Qu L, Lan X, et al. Myostatin (MSTN) gene indel variation and its associations with body traits in Shaanbei White Cashmere Goat. Animals. (2020) 10:168. doi: 10.3390/ani10010168
20. Wei Z, Wang K, Wu H, Wang Z, Pan C, Chen H, et al. Detection of 15-bp deletion mutation within PLAG1 gene and its effects on growth traits in goats. Animals. (2021) 11:2064. doi: 10.3390/ani11072064
21. Cai HF, Chen Z, Luo WX. Associations between polymorphisms of the GFI1B gene and growth traits of indigenous Chinese goats. Genet Mol Res. (2014) 13:872–80. doi: 10.4238/2014.February.13.5
22. Kholif AE, Gouda GA, Hamdon HA. Performance and milk composition of Nubian goats as affected by increasing level of nannochloropsis oculata microalgae. Animals. (2020) 10:2453. doi: 10.3390/ani10122453
23. Li X, Ding X, Liu L, Yang P, Yao Z, Lei C, et al. Copy number variation of bovine DYNC1I2 gene is associated with body conformation traits in Chinese beef cattle. Gene. (2022) 810:146060. doi: 10.1016/j.gene.2021.146060
24. Aljanabi SM, Martinez I. Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques. Nucleic Acids Res. (1997) 25:4692–3. doi: 10.1093/nar/25.22.4692
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Cui Y, Chen R, Lv X, Pan C. Detección de la secuencia codificante, expresión de ARNm y tres inserciones/deleciones (indeles) del gen KDM6A en cerdos machos. Teriogenología. (2019) 133:10–21. doi: 10.1016/j.theriogenology.2019.04.023
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Zhang X, Yu S, Yang Q, Wang K, Zhang S, Pan C, et al. Bola caprina: identificación de isoformas, expresión de ARNm en testículos y estudio funcional y perfiles de metilación promotora. Teriogenología. (2018) 116:53–63. doi: 10.1016/j.theriogenology.2018.05.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Fu W, Wang R, Yu J, Hu D, Cai Y, Shao J, et al. Una base de datos de variación del genoma de cabra para rastrear el proceso evolutivo dinámico de firmas selectivas e introgresiones antiguas. J Genet Genom. (2021) 48:248–56. doi: 10.1016/j.jgg.2021.03.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Wang Q, Bi Y, Wang Z, Zhu H, Liu M, Wu X, et al. SNX29 de cabra: expresión de ARNm, detección de indel y CNV, y sus asociaciones con el tamaño de la camada. Front Vet Sci. (2022) 9:981315. DOI: 10.3389/FVETS.2022.981315
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Li J, Zhang S, Erdenee S, Sun X, Dang R, Huang Y, et al. Variantes de nucleótidos en el gen de la proteína relacionada con priones (específica del testículo) (PRNT) y efectos sobre los fenotipos de ovejas chinas y mongolas. Prion. (2018) 12:185–96. doi: 10.1080/19336896.2018.1467193
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Yang Q, Zhang S, Li J, Wang X, Peng K, Lan X, et al. Desarrollo de un método de PCR multiplex de toma para detectar simultáneamente rápidamente tres nuevas inserciones/deleciones (indeles) dentro de un gen: un ejemplo para el gen GHR de cabra. Anim Biotechnol. (2019) 30:366–71. doi: 10.1080/10495398.2018.1517770
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Bi Y, Feng W, Kang Y, Wang K, Yang Y, Qu L, et al. Detección de la expresión de ARNm y variaciones en el número de copias dentro del gen FecB de cabra asociado con el tamaño de la camada. Front Vet Sci. (2021) 8:758705. doi: 10.3389/fvets.2021.758705
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Yang Y, Hu H, Mao C, Jiang F, Lu X, Han X, et al. Detección de la mutación de la secuencia de nucleótidos 23 pb en el gen del receptor huérfano alfa relacionado con el receptor de ácido retinoide (RORA) y su efecto sobre el tamaño de la camada ovina. Anim Biotechnol. (2020) 33:70–8. doi: 10.1080/10495398.2020.1770273
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Liu H, Xu H, Lan X, Cao X, Pan C. Las variantes InDel del del gen IGF2BP1 de ovejas están asociadas con rasgos de crecimiento. Anim Biotechnol. (2021) 13:1–9. doi: 10.1080/10495398.2021.1942029
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Edea Z, Hong JK, Jung JH, Kim DW, Kim YM, Kim ES, et al. Detección de firmas de selección entre poblaciones de cerdos sintéticos Duroc y Duroc utilizando chip SNP de alta densidad. Anim Genet. (2017) 48:473–7. doi: 10.1111/age.12559
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Lin S, Zhang H, Hou Y, Liu L, Li W, Jiang J, et al. Descubrimiento e identificación de genes candidatos funcionales para la composición de la leche basada en la resecuenciación del genoma completo de toros Holstein con valores de reproducción extremadamente altos y bajos. PLoS ONE. (2019) 14:E0220629. doi: 10.1371/journal.pone.0220629
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Muirhead G, Dev KK. La expresión de la nexina 8 de clasificación neuronal (SNX8) exacerba los niveles anormales de colesterol. J Mol Neurosci. (2014) 53:125–34. DOI: 10.1007/S12031-013-0209-Z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. An B, Xu L, Xia J, Wang X, Miao J, Chang T, et al. Análisis de asociación múltiple de loci y genes candidatos que regulan el tamaño corporal en tres etapas de crecimiento en ganado de carne Simmental. BMC Genet. (2020) 21:32. DOI: 10.1186/S12863-020-0837-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Castillejo-Lopez C, Pjanic M, Pirona AC, Hetty S, Wabitsch M, Wadelius C, et al. La caracterización funcional detallada de un locus de relación cintura-cadera en 7p15.2 define un potenciador que controla la diferenciación de los adipocitos. iScience. (2019) 20:42–59. doi: 10.1016/j.isci.2019.09.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Guo L, Sun H, Zhao Q, Xu Z, Zhang Z, Liu D, et al. Firmas de selección positivas en la población de cerdos blancos de seis extremos de Anqing basadas en datos de secuenciación del genoma de representación reducida. Anim Genet. (2021) 52:143–54. doi: 10.1111/age.13034
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Schultz J, Copley RR, Doerks T, Ponting CP, Bork P. SMART a web-based tool for the study of genetically mobile domains. Ácidos nucleicos res. (2000) 28:231–4. doi: 10.1093/nar/28.1.231
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Zhang Y, Wang K, Liu J, Zhu H, Qu L, Chen H, et al. Un polimorfismo de Indel de 11 pb dentro del gen CSN1S1 se asocia con el rendimiento de la leche y los rasgos de medición corporal en cabras chinas. Animales. (2019) 9:1114. DOI: 10.3390/ANI9121114
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Huang Y, Su P, Akhatayeva Z, Pan C, Zhang Q, Lan X, et al. Las nuevas variaciones InDel del gen Cry2 están asociadas con el tamaño de la camada en ovejas blancas australianas. Teriogenología. (2022) 179:155–61. doi: 10.1016/j.theriogenology.2021.11.023
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Zhou T, Wei H, Li D, Yang W, Cui Y, Gao J, et al. La nueva mutación sin sentido dentro del dominio del gen de la lisina desmetilasa 4D (KDM4D) está fuertemente asociada con los rasgos morfológicos de los testículos en cerdos. Anim Biotechnol. (2020) 31:52–8. doi: 10.1080/10495398.2018.1531880
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Wang Z, Ma H, Xu L, Zhu B, Liu Y, Bordbar F, et al. El escaneo de todo el genoma identifica firmas de selección en ganado Wagyu chino utilizando una matriz de SNP de alta densidad. Animales. (2019) 9:296. DOI: 10.3390/ANI9060296
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Liu M, Woodward-Greene J, Kang X, Pan MG, Rosen B, Van Tassell CP, et al. El análisis de CNV de todo el genoma reveló variantes asociadas con rasgos de crecimiento en cabras indígenas africanas. Genómica. (2020) 112:1477–80. doi: 10.1016/j.ygeno.2019.08.018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Bi Y, Chen Y, Xin D, Liu T, He L, Kang Y, et al. Efecto de las variantes indel dentro del gen de clasificación de nexina 29 (SNX29) sobre los rasgos de crecimiento de cabras. Anim Biotechnol. (2020) 19:1–6. doi: 10.1080/10495398.2020.1846547
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Dong Q, Liu H, Li X, Wei W, Zhao S, Cao JA, et al. Estudio de asociación de todo el genoma de cinco rasgos de calidad de la carne en cerdos de Yorkshire. Frente Agric Sci Eng. (2014) 1:137–43. doi: 10.15302/J-FASE-2014014
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: clasificación del gen nexina 29 (SNX29), variación del número de copias (CNV), rasgos de crecimiento, cabras, selección asistida por marcadores (MAS)
Cita: Wang Q, Song X, Bi Y, Zhu H, Wu X, Guo Z, Liu M y Pan C (2023) Distribución de detección de CNV de SNX29 en tres razas de cabras y sus asociaciones con rasgos de crecimiento. Frente. Vet. Sci. 10:1132833. doi: 10.3389/fvets.2023.1132833
Recibido: 28 de diciembre de 2022; Aprobado: 17 de julio de 2023;
Publicado: 29 agosto 2023.
Editado por:
Anupama Mukherjee, Consejo Indio de Investigación Agrícola (ICAR), India
Revisado por:
Zhuanjian Li, Universidad Agrícola de Henan, China
Tatiana Deniskova, L.K. Ernst Federal Science Center for Animal Husbandry (RAS), Rusia
Derechos de autor © 2023 Wang, Song, Bi, Zhu, Wu, Guo, Liu y Pan. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Chuanying Pan, chuanyingpan@126.com; Mei Liu, Mei.Liu@hunau.edu.cn
†Estos autores han contribuido igualmente a este trabajo
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía