Metabonómica plasmática de cerdos infectados por el virus de la peste porcina clásica
Metabonómica plasmática de cerdos infectados por el virus de la peste porcina clásica
 Jiedan Liao1,2
Jiedan Liao1,2  Wenshuo Hu2
Wenshuo Hu2  Weijun Wang2
Weijun Wang2  Xinyan Wang2
Xinyan Wang2  Shu Yu2
Shu Yu2  Xinni Niu2
Xinni Niu2  Wenhui Zhu2
Wenhui Zhu2  Bolun Zhou2
Bolun Zhou2  Canción de Yiwan2
Canción de Yiwan2  Weijun Zeng2
Weijun Zeng2  Zhimin Lu2
Zhimin Lu2  Jinding Chen2*
Jinding Chen2*- 1Facultad de Ciencias de la Vida e Ingeniería, Universidad de Foshan, Foshan, China
- 2Facultad de Medicina Veterinaria, Universidad Agrícola del Sur de China, Guangzhou, China
La peste porcina clásica (PPC) es una enfermedad infecciosa causada por el virus de la peste porcina clásica (PPC), que se caracteriza por depresión, fiebre alta, hemorragia cutánea extensa, leucopenia, anorexia, estreñimiento alterno y diarrea. El infarto hemorrágico del bazo es el principal cambio patológico característico tras la infección por el virus de la peste porcina clásica. Los brotes a gran escala de peste porcina clásica son raros en China y se distribuyen principalmente a nivel regional. Los síntomas clínicos del LCR no son evidentes y muestran variaciones de síntomas típicos a atípicos, lo que dificulta el diagnóstico basado en los síntomas clínicos y la patología. En los últimos años, la incidencia de granjas porcinas inmunizadas con peste porcina clásica en China ha aumentado y han aparecido nuevos subtipos de genes del virus de la peste porcina clásica, lo que plantea nuevos desafíos para la prevención y el control de la peste porcina clásica en China. Los cambios en los metabolitos causados por la infección viral reflejan el proceso patogénico. La metabonómica puede revelar los metabolitos traza de los organismos; sin embargo, rara vez se ha investigado la metabonómica plasmática de cerdos infectados con el virus de la peste porcina clásica. Por lo tanto, utilizamos un modelo establecido de infección por el virus de la peste porcina clásica para estudiar los cambios en los metabolitos plasmáticos. Los resultados mostraron diferencias significativas en cuarenta y cinco metabolitos plasmáticos en diferentes períodos de tiempo después de la infección por el virus de la peste porcina clásica en cerdos, con un aumento en veinticinco metabolitos y una disminución en veinte metabolitos. Estos metabolitos modificados se atribuyeron principalmente al ciclo del ácido tricarboxílico, el ciclo de los aminoácidos, el metabolismo del azúcar y el metabolismo de las grasas. Trece vías metabólicas cambiaron significativamente en cerdos infectados con PPC, incluido el ciclo del ácido tricarboxílico, el metabolismo del fosfato de inositol, el metabolismo de la glicina, la serina y la treonina, la degradación de la lisina, el metabolismo de la alanina, el aspartato y el ácido glutámico, la biosíntesis de pantotenato y CoA, el metabolismo de la β-alanina, la degradación de la lisina, el metabolismo de la arginina y la prolina, el metabolismo de los glicerolípidos, el metabolismo de la fenilalanina, el metabolismo del ácido araquidónico y el metabolismo del ácido linoleico. Entre estos, los cambios en la biosíntesis y el metabolismo de los ácidos grasos ocurrieron en todos los períodos de tiempo posteriores a la infección. Estos resultados indican que la infección por el virus de la peste porcina clásica en cerdos podría alterar seriamente las vías metabólicas.
Introducción
Los virus pueden utilizar la glucólisis de la célula huésped, el ciclo del ácido tricarboxílico, el metabolismo de los lípidos, el metabolismo de los aminoácidos y el metabolismo de los nucleótidos para promover su propia replicación en el huésped (1). Estudios recientes han demostrado que el citomegalovirus humano (HCMV) puede alterar muchos aspectos del metabolismo de la célula huésped, incluidas las vías metabólicas que son cruciales para la replicación viral (2). El virus de la hepatitis C puede utilizar la glucosa del huésped e inducir la desregulación de la biosíntesis lipídica después de establecer la infección (3). Cuando el cuerpo está infectado con un virus, la vía metabólica celular cambia para facilitar la replicación viral (4). En un estudio con ratones transgénicos del virus de la hepatitis B (VHB), se descubrió que el antígeno del VHB interfiere con el metabolismo de los lípidos en el hígado (5). Se descubrió que la envoltura viral del VHB absorbe los lípidos externos de las células huésped para satisfacer las necesidades de las membranas lipídicas virales (6).
La metabonómica es un enfoque que se basa en metabolitos traza para el análisis y la evaluación integrales, sistemáticos, cuantitativos y de alto rendimiento de organismos vivos en respuesta a un estímulo (como la infección por patógenos), lo que muestra un gran potencial en el descubrimiento de biomarcadores de enfermedades (7, 8). La compleja relación entre los patógenos y los huéspedes puede explicarse mejor mediante la genómica, la proteómica y la metabonómica. Las técnicas de análisis metabólico como la cromatografía de gases-espectrometría de masas (GC-MS), la cromatografía líquida-espectrometría de masas y la resonancia magnética nuclear combinadas con análisis estadísticos multivariados se utilizan ampliamente para proporcionar información valiosa, como los trastornos metabólicos inducidos por la estimulación después de la infección por virus (9). El análisis de metabolitos clave en los fluidos corporales se ha convertido en una parte importante para mejorar el diagnóstico, el tratamiento y el pronóstico de diversas enfermedades (10).
La peste porcina clásica (PPC) es una enfermedad infecciosa altamente contagiosa en cerdos y jabalíes causada por el virus de la peste porcina clásica (PPC). El LCR se caracteriza por inmunosupresión como hemorragia sistémica, septicemia y leucopenia (10). Nuestro estudio anterior mostró que la infección por el virus de la peste porcina clásica puede causar una gran reducción en los subconjuntos de leucocitos, acompañada de cambios en los índices metabólicos hepáticos y renales, lo que sugiere daño hepático y renal. Los estudios anatomopatológicos e histopatológicos han confirmado que algunos tejidos y órganos de cerdos infectados con el virus de la peste porcina clásica presentan inflamación y lesiones. Sobre la base de estos hallazgos, especulamos que la infección por el virus de la peste porcina clásica puede causar cambios en los metabolitos plasmáticos. Para comprender más profundamente la patogénesis del virus de la peste porcina clásica y explorar si los metabolitos plasmáticos después de la infección por el virus de la peste porcina clásica pueden servir como biomarcadores potenciales con valor diagnóstico, en el presente estudio, realizamos metabonómica plasmática para comparar cuantitativamente los metabolitos de cerdos no infectados e infectados por el virus de la peste porcina clásica en diferentes momentos posteriores a la infección. Este estudio podría ayudar a identificar nuevos biomarcadores del virus de la peste porcina clásica y proporciona una referencia para la patogénesis del virus de la peste porcina clásica y el establecimiento de nuevos métodos de diagnóstico clínico de enfermedades.
Materiales y métodos
Virus
Se utilizó veneno en la sangre de la cepa Shimen del virus de la peste porcina clásica como fuente de infección, que se conserva en el Departamento de Microbiología e Inmunología de la Facultad de Medicina Veterinaria de la Universidad Agrícola del Sur de China.
Anticuerpo
El anticuerpo monoclonal E2 anti-CSFV (9011) se compró a JBT Company of Korea. El anticuerpo secundario IgG (A0428) anti-ratón de cabra marcado con Alexa 488 se obtuvo de Beyotime.
Proliferación de virus y determinación de títulos
Cuando la monocapa de células PK-15 creció hasta una fusión del 80-90%, las células se lavaron dos veces con el medio Eagle modificado de Dulbecco (DMEM) sin suero ni antibióticos, se inocularon con sangre infectada con el virus de la peste porcina clásica y se adsorbieron a 37 °C durante 2 h. El frasco de cultivo se agitó suavemente cada 20 minutos para garantizar que la solución de VPC se distribuyera uniformemente por la superficie de la monocapa celular. Después de la adsorción, las células se lavaron dos veces con una solución de cultivo celular DMEM sin suero ni antibióticos, se incubaron con la solución de cultivo de mantenimiento celular y se cultivaron a 37 °C en un CO al 5%2 incubadora. Después de 72 h, la solución viral se cosechó y almacenó a -80 °C para su uso posterior.
Las células se introdujeron en una placa de cultivo celular de 96 pocillos y se inocularon con CSRF cuando la monocapa celular creció hasta alcanzar una confluencia del 80-90%. La solución vírica obtenida se diluyó a 10−10 con solución de cultivo celular DMEM sin suero ni antibióticos, y luego se inoculó la solución de VPC de cada gradiente en cuatro pocillos de la placa de cultivo celular a 0,1 mL/pocillo; Se utilizaron pozos en blanco como control. Después de la inoculación, las células se cultivaron de forma continua durante 2 días, y luego se retiró la placa de cultivo celular para la detección de inmunofluorescencia indirecta para calcular el título de infección por el virus de la peste porcina clásica. En detalle, el medio de cultivo se retiró cuidadosamente, se lavó con solución salina tamponada con fosfato (PBS) tres veces, se agregó etanol absoluto preenfriado a −20 °C y las celdas se fijaron a −20 °C durante 30 min. El etanol absoluto se desechó y las células se lavaron con PBS tres veces. Las células se incubaron con el anticuerpo de proteína E2 del virus de la peste porcina clásica (diluido con 0,01 mol/L de PBS; dilución 1:100) a 37 °C durante 1 h. Después de lavar con PBS tres veces, se añadió a la oscuridad IgG anti-ratón de cabra etiquetada con Alexa 488 (diluida con 0,01 mol/L de PBS; dilución 1:400) y se incubó a 37 °C durante 1 h. Las células se lavaron de nuevo con PBS tres veces y se observó la placa de cultivo celular bajo un microscopio de fluorescencia. El control en blanco se estableció para establecer la presencia de focos fluorescentes como positivos para el virus de la peste porcina clásica y la ausencia de focos fluorescentes como negativos para el virus de la peste porcina clásica. Se registró el número de pocillos con focos fluorescentes y se calculó el título de infección por el virus de la peste porcina clásica como la mediana de la dosis infecciosa del cultivo de tejidos (TCID50)/mL.
Agrupamiento de animales e infección
Se compraron diez cerdos miniatura tibetanos de 4 meses de edad y con un peso de entre 20 y 25 kg en el Centro de Animales Experimentales de la Universidad Médica del Sur. Se utilizó la reacción en cadena de la polimerasa con transcriptasa inversa y el inmunoensayo ligado a enzimas para detectar ácidos nucleicos y anticuerpos contra enfermedades infecciosas comunes en cerdos, incluidos el virus de la peste porcina clásica, el virus de la pseudorrabia, el virus del síndrome respiratorio y reproductivo porcino, el circovirus porcino tipo II, el parvovirus porcino y el virus de la fiebre aftosa tipo O. Se seleccionaron cerdos con índices negativos para todos los patógenos como cerdos experimentales.
Diez cerdos miniatura tibetanos que se confirmó que eran completamente negativos para los patógenos anteriores se dividieron aleatoriamente en dos grupos y se alimentaron por separado, con cinco cerdos en el grupo infectado por el virus de la peste porcina clásica (tres hembras, dos machos) y cinco cerdos en el grupo infectado simulado (tres hembras, dos machos). Los cerdos fueron observados y tratados con medicamentos antiparasitarios durante 1 semana. Antes del experimento, la temperatura corporal se midió a un tiempo fijo durante varios días consecutivos, y el valor promedio se tomó como la temperatura corporal basal. Se seleccionaron cerdos en buen estado de salud para experimentos formales.
Antes de la extracción de sangre, los cerdos fueron ayunados durante 12 h y se les permitió beber libremente. El día de la infección por el virus de la peste porcina clásica, se recogieron muestras de sangre y se inyectó sangre infectada con la cepa Shimen del virus de la peste porcina clásica a 1 × 105 TCID50/cabeza en los músculos del cuello, y a los otros cinco infectados simulados se les inyectó un volumen igual de solución salina normal en los músculos del cuello. El día de infección se estableció en 0 días después de la inoculación (DPI) y el primer día después de la infección fue de 1 DPI. Se recogieron muestras de sangre de cerdos infectados con el virus de la peste porcina clásica y el virus de la peste porcina clásica a una hora fija cada dos días.
Este experimento fue aprobado por el Comité de Ética de Animales Experimentales de la Universidad Agrícola del Sur de China, y todos los procedimientos estaban estrictamente de acuerdo con los requisitos de los experimentos con animales.
Recogida de muestras de plasma
Los resultados del experimento preliminar mostraron que no había diferencias significativas en los metabolitos plasmáticos entre los grupos infectados simulados. Con el fin de acercarse a los puntos de extracción de sangre del grupo infectado por el virus de la peste porcina clásica, se seleccionaron dos puntos temporales mg1 (1DPI) y mg2 (3DPI) para los grupos infectados simulados. La muestra de sangre (4 mL) se centrifugó a 4000 rpm durante 10 min, y el sobrenadante se recolectó y almacenó a -80 °C para su análisis. Se tomaron muestras de sangre en dos puntos para el grupo infectado simulado mg1 y mg2, mientras que las muestras de sangre se tomaron en cuatro puntos en el grupo infectado por el virus de la peste porcina clásica: cs-1DPI, cs-3DPI, cs-5DPI y cs-7DPI para la detección y comparación de metabolitos.
Extracción de metabolitos y derivatización de muestras
Veinte microlitros de muestras de plasma descongeladas y 80 μL de metanol helado se añadieron secuencialmente a un tubo electroproducitorio. La mezcla se agitó en vórtice durante 30 s, se almacenó a -20 °C durante 1 h y luego se centrifugó a 4 °C durante 15 min a 16.000 g. El sobrenadante (80 μL) se transfirió a 10 μL de patrón interno (0,02 mg/mL de L-fenilalanina-13C9–15N, 0,05 mg/mL de galactitol, L-leucina-13C6 y L-isoleucina-13C6–15N, 0,1 mg/ml de L-valina-13C5–15N y L-alanina-13C3–15N). La mezcla se secó bajo un flujo de nitrógeno suave y se añadieron 30 μL de 20 mg/mL de clorhidrato de metoxiamina (piridina anhidra) al residuo seco. La mezcla se agitó en vórtice durante 30 s y luego se incubó a 37°C durante 90 min. Se añadieron treinta microlitros de N,O-bis (trimetilsilil)trifluoroacetamida (que contenía 1% de trimetilclorosilano) a la mezcla y se derivatizó a 70°C durante 60 min.
Detección de GC-MS
Las muestras se analizaron con un cromatógrafo de gases Agilent 7890A y un espectrómetro de masas Agilent 5975C. Las condiciones cromatográficas fueron las siguientes: columna cromatográfica HP-5MS (30 m × 0,25 mm × 0,25 μm), helio (>99,999%) como gas portador y el caudal de la columna fue de 1 mL/min. El volumen de carga fue de 1 μL y el tiempo de retardo del disolvente fue de 6 min. La temperatura se mantuvo a 70 °C durante 2 min, se aumentó a 160 °C a una velocidad de 6 °C/min, se aumentó a 240 °C a una velocidad de 10 °C/min, se aumentó a 300 °C a una velocidad de 20 °C/min y finalmente se mantuvo a 300 °C durante 6 min. Las condiciones de la EM fueron las siguientes: temperatura de interfaz, 250 °C; temperatura de la varilla de la cuarta etapa, 290 °C; y la temperatura de la fuente de iones, 230 °C. El voltaje de ionización fue de 70 eV y se utilizó el modo de escaneo completo (m/z: 50-600) para recopilar datos.
Preprocesamiento de datos y análisis estadístico
Para corregir la respuesta del espectro de masas, se normalizaron la concentración y el área de pico total de cada muestra. El archivo de la tabla de picos (matriz X) se importó a Simca-P V11.0 (Umetrics AB, Suecia) para su centralización, y los datos se promediaron según la varianza unitaria. Después de la estandarización, se realizó un análisis estadístico multivariado, incluyendo el análisis de componentes principales (ACP) y el análisis discriminante de mínimos cuadrados parciales (PLS-DA). La calidad del modelo se describe mediante R2X (la tasa explicativa del modelo X) o R2Y (la cantidad observada de Y es la tasa explicativa de las diferencias entre los grupos) y Q2 (la tasa predictiva). R2X (PCA) o R2Y (PLS-DA) se define como la proporción de varianza en los datos explicados por el modelo e indica la bondad del ajuste. Q2 se define como la proporción de varianza en los datos que el modelo puede predecir e indica la predictibilidad del modelo actual, que se calcula a través del proceso de validación cruzada. Para evitar el sobreajuste del modelo, se realizó la validación cruzada predeterminada de 7 rondas, que proporcionó el mejor número de componentes principales según lo determinado en Simca-P. La R2X, R2Y y Q2 Los valores se utilizaron como indicadores para evaluar los principales parámetros simbólicos del modelo de reconocimiento de patrones. La significación estadística de los metabolitos diferenciales entre los grupos infectados y no infectados se evaluó mediante la prueba t de Student (p < 0,05).
Resultados
Inspección de datos
Los datos de titulación del virus de la peste porcina clásica para la misma cepa se han publicado anteriormente (11). La estabilidad del método de detección es un requisito previo importante para obtener datos metabolómicos fiables. En primer lugar, se sometió a cromatografía iónica total (TIC) a un grupo de muestras de plasma infectadas con el virus de la peste porcina clásica y a dos grupos de muestras de plasma infectadas con el virus de la peste porcina clásica. El TIC de todas las muestras mostró características de una señal fuerte, gran capacidad de pico y buena reproducibilidad del tiempo de retención, lo que indica que el método GC-MS adoptado en este estudio tiene buena reproducibilidad y estabilidad, y podría usarse para el análisis de alto rendimiento de muestras posteriores. En la Figura 1 se muestran los resultados representativos de las TIC para cada grupo.
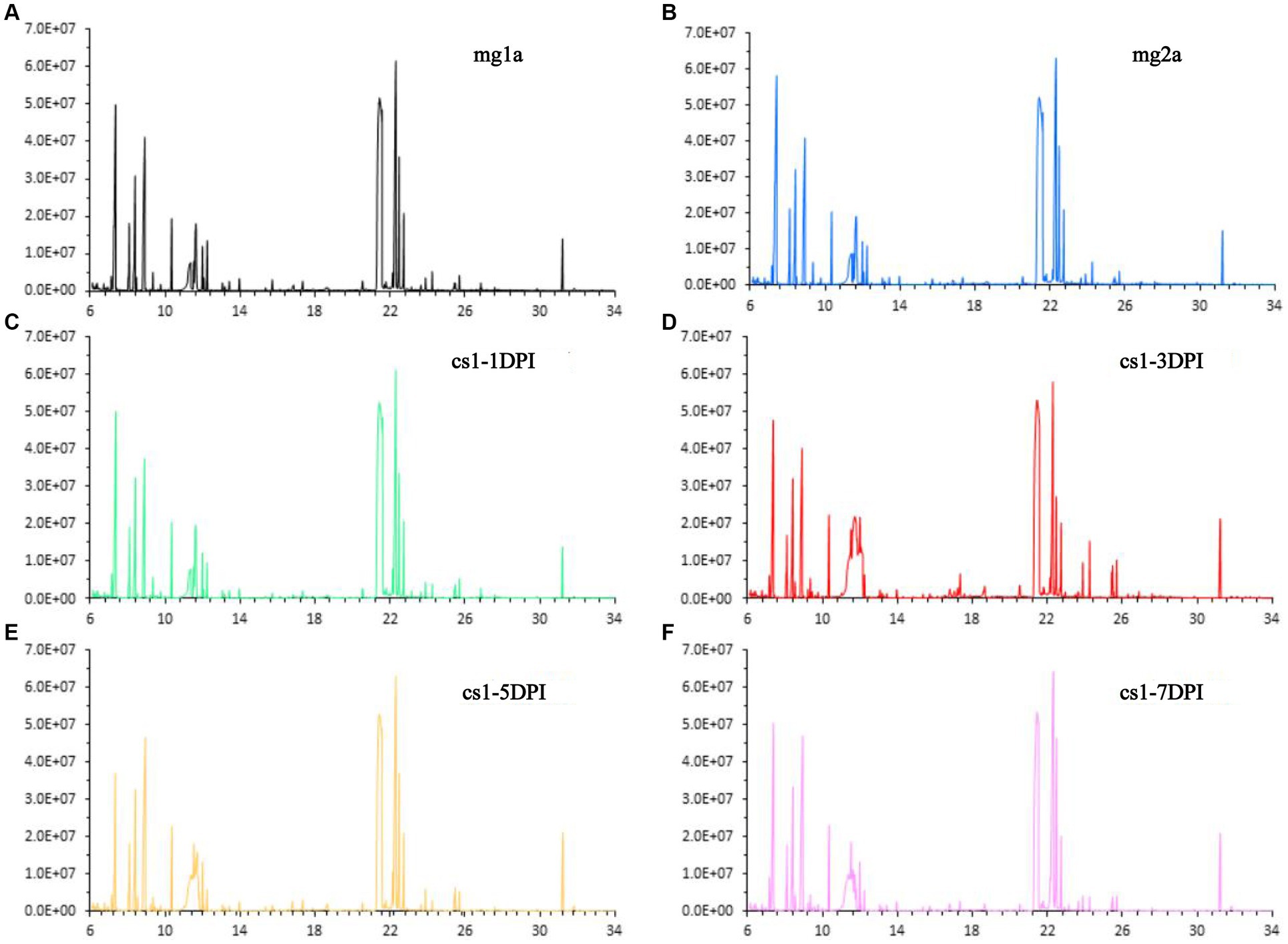 Figura 1. Cromatogramas iónicos totales representativos. (A) A-1DPI infectado simuladamente. (B) A-3DPI infectado simuladamente. (C) 1-1DPI infectado con el virus de la peste porcina clásica. (D) 1-3DPI infectado por el virus de la peste porcina clásica. (E) 1-5DPI infectado con CSFV. (F) 1-7DPI infectado con CSFV.
Figura 1. Cromatogramas iónicos totales representativos. (A) A-1DPI infectado simuladamente. (B) A-3DPI infectado simuladamente. (C) 1-1DPI infectado con el virus de la peste porcina clásica. (D) 1-3DPI infectado por el virus de la peste porcina clásica. (E) 1-5DPI infectado con CSFV. (F) 1-7DPI infectado con CSFV.
Análisis de datos multivariante
PCA
En primer lugar, utilizamos el ACP para revelar las diferencias en el agrupamiento y el patrón metabólico entre las muestras infectadas por el virus de la peste porcina clásica y las infectadas simuladas. El principal parámetro utilizado para juzgar la calidad del modelo de ACP fue la tasa explicativa acumulada R2X. Si R2X es mayor que 0.5, el modelo puede considerarse confiable. En la Figura 2A se muestra un gráfico de puntuación de la ACP y se obtuvieron cuatro componentes principales. La tasa explicativa acumulada del modelo fue R2X = 0,58, Q2 = 0,337. En general, una R2Un valor X mayor que 0,5 indica que el modelo es fiable; por lo tanto, el modelo de PCA obtenido se puede utilizar de manera confiable para reflejar las diferencias metabólicas entre los dos grupos de muestras. Todas las muestras estaban dentro del intervalo de confianza del 95% (elipse T2 de Hotelling) y no hubo muestras anormales. A partir de la gráfica de puntuación de PCA (Figura 2A, la abscisa indica el primer componente principal PC1, que está representado por t[1], y la ordenada indica el 4º componente principal PC4, que está representado por t[4]), no hubo una separación significativa entre las muestras de plasma de los mg1 y mg2 infectados con simulacros, mientras que hubo una separación significativa entre las muestras de plasma de los cs-1DPI infectados con el virus de la peste porcina clásica, cs-3DPI, cs-5DPI y cs-7DPI en PC1 y PC4, lo que indicó que se produjeron cambios metabólicos significativos a lo largo del tiempo después de la infección.
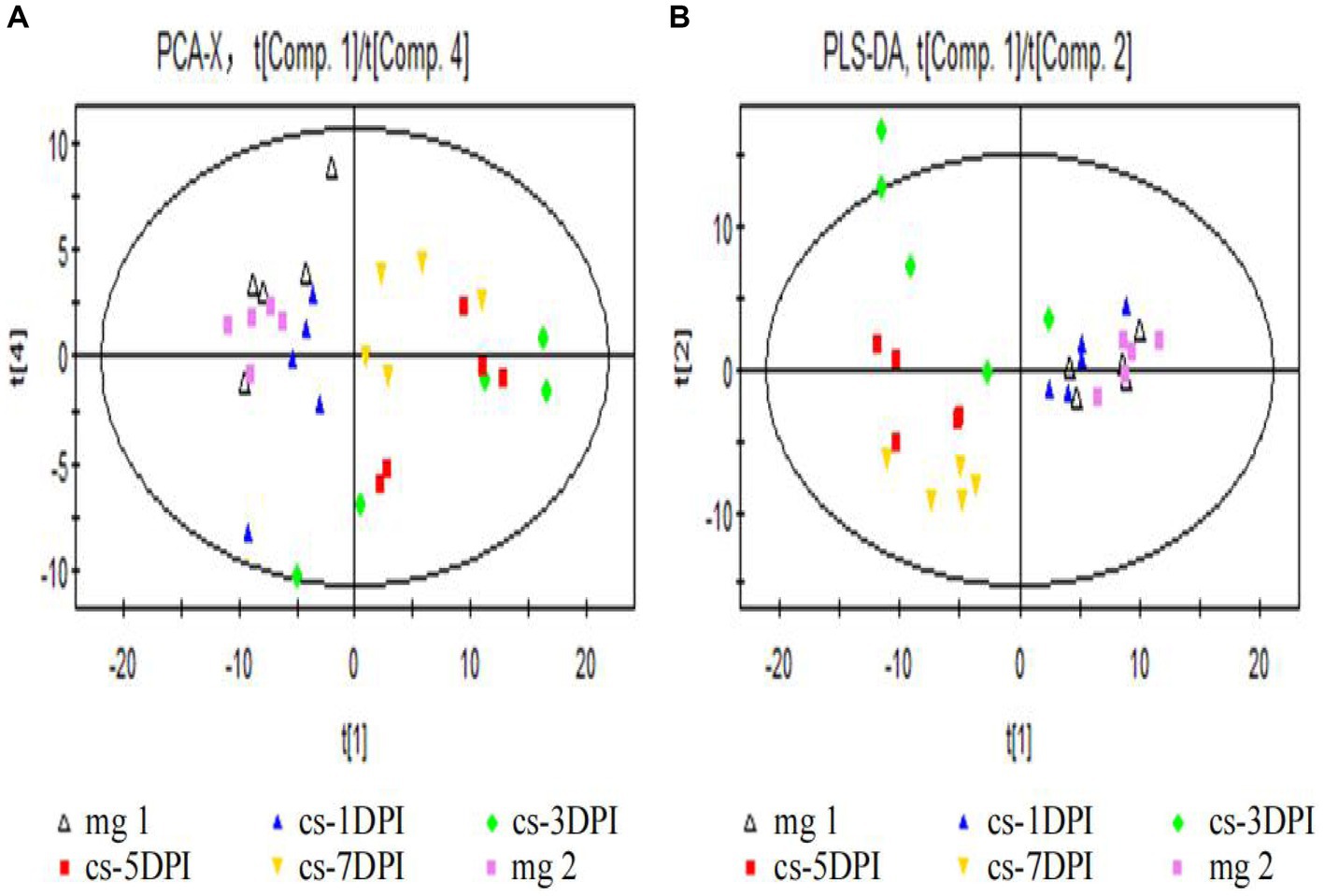 Figura 2. Puntuaciones generales de PCA (A) y PLS-DA (B) entre los grupos infectados por el virus de la peste porcina clásica y el virus de la peste porcina clásica. En todas las imágenes, mg1 y mg2 representaban grupos infectados por Mock (mg1-1DPI, mg2-3DPI), cs representaban grupos infectados por el virus de la peste porcina clásica.
Figura 2. Puntuaciones generales de PCA (A) y PLS-DA (B) entre los grupos infectados por el virus de la peste porcina clásica y el virus de la peste porcina clásica. En todas las imágenes, mg1 y mg2 representaban grupos infectados por Mock (mg1-1DPI, mg2-3DPI), cs representaban grupos infectados por el virus de la peste porcina clásica.
PLS-da
Para eliminar la influencia del ruido de fondo, se utilizó un análisis estadístico multidimensional supervisado por PLS-DA para analizar los modelos de los dos grupos de muestras. Este tipo de análisis se utiliza principalmente para eliminar la variación intragrupo, resaltando así las diferencias entre los grupos. En este estudio se obtuvieron siete componentes principales: R2X = 0,687, R2Y = 0,781 y Q2 = 0,272. El gráfico de puntuación se muestra en la Figura 2B (la abscisa indica el primer componente principal PC1, que está representado por t[1], y la ordenada representa el segundo componente principal PC2, representado por t[2]). Entre ellos, las muestras de mg1, mg2 y cs-1DPI infectadas simuladamente están en el lado derecho de PC1 (representadas por t[1]), mientras que las muestras cs-5DPI y cs-7DPI están en el lado izquierdo de PC1, y cinco muestras en cs-3DPI están en transición. La variación intragrupo de estas cinco muestras fue muy grande, lo que sugiere que el grupo cs-3DPI se encuentra en una etapa de cambio drástico. La tasa de explicación del modelo R2Y demostró que el modelo PLS-DA puede explicar las diferencias entre los cinco grupos de muestras. No hubo separación espectral significativa en PCA y PLS-DA entre los grupos infectados mg1 y mg2, lo que indica que no hubo diferencias metabólicas significativas entre los grupos. Con el fin de acercarse a la respuesta al estrés de los animales muestreados en el grupo infectado por el virus de la peste porcina clásica, se compararon las diferencias de metabolitos en el mg2 infectado por el virus de la peste porcina clásica con otros puntos temporales en el grupo infectado por el virus de la peste porcina clásica.
Puntuaciones de PCA y PLS-DA de grupos infectados por el virus de la peste porcina clásica y de simulacros en diferentes momentos
Día uno
En la Figura 3A se muestra el gráfico de puntuación de PCA para muestras infectadas por el virus simulado y el virus de la peste porcina clásica a 1 DPI, con dos componentes principales obtenidos. Las tasas explicativas acumuladas del modelo fueron R2X = 0,486 y Q2 = 0,0127. Por lo tanto, este modelo de PCA puede explicar de forma fiable las diferencias metabólicas entre los dos grupos de muestras. El cs-1DPI y el mg2 mostraron una separación evidente, lo que indica claras diferencias metabólicas entre los dos grupos.
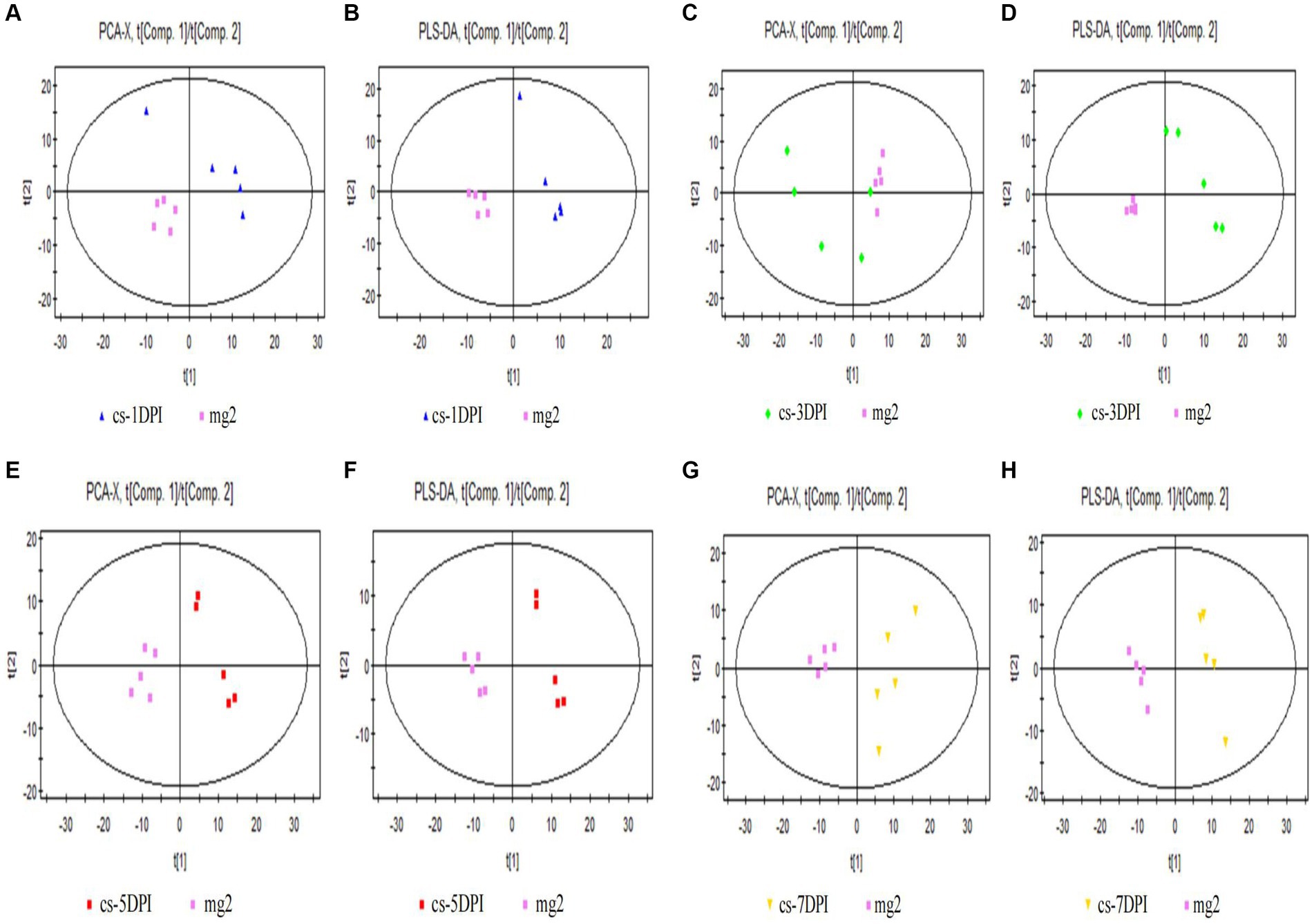 Figura 3. Gráficos de puntuación general de PCA y PLS-DA de infectados por el virus de la peste porcina clásica y mg2 en 1DPI (A,B), 3DPI (C,D), 5DPI (E,F) y 7DPI (G,H), respectivamente.
Figura 3. Gráficos de puntuación general de PCA y PLS-DA de infectados por el virus de la peste porcina clásica y mg2 en 1DPI (A,B), 3DPI (C,D), 5DPI (E,F) y 7DPI (G,H), respectivamente.
También se observaron diferencias significativas entre estos dos grupos en la tabla de puntuaciones PLS-DA. Se obtuvieron tres componentes principales: R2X = 0,562, R2Y = 0,998 y Q2 = 0,809. El gráfico de puntuación se muestra en la Figura 3B. Tanto la tasa de explicación del modelo R2Y y la tasa de predicción Q2 fueron muy altas, lo que indica que el modelo PLS-DA puede explicar y predecir las diferencias entre los dos grupos de muestras.
Tercer día
En la Figura 3C se muestra el gráfico de puntuación de PCA para muestras infectadas por el virus de la peste porcina clásica y simuladas a 3 DPI, con dos componentes principales obtenidos. Las tasas explicativas acumuladas del modelo fueron R2X = 0,578 y Q2 = 0,197. Por lo tanto, este modelo de PCA puede explicar de forma fiable las diferencias metabólicas entre los dos grupos de muestras. Los cs-3DPI y mg2 mostraron una separación significativa, lo que indica claras diferencias entre los dos grupos de muestras.
También hubo diferencias significativas (separación espectral) en las puntuaciones de PLS-DA entre estos dos grupos. Se obtuvieron dos componentes principales: R2X = 0,548, R2Y = 0,987 y Q2 = 0,873. El gráfico de puntuación se muestra en la Figura 3D. Tanto la tasa de explicación del modelo R2Y y la tasa de predicción Q2 fueron muy altas, lo que indica que el modelo PLS-DA puede explicar y predecir las diferencias entre los dos grupos de muestras.
Día cinco
En el panel izquierdo de la Figura 3E se muestra el gráfico de puntuación de PCA para muestras infectadas por el virus simulado y el virus de la peste porcina clásica a 5 DPI, con dos componentes principales obtenidos. Las tasas explicativas acumuladas del modelo fueron R2X = 0,565 y Q2 = 0,239. Por lo tanto, este modelo de PCA puede explicar de forma fiable las diferencias metabólicas entre los dos grupos de muestras. Los grupos cs-5DPI y mg2 se encuentran en dos posiciones distintas, lo que indica una diferencia significativa entre los dos grupos de muestras.
También hubo diferencias significativas (separación espectral) en las puntuaciones de PLS-DA entre estos dos grupos. Se obtuvieron dos componentes principales: R2X = 0,552, R2Y = 0,99 y Q2 = 0,924. El gráfico de puntuación se muestra en la Figura 3F. Tanto la tasa de explicación del modelo R2Y y la tasa de predicción Q2 fueron muy altos (cercanos a 1), lo que indica que el modelo PLS-DA se puede utilizar para explicar y predecir las diferencias entre los dos grupos de muestras de manera muy confiable.
Séptimo día
En la Figura 3G se muestra el gráfico de puntuación de PCA para muestras infectadas con PCA e infecciones por PCF a 7 DPI, con dos componentes principales obtenidos. La tasa explicativa acumulada del modelo fue R2X = 0,58, Q2 = 0,204. Por lo tanto, este modelo de PCA puede explicar de forma fiable las diferencias metabólicas entre los dos grupos de muestras. Los grupos cs-7DPI y mg2 se encontraban en dos posiciones diferentes, lo que indica una diferencia significativa entre los dos grupos.
También hubo diferencias significativas (separación espectral) en las puntuaciones de PLS-DA entre estos dos grupos. Se obtuvieron dos componentes principales: R2X = 0,558, R2Y = 0,993 y Q2 = 0,957. El gráfico de puntuación se muestra en la Figura 3H. Tanto la tasa de explicación del modelo R2Y y la tasa de predicción Q2 fueron muy altos (cercanos a 1), lo que indica que el modelo PLS-DA puede explicar y predecir bien las diferencias entre los dos grupos de muestras.
Análisis de la vía de metabolitos diferenciales entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados en diferentes momentos
Día uno
Metabolitos diferenciales
Al buscar metabolitos diferenciales, se examinó la significación entre los grupos infectados por el virus de la peste porcina clásica y los infectados por el virus de la peste porcina clásica mediante la prueba t de Student (con una p < 0,05). Se utilizó la primera variable componente principal de importancia en el valor de proyección (VIP) (umbral >1) del modelo PLS-DA para identificar los metabolitos expresados diferencialmente. Para la evaluación cualitativa de los cambios metabólicos, primero se buscó una base de datos de material de referencia de construcción propia, que incluyera la biblioteca Fiehn GC/MS Metabolomics RTL, la base de datos de metabolomas de Golm y la base de datos comercial del NIST. Los datos de metabolitos se presentan en la Tabla 1. En este estudio se examinaron e identificaron un total de treinta y nueve sustancias diferentes, incluidos veinte metabolitos con abundancia disminuida y diecinueve metabolitos con abundancia aumentada después de la infección.
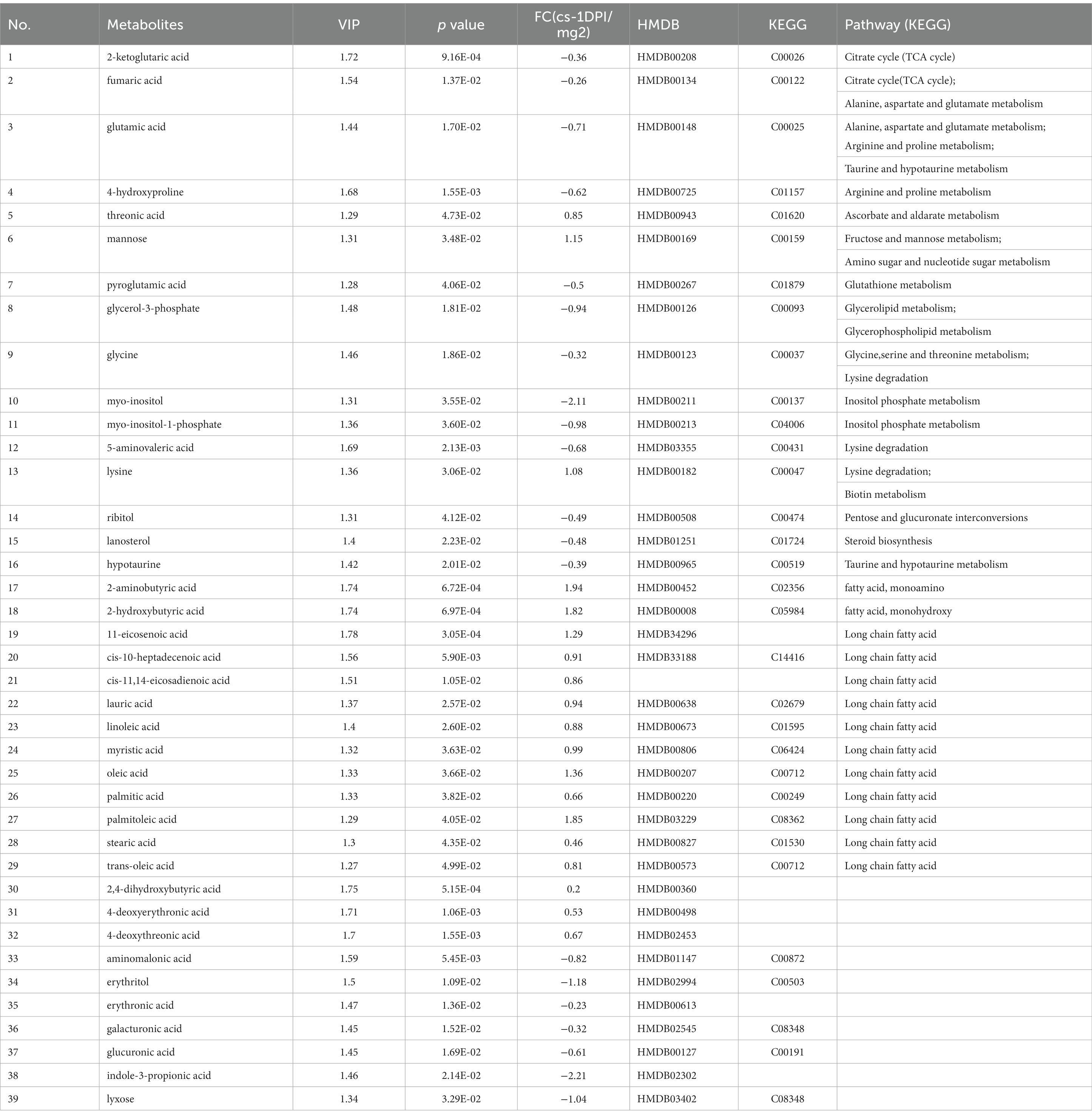 Tabla 1. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los infectados simulados al día siguiente de la infección.
Tabla 1. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los infectados simulados al día siguiente de la infección.
Como se muestra en la Tabla 1, para el DPI cs-1, las concentraciones de ácidos grasos aumentaron significativamente, mientras que las concentraciones de intermediarios del metabolismo lipídico, incluyendo glicerol-3-fosfato (fosfoglicerol deshidrogenasa) y mio-inositol-1-fosfato (inositol-1-fosfoglicerol), disminuyeron significativamente. Estos hallazgos indicaron que el anabolismo de los ácidos grasos a 1 DPI aumentó significativamente, lo que resultó en un gran consumo de dos materiales precursores utilizados para la síntesis de lípidos.
Análisis de vías
Para explorar las vías metabólicas que experimentaron cambios significativos en el plasma después de la infección por el virus de la peste porcina clásica, utilizamos el software MetaboAnalyst para realizar un análisis preliminar de las vías de los dos grupos de metabolitos diferentes. Como se muestra en la Figura 4A, la infección por LCR a 1 DPI causó cambios en ocho vías metabólicas: biosíntesis de ácidos grasos, metabolismo de ascorbato y aldarato, metabolismo del fosfato de inositol, degradación de la lisina, ciclo del ácido tricarboxílico, interconversión de pentosas y glucuronato, metabolismo del ácido linoleico y metabolismo de glicina, serina y treonina. Hubo cambios significativos en el ciclo del ácido tricarboxílico, el metabolismo del fosfato de inositol, el metabolismo de la glicina, la serina y la treonina, la degradación de la lisina (impacto >0,1). Los datos detallados de la vía se muestran en la Tabla 2, donde «total» indica el número total de metabolitos en la vía, «hits» indica el número de metabolitos diferenciales en esta vía, «–log(p)» indica el valor de ordenadas de la Figura 4A e «impacto» indica el valor de abscisa de la Figura 4A.
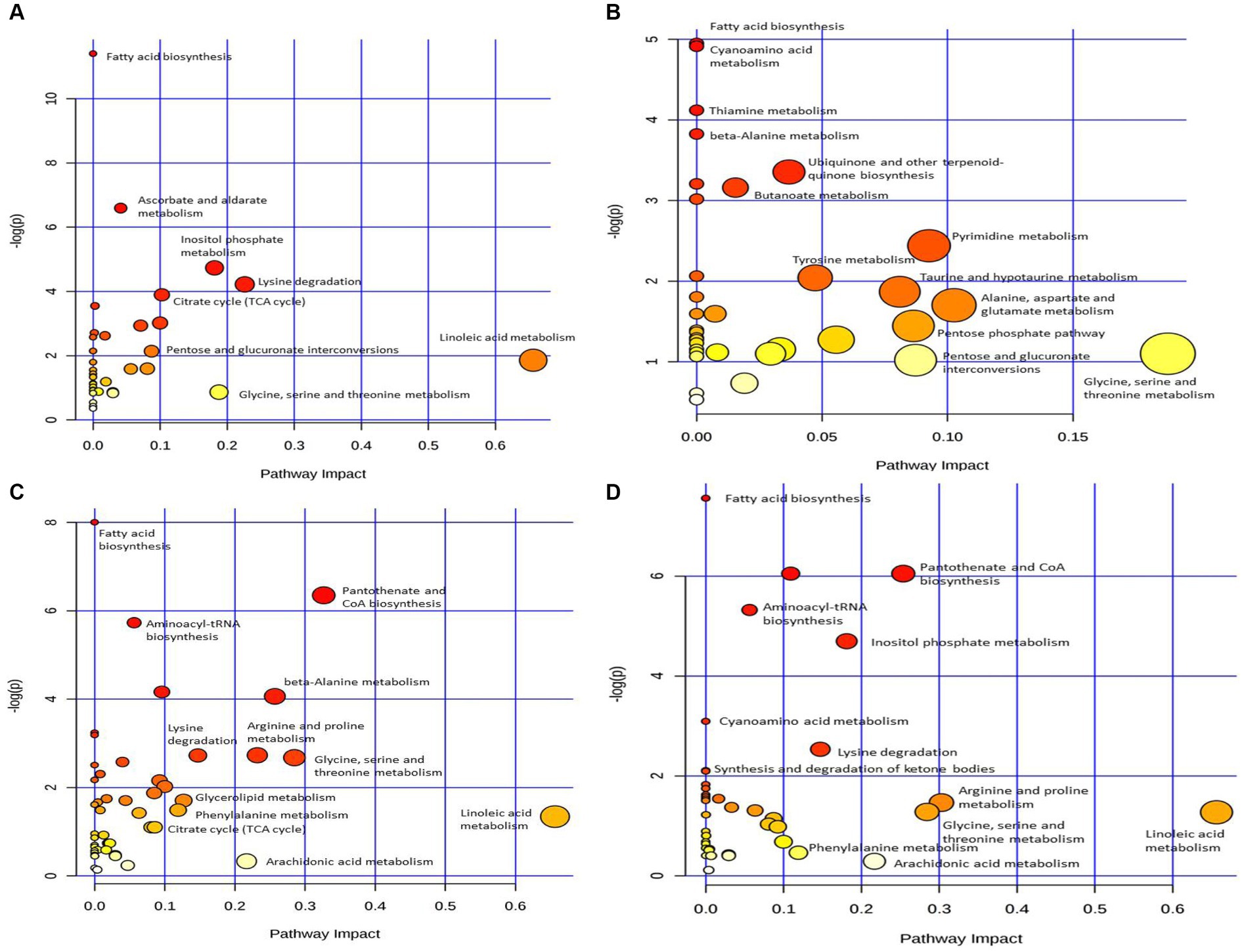 Figura 4. Análisis de la vía del metabolismo diferencial entre los infectados por el virus de la peste porcina clásica y los mg2 en 1DPI (A), 3DPI (B), 5DPI (C) y 7DPI (D), respectivamente.
Figura 4. Análisis de la vía del metabolismo diferencial entre los infectados por el virus de la peste porcina clásica y los mg2 en 1DPI (A), 3DPI (B), 5DPI (C) y 7DPI (D), respectivamente.
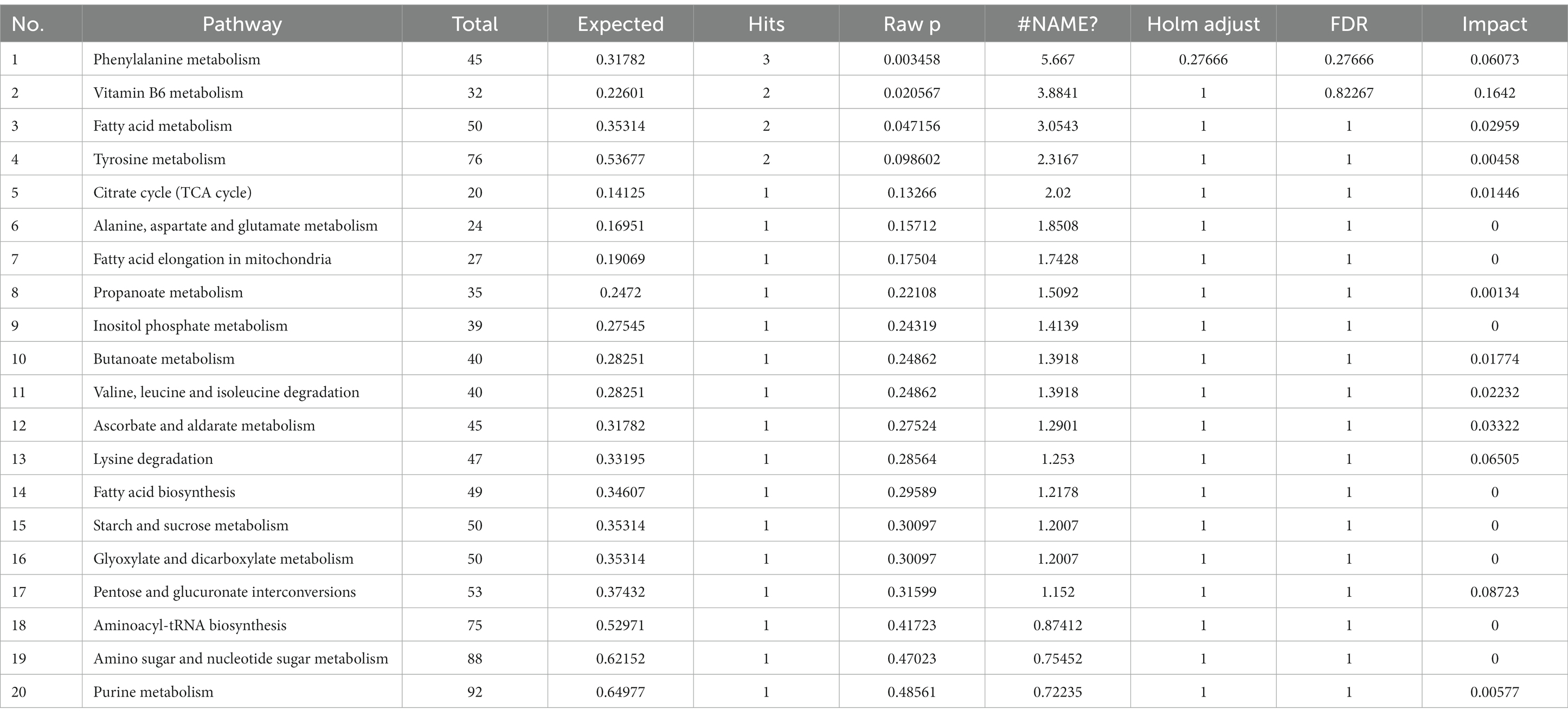 Tabla 2. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los infectados simulados 1 día después de la infección.
Tabla 2. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los infectados simulados 1 día después de la infección.
Tercer día
Metabolitos diferenciales
Los datos de los metabolitos que cambiaron a 3 DPI se presentan en la Tabla 3. Se examinaron e identificaron un total de veintiséis sustancias diferentes, incluidos ocho metabolitos con abundancia disminuida y dieciocho metabolitos con abundancia aumentada a 3 DPI.
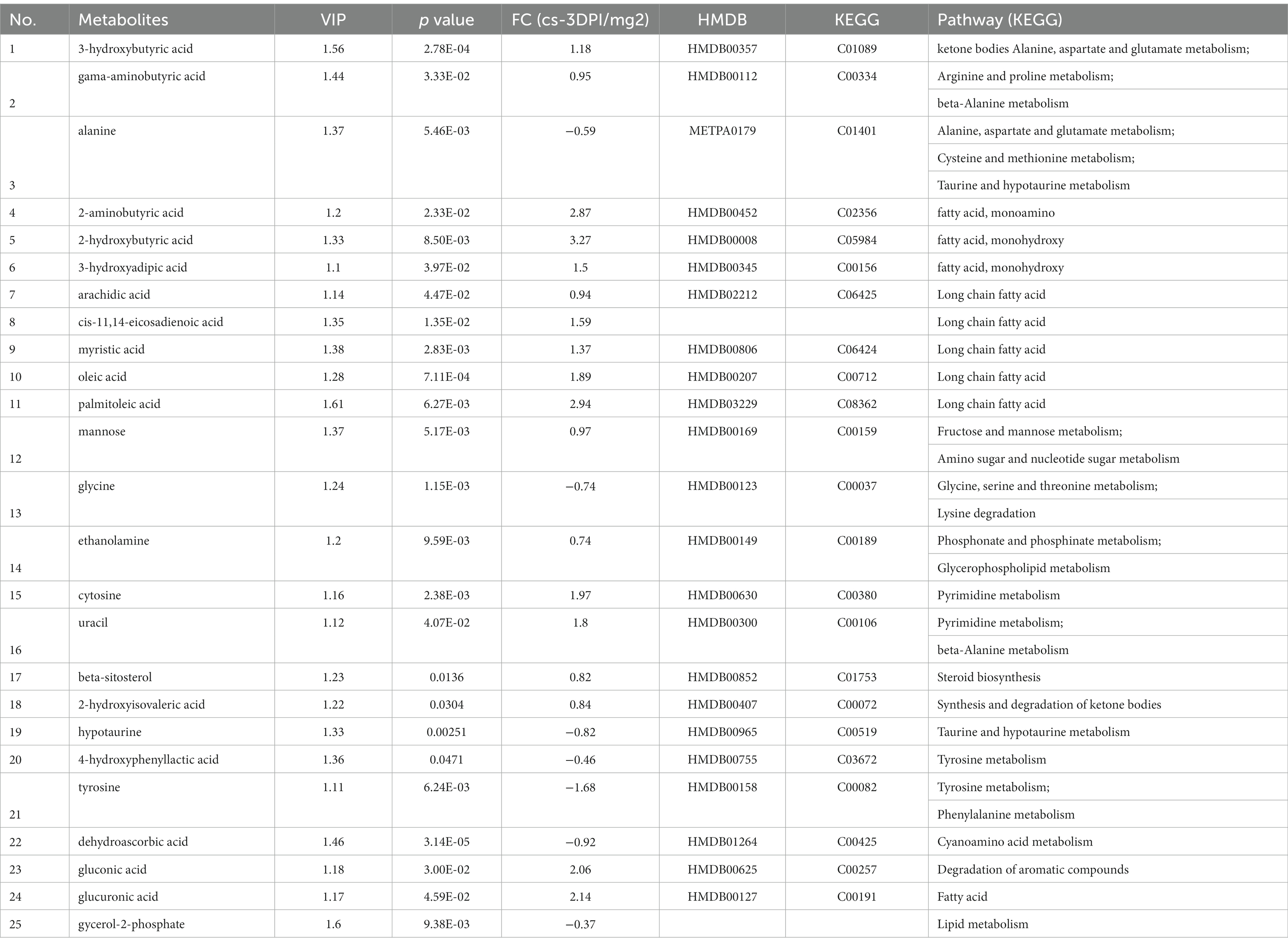 Tabla 3. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 3 días después de la infección.
Tabla 3. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 3 días después de la infección.
Análisis de vías
La figura 4B muestra que la infección por el virus de la peste porcina clásica a 3 DPI causó cambios en la biosíntesis de ácidos grasos, el metabolismo de los cianoaminoácidos, el metabolismo de la tiamina, el metabolismo de la β-alanina, la biosíntesis de ubiquinona y otros terpenoides quinonas, el metabolismo del butanoato, el metabolismo de la tirosina, el metabolismo de la pirimidina, el metabolismo de la taurna y la hipotaurina, la vía de las pentosas fosfato, las interconversiones de pentosa y glucuronato, el metabolismo de la alanina, el aspartato y el ácido glutámico, el metabolismo de la glicina, la serina y la treonina. Entre estas vías, hay cambios significativos en el metabolismo de la alanina, el aspartato y el ácido glutámico, el metabolismo de la glicina, la serina y la treonina (impacto >0,1). El análisis del diagrama de la vía metabólica se presenta en la Tabla 4.
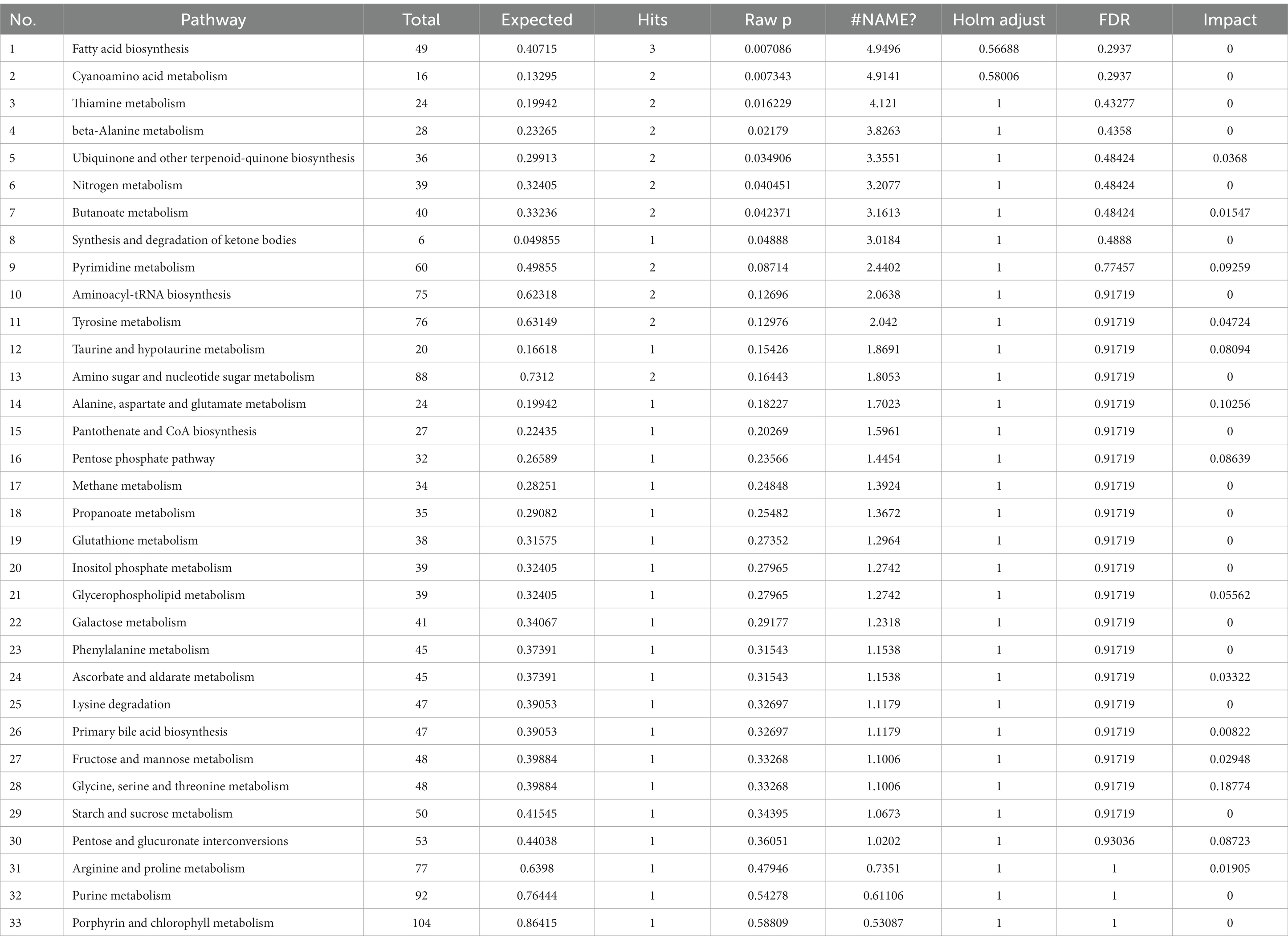 Tabla 4. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 3 días después de la infección.
Tabla 4. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 3 días después de la infección.
Día cinco
Metabolitos diferenciales
Los datos de los metabolitos que cambiaron a 5 DPI se presentan en la Tabla 5. Se examinaron e identificaron un total de sesenta y cinco sustancias diferentes, incluidos veinte metabolitos con abundancia disminuida y cuarenta y cinco metabolitos con abundancia aumentada a 5 DPI.
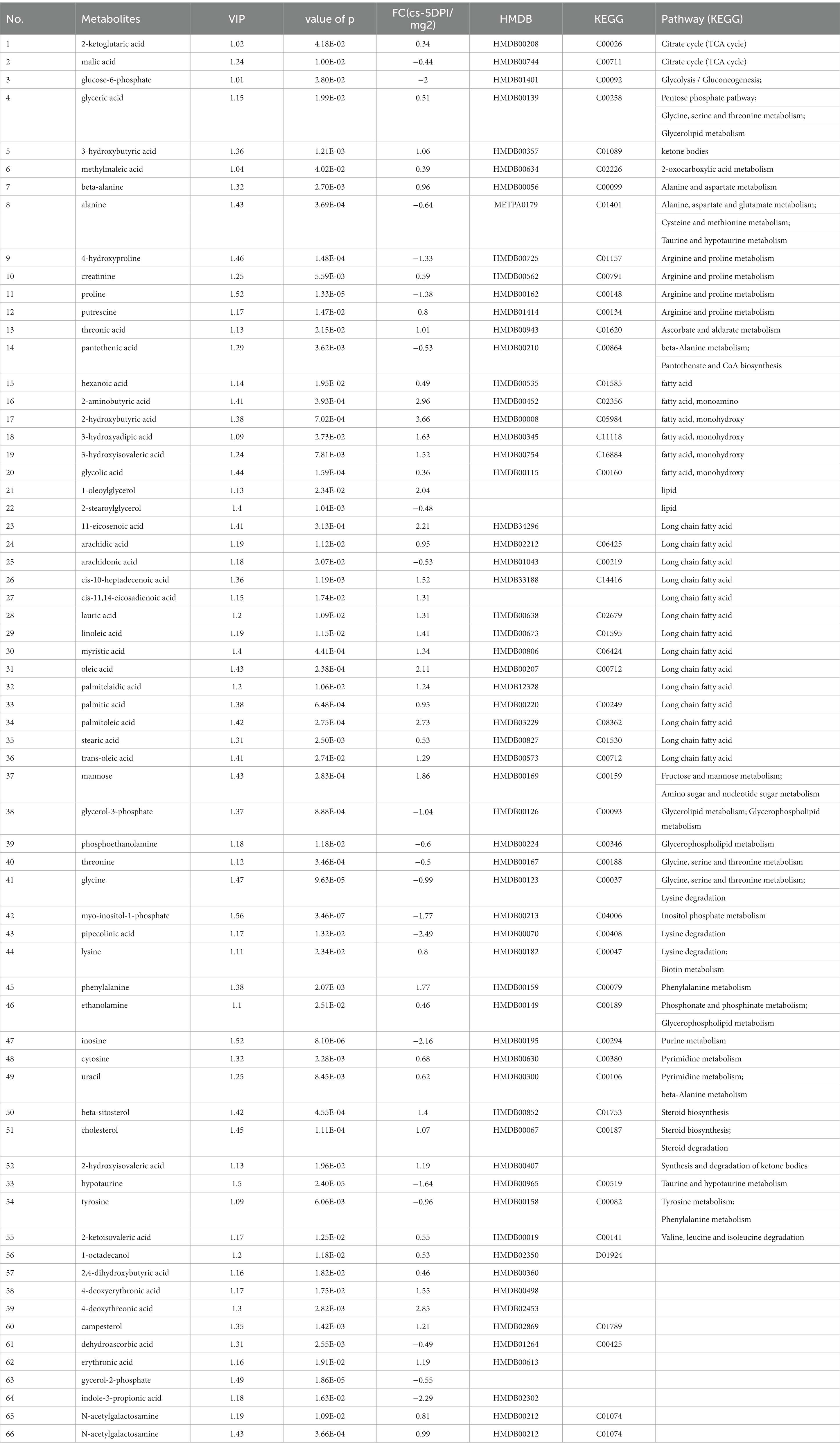 Tabla 5. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 5 días después de la infección.
Tabla 5. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 5 días después de la infección.
Análisis de vías
La figura 4C muestra que la infección por VPC a 5 DPI causó cambios en doce vías metabólicas, incluida la biosíntesis de ácidos grasos, la biosíntesis de pantotenato y CoA, la biosíntesis de aminoacil-ARNt, el metabolismo de la β-alanina, la degradación de la lisina, el metabolismo de la arginina y la prolina, el metabolismo de la glicina, la serina y la treonina, el metabolismo de los glicerolípidos, el metabolismo de la fenilalanina, el ciclo del citrato, el metabolismo del ácido araquidónico y el metabolismo del ácido linoleico. En las vías metabólicas anteriores, excepto la biosíntesis de ácidos grasos, la biosíntesis de aminoacil-ARNt y el ciclo del citrato, otras nueve vías metabólicas son diferencias significativas (impacto >0,1). Los datos del análisis de la vía se muestran en la Tabla 6.
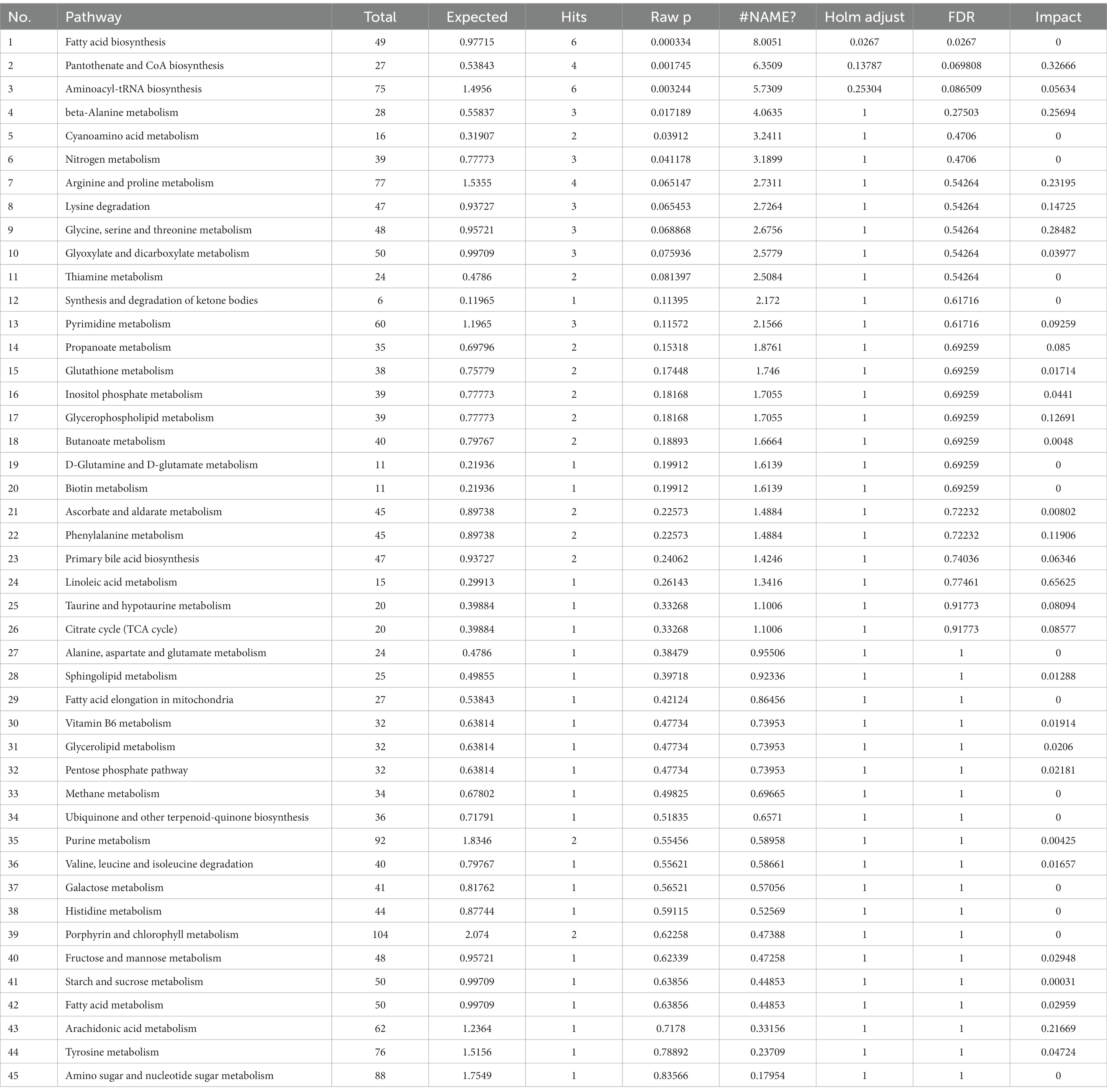 Tabla 6. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 5 días después de la infección.
Tabla 6. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 5 días después de la infección.
Séptimo día
Metabolitos diferenciales
Los datos de los metabolitos que cambiaron a 7 DPI se presentan en la Tabla 7. Se examinaron e identificaron un total de sesenta y nueve sustancias diferentes, incluidos veintiséis metabolitos con abundancia disminuida y cuarenta y tres metabolitos con abundancia aumentada a 7 DPI.
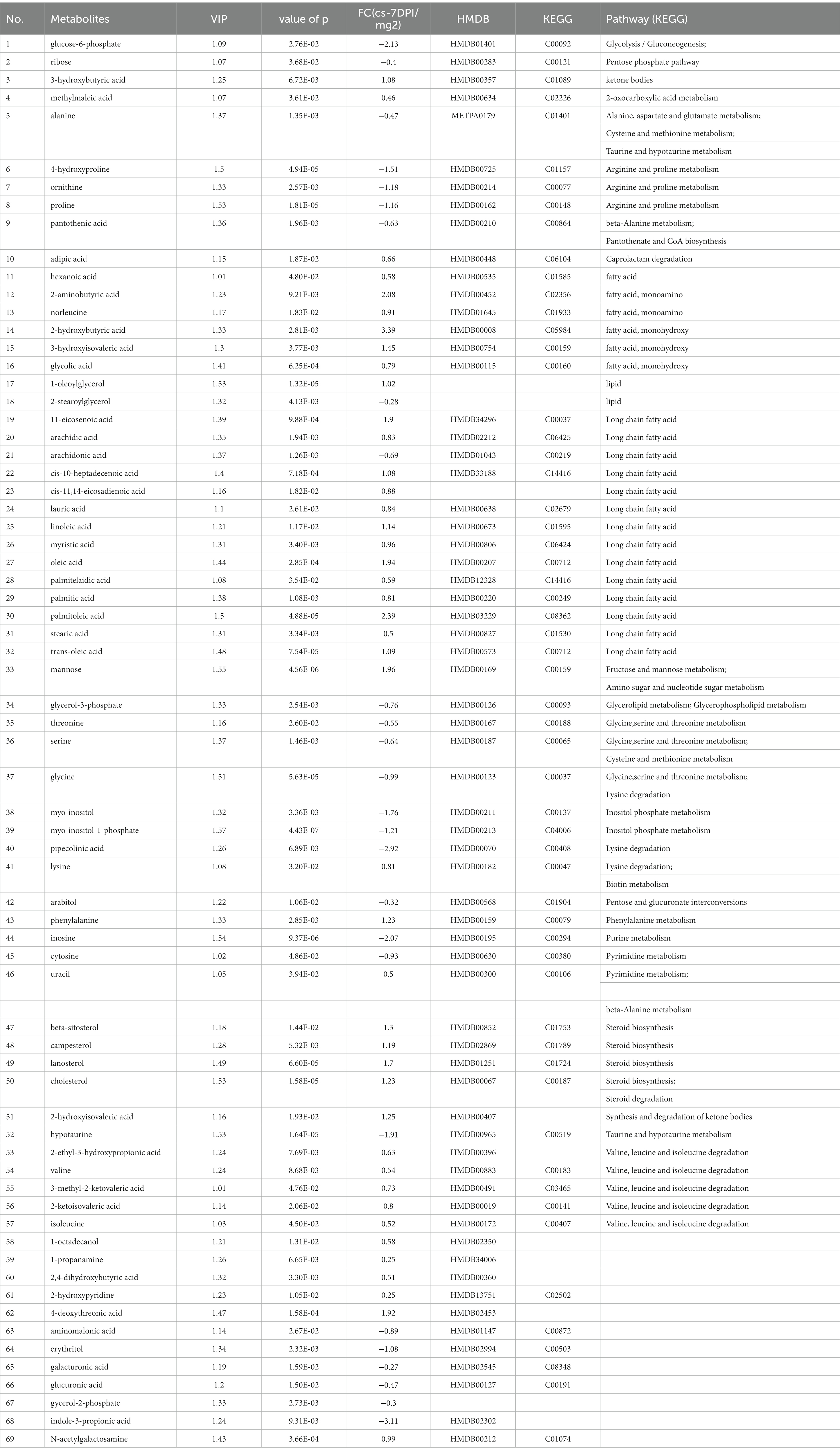 Tabla 7. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los infectados simulados a los 7 días después de la infección.
Tabla 7. Las diferencias de metabolitos entre los grupos infectados por el virus de la peste porcina clásica y los infectados simulados a los 7 días después de la infección.
Análisis de vías
La Figura 4D muestra que 7 DPI del virus de la peste porcina clásica causaron cambios en doce vías metabólicas, incluida la biosíntesis de ácidos grasos, la biosíntesis de pantotenato y CoA, la biosíntesis de aminoacil-ARNt, el metabolismo del fosfato de inositol, el metabolismo de los cianoaminoácidos, la degradación de la lisina, la síntesis y degradación de los cuerpos cetónicos, el metabolismo de la arginina y la prolina, el metabolismo de la glicina, la serina y la treonina, el metabolismo del ácido linoleico, el metabolismo de la fenilalanina y el metabolismo del ácido araquidónico. Además de la biosíntesis de ácidos grasos, la biosíntesis de aminoacil-ARNt y el metabolismo de los cianoaminoácidos, las otras nueve vías metabólicas mostraron diferencias significativas (impacto >0,1). Los datos del análisis de vías se muestran en la Tabla 8.
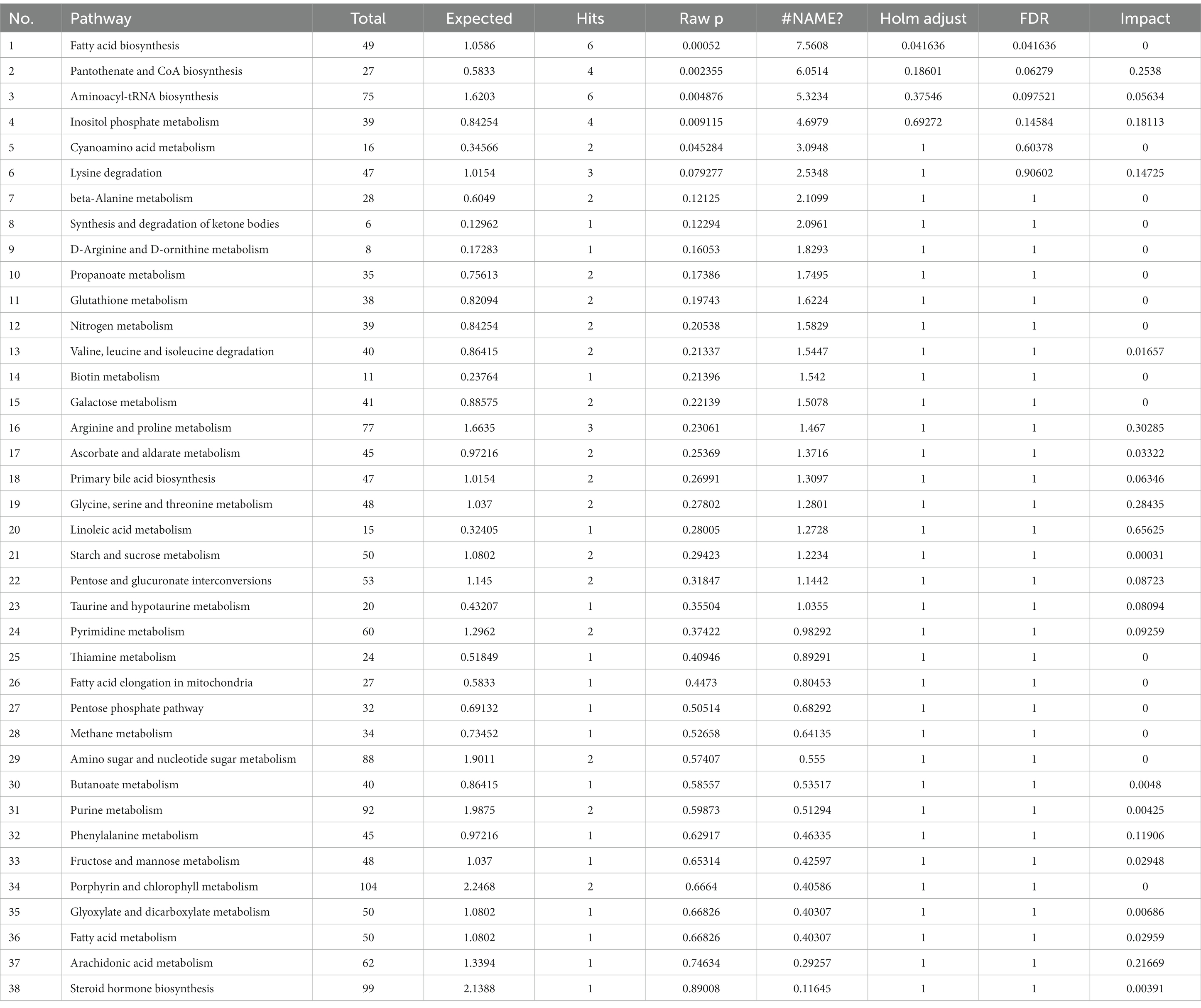 Tabla 8. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 7 días después de la infección.
Tabla 8. Análisis de la vía entre los grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados a los 7 días después de la infección.
Al establecer las conexiones entre las vías metabólicas relacionales, se construyeron las redes metabólicas en los grupos infectados por el virus de la peste porcina clásica (Figura 5).
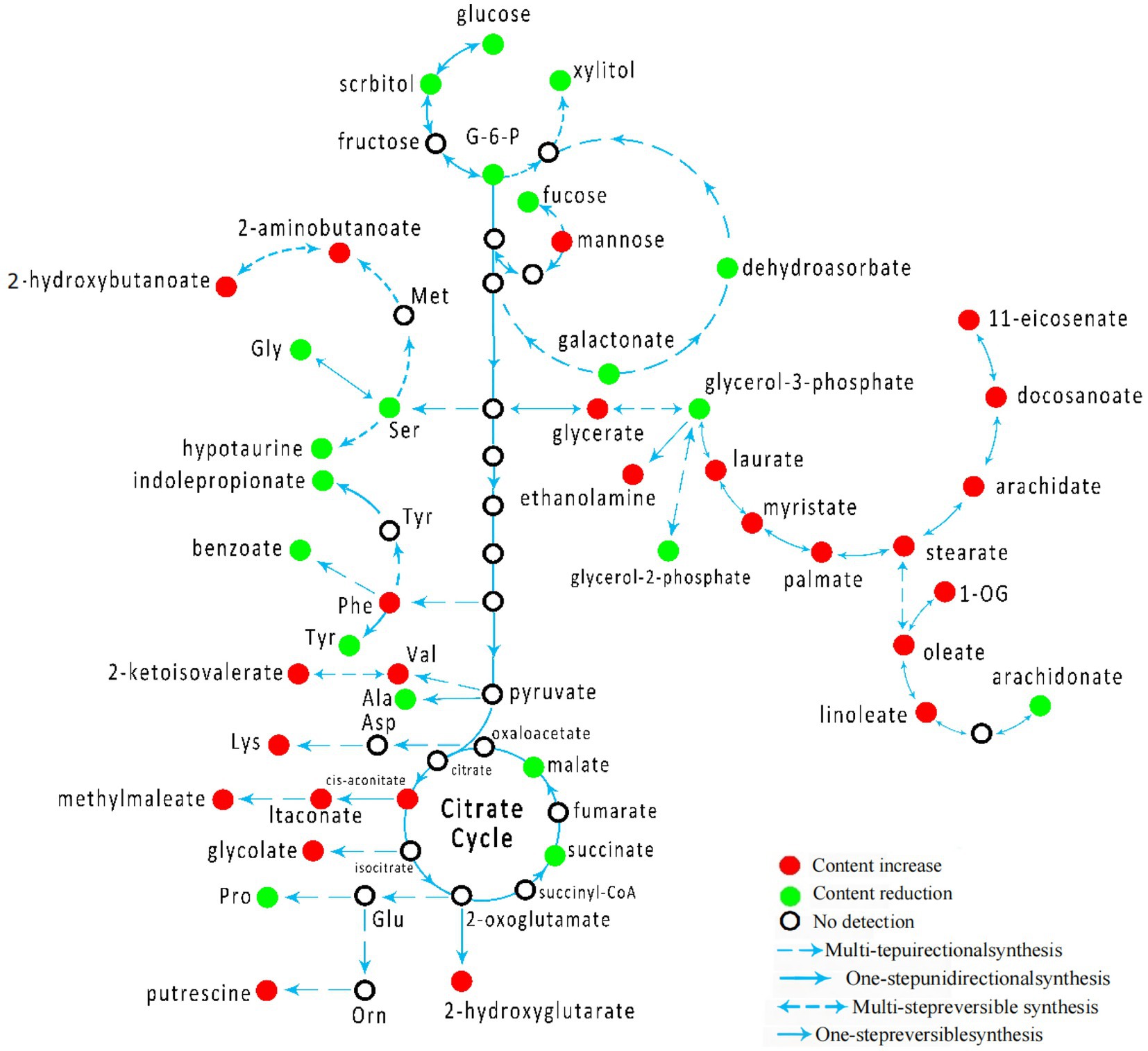 Figura 5. Resumen esquemático de la red metabólica en un grupo infectado por el virus de la peste porcina clásica.
Figura 5. Resumen esquemático de la red metabólica en un grupo infectado por el virus de la peste porcina clásica.
Matriz de correlación de diferentes metabolitos entre grupos infectados por el virus de la peste porcina clásica y los grupos infectados simulados en diferentes momentos
Para caracterizar las correlaciones de concentración entre diferentes metabolitos, se realizó un análisis de correlación de Pearson sobre la información cuantitativa de estas sustancias, como se muestra en la Figura 5. Las filas y columnas de la figura indican estas diferencias. El valor medido del coeficiente de correlación se muestra en el lado derecho de la figura. La profundidad de color del cuadrado en la imagen está relacionada con la correlación entre las diferencias. A partir de la distribución de colores, se puede ver que existe una alta correlación entre 1 DPI (Figura 6A), 3 DPI (Figura 6B), 5 DPI (Figura 6C) y 7 DPI (Figura 6D) y los cambios en la concentración de metabolitos con diferentes metabolitos en la misma vía metabólica o funciones relacionadas en el grupo infectado simulado, lo que indicó cambios en las vías metabólicas de diferentes metabolitos en todos los momentos después de la infección por VPC del grupo infectado simulado. en lugar de cambios en metabolitos individuales.
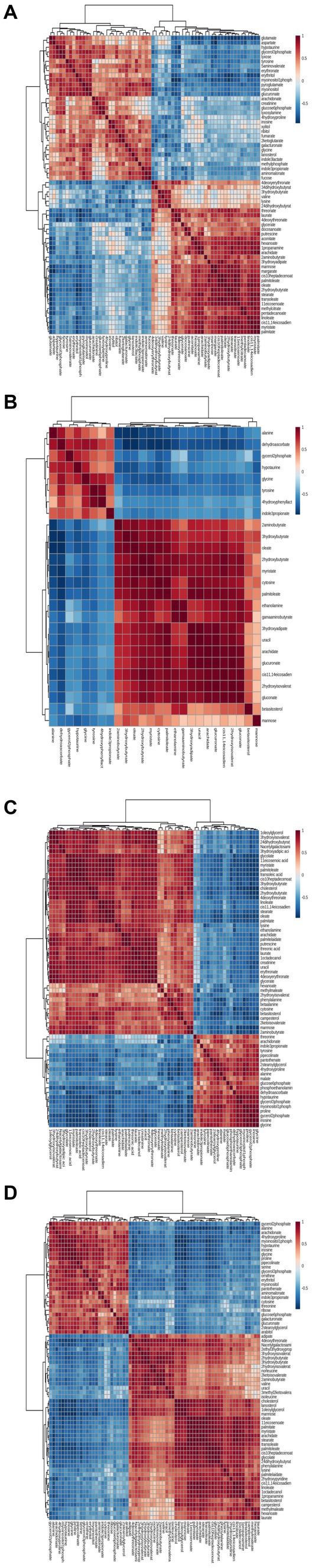 Figura 6. Análisis de correlación de vías metabólicas diferenciadas entre infectados por el virus de la peste porcina clásica y mg2 en 1DPI (A), 3DPI (B), 5DPI (C) y 7DPI (D), respectivamente.
Figura 6. Análisis de correlación de vías metabólicas diferenciadas entre infectados por el virus de la peste porcina clásica y mg2 en 1DPI (A), 3DPI (B), 5DPI (C) y 7DPI (D), respectivamente.
Análisis de mapas de calor de metabolitos diferenciales en grupos infectados por el virus de la peste porcina clásica y simulados en diferentes momentos
Para evaluar la relación entre los metabolitos diferenciales de los infectados por el virus de la peste porcina clásica y los infectados simulados, se analizó la información cuantitativa de estas sustancias mediante un mapa de calor, como se muestra en la Figura 6. Las filas representan diferentes metabolitos, las columnas representan los números de muestra, la estructura de árbol superior representa la relación de similitud entre las muestras y la estructura de árbol de la izquierda representa la relación de agrupamiento de similitud entre diferentes metabolitos. El análisis comparó los grupos infectados por el virus de la peste porcina clásica cs-1DPI (Figura 7A), cs-3DPI (Figura 7B), cs-5DPI (Figura 7C), cs-7DPI (Figura 7D) con mg2. Los diferentes metabolitos entre las muestras mostraron buenas relaciones de asentamiento de similitud en todos los momentos posteriores a la infección, lo que indicó que las diferencias intragrupo en los cambios en las vías metabólicas causadas por la infección por el virus de la peste porcina clásica eran pequeñas, y las diferencias entre los grupos son obvias (Figuras 7A-D).
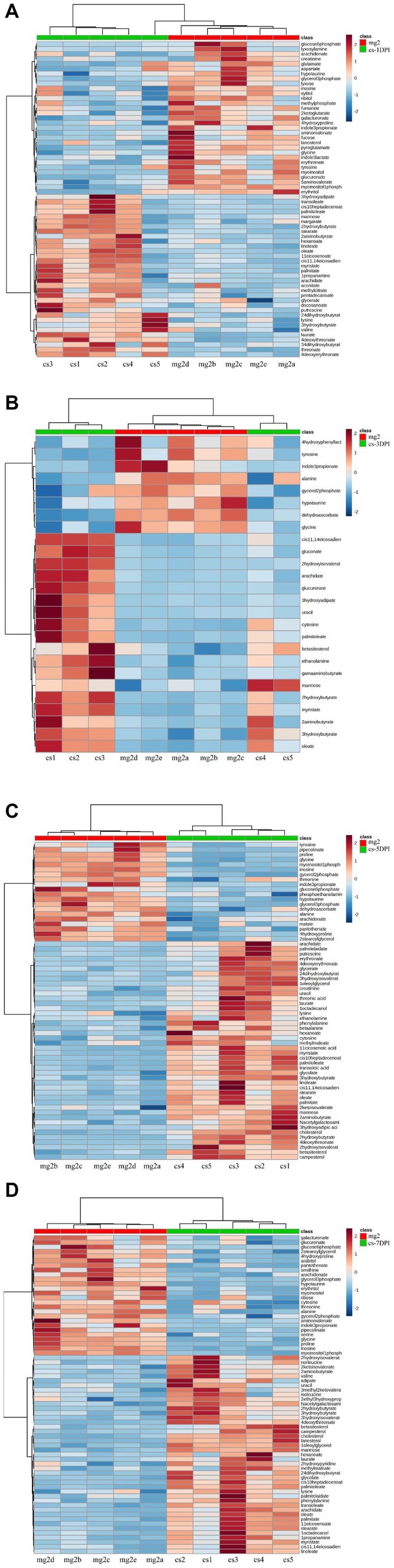 Figura 7. Mapa de calor de las vías metabólicas diferenciadas entre los infectados por el virus de la peste porcina clásica y mg2 en 1DPI (A), 3DPI (B), 5DPI (C) y 7DPI (D), respectivamente.
Figura 7. Mapa de calor de las vías metabólicas diferenciadas entre los infectados por el virus de la peste porcina clásica y mg2 en 1DPI (A), 3DPI (B), 5DPI (C) y 7DPI (D), respectivamente.
Discusión
La comparación de los metabolitos plasmáticos entre los grupos infectados por el virus de la peste porcina clásica y los infectados por simulacro mostró diferencias significativas en cuarenta y cinco metabolitos, entre los cuales veinticinco metabolitos tenían mayor abundancia que los del grupo no infectado y veinte metabolitos tenían menor abundancia que los del grupo infectado simulado. Estos metabolitos se distribuyeron principalmente en el ciclo del ácido tricarboxílico, el ciclo de aminoácidos, el metabolismo del azúcar y las vías del metabolismo de las grasas. Después de la infección por el virus de la peste porcina clásica, se alteró el ciclo del ácido tricarboxílico, la biosíntesis de ácidos grasos, el metabolismo del fosfato de inositol, la interconversión de pentosas y glucuronatos, el metabolismo de la glicina, la serina y la treonina, el metabolismo del ácido linoleico, el metabolismo del β-alanina, el metabolismo de la lisina, el metabolismo de los cianoaminoácidos, la biosíntesis de pantotenato-CoA, la biosíntesis de aminoacil-ARNt, el metabolismo de la arginina-prolina, el metabolismo de la fenilalanina, el metabolismo del ácido linoleico, el metabolismo del pantotenato y el metabolismo del ácido araquidónico. Después de la infección por el virus de la peste porcina clásica, el contenido de glucosa 6-fosfato, xilitol y fructosa en el metabolismo del azúcar disminuyó, lo que sugiere que la infección por el virus de la peste porcina clásica tiene un impacto en el metabolismo del azúcar. Después de la infección por el virus de la peste porcina clásica, se inhibió la producción de ácido málico y ácido succínico en el ciclo del ácido tricarboxílico, y se aumentó la vía metabólica del ácido cis-aconítico, lo que sugiere que la infección por el virus de la peste porcina clásica afectó el ciclo del ácido tricarboxílico. La infección por el virus de la peste porcina clásica también afectó claramente al metabolismo de los nucleótidos. Por un lado, la infección por el virus de la peste porcina clásica promovió el metabolismo de la fenilalanina, la valina y la lisina; por otro lado, la infección por el virus de la peste porcina clásica inhibió el metabolismo de la serina, la alanina y la tirosina. Así, la infección por el virus de la peste porcina clásica conduce a un trastorno en el metabolismo de los lípidos, en el que la vía del metabolismo del ácido araquidónico tiene una gran influencia.
Muchos virus parasitan las células y causan enfermedades en el cuerpo, y casi todos los patógenos requieren la ayuda de los metabolitos de su huésped para completar la replicación y la reproducción. Los metabolitos difieren del ARN y las proteínas. En lugar de estar codificados directamente en el genoma, los metabolitos son los productos de las vías bioquímicas utilizadas por las células o los tejidos para promover su propia supervivencia o metabolismo fisiológico normal. El uso de la metabonómica para analizar los cambios en los metabolitos en células, tejidos u organismos puede revelar los metabolitos específicos de algunas enfermedades y las diferentes sustancias que proporcionan posibles vías metabólicas, lo que puede ofrecer una mejor comprensión del mecanismo mutuo entre la vida y las enfermedades. La distribución y los cambios en los metabolitos después de la infección por virus proporcionan la evidencia más directa de la interacción entre un huésped y un patógeno; Sin embargo, la investigación sobre el análisis diferencial de metabolitos entre un huésped y un virus está en sus inicios (12). Con el rápido desarrollo de las técnicas analíticas y los métodos de reconocimiento de patrones, la metabonómica se convertirá en una poderosa herramienta para aclarar las complejas interacciones entre huéspedes y patógenos. Este enfoque puede evaluar y analizar sistemáticamente metabolitos de bajo peso molecular con alto rendimiento e identificar biomarcadores característicos de enfermedades.
Debido a que el virus de la peste porcina clásica tiene afinidad por los macrófagos, las células endoteliales (13, 14) y otras células auxiliares como las células dendríticas foliculares (15), el virus también puede utilizar algunos metabolitos de las células huésped para sintetizar su propia replicación y reproducción. Sin embargo, hasta la fecha, se ha investigado poco sobre el análisis diferencial de los metabolitos in vivo y los cambios en los metabolitos intracelulares después de la infección por el virus de la peste porcina clásica utilizando metabonómica. En este estudio, se seleccionó un modelo animal de infección experimental por VPC en cerdos miniatura tibetanos, se estudiaron dinámicamente las sustancias metabólicas en plasma infectado por VPC y se analizaron por primera vez las diferencias mediante metabonómica.
Los datos experimentales y los resultados de la primera parte de este estudio confirmaron que el modelo animal de infección por el virus de la peste porcina clásica se había establecido con éxito. A través de la detección dinámica de metabolitos plasmáticos en grupos infectados e infectados simulados, encontramos 45 metabolitos específicos relacionados con la infección por el virus de la peste porcina clásica, que se distribuyeron principalmente en las vías metabólicas del ciclo del ácido tricarboxílico, el ciclo de aminoácidos, el metabolismo del azúcar y el metabolismo de las grasas. Las concentraciones de 25 metabolitos fueron más altas que las del grupo infectado simulado y las concentraciones de 20 metabolitos fueron más bajas que las del grupo infectado simulado. En el grupo modelo después de la infección por el virus de la peste porcina clásica, varios metabolitos estaban regulados al alza o a la baja, lo que indica un metabolismo anormal. El análisis diferencial de los metabolitos de la infección por el virus de la peste porcina clásica se puede inferir a partir del cambio en los intermediarios en el proceso de metabolismo de la sustancia. Debido a que los metabolitos de la infección por el virus de la peste porcina clásica son complejos y diversos, algunos están relacionados con varias vías metabólicas. Podemos especular sobre su papel en algunas vías metabólicas basándonos en las diferencias entre los diferentes metabolitos. Además, para describir con mayor claridad el cambio relativo de la concentración de los metabolitos diana del virus de la peste porcina clásica, analizamos más a fondo las diferencias en los metabolitos entre los grupos infectados y los infectados simulados. Para estudiar la influencia de la infección por el virus de la peste porcina clásica en diferentes vías metabólicas, comparamos el espectro metabólico de los animales infectados por el virus de la peste porcina clásica y los infectados simulados. Los resultados mostraron que los metabolitos en plasma después de la infección por el virus de la peste porcina clásica causaron cambios en el ciclo del ácido tricarboxílico, la biosíntesis de ácidos grasos, el metabolismo del fosfato de inositol, la conversión mutua de pentosa y glucosa, el metabolismo del ácido glutámico, la serina y el metabolismo de la treonina, el metabolismo del ácido linoleico, el metabolismo de la β-alanina, el metabolismo de la lisina, el metabolismo de los cianoaminoácidos, la biosíntesis de pantotenato-CoA, la biosíntesis de aminoacil-ARNt, el metabolismo de la arginina y la prolina, el metabolismo de la fenilalanina, el ácido linoleico metabolismo, y metabolismo del pantotenato. En particular, se observó que la biosíntesis anormal de ácidos grasos se producía en todos los momentos después de la infección por el virus de la peste porcina clásica.
Estudios previos han demostrado que la replicación viral está estrechamente asociada con el metabolismo lipídico intracelular (10, 16). El VHB es un virus con envoltura. Para satisfacer las necesidades de su propia membrana lipídica, absorbe más lípidos externos de las células huésped para la biosíntesis de la envoltura. Las diferencias metabólicas en los pacientes con infección por VHB se manifiestan principalmente en el aumento de los niveles de metabolismo de lípidos y glucosa, y la disminución de los niveles de lactato, alanina, valina, glutamina y fosfato/colina de colina. Un estudio reciente demostró que la proteína X del VHB puede activar SREBP1 y PPAR para promover la biosíntesis de los lípidos del huésped y luego regular al alza la síntesis de lípidos en el VHB (17, 18). El antígeno de la hepatitis B puede interferir con el metabolismo de los lípidos en el hígado (5, 18). También se ha informado que la replicación del HCMV mejora la biosíntesis mediante la inducción de ácidos nucleicos y proteínas en las células huésped. La infección por HCMV estimula la glucólisis y el ciclo del ácido tricarboxílico, y promueve la síntesis de pirimidina (9). Los ácidos grasos saturados de cadena larga son esenciales para la producción de progenies infectadas por el VHC (19). También se ha informado que tanto Salmonella typhi como Salmonella pueden usar fosfolípidos en la bilis como fuentes importantes de carbono y energía (20). La infección por el virus del dengue (DENV) también conduce a la acumulación de ácidos grasos de cadena larga en el suero de los pacientes (10). La infección por DENV redistribuye la sintasa de ácidos grasos al sitio de replicación viral a través de la interacción entre la proteína no estructural 3 (NSP3) y la sintasa de ácidos grasos, promoviendo así la síntesis celular de ácidos grasos (21). Nuestro estudio encontró que la infección por el virus de la peste porcina clásica mejoró la biosíntesis de ácidos grasos, incluido el metabolismo del ácido linoleico y el ácido araquidónico. Entre ellos, la síntesis de ácidos grasos de cadena larga, incluidos el ácido láurico, el miristato, el ácido palmítico, el ácido esteárico, el ácido oleico y el ácido linoleico, aumentó, mientras que el metabolismo del ácido araquidónico disminuyó. La promoción de la biosíntesis de ácidos grasos por el virus de la peste porcina clásica puede promover la formación de un complejo de replicación del virus de membrana y, a su vez, facilitar la transmisión del virus. El ácido málico y el ácido succínico son intermediarios del ciclo del ácido tricarboxílico en el ATP, y sus concentraciones en el suero de los cerdos infectados fueron inferiores a las del grupo infectado simulado, mientras que el contenido de ácido cis-acónico fue mayor después de la infección que en el grupo infectado simulado. Estos hallazgos indican que la cepa altamente tóxica de CSRFV Shimen inhibió el ciclo de producción de energía del ácido tricarboxílico. Los niveles de fenilalanina y valina, que representan el metabolismo de los aminoácidos, fueron más altos que los del grupo infectado simulado, mientras que los niveles de serina, glicina y tirosina se redujeron después de la infección. Por lo tanto, nuestros resultados demostraron que el metabolismo de los aminoácidos después de la infección por el virus de la peste porcina clásica estaba desequilibrado.
En resumen, investigamos los mecanismos patogénicos de la infección por el virus de la peste porcina clásica en un modelo animal utilizando cerdos tibetanos en miniatura. Los resultados de la metabonómica mostraron que muchas vías metabólicas diferentes se alteran después de la infección por el virus de la peste porcina clásica. Por lo tanto, el análisis diferencial de metabolitos puede jugar un papel importante en la mejora del diagnóstico, la prevención y el tratamiento de enfermedades (10). Este estudio representa el primer análisis de los cambios en los metabolitos y las vías metabólicas en el plasma después de la infección por el virus de la peste porcina clásica, demostrando que el virus de la peste porcina clásica promueve la replicación viral y el escape inmunológico mediante la regulación de las vías metabólicas del azúcar, las vías metabólicas de los aminoácidos, el ciclo del ácido tricarboxílico y las vías metabólicas de los lípidos en el huésped. Los datos metabonómicos de la infección por el virus de la peste porcina clásica proporcionados en este artículo tienen un importante valor de referencia para el estudio posterior de la patogénesis del líquido cefalorraquídeo y el desarrollo de nuevos métodos de diagnóstico.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio se incluyen en el artículo/Material complementario. Otras consultas pueden dirigirse al autor de correspondencia.
Contribuciones de los autores
JL, WH y WW realizaron los experimentos y escribieron el manuscrito. XW, SY, XN, WenZ, BZ, YS, WeiZ y ZL analizaron los datos. JC concibió y diseñó los experimentos. Todos los autores contribuyeron al artículo y aprobaron la versión enviada.
Financiación
Este trabajo fue financiado por el Programa de la Fundación Nacional de Ciencias Naturales de China (NO.32172824 y NO.32102643), el Programa de Ciencia y Tecnología de Guangzhou, China (No.202206010161), los Proyectos de Investigación Clave de las Universidades de la Provincia de Guangdong (No.2019KZDXM026) y el Proyecto de Mejora de la Calidad y la Eficiencia de la Universidad Agrícola del Sur de China (No.C18).
Reconocimientos
Los autores agradecen a Shanghai ProfLeader Biotech Co., Ltd. por su ayuda con los experimentos metabolómicos de GC-MS. Nos gustaría agradecer a Editage (www.editage.cn) por la edición en inglés.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1171750/full#supplementary-material
Referencias
1. Chen, HY, Di Mascio, M, Perelson, AS, Ho, DD y Zhang, L. Determinación del tamaño de la ráfaga del virus in vivo utilizando un SIV de ciclo único en macacos rhesus. Proc Natl Acad Sci U S A. (2007) 104:19079–84. doi: 10.1073/pnas.0707449104
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Chambers, JW, Maguire, TG y Alwine, JC. El metabolismo de la glutamina es esencial para la infección por citomegalovirus humano. J Virol. (2010) 84:1867–73. doi: 10.1128/JVI.02123-09
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Blackham, S, Baillie, A, Al-Hababi, F, Remlinger, K, You, S, Hamatake, R, et al. El perfil de expresión génica indica el papel del estrés oxidativo, la apoptosis, el metabolismo de los lípidos y los genes de transporte intracelular del huésped en la replicación del virus de la hepatitis C. J Virol. (2010) 84:5404–14. doi: 10.1128/JVI.02529-09
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Hollenbaugh, JA, Munger, J y Kim, B. Perfiles de metabolitos de células T CD4+ infectadas por virus de inmunodeficiencia humana y macrófagos mediante análisis LC-MS/MS. Virología. (2011) 415:153–9. doi: 10.1016/j.virol.2011.04.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Yang, F, Yan, S, He, Y, Wang, F, Song, S, Guo, Y, et al. La expresión de proteínas del virus de la hepatitis B en ratones transgénicos altera el metabolismo lipídico e induce estrés oxidativo en el hígado. J Hepatol. (2008) 48:12–9. doi: 10.1016/j.jhep.2007.06.021
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Peinado, RDS, Eberle, RJ, Pacca, CC, Arni, RK y Coronado, MA. Revisión de estudios -ómicos sobre virus transmitidos por mosquitos del género Flavivirus. Virus Res. (2022) 307:198610. doi: 10.1016/j.virusres.2021.198610
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Yao, L, Hu, Q, Zhang, C, Ghonaim, AH, Cheng, Y, Ma, H, et al. Perfil metabolómico no dirigido basado en LC-MS de iPAM para investigar las alternancias de las vías metabólicas de los lípidos inducidas por diferentes cepas del virus de la pseudorrabia. Microbiol veterinario. (2021) 256:109041. doi: 10.1016/j.vetmic.2021.109041
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Huang, Z, Zhang, M, He, D, Song, P, Mo, C, Cheng, X, et al. Metabolómica sérica de cerdos miniatura Bama mordidos por Bungarus multicinctus. Toxicol Lett. (2021) 350:225–39. doi: 10.1016/j.toxlet.2021.07.017
9. Munger, J, Bajad, SU, Coller, HA, Shenk, T, y Rabinowitz, JD. Dinámica del metaboloma celular durante la infección por citomegalovirus humano. PLoS Pathog. (2006) 2:E132. doi: 10.1371/journal.ppat.0020132
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Sol, SQ, Yin, SH, Guo, HC, Jin, Y, Shang, YJ y Liu, XT. Tipificación genética de aislados del virus de la peste porcina clásica procedentes de China. Transbound Emerg Dis. (2013) 60:370–5. doi: 10.1111/j.1865-1682.2012.01346.x
11. Gou, H, Zhao, M, Yuan, J, Xu, H, Ding, H y Chen, J. Perfiles metabólicos en líneas celulares infectadas con el virus de la peste porcina clásica. Microbiol frontal. (2017) 8:691. doi: 10.3389/fmicb.2017.00691
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Aldridge, BB, y Rhee, KY. Metabolómica microbiana: innovación, aplicación, conocimiento. Curr Opin Microbiol. (2014) 19:90–6. doi: 10.1016/j.mib.2014.06.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Cheville, NF, y Mengeling, WL. La patogenia del cólera porcino crónico (peste porcina). Estudios histológicos, inmunofluorescentes y de microscopía electrónica. Lab Invest. (1969) 20:261–74.
14. Ressang, AA. Estudios sobre la patogenia del cólera porcino. II. Distribución del virus en los tejidos y morfología de la respuesta inmunitaria. Zentralbl Veterinarmed B. (1973) 20:272–88. doi: 10.1111/j.1439-0450.1973.tb01127.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Susa, M, König, M, Saalmuller, A, Reddehase, MJ, y Thiel, HJ. Patogenia de la peste porcina clásica: Deficiencia de linfocitos B causada por el virus del cólera porcino. J Virol. (1992) 66:1171–5. doi: 10.1128/jvi.66.2.1171-1175.1992
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Tamura, T, Nagashima, N, Ruggli, N, Summerfield, A, Kida, H y Sakoda, Y. El virus de la peste porcina clásica contribuye a la patogenicidad en los cerdos al prevenir la inducción de interferón tipo I en los sitios de replicación locales. Res. Veterinaria. (2014) 45:47. doi: 10.1186/1297-9716-45-47
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Kim, KH, Shin, HJ, Kim, K, Choi, HM, Rhee, SH, Moon, HB, et al. La proteína X del virus de la hepatitis B induce esteatosis hepática a través de la activación transcripcional de SREBP1 y PPARgamma. Gastroenterología. (2007) 132:1955–67. doi: 10.1053/j.gastro.2007.03.039
18. Kim, K, Kim, KH, Kim, HH y Cheong, J. La proteína X del virus de la hepatitis B induce el factor de transcripción lipogénico SREBP1 y la sintasa de ácidos grasos a través de la activación del receptor nuclear LXRalfa. Biochem J. (2008) 416:219–30. doi: 10.1042/BJ20081336
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Koyuncu, E, Purdy, JG, Rabinowitz, JD y Shenk, T. Los ácidos grasos saturados de cadena muy larga son necesarios para la producción de la progenie infecciosa del citomegalovirus humano. PLoS Pathog. (2013) 9:e1003333. doi: 10.1371/journal.ppat.1003333
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Antunes, LC, Andersen, SK, Menéndez, A, Arena, ET, Han, J, Ferreira, RB, et al. La metabolómica revela que los fosfolípidos son fuentes importantes de nutrientes durante el crecimiento de la salmonela en la bilis, in vitro e in vivo. J Bacteriol. (2011) 193:4719–25. doi: 10.1128/JB.05132-11
Palabras clave: virus de la peste porcina clásica, metabonómica, vía metabólica, ciclo del ácido tricarboxílico, mapa de calor
Cita: Liao J, Hu W, Wang W, Wang X, Yu S, Niu X, Zhu W, Zhou B, Song Y, Zeng W, Lu Z y Chen J (2023) Metabonómica plasmática de cerdos infectados por el virus de la peste porcina clásica. Frente. Vet. Sci. 10:1171750. doi: 10.3389/fvets.2023.1171750
Recibido: 22 de febrero de 2023; Aceptado: 19 de octubre de 2023;
Publicado: 06 Diciembre 2023.
Editado por:
Michael Kogut, Departamento de Agricultura de los Estados Unidos, Estados Unidos
Revisado por:
Hongbin Si, Universidad de Guangxi, China Yi Wu, Universidad Agrícola de Nanjing, China
Derechos de autor © 2023 Liao, Hu, Wang, Wang, Yu, Niu, Zhu, Zhou, Song, Zeng, Lu y Chen. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Jinding Chen, jdchen@scau.edu.cn
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

