Análisis de expresión tisular, clonación y caracterización de la región reguladora 5′ del gen LATS1 bovino

Análisis de expresión tisular, clonación y caracterización de la región reguladora 5′ del gen LATS1 bovino
 Sayed Haidar Abbas Raza3 ,
Sayed Haidar Abbas Raza3 , Xingping Wang1,2,
Xingping Wang1,2,  Rajwali Khan4, Zhaoxiong Lei1, Guijie Zhang1,2*, Jiupan Zhang5, Zhuoma Luoreng1,2, Yun Ma1,2, Muna O. Alamoudi6, Bandar Hamad Aloufi6, Ahmed Mohajja Alshammari6,
Rajwali Khan4, Zhaoxiong Lei1, Guijie Zhang1,2*, Jiupan Zhang5, Zhuoma Luoreng1,2, Yun Ma1,2, Muna O. Alamoudi6, Bandar Hamad Aloufi6, Ahmed Mohajja Alshammari6,  Ayman Hassan Abd El-Aziz7, Majid Alhomrani8,9 y Abdulhakeem S. Alamri8,9
Ayman Hassan Abd El-Aziz7, Majid Alhomrani8,9 y Abdulhakeem S. Alamri8,9- 1Escuela de Agricultura, Universidad de Ningxia, Yinchuan, China
- 2Laboratorio clave de Cría de Células Moleculares Ruminantes, Región Autónoma de Ningxia Hui, Yinchuan, China
- 3Colegio de Ciencia y Tecnología Animal, Universidad Northwest A&F, Yangling, China
- 4Departamento de Gestión Ganadera, Cría y Genética, Universidad de Agricultura, Peshawar, Pakistán
- 5Instituto de Ciencias Animales, Academia de Ciencias Agrícolas y Forestales de Ningxia, Yinchuan, China
- 6Departamento de Biología, Facultad de Ciencias, Universidad de Hail, Hail, Arabia Saudí
- 7Departamento de Alimentación Animal y Desarrollo de la Riqueza Animal, Facultad de Medicina Veterinaria, Universidad de Damanhour, Damanhour, Egipto
- 8Departamento de Laboratorios Clínicos de Ciencias, Facultad de Ciencias Médicas Aplicadas, Universidad de Taif, Taif, Arabia Saudí
- 9Centro de Investigación en Ciencias Biomédicas (CBSR), Decanato de Investigación Científica, Universidad de Taif, Taif, Arabia Saudí
Como miembro de la familia de genes supresores de tumores grandes (LATS), LATS1desempeña un papel importante en la regulación del crecimiento y el desarrollo muscular. En este estudio, determinamos los distintos patrones de exposición de expresión tisular del LATS1 bovino. Además, determinamos el promotor mínimo proximal funcional de LATS1 bovino e identificamos los factores de transcripción clave en la región del promotor central para dilucidar su mecanismo de regulación molecular. Los resultados mostraron que el LATS1 bovino estaba muy expresado en el torácico de mus y la regulación ascendente en el músculo de la infancia. Un ensayo de cambio de movilidad electroforética (EMSA) y un ensayo de inmunoprecipitación de cromatina (ChIP) en combinación con la mutación dirigida al sitio y la interferencia del ARN interferente de pequeña (siRNA) demostró que la diferenciación miogénica 1 (Myod1) y la unión al factor potenciador de miocitos 2A (MEF2A En conjunto, estas interacciones proporcionan información sobre los mecanismos reguladores de la transcripción de LATS1 en la mediación del crecimiento del músculo esquelético en el ganado.
Introducción
La industria del ganado vacuno en China seguramente ha tenido un rápido impulso de desarrollo, aunque comenzó tarde y ha estado en un nivel bajo en la última década. En la actualidad, el consumo de carne de vacuno en China escaso y el precio sigue funcionando a un nivel alto. Por lo tanto, China tiene que importar una gran cantidad de carne de vacuno del extranjero. En 2020, el volumen de importación de carne de vacuno alcanzó los 2,12 millones de toneladas, ocupando el primer lugar en el mundo. Se prevé que en los próximos 5 años, el consumo anual de carne de vacuno en China superará los 10 millones de toneladas. Según el nivel de producción actual, hay una brecha de al menos 3 millones de toneladas. Por lo tanto, todavía hay mucha necesidad para el desarrollo de la industria del ganado vacuno en China (1).
Los estudios han demostrado que la formación de tejido muscular esquelético en animales no solo está estrechamente relacionada con los niveles genéticos, ambientales y nutricionales, sino que también depende más del control genético (2, 3). Por lo tanto, es de gran importancia mejorar el crecimiento y el desarrollo del músculo esquelético y la calidad de la carne mediante la detección de genes y la identificación funcional (4). Como uno de los principales miembros de la vía del hipopótamo, el gen supresor de tumores grandes 1 (LATS1) desempeña un papel importante en la formación de tejidos y órganos y en la proliferación y diferenciación de las células madre embrionarias (5, 6). Además, la vía de señalización de la proteína asociada a Hiposí (YAP)-LATS1 ha surgido como una de las vías críticas conservadas que regulan la proliferación celular y la apoptosis en respuesta a las señales ambientales y de desarrollo (6-8). Se requiere la fosforilación de LATS1/2 para la activación de YAP que desempeña funciones en la regulación del mantenimiento de la pluripotencia de células madre, la tumorogénesis y el tamaño de los órganos (9, 10). Se ha descubierto que en muchas moléculas, LATS1 está involucrado en la vía Hippo, pero los mecanismos que rigen la activación de estas proteínas y la activación de la vía, en general, actualmente no están claros. En la actualidad, el estudio sobreLATS1 se centra principalmente en el mecanismo de la proliferación de células tumorales (11-13). En la actualidad, no hay ningún informe sobre el gen LATS1 que regula el crecimiento muscular y el desarrollo del ganado.
En nuestro estudio anterior, identificamos el gen LATS1 relacionado con los rasgos de crecimiento en el ganado Qinchuan. Especulamos que LATS1 desempeña un importante papel regulador en el proceso de diferenciación de las células musculares, mientras que el mecanismo de regulación de la actividad promotora de LATS1 durante la diferenciación, especialmente para la transcripción, aún no está claro. Aquí, analizamos los mecanismos moleculares de la regulación del gen LATS1 a través de la región promotora. Además, se determinó el patrón de expresión de ARN mensajero relativo (ARNm) de LATS1 bovino en el tejido. Nuestros resultados proporcionan una base sólida para obtener más información sobre los mecanismos reguladores transcripcionales para LATS1 en la mediación del crecimiento del músculo esquelético en el ganado.
Materiales y métodos
Declaración ética
Todos los procedimientos de operación de animales están en línea con el Consejo de China para el Cuidado de los Animales, así como con los estándares establecidos por la Universidad de Ningxia de prácticas experimentales de manejo de animales.
Análisis cuantitativo de PCR de patrones de expresión génica
Para comparar el nivel de expresión génica entre diferentes tejidos y la expresión temporal del gen bovineLATS1, se obtuvieron doce tejidos (corazón, hígado, bazo, pulmón, riñón, intestino delgado, intestino grueso, grasa abdominal, torácico de musgo, rumen, testículos y tejido cerebral) de tres bovinos adultos Qinchuan. Además, recogimos muestras de longissimus thoracis durante seis etapas de desarrollo del ganado macho Qinchuan, a saber, 0, 6, 12, 24 y 36 meses después del nacimiento. Aprobamos la expresión génica por triplicado para cada muestra y normalizamos los datos utilizando el gen de limpieza glicerinadehído-3-fosfato deshidrogenasa (GAPDH). Los cebadores cuantitativos de LATS1 y GAPDH se diseñaron y dieron en la Tabla 1.Brevemente, el ARN total en diferentes tejidos se extrajo utilizando el Kit de Extracción Universal de ARN MiniBEST (TaKaRa) y el ADN complementario (ADNc) se sintetizó a partir de 1 μl del ARN extraído utilizando el Kit de Reactivo PrimeScriptTM RT (TaKaRa) de acuerdo con los protocolos del fabricante. Se utilizaron 2 � Los niveles de expresión relativos de los ARNm objetivo se calcularon utilizando el método 2−ΔΔCt (14).
Clonación y análisis de secuencias
Obtuvimos la secuencia de la región reguladora de 5′ -1.783/+167 de LATS1 bovino de acuerdo con la información de la secuencia en GenBank (NC_037345, de 22618410 a 22620410). Las amplificaciones de PCR se realizaron con ADN genómico utilizando sangre de ganado Qinchuan como plantilla con los cebadores correspondientes PF y PR (Tabla 1). Sin embargo, el producto PCR produjo secuencias de región reguladoras de 5′, según lo verificado por el análisis. La estructura y la constitución del aminoácido del gen LATS1 bovino se analizaron mediante una serie de software de bioinformática. Además, los sitios de unión del factor de transcripción potencial (TF) y las islas de citosina Guanina (dinucleótido lineal) (CpG) se analizaron utilizando Genomatix (http://www.genomatix.de/) (15) y MethPrimer (http://www.urogene.org/methprimer/) (16), respectivamente. El umbral para los posibles sitios de unión de los factores de transcripción de detección fue >0,9.
Constructos de Luciferase Reporter
Siete cebadores específicos que incluían los sitios de restricción de KpnI y BglII se diseñaron para amplificar las eliminaciones unidireccionales del promotor bovino LATS1. Los productos de PCR se procesaron mediante extracción de gel, purificación y se digirieron con la enzima de restricción KpnI y BglII (New England BioLabs Incorporation, EE. UU.) siguiendo el protocolo del fabricante. Luego, los fragmentos se ligaron en la construcción del reportero de luciferasa pGL3- vector básico digerido con las mismas enzimas de restricción (KpnI y BglII), respectivamente. Estos plásmidos se llamaron pGL3-LATS1-P1, pGL3-LATS1-P2, pGL3-LATS1-P3, pGL3-LATS1-P4, pGL3-LATS1-P5, pGL3-LATS1-P6 y pGL3-LATS1-P7.
Cultivo y transfección de células
Las células mioblásticas del ganado Qinchuan (QCMC) se aislaron bajo un microscopio de disección de las muestras de músculo bovino fetal de Qinchuan, como se describió anteriormente (17). Los QCMC se cultivaron y cultivaron en placas de 24 pocillos por triplicado hasta alcanzar una densidad del 70 % a 37 °C y el 5 % de CO2. En pocas palabras, 800 ng de las siete construcciones de luciferasa pGL3-LATS1-P1 a pGL3-LATS1-P7 se cotransfectaron con 10 ng de vector de normalización pRL-TK utilizando Lip3000 (Invitrogen) de acuerdo con las instrucciones del fabricante. Después de 6 horas después de la transfección, el medio de cultivo de QCMC [10% de suero bovino fetal (FBS) + 90% de medio de águila modificado de Dulbecco (DMEM)] fue reemplazado por medio de diferenciación (2% de suero de caballo + 98% de DMEM) para inducir la diferenciación de los mioblastos en mis tubos como El vector básico pGL3 se utilizó como control negativo. Cuarenta y ocho horas después de la transfección, se recogieron lisados de células y se siguieron la cuantificación de la expresión de luciferasa y luciferasa de Renville mediante la realización de ensayos de actividad de la dobleluciferasa en la NanoQuant PlateTM (Tecan, Infinite M200PRO).
Mutagengénesis dirigida por el sitio
Los posibles sitios de unión a TF se analizaron utilizando el software Genomatix y se mutaron con los cebadores correspondientes (Tabla 1) utilizando el kit de mutagénesis de dirección rápida (TaKaRa) de acuerdo con el protocolo del fabricante. En pocas palabras, se identificaron 800 ng de los vectores de mutación y se cotransfectaron en QCMC con 10 ng de vector normalizador pRL-TK. El plásmido de tipo salvaje sirvió como control negativo. El método de operación específico era el mismo que el anterior.
Diferenciación miogénica 1 y reducción del factor potenciador de miocitos 2A
Los pequeños ARNs interferentes (siRNA) específicos contra la diferenciación miogénica 1 de TF objetivo (Myod1) o el factor potenciador de miocitos 2A (MEF2A) se sintetizaron y se cotransfectaron con el vector de plásmido promotor del núcleo pGL3-LATS1, respectivamente. El siRNA (UUCCGAACGUGACGUTT) sirvió como control negativo. En resumen, 800 ng de plásmido promotor del núcleo pGL3-LATS1 y 50 nM (concentración final) de cada dúplex de ARNs se cotransfectaron en QCMC utilizando Lip3000 de acuerdo con las instrucciones del fabricante. Veinticuatro horas después, la actividad de la luciferasa se midió utilizando el ensayo de reportero de la dobleluciferasa (E1910, Promega).
Ensayos de desplazamiento de movilidad electroforética
Las interacciones proteína-ADN se evaluaron mediante ensayos de cambio de movilidad electroforético (EMSA) (Thermo Fisher Scientific), como se describió anteriormente (18). En pocas palabras, se incubaron 200 fmol de sonda Myod1 marcada con 5′-biotina (Tabla 1) con extractos nucleares de 10 μg de QCMC, 2 μl de tampón de unión 10X y 1 μl de poli (dI-dC) en un volumen de 20 μl incubado durante 10 Para los ensayos de superdesplazamiento y competencia, se incubaron 2 μg del anti-Myod1 (sc-31940, Santa Cruz Biotechnology) o anti-MEF2A (EP1706Y, Abcam) o un exceso de los respectivos oligonucleótidos de doble cadena sin etiquetar se preincubaron con las proteínas de extracto nuclear indicadas durante 10 Después, las muestras se resolvieron mediante electroforesis en gel de tampón-poliacrilamida de ácido TRIS-borico nativo del 5 % y se analizaron mediante el generador de imágenes moleculares ChemiDocTM XRS+ System (Bio-Rad).
Ensayo de inmunoprecipitación de cromatina
Se utilizaron ensayos de inmunoprecipitación de cromatina (ChIP) para analizar la unión de Myod1 y MEF2A al promotor del gen LATS1 in vivo utilizando el Kit IP de cromatina enzimática SimpleChIP® (CST, Massachusetts, EE. UU.). Para la inmunoprecipitación, los complejos de proteína-ADN de los QCMC se digirieron con nucleasa microcócica en fragmentos de ~150-900 pb de longitud. La suspensión se dividió en dos partes: una de ellas se aclaró y se recogió con un tampón de dilución ChIP como entrada. Además, otro fue inmunoprecipitado utilizando 4 μl de anti-Myod1 (sc-31940, Santa Cruz Biotechnology) o anti-MEF2A (EP1706Y, Abcam) con una mezcla suave durante la noche a 4 °C. Los productos inmunoprecipitados se aislaron con cuentas de proteína G agarosa para el análisis de ChIP-PCR. La reacción se realizó en una serie de objetivos genéticos identificados para confirmar el enriquecimiento específico del ADN vs. Muestra de control negativo ChIP y se procesó como se describió anteriormente (19,20). El porcentaje de entrada se calculó de la siguiente manera: % Entrada =2[−ΔCt(Ct[ChIP]−(Ct[Input]−Log2(InputDilutionFactor)))] (21). La inmunoglobulina G (IgG) de conejo normal y el fragmento de ADN intragénico del exón LATS1 3 se utilizaron como controles negativos.
Análisis estadístico
Los datos se expresan como media ± SD de tres experimentos independientes. Los niveles de expresión génica basados en tres mismas condiciones de alimentación por parte de los individuos y la ANOVA unidireccional seguida de la ANOVA posterior se utilizaron para detectar diferencias significativas. Diferentes mayúsculas y minúsculas representan p < 0,01 y p < 0,05, respectivamente. Los ensayos de luciferasa y ChIP basados en tres experimentos independientes y la prueba t de estudiante de dos colas se utilizaron para detectar diferencias significativas. «*» p < 0,05 y «**» p < 0,01.
Resultados
Detección del patrón de expresión del supresor 1 del tumor grande
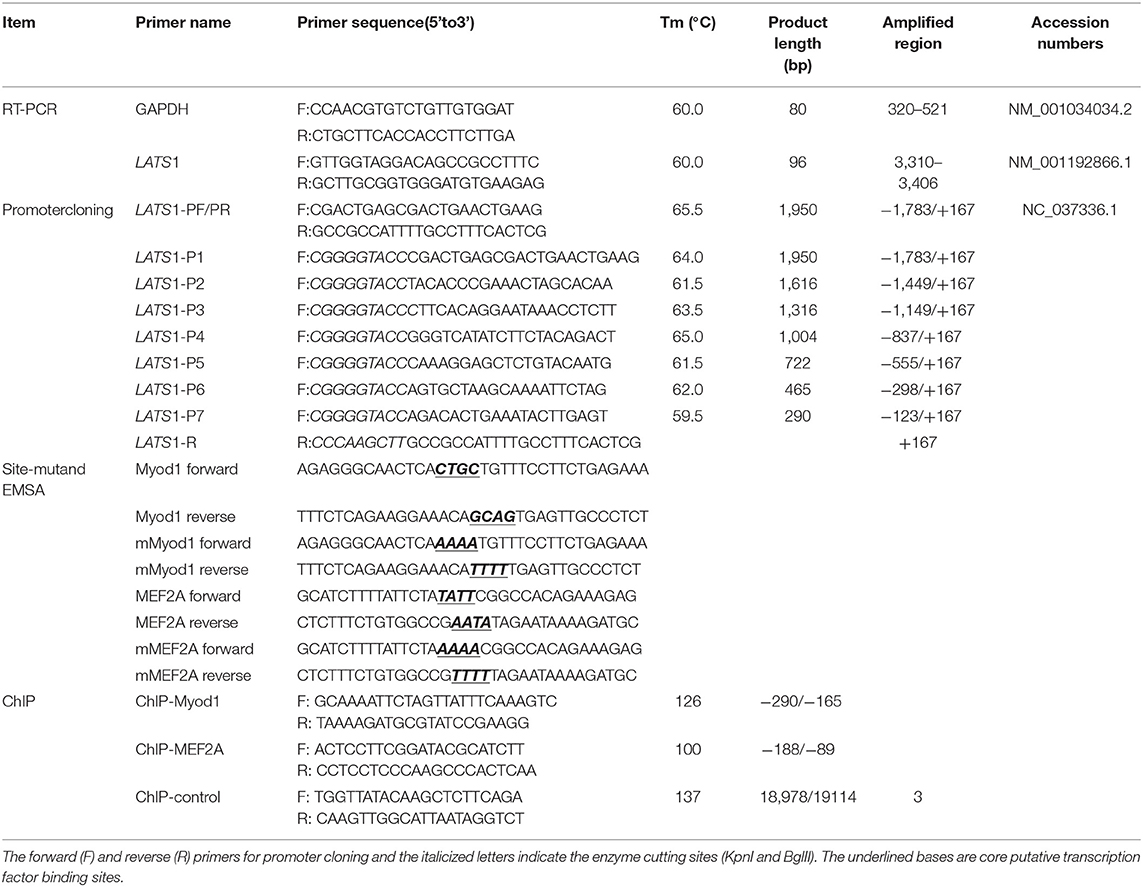
La distribución del ARNm LATS1 bovino se determinó a través de la qPCR utilizando ADNc de 12 tejidos diferentes (Figura 1A). La expresión del gen LATS1 fue significativamente mayor (p < 0,01) en el hígado y el cerebro que en otros tejidos y se expresó altamente (p < 0,05) en longissimus dorsi, intestino grueso y grasa abdominal. Sin embargo, era más bajo en el corazón, el bazo, el pulmón, los testículos, el intestino delgado, el rumen y el riñón. Para determinar aún más la expresión temporal del gen LATS1 bovino en seis etapas de longissimus thoracis a los 0, 6, 12, 24, 36 y 48 meses. Se procesó la qPCR y los resultados mostraron que la expresión relativa del gen LATS1 bovino en el torácico de longissimus a los 0, 6 y 12 meses de edad fue significativamente mayor (p < 0,01) que durante cualquier otra etapa (Figura 1B). Sin embargo, el nivel de expresión se redujo drásticamente después de 24 meses (Figura 1B). Estos resultados sugieren que el gen LATS1 está estrechamente relacionado con el desarrollo de tejidos y órganos en el ganado Qinchuan, especialmente en el desarrollo del cerebro y el tejido muscular.
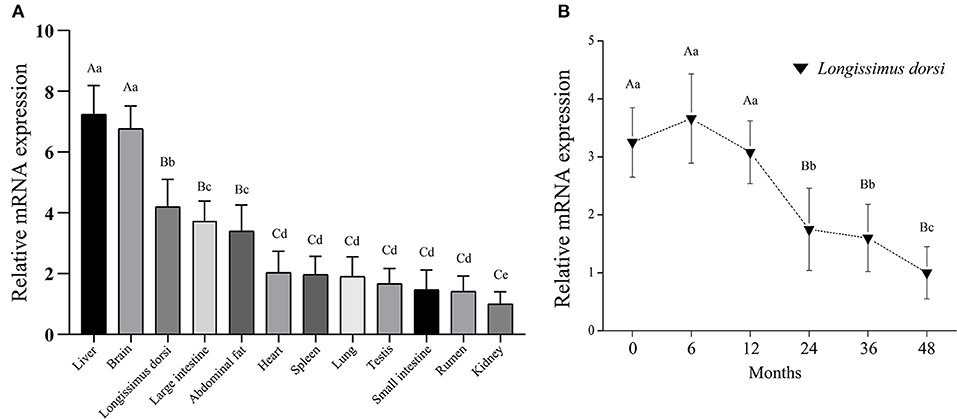 FIGURA 1. Análisis del patrón de expresión del supresor de tumores grandes bovinos 1 (LATS1). (A) Análisis del patrón de expresión de LATS1 bovino en tejidos y órganos. (B) Patrón de expresión del ARN mensajero LATS1 bovino (ARNm) en el torácico de mus mus en diferentes etapas de desarrollo. El valor de cada columna representa la media ± SD de tres experimentos independientes. Diferentes mayúsculas y minúsculas representan p < 0,01 y p < 0,05, respectivamente.
FIGURA 1. Análisis del patrón de expresión del supresor de tumores grandes bovinos 1 (LATS1). (A) Análisis del patrón de expresión de LATS1 bovino en tejidos y órganos. (B) Patrón de expresión del ARN mensajero LATS1 bovino (ARNm) en el torácico de mus mus en diferentes etapas de desarrollo. El valor de cada columna representa la media ± SD de tres experimentos independientes. Diferentes mayúsculas y minúsculas representan p < 0,01 y p < 0,05, respectivamente.
Construcción del gen supresor 1 del supresor de tumores grandes bovinos
Según la información de la secuencia, el gen LATS1 bovino se localizó en el cromosoma 9 (ENSBTAG00000015076, 86860562-86897588), con una longitud total de 37.027 pb e incluyendo ocho exones y siete intrones. De acuerdo con la secuencia de ADN bovino LATS1, se verificó un marco de lectura abierto (ORF) de 3.372 pb, que tenía 1.123 aminoácidos (aa) (Figuras 2A,B) con una fórmula química de C5, 542H8, 646N1, 602O1, 694S43 y un peso molecular de 126, Además, la región promotora del gen LATS1 bovino se clonó del ADN del genoma y se analizó la secuencia y las características estructurales. Los resultados mostraron que la región promotora del gen LATS1 bovino estaba compuesta por 647 en A, 544 en T, 368 en G y 441 en C, lo que resultó en un alto contenido de GC del 40,45 % y dos islas CpG (Figura 2C).
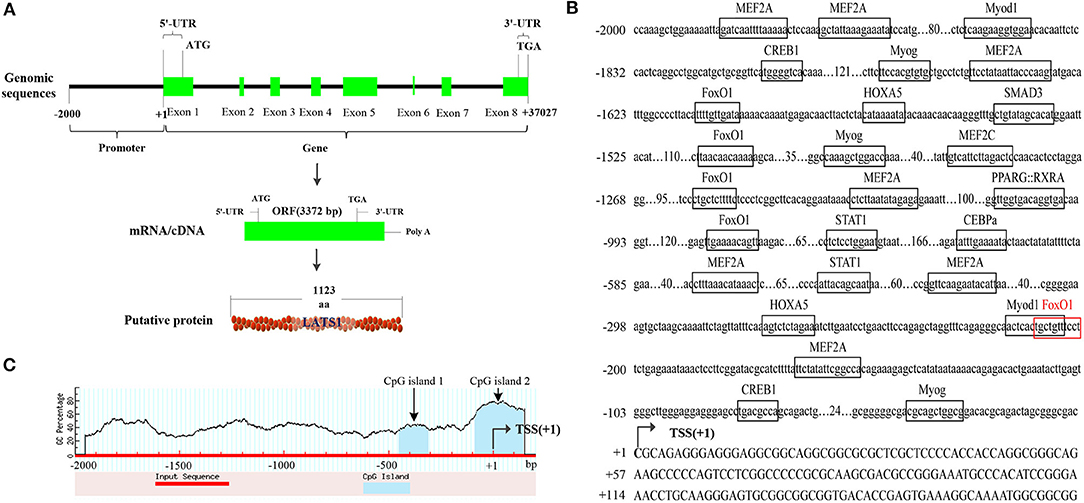 FIGURA 2. Características estructurales del gen bovineLATS1. (A) Los componentes detallados de genómico, ARNm y de proteínas contienen la región no traducida de 5′/3′ (5′/3′-UTR) y el marco de lectura abierto (ORF). (B) Secuencia de la región reguladora de 5 ′ del gen LATS1 bovino. Las flechas marcan los sitios de inicio de la transcripción. Los sitios de unión del factor de transcripción están en caja. (C) Las líneas discontinuas indican el porcentaje de GC representado en el eje y y el eje x denota la posición bp en la región no traducida de 5′. Las coordenadas se dan en relación con el sitio de inicio traslacional (se muestra como + 1).
FIGURA 2. Características estructurales del gen bovineLATS1. (A) Los componentes detallados de genómico, ARNm y de proteínas contienen la región no traducida de 5′/3′ (5′/3′-UTR) y el marco de lectura abierto (ORF). (B) Secuencia de la región reguladora de 5 ′ del gen LATS1 bovino. Las flechas marcan los sitios de inicio de la transcripción. Los sitios de unión del factor de transcripción están en caja. (C) Las líneas discontinuas indican el porcentaje de GC representado en el eje y y el eje x denota la posición bp en la región no traducida de 5′. Las coordenadas se dan en relación con el sitio de inicio traslacional (se muestra como + 1).
Además, las secuencias de proteínas LATS1 de diferentes especies fueron comparadas por el software MEGA (Figura 3). Los resultados mostraron que LATS1 tenía una gran similitud en diferentes especies, especialmente entre los animales estomacales compuestos como el ganado, las ovejas y las cabras, pero también en los animales monogástricos. Este resultado muestra que la función del gen LATS1 es muy importante en diferentes especies y es extremadamente conservadora en el proceso de evolución.
 FIGURA 3. La construcción del árbol filogenético de la proteína LATS1. En el lado izquierdo, está la agrupación de la proteína LATS1 en diferentes especies y en el lado derecho, está la arquitectura y las características de la longitud del aminoácido de la proteína LATS1 en las especies correspondientes.
FIGURA 3. La construcción del árbol filogenético de la proteína LATS1. En el lado izquierdo, está la agrupación de la proteína LATS1 en diferentes especies y en el lado derecho, está la arquitectura y las características de la longitud del aminoácido de la proteína LATS1 en las especies correspondientes.
Determinación del supresor de tumores grandes bovinos 1 Gene Core Promoter
Se obtuvieron siete fragmentos del promotor de genes LATS1 bovino utilizando la PCR de deleciones unidireccionales y se ligaron con el vector básico pGL3 para construir el plásmido recombinante. Además, el plásmido correspondiente fue identificado y transfectado en líneas celulares de mioblastos y miotubos por Lip3000 para determinar la actividad relativa de la luciferasa. Los resultados mostraron que la actividad de pLATS1–1,783/+167 fue significativamente mayor que en el control negativo básico pGL3- en las dos células (11,8 y 5,3 veces mayor que en el grupo de control negativo, respectivamente, Figura 4). Se encontró que la actividad relativa de la luciferasa en pLATS1-123/+167 fue significativamente menor (p < 0,01) que la de pLATS1-298/+167 tanto en las líneas celulares de mioblastos como de miotubo cuando se eliminó la secuencia de la región promotora −298/−123 bp del gen LATS Estos resultados sugieren que la secuencia de 5′ aguas arriba de 1,7 kb del gen LATS1 es una región promotora y tiene la función de regular la actividad transcripcional del gen. La región promotora de -298/−123 bp es la región transcripcional central del gen LATS1. La actividad transcripcional del gen LATS1en el mioblasto fue mayor que en el miotubo, lo que indicó que el mioblasto era más adecuado para la determinación de la actividad transcripcional del gen LATS1.
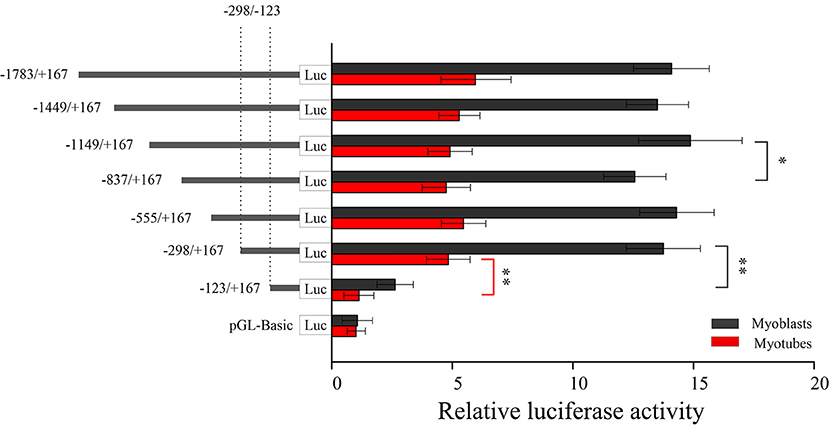 FIGURA 4. Aislamiento y análisis del promotor mínimo proximal funcional de LATS1. Una serie de plásmidos que contenían deleciones unidireccionales de 5′ de la región promotora del gen LATS1 fusionados en el marco del gen de la luciferasa se transfirieron en mioblastos y células de miotubo. Los resultados se expresan como las medias ± SD en unidades arbitrarias basadas en la actividad de luciferasa de lucivola/Renilla en transfecciones por triplicado. «*» p < 0,05 y «**» p < 0,01.
FIGURA 4. Aislamiento y análisis del promotor mínimo proximal funcional de LATS1. Una serie de plásmidos que contenían deleciones unidireccionales de 5′ de la región promotora del gen LATS1 fusionados en el marco del gen de la luciferasa se transfirieron en mioblastos y células de miotubo. Los resultados se expresan como las medias ± SD en unidades arbitrarias basadas en la actividad de luciferasa de lucivola/Renilla en transfecciones por triplicado. «*» p < 0,05 y «**» p < 0,01.
Identificación de los factores clave de transcripción
Muchos factores de transcripción contenían homeobox A5 (HOXA5), diferenciación miogénica 1 (Myod1), caja de cabeza de horquilla O1 (FoxO1) y factor potenciador de miocitos 2A (MEF2A) se predijo de la unión en la región promotora central (−298/−123 bp) del gen Para determinar los efectos de estos sitios transcripcionales en la regulación de LATS1, se crearon mutaciones de punto de 4 pb en los motivos HOXA5, Myod1, FoxO1 y MEF2A en plásmidos pLATS1–298/+167 y se transfirieron en QCMC. Como se muestra en la Figura 5A, la mutación del sitio Myod1 y MEF2A puede disminuir notablemente las actividades de la construcción pLATS1-298/+167 a 48,9 (p < 0,01) y 43,9% (p < 0,01) en comparación con el grupo de control, respectivamente. Sin embargo, no hubo ninguna diferencia significativa (p > 0,05) en la inhibición o simulación después de la mutación del sitio de unión HOXA5 y FoxO1.
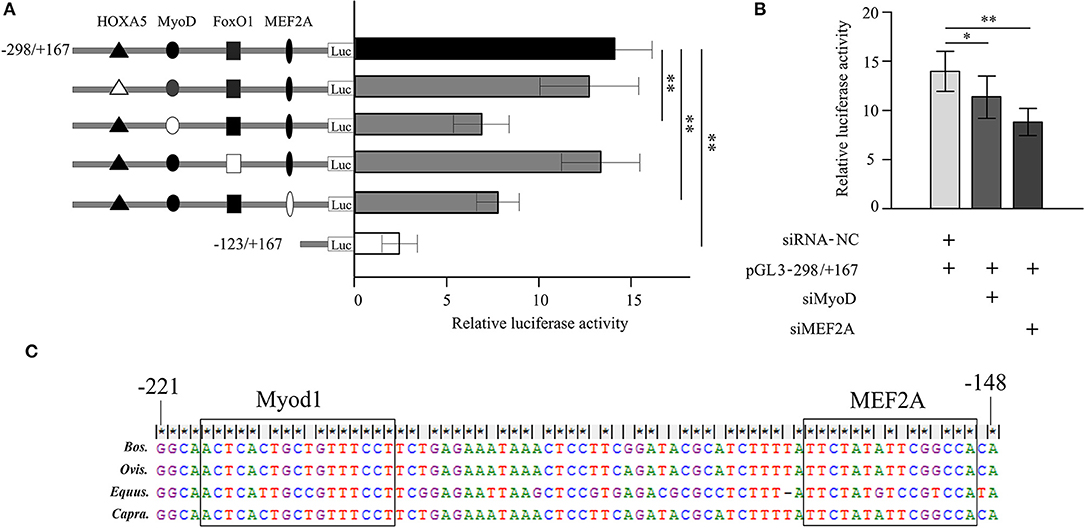 FIGURA 5. Análisis de la actividad transcripcional del factor de transcripción correspondiente en las células mioblásticas del ganado Qinchuan (QCMC). (A) La mutagénesis dirigida por el sitio para homeobox A5 (HOXA5), la diferenciación miogénica 1 (Myod1), la caja de cabeza de horquilla O1 (FoxO1) y el factor potenciador de miocitos 2A (MEF2A) se llevaron a cabo en la construcción pGL−298/+167. Las construcciones de correspondencia se transfirieron transitoriamente en QCMC y se utilizó la construcción pGL-123/+167 como control negativo.(B) Derribo de Myod1 y MEF2A por ARN interferente pequeño (siRNA) co-transfectado con pGL-298/+167 en QCMC. El siRNA NC se utilizó como control negativo. Los resultados se expresan como las medias ± SD en unidades arbitrarias basadas en la actividad de luciferasa de lucivola/Renilla para transfecciones triplicadas. (C) Análisis de la secuencia de alineación múltiple de los sitios de unión del factor de transcripción Myod1 y MEF2A en la región promotora del gen LATS1 en ganado, ovino, caballo y cabra. «*» p < 0,05 y «**» p < 0,01.
FIGURA 5. Análisis de la actividad transcripcional del factor de transcripción correspondiente en las células mioblásticas del ganado Qinchuan (QCMC). (A) La mutagénesis dirigida por el sitio para homeobox A5 (HOXA5), la diferenciación miogénica 1 (Myod1), la caja de cabeza de horquilla O1 (FoxO1) y el factor potenciador de miocitos 2A (MEF2A) se llevaron a cabo en la construcción pGL−298/+167. Las construcciones de correspondencia se transfirieron transitoriamente en QCMC y se utilizó la construcción pGL-123/+167 como control negativo.(B) Derribo de Myod1 y MEF2A por ARN interferente pequeño (siRNA) co-transfectado con pGL-298/+167 en QCMC. El siRNA NC se utilizó como control negativo. Los resultados se expresan como las medias ± SD en unidades arbitrarias basadas en la actividad de luciferasa de lucivola/Renilla para transfecciones triplicadas. (C) Análisis de la secuencia de alineación múltiple de los sitios de unión del factor de transcripción Myod1 y MEF2A en la región promotora del gen LATS1 en ganado, ovino, caballo y cabra. «*» p < 0,05 y «**» p < 0,01.
Para verificar aún más el papel de estos TF potenciales en el proceso de transcripción, siRNA-Myod1 y siRNA-MEF2A se transfectaron conjuntamente con plásmidos pLATS1-298/+167. Los resultados mostraron que el ARNs expresados transitoriamente podría interferir específicamente con los niveles de transcripción de LATS1, que disminuyen en 20,4 (p < 0,05) y 38,9 % (p < 0,01) (Figura 5B). Además, determinamos la conservación de los elementos Myod1 y MEF2A en diferentes especies, como en ganado, ovino, caballo y cabra (Figura 5C). Estos resultados indicaron que los motivos de Myod1 y MEF2A desempeñan un papel importante como sitios de unión para los TF de Myod1 y MEF2A en la regulación de la actividad transcripcional de LATS1.
La diferenciación miogénica 1 y el factor potenciador de miocitos 2A se unen a la región promotora
Se realizó un experimento de ensayos de cambio de movilidad electroforética para determinar la unión de Myod1 y MEF2A en el promotor central LATS1. Como se muestra en las figuras 6A, B, la proteína nuclear de QCMC se ató a las sondas Myod1/MEF2A con la etiqueta 5′-biotina y formó un complejo principal (la 2, Figuras 6A, B). Cuando se mezclaban tipos salvajes y mutantes no etiquetados de Myod1/MEF2A, apenas se podía ver una banda desplazada que se asemejaba al complejo (la 6-3, figuras 6A, B). Además, la adición de anticuerpo anti-Myod1/MEF2A condujo a una fuerte disminución de la banda desplazada hacia arriba (la 5, Figuras 6A, B). Sin embargo, el experimento EMSA de MEF2A no reveló ningún producto supershifted en los sitios de unión. Los resultados sugirieron que el superdesplazamiento puede estar formado por un polímero de alto peso molecular y estar atascado en la parte superior del pozo, causando una reducción en el desplazamiento de movilidad del gel (carril 5, Figura 6B).
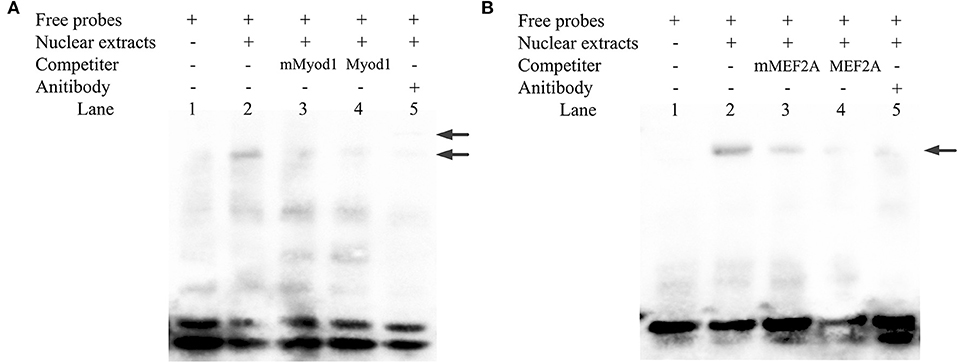 FIGURA 6. Identificación de Myod1 y MEF2A se unen al promotor mínimo proximal de LATS1 mediante un ensayo de desplazamiento de movilidad electroforética (EMSA) in vitro.( A,B) Los extractos de proteínas nucleares se incubaron con una sonda etiquetada con 5′-biotina que contenía el sitio de unión a Myod1- y MEF2A en presencia o ausencia de un competidor (ladro 2), sonda de mutación 50X (ladro 3) y sondas no etiquetadas 50X (ladro 4). El ensayo de superdesplazamiento se realizó utilizando 10 μg de anticuerpos anti-Myod1 y anti-MEF2A (la 5). Las flechas marcan los complejos principales y superdesplazadas, respectivamente.
FIGURA 6. Identificación de Myod1 y MEF2A se unen al promotor mínimo proximal de LATS1 mediante un ensayo de desplazamiento de movilidad electroforética (EMSA) in vitro.( A,B) Los extractos de proteínas nucleares se incubaron con una sonda etiquetada con 5′-biotina que contenía el sitio de unión a Myod1- y MEF2A en presencia o ausencia de un competidor (ladro 2), sonda de mutación 50X (ladro 3) y sondas no etiquetadas 50X (ladro 4). El ensayo de superdesplazamiento se realizó utilizando 10 μg de anticuerpos anti-Myod1 y anti-MEF2A (la 5). Las flechas marcan los complejos principales y superdesplazadas, respectivamente.
Además, se llevó a cabo un experimento ChIP para probar si Myod1 y MEF2A se pueden combinar en la región correspondiente del LATS1 in vivo. Los resultados mostraron que los niveles de enriquecimiento relativos fueron de ~9,2 y ~5,2 veces superiores al control de la inmunoglobulina G (IgG), respectivamente (Figura 7). Estos resultados sugieren que los TF de Myod1 y MEF2A se unen a la región promotora in vitro y vivo y desempeñan un papel importante en la regulación de la actividad transcripcional del gen LATS1.
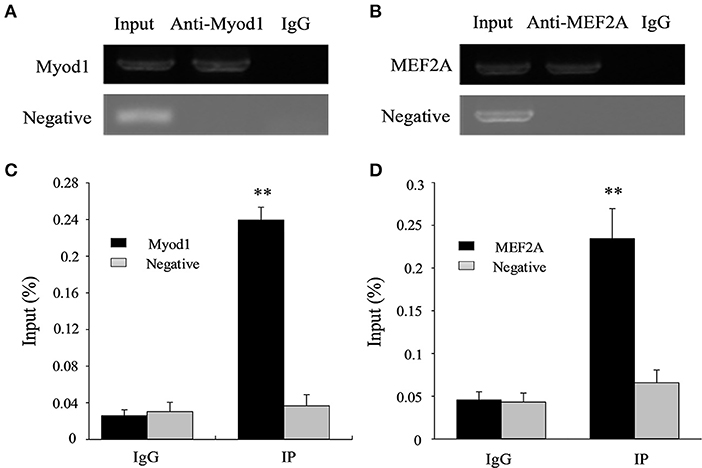 FIGURA 7. Ensayo de inmunoprecipitación de cromatina (ChIP) de Myod1 y MEF2A que se une al promotor de LATS1 in vivo. Los productos ChIP-PCR de entrada e inmunoprecipitados de Myod1 (A) y MEF2A (B) se amplificaron e identificaron mediante electroforesis de agarosa al 1 % del músculo. Los ensayos de ChIP-qPCR detectaron el enriquecimiento de fragmentos de ADN en muestras inmunoprecipitadas con anticuerpos Myod1 (C) y MEF2A (D). Utilizamos la entrada era cromatina total del músculo, mientras que los productos de la antiinmunoglobulina G (IgG) de conejo y un fragmento de ADN intragénico del exón LATS1 3. «**» p < 0,01.
FIGURA 7. Ensayo de inmunoprecipitación de cromatina (ChIP) de Myod1 y MEF2A que se une al promotor de LATS1 in vivo. Los productos ChIP-PCR de entrada e inmunoprecipitados de Myod1 (A) y MEF2A (B) se amplificaron e identificaron mediante electroforesis de agarosa al 1 % del músculo. Los ensayos de ChIP-qPCR detectaron el enriquecimiento de fragmentos de ADN en muestras inmunoprecipitadas con anticuerpos Myod1 (C) y MEF2A (D). Utilizamos la entrada era cromatina total del músculo, mientras que los productos de la antiinmunoglobulina G (IgG) de conejo y un fragmento de ADN intragénico del exón LATS1 3. «**» p < 0,01.
Discusión
El músculo esquelético es un tejido complicado y dinámico y afecta directamente a la eficiencia y calidad de la producción animal. Los factores de transcripción son los componentes clave en la maquinaria de transcripción de genes específicos, mientras que muchos cofactores de transcripción se combinan con los factores de transcripción para orquestar todo el evento de transcripción. Las expresiones inducidas de muchos genes están controladas por factores de transcripción específicos y elementos de acción cis (19, 22, 23). Como objetivo aguas abajo de la vía de Hipopótamo, LATS1 juega un papel importante en la proliferación de mioblastos, la regulación de la atrofia/hipertrofia y la transferencia de señales mecánicas a las respuestas transcripcionales (24, 25). Estudios recientes han encontrado que la familia de genes LATS no solo puede regular la proliferación y diferenciación de las células cancerosas, sino también el crecimiento muscular y el desarrollo de los animales (26-29). En este estudio, se encontró que el gen LATS1 estaba muy expresado en el hígado, el cerebro, el longissimus dorsi, el intestino grueso y los tejidos grasos abdominales del ganado y su proteína estaba muy conservada en la evolución de diferentes especies. El gen LATS1 desempeña un papel importante en el proceso de crecimiento y desarrollo individual.
En este estudio, se obtuvo la secuencia del promotor del gen LATS1 bovino y se encontraron dos islas CpG en su promotor de 2 kb mediante el análisis de software en línea. Aproximadamente la mitad de los genes de los vertebrados tienen secuencias ricas en CpG en sus promotores, a saber, las islas CpG (CGI). Los estudios han demostrado que una gran clase de CGI se encuentra cerca de la región reguladora de transcripción, especialmente lejos de los sitios de inicio de transcripción anotados (TSS). La metilación del ADN generalmente se produce en la secuencia del genoma del promotor y del exón 1 CGI y puede regular la transcripción génica (30-32). En general, se cree que la metilación de CGI puede inhibir la transcripción génica al unirse a los factores de transcripción o cambiar la estructura de la cromatina (30, 31). Por lo tanto, especulamos queLATS1 era esencial en la modificación epigenética durante el desarrollo de embriones o tejidos y órganos individuales.
La región central del gen LATS1 se detectó mediante el método de eliminación de la actividad de la doble luciferasa paso a paso y se encontró que la región central se encontraba en -298/−123 pb. Después del análisis con el software de predicción en línea, encontramos los posibles sitios de unión al factor de transcripción Myod1 y MEF2A en la secuencia de nucleótido −221 a −148 pb para conservar en animales domésticos. Myod1 es un miembro de la familia de factores reguladores miogénicos (MRF) (33, 34) y desempeña un importante regulador de la diferenciación de las células musculares y la formación de fibra muscular. Puede estar involucrado en la proliferación, diferenciación y regeneración de las células musculares (35). La expresión del gen Myod1 reguló el crecimiento y la diferenciación del músculo y afectó a la calidad de la carne (36, 37). Además, se encontró que la sobreexpresión del gen Myod1 se correlacionó positivamente con el porcentaje de fibra de tipo II B en el desarrollo muscular, lo que indica que Myod1 puede tener la función potencial de regular la diferenciación muscular en el proceso de formación de miocitos secundarios en el desarrollo muscular (38, 39). Muchos estudios han demostrado que el gen Myod1 desempeña un papel importante en el crecimiento y desarrollo muscular. En este estudio, determinamos la unión de Myod1 al promotor central del gen LATS1 bovino mediante el uso de EMSA y ChIP, lo que sugirió que Myod1 desempeña un papel importante en la regulación de la transcripción del gen LATS1.
El factor 2A potenciador de miocitos es un miembro de la familia de genes MEF2, que también incluye MEF2B, MEF2C y MEF2D (40). MEF2A es un factor de transcripción con el dominio básico hélice-bucle-hélice y es abundante en el cerebro, el músculo esquelético y el miocardio. Desempeña un papel importante en la promoción de la formación de fibra muscular, la homeostasis de la fibra muscular, la proliferación de cardiomiocitos, la diferenciación y la regeneración de las células madre y los mioblastos del músculo esquelético (17, 41, 42). El último estudio encontró que el silenciamiento del gen MEF2A bovino inhibió la diferenciación de los mioblastos y la reducción de la expresión de la miozenina 2 (17). En resumen, el MEF2A desempeña un papel importante en la regulación del crecimiento y el desarrollo muscular. En este estudio, la mutación y el derribo de MEF 2A disminuyeron la actividad del promotor basal de LATS1. Los resultados de EMSA y ChIP mostraron que MEF2A era capaz de tener una alta afinidad con LATS1, lo que sugiere que MEF2A desempeña un papel importante en la regulación de la actividad transcripcional del gen LATS1. En la Figura 8 se muestra un resumen que aclara el mecanismo de regulación transcripcional del gen LATS1 bovino.
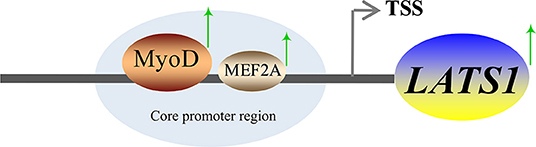 FIGURA 8. Un resumen esquemático propuesto de la regulación de la expresión LATS1 por los factores de transcripción (TF) de Myod1 y MEF2A.
FIGURA 8. Un resumen esquemático propuesto de la regulación de la expresión LATS1 por los factores de transcripción (TF) de Myod1 y MEF2A.
Conclusión
En resumen, este estudio identificó que la expresión relativa del gen LATS1 bovino estaba altamente expresada en el pulmón, el riñón, el corazón, la grasa subcutánea y el músculo longissimus dorsi. La región central del gen LATS1 bovino se encontraba en -298/−123 pb y está regulada por los TF de Myod1 y MEF2A. Estos resultados sentaron una base teórica para explorar el mecanismo de regulación molecular del gen LATS1 bovino en el crecimiento muscular.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio se incluyen en el artículo. Las consultas adicionales pueden dirigirse al/los) autor(s) correspondiente(s).
Declaración ética
El estudio de animales fue revisado y aprobado por todos los procedimientos de operación con animales que están en línea con el Consejo de China para el Cuidado de los Animales, así como con los estándares establecidos por la Universidad de Ningxia de prácticas experimentales de manejo de animales.
Contribuciones del autor
DW y SR diseñaron el experimento, realizaron los experimentos y escribieron el manuscrito. XW, RK, MAla y BA ayudaron principalmente a analizar los datos. AAls, AAb, ZL, JZ y YM proporcionaron sugerencias constructivas para el debate. GZ, DW y ZL contribuyeron a la administración y supervisión del proyecto. MAlh y AAla contribuyeron en el análisis de datos, validación, redacción, revisión y edición. Todos los autores han leído y aceptado la versión publicada del manuscrito.
Financiación
Este estudio contó con el apoyo de la Fundación de Ciencias Naturales de China (32060744 y 31960672), la Fundación de Ciencias Naturales de Ningxia (2021AAC05007 y 2020AAC03079), los Proyectos Clave de I+D en la Región Autónoma de Ningxia Hui (2019BEF02004 y 2020BEF02011), el Proyecto de Construcción de la Primera Disciplina de Ciencias de la Plata
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
El revisor CM declaró una afiliación compartida con el autor SR al editor de manejo en el momento de la revisión.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Agradecimientos
MA extiende su agradecimiento a la Universidad de Taif por financiar este estudio por parte de los investigadores de la Universidad de Taif Supporting Project Number (TURSP-2020/257), Universidad de Taif, Taif, Arabia Saudí.
Referencias
1. Li XZ, Yan CG, Zan LS. Situación actual y perspectivas futuras para la producción de carne de vacuno en China: una revisión. Asian-Australas J Anim Sci. (2018) 31:984–91. doi: 10.5713/ajas.18.0212
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
2. Lecker SH, Jagoe RT, Gilbert A, Gomes M, Baracos V, Bailey J, et al. Múltiples tipos de atrofia muscular esquelética implican un programa común de cambios en la expresión génica. FASEB J. (2004) 18:39–51. doi: 10.1096/fj.03-0610com
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
3. Fiszman MY. [Estrategias moleculars involucradas en el control de la expresión génica durante la diferenciación de las células musculares]. Reprod Nutr Dev. (1988) 28:703-13. doi: 10.1051/rnd:19880504
4. Listrat A, Lebret B, Louveau I, Astruc T, Bonnet M, Lefaucheur L, et al. Cómo la estructura y la composición muscular influyen en la calidad de la carne y la carne. Mundo de ciencia ficción J. (2016) 2016:3182746. doi: 10.1155/2016/3182746
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
5. Yi J, Lu L, Yanger K, Wang W, Sohn BH, Stanger BZ, et al. Los homólogos de supresores de tumores grandes 1 y 2 regulan la proliferación y maduración de las células progenitoras del hígado de ratón a través del antagonismo de los coactivadores YAP y TAZ. Hepatología. (2016) 64:1757-72. doi: 10.1002/hep.28768
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
6. Heallen T, Zhang M, Wang J, Bonilla-Claudio M, Klysik E, Johnson RL, et al. La vía del hipopótamo inhibe la señalización wnt para restringir la proliferación de cardiomiocitos y el tamaño del corazón. Ciencia.(2011) 332:458–61. doi: 10.1126/science.1199010
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
7. Lian I, Kim J, Okazawa H, Zhao J, Zhao B, Yu J, et al. El papel del coactivador de transcripción YAP en la regulación de la autorrenovación y diferenciación de las células madre. Genes Dev. (2010) 24:1106–18. doi: 10.1101/gad.1903310
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
8. Harvey K, Tapon N. La vía Salvador-Warts-Hippo: una red emergente de supresores de tumores. Nat Rev Cancer. (2007) 7:182–91. doi: 10.1038/nrc2070
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
9. Lu L, Li Y, Kim SM, Bossuyt W, Liu P, Qiu Q, et al. La señalización del hipopótamo es una potente vía de crecimiento in vivo y supresora de tumores en el hígado de los mamíferos. Proc Natl Acad Sci U S A.(2010) 107:1437–42. doi: 10.1073/pnas.0911427107
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
10. Song H, Mak KK, Topol L, Yun K, Hu J, Garrett L, et al. Las quinasas Mst1 y Mst2 de mamíferos desempeñan un papel esencial en el control del tamaño de los órganos y la supresión de tumores. Proc Natl Acad Sci U S A. (2010) 107:1431–6. doi: 10.1073/pnas.0911409107
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
11. Harvey KF, Zhang X, Thomas DM. La vía del hipopótamo y el cáncer humano. Nat Rev Cancer. (2013) 13:246–57. doi: 10.1038/nrc3458
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
12. Moroishi T, Hayashi T, Pan WW, Fujita Y, Holt MV, Qin J, et al. Las quinasas de la vía del hipopótamo LATS1/2 suprimen la inmunidad al cáncer. Cell. (2016) 167:1525–39 e17. doi: 10.1016/j.cell.2016.11.005
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
13. Furth N, Aylon Y. Los supresores de tumores LATS1 y LATS2: más allá de la vía del hipopótamo. La Muerte Celular Difiere. (2017) 24:1488–501. doi: 10.1038/cdd.2017.99
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
14. Livak KJ, Schmittgen TD. Análisis de los datos relativos de expresión génica utilizando PCR cuantitativa en tiempo real y el método 2(-Delta Delta C(T)). Métodos. (2001) 25:402–8. doi: 10.1006/meth.2001.1262
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
15. Cartharius K, Frech K, Grote K, Klocke B, Haltmeier M, Klingenhoff A, et al. MatInspector y más allá: análisis del promotor basado en sitios de unión del factor de transcripción. Bioinformática. (2005) 21:2933–42. doi: 10.1093/bioinformática/bti473
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
16. Li LC, Dahiya R. MethPrimer: diseño de cebadores para PCR de metilación. Bioinformática. (2002) 18:1427-31. doi: 10.1093/bioinformática/18.11.1427
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
17. Wang YN, Yang WC Li PW, Wang HB, Zhang YY, Zan LS. El factor 2a del potenciador de miocitos promueve la proliferación y su inhibición atenúa la diferenciación miogénica a través de la miozenina 2 en el mioblasto del músculo esquelético bovino. PLoS ONE. (2018) 13:e0196255. doi: 10.1371/journal.pone.0196255
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
18. Wei D, Raza SHA, Zhang J, Wang X, Khan R, Ma Y, et al. Caracterización de la región promotora del gen IRX3 bovino: funciones de SREBF2 y PPARG. Genómica física. (2020) 52:160–7. doi: 10.1152/physiolgenomics.00091.2019
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
19. Wei DW, Gui LS, Raza SHA, Zhang S, Khan R, Wang L, et al. NRF1 y ZSCAN10 se unen a la región promotora del gen SIX1 y sus efectos en las mediciones corporales del ganado Qinchuan. Representante de ciencia ficción (2017) 7:7867. doi: 10.1038/s41598-017-08384-1
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
20. Wei D, Li A, Zhao C, Wang H, Mei C, Khan R, et al. Regulación transcripcional por metilación de los sitios de CpG en la región promotora central del gen bovino SIX1: roles de la histona H4 y E2F2. Int J Mol Sci.(2018) 19. doi: 10.3390/ijms19010213
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
21. Chakrabarti SK, James JC, Mirmira RG. Evaluación cuantitativa del gen dirigido in vitro e in vivo por el factor de transcripción pancreática, Pdx1. Importancia de la estructura de cromatina en la dirección del enlace del promotor. J Biol Chem. (2002) 277:13286–93. doi: 10.1074/jbc.M111857200
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
22. Wei DW, Ma XY, Zhang S, Hong JY, Gui LS, Mei CG, et al. Caracterización de la región promotora del gen SIX1 bovino: Roles de MyoD, PAX7, CREB y MyoG. Representante de ciencia ficción (2017) 7:12599. doi: 10.1038/s41598-017-12787-5
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
23. Wei D, Feng L, Zhang W, Ma X, Cheng G, Li S, et al. Caracterización de la región promotora de SIX4 bovino: Funciones de la caja E y la mioD en la regulación de la transcripción basal. Biochem Biophys Res Commun. (2018) 496:44-50. doi: 10.1016/j.bbrc.2017.12.162
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
24. Halder G, Johnson RL. Señalización de hipopótamos: control del crecimiento y más allá. Desarrollo.(2011) 138:9-22. doi: 10.1242/dev.045500
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
25. Yu FX, Zhao B, Panupinthu N, Jewell JL, Lian I, Wang LH, et al. Regulación de la vía Hippo-YAP mediante la señalización del receptor acoplado a la proteína G. Celda. (2012) 150:780–91. doi: 10.1016/j.cell.2012.06.037
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
26. Zhang L, Noguchi YT, Nakayama H, Kaji T, Tsujikawa K, Ikemoto-Uezumi M, et al. El eje CalcR-PKA-Yap1 es fundamental para mantener la quiescencia en las células madre musculares. Cellular Rep. (2019) 29:2154–63 e5. doi: 10.1016/j.celrep.2019.10.057
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
27. McNeill H, Reginensi A. LATS 1/2 Regula Yap/Taz para controlar la epitelización del progenitor de nefrón e inhibir la formación de miofibroblastos. Soy Soc Nephrol. (2017) 28:852–61. doi: 10.1681/ASN.2016060611
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
28. Ikeda S, Sadoshima J. Regulación del crecimiento y la muerte de las células miocárdicas por la vía del hipopótamo. Circ J. (2016) 80:1511–9. doi: 10.1253/circj.CJ-16-0476
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
29. Guan H, Zhao L, Liu H, Xie D, Liu Y, Zhang G, et al. Efectos de la presión intermitente que imita la manipulación rodante en la medicina tradicional china en la ultraestructura y el metabolismo de las células musculares esqueléticas humanas lesionadas. Am J Transl Res. (2020) 12:248-60.
30. Deaton AM, Bird A. Islas CpG y la regulación de la transcripción. Genes Dev. (2011) 25:1010-22. doi: 10.1101/gad.2037511
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
31. Smith ZD, Meissner A. Metilización del ADN: funciones en el desarrollo de los mamíferos. Nat Rev Genet.(2013) 14:204–20. doi: 10.1038/nrg3354
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
32. Maunakea AK, Nagarajan RP, Bilenky M, Ballinger TJ, D’Souza C, Fouse SD, et al. Papel conservado de la metilación intragénica del ADN en la regulación de promotores alternativos. Naturaleza. (2010) 466:253–7. doi: 10.1038/nature09165
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
33. Zammit PS. Función de los factores reguladores miogénicos Myf5, MyoD, Myogenina y MRF4 en el músculo esquelético, las células satélite y la miogénesis regenerativa. Semin Cell Dev Biol. (2017) 72:19-32. doi: 10.1016/j.semcdb.2017.11.011
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
34. Hernández-Hernandez JM, García-Gonzalez EG, Brun CE, Rudnicki MA. Los factores reguladores miogénicos, los determinantes del desarrollo muscular, la identidad celular y la regeneración. Semin Cell Dev Biol. (2017) 72:10–8. doi: 10.1016/j.semcdb.2017.11.010
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
35. Yamamoto M, Legendre NP, Biswas AA, Lawton A, Yamamoto S, Tajbakhsh S, et al. La pérdida de MyoD y Myf5 en las células madre del músculo esquelético da lugar a una programación miogénica alterada y a una regeneración fallida. Rep. de células madre (2018) 10:956–69. doi: 10.1016/j.stemcr.2018.01.027
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
36. Ding S, Nie Y, Zhang X, Liu X, Wang C, Yuan R, et al. Los SNP en el gen myoD de individuos en desarrollo muscular normal no tienen ningún efecto sobre la masa muscular. BMC Genet. (2019) 20:72. doi: 10.1186/s12863-019-0772-6
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
37. Cho IC, Park HB, Ahn JS, Han SH, Lee JB, Lim HT, et al. Una variante reguladora funcional de MYH3 influye en la composición de fibra muscular y el contenido de grasa intramuscular en los cerdos. PLoS GENET. (2019) 15:e1008279. doi: 10.1371/journal.pgen.1008279
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
38. Bar-Nur O, Gerli MFM, Di Stefano B, Almada AE, Galvin A, Coffey A, et al. Reprogramación directa de fibroblastos de ratón en progenitores del músculo esquelético funcional. Rep. de células madre (2018) 10:1505–21. doi: 10.1016/j.stemcr.2018.04.009
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
39. Du J, Shen L, Zhang P, Tan Z, Cheng X, Luo J, et al. La regulación de la composición de la fibra muscular esquelética por betaína está asociada con NFATc1/MyoD. J Mol Med. (2018) 96:685–700. doi: 10.1007/s00109-018-1657-2
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
40. Estrella NL, Desjardins CA, Nocco SE, Clark AL, Maksimenko Y, Naya FJ. Los factores de transcripción MEF2 regulan distintos programas genéticos en la diferenciación del músculo esquelético de los mamíferos. J Biol Chem. (2015) 290:1256–68. doi: 10.1074/jbc.M114.589838
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
41. Wang Y, Mei C, Su X, Wang H, Yang W, Zan L. MEF2A regula la señalización PP2A dirigida al megagrupo de miARN MEG3-DIO3 en la diferenciación de mioblastos esqueléticos bovinos. Int J Mol Sci. (2019) 20. doi: 10.3390/ijms20112748
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
42. Schiaffino S, Dyar KA, Calabria E. La masa muscular esquelética está controlada por el eje MRF4-MEF2.Cuidado del metab de Nutr de la nuez de Curr Opin. (2018) 21:164-7. doi: 10.1097/MCO.0000000000000456
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Palabras clave: gen LATS1, expresión, promotor central, transcripción, factor
Cita: Wei D, Raza SHA, Wang X, Khan R, Lei Z, Zhang G, Zhang J, Luoreng Z, Ma Y, Alamoudi MO, Aloufi BH, Alshammari AM, Abd El-Aziz AH, Alhomrani M y Alamri AS (2022) Análisis de expresión tisular, clonación y caracterización de la Frente. Veterinario. Sci.9:853819. doi: 10.3389/fvets.2022.853819
Editado por:
Aline Silva Mello Cesar, Universidad de São Paulo, Brasil
Revisado por:
Junaith S. Mohamed, Centro de Ciencias de la Salud de la Universidad de Tennessee (UTHSC), Estados Unidos
Chugang Mei, Universidad Northwest A&F, China
Ali Raza Jahejo, Universidad Agrícola de Shanxi, China
Guobo Quan, Instituto de Ciencias y Veterinaria de los Animales de Yunnan, China
Rui-Si Hu, Universidad de Ciencia y Tecnología Electrónica de China, China
Copyright © 2022 Wei, Raza, Wang, Khan, Lei, Zhang, Zhang, Luoreng, Ma, Alamoudi, Aloufi, Alshammari, Abd El-Aziz, Alhomrani y Alamri. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución de Creative Commons (CC BY). *Correspondencia: Guijie Zhang, guijiezhang@nxu.edu.cn
Descargo de responsabilidad: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamación que pueda ser fabricado por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía