Análisis metagenómico de la microbiota fecal de la diarrea similar a la disentería en una granja porcina
 Análisis metagenómico de la microbiota fecal de la diarrea similar a la disentería en una granja porcina mediante secuenciación de nueva generación
Análisis metagenómico de la microbiota fecal de la diarrea similar a la disentería en una granja porcina mediante secuenciación de nueva generación
 Xi Chen1
Xi Chen1  Qing Guo2
Qing Guo2  Ying-Ying Li1
Ying-Ying Li1  Canción de Tie-Ying1
Canción de Tie-Ying1  Jun-Qing Ge1*
Jun-Qing Ge1*- 1Instituto de Biotecnología, Academia de Ciencias Agrícolas de Fujian, Fuzhou, Fujian, China
- 2Instituto de Ganadería y Medicina Veterinaria, Academia de Ciencias Agrícolas de Fujian, Fuzhou, Fujian, China
Las enfermedades entéricas porcinas, incluida la disentería porcina, implican una amplia gama de etiologías posibles y dañan gravemente el intestino de los cerdos de todas las edades. La secuenciación metagenómica de nueva generación se utiliza habitualmente en la investigación para detectar y analizar patógenos. En este estudio, se recogieron las heces de cerdos de una granja porcina comercial con diarrea similar a la disentería y se utilizaron para el análisis de la microbiota mediante secuenciación de nueva generación. Mientras que Brachyspira spp. no se detectó en muestras fecales diarreicas de cerdos, lo que indica que la enfermedad no era disentería porcina. La cantidad de población microbiana se redujo considerablemente y la composición bacteriana se alteró con una reducción en la abundancia relativa de los organismos probióticos, Firmicutes y Bacteroidetes, con un aumento de patógenos como Fusobacterium y Proteobacteria, en los que se identificaron las bacterias específicas a nivel de especie. También se detectaron patógenos virales, circovirus porcino tipo 2, herpesvirus linfotrópico porcino 1 y mastadenovirus porcino A en niveles bastante bajos. El análisis de enzimas activas de carbohidratos (CAZy) indicó que la constitución de Firmicutes y Bacteroidete también se modificó. Además, el análisis de alineación de la Enciclopedia de Genes y Genomas de Kioto (KEGG) indicó que la microbiota de los cerdos diarreicos tenía una menor capacidad para utilizar fuentes de energía, pero estaba enriquecida en vías de resistencia a múltiples fármacos. El análisis exhaustivo de la Base de Datos de Resistencia a los Antibióticos (CARD) y de los Factores de Virulencia de las Bacterias Patógenas (VFDB) indicó que los genes de resistencia a la elfamicina y la sulfonamida y el sistema de absorción de hierro estaban enriquecidos en los cerdos diarreicos. Esto reveló una posible infección bacteriana y puede guiar la selección de antibióticos para tratar la disentería. En general, nuestros datos sugirieron que las alteraciones tanto en la población como en los atributos funcionales de la microbiota en cerdos diarreicos con disminución de probióticos y aumento de microorganismos patógenos. Estos resultados ayudarán a dilucidar el mecanismo de la diarrea similar a la disentería y al desarrollo de enfoques para controlar la enfermedad.
1. Introducción
Las enfermedades entéricas porcinas, que se asociaron con diversos agentes etiológicos (1), podrían dar lugar a diarrea, bajo rendimiento del crecimiento y mortalidad variable (2). La disentería porcina es una de las enfermedades entéricas más graves (3), que a menudo se observa en cerdos de engorde, causando una tiflocolitis mucohemorrágica significativa que representa una de las mayores pérdidas económicas para los productores de cerdos (4). Aunque la disentería porcina se considera endémica en muchas regiones del mundo (5), puede controlarse con tratamientos consolidados y eficaces. Sin embargo, en los últimos años, algunas granjas porcinas comerciales en Fujian, China, a menudo desarrollaban una presunta diarrea similar a la disentería en cerdos en crecimiento, que era extremadamente difícil de curar.
Aunque algunos estudios sugirieron que la infección por disentería porcina estaba asociada con la cepa beta-hemolítica Brachyspirahyo dysenteriae (3), se requirió la colonización combinada de varios anaerobios para la aparición de disentería porcina (6, 7). Además, otros patógenos entéricos, como Salmonella enterica (8, 9), Lawsonia intracellularis (10) y Escherichia coli, también pueden causar diarrea con síntomas similares. Por lo tanto, se ha sospechado que más de un agente etiológico está implicado en la enfermedad. La secuenciación metagenómica ha demostrado numerosas ventajas sobre las tecnologías convencionales de detección dirigida al detectar y caracterizar patógenos múltiples, inesperados o nuevos, e incluso especies microbianas no cultivables (11-13). Además, el análisis de secuenciación metagenómica mostró un mayor nivel de resolución de filotipos en comparación con la secuenciación del gen 16S rRNA, lo que proporciona información más útil para descubrir posibles patógenos de infección (14), como el circovirus porcino tipo 2 (PCV2), el sapelovirus porcino (PSV), el enterovirus porcino, Escherichia coli y Mycoplasma hyorhinis (15-19).
En este estudio, se utilizó el análisis metagenómico para caracterizar la microbiota fecal de cerdos con sospecha de diarrea similar a la disentería. El objetivo principal fue explorar si la diarrea similar a la disentería era causada por B. dysenteriae, y los cambios en la composición y la capacidad funcional de la microbiota, que pueden guiar la terapia antibiótica de la enfermedad de disentería, y la estrategia para mantener la salud intestinal en cerdos.
2. Materiales y métodos
2.1. Diagnóstico clínico y recogida de muestras
Desde octubre de 2022 se observó diarrea similar a la disentería en cerdos de engorde en una granja porcina comercial ubicada en Fujian, China. A los cerdos se les administró una vacunación regular contra el circovirus porcino tipo 2 (PCV2), el virus de la fiebre aftosa y otros patógenos comunes. Las lesiones clínicas incluían un colon flácido a lleno de líquido con hiperemia serosa y expansión variable del mesocolon por edema. Las lesiones eran multifocales y se observaban con frecuencia en las espirales centrípetas y en el ápice del colon espiral, pero también se extendían a través de la porción centrífuga hasta las caras terminales con el tiempo. El análisis microscópico de las huellas de las heces no reveló el patógeno parasitario. Se inició tratamiento con antibióticos como mequindox o cefotaxima sódica inyectable, pero no tuvo efecto terapéutico. En noviembre de 2022, se recogieron 4 muestras de heces tanto de cerdos diarreicos (grupo DP) como de cerdos sanos (grupo HP) de alrededor de 90 días de edad, respectivamente. Todos los cerdos muestreados fueron diagnosticados recientemente y nunca habían recibido antibióticos u otros tratamientos. Las muestras se homogeneizaron inmediatamente después de la recolección y se almacenaron a -80 °C hasta su posterior procesamiento.
2.2. Aislamiento del ADN
El ADN genómico se extrajo de cada muestra de heces utilizando un Qiamp Fast DNA Stool Mini Kit (Qiagen, Alemania) de acuerdo con las instrucciones del fabricante. La calidad del ADN aislado se evaluó mediante un instrumento NanoDrop One (Thermo Fisher Scientific, Estados Unidos). El ADN aislado se cuantificó utilizando un fluorómetro de sobremesa Qubit 3.0 (Thermo Fisher Scientific, Estados Unidos) y se almacenó a -80 °C hasta su uso.
2.3. Construcción de bibliotecas y secuenciación metagenómica
Las bibliotecas de secuenciación se construyeron a partir de muestras de ADN aisladas utilizando un kit de preparación de bibliotecas de ADN ALFA-SEQ (Fangzhou Biological Technology Co, China) de acuerdo con los protocolos del fabricante. La calidad de la biblioteca se evaluó en el fluorómetro Qubit 4.0 (Life Technologies, NY) y en el sistema de análisis de proteínas de ácidos nucleicos de alto rendimiento Qsep400 (Houze Biological Technology Co, China). Por último, la biblioteca se secuenció en una plataforma Illumina NovaSeq 6.000 que empleaba una secuenciación de extremos emparejados de 150 pb.
2.4. Ensamblaje del metagenoma y procesamiento estadístico de datos
Las lecturas en bruto de los datos de secuenciación se convirtieron en archivos FASTQ utilizando el software Casava (versión 1.8.2). La evaluación de la calidad se realizó con Trimmomactic (Versión 0.36) para obtener lecturas limpias. Estas lecturas limpias se agruparon por tema y se ensamblaron usando MEGAHIT (Versión 1.0.6). Después del ensamblaje mixto, se obtuvieron los Scaftigs y se filtraron para su análisis estadístico. Los Scaftigs (≥500 pb) ensamblados a partir de simples y mixtos se utilizaron para predecir el ORF y se filtraron mediante MetaGeneMark (versión 3.38). Se empleó el CD-HIT (Versión 4.7) para eliminar la redundancia y obtener el catálogo inicial único de genes (Unigenes). Los datos limpios de cada muestra se asignaron al catálogo inicial de genes utilizando el software BBMAP (versión 38.79) para obtener la información de abundancia de genes individuales en cada muestra.
2.5. Predicción taxonómica y análisis de abundancia
Se utilizó el software DIAMOND (versión 0.9.30) para el análisis de los unógenos de bacterias, hongos, arqueas y virus extraídos de la base de datos NR del NCBI. Como cada secuencia puede tener varios resultados alineados, elija el resultado del cual el valor e 1e−10 tomar el algoritmo LCA que se aplica a la clasificación del sistema del software MEGAN (versión 6.22.1) para asegurarse de que la información de anotación de especies de las secuencias. Los resultados del análisis de abundancia relativa y de coordenadas principales (ACP) se basaron en la tabla de abundancia de cada jerarquía taxonómica. La composición microbiana en cada nivel taxonómico con disimilitudes significativas se exhibió mediante mapas de calor sobre abundancias relativas y se analizó posteriormente mediante la prueba de suma de rangos de Wilcoxon para mostrar diferencias entre los organismos. P ≤ 0,05 se consideró una diferencia significativa.
2.6. Anotaciones de bases de datos funcionales
Se utilizó el software DIAMOND (versión 0.9.30) para realizar el análisis de brusone Unigenes con bases de datos funcionales que incluyen la base de datos de enzimas activas de carbohidratos (CAZy), la base de datos de la Enciclopedia de Genes y Genomas de Kioto (KEGG), la Base de Datos Integral de Resistencia a los Antibióticos (CARD) y la base de datos de Factores de Virulencia de Bacterias Patógenas (VFDB). La abundancia relativa de cada jerarquía funcional fue igual a la suma de la abundancia relativa anotada a ese nivel funcional. La tabla de números de genes de cada muestra en cada jerarquía taxonómica se obtuvo a partir del resultado de la anotación de funciones y la tabla de abundancia de genes. Se utilizaron mapas de calor y análisis de tamaño del efecto de análisis discriminante lineal (LEfSe versión 1.1.01) (la puntuación LDA fue de 3) para encontrar las diferencias en las funciones del microbioma entre los dos grupos.
3. Resultados
3.1. Cambios en las frecuencias unigénicas y la diversidad microbiana entre cerdos diarreicos y cerdos sanos
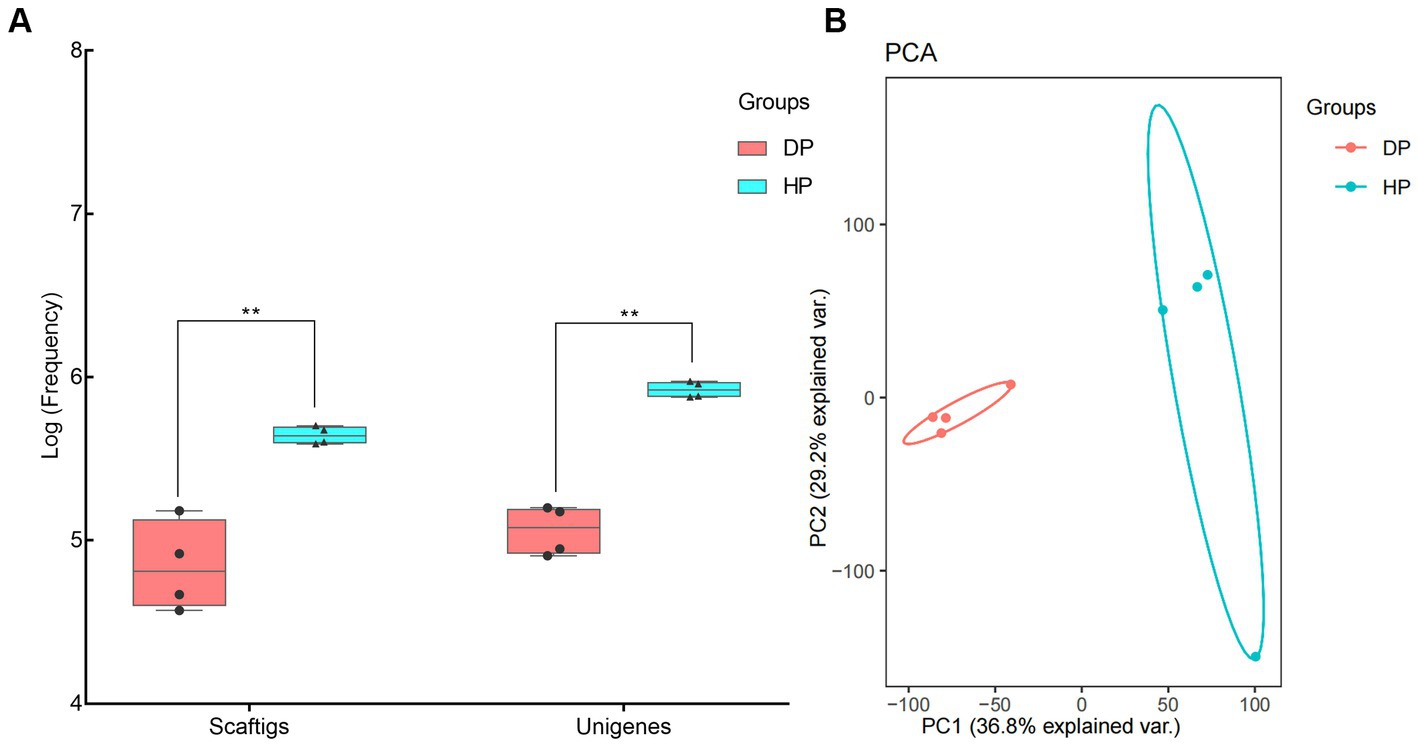
El ADN genómico se extrajo de muestras de heces de cerdos diarreicos y sanos, y se utilizó para la secuenciación metagenómica. Se obtuvo un promedio de 16, 976, 998 y 58, 985, 502 lecturas limpias del grupo diarreico y del grupo sano, respectivamente. El análisis posterior de Blast indicó que el 64,5% de las lecturas limpias del grupo diarreico pertenecían al huésped, mientras que solo el 3,2% en cerdos sanos era solo el 79,623%. Se armó un promedio de 441, 611 y 119, 426 Scaftigs del grupo diarreico y del grupo sano, respectivamente, representando 840, 750 y 1, 1 Unigenes (Figura <>A). Las frecuencias de Scaftigs y Unigenes tuvieron diferencias significativas entre cerdos diarreicos y sanos. El análisis posterior de componentes principales (ACP) verificó esta diferencia en la estructura de la microbiota fecal entre los dos grupos (Figura <>B).
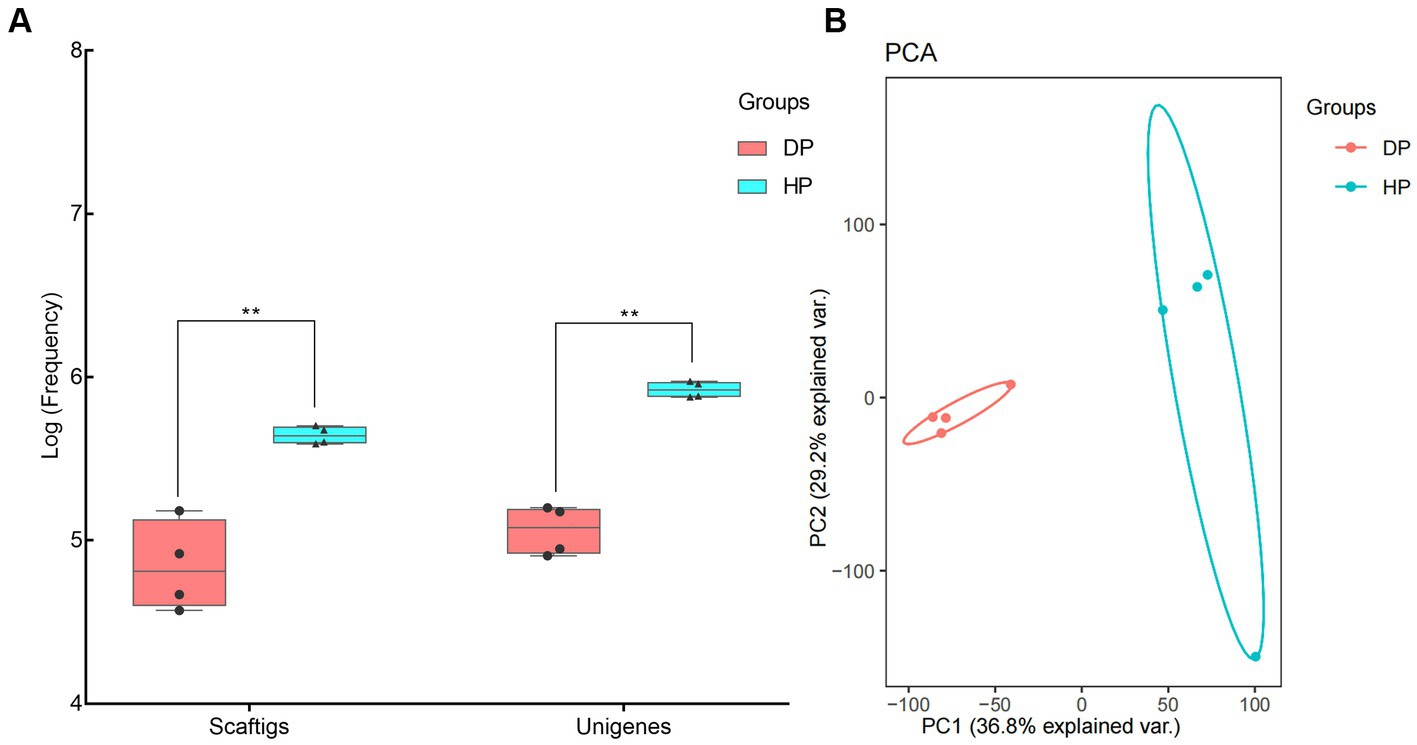 Figura 1. Identificación de la microbiota fecal de cerdos asociada a disentería mediante secuenciación metagenómica. (A) Valores de frecuencia de Scaftigs y Unigenes identificados en cerdos diarreicos y sanos, (B) Análisis de componentes principales (PCA) de la microbiota fecal entre cerdos diarreicos y sanos. DP: cerdos diarreicos, HP: cerdos sanos, **valores de p representados <0,01.
Figura 1. Identificación de la microbiota fecal de cerdos asociada a disentería mediante secuenciación metagenómica. (A) Valores de frecuencia de Scaftigs y Unigenes identificados en cerdos diarreicos y sanos, (B) Análisis de componentes principales (PCA) de la microbiota fecal entre cerdos diarreicos y sanos. DP: cerdos diarreicos, HP: cerdos sanos, **valores de p representados <0,01.
3.2. Comparación de la composición bacteriana entre cerdos diarreicos y sanos
Se evaluaron muestras de cerdos diarreicos y sanos para evaluar la composición bacteriana a nivel de filo, género y especie. Firmicutes, Bacteroidetes, Proteobacterias, Spirochaetes y Actinobacteria fueron los filos más abundantes de la microbiota tanto en cerdos diarreicos como sanos (Figura 2A). Sin embargo, se encontró una mayor abundancia de bacterias pertenecientes a Firmicutes, Bacteroidetes y Spirochaetes en cerdos sanos, mientras que las bacterias pertenecientes a Proteobacteria se encontraron en mayor abundancia en cerdos diarreicos. Cabe destacar que las bacterias pertenecientes a Fusobacteria se observaron en un nivel extremadamente mayor en los cerdos diarreicos (1,21% en promedio) que en los cerdos sanos (0,08% en promedio) (Figura 2A). Entre todos los géneros identificados, se encontraron diferencias estadísticamente significativas en los representantes de Prevotella, Clostridium, Bacteroides, Porphyromonas, Actinobacillus, Moraxella, Ruminococcus y Oscillibacter. La abundancia de Porphyromonas, Actinobacillus y Moraxella en el grupo diarreico aumentó a 5,73, 5,37 y 4,57%, respectivamente, mientras que la abundancia de Porphyromonas, Actinobacillus y Moraxella en los animales sanos fue de 0,08, 0,02 y 0,02%, respectivamente. Además, las abundancias relativas de Prevotella, Clostridium, Bacteroides, Ruminococcus y Oscillibacter se redujeron en cerdos diarreicos (Figura 2B).
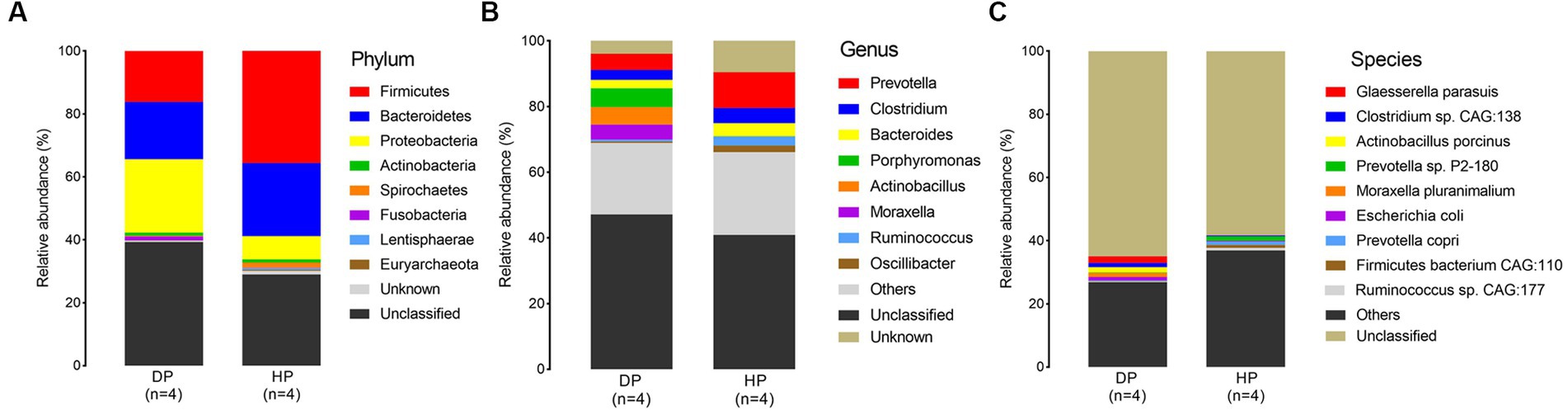 Figura 2. Composición microbiana de la microbiota fecal en cerdos diarreicos y sanos mediante análisis metagenómico. (A) nivel de filo, (B) nivel de género y (C) nivel de especie. DP: cerdos diarreicos, HP: cerdos sanos.
Figura 2. Composición microbiana de la microbiota fecal en cerdos diarreicos y sanos mediante análisis metagenómico. (A) nivel de filo, (B) nivel de género y (C) nivel de especie. DP: cerdos diarreicos, HP: cerdos sanos.
A nivel de especie, Glaesserella parasuis, Clostridium sp. CAG:138, Actinobacillus porcinus, Moraxella pluranimalium y Escherichia coli mostraron una abundancia extremadamente mayor en cerdos diarreicos, mientras que la abundancia de Prevotella sp. P2-180, Prevotella copri, Firmicutes bacterium CAG:110 y Ruminococcus sp. CAG:177 fue menor en cerdos diarreicos (Figura 2C ). El análisis posterior de la prueba Wilcoxon-Rank indicó que se sospechaba que la mayoría de las especies enriquecidas en cerdos diarreicos eran bacterias patógenas, como las pertenecientes a Moraxella, Actinobacillus (2 especies cada una), así como Clostridium sp. CAG:138, Glaesserella parasuis, Escherichia coli y Streptococcus suis. Además, se anotó que las especies con mayor abundancia en los cerdos sanos eran especies probióticas, como 5 especies en Firmicutes, 4 especies en Prevotella, Ruminococcus flavefaciens, Butyricicoccus porcorum y Streptococcus gallolyticus (Figura 3).
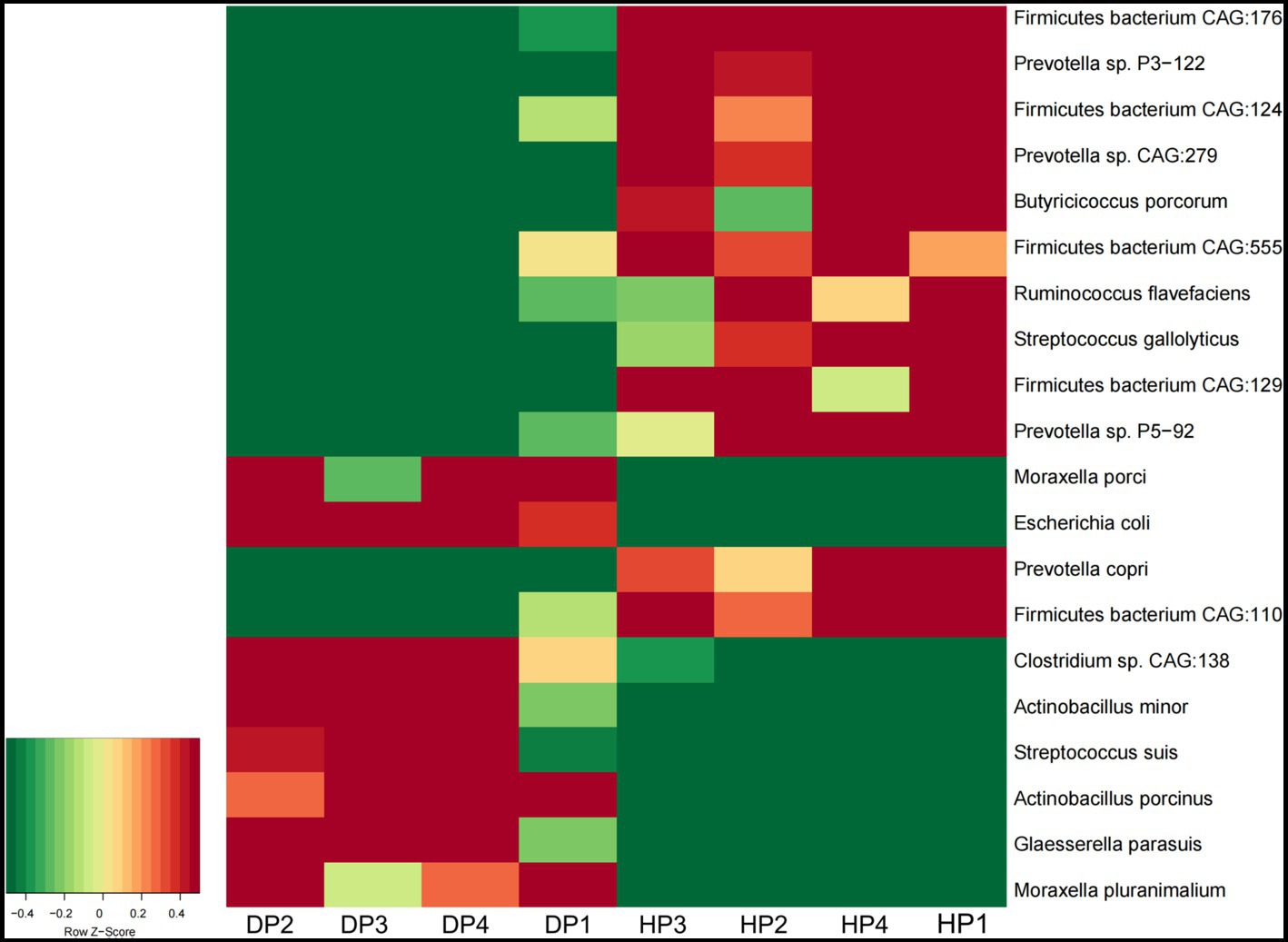 Figura 3. La abundancia de la microbiota fecal en cerdos diarreicos y cerdos sanos analizada mediante la prueba de Wilcoxon-Rank. La escala de colores representa un valor de expresión bajo en verde y un valor de expresión alto en rojo. DP: cerdos diarreicos, HP: cerdos sanos.
Figura 3. La abundancia de la microbiota fecal en cerdos diarreicos y cerdos sanos analizada mediante la prueba de Wilcoxon-Rank. La escala de colores representa un valor de expresión bajo en verde y un valor de expresión alto en rojo. DP: cerdos diarreicos, HP: cerdos sanos.
3.3. Identificación de Brachyspira hyodysenteriae, Salmonella enterica, Lawsonia intracellularis y virus porcinos en cerdos diarreicos y sanos
Dado que algunos patógenos potenciales sospechosos, como B. hyodysenteriae, S. enterica, L. intracellularis y virus porcinos, no fueron identificados por el análisis de composición bacteriana, realizamos una exploración independiente adicional para examinar la presencia de estos agentes etiológicos. Los resultados mostraron que, si bien no se detectó B. hyodysenteriae, se identificaron S. enterica, L. intracellularis, circovirus porcino tipo 2 (PCV-2), herpesvirus linfotrópico porcino 1 (PLHV-1) y mastadenovirus porcino A (PAdV-A) tanto en los cerdos diarreicos como en los sanos. Dado que estas bacterias (S. enterica y L. intracellularis) (datos no mostrados) y los virus (PCV-2, PLHV-1 y PAdV-A) (Figura 4) eran significativamente bajos en abundancia con un rango de 0,0001 a 0,005%, y no tenían diferencias entre cerdos diarreicos y sanos, indicó que estos microorganismos identificados podrían no ser los patógenos causantes de la disentería.
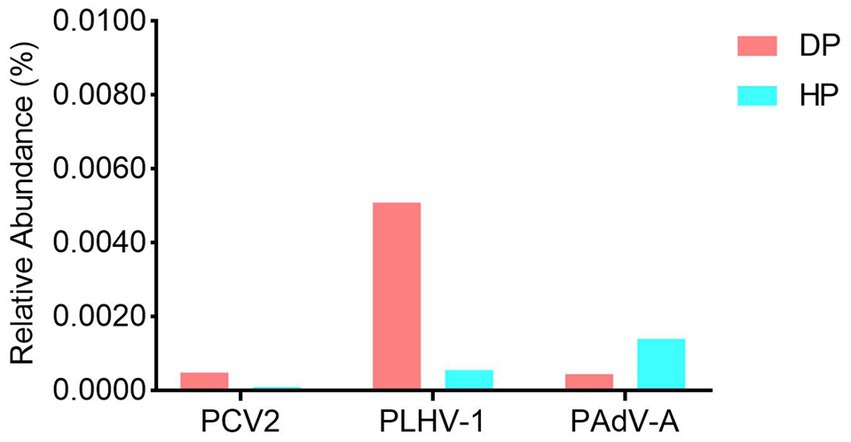 Figura 4. Análisis del perfil viral de la microbiota fecal porcina en cerdos diarreicos y sanos. DP: cerdos diarreicos, HP: cerdos sanos.
Figura 4. Análisis del perfil viral de la microbiota fecal porcina en cerdos diarreicos y sanos. DP: cerdos diarreicos, HP: cerdos sanos.
3.4. Comparación del análisis funcional de la microbiota entre cerdos diarreicos y sanos
Se realizaron análisis CAZy y KEGG para investigar la asociación de la capacidad funcional del microbioma intestinal de los cerdos con la disentería. Se identificaron 32.852 enzimas mediante el análisis CAZy, de las cuales 19.875 se clasificaron como glucósidos hidrolasas (GH) y 8.373 pertenecían a glicosiltransferasas (GT). Se observaron cambios significativos en 35 de las enzimas CAZy más abundantes. Principalmente, 17 enzimas fueron más abundantes en cerdos sanos que en cerdos diarreicos, la mayoría de las cuales pertenecían a glucósidos hidrolasas. De las 18 enzimas con mayor abundancia en cerdos diarreicos, la mayoría de ellas se clasificaron como glicosiltransferasas (GT4, GT9, GT13, GT19, GT23, GT28, GT35, GT51) y módulos de unión a carbohidratos (CBM32, CBM48, CBM50) (Figura 5A). Entre las 35 vías KEGG más abundantes identificadas, 33 estaban enriquecidas en la replicación del ADN, el metabolismo de los nutrientes y la vía de biosíntesis (por ejemplo, el metabolismo del carbono, el metabolismo del almidón y la sacarosa, el metabolismo de la glicina, la serina y la treonina, la exportación de proteínas, la biosíntesis de aminoácidos y la biosíntesis de metabolitos secundarios). Estas vías tuvieron una mayor abundancia en el grupo sano, mientras que solo 2 vías, los transportadores de casetes de unión a ATP (ABC) y el ciclo del citrato (TCA) se enriquecieron en cerdos diarreicos (Figura 5B).
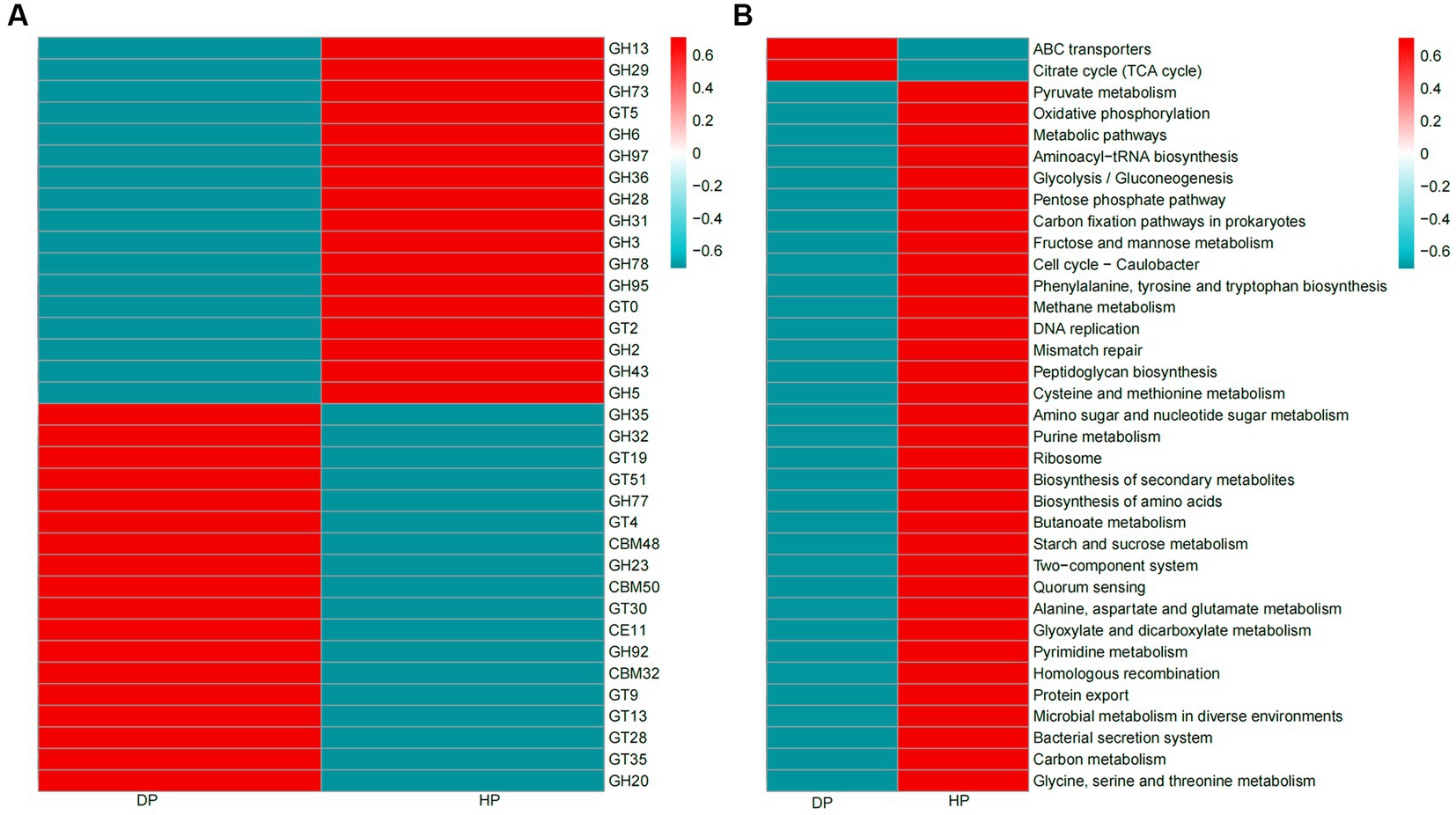 Figura 5. Mapas de calor de las enzimas CAZy y de las vías KEGG de la microbiota fecal de cerdos diarreicos y sanos. (A) enzimas CAZy, (B) vías KEGG. La escala de colores representa un valor de expresión bajo en azul y un valor de expresión alto en rojo. DP: cerdos diarreicos, HP: cerdos sanos.
Figura 5. Mapas de calor de las enzimas CAZy y de las vías KEGG de la microbiota fecal de cerdos diarreicos y sanos. (A) enzimas CAZy, (B) vías KEGG. La escala de colores representa un valor de expresión bajo en azul y un valor de expresión alto en rojo. DP: cerdos diarreicos, HP: cerdos sanos.
Dado que las alteraciones en los transportadores ABC y el ciclo del TCA podrían estar asociadas con la resistencia bacteriana a múltiples fármacos, se realizó una investigación adicional mediante el análisis de blastos de los Unigenes en las bases de datos CARD y VFDB. El análisis de CARD identificó 30 genes que codificaban la resistencia a los antibióticos. Los cambios entre el grupo diarreico y el grupo sano fueron analizados más a fondo por LEfSe. Los resultados demostraron que cinco genes de resistencia a tetraciclina, oxazolidinona, pleuromutilina, fluoroquinolona y aminocumarina, estaban enriquecidos en cerdos sanos, mientras que dos genes de resistencia, que representan sulfonamida y elfamicina, estaban enriquecidos en cerdos diarreicos (Figura 6A). Los alineamientos con VFDB identificaron 14 grupos de genes en los que se observaron cambios significativos en la abundancia de 5 genes. Estos genes estaban relacionados con el sistema inmunitario (antifagocitosis, resistencia sérica, proteasa del complemento y proteasa Ig) y la asimilación de micronutrientes (sistema de absorción de hierro). Entre estos, se encontró que los genes que representan la proteasa del complemento, la proteasa Ig y el sistema de captación de hierro están enriquecidos en cerdos diarreicos (Figura 6B).
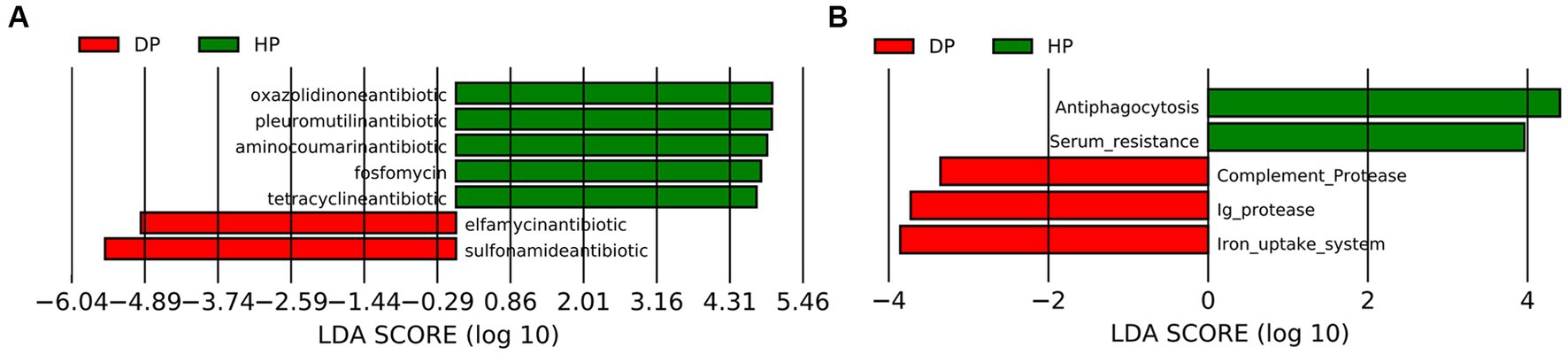 Figura 6. Análisis de la microbiota fecal en cerdos diarreicos y sanos mediante análisis LEfSe. (A) Análisis de la base de datos CARD, (B) Análisis de la base de datos VFDB. DP: cerdos diarreicos, HP: cerdos sanos. El eje X muestra las puntuaciones LDA.
Figura 6. Análisis de la microbiota fecal en cerdos diarreicos y sanos mediante análisis LEfSe. (A) Análisis de la base de datos CARD, (B) Análisis de la base de datos VFDB. DP: cerdos diarreicos, HP: cerdos sanos. El eje X muestra las puntuaciones LDA.
4. Discusión
Las enfermedades entéricas porcinas, como la disentería porcina, son problemas importantes que causan enormes pérdidas económicas en todo el mundo y son una de las principales razones del uso extensivo de antibióticos (2). B. hyodysenteriae se considera el agente etiológico de la disentería porcina, que puede aparecer en las heces de los cerdos de 1 a 4 días antes de la observación de los signos clínicos (20). Estas bacterias pueden identificarse fácilmente mediante el cultivo en medios selectivos (21). También pueden someterse a amplificación por PCR con los cebadores dirigidos al gen 16S rRNA (22), el gen nox (23) o el gen de la hemolisina tlyA (24) para su identificación. En este estudio, no se observaron resultados positivos con ninguno de los métodos cuando se analizaron las heces recolectadas de los cerdos diarreicos (los datos se muestran ahora), y el análisis de secuenciación basado en metagenómica adicional tampoco detectó Brachyspira spp. Por lo tanto, la enfermedad no era disentería porcina y debía estar relacionada con otros agentes etiológicos.
La microbiota intestinal se considera un factor clave de susceptibilidad a los trastornos intestinales (25). Estudios anteriores sobre la microbiota también demostraron que la diversidad de la comunidad microbiana podría verse afectada comúnmente por la diarrea (26). Los resultados de este estudio también demostraron que la diarrea similar a la disentería dio lugar a alteraciones notables en la comunidad microbiana, y que la abundancia de la población microbiana se vio significativamente modificada por una disminución de Unigenes en cerdos diarreicos. Sin embargo, los cambios en la composición de las bacterias fueron bastante diferentes de los estudios metagenómicos previos sobre la disentería porcina (27-29). Una de las características obvias fue la disminución de la abundancia de especies probióticas, como Firmicutes y Bacteroidetes. Los patrones de abundancia relativa de estos dos filos se revelaron de manera más distintiva a nivel de género y especie. Las especies de Firmicutes son miembros constantes de la microbiota intestinal normal de los cerdos (30), que pueden producir ácidos grasos de cadena corta, regular las respuestas inmunitarias sistémicas, mantener el equilibrio energético, inhibir los patógenos oportunistas y suprimir la inflamación intestinal excesiva (31). También a diferencia de estudios anteriores (29, 32), se observó que Prevotella, que representaba una parte importante de Bacteroidetes, estaba disminuida en los cerdos diarreicos. Como otra especie bacteriana probiótica común, Prevotella se detecta con frecuencia en el contenido fecal de los cerdos (30). Tiene la capacidad de degradar los polisacáridos vegetales (33), aumentar la acumulación de grasa (34) y mediar reacciones inflamatorias (35) La bacteria Prevotella también tiene una correlación negativa con el grupo Escherichia (36). La reducción de Firmicutes y Bacteroidetes podría afectar la absorción de nutrientes y la regulación antiinflamatoria del huésped y causar un mayor daño a la diarrea.
El aumento en la abundancia de Fusobacterias y Proteobacterias patógenas en este estudio es consistente con otros estudios (28, 37). Tanto Fusobacterium como Proteobacteria están asociadas con trastornos inflamatorios intestinales y se sabe que desempeñan funciones patógenas en el desarrollo de la diarrea porcina (38) En este estudio se detectaron especies específicas de Proteobacteria relacionadas con la disentería. M. porci y M. pluranimalium se consideraron patógenos oportunistas y fuentes potenciales de determinantes de resistencia a polimixinas similares a MCR (39). Además, A. minor y A. porcinus podrían producir citotoxinas virulentas en cerdos (40). E. coli es un factor de riesgo muy importante para el desarrollo de diarrea, y también está fuertemente correlacionado con la disbiosis intestinal predispuesta a la mortalidad (41). Además, G. parasuis puede desencadenar una respuesta proinflamatoria con poliserositis, artritis y sepsis en cerdos (42). También se demostró que S. suis induce una respuesta proinflamatoria con signos clínicos como artritis, neumonía o endocarditis (43). Combinando estos hallazgos, estas especies de Proteobacteria podrían estar asociadas con la diarrea similar a la disentería, pero sus funciones específicas en la aparición de la disentería necesitan más investigación. Además, aunque S. enterica y L. intracellularis son patógenos intestinales muy comunes en los cerdos en China, ambos se detectaron con una abundancia bastante baja tanto en cerdos diarreicos como sanos, su papel en la microbiota diarreica necesita más investigación.
La microbiota intestinal del cerdo podría secretar un gran repertorio de CAZymes que pueden descomponer y metabolizar los polisacáridos (44). Estas CAZymes se atribuyen principalmente a las especies probióticas Firmicutes y Bacteroidetes (45, 46). En concreto, las enzimas GH representan generalmente la capacidad de la microbiota para degradar los hidratos de carbono (47), mientras que las enzimas GT y CBM podrían estar implicadas en la biosíntesis de los hidratos de carbono (48, 49). Por lo tanto, es probable que la regulación a la baja de los GH y la regulación al alza de los GT y las medidas de fomento de la confianza en los cerdos diarreicos se atribuyan a la reducción de las especies originales y al aumento de nuevas especies de Firmicutes y Bacteroidetes.
La capacidad funcional del microbioma intestinal asociado con la disentería se exploró más a fondo a través de análisis KEGG. Los resultados mostraron que la capacidad de la microbiota fecal para asimilar y utilizar múltiples fuentes de energía se redujo drásticamente, lo que fue consistente con el hallazgo de que la población microbiana se redujo significativamente en los cerdos diarreicos. La microbiota fecal de los cerdos diarreicos se enriqueció en genes transportadores ABC y genes del ciclo TCA. Se ha considerado que los transportadores ABC desempeñan un papel importante en la resistencia a los medicamentos de las bacterias patógenas (50), y el ciclo del TCA se relacionó con el número y la formación de células persistentes, que son variantes fenotípicas en las poblaciones bacterianas (51). Estos resultados podrían estar relacionados con el aumento de la capacidad de resistencia a múltiples fármacos de las bacterias en cerdos con disentería.
Se realizó un análisis CARD para explorar posibles genes de resistencia a los antibióticos. Se observó una mayor abundancia de genes de resistencia a tetraciclina, oxazolidinona, pleuromutilina y fluoroquinolonas en la microbiota de cerdos sanos, lo que es consistente con estudios previos (52, 53). Sin embargo, se encontró que los genes de resistencia a la elfamicina y los genes de resistencia a la sulfonamida estaban enriquecidos en cerdos diarreicos. La elfamicina es un grupo de antibióticos estructuralmente diversos comúnmente producidos por cepas de Streptomyces (54). La resistencia a la sulfonamida suele estar presente en Enterococcus, Bacillus spp. y E. coli (55-57). Por lo tanto, nuestros resultados deberían llamar la atención para ajustar la aplicación de estos antibióticos. También es una advertencia sobre la propagación de estos patógenos potenciales.
Las variaciones en los factores de virulencia también podrían ayudar a prevenir patógenos potenciales (58). Dada una amplia gama de factores, es difícil establecer una conexión entre los factores de virulencia relacionados con el sistema inmunitario y los patógenos específicos. Además, las diferencias significativas entre los dos grupos podrían representar los cambios en las especies de bacterias patógenas. Además, el sistema de absorción de hierro es el mecanismo para que algunas bacterias patógenas obtengan hierro de sus huéspedes (59), lo cual es esencial para el crecimiento y la colonización exitosa de estos patógenos (60). Las especies con sistema de absorción de hierro abarcaron tanto bacterias Gram-positivas como Gram-negativas, los patógenos relacionados con los transportadores ABC en la microbiota porcina fueron limitados, como Campylobacter jejuni, Yersinia enterocolitica y E. coli. Por lo tanto, estos patógenos también podrían contribuir al desarrollo de la disentería.
5. Conclusión
En este estudio, confirmamos que Brachyspira spp. no era la causa patogénica de la diarrea porcina local, aunque los síntomas clínicos observados de la enfermedad eran similares a la disentería porcina. Encontramos que la microbiota fecal de los cerdos con disentería se modificó significativamente, incluyendo (i) una cantidad extremadamente baja de la población microbiana; ii) una reducción de las especies probióticas de Firmicutes y Bacteroidetes, así como un aumento de las especies patógenas de Fusobacterium y Proteobacteria; (iii) presencia de los patógenos virales PCV-2, PLHV-1 y PAdV-A. Un análisis posterior de CAZymes indicó que la estructura de Firmicutes y Bacteroidete también se modificó. La investigación de la capacidad funcional demostró que la microbiota fecal de los cerdos diarreicos tenía una menor capacidad para utilizar las fuentes de energía y una mayor resistencia a múltiples fármacos, lo que se confirmó por las modificaciones en los genes de resistencia a los antibióticos y los genes del factor de virulencia. Nuestros resultados pueden ayudar en la prevención de los patógenos y en la selección de antibióticos adecuados para el tratamiento de la diarrea similar a la disentería en cerdos.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/, PRJNA991276.
Declaración ética
El estudio en animales fue aprobado por el Instituto de Biotecnología de la Academia de Ciencias Agrícolas de Fujian. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
XC: Conceptualización, Análisis formal, Metodología, Redacción – borrador original. QG: Conceptualización, Metodología, Redacción – revisión y edición. Y-YL: Metodología, Redacción – revisión y edición. T-YS: Conceptualización, Escritura – revisión y edición. J-QG: Conceptualización, Obtención de fondos, Supervisión, Redacción, revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Esta investigación fue financiada por el Proyecto Principal de Ciencia y Tecnología de Fujian (2021NZ029023) y el Proyecto de Innovación Colaborativa «5511» de la Academia de Ciencias Agrícolas de Fujian (XTCXGC2021013).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Robbins, RC, Almond, G, y Byers, E. Enfermedades y trastornos porcinos. Sistema Agroalimentario de la Encíclica. (2014) 5:261–76. doi: 10.1016/B978-0-444-52512-3.00134-0
2. Cornelison, AS, Karriker, LA, Williams, NH, Haberl, BJ, Stalder, KJ, Schulz, LL, et al. Impacto de los desafíos sanitarios en el rendimiento del crecimiento de los cerdos, las características de la canal y los rendimientos netos en condiciones comerciales. Transl Anim Sci. (2018) 2:50–61. doi: 10.1093/tas/txx0005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Burrough, ER. Disentería porcina: etiopatogenia y diagnóstico de una enfermedad reemergente. Veterinario Pathol. (2017) 54:22–31. doi: 10.1177/0300985816653795
4. Mirajkar, NS, y Gebhart, CJ. Comprensión de la epidemiología molecular y las relaciones globales de Brachyspira hyodysenteriae de piaras porcinas en los Estados Unidos: un enfoque de tipificación de secuencias de múltiples locus. PLoS Uno. (2014) 9:E107176. doi: 10.1371/journal.pone.0107176
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Hartnack, S, Nathues, C, Nathues, H, Grosse, BE, y Lewis, FI. Estimación de la precisión de las pruebas diagnósticas para Brachyspira hyodysenteriae teniendo en cuenta las complejidades de la estructura poblacional en animales destinados a la alimentación. PLoS Uno. (2014) 9:e98534. doi: 10.1371/journal.pone.0098534
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Neef, NA, Lysons, RJ, Trott, DJ, Hampson, DJ, Jones, PW y Morgan, JH. Patogenicidad de espiroquetas intestinales porcinas en cerdos gnotobióticos. Infectar a Immun. (1994) 62:2395–403. doi: 10.1128/iai.62.6.2395-2403.1994
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Whipp, SC, Robinson, IM, Harris, DL, Glock, RD, Matthews, PJ y Alexander, TJ. Sinergismo patogénico entre Treponema hyodysenteriae y otros anaerobios seleccionados en cerdos gnotobióticos. Infectar a Immun. (1979) 26:1042–7. doi: 10.1128/iai.26.3.1042-1047.1979
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Li, Y, Ed-Dra, A, Tang, B, Kang, X, Müller, A, Kehrenberg, C, et al. Mayor tolerancia de los serovares predominantes de Salmonella que circulan en las granjas de piensos libres de antibióticos a las tensiones ambientales. J Hazard Mater. (2022) 438:129476. doi: 10.1016/j.jhazmat.2022.129476
9. Tang, B, Elbediwi, M, Nambiar, RB, Yang, H, Lin, J y Yue, M. Caracterización genómica de Salmonella enterica resistente a los antimicrobianos en granjas de patos, pollos y cerdos y mercados minoristas en el este de China. Microbiol Spectr. (2022) 10:e0125722. doi: 10.1128/spectrum.01257-22
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Vannucci, FA, y Gebhart, CJ. Avances recientes en la comprensión de la patogenia de las infecciones por Lawsonia intracellularis. Veterinario Pathol. (2014) 51:465–77. doi: 10.1177/0300985813520249
11. Vibin, J, Chamings, A, Klaassen, M, Bhatta, TR y Alexandersen, S. Caracterización metagenómica de parvovirus aviares y picornavirus de patos silvestres australianos. Sci Rep. (2020) 10:12800. doi: 10.1038/s41598-020-69557-z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Glendinning, L, Stewart, RD, Pallen, MJ, Watson, KA y Watson, M. Ensamblaje de cientos de nuevos genomas bacterianos del ciego de pollo. Genoma Biol. (2020) 21:34. doi: 10.1186/s13059-020-1947-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Bhatta, TR, Chamings, A, Vibin, J y Alexandersen, S. Detección y caracterización de astrovirus canino, parvovirus canino y virus del papiloma canino en cachorros mediante secuenciación de nueva generación. Sci Rep. (2019) 9:4602. doi: 10.1038/s41598-019-41045-z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Jiang, H, Fang, S, Yang, H y Chen, C. Identificación de la relación entre el microbioma intestinal y la eficiencia alimenticia en una cohorte de cerdos comerciales. J Anim Sci. (2021) 99:99. doi: 10.1093/jas/skab045
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Zhang, Y, Gan, Y, Bao, H y Wang, R. Perturbaciones del microbioma intestinal y el metaboloma de cerdos infectados con Mycoplasma hyorhinis. J Sci Food Agric. (2023) 103:6219–32. doi: 10.1002/jsfa.12690
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Wang, L, Wu, Y, Xu, J, Huang, Q, Zhao, Y, Dong, S, et al. Colicinas de Escherichia coli Conducen a la resistencia contra el patógeno causante de diarrea E. coli enterotoxigénica en cerdos. Microbiol Spectr. (2022) 10:e139622. doi: 10.1128/spectrum.01396-22
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Harima, H, Kajihara, M, Simulundu, E, Bwalya, E, Qiu, Y, Isono, M, et al. Diversidad genética y biológica de los sapelovirus porcinos predominantes en Zambia. Virus. (2020) 12:12. doi: 10.3390/v12020180
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Tsuchiaka, S, Naoi, Y, Imai, R, Masuda, T, Ito, M, Akagami, M, et al. Diversidad genética y recombinación de cepas de enterovirus G en cerdos japoneses: alta prevalencia de cepas portadoras de una secuencia de cisteína proteasa similar a la papaína en la población de enterovirus G. PLoS Uno. (2018) 13:e190819:e0190819. doi: 10.1371/journal.pone.0190819
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Blomstrom, AL, Fossum, C, Wallgren, P, y Berg, M. El análisis metagenómico viral muestra la situación de coinfección en cerdos sanos y afectados por PMWS. PLoS Uno. (2016) 11:e166863. doi: 10.1371/journal.pone.0166863
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Kinyon, JM, Harris, DL y Glock, RD. Enteropatogenicidad de varios aislados de Treponema hyodysenteriae. Infectar a Immun. (1977) 15:638–46. doi: 10.1128/iai.15.2.638-646.1977
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Calderaro, A, Merialdi, G, Perini, S, Ragni, P, Guegan, R, Dettori, G, et al. Un método novedoso para el aislamiento de Brachyspira (Serpulina) hyodysenteriae de cerdos con disentería porcina en Italia. Microbiol veterinario. (2001) 80:47–52. doi: 10.1016/s0378-1135(00)00374-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Phillips, ND, La, T y Hampson, DJ. Desarrollo de un ensayo de PCR dúplex anidado en dos pasos para la detección rápida de Brachyspira pilosicoli y Brachyspira intermedia en heces de pollo. Microbiol veterinario. (2006) 116:239–45. doi: 10.1016/j.vetmic.2006.03.020
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Weissenbock, H, Maderner, A, Herzog, AM, Lussy, H y Nowotny, N. La amplificación y secuenciación de porciones específicas de Brachyspira spp. de NOx utilizando muestras de tejido incluidas en parafina de colitis clínica en cerdos austriacos muestra una presencia frecuente y solitaria de Brachyspiramurdochii. Microbiol veterinario. (2005) 111:67–75. doi: 10.1016/j.vetmic.2005.09.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Fellstrom, C, Zimmerman, U, Aspan, A, y Gunnarsson, A. El uso de cultivo, muestras agrupadas y PCR para la identificación de rebaños infectados con Brachyspira hyodysenteriae. Anim Health Res Rev. (2001) 2:37–43. doi: 10.1079/AHRR200120
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Ley, RE, Hamady, M, Lozupone, C, Turnbaugh, PJ, Ramey, RR, Bircher, JS, et al. Evolución de los mamíferos y sus microbios intestinales. Ciencia. (2008) 320:1647–51. doi: 10.1126/science.1155725
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Gryaznova, MV, Dvoretskaya, YD, Syromyatnikov, MY, Shabunin, SV, Parshin, PA, Mikhaylov, EV, et al. Cambios en el perfil del microbioma en diferentes partes del intestino en lechones con diarrea. Animales (Basilea). (2022) 12:320. doi: 10.3390/ani12030320
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Costa, MO, Chaban, B, Harding, JC, y Hill, JE. Caracterización de la microbiota fecal de cerdos antes y después de la inoculación con «Brachyspira hampsonii«. PLoS Uno. (2014) 9:e106399. doi: 10.1371/journal.pone.0106399
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Burrough, ER, Arruda, BL, y Plummer, PJ. Comparación de la microbiota luminal y asociada a la mucosa en el colon de cerdos con y sin disentería porcina. Front Vet Sci. (2017) 4:139. doi: 10.3389/fvets.2017.00139
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Daniel, A, Pereira, C, Dorella, F, Pereira, FL, Laub, RP, Andrade, MR, et al. Efecto sinérgico de la coinfección por Brachyspira hyodysenteriae y Lawsonia intracellularis: evaluación anatomopatológica y del microbioma. Animales (Basilea). (2023) 13:2611. doi: 10.3390/ani13162611
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Slifierz, MJ, Amistad, RM, y Weese, JS. Estudio longitudinal de la microbiota fecal y nasal de los primeros años de vida del cerdo doméstico. BMC Microbiol. (2015) 15:184. doi: 10.1186/s12866-015-0512-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Atarashi, K, Tanoue, T, Shima, T, Imaoka, A, Kuwahara, T, Momose, Y, et al. Inducción de células T reguladoras del colon por especies autóctonas de Clostridium. Ciencia. (2011) 331:337–41. doi: 10.1126/science.1198469
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Helm, ET, Gabler, NK y Burrough, ER. La fibra altamente fermentable altera la microbiota fecal y mitiga la disentería porcina inducida por Brachyspira hyodysenteriae. Animales (Basilea). (2021) 11:11. doi: 10.3390/ani11020396
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Ivarsson, E, Roos, S, Liu, HY y Lindberg, JE. Los polisacáridos fermentables sin almidón aumentan la abundancia de Bacteroides-Prevotella-Porphyromonas en la comunidad microbiana ileal de cerdos en crecimiento. Animal. (2014) 8:1777–87. doi: 10.1017/S1751731114001827
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Chen, C, Fang, S, Wei, H, He, M, Fu, H, Xiong, X, et al. Prevotella copri aumenta la acumulación de grasa en cerdos alimentados con dietas de fórmula. Microbioma. (2021) 9:175. doi: 10.1186/s40168-021-01110-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Pianta, A, Arvikar, S, Strle, K, Drouin, EE, Wang, Q, Costello, CE, et al. Evidencia de la relevancia inmunológica de la prevotella copri, un microbio intestinal, en pacientes con artritis reumatoide. Artritis reumatológica. (2017) 69:964–75. doi: 10.1002/art.40003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Kang, DW, Park, JG, Ilhan, ZE, Wallstrom, G, Labaer, J, Adams, JB, et al. Reducción de la incidencia de Prevotella y otros fermentadores en la microflora intestinal de niños autistas. PLoS Uno. (2013) 8:e68322. doi: 10.1371/journal.pone.0068322
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Koh, HW, Kim, MS, Lee, JS, Kim, H, y Park, SJ. Cambios en la microbiota intestinal porcina en respuesta a la infección por diarrea epidémica porcina. Microbios Ambiente. (2015) 30:284–7. doi: 10.1264/jsme2. ME15046
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Nosho, K, Sukawa, Y, Adachi, Y, Ito, M, Mitsuhashi, K, Kurihara, H, et al. Asociación de Fusobacterium nucleatum con inmunidad y alteraciones moleculares en cáncer colorrectal. Mundo J Gastroenterol. (2016) 22:557–66. doi: 10.3748/wjg.v22.i2.557
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Kieffer, N, Nordmann, P, y Poirel, L. Especies de Moraxella como fuentes potenciales de determinantes de resistencia a polimixinas similares a MCR. Agentes antimicrobianos quimiotera. (2017) 61:61. doi: 10.1128/AAC.00129-17
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Arya, G, y Niven, DF. Producción de hemolisinas por cepas del complejo Actinobacillus minor/»porcitonsillarum». Microbiol veterinario. (2010) 141:332–41. doi: 10.1016/j.vetmic.2009.09.030
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Claud, EC, Keegan, KP, Brulc, JM, Lu, L, Bartels, D, Glass, E, et al. Estructura de la comunidad bacteriana y contribuciones funcionales a la aparición de enterocolitis necrotizante o de salud en recién nacidos prematuros. Microbioma. (2013) 1:20. doi: 10.1186/2049-2618-1-20
Resumen de PubMed | Texto completo de CrossRef | Google Académico
42. Di Sante, G, Gremese, E, Tolusso, B, Cattani, P, Di Mario, C, Marchetti, S, et al. Haemophilus parasuis (Glaesserella parasuis) como posible impulsor del mimetismo molecular y la inflamación en la artritis reumatoide. Front Med (Lausana). (2021) 8:671018. doi: 10.3389/fmed.2021.671018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Haas, B, y Grenier, D. Comprender la virulencia de Streptococcus suis: un desafío veterinario, médico y económico. Med Mal Infect. (2018) 48:159–66. doi: 10.1016/j.medmal.2017.10.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Navarro, D, Abelilla, JJ, y Stein, HH. Estructuras y características de los hidratos de carbono en las dietas alimentadas a cerdos: una revisión. J Anim Sci Biotechnol. (2019) 10:39. doi: 10.1186/s40104-019-0345-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Holman, DB, Kommadath, A, Tingley, JP, y Abbott, DW. Nuevos conocimientos sobre el microbioma intestinal del cerdo utilizando genomas ensamblados con metagenomas. Microbiol Spectr. (2022) 10:e238022:e0238022. doi: 10.1128/spectrum.02380-22
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Gaio, D, DeMaere, MZ, Anantanawat, K, Chapman, TA, Djordjevic, SP, y Darling, AE. Cambios posteriores al destete en la composición y el metabolismo del microbioma revelados por más de 25 000 genomas ensamblados con metagenoma intestinal de cerdo. Genoma Microb. (2021) 7:7. doi: 10.1099/mgen.0.000501
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Weijers, CA, Franssen, MC y Visser, GM. Síntesis catalizada por glicosiltransferasa de oligosacáridos bioactivos. Biotechnol Adv. (2008) 26:436–56. doi: 10.1016/j.biotechadv.2008.05.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Losey, HC, Peczuh, MW, Chen, Z, Eggert, US, Dong, SD, Pelczer, I, et al. Acción tándem de las glicosiltransferasas en la maduración de las agliconas de vancomicina y teicoplanina: nuevos glicopéptidos. Bioquímica. (2001) 40:4745–55. DOI: 10.1021/BI010050W
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Bernardes, A, Pellegrini, V, Curtolo, F, Camilo, CM, Mello, BL, Johns, MA, et al. Los módulos de unión de carbohidratos mejoran la hidrólisis enzimática de la celulosa al aumentar el acceso de las celulasas al sustrato. Carbohydr Polym. (2019) 211:57–68. doi: 10.1016/j.carbpol.2019.01.108
50. Robertson, GT, Doyle, TB y Lynch, AS. Uso de un panel de cepas de Streptococcus pneumoniae deficiente en eflujo para identificar transportadores de múltiples fármacos de clase ABC implicados en la resistencia intrínseca a los agentes antimicrobianos. Agentes antimicrobianos quimiotera. (2005) 49:4781–3. doi: 10.1128/AAC.49.11.4781-4783.2005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Matsumoto, Y, Nakashima, T, Cho, O, Ohkubo, T, Kato, J y Sugita, T. La regulación del ciclo de TCA activada por piruvato en Staphylococcus aureus promueve la tolerancia al valerato de betametasona. Biochem Biophys Res Commun. (2020) 528:318–21. doi: 10.1016/j.bbrc.2020.05.035
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Zhou, Y, Fu, H, Yang, H, Wu, J, Chen, Z, Jiang, H, et al. Análisis metagenómico exhaustivo del resistoma intestinal porcino para identificar indicadores que reflejen resistencia a los antimicrobianos. Microbioma. (2022) 10:39. doi: 10.1186/s40168-022-01241-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
53. Tunsagool, P, Mhuantong, W, Tangphatsornruang, S, Am-In, N, Chuanchuen, R, Luangtongkum, T, et al. Metagenómica de la resistencia a los antimicrobianos y a los metales pesados en el microbioma cecal de cerdos de engorde criados sin antibióticos. Appl Environ Microbiol. (2021) 87:e02684-20. doi: 10.1128/AEM.02684-20
Resumen de PubMed | Texto completo de CrossRef | Google Académico
54. Yarlagadda, V, Medina, R, Johnson, TA, Koteva, KP, Cox, G, Thaker, MN, et al. Descubrimiento guiado por resistencia de productores de antibióticos elfamicina con actividad antigonocócica. ACS infecta dis. (2020) 6:3163–73. doi: 10.1021/acsinfecdis.0c00467
Resumen de PubMed | Texto completo de CrossRef | Google Académico
55. Yang, H, Wei, SH, Hobman, JL y Dodd, C. Resistencia a antibióticos y metales en Escherichia coli aislada de mataderos de cerdos en el Reino Unido. Antibióticos (Basilea). (2020) 9:746. doi: 10.3390/antibióticos9110746
Resumen de PubMed | Texto completo de CrossRef | Google Académico
56. Wang, N, Yang, X, Jiao, S, Zhang, J, Ye, B y Gao, S. Bacterias resistentes a la sulfonamida y sus genes de resistencia en suelos fertilizados con estiércol de la provincia de Jiangsu, sureste de China. Plos Uno. (2014) 9:e112626. doi: 10.1371/journal.pone.0112626
Resumen de PubMed | Texto completo de CrossRef | Google Académico
57. Byrne-Bailey, KG, Gaze, WH, Kay, P, Boxall, AB, Hawkey, PM, y Wellington, EM. Prevalencia de genes de resistencia a sulfonamida en aislados bacterianos de suelos agrícolas abonados y purines de cerdo en el Reino Unido. Agentes antimicrobianos quimiotera. (2009) 53:696–702. doi: 10.1128/AAC.00652-07
Resumen de PubMed | Texto completo de CrossRef | Google Académico
58. Liu, B, Zheng, D, Jin, Q, Chen, L y Yang, J. VFDB 2019: una plataforma patogénica comparativa con una interfaz web interactiva. Ácidos nucleicos Res. (2019) 47:D687-92. doi: 10.1093/nar/gky1080
Resumen de PubMed | Texto completo de CrossRef | Google Académico
59. Skaar, EP, y Raffatellu, M. Metales en enfermedades infecciosas e inmunidad nutricional. Metalómica. (2015) 7:926–8. DOI: 10.1039/C5MT90021B
Palabras clave: diarrea tipo disentería, análisis metagenómico, microbiota fecal, enzima hidrahidrato activo de carbono, resistencia a antibióticos, factor de virulencia
Cita: Chen X, Guo Q, Li Y-Y, Song T-Y y Ge J-Q (2023) Análisis metagenómico de la microbiota fecal de la diarrea similar a la disentería en una granja porcina mediante secuenciación de nueva generación. Frente. Vet. Sci. 10:1257573. doi: 10.3389/fvets.2023.1257573
Recibido: 12 de julio de 2023; Aceptado: 04 de octubre de 2023;
Publicado: 17 octubre 2023.
Editado por:
Min Yue, Universidad de Zhejiang, China
Revisado por:
Marja-Liisa Hänninen, Universidad de Helsinki, Finlandia
Ziyao Zhou, Universidad Agrícola de Sichuan, China
Derechos de autor © 2023 Chen, Guo, Li, Song y Ge. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Jun-Qing Ge, jqge@163.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

