Análisis transcriptómico exhaustivo que revela la dinámica reguladora y las redes del eje hipófisis-testículo en ovejas
 Análisis transcriptómico exhaustivo que revela la dinámica reguladora y las redes del eje hipófisis-testículo en ovejas a lo largo de las etapas de desarrollo
Análisis transcriptómico exhaustivo que revela la dinámica reguladora y las redes del eje hipófisis-testículo en ovejas a lo largo de las etapas de desarrollo
 Shanglai Li†
Shanglai Li†  Bingru Zhao†
Bingru Zhao†  Hua Yang
Hua Yang  Keke Dai
Keke Dai  Yu Cai Hui
Yu Cai Hui  Xu
Xu  Peiyong Chen
Peiyong Chen  Feng Wang
Feng Wang  Yanli Zhang*
Yanli Zhang*- Laboratorio de Ingeniería de Embriones Ganaderos de Jiangsu, Universidad Agrícola de Nanjing, Nanjing, China
La espermatogénesis es un proceso complejo intrincadamente regulado por el eje hipotálamo-hipófisis-testículo (HPT). Sin embargo, la investigación sobre los factores regulatorios que rigen el eje HPT sigue siendo limitada. Este estudio aborda esta brecha mediante la realización de un análisis exhaustivo de los transcriptomas de los tejidos de la hipófisis y los testículos en varias etapas de desarrollo, que abarcan el día embrionario (E120), el período neonatal (P0), la prepubertad (P90) y el día posterior a la pubertad (P270). Utilizando edgeR y WGCNA, identificamos genes específicos de la etapa tanto en la hipófisis como en los testículos a lo largo de las cuatro etapas de desarrollo. En particular, se identificaron 380, 242, 34 y 479 genes específicos de la etapa en la hipófisis, mientras que 886, 297, 201 y 3.678 genes se identificaron en los testículos. Los análisis posteriores revelaron asociaciones entre estos genes específicos de la etapa y vías cruciales como la vía de señalización del AMPc, la secreción de GnRH y la generación de gametos masculinos. Además, aprovechando los datos de una sola célula de la hipófisis y los testículos, identificamos algunas vías de señalización que involucran BMP, HGF, IGF y TGF-β, destacando la regulación mutua entre la hipófisis y los testículos en diferentes etapas de desarrollo. Este estudio arroja luz sobre el papel fundamental del eje hipófisis-testículo en el proceso reproductivo de las ovejas en cuatro etapas de desarrollo distintas. Además, profundiza en las intrincadas redes reguladoras que rigen la reproducción, ofreciendo nuevos conocimientos sobre la dinámica del eje hipófisis-testículo dentro del sistema reproductivo.
1 Introducción
En los animales machos, el intrincado proceso de la espermatogénesis implica la acción orquestada de productos genéticos específicos de la célula y la colaboración entre diferentes células. Este proceso se lleva a cabo bajo la estricta gobernanza de las normas de desarrollo, que influyen profundamente en la salud reproductiva masculina (1). El testículo, que sirve como el sitio principal de producción de espermatozoides, pasa por distintas etapas de desarrollo en los animales machos, incluido el período embrionario, la fase neonatal, la etapa prepuberal y la madurez sexual. Durante el desarrollo embrionario, las células germinales primordiales se diferencian en células de soporte, células esteroidogénicas y células germinales dentro de los testículos en desarrollo. En las etapas neonatal y prepuberal, la espermatogénesis permanece latente y no se produce el inicio de la producción de espermatozoides. La producción de espermatozoides comienza en la madurez sexual, generalmente alrededor de los 6 a 9 meses de edad. El eje hipotálamo-hipófisis-gonadal (HPG) es un sistema regulador preciso que regula el desarrollo del sistema reproductor (2). El eje hipotálamo-hipófisis-testicular (HPT) es un componente crítico en el proceso de producción de espermatozoides. La glándula pituitaria desempeña un papel crucial en la secreción de gonadotropina (3), que secreta la hormona luteinizante (LH) y la hormona foliculoestimulante (FSH). Estas hormonas son esenciales para mantener la función espermatogénica normal al regular sinérgicamente el desarrollo de las células espermatogénicas, las células de Leydig y las células de Sertoli en los testículos, así como la síntesis y secreción de hormonas esteroides gonadales. Además, la investigación encontró que la supresión de PRL aumenta las concentraciones séricas de LH y testosterona en cabras (4). Por lo tanto, el desarrollo y la función de los testículos están estrechamente vinculados a los mecanismos reguladores de la glándula pituitaria.
Recientemente, los estudios genómicos en el eje hipófisis-testículo han revelado genes candidatos y vías que son potencialmente fundamentales en la regulación de los rasgos reproductivos en los animales. Por ejemplo, la secuenciación transcriptómica se utilizó para identificar el importante papel de las vías de señalización de GnRH y AMPc en carneros con diferentes comportamientos sexuales (5). Quince genes implicados en las vías neuroactivas de señalización ligando-receptor y WNT han sido identificados como potencialmente cruciales para el desarrollo de los genitales externos de los gansos (6). Un estudio ha demostrado los patrones de expresión de lincRNAs durante el desarrollo testicular postnatal de cabras (7). Sin embargo, pocos estudios se centraron en los mecanismos interconectados que controlan el desarrollo de la hipófisis y los testículos, particularmente en ovejas macho. Esta brecha pone de relieve la necesidad de realizar investigaciones exhaustivas para dilucidar la regulación coordinada del eje hipófisis-testículo, avanzando así en nuestra comprensión de la salud reproductiva masculina en los animales.
Las ovejas Hu, famosas por su excepcional prolificidad en China (8), sirven como modelo ideal para investigar los mecanismos de fecundidad. Para dilucidar los nuevos genes y las redes reguladoras del eje hipófisis-testículo durante varias etapas de desarrollo en ovejas Hu, realizamos un análisis exhaustivo de los transcriptomas de los tejidos de la hipófisis y los testículos mediante secuenciación de ARN (RNA-seq). Este estudio abarcó cuatro etapas críticas: desarrollo embrionario, período neonatal, prepubertad y postpubertad. Nuestro objetivo era proporcionar nuevos conocimientos sobre el funcionamiento del eje hipófisis-testículo a través de estas diversas etapas de desarrollo. Nuestro estudio identificó reguladores clave y vías de señalización dentro de estas etapas de desarrollo de los testículos para avanzar en nuestra comprensión de la espermatogénesis a nivel molecular. Los hallazgos de este estudio no solo mejoran nuestra comprensión de los intrincados procesos involucrados en la salud reproductiva masculina, sino que también contribuyen a la identificación de genes candidatos cruciales para la capacidad reproductiva masculina en ovejas Hu.
2 Materiales y métodos
2.1 Recogida y preparación de muestras
2.1.1 Recolección de animales y tejidos
Todos los animales experimentales se obtuvieron de Jiangsu Qianbao Sheep Industry Limited Company, Yancheng, China. El período de desarrollo del embrión de cordero puede estimarse en función de los registros de apareamiento de la oveja. Los embriones (E120) se recolectaron en ovejas gestantes a los 120 días embrionarios (n = 5). En las otras tres etapas, se colectaron tres repeticiones basadas en el crecimiento de los corderos (n = 3). La hipófisis y cada testículo derecho de cada carnero se recolectaron y se congelaron en nitrógeno líquido inmediatamente para la extracción de ARN.
2.1.2 Aislamiento y cualificación del ARN
El ARN total se extrajo mediante el método TRIzol (Invitrogen, CA, EE. UU.) y se trató con DNasa I libre de ARNasa (Takara, Kusatsu, Japón). La degradación y contaminación del ARN se controló en geles de agarosa al 1%. La concentración y pureza del ARN se midió con el espectrofotómetro NanoDrop (Thermo Scientific, DE, EE. UU.). La integridad del ARN se evaluó utilizando el bioanalizador Agilent 2100 (Agilent Technologies, CA, EE. UU.).
2.1.3 Preparación de la biblioteca para la secuenciación del transcriptoma
La purificación del ARN, la transcripción inversa, la construcción de bibliotecas y la secuenciación se realizaron en Beijing Allwegene Technology Company Limited (Beijing, China) de acuerdo con las instrucciones del fabricante (Illumina, San Diego, CA).
2.2 Análisis de datos
2.2.1 Control de calidad
Los datos en bruto (lecturas en bruto) del formato fastq se procesaron primero a través de scripts perl internos. En este paso, los datos limpios (lecturas limpias) se obtuvieron eliminando las lecturas que contenían adaptador, las lecturas que contenían ploy-N y las lecturas de baja calidad de los datos sin procesar. Al mismo tiempo, se calcularon Q20, Q30, el contenido de GC y el nivel de duplicación de secuencia de los datos limpios. Todos los análisis posteriores se basaron en datos limpios y de alta calidad.
2.2.2 Análisis cartográfico
Las secuencias de adaptadores y las lecturas de secuencias de baja calidad se eliminaron de los conjuntos de datos. Las secuencias sin procesar se transformaron en lecturas limpias después del procesamiento de datos. Estas lecturas limpias fueron mapeadas a la secuencia del genoma de referencia (Ovis_aries, ARS-UI_Ramb_V2.0) por STAR (9). Solo las lecturas con una coincidencia perfecta o una discrepancia se analizaron y anotaron más a fondo en función del genoma de referencia.
2.2.3 Identificación de genes específicos de estadio
Los genes específicos de la etapa (FDR < 0,05) para la hipófisis y los testículos se identificaron entre una etapa y otras utilizando el paquete R edgeR (10) y Mfuzz (11). Todos los análisis de enriquecimiento funcional se llevaron a cabo para cada tipo de gen específico de la etapa utilizando el paquete R clusterProfile (12).
2.2.4 Análisis de redes de coexpresión génica ponderada (WGCNA)
Para investigar los patrones de coexpresión de genes en los tejidos de la hipófisis y los testículos en cuatro etapas de desarrollo distintas, utilizamos el paquete R WGCNA (13) para construir redes de coexpresión génica. El análisis implicó la construcción de una red de coexpresión génica basada en correlaciones por pares entre perfiles de expresión génica. Para lograr esto, se seleccionó un poder de umbral suave para enfatizar las correlaciones fuertes y mitigar las más débiles. Se identificaron módulos de genes altamente correlacionados, y se calculó el gen propio de cada módulo para representar el patrón de expresión general del módulo. A continuación, se evaluaron las asociaciones módulo-rasgo para vincular los módulos de coexpresión con etapas específicas del desarrollo. Este enfoque integral de WGCNA facilitó la exploración de patrones dinámicos de expresión génica y posibles módulos reguladores que gobiernan las transiciones de desarrollo tanto en los tejidos hipofisarios como en los testículos. El análisis de enriquecimiento de motivos de secuencia de promotores de genes en módulos concentradores se llevó a cabo mediante MEME v.5.5.4 (14), basado en los motivos de vertebrados no redundantes del núcleo JASPAR (2020) de Tomtom (15).
2.2.5 Exploración de los patrones de interacción entre células pituitarias y testículos
Los datos de secuenciación de ARN de una sola célula de los testículos para ovejas Hu se descargaron de la base de datos GEO (GSE184343), mientras que los datos de la hipófisis se originaron a partir de nuestro conjunto de datos no publicado. Estos dos conjuntos de datos se fusionaron para su análisis, y se empleó el paquete de R «Harmony (16)» para eliminar los efectos de lote. Utilizando genes marcadores, los grupos se delinearon en 21 grupos distintos, con ocho grupos que representan células pituitarias y 13 grupos que representan células testiculares. Posteriormente, utilizamos CellChat (17) para explorar la dinámica de la comunicación intercelular entre las células pituitarias y testiculares.
3 Resultados
3.1 Caracterización de los perfiles transcripcionales en el desarrollo de la glándula pituitaria y los testículos de las ovejas
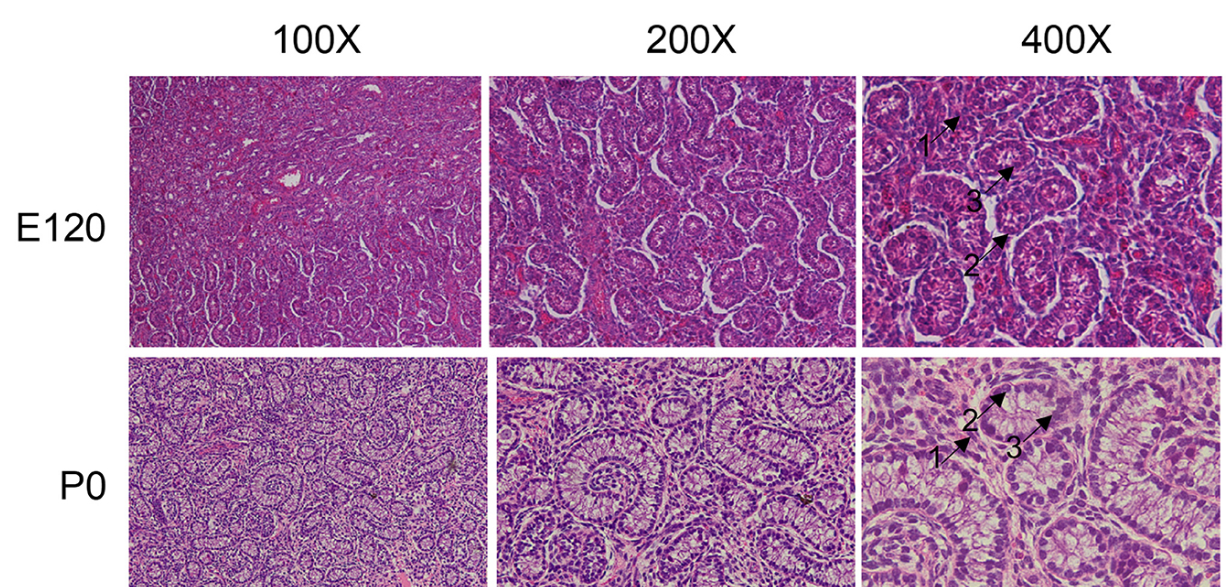
Se recolectaron tejidos de la hipófisis y los testículos de embriones (E120), recién nacidos (P0), prepubertad (P90) y postpubertad (P270) para la secuenciación del transcriptoma (Figura 1A). En total, integramos 28 conjuntos de datos distintos de secuenciación de ARN en cuatro etapas de desarrollo en hipófisis y testículos. Además, integramos dos conjuntos de datos de secuenciación de una sola célula para el análisis, un resumen del flujo de trabajo de análisis al que se hace referencia en la Figura 1B. La etapa de desarrollo fue consistentemente el principal factor que distinguió a las 14 muestras en función de sus perfiles moleculares en el análisis de componentes principales (PCA). Además, se examinó la morfología histológica de la glándula pituitaria y los testículos. En los testículos de los grupos P0 y E120, solo había unas pocas células de Sertoli (Sg), células de Leydig (LC) y células de espermatogonia (SC). Los espermatocitos aparecieron en la etapa P90 y los espermatozoides completamente desarrollados se observaron en la etapa P270 (Figura 2). No se observaron cambios histológicos significativos en la glándula pituitaria (Figura suplementaria S1).
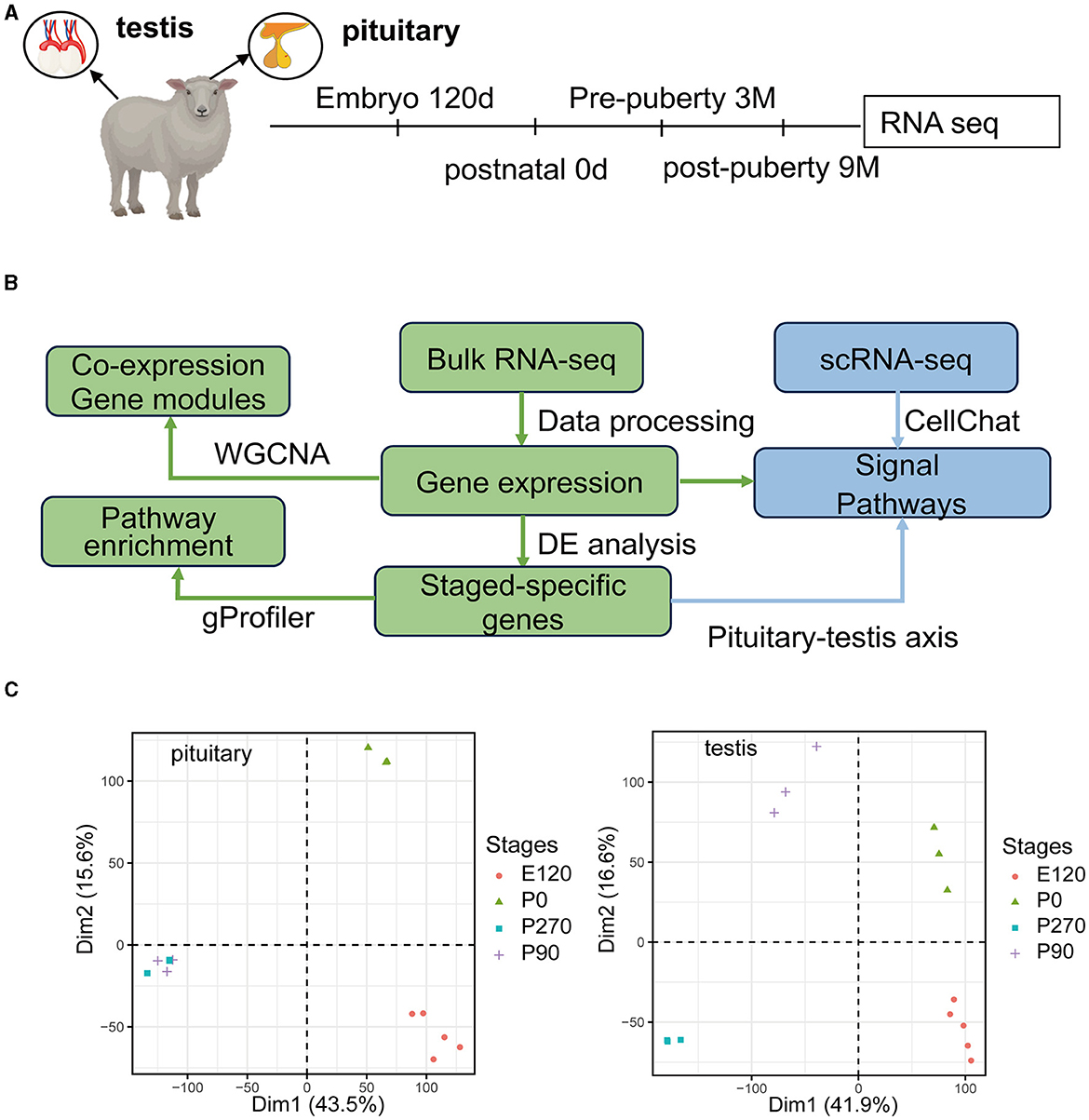 Figura 1. Visión general del transcriptoma de la hipófisis y los testículos durante cuatro etapas de desarrollo. (A) Esquema del diseño experimental. Las marcas en la línea de tiempo denotan la edad en la que se recolectaron la glándula pituitaria y los testículos. (B) Esquema del flujo de trabajo de análisis. (C) Gráfico de PCA para muestras de glándulas pituitarias y testículos basado en la expresión génica. Después de filtrar los genes de recuento bajo, logre2(normCounts+1) para realizar el análisis de componentes principales (ACP).
Figura 1. Visión general del transcriptoma de la hipófisis y los testículos durante cuatro etapas de desarrollo. (A) Esquema del diseño experimental. Las marcas en la línea de tiempo denotan la edad en la que se recolectaron la glándula pituitaria y los testículos. (B) Esquema del flujo de trabajo de análisis. (C) Gráfico de PCA para muestras de glándulas pituitarias y testículos basado en la expresión génica. Después de filtrar los genes de recuento bajo, logre2(normCounts+1) para realizar el análisis de componentes principales (ACP).
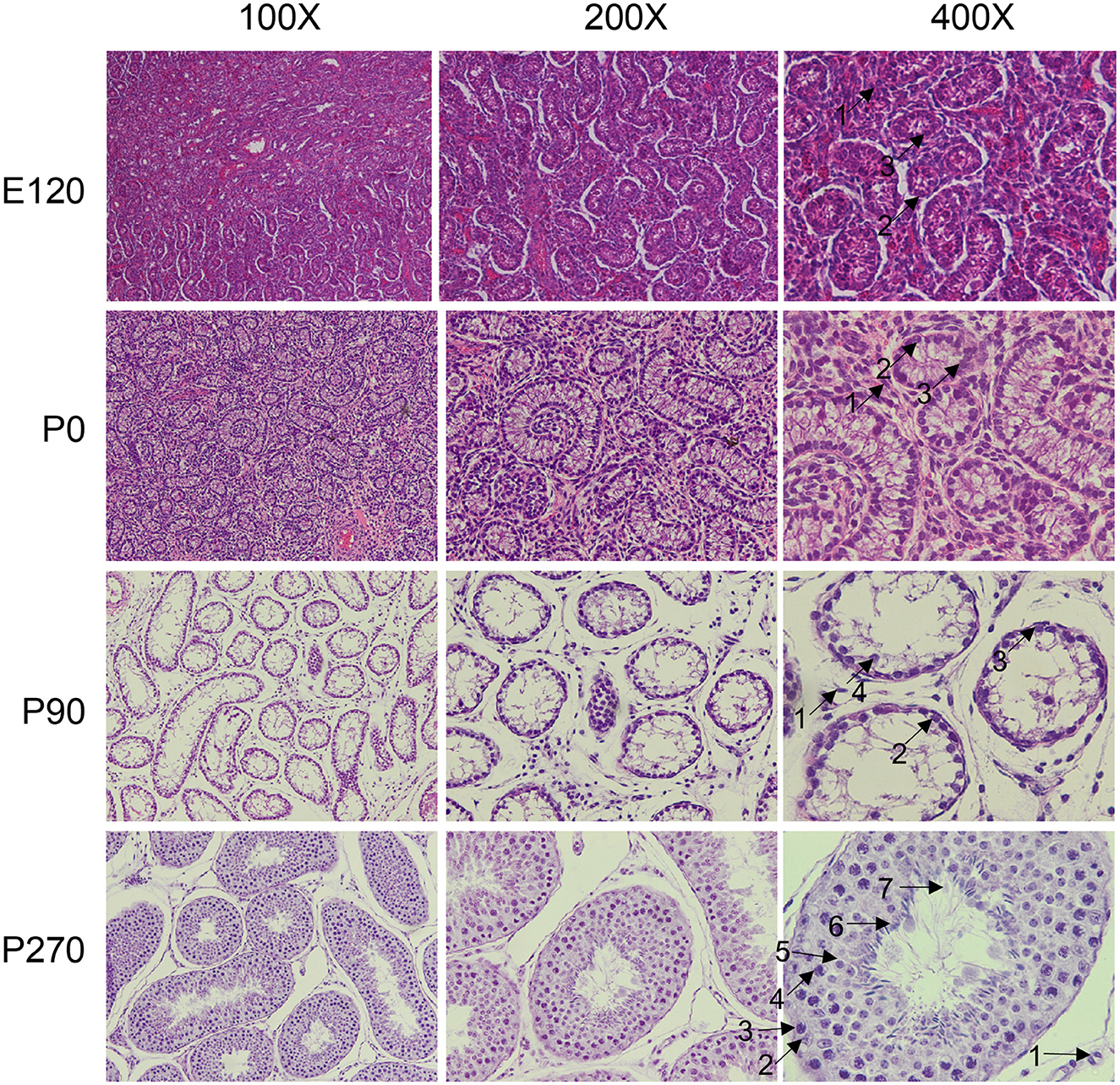 Figura 2. Análisis histológico de testículos en diferentes estadios de desarrollo. Los tejidos testiculares se observaron al microscopio a × 100, × 200 y × 400 aumentos. Los diferentes tipos de celdas se indicaron con flechas negras y se numeraron en consecuencia. 1: células de Leydig, 2: células de Sertoli, 3: espermatogonias, 4: espermatocitos, 5: espermátidas redondas, 6: espermátidas alargadas, 7: espermatozoides.
Figura 2. Análisis histológico de testículos en diferentes estadios de desarrollo. Los tejidos testiculares se observaron al microscopio a × 100, × 200 y × 400 aumentos. Los diferentes tipos de celdas se indicaron con flechas negras y se numeraron en consecuencia. 1: células de Leydig, 2: células de Sertoli, 3: espermatogonias, 4: espermatocitos, 5: espermátidas redondas, 6: espermátidas alargadas, 7: espermatozoides.
3.2 Genes específicos de la etapa en la hipófisis y los testículos en todas las etapas del desarrollo
Para investigar genes clave en la hipófisis y los testículos a lo largo de varias etapas de desarrollo en ovejas Hu, realizamos un análisis detallado de los genes regulados al alza en cada etapa y, posteriormente, realizamos un análisis de enriquecimiento de GO en estos genes. Nuestros resultados demostraron que durante las etapas embrionarias y neonatales del desarrollo hipofisario, los genes regulados al alza se enriquecieron significativamente en el ciclo celular mitótico y en los procesos metabólicos de ATP. A medida que avanzaba la edad, se produjo un cambio notable en el perfil de expresión génica hipofisaria, con un enriquecimiento de los procesos relacionados con la secreción de hormonas, la regulación del crecimiento y las características sexuales masculinas (Figura 3A).
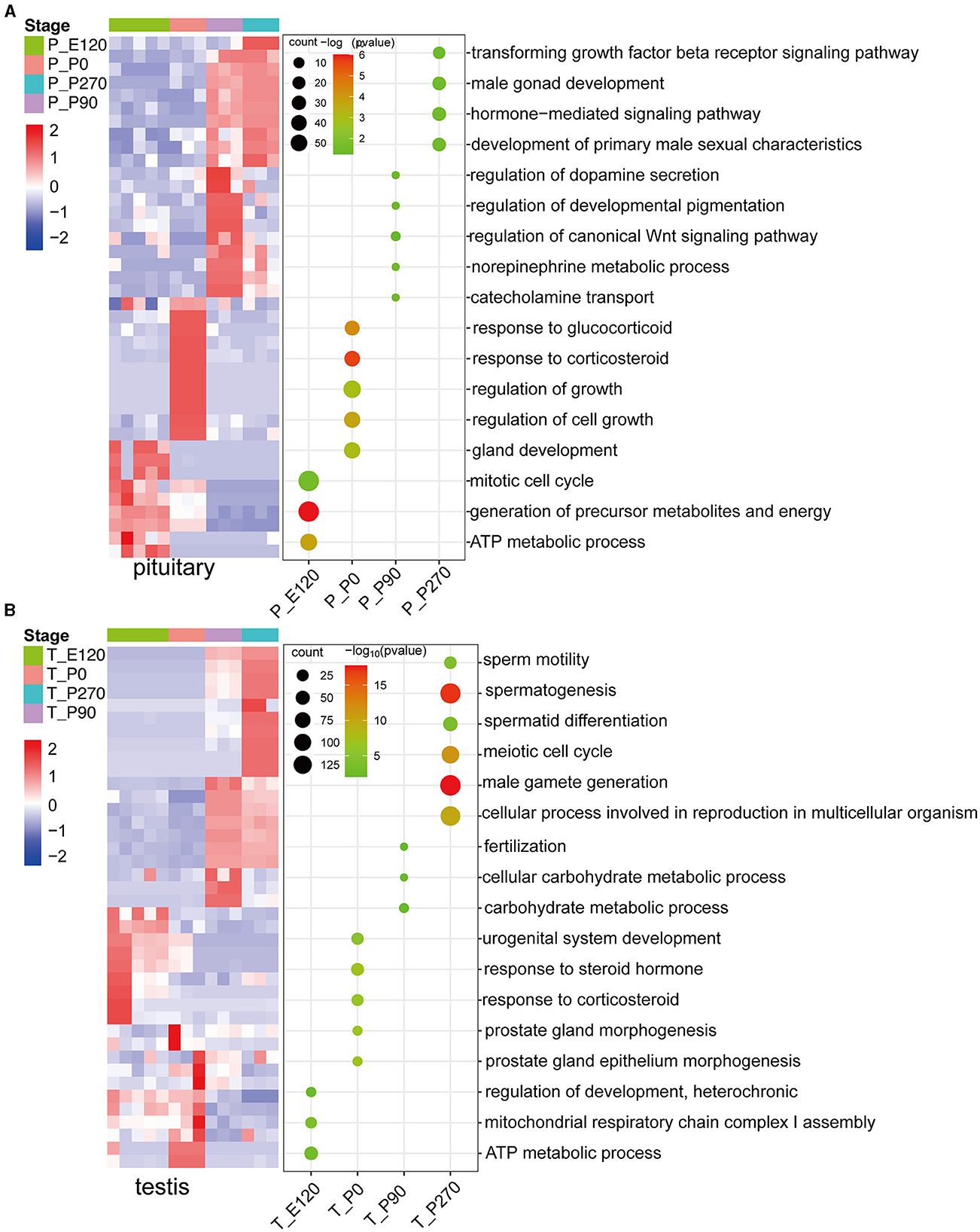 Figura 3. Los genes específicos de la etapa están regulados al alza en las etapas de desarrollo, la hipófisis y los testículos. (A) El mapa de calor (izquierda) muestra el nivel de expresión [log2(TPM+ 1)] de los 10 genes específicos de la etapa más alta regulados al alza en las etapas de desarrollo de la hipófisis; Los mapas de calor (derecha) muestran las puntuaciones de consenso normalizadas de términos de Gene Ontology (GO) significativamente enriquecidos (FDR < 0,05) para todos los genes regulados al alza en cada etapa. El eje horizontal representa las distintas etapas de la hipófisis. (B) Similar a (A), pero para los testículos.
Figura 3. Los genes específicos de la etapa están regulados al alza en las etapas de desarrollo, la hipófisis y los testículos. (A) El mapa de calor (izquierda) muestra el nivel de expresión [log2(TPM+ 1)] de los 10 genes específicos de la etapa más alta regulados al alza en las etapas de desarrollo de la hipófisis; Los mapas de calor (derecha) muestran las puntuaciones de consenso normalizadas de términos de Gene Ontology (GO) significativamente enriquecidos (FDR < 0,05) para todos los genes regulados al alza en cada etapa. El eje horizontal representa las distintas etapas de la hipófisis. (B) Similar a (A), pero para los testículos.
En un análisis paralelo del desarrollo de los testículos, observamos que los genes regulados al alza durante las etapas embrionaria y neonatal se enriquecían principalmente en el metabolismo energético y en la regulación de los procesos metabólicos celulares, incluida la síntesis de ATP. En particular, al alcanzar la madurez sexual, una regulación positiva significativa de los genes en los testículos se asoció predominantemente con procesos reproductivos como la fertilización y la generación de gametos masculinos (Figura 3B).
3.3 Patrones dinámicos de expresión de genes durante el desarrollo de la hipófisis y los testículos
Para analizar la tendencia de variación de los genes diferenciales, clasificamos todos los genes expresados diferencialmente en cinco grupos en función de sus niveles de expresión. En la hipófisis, los genes del grupo 1 demostraron una disminución gradual en los niveles de expresión a lo largo de las etapas de desarrollo, con un enriquecimiento significativo (P < 0,01) en el desarrollo de los órganos embrionarios y los procesos metabólicos de ATP. Por el contrario, los genes del grupo 3 mostraron un aumento progresivo en los niveles de expresión a lo largo de las etapas de desarrollo y se enriquecieron significativamente en la secreción de hormonas. Los genes del grupo 4 mostraron los niveles más altos de expresión en P0 y se enriquecieron significativamente (P < 0,01) en la regulación del crecimiento (Figura 4A).
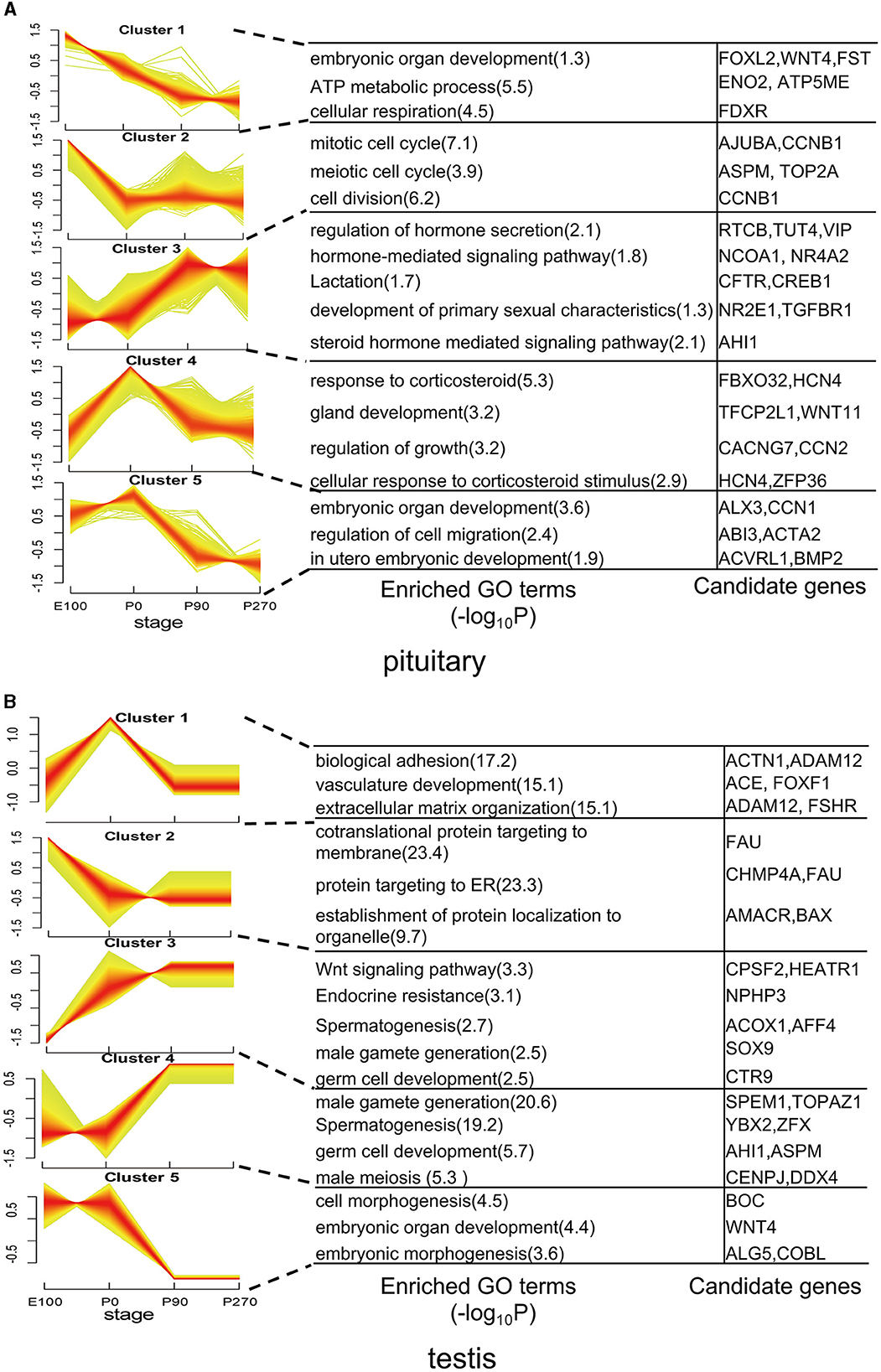 Figura 4. Patrones dinámicos de expresión de genes durante el desarrollo de la hipófisis y los testículos. (A) Los grupos de genes expresados diferencialmente en la hipófisis. Los procesos biológicos correspondientes se muestran junto a cada grupo. (B) Similar a (A), pero para los testículos.
Figura 4. Patrones dinámicos de expresión de genes durante el desarrollo de la hipófisis y los testículos. (A) Los grupos de genes expresados diferencialmente en la hipófisis. Los procesos biológicos correspondientes se muestran junto a cada grupo. (B) Similar a (A), pero para los testículos.
En los testículos, los genes del grupo 1 exhibieron niveles elevados de expresión principalmente durante las primeras etapas del desarrollo, particularmente al nacer. En particular, genes como ACTN1, ECA y FSHR dentro de este grupo se enriquecieron significativamente en vías relacionadas con la adhesión biológica y el desarrollo de la vasculatura. Por el contrario, los Clusters 2 y 3 mostraron patrones de expresión contrastantes, mientras que los Clusters 4 y 5 exhibieron tendencias opuestas en la expresión génica. Además, estos grupos diferían en las vías funcionales enriquecidas, lo que subraya su papel distintivo en la fisiología testicular (Figura 4B).
3.4 El gen WGCNA y el gen específico de la etapa revelan genes hup asociados con diferentes etapas
Creamos redes de coexpresión génica para diferentes etapas de desarrollo e identificamos once módulos utilizando el análisis de redes de coexpresión génica ponderada (WGCNA) (Figura 5A). A continuación, se analizó la correlación entre cada módulo y las diferentes etapas del desarrollo tisular (Figura 5B). Para determinar si ciertos factores de transcripción (TF) estaban regulando los genes en estos módulos específicos de la etapa, realizamos un análisis de enriquecimiento de motivos en los promotores de genes específicos de la etapa por separado (Figura 5C). Descubrimos que estos TF también exhibían una expresión específica de la etapa, lo que indica que podrían desempeñar un papel crucial en el desarrollo de embriones y órganos.
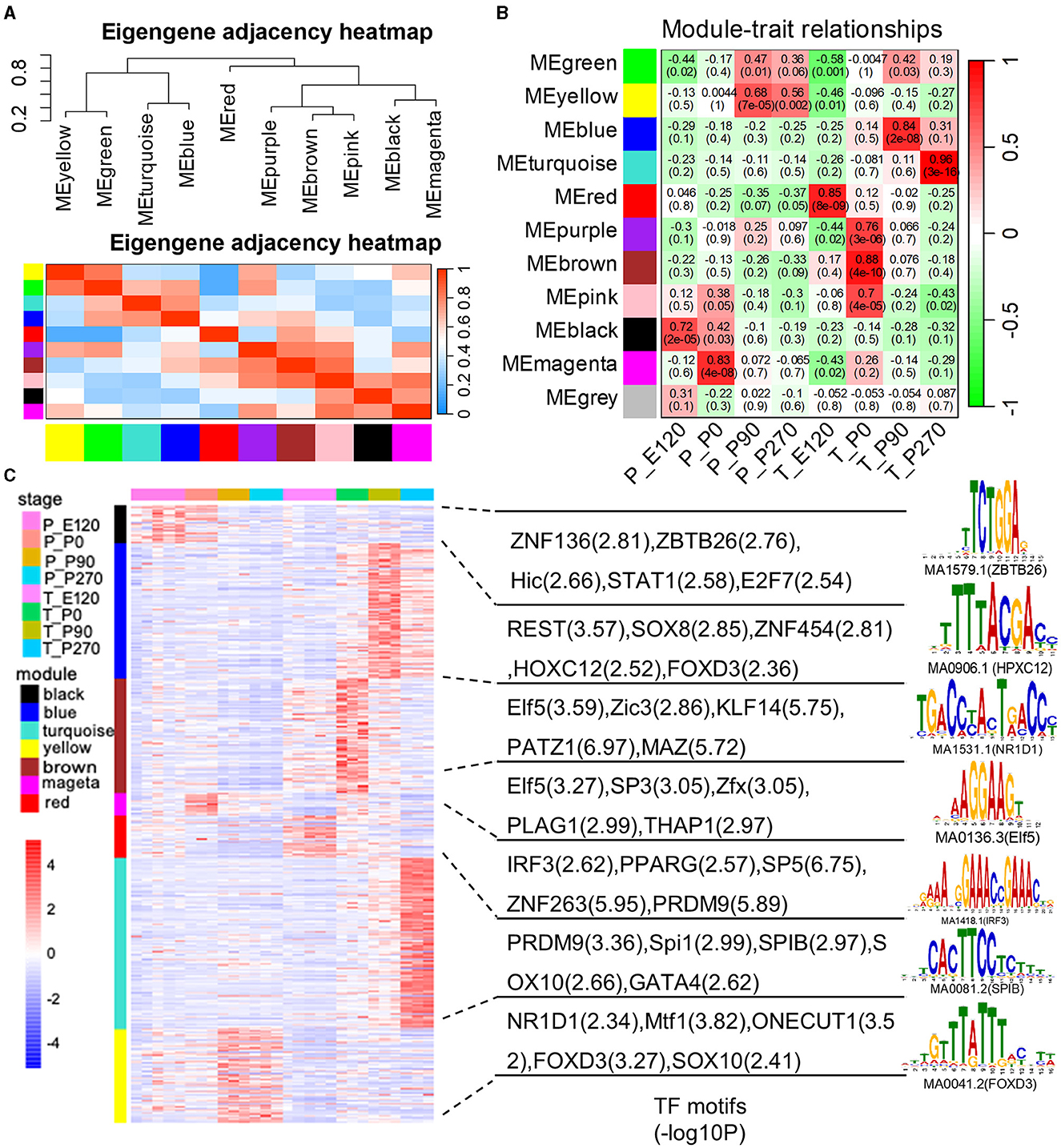 Figura 5. El análisis de redes de coexpresión génica ponderada (WGCNA). (A) El dendrograma de los genes propios y el mapa de calor identifican grupos de módulos cruzados de genes propios correlacionados. (B) Correlaciones entre los módulos genéticos y las etapas de desarrollo. La significación estadística de la relación entre el modelo y la etapa de desarrollo se corrige para múltiples pruebas utilizando el método FDR. Las estrellas amarillas denotan FDR < 0.05. Cada celda contiene la correlación y el valor FDR correspondiente entre paréntesis. (C) El mapa de calor muestra la expresión génica normalizada de los genes en los siete módulos significativos superiores. La expresión génica normalizada para los TF enriquecidos con módulos y el motivo de secuencia más representativo se muestran junto a cada módulo.
Figura 5. El análisis de redes de coexpresión génica ponderada (WGCNA). (A) El dendrograma de los genes propios y el mapa de calor identifican grupos de módulos cruzados de genes propios correlacionados. (B) Correlaciones entre los módulos genéticos y las etapas de desarrollo. La significación estadística de la relación entre el modelo y la etapa de desarrollo se corrige para múltiples pruebas utilizando el método FDR. Las estrellas amarillas denotan FDR < 0.05. Cada celda contiene la correlación y el valor FDR correspondiente entre paréntesis. (C) El mapa de calor muestra la expresión génica normalizada de los genes en los siete módulos significativos superiores. La expresión génica normalizada para los TF enriquecidos con módulos y el motivo de secuencia más representativo se muestran junto a cada módulo.
A continuación, generamos diagramas de Venn para intersectar genes de módulos específicos de la etapa con los regulados al alza en cada etapa del desarrollo. A través de los análisis de enriquecimiento funcional de KEGG, observamos un enriquecimiento predominante de estos genes en las vías de señalización reproductiva, incluida la vía de señalización de AMPc, la vía de señalización de TNF y la vía de señalización de Notch (Figura suplementaria S2). Posteriormente, empleamos estos genes para construir una red de interacción proteína-proteína (PPI). Se aplicó el algoritmo MCODE para extraer subredes pivotales, identificándolas como módulos centrales. Por ejemplo, en la hipófisis de E120, se identificó una red entre ASCL1, OTX2, FOXN4, LHX2 y FEZF1. En el grupo P0 se estableció una red compuesta por ACSL1, ELOVL5, FABP4 y PDK4. En el grupo P270, se construyeron redes entre genes como PTAR1, CHML, ARL5B y otros. Sin embargo, para el grupo P90, no se estableció ninguna red de interacción debido a las interacciones mínimas (Figura 6A). En los testículos, construimos cuatro módulos específicos de la etapa. El módulo del grupo E120 comprendía doce genes, incluidos MRPL30 y NME3. El módulo del grupo P0 constaba de quince genes, incluidos TPM4 y MYH1. El módulo del grupo P90 incluía cuatro genes, como FATE1, SLC4A5, VSIG1 y ADAM11. El módulo del grupo P270 comprendía seis genes, incluidos NUTM1, PHF7, ADGB y ENO4 (Figura 6B). Es importante destacar que los genes dentro de estas subredes demostraron correlaciones significativas entre sí, lo que sugiere una red estrechamente regulada de interacciones que son críticas en cada etapa del desarrollo.
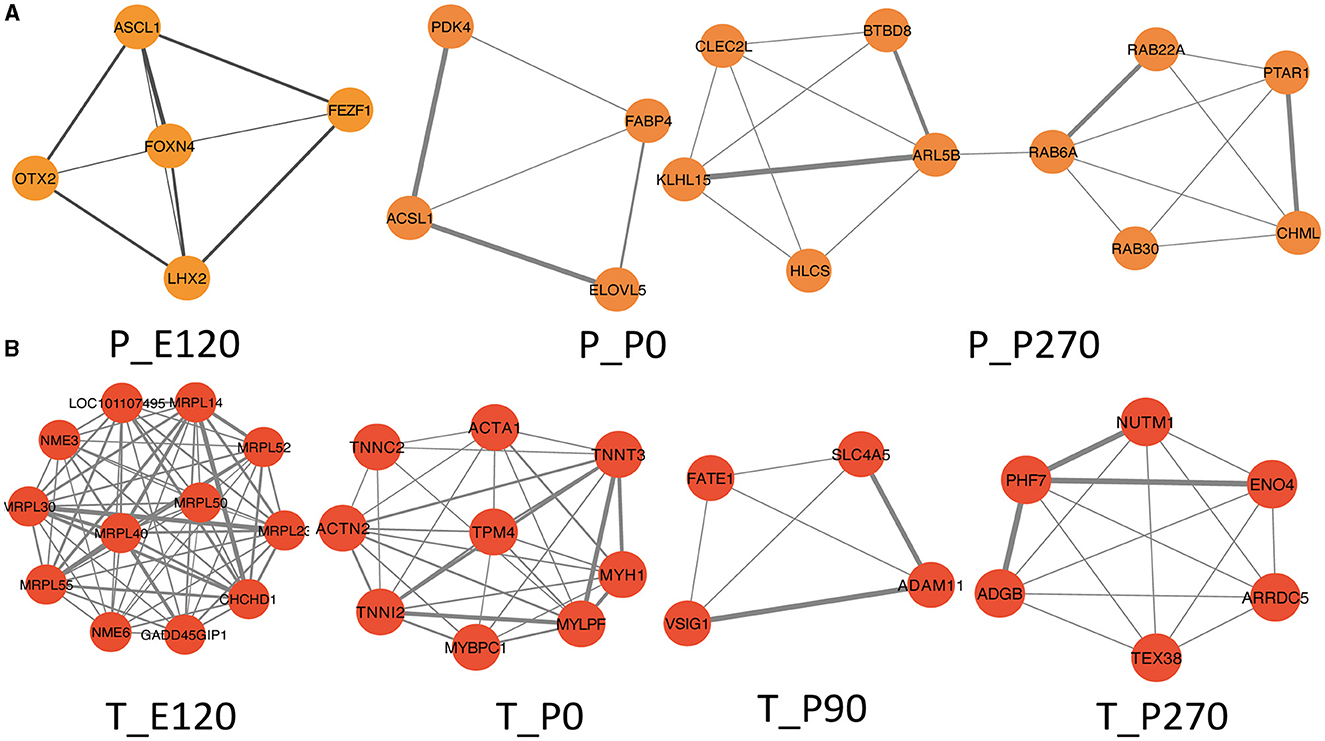 Figura 6. Construcción de la red Hup en diferentes etapas. (A) Subredes examinadas por MCODE entre diferentes estadios en hipófisis, puntuación MCODE = 4,5, 3,3, 4,2. (B) Subredes examinadas por MCODE entre diferentes estadios en testículos, puntuación MCODE = 12, 8, 5, 7.
Figura 6. Construcción de la red Hup en diferentes etapas. (A) Subredes examinadas por MCODE entre diferentes estadios en hipófisis, puntuación MCODE = 4,5, 3,3, 4,2. (B) Subredes examinadas por MCODE entre diferentes estadios en testículos, puntuación MCODE = 12, 8, 5, 7.
3.5 Exploración de los patrones de interacción célula pituitaria y testicular
Analizamos los datos de secuenciación de ARN de una sola célula para explorar las interacciones entre la hipófisis y los testículos. Después de un riguroso procesamiento y filtración de datos sin procesar, se identificaron 23.837 células en total, entre las cuales, 10.927 células testicular y 12.910 células pituitarias se utilizaron para su posterior análisis. El agrupamiento celular reveló una clara separación entre los grupos de células hipofisarias y los grupos de células testículos (Figura suplementaria S3A), lo que indica la alta calidad de los datos utilizados para el análisis. Después de realizar más análisis, se descubrió que hay ocho tipos diferentes de células hipofisarias y 13 tipos diferentes de células testiculares (Figura suplementaria S3B).
Luego, predijimos las principales entradas y salidas de señalización para diferentes grupos de células. Identificamos una lista de pares ligando-receptor entre los grupos celulares, como las vías TGFβ, FSH, PTN, BMP, NPR2, HGF e IGF. Se calcularon y detallaron los números y pesos de los pares ligando-receptor (Figura 7A). Entre todos los tipos de células, las células foliculoteladas, las células gonadotropas, las células adark, las células de Leydig y los somatotropos exhibieron una actividad relativamente alta. Analizamos los patrones de comunicación celular entre varios tipos celulares, considerando la similitud en la expresión de ligandos y receptores (Figura 7B). Nuestra observación indicó que los patrones de ligandos de las células secretoras se agrupaban automáticamente en función de tipos de células similares.
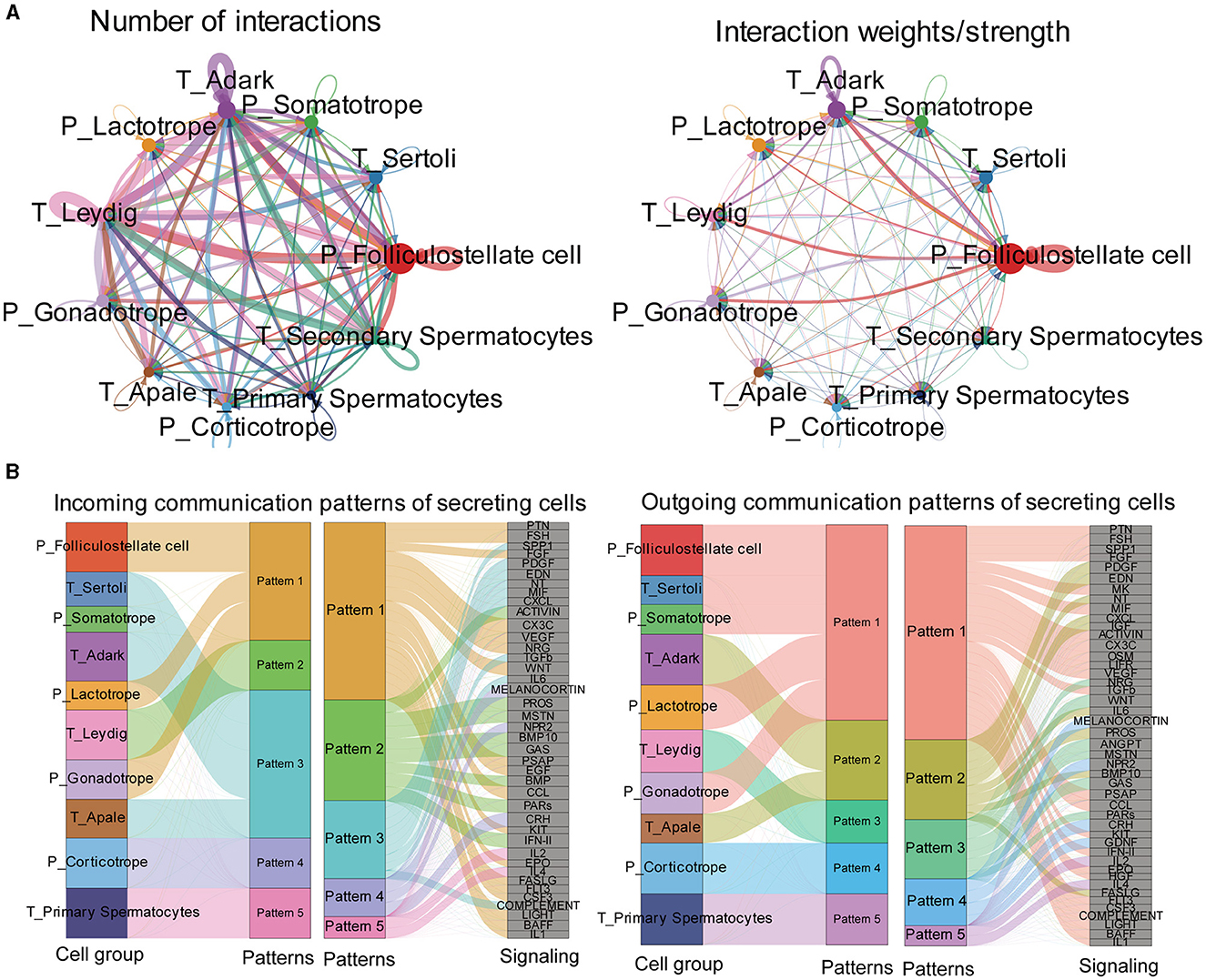 Figura 7. Exploración de los patrones de interacción célula pituitaria y testículo-célula. (A) Diagrama circular de los pares ligando-receptor de todos los tipos celulares, con el grosor de la cadena representando el número de pares ligando-receptor. (B) El diagrama del río muestra la contribución de ligandos y receptores a diferentes patrones y la contribución de patrones a diferentes tipos de células.
Figura 7. Exploración de los patrones de interacción célula pituitaria y testículo-célula. (A) Diagrama circular de los pares ligando-receptor de todos los tipos celulares, con el grosor de la cadena representando el número de pares ligando-receptor. (B) El diagrama del río muestra la contribución de ligandos y receptores a diferentes patrones y la contribución de patrones a diferentes tipos de células.
3.6 Red de señalización BMP, HGF, IGF y TGFβ entre todos los tipos de células
Para investigar las redes reguladoras entre la hipófisis y los testículos en varias etapas del desarrollo, realizamos un análisis de las vías de señalización que influyen en la regulación testicular de la hipófisis (Figura 8A). Nos centramos en las vías de señal, incluidas BMP, HGF, IGF y TGFβ, para las que calculamos la probabilidad de comunicación entre cada par de tipos de células implicadas. Nuestros hallazgos indicaron que la vía de señalización BMP funciona predominantemente dentro de las células de Leydig del testículo. La red de señalización de HGF exhibió una actividad máxima entre las células foliculoseladoras, las células oscuras de Leydig y los corticotropos. Para la red de señalización de IGF, se observó una alta actividad entre las células foliculoteladas, las células oscuras y las células somatotrópicas. En la vía de señalización del TGFβ, las células foliculoselas de la hipófisis actúan principalmente como emisoras, mientras que las células oscuras del testículo funcionan predominantemente como receptoras y otras células actúan como influenciadoras en esta vía (Figura 8B).
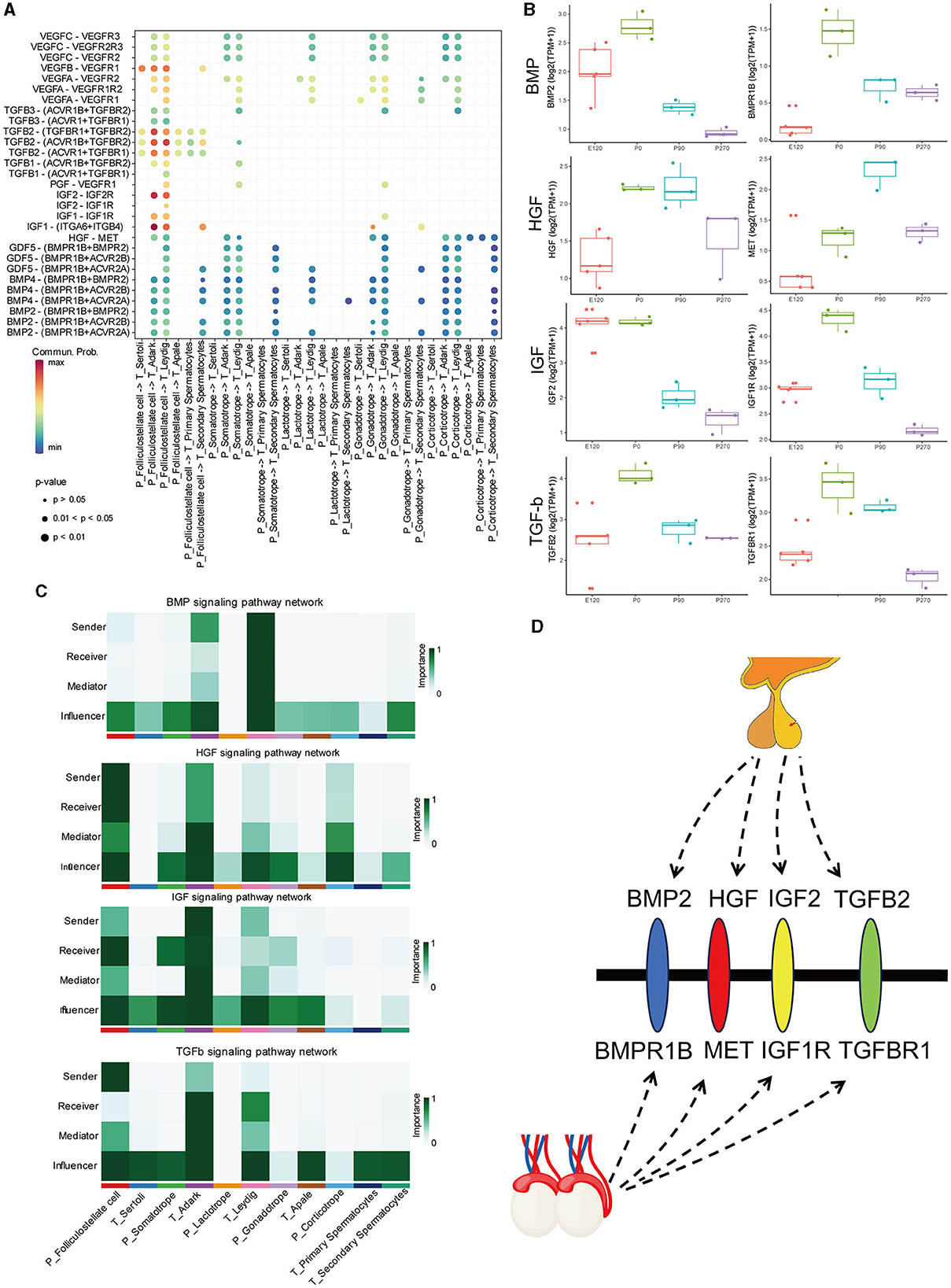 Figura 8. Red de señalización BMP, HGF, IGF y TGFβ entre todos los tipos de células. (A) Diagrama de burbujas de los pares ligando-receptor de algunos tipos celulares. (B) El diagrama de caja muestra la expresión de ligandos y receptores en las vías de señalización BMP, HGF, IGF y TGFβ en diferentes etapas de la hipófisis y los testículos. (C) El mapa de calor muestra la importancia relativa de cada grupo de células en función de las cuatro medidas de centralidad de red calculadas de la red de señalización BMP, HGF, IGF y TGFβ, respectivamente. (D) Hipótesis del eje hipófisis-testículo.
Figura 8. Red de señalización BMP, HGF, IGF y TGFβ entre todos los tipos de células. (A) Diagrama de burbujas de los pares ligando-receptor de algunos tipos celulares. (B) El diagrama de caja muestra la expresión de ligandos y receptores en las vías de señalización BMP, HGF, IGF y TGFβ en diferentes etapas de la hipófisis y los testículos. (C) El mapa de calor muestra la importancia relativa de cada grupo de células en función de las cuatro medidas de centralidad de red calculadas de la red de señalización BMP, HGF, IGF y TGFβ, respectivamente. (D) Hipótesis del eje hipófisis-testículo.
Además, analizamos los patrones de expresión de ligandos clave y genes receptores dentro de estas vías de señalización en la hipófisis y los testículos en diferentes etapas. Observamos que las tendencias de expresión de estos genes ligandos y receptores eran en gran medida consistentes tanto en la hipófisis como en los testículos. Específicamente, en la vía de señalización de HGF, la expresión génica del ligando HGF mostró una expresión relativamente mayor en la hipófisis al nacer y a los 90 días en comparación con otras etapas. Por el contrario, su gen receptor MET exhibe la mayor expresión en el testículo a los 90 días. De manera similar, en la vía de señalización de TGF, el gen ligando TGFB2 y el gen receptor TGFBR1 exhibieron altos niveles de expresión en la hipófisis y los testículos al nacer, respectivamente (Figura 8B).
4 Discusión
El eje HPT desempeña un papel importante en la reproducción, con una amplia investigación centrada en el hipotálamo (18, 19), la hipófisis (20) y los testículos (21) en varias etapas de desarrollo. La glándula pituitaria es fundamental en la reproducción y el desarrollo del crecimiento de los mamíferos mediante la secreción de hormonas como GH, FSH, LH y PRL. Cabe destacar que las células endocrinas aparecen a los 60 días de gestación en la hipófisis de las ovejas (22). Un estudio identificó células FSHβ en la hipófisis fetal a los 100 días, acompañadas de un aumento en número a medida que la glándula madura (23). En nuestro estudio, exploramos el desarrollo hipofisario en ovejas a los 120 días de la etapa embrionaria y las otras tres etapas postnatales utilizando RNA-seq. Identificamos 380, 242, 34 y 479 genes en etapa específica en la hipófisis entre las cuatro etapas de desarrollo, respectivamente. La dinámica del transcriptoma regula principalmente el desarrollo de los testículos y la espermatogénesis de una manera dinámica y específica para cada estadio (24). Los tipos de células en los testículos difieren en varias etapas de desarrollo a través del análisis morfológico. De acuerdo con esta observación, se identificaron 886, 297, 201 y 3.678 genes en estadio específico en los testículos. Durante su desarrollo, los testículos de carnero experimentan tres cambios histológicos: prepubertad, madurez y edad adulta. Durante la etapa de prepubertad, que dura desde el primer mes hasta el quinto mes, no comienza el proceso de espermatogénesis. Estos resultados son consistentes con los observados por la morfología histológica. La etapa de la espermatogénesis comienza entre los 6 y 9 meses de edad, cuando se inicia la producción de espermatozoides (25). En particular, el aumento del número de genes identificados en la etapa P270 sugiere un vínculo potencial con la espermatogénesis y la maduración sexual.
El desarrollo de la hipófisis y los testículos está regulado por genes centrales (26). En este estudio, utilizamos varios métodos analíticos para construir la red de genes específicos en cada etapa. En la hipófisis, la mayoría de los genes específicos de las etapas se enriquecieron en el desarrollo de los órganos embrionarios y el ciclo celular mitótico en los grupos E120 y P0. Y la red de genes específicos contenía principalmente OTX2, ASCL1, FOXN4 y otros genes. Estudios previos han sugerido que estos genes juegan un papel crucial tanto en el desarrollo de la hipófisis como en la diferenciación de las células madre (27, 28). Un análisis de secuenciación de ARN de una sola célula de la glándula pituitaria humana en desarrollo reveló que la expresión de ASCL1 era una característica prominente de las células Pro.PIT1_all (29). Indica que las glándulas pituitarias de embriones y recién nacidos poseen propiedades de células madre y desempeñan un papel vital en la regulación de la proliferación y diferenciación de las células endocrinas. Además, identificamos genes clave en la hipófisis de P90 y P270, pero solo los genes de P270 conducían una red. Las principales funciones de estos genes son el transporte mediado por vesículas y el metabolismo de las proteínas. Esto puede estar relacionado con la secreción de hormonas hipofisarias. En la etapa embrionaria, los genes de las proteínas ribosómicas mitocondriales son los genes centrales, lo que indica que el metabolismo energético ocurre principalmente en los testículos. En el grupo de recién nacidos, la red central comprendía principalmente genes asociados con la estructura muscular (30). A los días P90 y P270, la red central consiste principalmente en genes relacionados con la espermatogénesis. Por ejemplo, VSIG1, un miembro de la familia de moléculas de adhesión de unión, se expresa principalmente en el estómago y los testículos, y desempeña un papel importante durante la espermatogénesis (31). Estudios anteriores han destacado que la ENO4 es la enolasa primaria en los espermatozoides de ratón, y su ausencia conduce a anomalías estructurales en los espermatozoides (32). Esto subraya la ausencia de espermatozoides maduros en los testículos a los 90 días, con predominio de las funciones espermatogénicas. A los 270 días, los espermatozoides maduros son evidentes en los testículos, con la mayoría de los genes clave participando en la regulación de la fertilización de los espermatozoides. Durante el desarrollo de la hipófisis y los testículos, los genes clave juegan un papel crucial en la producción de espermatozoides.
La espermatogénesis está regulada por la acción endocrina coordinada del eje HPT. La GnRH secretada por el hipotálamo es transportada a la hipófisis anterior, que estimula a la gonadotropina para secretar LH y FSH. La LH estimula las células de Leydig para producir testosterona, mientras que la FSH regula la espermatogénesis a través de sus efectos sobre las células de Sertoli en los túbulos seminíferos (33). Muchas otras vías de señalización juegan un papel crucial en la regulación de este proceso. Por lo tanto, utilizamos datos de secuenciación de ARN de una sola célula de la hipófisis y los testículos para investigar las vías de señalización que actúan sobre el testículo e identificar posibles mecanismos subyacentes. Las vías de señalización, como BMP, HGF, IGF y TGFβ fueron muy activas en los tipos de células hipofisarias y testículos. Experimentos previos en testículos de ratón han demostrado que la señalización de BMPR-1B inhibe la producción de testosterona mediante la regulación de las isoformas de HSD y la aromatasa, tanto in vivo como in vitro (34). Se ha demostrado en estudios previos que las proteínas morfogenéticas óseas (BMPs) juegan un papel crucial en la reproducción masculina, junto con la LH y la FSH (35-37). En el presente estudio, se descubrió que el gonadotropo hipofisario está ligado a los espermatocitos en el testículo a través de la vía BMP. La vía de señalización HGF se expresa activamente en todas las etapas del desarrollo testicular (38, 39). Esto fue confirmado por la expresión de MET en este experimento. Además, el HGF puede estimular la secreción de proteasa en las células estromales, mejorar la actividad del TGF-β, modular las uniones estrechas entre las células de soporte e influir en la formación de la barrera hematotesticular (40). In vitro, se observó que las células estromales testiculares cultivadas sintetizan testosterona, desarrollan células estromales testiculares embrionarias y aumentan la supervivencia de las células estromales testiculares cuando son estimuladas por HGF (41). Un informe reciente destacó la importancia de los IGF como el principal factor de crecimiento en la regulación del número de SC y el tamaño de los testículos (42). Las SC son las únicas células somáticas presentes en el epitelio seminífero y desempeñan un papel crucial en el apoyo a la espermatogénesis (43). Los experimentos in vivo han demostrado que los efectos proliferativos de la FSH en las células madre inmaduras durante la etapa neonatal están mediados por la vía de señalización insulina/IGF. Esto indica la participación de la señalización insulina/IGF en la facilitación de las acciones de la FSH sobre la proliferación de SC (42). Los ligandos de la superfamilia TGF-β regulan el desarrollo de los testículos mediante el control de la especificación del destino de la línea germinal y la reorganización celular que subyace a la formación de los testículos en los embriones. Además, puede influir tanto en las células somáticas como en las germinales durante el inicio de la espermatogénesis en el crecimiento de los testículos juveniles (44). Las activinas e inhibinas en las vías del TGFβ desempeñan un papel clave en la regulación del eje hipotálamo-hipófisis-gonadal en animales domésticos. En resumen, el eje hipófisis-testicular está significativamente influenciado por estas cuatro vías de señalización.
5 Conclusión
En resumen, caracterizamos los cambios globales del transcriptoma a lo largo de cuatro etapas de desarrollo de la hipófisis y los testículos en ovejas. Además, de acuerdo con los datos de scRNA, proporcionamos evidencia fundamental de que la hipófisis podría regular las funciones de los testículos a través de múltiples vías de señalización, el mecanismo asociado con nuestros hallazgos debería validarse en el futuro.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están a disposición del público. Estos datos se pueden encontrar aquí: https://www.ncbi.nlm.nih.gov/bioproject/; PRJNA1066045.
Declaración ética
El estudio en animales fue aprobado por el Comité de Cuidado y Uso de Animales de la Universidad Agrícola de Nanjing. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
SL: Conceptualización, Investigación, Redacción, borrador original, Redacción, revisión y edición. BZ: Curación de datos, Análisis formal, Software, Redacción, revisión y edición. HY: Curación de datos, investigación, redacción, revisión y edición. KD: Curación de datos, investigación, redacción, revisión y edición. YC: Escribir, revisar y editar. HX: Escritura, revisión y edición. CP: Curación de datos, investigación, redacción, revisión y edición. FW: Adquisición de fondos, supervisión, redacción, revisión y edición. YZ: Conceptualización, Obtención de Fondos, Administración de Proyectos, Supervisión, Redacción, Revisión y Edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo contó con el apoyo del Programa Nacional Clave de Investigación y Desarrollo (No. 2021YFF1000702), el Proyecto de Revitalización de la Industria de Semillas en la Provincia de Jiangsu (JBGS [2021]113) y el Programa Postdoctoral Excelente 2023 en la Provincia de Jiangsu, China (No. 2023ZB584).
Reconocimientos
Los autores desean expresar su sincero agradecimiento a todos los miembros del Laboratorio de Ingeniería de Embriones Ganaderos de Jiangsu por la recolección de muestras y la asistencia técnica durante la preparación de esta investigación.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1367730/full#supplementary-material
Referencias
1. Makela JA, Koskenniemi JJ, Virtanen HE, Toppari J. Desarrollo de testículos. Endocr Rev. (2019) 40:857–905. doi: 10.1210/er.2018-00140
2. Jin JM, Yang WX. Regulación molecular del eje hipotálamo-hipófisis-gónadas en varones. Gen. (2014) 551:15–25. doi: 10.1016/j.gene.2014.08.048
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Rizzoti K. Regulación genética del desarrollo de la hipófisis murina. J Mol Endocrinol. (2015) 54:R55–73. doi: 10.1530/JME-14-0237
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Chen M, Duan C, Yin X, Li X, Liu X, Zhang L, et al. El inhibidor de la prolactina cambia la producción de testosterona, la morfología testicular y la expresión de genes relacionados en cabras de cachemira. Front Vet Sci. (2023) 10:1249189. doi: 10.3389/fvets.2023.1249189
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Lakhssassi K, Sarto MP, Marin B, Lahoz B, Folch J, Alabart JL, et al. Explorando genes expresados diferencialmente en transcriptomas hipotálamos, pars tuberalis y glándula pineal en diferentes fenotipos de comportamiento sexual en carneros utilizando RNA-Seq. J Anim Sci 101. (2023). doi: 10.1093/jas/skac365
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Tang B, Hu S, Ouyang Q, Wu T, Lu Y, Hu J, et al. El análisis comparativo del transcriptoma identifica genes candidatos cruciales y vías en el eje hipotálamo-hipófisis-gonadal durante el desarrollo de los genitales externos de los gansos machos. BMC Genómica. (2022) 23:136. doi: 10.1186/s12864-022-08374-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Bo D, Jiang X, Liu G, Hu R, Chong Y. RNA-seq implica patrones de regulación divergentes de LincRNA en la espermatogénesis y el crecimiento de los testículos en cabras. Animales (Basilea). (2021) 11:625. doi: 10.3390/ani11030625
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Li J, Zhu X, Ma L, Xu H, Cao X, Luo R, et al. Detección de una nueva inserción/deleción de 20 pb (indel) en el gen PRND de ovejas mediante el método de expectativa matemática (EM). Prion. (2017) 11:143–50. doi: 10.1080/19336896.2017.1300740
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Dobin A, Davis CA, Schlesinger F, Drenkow J, Zaleski C, Jha S, et al. STAR: alineador de secuenciación de ARN universal ultrarrápido. Bioinformática. (2013) 29:15–21. doi: 10.1093/bioinformatics/bts635
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Robinson MD, McCarthy DJ, Smyth GK. edgeR: un paquete de bioconductores para el análisis de expresión diferencial de datos digitales de expresión génica. Bioinformática. (2010) 26:139–40. doi: 10.1093/bioinformatics/BTP616
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Kumar L. Mfuzz: un paquete de software para la agrupación en clústeres de datos de microarrays. Bioinformación. (2007) 2:5–7. doi: 10.6026/97320630002005
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Yu G, Wang LG, Han Y, He QY. clusterProfiler: un paquete de R para comparar temas biológicos entre grupos de genes. ÓMICAS. (2012) 16:284–7. doi: 10.1089/omi.2011.0118
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Langfelder P, Horvath S, WGCNA. un paquete R para el análisis de redes de correlación ponderada. BMC Bioinformática. (2008) 9:559. doi: 10.1186/1471-2105-9-559
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Amith MT, Fujimoto K, Tao C. NET-EXPO: un complemento gephi para el análisis de redes sociales de la exposición de la red para gráficos unipartitos y bipartitos. Carteles de HCI Int. (2019) 1034:3–12. doi: 10.1007/978-3-030-23525-3_1
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Fornes O, Castro-Mondragon JA, Khan A, van der Lee R, Zhang X, Richmond PA, et al. JASPAR 2020: actualización de la base de datos de acceso abierto de perfiles de unión de factores de transcripción. Ácidos nucleicos Res. (2020) 48:D87-92. doi: 10.1093/nar/gkz1001
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Korsunsky I, Millard N, Fan J, Slowikowski K, Zhang F, Wei K, et al. Integración rápida, sensible y precisa de datos de una sola celda con Harmony. Métodos Nat. (2019) 16:1289. doi: 10.1038/s41592-019-0619-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Jin S, Guerrero-Juárez CF, Zhang L, Chang I, Ramos R, Kuan CH, et al. Inferencia y análisis de la comunicación célula-célula utilizando CellChat. Nat Commun. (2021) 12:1088. doi: 10.1038/s41467-021-21246-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Gao X, Ye J, Yang C, Zhang K, Li X, Luo L, et al. Cribado y evaluación de ARN largos no codificantes en la pubertad de cabras. BMC Genómica. (2017) 18:164. doi: 10.1186/s12864-017-3578-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Gao X, Ye J, Yang C, Luo L, Liu Y, Ding J, et al. Análisis de secuenciación de ARN de la expresión génica del desarrollo controlada por lncRNA durante la pubertad en cabras y ratas. BMC Genet. (2018) 19:19. doi: 10.1186/s12863-018-0608-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Yang H, Ma J, Wang Z, Yao X, Zhao J, Zhao X, et al. Análisis de todo el genoma y predicción de la función de ARN largos no codificantes en la glándula pituitaria de oveja asociados con la maduración sexual. Genes (Basilea) 11. (2020). doi: 10.3390/genes11030320
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Ran M, Chen B, Li Z, Wu M, Liu X, He C, et al. Identificación sistemática de ARN largos no codificantes en testículos porcinos inmaduros y maduros. Biol Reprod. (2016) 94:77. doi: 10.1095/biolreprod.115.136911
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Liu J, Liu Z, Yi S, Cui S. Expresión del islote-1 y su colocalización con la hormona luteinizante, la hormona estimulante de la tiroides y el receptor de estrógeno alfa en la glándula pituitaria en desarrollo del feto de oveja. J Neuroendocrinol. (2005) 17:773–80. doi: 10.1111/j.1365-2826.2005.01364.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Thomas GB, McNeilly AS, Brooks AN. Desarrollo de gonadotrofos y tirotrofos en la hipófisis fetal de ovejas hembras: estudios de localización inmunocitoquímica. J Neuroendocrinol. (1993) 5:157–61. doi: 10.1111/j.1365-2826.1993.tb00376.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Gong W, Pan L, Lin Q, Zhou Y, Xin C, Yu X, et al. Perfil del transcriptoma del testículo de ratón postnatal en desarrollo mediante secuenciación de nueva generación. Sci China Life Sci. (2013) 56:1–12. doi: 10.1007/s11427-012-4411-y
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Yang H, Wang F, Li F, Ren C, Pang J, Wan Y, et al. Análisis exhaustivo de los patrones de expresión de ARN largo no codificante y ARNm en la maduración testicular de ovejas. Biol Reprod. (2018) 99:650–61. doi: 10.1093/biolre/ioy088
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Wu J, Xiong S, Jing J, Chen X, Wang W, Gui JF, et al. Análisis comparativo del transcriptoma de genes expresados diferencialmente y vías de señalización entre los testículos XY e YY en bagre amarillo. PLoS UNO. (2015) 10:e0134626. doi: 10.1371/journal.pone.0134626
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Mortensen AH, Schade V, Lamonerie T, Camper SA. La deleción de OTX2 en el ectodermo neural retrasa el desarrollo de la hipófisis anterior. Hum Mol Genet. (2015) 24:939–53. DOI: 10.1093/HMG/DDU506
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Xiang M, Li S. Foxn4: un regulador transcripcional multifacético de los destinos celulares en el desarrollo de vertebrados. Sci China Life Sci. (2013) 56:985–93. doi: 10.1007/s11427-013-4543-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Zhang S, Cui Y, Ma X, Yong J, Yan L, Yang M, et al. La transcriptómica unicelular identifica trayectorias divergentes de linaje de desarrollo durante el desarrollo de la hipófisis humana. Nat Commun. (2020) 11:5275. doi: 10.1038/s41467-020-19012-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Stockl JB, Schmid N, Flenkenthaler F, Drummer C, Behr R, Mayerhofer A, et al. Alteraciones relacionadas con la edad en el proteoma testicular de un primate no humano. Celdas 10. (2021). doi: 10.3390/cells10061306
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Kim E, Lee Y, Kim J-S, Song B-S, Kim S-U, Huh J-W, et al. El dominio extracelular de V-set y el dominio de inmunoglobulina que contiene 1 (VSIG1) interactúa con la proteína de la membrana celular de Sertoli, mientras que su motivo de unión a PDZ forma un complejo con ZO-1. Células molares. (2010) 30:443–8. doi: 10.1007/s10059-010-0138-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Nakamura N, Dai Q, Williams J, Goulding EH, Willis WD, Brown PR, et al. La alteración de un gen de la enolasa 4 (Eno4) de ratón específico de células espermatogénicas causa defectos estructurales en los espermatozoides e infertilidad masculina1. Biología de la reproducción. (2013) 88:107128. doi: 10.1095/biolreprod.112.107128
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Chimento A, Sirianni R, Casaburi I, Pezzi V. Papel de los receptores de estrógeno y el receptor de estrógeno acoplado a proteína g en la regulación del eje hipotálamo-hipófisis-testículo y la espermatogénesis. Endocrinol frontal (Lausana). (2014) 5:1. doi: 10.3389/fendo.2014.00001
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Ciller I, Palanisamy S, Ciller U, Al-Ali I, Coumans J, McFarlane J. La expresión génica de enzimas esteroidogénicas y la producción de testosterona están moduladas por el desarrollo del receptor de proteína morfogenética ósea-1B en testículos de ratón. Physiol Res. (2023) 72:359–69. doi: 10.33549/physiolres.935014
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Pellegrini M, Grimaldi P, Rossi P, Geremia R, Dolci S. Expresión del desarrollo de la vía de señalización BMP4 / ALK3 / SMAD5 en los testículos de ratón: un papel potencial de BMP4 en la diferenciación de espermatogonias. J Cell Sci. (2003) 116:3363–72. doi: 10.1242/jcs.00650
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Puglisi R, Montanari M, Chiarella P, Stefanini M, Boitani C. Papel regulador de BMP2 y BMP7 en la proliferación de células de espermatogonia y Sertoli en el ratón inmaduro. Eur J Endocrinol. (2004) 151:511–20. doi: 10.1530/eje.0.1510511
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Zhao GQ, Deng K, Labosky PA, Liaw L, Hogan BL. El gen que codifica la proteína morfogenética ósea 8B es necesario para el inicio y mantenimiento de la espermatogénesis en el ratón. Genes Dev. (1996) 10:1657–69. doi: 10.1101/gad.10.13.1657
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Zachow R, Uzumcu M. El sistema del factor de crecimiento de los hepatocitos como regulador de la función gonadal femenina y masculina. J Endocrinol. (2007) 195:359–71. doi: 10.1677/JOE-07-0466
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Scheri KC, Leonetti E, Laino L, Gigantino V, Gesualdi L, Grammatico P, et al. Receptor c-MET como posible biomarcador y molécula diana para tumores malignos de células germinales testiculares. Oncotarget. (2018) 9:31842–60. doi: 10.18632/oncotarget.25867
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Catizone A, Ricci G, Caruso M, Galdieri M, Scheri KC, Di Paolo V, et al. HGF modula la remodelación y contracción del citoesqueleto de actina en células mioides testiculares. Biomedicinas. (2015) 3:89–109. doi: 10.3390/biomedicines3010089
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Gumustekin M, Arici A, Cilaker Micili S, Karaman M, Guneli ME, Tekmen I. La vía Hgf/C-met tiene un papel en el daño testicular en la diabetes inducida por la estreptozotocina. Acta Endocrinol (Buchar). (2017) 13:17–22. doi: 10.4183/aeb.2017.17
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Pitetti JL, Calvel P, Zimmermann C, Conne B, Papaioannou MD, Aubry F, et al. Un papel esencial para los receptores de insulina e IGF1 en la regulación de la proliferación de células de Sertoli, el tamaño de los testículos y la acción de la FSH en ratones. Mol Endocrinol. (2013) 27:814–27. doi: 10.1210/me.2012-1258
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Griffeth RJ, Bianda V, Nef S. El papel emergente de los factores de crecimiento similares a la insulina en el desarrollo y la función de los testículos. Clin Androl básico. (2014) 24:12. doi: 10.1186/2051-4190-24-12
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Young JC, Wakitani S, Loveland KL. Señalización de la superfamilia TGF-beta en la formación de testículos y el desarrollo temprano de la línea germinal masculina. Semin Cell Dev Biol. (2015) 45:94–103. doi: 10.1016/j.semcdb.2015.10.029
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: RNA-seq, eje hipófisis-testículo, ovejas, red reguladora, etapas de desarrollo
Cita: Li S, Zhao B, Yang H, Dai K, Cai Y, Xu H, Chen P, Wang F y Zhang Y (2024) Análisis transcriptómico exhaustivo que revela la dinámica reguladora y las redes del eje hipófisis-testículo en ovejas a lo largo de las etapas de desarrollo. Frente. Vet. Sci. 11:1367730. doi: 10.3389/fvets.2024.1367730
Recibido: 09 de enero de 2024; Aceptado: 05 de febrero de 2024;
Publicado: 19 febrero 2024.
Editado por:
Abdolrahman Khezri, Universidad de Ciencias Aplicadas del Interior de Noruega, Noruega
Revisado por:
Gan Shangquan, Academia de Ciencias Agrícolas y de Recuperación de Xinjiang (XAARS), China Feng Su, Universidad Agrícola de Shandong, China
Derechos de autor © 2024 Li, Zhao, Yang, Dai, Cai, Xu, Chen, Wang y Zhang. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY). S
*Correspondencia: Yanli Zhang, zhangyanli@njau.edu.cn
†Estos autores han contribuido igualmente a este trabajo
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

