Circovirus canino: ¿un virus de enteritis emergente o endémico no diagnosticado?

Circovirus canino: ¿un virus de enteritis emergente o endémico no diagnosticado?
 Diana Gómez-Betancur1,
Diana Gómez-Betancur1,  Diana S. Vargas-Bermúdez2,
Diana S. Vargas-Bermúdez2,  Sebastián Giraldo-Ramírez3,
Sebastián Giraldo-Ramírez3,  Jairo Jaime2 y
Jairo Jaime2 y  Julián Ruiz-Sáenz1*
Julián Ruiz-Sáenz1*- 1Grupo de Investigación en Ciencias Animales—GRICA, Facultad de Medicina Veterinaria y Zootecnia, Universidad Cooperativa de Colombia, Bucaramanga, Colombia
- 2Universidad Nacional de Colombia, Facultad de Medicina Veterinaria y de Zootecnia, Centro de investigación en Infectología e Inmunología Veterinaria (CI3V), Sede Bogotá, Bogotá, Colombia
- 3Facultad de Medicina Veterinaria y Zootecnia, Fundación Universitaria Autónoma de las Américas, Medellín, Colombia
El Circovirus Canino (CanineCV) pertenece a la familia Circoviridae. Es un virus emergente descrito por primera vez en 2011; Desde entonces, se ha detectado en diferentes países y se puede definir como virus de distribución mundial. CanineCV infecta cánidos domésticos y silvestres y se relaciona principalmente con enteritis hemorrágica en caninos. Sin embargo, se ha identificado en muestras fecales de animales aparentemente sanos, donde en la mayoría de los casos se encuentra en coinfección con otros agentes virales como el parvovirus canino tipo 2 (CPV). La prevalencia/frecuencia estimada de CanineCV ha sido variable en las poblaciones y países donde se ha evaluado, llegando del 1 al 30%, y todavía hay muchos conceptos para definir las características epidemiológicas del virus. La caracterización molecular y los análisis filo-evolutivos que permiten postular el origen silvestre y la distribución intercontinental del virus. Esta revisión se centra en la importancia de continuar la investigación y establecer sistemas de vigilancia para este virus emergente.
1. Introducción
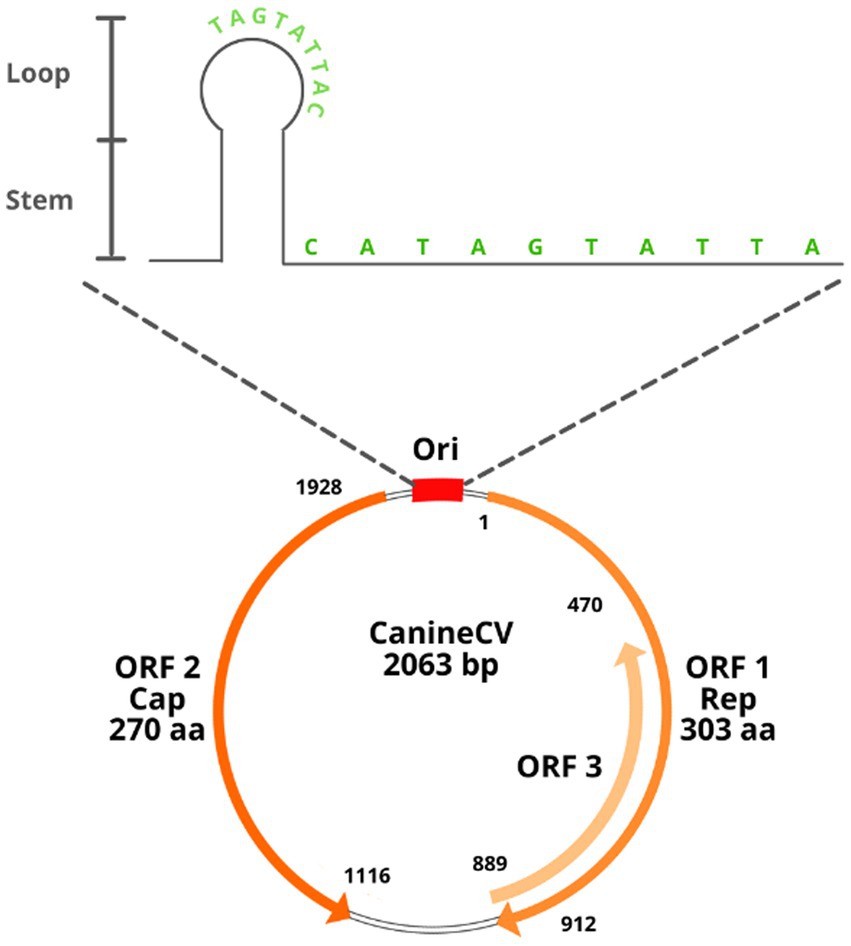
La familia Circoviridae comprende virus sin envoltura con un diámetro de 15-25 nm y un genoma de ADN circular monocatenario de menos de 2.500 nt de longitud. Según el Comité Internacional de Taxonomía de Virus (ICTV)1), dos géneros han sido reconocidos dentro de esta familia: Circovirus y Cyclovirus (1-3). Dentro del género Circovirus, se han identificado casi 50 especies que infectan aves y mamíferos (1), incluyendo la enfermedad clínicamente relevante del pico y las plumas (BFDV), circovirus del ganso, circovirus de paloma, circovirus canario, circovirus porcino y el circovirus canino más recientemente reconocido (CanineCV) (4-6).
CanineCV tiene un genoma de ADN monocatenario covalentemente cerrado de 2062-2063 nt (7, 8). Tiene un genoma ambisentido con dos ORFs principales dispuestos inversamente; ORF1 codifica el gen de la replicasa viral (Rep) (nt 1-912) necesario para el inicio del ciclo de replicación, y ORF2 codifica la proteína de la cápside (Cap) (nt 1,116-1928), que participa en la respuesta inmune del huésped (1, 7). Los genes Cap y Rep de CanineCV comparten, respectivamente, entre un 25 y un 50% de identidad con los otros circovirus animales conocidos (2). Se ha propuesto un tercer ORF (ORF3) en muestras de Tailandia; este ORF se encuentra entre nt 470 y 889 en la región antisentido en ORF1 (5). Los estudios en circovirus porcino tipo 2 (PCV2) relacionan ORF3 con una proteína asociada con la patogénesis viral y la inducción de apoptosis; sin embargo, en el caso de CanineCV, su función aún no se está aclarando (5, 9).
2. Replicación de virus
Al igual que con otros circovirus, CanineCV tiene 2 regiones intergénicas no codificantes. El primero corresponde a la región 5′-intergénica de 135 nt situada entre los codones de inicio de los 2 ORFs y comprende el origen de la replicación (Ori). Este último es una estructura tallo-bucle conservada termodinámicamente estable con una secuencia característica de 9 nt para el inicio de la replicación (TAGTATTAC), una secuencia palindrómica de 12 pares de nt y un bucle abierto de 10 nt (CATAGTATTA) (2). La segunda región intergénica es la región 3′ de 203 nt, ubicada entre los codones de terminación de los dos ORFs principales (Figura 1). Estas secuencias altamente conservadas en circovirus están asociadas con la replicación del círculo rodante (2, 10).
El mecanismo del círculo rodante constituye el proceso de replicación de los genomas circulares del ADN. También se ha descrito en plásmidos, virus de plantas y otras familias de virus animales, como Geminiviridae y Nanoviridae. Para los circovirus, el modelo de estudio ha sido principalmente el del circovirus porcino (PCV). Debido al tamaño reducido del genoma viral que limita su capacidad de codificación, este tipo de replicación depende en gran medida de los factores de la célula huésped y requiere enzimas celulares expresadas durante la fase S del ciclo celular (11, 12). Se estima que el virus utiliza glicosaminoglicanos como receptores de unión a las células diana y penetra en la célula por endocitosis mediada por clatrina (9). El genoma ssDNA se transporta al núcleo, donde se convierte en una doble cadena intermedia superenrollada que servirá como plantilla para generar ADN viral. En las especies de Geminivirus y Nanovirus, el cebador para la cadena negativa está compuesto de ADN o ARN con múltiples ribonucleótidos 5′ o ARN sintetizados por el huésped después de la infección; Sin embargo, aún no se ha determinado el cebador para la cadena negativa en circovirus (12).
Una vez que se forma la doble cadena del genoma, un complejo de iniciación compuesto por proteínas Rep y Rep’ que tienen actividad endonucleasa se une a la estructura tallo-bucle para dividir el bucle, generando un extremo 3’OH libre al que la ADN polimerasa celular se une para iniciar la replicación. Cuando una unidad del genoma se ha replicado, el complejo Rep cierra el bucle covalentemente, lo que lleva a la liberación de una molécula madre monocatenaria circular positiva y una molécula circular de ADN bicatenario, esta última compuesta por un híbrido de una hebra parental negativa y una hebra positiva recién sintetizada (11, 12). La molécula de ADN monocatenario puede encapsularse o participar en un segundo ciclo de replicación. En estudios in vitro en PCV, se han sugerido dos vías diferentes para la liberación del virus de la célula al final del ciclo de replicación: la primera es la gemación de grupos de viriones, y la segunda es la lisis celular por apoptosis; sin embargo, el mecanismo in vivo aún no se conoce bien [Figura 2; (11, 13)].
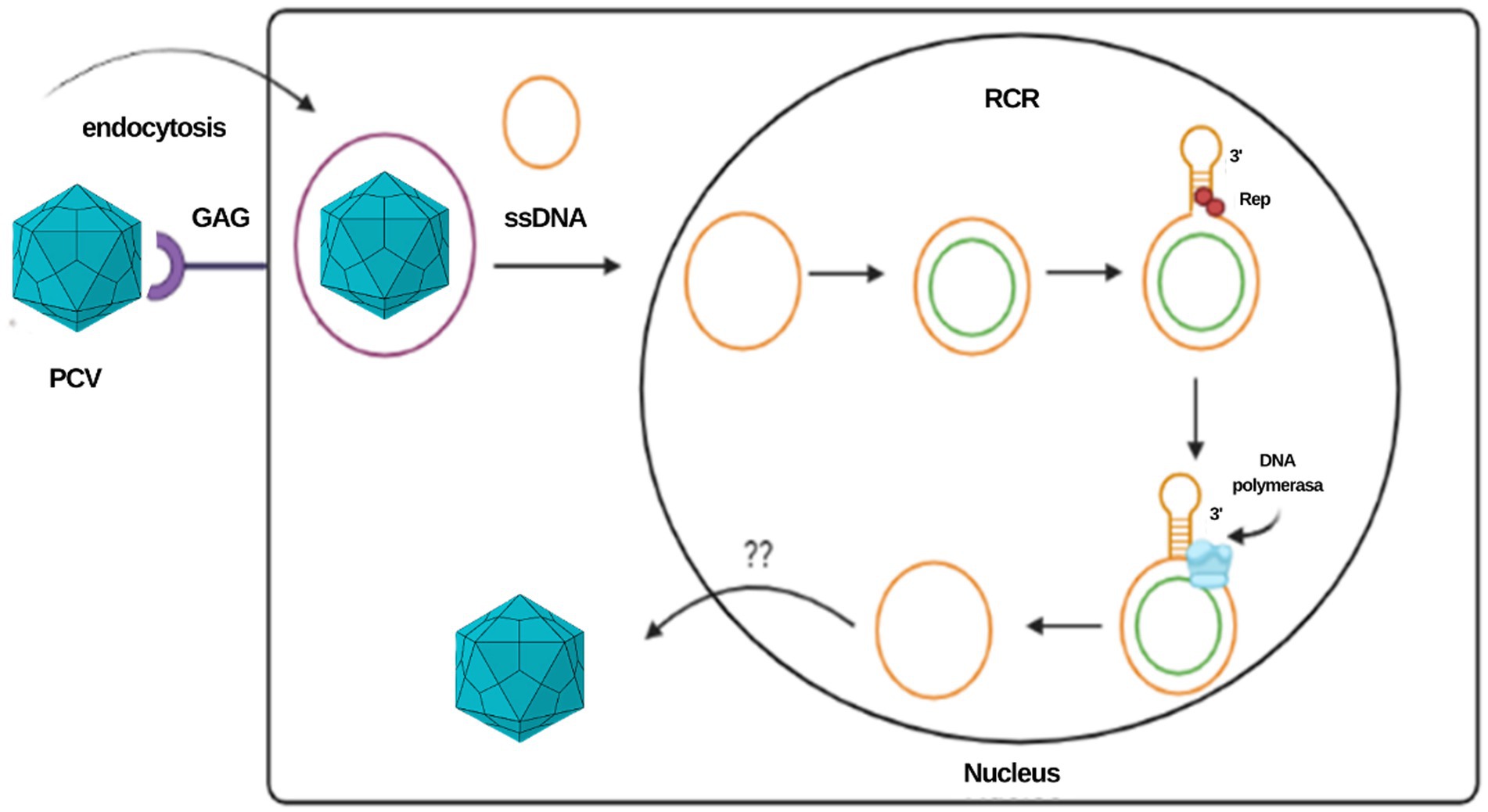 Figura 2. Representación del ciclo de replicación PCV y el mecanismo de círculo rodante. Este mismo mecanismo se propone para el CanineCV.
Figura 2. Representación del ciclo de replicación PCV y el mecanismo de círculo rodante. Este mismo mecanismo se propone para el CanineCV.
El modelo de «crisol» se ha propuesto para explicar la formación del intermediario de doble cadena. En este modelo, las proteínas Rep se unen a secuencias palindrómicas, generando una esfera de inestabilidad conocida como crisol. En este entorno, las cuatro cadenas repetidas invertidas presentan un estado «fusionado» ya que hay una ausencia de enlaces de hidrógeno entre los genomas positivo y negativo. En esta estructura, no se puede mantener ninguna formación de doble hélice, pero las cuatro hebras permanecen muy cerca una de la otra, yuxtapuestas en una estructura terciaria que permite un posible intercambio entre las hebras plantilla durante la síntesis del genoma, lo que proporciona dos plantillas para la síntesis de las secuencias palindrómicas durante el inicio y terminación de la replicación (11, 13). La disponibilidad de dos cadenas de plantillas durante la síntesis de la cadena principal contribuye a la flexibilidad y al aumento de la frecuencia de mutaciones en los Ori; sin embargo, el mecanismo de corrección inherente al crisol permite eliminar, corregir o regenerar mutaciones en las secuencias palindrómicas a través del mecanismo de cambio de plantilla, permitiendo la integración de la secuencia nt en el Ori (11).
3. Epidemiología molecular
CanineCV fue identificado por primera vez en 2011 por Kapoor et al. (2) en muestras de suero de seis de 205 perros clínicamente sanos, confirmando el primer circovirus no porcino en infectar mamíferos (2, 4, 6). Desde este primer informe y hasta la fecha, se han llevado a cabo diferentes estudios de detección, secuenciación genética y análisis filogenético en diferentes países del mundo, tanto en perros domésticos como en cánidos salvajes. El objetivo general de tales investigaciones ha sido principalmente establecer la circulación de CanineCV a nivel demográfico en la región o área (2, 4). Los estudios de detección de virus se han basado en técnicas moleculares que incluyen PCR convencional y cuantitativa (qPCR) (14, 15). El aislamiento del virus no ha tenido éxito. En las células MDCK infectadas con la cepa CanineCV D1056 de Brasil, solo se detectó ADN por qPCR en cantidades bajas durante la fase de inoculación, tal vez debido a que la cantidad de ADN viral encontrado por qPCR para CanineCV fue baja (16). El mismo resultado negativo se ha reportado utilizando muestras italianas en células MDCK y D17 (8). Sin embargo, es bien sabido que la mayoría de los circovirus, como la PCV, generalmente se consideran no citopatógenos y la baja eficiencia de crecimiento de las PCV en células cultivadas se ha convertido en un obstáculo importante para el establecimiento de métodos de diagnóstico (17). Sin embargo, pocos informes han mostrado el efecto citopático de PCV2 en líneas celulares específicas (18, 19).
CanineCV se define como un agente de distribución mundial ya que su circulación ha sido reportada en 4 de los 5 continentes. Los análisis filogenéticos de las cepas reportadas hasta la fecha se han preparado en base al genoma completo y con las secuencias de nucleótidos o aminoácidos concatenados de las proteínas Rep y Cap (3, 4, 7). Se han hecho múltiples esfuerzos para establecer un sistema de clasificación que ayude a comprender el origen y la evolución del virus. Sun et al. en China en 2018 basaron el análisis filogenético de las cepas de CanineCV detectadas en secuencias ORF2 y propusieron una clasificación en dos grupos: CanineCV-1 y CanineCV-2 (10). Posteriormente, se informó que las cepas existentes de CanineCV a nivel mundial podrían dividirse en cuatro grupos diferentes: CanineCV-1, que incluye cepas de Italia, cuatro de los Estados Unidos, dos de Alemania, una de Argentina y una de China. Cepas del grupo CanineCV-2 de China, de las cuales tres se detectaron en 2014, 13 en 2015 y 24 en 2017. CanineCV-3 incluye cepas de China identificadas entre 2014 y 2015, y CanineCV-4 contiene una cepa de los Estados Unidos. tres cepas de la provincia de Guangxi e incluyeron las cinco cepas detectadas en su investigación. Los autores indican que para diferenciar los 4 grupos, es posible utilizar polimorfismos de secuencias de nucleótidos individuales (20). Por otro lado, en el análisis realizado por Piewbang et al. en 2018 (5), se concluyó que las secuencias CanineCV detectadas en su estudio eran diferentes de la mayoría de las secuencias reportadas y estaban relacionadas principalmente con la cepa UCD3-478 de Estados Unidos. Además, los autores destacan que los árboles filogenéticos basados en los genes Rep y Cap de los genomas CanineCV que circulan en Tailandia son divergentes y revelan patrones desiguales al comparar ambos genes, lo que se asocia con eventos de recombinación (5).
En América del Sur, CanineCV se ha detectado en Argentina, Brasil y Colombia. Kotsias F et al. (21) detectaron la cepa UBA-Baires de CanineCV en un brote de enfermedad entérica en cachorros entre 2014 y 2015 en Argentina. La evaluación filogenética de las secuencias del genoma completo mostró que esta cepa se agrupa con virus de Europa, principalmente de Italia, y difiere considerablemente de las cepas reportadas en los Estados Unidos y China (21). En contraste, la cepa D1056 reportada en Brasil por Cruz et al. (16) mostró una alta similitud (<80%) con la cepa NY214 reportada en los Estados Unidos. Giraldo-Ramírez et al. Colombia confirmó la separación de las cepas CanineCV en cuatro genotipos diferentes, designando clados de acuerdo con la distribución geográfica y con las nuevas secuencias reportadas de la siguiente manera: El genotipo China incluye secuencias encontradas en China, el genotipo Asia-1 y Asia-2 incluye secuencias de Tailandia y China, y el genotipo cosmopolita contiene secuencias de Italia, Alemania, China, Estados Unidos, Argentina y la cepa encontrada en Colombia, definiendo también un clado de secuencias CanineCV identificadas en zorros [Figura 3; (22)].
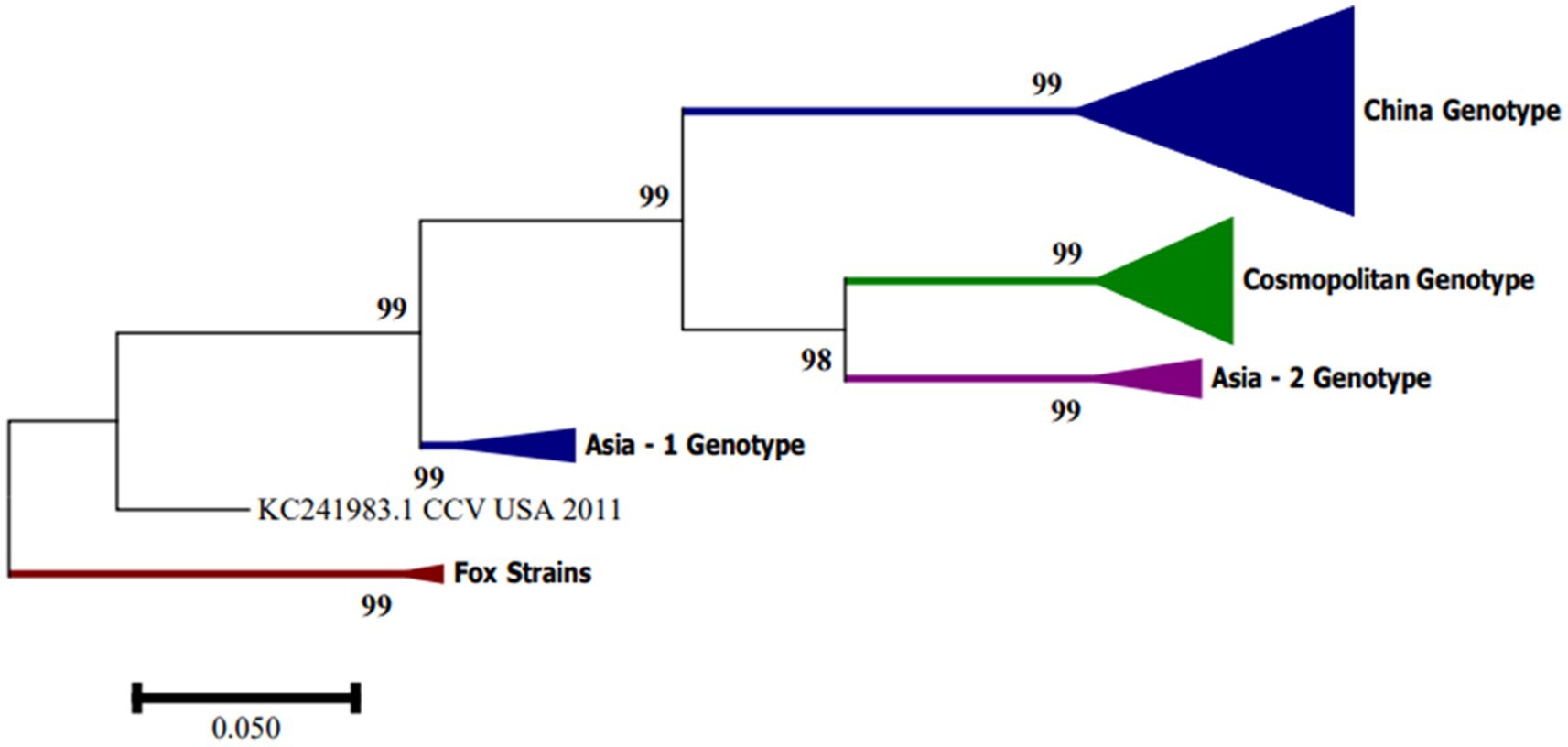 Figura 3. Árbol filogenético basado en secuencias completas del genoma CanineCV. El análisis filogenético se realizó utilizando MEGA 7.0 para Windows por máxima verosimilitud con 1.000 réplicas bootstrap y el modelo de sustitución de nucleótidos Tamura de 3 parámetros.
Figura 3. Árbol filogenético basado en secuencias completas del genoma CanineCV. El análisis filogenético se realizó utilizando MEGA 7.0 para Windows por máxima verosimilitud con 1.000 réplicas bootstrap y el modelo de sustitución de nucleótidos Tamura de 3 parámetros.
El análisis filogenético de secuencias CanineCV identificadas en zorros árticos y rojos en Noruega (23) mostró una agrupación distinguible en cinco grupos en el árbol construido con secuencias de genomas completos y en el árbol de aminoácidos concatenados de las proteínas Rep y Cap. El grupo 1 incluyó secuencias identificadas en perros, lobos y un tejón encontrado en Europa, Asia y los Estados Unidos. Los grupos 2, 3 y 4 incluyeron secuencias identificadas en perros en Asia, y la secuencia 09-10F de 2011 de un zorro rojo en Italia, Grupo 5, se compuso de secuencias identificadas en zorros. La cepa UCD3-478 no encajó en ninguno de los grupos anteriores (23). La presencia de la secuencia 09-10F/2011 en el Grupo 4 indica que la segregación de los grupos no ocurre en función del huésped donde fueron identificados. Los autores también definen que la cepa UCD3-478 puede representar un intermediario entre los grupos 4 y 5 o la única secuencia de un posible sexto grupo (23).
Análisis recientes de Oriente Medio, incluyendo muestras turcas e iraníes pertenecientes a perros diarreicos a través del análisis de las secuencias parciales Rep y Cap nt, habían encontrado que las cepas CanineCV iraníes estaban más estrechamente relacionadas con las cepas detectadas en Turquía, lo que permite plantear la hipótesis de una posible introducción del virus desde este país vecino (24-26). Sin embargo, el análisis más reciente de secuencias completas de nucleótidos del genoma de cepas CanineCV obtenidas de perros iraníes pertenece a un clado independiente separado (24).
Adicionalmente, en 2020 se realizó el primer informe sistemático sobre el uso de codones y adaptación a los hospedadores de CanineCV en el que se utilizaron todas las secuencias disponibles. Los resultados sugieren que CanineCV desciende del murciélago y que las secuencias han estado dominadas principalmente por la selección natural, lo que permite proponer una hipótesis de transmisión entre especies en las que CanineCV puede haber evolucionado a partir de murciélagos (análisis de origen) y, posteriormente, haberse adaptado a cánidos domésticos y silvestres (27).
4. Enfermedad en perros
En 2013, Li et al. caracterizaron el genoma completo de CanineCV del hígado de un perro con gastroenteritis hemorrágica severa, linfadenitis granulomatosa y vasculitis. Desde entonces, la infección CanineCV se ha asociado con perfiles clínicos de gastroenteritis aguda, diarrea hemorrágica, signos de vasculitis, linfadenitis, trombocitopenia, neutropenia y linfopenia, que se relaciona con la actividad inmunosupresora debido al daño del tejido linfoide; este último también se ha descrito en la infección por PCV en cerdos y en aves que sufren el virus de la enfermedad del pico y las plumas (BFDV) (3, 6, 28). Otros autores también han asociado CanineCV con signos respiratorios como disnea, secreción nasal, tos o estertores en los pulmones (5). Sin embargo, el virus también se ha detectado en muestras de caninos asintomáticos, por lo que su patogénesis y epidemiología aún no se pueden definir claramente.
Se podrían sugerir diferentes formas de infección, como la subclínica, clínica o coinfección con otros agentes infecciosos, y la vía fecal como principal vía de transmisión (3, 6, 28). Con el objetivo de comprender y establecer el comportamiento de la infección CanineCV, además de su prevalencia y diversidad genética, se han llevado a cabo múltiples investigaciones a nivel mundial en perros con síntomas compatibles con los descritos y en perros aparentemente sanos.
La prevalencia de CanineCV en diferentes países, tanto en muestras de pacientes con signos compatibles como en animales asintomáticos, es variable. Li et al. en 2013 en Estados Unidos (3) establecieron una prevalencia en muestras de heces del 11,3% en perros con diarrea, del 6,9% en perros sanos y del 3,3% en muestras de suero en perros como signos de trombocitopenia, neutropenia o antecedentes de garrapatas. La prevalencia entre muestras positivas de animales con diarrea y muestras de perros sanos no fue significativa (3). En Asia, se han realizado varios estudios en diferentes países. En Taiwán, Hsu et al. (4) encontraron una correlación altamente significativa entre la presencia de diarrea y CanineCV, con una prevalencia de 28,02% en perros con diarrea y 11,9% en perros sanos; los autores sugirieron que la diferencia en los resultados con respecto a los EE.UU. puede estar relacionada con la distribución geográfica, la técnica molecular o el diseño del cebador (4). En el noreste de China, se encontró una prevalencia de CanineCV del 15,6% en perros con diarrea y del 6,7% en perros sanos; Además, los autores encontraron una diferencia significativa entre ambas prevalencias (20).
Sun W et al. en 2019, en la región sur de Guangxi establecieron una prevalencia molecular de CanineCV en muestras de suero de perros con diarrea de 8.75% y en perros sanos de 9.25% (10). En Alemania, se estableció una prevalencia de CV canino del 20,1% en perros con diarrea y del 7,3% en perros sanos, lo que indica que el virus se puede encontrar significativamente más frecuentemente en perros con diarrea (29). Sin embargo, tampoco se ha encontrado en Alemania diferencias significativas entre la detección de CanineCV en perros sanos y perros con diarrea hemorrágica, con tasas de prevalencia de 4,5 y 3,6%, respectivamente (28). En Vietnam, la evaluación de CanineCV se realizó en 81 muestras fecales positivas para CPV-2c, estableciendo que 19,8% de las muestras fueron positivas para CanineCV en cachorros diarreicos menores de 7 meses de edad (30).
En la región de Oriente Medio, la presencia de CanineCv se ha estudiado en Turquía e Irán. En Turquía, de un total de 150 muestras de hisopos rectales recogidas de animales con manifestaciones de enfermedad gastrointestinal, se obtuvieron nueve muestras positivas (6%) con coinfección con los otros dos virus entre 2 y 5% (26). Para investigar la prevalencia y las características genómicas de CanineCV en perros iraníes, se recogieron un total de 203 muestras fecales de perros entre febrero y noviembre de 2018 de cinco regiones geográficas diferentes y se examinaron mediante PCR en tiempo real (qPCR). Trece perros (6,4%) dieron positivo para el ADN de CanineCV, y todos fueron detectados en coinfección con CPV-2. Más tarde, en 2022, se llevó a cabo un segundo estudio de la misma manera utilizando PCR en tiempo real en 156 hisopos rectales de caninos clínicamente sanos. Se obtuvieron 14 muestras positivas para CanineCV (8,9% de prevalencia). La circulación de CanineCV en perros no diarreicos en Irán destaca la necesidad de más investigaciones epidemiológicas (24, 25).
El único estudio reportado hasta la fecha sobre la seroprevalencia de CanineCV se realizó con muestras tomadas entre 2016 y 2017 en el noreste de China (31). Los autores encontraron una seroprevalencia del 39,82%, con diferencias entre las diferentes ciudades que oscilan entre el 23 y el 43%. Del mismo modo, el estudio mostró una diferencia significativa en la prevalencia entre perros con diarrea y perros sanos (31). En la Tabla 1 se muestra una recopilación de los estudios realizados hasta la fecha en diferentes países y obtenidos de bases de datos como PubMed y Science Direct, con el propósito de la detección de CanineCV en perros domésticos.
La prevalencia de CanineCV varía en un amplio rango. Las diferencias entre los estudios pueden deberse a múltiples factores como se mencionó anteriormente. Además, todavía no es posible definir si el virus se encuentra con mayor frecuencia en perros con diarrea que en animales sanos, como lo demuestran las variaciones en los resultados de los informes publicados hasta la fecha. Por lo tanto, es importante realizar investigaciones moleculares y que definan claramente las características epidemiológicas del virus y su papel como agente infeccioso en la enfermedad clínica gastrointestinal en perros domésticos.
5. Hallazgos histopatológicos
La necropsia e inmunohistoquímica de casos retrospectivos de perros con signos compatibles con CanineCV ha mostrado lesiones críticas, como vasculitis necrotizante en intestino y bazo, linfadenitis granulomatosa y drenaje histiocítico en parches y ganglios linfáticos de Peyer, y lesiones microscópicas en el riñón (3). Los hallazgos clínico-histopatológicos en un informe de caso en Connecticut, Estados Unidos, mostraron membranas mucosas macroscópicamente pálidas, intestino delgado azul-negro, heces negras en el colon, esplenomegalia y múltiples ganglios linfáticos agrandados y hemorrágicos. A nivel microscópico, se observó vasculitis transmural circunferencial y necrosis de células de cripta y necrosis linfocitaria en los ganglios linfáticos y el bazo en el intestino, y también se encontró vasculitis en el hígado, los riñones, el pulmón, las meninges y el cerebro (33). Del mismo modo, se han reportado lesiones como friabilidad del intestino delgado, congestión focal de los parches de Peyer, contenido acuoso y hemorrágico en el intestino delgado, intestino grueso con petequias multifocales y melena en el interior (6).
Histológicamente, se encontró pérdida de microvellosidades, necrosis en las células de la cripta, necrosis linfoide en los parches de Peyer en el íleon, ganglios linfáticos mesentéricos y bazo, inflamación granulomatosa y aumento de histiocitos en los ganglios linfáticos (6). Recopilando lo anterior, es posible establecer que los hallazgos histopatológicos asociados a CanineCV tienen una estrecha similitud, tanto en los órganos afectados como en las lesiones encontradas, siendo las más comunes la coloración azul-negra en el intestino, la necrosis linfocitaria y la inflamación granulomatosa. También se ha descrito un perfil histopatológico similar en la infección por PCV2 en cerdos; Sin embargo, todavía faltan estudios de causalidad que demuestren mediante hibridación in situ o inmunohistoquímica la presencia de este agente viral en lesiones compatibles (33).
6. Infección en carnívoros salvajes
Diferentes estudios han confirmado la presencia de CanineCV en carnívoros salvajes. Un análisis retrospectivo de muestras tomadas entre 2013 y 2014, detectó y caracterizó molecularmente CanineCV en muestras de órganos de perros domésticos, lobos, zorros y tejones recolectados en regiones del centro y sur de Italia; CanineCV se encontró en nueve de 34 lobos, ocho de 209 perros y uno de cada 10 tejones, lo que confirma que la circulación del virus no está restringida a los perros domésticos (8).
CanineCV también se ha confirmado en zorros en el Reino Unido, a partir de muestras tomadas de zorros que sufrieron meningoencefalitis entre 2009 y 2013. Altas frecuencias de infección en muestras histológicas y en suero de zorros medidas por PCR en tiempo real. Además, el análisis filogenético reveló una estrecha relación con CanineCV identificada en 2011 en los EE.UU. con una identidad de 92 y 89% en la secuencia de aminoácidos en la proteína Rep y en la secuencia nt, respectivamente; sin embargo, las secuencias de CanineCV de perros y CanineCV de zorros agrupados en clados separados (34). En particular, la infección por circovirus en zorros salvajes se asocia con signos neurológicos, en contraste con la enfermedad del perro doméstico que se asoció con gastrointestinal, respiratoria y vasculitis. El hecho de que los circovirus que infectan a los zorros y los que infectan a los perros domésticos sean virus muy similares pero con síntomas diferentes plantea varias preguntas para futuras investigaciones sobre la transmisión y patogénesis de ambos virus entre especies de la misma familia (34). Además, buscando evaluar la presencia de CanineCV en zorros rojos (Vulpes vulpes) de Italia mediante el uso de enfoques moleculares y filogenéticos, se detectó ADN CanineCV en una de las 32 muestras de heces procesadas; confirmando que los perros y los zorros podrían compartir circovirus filogenéticos relacionados (35).
En otro análisis retrospectivo en muestras de 51 zorros árticos (Vulpes lagopus) del archipiélago de Svalbard, Noruega y 59 zorros rojos (Vulpes vulpes) del norte de Noruega (continente) muestreados de 1996 a 2018, se detectó ADN CanineCV en 10 zorros rojos y 11 zorros árticos. Se encontró ADN positivo incluso en muestras de zorros árticos muestreados en 1996, quince años antes del primer informe en perros domésticos en Estados Unidos, lo que sugiere que los carnívoros salvajes podrían haber albergado un ancestro de CanineCV que luego podría transmitirse a los cánidos domésticos, destacando así el papel de los carnívoros salvajes en la transmisión de patógenos a las poblaciones de perros domésticos (23).
7. Coinfección y análisis comparativo con PCV
En los estudios sobre la detección de CanineCV realizados hasta la fecha, también se ha analizado la coinfección con otros agentes infecciosos. Entre estas, la presencia de bacterias como Salmonella sp., Campylobacter sp. y enterotoxinas de Clostridium sp.; parásitos como Giardia sp. y Cryptosporidium sp.; y se han encontrado virus como el parvovirus canino tipo 2 (CPV-2), el coronavirus canino y el moquillo canino (3, 6, 7). De manera similar, la coinfección entre varios patógenos y circovirus ya se ha descrito en cerdos, donde es relativamente común (36). Las PCV se han estudiado ampliamente durante varias décadas, y se han identificado cuatro subtipos diferentes: PCV1, PCV2, PCV3 y PCV4 (36-38). PCV1 corresponde a un tipo no patógeno, descrito en 1974 como un contaminante en líneas celulares de riñón de cerdo PK-15 (39, 40). La PCV2 es altamente patógena, y se ha asociado con una serie de síndromes y patologías clasificadas en cuatro formas de presentación que afectan la producción porcina a nivel mundial, generando innumerables pérdidas económicas en la industria (41). La PCV3 se asocia con dos presentaciones clínicas llamadas forma sistémica y forma reproductiva (42). Finalmente, PCV4 es el descubrimiento más reciente y se ha reportado solo en China (43).
La PCV2 se asoció inicialmente con el síndrome de desgaste multisistémico (PMWS); sin embargo, después de múltiples investigaciones, se estableció la asociación de PCV2 con otras condiciones clínicas, como hepatitis, insuficiencia reproductiva, enfermedad respiratoria y nefropatía (PDNS). También se ha asociado con la enfermedad del complejo respiratorio porcino (PRDC) (36, 38). Actualmente, se reconoce como agente causal de algunas afecciones conocidas como enfermedades asociadas al circovirus porcino (PCVAD), que afectan principalmente a cerdos de engorde de 7 a 16 semanas de edad, donde existen manifestaciones clínicas como pérdida de peso, hipertrofia de ganglios linfáticos, ictericia, retraso del crecimiento, disnea y abortos. Los hallazgos histopatológicos incluyen depleción linfoide, reemplazo por histiocitos en tejido linfoide, bronquiolitis y neumonía intersticial con infiltrado mononuclear en los pulmones (41). Existen varios informes y estudios sobre el papel de la coinfección con otros agentes infecciosos, ya que es inusual que la PCV2 genere cuadros clínicos como un solo agente (36). Generalmente se encuentra junto con otros microorganismos, como Salmonella sp., Mycoplasma hyopneumoniae, virus de la influenza porcina (SIV), virus del síndrome reproductivo y respiratorio porcino (PRRSV) y parvovirus porcino (PPV). Incluso se ha descrito coinfección entre diferentes genotipos de PCV2. Además, se ha sugerido que las coinfecciones se asocian con una mayor gravedad de la infección (41, 44).
El efecto adverso de las coinfecciones por PCV2 con los agentes antes mencionados podría explicarse por las repercusiones en el sistema inmunológico de los cerdos, ya que el virus se dirige a los tejidos linfoides. La replicación de PCV2 destruye la arquitectura normal de los folículos linfoides, causando el agotamiento linfoide que luego es reemplazado por histiocitos. La replicación del virus se ha evidenciado en órganos como el bazo, las amígdalas, los pulmones, el riñón, el hígado y el timo; en este último, la PCV2 altera la selección de linfocitos T, particularmente en animales más jóvenes. También infecta las células dendríticas, limitando su función; Tales efectos generan una depleción linfoide severa e inmunosupresión severa en cerdos, aumentando la probabilidad de contraer otras infecciones (41, 44).
También hay informes de la detección de PCV2 y PCV3 en perros. Hasta la fecha, se ha descubierto PCV2 en otros reservorios, como terneros, cabras, ratones y en carnívoros, como visones y zorros, lo que indica que la transmisión a huéspedes no porcinos es posible. El contacto cercano entre animales es probablemente la principal vía de transmisión, particularmente en granjas multiespecies y sitios accesibles a la vida silvestre (45, 46). Los estudios reportados de PCV2 en huéspedes no porcinos han asociado la presencia del virus con signos gastrointestinales en perros y con esterilidad en mapaches. Sin embargo, los autores concluyen en la necesidad de continuar con estudios que permitan definir la asociación directa entre PCV2 y el perfil clínico descrito. Del mismo modo, el genoma PCV3 se ha detectado en poblaciones de perros domésticos, lo que indica su transmisión a huéspedes no porcinos, e incluso se ha encontrado coinfección CPV / PCV3 en perros (47, 48).
Al igual que en la infección por PCV2, la patogénesis de CanineCV y su participación como agente coinfectante aún no está claramente definida. La investigación actual sugiere que su papel como patógeno puede ser exacerbar las condiciones clínicas, como la infección por PCV2, debilitando el sistema inmunológico y favoreciendo la entrada y diseminación de otros virus y bacterias. Esto se debe a que, en la mayoría de las detecciones de CanineCV realizadas, se ha encontrado que el virus está acompañado de otros agentes infecciosos, como CPV con tasas de coinfección de hasta el 100% (22). Hasta la fecha, pocos informes confirmaron CanineCV como un agente único en perros que sufren gastroenteritis aguda o diarrea hemorrágica (3, 21).
A partir de los hallazgos del material genético de CanineCV en tejidos linfoides, se han propuesto diferentes hipótesis respecto a la manifestación clínica de la infección CanineCV (6, 28). En primer lugar, la replicación de CanineCV en tejidos linfoides podría verse favorecida por una infección previa con CPV, sin manifestación clínica aparente o generación de enfermedad. En segundo lugar, la presencia de un efecto sinérgico donde la infección CanineCV causa inmunosupresión y permite el desarrollo de la enfermedad clínica por CPV, incluso en individuos vacunados, una situación que se ha descrito en cerdos como se mencionó anteriormente.
También se ha evaluado el posible papel de CanineCV en el síndrome de diarrea hemorrágica aguda. De acuerdo con los resultados, se ha reportado una baja probabilidad de que CanineCV actúe como un agente putativo del síndrome, pero CanineCV puede ser un elemento predisponente para infecciones bacterianas o virales. La prevalencia de CanineCV en el estudio fue del 4,6%, lo que indica que podría ser solo un hallazgo incidental donde el virus formaría parte de la microbiota intestinal normal de los caninos (28); esto también se ha descrito en cerdos donde PCV1 está presente en animales asintomáticos como agente no patógeno (49). El hallazgo más significativo del estudio fue el hallazgo de una mayor mortalidad en perros que presentaron coinfección CPV2/CanineCV. Hay informes previos donde se encontró una mayor susceptibilidad o una mayor gravedad de la infección cuando CPV2 se manifiesta en coinfección con otros patógenos, como el coronavirus entérico. Los autores sugieren que lo mismo puede suceder con CanineCV, como se ha observado y reportado en cerdos en infección por PCV2.
8. Observaciones finales
El descubrimiento de virus emergentes como CanineCV plantea un importante tema de investigación en animales domésticos y salvajes. La evidencia de múltiples lagunas en el conocimiento de este virus en aspectos como las características patogénicas y epidemiológicas plantea la necesidad de crear y continuar con estudios que permitan ampliar y definir el conocimiento sobre la infección por CanineCV. La evidencia científica respalda que este virus representa un riesgo para la salud y el bienestar de la población canina, por lo que se debe trabajar para definir muchos conceptos y lagunas en la comprensión de este patógeno emergente. Por lo tanto, es necesario fortalecer el conocimiento de los veterinarios y clínicos sobre las posibles manifestaciones como enfermedad no solo como agente único sino también en la coinfección con otros patógenos. Adicionalmente, cabe destacar el papel de los cánidos silvestres, ya que, según investigaciones recientes, es evidente que en esta población, CanineCV parece ser más patógeno, y su participación en la transmisión del virus a caninos domésticos parece ser relevante, especialmente porque la invasión humana de hábitats silvestres favorece el contacto entre ambas poblaciones, lo que contribuye a la transmisión de patógenos entre especies.
La función del CV Canino como agente primario o coinfectante en enfermedades entéricas en caninos sigue siendo incierta; La investigación existente sugiere la importancia de continuar con otros modelos de estudio que aporten nueva información para definir los conceptos que siguen siendo dudosos hasta ahora. Como se puede observar en el desarrollo de este artículo, CanineCV podría tener diferentes vías de desarrollo de la enfermedad en caninos, ya sea como agente primario que conduce a generar los signos clínicos descritos o para causar infección subclínica donde no hay signos pero donde el animal podría propagar el virus o contribuir a la gravedad de la enfermedad en la coinfección con otros agentes. Además, CanineCV, al igual que los otros circovirus, podría jugar un papel importante en la respuesta inmune del animal, favoreciendo la entrada de otros agentes infecciosos o impidiendo su recuperación (Figura 4).
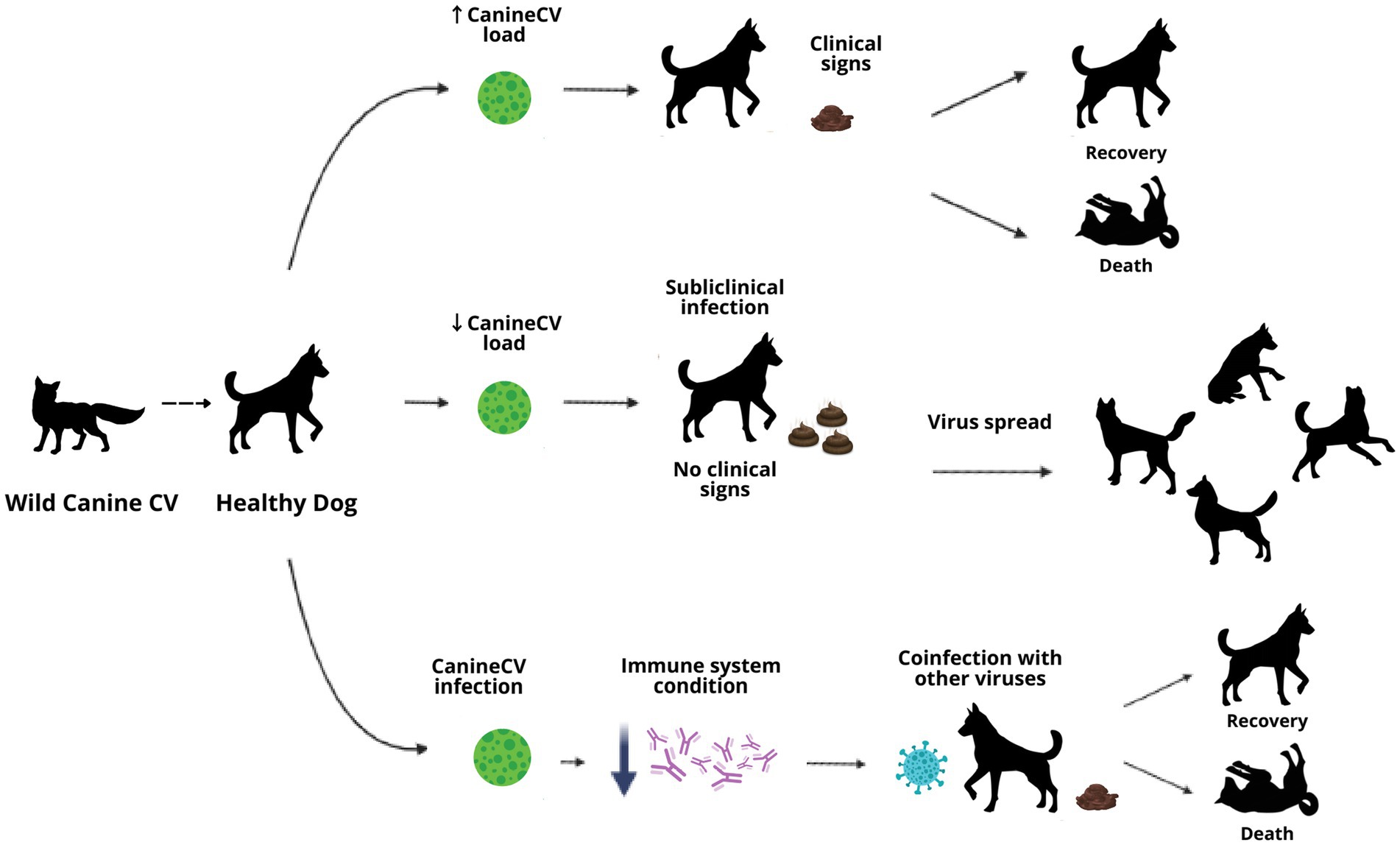 Figura 4. Diagrama de posibles rutas de enfermedad en la infección por CanineCV en perros domésticos.
Figura 4. Diagrama de posibles rutas de enfermedad en la infección por CanineCV en perros domésticos.
9. Conclusión
Las características patogénicas y epidemiológicas de CanineCV son actualmente poco conocidas, por lo que CanineCV representa un reto de exploración multidisciplinar tanto en el campo de la investigación como en la clínica. Comprender las particularidades de este nuevo virus, ya sea en animales sanos o coinfectados con otros agentes, podría proporcionar información valiosa sobre las prácticas profilácticas o terapéuticas en enfermedades entéricas en caninos, contribuyendo a la salud y el bienestar de las poblaciones caninas. Así, es necesario seguir realizando investigaciones en diferentes poblaciones caninas que permitan evaluar la epidemiología del CV Canino en sitios geográficos aún no explorados y su comportamiento en animales sanos o coinfectados, además, de monitorizar la evolución clínica de los animales infectados para ayudar a comprender la patogénesis del CV Canino.
Contribuciones del autor
JR-S y JJ: conceptualización, validación, recursos y curación de datos. DG-B y DV-B: metodología, análisis formal e investigación. DG-B: redacción: preparación original del proyecto. DG-B, DV-B, SG-R, JJ y JR-S: redacción, revisión y edición. SG-R: software y visualización. JR-S: supervisión, administración de proyectos y adquisición de fondos. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Esta investigación fue financiada por una subvención de CONADI-UCC a JR-S y el APC fue financiado por CONADI-UCC.
Reconocimientos
Authors quiere agradecer a los miembros del laboratorio GRICA en Medellín por sus discusiones y comentarios significativos.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Notas
1. ^ http://www.ictvonline.org/virustaxonomy.asp
Referencias
1. Breitbart, M , Delwart, E , Rosario, K , Segales, J , Varsani, A , e Informe ICTV, C . Perfil taxonómico del virus ICTV: Circoviridae. J Gen Virol. (2017) 98:1997–8. DOI: 10.1099/jgv.0.000871
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Kapoor, A , Dubovi, EJ , Henríquez-Rivera, JA , y Lipkin, WI . Secuencia completa del genoma del primer circovirus canino. J Virol. (2012) 86:7018. doi: 10.1128/JVI.00791-12
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Li, L , McGraw, S , Zhu, K , Leutenegger, CM , Marcas, S.L. , Kubiski, S, et al. Circovirus en tejidos de perros con vasculitis y hemorragia. Emerg infectar dis. (2013) 19:534–41. doi: 10.3201/eid1904.121390
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Hsu, SA , Lin, TH , Wu, HY , Lin, LS , Chung, CS , Chiou, MT, et al. Alta tasa de detección de circovirus canino en perros diarreicos. BMC Vet Res. (2016) 12:116. DOI: 10.1186/S12917-016-0722-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Piewbang, C , Jo, WK , Soplo, C , Van der Vries, E , Kesdangsakonwut, S , Rungsipipat, A, et al. Nuevas cepas de circovirus canino de Tailandia: evidencia de recombinación genética. Sci Rep. (2018) 8:7524. DOI: 10.1038/S41598-018-25936-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Thaiwong, T , Sabio, AG , Maes, RK , Mullaney, T , y Kiupel, M . Circovirus canino 1 (CaCV-1) y parvovirus canino 2 (CPV-2): infecciones duales recurrentes en una colonia de cría de Papillon. Veterinario Pathol. (2016) 53:1204–9. doi: 10.1177/0300985816646430
7. Decaro, N , Martella, V , Desario, C , Lanave, G , Circella, E , Cavalli, A, et al. Caracterización genómica de un circovirus asociado a enteritis hemorrágica mortal en perro. Italia PLoS One. (2014) 9:E105909. doi: 10.1371/journal.pone.0105909
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Zaccaria, G , Malatesta, D , Scipioni, G , Di Felice, E , Campolo, M , Casaccia, C, et al. Circovirus en carnívoros domésticos y salvajes: ¿un importante agente oportunista? Virología. (2016) 490:69–74. doi: 10.1016/j.virol.2016.01.007
9. Zhai, SL , Chen, SN , Xu, ZH , Tang, MH , Wang, FG , Li, XJ, et al. Circovirus porcino tipo 2 en China: una actualización y conocimientos sobre su prevalencia y control. Virol J. (2014) 11:88. doi: 10.1186/1743-422X-11-88
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Sol, W , Zhang, H , Zheng, M , Cao, H , Lu, H , Zhao, G, et al. La detección de circovirus canino en Guangxi, China. Virus Res. (2019) 259:85–9. doi: 10.1016/j.virusres.2018.10.021
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Cheung, AK . Circovirus porcino: transcripción y replicación del ADN. Virus Res. (2012) 164:46–53. doi: 10.1016/j.virusres.2011.10.012
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Faurez, F , Dory, D , Grasland, B , y Jestin, A . Replicación de circovirus porcinos. Virol J. (2009) 6:60. doi: 10.1186/1743-422X-6-60
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Rodríguez-Carino, C , Sánchez-Chardi, A , y Segales, J . Inmunolocalización subcelular del circovirus porcino tipo 2 (PCV2) en ganglios linfáticos de cerdos con síndrome de desgaste multisistémico post-destete (PMWS). J Comp Pathol. (2010) 142:291–9. doi: 10.1016/j.jcpa.2009.12.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Caddy, S.L. . Nuevos virus asociados con gastroenteritis canina. Veterinario J. (2018) 232:57–64. doi: 10.1016/j.tvjl.2017.12.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Pesavento, PA , y Murphy, BG . Enfermedades infecciosas comunes y emergentes en el refugio de animales. Veterinario Pathol. (2014) 51:478–91. doi: 10.1177/0300985813511129
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Cruz, TF , Batista, TN , Vieira, EM , Portela, LMF , Baccarin, AM , Gradiz, JJ, et al. Caracterización genómica del circovirus canino detectado en un perro con gastroenteritis hemorrágica intermitente en Brasil. Ciência Rural. (2020) 50:e20190909. doi: 10.1590/0103-8478cr20190909
17. Misinzo, G , Delputte, PL , Lefebvre, DJ , y Nauwynck, HJ . Aumento del rendimiento de circovirus-2 porcino mediante un tratamiento combinado de células PK-15 con interferón-gamma e inhibidores de la acidificación del sistema endosómico-lisosomal. Arco Virol. (2008) 153:337–42. DOI: 10.1007/S00705-007-1092-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Chen, H-CG , Kuo, T-Y , Yang, Y-C , Wu, C-C , y Lai, S-S . Subclon altamente permisivo de la línea celular de riñón porcino para la producción de circovirus porcino tipo 2. J Virol Métodos. (2013) 187:380–3. doi: 10.1016/j.jviromet.2012.11.013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Hosono, S , Shiokawa, M , Kobayashi, T , Fukusho, A , y Aoki, H . El circovirus porcino tipo 2 induce un fuerte efecto citopático en la línea celular de cultivo libre de suero CPK-NS. J Virol Métodos. (2019) 273:113706. doi: 10.1016/j.jviromet.2019.113706
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Niu, L , Wang, Z , Zhao, L , Wang, Y , Cui, X , Shi, Y, et al. Detección y caracterización molecular del circovirus canino circulando en el noreste de China durante 2014-2016. Arco Virol. (2020) 165:137–43. DOI: 10.1007/S00705-019-04433-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Kotsias, F , Bucafusco, D , Núñez, DA , Lago Borisovsky, LA , Rodríguez, M , y Bratanich, AC . Caracterización genómica del circovirus canino asociado a enfermedad mortal en perros en Sudamérica. PLoS One. (2019) 14:E0218735. doi: 10.1371/journal.pone.0218735
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Giraldo-Ramírez, S , Rendón-Marín, S , Vargas-Bermúdez, DS , Jaime, J , y Ruiz-Saenz, J . Primera detección y análisis genómico completo del circovirus canino en perros infectados con CPV-2 en Colombia. Sudamérica Sci Rep. (2020) 10:17579. DOI: 10.1038/S41598-020-74630-8
23. Urbani, L , Tryland, M , Ehrich, D , Fuglei, E , Battilani, M , y Balboni, A . Origen antiguo y segregación genética del circovirus canino que infecta zorros árticos (Vulpes lagopus) en Svalbard y zorros rojos (Vulpes vulpes) en el norte de Noruega. Transbound Emerg Dis. (2021) 68:1283–93. doi: 10.1111/tbed.13783
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Beikpour, F , Ndiana, LA , Sazmand, A , Capozza, P , Nemati, F , Pellegrini, F, et al. Detección y caracterización genómica del Circovirus canino en Irán. Animales. (2022) 12:507. doi: 10.3390/ani12040507
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Faraji, R , Sadeghi, M , Mozhgani, S-H , Vasinioti, V , Ndiana, LA , Desario, C, et al. Detección de circovirus canino en perros infectados con parvovirus canino. Acta Trop. (2022) 235:106646. doi: 10.1016/j.actatropica.2022.106646
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Turan, T , e Işıdan, H . Caracterización molecular de astrovirus canino, vesivirus y circovirus, aislados de perros diarreicos en Turquía. Iraní J vet res. (2020) 21:172–9.
27. Wang, L , Li, Y , Guo, Z , Yi, Y , Zhang, H , Shangguan, H, et al. Cambios genéticos y análisis evolutivo del circovirus canino. Arco Virol. (2021) 166:2235–47. doi: 10.1007/s00705-021-05125-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Anderson, A , Hartmann, K , Leutenegger, CM , Proksch, AL , Mueller, RS , y Unterer, S . Papel del circovirus canino en perros con diarrea hemorrágica aguda. Veterinario Rec. (2017) 180:542. DOI: 10.1136/VR.103926
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Gentil, M , Gruber, AD , y Müller, E . Nachweishäufigkeit von dog circovirus bei gesunden und an Durchfall erkrankten Hunden. Tierarztl prax ausg k klientiere heimtiere. (2017) 45:89–94. doi: 10.15654/TPK-160701
30. Tuong, Nuevo México , Piewbang, C , Rungsipipat, A , y Techangamsuwan, S . Detección y caracterización molecular de dos genotipos de circovirus caninos cocirculantes en Vietnam. Veterinario Q. (2021) 41:232–41. doi: 10.1080/01652176.2021.1967511
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Wang, Z , Shi, Y , Wang, Y , Zhao, L , Cui, X , Wen, S, et al. Detección de anticuerpos contra el circovirus canino en caninos infectados de forma natural y experimental mediante ensayo inmunoabsorbente ligado a enzimas de la cápside recombinante. Front Vet Sci. (2020) 7:294. DOI: 10.3389/FVETS.2020.00294
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Dowgier, G , Lorusso, E , Decaro, N , Desario, C , Mari, V , Lucente, MS, et al. Un estudio molecular para enteropatógenos virales seleccionados reveló un papel limitado del circovirus canino en el desarrollo de gastroenteritis aguda canina. Veterinario Microbiol. (2017) 204:54–8. doi: 10.1016/j.vetmic.2017.04.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Van Kruiningen, HJ , Heishima, M , Kerr, KM , Garmendia, AE , Helal, Z , y Smyth, JA . Enteritis hemorrágica circoviral canina en un perro en Connecticut. J Vet Diagn Investig. (2019) 31:732–6. DOI: 10.1177/1040638719863102
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Bexton, S , Wiersma, LC , Getu, S , van Run, PR , Verjans, GM , Schipper, D, et al. Detección de Circovirus en zorros con meningoencefalitis, Reino Unido, 2009-2013. Emerg infectar dis. (2015) 21:1205–8. DOI: 10.3201/EID2107.150228
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. De Arcangeli, S , Balboni, A , Kaehler, E , Urbani, L , Verín, R , y Battilani, M . Caracterización genómica del Circovirus canino detectado en zorros rojos (Vulpes vulpes) de Italia mediante un nuevo ensayo de PCR en tiempo real. J Wildl Dis. (2020) 56:239–42. doi: 10.7589/2018-11-270
36. Ouyang, T , Zhang, X , Liu, X , y Ren, L . Coinfección de cerdos con Circovirus porcino tipo 2 y otros virus porcinos. Virus. (2019) 11:185. DOI: 10.3390/v11020185
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Liu, Y , Gong, Q-L , Nie, L-B , Wang, Q , Ge, G-Y , Li, D-L, et al. Prevalencia del circovirus porcino 2 en toda China en 2015-2019: una revisión sistemática y metanálisis. Microb Pathog. (2020) 149:104490. doi: 10.1016/j.micpath.2020.104490
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Segales, J . Infecciones por circovirus porcino tipo 2 (PCV2): signos clínicos, patología y diagnóstico de laboratorio. Virus Res. (2012) 164:10–9. doi: 10.1016/j.virusres.2011.10.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Tischer, I , Gelderblom, H , Vettermann, W , y Koch, MA . Un virus porcino muy pequeño con ADN monocatenario circular. Naturaleza. (1982) 295:64–6. DOI: 10.1038/295064A0
40. Tischer, I , Rasch, R , y Tochtermann, G . Caracterización de partículas similares a papovavirus y picornavirus en líneas celulares permanentes de riñón de cerdo. Zentralbl Bakteriol Orig A. (1974) 226:153–67.
41. Meng, XJ . Circovirus porcino tipo 2 (PCV2): patogénesis e interacción con el sistema inmunitario. Annu Rev Anim Biosci. (2013) 1:43–64. doi: 10.1146/annurev-animal-031412-103720
42. Palinski, R , Piñeyro, P , Shang, P , Yuan, F , Guo, R , Fang, Y, et al. Un nuevo circovirus porcino lejanamente relacionado con circovirus conocidos se asocia con dermatitis porcina y síndrome de nefropatía y fracaso reproductivo. J Virol. (2017) 91:E01879–16. doi: 10.1128/JVI.01879-16
43. Zhang, H-H , Hu, W-Q , Li, J-Y , Liu, T-N , Zhou, J-Y , Opriessnig, T, et al. Nuevas especies de circovirus identificadas en cerdos de granja designadas como circovirus porcino 4, provincia de Hunan, China. Transbound Emerg Dis. (2020) 67:1057–61. doi: 10.1111/tbed.13446
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Ren, L , Chen, X , y Ouyang, H . Interacciones del circovirus porcino 2 con sus huéspedes. Genes de virus. (2016) 52:437–44. DOI: 10.1007/S11262-016-1326-X
45. Herbst, W , y Willems, H . Detección de partículas de virus parecidas a secuencias de circovirus y circovirus porcino 2a (PCV2a) en heces de perros. Res Vet Sci. (2017) 115:51–3. doi: 10.1016/j.rvsc.2017.01.014
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Canción, T , Hao, J , Zhang, R , Tang, M , Li, W , Hui, W, et al. Primera detección y análisis filogenético del circovirus porcino tipo 2 en perros mapaches. BMC Vet Res. (2019) 15:107. DOI: 10.1186/S12917-019-1856-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Sol, W , Wang, W , Xin, J , Cao, L , Zhuang, X , Zhang, C, et al. Una investigación epidemiológica de la infección por circovirus porcino 3 en perros en la provincia de Guangxi de 2015 a 2017. China Virus Res. (2019) 270:197663. doi: 10.1016/j.virusres.2019.197663
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Zhang, J , Liu, Z , Zou, Y , Zhang, N , Wang, D , Tu, D, et al. Primera detección molecular de circovirus porcino tipo 3 en perros en China. Genes de virus. (2018) 54:140–4. DOI: 10.1007/S11262-017-1509-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Ellis, J , Clark, E , Haines, D , Oeste, K , Cracovia, S , Kennedy, S, et al. Circovirus-2 porcino e infecciones concurrentes en el campo. Veterinario Microbiol. (2004) 98:159–63. doi: 10.1016/j.vetmic.2003.10.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cita: Gomez-Betancur D, Vargas-Bermudez DS, Giraldo-Ramírez S, Jaime J y Ruiz-Saenz J (2023) Circovirus canino: ¿un virus de enteritis no diagnosticado emergente o endémico? Frente. Vet. Sci. 10:1150636. doi: 10.3389/fvets.2023.1150636
Recibido: 24 de enero de 2023; Aprobado: 28 de marzo de 2023;
Publicado: 17 abril 2023.
Editado por:
Mrigendra Rajput, Universidad de Dayton, Estados Unidos
Revisado por:
Tereza Cristina Cardoso, Universidade Estadual de São Paulo, Brasil
Olusegun A. Fagbohun, Universidad de Ibadan, Nigeria
Copyright © 2023 Gomez-Betancur, Vargas-Bermudez, Giraldo-Ramírez, Jaime y Ruiz-Saenz. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Julián Ruiz-Sáenz, julian.ruizs@campusucc.edu.co
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía