Construcción y caracterización de un sistema de genética inversa del virus de la parainfluenza bovina
Construcción y caracterización de un sistema de genética inversa del virus de la parainfluenza bovina tipo 3c como herramienta para el cribado rápido de antivirales in vitro
 Yu Han1
Yu Han1  Kejia Lu1
Kejia Lu1  Riteng Zhang1
Riteng Zhang1  Xi Wei1
Xi Wei1  Hanwei Guo1
Hanwei Guo1  Lina Tong1,2
Lina Tong1,2  Xinglong Wag1
Xinglong Wag1  Sa Xiao1
Sa Xiao1  Haijin Liu1*
Haijin Liu1*  Zengqi Yang1*
Zengqi Yang1*- 1Facultad de Medicina Veterinaria, Universidad Northwest A&F, Yangling, China
- 2Facultad de Agricultura y Ganadería, Universidad de Qinghai, Xining, China
El virus de la parainfluenza bovina tipo 3 (BPIV3) es un patógeno clave asociado con el complejo de enfermedades respiratorias bovinas (BRDC). Sin embargo, sus mecanismos patogénicos específicos no se han dilucidado por completo. La genética inversa proporciona un método útil para comprender el mecanismo patogénico de BPIV3. Para garantizar la funcionalidad de las plataformas de rescate, primero construimos un sistema de minigenoma (MG) de BPIV3 utilizando un sistema de 5 plásmidos en esta investigación. A continuación, se obtuvo un clon de infección completa de BPIV3 de la cepa SX-2021 y se emplearon diferentes métodos para identificar el virus rescatado. Además, recuperamos un BPIV3 recombinante utilizando el sistema de genética inversa que podría expresar la proteína de fluorescencia verde mejorada (eGFP). A través de los ensayos de curva de crecimiento, se encontró que la capacidad de replicación de rBPIV3-SX-EGFP era similar a la del virus parental. Posteriormente, se utilizó el rBPIV3-SX-EGFP para determinar la actividad antiviral de la ribavirina. Los resultados mostraron que la ribavirina tenía un efecto anti-BPIV3 en las células MDBK. En conclusión, el desarrollo exitoso de un sistema genético inverso para la cepa SX-2021 sienta las bases para futuros estudios sobre BPIV3, incluidas investigaciones sobre su mecanismo patogénico, función génica y propiedades de detección antiviral.
1 Introducción
El BPIV3 es reconocido como un patógeno importante que contribuye a la BRDC, induciendo un espectro de síntomas respiratorios de leves a graves (1, 2). BPIV3 es un virus de ARN de cadena negativa no segmentado y con envoltura que es miembro del género Respirovirus dentro de la familia Paramyxovirdae (2). En los últimos años, ha habido un número creciente de brotes de enfermedades respiratorias inducidas por BPIV3 en el ganado, lo que ha provocado pérdidas financieras sustanciales dentro de la industria ganadera mundial (3-5). BPIV3 se ha clasificado en tres genotipos, y el primer informe de BPIV3 genotipo C como un nuevo genotipo se identificó en China (6). Sin embargo, la BPIV3c carece de una terapia reconocida y su mecanismo patogénico sigue siendo difícil de alcanzar.
Hasta la fecha, solo se han reportado estudios limitados sobre la genética inversa del genotipo A de BPIV3 (7, 8). Por lo tanto, es crucial establecer un sistema de genética inversa para BPIV3c para investigar los mecanismos patogénicos y facilitar el desarrollo de una vacuna de virus vivo modificado. Los sistemas de minigenoma (MG), también conocidos como tecnología de mini replicón, se utilizan comúnmente para explorar el ciclo de vida de los virus dentro de la familia Paramyxoviridae (9-11). Anteriormente, los virus reporteros se han empleado eficazmente para examinar la fisiopatología de los paramixovirus y detectar medicamentos antivirales con alta sensibilidad. La ribavirina, un fármaco antiviral de amplio espectro, se utiliza habitualmente en pruebas antivirales (12).
En este estudio, desarrollamos con éxito el primer sistema genético inverso para el genotipo C de BPIV3 utilizando un sistema de 5 plásmidos. Además, se empleó el rBPIV3-SX-EGFP como método de cribado de antivirales in vitro.
2 Materiales y métodos
2.1 Células, virus, plásmidos y anticuerpos
Las células MDBK y BHK-21 se cultivaron en el medio de Eagle modificado de Dulbecco (DMEM) (Gibco, Carlsbad, CA), suplementado con suero fetal bovino (FBS) al 10% (Gibco, Carlsbad, CA), a 37 °C con 5% de CO2. La cepa BPIV3 se aisló a partir de una muestra de tejido pulmonar BPIV3 positivo, designada como BPIV3/SX/2021 (GenBank: ON804787). Los plásmidos pCMV y pCI-neo se utilizaron para construir el clon de ADNc rBPIV3-SX y tres plásmidos auxiliares, respectivamente. El plásmido pGAGGS-T7, que contiene ARN polimerasa T7, fue construido en nuestro laboratorio. En nuestro laboratorio se prepararon anticuerpos policlonales frente a NP y la especificidad se ha comprobado en nuestro estudio previo (13). Se compró un anticuerpo anti-EGFP (ab184601) a Abcam. Se compró IgG anti-ratón de cabra conjugada con HRP a Invitrogen. Se compró IgG (H + L) antiratón de cabra con la etiqueta FITC a Beyotime.
2.2 Construcción de plásmidos
Se diseñó un conjunto de cebadores para construir un sistema MG para BPIV3 basado en las regiones genómicas conservadas de la región viral 5′ no traducida (5′ UTR), EGFP y la viral 3′ UTR. Los tres productos de PCR se ensamblaron por superposición y se insertaron en la orientación antisentido entre las secuencias promotoras y terminadoras de T7, lo que dio como resultado una construcción denominada pT7-MG.
Para construir un clon de ADNc de longitud completa de la cepa SX-2021, se diseñó un conjunto de cebadores para amplificar el genoma del ARN viral mediante la reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR). Los productos de PCR se secuenciaron mediante secuenciación Sanger con tres pruebas paralelas independientes. Se ensamblaron seis fragmentos separados (de la A a la F) utilizando sitios enzimáticos de restricción únicos. Se insertó un promotor T7 aguas arriba del fragmento A y tres residuos G que precedieron a la secuencia promotora T7. El fragmento F contenía una ribozima parcial de HDV de 24 nucleótidos (nt) (Tabla suplementaria S1). Además, se creó un marcador molecular para diferenciar el virus rescatado del virus parental. Los cebadores correspondientes a los fragmentos A y B fueron modificados para eliminar el sitio NcoI innecesario. Los fragmentos de (A-C) y (D-F) se insertaron, respectivamente, en el plásmido pCMV; Los plásmidos pCMV (A-C) y pCMV (D-F) fueron digeridos por NcoI y RsrII. A continuación, se unieron estos dos fragmentos y se ensambló el ADNc BPIV3 de longitud completa, denominado pBPIV3-SX (Figura 1A). Los ORF NP, P y L se subclonaron por separado en el plásmido pCI-neo para construir los tres plásmidos auxiliares, pCI-NP, pCI-P y pCI-L.
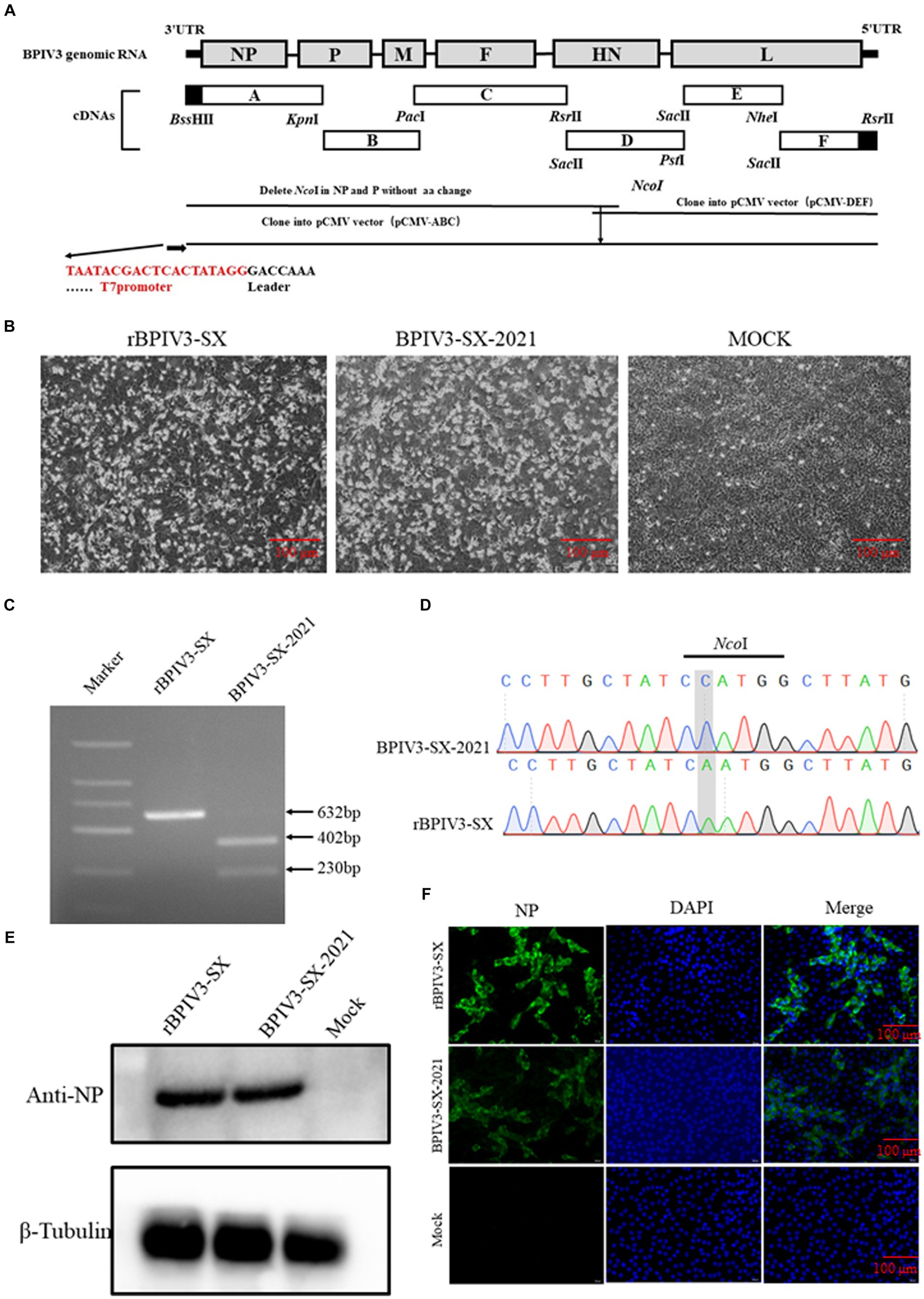 Figura 1. Identificación del ensayo del virus rBPIV3-SX rescatado. (A) Representación esquemática que muestra la construcción de pBPIV3-SX. (B) Efecto citopático específico (CPE) de BPIV3 del virus rescatado. Los cultivos celulares de MDBK se recolectaron a las 48 h después de la infección con el virus de rescate y parental. (C) Identificación del marcador genético en el virus rescatado. El sitio de la enzima de restricción NcoI se mutó en el plásmido recombinante para crear un marcador genético que distinguiera el virus rescatado del virus parental. (D) La mutación del marcador genético se identificó por secuenciación. (E) Expresión de NP en células infectadas por rBPIV3-SX. Las células MDBK infectadas con el virus de rescate y parental se detectaron mediante Western blot utilizando el anticuerpo policlonal anti-BPIV3 NP a las 24 h. (F) Análisis de inmunofluorescencia del virus rescatado. Las células MDBK se infectaron con el virus de rescate y parental, y las células infectadas se tiñeron con el anticuerpo policlonal anti-BPIV3 NP.
Figura 1. Identificación del ensayo del virus rBPIV3-SX rescatado. (A) Representación esquemática que muestra la construcción de pBPIV3-SX. (B) Efecto citopático específico (CPE) de BPIV3 del virus rescatado. Los cultivos celulares de MDBK se recolectaron a las 48 h después de la infección con el virus de rescate y parental. (C) Identificación del marcador genético en el virus rescatado. El sitio de la enzima de restricción NcoI se mutó en el plásmido recombinante para crear un marcador genético que distinguiera el virus rescatado del virus parental. (D) La mutación del marcador genético se identificó por secuenciación. (E) Expresión de NP en células infectadas por rBPIV3-SX. Las células MDBK infectadas con el virus de rescate y parental se detectaron mediante Western blot utilizando el anticuerpo policlonal anti-BPIV3 NP a las 24 h. (F) Análisis de inmunofluorescencia del virus rescatado. Las células MDBK se infectaron con el virus de rescate y parental, y las células infectadas se tiñeron con el anticuerpo policlonal anti-BPIV3 NP.
Para construir un virus de rescate que contenía eGFP, se utilizó un plásmido que contenía la secuencia completa de BPIV3, con una secuencia de fusión de eGFP. La secuencia de fusión, flanqueada por los sitios BssHII y PacI, se subclonó en el pBPIV3-SX, dando como resultado el plásmido denominado pBPIV3-SX-EGFP.
2.3 Transfección y rescate de virus recombinantes
Las células BHK-21 se sembraron en una placa de 12 pocillos y se transfectaron con un plásmido del minigenoma o clon de ADNc de longitud completa, junto con pCI-NP, pCI-P, pCI-L y pCAGGS-T7 en una proporción de 0,5 μg: 0,5 μg: 0,1 μg: 0,05 μg: 0,35 μg, utilizando el reactivo TurboFect™ (Invitrogen, Carlsbad, CA). La replicación de este minigenoma se observó en varios puntos de tiempo bajo un microscopio fluorescente. Después de 60 h después de la transfección con el clon de ADNc de longitud completa, se cosechó el sobrenadante celular de las células BHK-21 y se transfirió a las células MDBK. Los virus de rescate se recolectaron entre 48 y 60 h después de la infección, cuando se observó un CPE significativo. Además, los virus de rescate se sometieron a 20 pases en serie en células MDBK, utilizando una multiplicidad de infección (MOI) de 0,1. El virus se adsorbió en monocapas de células confluentes en placas celulares de 60 mm durante 1 h a 37 °C con balanceo frecuente. Después de la adsorción, se retiró el inóculo, las células se lavaron con DMEM, se agregaron 4 mL de medios de mantenimiento y las placas se incubaron a 37 °C. El medio de cada pocillo se cosechó a 72 hpi y se almacenó en alícuotas a -80 °C. Después de cada paso, la cosecha viral fue cuantificada por TCID50 ensayo en células MDBK y diluido para producir un MOI de 0,1 para cada paso posterior.
2.4 Identificación de virus recombinantes
Para determinar el marcador molecular (mutación C341 a A341 en el sitio NcoI) en el virus rescatado, rBPIV3/SX, SE REALIZÓ RT-PCR en la región genómica que contiene la mutación específica. Para ello, se extrajo ARN viral del pasaje 5 del virus rBPIV3-SX utilizando el reactivo TRIzol (Takara) y se transcribió inversamente en ADNc utilizando un kit RT StarScript II (Genstar, Pekín, China). Se empleó RT-PCR para amplificar la región genómica viral relevante utilizando el par de cebadores, Nco-F/Nco-R (Tabla suplementaria S1). El producto de la PCR se purificó en gel, se digirió con NcoI y se secuenció el ADN para confirmar la presencia de la mutación C341 a A341.
2.5 Análisis de Western blot
Las células MDBK se sembraron en una placa de 12 pocillos y se infectaron con BPIV3-SX-2021, rBPIV3-SX, rBPIV3-SX-EGFP o se infectaron simuladamente con un MOI de 0,1. Los lisados celulares se cosecharon utilizando un tampón de lisis de 160 μL/pocillo a 24 hpi, y el lisado celular se desnaturalizó a 95 °C durante 10 min en un tampón de carga 5x (Bio-Rad, Hercules, CA). Las proteínas se analizaron en un SDS-PAGE al 10% y luego se transfirieron a una membrana de PVDF. La membrana se bloqueó con leche descremada al 5% durante 2 h a temperatura ambiente. Para identificar la expresión de BPIV3 NP o eGFP, la membrana se incubó durante la noche a 4°C con anticuerpos primarios. A continuación, la membrana se lavó tres veces con TBST, seguida de una incubación con anticuerpos secundarios a temperatura ambiente durante 1 h. Las proteínas diana se analizaron con BeyoECL Plus (Beyotime, Pekín, China).
2.6 Ensayo de inmunofluorescencia
Las células MDBK se sembraron en una placa de 24 pocillos y se infectaron con BPIV3-SX-2021, rBPIV3-SX y rBPIV3-SX-EGFP con un MOI de 0,1 o se infectaron simuladamente. A 24 hpi, las monocapas celulares se fijaron con paraformaldehído al 4% a temperatura ambiente durante 30 min y luego se bloquearon con PBS que contenía albúmina sérica bovina (BSA) al 1% durante 30 min y luego se permeabilizaron con Triton X-100 al 0,1% a los 5 min. Las monocapas celulares se incubaron con pAb contra NP durante 1 h y luego se lavaron con PBS tres veces. Las monocapas celulares se incubaron durante 1 h con un anticuerpo secundario. Después de lavar tres veces, las células fueron analizadas bajo un microscopio fluorescente (IX73; OLIMPO, Tokio, Japón).
2.7 TCID50 Ensayo y cinética de crecimiento del virus
Las células MDBK se sembraron en una placa de 6 pocillos y se infectaron con BPIV3-SX-2021, rBPIV3-SX y rBPIV3-SX-EGFP a un MOI de 0,1. Las placas se incubaron a 37°C durante 1 h, y el sobrenadante del cultivo celular se retiró y se reemplazó con DMEM suplementado que contenía 2% de FBS. Los sobrenadantes de cultivo celular se recolectaron a 12, 24, 36, 48, 60 y 72 hpi, y los títulos virales se midieron utilizando el TCID50 ensayo en células MDBK.
2.8 Ensayos anti-rBPIV3-SX de ribavirina
Para determinar el efecto de la concentración tratada con ribavirina sobre la viabilidad de las células MDBK, se empleó un ensayo CCK-8 (Solarbio, CA1210). Las células MDBK se sembraron en una placa de 96 pocillos, y los sobrenadantes se suplementaron con DMEM que contenía ribavirina en varias dosis. Después de incubar las células tratadas con ribavirina durante 24 h, se añadió la solución de CCK-8 (10 μL/pocillo) a cada pocillo. La placa se examinó utilizando un lector de microplacas a 450 nm después de 4 h de incubación.
Las células MDBK se sembraron en una placa de 12 pocillos para evaluar las actividades antivirales de la ribavirina en varias dosis. Las células se infectaron con rBPIV3-SX-EGFP a un MOI de 1 durante 1 h a 37 °C. Los sobrenadantes se reemplazaron y se agregaron al DMEM que contenía ribavirina en varias dosis. Se utilizó un microscopio fluorescente para examinar las células tratadas con ribavirina después de 24 h. Los sobrenadantes se recolectaron para la determinación del título viral utilizando el TCID50 ensayo de rBPIV3-SX-EGFP.
3 Resultados
3.1 Construcción de un sistema minigenómico de BPIV3
Además de los virus reporteros, un sistema de minigenoma es una herramienta poderosa para los estudios virales. En consecuencia, construimos un sistema de minigenoma, con las regiones no codificantes 5′ y 3′ del genoma BPIV3 constituyendo la secuencia de remolque y líder. Las estructuras genómicas de dos genes estaban flanqueadas por secuencias de inicio de genes (GS) y finales de genes (GE). El plásmido reportero contiene el gen reportero eGFP insertado entre las regiones líder y remolque y está flanqueado por el promotor T7 y la ribozima del VHD. La secuencia MG de BPIV3 se mostró esquemáticamente en la Figura 2A. Además del plásmido MG, se utilizaron los tres plásmidos auxiliares en el sistema del minigenoma (Figura 2B). El plásmido (pT7-MG) y el plásmido pCAGGS-T7, junto con los tres plásmidos auxiliares, se transfectaron conjuntamente en células BHK-21. La replicación de este minigenoma (MG) y la actividad de eGFP se evaluaron en varios momentos. A 24 hpi, unas pocas células exhibieron fluorescencia verde, y la proporción de dichas células aumentó con el tiempo. La actividad de eGFP aumentó sustancialmente de 24 a 72 h después de la transfección, lo que indica que nuestro sistema MG se replicó de manera eficiente (Figura 2C).
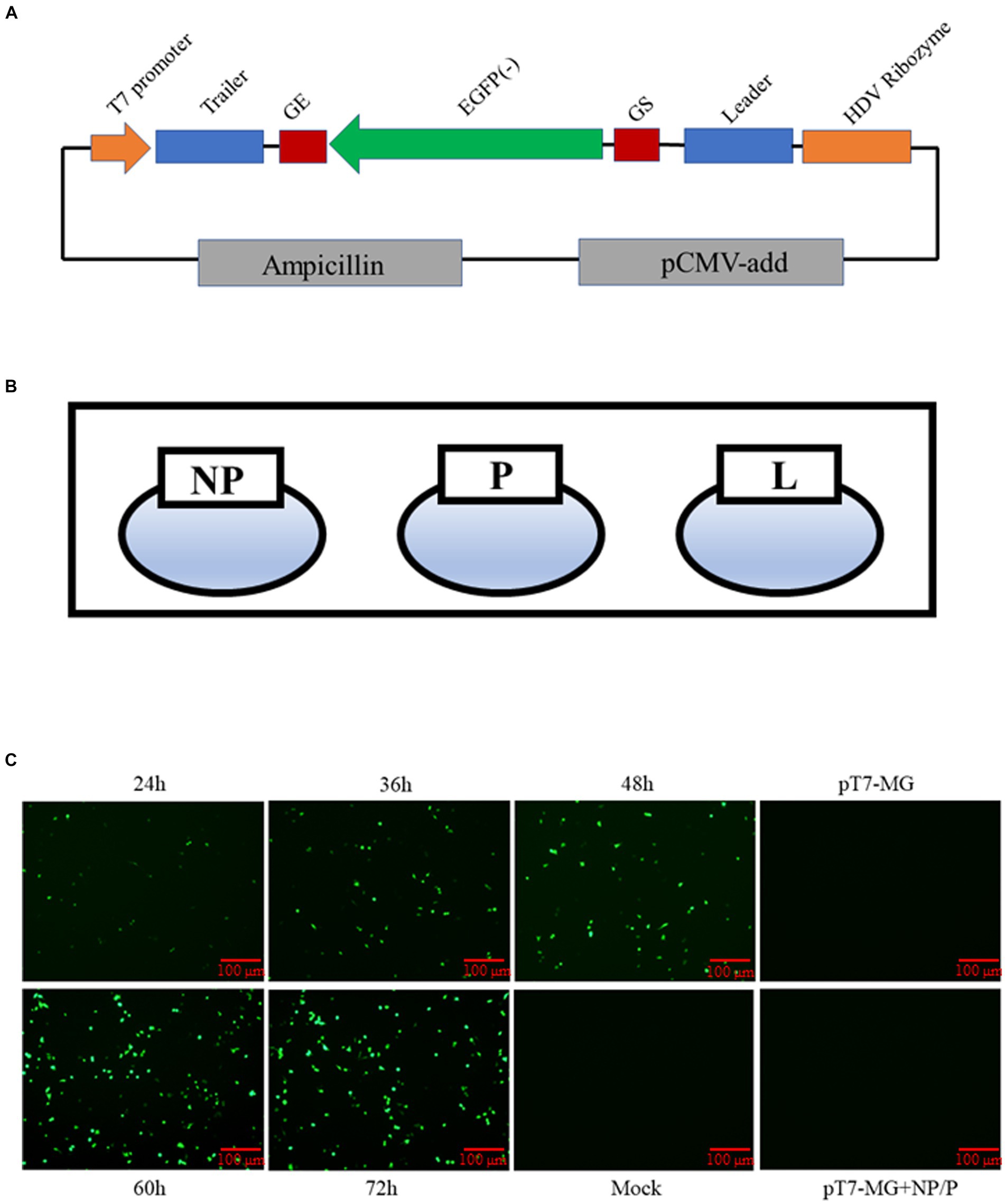 Figura 2. Construcción del sistema minigenómico BPIV3. (A) Diagrama que muestra la estructura del sistema del minigenoma BPIV3. Este plásmido reportero contiene el gen reportero eGFP insertado entre las regiones líder y remolque y luego flanqueado por el promotor T7 y la ribozima del VHD. (B) Mapa esquemático de los tres plásmidos auxiliares para el virus de rescate. (C) Células BHK-21 cotransfectadas con cinco plásmidos a las 24 h, 36 h, 48 h, 60 h y 72 h. Los niveles de expresión de eGFP se determinaron bajo el microscopio de fluorescencia.
Figura 2. Construcción del sistema minigenómico BPIV3. (A) Diagrama que muestra la estructura del sistema del minigenoma BPIV3. Este plásmido reportero contiene el gen reportero eGFP insertado entre las regiones líder y remolque y luego flanqueado por el promotor T7 y la ribozima del VHD. (B) Mapa esquemático de los tres plásmidos auxiliares para el virus de rescate. (C) Células BHK-21 cotransfectadas con cinco plásmidos a las 24 h, 36 h, 48 h, 60 h y 72 h. Los niveles de expresión de eGFP se determinaron bajo el microscopio de fluorescencia.
3.2 Rescate e identificación de rBPIV3
Para rescatar el virus, se construyó el plásmido clon de infección de longitud completa de rBPIV3-SX y se cotransfectó en células BHK-21 con los plásmidos auxiliares. Después de 72 h de transfección, los sobrenadantes de las células transfectadas se recolectaron y posteriormente se transfirieron a las células MDBK. Simultáneamente, se observaron CPE típicas en monocapas celulares infectadas por virus a las 48 h (Figura 1B). El ARN total se extrajo de rBPIV3-SX o del virus parental, y el fragmento que contenía el marcador molecular NcoI en rBPIV3-SX se analizó mediante RT-PCR utilizando el cebador Nco-F/R. Los productos de PCR fueron purificados en gel y digeridos por NcoI. El virus parental produjo un fragmento de 402 pb y otro de 230 pb después de la digestión de NcoI, mientras que el virus rescatado no pudo lograrlo (Figura 1C). Además, se secuenciaron los productos de PCR para confirmar la presencia de la mutación en el sitio NcoI (Figura 1D). La expresión de la proteína NP en células infectadas con rBPIV3-SX se analizó mediante Western blot. El resultado mostró que la proteína NP (69 kDa) se expresó en células infectadas con el rBPIV3-SX (Figura 1E). La expresión de BPIV3 NP se evaluó además utilizando un pAb contra la proteína NP por IFA. La monocapa celular infectada por el virus era visible en fluorescencia verde brillante, pero no en las células simuladas (Figura 1F).
3.3 Construcción de clones de infección que contienen un gen reportero
Además, generamos un clon infeccioso de longitud completa de BPIV3 que contiene un reportero de eGFP. Se ha demostrado que el sitio intergénico P-M es preferible para la expresión óptima de proteínas extrañas (14). Utilizando el clon de ADNc rBPIV3 (SX-2021) como columna vertebral del clon de ADNc rBPIV3-SX-EGFP, se insertó un gen reportero de EGFP entre los genes BPIV3 P y M del clon de ADNc rBPIV3-SX utilizando tecnología de superposición (Figura 3A). Para rescatar el rBPIV3-SX-EGFP, cotransfectamos el plásmido de longitud completa y los plásmidos auxiliares en células BHK-21. Como se muestra en la Figura 3B, para caracterizar el virus recombinante, la proteína viral NP se confirmó mediante microscopía inmunofluorescente y las proteínas eGFP y NP se expresaron en células infectadas por el virus rBPIV3-SX-EGFP. Para determinar aún más la expresión de las proteínas NP y eGFP utilizando el Western blot, los resultados mostraron que las proteínas NP (69 kDa) y eGFP (27 kDa) se expresaron en células infectadas con rBPIV3-SX-EGFP. Sin embargo, solo la proteína NP se expresó en las células infectadas con el virus parental (Figura 3C). Para evaluar el impacto de la expresión de eGFP en la replicación viral, se compararon la cinética de crecimiento de rBPIV3-SX-EGFP con la de rBPIV3-SX y el virus parental. Los resultados han mostrado que todos los virus mostraron un comportamiento de crecimiento similar durante 72 h (Figura 3D). Para evaluar la estabilidad genética del virus recombinante, rBPIV3-SX-EGFP se sometió a 20 pases seriados en células MDBK. Como se muestra en la Figura 3E, la eGFP se expresó consistentemente a un nivel alto durante los pasajes.
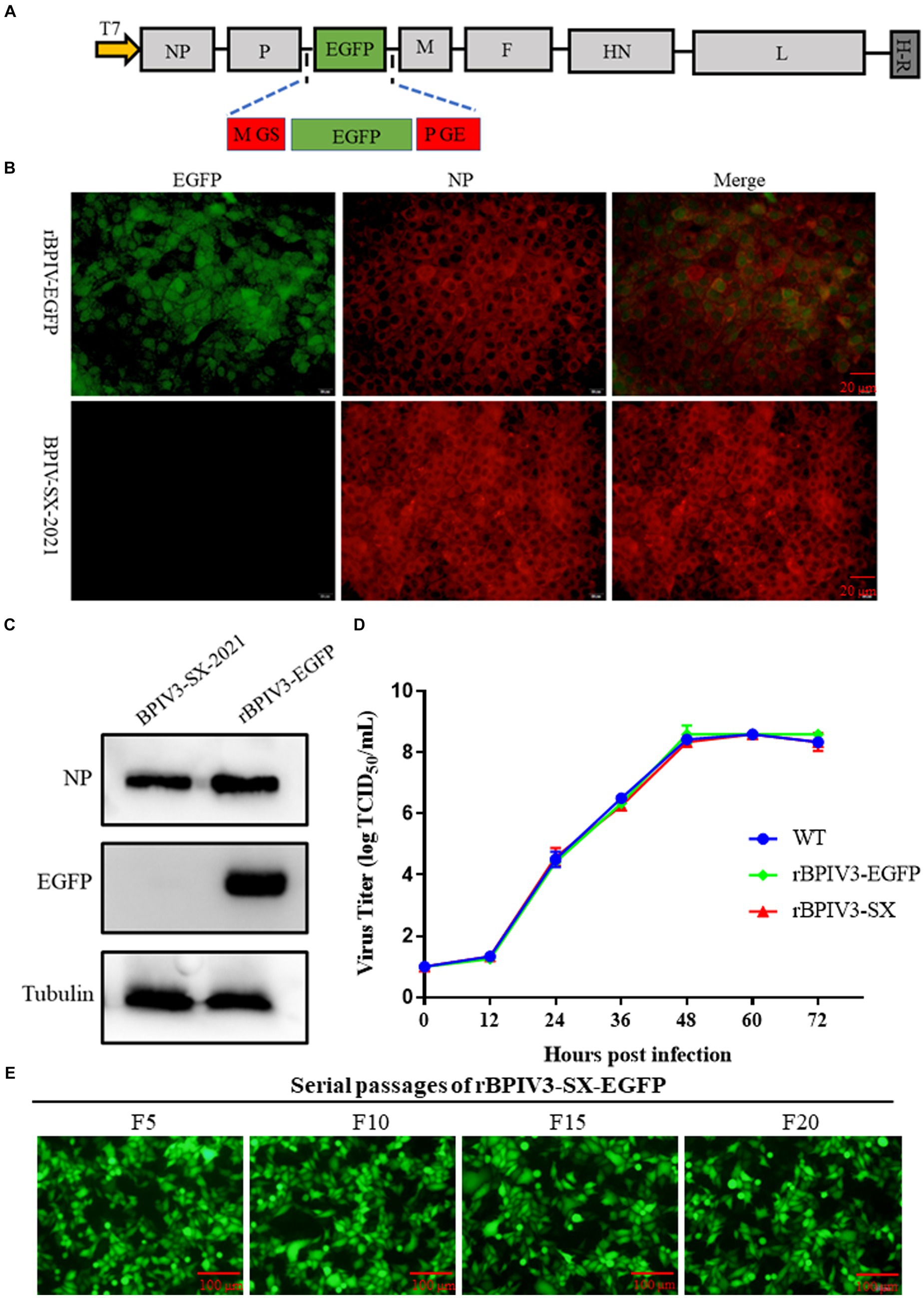 Figura 3. Construcción, identificación y caracterización del virus reportero BPIV3 de longitud completa. (A) Diagrama que muestra la estructura del virus reportero BPIV3 de longitud completa. La H-R significa ribozima HDV. (B) Análisis de inmunofluorescencia del virus rescatado. Las células MDBK estaban infectadas con rBPIV3-SX-EGFP y virus parental; las células infectadas se tiñeron con el anticuerpo policlonal anti-BPIV3 NP. (C) Expresión de NP y eGFP en células infectadas con rBPIV3-SX-EGFP. Las células MDBK infectadas con rBPIV3-SX-EGFP y el virus parental se detectaron mediante Western blot utilizando el anticuerpo policlonal anti-BPIV3 NP y el anticuerpo monoclonal anti-EGFP a las 24 h. (D) La cinética de crecimiento de los virus rescatados. Las células MDBK en placas de seis pocillos se infectaron con rBPIV3-SX, rBPIV3-SX-EGFP y virus parentales. El sobrenadante se cosechó a las 12, 24, 36, 48, 60 y 72 h y se tituló en células MDBK. (E) Pasajes seriados de rBPIV3-SX-EGFP. Las células MDBK se infectaron con diferentes conductos de rBPIV3-SX-EGFP, y la expresión de eGFP se determinó bajo el microscopio de fluorescencia.
Figura 3. Construcción, identificación y caracterización del virus reportero BPIV3 de longitud completa. (A) Diagrama que muestra la estructura del virus reportero BPIV3 de longitud completa. La H-R significa ribozima HDV. (B) Análisis de inmunofluorescencia del virus rescatado. Las células MDBK estaban infectadas con rBPIV3-SX-EGFP y virus parental; las células infectadas se tiñeron con el anticuerpo policlonal anti-BPIV3 NP. (C) Expresión de NP y eGFP en células infectadas con rBPIV3-SX-EGFP. Las células MDBK infectadas con rBPIV3-SX-EGFP y el virus parental se detectaron mediante Western blot utilizando el anticuerpo policlonal anti-BPIV3 NP y el anticuerpo monoclonal anti-EGFP a las 24 h. (D) La cinética de crecimiento de los virus rescatados. Las células MDBK en placas de seis pocillos se infectaron con rBPIV3-SX, rBPIV3-SX-EGFP y virus parentales. El sobrenadante se cosechó a las 12, 24, 36, 48, 60 y 72 h y se tituló en células MDBK. (E) Pasajes seriados de rBPIV3-SX-EGFP. Las células MDBK se infectaron con diferentes conductos de rBPIV3-SX-EGFP, y la expresión de eGFP se determinó bajo el microscopio de fluorescencia.
3.4 Virus reportero para pruebas antivirales
Utilizando el ensayo Cell Counting Kit 8, evaluamos el impacto de la ribavirina en la viabilidad de las células MDBK. Los resultados mostraron que el tratamiento con ribavirina no afectó a la viabilidad de las células (Figura 4A). Las monocapas celulares tratadas con ribavirina se monitorizaron a 24 hpi en un microscopio de fluorescencia de campo de visión seleccionado al azar para evaluar la tendencia a la disminución de la expresión de eGFP. Los resultados mostraron que la fracción de células verdes disminuyó gradualmente con el aumento de la concentración del fármaco en el grupo tratado con ribavirina (Figura 4B). A 24 hpi, todos los cultivos celulares tratados con ribavirina se valoraron utilizando la TCID50 y el título viral disminuyó constantemente con el aumento de la concentración de ribavirina, como se observó bajo el microscopio de fluorescencia (Figura 4C).
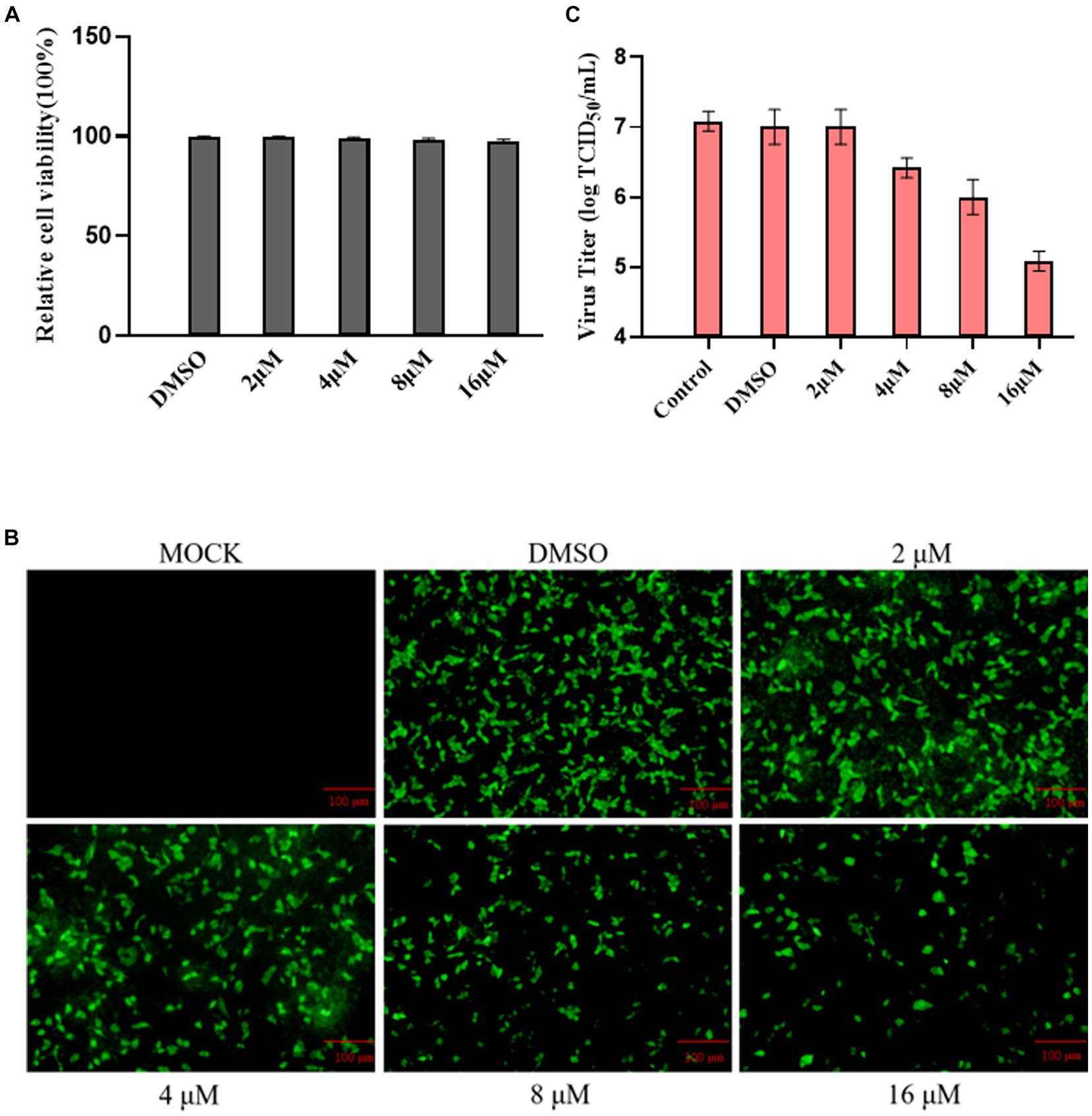 Figura 4. Inhibiciones de ribavirina de la replicación rescatada de rBPIV3-SX-EGFP. (A) El efecto citotóxico del tratamiento con ribavirina en las células MDBK. Las células fueron tratadas con 0, 2, 4, 8 o 16 μM. La viabilidad celular relativa se determinó mediante el ensayo CCK-8. (B) Perfiles de eGFP expresada por rBPIV3-SX-EGFP a las 24 h después de los tratamientos por separado con ribavirina a diferentes concentraciones. (C) El título viral se detectó con diferentes concentraciones de tratamiento con ribavirina mediante el ensayo Reed-Muench.
Figura 4. Inhibiciones de ribavirina de la replicación rescatada de rBPIV3-SX-EGFP. (A) El efecto citotóxico del tratamiento con ribavirina en las células MDBK. Las células fueron tratadas con 0, 2, 4, 8 o 16 μM. La viabilidad celular relativa se determinó mediante el ensayo CCK-8. (B) Perfiles de eGFP expresada por rBPIV3-SX-EGFP a las 24 h después de los tratamientos por separado con ribavirina a diferentes concentraciones. (C) El título viral se detectó con diferentes concentraciones de tratamiento con ribavirina mediante el ensayo Reed-Muench.
4 Discusión
La infección por BPIV3 induce principalmente síntomas respiratorios tanto en bovinos jóvenes como adultos, como tos, fiebre y secreción nasal, particularmente en bovinos de corral de engorde (15, 16). El genotipo C de BPIV3 como un genotipo potencialmente nuevo fue aislado e identificado en China en 2008 (6). Posteriormente, se ha identificado BPIV3c en los principales países productores de ganado, incluidos Estados Unidos, Australia, Japón y Corea. Su presencia ha provocado importantes pérdidas económicas en la industria ganadera mundial (4, 5, 17).
Estudios filogenéticos previos han revelado una diversidad genética significativa entre los diferentes genotipos de BPIV3, con posibles rangos geográficos y temporales (6). Por lo tanto, la construcción de los sistemas genéticos inversos para diferentes cepas virales podría ser útil para evaluar las evaluaciones evolutivas y ecológicas de BPIV3. El sistema de genética inversa para BPIV3 se remonta al año 2000, cuando se estableció como un vector de virus de ARN para expresar y distribuir antígenos extraños, lo que contribuye a la prevención de enfermedades en humanos (7). En este estudio, primero construimos con éxito un sistema genético inverso para la cepa BPIV3c, SX-2021. Para construir el sistema genético inverso, primero desarrollamos un sistema de minigenoma (MG) para probar la utilidad de los plásmidos auxiliares. Además, el sistema MG es una poderosa herramienta comúnmente utilizada para estudiar el ciclo de vida de los miembros de la familia Paramyxoviridae. Además, se ha demostrado que la proporción de plásmidos auxiliares influye en la eficiencia de la expresión génica reportera de MG (18). Sin embargo, el sistema MG, al ser más corto que el genoma completo, tiene limitaciones inherentes que deben reconocerse. Por lo tanto, el rescate de un nuevo virus recombinante ha proporcionado ventajas significativas para estudiar más a fondo la función de los genes virales. Para rescatar el virus recombinante, construimos un clon estable de ADNc de longitud completa que lleva un marcador genético. A continuación, este clon se transfectó conjuntamente con el clon de la infección en células BHK-21 junto con los plásmidos auxiliares. El rBPIV3-SX rescatado y el BPIV3 parental mostraron características idénticas en los ensayos de Western blot e inmunofluorescencia. Para determinar la estabilidad del marcador genético de la rBPIV3-SX rescatada, empleamos el análisis enzimático de restricción y la secuenciación de Sanger.
Una aplicación fundamental del sistema genético inverso consiste en utilizar la columna vertebral viral para expresar genes extraños (18). En un estudio previo con la cepa BN-1, se construyó un virus recombinante que contenía el gen reportero de eGFP y se insertó entre los genes N y P (8). Sin embargo, el virus reportero solo se utilizó para examinar el tropismo y la patogénesis de BPIV3 en hámsters. Estudios previos sobre otros paramixovirus han indicado que la expresión de proteínas extrañas, con diferentes tamaños de inserción, podría afectar la replicación del virus (19-21). En este estudio, insertamos secuencias de genes de proteínas reporteras entre las proteínas P y M en BPIV3, fusionadas inmediatamente con secuencias de inicio de genes (GS) y genes finales (GE) (14, 22). Al comparar la replicación de rBPIV3-SX-EGFP con BPIV3 parental de tipo salvaje, nuestros resultados demostraron que rBPIV3-SX-EGFP exhibió una cinética de crecimiento similar a la del virus parental. Además, la utilización de genes reporteros de la expresión del virus sirve como una herramienta poderosa para la detección y prueba de medicamentos antivirales (23-25). La ribavirina es un derivado de nucleósido de purina con actividad antiviral de amplio espectro. En este estudio, empleamos rBPIV3-SX-EGFP para pruebas de citotoxicidad de fármacos e infección viral para evaluar su potencial en el descubrimiento de nuevos fármacos antivirales y en la realización de cribado de fármacos anti-BPIV3 de alto rendimiento. Estos hallazgos sugieren que el rBPIV3-SX-EGFP es prometedor para descubrir nuevos fármacos antivirales y realizar un cribado de fármacos anti-BPIV3 de alto rendimiento.
En resumen, nuestro éxito en el desarrollo de un sistema de minigenoma y un genoma completo para un BPIV3c emergente representa un logro significativo. Además, rescatamos con éxito un virus recombinante que contenía un gen reportero de eGFP. Esta herramienta será fundamental para realizar pruebas de medicamentos antivirales. Creemos que el sistema del minigenoma, el genoma completo y el virus recombinante con un gen reportero de eGFP pueden contribuir colectivamente a futuras investigaciones sobre la biología, el tropismo tisular y la dinámica de transmisión de BPIV3.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en el artículo/Material complementario.
Contribuciones de los autores
YH: Escritura – borrador original. KL: Investigación, Escritura – borrador original. RZ: Software, Escritura – borrador original. XW: Validación, Escritura – borrador original. HG: Recursos, Escritura – borrador original. LT: Investigación, Escritura – borrador original. XLW: Recursos, Escritura – borrador original. SX: Curación de datos, Escritura – borrador original. NS: Análisis formal, Redacción – borrador original. ZY: Escritura – revisión y edición.
Financiación
El/los autor/es declaran/n que no se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1336663/full#supplementary-material
Referencias
1. Mahony, TJ , McCarthy, FM , Grava, JL , Oeste, L y Young, PL . Construcción y manipulación de un clon infeccioso del genoma del herpesvirus bovino 1 mantenido como cromosoma artificial bacteriano. J Virol. (2002) 76:6660–8. doi: 10.1128/jvi.76.13.6660-6668
2. Horwood, PF , Grava, JL y Mahony, TJ . Identificación de dos genotipos distintos del virus de la parainfluenza bovina tipo 3. J Gen Virol. (2008) 89:1643–8. doi: 10.1099/vir.0.2008/000026-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Konishi, M , Ohkura, T , Shimizu, M , Akiyama, M , Kameyama, K y Takeuchi, K . Secuencia completa del genoma del primer aislado del virus de la parainfluenza bovina tipo 3 genotipo C en Japón. Anuncio del genoma. (2014) 2:e01215–4. doi: 10.1128/genomaA.01215-14
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. OEM, JK , Lee, EY , Lee, KK , Kim, SH , Lee, MH y Hyun, BH . Caracterización molecular de un aislado del virus de la parainfluenza bovina tipo 3 coreano. Microbiol veterinario. (2013) 162:224–7. doi: 10.1016/j.vetmic.2012.10.013
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Neill, JD , Ridpath, JF y Valayudhan, BT . Identificación y caracterización genómica de los virus de la parainfluenza bovina tipo 3 de genotipo B y genotipo C aislados en los Estados Unidos. BMC Vet Res. (2015) 11:112. doi: 10.1186/s12917-015-0431-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Zhu, YM , Shi, HF , Gao, YR , Xin, JQ , Liu, NH , Xiang, WH, et al. Aislamiento y caracterización genética del virus de la parainfluenza bovina tipo 3 de bovinos en China. Microbiol veterinario. (2011) 149:446–51. doi: 10.1016/j.vetmic.2010.11.011
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Haller, AA , Molinero, T , Mitiku, M y Coelingh, K . Expresión de las glicoproteínas de superficie del virus de la parainfluenza humana tipo 3 por el virus de la parainfluenza bovina tipo 3, un nuevo vector vacunal de virus atenuado. J Virol. (2000) 74:11626–35. doi: 10.1128/jvi.74.24.11626-11635
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Ohkura, T , Minakuchi, M , Sagai, M , Kokuho, T , Konishi, M , Kameyama, KI, et al. Infección del tracto respiratorio superior de hámsters por la cepa BN-1 del virus de la parainfluenza bovina tipo 3 que expresa proteína fluorescente verde mejorada. Virología. (2015) 476:134–40. doi: 10.1016/j.virol.2014.12.015
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Minet, C , Yami, M , Egzabhier, B , Gil, P , Picante, F , Brémont, M, et al. Análisis de la secuencia del gen de la polimerasa grande (L) y del remolque de la cepa vacunal del virus de la peste de los pequeños rumiantes Nigeria 75/1: expresión y uso de la proteína L en genética inversa. Virus Res. (2009) 145:9–17. doi: 10.1016/j.virusres.2009.06.002
10. Iwasaki, M , Takeda, M , Shirogane, Y , Nakatsu, Y , Nakamura, T y Yanagi, Y . La proteína de la matriz del virus del sarampión regula la síntesis y el ensamblaje del ARN viral al interactuar con la proteína de la nucleocápside. J Virol. (2009) 83:10374–83. doi: 10.1128/JVI.01056-09
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Blondot, ML , Dubosclard, V , Arreglar, J , Lassoued, S , Aumont-Nicaise, M , Bontems, F, et al. Estructura y análisis funcional del dominio de unión a ARN y fosfoproteína viral de la proteína M2-1 del virus respiratorio sincitial. PLoS Pathog. (2012) 8:e1002734. doi: 10.1371/journal.ppat.1002734
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Graci, JD y Cameron, CE . Mecanismos de acción de la ribavirina frente a distintos virus. Rev Med Virol. (2006) 16:37–48. DOI: 10.1002/rmv.483
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Han, Y , Adam, FEA , Zhang, R , Gao, Y , Lei, J , Lu, K, et al. Aislamiento e identificación de un genotipo C del virus de la parainfluenza bovina tipo 3 y su patogenicidad en cobayas albinas. Transbound Emerg Dis. (2023) 2023:1–10. doi: 10.1155/2023/8854528
14. Carnero, E , Li, W , Borderia, AV , Moltedo, B , Moran, T y García-Sastre, A . Optimización de la expresión de gag del virus de la inmunodeficiencia humana por vectores del virus de la enfermedad de Newcastle para la inducción de potentes respuestas inmunitarias. J Virol. (2009) 83:584–97. doi: 10.1128/JVI.01443-08
15. Autio, T , Pohjanvirta, T , Holopainen, R , Rikula, U , Pentikäinen, J , Huovilainen, A, et al. Etiología de la enfermedad respiratoria en terneros no vacunados y no medicados en rebaños de cría. Microbiol veterinario. (2007) 119:256–65. doi: 10.1016/j.vetmic.2006.10.001
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Snowder, GD , Van Vleck, LD , Cundiff, LV , Bennett, GL , Koohmaraie, M y Dikeman, ME . Enfermedad respiratoria bovina en bovinos de engorde: correlaciones fenotípicas, ambientales y genéticas con los rasgos de crecimiento, canal y palatabilidad muscular longissimus. J Anim Sci. (2007) 85:1885–92. doi: 10.2527/jas.2007-0008
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Kumagai, A , Kanno, T , Kawauchi, K , Tanaka, K , Ishihara, R y Hatama, S . Análisis filogenético y antigénico del virus de la parainfluenza bovina tipo 3 aislado en Japón entre 2002 y 2019. Microbiol veterinario. (2020) 247:108774. doi: 10.1016/j.vetmic.2020.108774
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Su, J , Dou, Y , Tú, Y y Cai, X . Aplicación de la tecnología del minigenoma en la investigación virológica de la familia Paramyxoviridae. J Microbiol Immunol Infect. (2015) 48:123–9. doi: 10.1016/j.jmii.2014.02.008
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Liang, B , Ngwuta, JO , Herbert, R , Swerczek, J , Dorward, DW , Amaro-Carambot, E, et al. El empaquetamiento y la estabilización previa a la fusión aumentan por separado y de forma aditiva la cantidad y la calidad de los anticuerpos neutralizantes del virus respiratorio sincitial (VRS) inducidos por una proteína de fusión del VRS expresada por un vector del virus de la paragripe. J Virol. (2016) 90:10022–38. doi: 10.1128/JVI.01196-16
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Liu, F , Li, L , Liu, Y , Dom, C , Liu, C , Wu, X, et al. Desarrollo de un sistema de genética inversa para morbillivirus de pequeños rumiantes: rescate de virus recombinantes para expresar el antígeno EG95 de Echinococcus granulosus. Virus Res. (2019) 261:50–5. doi: 10.1016/j.virusres.2018.12.008
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Liu, F , Zhang, Y , Li, L , Zuo, Y , Dom, C , Xiaodong, W, et al. Rescate de morbillivirus de pequeños rumiantes que expresan eGFP para identificar susceptibilidades de ocho líneas celulares de mamíferos a su infección. Virus Res. (2019) 261:60–4. doi: 10.1016/j.virusres.2018.12.011
22. Chen, C , Zhou, M , Yan, XG , Chen, YX , Cui, M , Chen, HC, et al. Un virus recombinante del moquillo canino que expresa interleucina-7 mejora la inmunidad humoral. J Gen Virol. (2019) 100:602–15. doi: 10.1099/jgv.0.001247
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Liu, F , Wang, Q y Shan, H . Rescate del virus de la parainfluenza 5 marcado con doble reportero como herramienta para el cribado rápido de antivirales in vitro. Microbiol veterinario. (2021) 259:109154. doi: 10.1016/j.vetmic.2021.109154
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Ren, T , Zhou, L , Mín., X , Sui, M , Zhi, X , Mo, Y, et al. Desarrollo de un virus Getah reportero recombinante para ensayos de cribado de fármacos antivirales. Microbiol veterinario. (2023) 281:109742. doi: 10.1016/j.vetmic.2023.109742
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Liu, T , Liao, Y , Du, Y , Dong, Q , Zhang, W , Qiao, C, et al. Inserción de genes exógenos dentro de la región codificante ORF1b del astrovirus porcino. Microbiol veterinario. (2023) 280:109675. doi: 10.1016/j.vetmic.2023.109675
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: virus de la parainfluenza bovina, sistema de genética inversa, rescate de virus, medicamentos antivirales, ribavirina
Cita: Han Y, Lu K, Zhang R, Wei X, Guo H, Tong L, Wang X, Xiao S, Liu H y Yang Z (2024) Construcción y caracterización de un sistema de genética inversa del virus de la parainfluenza bovina tipo 3c como herramienta para el cribado rápido de antivirales in vitro. Frente. Vet. Sci. 11:1336663. doi: 10.3389/fvets.2024.1336663
Recibido: 11 de noviembre de 2023; Aceptado: 26 de febrero de 2024;
Publicado: 12 marzo 2024.
Editado por:
Kaori Sano, Instituto Nacional de Enfermedades Infecciosas (NIID), Japón
Revisado por:
Kang Ouyang, Universidad de Guangxi, China
Xin Yin, Academia China de Ciencias Agrícolas (CAAS), China
Kohei Oishi, Escuela de Medicina Icahn en Mount Sinai, Estados Unidos
Derechos de autor © 2024 Han, Lu, Zhang, Wei, Guo, Tong, Wang, Xiao, Liu y Yang. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Haijin Liu, de liuhaijin@nwafu.edu.cn años; Zengqi Yang, yzq8162@126.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía


