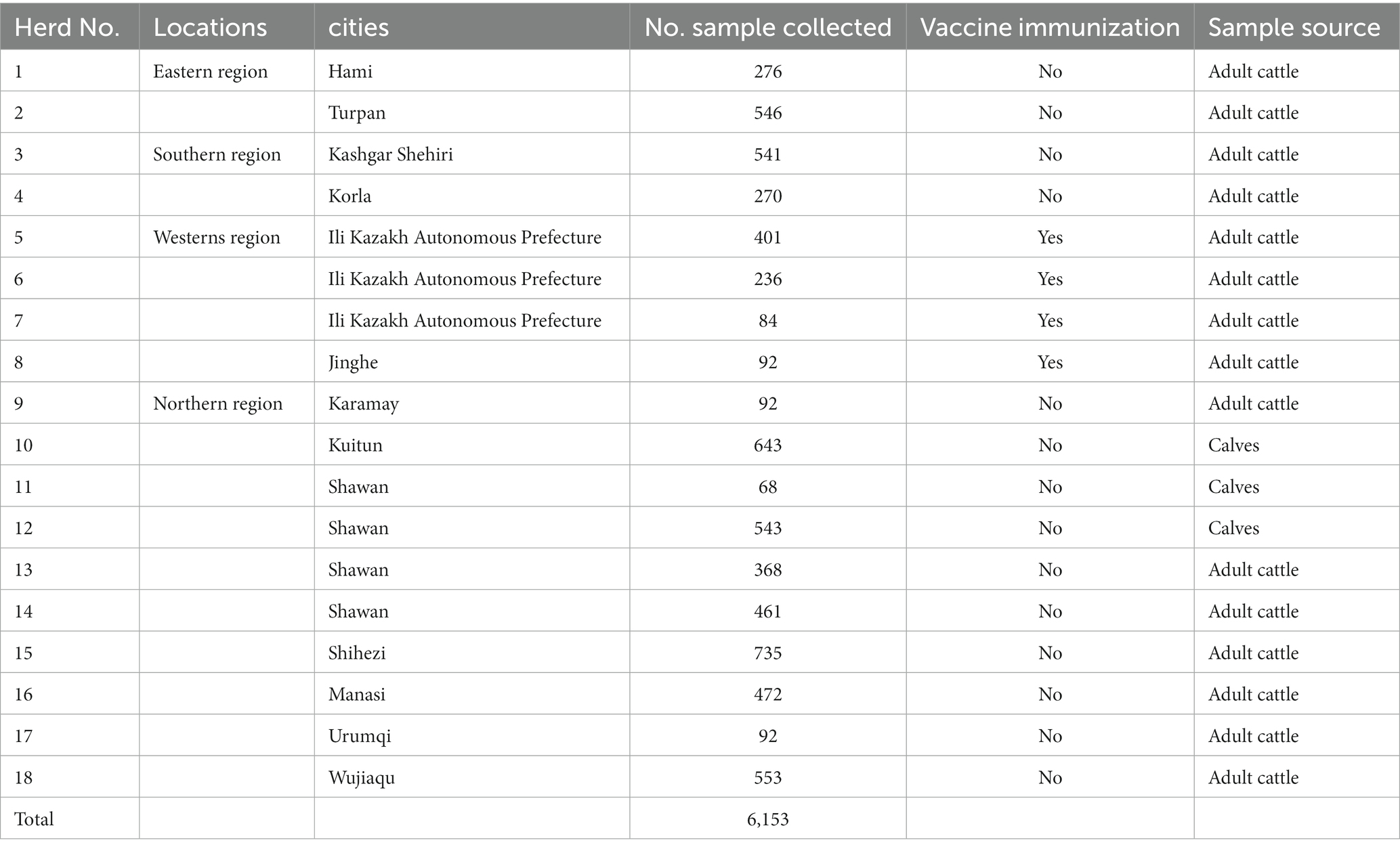
Detección de Pestivirus emergente similar a HoBi (BVD-3)

Detección de Pestivirus emergente similar a HoBi (BVD-3) durante una investigación epidemiológica del virus de la diarrea viral bovina en Xinjiang: un informe único en su tipo
 Ningning Yang1†,
Ningning Yang1†,  Mingguo Xu1†,
Mingguo Xu1†,  Zhenguo Ma1,
Zhenguo Ma1,  Honghuan Li1,
Honghuan Li1,  Canción Shengnan1,
Canción Shengnan1,  Xiaoxiao Gu1,
Xiaoxiao Gu1,  Jingnan Liu1,
Jingnan Liu1,  Zhonglian Yang1,
Zhonglian Yang1,  Hongji Zhu1,
Hongji Zhu1,  Hailong Ma1,
Hailong Ma1,  Jihai Yi1,
Jihai Yi1,  Yong Wang1,
Yong Wang1,  Zhen Wang1,
Zhen Wang1,  Jinliang Sheng1* y
Jinliang Sheng1* y  Chuangfu Chen1,2,3*
Chuangfu Chen1,2,3*- 1Facultad de Ciencia y Tecnología Animal, Universidad Shihezi, Shihezi, China
- 2Laboratorio clave de control y prevención de enfermedades animales, Cuerpo de Producción y Construcción de Xinjiang, Shihezi, China
- 3Centro de Co-Innovación para Enfermedades Infecciosas Zoonóticas en la Región Occidental, Shihezi, China
El área de pastoreo de Xinjiang es la segunda área de pastoreo más grande de China, representando el 26,8% del área de pastizales disponibles en el país, y la ventaja geográfica de la industria ganadera es muy obvia. El virus de la diarrea viral bovina (BVDV) siempre ha sido una de las enfermedades virales importantes que han plagado el desarrollo de la industria ganadera en el mundo. Como una de las principales áreas de pastoreo de la industria ganadera de China, el área pastoral de Xinjiang también se ha visto profundamente afectada. En este estudio, se recolectaron 6.153 muestras de suero bovino de 18 granjas ganaderas a gran escala en 13 ciudades de Xinjiang. Los anticuerpos y antígenos de 6.153 y 588 muestras de suero se detectaron mediante métodos de detección serológica, respectivamente. Diez muestras de suero, que fueron positivas para antígeno por ELISA, se seleccionaron aleatoriamente para la detección de RT-PCR, secuenciación y análisis filogenético de cepas sospechosas de Pestivirus similar a HoBi (HoBiPeV). Los resultados mostraron que las tasas positivas de anticuerpos y antígenos BVDV fueron 53,68% (3.303/6.153) y 6,12% (36/588), respectivamente. Una de las 10 muestras seropositivas seleccionadas al azar fue infectada con la cepa HoBiPeV. HoBiPeV, también conocido como BVDV-3, es un Pestivirus atípico emergente que ocurre en bovinos y pequeños rumiantes, y sus signos clínicos son similares a los de la infección por BVDV. Basado en el genoma completo de la cepa de referencia BVDV-3 (JS12/01) en el GenBank, la homología de la cepa detectada fue del 96,02%. La secuencia completa de nucleótidos del genoma se envió a la base de datos GenBank, y se obtuvo el número de acceso del gen: OP210314. El genoma completo del aislado OP210314 era de 12,239 nucleótidos y contenía un 5′-UTR de 340 nucleótidos, un 3′-UTR de 199 nucleótidos y un gran marco de lectura abierto (ORF) que codificaba una poliproteína que consta de 3.899 aminoácidos. En conclusión, la tasa de prevalencia de la infección por BVDV en las vacas lecheras de Xinjiang es alta y la diversidad genética está aumentando. Este estudio identificó y aisló con éxito HoBiPeV en Xinjiang por primera vez, lo que representa una amenaza potencial para la industria ganadera en Xinjiang.
Introducción
La diarrea viral bovina/enfermedad mucosa (BVD/MD) es una enfermedad infecciosa causada por el virus de la diarrea viral bovina (BVDV; Richter et al., 2017; Deng et al., 2020). El BVDV puede infectar a varios animales, como vacas, cerdos, ovejas y ciervos (Krametter-Froetscher et al., 2010; Blome et al., 2017; Chen et al., 2021; Zhang et al., 2022). BVDV infecta principalmente al ganado en su entorno natural, siendo los terneros los huéspedes más vulnerables y susceptibles. Los animales infectados con BVDV pueden experimentar síntomas como fiebre, diarrea, síntomas respiratorios, erosión de la mucosa, y los animales preñados pueden experimentar aborto, muerte fetal o fetos anormales (Baker, 1995; Blome et al., 2017; Zhou et al., 2022). La Organización Mundial de Sanidad Animal (WOAH) clasifica el BVDV como una enfermedad infecciosa de Clase III, según el grado de daño a la salud animal y la salud pública.
BVDV es un miembro de la familia Flaviviridae, perteneciente al género Pestivirus junto con el virus de la peste porcina clásica (PPC) y el virus de la enfermedad fronteriza (BDV; Chen et al., 2021; Yang et al., 2022). De acuerdo con las recomendaciones del Grupo de Estudio Flaviviridae del Comité Internacional de Taxonomía de Virus (ICTV), BVDV se puede dividir en tres genotipos: BVDV-1 (Pestivirus A), BVDV-2 (Pestivirus B) y Pestivirus atípico o BVDV-3 (Pestivirus H; Smith et al., 2017). BVDV-1 se puede dividir en al menos 23 subtipos (1a-1w), BVDV-2 se puede dividir en cuatro subtipos (2a-2d) y BVDV-3 se puede dividir en fuentes brasileñas, tailandesas, italianas y otras (Smith et al., 2017; Yeşilbağ et al., 2017; Deng et al., 2020). Entre ellos, las cepas BVDV-1 son las más prevalentes en todo el mundo y a menudo se utilizan en estudios de laboratorio y desarrollo de vacunas. BVDV se puede dividir en tipos citopáticos (CP) y no citopáticos (NCP), dependiendo de si pueden causar efecto citopático (CPE) en las células huésped, y ambos biotipos son patógenos. Sin embargo, sólo las cepas NCP pueden causar infección persistente (IP) en el ganado (Lackner et al., 2004; Yang et al., 2022). La existencia de ganado PI es también una de las principales razones por las que el BVDV es difícil de prevenir y eliminar.
El BVDV se descubrió inicialmente en 1946 en bovinos que presentaban síntomas como úlceras pépticas y disentería (Olafson et al., 1946). Fue aislado con éxito en 1957 (Lee y Gillespie, 1957). En 1980, el BVDV se aisló por primera vez en China, confirmando la presencia del virus en el país (Li et al., 1983). De 1980 a 2013, las investigaciones serológicas del BVDV realizadas por investigadores prominentes como Zheng et al. (1991), Wang et al. (1996), Qiu et al. (1998, 2000) y Shang et al. (2013) revelaron que la infección por BVDV era prevalente en la mayoría de las regiones de China, como Mongolia Interior, Shaanxi, Gansu, Ningxia, Qinghai, Xinjiang, Heilongjiang, Liaoning, Jilin y Henan, donde las vacas exhibieron la tasa positiva de anticuerpos más alta. Sin embargo, hubo disparidades notables en la situación de infección entre diferentes regiones o granjas dentro de la misma región.
Xinjiang cuenta con vastos pastizales naturales, con aproximadamente 57 millones de hectáreas de pastizales naturales utilizables y una amplia gama de ganado (Red Geofísica de Xinjiang, 2014). Es una de las cuatro principales áreas pastorales en China. La cría de ganado en Xinjiang tiene ventajas geográficas únicas, y la proporción de ganado en la población total de especies de ganado es superada solo por las ovejas, ocupando el segundo lugar en Xinjiang (Chao et al., 2016; Zhou, 2018). Con el rápido desarrollo de la cría de animales en Xinjiang, el modo tradicional de cultivo al aire libre ha dado paso a un modo de cultivo a gran escala, intensivo y saludable. La escala de la agricultura se está expandiendo, y el número de granjas está aumentando. Sin embargo, con la continua expansión de la escala de reproducción, han surgido varias epidemias, y entre ellas, BVDV es una de las enfermedades importantes que afectan seriamente el desarrollo saludable de la industria ganadera en Xinjiang.
En los últimos años, los datos de la encuesta demuestran una infección grave por BVDV en Xinjiang. Guo (2007) realizó una encuesta epidemiológica del BVDV en la región norte de Xinjiang y encontró que la prevalencia promedio de BVDV era del 35,40%, y todas las muestras recolectadas eran del tipo BVDV-1. Li et al. (2009) encontraron una tasa positiva de ácido nucleico del 39,06% en la encuesta epidemiológica del BVDV en la región de Shihezi, e identificaron la cepa prevalente como el subtipo BVDV-1b. En el período de 2010 a 2012, nuestro equipo de investigación realizó una encuesta epidemiológica sobre BVDV de siete áreas, incluidas Shihezi, Manas, Kashgar y Aksu, y encontró que la tasa de prevalencia promedio de BVDV era de 18.61%, y encontró que la cepa prevalente era principalmente BVDV-1b subtipo (Ren, 2010). Wang et al. (2015) llevaron a cabo la detección serológica del BVDV en granjas ganaderas en Xinjiang, y aislaron 17 cepas de BVDV-I y 4 cepas de BVDV-II. En el período de 2016 a 2018, nuestro equipo de investigación realizó una encuesta epidemiológica del BVDV en granjas de ganado en algunas áreas de Xinjiang, y descubrió que el BVDV está muy extendido en Xinjiang, entre los cuales la cepa 1q es la más prevalente (He et al., 2020). En resumen, los resultados de esta encuesta indican que existe un grave fenómeno de infección por BVDV en la industria ganadera de Xinjiang, y los tipos epidémicos del virus son diversos, lo que ha traído graves pérdidas económicas a la cría de animales de Xinjiang. Sin embargo, las pérdidas económicas digitales específicas aún necesitan más investigación e investigación. Sin embargo, se ha informado que la pérdida de producción anual promedio causada por la infección por BVDV es de € 42.14-€ 67.19 por animal (Pinior et al., 2019). En Australia, la pérdida económica causada por BVDV puede alcanzar hasta AUD 50.9 millones anuales (McGowan et al., 2020).
La prevención y el control del BVDV son cruciales para la industria ganadera, y la vacunación y el sacrificio de animales infectados persistentemente son dos medidas importantes en este sentido. Sin embargo, debido a la rápida variación de las cepas de BVDV, aún no se dispone de una vacuna efectiva. Algunos países europeos han lanzado programas de erradicación del BVDV para combatir este problema, pero nuestro país no ha implementado dicho programa (Yue et al., 2022). Por lo tanto, es imperativo implementar un monitoreo continuo y a largo plazo del BVDV en el ganado para evaluar la eficacia de los métodos de prevención y control, incluida la vacunación y el rastreo de la fuente de infección. El objetivo de esta encuesta es evaluar la prevalencia del BVDV en Xinjiang, así como aislar e identificar cualquier nueva cepa que pueda estar surgiendo en la región, Este estudio es crucial para el desarrollo saludable de la industria ganadera, ya que proporcionará información sobre el estado actual del BVDV y ayudará a desarrollar estrategias de prevención y control más efectivas.
Materiales y métodos
Declaración ética
Este estudio fue aprobado por el Comité de Ética Animal Experimental del Primer Hospital Afiliado del Colegio Médico de la Universidad de Shihezi, y obtuvo el consentimiento informado por escrito del propietario para la participación de su ganado. El formulario de consentimiento delineó explícitamente los objetivos, procedimientos, riesgos potenciales y beneficios de la investigación. Además, nos adherimos meticulosamente a los principios de bienestar animal y utilizamos las medidas apropiadas para minimizar la angustia y la incomodidad experimentadas por los animales.
Área de estudio y recolección de muestras
De 2017 a 2020, nuestro equipo recolectó un total de 6,153 muestras de sangre total de 18 granjas ganaderas distintas ubicadas en 13 ciudades de Xinjiang, China (Figura 1; Tabla 1). Las muestras de sangre entera recolectadas se transportaron cuidadosamente al Laboratorio de Patología de la Escuela de Ciencia y Tecnología Animal de la Universidad Shihezi para la separación del suero, y posteriormente se almacenaron a una temperatura de -20 ° C para su preservación.
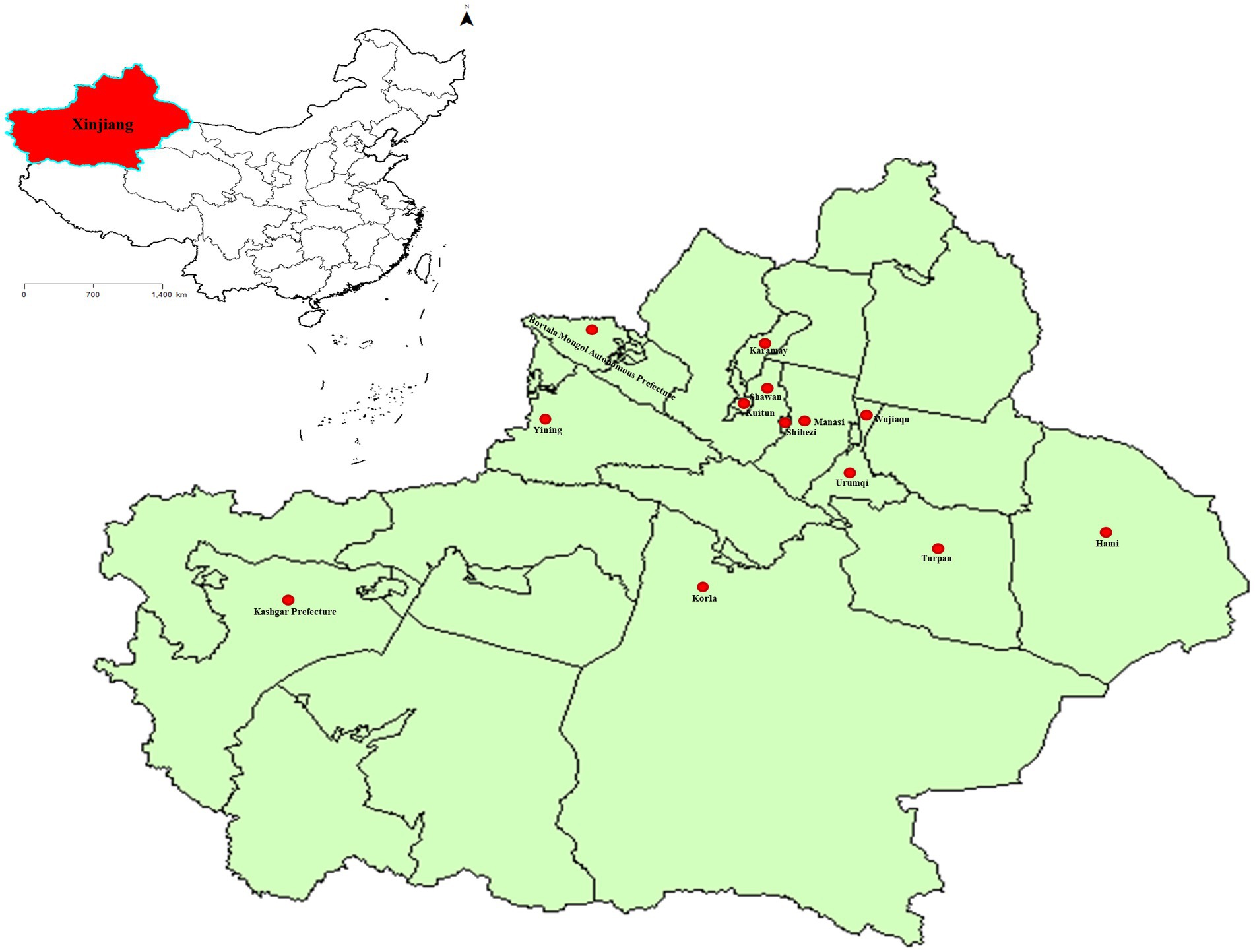 Figura 1. Los puntos de muestreo de BVDV en Xinjiang, China. El mapa estándar de China se obtuvo del servicio nacional de mapas estándar (http://bzdt.ch.mnr.gov.cn/). El área roja representa la ubicación de Xinjiang en China, mientras que el punto rojo marca las ciudades donde se recolectaron las muestras en Xinjiang.
Figura 1. Los puntos de muestreo de BVDV en Xinjiang, China. El mapa estándar de China se obtuvo del servicio nacional de mapas estándar (http://bzdt.ch.mnr.gov.cn/). El área roja representa la ubicación de Xinjiang en China, mientras que el punto rojo marca las ciudades donde se recolectaron las muestras en Xinjiang.
Detección de anticuerpos y antígenos contra el BVDV
Las muestras de suero recolectadas se sometieron a un cribado de anticuerpos y antígenos utilizando el kit de prueba de anticuerpos ELISA indirectos BVDV desarrollado por nuestro equipo de investigación (datos aún no publicados) y la prueba IDEXX BVDV Ag / Serum plus (Estados Unidos), respectivamente.
RT-PCR
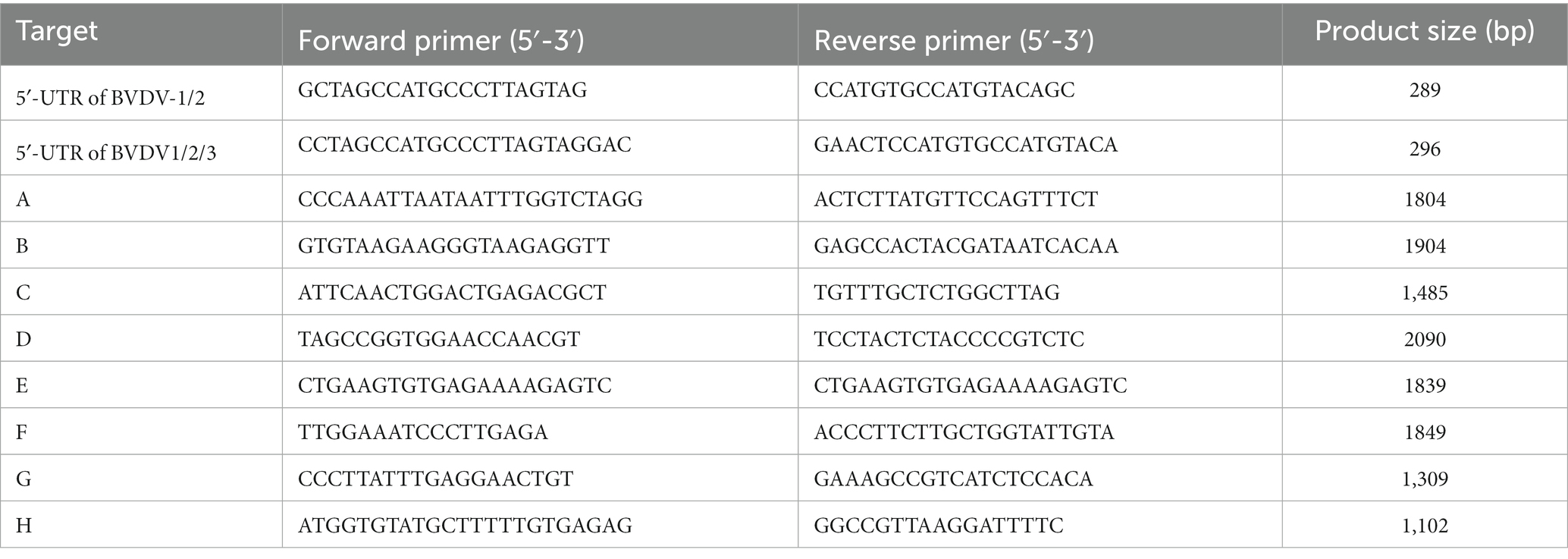
Diez muestras que mostraban antígeno positivo detectado por ELISA fueron seleccionadas aleatoriamente y posteriormente verificadas por RT-PCR. En resumen, el genoma del ARN viral se extrajo utilizando un kit de extracción de ARN del virus biospin (Bioer Technology, China) y se transcribió inversamente para sintetizar ADNc (CWBIO, China), siguiendo las instrucciones del fabricante. Las secuencias de cebadores fueron delineadas en la Tabla 2. Los productos de amplificación se sometieron a electroforesis con geles de agarosa al 1% y fueron examinados bajo un sistema de análisis de imágenes en gel (Bio-Rad, Estados Unidos). Las bandas intencionales se extirparon individualmente del gel, se recuperaron y purificaron utilizando un kit de purificación TIANgel (TIANGEN, China). Los productos purificados fueron posteriormente clonados en el vector pMD-19-T (TaKaRa, Japón), y los clones positivos (tres por muestra) fueron secuenciados por Rui Biotech (Beijing, China).
Análisis filogenético
Las secuencias de nucleótidos se alinearon y examinaron sistemáticamente utilizando la herramienta SeqMan integrada en el paquete de software DNAStar (DNAStar Inc., Estados Unidos; Deng et al., 2015; Kumar et al., 2018). La reconstrucción filogenética de las secuencias 5′-UTR y completas del genoma se realizó utilizando el método de unión de vecinos en el software MEGA 7.0 (Tamura et al., 2004). La robustez estadística del árbol filogenético se evaluó arrancando el conjunto de datos con 1.000 réplicas.
Cultivo celular y aislamiento de virus
Las células de riñón bovino Madin-Darby (MDBK) se obtuvieron de la Colección Nacional de Autenticados (Shanghai, China) y se propagaron en el medio de águila modificado de Dulbecco (DMEM; Gibco, Estados Unidos) suplementado con suplementado con suero fetal bovino inactivado por calor al 10% (FBS; Gibco, Estados Unidos) a 37°C con 5% de CO2. Para aislar el virus (Zhu et al., 2016; Chen et al., 2021), 200 μl de suero BVDV positivo después de la filtración se inoculó en células MDBK con DMEM pero sin FBS. Después de 2 h de infección, se descartó el medio de cultivo celular y se complementó FBS fresco al 1%. Posteriormente, los cultivos celulares que contenían virus se recolectaron después de 5 días y se sometieron a tres ciclos de congelación y descongelación repetidas a -80 ° C. El procedimiento descrito anteriormente se repitió con 200 μl de cultivos de virus inoculados en células MDBK hasta el paso 10 (P10), con un intervalo de paso de 5 días. El CPE en células MDBK se monitorizó bajo un microscopio invertido (Nikon, Japón).
Ensayo de inmunofluorescencia indirecta
Las células MDBK se sometieron a infección por BVDV en una multiplicidad de infección (MOI) de 10, y se incubaron durante 36 h, sirviendo como grupo experimental (grupo MDBK infectado por BVDV). Las células MDBK cultivadas en DMEM se utilizaron como grupo control (grupo MDBK infectado simulado). El IFA se realizó como se describió anteriormente (Zhang et al., 2022). El protocolo IFA se modificó ligeramente, por lo que las células MDBK se incubaron primero con suero BVDV positivo (diluido 1:300) como anticuerpo primario, seguido de IgG antibovino de conejo marcado con isotiocianato de fluoresceína (FITC) (diluido 1:500; Solarbio, China) como el anticuerpo secundario. Finalmente, las células se observaron bajo un microscopio de barrido láser confocal (microscopio confocal C2 +, Nikon, Japón), y las imágenes se analizaron utilizando el software NIS-Elements Viewer (Nikon, Japón).
Análisis estadístico
El análisis estadístico de los datos se realizó mediante el método de prueba Chi-cuadrado en SPSS 17.0 (SPSS Inc., Chicago), y se consideró un valor de p <0,05 para indicar diferencias estadísticamente significativas. El software GraphPad Prism 8.0 (Graph-Pad Software Inc., Estados Unidos) se empleó para la visualización de datos.
Resultados
Detección de anticuerpos y antígenos contra el BVDV en muestras de suero
Se recogieron sistemáticamente un total de 6.153 muestras de suero bovino de 18 rebaños que abarcaban 13 ciudades de Xinjiang (Figuras 1, 2A; Tabla 1). La tasa media positiva de anticuerpos contra el BVDV en bovinos se calculó en 53,68% (3.303/6.153). Se observó que la tasa positiva de anticuerpos de la región oriental fue comparativamente menor en 19,46%, que fue significativamente diferente de la región sur (44,88%), la región norte (58,60%) y la región occidental (84,99%, Figura 2B). La tasa positiva de anticuerpos de las regiones sur y norte fue significativamente menor que la de la región occidental. Por lo tanto, la región occidental tuvo la tasa positiva de anticuerpos más alta, posiblemente porque la región había vacunado a su ganado con la vacuna BVDV 4 meses antes del muestreo. La tasa positiva de anticuerpos del ganado vacuno inmunizado fue significativamente mayor que la del ganado no inmunizado (Figura 2C). Los niveles de antígenos de la región occidental no se probaron porque ya se habían vacunado. Curiosamente, los niveles de anticuerpos y antígenos de los terneros fueron significativamente más altos que los del ganado adulto (Figuras 2D, E), lo que podría deberse a anticuerpos maternos o transmisión vertical. Como se indica en la Figura 2F, la región oriental tuvo la tasa más baja de antígenos positivos con un 3,88%, seguida de la región sur (4,08%), mientras que la región norte tuvo la tasa más alta de antígenos positivos de 7,24%. Sin embargo, no hubo diferencias significativas en la tasa de antígenos positivos entre las tres regiones.
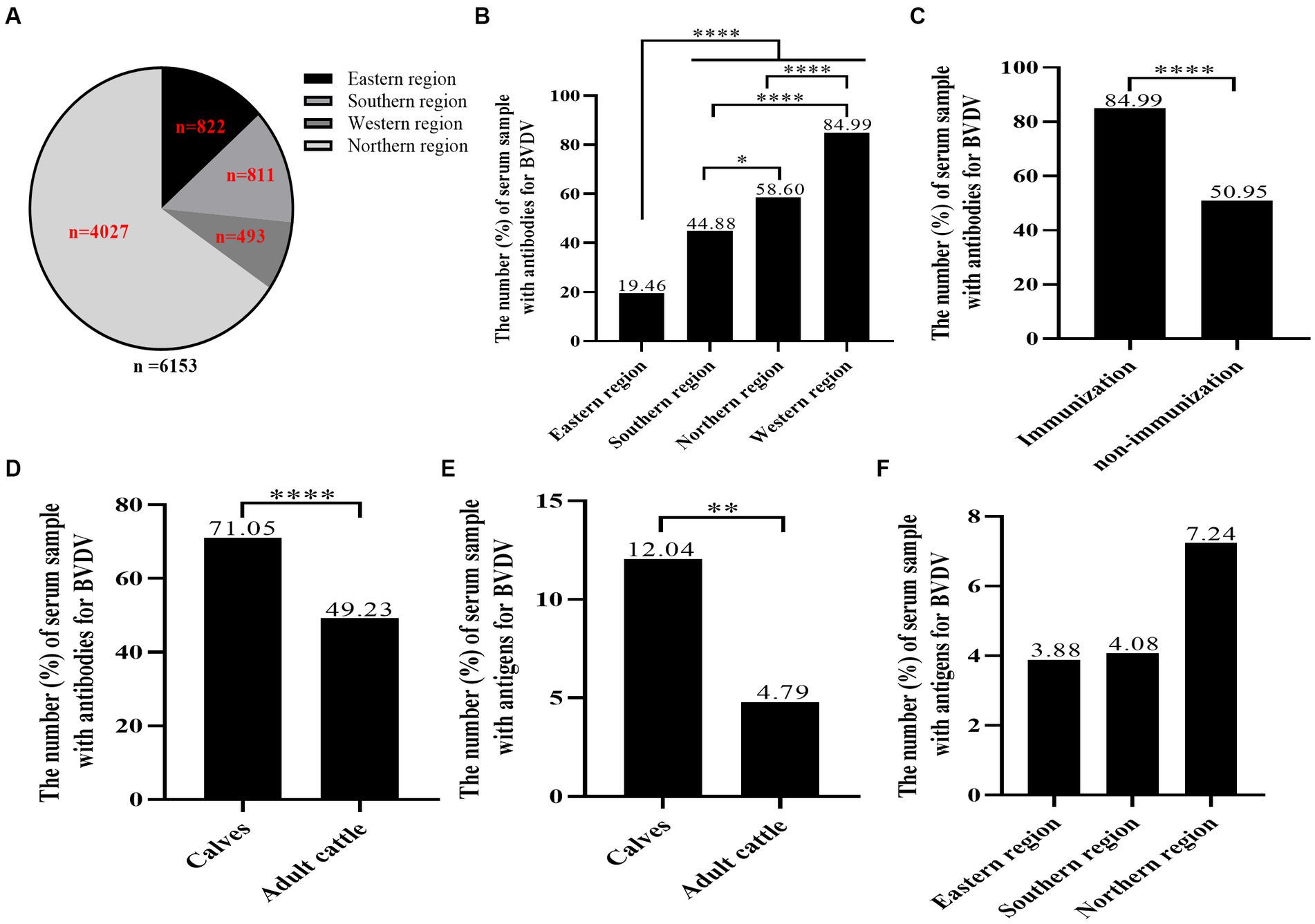 Figura 2. La seroprevalencia del BVDV en animales individuales. Se recogieron un total de 6.153 muestras de sangre bovina de 18 granjas, que comprendían terneros y ganado adulto, en 13 ciudades de cuatro regiones distintas. Los anticuerpos y antígenos del BVDV se identificaron a través del kit de prueba de anticuerpos ELISA indirectos BVDV de nuestro equipo de investigación y la prueba IDEXX BVDV Ag / Serum Plus Test, respectivamente. (A) La distribución de muestras de BVDV por región. (B) El análisis de la prevalencia de anticuerpos BVDV en las cuatro regiones. (C) El análisis de la tasa positiva de anticuerpos contra el BVDV en bovinos inmunizados y no inmunizados. (D,E) El análisis de los niveles de anticuerpos y antígenos del BVDV de terneros. (F) El análisis de los niveles de antígeno BVDV de la región oriental, la región sur y la región norte. La significación estadística se fijó en p < 0,05 (*), p < 0,01 (**) y p < 0,0001 (****).
Figura 2. La seroprevalencia del BVDV en animales individuales. Se recogieron un total de 6.153 muestras de sangre bovina de 18 granjas, que comprendían terneros y ganado adulto, en 13 ciudades de cuatro regiones distintas. Los anticuerpos y antígenos del BVDV se identificaron a través del kit de prueba de anticuerpos ELISA indirectos BVDV de nuestro equipo de investigación y la prueba IDEXX BVDV Ag / Serum Plus Test, respectivamente. (A) La distribución de muestras de BVDV por región. (B) El análisis de la prevalencia de anticuerpos BVDV en las cuatro regiones. (C) El análisis de la tasa positiva de anticuerpos contra el BVDV en bovinos inmunizados y no inmunizados. (D,E) El análisis de los niveles de anticuerpos y antígenos del BVDV de terneros. (F) El análisis de los niveles de antígeno BVDV de la región oriental, la región sur y la región norte. La significación estadística se fijó en p < 0,05 (*), p < 0,01 (**) y p < 0,0001 (****).
RT-PCR
Diez muestras positivas fueron seleccionadas aleatoriamente para la verificación de RT-PCR. Para la detección se utilizó el cebador BVDV-1/2, y se observó que todas las muestras, excepto la muestra No.5, exhibieron los fragmentos anticipados (Figura 3A), lo que implica que la muestra No.5 podría no haber sido causada por la infección por BVDV-1/2 o podría ser un falso positivo. Por lo tanto, se realizaron pruebas adicionales utilizando los cebadores de BVDV-1/2/3, que revelaron la presencia del fragmento esperado (Figura 3B), lo que indica que BVDV-3 podría ser el patógeno causante en la muestra No.5. Para confirmar este hallazgo, se diseñaron cebadores de amplificación segmentaria de longitud completa para la muestra No.5, con referencia a la secuencia completa de la cepa BVDV-3 PB22487 (GenBank: KY762287.2). Los resultados demostraron la amplificación exitosa de los fragmentos del gen diana a través de la amplificación segmentaria (Figura 3C). Los resultados de la RT-PCR mostraron que la muestra No.5 estaba infectada con la cepa BVDV-3.
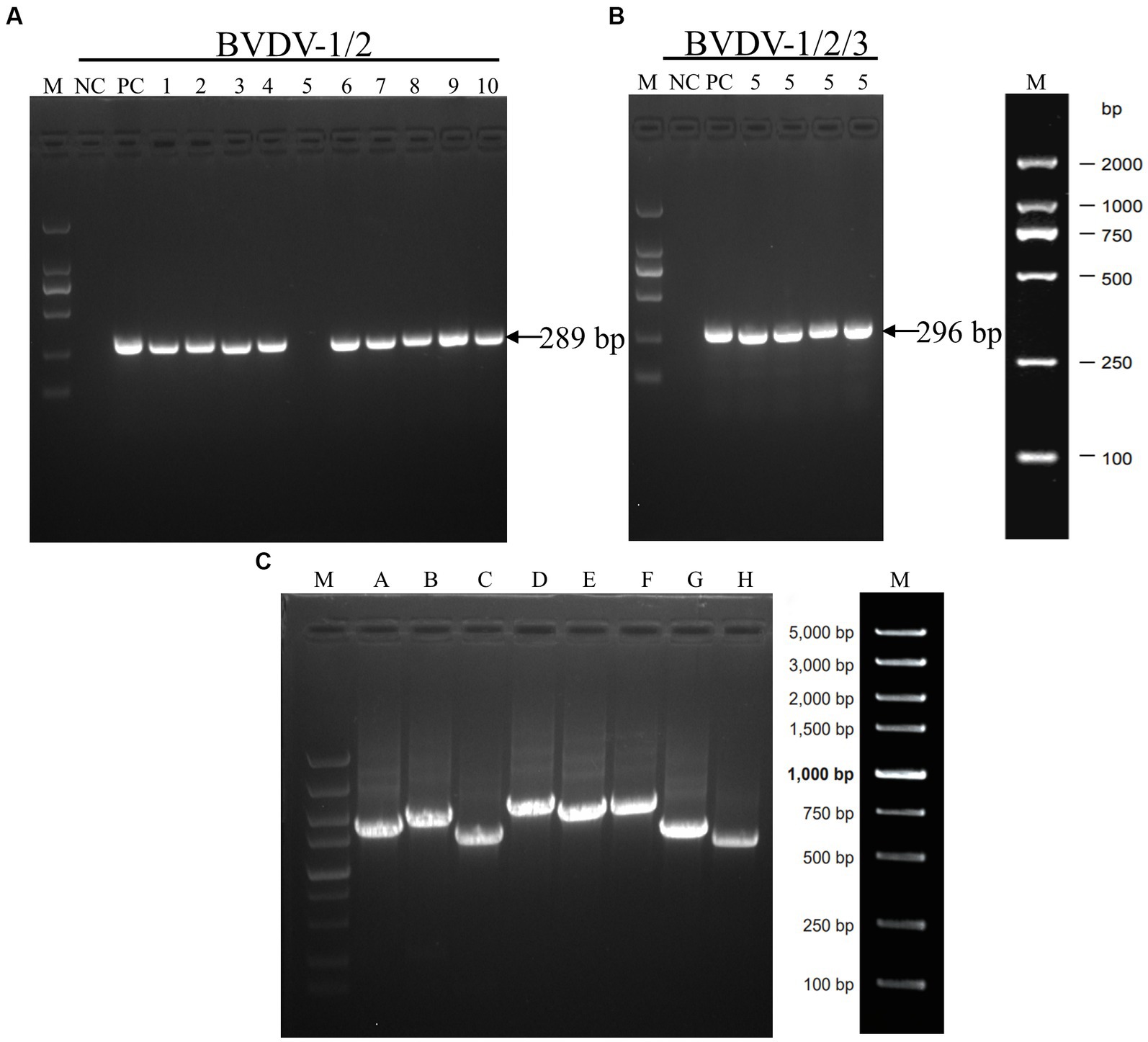 Figura 3. Los resultados de las pruebas RT-PCR realizadas en algunas muestras que dieron positivo para BVDV usando ELISA. (A) Los resultados del análisis por RT-PCR del fragmento genómico BVDV-1/2 5′-UTR. Se analizaron muestras de suero numeradas del 1 al 10. (B) El análisis de RT-PCR se realizó en el fragmento genómico BVDV1/2/3 5′-UTR utilizando la muestra sérica número 5. (C) La amplificación de fragmentos del genoma completo utilizando cebadores segmentados (A-H). El marcador de ADN está representado por la letra M, mientras que los controles negativos y positivos se indican por NC y PC, respectivamente.
Figura 3. Los resultados de las pruebas RT-PCR realizadas en algunas muestras que dieron positivo para BVDV usando ELISA. (A) Los resultados del análisis por RT-PCR del fragmento genómico BVDV-1/2 5′-UTR. Se analizaron muestras de suero numeradas del 1 al 10. (B) El análisis de RT-PCR se realizó en el fragmento genómico BVDV1/2/3 5′-UTR utilizando la muestra sérica número 5. (C) La amplificación de fragmentos del genoma completo utilizando cebadores segmentados (A-H). El marcador de ADN está representado por la letra M, mientras que los controles negativos y positivos se indican por NC y PC, respectivamente.
Secuenciación y análisis filogenético
Los segmentos mencionados anteriormente se ligaron al vector PMD-19-T y se enviaron a Rui Biotech para su secuenciación. Se generaron tres réplicas para cada muestra para garantizar la precisión de los resultados. Posteriormente, los datos de secuenciación fueron corregidos y empalmados utilizando el paquete de programas informáticos DNAStar. El análisis reveló que la cepa BVDV comprendía 12.239 nucleótidos y 3.899 aminoácidos. Para obtener información sobre las relaciones evolutivas de los aislados de BVDV, se realizó un análisis filogenético utilizando el software MEGA 7.0. La secuencia del genoma completo se utilizó para construir un árbol filogenético utilizando el método de unión de vecinos. Los resultados indicaron que la cepa BVDV se agrupó en BVDV-3 junto con la cepa JS12/01 (Figura 4). La cepa BVDV recién identificada fue nombrada XJ-BVDV-3. La información de la secuencia del genoma completo se cargó en el Centro Nacional de Información Biotecnológica (NCBI) y se obtuvo un número de inicio de sesión de GenBank (GenBank: OP210314).
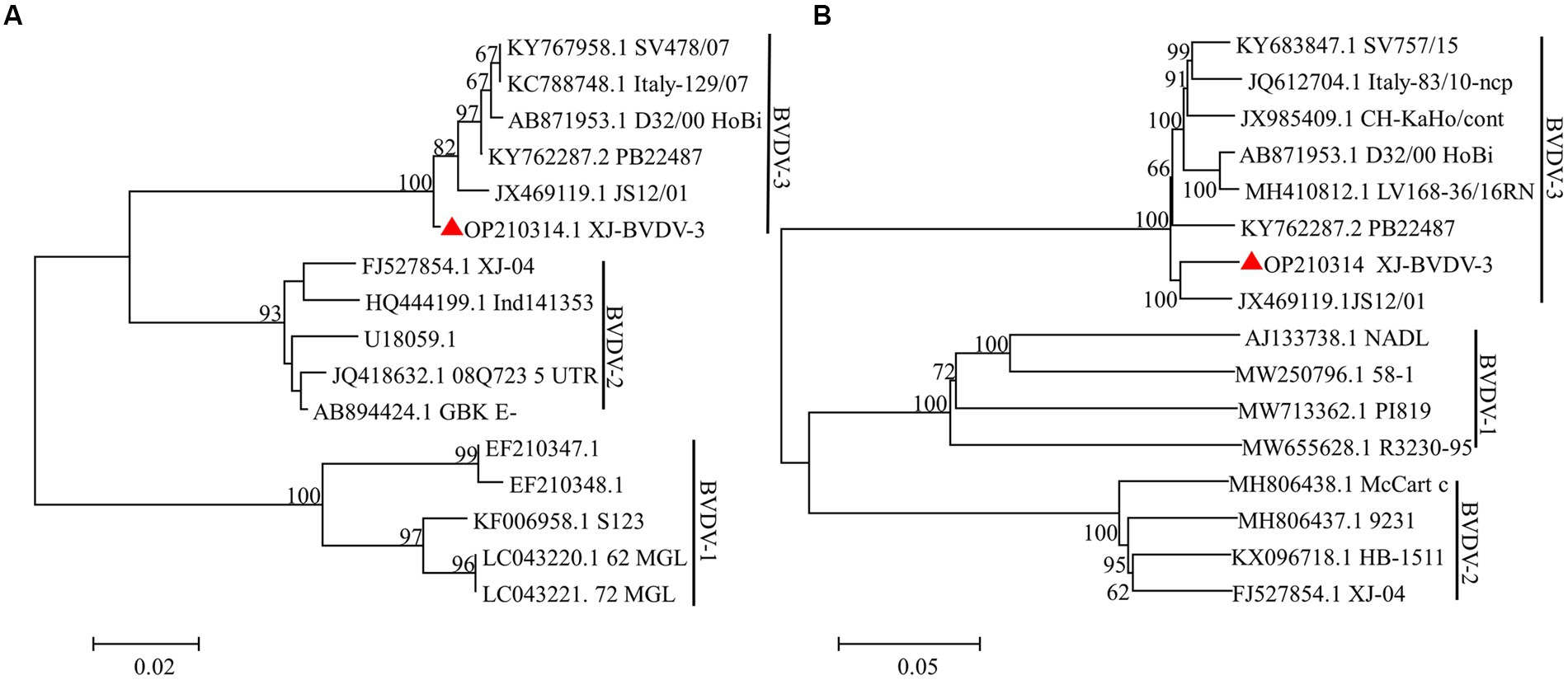 Figura 4. Los árboles filogenéticos basados en el análisis de las secuencias del 5′-UTR y el genoma completo. Los árboles se construyeron utilizando el método de unión de vecinos y pruebas de bootstrap. Los números sobre las ramas indican el porcentaje de 1.000 réplicas de bootstrap que soportan cada rama filogenética. (A) Los árboles filogenéticos basados en el análisis de las secuencias del 5′-UTR. (B) Los árboles filogenéticos basados en el análisis de las secuencias de todo el genoma. El triángulo rojo representa las secuencias identificadas en este estudio.
Figura 4. Los árboles filogenéticos basados en el análisis de las secuencias del 5′-UTR y el genoma completo. Los árboles se construyeron utilizando el método de unión de vecinos y pruebas de bootstrap. Los números sobre las ramas indican el porcentaje de 1.000 réplicas de bootstrap que soportan cada rama filogenética. (A) Los árboles filogenéticos basados en el análisis de las secuencias del 5′-UTR. (B) Los árboles filogenéticos basados en el análisis de las secuencias de todo el genoma. El triángulo rojo representa las secuencias identificadas en este estudio.
Ensayo de inmunofluorescencia indirecta
Las células MDBK todavía no tenían CPE cuando se infectaron con P10 XJ-BVDV-3, lo que indica que XJ-BVDV-3 es un BVDV no citopatogénico. Los resultados de IFA mostraron que las células MDBK infectadas con XJ-BVDV-3 mostraron fluorescencia verde, mientras que las células MDBK infectadas simuladas no mostraron fluorescencia verde (Figura 5).
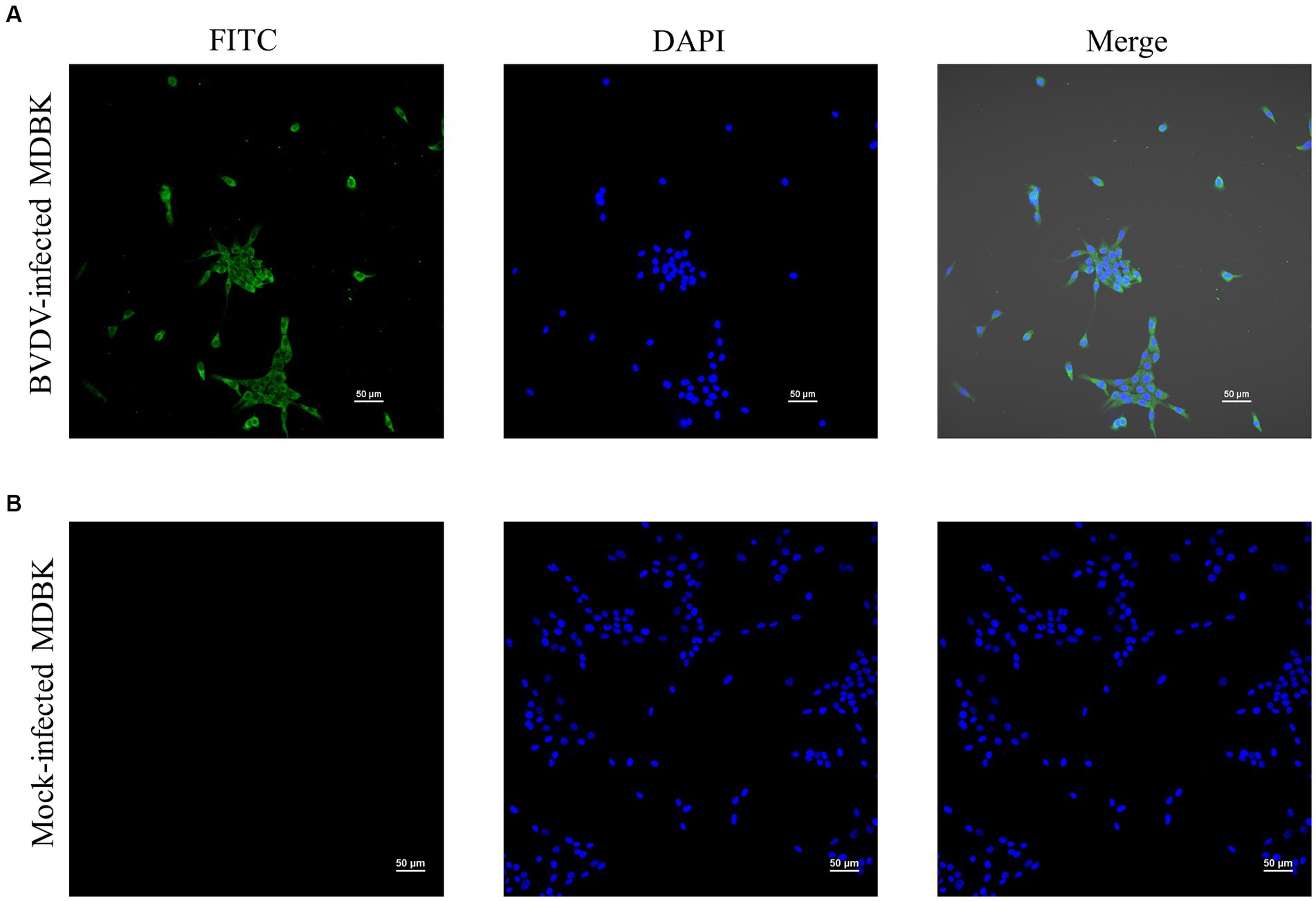 Figura 5. La identificación del virus aislado cultivado en células MDBK a través de IFA. (A) Células MDBK infectadas por BVDV. (B) Células MDBK infectadas simuladas.
Figura 5. La identificación del virus aislado cultivado en células MDBK a través de IFA. (A) Células MDBK infectadas por BVDV. (B) Células MDBK infectadas simuladas.
Discusión
BVDV se ha convertido en una amenaza global, causando estragos en la salud animal y el rendimiento de la producción, y causando pérdidas económicas significativas en la industria ganadera en todo el mundo (Ran et al., 2019). Con la creciente demanda de productos lácteos y de carne, ha habido un aumento en las inversiones en la industria ganadera en muchos países, incluida China, lo que lleva al transporte frecuente de ganado y contribuye a la rápida propagación del BVDV en todo el país. Un estudio realizado por Deng et al. (2015) reveló que incluso el ganado sano observado clínicamente (como vacas, vacas, búfalos y yaks) tenía una tasa positiva total de anticuerpos contra el BVDV del 58,09%. Además, otros estudios han informado que más del 46,7% de las granjas de ganado en China dieron positivo para antígenos del BVDV, con una tasa de infección persistente del 2,2% en los rebaños de ganado, significativamente más alta que la de la mayoría de los países asiáticos e incluso más alta que la de los países europeos y americanos (Zhong et al., 2011; Guo, 2018; He et al., 2020).
En la actualidad, se han identificado numerosos subtipos de BVDV en China, incluidos 1a, 1b, 1c, 1d, 1 m, 1o, 1p, 1q, 1u, 2a, 2b, así como dos subtipos novedosos, 1v y 1w, junto con HoBiPeV (Deng et al., 2015; Yeşilbağ et al., 2017; Deng et al., 2020; Chen et al., 2021). Se sabe que el BVDV-II es más virulento, causa síntomas clínicos más graves, y con frecuencia es el culpable de los brotes en Europa y América (Nie et al., 2012; Zhao, 2015). HoBiPeV se descubrió por primera vez en FBS originario de Brasil y posteriormente se descubrió que infectaba ganado en China también (Schirrmeier et al., 2004; Chen et al., 2021). En resumen, existe una epidemia grave y compleja de BVDV en China con genotipos cada vez más diversos, lo que representa una amenaza significativa para el desarrollo sostenible de la industria ganadera. En consecuencia, se deben tomar las medidas adecuadas para prevenir y controlar esta enfermedad.
En este estudio, obtuvimos 6.153 muestras de sangre de 18 granjas de ganado, que abarcan 13 ciudades en cuatro regiones de Xinjiang (Figura 1; Tabla 1). Inicialmente, analizamos las 6.153 muestras de suero para detectar anticuerpos contra el BVDV. Los resultados indicaron que la tasa positiva de anticuerpos fue la más baja en la región oriental (19,46%) y la más alta en la región occidental (84,99%; Figura 2B). Esta observación puede atribuirse al hecho de que la región occidental había administrado vacunas contra el BVDV a su ganado 4 meses antes del muestreo. Además, el nivel de anticuerpos séricos de los terneros fue significativamente mayor que el del ganado adulto (Figura 2D), debido plausiblemente a la presencia de anticuerpos maternos robustos en los terneros. Se encontró que la tasa positiva general de anticuerpos contra el BVDV fue del 53,68%, lo que es consistente con la tasa de prevalencia del BVDV en el ganado lechero reportada en China (53,0%, Ran et al., 2019). Posteriormente, se examinaron 588 muestras de suero para detectar el antígeno BVDV. Curiosamente, el nivel de antígeno de los terneros fue significativamente mayor que el del ganado adulto (Figura 2E), lo que sugiere que los terneros son más susceptibles a la infección por BVDV que el ganado adulto. Como la región occidental ya ha sido vacunada, no probamos el antígeno en esta área. Las tasas positivas del antígeno BVDV en las tres regiones restantes, de baja a alta, fueron la región oriental (3,88%), la región sur (4,08%) y la región norte (7,24%), pero no se observaron diferencias significativas entre las tres regiones (Figura 2F). Debido a que el sitio de muestreo no cubrió completamente toda la región de Xinjiang. Por lo tanto, estos datos pueden no reflejar con precisión la prevalencia del BVDV en varias regiones de Xinjiang.
Se empleó una selección aleatoria de 10 muestras de suero que dieron positivo para el antígeno BVDV por ELISA, para la detección de RT-PCR. Como el BVDV-3 nunca se había informado en Xinjiang, inicialmente utilizamos el cebador BVDV-1/2 para la detección. Los resultados mostraron que solo la muestra No.5 no produjo la banda esperada (Figura 3A). Posteriormente, utilizamos cebadores BVDV-1/2/3 para probar más a fondo la muestra No.5, que generó la banda esperada a 296 pb, confirmando tentativamente que la muestra No.5 estaba infectada con BVDV-3 (Figura 3B). Debido a la dificultad del aislamiento del virus, la mayor parte de la investigación se centra en la detección de ácidos nucleicos en regiones 5′-UTR para la identificación y clasificación del virus, lo que conduce a la falta de evidencia confiable para el genotipado del BVDV y el análisis exhaustivo de las características genéticas (Hou et al., 2019; Chang et al., 2021; Tian et al., 2021; Afify et al., 2022). Para obtener resultados concluyentes con respecto a la infección de la muestra No.5 con BVDV-3, diseñamos cebadores de amplificación de BVDV de longitud completa con referencia a la secuencia de la cepa BVDV-3 PB22487 (GenBank: KY762287.2), y amplificamos con éxito el fragmento objetivo de manera segmentada (Figura 3C). La secuencia resultante se empalmó y corrigió utilizando el paquete de programas informáticos DNAStar, que determinó la longitud total del genoma del virus como 12.239 nt. La comparación con la base de datos del NCBI confirmó que la muestra Nº 5 era del tipo BVDV-3, designada como XJ-BVDV-3 (Figura 4), y los datos obtenidos se cargaron en la base de datos del NCBI para obtener el número de inicio de sesión: OP210314.1. En conclusión, este estudio confirmó el nuevo genotipo BVDV-3 basado en el análisis filogenético de todo el genoma, lo que indica la fiabilidad de identificar el nuevo genotipo BVDV-3.
Dado que el aislamiento del virus generalmente se considera el estándar de oro para el diagnóstico del BVDV (Saliki et al., 2000; Hou et al., 2019), aislamos y cultivamos el virus de la muestra No.5 utilizando células MDBK. Utilizamos IFA para detectar el virus que se transmitió a los 10ésimo generación. Nuestros resultados demostraron que las células MDBK infectadas con XJ-BVDV-3 mostraron fluorescencia verde, mientras que las células no infectadas no mostraron fluorescencia (Figura 5), lo que indica que obtuvimos una cepa viral XJ-BVDV-3 proliferante estable en células MDBK. La virulencia viral es importante para comprender el mecanismo patogénico de los virus y seleccionar cepas desafiantes para evaluar las vacunas. Sin embargo, considerando el costo experimental, este estudio no determinó las características patogénicas y la virulencia de XJ-BVDV-3, y aún se desconoce si infecta a otros animales además del ganado, como cerdos, ovejas, etc. Dado que informes anteriores han demostrado que HoBiPeV puede infectar en ganado y pequeños rumiantes (Decaro et al., 2012; Bauermann et al., 2015; Bauermann y Ridpath, 2015). Podemos plantear la hipótesis de que las cepas XJ-BVDV-3 también pueden infectar a cerdos, ovejas y otros pequeños rumiantes, y se necesitan más experimentos con animales para verificar la situación específica.
Nuestra investigación epidemiológica del BVDV en algunas áreas de Xinjiang reveló un grave fenómeno de infección por BVDV en la región. Durante nuestra investigación, aislamos la cepa XJ-BVDV-3 por primera vez. El análisis filogenético y de secuencia del gen 5′-UTR y de todo el genoma reveló que la cepa XJ-BVDV-3 pertenece al genotipo BVDV-3, lo que indica que la cepa BVDV-3 está presente en Xinjiang. En conclusión, el monitoreo en tiempo real de la propagación de cepas de BVDV en rebaños de ganado puede proporcionar un apoyo teórico confiable para el desarrollo de reactivos de diagnóstico y vacunas
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio se incluyen en el artículo / material complementario, las consultas adicionales pueden dirigirse a los autores correspondientes.
Contribuciones del autor
NY, MX, JS y CC concibieron y diseñaron el proyecto. NY, MX, ZM, HL, SS, XG, JL, ZY, HZ, HM, JY, YW y ZW recolectaron las muestras. NY, MX y ZM realizaron los experimentos. NY, MX y ZM realizaron los experimentos. NY y MX analizaron e interpretaron los datos y redactaron el manuscrito inicial. JS y CC revisaron y revisaron críticamente el borrador inicial. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Los autores declaran que este estudio recibió fondos de la Transformación y demostración de aplicación de los logros de la tecnología de detección rápida para enfermedades animales importantes en la cría intensiva (subvención no. 21322912D) y el Fondo Especial para el Desarrollo de Ciencia y Tecnología del Cuerpo de Producción y Construcción de Xinjiang «Investigación y aplicación del kit de diagnóstico rápido para la diarrea viral bovina / enfermedad mucosa (BVDV / MD)» (subvención no. 2017BA044). El financiador no participó en el diseño del estudio, la recopilación, el análisis, la interpretación de los datos, la redacción de este artículo o la decisión de enviarlo para su publicación.
Reconocimientos
Todos estamos agradecidos a los revisores por sus valiosos comentarios sobre la mejora de este manuscrito.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pueda interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
Afify, F., Hassanien, T., Abdelmegeed, K., Abouelyazeed, A., Ali, H., Abdelwahed, A., et al. (2022). Primera detección del emergente Pestivirus HoBi-1ike (BVD-3) entre algunos rebaños lecheros persistentes1y infectados catt1e en Egipto. Trop. Anim. Salud Prod. 54, 1–9. DOI: 10.1007/S11250-022-03332-2
Baker, J. (1995). Las manifestaciones clínicas de la infección por diarrea viral bovina. Veterinario Clin. North Am. Food Anim. Pract. 11, 425–445. doi: 10.1016/S0749-0720(15)30460-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bauermann, F., Falkenberg, M., Decaro, N., Flores, E. y Ridpath, J. (2015). Infección experimental de terneros, ovinos, caprinos y cerdos con virus similares a HoBi por inoculación directa o exposición a terneros persistentemente infectados. Veterinario Microbiol. 181, 289–293. doi: 10.1016/j.vetmic.2015.10.011
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bauermann, F. y Ridpath, J. (2015). Virus similares a HoBi: el típico pestivirus bovino atípico. 16, págs. 64–69. doi: 10.1017/S146625231500002X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Blome, S., Beer, M. y Wernike, K. (2017). Nuevas hojas en el creciente árbol de Pestivirus. Adv. Virus Res. 99, 139–160. doi: 10.1016/bs.aivir.2017.07.003
Chang, L., Qi, Y., Liu, D., Du, Q., Zhao, X. y Tong, D. (2021). Detección molecular y genotipado del virus de la diarrea viral bovina en el oeste de China. BMC Vet. 17, 1–7. DOI: 10.1186/S12917-021-02747-7
Chao, X., Ma, B., Yin, J., Xu, M., Chen, N. y Gan, S. (2016). Investigación, análisis y contramedidas para el estado actual de la industria ganadera en Xinjiang. Oasis Agric. Sci. Ing. 3, 9–15.
Chen, M., Liu, M., Liu, S. y Shang, Y. (2021). La infección por pestivirus tipo HoBi conduce a la muerte bovina y a enfermedades respiratorias graves en China. Transbound. Emerg. Dis. 68, 1069–1074. doi: 10.1111/tbed.13832
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Decaro, N., Mari, V., Lucente, M., Sciarretta, R., Moreno, A., Armenise, C., et al. (2012). Infección experimental de bovinos, ovinos y porcinos con pestivirus tipo ‘Hobi’. Veterinario Microbiol. 155, 165–171. doi: 10.1016/j.vetmic.2011.08.030
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Deng, M., Chen, N., Guidarini, C., Xu, Z., Zhang, J., Cai, L., et al. (2020). Prevalencia y diversidad genética del virus de la diarrea viral bovina en rebaños lecheros de China. Veterinario Microbiol. 242:108565. doi: 10.1016/j.vetmic.2019.108565
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Deng, M., Ji, S., Fei, W., Raza, S., He, C., Chen, Y., et al. (2015). Estudio de prevalencia y tipificación genética del virus de la diarrea viral bovina (BVDV) en cuatro especies bovinas en China. PLoS One 10:e0121718. doi: 10.1371/journal.pone.0121718
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Guo, Y. (2007). Xinjiang frontera norte intensificación parcial granja de ganado Virus de la diarrea viral bovina Investigación epidemiológica molecular. [disertación/tesis de maestría]. [Shihezi (Región Autónoma Uigur de Xinjiang)]: Universidad Shihezi.
Guo, A. (2018). Los últimos datos de la encuesta epidemiológica del virus de la diarrea viral bovina (BVDV) en China publicados en Shanghai. China Dairy Cattle 342:35. doi: cnki: sun: zgnn.0.2018-10-010
He, Y., Ma, X., Huang, X., Sheng, J., Zhong, F., Zhao, X., et al. (2020). Detección de BVDV 1q en China: caracterización genética e infección experimental para la investigación de su patogenicidad. Kafkas Univ. Erg. 26, 377–384. DOI: 10.9775/KVFD.2019.23273
Hou, P., Zhao, G., Wang, H. y He, H. (2019). Prevalencia del virus de la diarrea viral bovina en rebaños de ganado lechero en el este de China. Trop. Anim. Salud Prod. 51, 791–798. DOI: 10.1007/S11250-018-1751-Z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Krametter-Froetscher, R., Duenser, M., Preyler, B., Theiner, A., Benetka, V., Moestl, K., et al. (2010). Infección por pestivirus en ovejas y cabras en Austria occidental. Veterinario J. 186, 342–346. doi: 10.1016/j.tvjl.2009.09.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Kumar, S., Stecher, G., Li, M., Knyaz, C. y Tamura, K. (2018). MEGA X: análisis de genética evolutiva molecular a través de plataformas informáticas. Mol. Biol. Evol. 35, 1547–1549. doi: 10.1093/molbev/msy096
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Lackner, T., Müller, A., Pankraz, A., Becher, P., Thiel, H., Gorbalenya, A., et al. (2004). La modulación temporal de una autoproteasa es crucial para la replicación y patogenicidad de un virus de ARN. J. Virol. 78, 10765–10775. doi: 10.1128/JVI.78.19.10765-10775.2004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Lee, K., y Gillespie, J. (1957). Propagación del virus diahea virus del ganado bovino en cultivo de tejidos. Am. J. Vet. 18, 952–953.
Li, N., Han, M., Huang, X., Bo, X., Wang, X., Zhao, Y., et al. (2009). Investigación epidemiológica del virus de la diarrea viral bovina en Shihezi, Xinjiang. Universidad J. Shihezi 27, 706–711. doi: 10.13880/j.cnki.65-1174/n.2009.06.018
Li, Y., Liu, Z. y Wu, Y. (1983). Aislamiento e identificación de la cepa del virus de la enfermedad de la mucosa de la enfermedad viral bovina (Changchun 184). Chin. J. Vet. 3, 113–120. doi: 10.16303/j.cnki.1005-4545.1983.02.001
McGowan, M., McCosker, K., Fordyce, G. y Kirkland, P. (2020). Epidemiología y manejo del BVDV en rebaños de cría de carne de corral en el norte de Australia. Virus 12:1063. DOI: 10.3390/v12101063
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Nie, Z., Tian, F. y Jiang., S., (2012). Aislamiento, identificación y purificación de un virus de la diarrea viral bovina de genotipo 2 en células MDBK. Acta Agric. Boreali-occidentalis Sin. 21, 21–25. doi: 10.3969/j.issn.1004-1389.2012.01.004
Olafson, P., Maccallum, A., y Fox, F. (1946). Una enfermedad transmisible aparentemente nueva del ganado. Veterinario de Cornell. 36, 205–213.
Pinior, B., García, S., Minviel, J. y Raboisson, D. (2019). Factores epidemiológicos y medidas de mitigación que influyen en las pérdidas de producción en el ganado bovino debido a la infección por el virus de la diarrea viral bovina: un metanálisis. Transbound. Emerg. Dis. 66, 2426–2439. doi: 10.1111/tbed.13300
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Qiu, C., Gao, S., Zhou, J., Cheng, S., Li, Y., Chen, G., et al. (1998). Monitoreo del estado de prevalencia del BDV-MD en rebaños de ganado vacuno en China. Mentón. Vet. Sci. 08, 15–16. doi: 10.16656/j.issn.1673-4696
Qiu, C., Guo, H., Cheng, S., Wang, Y., Gao, S., Zhou, J., et al. (2000). Monitoreo serológico de la diarrea viral bovina / enfermedad mucosa en búfalos en las regiones parciales de las provincias de Anhui, Jiangsu y Guangxi. Chin. J. Preventive Vet. Med. 06, 54–55. doi: 10.3969/j.issn.1008-0589.2000.06.017
Ran, X., Chen, X., Ma, L., Wen, X., Zhai, J., Wang, M., et al. (2019). Una revisión sistemática y metanálisis de la epidemiología de la infección por el virus de la diarrea viral bovina (BVDV) en ganado lechero en China. Acta Trop. 190, 296–303. doi: 10.1016/j.actatropica.2018.08.031
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ren, Y. (2010). Mecanismo molecular de la proteína E2 de la envoltura del virus de la diarrea viral bovina que interactúa con las células del trofoblasto bovino. [disertación/tesis doctoral]. [Shihezi (Región Autónoma Uigur de Xinjiang)]: Universidad Shihezi
Richter, V., Lebl, K., Baumgartner, W., Obritzhauser, W., Käsbohrer, A. y Pinior, B. (2017). Una revisión mundial sistemática de las pérdidas monetarias directas en el ganado debido a la infección por el virus de la diarrea viral bovina. Veterinario J. 220, 80–87. doi: 10.1016/j.tvjl.2017.01.005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Saliki, J., Huchzermeier, R. y Dubovi, J. (2000). Evaluación de un nuevo kit ELISA sándwich que utiliza suero para la detección de ganado persistentemente infectado con el virus BVD. Ann. N. Y. Acad. 916, 358–363. doi: 10.1111/j.1749-6632.2000.tb05313.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Schirrmeier, H., Strebelow, G., Depner, K., Hoffmann, B. y Beer, M. (2004). Caracterización genética y antigénica de un aislado de pestivirus atípico, un miembro putativo de una nueva especie de pestivirus. J. General Virol. 85, 3647–3652. doi: 10.1099/vir.0.80238-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Shang, Y., Liu, H., Zhang, H., Gao, M., Zhang, W. y Wang, J. (2013). Investigación serológica de la diarrea viral bovina-enfermedad mucosa en granjas lecheras a escala en el noreste de China. Chin. J. Preventive Vet. Med. 35, 559–561. doi: 10.3969/j.issn.1008-0589.2013.07.11
Smith, D., Meyers, G., Bukh, J., Gould, E., Monath, T., Scott, M., et al. (2017). Propuesta de revisión de la taxonomía del género Pestivirus, familia Flaviviridae. J. General Virol. 98, 2106–2112. DOI: 10.1099/jgv.0.000873
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Tamura, K., Nei, M. y Kumar, S. (2004). Perspectivas para inferir filogenias muy grandes utilizando el método de unión de vecinos. Proc. Natl. Acad. Sci. U. S. A. 101, 11030–11035. DOI: 10.1073/PNAS.0404206101
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Tian, B., Cai, D., Li, W., Bu, Q., Wang, M., Ye, G., et al. (2021). Identificación y genotipado de un nuevo subtipo de virus de la diarrea viral bovina 1 aislado de bovinos con diarrea. Arq. Virol. 166, 1259–1262. DOI: 10.1007/S00705-021-04990-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Wang, L., Liang, X., Ji, X., Shi, Q. y Rang, D. (2015). Identificación del aislamiento del virus de la diarrea viral bovina e investigación de epidemiología molecular en algunas áreas de Xinjiang. Xinjiang Agric. 52, 334–338. doi: 10.6048/j.issn.1001-4330.2015.02.024
Wang, X., Tu, C., Li, H., Jin, K., Xuan, H., Chang, G., et al. (1996). Cerdos naturalmente infectados por el virus de la diarrea bovina PresentSignos que se asemejan al cólera porcino. Chino. J. Vet. Sci. 16, 341–345. doi: 10.16303/j.cnki.1005-4545
Red Geofísica de Xinjiang (2014). Visión general de los recursos naturales en Xinjiang. Disponible en: https://www.xjtonglan.com/qjgk/zrhj/1114.shtml (consultado el 31 de agosto de 2014).
Yang, N., Zhang, J., Xu, M., Yi, J., Wang, Z., Wang, Y., et al. (2022). Las vacunas de partículas similares a virus basadas en glicoproteínas E0 y E2 del virus de la diarrea viral bovina inducen respuestas humorales. Frente. Microbiol. 13:1047001. doi: 10.3389/fmicb.2022.1047001
Yeşilbağ, K., Alpay, G. y Becher, P. (2017). Variabilidad y distribución global de los subgenotipos del virus de la diarrea viral bovina. Virus 9:128. DOI: 10.3390/V9060128
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Yue, X., Wu, J., Van, D., Steeneveld, W. y Hogeveen, H. (2022). Estimación del efecto de un programa de control del virus de la diarrea viral bovina: un estudio empírico sobre el rendimiento de los rebaños lecheros holandeses. Frente. Vet. Sci. 9:962. doi: 10.3389/fvets.2022.892928
Zhang, K., Zhang, J., Qiu, Z., Zhang, K., Liang, F., Zhou, Q., et al. (2022). Prevalencia característica del BVDV en algunas granjas lecheras a gran escala en el oeste de China. Frente. Vet. Sci. 9:961337. DOI: 10.3389/FVETS.2022.961337
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhao, S. (2015). La patogénesis y los síntomas clínicos de la diarrea viral bovina. Moderno An. Husb. Sci. Technol. 1:107. doi: 10.3969/j.issn.1673-1921.2015.01.093
Zheng, Z., Liu, P., Zheng, Z., Wang, Y. y Du, Y. (1991). Informe de investigación sobre anticuerpos neutralizantes del suero contra la diarrea viral bovina/enfermedad mucosa. Chin. An. Health Inspection 05, 42–44.
Zhong, F., Li, N., Huang, X., Guo, Y., Chen, H., Wang, X., et al. (2011). Tipificación genética y observación epidemiológica del virus de la diarrea viral bovina en el oeste de China. Virus Genes 42, 204–207. DOI: 10.1007/S11262-010-0558-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhou, Y. (2018). Estudio sobre el modelo de gestión de las cooperativas profesionales de pastores, basado en la investigación de las cuatro áreas de pastoreo de pastizales en Mongolia Interior, Xinjiang, Qinghai, Gansu. [disertación/tesis de maestría]. [Lanzhou (Gansu)]: Universidad Lanzhou Jiaotong.
Zhou, Y., Ren, Y., Dai, G., Li, X., Xiang, Y., Zhang, J., et al. (2022). Caracterización genética y características clínicas de los virus de la diarrea viral bovina en rebaños bovinos de la provincia de Heilongjiang, China. Irán. J. Vet. 23, 69–73. doi: 10.22099/IJVR.2021.38650.5625
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhu, L., Lu, H., Cao, Y., Gai, X., Guo, C., Liu, Y., et al. (2016). Caracterización molecular de un nuevo aislado de virus de la diarrea viral bovina SD-15. PLoS One 11:e0165044. doi: 10.1371/journal.pone.0165044
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cita: Yang N, Xu M, Ma Z, Li H, Song S, Gu X, Liu J, Yang Z, Zhu H, Ma H, Yi J, Wang Y, Wang Z, Sheng J y Chen C (2023) Detección de Pestivirus emergente similar a HoBi (BVD-3) durante una investigación epidemiológica del virus de la diarrea viral bovina en Xinjiang: un informe primero en su tipo. Frente. Microbiol. 14:1222292. doi: 10.3389/fmicb.2023.1222292
Recibido: 14 de mayo de 2023; Aprobado: 20 de junio de 2023;
Publicado: 04 Julio 2023.
Editado por:
Qin Zhao, Universidad Northwest A&F, China
Revisado por:
Beibei Zhang, Universidad de Xinjiang, China
Gaopeng Hou, Universidad de Washington en St. Louis, Estados Unidos
Derechos de autor © 2023 Yang, Xu, Ma, Li, Song, Gu, Liu, Yang, Zhu, Ma, Yi, Wang, Wang, Sheng y Chen. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Chuangfu Chen, ccf@shzu.edu.cn; Jinliang Sheng, 1572621211@qq.com
†Estos autores han contribuido igualmente a este trabajo
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.