El análisis de secuenciación de ARN reveló genes expresados diferencialmente y su anotación funcional en el músculo longissimus dorsi porcino
El análisis de secuenciación de ARN reveló genes expresados diferencialmente y su anotación funcional en el músculo longissimus dorsi porcino afectado por la restricción de lisina en la dieta
 Dr. Shamimul Hasan1
Dr. Shamimul Hasan1  Ying Wang2
Ying Wang2  Jean M. Feugnag1
Jean M. Feugnag1  Huaijun Zhou2
Huaijun Zhou2  Shengfa F. Liao1*
Shengfa F. Liao1*- 1Departamento de Ciencias Animales y Lecheras, Universidad Estatal de Mississippi, Starkville, MS, Estados Unidos
- 2Departamento de Ciencia Animal, Universidad de California, Davis, Davis, CA, Estados Unidos
El objetivo de este estudio fue investigar los efectos de la restricción de lisina en la dieta sobre el perfil global de expresión génica del músculo esquelético en cerdos en crecimiento. Doce túmulos cruzados (Yorkshire × Landrace) (peso corporal inicial 22,6 ± 2,04 kg) fueron asignados aleatoriamente a dos tratamientos dietéticos (LDD: una dieta deficiente en lisina; LAD: una dieta adecuada a la lisina) según un diseño experimental completamente aleatorizado (n = 6). Después de la alimentación durante 8 semanas, se tomaron muestras de músculo esquelético del longissimus dorsi de cerdos individuales. Se aisló el ARN total del músculo y se prepararon bibliotecas de ADNc para el análisis de secuenciación de ARN (RNA-Seq). Los datos de RNA-Seq obtenidos se analizaron utilizando CLC Genomics Workbench para identificar genes expresados diferencialmente (DEG). Un total de 80 genes (padj ≤ 0,05) se expresaron diferencialmente en el músculo longissimus dorsi de los cerdos alimentados con LDD frente a LAD, de los cuales 46 genes estaban regulados a la baja y 34 genes estaban regulados al alza. El análisis de ontología génica (GO) de los DEG (padj ≤ 0,05) para la anotación funcional identificó aquellos términos GO que se asocian principalmente con las funciones moleculares de las moléculas estructurales y las enzimas metabólicas (p. ej., oxidorreductasa y endopeptidasa), el proceso biológico de respuesta de fase aguda y el metabolismo de los aminoácidos, incluida la síntesis y degradación en la región de la matriz extracelular. En conjunto, los resultados de este estudio han proporcionado información novedosa sobre los mecanismos moleculares del crecimiento muscular que están asociados con el suministro de lisina en la dieta.
1. Introducción
La lisina, un nutriente esencial para los cerdos, suele ser el primer aminoácido limitante (AA) en las dietas porcinas comunes a base de granos. Los estudios han demostrado que la deficiencia dietética de lisina puede conducir a una reducción del rendimiento del crecimiento en los cerdos debido a la disminución de la síntesis de proteínas corporales y al aumento de la deposición de grasa (1, 2). Por lo tanto, en la industria porcina, las dietas de los cerdos se suministran comúnmente con la lisina libre no unida a proteínas (llamada lisina cristalina) para garantizar que los cerdos reciban suficiente lisina para su crecimiento óptimo (3).
A nivel biológico molecular, la lisina tiene efectos reguladores sobre la expresión génica, lo que puede alterar las vías metabólicas y de señalización animal asociadas con el metabolismo de proteínas y lípidos (4). Por ejemplo, Wang et al. (2) informaron que la deficiencia de lisina en la dieta puede activar la vía de ubiquitinación para aumentar la degradación de proteínas musculares y regular al alza la expresión de genes asociados con la biosíntesis de lípidos en cerdos de engorde. Además, Jin et al. (4, 5) también encontraron que la deficiencia de lisina en la dieta inhibía la proliferación de células satélite y reducía la síntesis de proteínas a través de la inhibición de la vía de señalización mTOR en el músculo longissimus dorsi de cerdos destetados. Sin embargo, no se ha informado del perfil global de expresión génica ni de las respuestas transcripcionales a la restricción o deficiencia de lisina en la dieta en cerdos en crecimiento.
El transcriptoma de un tejido específico bajo una determinada condición ambiental, nutricional y fisiológica comprende todos los tipos de ARN que incluyen ARN codificantes y no codificantes (6, 7). En los últimos años, la secuenciación de ARN (RNA-Seq), que utiliza tecnología de secuenciación de próxima generación, se ha empleado como una poderosa herramienta para realizar un análisis y cuantificación exhaustivos de todas las especies de ARN expresadas en tejidos o células (7-9). El objetivo principal de este estudio fue utilizar RNA-Seq para investigar los efectos de la restricción de lisina en la dieta sobre el perfil global de expresión génica en el músculo longissimus dorsi de cerdos jóvenes en crecimiento. También se anotaron las funciones de esos genes expresados diferencialmente (DEGs).
2. Materiales y métodos
2.1. Animales y tratamientos dietéticos
Se compraron doce túmulos de crecimiento cruzados (Yorkshire × Landrace) (peso inicial de 22,6 ± 2,04 kg) de Prestage Farms of Mississippi (West Point, MS), y se alojaron en un establo de cerdos con control ambiental en el Centro de Investigación de Animales Leveck de la Universidad Estatal de Mississippi. Los cerdos se distribuyeron aleatoriamente en 12 corrales de alimentación individuales y se les permitió aclimatarse al entorno del establo durante 1 semana, mientras se alimentaba con una dieta comercial para cerdos en crecimiento (ad libitum). Posteriormente, los cerdos fueron asignados a 2 tratamientos dietéticos (n = 6) de acuerdo con un diseño experimental completamente aleatorizado con corral o cerdo como unidad experimental.
Una dieta basada en harina de maíz y soja (una dieta deficiente en lisina; LDD) se formuló (Tabla 1) para cumplir o superar los requisitos recomendados por el NRC (10) para varios nutrientes, incluida la proteína bruta y varios AA esenciales, pero no la lisina. Una dieta de control (una dieta adecuada en lisina; LAD) se formuló mediante la adición de monoclorhidrato de L-lisina (la forma comercial comúnmente utilizada de lisina cristalina) a la LDD a una tasa del 0,40%. No se hizo ningún esfuerzo para mantener las proporciones constantes de otros AA esenciales de la dieta en relación con la lisina. Las muestras de dietas se recogieron varias veces durante el ensayo de alimentación, se mezclaron, se submuestrearon y se enviaron al Laboratorio de Nutrición Animal Essig de la Universidad Estatal de Mississippi para su análisis aproximado con el fin de confirmar el contenido de los principales nutrientes y energía. Además, se analizó la composición de AA de las dietas en un laboratorio analítico de Ajinomoto Heartland, Inc. (Chicago, IL). Los nutrientes analizados y la composición de AA de las dos dietas experimentales se presentan en la Tabla 2.
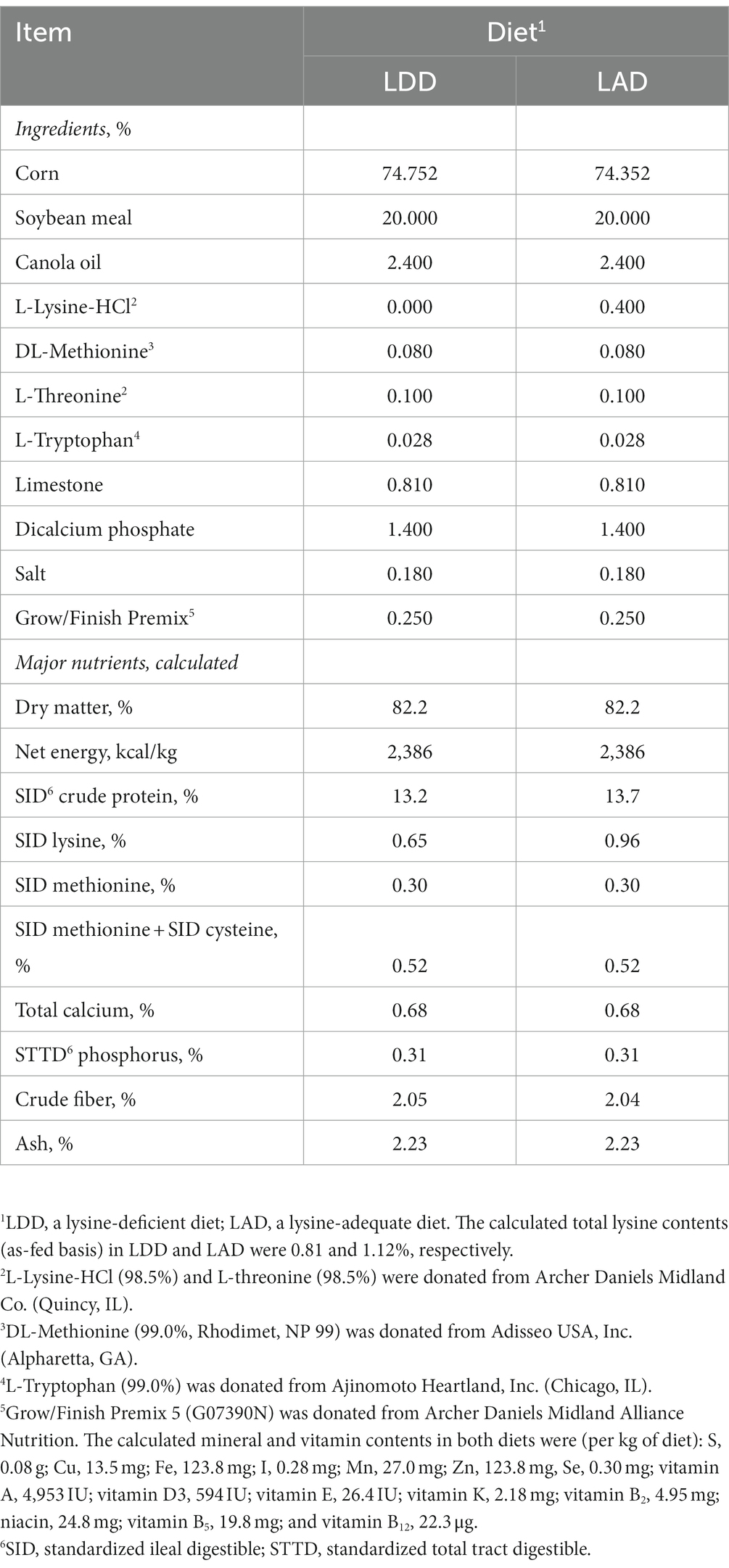 Tabla 1. Composición de las dos dietas experimentales suministradas a los cerdos en crecimiento (sobre la base de la alimentación).
Tabla 1. Composición de las dos dietas experimentales suministradas a los cerdos en crecimiento (sobre la base de la alimentación).
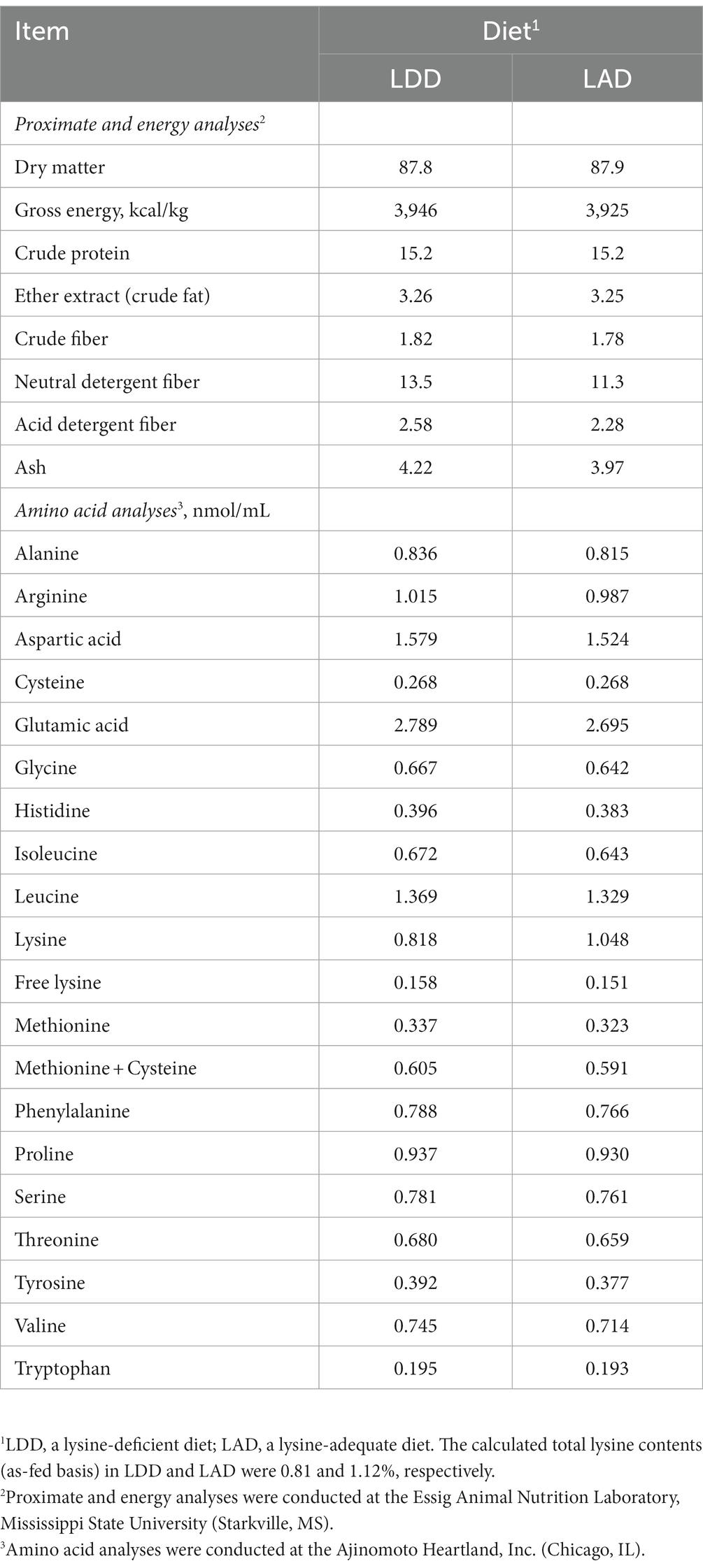 Tabla 2. Se analizó la composición de nutrientes (%, o según se indique) de las dos dietas experimentales administradas a los cerdos en crecimiento (sobre la base de la alimentación).
Tabla 2. Se analizó la composición de nutrientes (%, o según se indique) de las dos dietas experimentales administradas a los cerdos en crecimiento (sobre la base de la alimentación).
2.2. Ensayo con animales y recogida de muestras
El ensayo de alimentación animal duró 8 semanas, y los cerdos tuvieron acceso ad libitum a las dietas experimentales y al agua dulce durante el período de 8 semanas. Todos los cerdos, comederos y bebederos fueron revisados de 2 a 3 veces al día (0600 a 2000 h). Al final del ensayo, los cerdos fueron sacrificados en el Laboratorio de Ciencia de la Carne y Biología Muscular de la Universidad Estatal de Mississippi. Se recogieron muestras de músculo esquelético (aproximadamente 2 g/cerdo) de la porción media del longissimus dorsi (entre las costillas 10ª y 12ª) de cada cerdo, e inmediatamente se congelaron en nitrógeno líquido. A continuación, las muestras de músculo congeladas se almacenaron a -80 °C en un congelador hasta que se inició el análisis de RNA-Seq. Todos los protocolos experimentales relacionados con animales (por ejemplo, cuidado, manejo y tratamiento de cerdos) fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) de la Universidad Estatal de Mississippi.
2.3. Aislamiento de ARNm y construcción y secuenciación de bibliotecas de ADNc
El ARN total se extrajo de las muestras de músculo congeladas utilizando el reactivo TRIzol (Invitrogen Corporation, Carlsbad, CA) de acuerdo con las instrucciones del fabricante. Las muestras de ARN se purificaron aún más mediante un tratamiento con DNasa I (Ambion, Austin, TX) para evitar cualquier contaminación del ADN. Posteriormente, se evaluó la integridad del ARN (RIN) de las muestras de ARN purificadas utilizando el Bioanalizador Agilent 2100 (Agilent Technologies, Santa Clara, CA), y solo las muestras con valores RIN ≥7 se utilizaron para la preparación de la biblioteca de ADNc. Las concentraciones y la pureza de las muestras de ARN también se determinaron utilizando un espectrofotómetro NanoDrop ND-1000 (NanoDrop Technologies, Wilmington, DE).
Después de los análisis de control de calidad, se construyeron bibliotecas de ADNc de extremos emparejados (es decir, 250 pb) para cada muestra utilizando el NEBNext Ultra®™ Kit de preparación de la biblioteca de ARN direccional (New England Biolabs, Inc., Ipswich, MA) siguiendo los protocolos del fabricante. Para generar lecturas de extremos emparejados de 100 pb en formato FASTQ, se llevó a cabo una secuenciación múltiplex de las bibliotecas de ADNc en un analizador de secuencias HiSeq 4000 (llumina, San Diego, CA) en el Centro del Genoma del Núcleo de Tecnologías de ADN de la Universidad de California, Davis, CA.
2.4. Análisis bioinformático e interpretación de datos
2.4.1. Mapeo de secuencias y expresión génica diferencial
Los datos brutos de RNA-Seq se analizaron utilizando CLC Genomic Workbench (versión 22.0; Qiagen, Germantown, MD). La calidad de las lecturas de la secuencia se comprobó primero mediante la canalización de control de calidad con los parámetros predeterminados del programa, y las lecturas de alta calidad se compararon con la versión reciente del genoma de referencia porcino, Sus scrofa 11.1, alojada en una base de datos genómica del Centro Nacional de Información Biotecnológica (NCBI).1 A continuación, los datos de expresión génica se normalizaron mediante el cálculo de los fragmentos por kilobase por millón de lecturas mapeadas (FPKM). A continuación, se utilizó la herramienta analítica DEG del CLC Genomic Workbench para realizar análisis estadísticos, para lo cual se implementó una ‘Prueba Exacta’ modificada, desarrollada por Robinson y Smyth (11) e incorporada en el paquete Edge R Bioconductor por Robinson et al. (12). Los valores p obtenidos se corrigieron mediante el procedimiento de prueba múltiple de Benjamini-Hochberg (B-H).
2.4.2. Enriquecimiento génico y análisis funcional de genes expresados diferencialmente
Para el análisis de Gene Ontology (GO), se utilizó la herramienta de análisis funcional basada en la web, el programa bioinformático DAVID (Database for Annotation, Visualization, and Integrated Discovery), para analizar el enriquecimiento génico y la anotación funcional del DEG. Brevemente, se cargó una lista de nombres de DEG y se almacenó en el panel centralizado de administración de listas de DAVID. Dado que el programa es una herramienta que no distingue entre mayúsculas y minúsculas para todas las accesiones (es decir, las identificaciones), las identificaciones de DEG se convirtieron primero en identificaciones del gen DAVID; Se utilizaron un total de 78 (80) ID de DAVID para un análisis funcional posterior, mientras que la anotación del genoma de Sus scrofa se seleccionó como especie de fondo.
Se generaron informes para el gráfico de anotaciones funcionales para las Funciones Moleculares (MF), los Procesos Biológicos (BP), los Componentes Celulares (CC) y el enriquecimiento de la vía de la Enciclopedia de Genes y Genomas de Kioto (KEGG). Se utilizó la prueba exacta de Fisher con los identificadores del gen DAVID, y solo se mostraron en la tabla los resultados que cumplían con criterios específicos (como una probabilidad máxima de 0,1 y un recuento mínimo de 2). El umbral EASE Score, que es un valor exacto de Fisher modificado de p, se utilizó para analizar esos genes y vías enriquecidas.
3. Resultados
3.1. Mapeo e identificación de genes expresados diferencialmente
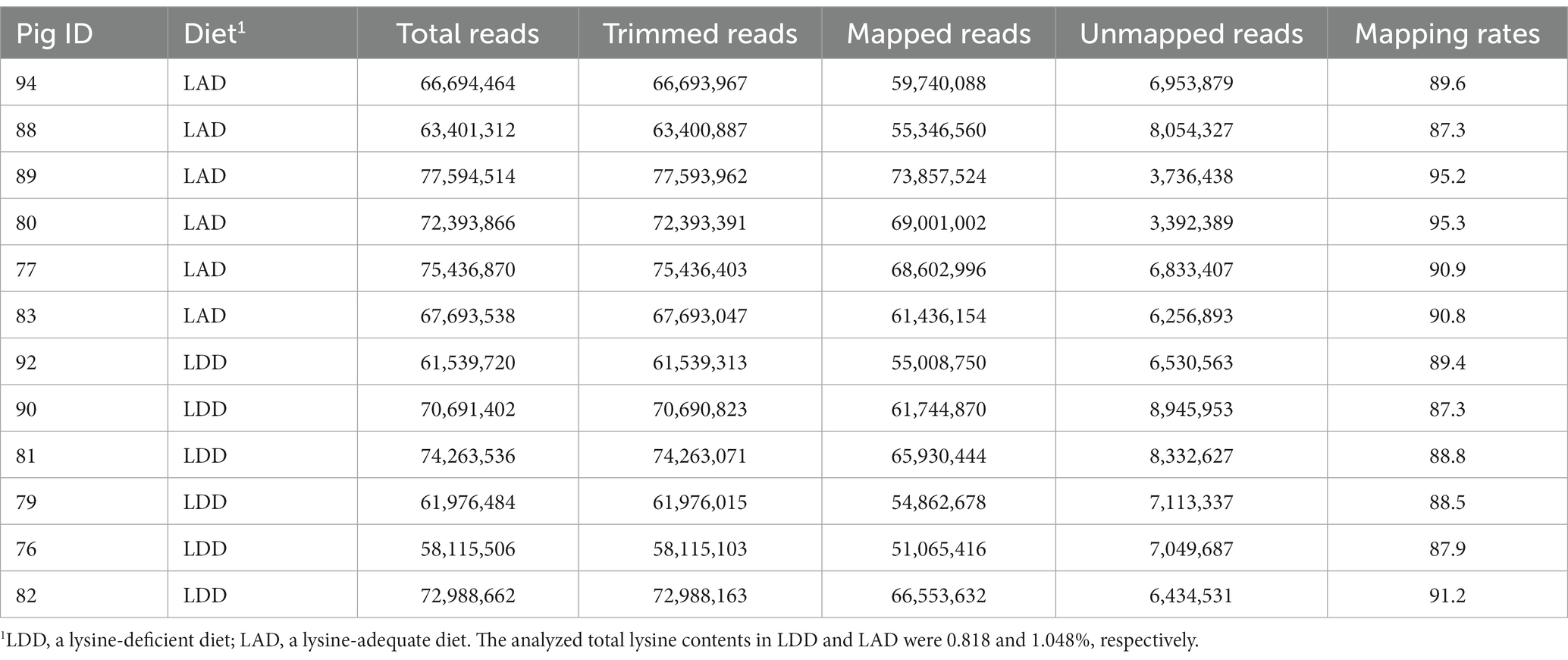
Como se muestra en la Tabla 3, el análisis de RNA-Seq generó aproximadamente 822 millones de lecturas sin procesar de las doce muestras de tejido de longissimus dorsi (es decir, 6 cerdos en el grupo de LAD y 6 cerdos en el grupo de LDD), y estos datos de secuencia sin procesar se han enviado al Archivo de Lectura de Secuencias (SRA) del NCBI y están disponibles bajo el número de acceso de BioProject PRJNA975688. Tras el control de calidad, se eliminaron aproximadamente 59 millones de lecturas de secuencias ambiguas y de baja calidad, y las lecturas de secuencias limpias resultantes se utilizaron para el análisis. La alineación de la secuencia con el genoma del cerdo produjo tasas de mapeo del 87,3 al 95,3% de las lecturas de secuencia alineadas de forma única en pares. Las lecturas de secuencia alineadas en pares rotos y las no mapeadas se excluyeron del análisis posterior.
Los DEG se identificaron utilizando el modo de «contra el grupo de control» en el CLC Genomic Workbench. Este modo prueba las diferencias en el nivel de expresión génica mediante «pruebas de Wald» entre el grupo experimental (es decir, el grupo LDD) y el grupo de control (es decir, el grupo LAD). Se identificaron un total de 943 DEGs (p ≤ 0,05) que incluyen 101 genes regulados al alza (Log2 Cambio de pliegue (FC) ≥ 1) y 337 genes regulados a la baja (Log2 FC ≤ −1) en el músculo longissimus dorsi de los cerdos del grupo LDD (Figura 1). A continuación, se ajustaron los valores de p (padj) mediante el método de Benjamini-Hochberg (B-H). Se encontró que un total de 80 genes (padj ≤ 0,05) se expresaban diferencialmente debido a la restricción de lisina en la dieta. Los nombres de los genes, las descripciones, los números de identificación de Ensembl y los cambios en el plegamiento se enumeran en la Tabla Suplementaria S1.
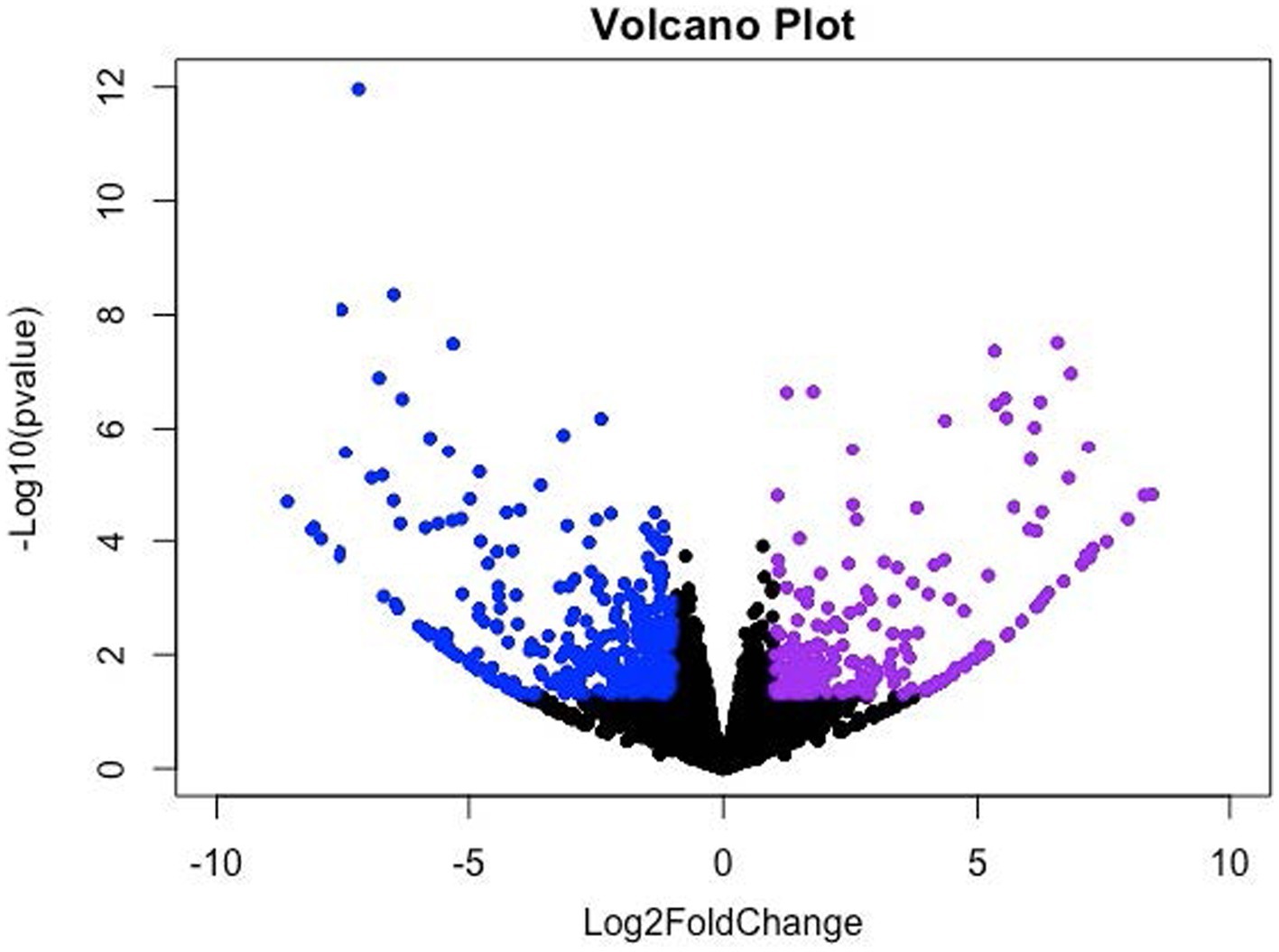 Figura 1. Diagrama de volcán (dispersión) para genes significativamente regulados al alza y a la baja. El eje X y el eje Y denotan el registro2 Cambio de pliegue (FC) y −log10 de los valores p ajustados (Padj), respectivamente; donde −1 ≥ log2FC ≥ 1.0 y padj < 0.05 se consideraron cambios significativos, los cuales se indican en color violeta (regulado al alza) y azul (regulado a la baja).
Figura 1. Diagrama de volcán (dispersión) para genes significativamente regulados al alza y a la baja. El eje X y el eje Y denotan el registro2 Cambio de pliegue (FC) y −log10 de los valores p ajustados (Padj), respectivamente; donde −1 ≥ log2FC ≥ 1.0 y padj < 0.05 se consideraron cambios significativos, los cuales se indican en color violeta (regulado al alza) y azul (regulado a la baja).
De estos 80 genes, 46 genes estaban regulados a la baja y 34 genes estaban regulados al alza (Figura 2). Los 25 genes principales regulados a la baja y los 25 genes regulados al alza se muestran en las Tablas 4 y 5, respectivamente. Los genes regulados a la baja codifican varias proteínas funcionales, como proteínas de transporte (TTR, ALB), lipoproteínas (APOE, APOC3, APOH), proteínas de la familia del citocromo p450 (CYP3A22, CYP2E1), enzimas metabólicas AA (HAP, TAT) y fibrinógenos (FGA, FGB). Por otro lado, los genes regulados al alza codifican aquellas proteínas que incluyen principalmente proteínas estructurales (isoformas de queratina: KRT 25, 31, 74), proteoglicanos (HAPLN1, ACAN), proteínas de la familia del colágeno (COL2A1, COL9A1), y también las enzimas relacionadas con AA y el metabolismo energético (PHGDH, PSAT1).
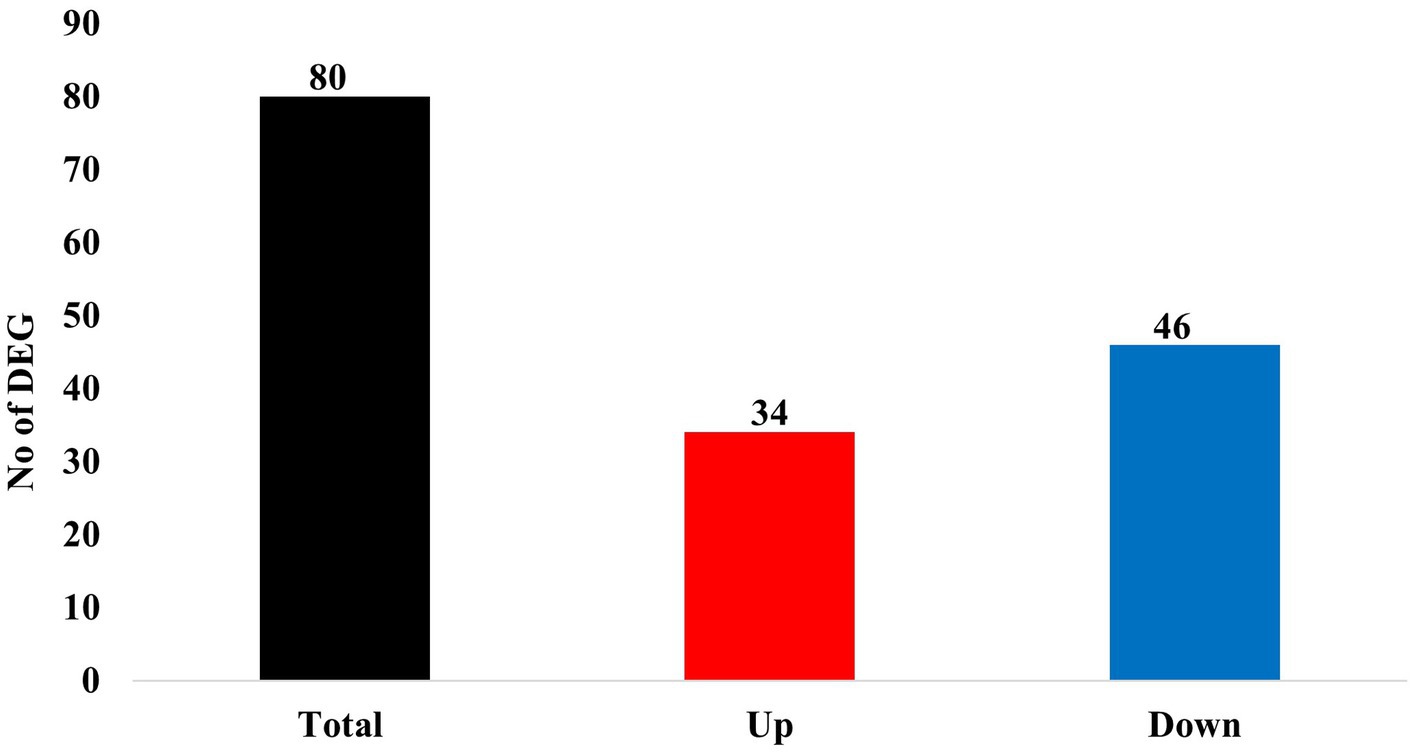 Figura 2. Resumen de los genes expresados diferencialmente (DEGs) en el músculo esquelético de cerdos en crecimiento afectados por la restricción de lisina en la dieta (padj ≤ 0,05).
Figura 2. Resumen de los genes expresados diferencialmente (DEGs) en el músculo esquelético de cerdos en crecimiento afectados por la restricción de lisina en la dieta (padj ≤ 0,05).
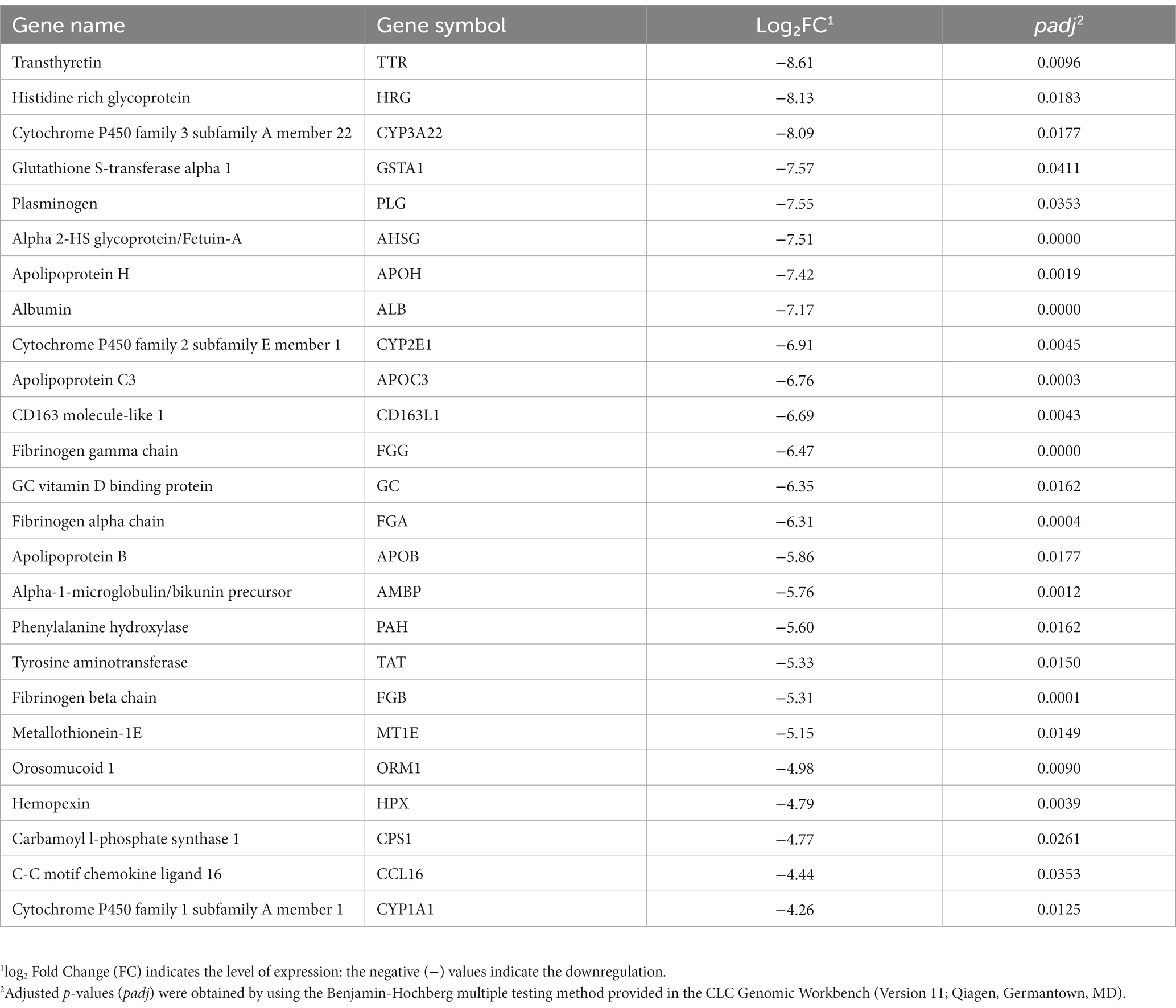 Tabla 4. Los 25 genes principales regulados a la baja en el músculo longissimus dorsi de los cerdos en crecimiento alimentados con una dieta deficiente en lisina frente a una dieta adecuada en lisina.
Tabla 4. Los 25 genes principales regulados a la baja en el músculo longissimus dorsi de los cerdos en crecimiento alimentados con una dieta deficiente en lisina frente a una dieta adecuada en lisina.
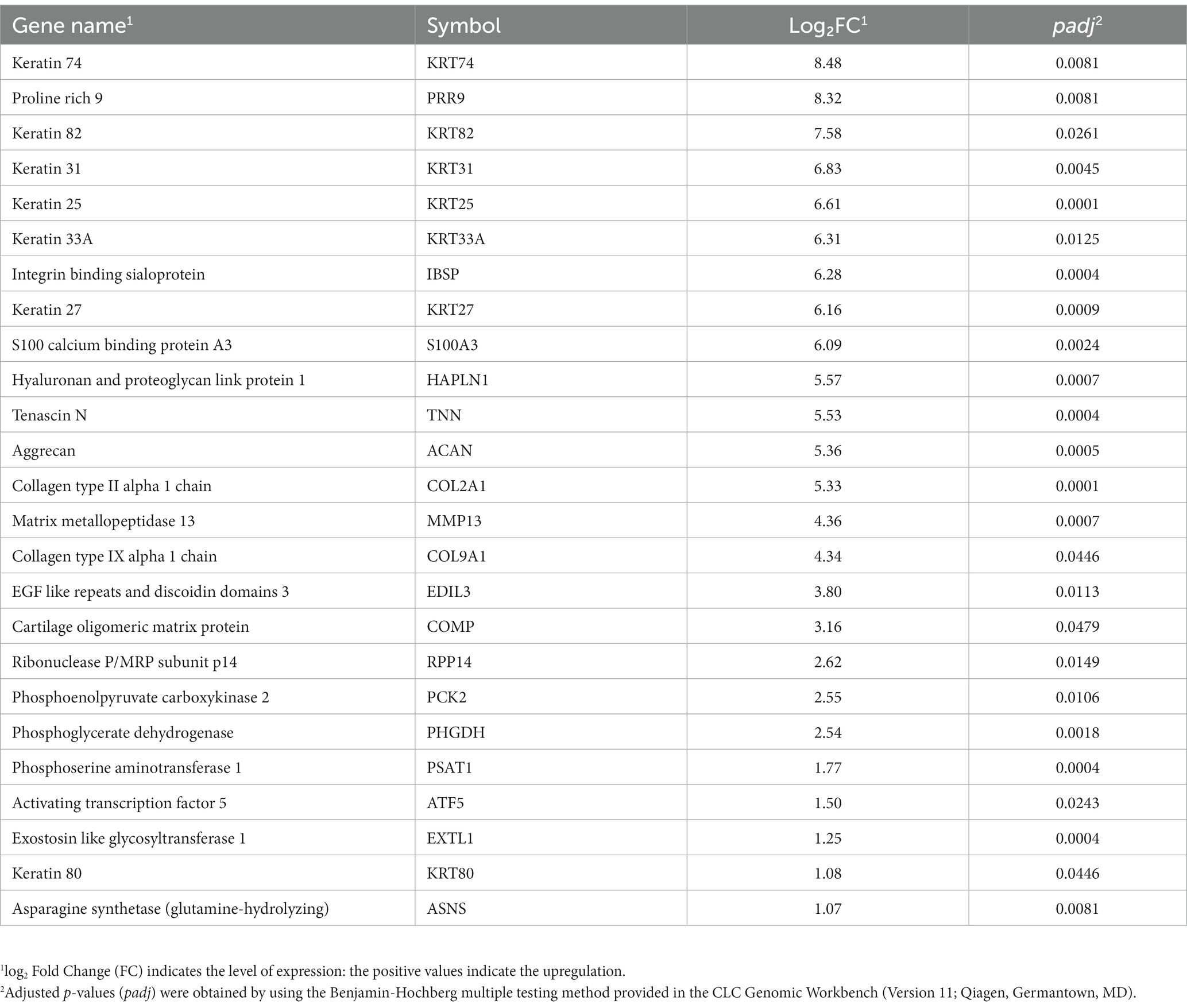 Tabla 5. Los 25 genes principales regulados al alza en el músculo longissimus dorsi de los cerdos en crecimiento alimentados con una dieta restringida en lisina frente a una dieta adecuada en lisina.
Tabla 5. Los 25 genes principales regulados al alza en el músculo longissimus dorsi de los cerdos en crecimiento alimentados con una dieta restringida en lisina frente a una dieta adecuada en lisina.
3.2. Anotación funcional de los genes expresados diferencialmente
Para comprender las funciones biológicas relacionadas con estos DEGs, se realizó un análisis de anotación funcional con el programa de bioinformática DAVID. La herramienta de conversión de accesión génica en DAVID fue capaz de convertir 78 de 80 DEG (97,5%) en ID de gen DAVID. Los resultados del análisis GO de los DEG demuestran que la alteración de los niveles de expresión génica por la restricción de lisina en la dieta puede afectar a las vías celulares BP, MF y KEGG. Como se muestra en la Tabla Suplementaria S2, los resultados de la anotación funcional génica se clasificaron en las vías MF, BP, CC y KEGG y se describieron en tipos y términos de GO. El análisis de enriquecimiento para MF muestra que el 60% (47 de 78) genes están enriquecidos en varios términos de GO relacionados con MF. Se encontraron 61 registros de términos MF GO, de los cuales el 78% coinciden con los antecedentes de la población porcina. Como se muestra en la Figura 3, los principales términos de GO relacionados con MF incluyen la actividad de la molécula estructural, la actividad inhibidora de la endopeptidasa de tipo serina, la unión a moléculas pequeñas, la unión al hemo, la actividad oxidorreductasa, el constituyente estructural de la matriz extracelular (MEC), la unión al proteoglicano de sulfato de heparán y la actividad inhibidora de las endopeptidasas de tipo cisteína.
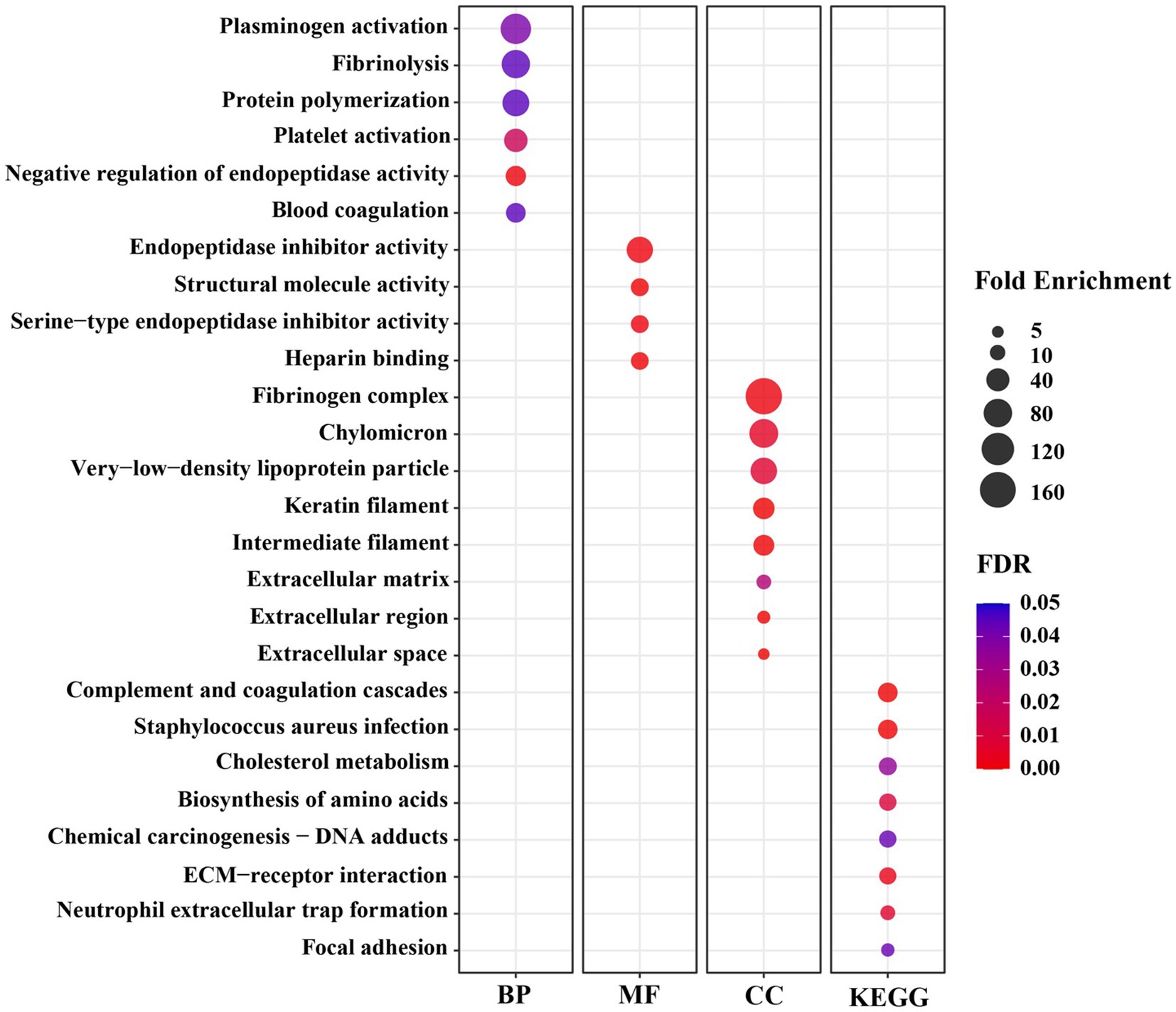 Figura 3. Los términos significativos de la Ontología Génica (GO) enriquecidos por genes expresados diferencialmente (DEGs) en el músculo longissimus dorsi de cerdos en crecimiento alimentados con una dieta restringida en lisina frente a una dieta adecuada a la lisina. BP, Procesos Biológicos; MF, Funciones Moleculares; CC: Componentes Celulares; KEGG, Enciclopedia de Genes y Genomas de Kioto.
Figura 3. Los términos significativos de la Ontología Génica (GO) enriquecidos por genes expresados diferencialmente (DEGs) en el músculo longissimus dorsi de cerdos en crecimiento alimentados con una dieta restringida en lisina frente a una dieta adecuada a la lisina. BP, Procesos Biológicos; MF, Funciones Moleculares; CC: Componentes Celulares; KEGG, Enciclopedia de Genes y Genomas de Kioto.
El 57% de los DEG (Figura 3) se enriquecieron significativamente en diferentes términos de GO relacionados con la PA (Figura <>), con términos que incluyen la respuesta de fase aguda, la adhesión celular, la regulación positiva de la fagocitosis, la regulación negativa de la actividad endopeptidasa, el proceso metabólico heterociclo, el transporte hemo, el proceso biosintético de L-serina, el desarrollo del cartílago de la placa de crecimiento, la regulación negativa del crecimiento, la regulación de la fosforilación de peptidil-tirosina, la polimerización de proteínas, la lipoproteína de alta densidad remodelación de partículas, y proceso metabólico de glutamina.
Los genes indicadores de CC (58% de los DEGs) se detectaron como altamente enriquecidos con los siguientes términos GO (Figura 3): micropartícula sanguínea, espacio extracelular, región extracelular, exosoma extracelular, matriz extracelular, filamento de queratina, filamento intermedio y complejo fibrinógeno. El análisis de la vía KEGG de estas 78 ID de DAVID reveló que varias vías metabólicas y de señalización celular podrían verse afectadas por la restricción de lisina en la dieta (Figura 3), de las cuales las 5 principales son responsables de las cascadas del complemento y la coagulación, la carcinogénesis química, la biosíntesis de aminoácidos, el metabolismo de los xenobióticos por el citocromo P450 y las vías de biosíntesis de hormonas esteroideas.
4. Discusión
La lisina suele ser el primer AA esencial limitante en las dietas porcinas a base de granos. Se sabe que la deficiencia de lisina en la dieta afectará negativamente no solo al depósito de proteínas del músculo de los cerdos, la mayor parte de la masa corporal, sino también a la salud general y al rendimiento del crecimiento (1, 2). Los datos de rendimiento de crecimiento generados a partir de este estudio se han informado anteriormente, lo que mostró que la ganancia de peso diaria promedio y la relación ganancia a alimento disminuyeron significativamente en los cerdos alimentados con LDD que en los alimentados con LAD, aunque no hubo diferencia en la ingesta diaria promedio de alimento entre los dos grupos de cerdos (3). La reducción de peso observada en este estudio debe ser consecuencia de la mala utilización de los nutrientes de la dieta por parte de los cerdos y, especialmente del AA proteinogénico, para la biosíntesis de proteínas corporales y el crecimiento muscular, ya que el músculo esquelético es el órgano más grande, así como la mayor reserva de proteínas, en el cuerpo (1, 3). Para explorar más a fondo el mecanismo molecular responsable de la reducción del crecimiento (principalmente el crecimiento muscular) en respuesta a la restricción de lisina en la dieta, realizamos este perfil de expresión génica global para identificar los DEG que pueden tener diversas funciones moleculares asociadas con el crecimiento y desarrollo del músculo esquelético.
A través de este estudio, encontramos que la restricción de lisina en la dieta regulaba a la baja algunos genes que codifican proteínas transportadoras (Tabla 4), como la transtiretina (TTR) y la albúmina (ALB). Aunque los genes TTR y ALB se expresan en gran medida en el hígado de acuerdo con la anotación de genes humanos, Soprano et al. (13) y Wagatsuma et al. (14) informaron que estos genes también se expresaban en el músculo esquelético de los animales. El producto del gen TTR es una proteína transportadora homotetraérica de 55 kDa para T3 y T4 (ambas son hormonas tiroideas) en la sangre (15). Según Monk et al. (16), los ratones deficientes en TTR muestran una transición más lenta de la lactancia al destete, un crecimiento más lento, menos masa muscular y un crecimiento óseo deficiente. Además, algunos otros estudios demostraron que el TTR desempeña un papel en la promoción de la diferenciación de las células musculares y la reparación del tejido muscular después de una lesión (15, 17, 18). El producto del gen ALB es otro reactante de fase aguda y un transportador de proteínas no específico para numerosas hormonas existentes en la sangre. Tiene la mayor afinidad de unión para T3 y T4 seguida de TTR, y determina el tiempo de transporte y la distribución de las hormonas tiroideas a los tejidos diana. La hipoalbuminemia puede ser causada por una mala nutrición y una inflamación continua en los animales (19). Un estudio transversal reportó una asociación significativa entre la baja albúmina sérica y la baja fuerza muscular (20). Estos resultados sugieren que la reducción del recambio de proteínas del músculo esquelético y la alteración del metabolismo muscular local de las hormonas tiroideas pueden conducir a una disminución de la disponibilidad intracelular de T3 (21). Por lo tanto, la regulación negativa de los genes TTR y ALB en este estudio implica que una reducción en la expresión de los genes TTR y ALB puede ser un factor en el impacto negativo de una dieta restringida en lisina en el crecimiento muscular. Esto podría deberse a un impacto en aquellas proteínas encargadas de transportar hormonas como las hormonas tiroideas y otras moléculas de señalización.
La expresión de varios genes que codifican para los miembros de la familia del citocromo P450, como los miembros 450 y 1 de la subfamilia A del citocromo P1 de la familia 22 (CYP1A1 y CYP1A22) y el miembro 450 de la subfamilia E de la subfamilia E del citocromo P2 P1 (CYP2E1), disminuyó en los cerdos alimentados con la dieta de restricción de lisina (Tabla 4). Se sabe que las enzimas CYP1A1 y CYP2E1 metabolizan los ácidos grasos poliinsaturados para sintetizar moléculas de señalización celular intracelular biológicamente activas o mediadores lipídicos, como las resolvinas derivadas de ácidos grasos ω-3 y las maresinas (22). Algunos de los mediadores lipídicos son bien conocidos por su función en el mantenimiento de la masa muscular esquelética. Durante el estrés nutricional (por ejemplo, deficiencia), estas moléculas antiinflamatorias desempeñan un papel importante en la reducción del desgaste del músculo esquelético (23). En consecuencia, la disminución de la expresión de los genes que están asociados con el metabolismo de los ácidos grasos poliinsaturados y la síntesis de moléculas antiinflamatorias, puede contribuir negativamente al mantenimiento y la homeostasis del músculo esquelético.
De los genes regulados a la baja, la fenilalanina hidroxilasa (HAP) y la tirosina amino transferasa (TAT) están asociadas con el metabolismo del AA. La enzima PAH cataliza la conversión de L-fenilalanina en L-tirosina, lo que indica que la restricción de lisina en la dieta puede suprimir la actividad de la enzima PAH en cerdos. Durante mucho tiempo se ha descubierto que una actividad deprimida de HAP se asocia con una disminución de la capacidad para eliminar la carga de fenilalanina, lo que conduce a una formación de tirosina alterada (24). La tirosina se utiliza no solo en la síntesis de proteínas, sino también en la síntesis de algunos neurotransmisores (por ejemplo, dopamina, epinefrina y nor-epinefrina) y hormonas (por ejemplo, la hormona tiroidea). Además, la tirosina actúa como sustrato en reacción con el oxoglutarato catalizado por la enzima TAT para producir 4-hidroxifenilpiruvato y glutamato. El glutamato puede actuar como precursor de varios neurotransmisores (por ejemplo, GABA) (25). Por lo tanto, una disminución en la expresión de HAP y TAT en respuesta a la restricción de lisina en la dieta puede afectar indirectamente la producción de algunos neurotransmisores y hormonas asociados con el crecimiento animal.
El término «queratina» se conoce como proteínas intermedias formadoras de filamentos en el citoesqueleto celular, que pertenecen a las proteínas fibrilares. La queratina influye fundamentalmente en la arquitectura (por ejemplo, la polaridad celular y la forma celular) y la actividad mitótica de las células epiteliales para mantener su estrés mecánico, mantener su integridad estructural y establecer la polaridad celular (26, 27). Este estudio reveló que la restricción de lisina en la dieta reguló al alza la expresión de varias isoformas de genes de queratina, incluyendo la queratina 74 (KRT74), la queratina 82 (KRT82), la queratina 31 (KRT31), la queratina 25 (KRT25) y la queratina 33A (KRT33A) (Tabla 5). Como es sabido, la mayoría de las isoformas de queratina se anotan como específicas del cabello y la piel en humanos, una contaminación de nuestras muestras musculares por tejido de cabello o piel es muy poco probable debido a nuestro riguroso protocolo de preparación de muestras. Además, algunos estudios previos habían reportado la presencia de varias proteínas de filamentos intermedios tipo I y tipo II (conocidas como queratinas) en el músculo en desarrollo y estriado (28, 29). Por ejemplo, la queratina 19 (K19) y la queratina 8 (K8) son queratinas prominentes expresadas en el músculo estriado maduro, donde desempeñan un papel fundamental en el mantenimiento de la integridad estructural de las fibras musculares (30).
Sin embargo, no hay investigaciones previas que hayan demostrado un efecto regulador de la lisina sobre la transcripción y traducción del gen de la queratina in vivo. Es interesante encontrar que la lisina dietética puede desempeñar un papel en la actividad estructural de la queratina, lo que justifica una mayor investigación.
La MEC del músculo esquelético, una malla compleja que consta de colágenos, glicoproteínas, proteoglicanos y elastina, mantiene la estructura e integridad del tejido (31). En este estudio se incrementó la abundancia de ARNm de varios genes que codifican colágenos, como la cadena alfa 1 del colágeno tipo II (COL2A1) y la cadena alfa 1 del colágeno tipo IX (COL9A1), los proteoglicanos, como la proteína 1 de enlace de hialuronano y proteoglicano (HAPLN1) y el agrecano (ACAN), y las glicoproteínas, como la tenascina N (TNN). El colágeno es la proteína principal en la MEC. Tiene una tasa sintética fraccional más baja que las proteínas miofibrilares, y su concentración en el músculo esquelético es solo del 15 al 20% de las proteínas miofibrilares. La condición nutricional subóptima, como la deficiencia de AA, puede aumentar la tasa de síntesis fraccional de colágeno mientras disminuye la síntesis de proteínas miofibrilares y sarcoplásmicas (32). Por lo tanto, la restricción de lisina en la dieta puede ser una condición nutricional subóptima que regula al alza la síntesis de colágeno en los cerdos.
Por otro lado, las proteínas de enlace de proteoglicanos del cartílago son los principales componentes no colágenos del cartílago de la MEC (33). En este estudio, la expresión de los genes agrecano (ACAN) y de la proteína 1 de enlace de hialuronano y proteoglicano (HAPLN1) aumentó en respuesta a la restricción de lisina en la dieta. ACAN es mejor conocido como un componente de cartílago importante, esencial y definitorio cuantitativamente. De acuerdo con Mundlos et al. (34), los patrones de expresión génica del agrecano y de la proteína de enlace son idénticos en diferentes tejidos, lo que concuerda con nuestro hallazgo. El mecanismo regulador que promueve una alteración tan drástica en la expresión del ARNm en respuesta a la restricción de lisina sigue siendo incierto y estará sujeto a más estudios.
La biosíntesis de novo de serina y glicina puede generar unidades de carbono que satisfacen muchas demandas metabólicas, incluidos los precursores de nucleótidos, el mantenimiento redox y los sustratos para la reacción de metilación (35, 36). De los varios pasos de reacción enzimática en la vía de biosíntesis de serina, la fosfoglicerato deshidrogenasa (PHGDH) cataliza la conversión de 3-fosfoglicerato en 3-fosfohidroxipiruvato. La fosfoserina aminotransferasa 1 (PSAT1) es una enzima fundamental que regula la producción de dos metabolitos, la serina y el α-cetoglutarato (α-KG), que están involucrados en el metabolismo de un carbono y en el ciclo del TCA, respectivamente (1, 37). Este estudio muestra que los genes PHGDH y PSAT1 estaban regulados al alza en el músculo longissimus dorsi de los cerdos alimentados con una dieta restringida en lisina. Esto concuerda con uno de nuestros estudios previos, que reveló que la deficiencia de lisina en la dieta disminuyó la expresión de PHGDH y PSAT1 en el músculo longissimus dorsi de cerdos de engorde (2). La sobreexpresión de estos genes indica que el nivel de lisina en la dieta podría afectar el equilibrio del metabolismo de un carbono y sus vías metabólicas asociadas.
El factor de transcripción activador (ATF5) ampliamente expresado regula el ciclo celular, la diferenciación, la homeostasis y la supervivencia (38, 39). Los resultados de este estudio indican que la expresión de ATF5 aumentó en los cerdos alimentados con la dieta LDD en comparación con la dieta LAD. Hallazgos similares en estudios anteriores informaron que la expresión de ATF5 aumentó en respuesta a condiciones de estrés celular como la limitación de AA, el estrés por calor o el estrés oxidativo (39, 40). El aumento de la activación de ATF5 puede regular al alza la fosforilación de las proteínas quinasas de EIF2α y conducir a una reducción global en la traducción de proteínas.
Seis términos de BP se enriquecieron significativamente bajo el estado de restricción de lisina (Figura 3), de los cuales la polimerización de proteínas (66 veces), la fibrinólisis (79 veces) y la activación del plasminógeno (99 veces) estuvieron altamente enriquecidas. Con base en estos términos, se puede ver que algunos DEG se asocian principalmente con la homeostasis, la cicatrización de heridas, la inflamación, la angiogénesis y varias otras funciones biológicas (41, 42). Por ejemplo, el fibrinógeno, una glicoproteína proteolítica, compuesta por tres polipéptidos estrechamente ligados codificados a partir de los genes FGA, FGB y FGG. La restricción de lisina en la dieta reguló a la baja estos genes que pueden afectar la tasa de síntesis de fibrinógeno y dar lugar a anomalías en la circulación sanguínea (42), lo que a su vez puede afectar indirectamente el transporte de sangre oxigenada o nutrientes al músculo.
En segundo lugar, hubo 4 términos MF que se enriquecieron significativamente (Figura 3), de los cuales, el altamente enriquecido fue la actividad inhibidora de la endopeptidasa (62 veces). Las endopeptidasas, incluidas la tripsina, la quimotripsina, la elastasa y la pepsina, son enzimas proteolíticas que rompen los enlaces peptídicos de los AA no terminales (43, 44) y están asociadas con una amplia gama de funciones biológicas, como los procesos de desarrollo, la digestión, la fertilización, la coagulación de la sangre, la apoptosis, la fibrinólisis y la defensa inmunitaria (45). El efecto negativo de la restricción de lisina en la dieta sobre el rendimiento del crecimiento de los cerdos también puede estar asociado con la inhibición de la endopeptidasa.
En tercer lugar, hubo 8 términos CC que se enriquecieron significativamente (Figura 3), de los cuales el complejo fibrinógeno se enriqueció en 166 veces. Los términos CC en el análisis de GO demuestran la ubicación subcelular o la estructura del complejo macromolecular de productos génicos (46). Como se muestra en la Figura 3, un alto porcentaje de los productos génicos entre los DEG forman parte del complejo fibrinógeno, el filamento intermedio y la MEC. El fibrinógeno, un componente importante de la MEC, es una proteína compleja grande compuesta por tres cadenas polipeptídicas, como A, B y G, que están codificadas por los genes FGA, FGB y FGG, respectivamente (47, 48). El resultado de DEG de este estudio indica que la restricción de lisina en la dieta disminuyó la expresión génica del fibrinógeno (Tabla 4) lo que puede conducir a una reducción en la cantidad de fibrinógeno disponible para ser incorporado a la MEC, lo que conduce a una MEC más débil y una reducción en el proceso de angiogénesis, lo que podría afectar la integridad de la MEC y el crecimiento muscular (49, 50).
Por último, los resultados de los términos GO del enriquecimiento de la vía KEGG indican que el impacto negativo de la alimentación de los cerdos con una dieta restringida en lisina sobre el crecimiento puede estar mediado por las vías metabólicas y de señalización que incluyen la biosíntesis de AA, el metabolismo del colesterol, la interacción con los receptores y las cascadas del complemento y la coagulación (Figura 3). Por ejemplo, el gen de la carbamoil l-fosfato sintasa 1 (CPS1) codifica una enzima involucrada en el metabolismo del nitrógeno que elimina los productos de desecho de nitrógeno del cuerpo (51). La disminución en la expresión de CPS1 se observó en este estudio, lo que sugiere que puede causar alteraciones en el equilibrio de la síntesis y degradación de proteínas musculares, lo que lleva a la pérdida de masa muscular. Es importante tener en cuenta que el metabolismo AA es un proceso metabólico muy complejo cuyo mecanismo regulador justifica una mayor investigación.
Al final, a los autores les gustaría señalar que la abundancia de ARNm no siempre se correlaciona con la abundancia de proteínas, a pesar de que muchos científicos utilizan la abundancia de ARNm para indicar la abundancia de proteínas porque en muchos casos más ARNm traduciría más proteína. Además, hay que tener en cuenta que los datos globales de expresión génica en general no son fáciles de interpretar simplemente por las complejidades no sólo de los propios datos, sino también del aspecto de los procesos biológicos animales.
5. Conclusión
Un análisis del transcriptoma mediante tecnología RNA-Seq reveló que la restricción de lisina en la dieta alteró el perfil de expresión génica en el músculo longissimus dorsi de cerdos en crecimiento. Encontramos que 80 genes se expresaban diferencialmente, de los cuales 46 genes estaban regulados a la baja y 34 genes estaban regulados al alza. Algunos de los genes regulados a la baja (p. ej., TTR, ALB) están asociados con el transporte de moléculas de señalización (p. ej., hormonas y neurotransmisores) y el metabolismo AA (p. ej., HAP, TAT). Por otro lado, los genes regulados a la baja, como la queratina, las isoformas ACAN y COL, están asociados con las actividades de las moléculas estructurales celulares y el metabolismo de AA (por ejemplo, PHGDH y PSAT1). Los resultados del análisis de GO sugieren que la restricción de lisina en la dieta puede influir en algunos procesos biológicos, como la coagulación de la sangre y la inflamación, algunas funciones moleculares, como la actividad inhibidora de la endopeptidasa; y algunas vías KEGG, como la vía del complemento y la vía de la coagulación. En conjunto, los resultados generados a partir de este estudio han proporcionado una nueva visión crítica sobre los mecanismos moleculares del crecimiento muscular que están asociados con el suministro de lisina en la dieta.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en el artículo/Material complementario.
Declaración ética
El estudio en animales fue aprobado por el Comité Institucional de Cuidado y Uso de Animales (IACUC, por sus siglas en inglés) de la Universidad Estatal de Mississippi. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
SL conceptualizó el estudio, obtuvo fondos para la investigación y coordinó todo el proyecto. JF y HZ participaron en el diseño del experimento y en la preparación del manuscrito. MH y SL llevaron a cabo el ensayo con animales, analizaron los datos y prepararon el manuscrito. MH y YW realizaron los análisis de las muestras de laboratorio. Todos los autores aprobaron la versión final del manuscrito.
Financiación
Esta investigación fue financiada en parte por el Proyecto Hatch/Multiestatal del Instituto Nacional de Alimentos y Agricultura del USDA (MIS-351130) y la Beca Doctoral del Director MAFES/FWRC de la Universidad Estatal de Mississippi (2015-19).
Reconocimientos
Las donaciones de varios ingredientes para piensos de Ajinomoto Heartland (Chicago, IL), ADM Alliance Nutrition (Quincy, IL) y Adisseo USA (Alpharetta, GA) son muy apreciadas. Los autores desean agradecer a William White (gerente de la granja, Centro de Investigación Animal Leeck, Universidad Estatal de Mississippi) y a todos los profesionales de la granja por su excelente asistencia en el manejo de las instalaciones y los animales.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1233292/full#supplementary-material
Notas
1. ^ https://www.ncbi.nlm.nih.gov
Referencias
1. Liao, SF, Wang, T y Regmi, N. Nutrición de lisina en cerdos y animales monogástricos relacionados: biosíntesis de proteínas musculares y más allá. Springerplus. (2015) 4:147. doi: 10.1186/s40064-015-0927-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Wang, T, Feugang, JM, Crenshaw, MA, Regmi, N, Blanton, JR y Liao, SF. Un enfoque de biología de sistemas que utiliza datos transcriptómicos revela genes y vías en el músculo esquelético porcino afectados por la lisina dietética. Int J Mol Sci. (2017) 18:18. doi: 10.3390/ijms18040885
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Hasan, MS, Crenshaw, MA, y Liao, SF. La lisina dietética afecta el metabolismo de los aminoácidos y el rendimiento del crecimiento, que puede no involucrar el eje GH / IGF-1, en cerdos jóvenes en crecimiento. J Anim Sci. (2020) 98:skaa 004. doi: 10.1093/jas/skaa004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Jin, CL, Ye, JL, Yang, J, Gao, CQ, Yan, HC, Li, HC, et al. mTORC1 media la activación de células satélite inducida por lisina para promover el crecimiento del músculo esquelético. Células. (2019) 8:1549. doi: 10.3390/cells8121549
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Jin, CL, Zhang, ZM, Song, ZW, Gao, CQ, Yan, HC y Wang, XQ. La diferenciación de células satélite mediada por mTORC1 es necesaria para el crecimiento del músculo esquelético inducido por lisina. J Agric Food Chem. (2020) 68:4884–92. doi: 10.1021/acs.jafc.0c01275
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Mortazavi, A, Williams, BA, McCue, K, Schaeffer, L, y Wold, B. Mapeo y cuantificación de transcriptomas de mamíferos por RNA-Seq. Métodos Nat. (2008) 5:621–8. doi: 10.1038/nmeth.1226
7. Wang, Z, Gerstein, M y Snyder, M. RNA-Seq: una herramienta revolucionaria para la transcriptómica. Nat Rev Genet. (2009) 10:57–63. doi: 10.1038/NRG2484
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Hasan, MS, Feugang, JM, y Liao, SF. Un enfoque de nutrigenómica que utiliza tecnología de secuenciación de ARN para estudiar las interacciones nutriente-gen en animales de granja. Curr Dev Nutr. (2019) 3:nzz082. doi: 10.1093/cdn/nzz082
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Stefaniuk, M y Ropka-Molik, K. Secuenciación de ARN como una poderosa herramienta en la búsqueda de genes que influyen en la salud y los rasgos de rendimiento de los caballos. J Appl Genética. (2016) 57:199–206. doi: 10.1007/s13353-015-0320-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Consejo Nacional de Investigación (NRC). Proteínas y aminoácidos En: Requerimientos de nutrientes de los cerdos. 11ª ed. revisada, Washington: The National Academies Press (2012). 15–44.
11. Robinson, MD, y Smyth, GK. Estimación de muestras pequeñas de dispersión binomial negativa, con aplicaciones a datos SAGE. Bioestadística. (2007) 9:321–32. doi: 10.1093/biostatistics/kxm030
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Robinson, MD, McCarthy, DJ y Smyth, GK. edge R: un paquete de bioconductores para el análisis de expresión diferencial de datos digitales de expresión génica. Bioinformática. (2010) 26:139–40. doi: 10.1093/bioinformatics/BTP616
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Soprano, DR, Herbert, J, Soprano, KJ, Schon, EA y Goodman, DS. Demostración del ARNm de la transtiretina en el cerebro y otros tejidos extrahepáticos de la rata. J Biol Chem. (1985) 260:11793–8. doi: 10.1016/S0021-9258(17)39100-7
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Wagatsuma, A, Yamazaki, Y, Mizuno, K y Yamada, S. Propiedades moleculares y expresión génica de la albúmina en el músculo esquelético después de la inmovilización de las extremidades posteriores en una posición acortada. Acta Neuropathol. (2001) 101:540–6. doi: 10.1007/s004010000311
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Lee, EJ, Shaikh, S, Choi, D, Ahmad, K, Baig, MH, Lim, JH, et al. La transtiretina mantiene la homeostasis muscular a través de la novedosa vía de lanzadera de las hormonas tiroideas durante la diferenciación de los mioblastos. Células. (2019) 8:1565. doi: 10.3390/cells8121565
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Monk, JA, Sims, NA, Dziegielewska, KM, Weiss, RE, Ramsay, RG y Richardson, SJ. Retraso en el desarrollo de eventos específicos regulados por la hormona tiroidea en ratones con transtiretina nula. Am J Physiol Endocrinol Metab. (2013) 304:E23–31. doi: 10.1152/ajpendo.00216.2012
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Lee, EJ, Bhat, AR, Kamli, MR, Pokharel, S, Chun, T, Lee, YH, et al. La transtiretina es un regulador clave de la diferenciación de los mioblastos. PLoS Uno. (2013) 8:E63627. doi: 10.1371/journal.pone.0063627
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Lee, E, Pokharel, S, Jan, A, Huh, S, Galope, R, Lim, J, et al. Transtiretina: proteína transportadora esencial para la proliferación de mioblastos en el programa miogénico. Int J Mol Sci. (2017) 18:115. doi: 10.3390/ijms18010115
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Sullivan, DH. Editorial invitado: ¿qué nos dicen las proteínas séricas sobre nuestros pacientes ancianos? J Gerontol. (2001) 56:M71-4. doi: 10.1093/gerona/56.2.M71
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Kwon, J, Suzuki, T, Yoshida, H, Kim, H, Yoshida, Y e Iwasa, H. Los niveles concomitantes más bajos de albúmina sérica y vitamina D se asocian con una disminución del rendimiento físico objetivo entre los ancianos japoneses que viven en la comunidad. Gerontología. (2007) 53:322–8. doi: 10.1159/000103257
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Calonne, J, Isacco, L, Miles-Chan, J, Arsenijevic, D, Montani, JP, Guillet, C, et al. Reducción del recambio proteico del músculo esquelético y del metabolismo de la hormona tiroidea en la termogénesis adaptativa que facilita la recuperación de la grasa corporal durante la recuperación de peso. Endocrinol frontal. (2019) 10:119. doi: 10.3389/fendo.2019.00119
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Divanovic, S, Dalli, J, Jorge-Nebert, LF, Flick, LM, Gálvez-Peralta, M, Boespflug, ND, et al. Contribuciones de las tres monooxigenasas CYP1 a las vías mediadoras lipídicas proinflamatorias y de resolución de inflamación. J Immunol. (2013) 191:3347–57. doi: 10.4049/jimmunol.1300699
23. Tachtsis, B, Camera, D, y Lacham-Kaplan, O. Roles potenciales de los AGPI n-3 durante el crecimiento y la regeneración del músculo esquelético. Nutrientes. (2018) 10:E309. doi: 10.3390/nu10030309
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Williams, RA, Mamotte, CDS y Burnett, JR. Fenilcetonuria: un error congénito del metabolismo de la fenilalanina. Clin Biochem Rev. (2008) 29:31–41. PMID:18566668
25. Fernstrom, JD, y Fernstrom, MH. Síntesis y función de tirosina, fenilalanina y catecolaminas en el cerebro. J Nutr. (2007) 137:1539S–47S. doi: 10.1093/jn/137.6.1539S
26. Magin, TM, Vijayaraj, P, y Leube, RE. Funciones estructurales y reguladoras de las queratinas. Exp Cell Res. (2007) 313:2021–32. doi: 10.1016/j.yexcr.2007.03.005
27. Bragulla, HH, y Homberger, DG. Estructura y funciones de las proteínas de queratina en epitelios simples, estratificados, queratinizados y cornificados. J Anat. (2009) 214:516–59. doi: 10.1111/j.1469-7580.2009.01066.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. O’Neill, A, Williams, MW, Resneck, WG, Milner, DJ, Capetanaki, Y, y Bloch, RJ. Organización sarcolemal en músculo esquelético carente de desmina: evidencia de citoqueratinas asociadas con el esqueleto de la membrana en los costámeros. Mol Biol Cell. (2002) 13:2347–59. doi: 10.1091/mbc.01-12-0576
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Ursitti, JA, Lee, PC, Resneck, WG, McNally, MM, Bowman, AL, O’Neill, A, et al. Clonación y caracterización de citoqueratinas 8 y 19 en músculo estriado de rata adulta. J Biol Chem. (2004) 279:41830–8. doi: 10.1074/jbc. M400128200
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Shah, SB, Love, JM, O’Neill, A, Lovering, RM y Bloch, RJ. Influencias de la desmina y la queratina 19 en las propiedades biomecánicas pasivas del músculo esquelético del ratón. J Biomed Biotechnol. (2012) 2012:1–12. doi: 10.1155/2012/704061
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Csapo, R, Gumpenberger, M y Wessner, B. Matriz extracelular del músculo esquelético: ¿qué sabemos sobre su composición, regulación y funciones fisiológicas? Una revisión narrativa. Fisiola delantera. (2020) 11:253. doi: 10.3389/fphys.2020.00253
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Babraj, JA, Cuthbertson, DJR, Smith, K, Langberg, H, Miller, B, Krogsgaard, MR, et al. Síntesis de colágeno en tejidos musculoesqueléticos humanos y piel. Am J Physiol Endocrinol Metab. (2005) 289:E864-9. doi: 10.1152/ajpendo.00243.2005
33. Brandan, E, y Gutierrez, J. Papel de los proteoglicanos en la regulación de la respuesta fibrótica del músculo esquelético. FEBS J. (2013) 280:4109–17. doi: 10.1111/febs.12278
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Mundlos, S, Meyer, R, Yamada, Y y Zabel, B. Distribución de la proteína central del proteoglicano del cartílago (Aggrecan) y la expresión génica de la proteína de enlace durante el desarrollo esquelético humano. Matriz. (1991) 11:339–46. doi: 10.1016/S0934-8832(11)80205-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Yang, M, y Vousden, KH. Serina y metabolismo de un carbono en el cáncer. Nat Rev Cáncer. (2016) 16:650–62. doi: 10.1038/nrc.2016.81
36. Reid, MA, Allen, AE, Liu, S, Liberti, MV, Liu, P, Liu, X, et al. La síntesis de serina a través de PHGDH coordina los niveles de nucleótidos manteniendo el metabolismo central del carbono. Nat Commun. (2018) 9:5442. doi: 10.1038/s41467-018-07868-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Huang, SP, Chan, YC, Huang, SY y Lin, YF. La sobreexpresión del gen PSAT1 es un marcador pronóstico favorable en los gliomas de grado más bajo y predice un desenlace favorable en pacientes con mutaciones en IDH1 y codeleción del cromosoma 1p19q. Cánceres. (2019) 12:E13. doi: 10.3390/cancers12010013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Madarampalli, B, Yuan, Y, Liu, D, Lengel, K, Xu, Y, Li, G, et al. ATF5 conecta los materiales pericentriolares con el extremo proximal del centriolo madre. Células. (2015) 162:580–92. doi: 10.1016/j.cell.2015.06.055
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Gaither, KA, Watson, CJW, Madarampalli, B y Lazarus, P. La expresión del factor de transcripción activador 5 (ATF5) está mediada por micro RNA-520b-3p bajo estrés celular diverso en células cancerosas. PLoS Uno. (2020) 15:e0225044. doi: 10.1371/journal.pone.0225044
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Zhou, D, Palam, LR, Jiang, L, Narasimhan, J, Staschke, KA y Wek, RC. La fosforilación de eIF2 dirige el control traduccional de ATF5 en respuesta a diversas condiciones de estrés. J Biol Chem. (2008) 283:7064–73. doi: 10.1074/jbc. M708530200
41. Weisel, JW, y Litvinov, RI. Formación, estructura y propiedades de la fibrina En: DAD Parry y JM Squire, editores. Proteínas fibrosas: estructuras y mecanismos. Bioquímica subcelular. Cham: Springer International Publishing (2017). 405–56.
42. Choe, HM, Quan, B, Paek, H, Luo, Z, Gao, K, Han, S, et al. Alteración del nivel de fibrinógeno y de la estructura del coágulo de fibrina en cerdos mutantes homocigotos con miostatina. Anim Grupos sanguíneos Biochem Genet. (2022) 53:307–16. doi: 10.1111/age.13187
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Yamauchi, Y, Ejiri, Y, Sugimoto, T, Sueyoshi, K, Oji, Y y Tanaka, K. Una glutamil endopeptidasa de alto peso molecular y sus inhibidores endógenos de las hojas de pepino. J Biochem. (2001) 130:257–61. doi: 10.1093/oxfordjournals.jbchem.a002980
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Park, K, y Kwak, IS. Alteración del desarrollo inducida por cadmio y regulación positiva de las transcripciones de endopeptidasa de tipo serina en poblaciones silvestres de agua dulce de Chironomus plumosus. Ecotoxicol Environ Saf. (2020) 192:110240. doi: 10.1016/j.ecoenv.2020.110240
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Alves, CR, Souza, RS, Charret, KDS, Côrtes, LMC, Sá-Silva, MP, Barral-Veloso, L, et al. Comprender las implicaciones de las serina proteasas en el ciclo de vida de Leishmania spp. Exp parasitol. (2018) 184:67–81. doi: 10.1016/j.exppara.2017.11.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Roncaglia, P, Martone, ME, Hill, DP, Berardini, TZ, Foulger, RE, Imam, FT, et al. La ontología génica (GO) ontología del componente celular: integración con SAO (ontología de anatomía subcelular) y otros desarrollos recientes. J Biomed Semántica. (2013) 4:20. doi: 10.1186/2041-1480-4-20
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Pereira, M, Rybarczyk, BJ, Odrljin, TM, Hocking, DC, Sottile, J, y Simpson-Haidaris, PJ. La incorporación de fibrinógeno en la matriz extracelular depende del ensamblaje activo de una matriz de fibronectina. J Cell Sci. (2002) 115:609–17. doi: 10.1242/jcs.115.3.609
Resumen de PubMed | Texto completo de CrossRef | Google Académico
48. Fish, RJ y Neerman-Arbez, M. Regulación génica del fibrinógeno. Hemaste del trombo. (2012) 108:419–26. doi: 10.1160/TH12-04-0273
49. Lapidos, KA, Kakkar, R, y McNally, EM. El complejo glicoproteico de la distrofina: fuerza de señalización e integridad del sarcolema. Circ Res. (2004) 94:1023–31. doi: 10.1161/01.RES.0000126574.61061.25
50. Liu, J, Burkin, DJ, y Kaufman, SJ. El aumento de la alfa 7 beta 1-integrina promueve la proliferación de células musculares, la adhesión y la resistencia a la apoptosis sin cambiar la expresión génica. Am J Physiol Cell Physiol. (2008) 294:C627-40. doi: 10.1152/ajpcell.00329.2007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Yefimenko, I, Fresquet, V, Marco-Marín, C, Rubio, V, y Cervera, J. Comprensión de la deficiencia de carbamoil fosfato sintetasa: impacto de las mutaciones clínicas en la funcionalidad enzimática. J Mol Biol. (2005) 349:127–41. doi: 10.1016/j.jmb.2005.03.078
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: lisina, músculo esquelético, secuenciación de ARN, análisis transcriptómico, porcino
Cita: Hasan MdS, Wang Y, Feugnag JM, Zhou H y Liao SF (2023) El análisis de secuenciación de ARN reveló genes expresados diferencialmente y su anotación funcional en el músculo longissimus dorsi porcino afectado por la restricción de lisina en la dieta. Frente. Vet. Sci. 10:1233292. doi: 10.3389/fvets.2023.1233292
Recibido: 05 de junio de 2023; Aceptado: 24 de octubre de 2023;
Publicado: 08 Noviembre 2023.
Editado por:
Kai Wang, Academia China de Ciencias Agrícolas (CAAS), China
Revisado por:
Steffen Maak, Instituto Leibniz de Biología de Animales de Granja (FBN), Alemania
Agus Suryawan, Baylor College of Medicine, Estados Unidos
Derechos de autor © 2023 Hasan, Wang, Feugnag, Zhou y Liao. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Shengfa F. Liao, s.liao@msstate.edu
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía