Ensayo multiplex basado en perlas Luminex de alto rendimiento para la identificación patógenos bacterianos principales de mastitis

Ensayo multiplex basado en perlas Luminex de alto rendimiento para la identificación de seis patógenos bacterianos principales de mastitis en animales lecheros
 Garima Shrinet1,
Garima Shrinet1,  Rajesh Chhabra2*,
Rajesh Chhabra2*,  Archana Sharma1,
Archana Sharma1,  Kanisht Batra3,
Kanisht Batra3,  Saurabh Jyoti Talukdar2 y
Saurabh Jyoti Talukdar2 y  Sushila Maan3*
Sushila Maan3*- 1Departamento de Microbiología Veterinaria, Lala Lajpat Rai Universidad de Ciencias Veterinarias y Animales, Hisar, Haryana, India
- 2College Central Laboratory, Lala Lajpat Rai University of Veterinary and Animal Sciences, Hisar, Haryana, India
- 3Departamento de Biotecnología Animal, Lala Lajpat Rai Universidad de Ciencias Veterinarias y Animales, Hisar, Haryana, India
Introducción: La mastitis bovina es causada por más de 150 microorganismos diferentes. La identificación y cuantificación específicas de múltiples bacterias en una sola muestra de leche se vuelve esencial para una intervención rápida.
Métodos: En el presente estudio se desarrolló un ensayo multiplex basado en perlas de Luminex que enfatiza la identificación precisa de seis patógenos bacterianos principales de mastitis. El ensayo se desarrolló en dos conjuntos triplex, triplex 1 compuesto por Streptococcus agalactiae, Streptococcus dysgalactiae y Streptococcus uberis, mientras que triplex 2 consistió en Staphylococcus aureus, E. coli y Klebsiella pneumoniae.
Resultados: La sensibilidad analítica fue de 10 a 6 copias por mezcla de reacción para las seis bacterias. Se observó una especificidad analítica del 100% para la detección simultánea de estas bacterias. Se analizaron muestras clínicas de leche de 100 cuartos bovinos para su validación.
Discusión: La sensibilidad analítica fue similar a los hallazgos informados anteriormente en el ensayo múltiple de PCR en tiempo real dirigido al ADN de las 11 especies o grupos bacterianos más comunes en la mastitis. La especificidad analítica del ensayo optimizado fue 100% similar a la reportada anteriormente para la detección simultánea de Mycoplasma spp. y para siete virus entrónicos de humanos. El ensayo desarrollado indica una prueba de concepto de una herramienta de diagnóstico de alto rendimiento rápida y rentable para la identificación de las principales bacterias que causan mastitis.
1 Introducción
La mastitis bovina es la enfermedad más prevalente en la industria láctea caracterizada por la inflamación de las glándulas mamarias; Está afectando gravemente a las granjas lecheras en todo el mundo. Las pérdidas económicas se deben a la reducción de la producción de leche, la producción de leche de baja calidad, el costo de los medicamentos y los servicios veterinarios, el aumento de la tasa de sacrificio y la reducción de la eficiencia reproductiva (Down et al., 2017). La pérdida económica solo en la India se estima en 1390 INR por lactancia debido a la mastitis bovina (Sinha et al., 2014). Además, el uso extensivo de antibióticos en el tratamiento y control de la mastitis también tiene posibles implicaciones en la salud humana. Esto se debe a un mayor riesgo de que surjan cepas de bacterias resistentes a los antibióticos que pueden entrar en la cadena alimentaria (White y McDermott, 2001). La alta prevalencia de resistencia a los antimicrobianos (RAM) en patógenos de mastitis se ha registrado en diferentes granjas lecheras, lo que sugiere que la mastitis bovina potencialmente pone en peligro tanto la eficacia antimicrobiana como la salud pública (Chandrasekaran et al., 2014; Cheng et al., 2019; Abdi y otros, 2021; Pascu et al., 2022).
La mastitis es una enfermedad multietiológica generalmente representada por coinfección o infección mixta. La etiología de la mastitis consiste en un amplio espectro de agentes patógenos que penetran en el canal del pezón y se multiplican dentro de la cisterna de la ubre, producen toxinas que causan daño tisular. La mayoría de los casos de mastitis son producidos por un grupo relativamente pequeño de bacterias, que comprende Staphylococcus aureus, Streptococcus agalactiae, Streptococcus dysgalactiae, Streptococcus uberis, E. coli y Klebsiellaspp (Shome et al., 2011) Por lo tanto, para diseñar un programa de prevención y control de mastitis, vale la pena tener en cuenta no solo las prácticas de manejo sino también la identificación específica del agente etiológico. y la prevalencia a nivel de rebaño de patógenos de mastitis contagiosa.
La prueba estándar de oro para la mastitis es el cultivo de leche para el aislamiento y la identificación de bacterias, puede tomar tiempo entre 5 y 7 días para la identificación de especies específicas (Dingwell et al., 2003). También puede haber posibilidad de perder colonias con fenotipo similar a las predominantes (Bradley et al., 2007). También en más del 26,5% de las muestras de leche de mastitis bovina clínica y subclínica, las bacterias no crecen incluso después de 48 horas en cultivo convencional (Bradley et al., 2007). Por lo tanto, se desea la identificación múltiple o simultánea, así como la cuantificación de las principales bacterias asociadas con la mastitis en un gran número de muestras de leche de una sola vez.
El desarrollo de métodos basados en PCR proporcionó una opción prometedora para la identificación rápida de bacterias de casos de mastitis (Phuektes et al., 2001; Shome et al., 2011; Ashraf et al., 2017; Calonzi et al., 2020). Las desventajas incluyen una alta sensibilidad que puede conducir a falsos positivos, contaminantes menores en muestras que conducen a diagnósticos erróneos (Phuektes et al., 2001), requieren electroforesis en gel de agarosa para su interpretación y no se puede procesar un gran número de muestras al mismo tiempo (Christopher et al., 2013). Sobre todo, el número de patógenos identificados en la PCR múltiple o PCR en tiempo real es limitado, lo que es un desafío teniendo en cuenta la diversa lista de patógenos de la mastitis (Shome et al., 2011).
En consecuencia, un ensayo multiplex basado en perlas Luminex diseñado con tecnología xMAP reduce el tiempo, el volumen de muestra, tiene multiplexación flexible de hasta 500 objetivos, lo que permite pruebas de alto rendimiento y también es un ensayo cuantitativo (Deregt et al., 2006; Reslova et al., 2017). Puede resultar eficaz en la evaluación de infecciones mixtas como la mastitis. Actualmente muy pocos ensayos están disponibles comercialmente para aplicaciones veterinarias utilizando ensayos multiplex basados en perlas, entre los cuales muy pocos están basados en ácidos nucleicos para la detección de patógenos de una enfermedad (Deregt et al., 2006; LeBlanc y otros, 2009; Righter et al., 2011; Reslova et al., 2017). En general, la mayoría de los ensayos basados en Luminex son inmunoensayos para identificar antígenos, proteínas, producciones de anticuerpos y diferenciación de animales vacunados de infectados (Woolhouse y Alex, 2001; Hsu y otros, 2005; Hsu y otros, 2007; Watson y otros, 2009; Ros et al., 2012).
Aquí informamos sobre el desarrollo de un ensayo multiplex de Luminex basado en ácidos nucleicos para la identificación cualitativa de seis bacterias principales que causan mastitis en animales lecheros, a saber, Staphylococcus aureus, Streptococcus agalactiae, Streptococcus dysgalactiae, Streptococcus uberis, E. coli y Klebsiella pneumoniae.
2 Material y métodos
2.1 Cepas bacterianas
Las cepas ATCC de las seis bacterias se utilizaron como estándares para la optimización del ensayo. El panel incluyó cepas Staphylococcus aureus ATCC 12600, Streptococcus agalactiae ATCC 12386, Streptococcus dysgalactiae ATCC 12394, Streptococcus uberis ATCC 700407, Klebsiella pneumoniae ATCC 1706 y E. coli ATCC 10536. Estas cepas se cultivaron en placas de agar sangre de oveja al 5% a 37°C. Las cepas también se almacenaron en existencias de glicerol al 30%, así como liofilizadas para su uso posterior.
2.2 Diseño y modificación de cebadores y sondas
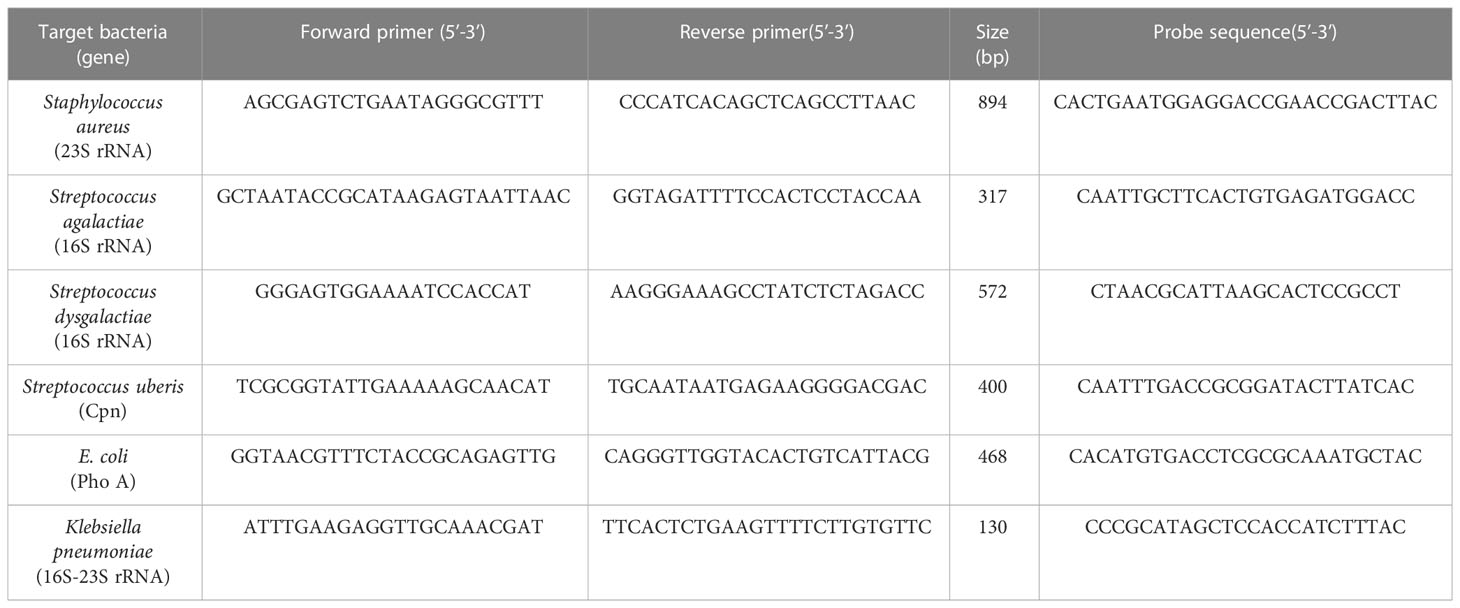
Se seleccionaron secuencias de cebadores publicadas dirigidas al gen 23S rRNA de Staphylococcus aureus, el gen 16S rRNA de Streptococcus agalactiae y Streptococcus dysgalactiae, la secuencia parcial 16-23SrRNA de Klebsiella pnuemoniae, el gen Cpn de Streptococcus uberis y el gen PhoA de E. coli que son genes altamente conservados en la bacteria (Shome et al., 2011 ). Se utilizaron la modificación de biotina 5′ de cebadores inversos purificados por HPLC dependiendo de la hebra complementaria a la secuencia de la sonda y los cebadores hacia adelante purificados PAGE a una concentración de 50 nmoles preparados por Integrated DNA Technology (Coralville, LA, EUA).
Las sondas de captura específicas que eran complementarias a las secuencias específicas del organismo disponibles en Genbank (número de acceso: X68425.1, DQ232512.1, AB002488.1, AF485804.1, FJ546461.1 y DQ399570.1) se diseñaron utilizando el software BioEdit versión 7.2. Las sondas tenían una modificación del espaciador 5′ amino C12 y un grado de purificación HPLC. La secuencia de cebador y sonda utilizada se enumeran en la Tabla 1.
2.3 Extracción de ADN genómico bacteriano de muestras de leche
El ADN genómico bacteriano (ADNg) se extrajo de 100 μL de leche probada como estéril mediante cultivo en caldo LB. Fue pinchado con 108 UFC/ml de cepas ATCC de las seis bacterias para la optimización del ensayo desarrollado. Se utilizó el kit de aislamiento de ADN bacteriano de leche (Norgen Biotek Corp., ON, Canadá) de acuerdo con el protocolo para cepas desconocidas de bacterias. La concentración de dsDNA y la pureza de los seis ADN bacteriano se midieron utilizando el Scandrop2 (Analytik Jena, Jena · Alemania). Cada especie bacteriana se confirmó mediante amplificación utilizando los cebadores enumerados en la Tabla 1 y visualización en gel de agarosa al 1,5%.
2.4 Optimización de la PCR multiplex
El ensayo de PCR multiplex se optimizó en dos combinaciones triplex. El triplex 1 consistió en Streptococcus agalactiae, Streptococcus dysgalactiae y Streptococcus uberis. Mientras que el triplex 2 compuesto por Staphylococcus aureus, E. coli y Klebsiella pneumoniae.
Las condiciones para la PCR multiplex se optimizaron mediante PCR de gradiente (datos no mostrados). Los parámetros de ciclismo para triplex 1 consistieron en un arranque en caliente a 95 °C durante 10 minutos, un paso de desnaturalización inicial de 95 °C durante 7 minutos seguido de 30 ciclos de 95 °C durante 30 segundos, 58 °C durante 46 segundos y 72 °C durante 46 segundos con una extensión final a 72 °C durante 10 minutos. El parámetro de ciclismo para triplex 2 fue el arranque en caliente a 95 °C durante 10 minutos, el paso de desnaturalización inicial de 95 °C durante 10 minutos, seguido de 30 ciclos de 95 °C durante 30 segundos, 57 °C durante 45 segundos y 72 °C durante 45 segundos con una extensión final a 72 °C durante 10 minutos.
2.5 Acoplamiento y recuento de cuentas
Las sondas estaban unidas a tres perlas fluorescentes espectralmente únicas diferentes para el triplex 1 y 2. El protocolo de acoplamiento de perlas y el recuento de perlas por hemocitómetro se realizaron según el libro de cocina xMAP (Angeloni et al., 2013). La concentración de perlas utilizadas fue de 12,5 millones/ml. El volumen de perlas se optimizó en 1,25 millones de perlas por sonda específica (10 pmole) que era tres veces menor que la concentración recomendada de perlas.
2.6 Hibridación de la sonda
Los productos de PCR biotinilados se hibridaron a las perlas acopladas a la sonda en 1.5 X TMACsolución. Se optimizó un volumen de reacción total de 50 μL que comprende 2,5 μL del producto de PCR biotinilado con 33 μL de la solución 1,5 X TMAC de trabajo, 1 μL de cada perlas marcadas del conjunto triplex específico y 11,5 μL de agua libre de nucleasas como mezcla de trabajo del ensayo. Se probaron tres temperaturas de hibridación diferentes (50 ° C, 52 ° C y 55 ° C) y tiempo (10, 15 y 30 minutos) para ambos conjuntos triplex. El colorante de detección, estreptavidina-ficoeritrina (SAPE) también se optimizó a 1 μL en 1000 μL (Sigma, 1 mg / ml).
2.7 Evaluación de la sensibilidad analítica y la especificidad del ensayo
Las reacciones de ensayo se optimizaron primero en el formato monoplex utilizando diluciones seriadas diez veces mayores para cada bacteria seguidas del formato triplex. Se utilizó el protocolo de lavado para la hibridación. Los resultados se observaron en la versión 4.3 del software Luminex xPONENT y se expresaron en términos de MFI (intensidad media de fluorescencia). El límite de detección (LOD) se expresó en términos de número de copias. Las bacterias patógenas vivas se utilizaron para el cálculo del número de copias en la concentración definida, como Streptococcus agalactiae (7 X 108 UFC/ml), Streptococcus dysgalactiae (5.7 X 108 UFC/ml) y Streptococcus uberis (9 X 108 UFC/ml), Staphylococcus aureus (6 X 108 UFC/ml), E. coli (7 X 108 UFC/ml) y Klebsiella pneumoniae (5 X 108 UFC/ml). La UFC es indicativa de carga patógena de bacterias con el número genómico presente en la muestra. El número de copias se calculó utilizando la concentración cuantificada de ADN de acuerdo con las siguientes fórmulas para el número de copias = A X No/Longitud en par de bases X 109 X 650. Donde A es la concentración de ADN en ng/μL, No es el número de Avogadro (6.022 X 1023) y Length es el tamaño del amplicón.
La especificidad del ensayo se evaluó utilizando el ADNg de las cepas ATCC estándar de las seis bacterias objetivo. También se incluyó un control negativo consistente en perlas marcadas con agua libre de nucleasa y SAPE. Se registró la IMF de las sondas acopladas a perlas y el producto de PCR marcado con biotina. Además, también se realizó una comparación in silico para la reactividad cruzada de los cebadores y las secuencias de la sonda con especies estrechamente relacionadas con las bacterias objetivo utilizando el editor de alineación de secuencias biológicas (BioEdit 7.2) y la herramienta básica de búsqueda de alineación local (BLAST).
2.8 Repetibilidad del ensayo
Se determinó la repetibilidad intra e interensayo. Se realizaron tres ejecuciones separadas para determinar la repetibilidad intraensayo con 2 réplicas y se realizaron 5 ejecuciones separadas para determinar la repetibilidad entre ensayos según el libro de cocina Luminex xMAP (Basile et al., 2010). El rango aceptable de coeficiente de varianza (CV) para la repetibilidad intraensayo es < 10% y la repetibilidad entre ensayos es < 20% (Phuektes et al., 2001).
2.9 Validación del ensayo
Para validar preliminarmente el ensayo estandarizado, se probó en muestras de leche bovina de 40 cuartas partes de casos sospechosos de mastitis llevados al Laboratorio Central del Colegio Lala Lajpat Rai de la Universidad de Ciencias Veterinarias y Animales, Hisar, India. Cada muestra de leche también se cultivó en agar sangre y agar lactosa MacConkey (MLA) simultáneamente, para aislar e identificar bacterias por método de cultivo convencional y PCR.
3 Resultados
3.1 Volumen de reacción de ensayo optimizado, condiciones y análisis
Las condiciones de hibridación para triplex 1 se optimizaron como desnaturalización a 96 °C durante 2 minutos y recocido a 55 °C durante 30 minutos. Mientras que la condición de hibridación para triplex 2 se optimizó a 98 ° C durante 5 minutos y 50 ° C durante 30 minutos. La mezcla del reportero se incubó a 50 ° C durante 5 minutos y se analizó con el lector de multiplex Magpix. Los resultados se observaron en la versión 4.3 del software Luminex xPONENT (Figuras S1, S2). Se observó una MFI baja con una dilución más baja que luego se volvió constante en la dilución adicional para Staphylococcus aureus y E. coli en triplex 2.
3.2 Especificidad analítica del ensayo
En este ensayo, no se encontró reactividad cruzada con los otros cuatro patógenos para ninguna de las sondas. Tampoco se observó ninguna señal de colorante correspondiente al control negativo (Figura 1). Con el fin de evaluar aún más el rendimiento del ensayo desarrollado, también se probó en diferentes combinaciones de objetivos. Para ello se incluyeron seis conjuntos de muestras mixtas similares a los casos de infección natural y un control negativo. Esto mostró una alta especificidad del ensayo desarrollado para la detección del objetivo con una barra de error al 5% (Figura 2).
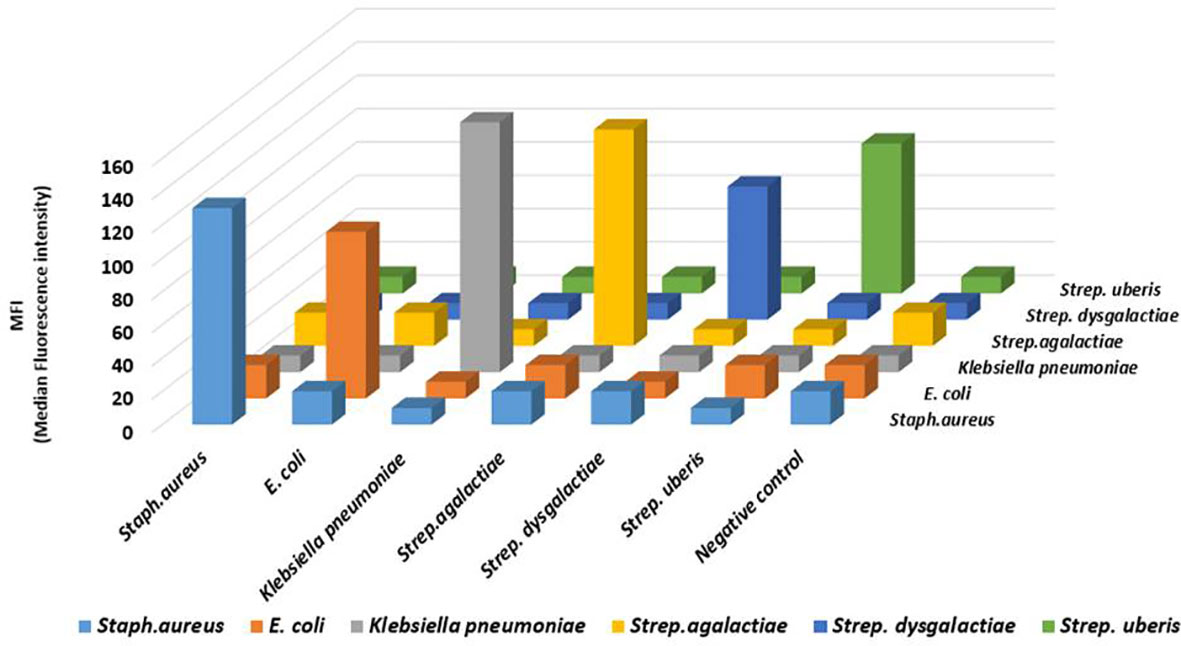 Figura 1 El resultado analítico de la especificidad del ensayo multiplex basado en perlas de Luminex desarrollado para bacterias causantes de mastitis bovina: El análisis de la especificidad se llevó a cabo para Staphylococcus aureus, Klebsiella pneumoniae, E. coli, Streptococcus agalactiae, Streptococcus dysgalactiae y Streptococcus uberis. Los productos de PCR marcados con biotina se separaron mediante perlas acopladas a sonda y se presentan en términos de intensidad de fluorescencia mediana (MFI) de la señal de colorante en unidades arbitrarias en el eje y. Cada pico fue identificado por perlas acopladas con sondas de captura específicas y se indica en el eje z.
Figura 1 El resultado analítico de la especificidad del ensayo multiplex basado en perlas de Luminex desarrollado para bacterias causantes de mastitis bovina: El análisis de la especificidad se llevó a cabo para Staphylococcus aureus, Klebsiella pneumoniae, E. coli, Streptococcus agalactiae, Streptococcus dysgalactiae y Streptococcus uberis. Los productos de PCR marcados con biotina se separaron mediante perlas acopladas a sonda y se presentan en términos de intensidad de fluorescencia mediana (MFI) de la señal de colorante en unidades arbitrarias en el eje y. Cada pico fue identificado por perlas acopladas con sondas de captura específicas y se indica en el eje z.
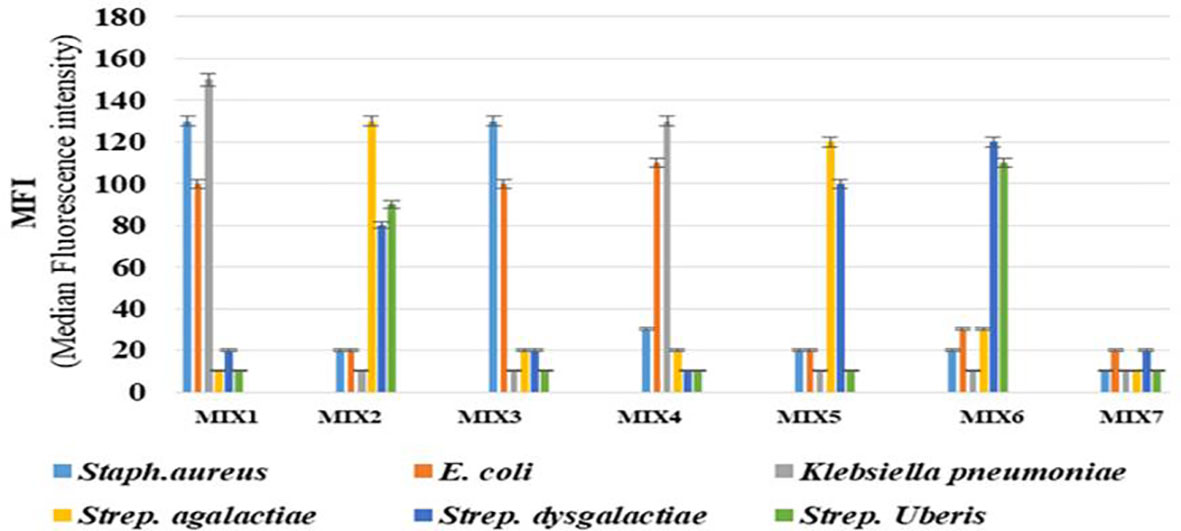 Figura 2 El análisis de la capacidad de detección de muestras mixtas del ensayo multiplex basado en perlas Luminex desarrollado para bacterias causantes de mastitis bovina: La capacidad de detección de muestras mixtas del ensayo multiplex basado en perlas Luminex se llevó a cabo utilizando seis conjuntos de muestras mezcladas aleatoriamente y control negativo. Las barras de error representan el 5% del valor de la IMF. Mezcla 1: Staphylococcus aureus, E. coli y Klebsiella pneumoniae; Mezcla 2: Streptococcus agalactiae, Streptococcus dysgalactiae&Streptococcus uberis; Mezcla 3: Staphylococcus aureus & E. coli; Mezcla 4: E. coli y Klebsiella pneumoniae; Mezcla 5: Streptococcus agalactia&, Streptococcus dysgalactiae; Mix 6: Streptococcus dysgalactiae & Streptococcus uberis y Mix 7 o Control negativo: Bead + NFW + SAPE.
Figura 2 El análisis de la capacidad de detección de muestras mixtas del ensayo multiplex basado en perlas Luminex desarrollado para bacterias causantes de mastitis bovina: La capacidad de detección de muestras mixtas del ensayo multiplex basado en perlas Luminex se llevó a cabo utilizando seis conjuntos de muestras mezcladas aleatoriamente y control negativo. Las barras de error representan el 5% del valor de la IMF. Mezcla 1: Staphylococcus aureus, E. coli y Klebsiella pneumoniae; Mezcla 2: Streptococcus agalactiae, Streptococcus dysgalactiae&Streptococcus uberis; Mezcla 3: Staphylococcus aureus & E. coli; Mezcla 4: E. coli y Klebsiella pneumoniae; Mezcla 5: Streptococcus agalactia&, Streptococcus dysgalactiae; Mix 6: Streptococcus dysgalactiae & Streptococcus uberis y Mix 7 o Control negativo: Bead + NFW + SAPE.
3.3 Sensibilidad analítica del ensayo
La dilución limitante fue de 10-6 para cada bacteria en ambos conjuntos triplex (Figuras S1, S2). El LOD de cada bacteria en formatos triplex 1 y triplex 2 fue de 106 copias por reacción, como se muestra en los cuadros suplementarios 1 a 3, respectivamente.
3.4 Repetibilidad del ensayo
El CV para cada bacteria diana en ambos triplex dentro de una corrida varió de 4-7% y 6-9%, mientras que entre corridas varió de 7-11% y 8-12% para triplex 1 y 2, respectivamente (Tablas suplementarias 4, 5).
3.5 Validación del ensayo en muestras clínicas de leche
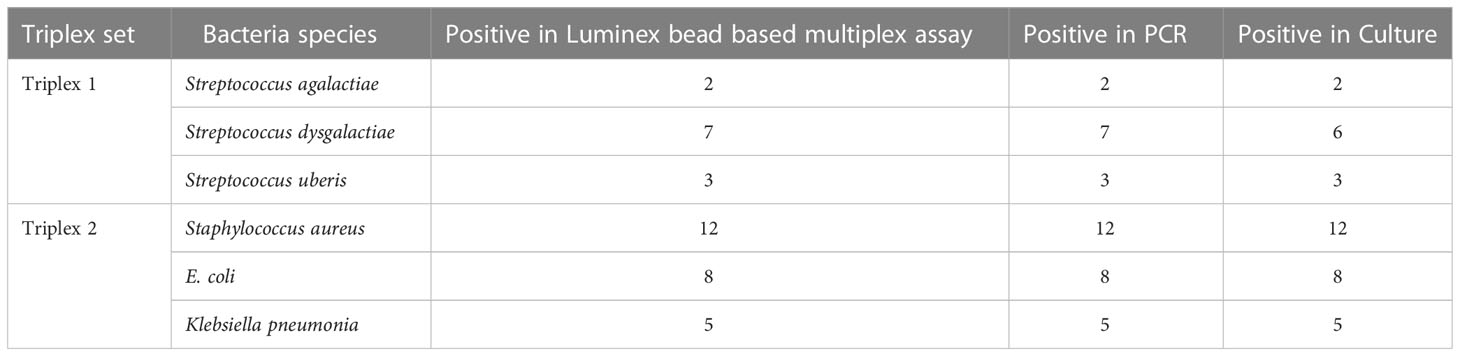
Para iniciar la validación preliminar, se utilizó muestra clínica de leche de 100 cuartos bovinos. El resultado mostró que la sensibilidad analítica del ensayo fue del 100% para Staphylococcus aureus, Streptococcus agalactiae, Streptococcus uberis, E. coli y Klebsiella pneumoniae.Sin embargo, entre los 7 aislamientos analizados paraStreptococcus dysgalactiae utilizando el método de cultivo y el ensayo basado en luminex, un solo aislado permanece sin identificar en el método de cultivo pero identificado mediante el ensayo basado en luminex. Los resultados de identificación de especies por los diferentes métodos se dan en la Tabla 2. Los resultados sugieren que no hay diferencias significativas en la identificación de especies mediante el ensayo desarrollado y la PCR, mientras que los resultados de identificación del ensayo desarrollado y el método de cultivo convencional mostraron una ligera diferencia en las especies de Streptococcus dysgalactiae.
4 Discusión
La mastitis es una enfermedad multietiológica compleja de la glándula mamaria, que afecta gravemente a la industria láctea y ganadera en todo el mundo. También conduce a un reservorio de infección para los seres humanos y al aumento de la resistencia a los antimicrobianos en los animales tratados (Cheng et al., 2019). La identificación precisa de las especies bacterianas es esencial para permitir estrategias de manejo exitosas, una intervención rápida con el uso de un tratamiento adecuado y la prevención de casos crónicos. También es esencial para la investigación de patógenos de mastitis (Chandrasekaran et al., 2014). Además, las muestras «sin crecimiento» son problemáticas para los laboratorios de mastitis, los veterinarios y los productores lácteos (Shome et al., 2011). Esto puede deberse a un menor número de bacterias en la leche, que no crecen en medios estándar o sustancias en la leche que inhiben su crecimiento (Gangwal et al., 2017). Los métodos de identificación fiables que sean rápidos y precisos siguen siendo una necesidad. Recientemente, en el pasado, se han desarrollado varios métodos de detección, incluyendo PCR, PCR múltiple y PCR múltiple en tiempo real para la identificación de bacterias causantes de mastitis (Phuektes et al., 2001; Ashraf et al., 2017; Calonzi et al., 2020). Estos ensayos moleculares multiplexados están sujetos a preocupaciones como amplificación incompatible o inadecuada, disminución de la sensibilidad y la especificidad relacionadas con la amplificación inespecífica, conjuntos de cebadores incompatibles o de reacción cruzada, alto fondo o ruido y mala reproducibilidad (Koskinen et al., 2009). Los estudios anteriores que implicaron el desarrollo de un ensayo multiplex basado en perlas Luminex acoplado a PCR múltiple sugirieron que la detección microbiana puede ser altamente sensible y específica y es un método eficiente para detectar múltiples patógenos en un solo ensayo y excedió la capacidad de PCR en tiempo real ().
La presente investigación hizo hincapié en la identificación precisa de seis patógenos bacterianos de mastitis en una sola muestra, ya que generalmente se trata de una infección mixta. El protocolo se puede hacer de alto rendimiento con la prueba de 96 muestras extraídas en pocas horas (Angeloni et al., 2013). Por lo tanto, el ensayo se optimizó utilizando ADN de concentración conocida (ng / μL) de cepas ATCC de las seis bacterias asociadas con mastitis en dos conjuntos triplex; triplex 1 compuesto por Streptococcus agalactiae, Streptococcus dysgalactiae y Streptococcus uberis, mientras que triplex 2 compuesto por Staphylococcus aureus, E. coli y Klebsiella pneumoniae. Las perlas magnéticas Luminex de diferente dirección espectral se acoplaron a la sonda específica de secuencia utilizando el protocolo de acoplamiento de cuentas del libro de cocina xMAP, 2013 con algunas modificaciones. Esta reacción de acoplamiento se optimizó utilizando solo 5 millones de perlas que es menor que el volumen recomendado, lo que reduce el costo efectivo del ensayo (Capurro et al., 2009).
La sensibilidad analítica (LOD) en términos de número de copias por reacción se calculó de manera similar al estudio de detección de siete virus entéricos (Bruse et al., 2008). Se encontró que eran 106 por reacción para las seis bacterias en ensayos monoplex y triplex. Esto fue similar al registrado en el estudio para la detección de diferentes Mycoplasma spp. donde no se observaron diferencias en la LOD en el ensayo monoplex y multiplex (Righter et al., 2011).
Se observó una MFI reducida con menor dilución de Staphylococcus aureus y E. coli que tras una dilución adicional aumentó y luego se volvió constante. Esto podría explicarse por el «efecto matriz». Esto implica el hecho de que las muestras biológicas complejas concentradas, como el suero, el plasma o los lisados tisulares, pueden provocar un efecto de matriz que conduce a interferencia o aglutinación de microesferas, recuperación deficiente de perlas, señales bajas y resultados variables (Angeloni et al., 2013). Además, también debe conocerse el rango biológico de cada analito, la especificidad de unión de los reactivos de ensayo y la composición única de la muestra. La dilución de la muestra y la adición de un paso de lavado adicional antes de la hibridación pueden eliminar la interferencia del efecto matriz en los resultados (Christopher et al., 2013). Por lo tanto, en el estudio se realizó el protocolo de hibridación lavada y dilución del analito.
No se observó reactividad cruzada similar a la especificidad analítica del 100% reportada en el ensayo para la detección simultánea de Mycoplasma spp (Righter et al., 2011). así como en el estudio para la identificación de siete virus entéricos de humanos (Bruse et al., 2008). La comparación in silico de los conjuntos de secuencias de cebador y sonda con especies estrechamente relacionadas que también están asociadas con mastitis mostró una especificidad del 100% de los conjuntos con el patógeno bacteriano específico similar al hallazgo reportado en otro estudio (Taponen et al., 2009).
El rendimiento del ensayo también se probó determinando la capacidad de detección de muestras mixtas del ensayo múltiplex basado en perlas para triplex 1 y 2. Esto fue importante ya que en la infección natural están involucradas múltiples combinaciones de patógenos bacterianos, por lo tanto, puede ser difícil de detectar. Se encontró que cada pico fue identificado por cuentas acopladas con sondas de captura específicas. La repetibilidad del ensayo también se evaluó en que el coeficiente de varianza inter e intra ensayo se encontró dentro del rango aceptable según el libro de recetas de Luminex xMAP (Angeloni et al., 2013).
Cualquier ensayo de diagnóstico debe abordar si el ensayo es «adecuado para su propósito», como se describe en el flujo de trabajo de validación del Manual Terrestre de la Organización Mundial de Sanidad Animal (OIE), 2012.
Por lo tanto, la validación preliminar del ensayo desarrollado se realizó mediante la evaluación de muestras clínicas de leche de casos sospechosos de mastitis. El resultado mostró que el porcentaje de concordancia para la identificación fue del 100% para Staphylococcus aureus, Streptococcus uberis, E. coli y Klebsiella pneumoniae entre el ensayo desarrollado, el método de cultivo y la PCR. Un solo aislado de Streptococcus dysgalactiae no fue identificado en el método convencional, probablemente debido a la pérdida de la colonia debido a la similitud fenotípica entre varios Streptococcus spp. involucrados en la mastitis (Koskinen et al., 2009).
La concordancia entre el ensayo desarrollado y los resultados de PCR convencional para todas las bacterias fue del 100% que coincidió con los registrados anteriormente para la detección simultánea de virus entéricos (Bruse et al., 2008). El ensayo desarrollado coincidió con el método convencional de identificación bacteriana de la leche en un 92,30%, lo que hace que el ensayo sea más sensible. Aunque el ensayo desarrollado tenía la limitación de la detección de solo seis bacterias en dos conjuntos diferentes, podría ampliarse fácilmente al ensayo de seis plex dependiendo de la disponibilidad de perlas, ya que el estudio de la reactividad cruzada de seis cebadores y sondas se ha realizado en el presente estudio. Además, ya se sabe que la mastitis es causada por 150 microorganismos diferentes, el ensayo desarrollado también se puede ampliar aún más para incluir sondas más específicas para identificar otras bacterias asociadas con la mastitis a la plataforma existente, reduciendo así aún más el costo de la prueba.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio se incluyen en el artículo/Material Suplementario. Las consultas adicionales pueden dirigirse a los autores correspondientes.
Declaración ética
Las muestras de leche utilizadas en el estudio se recibieron directamente en el laboratorio de los propietarios de los animales para el aislamiento bacteriano y las pruebas de sensibilidad a los antibióticos. La historia del animal con los síntomas de mastitis se registró en el momento de la presentación de la muestra. El procedimiento de ordeño / recolección de muestras de leche no implica un procedimiento invasivo, por lo tanto, no se indican permisos éticos.
Contribuciones del autor
GS desarrolló las hipótesis, diseñó y realizó los experimentos, analizó e interpretó los datos, y redactó el manuscrito como parte de su trabajo de investigación de doctorado; RC administró el proyecto de investigación general, supervisó los experimentos, el análisis y la interpretación de los datos y revisó el manuscrito; AS revisó críticamente el manuscrito; KB ayudó con los experimentos; ST ayudó en el aislamiento genómico de ADN de colonias bacterianas y muestras de leche; SM ayudó en el diseño de la sonda y proporcionó experiencia en el desarrollo del ensayo Luminex y revisó el manuscrito. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Reconocimientos
La primera autora reconoce al Consejo Indio de Investigación Médica por proporcionar una beca de investigación senior durante su programa de doctorado en la Universidad Lala Lajpat Rai de Ciencias Veterinarias y Animales, Hisar, India.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fcimb.2023.1125562/full#supplementary-material
Referencias
Abdi, R. D., Gillespie, B. E., Ivey, S., Pighetti, G. M., Almeida, R. A., Kerro Dego, O. (2021). Resistencia antimicrobiana de los principales patógenos bacterianos de vacas lecheras con alto recuento de células somáticas y mastitis clínica. Animales 11 (1), 131. doi: 10.3390/ani11010131
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Angeloni, S., Cordes, R., Dunbar, S., Garcia, C., Gibson, G., Martin, C., et al. (2013). xMAP cookbook: a collection of methods and protocols for developing multiplex assays with xMAP technology (Austin, TX, USA: Luminex).
Ashraf, A., Imran, M., Yaqub, T., Tayyab, M., Shehzad, W., Thomson, P. C. (2017). Un nuevo ensayo de PCR multiplex para la detección simultánea de nueve patógenos bacterianos clínicamente significativos asociados con la mastitis bovina. Mol. Sondas 33, 57–64. doi: 10.1016/j.mcp.2017.03.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Basile, A. J., Brad, J. B., Olga, L. K., Shilpa, R. J., Nicholas, A. P., Ann, M. P., et al. (2010). Eliminación de las restricciones de especies en la detección de anticuerpos. Clin. Vac. Immunol. 17, 56–61. doi: 10.1128/CVI.00291-09
Bradley, A. J., Leach, K. A., Breen, J. E., Green, L. E., Green, M. J. (2007). Estudio de la incidencia y etiología de la mastitis en granjas lecheras en Inglaterra y Gales. Rec. 160, 253–258. DOI: 10.1136/VR.160.8.253
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bruse, S. E., Michael, P. M., Marco, A. A., Ray, Z., Linda, M. B. (2008). Mejoras en los ensayos de genotipado de SNP de ligadura de oligonucleótidos basados en perlas. Biotechniq 45, 559–571. DOI: 10.2144/000112960
Calonzi, D., Romano, A., Monistero, V., Moroni, P., Luini, M. V., Biscarini, F., et al. (2020). Nota técnica: desarrollo de ensayos de PCR multiplex para la caracterización molecular de cepas de Streptococcus uberis aisladas de mastitis bovina. J. Dairy Sci. 103 (1), 915–921. DOI: 10.3168/JDS.2019-16823
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Capurro, A. K., Artursson, K., Waller, P., Bengtsson, B., Ericsson-Unnerstad, H., Aspán, A. (2009). Comparación de un sistema de fenotipado comercializado, pruebas de susceptibilidad antimicrobiana y genotipado basado en la secuencia del gen tuf para la identificación a nivel de especie de estafilococos coagulasa negativos aislados de casos de mastitis bovina. Veterinario Microbiol. 134, 327–333. doi: 10.1016/j.vetmic.2008.08.028
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chandrasekaran, D., Venkatesan, P., Tirumurugaan, K. G., Nambi, A. P., Thirunavukkarasu, P. S., Kumanan, K., et al. (2014). Patrón de mastitis resistente a antibióticos en vacas lecheras. Mundo Veterinario 7 (6), 389–394. doi: 10.14202/vetworld.2014.389-394
Cheng, J., Qu, W., Barkema, H. W., Nobrega, D. B., Gao, J., Liu, G., et al. (2019). Perfiles de resistencia antimicrobiana de 5 patógenos comunes de mastitis bovina en grandes rebaños lecheros chinos. J. Dairy Sci. 102, 2416–2426. DOI: 10.3168/JDS.2018-15135
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Christopher-Hennings, J., Karla, P. C. A., Carlos, J. H., Ying, F., Steven, L., et al. (2013). Oportunidades para ensayos multiplex basados en cuentas en laboratorios de diagnóstico veterinario. J. Vet. Diag. Invest. 25, 671–691. DOI: 10.1177/1040638713507256
Deregt, D., Scott, A., Gilbert, S. D., John, P., Shailja, B., Kimberley, M. B., et al. (2006). Un microarray de suspensión de ADN multiplex para la detección y diferenciación simultáneas del virus de la peste porcina clásica y otros pestivirus. J. Virol. Métodos 136, 17–23. doi: 10.1016/j.jviromet.2006.03.025
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dingwell, R. T., Leslie, K. E., Schukken, Y. H., Sargeant, J. M., Timms, L. L. (2003). Evaluación de la prueba de mastitis de California para detectar una infección intramamaria con un patógeno importante en vacas lecheras de lactancia temprana. Can. Vet. J. 44 (5), 413–415.
Down, P. M., Andrew, J., Bradley, J. E., Martin, J. (2017). Factores que afectan la rentabilidad del cultivo en granja antes del tratamiento de la mastitis clínica en vacas lecheras. Prev. Vet. Med. 145, 91–99. doi: 10.1016/j.prevetmed.2017.07.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Gangwal, A., Kashyap, S. K., Katiyar, S., Meena, D., Boyal, P., Gupta, K. (2017). Aislamiento e identificación de patógenos causantes de mastitis comunes a partir de leche mastítica bovina clínica. J. Pure App. Microbiol. 11, 329–333. doi: 10.22207/JPAM.11.1.41
Hsu, C. C., Christiane, E. W., Earl, K. S., Lela, K. R., Robert, S. L. (2005). Desarrollo de un inmunoensayo fluorescente multiplexado serológico basado en microesferas y un ensayo de PCR con transcriptasa inversa para detectar la infección por norovirus murino 1 en ratones. Clin. Vacuna Immunol. 12, 1145–1151. doi: 10.1128/CDLI.12.10.1145-1151.2005
Hsu, C. C., Craig, F., Lela, K. R. (2007). Inmunoensayo fluorescente multiplex para la detección simultánea de anticuerpos séricos contra múltiples patógenos de roedores. Laboratorio. Ani. 36, 36–38. DOI: 10.1038/LABAN0907-36
Koskinen, M. T., Holopainen, J., Pyörälä, S., Bredbacka, P., Pitkälä, A., Barkema, H. W., et al. (2009). Especificidad analítica y sensibilidad de un ensayo de reacción en cadena de la polimerasa en tiempo real para la identificación de patógenos de mastitis bovina. J. Dairy Sci. 92, 3952–3959. DOI: 10.3168/JDS.2008-1549
Leblanc, N., Gantelius, J., Schwenk, J. M., Ståhl, K., Blomberg, J., Andersson-Svahn, H., et al. (2009). Desarrollo de un microarray de perlas magnéticas para la detección simultánea y sencilla de cuatro pestivirus. J. Virol. Métodos 155 (1), 1–9. doi: 10.1016/j.jviromet.2008.04.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Pascu, C., Herman, V., Iancu, I., Costinar, L. (2022). Etiología de la mastitis y la resistencia a los antimicrobianos en granjas de ganado lechero en la parte occidental de Rumania. Antibióticos (Basilea) 11 (1), 57. doi: 10.3390/antibióticos11010057
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Phuektes, P. P., Mansell, D., Browning, G. F. (2001). Ensayo de reacción en cadena de la polimerasa multiplex para la detección simultánea de Staphylococcus aureus y causas estreptocócicas de mastitis bovina. J. Dairy Sci. 84, 1140–1148. DOI: 10.3168/JDS. S0022-0302(01)74574-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Reslova, N., Michna, V., Kasny, M., Mikel, P., Kralik, P. (2017). Tecnología xMAP: aplicaciones en detección de patógenos. Frente. Microbiol. 8, 55. DOI: 10.3389/fmicb.2017.00055
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Righter, D. J., Fred, R. R., Douglas, R. C., Terry, F. M. (2011). Desarrollo de un ensayo de PCR multiplex basado en perlas para la detección simultánea de múltiples especies de Mycoplasma. Veterinario Microbiol. 153, 246–256. doi: 10.1016/j.vetmic.2011.06.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ros, G. A., García-Pérez, A. L., Verdera, J., Juste, R. A., Hurtado, A. (2012). Seguimiento de la infección por piroplasmas en tres explotaciones ganaderas de Menorca (Islas Baleares, España) con antecedentes de piroplamosis clínica. Veterinario Parasito. 90, 318–325. doi: 10.1016/j.vetpar.2012.07.024
Shome, B. R., Das Mitra, S., Bhuvana, M., Krithiga, N., Velu, D., Shome, R., et al. (2011). Ensayo de PCR multiplex para la identificación de especies de patógenos de mastitis bovina. J. App. Microbiol. 111, 1349–1356. doi: 10.1111/j.1365-2672.2011.05169.x
Sinha, M. K., Thombare, N. N., Mondal, B. (2014). Mastitis subclínica en animales lecheros: incidencia, economía y factores predisponentes. Sci. Mundo J. 523984. doi: 10.1155/2014/523984
Taponen, S., Salmikivi, L., Simojoki, H., Koskinen, M. T., Pyorala, S. (2009). Identificación basada en la reacción en cadena de la polimerasa en tiempo real de bacterias en muestras de leche de mastitis clínica bovina sin crecimiento en cultivos convencionales. J. Dairy Sci. 92, 2610–2617. DOI: 10.3168/JDS.2008-1729
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Watson, D. S., Sanjay, M. R., Vinayak, B., Blanca, L. (2009). Inmunoensayo multiplexado para la detección de anticuerpos contra el virus de la influenza aviar. J. Immunol. Métodos 340, 123–131. doi: 10.1016/j.jim.2008.10.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
White, D. G., McDermott, P. F. (2001). Aparición y transferencia de resistencia antibacteriana. J. Dairy Sci. 84, E151–E155. DOI: 10.3168/JDS. S0022-0302(01)70209-3
Woolhouse, M., Alex, D. (2001). Gestión de la fiebre aftosa. Nature 410, 515–516. doi: 10.1038/35069250
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cita: Shrinet G, Chhabra R, Sharma A, Batra K, Talukdar SJ y Maan S (2023) High throughput Luminex beads based multiplex assay for identification of six major bacterial pathogens of mastitis in dairy animals. Frente. Celda. Infecta. Microbiol. 13:1125562. doi: 10.3389/fcimb.2023.1125562
Recibido: 16 de diciembre de 2022; Aprobado: 03 Julio 2023;
Publicado: 18 julio 2023.
Editado por:
Baljit Singh, Universidad de Saskatchewan, Canadá
Revisado por:
Nagendra R. Hegde, Instituto Nacional de Biotecnología Animal (NIAB), India
Sunghyun Yoon, Centro Nacional de Investigación Toxicológica (FDA), Estados Unidos
Derechos de autor © 2023 Shrinet, Chhabra, Sharma, Batra, Talukdar y Maan. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Rajesh Chhabra, rchhabra59@rediffmail.com; rajesh.chhabra@luvas.edu.in; Sushila Maan, sushilamaan105@googlemail.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía