Evaluación metagenómica y química emparejada de croquetas de perro contaminadas con aflatoxinas
Evaluación metagenómica y química emparejada de croquetas de perro contaminadas con aflatoxinas
 Andrea Ottesen1*
Andrea Ottesen1*  Brandon Kocurek1
Brandon Kocurek1  Elizabeth Reed2
Elizabeth Reed2  Seth Commichaux2
Seth Commichaux2  Marcos Mammel2
Marcos Mammel2  Padmini Ramachandran2
Padmini Ramachandran2  Patricio McDermott1
Patricio McDermott1  Brenna M. Flannery2
Brenna M. Flannery2  Cepa Errol2
Cepa Errol2- 1Centro de Medicina Veterinaria (CVM), Administración de Alimentos y Medicamentos de EE. UU., Laurel, MD, Estados Unidos
- 2Centro de Seguridad Alimentaria y Nutrición Aplicada (CFSAN), Administración de Alimentos y Medicamentos de EE. UU., College Park, MD, Estados Unidos
Introducción: La identificación de toxinas químicas de alimentos complejos o altamente procesados puede presentar desafíos de «aguja en el pajar» para los químicos. Los datos metagenómicos se pueden utilizar para guiar las evaluaciones de toxicidad química al proporcionar una descripción basada en el ADN de la composición holística (eucariota, bacteriana, protozoaria, viral y resistencia a los antimicrobianos) de los alimentos que se sospecha que albergan toxinas, alérgenos o patógenos. Este tipo de información puede centrar los diagnósticos basados en la química, mejorar la caracterización de los peligros y la evaluación de riesgos, y abordar las lagunas de datos. Además, cada vez se reconoce más que las micotoxinas concurrentes simultáneamente, ya sea de una o varias especies, pueden afectar la exposición a la toxicidad de la dieta. Los datos metagenómicos proporcionan una forma de abordar las lagunas de datos relacionadas con la co-ocurrencia de múltiples especies de hongos.
Métodos: Se utilizaron datos metagenómicos y químicos emparejados para evaluar croquetas contaminadas con aflatoxinas con niveles conocidos de micotoxinas específicas. Las croquetas se molieron hasta convertirlas en un polvo fino para análisis químicos y moleculares. Los análisis químicos se realizaron con Cromatografía Líquida por Espectrometría de Masas (LCMS) y de acuerdo con el método oficial de la AOAC 2005.08: Aflatoxinas en Maíz, Maní Crudo y Mantequilla de Maní utilizando Cromatografía Líquida con Derivatización Fotoquímica Post-Columna. Los metagenomas se crearon a partir de ADN extraído de croquetas molidas y secuenciadas en un Illumina NextSeq 2000 con una profundidad de secuencia promedio de 180 millones de lecturas por réplica.
Resultados y discusión: Los datos metagenómicos demostraron que la abundancia de ADN de Aspergillus spp. aflatoxigénico putativo se correlacionó con los niveles de aflatoxina cuantificados por LCMS. Los datos metagenómicos también identificaron una amplia gama de taxones fúngicos coexistentes que pueden producir micotoxinas adicionales. Los datos de ADN, junto con los datos químicos, proporcionan una modalidad novedosa para abordar las lagunas de datos actuales en torno a la exposición a micotoxinas en la dieta, la taxonomía de los hongos toxigénicos y las micotoxinas de preocupación emergente.
Introducción
La evaluación de nuevos enfoques metodológicos para identificar los peligros microbiológicos y químicos en los alimentos para humanos y animales es un enfoque central de la investigación de la FDA (1). El avance de la ciencia para mejorar la política regulatoria es un dogma central que sustenta la misión de salud pública de la Agencia. En la actualidad, los datos genómicos y metagenómicos se utilizan para aplicaciones de seguridad alimentaria, como el seguimiento de patógenos en origen (2, 3), la identificación de adulterantes, contaminantes, toxinas y alérgenos (4) y la detección y predicción de la resistencia a los antimicrobianos (RAM) (5). Una nueva frontera de utilidad para los datos metagenómicos incluye el apoyo a las evaluaciones de toxicidad química a través de la provisión de perfiles holísticos basados en el ADN de componentes macro (plantas, animales e insectos) y micro (bacterias, hongos, virales y RAM) de alimentos humanos y animales no procesados y altamente procesados.
Las herramientas moleculares han facilitado un cambio de paradigma en nuestra comprensión de la ecología de la patogenicidad asociada a los alimentos humanos y animales (6). Una tendencia similar en la evaluación del riesgo de micotoxinas ha evolucionado para considerar la coexposición simultánea a diversas micotoxinas (7). Las micotoxinas son metabolitos secundarios producidos por hongos. Hay cientos de fracciones diferentes producidas por una o varias especies. Es raro que exista una sola micotoxina en cualquier cultivo o alimento debido a la compleja biodiversidad de las ecologías agrícolas (8). Las especies de hongos reportadas para el maíz solo incluyen Fusarium proliferatum, Trichoderma gamsii, T. longibrachiatum, Penicillium oxalicum, P. aurantiogriseum, P. polonicum, Bipolaris zeicola, Sarocladium zeae, Chaetomium murorum, Botrytrichum murorum, Cladosporium cladosporioides, C. sphaerospermum, Aspergillus niger, A. flavus, Alternaria alternata y Rhizopus microsporus (9), de los cuales se sabe que al menos la mitad producen toxinas importantes para la salud humana y animal.
Hasta la fecha, la evaluación del riesgo de las micotoxinas en los alimentos se ha centrado principalmente en un pequeño número de toxinas importantes con efectos adversos críticos, sin tener en cuenta ampliamente la coexposición a múltiples compuestos (7), a pesar de que la presencia de múltiples micotoxinas es más común que la presencia de una sola micotoxina. Estudios recientes sobre la alimentación animal en Europa revelaron que entre el 75 y el 100 % de los piensos examinados contenían más de una micotoxina (10). La exposición a una sola toxina o a múltiples micotoxinas puede provocar efectos adversos (7). Si bien se ha realizado mucho trabajo para describir la coexposición a las micotoxinas, todavía hay más que aprender sobre las toxicidades dietéticas asociadas con las micotoxinas concurrentes.
Aspergillus flavus y A. parasiticus son dos de las especies aflatoxigénicas más tradicionalmente reconocidas en productos básicos antes y después de la cosecha, pero hay muchas otras especies de Aspergillus productoras de aflatoxinas, e incluso se ha propuesto que las especies de Fusarium, Penicillium, Claviceps y Alternaria pueden producir aflatoxinas (11, 12). La Agencia Internacional para la Investigación del Cáncer (IARC, por sus siglas en inglés) ha clasificado las aflatoxinas como los carcinógenos naturales más potentes conocidos por la humanidad y se estima que contaminan el 25% de los cultivos en todo el mundo (13). Muchos ingredientes comúnmente utilizados para alimentos de origen animal, como el maíz, el trigo y el arroz, son susceptibles a la contaminación por micotoxinas.
La secuenciación del genoma completo (WGS, por sus siglas en inglés) y la secuenciación metagenómica (MGS, por sus siglas en inglés) se han utilizado para describir cómo los patógenos y las toxinas se asocian con los alimentos humanos y animales (6, 14-19), pero una nueva frontera de datos químicos y metagenómicos integrados está en el horizonte para la evaluación modernizada de las toxinas químicas en los alimentos. Aquí utilizamos datos de MGS para describir cómo la abundancia taxonómica (cantidad de ADN) de la especie de hongo aflatoxigénico putativo, Aspergillus, se correlacionó con los niveles de aflatoxina cuantificados por espectrometría de masas por cromatografía líquida (LCMS). Se demostró que dos niveles de croquetas contaminadas con aflatoxinas, uno a 15 ppb y otro a 522 ppb, se correlacionan con una abundancia baja y alta (respectivamente) de ADN de Aspergillus. Además, demostramos que una amplia gama de especies que pueden producir micotoxinas adicionales podrían identificarse mediante datos metagenómicos, para abordar mejor las brechas de datos actuales y modernizar la evaluación del riesgo de micotoxinas.
Materiales y métodos
Evaluación de micotoxinas
Se midieron niveles bajos (15 ppb) y altos (522 ppb) de croquetas contaminadas con aflatoxinas de acuerdo con los procedimientos operativos estándar de la División de Industrias Vegetales del Departamento de Agricultura de Missouri, que utiliza el método oficial AOAC 2005.08: Aflatoxinas en maíz, maní crudo y mantequilla de maní (cromatografía líquida con derivatización fotoquímica posterior a la columna). Posteriormente, las muestras se enviaron al Centro de Medicina Veterinaria de la FDA para su análisis metagenómico. Las croquetas que miden 15 ppb de aflatoxina se consideraron «bajas» y las croquetas que miden 522 ppb se consideraron muestras de aflatoxina de nivel «alto» para los fines de este estudio. La aflatoxina a 15 ppb en las croquetas está por debajo del nivel de acción de la FDA para las aflatoxinas de 20 ppb en los alimentos para mascotas (20). Un laboratorio de evaluación de piensos de terceros llevó a cabo una evaluación adicional de micotoxinas múltiples de LCMS (Cumberland Valley Analytical Services, Zullinger, PA). Las micotoxinas evaluadas incluyeron aflatoxina, fumonisina, ocratoxina A y zearalenona en croquetas de control y contaminadas.
Se tomaron muestras de «control» de exactamente la misma marca de alimento para perros, pero con un número de lote diferente (que no forma parte de las croquetas contaminadas con aflatoxinas retiradas del mercado) para representar una línea de base. Los ingredientes de las croquetas enumerados son harina de subproductos de pollo, maíz, trigo, harina de carne, salvado de arroz, grasa de pollo, pulpa de remolacha seca, harina de pescado blanco, linaza, sal, cloruro de potasio, cloruro de colina, vitaminas y minerales. La etiqueta describía una composición de al menos 26% de proteína, 15% de grasa con un máximo de fibra al 6%, humedad al 10% y 3.645 kcal/kg.
Preparación del metagenoma
Tres porciones de 2,5 g de cada nivel de croquetas contaminadas con aflatoxinas y control se molieron hasta obtener un polvo fino en un Qiagen Tissue Lyser II a 30 Hz (1800 oscilaciones por minuto) durante 1,3 min por muestra. Para la extracción de ADN se utilizaron réplicas de 100 mg de polvo de la mezcla combinada (7,5 gramos). La extracción de ADN se realizó utilizando el kit de extracción de ADN de alto peso molecular Zymo de acuerdo con las especificaciones del fabricante, incluida una etapa de lisis adicional con PBS y lisozima (21). Las bibliotecas de ADN se crearon utilizando el kit de preparación de bibliotecas de ADN de Illumina de acuerdo con los protocolos especificados por el fabricante y la secuenciación se realizó en un NextSeq 2000 utilizando un kit de alto rendimiento de acuerdo con los métodos descritos anteriormente (22). Se realizó una ejecución adicional de secuenciación de alto rendimiento NextSeq 2000 con solo dos réplicas de muestras de bajo y alto nivel para lograr una profundidad de secuenciación de entre 100 y 250 millones de lecturas por réplica para evaluar cómo el aumento de la profundidad de lectura afectó la incidencia de especies clave.
Análisis bioinformáticos
Los datos de la secuencia se examinaron para determinar las métricas de calidad utilizando Trimmomatic (23) y se analizaron utilizando tuberías y bases de datos internas de la FDA, como se describió anteriormente (22). La determinación de la composición bacteriana y fúngica a partir de la secuenciación de escopeta se llevó a cabo utilizando programas personalizados de C++ desarrollados para compilar una base de datos de firmas k-mer que contiene múltiples secuencias únicas de 30 pb por especie y luego identificar cada lectura en el archivo de entrada utilizando las sondas de 30 pb. Para cada especie o subespecie bacteriana o fúngica, cada 30 meros no duplicado de una secuencia de genoma completo de referencia se colocó en una base de datos. Se eliminaron los k-mers que no se encontraban en al menos 2/3 de un conjunto de secuencias genómicas adicionales de la misma especie y se eliminaron los k-mers encontrados en genomas de otras especies. La normalización se realizó para corregir el sesgo debido al diferente número de k-mers por entrada de la base de datos y los resultados se tabularon como porcentaje de lecturas identificadas (contribución a la población microbiana de las especies identificadas) para cada entrada de la base de datos. Los resultados se pueden expresar por golpes brutos o como abundancia relativa. Ambos son útiles cuando se detectan organismos de baja abundancia en metagenomas complejos. La anotación también se realizó utilizando la aplicación basada en la nube Cosmos ID (CosmosID Metagenomics Cloud, app.cosmosid.com, CosmosID Inc., www.cosmosid.com) con Fungal Database Version 1.2 (24). LEfSe (Linear discriminant analysis effect size) por la biopanadería Huttenhower1 (25) se calculó en la aplicación COSMOSID. LEfSe (Linear discriminant analysis effect size) es un algoritmo utilizado para el descubrimiento de biomarcadores biológicos. Se identificaron características como genes, vías o taxones para cada tratamiento y se utilizó la prueba factorial no paramétrica de rangos de suma de Kruskal-Wallis (KW) (26) para identificar una abundancia diferencial significativa de características específicas entre los tratamientos. Se utilizó el Análisis Discriminante Lineal (LDA) para estimar el tamaño del efecto de cada característica diferencialmente abundante y clasificar la característica en consecuencia. Para el gráfico de barras presentado en la sección de resultados, el LDA varió de 2,9 a 5,27 y el valor de p varió de 0,022 a 0,035. Las anotaciones de datos de secuencia se visualizaron utilizando gráficos creados por el paquete R Tidyverse.2
Identificación de la vía de las micotoxinas
Las lecturas metagenómicas se mapearon con Diamond (v2.0.5) (27) BLASTX (≥95% de identidad y ≥90% de cobertura de lectura) a una base de datos de genes involucrados en las vías biosintéticas de micotoxinas: aflatoxina, deoxinivalenol, nivalenol, ocratoxina, patulina, esterigmatocistina, T2_toxin y tenuazonic_acid. Los genes fueron identificados y descargados de la base de datos MetaCyc (28).3 La identidad de las lecturas que se alinean con los genes de micotoxinas fue confirmada por BLASTX, alineándolas con la base de datos NCBI nr en línea.
Resultados
Ingredientes de croquetas identificados mediante secuenciación metagenómica
Para describir la composición y la abundancia relativa de los ingredientes, se crearon datos metagenómicos para tres réplicas de control, tres réplicas de croquetas contaminadas con aflatoxinas de baja concentración (15 ppb) y tres réplicas de croquetas contaminadas con aflatoxinas de alta concentración (522 ppb). Se utilizó un promedio de 182 millones de secuencias por réplica en los análisis posteriores. La abundancia relativa de macroingredientes [anotados por el ADN mitocondrial (29) y que ocurren en más del 1% de los datos normalizados] incluyeron Zea (maíz), Gallus (pollo), Triticum (trigo), soja (soja), Bos (vaca, buey, toro, yak, ganado) y levadura. Un mayor refinamiento de las anotaciones de las especies describió Z. mays, G. gallus, G. gallus ssp. spadiceus, T. aestivum, B. taurus, varias especies de Saccharomyces (cerevisiae, pastorianus y pastorianus Weihenstephan) y Fusarium verticillioides.
Especies de Aspergillus en croquetas
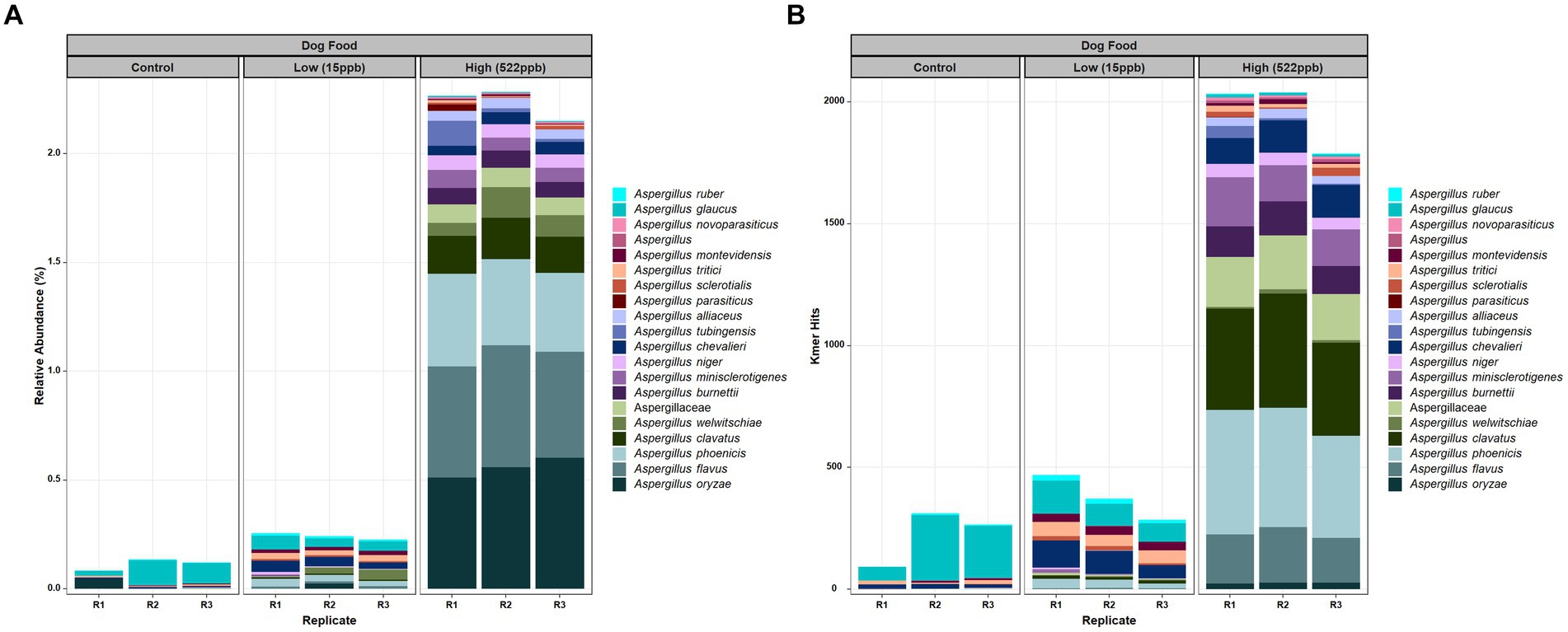
La evaluación de los datos metagenómicos de las croquetas de control, de bajo y alto nivel que contenían aflatoxinas se correlacionó con la abundancia relativa de ADN de las especies de Aspergillus, es decir, las croquetas contaminadas con aflatoxinas de bajo nivel tenían una baja abundancia de ADN de las especies de Aspergillus y las croquetas de alto nivel tenían una mayor abundancia relativa de Aspergillus especie. La incidencia de Aspergillus o de cualquier otro taxón puede expresarse mediante aciertos brutos en el genoma de interés o como abundancia relativa después de la normalización por recuento de k-mer por organismo y número de lecturas de secuencia. Casi no se detectó ADN de Aspergillus en los controles (Figura 1). La especie de Aspergillus identificada en las croquetas de control fue predominantemente A. glaucus. La Figura 1A muestra las veinte especies de Aspergillus más abundantes identificadas en los niveles control, bajos (15 ppb) y altos (522 ppb) de croquetas para perros contaminadas con aflatoxinas utilizando la normalización de abundancia relativa. La Figura 1B muestra los impactos de k-mer en bruto a las 20 principales especies de Aspergillus observadas en los metagenomas. Ambos enfoques, abundancia relativa y aciertos de k-mer en bruto, son útiles cuando se intenta detectar ADN de baja abundancia de organismos de baja abundancia. Hubo un extenso rango taxonómico de especies de Aspergillus identificadas en las croquetas. La producción de aflatoxina puede haber estado asociada con una sola especie o potencialmente con varias especies. Aspergillus oryzae, flavus y phoenicis fueron las especies más abundantes observadas en croquetas contaminadas con aflatoxinas de alto nivel (522 ppb). Aspergillus flavus y A. parasiticus son dos de los productores de aflatoxina más tradicionalmente reconocidos en productos básicos antes y después de la cosecha (12). Es probable que estas especies desempeñaran un papel en la producción de aflatoxinas en las croquetas, ya que no se sabe que A. oryzae y A. phoenicis produzcan aflatoxinas (30, 31).
Figura 1. Especies de Aspergillus en croquetas contaminadas con aflatoxinas de bajo y alto nivel de control. (A) Abundancia relativa de las veinte especies principales de Aspergillus y (B) Golpes de k-mer crudos (sin normalización) a las especies de Aspergillus. El Aspergillus se anotó utilizando una base de datos de hongos interna de la FDA, como se describe en Métodos.
Detección adicional de micotoxinas y especies asociadas
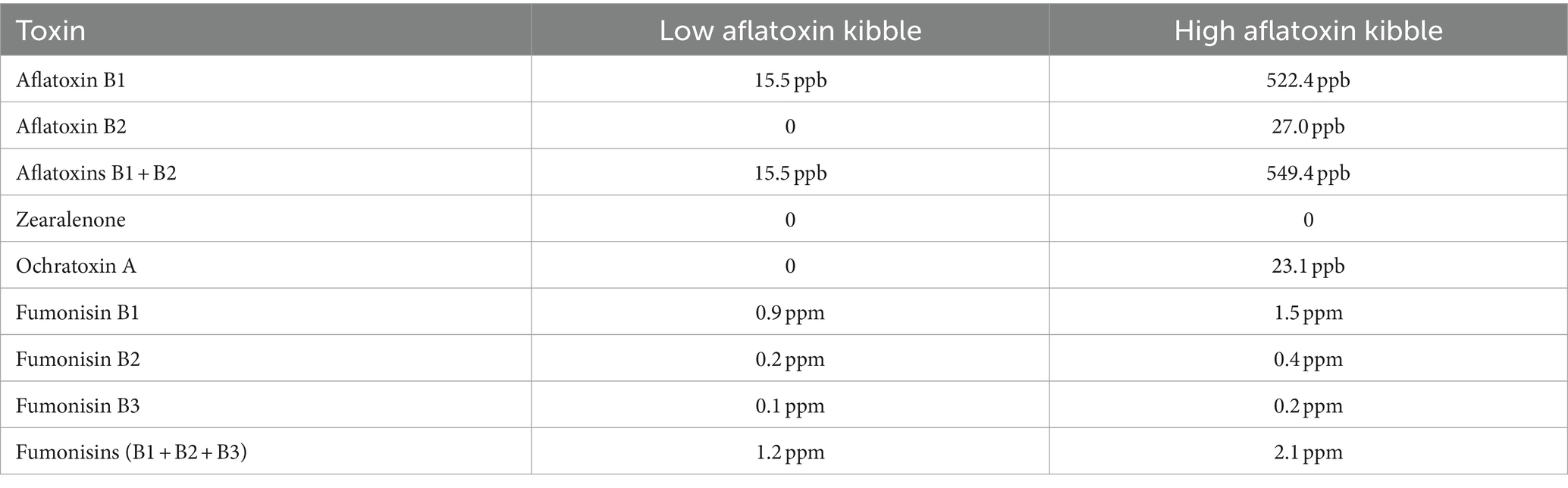
Si bien la aflatoxina B1 fue el foco principal de la evaluación química de las croquetas debido a su toxicidad aguda, las concentraciones de fumonisinas (B1, B2, B3) y ocratoxina A también fueron detectadas por LCMS (Tabla 1).
Tabla 1. Concentraciones químicas de micotoxinas en niveles bajos y altos de croquetas para perros contaminadas con aflatoxinas.
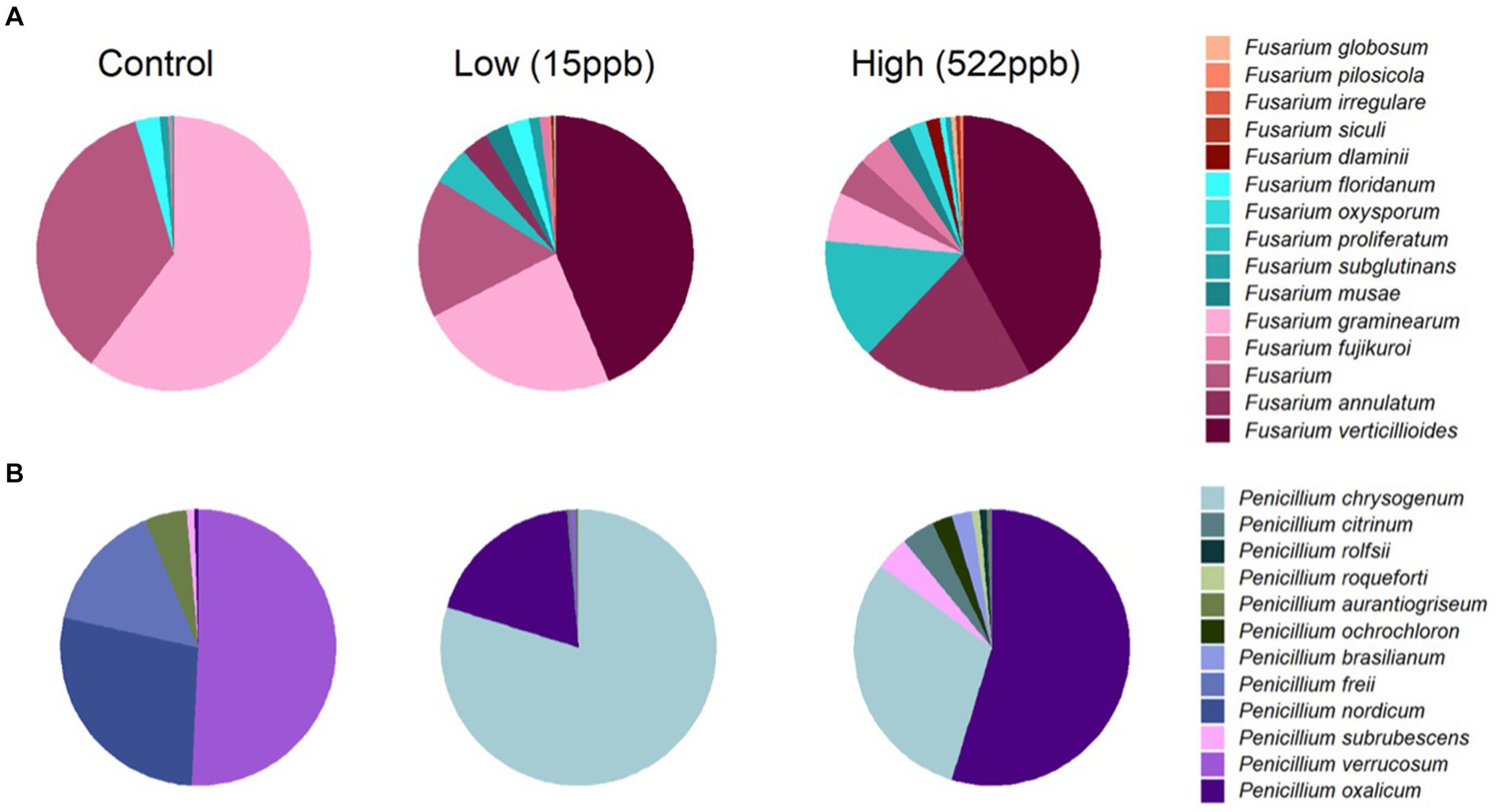
Se ha informado que Fusarium verticillioides produce fumonisinas y zearalenona y se observó en muestras bajas y altas de croquetas, pero no en controles (Figura 2A). Otras especies de Fusarium, como F. annulatum, proliferatum, dlaminii, siculi, irregulare, pilosicola y globosum, se asociaron principalmente con croquetas contaminadas y no se observaron en los controles (Figura 2A). El deoxinivalenol también es producido por especies de Fusarium, pero no se midió en este estudio.
Figura 2. Abundancia relativa de especies de Fusarium y Penicillium en croquetas contaminadas con aflatoxinas de control, niveles bajos y altos. (A) Muestra la abundancia relativa promedio de especies de Fusarium de tres réplicas de cada tipo de croquetas (control, contaminación por aflatoxinas de bajo y alto nivel) anotadas utilizando una base de datos de hongos de la FDA desarrollada para datos metagenómicos. (B) Muestra la abundancia relativa promedio de especies de Penicillium en cada tipo de croquetas. Las croquetas contaminadas tuvieron una incidencia distintiva de Fusarium verticillioides, Penicillium oxalicum y P. chrysogenum, que no se observó en las croquetas de control.
Penicillium spp. Se ha reportado que producen ocratoxina A y patulina (32). Penicillium oxalicum, subrubescens, brasilianum, ochrochloron, roqueforti y citrinum se observaron principalmente en muestras contaminadas, mientras que P. verrucosum, nordicum y freii fueron las especies más abundantes observadas en los controles (Figura 2B). Es posible que P. oxalicum o P. subrescens desempeñaran un papel en los niveles de ocratoxina observados (23 ppb) en muestras «altas». Sin embargo, P. oxalicum también se observó en muestras de bajo nivel para las cuales no se cuantificó ocratoxina, por lo que quizás otros géneros que se enriquecieron diferencialmente en muestras de alto nivel fueron responsables de la ocratoxina observada por LCMS (Tabla 1).
Descubrimiento de biomarcadores
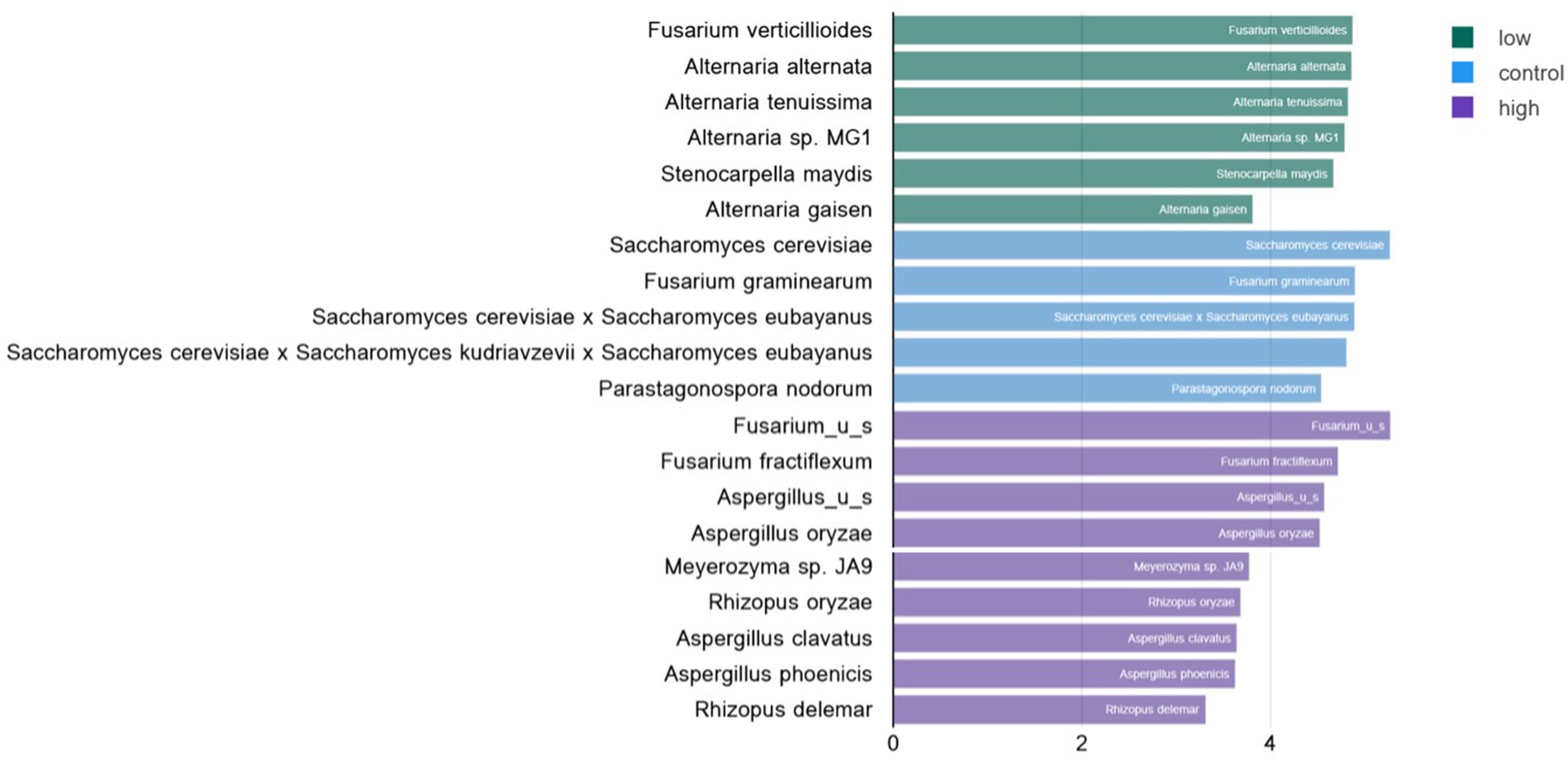
Se calculó el análisis discriminante lineal de especies fúngicas en croquetas contaminadas de bajo y alto nivel utilizando las herramientas de biopanadería de Huttenhowever (25) disponibles en la línea de análisis de COSMOSID (CosmosID Metagenomics Cloud, app.cosmosid.com, CosmosID Inc., www.cosmosid.com). Esta herramienta funciona como una herramienta de descubrimiento de biomarcadores, comparando la abundancia diferencial de taxonomía o genes funcionales en diferentes grupos de tratamiento. Utilizado con la taxonomía fúngica para el control, las croquetas contaminadas con aflatoxinas de bajo y alto nivel, quedó claro que las croquetas de alto nivel contenían una mayor abundancia de especies de Aspergillus que las croquetas de bajo nivel (Figura 3). Curiosamente, las croquetas de control tenían significativamente más especies de Saccharomyces que las croquetas de alto nivel (p < 0,022 a 0,035). Es una práctica común agregar levadura a los ingredientes que contienen aflatoxina. Se ha demostrado que las especies de levadura se unen a la aflatoxina B y, por lo tanto, reducen el impacto tóxico del consumo de alimentos contaminados con aflatoxinas (33). Se ha demostrado que este enfoque proporciona un efecto protector a los pollos de engorde que consumen alimentos contaminados con aflatoxina B1 (34). La levadura figuraba como ingrediente en todas las croquetas para perros.
Figura 3. Análisis discriminado lineal (LDA) de abundancia diferencial significativa de especies fúngicas en croquetas para perros contaminadas con aflatoxinas de bajo, control y alto nivel. Utilizando la herramienta LefSE disponible en la tubería de anotación COSOMOSIDA se describieron los taxones que se enriquecieron significativamente en cada tipo de croqueta (control, bajo, alto). El umbral LDA osciló entre 2,9 y 5,27 con un rango de valores de p de 0,022 a 0,035. Este enfoque proporciona biomarcadores que pueden correlacionarse con el riesgo de contaminación por aflatoxinas o que pueden correlacionarse con croquetas seguras o de bajo riesgo.
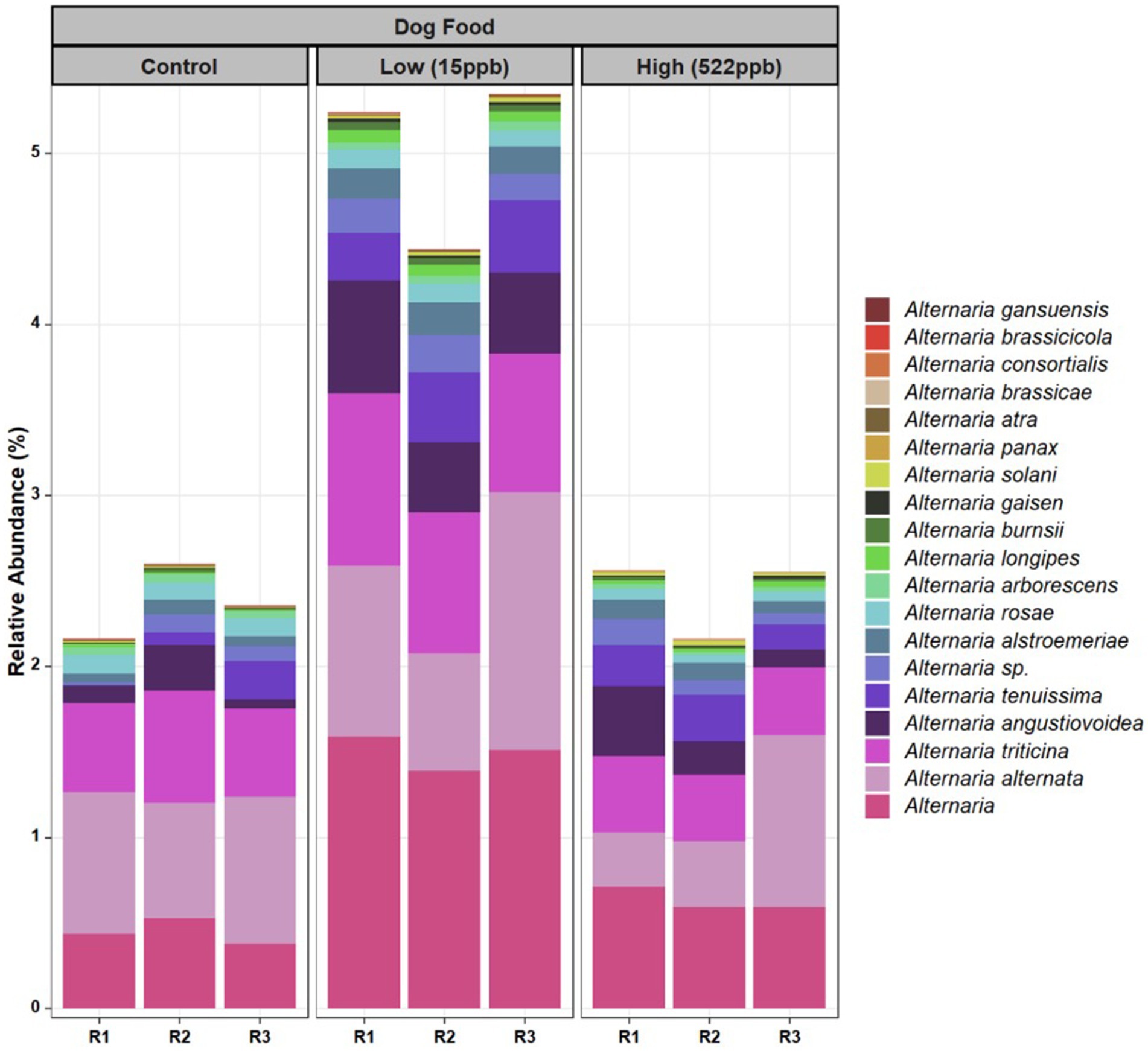
Además, se observó una mayor abundancia relativa de especies de Alternaria en croquetas contaminadas con aflatoxinas de bajo nivel. Si bien se observaron especies de Alternaria en los datos metagenómicos, en este estudio no se midieron las toxinas asociadas a Alternaria (altenueno, alternariol, monometil éter alternariol, tentoxina y ácido tenuazónico). La Figura 4 proporciona un desglose de las especies de Alternaria en cada tipo de croquetas.
Figura 4. Especies de Alternaria identificadas en croquetas contaminadas con aflatoxinas de bajo y alto nivel de control. Las especies de Alternaria observadas en las croquetas contaminadas con aflatoxinas de bajo y alto nivel de control se muestran para cada una de las tres repeticiones. En su mayor parte, se observaron especies similares en todos los tipos de croquetas, sin embargo, se observó una mayor abundancia relativa en las muestras de bajo nivel.
En el caso de Alternaria, la composición de especies desde el control hasta las croquetas contaminadas fue más estable que la observada para géneros como Aspergillus, Penicillium y Fusarium (Figuras 1, 2). Las huellas dactilares de la abundancia diferencial de taxones fúngicos (Figura 5) podrían utilizarse para detectar rápidamente una gran cantidad de piensos o ingredientes que pueden tener un mayor riesgo de exposición a aflatoxinas u otras micotoxinas. Cuando las especies de Saccharomyces (utilizadas para unirse a la aflatoxina) se han agotado, es probable que exista un mayor riesgo de exposición a la toxicidad.
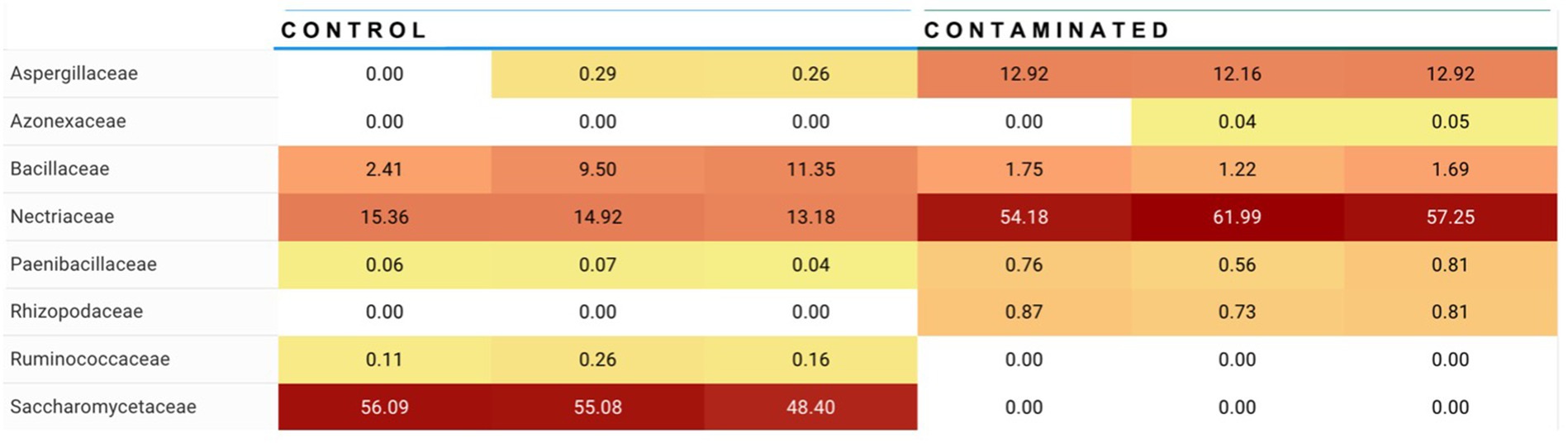 Figura 5. Resumen de la abundancia diferencial a nivel de familia fúngica. Las familias Apergillaceae (que contienen los géneros Aspergillus) y Nectriaceae (que contienen los géneros Fusarium) se observaron en una abundancia significativamente mayor en las croquetas contaminadas (aflatoxina de alto nivel) que en las croquetas de control, y las Saccharomycetaceae (que contienen los géneros Saccharomyces, es decir, levadura) tienen una abundancia significativamente mayor en las croquetas de control en comparación con las croquetas contaminadas.
Figura 5. Resumen de la abundancia diferencial a nivel de familia fúngica. Las familias Apergillaceae (que contienen los géneros Aspergillus) y Nectriaceae (que contienen los géneros Fusarium) se observaron en una abundancia significativamente mayor en las croquetas contaminadas (aflatoxina de alto nivel) que en las croquetas de control, y las Saccharomycetaceae (que contienen los géneros Saccharomyces, es decir, levadura) tienen una abundancia significativamente mayor en las croquetas de control en comparación con las croquetas contaminadas.
Identificación de genes funcionales en las vías de las micotoxinas
Más allá de la identificación de la estructura taxonómica de las croquetas, los datos metagenómicos también describieron genes funcionales implicados en la producción de micotoxinas y, por lo tanto, proporcionaron una confirmación adicional de las vías de micotoxinas como el deoxinivalenol, el nivalenol y la aflatoxina. No es sorprendente que los impactos más numerosos en los genes implicados en la vía de producción de aflatoxinas se observaran en las muestras contaminadas con aflatoxinas de alto nivel (Tabla 2). Los aciertos dobles y únicos de nivalenol (VNI) y deoxinivalenol (DON) en muestras de control pueden ser indicativos de niveles bajos de VNI o DON en estas muestras.
 Tabla 2. Impactos en genes en las vías de deoxinivalenol, nivalenol y aflatoxinas en datos metagenómicos de croquetas.
Tabla 2. Impactos en genes en las vías de deoxinivalenol, nivalenol y aflatoxinas en datos metagenómicos de croquetas.
Otras observaciones interesantes
Stenocarpella maydis, otro potencial productor de toxinas, se observó en las muestras de croquetas contaminadas, pero no en los controles. Stenocarpella se identificó como un biomarcador para muestras de aflatoxinas de bajo nivel donde se enriqueció significativamente (Figura 3). Los metabolitos tóxicos, como la diplodiatoxina, las quetoglobosinas K y L, y (all-E)-trideca-4,6,10,12-tetraeno-2,8-diol asociados con Stenocarpella no se evalúan con frecuencia y no se dispone fácilmente de ELISA simple u otras pruebas basadas en HPLC.
Especies bacterianas en los metagenomas de las croquetas de perro
En la Figura 6 se muestran los perfiles bacterianos de los controles y las croquetas para perros contaminadas con aflatoxinas. Se promediaron tres réplicas de croquetas de perro contaminadas con aflatoxinas de control, de bajo y alto nivel y se graficaron taxones que se encontraban en más del 3% de los datos totales para resumir las características bacterianas de cada croqueta. Los géneros Enterobacter, Serratia y Kosakonia se observaron en croquetas contaminadas con toxinas y no en croquetas de control.
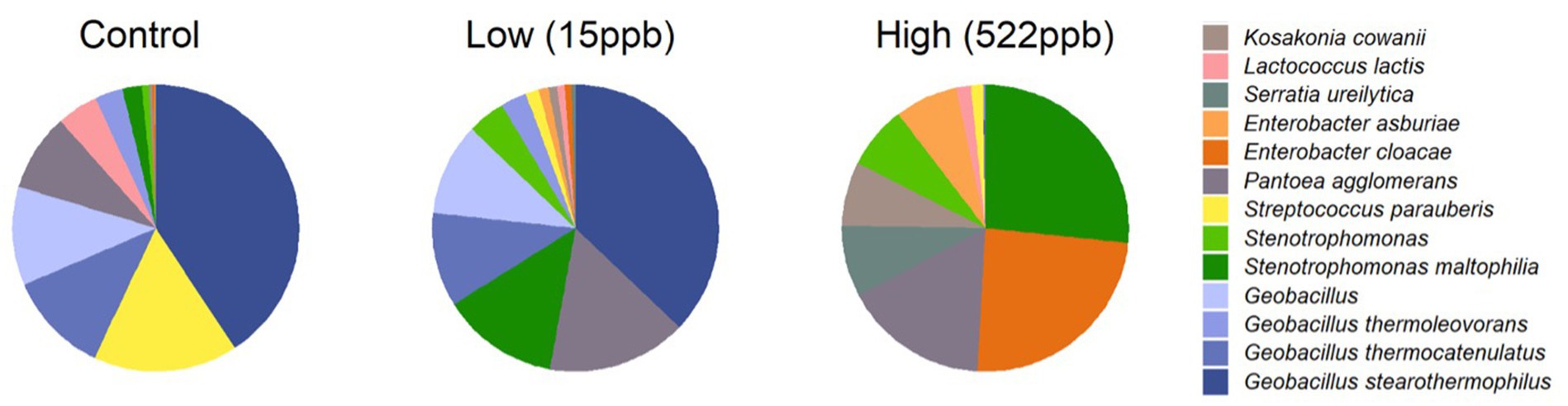 Figura 6. Especies bacterianas en croquetas contaminadas con aflatoxinas de bajo y alto nivel de control. Aquí se muestran los taxones bacterianos de croquetas para perros contaminadas con aflatoxinas de bajo y alto nivel. Las croquetas de alto nivel tuvieron una fuerte incidencia de Enterobacter cloaceae. Stenotrophomonas, que se ha descrito como un patógeno global emergente (a veces resistente a múltiples fármacos), también se observó en las croquetas de alto nivel.
Figura 6. Especies bacterianas en croquetas contaminadas con aflatoxinas de bajo y alto nivel de control. Aquí se muestran los taxones bacterianos de croquetas para perros contaminadas con aflatoxinas de bajo y alto nivel. Las croquetas de alto nivel tuvieron una fuerte incidencia de Enterobacter cloaceae. Stenotrophomonas, que se ha descrito como un patógeno global emergente (a veces resistente a múltiples fármacos), también se observó en las croquetas de alto nivel.
Resistencia a los antimicrobianos en croquetas
Las croquetas de control tenían genes de resistencia a los antimicrobianos diferentes a los observados en las croquetas contaminadas con aflatoxinas. Lo más probable es que esto se deba a las diferentes bacterias que estaban presentes en las muestras contaminadas en comparación con los controles. Los genes implicados en la resistencia a los macrólidos se observaron en las croquetas contaminadas, así como los genes betalactámicos bla R1 y bla I, que influyen en la expresión de blaZ y la proteína de unión a la penicilina 2a (PBP 2a) y contribuyen a la resistencia a la meticilina (35, 36). El gen bla 1 se observó en muestras de control, típicamente asociado con resistencia a la ampicilina.
Discusión
Una de las principales misiones del Centro de Medicina Veterinaria (CVM) es proteger la salud animal garantizando la inocuidad de los alimentos para animales. La aplicación de datos metagenómicos para complementar las evaluaciones de identificación de toxicidad (TIE) presentadas aquí es un enfoque metodológico novedoso para apoyar la misión de salvaguardar los alimentos. Los datos metagenómicos proporcionan una identificación completa basada en el ADN de todos los constituyentes eucariotas (insectos, plantas y animales) y procariotas (bacterias, hongos y protistas) de cualquier alimento, incluidos adulterantes, alérgenos, patógenos, determinantes de resistencia a los antimicrobianos y biomarcadores. Este tipo de información puede ayudar a los químicos a enfocar los parámetros de búsqueda cuando toxinas químicas desconocidas han causado enfermedades transmitidas por los alimentos.
En este trabajo, demostramos que los datos metagenómicos de croquetas con niveles bajos y altos de contaminación por aflatoxinas se correlacionaron con la incidencia y abundancia de ADN de especies de Aspergillus. Esta observación apoya la hipótesis de que las especies de Aspergillus desempeñaron un papel en la producción de aflatoxinas que condujo a los niveles observados en las croquetas altas. Además, los genes en la vía de producción de aflatoxinas solo se identificaron en croquetas de alto nivel, lo que respalda la utilidad de los datos metagenómicos para identificar aflatoxinas en los alimentos. Los biomarcadores estadísticamente significativos asociados con aflatoxinas de alto nivel incluyeron cuatro taxones de Aspergillus enriquecidos diferencialmente y taxones distintivos de Rhizopus y Fusarium. Los biomarcadores estadísticamente significativos asociados con las croquetas de control incluyeron cuatro taxones de Saccharomyceenriquecidos diferencialmente, que, como se describió anteriormente, a menudo se agregan a los alimentos para unirse a la aflatoxina y hacer que los alimentos sean más seguros para el consumo. Los datos metagenómicos podrían servir como una evaluación de puntos de control críticos para identificar poblaciones robustas de Saccharomyces en croquetas antes de que se vendan a los consumidores. Los biomarcadores también pueden informar sobre los orígenes regionales y/o temporales de los ingredientes (31). Comprender exactamente cómo funcionan los biomarcadores de ADN como predictores de toxicidad requerirá futuros trabajos de validación, pero estos resultados preliminares son muy prometedores.
Los datos metagenómicos utilizados junto con el perfil químico no solo son útiles para detectar la contaminación por especies aflatoxigénicas conocidas, sino también para describir especies desconocidas de hongos micotoxigénicos en alimentos humanos y animales. Actualmente, la FDA monitorea las aflatoxinas, fumonisinas, vomitoxina, zearalenona y ocratoxina A en alimentos para humanos y animales utilizando métodos de un solo objetivo (37, 38), aunque la FDA ha iniciado recientemente un enfoque de vigilancia de múltiples micotoxinas para alimentos para bebés y niños pequeños (39). La metagenómica proporciona un enfoque no dirigido que puede describir los hongos micotoxigénicos que pueden producir toxinas que actualmente no son monitoreadas por la FDA y los datos podrían indicar que es posible que se necesiten pruebas adicionales de micotoxinas para caracterizar con precisión los peligros y evaluar el riesgo.
Los datos metagenómicos aquí descritos identificaron múltiples especies de Penicillium, que pueden producir toxinas como brevianamid A, citreoviridina, citrinina, ácido ciclopiazónico, fumitremorgina B, griseofulvina, luteoskyrina, ácido penicilico, penitrem A, toxina PR, roquefortina, rugulosina, verrucosidina, verruculógeno, viridicarumtoxina y xantomegnina (40). Las pruebas químicas concordantes podrían establecer si estos compuestos estaban presentes o no. Los datos metagenómicos también identificaron Stenocarpella maydis, Rhizopus delemar y R. oryzae en croquetas contaminadas y no en croquetas de control. Stenocarpella maydis es un patógeno fúngico del maíz y sus metabolitos tóxicos incluyen diplodiatoxina, quetoglobosinas K y L, y (all-E)-trideca-4,6,10,12-tetraeno-2,8-diol (41, 42). Los sinónimos de S. maydis incluyen Diplodia zeae, Diplodia maydis, Sphaeria maydis, S. zeae, Macrodiplodia zeae y Dothiora zeae, que se han relacionado con la toxicidad por diplodia (diplodiosis). La diplodiosis se caracteriza por temblores musculares, falta de coordinación, cuartos traseros atáxicos, parálisis y muerte de bovinos, ovejas, ratas y patitos, con informes de mortalidad de ganado que se remontan a 1919 (43).
Utilizando un estudio de caso limitado con croquetas retiradas del mercado con dos niveles conocidos de aflatoxina (15 ppb y 522 ppb), demostramos que el ADN de los productores putativos de aflatoxinas se enriquecía diferencialmente en croquetas de bajo y alto nivel (es decir, se observaron cantidades comparativamente altas de ADN de Aspergillus en las croquetas contaminadas con aflatoxinas de alto nivel en contraste con las de bajo nivel). También documentamos la vía de producción de aflatoxinas mediante la identificación de genes en esa vía que también solo se observaron en el ADN de croquetas de aflatoxinas de alto nivel. La identificación taxonómica de otros posibles productores de toxinas podría servir para guiar las pruebas químicas adicionales para comprender mejor si otras toxinas estaban presentes en los alimentos y qué especies pueden haberlas producido. Los datos metagenómicos pueden describir la composición total (género, especie, subespecie e incluso serovares y variedades) de plantas, animales, insectos, bacterias, hongos, virus, plásmidos y resistencia a los antimicrobianos. Los datos combinados de productos químicos y MGS son una frontera emocionante que, sin duda, proporcionará información valiosa para respaldar la evaluación de riesgos modernizada para los alimentos humanos y animales.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/bioproject/PRJNA1062328/.
Contribuciones de los autores
AO: Redacción – borrador original, Visualización, Supervisión, Administración de proyectos, Metodología, Investigación, Análisis formal, Curación de datos, Conceptualización. BK: Redacción – revisión y edición, Visualización, Administración de proyectos, Metodología, Análisis formal, Curación de datos. ER: Escritura – revisión y edición, Visualización, Software, Análisis formal. SC: Redacción – revisión y edición, Metodología, Análisis formal. MM: Redacción – revisión y edición, Metodología, Análisis formal. PR: Redacción – revisión y edición. PM: Redacción – revisión y edición. BF: Escritura, revisión y edición. ES: Redacción – revisión y edición.
Financiación
El/los autor/es declaran/n que no se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo.
Reconocimientos
Nos gustaría agradecer a Stan Cook, Madison Fink, Quintin Muenks y Mary Koestner del Departamento de Agricultura de Missouri – Industrias Vegetales, Oficina de Piensos y Semillas por proporcionar muestras de croquetas de investigación.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Descargo de responsabilidad del autor
Las opiniones expresadas en este manuscrito son las de los autores y no reflejan necesariamente la política oficial del Departamento de Salud y Servicios Humanos, la Administración de Alimentos y Medicamentos de los Estados Unidos o el Gobierno de los Estados Unidos. La referencia a cualquier material, equipo o proceso comercial no constituye de ninguna manera aprobación, respaldo o recomendación por parte de la Administración de Alimentos y Medicamentos.
Notas
1. ^ https://github.com/biobakery/biobakery
2. ^ https://cran.r-project.org/web/packages/tidyverse/index.html
3. ^ https://metacyc.org/
Referencias
2. Ottesen, A, Ramachandran, P, Chen, Y, Brown, E, Reed, E y Strain, E. Seguimiento cuasimetagenómico de la fuente de Listeria monocytogenes a partir de helados contaminados de forma natural. BMC infecta dis. (2020) 20:83. doi: 10.1186/s12879-019-4747-z
3. Stevens, EL, Carleton, HA, Beal, J, Tillman, GE, Lindsey, RL, Lauer, AC, et al. Uso de la secuenciación del genoma completo por parte de la colaboración interinstitucional federal para la genómica para la inocuidad de los alimentos y los piensos en los Estados Unidos. J Food Prot. (2022) 85:755–72. doi: 10.4315/jfp-21-437
4. Handy, SM, Pawar, RS, Ottesen, AR, Ramachandran, P, Sagi, S, Zhang, N, et al. HPLC-UV, metabarcoding y desnatados del genoma de suplementos dietéticos botánicos: un estudio de caso en Echinacea. Planta Med. (2021) 87:314–24. doi: 10.1055/a-1336-1685
5. Ottesen, A, Kocurek, B, Ramachandran, P, Reed, E, Commichaux, S, Engelbach, G, et al. Avanzar en el monitoreo de la resistencia a los antimicrobianos en aguas superficiales con métodos metagenómicos y cuasimetagenómicos. PLoS Agua. (2022) 1:e0000067. doi: 10.1371/journal.pwat.0000067
6. Ottesen, A, y Ramachandran, P. Microbiología de los alimentos: fundamentos y fronteras En: Microbiomas alimentarios: un nuevo paradigma para los alimentos y la ecología de los alimentos. 5ª ed. Washington, DC: ASM Press (2019).
7. Van den Brand, AD, Bokkers, BGH, Te Biesebeek, JD, y Mengelers, MJB. Exposición combinada a múltiples micotoxinas: un ejemplo del uso de un enfoque escalonado en una evaluación del riesgo de la mezcla. Toxinas. (2022) 14:303. doi: 10.3390/toxinas14050303
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Palumbo, R, Crisci, A, Venâncio, AA-O, Cortiñas Abrahantes, J, Dorne, JL, Battilani, PA-O, et al. Aparición y co-ocurrencia de micotoxinas en piensos y alimentos a base de cereales. Microorganismos. (2020) 8:74. doi: 10.3390/microorganismos8010074
9. Xing, HQ, Ma, JC, Xu, BL, Zhang, SW, Wang, J, Cao, L, et al. Micobiota de semillas de maíz revelada mediante análisis de secuencia de ADNr-ITS de muestras con diferentes tiempos de almacenamiento. Microbiología. (2018) 7:E00609. DOI: 10.1002/MBO3.609
10. Tolosa, J, Rodríguez-Carrasco, Y, Ruiz, MJ y Vila-Donat, P. Aparición de múltiples micotoxinas en los piensos, el metabolismo y el arrastre a los productos alimenticios de origen animal: una revisión. Toxicol químico alimentario. (2021) 158:112661. doi: 10.1016/j.fct.2021.112661
11. Saleemi MKR, A, Khatoon, A, Zubair, M, Yongping, X, Murtaza, B, Li, X, et al. Efectos tóxicos de la aflatoxina B1 sobre parámetros hematobioquímicos e histopatológicos de machos juveniles de Leghorn blanco y su mejoría con vitamina E y Moringa oleifera. Pak Vet J. (2023) 23:53. doi: 10.29261/pakvetj/2023.053
12. Ward, SA, Sarah y Vaughan, R. Guía de micotoxinas que se encuentran comúnmente en alimentos para animales/aflatoxinas. Publicaciones de Extensión del Estado de Carolina del Norte (2021).
13. Armendáriz, CR, Fernández, ÁJG, Gironés, MCLR, y de la Torre, AH. Micotoxinas en: P Wexler editor. Enciclopedia de toxicología. 3ª ed. Oxford: Academic Press (2014). 424–7.
14. Andrea Ottesen, AT, White, JR, Newell, MJ, Pahl, D, Brown, EW, Musser, S, et al. Bacterias de tomates gestionadas con agua de pozo y agua de estanque: impacto de las fuentes de agua agrícola en la microbiota de la carposfera. Int J Environ Agric Res. (2016) 2. Disponible en: https://ijoear.com/assets/articles_menuscripts/file/IJOEAR-JUN-2016-20.pdf
15. Ottesen, A, Skaltsas, D, White, JR, Gorham, S, Ramachandran, P, Brown, E, et al. Utilización de la secuenciación de nueva generación para describir la microbiota epífita asociada a manzanas ecológicas y gestionadas de forma convencional. Int J Environ Agric Res. (2016). Disponible en: https://ijoear.com/assets/articles_menuscripts/file/IJOEAR-SEP-2016-28.pdf
16. Ottesen, A, White, J, Gorham, S, Reed, E, Newell, M, Burrows, E, et al. Filosfera envirosfera En: Fitopatología, vol. 104. St Paul, MN: Sociedad Americana de Fitopatología (2014). 87–8.
17. Ottesen, AR, Gorham, S, Pettengill, JB, Rideout, S, Evans, P, y Brown, E. El impacto de las aplicaciones sistémicas y de plaguicidas de cobre en la microflora filosférica de los tomates. J Sci Food Agric. (2015) 95:1116–25. doi: 10.1002/jsfa.7010
18. Reed, E, Ramachandran, P, Ottesen, A, Brown, E y Zheng, J. Caracterización metagenómica del agua de riego gastada de brotes de alfalfa a partir de semillas contaminadas con Salmonella En: Reunión anual de la IAFP 2018 : IAFP (2018)
19. Telias, A, White, JR, Pahl, DM, Ottesen, AR, y Walsh, CS. Diversidad y variación de la comunidad bacteriana en las fuentes de agua de pulverización y la superficie del fruto del tomate. BMC Microbiol. (2011) 11:81. doi: 10.1186/1471-2180-11-81
20. Staff PaR. Sec. 683.100 Niveles de acción de las aflatoxinas en los alimentos para animales. Guía de Políticas de Cumplimiento Guía para el Personal de la FDA. (2019).
22. Kocurek, B, Behling, S, Martin, G, Ramachandran, P, Reed, E, Grim, C, et al. Estudio metagenómico de la resistencia a los antimicrobianos (RAM) en las aguas superficiales de Maryland diferenciadas por su alto y bajo impacto humano. Anuncio de Microbiol Resour. (2024) 13:e0047723. doi: 10.1128/mra.00477-23
23. Bolger, AM, Lohse, M y Usadel, B. Trimmomatic: un recortador flexible para datos de secuencia de Illumina. Bioinformática. (2014) 30:2114–20. doi: 10.1093/bioinformatics/btu170
25. McIver, LJ, Abu-Ali, G, Franzosa, EA, Schwager, R, Morgan, XC, Waldron, L, et al. bioBakery: un entorno de análisis meta’ómico. Bioinformática. (2017) 34:1235–7. doi: 10.1093/bioinformatics/btx754
26. Kruskal, WH, y Wallis, WA. Uso de rangos en el análisis de varianza de un criterio. J Am Stat Assoc. (1952) 47:583–621. doi: 10.1080/01621459.1952.10483441
27. Buchfink, B, Reuter, K, y Drost, H-G. Alineaciones de proteínas sensibles a escala de árbol de vida utilizando DIAMOND. Métodos Nat. (2021) 18:366–8. doi: 10.1038/s41592-021-01101-x
28. Karp, PD, Billington, R, Caspi, R, Fulcher, CA, Latendresse, M, Kothari, A, et al. La colección bio Cyc de genomas microbianos y vías metabólicas. (1477-4054 [electrónico]). (2019).
29. Ramachandran, P, Mammel, M, Ottesen, A, y Pava-Ripoll, M. MitochonTrakr: una colección de referencia de genomas mitocondriales de alta calidad para la detección de especies de insectos en productos alimenticios. ADN mitocondrial Parte B. (2019) 4:292–3. doi: 10.1080/23802359.2018.1542986
30. Kiyota, T, Hamada, R, Sakamoto, K, Iwashita, K, Yamada, O y Mikami, S. No productividad de aflatoxinas de Aspergillus oryzae causada por la pérdida de función en el producto del gen aflJ. J Biosci Bioeng. (2011) 111:512–7. doi: 10.1016/j.jbiosc.2010.12.022
31. Navale, V, Vamkudoth, KR, Ajmera, S, y Dhuri, V. Micotoxinas derivadas de Aspergillus en los alimentos y el medio ambiente: prevalencia, detección y toxicidad. Toxicol Rep. (2021) 8:1008–30. doi: 10.1016/j.toxrep.2021.04.013
33. Liu, L, Xie, M y Wei, D. Desintoxicación biológica de micotoxinas: estado actual y avances futuros. Int J Mol Sci. (2022) 23. doi: 10.3390/ijms23031064
34. Arif, M, Iram, A, Bhutta, MAK, Naiel, MAE, Abd El-Hack, ME, Othman, SI, et al. El papel de la biodegradación de Saccharomyces cerevisiae contra los efectos nocivos de las dietas contaminadas con micotoxinas sobre el rendimiento de los pollos de engorde, el estado de inmunidad y las características de la canal. Animales. (2020) 10:238. doi: 10.3390/ani10020238
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Frederick, TE, y Peng, JW. Un inductor gratuito de β-lactamasas descubre la dinámica del sitio activo oculto del dominio sensor BlaR1 de Staphylococcus aureus. PLoS Uno. (2018) 13:e0197241. doi: 10.1371/journal.pone.0197241
36. Rocha, GD, Nogueira, JF, Gomes dos Santos, MV, Boaventura, JA, Nunes Soares, RA, de Simoni, J, et al. Impacto de polimorfismos en los genes Bla Z, blaR1 y blaI y su relación con la resistencia a β-lactámicos en cepas de S. aureus aisladas de mastitis bovina. Microb Pathog. (2022) 165:105453. doi: 10.1016/j.micpath.2022.105453
38. Administración de Alimentos y Medicamentos (FDA, por sus siglas en Manual de cumplimiento de la Administración de Alimentos y Medicamentos. Biología Molecular y Toxinas Naturales (2016).
39. Zhang, K, Flannery, BM, Oles, CJ y Adeuya, A. Micotoxinas en alimentos para bebés/niños pequeños y cereales para el desayuno en el mercado minorista de EE. UU. Aditivo alimentario Contaminar Parte B. (2018) 11:183–90. doi: 10.1080/19393210.2018.1451397
40. El-banna, AA, Pitt, JI y Leistner, L. Producción de micotoxinas por especies de Penicillium. Syst Appl Microbiol. (1987) 10:42–6. doi: 10.1016/S0723-2020(87)80008-5
41. Rogers, KD, Cannistra, JC, Gloer, JB y Wicklow, DT. Diplodiatoxina, quetoglobosinas y diplonina asociadas con un brote de campo de pudrición de la mazorca por Stenocarpella en Illinois. Micotoxinas Res. (2014) 30:61–70. doi: 10.1007/s12550-014-0188-0
42. Wicklow, DT, Rogers, KD, Dowd, PF y Gloer, JB. Metabolitos bioactivos de Stenocarpella maydis, un patógeno de la pudrición del tallo y la mazorca del maíz. Fúngico Biol. (2011) 115:133–42. doi: 10.1016/j.funbio.2010.11.003
Palabras clave: metagenoma, metagenómica, química, aflatoxina, croquetas, Aspergillus
Cita: Ottesen A, Kocurek B, Reed E, Commichaux S, Mammel M, Ramachandran P, McDermott P, Flannery BM y Strain E (2024) Evaluación metagenómica y química emparejada de croquetas de perro contaminadas con aflatoxinas. Frente. Vet. Sci. 11:1374839. doi: 10.3389/fvets.2024.1374839
Recibido: 22 de enero de 2024; Aceptado: 25 de marzo de 2024;
Publicado: 11 abril 2024.
Editado por:
Kai Wang, Academia China de Ciencias Agrícolas, China
Revisado por:
Aisha Khatoon, Universidad de Agricultura, Faisalabad, Pakistán
Bishwo Adhikari, Servicio de Inspección de Sanidad Animal y Vegetal (USDA), Estados Unidos
Derechos de autor © 2024 Ottesen, Kocurek, Reed, Commichaux, Mammel, Ramachandran, McDermott, Flannery y Strain. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Andrea Ottesen, Andrea.Ottesen@fda.hhs.gov
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía