Expresión génica y perfil de ácidos grasos en músculo Longissimus thoracis, grasa subcutánea e hígado de corderos

Expresión génica y perfil de ácidos grasos en músculo Longissimus thoracis, grasa subcutánea e hígado de corderos ligeros en respuesta al pastoreo concentrado o alfalfa
 Elda Dervishi1,
Elda Dervishi1,  Laura González-Calvo2,
Laura González-Calvo2,  Mireia Blanco2,
Mireia Blanco2,  Margalida Alegría2,
Margalida Alegría2,  Pilar Sarto2,
Pilar Sarto2,  R. Martín-Hernández3,
R. Martín-Hernández3,  José M. Ordovás4,
José M. Ordovás4,  Magdalena Serrano5 y
Magdalena Serrano5 y  Jorge H. Calvo2,6*
Jorge H. Calvo2,6*- 1Livestock Gentec, Universidad de Alberta, Edmonton, AB, Canadá
- 2Unidad de Producción y Sanidad Animal, Centro de Investigación y Tecnología Agroalimentaria de Aragón (CITA)-Instituto Agroalimentario de Aragón (IA2) (CITA-Universidad de Zaragoza), Zaragoza, España
- 3Precision Nutrition and Obesity, IMDEA-Alimentación, Madrid, España
- 4Jean Mayer-USDA Human Nutrition Research Center on Aging, Tufts University, Boston, MA, Estados Unidos
- 5Departamento de Mejora Genética Animal, INIA, Madrid, España
- 6ARAID, Zaragoza, España
Una mejor comprensión de la expresión génica y las vías metabólicas en respuesta a un sistema de alimentación es fundamental para identificar los procesos fisiológicos clave y los genes asociados con el contenido de ácidos grasos poliinsaturados (PUFA) en la carne de cordero. El objetivo principal de este estudio fue investigar los cambios transcripcionales en el músculo L. thoracis (LT), el hígado y la grasa subcutánea (SF) de corderos que pastaron alfalfa (ALF) y concentrados (CON) sacrificados a 23 kg y utilizando la matriz del genoma completo Affymetrix Ovine Gene 1.1 ST. El estudio también evaluó la relación entre los rasgos de la carne en el músculo LT, incluido el color, los pigmentos y la oxidación de lípidos durante 7 días de exhibición, el contenido de α-tocoferol, el contenido de grasa intramuscular (IMF) y el perfil de ácidos grasos (FA). Los corderos que pastaban en alfalfa tenían una mayor concentración de α-tocoferol en plasma que los corderos CON (P < 0,05). El tratamiento no afectó el contenido de IMF, el color de la carne o los pigmentos (P > 0,05). El pastoreo aumentó el contenido de α-tocoferol (P < 0,001) y disminuyó la oxidación lipídica en el día 7 de exhibición (P < 0,05) en el músculo LT. El grupo ALF contenía una mayor cantidad de ácido linoleico conjugado (CLA), C18:3 n−3, C20:5 n−3, C22:5 n−3 y C22:6 n−3 que el grupo CON (P < 0,05). Identificamos 41, 96 y cuatro genes expresados diferencialmente en LT músculo, hígado y grasa subcutánea, respectivamente. Los procesos biológicos más enriquecidos en el músculo LT fueron el desarrollo del tejido muscular esquelético, siendo los genes relacionados con los procesos catabólicos y lipídicos regulados a la baja, excepto CPT1B, que fue regulado al alza en los corderos ALF. Los animales que pastaban alfalfa tenían una menor expresión de enzimas desaturasas en el hígado (FADS1 y FADS2), que regulan la insaturación de ácidos grasos y están directamente involucradas en el metabolismo de la serie n−3 PUFA. Los resultados encontrados en el estudio actual mostraron que ingerir dietas más ricas en AGPI n-3 podría tener efectos negativos en la síntesis de novo de AGPI n-3 al regular negativamente la expresión de FADS1 y FADS2. Sin embargo, la alimentación con dietas más pobres en AGPI n-3 puede promover la desaturación de ácidos grasos, lo que hace que estos dos genes sean candidatos atractivos para alterar el contenido de AGPI en la carne.
Introducción
Las políticas de salud pública recomiendan un aumento en la ingesta de la serie de ácidos grasos poliinsaturados n-3 (PUFA) debido al impacto positivo que estas moléculas tienen en la salud humana. Además, se recomienda una disminución en el consumo de ácidos grasos trans y ácidos grasos saturados (SFA) porque se han asociado con un aumento de los niveles de colesterol (Takeuchi y Sugano, 2017; Zhu et al., 2019). Otros ácidos grasos, como los ácidos linoleicos conjugados (CLA), también han recibido una atención creciente debido a sus posibles efectos beneficiosos sobre la salud humana (Lehnen et al., 2015; Lee et al., 2018).
Actualmente, aumentar la eficiencia alimenticia y producir carne magra sin reducir el valor nutricional de la carne son los principales desafíos de la industria cárnica. El valor nutricional de la carne puede verse influenciado por los efectos dietéticos y genéticos (Scollan et al., 2014). La alimentación con pasto mejora los contenidos de ácido eicosapentaenoico (20:5n−3, EPA), ácido docosapentaenoico (DPA, 22:5n−3) y ácido docosahexaenoico (22:6n−3, DHA) en el músculo (Fisher et al., 2000; Dervishi et al., 2010; Dervishi et al., 2011) a medida que el forraje aumenta el contenido de ácido alfa-linolénico (18:3n−3), el precursor de la producción de DHA y EPA (Kitessa et al., 2010). Se ha demostrado que la dieta tiene un gran impacto en el perfil intramuscular de AF del músculo de corderos ligeros (Dervishi et al., 2010; González-Calvo et al., 2015a); El pastoreo aumenta el contenido de AGPI de la serie n-3 y los ácidos grasos conjugados (CLA) en comparación con la alimentación concentrada. En el músculo semitendinoso, los genes relacionados con la adipogénesis están regulados al alza en corderos alimentados con concentrado, mientras que la expresión del gen CPT1B, relacionada con el proceso de oxidación β, está regulada al alza en corderos de pastoreo (Dervishi et al., 2011). Sin embargo, la expresión de genes implicados en el metabolismo lipídico no es similar en el músculo longissimus de corderos de pastoreo y alimentados con concentrado (González-Calvo et al., 2015a). Estos resultados demuestran que el sistema de dieta/alimentación tiene un efecto diferencial sobre la expresión génica en diferentes tejidos animales. También se ha demostrado que la composición del tipo de fibra en el músculo esquelético (las cantidades relativas de fibras de contracción rápida versus lenta) afectó los perfiles de expresión génica entre diferentes músculos en el mismo entorno (Terry et al., 2018). Por lo tanto, una mejor comprensión de los genes y las vías metabólicas en respuesta al sistema de alimentación es fundamental para identificar los procesos fisiológicos clave y los genes asociados con el metabolismo de los lípidos, especialmente para la serie n-3 AGPI. Una comprensión más profunda de la regulación genética de los niveles de n-3 en la carne de cordero puede ayudar a diseñar nuevas estrategias para la producción de carne más saludable y satisfacer la demanda de los consumidores.
La combinación de tecnologías como los ácidos grasos y el perfil genético proporciona una herramienta poderosa para descubrir cambios en la expresión génica asociados con los rasgos de calidad de la carne y para descubrir genes que contribuyen a la variación del contenido de ácidos grasos en la carne. El objetivo principal de este estudio fue investigar el perfil de ácidos grasos y los cambios transcripcionales en el músculo LT, el hígado y la grasa subcutánea (SF) de corderos que pastan en pastos de alfalfa y reciben concentrado utilizando la matriz de genoma completo Affymetrix Ovine Gene 1.1 ST. Además, nuestro objetivo fue identificar nuevos genes que pueden desempeñar un papel importante en el metabolismo de los AGPI que pueden estar asociados con los rasgos de calidad de la carne.
Material y métodos
Declaración ética
Todos los procedimientos experimentales, incluidos el cuidado de animales y la eutanasia, se realizaron de acuerdo con las directrices de la normativa de la Unión Europea y española para el uso y cuidado de animales en investigación y fueron aprobados por el Comité de Bienestar Animal del Centro de Investigación y Tecnología Agroalimentaria (CITA) (número de protocolo 2009-01_MJT). En todos los casos, la eutanasia se realizó mediante un rayo cautivo penetrante seguido de una exanguinación inmediata.
Recolección de animales y muestras
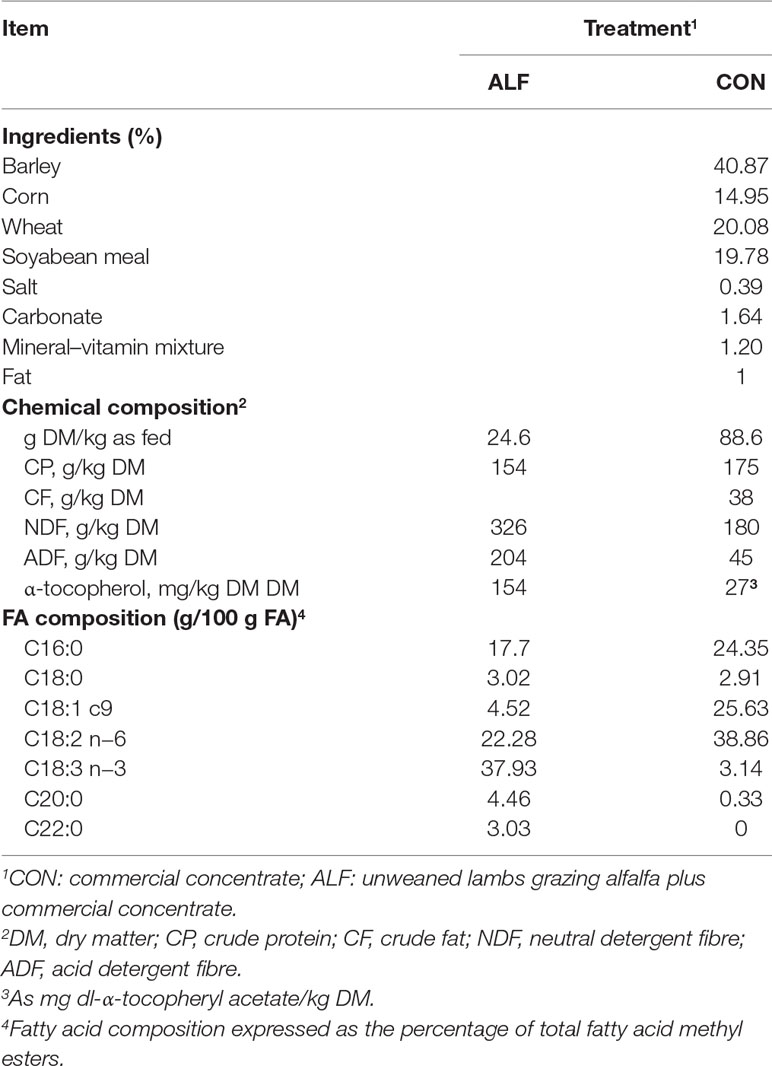
Catorce parejas de corderos machos criados de oveja de la raza Rasa Aragonesa pastaron continuamente durante la lactancia en pastos de alfalfa. Los corderos tuvieron acceso ad libitum a un concentrado durante la lactancia. Siete parejas de corderos de oveja no fueron destetados, sino que permanecieron pastando alfalfa con sus madres desde el nacimiento hasta el sacrificio de los corderos (23 ± 0,4 kg) (grupo ALF). Los otros siete corderos fueron destetados (48 ± 0,9 días de edad) y luego alimentados con un concentrado basal durante 24 (± 2,6) días hasta el sacrificio a los 23 kg (grupo CON). Estos corderos eran los mismos que los descritos en González-Calvo et al. (2017), y fueron criados junto al grupo ALF. Los corderos pertenecientes al tratamiento ALF recibieron leche de madre, alfalfa fresca (pastoreo) y concentrado comercial, el mismo que se ofreció al tratamiento CON durante el período experimental. La ingesta media de concentrados de los grupos CON y ALF durante el período experimental fue de 24,3 y 7,4 kg por cordero, respectivamente. El peso al destete de los animales tratados con CON fue de 11,6 ± 1,91 kg de peso corporal y el peso de los corderos de alfalfa en el mismo momento del destete del tratamiento con CON fue de 12,8 ± 1,35 kg de peso corporal. Los ingredientes, la composición química y la composición FA de los piensos se muestran en la Tabla 1. Los procedimientos experimentales, la composición de las dietas, el manejo de los animales y los detalles de la muestra para cada grupo se describen en detalle en Ripoll et al. (2013). Las muestras de sangre se obtuvieron semanalmente en tubos de ensayo que contenían heparina de la vena yugular. Las muestras se centrifugaron a 3.500 rpm durante 20 min, y el plasma se almacenó a -80 ° C hasta que se analizaron α-tocoferol y triacilgliceroles (TG), colesterol, colesterol de lipoproteínas de baja densidad (colesterol LDL) y colesterol de lipoproteínas de alta densidad (colesterol HDL).
Todos los corderos fueron sacrificados cuando alcanzaron los 22-24 kg de peso de sacrificio (SW) según las especificaciones de la Indicación Geográfica Protegida Ternasco de Aragón (Reglamento (CE) Nº 1107/96) que estipula que los corderos deben tener menos de 90 días de edad con un SW entre 22 y 24 kg. Los corderos fueron sacrificados utilizando las leyes de la UE en el mismo matadero comercial, y las canales fueron colgadas por el tendón de Aquiles y enfriadas durante 24 h a 4 ° C en total oscuridad. La edad de sacrificio, el peso de sacrificio y la tasa de crecimiento de las dos estrategias de manejo se presentan en la Tabla Suplementaria 1.
Justo después del sacrificio, se extirpó una muestra del músculo LT de la 12ª vértebra torácica, una muestra de SF entre las vértebras cervicales del atlas y el eje y una muestra del hígado, se congelaron en nitrógeno líquido y se almacenaron a -80 ° C hasta el aislamiento del ARN.
Análisis químicos
Grasa intramuscular (IMF)
El contenido de grasa intramuscular se cuantificó mediante el procedimiento Ankom (AOAC, 2000) con un extractor Ankom (modelo XT10, Ankom Technology, Nueva York, EE.UU.).
Determinación de ácidos grasos
Se determinaron tanto los ácidos grasos musculares como los alimentarios como se describe en González-Calvo et al. (2015a). Se extrajeron muestras de alimento de Soxhlet (Sukhija y Palmquist, 1988), y se determinaron muestras musculares según Bligh y Dyer (1959) con las modificaciones descritas en González-Calvo et al. (2015a). Los contenidos individuales de AF se expresaron como porcentajes de peso (g/100 g de FAME). Se determinó la cantidad total de AGS, AGF monoinsaturada (AGPI), AGPI, AGPI n-6 y AGPI n-3 y sus proporciones asociadas (AGPI: AGS y n-6:n−3).
Análisis de α-tocoferol, TG, colesterol LDL, colesterol HDL y colesterol en plasma
El alfa-tocoferol en plasma se determinó por extracción líquida por duplicado como se describe en González-Calvo et al. (2015b). Los triacilgliceroles, el colesterol, el colesterol LDL y el colesterol HDL se determinaron mediante un analizador automático (Gernonstar, RAL, Barcelona, España). El fabricante del reactivo fue RAL (Técnica para el Laboratorio, S.A. Sant Joan Despí, Barcelona, España). Los coeficientes de variación intraensayo medios fueron 0,99-1,57%, 0,76-1,22%, 0,63-0,67% y 0,8-1,06% para TG, colesterol, colesterol LDL y colesterol HDL, respectivamente. Los coeficientes de variación entre ensayos fueron 3,15-7,77%, 4,36-6,91%, 1,29-1,45% y 2,71-4,60% para los mismos metabolitos.
Análisis de la concentración de α-tocoferol, TBARS y formación de metmioglobina en el músculo
Después de enfriarse, un pedazo del músculo LT entre la 4ª y la 6ª vértebras lumbares se envasó al vacío y se mantuvo a -20 ° C en la oscuridad hasta el análisis de α-tocoferol. La concentración de α-tocoferol se determinó mediante extracción líquida como se describe en González-Calvo et al. (2015b). Se utilizó una porción del lomo entre la 7ª y la 13ª vértebra torácica para medir el color (contenido de metmioglobina, MMb) y el análisis de oxidación lipídica (sustancia reactiva al ácido tiobarbitúrico, TBARS), y se cuantificaron a los 7 días después de mantenerse en la oscuridad a 4 ° C. El color muscular LT y el análisis TBARS de grasa intramuscular LT se midieron como se describe en González-Calvo et al. (2015b). Brevemente, el contenido relativo de metmioglobina (MMb) fue estimado por el K/S572/525 ratio (Hunt, 1980). Esta proporción disminuye cuando aumenta el contenido de MMb. El análisis TBARS se realizó utilizando el procedimiento descrito por Pfalzgraf et al. (1995). Los valores de TBARS se expresan como miligramos de malonaldehído (MDA) kg−1 de músculo.
Aislamiento de ARN y evaluación de la integridad del ARN
El ARN total se extrajo de aproximadamente 500 mg de LT muscular, SF e hígado utilizando mini kits RNeasy Tissue (QIAGEN, Madrid, España) siguiendo el protocolo del fabricante. Antes del análisis de microarrays, la integridad y la calidad del ARN se evaluaron mediante un RNA 6000 Nano LabChip en un bioanalizador Agilent 2100 y se cuantificaron utilizando un espectrofotómetro nanofotométrico (Implen, Madrid, España). Todos los valores del número de integridad del ARN (RIN) fueron superiores a 8.
Hibridación de microarrays y procesamiento de datos
Las muestras de ARN (n = 14, siete muestras de cada tratamiento) se analizaron utilizando el Ovine Gene 1.1 ST Array Strip (Affymetrix, High Wycombe, Reino Unido). La hibridación y el escaneo de microarrays se realizaron en el Functional Genomics Core Facility (Instituto de Investigación Biomédica, IRB Barcelona, España) siguiendo las recomendaciones del fabricante. Las imágenes escaneadas (archivos DAT) fueron transformadas en intensidades (archivos CEL) por Affymetrix GeneChip Operating Software (GCOS). La intensidad general de la matriz se normalizó entre las matrices para corregir el sesgo sistemático en los datos y eliminar el impacto de las influencias no biológicas en los datos biológicos. Los datos importados se analizaron a nivel de gen, con exones resumidos en genes, utilizando la expresión media de todos los exones de un gen. La normalización se llevó a cabo con el algoritmo Robust Multi-Array Average (RMA) utilizando normalización cuantil, resumen de sonda de pulido mediano y transformación de sonda log2. Los conjuntos de datos que respaldan los resultados y se discuten en esta publicación se han depositado en el repositorio NCBI Gene Expression Omnibus (Barrett et al., 2012) y son accesibles a través de los números de acceso de la serie GEO GSE63774 (músculo LT y SF) y GSE125661 (hígado). Los conjuntos de datos para músculo LT y SF en el grupo CON se presentaron previamente en González-Calvo et al. (2017).
Validación de datos de microarrays mediante análisis cuantitativo de PCR en tiempo real (RT-qPCR)
Se trató un microgramo de ARN de cada muestra con DNAse (Invitrogen, Carlsbad, CA, EUA), y se sintetizó ADNc monocatenario utilizando el kit de transcriptasa inversa SuperScriptIII (Invitrogen, Carlsbad, CA, EUA), siguiendo las recomendaciones del fabricante. Se generaron cebadores específicos que abarcan exones para genes y se confirmó su especificidad utilizando BLAST (Centro Nacional de Información Biotecnológica: http://www.ncbi.nlm.nih.gov/BLAST/). Antes de realizar las PCR en tiempo real, se realizó una PCR convencional para todos los genes para probar los cebadores y verificar los productos amplificados. Los productos de PCR se secuenciaron para confirmar la identidad génica utilizando un ABI Prism 3700 (Applied Biosystems, Madrid, España) con protocolos estándar. Se realizaron búsquedas de homología con BLAST para verificar la identidad de los fragmentos amplificados. La PCR en tiempo real se llevó a cabo en una mezcla de reacción total de PCR de 10 μl que contenía SYBR Green Master Mix: SYBR Premix Ex Taq II (Tli RNase H Plus, Takara, Sumalsa, Zaragoza, España). Las reacciones se ejecutaron por triplicado en una plataforma ABI Prism 7500 (Applied Biosystem, Madrid, España) siguiendo los parámetros de ciclo del fabricante. Se generaron curvas estándar para cada gen para calcular la eficiencia de amplificación a través de una dilución en serie de 4 veces de ADNc agrupado a partir de músculo LT, hígado y SF. La eficiencia (E) de la amplificación por PCR para cada gen se calculó utilizando el método de curva estándar (E = 10®(−1/pendiente)). Se replicaron dos «muestras de conectores» en todas las placas para eliminar la variación técnica de esta fuente de variabilidad. Las temperaturas de recocido, las concentraciones de cebador y las secuencias de cebadores para GOI (Genes de interés: CPT1B, MYOD1, MSTN, ABCC4, IGF1R y PLA2G16 para músculo LT; METTL1 para SF; y FADS1, FADS2, ACACA, SCD, SQLE, IER3, SLC19A1 y THRSP para el tejido hepático) y los genes de referencia (GUSB y YWHAZ para el músculo LT y SF; y RPL37, GUSB y RPL19 para el hígado) se describen en la Tabla complementaria 2. Estos genes de referencia para músculo LT y SF fueron elegidos porque eran los más estables en estos tejidos en estudios previos (González-Calvo et al., 2014). Se analizaron cinco genes candidatos de referencia (B2M, YWHAZ, RPL37, RPL19 y GUSB) para detectar tejido hepático. Las determinaciones de la estabilidad de la expresión génica de los genes hepáticos incluidos en este estudio se calcularon utilizando NormFinder para seleccionar los mejores genes de referencia (Andersen et al., 2004).
Análisis estadístico
Análisis estadístico del rendimiento, concentraciones de TG, colesterol LDL, colesterol HDL y colesterol en plasma, y características de calidad de la carne en músculo LT
El análisis estadístico del rendimiento, los metabolitos plasmáticos y la oxidación lipídica del músculo LT (TBARS) se realizó utilizando el paquete estadístico SAS v. 9.3 (SAS Institute, Cary NC, USA). La concentración de analitos en plasma, los niveles de oxidación lipídica y el color y pigmentos de la carne se analizaron utilizando modelos mixtos para mediciones repetidas basadas en la solución de grados de libertad ajustados de Kenward-Roger para medidas repetidas, incluida la estrategia de manejo (CON y ALF), la semana / hora de visualización y su interacción como efectos fijos y el cordero como efecto aleatorio. Se utilizó una estructura autorregresiva de primer orden con varianzas heterogéneas para cada fecha para modelar el error residual heterogéneo.
El aumento de peso, la edad, el peso al sacrificio y la IMF en músculo LT se analizaron mediante un modelo lineal general (GLM) con el tratamiento como factor fijo. El contenido de α-tocoferol y el perfil de ácidos grasos del músculo LT se analizaron con un GLM con el tratamiento como factor fijo y la edad de sacrificio (SA) como covariable. Los resultados se expresaron como medias cuadráticas mínimas (LSM) ± los valores de error estándar (EE), y las diferencias se probaron a un nivel de significancia de 0,05 con la estadística t. Se utilizó la prueba post hoc de Tukey para evaluar las diferencias entre los tratamientos.
Análisis estadístico de la expresión génica de microarrays
Identificación de genes expresados diferencialmente mediante análisis de microarrays en músculo LT y SF
Los datos normalizados se analizaron más a fondo utilizando el software Babelomics (http://babelomics.bioinfo.cipf.es/graph.html) y MetaboAnalyst (Xia et al., 2009). Los genes que mostraban un valor estadísticamente significativo de la prueba de Limma (P < 0,01) se seleccionaron como expresados diferencialmente entre los tratamientos. Se anotaron genes significativos en función de las puntuaciones de similitud en las comparaciones de blastn de secuencias de conglomerados de transcripción de Affymetrix contra secuencias ovinas en GenBank. Se utilizó un segundo método, el análisis de significación de microarrays (SAM), para identificar y reconfirmar genes expresados diferencialmente en comparaciones ALF-CON. Los detalles del protocolo se describen en González-Calvo et al. (2017).
Análisis Multivariante de Expresión Génica y Análisis de Agrupamiento Jerárquico (HCA)
El análisis multivariante y de conglomerados se realizó utilizando MetaboAnalyst según Xia et al. (2009). El análisis de componentes principales (ACP) se utilizó para agrupar las muestras en función del perfil de expresión génica seleccionado para cada tejido. El análisis de agrupamiento jerárquico para la expresión génica se realizó utilizando todos los genes y solo los genes significativos para cada tejido. Los detalles se describen en González-Calvo et al. (2017).
Análisis estadístico de la expresión génica validado por RT-qPCR
Los niveles de ARNm correspondientes se midieron y analizaron mediante su ciclo de cuantificación (Cq). La metodología estadística para analizar las diferencias en la tasa de expresión se llevó a cabo siguiendo el método propuesto por Steibel et al. (2009). El modelo mixto ajustado fue el siguiente:
donde yrigkm es la Cq valor (datos transformados teniendo en cuenta E < 2) obtenidos del software termociclador para el gen gth (GOI y genes de referencia) del pocillo rth (las reacciones se realizaron por triplicado) en la placa k-ésima correspondiente al animal mth y al tratamiento ith (CON y ALF); TGGi es la interacción fija entre el tratamiento i-ésimo y el gen gth (T es el efecto del tratamiento i-ésimo, y G es el efecto del gen gth); Pk es el efecto fijo de la placa k-ésima; FMIm y SAm son los efectos de la grasa intramuscular (sólo utilizada en la expresión génica del tejido muscular LT) y la edad de sacrificio del animal Mth, respectivamente, incluidos como covariables; Unm es el efecto aleatorio del animal mth del que se recogieron las muestras (Am∼(0,σ2Un)); y erigkm es el residuo aleatorio. La varianza residual específica del gen (residuo heterogéneo) se ajustó al gen por efecto del tratamiento (erigkm∼N(0, σ2EGI).
Para probar las diferencias (diffGOI) en la tasa de expresión de los genes diana entre tratamientos y para obtener valores de cambio de pliegue (FC) a partir de las diferencias TG estimadas, se utilizó el enfoque sugerido en Steibel et al. (2009). El significado de la diferenciaGOI Las estimaciones se determinaron con el estadístico T. Además, se calcularon intervalos de confianza asimétricos del 95% (superior e inferior) para cada valor de FC utilizando el error estándar (SE) de diffGOI.
Análisis de anotaciones funcionales
La Database for Annotation, Visualization and Integrated Discovery (DAVID) v6.7b (Huang et al., 2008) se utilizó para determinar vías y procesos de mayor importancia biológica e importancia a través de la herramienta Functional Annotation Cluster (FAC) basada en la función de anotación Gene Ontology (GO). El análisis DAVID FAC se realizó con las listas de genes obtenidas tras el análisis SAM. Se seleccionaron los parámetros de puntuación EASE de rigor medio para indicar puntuaciones de enriquecimiento seguras de significación funcional e importancia de las vías y procesos investigados dados. Se empleó una puntuación de enriquecimiento de 1,3 como umbral para la significación del conglomerado. El plug-in ClueGO (Bindea et al. 2009) y el programa Cytoscape (Shannon et al., 2003) se utilizaron para agrupar genes según la similitud de los procesos biológicos en los que están involucrados. Las relaciones entre la serie de AGPI n-3 en el músculo y la expresión génica en el hígado se visualizaron utilizando el complemento Metscape (Karnovsky et al., 2012) en Cytoscape (Shanon et al., 2003). Treinta genes significativos (de 96) y nueve (de 13) compuestos significativos fueron mapeados a KEGG IDs. El archivo que contiene la lista de genes y metabolitos, su cambio de pliegue y los valores P se cargó en Metscape para generar una red de genes compuestos.
Resultados
Rendimiento de los corderos y concentraciones de α-tocoferol, TG, colesterol LDL, colesterol HDL y colesterol en plasma
No se encontraron diferencias en el peso y la edad de sacrificio (SA) o la ganancia diaria promedio (ADG) desde el nacimiento hasta el sacrificio entre los tratamientos (Tabla complementaria 1). Las pruebas de tipo III de los efectos fijos (tratamiento y día) sobre los parámetros sanguíneos se muestran en la Tabla complementaria 3.
Las concentraciones de α-tocoferol, colesterol, colesterol LDL y colesterol HDL en plasma se vieron afectadas por la interacción entre el tratamiento y el día (P < 0,05 a < 0,01). Mientras tanto, la concentración de TG en plasma se vio afectada solo por el tratamiento (P < 0,01).
Los animales de pastoreo (grupo ALF) tuvieron una mayor concentración de contenidos de α-tocoferol (P < 0,0001) y colesterol similar (P = 0,056) durante todo el período experimental en comparación con los del grupo CON (Figuras 1A, B). El contenido de colesterol HDL fue similar entre los tratamientos, excepto en el día 8 después del destete, y el grupo de IHA tuvo un contenido mayor en comparación con el grupo CON (0,58 ± 0,03 mmol / l frente a 0,34 ± 0,03 mmol / l; P < 0,05). Del mismo modo, los corderos ALF presentaron una mayor concentración de TG en los días 8 y 28 postdestete (0,58 ± 0,03 vs. 0,67 ± 0,05 y 0,25 ± 0,03 vs. 0,3073 ± 0,07 mmol/l; P < 0,05). El contenido de colesterol LDL fue mayor en el grupo ALF solo en el día 0 (equivalente al día del destete en el grupo CON) en comparación con el grupo CON (0.48 ± 0.05 vs. 0.18 ± 0.05 mmol / l; P < 0,05) (Figura 1).
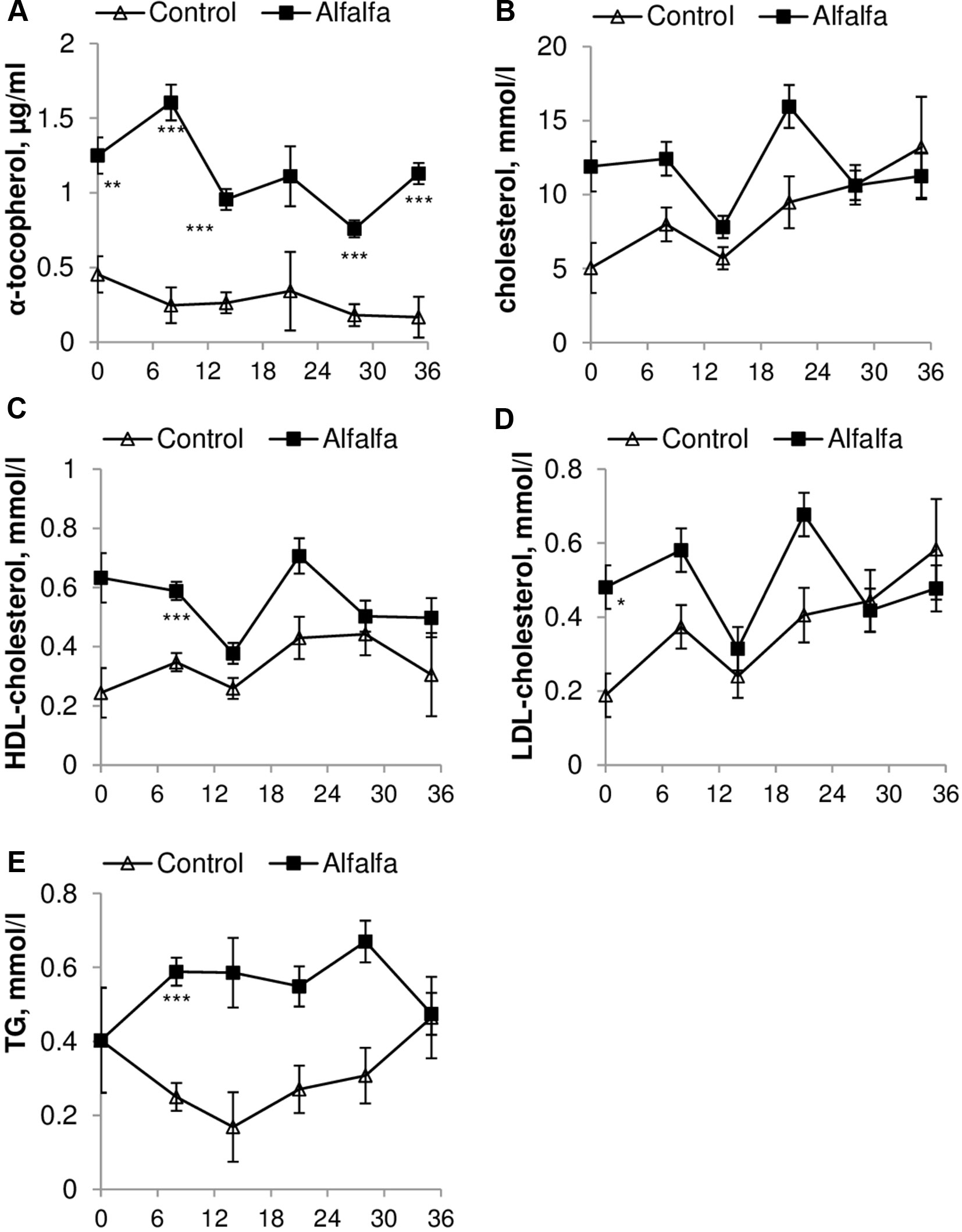 FIGURA 1 Concentración de (A) α-tocoferol, (B) colesterol, (C) colesterol HDL, (D) colesterol LDL y (E) TG durante todo el período experimental en comparación con los del grupo CON.
FIGURA 1 Concentración de (A) α-tocoferol, (B) colesterol, (C) colesterol HDL, (D) colesterol LDL y (E) TG durante todo el período experimental en comparación con los del grupo CON.
Características de la carne en el músculo Longissimus thoracis
El contenido de grasa intramuscular, el color de la carne y los pigmentos no fueron diferentes entre los tratamientos (Tabla complementaria 4; P > 0,05); sin embargo, los corderos ALF tuvieron un mayor contenido de α-tocoferol en el músculo LT en comparación con el del grupo CON (2,38 ± 0,17 vs. 0,48 ± 0,17; P < 0,05) (Tabla complementaria 3). La oxidación lipídica se vio afectada por la interacción entre el tratamiento y el día de visualización (P < 0,01). La oxidación lipídica fue similar en los primeros días de visualización (P > 0,05), pero los corderos ALF tuvieron una oxidación menor que los corderos CON en el día 7 de exhibición (P < 0,05).
En cuanto al perfil de ácidos grasos, el músculo LT del grupo ALF tuvo un mayor contenido de ácidos cáprico (C10:0) y araquídico (C20:0) (Tabla 2; P < 0,05) y tendían a tener un mayor contenido de ácido margárico (C17:0) y ácido esteárico (C18:0) (0,1 < P > 0,05). Sin embargo, el contenido total de AGS no fue diferente entre los tratamientos (P > 0,05; Tabla 2).
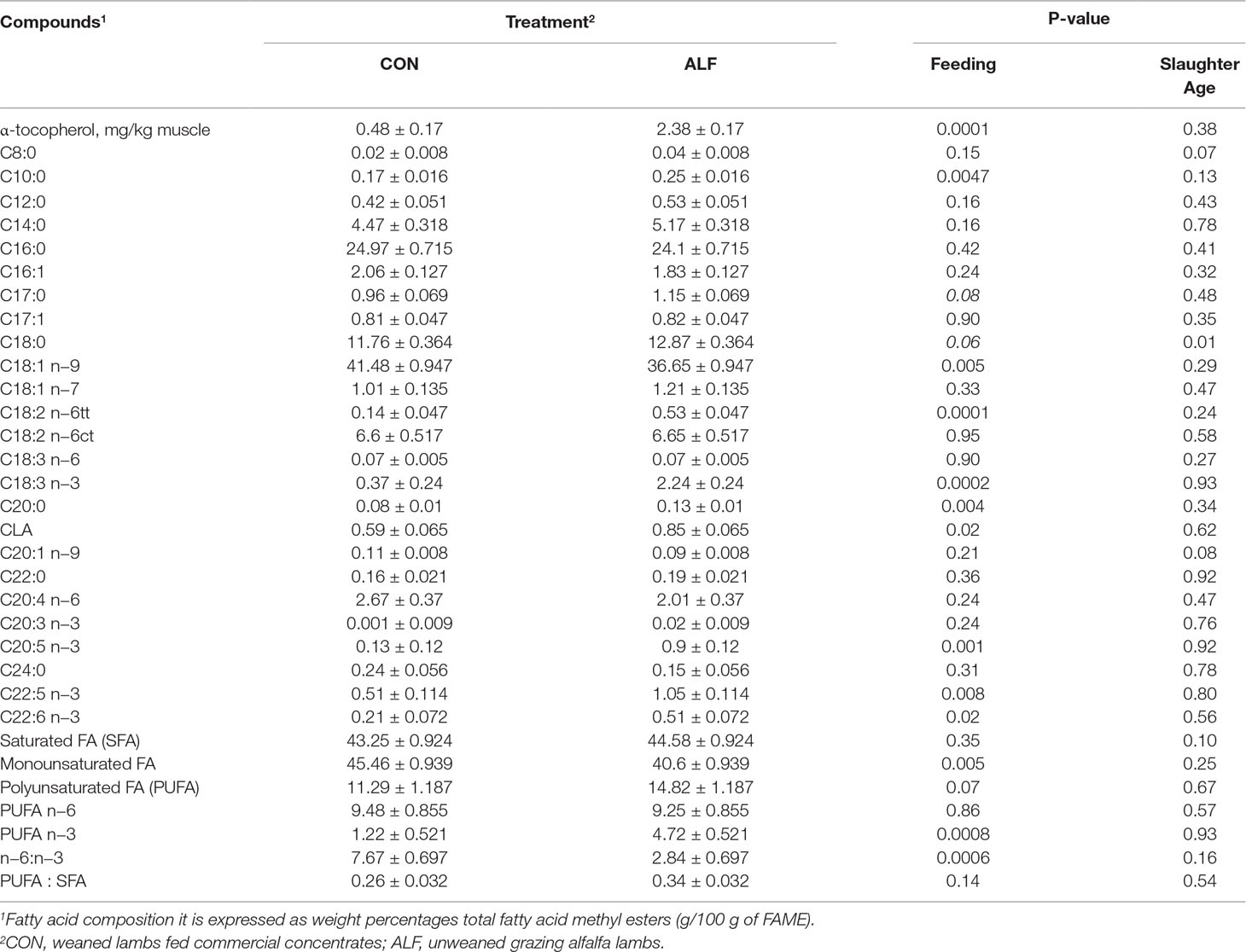 CUADRO 2 Efecto del tratamiento sobre el contenido de composición de α-tocoferol y ácidos grasos (FA) del músculo LT en corderos Rasa Aragonesa.
CUADRO 2 Efecto del tratamiento sobre el contenido de composición de α-tocoferol y ácidos grasos (FA) del músculo LT en corderos Rasa Aragonesa.
El tratamiento no afectó al ácido palmitoleico (C16:1; P > 0,05), ácido vaccénico (C18:1 n−7; P > 0,05) y ácido eicosenoico (C20:1 n−9; P > 0,05) pero afectó al contenido de ácido oleico (C18:1 n−9; P < 0,05) y MUFA totales, que fue mayor en CON que en corderos ALF (P < 0,05; Tabla 2). Con respecto a los contenidos individuales de AGPI, los contenidos de ácido linoleico (C18:2 n−6), ácido linolénico (C18:3 n−3), EPA (C20:5 n−3), ácido docosapentaenoico (C22:5 n−3) y DHA (C22:6 n−3) fueron mayores en el grupo ALF que en el grupo CON (P < 0,05). Además, el músculo LT de los corderos ALF tuvo un mayor contenido de AGPI n-3 y una menor relación n-6:n−3 (P < 0,001) en comparación con los de los corderos CON, pero no afectó el contenido total de AGPI (P = 0,07).
Resultados de la expresión génica de microarrays
Identificación y clasificación de genes expresados diferencialmente en músculo LT, hígado y SF
Cuarenta y uno, cuatro y 96 genes se expresaron diferencialmente en músculo LT, SF e hígado, respectivamente, después del análisis SAM (Figura suplementaria 1). En el músculo LT, 41 genes se expresaron diferencialmente con un FDR = 0,002 (Tabla 3), de los cuales 32 fueron regulados negativamente y nueve genes fueron regulados al alza. En el hígado, 96 genes se expresaron diferencialmente (Tabla complementaria 5), entre los cuales cuatro genes fueron regulados al alza y 92 genes fueron regulados negativamente con el tratamiento de IHA (FDR = 0,002). Los 20 genes principales significativos en el hígado se muestran en la Tabla 4.
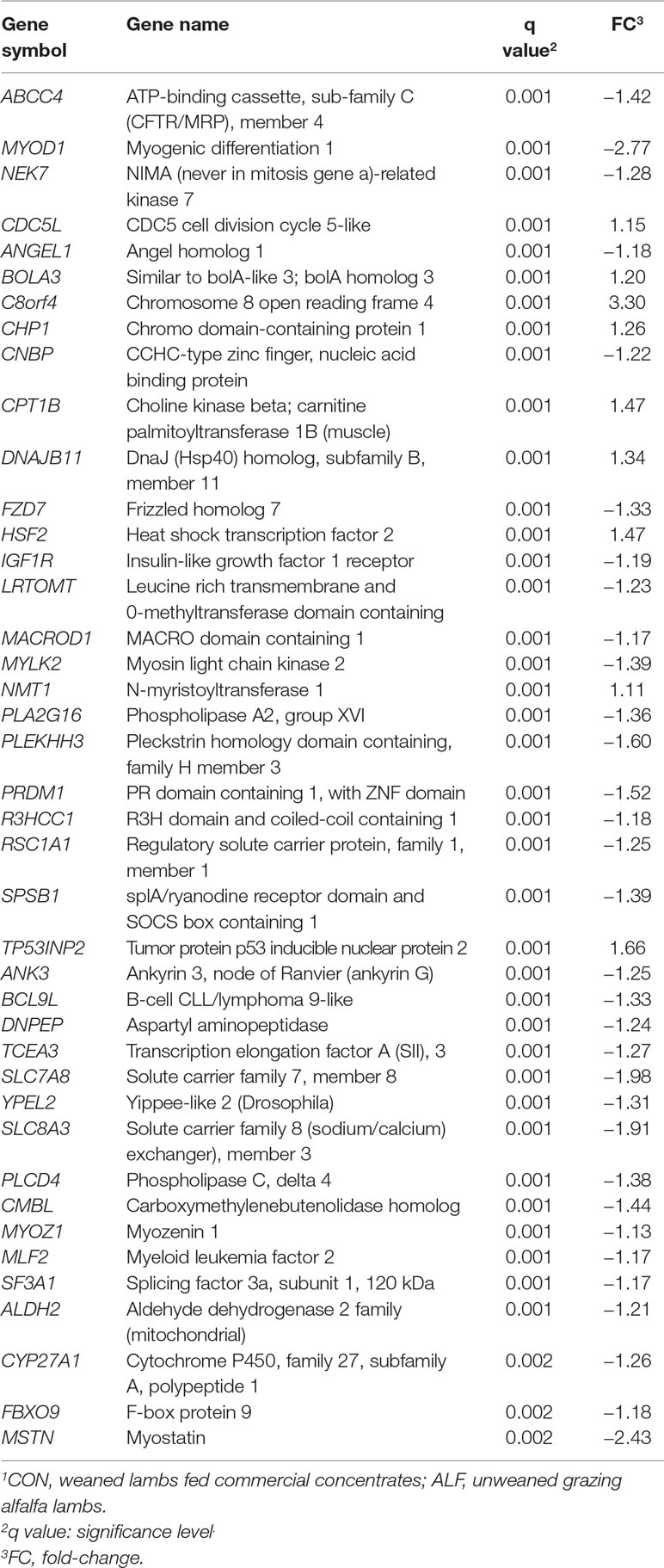 CUADRO 3 Genes expresados diferencialmente en el músculo Longissimus thoracis y cambio de pliegue en ALF-CON1 contraste.
CUADRO 3 Genes expresados diferencialmente en el músculo Longissimus thoracis y cambio de pliegue en ALF-CON1 contraste.
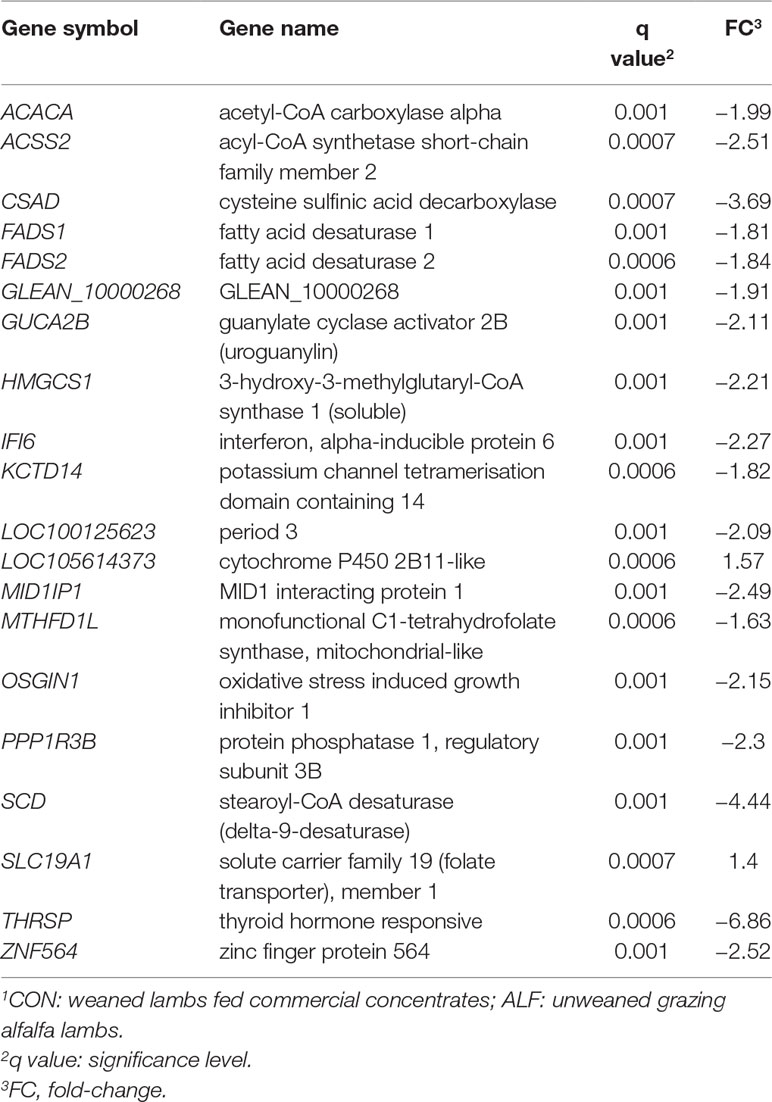 CUADRO 4 Los 20 genes principales expresados diferencialmente en el hígado y el cambio de pliegue en ALF-CON1 contraste.
CUADRO 4 Los 20 genes principales expresados diferencialmente en el hígado y el cambio de pliegue en ALF-CON1 contraste.
En cuanto a la SF, cuando se comparó el tratamiento de la IHA con la CON, sólo cuatro genes se expresaron diferencialmente con una FDR = 0,051, y todos ellos fueron regulados al alza en el grupo de IHA (Tabla 5).
 CUADRO 5 Genes expresados diferencialmente significativos en la grasa subcutánea y el cambio de pliegue en ALF-CON1 contraste.
CUADRO 5 Genes expresados diferencialmente significativos en la grasa subcutánea y el cambio de pliegue en ALF-CON1 contraste.
Resultados del análisis multivariante dependiente del tratamiento de la expresión génica en músculo, hígado y grasa subcutánea de Longissimus thoracis
El análisis de componentes principales (PC) del conjunto completo mostró que los dos primeros PC cubrieron el 81,1% de la varianza observada del conjunto de muestras en el músculo LT (Figura 2A). Los grupos correspondientes a los perfiles de expresión génica de los grupos ALF y CON estaban claramente separados entre sí. Se obtuvieron resultados muy similares en el hígado (Figura 2B), pero en SF, esta separación fue menos clara (Figura 2C).
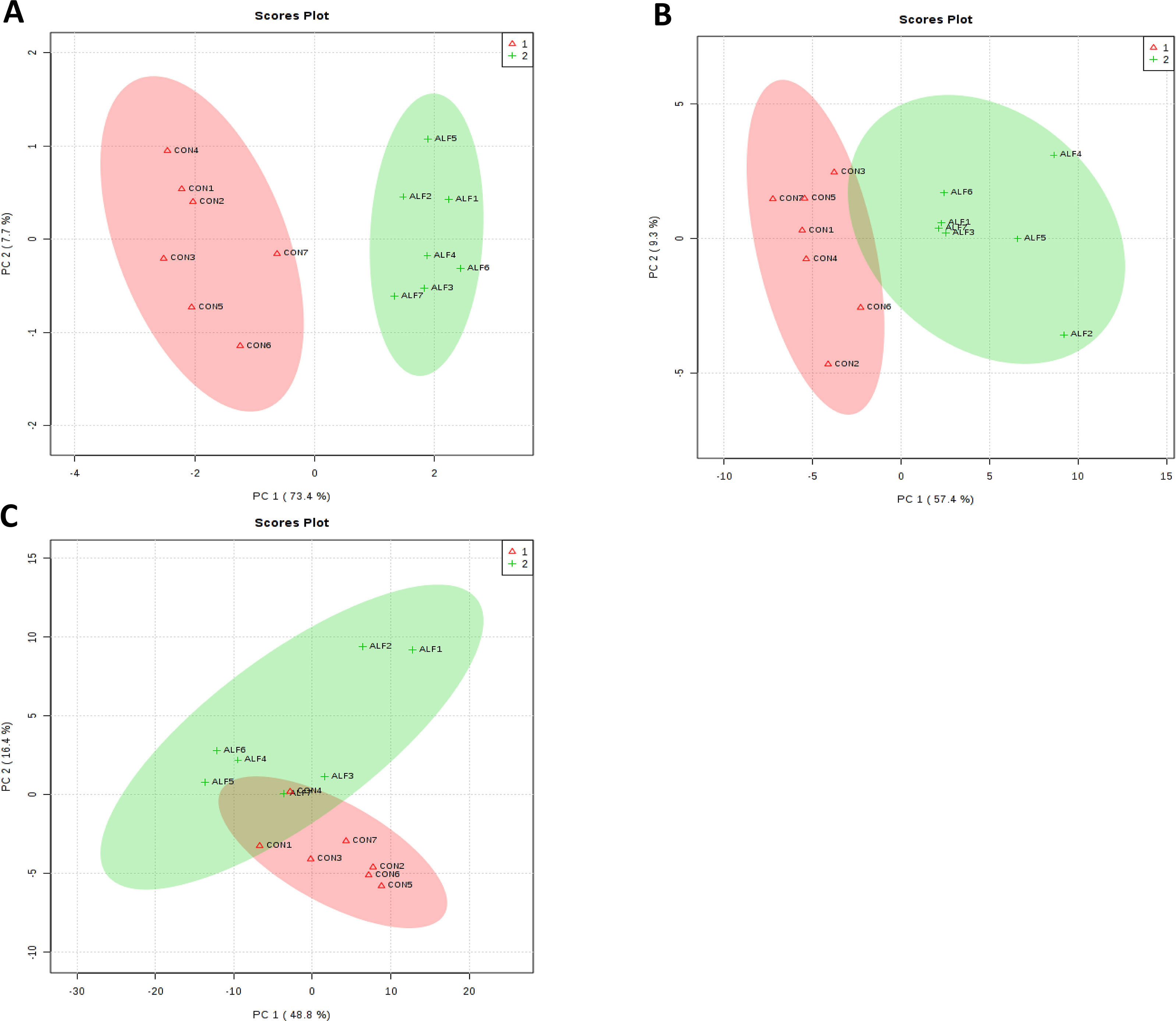 FIGURA 2 Análisis de componentes principales (PCA) basado en datos del perfil de expresión génica para el músculo (A) LT (B) hígado y (C) grasa subcutánea. El análisis de componentes principales puntúa las gráficas que discriminan entre el músculo LT de corderos alimentados con concentrado CON (+) y alfalfa (triángulo).
FIGURA 2 Análisis de componentes principales (PCA) basado en datos del perfil de expresión génica para el músculo (A) LT (B) hígado y (C) grasa subcutánea. El análisis de componentes principales puntúa las gráficas que discriminan entre el músculo LT de corderos alimentados con concentrado CON (+) y alfalfa (triángulo).
Análisis de agrupamiento jerárquico (HCA) en músculo e hígado de Longissimus Thoracis
El análisis de agrupamiento jerárquico para la expresión génica se realizó utilizando todos los genes y solo los genes significativos para cada tejido. Debido a que solo cuatro genes fueron significativos en SF, los resultados del análisis de conglomerados no se incluyen. Los resultados de HCA utilizando solo los genes significativos para el músculo LT y el hígado se presentan en la Figura 3. El perfil de expresión de estos genes fue capaz de agrupar y clasificar correctamente las muestras dentro de su grupo correspondiente. El mapa de calor muestra la presencia de dos grupos diferentes en ambos tejidos. Estos dos grupos distinguieron claramente al grupo ALF del grupo CON, ya que ambos grupos mostraron patrones de expresión génica muy diferentes. Por ejemplo, en el músculo LT, los genes BOLA, HSF2, CHP1, DNAJB11, CDC5L, TP53INP2, CPT1B, C8ORF4 y NMT1 se regularon al alza en el grupo ALF. Además, se encontró que un segundo grupo que incluía el resto de los genes estaba regulado a la baja en el grupo ALF (Figura 3A). En el hígado, los genes BHMT, LOC105614373 y SLC19A1 fueron regulados al alza en el grupo ALF, y un segundo grupo, incluyendo los genes restantes, fue regulado a la baja en el grupo ALF (Figura 3B).
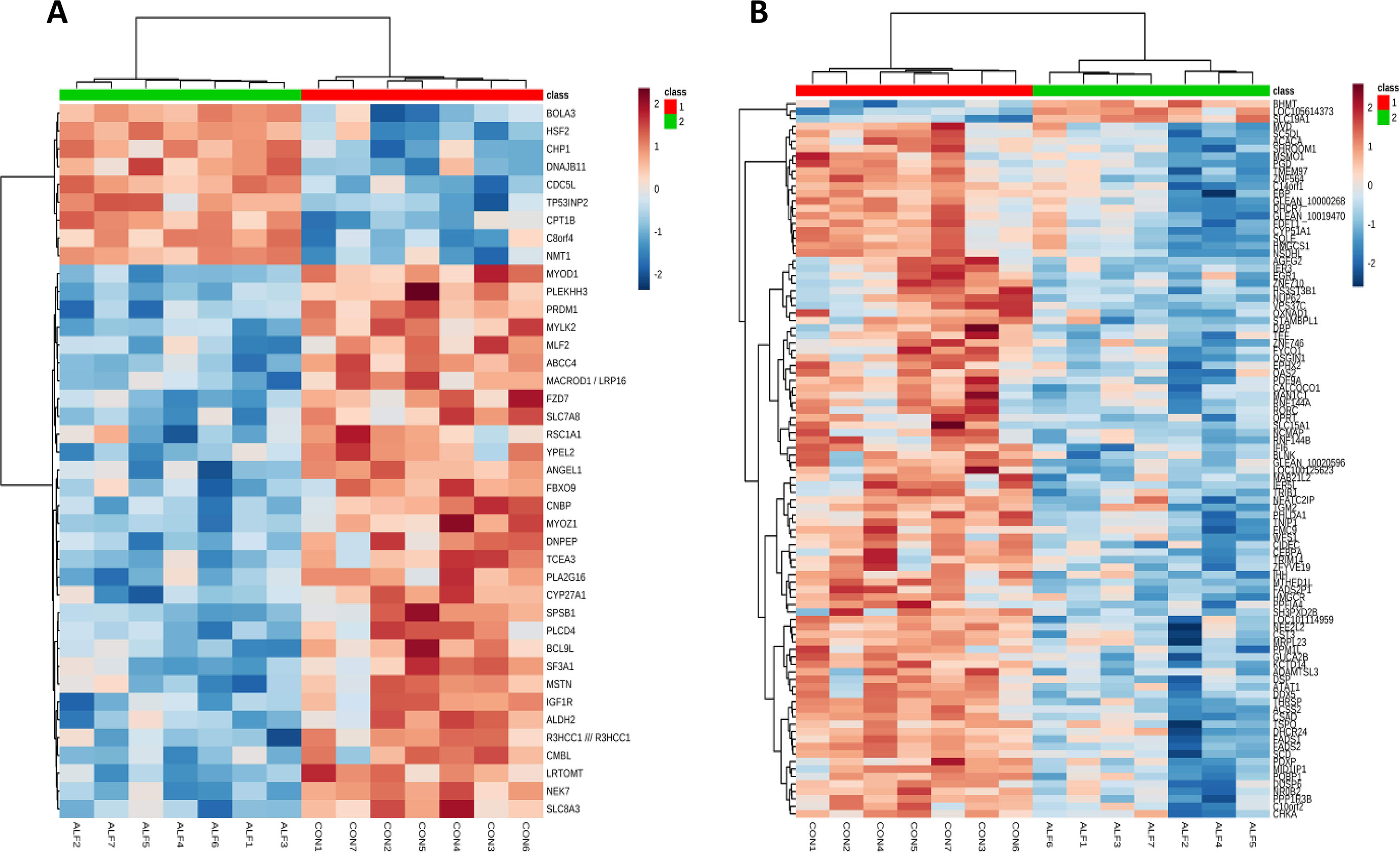 FIGURA 3 Análisis de agrupamiento jerárquico de expresión génica significativa en el músculo (A) LT y (B) hígado de corderos CON (1) y corderos ALF (2). Las celdas se colorean en función de la intensidad de la señal medida. El marrón oscuro representa altos niveles de expresión génica, el azul indica baja intensidad de señal y las células grises representan el nivel intermedio.
FIGURA 3 Análisis de agrupamiento jerárquico de expresión génica significativa en el músculo (A) LT y (B) hígado de corderos CON (1) y corderos ALF (2). Las celdas se colorean en función de la intensidad de la señal medida. El marrón oscuro representa altos niveles de expresión génica, el azul indica baja intensidad de señal y las células grises representan el nivel intermedio.
Anotación de agrupación en clústeres funcionales
Longissimus thoracis Músculo
Para obtener información sobre los procesos biológicos que se regulan diferencialmente entre los tratamientos dietéticos, realizamos análisis de enriquecimiento utilizando DAVID y ClueGo. Los resultados de DAVID functional annotation clustering (FAC) revelaron que los clusters funcionales más enriquecidos estaban asociados con «procesos lipídicos y catabólicos» (CPT1B, PLA2G16, SPSB1, LRTOMT, PLCD4, FBXO9, CNBP y CYP27A1) y 14 genes relacionados con el «desarrollo muscular» (ALDH2, ANK3, CPT1B, FZD7, HSF2, IGF1R, LRTOMT, MSTN, MYLK2, MYOD1, MYOZ1, NMT1, PRDM1 y RSC1) (Tabla complementaria 5). Todos estos genes fueron regulados a la baja en el grupo ALF excepto CPT1B, HSF2 y NMT1, aunque las puntuaciones de enriquecimiento seguro fueron inferiores a 1,3 en ambos casos. Los roles biológicos de los genes regulados a la baja en el músculo LT también se visualizaron con ClueGO (Figura 4). El tamaño de los nodos refleja la significación estadística del término. El proceso biológico más enriquecido fue el del «desarrollo del tejido muscular esquelético» con cuatro genes, MSTN, MYLK2, MYOD1 y BCL9L.
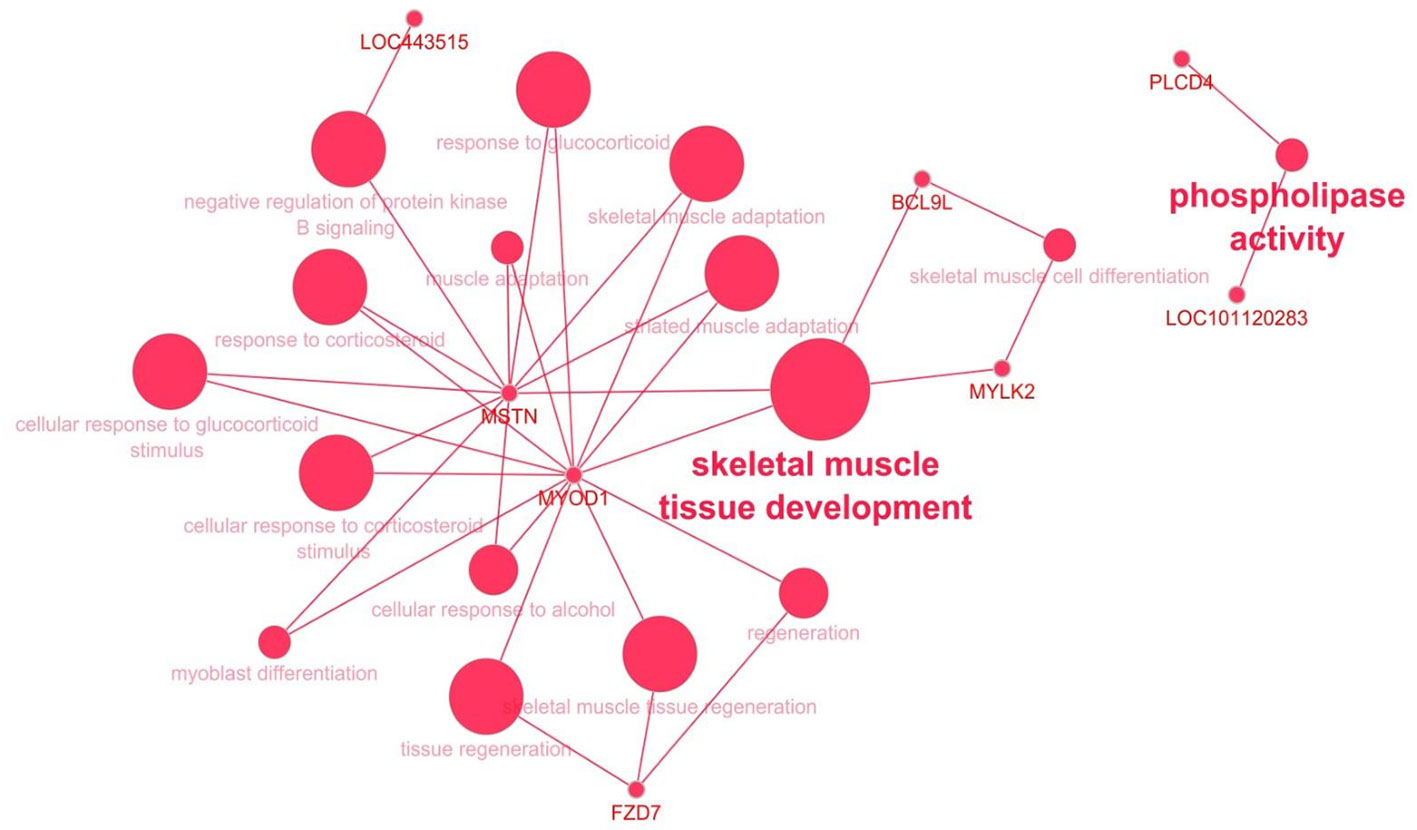 FIGURA 4 El papel biológico de los genes regulados hacia arriba (verde) y hacia abajo (rojo) en el músculo LT de animales ALF visualizado con ClueGO. El tamaño de los nodos refleja la significación estadística del término.
FIGURA 4 El papel biológico de los genes regulados hacia arriba (verde) y hacia abajo (rojo) en el músculo LT de animales ALF visualizado con ClueGO. El tamaño de los nodos refleja la significación estadística del término.
Hígado
Los resultados de DAVID revelaron dos grupos principales de genes asociados con la «biosíntesis de esteroles» (EBP, MVD, HMGCR, CYP51A1, HMGCS1, NR0B2, C14ORF1, FDFT1, SQLE, DHCR7, SC5DL, DHCR24 y NSDHL), «proceso biosintético de lípidos» (ACACA, CYP51A1, FADS1, FADS2, SCD y SC5DL), y «proceso metabólico del colesterol» (EBP, MVD, HMGCR, CYP51A1, SQLE, DHCR7, HMGCS1, NR0B2, DHCR24, FDFT1 y NSDHL) (Tabla suplementaria 6). Resultados similares se obtuvieron con ClueGo, donde los procesos biológicos más enriquecidos fueron «proceso biosintético de ácidos grasos», «proceso metabólico de esteroles», «procesos metabólicos de cofactor» y «procesos metabólicos de coenzimas» (Figura 5). Todos estos genes fueron regulados a la baja en el tratamiento de la IHA.
 FIGURA 5 El papel biológico de los genes up- (verde) y downregulated (rojo) en animales ALF visualizados con ClueGO en el hígado. El tamaño de los nodos refleja la significación estadística del término.
FIGURA 5 El papel biológico de los genes up- (verde) y downregulated (rojo) en animales ALF visualizados con ClueGO en el hígado. El tamaño de los nodos refleja la significación estadística del término.
Grasa subcutánea
Solo 4 genes fueron significativos en SF, y no se encontró ningún grupo con DAVID FAC.
Validación de resultados de microarrays mediante qPCR
El conjunto de genes seleccionado para validar los resultados de microarrays por qPCR incluyó los siguientes 15 genes: CPT1B, MYOD1, MSTN, ABCC4, IGF1R y PLA2G16 para músculo LT; FADS1, FADS2, ACACA, SCD, SQLE, IER3, SLC19A1 y THRSP para hígado; y METTL1 para SF. Los genes fueron seleccionados porque se expresaron significativamente diferencialmente entre los grupos. La expresión de estos genes mediante tecnología de microarrays y qPCR se muestra en la Tabla 6. Los genes de limpieza GUSB y YWHAZ se utilizaron para normalizar los resultados para el músculo LT y SF. En el hígado, se probaron cinco genes de referencia candidatos, y los genes más estables que exhibieron el valor de estabilidad de expresión (M) más bajo fueron RPL37 (M = 0.182), GUSB (M = 0.226) y RPL19 (M = 0.296). Los tres genes de referencia fueron más estables que los GOI. La magnitud del cambio de pliegue obtenido por microarray y qPCR fue ligeramente diferente en algunos casos, pero los resultados de qPCR demostraron una tendencia similar en comparación con los resultados de microarrays de estos genes (Tabla 6).
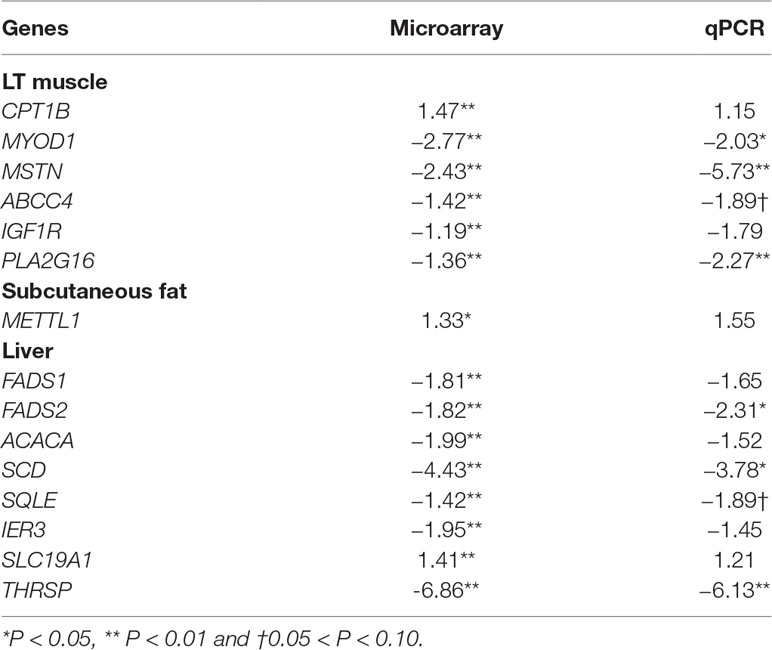 CUADRO 6 Confirmación por PCR en tiempo real de los resultados del microarray. Cambios en la expresión génica en músculo LT, hígado y grasa subcutánea en la comparación de IHA vs CON, y el cambio de pliegue (FC) obtenido con datos de microarrays y qPCR.
CUADRO 6 Confirmación por PCR en tiempo real de los resultados del microarray. Cambios en la expresión génica en músculo LT, hígado y grasa subcutánea en la comparación de IHA vs CON, y el cambio de pliegue (FC) obtenido con datos de microarrays y qPCR.
Discusión
En este estudio, investigamos el perfil de ácidos grasos y la expresión génica utilizando un microarray en el músculo LT, hígado y SF de corderos alimentados con concentrado o alfalfa. Como era de esperar, los animales con IHA contenían mayores CLAs y una mayor proporción de AGPI n−3 en el músculo, como ácido linolénico (C18:3 n−3), EPA (C20:5 n−3), ácido docosapentaenoico (C22:5 n−3) y DHA (C22:6 n−3), en comparación con los niveles en el grupo CON. Muchos estudios han reportado el impacto del pastoreo en el perfil de ácidos grasos en corderos de carne, particularmente los ácidos grasos de la serie n-3 (Fisher et al., 2000; Dervishi et al., 2010; Vasta et al., 2012). Zhang et al. (2017) sugirieron que compuestos específicos en la dieta pueden transferirse a la carne. En nuestro experimento, la composición de ácidos grasos, especialmente la de la serie n-3, y el α-tocoferol son probablemente un reflejo de la composición de la dieta. Los corderos lechales son funcionalmente no rumiantes, y su perfil de AF de carne debe reflejar el perfil de AF de la leche amamantada (Napolitano et al., 2002; Valvo y otros, 2005). Por lo tanto, las presas de pastoreo son una alternativa aconsejable para aumentar los AGPI en la carne de cordero lechal porque el pasto fresco tiene una alta concentración de ácido linolénico (C18: 3n-3), lo que aumenta el contenido de ácido vaccénico (C18: 1t-11), isómeros conjugados de ácido linoleico (CLA) y AGPI n-3 en la leche en comparación con las dietas que comprenden forraje concentrado o conservado (Nudda et al., 2005; Joy et al., 2012). El alto valor de C18:3n−3 en la carne de cordero ALF podría deberse al C18:3n−3 proporcionado por los pastos que, como aún no están completamente destetados, no están biohidrogenados por la microbiota ruminal. Además, la efectividad relativamente baja de la leche para afectar la composición de ácidos grasos de la carne, podría explicar la ligera diferencia entre CON y ALF en CLA y VA. Por lo tanto, los corderos a los que se les permitió pastar dieron como resultado un perfil de ácidos grasos de la carne que es más rico en ácidos grasos de la serie n-3, principalmente debido a la leche de la madre que pastaba continuamente durante la lactancia en los pastos de alfalfa. Según Álvarez-Rodriguez et al. (2018) la alfalfa dietética, pero no el suministro de leche, mejoró el CLA y el contenido de AGPI n-3 en la carne de cordero. La composición de FA de los corderos ALF estaba más relacionada con la leche de oveja que con el forraje fresco (Dervishi et al., 2010). Estudios previos han demostrado que el pastoreo aumenta el contenido de AGPI en la leche, particularmente el ácido linolénico (C18: 3n-3), mientras que los concentrados modifican el tiempo de retención ruminal del alimento, aumentan la ingesta de ácido linoleico (C18: 2n-6) y alteran las vías de biohidrogenación hacia un menor contenido de AGPI y CLA n-3, lo que lleva a un menor contenido de estos compuestos en la leche (Elgersma, 2015).). Además, estos animales tenían mayor α-tocoferol en músculo y plasma. La vitamina E es un poderoso antioxidante soluble en grasa que desempeña un papel importante en la eliminación de los radicales libres y la función neurológica (Wang y Quinn, 2000; Traber y Atkinson, 2007). En este estudio, encontramos que la oxidación lipídica fue menor en los corderos ALF en el día 7 de visualización en comparación con los niveles en corderos CON. Estos resultados están en concordancia con estudios previos en los que informamos que la adición de vitamina E a la dieta aumentó el contenido del músculo α-tocoferol y disminuyó drásticamente la oxidación lipídica de la carne (Kasapidou et al., 2012; González-Calvo et al., 2015b; Ponnampalam et al., 2017).
Además, investigamos cómo el sistema de alimentación impactó la expresión génica en el músculo LT, el hígado y SF en ambos grupos de tratamiento. De hecho, encontramos que ambos grupos diferían en su perfil de expresión génica, principalmente en el músculo LT y el hígado, con el mayor impacto en el hígado. Se ha informado que la intervención dietética puede conducir a cambios importantes en la expresión génica en el músculo y el hígado (Dervishi et al., 2011; Cui et al, 2018). En el grupo ALF, los procesos biológicos más enriquecidos en el músculo LT fueron el desarrollo del tejido muscular esquelético (MYOD1, MYLK2 y MSTN) (Figura 4 y Tabla complementaria 5). Estos genes fueron regulados a la baja en el grupo ALF, siendo MYOD1 y MSTN los genes más regulados (el FC más bajo). El rendimiento de la carne vendible y la calidad de la carne, y por lo tanto la rentabilidad de las operaciones ganaderas, están muy influenciados por el crecimiento durante el período postnatal. Por lo tanto, la identificación de genes que desempeñan un papel en el crecimiento muscular en ovejas es un paso importante para mejorar la producción de carne de ovino por selección. En este sentido, en las especies ganaderas, MYOD1 y MSTN se consideran genes candidatos para la calidad de la carne y los rasgos de la canal (Ibeagha-Awemu et al., 2008; Bhuiyan y otros, 2009). MYOD1 regula la diferenciación, el crecimiento y el desarrollo de las células musculares y también está involucrado en la regeneración muscular (Kitzmann et al., 1998). Por ejemplo, los polimorfismos de MYOD1 se han asociado con el peso, varias características de la fibra muscular, el área del ojo del lomo y la ligereza en poblaciones de yak, cerdo y ganado (Chu et al., 2012; Lee et al., 2012; Du et al., 2013). Además, los bajos niveles de expresión de MYOD1 se relacionaron con una baja fuerza de cizallamiento de Warner-Bratzler medida en el músculo longissimus dorsi de la carne de res (Tizioto et al., 2014) y, por lo tanto, con una mayor ternura. En ovejas, se encontró una correlación positiva entre la expresión de MYOD1 y el rendimiento de canales frías (Lôbo et al., 2012). Los autores propusieron que los animales con una mayor expresión de MYOD1 fueron más eficientes durante el crecimiento postnatal y tuvieron un mayor peso del longissimus dorsi y un mejor rendimiento de canales frías y calientes. En nuestro estudio, no observamos diferencias en el peso de sacrificio o la ganancia diaria promedio entre los grupos ALF y CON. Estas discrepancias pueden deberse a diferentes edades de sacrificio entre ambos estudios. Sacrificamos a nuestros animales a los 67-72 días, y otros estudios compararon corderos pesados (a un promedio de 200 días) alimentados con concentrado o pastoreo limitado (Lôbo et al., 2012), mientras que en el presente estudio, los corderos de pastoreo tenían acceso libre a forraje, concentrado y leche de madre. A pesar de los diferentes resultados, es necesaria una mayor investigación sobre el papel de MYOD1 en la canal de oveja y los rasgos de calidad de la carne en ovejas para una selección efectiva asistida por marcadores. Otro gen que fue regulado a la baja en el grupo ALF fue MSTN. La miostatina es una citoquina extracelular que se expresa principalmente en los músculos esqueléticos y se sabe que desempeña un papel crucial en la regulación negativa de la masa muscular (Elkina et al., 2011). Este efecto se debe a un aumento tanto en el número de fibras musculares (hiperplasia) como en la masa (hipertrofia). Por ejemplo, Ji et al. (1998) demuestranEs que la expresión de miostatina en el músculo esquelético alcanza su punto máximo prenatal y que una mayor expresión durante el período prenatal se asocia con bajo peso al nacer en cerdos. Las mutaciones en el gen de la miostatina con inactivación funcional en el ganado vacuno de carne aumentan la masa muscular en el fenotipo de doble músculo y conducen a adipocitos más pequeños y menos islas de grasa en el músculo (Cassar-Malek et al., 2007). Además, en diferentes razas de ganado, las mutaciones en MSTN se han asociado con reducciones significativas en la fuerza de cizallamiento y una disminución en el contenido total de colágeno (Ngapo et al., 2002; Lines et al., 2009). Además, las mutaciones en MSTN en ovejas se asociaron con musculación y reducción de la grasa intramuscular (Kijas et al., 2007) y un mayor porcentaje de miofibras glucolíticas rápidas (Laville et al., 2004). En nuestro experimento, el grupo ALF mostró genes MYOD1 y MSTN regulados a la baja, que pueden ser beneficiosos para aumentar la ternura de la carne y el rendimiento de la canal fría en animales más pesados. Sin embargo, la regulación simultánea a la baja de los genes MYOD1 y MSTN en el grupo ALF podría determinar un efecto opuesto sobre el rendimiento del animal, justificando así la falta de rendimiento diferente entre dos grupos.
Los resultados del análisis funcional mostraron que los genes relacionados con los procesos catabólicos y lipídicos en el músculo LT estaban regulados a la baja (PLA2G16, SPSB1, LRTOMT, PLCD4, FBXO9, CNBP y CYP27A1), excepto CPT1B, que estaba regulado al alza en el grupo ALF (Tabla complementaria 5). La carnitina palmitoiltransferasa I (M-CPT 1), codificada por el gen CPT1B, forma parte del sistema de transporte mitocondrial y es una enzima clave en el control de la oxidación de ácidos grasos de cadena larga (Bartelds et al., 2004). Estos resultados están de acuerdo con los obtenidos previamente por Dervishi et al. (2011, 2012) en los que los sistemas de pastoreo promovieron niveles más altos de expresión génica CPT1B en el músculo semitendinoso y la glándula mamaria. Según lo informado por Dervishi et al. (2010), la alimentación concentrada promueve la regulación positiva de genes relacionados con la adipogénesis, mientras que el sistema de pastoreo promueve niveles más altos de genes implicados en la oxidación de ácidos grasos.
El impacto del sistema de alimentación fue más pronunciado en la expresión génica del hígado, donde 96 genes cambiaron significativamente en comparación con los del músculo LT (41 genes) y SF (4 genes). Los principales sitios de síntesis de ácidos grasos son el tejido adiposo y el hígado. Sin embargo, los resultados de la expresión génica en estos tres tejidos diferentes sugieren que en corderos jóvenes, el sitio principal del metabolismo de los lípidos es el hígado en lugar de la grasa subcutánea.
Intentamos vincular los ácidos grasos significativos en el músculo y los metabolitos en el plasma con los resultados de la expresión génica para obtener una mejor comprensión de los procesos metabólicos subyacentes asociados con diferentes sistemas de alimentación. La relación entre n-3 FA en el músculo y la expresión génica en el hígado mapeó 30 genes significativos (de 92) y 9 compuestos significativos (de 13) a KEGG ID. Se generó una red de genes compuestos (Figura 6 y Figura complementaria 2). Además, este enfoque nos ayudó a identificar genes relacionados con procesos biológicos enriquecidos y ciertos resultados deseados, por ejemplo, n−3 series de AGPI que son deseables para la salud humana (Simopoulos, 2008; Cabo et al., 2012; Liu y Ma, 2014). De hecho, los animales que pastaban alfalfa tenían un mayor contenido de ácidos grasos de la serie n−3 como ácido linolénico (C18:3 n−3), EPA (C20:5 n−3), ácido docosapentaenoico (C22:5 n−3) y DHA (C22:6 n−3) en músculo LT y menor expresión de FADS1 y FADS2 en hígado (Figura 6). Vale la pena mencionar que FADS1 y FADS2 en el grupo de «proceso biosintético de ácidos grasos» son genes clave en el metabolismo de la serie de AGPI n-3. Las proteínas codificadas por estos genes (FADS1 y FADS2) son miembros de la familia de genes de la desaturasa de ácidos grasos (FADS). Las enzimas desaturasas regulan la insaturación de ácidos grasos a través de la introducción de dobles enlaces entre carbonos definidos de la cadena de acilo graso (Nakamura y Nara, 2004). Además, estos animales se caracterizaron por una disminución en la expresión de genes relacionados con el metabolismo del colesterol (DHCR7, SC5DL, EBP, NSDHL, MTHFD1L y CYP51A1; Figura complementaria 2).
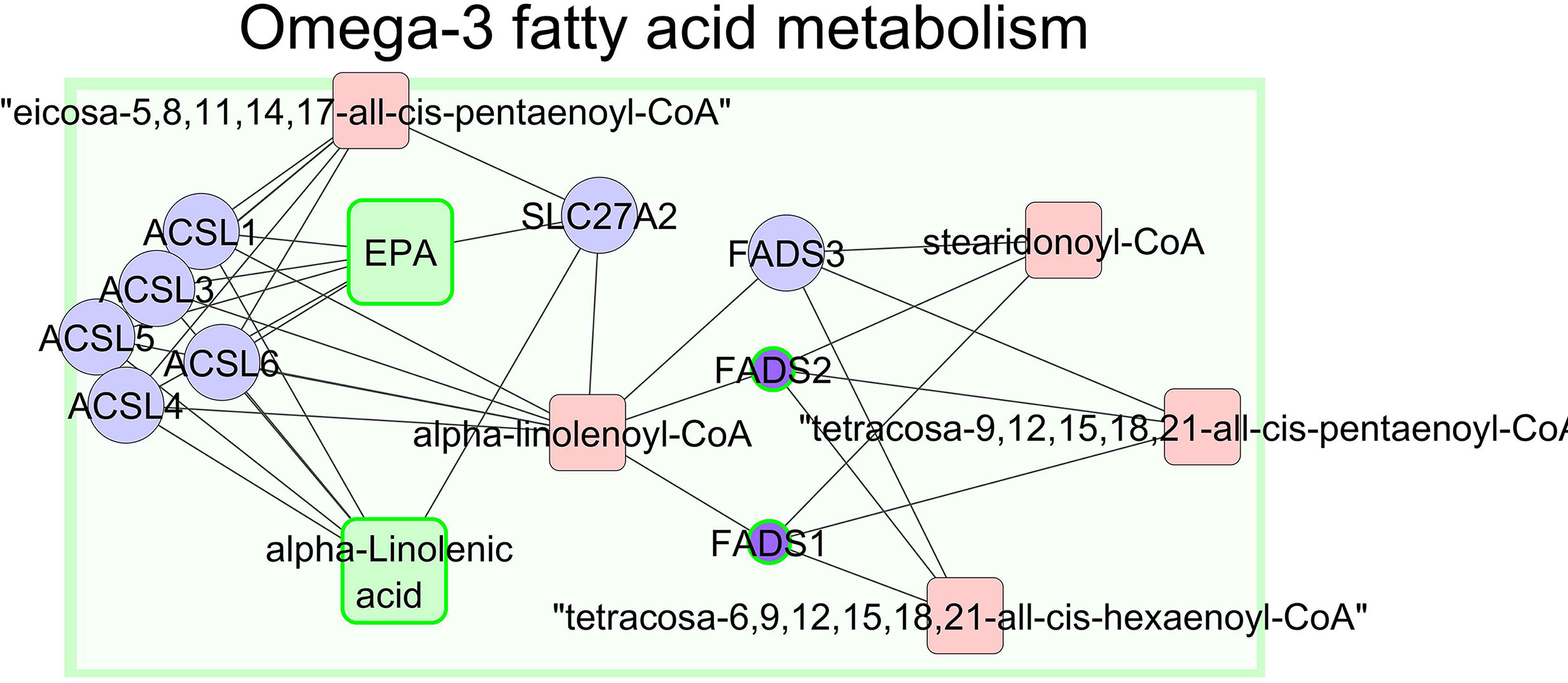 FIGURA 6 La red de metabolitos y genes implicados en el metabolismo de los ácidos grasos omega-3 en corderos ALF. Los metabolitos significativos con datos experimentales se muestran en cuadrados verdes, y los genes significativos con datos experimentales se muestran en círculos púrpuras con bordes verdes. El tamaño de los nodos representa la dirección del cambio. Un pequeño círculo púrpura con un borde verde indica genes regulados a la baja, y grandes nodos cuadrados verdes apuntan a compuestos regulados al alza.
FIGURA 6 La red de metabolitos y genes implicados en el metabolismo de los ácidos grasos omega-3 en corderos ALF. Los metabolitos significativos con datos experimentales se muestran en cuadrados verdes, y los genes significativos con datos experimentales se muestran en círculos púrpuras con bordes verdes. El tamaño de los nodos representa la dirección del cambio. Un pequeño círculo púrpura con un borde verde indica genes regulados a la baja, y grandes nodos cuadrados verdes apuntan a compuestos regulados al alza.
La nutrición es una estrategia importante para alterar la expresión génica y el perfil de ácidos grasos de la carne. Se ha informado ampliamente que los animales de pastoreo tienen un mayor contenido de la serie de AGPI n-3 en suero, hígado y músculo y una menor relación n-6:n-3, de acuerdo con el presente estudio. Curiosamente, también encontramos que la expresión de dos genes relacionados con el metabolismo de los AGPI n-3 estaba regulada a la baja en los hígados de los animales con IHA. Los genes de la desaturasa de ácidos grasos 1 (FADS1) y 2 (FADS2) codifican las desaturasas delta-5 y delta-6, respectivamente, que son enzimas limitantes de la velocidad en la síntesis de ácidos grasos omega-3 y omega-6 FA. Se ha demostrado que los FA dietéticos regulan la actividad de la desaturasa (Nakamura y Nara, 2004). La expresión génica de FADS1 y FADS2 se reduce por los AGPI en varios modelos hepáticos (Reardon et al., 2013; Cho y otros, 1999a; Cho y otros, 1999b). Además, la expresión génica de FADS1 y FADS2 se redujo por EPA y AA en adipocitos 3T3-L1 (Ralston et al., 2015). En nuestro estudio encontramos que los corderos ALF tienen una mayor cantidad de EPA en su músculo principalmente debido a su dieta. Los corderos ALF ingirieron dietas ricas en AGPI (alfalfa fresca y principalmente leche de madre), que a su vez podrían tener una expresión génica FADS1 y FADS2 regulada a la baja en el hígado. En apoyo a nuestra especulación, da Costa et al. (2014) encontraron que los altos niveles de AGPI n-3 en el hígado de ganado regulaban a la baja la expresión de los genes FADS1 y FADS2.
Los resultados encontrados en el estudio actual mostraron que ingerir dietas más ricas en AGPI n-3 podría tener efectos negativos en la síntesis de novo de AGPI n-3 por las enzimas FADS1 y FADS2. Sin embargo, la alimentación con dietas más pobres en AGPI n-3 puede promover la desaturación de ácidos grasos, lo que hace que estos dos genes sean candidatos atractivos para alterar el contenido de AGPI en la carne, al buscar polimorfismos que puedan afectar la funcionalidad y eficiencia de estas enzimas y alterar el perfil de ácidos grasos en la carne de cordero. Los SNP funcionales pueden proporcionar un recurso adicional como marcadores genéticos potenciales en los programas de mejoramiento. En este sentido, en humanos, numerosos estudios han replicado consistentemente las asociaciones entre polimorfismos en los genes FADS1 y FADS2 y la concentración de PUFA (Corella y Ordovas, 2012). En porcinos, un polimorfismo en el exón 3 del cerdo FADS2 se ha asociado con el contenido de C20:4 y grasa intramuscular (IMF) (Renaville et al., 2013; Gol et al., 2018). En vacas lecheras, Ibeagha-Awemu et al. (2014) demostraron asociaciones positivas entre tres SNP dentro de FADS1 y FADS2 con tres AGPI de leche. Mientras tanto, se han reportado resultados contradictorios en ovejas. Por ejemplo, un SNP en FADS2 se asoció significativamente con niveles intramusculares de EPA (C20: 5n-3) y DHA (C22: 6n-3) (Malau-Aduli et al., 2011), pero en un informe diferente, ningún SNP dentro de las regiones del gen FADS1 y FADS2 se asoció con los niveles de n-3 del músculo de cordero (Knight et al., 2012). Nuestro estudio apunta además a la importancia de la modulación nutricional de la expresión génica de FADS1 y FADS2 y el perfil de ácidos grasos en ovejas.
Conclusión
Los corderos de pastoreo presentaron un mayor contenido de CLA y series de AGPI n−3 y mostraron una menor relación n−6/n−3, lo que es favorable para la salud humana actual. El sistema de alimentación es el principal factor que afecta la composición de ácidos grasos y la expresión génica en el músculo LT y el hígado. La expresión génica da como resultado que los tres tejidos diferentes sugieran que el sitio principal del metabolismo lipídico es el hígado en lugar de la grasa subcutánea en corderos jóvenes de la raza Rasa Aragonesa. La expresión génica de FADS1 y FADS2 juega un papel importante en la síntesis de la serie de AGPI n-3, lo que a su vez hace que estos dos genes sean candidatos atractivos para alterar el contenido de AGPI en la carne. Se necesitarán más estudios para dilucidar los efectos del sistema de alimentación sobre la expresión de FADS1 y FADS2 en otros tejidos de interés o para buscar mutaciones o SNP funcionales que puedan usarse en el futuro como herramienta para mejorar el perfil de ácidos grasos en la carne de cordero.
Declaración de disponibilidad de datos
Los conjuntos de datos generados para este estudio se pueden encontrar en el repositorio NCBI Gene Expression Omnibus, GSE63774 (músculo LT y SF) y GSE125661 (hígado).
Declaración ética
Todos los procedimientos experimentales, incluido el cuidado de los animales y la eutanasia, se realizaron de acuerdo con las directrices de la normativa de la Unión Europea y española para el uso y cuidado de animales en investigación y fueron aprobados por el Comité de Bienestar Animal del Centro de Investigación y Tecnología Agroalimentaria (CITA) (Protocolo número 2009-01_MJT). En todos los casos, la eutanasia se realizó mediante un rayo cautivo penetrante seguido de una exanguinación inmediata.
Contribuciones del autor
LG-C, PS y MB realizaron los experimentos. MJ, MS y JC diseñaron la investigación y obtuvieron fondos para esta investigación. ED, MS, MB y JC escribieron el artículo. ED, LG-C, MS, MB, MJ, RM-H, JO y JC analizaron los datos. MJ proporcionó animales. JC tenía la responsabilidad principal del contenido final. Todos los autores contribuyeron a la discusión del manuscrito. Todos los autores leyeron y aprobaron el manuscrito final.
Financiación
Este estudio ha contado con el apoyo del Ministerio de Economía y Competitividad de España, los Fondos de Desarrollo Regional de la Unión Europea (INIA RTA2012-080-00, INIA RZP2017-00001-00) y los Fondos del Grupo de Investigación del Gobierno de Aragón (A14_17R).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Reconocimientos
Se agradece al personal de CITA de Aragón su ayuda en la recopilación de datos. Agradecemos especialmente a F. Molino y G. Ripoll por su asistencia de laboratorio
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fgene.2019.01070/full#supplementary-material
Referencias
Álvarez-Rodríguez, J., Ripoll, G., Lobón, S., Sanz, A., Blanco, M., Joy, M. (2018). La alfalfa, pero no la leche en la dieta del cordero, mejora el perfil de ácidos grasos de la carne y el contenido de α-tocoferol. Food Res. Int. 107, 708–716. doi: 10.1016/j.foodres.2018.03.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Andersen, C. L., Jensen, J. L., Ørntoft, T. F. (2004). Normalización de datos cuantitativos de PCR con transcripción inversa en tiempo real: un enfoque de estimación de varianza basado en modelos para identificar genes adecuados para la normalización, aplicado a conjuntos de datos de cáncer de vejiga y colon. Cáncer Res. 64, 5245–5250. doi: 10.1158/0008-5472.CAN-04-0496
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Barrett, T., Wilhite, S. E., Ledoux, P., Evangelista, C., Kim, I. F., Tomashevsky, M., et al. (2012). NCBI GEO: archivo para la actualización de conjuntos de datos de genómica funcional. Ácidos nucleicos res. 41, D991–D995. doi: 10.1093/nar/gks1193
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bartelds, B., Takens, J., Smid, G. B., Zammit, V. A., Prip-Buus, C., Kuipers, J. R. G., et al. (2004). Expresión de la carnitina palmitoiltransferasa I miocárdica y oxidación de ácidos grasos de cadena larga en corderos fetales y recién nacidos. Am. J. Physiol. Circ. Physiol. 286, H2243–H2248. doi: 10.1152/ajpheart.00864.2003
Bhuiyan, M. S. A., Kim, N. K., Cho, Y. M., Yoon, D., Kim, K. S., Jeon, J. T., et al. (2009). Identificación de SNPs en la familia de genes MYOD y sus asociaciones con rasgos de canal en bovinos. Livest. Sci. 126, 292–297. doi: 10.1016/J.LIVSCI.2009.05.019
Bindea, G., Mlecnik, B., Hackl, H., Charoentong, P., Tosolini, M., Kirilovsky, A., et al. (2009). ClueGO: un plug-in de Cytoscape para descifrar ontologías de genes agrupados funcionalmente y redes de anotación de vías. Bioinformática 25 (8), 1091–1093. DOI: 10.1093/Bioinformatics/BTP101
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bligh, E. G., Dyer, W. J. (1959). Un método rápido de extracción y purificación total de lípidos. Can. J. Biochem. Physiol. 37, 911–917. DOI: 10.1139/O59-099
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cassar-Malek, I., Passelaigue, F., Bernard, C., Léger, J., Hocquette, J.-F. (2007). Genes diana de pérdida de función de miostatina en músculos de fetos bovinos tardíos. BMC Genómica 8, 63. doi: 10.1186/1471-2164-8-63
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cabo, J., Alonso, R., Mata, P. (2012). Ácidos grasos omega-3 y presión arterial. Fr. J. Nutr. 107, S195–S200. doi: 10.1017/S0007114512001584
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cho, H. P., Nakamura, M., Clarke, S. D. (1999a). Clonación, expresión y regulación de ácidos grasos de la desaturasa delta-5 humana. J. Biol. Chem. 274, 37335–37339. DOI: 10.1074/JBC.274.52.37335
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cho, H. P., Nakamura, M. T., Clarke, S. D. (1999b). Clonación, expresión y regulación nutricional de la desaturasa delta-6 de mamíferos. J. Biol. Chem. 274, 471–477. DOI: 10.1074/JBC.274.1.471
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chu, M., Yan, P., Liang, C. N., Guo, X., Pei, J., Ding, X. Z., et al. (2012). Asociación entre el polimorfismo del gen MyoD1 y los rasgos de crecimiento en Yak. China Anim. Husb. Vet. Med 29, 111–113.
Corella, D., Ordovás, J. M. (2012). Interacciones entre los ácidos grasos n-3 dietéticos y las variantes genéticas y el riesgo de enfermedad. Fr. J. Nutr. 107 Suppl 2(0 2), S271–S283. doi: 10.1017/S0007114512001651
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cui, K., Wang, B., Ma, T., Si, B. W., Zhang, N. F., Tu, Y., et al. (2018). Efectos de la restricción de proteínas en la dieta seguida de realimentación sobre el rendimiento del crecimiento y las alteraciones del transcriptoma hepático del cordero. Informes científicos. 8, 15185. DOI: 10.1038/S41598-018-33407-W
da Costa, A. S., Bessa, R. J., Pires, V. M., Rolo, E. A., Pinto, R. M., Andrade Fontes, C. M., et al. (2014). ¿El metabolismo lipídico hepático del ganado vacuno de carne está influenciado por la raza y el nivel de ensilaje dietético? BMC Vet. 10, 65. doi: 10.1186/1746-6148-10-65
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dervishi, E., Joy, M., Alvarez-Rodriguez, J., Serrano, M., Calvo, J. H. (2012). El tipo de forraje (pastoreo versus pasto de heno) alimentado a las ovejas y el sexo del cordero afectan el perfil de ácidos grasos y la expresión génica lipogénica en el músculo longissimus de los corderos lactantes1. J. Anim. 90, 54–66. DOI: 10.2527/jas.2011-4057
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dervishi, E., Serrano, C., Joy, M., Serrano, M., Rodellar, C., Calvo, J. H. (2010). Efecto del sistema de alimentación sobre la composición de ácidos grasos, expresión de los genes alfa, gamma y proteína 9 de unión al elemento regulador de éster Δ1-desaturasa, proliferador de peroxisomas en el músculo semitendinoso de corderos ligeros de R. BMC Vet. 6, 40. doi: 10.1186/1746-6148-6-40
Dervishi, E., Serrano, C., Joy, M., Serrano, M., Rodellar, C., Calvo, J. H. (2011). The effect of feeding system in the expression of genes related with fat metabolism in semitendinous muscle in sheep. Meat Sci. 89, 91–97. doi: 10.1016/j.meatsci.2011.04.003
Du, X. H., Gan, Q. F., Yuan, Z. R., Gao, X., Zhang, L. P., Gao, H. J., et al. (2013). Polymorphism of MyoD1 and Myf6 genes and associations with carcass and meat quality traits in beef cattle. Genet. Mol. Res. 12, 6708–6717. doi: 10.4238/2013.December.13.4
Elgersma, A. (2015). Grazing increases the unsaturated fatty acid concentration of milk from grass-fed cows: a review of the contributing factors, challenges and future perspectives. Eur. J. Lipid Sci. Technol. 117, 1345–1369. doi: 10.1002/ejlt.201400469
Elkina, Y., von Haehling, S., Anker, S. D., Springer, J. (2011). The role of myostatin in muscle wasting: an overview. J. Cachexia. Sarcopenia Muscle 2, 143–151. doi: 10.1007/s13539-011-0035-5
Fisher, A. V., Enser, M., Richardson, R. I., Wood, J. D., Nute, G. R., Kurt, E., et al. (2000). Fatty acid composition and eating quality of lamb types derived from four diverse breed × production systems. Meat Sci. 55, 141–147.
Gol, S., Pena, R. N., Rothschild, M. F., Tor, M., Estany, J. (2018). A polymorphism in the fatty acid desaturase-2 gene is associated with the arachidonic acid metabolism in pigs. Sci. Rep. 8. doi: 10.1038/s41598-018-32710-w
González-Calvo, L., Dervishi, E., Joy, M., Sarto, P., Martin-Hernandez, R., Serrano, M., et al. (2017). Genome-wide expression profiling in muscle and subcutaneous fat of lambs in response to the intake of concentrate supplemented with vitamin E. BMC Genomics 18, 92. doi: 10.1186/s12864-016-3405-8
González-Calvo, L., Joy, M., Alberti, C., Ripoll, G., Molino, F., Serrano, M., et al. (2014). Effect of finishing period length with α-tocopherol supplementation on the expression of vitamin E-related genes in the muscle and subcutaneous fat of light lambs. Gene 552, 225–233. doi: 10.1016/j.gene.2014.09.037
González-Calvo, L., Joy, M., Blanco, M., Dervishi, E., Molino, F., Sarto, P., et al. (2015a). Effect of vitamin E supplementation or alfalfa grazing on fatty acid composition and expression of genes related to lipid metabolism in lambs1. J. Anim. Sci. 93, 3044–3054. doi: 10.2527/jas.2014-8758
González-Calvo, L., Ripoll, G., Molino, F., Calvo, J. H., Joy, M. (2015b). The relationship between muscle α -tocopherol concentration and meat oxidation in light lambs fed vitamin E supplements prior to slaughter. J. Sci. Food Agric. 95, 103–110. doi: 10.1002/jsfa.6688
Huang, D. W., Sherman, B. T., Lempicki, R. A. (2009). Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nat. Protoc. 4, 44–57. doi: 10.1038/nprot.2008.211
Hunt, M. (1980). Meat color measurements. Proceedings of 33th Reciprocal Meat Conference (1980). Reciprocal Meat Conf. Proc. 33, 41–46. Avalaible at: https://meatscience.org/docs/default-source/publications-resources/rmc/1980/meat-color-measurements.pdf?sfvrsn=ae05bbb3_2. [Accessed October 18, 2019].
Ibeagha-Awemu, E. M., Akwanji, K. A., Beaudoin, F., Zhao, X. (2014). Associations between variants of FADS genes and omega-3 and omega-6 milk fatty acids of Canadian Holstein cows. BMC Genet. 15, 25. doi: 10.1186/1471-2156-15-25
Ibeagha-Awemu, E. M., Kgwatalala, P., Zhao, X. (2008). A critical analysis of production-associated DNA polymorphisms in the genes of cattle, goat, sheep, and pig. Mamm. Genome 19, 591–617. doi: 10.1007/s00335-008-9141-x
Joy, M., Ripoll, G., Molino, F., Dervishi, E., Álvarez-Rodriguez, J. (2012). Influence of the type of forage supplied to ewes in pre- and post-partum periods on the meat fatty acids of suckling lambs. Meat Sci. 90, 775–782. doi: 10.1016/J.MEATSCI.2011.11.013
Ji, S., Losinski, R. L., Cornelius, S. G., Frank, G. R., Willis, G. M., Gerrard, D. E., et al. (1998). Myostatin expression in porcine tissues: tissue specificity and developmental and postnatal regulation. Am. J. Physiol. Integr. Comp. Physiol. 275, R1265–R1273. doi: 10.1152/ajpregu.1998.275.4.R1265
Karnovsky, A., Weymouth, T., Hull, T., Tarcea, V. G., Scardoni, G., Laudanna, C., et al. (2012). Metscape 2 bioinformatics tool for the analysis and visualization of metabolomics and gene expression data. Bioinformatics 28, 373–380. doi: 10.1093/bioinformatics/btr661
Kasapidou, E., Wood, J. D., Richardson, R. I., Sinclair, L. A., Wilkinson, R. G., Enser, M. (2012). Effect of vitamin E supplementation and diet on fatty acid composition and on meat colour and lipid oxidation of lamb leg steaks displayed in modified atmosphere packs. Meat Sci. 90, 908–916. doi: 10.1016/j.meatsci.2011.11.031
Kijas, J. W., McCulloch, R., Edwards, J. E. H., Oddy, V. H., Lee, S. H., van der Werf, J. (2007). Evidence for multiple alleles effecting muscling and fatness at the Ovine GDF8 locus. BMC Genet. 8, 80. doi: 10.1186/1471-2156-8-80
Kitessa, S., Liu, S., Briegel, J., Pethick, D., Gardner, G., Ferguson, M., et al. (2010). Effects of intensive or pasture finishing in spring and linseed supplementation in autumn on the omega-3 content of lamb meat and its carcass distribution. Anim. Prod. Sci. 50, 130. doi: 10.1071/AN09095
Kitzmann, M., Carnac, G., Vandromme, M., Primig, M., Lamb, N. J., Fernandez, A. (1998). The muscle regulatory factors MyoD and myf-5 undergo distinct cell cycle-specific expression in muscle cells. J. Cell Biol. 142, 1447–1459.
Knight, M. I., Daetwyler, H. D., Hayes, B. J., Hayden, M. J., Ball, A. J., Pethick, D. W., et al. (2012). Descubrimiento y asociación de rasgos de polimorfismos de nucleótido único de regiones genéticas de influencia en la ternura de la carne y el contenido de ácidos grasos omega-3 de cadena larga en cordero australiano. Anim. Prod. Sci. 52, 591. doi: 10.1071/AN11229
Laville, E., Bouix, J., Sayd, T., Elsen, J. M., Larzul, C., et al. (2004). Efectos de un locus de rasgo cuantitativo para la hipertrofia muscular de ovejas belgas Texel sobre la conformación de la canal y la musculatura. J. Anim. 82, 3128–3137. doi: 10.2527/2004.82113128x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Lee, E. A., Kim, J. M., Lim, K. S., Ryu, Y. C., Jeon, W. M., Hong, K. C. (2012). Efectos de la variación en el gen MYOD1 porcino sobre las características de la fibra muscular, la producción de carne magra y los rasgos de calidad de la carne. Carne Sci. 92, 36–43. doi: 10.1016/J.MEATSCI.2012.03.018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Lehnen, T. E., da Silva, M. R., Camacho, A., Marcadenti, A., Lehnen, A. M. (2015). Una revisión sobre los efectos del ácido graso linoleico conjugado (CLA) sobre la composición corporal y el metabolismo energético. J. Int. Soc. Sports Nutr. 12, 36. DOI: 10.1186/S12970-015-0097-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Li, K., Sinclair, A. J., Zhao, F., Li, D. (2018). Ácidos grasos poco comunes y salud cardiometabólica. Nutrientes 10 (10), 1559. doi: 10.3390/nu10101559
Lines, D. S., Pitchford, W. S., Kruk, Z. A., Bottema, C. D. K. (2009). La variante de miostatina F94L de Limousin afecta la sensibilidad semitendinosa. Carne Sci. 81, 126–131. doi: 10.1016/j.meatsci.2008.07.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Liu, J., Ma, D. (2014). El papel de los ácidos grasos poliinsaturados n-3 en la prevención y el tratamiento del cáncer de mama. Nutrientes 6, 5184–5223. doi: 10.3390/nu6115184
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Lôbo, A. M. B. O., Guimarães, S. E. F., Paiva, S. R., Cardoso, F. F., Silva, F. F., Júnior, G. A. F., et al. (2012). Genes transcritos diferencialmente en músculo esquelético de corderos. Livest. Sci. 150, 31–41. doi: 10.1016/J.LIVSCI.2012.07.027
Malau-Aduli, A. E. O., Bignell, C. W., McCulloch, R., Nichols, P. D., Kijas, J. W., (2011). Asociación genética del marcador molecular polimórfico polimórfico de ácido graso desaturasa delta-seis y ácidos grasos omega-3 de cadena larga muscular en cordero australiano. Actas del 57º Congreso Internacional de Ciencia y Tecnología de la Carne. págs. 141–144. Gante, Bélgica. Disponible en: https://isgcdata.agresearch.co.nz/ [consultado el 25 de enero de 2019].
Nakamura, M. T., Nara, T. Y. (2004). Estructura, función y regulación dietética de las desaturasas Δ6, Δ5 y Δ9. Nutr. 24, 345–376. doi: 10.1146/annurev.nutr.24.121803.063211
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Napolitano, F., Cifuni, G. F., Pacelli, C., Riviezzi, A. M., Girolami, A. (2002). Efecto de la cría artificial sobre el bienestar del cordero y la calidad de la carne. Carne Sci. 60, 307–315.
Ngapo, T. M., Berge, P., Culioli, J., Dransfield, E., De Smet, S., Claeys, E. (2002). Reticulación perimisial de colágeno y ternura de la carne en ganado bovino de doble musculatura azul belga. Carne Sci. 61, 91–102.
Nudda, A., McGuire, M. A., Battacone, G., Pulina, G. (2005). Variación estacional del ácido linoleico conjugado y ácido vaccénico en la grasa láctea de oveja y su transferencia al queso y ricotta. J. Dairy Sci. 88, 1311–1319. DOI: 10.3168/JDS. S0022-0302(05)72797-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Pfalzgraf, A., Frigg, M., Steinhart, H. (1995). Contenido de alfa-tocoferol y oxidación lipídica en músculo de cerdo y tejido adiposo durante el almacenamiento. J. Agric. Food Chem. 43, 1339–1342. DOI: 10.1021/JF00053A039
Ponnampalam, E. N., Plozza, T., Kerr, M. G., Linden, N., Mitchell, M., Bekhit, A. E.-D. A., et al. (2017). Interacción de la dieta y el largo período de envejecimiento en la oxidación lipídica y la estabilidad del color de la carne de cordero. Carne Sci. 129, 43–49. doi: 10.1016/j.meatsci.2017.02.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ralston, J. C., Matravadia, S., Gaudio, N., Holloway, G. P., Mutch, D. M. (2015). Regulación de ácidos grasos poliinsaturados de la expresión y función de los adipocitos FADS1 y FADS2. Obesidad (Silver Spring). 23, 725–728. doi: 10.1002/oby.21035
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Reardon, H. T., Hsieh, A. T., Park, W. J., Kothapalli, K. S. D., Anthony, J. C., Nathanielsz, P. W., et al. (2013). Los ácidos grasos poliinsaturados de cadena larga en la dieta regulan al alza la expresión de las transcripciones FADS3. Prostaglandinas. Leukot. Essent. Ácidos grasos 88, 15–19. doi: 10.1016/j.plefa.2012.02.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Renaville, B., Prandi, A., Fan, B., Sepulcri, A., Rothschild, M. F., Piasentier, E. (2013). Asociaciones de marcadores genéticos candidatos con perfiles de ácidos grasos en cerdos pesados. Carne Sci. 93, 495–500. doi: 10.1016/J.MEATSCI.2012.11.019
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ripoll, G., González-Calvo, L., Molino, F., Calvo, J. H., Joy, M. (2013). Efectos de la duración del período de acabado con suplementos de vitamina E y pastoreo de alfalfa sobre el color de la canal y la evolución del color de la carne y la oxidación lipídica de corderos ligeros. Carne Sci. 93, 906–913. doi: 10.1016/j.meatsci.2012.09.017
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Scollan, N. D., Dannenberger, D., Nuernberg, K., Richardson, I., MacKintosh, S., Hocquette, J.-F., et al. (2014). Mejorar el valor nutricional y para la salud de los lípidos de la carne de vacuno y su relación con la calidad de la carne. Carne Sci. 97, 384–394. doi: 10.1016/j.meatsci.2014.02.015
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Shannon, P., Markiel, A., Ozier, O., Baliga, N. S., Wang, J. T., Ramage, D., et al.. (2003). Cytoscape: un entorno de software para modelos integrados de redes de interacción biomolecular. Genoma Res. 13, 2498–2504. DOI: 10.1101/GR.1239303
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Simopoulos, A. P. (2008). La importancia de la relación ácidos grasos omega-6/omega-3 en las enfermedades cardiovasculares y otras enfermedades crónicas. Exp. Biol. Med. 233, 674–688. doi: 10.3181/0711-MR-311
Steibel, J. P., Poletto, R., Coussens, P. M., Rosa, G. J. M. (2009). Un marco de modelo mixto lineal potente y flexible para el análisis de datos de cuantificación relativa de RT-PCR. Genómica 94, 146-152. doi: 10.1016/j.ygeno.2009.04.008
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Sukhija, P. S., Palmquist, D. L. (1988). Método rápido para la determinación del contenido total de ácidos grasos y la composición de los piensos y las heces. J. Agric. Food Chem. 36, 1202–1206. DOI: 10.1021/JF00084A019
Takeuchi, H., Sugano, M. (2017). Ácidos grasos trans industriales y colesterol sérico: el nivel dietético permitido. J. Lipids 2017, 9751756. doi: 10.1155/2017/9751756
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Terry, E. E., Zhang, X., Hoffmann, C., Hughes, L. D., Lewis, S. A., Li, J., et al. (2018). El perfil transcripcional revela una extraordinaria diversidad entre los tejidos del músculo esquelético. Elife 7. eLife. 7, e34613. doi: 10.7554/eLife.34613
Tizioto, P., Gasparin, G., Coutinho, L., Mourao, G., Mudadu, M., Souza, M., et al. (2014). MyoD1 expression levels affect meat tenderness in Nellore beef cattle. Proceedings of 10th World Congress of Genetics Applied to Livestock Production. pp. 601. Vancouver. Canada. Avalaible at: http://www.alice.cnptia.embrapa.br/alice/handle/doc/998063 [Accessed October 18, 2019].
Traber, M. G., Atkinson, J. (2007). Vitamin E, antioxidant and nothing more. Free Radic. Biol. Med. 43, 4–15. doi: 10.1016/j.freeradbiomed.2007.03.024
Vasta, V., Pagano, R. I., Luciano, G., Scerra, M., Caparra, P., Foti, F., et al. (2012). Efecto del pastoreo matutino vs. vespertino sobre la composición intramuscular de ácidos grasos en cordero. Carne Sci. 90, 93–98. doi: 10.1016/j.meatsci.2011.06.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Valvo, M. A., Lanza, M., Bella, M., Fasone, V., Scerra, M., Biondi, L., et al. (2005). Efecto del sistema de alimentación de oveja (pasto v. concentrado) sobre los ácidos grasos intramusculares de corderos criados exclusivamente con leche materna. Anim. Sci. 81, 431–436. doi: 10.1079/ASC50480431
Wang, X., Quinn, P. J. (2000). La ubicación y función de la vitamina E en las membranas (revisión). Mol. Membr. Biol. 17, 143–156. DOI: 10.1080/09687680010000311
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Xia, J., Psychogios, N., Young, N., Wishart, D. S. (2009). MetaboAnalyst: un servidor web para el análisis e interpretación de datos metabolómicos. Ácidos nucleicos res. 37, W652–W660. doi: 10.1093/nar/gkp356
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhang, X. Q., Jin, Y. M., Badgery, W. B., Tana, (2017). Selección de la dieta y depósito de ácidos grasos poliinsaturados n-3 en corderos afectados por el tiempo restringido en el pasto. 7, 15641. DOI: 10.1038/S41598-017-15875-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhu, Y., Bo, Y., Liu, Y. (2019). Grasa total en la dieta, ingesta de ácidos grasos y riesgo de enfermedad cardiovascular: un metanálisis dosis-respuesta de estudios de cohortes. Lipids Health Dis. 18, 91. DOI: 10.1186/S12944-019-1035-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cita: Dervishi E, González-Calvo L, Blanco M, Joy M, Sarto P, Martin-Hernandez R, Ordovás JM, Serrano M y Calvo JH (2019) Expresión génica y perfil de ácidos grasos en músculo Longissimus thoracis, grasa subcutánea e hígado de corderos ligeros en respuesta al concentrado o al pastoreo de alfalfa. Frente. Jineta. 10:1070. doi: 10.3389/fgene.2019.01070
Recibido: 09 Abril 2019; Aprobado: 04 Octubre 2019;
Publicado: 31 octubre 2019.
Editado por:
Robert J. Schaefer, Universidad de Minnesota Twin Cities, Estados Unidos
Revisado por:
Andrea Serra, Universidad de Pisa, Italia
Paula Alexandra Lopes, Universidad de Lisboa, Portugal
Copyright © 2019 Dervishi, González-Calvo, Blanco, Joy, Sarto, Martin-Hernandez, Ordovás, Serrano y Calvo. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Jorge H. Calvo, jhcalvo@aragon.es
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía