FADD promueve la producción de interferón tipo I para suprimir la infección por el virus del síndrome respiratorio y reproductivo porcino
FADD promueve la producción de interferón tipo I para suprimir la infección por el virus del síndrome respiratorio y reproductivo porcino
 Xiaobo Chang1,2
Xiaobo Chang1,2  Mengqi Wang2
Mengqi Wang2  Zhaopeng Li2
Zhaopeng Li2  Lei Wang2
Lei Wang2  Pegando a Zhang3
Pegando a Zhang3  Yafei Chang1,2*
Yafei Chang1,2*  Jianhe Hu1,2*
Jianhe Hu1,2*- 1Base de Prácticas de Innovación Postdoctoral, Facultad de Ciencia Animal y Medicina Veterinaria, Instituto de Ciencia y Tecnología de Henan, Xinxiang, China
- 2Facultad de Ciencia Animal y Medicina Veterinaria, Instituto de Ciencia y Tecnología de Henan, Xinxiang, China
- 3Facultad de Medicina Veterinaria, Universidad Agrícola de Henan, Zhengzhou, China
El síndrome respiratorio y reproductivo porcino (PRRS) es una enfermedad infecciosa animal epidémica en todo el mundo, que causa enormes pérdidas económicas a la industria porcina mundial. El dominio de muerte asociado a Fas (FADD) se informó previamente que es una proteína adaptadora que funciona en la transferencia de las señales apoptóticas reguladas por los receptores de muerte. En el presente estudio, desentrañamos su papel no identificado en la promoción de la producción de interferón tipo I (IFN) durante la infección por el virus del PRRS (PRRSV). Identificamos que FADD inhibió la infección por PRRSV a través de la promoción de la transcripción de IFN tipo I. La sobreexpresión de FADD suprimió la replicación del PRRSV, mientras que la eliminación de FADD aumentó el título viral y la expresión de la proteína de la nucleocápside. Mecánicamente, FADD promovió la producción de IFN-β mediada por la proteína de señalización antiviral mitocondrial (MAVS) y algunos genes estimulados por IFN (ISG). Además, FADD ejerció efectos anti-PRRSV de una manera dependiente de MAVS y aumentó la señalización de IFN tipo I durante la infección por PRRSV. Este estudio destaca la importancia de FADD en la replicación del PRRSV, lo que puede tener implicaciones para el control futuro del PRRS.
Introducción
Síndrome reproductivo y respiratorio porcino (PRRS), una importante enfermedad epidémica, caracterizada por un fallo reproductivo tardío en la gestación en las cerdas y síntomas respiratorios generales en cerdos de todas las edades y sexos, lo que causa enormes pérdidas económicas a la industria porcina (1-4). El PRRSV, el agente causal del PRRS, es un virus envuelto con un genoma de ARN de cadena positiva de 15 kb que contiene al menos 10 marcos de lectura abiertos (ORF), perteneciente a la familia Arteriviridae, orden Nidovirales (5-7). El PRRSV se divide en PRRSV-1 y PRRSV-2, y las cepas de PRRSV-2 son predominantemente prevalentes en China (8). Además, la infección por PRRSV puede desencadenar una respuesta inmunitaria innata del huésped (9).
Los IFN de tipo I (IFN-α/β) desempeñan un papel fundamental en la respuesta antiviral. Tras la infección por virus, los ácidos nucleicos virales como patrones moleculares asociados a patógenos (PAMP) pueden ser detectados por los receptores celulares, como los receptores similares al gen I (RIG-I) del sensor de ARN inducible por ácido retinoico (RIG-I), que reclutan la señalización antiviral mitocondrial (MAVS), lo que conduce a la fosforilación dependiente de TBK1 de IRF3 y a la transcripción de IFN e ISG de tipo I (10, 11). Además, se ha confirmado que los IFN de tipo I y numerosos ISG tienen efectos antivirales contra el PRRSV (12-15).
La FADD, descrita inicialmente como una proteína adaptadora clave, interactuó con los receptores de muerte (DR), como el FAS y el receptor 1 del factor de necrosis tumoral (TNF-R1), lo que condujo al reclutamiento y la activación de la procaspasa-8 y la consiguiente apoptosis de la célula (16, 17). Sin embargo, en los últimos años, se ha informado que la FADD está involucrada en una variedad de procesos no apoptóticos, incluida la progresión del ciclo celular, el desarrollo embrionario, la hematopoyesis y la progresión y supervivencia del ciclo celular de los linfocitos (18, 19). Además, FADD se ha convertido en un actor de la inmunidad innata y la inflamación. En particular, FADD y RIPK1 podrían formar el inteosoma en respuesta al ARN viral reconocido por los receptores intracelulares, regulando así la activación mediada por TBK1 de la producción de IRF3 e IFN-β (17, 20). Y varios estudios han demostrado que la replicación de los virus de ARN, como el VSV, el ZIKV, el virus de la influenza y el virus de la encefalomiocarditis (EMCV), se facilita en gran medida en fibroblastos humanos y de ratón deficientes en FADD, lo que destaca la importancia de FADD en la respuesta inmune antiviral (21-24). Sin embargo, no se han dilucidado las funciones y el mecanismo de FADD en respuesta al PRRSV.
En este estudio, encontramos que la sobreexpresión de FADD restringió la replicación del PRRSV, mientras que el silencio de FADD promovió la replicación del PRRSV. Además, FADD reguló positivamente la vía de señalización de IFN tipo I mediada por MAVS. Más importante aún, MAVS fue esencial para que FADD reprimiera la replicación del PRRSV, y la actividad antiviral de FADD dependía de la transcripción de IFN tipo I. Estos hallazgos pueden contribuir a la comprensión de las proteínas celulares en el papel de la regulación de la replicación del PRRSV.
Materiales y métodos
Células y virus
Las células MARC-145 (una línea celular permisiva del PRRSV subclonada a partir de MA-104) y las células HEK-293T se cultivaron en el medio de Eagle modificado de Dulbecco (DMEM, Solarbio) suplementado con suero fetal bovino al 10% (FBS, Gibco), 100 U/mL de penicilina (Gibco) y 100 μg/mL de estreptomicina (Gibco) a 37 °C en una atmósfera humidificada de 5% de CO2. La cepa BJ-4 del PRRSV (GenBank No. AF331831) almacenado en el Laboratorio Clave Provincial de Inmunología Animal de Henan fue un amable regalo del Prof. Hanchun Yang (Universidad Agrícola de China).
Anticuerpos y reactivos
El anticuerpo anti-FADD de conejo y el anticuerpo anti-MAVS de conejo se compraron a Proteintech. El anticuerpo monoclonal anti-Flag M2 de ratón y el anticuerpo monoclonal anti-c-Myc de ratón se compraron a Sigma-Aldrich. El anticuerpo de la proteína N del PRRSV se compró a GeneTex. El anticuerpo monoclonal anti-β-actina de ratón se compró a GenScript. Los anticuerpos secundarios conjugados con HRP se compraron a Proteintech. El ácido poliinosínico-policitidílico (polyI:C) se compró a Sigma-Aldrich. El reactivo de transfección Lipofectamine 2000 y el reactivo de transfección Lipofectamine RNAiMAX se compraron a Invitrogen.
Construcción de plásmidos
El ADNc de FADD se clonó en pCMV-Myc, denominado FADD-Myc. Los genes de RIG-I, MDA5, MAVS, TBK1 e IRF3 se clonaron en pCMV-Flag, que se denominaron RIG-I-Flag, MDA5-Flag, MAVS-Flag, TBK1-Flag e IRF3-Flag, respectivamente. El plásmido indicador de luciferasa IFN-β (IFN-β-Luc), se ha construido como se describió anteriormente (25). El pRL-TK (Promega), que contiene un gen reportero Renilla-luciferasa, se utilizó como control endógeno en el sistema de ensayo de doble indicador de luciferasa. Todas las imprimaciones utilizadas se enumeran en la Tabla 1.
Interferencia de ARN
GenePharma diseñó y sintetizó pequeños ARN interferentes (siRNA) dirigidos a FADD (siFADD: 5′-GGAAGAACGCAGAGAAGAATT-3′), MAVS (siMAVS: 5′-GCAUCUCUUCAGUACCCUUTT-3′) y el siRNA de control no diana (siNC: 5′-UUCUCCGAACGUGUCACGUTT-3′). Las células MARC-145 se transfectaron con siRNA de 50 nM utilizando el reactivo de transfección Lipofectamine RNAiMAX. Veinticuatro horas después, las células estaban infectadas con PRRSV con una multiplicidad de infección (MOI) de 0,1. Y luego, las células o los sobrenadantes se cosecharon en los tiempos indicados (24, 36 y 48 h) para experimentos posteriores.
Análisis cuantitativo de PCR en tiempo real
Los ARN totales se extrajeron utilizando el kit de aislamiento de ARN total de células/tejidos (Vazyme), y los ADNc de transcripción inversa se prepararon a partir de ARN totales utilizando el HiScript III All-in-one RT SuperMix Perfect for qPCR (Vazyme). Los ADNc se amplificaron mediante PCR cuantitativa en tiempo real (qPCR) utilizando ChamQ Universal SYBR qPCR Master Mix (Vazyme). El nivel relativo de expresión génica del ARNm se evaluó mediante el método 2®−ΔΔCT método con ARNm de gliceraldehído-3-fosfato deshidrogenasa (GAPDH) como control endógeno. Los cebadores para la qPCR se enumeran en la Tabla 2.
Ensayos reporteros de luciferasa dual
Los ensayos de luciferasa fueron realizados por el Sistema de Ensayo Reportero de Luciferasa Dual de acuerdo con las instrucciones de Promega. En resumen, las células MARC-145 o las células HEK-293T se transfectaron con plásmidos de expresión indicada, plásmido indicador de luciferasa IFN-β y plásmido indicador de luciferasa renilla pRL-TK mediante el uso del reactivo de transfección Lipofectamine 2000 (Invitrogen). A 36 hpt, las células se transfectaron con poliI:C (10 μg/mL) durante 12 h. PolyI:C, el análogo sintético del ARN bicatenario (dsRNA), se utiliza experimentalmente para desencadenar la producción de IFN tipo I (26). A continuación, las células transfectadas se lisaron en tampón de lisis pasiva y se sometieron a la medición de la actividad de la luciferasa utilizando el sistema de ensayo reportero de luciferasa dual (Promega).®
Análisis de Western blot
Las células MARC-145 tratadas o simuladas se recolectaron y lavaron tres veces con PBS frío, y luego se lisaron en tampón RIPA (Sigma-Aldrich) que contenía un cóctel inhibidor de la proteasa (Roche). La concentración de proteínas se determinó utilizando el kit de ensayo de proteínas Micro BCA (Thermo Fisher Scientific). Se analizaron cantidades iguales de lisados de proteínas con tampón de carga SDS-PAGE en geles SDS-PAGE al 10% y se transfirieron a membranas de PVDF de 0,22 μm. A continuación, las membranas se bloquearon en leche desnatada al 5% y se incubaron con el anticuerpo primario indicado durante la noche a 4 °C, seguido de la incubación con anticuerpos secundarios conjugados con HRP durante 2 h a temperatura ambiente. Posteriormente, se detectaron señales luminiscentes con SuperSignal™ sustratos HRP quimioluminiscentes (Thermo Fisher Scientific). Los niveles relativos de las proteínas diana normalizadas a β-actina se analizaron utilizando el software Image J.
Titulación de virus
Los títulos de virus se determinaron de acuerdo con un informe previo (27). Brevemente, las células MARC-145, cultivadas en placas de 96 pocillos, se infectaron con una dilución en serie de muestras 10 veces. Después de 1 h de incubación a 37 °C, los sobrenadantes se reemplazaron con DMEM fresco que contenía 2% de FBS. Cinco días después de la infección, el efecto citopático (CPE) caracterizado por aglomeración y contracción de las células fue visible en las células MARC-145 y los títulos virales se calcularon mediante una dosis infecciosa de cultivo de tejidos al 50% (TCID50) según el método de Reed-Muench (28).
Análisis estadístico
Todos los experimentos se repitieron de forma independiente al menos tres veces. Los datos experimentales se presentaron como media de grupo y desviación estándar (DE) y se analizaron mediante la prueba t de Student de dos colas no apareadas con GraphPad. Los asteriscos de las figuras indican diferencias significativas (p < 0,05, p < 0,01 y p < 0,001).******
Resultados
La sobreexpresión de FADD suprime la replicación del PRRSV
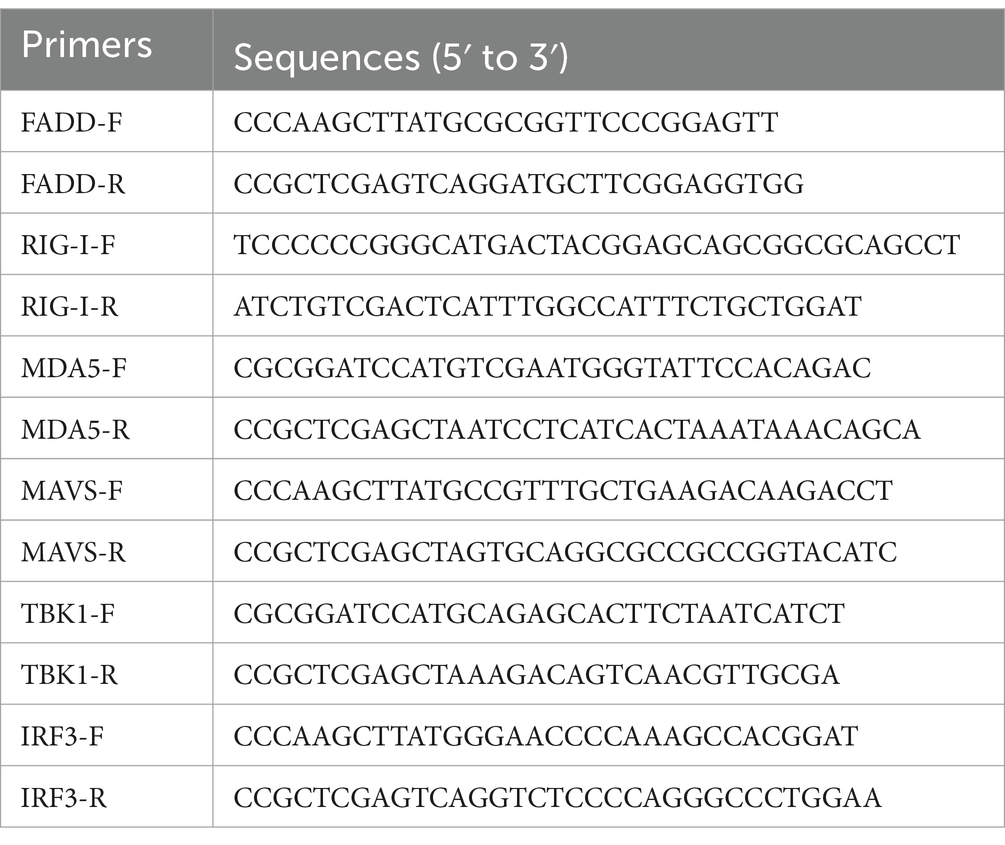
En el estudio anterior, informamos que FADD es un objetivo de miR-382-5p aumentado durante la infección por PRRSV (29). Para verificar la importancia biológica de FADD durante la infección por PRRSV, examinamos el efecto de FADD en la replicación del PRRSV. FADD se sobreexpresó en las células MARC-145 por transfección con FADD-Myc. Como se muestra en la Figura 1, cuando se sobreexpresó FADD, los niveles de ARNm del gen N del PRRSV fueron más bajos que los de las células de control a las 24-48 h (Figura 1A). Posteriormente, se midieron los títulos virales de los sobrenadantes de las células FADD sobreexpresadas o control. La sobreexpresión de FADD redujo la liberación de PRRSV, como lo muestra TCID50 (Figura 1B). Además, la sobreexpresión de FADD disminuyó notablemente la abundancia de la proteína N del PRRSV (Figuras 1C-E), lo que indicó que la sobreexpresión de FADD mostró efectos inhibidores sobre el PRRSV.
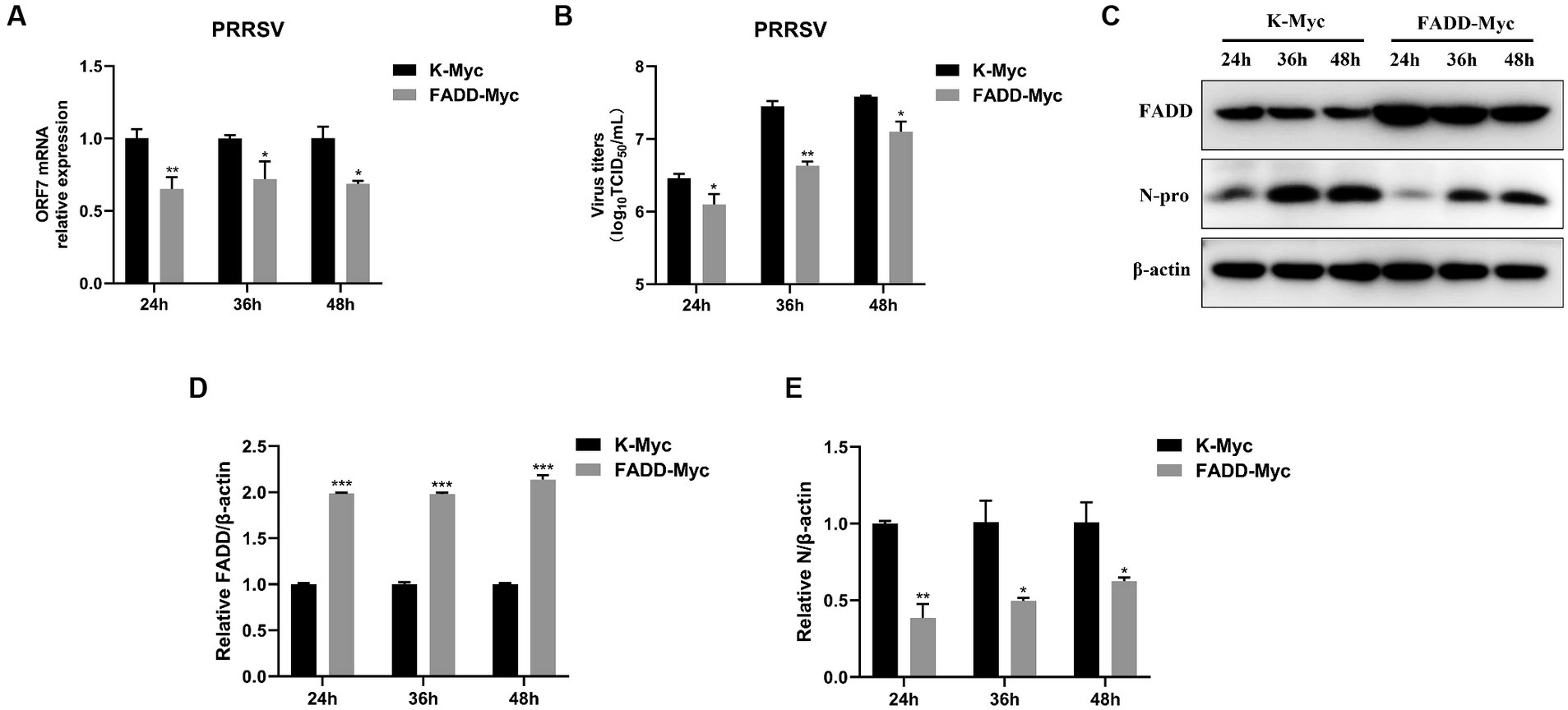 Figura 1. La sobreexpresión de FADD inhibe la replicación del PRRSV. Las células MARC-145 se transfectaron con FADD-Myc o vector control durante 24 h, y luego se infectaron con PRRSV a un MOI de 0,1 durante otras 24, 36 y 48 h. (A) Los niveles de ARN del PRRSV se evaluaron mediante qPCR. (B) Los títulos virales en el sobrenadante de cultivo celular fueron ensayados y presentados por TCID50. (C) Los niveles de expresión de FADD y proteína N del PRRSV se analizaron mediante western blot. (D,E) Las proporciones de intensidad relativa de FADD y proteína N también se mostraron en esta figura utilizando el software Image J. Todos los experimentos se repitieron al menos tres veces con resultados similares. p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Figura 1. La sobreexpresión de FADD inhibe la replicación del PRRSV. Las células MARC-145 se transfectaron con FADD-Myc o vector control durante 24 h, y luego se infectaron con PRRSV a un MOI de 0,1 durante otras 24, 36 y 48 h. (A) Los niveles de ARN del PRRSV se evaluaron mediante qPCR. (B) Los títulos virales en el sobrenadante de cultivo celular fueron ensayados y presentados por TCID50. (C) Los niveles de expresión de FADD y proteína N del PRRSV se analizaron mediante western blot. (D,E) Las proporciones de intensidad relativa de FADD y proteína N también se mostraron en esta figura utilizando el software Image J. Todos los experimentos se repitieron al menos tres veces con resultados similares. p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
La eliminación de FADD promueve la infección por PRRSV
Por el contrario, examinamos los efectos de la eliminación de FADD en la infección por PRRSV. Utilizamos siRNA específico para regular a la baja la expresión de FADD endógeno, lo que podría reducir eficientemente la expresión de FADD (Figuras 2A, D, E). Luego, las células MARC-145 transfectadas con siRNAs se infectaron con PRRSV durante 24, 36 y 48 h, en comparación con las células transfectadas con siRNA de control no dirigido (siNC), los niveles de ARN de PRRSV y los títulos virales en las células knockdown de FADD aumentaron notablemente a 24, 36 y 48 hpi (Figuras 2B, C). Del mismo modo, el silenciamiento de FADD mejoró significativamente los niveles de expresión de la proteína N (Figuras 2D-F). Estos datos sugieren que FADD está implicado en la infección por PRRSV y es un factor antiviral celular contra el PRRSV.
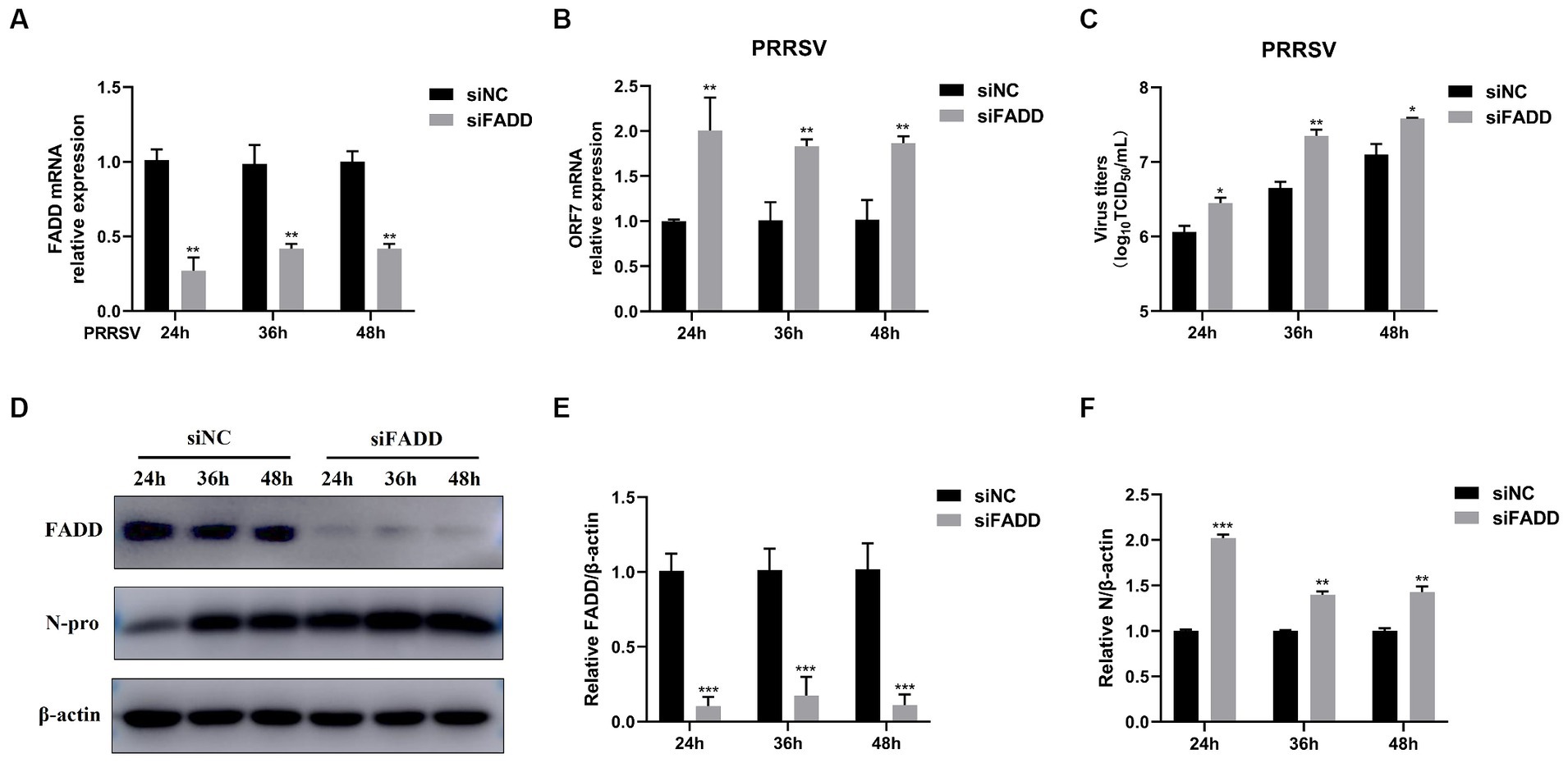 Figura 2. La eliminación de FADD promueve la infección por PRRSV. Las células MARC-145 se transfectaron con siFADD o un control no dirigido (siNC) durante 24 h, luego las células transfectadas con siFADD o siNC se infectaron con PRRSV a un MOI de 0,1 durante 24, 36, 48 h. (A) La eficiencia de eliminación de FADD y (B) los niveles de ARNm de PRRSV ORF7 se examinaron mediante qPCR. (C) Los títulos de virus fueron analizados por TCID50. (D) Los niveles de expresión de la proteína N se detectaron mediante western blot con un anticuerpo policlonal anti-proteína N anti-PRRSV de conejo. (E,F) Los niveles relativos de expresión de FADD y proteína N se analizaron mediante el software Image J. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Figura 2. La eliminación de FADD promueve la infección por PRRSV. Las células MARC-145 se transfectaron con siFADD o un control no dirigido (siNC) durante 24 h, luego las células transfectadas con siFADD o siNC se infectaron con PRRSV a un MOI de 0,1 durante 24, 36, 48 h. (A) La eficiencia de eliminación de FADD y (B) los niveles de ARNm de PRRSV ORF7 se examinaron mediante qPCR. (C) Los títulos de virus fueron analizados por TCID50. (D) Los niveles de expresión de la proteína N se detectaron mediante western blot con un anticuerpo policlonal anti-proteína N anti-PRRSV de conejo. (E,F) Los niveles relativos de expresión de FADD y proteína N se analizaron mediante el software Image J. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
FADD promueve la producción de IFN tipo I después de la estimulación de poliI:C
FADD y RIPK1 podrían formar un proteosoma innato en respuesta al dsRNA viral y luego regular la producción de IFN-β mediada por TBK1 (17). Así que a continuación nos preguntamos si FADD podría regular la producción de IFN tipo I mediada por poliI:C. El poli (I:C), el análogo sintético del ARN bicatenario (dsRNA), se utiliza experimentalmente para desencadenar la producción de IFN tipo I (30). Los resultados mostraron que la sobreexpresión de FADD en células MARC-145 aumenta la actividad del promotor de IFN-β (Figura 3A) y los niveles transcripcionales de IFN-β desencadenados por poliI:C (Figura 3B). Estos datos sugieren que FADD participa en la promoción de la señalización de IFN tipo I.
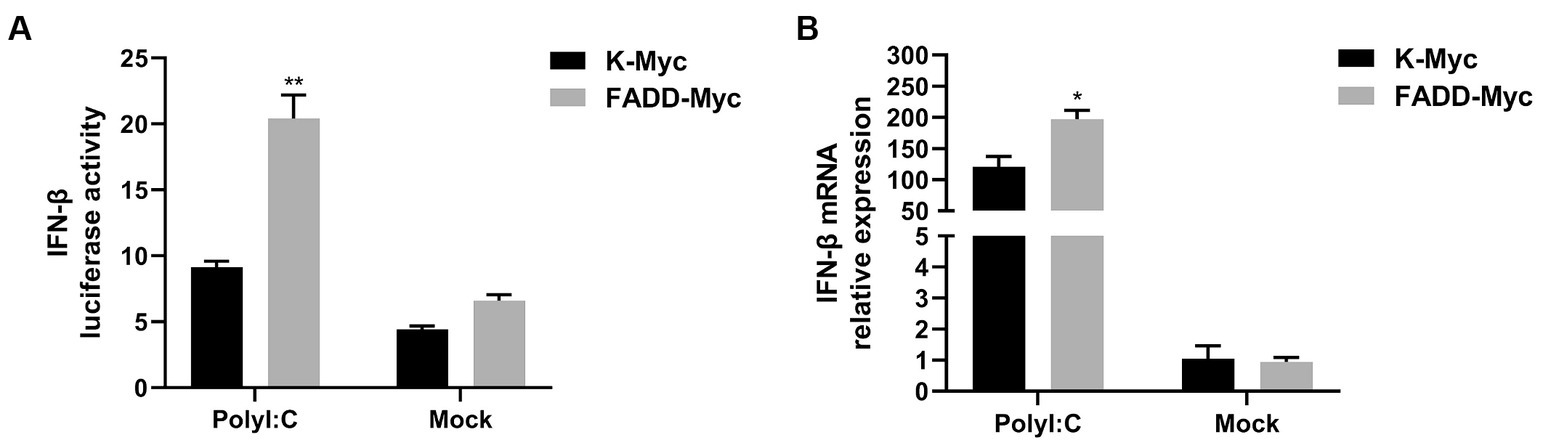 Figura 3. FADD promueve la producción de IFN-β mediada por estratagemaI:C. (A) Las células HEK-293T se transfectaron con plásmido indicador de IFN-β y FADD-Myc o vector de control, y 36 h después, las células se transfectaron con poliI:C (10 μg/mL) durante 12 h, y luego las actividades luciferasas de IFN-β se analizaron mediante un ensayo de informe de luciferasa dual. (B) Las células MARC-145 se transfectaron con FADD-Myc o vector de control durante 36 h, luego se transfectaron con poliI:C durante 12 h, los niveles de ARNm de IFN-β se midieron mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05 y p < 0,01. La significación estadística de las diferencias se determinó mediante la prueba t de Student.***
Figura 3. FADD promueve la producción de IFN-β mediada por estratagemaI:C. (A) Las células HEK-293T se transfectaron con plásmido indicador de IFN-β y FADD-Myc o vector de control, y 36 h después, las células se transfectaron con poliI:C (10 μg/mL) durante 12 h, y luego las actividades luciferasas de IFN-β se analizaron mediante un ensayo de informe de luciferasa dual. (B) Las células MARC-145 se transfectaron con FADD-Myc o vector de control durante 36 h, luego se transfectaron con poliI:C durante 12 h, los niveles de ARNm de IFN-β se midieron mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05 y p < 0,01. La significación estadística de las diferencias se determinó mediante la prueba t de Student.***
FADD promueve la señalización de IFN tipo I mediada por MAVS
Dado que FADD gobierna la activación de IFN-β activada por poliI:C, investigamos el papel y el mecanismo de FADD en la vía de señalización RLR-MAVS. Las células HEK-293T se transfectaron conjuntamente con plásmidos que codifican FADD, componentes de la vía RIG-I y plásmido indicador de IFN-β. Los resultados mostraron que FADD mejoró en gran medida la actividad del promotor de IFN-β inducida por MAVS y TBK1 (Figura 4A). Y los niveles transcripcionales de ARNm de IFN-β aumentaron drásticamente en las células cotransfectadas con FADD y MAVS, y aumentaron ligeramente en las células cotransfectadas con FADD e IRF3 (Figura 4B). Mientras tanto, FADD también reguló al alza las expresiones de ARNm inducidas por MAVS de ISG15, ISG56 y OAS1 (Figuras 4C-E). Además, las expresiones de ARNm de OAS1 también aumentaron con RIG-I y TBK1 en células transfectadas con FADD (Figura 4E). Posteriormente, las células se cotransfectaron con MAVS, plásmido indicador de IFN-β y diferentes concentraciones de FADD, y encontramos que FADD aumentó la actividad del promotor de IFN-β inducida por MAVS (Figura 5A) y los niveles de transcripción de ARNm de IFN-β, ISG15, ISG56 y OAS1 de manera dependiente de la dosis (Figuras 5B-E).
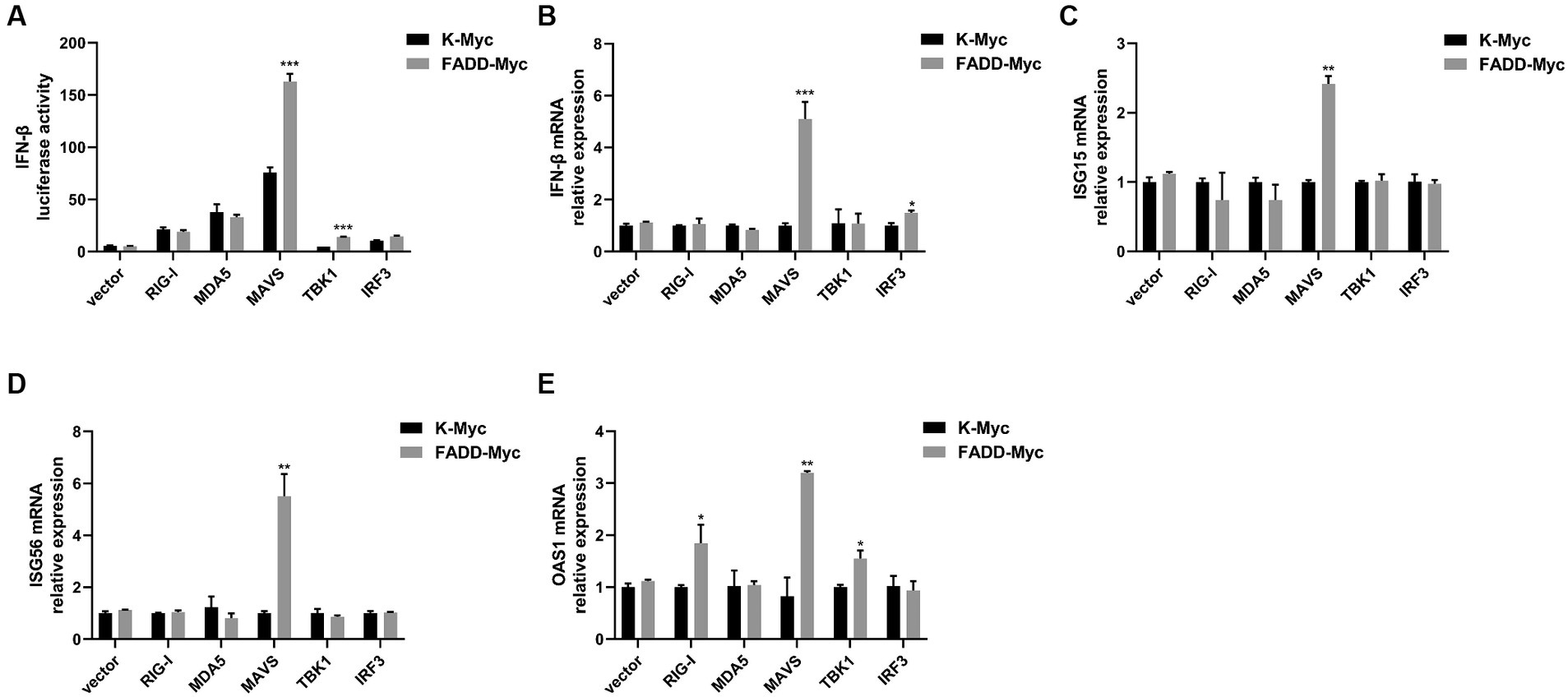 Figura 4. FADD promueve la señalización de IFN tipo I mediada por MAVS. (A) Las células HEK-293T se transfectaron con plásmido reportero de IFN-β, FADD-Myc y plásmidos RIG-I, MDA5, MAVS, TBK1 o IRF3. Los ensayos de luciferasa se realizaron a las 48 h. (B-E) Las células MARC-145 se transfectaron con plásmidos FADD-Myc y RIG-I, MDA5, MAVS, TBK1 o IRF3. Y 48 h después, los niveles transcripcionales de IFN-β (B), ISG15 (C), ISG56 (D) y OAS1 (E) se detectaron mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Figura 4. FADD promueve la señalización de IFN tipo I mediada por MAVS. (A) Las células HEK-293T se transfectaron con plásmido reportero de IFN-β, FADD-Myc y plásmidos RIG-I, MDA5, MAVS, TBK1 o IRF3. Los ensayos de luciferasa se realizaron a las 48 h. (B-E) Las células MARC-145 se transfectaron con plásmidos FADD-Myc y RIG-I, MDA5, MAVS, TBK1 o IRF3. Y 48 h después, los niveles transcripcionales de IFN-β (B), ISG15 (C), ISG56 (D) y OAS1 (E) se detectaron mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
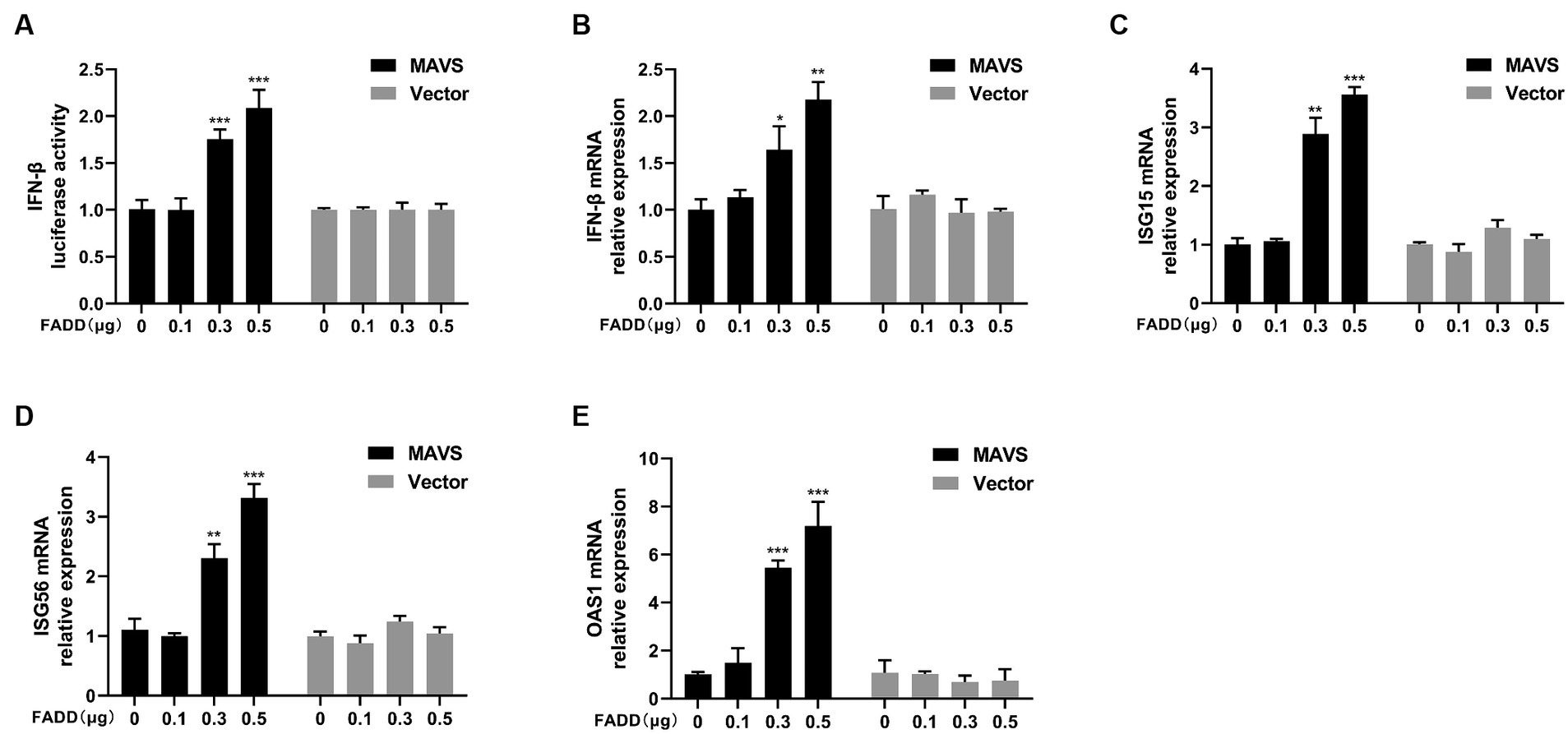 Figura 5. FADD promueve la señalización de IFN tipo I mediada por MAVS de una manera dependiente de la dosis. (A) Las células HEK-293T se transfectaron con MAVS, aumentando las cantidades de plásmido reportero de FADD-Myc e IFN-β, a 48 hpt, las actividades de luciferasa se analizaron utilizando un ensayo reportero de luciferasa dual. (B-E) Las células MARC-145 se transfectaron con MAVS y se detectaron cantidades crecientes de FADD-Myc, los niveles de ARNm de IFN-β (B), ISG15 (C), ISG56 (D) y OAS1 (E) mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Figura 5. FADD promueve la señalización de IFN tipo I mediada por MAVS de una manera dependiente de la dosis. (A) Las células HEK-293T se transfectaron con MAVS, aumentando las cantidades de plásmido reportero de FADD-Myc e IFN-β, a 48 hpt, las actividades de luciferasa se analizaron utilizando un ensayo reportero de luciferasa dual. (B-E) Las células MARC-145 se transfectaron con MAVS y se detectaron cantidades crecientes de FADD-Myc, los niveles de ARNm de IFN-β (B), ISG15 (C), ISG56 (D) y OAS1 (E) mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Dado que FADD podría mejorar la señalización de IFN tipo I mediada por MAVS, para identificar si MAVS es indispensable para que FADD regule la producción de IFN tipo I, las células MARC-145 se transfectaron con siMAVS, FADD-Myc y plásmido indicador de IFN-β, y 36 h después, las células se estimularon con poliI:C. Como se muestra en la Figura 6, FADD promovió las expresiones de IFN-β e ISG inducidas por poliI:C (ISG15, ISG56 y OAS1) en células siNC. Sin embargo, no pudo aumentar la producción mediada por poliI:C de IFN tipo I e ISG tras el silenciamiento de MAVS. En conjunto, estos resultados sugieren que FADD regula positivamente la señalización de IFN tipo I mediada por MAVS.
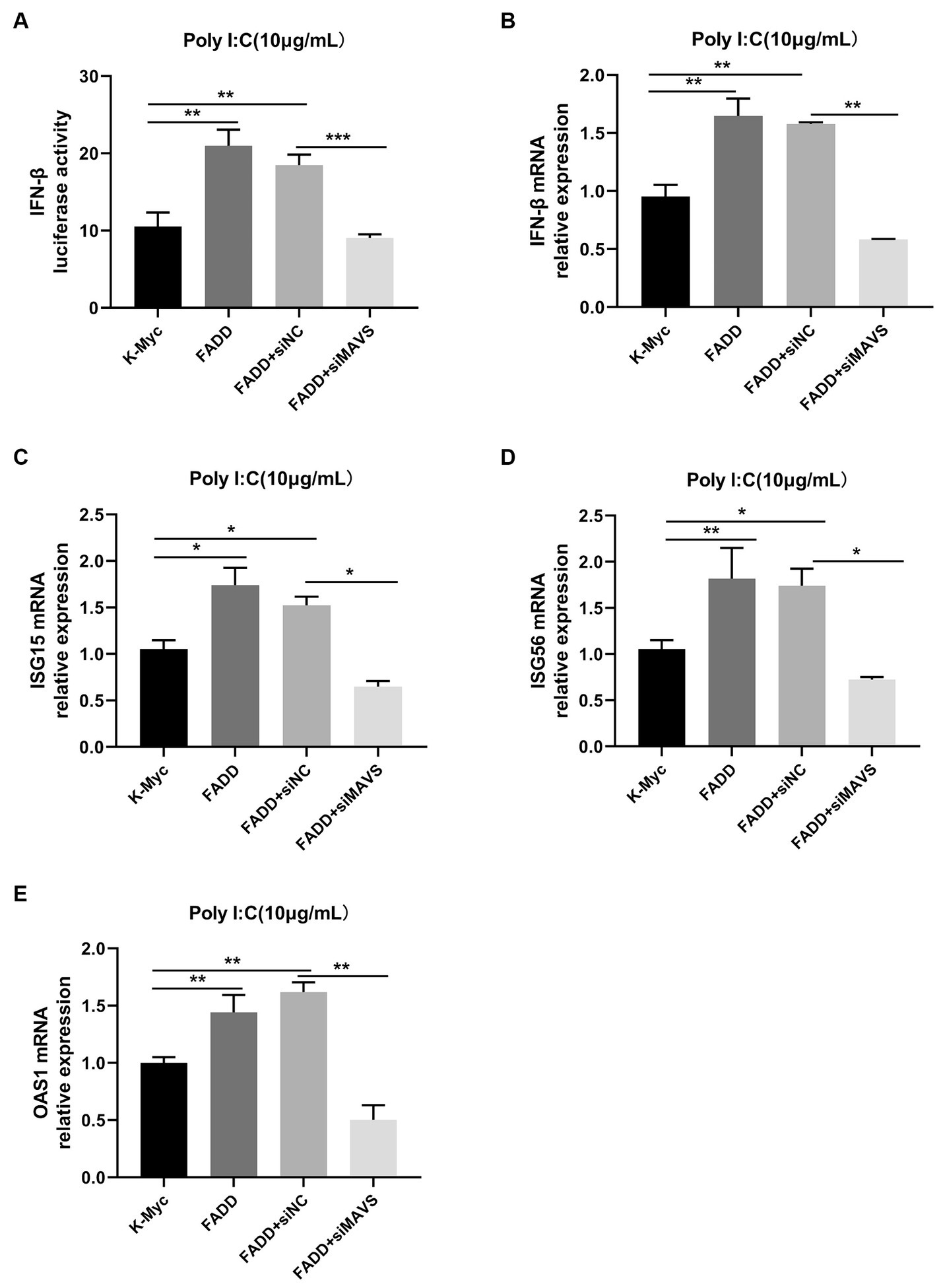 Figura 6. FADD promueve la señalización de IFN tipo I de una manera dependiente de MAVS. (A) Las células MARC-145 se transfectaron con plásmido indicador de IFN-β, FADD-Myc y siNC o siMAVS durante 36 h, luego las células se estimularon con poliI:C y, 12 h después, las actividades luciferasas de IFN-β se analizaron mediante ensayo reportero de luciferasa dual. (B-E) Las células MARC-145 se transfectaron con FADD-Myc y siNC o siMAVS durante 36 h, y luego las células se estimularon con poliI:C, y 12 h después, se detectaron los niveles de ARNm de IFN-β (B), ISG15 (C), ISG56 (D) y OAS1 (E) mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Figura 6. FADD promueve la señalización de IFN tipo I de una manera dependiente de MAVS. (A) Las células MARC-145 se transfectaron con plásmido indicador de IFN-β, FADD-Myc y siNC o siMAVS durante 36 h, luego las células se estimularon con poliI:C y, 12 h después, las actividades luciferasas de IFN-β se analizaron mediante ensayo reportero de luciferasa dual. (B-E) Las células MARC-145 se transfectaron con FADD-Myc y siNC o siMAVS durante 36 h, y luego las células se estimularon con poliI:C, y 12 h después, se detectaron los niveles de ARNm de IFN-β (B), ISG15 (C), ISG56 (D) y OAS1 (E) mediante qPCR. Los resultados son representativos de tres experimentos independientes (las medias ± DE). p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
FADD inhibe la infección por PRRSV a través de la mejora de la señalización de MAVS-IFN
En vista del hecho de que FADD posee una actividad antiviral significativa y mejora la señalización de IFN tipo I mediada por MAVS, para investigar si la actividad antiviral de FADD depende de MAVS, las células MARC-145 se transfectaron con siRNAs de MAVS (siMAVS) y FADD-Myc. Como se muestra en la Figura 7, se observó una marcada reducción de la expresión de MAVS en las células silenciadas de MAVS (Figuras 7A, D, E). Y FADD no pudo disminuir los niveles de ARN del PRRSV (Figura 7B) y los títulos virales (Figura 7C) en las células silenciadas por MAVS. Mientras tanto, no hubo diferencia en la proteína N del PRRSV entre las células knockdown de MAVS transfectadas con K-Myc y FADD-Myc (Figuras 7D, F, G). Estos datos indicaron que la actividad anti-PRRSV de FADD necesita la participación de MAVS.
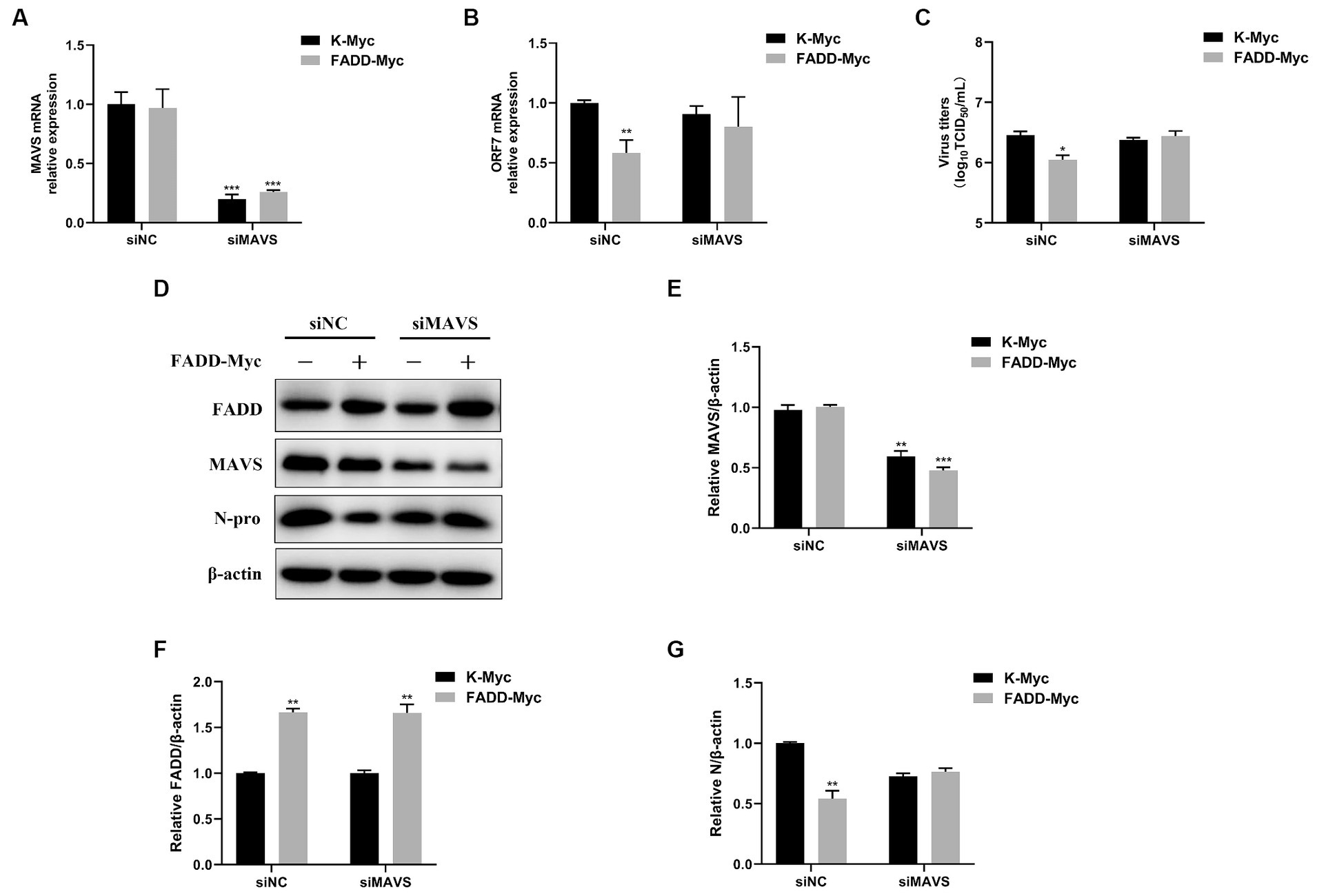 Figura 7. La actividad antiviral de FADD depende de MAVS. Las células MARC-145 se transfectaron con siMAVS y FADD-Myc, y 24 h después, y las células se infectaron con PRRSV a un MOI de 0,1 durante 24 h. Luego, se recolectaron las células, se determinó la eficiencia de silenciamiento de MAVS (A) y los niveles de ARN de PRRSV (B) mediante qPCR. (C) Los títulos de virus de sobrenadantes celulares se analizaron mediante TCID50. (D) Los niveles de expresión de la proteína N se detectaron mediante Western blot. (E–G) Las relaciones de intensidad relativa de MAVS, FADD y proteína N se analizaron utilizando el software Image J. p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Figura 7. La actividad antiviral de FADD depende de MAVS. Las células MARC-145 se transfectaron con siMAVS y FADD-Myc, y 24 h después, y las células se infectaron con PRRSV a un MOI de 0,1 durante 24 h. Luego, se recolectaron las células, se determinó la eficiencia de silenciamiento de MAVS (A) y los niveles de ARN de PRRSV (B) mediante qPCR. (C) Los títulos de virus de sobrenadantes celulares se analizaron mediante TCID50. (D) Los niveles de expresión de la proteína N se detectaron mediante Western blot. (E–G) Las relaciones de intensidad relativa de MAVS, FADD y proteína N se analizaron utilizando el software Image J. p < 0,05, p < 0,01 y p < 0,001. La significación estadística de las diferencias se determinó mediante la prueba t de Student.******
Además, investigamos si FADD regula la producción de IFN tipo I e ISG durante la infección por PRRSV. Las células MARC-145 se transfectaron con FADD-Myc o vector control durante 24 h, luego las células se infectaron con PRRSV durante 24, 36 y 48 h. En comparación con las células control, las células con sobreexpresión de FADD mejoraron significativamente la expresión de IFN-β a las 24, 36 y 48 h (Figura 8A). Mientras tanto, las expresiones de ARNm de ISG15, ISG56 y OAS1 también aumentaron en las células sobreexpresadas por FADD (Figuras 8B-D). En general, FADD promueve la respuesta anti-PRRSV a través de la regulación del IFN-β y la expresión génica estimulada por interferón mediada por MAVS.
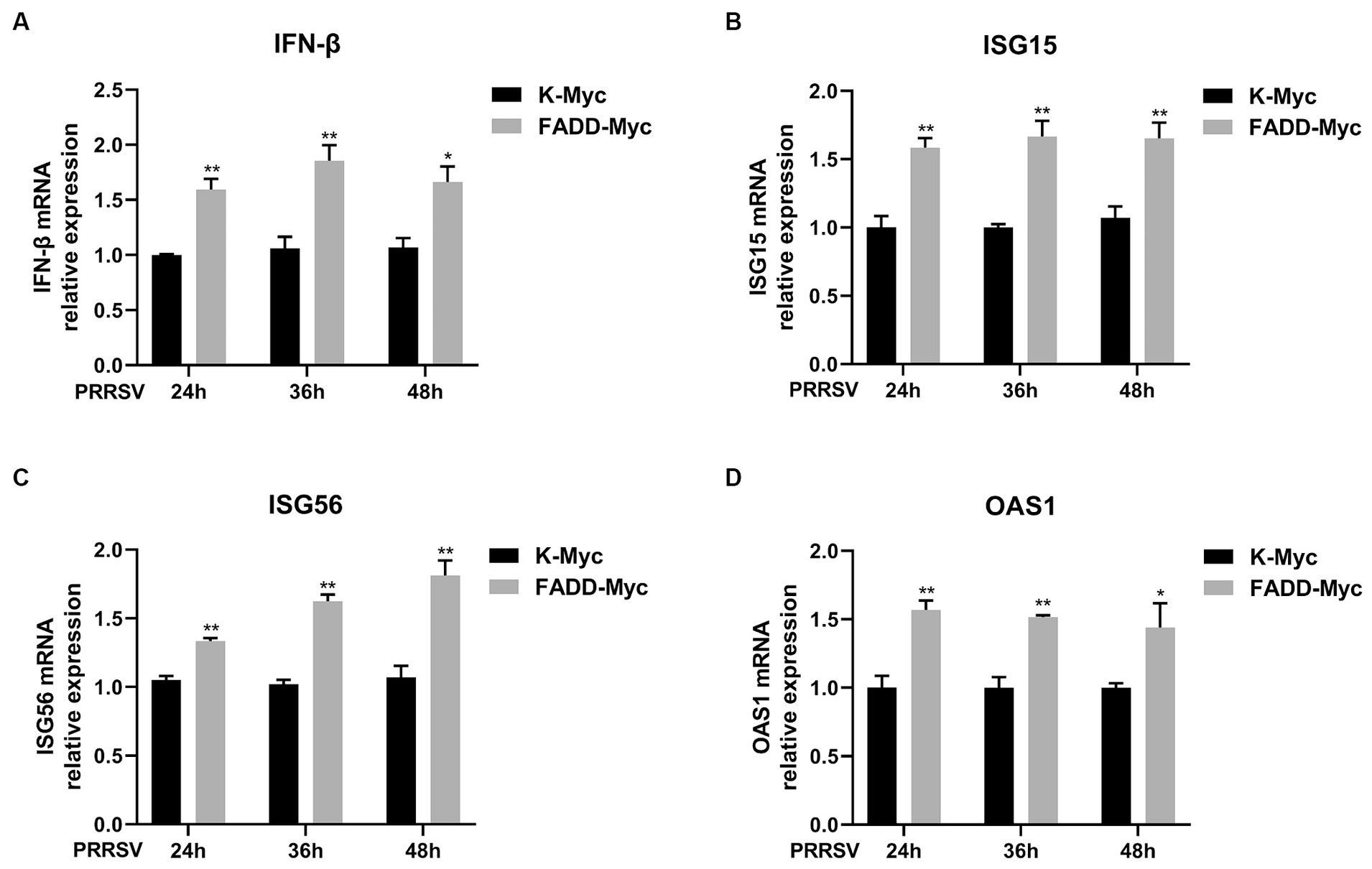 Figura 8. FADD mejora la señalización de IFN tipo I durante la infección por PRRSV. Las células MARC-145 se transfectaron con FADD-Myc o vector control durante 24 h y se infectaron con PRRSV a un MOI de 0,1 durante otras 24, 36 y 48 h. A continuación, se recolectaron las células y se detectaron los niveles de ARNm de IFN-β (A), ISG15 (B), ISG56 (C) y OAS1 (D) mediante qPCR. p < 0,05 y p < 0,01. La significación estadística de las diferencias se determinó mediante la prueba t de Student.***
Figura 8. FADD mejora la señalización de IFN tipo I durante la infección por PRRSV. Las células MARC-145 se transfectaron con FADD-Myc o vector control durante 24 h y se infectaron con PRRSV a un MOI de 0,1 durante otras 24, 36 y 48 h. A continuación, se recolectaron las células y se detectaron los niveles de ARNm de IFN-β (A), ISG15 (B), ISG56 (C) y OAS1 (D) mediante qPCR. p < 0,05 y p < 0,01. La significación estadística de las diferencias se determinó mediante la prueba t de Student.***
Discusión
El PRRSV se ha convertido en una de las principales amenazas para la industria porcina mundial desde su brote a finales de la década de 1980 (31). Aunque las vacunas disponibles en el mercado se utilizan ampliamente, el PRRSV aún no se controla eficazmente debido a la deriva antigénica. La relación entre el PRRSV y su huésped es compleja y delicada. Por un lado, el PRRSV ha desarrollado múltiples estrategias para suprimir la producción de IFN de tipo I. Por otro lado, la inmunidad innata ejerce un papel clave en la respuesta al PRRSV (32, 33). Aquí, se revela una estrategia novedosa en la que la proteína del huésped FADD suprime el PRRSV mediante la regulación positiva de la respuesta inmunitaria innata del huésped.
En primer lugar, se investigó el papel de FADD en la infección por PRRSV, y los resultados mostraron que la sobreexpresión de FADD podría suprimir significativamente la replicación del PRRSV (Figura 1), y el silenciamiento de la expresión de FADD endógeno por pequeños ARN interferentes condujo a la promoción de la replicación del PRRSV (Figura 2). Posteriormente, demostramos que FADD mejora notablemente la señalización de IFN tipo I mediada por MAVS (Figura 5). Además, la capacidad de FADD para antagonizar la replicación del PRRSV depende de los MAVS (Figura 7), y durante la infección por PRRSV, FADD puede promover la producción de IFN tipo I e ISG (ISG15, ISG56 y OAS1) (Figura 8). En conclusión, nuestro estudio descubrió que la FADD desempeña un papel importante en la respuesta inmunitaria antiviral.
FADD es una proteína adaptadora conocida por ser crucial para la vía extrínseca de la apoptosis de las células de mamíferos (34). Además de su papel fundamental en la señalización de los receptores de muerte de la apoptosis, FADD ha surgido como un nuevo actor en el desarrollo del cáncer, la inflamación, la inmunidad innata y la infección viral. Se ha informado que FADD−/− Los MEF son muy sensibles a la infección por muchos virus de ARN, incluidos el virus de la gripe, el VSV y el EMCV, debido a la producción defectuosa de interferón (21). En este estudio, demostramos que FADD juega un papel importante en la inhibición de la replicación del PRRSV, y fue la primera vez que dimos evidencia de que FADD era una proteína huésped contra el PRRSV. Además, muchos estudios han demostrado que el PRRSV induce la apoptosis tanto in vitro como in vivo. La apoptosis a menudo se considera como un mecanismo de defensa innato que limita la infección por virus mediante la eliminación de las células infectadas (32). Y el PRRSV podría aumentar los niveles de expresión de TNFR1/FasL (35), por lo que tal vez el FADD tenga un papel importante en la apoptosis después de la infección por PRRSV. Además, encontramos que la inhibición de FADD de la replicación del PRRSV dependía de MAVS, y FADD podría promover la producción de IFN-β mediada por MAVS e ISG posteriores, lo que indica que la vía de IFN tipo I mediada por MAVS puede desempeñar un papel clave en la supresión de la replicación de PRRSV por FADD.
MAVS es un adaptador crítico en la vía de señalización RLR que vincula el reconocimiento ascendente del ARN viral con la transducción de señales aguas abajo en la respuesta antiviral (36). Es bien sabido que la agregación de MAVS se considera un sello distintivo de su activación y que es esencial para su función antiviral. Se demostró que la ubiquitinación de RIG-I es un elemento crítico para que active MAVS, RIG-I se une a cadenas de poliubiquitina de lisina-63 (K63) no ancladas y que esta unión es importante para la agregación y activación de MAVS (37). SNX8 se asoció con VISA y aumentó la agregación de VISA, lo que llevó a la activación de la señalización descendente (38). También se ha informado que la fosforilación y la desfosforilación desempeñan un papel fundamental en la inmunidad innata antiviral. c-Abl regula positivamente la señalización de RLR mediante la fosforilación de MAVS en Y9, Y30 e Y71 (39, 40). Además, la succinilación de proteínas causada por el succinil-CoA es una nueva modificación postraduccional recientemente descubierta (41). La sirtuina5 desuccinilla MAVS en K7 para disminuir y antagonizar la agregación de MAVS y la respuesta antiviral (42). Dado que rara vez se explora el papel de FADD en los RLR o en la señalización ascendente mediada por MAVS, es necesario realizar más estudios sobre la agregación de MAVS y las modificaciones postraduccionales de MAVS por FADD para aclarar el mecanismo detallado de FADD que regula MAVS.
Además, también encontramos que FADD podría mejorar la expresión de OAS1 mediada por RIG-I y TBK1, pero no afectó la expresión de IFN-β, ISG15 e ISG56. Estudios previos han identificado que después de la infección viral, la señalización de IFN tipo I induce la producción de la familia OAS que consiste en OAS1, OAS2, OAS3 y proteínas similares a OAS (OASL). Los dominios similares a la ubiquitina de OASL podrían sustituir a la poliubiquitina ligada a K63 e interactuar con los CARD de RIG-I y, por lo tanto, mejorar la señalización de RIG-I. Y la estructura cristalina del dominio similar a OAS muestra una sorprendente similitud con OAS1 (43, 44), por lo que los roles de FADD en el eje de señalización RIG-I-OAS1 aún no se han estudiado más a fondo.
En resumen, nuestro estudio destaca la importancia de FADD en la replicación del PRRSV. Informamos que FADD promueve la vía de IFN tipo I mediada por MAVS y la expresión de genes posteriores. Además, FADD inhibe la infección por PRRSV a través de la vía MAVS-IFN, lo que contribuye a comprender el mecanismo antiviral del huésped y puede tener implicaciones para el control futuro del PRRS.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo/material complementario, las consultas adicionales pueden dirigirse a los autores correspondientes.
Declaración ética
No se requirió aprobación ética para los estudios en seres humanos de acuerdo con la legislación local y los requisitos institucionales, ya que solo se utilizaron líneas celulares establecidas disponibles comercialmente. No se requirió aprobación ética para los estudios en animales de acuerdo con la legislación local y los requisitos institucionales, ya que solo se utilizaron líneas celulares establecidas disponibles comercialmente.
Contribuciones de los autores
XC: Curación de datos, Análisis formal, Metodología, Redacción – revisión y edición, Redacción – borrador original. MW: Curación de datos, Análisis formal, Redacción – borrador original. ZL: Curación de datos, Análisis formal, Redacción – borrador original. LW: Análisis formal, Metodología, Redacción – borrador original. GZ: Supervisión, Redacción – borrador original. YC: Supervisión, Redacción – borrador original. JH: Supervisión, Redacción – borrador original.
Financiación
El/los autor/es declaran/n que se recibió apoyo financiero para la investigación, autoría y/o publicación de este artículo. La investigación contó con el apoyo del Fondo Juvenil de la Fundación de Ciencias Naturales de la Provincia de Henan (222300420157), el Proyecto de Investigación Científica y Tecnológica de la Provincia de Henan (222102110387), la Fundación Nacional de Ciencias Naturales de China (32172862), la Fundación de la Juventud Sobresaliente del Comité Científico de Henan (222300420043) y los Principales Talentos de la Innovación Científica y Tecnológica en las Llanuras Centrales (224200510024).
Reconocimientos
Los autores quieren agradecer a Hanchun Yang por proporcionarnos la cepa BJ-4 del PRRSV.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Albina, E. Epidemiología del síndrome respiratorio y reproductivo porcino (PRRS): una visión general. Microbiol veterinario. (1997) 55:309–16. doi: 10.1016/S0378-1135(96)01322-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Holtkamp, DJ, Kliebenstein, JB, Neumann, EJ, Zimmerman, JJ, Rotto, HF, Yoder, TK, et al. Evaluación del impacto económico del virus del síndrome respiratorio y reproductivo porcino en los productores de carne de cerdo de los Estados Unidos. J Salud Porcina Prod. (2013) 21:72–84.
3. Nieuwenhuis, N, Duinhof, TF y van Nes, A. Análisis económico de los brotes del virus del síndrome respiratorio y reproductivo porcino en nueve rebaños de cerdas. Rec. Veterinario (2012) 170:225. doi: 10.1136/vr.100101
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Pejsak, Z, Stadejek, T, y Markowska-Daniel, I. Signos clínicos y pérdidas económicas causadas por el virus del síndrome respiratorio y reproductivo porcino en una gran granja de cría. Microbiol veterinario. (1997) 55:317–22. doi: 10.1016/S0378-1135(96)01326-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Han, MY, y Yoo, DW. Ingeniería del genoma del virus del PRRS: actualizaciones y perspectivas. Microbiol veterinario. (2014) 174:279–95. doi: 10.1016/j.vetmic.2014.10.007
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Johnson, CR, Griggs, TF, Gnanandarajah, J, y Murtaugh, MP. Nueva proteína estructural en el virus del síndrome respiratorio y reproductivo porcino codificada por una alternativa ORF5 presente en todos los arterivirus. J Gen Virol. (2011) 92:1107–16. doi: 10.1099/vir.0.030213-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Música, N, y Gagnon, CA. El papel de las proteínas estructurales y no estructurales del virus del síndrome respiratorio y reproductivo porcino (PRRS) en la patogénesis del virus. Anim Health Res Rev. (2010) 11:135–63. doi: 10.1017/S1466252310000034
8. Adams, MJ, Lefkowitz, EJ, King, AM, Harrach, B, Harrison, RL, Knowles, NJ, et al. Votación de ratificación de propuestas taxonómicas al comité internacional de taxonomía de virus (2016). Arch Virol. (2016) 161:2921–49. doi: 10.1007/s00705-016-2977-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Lunney, JK, Fang, Y, Ladinig, A, Chen, N, Li, Y, Rowland, B, et al. Virus del síndrome respiratorio y reproductivo porcino (PRRSV): patogenia e interacción con el sistema inmune. Annu Rev Anim Biosci. (2016) 4:129–54. doi: 10.1146/annurev-animal-022114-111025
10. Bowie, AG y Unterholzner, L. Evasión viral y subversión de la señalización del receptor de reconocimiento de patrones. Nat Rev Immunol. (2008) 8:911–22. doi: 10.1038/nri2436
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Nakhaei, P, Genin, P, Civas, A y Hiscott, J. Receptores similares a RIG-I: detección y respuesta a la infección por virus de ARN. Semin Immunol. (2009) 21:215–22. doi: 10.1016/j.smim.2009.05.001
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Overend, C, Mitchell, R, He, D, Rompato, G, Grubman, M y Garmendia, A. El interferón beta porcino recombinante protege a los macrófagos alveolares porcinos y a las células MARC-145 de la infección por el virus del síndrome respiratorio y reproductivo porcino. J Gen Virol. (2007) 88:925–31. doi: 10.1099/vir.0.82585-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Schneider, WM, Chevillotte, MD y Rice, CM. Genes estimulados por interferón: una compleja red de defensas del huésped. Annu Rev Immunol. (2014) 32:513–45. doi: 10.1146/annurev-immunol-032713-120231
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Wang, H, Bai, J, Fan, B, Li, Y, Zhang, Q y Jiang, P. El Mx2 inducido por interferón inhibe la replicación del virus del síndrome respiratorio y reproductivo porcino. J Interf Citoquinas Res. (2016) 36:129–39. doi: 10.1089/jir.2015.0077
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Zhao, J, Feng, N, Li, Z, Wang, P, Qi, Z, Liang, W, et al. La 2′,5′-oligoadenilato sintetasa 1 (OAS1) inhibe la replicación del PRRSV en las células MARC-145. Antivir Res. (2016) 132:268–73. doi: 10.1016/j.antiviral.2016.07.001
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Carrington, P, Sandu, C, Wei, Y, Hill, J, Morisawa, G, Huang, T, et al. La estructura de FADD y su modo de interacción con la procaspasa-8. Célula molar. (2006) 22:599–610. doi: 10.1016/j.molcel.2006.04.018
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Mouasni, S, y Tourneur, L. FADD en la encrucijada entre el cáncer y la inflamación. Tendencias Immunol. (2018) 39:1036–53. doi: 10.1016/j.it.2018.10.005
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Wilson, NS, Dixit, V y Ashkenazi, A. Transductores de señales del receptor de muerte: nodos de coordinación en redes de señalización inmune. Nat Immunol. (2009) 10:348–55. doi: 10.1038/ni.1714
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Zhuang, HQ, Wang, XS, Zha, DL, Gan, ZY, Cai, FF, Du, P, et al. FADD es un regulador clave del metabolismo de los lípidos. EMBO Mol Med. (2016) 8:895–918. doi: 10.15252/emmm.201505924
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Tourneur, L, y Chiocchia, G. FADD: un regulador de la vida y la muerte. Tendencias Immunol. (2010) 31:260–9. doi: 10.1016/j.it.2010.05.005
21. Balachandran, S, Thomas, E, y Barber, G. Mecanismo inmunitario innato dependiente de FADD en células de mamíferos. Naturaleza. (2004) 432:401–5. doi: 10.1038/nature03124
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Balachandran, S, Venkataraman, T, Fisher, P y Barber, G. La señalización inmunitaria innata antiviral mediada por proteínas que contiene el dominio de muerte asociado a Fas implica la regulación de IRF 7. J Immunol. (2007) 178:2429–39. doi: 10.4049/jimmunol.178.4.2429
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Michallet, M, Meylan, E, Ermolaeva, M, Vázquez, J, Rebsamen, M, Curran, J, et al. La proteína Tradd es un componente esencial de la vía antiviral helicasa similar a RIG. Inmunidad. (2008) 28:651–61. doi: 10.1016/j.immuni.2008.03.013
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Young, J, Sermwittayawong, D, Kim, H, Nandu, S, An, N, Erdjument-Bromage, H, et al. El dominio de muerte asociado a Fas (FADD) y la ubiquitina-proteína ligasa E3 TRIM21 interactúan para regular negativamente la producción de interferón inducida por el virus. J Biol Chem. (2011) 286:6521–31. doi: 10.1074/jbc. M110.172288
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Shi, XB, Wang, L, Zhi, YB, Xing, GX, Zhao, D, Deng, RG, et al. El virus del síndrome respiratorio y reproductivo porcino (PRRSV) pudo ser detectado por el sistema profesional productor de interferón beta y tenía mecanismos para inhibir esta acción en las células MARC-145. Virus Res. (2010) 153:151–6. doi: 10.1016/j.virusres.2010.07.028
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Chang, X, Shi, X, Zhang, X, Wang, L, Li, X, Wang, A, et al. IFI16 inhibe la replicación del virus del síndrome respiratorio y reproductivo porcino 2 de manera dependiente de MAVS en células MARC-145. Virus. (2019) 11:1160. doi: 10.3390/v11121160
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Chang, XB, Yang, YQ, Gao, JC, Zhao, K, Guo, JC, Ye, C, et al. La anexina A2 se une a la vimentina y contribuye a la multiplicación del virus del síndrome respiratorio y reproductivo porcino. Res. Veterinaria. (2018) 49:75. doi: 10.1186/s13567-018-0571-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Caña, LJ. Un método simple para estimar el cincuenta por ciento de los criterios de valoración. Am J Epidemiol. (1938) 27:493–7. doi: 10.1093/oxfordjournals.aje.a118408
29. Chang, X, Shi, X, Zhang, X, Chen, J, Fan, X, Yang, Y, et al. miR-382-5p promueve la replicación del virus del síndrome respiratorio y reproductivo porcino (PRRSV) al regular negativamente la inducción del interferón tipo I. FASEB J. (2020) 34:4497–511. doi: 10.1096/fj.201902031RRR
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Cheng, Y y Xu, F. Función anticancerígena del ácido poliinosínico-policitidílico. Cáncer Biol Ther. (2010) 10:1219–23. doi: 10.4161/cbt.10.12.13450
31. Shi, M, Lam, TTY, Hon, CC, Hui, RKH, Faaberg, KS, Wennblom, T, et al. Epidemiología molecular del PRRSV: una perspectiva filogenética. Virus Res. (2010) 154:7–17. doi: 10.1016/j.virusres.2010.08.014
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. An, T, Li, J, Su, C y Yoo, D. Mecanismos moleculares y celulares para la patogénesis del PRRSV y la respuesta del huésped a la infección. Virus Res. (2020) 286:197980. doi: 10.1016/j.virusres.2020.197980
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Sun, Y, Han, M, Kim, C, Calvert, JG y Yoo, D. Interacción entre la inmunidad innata mediada por interferón y el virus del síndrome respiratorio y reproductivo porcino. Virus. (2012) 4:424–46. doi: 10.3390/v4040424
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Chinnaiyan, A, O’Rourke, K, Tewari, M y Dixit, V. FADD, una nueva proteína que contiene el dominio de la muerte, interactúa con el dominio de la muerte de Fas e inicia la apoptosis. Célula. (1995) 81:505–12. doi: 10.1016/0092-8674(95)90071-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Chang, H, Jeng, C, Lin, C, Liu, J, Chang, C, Tsai, Y, et al. La implicación de la interacción Fas/FasL en la apoptosis in vitro de los linfocitos asociada al circovirus porcino tipo 2 y en la apoptosis linfocitaria asociada a la coinoculación del virus del síndrome respiratorio y reproductivo porcino. Microbiol veterinario. (2007) 122:72–82. doi: 10.1016/j.vetmic.2007.01.013
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Liu, B, y Gao, C. Regulación de la activación de MAVS a través de modificaciones postraduccionales. Curr Opin Immunol. (2018) 50:75–81. doi: 10.1016/j.coi.2017.12.002
37. Hou, F, Sun, L, Zheng, H, Skaug, B, Jiang, QX y Chen, ZJ. MAVS forma agregados funcionales similares a priones para activar y propagar la respuesta inmunitaria innata antiviral. Célula. (2011) 146:448–61. doi: 10.1016/j.cell.2011.06.041
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Guo, W, Wei, J, Zhong, X, Zang, R, Lian, H, Hu, M, et al. SNX8 modula la respuesta inmune innata a los virus de ARN mediante la regulación de la agregación de VISA. Cell Mol Immunol. (2020) 17:1126–35. doi: 10.1038/s41423-019-0285-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Chen, Y, Shi, Y, Wu, J y Qi, N. MAVS: una tarjeta de dos caras que media la señalización inmunitaria innata antiviral y regula la homeostasis inmunitaria. Microbiol frontal. (2021) 12:12. doi: 10.3389/fmicb.2021.744348
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Cheng, J, Liao, Y, Xiao, L, Wu, R, Zhao, S, Chen, H, et al. La autofagia regula la activación de la señalización de MAVS de una manera dependiente de la fosforilación en la microglía. La muerte celular difiere. (2016) 24:276–87. doi: 10.1038/cdd.2016.121
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Yang, Y, y Gibson, G. La succinilación vincula el metabolismo con las funciones de las proteínas. Neurochem Res. (2019) 44:2346–59. DOI: 10.1007/S11064-019-02780-X
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Liu, X, Zhu, C, Zha, H, Tang, J, Rong, F, Chen, X, et al. SIRT5 perjudica la agregación y activación del adaptador de señalización MAVS a través de la catalización de la desuccinilación de lisina. EMBO J. (2020) 39:e103285. doi: 10.15252/embj.2019103285
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Ibsen, M, Gad, H, Andersen, L, Hornung, V, Julkunen, I, Sarkar, S, et al. El análisis estructural y funcional revela que el OASL humano se une al dsRNA para mejorar la señalización RIG-I. Ácidos nucleicos Res. (2015) 43:5236–48. doi: 10.1093/nar/gkv389
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Leisching, G, Wiid, I, y Baker, B. La asociación de OASL e interferones tipo I en la patogénesis y supervivencia de especies bacterianas replicantes intracelulares. Las células frontales infectan el microbiol. (2017) 7:196. doi: 10.3389/fcimb.2017.00196
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: PRRSV, FADD, MAVS, señalización de IFN tipo I, respuesta antiviral
Cita: Chang X, Wang M, Li Z, Wang L, Zhang G, Chang Y y Hu J (2024) FADD promueve la producción de interferón tipo I para suprimir la infección por el virus del síndrome respiratorio y reproductivo porcino. Frente. Vet. Sci. 11:1380144. doi: 10.3389/fvets.2024.1380144
Recibido: 01 de febrero de 2024; Aceptado: 26 de marzo de 2024;
Publicado: 08 Abril 2024.
Editado por:
Zhenbang Zhu, Universidad de Yangzhou, China
Revisado por:
Kuan Zhao, Universidad Agrícola de Hebei, China
Wei Wen, Universidad Agrícola de Huazhong, China
Derechos de autor © 2024 Chang, Wang, Li, Wang, Zhang, Chang y Hu. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Yafei Chang, changyafei1990@163.com; Jianhe Hu, xxjianhe@126.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía