Fracaso en la infección experimental de lechones de 10 días de edad
 Fracaso en la infección experimental de lechones de 10 días de edad con una cepa infecciosa propagada por cultivo celular de un virus de la diarrea epidémica porcina de genotipo 1a clásico
Fracaso en la infección experimental de lechones de 10 días de edad con una cepa infecciosa propagada por cultivo celular de un virus de la diarrea epidémica porcina de genotipo 1a clásico
 Priscilla F. Gerber1*
Priscilla F. Gerber1*  Dianjun Cao2
Dianjun Cao2  Chao-Ting Xiao3
Chao-Ting Xiao3  Qi Chen4
Qi Chen4  Kelly Lager5
Kelly Lager5  Berend Jan Bosch6
Berend Jan Bosch6  Xiang-Jin Meng7
Xiang-Jin Meng7  Patricio G. Halbur4
Patricio G. Halbur4  Tanja Opriessnig4,8*
Tanja Opriessnig4,8*- 1Departamento de Enfermedades Infecciosas y Salud Pública, Universidad de la Ciudad de Hong Kong, Kowloon, RAE de Hong Kong, China
- 2Facultad de Medicina Veterinaria, Universidad de Long Island, Nueva York, NY, Estados Unidos
- 3Laboratorio Provincial Clave de Virología Médica de Hunan, Instituto de Biología e Inmunología de Patógenos, Facultad de Biología, Universidad de Hunan, Changsha, China
- 4Departamento de Diagnóstico Veterinario y Medicina de Animales de Producción, Facultad de Medicina Veterinaria, Universidad Estatal de Iowa, Ames, IA, Estados Unidos
- 5Centro Nacional de Enfermedades Animales, Departamento de Agricultura de los Estados Unidos-Servicios de Investigación Agrícola, Ames, IA, Estados Unidos
- 6Sección de Virología, División de Enfermedades Infecciosas e Inmunología, Departamento de Ciencias Biomoleculares de la Salud, Facultad de Medicina Veterinaria, Universidad de Utrecht, Utrecht, Países Bajos
- 7Departamento de Ciencias Biomédicas y Patobiología, Facultad de Medicina Veterinaria, Instituto Politécnico y Universidad Estatal de Virginia, Blacksburg, VA, Estados Unidos
- 8Departamento de Vacunas y Diagnóstico, Instituto de Investigación Moredun, Penicuik, Reino Unido
Introducción: El virus de la diarrea epidémica porcina (PEDV, por sus siglas en inglés) causa enfermedad entérica en cerdos de todas las edades. El PEDV se puede agrupar en G1 (cepas clásicas) y G2 (cepas variantes) en función de las diferencias de secuencia en el gen de la espícula. Aunque hasta la fecha se han llevado a cabo varios estudios de patogénesis utilizando cepas contemporáneas de PEDV, existe información limitada sobre la patogénesis de las cepas históricas de PEDV en cerdos contemporáneos. El objetivo de este estudio fue investigar el curso clínico de la enfermedad en cerdos de 10 días de edad infectados con una cepa clásica europea G1a de PEDV de la década de 1980, que se transmitió por última vez en cerdos en 1994.
Métodos: Los resultados de la secuenciación confirmaron que el inóculo del virus era una cepa de PEDV estrechamente relacionada con la cepa prototipo CV777. La cepa de PEDV se pasó en serie tres veces en células Vero, y la cepa de virus infeccioso P3 se utilizó para inocular a los cerdos. Un total de 40 cerdos fueron inoculados por vía oral.
Resultados: Los cerdos no mostraron signos de enfermedad entérica y no se detectó excreción de PEDV durante 44 días después de la inoculación (dpi). En la necropsia a los 3 (5 cerdos) o a los 7 dpi (5 cerdos), no se observaron lesiones en los cortes intestinales, que fueron negativos para el antígeno PEDV por inmunohistoquímica. Además, la ausencia de anticuerpos específicos contra IgG o IgA contra el PEDV en muestras séricas o fecales durante 35 ppp indica además la ausencia de infección. La titulación del inóculo sobrante del stock del virus PEDV descongelado y recongelado mostró que el stock del virus conservó su infectividad en el cultivo de células Vero y en la línea celular de enterocitos del intestino delgado porcino IPEC-J2.
Discusión: Se desconocen las razones de la pérdida de infectividad en los cerdos. En conclusión, demostramos que una cepa clásica de PEDV G1a propagada con éxito en cultivos celulares no podía infectar oralmente a 40 lechones.
1 Introducción
El virus de la diarrea epidémica porcina (PEDV), miembro del género Alphacoronavirus de la familia Coronaviridae, es altamente contagioso y causa una enfermedad entérica caracterizada por un inicio agudo de vómitos y diarrea en cerdos de todas las edades (1). Los coronavirus consisten en un ARN monocatenario grande (~30 kb) de sentido positivo, con los primeros dos tercios del genoma codificando proteínas no estructurales involucradas en la replicación del virus y las interacciones del huésped (por ejemplo, evasión inmune), mientras que el tercio restante codifica para cuatro proteínas estructurales principales: espícula (S), envoltura (E), membrana (M) y nucleocápside (N) (2). Sobre la base de las diferencias de aminoácidos en el dominio N-terminal de la proteína S, el PEDV se divide en dos genogrupos, G1 y G2, que pueden dividirse a su vez en G1a (CV777 y otras cepas clásicas), G1b (cepas adaptadas al cultivo celular y otras cepas recombinantes), G2a (cepas de PEDV pandémicas similares a las de EE. UU.), G2b (cepas asiáticas) y G2c [cepas S-INDEL (proteína S variante que contiene inserciones y deleciones) de los EE. UU., Europa y China] (3, 4).
A finales de la década de 1970, se describió en Europa un síndrome diarreico caracterizado por diarrea acuosa aguda en cerdos de todas las edades, la diarrea epidémica porcina (PED), que causó brotes con tasas de mortalidad en lechones lactantes que oscilaron entre el 0% y el 100% (promedio del 50%) y la gravedad de la enfermedad dependió de la camada y la granja (5). Uno de los aislados de PEDV de la década de 1970, CV777, se convirtió en la cepa prototipo de PEDV y se utilizó para estudios de patogénesis en cerdos privados de calostro (6). El PEDV se extendió a Asia en la década de 1980 y, desde 2010, ha causado graves epidemias en muchos países asiáticos, principalmente asociadas a las cepas G2 del PEDV (3, 4, 7).
El G1b se notificó por primera vez en China en 2011, se propagó por Asia y se atribuyó a los brotes de 2015-2016 en granjas de engorde y engorde en Europa (3). En 2013 y 2014 se introdujeron en las Américas, el Caribe y Ucrania cepas aisladas de PEDV G2b y G1b similares a las cepas de PEDV que circulaban en China en 2012 (3, 8). Se han realizado varios estudios de patogénesis con cepas G1b y G2b (9-14), lo que indica que las cepas G1b son menos patógenas que las cepas G2b, aunque los signos clínicos varían considerablemente. Sin embargo, existe información limitada sobre la patogénesis de las cepas clásicas de PEDV en cerdos contemporáneos (15). Este estudio tuvo como objetivo investigar el curso clínico de la enfermedad en cerdos de 10 días de edad inoculados experimentalmente con una cepa clásica de PEDV G1a similar a CV777.
2 Métodos
2.1 Origen del virus
Una cepa G1a de PEDV designada como CV777 se aisló originalmente en Bélgica en la década de 1980 y se obtuvo para este estudio de la Universidad de Utrecht, Países Bajos (16). En 1994, este aislado se propagó por última vez en lechones derivados de cesárea y privados de calostro, los cerdos mostraron diarrea después de la infección y la perfusión intestinal se recolectó y almacenó a -80 °C.
2.2 Propagación y valoración in vitro de virus
Todo el trabajo de laboratorio fue aprobado por el Comité de Bioseguridad de la Universidad Estatal de Iowa (número de aprobación: 14-I-0018-A). El stock viral de PEDV se pasó tres veces (CV777 P3) en células Vero como se describió anteriormente (17), y se produjo un volumen total de 0,6 L de stock de inóculo de virus infeccioso. Para la propagación del virus se utilizó un medio esencial mínimo (MEM) suplementado con suero bovino fetal al 10%, 2 mM de L-glutamina, 0,05 mg/ml de gentamicina, 10 unidades/ml de penicilina, 10 μg/ml de estreptomicina y 0,25 μg/ml de anfotericina, caldo de fosfato de triptosa (0,3%), extracto de levadura (0,02%) y tripsina 250 (5 μg/mL) (17). El stock de virus infeccioso se tituló mediante el método de la unidad formadora de placa (PFU) con un título infeccioso de 5 × 104 UFP/ml y por un método de unidad formadora de fluorescencia (UFF) con un título infeccioso de 3,4 × 104 UFC/ml (17).
2.3 Secuenciación y análisis genómico
Para amplificar la secuencia genómica del stock de virus CV777 P3 de PEDV, se diseñaron cebadores de oligonucleótidos (Tablas suplementarias S1, S2) y se sintetizaron en función de las regiones conservadas entre las secuencias del prototipo CV777 (AF353511) y los aislamientos recientes de PEDV en EE. UU. Los fragmentos que cubren la mayor parte del genoma de PEDV para la cepa CV777 P3 se amplificaron mediante RT-PCR utilizando estos cebadores con ADN polimerasa de alta fidelidad PfuUltra II (Agilent, Santa Clara, CA). Los productos de RT-PCR se purificaron a partir del gel de agarosa con el kit de recuperación de ADN en gel Zymoclean™ (Zymo Research Corp, CA) y se secuenciaron directamente mediante el método Sanger. Los contigs de secuencia se ensamblaron y analizaron utilizando el paquete Lasergene (DNAStar, Inc., Madison, WI). El árbol filogenético de la proteína S se construyó utilizando el método de máxima verosimilitud con pruebas bootstrap de 1.000 réplicas utilizando DNAStar.
2.4 Infección in vitro de células porcinas del intestino delgado
Se utilizó una línea celular epitelial de yeyuno porcino inmortalizada IPEC-J2 (ACC 701, DSMZ, Braunschweig, Alemania) como modelo de infección in vitro. Se inoculó el cincuenta por ciento de cobertura de la monocapa celular IPEC-J2 (paso celular 33) en la pared inferior de placas de 24 pocillos (Corning, Corning, Nueva York, Estados Unidos) con 1 × 104 FFU de PEDV CV777 P3 diluido en 1 mL de medio de propagación PEDV como se ha descrito anteriormente (17), y sección 2.2. Después de 48 h de incubación, se examinó la aparición del efecto citopático (CPE) y posteriormente las células se fijaron con acetona al 80% y se tiñeron con el anticuerpo monoclonal específico de PEDV SD6-29 conjugado con FITC (Medgene, Brookings, Dakota del Sur) en un ensayo de inmunofluorescencia (IFA) (18).
2.5 Infección in vivo en cerdos de 10 días de edad
El protocolo experimental fue aprobado por el Comité Institucional de Cuidado y Uso de Animales de la Universidad Estatal de Iowa (número de aprobación 2-14-7804-S) y por el Centro Nacional de Enfermedades Animales, Comité Institucional de Cuidado y Uso de Animales del USDA-ARS. Cuarenta lechones cruzados de 10 días de edad negativos para PEDV por ELISA (anti-PEDV IgA e IgG) y PCR criados en el Centro Nacional de Enfermedades Animales, USDA-ARS en Ames, Iowa, fueron inoculados con la cepa de virus infecciosos PEDV CV777 P3 como parte de otro estudio (19). Cada cerdo recibió 10 mL de la cepa de inóculo a una dosis de PFU de 5 × 104 por ml por vía oral goteando lentamente el inóculo en la cavidad bucal del cerdo. Después de la inoculación, aproximadamente 10 ml del inóculo del virus restante se congelaron y almacenaron a -80 °C. El grupo no infectado (n = 40, sin exposición al PEDV) y el grupo infectado con la cepa G2b del PEDV US/Colorado/2013 a los 10 días de edad (n = 43) descritos en el estudio original (19) sirvieron como controles negativos y positivos, respectivamente, para el presente estudio.
2.6 Recogida y almacenamiento de muestras
Las muestras de sangre se recogieron en tubos separadores de suero (Fisher Scientific, Pittsburgh, Pensilvania, Estados Unidos) al día siguiente de la inoculación con PEDV (dpi) 0, 7, 14, 24 y 35 centrifugados a 3000 × g durante 10 min a 4 °C. Se recogieron muestras fecales a 35 ppp y se analizaron para detectar anticuerpos IgA anti-PEDV. Los hisopos fecales se recolectaron diariamente con hisopos de poliéster de 0 a 33 y 44 ppp y se almacenaron en tubos de plástico de 5 ml que contenían 1 ml de solución salina estéril. Todas las muestras se almacenaron a -80 °C hasta nuevas pruebas.
2.7 Ensayo de inmunoabsorción ligado a enzimas
Las muestras de suero se analizaron para detectar anticuerpos anti-PEDV IgA e IgG mediante un ensayo interno de inmunoadsorción enzimática indirecta (ELISA) basado en S1 (20, 21) y mediante un ELISA interno basado en células enteras G2b ofrecido en el Laboratorio de Diagnóstico Veterinario de la Universidad Estatal de Iowa (ISU-VDL) (18). Las muestras fecales se analizaron en busca de anticuerpos IgA anti-PEDV mediante un ELISA indirecto basado en S1 (21).
2.8 Extracción de ARN y ensayos de PCR
El ARN se extrajo de suspensiones de hisopos fecales utilizando el kit de ARN/ADN de patógenos MagMax (Applied Biosystems, Life Technologies, Carlsbad, California, Estados Unidos) y un sistema automatizado de extracción de ADN/ARN (Thermo Scientific Kingfisher Flex, Thermo Fisher Scientific, Pittsburgh, Pensilvania, Estados Unidos) de acuerdo con las instrucciones del fabricante. Los extractos se analizaron para detectar el ARN del PEDV mediante una PCR cuantitativa con transcriptasa inversa (RT) basada en el gen N ofrecida en ISU-VDL y mediante una RT-PCR diferencial en tiempo real PEDV G1 y G2 del gen N utilizando el siguiente par de cebadores de detección (PED-NDF: 5′-CGATGATCTGGTGGCTGCTGTGTGT-3′ y PED-NDR: 5′-GGGATGTCTTTGAGGTCACGTTC-3′) y sondas TaqMan (PEDV-G1prob(CV777) CAL Fluor Orange 560-5′-TAAGCAGGAAAAGTCTGACAACAGCGGC-3′-BHQ y PEDV-G2prob FAM-5′-CAAACAGGAAAGGTCTGACAGCAGCGG-3′-BHQ).
2.9 Signos clínicos
Se obtuvo una puntuación diaria de diarrea en cada cerdo observando a los cerdos una vez cada mañana de 0 a 8 y en 10, 12, 14, 17, 18, 21, 24 y 35. La composición fecal se puntuó de 0 a 3 (0 = heces normales, 1 = heces húmedas, 2 = heces pastosas, 3 = heces acuosas).
2.10 Necropsia y recogida de muestras
Cinco cerdos fueron sacrificados a los ppp 3, 7 y 14 y los cerdos restantes fueron sacrificados a los ppp 35 por sobredosis de pentobarbital sódico (100 mg/kg de Fatal-plus, Vortech Pharmaceuticals, LTD, Dearborn, Michigan, Estados Unidos). La muerte se confirmó asegurando el cese de los movimientos respiratorios y cardiovasculares mediante observación antes de la necropsia de los cerdos. En la necropsia se recogieron ocho secciones de intestino delgado y tres secciones de intestino grueso y se fijaron en formol tamponado neutro al 10% y se procesaron de forma rutinaria para su examen histológico.
2.11 Lesiones microscópicas e inmunohistoquímica
Las lesiones microscópicas fueron evaluadas por un patólogo veterinario ciego al estado de tratamiento (PGH) y evaluadas para detectar la presencia de inflamación, atrofia de vellosidades y necrosis. El antígeno específico de PEDV fue detectado por una IHQ en secciones seleccionadas de secciones intestinales fijadas en formol e incluidas en parafina utilizando anticuerpos monoclonales específicos para PEDV (BioNote, Hwaseong-si, Gyeonggi-do, República de Corea) como se describe (22, 23).
3 Resultados
3.1 El inóculo del virus G1a PEDV utilizado en este estudio es una cepa clásica relacionada con CV777
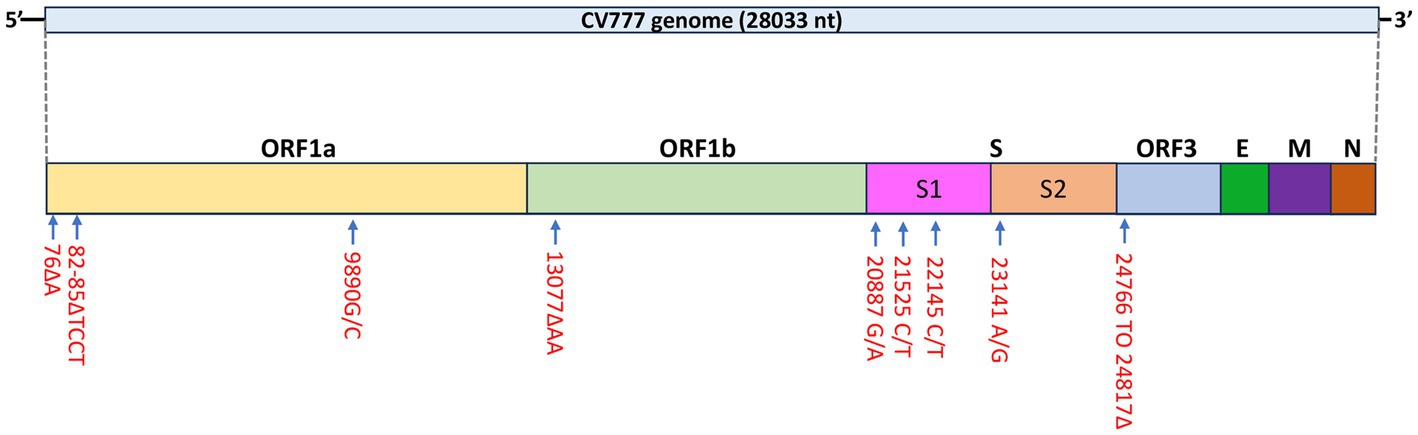
La secuencia obtenida para el genoma casi completo del PEDV (GenBank No. OR348434) indica que el virus G1a PEDV del pasaje 3 está estrechamente relacionado con el prototipo CV777 [GenBank No. AF353511, 99,9% de identidad de nucleótidos (nt)], y en menor medida a aislados asiáticos de PEDV G1a como CHM2013 (KM887144,1, China, 2013, 99,4% de identidad nt) y AVCT12 (LC053455,1, Tailandia, 2015, 99,3% de identidad nt). Al utilizar la secuencia clásica de la cepa CV777 como referencia, se identificaron deleciones en la región no codificante 5′ del genoma, incluida una deleción en la posición nt 72 y una deleción TCCT en la posición nt 82-85, que es similar a la cepa G1a CHM2013 y la cepa AVCT12. Hubo dos poblaciones virales en el 9890 nt del ORF1a en el stock CV777 P3. La mayoría de la población de la secuencia tenía una mutación G a C, que da lugar a una mutación de aminoácidos Glu (E) a Asp (D) en la proteasa similar a 3C. Una búsqueda en GenBank BLAST del aminoácido proteasa similar a CV777 P3 3C mostró que la mutación E/D en el virus CV777 P3 es única entre los genomas conocidos de PEDV. Se encuentra dentro de una cadena helicoidal en lugar de en el sitio catalítico. La Figura 1 muestra su posición en la estructura 3D de la proteasa similar a PEDV 3C, así como una alineación de fragmentos de ADNc de virus relevantes adaptados de estudios previos (24, 25). Además, el ORF1b también tuvo una deleción en 13077-13078.
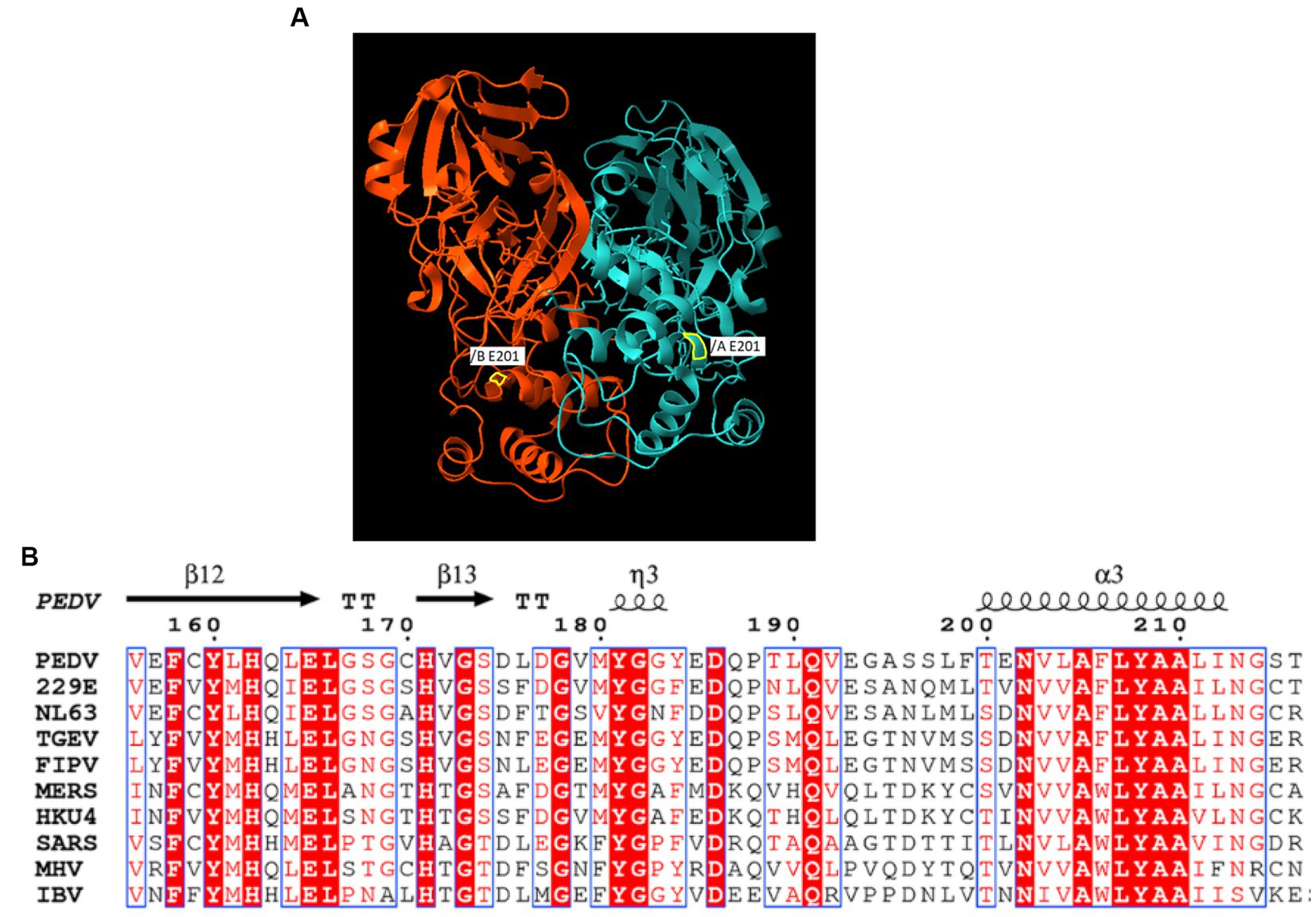 Figura 1. (A) La estructura cristalina de rayos X de la estructura de proteasa similar a PEDV-3C (entrada PDB 5HYO). Una línea de borde amarilla indica la ubicación de la mutación E/D. (B) Alineación de la región adyacente de la mutación E/D de la proteasa similar a 3C de los coronavirus.
Figura 1. (A) La estructura cristalina de rayos X de la estructura de proteasa similar a PEDV-3C (entrada PDB 5HYO). Una línea de borde amarilla indica la ubicación de la mutación E/D. (B) Alineación de la región adyacente de la mutación E/D de la proteasa similar a 3C de los coronavirus.
Se encontraron un total de cuatro mutaciones en el gen S: 20887 G/A, 21525 C/T, 22145 C/T y 23141 A/G y todas las mutaciones dieron lugar a cambios de aminoácidos excepto la mutación en la posición nt 21.525 (Figura 2).
Al igual que las cepas CHM2013 y AVCT12, hubo una deleción de 52 nt entre las posiciones nt 24766 y 24817, lo que resultó en el truncamiento de la proteína S por 7 aminoácidos y la deleción del codón de inicio de ORF3. De acuerdo con la secuencia, la deleción hace que ORF3 se trunque en 70 aminoácidos, lo que lleva a la producción de péptidos que constan de 154 aminoácidos. También se encontró una mutación C/T en la posición nt 25523. En la Figura 3 se muestra un árbol filogenético basado en las secuencias de aminoácidos de la proteína S.
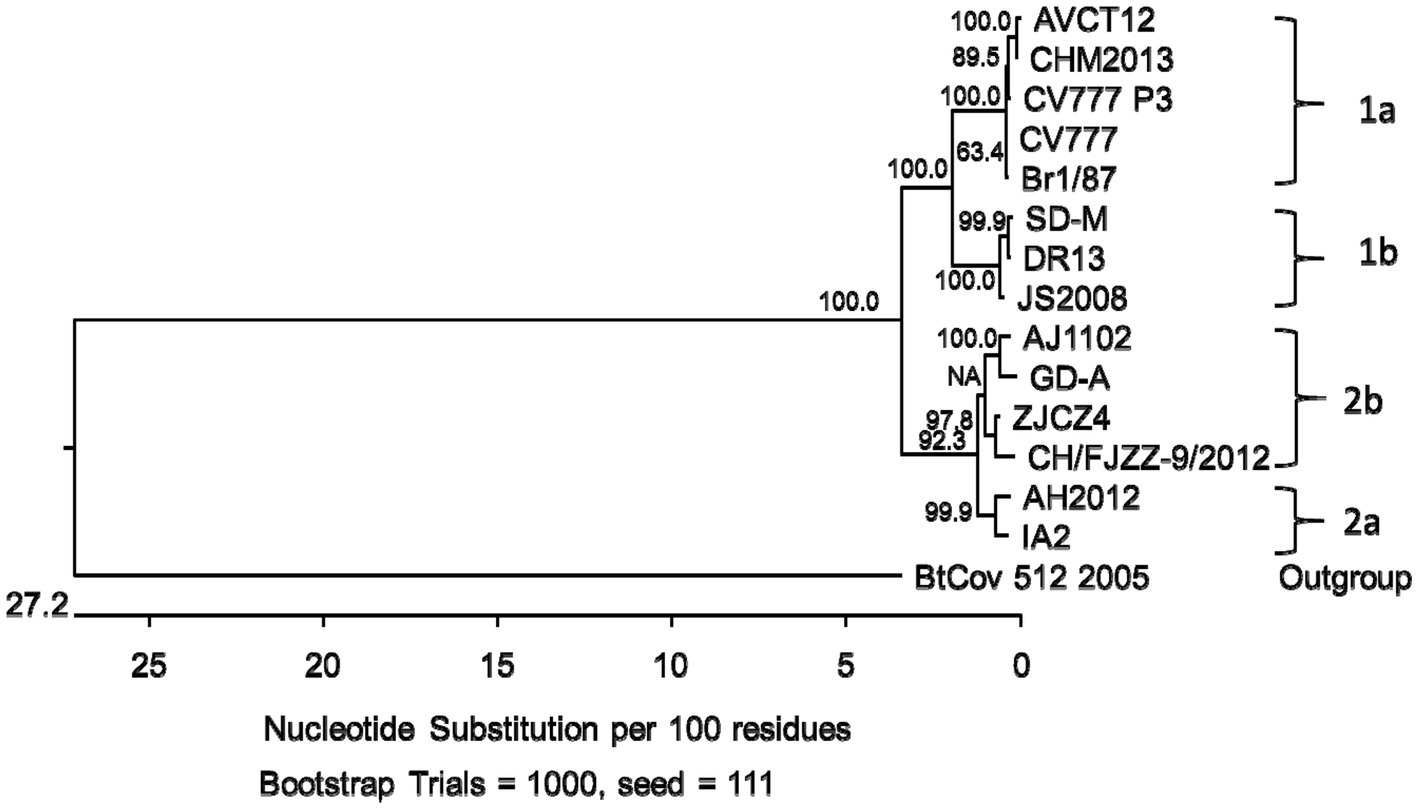 Figura 3. Árbol filogenético basado en el gen de la espícula (S) traducido secuencias de aminoácidos de cepas del virus de la diarrea epidémica porcina de los genogrupos 1a, 1b, 2a y 2b. Los valores de arranque se representan en los nodos clave.
Figura 3. Árbol filogenético basado en el gen de la espícula (S) traducido secuencias de aminoácidos de cepas del virus de la diarrea epidémica porcina de los genogrupos 1a, 1b, 2a y 2b. Los valores de arranque se representan en los nodos clave.
3.2 La cepa G1a de PEDV infectó células epiteliales del intestino delgado porcina in vitro
El PEDV CV777 P3 fue infeccioso en células Vero y en células epiteliales del intestino delgado porcino IPEC-J2. Después de 48 h de inoculación, se observó una ECP típica inducida por PEDV, sincitios de células multifocales que se presentan como células multinucleadas coalescentes con citoplasma agrandado, en la monocapa de células IPEC-J2 inoculadas con PEDV CV777 (Figura 4). Además, las proteínas virales de PEDV localizadas en el citoplasma de la célula IPEC-J2 infectada se marcaron y visualizaron con anticuerpos monoclonales específicos de PEDV conjugados con FITC (Figura 4).
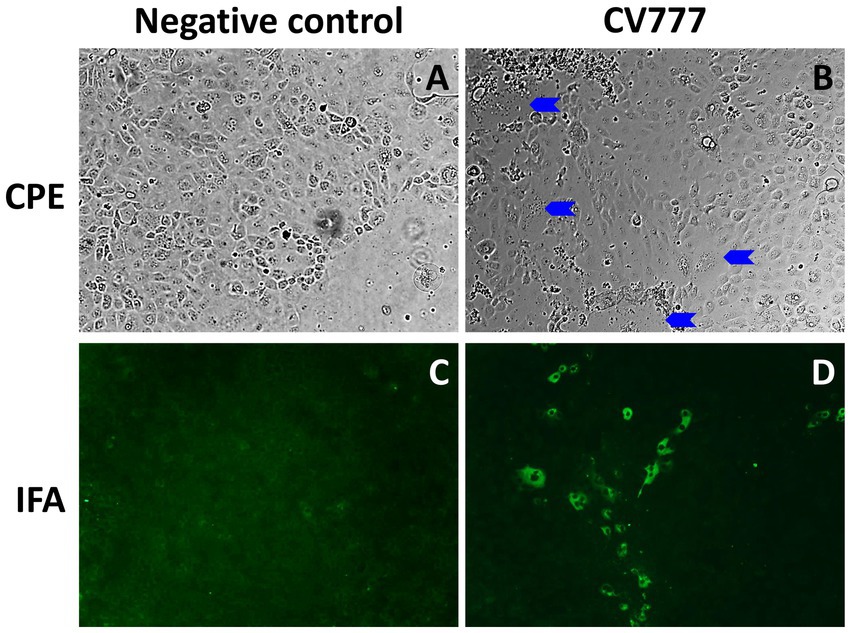 Figura 4. Tinción con efecto citopático (CPE) y tinción por inmunofluorescencia (IFA) en células IPEC-J2 a las 48 h después de la inoculación. En las células IPEC-J2 inoculadas simuladamente no se observó tinción de CPE (A) o IFA (C). La célula IPEC-J2 infectada con PEDV CV777 presentó CPE (B, flechas azules: cuerpos sincitiales) y tinción de IFA (D) (aumento de ×40).
Figura 4. Tinción con efecto citopático (CPE) y tinción por inmunofluorescencia (IFA) en células IPEC-J2 a las 48 h después de la inoculación. En las células IPEC-J2 inoculadas simuladamente no se observó tinción de CPE (A) o IFA (C). La célula IPEC-J2 infectada con PEDV CV777 presentó CPE (B, flechas azules: cuerpos sincitiales) y tinción de IFA (D) (aumento de ×40).
3.3 Fracaso en la infección experimental de cerdos con la cepa G1a PEDV
Después de la inoculación, los cerdos fueron monitoreados diariamente para detectar signos clínicos y no hubo diarrea u otro signo de infección por PEDV en los lechones inoculados de acuerdo con la ausencia de detección de ARN de PEDV en hisopos fecales durante 35 días después de la inoculación. No se observaron lesiones compatibles con enfermedad entérica en la necropsia ni en el examen microscópico de los ppp 3 o 7. Además, no se detectó antígeno específico de PEDV en las secciones intestinales. No se detectaron anticuerpos específicos contra IgG o IgA contra el PEDV mediante dpi 35 en muestras de suero y no se detectaron anticuerpos específicos contra el PEDV IgA mediante el dpi 35 en las heces. En conjunto, estos resultados indican que los lechones no estaban infectados con la cepa relacionada con PEDV CV777 P3.
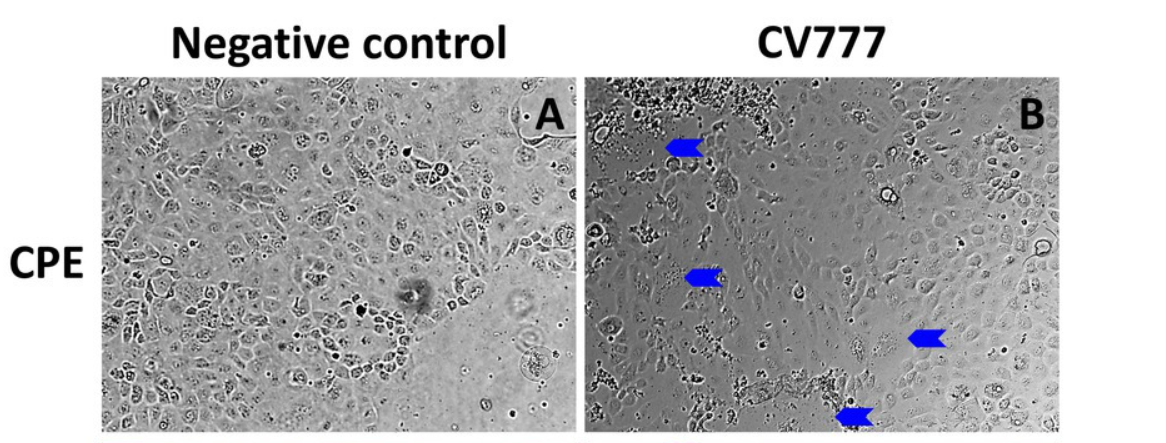
Para descartar la posibilidad de una posible pérdida de infectividad de la reserva viral durante el almacenamiento, la descongelación y la manipulación, probamos y volvimos a ajustar el inóculo sobrante descongelado y recongelado en células Vero en paralelo con el inóculo viral congelado. El inóculo sobrante descongelado aún conservaba su infectividad en las células Vero con títulos infecciosos similares en comparación con el stock viral no congelado (Figura 5).
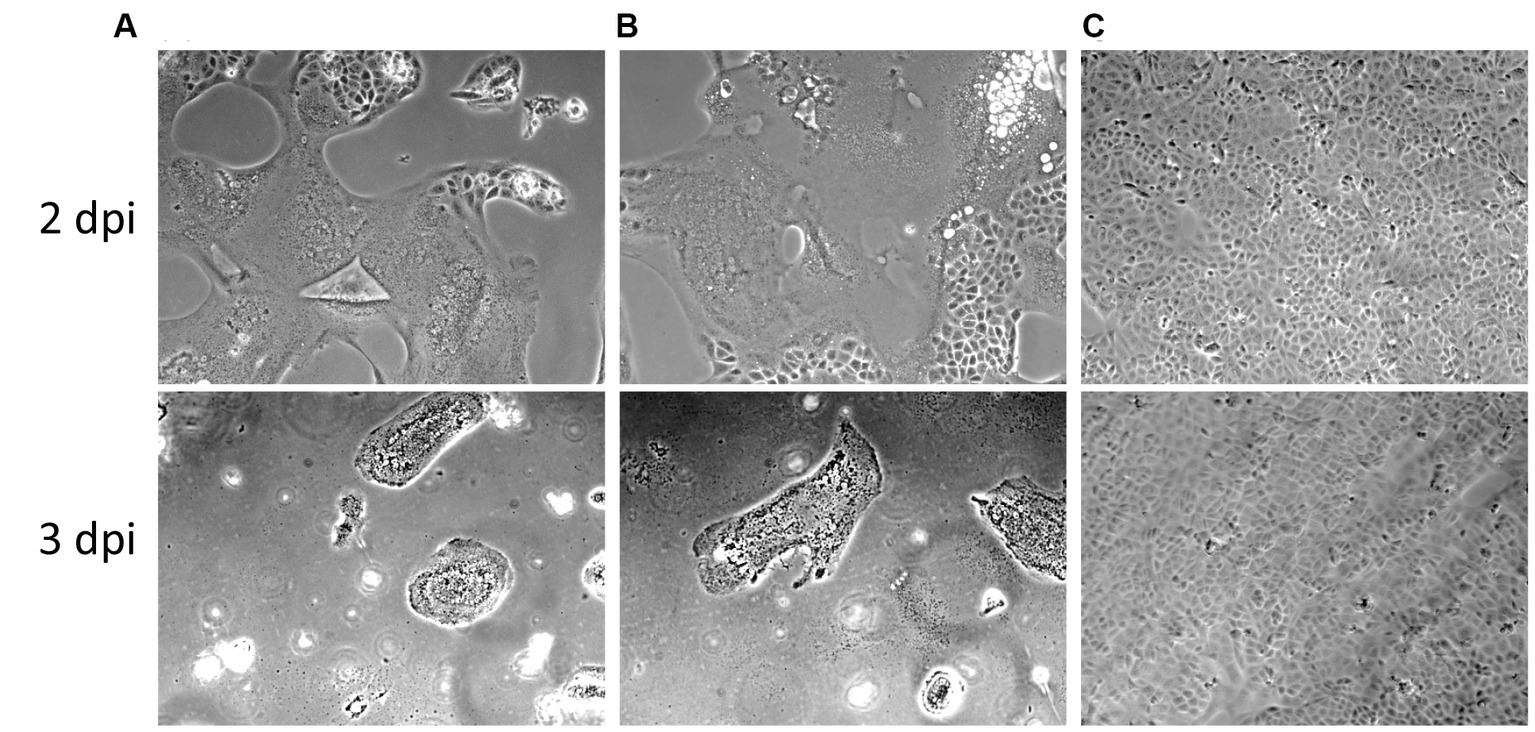 Figura 5. Efectos citopáticos del PEDV CV777 P3 utilizado en este estudio en células Vero a los días post inoculación (dpi) 2 y 3. (A) Stock viral original. (B) Restos de inóculo previamente utilizados para inocular 40 cerdos de 10 días de edad. (C) Simular células infectadas.
Figura 5. Efectos citopáticos del PEDV CV777 P3 utilizado en este estudio en células Vero a los días post inoculación (dpi) 2 y 3. (A) Stock viral original. (B) Restos de inóculo previamente utilizados para inocular 40 cerdos de 10 días de edad. (C) Simular células infectadas.
4 Discusión
En este estudio realizado en 2014, descubrimos que una cepa europea de PEDV aislada inicialmente de cerdos belgas en la década de 1970 (16), posteriormente suministrada a laboratorios de investigación para trabajos in vitro, no logró infectar a ninguno de los 40 lechones de 10 días de edad inoculados experimentalmente con una cepa infecciosa recién preparada del virus. La titulación de la cepa de virus PEDV que se utilizó para inocular a los lechones y que se había congelado demostró que la cepa de virus conservaba su infectividad in vitro cuando se valoraba en células Vero. Se ha informado de la resistencia de la edad a la enfermedad inducida por la infección por PEDV y los lechones neonatos (de 1 a 9 días de edad) a menudo muestran signos clínicos más graves que los cerdos destetados (de 3 a 4 semanas de edad) (10, 18). Los lechones (cerdos cruzados de 10 días de edad) utilizados en este documento deben ser muy susceptibles a la infección por PEDV. Además, lechones contemporáneos de la misma fuente y alojados en las mismas instalaciones se infectaron con éxito con la cepa G2b Colorado PEDV cuando se utilizó una dosis y una ruta infecciosa similares en un estudio anterior (19). La razón de la falta de infectividad del PEDV relacionada con el CV777 en los cerdos es desconocida e inesperada. La cepa G1a Br1/87 aislada en 1987 en Gran Bretaña, estrechamente relacionada con el prototipo CV777 (26), se utilizó con éxito para infectar por vía oral a cinco cerdos convencionales de 5 semanas de edad en 2014 (15). Los cerdos infectados experimentalmente con la cepa Br1/87 tuvieron diarrea leve a moderada y bajos niveles de excreción viral intermitente en hisopos fecales durante 1 a 3 semanas (15). Además, la cepa CV777 atenuada en cultivo celular y de tipo salvaje a una dosis similar (2,55 × 105 FFU/cerdo) utilizados en este documento se han utilizado con éxito para infectar lechones convencionales de 11 días de edad a principios de la década de 2000 (27).
Los estudios realizados a finales de la década de 1970 y principios de la década de 1980 con cepas clásicas de G1a (6, 28) y un estudio con una cepa asiática de PEDV KPEDV-9 de la década de 1990 (29) informaron resultados clínicos similares a los que se han reportado en los últimos años para las cepas contemporáneas de PEDV G1b y G2 (9, 10). En lechones CDCD de 2 a 3 días de edad infectados experimentalmente con la cepa CV777 de PEDV en la década de 1980, se observaron partículas virales en el yeyuno a partir de las 18 h después de la infección (HPI) y se observaron signos clínicos caracterizados por diarrea acuosa profusa a partir de las 24 a 36 hpi (6, 28). Dos horas después del inicio de los signos clínicos (aproximadamente 24 hpi), se pudo observar una exfoliación de los enterocitos que condujo a una atrofia vellositaria severa, mientras que el epitelio de las criptas no se vio afectado (28). Los cerdos que no murieron por deshidratación de 2 a 4 días después del inicio de la diarrea se recuperaron después de una semana (6).
Aunque el virus CV777 P3 está estrechamente relacionado con la tinción prototipo de PEDV G1a CV777, las mutaciones observadas en el gen de la espícula y en la región no codificante 5′ son similares a las observadas en las cepas asiáticas contemporáneas CHM2013 y AVCT12, lo que no es sorprendente ya que desde la década de 1990 se han utilizado en Asia vacunas vivas atenuadas modificadas similares a CV777 y Br1/87 y estas cepas siguen circulando en esa región (3). Es posible que se hayan adquirido mutaciones durante los pasajes secuenciales in vivo a los que se sometió la cepa CV777 original a lo largo de los años. Sin embargo, se desconoce por qué los lechones de 10 días de edad eran resistentes a la cepa similar a CV777 después de haber sido pasados solo tres veces in vitro, especialmente porque el virus CV777 P3 que se utilizó en el estudio con cerdos es totalmente infeccioso en las células Vero y en las células epiteliales del intestino delgado IPEC-J2. Se ha demostrado que un PEDV modificado genéticamente con un ORF3 delecionado, similar al patrón de ORF3 delecionado encontrado en el presente estudio, infecta con éxito a lechones gnotobióticos produciendo resultados letales de la enfermedad e infecta células Vero (30), lo que indica que el ORF3 no es esencial para la replicación in vivo o in vitro. Además, se ha demostrado que la pérdida de la porción del dominio N de la proteína S se correlaciona con una pérdida de tropismo entérico para algunos coronavirus (31). Por ejemplo, una variante natural del virus de la gastroenteritis transmisible (TGEV), el coronavirus respiratorio porcino (PRCV) perdió su tropismo entérico y se replica principalmente en el tracto respiratorio después de la pérdida de la porción del dominio N de la proteína S (32). La pérdida de tropismo entérico para el TGEV se asoció específicamente con la pérdida de la actividad de unión siálica que reside en este dominio (31).
Curiosamente, se pudieron encontrar dos poblaciones de virus en el stock de virus CV777 P3, y la mayoría presentó una mutación G a C en la posición 9890, lo que resultó en una mutación en el gen de la proteinasa similar a 3C que condujo a una proteína truncada. Esta mutación es similar a la mutación descrita en el virus de la peritonitis infecciosa felina (FIPV), que se replica sistémicamente en gatos y ha evolucionado como una deleción de la porción del dominio N de la proteína S del coronavirus entérico felino (FECV) que se replica en enterocitos (33). Se ha demostrado que se requiere una proteinasa completa similar a la 3C para la replicación del FECV en el epitelio intestinal. Aunque se desconoce el efecto de esta mutación sobre la infectividad del PEDV en cerdos, puede haber afectado al tropismo del virus y contribuido al fracaso de la infección oral en cerdos. Por lo tanto, sería interesante determinar aún más la función de la mutación E/D en la proteinasa similar a 3C de la cepa de virus CV777 P3 y si afecta a la infección por virus en cerdos.
5 Conclusión
Demostramos que una cepa clásica de PEDV G1a, aunque infecciosa en células Vero e IPEC-J2 in vitro, fue incapaz de infectar a 40 lechones por vía oral de inoculación. El mecanismo de la pérdida de infectividad en su huésped natural sigue siendo desconocido.
Declaración de disponibilidad de datos
Los conjuntos de datos de secuencias presentados en este estudio se pueden encontrar en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar a continuación: https://www.ncbi.nlm.nih.gov/ y OR348434. Otros conjuntos de datos presentados están disponibles a pedido.
Declaración ética
El estudio en animales fue aprobado por el Comité Institucional de Cuidado y Uso de Animales de la Universidad Estatal de Iowa y el Comité Institucional de Cuidado y Uso de Animales del USDA-ARS. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
PG: Metodología, Redacción – borrador original, Redacción – revisión y edición. DC: Análisis formal, Metodología, Redacción – revisión y edición. C-TX: Metodología, Redacción – revisión y edición. QC: Metodología, Redacción – revisión y edición. KL: Conceptualización, Obtención de Financiamiento, Metodología, Recursos, Redacción – revisión y edición. BB: Recursos, Redacción – revisión y edición. X-JM: Metodología, Redacción – revisión y edición. PH: Metodología, Redacción – revisión y edición. TO: Conceptualización, Metodología, Administración de Proyectos, Supervisión, Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. El financiamiento para este estudio fue proporcionado por el USDA-ARS (KL) y un Acuerdo Interagencial USDA-APHIS 14-9419-340 (KL).
Reconocimientos
Los autores agradecen a Deborah Adolphson, Brett Burke, Karina Sonalio y Melisa Spadaro por su ayuda con el trabajo con animales.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
El/los autor/es declararon, en el momento de la presentación, ser miembro del consejo editorial de Frontiers. Esto no tuvo ningún impacto en el proceso de revisión por pares ni en la decisión final.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1279162/full#supplementary-material
Referencias
1. Jung, K, Saif, LJ y Wang, Q. Virus de la diarrea epidémica porcina (PEDV): una actualización sobre etiología, transmisión, patogénesis y prevención y control. Virus Res. (2020) 286:198045. doi: 10.1016/j.virusres.2020.198045
Resumen de PubMed | Texto completo de CrossRef | Google Académico
2. Woo, PCY, de Groot, RJ, Haagmans, B, Lau, SKP, Neuman, BW, Perlman, S, et al. Perfil de taxonomía de virus ICTV: Coronaviridae 2023. J Gen Virol. (2023) 104. doi: 10.1099/jgv.0.001843
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Guo, J, Fang, L, Ye, X, Chen, J, Xu, S, Zhu, X, et al. Análisis evolutivo y genotípico de cepas del virus de la diarrea epidémica porcina mundial. Transbound Emerg Dis. (2019) 66:111–8. doi: 10.1111/tbed.12991
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. Kim, SJ, Nguyen, VG, Huynh, TM, Park, YH, Park, BK y Chung, HC. Caracterización molecular del virus de la diarrea epidémica porcina y su nueva clasificación genética basada en el gen de la nucleocápside. Virus. (2020) 12:790. doi: 10.3390/v12080790
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Pensaert, MB y Martelli, P. Diarrea epidémica porcina: una retrospectiva desde Europa y temas de debate. Virus Res. (2016) 226:1–6. doi: 10.1016/j.virusres.2016.05.030
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Debouck, P, y Pensaert, M. Infección experimental de cerdos con un nuevo coronavirus entérico porcino, CV 777. Am J Vet Res. (1980) 41:219–23.
7. Wang, D, Fang, L y Xiao, S. Diarrea epidémica porcina en China. Virus Res. (2016) 226:7–13. doi: 10.1016/j.virusres.2016.05.026
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Hanke, D, Pohlmann, A, Sauter-Louis, C, Hoper, D, Stadler, J, Ritzmann, M, et al. Diarrea epidémica porcina en Europa: análisis detallados de la dinámica de la enfermedad y epidemiología molecular. Virus. (2017) 9:177. doi: 10.3390/v9070177
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Chen, Q, Gauger, PC, Stafne, MR, Thomas, JT, Madson, DM, Huang, H, et al. Comparación de la patogénesis entre el prototipo del virus de la diarrea epidémica porcina de los Estados Unidos y las cepas de la variante S-INDEL en lechones neonatos convencionales. J Gen Virol. (2016) 97:1107–21. doi: 10.1099/jgv.0.000419
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Jung, K, Annamalai, T, Lu, Z y Saif, LJ. Patogénesis comparativa de la cepa PC21A del virus de la diarrea epidémica porcina (PEDV) de EE. UU. en lechones lactantes convencionales de 9 días de edad frente a cerdos destetados de 26 días de edad. Microbiol veterinario. (2015) 178:31–40. doi: 10.1016/j.vetmic.2015.04.022
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Lin, CM, Annamalai, T, Liu, X, Gao, X, Lu, Z, El-Tholoth, M, et al. Infección experimental de un virus de diarrea epidémica porcina con deleción por inserción de espiga en lechones lactantes convencionales y protección cruzada con la infección original por PEDV de EE. UU. Res. Veterinaria. (2015) 46:134. doi: 10.1186/s13567-015-0278-9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Yamamoto, R, Soma, J, Nakanishi, M, Yamaguchi, R, y Niinuma, S. Aislamiento e inoculación experimental de una cepa S INDEL del virus de la diarrea epidémica porcina en Japón. Res Vet Sci. (2015) 103:103–6. doi: 10.1016/j.rvsc.2015.09.024
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Lazov, CM, Lohse, L, Belsham, GJ, Rasmussen, TB y Botner, A. Infección experimental de cerdos con virus de diarrea epidémica porcina europea reciente. Virus. (2022) 14:2751. doi: 10.3390/v14122751
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Gallien, S, Moro, A, Lediguerher, G, Catinot, V, Paboeuf, F, Bigault, L, et al. Diseminación limitada de una cepa S-InDel del virus de la diarrea epidémica porcina (PEDV) en el semen y preguntas sobre la infectividad del virus detectado. Microbiol veterinario. (2019) 228:20–5. doi: 10.1016/j.vetmic.2018.09.025
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Lohse, L, Krog, JS, Strandbygaard, B, Rasmussen, TB, Kjaer, J, Belsham, GJ, et al. Infección experimental de cerdos jóvenes con una cepa europea temprana del virus de la diarrea epidémica porcina y una cepa reciente de EE. UU. Transbound Emerg Dis. (2017) 64:1380–6. doi: 10.1111/tbed.12509
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Egberink, HF, Ederveen, J, Callebaut, P, y Horzinek, MC. Caracterización de las proteínas estructurales del virus de la diarrea epizoótica porcina, cepa CV777. Am J Vet Res. (1988) 49:1320–4.
17. Chen, Q, Li, G, Stasko, J, Thomas, JT, Stensland, WR, Pillatzki, AE, et al. Aislamiento y caracterización de los virus de la diarrea epidémica porcina asociados con el brote de la enfermedad de 2013 entre los cerdos en los Estados Unidos. J Clin Microbiol. (2014) 52:234–43. doi: 10.1128/JCM.02820-13
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Thomas, JT, Chen, Q, Gauger, PC, Gimenez-Lirola, LG, Sinha, A, Harmon, KM, et al. Efecto de las dosis infecciosas del virus de la diarrea epidémica porcina en los resultados de la infección en cerdos neonatos y destetados convencionales naïve. PLoS Uno. (2015) 10:e0139266. doi: 10.1371/journal.pone.0139266
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Gerber, PF, Xiao, CT, Lager, K, Crawford, K, Kulshreshtha, V, Cao, D, et al. Aumento de la frecuencia de diseminación y lesiones del virus de la diarrea epidémica porcina en lechones en comparación con los lechones de destete e inmunidad protectora en cerdos de destete después de la reexposición homóloga. Res. Veterinaria. (2016) 47:118. doi: 10.1186/s13567-016-0402-5
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Gerber, PF, Gong, Q, Huang, YW, Wang, C, Holtkamp, D, y Opriessnig, T. Detección de anticuerpos contra el virus de la diarrea epidémica porcina en suero y calostro mediante ELISA indirecto. Veterinario J. (2014) 202:33–6. doi: 10.1016/j.tvjl.2014.07.018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Gerber, PF y Opriessnig, T. Detección de anticuerpos de inmunoglobulina (Ig) a contra el virus de la diarrea epidémica porcina (PEDV) en muestras fecales y séricas. MétodosX. (2015) 2:368–73. doi: 10.1016/j.mex.2015.10.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Stevenson, GW, Hoang, H, Schwartz, KJ, Burrough, ER, Sun, D, Madson, D, et al. Aparición del virus de la diarrea epidémica porcina en los Estados Unidos: signos clínicos, lesiones y secuencias genómicas virales. J Vet Diagn Investig. (2013) 25:649–54. doi: 10.1177/1040638713501675
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Kim, O, Chae, C, y Kweon, C-H. Detección inmunohistoquímica basada en anticuerpos monoclonales del antígeno del virus de la diarrea epidémica porcina en tejidos intestinales fijados en formol e incluidos en parafina. J Vet Diagn Investig. (1999) 11:458–62. doi: 10.1177/104063879901100512
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. St John, SE, Anson, BJ y Mesecar, AD. Estructura de rayos X e inhibición de la proteasa similar a 3C del virus de la diarrea epidémica porcina. Sci Rep. (2016) 6:25961. doi: 10.1038/srep25961
25. Ye, G, Wang, X, Tong, X, Shi, Y, Fu, ZF y Peng, G. Base estructural para inhibir la replicación del virus de la diarrea epidémica porcina con el inhibidor de la proteasa similar a 3C GC376. Virus. (2020) 12:240. doi: 10.3390/v12020240
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Bridgen, A, Duarte, M, Tobler, K, Laude, H y Ackermann, M. La determinación de la secuencia del gen de la proteína de la nucleocápside del virus de la diarrea epidémica porcina confirma que este virus es un coronavirus relacionado con el coronavirus humano 229E y el virus de la gastroenteritis transmisible porcina. J Gen Virol. (1993) 74:1795–804. doi: 10.1099/0022-1317-74-9-1795
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. de Arriba, ML, Carvajal, A, Pozo, J, y Rubio, P. Respuestas y protección de anticuerpos específicos de isotipo sistémico y mucosa en cerdos convencionales expuestos al virus de la diarrea epidémica porcina virulenta o atenuada. Veterinario Inmunol Inmunopatíaco. (2002) 85:85–97. doi: 10.1016/s0165-2427(01)00417-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Coussement, W, Ducatelle, R, Debouck, P y Hoorens, J. Patología de la enteritis experimental por coronavirus CV777 en lechones. I. Estudio histológico e histoquímico. Veterinario Pathol. (1982) 19:46–56. doi: 10.1177/030098588201900108
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Kweon, CH, Kwon, BJ, Lee, JG, Kwon, GO y Kang, YB. Derivación del virus de la diarrea epidémica porcina atenuada (PEDV) como candidato vacunal. Vacuna. (1999) 17:2546–53. doi: 10.1016/s0264-410x(99)00059-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Beall, A, Yount, B, Lin, CM, Hou, Y, Wang, Q, Saif, L, et al. Caracterización de un clon patogénico de ADNc de longitud completa y modelo de transmisión para la cepa PC22A del virus de la diarrea epidémica porcina. mBio. (2016) 7:E01451–15. doi: 10.1128/mBio.01451-15
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Li, W, van Kuppeveld, FJM, He, Q, Rottier, PJM, y Bosch, BJ. Entrada celular del virus de la diarrea epidémica porcina. Virus Res. (2016) 226:117–27. doi: 10.1016/j.virusres.2016.05.031
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Cox, E, Pensaert, MB, Callebaut, P, y van Deun, K. Replicación intestinal de un coronavirus respiratorio porcino estrechamente relacionado antigénicamente con el virus de la gastroenteritis transmisible entérica. Microbiol veterinario. (1990) 23:237–43. doi: 10.1016/0378-1135(90)90154-N
33. Kipar, A, y Meli, ML. Peritonitis infecciosa felina: ¿sigue siendo un enigma? Veterinario Pathol. (2014) 51:505–26. doi: 10.1177/0300985814522077
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: virus de la diarrea epidémica porcina, PEDV, cepa CV777, atenuación del virus, modelo porcino
Cita: Gerber PF, Cao D, Xiao C-T, Chen Q, Lager K, Bosch BJ, Meng X-J, Halbur PG y Opriessnig T (2023) Fracaso al infectar experimentalmente lechones de 10 días de edad con un stock infeccioso propagado por cultivo celular de un virus de diarrea epidémica porcina de genotipo 1a clásico. Frente. Vet. Sci. 10:1279162. doi: 10.3389/fvets.2023.1279162
Recibido: 17 de agosto de 2023; Aceptado: 20 de octubre de 2023;
Publicado: 16 noviembre 2023.
Editado por:
Shixing Yang, Universidad de Jiangsu, China
Revisado por:
Naidong Wang, Universidad Agrícola de Hunan, China
Livia De Paolis, Instituto Zooprofiláctico Experimental para Piamonte, Liguria y Valle de Aosta (IZSPLVA), Italia
Derechos de autor © 2023 Gerber, Cao, Xiao, Chen, Lager, Bosch, Meng, Halbur y Opriessnig. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Priscilla F. Gerber, de priscilla.gerber@cityu.edu.hk; Tanja Opriessnig, de Tanja.opriessnig@moredun.ac.uk años; tanjaopr@iastate.edu
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía