Generación y caracterización de un anticuerpo monoclonal contra una proteína del virus de la peste porcina africana
Generación y caracterización de un anticuerpo monoclonal contra una proteína del virus de la peste porcina africana codificada por el gen A137R
 Carissa Embury-Hyatt
Carissa Embury-Hyatt  Estella Moffat
Estella Moffat  Dmytro Zhmendak
Dmytro Zhmendak  Cassidy N. G. Erdelyan
Cassidy N. G. Erdelyan  Brad Collignon
Brad Collignon  Kalhari Goonewardene
Kalhari Goonewardene  Aruna Ambagala*
Aruna Ambagala*  Ming Yang*
Ming Yang*- Centro Nacional de Enfermedades de Animales Extranjeros, Winnipeg, MB, Canadá
La actual pandemia de peste porcina africana (PPA) sigue teniendo un gran impacto en la producción y el comercio mundial de carne de cerdo. Dado que la PPA no se puede distinguir clínicamente de otras fiebres hemorrágicas porcinas, el diagnóstico de laboratorio específico de la PPA es fundamental. Por lo tanto, los anticuerpos monoclonales (mAb) específicos del virus de la PPA (PPA) son fundamentales para el desarrollo de diagnósticos de laboratorio. En este estudio, reportamos un mAb específico para el virus de la peste porcina africana, F88ASF-55, que fue generado y caracterizado. Este mAb reconoce la proteína codificada por ASFV A137R (pA137R). Los resultados del mapeo de epítopos revelaron un epítopo lineal altamente conservado reconocido por este mAb, correspondiente a los aminoácidos 111-125 de pA137R. Exploramos el uso potencial de este mAb en aplicaciones diagnósticas. Utilizando F88ASF-55 como anticuerpo de detección, se detectaron seis cepas de PPA en un ensayo de inmunoadsorción enzimática (ELISA) con un fondo bajo. En los ensayos de inmunohistoquímica (IHQ), este AcM reconoció específicamente los antígenos del virus de la PPA en los ganglios linfáticos submandibulares de animales infectados experimentalmente con diferentes cepas del virus de la PPA. Aunque no todos los genotipos del virus de la PPA se analizaron en este estudio, según el epítopo conservado del virus de la PPA al que se dirige F88ASF-55, tiene el potencial de detectar múltiples genotipos del virus de la PPA. En conclusión, este mAb específico de pA137R de PPA recientemente generado tiene un valor potencial en el desarrollo de herramientas de diagnóstico de PPA. Se puede utilizar en ELISA, IHQ y, posiblemente, ensayos de tiras inmunocromatográficas para la detección del virus de la PPA. También sugiere que pA137R puede ser una buena diana para los ensayos de diagnóstico para detectar la infección por el virus de la PPA.
Introducción
La peste porcina africana (PPA) es una enfermedad hemorrágica viral letal de los cerdos domésticos, y se notificó por primera vez en África oriental en 1921. Esta propagación mundial continua e ininterrumpida sigue teniendo un gran impacto en la producción y el comercio mundial de carne de cerdo (1). La PPA es causada por el virus de la peste porcina africana (PPA), el único virus perteneciente a la familia Asfarviridae del género Asfivirus (2, 3). El virus de la PPA es un virus de ADN bicatenario grande y estructuralmente complejo, con una simetría icosaédrica y una envoltura lipídica interna y externa (3). La partícula viral de PPA consta de varias capas concéntricas que contribuyen a su estructura; a partir del nucleoide que contiene el material nuclear, que está rodeado por la gruesa capa del núcleo de la proteína, y una envoltura lipídica interna circundante que finalmente está encerrada por una cápside (4). El virus de la peste porcina africana se clasifica en 24 genotipos basados en B646L, que codifican la proteína estructural p72. Las cepas del virus de la peste porcina africana pueden dividirse en ocho serogrupos en función de la inhibición de la hemadsorción mediada por anticuerpos (5). Hasta el momento, no hay ninguna vacuna o antiviral disponible comercialmente (6). El virus de la peste porcina africana se originó en el África subsahariana, donde sigue siendo endémico. Sin embargo, tras la introducción en Georgia en 2007, el virus de la PPA se extendió posteriormente a Rusia y Europa (1). En 2018, China notificó el primer brote de PPA; desde entonces, 16 países de Asia-Pacífico han notificado hasta ahora esta enfermedad porcina letal (7). El genotipo II del virus de la peste porcina africana es la cepa pandémica que circula actualmente y causa brotes en Europa, la Federación de Rusia, el Sudeste Asiático, la República Dominicana y Haití (1, 8, 9). Además, recientemente se notificó por primera vez la presencia del virus de la peste porcina africana de genotipo II en Nigeria y África occidental (10).
Dado que la PPA no se puede diferenciar de otras fiebres hemorrágicas porcinas clínicamente o mediante un examen postmortem, las herramientas de diagnóstico de laboratorio específicas de la PPA son fundamentales. La detección rápida y precoz de la infección por el virus de la PPA es uno de los componentes clave en el control de esta enfermedad. Los anticuerpos monoclonales (AcM) específicos del virus de la peste porcina africana son esenciales para el desarrollo de ensayos diagnósticos de laboratorio. Anteriormente se han generado varios anticuerpos monoclonales específicos para el virus de la peste porcina africana; sin embargo, todos los anticuerpos monoclonales se generaron a partir de animales inmunizados con proteínas recombinantes como p30 (11-13) o p72 (14, 15). Los inmunógenos de proteínas recombinantes se pueden utilizar para generar anticuerpos altamente específicos para detectar una sola proteína. Cuando se utilizan virus enteros como inmunógenos, se pueden generar anticuerpos monoclonales contra varias proteínas virales. Por lo tanto, los mAbs generados pueden tener múltiples aplicaciones en diferentes inmunoensayos. Además, los epítopos pueden conservarse en secuencias para que los anticuerpos individuales puedan tener reactividad en varios genotipos de virus.
En este estudio, se utilizó el virus de la PPA completa como inmunógeno y se generó y caracterizó un mAb F88ASF-55. El epítopo reconocido por el mAb se identificó mediante una matriz de péptidos. También se examinó la aplicación diagnóstica de este mAb. Los resultados indicaron que F88ASF-55 se puede utilizar para detectar el virus de la PPA en inmunoensayos, como ELISA y ensayos de inmunohistoquímica.
Materiales y métodos
Preparación del virus de la peste porcina africana
Todas las cepas de PPA utilizadas en este estudio se enumeran en la Tabla 1. Para generar la partícula intacta del virus de la PPA para la inmunización de ratones, se utilizó el virus de la PPA Lisbon/61, adaptado en células de riñón de mono verde africano (Vero-76) (16). Brevemente, las células Vero-76 (ATCC) cultivadas en matraces de cultivo celular T-75 se infectaron con el virus de la PPA Lisboa/61 con una multiplicidad de infección (MOI) de 0,1. Los cultivos infectados se incubaron a 37°C en un CO al 5%2 incubadora durante 5-7 días hasta que el 90-100% de las células mostraron efectos citopáticos. A continuación, los frascos se congelaron durante la noche a -20 °C, se descongelaron al día siguiente y el contenido se cosechó y luego se sonicó durante 30 s tres veces en hielo. La suspensión celular resultante se centrifugó a 3.600 × g durante 10 min a 4°C para eliminar los restos celulares. A continuación, el sobrenadante se ultracentrifugó (rotor Beckmann JA20) a 8500 rpm durante 6 h a 4 °C. El gránulo resultante se resuspendió en DMEM, se colocó en capas sobre un cojín de sacarosa al 40% y se centrifugó a 20.000 rpm (49.400 × g) durante 45 minutos a 4 °C utilizando un rotor Beckman SW-41Ti. El gránulo se resuspendió en PBS y se alícuota en crioviales de 2 mL. A continuación, los viales se sumergieron en un baño de agua a 70 °C durante 60 minutos y posteriormente se expusieron a irradiación gamma (5 millones de rad) para inactivar el virus. El virus de la peste porcina africana tratado se utilizó para la inmunización en ratones. Otros virus de la PPA utilizados en este estudio se propagaron y almacenaron como se describió anteriormente (17).
 Tabla 1. Detección del virus de la peste porcina africana mediante el ELISA de doble sándwich y el ensayo de inmunohistoquímica.
Tabla 1. Detección del virus de la peste porcina africana mediante el ELISA de doble sándwich y el ensayo de inmunohistoquímica.
Generación de anticuerpos monoclonales
Los anticuerpos monoclonales contra el virus de la PPA se produjeron como se describió anteriormente (18, 19). Brevemente, ratones hembra BALB/c fueron inoculados con ASFV Lisbon/61 irradiado y concentrado (~20 μg/ratón) en un volumen igual de TiterMax Gold (TiterMax Inc., Estados Unidos) por vía subcutánea. Un total de seis ratones fueron inmunizados y reforzados cuatro veces después de la inmunización inicial. De tres a 4 días antes de la fusión, los ratones fueron reforzados con el mismo antígeno en solución salina tamponada con fosfato (PBS) mediante inyección intravenosa. A continuación, las células inmunizadas del bazo se fusionaron con células de mieloma (P3X63 Ag8.653). Después de 2 semanas, los sobrenadantes de hibridoma se examinaron mediante un ELISA. Los clones positivos se subclonaron utilizando un método de dilución limitante. Los anticuerpos monoclonales se isotiparon utilizando un kit de isotipado de anticuerpos monoclonales de ratón (Roche, Indianápolis, IN, Estados Unidos).
ELISA sándwich de doble anticuerpo
El suero porcino anti-PPA se recolectó 48 días después de la inoculación (dpi) con PPA (Estonia 2014). Las placas de microtitulación (Nunc-Immunoplate Maxisorp, Roskilde, Dinamarca) se recubrieron con suero policlonal porcino anti-PPA (Estonia 2014) diluido 1:5000 en un tampón de carbonato/bicarbonato, pH 9,6 durante la noche a 4 °C. Después del bloqueo en el tampón de bloqueo de caseína (Sigma-Aldrich, Estados Unidos), se añadieron los virus de la PPA (Tabla 1). Se utilizó el virus de la peste porcina africana lisboa/61 concentrado e irradiado como antígeno para el cribado de hibridomas. A continuación, se añadieron sobrenadantes de cultivo de hibridomas. Después de la incubación, se añadió IgG anti-ratón conjugada con peroxidasa de rábano picante (HRP) (1:2000, Jackson ImmunoResearch Laboratories, West Grove, PA, Estados Unidos), seguida de -3,3′,5,5′-Tetrametilbencidina (TMB, Pierce Biotechnology, Inc. Rockford, Illinois, Estados Unidos). Después de la parada, la densidad óptica (DO) se midió a 450 nm utilizando un lector de microplacas Emax (Molecular Devices, San José, CA, Estados Unidos). Cada etapa de incubación fue de 60 min a 37°C con agitación suave, seguida de cinco lavados (PBS con 0,1% Tween 20).
Análisis de Western blot de la proteína del virus de la PPA
El virus de la PPA irradiado y concentrado (Lisbon/61) se separó en un gel NuPAGE Novex Bis-Tris al 4%-12% (Invitrogen, Carlsbad, Estados Unidos); Las bandas de proteínas separadas se transfirieron a membranas de nitrocelulosa (NC) utilizando el dispositivo de transferencia de gel iBlot (Invitrogen, Carlsbad, Estados Unidos). A continuación, las membranas se bloquearon con tampón bloqueador de caseína a temperatura ambiente durante 1 h y se sondearon con suero de cerdo positivo o mAbs diluidos en tampón bloqueante de caseína. Después de la incubación primaria de anticuerpos, las transferencias se incubaron con anticuerpos secundarios anti-cerdos o anti-ratones conjugados con HRP (1:2000; Jackson ImmunoResearch Laboratories, West Grove, Estados Unidos) durante 1 h a temperatura ambiente. Los blots se desarrollaron utilizando el sustrato 3,3′-diaminobenzidina (Sigma-Aldrich, Santa Lucía, Estados Unidos).
Identificación de proteínas de unión a anticuerpos monoclonales mediante cromatografía líquida-espectrometría de masas
Las rodajas de gel SDS-PAGE se eliminaron mediante una combinación de lavados alternos con bicarbonato amónico de 100 mM y acetonitrilo (Thermo Fisher Scientific, MA, Estados Unidos). Una vez eliminada la tinción azul de Coomassie, las proteínas dentro de las rodajas de gel se redujeron y alquilaron mediante la adición de 10 mM de ditiotreitol y 66 mM de yodoacetamida (Sigma Aldrich, MO, Estados Unidos), respectivamente. A continuación, las proteínas de las rodajas de gel se digirieron utilizando tripsina (Thermo Fisher Scientific, MA, Estados Unidos), y los péptidos se extrajeron de las rodajas de gel utilizando una combinación de lavados alternos con ácido fórmico al 1% (Millipore Sigma, MA, Estados Unidos) y acetonitrilo. La solución que contenía los péptidos trípticos se secó casi por completo y los péptidos se resuspendieron en acetonitrilo al 2% y ácido fórmico al 0,1% (tampón A).
Las muestras de péptidos digeridos se analizaron utilizando un Easy nLC 1200 de nanoflujo conectado en línea a un espectrómetro de masas Orbitrap Fusion Lumos con una fuente de iones de nanoelectrospray (2,3 kV) y una interfaz FAIMS Pro (Thermo Fisher Scientific, MA, Estados Unidos). Las muestras se cargaron (2 μL) en una columna Easy Spray de fase reversa C18 (50 cm de largo, 75 μm de diámetro interior, 2 μm de partículas, (Thermo Fisher Scientific) con 100 % de tampón A. A continuación, los péptidos se eluyeron utilizando un gradiente lineal de 3 % a 27 % de tampón B (80 % de acetonitrilo, 0,1 % de ácido fórmico) durante 50 min, 27 % a 39 % de tampón B durante 15 min. 39%-100% tampón B durante 5 min y un lavado al 100% tampón B durante 10 min a un caudal constante de 250 nL/min). El tiempo de ejecución total de cromatografía líquida-espectrometría de masas (LC-MS/MS) fue de 100 min. La adquisición dependiente de los datos se utilizó con tres configuraciones CV diferentes en el FAIMS Pro, (−40, −60, −80) 1 s cada una durante un tiempo de ciclo total de 3 s. Los iones precursores más abundantes en los escaneos de sondeo de un segundo se aislaron selectivamente en el cuadrupolo (ancho de aislamiento de 1,6 m/z) y se fragmentaron mediante disociación colisional (HCD) de mayor energía (35% de energía de colisión normalizada). Los escaneos de sondeo se adquirieron en el Orbitrap en un rango de m/z 375-1.500 con una resolución objetivo de 120.000 a m/z 200, y los escaneos de iones de fragmentos se adquirieron en la trampa de iones lineal con la velocidad de escaneo establecida en «Rápido».
Los datos de espectrometría de masas sin procesar se convirtieron a archivos .mgf utilizando msconvert (ProteoWizard versión 3.0.19254). Los datos se buscaron utilizando Mascot (v3.1, Matrix Science) en una base de datos FASTA de secuencias de proteínas del virus de la peste porcina africana y una base de datos de proteínas contaminantes comunes. Los archivos Mascot.dat se importaron a Scaffold (v 5.1.2) para generar una lista de identificaciones de proteínas.
ELISA peptídico
Se sintetizaron un total de 14 péptidos superpuestos [15 aminoácidos (aa) de longitud, superpuestos entre sí por 10 aa] en base a la secuencia de proteínas codificadas por ASFV A137R (pA137R) (acceso GenBank #AYW34014.1, tecnología ABclonal, MA, Estados Unidos). Los ELISA peptídicos se realizaron como se describió anteriormente (18, 19). Brevemente, las placas de microtitulación (Nunc Maxisorb) se recubrieron con péptidos (1 μg/pocillo). Después del bloqueo, se añadió a la placa un sobrenadante diluido para el cultivo de hibridomas. Después de la incubación, se añadió IgG anti-ratón conjugada con HRP (1:2000, Jackson ImmunoResearch Laboratories, West Grove, Pensilvania, Estados Unidos). A continuación, se añadió TMB y se incubó durante 15 min. Después de detenerse, el diámetro exterior se midió a 450 nm utilizando un lector de microplacas Emax. Cada etapa de incubación fue de 60 minutos a 37 °C con una agitación suave, seguida de cinco lavados con un tampón de lavado.
Tejidos de animales infectados por el virus de la PPA
Se obtuvieron tejidos archivados fijados en formol e incluidos en parafina de cerdos inoculados experimentalmente con PPA en el Centro Nacional de Enfermedades de Animales Extranjeros (NCFAD). Ganglios linfáticos de animales infectados con Gasson (7 dpi), Lillie SI-85 (5 dpi), Georgia 2007/1 (7 dpi) y Estonia 2014 (10 dpi). Los tejidos se seccionaron a 5 μm en portaobjetos cargados positivamente para realizar pruebas adicionales en ensayos de inmunohistoquímica (IHQ) e hibridación in situ (ISH).
Ensayo de inmunohistoquímica
Las secciones de tejido se enfriaron durante 10 min en peróxido de hidrógeno acuoso al 3%. Los epítopos se recuperaron utilizando una solución interna de recuperación de glicanos en una cámara de desobstrucción médica de Biocare. El anticuerpo primario F88ASF-55 se aplicó a secciones en diluciones pretituladas y se incubó a temperatura ambiente durante 30 min. Las reacciones se visualizaron utilizando el polímero antiratón conjugado con HRP Envision+ System (Agilent Technologies, CO, Estados Unidos) y se desarrollaron con el reactivo cromogénico diaminobenzidina (DAB). A continuación, las secciones se contratiñeron con hematoxilina de Gill. Los ganglios linfáticos de animales no infectados se analizaron como controles negativos en el ensayo IHQ y no se observó inmunotinción.®
Hibridación in situ
Para el ensayo de hibridación in situ (ISH) se utilizó el kit RNAScope 2.5 HD Detection Reagent-Red (Advanced Cell Diagnostics, Inc., CA, Estados Unidos). El procedimiento se realizó siguiendo las instrucciones del fabricante. Brevemente, las secciones de tejido se limpiaron e hidrataron en xileno y etanol al 100% y luego se secaron al aire. Las secciones se enfriaron durante 10 min en H acuoso2O2, hervido en solución de recuperación de objetivos durante 15 min, enjuagado en etanol al 100% y secado al aire nuevamente. A continuación, se aplicó un pretratamiento final de proteasa más enzima durante 15 min a 40°C. Se aplicó la sonda V-AFSV-01 (Advanced Cell Diagnostics, Inc., CA, Estados Unidos) y se incubó a 40°C durante 2 h. A continuación, las secciones se lavaron dos veces en un tampón de lavado de 1×. Después de cada uno de los pasos de hibridación posteriores, las secciones se lavaron dos veces más. La señal se visualizó con el cromógeno Fast Red. A continuación, las secciones se contratiñeron con hematoxilina 1 de Gill, se secaron, se cubrieron con EcoMount (BioCare Medical, CA, Estados Unidos) y el patólogo las examinó. Se analizó un ganglio linfático de control negativo de un animal no infectado en un ensayo de ISH y no se observó inmunotinción. Además, se analizó una sonda de control negativo en tejido positivo (ganglio linfático de un animal infectado por el virus de la PPA Georgia 2007/1) y no se observó tinción.
Resultados
Generación de anticuerpos monoclonales contra el virus de la peste porcina africana
Tras la inmunización en ratones con PPA concentrada Lisbon/61, se recolectaron esplenocitos y se fusionaron con células de mieloma. Los sobrenadantes de los hibridomas resultantes se examinaron utilizando el virus de la peste porcina africana Lisbon/61 como antígeno en un ELISA. Los clones positivos fueron seleccionados y luego subclonados. Para determinar la especificidad de los clones, los sobrenadantes de cultivo de hibridomas subclonados se probaron en un ELISA utilizando el virus de la fiebre aftosa y la proteína E2 de la peste porcina clásica recombinante como controles negativos. Solo un clon de hibridoma, F88ASF-55, mostró especificidad para el virus de la peste porcina africana y ninguna reactividad cruzada con otros antígenos (datos no mostrados). El mAb F88ASF-55 es un isotipo IgG1 y contiene una cadena ligera ƙ.
Determinación de la proteína de unión a anticuerpos monoclonales del virus de la peste porcina africana
Para identificar la proteína de unión del mAb, se realizó un análisis de Western blot. La PPA lisbo/61 irradiada y concentrada se separó en geles NuPAGE Novex Bis-Tris y las proteínas se transfirieron a membranas de nitrocelulosa. Las proteínas del virus de la PPA se detectaron con suero porcino positivo (Figura 1A) y F88ASF-55 (Figura 1B). El suero porcino positivo para el virus de la PPA detectó proteínas del virus de la peste porcina africana con pesos moleculares (MW) de aproximadamente 30, 54, 72 y 17 kDa, correspondientes a las proteínas estructurales p30, p54, p72 y k145Rp del virus de la peste porcina africana, respectivamente. El suero policlonal porcino mostró una fuerte reacción a p30, pero fue débil a p54, p72 y k145Rp (Figura 1A). Además, el suero policlonal mostró una reacción débil a una proteína de 14 kDa. Mientras que F88ASF-55 reaccionó fuertemente a una proteína con un MW de 14 kDa y débilmente a una proteína con un MW de 33 kDa (Figura 1B). El epítopo reconocido por F88ASF-55 es lineal porque el mAb reaccionó con la proteína desnaturalizada del virus de la PPA en el análisis de Western blot.
 Figura 1. Reactividad de anticuerpos a proteínas estructurales del virus de la PPA en el análisis de Western blot. Las proteínas concentradas del virus de la peste porcina africana (Lisboa/61) se separaron en geles NuPAGE Novex Bis-Tris al 4%-12% y se transfirieron a membranas de nitrocelulosa. Las proteínas del virus de la peste porcina africana se detectaron utilizando un suero positivo para el virus de la peste porcina africana (A) y un mAb F88ASF-55 (B). Los blots se incubaron con IgG anti-cerdo conjugada con HRP o HRP anti-ratón. utilizando 3, 3’–Se utilizó diaminobenzidina para la visualización. Carriles 1 y 3: marcador MW; y carriles 2 y 4: proteína del virus de la PPA.
Figura 1. Reactividad de anticuerpos a proteínas estructurales del virus de la PPA en el análisis de Western blot. Las proteínas concentradas del virus de la peste porcina africana (Lisboa/61) se separaron en geles NuPAGE Novex Bis-Tris al 4%-12% y se transfirieron a membranas de nitrocelulosa. Las proteínas del virus de la peste porcina africana se detectaron utilizando un suero positivo para el virus de la peste porcina africana (A) y un mAb F88ASF-55 (B). Los blots se incubaron con IgG anti-cerdo conjugada con HRP o HRP anti-ratón. utilizando 3, 3’–Se utilizó diaminobenzidina para la visualización. Carriles 1 y 3: marcador MW; y carriles 2 y 4: proteína del virus de la PPA.
Se realizó un enfoque proteómico para determinar las identidades de secuencia de la banda de proteínas (14 kDa) reconocida por F88ASF-55. La banda proteica teñida fue extirpada, digerida y analizada por LC-MS/MS. Se identificaron un total de 10 péptidos únicos exclusivos y 15 espectros únicos exclusivos (datos no mostrados). La cobertura de aminoácidos (88/137) de la proteína estructural codificada por A137R fue del 64,2% (Figura 2). Los resultados confirmaron que el epítopo de unión a mAb se encuentra en la proteína codificada por A137R (anteriormente conocida como p11.5).
 Figura 2. Secuencia de la proteína A137R (p11.5) codificada por A137R con péptidos trípticos únicos identificados. Las bandas proteicas (14 kDa) correspondientes a la posición indicada en el western blot se extirparon del SDS-PAGE, se detuvieron, se digierieron con tripsina y se identificaron mediante la identificación de la masa peptídica de las proteínas estructurales del virus de la PPA. La figura muestra la secuencia de aminoácidos de la poliproteína A137R del virus de la peste porcina africana. Los aminoácidos resaltados se identificaron como péptidos trípticos únicos que abarcan la secuencia de proteínas. La cobertura de aminoácidos de la proteína estructural codificada por A137R fue del 64,2% (88/137).
Figura 2. Secuencia de la proteína A137R (p11.5) codificada por A137R con péptidos trípticos únicos identificados. Las bandas proteicas (14 kDa) correspondientes a la posición indicada en el western blot se extirparon del SDS-PAGE, se detuvieron, se digierieron con tripsina y se identificaron mediante la identificación de la masa peptídica de las proteínas estructurales del virus de la PPA. La figura muestra la secuencia de aminoácidos de la poliproteína A137R del virus de la peste porcina africana. Los aminoácidos resaltados se identificaron como péptidos trípticos únicos que abarcan la secuencia de proteínas. La cobertura de aminoácidos de la proteína estructural codificada por A137R fue del 64,2% (88/137).
Identificación del epítopo de unión a mAb
Para localizar aún más los epítopos de unión, se sintetizaron 14 péptidos superpuestos que representan el polipéptido completo codificado por A137R. La reactividad del AcM a los péptidos pA137R del virus de la PPA se examinó mediante un ELISA peptídico. El mAb reaccionó con el péptido #12, correspondiente a los aminoácidos 111-125 (VDFQANIENMDDLQK) (Figura 3). Las secuencias de esta región (111-125 aa) se alinearon con los aislamientos del virus de la PPA entre 1950 y 2008 (Tabla 2). Los resultados mostraron que el epítopo de unión a mAb era 100% idéntico en los 12 genotipos examinados por el virus de la PPA.
 Figura 3. Péptido ELISA a péptidos proteicos del virus de la PPA codificados por A137R. La reactividad de mAb con péptidos pA137R en ELISA. Se sintetizaron un total de 14 péptidos superpuestos [15 aminoácidos (aa) de longitud, superpuestos por 10 aa] basados en la secuencia pA137R del virus de la PPA y se recubrieron en una placa de 96 pocillos. Se detectó la unión peptídica por mAb F88ASF-55 con un conjugado IgG-HRP anti-ratón de conejo. Los valores de densidad óptica (DO) son duplicados medios.
Figura 3. Péptido ELISA a péptidos proteicos del virus de la PPA codificados por A137R. La reactividad de mAb con péptidos pA137R en ELISA. Se sintetizaron un total de 14 péptidos superpuestos [15 aminoácidos (aa) de longitud, superpuestos por 10 aa] basados en la secuencia pA137R del virus de la PPA y se recubrieron en una placa de 96 pocillos. Se detectó la unión peptídica por mAb F88ASF-55 con un conjugado IgG-HRP anti-ratón de conejo. Los valores de densidad óptica (DO) son duplicados medios.
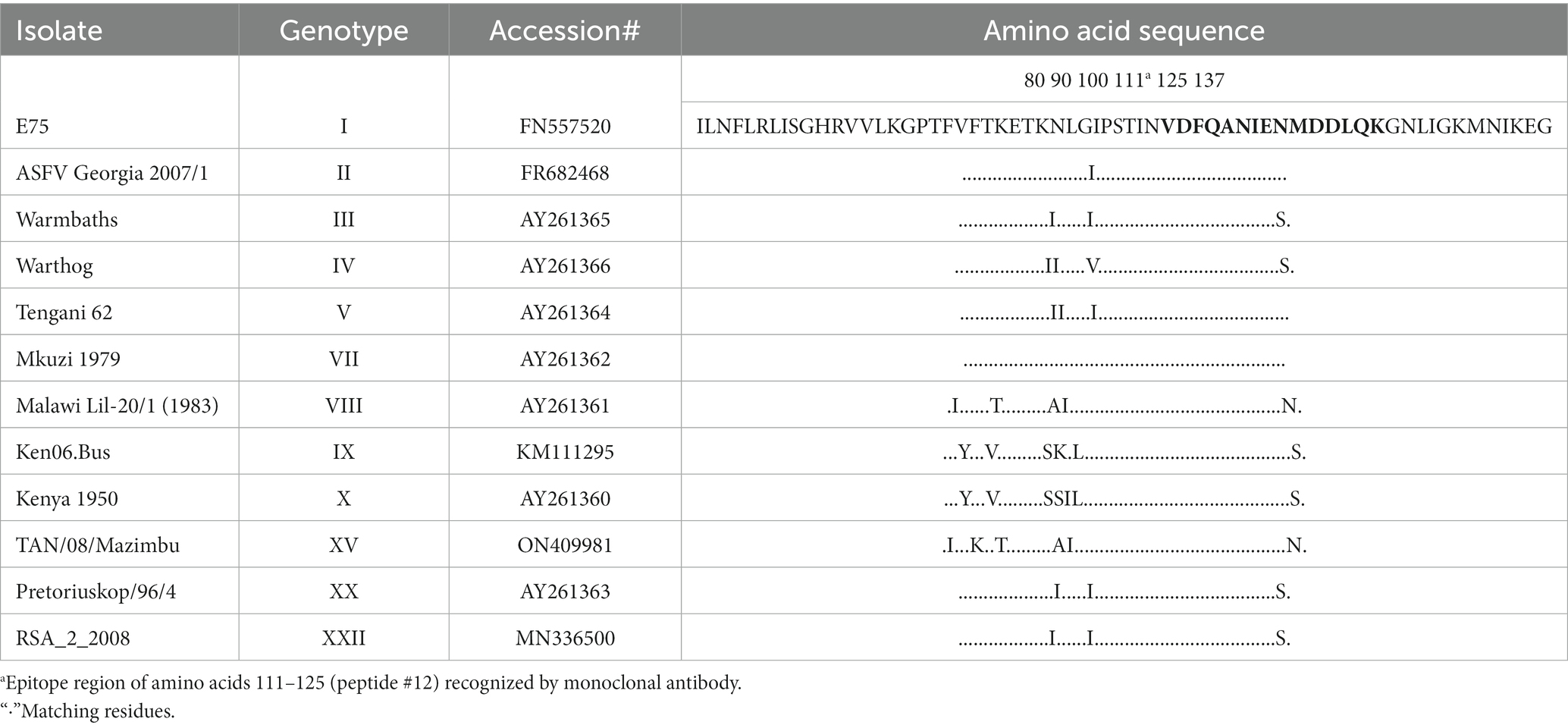 Tabla 2. Alineación parcial de la secuencia de aminoácidos (74-137) de la proteína pA137R (p11.5) del virus de la peste porcina africana con doce genotipos de aislados del virus de la peste porcina africana desde 1950 hasta 2008.
Tabla 2. Alineación parcial de la secuencia de aminoácidos (74-137) de la proteína pA137R (p11.5) del virus de la peste porcina africana con doce genotipos de aislados del virus de la peste porcina africana desde 1950 hasta 2008.
Aunque numerosos genotipos no tienen información de secuencia del genoma completo disponible, este epítopo es 100% idéntico en 256 de las 258 secuencias en GenBank que abarcan los años 1950-2023. La secuencia de epítopos de dos cepas, Uvira B53 (Genotipo X; 2019; MT956648) y BUR/18/Rutana (Genotipo X; 2018; MW856067) varió en un aminoácido (Q124E; péptido Q14E). Además, el epítopo reconocido por F88ASF-55 no se puede encontrar en ningún otro organismo.
ELISA sándwich de doble anticuerpo para la detección del virus de la peste porcina africana
Se utilizó un ELISA de DAS para determinar si el AcM podía detectar el virus de la PPA. En el ELISA se utilizó como agente de captura el anti-virus porcino policlonal de la PPA y como agente de detección el mAb F88ASF-55. El ELISA DAS identificó 5 cepas de virus de los genotipos I y II del virus de la PPA, además de Lisbon/61 (Figura 4). Se espera que el ELISA sea específico para el virus de la peste porcina africana, ya que los sobrenadantes de cultivos celulares y los controles libres de virus mostraron un fondo muy bajo. No se analizaron todos los genotipos del virus de la PPA debido a la limitada información de secuencia disponible para otros genotipos. F88ASF-55 tiene el potencial de detectar numerosos genotipos de PPA en ELISA porque este AcM se dirige a un epítopo conservado compartido entre los genotipos de PPA con secuencias A137R, como se muestra en la Tabla 2. Sin embargo, se requiere una evaluación completa.
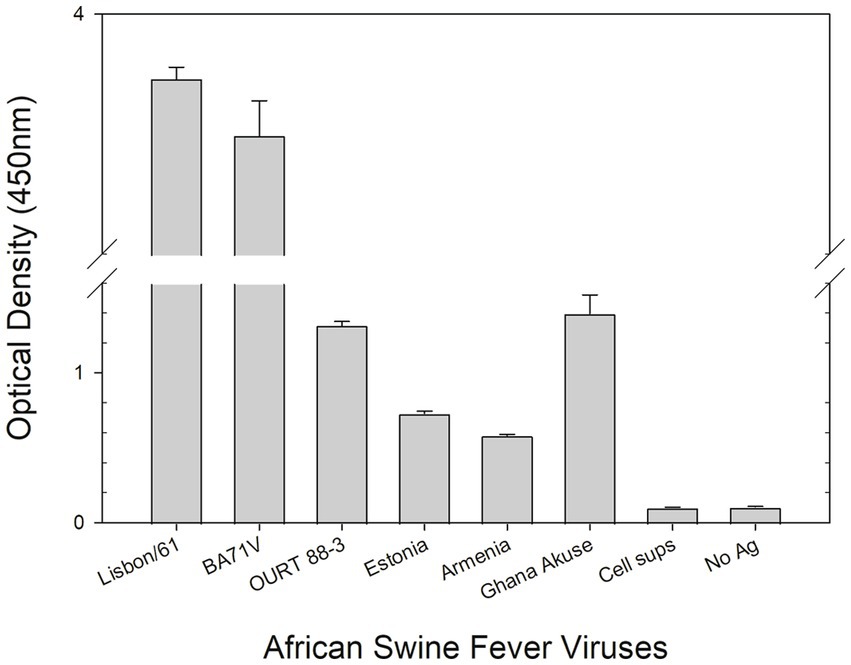 Figura 4. DAS ELISA para la detección del virus porcino africano. En el ELISA, el anticuerpo policlonal anti-PPA se recubrió en placas de microtitulación. Los virus en sobrenadantes de cultivos celulares (Lisbon/61, BA71V, OURT 88/3, Estonia 2014, Armenia/07 y Ghana Akuse/2020) se añadieron a las placas. Luego se agregó sobrenadante de cultivo de hibridoma (F88ASF-55). La unión de los anticuerpos se detectó con un conjugado IgG-HRP anti-ratón de conejo. Los valores de densidad óptica (DO) son duplicados medios.
Figura 4. DAS ELISA para la detección del virus porcino africano. En el ELISA, el anticuerpo policlonal anti-PPA se recubrió en placas de microtitulación. Los virus en sobrenadantes de cultivos celulares (Lisbon/61, BA71V, OURT 88/3, Estonia 2014, Armenia/07 y Ghana Akuse/2020) se añadieron a las placas. Luego se agregó sobrenadante de cultivo de hibridoma (F88ASF-55). La unión de los anticuerpos se detectó con un conjugado IgG-HRP anti-ratón de conejo. Los valores de densidad óptica (DO) son duplicados medios.
Detección del antígeno del virus de la PPA mediante inmunohistoquímica e hibridación in situ
El AcM se utilizó en el ensayo IHQ para detectar el virus de la PPA en tejidos porcinos infectados. Los resultados de IHQ indicaron que F88ASF-55 fue capaz de reconocer específicamente el antígeno viral con baja tinción de fondo inespecífica en ganglios linfáticos submandibulares de animales infectados experimentalmente con PPA (Figura 5). La inmunotinción observada fue intensa y específica, con una excelente relación señal-ruido. El mAb no mostró reactividad cruzada con el tejido control negativo (datos no mostrados).
 Figura 5. Análisis de inmunohistoquímica (IHQ) e hibridación in situ (ISH) de tejidos infectados por el virus de la PPA. Ganglios linfáticos submandibulares de animales infectados experimentalmente con cepas del virus de la peste porcina africana (A) Lillie SI-85; b) Gasson; (C) Georgia 2007/1 y (D) Estonia 2014 se incluyeron en parafina y se analizaron mediante IHQ e ISH. En la IHQ, el antígeno del virus de la PPA se detectó mediante mAb F88ASF-55 (A1–D1). En ISH, el ARN del virus de la PPA se detectó utilizando la sonda V-AFSV-01 (A2-D2). No se muestran los controles negativos de IHQ e ISH.
Figura 5. Análisis de inmunohistoquímica (IHQ) e hibridación in situ (ISH) de tejidos infectados por el virus de la PPA. Ganglios linfáticos submandibulares de animales infectados experimentalmente con cepas del virus de la peste porcina africana (A) Lillie SI-85; b) Gasson; (C) Georgia 2007/1 y (D) Estonia 2014 se incluyeron en parafina y se analizaron mediante IHQ e ISH. En la IHQ, el antígeno del virus de la PPA se detectó mediante mAb F88ASF-55 (A1–D1). En ISH, el ARN del virus de la PPA se detectó utilizando la sonda V-AFSV-01 (A2-D2). No se muestran los controles negativos de IHQ e ISH.
Para confirmar la especificidad de los resultados del ensayo IHQ, se realizó una ISH en paralelo utilizando los mismos tejidos (Figura 5). El ARN del virus de la peste porcina africana se pudo detectar con una distribución similar a los resultados del ensayo de IHQ, lo que indica que el ensayo de IHQ que utilizó F88ASF-55 detectó un antígeno específico del virus de la PPA. Sin embargo, la detección de IHQ mostró señales más fuertes que la ISH para las cuatro cepas de PPA analizadas. Las concentraciones de antígeno viral en los tejidos a 5-10 dpi pueden ser más altas que el ARN viral detectado por ISH.
Discusión
Recientemente se han producido brotes de peste porcina africana en los principales países productores de cerdos de Europa, además de China y Mongolia. Por lo tanto, el virus de la PPA representa la amenaza más importante para la industria porcina mundial. La detección rápida y precoz de la infección por el virus de la PPA es uno de los componentes clave en el control de esta enfermedad. Con el fin de desarrollar inmunoensayos valiosos para la detección del virus de la peste porcina africana, se produjo un mAb específico para el virus de la peste porcina africana. Se caracterizó el epítopo de unión al mAb y se evaluaron las aplicaciones diagnósticas del mAb.
Se ha informado previamente que los anticuerpos monoclonales específicos contra el virus de la PPA fueron producidos por ratones inmunizados con proteínas recombinantes (11-15), y que los anticuerpos monoclonales mAb son altamente específicos para una sola proteína. En nuestro laboratorio, producimos anticuerpos monoclonales de ratones inmunizados con ASV recombinante p534, p72 y p30, sin embargo, estos anticuerpos monoclonales no reaccionaron con partículas nativas de PPA (datos no publicados). Una posible explicación es que, aunque la proteína recombinante tiene la secuencia correcta, el pliegue proteico puede ser diferente al del virus nativo, por lo que el anticuerpo monoclonal no puede unirse a las partículas virales. Por lo tanto, estos anticuerpos monoclonales no se pueden utilizar en inmunoensayos para la detección de virus. Para superar este problema, en este estudio, se generaron anticuerpos monoclonales a partir de ratones inmunizados con partículas del virus de la PPA. Se demostró que un mAb F88ASF-55 reacciona solo con el virus de la PPA, sin reacción cruzada a la proteína E2 de la peste porcina clásica recombinante no relacionada y al virus de la fiebre aftosa (FMDV). Se ha reportado que p30 es inmunogénico y estimula la mayor respuesta de anticuerpos virales durante la infección por PPA (20). Sin embargo, mAb F88ASF-55 no respondió a p30 y otras proteínas estructurales altamente inmunogénicas importantes como p54 y p72. A pesar del uso del genotipo I histórico de la PPA aislada de Lisboa/61, el mAb F88ASF-55 generado reconoció diferentes genotipos y aislados de PPA. De manera inusual, solo se obtuvo un mAb específico para el virus de la PPA después de varios intentos. La razón por la que solo se obtuvo un AcM específico para el virus de la peste porcina africana a partir de las fusiones puede deberse al método de cribado de hibridomas que utilizamos. En primer lugar, dado que los ratones están inmunizados con partículas de virus, es mejor utilizar el virus completo como antígeno para la detección, pero el virus de la PPA purificada no estaba disponible en el laboratorio. En segundo lugar, se utilizó suero policlonal no purificado positivo para el virus de la PPA (48 dpi) como agente de captura, lo que indujo una unión inespecífica. En tercer lugar, en el primer cribado, no se incluyeron controles sin antígeno negativo, por lo que fue imposible distinguir si la unión del AcM era al virus de la PPA o al polisuero, por lo que la mayoría de los clones seleccionados fueron falsos positivos para el virus de la peste porcina africana.
Dado que el virus de la PPA consta de más de 50 polipéptidos (21, 22), fue necesario determinar la proteína de unión y el epítopo reconocidos por F88ASF-55. Se realizó un abordaje proteómico para identificar la proteína de unión. Los resultados de LC-MS/MS confirmaron que el epítopo reconocido por F88ASF-55 es una proteína estructural del virus de la PPA codificada por A137R (pA137R). Dado el alto nivel de expresión del A137R, puede ser importante a lo largo de la infección, lo que lo convierte en un candidato interesante para la detección (23). Recientemente, se ha informado que el pA137R del virus de la PPA inhibe la producción de interferón tipo I (24). Además, la atenuación del virus de la peste porcina africana por deleción de A137R en una cepa pandémica de PPA ofreció protección contra la cepa parental virulenta (25). En este contexto, pA137R es potencialmente un antígeno diana para el diagnóstico de la PPA.
El epítopo unido por F88ASF-55 es lineal ya que el mAb reaccionó con pA137R bajo las condiciones de desnaturalización de SDS-PAGE según lo determinado por el análisis de Western blot. El epítopo reconocido por F88ASF-55 se dilucidó utilizando 14 péptidos superpuestos que representan el polipéptido completo de pA137R del virus de la PPA. F88ASF-55 reaccionó con el péptido #12, correspondiente a los aminoácidos 111-125 (VDFQANIENMDDLQK). Los resultados mostraron que los epítopos de unión a mAb de los aislados de PPA de 1950 a 2008 eran 100% idénticos en todos los genotipos de PPA con secuencias A137R disponibles. Este hallazgo es consistente con el informe de Yin et al. (23). Desarrollaron un ensayo de RT-PCR dirigido a A137R basado en la alineación de 100 secuencias de codificación completas disponibles en GenBank. Aunque la tasa general de mutación del genoma del virus de la PPA es relativamente baja, el virus de la PPA sigue mostrando diversidad genética y antigénica. Se han notificado múltiples desajustes de pares de bases cuando se utilizan cebadores y sondas recomendados por la OMSA dirigidos a p72, lo que pone de relieve la necesidad de vigilar la pertinencia diagnóstica de las cepas circulantes (26). Como demostraron Yin et al. (23), la detección por PCR en tiempo real de A137R funcionó mejor que el ensayo de RT-PCR recomendado por la OMSA dirigido a p72 para la detección del virus de la PPA en muestras clínicas (23). Recientemente, un informe publicado describió un nuevo ELISA serológico indirecto que utiliza la proteína recombinante p11.5 (pA137R) como antígeno (27). Se apoya que pA137R es una buena diana para los ensayos de diagnóstico para detectar la infección por el virus de la PPA.
Debido a la robustez de los inmunoensayos y a su reducido coste, ELISA sigue siendo una de las principales plataformas utilizadas para la detección de virus. Sin embargo, su uso tiene inconvenientes, como la reacción cruzada de anticuerpos con otros virus coinfecciosos, lo que conduce a falsos positivos o resultados inexactos. El uso de anticuerpos monoclonales podría mejorar la especificidad del ELISA. Hutchings y Ferri informaron de un ELISA sándwich utilizando un mAb contra p72, y suero policlonal de conejo para la detección antigénica del virus de la PPA (28). Sin embargo, el informe indicó que la prueba ELISA no era tan sensible como la prueba de hemadsorción y que podría no detectar concentraciones bajas de PPA. Utilizando mAb F88ASF-55, que reconoce pA137R pero no p72, la sensibilidad del ELISA puede ser mayor que los resultados informados anteriormente. Aunque la secuencia A137R de numerosos genotipos sigue siendo desconocida, según las secuencias actuales en GenBank, F88ASF-55 se une a un epítopo altamente conservado en un amplio rango temporal (1950-2008) y al menos 4 genotipos, por lo que puede detectar nuevos aislados virales. Dado que el presente estudio tenía como objetivo demostrar que este mAb puede utilizarse para la detección del antígeno del virus de la PPA en ELISA, no se ha establecido la especificidad diagnóstica y la sensibilidad del ELISA de detección de antígenos. La validación completa del ELISA se realizará y se publicará en un informe separado.
Los análisis IHQ e ISH se utilizan comúnmente para la identificación de virus específicos. Ambas tecnologías permiten visualizar la distribución y localización del virus en células o tejidos infectados. Varios investigadores informan sobre el uso de IHQ con anticuerpos policlonales para la detección del virus de la PPA (12, 13). Los anticuerpos policlonales pueden tener un fondo alto y una especificidad baja, por lo que se preferiría un AcM. Previamente se generó un AcM contra p30 del virus de la PPA para su uso en IHQ (11). Los aislados de países de África oriental y meridional pueden tener variabilidad en la región antigénica de p30. Debido a esta variabilidad entre los diferentes aislamientos del virus de la PPA, los anticuerpos monoclonales específicos de p30 pueden no detectar el antígeno viral, especialmente los aislados con diferentes orígenes geográficos (29). En otro informe se detectó el genotipo I del virus de la PPA mediante IHQ con un AcM frente a p72 del virus de la PPA (15), pero no se identificó el epítopo exacto de unión al AbM. Dado que el virus de la peste porcina africana se clasifica en 24 genotipos basados en B646L que codifica p72 (5, 30), es posible que los anticuerpos monoclonales generados en un genotipo no detecten otros p72 de otros genotipos. Se informó que los anticuerpos monoclonales contra pB602L, una proteína no estructural, podían detectar el virus de la peste porcina africana en el tejido infectado por el virus de la peste porcina africana mediante IHQ, sin embargo, su rendimiento solo se evaluó con una cepa (31). Aunque varios de estos anticuerpos monoclonales funcionaron bien en la IHQ, un mayor número de anticuerpos monoclonales disponibles para la IHQ permitiría una detección más fiable del virus de la peste porcina africana. En este estudio, el mAb (F88-ASF55) detectó antígeno viral de PPA en tejidos infectados sin tinción de fondo inespecífica en los ensayos de IHQ. Aunque en este estudio no se examinaron los 24 genotipos del virus de la PPA, se detectaron múltiples cepas diferentes de virus de genotipo II tanto en los ensayos de IHQ como en los ELISA que utilizaron este AcM. Esto es importante si se tiene en cuenta que el virus de la PPA genotipo II se ha notificado en Europa, la Federación de Rusia, el sudeste asiático, la República Dominicana y Haití, así como en Nigeria (1, 8-10). Este mAb podría detectar todos los genotipos del virus de la PPA en función de que el epítopo reconocido esté altamente conservado. Observamos una tinción más abundante dependiendo de la cepa del virus de la PPA, lo que probablemente refleja la carga viral, lo que podría deberse a una diferencia en la virulencia, la susceptibilidad de los animales individuales o la fecha de muestreo.
El virus de la peste porcina africana en tejidos de cerdos infectados con el aislado altamente virulento de la PPA en Malawi se detectó principalmente en células del sistema fagocítico mononuclear cuando se utilizaron IHQ e ISH (30). En nuestro informe, el ARN del virus de la peste porcina africana detectado por ISH tenía una distribución de antígenos del virus de la peste porcina africana similar a la detectada por IHQ, lo que sugiere que ambos ensayos pueden utilizarse con éxito como pruebas diagnósticas. Sin embargo, el ensayo IHQ fue considerablemente más fácil de realizar, tardando aproximadamente 4 h en completarse, en comparación con el ISH no radiactivo, que tarda 2 días (30). Curiosamente, el ensayo IHQ pareció ser más sensible para todas las cepas probadas, detectando más antígeno viral que ARN en los tejidos. Esto puede estar relacionado con la duración de la infección, lo que refleja una acumulación de antígeno viral en comparación con el ARNm viral. Sin embargo, dado el rango de puntos de tiempo de 5 a 10 ppp, es más probable que el ensayo IHQ tenga una sensibilidad más alta que el ensayo ISH. Dado que es difícil obtener o producir un gran número de muestras positivas para múltiples cepas diferentes, no pudimos evaluar la sensibilidad y la especificidad diagnósticas en este momento. Se requiere una evaluación completa para ambos enfoques antes de llegar a una conclusión final.
Conclusión
En resumen, se generó y caracterizó un mAb (F88ASF-55) frente al virus de la PPA. Este mAb reconoce el pA137R, y el epítopo ha sido mapeado utilizando una matriz de péptidos. Identificamos que F88ASF-55 se une a un epítopo altamente conservado, lo cual es importante en aplicaciones de diagnóstico. Los resultados indican que F88ASF-55 podría utilizarse en ensayos ELISA e IHQ para la detección del antígeno del virus de la PPA y, posiblemente, en ensayos de tiras inmunocromatográficas.
Declaración de disponibilidad de datos
Los datos presentados en el estudio se depositan en https://massive.ucsd.edu, números de acceso MassIVE MSV000092995 y ProteomeXchange PXD045773.
Declaración ética
Todos los tejidos de archivo utilizados en este estudio se obtuvieron de experimentos realizados de acuerdo con las directrices del Consejo Canadiense para el Cuidado de los Animales. Todos los experimentos fueron aprobados por el comité institucional de cuidado animal y se realizaron en el Centro Nacional de Enfermedades de Animales Extraños.
Contribuciones de los autores
CE-H: Curación de datos, Análisis formal, Investigación, Metodología, Supervisión, Redacción – borrador original, Redacción – revisión y edición. EM: Curación de datos, metodología, redacción, revisión y edición. DZ: Curación de datos, metodología, redacción, revisión y edición. CE: Curación de datos, Análisis formal, Metodología, Validación, Redacción – revisión y edición. BC: Análisis formal, Metodología, Redacción – revisión y edición. KG: Escritura – borrador original. AA: Adquisición de fondos, administración de proyectos, recursos, supervisión, redacción, revisión y edición. MY: Conceptualización, Curación de datos, Análisis formal, Adquisición de fondos, Investigación, Administración de proyectos, Recursos, Supervisión, Validación, Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo fue apoyado por los fondos suplementarios de la PPA de la CFIA y el proyecto de TD «Mejora de los diagnósticos serológicos de la PPA en el NCFAD-N-000404».
Reconocimientos
Los autores desean agradecer a la sección de Servicios Básicos de Espectrometría de Masas y Proteómica del Laboratorio Nacional de Microbiología de la Agencia de Salud Pública de Canadá por proporcionar el análisis LC-MS/MS para la identificación de secuencias de proteínas. Los autores también agradecen a Melissa Goolia y Orlando Pérez por su asistencia técnica, y al personal de cuidado de animales por su apoyo experto. El apoyo financiero fue proporcionado por el Organismo Canadiense de Inspección de Alimentos.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Organización Mundial de Sanidad Animal (OMSA) , Peste porcina africana. Disponible en: https://www.woah.org/en/disease/african-swine-fever/#ui-id-2. A. Informe de seguimiento de la peste porcina africana en Georgia 1. Disponible en: https://wahis.woah.org/#/in-review/304.
2. Dixon, LK , Chapman, DA , Netherton, CL y Upton, C . Replicación y genómica del virus de la peste porcina africana. Virus Res. (2013) 173:3–14. doi: 10.1016/j.virusres.2012.10.020
3. Dixon, L , Escribano, J , Martins, C , Roca, D , Salas, M y Wilkinson, P. Los Asfarviridae. En Taxonomía de Virus: VIII Informe del Comité Internacional para la Taxonomía de Virus. eds. CM Fauquet, MA Mayo y J Maniloff. Londres: Elsevier/Academic Press (2005). 135–143.
4. Wang, Y , Kang, W , Yang, W , Zhang, J , Li, D y Zheng, H . Estructura del virus de la peste porcina africana y mecanismos moleculares asociados subyacentes a la infección y la inmunosupresión: una revisión. Frente Immunol. (2021) 12:715582. doi: 10.3389/fimmu.2021.715582
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Qu, H , Ge, S , Zhang, Y , Wu, X y Wang, Z . Una revisión sistemática de los genotipos y serogrupos del virus de la peste porcina africana. Genes de virus. (2022) 58:77–87. doi: 10.1007/s11262-021-01879-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Urbano, A.C. y Ferreira, F . Control y prevención de la peste porcina africana: actualización sobre el desarrollo de vacunas. Los microbios emergentes infectan. (2022) 11:2021–33. doi: 10.1080/22221751.2022.2108342
7. Penrith, ML y Kivaria, FM . Cien años de peste porcina africana en África: ¿dónde hemos estado, dónde estamos ahora, hacia dónde vamos? Transbound Emerg Dis. (2022) 69:E1179–200. doi: 10.1111/tbed.14466
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Jean-Pierre, RP , Hagerman, AD y Rich, KM . Un análisis de las consecuencias de la peste porcina africana en las economías rurales y los pequeños productores porcinos en Haití. Front Vet Sci. (2022) 9:960344. doi: 10.3389/fvets.2022.960344
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Njau, EP , Domelevo Entfellner, J , Machuka, EM , Bochere, ES , Cleaveland, S , Shirima, GM, et al. El primer virus de la peste porcina africana de genotipo II aislado en África proporciona información sobre la actual pandemia euroasiática. Sci Rep. (2021) 11:13081. doi: 10.1038/s41598-021-92593-2
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Adedeji, AJ , Luka, PD , Atai, RB , Olubade, TA , Hambolu, DA , Ogunleye, MA, et al. Presencia por primera vez del virus de la peste porcina africana genotipo II en Nigeria. Anuncio de Microbiol Resour. (2021) 10:E00350–21. doi: 10.1128/MRA.00350-21
11. Petrovan, V , Yuan, F , Li, Y , Shang, P , Murgia, MV , Misra, S, et al. Desarrollo y caracterización de anticuerpos monoclonales contra la proteína p30 del virus de la peste porcina africana. Virus Res. (2019) 269:197632. doi: 10.1016/j.virusres.2019.05.010
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Izzati, UZ , Inanaga, M , Hoa, NT , Nueangphuet, P , Myint, O , Truong, QL, et al. Investigación patológica y distribución de antígenos virales de la peste porcina africana emergente en Vietnam. Transbound Emerg Dis. (2021) 68:2039–50. doi: 10.1111/tbed.13851
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Nguyen, HN , Nguyen, QT , Nguyen, BT , Tran, TH , Hacer, DT y Hoang, HT . Detección del virus de la peste porcina africana en lechones neonatos con temblores congénitos. Arch Virol. (2022) 167:1131–5. doi: 10.1007/s00705-022-05378-x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Cobbold, C , Whittle, JT y Wileman, T . Implicación del retículo endoplásmico en el ensamblaje y envoltura del virus de la peste porcina africana. J Virol. (1996) 70:8382–90. doi: 10.1128/jvi.70.12.8382-8390.1996
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Pérez, J , Fernández, A , Sierra, M , Herraez, P y Fernández, A . Estudio serológico e inmunohistoquímico de la peste porcina africana en jabalíes en España. Rec. Veterinario (1998) 143:136–9. doi: 10.1136/vr.143.5.136
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Weingartl, HM , Sabara, M y Pasick, J . Líneas celulares porcinas continuas desarrolladas a partir de macrófagos alveolares: caracterización parcial y susceptibilidad al virus. Métodos J Virol. (2002) 104:203–16. doi: 10.1016/S0166-0934(02)00085-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Onyilagha, C , Nash, M , Pérez, O , Goolia, M , Clavijo, A , Richt, JA, et al. Exudado de carne para la detección de material genómico del virus de la peste porcina africana y anticuerpos anti-PPA. Virus. (2021) 13:1744. doi: 10.3390/v13091744
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Yang, M , Holanda, H y Clavijo, A . Producción de anticuerpos monoclonales contra partículas virales enteras de los serotipos O y A del virus de la fiebre aftosa y su posible uso en la cuantificación del virus intacto para la fabricación de vacunas. Vacuna. (2008) 26:3377–82. doi: 10.1016/j.vaccine.2008.04.062
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Yang, M , Berhane, Y , Salo, T , Li, M , Agujero, K y Clavijo, A . Desarrollo y aplicación de anticuerpos monoclonales contra la nucleoproteína del virus de la influenza aviar. Métodos J Virol. (2008) 147:265–74. doi: 10.1016/j.jviromet.2007.09.016
20. Giménez-Lirola, LG , Mur, L , Rivera, B , Mogler, M , Sol, Y , Lizano, S, et al. Detección de anticuerpos contra el virus de la peste porcina africana en muestras de suero y fluidos orales mediante un ELISA indirecto de matriz dual de proteína recombinante 30 (p30). PLoS Uno. (2016) 11:e0161230. doi: 10.1371/journal.pone.0161230
21. Alejo, A , Matamoros, T , Guerra, M y Andrés, G . Atlas proteómico de la partícula del virus de la peste porcina africana. J Virol. (2018) 92:e01293–18. doi: 10.1128/JVI.01293-18
22. Esteves, A , Marques, MI y Costa, JV . Análisis bidimensional de las proteínas del virus de la peste porcina africana y de las proteínas inducidas en células infectadas. Virología. (1986) 152:192–206. doi: 10.1016/0042-6822(86)90384-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Yin, D , Geng, R , Lv, H , Bao, C , Shao, H , Ye, J, et al. Desarrollo de PCR en tiempo real basada en el gen A137R para la detección del virus de la peste porcina africana. Front Vet Sci. (2021) 8:753967. doi: 10.3389/fvets.2021.753967
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Sol, M , Yu, S , Ge, H , Wang, T , Li, Y , Zhou, P, et al. La proteína A137R del virus de la peste porcina africana inhibe la producción de interferón tipo I a través de la degradación lisosomal mediada por autofagia de TBK1. J Virol. (2022) 96:e01957–21. doi: 10.1128/jvi.01957-21
25. Gladue, DP , Ramírez-Medina, E , Vuono, E , Silva, E , Rai, A , Pruitt, S, et al. La eliminación del gen A137R de la cepa pandémica del virus de la peste porcina africana atenúa la cepa y ofrece protección contra el virulento virus pandémico. J Virol. (2021) 95:e01139–21. doi: 10.1128/JVI.01139-21
26. Tran, HTT , Dang, AK , Ly, DV , Vu, HT , Hoang, TV , Nguyen, CT, et al. Se requiere una mejora del sistema de reacción en cadena de la polimerasa en tiempo real basado en la modificación de la sonda para la detección precisa del virus de la peste porcina africana en muestras clínicas en Vietnam. Australas Asiáticas J Anim Sci. (2020) 33:1683–90. doi: 10.5713/ajas.19.0525
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Watanabe, M , Kitamura, T , Nagata, K , Ikezawa, M , Kameyama, KI , Masujin, K, et al. Desarrollo de un nuevo ELISA indirecto para el diagnóstico serológico de la peste porcina africana utilizando la proteína p11.5 como antígeno diana. Patógenos. (2023) 12:774. doi: 10.3390/pathogens12060774
28. Conejeras, GH y Ferris, NP . ELISA sándwich indirecto para la detección de antígenos del virus de la peste porcina africana: comparación de anticuerpos policlonales y monoclonales. Métodos J Virol. (2006) 131:213–7. doi: 10.1016/j.jviromet.2005.08.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Pérez-Filgueira, D , González-Camacho, F , Gallardo, C , Resino-Talavan, P , Blanco, E , Gómez-Casado, E, et al. Optimización y validación de pruebas serológicas recombinantes para el diagnóstico de peste porcina africana basadas en la detección de la proteína p30 producida en larvas de Trichoplusia ni. J Clin Microbiol. (2006) 44:3114–21. doi: 10.1128/JCM.00406-06
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Oura, C , Powell, P y Parkhouse, R . Detección del virus de la peste porcina africana en tejidos infectados de cerdo mediante inmunocitoquímica e hibridación in situ. Métodos J Virol. (1998) 72:205–17. doi: 10.1016/S0166-0934(98)00029-9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: Virus de la peste porcina africana, anticuerpo monoclonal, epítopo de unión a anticuerpos, ELISA sándwich de doble anticuerpo, inmunohistoquímica, hibridación in situ
Cita: Embury-Hyatt C, Moffat E, Zhmendak D, Erdelyan CNG, Collignon B, Goonewardene K, Ambagala A y Yang M (2023) Generación y caracterización de un anticuerpo monoclonal contra una proteína del virus de la peste porcina africana codificada por el gen A137R. Frente. Vet. Sci. 10:1286906. doi: 10.3389/fvets.2023.1286906
Recibido: 31 de agosto de 2023; Aceptado: 29 de septiembre de 2023;
Publicado: 19 octubre 2023.
Editado por:
Rohana P. Dassanayake, Servicio de Investigación Agrícola (USDA), Estados Unidos
Revisado por:
Gaber S. Abdellrazeq, Universidad Estatal de Washington, Estados
Unidos Yun Tian, Hospital General de Massachusetts y Facultad de Medicina de Harvard, Estados Unidos
Derechos de autor © 2023 Embury-Hyatt, Moffat, Zhmendak, Erdelyan, Collignon, Goonewardene, Ambagala y Yang. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Aruna Ambagala, aruna.ambagala@inspection.gc.ca; Ming Yang, ming.yang@inspection.gc.ca
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía


