Los antecedentes genéticos influyen en las respuestas de los cerdos al virus del síndrome respiratorio y reproductivo porcino
 Los antecedentes genéticos influyen en las respuestas de los cerdos al virus del síndrome respiratorio y reproductivo porcino
Los antecedentes genéticos influyen en las respuestas de los cerdos al virus del síndrome respiratorio y reproductivo porcino
 Yangli Pei
Yangli Pei  Chenghong Lin
Chenghong Lin  Hua Li
Hua Li  Zheng Feng*
Zheng Feng*- Laboratorio Clave Provincial de Guangdong de Diseño Molecular Animal y Cría Precisa, Laboratorio Clave de Diseño Molecular Animal y Cría Precisa de los Institutos de Educación Superior de Guangdong, Escuela de Ciencias de la Vida e Ingeniería, Universidad de Foshan, Foshan, China
El virus del síndrome respiratorio y reproductivo porcino (PRRSV) es un virus altamente infeccioso y económicamente significativo que causa enfermedades respiratorias y reproductivas en los cerdos. Da como resultado una reducción de la productividad y un aumento de la mortalidad en los cerdos, causando pérdidas económicas sustanciales en la industria. Comprender los factores que afectan las respuestas de los cerdos al PRRSV es crucial para desarrollar estrategias de control efectivas. Los antecedentes genéticos se han convertido en un determinante importante de la susceptibilidad y resistencia al PRRSV en cerdos. Esta revisión proporciona una visión general del proceso básico de infección por PRRSV en cerdos, los síntomas asociados, los mecanismos inmunitarios subyacentes y las funciones del ARN no codificante y el empalme alternativo en la infección por PRRSV. Además, hizo hincapié en las variaciones específicas de cada raza en estos aspectos que pueden tener implicaciones para las opciones de tratamiento individuales.
1. Introducción
El síndrome reproductivo y respiratorio porcino (PRRS, por sus siglas en inglés) es una enfermedad altamente destructiva que se identificó por primera vez en los Estados Unidos en 1987 y luego se extendió a Europa en 1990 (1, 2). Supone un riesgo económico considerable para la industria porcina (3-5). El impacto económico estimado del PRRS en todo el rebaño de cuatro granjas chinas que experimentan brotes es de 1424,37 yenes por cerda (4). El virus del PRRS (PRRSV) es el patógeno responsable de causar el PRRS, caracterizado por su naturaleza de ARN de cadena positiva y su estructura envuelta (6). Pertenece al orden Nidovirales y a la familia Arteriviridae (6). Los PRRSV se clasifican en genotipos PRRSV-1 y -2 que se encuentran en Europa y América del Norte, respectivamente (7). PRRSV-1 y -2 comparten aproximadamente un 60% de identidad nucleotídica (1, 8); sin embargo, se cree que siguieron caminos evolutivos separados, originados en un ancestro común lejano (9). El PRRSV-2 se dirige principalmente a las células del linaje monocitos/macrófagos, en particular a los macrófagos alveolares porcinos (AMP) (10).
La prevención y el control del PRRS plantean un reto importante. Las estrategias actuales incluyen la vacunación, el manejo del rebaño, la bioseguridad y el tratamiento antiviral (11). Sin embargo, la eficacia de estas medidas varía en función de las circunstancias específicas y de las estrategias de aplicación. La vacunación muestra resultados prometedores en la reducción de la incidencia y la gravedad del PRRS; sin embargo, no ofrece una solución completa (12). El manejo eficaz del rebaño (incluido el monitoreo y el control del movimiento de los cerdos) puede ayudar a reducir la propagación de la enfermedad. Además, las medidas de bioseguridad, como la desinfección y la limpieza de las instalaciones, pueden ayudar a prevenir la transmisión de enfermedades (11). Aunque el tratamiento antiviral puede reducir la gravedad de la enfermedad, aunque no es una cura permanente y puede no ser rentable en todas las situaciones (13). Por lo tanto, es necesario seguir investigando para identificar y desarrollar métodos más eficaces para prevenir y controlar el PRRS.
Los antecedentes genéticos de los cerdos son un factor importante que determina su respuesta al PRRSV. Varias razas y líneas de cerdos exhiben diferentes niveles de resistencia a la infección por PRRSV (14, 15). Las razas Meishan y Tongcheng (TC), conocidas por su elevada resistencia al PRRSV, son menos susceptibles a la infección en comparación con otras razas, como el Large White (LW) (16-18). Además, tejidos específicos de cerdo, como los pulmones y los ganglios linfáticos, pueden presentar diversas susceptibilidades al PRRSV, que pueden estar influenciadas por factores genéticos (19). Un mejor conocimiento de los factores genéticos que contribuyen a las diferencias en la resistencia al PRRSV entre las razas porcinas mejora potencialmente la salud y el bienestar de los cerdos, reduciendo en última instancia las pérdidas económicas asociadas con la infección por PRRSV.
En general, el control del PRRS sigue siendo un reto, debido a las complejas interacciones huésped-patógeno a pesar de los extensos esfuerzos de investigación. Se requiere más investigación para desarrollar contramedidas efectivas contra el virus. Esta revisión se centra en la influencia de los factores genéticos en las respuestas de los cerdos al PRRSV, incluidas las diferencias entre razas y líneas de cerdos. Además, investigamos los mecanismos de infección por PRRSV y los factores que afectan a la respuesta del huésped, como el sistema inmunitario innato y adaptativo. Además, se exploraron eventos de empalme alternativos y ARN no codificantes implicados en la infección y replicación por PRRSV. Las implicaciones potenciales de esta investigación fueron el desarrollo de estrategias de control efectivas y programas de cría que utilizan información genética para mejorar la salud y la productividad de los cerdos. Este amplio conocimiento mejorará potencialmente el bienestar de los cerdos, aumentará la productividad y promoverá la sostenibilidad de la cría de cerdos en todo el mundo.
2. Respuestas variadas de los receptores en diferentes razas porcinas tras la invasión del PRRSV
2.1. Mecanismos de entrada de la célula huésped
El genoma del PRRSV tiene un tamaño aproximado de 15 kbp y una organización específica. Los genes de la replicasa están situados en el extremo 5′ del genoma, mientras que los genes que codifican proteínas estructurales se encuentran en el extremo 3′ (20, 21). El genoma viral consta de más de 10 marcos de lectura abiertos (ORF, por sus siglas en inglés). Más del 66% del genoma viral está compuesto por ORF1a y ORF1b, que codifican proteínas no estructurales que cumplen funciones cruciales, incluidas las actividades de proteasa y replicasa. Estas proteínas también modulan los genes del huésped que son vitales para la replicación del virus. Por el contrario, los ORF 2-7 codifican las proteínas estructurales necesarias para la formación del virus (21).
Procesos de PRRSV de ocho proteínas estructurales, que incluyen una pequeña proteína no glicosilada y un grupo de proteínas glicosiladas: glicoproteína (GP) 2ab, GP3, GP4, GP5, GP5a, matriz (M) y nucleocápside (N) (21, 22). Las proteínas estructurales primarias codificadas por los ORF 5, 6 y 7 son GP5, M y N, respectivamente. Mientras que GP5 típicamente forma un heterodímero con M, ha habido informes de homodímeros GP5 (23). Entre las glicoproteínas de superficie, GP2, GP3 y GP4, derivadas de los ORF 2, 3 y 4, respectivamente, actúan como componentes menores. Además, dos proteínas no glicosiladas muy pequeñas, designadas como 2b o E y 5a, se traducen de ORF2b y ORF5a, respectivamente (24, 25). El exterior liso del virión del PRRSV se atribuye principalmente a la presencia de secuencias peptídicas cortas en los ectodominios de M y GP5. Sin embargo, los ectodominios más grandes de GP2, GP3 y GP4 también pueden dar lugar a algunas protuberancias en la superficie del virus (21).
Los macrófagos son las principales células diana de la infección por PRRSV (10, 26-28), desempeñando un papel crucial en la modulación inmunitaria y contribuyendo a la dificultad respiratoria observada en los cerdos afectados por el complejo de enfermedades respiratorias porcinas. La siguiente sección proporciona una visión general del reconocimiento del virus por parte de las células receptoras (Tabla 1) y los mecanismos implicados.
CD163 sirve como un receptor crucial para el PRRSV y desempeña un papel fundamental en la determinación de la susceptibilidad celular al virus (29). Es una glicoproteína receptora depuradora que se encuentra predominantemente en macrófagos y monocitos maduros. La porción extracelular de CD163 comprende nueve dominios ricos en cisteína del receptor eliminador (SRCR) y dos motivos ricos en prolina-serina-treonina (PST), que se repiten varias veces (56). Las proteínas heterotriméricas GP2, GP3 y GP4 del PRRSV se unen a CD163, y GP2 y GP4 forman múltiples interacciones con diferentes receptores (30, 56). Los cerdos con un gen CD163 knockout (KO) no son permisivos a la infección por PRRSV-2 (31), y sus macrófagos muestran resistencia a PRRSV-1 y -2 (32). Estudios recientes muestran que los cerdos deficientes en CD163 SRCR5 son resistentes a cepas específicas de PRRSV-2 (33, 34). Los cerdos modificados genéticamente con un dominio CD163 SRCR5 modificado muestran un crecimiento normal en condiciones estándar (34, 35). Esto sugiere que las técnicas de edición genética dirigidas a CD163 pueden controlar y erradicar los brotes de PRRS.
CD163 participa en el mimetismo apoptótico viral, una estrategia empleada por ciertos virus para infectar las células huésped (57). Este mecanismo implica que los virus se disfrazan de desechos apoptóticos y se involucran con receptores en la superficie de los fagocitos que reconocen la fosfatidilserina (PtdSer), un marcador de apoptosis (58). Estas interacciones activan cascadas de señalización y conducen a reordenamientos de actina para facilitar el engullimiento endocítico y la posterior degradación de las partículas virales (59). Los virus adoptan distintos mecanismos de mimetismo apoptótico, dando lugar a los conceptos de mimetismo apoptótico clásico y no clásico (60). El PRRSV aprovecha el mimetismo apoptótico viral para inducir la macropinocitosis a través de la participación de la inmunoglobulina de células T y las proteínas de dominio mucina TIM-1 y -4 (57). Durante la infección por PRRSV, CD163 desempeña un papel vital en la facilitación de la macropinocitosis inducida por TIM (61). En consecuencia, el PRRSV adopta una vía alternativa de infección a través de la actividad de los macrófagos que involucra a CD163.
Las células de mamíferos contienen sulfato de heparina (HS) como glicosaminoglicano en su superficie y en su matriz extracelular (62). El sulfato de heparina desempeña un papel importante en la adhesión durante la infección por PRRSV al interactuar con las proteínas M y GP5-M durante la infección por PRRSV (37). Aunque no es esencial para la invasión del PRRSV de los macrófagos alveolares porcinos (PAM), la HS permite que el PRRSV se adhiera a líneas celulares no susceptibles sin completar los pasos posteriores de la infección (38). El PRRSV-1 y el PRRSV-2 presentan diferentes sensibilidades a la SA (37). El tratamiento de las PAMs con heparinasa (que degrada la HS) reduce la infección por PRRSV (37). Además, la infección por PRRSV puede activar NF-κB y catepsina L, lo que resulta en una regulación y procesamiento al alza de la heparinasa, una reducción en la expresión superficial de HS y la promoción de la replicación y liberación viral (63).
La sialoadhesina (Sn), también conocida como CD169 o SIGLIC-1, actúa como correceptor de la invasión del PRRSV. El dominio de inmunoglobulina N-terminal del Sn porcino es necesario y suficiente (39, 40). Las células que expresan Sn facilitan la internalización del PRRSV, pero no promueven el desrecubrimiento viral (40). La colaboración entre Sn y otros receptores, como CD163, sensibiliza a las células a la infección por PRRSV, promoviendo la adhesión, internalización y desensamblaje efectivos de las partículas virales (41, 42). La ausencia de Sn en cerdos editados genéticamente no altera la adhesión/internalización del PRRSV ni tiene ningún efecto sobre la progresión de la enfermedad o la histopatología (64). Esta discrepancia entre los modelos in vitro e in vivo de los receptores del PRRSV indica resultados contradictorios. Estos hallazgos sugieren que el Sn desempeña un papel principal en la unión del PRRSV a la superficie de los macrófagos en lugar de facilitar la internalización viral.
La vimentina (VIM) es un componente crucial del complejo receptor del PRRSV y desempeña un papel clave en la replicación intracelular y la metástasis del PRRSV (45, 46). Forma un polímero con otros microfilamentos de estructura ósea de células finas e interactúa con la proteína de la nucleocápside del PRRSV (47). La vimentina puede hacer que las líneas celulares normalmente no susceptibles sean susceptibles a la infección por PRRSV. Esto pone de manifiesto su implicación en el complejo receptor viral (45). Tras la entrada en el PRRSV, la vimentina experimenta una reorganización facilitada por la fosforilación de la serina 38 por la proteína quinasa II gamma dependiente de calmodulina cálcica (48). Como resultado de esta reorganización, se forman estructuras en forma de jaula alrededor de los complejos de replicación del PRRSV dentro del núcleo.
El miembro de la superfamilia CD151 de la tetraspanina es una proteína de unión al ARN que interactúa con la 3′-UTR del genoma del PRRSV y funciona como una proteína de unión al ARN (49). Al silenciar el gen CD151 en las células MARC-145, la infección por PRRSV disminuye significativamente y los anticuerpos contra CD151 la previenen por completo (49). La expresión de CD151 puede ser regulada por microARN (como miR-506) que conducen a una disminución en los niveles de ARNm y proteínas de CD151, lo que resulta en la inhibición de la replicación del PRRSV y la liberación viral en las células MARC-145 (50). CD151 posee sitios de N-glicosilación y palmitoilación (65); su participación en la regulación de la infección por PRRSV requiere más investigación.
La cadena pesada de miosina no muscular 9 (MYH9) desempeña varios papeles en la adhesión, polarización, morfogénesis y migración celular (66, 67). Interactúa con PRRSV GP5, que es crucial para la internalización y propagación intercelular del PRRSV. El dominio C-terminal de MYH9 se une directamente al primer ectodominio de GP5 viral, y la interrupción de esta interacción reduce la internalización del PRRSV (51). Se cree que los residuos de aminoácidos específicos dentro del dominio C-terminal de MYH9 son sitios de unión clave para GP5 (52). Además, MYH9 sufre una reorganización tras la infección por PRRSV, formando estructuras en forma de jaula alrededor del complejo de replicación del PRRSV (53). La coexpresión de MYH9 con CD163 aumenta la infección por PRRSV (51).
El miembro 8 de la proteína de choque térmico (HSPA8) desempeña un papel en varias infecciones virales al regular la entrada, replicación y ensamblaje viral (68). La inhibición de HSPA8 endógeno reduce la replicación del PRRSV al disminuir la adhesión viral y la internalización (54). HSPA8 interactúa con la clatrina y está implicada en la endocitosis mediada por clatrina (EMC) (69). El dominio PB de HSPA8 interactúa con PRRSV GP4, y la actividad de la ATPasa HSPA8 es necesaria para la infección por PRRSV a través de CME (54). Además, HSPA8 se colocaliza con PRRSV en la superficie celular y facilita la fusión y entrada viral (54). Las terapias basadas en HSPA8 son prometedoras en ensayos clínicos y en el desarrollo de vacunas para otras enfermedades. Por ejemplo, el HSPA8 recombinante fusionado con GP3 y GP4 aumenta las respuestas inmunitarias y confiere efectos protectores contra la infección altamente patógena por PRRSV en cerdos (55). Por lo tanto, las estrategias basadas en HSPA8 tienen el potencial para el desarrollo de vacunas.
El genoma viral contiene más de 10 ORF. Estos ORF codifican proteínas no estructurales cruciales para la replicación viral. Los ORF 2-7 codifican proteínas estructurales que desempeñan un papel vital en la formación viral y son necesarias para el ensamblaje de partículas virales. El PRRSV utiliza múltiples receptores para entrar en las células huésped, incluidos CD163, Sn, HS, VIM, MYH9, CD151, CD209 y HSPA8. La comprensión de las interacciones entre el PRRSV y estos receptores ofrece objetivos potenciales para controlar y erradicar los brotes de PRRSV, y para desarrollar vacunas y estrategias terapéuticas.
2.2. Diferentes razas porcinas muestran una expresión variable de los genes receptores del PRRSV en los tejidos pulmonares tras la infección por PRRSV
Los cerdos chinos de Dapulia (DPL) exhiben una mayor resistencia al PRRSV en comparación con los cerdos comerciales cruzados Duroc×Landrace×Yorkshire (DLY), como lo demuestran las temperaturas rectales más bajas y el número sérico de copias del PRRSV (70). El análisis de muestras de tejido pulmonar de cerdos DPL y DLY no infectados con PRRSV muestra patrones de expresión variados de cinco genes mediadores del PRRSV (NMMHC-IIA, SIGLEC1, CD163, HSPG2 y VIM), con niveles de expresión de ARNm significativamente más altos de SIGLEC1, NMMHC-IIA, CD163 y VIM en cerdos DLY que en cerdos DPL (70). Otro estudio reveló que el nivel de ARNm de CD163 en los PAM de los cerdos de Dingyuan es significativamente más bajo que el de los cerdos de Jiangquhai dentro de las 24 h posteriores a la infección (hpi); esto puede explicar la alta resistencia de los cerdos de Jiangquhai al PRRSV (71). Además, la expresión de Sn en las PAM de los cerdos de Dingyuan aumenta a un ritmo más rápido que en las PAM de los cerdos de Jiangquhai después de la infección viral (71). Este hallazgo se corresponde con el alto contenido de PRRSV en los PAM de los cerdos Dingyuan (71). El aumento de los niveles de expresión de ARNm de CD163 y Sn puede contribuir a una invasión viral más rápida y a sitios de penetración más amplios in vivo e in vitro (72). La expresión de los receptores virales varía entre las diferentes razas de cerdos después de la infección por PRRSV, y existen variaciones en la expresión de los receptores de PRRSV (HS, Sn, CD163, CD151 y VIM) en los tejidos pulmonares de diferentes razas de cerdos, incluso en condiciones fisiológicas normales (47, 71, 73).
Las variaciones en la expresión de los receptores y la abundancia de ARN pueden conducir a la activación o inhibición de varias vías, lo que da lugar a respuestas distintas a la infección por PRRSV en diferentes razas de cerdos. Una mayor exploración de los mecanismos subyacentes a estas diferencias de susceptibilidad podría allanar el camino para el desarrollo de enfoques innovadores para controlar la infección por PRRSV en las poblaciones porcinas.
3. Variación de las manifestaciones en diferentes razas porcinas durante la infección por PRRSV
3.1. Síntomas y lesiones tras la infección por PRRSV
Las manifestaciones clínicas del PRRS están influenciadas por múltiples factores, como la cepa del virus, la edad y el estado inmunitario del huésped, el entorno de producción, el estado productivo y cepas específicas del PRRSV. Los síntomas típicos durante la fase aguda de la enfermedad incluyen pérdida de apetito, debilidad, fiebre y dificultades respiratorias. La disnea respiratoria se observa comúnmente en todos los grupos de edad de los cerdos, aunque las cerdas gestantes infectadas pueden presentar síntomas más graves.
Las cerdas gestantes son muy susceptibles a la infección por PRRSV y pueden presentar diversos síntomas clínicos. Esto incluye pérdida de apetito, abortos, decoloración transitoria de las orejas (comúnmente conocida como enfermedad de la oreja azul, que afecta aproximadamente al 2% de las cerdas), parto prematuro, anestro prolongado, retraso en el retorno al celo después del destete, tos y signos respiratorios (74).
Los síntomas en lechones destetados y engordados pueden ser significativos y pueden incluir pérdida de cabello, pérdida leve de apetito, problemas respiratorios leves (como tos) y enrojecimiento localizado de la piel. La tasa de mortalidad durante esta etapa oscila entre el 10 y el 20% y está influenciada por la higiene y el manejo operativo. La presencia de otros microorganismos dentro de un rebaño puede aumentar las tasas de mortalidad. Los cerdos de 4 a 12 semanas nacidos de cerdas infectadas presentan síntomas clínicos similares a los de los lechones, incluyendo pérdida de apetito, malabsorción, emaciación, tos y neumonía, y una tasa de mortalidad post-destete un 12% más alta (75, 76). Las infecciones bacterianas secundarias pueden provocar abscesos pulmonares y sistémicos, cojera relacionada con abscesos o crecimiento deficiente (77, 78).
Una serie de síntomas clínicos indican posibles problemas en las cerdas en parto. Estos síntomas incluyen anorexia, disminución de la ingesta de agua, reducción de la producción de leche, mastitis, parto prematuro de lechones, decoloración de la piel (por ejemplo, marchitez por verticillium o vulva y orejas azules), úlceras por presión, letargo, síntomas respiratorios (por ejemplo, tos y neumonía), lechones momificados, lechones nacidos muertos y lechones débiles al nacer (77, 79).
Las enfermedades respiratorias graves y la disminución de las tasas de supervivencia son los problemas más comunes en los lechones. Otros síntomas clínicos incluyeron hinchazón de los párpados, conjuntivitis, apatía, pérdida significativa de peso, diarrea, pelaje áspero y descuidado, decoloración púrpura de la oreja y comportamiento anormal (76, 77).
3.2. Características clínicas de los cerdos infectados por el PRRSV
La infección artificial de cerdos TC y cerdos LW con HP-PRRSV produce síntomas similares de fiebre alta. Los cerdos de PV tienen una temperatura superior a 40,5 °C de 0 a 3 días después del contacto (dpc) y superior a 41,0 °C de 4 a 7 dpc, mientras que los cerdos TC tienen una temperatura superior a 40,5 °C de 1 a 3 dpc y superior a 41,0 °C de 4 a 6 dpc (18). Sin embargo, los signos clínicos son menos graves en los cerdos TC que en los cerdos LW, mostrando cambios en el comportamiento de mentira, menos depresión, respiración profunda, enrojecimiento de la piel y reducción de la ingesta de alimentos. Además, los cerdos TC tienen significativamente menos exudación inflamatoria (p < 0,01) y engrosamiento de la pared alveolar (p < 0,05) en comparación con los cerdos LW. El análisis post-mortem reveló diversos grados de hinchazón y sangrado en el cerebro, el hígado y el bazo, con bordes dentados en el bazo (18).
De manera similar, un estudio en el que participaron lechones tibetanos, negros ZangMei (ZM) y LW de 4 a 6 semanas de edad desafiados con HP-PRRSV (JXA1) mostró que los lechones LW tenían un aumento significativo de la temperatura rectal desde 2 ppp que permanecieron elevados hasta 15 ppp (40,4 °C ± 0,55). Los lechones ZM exhibieron un aumento significativo de la temperatura rectal durante 4 días (2-5 dpi) sin lecturas superiores a 40 °C, y los lechones tibetanos no mostraron ninguna lectura de temperatura rectal superior a 40 °C. La anorexia, los estornudos, la tos y la diarrea aparecieron en los lechones ZM y LW afectados en un plazo de 2 a 3 dpi; sin embargo, los lechones LW experimentaron síntomas más graves, incluido un aumento de los escalofríos, la hiperespasmia y la frecuencia respiratoria de 6 a 8 dpi. Algunos de los lechones LW desafiados murieron a las 9, 11 y 13 ppp, mientras que los lechones tibetanos no mostraron signos típicos de muerte durante el período de 28 días (80). Los signos clínicos observados se alinearon con los cambios correspondientes en la temperatura y el aumento de peso corporal. Los lechones LW mostraron una disminución del peso corporal durante la segunda semana después del desafío, los lechones ZM experimentaron pérdida de peso durante la primera semana y los lechones tibetanos mostraron un aumento de peso constante a lo largo de las 4 semanas (80).
La infección por el virus del síndrome respiratorio y reproductivo porcino tiene un efecto notable en la presentación clínica y el rendimiento del crecimiento de los cerdos, que puede variar significativamente entre razas. Por ejemplo, los cerdos de peso muerto experimentan pérdida de peso y mortalidad después de la infección por PRRSV, mientras que los cerdos ZM no muestran cambios de peso. Por el contrario, los cerdos tibetanos (conocidos por su robusta resistencia a las enfermedades) continúan exhibiendo un aumento de peso a lo largo de la infección (80).
3.3. Carga viral tras la infección por PRRSV
Los cerdos TC muestran cargas virales significativamente más bajas que los cerdos LW después de la infección artificial por HP-PRRSV. Además, los cerdos TC muestran una carga viral máxima reducida y mantienen un declive constante durante todo el período de infección. La cantidad máxima de partículas de PRRSV en los cerdos TC es 0,4 veces mayor que la encontrada en los cerdos LW; esto sugiere una capacidad superior para controlar la replicación viral en cerdos TC (18).
Un estudio de 100 cerdos del NEI (una población compuesta de raza blanca y autóctona grande) y 100 cerdos de un cruce entre la línea Hampshire y Duroc (HD) inoculados con PRRSV (cepa 97-7895) indicó que el título de viremia fue mayor en los cerdos HD que en los cerdos NEI en los días 4, 7 y 14, mientras que los títulos virales en los pulmones y los ganglios linfáticos bronquiales fueron significativamente más altos en los cerdos HD (14).
Un estudio en el que participaron siete cerdos miniatura (MI) y ocho cerdos Pietrain (PI) comerciales desafiados con una cepa atenuada de PRRSV muestra que la viremia alcanza su punto máximo a los 6 dpi, con un 100% de viremia observada en los cerdos PI y a los 12 dpi, con un 87% de viremia observada en los cerdos IM (81). Los cerdos con IM tienen una duración reducida de la viremia. Las diferentes susceptibilidades genéticas al PRRSV pueden contribuir a las variaciones en la producción de anticuerpos (81).
La inoculación de ocho verracos de raza pura (dos Landrace, tres Yorkshire y tres Hampshire) con VR-2332 detectó ARN del PRRSV en el suero y PBMC entre 4 y 11 dpi. El virus no se detecta en los tejidos linfoides de los cerdos Landrace a 47 y 88 dpi, mientras que los cerdos Yorkshire y Hampshire muestran cargas virales variables en los tejidos linfoides. Los verracos de Yorkshire y Hampshire exhiben una mayor resistencia a la excreción del PRRSV en el semen que los verracos Landrace (82).
En 2015, los lechones tibetanos, Zang Mei y Large White fueron desafiados con HP-PRRSV (JXA1). Durante el desafío, la carga viral sérica en lechones LW alcanzó un máximo de 7 dpi, que disminuyó gradualmente hasta los 28 dpi. Los lechones Zang Mei tuvieron su pico viral de virus a los 4 dpi, que disminuyó rápidamente, lo que resultó en cargas virales significativamente más bajas en comparación con los lechones LW a los 14 y 21 dpi (80). Los cerdos tibetanos exhibieron consistentemente cargas virales más bajas que las otras dos razas a lo largo del desafío (80).
El título del virus y la abundancia de ARNm en los sobrenadantes de PAM después de la inoculación con PRRSV NJGC in vitro muestran tendencias similares entre las razas Landrace, Erhualian, Suzhong, Jiangquhuai, Dingyuan y Meishan (71).
Las diferentes razas exhiben variaciones significativas en las características clínicas, el rendimiento del crecimiento y los títulos virales debido a variaciones genéticas que, en última instancia, afectan su capacidad para combatir el PRRSV. Algunas razas, como Meishan y Tongcheng, demuestran una ventaja genética en la lucha contra el virus que resulta en una reducción en la duración de la viremia.
4. Respuestas inmunitarias variadas en diferentes razas porcinas infectadas por el PRRSV
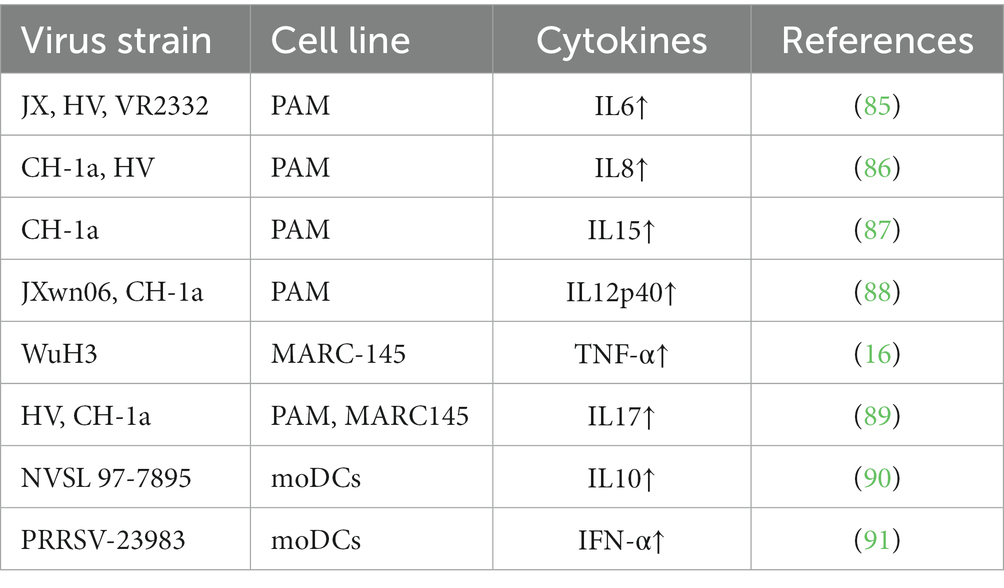
La respuesta inmunitaria innata sirve como defensa inicial contra el PRRSV, que es conocido por su capacidad para evadir el sistema inmunitario del huésped mediante la regulación negativa de los receptores de reconocimiento de patrones (PRR) (83). A pesar de las estrategias de evasión viral, la respuesta inmunitaria innata desempeña un papel vital en el control de la propagación viral, minimizando el daño tisular e iniciando la respuesta inmunitaria adaptativa (84). Las citocinas esenciales [incluidas la interleucina-1 (IL-1), la interleucina-6 (IL-6), la interleucina-10 (IL-10) y el factor de necrosis tumoral alfa (TNF-α)] tienen funciones multifacéticas en la influencia del resultado de la infección por PRRSV (Tabla 2) (16, 85–91). Estas citocinas modulan la inflamación, ejercen efectos antivirales y activan las células inmunitarias (92).
Tabla 2. Resumen de diferentes cepas de virus que afectan a diferentes citocinas celulares.
El sistema inmunitario adaptativo (incluidas las células presentadoras de antígenos) ofrece un reconocimiento más amplio y sofisticado de los antígenos (93). Se basa en el reconocimiento específico de antígenos por parte de las células T y B que se generan a través de reordenamientos genéticos durante el desarrollo de los linfocitos, lo que da lugar a una especificidad única pero limitada (93). El sistema inmunitario innato es vital para eliminar los virus; sin embargo, puede no ser siempre adecuado para erradicar patógenos (93). Las funciones inmunitarias adquiridas son responsables de eliminar los virus y proporcionar inmunidad a largo plazo.
La respuesta inmunitaria adaptativa al PRRSV implica la activación de las células T y B, lo que da lugar a la producción de anticuerpos dirigidos y al desarrollo de respuestas inmunitarias celulares (94). Estas respuestas inmunitarias son cruciales para erradicar el virus y proteger contra la reinfección. Los intrincados mecanismos que subyacen a la respuesta inmunitaria adaptativa al PRRSV se exploraron ampliamente en revisiones anteriores (94, 95).
Se observaron diferencias en las respuestas inmunitarias entre varias razas de cerdos infectadas con PRRSV (14, 15, 17-19, 47, 82, 96-101). Por ejemplo, los cerdos TC demuestran una mayor resistencia al PRRSV con síntomas clínicos más leves, menos lesiones pulmonares y niveles más bajos de viremia en comparación con otras razas, como los cerdos LW. Estas distinciones pueden atribuirse a factores genéticos y variaciones en los niveles de citoquinas. Los cerdos TC muestran niveles séricos más altos de interferón-gamma (IFN-γ) que se asocia con una respuesta inmune celular mediada por células T, mientras que los cerdos LW muestran niveles elevados de interleucina-10 (IL-10) que puede inhibir la eliminación viral e impedir la respuesta inmune (17, 18). Estos indican la presencia de variaciones genéticas que influyen en la resistencia o susceptibilidad viral.
Un análisis más detallado de los ganglios linfáticos de cerdos TC y LW infectados con PRRSV reveló diferencias genéticas en la presentación de antígenos, el metabolismo y la activación inmunitaria; Esto sugiere que las variaciones genéticas contribuyen a respuestas inmunitarias divergentes (17). El análisis integrado de los datos transcriptómicos y metabolómicos proporciona información adicional sobre la respuesta inmunitaria a la infección por PRRSV al destacar la importancia de la activación inmunitaria, la capacidad de reconocimiento de antígenos, el metabolismo celular y el ciclo celular en la eliminación del PRRSV. Este análisis revela diferencias en el metabolismo de los lípidos y las vías de los aminoácidos entre los cerdos resistentes y susceptibles, e ilustra aún más el impacto de los factores genéticos en la respuesta inmunitaria (98).
Una comparación de las respuestas inmunitarias innatas de los cerdos Yorkshire convencionales y libres de patógenos específicos (SPF) al PRRSV muestra que los cerdos SPF tienen niveles elevados de citocinas proinflamatorias, como IL-1β, IL-6 y TNF-β, y concentraciones más altas de IFN-β en comparación con las razas de cerdos convencionales (102). Además, los cerdos con SPF exhiben niveles más bajos de ARN viral y síntomas clínicos menos graves en respuesta a las infecciones por PRRSV (102). Mientras tanto, un estudio de las respuestas inmunitarias innatas de los cerdos LW y Meishan al PRRSV reveló que la raza Meishan produce una mayor concentración de IFN-α y tiene cargas virales más bajas en comparación con la raza LW; esto sugiere una mayor resistencia al PRRSV debido a las diferencias en su respuesta inmune innata (16).
Además, la diversidad genómica viral del PRRSV (incluidas las diferencias en los epítopos inmunitarios) contribuye a su capacidad para evadir el sistema inmunitario (22). La variabilidad genética entre las cepas de PRRSV afecta a la respuesta de las razas porcinas a las infecciones. Las razas autóctonas, Large White y Yorkshire son más susceptibles al PRRSV, mientras que las razas Pietrain, Meishan y Hampshire son relativamente resistentes debido a las respuestas inmunitarias innatas más fuertes (82, 103). Las células dendríticas derivadas de cerdos Pietrain provocan una respuesta de células T más potente; Esto subraya la importancia de la inmunidad innata en la inmunidad adaptativa (104). La susceptibilidad de las razas porcinas a la infección por PRRSV está determinada principalmente por factores genéticos más que por factores ambientales o de cría.
Se identifican variaciones en los genes asociados con la respuesta inmune innata y la resistencia al PRRSV. Por ejemplo, los polimorfismos de un solo nucleótido (SNP) en genes como EIF2AK2, CD163, CD169 y RGS16 están relacionados con una mayor resistencia contra la infección por PRRSV (105).
Las diferencias en la inmunidad innata observadas entre varias razas de cerdos sugieren disparidades en su composición genética. La implementación de iniciativas de cría destinadas a mejorar la inmunidad innata puede ser una estrategia valiosa para mejorar la salud y la productividad de las piaras porcinas. Los criadores e investigadores pueden identificar los marcadores genéticos responsables del aumento de la inmunidad innata y el aumento de la resistencia al PRRSV profundizando en los mecanismos que regulan la inmunidad innata.
5. Eventos de empalme alternativos diferenciales desencadenados por la invasión del PRRSV en diferentes razas porcinas
El empalme alternativo (AS) es un mecanismo crucial para el procesamiento postranscripcional del ARN y es responsable de la modificación significativa de las secuencias de transcripción (106, 107). Es un mecanismo crítico en la regulación de la expresión génica eucariota a través del control transcripcional que mejora la versatilidad y diversidad de transcriptomas y proteomas (108-111). Esto da como resultado varios eventos de empalme alternativos (ASE), incluidos los exones omitidos (SE), los intrones retenidos (RI), los sitios de empalme alternativos 5′ y 3′ (A5SS y A3SS) y los eventos mutuamente excluyentes (ME) (112, 113).
La infección por el virus del síndrome respiratorio y reproductivo porcino afecta profundamente al empalme alternativo en cerdos. Específicamente, los genes relacionados con la respuesta inmune (como los genes estimulados por interferón) exhiben un empalme alternativo después de la infección por PRRSV (114). Esto sugiere que el empalme alternativo puede desempeñar un papel en la regulación de la respuesta del huésped a las infecciones virales, como el PRRSV. Además, la infección por PRRSV puede desencadenar eventos generalizados de EA en el bazo y los ganglios linfáticos inguinales (ILN) de cerdos TC y LW. La infección por PRRSV dio lugar a que 373 y 595 genes mostraran ASE diferenciales en el bazo y la ILN en cerdos TC, respectivamente (114). Mientras tanto, 458 y 560 genes exhiben ASE diferenciales en el bazo y la ILN, respectivamente en cerdos LW. El análisis funcional de la ontología génica reveló que estos genes son importantes para las respuestas inmunes, la regulación transcripcional, el metabolismo y la apoptosis (114). Además, la respuesta al PRRSV en términos de empalme alternativo difirió significativamente entre los cerdos TC y LW. Esto sugiere un posible vínculo entre la infección por PRRSV y la variación genética en estas dos razas de cerdos.
6. Funciones de los ARN no codificantes del huésped en la infección y replicación por PRRSV
Los ARN no codificantes (ncRNAs) desempeñan diversas funciones en la infección y replicación del PRRSV. El PRRSV es un virus de ARN que posee una región larga no traducida (UTR) aguas abajo de su marco de lectura abierto, 1ab (21). La investigación sugiere que el 3′-UTR desempeña un papel importante en la modulación de los ARNm dirigidos en animales (115, 116). Esto indica que la UTR extendida en el PRRSV podría servir potencialmente como un conjunto de dianas para los miARN del huésped.
Los miRNAs desempeñan un papel fundamental en la regulación de la replicación viral y la infección de la respuesta inmunitaria del huésped durante el PRRSV. Por ejemplo, miR-181 inhibe el PRRSV in vivo e in vitro, y la administración terapéutica de miR-181 alivia los síntomas y prolonga la supervivencia de los cerdos infectados por PRRSV altamente patógenos (117). Además, regula a la baja el receptor CD163 del PRRSV, lo que dificulta eficazmente la infección por PRRSV. La construcción de un PRRSV mutado en el sitio diana de miR-181 demostró que miR-181 bloqueó eficazmente la invasión del PRRSV de tipo salvaje en la etapa tardía, lo que sugiere su impacto significativo en la infección y replicación del PRRSV in vivo. Además, el miR-23 celular inhibe la replicación del PRRSV al dirigirse directamente al ARN del PRRSV y potencialmente al alza el interferón tipo I (118). MiR-378 y miR-505 suprimen la replicación del PRRSV al dirigirse directamente al ARN del PRRSV (118), mientras que miR-10a-5p inhibe la replicación del PRRSV al suprimir la expresión de SRP14 (119).
Por el contrario, el miR-142-5p inducido por PRRSV promueve significativamente la replicación viral al dirigirse directamente a FAM134B (120). Por el contrario, los miRNAs de la familia let-7 inhiben la replicación del PRRSV al dirigirse a la 3′-UTR del genoma del PRRSV-2 y a la IL-6 porcina (121). La expresión de MiR-146a aumenta en los macrófagos durante la infección por PRRSV, y afecta positivamente a la respuesta inmune mediante la regulación de la expresión de genes, como C1QTNF3 y MAFB (122). En particular, ni las células diana infectadas por el PRRSV ni los cerdos huéspedes inducen la producción de proteínas de interferón tipo I (IFN) in vivo o in vitro (123). Los miRNAs pueden ser inducidos o reprimidos por el IFN tipo I, aunque también pueden desempeñar un papel clave en la regulación de las respuestas inmunitarias innatas mediante la modulación de la producción de IFN tipo I y otras vías moleculares importantes (124). En particular, miR-331-3p/miR-210 está implicado en la inflamación pulmonar al dirigirse a ORF1b y regular a la baja STAT1/TNF-α (16).
Let-7b, miR-26a, miR-34a y miR-145 se dirigen directamente a secuencias dentro de las regiones β-UTR de IFN-β 3 porcino en las posiciones 160-181, 9-31, 27-47 y 12-32 pb, respectivamente, para inhibir la expresión de la proteína IFN-β en las PAM primarias (125). Además, se sugiere que el PRRSV puede suprimir la expresión postranscripcional de la proteína IFN-β mediante la regulación positiva de estos cuatro miARN en PAM cultivados (126). MiR-199a-3p regula a la baja la expresión proteica de CD151, un receptor utilizado por el PRRSV (125). Además, miR-199a-3p se expresa diferencialmente en los tejidos pulmonares de diferentes razas de cerdos (como Tongcheng y Landrace) en los grupos de control e infección; esto sugiere su papel crucial en la regulación de la infección por PRRSV en cerdos (127). Además, miR-378 y miR-10a-5p están regulados al alza en el grupo de control de cerdos Tongcheng en comparación con los del grupo de control de cerdos Landrace, y ambos miRNAs mostraron efectos inhibidores sobre la replicación del PRRSV (126).
Numerosos estudios han explorado el papel de los miRNAs en el proceso del PRRSV, arrojando luz sobre sus mecanismos reguladores en la infección viral y revelando variaciones en la expresión de miRNAs entre diferentes razas de cerdos. Estos hallazgos proporcionan nuevos conocimientos sobre la interacción entre el PRRSV y el huésped, y presentan vías prometedoras para desarrollar estrategias antivirales contra la infección por PRRSV.
7. Perspectivas de futuro
La investigación futura debe centrarse en varias áreas clave para avanzar en nuestra comprensión de las infecciones por PRRSV e impulsar el desarrollo de estrategias de control efectivas.
En primer lugar, se requieren estudios exhaustivos para investigar los factores genéticos que subyacen a las diversas respuestas a la infección por PRRSV entre diferentes razas de cerdos. La identificación de genes específicos y marcadores genéticos asociados con la resistencia o susceptibilidad al PRRSV proporcionará información valiosa sobre los programas de mejoramiento específicos y el desarrollo de enfoques de medicina de precisión. A través de la mejora genética y los métodos de cría selectiva, es posible criar razas de cerdos que presentan una mayor resistencia al PRRSV. Aprovechando las modernas tecnologías de ingeniería genética y los métodos de cría selectiva, se pueden seleccionar individuos con respuestas inmunitarias robustas para la reproducción, mejorando así gradualmente la resistencia general al PRRSV en toda la población porcina.
En segundo lugar, los rápidos avances en la secuenciación del transcriptoma de una sola célula y la transcriptómica espacial presentan oportunidades para obtener información molecular detallada sobre la infección por PRRSV. La investigación adicional en estas áreas puede proporcionar una comprensión integral de la propagación viral dentro de los tejidos, la dinámica de las interacciones huésped-virus y cómo varios tipos de células huésped contribuyen a la patogénesis y la respuesta inmunitaria contra el PRRSV.
Además, se debe explorar el papel funcional de los ncRNAs en la infección y replicación del PRRSV. Investigaciones adicionales sobre los mecanismos reguladores de los ncRNAs del huésped en la modulación de la replicación viral, las respuestas inmunitarias y los resultados de la enfermedad podrían conducir al desarrollo de nuevas intervenciones terapéuticas y a la identificación de posibles biomarcadores con fines diagnósticos.
Además, la incorporación de enfoques multiómicos (como la transcriptómica, la genómica, la proteómica y la epigenómica) proporcionará una comprensión más completa de la compleja interacción molecular entre el PRRSV y el huésped. La integración de estos conjuntos de datos ómicos podría descubrir interacciones, vías y redes cruciales involucradas en la infección por PRRSV y las respuestas del huésped que, en última instancia, conducen a estrategias preventivas y terapéuticas más efectivas.
Por último, los esfuerzos deben seguir centrándose en prácticas sostenibles de cría de cerdos que reduzcan la dependencia de los antibióticos y mitiguen los impactos ambientales. Esto incluye la integración de la información genética en los programas de mejoramiento genético para seleccionar rasgos de resistencia a las enfermedades, el avance de las tecnologías de agricultura de precisión para la detección temprana y la intervención, y la promoción de medidas de bioseguridad para minimizar el riesgo de transmisión de patógenos.
En conclusión, los futuros estudios sobre el PRRSV deberían avanzar en nuestra comprensión de los mecanismos genéticos y moleculares que subyacen a las interacciones entre el huésped y el virus, desarrollar estrategias de control específicas y promover prácticas sostenibles de cría de cerdos. El aprovechamiento de estos conocimientos facilitará una prevención, gestión y control más eficaces del PRRSV que beneficien la salud y la productividad de los cerdos.
8. Conclusión
En general, los hallazgos de esta revisión contribuyen a una comprensión integral de los factores genéticos que subyacen a las diversas respuestas al PRRSV en diferentes razas de cerdos a la infección por PRRSV. Este conocimiento se puede utilizar para formular medidas de control más efectivas para el PRRSV, como el diseño de programas de mejoramiento genético para seleccionar rasgos de resistencia y el desarrollo de vacunas dirigidas. Además, la exploración del potencial de las intervenciones basadas en la genética y la investigación adicional sobre las interacciones huésped-virus a nivel molecular son prometedoras para futuros desarrollos en el control y la prevención del PRRSV.
Contribuciones de los autores
YP: Escritura – borrador original, Escritura – revisión y edición. CL: Escritura – borrador original. NS: Redacción – revisión y edición. ZF: Redacción – borrador original, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Esta investigación fue financiada por la Fundación de Investigación Básica y Básica Aplicada de Guangdong (2020B1515120016), Programa de Investigación y Desarrollo de Tecnologías Clave de la Provincia de Guangdong (2022B0202090001).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Wensvoort, G, Terpstra, C, Pol, JMA, ter Laak, EA, Bloemraad, M, de Kluyver, EP, et al. Mystery swine disease in the Netherlands: the isolation of Lelystad virus. Vet Q. (1991) 13:121–30. doi: 10.1080/01652176.1991.9694296
2. Benfield, DA, Nelson, E, Collins, JE, Harris, L, Goyal, SM, Robison, D, et al. Characterization of swine infertility and respiratory syndrome (SIRS) virus (isolate ATCC VR-2332). J Vet Diagn Investig. (1992) 4:127–33. doi: 10.1177/104063879200400202
3. Neumann, EJ, Kliebenstein, JB, Johnson, CD, Mabry, JW, Bush, EJ, Seitzinger, AH, et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States. J Am Vet Med Assoc. (2005) 227:385–92. doi: 10.2460/javma.2005.227.385
4. Zhang, Z, Li, Z, Li, H, Yang, S, Ren, F, Bian, T, et al. The economic impact of porcine reproductive and respiratory syndrome outbreak in four Chinese farms: based on cost and revenue analysis. Front Vet Sci. (2022) 9:1024720. doi: 10.3389/fvets.2022.1024720
5. Renken, C, Nathues, C, Swam, H, Fiebig, K, Weiss, C, Eddicks, M, et al. Application of an economic calculator to determine the cost of porcine reproductive and respiratory syndrome at farm-level in 21 pig herds in Germany. Porcine Health Manag. (2021) 7:3. doi: 10.1186/s40813-020-00183-x
6. Dea, S, Gagnon, CA, Mardassi, H, Pirzadeh, B, and Rogan, D. Current knowledge on the structural proteins of porcine reproductive and respiratory syndrome (PRRS) virus: comparison of the north American and European isolates. Arch Virol. (2000) 145:659–88. doi: 10.1007/s007050050662
Resumen de PubMed | Texto completo de CrossRef | Google Académico
7. Murtaugh, MP, Shi, M, Brar, MS, y Leung, FCC. Diversificación evolutiva del virus del síndrome respiratorio y reproductivo porcino tipo 2. J Gen Virol. (2015) 96:1570–80. doi: 10.1099/vir.0.000104
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Allende, R, Lewis, TL, Lu, Z, Rock, DL, Kutish, GF, Ali, A, et al. Los virus del síndrome respiratorio y reproductivo porcino de América del Norte y Europa difieren en las regiones codificantes de proteínas no estructurales. J Gen Virol. (1999) 80:307–15. doi: 10.1099/0022-1317-80-2-307
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Plagemann, PG . Virus del síndrome respiratorio y reproductivo porcino: hipótesis de origen. Emerg Infect Dis. (2003) 9:903–8. doi: 10.3201/eid0908.030232
10. Su, CM, Rowland, RRR y Yoo, D. Avances recientes en los receptores del virus del PRRS y la focalización del receptor-ligando para el control. Vacunas (Basilea). (2021) 9:354. doi: 10.3390/vaccines9040354
11. du, T, Nan, Y, Xiao, S, Zhao, Q y Zhou, EM. Estrategias antivirales contra la infección por PRRSV. Tendencias Microbiol. (2017) 25:968–79. doi: 10.1016/j.tim.2017.06.001
12. Kick, AR, Grete, AF, Crisci, E, Almond, GW y Käser, T. Correlatos inmunitarios candidatos comprobables de protección para la vacunación contra el virus del síndrome respiratorio y reproductivo porcino. Vacunas (Basilea). (2023) 11:594. doi: 10.3390/vacunas11030594
13. Li, Z, Li, L, Zhao, S, Li, J, Zhou, H, Zhang, Y, et al. Reentender la estrategia antigripal: conceder la misma importancia a las terapias antivirales y antiinflamatorias. J Dis. de tórax. (2018) 10:S2248–59. doi: 10.21037/jtd.2018.03.169
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Petry, DB, Holl, JW, Weber, JS, Doster, AR, Osorio, FA y Johnson, RK. Respuestas biológicas al virus del síndrome respiratorio y reproductivo porcino en cerdos de dos poblaciones genéticas. J Anim Sci. (2005) 83:1494–502. doi: 10.2527/2005.8371494x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Vincent, AL, Thacker, BJ, Halbur, PG, Rothschild, MF y Thacker, EL. La susceptibilidad in vitro de los macrófagos al virus del síndrome respiratorio y reproductivo porcino varía entre líneas de cerdos genéticamente diversas. Inmunología Viral. (2005) 18:506–12. doi: 10.1089/vim.2005.18.506
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Tú, X, Qu, Y, Zhang, Y, Huang, J, Gao, X, Huang, C, et al. Mir-331-3p inhibe la replicación del PRRSV-2 y la lesión pulmonar al dirigirse al PRRSV-2 ORF1b y al TNF-alfa porcino. Frente Immunol. (2020) 11:547144. doi: 10.3389/fimmu.2020.547144
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Liang, W, Meng, X, Zhen, Y, Zhang, Y, Hu, X, Zhang, Q, et al. La integración del transcriptoma y el proteoma en los ganglios linfáticos revela las diferentes respuestas inmunitarias al PRRSV entre los cerdos Tongcheng resistentes al PRRSV y los cerdos blancos grandes susceptibles al PRRSV. Frente Genet. (2022) 13:800178. doi: 10.3389/fgene.2022.800178
18. Liang, W, Li, Z, Wang, P, Fan, P, Zhang, Y, Zhang, Q, et al. Differences of immune responses between Tongcheng (Chinese local breed) and large white pigs after artificial infection with highly pathogenic porcine reproductive and respiratory syndrome virus. Virus Res. (2016) 215:84–93. doi: 10.1016/j.virusres.2016.02.004
19. Bates, JS, Petry, DB, Eudy, J, Bough, L, and Johnson, RK. Differential expression in lung and bronchial lymph node of pigs with high and low responses to infection with porcine reproductive and respiratory syndrome virus. J Anim Sci. (2008) 86:3279–89. doi: 10.2527/jas.2007-0685
20. Snijder, EJ, and Meulenberg, JJ. The molecular biology of arteriviruses. J Gen Virol. (1998) 79:961–79.
21. Dokland, T . The structural biology of PRRSV. Virus Res. (2010) 154:86–97. doi: 10.1016/j.virusres.2010.07.029
22. Huang, C, Zhang, Q, and Feng, WH. Regulation and evasion of antiviral immune responses by porcine reproductive and respiratory syndrome virus. Virus Res. (2015) 202:101–11. doi: 10.1016/j.virusres.2014.12.014
23. Matanin, BM, Huang, Y, Meng, XJ, and Zhang, C. Purification of the major envelop protein GP5 of porcine reproductive and respiratory syndrome virus (PRRSV) from native virions. J Virol Methods. (2008) 147:127–35. doi: 10.1016/j.jviromet.2007.08.018
24. Johnson, CR, Griggs, TF, Gnanandarajah, J, and Murtaugh, MP. Novel structural protein in porcine reproductive and respiratory syndrome virus encoded by an alternative ORF5 present in all arteriviruses. J Gen Virol. (2011) 92:1107–16. doi: 10.1099/vir.0.030213-0
25. Wu, WH, Fang, Y, Farwell, R, Steffen-Bien, M, Rowland, RRR, Christopher-Hennings, J, et al. A 10-kDa structural protein of porcine reproductive and respiratory syndrome virus encoded by ORF2b. Virology. (2001) 287:183–91. doi: 10.1006/viro.2001.1034
26. Lawson, SR, Rossow, KD, Collins, JE, Benfield, DA, and Rowland, RRR. Porcine reproductive and respiratory syndrome virus infection of gnotobiotic pigs: sites of virus replication and co-localization with MAC-387 staining at 21 days post-infection. Virus Res. (1997) 51:105–13. doi: 10.1016/S0168-1702(97)00086-5
27. Duan, X, Nauwynck, HJ, and Pensaert, MB. Virus quantification and identification of cellular targets in the lungs and lymphoid tissues of pigs at different time intervals after inoculation with porcine reproductive and respiratory syndrome virus (PRRSV). Vet Microbiol. (1997) 56:9–19. doi: 10.1016/S0378-1135(96)01347-8
28. Kim, HS, Kwang, J, Yoon, IJ, Joo, HS, and Frey, ML. Enhanced replication of porcine reproductive and respiratory syndrome (PRRS) virus in a homogeneous subpopulation of MA-104 cell line. Arch Virol. (1993) 133:477–83. doi: 10.1007/BF01313785
29. Calvert, JG, Slade, DE, Shields, SL, Jolie, R, Mannan, RM, Ankenbauer, RG, et al. CD163 expression confers susceptibility to porcine reproductive and respiratory syndrome viruses. J Virol. (2007) 81:7371–9. doi: 10.1128/JVI.00513-07
30. Stoian, AMM, and Rowland, RRR. Challenges for porcine reproductive and respiratory syndrome (PRRS) vaccine design: reviewing virus glycoprotein interactions with CD163 and targets of virus neutralization. Vet Sci. (2019) 6:9. doi: 10.3390/vetsci6010009
31. Whitworth, KM, Rowland, RRR, Ewen, CL, Trible, BR, Kerrigan, MA, Cino-Ozuna, AG, et al. Gene-edited pigs are protected from porcine reproductive and respiratory syndrome virus. Nat Biotechnol. (2016) 34:20–2. doi: 10.1038/nbt.3434
32. Burkard, C, Lillico, SG, Reid, E, Jackson, B, Mileham, AJ, Ait-Ali, T, et al. Precision engineering for PRRSV resistance in pigs: macrophages from genome edited pigs lacking CD163 SRCR5 domain are fully resistant to both PRRSV genotypes while maintaining biological function. PLoS Pathog. (2017) 13:e1006206. doi: 10.1371/journal.ppat.1006206
33. Xu, K, Zhou, Y, Mu, Y, Liu, Z, Hou, S, Xiong, Y, et al. CD163 and pAPN double-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance. elife. (2020) 9:9. doi: 10.7554/eLife.57132
34. Stoian, AMM, Rowland, RRR, and Brandariz-Nunez, A. Mutations within scavenger receptor cysteine-rich (SRCR) protein domain 5 of porcine CD163 involved in infection with porcine reproductive and respiratory syndrome virus (PRRS). J Gen Virol. (2022) 103:001740. doi: 10.1099/jgv.0.001740
35. Guo, CH, Wang, M, Zhu, Z, He, S, Liu, H, Liu, X, et al. Highly efficient generation of pigs harboring a partial deletion of the CD163 SRCR5 domain, which are fully resistant to porcine reproductive and respiratory syndrome virus 2 infection. Front Immunol. (2019) 10:1846. doi: 10.3389/fimmu.2019.01846
36. Ye, N, Wang, B, Feng, W, Tang, D, and Zeng, Z. PRRS virus receptors and an alternative pathway for viral invasion. Virus Res. (2022) 320:198885. doi: 10.1016/j.virusres.2022.198885
37. Delputte, PL, Vanderheijden, N, Nauwynck, HJ, and Pensaert, MB. Involvement of the matrix protein in attachment of porcine reproductive and respiratory syndrome virus to a heparinlike receptor on porcine alveolar macrophages. J Virol. (2002) 76:4312–20. doi: 10.1128/JVI.76.9.4312-4320.2002
38. Delputte, PL, Costers, S, and Nauwynck, HJ. Analysis of porcine reproductive and respiratory syndrome virus attachment and internalization: distinctive roles for heparan sulphate and sialoadhesin. J Gen Virol. (2005) 86:1441–5. doi: 10.1099/vir.0.80675-0
39. Delputte, PL, van Breedam, W, Delrue, I, Oetke, C, Crocker, PR, and Nauwynck, HJ. Porcine arterivirus attachment to the macrophage-specific receptor sialoadhesin is dependent on the sialic acid-binding activity of the N-terminal immunoglobulin domain of sialoadhesin. J Virol. (2007) 81:9546–50. doi: 10.1128/JVI.00569-07
40. An, TQ, Tian, ZJ, He, YX, Xiao, Y, Jiang, YF, Peng, JM, et al. Porcine reproductive and respiratory syndrome virus attachment is mediated by the N-terminal domain of the sialoadhesin receptor. Vet Microbiol. (2010) 143:371–8. doi: 10.1016/j.vetmic.2009.11.006
41. Delrue, I, van Gorp, H, van Doorsselaere, J, Delputte, PL, and Nauwynck, HJ. Susceptible cell lines for the production of porcine reproductive and respiratory syndrome virus by stable transfection of sialoadhesin and CD163. BMC Biotechnol. (2010) 10:48. doi: 10.1186/1472-6750-10-48
42. Xie, J, Vereecke, N, Theuns, S, Oh, D, Vanderheijden, N, Trus, I, et al. Comparison of primary virus isolation in pulmonary alveolar macrophages and four different continuous cell lines for type 1 and type 2 porcine reproductive and respiratory syndrome virus. Vaccines (Basel). (2021) 9:594. doi: 10.3390/vaccines9060594
43. Vanderheijden, N, Delputte, PL, Favoreel, HW, Vandekerckhove, J̈, van Damme, J, van Woensel, PA, et al. Implicación de sialoadhesina en la entrada del virus del síndrome respiratorio y reproductivo porcino en macrófagos alveolares porcinos. J Virol. (2003) 77:8207–15. doi: 10.1128/JVI.77.15.8207-8215.2003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. van Breedam, W, van Gorp, H, Zhang, JQ, Crocker, PR, Delputte, PL, y Nauwynck, HJ. El complejo glicoproteico M/GP(5) del virus del síndrome respiratorio y reproductivo porcino se une al receptor de sialoadhesina de una manera dependiente del ácido siálico. PLoS Pathog. (2010) 6:E1000730. doi: 10.1371/journal.ppat.1000730
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Kim, JK, Fahad, AM, Shanmukhappa, K y Kapil, S. Definición de la(s) diana(s) celular(es) del anticuerpo monoclonal 7G10 que bloquea el virus del síndrome respiratorio y reproductivo porcino. J Virol. (2006) 80:689–96. doi: 10.1128/JVI.80.2.689-696.2006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Wang, WW, Zhang, L, Ma, XC, Gao, JM, Xiao, YH y Zhou, EM. El papel de la vimentina durante la infección por PRRSV de las células Marc-145. Bing Du Xue Bao. (2011) 27:456–61. doi: 10.13242/j.cnki.bingduxuebao.002205
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Shi, C, Liu, Y, Ding, Y, Zhang, Y y Zhang, J. Receptores del PRRSV y su papel en la infección por virus. Arch Microbiol. (2015) 197:503–12. doi: 10.1007/s00203-015-1088-1
48. Zheng, XX, Li, R, Qiao, S, Chen, XX, Zhang, L, Lu, Q, et al. El reordenamiento de la vimentina por fosforilación es beneficioso para la replicación in vitro del virus del síndrome respiratorio y reproductivo porcino. Microbiol veterinario. (2021) 259:109133. doi: 10.1016/j.vetmic.2021.109133
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Shanmukhappa, K, Kim, JK y Kapil, S. Papel de CD151, una tetraspanina, en la infección por el virus del síndrome respiratorio y reproductivo porcino. Virol J. (2007) 4:62. doi: 10.1186/1743-422X-4-62
50. Wu, J, Peng, X, Zhou, A, Qiao, M, Wu, H, Xiao, H, et al. MiR-506 inhibe la replicación del PRRSV en las células MARC-145 a través de CD151. Bioquímica de células molinos. (2014) 394:275–81. doi: 10.1007/s11010-014-2103-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
51. Gao, J, Xiao, S, Xiao, Y, Wang, X, Zhang, C, Zhao, Q, et al. MYH9 es un factor esencial para la infección por el virus del síndrome respiratorio y reproductivo porcino. Sci Rep. (2016) 6:25120. doi: 10.1038/srep25120
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Xue, B, Hou, G, Zhang, G, Huang, J, Li, L, Nan, Y, et al. La agregación de MYH9 inducida por la interacción directa con el ectodominio PRRSV GP5 facilita la internalización viral por parte de las células permisivas. Microbiol frontal. (2019) 10:2313. doi: 10.3389/fmicb.2019.02313
Resumen de PubMed | Texto completo de CrossRef | Google Académico
53. Li, L, Zhang, L, Hu, Q, Zhao, L, Nan, Y, Hou, G, et al. Los residuos de aminoácidos clave de MYH9 identificados por el anticuerpo antiidiotípico contra la glicoproteína 5 del virus del síndrome respiratorio y reproductivo porcino participan en la internalización del virus por macrófagos alveolares porcinos. Virus. (2020) 12:40. doi: 10.3390/v12010040
54. Wang, L, Li, R, Geng, R, Zhang, L, Chen, XX, Qiao, S, et al. Heat shock protein member 8 (HSPA8) is involved in porcine reproductive and respiratory syndrome virus attachment and internalization. Microbiol Spectr. (2022) 10:e0186021. doi: 10.1128/spectrum.01860-21
55. Li, J, Jiang, P, Li, Y, Wang, X, Cao, J, Wang, X, et al. HSP70 fused with GP3 and GP5 of porcine reproductive and respiratory syndrome virus enhanced the immune responses and protective efficacy against virulent PRRSV challenge in pigs. Vaccine. (2009) 27:825–32. doi: 10.1016/j.vaccine.2008.11.088
56. Stoian, AMM, Rowland, RRR, and Brandariz-Nuñez, A. Identification of CD163 regions that are required for porcine reproductive and respiratory syndrome virus (PRRSV) infection but not for binding to viral envelope glycoproteins. Virology. (2022) 574:71–83. doi: 10.1016/j.virol.2022.07.012
57. Wei, X, Li, R, Qiao, S, Chen, XX, Xing, G, and Zhang, G. Porcine reproductive and respiratory syndrome virus utilizes viral apoptotic mimicry as an alternative pathway to infect host cells. J Virol. (2020) 94:e00709-20. doi: 10.1128/JVI.00709-20
58. Segawa, K, and Nagata, S. An apoptotic ‘Eat Me’ signal: phosphatidylserine exposure. Trends Cell Biol. (2015) 25:639–50. doi: 10.1016/j.tcb.2015.08.003
59. Cvetanovic, M, and Ucker, DS. Innate immune discrimination of apoptotic cells: repression of proinflammatory macrophage transcription is coupled directly to specific recognition. J Immunol. (2004) 172:880–9. doi: 10.4049/jimmunol.172.2.880
60. Amara, A, and Mercer, J. Viral apoptotic mimicry. Nat Rev Microbiol. (2015) 13:461–9. doi: 10.1038/nrmicro3469
61. Diaz-Griffero, F, Jackson, AP, and Brojatsch, J. Cellular uptake of avian leukosis virus subgroup B is mediated by clathrin. Virology. (2005) 337:45–54. doi: 10.1016/j.virol.2005.02.027
62. Mulloy, B, Lever, R, and Page, CP. Mast cell glycosaminoglycans. Glycoconj J. (2017) 34:351–61. doi: 10.1007/s10719-016-9749-0
63. Guo, C, Zhu, Z, Guo, Y, Wang, X, Yu, P, Xiao, S, et al. Heparanase upregulation contributes to porcine reproductive and respiratory syndrome virus release. J Virol. (2017) 91:e00625-17. doi: 10.1128/JVI.00625-17
64. Prather, RS, Rowland, RRR, Ewen, C, Trible, B, Kerrigan, M, Bawa, B, et al. An intact sialoadhesin (Sn/SIGLEC1/CD169) is not required for attachment/internalization of the porcine reproductive and respiratory syndrome virus. J Virol. (2013) 87:9538–46. doi: 10.1128/JVI.00177-13
65. Hasegawa, H, Watanabe, H, Nomura, T, Utsunomiya, Y, Yanagisawa, K y Fujita, S. Clonación molecular y expresión del homólogo de ratón de SFA-1 / PETA-3 (CD151), un miembro de la superfamilia transmembrana 4. Biochim Biophys Acta. (1997) 1353:125–30. doi: 10.1016/S0167-4781(97)00095-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
66. Li, L, Xue, B, Sun, W, Gu, G, Hou, G, Zhang, L, et al. El dominio C-terminal de la proteína MYH9 recombinante bloquea la internalización del virus del síndrome respiratorio y reproductivo porcino mediante la interacción directa con la glicoproteína viral 5. Antivir Res. (2018) 156:10–20. doi: 10.1016/j.antiviral.2018.06.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
67. Vicente-Manzanares, M, Ma, X, Adelstein, RS, y Horwitz, AR. La miosina II no muscular ocupa un lugar central en la adhesión y migración celular. Nat Rev Mol Cell Biol. (2009) 10:778–90. DOI: 10.1038/NRM2786
Resumen de PubMed | Texto completo de CrossRef | Google Académico
68. Vega-Almeida, TO, Salas-Benito, M, de Nova-Ocampo, MA, del Ángel, RM, y Salas-Benito, JS. Proteínas de superficie de las células C6/36 implicadas en la unión y entrada del virus del dengue 4. Arch Virol. (2013) 158:1189–207. doi: 10.1007/s00705-012-1596-0
Resumen de PubMed | Texto completo de CrossRef | Google Académico
69. Yu, A, Shibata, Y, Shah, B, Calamini, B, Lo, DC y Morimoto, RI. La agregación de proteínas puede inhibir la endocitosis mediada por clatrina por la competencia de chaperonas. Proc Natl Acad Sci U S A. (2014) 111:E1481–90. doi: 10.1073/pnas.1321811111
Resumen de PubMed | Texto completo de CrossRef | Google Académico
70. Jiang, C, Xing, F, Xing, J, Jiang, Y y Zhou, E. Diferentes patrones de expresión de genes mediadores del PRRSV en los tejidos pulmonares de cerdos resistentes y susceptibles al PRRSV. Dev Comp Immunol. (2013) 39:127–31. doi: 10.1016/j.dci.2012.01.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
71. Meng, CH, Su, L, Li, Y, Zhu, Q, Li, J, Wang, H, et al. Diferente susceptibilidad a la infección por el virus del síndrome respiratorio y reproductivo porcino entre las razas de cerdos autóctonas chinas. Arch Virol. (2018) 163:2155–64. doi: 10.1007/s00705-018-3821-y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
72. Wang, F, Qiu, H, Zhang, Q, Peng, Z y Liu, B. Asociación de dos genes receptores del virus del síndrome respiratorio y reproductivo porcino (PRRSV), CD163 y SN con rasgos inmunológicos. Mol Biol Rep. (2012) 39:3971–6. doi: 10.1007/s11033-011-1177-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
73. You, X, Li, G, Lei, Y, Xu, Z, Zhang, P y Yang, Y. Papel de los factores genéticos en diferentes razas porcinas que exhiben diferentes niveles de resistencia/susceptibilidad al PRRSV. Virus Res. (2023) 326:199057. doi: 10.1016/j.virusres.2023.199057
Resumen de PubMed | Texto completo de CrossRef | Google Académico
74. Nodelijk, G . Síndrome reproductivo y respiratorio porcino (PRRS) con especial referencia a los aspectos clínicos y diagnósticos. Una reseña Vet Q. (2002) 24:95–100. doi: 10.1080/01652176.2002.9695128
75. Salguero, FJ, Frossard, JP, Rebel, JMJ, Stadejek, T, Morgan, SB, Graham, SP, et al. Interacciones huésped-patógeno durante la infección porcina por el virus del síndrome respiratorio y reproductivo 1 en lechones. Virus Res. (2015) 202:135–43. doi: 10.1016/j.virusres.2014.12.026
76. Clilverd, H, Martín-Valls, G, Li, Y, Martín, M, Cortey, M, and Mateu, E. Infection dynamics, transmission, and evolution after an outbreak of porcine reproductive and respiratory syndrome virus. Front Microbiol. (2023) 14:1109881. doi: 10.3389/fmicb.2023.1109881
77. Done, SH, and Paton, DJ. Porcine reproductive and respiratory syndrome: clinical disease, pathology and immunosuppression. Vet Rec. (1995) 136:32–5. doi: 10.1136/vr.136.2.32
78. Paiva, RC, Moura, CA, Thomas, P, Haberl, B, Greiner, L, Rademacher, CJ, et al. Risk factors associated with sow mortality in breeding herds under one production system in the midwestern United States. Prev Vet Med. (2023) 213:105883. doi: 10.1016/j.prevetmed.2023.105883
79. Pejsak, Z, Stadejek, T, and Markowska-Daniel, I. Clinical signs and economic losses caused by porcine reproductive and respiratory syndrome virus in a large breeding farm. Vet Microbiol. (1997) 55:317–22. doi: 10.1016/s0378-1135(96)01326-0
80. Kang, RM, Ji, G, Yang, X, Lv, X, Zhang, Y, Ge, M, et al. Investigation on host susceptibility of Tibetan pig to infection of porcine reproductive and respiratory syndrome virus through viral challenge study. Vet Microbiol. (2016) 183:62–8. doi: 10.1016/j.vetmic.2015.11.035
81. Reiner, G, Willems, H, Pesch, S, and Ohlinger, VF. Variation in resistance to the porcine reproductive and respiratory syndrome virus (PRRSV) in Pietrain and miniature pigs. J Anim Breed Genet. (2010) 127:100–6. doi: 10.1111/j.1439-0388.2009.00818.x
82. Christopher-Hennings, J, Holler, LD, Benfield, DA, and Nelson, EA. Detection and duration of porcine reproductive and respiratory syndrome virus in semen, serum, peripheral blood mononuclear cells, and tissues from Yorkshire, Hampshire, and landrace boars. J Vet Diagn Investig. (2001) 13:133–42. doi: 10.1177/104063870101300207
83. Fitzgerald, KA, and Kagan, JC. Toll-like receptors and the control of immunity. Cells. (2020) 180:1044–66. doi: 10.1016/j.cell.2020.02.041
84. Ishii, KJ, Koyama, S, Nakagawa, A, Coban, C, and Akira, S. Host innate immune receptors and beyond: making sense of microbial infections. Cell Host Microbe. (2008) 3:352–63. doi: 10.1016/j.chom.2008.05.003
85. Xu, Y, Wang, H, Zhang, X, Zheng, X, Zhu, Y, Han, H, et al. Highly pathogenic porcine reproductive and respiratory syndrome virus (HP-PRRSV) induces IL-6 production through TAK-1/JNK/AP-1 and TAK-1/NF-kappaB signaling pathways. Vet Microbiol. (2021) 256:109061. doi: 10.1016/j.vetmic.2021.109061
86. Liu, Y, du, Y, Wang, H, du, L, and Feng, WH. Porcine reproductive and respiratory syndrome virus (PRRSV) up-regulates IL-8 expression through TAK-1/JNK/AP-1 pathways. Virology. (2017) 506:64–72. doi: 10.1016/j.virol.2017.03.009
87. du, L, Liu, Y, du, Y, Wang, H, Zhang, M, du, Y, et al. El virus del síndrome respiratorio y reproductivo porcino (PRRSV) regula al alza la IL-15 a través de la vía de señalización PKCbeta1-TAK1-NF-kappaB. Virología. (2016) 496:166–74. doi: 10.1016/j.virol.2016.06.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
88. Yu, Z, Huang, C, Zhang, Q y Feng, WH. El virus del síndrome respiratorio y reproductivo porcino (PRRSV) induce la producción de IL-12p40 a través de las vías de señalización JNK-AP-1 y NF-kappaB. Virus Res. (2016) 225:73–81. doi: 10.1016/j.virusres.2016.09.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
89. Wang, H, du, L, Liu, F, Wei, Z, Gao, L y Feng, WH. El virus del síndrome respiratorio y reproductivo porcino altamente patógeno induce la producción de interleucina-17 a través de la activación de las vías IRAK1-PI3K-p38MAPK-C/EBPbeta/CREB. J Virol. (2019) 93:E01100-19. doi: 10.1128/JVI.01100-19
Resumen de PubMed | Texto completo de CrossRef | Google Académico
90. Flores-Mendoza, L, Silva-Campa, E, Reséndiz, Ḿ, Osorio, FA, y Hernández, J́. El virus del síndrome respiratorio y reproductivo porcino infecta las células dendríticas porcinas maduras y regula al alza la producción de interleucina-10. Clin Vacuna Immunol. (2008) 15:720–5. doi: 10.1128/CVI.00224-07
Resumen de PubMed | Texto completo de CrossRef | Google Académico
91. Zhang, H, Guo, X, Nelson, E, Christopher-Hennings, J y Wang, X. El virus del síndrome respiratorio y reproductivo porcino activa la transcripción del interferón alfa/beta (IFN-alfa/beta) en las células dendríticas derivadas de monocitos (Mo-DC). Microbiol veterinario. (2012) 159:494–8. doi: 10.1016/j.vetmic.2012.04.025
Resumen de PubMed | Texto completo de CrossRef | Google Académico
92. Bi, J, Song, S, Fang, L, Wang, D, Jing, H, Gao, L, et al. El virus del síndrome respiratorio y reproductivo porcino induce la producción de IL-1 ss dependiendo de la vía TLR4/MyD88 y del inflamasoma NLRP3 en macrófagos alveolares porcinos primarios. Mediat Inflamm. (2014) 2014:1–14. doi: 10.1155/2014/403515
Resumen de PubMed | Texto completo de CrossRef | Google Académico
93. Bonilla, FA, y Oettgen, HC. Inmunidad adaptativa. J Alergia Clin Immunol. (2010) 125:S33-40. doi: 10.1016/j.jaci.2009.09.017
94. Sang, Y, Rowland, RR y Blecha, F. Interacción entre la inmunidad innata y el virus del síndrome respiratorio y reproductivo porcino. Anim Health Res Rev. (2011) 12:149–67. doi: 10.1017/S1466252311000144
95. Loving, CL, Osorio, FA, Murtaugh, MP y Zuckermann, FA. Inmunidad innata y adaptativa contra el virus del síndrome respiratorio y reproductivo porcino. Veterinario Inmunol Inmunopatíaco. (2015) 167:1–14. doi: 10.1016/j.vetimm.2015.07.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
96. Thanawongnuwech, R, Halbur, PG, Ackermann, MR, Thacker, EL, y Royer, RL. Efectos de las cepas de baja (vacuna de virus vivo modificado) y alta virulencia (VR-2385) del virus del síndrome respiratorio y reproductivo porcino sobre el aclaramiento pulmonar de partículas de cobre en cerdos. Veterinario Pathol. (1998) 35:398–406. doi: 10.1177/030098589803500509
Resumen de PubMed | Texto completo de CrossRef | Google Académico
97. Drigo, M, Giacomini, E, Lazzaro, M, Pasotto, D, Bilato, D, Ruggeri, J, et al. Evaluación comparativa de las respuestas inmunitarias de cerdos en rebaños estables e inestables con PRRS. Veterinario Inmunol Inmunopatíaco. (2018) 200:32–9. doi: 10.1016/j.vetimm.2018.04.007
98. Wu, Q, Han, Y, Wu, X, Wang, Y, Su, Q, Shen, Y, et al. Integrated time-series transcriptomic and metabolomic analyses reveal different inflammatory and adaptive immune responses contributing to host resistance to PRRSV. Front Immunol. (2022) 13:960709. doi: 10.3389/fimmu.2022.960709
99. Xu, Y, Zhang, Y, Sun, S, Luo, J, Jiang, S, Zhang, J, et al. The innate immune DNA sensing cGAS-STING signaling pathway mediates anti-PRRSV function. Viruses. (2021) 13:1829. doi: 10.3390/v13091829
100. Xu, Y, Ye, M, Zhang, Y, Sun, S, Luo, J, Jiang, S, et al. Screening of porcine innate immune adaptor signaling revealed several anti-PRRSV signaling pathways. Vaccines (Basel). (2021) 9:1176. doi: 10.3390/vaccines9101176
101. Zhu, Z, Zhang, X, Dong, W, Wang, X, He, S, Zhang, H, et al. TREM2 suppresses the proinflammatory response to facilitate PRRSV infection via PI3K/NF-kappaB signaling. PLoS Pathog. (2020) 16:e1008543. doi: 10.1371/journal.ppat.1008543
102. Eclercy, J, Larcher, T, Andraud, M, Renson, P, Bernard, C, Bigault, L, et al. PCV2 co-infection does not impact PRRSV MLV1 safety but enhances virulence of a PRRSV MLV1-like strain in infected SPF pigs. Vet Microbiol. (2020) 244:108656. doi: 10.1016/j.vetmic.2020.108656
103. Wang, SJ, Liu, WJ, Yang, LG, Sargent, CA, Liu, HB, Wang, C, et al. Effects of FUT1 gene mutation on resistance to infectious disease. Mol Biol Rep. (2012) 39:2805–10. doi: 10.1007/s11033-011-1039-0
104. Islam, MA, Große-Brinkhaus, C, Pröll, MJ, Uddin, MJ, Aqter Rony, S, Tesfaye, D, et al. Los perfiles del transcriptoma de PBMC identifican posibles genes candidatos y redes funcionales que controlan la respuesta inmunitaria innata y adaptativa a la vacuna contra el PRRSV en el cerdo Pietrain. PLoS Uno. (2017) 12:e0171828. doi: 10.1371/journal.pone.0171828
Resumen de PubMed | Texto completo de CrossRef | Google Académico
105. Dong, Q, Dunkelberger, J, Lim, KS, Lunney, JK, Tuggle, CK, Rowland, RRR, et al. Asociaciones de variación natural en el CD163 y otros genes candidatos en la respuesta del huésped de cerdos de destete a la infección por el virus del síndrome respiratorio y reproductivo porcino. J Anim Sci. (2021) 99:skab274. doi: 10.1093/jas/skab274
Resumen de PubMed | Texto completo de CrossRef | Google Académico
106. Fica, SM y Nagai, K. Instantáneas de criomicroscopía electrónica del spliceosoma: información estructural sobre una máquina dinámica de ribonucleoproteínas. Nat Struct Mol Biol. (2017) 24:791–9. doi: 10.1038/nsmb.3463
107. Papasaikas, P, y Valcárcel, J. El spliceosoma: el último acompañante y escultor de ARN. Tendencias Biochem Sci. (2016) 41:33–45. doi: 10.1016/j.tibs.2015.11.003
108. Hu, B, Li, X, Huo, Y, Yu, Y, Zhang, Q, Chen, G, et al. Las respuestas celulares a la infección por VHS-1 están relacionadas con tipos específicos de alteraciones en el transcriptoma del huésped. Sci Rep. (2016) 6:28075. doi: 10.1038/srep28075
Resumen de PubMed | Texto completo de CrossRef | Google Académico
109. Boudreault, S, Martenon-Brodeur, C, Caron, M, Garant, JM, Tremblay, MP, Armero, VES, et al. Perfil global del panorama de empalme de ARN alternativo celular durante las interacciones virus-huésped. PLoS Uno. (2016) 11:E0161914. doi: 10.1371/journal.pone.0161914
Resumen de PubMed | Texto completo de CrossRef | Google Académico
110. Bonenfant, G, Meng, R, Shotwell, C, Badu, P, Payne, AF, Ciota, AT, et al. El aislado del virus del Zika asiático cambia significativamente el perfil transcripcional y los eventos alternativos de empalme de ARN en una línea celular de neuroblastoma. Virus. (2020) 12:510. doi: 10.3390/v12050510
Resumen de PubMed | Texto completo de CrossRef | Google Académico
111. Fabozzi, G, Oler, AJ, Liu, P, Chen, Y, Mindaye, S, Dolan, MA, et al. La secuenciación de ARN dual específica de la cadena de células epiteliales bronquiales infectadas con los virus de la influenza A/H3N2 revela el empalme del segmento 6 del gen y las nuevas interacciones huésped-virus. J Virol. (2018) 92:E00518-18. doi: 10.1128/JVI.00518-18
Resumen de PubMed | Texto completo de CrossRef | Google Académico
112. Ule, J, y Blencowe, BJ. Redes reguladoras de empalme alternativo: funciones, mecanismos y evolución. Célula molar. (2019) 76:329–45. doi: 10.1016/j.molcel.2019.09.017
Resumen de PubMed | Texto completo de CrossRef | Google Académico
113. Baralle, FE y Giudice, J. Empalme alternativo como regulador del desarrollo y la identidad tisular. Nat Rev Mol Cell Biol. (2017) 18:437–51. doi: 10.1038/nrm.2017.27
Resumen de PubMed | Texto completo de CrossRef | Google Académico
114. Zhang, Y, Xue, L, Xu, H, Liang, W, Wu, Q, Zhang, Q, et al. Análisis global de la diferencia de empalme alternativo en órganos inmunes periféricos entre cerdos Tongcheng y cerdos blancos grandes infectados artificialmente con PRRSV in vivo. Biomed Res Int. (2020) 2020:4045204. doi: 10.1155/2020/4045204
115. Pasquinelli, AE . NON-CODING RNA MicroRNAs and their targets: recognition, regulation and an emerging reciprocal relationship. Nat Rev Genet. (2012) 13:271–82. doi: 10.1038/nrg3162
116. Guo, XK, Zhang, Q, Gao, L, Li, N, Chen, XX, and Feng, WH. Increasing expression of MicroRNA 181 inhibits porcine reproductive and respiratory syndrome virus replication and has implications for controlling virus infection. J Virol. (2013) 87:1159–71. doi: 10.1128/JVI.02386-12
117. Gao, L, Guo, XK, Wang, L, Zhang, Q, Li, N, Chen, XX, et al. MicroRNA 181 suppresses porcine reproductive and respiratory syndrome virus (PRRSV) infection by targeting PRRSV receptor CD163. J Virol. (2013) 87:8808–12. doi: 10.1128/JVI.00718-13
118. Zhang, Q, Guo, XK, Gao, L, Huang, C, Li, N, Jia, X, et al. MicroRNA-23 inhibits PRRSV replication by directly targeting PRRSV RNA and possibly by upregulating type I interferons. Virology. (2014) 450-451:182–95. doi: 10.1016/j.virol.2013.12.020
119. Zhao, G, Hou, J, Xu, G, Xiang, A, Kang, Y, Yan, Y, et al. Cellular microRNA miR-10a-5p inhibits replication of porcine reproductive and respiratory syndrome virus by targeting the host factor signal recognition particle 14. J Gen Virol. (2017) 98:624–32. doi: 10.1099/jgv.0.000708
120. Guan, K, Su, Q, Kuang, K, Meng, X, Zhou, X, and Liu, B. MiR-142-5p/FAM134B Axis manipulates ER-Phagy to control PRRSV replication. Front Immunol. (2022) 13:842077. doi: 10.3389/fimmu.2022.842077
121. You, X, Liu, M, Liu, Q, Li, H, Qu, Y, Gao, X, et al. miRNA let-7 family regulated by NEAT1 and ARID3A/NF-kappaB inhibits PRRSV-2 replication in vitro and in vivo. PLoS Pathog. (2022) 18:e1010820. doi: 10.1371/journal.ppat.1010820
122. Hicks, JA, Yoo, D, and Liu, HC. Characterization of the microRNAome in porcine reproductive and respiratory syndrome virus infected macrophages. PLoS One. (2013) 8:e82054. doi: 10.1371/journal.pone.0082054
123. Forster, SC, Tate, MD, and Hertzog, PJ. MicroRNA as type I interferon-regulated transcripts and modulators of the innate immune response. Front Immunol. (2015) 6:1–9. doi: 10.3389/fimmu.2015.00334
124. Wang, LL, Zhou, L, Hu, D, Ge, X, Guo, X, and Yang, H. Porcine reproductive and respiratory syndrome virus suppresses post-transcriptionally the protein expression of IFN-beta by upregulating cellular microRNAs in porcine alveolar macrophages in vitro. Exp Ther Med. (2018) 15:115–26. doi: 10.3892/etm.2017.5397
125. Zhen, Y, Wang, F, Liang, W, Liu, J, Gao, G, Wang, Y, et al. Identification of differentially expressed non-coding RNA in porcine alveolar macrophages from Tongcheng and large white pigs responded to PRRSV. Sci Rep. (2018) 8:15621. doi: 10.1038/s41598-018-33891-0
126. Li, J, Chen, Z, Zhao, J, Fang, L, Fang, R, Xiao, J, et al. Difference in microRNA expression and editing profile of lung tissues from different pig breeds related to immune responses to HP-PRRSV. Sci Rep. (2015) 5:9549. doi: 10.1038/srep09549
Keywords: porcine reproductive and respiratory syndrome, pig breeds, genetic backgrounds, PRRSV, PRRSV receptors, innate immunity, acquired immunity
Citation: Pei Y, Lin C, Li H and Feng Z (2023) Genetic background influences pig responses to porcine reproductive and respiratory syndrome virus. Front. Vet. Sci. 10:1289570. doi: 10.3389/fvets.2023.1289570
Received: 06 September 2023; Accepted: 09 October 2023;
Published: 20 October 2023.
Editado por:
Lingxue Yu, Academia China de Ciencias Agrícolas, China
Revisado por:
Pu Sun, Academia China de Ciencias Agrícolas, China
Qingkui Jiang, Universidad de Rutgers, Newark, Estados Unidos
Derechos de autor © 2023 Pei, Lin, Li y Feng. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Zheng Feng, greatfz@126.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía