Identificación y caracterización de los agentes causantes de la dermatitis ulcerativa focal en gallinas ponedoras comerciales
Identificación y caracterización de los agentes causantes de la dermatitis ulcerativa focal en gallinas ponedoras comerciales
- Centro de Nutrición Animal Purina, Land O’ Lakes, Gray Summit, MO, Estados Unidos
La dermatitis ulcerosa focal (FUDS) es una enfermedad dermatológica emergente que afecta a las bandadas de puestas libres de jaulas, se caracteriza por el desarrollo de una lesión en el dorso de las aves; el FUDS es de naturaleza esporádica y puede resultar en una caída en la producción de huevos y hasta el 50 % de la mortalidad acumulada. En este estudio se tomaron muestras de un total de dos rebaños libres de jaula (rebando 1: sin antecedentes de FUDS; rebaño 2: aves afectadas por FUDS) de una operación comercial de gallinas ponedoras en el medio oeste de los Estados Unidos. La composición microbiana de las muestras cutáneas, cloacas, cécales e ileales de cada ave se caracterizó a través de la secuenciación de próxima generación (NGS). Los resultados identificaron Staphylococcus aureus y Staphylococcus agnetis como los posibles agentes causantes de FUDS, siendo los más predominantes en las aves FUDS positivas. Estos resultados se confirmaron mediante el chapado, con ambos estafilococos como los únicos patógenos aislados de lesiones de aves FUDS positivas. Un total de 68 aislados de estafilococos confirmados de muestras cutáneas y ambientales fueron analizados mediante la secuenciación del genoma completo (WGS) para detectar la presencia de genes de resistencia antimicrobiana (AMR) y factores de virulencia que podrían haber contribuido al desarrollo de FUDS. Cuarenta y cuatro puntos uno y dos por ciento de los aislados tenían entre uno y cuatro genes AMR adquiridos que codifican la resistencia a los macrólidos, lincosamidas, espectrogratinas y betalactámicos. Se identificaron seis clases de factores de virulencia asociados con la adherencia, la enzima, la evasión inmune, el sistema de secreción, la toxina y la absorción de hierro. El efecto antimicrobiano de 4 combinaciones patentadas de Bacillus Direct Fed Microbial (DFM) se evaluó contra los aislados de Staphylococcus aureus y Staphylococcus agnetis, mediante el ensayo de difusión de pozos de agar (AWD) y la exclusión competitiva (CE) en el cultivo de caldo. A través de esta detección antimicrobiana, se identificó una combinación particular de dos cepas de Bacillus pumilus como el inhibidor más eficaz de ambos estafilococos. Un producto personalizado de Bacillus pumilus se está utilizando en diferentes granjas con antecedentes de FUDS, lo que resulta en la inhibición exitosa tanto de Staphylococcus aureus como de Staphylococcus agnetis, disminuyendo la mortalidad por FUDS y mejorando los huevos cosechables.
Introducción
En los Estados Unidos, la demanda de consumidores y minoristas sobre cómo se gestionan las gallinas ponedoras ha cambiado en la última década, con un mayor interés en el uso de sistemas de producción alternativos en comparación con los sistemas convencionales (1). En 2019, el 81,6 % de las aves ponedora de huevos en los EE. UU. estaban alojadas en jaulas convencionales, mientras que el 13,3 % estaban libres de jaulas (2). El 1 de diciembre de 2020, el rebaño total libre de jaulas de EE. UU. tenía 80,1 millones de gallinas, un aumento de 9,3 millones de cabezas desde la misma fecha en 2019 (3). Se estima que estos números aumentarán con más de la mitad de la vivienda de gallina de EE. UU. que se espera que esté libre de jaulas para 2025 (4). Debido a la naturaleza de su diseño de producción, los sistemas de vivienda sin jaulas tienen una mayor incidencia de infecciones bacterianas (5, 6). El síndrome de dermatitis ulcerosa focal (FUDS) es una de las enfermedades emergentes que experimentan las rebaños sin jaulas. El FUDS se describió por primera vez en 2009 en una operación de gallina de la cría en el Medio Oeste de los Estados Unidos, el síndrome se caracteriza por el desarrollo de lesiones en el dorso de las aves, solo craneal a la glándula uropygial. El inicio de la afección suele ocurrir entre las 24 y las 50 semanas de edad, carece de estacionalidad y se observa principalmente en casas con pisos de listones (7). Los brotes son de naturaleza relativamente esporádica; sin embargo, cuando están presentes pueden resultar en una mortalidad acumulada de hasta el 50 %.
Las lesiones típicas de FUDS recuerdan a la epidermitis exudativa porcina, una enfermedad causada por Staphylococcus hyicus. Los estafilococos son omnipresentes en entornos avícolas y forman parte de la microbiota normal de la piel y las membranas mucosas de las aves; sin embargo, cuando la integridad de la piel u otras membranas mucosas se ve comprometida, algunas especies de Staphylococcus pueden convertirse en patógenos oportunistas que causan infecciones localizadas o sistémicas (8, 9). Las cepas de S. hyicus han sido implicadas como agentes causantes secundarios de dermatosis preexistentes (10), producen toxinas exfoliativas que inducen dermatitis, lo que resulta en un engrosamiento de la piel (11). Aunque se ha realizado poca investigación sobre la etiología de FUDS, se ha observado una correlación entre la enfermedad y la abundancia de S. hyicus (7). Las pérdidas de producción debido a las infecciones por Staphylococcus en aves de corral (dejar óvulos o aves de carne) se han asociado con la cojera, la disminución de la producción de huevos (9, 12), el aumento de la mortalidad y la condena de los cadáveres en las instalaciones de sacrificio (8).
Los antibióticos se han utilizado ampliamente en la industria avícola para promover el crecimiento (13) y controlar los patógenos, incluido Staphylococcus spp., maximizando así la producción. Sin embargo, el aumento del uso de antibióticos en la producción ganadera ha llevado a una rápida propagación de la resistencia a los antimicrobianos (AMR) entre los aislados bacterianos y a una creciente preocupación pública por los efectos de la RAM en la salud humana (14), lo que ha dado lugar a una lenta disminución en el uso de antibióticos en la producción animal (13). Estas tendencias, junto con el aumento de la demanda de los minoristas y consumidores de producción de aves de corral libres de antibióticos, han puesto de relieve la importancia de desarrollar tecnologías alternativas para reducir/inhibir los patógenos bacterianos sin impactos negativos en el rendimiento de los animales.
Los microbianos alimentados directamente (DFM) han surgido como una alternativa viable al uso de antibióticos en la avicultura (15, 16). Son microorganismos vivos que, cuando se alimentan en cantidades adecuadas, confieren beneficios al huésped (17); entre los beneficios se encuentran una mejor inmunidad, un crecimiento mejorado y un aumento general en el rendimiento de la puesta (14). Se han utilizado múltiples géneros bacterianos en la producción avícola, siendo Lactobacillus, Bacillus y Bifidobacterium los más comunes. Cada cepa DFM confiere una acción específica que afecta a la ecología del huésped y microbiana de la vía gastrointestinal. Su modo de acción incluye la producción de compuestos antimicrobianos (es decir, bacteriocinas, ácidos orgánicos), mecanismos de exclusión competitivos y la producción de productos de fermentación beneficiosos como los ácidos grasos volátiles (18). Este estudio tenía como objetivo caracterizar la microbiota cecal, ileal, cloaca y cutánea de las gallinas ponedoras FUDS positivas y FUDS negativas a través de la secuenciación de próxima generación (NGS). Los objetivos principales eran (1) identificar los patógenos que causan el FUDS, (2) aislar los patógenos, (3) identificar la asociación entre los aislados y 4) desarrollar una combinación de DFM inhibidora contra los agentes causantes de los FUDS.
Metodología
Descripciones de los Rebaños y puntuación de las lesiones
Para este estudio se probó una operación comercial de gallinas ponedoras en el medio oeste de los Estados Unidos con un historial de FUDS. Dentro de la operación, se seleccionaron dos rebaños libres de jaula: (1) rebaño 1; sin antecedentes de la enfermedad (Control) y (2) rebaño 2; aves afectadas por FUDS. Ambas bandadas se originaron en la misma incubadora, se alojaron en un sistema de producción similar sin jaulas, y se tomaron muestras a las 73 semanas de edad. Para este estudio se tomaron muestras de un total de 20 aves del rebaño de control y 39 aves del rebaño afectado por el FUDS [18 aves que no muestran síntomas de FUDS (FUDS-) y 21 aves que muestran síntomas visibles de FUDS (FUDS +)].
Antes de la recolección de muestras, las aves del rebaño afectado por los FUDS fueron puntuadas para las lesiones de acuerdo con el tamaño de la lesión ubicada en el craneal de la glándula uropygial, como se ilustra en la Figura 1. Las lesiones se evaluaron por el tamaño y la participación del tejido subyacente con puntuaciones que oscilaron entre 0 y 3: 0, sin lesión; 1, tamaño de la lesión < 2 cm; 2, tamaño de la lesión de 2 a 6 cm; 3, lesiones > 6 cm.
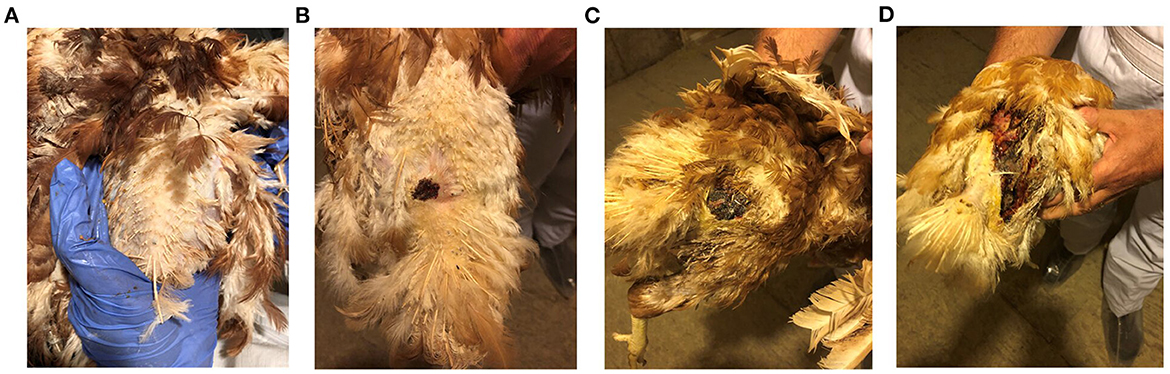 Figura 1. puntuación de la lesión de FUDS. Las puntuaciones se evaluaron en función del tamaño y la participación en el tejido subyacente. Las puntuaciones oscilaron entre 0 y 3: (A) 0, sin lesión; (B) 1, tamaño de la lesión < 2 cm; (C) 2, tamaño de la lesión de 2 a 6 cm; (D) 3, lesiones > 6 cm.
Figura 1. puntuación de la lesión de FUDS. Las puntuaciones se evaluaron en función del tamaño y la participación en el tejido subyacente. Las puntuaciones oscilaron entre 0 y 3: (A) 0, sin lesión; (B) 1, tamaño de la lesión < 2 cm; (C) 2, tamaño de la lesión de 2 a 6 cm; (D) 3, lesiones > 6 cm.
Recogida de muestras para el análisis del microbioma
En este estudio se recogieron un total de cuatro tipos de muestras de cada ave: piel, cloaca, cecum e íleon. Las muestras se recogieron de forma aséptica para evitar la contaminación cruzada. Las muestras de piel se tomaron utilizando un hisopo flocado (Puritan, Guilford, ME, EE. UU.) que se giró y se frotó en un área de 2 cm2 de piel afectada cranial a la glándula uropygial para FUDS + aves y la misma área de piel para Control y FUDS-aves. Las muestras cloacas se recogieron insertando un hisopo flocado ~1 cm en la cloaca, el hisopo se giró para permitir la recolección de suficiente material cloaca. Para la recolección de muestras de cecum e íleon, se abrió la cavidad toraco-abdominal para revelar el tracto gastrointestinal. Se hizo una abertura de 1 cm en el medio del íleon y luego se giró un hisopo flocado dentro del lumen. El proceso se repitió con un nuevo hisopo para la recolección de contenido cécale. Después de la recolección de muestras, todos los hisopos se colocaron en tubos microcentrífugos estériles que contenían 1 ml de escudo de ADN/ARN (Zymo Research, Irvine CA), e inmediatamente se colocaron en hielo después de la recolección, y se enviaron durante la noche al Centro de Tecnología Emergente del Centro de Nutrición Animal Purina (ETC: PANC) para su posterior procesamiento.
Extracción de ADN del microbioma 16S y preparación de la biblioteca
El ADN de la comunidad microbiana total se extrajo de todos los tipos de muestras y se analizó utilizando el Quick-DNA Fecal/Soil Microbe miniprep (Zymo Research Corp, Irvine, CA, EE. UU.) siguiendo las recomendaciones del fabricante. El ADN puro se cuantificó con un fluorómetro Qubit 2.0. Se utilizaron muestras de ADN a una concentración de 5 ng/μl para preparar bibliotecas utilizando el protocolo de preparación de bibliotecas Illumina 16S-metagenomics. La región hipervariable V3-V4 del gen rRNA bacteriano 16S se amplió utilizando los cebadores 341F (5′-CCTACGGGNGGCWGCAG-3′) y 805R (5′-GACTACHVGGGTATCTAATCC-3′) que contienen adaptadores de Illumina como se ilustra en Klindworth (19). Los amplificadores de PCR se comprobaron en geles de agarosa y se purificaron con cuentas AMPure XP (Beckman Coulter Inc) según las recomendaciones del fabricante. Luego se indexaron los amplificadores purificados utilizando el conjunto de índices Nextera UD (Illumina, San Diego, CA, EE. UU.). Las bibliotecas se cuantificaron por triplicado utilizando un fluorómetro Qubit 2.0 con un kit de ensayo HS dsDNA (Invitrogen, Carlsbad, CA, EE. UU.), agrupado en concentraciones iguales (4 nM) para generar un número equivalente de lecturas en bruto, y diluido a una concentración final de 6 pM. Las bibliotecas de Amplicon se vieron con un 5 % de control PhiX (Illumina, San Diego, CA, EE. UU.) de acuerdo con las recomendaciones del fabricante. Las muestras se secuenciaron utilizando un kit de reactivo MiSeq v3 (ciclo de 600) en una plataforma Illumina MiSeq (Illumina, San Diego, CA, EE. UU.).
Análisis de datos 16S y bioinformática
Los datos de secuencias en bruto se analizaron utilizando conocimientos cuantitativos sobre la canalización de la ecología microbiana (QIIME2) (20). Usando el software de reportero MiSeq (Illumina, San Diego, CA, EE. UU.), se recortaron los cebadores de amplificación y los adaptadores Illumina, se desmultiplexaron las muestras y se generaron archivos fastq.gz. Los datos de secuencia sin procesar se filtraron y procesaron utilizando la tubería DADA2 en QIIME2. Las secuencias del gen rRNA 16S se agruparon en variantes de secuencia de amplicon (ASV) y la taxonomía se asignó en base a la comparación con la base de datos SILVA (21). Los índices de riqueza y diversidad de especies se calcularon utilizando QIIME. La diversidad beta se determinó utilizando la distancia ponderada UniFrac con un análisis permutacional multivariable de la varianza (PERMANOVA) para determinar las diferencias entre las comunidades microbianas en función de la relación fleogénica de comunidades enteras.
Análisis estadístico
El tamaño del efecto del análisis discriminante lineal (LDA) (LEfSe) (22) se utilizó en este estudio para identificar taxones estadísticamente significativos para caracterizar las diferencias entre los grupos FUDS+, FUDS- y Control. La prueba de rango de suma factorial no paramétrica de Kruskal-Wallis fue utilizada por el método LEfSe para identificar las diferencias de abundancia de unidades taxonómicas operativas (OTU) entre dos grupos. Las pruebas en pares entre los grupos se realizaron utilizando la prueba de suma de rango de Wilcoxon, seguida de la LDA en abundancias para estimar el tamaño del efecto de cada OTU abundante diferencialmente significativa. Las OTU se consideraban significativamente diferentes cuando las diferencias tenían p < 0,05 y una puntuación de LDA (log10) > 3,5. Se realizó un segundo análisis de LEfSe en Staphylococcus y Lactobacillus para identificar especies específicas asociadas con el estado de salud de las aves, las OTU se consideraron significativamente diferentes cuando p < 0,05 y una puntuación de LDA (log10) era > 3,5.
Recogida de muestras para el aislamiento bacteriano
Se seleccionaron un total de 10 FUDS+, 10 FUDS y 10 aves de control en función de la presencia/ausencia de lesiones cutáneas para la recolección de muestras. Se eliminaron las plumas alrededor de la glándula preen y se frotó una superficie de 5 cm2de la piel con hisopos de esponja pre-inhmemados (Whirl-Pak, Madison, WI). Además, se recogieron un total de 3 tipos de muestras ambientales, por rebaño, incluyendo desde la parte superior de las cajas nido, perchas y áreas de rascado. Una superficie de 5 cm2 se frotó a un intervalo par y se distribuyó por todas las casas. Las muestras se enviaron durante la noche al ETC: PANC para cultivo y aislamiento bacteriano.
Aislamiento bacteriano y condiciones de crecimiento
A su llegada a ETC: PANC, las muestras se homogeneizaron en un estómago (Stomacher® 400, Seward) durante 3 minutos a 230 rpm. Se recogió una alícuota de 1 ml y se añadió a 9 ml de base de caldo de enriquecimiento de Staphylococcus de Baird (Sigma-Aldrich, St. Louis, MO) suplementado con 0,1 ml de solución de telurito de potasio (Sigma-Aldrich, St. Louis, MO), los tubos se incubaron aeróbicamente a 37 °C durante 24 horas. Después de la incubación, las muestras se diluyeron en serie en 9 ml de agua de peptona tamponada (BPW, Sigma-Aldrich, St. Louis, MO), se extendió sobre las placas de agar de Staphylococcus CHROMagar (CHROMagar, París, Francia), e incubado aeróbicamente a 37 °C durante 24 horas. Según la morfología de la colonia, se seleccionaron hasta 4 colonias representativas de cada placa, estrías en placas de agar de soja tripptica (TSA, Sigma-Aldrich, St. Louis, MO), e incubado a 37 °C durante 24 horas. Se seleccionó una sola colonia bien aislada de cada placa de TSA, cultivada en 9 ml de caldo de soja triptíptico (TSB, Sigma-Aldrich, St. Louis, MO) e incubado a 37 °C durante 24 horas (cultivo nocturno). Se utilizó una alicuota de 1,5 ml del cultivo nocturno para la extracción de ADNg y se añadió una aliquot de 500 μl al 50 % de glicerol y se almacenó a -80 °C para realizar más pruebas.
Amplificación de PCR 16S para la especiación bacteriana
Se utilizó una alícuota de 1,5 ml del cultivo nocturno para la extracción de ADN g utilizando el kit de extracción de ADN Invitrogen PureLink (Thermo Fisher Scientific, Waltham, MA, EE. UU.). Las muestras de ADN se cuantificaron utilizando un fluorómetro Qubit 2.0 (Life Technologies, CA, Estados Unidos) y se diluyeron a 20 ng/μl para la amplificación de PCR.
Una región de 1.483 pares de bases (bp) de la subunidad ribosómica 16S se amplió con PCR utilizando imprimación hacia adelante 5′-AGAGTTTGATCCTGGCTCAG e imprimación inversa 5′-GGTTACCTTGTTACGACTT. La amplificación de PCR se llevó a cabo utilizando ZymoTaq PreMix de acuerdo con Después de la reacción, los productos de PCR se purificaron utilizando un kit de limpieza y concentrador de ADN (Zymo Research, Irvine CA), de acuerdo con las instrucciones del fabricante. Luego se enviaron productos purificados a la instalación de secuenciación central de la Universidad de Missouri para la secuenciación de sangría. El ID taxonómico de cada dato de secuencia sin procesar se asignó en base a los resultados de una consulta de la herramienta de búsqueda de alineación local básica (BLAST) del Centro Nacional de Información Biotecnológica (NCBI) (https://blast.ncbi.nlm.nih.gov/Blast.cgi).
Secuenciación del genoma completo
Se seleccionaron un total de 68 aislados de Staphylococcus confirmados por PCR de las muestras ambientales y cutáneas de las aves FUDS+ y FUDS para un análisis genotíptico adicional. Los aislados de Staphylococcus se cultivaron durante la noche en 9 ml de TSB y se incubaron a 37 °C durante 24 horas. El ADN genómico total se aisló utilizando el kit de extracción de ADN Invitrogen Purelink. El ADN gDNA puro se cuantificó utilizando un fluorómetro Qubit 2.0 y se utilizó para la preparación de la biblioteca con el kit Nextera XT v2.0 (San Diego, CA, Estados Unidos) según las recomendaciones del fabricante. Las bibliotecas de ADN se secuenciaron con el extremo emparejado utilizando el kit de secuenciación 2 × 300 bp v3 en una plataforma Illumina MiSeq. Las lecturas en bruto se preprocesaron y filtraron usando la versión Trimmomatic 0.36 (23), seguida del ensamblaje de novo usando SPAdes versión 11 (24).
Análisis bioinformático de WGS
Se utilizaron genomas ensamblados para la identificación de factores de virulencia y genes de resistencia antimicrobiana potencial (AMR) comparándolos con las bases de datos Virulence Finder y ResFinder, respectivamente (25, 26). Las alineaciones se identificaron como positivas cuando el porcentaje de identidad era del 95 % o más. Se generó un árbol filogenético utilizando la alineación concatenada en RAxML (Randomized Axelerated Maximum Probabillihood) (27).
Selección de DFM y condiciones de crecimiento
Se seleccionaron un total de 4 combinaciones patentadas de Bacillus DFM de un conjunto de más de 500 nuevos DFM para este estudio en función de su efecto antagonista contra un conjunto de patógenos bacterianos, incluidos Salmonella, Staphylococcus aureus, E. coli, entre otros. Las muestras de 1 g de cada combinación de DFM liofilizada se cultivaron individualmente en 9 ml de caldo de Luria-Bertani (LB, ThermoFisher Scientific, Waltham, MA) a 37 °C durante 24 horas. Los cultivos nocturnos se utilizaron para el ensayo de difusión de pozos de agar, como se describe a continuación, y se diluyeron en serie en 9 ml de BPW y se platean aeróbicamente en placas de agar LB (ThermoFisher Scientific, Waltham, MA) a 37 °C durante 24 horas para la enumeración.
Detección de inhibición de patógenos mediante ensayo de difusión de pozo de agar
El método de difusión de pozos de agar, descrito por Vinderola y otros (28) con ligeras modificaciones (29), se utilizó para determinar la actividad antimicrobiana de un conjunto de combinaciones de Bacillus DFM a partir de una colección de cultivos de stock, mantenida en ETC: PANC, contra aislados de Staphylococcus. Los aislados de S. agnetis y S. aureus recuperados de la piel de las aves FUDS+ se incubaron durante la noche en TSB durante 18-24 horas a 37 °C y luego se diluiron a 106 UFC/mL. Se frotaron 100 μl de la última dilución en el agar nutritivo (Sigma-Aldrich, St. Louis, MO) placas para crear un césped a una concentración final de 105 UFC/mL. Las placas se secaron durante 5 minutos en un gabinete de bioseguridad, luego se hicieron pozos de 6 mm de ancho en el agar y cada duplicado del pozo se llenó con alícuotas de 100 μl de una de las 4 combinaciones patentadas de Bacillus DFM (108 UFC/mL) y un solo pozo con 100 μl de Las placas se incubaron a 4 °C durante 2 horas para permitir que las suspensiones se difundaran en el agar seguido de una incubación de 24 horas a 37 °C. La actividad antimicrobiana se evaluó midiendo la zona clara de inhibición alrededor de cada pozo. Las combinaciones de DFM se clasificaron por su actividad antimicrobiana general [descrita por Ayala y otros (29)] calculando la suma de las puntuaciones de inhibición individual de DFM (mm) en todos los aislados de Staphylococcus probados.
Ensayo de cultivo de caldo de exclusión competitiva
Los cultivos de Staphylococcus y Bacillus DFM se cultivaron de la noche a la mañana como se describió anteriormente, se coincularon a 105 y 106 UFC/mL respectivamente en caldo de nutrientes (ThermoFisher Scientific, Waltham, MA) e incubados con agitación (130 rpm) a 37 °C durante 24 horas (29). Después de la incubación, los cocultivos se diluyeron en serie y se platearon en placas de agar CHROMagarStaphylococcus y se incubaron a 37 °C durante 24 horas para la enumeración de Staphylococcus Staphylococcus. La actividad antimicrobiana se evaluó mediante la reducción de Staphylococcus (log10 UFC/mL) con respecto a las muestras de control (cosecos de Staphylococcus sin DFM) después de 24 horas de coinoculación. Los cultivos de DFM se clasificaron por su efecto antagónico sumando las reducciones generales (log10 UFC/mL) en todos los aislados de Staphylococcus probados.
Resultados
Puntuación de la lesión
Las aves de control y los grupos FUDS tenían una puntuación de lesión de 0, mientras que las aves del grupo FUDS+ tenían puntuaciones de lesiones que oscilaban entre 1 y 3 (1 = 14 aves, 2 = 5 aves y 3 = 2 aves), como se muestra en las Figuras 1A–D.
Amplificación de PCR 16S para la especiación bacteriana
La identificación taxonómica de los aislados de Staphylococcus de muestras ambientales recogidas del Control; y las bandadas afectadas por FUDS se muestra en la Tabla 1. Se recuperaron un total de 11 especies de Staphylococcusococcus de ambos entornos, Staphylococcus simulans fue el más comúnmente aislado, encontrado en todas las muestras ambientales de ambas bandadas (Tabla 1).
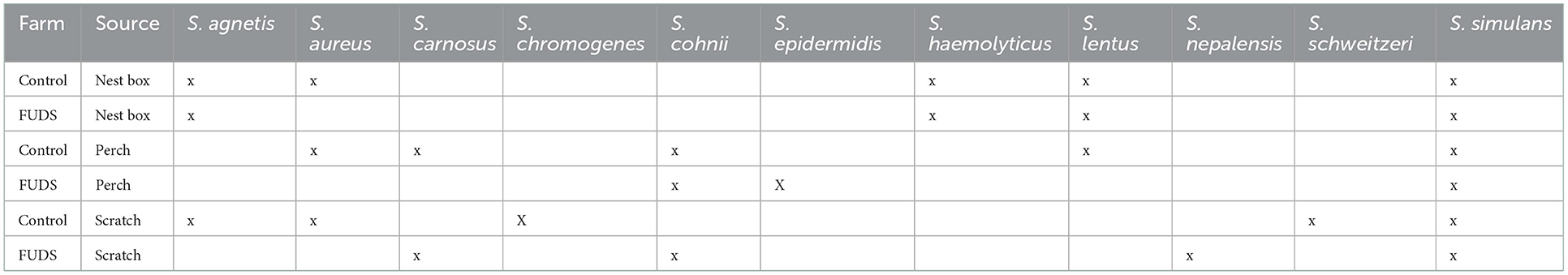 Tabla 1. Identificación taxonómica de los aislados de Staphylococcus de muestras ambientales recogidas de las bandadas afectadas por el Control y los FUDS.
Tabla 1. Identificación taxonómica de los aislados de Staphylococcus de muestras ambientales recogidas de las bandadas afectadas por el Control y los FUDS.
La identificación taxonómica de los aislados de Staphylococcus de los hisopos de piel recogidos de las bandadas afectadas por el Control y el FUDS se muestra en la Tabla 2.Se identificaron un total de 10 especies de Staphylococcus, con hisopos de piel de aves no afectadas que muestran más variación de especies de Staphylococcus.Staphylococcus aureus y Staphylococcus agnetis fueron las dos únicas especies de Staphylococcus identificadas en los hisopos de piel FUDS + (Tabla 2).
 Tabla 2. Identificación taxonómica de los aislados de Staphylococcus de los hisopos de piel recogidos de las aves FUDS y FUDS + de las bandadas afectadas por el FUDS.
Tabla 2. Identificación taxonómica de los aislados de Staphylococcus de los hisopos de piel recogidos de las aves FUDS y FUDS + de las bandadas afectadas por el FUDS.
Análisis de metagenómica 16S
El conjunto completo de datos de rRNA 16S analizado en este estudio está disponible en el repositorio NCBI Sequence Read Archive con acceso BioProject ID PRJNA884648.
Composición microbiana por tipo de muestra
La composición de las comunidades cecum, ileum, cloaca y bacterianas de la piel se exploró a través de diagramas de análisis de coordenadas principales (PCoA) utilizando el índice de Bray-Curtis. Como se muestra en el gráfico de PCoA (Figura 2), las muestras tendían a agruparse por tipo de muestra y subagruparadas por bandada de origen con una composición microbiana uniformemente divergente observada en el control y los FUDS afectados. Dentro del rebaño afectado por el FUDS, las muestras se agrupan por estado de enfermedad (Figura 2). Para las aves FUDS +, las muestras de piel mostraron una gran desviación del control y de las aves FUDS, sin embargo, las muestras de ileum no estaban fuertemente agrupadas por el estado de rebaño y/o enfermedad. Las métricas de distancia ponderadas de UniFrac se utilizaron para determinar la variación entre las comunidades microbianas del cecum, ileum, cloaca y piel. De acuerdo con los gráficos de PCoA, la microbiota de cada tipo de muestra se encontró en términos de composición distinta de la microbiota en otros tipos de muestra (p < 0,001).
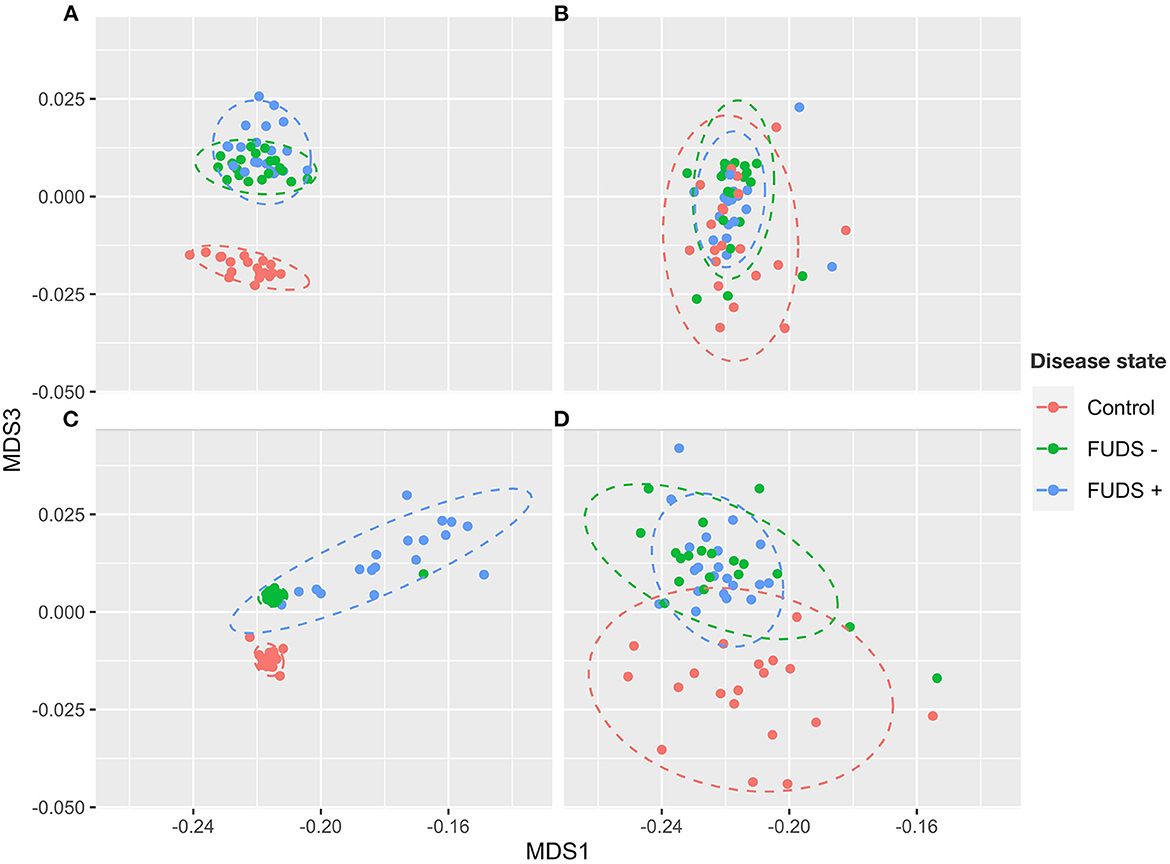 Figura 2. Análisis de componentes principales (PCoA) para las bandadas de control, FUDS y FUDS + por tipo de muestra. (A) Cecum; (B) íleon; (C) piel; y (D) cloaca. La trama se generó utilizando las métricas de Bray-Curtis.
Figura 2. Análisis de componentes principales (PCoA) para las bandadas de control, FUDS y FUDS + por tipo de muestra. (A) Cecum; (B) íleon; (C) piel; y (D) cloaca. La trama se generó utilizando las métricas de Bray-Curtis.
Abundancia relativa por filo
A nivel de filo para todas las bandadas, los seis filos más predominantes por tipo de muestra se muestran en la Figura 3. Las muestras de Ileum estaban dominadas por Firmicutes, Proteobacterias, Bacteroidetes, Actinobacterias, Cyanobacterias y Fusobacterias, con estos filos representando el 99 % de la abundancia total, y Firmicutes solos que comprenden el 97 % de las bacterias totales presentes. Las muestras de Cecum fueron compuestas principalmente por Firmicutes, Bacteroidetes, Proteobacteria, Fusobacteria, Cyanobacteria y Verrucomicrobia, y estos filos representan el 96 % de todas las bacterias presentes. Las muestras cloacales estaban dominadas por Firmicutes, Fusobacterias, Proteobacterias, Bacteroidetes, Actinobacterias y Synergistetes, y estos seis filos representan el 98 % de la abundancia relativa total. Las muestras de piel fueron compuestas principalmente por Firmicutes, Bacteroidetes, Proteobacteria, Actinobacteria, Fusobacteria y Cyanobacteria; estos filos comprendían el 98 % de la comunidad bacteriana total presente.
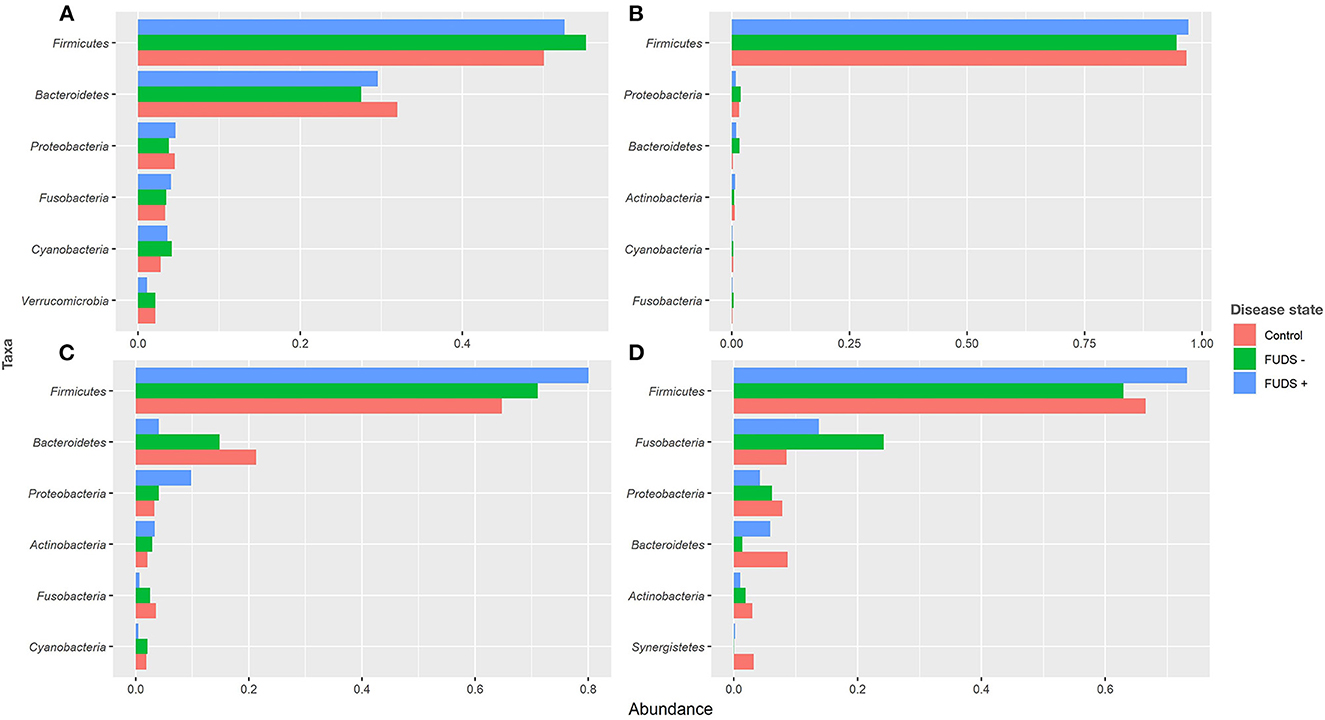 Figura 3. Parcela de abundancia relativa de los seis taxones superiores a nivel de filo para las bandadas de control, FUDS y FUDS + por tipo de muestra. (A) Cecum; (B) íleon; (C) piel; y (D) cloaca.
Figura 3. Parcela de abundancia relativa de los seis taxones superiores a nivel de filo para las bandadas de control, FUDS y FUDS + por tipo de muestra. (A) Cecum; (B) íleon; (C) piel; y (D) cloaca.
Comparaciones de abundancia relativa de Phyla entre las rebaños
Los firmicutes y los bacterioides fueron los dos filos más abundantes en el cecum a través de las bandadas. Firmicutes se encontró en 52,62, 55,27, 50,12% en bandadas de FUDS+, FUDS y de control, respectivamente, mientras que Bacteroidetes se encontró en 29,59, 27,52 y 32,97 para FUDS+, FUDS y rebaños de control, respectivamente.
Las muestras de Ileum fueron dominadas por Firmicutes con abundancias relativas similares en las rebaños y el estado de enfermedad. Se encontró en 97,10, 94,56 y 96,67 % para FUDS+, FUDS y rebaños de control, respectivamente.
Las muestras cloacales tenían diferencias notables de abundancia entre las muestras. Se observaron las principales diferencias para los cuatro primeros filos: Firmicutes, Fusobacterias, Proteobacterias y Baccteroidetes. Las aves FUDS+ tenían la mayor abundancia relativa de Firmicutes, representando 73,18, 62,92 y 66,52% para FUDS +, FUDS- y las aves de control, respectivamente. Las fusobacterias se encontraron en 13,73, 24,23 y 8,52 % en FUDS+, FUDS y aves de control, respectivamente. Las proteobacterias mostraron una tendencia leve con las aves FUDS+ que tenían la menor abundancia relativa en comparación con las aves asintomáticas y sanas. Las proteobacterias estaban presentes en 4,21, 6,12 y 7,86 % en FUDS+, FUDS y aves de control, respectivamente.
La microbiota de las muestras de piel estaba dominada por Firmicutes, Bacteroidetes yProteobacterias. En todo el estado de la enfermedad, el microbioma de la piel de las aves enfermas tenía mayores abundancias relativas de Firmicutes y Proteobacterias, y menor abundancias relativas de Baccteroidetes en comparación con las aves sanas. Las abundancias relativas de Firmicutes fueron en 800.10, 71,11 y 64,79 % para FUDS+, FUDS- y aves de control, respectivamente. Los bacteroidetes se encontraron en 4,11, 14,84 y 21,31 % para FUDS+, FUDS y aves de control, respectivamente. La abundancia relativa de Proteobacterias se encontró en 9,87, 4,08 y 3,33% para FUDS+, FUDS y aves de control, respectivamente.
Abundancia relativa por géneros
A nivel de género para todas las bandadas, los seis géneros más predominantes por tipo de muestra se muestran en la Figura 4. Las muestras de Ileum estaban compuestas principalmente por Lactobacillus, Rombustia, Turnibacter, Clostridium, Tyzzerella y Streptococcus, y estos géneros representaban el 91 % de la población bacteriana total. Las muestras de Cecum estuvieron dominadas por Baccteroides, Lactobacillus, Fusobacterium, Ruminococcaceae UCG.005, Alistipes y Lachnospiraceae, y estos géneros representan el 36 % de las bacterias totales. Los seis principales géneros de muestras cloacas fueron Fusobacterium, Turnibacter, Lactobacillus, Rombustia, Clostridium y Bacteroides, y estos géneros representan el 63 % del total de bacterias presentes. Las muestras de piel estaban compuestas principalmente por Staphylococcus, Lactobacillus, Bacteroides, Turnibacter, Rombustia y Ruminococcus, y estos géneros representan el 48 % de la microbiota cutánea total.
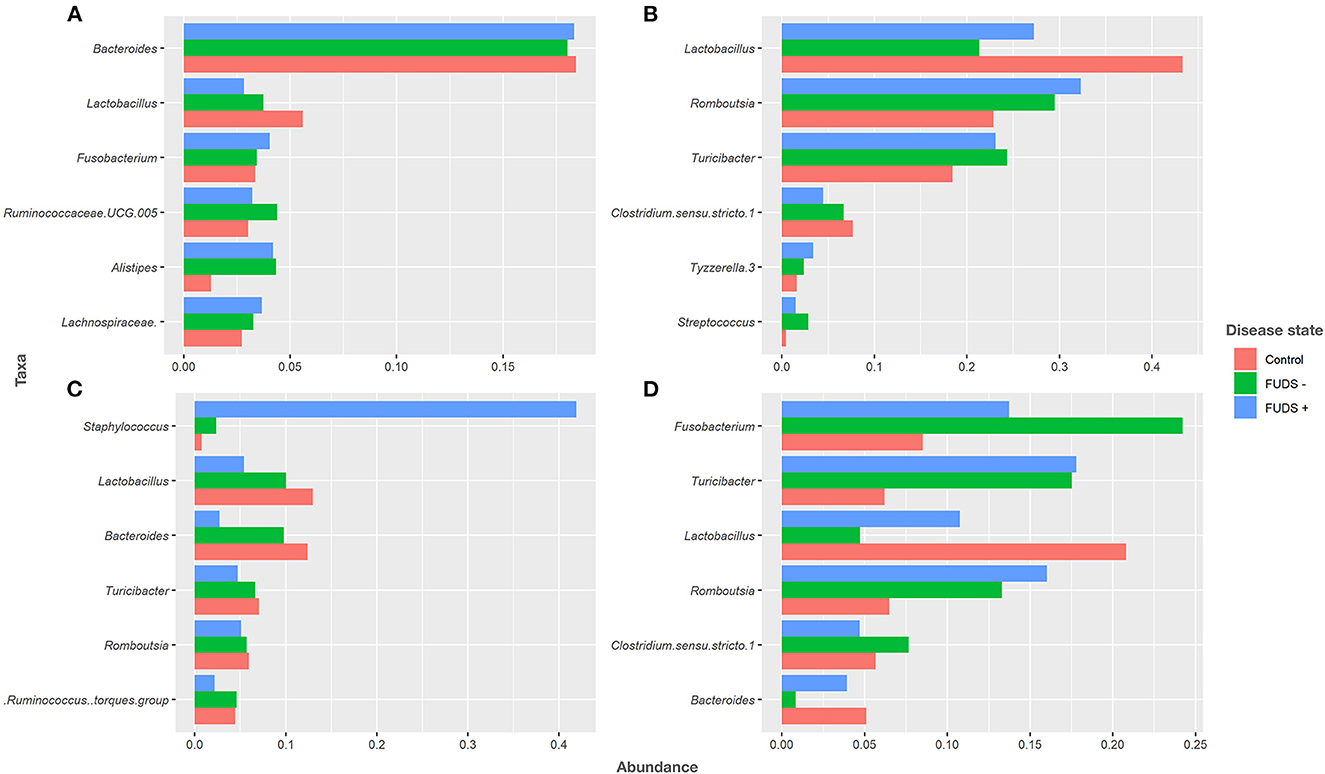 Figura 4. Parcela de abundancia relativa de los seis taxones principales a nivel de género para las bandadas de control, FUDS y FUDS + por tipo de muestra. (A) Cecum; (B) íleon; (C) piel; y (D) cloaca.
Figura 4. Parcela de abundancia relativa de los seis taxones principales a nivel de género para las bandadas de control, FUDS y FUDS + por tipo de muestra. (A) Cecum; (B) íleon; (C) piel; y (D) cloaca.
Tasas de muestras de piel, abundancia relativa
Mirando específicamente la lesión, la abundancia de Staphylococcus fue significativamente mayor (p < 0,001) en la piel de las aves FUDS + en comparación con las aves FUDS, lo que destaca una posible asociación con el desarrollo de FUDS. La abundancia relativa por género de muestras de piel para el control y las bandadas de FUDS se muestra en la Figura 5. La abundancia de Staphylococcus spp. aumentó a medida que progresaba la lesión por FUDS. En las muestras del rebaño de control (puntuación de lesión = 0), Staphylococcus spp. representó el 0,7 % de la abundancia relativa total, mientras que, en las muestras del rebaño FUDS+, la concentración de Staphylococcus spp. fue de 2,1, 41,6, 45,6 y 60,8%, en los puntajes de lesión 0, 1, 2 y 3, respectivamente El estafiloco se dividió en dos grupos de OTU igualmente abundantes cuando se accedió a nivel de especie. Sin embargo, la identificación taxonómica de Silva fue capaz de especiar Staphylococcus agnetis para un grupo OTU, mientras que el segundo grupo no se identificó por debajo del nivel de género. Un análisis adicional a través de la explosión de NCBI de las secuencias asociadas con el segundo grupo de Staphylococcus las identificó como Staphylococcus aureus. El Staphylococcus aureus OTU predominante estuvo presente en 0,71, 2,06, 21,65 y 37,79 % para las puntuaciones de lesiones 0, 1, 2 y 3, respectivamente. Staphylococcus agnetis estuvo presente en 0,04, 19,97, 18,56 y 23,13 % para las puntuaciones de lesiones 0, 1, 2 y 3 respectivamente.
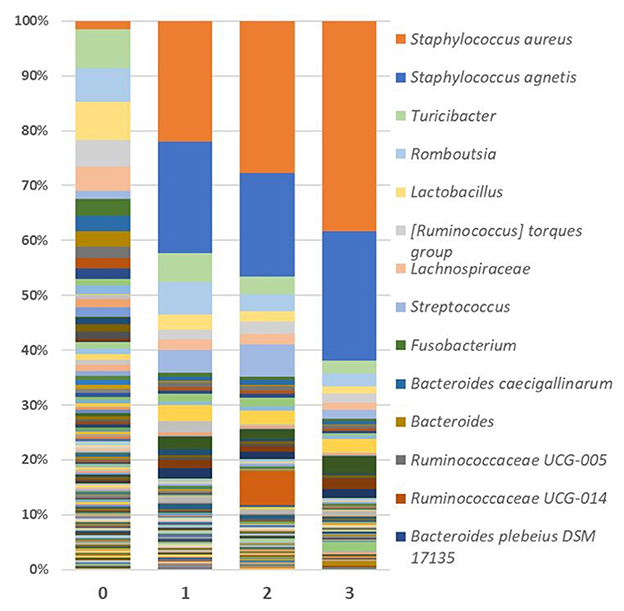 Figura 5. Abundancia relativa como porcentaje promedio de poblaciones de taxones bacterianos para muestras de piel agrupadas por puntuación de lesión. El Staphylococcus aureus OTU predominante estuvo presente en 0,709, 2.063, 21,647 y 37,787% para las puntuaciones de lesiones 0, 1, 2 y 3, respectivamente. Staphylococcusococcus agnetis estuvo presente en 0,043, 19,969, 18.564 y 23,125% para las puntuaciones de lesiones 0, 1, 2 y 3, respectivamente.
Figura 5. Abundancia relativa como porcentaje promedio de poblaciones de taxones bacterianos para muestras de piel agrupadas por puntuación de lesión. El Staphylococcus aureus OTU predominante estuvo presente en 0,709, 2.063, 21,647 y 37,787% para las puntuaciones de lesiones 0, 1, 2 y 3, respectivamente. Staphylococcusococcus agnetis estuvo presente en 0,043, 19,969, 18.564 y 23,125% para las puntuaciones de lesiones 0, 1, 2 y 3, respectivamente.
Genera comparaciones de abundancia relativa entre rebaños
Bacteroides y Lactobacillus fueron los dos géneros más abundantes en muestras de cecum en todas las bandadas. La abundancia relativa de lactobacillus se encontró en niveles aumentados de control (5,62%), seguido de FUDS- (3,7 %) y FUDS + (2,2 %) aves, respectivamente. Las abundancias relativas de Bacteroides fueron similares entre las bandadas, encontradas en 18,35, 18,03 y 18,04 % en FUDS+, FUDS y aves de control, respectivamente.
Lactobacillus fue el taxón más abundante en las muestras de íleon, con una mayor abundancia en las muestras de control. Lactobacillus se encontró en 27,24, 21,35 y 43,38% en FUDS+, FUDS- y aves de control, respectivamente. Romboutsia bajó la tendencia a la baja después del estado de enfermedad, siendo el más alto en las aves FUDS+ con un 32,36 %, seguido del 29,47 % en el FUDS – y el 18,437% en las aves de control.
Las muestras cloacales mostraron las mayores diferencias entre las bandadas y el estado de enfermedad, siendo Fusobactereium, Turicibacter y Lactobacillus los tres taxones más abundantes. Fusobacterium estuvo presente en 13,72, 24,23 y 8,8 % en FUDS+, FUDS y aves de control, respectivamente. El Turicibacter era notablemente diferente entre las bandadas, pero no entre el estado de enfermedad dentro de una bandada. El Turicibacter consentía un 17,78 % y un 17,52 % de la composición microbiana de las aves FUDS+ y FUDS, mientras que solo comprende el 6,27 % de la microbiota cloaca de las aves de control. Lactobacillus también era diferencialmente abundante en todas las rebaños y el estado de enfermedad, con su máxima representación en el rebaño de control. Lactobacillus se encontró en 10,75, 4,74 y 21,61 % en FUDS+, FUDS y aves de control, respectivamente.
Las muestras de piel fueron dominadas por Staphylococcus, Lactobacillus y Bacteroides, con tendencias notables en todo el estado de la enfermedad y las bandadas. Staphylococcus comprendía el 41,96 % de FUDS+, el 2,35% de FUDS- y el 0,77 % de las muestras de piel de control. Lactobacillus exhibió su representación más baja en las muestras de FUDS+. Lactobacillus estuvo presente en 5,39, 10.02 y 13,05 % en las muestras de FUDS+, FUDS y control, respectivamente. Bacteroides mostró una tendencia similar a la de Lactobacillus entre los estados de la enfermedad. Bacteroides se encontró en 2,72, 9,79, 12,01 % en FUDS+, FUDS- y muestras de control, respectivamente.
Abundancia relativa de Staphylococcus por tipo de muestra
La abundancia de Staphylococcus en la piel se encontró en 41,96, 2,63 y 0,77%, y para FUDS+, FUDS – y las bandadas de control, respectivamente. En el íleon estaba en 0,032, 0,052 y 0,011 % para FUDS+, FUDS- y rebaños de control, respectivamente. Se encontró estafilocoqueo en la cloaca en 0,058, 0,075 y 0,654%, para FUDS+, FUDS- y bandadas de control, respectivamente. La menor abundancia de Staphylococcus se encontró en el cecum, que se encuentra en 0,001, 0,003 y 0 % para FUDS+, FUDS- y bandadas de control, respectivamente.
Análisis de análisis de discriminación lineal, análisis de tamaño de efectos
El análisis LEfSe se realizó para identificar las características microbianas diferenciales entre las bandadas afectadas por el Control y el FUDS (Figura 6). Para la piel, el género Staphylococcus se enriqueció significativamente (p < 0,05) en el rebaño afectado por los FUDS y Bacteroides y Lactalos se enriqueció significativamente (p < 0,05) en el rebaño de control (saludable). Se realizó un segundo análisis de LEfSe solo en Staphylococcus para identificar especies específicas asociadas con el estado de salud de las aves (Figura 7). S. aureus y S. agnetis se identificaron como los únicos dos taxones asociados con las aves FUDS + (p < 0,05), mientras que S. cohnii se enriqueció significativamente en el rebaño de control. Lactobacillus se encontró como un taxón significativo asociado con el grupo sano en todos los tipos de muestra analizados en este estudio. Un análisis más adicional de LEfSe sobre el diferencial Lactobacillus spp. mostró una alta diversidad de Lactobacillus asociado con aves sanas, con L. alvi, L. gallinarum, L. johnsonii y L. acidophilus diferencialmente abundantes en el rebaño de control en comparación con el rebaño de FUDS afectado.
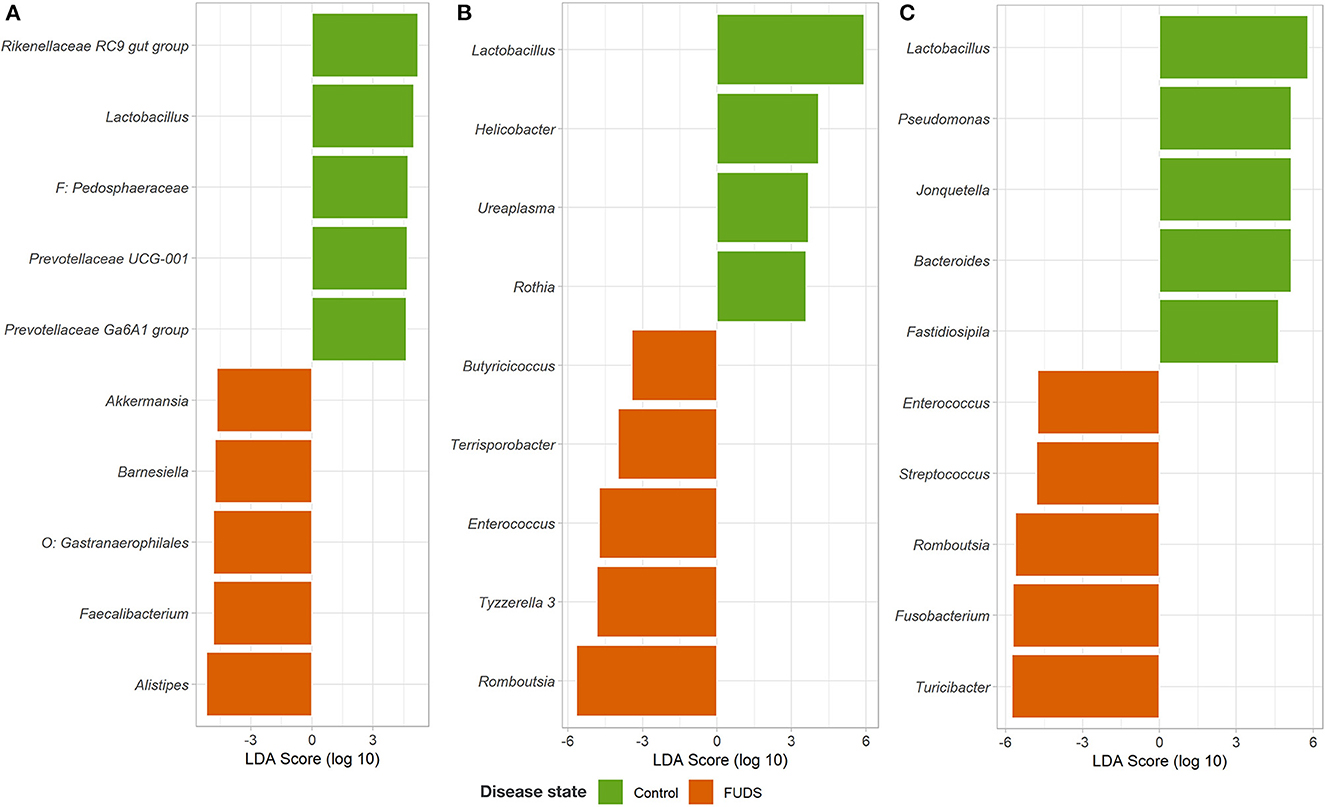 Figura 6. Tamaño del efecto del análisis discriminante lineal (LDA) (LEfSe) para la abundancia diferencial microbiana entre las bandadas afectadas por Control y FUDS. (A) Cecum, (B) ileum y (C) cloaca.
Figura 6. Tamaño del efecto del análisis discriminante lineal (LDA) (LEfSe) para la abundancia diferencial microbiana entre las bandadas afectadas por Control y FUDS. (A) Cecum, (B) ileum y (C) cloaca.
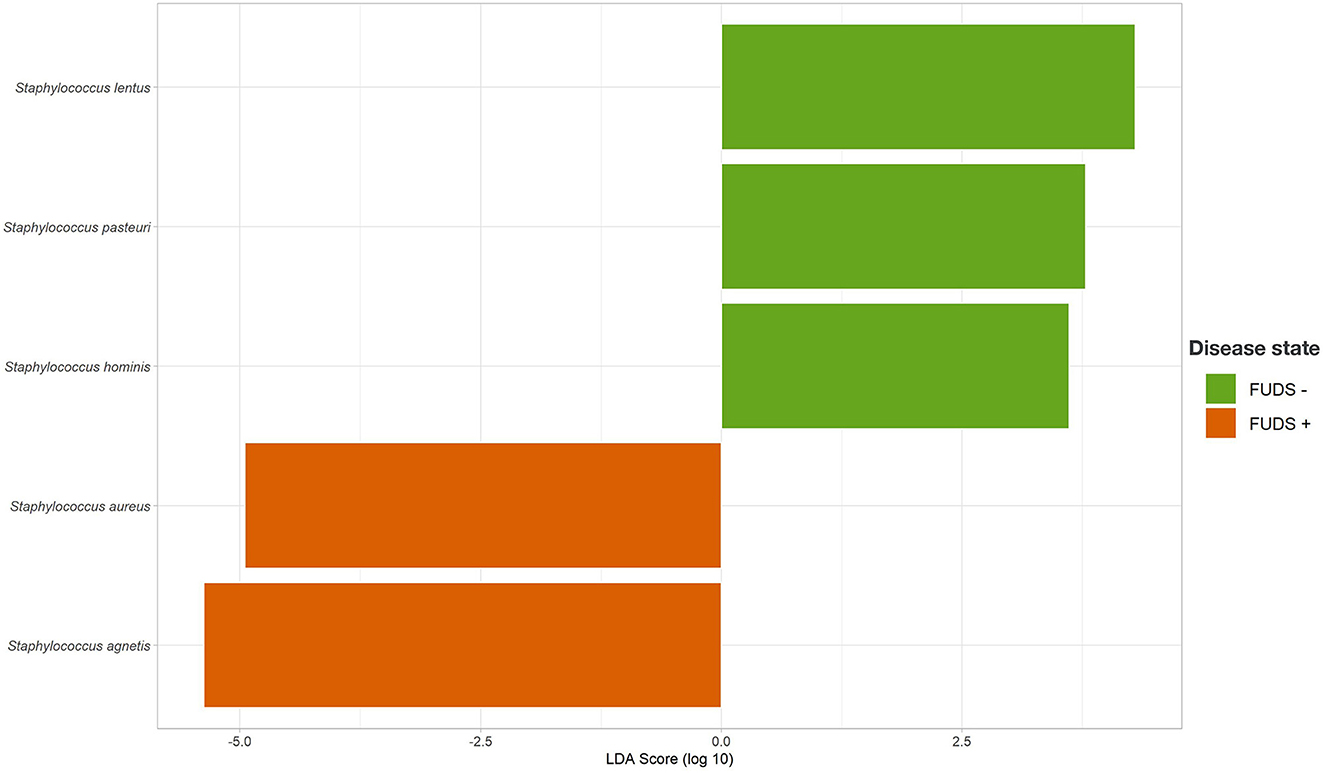 Figura 7. Tamaño del efecto del análisis discriminante lineal (LDA) (LEfSe) para la abundancia diferencial de Staphylococcus entre FUDS + y FUDS-locks.
Figura 7. Tamaño del efecto del análisis discriminante lineal (LDA) (LEfSe) para la abundancia diferencial de Staphylococcus entre FUDS + y FUDS-locks.
Análisis de la secuencia del genoma completo
El conjunto de datos WGS analizado en este estudio está disponible en el repositorio NCBI Sequence Read Archive con acceso BioProject ID PRJNA884648. Las especies de Staphylococcus de los 68 aislados seleccionados fueron confirmadas por WGS, tres aislados identificados taxonómicamente por la amplificación de PCR 16S como S. fleuretti, S. simulans y S. pasteuri fueron identificados por WGS como S. sciuri, S. aureus y S. warneri, respectivamente (Tabla 3, Figura 8).
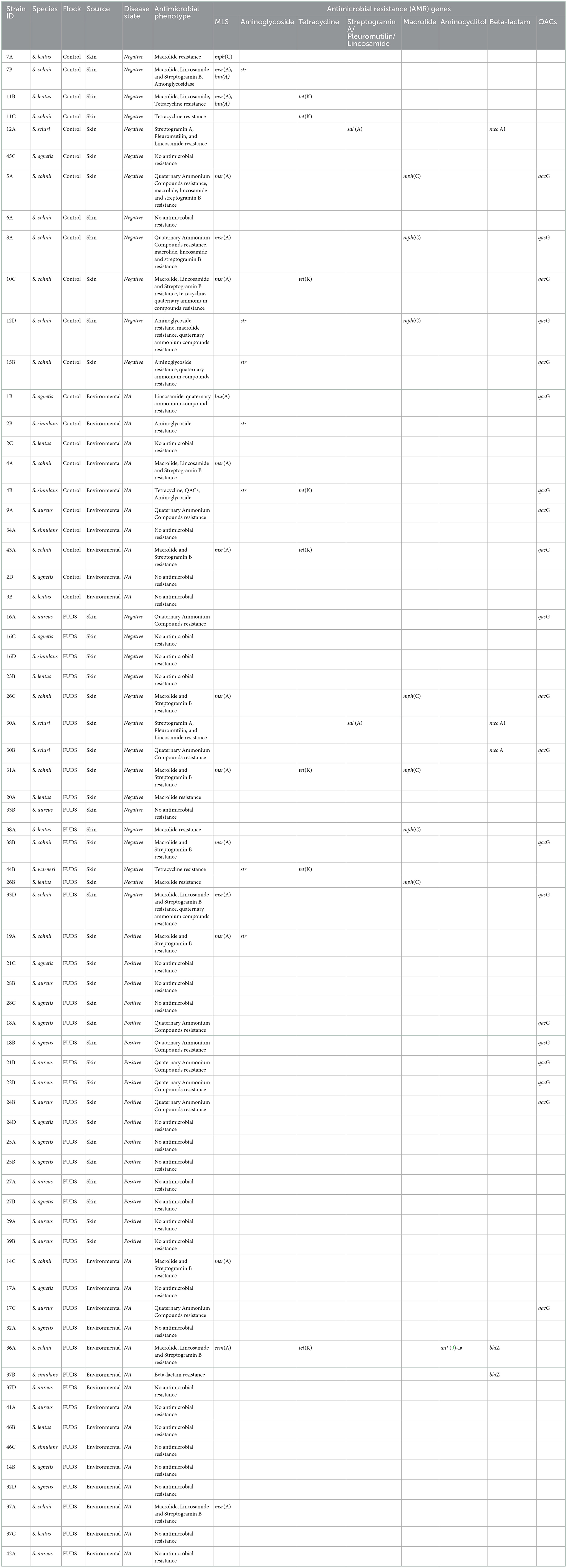 Tabla 3. Análisis de secuenciación del genoma completo de aislados de estafilococos de bandadas afectadas por FUDS, análisis bioinformático para la identificación de genes AMR.
Tabla 3. Análisis de secuenciación del genoma completo de aislados de estafilococos de bandadas afectadas por FUDS, análisis bioinformático para la identificación de genes AMR.
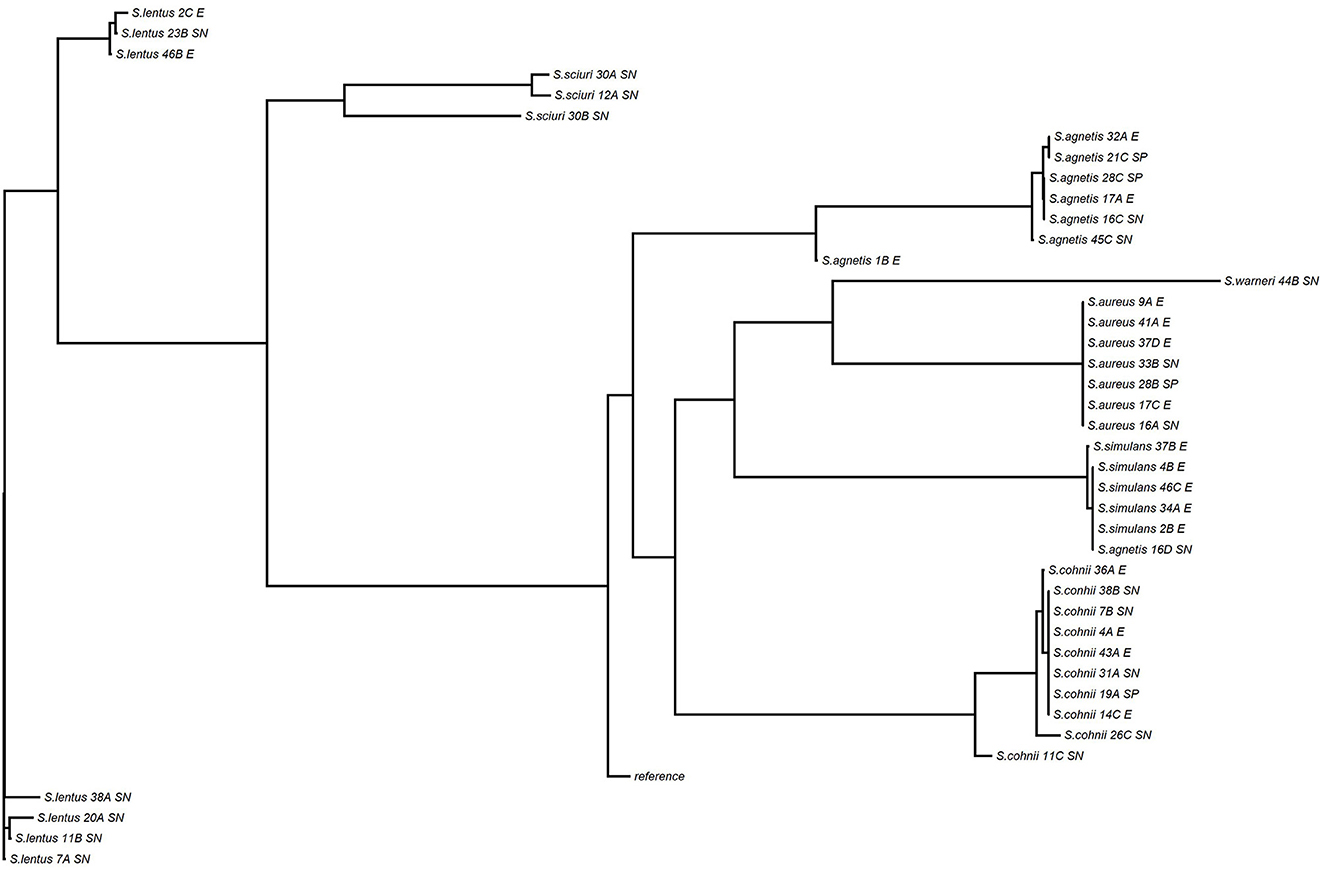 Figura 8. Árbol filogenético de sístos de Staphylococcus aislados de muestras cutáneas y ambientales de bandadas de Control y FUDS.
Figura 8. Árbol filogenético de sístos de Staphylococcus aislados de muestras cutáneas y ambientales de bandadas de Control y FUDS.
Caracterización de la resistencia a los antimicrobianos y la resistencia al desinfectante
Un total de 30 aislados de estafilocofiloco (44,1 %) de los 68 secuenciados tenían entre uno y cuatro genes AMR adquiridos que codifican macrólidos, lincosamidas, espectrograninas, aminoglucósidos, tetraciclinas y resistencia a los betalactámicos (Tabla 3). El gen msrA fue el gen adquirido por AMR más común identificado en el conjunto, que codifica la resistencia a los macrolidos, la lincosamida y la estreptogramina B. Fue transportado por 14 aislados (13 aislados de S. cohnii, 1 S. lentus) y se encontró solo (n = 6) o en combinación con mphC (n = 3), tetK (n = 2), lnuA (n = 2) y spr (n = 1). Los genes tetK ymphC que codifican la proteína de eflujo de tetraciclina y la proteína macrolide fosfotransferasa C, respectivamente, fueron el segundo y tercer genes AMR adquiridos más comunes identificados por 9 (5 S. cohnii, 2 S. lentus, 1 S. warneri y 1 S. simulans) y 8 (5 S. El gen tetK se encontró solo (n = 1), o en combinación con msrA (n = 4), str (n = 2) y mphC (n = 2). El gen mphC se encontró solo (n = 2), o en combinación con msrA (n = 4), tetK (n = 1) y str (n = 1).
Control del rebaño
Un total de 22 aislados de Staphylococcus (32,35%) de los 68 secuenciados se aislaron de la piel (n = 12; 54,44 %) y del medio ambiente (n = 10; 45,45 %). Diez (83,33 %) de los aislados de piel tenían genes AMR en sus genomas, con los genes mphC, msrA y lnuAasociados con la resistencia a la MLS (macrolida, lincosamida y estreptogramína B) (n = 6), como los más prevalentes; 2 aislados (16,67%) no tenían genes AMR en Cinco (50%) de los aislados ambientales tenían genes AMR asociados con MLS, aminoglucósidos y resistencia a la tetraciclina, los 5 aislados ambientales restantes no tenían genes AMR en sus genomas (Tabla 3).
El rebaño afectado por los FUDS
Un total de 46 aislados de Staphylococcus (67,64 %) de los 68 secuenciados se aislaron de la piel de FUDS (n = 15; 32,61%), FUDS + piel (n = 16; 34,78 %) y del medio ambiente (n = 15; 32,61 %). Se observaron diferencias en el contenido de genes de AMR entre los aislados de piel FUDS y FUDS +. Un total de 11 (73,33%) de aislados de FUDS-piel tenían genes de resistencia a la AMR asociados con la resistencia a la MLS y a la tetraciclina, siendo el msrA como el más común; los 4 (26,77%) aislados restantes no tenían genes de resistencia a la AMR en sus genomas. Los aislados de la piel FUDS + estaban dominados por S. aureus y S. agnetis, todos los S. aureus (n = 7) y S. agnetis (n = 8) seleccionados para WGS no tenían genes AMR en sus genomas. Un aislado, S. cohnii, de muestras de piel FUDS+ tenía el gen msrA. Los aislados ambientales seleccionados estaban compuestos por S. aureus (n = 4), S. agnetis (n = 4), S. cohnii (n = 3), S. simulans (n = 2), S. lentus (2). Un total de 11 (73,33 %) de los aislados seleccionados no tenían genes AMR en sus genomas, de manera similar a los aislados de piel +, los aislados de S. aureus y S. agnetis no tenían genes de AMR en sus genomas. Los 4 (26,67%) aislados ambientales restantes tenían genes AMR asociados con MLS, tetraciclina y resistencia a la betalactámica (Tabla 3).
Resistencia al desinfectante
Un total de 20 aislados (29,41 %), de los 68 secuenciados, tenían el gen qacG que codificaba resistencia a los compuestos de amonio cuaternario (QAC). Los aislados resistentes a QACs estaban compuestos por 5 (25 %) de la piel en el grupo de control, 4 (20%) del entorno del grupo de control, 5 (25%) de FUDS + piel, 5 (25%) de FUDS-piel y 1 (5%) del entorno doméstico de FUDS. Ocho (40%) aislados tenían el gen quaG solo, los 12 (60%) restantes tenían el gen quaG en combinación con uno (n = 5), o dos (n = 7) genes AMR adquiridos. El fenotipo más común identificado en los aislados que exhiben resistencia a QACs fue msrA– mphC– qacG (n = 3), msrA– tetK– qacG (n = 2), msrA– qacG (n = 2) (Tabla 3).
Identificación de los factores de virulencia del estafilococo
Se identificaron seis clases de Staphylococcus VF, incluyendo adherencia, enzima, evasión inmune, sistema de secreción, toxina y absorción de hierro. Además, se identificó VF con >50% de homología con especies no estaphylococcus; esta última incluyó las siguientes 7 clases de VF: proteínas de adhesión, regulación, anclaje de proteínas superficiales, antifagocitosis, supervivencia intracelular, resistencia séricas y detención de fargos (Tabla complementaria 1).
La clase de VF con la mayoría de los genes identificados fueron la enzima (n = 171) y la evasión inmune, seguida de la adherencia (n = 84) y la toxina (n = 84). Entre los genes que codifican las enzimas, geh y lip, que codifican para la lipasa, fueron los más prevalentes (n = 54), seguidos de spA (n = 39), que codifican para la proteasa de serina V8, y hysA (n = 26), que codifica para la liasis de hialuronato (Tabla 1 suplementaria Los genes geh y labio se identificaron en todas las especies de Staphylococcus analizadas en este estudio, excepto en S. sciuri y S. lentus, sspA se encontró en todas las especies de Staphylococcus, y hysA solo en los aislados de S. aureus y S. agnetis. En la categoría de evasión inmune, la VF más común incluyó capB (n = 36), que codifica para la cápsula de ácido poliglutámico, la codificación adsA (n = 35) para la adenosina sintasa (A) y la galE (n = 22) que codifica para la cápsula de polisacárido. El capB se encontró en todas las especies de Staphylococcus analizadas en este estudio, excepto en S. aureus, S. sciuri y S. lentus; adsA solo estaba presente en S. aureus, S. agnetis y S. simulans, y galE presente en todas las especies excepto en S. aureus, S. sciuri y S. warneri.
En la categoría de adherencia, la codificación del gen ebp para la proteína de unión a la elastina fue la más común (n = 19), seguido de icaA, icaB y icaC, los genes asociados con la adhesión intercelular (n = 17), otros genes encontrados fueron fnbB (n = 13) que codifica la proteína de unión a la fibron El ebp se encontró solo en S. aureus y S. cohnii; los genes icaA, icaB y icaC solo estaban presentes en S. aureus, S. sciuri; los genes cna estaban presentes solo en S. agnetis, y el atl solo estaba presente en S. aureus. En la categoría de toxina, los genes conjuntos que codifican para la exotoxina fueron los más comunes (n = 25), seguidos de los genes hlgA, hlgB y hlgC asociados con la hemolisina gamma (n = 14) y hlb (n = 14) que codifican la hemolisina B. Los genes establecidos solo se encontraron en S. aureus y S. agnetis. Los genes hlgA, hlgB y hlgC se encontraron solo en S. aureus; adsA solo estaba presente en S. aureus, S. agnetis y S. simulans, y galE presente en todas las especies excepto en S. aureus, S. sciuri y S. warneri.
Efecto antimicrobiano mediante el ensayo de difusión de pozo de agar
Los resultados inhibitorio de las cuatro combinaciones patentadas de Bacillus DFM contra S. aureus y S. agnetis aisladas de lesiones cutáneas de aves afectadas por FUDS se muestran en la Tabla 4. Todos los DFM probados en este estudio se encontraron inhibidores tanto contra Staphylococcus aureus como contra Staphylococcus agnetis.La inhibición de S. aureus osciló de 6 a 22 mm con DFM Combo 4 y DFM Combo 1, respectivamente. La inhibición de S. agnetis osciló entre 14,5 y 25 mm con DFM Combo 4 y DFM Combo 1, respectivamente. Bacillus DFM Combo 1 produjo el efecto inhibitorio más alto con zonas claras de inhibición que oscilaban entre 20 y 22 mm para los aislados de S. aureus y 22 a 25 mm para los aislados de S. agnetis ( DFM Combo 4 fue el menos inhibidor con zonas de inhibiciones que oscilaban entre 6 y 11,5 mm para los aislados de S. aureus y de 14,5 a 16 mm para los aislados de S. agnetis recuperados de las lesiones.
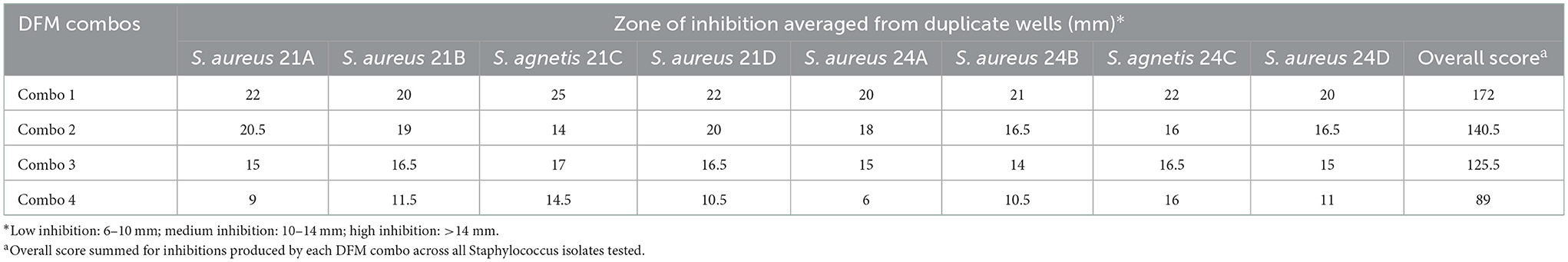 Tabla 4. Actividad antimicrobiana, por difusión de pozo de agar, de la combinación patentada de DFM contra S. agnetis y S. aureus aislados de la piel de las aves FUDS +.
Tabla 4. Actividad antimicrobiana, por difusión de pozo de agar, de la combinación patentada de DFM contra S. agnetis y S. aureus aislados de la piel de las aves FUDS +.
Efecto antimicrobiano por exclusión competitiva en el caldo
El efecto antimicrobiano de las 4 combinaciones de Bacillus DFM por exclusión competitiva en el caldo se muestra en la Tabla 5. Se encontraron que todos los combos de DFM eran inhibidores, las reducciones de S. aureus oscilaron entre 0,24 y 1,55 log10UFC/mL con DFM Combo 4 y Combo 2, respectivamente. Las reducciones de S. agnetis y S. aureus (log10 UFC/mL) se evaluaron 24 horas después de la coinoculación con respecto a las muestras de control. Las reducciones de S. agnetis oscilaron entre -0,05 y 1,17 log10 UFC/mL con DFM Combo 4 y Combo 2, respectivamente. Las reducciones se suman en todos los aislados de Staphylococcus y los combos de DFM se clasificaron del más alto al más bajo efecto antimicrobiano, el combo DFM 2 mostró las inhibiciones más altas en todos los aislados de Staphylococcus probados.
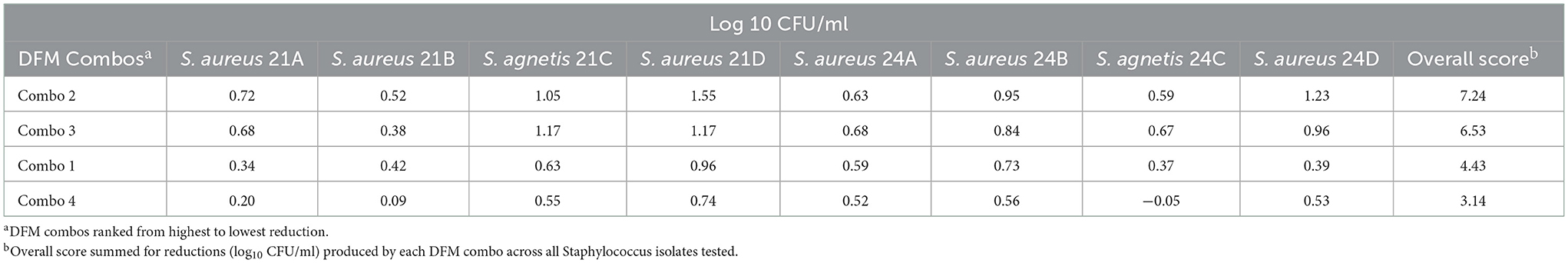 Tabla 5. Reducción de S. agnetis y S. aureus (log10UFC/ml) por combos DFM co-cultivados 24 h en caldo de nutrientes.
Tabla 5. Reducción de S. agnetis y S. aureus (log10UFC/ml) por combos DFM co-cultivados 24 h en caldo de nutrientes.
La puntuación general de difusión de pozo de agar (AWD) se añadió a la puntuación general de exclusión competitiva (CE) para clasificar las combinaciones de DFM con mayor efecto antimicrobiano (Tabla 6). DFM Combo 1 tuvo el mayor efecto en todos los aislados probados.
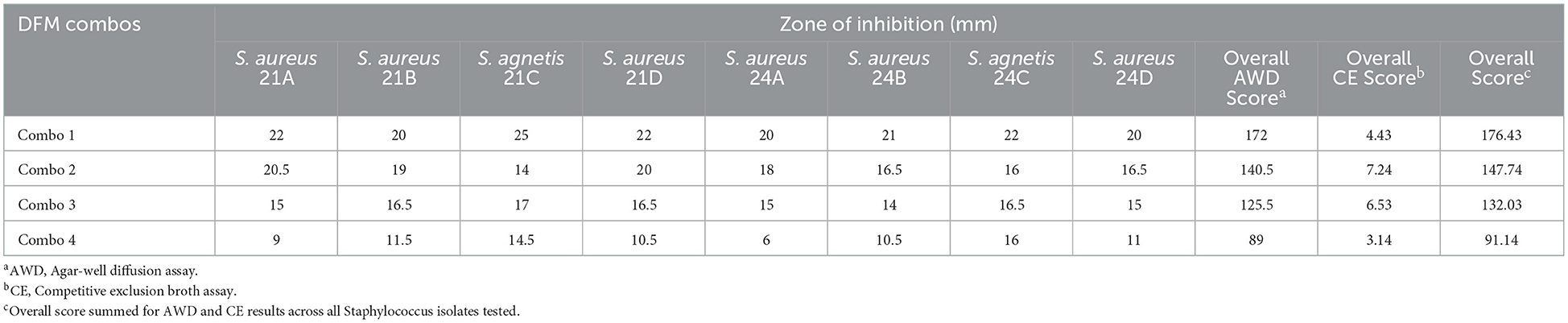 Tabla 6. Puntuaciones combinadas de difusión y exclusión competitiva de DFM examinadas en todos los aislados de Staphylococcus probados.
Tabla 6. Puntuaciones combinadas de difusión y exclusión competitiva de DFM examinadas en todos los aislados de Staphylococcus probados.
Discusión
La transición de los sistemas de producción convencionales a los libres de jaulas ha presentado algunos desafíos para los productores de huevos, uno de estos desafíos es el aumento de las infecciones bacterianas que se han descrito como una de las causas más comunes de mortalidad en aves criadas en sistemas libres de jaulas y basados en camadas (5). Las infecciones de la piel en aves de corral comerciales tienen graves impactos en el crecimiento, la producción de huevos, la conversión de piensos y el rendimiento general. Los miembros del género Staphylococcus han sido descritos como agentes causantes comunes de estas afecciones (30, 31). Las fuentes de agentes infecciosos incluyen el suelo, la basura, la contaminación cruzada entre aves infectadas y los fomites como el equipo contaminado.
En este estudio, se utilizó la secuenciación de próxima generación para caracterizar la composición microbioma de los sitios en el tracto gastrointestinal (GIT) y la piel de las gallinas ponedoras con signos de dermatitis ulcerosa focal (FUDS) y compararla con la composición microbioma de las aves sanas con el objetivo general de identificar los agentes causantes de FUDS y las posibles La comparación de la estructura de la comunidad microbiana entre poblaciones sanas y enfermas es crucial para identificar lo que constituye una microbiota intestinal/piel saludable e identificar la disbiosis potencial, proporcionando así oportunidades para modularla y mejorar la salud animal en general (32). La composición microbiana puede verse afectada por muchos factores, como la edad, la raza, la dieta, el tipo de muestra (es decir, el sitio GIT), el estado de la enfermedad, el sistema de vivienda y el manejo. En este estudio se observó una variación significativa en la población microbiana atribuida al estado de salud del rebaño y el tipo de muestra. Por ejemplo, un género identificado con una abundancia significativamente mayor en la piel y confirmado como presente en las aves FUDS + fue Staphylococcus. La abundancia de Staphylococcus en la piel se encontró en 2,63 y 0,77 % en FUDS, y bandadas de control, respectivamente, en comparación con FUDS + bandada (>41,96 %).
Los estafilococos son omnipresentes en las granjas avícolas y criaderos y son habitantes naturales de la piel y las membranas mucosas del pollo, pero también se han asociado con infecciones secundarias (12). Los resultados de metagenómica y aislamiento de 16S en este estudio mostraron una alta diversidad de especies de Staphylococcus en muestras cutáneas y ambientales de bandadas de FUDS negativas y de control en comparación con el grupo FUDS positivo. De acuerdo con la naturaleza ubicua de los estafilococos en la población microbiana de las aves de corral, las muestras ambientales analizadas en este estudio mostraron una gran diversidad de especies de Staphylococcus, incluyendo S. cohnii, S. lentus, S. simulans, S. agnetis y S. aureus. Estudios recientes han asociado la abundancia de estafilococos coagulas negativos (CoNS) como S. conhii y S. simulans con piel sana en modelos humanos y de ratones (33-35). En 2020, Brown et al. (34) informaron que el comensal S. simulans bloqueó efectivamente la detección de quórum de Staphylococcus aureus resistente a la meticilina (MRSA) en un modelo de infección en ratones. La resistencia observada a la colonización patógena fue el resultado de la producción de un péptido autoinductor que actuó como inhibidor del regulador del gen accesorio (agr) del MRSA S. aureus que protegía al huésped de la infección invasiva. En este estudio, S. simulans se aisló de todos los tipos de muestras ambientales y también se aisló de muestras cutáneas de grupos FUDS negativos y de control. Este resultado podría sugerir un posible efecto protector de esta especie en el desarrollo de la enfermedad.
Staphylococcus cohnii fue otro estafilococo comensal identificado en muestras de la piel de aves FUDS negativas y de control y no presente en muestras de piel FUDS positivas. También fue identificado por el análisis de LEfSe como el taxón más enriquecido en el rebaño de control (saludable) no presente en el rebaño de FUDS. Al igual que S. simulans, S. cohnii se ha asociado con una piel sana. En un análisis del microbioma de la piel de ratones con dermatitis se encontró que el commensal S. cohnii aislado de ratones enfermos tenía efectos preventivos y terapéuticos sobre la inflamación de la piel mediada por S. aureus (33). Se necesita más investigación para determinar el posible efecto protector de los estafilococos comensales encontrados en las muestras de control y FUDS y no aislados de las muestras de piel de FUDS +, en el desarrollo de FUDS.
El género Lactobacillus se encontró de forma ubicua en todos los tipos de muestra asociados con las aves sanas. Nuestros hallazgos son similares a los observados por Ngunjiri et al. (36), que estudiaron el efecto de la edad y el sitio del cuerpo en la composición microbiana de los pollos de capa comercial. Los autores encontraron que Lactobacillus era omnipresente en las muestras de tráquea, nasal, íleon y ceco analizadas. Lactobacillus fue el taxón más significativo en la cloaca e íleon del rebaño de control, lo que sugiere un posible efecto beneficioso/protector para las aves. A menudo se ha atribuido a Lactobacillus un efecto protector a través de la producción de ácido láctico que promueve la inhibición de las bacterias patógenas.
La contribución de la microbiota de la piel al desarrollo de FUDS se analizó a través de la secuenciación metagennómica 16S, y los resultados mostraron una mayor carga de Staphylococcus (>60% de todas las bacterias presentes) a medida que las lesiones cutáneas progresaron de la puntuación de lesión 0 a 3 que en las muestras de FUDS negativas y de control. La disbiosis revelada se caracterizó por un aumento de la abundancia de S. aureus y S. agnetis. Estos resultados se confirmaron mediante la enumeración del recuento de placas, siendo S. aureus y S. agnetis los únicos estafilococos aislados de la piel de las aves FUDS positivas. Nuestros resultados coinciden con los observados por Ito et al. (33) en su análisis comparativo del microbioma de la piel en ratones para identificar cepas comensales con efecto protector de las enfermedades inflamatorias de la piel. Los autores observaron que la gravedad de la dermatitis en ratones se asoció con un aumento de la abundancia de S. aureus y Corynebacterium mastitidis y se asoció negativamente con la abundancia de S. cohnii (33).
Staphylococcus aureus se ha asociado con el desarrollo de múltiples infecciones localizadas en aves de corral, incluyendo abejorro, infecciones articulares, osteomielitis y abscesos de la piel (9, 37, 38). También es el patógeno bacteriano noclostridial más común asociado con la dermatitis gangrenosa, una infección sistémica en pollos (31). En nuestro estudio, se recuperaron los aislados de S. aureus de todas las muestras ambientales y muestras de piel de las aves FUDS +, lo que demuestra una amplia distribución del patógeno en la casa avícola probada. Meyer et al. (9) informaron que el S. aureus fue el principal patógeno bacteriano aislado de diferentes tejidos (pulmones, hígado, lesiones de la mella, entre otros) de múltiples bandadas de capas comerciales, experimentando un aumento de la mortalidad y una caída en la producción de huevos entre abril y noviembre de 2018. A pesar de que S. aureus fue la bacteria predominante aislada, un estudio desafiante, para dilucidar los mecanismos por los cuales S. aureus podría haber estado causando la infección sistémica, no fue capaz de replicar la enfermedad, destacando el papel potencial de otros agentes infecciosos en el desarrollo de la enfermedad (9). En nuestro estudio, S. aureus y S. agnetis se encontraron en combinación en muestras de piel de aves FUDS +, lo que sugiere una posible interacción sinérgica de ambos estafilococos en la patogénesis de la enfermedad.
Staphylococcus agnetis, un Staphylococcus coagulasa variable, se identificó como una especie separada en 2012, asociada con casos clínicos y subclínicos de mastitis bovina (39). Desde su descubrimiento, ha sido reconocido como el agente causal de las enfermedades avícolas como la condronecrnecrosis bacteriana con osteomielitis (COB) (40), endocarditis valvular y septicemia en rebaños de criadores de pollos de ganado (41). En nuestro estudio, los aislados de S. agnetis se recuperaron de muestras ambientales de las casas de control (caja de nido, arañazo) y FUDS (caja de nido); similar a S. aureus, se correlacionó positivamente con la gravedad de la lesión con el aumento de la abundancia de muestras de aves FUDS + con una puntuación de lesión de 3. Se encontraron aislados de S. agnetis agrupados, lo que sugiere que un solo clon circulaba en el entorno de la granja.
El FUDS es una enfermedad emergente de las bandadas de puestas sin jaulas, se han identificado casos tanto en los marrones de Bovan como en Hy-Line. Los análisis bacterianos anteriores de hisopos de lesiones de FUDS identificaron a S. hyicus, una bacteria comensal cutánea asociada con infecciones oportunistas, como el principal patógeno presente en las lesiones (42). En nuestro estudio, el análisis del microbioma 16S y el aislamiento bacteriano de los aislados de la piel identificaron S. agnetis y S. aureus en aves FUDS positivas. Los aislados de S. agnetis recuperados los confirmaron como S. agnetis. Como identificaron Taponen y otros (39), los aislados de S. agnetis están estrechamente relacionados con S. hyicus y S. chromogenes, su diferenciación utilizando métodos fenotípicos y la amplificación de PCR 16S no ha tenido éxito, lo que ha llevado a una identificación errónea de estas especies (8). Además, se plantea la hipótesis de que S. agnetis se desvió de S. hyicus y que algunas de las infecciones atribuidas a S. hyicus podrían haber sido causadas por S. agnetis (8).
Se realizó una caracterización adicional para identificar genes de resistencia antimicrobiana y factores virulentos que podrían haber contribuido al desarrollo de FUDS en un conjunto seleccionado de aislados de estafilococos recuperados en este estudio. El uso de antibióticos en el ganado para el control de enfermedades y la promoción del crecimiento ha llevado a la aparición de genes de la RAM y a una preocupación de salud pública mundial, debido a su posible difusión a bacterias comensales (43). Los genes de resistencia antimicrobiana se propagaron ampliamente en los aislados examinados, con un 44,12 % de ellos albergando entre uno y cuatro genes AMR adquiridos, se encontraron igualmente distribuidos en bandadas de control y FUDS, en muestras cutáneas y ambientales. La resistencia al macroidal, la lincosamida y la espectrogratina fue la más común, caracterizada por la presencia de los genes msrA y mphC en los genotipos de los aislados, el gen msrA estaba albergado por el 46,66 % de los aislados (13 aislados de S. cohnii, 1 S. lentus). Nuestros resultados están de acuerdo con los observados por Zmantar y otros (44) en un estudio para determinar la susceptibilidad antimicrobiana y la resistencia a los compuestos de amonio cuaternario (QAC) en S. aureus clínico y coagulase negativoStaphylococcus (CoNS). Los autores encontraron que el gen msrA es uno de los más prevalentes, identificado en el 41 % de CoNS y en el 10,2% de las cepas de S. aureus. Sorprendentemente, en nuestro estudio, allS. aureus y el 93,333% (n = 14/15) de los aislados de S. agnetis no llevaban ningún gen de AMR en sus genotipos. El gen msrA codifica para una bomba de eflujo dependiente de ATP que transporta eritromicina y estreptogramina B de la resistencia inductora celular a estos antibióticos; y se ha asociado principalmente a Staphylococcus y se ha descubierto recientemente en otros géneros, incluyendo Streptococcus, Enterococcus y Pseudomonas (45).
El gen tetK fue el segundo gen AMR más común identificado en nuestro estudio, albergado por el 30 % de los aislados. Codifica una bomba de eflujo de tetraciclina que confiere resistencia, esta resistencia se observa comúnmente en aislados de estafilococos, independientemente de la fuente debido a la difusión de los genes tetK/tetM (46). Ali Syed et al. (47) encontraron resultados similares, con los genes tetK/tetM presentes en el 58,8 % de los estafilococos de pollos de granja. Al igual que nuestros resultados, los autores encontraron los genes albergados por CoNS. La amplia distribución de los genes de la RAM entre los estafilococos no asociados con el desarrollo de FUDS constituye un riesgo potencial de transmisión de genes de la RAM a estafilococos patógenos o bacterias comensales, lo que destaca la importancia de monitorear la resistencia a los antibióticos en las instalaciones avícolas.
En este estudio también se analizó el refugio de genes de resistencia al desinfectante. Los desinfectantes son ampliamente utilizados en la avicultura y consisten en dos o más componentes activos con compuestos de amonio cuaternario (QAC), el componente más utilizado (48). La aplicación de concentraciones subcontraitorias ha reducido el efecto bactericida de los desinfectantes, lo que ha llevado a una mayor resistencia a los desinfectantes basados en QAC y a un posible aumento de la resistencia a los antibióticos. La resistencia a los QACs está asociada a la presencia de los genes qac (qacA, qacB, qacC, qacD y qacG) y smr. En este estudio, el 29,41 % de los aislados secuenciados tenían el gen qacG presente, el 40 % de los aislados (exclusivamente S. aureus y S. agnetis) tenían el gen qacG presente y ningún otro gen AMR. El 60% restante de los aislados de estafilococos examinados tenían el gen qacGpresente en combinación en uno o dos genes AMR con resistencia a los macrólidos más comunes. El Zayed Zaki et al. (49) evaluaron la prevalencia de genes qac y smr en aislados clínicos de S. aureus de infecciones adquiridas en el hospital y su susceptibilidad a los desinfectantes y antibióticos basados en QAC. Los autores encontraron que el gen qacG es uno de los genes más prevalentes, presente en el 17,8 % de los aislados, los resultados estaban fuertemente correlacionados con la reducción de la susceptibilidad a los desinfectantes basados en QAC. En nuestro estudio, S. aureus y S. agnetis de las aves FUDS + no mostraba resistencia a los QAC.
El análisis de la virulencia es un paso importante a la hora de evaluar las intervenciones para inhibir los agentes causantes de una enfermedad. Está bien establecido que los estafilococos expresan una amplia variedad de factores de virulencia que les permiten evadir la respuesta inmune y aumentar su infectividad (50). Los aislados de estafilococos examinados en este estudio albergaron una serie de genes de virulencia que codifican las enzimas, la evasión inmune, la adherencia y las toxinas que podrían tener un efecto aditivo en el desarrollo de FUDS. Los genes geh y labio que codifican para la lipasa fueron los genes codificantes de enzimas más prevalentes identificados, ambos se encontraron ampliamente difundidos en los aislados de Staphylococcus examinados en este estudio. Las lipasas son factores importantes para la colonización y persistencia de cepas de estafilococos en la piel y tienen un papel potencial en el crecimiento bacteriano por la liberación de ácidos grasos del huésped durante la patogénesis. Delekta et al. (51) demostraron que la inactivación del gen geh redujo la capacidad de las cepas de S. aureus para utilizar las LDL del huésped como fuente de ácidos grasos exógenos, con un impacto potencial en la patogénesis (51). El gen hysA que codifica la lyasa de hialuronato fue otro factor de virulencia común identificado, solo presente en S. aureus y S. agnetis. Las hialuronidasas son enzimas bacterianas que se rompen el enlace glucosídico β-1,4 del ácido hialurónico, una sustancia natural abundante en la piel, las articulaciones y los tejidos. Estos factores de virulencia están asociados con una rápida diseminación patógena a través de los tejidos que provoca un aumento del tamaño de la lesión durante la infección. Los tejidos con altas concentraciones de ácido hialurónico a menudo se han encontrado infectados con S. aureus (52). Ibberson et al. (52) demostraron una reducción en el daño tisular del pulmón de los ratones cuando se desafía usar una cepa mutante de S. aureus hysA, haciendo hincapié en el papel de hysA en la infección bacteriana de S. aureus. En nuestro estudio, el gen hysA se difundió ampliamente entre los aislados de S. aureus y S. agnetis de las aves FUDS +, lo que sugiere un papel potencial en su difusión, patogenicidad y desarrollo de FUDS.
Los estafilococos albergan proteínas de la pared celular involucradas en la adhesión junto con la evasión de tejido y defensa inmune (50). Las proteínas de la pared celular interactúan directa o indirectamente con los receptores del huésped para iniciar la unión a las células epiteliales. Se ha descubierto que esta interacción entre el patógeno y las células huésped es dependiente de la especie y la cepa (53). La proteína S. aureus A codificada por el gen spA se ha asociado comúnmente con la evasión de la respuesta inmunitaria. En nuestro estudio, se encontró en 3 aislados de S. aureus (2 de muestras ambientales de FUDS y 1 de muestras de FUDS + piel). Otras proteínas, como thefnbB, que codifican para las proteínas de unión a la fibronectina y la codificación de cna para la adhesina de colágeno, se encontraron solo en los aislados de S. agnetis (n = 13). Poulsen et al. (41) analizaron los genomas de tres aislados de S. agnetis de aves con endocarditis valvular e identificaron el gen fnbB junto con otros seis factores de virulencia asociados con las proteínas de unión a la fibronectina. La presencia de estos genes destaca un papel potencial en la adhesión celular, la difusión y la invasión de los tejidos del huésped. La producción de toxinas es otro factor importante que contribuye a la patogénesis por la interrupción de las membranas eucariotas, la intección con los receptores y la causa de la lisis celular (50). S. aureus y S. agnetis en este estudio, albergaron genes que codifican exotoxinas (génesis de conjunto), toxinas formadoras de alfa y betaporina y toxina exfoliativa tipo A. Estas toxinas han estado implicadas en el desarrollo de dermatosis inducidas por Staphylococcus, como la dermatitis acantolítica en humanos (10), y la dermatitis gangrenosa en aves de corral (31). Nuestros resultados están de acuerdo con los observados por Al-Rubaye et al. (40), quienes identificaron S. agnetis como un agente causal de la cojera en pollos, y los autores también identificaron un repertorio de determinantes de la virulencia, incluyendo la toxina exfoliativa A, proteínas de unión a la fibronectina y varias otras asociadas con la adherencia, Nuestros hallazgos sugieren que el desarrollo de FUDS se produce como resultado de una combinación de mecanismos «de lado hacia fuera» por los cuales ambos estafilococos se translocan a través de los tejidos anfitriones utilizando los factores de virulencia cooperativos que expresan y proliferan en la piel desarrollando lesiones típicas de FUDS. Las lesiones expuestas son una fuente de contaminación cruzada para otros animales y el medio ambiente.
Una vez que se identificaron y caracterizaron completamente los patógenos, el siguiente paso fue el desarrollo de un producto DFM personalizado para inhibir/reducir su crecimiento. El producto desarrollado se basó en el uso de nuevos microbios alimentados directamente de propiedad. Los DFM pueden ser una alternativa natural al uso subterapéutico de antibióticos en la producción animal. Hay un mayor interés en su uso para reducir la propagación de la resistencia a los antimicrobianos al tiempo que se apoya una mejor eficiencia de la producción. En este estudio, se desarrollaron cuatro combinaciones patentadas de Bacillus basadas en la detección antagónica previa contra múltiples patógenos, incluido Staphylococcus spp. El efecto antimicrobiano de las combinaciones se determinó en base a la reducción general de los aislados de S. aureus y S. agnetis por difusión de pozos de agar y pruebas de detección antimicrobianas de exclusión competitiva. El combo DFM 1, una combinación de dos Bacillus pumilus, se encontró como la más eficaz para inhibir el crecimiento de ambos estafilococos aislados de la piel de las aves enfermas en comparación con las otras combinaciones probadas. Como se observó en otros estudios, hubo una fuerte asociación entre las especies de Bacillus y el efecto antagonista, con las mayores inhibiciones logradas con la combinación Bacillus pumilus en el combo DFM 1, seguido del combo 2 que contiene dos B. pumilus, un B. amyloliquifaciens y una cepa B. subtilis.
Los bacilli son bacterias formadoras de endospora ampliamente utilizadas en las preparaciones de DFM debido a su capacidad para sobrevivir a altas temperaturas, entornos ácidos y desecación, aumentando así su viabilidad durante los procesos de fabricación y peletización y mejorando su estabilidad en el GIT del huésped (54, 55). Se sabe que promueven la salud intestinal mediante la producción de enzimas, péptidos antimicrobianos y otros metabolitos que inhiben/reducen el crecimiento de patógenos bacterianos (56). En nuestro estudio, la combinación de B. pumilus ejerció las inhibiciones mejoradas con zonas de inhibición que oscilaban entre 20 y 22 y 22 y 25 mm, para las cepas de S. aureus y S. agnetis, respectivamente. Estas inhibiciones fueron más altas que las observadas por Ouoba y otros (57) que informaron que, en un ensayo de difusión de puntos de agar, las cepas B. pumilus B6 y B10 inhibieron los aislados del indicador S. aureus con zonas de inhibición < 3 mm, 24 horas después de la incubación (57).
Al igual que otros Bacilli, B. pumilus produce péptidos antimicrobianos como bactericinas y otros compuestos, incluidas las amicoumacinas con propiedades antimicrobianas, antifúngicas y antiinflamatorias (58). Aunpad y Bangchang (59) informaron sobre el efecto antimicrobiano mejorado de la bacteriocina pumilicina 4, contra S. aureus resistente a la meticilina (MRSA). En 2012, Hashimoto y otros (60) informaron de un fuerte efecto anti-MRSA por los derivados de las amicoumacinas A y B con concentraciones mínimas inhibitorias (MIC) entre 0,6 y 256 ug/ml que no permiten un crecimiento visible de MRSA. En nuestro estudio, el co-cultivo de B. pumilus combo 1 con Staphylococcus aureus y Staphylococcus agnetis permitió reducciones entre 0,3-4-80,96 log10 UFC/ml, y 0,37–0.63 log10 UFC/ml, respectivamente, después de 24 horas de co-inoculación. Es importante destacar que las concentraciones del Bacillus pumilus probado se encontraron a 108CFU/ml, 24 horas después de la co-inoculación.
El control de los patógenos es uno de los principales intereses de la industria avícola desde el punto de vista económico y de salud pública. La suplementación de Bacillus DFM de las dietas de gallina en capas ha demostrado una mejor producción de puesta al mejorar la salud intestinal, aumentar la fermentación intestinal y la asimilación de minerales, y mejorar la ingesta diaria general de piensos (14, 61, 62). El mecanismo exacto por el cual la suplementación de Bacillus DFM inhibe a los patógenos no se entiende bien; la inhibición directa por la producción de metabolitos antimicrobianos o la exclusión competitiva se han sugerido como los principales modos de acción, también se han sugerido otros mecanismos como la mejora de la capa de la mucosa intestinal para evitar la difusión microbiana a través de la membrana (14) La combinación específica de B. pumilus desarrollada para este estudio, inhibió ambos patógenos in vitro y se está utilizando en diferentes granjas con antecedentes de FUDS con resultados exitosos en: (1) inhibición del efecto sinérgico de S. agnetis y S. aureus, (2) disminución de la mortalidad por FUDS y (3) mejora de los huevos cosechables (datos Se realizarán más análisis para determinar el efecto de la suplementación de Bacillus DFM en un posible cambio de la población microbiana de las gallinas ponedoras en granjas previamente afectadas por FUDS.
Conclusión
Staphylococcus aureus y Staphylococcus agnetis se identificaron como los agentes causantes de FUDS en las gallinas ponedoras. Ambos patógenos se encontraron como especies bacterianas estadísticamente significativas presentes solo en la piel de las aves FUDS +, este resultado se confirmó mediante el recubrimiento con ambos estafilococos como los únicos patógenos aislados de la piel. Tanto S. aureus como S. agnetis tenían un alto número de factores de virulencia asociados con la adherencia, la producción de toxinas y la evasión inmune que podrían haber contribuido a su patogénesis y al desarrollo de FUDS. El producto personalizado Bacillus pumilus desarrollado en este estudio se encontró exitoso en la inhibición de ambos patógenos in vitro y actualmente se está alimentando en instalaciones avícolas con pollos afectados por FUDS con mejores resultados en la reducción de la mortalidad y una mayor producción de huevos.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio/de los repositorios y el(los) número(s) de acceso se pueden encontrar en el artículo/material complementario.
Contribuciones del autor
DA, DG y BT realizaron todos los experimentos del estudio. NE recogió muestras para su análisis, anotó las aves en función de la progresión de la lesión y escribió la metodología de recolección de muestreo. DA, ML y DG contribuyeron a los análisis bioinformáticos. TK y CN concibieron el estudio y desarrollaron el diseño del estudio. DA escribió el primer borrador del manuscrito. DG, BT, KR y EK contribuyeron a escribir. Todos los autores contribuyeron a la edición de la versión final del manuscrito.
Financiación
Los autores declaran que este estudio recibió financiación de Purina Animal Nutrition. El financiador no participó en el diseño del estudio, la recopilación, el análisis, la interpretación de los datos, la redacción de este artículo o la decisión de presentarlo para su publicación.
Conflicto de intereses
DA, DG, NE, KR, EK, BT, ML, CN y TK fueron empleados por Purina Animal Nutrition Center, Land O’ Lakes.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1110573/full#supplementary-material
Referencias
1. Brannan KE, Anderson KE. Examen del impacto del alcance, los sistemas modificados y sin jaulas y los entornos de jaula convencionales en los insumos de mano de obra comprometidos con el cuidado de aves para tres cepas de capa de huevo de óvulo pardo. J Appl Poult Res. (2021) 30:1–7. doi: 10.1016/j.japr.2020.100118
2. Departamento de Agricultura de los Estados Unidos. Servicio Nacional de Estadísticas Agrícolas (USDA-NASS); Washington DC: Pollo y huevos (2019). p. 1948-9064.
3. Departamento de Agricultura de los Estados Unidos. Informe de huevos de cáscara sin jaula del USDA: Resumen de pollos y huevos 2020 (febrero de 2021). USDA, Servicio Nacional de Estadísticas Agrícolas (2020).
4. van Goor A, Redweik GAJ, Stromberg ZR, Treadwell CG, Xin H, Mellata M. El microbioma y los marcadores biológicos de la sangre cambian en las gallinas en diferentes etapas de puesta en viviendas convencionales y libres de jaulas. Poult Sci. (2020) 99:2362–74. doi: 10.1016/j.psj.2020.01.011
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
5. Lay DC, Fulton RM, Hester PY, Karcher DM, Kjaer JB, Mench JA, et al. Bienestar de gallinas en diferentes sistemas de vivienda. Poult Sci. (2011) 90:278-94. doi: 10.3382/ps.2010-00962
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
6. Hartcher KM, Jones B. El bienestar de las gallinas de capa en jaulas y sistemas de vivienda sin jaulas. Worlds Poult Sci J. (2017) 73:767–81. doi: 10.1017/S0043933917000812
7. Gingerich E. El estado actual de la salud de Pullet y Layer en los Estados Unidos. En: Actas de la Federación de Aves del Medio Oeste (2020). Disponible en línea en: https://midwestpoultry.com/wp-content/uploads/2020/08/Gingerich-Eric.pdf (consultado del 12 al 14 de agosto de 2020).
8. Szafraniec GM, Szeleszczuk P, Dolka B. Una revisión de los conocimientos actuales sobre staphylococcus agnetis en aves de corral. Animales. (2020) 10:1-19. doi: 10.3390/ani10081421
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
9. Meyer, M, Bobeck, E, Sato, Y, El-Gazzar, B,. Comparación de la infección natural frente a la infección experimental de Staphylococcus aureus Seppticemia en gallinas laces en dos grupos de edad diferentes. Disponible en línea en: http://meridian.allenpress.com/avian-diseases/article-pdf/65/2/310/2868161/i0005-2086-65-2-310.pdf (consultado el 7 de octubre de 2021).
10. Chénier S, Lallier L. Folliculitis aconlítica y epidermitis asociadas con Staphylococcus hyicus en una línea de pollos ponedores de cuernos de pierna blanca. Vet Pathol. (2012) 49:284–7. doi: 10.1177/0300985811415705
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
11. Ahrens P, Andresen LO. Clonación y análisis de secuencia de genes que codifican los tipos de toxina exfoliativa Staphylococcus hyicus A, B, C y D. J Bacteriol. (2004) 186:1833–7. doi: 10.1128/JB.186.6.1833-1837.2004
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
12. Fries-Craft K, Meyer MM, Sato Y, El-Gazzar M, Bobeck EA. La edad y la vía de inoculación de Staphylococcus aureus alteran diferencialmente el potencial metabólico y las poblaciones de células inmunitarias en las gallinas ponedoras. Vet Vet frontal Sci. (2021) 8:1-13. doi: 10.3389/fvets.2021.653129
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
13. Johnson TA, Sylte MJ, Looft T. El disalicato de bacitracina de metileno en alimentación modula la microbiota del pavo y el metaboloma de una manera dependiente de la dosis. Sci Rep. (2019) 9:8212. doi: 10.1038/s41598-019-44338-5
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
14. Jha R, Das R, Oak S, Mishra P. Probióticos (bi microbios alimentados directamente) en la nutrición de las aves de corral y sus efectos sobre la utilización de nutrientes, el crecimiento y el rendimiento de la puesta, y la salud intestinal: una revisión sistemática. Animales. (2020) 10:1-19. doi: 10.3390/ani10101863
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
15. Krysiak K, Konkol D, Korczyński M. Revise la descripción general del uso de probióticos en la producción avícola. Animales. (2021) 11:1620. doi: 10.3390/ani11061620
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
16. Shivaramaiah S, Pumford NR, Morgan MJ, Wolfenden RE, Wolfenden AD, Torres-Rodriguez A, et al. Evaluación de las especies de bacilos como posibles candidatos para microbios alimentados directamente en aves de corral comerciales. Poult Sci. (2011) 90:1574-80. doi: 10.3382/ps.2010-00745
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
17. Fuller R. Historia y desarrollo de los probióticos. En: Probióticos. Países Bajos: Springer (1992). p. 1–8.
18. Hernandez-Patlan D, Solis-Cruz B, Pontin KP, Hernandez-Velasco X, Merino-Guzman R, Adhikari B, et al. Impacto de un bacilo microbio alimentado directamente en el rendimiento del crecimiento, la integridad de la barrera intestinal, las lesiones por enteritis necrótica y la microbiota ileal en pollos de pollos de engorde utilizando un modelo de desafío de laboratorio. Vet Vet frontal Sci. (2019) 6:108. doi: 10.3389/fvets.2019.00108
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
19. Klindworth A, Pruesse E, Schweer T, Peplies J, Quast C, Horn M, et al. Evaluación de los cebadores generales de PCR del gen de ARN ribosómico 16S para estudios de diversidad basados en la secuenciación clásicos y de próxima generación. Nucleic Acids Res. (2013) 41:e1. doi: 10.1093/nar/gks808
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
20. Bolyen E, Rideout JR, Dillon MR, Bokulich NA, Abnet CC, Al-Ghalith GA, et al. Ciencia de datos del microbioma reproducible, interactiva, escalable y extensible utilizando QIIME 2. Nat Biotechnol. (2019) 37:852–7. doi: 10.1038/s41587-019-0209-9
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
21. Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, et al. El proyecto de base de datos de genes de ARN ribosómico SILVA: procesamiento de datos mejorado y herramientas basadas en la web. Ácidos Nucleicos Res. (2013) 41:D590–6. doi: 10.1093/nar/gks1219
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
22. Segata N, Izard J, Waldron L, Gevers D, Miropolsky L, Garrett WS, et al. Descubrimiento y explicación de biomarcadores metanómicos. Genome Biol. (2011) 12:R60. doi: 10.1186/gb-2011-12-6-r60
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
23. Bolger AM, Lohse M, Usadel B. Trimmomático: un recortador flexible para datos de secuencia de Illumina. Bioinformática. (2014) 30:2114–20. doi: 10.1093/bioinformática/btu170
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
24. Bankevich A, Nurk S, Antipov D, Gurevich AA, Dvorkin M, Kulikov AS, et al. SPAdes: un nuevo algoritmo de ensamblaje del genoma y sus aplicaciones a la secuenciación unicelular. J Comput Biol. (2012) 19:455–77. doi: 10.1089/cmb.2012.0021
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
25. Zankari E, Hasman H, Cosentino S, Vestergaard M, Rasmussen S, Lund O, et al. Identificación de los genes de resistencia antimicrobiana adquiridos. J Antimicrob Chemother. (2012) 67:2640–4. doi: 10.1093/jac/dks261
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
26. Chen L, Yang J, Yu J, Yao Z, Sun L, Shen Y, et al. VFDB: una base de datos de referencia para los factores de virulencia bacteriana. Ácidos Nucleicos Res. (2005) 33: D325-8. doi: 10.1093/nar/gki008
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
27. Stamatakis A. RAxML versión 8: una herramienta para el análisis filogenético y el postanálisis de grandes filogenias. Bioinformática. (2014) 30:1312–3. doi: 10.1093/bioinformática/btu033
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
28. Vinderola G, Capellini B, Villarreal F, Suárez V, Quiberoni A, Reinheimer J. Utilidad de un conjunto de pruebas simples in vitro para la detección e identificación de cepas candidatas probióticas para uso lácteo. LWT. (2008) 41:1678–88. doi: 10.1016/j.lwt.2007.10.008
29. Ayala DI, Cook PW, Franco JG, Bugarel M, Kottapalli KR, Loneragan GH, et al. Un enfoque sistemático para identificar y caracterizar la eficacia y seguridad de las nuevas cepas probióticas para controlar los patógenos transmitidos por los alimentos. Microbiol frontal. (2019) 10:1108. doi: 10.3389/fmicb.2019.01108
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
30. Szafraniec GM, Szeleszczuk P, Dolka B. Revisión de los trastornos esqueléticos causados por Staphylococcus spp. en aves de corral. Vet Quart. (2022) 42:21–40. doi: 10.1080/01652176.2022.2033880
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
31. Gornatti-Churria CD, Crispo M, Shivaprasad HL, Uzal FA. Dermatitis gangrenosa en pollos y pavos. J Vet Diagn Invest. (2018) 30:188–96. doi: 10.1177/1040638717742435
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
32. Carrasco JMD, Casanova NA, Miyakawa MEF. Microbiota, salud intestinal y productividad del pollo: ¿Cuál es la conexión? Microorganismos. (2019) 7:374. doi: 10.3390/microorganismos7100374
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
33. Ito Y, Sasaki T, Li Y, Tanoue T, Sugiura Y, Skelly AN, et al. Staphylococcus cohnii es un comensal potencialmente bioterapéutico de la piel que alivia la inflamación de la piel. Cell Rep. (2021) 35:109052. doi: 10.1016/j.celrep.2021.109052
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
34. Brown MM, Kwiecinski JM, Cruz LM, Shahbandi A, Todd DA, Cech NB, et al. El nuevo péptido de Staphylococcus simulns comensal bloquea la detección de quórum de Staphylococcus aureus resistente a la meticilina y protege la piel del huésped del daño. Agentes antimicrobianos Chemother. (2020) 64:e00172-20. doi: 10.1128/AAC.00172-20
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
35. Byrd AL, Belkaid Y, Segre JA. El microbioma de la piel humana. Nat Rev Microbiol. (2018) 16:143-55. doi: 10.1038/nrmicro.2017.157
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
36. Ngunjiri JM, Taylor KJM, Abundo MC, Jang H, Elaish M, Mahesh KC, et al. La etapa de la granja, la edad de las aves y el sitio corporal afectan predominantemente la cantidad, la composición taxonómica y la dinámica de la microbiota respiratoria e intestinal de los pollos de capa comercial. Appl Environ Microbiol. (2019) 85:e03137-18. doi: 10.1128/AEM.03137-18
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
37. Jiang T, Mandal RK, Wideman RF, Khatiwara A, Pevzner I, Kwon YM. Estudio molecular de las comunidades bacterianas asociadas con la condronecrosis bacteriana con osteomielitis (OB) en pollos de engordería. PLoS ONE. (2015) 10:e0124403. doi: 10.1371/journal.pone.0124403
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
38. Andreasen CB. Staphylococcosis. En:Swayne DE, , editor. 14a edición. Enfermedades de las aves de corral. Hoboken, NJ: John Wiley and Sons, Ltd. (2020). p. 995–1003.
39. Taponen S, Supré K, Piessens V, van Coillie E, de Vliegher S, Koort JMK. Staphylococcus agnetis sp. nov, una especie variable de coagulasa de mastitis subclínica y clínica leve bovina. Int J Syst Evol Microbiol. (2011) 62:61–5. doi: 10.1099/ijs.0.028365-0
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
40. Alrubaye AAK, Ekesi NS, Hasan A, Koltes DA, Wideman RF, Rhoads DD. Chondroncrosis con osteomielitis en pollos de engorde: definición adicional de un modelo de desafío bacteriano utilizando pisos de arena estándar y protección con probióticos. Poult Sci. (2020) 99:6474–80. doi: 10.1016/j.psj.2020.08.067
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
41. Poulsen LL, Thøfner I, Bisgaard M, Olsen RH, Christensen JP, Christensen H. Staphylococcus agnetis, un patógeno potencial en los criadores de pollos de engorde. Microbiol veterinario.(2017) 212:1–6. doi: 10.1016/j.vetmic.2017.10.018
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
42. Lossie G, Beilke E, Gingerich E, Wakenell P. Un caso único de dermatitis ulcerosa en una bandada de capas marrones comerciales y sin jaula. En: Actas anuales de la Asociación Americana de Patólogos Aviares. (2016). p. 5-6. Disponible en línea en: https://aaap.memberclicks.net/assets/2016_Annual_Meeting/aaap%20proceedings%202016%20v2.pdf (consultado el 7 de octubre de 2020).
43. Peralta-Sánchez JM, Martín-Platero AM, Ariza-Romero JJ, Rabelo-Ruiz M, Zurita-González MJ, Baños A, et al. La producción de huevos en la avicultura se ve mejorada por las bacterias probióticas. Microbiol frontal. (2019) 10:1042. doi: 10.3389/fmicb.2019.01042
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
44. Zmantar T, Kouidhi B, Miladi H, Bakhrouf A. Detección de genes de resistencia a los macrólidos y al desinfectante en Staphylococcus aureus clínico y estafilocococos negativos a la coagulasa. Notas de BMC Res. (2011) 4:453. doi: 10.1186/1756-0500-4-453
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
45. Ojo KK, Striplin MJ, Ulep CC, Close NS, Zittle J, Luis H, et al. Gene de eflujo de Staphylococcus msr(A) caracterizado en aislados de Streptococcus, Enterococcus, Corynebacterium y Pseudomonas. Agentes antimicrobianos Chemother. (2006) 50:1089–91. doi: 10.1128/AAC.50.3.1089-1091.2006
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
46. Leroy S, Christieans S, Talon R. Transferencia del gen de la tetraciclina en staphylococcus xylosus in situ durante la fermentación de la salchicha. Microbiol frontal. (2019) 10:392. doi: 10.3389/fmicb.2019.00392
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
47. Syed MA, Ullah H, Tabassum S, Fatima B, Woodley TA, Ramadan H, et al. Staphylococci en los intestinos de aves de corral: una comparación entre pollos de granja y domésticos. Poult Sci. (2020) 99:4549–57. doi: 10.1016/j.psj.2020.05.051
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
48. Maertens H, Demeyere K, de Reu K, Dewulf J, Vanhauteghem D, van Coillie E, et al. Efecto de la exposición subhibitoria a compuestos de amonio cuaternario en la susceptibilidad a la ciprofloxacina de las cepas de Escherichia coli en la ganadería. BMC Microbiol. (2020) 20:155. doi: 10.1186/s12866-020-01818-3
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
49. el Sayed Zaki M. Estudio molecular de la resistencia de Staphylococcus aureus a los compuestos antisépticos de amonio cuaternario. Resistencia antimicrobiana J Glob. (2019) 17:94–7. doi: 10.1016/j.jgar.2018.11.022
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
50. González-Martín M, Corbera JA, Suárez-Bonnet A, Tejedor-Junco MT. Factores de virulencia en estafilococos coagulas positivos de interés veterinario que no sean Staphylococcus aureus. Vet Quart. (2020) 40:118-31. doi: 10.1080/01652176.2020.1748253
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
51. Delekta PC, Shook JC, Lydic TA, Mulks MH, Hammer ND. Staphylococcus aureus utiliza partículas de lipoproteínas derivadas del huésped como fuentes de ácidos grasos. J Bacteriol. (2018) 200:e00728-17. doi: 10.1128/JB.00728-17
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
52. Ibberson CB, Jones CL, Singh S, Wise MC, Hart ME, Zurawski D, et al. La hialuronidasa Staphylococcus aureus es un factor de virulencia regulado por CodY. Infectar Inmune. (2014) 82:4253-64. doi: 10.1128/IAI.01710-14
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
53. Garciarena CD, McHale TM, Watkin RL, Kerrigan SW. La diafonía molecular coordinada entre el estafilococo aureus, las células endoteliales y las plaquetas en la infección del torrente sanguíneo. Patógenos. (2015) 4:869-82. doi: 10.3390/pathogens4040869
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
54. Nishiyama T, Nakagawa K, Imabayashi T, Iwatani S, Yamamoto N, Tsushima N. El bacilo probiótico subtilis c-3102 mejora la calidad de la cáscara de huevo después de la muda forzada en gallinas ponedoras envejecidas. J. Poult. Ciencia. (2021) 58:230–7. doi: 10.2141/jpsa.0200081
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
55. Susanti D, Volland A, Tawari N, Baxter N, Gangaiah D, Plata G, et al. Caracterización multiómica de los probióticos de Bacillus spp. derivados del huésped para mejorar el rendimiento de crecimiento en las aves de corral. Microbiol frontal. (2021) 12:747845. doi: 10.3389/fmicb.2021.747845
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
56. Grant A, Gay CG, Lillehoj HS. Bacillus spp. como alternativas de antibióticos microbianos alimentados directamente para mejorar el crecimiento, la inmunidad y la salud intestinal en las aves de corral. Pato Aviar. (2018) 47:339-51. doi: 10.1080/03079457.2018.1464117
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
57. Ouoba LII, Diawara B, Jespersen L, Jakobsen M. Actividad antimicrobiana de Bacillus subtilis y Baccillus pumilus durante la fermentación de la langosta africana (Parkia biglobosa) para la producción de Soumbala. J Appl Microbiol. (2007) 102:963–70. doi: 10.1111/j.1365-2672.2006.03156.x
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
58. Tran C, Cock IE, Chen X, Feng Y. Bacilto antimicrobiano: metabolitos y su modo de acción. Antibióticos. (2022) 11:88. doi: 10.3390/antibiotics11010088
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
59. Aunpad RN-BK. Pumilicina 4, una nueva bacteriocina con actividad anti-MRSA y anti-VRE producida por la cepa WAPB4 de bacterias bacillus pumilus recientemente aisladas. Curr Microbiol. (2007) 55:308–13. doi: 10.1007/s00284-006-0632-2
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
60. Hashimoto M, Taguchi T, Nishida S, Ueno K, Koizumi K, Aburada M, et al. Aislamiento de derivados de éster de 8-fosfatos de amicoumacinas: relación estructura-actividad de la porción de hidroxi aminoácidos. J Antibiot. (2007) 60:752–6. doi: 10.1038/ja.2007.99
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
61. Abd El-Hack ME, El-Saadony MT, Shafi ME, Qattan SYA, Batiha GE, Khafaga AF, et al. Probióticos en la alimentación avícola: una revisión exhaustiva. J Anim Physiol Anim Nutr.(2020) 104:1835–50. doi: 10.1111/jpn.13454
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
62. Mazanko MS, Gorlov IF, Prazdnova EV, Makarenko MS, Usatov AV, Bren AB, et al. Las suplementos probióticas de Bacillus mejoran el rendimiento de la puesta, la calidad de los óvulos, la eclosión de gallinas ponedoras y la calidad del esperma de los gallos. Probióticos Proteicas Antimicrobianas. (2018) 10:367-73. doi: 10.1007/s12602-017-9369-4
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Palabras clave: gallinas ponedoras comerciales, microbiota intestinal, microbiota cutánea, FUDS, desarrollo de DFM
Cita: Ayala DI, Grum DS, Evans NP, Russo KN, Kimminau EA, Trible BR, Lahoti MM, Novak CL y Karnezos TP (2023) Identificación y caracterización de los agentes causantes de la dermatitis ulcerosa focal en gallinas ponedoras comerciales. Frente. Veterinario. Ciencia. 10:1110573. doi: 10.3389/fvets.2023.1110573
Recibido: 29 de noviembre de 2022; Aceptado: 13 de enero de 2023;
Publicado: 08 de febrero de 2023.
Editado por:
Bruno Solis-Cruz, Facultad de Estudios Superiores Cuautitlán, Universidad Nacional Autónoma de México, México
Revisado por:
Roberto Senas Cuesta, Universidad de Arkansas, Estados Unidos
Muhammad Zahoor, Universidad de Agricultura, Dera Ismail Khan, Pakistán
Copyright © 2023 Ayala, Grum, Evans, Russo, Kimminau, Trible, Lahoti, Novak y Karnezos.
*Correspondencia: Diana I. Ayala,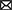 dayala@landolakes.com
dayala@landolakes.com
Descargo de responsabilidad: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía


