Identificación y discriminación precisa de Salmonella enterica serovar Gallinarum biovars Gallinarum y Pullorum mediante una PCR multiplex

Identificación y discriminación precisa de Salmonella enterica serovar Gallinarum biovars Gallinarum y Pullorum mediante una PCR multiplex basada en los nuevos genes de torT y I137_14430
 Li Song1,2,3,
Li Song1,2,3,  Ruimeng Tan1,2,3,
Ruimeng Tan1,2,3,  Dan Xiong1,2,3,
Dan Xiong1,2,3,  Xinan Jiao1,2,3* y
Xinan Jiao1,2,3* y  Sartén Zhiming1,2,3*
Sartén Zhiming1,2,3*- 1Laboratorio clave de Zoonosis de Jiangsu, Universidad de Yangzhou, Yangzhou, China
- 2Jiangsu Co-innovation Center for Prevention and Control of Important Animal Infectious Diseases and Zoonosis, Yangzhou University, Yangzhou, China
- 3Laboratorio clave de prevención y control de factores de peligro biológicos (origen animal) para la inocuidad y calidad agroalimentaria, Ministerio de Agricultura y Asuntos Rurales, Universidad de Yangzhou, Yangzhou, China
La mayoría de los casos de salmonelosis de pollo son causados por Salmonella enterica serovar Gallinarum biovars Gallinarum y Pullorum, que conducen a una tasa significativa de morbilidad y mortalidad. Aunque el esquema convencional de Kaufmann-White es el método confiable para el serotipado de Salmonella, no distingue entre biotipos estrechamente relacionados como S. Pullorum y S. Gallinarum. En este documento, realizamos un ensayo de PCR múltiple de un solo paso que puede identificar y distinguir entre S. Pullorum y S. Gallinarum de manera precisa. Este método de PCR se basó en tres genes, incluyendo torT para S. Identificación de Pullorum, I137_14430 para S. Identificación de Gallinarum, y stn como el gen de referencia a nivel de género para Salmonella. Comparando S. Pullorum a S. Gallinarum y otros serovares de Salmonella, en estudio in silico reveló que sólo el primero tiene una deleción de 126 pb-región en el extremo carboxilo de torT. El gen I137_14430 no existe en S. Gallinarum. Sin embargo, está presente en todos los demás serotipos de Salmonella. El enfoque de PCR múltiple utiliza conjuntos únicos de cebadores que están destinados a dirigirse específicamente a estos tres genes diferentes. El método de PCR establecido fue capaz de distinguir entre los biovars Pullorum y Gallinarum de los 29 serotipos distintos de Salmonella, así como los 50 patógenos distintos que no son Salmonella, mostrando una excelente especificidad y exclusividad. La cantidad mínima de células bacterianas requeridas para la detección por PCR fue de 100 UFC, mientras que el nivel más bajo de ADN genómico requerido fue de 27,5 pg/μL para ambas S. Pullorum y S. Gallinarum. Después de ser implementada en los aislados clínicos de Salmonella recolectados de una granja avícola, la prueba PCR fue capaz de distinguir los dos biovares Pullorum y Gallinarum de las otras cepas de Salmonella. Los hallazgos del ensayo de PCR estuvieron en línea con los de los métodos tradicionales de serotipado e identificación bioquímica. Esta nueva PCR multiplex podría utilizarse como una herramienta novedosa para reforzar el diagnóstico clínico y la diferenciación de S. Pullorum y S. Gallinarum, particularmente en situaciones de detección de alto rendimiento, brinda la oportunidad de una detección temprana de infecciones y, como resultado, un manejo más efectivo de la enfermedad entre las parvadas.
1. Introducción
La Salmonella enterica es uno de los patógenos transmitidos por los alimentos más importantes. Basado en el método de White-Kauffmann-Le Minor, se han identificado más de 2.650 serotipos distintos de Salmonella por sus combinaciones distintivas de antígenos somáticos (O) y flagelares (H) (1, 2). La infección de Salmonella enterica, como S. Enteritidis, S. Typhimurium, S. Infantis y S. Kentucky, en aves de corral y productos avícolas está en aumento (3, 4). Las aves son el huésped específico de Salmonella enterica serovar Gallinarum biovars Pullorum y Gallinarum. S. Pullorum y S. gallinarum se consideran actualmente biovares de serovar Gallinarum dentro del serogrupo D, que causan la enfermedad de pullorum (EP) y la fiebre tifoidea aviar (FT), respectivamente (5). S. pullorum puede transmitirse verticalmente a las crías recién nacidas y horizontalmente a otras aves y causar una grave carga económica para la industria avícola (6).
Los pollitos de < 3 semanas son más susceptibles a contraer la enfermedad de Pullorum, que es una enfermedad sistémica. La diarrea viscosa blanca y la septicemia aguda son características de la enfermedad de Pullorum, que se asocia con una alta tasa de morbilidad y mortalidad (alcanzando el 100% entre los pollitos jóvenes) (7). Los síntomas de infecciones en aves adultas incluyen desnutrición, reducción de la producción de huevos, diarrea, deformidades del sistema reproductivo y más (8, 9). Los animales que sobreviven pueden convertirse en portadores, pueden no cumplir con los requisitos esperados de producción animal y podrían producir huevos contaminados (10). En aves de corral, S. Gallinarum con mayor frecuencia resulta en fiebre tifoidea aviar, que puede manifestarse como septicemia crónica o aguda en aves jóvenes y adultas (11). Las enfermedades siguen siendo una carga económica significativa para el negocio avícola en varias naciones de América del Sur, América Central, Asia y África (12).
Los subtipos de Salmonella por debajo de la clasificación de subespecies se han determinado tradicionalmente utilizando el serotipado, que puede ayudar a identificar los orígenes de los aislados de Salmonella, determinar la gravedad de la enfermedad y evaluar si son o no resistentes a los antibióticos (13). Por lo tanto, la determinación de los serotipos de Salmonella sigue siendo una necesidad diagnóstica crucial en la promoción de la salud (14). Sin embargo, el serotipado tradicional de Salmonella es un método laborioso que implica varios antisueros de tipificación diferentes, además de tomar entre 5 y 6 días para terminar el experimento general (15). Por otra parte, los biovars S. Pullorum y S. Gallinarum son antigénicamente similares, por lo que es difícil distinguir entre ellos después del aislamiento de serovar Gallinarum. A pesar de esta similitud, cada uno produce enfermedades con distintas presentaciones clínicas y patrones de transmisión, y por lo tanto, es crucial distinguir entre los dos biovares. Los ensayos bioquímicos se realizaron en un estudio previo para distinguir entre las dos cepas de bacterias mediante la observación de cómo fermentaban ornitina, espironolactona, dulcitol y maltosa (16). Esto se puede lograr bioquímicamente, pero tomará entre 2 y 3 días (17). Por lo tanto, existe una necesidad inmediata de una técnica de diagnóstico que sea rápida y económica para reconocer y distinguir entre S. Gallinarum y S. Pullorum, ya que la identificación temprana del patógeno ayuda a dirigir la prevención y el control de patógenos (18).
La gran especificidad y sensibilidad de la reacción en cadena de la polimerasa (PCR) ha demostrado un potencial considerable en la detección de muchas infecciones debido al avance de los métodos biológicos moleculares. En particular, incluso cepas y variantes de bacterias altamente relacionadas se han identificado y diferenciado con éxito mediante PCR (19). La efectividad de varias pruebas de PCR para distinguir varios serovariedades de Salmonella se ha estudiado en investigaciones separadas, que demostraron la notable sensibilidad y especificidad del ensayo de PCR (20, 21).
En el presente estudio, desarrollamos y validamos un ensayo preciso de PCR multiplex para detectar y diferenciar simultáneamente biovars S. Gallinarum y S. Pullorum. La PCR multiplex se basó en tres genes específicos de torT, I137_14430 y stn. La especificidad y la sensibilidad del ensayo de PCR múltiple se evaluaron para este ensayo. Este método recientemente desarrollado se aplicó para identificar los dos biovares S. Gallinarum y S. Pullorum en muestras clínicas de una granja de pollos.
2. Materiales y métodos
2.1. Cepas bacterianas
Los ensayos se realizaron con cepas puras caracterizadas serológicamente para estudios adicionales. La Tabla Suplementaria S1 enumera los patógenos de Salmonella y no Salmonella utilizados para desarrollar y validar la técnica de PCR múltiple. Un total de 75 aislados de Salmonella, incluidos 29 serovares diferentes y 50 patógenos no relacionados con Salmonella, se obtuvieron en el trabajo de laboratorio de rutina y se utilizaron en el presente estudio. Se utilizaron antisueros de diagnóstico de aglutinación rápida (Tianrun Bio-Pharmaceutical, Ningbo, China) para serotipar todas las cepas identificadas de Salmonella. Los biovars Gallinarum y Pullorum se distinguieron entre sí con la ayuda de la fermentación de dulcitol y la ornitina descarboxilante.
2.2. Crecimiento bacteriano y extracción de ADN genómico
Las existencias congeladas de los aislados se recuperaron en agar Luria-Bertani (LB) (Oxoid, Basingstoke, Reino Unido) o agar de infusión cerebral y corazón (BHI) (Becton, Dickinson and Company, Sparks, MD, Estados Unidos) durante 18 h a 37 °C para la purificación del ADN. Las colonias se inocularon en caldo LB o BHI y se incubaron durante la noche a 37 ° C con agitación continua a 180 rpm.
Siguiendo el protocolo descrito por el fabricante de un kit de extracción de ADN (Tiangen, Beijing, China), se aisló ADN genómico de las bacterias cultivadas. Posteriormente, el ADN se resuspendió con 100 μL de agua estéril destilada doble. Se utilizó un NanoDrop ND-1000 (Thermo Scientific, Wilmington, DE) para analizar el nivel y la pureza del ADN genómico extraído. El ADN extraído se almacenó a -20 ° C en preparación para las pruebas de PCR.
2.3. Análisis in silico y diseño específico de imprimación
Se examinó la especificidad de dos genes candidatos, torT y I137_14430, para establecer un enfoque fácil de usar, rápido y reproducible para identificar y distinguir S. Gallinarum y S. Pullorum. Los genes torT (GenBank acc. no. AM933173.1, región 3797861-3798901) y I137_14430 (GenBank acc. no. CP006575.1, región 3085007-3085765) se identificaron mediante el cribado de la base de datos para la recolección de nucleótidos no redundantes (nr/nt) utilizando la herramienta básica de búsqueda de alineación local (BLAST). Todas las demás configuraciones se dejaron en sus valores predeterminados, excepto el límite superior de cuántas secuencias alineadas se mostrarán, que se estableció en el máximo de 5,000. Alineaciones de secuencia de I137_14430 y torT de S. Gallinarum, S. Pullorum y otros serovares fueron realizados por Clustal W. La búsqueda BLAST se realizó utilizando S. Secuencias de nucleótidos Pullorum torT, I137_14430 y stn contra las secuencias torT o genómicas de la cepa FDAARGOS_549 Citrobacter freundii (acceso GenBank no. NZ_CP033744.1).
Se diseñaron tres juegos de cebadores de torT, I137_14430 y stn. Las posiciones de los cebadores se basaron en la región deficiente de torT, la secuencia única de I137_14430 y la secuencia conservada de stn para el género Salmonella. El primer conjunto de cebadores, troT-F/R, fue diseñado para amplificar un fragmento de 801 pb para permitir la identificación específica de S. Pullorum. El segundo juego de cebadores, I137_14430-F/R, fue diseñado para distinguir S. Gallinarum de otras cepas, mientras que el tercer conjunto de cebadores, stn-F/R, se utilizó como gen de referencia para identificar el género Salmonella (Tabla 1). La especificidad de los cebadores se evaluó mediante el uso de BLAST y los cebadores fueron sintetizados comercialmente por GenScript (Nanjing, China).
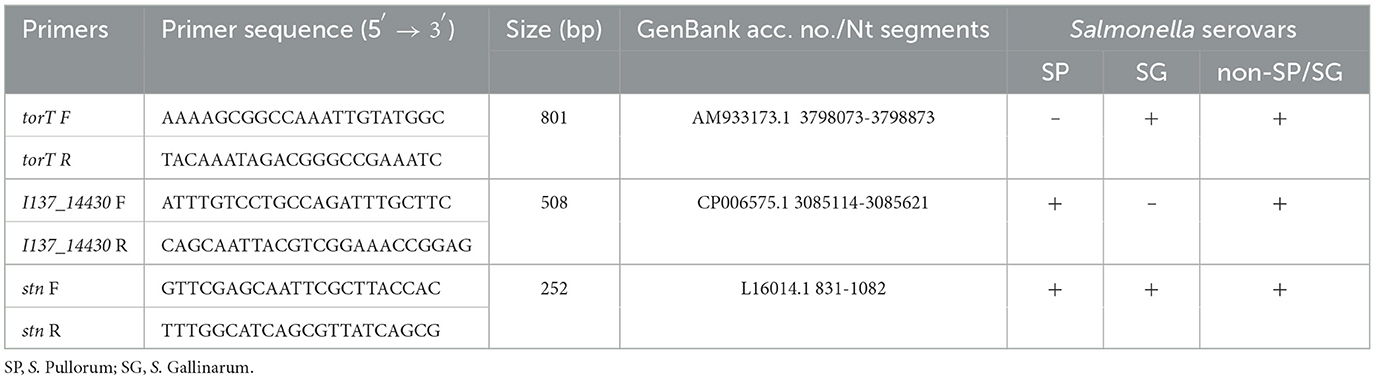 Tabla 1. Secuencias de cebadores para la detección y diferenciación específica de S. Gallinarum y S. Pullorum con el sistema de PCR multiplex.
Tabla 1. Secuencias de cebadores para la detección y diferenciación específica de S. Gallinarum y S. Pullorum con el sistema de PCR multiplex.
2.4. Optimización y desarrollo del ensayo de PCR multiplex
Las pruebas de PCR se realizaron en una solución de reacción de 25 μL compuesta de 12,5 μL de 2 × mezcla Taq Master (Vazyme, Nanjing, China), concentraciones de cebador que incluían 80 nM stn, 40 nM torT y 40 nM I137_14430, ADN genómico de bacterias (100 ng) y agua altamente purificada hasta que la cantidad total alcanzó los 25 μL. La amplificación por PCR se realizó en un termociclador T100 (Bio-Rad, Hercules, CA, EUA) con una desnaturalización inicial de 94 °C durante 3 min, 25 ciclos de 94 °C durante 45 s, 60 °C durante 30 s y 72 °C durante 60 s, seguida de una extensión final a 72 °C durante 10 min. Después de la electroforesis, los productos de PCR se examinaron en un gel de agarosa al 1% antes de la tinción con tinción de gel de ácido nucleico GelRed (Biotium, Fremont, CA, EUA), y la posterior visualización bajo luz UV utilizando un sistema de documentación de gel GelDoc XR (Bio-Rad).
2.5. Especificidad del ensayo de PCR multiplex
Se tomó ADN genómico de 75 cepas de Salmonella que representan 29 serovariedades distintas y 50 patógenos no Salmonella presentados en la Tabla Suplementaria S1, con el fin de probar la especificidad y la compatibilidad de las secuencias de cebadores en la PCR múltiple establecida. Especificidad de la PCR multiplex de un solo paso para la detección y diferenciación de S. Pullorum y S. Gallinarum fue validado en otro laboratorio. Se seleccionaron dos cepas al azar para cada serotipo de Salmonella y patógenos no relacionados con Salmonella.
2.6. Sensibilidad del ensayo de PCR
Para determinar el límite de detección en la prueba PCR, se evaluó su sensibilidad. S. Pullorum cepa S06004 y S. La cepa SG9 de Gallinarum se cultivó durante la noche en el medio LB y el ADN se extrajo con un kit de extracción de ADN genómico bacteriano. El ADN genómico se diluyó consecutivamente de 27,5 ng/μL a 2,75 pg/μL en agua estéril y sirvió como plantilla para la PCR múltiple. Se utilizaron dos lavados con PBS para enjuagar el cultivo bacteriano de S. Gallinarum y S. Pullorum, ajustado para que coincida con los niveles de concentración finales de 2 × 107 a 2 × 103 CFU / ml y ADN de genomas bacterianos se extrajeron hirviendo durante 10 minutos en un baño de agua. Finalmente, 5 μL de cada concentración recuperada a través de la centrifugación se utilizaron para esta prueba de PCR para determinar el número más bajo de S. Pullorum y S. Células de Gallinarum.
2.7. Aplicación de la prueba PCR multiplex en muestras de huevo de gallina
Las cepas clínicas de Salmonella se aislaron de huevos muertos obtenidos de una granja avícola en Jiangsu, China. La salmonela se aisló de muestras siguiendo procedimientos previamente establecidos para el procesamiento, enriquecimiento y aislamiento de muestras (22, 23). Brevemente, todas las muestras se sometieron a un preenriquecimiento en 50 ml de agua de peptona tamponada (Difco, BD, Sparks, MD, Estados Unidos) durante 24 h a 37°C. Después de ser rayado sobre el agar xilosa lisina tergitol 4 (Difco, BD), el cultivo bacteriano se sometió a incubación durante 16 h a 37°C. El enfoque de PCR múltiple establecido se empleó para detectar el ADN de las colonias putativas de Salmonella. Además, cada muestra también se sometió a los procedimientos estándar de cultivo bacteriano, así como a la prueba tradicional de aglutinación sérica.
3. Resultados y discusión
3.1. Evaluación de la alineación de secuencias y desarrollo de cebadores
Los pavos, pollos y algunos otros tipos de aves son susceptibles a dos cepas diferentes de Salmonella, llamadas Gallinarum y Pullorum, que causan la fiebre tifoidea de las aves y la enfermedad de Pullorum (8). Desde S. Pullorum y S. Gallinarum son ambos miembros del mismo serovar, pero pertenecen a biovares separados, las características bioquímicas son la base principal para identificar y distinguir entre ellos. Aunque la identificación bioquímica es el método más común, analizar un gran número de muestras rápidamente puede ser difícil, costoso y lento. En consecuencia, se requerían enfoques basados en el ADN, en particular métodos basados en PCR, para distinguir entre los dos biovares estrechamente relacionados. El cribado molecular con el uso de PCR es el método más eficiente en el tiempo, ya que tiene un alto grado de especificidad y sensibilidad y podría aplicarse para la detección y caracterización rápidas de tipos particulares de infección microbiana patógena (24, 25).
La selección del objetivo es uno de los aspectos más cruciales involucrados en el desarrollo de este tipo de ensayo de detección. Con base en el análisis bioinformático, encontramos que una región de deleción de 126 pb en el carboxilo-terminal de torT se observó solo en S. Pullorum y no en otros serovares de Salmonella incluyendo S. Gallinarum, que puede emplearse para identificar con precisión S. Pullorum (Figura 1A; Figura suplementaria S1). El gen I137_14430, que se encuentra en todos los serovares de Salmonella además de S. Gallinarum, podría utilizarse para identificar este biovar (Figura 1B; Figura suplementaria S2). La similitud y la existencia de los tres objetivos también se examinaron en C. freundii. Los resultados muestran que la longitud de S. El gen Pullorum torT es 923 pb. Sin embargo, la longitud del gen torT de C. freundii es de 1.032 pb. Además, no se encontró similitud significativa en las secuencias torT entre las dos especies (Figura Suplementaria S3A). I137_14430 y stn genes de S. Pullorum se alinearon con el genoma de C. freundii, y no se encontró similitud significativa en el genoma de C. freundii tanto para la secuencia I137_14430 (Figura Suplementaria S3B) como para la secuencia stn (Figura Suplementaria S3C). Además, el gen stn se emplea ampliamente como el control de referencia del género Salmonella (26-28). Por lo tanto, en base a las características de la secuencia, se diseñaron tres conjuntos de cebadores de torT, I137_14430 y stn. Los tres pares de cebadores amplificaron tres productos específicos para torT (801 pb), I137_14430 (508 pb) y stn (252 pb) (Tabla 1).
 Figura 1. Alineación de secuencias de los genes torT y I137_14430 de S. Gallinarum, S. Pullorum y otros serovares. (A) Se observó una región de deleción de 126 pb en el terminal carboxilo de torT solo en S. Pullorum después de la comparación con la de S. Gallinarum y otros serovares de Salmonella, que podrían utilizarse para la identificación específica de S. Pullorum. (B) El gen I137_14430 está presente en todos los serovares de Salmonella excepto en S. Gallinarum, y esta discrepancia podría usarse para la identificación de S. Gallinarum. Los cebadores diseñados se indican con las flechas negras. CE 1, S. Gallinarum str. 9184 (GenBank acc. no. CP019035.1); CE 2, S. Gallinarum str. 07Q015 (GenBank acc. no. CP077760.1); CE 3, S. Gallinarum str. 287/91 (GenBank acc. no. AM933173.1); SP1, S. Pullorum str. S06004 (GenBank acc. no. CP006575.1); SP2, S. Pullorum str. R51 (GenBank acc. no. CP068386.1); SP3, S. Pullorum str. QJ-2D-Sal (GenBank acc. no. CP022963.1); STy, S. Typhimurium str. SCPM-O-B-4515 (GenBank acc. no. CP088136.1); SE, S. Enteritidis str. SE95 (GenBank acc. no. CP050716.1); STh, S. Thompson str. SH11G0791 (GenBank acc. no. CP041171.1); SDe, S. Derby str. FDA161736 (GenBank acc. no. CP075036.1); SN, S. Newport str. SAP18-8729 (GenBank acc. no. CP041208.1); SR, S. Rissen str. GJ0703-2 (GenBank acc. no. CP043509.1); SDu, S. Dublin str. USMARC-69807 (GenBank acc. no. CP032379.1); SL, S. London str. L1 (GenBank acc. no. CP117698.1).
Figura 1. Alineación de secuencias de los genes torT y I137_14430 de S. Gallinarum, S. Pullorum y otros serovares. (A) Se observó una región de deleción de 126 pb en el terminal carboxilo de torT solo en S. Pullorum después de la comparación con la de S. Gallinarum y otros serovares de Salmonella, que podrían utilizarse para la identificación específica de S. Pullorum. (B) El gen I137_14430 está presente en todos los serovares de Salmonella excepto en S. Gallinarum, y esta discrepancia podría usarse para la identificación de S. Gallinarum. Los cebadores diseñados se indican con las flechas negras. CE 1, S. Gallinarum str. 9184 (GenBank acc. no. CP019035.1); CE 2, S. Gallinarum str. 07Q015 (GenBank acc. no. CP077760.1); CE 3, S. Gallinarum str. 287/91 (GenBank acc. no. AM933173.1); SP1, S. Pullorum str. S06004 (GenBank acc. no. CP006575.1); SP2, S. Pullorum str. R51 (GenBank acc. no. CP068386.1); SP3, S. Pullorum str. QJ-2D-Sal (GenBank acc. no. CP022963.1); STy, S. Typhimurium str. SCPM-O-B-4515 (GenBank acc. no. CP088136.1); SE, S. Enteritidis str. SE95 (GenBank acc. no. CP050716.1); STh, S. Thompson str. SH11G0791 (GenBank acc. no. CP041171.1); SDe, S. Derby str. FDA161736 (GenBank acc. no. CP075036.1); SN, S. Newport str. SAP18-8729 (GenBank acc. no. CP041208.1); SR, S. Rissen str. GJ0703-2 (GenBank acc. no. CP043509.1); SDu, S. Dublin str. USMARC-69807 (GenBank acc. no. CP032379.1); SL, S. London str. L1 (GenBank acc. no. CP117698.1).
También se han publicado nuevas pruebas basadas en genes para identificar Salmonella. Por ejemplo, al examinar los datos de secuenciación del genoma completo de S. Los serotipos Pullorum, Xu et al. (29) diseñaron efectivamente la prueba PCR para el reconocimiento de S. Pullorum se basa en el nuevo gen ipaJ de S. Pullorum. Sin embargo, no pudo diferenciar los dos biovares de S. Gallinarum y S. Pullorum. En particular, nuestra investigación identificó y diferenció con éxito S. Pullorum y S. Gallinarum por primera vez dirigiéndose a los nuevos genes de torT y I137_14430.
3.2. Especificidad de la prueba de PCR multiplex para distinguir S. Gallinarum y S. Biovares de Pullorum
Es necesario identificar y serotipar la salmonela para ofrecer detalles adicionales que puedan utilizarse para determinar las fuentes de infección durante las investigaciones de brotes y para la identificación de cepas (30). Sin embargo, los hallazgos de la mayoría de las técnicas de genotipado, como el análisis del perfil plásmido, la electroforesis en gel de campo pulsado (PFGE), el ribotipado y el polimorfismo amplificado de longitud de fragmento (AFLP), no muestran una correlación entre el genotipo y el serotipo del serogrupo Salmonella (31).
Para evaluar la especificidad de los tres conjuntos de cebadores, primero se optimizó la PCR multiplex y luego se probó con plantillas de ADN preparadas a partir de los 50 patógenos no relacionados con Salmonella y 75 patógenos de Salmonella que figuran en la Tabla Suplementaria S1. Los resultados mostraron que solo dos productos específicos de 508 pb I137_14430 y 252 pb stn fueron amplificados para S. Pullorum. Sólo se generaron dos productos de 801-bp torT y 252-bp stn para S. Gallinarum. Los tres productos de torT, I137_14430 y stn se amplificaron para otros serovares de Salmonella. Sin embargo, no se generó una sola banda para ninguno de los patógenos que no fueran Salmonella (Figura 2). Las tres dianas específicas no pudieron ser amplificadas en cuatro cepas de C. freundii conservadas en nuestro laboratorio utilizando los cebadores diseñados en este estudio (Figura 3), lo que fue consistente con los del análisis bioinformático. Se realizó la validación cruzada de las pruebas entre laboratorios para verificar la precisión del ensayo de PCR múltiple. Se seleccionaron dos cepas al azar para cada serotipo de Salmonella y patógenos no Salmonella (incluyendo Citrobacter spp.). Los resultados fueron consistentes con los de nuestro laboratorio, y este método de PCR múltiple pudo identificar y distinguir con precisión S. Gallinarum y S. Pullorum (Figura suplementaria S4), mostrando que este método tiene buena reproducibilidad. Además, no se crearon falsos positivos o negativos en el método de PCR establecido utilizando los tres pares de cebadores específicos. Mientras tanto, la PCR múltiplex no indicó ninguna reacción cruzada con una especificidad del 100%, en línea con los resultados de BLAST. Esta prueba PCR tuvo una excelente especificidad y pudo identificar y diferenciar efectivamente entre S. Pullorum y S. Gallinarum.
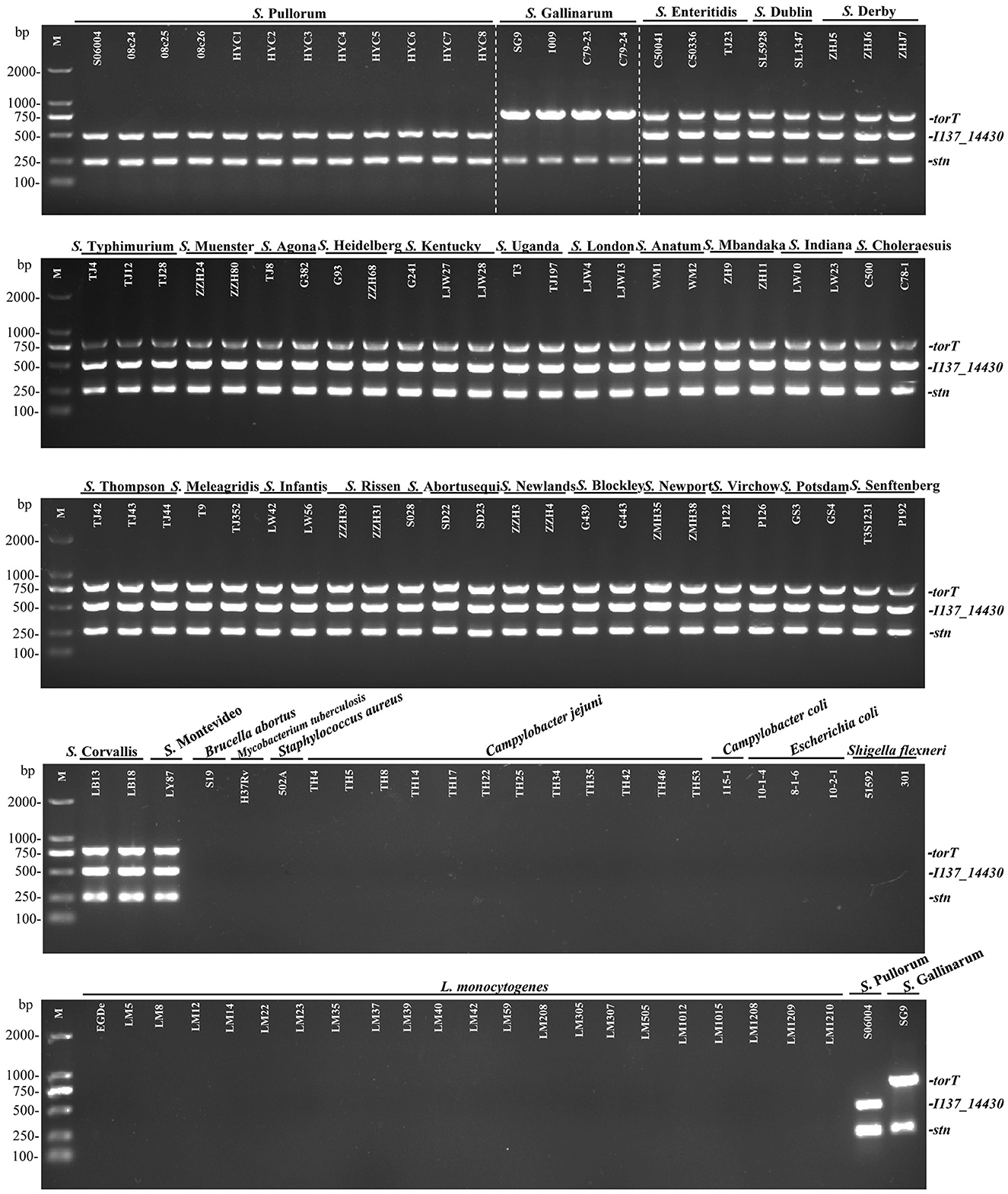 Figura 2. Especificidad de la PCR multiplex de un solo paso para la detección y diferenciación de S. Pullorum y S. Gallinarum. Se tomó ADN genómico de 75 cepas de Salmonella que representan 29 serovares distintos y 43 patógenos no Salmonella presentados en la Tabla Suplementaria S1, con el fin de probar la especificidad y la compatibilidad de los nuevos conjuntos de cebadores de I137_14430, torT y stn. La PCR amplifica solo dos productos específicos de 508 pb I137_14430 y 252 pb stn para S. Pullorum. Sólo se generaron dos productos de 801-bp torT y 252-bp stn para S. Gallinarum. Los tres productos de torT, I137-14430 y stn se amplificaron para otros serovares de Salmonella. No obstante, no se generó una sola banda para ningún otro patógeno que no fuera de Salmonella. Lane M: DL2000 DNA marker (Takara Biotechnology Co., Dalian, China).
Figura 2. Especificidad de la PCR multiplex de un solo paso para la detección y diferenciación de S. Pullorum y S. Gallinarum. Se tomó ADN genómico de 75 cepas de Salmonella que representan 29 serovares distintos y 43 patógenos no Salmonella presentados en la Tabla Suplementaria S1, con el fin de probar la especificidad y la compatibilidad de los nuevos conjuntos de cebadores de I137_14430, torT y stn. La PCR amplifica solo dos productos específicos de 508 pb I137_14430 y 252 pb stn para S. Pullorum. Sólo se generaron dos productos de 801-bp torT y 252-bp stn para S. Gallinarum. Los tres productos de torT, I137-14430 y stn se amplificaron para otros serovares de Salmonella. No obstante, no se generó una sola banda para ningún otro patógeno que no fuera de Salmonella. Lane M: DL2000 DNA marker (Takara Biotechnology Co., Dalian, China).
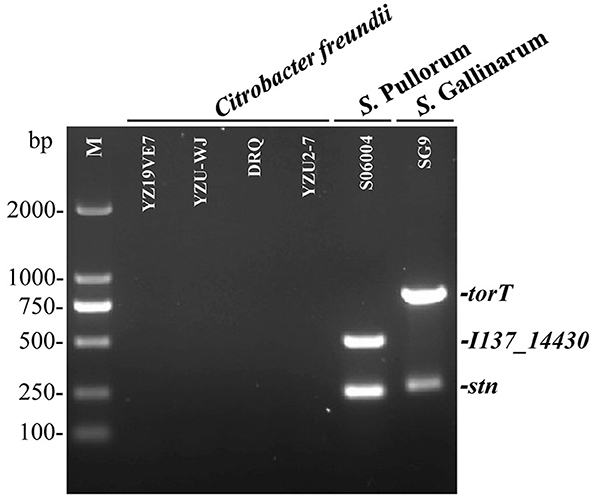 Figura 3. Determinación de la precisión de la PCR multiplex de un solo paso para la identificación de S. Pullorum y S. Gallinarum. Se tomó ADN genómico de cuatro cepas de C. freundii conservadas en nuestro laboratorio presentadas en la Tabla Suplementaria S1, con el fin de probar la especificidad de los conjuntos de cebadores diseñados de I137_14430, torT y stn. S. Pullorum cepa S06004 y S. Se utilizó la cepa SG9 de Gallinarum como controles positivos. La PCR amplifica solo dos productos específicos de 508 pb I137_14430 y 252 pb stn para S. Pullorum. Sólo se generaron dos productos de 801-bp torT y 252-bp stn para S. Gallinarum. Sin embargo, los tres objetivos específicos no pudieron amplificarse en cuatro cepas de C. freundii utilizando los cebadores diseñados.
Figura 3. Determinación de la precisión de la PCR multiplex de un solo paso para la identificación de S. Pullorum y S. Gallinarum. Se tomó ADN genómico de cuatro cepas de C. freundii conservadas en nuestro laboratorio presentadas en la Tabla Suplementaria S1, con el fin de probar la especificidad de los conjuntos de cebadores diseñados de I137_14430, torT y stn. S. Pullorum cepa S06004 y S. Se utilizó la cepa SG9 de Gallinarum como controles positivos. La PCR amplifica solo dos productos específicos de 508 pb I137_14430 y 252 pb stn para S. Pullorum. Sólo se generaron dos productos de 801-bp torT y 252-bp stn para S. Gallinarum. Sin embargo, los tres objetivos específicos no pudieron amplificarse en cuatro cepas de C. freundii utilizando los cebadores diseñados.
Tradicionalmente, S. Pullorum se distinguió de S. Gallinarum utiliza enfoques como el polimorfismo de longitud de fragmento de restricción de PCR (RFLP) y el polimorfismo conformacional de cadena simple (SSCP) que emplean secciones cambiables de un gen o un polimorfismo de nucleótido único (SNP) (32, 33). Esta investigación, sin embargo, utilizó los genes torT y I137_14430 para decir definitivamente S. Pullorum y S. Gallinarum aparte. Además, como los cebadores del gen torT son específicos para S. Pullorum, y los cebadores de I137_14430 son específicos para S. Gallinarum, sería posible distinguir los dos biovares utilizando los dos objetivos específicos de forma independiente. Es posible un ahorro significativo de tiempo y mano de obra, ya que la PCR múltiple establecida puede amplificar secuencias específicas de ADN y distinguir entre los dos biovares de Salmonella simultáneamente.
3.3. Sensibilidad del ensayo de PCR multiplex para la identificación de los biovares Pullorum y Gallinarum
Se utilizaron dos tipos distintos de plantillas para determinar la sensibilidad de la PCR múltiple. El límite de detección de la técnica de PCR se evaluó diluyendo en serie S. Gallinarum y S. ADN genómico de Pullorum de 27,5 ng/μL a 2,75 pg/μL. Se determinó mediante pruebas de sensibilidad que se necesitaba un mínimo de 27,5 pg/μL de ADN genómico para la identificación de S. Pullorum y S. Gallinarum después de la detección por PCR multiplex (Figura 4A). La sensibilidad de la PCR múltiple desarrollada es mayor que la de la prueba HRM-PCR, que tiene una sensibilidad de 126,2 pg/μL (34).
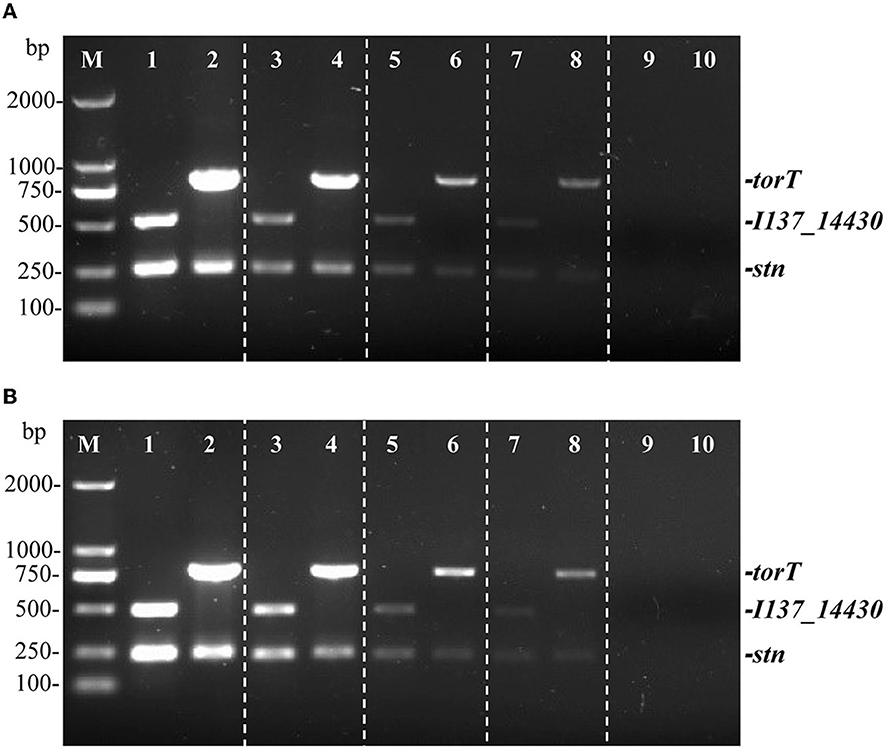 Figura 4. Límite de identificación del método de PCR multiplex para la detección de ADN genómico y células de S. Pullorum (S06004) y S. Gallinarum (CE 9). La PCR multiplex amplifica tres bandas específicas de torT (801 pb), I137_14430 (508 pb) y stn (252 pb). La PCR multiplex para la detección de ADN genómico (A) y células de Salmonella (B), carriles 1, 3, 5, 7, 9 (S. Pullorum) y 2, 4, 6, 8, 10 (S. Gallinarum): las plantillas de ADN genómico en las siguientes concentraciones, respectivamente: 27,5, 2,75 ng/μL, 275, 27,5, 2,75 pg/μL; el número de células por ensayo de PCR a las siguientes concentraciones, respectivamente: 105 UFC, 104 UFC, 103 UFC, 102 UFC y 101 UFC.
Figura 4. Límite de identificación del método de PCR multiplex para la detección de ADN genómico y células de S. Pullorum (S06004) y S. Gallinarum (CE 9). La PCR multiplex amplifica tres bandas específicas de torT (801 pb), I137_14430 (508 pb) y stn (252 pb). La PCR multiplex para la detección de ADN genómico (A) y células de Salmonella (B), carriles 1, 3, 5, 7, 9 (S. Pullorum) y 2, 4, 6, 8, 10 (S. Gallinarum): las plantillas de ADN genómico en las siguientes concentraciones, respectivamente: 27,5, 2,75 ng/μL, 275, 27,5, 2,75 pg/μL; el número de células por ensayo de PCR a las siguientes concentraciones, respectivamente: 105 UFC, 104 UFC, 103 UFC, 102 UFC y 101 UFC.
Además, una dilución en serie diez veces mayor de S. Pullorum y S. Células de Gallinarum que variaron de 105 UFC a 101 Se utilizó UFC para determinar el límite detectable de células bacterianas. Los resultados de tres conjuntos de cebadores confirmaron que 100 UFC era la concentración más baja en la que S. Gallinarum y S. Pullorum pudo ser detectado (Figura 4B). Estos resultados de PCR mostraron un umbral más bajo para el reconocimiento de células bacterianas que los de la PCR basada en el gen sefA (400 UFC) (35), comparable a la PCR multiplex centrada en los genes flhB, lygD y tcpS (100 UFC) (36), y la PCR basada en el gen ipaJ (100 UFC) (29). Estos hallazgos confirmaron que la técnica de PCR tenía un límite superior de detección, permitiendo la identificación de S. Pullorum y S. Células de Gallinarum en cantidades muy bajas. Además, este ensayo de PCR también fue muy rápido, con un procedimiento de PCR de una sola ronda que duró < 2 h.
3.4. Aplicación de la PCR multiplex en la identificación y distinción entre biovars Pullorum y Gallinarum
Tanto la EP (que es causada por S. Pullorum) y FT (que es causada por S. Gallinarum) tienden a causar graves pérdidas económicas de ganado (37). S. Pullorum y S. Gallinarum son dos de las infecciones bacterianas más importantes que afectan al pollo en China (38). La identificación precisa y la posterior eliminación de las aves enfermas son esenciales para la prevención y el éxito en la eliminación de S. Gallinarum y S. Pullorum en aves de corral.
Para evaluar el rendimiento diagnóstico en términos de la precisión de nuestro enfoque de PCR multiplex, se utilizaron muestras clínicas adicionales obtenidas de muestras de huevo de gallina contaminadas naturalmente. Los hallazgos de la PCR demostraron que diez de las veinticuatro muestras solo incluían el objetivo único de 508 pb de I137_14430 y el objetivo de 252 pb de stn, lo que implicaba que los aislados de Ch4, Ch5, Ch9, Ch10, Ch11, Ch12, Ch16, Ch17, Ch18 y Ch24 eran S. Pullorum. Tanto el único torT de 801 pb como el stn de 252 pb fueron generados por dos aislados diferentes, lo que confirma que los dos aislados de Ch22 y Ch23 eran S. Gallinarum. Los resultados del experimento de PCR fueron consistentes con los hallazgos obtenidos utilizando reacciones bioquímicas tradicionales y técnicas de serotipado (Tabla 2).
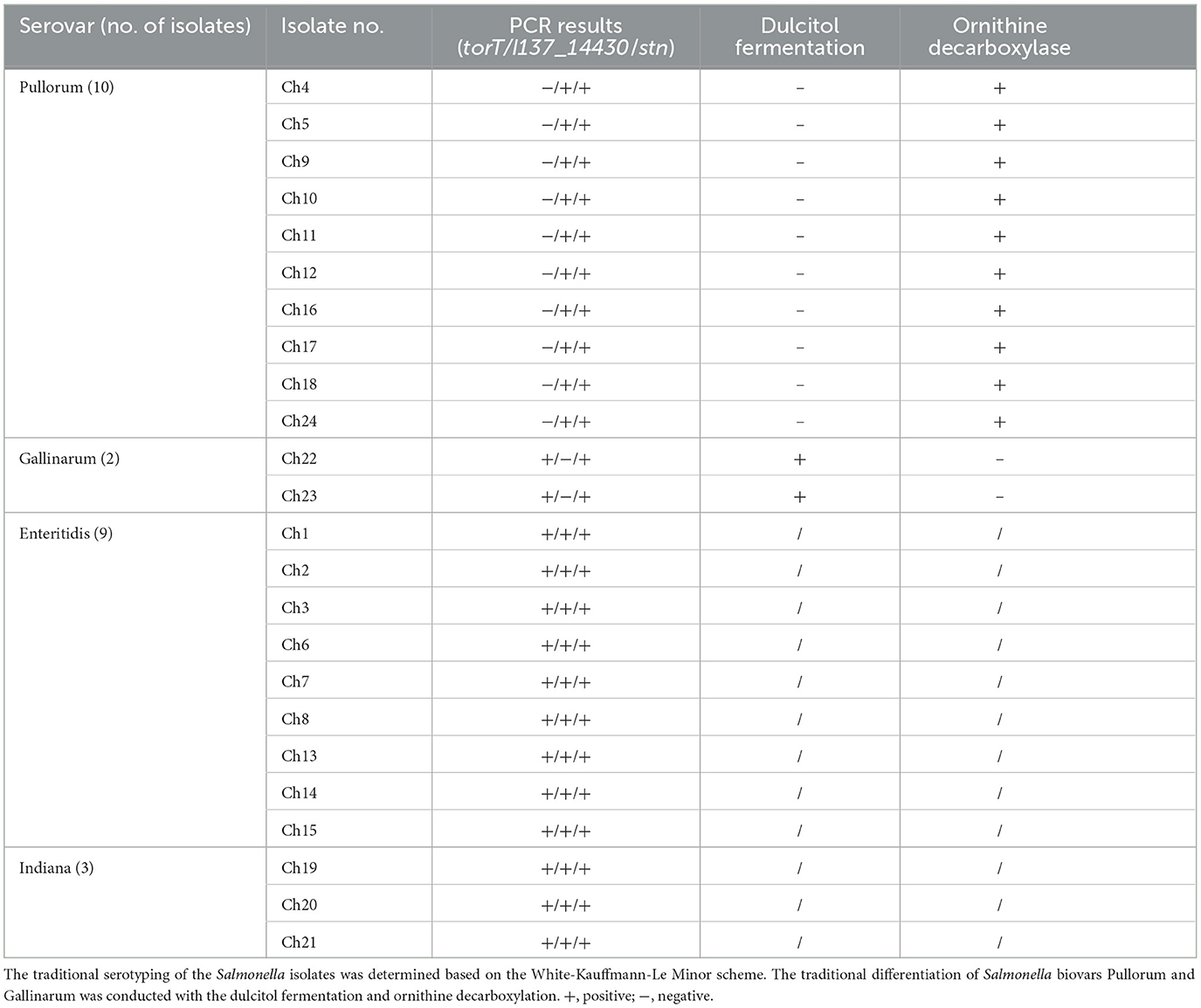 Tabla 2. Se aplicó el método de PCR múltiple desarrollado para la identificación de S. Pullorum y S. Gallinarum se aísla de una granja de pollos.
Tabla 2. Se aplicó el método de PCR múltiple desarrollado para la identificación de S. Pullorum y S. Gallinarum se aísla de una granja de pollos.
Las técnicas microbiológicas clásicas han demostrado ser menos eficientes y menos sensibles que las pruebas basadas en PCR para identificar serovariedades de Salmonella (5). Las técnicas moleculares, como la PCR acompañada de RFLP basada en los genes speC o fliC, se han utilizado para distinguir entre S. Gallinarum y S. Pullorum para identificar estas infecciones (10, 32). Otros métodos, como una PCR dúplex y una PCR multiplex en tiempo real, han sido diseñados para distinguir entre S. Pullorum y S. Gallinarum (2, 39). Sin embargo, estos métodos eran caros y requerían equipo especial. La PCR multiplex establecida en este estudio es un ajuste perfecto para esta necesidad, y ofrece asistencia tecnológica para eliminar el serovar Gallinarum en granjas avícolas comerciales.
Estos hallazgos validaron los genes torT, I137_14430 y stn como candidatos prometedores para S. Gallinarum y S. Identificación y diferenciación de Pullorum. El serotipado de Salmonella utilizando el método serológico tradicional requiere mucho tiempo, requiere mucha mano de obra y es costoso. La tecnología desarrollada en este estudio, que se basa en la PCR múltiple, permite distinguir S. Gallinarum de S. Pullorum de forma rápida y precisa. Como las aves de corral también pueden infectarse con S. Enteritidis, S. Typhimurium, y otros serovares, la PCR multiplex puede ser capaz de diferenciar entre S. Biovares de Gallinarum y otros serovares de Salmonella. Esta prueba podría permitir el diagnóstico temprano de infecciones, lo que permite un manejo más eficiente de la enfermedad en las aves de corral, así como la detección temprana de infecciones.
4. Conclusión
En resumen, este estudio desarrolló por primera vez un nuevo método de PCR múltiple basado en tres genes específicos de torT, I137_14430 y stn. La PCR múltiplex podría detectar y diferenciar simultáneamente la S prevalente. Pullorum y S. Gallinarum, que exhibió una capacidad eficiente de identificación y discriminación en bacterias cultivadas y muestras clínicas de huevos de gallina. La PCR múltiple desarrollada representa un procedimiento económico y de un solo paso para la detección rápida, específica y sensible de ambas S. Pullorum y S. Gallinarum. Puede contribuir a medidas de control más oportunas y eficientes tanto en DP como en FT.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres del repositorio / repositorios y el número (s) de acceso se pueden encontrar en el artículo / material complementario.
Contribuciones del autor
LS, XJ y ZP concibieron el estudio y escribieron el artículo. LS diseñó los experimentos, realizó los ensayos y analizó los resultados. LS, RT y DX realizaron los experimentos y analizaron los resultados. Todos los autores han contribuido al manuscrito, leído y aprobado el manuscrito final.
Financiación
Esta investigación fue financiada por la Fundación Nacional de Ciencias Naturales de China (Números de subvención 32102679, 31972685 y 31920103015), la Fundación de Ciencias Postdoctorales de China (Número de subvención 2020M681741), el Proyecto 111 (D18007) y el Desarrollo del Programa Académico Prioritario de las Instituciones de Educación Superior de Jiangsu (Número de Subvención PAPD).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1220118/full#supplementary-material
Referencias
1. Grimont PAD, Weill FX. Fórmulas antigénicas de los serovares de Salmonella. 9ª ed. París: Centro Colaborador de la Organización Mundial de la Salud para la Referencia e Investigación sobre Salmonella, Instituto Pasteur (2007).
2. Rubio MDS, Penha Filho RAC, Almeida AM, Berchieri Junior A. Desarrollo de una qPCR multiplex en tiempo real para cuantificación y diagnóstico diferencial de Salmonella Gallinarum y Salmonella Pullorum. Pathol aviar. (2017) 46:644–51. doi: 10.1080/03079457.2017.1339866
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Leati M, Zaccherini A, Ruocco L, D’Amato S, Busani L, Villa L, et al. La difícil tarea de seleccionar serovariedades objetivo de Salmonella en aves de corral: el punto de vista italiano. Epidemiol Infect. (2021) 149:e160. doi: 10.1017/S0950268821001230
Resumen de PubMed | Texto completo de CrossRef | Google Académico
4. El Hage R, El Rayess Y, Bonifait L, El Hafi B, Baugé L, Viscogliosi E, et al. Un estudio nacional a través de un enfoque ‘de la granja a la mesa’ para determinar la diseminación de Salmonella junto con la cadena de producción avícola libanesa. Zoonosis Salud Pública. (2022) 69:499–513. doi: 10.1111/zph.12939
Resumen de PubMed | Texto completo de CrossRef | Google Académico
5. Soria MC, Soria MA, Bueno DJ. Comparación de 2 métodos de cultivo y ensayos de PCR para la detección de Salmonella en heces de aves de corral. Poult Sci. (2012) 91:616–26. DOI: 10.3382/PS.2011-01831
Resumen de PubMed | Texto completo de CrossRef | Google Académico
6. Shivaprasad HL, Barrow PA. Enfermedad de Pullorum y fiebre tifoidea aviar. En:Saif YM, , editores. Enfermedad de las aves de corral, 12th Edn. Ames, IA: Iowa State Press (2008). págs. 620–35.
7. Rettger LF. Estudios adicionales sobre la septicemia mortal en pollos jóvenes. o «diarrea blanca». J Med Res. (1909) 21:115–23.
8. Shivaprasad HL. Fiebre tifoidea aviar y enfermedad del pullorum. Rev Sci Tech. (2000) 19:405–24. doi: 10.20506/rst.19.2.1222
Resumen de PubMed | Texto completo de CrossRef | Google Académico
9. Barrow PA, Freitas Neto OC. Pullorum disease and fowl typhoid: new thoughts on old diseases: a review. Pathol aviar. (2011) 40:1–13. doi: 10.1080/03079457.2010.542575
Resumen de PubMed | Texto completo de CrossRef | Google Académico
10. Ribeiro SA, de Paiva JB, Zotesso F, Lemos MV, Berchieri Jánior A. Molecular differentiation between Salmonella enterica subsp enterica serovar pullorum and Salmonella enterica subsp enterica serovar gallinarum. Braz J Microbiol. (2009) 40:184–8. doi: 10.1590/S1517-83822009000100032
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Yin J, Xiong W, Yuan X, Li S, Zhi L, Pan P, et al. Salmonella Pullorum que carece de srfA es atenuada, inmunogénica y protectora en pollos. Microb Pathog. (2021) 161:105230. doi: 10.1016/j.micpath.2021.105230
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Lee HJ, Jeong JY, Jeong OM, Youn SY, Kim JH, Kim DW, et al. Impacto de la infestación de Dermanyssus gallinae en brotes persistentes de fiebre tifoidea aviar en granjas comerciales de pollos ponedores. Poult Sci. (2020) 99:6533–41. doi: 10.1016/j.psj.2020.09.035
Resumen de PubMed | Texto completo de CrossRef | Google Académico
13. Guibourdenche M, Roggentin P, Mikoleit M, Fields PI, Bockemühl J, Grimont PA, et al. Suplemento 2003-2007 (Nº 47) del plan White-Kauffmann-Le minor. Res Microbiol. (2010) 161:26–9. doi: 10.1016/j.resmic.2009.10.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Grossi JL, Yamatogi RS, Call DR, Nero LA. Alta prevalencia de resistencia intermedia a ciprofloxacino en Salmonella enterica aislada de una cadena de producción avícola brasileña, localizada en el estado de Minas Gerais. Int J Food Microbiol. (2023) 394:110180. doi: 10.1016/j.ijfoodmicro.2023.110180
Resumen de PubMed | Texto completo de CrossRef | Google Académico
15. Bell RL, Jarvis KG, Ottesen AR, McFarland MA, Brown EW. Innovaciones recientes y emergentes en la detección de Salmonella: una perspectiva alimentaria y ambiental. Microbio Biotechnol. (2016) 9:279–92. doi: 10.1111/1751-7915.12359
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Crichton PB, Antiguo DC. Salmonelas de los serotipos gallinarum y pullorum agrupadas por biotipado y sondeo de genes fimbriales. J Med Microbiol. (1990) 32:145–52. doi: 10.1099/00222615-32-3-145
Resumen de PubMed | Texto completo de CrossRef | Google Académico
17. Batista DF, de Freitas Neto OC, Lopes PD, de Almeida AM, Barrow PA, Berchieri AJr. El ensayo de reacción en cadena de la polimerasa basado en el gen ratA permite diferenciar entre Salmonella enterica subsp. enterica serovar gallinarum biovars gallinarum y pullorum. J Vet Diagn Invest. (2013) 25:259–62. DOI: 10.1177/1040638713479361
Resumen de PubMed | Texto completo de CrossRef | Google Académico
18. Yan SS, Pendrak ML, Abela-Ridder B, Punderson JW, Pedorko PD, Foley SL. Una visión general de la tipificación de Salmonella: perspectivas de salud pública. Clin Appl Immunol Rev. (2004) 4:189–204. doi: 10.1016/j.cair.2003.11.002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
19. Hoorfar J, Koláčková I, Johannessen GS, Garofolo G, Marotta F, Wieczorek K, et al. Una propuesta multicéntrica para una herramienta rápida para detectar el patógeno transmitido por los alimentos Campylobacter en parvadas de pollos bioseguras. Aplicación Environ Microbiol. (2020) 86:E01051–E01020. doi: 10.1128/AEM.01051-20
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. El-Sayed Ahmed KA, El-Shishtawy M, El-Taweel F, El-Mansoury H. Multiplex PCR for diagnosis of Salmonella enterica serovar Typhi. Laboratorio Clin. (2015) 61:1537–43. doi: 10.7754/Clin.Lab.2015.150115
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Prabagaran SR, Kalaiselvi V, Chandramouleeswaran N, Deepthi KNG, Brahmadathan KN, Mani M. Molecular diagnosis of Salmonella typhi and its virulence in suspect typhoid blood samples through nested multiplex PCR. J Métodos de microbiol. (2017) 139:150–4. doi: 10.1016/j.mimet.2017.05.013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
22. Cai Y, Tao J, Jiao Y, Fei X, Zhou L, Wang Y, et al. Características fenotípicas y correlación genotípica entre aislados de Salmonella de un matadero y mercados minoristas en Yangzhou, China. Int J Food Microbiol. (2016) 222:56–64. doi: 10.1016/j.ijfoodmicro.2016.01.020
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Zhou Z, Li J, Zheng H, Jin X, Shen Y, Lei T, et al. Diversidad de aislados de Salmonella y su distribución en un matadero de cerdos en Huaian, China. Control de alimentos. (2017) 78:238–46. doi: 10.1016/j.foodcont.2017.02.064
24. Zhai L, Yu Q, Bie X, Lu Z, Lv F, Zhang C, et al. Desarrollo de un sistema de prueba PCR para la detección específica de Salmonella paratyphi B en alimentos. FEMS Microbiol Lett. (2014) 355:83–9. doi: 10.1111/1574-6968.12443
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Ogunremi D, Nadin-Davis S, Dupras AA, Márquez IG, Omidi K, Pope L, et al. Evaluación de un ensayo de PCR multiplex para la identificación de Salmonella serovars enteritidis y typhimurium utilizando muestras minoristas y de matadero. J Food Prot. (2017) 80:295–301. doi: 10.4315/0362-028X. JFP-16-167
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Moore MM, Feist MD. Método de PCR en tiempo real para Salmonella spp. dirigido al gen stn. J Appl Microbiol. (2007) 102:516–30. doi: 10.1111/j.1365-2672.2006.03079.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Srisawat M, Panbangred W. Detección eficiente y específica de Salmonella en muestras de alimentos utilizando un método de amplificación isotérmica mediada por bucle basado en stn. Biomed Res Int. (2015) 2015:356401. DOI: 10.1155/2015/356401
Resumen de PubMed | Texto completo de CrossRef | Google Académico
28. Yamasaki E, Matsuzawa S, Takeuchi K, Morimoto Y, Ikeda T, Okumura K, et al. Serotipado rápido de aislados de Salmonella basados en perfiles de secuencia similares a polimorfismos de un solo nucleótido de un gen específico de Salmonella. Foodborne Pathog Dis. (2021) 18:31–40. DOI: 10.1089/FPD.2020.2823
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Xu L, Liu Z, Li Y, Yin C, Hu Y, Xie X, et al. Un método rápido para identificar Salmonella enterica serovar Gallinarum biovar Pullorum utilizando un gen diana específico ipaJ. Pathol aviar. (2018) 47:238–44. doi: 10.1080/03079457.2017.1412084
Resumen de PubMed | Texto completo de CrossRef | Google Académico
30. Gebreyes WA, Altier C, Thakur S. Molecular epidemiology and diversity of Salmonella serovar Typhimurium in pigs using phenotypic and genotypic approaches. Epidemiol Infect. (2006) 134:187–98. doi: 10.1017/S0950268805004723
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Turki Y, Mehri I, Fhoula I, Hassen A, Ouzari H. Comparison of five molecular subtyping methods for differentiation of Salmonella Kentucky isolates in Tunisia. Mundo J Microbiol Biotechnol. (2014) 30:87–98. DOI: 10.1007/S11274-013-1414-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
32. Kwon HJ, Park KY, Yoo HS, Park JY, Park YH, Kim SJ. Diferenciación del biotipo pullorum de Salmonella enterica serotipo gallinarum del biotipo gallinarum del biotipo gallinarum mediante el análisis del gen fase 1 de la flagelina C (fliC). J Métodos de microbiol. (2000) 40:33–8. doi: 10.1016/S0167-7012(99)00129-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Kisiela D, Kuczkowski M, Kiczak L, Wieliczko A, Ugorski M. Diferenciación de Salmonella Gallinarum biovar Gallinarum de Salmonella Gallinarum biovar Pullorum por PCR-RFLP del gen fimH. J Vet Med B Infect Dis Vet Public Health. (2005) 52:214–8. doi: 10.1111/j.1439-0450.2005.00846.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Ren X, Fu Y, Xu C, Feng Z, Li M, Zhang L, et al. Análisis de fusión de alta resolución (HRM) como una nueva herramienta para la identificación rápida de Salmonella enterica serovar gallinarum biovars pullorum y gallinarum. Poult Sci. (2017) 96:1088–93. DOI: 10.3382/ps/pew400
Resumen de PubMed | Texto completo de CrossRef | Google Académico
35. Gong J, Zhuang L, Zhu C, Shi S, Zhang D, Zhang L, et al. Amplificación isotérmica mediada por bucle del gen sefA para la detección rápida de Salmonella enteritidis y Salmonella gallinarum en pollos. Foodborne Pathog Dis. (2016) 13:177–81. DOI: 10.1089/FPD.2015.2082
Resumen de PubMed | Texto completo de CrossRef | Google Académico
36. Xiong D, Song L, Tao J, Zheng H, Zhou Z, Geng S, et al. Un ensayo eficiente basado en PCR multiplex como una herramienta novedosa para la discriminación interserovar precisa de Salmonella Enteritidis, S. pullorum/gallinarum y S dublín. Microbiol frontal. (2017) 8:420. DOI: 10.3389/fmicb.2017.00420
Resumen de PubMed | Texto completo de CrossRef | Google Académico
37. Munir A, Ilyas SZ, Tahir H, Basit A, Haider Z, Rehman SU. Detección temprana basada en PCR y patrón de resistencia a antibióticos de aislados de Salmonella Gallinarum de aves de corral de Pakistán. J Métodos de microbiol. (2023) 2:106709. doi: 10.1016/j.mimet.2023.106709
Resumen de PubMed | Texto completo de CrossRef | Google Académico
38. Guo X, Wang H, Cheng Y, Zhang W, Luo Q, Wen G, et al. Fenotipo de resistencia a quinolonas y caracterización genética de aislados de Salmonella enterica serovar pullorum en China, durante 2011 a 2016. BMC Microbiol. (2018) 18:225. DOI: 10.1186/S12866-018-1368-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Batista DF, de Freitas Neto OC, de Almeida AM, Barrow PA, de Oliveira Barbosa F, Berchieri Junior A. Molecular identification of Salmonella enterica subsp. enterica serovar gallinarum biovars gallinarum and pullorum by a duplex PCR assay. J Vet Diagn Invest. (2016) 28:419–22. DOI: 10.1177/1040638716651466
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: Salmonella Pullorum, Salmonella gallinarum, PCR múltiple, torT, I137_14430, discriminación precisa
Cita: Song L, Tan R, Xiong D, Jiao X y Pan Z (2023) Identificación precisa y discriminación de Salmonella enterica serovar Gallinarum biovars Gallinarum y Pullorum mediante una PCR multiplex basada en los nuevos genes de torT y I137_14430. Frente. Vet. Sci. 10:1220118. doi: 10.3389/fvets.2023.1220118
Recibido: 12 de mayo de 2023; Aprobado: 20 de junio de 2023;
Publicado: 05 julio 2023.
Editado por:
Fabrizio Bertelloni, Universidad de Pisa, Italia
Revisado por:
Rajesh Kumar Vaid, Centro Nacional de Investigación sobre Equinos (ICAR), India
Angelo Berchieri Junior, Universidad Estatal de São Paulo, Brasil
Derechos de autor © 2023 Canción, Tan, Xiong, Jiao y Pan. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Xinan Jiao, jiao@yzu.edu.cn; Zhiming Pan, zmpan@yzu.edu.cn
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

