Inactivación de virus ganaderos y aviares altamente transmisibles
Inactivación de virus ganaderos y aviares altamente transmisibles, incluidos la gripe A y el virus de la enfermedad de Newcastle, para el diagnóstico molecular
 Jennifer L. Welch
Jennifer L. Welch  Ram Shrestha
Ram Shrestha  Heather Hutchings
Heather Hutchings  Narinder Pal
Narinder Pal  Randall Levings
Randall Levings  Suelee Robbe-Austerman
Suelee Robbe-Austerman  Rachel Palinski
Rachel Palinski  Karthik K. Shanmuganatham*
Karthik K. Shanmuganatham*- Laboratorios de Servicios Veterinarios Nacionales, Servicios Veterinarios, Servicio de Inspección de Sanidad Animal y Vegetal, Departamento de Agricultura de los Estados Unidos, Ames, IA, Estados Unidos
Existe una necesidad crítica de un método de inactivación que inactive completamente los patógenos en el momento de la recolección de la muestra, manteniendo al mismo tiempo la calidad de ácido nucleico requerida para las pruebas de PCR de diagnóstico. Este método de inactivación es necesario para aliviar las preocupaciones sobre el potencial de transmisión, minimizar las complicaciones y el costo del envío, y permitir la realización de pruebas en laboratorios de menor contención, mejorando así el diagnóstico de enfermedades a través de un mejor tiempo de respuesta. Este estudio evaluó un panel de 10 virus sustitutos que representan enfermedades animales altamente patógenas. Estos resultados mostraron que un medio de transporte molecular (PSMTM) comercial de PrimeStore® inactivó completamente todos los virus probados en un >99,99%, según lo determinado por ensayos de infectividad y paso en serie. Sin embargo, la detección de ácido nucleico viral mediante qRT-PCR fue comparable en condiciones de PSMTM y controladas. Estos resultados fueron consistentes cuando los virus se evaluaron en presencia de material biológico como sueros e hisopos cloacales para imitar las condiciones de diagnóstico de las muestras de virus aviares y no aviares, respectivamente. Los resultados de este estudio pueden ser utilizados por los laboratorios de pruebas diagnósticas para agentes altamente patógenos que afectan a poblaciones animales y humanas. Estos resultados se pueden utilizar para revisar las directrices para las pruebas diagnósticas de determinados agentes y el envío de sustancias infecciosas.
Introducción
Se están llevando a cabo esfuerzos de prevención para proteger a los animales en los Estados Unidos de enfermedades altamente contagiosas causadas por el virus de la peste porcina clásica (PPC), el virus de la peste porcina africana (PPA), el virus de la fiebre aftosa (FMDV), el virus de la encefalitis equina oriental (EEEV), el virus de la enfermedad de Newcastle (NDV) y el virus de la influenza aviar A altamente patógena (IAAP), entre otros (1–3). Cuando no se dispone de una vacuna o un tratamiento aprobados, la despoblación se convierte en el curso de acción obligatorio para las explotaciones afectadas (4). Esta cuestión subraya la importancia crítica de los esfuerzos de prevención, ya que la despoblación perturba el suministro de alimentos y tiene importantes implicaciones financieras para los productores y los consumidores (1). Por ejemplo, entre 2018 y 2019 se notificó en China el mayor brote de PPA hasta la fecha, lo que supuso unas pérdidas financieras totales estimadas en más de 111.200 millones de dólares (5). El brote actual de gripe aviar altamente patógena, identificado por primera vez en los Estados Unidos en 2022, ha provocado pérdidas económicas actuales en los EE. UU. estimadas en un rango de USD 2.500 a 3 mil millones de dólares (6). Además, algunos de estos virus, como el FMDV, EEEV, NDV y la IAAP, son zoonóticos, lo que los convierte en una preocupación para la salud pública (7-10). Los esfuerzos de prevención incluyen pruebas de vigilancia activa (11).
Las pruebas de vigilancia activa requieren el envío de muestras sospechosas de enfermedades altamente contagiosas a las instalaciones de alta contención BSL3+ para realizar pruebas de confirmación por PCR a fin de cumplir con los requisitos de agentes seleccionados (12, 13). El envío de sustancias potencialmente infecciosas es costoso y urgente, y requiere contenedores de transporte especializados y una cadena de frío para evitar el deterioro de las muestras para las pruebas de PCR (14). Se necesita urgentemente un método de inactivación que proteja el contenido de ácidos nucleicos esencial para las pruebas de PCR, al tiempo que elimina la amenaza de transmisión y minimiza el requisito de temperatura, para minimizar los costos de envío y reducir la restricción de las pruebas de confirmación a instalaciones de menor nivel de bioseguridad (BSL2). La implementación de un método de inactivación aliviará la presión que la alta demanda de pruebas de PCR está ejerciendo sobre las limitadas instalaciones BSL3 en los Estados Unidos, disminuyendo así el tiempo de respuesta de las pruebas y mejorando las dificultades prácticas. Esto incluye mejorar las complicaciones de compartir reactivos entre laboratorios internacionales (15). El envío accidental de ántrax vivo a laboratorios dentro de los Estados Unidos debido a una inactivación incompleta resalta la importancia de un método de inactivación que proporcione un amortiguador de seguridad para su uso con patógenos de altas consecuencias (16).
Los tubos de medios de transporte molecular (PSMTM) de PrimeStore® contienen reactivos patentados que inactivan tanto los virus como las bacterias al alterar las membranas lipídicas e inhibir la maquinaria de replicación al tiempo que estabilizan los ácidos nucleicos (PrimeStore, Longhorn Vaccines and Diagnostics) (17, 18). Como se indica en la ficha de datos de seguridad disponible públicamente, este reactivo contiene una mezcla de tiocianato de guanidina, etanol y n-lauroilsarcosina (19). Anteriormente se ha demostrado que los reactivos a base de tiocianato de guanidina inactivan eficazmente el poliovirus y el virus de la fiebre aftosa, aunque la comparación de los resultados debe interpretarse con precaución, ya que la formulación exacta de la PSMTM es patentada (20, 21). Estudios previos han demostrado que el etanol y la n-lauroilsarcosina inactivan varios virus con envoltura y algunos sin envoltura al interactuar con lípidos y desnaturalizar proteínas (22).®
El PSMTM está autorizado por la Administración de Alimentos y Medicamentos (FDA, por sus siglas en inglés) como un dispositivo de Clase II para la recolección de muestras sospechosas de contener el virus de la influenza A (IAV) y Mycobacterium tuberculosis (17). Otros estudios han demostrado que la PSMTM inactiva eficazmente el SARS-CoV-2, el adenovirus tipo 5, la influenza A H3N2 y la HPAI H5N1 a temperatura ambiente (17, 23). Los estudios de influenza A H1N1 mostraron que el ARN viral se conservó en PSMTM durante 30 días a 25 °C, y los valores del umbral del ciclo (CT) se redujeron mínimamente de los días 0 a 30 (23). Además, los tubos PSMTM se utilizaron durante la pandemia de COVID-19 para las pruebas clínicas del SARS-CoV-2 (17, 24). Los estudios demostraron que un tiempo de contacto de 10 minutos era suficiente para inactivar completamente el SARS-CoV-2 y no se detectó ningún virus en los ensayos de temblor o después del paso en serie en células susceptibles (24). En conjunto, estos datos respaldan que la PSMTM inactiva los patógenos virales y conserva los ácidos nucleicos a temperatura ambiente, lo que permite los procedimientos normales de envío sin interrumpir los flujos de trabajo normales de las pruebas de PCR.
En este estudio, evaluamos la efectividad de la PSMTM en la inactivación de virus sustitutos que representan virus de agentes seleccionados por los Servicios Veterinarios (VS) del USDA, algunos de los cuales se consideran agentes de enfermedades animales extrañas, incluidos el virus de la peste porcina africana, el virus de la peste porcina clásica y el virus de la fiebre aftosa (Tabla 1) (3). En este estudio se utilizaron virus sustitutos, ya que las pruebas de inactivación se completaron en condiciones BSL2. El virus de la PPA es el único miembro de la familia Asfarviridae, lo que hace que la selección sustituta sea un desafío. Para abordar esto, utilizamos el virus de la viruela porcina relevante para la especie y el virus vaccinia genéticamente similar (32-34). Tanto la viruela porcina como el virus vaccinia son miembros de la familia Poxvirus y, al igual que Asfarviridae, son virus nucleocitoplasmáticos de ADN grande (NCLDV) que comparten un ancestro común (35). Otras personas identificaron previamente selecciones indirectas para otros virus diana e incluyeron el virus de la diarrea viral bovina (BVDV) como sustituto del virus de la peste porcina clásica y el sénecavirus A (SVA) como sustituto del virus de la fiebre aftosa (36, 37). Además, cuando fue posible, se utilizaron cepas menos patógenas de virus diana como sustitutos, como la influenza aviar de baja patogenicidad (IAAP) H7N3, la influenza aviar LPAI H6N1 y la gripe aviar LPAI H5N3 como sustitutos de la IAV, el VEN lentógeno como sustituto del VEN velogénico, el virus de la influenza porcina (VIS) como sustituto del virus IAV y la quimera del virus EEEV/Sindbis como sustituto del EEEV (38). Por lo tanto, las selecciones de virus sustitutos se ajustaron mejor a los virus diana para el VEN, la IAAP y la IAV (misma especie, género y familia), seguidos de EEEV (quimera del virus objetivo y mismo género), LCR (mismo género y familia), FMDV (misma familia) y PPA (ancestro común).
 Tabla 1. Características y especificaciones del virus sustituto utilizado para los estudios de inactivación de PSMTM.
Tabla 1. Características y especificaciones del virus sustituto utilizado para los estudios de inactivación de PSMTM.
Varios otros estudios han evaluado la compatibilidad de los medios de transporte comerciales con las pruebas de ácido nucleico (39). Estos estudios han demostrado que el PSMTM y otros medios de transporte disponibles comercialmente inactivan varios virus aviares, así como el SARS-CoV-2 (14, 17, 23, 39-43). Sin embargo, los resultados de la preservación de ácidos nucleicos no estaban disponibles en todos estos estudios. Existe una escasez de medios de transporte comerciales evaluados minuciosamente que inactiven los virus en múltiples familias mientras protegen el ácido nucleico viral para su uso posterior en pruebas diagnósticas. Los virus sustitutos seleccionados utilizados en este estudio representan virus con diversas clasificaciones en la estructura exterior (envoltura vs. no envoltura), composición de ácidos nucleicos (ADN vs. ARN) y estructura de ácidos nucleicos (cadena simple vs. bicatenaria) (Tabla 1). Se utilizó al menos un virus representativo para cada virus objetivo y se seleccionó en función de la facilidad de crecimiento a títulos altos. Las muestras diagnósticas se recogen de material biológico que puede influir en la eficacia de la inactivación. Estudios previos identificaron que la presencia de sangre puede interferir con la actividad viricida de los desinfectantes (44); Sin embargo, otros estudios han demostrado que los virus siguen siendo sensibles a la inactivación química en presencia de sangre (45). Por lo tanto, debido a la alta patogenicidad y las consecuencias de transmisión de estos virus, es importante validar la PSMTM no solo con virus de diferentes familias, sino también con múltiples tipos de muestras y condiciones de recolección, como el volumen de PSMTM y el tiempo de incubación. En este estudio, se evaluó la detección por PCR y la inactivación de la replicación del virus después del tratamiento con PSMTM. Además, también se evaluó la inactivación de sueros e hisopos cloacales.
Materiales y métodos
Virus
La quimera del virus EEEV/Sindbis, la viruela porcina, el SVA, la LPAI H6N1, la LPAI H7N3, la LPAI H5N3, el SIV H3N2, el NDV lentógeno y el BVDV 1a fueron proporcionados por el Laboratorio de Virología Diagnóstica (DVL) del USDA. El virus vaccinia fue comprado a la American Type Culture Collection (ATCC). Los virus se propagaron y se propagaron mediante la mediana de la dosis infecciosa de cultivo de tejidos (TCID50) en sistemas apropiados de huevos o células (Tabla 1), y todos los huevos y células utilizados para la propagación y la titulación fueron proporcionados por la sección de Pruebas de Aptitud y Reactivos (PTR) del Laboratorio de Diagnóstico Bioanalítico y Reactivos (DBRL) del USDA. El uso de productos de origen animal fue aprobado por el Comité Institucional de Cuidado y Uso de Animales de los Laboratorios de Servicios Veterinarios y el Centro de Productos Biológicos Veterinarios (APH-22-1033). El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales. Los virus se propagaron en medios esenciales mínimos (MEM) con 5% de suero fetal bovino, 4 mM de L-glutamina (Gibco), 0,015 g/L de penicilina, 0,1 g/L de estreptomicina y 1x aminoácidos no esenciales (Gibco). Las titulaciones de virus se realizaron en MEM con suero fetal bovino al 2% y los mismos suplementos indicados para las propagaciones.
Inactivación de la descomposición natural del virus
Se añadieron 200 uL de virus a cada pocillo de una placa de cultivo de tejidos de 24 pocillos por triplicado para cada punto de tiempo. Los virus se incubaron a temperatura ambiente hasta el momento indicado. Para mitigar los efectos de la evaporación, los virus se recolectaron en el punto de tiempo indicado agregando 200 μL de medios de propagación del virus a cada pocillo y realizando pipeteos repetidos. A continuación, los virus recolectados se congelaron una vez a -80 °C hasta la valoración mediante TCID50 en células susceptibles (Tabla 1).
Inactivación del virus PSMTM
Los virus se trataron con medios de transporte molecular PrimeStore® (PSMTM) de acuerdo con las instrucciones del fabricante. Se combinó una parte de virus con tres partes de PSMTM y se incubó durante 1 h a temperatura ambiente. Infectividad del virus (TCID50) se comparó con el control sin inactivación, en el que la solución salina tamponada con fosfato (PBS) sustituyó a la MSTPM y se trató de la misma manera. Para imitar la relevancia de las muestras de diagnóstico animal recogidas a partir de material biológico, los virus se combinaron con suero o hisopos apropiados para la especie. Los virus combinados con suero fueron los siguientes: VIS, vaccinia, viruela porcina, quimera del virus EEEV/Sindbis, SVA y BVDV. Los virus combinados con hisopos fueron los siguientes: LPAI H7N3, LPAI H6N1, LPAI H5N3 y NDV. En los estudios de sueros, el virus se diluyó 1:1 con suero porcino o bovino antes del tratamiento con PSMTM o PBS. Para los estudios con hisopos, cada virus en medios líquidos se agregó a un tubo que contenía un hisopo cloacal recolectado de un pollo no infectado y luego se incubó a temperatura ambiente durante aproximadamente 10 minutos. A continuación, el virus en medios líquidos se recogió con una pipeta y se trató con PSMTM o PBS control, como se ha descrito anteriormente. Todos los procedimientos de control e inactivación se completaron en tres réplicas experimentales. Se mantuvieron tres partes de control de PSMTM o PBS por una parte de proporciones de virus para todos los experimentos, excepto para los experimentos de proporción variable. También se evaluaron las variaciones de la proporción de 1 parte de control de PSMTM o PBS por 1 parte de virus, 1 parte de control de PSMTM o PBS por 3 partes de virus, 1 parte de control de PSMTM o PBS por 10 partes de virus y 1 parte de control de PSMTM o PBS por 100 partes de virus con quimera del virus EEEV/Sindbis para evaluar la inactivación de PSMTM si no se mantiene la proporción recomendada. Todos los TCID50 Los resultados se completaron con diluciones seriadas de 10 veces.
Paso de la serie del virus
Los virus tratados con control PSMTM o PBS se pasaron a células susceptibles (Tabla 1) durante un mínimo de tres pases seriados. La PSMTM es altamente citotóxica para las células (Figura suplementaria S1). Para minimizar la citotoxicidad de la PSMTM, se completó el intercambio de tampones antes del primer paso de los virus tratados con PSMTM y PBS control. El material viral tratado con PSMTM y PBS se diluyó a una proporción de 1:10 en MEM sin suplementos. A continuación, se añadió material diluido a un filtro centrífugo de corte Amicon® de 50 kDa (Millipore) y se centrifugó a 1500 x g hasta que el material diluido se concentró en el volumen original (aproximadamente 10 min). Se eligió este punto de corte de filtro porque todos los virus incluidos en este estudio tenían más de 50 kDa y quedarían retenidos en el depósito del filtro. A continuación, el material concentrado se diluyó en una proporción de 1:10 en MEM y se repitieron los pasos de concentración de 50 kDa. Después de la concentración final al volumen original, los virus tratados con PSMTM y PBS se diluyeron a una proporción de 1:10 en MEM para la propagación del virus, y el volumen total (2,0 ml) se agregó a un pocillo de una placa de 6 pocillos. Las células se incubaron con virus tratados con PSMTM o PBS hasta que se observó un efecto citopático (CPE) en pocillos de virus tratados con control de PBS o durante un máximo de 7 días. Los productos transitados se recolectaron mediante tres ciclos de congelación/descongelación de células y sobrenadante juntos. A continuación, los productos pasados se centrifugaron a 1500 x g durante 10 min para eliminar los restos celulares. Una fracción (10%) del producto clarificado se diluyó en una proporción de 1:10 en MEM para la propagación del virus, y el volumen total (2,0 mL) se agregó a las células vírgenes (un pocillo de una placa de 6 pocillos) para cada paso posterior. La colección de virus inactivados o tratados con PSMTM a partir de células susceptibles se designó como virus de paso.
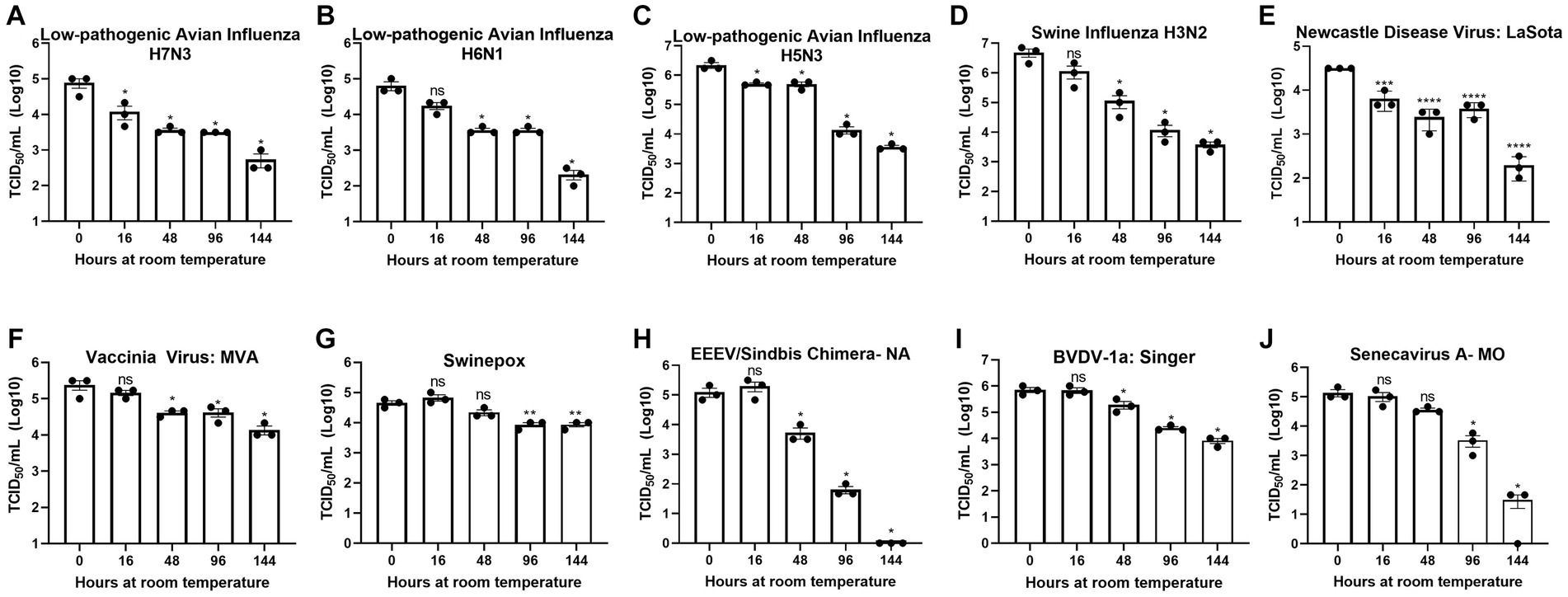 Figura 1. Diversos virus son estables a temperatura ambiente. Recuperación del virus a lo largo del tiempo depositado en una superficie de plástico a temperatura ambiente para (A) virus de la influenza aviar de baja patogenicidad (LPAI) H7N3; (B) LPAI H6N1; (C) LPAI H5N3; D) virus de la influenza porcina (VIS) H3N2; E) Virus de la enfermedad de Newcastle (VEN); (F) vaccinia; (G) viruela porcina; (H) virus de la encefalitis equina oriental (EEEV)/quimera de Sindbis; (I) virus de la diarrea viral bovina (BVDV); y (J) Senecavirus A. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. Cada punto de tiempo se comparó con el punto de tiempo 0. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado.**********
Figura 1. Diversos virus son estables a temperatura ambiente. Recuperación del virus a lo largo del tiempo depositado en una superficie de plástico a temperatura ambiente para (A) virus de la influenza aviar de baja patogenicidad (LPAI) H7N3; (B) LPAI H6N1; (C) LPAI H5N3; D) virus de la influenza porcina (VIS) H3N2; E) Virus de la enfermedad de Newcastle (VEN); (F) vaccinia; (G) viruela porcina; (H) virus de la encefalitis equina oriental (EEEV)/quimera de Sindbis; (I) virus de la diarrea viral bovina (BVDV); y (J) Senecavirus A. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. Cada punto de tiempo se comparó con el punto de tiempo 0. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado.**********
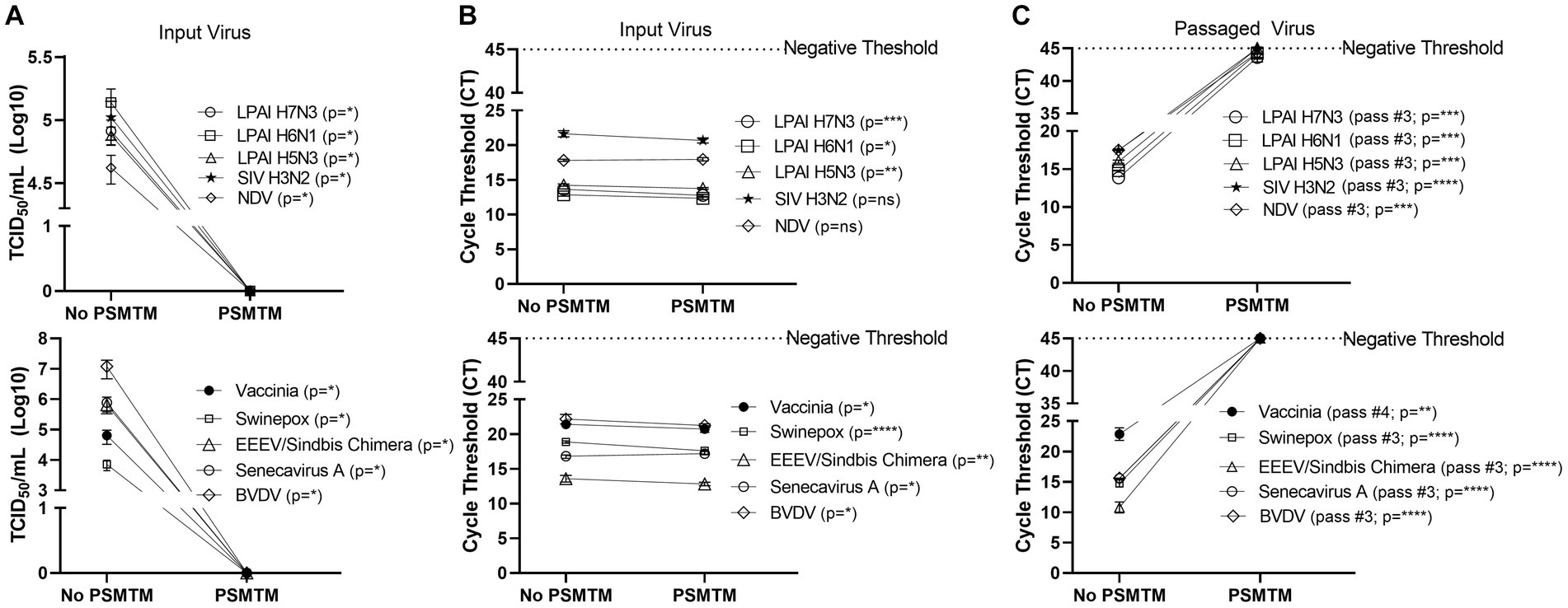 Figura 2. PSMTM inactiva eficazmente diversos virus animales mientras mantiene la detección de ácido nucleico. (A) Recuperación de virus después de la inactivación de los medios de transporte molecular (PSMTM) de PrimeStore® o el control de no inactivación de PBS. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. (B) Detección del umbral del ciclo de ácido nucleico (CT) después de la inactivación de PSMTM o el control de no inactivación de PBS. (C) Detección del umbral del ciclo de ácido nucleico (CT) del punto final de paso en serie después de la inactivación de PSMTM o la no inactivación de PBS. El pasaje serial se completó utilizando la línea celular de la Tabla 1 y la descripción descrita en Métodos. El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en la sección Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM, y la inactivación se evaluó utilizando las condiciones recomendadas por el fabricante. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la influenza aviar de baja patogenicidad (LPAI); virus de la influenza porcina (VIS); Virus de la enfermedad de Newcastle (VEN); virus de la encefalitis equina oriental (EEEV); y el virus de la diarrea viral bovina (BVDV).**********
Figura 2. PSMTM inactiva eficazmente diversos virus animales mientras mantiene la detección de ácido nucleico. (A) Recuperación de virus después de la inactivación de los medios de transporte molecular (PSMTM) de PrimeStore® o el control de no inactivación de PBS. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. (B) Detección del umbral del ciclo de ácido nucleico (CT) después de la inactivación de PSMTM o el control de no inactivación de PBS. (C) Detección del umbral del ciclo de ácido nucleico (CT) del punto final de paso en serie después de la inactivación de PSMTM o la no inactivación de PBS. El pasaje serial se completó utilizando la línea celular de la Tabla 1 y la descripción descrita en Métodos. El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en la sección Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM, y la inactivación se evaluó utilizando las condiciones recomendadas por el fabricante. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la influenza aviar de baja patogenicidad (LPAI); virus de la influenza porcina (VIS); Virus de la enfermedad de Newcastle (VEN); virus de la encefalitis equina oriental (EEEV); y el virus de la diarrea viral bovina (BVDV).**********
 Figura 3. La presencia de material biológico no afecta la efectividad de PSMTM en la inactivación o detección de ácidos nucleicos de diversos virus. Los virus se combinaron con sueros o hisopos cloacales apropiados para la especie, como se describe en la sección Métodos. (A) Recuperación de virus después de la inactivación de los medios de transporte molecular (PSMTM) de PrimeStore® o el control de no inactivación de PBS. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. (B) Detección del umbral del ciclo de ácido nucleico (CT) después de la inactivación de PSMTM o el control de no inactivación de PBS. (C) Detección del umbral del ciclo de ácido nucleico (CT) del punto final de paso en serie después de la inactivación de PSMTM o la no inactivación de PBS. El pasaje en serie se completó utilizando la línea celular de la Tabla 1 y la descripción descrita en la sección Métodos. El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM, y la inactivación se evaluó utilizando las condiciones recomendadas por el fabricante. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la influenza aviar de baja patogenicidad (LPAI); virus de la influenza porcina (VIS); Virus de la enfermedad de Newcastle (VEN); virus de la encefalitis equina oriental (EEEV); y el virus de la diarrea viral bovina (BVDV).**********
Figura 3. La presencia de material biológico no afecta la efectividad de PSMTM en la inactivación o detección de ácidos nucleicos de diversos virus. Los virus se combinaron con sueros o hisopos cloacales apropiados para la especie, como se describe en la sección Métodos. (A) Recuperación de virus después de la inactivación de los medios de transporte molecular (PSMTM) de PrimeStore® o el control de no inactivación de PBS. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. (B) Detección del umbral del ciclo de ácido nucleico (CT) después de la inactivación de PSMTM o el control de no inactivación de PBS. (C) Detección del umbral del ciclo de ácido nucleico (CT) del punto final de paso en serie después de la inactivación de PSMTM o la no inactivación de PBS. El pasaje en serie se completó utilizando la línea celular de la Tabla 1 y la descripción descrita en la sección Métodos. El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM, y la inactivación se evaluó utilizando las condiciones recomendadas por el fabricante. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la influenza aviar de baja patogenicidad (LPAI); virus de la influenza porcina (VIS); Virus de la enfermedad de Newcastle (VEN); virus de la encefalitis equina oriental (EEEV); y el virus de la diarrea viral bovina (BVDV).**********
Detección de PCR de virus
El ácido nucleico se extrajo de 200 uL de material vírico tratado con PSMTM o PBS, así como de cada producto de paso de los pasajes seriados descritos anteriormente. El ácido nucleico se extrajo utilizando el kit de purificación de ácido nucleico MagMax™ CORE (Applied Biosystems) en un instrumento KingFisher Flex (Thermo Scientific) con el guión de MagMax_CORE_Flex proporcionado (con calor) de acuerdo con las instrucciones del fabricante. El ácido nucleico se detectó mediante un cebador/sonda específico del virus de acuerdo con las secuencias de referencia indicadas (Tabla 1), utilizando el instrumento de PCR en tiempo real TaqMan™ Fast Virus 1-Step Master Mix (Applied Biosystems) y QuantStudio™ 5 (Thermo Scientific). Las condiciones cíclicas para LPAI H6N1, LPAI H7N3, LPAI H5N3, SIV H3N2, NDV y BVDV fueron las siguientes: 50 °C durante 5 min, 95 °C durante 20 s, seguidos de 45 ciclos de 95 °C durante 15 s y 60 °C durante 45 s. Las condiciones cíclicas para el virus vaccinia fueron las siguientes: 95 °C durante 20 s, seguido de 45 ciclos de 95 °C durante 10 s y 60 °C durante 30 s. Las condiciones cíclicas para la quimera del virus EEEV/Sindbis y la SVA fueron las siguientes: 50 °C durante 15 min, 95 °C durante 2 min, seguidos de 45 ciclos de 95 °C durante 15 s y 60 °C durante 30 s. Las condiciones cíclicas para la viruela porcina fueron las siguientes: 50 °C durante 5 min, 95 °C durante 20 s, seguidas de 45 ciclos de 95 °C durante 15 s y 53 °C durante 1 min. Los datos se analizaron utilizando el software de diseño y análisis (versión 2.6, Thermo Scientific), donde los valores umbral se establecieron en 0,100. Se utilizó un TC de 45 como umbral negativo sin amplificación. La comparación de los TCID de virus de stock50 y los valores de TC revelaron tendencias similares, validando los parámetros del ensayo (Figura Suplementaria S2). Todos los procedimientos de control e inactivación se completaron en tres réplicas experimentales, y cada PCR se completó con tres réplicas técnicas.
Estabilidad de la PCR de virus
Se añadieron 200 μL de virus tratados con control PSMTM o PBS a los tubos con tapón de rosca por triplicado para cada punto de tiempo. Se utilizaron tubos con tapón de rosca para imitar las condiciones del tubo del fabricante de PSMTM que se pueden utilizar en la recolección de muestras de campo. Los puntos de tiempo se recolectaron durante un máximo de 21 días para imitar el tiempo máximo de tránsito anticipado una vez que se recolecta una muestra de campo hasta que llega a un laboratorio para pruebas de diagnóstico. Los virus tratados con control PSMTM o PBS se incubaron a temperatura ambiente hasta el punto de tiempo indicado, donde los virus se recolectaron agregando 200 μL de medio de propagación del virus a cada tubo y realizando pipeteos repetidos. Los virus recolectados se congelaron una vez a -80 °C hasta la titulación por TCID50 en células susceptibles (Tabla 1), y la detección por PCR se realizó como se describió anteriormente.
Viabilidad celular
La viabilidad celular se determinó mediante un ensayo MTT (Invitrogen), donde las líneas celulares utilizadas en este estudio se sembraron en placas de 96 pocillos y se incubaron con diluciones de control PSMTM o PBS. La PSMTM o la PBS se diluyeron inicialmente a la proporción recomendada por el fabricante utilizada para los estudios de inactivación de la PSMTM descritos anteriormente, en los que se sustituyó una parte del virus por medios de propagación del virus (que no contenían virus) para evaluar el efecto de la PSMTM sola (sin virus) sobre la viabilidad celular, ya que estos virus inducen ECP y confundirían la evaluación del efecto de la PSMTM sobre la viabilidad celular. A continuación, los medios tratados con PSMTM o PBS se diluyeron en serie 1:10 y se incubaron con células durante 48 h. A continuación, las células se incubaron con 5 mg/ml de reactivo MTT en la oscuridad durante 3,5 h a 37 °C antes de la adición de 4 mM de HCl en isopropanol y la disrupción de la monocapa mediante pipeteo. La absorbancia se midió a 490 nm utilizando un lector de placas BioTek. Los valores de viabilidad celular de PSMTM se normalizaron a una dilución de PBS equivalente, establecida en 100%.
Estadística
La significación estadística se determinó mediante el software GraphPad v9.3.1 (GraphPad Prism 9) utilizando una prueba t de Student de dos colas. Los valores de p de <0,05 se consideraron significativos. Las barras de error representan el error estándar de la media (SEM) a partir de tres réplicas biológicas.
Resultados
Diversos virus muestran patrones similares de estabilidad a temperatura ambiente
Para evaluar el riesgo potencial de enviar muestras transmisibles de alta contención sin un método de inactivación aplicado en el momento de la recolección de la muestra, evaluamos la descomposición natural de los virus utilizados en este estudio a lo largo del tiempo. La infectividad del virus en cada punto de tiempo se comparó con el punto de tiempo 0. El punto de tiempo 0 muestra que se depositaron y recuperaron un mínimo de 4 logaritmos de virus para todos los virus analizados (Figuras 1A-J).
 Figura 4. La proporción de PSMTM a virus determina la efectividad de la inactivación. La recuperación del virus y la detección de ácido nucleico se determinaron después de la inactivación de los medios de transporte molecular (PSMTM) de PrimeStore® o el control de no inactivación de PBS utilizando varias condiciones de tratamiento a la relación entre virus: (A) relación 1:1; (B) Proporción 1:3; (C) Proporción 1:10; y (D) proporción 1:100. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en la sección Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la encefalitis equina oriental (EEEV).**********
Figura 4. La proporción de PSMTM a virus determina la efectividad de la inactivación. La recuperación del virus y la detección de ácido nucleico se determinaron después de la inactivación de los medios de transporte molecular (PSMTM) de PrimeStore® o el control de no inactivación de PBS utilizando varias condiciones de tratamiento a la relación entre virus: (A) relación 1:1; (B) Proporción 1:3; (C) Proporción 1:10; y (D) proporción 1:100. La recuperación del virus se evaluó mediante TCID50 utilizando la línea celular descrita en la Tabla 1. El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en la sección Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; p < 0,001; p < 0,0001; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la encefalitis equina oriental (EEEV).**********
Aunque la descomposición natural varió entre los virus individuales, los resultados muestran una reducción máxima de 1 log después de 48 h a temperatura ambiente y la supervivencia de todos los virus después de 144 h a temperatura ambiente, con la excepción de la quimera del virus EEEV/Sindbis, que se inactivó completamente a las 144 h (Figuras 1A-J). Estos datos indican que los virus altamente patógenos pueden sobrevivir períodos prolongados de tiempo en condiciones naturales y demuestran la necesidad de un método de inactivación que pueda utilizarse en el momento de la recolección de muestras para reducir el riesgo de transmisión.
El PSMTM inactiva completamente los virus que representan enfermedades animales altamente contagiosas
Los virus se trataron con PSMTM de acuerdo con las condiciones recomendadas por el fabricante o PBS (no-PSMTM) para la comparación de control. La replicación viral se evaluó mediante TCID50 y el paso serial de células susceptibles, y el ácido nucleico se evaluó mediante qRT-PCR. El tratamiento con PSMTM inactivó completamente todos los virus probados en comparación con el control de PBS. No persistió la infectividad en los virus tratados con PSMTM, y los títulos de virus se redujeron >4-log (99,99%) (Figura 2A; Tabla 2). A pesar de que no quedaba infectividad del virus después del tratamiento con PSMTM, los valores de CT de los virus tratados con PSMTM en comparación con el control sin PSMTM fueron muy comparables y variaron en menos de 1,2 valores de CT (Figura 2B; Tabla 2). Se considera que un cambio en el valor de CT de 3,3 representa un cambio de 1 log (10 veces) (46). Para garantizar la inactivación completa, los virus tratados con PSMTM y control se pasaron en serie a las células susceptibles (Tabla 1). Los valores de CT de los virus tratados con PSMTM alcanzaron un umbral negativo sin infectividad celular observable por un criterio de valoración de un mínimo de tres pasajes, lo que indica que no hay amplificación de ácidos nucleicos ni replicación del virus (Figura 2C; Figuras suplementarias S3, S4). Por el contrario, los valores de CT de los virus tratados con control fueron estables o mostraron una reducción de CT (aumento de ácido nucleico) en el punto final de paso en comparación con la entrada (Figura 2C; Figura complementaria S3). También se observó infectividad celular en el punto final de paso para los virus tratados con control (Figura suplementaria S3). En conjunto, estos datos muestran una inactivación completa de la replicación del virus tratado con PSMTM sin deterioro del ácido nucleico que se necesita para las pruebas de PCR.
 Tabla 2. Log10 TCID50un reducción y cambio del umbral del ciclo (TC) de qRT-PCR en PSMTMb Tratamiento vs. virus de tratamiento de control.
Tabla 2. Log10 TCID50un reducción y cambio del umbral del ciclo (TC) de qRT-PCR en PSMTMb Tratamiento vs. virus de tratamiento de control.
Se evaluó la inactivación de virus por PSMTM en presencia de sueros relevantes para la especie o, en el caso de sustitutos aviares, hisopos cloacales. De acuerdo con nuestros hallazgos sin materiales biológicos añadidos, el tratamiento con PSMTM inactivó completamente todos los sueros o hisopos cloacales con virus (Figura 3A). Los valores de TC de los sueros con picos de virus o de los hisopos cloacales con virus tratados con PSMTM fueron comparables a los del control sin PSMTM (Figura 3B). Es importante destacar que el paso en serie de los virus tratados con PSMTM en presencia de suero o hisopo se acercó al umbral negativo por criterio de valoración de paso, mientras que los virus tratados con control no lo hicieron (Figura 3C; Figura complementaria S3). Estos datos muestran que la presencia de material biológico no afecta a la inactivación de los virus por PSMTM.
Se requiere el mantenimiento de una proporción adecuada de PSMTM para una inactivación efectiva del virus
Es posible que la recolección de muestras diagnósticas no siempre logre la proporción exacta de inactivación del virus 3 PSMTM:1 recomendada por el fabricante. Para evaluar la seguridad del uso de la inactivación de PSMTM si no se mantiene la proporción recomendada, se evaluó la infectividad del virus y el paso en serie de los virus tratados con cantidades menores de PSMTM. Se evaluó la quimera del virus EEEV/Sindbis debido a su alto crecimiento de títulos. Los resultados muestran que la relación entre 1 PSMTM: 1 virus completamente inactivado >6-log y el paso en serie del virus alcanzó un umbral negativo por punto final de paso, mientras que el virus tratado con el control no logró resultados similares (Figura 4A). Sin embargo, aunque la relación de infectividad del virus inactivado de 1 PSMTM: 3 >6-log, los resultados de paso en serie del virus revelaron que el virus no estaba completamente inactivado, donde los valores de CT del virus tratados con PSMTM fueron comparables a los valores de CT del virus tratado con el control por criterio de valoración de paso (Figura 4B). Los resultados de 1 PSMTM: 10 virus y 1 PSMTM: 100 alteraciones en la proporción de virus mostraron además que el virus no estaba inactivado según lo medido por ensayos de infectividad y paso en serie (Figuras 4C, D). Estos resultados indican que las variaciones mínimas en la tasa de inactivación de PSMTM recomendada por el fabricante aún pueden inactivar completamente el virus. Sin embargo, se debe tener precaución durante la recolección de muestras para mantener la proporción adecuada para garantizar la inactivación completa.
La PSMTM puede mejorar modestamente la estabilidad del contenido de ácidos nucleicos virales requerido para las pruebas de PCR
Durante el envío de muestras, que puede tardar varios días en llegar a los laboratorios para realizar pruebas diagnósticas, el ácido nucleico puede deteriorarse o las condiciones ambientales, como la evaporación, pueden influir en la eficacia de la inactivación (47). Evaluamos el efecto de PSMTM sobre la estabilidad de la inactivación y la detección de ácidos nucleicos del virus a lo largo del tiempo. La LPAI H7N3 y el virus vaccinia se evaluaron debido a su moderada y alta estabilidad, respectivamente, de la infectividad a lo largo del tiempo a temperatura ambiente (Figuras 1A, F). Los virus tratados con PSMTM o con control se depositaron en tubos con tapón de rosca para imitar los tubos de recolección de muestras y se recolectaron en el momento indicado. La infectividad del virus para cada punto de tiempo se comparó con el punto de tiempo 0 de cada tratamiento respectivo.
Los resultados de infectividad muestran que los virus tratados con PSMTM se inactivaron completamente en todos los puntos de tiempo en comparación con los virus tratados con control (Figuras 5A, C). Estos datos indican que la inactivación de PSMTM es completa y que la infectividad del virus no se ha recuperado con el tiempo. Los resultados de infectividad del virus tratado con control revelan tendencias similares a los hallazgos de descomposición natural (Figuras 1A, F), donde el virus H7N3 de la LPAI se mantuvo moderadamente estable hasta que se produjo la pérdida completa de la infectividad en el día 14 (Figura 5A). Sin embargo, el ácido nucleico H7N3 de la LPAI se detectó durante 21 días a pesar de la pérdida de infectividad (Figuras 5A, B). La comparación de los virus H7N3 de la LPAI tratados con PSMTM y el control muestra valores comparables de CT en cada punto de tiempo, con solo un aumento moderado en la estabilidad de la TC (reducción del valor de CT o aumento del ácido nucleico) en PSMTM en puntos de tiempo posteriores (Figura 5B). El virus vaccinia tratado con el control fue altamente estable y la infectividad solo se redujo <2 logaritmos en 21 días a temperatura ambiente (Figura 5C). El ácido nucleico del virus Vaccinia se detectó durante 21 días, y los valores de CT fueron comparables para los virus tratados con PSMTM y control, con un ligero aumento de la estabilidad de la CT (reducción del valor de CT o aumento del ácido nucleico) en PSMTM en puntos temporales posteriores (Figura 5D). Estos datos indican que la PSMTM puede mejorar la estabilidad del ácido nucleico viral en puntos temporales posteriores, aunque el efecto es modesto.
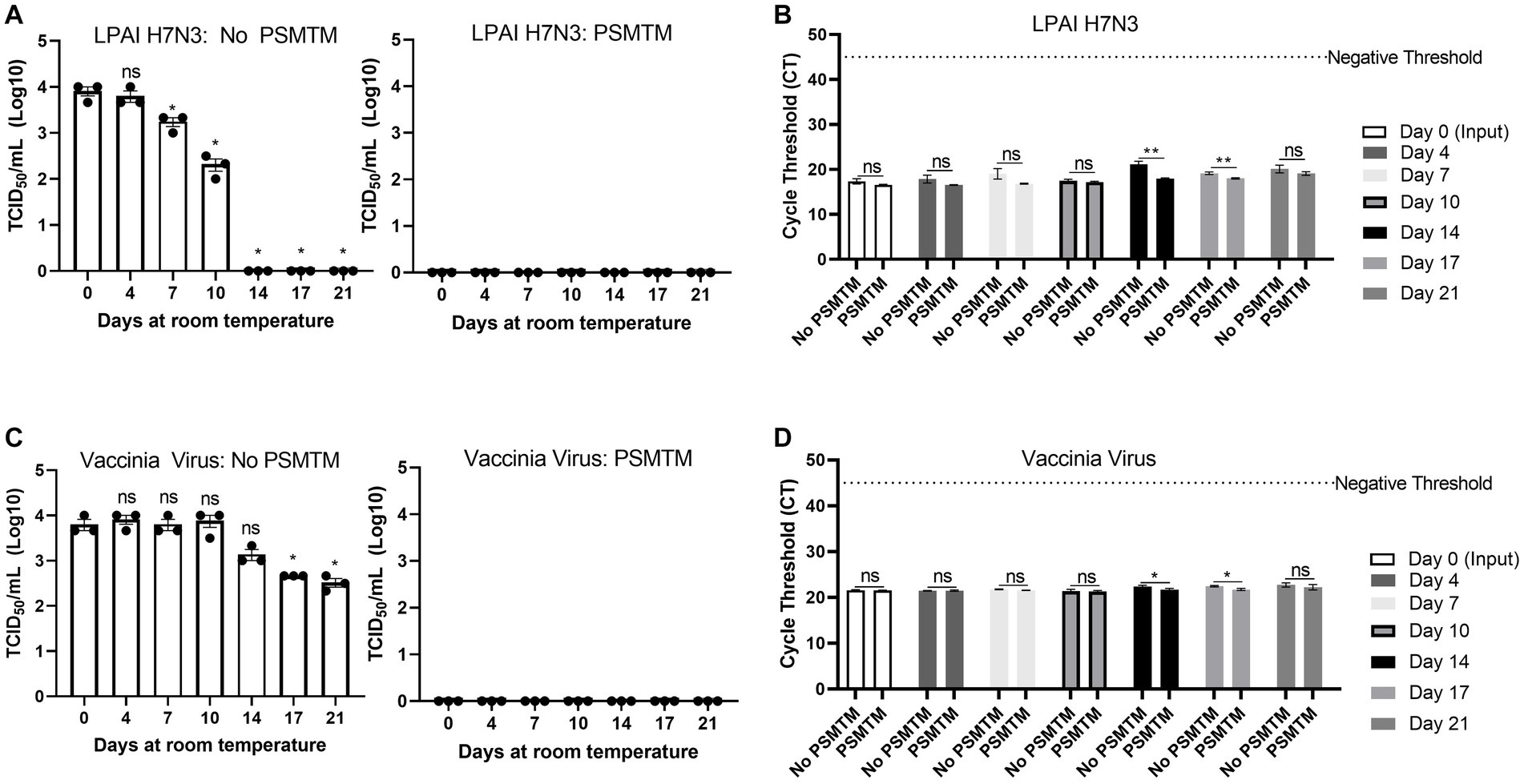 Figura 5. La inactivación del virus y la detección de ácidos nucleicos son estables a lo largo del tiempo en PSMTM. Recuperación de virus y detección de ácidos nucleicos durante 21 días para virus depositados en un tubo de plástico con tapón de rosca a temperatura ambiente. La inactivación se evaluó utilizando las condiciones recomendadas por el fabricante. (A) La recuperación del virus fue evaluada por TCID50 para la LPAI H7N3 tratada con control de inactivación PBS (izquierda) y la LPAI H7N3 tratada con medios de transporte molecular PrimeStore® (PSMTM) (derecha). (B) Detección del umbral del ciclo de ácido nucleico (CT) del virus para el control de la no inactivación de PBS y la LPAI H7N3 tratada con PSMTM. (C) La recuperación del virus fue evaluada por TCID50 para el virus vaccinia tratado con control de inactivación PBS (izquierda) y el virus vaccinia tratado con PSMTM (derecha). (D) Detección del umbral del ciclo de ácido nucleico (CT) del virus para el control de no inactivación de PBS y el virus vaccinia tratado con PSMTM. (A,C) TCID50 El título se determinó en la línea celular apropiada como se identifica en la Tabla 1. Cada punto de tiempo se comparó con el punto de tiempo 0 para cada tratamiento respectivo. (B,D) El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la influenza aviar de baja patogenicidad (LPAI, por sus siglas en inglés).***
Figura 5. La inactivación del virus y la detección de ácidos nucleicos son estables a lo largo del tiempo en PSMTM. Recuperación de virus y detección de ácidos nucleicos durante 21 días para virus depositados en un tubo de plástico con tapón de rosca a temperatura ambiente. La inactivación se evaluó utilizando las condiciones recomendadas por el fabricante. (A) La recuperación del virus fue evaluada por TCID50 para la LPAI H7N3 tratada con control de inactivación PBS (izquierda) y la LPAI H7N3 tratada con medios de transporte molecular PrimeStore® (PSMTM) (derecha). (B) Detección del umbral del ciclo de ácido nucleico (CT) del virus para el control de la no inactivación de PBS y la LPAI H7N3 tratada con PSMTM. (C) La recuperación del virus fue evaluada por TCID50 para el virus vaccinia tratado con control de inactivación PBS (izquierda) y el virus vaccinia tratado con PSMTM (derecha). (D) Detección del umbral del ciclo de ácido nucleico (CT) del virus para el control de no inactivación de PBS y el virus vaccinia tratado con PSMTM. (A,C) TCID50 El título se determinó en la línea celular apropiada como se identifica en la Tabla 1. Cada punto de tiempo se comparó con el punto de tiempo 0 para cada tratamiento respectivo. (B,D) El ácido nucleico se detectó de acuerdo con la referencia de la Tabla 1 y la descripción en Métodos. Los virus tratados con PSMTM se compararon con el control correspondiente sin PSMTM. La significación se determinó mediante la prueba t de Student. p < 0,05; p < 0,01; De hecho, la mayoría de las personas que no son significativas son Las barras de error representan el error estándar de la media (SEM) de los experimentos por triplicado. Virus de la influenza aviar de baja patogenicidad (LPAI, por sus siglas en inglés).***
Discusión
Los brotes de enfermedades altamente patógenas, incluido el brote de gripe aviar altamente patógena en EE. UU. de 2022 y la pandemia de COVID-19, ponen a prueba la capacidad de diagnóstico de las ya limitadas instalaciones de alta contención. Existe la necesidad de un reactivo que proteja el ácido nucleico necesario para las pruebas diagnósticas y, al mismo tiempo, inactive los patógenos para permitir las pruebas en instalaciones de menor contención. La PSMTM se utilizó anteriormente durante la pandemia de COVID-19 para pruebas diagnósticas de RT-PCR (24) y está autorizada por la FDA para la recolección de muestras de IAV y Mycobacterium tuberculosis (17). Sin embargo, se necesitan estudios adicionales para determinar la inactivación de una amplia gama de virus con una evaluación emparejada del ácido nucleico viral necesario para la prueba. En este estudio, evaluamos la inactivación de diversos sustitutos de virus animales, incluidos cuatro virus de la influenza A, a través de ensayos de titulación y pasaje en serie, y evaluamos la preservación de ácidos nucleicos a través de qRT-PCR. Este es uno de los estudios más grandes realizados hasta la fecha que incluye virus animales de alta consecuencia de diferentes familias. Este reactivo de inactivación es uno de los únicos reactivos disponibles para muestras de diagnóstico veterinario que elimina la necesidad de control de temperatura, lo que puede aumentar las pruebas de vigilancia.
Los virus utilizados en este estudio mostraron una replicación reducida pero detectable después de varios días a temperatura ambiente en medios líquidos (Figura 1). Aunque las condiciones pueden variar considerablemente entre los estudios, estas tendencias de descomposición del virus generalmente concuerdan con las informadas por otros (48-55). Por lo tanto, un reactivo de inactivación utilizado en el momento de la recolección de la muestra es ideal para aliviar las preocupaciones sobre la transmisión durante los procedimientos de envío y facilitar las pruebas en instalaciones de contención inferiores. Los 10 virus analizados en este estudio, incluidos cuatro virus de influenza A de origen aviar y porcino, fueron completamente inactivados (reducción del ≥99,99 %) por PSMTM cuando se utilizaron de acuerdo con las recomendaciones del fabricante (Figura 2). El paso en serie de los virus tratados con PSMTM no reveló ningún virus replicante (Figura 2). La inactivación no dependió del título del virus, ya que los títulos del virus utilizados en este estudio variaron de 4 a 7 log/ml (Figuras 2 y 3). Estos títulos son biológicamente relevantes, ya que los estudios en modelos animales mostraron títulos de ≤5log unidades formadoras de placa por ml (PFU/mL) para el virus de la fiebre aftosa (56), una mediana de 6-8log de la dosis de hemadsorbencia por mililitro (HAD50/ml) para la PPA (57), TCID de 5–7 log50/ml para LCR (58), ≤7log de UFP por gramo de tejido para EEEV (59) y IAV (60), y ≤ 7log y ≤ 8log TCID de tejido por gramo de tejido para NDV (61) y HPAI (62), respectivamente. Los resultados de inactivación se verificaron en presencia de material biológico (sueros con virus o hisopos cloacales con virus con virus) (Figura 3) y son consistentes con los hallazgos de otros, lo que demuestra que los métodos de inactivación química no son sensibles a la presencia de material biológico (45). Sin embargo, se necesitan estudios de infección natural para confirmar que la inactivación de PSMTM no es sensible a la saturación de concentraciones de material biológico.
Estudios anteriores han demostrado que la inactivación de PSMTM es completa incluso con un tiempo de contacto reducido de la duración recomendada por el fabricante de 60 minutos, donde se identificó un tiempo de contacto de tan solo 2 minutos como suficiente (63). Sin embargo, estudios previos no evaluaron la importancia de la relación volumétrica de los medios de inactivación con respecto al volumen de la muestra que contiene el virus. La variación de la proporción de medios de inactivación en nuestros resultados mostró que la inactivación era incompleta, con una desviación modesta de la proporción recomendada por el fabricante de 3:1 (Figura 4). Los resultados de titulación y paso en serie revelaron que la inactivación se completó cuando la relación se modificó de la proporción recomendada por el fabricante de 3:1 a 1:1; sin embargo, la inactivación fue incompleta con una disminución adicional en la proporción de los medios de inactivación con respecto a los medios de muestra del virus (Figura 4). La inactivación incompleta no se atribuyó a un aumento significativo en el título de los medios de muestra del virus, ya que el PBS sin condiciones de control de inactivación de PSMTM se mantuvo dentro de 1 logaritmo para todas las desviaciones de la relación (Figura 4). Si bien estudios anteriores indicaron que el tiempo de contacto se puede reducir sin comprometer la inactivación, estos resultados indican que el cumplimiento de la relación volumétrica recomendada por el fabricante proporciona un margen de seguridad confiable para garantizar una inactivación completa.
Es importante destacar que los valores de CT de ácidos nucleicos estuvieron dentro de 1,5 para todos los virus en condiciones de control sin inactivación de PSMTM y PBS, lo que indica la preservación de los ácidos nucleicos a pesar de la inactivación de la replicación del virus (Figuras 2, 3). Otros estudios han demostrado que los valores de CT fueron comparables para la detección de virus respiratorios humanos recogidos en PSMTM en comparación con otros medios (23, 64). En conjunto, los resultados de este estudio y los de estudios anteriores de otros muestran que la PSMTM mantiene la estabilidad tanto del ARN viral como del ADN viral para los virus humanos y animales a pesar de la estabilidad ambiental reducida del ARN viral (23, 64, 65). Además, los resultados de este estudio muestran que la inactivación y la estabilidad de los ácidos nucleicos parecen mantenerse durante largos períodos de tiempo. La incubación de la LPAI H7N3 y el virus vaccinia durante 21 días reveló que la inactivación de PSMTM fue completa sin observación de replicación del virus, a pesar de una detección de ácido nucleico comparable o modestamente mejorada para PSMTM en comparación con el control sin inactivación de PBS (Figura 5). El criterio de valoración de este estudio fue de 21 días; sin embargo, otros estudios mostraron que el ácido nucleico se detectó en muestras en PSMTM hasta 196 días después de la recolección de la muestra (64).
En este estudio, el PSMTM fue completamente eficaz en la inactivación de todos los virus probados. Nuestros resultados son consistentes con otros que evaluaron el uso de PSMTM con virus humanos (23, 64). Sin embargo, los virus utilizados en este estudio fueron virus sustitutos, y se requieren estudios adicionales con los virus objetivo representados en este estudio para validar la inactivación con virus reales de agentes seleccionados. Nuestros datos indican que el uso de este medio de transporte viral, de acuerdo con los procedimientos recomendados por el fabricante, inactiva con éxito todos los virus probados, y los procedimientos alternativos, incluidos los diferentes tipos de muestras, requerirían una evaluación adicional para garantizar la inactivación. Este método de inactivación puede mejorar el tiempo de respuesta de las pruebas durante los brotes de enfermedades altamente patógenas al eliminar las restricciones de las pruebas diagnósticas. Este método también se puede utilizar para garantizar que la calidad de la muestra se mantenga durante los procedimientos normales de envío. Los resultados de este estudio pueden aplicarse a otros patógenos que requieren pruebas moleculares y pueden ser sensibles a las condiciones ambientales, como la temperatura y el tiempo de prueba, y en escenarios en los que las muestras pueden recogerse en sitios de campo con recursos limitados. Sin embargo, las pruebas moleculares solo se evaluaron en este estudio mediante qRT-PCR, y otros métodos basados en moléculas requerirían una validación adicional.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están incluidas en el artículo/Material complementario, las consultas posteriores pueden dirigirse al autor correspondiente.
Declaración ética
El estudio en animales fue aprobado por el Comité Institucional de Cuidado y Uso de Animales APH-22-1033 de los Laboratorios Nacionales de Servicios Veterinarios/Centro de Productos Biológicos Veterinarios. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
JW: Conceptualización, Escritura – borrador original, Escritura – revisión y edición, Investigación. RS: Escritura – revisión y edición, Investigación. HH: Escritura – revisión y edición, Recursos. NP: Escritura – revisión y edición, Investigación. RL: Escritura – revisión y edición, conceptualización. SR-A: Escritura – revisión y edición, conceptualización. RP: Escritura – borrador original, Escritura – revisión y edición. KS: Conceptualización, Redacción – borrador original, Redacción – revisión y edición, Supervisión.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este estudio fue apoyado en parte por un nombramiento (JW) en el Servicio de Inspección de Sanidad Animal y Vegetal (APHIS, por sus siglas en inglés) administrado por el Instituto Oak Ridge para la Ciencia y la Educación (ORISE, por sus siglas en inglés) a través de un acuerdo interinstitucional entre el Departamento de Energía de los Estados Unidos (DOE, por sus siglas en inglés) y el Departamento de Agricultura de los Estados Unidos (USDA, por sus siglas en inglés). ORISE es administrada por ORAU bajo el número de contrato del DOE DE-SC0014664. Los financiadores no tuvieron ningún papel en las opiniones expresadas en este documento. Los datos presentados en este estudio son solo para fines informativos, y las referencias a productos que no son del USDA no son un respaldo del USDA.
Reconocimientos
Los autores agradecen al grupo de citología dentro de la sección de Pruebas de Aptitud y Reactivos de Laboratorio de Diagnóstico, Bioanálisis y Reactivos de los Laboratorios Nacionales de Servicios Veterinarios del USDA por el cultivo celular. Los autores agradecen a June Meichsner y Alethea Fry, del Centro de Productos Biológicos Veterinarios del USDA, por proporcionar células CEF primarias. Los autores agradecen a Denise Chapman de la Unidad de Recursos Animales del USDA por la recolección de hisopos cloacales. Agradecemos al Laboratorio de Virología Diagnóstica de los Laboratorios de Servicios Veterinarios Nacionales del USDA por proporcionar los virus fuente.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1304022/full#supplementary-material
Referencias
1. Belay, ED, Kile, JC, Hall, AJ, Barton-Behravesh, C, Parsons, MB, Salyer, S, et al. Programas de enfermedades zoonóticas para mejorar la seguridad sanitaria mundial. Emerg Infect Dis. (2017) 23:S65–70. doi: 10.3201/eid2313.170544
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Poderes, AM . Resurgimiento del interés en el desarrollo de vacunas contra el virus de la encefalitis equina oriental. J Med Entomol. (2022) 59:20–6. doi: 10.1093/jme/tjab135
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Brown, VR, Miller, RS, McKee, SC, Ernst, KH, Didero, NM, Maison, RM, et al. Riesgos de introducción y consecuencias económicas asociadas a la peste porcina africana, la peste porcina clásica y la fiebre aftosa: revisión de la bibliografía. Transbound Emerg Dis. (2021) 68:1910–65. doi: 10.1111/tbed.13919
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Thornber, PM, Rubira, RJ y Styles, DK. Matanza humanitaria de animales con fines de control de enfermedades. Rev Sci Tech. (2014) 33:303–10. doi: 10.20506/rst.33.1.2279
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Tú, S, Liu, T, Zhang, M, Zhao, X, Dong, Y, Wu, B, et al. Los brotes de peste porcina africana en China provocaron pérdidas económicas y del producto interior bruto. Alimentos de la naturaleza. (2021) 2:802–8. doi: 10.1038/s43016-021-00362-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Farahat, RA, Khan, SH, Rabaan, AA, y Al-Tawfiq, JA. El resurgimiento de la influenza aviar y la infección humana: una breve perspectiva. Nuevos microbios, nuevas infectas. (2023) 53:101122. doi: 10.1016/j.nmni.2023.101122
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Prempeh, H, Smith, R, y Muller, B. Fiebre aftosa: las consecuencias humanas. Las consecuencias para la salud son leves, las económicas enormes. BMJ. (2001) 322:565–6. doi: 10.1136/bmj.322.7286.565
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Corrin, T, Ackford, R, Mascarenhas, M, Greig, J y Waddell, LA. Virus de la encefalitis equina oriental: una revisión exploratoria de la evidencia global. Vector Transmitido Zoonótico Dis. (2021) 21:305–20. doi: 10.1089/vbz.2020.2671
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Ul-Rahman, A, Ishaq, HM, Raza, MA, y Shabbir, MZ. Potencial zoonótico del virus de la enfermedad de Newcastle: perspectivas antiguas y novedosas relacionadas con la salud pública. Rev Med Virol. (2022) 32:E2246. doi: 10.1002/rmv.2246
10. Horman, WSJ, Nguyen, THO, Kedzierska, K, Bean, AGD y Layton, DS. Los impulsores de la patología en la influenza aviar zoonótica: la interacción entre el huésped y el patógeno. Frente Immunol. (2018) 9:1812. doi: 10.3389/fimmu.2018.01812
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Charlier, J, Barkema, HW, Becher, P, De Benedictis, P, Hansson, I, Hennig-Pauka, I, et al. Herramientas de control de enfermedades para garantizar la salud animal y pública en un mundo densamente poblado. Lancet Planet Health. (2022) 6:E812–24. doi: 10.1016/S2542-5196(22)00147-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. Morse, S.A., y Quigley, BR. Regulaciones de agentes selectos. Microbial Forensics. 3ª edición. (2020):425–39. doi: 10.1016/B978-0-12-815379-6.00028-3
13. Waldrup, KA, y Conger, TH. Mantener una vigilancia de las enfermedades foráneas de los animales. Vet Clin North Am Food Anim Pract. (2002) 18:379–87. doi: 10.1016/S0749-0720(02)00053-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Saini, V, Kalra, P, Sharma, M, Rai, C, Saini, V, Gautam, K, et al. Un medio de recolección y transporte de muestras independiente de la cadena de frío mejora la sensibilidad diagnóstica y minimiza los desafíos de bioseguridad del diagnóstico molecular de COVID-19. Microbiol Spectr. (2021) 9:E0110821. doi: 10.1128/Spectrum.01108-21
15. Xia, H, y Yuan, Z. Instalaciones de alta contención y el papel que desempeñan en la seguridad sanitaria mundial. J Bioseguridad Biosecur. (2022) 4:1–4. doi: 10.1016/j.jobb.2021.11.005
16. Enserink, M, y Kaiser, J. Investigación en biodefensa. Un cargamento accidental de ántrax genera un debate sobre la seguridad. Ciencia. (2004) 304:1726–7. doi: 10.1126/science.304.5678.1726a
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Daum, LT, y Fischer, GW. Detección rápida y segura del ARN del SARS-CoV-2 y del virus de la gripe mediante pruebas diagnósticas cuantitativas de PCR in situ a partir de muestras clínicas recogidas en medio de transporte molecular. J Appl Lab Med. (2021) 6:1409–16. doi: 10.1093/jalm/jfab073
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Clarke, C, Smith, K, Goldswain, SJ, Helm, C, Cooper, DV, Kerr, TJ, et al. El nuevo medio de transporte molecular utilizado en combinación con Xpert MTB/RIF ultra proporciona una rápida detección de Mycobacterium bovis en búfalos africanos. Sci Rep. (2021) 11:7061. doi: 10.1038/s41598-021-86682-5
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. LHNVD . Medio de transporte® molecular (MTM) de PrimeStore: Longhorn Vaccines and Diagnostics LLC: [Disponible en: https://static1.squarespace.com/static/5f7dde76e3d6f427822285d4/t/5fbe6052fa04221c71f07ecd/1606312018859/US_SDS_Version02_Oct29_2020.pdf
20. Honeywood, MJ, Jeffries-Miles, S, Wong, K, Harrington, C, Burns, CC, Oberste, MS, et al. Uso de tampones de extracción de ácidos nucleicos basados en tiocianato de guanidina para inactivar el virus de la poliomielitis en materiales potencialmente infecciosos. Métodos J Virol. (2021) 297:114262. doi: 10.1016/j.jviromet.2021.114262
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Wood, BA, Mioulet, V, Henry, E, Gray, A, Azhar, M, Thapa, B, et al. Inactivación del virus de la fiebre aftosa a/IRN/8/2015 con tampones de lisis disponibles en el mercado. Métodos J Virol. (2020) 278:113835. doi: 10.1016/j.jviromet.2020.113835
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Lin, Q, Lim, JYC, Xue, K, Yew, PYM, Owh, C, Chee, PL, et al. Agentes higienizantes para la inactivación y desinfección de virus. Vista. (2020) 1:e16. doi: 10.1002/viw2.16
23. Daum, LT, Worthy, SA, Yim, KC, Nogueras, M, Schuman, RF, Choi, YW, et al. Un medio de recolección y transporte de muestras clínicas para aplicaciones genómicas y de diagnóstico molecular. Epidemiol Infect. (2011) 139:1764–73. doi: 10.1017/S0950268810002384
24. Welch, SR, Davies, KA, Buczkowski, H, Hettiarachchi, N, Green, N, Arnold, U, et al. Análisis de la inactivación del SARS-CoV-2 por medios de transporte de muestras, reactivos de extracción de ácidos nucleicos, detergentes y fijadores. J Clin Microbiol. (2020) 58:e01713-20. doi: 10.1128/JCM.01713-20
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Baker, JL y Ward, BM. Desarrollo y comparación de un ensayo cuantitativo de PCR en tiempo real TaqMan-MGB con otros tres métodos de cuantificación de viriones vaccinia. Métodos J Virol. (2014) 196:126–32. doi: 10.1016/j.jviromet.2013.10.026
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Kaiser, FK, Wiedemann, A, Kuhl, B, Menke, L, Beineke, A, Baumgartner, W, et al. Las cepas del virus de la viruela porcina aisladas de cerdos domésticos y jabalíes en Alemania muestran una capacidad de codificación alterada en la región terminal del genoma que codifica genes específicos de la especie. Virus. (2021) 13:2038. doi: 10.3390/v13102038
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Baxi, M, McRae, D, Baxi, S, Greiser-Wilke, I, Vilcek, S, Amoako, K, et al. Una RT-PCR multiplexada en tiempo real de un solo paso para la detección y tipificación de los virus de la diarrea viral bovina. Microbiol veterinario. (2006) 116:37–44. doi: 10.1016/j.vetmic.2006.03.026
28. Richt, JA, Lager, KM, Clouser, DF, Spackman, E, Suárez, DL y Yoon, KJ. Ensayos de reacción en cadena de la polimerasa con transcriptasa inversa en tiempo real para la detección y diferenciación de los virus de la influenza porcina de América del Norte. J Vet Diagn Invest. (2004) 16:367–73. doi: 10.1177/104063870401600501
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Joshi, VG, Chaudhary, D, Bansal, N, Singh, R, Maan, S, Mahajan, NK, et al. Prevalencia del virus de la enfermedad de Newcastle en aves de corral comerciales y domésticas en Haryana, India. Front Vet Sci. (2021) 8:725232. doi: 10.3389/fvets.2021.725232
30. Armstrong, PM, Prince, N, y Andreadis, TG. Desarrollo de un ensayo TaqMan multiobjetivo para detectar variantes del virus de la encefalitis equina oriental en mosquitos. Vector Transmitido Zoonótico Dis. (2012) 12:872–6. doi: 10.1089/vbz.2012.1008
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Dall Agnol, AM, Otonel, RAA, Leme, RA, Alfieri, AA, y Alfieri, AF. Un ensayo qRT-PCR basado en TaqMan para la detección de Senecavirus, una detección en muestras de tejido de lechones neonatos. Sondas de células molares. (2017) 33:28–31. doi: 10.1016/j.mcp.2017.03.002
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Zhu, JJ . Vacunología de la peste porcina africana: los desafíos biológicos desde perspectivas inmunológicas. Virus. (2022) 14:2021. doi: 10.3390/v14092021
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Ebling, R, Paim, WP, Turner, J, Flory, G, Seiger, J, Whitcomb, C, et al. Viabilidad del virus en tejido de médula ósea porcina con púas durante el método de enterramiento sobre el suelo y en condiciones in vitro. Transbound Emerg Dis. (2022) 69:2987–95. doi: 10.1111/tbed.14462
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Rhee, CH, Her, M y Jeong, W. Virus vaccinia modificado de Ankara como posible sustituto de nivel 2 de bioseguridad para el virus de la peste porcina africana en pruebas de eficacia de desinfectantes. Patógenos. (2022) 11:320. doi: 10.3390/pathogens11030320
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Koonin, EV y Yutin, N. Origen y evolución de los virus eucariotas de ADN nucleocitoplasmático grande. Intervirología. (2010) 53:284–92. doi: 10.1159/000312913
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Dee, SA, Bauermann, FV, Niederwerder, MC, Singrey, A, Clement, T, de Lima, M, et al. Supervivencia de patógenos virales en ingredientes de alimentación animal bajo modelos de transporte marítimo transfronterizo. PLoS Uno. (2018) 13:e0194509. doi: 10.1371/journal.pone.0194509
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Mahy, BWJ, y Van Regenmortel, MHV. Enciclopedia de escritorio de virología animal y bacteriana. Amsterdam; Londres: Academic (2010).
38. Wang, E, Petrakova, O, Adams, AP, Aguilar, PV, Kang, WL, Paessler, S, et al. Las vacunas candidatas contra la encefalitis quimérica sindbis/equina oriental están muy atenuadas y son inmunogénicas en ratones. Vacuna. (2007) 25:7573–81. doi: 10.1016/j.vaccine.2007.07.061
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Dsa, OC, Kadni, TS y N, De la cadena de frío a la temperatura ambiente: transporte de muestras virales: una revisión. Ann Med (2023); 55,:2257711. doi: 10.1080/07853890.2023.2257711
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Perozo, F, Villegas, P, Estevez, C, Alvarado, I, y Purvis, LB. Uso de papel de filtro FTA para la detección molecular del virus de la enfermedad de Newcastle. Pathol aviar. (2006) 35:93–8. doi: 10.1080/03079450600597410
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Moscoso, H, Raybon, EO, Thayer, SG y Hofacre, CL. Detección molecular y serotipificación del virus de la bronquitis infecciosa a partir de papel de filtro FTA. Avian Dis. (2005) 49:24–9. doi: 10.1637/7220
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Abdelwhab, EM, Luschow, D, Harder, TC, y Hafez, HM. El uso de papeles de filtro FTA(R) para el diagnóstico del virus de la influenza aviar. Métodos J Virol. (2011) 174:120–2. doi: 10.1016/j.jviromet.2011.03.017
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Luinstra, K, Petrich, A, Castriciano, S, Ackerman, M, Chong, S, Carruthers, S, et al. Evaluación y validación clínica de un medio de transporte a base de alcohol para la conservación e inactivación de virus respiratorios. J Clin Microbiol. (2011) 49:2138–42. doi: 10.1128/JCM.00327-11
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Weber, DJ, Barbee, SL, Sobsey, MD, y Rutala, WA. Efecto de la sangre sobre la actividad antiviral del hipoclorito de sodio, un compuesto fenólico y un compuesto de amonio cuaternario. Infect Control Hosp Epidemiol. (1999) 20:821–7. doi: 10.1086/501591
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. Welch, JL, Xiang, J, Mackin, SR, Perlman, S, Thorne, P, O’Shaughnessy, P, et al. Inactivación del virus respiratorio agudo grave del coronavirus 2 (SARS-CoV-2) y diversos virus de ARN y ADN en materiales de mascarillas quirúrgicas impresas tridimensionalmente. Infect Control Hosp Epidemiol. (2021) 42:253–60. doi: 10.1017/ice.2020.417
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. Al Bayat, S, Mundodan, J, Hasnain, S, Sallam, M, Khogali, H, Ali, D, et al. ¿Puede el valor del umbral del ciclo (Ct) de la prueba RT-PCR para SARS CoV2 predecir la infectividad entre los contactos cercanos? J Infectar la salud pública. (2021) 14:1201–5. doi: 10.1016/j.jiph.2021.08.013
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Morris, DH, Yinda, KC, Gamble, A, Rossine, FW, Huang, Q, Bushmaker, T, et al. La teoría mecanicista predice los efectos de la temperatura y la humedad en la inactivación del SARS-CoV-2 y otros virus con envoltura. elife. (2021) 10:10. doi: 10.7554/eLife.65902
48. Tuladhar, E, De Koning, MC, Fundeanu, I, Beumer, R, y Duizer, E. Diferentes actividades virucidas de recubrimientos hiperramificados de amonio cuaternario en el virus de la poliomielitis y el virus de la influenza. Appl Environ Microbiol. (2012) 78:2456–8. doi: 10.1128/AEM.07738-11
49. Osman, N, Goovaerts, D, Sultan, S, Salt, J y Grund, C. La calidad de la vacuna es un factor clave para determinar la estabilidad térmica de las vacunas comerciales contra la enfermedad de Newcastle (ND). Vacunas. (2021) 9:363. doi: 10.3390/vaccines9040363
50. Choo, JJY, McMillan, CLD, Fernando, GJP, Hall, RA, Young, PR, Hobson-Peters, J, et al. Desarrollo de una formulación estabilizadora de una vacuna viva contra el virus quimérico del dengue recubierta en seco en un parche de microarrays de alta densidad. Vacunas. (2021) 9:1301. doi: 10.3390/vacunas9111301
Resumen de PubMed | Texto completo de Crossref | Google Académico
51. Newman, FK, Frey, SE, Blevins, TP, Yan, L, y Belshe, RB. Estabilidad de la vacuna contra el virus vaccinia sin diluir y diluida, Dryvax. J Infectar Dis. (2003) 187:1319–22. doi: 10.1086/374564
Resumen de PubMed | Texto completo de Crossref | Google Académico
52. Caserta, LC, Noll, JCG, Singrey, A, Niederwerder, MC, Dee, S, Nelson, EA, et al. Estabilidad del Senecavirus a en los ingredientes de la alimentación animal y la infección tras el consumo de piensos contaminados. Transbound Emerg Dis. (2022) 69:88–96. doi: 10.1111/tbed.14310
Resumen de PubMed | Texto completo de Crossref | Google Académico
53. Muhsen, M, Aoki, H, Ikeda, H y Fukusho, A. Propiedades biológicas de las cuasiespecies del virus de la diarrea viral bovina detectadas en la línea celular RK13. Arch Virol. (2013) 158:753–63. doi: 10.1007/s00705-012-1538-x
54. Cardoso, N, Franco-Mahecha, OL, Czepluch, W, Quintana, ME, Malacari, DA, Trotta, MV, et al. El virus de la diarrea viral bovina infecta las células dendríticas bovinas derivadas de monocitos mediante un mecanismo mediado por la glicoproteína E2 y altera transitoriamente la presentación del antígeno. Inmunología Viral. (2016) 29:417–29. doi: 10.1089/vim.2016.0047
Resumen de PubMed | Texto completo de Crossref | Google Académico
56. Colenutt, C, Brown, E, Nelson, N, Paton, DJ, Eble, P, Dekker, A, et al. Cuantificar la transmisión del virus de la fiebre aftosa en el ganado a través de un medio contaminado. MBio. (2020) 11:E00381-20. doi: 10.1128/mBio.00381-20
Resumen de PubMed | Texto completo de Crossref | Google Académico
57. Guinat, C, Reis, AL, Netherton, CL, Goatley, L, Pfeiffer, DU y Dixon, L. Dinámica de la excreción y excreción del virus de la peste porcina africana en cerdos domésticos infectados por inoculación intramuscular y transmisión por contacto. Res. Veterinaria. (2014) 45:93. doi: 10.1186/s13567-014-0093-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
58. Muñoz-González, S, Ruggli, N, Rosell, R, Pérez, LJ, Frías-Leuporeau, MT, Fraile, L, et al. Infección postnatal persistente por el virus de la peste porcina clásica y sus implicaciones inmunológicas. PLoS Uno. (2015) 10:e0125692. doi: 10.1371/journal.pone.0125692
Resumen de PubMed | Texto completo de Crossref | Google Académico
59. Honnold, SP, Mossel, EC, Bakken, RR, Lind, CM, Cohen, JW, Eccleston, LT, et al. Virus de la encefalitis equina oriental en ratones II: la patogenia depende de la vía de exposición. Virol J. (2015) 12:154. doi: 10.1186/s12985-015-0385-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
60. Jacobsen, H, Walendy-Gnirss, K, Tekin-Bubenheim, N, Kouassi, NM, Ben-Batalla, I, Berenbrok, N, et al. Las crías nacidas con ratones preñados infectados con el virus de la influenza A tienen una mayor susceptibilidad a las infecciones virales y bacterianas en los primeros años de vida. Nat Commun. (2021) 12:4957. doi: 10.1038/s41467-021-25220-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
61. Hu, Z, Hu, J, Hu, S, Song, Q, Ding, P, Zhu, J, et al. Los altos niveles de replicación del virus y una intensa respuesta inflamatoria contribuyen a la patología grave en los tejidos linfoides causada por el genotipo VIId del virus de la enfermedad de Newcastle. Arch Virol. (2015) 160:639–48. doi: 10.1007/s00705-014-2301-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
62. Kandeil, A, Patton, C, Jones, JC, Jeevan, T, Harrington, WN, Trifkovic, S, et al. Rápida evolución de los virus de la influenza a(H5N1) después de la propagación intercontinental a América del Norte. Nat Commun. (2023) 14:3082. doi: 10.1038/s41467-023-38415-7
Resumen de PubMed | Texto completo de Crossref | Google Académico
63. van Bockel, D, Munier, CML, Turville, S, Badman, SG, Walker, G, Stella, AO, et al. Evaluación del medio de transporte viral (VTM) disponible comercialmente para la inactivación del SARS-CoV-2 y su uso en pruebas en el punto de atención (POC). Virus. (2020) 12:1208. doi: 10.3390/v12111208
64. Schlaudecker, EP, Heck, JP, MacIntyre, ET, Martínez, R, Dodd, CN, McNeal, MM, et al. Comparación de un nuevo medio de transporte con un medio de transporte universal en un campo tropical. Diagn Microbiol Infect Dis. (2014) 80:107–10. doi: 10.1016/j.diagmicrobio.2014.05.018
Resumen de PubMed | Texto completo de Crossref | Google Académico
65. Kockler, ZW, y Gordenin, DA. Del mundo del ARN al SARS-CoV-2: la historia editada de la evolución viral del ARN. Célula. (2021) 10:1557. doi: 10.3390/cells10061557
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: virus animal, diagnóstico molecular, inactivación, virus de la gripe, medios moleculares de transporte
Cita: Welch JL, Shrestha R, Hutchings H, Pal N, Levings R, Robbe-Austerman S, Palinski R y Shanmuganatham KK (2024) Inactivación de virus aviares y ganaderos altamente transmisibles, incluidos la influenza A y el virus de la enfermedad de Newcastle para diagnósticos moleculares. Frente. Vet. Sci. 11:1304022. doi: 10.3389/fvets.2024.1304022
Recibido: 29 de septiembre de 2023; Aceptado: 06 de febrero de 2024;
Publicado: 07 Marzo 2024.
Editado por:
Binu Velayudhan, Universidad de Georgia, Estados Unidos
Revisado por:
Grazieli Maboni, Universidad de Georgia, Estados
Unidos Nagendrakumar Singanallur Balasubramanian, Laboratorio Australiano de Sanidad Animal (CSIRO), Australia
Derechos de autor © 2024 Welch, Shrestha, Hutchings, Pal, Levings, Robbe-Austerman, Palinski y Shanmuganatham. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Karthik K. Shanmuganatham, karthik.k.shanmuganatham@usda.gov
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

