Inhibición de la respuesta del IFN por el virus de la lengua azul

Inhibición de la respuesta del IFN por el virus de la lengua azul: la historia hasta ahora
 José Manuel Rojas*,
José Manuel Rojas*,  Miguel Avia,
Miguel Avia,  Verónica Martín y
Verónica Martín y  Noemí Sevilla*
Noemí Sevilla*Centro de Investigación en Sanidad Animal (CISA-INIA), Centro Nacional Instituto de Investigación y Tecnología Agraria y Alimentaria, Consejo Superior de Investigaciones Científicas (CSIC), Madrid, España
El virus de la lengua azul (BTV) es el orbivirus prototípico que pertenece a la familia Reoviridae. La infección por el virus de la lengua azul produce una enfermedad en los rumiantes, particularmente en las ovejas, que resulta en pérdidas económicas a través de la reducción de la productividad. El virus de la lengua azul se transmite por la picadura de los mosquitos Culicoides spp. y hoy en día se distribuye globalmente en regiones subtropicales e incluso templadas. Como la mayoría de los virus, el virus de la lengua azul es susceptible a la respuesta al IFN, la primera línea de defensa empleada por el sistema inmunitario para combatir las infecciones virales. A su vez, el BTV ha desarrollado estrategias para contrarrestar la respuesta del IFN y promover su replicación. En la presente revisión revisaremos los trabajos que describen cómo el virus de la lengua azul interfiere con la respuesta del IFN.
Introducción
El sistema de interferón (IFN) está presente en todos los vertebrados y es fundamental para la inmunidad antiviral. Las células pueden responder a la infección viral secretando IFN que advierten a las células vecinas del riesgo continuo y desencadenan una programación que hace que las células sean refractarias a la infección. La exposición celular al IFN provoca la expresión de más de 2,000 genes estimulados por interferón (ISG) y reprime la expresión de casi 1,500 genes en humanos que finalmente promueve un estado antiviral (Shaw et al., 2017). El estado antiviral es esencial para la protección contra la infección viral, ya que los defectos en las vías de señalización involucradas en la inducción o señalización de IFN conducen a una mayor susceptibilidad a los patógenos virales. Las respuestas de IFN también son críticas para el desarrollo de una inmunidad adaptativa adecuada.
Las células pueden detectar la presencia viral a través de receptores de reconocimiento de patrones (PRR) que reconocen patrones moleculares asociados a patógenos (PAMP) que incluyen ácidos nucleicos, proteínas y carbohidratos relacionados con patógenos (Goubau et al., 2013; Ma y Damania, 2016; McFadden et al., 2017; Abe et al., 2019). Típicamente, uno de los principales PAMP reconocidos durante la infección viral consiste en los ácidos nucleicos virales y sus intermedios producidos durante la replicación viral (Chan y Gack, 2016; Rojas et al., 2021a). La activación de PRRs por PAMPs desencadena varias cascadas de señalización entre las que se incluyen vías que resultan en la inducción de IFN. Una vez que se producen los IFN, pueden enviar señales a las células vecinas y desencadenar el estado antiviral. Los IFN se pueden clasificar en tres grupos de acuerdo con los receptores que utilizan para la señalización (revisado en Pestka et al., 2004). Los IFN de tipo I (IFN-I), que incluyen IFN-αs e IFN-β, se envían señales a través del receptor IFN tipo I (IFNAR) expresado ubicuamente. El IFN tipo II (IFN-II), que solo incluye IFN-γ y es producido principalmente por células inmunes, envía señales a través del receptor de IFN tipo II (IFNGR). Finalmente, los IFN tipo III (IFN-III), o IFN-λs, señalan a través del receptor IFN tipo III que se expresa principalmente en las células epiteliales. El rango de actividades de IFN-I e IFN-III se centra principalmente en la inmunidad antiviral, mientras que el IFN-γ está involucrado principalmente en la modulación de la inmunidad adaptativa, aunque también posee propiedades antivirales (McNab et al., 2015).
El objetivo de la presente revisión no es describir completamente estas vías de activación, sino abordarlas de manera concisa en el contexto de la infección por el virus de la lengua azul (BTV). Se han publicado extensas revisiones sobre la detección de virus y los eventos que conducen a la inducción y señalización de IFN en otros lugares si el lector busca información más detallada (Goubau et al., 2013; McNab et al., 2015; Chan y Gack, 2016; Ma y Damania, 2016; McFadden et al., 2017; Abe et al., 2019; Rojas et al., 2021a).
El sistema IFN: de la inducción de IFN a la producción de ISG
Como se mencionó anteriormente, las células pueden detectar el genoma viral a través de PRR. Estos se pueden clasificar ampliamente en cinco grupos: receptores tipo Toll (revisados en Finberg et al., 2007), receptores similares al gen I INDUCIBLES POR ÁCIDO RETINOICO (RIG-I) (RLR) (revisados en Rehwinkel y Gack, 2020), receptores similares a dominios de oligomerización de nucleótidos (NLR) (revisados en Jacobs y Damania, 2012); helicasas no RLR DEXD/H-box (revisadas en Chow et al., 2018); y sensores de ADN citosólico (revisado en Ma et al., 2018). Los virus de ARN bicatenario (dsRNA), como el BTV, son típicamente detectados por RLR en el citoplasma (Figura 1). Estos incluyen RIG-I y el factor 5 asociado a la diferenciación del melanoma (MDA-5), los cuales detectan diferentes motivos de dsRNA (Rehwinkel y Gack, 2020). Mientras que las RLR se expresan ubicuamente, la expresión de TLR se limita principalmente a las células del linaje hematopoyético. TLR3 es el sensor prototípico de dsRNA que reconoce este material genético en endosomas (Finberg et al., 2007). El reconocimiento del material genético viral desencadena una cascada de señalización que generalmente conduce a la inducción de IFN-I y la producción de citoquinas proinflamatorias. RIG-I y MDA-5 poseen dos dominios de activación y reclutamiento de caspasas (CARD) que se liberan con la activación de RLR. Por lo tanto, las CARD RLR liberadas pueden interactuar con la CARD de la proteína de señalización antiviral mitocondrial (MAVS). Esto forma agregados similares a priones que son esenciales para el reclutamiento MAVS de los factores asociados al receptor del factor de necrosis tumoral (TRAF) responsables de la señalización aguas abajo (Ren et al., 2020). Los TRAF promueven la activación del complejo TANK binding kinase 1 (TBK1) [compuesto por TBK1, IκB quinasa ε (IKKε) y NF-κB essential modulator (NEMO)], que inducen la inducción de IFN, y el complejo IKK (compuesto por IKKα y β, y NEMO) que activa NF-κB y la transcripción de citoquinas proinflamatorias (Yu et al., 2012). El complejo TBK1 activado puede entonces mediar la fosforilación y homodimerización de los factores reguladores de IFN (IRF) 3/7 que, en consecuencia, se translocan al núcleo para desencadenar la transcripción de genes de respuesta temprana de IFN, como IFN-I.
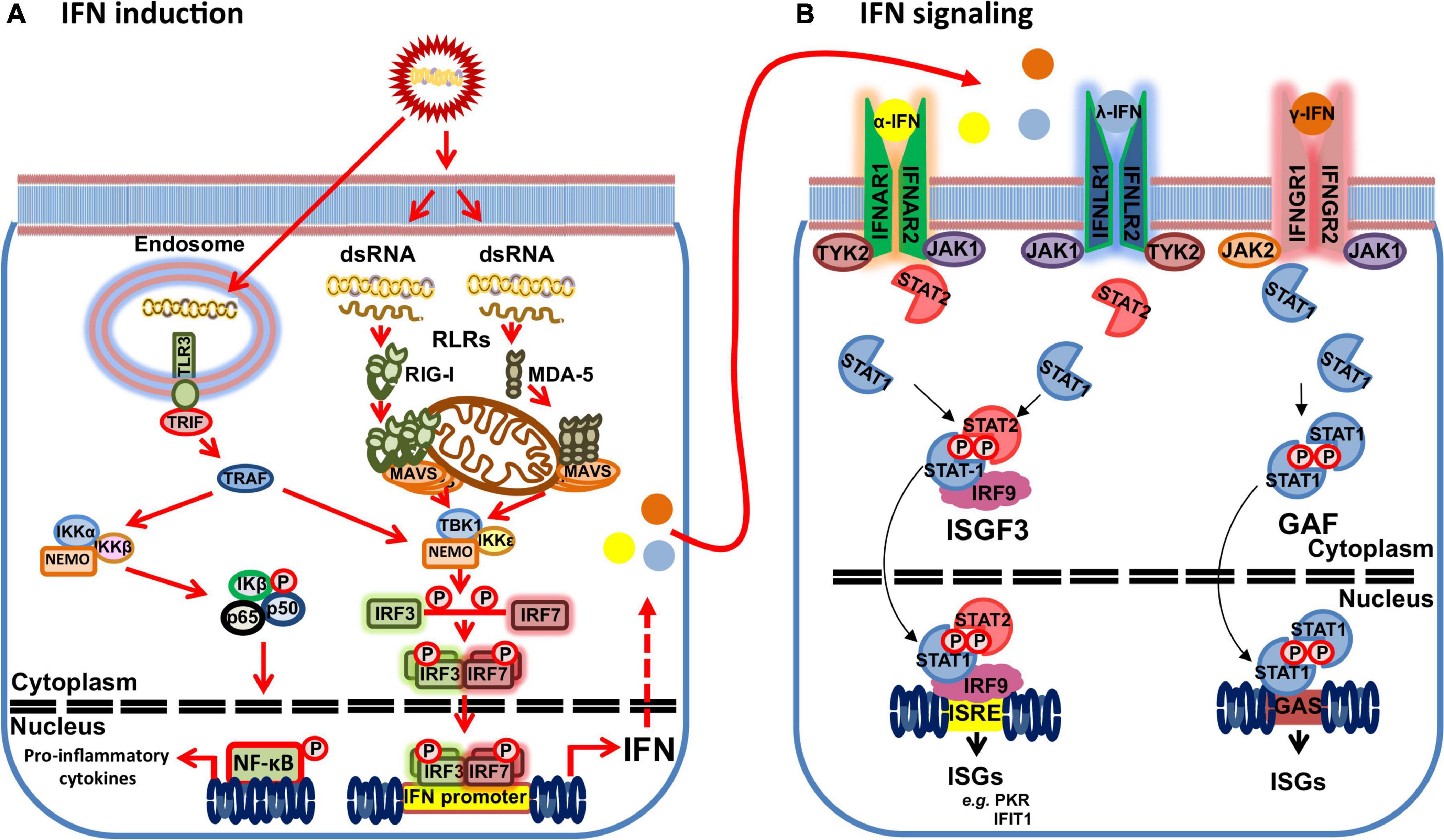 FIGURA 1. Esquema de la respuesta de IFN a los virus dsRNA. (A) Los motivos de dsRNA pueden ser reconocidos por receptores similares al gen I INDUCIBLES POR ÁCIDO RETINOICO (RIG-I) (RLR) (RIG-I y MDA-5) en el citoplasma o receptores tipo Toll (TLR) 3 en los endosomas que desencadenan la producción de IFN y citoquinas proinflamatorias. Los RLR activados se reclutan para la proteína de señalización antiviral mitocondrial (MAVS), que a su vez recluta factores asociados al receptor del factor de necrosis tumoral (TRAF). TLR3 señala a través de su adaptador adaptador que contiene el dominio Toll/IL-1R que induce al IFN-β (TRIF) a activar los TRAF. Los TRAF promueven la activación del complejo TANK binding kinase 1 (TBK1) [compuesto por TBK1, IκB quinasa ε (IKKε) y NF-κB essential modulator (NEMO)] que conduce a la activación de TBK1 que media la fosforilación y homodimerización de los factores reguladores de IFN (IRF) 3/7. Los dímeros IRF3/7 activados se translocan al núcleo para desencadenar la transcripción del gen IFN. La activación de TRAF también promueve la activación del complejo IKK (compuesto por IKKα y β, y NEMO) que activa NF-κB y la transcripción de citoquinas proinflamatorias. (B) señales de IFNs a través de sus receptores de superficie heterodiméricos (IFNAR1 y 2 para α-IFN; IFNLR 1 y 2 para λ-IFN; e IFNGR 1 y 2 para γ-IFN) que conducen a la activación de las vías intracelulares Janus quinasa (JAK)/transductor de señal y activador de la transcripción (STAT) y a la transcripción de genes estimulados por IFN (ISG). La unión del α-IFN a su receptor activa las quinasas JAK JAK1 y TYK2, que a su vez conduce a la fosforilación de STAT1 y STAT2. STAT1 y STAT2 fosforilados dimerizan y se unen a IRF9 para formar el factor transcripcional complejo IFN estimulado factor génico (ISGF) 3 que se transloca al núcleo para unirse a los elementos de respuesta IFN (ISRE) en promotores ISG e impulsar la expresión de productos ISG, como la proteína quinasa R (PKR) o proteínas inducidas por interferón con repeticiones de tetratricopéptidos (IFIT) 1. La señalización de λ-IFN sigue esta misma vía canónica que la señalización de α-IFN. γ-IFN señala a través de su receptor mediante la activación de las quinasas JAK JAK1 y JAK2 que conduce a la fosforilación de STAT1 y la homodimerización en el factor de transcripción denominado factor activado por interferón gamma (GAF). GAF luego se une al sitio activado por interferón gamma (GAS) para impulsar la transcripción de ISG dependientes de γ-IFN.
FIGURA 1. Esquema de la respuesta de IFN a los virus dsRNA. (A) Los motivos de dsRNA pueden ser reconocidos por receptores similares al gen I INDUCIBLES POR ÁCIDO RETINOICO (RIG-I) (RLR) (RIG-I y MDA-5) en el citoplasma o receptores tipo Toll (TLR) 3 en los endosomas que desencadenan la producción de IFN y citoquinas proinflamatorias. Los RLR activados se reclutan para la proteína de señalización antiviral mitocondrial (MAVS), que a su vez recluta factores asociados al receptor del factor de necrosis tumoral (TRAF). TLR3 señala a través de su adaptador adaptador que contiene el dominio Toll/IL-1R que induce al IFN-β (TRIF) a activar los TRAF. Los TRAF promueven la activación del complejo TANK binding kinase 1 (TBK1) [compuesto por TBK1, IκB quinasa ε (IKKε) y NF-κB essential modulator (NEMO)] que conduce a la activación de TBK1 que media la fosforilación y homodimerización de los factores reguladores de IFN (IRF) 3/7. Los dímeros IRF3/7 activados se translocan al núcleo para desencadenar la transcripción del gen IFN. La activación de TRAF también promueve la activación del complejo IKK (compuesto por IKKα y β, y NEMO) que activa NF-κB y la transcripción de citoquinas proinflamatorias. (B) señales de IFNs a través de sus receptores de superficie heterodiméricos (IFNAR1 y 2 para α-IFN; IFNLR 1 y 2 para λ-IFN; e IFNGR 1 y 2 para γ-IFN) que conducen a la activación de las vías intracelulares Janus quinasa (JAK)/transductor de señal y activador de la transcripción (STAT) y a la transcripción de genes estimulados por IFN (ISG). La unión del α-IFN a su receptor activa las quinasas JAK JAK1 y TYK2, que a su vez conduce a la fosforilación de STAT1 y STAT2. STAT1 y STAT2 fosforilados dimerizan y se unen a IRF9 para formar el factor transcripcional complejo IFN estimulado factor génico (ISGF) 3 que se transloca al núcleo para unirse a los elementos de respuesta IFN (ISRE) en promotores ISG e impulsar la expresión de productos ISG, como la proteína quinasa R (PKR) o proteínas inducidas por interferón con repeticiones de tetratricopéptidos (IFIT) 1. La señalización de λ-IFN sigue esta misma vía canónica que la señalización de α-IFN. γ-IFN señala a través de su receptor mediante la activación de las quinasas JAK JAK1 y JAK2 que conduce a la fosforilación de STAT1 y la homodimerización en el factor de transcripción denominado factor activado por interferón gamma (GAF). GAF luego se une al sitio activado por interferón gamma (GAS) para impulsar la transcripción de ISG dependientes de γ-IFN.
La producción de IFN-I actúa como una señal de advertencia en las células vecinas que provoca un estado antiviral que protege de la infección. Señal de IFN-I a través del receptor heterodimérico de IFNα (IFNAR) compuesto por α y β subunidades transmembrana (Pestka et al., 2004). La unión del IFN al receptor provoca una cascada de señalización a través de las vías de la familia Janus quinasa (JAK)/transductor de señal y activador de la transcripción (STAT) (Figura 1; Majoros et al., 2017; Nan et al., 2017). La unión de IFN-I conduce a la fosforilación secuencial de las quinasas JAK Jak1 y Tyk2, que a su vez fosforilan STAT1 y STAT2. STAT1 y STAT2 fosforilados heterodimerizan y se unen a IRF9 para formar el complejo de factor transcripcional IFN estimulado por el factor gen (ISGF) 3. ISGF3 luego se transloca al núcleo y se une a los elementos de respuesta de IFN (ISRE) en los promotores de ISG que conducen a la expresión de productos ISG. Del mismo modo, la señalización de IFN-II se transduce a través de las vías JAK/STAT. La unión de IFN-II a su receptor desencadena la fosforilación de Jak1 y Jak2, que a su vez conduce a la fosforilación de STAT1. El STAT1 fosforilado homodimeriza y se transloca al núcleo para unirse al sitio activado por interferón gamma (GAS) y, por lo tanto, impulsar la transcripción de ISG dependientes de IFN-II (Majoros et al., 2017).
La expresión de ISG proporciona a la célula mecanismos para combatir la infección viral como resultado de la estimulación de IFN (revisado en Schneider et al., 2014). Las respuestas de IFN también reprimen parte de la expresión génica (Shaw et al., 2017), aunque estas no están tan ampliamente estudiadas y no se discutirán en la presente revisión. También es de destacar que la expresión de ISG difiere entre especies (Shaw et al., 2017), y esto debe considerarse al analizar la respuesta de IFN utilizando herramientas in vitro basadas en especies que difieren del huésped natural de la enfermedad. Los productos ISG promueven múltiples aspectos de la respuesta antiviral celular. Pueden, entre otras cosas, cooperar en el reconocimiento PRR de PAMP virales, bloquear la entrada del virus, estabilizar complejos de señalización, obstaculizar la formación de cápside viral o perjudicar la salida del virión de las células infectadas. Algunos productos ISG también modulan la respuesta de IFN para prevenir la toxicidad de estos potentes mediadores inmunes. Entre los ISG, la proteína quinasa R (PKR) que detecta el dsRNA citosólico pertenece a una de las llamadas vías clásicas de ISG. La forma activada de PKR regula el inicio de la traducción por medio de la fosforilación de la subunidad alfa del factor iniciador eIF2 eucariota (Williams, 1999). PKR también participa en otros mecanismos en el estado antiviral, como la inducción de apoptosis (Gil y Esteban, 2000), la regulación de la síntesis de IFN-β y de la vía de citoquinas proinflamatorias mediada por NF-κB (Kirchhoff et al., 1995; Kumar y otros, 1997; Chu et al., 1999), o modulación de la actividad STAT1 en la vía de señalización IFN-I (Wong et al., 1997). En general, el reconocimiento PKR de dsRNA limita la replicación viral a través de estos mecanismos. Otra familia de ISG con funciones antivirales son las proteínas inducidas por interferón con repeticiones de tetratricopéptidos (IFIT) que incluye cuatro miembros en humanos (IFIT1, 2, 3 y 5) y tres en ratones (IFIT1, 2 y 3) (revisado en Vladimer et al., 2014). La transcripción de estos genes aumenta rápidamente después de la activación por señalización de IFN, pero también después del reconocimiento de PAMPs virales. IFIT1 detecta la ausencia de 2′-O-metilación en especies de ARN, una metilación presente en el ARNm eucariota pero que carece de algún ARN viral (Habjan et al., 2013). IFIT1 también reconoce el extremo 5′-triP presente en algunos ARN virales (Abbas et al., 2013). IFIT1 puede secuestrar ARN virales para evitar su transcripción y también inhibir el inicio de la traducción de estas especies de ARN unidas a IFIT1 por el factor de iniciación de traducción 3, proporcionando así a las células medios para limitar la replicación viral.
Por lo tanto, la respuesta de IFN está mediada por un complejo sistema de reconocimiento de PAMP que desencadena vías de señalización que conducen a la traducción de productos ISG efectores que limitan la replicación viral. Los virus a menudo pueden interferir en múltiples puntos de estas vías para que puedan replicarse y continuar su ciclo infeccioso a pesar de la respuesta al IFN. En esta revisión, revisaremos el conocimiento actual sobre las interacciones del virus de la lengua azul (BTV) con la respuesta IFN del huésped mamífero.
Virus de la lengua azul (BTV)
El virus de la lengua azul (BTV) es el orbivirus prototípico que pertenece a la familia Reoviridae. Los orbivirus son virus transmitidos por artrópodos (arbovirus) que se replican en huéspedes artrópodos y vertebrados (revisado en Attoui et al., 2016). La infección del huésped vertebrado generalmente está mediada por la picadura de garrapatas o insectos hematófagos. En el caso del BTV, el virus se transmite al huésped rumiante por la picadura de mosquitos Culicoides spp. infectados (Baylis et al., 2008). Produce una enfermedad económicamente importante de declaración obligatoria a la OIE (Organización Mundial de Sanidad Animal) que limita la productividad en pequeños rumiantes (revisado en Rushton y Lyons, 2015). Durante los brotes, pueden producirse altas tasas de mortalidad en rebaños ingenuos, mientras que en las regiones endémicas la limitación del movimiento de animales, las pérdidas de producción (por ejemplo, las roturas de lana) y la reducción de la fertilidad pueden obstaculizar la rentabilidad. La existencia de múltiples serotipos que confieren poca protección cruzada en términos de inmunidad esterilizante entre serotipos, y la posibilidad de formación de reordenados cuando múltiples cepas de virus de la lengua azul están cocirculando dentro de un territorio complica el control de esta enfermedad. Como tal, el virus de la lengua azul se ha establecido como una enfermedad endémica en las latitudes del sur de Europa con frecuentes incursiones en las latitudes septentrionales a finales del verano y el otoño (Baylis et al., 2017).
El virus de la lengua azul posee un genoma segmentado que consta de 10 segmentos de dsRNA que codifican para 7 proteínas estructurales y 4-5 no estructurales (Figura 2). La partícula BTV consiste en una cápside externa formada por proteínas VP2 y VP5 que media la unión y entrada celular, y una cápside interna (núcleo) formada por proteínas VP7 y VP3 que encapsularon el material genético, así como la ARN polimerasa VP1, la enzima de recubrimiento de ARN y metil transferasa VP4, y la helicasa de ARN VP6 (revisado en Roy, 2005, 2017). El virus también codifica para al menos cuatro proteínas no estructurales denominadas NS1 a NS4. NS1 forma estructuras tubulares en el citoplasma y promueve la expresión de proteínas virales a través de un mecanismo que implica la transición de la forma tubular NS1 a una forma activa no tubular (Boyce et al., 2012; Kerviel et al., 2019). NS2 es una proteína de unión al ARN que es el componente principal de los cuerpos de inclusión viral (VIB) y desempeña un papel crítico en la formación del núcleo interno (Kar et al., 2007). NS3 (y la isoforma más corta NS3a que está codificada por un ORF alternativo en el marco) está involucrado en la salida del virión (Han y Harty, 2004; Wirblich y otros, 2006; Celma y Roy, 2009; Labadie et al., 2019). La NS3 también ha sido identificada como antagonista de la respuesta del IFN (Chauveau et al., 2013; Avia et al., 2019) como se discutirá en esta revisión. NS4 está codificado por un ORF alternativo en el segmento 9 (Belhouchet et al., 2011; Ratinier et al., 2011) y también actúa como antagonista del IFN (Ratinier et al., 2016). Finalmente, se ha identificado un ORF alternativo en el segmento 10 que podría codificar para una proteína NS5 no estructural adicional (Stewart et al., 2015), aunque su expresión durante la infección y su papel aún no se han aclarado.
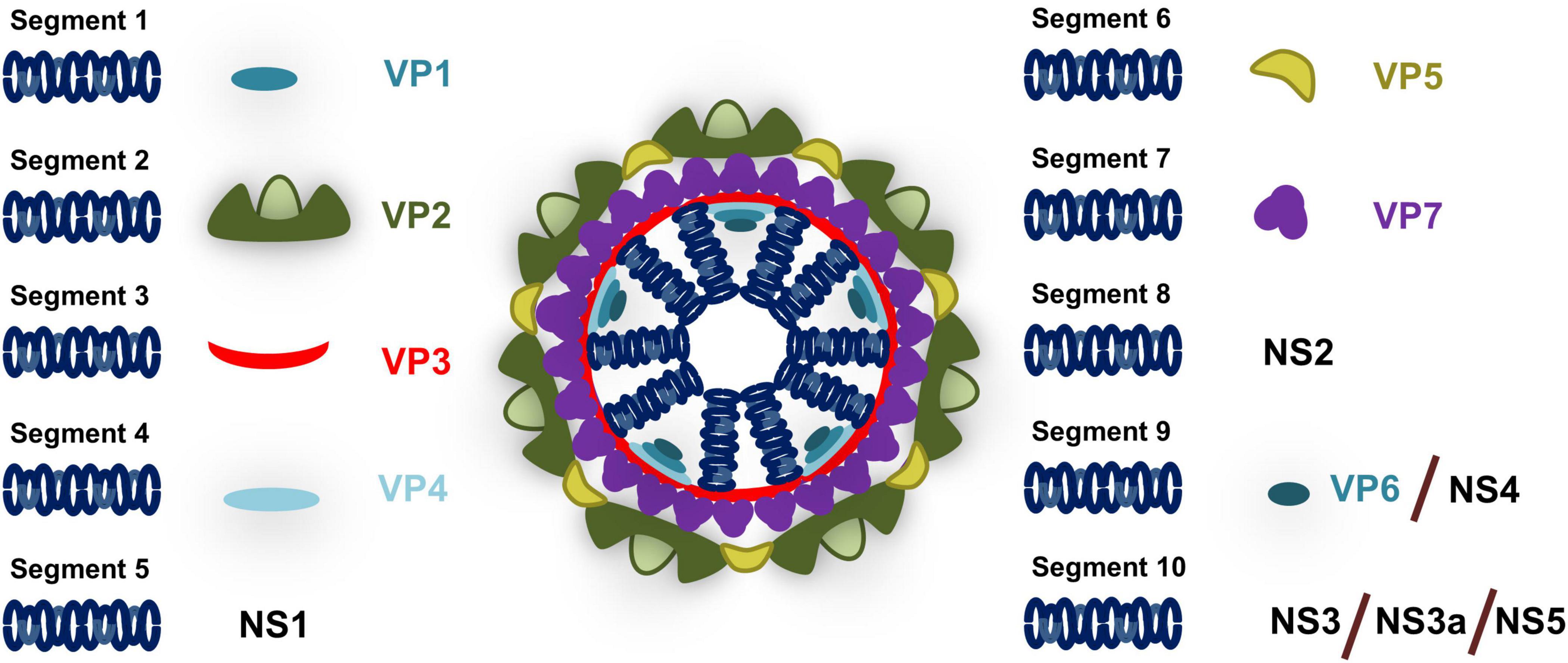 FIGURA 2. Esquema de la partícula de BTV. El genoma del virus de la lengua azul consta de 10 segmentos que codifican proteínas estructurales (VP) presentes en la partícula viral y proteínas no estructurales (NS) que se expresan durante la infección. El segmento 1 codifica para la ARN polimerasa VP1; Segmento 2 para la proteína VP2 de la cápside externa; Segmento 3 para la proteína del núcleo interno VP3; Segmento 4 para la enzima de recubrimiento de ARN y la metiltransferasa VP4; Segmento 5 para NS1 que es responsable de la formación de estructuras tubulares durante la infección; Segmento 6 para la proteína VP5 de la cápside externa; Segmento 7 para la proteína del núcleo interno VP7; Segmento 8 para la proteína de unión al ARN NS2 que participa en la formación del núcleo interno; Segmento 9 para la helicasa de ARN VP6 y para NS4 que actúa como antagonista del IFN; y el segmento 10 para NS3 (y su isoforma NS3a) que está involucrado en la salida del virión y el antagonismo de IFN y supuestamente para NS5 que podría estar involucrado en el apagado celular.
FIGURA 2. Esquema de la partícula de BTV. El genoma del virus de la lengua azul consta de 10 segmentos que codifican proteínas estructurales (VP) presentes en la partícula viral y proteínas no estructurales (NS) que se expresan durante la infección. El segmento 1 codifica para la ARN polimerasa VP1; Segmento 2 para la proteína VP2 de la cápside externa; Segmento 3 para la proteína del núcleo interno VP3; Segmento 4 para la enzima de recubrimiento de ARN y la metiltransferasa VP4; Segmento 5 para NS1 que es responsable de la formación de estructuras tubulares durante la infección; Segmento 6 para la proteína VP5 de la cápside externa; Segmento 7 para la proteína del núcleo interno VP7; Segmento 8 para la proteína de unión al ARN NS2 que participa en la formación del núcleo interno; Segmento 9 para la helicasa de ARN VP6 y para NS4 que actúa como antagonista del IFN; y el segmento 10 para NS3 (y su isoforma NS3a) que está involucrado en la salida del virión y el antagonismo de IFN y supuestamente para NS5 que podría estar involucrado en el apagado celular.
BTV y la respuesta del IFN
El virus de la lengua azul es sensible a la respuesta del IFN, pero puede superarla en rumiantes
Al igual que para la mayoría de los virus, la replicación del virus de la lengua azul puede verse afectada in vitro por la adición exógena de IFN-I. Doceul et al. (2014) mostraron que la adición de IFN-β a las células epiteliales A549 alveolares humanas y a las células Hela alteró la replicación viral en 2 serotipos de BTV. El tratamiento de las células CPT-Tert ovinas con IFN universal tipo I o IFN-τ ovino tipo I antes de las infecciones por BTV-1 o BTV-8 también alteró la replicación viral (Ratinier et al., 2011). Sin embargo, el virus de la lengua azul todavía es capaz de replicarse en presencia de IFN (particularmente en células derivadas de su huésped natural) (Ratinier et al., 2011), lo que indica que el virus posee mecanismos para superar los efectos del IFN. Existe evidencia del papel protector del IFN-I durante las infecciones por BTV in vivo. En modelos murinos, los progenitores hematopoyéticos y las células dendríticas (DC) son altamente susceptibles a la infección por BTV en ausencia del receptor IFN-I, mientras que en presencia del receptor resisten la infección (Rodriguez-Calvo et al., 2014). El papel protector del IFN-I durante las infecciones por BTV también se ejemplifica claramente por la susceptibilidad a la infección en ratones con señalización alterada de IFN-I, ya sea a través del knock-out del gen de cadena β del receptor de IFN tipo I [IFNAR(–/–) ratones] (Calvo-Pinilla et al., 2009a) o mediante bloqueo de anticuerpos del receptor (Saminathan et al., 2020). De hecho, el transgénico IFNAR(–/–) El modelo murino se utiliza ahora ampliamente para evaluar la capacidad protectora de las vacunas candidatas para el virus de la lengua azul (Calvo-Pinilla et al., 2009b, 2012; Martin et al., 2015; Marín-López et al., 2018; Rojas et al., 2021b), ya que recapitula algunos de los efectos de la infección aguda por virus de la lengua azul en el huésped natural (Marín-López et al., 2016). También hay evidencia de que el IFN-I limita la replicación del virus de la lengua azul en rumiantes. La inducción temprana de IFN-I probablemente limita la replicación del virus de la lengua azul en terneros infectados, ya que el título de lengua azul aumenta una vez que los niveles de IFN disminuyen en suero (MacLachlan y Thompson, 1985). También se han reportado picos de viremia difásica de BTV en la infección de ovejas, y esto coincidió con la respuesta de IFN-I. La viremia inicial aparece suprimida por la inducción de IFN-I, pero luego alcanza su punto máximo nuevamente una vez que la respuesta de IFN-I disminuye (Foster et al., 1991). Estos datos insinúan claramente un papel protector del IFN-I contra el virus de la lengua azul en el huésped natural. Sin embargo, el virus de la lengua azul es capaz de replicarse en el huésped natural a pesar de una respuesta concomitante de IFN. Melzi et al. (2016) mostraron que la presencia de virus de la lengua azul en el ganglio linfático causó una inducción temprana del ISG MX1, sin embargo, la infección viral progresó en las ovejas. De hecho, la susceptibilidad a la infección por el virus de la lengua azul podría explicarse parcialmente por las diferencias en la inducción de ISG entre especies (Shaw et al., 2017). Esto se ejemplifica por la capacidad del virus de la lengua azul para infectar IFNAR(–/–) ratones, pero no contrapartes de tipo salvaje, lo que indica que los ratones expresan naturalmente una gama de ISG que contrarrestan la replicación del virus de la lengua azul, mientras que los ISG de rumiantes no pueden contrarrestar completamente esto.
La infección por BTV induce IFN
La inducción de IFN durante la infección por el virus de la lengua azul se describió por primera vez en cultivo celular en 1969 utilizando una cepa atenuada de BTV-10 e infectando células embrionarias murinas (Huismans, 1969). Varios grupos confirmaron posteriormente la inducción de IFN en células de rumiantes (Rinaldo et al., 1975; Coen y otros, 1991; Russell y otros, 1996; Chauveau et al., 2012) y en otras células de mamíferos (Jameson y Grossberg, 1979, 1981; Fulton y Pearson, 1982). In vivo, el virus de la lengua azul ha sido descrito como un buen inductor de IFN en ratones (Jameson et al., 1978). La infección por virus de la lengua azul en bovinos y ovinos conduce a la producción de IFN (MacLachlan y Thompson, 1985; Foster y otros, 1991; Ruscanu et al., 2012). El análisis comparativo de la respuesta de IFN-I en células endoteliales bovinas y ovinas no sugiere diferencias importantes en la inducción de IFN in vitro (Russell et al., 1996). De hecho, las células ovinas parecen producir una mayor cantidad de IFN-I que su contraparte bovina. A pesar de esta respuesta de IFN, el virus de la lengua azul todavía era capaz de replicarse productivamente en las células endoteliales (Russell et al., 1996), lo que demuestra que el virus posee mecanismos para superar la respuesta del IFN.
Cabe señalar que la infección por BTV no solo desencadena la respuesta IFN-I en rumiantes, sino que también provoca la producción de citoquinas proinflamatorias in vivo (Channappanavar et al., 2012; Umeshappa y otros, 2012; Sánchez-Cordón et al., 2015; Rojas et al., 2017). La infección in vitro de células bovinas dirigidas por BTV, como células endoteliales o macrófagos, mostró un aumento en las citoquinas proinflamatorias características, como TNF-α, IL-1β e IL-8 (Drew et al., 2010). La respuesta proinflamatoria de citoquinas e IFN generada por la infección por el virus de la lengua azul que, por un lado, probablemente limita la replicación del virus de la lengua azul también podría, por otro lado, explicar algunas de las lesiones vasculares inflamatorias in vivo que normalmente produce la enfermedad. Esto a menudo se conoce como una «tormenta de citoquinas» y explica algunos de los efectos patógenos sobre la vasculatura que desencadena el virus de la lengua azul (Howerth, 2015). Los mediadores proinflamatorios desencadenados por la infección pueden modular la infección por el virus de la lengua azul de las células endoteliales y esto también depende del origen del material endotelial (DeMaula et al., 2001, 2002a, 2002b). Estos estudios también mostraron una sensibilidad diferencial a la muerte celular de las células endoteliales microvasculares pulmonares ovinas tras la exposición a mediadores proinflamatorios y al virus de la lengua azul en comparación con sus homólogos bovinos, lo que podría explicar algunos de los efectos patogénicos del virus de la lengua azul en la vasculatura ovina.
Los eventos tempranos de infección por virus de la lengua azul se han asociado con la infección por CC que permite la propagación del virus al tejido linfoide. Hemati et al. (2009) demostraron elegantemente usando la canulación del conducto linfático aferente en ovejas que la infección por BTV de DC convencional (cDC) en la piel contribuye a la diseminación del virus al ganglio linfático. La infección por cDC no pareció afectar la función de DC in vitro, ya que la activación de DC y la presentación adecuada del antígeno a las células T todavía ocurrieron en las cDC infectadas. Sin embargo, los experimentos in vivo han indicado que la infección por BTV tiene la capacidad de interrumpir la función folicular de DC en el ganglio linfático (Melzi et al., 2016). Melzi et al. (2016) confirmaron que el virus de la lengua azul se disemina al ganglio linfático a través de la linfa, y mostraron que esto se desencadenó como respuesta de IFN como lo demuestra la inducción del ISG MX1 en el ganglio linfático de ovejas infectadas. A pesar de esta respuesta de IFN, el virus de la lengua azul aún interrumpió la respuesta humoral, retrasando la respuesta de anticuerpos y disminuyendo la avidez de anticuerpos a un antígeno modelo, demostrando aún más que el virus de la lengua azul puede replicarse en presencia de mecanismos antivirales inducidos por el IFN (Melzi et al., 2016). Aunque los cDC son probablemente el principal tipo de célula responsable de la diseminación, es poco probable que sean la principal fuente de IFN in vivo. Ruscanu et al. (2012) mostraron que el plasma citoide purificado DC (pDC) de ovejas respondió al BTV produciendo IFN-I, mientras que cDC eran productores pobres de IFN-I. El virus de la lengua azul inactivado por UV también indujo IFN-I, lo que indica que la partícula viral posee PAMP que pueden ser reconocidos por los pDC incluso en ausencia de replicación. El análisis posterior de microarrays de pDC circulantes en ovejas infectadas confirmó que estas células adquieren un perfil proinflamatorio tras la infección que incluye la activación de IFN-I (Ruscanu et al., 2013); apuntando así a este tipo de célula como un sospechoso plausible para desencadenar la patogénesis relacionada con el sistema inmune en las infecciones por el virus de la lengua azul. Curiosamente, las PDC pierden su perfil proinflamatorio dentro de los ganglios linfáticos mientras mantienen su capacidad de respuesta al IFN, lo que ilustra los complejos mecanismos que desempeña el microambiente en la modulación de las respuestas de CC. Tomados en conjunto, estos estudios demuestran que el virus de la lengua azul se detecta fácilmente en caso de infección y que las respuestas de IFN-I están montadas.
Esto nos lleva a la siguiente pregunta: ¿qué PAMP de BTV son reconocidos por el sistema inmune innato? Como se discutió anteriormente, la replicación del virus de la lengua azul no es necesaria para desencadenar la respuesta IFN de las pDC, pero aumentó la respuesta de estas células (Ruscanu et al., 2012). Esto indica que la partícula BTV posee PAMPs fácilmente reconocibles por los PRR. Está bien documentado que el dsRNA de Reoviridae es suficiente para desencadenar la producción de IFN (Tytell et al., 1967; Abad y Danthi, 2020). BTV dsRNA puede inducir respuestas de IFN en ratones (Huismans, 1969), y la transfección con BTV dsRNA puede desencadenar la actividad del promotor IFN-β (Vitour et al., 2014). BTV dsRNA representa, por lo tanto, un motivo reconocible por los PRR celulares. Dado que la replicación del virus de la lengua azul se ha asociado con una mayor producción de IFN-I y la activación del promotor IFN-β (Chauveau et al., 2012; Ruscanu et al., 2012), es probable que los intermediarios de replicación viral también sean detectados por PRRs y esto mejora la respuesta de IFN. Si algunas proteínas del virus de la lengua azul también podrían ser detectadas por PRR, de manera similar a la hemaglutinina de algunos morbillivirus que activa TLR2 (Bieback et al., 2002; Rojas et al., 2021c), no se conoce.
A nivel molecular, se ha demostrado que el virus de la lengua azul activa los factores de transcripción NF-κB, IRF3 e IRF7 al principio de la infección en las células HeLa (Stewart y Roy, 2010), lo que confirma que las infecciones por el virus de la lengua azul tienen el potencial de desencadenar respuestas proinflamatorias mediadas por las respuestas NF-κB e IFN mediadas por IRF3/7. De hecho, Ratinier et al. (2016) mostraron que la infección por BTV de las células A549 desencadenó la translocación nuclear de los complejos IRF3 y NF-κB. Los sensores de ARN citosólico RIG-I y MDA-5 están involucrados en el reconocimiento del virus de la lengua azul durante la infección (Chauveau et al., 2012; Figura 3). Usando siRNA dirigido a RIG-I y MDA-5, Chauveau et al. (2012) mostraron en células A549 que la inducción de IFN-β depende de estas RLR. Además, la regulación positiva de RIG-I y MDA-5 también deterioró la replicación del virus de la lengua azul, lo que demuestra que estos PRR participan en el establecimiento del estado antiviral en las infecciones por el virus de la lengua azul. Chauveau et al. (2012) confirmaron la activación de NF-κB e IRF3 tras la infección por BTV, y mostraron que esta activación dependía de la señalización MAVS aguas arriba de los factores de transcripción y aguas abajo de los sensores citosólicos de ARN RIG-I y MDA-5.
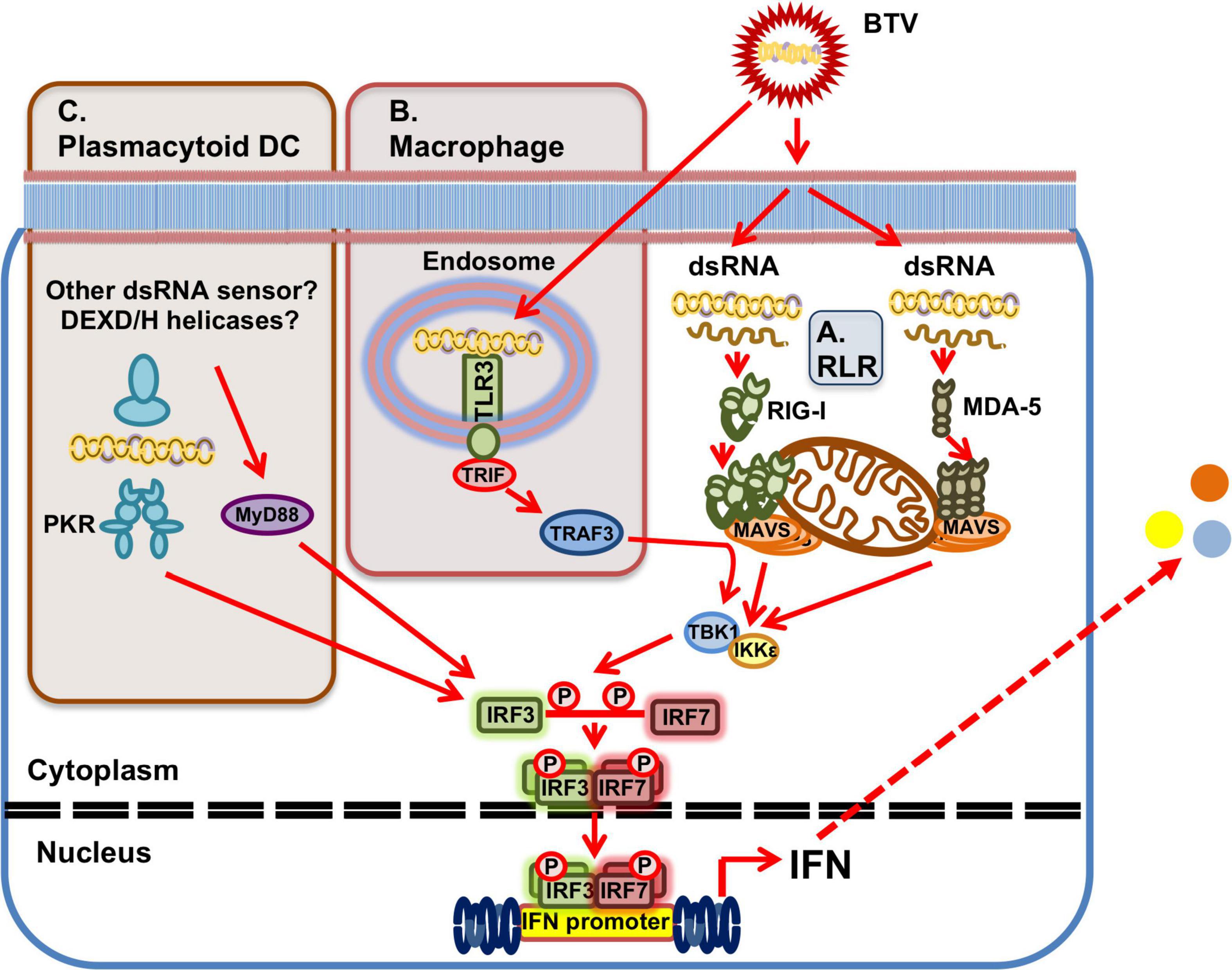 FIGURA 3. Detección de virus de la lengua azul por PRR. (A) Las RLR expresadas ubicuamente pueden detectar la presencia de ARNd del virus de la lengua azul en el citoplasma. RIG-I y MDA-5 están involucrados en el reconocimiento del virus de la lengua azul y esto desencadena una cascada de señalización que da lugar a la inducción de IFN a través de la activación de la vía MAVS/TBK1. (B) En macrófagos, TLR3 en endosomas también puede reconocer el dsRNA del virus de la lengua azul, lo que resulta en la inducción de IFN y la generación de un estado antiviral. (C) En DC, que no expresan TLR3, PKR está implicada en el reconocimiento de dsRNA en el citoplasma y la inducción de IFN. La inducción de IFN en estas células también depende del adaptador MyD88 para la inducción de IFN, pero el reconocimiento es independiente de TLR7. Aún no se ha determinado si otros sensores de dsRNA, como las helicasas DEXD / H-box, están involucrados aguas arriba de MyD88 para la detección de dsRNA en pDC.
FIGURA 3. Detección de virus de la lengua azul por PRR. (A) Las RLR expresadas ubicuamente pueden detectar la presencia de ARNd del virus de la lengua azul en el citoplasma. RIG-I y MDA-5 están involucrados en el reconocimiento del virus de la lengua azul y esto desencadena una cascada de señalización que da lugar a la inducción de IFN a través de la activación de la vía MAVS/TBK1. (B) En macrófagos, TLR3 en endosomas también puede reconocer el dsRNA del virus de la lengua azul, lo que resulta en la inducción de IFN y la generación de un estado antiviral. (C) En DC, que no expresan TLR3, PKR está implicada en el reconocimiento de dsRNA en el citoplasma y la inducción de IFN. La inducción de IFN en estas células también depende del adaptador MyD88 para la inducción de IFN, pero el reconocimiento es independiente de TLR7. Aún no se ha determinado si otros sensores de dsRNA, como las helicasas DEXD / H-box, están involucrados aguas arriba de MyD88 para la detección de dsRNA en pDC.
La detección de BTV PAMP probablemente también variará según los tipos de células. Mientras que los RLR se expresan ubicuamente, los TLR se expresan principalmente en las células inmunes. En cultivos de macrófagos primarios humanos, el virus de la lengua azul activó las respuestas de IFN-I a través del reconocimiento de TLR3 (Dai et al., 2015; Figura 3). Aunque el virus de la lengua azul no puede infectar productivamente estas células, el tratamiento con macrófagos humanos con virus de la lengua azul vivos o virus de la lengua azul inactivados con UV desencadenó un estado antiviral, lo que indica que el sensor de dsRNA TLR3 puede detectar material genético de la lengua azul en los macrófagos (Dai et al., 2015). Como se discutió anteriormente, las pDC ovinas producen IFN-I cuando se infectan con el virus de la lengua azul o cuando internalizan el virus inactivado por UV (Ruscanu et al., 2012). Ruscanu et al. (2012) encontraron que la inducción de IFN-I requería internalización a través del compartimiento endosomal tanto para el virus vivo como para el inactivado, lo que insinuaba la activación mediada por TLR del IFN-I. Sin embargo, los pDC no expresan TLR3 (Liu, 2005), el sensor típico para dsRNA en endosomas, y por lo tanto otros sensores de ARN probablemente están implicados en el reconocimiento de PAMPs de BTV en estas células. Ruscanu et al. (2012) mostraron en pDC ovinas que la señalización de inducción de IFN-I ocurrió, al menos en parte, a través del factor de diferenciación mieloide adaptador 88 (MyD88), que generalmente se asocia a la señalización TLR, pero fue independiente del sensor de ARN TLR7 (Figura 3). La inhibición farmacológica de PKR redujo la respuesta de IFN-I a BTV, lo que sugiere que este sensor está involucrado en el reconocimiento de BTV en estas células (Ruscanu et al., 2012). Otros sensores de ARN también podrían estar implicados en la detección de BTV, como los miembros de la familia de helicasas DEXD / H-box. Se ha demostrado que el sensor de ARN citosólico DHX33 reconoce el dsRNA reoviral y activa el inflamasoma en macrófagos humanos primarios (Mitoma et al., 2013), mientras que se ha demostrado que otros miembros de esta familia detectan dsRNA en CD mieloides (Zhang et al., 2011). Los mecanismos exactos del reconocimiento del virus de la lengua azul en la pDC aún no se han dilucidado completamente y pueden implicar varias vías de reconocimiento de ARN. Dado que las pDC han sido identificadas como las principales células productoras de IFN-I en la infección temprana (Ruscanu et al., 2012), la identificación del mecanismo exacto de inducción de IFN-I en estas células podría proporcionar pistas importantes sobre las interacciones huésped-patógeno.
El virus de la lengua azul emplea múltiples proteínas para perjudicar la respuesta del IFN del huésped
Como se mencionó anteriormente IFNAR(–/–) los ratones son susceptibles a la infección por el virus de la lengua azul (Calvo-Pinilla et al., 2009a) y, por lo tanto, pueden utilizarse para evaluar la patogenicidad de las cepas de virus de la lengua azul. Esto indica que la virulencia del virus de la lengua azul depende, al menos en parte, de la respuesta del IFN del huésped. Los estudios sobre los factores de virulencia que influyen en la patogénesis del virus de la lengua azul han implicado varias proteínas codificadas por el genoma viral (Caporale et al., 2011; Celma et al., 2014; Janowicz et al., 2015). Las proteínas de la cápside externa VP2 y VP5, que están involucradas en la entrada de partículas virales en células de mamíferos, y la ARN polimerasa VP1 se han identificado como determinantes de la virulencia en BTV-8 cuando se comparó una cepa de paso múltiple con la cepa parental (Janowicz et al., 2015). Estos cambios probablemente reflejan la adaptación de la cepa del virus a las condiciones de cultivo de tejidos, lo que a su vez condujo a una reducción de la infectividad en el IFNAR.(–/–) Modelo de ratón. En estos estudios, NS3 parece ser un jugador importante en la virulencia, ya que la única introducción del segmento de codificación NS3 de la cepa de paso múltiple en la cepa parental patógena deterioró significativamente la patogénesis. De hecho, utilizando la genética inversa, también se demostró que NS3 modula la cinética de replicación del virus de la lengua azul y los efectos citopáticos en células de mamíferos (Ftaich et al., 2015). La presencia de un residuo de prolina en la posición 24 redujo la vida media de la proteína NS3, lo que a su vez disminuyó la virulencia en IFNAR(–/–) ratones de un virus reordenado portadores de este segmento 10 (Ftaich et al., 2015). Sin embargo, NS3 no es el único responsable de la patogénesis, ya que un virus reordenado que expresa VP1, VP2, VP5 y NS3 de la cepa patógena en la columna vertebral de la cepa de paso múltiple no patógena no volvió al fenotipo patógeno (Janowicz et al., 2015). De hecho, la expresión adicional de la enzima taponadora y la metiltransferasa VP4, la proteína VP7 de la cápside interna y la helicasa de ARN VP6 y la proteína NS4 no estructural codificada por el segmento 9 fue esencial para revertir la atenuación de la cepa de paso múltiple (Janowicz et al., 2015). Por lo tanto, parece que el virus de la lengua azul utiliza múltiples proteínas para perjudicar las respuestas del huésped y promover su replicación.
Utilizando ensayos de reporteros, se ha demostrado que el virus de la lengua azul perjudica la inducción de IFN (Chauveau et al., 2013; Avia et al., 2019). También se ha demostrado que las infecciones por BTV inhiben la señalización de IFN-I y -II al prevenir la fosforilación de STAT1 y la posterior translocación al núcleo (Doceul et al., 2014; Avia et al., 2019). Doceul et al. (2014) encontraron que la infección por BTV redujo la expresión de JAK1 y TYK2, mientras que Avia et al. (2019) encontraron que la expresión de STAT2 disminuyó en las células infectadas. Al menos tres productos del gen BTV están involucrados en el deterioro de las respuestas de IFN (Figura 4). En la siguiente sección discutiremos el conocimiento actual de los mecanismos empleados por BTV para perjudicar la respuesta del IFN del huésped.
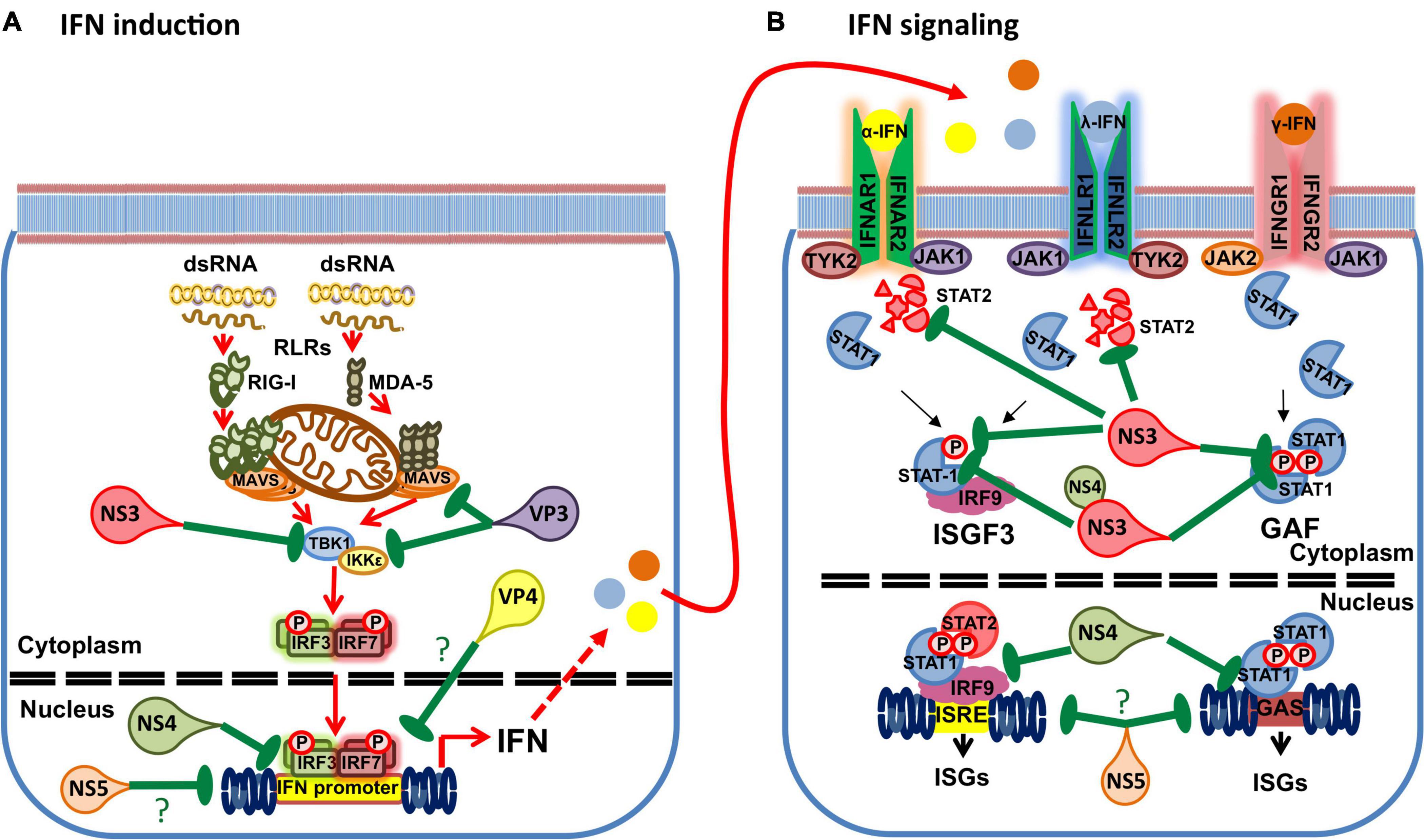 FIGURA 4. Interferencia de los productos del gen BTV con la respuesta IFN. (A) El virus de la lengua azul utiliza múltiples productos génicos para perjudicar la inducción de IFN. Se ha demostrado que NS3 perjudica la activación de TBK1 al dirigirse a la proteína de unión a ubiquitina Optineurin responsable de la translocación de TBK1 al aparato de Golgi, donde tiene lugar la activación de TBK1 por transautofosforilación. Se ha demostrado que NS4 perjudica la actividad del gen promotor del IFN en ensayos de reporteros. Se ha demostrado que VP3 interactúa con MAVS e IKKε para interferir con la inducción de IFN. Se ha informado que VP4 interfiere con la inducción de IFN, pero el mecanismo aún no se ha aclarado. Finalmente, se ha demostrado que el supuesto NS5 interfiere con la actividad de múltiples promotores, incluido el promotor IFN-β. (B) El virus de la lengua azul también utiliza múltiples productos génicos para interferir con la señalización del IFN. NS3 puede dirigirse a STAT2 para su degradación a través de una vía autofágica/lisosomal. NS3 también puede interferir con la fosforilación de STAT1. NS4 y NS3 parecen perjudicar coordinadamente la fosforilación de STAT1 al unirse al dominio STAT1 SH2. NS4 también puede interferir con la actividad promotora de ISRE y GAS. Finalmente, el supuesto NS5 puede perjudicar la actividad de múltiples promotores y, por lo tanto, posiblemente perjudicar la actividad promotora de ISRE y GAS. (?) denota mecanismos desconocidos o especulativos.
FIGURA 4. Interferencia de los productos del gen BTV con la respuesta IFN. (A) El virus de la lengua azul utiliza múltiples productos génicos para perjudicar la inducción de IFN. Se ha demostrado que NS3 perjudica la activación de TBK1 al dirigirse a la proteína de unión a ubiquitina Optineurin responsable de la translocación de TBK1 al aparato de Golgi, donde tiene lugar la activación de TBK1 por transautofosforilación. Se ha demostrado que NS4 perjudica la actividad del gen promotor del IFN en ensayos de reporteros. Se ha demostrado que VP3 interactúa con MAVS e IKKε para interferir con la inducción de IFN. Se ha informado que VP4 interfiere con la inducción de IFN, pero el mecanismo aún no se ha aclarado. Finalmente, se ha demostrado que el supuesto NS5 interfiere con la actividad de múltiples promotores, incluido el promotor IFN-β. (B) El virus de la lengua azul también utiliza múltiples productos génicos para interferir con la señalización del IFN. NS3 puede dirigirse a STAT2 para su degradación a través de una vía autofágica/lisosomal. NS3 también puede interferir con la fosforilación de STAT1. NS4 y NS3 parecen perjudicar coordinadamente la fosforilación de STAT1 al unirse al dominio STAT1 SH2. NS4 también puede interferir con la actividad promotora de ISRE y GAS. Finalmente, el supuesto NS5 puede perjudicar la actividad de múltiples promotores y, por lo tanto, posiblemente perjudicar la actividad promotora de ISRE y GAS. (?) denota mecanismos desconocidos o especulativos.
BTV-NS3
BTV-NS3 es la principal proteína viral implicada en la salida del virión en células de insectos y mamíferos (Hyatt et al., 1993; Beaton y otros, 2002; Labadie et al., 2020). NS3 es una proteína transmembrana que probablemente se sintetiza en el RE y trafica a través del aparato de Golgi para llegar a la membrana plasmática (Wu et al., 1992; Bansal y otros, 1998; Bhattacharya y otros, 2015; Labadie et al., 2020). La región C-terminal NS3 interactúa con la proteína VP2 de la cápside externa del virus de la lengua azul, que permite el transporte de viriones a la superficie celular (Celma y Roy, 2009). Recientemente se ha demostrado la presencia de NS3 en cuerpos de inclusión de virus (Mohl et al., 2020), y su correcto tráfico a la membrana celular es esencial para la maduración y liberación del virus (Labadie et al., 2019). NS3 también se ha identificado como una viroporina, ya que puede ensamblarse en forma de poros en las membranas (Chacko et al., 2015), lo que probablemente facilita la liberación de las partículas de BTV de la célula. De hecho, dado el papel de NS3 en la salida del virus, las partículas recombinantes de BTV deficientes en el segmento de codificación NS3 se han propuesto como una vacuna infecciosa infecciosa de un solo animal deshabilitada (van Rijn et al., 2017). Recientemente, también se demostró que NS3 activa la vía MAPK / ERK probablemente para promover la supervivencia celular y aumentar la traducción de proteínas, aumentando así la replicación del virus (Kundlacz et al., 2019).
Además de su papel en la liberación de viriones, NS3 ha sido identificado como un factor de virulencia (Janowicz et al., 2015), y podría estar involucrado en el cierre de la proteína del huésped celular. Stewart et al. (2015) mostraron que la transfección de NS3 podría disminuir la actividad de la luciferasa basal impulsada por una variedad de promotores. Avia et al. (2019) también encontraron que la transfección con altas cantidades de plásmido codificante de NS3 a veces puede producir un efecto similar sobre la actividad basal de la luciferasa. Finalmente, la NS3 también ha sido descrita como antagonista del IFN en varios estudios (Chauveau et al., 2013; Doceul et al., 2014; Avia et al., 2019; Li et al., 2021). En 2013, Chauveau et al. (2013) demostraron utilizando ensayos de reporteros que NS3 interfiere con la señalización RLR, bloqueando así la transcripción de los genes estimulados por IFN y NF-κB. Esta inhibición se produjo aguas abajo del reconocimiento de RLR y aguas arriba de la activación de IKKε. En un estudio posterior, se encontró que NS3 interfiere con la activación de TBK1 al interactuar con optineurina en el aparato de Golgi (Pourcelot et al., 2016). Tras la activación de RLR o TLR3, TBK1 se ubiquitina y se transporta al aparato de Golgi donde los complejos TBK1 se activan por transautofosforilación, lo que finalmente conduce a la fosforilación de IRF3 y la inducción de IFN (Pourcelot et al., 2016). La optineurina es una proteína de unión a ubiquitina que recluta TBK1 al aparato de Golgi y facilita la formación de los complejos de activación TBK1 (Ryan y Tumbarello, 2018). Al unirse a la optineurina en el aparato de Golgi, BTV-NS3 es, por lo tanto, capaz de amortiguar la fosforilación de TBK1 y, en consecuencia, la inducción de IFN (Pourcelot et al., 2016). Curiosamente, BTV-NS3 también está ubiquitinado (Avia et al., 2019) y se podría especular que NS3 podría actuar como un sustrato competidor para la unión de optineurina en el aparato de Golgi. Será necesario seguir trabajando en esta área para determinar el mecanismo exacto de la interferencia de NS3 con la actividad de la optineurina.
NS3 no solo interfiere con la inducción de IFN-I, sino que también inhibe la señalización de IFN-I e IFN-II. Se demostró que la transfección de NS3 en líneas celulares perjudica la fosforilación de STAT1 y la translocación al núcleo (Avia et al., 2019; Li et al., 2021). Es importante destacar que la infección con un virus de la lengua azul genético inversa que carece del segmento que codifica para NS3 solo podría afectar parcialmente la fosforilación y translocación de STAT1, lo que indica que NS3 no es el único responsable de estos mecanismos de interferencia (Avia et al., 2019). De hecho, se ha propuesto un papel colaborativo de otra proteína no estructural (NS4) con NS3 como un mecanismo que mejora la interferencia de STAT1 (Li et al., 2021). Mientras que Avia et al. (2019) encontraron que NS3 no interactuó con STAT1 en estudios de inmunoprecipitación; Li et al. (2021) informaron que NS3 interactúa con el dominio STAT1 SH2. Estas discrepancias podrían deberse a diferencias en los procedimientos de inmunoprecipitación: Avia et al. (2019) evaluaron los niveles endógenos de proteína STAT1 precipitada con NS3, mientras que Li et al. (2021) evaluaron esta interacción utilizando células transfectadas con NS3 y STAT1. Cabe señalar que estos estudios también emplearon productos génicos clonados de diferentes serotipos de virus de la lengua azul (BTV-8 para Avia et al., 2019 y BTV-1 para Li et al., 2021), que también podrían contribuir a las diferencias observadas. En general, NS3 interfiere con la fosforilación de STAT1 y la translocación nuclear, lo que perjudica la señalización de IFN, aunque el mecanismo aún no se ha dilucidado completamente.
También se ha demostrado que NS3 induce la degradación autofágica de STAT2, inhibiendo así la señalización de IFN-I (Avia et al., 2019). En la infección por el virus de la lengua azul que carecía del segmento que codifica para NS3, no se produjo degradación de STAT2, lo que demuestra que la NS3 es fundamental para este mecanismo. Al mutar los residuos de lisina NS3 13 y 15, Avia et al. (2019) también mostraron que NS3 es ubiquitinada y que la ubiquitinación en ambos residuos es esencial para la orientación de NS3 de STAT2 a la degradación autofágica. Además, el mutante NS3 incapaz de ser ubiquitinado no era capaz de inhibir la señalización de IFN en ensayos de reporteros, lo que indicó que la ubiquitinación de NS3 es crítica para su interferencia en la señalización de IFN. NS3 parecía estar poliubiquitinado con cadenas enlazadas con K63. En mamíferos, la modificación postraduccional de proteínas con ubiquitina unidas a través de los enlaces isopeptídicos K63 se asocia, entre otras cosas, con endocitosis y clasificación vesicular en los cuerpos multivesiculares (MVB) (Lauwers et al., 2009; Erpapazoglou et al., 2014). Por lo tanto, se podría especular que la ubiquitinación de NS3 permite la clasificación de NS3 dentro del sistema vesicular celular. En estudios de inmunofluorescencia, encontramos que el mutante NS3 que carece de sitios de ubiquitinación no colocalizó con el marcador MVB HGS (Figura 5), confirmando la importancia de esta modificación post-traduccional para el tráfico y la función de NS3. NS3 también posee un sitio de reclutamiento de ligasa E3 en su región N-terminal que está involucrado en la liberación del virus (Wirblich et al., 2006). Avia et al. (2019) también mostraron que este dominio era importante para la degradación autofágica mediada por NS3 de STAT2. Este mismo mutante tampoco pudo colocalizar con el marcador MVB HGS (Figura 5), lo que indica que este dominio debe permanecer intacto para que NS3 corrija el tráfico vesicular.
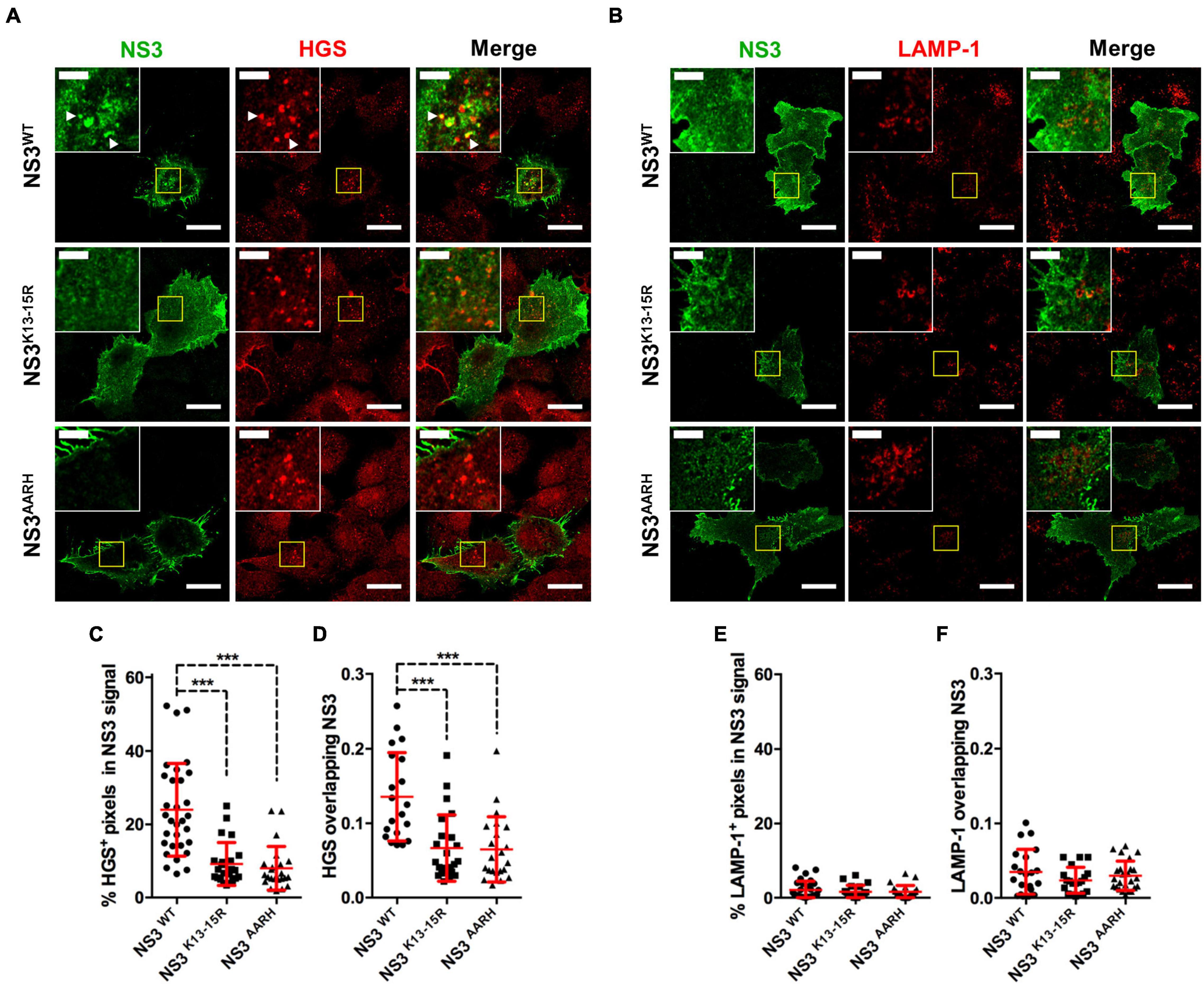 FIGURA 5. NS3 trafica a través del MVB. (A,B) Tinción de inmunofluorescencia representativa de células Vero transfectadas con plásmidos que expresan NS3 marcado con banderaWT, NS3K13–15R (mutación del sitio de ubiquitinación), o NS3UnARH (E3 ligase binding domain mutation) y teñido para NS3 con anticuerpos anti-FLAG tag [Cell Signaling, #14793 (D6W5B)], y para (A) el marcador MVB HGS (Abcam, ab72053) o (B) el marcador lisosómico LAMP-1 (BioLegend, 328612). Barra de escala = 20 μm. El recuadro muestra las áreas seleccionadas y las puntas de flecha indican la colocalización de píxeles. Barra de escala insertada = 5 μm. (C-F) Evaluación de la colocalización de píxeles mediante el software ImageJ (http://rsbweb.nih.gov/ij/ los Institutos Nacionales de Salud de EE. UU.). Media ± área porcentual SD por celda de (C) HGS + o (E) LAMP-1 + píxeles en señal NS3. Se muestra el coeficiente de Manders por celda para (D) HGS o (F) LAMP-1 que se superpone a la señal NS3 determinado por el complemento ImageJ JACOP. Datos acumulativos de 2 a 3 experimentos con 10-15 celdas analizadas por experimento. p < 0,001; ANOVA unidireccional con la post-prueba de Bonferroni. Los detalles del método para estos ensayos (construcción de plásmidos, transfección y microscopía confocal de inmunofluorescencia) se pueden encontrar en Avia et al. (2019). Para el análisis de colocalización de píxeles NS3 con señal HGS o LAMP-1, se capturaron imágenes de pila z (paso 0.5 μm) de celdas transfectadas para cada canal, se obtuvo la máscara celular (Mulens-Arias et al., 2015), se determinó la señal positiva para cada canal con la herramienta de umbral ImageJ y el porcentaje de píxeles superpuestos en máscaras de celda para cada plano z determinado con la herramienta calculadora de imágenes ImageJ (operación «AND»). El coeficiente de Manders en máscaras celulares se obtuvo utilizando el plugin JACOP ImageJ (Bolte y Cordelieres, 2006).
FIGURA 5. NS3 trafica a través del MVB. (A,B) Tinción de inmunofluorescencia representativa de células Vero transfectadas con plásmidos que expresan NS3 marcado con banderaWT, NS3K13–15R (mutación del sitio de ubiquitinación), o NS3UnARH (E3 ligase binding domain mutation) y teñido para NS3 con anticuerpos anti-FLAG tag [Cell Signaling, #14793 (D6W5B)], y para (A) el marcador MVB HGS (Abcam, ab72053) o (B) el marcador lisosómico LAMP-1 (BioLegend, 328612). Barra de escala = 20 μm. El recuadro muestra las áreas seleccionadas y las puntas de flecha indican la colocalización de píxeles. Barra de escala insertada = 5 μm. (C-F) Evaluación de la colocalización de píxeles mediante el software ImageJ (http://rsbweb.nih.gov/ij/ los Institutos Nacionales de Salud de EE. UU.). Media ± área porcentual SD por celda de (C) HGS + o (E) LAMP-1 + píxeles en señal NS3. Se muestra el coeficiente de Manders por celda para (D) HGS o (F) LAMP-1 que se superpone a la señal NS3 determinado por el complemento ImageJ JACOP. Datos acumulativos de 2 a 3 experimentos con 10-15 celdas analizadas por experimento. p < 0,001; ANOVA unidireccional con la post-prueba de Bonferroni. Los detalles del método para estos ensayos (construcción de plásmidos, transfección y microscopía confocal de inmunofluorescencia) se pueden encontrar en Avia et al. (2019). Para el análisis de colocalización de píxeles NS3 con señal HGS o LAMP-1, se capturaron imágenes de pila z (paso 0.5 μm) de celdas transfectadas para cada canal, se obtuvo la máscara celular (Mulens-Arias et al., 2015), se determinó la señal positiva para cada canal con la herramienta de umbral ImageJ y el porcentaje de píxeles superpuestos en máscaras de celda para cada plano z determinado con la herramienta calculadora de imágenes ImageJ (operación «AND»). El coeficiente de Manders en máscaras celulares se obtuvo utilizando el plugin JACOP ImageJ (Bolte y Cordelieres, 2006).
NS3 es, por lo tanto, una proteína polifuncional codificada por BTV. Permite la salida del virión en células de insectos y mamíferos, pero también interfiere con la inducción y señalización de IFN. Parece que la ubiquitinación de NS3 a través de cadenas K63 podría ser fundamental para su tráfico a través del sistema celular vesicular, dando así acceso a NS3 a la ubicación celular necesaria para la inducción de IFN como el aparato de Golgi donde afecta la activación de TBK1, y a las vías degradativas, como la vía autofágica / lisosomal a través de la cual media la degradación de STAT2. Una mayor subestimación de la biología de NS3 ayudará a arrojar más luz sobre los mecanismos exactos de interferencia que media esta proteína.
BTV-NS4
La proteína NS4 del virus de la lengua azul se propuso por primera vez como un ORF superpuesto mediante análisis bioinformático (Firth, 2008) y posteriormente se describió en 2011 (Belhouchet et al., 2011; Ratinier et al., 2011). NS4 está codificado por un marco de lectura abierto alternativo en el segmento 9. La expresión de NS4 se puede detectar en el citoplasma, así como en el núcleo de las células infectadas por BTV. Las estructuras putativas de bobinas enrolladas NS4 indican que puede asociarse con ácidos nucleicos y / o membranas, y de hecho se ha demostrado que se localiza con gotitas de lípidos en el citoplasma y con los nucléolos en el núcleo (Belhouchet et al., 2011). NS4 contiene una secuencia de residuos básicos de aminoácidos que impulsan la localización nuclear y dos supuestos dominios de cremallera de leucina (Ratinier et al., 2011). Se ha demostrado que NS4 se une a dsDNA (Belhouchet et al., 2011). Ratinier et al. (2011) demostraron que la expresión de NS4 no es esencial para la replicación del virus de la lengua azul y confirmaron la localización de NS4 en los nucléolos durante la infección. Un BTV-8 genético inverso que carecía de NS4 se atenuó en ovejas en comparación con las infecciones con una contraparte que expresó la proteína (Ratinier et al., 2016), lo que indica que NS4 modula las respuestas inmunes del huésped.
De hecho, los primeros estudios ya indicaban que NS4 podría interferir con la respuesta de IFN, proporcionando así una ventaja replicativa a BTV (Ratinier et al., 2011). NS4 puede antagonizar la inducción de IFN y la señalización de IFN-I y II (Ratinier et al., 2016; Avia et al., 2019; Li et al., 2021). El análisis transcriptómico mostró que 102 genes relacionados con las vías de IFN estaban regulados al alza en las infecciones con el virus defectuoso NS4 en comparación con la contraparte que expresa NS4 (Ratinier et al., 2016). En los ensayos de reporteros, NS4 fue capaz de inhibir no solo el promotor IFN-β y los promotores que contienen ISRE, sino también el promotor CMV (aunque en menor medida), lo que indica que NS4 podría alterar la actividad de una amplia gama de promotores (Ratinier et al., 2016). NS4 clonado de diferentes cepas de BTV (incluidas las atípicas BTV-25 y -26) podría afectar la expresión génica bajo el control de un promotor de CMV, excepto por un clon que carecía de la secuencia básica de aminoácidos responsable de la localización nuclear (Ratinier et al., 2016). Esto confirma además que una de las funciones de NS4 durante la infección por BTV es probable que implique la modulación de la expresión génica del huésped, incluidos los IFN e ISG después de la translocación en el núcleo.
También se ha demostrado que NS4 perjudica la señalización de IFN-I y II de manera dependiente de la dosis en ensayos de reporteros (Avia et al., 2019). La expresión de NS4 sola no afecta la fosforilación de STAT1 después de la estimulación de IFN (Avia et al., 2019; Li et al., 2021); sin embargo, su coexpresión con NS3 parece mejorar la capacidad de NS3 para perjudicar la fosforilación de STAT1 (Li et al., 2021). Mientras que Avia et al. (2019) no detectaron la interacción NS4 con STAT1; Li et al. (2021) informaron que NS4 interactuó con el dominio SH2 de STAT1. En cuanto a los ensayos de inmunoprecipitación NS3 en estos dos estudios, estas discrepancias podrían deberse a diferencias en los procedimientos experimentales, ya que en Avia et al. (2019) la interacción laboral se evaluó utilizando niveles endógenos de STAT1, mientras que Li et al. (2021) utilizaron la transfección STAT1. Alternativamente, la diferencia en la actividad de NS4 entre las diferentes cepas de BTV utilizadas en estos estudios también podría contribuir a estas observaciones. De hecho, Ratinier et al. (2011) mostraron que la actividad de NS4 en la respuesta de IFN varió entre los serotipos de BTV. La expresión de la proteína NS4 de BTV-8 permitió la replicación del virus de la lengua azul en las células tratadas con IFN, mientras que la NS4 de la BTV-1 no potenció este efecto (Ratinier et al., 2011), lo que indicó que la actividad de la NS4 en la respuesta del IFN puede variar dependiendo de los aislados del virus de la lengua azul. Li et al. (2021) también mostraron que la formación del complejo NS3 + NS4 + STAT1 redujo la heterodimerización de STAT1 y STAT2 que es necesaria para la señalización de IFN-I. La cotransfección de NS3 y NS4 también deterioró la translocación al núcleo de STAT1 fosforilada, confirmando aún más que NS4 podría potenciar los efectos de NS3 en la actividad de STAT1 (Li et al., 2021). Sin embargo, el mecanismo a través del cual la única expresión de NS4 media la inhibición de la señalización de IFN aún no se ha dilucidado. Si esto implica la interacción con componentes o adaptadores críticos para la señalización de IFN y / o la interferencia con la transcripción ISG es una pregunta abierta.
Todos los estudios hasta ahora coinciden en demostrar que NS4 es un antagonista de la inducción y señalización de IFN. Es probable que NS4 module la expresión génica en los nucléolos y podría afectar la señalización de IFN cuando está presente en el citoplasma. Su conservación a través de serotipos de BTV y en otros orbivirus (Belhouchet et al., 2011; Ratinier et al., 2011, 2016; Zwart et al., 2015; Boughan et al., 2020), y la observación de que su eliminación conduce a la atenuación también indica que proporciona al virus una ventaja replicativa.
El papel de otras proteínas del virus de la lengua azul
Hay indicios de que NS3 y NS4 no son las únicas proteínas BTV involucradas en el antagonismo de IFN. Usando ensayos de reporteros, se demostró que VP3 y VP4 interfieren con la inducción de IFN (Chauveau et al., 2013). También se demostró que NS1 y NS2 inhiben la inducción de IFN en ensayos de reporteros (Stewart y Roy, 2010), aunque esto no se confirmó en otros estudios (Chauveau et al., 2013; Avia et al., 2019). Varios factores podrían explicar estas discrepancias, como la diferencia en el sistema informador utilizado, así como los diversos estímulos utilizados para la inducción de IFN (transfección poli I: C (Stewart y Roy, 2010), transfección RIG-I constitutivamente activa (Chauveau et al., 2013) o infección por el virus Sendai (Avia et al., 2019).
Recientemente, la interferencia con la inducción de IFN mediada por la proteína del núcleo interno VP3 se caracterizó aún más (Pourcelot et al., 2021). VP3 es importante en el ensamblaje de virus y se ha demostrado que interactúa con NS2 en cuerpos de inclusión de virus (Kar et al., 2005; Mohl y Roy, 2014). VP3 podría perjudicar la actividad del promotor IFN-β de una manera dependiente de la dosis en los ensayos de reporteros, y limitar la inducción de ISG56 (Pourcelot et al., 2021). Se demostró que VP3 interactúa con MAVS e IKKε aguas abajo de la activación de RIG-I (Pourcelot et al., 2021). Más precisamente, Pourcelot et al. (2021) mapearon la interacción de VP3 con la región C-terminal de MAVS. Esta región contiene un motivo de interacción TRAF requerido para la activación correcta de la respuesta IFN, así como un sitio de unión regulatorio que recluta IKKε después de la ubiquitinación de MAVS en lisina 500 (Paz et al., 2011). El mecanismo exacto de la acción de VP3 en esta vía no está completamente dilucidado. Se podría especular que VP3 perjudica el reclutamiento de TRAF3 para MAVS y/o recluta la actividad reguladora de IKKε para amortiguar la inducción de IFN. Se necesitarán más estudios para comprender esta interacción del virus de la lengua azul con la vía de inducción del IFN.
Como se dijo anteriormente, se demostró que VP4 interfiere con la inducción de IFN en ensayos de reporteros (Chauveau et al., 2013). Esto no se observó en un estudio separado (Stewart y Roy, 2010), aunque debe tenerse en cuenta que se utilizó una estrategia diferente para la estimulación del IFN. La enzima de recubrimiento VP4 posee una actividad enzimática nucleósido-2′-O-metiltransferasa que es esencial para la replicación del virus (Stewart y Roy, 2015). También se ha demostrado que la actividad enzimática de la 2′-O-metilasa permite evadir el reconocimiento del ARN viral por parte de los PRR, como los miembros de la familia IFIT o TLR7 (Daffis et al., 2010; Jöckel et al., 2012). En el rotavirus, otro miembro de la familia Reoviridae, la enzima de recubrimiento VP3 posee una actividad 2′-O-metiltransferasa que limita el reconocimiento del ARN viral por sensores citosólicos (Chen et al., 1999; Uzri y Greenberg, 2013). Si la enzima de tapado de BTV VP4 actúa de manera similar requerirá más trabajo.
Finalmente, se ha descrito un ORF putativo en el segmento 10 que podría codificar para una quinta proteína no estructural (NS5) (Stewart et al., 2015). Aunque aún no se ha establecido evidencia de su expresión durante la infección por el virus de la lengua azul, la conservación de este ORF alternativo dentro del segmento 10 en más de 350 secuencias del virus de la lengua azul, así como su presencia en otros orbivirus (como el virus de la peste equina) indica que este ORF probablemente codifica para una proteína (Stewart et al., 2015). Stewart et al. (2015) también mostraron utilizando una construcción del segmento 10 que un gen reportero de luciferasa podría ser traducido por el ORF alternativo, proporcionando evidencia adicional de que este ORF alternativo podría codificar una proteína no estructural. La supuesta proteína NS5 posee una señal de localización nucleolar, y los estudios de inmunofluorescencia mostraron su localización en este espacio nuclear (Stewart et al., 2015). En los ensayos de reporteros, NS5 inhibió la expresión génica impulsada por varios promotores, incluido el promotor IFN-β, y esto dependía de la señal de localización nucleolar. Sin embargo, la genética inversa BTV-8 que carece del ORF putativo indujo niveles similares de IFN como su contraparte con el ORF, lo que sugiere que el NS5 putativo no es un antagonista del IFN. Este mutante genético inverso también tuvo una virulencia similar a la del virus de tipo salvaje en modelos murinos (Stewart et al., 2015). La localización nucleolar de NS5, su capacidad para perjudicar la expresión génica y su conservación en secuencias de virus de la lengua azul sugieren, sin embargo, que podría tener relevancia en el cierre celular producido por la lengua azul (Huismans, 1971). NS4 también se localiza en los nucléolos y afecta la expresión génica impulsada por varios promotores (Ratinier et al., 2016). Es tentador especular que ambas proteínas podrían participar en este fenómeno que probablemente contribuya a la evasión del virus de la lengua azul de los mecanismos antivirales.
Influencia vectorial en la respuesta del IFN
Un aspecto a menudo pasado por alto de la infección por el virus de la lengua azul son los efectos mediados por el vector sobre la inmunidad, ya que son difíciles de cuantificar. Curiosamente, la respuesta de IFN difirió en ovejas infectadas experimentalmente con Culicoides spp. portadoras de BTV o inyectadas con aguja con el virus (Pages et al., 2014). La infección por Culicoides spp. retrasó la respuesta al IFN y la producción de anticuerpos neutralizantes en comparación con la infección por inyección con aguja. De hecho, la cantidad de inflamación local debida a las picaduras de Culicoides spp. parece correlacionarse inversamente con la carga de viremia en ovejas infectadas (Pages et al., 2014). También se ha descrito que las infecciones con virus de la lengua azul cultivados en tejidos pueden diferir de la infección directa con animales infectados por la sangre en términos de virulencia (MacLachlan et al., 2008; Eschbaumer y otros, 2010; Caporale et al., 2014), y esto se ha correlacionado con una disminución de la variabilidad en la población de variantes de virus de la lengua azul aprobada en el cultivo de tejidos en células de mamíferos (Caporale et al., 2014). Los virus de ARN nunca existen como un genotipo único, sino más bien como una gama de variantes (o cuasiespecies, aunque hasta donde sabemos este concepto no se ha estudiado directamente en BTV). El aumento de la variabilidad podría facilitar la adaptación del virus de la lengua azul a la presión externa. Curiosamente, mientras que la secuencia de consenso de la lengua azul pasada en células de mamíferos o en una línea celular de Culicoides spp., el paso en la línea celular de Culicoides spp. aumentó la cantidad de variantes (Caporale et al., 2014). La interacción entre los factores del huésped y del vector puede influir claramente en la patogénesis del virus y, por extensión, en los pasos de reconocimiento temprano del virus por parte del sistema inmunitario. Múltiples factores probablemente participan en este efecto, como los productos de saliva Culicoides spp. que limitan la inflamación o la diversidad de variantes generadas en el huésped insecto. Sería interesante para la investigación del virus de la lengua azul establecer modelos experimentales de infección en rumiantes que imiten mejor las condiciones de campo para que se pueda estudiar una comprensión más profunda de la interacción del virus de la lengua azul con los primeros eventos de reconocimiento inmunitario.
Conclusión
Aunque el virus de la lengua azul se consideraba tradicionalmente como un buen inductor del IFN, ahora es evidente que posee múltiples mecanismos para perjudicar la respuesta del IFN y, en consecuencia, promueve su replicación. Las proteínas no estructurales NS3 y NS4 han surgido como los principales antagonistas del IFN en las infecciones por BTV, pero estudios recientes también han demostrado que las proteínas estructurales como VP3 pueden modular la respuesta del IFN. La interacción de las proteínas del virus de la lengua azul con el sistema IFN también es fundamental para la patogenicidad del virus y, por lo tanto, podría ayudar a avanzar en el diseño de la vacuna. Comprender estas interacciones celulares virus-huésped también arrojará algo de luz sobre la polifuncionalidad que muestran algunos productos del gen BTV. En última instancia, estudiar la interacción del virus de la lengua azul con el sistema de IFN ayudará a comprender mejor la compleja biología que tiene lugar durante las infecciones por la lengua azul.
Contribuciones del autor
Todos los autores hicieron una contribución intelectual directa a la obra. JR escribió el cuerpo principal del manuscrito. MA y JR diseñaron las figuras. Todos los autores contribuyeron a la revisión del manuscrito, leyeron y aprobaron la versión presentada.
Financiación
Este trabajo ha sido financiado por las Subvenciones AGL2015-64290R y RTI2018-094616-B-100 del Ministerio de Ciencia (España) y la Subvención S2018/BAA-4370-PLATESA2 de la Comunidad de Madrid (Fondo Europeo de Desarrollo Regional, FEDER). MA fue financiado por una subvención FPI (BES-2013-066406).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Reconocimientos
Queremos agradecer a Ana Villa por su ayuda técnica en microscopía confocal, y a todos los miembros del Laboratorio de Nuevas Estrategias para el Control de Patógenos Relevantes en Sanidad Animal por su asistencia técnica y útiles discusiones.
Referencias
Abad, A. T. y Danthi, P. (2020). Reconocimiento de ARN de reovirus por el sistema inmune innato. Virus 12:667. DOI: 10.3390/v12060667
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Abbas, Y. M., Pichlmair, A., Górna, M. W., Superti-Furga, G. y Nagar, B. (2013). Bases estructurales para el reconocimiento viral de 5′-PPP-RNA por proteínas IFIT humanas. Nature 494, 60–64. doi: 10.1038/nature11783
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Abe, T., Marutani, Y. y Shoji, I. (2019). Respuesta inmune citosólica sensible al ADN e infección viral. Microbiol. Immunol. 63, 51–64. doi: 10.1111/1348-0421.12669
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Attoui, H., Nomikou, K., Maan, S., Belaganahalli, M. y Mertens, P. P. C. (2016). «¿Orbivirus?», en Reference Module in Biomedical Sciences, (Amsterdam: Elsevier).
Avia, M., Rojas, J. M., Miorin, L., Pascual, E., Van Rijn, P. A., Martín, V., et al. (2019). Degradación autofágica inducida por virus de STAT2 como mecanismo para el bloqueo de señalización de interferón. EMBO Rep. 20:e48766.
Bansal, O. B., Stokes, A., Bansal, A., Bishop, D. y Roy, P. (1998). Organización de la membrana de la glicoproteína NS3 no estructural del virus de la lengua azul. J. Virol. 72, 3362–3369. DOI: 10.1128/JVI.72.4.3362-3369.1998
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Baylis, M., Caminade, C., Turner, J. y Jones, A. E. (2017). El papel del cambio climático en una amenaza en desarrollo: el caso de la lengua azul en Europa. Rev. Sci. Tech. 36, 467–478. DOI: 10.20506/RST.36.2.2667
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Baylis, M., O’Connell, L. y Mellor, P. S. (2008). Tasas de transmisión del virus de la lengua azul entre Culicoides sonorensis y ovejas. Entomol. 22, 228–237.
Beaton, A. R., Rodríguez, J., Reddy, Y. K. y Roy, P. (2002). La proteína de tráfico de membrana calpactina forma un complejo con la proteína NS3 del virus de la lengua azul y media la liberación del virus. Proc. Natl. Acad. Sci. U.S.A. 99, 13154–13159. doi: 10.1073/PNAS.192432299
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Belhouchet, M., Mohd Jaafar, F., Firth, A. E., Grimes, J. M., Mertens, P. P. y Attoui, H. (2011). Detección de una cuarta proteína no estructural de orbivirus. PLoS One 6:e25697. doi: 10.1371/journal.pone.0025697
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bhattacharya, B., Celma, C. C. y Roy, P. (2015). Influencia de la vía de tráfico celular en la infección por el virus de la lengua azul en células ovinas. Virus 7, 2378–2403. DOI: 10.3390/V7052378
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bieback, K., Lien, E., Klagge, I. M., Avota, E., Schneider-Schaulies, J., Duprex, W. P., et al. (2002). La proteína hemaglutinina del virus del sarampión de tipo salvaje activa la señalización del receptor 2 tipo toll. J. Virol. 76, 8729–8736. doi: 10.1128/jvi.76.17.8729-8736.2002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bolte, S., and Cordelieres, F. P. (2006). A guided tour into subcellular colocalization analysis in light microscopy. J. Microsc. 224(Pt 3), 213–232. doi: 10.1111/j.1365-2818.2006.01706.x
Boughan, S., Potgieter, A. C., and van Staden, V. (2020). African horse sickness virus NS4 is a nucleocytoplasmic protein that localizes to PML nuclear bodies. J. Gen. Virol. 101, 366–384.
Boyce, M., Celma, C. C. y Roy, P. (2012). La proteína no estructural 1 del virus de la lengua azul es un regulador positivo de la síntesis de proteínas virales. Virol. J. 9:178. doi: 10.1186/1743-422x-9-178
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Calvo-Pinilla, E., Navasa, N., Anguita, J. y Ortego, J. (2012). Protección multiserotipo provocada por una estrategia combinatoria de vacunación contra el virus de la lengua azul. PLoS One 7:e34735. doi: 10.1371/journal.pone.0034735
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Calvo-Pinilla, E., Rodríguez-Calvo, T., Anguita, J., Sevilla, N. y Ortego, J. (2009a). Establecimiento de un modelo de infección por el virus de la lengua azul en ratones que son deficientes en el receptor de interferón alfa/beta. PLoS One 4:e5171. doi: 10.1371/journal.pone.0005171
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Calvo-Pinilla, E., Rodríguez-Calvo, T., Sevilla, N. y Ortego, J. (2009b). Vacunación heteróloga de refuerzo principal con ADN y virus vaccinia modificado recombinante Ankara protege a los ratones IFNAR(-/-) contra la infección letal de la lengua azul. Vacuna 28, 437–445. doi: 10.1016/j.vaccine.2009.10.027
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Caporale, M., Di Gialleonorado, L., Janowicz, A., Wilkie, G., Shaw, A., Savini, G., et al. (2014). Factores del virus y del huésped que afectan el resultado clínico de la infección por el virus de la lengua azul. J. Virol. 88, 10399–10411. DOI: 10.1128/JVI.01641-14
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Caporale, M., Wash, R., Pini, A., Savini, G., Franchi, P., Golder, M., et al. (2011). Determinantes de la virulencia del virus de la lengua azul en modelos murinos de enfermedad. J. Virol. 85, 11479–11489. DOI: 10.1128/JVI.05226-11
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Celma, C. C. y Roy, P. (2009). Una proteína viral no estructural regula el tráfico y la liberación del virus de la lengua azul. J. Virol. 83, 6806–6816. DOI: 10.1128/JVI.00263-09
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Celma, C. C., Bhattacharya, B., Eschbaumer, M., Wernike, K., Beer, M. y Roy, P. (2014). Estudio de patogenicidad en ovejas utilizando virus de la lengua azul reordenados basados en la genética inversa. Veterinario Microbiol. 174, 139–147. doi: 10.1016/j.vetmic.2014.09.012
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chacko, N., Mohanty, N. N., Biswas, S. K., Chand, K., Yogisharadhya, R., Pandey, A. B., et al. (2015). Un motivo de bobina enrollada en la proteína no estructural 3 (NS3) del virus de la lengua azul forma un oligómero. Genes del virus 51, 244–251. DOI: 10.1007/S11262-015-1230-9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chan, Y. K. y Gack, M. U. (2016). Evasión viral de la detección intracelular de ADN y ARN. Nat. Rev. Microbiol. 14, 360–373. doi: 10.1038/nrmicro.2016.45
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Channappanavar, R., Singh, K. P., Singh, R., Umeshappa, C. S., Ingale, S. L. y Pandey, A. B. (2012). Aumento de la actividad de citoquinas proinflamatorias durante la infección experimental por el virus de la lengua azul-1 en ovejas nativas de la India. Veterinario Immunol. Inmunopatol. 145, 485–492. doi: 10.1016/j.vetimm.2011.10.013
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chauveau, E., Doceul, V., Lara, E., Adam, M., Breard, E., Sailleau, C., et al. (2012). Detección y control de la infección por el virus de la lengua azul en células epiteliales a través de helicasas RIG-I y MDA5. J. Virol. 86, 11789–11799. DOI: 10.1128/JVI.00430-12
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chauveau, E., Doceul, V., Lara, E., Breard, E., Sailleau, C., Vidalain, P. O., et al. (2013). La NS3 del virus de la lengua azul interfiere con la inducción del interferón tipo I. J. Virol. 87, 8241–8246. DOI: 10.1128/JVI.00678-13
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chen, D., Luongo, C. L., Nibert, M. L. y Patton, J. T. (1999). Los núcleos abiertos de rotavirus catalizan el tapado de 5′ y la metilación del ARN exógeno: evidencia de que VP3 es una metiltransferasa. Virology 265, 120–130. DOI: 10.1006/Viro.1999.0029
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chow, K. T., Gale, M. y Loo, Y. M. (2018). RIG-I y otros sensores de ARN en inmunidad antiviral. Annu. Rev. Immunol. 36, 667–694. doi: 10.1146/annurev-immunol-042617-053309
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Chu, W. M., Ostertag, D., Li, Z. W., Chang, L., Chen, Y., Hu, Y., et al. (1999). JNK2 e IKKbeta son necesarios para activar la respuesta innata a la infección viral. Inmunidad 11, 721–731. doi: 10.1016/s1074-7613(00)80146-6
Coen, M. L., Ellis, J. A., O’Toole, D. T. y Wilson, W. C. (1991). Modulación de citoquinas de la interacción entre el virus de la lengua azul y las células endoteliales in vitro. Veterinario Pathol. 28, 524–532. DOI: 10.1177/030098589102800610
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Daffis, S., Szretter, K. J., Schriewer, J., Li, J., Youn, S., Errett, J., et al. (2010). La metilación 2′-O de la tapa del ARNm viral evade la restricción del huésped por parte de los miembros de la familia IFIT. Nature 468, 452–456. doi: 10.1038/nature09489
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dai, M., Wang, X., Li, J. L., Zhou, Y., Sang, M., Liu, J. B., et al. (2015). La activación de la vía de señalización TLR3/interferón por el virus de la lengua azul da lugar a la inhibición del VIH en los macrófagos. FASEB J. 29, 4978–4988. DOI: 10.1096/FJ.15-273128
Resumen de PubMed | Texto completo de CrossRef | Google Académico
DeMaula, C. D., Jutila, M. A., Wilson, D. W. y MacLachlan, N. J. (2001). Cinética de infección, liberación de prostaciclina y modulación mediada por citoquinas del mecanismo de muerte celular durante la infección por el virus de la lengua azul de células endoteliales microvasculares ovinas y bovinas cultivadas de la arteria pulmonar y pulmón. J. General Virol. 82(Pt 4), 787–794. doi: 10.1099/0022-1317-82-4-787
Resumen de PubMed | Texto completo de CrossRef | Google Académico
DeMaula, C. D., Leutenegger, C. M., Bonneau, K. R. y MacLachlan, N. J. (2002a). El papel de los mediadores inflamatorios y vasoactivos derivados de células endoteliales en la patogénesis de la lengua azul. Virología 296, 330–337. DOI: 10.1006/Viro.2002.1476
Resumen de PubMed | Texto completo de CrossRef | Google Académico
DeMaula, C. D., Leutenegger, C. M., Jutila, M. A. y MacLachlan, N. J. (2002b). Activación inducida por el virus de la lengua azul de células endoteliales microvasculares primarias del pulmón bovino. Veterinario Immunol. Inmunopatol. 86, 147–157. doi: 10.1016/s0165-2427(02)00012-0
Doceul, V., Chauveau, E., Lara, E., Breard, E., Sailleau, C., Zientara, S., et al. (2014). Modulación dual de la respuesta de interferón tipo I por el virus de la lengua azul. J. Virol. 88, 10792–10802. DOI: 10.1128/JVI.01235-14
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Drew, C. P., Heller, M. C., Mayo, C., Watson, J. L. y Maclachlan, N. J. (2010). La infección por el virus de la lengua azul activa los macrófagos derivados de monocitos bovinos y las células endoteliales de la arteria pulmonar. Veterinario Immunol. Inmunopatol. 136, 292–296. doi: 10.1016/j.vetimm.2010.03.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Erpapazoglou, Z., Walker, O. y Haguenauer-Tsapis, R. (2014). Roles versátiles de las cadenas de ubiquitina ligadas a k63 en el tráfico. Celdas 3, 1027–1088. doi: 10.3390/cells3041027
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Eschbaumer, M., Wäckerlin, R., Rudolf, M., Keller, M., König, P., Zemke, J., et al. (2010). Virus infecciosos cultivados en sangre o cultivo: una comparación de los modelos de desafío del virus de la lengua azul. Veterinario Microbiol. 146, 150–154. doi: 10.1016/j.vetmic.2010.05.004
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Finberg, R. W., Wang, J. P. y Kurt-Jones, E. A. (2007). Toll como receptores y virus. Rev. Med. 17, 35–43.
Firth, A. E. (2008). El análisis bioinformático sugiere que el cistrón Orbivirus VP6 codifica un gen superpuesto. Virol. J. 5:48. doi: 10.1186/1743-422x-5-48
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Foster, N. M., Luedke, A. J., Parsonson, I. M. y Walton, T. E. (1991). Relaciones temporales de viremia, actividad de interferón y respuestas de anticuerpos de ovejas infectadas con varias cepas del virus de la lengua azul. Am. J. Vet. 52, 192–196.
Ftaich, N., Ciancia, C., Viarouge, C., Barry, G., Ratinier, M., van Rijn, P. A., et al. (2015). La tasa de renovación de las proteínas NS3 modula la cinética de replicación del virus de la lengua azul de una manera específica del huésped. J. Virol. 89, 10467–10481. DOI: 10.1128/JVI.01541-15
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Fulton, R. W., y Pearson, N. J. (1982). Inducción de interferón en cultivos monocapa bovinos y felinos por cuatro serotipos del virus de la lengua azul. Can. J. Comp. Med. 46, 100–102.
Gil, J., y Esteban, M. (2000). La proteína quinasa inducida por interferón (PKR) desencadena la apoptosis a través de la activación mediada por FADD de la caspasa 8 de una manera independiente de los receptores Fas y TNF-alfa. Oncogén 19, 3665–3674. doi: 10.1038/sj.onc.1203710
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Goubau, D., Deddouche, S. y Reis e Sousa, C. (2013). Detección citosólica de virus. Inmunidad 38, 855–869. doi: 10.1016/j.immuni.2013.05.007
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Habjan, M., Hubel, P., Lacerda, L., Benda, C., Holze, C., Eberl, C. H., et al. (2013). El secuestro por IFIT1 perjudica la traducción del ARN tapado 2’O-no metilado. PLoS Pathog. 9:E1003663. doi: 10.1371/journal.ppat.1003663
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Han, Z., y Harty, R. N. (2004). La proteína NS3 del virus de la lengua azul exhibe propiedades similares a la viroporina. J. Biol. Chem. 279, 43092–43097. doi: 10.1074/jbc.m403663200
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Hemati, B., Contreras, V., Urien, C., Bonneau, M., Takamatsu, H. H., Mertens, P. P., et al. (2009). El virus de la lengua azul se dirige a las células dendríticas convencionales en la linfa de la piel. J. Virol. 83, 8789–8799. DOI: 10.1128/JVI.00626-09
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Howerth, E. W. (2015). Liberación de citoquinas y disfunción endotelial: una tormenta perfecta en la patogénesis del orbivirus. Vet. Ital. 51, 275–281.
Huismans, H. (1969). Síntesis de interferón inducida por el virus de la lengua azul. Onderstepoort J. Vet. 36, 181–185.
Huismans, H. (1971). Síntesis de proteínas de la célula huésped después de la infección con el virus de la lengua azul y el reovirus. Virología 46, 500–503. DOI: 10.1016/0042-6822(71)90053-5
Hyatt, A. D., Zhao, Y. y Roy, P. (1993). La liberación de partículas similares al virus de la lengua azul de las células de insectos está mediada por la proteína no estructural NS3 / NS3A del virus de la lengua azul. Virología 193, 592–603. DOI: 10.1006/Viro.1993.1167
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Jacobs, S. R., y Damania, B. (2012). NLR, inflamasomas e infección viral. J. Leukoc. Biol. 92, 469–477. DOI: 10.1189/JLB.0312132
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Jameson, P., y Grossberg, S. E. (1979). Producción de interferón por líneas celulares tumorales humanas. Arq. Virol. 62, 209–219. DOI: 10.1007/BF01317553
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Jameson, P., y Grossberg, S. E. (1981). Inducción de interferón con el virus de la lengua azul en diversas células. Métodos Enzymol. 78(Pt A), 254–258. DOI: 10.1016/0076-6879(81)78126-6
Jameson, P., Schoenherr, C. K. y Grossberg, S. E. (1978). Virus de la lengua azul, un inductor de interferón excepcionalmente potente en ratones. Infectar. Inmune. 20, 321–323. DOI: 10.1128/IAI.20.1.321-323.1978
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Janowicz, A., Caporale, M., Shaw, A., Gulletta, S., Di Gialleonardo, L., Ratinier, M., et al. (2015). Múltiples segmentos del genoma determinan la virulencia del serotipo 8 del virus de la lengua azul. J. Virol. 89, 5238–5249. DOI: 10.1128/JVI.00395-15
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Jöckel, S., Nees, G., Sommer, R., Zhao, Y., Cherkasov, D., Hori, H., et al. (2012). El estado de 2′-O-metilación de una sola guanosina controla la activación o inhibición del receptor tipo Toll 7 mediada por ARN. J. Exp. Med. 209, 235–241. DOI: 10.1084/JEM.20111075
Kar, A. K., Bhattacharya, B., and Roy, P. (2007). Bluetongue virus RNA binding protein NS2 is a modulator of viral replication and assembly. BMC Mol. Biol. 8:4.
Kar, A. K., Iwatani, N., and Roy, P. (2005). Assembly and intracellular localization of the bluetongue virus core protein VP3. J. Virol. 79, 11487–11495. doi: 10.1128/jvi.79.17.11487-11495.2005
Kerviel, A., Ge, P., Lai, M., Jih, J., Boyce, M., Zhang, X., et al. (2019). Atomic structure of the translation regulatory protein NS1 of bluetongue virus. Nat. Microbiol. 4, 837–845. doi: 10.1038/s41564-019-0369-x
Kirchhoff, S., Koromilas, A. E., Schaper, F., Grashoff, M., Sonenberg, N., and Hauser, H. (1995). IRF-1 induced cell growth inhibition and interferon induction requires the activity of the protein kinase PKR. Oncogene 11, 439–445.
Kumar, A., Yang, Y. L., Flati, V., Der, S., Kadereit, S., Deb, A., et al. (1997). Deficient cytokine signaling in mouse embryo fibroblasts with a targeted deletion in the PKR gene: role of IRF-1 and NF-kappaB. EMBO J. 16, 406–416. doi: 10.1093/emboj/16.2.406
Kundlacz, C., Pourcelot, M., Fablet, A., Amaral, Da Silva, Moraes, R., et al. (2019). Novel function of bluetongue virus NS3 protein in regulation of the MAPK/ERK signaling pathway. J. Virol. 93:e00336-19.
Labadie, T., Jegouic, S., and Roy, P. (2019). Bluetongue virus nonstructural protein 3 orchestrates virus maturation and drives non-lytic egress via two polybasic motifs. Viruses 11:1107. doi: 10.3390/v11121107
Labadie, T., Sullivan, E., and Roy, P. (2020). Multiple routes of bluetongue virus egress. Microorganisms 8:965. doi: 10.3390/microorganisms8070965
Lauwers, E., Jacob, C., and Andre, B. (2009). K63-linked ubiquitin chains as a specific signal for protein sorting into the multivesicular body pathway. J. Cell Biol. 185, 493–502. doi: 10.1083/jcb.200810114
Li, Z., Lu, D., Yang, H., Li, Z., Zhu, P., Xie, J., et al. (2021). Bluetongue virus non-structural protein 3 (NS3) and NS4 coordinatively antagonize type ? interferon signaling by targeting STAT1. Vet. Microbiol. 254:108986. doi: 10.1016/j.vetmic.2021.108986
Liu, Y. J. (2005). IPC: professional type 1 interferon-producing cells and plasmacytoid dendritic cell precursors. Annu. Rev. Immunol. 23, 275–306. doi: 10.1146/annurev.immunol.23.021704.115633
Ma, Z., and Damania, B. (2016). The cGAS-STING defense pathway and its counteraction by viruses. Cell Host Microbe 19, 150–158. doi: 10.1016/j.chom.2016.01.010
Ma, Z., Ni, G., and Damania, B. (2018). Innate sensing of DNA virus genomes. Annu. Rev. Virol. 5, 341–362. doi: 10.1146/annurev-virology-092917-043244
MacLachlan, N. J., and Thompson, J. (1985). Bluetongue virus-induced interferon in cattle. Am. J. Vet. Res. 46, 1238–1241.
MacLachlan, N. J., Crafford, J. E., Vernau, W., Gardner, I. A., Goddard, A., Guthrie, A. J., et al. (2008). Experimental reproduction of severe bluetongue in sheep. Vet. Pathol. 45, 310–315. doi: 10.1354/vp.45-3-310
Majoros, A., Platanitis, E., Kernbauer-Hölzl, E., Rosebrock, F., Müller, M., and Decker, T. (2017). Canonical and non-canonical aspects of JAK–STAT signaling: lessons from interferons for cytokine responses. Front. Immunol. 8:29.
Marín-López, A., Bermúdez, R., Calvo-Pinilla, E., Moreno, S., Brun, A., and Ortego, J. (2016). Pathological characterization Of IFNAR(-/-) mice infected with bluetongue virus Serotype 4. Int. J. Biol. Sci. 12, 1448–1460. doi: 10.7150/ijbs.14967
Marín-López, A., Calvo-Pinilla, E., Barriales, D., Lorenzo, G., Brun, A., Anguita, J., et al. (2018). CD8 T Cell Responses to an immunodominant epitope within the nonstructural protein NS1 provide wide immunoprotection against bluetongue virus in IFNAR(-/-) Mice. J. Virol. 92:e00938-18.
Martin, V., Pascual, E., Avia, M., Pena, L., Valcarcel, F., and Sevilla, N. (2015). Protective efficacy in sheep of adenovirus-vectored vaccines against bluetongue virus is associated with specific T cell responses. PLoS One 10:e0143273. doi: 10.1371/journal.pone.0143273
McFadden, M. J., Gokhale, N. S., and Horner, S. M. (2017). Protect this house: cytosolic sensing of viruses. Curr. Opin. Virol. 22, 36–43. doi: 10.1016/j.coviro.2016.11.012
McNab, F., Mayer-Barber, K., Sher, A., Wack, A., and O’Garra, A. (2015). Type I interferons in infectious disease. Nat. Rev. Immunol. 15, 87–103. doi: 10.1038/nri3787
Melzi, E., Caporale, M., Rocchi, M., Martin, V., Gamino, V., di Provvido, A., et al. (2016). Follicular dendritic cell disruption as a novel mechanism of virus-induced immunosuppression. Proc. Natl. Acad. Sci. U.S.A. 113, E6238–E6247.
Mitoma, H., Hanabuchi, S., Kim, T., Bao, M., Zhang, Z., Sugimoto, N., et al. (2013). The DHX33 RNA helicase senses cytosolic RNA and activates the NLRP3 inflammasome. Immunity 39, 123–135. doi: 10.1016/j.immuni.2013.07.001
Mohl, B.-P., and Roy, P. (2014). Bluetongue virus capsid assembly and maturation. Viruses 6, 3250–3270. doi: 10.3390/v6083250
Mohl, B.-P., Kerviel, A., Labadie, T., Matsuo, E., and Roy, P. (2020). Differential localization of structural and non-structural proteins during the bluetongue virus replication cycle. Viruses 12:343. doi: 10.3390/v12030343
Mulens-Arias, V., Rojas, J. M., Pérez-Yagüe, S., Morales, M. P., and Barber, D. F. (2015). Polyethylenimine-coated SPIONs trigger macrophage activation through TLR-4 signaling and ROS production and modulate podosome dynamics. Biomaterials 52, 494–506. doi: 10.1016/j.biomaterials.2015.02.068
Nan, Y., Wu, C., and Zhang, Y. J. (2017). Interplay between Janus Kinase/signal transducer and activator of transcription signaling activated by Type I interferons and viral antagonism. Front. Immunol. 8:1758.
Pages, N., Bréard, E., Urien, C., Talavera, S., Viarouge, C., Lorca-Oro, C., et al. (2014). Culicoides midge bites modulate the host response and impact on bluetongue virus infection in sheep. PLoS One 9:e83683. doi: 10.1371/journal.pone.0083683
Paz, S., Vilasco, M., Werden, S. J., Arguello, M., Joseph-Pillai, D., Zhao, T., et al. (2011). A functional C-terminal TRAF3-binding site in MAVS participates in positive and negative regulation of the IFN antiviral response. Cell Res. 21, 895–910. doi: 10.1038/cr.2011.2
Pestka, S., Krause, C. D., and Walter, M. R. (2004). Interferons, interferon-like cytokines, and their receptors. Immunol. Rev. 202, 8–32. doi: 10.1111/j.0105-2896.2004.00204.x
Pourcelot, M., Amaral Moraes, R., Fablet, A., Bréard, E., Sailleau, C., Viarouge, C., et al. (2021). La proteína VP3 del virus de la lengua azul se asocia con el complejo MAVS e interfiere con la vía de señalización RIG-I. Virus 13:230. DOI: 10.3390/v13020230
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Pourcelot, M., Zemirli, N., Silva, Da Costa, L., Loyant, R., Garcin, D., et al. (2016). El aparato de Golgi actúa como una plataforma para la activación de TBK1 después de la detección de ARN viral. BMC Biol. 14:69.
Ratinier, M., Caporale, M., Golder, M., Franzoni, G., Allan, K., Nunes, S. F., et al. (2011). Identificación y caracterización de una nueva proteína no estructural del virus de la lengua azul. PLoS Pathog. 7:E1002477. doi: 10.1371/journal.ppat.1002477
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ratinier, M., Shaw, A. E., Barry, G., Gu, Q., Di Gialleonardo, L., Janowicz, A., et al. (2016). La proteína NS4 del virus de la lengua azul es un antagonista del interferón y un determinante de la virulencia del virus. J. Virol. 90, 5427–5439. DOI: 10.1128/JVI.00422-16
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Rehwinkel, J. y Gack, M. U. (2020). Receptores tipo RIG-I: su regulación y funciones en la detección de ARN. Nat. Rev. Immunol. 20, 537–551. DOI: 10.1038/S41577-020-0288-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ren, Z., Ding, T., Zuo, Z., Xu, Z., Deng, J. y Wei, Z. (2020). Regulación de la expresión de MAVS y la función de señalización en la respuesta inmune innata antiviral. Frente. Immunol. 11:1030.
Rinaldo, C. R., Overall, J. C., y Glasgow, L. A. (1975). Replicación viral y producción de interferón en leucocitos ovinos fetales y adultos y células del bazo. Infectar. Inmune. 12, 1070–1077. DOI: 10.1128/IAI.12.5.1070-1077.1975
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Rodriguez-Calvo, T., Rojas, J. M., Martin, V. y Sevilla, N. (2014). El interferón tipo I limita la capacidad del virus de la lengua azul para infectar precursores hematopoyéticos y células dendríticas in vitro e in vivo. J. Virol. 88, 859–867. DOI: 10.1128/JVI.02697-13
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Rojas, J. M., Alejo, A., Martín, V. y Sevilla, N. (2021a). Mecanismos inducidos por patógenos virales para antagonizar la vía de señalización del interferón en mamíferos (IFN). Cell Mol. Life Sci. 78, 1423–1444. DOI: 10.1007/S00018-020-03671-Z
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Rojas, J. M., Barba-Moreno, D., Avia, M., Sevilla, N. y Martín, V. (2021b). La vacunación con adenovirus recombinantes que expresan las subunidades VP7 y VP2 del virus de la lengua azul proporciona protección contra el desafío del virus heterólogo. Frente. Vet. Sci. 8:645561.
Rojas, J. M., Pascual, E., Wattegedera, S. R., Avia, M., Santiago, C., Martín, V., et al. (2021c). La proteína hemaglutinina del virus Peste des Petits Rumiants (PPRV) activa la respuesta inmune innata a través de la señalización del receptor tipo Toll 2. Virulencia 12, 690–703. doi: 10.1080/21505594.2021.1882246
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Rojas, J. M., Rodríguez-Calvo, T. y Sevilla, N. (2017). Recordemos que las respuestas de las células T al virus de la lengua azul producen un estrechamiento del repertorio de células T. Vet. 48:38.
Roy, P. (2005). Proteínas y partículas del virus de la lengua azul y su papel en la entrada, ensamblaje y liberación del virus. Adv. Virus Res. 64, 69–123. doi: 10.1016/s0065-3527(05)64004-3
Roy, P. (2017). Estructura y ensamblaje del virus de la lengua azul. Curr. Opinar. Virol. 24, 115–123. doi: 10.1016/j.coviro.2017.05.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ruscanu, S., Jouneau, L., Urien, C., Bourge, M., Lecardonnel, J., Moroldo, M., et al. (2013). Los subtipos de células dendríticas de los ganglios linfáticos y la sangre muestran programas de expresión génica contrastados tras la infección por el virus de la lengua azul. J. Virol. 87, 9333–9343. DOI: 10.1128/JVI.00631-13
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ruscanu, S., Pascale, F., Bourge, M., Hemati, B., Elhmouzi-Younes, J., Urien, C., et al. (2012). El virus de la lengua azul de ARN bicatenario induce interferón tipo I en células dendríticas plasmocitoides a través de una vía de señalización independiente de TLR88/7 dependiente de MYD8. J. Virol. 86, 5817–5828. DOI: 10.1128/JVI.06716-11
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Rushton, J. y Lyons, N. (2015). Impacto económico de la lengua azul: una revisión de los efectos sobre la producción. Vet. Ital. 51, 401–406.
Russell, H., O’Toole, D. T., Bardsley, K., Davis, W. C. y Ellis, J. A. (1996). Efectos comparativos de la infección por el virus de la lengua azul de células endoteliales ovinas y bovinas. Veterinario Pathol. 33, 319–331. doi: 10.1177/030098589603300309
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ryan, T. A., y Tumbarello, D. A. (2018). Optineurin: un coordinador del tráfico de carga asociado a membranas y la autofagia. Frente. Immunol. 9:1024.
Saminathan, M., Singh, K. P., Vineetha, S., Maity, M., Biswas, S. K., Manjunathareddy, G. B., et al. (2020). Hallazgos virológicos, inmunológicos y patológicos del serotipo 1 del virus de la lengua azul transmitido transplacentariamente en ratones bloqueados por IFNAR1 durante la gestación temprana y media. Rep. 10:2164.
Sánchez-Cordón, P. J., Pérez, de Diego, A. C., Gómez-Villamandos, J. C., Sánchez-Vizcaíno, J. M., Pleguezuelos, F. J., et al. (2015). Análisis comparativo de las respuestas inmunes celulares y los niveles de citoquinas en ovejas infectadas experimentalmente con el virus de la lengua azul serotipo 1 y 8. Veterinario Microbiol. 177, 95–105. doi: 10.1016/j.vetmic.2015.02.022
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Schneider, W. M., Chevillotte, M. D. y Rice, C. M. (2014). Genes estimulados por interferón: una compleja red de defensas del huésped. Annu. Rev. Immunol. 32, 513–545. doi: 10.1146/annurev-immunol-032713-120231
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Shaw, A. E., Hughes, J., Gu, Q., Behdenna, A., Singer, J. B., Dennis, T., et al. (2017). Propiedades fundamentales del sistema inmune innato de los mamíferos reveladas por la comparación multiespecie de las respuestas de interferón tipo I. PLoS Biol. 15:E2004086. doi: 10.1371/journal.pbio.2004086
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Stewart, M. E. y Roy, P. (2010). Papel de las caspasas celulares, factor nuclear-kappa B y factores reguladores de interferón en la infección por el virus de la lengua azul y el destino celular. Virol. J. 7:362.
Stewart, M. E. y Roy, P. (2015). Identificación basada en la estructura de residuos funcionales en el dominio nucleósido-2′-O-metilasa de la enzima VP4 del virus de la lengua azul. FEBS Open Bio 5, 138–146. doi: 10.1016/j.fob.2015.02.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Stewart, M., Hardy, A., Barry, G., Pinto, R. M., Caporale, M., Melzi, E., et al. (2015). Caracterización de un segundo marco de lectura abierto en el segmento 10 del genoma del virus de la lengua azul. J. General Virol. 96, 3280–3293. doi: 10.1099/jgv.0.000267
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Tytell, A. A., Lampson, G. P., Field, A. K., y Hilleman, M. R. (1967). Inductores de interferón y resistencia del huésped. 3. ARN bicatenario de viriones de reovirus tipo 3 (reo 3-RNA). Proc. Natl. Acad. Sci. U. S. A. 58, 1719–1722. DOI: 10.1073/PNAS.58.4.1719
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Umeshappa, C. S., Singh, K. P., Nanjundappa, R. H., Channappanavar, R., Maan, S. y Maan, N. S. (2012). Virus de la lengua azul: 23 estimula la expresión inducible de óxido nítrico sintasa y la producción de óxido nítrico en células mononucleares de sangre y / o órganos linfoides regionales. Vet. Res. Commun. 36, 245–250. DOI: 10.1007/S11259-012-9538-6
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Uzri, D. y Greenberg, H. B. (2013). Caracterización de ARN de rotavirus que activan la señalización inmune innata a través de los receptores tipo RIG-I. PLoS One 8:e69825. doi: 10.1371/journal.pone.0069825
Resumen de PubMed | Texto completo de CrossRef | Google Académico
van Rijn, P. A., Daus, F. J., Maris-Veldhuis, M. A., Feenstra, F. y van Gennip, R. G. P. (2017). Vacuna infecciosa de un solo animal (DISA) con discapacidad de la lengua azul: estudios sobre la ruta y la dosis óptimas en ovinos. Vacuna 35, 231–237. doi: 10.1016/j.vaccine.2016.11.081
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Vitour, D., Doceul, V., Ruscanu, S., Chauveau, E., Schwartz-Cornil, I. y Zientara, S. (2014). Inducción y control de la vía del interferón tipo I por el virus de la lengua azul. Virus Res. 182, 59–70. doi: 10.1016/j.virusres.2013.10.027
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Vladimer, G. I., Gorna, M. W. y Superti-Furga, G. (2014). IFITs: roles emergentes como proteínas antivirales clave. Frente. Immunol. 5:94.
Wirblich, C., Bhattacharya, B. y Roy, P. (2006). La proteína no estructural 3 del virus de la lengua azul ayuda a la liberación del virus mediante el reclutamiento de la proteína ESCRT-I Tsg101. J. Virol. 80, 460–473. DOI: 10.1128/JVI.80.1.460-473.2006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Wong, A. H., Tam, N. W., Yang, Y. L., Cuddihy, A. R., Li, S., Kirchhoff, S., et al. (1997). Asociación física entre STAT1 y la proteína quinasa PKR inducible por interferón e implicaciones para las vías de señalización de interferón y ARN bicatenario. EMBO J. 16, 1291–1304. doi: 10.1093/emboj/16.6.1291
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Wu, X., Chen, S. Y., Iwata, H., Compans, R. W. y Roy, P. (1992). Múltiples glicoproteínas sintetizadas por el segmento de ARN más pequeño (S10) del virus de la lengua azul. J. Virol. 66, 7104–7112. DOI: 10.1128/JVI.66.12.7104-7112.1992
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Yu, T., Yi, Y.-S., Yang, Y., Oh, J., Jeong, D. y Cho, J. Y. (2012). El papel fundamental de TBK1 en las respuestas inflamatorias mediadas por macrófagos. Mediadores inflamm. 2012:979105.
Zhang, Z., Kim, T., Bao, M., Facchinetti, V., Jung, S. Y., Ghaffari, A. A., et al. (2011). Las helicasas DDX1, DDX21 y DHX36 forman un complejo con la molécula adaptadora TRIF para detectar dsRNA en células dendríticas. Inmunidad 34, 866–878. doi: 10.1016/j.immuni.2011.03.027
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zwart, L., Potgieter, C. A., Clift, S. J. y van Staden, V. (2015). Caracterización de la proteína NS4 no estructural del virus de la peste equina africana. PLoS One 10:e0124281. doi: 10.1371/journal.pone.0124281
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: orbivirus, BTV-NS3, BTV-NS4, ARN bicatenario, STAT1, STAT2, TBK1, IFNAR–/– ratón
Cita: Rojas JM, Avia M, Martín V y Sevilla N (2021) Inhibición de la respuesta de IFN por el virus de la lengua azul: la historia hasta ahora. Frente. Microbiol. 12:692069. doi: 10.3389/fmicb.2021.692069
Recibido: 07 Abril 2021; Aprobado: 17 de mayo de 2021;
Publicado: 08 junio 2021.
Editado por:
Akio Adachi, Universidad Médica de Kansai, Japón
Revisado por:
Meredith Stewart, MRC-Centro de Investigación de Virus de la Universidad de Glasgow, Reino Unido
Gregory Caignard, INRA UMR 1161 Virologie, Francia
Molalegne Bitew, Instituto de Biotecnología de Etiopía (EBTi), Etiopía
Copyright © 2021 Rojas, Avia, Martín y Sevilla. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: José Manuel Rojas, rojas.jose@inia.es; Noemí Sevilla, sevilla@inia.es
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

