Investigación del impacto de la acidosis ruminal subaguda inducida por piensos en terneros jóvenes
Investigación del impacto de la acidosis ruminal subaguda inducida por piensos en el transcriptoma epimural y el metatranscriptoma del rumen en terneros jóvenes a las 8 y 17 semanas de edad
 Wenli Li1*
Wenli Li1*  Anna Larsen2,3
Anna Larsen2,3  Priscila Fregulia1,2
Priscila Fregulia1,2- 1Centro de Investigación de Forrajes Lácteos de EE. UU., Servicio de Investigación Agrícola del USDA, Madison, WI, Estados Unidos
- 2Instituto Oak Ridge para la Ciencia y la Educación, Oak Ridge, Tennessee, Estados Unidos
- 3Departamento de Ciencias Animales y Lácteas, Universidad de Wisconsin-Madison, Madison, WI, Estados Unidos
Introducción: Con el objetivo de maximizar la ingesta de una dieta alta en fermentación necesaria para satisfacer las necesidades energéticas durante el período de destete, los terneros corren el riesgo de sufrir acidosis ruminal. Utilizando los terneros del modelo previamente establecido de acidosis ruminal inducida por alimento en terneros jóvenes, nuestro objetivo fue investigar los cambios en el transcriptoma epimural del rumen y su metatranscriptoma microbiano al destete (8 semanas) y después del destete (17 semanas) en terneros Holstein canulados (que ocurrieron por primera vez a las 3 semanas de edad) con acidosis ruminal subaguda inducida por el alimento.
Métodos: Ocho terneros fueron asignados al azar a una dieta inductora de acidosis (Tratado, n = 4 ; peletizado, 42.7% de almidón, 15.1% de fibra detergente neutra [FDN] y 57.8% de carbohidratos sin fibra), mientras que el iniciador texturizado fue alimentado como control (Control, n = 4; 35.3% de almidón, 25.3% de FDN y 48.1% de carbohidratos sin fibra) a partir de 1 semana hasta 17 semanas. Los terneros alimentados con una dieta inductora de acidosis mostraron un peso corporal significativamente menor (p < 0,01) en el transcurso del experimento, además de un pH ruminal más bajo (p < 0,01) en comparación con el grupo control. Se recolectaron tejidos epiteliales del rumen (ER) a las 8 semanas (mediante biopsia) y a las 17 semanas (mediante eutanasia) y se realizó un seguimiento para el análisis de secuenciación del ARN del transcriptoma completo. El análisis de genes expresados diferencialmente (DEGs) se realizó mediante gemelos2 (cambio de pliegue ≥2 y p < 0,05) entre los grupos tratado y control a las 8 semanas de edad, y entre las 8 y las 17 semanas para el grupo tratado.
Resultados: A las 8 semanas de edad, los DEG entre los grupos de tratamiento mostraron un enriquecimiento de genes relacionados con la respuesta al lipopolisacárido (LPS) (p < 0,005). El impacto de la acidosis prolongada inducida por la alimentación se reflejó en la disminución de la expresión (p < 0,005) en los genes implicados en las vías relacionadas con la proliferación celular en el ER a las 17 semanas de edad en el grupo tratado. Se identificaron conjuntos únicos de taxones microbianos discriminantes entre los terneros de 8 y 17 semanas en el grupo tratado y los grupos de tratamiento a las 8 semanas, lo que indica que los cambios activos en la comunidad microbiana en el RE son una parte integral del desarrollo y la progresión de la acidosis ruminal.
Resúmenes
• La dieta alta fermentable en terneros jóvenes durante el período de destete induce acidosis ruminal.
• La acidosis ruminal prolongada inducida por el alimento afecta la expresión del transcriptoma en el epitelio ruminal.
• Los cambios en la comunidad microbiana en el epitelio ruminal son una parte integral del desarrollo y progresión de la acidosis ruminal.
• La respuesta al lipopolisacárido fue una respuesta importante en el epitelio ruminal a las 8 semanas de edad.
Introducción
En los rumiantes neonatos, el rumen está incompletamente desarrollado. A medida que el ternero consume más alimento de inicio, el pH ruminal disminuye, mientras que los ácidos grasos de cadena corta (AGCC), producidos por la fermentación ruminal, aumentan gradualmente. La administración intrarruminal de AGCC, como acetato, propionato y butirato, puede estimular el crecimiento de RE y masa ruminal en rumiantes jóvenes (1-3). Por lo tanto, se cree que la presencia y absorción de AGCC en el rumen son los estímulos químicos necesarios para la proliferación de las papilas funcionales del rumen. Sin embargo, se ha demostrado que las dietas altamente procesadas y ricas en almidón inducen acidosis en rumiantes (4, 5). Los terneros alimentados con una dieta alta en concentración mostraron una producción excesiva de AGCC y ácido láctico debido a la rápida descomposición microbiana de los carbohidratos, lo que conduce a una disminución significativa del pH del rumen (6). Una reducción general en el pH ruminal (7, 8) causada por la ingestión de dietas ricas en carbohidratos rápidamente fermentables con una cantidad insuficiente de fibra requerida para una amortiguación ruminal eficiente puede conducir a la acidosis ruminal subaguda (SARA), un trastorno metabólico común en el ganado lechero. El SARA es un trastorno bien reconocido y económicamente importante en el ganado lechero. En el ganado adulto, la reducción de la producción de leche, el sacrificio prematuro y el aumento de la mortalidad son algunas de las consecuencias directas de las disfunciones digestivas y metabólicas inducidas por SARA. Sin embargo, no se comprende bien el impacto a largo plazo del consumo de una dieta altamente fermentable desde el nacimiento hasta más allá del período de destete. El SARA se caracteriza en general por un pH ruminal bajo entre 5,2 y 6 durante períodos prolongados (9, 10). Sin embargo, los estudios han demostrado que existe una variación en la susceptibilidad de los animales a la acidosis inducida por el alimento y la gravedad de la depresión del pH requerida para causar síntomas de SARA varía entre las vacas (11-13). Los biomarcadores basados en la expresión génica se han investigado como biomarcadores predictivos para la detección de enfermedades (14-16). Estos biomarcadores tienen un alto potencial como alternativa o para ser utilizados junto con la medición del pH ruminal para el diagnóstico precoz o la prevención de la acidosis.
Hemos establecido un modelo de acidosis inducida por alimento en terneros jóvenes, donde se ofreció a los terneros una dieta altamente procesada y fermentable al nacer hasta las 16 semanas de edad para obtener una acidosis prolongada (17, 18). Con este modelo, observamos diferencias significativas en los valores medios de pH ruminal entre los grupos tratado y control. Lo más importante es que se observó un mayor grado de degradación tisular en los terneros acidóticos (p < 0,01) (18). Los principales objetivos de este trabajo son (1) investigar los cambios en el transcriptoma de RE y la comunidad microbiana asociada al destete (8 semanas de edad) entre los grupos de tratamiento; y (2) investigar la respuesta de la ER del huésped a la acidosis prolongada inducida por la alimentación, a través del análisis transcriptómico comparativo en la ER entre los dos puntos temporales, 8 y 17 semanas de edad, en el grupo tratado. Nuestra hipótesis es que la acidosis inducida por el alimento se acompaña de cambios significativos en la expresión génica en el epitelio ruminal de los terneros. Además, planteamos la hipótesis de que un cambio significativo en la microbiota asociada al epitelio ruminal, la microbiota epimural, es una parte integral de la respuesta del huésped a la acidosis inducida por el alimento, y que esta respuesta difiere en los períodos de destete y posdestete.
Materiales y métodos
Todos los protocolos para animales (A005848) fueron aprobados por el Comité de Cuidado y Uso de Animales de la Universidad de Wisconsin-Madison. Todos los procedimientos relacionados con el cuidado y uso de los animales en este estudio se implementaron de acuerdo con las pautas y regulaciones del Centro de Investigación de Forrajes Lecheros de EE. UU. Todos los terneros de toro Holstein incluidos en este estudio pertenecían al mismo estudio publicado anteriormente (17-20).
Diseño experimental, mediciones y recolección de muestras
Este estudio es parte de un estudio más amplio, en el que se han publicado otras partes del estudio. El procedimiento para la acidosis inducida por el alimento fue descrito anteriormente (17-20). En resumen, se inscribieron 8 terneros de toro Holstein para este experimento. Durante las primeras 8 semanas después del nacimiento, los terneros se alojaron en conejeras individuales (4,8 m2/ternero), luego fueron trasladados a superconejeras divididas (5.0 m2/ternero), donde los terneros tenían su propio corral individual, y solo podían acceder a su propio alimento y agua, hasta las 16 semanas de edad. Se administraron dos dietas a los terneros. Una de ellas era una dieta baja en fibra y granulada. Esta dieta fue diseñada para causar acidosis ruminal (tratada; peletizada, 42.7% de almidón, 15.1% de fibra detergente neutra [FDN] y 57.8% de carbohidratos sin fibra). El iniciador texturizado se administró como dieta control (35,3% de almidón, 25,3% de FDN y 48,1% de carbohidratos sin fibra). El pienso es fabricado por Vita Plus Cooperation (Wisconsin, Estados Unidos). La composición completa de nutrientes de cada dieta se proporciona en Gelsinger et al. (17), y los efectos de las dos dietas se informaron previamente (17, 18). Ocho terneros de toro Holstein fueron asignados al azar a una dieta inductora de acidosis (Tratado; n = 4) y la dieta de control (n = 4), a través de la herramienta generadora de números aleatorios de Graphpad.1 Los iniciadores de terneros se ofrecieron a terneros a la semana de edad y duraron hasta las 17 semanas, utilizando terneros nacidos entre el 17 de junio y el 5 de julio de 2017. Los terneros tuvieron acceso ad libitum al agua durante la duración del estudio.
A las 3 semanas de edad, se colocaron quirúrgicamente cánulas de goma blanda (28 mm de diámetro interior) en cada ternero siguiendo el método de Kristensen y coautores (21). Entre las 7 y las 9 semanas de edad, las cánulas de caucho blando más grandes (51 mm de diámetro interior; Bar-Diamond Inc., Parma, ID, Estados Unidos) se utilizaron para reemplazar las cánulas originales para adaptarse al aumento en el tamaño de la fístula. A las 8 semanas, se realizó una biopsia RE en el saco craneal utilizando una herramienta de biopsia uterina para cada ternero. A las 17 semanas, los terneros fueron sacrificados mediante aturdimiento con perno cautivo para la recolección de tejido del tracto digestivo, incluyendo RE, hígado, intestino delgado y ciego. Se fijaron tejidos de las papilas ruminales de los lados izquierdo y derecho del saco ventral craneal para su análisis histológico con el fin de evaluar las puntuaciones de la lesión (18). En ambos momentos, los tejidos RE se recolectaron y se enjuagaron en PBS 1X para eliminar las partículas restantes del alimento. Los tejidos limpios se congelaron inmediatamente en nitrógeno líquido y se almacenaron a -80 °C para su posterior extracción de ARN.
Se insertó un electrodo de pH calibrado (Oakton, Cole Parmer Instrument, Estados Unidos) en el rumen a través de la cánula antes de cada recolección durante 45 s para medir el pH ruminal. El pH ruminal se midió en siete puntos temporales (−8, −4, 0, 2, 4, 8 y 12 h en relación con la alimentación con grano) en un solo día cada dos semanas desde las 6 hasta las 16 semanas. El peso corporal se registró semanalmente. A las 0800 h se ofrecía diariamente una cantidad medida de arrancador y se determinaban diariamente los rechazos. Cuando se registró el rechazo <200 g en 2 días consecutivos, se incrementó la asignación diaria de iniciador de un ternero (18). Las puntuaciones de salud de los terneros se monitorizaron midiendo semanalmente la frecuencia cardíaca y respiratoria, la temperatura rectal, la consistencia fecal y las características navales.
Análisis del poder de secuenciación del ARN
Utilizando el paquete de bioconductores ssizeRNA (22), determinamos que 4 réplicas biológicas por tratamiento con 20 millones de lecturas por muestra serían suficientes para identificar genes expresados diferencialmente a partir de datos de secuenciación de ARN a una potencia de 0,90 utilizando los siguientes parámetros: corte estadístico = 0,8, número de genes = 20.000, número mínimo de DEG = 200, recuento medio de lecturas = 1.000, y fold change = 2, FDR = 0.05. Obtuvimos entre 50 y 60 millones de lecturas por muestra en el diseño de nuestro experimento. Por lo tanto, la profundidad de secuenciación garantizaría suficiente poder estadístico para el análisis del transcriptoma.
Extracción de ARN y preparación de la biblioteca de secuenciación
Para la extracción del ARN total del huésped, se homogeneizaron 50 mg de tejidos de papilas ruminales en el homogeneizador Precellys (Instrumento Bertin, Francia) a 7.500 RPM durante 30 s por ciclo, con 4 repeticiones. El homogeneizado de tejido se colocó en hielo durante 1 min entre ciclos. Después de la homogeneización, se extrajeron los ARN totales de ambos tipos de tejido siguiendo el protocolo miRNeasy con un instrumento QIAcube (Qiagen, Estados Unidos). La calidad de los ARN extraídos se evaluó utilizando el kit Bioanalyzer RNA 6000 nano (Agilent Technologies, Estados Unidos). Se buscaron muestras de ARN con un valor de número de integridad de ARN (RIN) ≥8 para la cuantificación de ARN utilizando Qubit (Thermo Fisher, Estados Unidos). La preparación de la biblioteca de secuenciación de ARN se realizó utilizando el kit Illumina TruSeq ribo-zero Gold siguiendo las instrucciones del fabricante. Para cada muestra, se utilizó 1 μg de ARN total para la preparación de la biblioteca de secuenciación. La cuantificación de las bibliotecas preparadas se realizó utilizando un kit de cuantificación Kapa siguiendo las instrucciones del fabricante (KK4873, Kapa systems, Roche, Suiza) con un instrumento QuanStudio 5 RT-qPCR (ThermoFisher Scientific, Estados Unidos). Utilizando la concentración generada por el kit Kapa, se preparó la agrupación secuencial de acuerdo con el cálculo ofrecido por la calculadora de agrupación.2 Las bibliotecas agrupadas se secuenciaron inicialmente utilizando un kit Illumina MiSeq nano de 300 ciclos. La agrupación se normalizó aún más para garantizar la misma profundidad de secuenciación de todas las bibliotecas, de acuerdo con las proporciones de índice generadas por la ejecución del nanokit. Las bibliotecas agrupadas finalmente normalizadas se secuenciaron en el instrumento Illumina NextSeq 500, utilizando un cartucho de 300 ciclos de alto rendimiento para generar lecturas de 2×150 bp en el extremo emparejado.
Análisis bioinformático de lecturas de secuenciación de RE y clasificación de taxones de lecturas microbianas
El análisis de los datos de secuenciación siguió el procedimiento descrito en la publicación del coautor (20). A partir de este estudio se generaron datos brutos de secuenciación de ARN para los tejidos ER a las 8 semanas de edad, los de las 17 semanas de edad se obtuvieron de nuestro trabajo publicado anteriormente (20). Brevemente, las lecturas brutas se asignaron al genoma de referencia del ganado bovino (ARS-UCD 1.2) utilizando STAR (2.5.2b) (23). El análisis de genes expresados diferencialmente (DEG) se realizó mediante el paquete de manguitos en gemelos2 (24). Para determinar significativamente los genes expresados diferencialmente, se utilizaron los siguientes valores de corte: FPKM ≥5, valor de ajuste de p <0,05 y cambio de pliegue (FC) ≥2. La ontología génica (GO) y el análisis de vías se realizaron mediante DAVID (25). Para estas comparaciones se realizó un análisis de DEG y un análisis de GO asociado: entre los grupos de tratamiento a las 8 semanas de edad y entre las 8 y las 17 semanas de edad en el grupo de tratamiento. Además, se obtuvieron DEGs entre las semanas 8 y 17 en terneros alimentados con dieta control para identificar genes expresados diferencialmente debido al crecimiento y madurez. Y estos DEG se filtraron de la lista de DEG entre los dos puntos temporales en el grupo tratado para identificar los DEG debidos a acidosis prolongada.
Las lecturas no mapeadas del genoma de referencia del ganado bovino se consideraron de origen microbiano. Para enriquecer aún más las lecturas microbianas de ARNr, se utilizó SortMeRNA (versión 2.1b) (26) para mapear las lecturas no mapeadas del genoma del huésped a las bases de datos de ARNr de referencia proporcionadas por SILVA (versión 138) (27) y Rfam 11.0 (28). Las lecturas de ARNr enriquecidas se utilizaron para la clasificación taxonómica bacteriana, por Kraken2 (v.2.0.8-beta) (29) utilizando la base de datos estándar que incluye bacterias y arqueas. Los recuentos de lecturas sin procesar en cada nivel taxonómico (es decir, filo y género) identificados por Kraken2 se normalizaron por el número total de lecturas clasificadas por muestra mediante estos pasos: (1) el número total de lecturas asignadas al nivel taxonómico dado (es decir, filo, género y especie) se dividió por 1.000.000 para obtener el «factor por millón»; (2) El número total de lecturas asignadas a cada nivel taxonómico específico se dividió por el «factor por millón» para obtener el recuento de lecturas normalizado. Se utilizaron recuentos de lectura normalizados a un nivel taxonómico dado como medida para el análisis de abundancia microbiana. Se utilizó el conteo de lectura normalizado de 2 o más para determinar la presencia/expresión de los taxones. El análisis de abundancia diferencial de taxones microbianos a nivel de género se realizó utilizando DEseq2 (con el método de Benjamini y Hochberg para pruebas múltiples) utilizando los recuentos de lectura brutos mapeados a los taxones dados (p < 0.05; recuento de lectura promedio ≥10 y cambio de plegamiento ≥2). Para los tejidos RE recolectados a las 17 semanas de edad, utilizamos recuentos de lecturas a nivel de género de nuestro trabajo publicado anteriormente (19). Se sabe que el desarrollo ruminal va acompañado de colonización microbiana (30). Estábamos interesados en identificar los cambios en la firma microbiana en el RE debido a la acidosis ruminal prolongada mediante la comparación de las comunidades microbianas en los terneros tratados entre las 8 y las 17 semanas. Por lo tanto, se eliminaron del análisis los géneros microbianos que mostraron cambios diferenciales significativos en la abundancia debido al desarrollo y la maduración entre los dos puntos temporales en el grupo control (44 de ellos).
Análisis de la comunidad microbiana y variables experimentales
Se utilizó el método multivariado mixMC implementado en el paquete mixOmics R (31) para identificar asociaciones específicas entre la abundancia de taxones microbianos y las variables experimentales. Identificamos firmas microbianas relacionadas con los siguientes tratamientos: (1) tratados vs. control para animales a las 8 semanas de edad; (2) muestras recogidas a las 8 y 17 semanas de edad para el grupo tratado, y a las 17 semanas para el grupo control. Los taxones microbianos que mostraron diferencias significativas de abundancia entre los dos puntos temporales (8 y 17 semanas) en el grupo control se consideraron afectados por el crecimiento y se eliminaron (44 de ellos) del análisis microbiano para muestras de 8 y 17 semanas en el grupo tratado. Dentro del marco estadístico del paquete mixMC, se utilizó el análisis discriminante de mínimos cuadrados parciales dispersos (sPLS-DA) (32) para realizar la selección de características mediante el supuesto de escasez, suponiendo que un pequeño número de características puede impulsar un evento biológico (33). En este análisis se incluyeron los taxones microbianos con abundancia relativa >0,01%. El número óptimo de componentes se seleccionó en función de la tasa de error de clasificación equilibrada promedio con una distancia máxima de más de 10 repeticiones de una validación cruzada de 3 veces de un modelo sPLS-DA. El número óptimo de variables para cada componente se eligió en función de la tasa de error de clasificación balanceada promedio más baja. Los géneros discriminantes se graficaron de acuerdo con su contribución al componente 1 de 2 de sPLS-DA.
Resultados
pH ruminal, peso corporal y degradación de las papilas ruminales en los grupos tratado y control
Los resultados detallados para el peso corporal, la ingesta de alimento, el pH ruminal y las papilas ruminales se informaron anteriormente (18, 20). Brevemente, el pH ruminal medio, que fue de 5,37 ± 0,24 (3,3, 7,2) y 5,63 ± 0,24 (3,5, 6,8) para los terneros tratados y control, respectivamente, difirió según la dieta (p < 0,01). El peso corporal y el consumo de alimento mostraron un aumento lineal con la edad (p < 0,01). Tanto a las 4 como a las 5 semanas de edad, los terneros alimentados con la dieta control alcanzaron un mayor peso corporal (p < 0,01) y consumieron una mayor cantidad de alimento (p < 0,01) en comparación con el grupo tratado. Estas diferencias se mantuvieron durante la semana 16 (p < 0,001). Para el análisis histológico de las papilas ruminales, se observó un mayor grado de degradación tisular para el grupo tratado en comparación con el control (p < 0.01) (18).
La secuenciación lee la alineación de los tejidos epiteliales del rumen
Para los tejidos ruminales recolectados a las 8 semanas de edad, el promedio del número total de lecturas es de 59,8 M ± 1,2 M, con una tasa promedio de mapeo a la referencia bovina del 76% ± 2%. Para este punto de tiempo, el número promedio de lecturas de ARNr asignadas a la base de datos de ARNr y utilizadas para la clasificación microbiana es de 5,7 M ± 0,6 M (media ± e.e.). Del mismo modo, se obtuvo una alta tasa de clasificación de Kraken para estas muestras de 99,2 ± 0,12 (media ± s.e.). Para los tejidos ruminales recolectados a las 17 semanas de edad, el promedio del número total de lecturas es de 57,5 M ± 0,4 M, con una tasa promedio de mapeo del genoma de referencia del ganado bovino del 56,8% ± 4,3%. Y para este punto de tiempo, el número promedio de lecturas de ARNr asignadas a la base de datos de ARNr y utilizadas para la clasificación microbiana de la pared del rumen es de 9,3 M ± 1,6 M (media ± e.e.). Se logró una alta tasa de clasificación para cada muestra, con una tasa media de clasificación de Kraken de 99,39 ± 0,13 (media ± s.e.).
Perfil de expresión génica en el RE a las 8 semanas
Un total de 82 genes mostraron cambios significativos en la expresión diferencial entre los grupos de tratamiento a las 8 semanas de edad (FPKM ≥5, FC ≥ 2 y valor de p ≤0,05). En comparación con el grupo control, 24 genes mostraron un aumento en la expresión y 58 de ellos mostraron una disminución en la expresión en el grupo tratado. El análisis de GO utilizando DEG identificados indicó que estos genes estaban enriquecidos en las vías relacionadas con la respuesta inmunitaria. Incluyen la respuesta inmune innata (GO:0045087, 10 genes, valor de p < 0,01), la respuesta celular al lipopolisacárido (LPS; GO:0071222, 5 genes, valor de p < 0,05) y vía de señalización medicada por quimiocinas (GO:0070098, 4 genes, valor de p < 0,05).
Para los 10 genes más expresados en el grupo tratado, observamos un enriquecimiento significativo en las vías celulares relacionadas con la respuesta al estrés (ISG15, S100A9, LGALS4, PPIF y PLIN2). En concreto, se identificaron 3 de estos genes con función anotada en respuesta a bacterias (ISG15, S100A9 y LGALS4).
Cambios en la expresión génica del rumen en el grupo tratado entre las 17 y las 8 semanas
Para el grupo tratado, comparamos los perfiles de expresión génica entre los puntos de tiempo de 8 y 17 semanas. Entre los dos puntos temporales en el grupo tratado, un total de 502 genes mostraron expresión diferencial significativa (FPKM ≥5, FC ≥ 2 y valor de p ≤0,05). En el grupo control, se identificaron un total de 313 DEG entre las 8 y las 17 semanas. En ausencia de dieta inductora de acidosis, estos DEG representaron cambios en el transcriptoma debidos al desarrollo, se eliminaron de la lista de DEG identificados en el grupo tratado. Después del filtrado, 357 DEG fueron únicos para la comparación en el grupo tratado y se les hizo un seguimiento para su posterior análisis. Para estos genes, 112 de ellos estaban regulados al alza y 245 de ellos estaban regulados a la baja a las 17 semanas de edad en comparación con las 8 semanas de edad. El análisis de los términos de GO utilizando los genes regulados al alza a las 17 semanas de edad indicó enriquecimientos predominantes en las vías relacionadas con la proliferación celular y la migración, y: espacio extracelular (GO:0005615, 24 genes, valor de p << 0,0001), adhesión celular (GO:0007155, 24 genes, p << 0,0001), proliferación celular (GO:0008283, 24 genes , valor de p << 0,0001), migración celular (GO:0016477, 19 genes; valor de p << 0,0001), regulación del proceso del sistema inmune (GO:0002682, 20 genes, valor de p < 0,01), lisosoma (GO:0005763, 11 genes, valor de p < 0,005), regulación de la activación leucocitaria (GO:0002694, 13 genes, valor de p < 0,005), regulación de la activación de linfocitos (GO:0051249, 12 genes, valor de p < 0,005) y complejo proteico MHC clase II (GO:0042613, 4 genes, valor de p < 0,05; Tabla 1). El análisis del término GO utilizando los genes regulados a la baja a las 17 semanas de edad indicó un enriquecimiento en los procesos metabólicos de lípidos y ácidos orgánicos. Incluyen el proceso metabólico lipídico (GO:0006629, 24 genes, valor de p < 0,00001), la biosíntesis de colesterol (GO:0006695; 8 genes, valor de p < 0,0001), el proceso metabólico de ácidos orgánicos (GO:0006082, 31 genes, valor de p < 0,05), el proceso biosintético del ácido carboxílico (GO:0046394; 31 genes, valor de p < 0,001) y el proceso metabólico de compuestos hidroxi orgánicos (GO:1901615, 19 genes, valor de p < 0,01; Tabla 2).
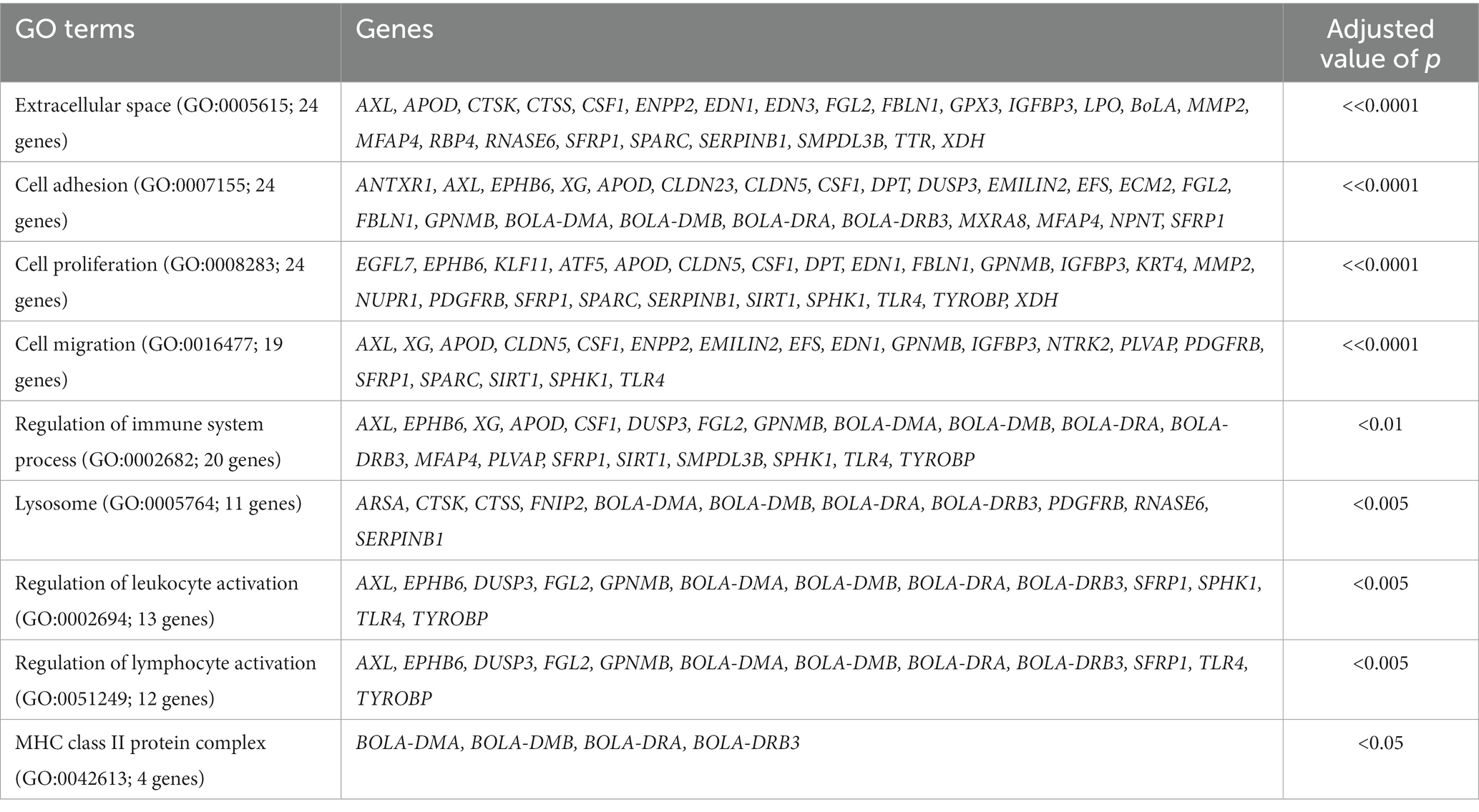 Tabla 1. Los términos GO y los genes asociados se identificaron utilizando genes regulados al alza a las 17 semanas de edad en comparación con las 8 semanas del grupo tratado.
Tabla 1. Los términos GO y los genes asociados se identificaron utilizando genes regulados al alza a las 17 semanas de edad en comparación con las 8 semanas del grupo tratado.
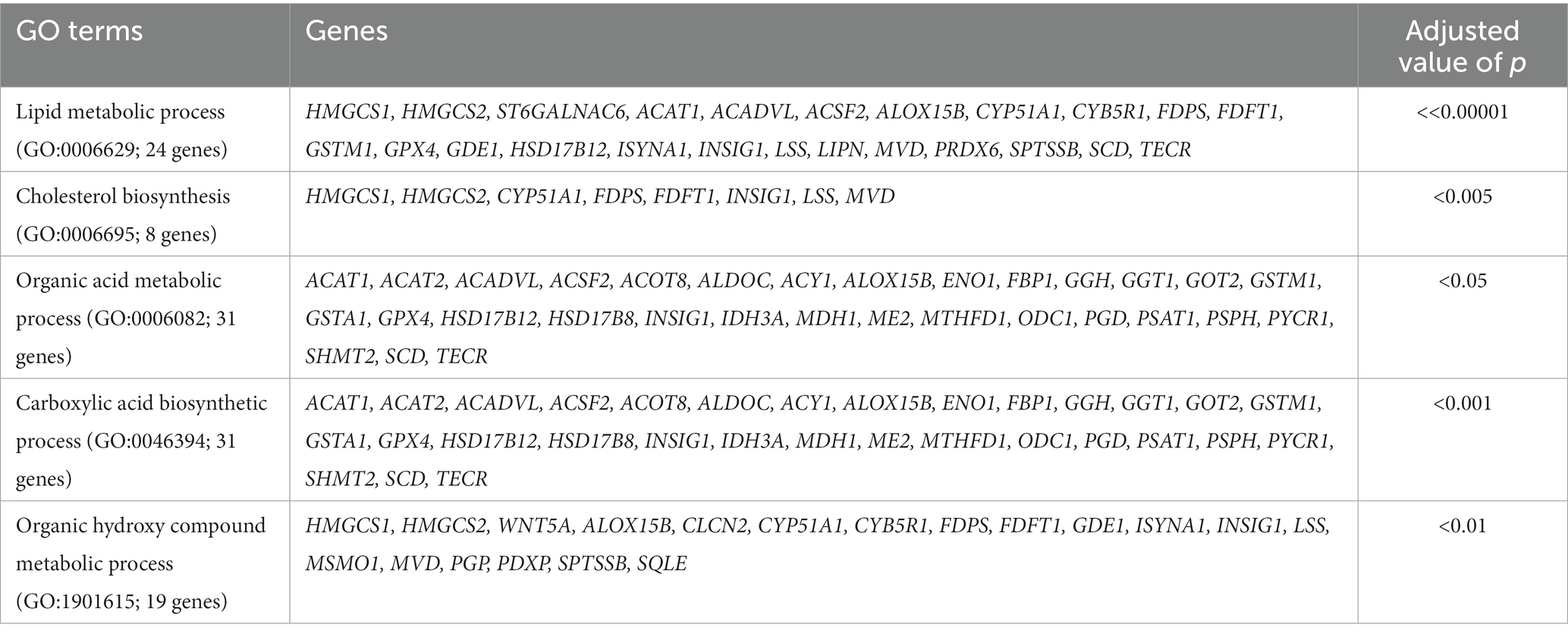 Tabla 2. Los términos GO y los genes asociados se identificaron utilizando genes regulados a la baja a las 17 semanas de edad en comparación con las 8 semanas en el grupo tratado.
Tabla 2. Los términos GO y los genes asociados se identificaron utilizando genes regulados a la baja a las 17 semanas de edad en comparación con las 8 semanas en el grupo tratado.
Finalmente, para los genes regulados al alza en el grupo tratado (URGT) tanto a las 8 como a las 17 semanas de edad, observamos un total de 10 genes con una expresión significativamente aumentada en ambos puntos temporales en el grupo tratado. Estos genes incluyen CNFN, EPHB6, KLK12, KRTDAP, NOXA1, PI3, PSORS1C2, RAB38, S100A9 y TMEM229B. El análisis de enriquecimiento de vías indicó que estos genes estaban enriquecidos en la membrana celular (GO:0016020; 4 genes, valor de p < 0,05). Para ambos puntos temporales, 8 genes mostraron una disminución significativa de la expresión en el grupo tratado. Entre ellos se encontraban BEST2, CCL20, CMPK2, ENTPD5, LGALS4, PRSS35, RBP4 y UGT2B10.
Perfil de la comunidad microbiana en el RE a las 8 y 17 semanas de edad
El análisis sPLS-DA reveló una clara separación en los perfiles microbianos entre la biopsia de los animales tratados (8 semanas), los animales tratados en el momento del sacrificio (17 semanas) y los animales de control en el momento del sacrificio (Figura 1A). Además, se identificó una clara separación para las siguientes comparaciones por pares: muestras de control y tratadas recolectadas a las 8 semanas de edad (Figura 1B); muestras recogidas a las 8 y 17 semanas para los animales tratados (Figura 1C). En general, el 40% de la firma bacteriana seleccionada en el componente 1 del sPLS-DA caracterizó el microbioma ruminal de los animales del grupo de control en el momento del sacrificio, que incluía los taxones bacterianos Gallibacterium, Mediterraneibacter, Ligilactobacillus, Massilistercora, Limosilactobacillus y L actobacillus. Por otro lado, el 33% de la firma microbiana caracterizó el microbioma ruminal de los animales del grupo tratado en la biopsia, y la firma de este grupo estuvo compuesta por Fusobacterium, Oscillibacter, Parabacteroides, Desultovibrio y Porphyromonas. Para el grupo tratado en el momento del sacrificio, la firma microbiana estaba compuesta por Olsenella, Cloacibacillus, Comamonas y Bacteroides (Figura 2). De acuerdo con el análisis sPLS-DA, el mapa de calor mostró una clara distinción entre los tratamientos (Figura 3).
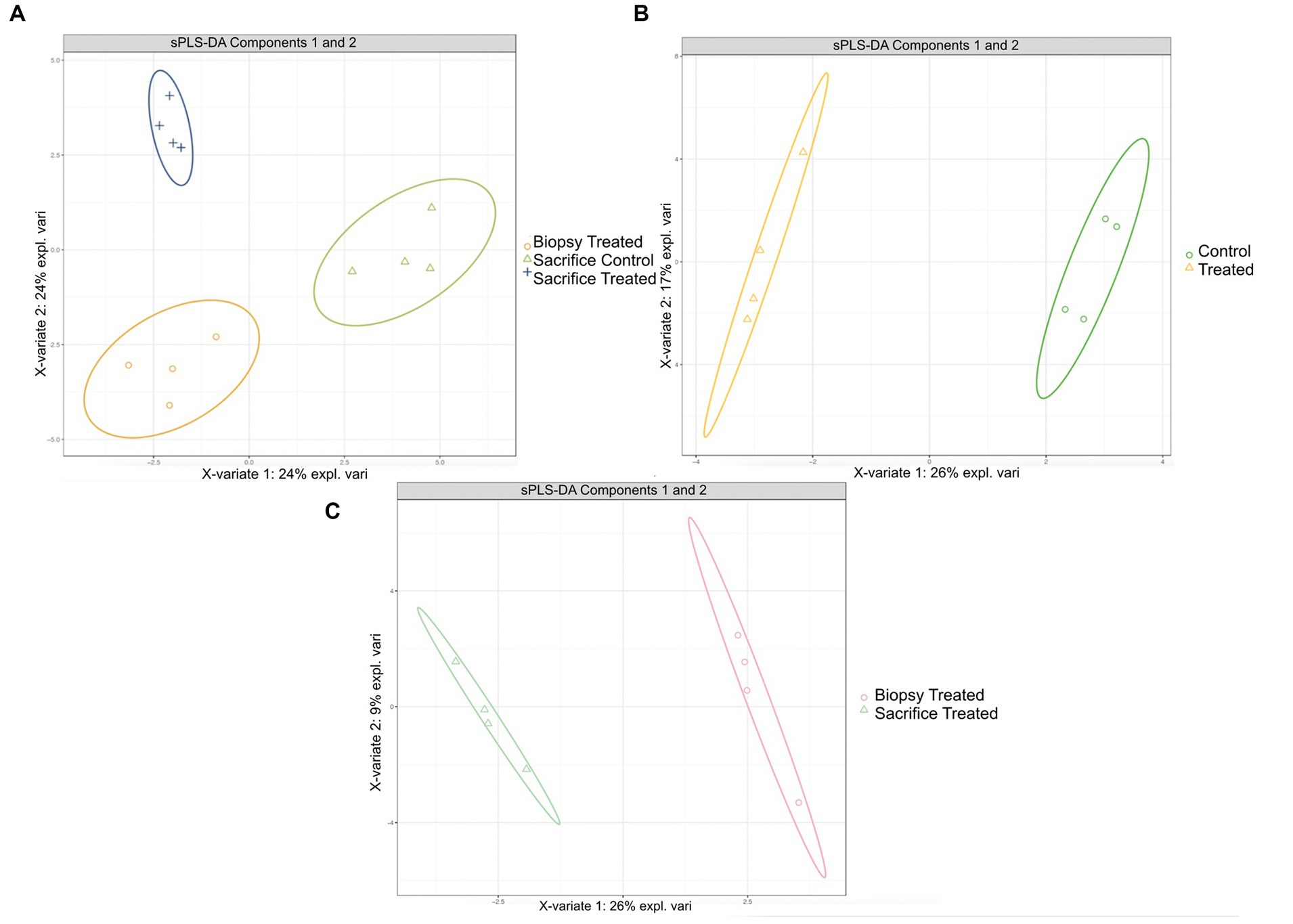 Figura 1. Resultados de sPLS-DA para el perfil microbiano a nivel de género en rumen de terneros. Gráfico de puntuación individual de las muestras a lo largo de los dos primeros componentes, con un nivel de confianza del 95%. (A) Perfiles microbianos de ER en terneros tratados a las 8 semanas, terneros tratados y control al sacrificio (17 semanas). (B) Perfiles microbianos para muestras de ER recogidas de terneros de control y tratados a las 8 semanas; (C) Perfiles microbianos para muestras de ER recogidas a las 8 semanas y a las 17 semanas para los terneros tratados.
Figura 1. Resultados de sPLS-DA para el perfil microbiano a nivel de género en rumen de terneros. Gráfico de puntuación individual de las muestras a lo largo de los dos primeros componentes, con un nivel de confianza del 95%. (A) Perfiles microbianos de ER en terneros tratados a las 8 semanas, terneros tratados y control al sacrificio (17 semanas). (B) Perfiles microbianos para muestras de ER recogidas de terneros de control y tratados a las 8 semanas; (C) Perfiles microbianos para muestras de ER recogidas a las 8 semanas y a las 17 semanas para los terneros tratados.
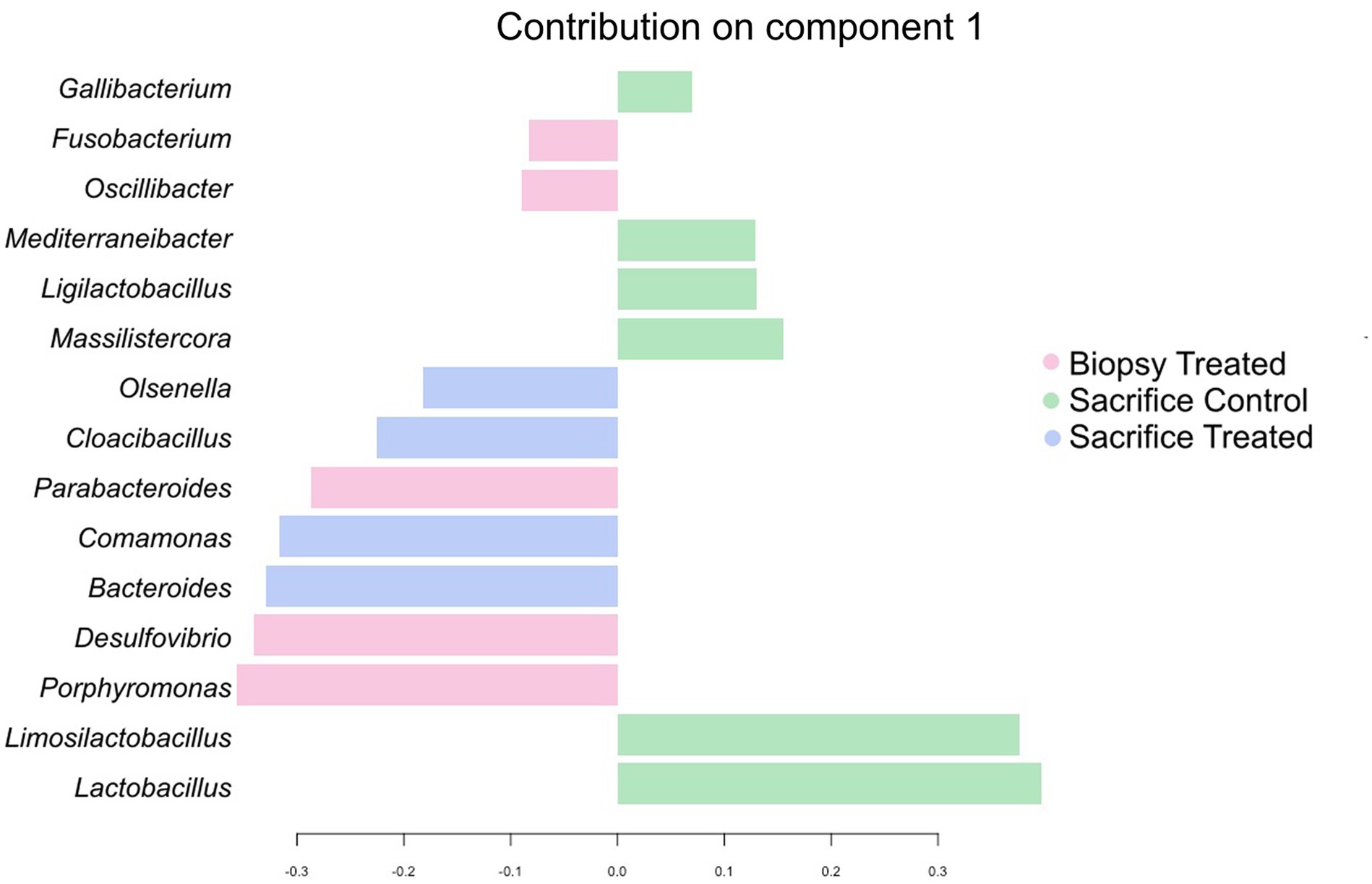 Figura 2. La contribución de los géneros se clasificó de abajo (la más importante) a la mayor para los grupos tratado y control recolectados a las 8 semanas de edad. Los colores indican el tratamiento en el que la característica fue más relevante.
Figura 2. La contribución de los géneros se clasificó de abajo (la más importante) a la mayor para los grupos tratado y control recolectados a las 8 semanas de edad. Los colores indican el tratamiento en el que la característica fue más relevante.
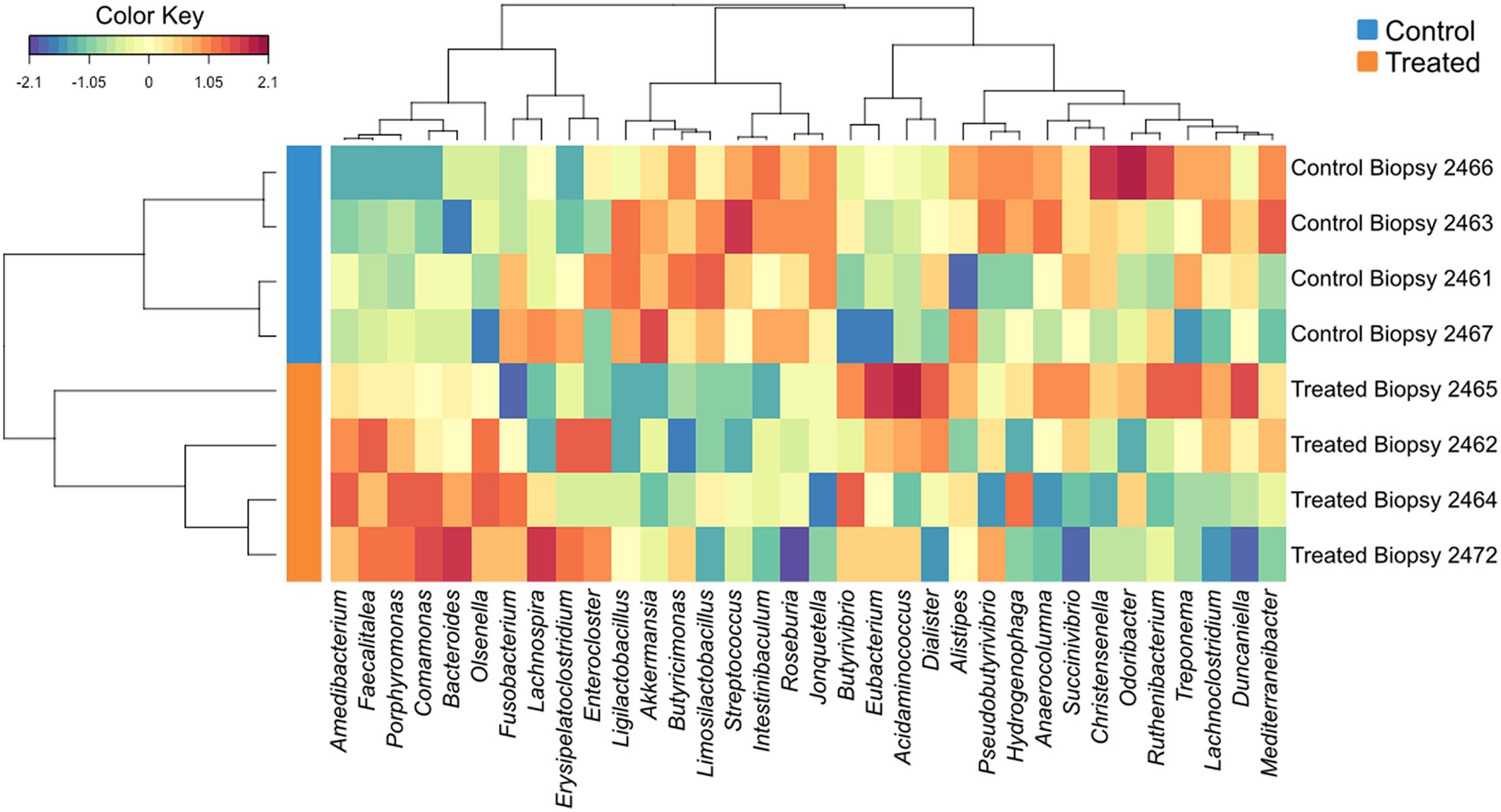 Figura 3. Agrupamiento jerárquico (distancia euclidiana, ligamiento de Ward) de los géneros seleccionados a partir de los resultados de sPLS-DA. Los grupos de control se indicaron con barras azules y los grupos tratados con barras naranjas.
Figura 3. Agrupamiento jerárquico (distancia euclidiana, ligamiento de Ward) de los géneros seleccionados a partir de los resultados de sPLS-DA. Los grupos de control se indicaron con barras azules y los grupos tratados con barras naranjas.
Discusión
Respuesta de la ER del huésped a la inducción de SARA a las 8 semanas de edad
En este experimento, monitoreamos las puntuaciones de salud de la cría midiendo la frecuencia cardíaca y respiratoria, la temperatura rectal, la consistencia fecal y las características navales semanalmente. Como era de esperar, no detectamos ninguna diferencia cualitativa en estas mediciones entre los grupos de tratamiento a las 8 semanas de edad (datos no publicados), ya que el SARA es un trastorno subclínico y los signos clínicos no se observan con frecuencia. Nuestro estudio indicó que la respuesta del huésped a la dieta acidótica fue capturada por los cambios en el transcriptoma en el epitelio ruminal. En particular, para los DEG, observamos un enriquecimiento de GO en la respuesta celular al lipopolisacárido a las 8 semanas de edad. A pesar de los animales canulados en este estudio, que potencialmente pueden influir en la fermentación ruminal y la respuesta inflamatoria (34), todos los terneros incluidos en el análisis comparativo fueron canulados, eliminando la diferencia debida a la canulación sola. En este estudio, la dieta de tratamiento es una dieta alta en concentrados, dirigida a inducir acidosis ruminal subaguda. En el ganado bovino, los modelos SARA inducidos por la dieta han dado lugar a cambios deletéreos en la digesta ruminal y la sangre periférica (35). Estos incluyen la depresión del pH ruminal, el aumento de la concentración de endotoxinas bacterianas [es decir, lipopolisacáridos (LPS)] y bioaminas (p. ej., histamina, tiramina y triptamina). Se ha informado que el SARA moderado reduce la función de barrera intestinal ruminal, lo que resulta en la translocación de estas toxinas bacterianas (36, 37). Las bacterias ruminales son predominantemente Gram negativas (38), que contienen LPS en su delgada pared celular de peptidoglicano. El LPS es importante para la integridad estructural y funcional de las bacterias (39). Por lo tanto, las bacterias Gram negativas son la principal fuente de LPS en el rumen. Se ha reportado un aumento significativo (de 4 a 16 carpetas más) de la concentración de LPS en el rumen en vacas con SARA en comparación con las que no lo tenían (40, 41). El LPS también puede translocarse de la función de barrera RE comprometida al torrente sanguíneo y otras partes del cuerpo, invocando una respuesta inflamatoria local y sistémica (36, 37, 42), al estimular la liberación de citoquinas proinflamatorias, como TNF-α, IL-1β e IL-6 (43). Además, se ha informado de un aumento de las proteínas de fase aguda, como la amiloide A sérica, la haptoglobina, la proteína C reactiva y la glicoproteína alfa-1 ácida en la sangre periférica del ganado bovino (40, 44).
De acuerdo con esto, los genes más expresados en el grupo tratado incluyeron 5 genes enriquecidos en la vía celular de respuesta al estrés (ISG15, S100A9, LGALS4, PPIF y PLIN2). Y se identificaron 3 de estos genes con función reportada en respuesta directa a bacterias (ISG15, S100A9 y LGALS4). En particular, se informó que el PPIF tiene un papel en la regulación de la necrosis tisular debida al estrés o la enfermedad (45). La expresión de estos genes no se ha estudiado antes por su potencial poder diagnóstico para detectar con SARA inducido por la dieta en bovinos. El manejo de la acidosis ruminal basado en la dieta es muy deseable, ya que es relativamente fácil de manejar y rentable en una granja sin el costo devastador de las intervenciones clínicas. Un seguimiento adicional de estos genes en una cohorte más grande puede ayudar a determinar si el aumento de la expresión en el sistema circulatorio en cualquiera de estos genes puede servir como biomarcadores eficaces de detección temprana para la propensión a desarrollar acidosis ruminal.
Respuesta del ER del huésped a la acidosis subaguda prolongada a las 17 semanas de edad en el grupo tratado
En nuestro experimento, los efectos de la alimentación prolongada con una dieta alta en concentrados fueron capturados por los cambios en el transcriptoma entre los terneros de 17 y 8 semanas de edad alimentados con una dieta inductora de acidosis. Observamos la tendencia al aumento de la expresión génica en genes relacionados con la proliferación celular, lo que indica que la acidosis prolongada inducida por el alimento puede afectar el desarrollo de la masa física ruminal. El rumen es la cámara más grande en el estómago del ganado y es el lugar principal donde el alimento no digerible se fermenta y se convierte en nutriente absorbible por el ganado. Los nutrientes, como los minerales y los AGCC, son absorbidos por la pared del rumen (46). Las papilas que sobresalen de la pared del RE aumentan en gran medida el área de superficie para la absorción de AGCC que representa el 75% del suministro de energía metabolizable (47). La longitud, el ancho y la densidad de las papilas ruminales contribuyen directamente al grosor del RE, y se ha reportado su asociación con la eficiencia de la alimentación del ganado (48, 49). Como respuesta adaptativa al aumento de los niveles de AGCC debido a una dieta alta en concentrados, se informó de un aumento del tamaño de las papilas en ovejas para maximizar el área de superficie para la absorción de AGCC (50). Se informó en ovejas y bovinos que el aumento del nivel de concentración de AGCC en el rumen puede conducir a una rápida proliferación del epitelio escamoso estratificado del rumen y su morfogénesis (3, 51).
Los genes que codifican el lisosoma y que se asocian con el proceso inmune mostraron un aumento en la expresión a las 17 semanas de edad. Los lisosomas son orgánulos unidos a la membrana con funciones en los procesos implicados en la degradación y el reciclaje de los residuos celulares y las células muertas, la señalización celular y el metabolismo energético. Como parte inherente del sistema de defensa intracelular contra los microbios, la biogénesis de los lisosomas es desencadenada por infecciones intracelulares de patógenos y desempeña un papel clave en la detección y señalización de patógenos (52). Por lo tanto, las actividades de los lisosomas están directamente relacionadas con la inmunidad innata del huésped (53-55). Yoon y sus coautores (56) informaron que los lisosomas aislados de la clara de huevo mostraron un efecto antimicrobiano significativo contra varios organismos microbianos, lo que sugiere que los lisosomas podrían ser una alternativa antibiótica prometedora. Se observó erosión epitelial ruminal con acidosis ruminal (57, 58). En nuestro estudio, se observó una mayor escala de degradación tisular en los tejidos de las papilas ruminales en terneros acidóticos (18). Con un pH ruminal bajo prolongado, los terneros tratados en nuestro estudio no mostraron ningún signo clínico de SARA (datos no publicados). Nuestra hipótesis es que gran parte de esto se debe a la regulación de la inmunidad innata del huésped y su capacidad para combatir patógenos potenciales en estrecho contacto con el epitelio ruminal.
En el caso de los genes regulados a la baja a las 17 semanas de edad, observamos un enriquecimiento en el metabolismo de los lípidos y en las vías relacionadas con la biosíntesis. En un rumen sano, los lípidos ruminales derivados de plantas y microbios son absorbidos a través del RE y utilizados para el crecimiento y la proliferación celular por las células epiteliales (59). Por lo tanto, una función importante de las ER es el metabolismo de los lípidos, como sugieren Zhao y sus coautores (60). En un estudio sobre los cambios en el transcriptoma en corderos alimentados con una dieta alta en concentrados y una dieta normal, Sun y sus coautores observaron cambios significativos en la expresión de los genes asociados al metabolismo de los lípidos (61), lo que sugiere que la capacidad de absorción de los lípidos ruminales a través del RE es fundamental para mantener la homeostasis ruminal. Steel y sus coautores observaron una reducción de la expresión en genes asociados con la biosíntesis de colesterol tanto en vacas secas como lactantes (58, 62). Los AGCC son precursores del colesterol. El aumento de la concentración de AGCC en terneros acidóticos significa que los sustratos disponibles para la síntesis de colesterol aumentan. Por lo tanto, la regulación a la baja de los genes relacionados con la biosíntesis del colesterol podría ser una forma eficaz de mantener la homeostasis del colesterol. Hasta ahora, todavía tenemos un conocimiento limitado sobre la capacidad del metabolismo lipídico del epitelio ruminal en el contexto de una dieta alta en concentrados y su contribución al desarrollo y la madurez ruminal en terneros jóvenes. El seguimiento funcional de los genes clave identificados en este estudio que están implicados en el metabolismo de los lípidos y la biosíntesis del colesterol puede proporcionar nuevos conocimientos en terneros con SARA inducida por el alimento.
Cambios en la comunidad microbiana de RE con acidosis prolongada que inducen el tratamiento dietético
Aunque se ha considerado que la dieta alta en concentrados es la causa principal de la acidosis ruminal en el ganado lechero, las vacas pueden mostrar una respuesta fenotípica diferente y una gravedad variada de la acidosis ruminal al tratamiento alimenticio. Los cambios microbiológicos intestinales se consideraron esenciales para modular la salud sistemática en el ganado. El microbioma RE se encuentra en la interfaz directa entre el contenido ruminal y el RE. Por lo tanto, se espera que el microbioma RE pueda desempeñar un papel indispensable en la protección del huésped de la intrusión de microbios dañinos mediante la formación de una biopelícula protectora. Sin embargo, el papel funcional del microbioma ER no se comprende bien. Estudios previos han reportado cambios significativos en la comunidad microbiana del rumen durante el SARA a través de la secuenciación del contenido ruminal (63-65). En el experimento de Wetzels y sus coautores (13), en el que se realizó un desafío de alimentación continuo a largo plazo que indujo SARA, las vacas se agruparon como respondedoras y no respondedoras. De acuerdo con nuestros resultados, Petri et al. (66) encontraron una alta abundancia de los géneros Olsenella y Comamonas en animales con acidosis ruminal inducida. El género Fusobacterium es un habitante normal del rumen del ganado, sin embargo, algunas especies de este género son patógenos oportunistas y causantes primarios de abscesos ruminales (67, 68). La presencia de este género microbiano en los animales tratados pone de manifiesto la posibilidad de utilizar taxones microbianos como biomarcadores de acidosis subaguda. Se observaron fuertes cambios específicos de la dieta en los miembros altamente abundantes de las comunidades epimurales del rumen. En este estudio, nuestro análisis identificó un conjunto único de taxones microbianos que se correlacionaron de manera diferente con las muestras de RE a los dos puntos de tiempo en el grupo tratado. Estas diferencias son esperables considerando la colonización microbiana y el desarrollo ruminal. Además, sugiere que las comunidades microbianas en el ER están evolucionando, potencialmente como un mecanismo para que el huésped defienda cualquier invasión de patógenos en el momento en que el ER es más vulnerable.
Conclusión
Utilizando un modelo de acidosis inducida por la alimentación en terneros jóvenes, nuestro análisis de secuenciación del transcriptoma completo indicó que la respuesta del huésped a la dieta inductora de acidosis se puede capturar a partir de las 8 semanas de edad. En este momento, la respuesta celular al lipopolisacárido es la respuesta principal en el epitelio ruminal. En el grupo tratado alimentado con una dieta inductora de acidosis, se identificaron 5 genes con mayor expresión a las 8 semanas de edad. Estos genes han reportado su papel en la respuesta directa a las bacterias. Se identificó un conjunto único de taxones microbianos como los taxones clave, difiriendo entre los grupos de tratamiento a las 8 semanas y entre los dos de 8 y 17 semanas de edad en el grupo tratado. Nuestros hallazgos sugirieron que los cambios en la comunidad microbiana en el epitelio ruminal son una parte integral y evolutiva de la respuesta del huésped a la acidosis inducida por el alimento. El seguimiento futuro de los nuevos genes y los taxones clave identificados en este estudio puede facilitar el desarrollo de nuevos biomarcadores para el diagnóstico precoz y la prevención de la acidosis inducida por piensos.
Declaración de disponibilidad de datos
Los conjuntos de datos presentados en este estudio se pueden encontrar en repositorios en línea. Los nombres de los repositorios y los números de acceso se pueden encontrar en: https://www.ncbi.nlm.nih.gov/, PRJNA948013, PRJNA1025110.
Declaración ética
El estudio en animales fue aprobado por el comité IACUC de la Universidad de Wisconsin-Madison. El estudio se llevó a cabo de acuerdo con la legislación local y los requisitos institucionales.
Contribuciones de los autores
WL: Conceptualización, Curación de datos, Análisis formal, Investigación, Metodología, Supervisión, Redacción – borrador original, Redacción – revisión y edición. AL: Metodología, Redacción – revisión y edición. PF: Análisis formal, Metodología, Redacción – revisión y edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. La financiación de este experimento proviene de los proyectos 5090-31000-024 y 5090-31000-026 del Servicio de Investigación Agrícola del Departamento de Agricultura de los Estados Unidos (USDA, por sus siglas en inglés). Esta investigación utilizó recursos proporcionados por el proyecto SCINet y el Centro de Excelencia de IA del Servicio de Investigación Agrícola del USDA, ARS número de proyecto 0500-00093-001-00-D.
Reconocimientos
Agradecemos a Sonia Gelsinger por diseñar la dieta de alimentación y realizar los ensayos de alimentación. Andrea Edwards ayudó con el procesamiento y la catalogación de muestras de tejido. El equipo de la granja en el Centro de Investigación de Forrajes Lácteos de EE. UU. ayudó con el manejo diario de los terneros.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Abreviaturas
SARA: acidosis ruminal subaguda; AGCC: ácidos grasos volátiles; DF: fibra detergente neutra; RIN: número de integridad del ARN; DEG: gen expresado diferencialmente; sPLS-DA, análisis discriminante de mínimos cuadrados parciales dispersos; FPKM, fragmentos por kilobase del modelo de exón por millón de lecturas mapeadas; URGT, genes regulados al alza en el grupo tratado; LPS: lipopolisacárido.
Notas
1. ^https://www.graphpad.com/quickcalcs/randomize1/
2. ^https://support.illumina.com/help/pooling-calculator/pooling-calculator.htm
Referencias
1. Gorka, P, Kowalski, ZM, Pietrzak, P, Kotunia, A, Kiljanczyk, R, Flaga, J, et al. Efecto de la suplementación con butirato de sodio en el sustituto de la leche y la dieta de inicio sobre el desarrollo ruminal en terneros. J Physiol Pharmacol. (2009) 60:47–53.
2. Malhi, M, Gui, H, Yao, L, Aschenbach, JR, Gabel, G y Shen, Z. El aumento del crecimiento de las papilas y el aumento de la absorción de ácidos grasos de cadena corta en el rumen de las cabras se asocian con aumentos transitorios en la expresión de ciclina D1 después de la infusión ruminal de butirato. J Lácteos Sci. (2013) 96:7603–16. doi: 10.3168/jds.2013-6700
Resumen de PubMed | Texto completo de Crossref | Google Académico
3. Shen, Z, Seyfert, HM, Lohrke, B, Schneider, F, Zitnan, R, Chudy, A, et al. Una dieta rica en energía provoca la proliferación de las papilas ruminales asociadas con más receptores de IGF tipo 1 y un aumento de las concentraciones plasmáticas de IGF-1 en cabras jóvenes. J Nutr. (2004) 134:11–7. doi: 10.1093/jn/134.1.11
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Keunen, JE, Plaizier, JC, Kyriazakis, L, Duffield, TF, Widowski, TM, Lindinger, MI, et al. Efectos de un modelo de acidosis ruminal subaguda en la selección de la dieta de vacas lecheras. J Lácteos Sci. (2002) 85:3304–13. doi: 10.3168/jds. S0022-0302(02)74419-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Larson, B. Acidosis y conexión de alimentación. Veterinario Bovino. (2023). Disponible en: https://www.bovinevetonline.com/news/education/acidosis-and-feed-connection
6. Laarman, AH y Oba, M. Comunicación corta: efecto del iniciador de terneros sobre el pH ruminal de terneros lecheros Holstein al destete. J Lácteos Sci. (2011) 94:5661–4. doi: 10.3168/jds.2011-4273
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Gozho, GN, Plaizier, JC, Krause, DO, Kennedy, AD y Wittenberg, KM. La acidosis ruminal subaguda induce la liberación de endotoxinas de lipopolisacáridos ruminales y desencadena una respuesta inflamatoria. J Lácteos Sci. (2005) 88:1399–403. doi: 10.3168/jds. S0022-0302(05)72807-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Zebeli, Q, Dijkstra, J, Tafaj, M, Steingass, H, Ametaj, BN y Drochner, W. Modelización de la adecuación de la fibra dietética en vacas lecheras en función de las respuestas del pH ruminal y la producción de grasa láctea a la composición de la dieta. J Lácteos Sci. (2008) 91:2046–66. doi: 10.3168/jds.2007-0572
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. Cooper, R, Klopfenstein, T, Stock, R, Parrott, C y Herold, D. Efectos de la rumensin y la variación de la ingesta de alimento en el pH ruminal. Informes de ganado vacuno de Nebraska. (1996) 430.
10. Li, S, Danscher, AM, y Plaizier, JC. Acidosis ruminal subactuante (SARA) en bovinos lecheros: nuevos desarrollos en aspectos diagnósticos y manejo de la alimentación. Can J Anim Sci. (2013) 94:353–64.
11. Khiaosa-ard, R, Pourazad, P, Aditya, S, Humer, E y Zebeli, Q. Factores relacionados con la variación en la susceptibilidad a la acidosis ruminal subaguda en vacas Simmental lactantes tempranas alimentadas con la misma dieta rica en granos. Anim Feed Sci Technol. (2018) 238:111–22. doi: 10.1016/j.anifeedsci.2018.02.005
12. Khorrami, B, Khiaosa-Ard, R y Zebeli, Q. Modelos para predecir el riesgo de acidosis ruminal subaguda en vacas lecheras basados en factores dietéticos y de vacas: un metaanálisis. J Lácteos Sci. (2021) 104:7761–80. doi: 10.3168/jds.2020-19890
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Wetzels, SU, Mann, E, Pourazad, P, Qumar, M, Pinior, B, Metzler-Zebeli, BU, et al. Estructura de la comunidad bacteriana epimural en el rumen de vacas Holstein con diferentes respuestas a un desafío dietético de acidosis ruminal subaguda a largo plazo. J Lácteos Sci. (2017) 100:1829–44. doi: 10.3168/jds.2016-11620
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Barbeira, AN, Dickinson, SP, Bonazzola, R, Zheng, J, Wheeler, HE, Torres, JM, et al. Exploración de las consecuencias fenotípicas de la variación de la expresión génica específica de los tejidos inferida a partir de las estadísticas resumidas de GWAS. Nat Commun. (2018) 9:1825. doi: 10.1038/s41467-018-03621-1
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Hoque, MN, Sarkar, MMH, Khan, MA, Hossain, MA, Hasan, MI, Rahman, MH, et al. El perfil diferencial de expresión génica revela posibles biomarcadores y compuestos farmacológicos contra el SARS-CoV-2: perspectivas de los enfoques de aprendizaje automático y bioinformática. Frente Immunol. (2022) 13:918692. doi: 10.3389/fimmu.2022.918692
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Momeni, M, Rashidifar, M, Balam, FH, Roointan, A, y Gholaminejad, A. Un análisis exhaustivo de los datos de perfiles de expresión génica en pacientes con COVID-19 para el descubrimiento de firmas de biomarcadores sanguíneos específicos y diferenciales. Sci Rep. (2023) 13:5599. doi: 10.1038/s41598-023-32268-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Gelsinger, SL, Coblentz, WK, Zanton, GI, Ogden, RK y Akins, MS. Desaparición ruminal in situ y digestión de todo el tracto de alimentos iniciadores en terneros antes, durante y después del destete. J Lácteos Sci. (2019) 102:2196–206. doi: 10.3168/jds.2018-15551
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Gelsinger, SL, Coblentz, WK, Zanton, GI, Ogden, RK y Akins, MS. Efectos fisiológicos de la acidosis ruminal inducida por el iniciador en terneros antes, durante y después del destete. J Lácteos Sci. (2020) 103:2762–72. doi: 10.3168/jds.2019-17494
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Li, W, Gelsinger, S, Edwards, A, Riehle, C y Koch, D. Cambios en el metatranscriptoma de la comunidad microbiana epimural del rumen y el transcriptoma hepático en terneros jóvenes con acidosis inducida por alimento. Sci Rep. (2019) 9:18967. doi: 10.1038/s41598-019-54055-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. Li, W, Gelsinger, S, Edwards, A, Riehle, C y Koch, D. Análisis del transcriptoma del epitelio ruminal y análisis del metatranscriptoma de la comunidad microbiana epimural del rumen en terneros jóvenes con acidosis inducida por alimento. Sci Rep. (2019b) 9:4744. doi: 10.1038/s41598-019-40375-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
21. Kristensen, NB, Engbaek, M, Vestergaard, M, y Harmon, DL. Nota técnica: técnica de canulación ruminal en terneros Holstein jóvenes: efectos de la canulación sobre la ingesta de alimento, la ganancia de peso corporal y el desarrollo ruminal a las seis semanas de edad. J Lácteos Sci. (2010) 93:737–42. doi: 10.3168/jds.2009-2488
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Bi, R y Liu, P. Cálculo del tamaño de la muestra mientras se controla la tasa de falsos descubrimientos para el análisis de expresión diferencial con experimentos de secuenciación de ARN. Bmc Bioinformática. (2016) 17:146. doi: 10.1186/s12859-016-0994-9
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Dobin, A, Davis, CA, Schlesinger, F, Drenkow, J, Zaleski, C, Jha, S, et al. STAR: alineador universal ultrarrápido de secuenciación de ARN. Bioinformática. (2013) 29:15–21. doi: 10.1093/bioinformatics/bts635
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. Trapnell, C, Roberts, A, Goff, L, Pertea, G, Kim, D, Kelley, DR, et al. Análisis diferencial de expresión génica y de transcripción de experimentos de RNA-seq con TopHat y gemelos. Nat Protoc. (2012) 7:562–78. doi: 10.1038/nprot.2012.016
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Jiao, X, Sherman, BT, Huang, DW, Stephens, R, Baseler, MW, Lane, HC, et al. DAVID-WS: un servicio web con estado para facilitar el análisis de listas de genes/proteínas. Bioinformática. (2012) 28:1805–6. doi: 10.1093/bioinformatics/bts251
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Kopylova, E, Noe, L y Touzet, H. SortMeRNA: filtrado rápido y preciso de ARN ribosómicos en datos metatranscriptómicos. Bioinformática. (2012) 28:3211–7. doi: 10.1093/bioinformatics/bts611
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Quast, C, Pruesse, E, Yilmaz, P, Gerken, J, Schweer, T, Yarza, P, et al. El proyecto de base de datos de genes de ARN ribosómico SILVA: mejora del procesamiento de datos y herramientas basadas en la web. Ácidos nucleicos Res. (2013) 41:D590-6. doi: 10.1093/nar/gks1219
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Burge, SW, Daub, J, Eberhardt, R, Tate, J, Barquist, L, Nawrocki, EP, et al. Rfam 11.0: 10 años de familias de ARN. Ácidos nucleicos Res. (2013) 41:D226-32. doi: 10.1093/nar/gks1005
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Wood, DE, y Salzberg, SL. Kraken: clasificación ultrarrápida de secuencias metagenómicas mediante alineamientos exactos. Genoma Biol. (2014) 15:R46. doi: 10.1186/gb-2014-15-3-r46
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Rey, M, Enjalbert, F, Combes, S, Cauquil, L, Bouchez, O, y Monteils, V. El establecimiento de la comunidad bacteriana ruminal en terneros lecheros desde el nacimiento hasta el destete es secuencial. J Appl Microbiol. (2014) 116:245–57. doi: 10.1111/jam.12405
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Rohart, F, Gautier, B, Singh, A, y Le Cao, KA. mixOmics: un paquete de R para la selección de características ómicas y la integración de múltiples datos. PLoS Comput Biol. (2017) 13:e1005752. doi: 10.1371/journal.pcbi.1005752
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Le Cao, KA, Boitard, S y Besse, P. Análisis discriminante PLS disperso: selección de características biológicamente relevantes y representaciones gráficas para problemas multiclase. BMC Bioinformática. (2011) 12:253. doi: 10.1186/1471-2105-12-253
Resumen de PubMed | Texto completo de Crossref | Google Académico
33. Ruiz-Pérez, D, Guan, H, Madhivanan, P, Mathee, K, y Narasimhan, G. ¿Así que crees que puedes PLS-DA? BMC Bioinformática. (2020) 21:2. doi: 10.1186/s12859-019-3310-7
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Wang, R, Wang, M, Zhang, XM, Wen, JN, Ma, ZY, Long, DL, et al. Efectos de la canulación ruminal sobre los gases disueltos y la comunidad de metanógenos en vacas lecheras. J Lácteos Sci. (2019) 102:2275–82. doi: 10.3168/jds.2018-15187
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Humer, E, Kroger, I, Neubauer, V, Schedle, K, Reisinger, N y Zebeli, Q. La suplementación de compuestos fitogénicos o levadura autolizada modula las aminas biógenas ruminales y el metaboloma plasmático en vacas secas que experimentan acidosis ruminal subaguda. J Lácteos Sci. (2018) 101:9559–74. doi: 10.3168/jds.2018-14744
36. Plaizier, JC, Khafipour, E, Li, S, Gozho, GN y Krause, DO. Acidosis ruminal subaguda (SARA), endotoxinas y consecuencias para la salud. Anim Feed Sci Technol. (2012) 172:9–21. doi: 10.1016/j.anifeedsci.2011.12.004
37. Zebeli, Q, y Ametaj, BN. Relaciones entre el lipopolisacárido ruminal y los mediadores de la respuesta inflamatoria con la producción y eficiencia de grasa láctea en vacas lecheras. J Lácteos Sci. (2009) 92:3800–9. doi: 10.3168/jds.2009-2178
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Nagaraja, TG, y Lechtenberg, KF. Acidosis en ganado de engorde. Vet Clin North Am Food Anim Pract. (2007) 23:333–50. doi: 10.1016/j.cvfa.2007.04.002
39. Guerville, M, y Boudry, G. Mecanismos gastrointestinales y hepáticos que limitan la entrada y diseminación de lipopolisacáridos en la circulación sistémica. Am J Physiol Gastrointest Liver Physiol. (2016) 311:G1–G15. doi: 10.1152/ajpgi.00098.2016
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Gozho, GN, Krause, DO, y Plaizier, JC. Concentración ruminal de lipopolisacáridos y respuesta inflamatoria durante la acidosis ruminal subaguda inducida por granos en vacas lecheras. J Lácteos Sci. (2007) 90:856–66. doi: 10.3168/jds. S0022-0302(07)71569-2
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Khafipour, E, Krause, DO, y Plaizier, JC. Un desafío de acidosis ruminal subaguda a base de granos causa la translocación de lipopolisacáridos y desencadena inflamación. J Lácteos Sci. (2009) 92:1060–70. doi: 10.3168/jds.2008-1389
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Eckel, EF y Ametaj, BN. Endotoxinas bacterianas y su papel en las enfermedades periparto de las vacas lecheras: perspectivas de la vacuna mucosa. Lácteos. (2020) 1:61–90. doi: 10.3390/dairy1010006
43. Xia, G, Sun, J, Fan, Y, Zhao, F, Ahmed, G, Jin, Y, et al. El beta-sitosterol atenúa el estrés inflamatorio inducido por la dieta alta en cereales y modifica la fermentación ruminal y la microbiota en ovejas. Animales (Basilea). (2020) 10:171. doi: 10.3390/ani10010171
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Eckel, EF, y Ametaj, BN. Revisión invitada: papel de las endotoxinas bacterianas en la etiopatogenia de las enfermedades periparto de las vacas lecheras en transición. J Lácteos Sci. (2016) 99:5967–90. doi: 10.3168/jds.2015-10727
Resumen de PubMed | Texto completo de Crossref | Google Académico
45. Zhu, X, Hogan, SP, Molkentin, JD y Zimmermann, N. La ciclofilina D regula la necrosis, pero no la apoptosis, de los eosinófilos murinos. Am J Physiol Gastrointest Liver Physiol. (2016) 310:G609–17. doi: 10.1152/ajpgi.00389.2015
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. Gabel, G, Aschenbach, JR y Muller, F. Transferencia de sustratos energéticos a través del epitelio ruminal: implicaciones y limitaciones. Anim Health Res Rev. (2002) 3:15–30. doi: 10.1079/ahrr200237
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Bergman, EN. Aportes energéticos de ácidos grasos volátiles del tracto gastrointestinal en diversas especies. Physiol Rev. (1990) 70:567–90. doi: 10.1152/physrev.1990.70.2.567
Resumen de PubMed | Texto completo de Crossref | Google Académico
48. Elolimy, AA, Abdelmegeid, MK, McCann, JC, Shike, DW y Loor, JJ. Consumo de alimento residual en ganado vacuno de carne y su asociación con rasgos de canal, bacterias de fracción sólida ruminal y expresión génica del epitelio. J Anim Sci Biotechnol. (2018) 9:67. doi: 10.1186/s40104-018-0283-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
49. Lam, S, Munro, JC, Zhou, M, Guan, LL, Schenkel, FS, Steele, MA, et al. Asociaciones de los parámetros ruminales con la eficiencia alimenticia y la rutina de muestreo en bovinos de carne. Animal. (2018) 12:1442–50. doi: 10.1017/S1751731117002750
Resumen de PubMed | Texto completo de Crossref | Google Académico
50. Melo, LQ, Costa, SF, Lopes, F, Guerreiro, MC, Armentano, LE, y Pereira, MN. Morfometría ruminal y efecto del pH y volumen de la digesta en la absorción de ácidos grasos volátiles. J Anim Sci. (2013) 91:1775–83. doi: 10.2527/jas.2011-4999
Resumen de PubMed | Texto completo de Crossref | Google Académico
51. Steele, MA, AlZahal, O, Hook, SE, Croom, J y McBride, BW. Acidosis ruminal y la rápida aparición de paraqueratosis ruminal en una vaca lechera madura: reporte de un caso. Acta Vet Scand. (2009) 51:39. doi: 10.1186/1751-0147-51-39
Resumen de PubMed | Texto completo de Crossref | Google Académico
52. Ballabio, A, y Bonifacino, JS. Los lisosomas como reguladores dinámicos de la homeostasis celular y del organismo. Nat Rev Mol Cell Biol. (2020) 21:101–18. doi: 10.1038/s41580-019-0185-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
53. Balka, KR y De Nardo, D. Comprensión de la señalización temprana de TLR a través del Myddosome. J Leukoc Biol. (2019) 105:339–51. doi: 10.1002/Jlb.Mr0318-096r
Resumen de PubMed | Texto completo de Crossref | Google Académico
54. Gutiérrez, MG, Master, SS, Singh, SB, Taylor, GA, Colombo, MI, y Deretic, V. La autofagia es un mecanismo de defensa que inhibe la supervivencia de BCG y Mycobacterium tuberculosis en macrófagos infectados. Célula. (2004) 119:753–66. doi: 10.1016/j.cell.2004.11.038
Resumen de PubMed | Texto completo de Crossref | Google Académico
55. Nakagawa, I, Amano, A, Mizushima, N, Yamamoto, A, Yamaguchi, H, Kamimoto, T, et al. La autofagia defiende a las células contra los estreptococos invasores del grupo A. Ciencia. (2004) 306:1037–40. doi: 10.1126/science.1103966
Resumen de PubMed | Texto completo de Crossref | Google Académico
56. Yoon, J, Park, JM, Kim, KJ, Kim, YH y Min, J. Actividad antimicrobiana de los orgánulos celulares, lisosomas aislados de clara de huevo. J Microbiol Biotechnol. (2009) 19:1364–8. doi: 10.4014/jmb.0905.05053
57. Nagaraja, TG y Titgemeyer, EC. Acidosis ruminal en ganado vacuno: las perspectivas microbiológicas y nutricionales actuales. J Lácteos Sci. (2007) 90:E17-38. doi: 10.3168/jds.2006-478
Resumen de PubMed | Texto completo de Crossref | Google Académico
58. Steele, MA, Croom, J, Kahler, M, AlZahal, O, Hook, SE, Plaizier, K, et al. El epitelio ruminal bovino experimenta rápidas adaptaciones estructurales durante la acidosis ruminal subaguda inducida por granos. Am J Physiol Regul Integr Comp Physiol. (2011) 300:R1515–23. doi: 10.1152/ajpregu.00120.2010
Resumen de PubMed | Texto completo de Crossref | Google Académico
59. Na, SW y Guan, LL. Comprender el papel de las interacciones huésped-microbio epitelial del rumen en la eficiencia de la alimentación del ganado. Anim Nutr. (2022) 10:41–53. doi: 10.1016/j.aninu.2022.04.002
Resumen de PubMed | Texto completo de Crossref | Google Académico
60. Zhao, K, Chen, YH, Penner, GB, Oba, M y Guan, LL. El análisis del transcriptoma de los epitelios ruminales reveló posibles mecanismos reguladores implicados en la adaptación del huésped a la transición gradual de la dieta de alta fermentabilidad en el ganado vacuno. BMC Genómica. (2017) 18:976. doi: 10.1186/s12864-017-4317-y
Resumen de PubMed | Texto completo de Crossref | Google Académico
61. Sun, D, Yin, Y, Guo, C, Liu, L, Mao, S, Zhu, W, et al. El análisis transcriptómico revela los mecanismos moleculares del desarrollo morfológico y funcional de la pared ruminal inducido por la introducción de diferentes dietas sólidas en un modelo de cordero. J Anim Sci Biotechnol. (2021) 12:33. doi: 10.1186/s40104-021-00556-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
62. Steele, MA, Dionissopoulos, L, AlZahal, O, Doelman, J, y McBride, BW. La adaptación epitelial ruminal a la acidosis ruminal en bovinos lactantes implica la expresión coordinada de proteínas de unión al factor de crecimiento similares a la insulina y una enzima colesterolgénica. J Lácteos Sci. (2012) 95:318–27. doi: 10.3168/jds.2011-4465
Resumen de PubMed | Texto completo de Crossref | Google Académico
63. Hook, SE, Steele, MA, Northwood, KS, Dijkstra, J, France, J, Wright, AD, et al. Impacto de la adaptación y recuperación de la acidosis ruminal subaguda (SARA) sobre la densidad y diversidad de bacterias en el rumen de vacas lecheras. FEMS Microbiol Ecol. (2011) 78:275–84. doi: 10.1111/j.1574-6941.2011.01154.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
64. Khafipour, E, Plaizier, JC, Aikman, PC y Krause, DO. Estructura poblacional de Escherichia coli ruminal asociada con acidosis ruminal subaguda (SARA) en ganado lechero. J Lácteos Sci. (2011) 94:351–60. doi: 10.3168/jds.2010-3435
Resumen de PubMed | Texto completo de Crossref | Google Académico
65. Mao, SY, Zhang, RY, Wang, DS, y Zhu, WY. Impacto de la adaptación a la acidosis ruminal subaguda (SARA) en la microbiota ruminal en ganado lechero mediante pirosecuenciación. Anaerobio. (2013) 24:12–9. doi: 10.1016/j.anaerobe.2013.08.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
66. Petri, RM, Schwaiger, T, Penner, GB, Beauchemin, KA, Forster, RJ, McKinnon, JJ, et al. Cambios en la diversidad bacteriana epimural del rumen del ganado vacuno de carne afectada por la dieta y la acidosis ruminal inducida. Appl Environ Microbiol. (2013) 79:3744–55. doi: 10.1128/AEM.03983-12
Resumen de PubMed | Texto completo de Crossref | Google Académico
67. Schwarz, C, Mathieu, J, Gómez, JL, Miller, MR, Tikhonova, M, Nagaraja, TG, et al. Hallazgo inesperado de Fusobacterium varium como la especie dominante de Fusobacterium en el rumen bovino: implicaciones potenciales para la etiología e intervenciones del absceso hepático. J Anim Sci. (2023) 101:skad130. doi: 10.1093/jas/skad130
Resumen de PubMed | Texto completo de Crossref | Google Académico
68. Tadepalli, S, Narayanan, S, Stewart, G, Chengappa, M, y Nagaraja, T. Fusobacterium necrophorum: una bacteria ruminal que invade el hígado para causar abscesos en el ganado. Anaerobio. (2009) 15:36–43. doi: 10.1016/j.anaerobe.2008.05.005
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: acidosis inducida por alimentación, terneros jóvenes, transcriptoma epitelial ruminal, etatranscriptoma epitelial ruminal, cambios en la comunidad microbiana
Cita: Li W, Larsen A y Fregulia P (2024) Investigación del impacto de la acidosis ruminal subaguda inducida por el alimento en el transcriptoma epimural y el metatranscriptoma del rumen en terneros jóvenes a las 8 y 17 semanas de edad. Frente. Vet. Sci. 11:1328539. doi: 10.3389/fvets.2024.1328539
Recibido: 26 de octubre de 2023; Aceptado: 07 de febrero de 2024;
Publicado: 21 de febrero de 2024.
Editado por:
Massimo Trabalza-Marinucci, Universidad de Perugia, Italia
Revisado por:
Rayudika Aprilia Patindra Purba, Universidad Tecnológica de Suranaree, Tailandia
Helen Marie Golder, Scibus, Australia
Derechos de autor © 2024 Li, Larsen y Fregulia. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Wenli Li, wenli.li@usda.gov
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía


