La resecuenciación del genoma completo de cabras lecheras nativas e importadas identifica genes asociados con la productividad y la inmunidad
 Jianqing Zhao1
Jianqing Zhao1 Yuanpan Mu1
Yuanpan Mu1 Ping Gong2
Ping Gong2 Baolong Liu1
Baolong Liu1 Fuhong Zhang1
Fuhong Zhang1 Lu Zhu1
Lu Zhu1 Chenbo Shi1
Chenbo Shi1 Xuefeng Lv2
Xuefeng Lv2 Jun Luo1*
Jun Luo1*- 1Laboratorio Clave de Biología Molecular para la Agricultura de Shaanxi, Facultad de Ciencia y Tecnología Animal, Universidad Northwest A&F, Xianyang, China
- número arábigoInstituto de Estándares de Calidad de la Ganadería, Academia de Ciencias Animales de Xinjiang, Urumqi, China
Comprender las diferencias en la variación genética entre las razas locales de cabras lecheras chinas y las razas importadas puede ayudar a la innovación en germoplasma y al mejoramiento molecular. Sin embargo, la investigación es limitada en esta área. En este estudio, se analizaron los datos de resecuenciación del genoma completo de 134 individuos de razas de cabras lecheras locales e importadas, y posteriormente se identificaron sus diferencias en la variación genética genómica, la diversidad genética y la estructura de la población. También analizamos genes candidatos asociados con rasgos importantes de las cabras lecheras, como la producción de leche (STK3, GHR, PRELID3B), la reproducción (ATP5E), el crecimiento y el desarrollo (CTSZ, GHR) y la función inmune (CTSZ, NELFCD). Además, examinamos las distribuciones de frecuencia alélica de los genes de interés y encontramos diferencias significativas entre las dos poblaciones. Este estudio proporciona valiosos recursos para el estudio de la diversidad genética en cabras lecheras y sienta las bases para la cría selectiva de cabras lecheras en el futuro.
Introducción
Las cabras (Capra hircus) fueron una de las primeras especies animales domesticadas, proporcionando a los humanos alimentos esenciales y recursos vivos, como carne, leche y pieles (1-4). Como raza lechera especializada, las cabras lecheras son una parte importante de la ganadería y las industrias lácteas de muchos países (5-7). China desempeña un papel destacado en la cría y producción mundial de leche caprina lechera, contribuyendo significativamente al suministro mundial de leche (8).
La historia de la cría de cabras lecheras en China se remonta a principios del siglo XX, con un enfoque principal en la mejora de la cabra lechera Saanen y su descendencia híbrida. Después de casi un siglo de cría, las razas actuales de cabras lecheras en China consisten principalmente en cabras lecheras Xinong Saanen, Guanzhong y Laoshan (9, 10). A medida que el nivel de vida de las personas ha mejorado en China en las últimas décadas, ha aumentado la demanda de productos lácteos de alta calidad. Para cumplir con estos requisitos, la cría de cabras lecheras se ha centrado principalmente en rasgos asociados con un alto rendimiento y calidad (11, 12). Actualmente, para mejorar las excelentes características de las razas de cabras lecheras en China, la mayoría de las granjas se centran en mejorar las razas de cabras lecheras a través de la cría selectiva y el cruce con razas importadas. Las razas importadas en China provienen principalmente de Nueva Zelanda y Australia, que comprenden cabras lecheras Saanen, Alpine y Toggenburg (13, 14). Entre ellas, el Saanen es la raza más comúnmente importada para mejorar la producción de leche, mientras que otras razas se utilizan principalmente para mejorar la calidad de la leche de las cabras lecheras (15, 16). Sin embargo, los rasgos siguen sin estar claros, lo que dificulta la verificación del proceso de reproducción. Mientras tanto, la importación de cabras lecheras se basa principalmente en la evaluación del fenotipo sin selección del nivel genómico, lo cual no es preciso ni muy efectivo.
Con la finalización del genoma de referencia de la cabra, se han logrado avances significativos en el estudio de la diversidad genética en cabras lecheras. Estos estudios no solo revelan variaciones genéticas en el genoma de la cabra, sino que también proporcionan pistas importantes para comprender las diferencias genéticas y los mecanismos de adaptación entre las diferentes razas (17). Sin embargo, estudios previos han revelado la asociación entre la variación genética y rasgos específicos en el genoma de la cabra, la investigación sobre la variación genética y la asociación de rasgos de esta población específica de cabras lecheras es todavía relativamente limitada (18-20). La investigación existente se centra principalmente en unas pocas razas principales, y la comprensión de las diferencias genéticas entre una gama más amplia de razas de cabras lecheras aún no es profunda. Por lo tanto, la exploración en profundidad de la diversidad genética, la estructura de la población y las variaciones genéticas asociadas con rasgos agronómicos importantes de las poblaciones de cabras lecheras es de gran importancia para el desarrollo sostenible de la industria caprina lechera. En este estudio, resecuenciamos 50 genomas locales de cabras lecheras a una profundidad de cobertura del 17,01×. Luego se combinó con los datos de otros 84 individuos de especies de cabras lecheras locales e importadas para un análisis más detallado para identificar sus diferencias en la variación genética genómica, la diversidad genética y la estructura de la población. El análisis de las corridas de islas de homocigosidad (ROH) y el escaneo selectivo identificaron regiones y genes específicos afectados por la selección, asociados con varios rasgos morfológicos y agronómicos significativos. Además, examinamos las distribuciones de frecuencia alélica de los genes dentro de las firmas selectivas. Nuestros hallazgos contribuyen a la comprensión de la diversidad genética y la estructura genética poblacional de las cabras lecheras. Además, reveló genes candidatos que están asociados con varios fenotipos, lo que es valioso para la futura importación y selección de cría de cabras lecheras.
Materiales y métodos
Recogida y secuenciación de muestras
Este estudio recolectó dos poblaciones de cabra lechera Xinong Saanen (XNG, n = 40) y cabra lechera Guanzhong (GZG, n = 10) de la provincia de Shaanxi. A cada cabra se le extrajeron 5 mL de sangre de la vena yugular y se extrajo el ADN utilizando el método estándar de fenol-cloroformo. Las muestras con concentraciones de ADN determinadas fueron sometidas a la secuenciación del genoma completo por parte de Huada Company, con bibliotecas de extremos emparejados construidas utilizando la plataforma Huada T7 con un tamaño de inserción promedio de 500 pb por individuo y una longitud de lectura promedio de 150 pb. Además, los datos de resecuenciación de 84 individuos se descargaron de bases de datos públicas, incluidas 8 razas de la siguiente manera: cabra lechera alpina australiana (AAG, n = 2), cabra lechera australiana Saanen (ASG, n = 6), cabra lechera Guanzhong (GZG, n = 26), cabra lechera Saanen de Nueva Zelanda (NSG, n = 6), cabra lechera alpina de Nueva Zelanda (NAG, n = 4), cabra lechera Laoshan (LSG, n = 9), cabra lechera de Nubia (NBY, n = 15), cabra lechera de Tugenburg (TGB, n = 16). Dando como resultado un total de 134 muestras en el estudio.
Asignación de lectura y llamada de variantes
El estudio utilizó Trimmomatic v0.38 (21) para filtrar las secuencias de extremos emparejados. A continuación, BWA-MEM (v0.7.15-r1140) (22) alineó los datos limpios con el genoma de referencia de la cabra ARS1.2 (GCF_001704415.2, https://www.ncbi.nlm.nih.gov/datasets/genome/GCF_001704415.2/). Posteriormente, se utilizó samtools (23) para construir un índice de archivos BAM para el mapeo. A continuación, se ordenaron los archivos BAM y se eliminaron las posibles lecturas duplicadas utilizando la herramienta Picard1. Después del mapeo, la llamada de SNP se realizó utilizando los módulos «Haplotype Caller», «Genotype GVCFs» y «Select Variants» en el paquete de herramientas de análisis genómico GATK (GATK, versión 3.8-1-0-gf15c1c3ef) (24). Los SNP iniciales se filtraron con el módulo «Filtración de variantes» basado en los parámetros: «QD < 2.0, FS > 60.0, MQ < 40.0, MQRankSum < −12.5, ReadPosRankSum < −8.0 y SOR > 3.0», y una profundidad de secuenciación promedio de variantes dentro del rango «<1/3× y >3×» para todos los individuos. Por último, se utilizó el software ANNOVAR (25) para la anotación funcional de los SNP.
Análisis de la diversidad genética
Después de aplicar el desequilibrio de ligamiento (LD) a la poda de SNP, el programa PLINK (26) se utiliza para calcular corridas de homocigosidad (ROH) con parámetros específicos de la siguiente manera: (a) 100 SNP homocigotos consecutivos, (b) mínimo de 50 SNP por ventana, (c) 500 kb de longitud homocigota contigua, (d) densidad mínima de 1 SNP por 50 kb, (e) tasa de superposición de ventana de 0,05, y (f) cada ventana contiene 1 llamada heterocigota y 2 llamadas faltantes. Los resultados del análisis posterior se clasifican en 0,5-1 Mb, 1-2 Mb y 2-4 Mb para su visualización. Se calcula la heterocigosidad de los SNPs para estimar el coeficiente de consanguinidad (Fhom). Las herramientas VCF (27) se utilizan para analizar la diversidad de nucleótidos para cada raza en ventanas de 50 kb no superpuestas. A continuación, se utiliza el software PopLDdecay (28) para calcular el decaimiento de LD para evaluar la distancia física entre SNPs dentro de bloques de haplotipos de diferentes razas. Por último, se calculan las frecuencias de las islas de homocigosidad (ROH) para las cabras lecheras de Xinong Saanen y las cabras lecheras de Guanzhong, y se determinan las regiones con frecuencias superiores al 20% como islas de puntos críticos de ROH.
Análisis genómico poblacional
Se utilizan tres métodos para el filtrado del formato de llamada variante (VCF) y la estimación de la estructura de la población: (a) construir un árbol de unión de vecinos con MEGA v10.2.6 (29) y visualizarlo utilizando iTOL (30); b) análisis de componentes principales (PCA) con el programa informático EIGENSOFT v5.0 (31); y (c) realizar análisis de la estructura poblacional utilizando ADMIXTURE v1.3.0 (32). La validación cruzada se emplea para calcular el error de validación cruzada y determinar el valor óptimo de K (suponiendo que el valor de K de la población ancestral oscila entre 2 y 8).
Detección de barridos selectivos
Con base en las características y orígenes de las razas de cabras lecheras, clasificamos 134 cabras lecheras en razas de cabras lecheras nativas (NDG) y cabras lecheras importadas (IDG). A continuación, comparamos los genomas de estas dos razas de cabras lecheras y estimamos las regiones de escaneo de señales utilizando una combinación de diversidad de nucleótidos (θπ NDG/IDG) e índice de fijación (FST) con un VCFtools, empleando ventanas correderas de 50 kb y pasos deslizantes de 25 kb. Además, se empleó el estadístico D (33) de Tajima y la prueba de razón de verosimilitud compuesta entre poblaciones (XP-CLR) (34) para identificar posibles diferencias regionales entre las diferentes razas. XP-CLR es un método de probabilidad para detectar barridos selectivos mediante el modelado conjunto de diferencias de frecuencia alélica multilocus entre dos grupos. Las regiones escaneadas detectadas a partir de la intersección de los dos parámetros con el umbral más alto del 5% se anotaron para identificar genes candidatos. Por último, se utilizó Bedtools (35) para anotar las regiones seleccionadas para su posterior análisis.
Análisis de enriquecimiento y visualización
Se utilizó el módulo de enriquecimiento en KOBAS (36) para identificar vías y términos de Gene Ontology (GO) que mostraron asociaciones estadísticamente significativas (con un nivel de significación de p ≤ 0.05). Posteriormente, se visualizaron los resultados a través de la herramienta OmicShare. número arábigo
Resultados
Secuenciación y llamada de variación
Para identificar la diferencia genómica entre las razas locales (XNG, GZG, LSG) e importadas de cabras lecheras (AAG, ASG, NSG, NAG, TGB, NBY), comparamos 134 conjuntos de datos de resecuenciación del genoma completo, incluidos 40 genomas de XNG y 10 genomas de GZG, combinados con 84 datos de genomas publicados que incluyen razas locales e importadas (GZG, LSG, AAG, ASG, NSG, NAG, TGB, NBY) (Figura 1). Los datos se alinearon con el genoma de referencia de la cabra ARS1.2 (GCF_001704415), logrando una tasa de alineación promedio de 97.74% y una profundidad media de secuenciación de 11.3×. Después de la eliminación de las secuencias de baja calidad, las lecturas limpias promedio para cada muestra fueron de 373.568.004 pb, con un contenido de GC que osciló entre el 41,82 y el 42,33%. La tasa media de alineamiento para las poblaciones estudiadas (XNG, GZG) es del 97,5%, acompañada de una profundidad media de secuenciación del 17,01×. Las poblaciones de razas importadas (AAG, ASG, NSG, NAG, TGB) exhibieron una tasa de alineación promedio de 97.04% y una profundidad de secuenciación promedio de 22.51× (Tabla Suplementaria S1).
Tras la detección de la variación de SNP y el análisis estadístico de los 134 conjuntos de datos del genoma de la cabra lechera obtenidos, se detectaron un total de 33.011.806 SNPs. Entre ellos, la proporción de SNPs intrónicos fue la más alta (32.705.301), representando el 55,04% del total; la proporción de SNPs exónicos fue de aproximadamente el 1,142%. Había 283.685 SNP sinónimos y 227.235 SNP con cambio de sentido. Se calculó y analizó la relación transición/transversión (Ti/Tv), revelando una relación de 2,344, consistente con el valor general para las poblaciones caprinas. Estos hallazgos indican una alta calidad de la secuenciación, por lo que los datos obtenidos cumplen con los requisitos para su posterior análisis (Tabla Suplementaria S2).
Análisis de la diversidad genética
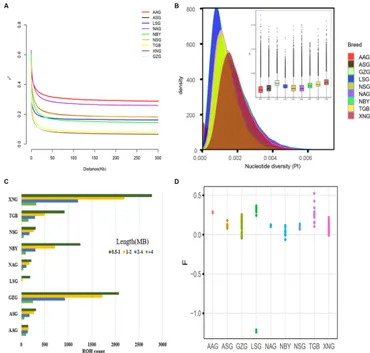
El análisis LD muestra que GZG y XNG exhiben la tasa de descomposición más rápida (Figura 2A), lo que sugiere niveles de domesticación más bajos y una mayor diversidad genética. El análisis de la diversidad de nucleótidos (Figura 2B) revela una mayor diversidad en las razas locales en comparación con las importadas. La ROH es un indicador de la proximidad del parentesco y la historia de endogamia, y el análisis revela los patrones genómicos de la historia demográfica reciente. El ROH corto indica que la endogamia antigua es mucho más frecuente en el XNG y GZG en comparación con otras razas importadas. Además, XNG exhibe un valor más alto de ROH, lo que indica una historia de reproducción más larga (Figura 2C). Mientras tanto, el coeficiente de consanguinidad debido a la consanguinidad reciente es más alto para cada genoma individual en TGB, con un rango de 0,1 a 0,51 (Figura 2D).
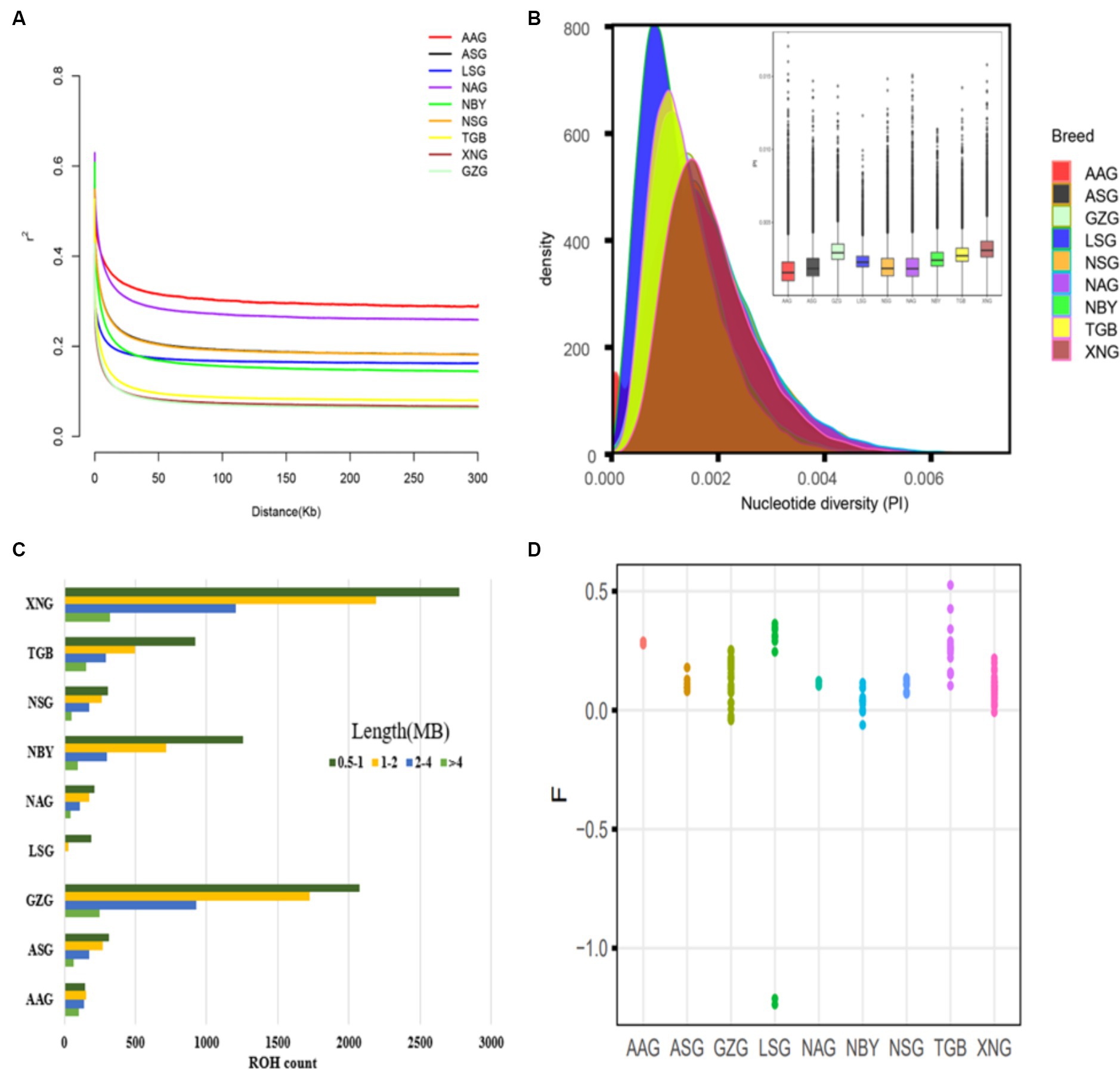
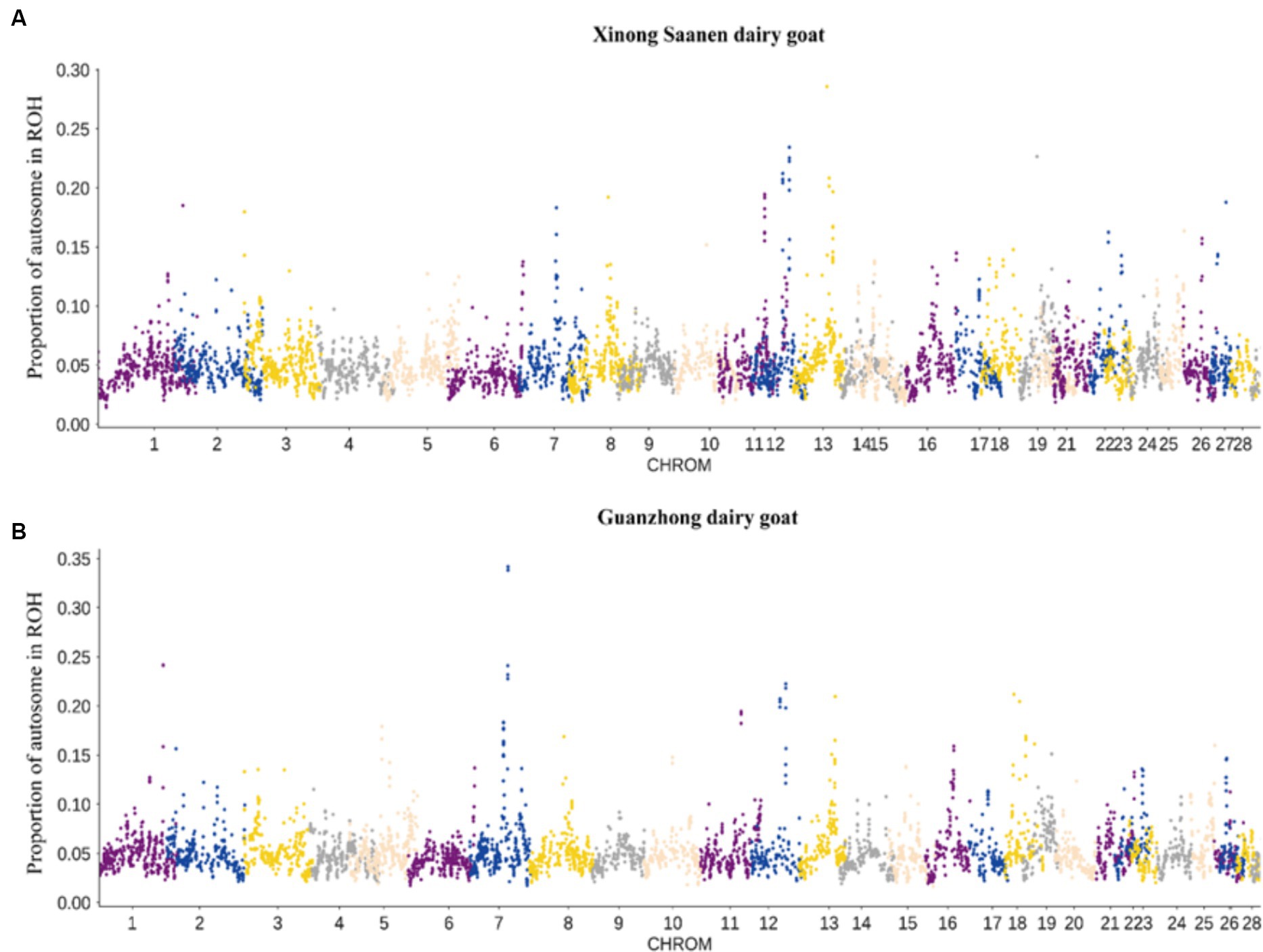
Este estudio llevó a cabo un análisis de la isla ROH en XNG y GZG. En las muestras de XNG, se identificaron 16 islas ROH con una frecuencia del 20% en los cromosomas 12, 13 y 19 (Figura 3A), anotando 31 genes. Del mismo modo, en los cromosomas 1, 7, 12, 13 y 18 del GZG, se identificaron 15 islas ROH con una frecuencia del 20% (Figura 3B), anotando 25 genes. El diagrama de Venn (Figura suplementaria S1) ilustra 4 genes comunes entre las dos razas (MPHOSPH8, LOC108637296, PSPC1, NBEA). Además, los análisis de enriquecimiento de GO y KEGG (Figura suplementaria S2) indican un enriquecimiento de los genes identificados en 23 procesos biológicos (BP), 9 funciones moleculares (MF) y 2 componentes celulares (CC). Las vías enriquecidas incluyen principalmente la regulación biológica, los procesos metabólicos, los procesos del sistema inmunitario y la regulación transcripcional. Además, el análisis KEGG revela enriquecimiento en degradación de valina, leucina e isoleucina, apoptosis, vía de señalización PI3K-Akt y vía de señalización Rap1.
Filogenia y estructura genética de poblaciones
Para comprender la estructura poblacional de las cabras lecheras locales en China, analizamos los resultados de SNP derivados de los datos de resecuenciación. Empleando razas importadas como grupos externos, construimos agrupamientos de diferenciación de SNP basados en varias poblaciones, cuyos resultados se pueden utilizar para verificar mutuamente con otros métodos de agrupamiento. Los porcentajes de PC1, PC2 y PC3 son de 7,91, 5,61 y 3,60%, respectivamente (Figura 4A; Figura complementaria S3). Hay tres clústeres principales en los que AAG, ASG, LSG, NAG, NSG y TGB están estrechamente agrupados, XNG y GZG forman otro clúster, mientras que NBY está lejos de ambos. El resultado indica que las razas locales se pueden diferenciar de las importadas, y existen diferencias en los patrones de agrupamiento entre las 9 poblaciones. Los datos genéticos de las poblaciones se utilizaron para calcular el grado de parentesco entre los individuos, construir una matriz de distancia genética y construir un árbol filogenético utilizando la matriz de distancia. Aunque GZG y XNG se superponen parcialmente en el árbol filogenético, el grupo XNG se agrupa por sí solo, y las razas importadas se pueden diferenciar de las razas locales, lo que es consistente con los resultados de la PCA (Figura 4B). Además, para la población de 9, el número de subgrupos (valor de K) se preestableció en 2-9 para la agrupación, y los resultados de la agrupación se validaron de forma cruzada. El número óptimo de subgrupos se identificó en función de la tasa mínima de error de validación cruzada. K = 4 se identificó como el número óptimo de subgrupos, y hubo diferencias genéticas significativas entre las poblaciones XNG, NBY, NAG y TGB. Independientemente del valor de K, mostró consistentemente diferenciación genética entre razas locales e importadas, lo que indica diferentes composiciones genéticas entre las poblaciones, y antecedentes genéticos relativamente consistentes entre las diferentes 9 poblaciones (Figura 4C).
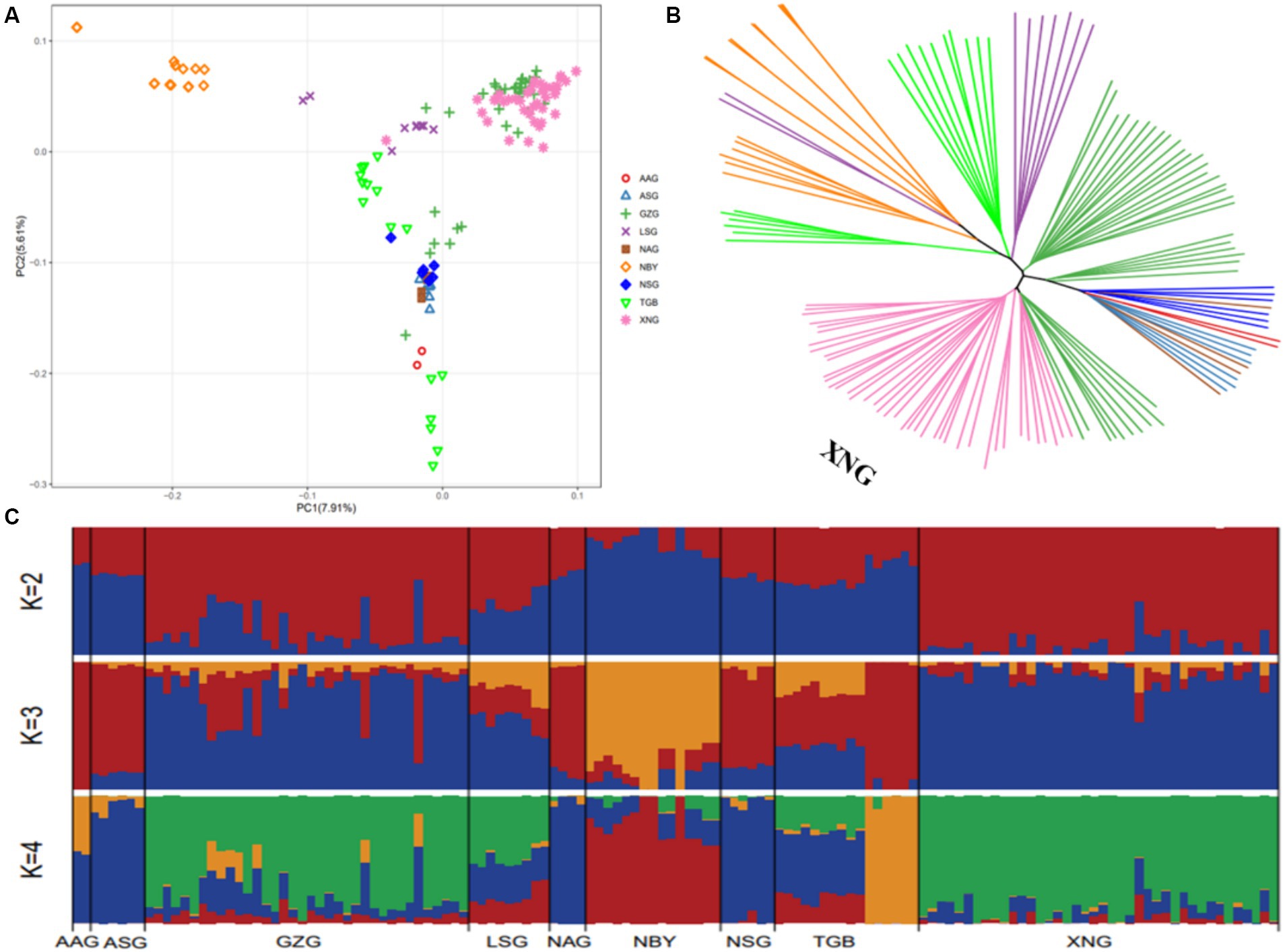
Prueba de barrido selectivo de todo el genoma
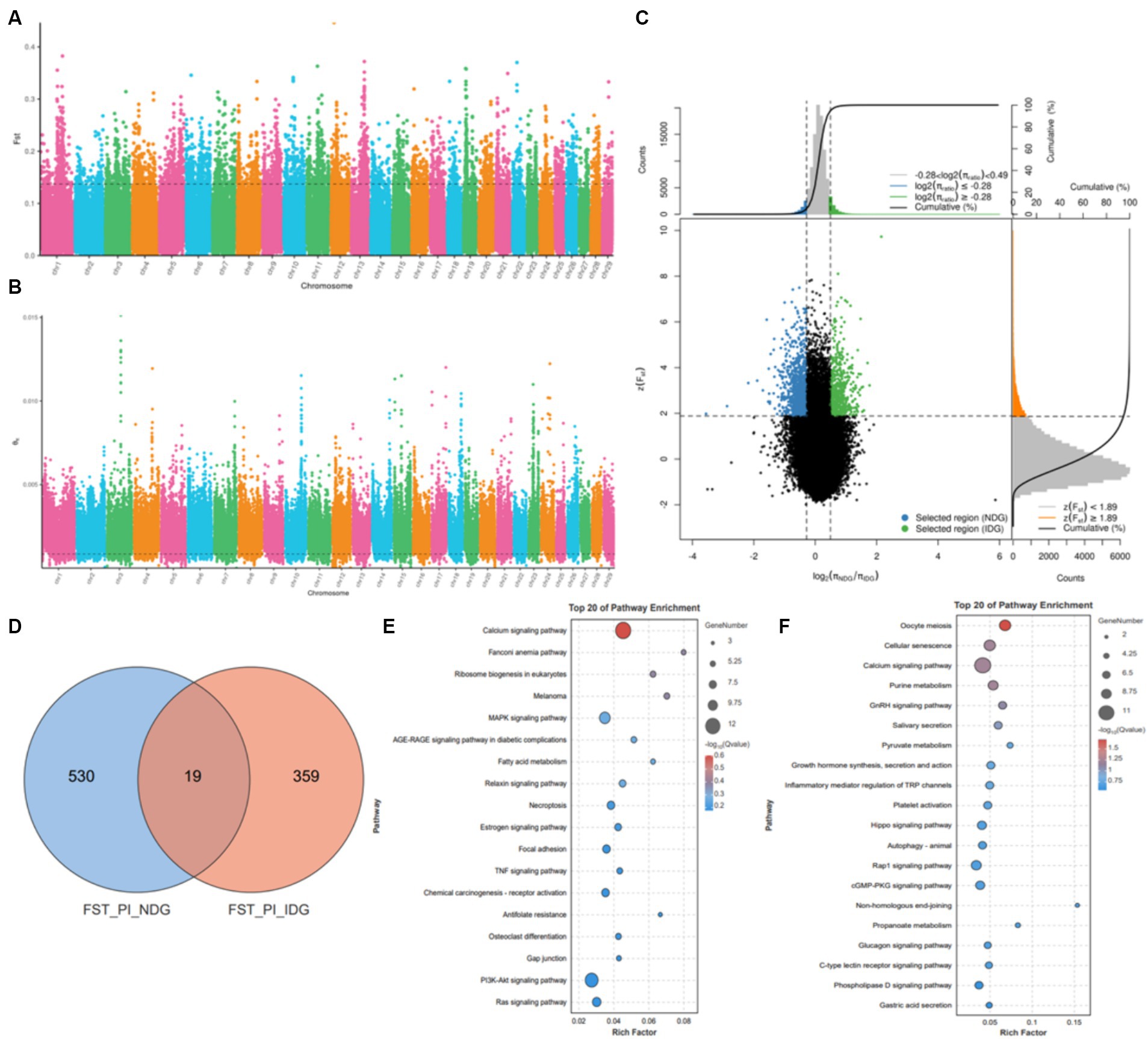
Para identificar las regiones de selección entre las razas autóctonas de cabras lecheras y las razas de cabras lecheras importadas, realizamos un análisis comparativo utilizando los datos de resecuenciación. A continuación, utilizando el 5% superior de los puntos de corte de FST (Figura 5A) y relación θπ (Figura 5B) (Figura 5C), observamos que las razas nativas de cabras lecheras exhibieron un total de 41,25 Mb en 607 regiones escaneadas selectivamente. Estas regiones abarcaron 549 genes (Figura 5D) y mostraron un enriquecimiento significativo en vías como la vía de señalización del calcio, la vía de señalización MAPK, el metabolismo de los ácidos grasos y la vía de señalización PI3K-Akt (Figura 5E). Por otro lado, las razas caprinas lecheras importadas mostraron 25,05 Mb en 385 regiones de barrido selectivo, que abarcaron 387 genes (Figura 5D). El análisis de enriquecimiento de la vía reveló que esos genes están implicados en la meiosis de los ovocitos, la senescencia celular, la vía de señalización del calcio, la vía de señalización de la GnRH y la síntesis y secreción de la vía de la hormona del crecimiento (Figura 5F). Estos hallazgos enfatizan las diferencias funcionales genéticas entre las razas locales e importadas (Figura Suplementaria S4).
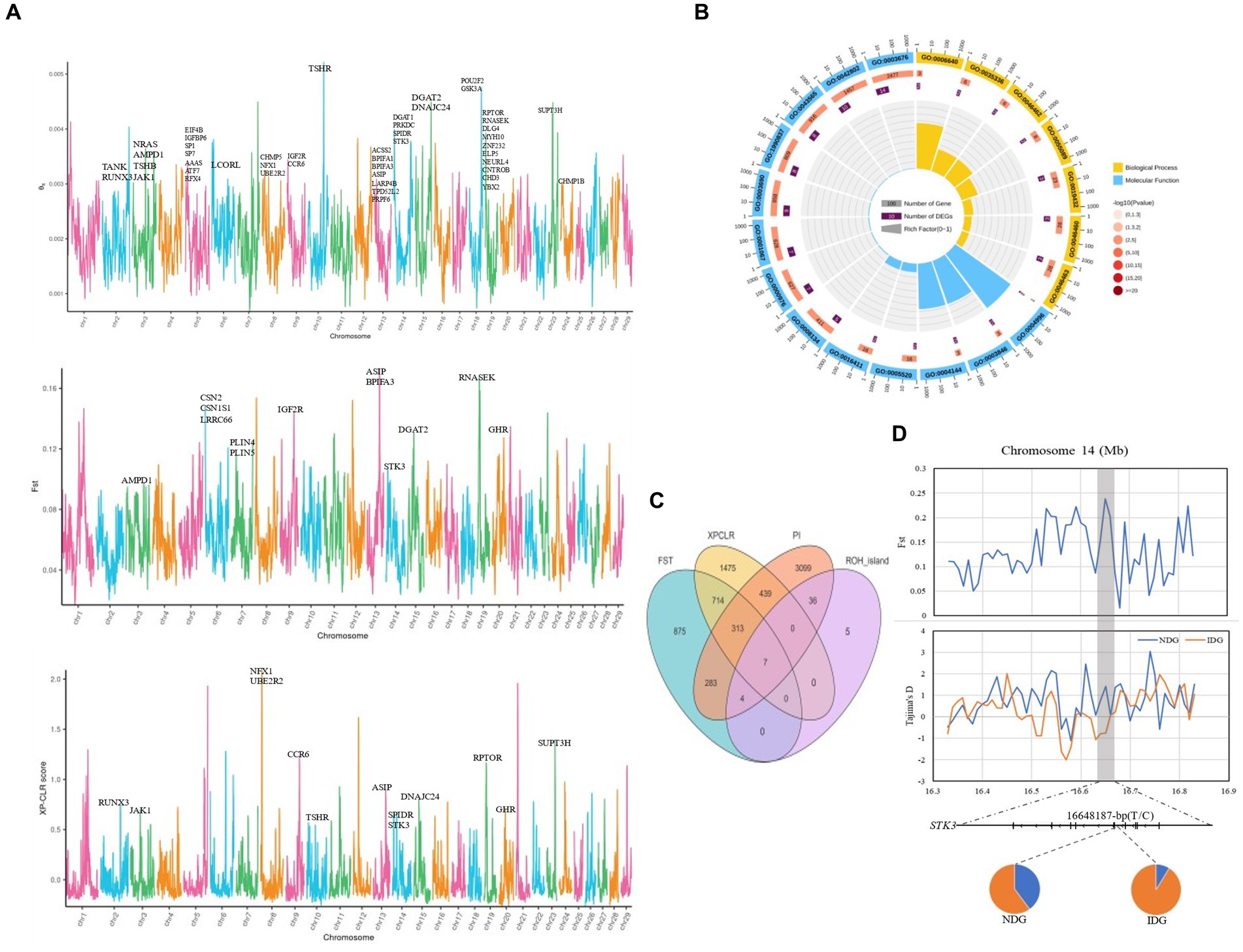
Firmas selectivas para rasgos
Para explorar más a fondo los rasgos correspondientes a las señales de selección, realizamos ensayos FST, PI y XP-CLR para descubrir una selección positiva en cabras lecheras. Mientras tanto, para una anotación más completa de los genes, integramos y anotamos la base de datos QTLdb y un análisis de asociación de todo el genoma publicado anteriormente sobre rasgos importantes de las cabras lecheras en nuestros resultados de señalización de selección (Figura 6A). Encontramos un gran número de genes asociados con rasgos de producción de leche en los cromosomas 5, 13 y 19 (Figura 6A), que estaban principalmente involucrados en el proceso biosintético de monoacilglicerol (GO:0006640), el proceso metabólico de cadena larga de acil-acil-CoA (GO:00353336), el proceso metabólico de monoacilglicerol (GO:0046462) y la homeostasis de ácidos grasos (GO:0055089) (Figura 6B). Además, estos genes también se enriquecieron significativamente en la vía de señalización PI3K-Akt, el metabolismo de los glicerolípidos, la digestión y absorción de grasas y la regulación de la lipólisis en las vías de señalización de los adipocitos (Figura suplementaria S5). El análisis integrativo (Fst, PI, XP-CLR) reveló dos genes superpuestos (Figura Suplementaria S6), ASIP y STK3, respectivamente. En particular, el análisis integrativo (Fst, PI, XP-CLR, ROH_island) de los genes obtenidos de los métodos analíticos anteriores reveló siete genes superpuestos, incluidos PRELID3B, ATP5E, CTSZ, NELFCD, LOC108637417, TRNAF-GAA y TRNAN-GUU (Figura 6C). A continuación, analizamos la distribución de frecuencias alélicas de las regiones seleccionadas para STK3, GHR y PRELID3B ya que muchos métodos han detectado estos tres genes, lo que mostró que las frecuencias génicas de los genes seleccionados diferían entre las razas locales e importadas (Figura 6D; Figura complementaria S7). A través de la función de selección, descubrimos que algunas regiones estaban fuertemente seleccionadas. Algunos genes candidatos se asociaron con rasgos de producción de leche, crecimiento y desarrollo, y rasgos inmunológicos.
Discusión
La detección de firmas de selección positiva recientes en animales domésticos que han pasado por selección artificial y natural puede proporcionar información sobre los sitios genómicos que pueden contribuir a la identificación de mutaciones beneficiosas y vías biológicas subyacentes para rasgos económicamente importantes. En este estudio, analizamos el genoma completo de 134 cabras lecheras de razas autóctonas e importadas. Además, las muestras se obtuvieron de poblaciones que también estaban ampliamente distribuidas en China, una característica que está ausente en estudios anteriores, lo que proporciona un fondo más confiable para el estudio. La detección de la variación genética, la diversidad genética y la estructura de la población mostró que las razas locales de cabras lecheras son más diversas genéticamente que las razas importadas. Estudios previos en bovinos (37, 38) y ovinos (17) han mostrado resultados similares. Este fenómeno sugiere que las razas locales han sido sometidas a una selección menos intensiva, lo que puede explicar por qué su rendimiento en la producción de leche no es sobresaliente. Por lo tanto, el cruce con razas importadas más la selección podría ser una forma prometedora de mejorar las razas locales. Sin embargo, se necesita información genética detallada para guiar dicho proceso de mejoramiento.
Los rasgos de calidad de la leche son un problema biológico complejo que implica la interacción de múltiples genes y vías biológicas. Los ácidos grasos, como uno de los principales componentes de la grasa láctea, tienen un impacto significativo en la calidad y las características de la leche (39). Analizando las señales de selección de razas locales e importadas para una selección positiva. identificamos cuatro genes que están involucrados en el metabolismo de los ácidos grasos (HSD17B12, ACOX3, CPT1B y CBR4) en las señales de selección de cabras lecheras locales. El gen HSD17B12 está implicado en la síntesis de ácidos grasos insaturados (40) y también se asocia con el rendimiento reproductivo (41). El gen ACOX3 está asociado con los rasgos de la grasa de la leche (42) y es un nuevo candidato para los rasgos de producción de leche (43). El gen CPT1B regula los ácidos grasos saturados de cadena larga y está implicado en la lipólisis (44, 45). La síntesis de ácidos grasos puede implicar CBR4 (46). En particular, se observaron diferentes enriquecimientos de vías en las razas importadas. ACADS, AGPAT4, PTDSS2 y SMOX están involucrados principalmente en el metabolismo de los lípidos y las vías del metabolismo de los aminoácidos. ACADS está involucrado en la vía del metabolismo de los ácidos grasos (47), AGPAT4 promueve la síntesis de triacilglicerol y la composición de ácidos grasos (48), PTDSS2 está involucrado en la vía del metabolismo de los glicerofosfolípidos (49) y SMOX es un gen candidato para el crecimiento, los rasgos de composición de la canal y los rasgos de producción de leche (50, 51). Además, los rasgos previamente reportados asociados con la inmunidad (BPIFA3, LRRC66) (52, 53), el color del pelaje (ASIP) (54), la composición de la leche (CSN1S1, DGAT2, PLIN4, PLIN5, IGF2R) (55) y la producción de leche (AMPD1) (56) también se encontraron en razas importadas. En conjunto, los resultados sugieren que los rasgos seleccionados se asociaron principalmente con la producción de leche, la reproducción y la inmunidad tanto para las razas locales como para las importadas de cabras lecheras, a pesar de la diferencia en los genes.
Las características de la producción de leche son las características económicas más importantes de las cabras lecheras. Este estudio incluye marcadores genéticos asociados a rasgos de producción de leche de la base de datos QTLdb para el análisis de señales de selección. Se identificó un gran número de genes seleccionados en los cromosomas 13 y 19, lo que es similar a los resultados de un estudio previo de asociación de genoma completo en cabras lecheras (15, 57). A través del análisis de Fst, PI y XP-CLR, aparecieron dos genes con las señales de selección más altas, incluidos ASIP y STK3. El ASIP es responsable de regular la pigmentación y se asocia con la deposición de grasa y la composición de ácidos grasos en las ovejas (58, 59). El gen se identificó como fuertemente seleccionado debido a la diferencia de color de pelaje entre las cabras lecheras alpinas (negras) y las cabras lecheras nativas (blancas). Mientras que el gen STK3 se identificó como un nuevo gen candidato para los rasgos de producción de leche (60), también mostró una fuerte señal de selección en este estudio. Incorporamos los resultados del análisis de ROH_island en el análisis de señalización selectiva e identificamos siete genes que se superponían, incluidos PRELID3B, ATP5E, CTSZ, NELFCD, LOC108637417, TRNAF-GAA y TRNAN-GUU. PRELID3B tiene un papel importante en la deposición excesiva de melanina (61) y regula la acumulación de lípidos en las mitocondrias (62). En los ovocitos MII de cordero, la regulación negativa de ATP5E y la regulación positiva de CUL1, MARCH7 y TRIM17 podrían causar una baja competencia de los embriones de cordero (63). Además, se ha informado de que ATP5E es un prometedor biomarcador candidato para la viabilidad de los ovocitos después de la MIV (64), por lo que el gen podría estar asociado con rasgos reproductivos en animales hembra. Muchos estudios han demostrado que la CTSZ está relacionada con varios rasgos, incluida la inmunidad (65), el embarazo (66), la regulación de la termogénesis en los adipocitos marrones (67), el crecimiento, la canal y la producción (68). NELFCD puede servir como un nuevo gen candidato para la inmunomodulación temprana (69) y está fuertemente relacionado con CTSZ (70). TRNAF-GAA y TRNAN-GUU son genes relacionados con el ARNt, que podrían estar asociados con rasgos inmunes (71) y reproductivos (72, 73). Sin embargo, la función del gen LOC108637417 sigue sin estar clara. Vale la pena señalar que el GHR no solo está fuertemente seleccionado, sino que también se asocia con varios rasgos económicos [producción de leche (74), crecimiento (75) y rasgos de calidad de la leche (76)]. En resumen, los genes identificados en este estudio podrían ser nuevos genes candidatos para la producción de leche, la reproducción y los rasgos inmunológicos en cabras lecheras.
Para investigar más a fondo las diferencias de los genes asociados con las regiones seleccionadas en diferentes razas, los genes clave STK3, GHR y PRELID3B se sometieron a un análisis de frecuencia alélica. Se encontró que las frecuencias genotípicas de los genes fuertemente seleccionados diferían significativamente entre las diferentes poblaciones. Resultados similares se encontraron en la investigación genómica de pollos (77) y ovejas (17). Al comparar las diferencias genéticas entre las diferentes variedades, podemos comprender mejor los mecanismos genéticos que subyacen a estas diferencias, proporcionando orientación para el mejoramiento futuro de las variedades y la protección de los recursos genéticos.
Conclusión
Este estudio analizó la diversidad genética, la estructura poblacional, las señales de selección y las frecuencias alélicas de cabras lecheras locales e importadas en China. Se encontró que existían diferencias genómicas significativas entre las dos poblaciones. Además, se identificaron genes candidatos relacionados con la producción de leche (STK3, GHR, PRELID3B), la reproducción (ATP5E), el crecimiento y el desarrollo (CTSZ, GHR) y los rasgos inmunes (CTSZ, NELFCD). Proporciona información valiosa sobre la diversidad genética de las cabras lecheras y, por lo tanto, pone el apoyo de datos para la futura cría y selección de cabras lecheras en China.
Declaración de disponibilidad de datos
Los datos que respaldan los hallazgos de este estudio están disponibles abiertamente en NCBI Sequence Read Archive en https://www.ncbi.nlm.nih.gov/, número de acceso: PRJNA1127047.
Declaración ética
Los estudios en animales fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad Northwest A&F. Los estudios se llevaron a cabo de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones de los autores
JZ: Visualización, Escritura – revisión y edición, Escritura – borrador original, Análisis formal. YM: Redacción – revisión y edición, curación de datos. PG: Redacción – revisión y edición, Recursos. BL: Redacción – revisión y edición, supervisión. FZ: Software, Redacción – revisión y edición, Metodología. LZ: Redacción – revisión y edición, Metodología. CS: Redacción – revisión y edición, Metodología. XL: Escritura – revisión y edición, Recursos. JL: Administración de proyectos, Redacción – borrador original, Redacción – revisión y edición, Supervisión, Conceptualización.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo contó con el apoyo del Proyecto Clave de Fusión de Doble Cadena de Cría de Ganado y Aves de Corral de Shaanxi de China (2022GD-TSLD-46-0201), el Proyecto del Plan de Ciencia y Tecnología de la Ciudad de Hohhot (Proyecto Especial Principal de Ciencia y Tecnología) en China (2023150103000025).
Reconocimientos
Este trabajo recibió el apoyo de los recursos de computación de alto rendimiento (HPC) de la Universidad del Noroeste A&F (NWAFU).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2024.1409282/full#supplementary-material
Notas
Referencias
1. Zeder, MA. Domesticación y agricultura temprana en la cuenca mediterránea: orígenes, difusión e impacto. Proc Natl Acad Sci USA. (2008) 105:11597–604. doi: 10.1073/pnas.0801317105
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Zhang, B, Chang, L, Lan, X, Asif, N, Guan, F, Fu, D, et al. La definición de barridos selectivos en todo el genoma revela evidencia molecular de domesticación impulsada por rasgos entre las razas de cabras de élite (especies de Capra) para la producción de lácteos, cachemira y carne. GigaCiencia. (2018) 7:GIY105. doi: 10.1093/gigascience/giy105
3. Pereira, F, y Amorim, A. (2010). Origen y difusión del pastoreo caprino. eLS. Disponible en: https://www.researchgate.net/profile/Filipe-Pereira-17/publication/222094707_Origin_and_Spread_of_Goat_Pastoralism/links/5a0485b5a6fdcceda02baefa/Origin-and-Spread-of-Goat-Pastoralism.pdf. (Consultado el 9 de diciembre de 2023)
4. Cao, Y, Feng, T, Wu, Y, Xu, Y, Du, L, Wang, T, et al. El microbioma multireino del tracto gastrointestinal de la cabra. Microbioma. (2023) 11:219. doi: 10.1186/s40168-023-01651-6
5. Pulina, G, Milán, MJ, Lavín, MP, Theodoridis, A, Morin, E, Capote, J, et al. Revisión invitada: tendencias actuales de producción, estructuras de las explotaciones y economía de los sectores de ovino y caprino lechero. J Dairy Sci. (2018) 101:6715–29. doi: 10.3168/jds.2017-14015
Resumen de PubMed | Texto completo de Crossref | Google Académico
6. Mónica, K. Influencia de las prácticas ganaderas lecheras en la producción de leche. Una revisión crítica de la literatura. Anim Salud J. (2022) 3:1–15. doi: 10.47941/ahj.771
7. Nayik, GA, Jagdale, YD, Gaikwad, SA, Devkatte, AN, Dar, AH, y Ansari, MJ. Perfil nutricional, procesamiento y productos potenciales: una revisión comparativa de la leche de cabra. Lácteos. (2022) 3:622–47. doi: 10.3390/dairy3030044
8. Xiaopa, G. Basado en los proyectos de construcción de la industria de cabras lecheras de Feihe, estudie el desarrollo de la industria de cabras lecheras del condado de Fuping en la provincia de Shaanxi. Feed Rev. (2015) Disponible en: https://api.semanticscholar.org/CorpusID:114375686
9. Korma, SA, Li, L, Wei, W, Liu, P, Zhang, X, Bakry, IA, et al. Estudio comparativo de la grasa láctea extraída de la leche de diferentes razas caprinas en China: ácidos grasos, triacilgliceroles y caracterización térmica y espectroscópica. Biomoléculas. (2022) 12:730. doi: 10.3390/biom12050730
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Zhang, L. Diversidad genética de cinco razas de cabras en China basada en marcadores microsatélites. Afr J Biotechnol. (2012) 11:54. doi: 10.5897/AJB12.539
11. Fan, L, Shen, J, Li, X, Li, H, Shao, Y, Lu, CD, et al. Análisis de los cambios temporales de la diversidad de la microbiota y las interacciones ambientales en cabras lecheras de Saanen. J Appl Anim Res. (2023) 51:749–63. doi: 10.1080/09712119.2023.2273945
12. Idamokoro, EM. La importancia de la leche de cabra en la mejora de la seguridad nutricional: una evaluación cienciométrica de los estudios de investigación desde 1966 hasta 2020. Agric Food Secur. (2023) 12:34. doi: 10.1186/s40066-023-00441-5
13. Rout, PK, y Behera, BK. Ganadería caprina y ovina En: PK Rout y BK Behera, editores. Sostenibilidad en la ganadería de rumiantes: gestión y comercialización. Singapur: Springer (2021). 33–76.
14. Harper, K, Tait, A, Li, X, Sullivan, M, Gaughan, J, Poppi, D, et al. Industrias ganaderas en Australia: sistemas de producción y gestión (2021). 79–139.
15. Massender, E, Brito, LF, Maignel, L, Oliveira, HR, Jafarikia, M, Baes, CF, et al. Evaluación genómica en un solo paso de los rasgos de producción de leche en cabras lecheras canadienses alpinas y saanen. J Dairy Sci. (2022) 105:2393–407. doi: 10.3168/jds.2021-20558
Resumen de PubMed | Texto completo de Crossref | Google Académico
16. Lima, ARC, Silveira, RMF, Castro, MSM, De Vecchi, LB, da Rocha Fernandes, MHM, y de Resende, KT. Relación entre el ambiente térmico, las respuestas termorreguladoras y el metabolismo energético en cabras: una revisión exhaustiva. J Therm Biol. (2022) 109:103324. doi: 10.1016/j.jtherbio.2022.103324
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Li, X, Yang, J, Shen, M, Xie, X-L, Liu, G-J, Xu, Y-X, et al. La resecuenciación del genoma completo de ovejas silvestres y domésticas identifica genes asociados con rasgos morfológicos y agronómicos. Nat Commun. (2020) 11:2815. doi: 10.1038/s41467-020-16485-1
18. Li, X, Liu, Q, Fu, C, Li, M, Li, C, Li, X, et al. La caracterización de las variantes estructurales basada en el genotipado gráfico proporciona información sobre la domesticación del cerdo y la adaptación local. J Genómica de Genet. (2023) S1673-8527:00241–2. doi: 10.1016/j.jgg.2023.11.005
19. Xia, X, Zhang, F, Li, S, Luo, X, Peng, L, Dong, Z, et al. La variación estructural y la introgresión de las poblaciones silvestres en los genomas del ganado bovino de Asia oriental confieren adaptación al medio ambiente local. Genoma Biol. (2023) 24:211. doi: 10.1186/s13059-023-03052-2
20. Chen, N, Xia, X, Hanif, Q, Zhang, F, Dang, R, Huang, B, et al. La diversidad genética global, la introgresión y la adaptación evolutiva del ganado indígena reveladas por la secuenciación del genoma completo. Nat Commun. (2023) 14:7803. doi: 10.1038/s41467-023-43626-z
21. Bolger, AM, Lohse, M y Usadel, B. Trimmomatic: un recortador flexible para los datos de secuencia de Illumina. Bioinformática. (2014) 30:2114–20. doi: 10.1093/bioinformatics/btu170
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Li, H y Durbin, R. Alineación de lectura corta rápida y precisa con la transformada de Burrows-Wheeler. Bioinformática. (2009) 25:1754–60. doi: 10.1093/bioinformatics/btp324
23. Li, H, Handsaker, B, Wysoker, A, Fennell, T, Ruan, J, Homer, N, et al. El formato de alineación/mapa de secuencia y SAMtools. Bioinformática. (2009) 25:2078–9. doi: 10.1093/bioinformatics/BTP352
Resumen de PubMed | Texto completo de Crossref | Google Académico
24. McKenna, A, Hanna, M, Banks, E, Sivachenko, A, Cibulskis, K, Kernytsky, A, et al. El kit de herramientas de análisis del genoma: un marco de MapReduce para analizar datos de secuenciación de ADN de próxima generación. Genoma Res. (2010) 20:1297–303. doi: 10.1101/gr.107524.110
Resumen de PubMed | Texto completo de Crossref | Google Académico
25. Wang, K, Li, M y Hakonarson, H. ANNOVAR: anotación funcional de variantes genéticas a partir de datos de secuenciación de alto rendimiento. Ácidos nucleicos Res. (2010) 38:E164. doi: 10.1093/nar/gkq603
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Purcell, S, Neale, B, Todd-Brown, K, Thomas, L, Ferreira, MAR, Bender, D, et al. PLINK: un conjunto de herramientas para la asociación del genoma completo y los análisis de ligamiento basados en la población. Am J Hum Genet. (2007) 81:559–75. doi: 10.1086/519795
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. Danecek, P, Auton, A, Abecasis, G, Albers, CA, Banks, E, DePristo, MA, et al. El formato de llamada variante y VCFtools. Bioinformática. (2011) 27:2156–8. doi: 10.1093/bioinformatics/btr330
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. Zhang, C, Dong, S-S, Xu, J-Y, He, W-M y Yang, T-L. PopLDdecay: una herramienta rápida y eficaz para el análisis de la caída del desequilibrio del enlace basada en archivos de formato de llamada variante. Bioinformática. (2019) 35:1786–8. doi: 10.1093/bioinformatics/bty875
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Kumar, S, Stecher, G, Li, M, Knyaz, C y Tamura, K. MEGA X: análisis de genética evolutiva molecular a través de plataformas informáticas. Mol Biol Evol. (2018) 35:1547–9. DOI: 10.1093/molbev/msy096
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Letunic, I, y Bork, P. Interactive Tree Of Life (iTOL) v5: una herramienta en línea para la visualización y anotación de árboles filogenéticos. Ácidos nucleicos Res. (2021) 49:W293–6. doi: 10.1093/nar/gkab301
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Patterson, N, Price, AL, y Reich, D. Estructura de la población y análisis propio. PLoS Genet. (2006) 2:E190. doi: 10.1371/journal.pgen.0020190
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Alexander, DH y Lange, K. Mejoras en el algoritmo ADMIXTURE para la estimación de ascendencia individual. BMC Bioinformática. (2011) 12:246. doi: 10.1186/1471-2105-12-246
33. Korneliussen, TS, Moltke, I, Albrechtsen, A, y Nielsen, R. Cálculo de las estadísticas de la prueba D de Tajima y otras pruebas de neutralidad a partir de datos de secuenciación de próxima generación de baja profundidad. BMC Bioinformática. (2013) 14:289. doi: 10.1186/1471-2105-14-289
34. Chen, H, Patterson, N, y Reich, D. Diferenciación poblacional como prueba para barridos selectivos. Genoma Res. (2010) 20:393–402. doi: 10.1101/gr.100545.109
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Quinlan, AR, y Hall, IM. BEDTools: un conjunto flexible de utilidades para comparar características genómicas. Bioinformática. (2010) 26:841–2. doi: 10.1093/bioinformatics/btq033
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. Bu, D, Luo, H, Huo, P, Wang, Z, Zhang, S, He, Z, et al. KOBAS-i: priorización inteligente y visualización exploratoria de funciones biológicas para el análisis de enriquecimiento de genes. Ácidos nucleicos Res. (2021) 49:W317–25. doi: 10.1093/nar/gkab447
Resumen de PubMed | Texto completo de Crossref | Google Académico
37. Jin, L, Qu, K, Hanif, Q, Zhang, J, Liu, J, Chen, N, et al. La secuenciación del genoma completo del ganado de Dengchuan, en peligro de extinción, revela su diversidad genómica y sus firmas de selección. Frente Genet. (2022) 13:833475. doi: 10.3389/fgene.2022.833475
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Jin, L, Zhang, B, Luo, J, Li, J, Liang, J, Wu, W, et al. Genómica, origen y señales de selección del ganado Loudi en Hunan Central. Biología. (2022) 11:1775. doi: 10.3390/biology11121775
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Palombo, V, Milanesi, M, Sgorlon, S, Capomaccio, S, Mele, M, Nicolazzi, E, et al. Estudio de asociación de todo el genoma de la composición de ácidos grasos de la leche en vacas italianas Simmental e italianas Holstein utilizando matrices de polimorfismo de un solo nucleótido. J Dairy Sci. (2018) 101:11004–19. doi: 10.3168/jds.2018-14413
Resumen de PubMed | Texto completo de Crossref | Google Académico
40. Liu, T, Feng, H, Yousuf, S, Xie, L y Miao, X. Regulación diferencial de ARNm y ARN lnc relacionados con el metabolismo lipídico en ovejas Duolang y Han de cola pequeña. Sci Rep. (2022) 12:11157. doi: 10.1038/s41598-022-15318-z
41. Juengel, JL, Mosaad, EMO, Mitchell, MD, Phyn, CVC, French, MC, Meenken, ED, et al. Relaciones entre las concentraciones de prostaglandinas, un polimorfismo de un solo nucleótido en HSD17B12 y el rendimiento reproductivo en vacas lecheras. J Dairy Sci. (2022) 105:4643–52. doi: 10.3168/jds.2021-21298
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Ariyarathne, HBPC, Correa-Luna, M, Blair, HT, Garrick, DJ y López-Villalobos, N. Identificación de regiones genómicas asociadas con las concentraciones de grasa láctea, proteína, urea y eficiencia de la utilización de proteína cruda en vacas lecheras en pastoreo. Genes. (2021) 12:456. doi: 10.3390/genes12030456
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Ibeagha-Awemu, EM, Peters, SO, Akwanji, KA, Imumorin, IG y Zhao, X. El genotipado de genoma completo de alta densidad por secuenciación y asociación identifica SNP comunes y de baja frecuencia, y nuevos genes candidatos que influyen en los rasgos de la leche de vaca. Sci Rep. (2016) 6:31109. doi: 10.1038/srep31109
44. Dervishi, E, Joy, M, Sanz, A, Alvarez-Rodriguez, J, Molino, F, y Calvo, JH. La conservación del forraje (pastoreo vs. heno) con la que se alimenta a las ovejas afecta el perfil de ácidos grasos de la leche y la expresión génica de CPT1B en la glándula mamaria de las ovejas. BMC Vet Res. (2012) 8:106. doi: 10.1186/1746-6148-8-106
45. Fang, X, Zhao, Z, Jiang, P, Yu, H, Xiao, H y Yang, R. Identificación de los perfiles de expresión génica de HSL bovino y su asociación con la composición de ácidos grasos y los rasgos de deposición de grasa. Ciencia de la carne. (2017) 131:107–18. doi: 10.1016/j.meatsci.2017.05.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. Cai, C, Li, M, Zhang, Y, Meng, S, Yang, Y, Gao, P, et al. Análisis comparativos del transcriptoma de Longissimus thoracis entre razas de cerdos que difieren en características musculares. Frente Genet. (2020) 11:526309. doi: 10.3389/fgene.2020.526309
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Alaedin, M, Ghaffari, MH, Sadri, H, Meyer, J, Dänicke, S, Frahm, J, et al. Efectos de la suplementación dietética con l-carnitina en la respuesta a un desafío inflamatorio en vacas lecheras en lactancia media: abundancia hepática de ARNm de genes implicados en el metabolismo de los ácidos grasos. J Dairy Sci. (2021) 104:11193–209. doi: 10.3168/jds.2021-20226
Resumen de PubMed | Texto completo de Crossref | Google Académico
48. Li, Z, Li, R, Ren, H, Qin, C, Su, J, Song, X, et al. Papel de diferentes miembros de la familia de genes AGPAT en la síntesis de grasa láctea en Bubalus bubalis. Genes. (2023) 14:2072. doi: 10.3390/genes14112072
Resumen de PubMed | Texto completo de Crossref | Google Académico
49. Lin, X, Wei, Y, Li, Y, Xiong, Y, Fang, B, Li, C, et al. El ácido tormentico mejora la fibrosis hepática in vivo mediante la inhibición del metabolismo de los glicerofosfolípidos y de las vías PI3K/Akt/mTOR y NF-κB: según la transcriptómica y la metabolómica. Frente Pharmacol. (2022) 13:801982. doi: 10.3389/fphar.2022.801982
Resumen de PubMed | Texto completo de Crossref | Google Académico
50. Yilmaz, O, Kizilaslan, M, Arzik, Y, Behrem, S, Ata, N, Karaca, O, et al. Estudios de asociación de todo el genoma de los rasgos de crecimiento predestete y composición de la canal in vivo en ovejas Esme. J Anim Raza Genet. (2022) 139:26–39. doi: 10.1111/jbg.12640
Resumen de PubMed | Texto completo de Crossref | Google Académico
51. Li, X, Yuan, L, Wang, W, Zhang, D, Zhao, Y, Chen, J, et al. La resecuenciación del genoma completo revela la selección artificial y natural de los rasgos de la leche en las ovejas de Frisona Oriental. Front Vet Sci. (2022) 9:1034211. doi: 10.3389/fvets.2022.1034211
52. Guan, D, Landi, V, Luigi-Sierra, MG, Delgado, JV, Such, X, Castelló, A, et al. Análisis de la arquitectura genómica y transcriptómica de las características de la leche en cabras murciano-granadinas. J Anim Sci Biotechnol. (2020) 11:35. doi: 10.1186/s40104-020-00435-4
53. Lien, Y-C, Wang, PZ, Lu, XM y Simmons, RA. Alteración de la unión al factor de transcripción y bivalencia génica en islotes de ratas con retraso en el crecimiento intrauterino. Células. (2020) 9:1435. doi: 10.3390/cells9061435
Resumen de PubMed | Texto completo de Crossref | Google Académico
54. Liu, M, Zhou, Y, Rosen, BD, Van Tassell, CP, Stella, A, Tosser-Klopp, G, et al. Diversidad de variaciones en el número de ejemplares en la población caprina mundial. Herencia. (2019) 122:636–46. doi: 10.1038/s41437-018-0150-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
55. Xiong, J, Bao, J, Hu, W, Shang, M y Zhang, L. La resecuenciación del genoma completo revela la diversidad genética y las características de selección de la cabra lechera. Frente Genet. (2023) 13:1044017. doi: 10.3389/fgene.2022.1044017
Resumen de PubMed | Texto completo de Crossref | Google Académico
56. Wei, CB, He, F, Tang, JC, Yang, YH, Deng, XL y Liu, FX. El polimorfismo de la secuencia de ADN dentro de la adenosina monofosfato desaminasa 1 bovina (AMPD1) se asocia con rasgos de producción en el ganado chino. Genet Mol Res. (2015) 14:1025–33. doi: 10.4238/2015.febrero.6.6
57. Scholtens, M, Jiang, A, Smith, A, Littlejohn, M, Lehnert, K, Snell, R, et al. Estudios de asociación de todo el genoma de los rendimientos de leche, grasa, proteína y puntuación de células somáticas en lactancia en cabras lecheras de Nueva Zelanda. J Anim Sci Biotechnol. (2020) 11:55. doi: 10.1186/s40104-020-00453-2
58. Norris, BJ, y Whan, VA. Una duplicación génica que afecta la expresión del gen ASIP ovino es responsable de las ovejas blancas y negras. Genoma Res. (2008) 18:1282–93. doi: 10.1101/gr.072090.107
Resumen de PubMed | Texto completo de Crossref | Google Académico
59. Rochus, CM, Westberg Sunesson, K, Jonas, E, Mikko, S, y Johansson, AM. Mutaciones en ASIP y MC1R: los alelos negro dominante y negro recesivo se segregan en las poblaciones nativas de ovejas suecas. Anim Genet. (2019) 50:712–7. doi: 10.1111/age.12837
Resumen de PubMed | Texto completo de Crossref | Google Académico
60. Teng, J, Wang, D, Zhao, C, Zhang, X, Chen, Z, Liu, J, et al. Estudios longitudinales de asociación de todo el genoma de los rasgos de producción de leche en bovinos Holstein utilizando datos de secuencia del genoma completo imputados a partir de datos de chips de densidad media. J Dairy Sci. (2023) 106:2535–50. doi: 10.3168/jds.2022-22277
Resumen de PubMed | Texto completo de Crossref | Google Académico
61. Li, D, Sun, G, Zhang, M, Cao, Y, Zhang, C, Fu, Y, et al. Historia de reproducción y genes candidatos responsables de la piel negra del pollo de hueso negro de Xichuan. BMC Genómica. (2020) 21:511. doi: 10.1186/s12864-020-06900-8
62. Miliara, X, Tatsuta, T, Berry, J-L, Rouse, SL, Solak, K, Chorev, DS, et al. Determinantes estructurales de la especificidad lipídica dentro de las proteínas de transferencia de lípidos ups/PRELI. Nat Commun. (2019) 10:1130. doi: 10.1038/s41467-019-09089-x
63. Ying, C, Yangsheng, W, Jiapeng, L, Liqin, W, Xiaolin, L, Mingjun, L, et al. Perfiles transcriptómicos de ovocitos ovinos madurados in vitro prepuberales y adultos obtenidos de animales estimulados por FSH. Reprod Domest Anim. (2021) 56:1085–94. doi: 10.1111/rda.13951
Resumen de PubMed | Texto completo de Crossref | Google Académico
64. Gao, L, Jia, G, Li, A, Ma, H, Huang, Z, Zhu, S, et al. Perfil del transcriptoma RNA-Seq de ovocitos de ratón después de la maduración y/o vitrificación in vitro. Sci Rep. (2017) 7:13245. doi: 10.1038/s41598-017-13381-5
65. Hu, Y, Yu, L, Fan, H, Huang, G, Wu, Q, Nie, Y, et al. Firmas genómicas de la coevolución entre mamíferos no modelo y nematodos parásitos. Mol Biol Evol. (2021) 38:531–44. DOI: 10.1093/molbev/msaa243
Resumen de PubMed | Texto completo de Crossref | Google Académico
66. Song, G, Spencer, TE, y Bazer, FW. Catepsinas en el útero ovino: regulación por embarazo, progesterona e interferón tau. Endocrinología. (2005) 146:4825–33. doi: 10.1210/en.2005-0768
Resumen de PubMed | Texto completo de Crossref | Google Académico
67. Shen, Y, Su, Y, Silva, FJ, Weller, AH, Sostre-Colón, J, Titchenell, PM, et al. Los genes diana PPARα/γ compartidos regulan la función termogénica de los adipocitos marrones. Rep. de Célula (2020) 30:3079–3091.e5. doi: 10.1016/j.celrep.2020.02.032
Resumen de PubMed | Texto completo de Crossref | Google Académico
68. Russo, V, Fontanesi, L, Scotti, E, Beretti, F, Davoli, R, Nanni Costa, L, et al. Los polimorfismos de un solo nucleótido en varios genes de catepsina porcina se asocian con rasgos de crecimiento, canal y producción en cerdos blancos grandes italianos. J Anim Sci. (2008) 86:3300–14. doi: 10.2527/jas.2008-0920
Resumen de PubMed | Texto completo de Crossref | Google Académico
69. van Bilsen, JHM, Dulos, R, van Stee, MF, Meima, MY, Rouhani Rankouhi, T, Neergaard Jacobsen, L, et al. Búsqueda de ventanas de oportunidad para dar forma a la salud inmunitaria a lo largo de la vida: una estrategia basada en la red para predecir y priorizar los marcadores de la modulación inmunitaria en los primeros años de vida. Frente Immunol. (2020) 11:644. doi: 10.3389/fimmu.2020.00644
70. Nishida, N, Aiba, Y, Hitomi, Y, Kawashima, M, Kojima, K, Kawai, Y, et al. Los loci NELFCD y CTSZ se asocian con la progresión de la etapa de ictericia en la colangitis biliar primaria en la población japonesa. Sci Rep. (2018) 8:8071. doi: 10.1038/s41598-018-26369-6
71. Mailu, BM, Li, L, Arthur, J, Nelson, TM, Ramasamy, G, Fritz-Wolf, K, et al. La biosíntesis de Plasmodium apicoplast Gln-tRNAGln utiliza una amidotransferasa GatAB única esencial para los parásitos en etapa eritrocítica. J Biol Chem. (2015) 290:29629–41. doi: 10.1074/jbc. M115.655100
Resumen de PubMed | Texto completo de Crossref | Google Académico
72. Wang, D, Ning, C, Xiang, H, Zheng, X, Kong, M, Yin, T, et al. Polimorfismo de los genes del ARNt mitocondrial asociado con el número de cerdos nacidos vivos. J Anim Sci Biotechnol. (2018) 9:86. doi: 10.1186/s40104-018-0299-0
73. Zhang, Y, Labrecque, R, Tremblay, P, Plessis, C, Dufour, P, Martin, H, et al. Análisis de tsRNAs y miRNAs transmitidos por espermatozoides en relación con la fertilidad del ganado lechero. Teriogenología. (2023) 215:241–8. doi: 10.1016/j.theriogenology.2023.11.029
74. Dettori, ML, Pazzola, M, Paschino, P, Amills, M y Vacca, GM. Asociación entre los polimorfismos de los genes GHR, GHRHR e IGF1 y los rasgos de producción y calidad de la leche en ovejas Sarda. J Dairy Sci. (2018) 101:9978–86. doi: 10.3168/jds.2018-14914
Resumen de PubMed | Texto completo de Crossref | Google Académico
75. Wu, M, Zhao, H, Tang, X, Li, Q, Yi, X, Liu, S, et al. Novel InDels of GHR, GHRH, GHRHR y su asociación con rasgos de crecimiento en siete razas de ovejas chinas. Animales. (2020) 10:1883. doi: 10.3390/ani10101883
Resumen de PubMed | Texto completo de Crossref | Google Académico
76. Pazzola, M, Vacca, GM, Paschino, P, Bittante, G y Dettori, ML. Nuevos genes asociados con rasgos lácteos en ovejas Sarda. Animales. (2021) 11:2207. doi: 10.3390/ani11082207
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: cabra lechera, secuencia del genoma completo, diversidad genética, señal selectiva, características de producción
Cita: Zhao J, Mu Y, Gong P, Liu B, Zhang F, Zhu L, Shi C, Lv X y Luo J (2024) La resecuenciación del genoma completo de cabras lecheras nativas e importadas identifica genes asociados con la productividad y la inmunidad. Frente. Vet. Sci. 11:1409282. doi: 10.3389/fvets.2024.1409282
Recibido: 29 de marzo de 2024; Aceptado: 23 de mayo de 2024;
Publicado: 08 Julio 2024.
Editado por:
Shijie Lyu, Academia de Ciencias Agrícolas de Henan (HNAAS), China
Revisado por:
Gan Shangquan, Academia de Ciencias Agrícolas y de la Recuperación de Xinjiang (XAARS), China
Marina Mortati Dias Barbero, Universidad Federal Rural de Río de Janeiro, Brasil
Derechos de autor © 2024 Zhao, Mu, Gong, Liu, Zhang, Zhu, Shi, Lv y Luo. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Jun Luo, luojun@nwafu.edu.cn
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente a las de sus organizaciones afiliadas, o las del editor, de los editores y de los revisores. Cualquier producto que puede ser evaluada en este artículo o afirmación que puede ser hecha por su El fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía