Lactobacillus rhamnosus GG ATCC53103 y Lactobacillus plantarum JL01 mejoraron el metabolismo del nitrógeno en lechones destetados

Lactobacillus rhamnosus GG ATCC53103 y Lactobacillus plantarum JL01 mejoraron el metabolismo del nitrógeno en lechones destetados mediante la regulación de la estructura de la flora intestinal y los metabolitos de la vena porta
 Feng He1,2,3,
Feng He1,2,3,  Xueying Jin1,2,3,
Xueying Jin1,2,3,  Chunfeng Wang1,2,3,
Chunfeng Wang1,2,3,  Jingtao Hu1,2,3,
Jingtao Hu1,2,3,  Shuai Su1,2,3,
Shuai Su1,2,3,  Lei Zhao4,
Lei Zhao4,  Tingting Geng1,2,3,
Tingting Geng1,2,3,  Yuan Zhao1,2,3,
Yuan Zhao1,2,3,  Li Pan1,2,3,
Li Pan1,2,3,  Nan Bao1,2,3 y
Nan Bao1,2,3 y  Hui Sun1,2,3*
Hui Sun1,2,3*- 1Facultad de Ciencia y Tecnología Animal, Universidad Agrícola de Jilin, Changchun, China
- 2Ministerio de Educación Laboratorio de Producción Animal y Seguridad de la Calidad, Universidad Agrícola de Jilin, Changchun, China
- 3Laboratorio Provincial Clave de Jilin de Nutrición Animal y Ciencia de los Piensos, Universidad Agrícola de Jilin, Changchun, China
- 4Facultad de Ciencia Animal y Medicina Veterinaria, Universidad Agrícola Heilongjiang Bayi, Daqing, Heilongjiang, China
En la actualidad, la mayoría de los estudios han demostrado que los probióticos tienen un efecto regulador positivo sobre el metabolismo nutricional del cuerpo, pero el mecanismo aún no está claro. Aquí, 48 lechones se dividieron en cuatro grupos. El grupo de control no fue alimentado con probióticos, el grupo Lac fue alimentado con L. Rhamnosus GG ATCC53103, el grupo Rha fue alimentado con L. Plantarum JL01, y el grupo mixto fue alimentado con dos tipos de probióticos. El metabolismo del nitrógeno y los niveles de ARNm de mTOR y S6K en el músculo esquelético se observaron en cada grupo. Luego, se utilizó metagenoma y metabonómica no dirigida para observar los cambios de microorganismos intestinales y metabolitos plasmáticos en los canales portales después de la alimentación con probióticos. Finalmente, combinamos los resultados del análisis ómico para revelar el mecanismo de los probióticos en el metabolismo del nitrógeno en lechones destetados. Los resultados mostraron que L. Rhmnosus GG ATCC53103 y L. Plantarum JL01 aumentaron la digestibilidad aparente del nitrógeno, la tasa de deposición de nitrógeno y la tasa de utilización de nitrógeno de los lechones destetados (P < 0,05); la expresión relativa de mTOR y ARNm SK6 en el músculo esquelético aumentó significativamente (P < 0,05). Cuando se combinaron L. rhamnosus GG ATCC53103 y L. plantarum JL01, encontramos que Clostridium y Prevotella aumentaron significativamente en el yeyuno (P < 0,05). La abundancia relativa de Lactobacillus, Ruminococcus, Streptococcus y Prevotella en el íleon aumentó significativamente (P < 0,05). En comparación con el grupo control, L-triptófano, 3-fosfoniloxipiruvato, cis-aconitato y fosfato de carbamoil aumentaron significativamente en la vena porta del grupo mixto. El resultado del análisis combinatorio mostró que los microorganismos significativamente aumentados podrían codificar los genes de la enzima para la síntesis de L-triptófano, 3-fosfonooxipiruvato, cis-aconitato y fosfato de carbamoil. En resumen, nuestros resultados demostraron que L. rhamnosus GG ATCC53103 y L. plantarum JL01 podrían estimular la expresión de genes de síntesis de proteínas del músculo esquelético de lechones destetados modulando la estructura de la microbiota intestinal y sus metabolitos, mejorando así el metabolismo del nitrógeno en lechones destetados.
1. Introducción
La comunidad microbiana gastrointestinal es diversa y vibrante y desempeña un papel crucial en la salud y la nutrición del huésped (Roager y Licht, 2018). En estudios anteriores, el metabolismo y la utilización del nitrógeno generalmente se consideraron el papel de las enzimas digestivas en el tracto digestivo. Sin embargo, los microbios intestinales son ricos en genes funcionales que codifican el metabolismo de los aminoácidos y la síntesis de proteínas. Prevotella ruminicola, Butyrivibrio fibrisolvens, Mitsuokella multiacidas y Streptococcus bovis pueden secretar dipeptidil peptidasa y dipeptidasa altamente activas para la digestión y absorción de proteínas. Por lo tanto, los microbios intestinales desempeñan un papel vital en el metabolismo de los aminoácidos (Krajmalnik-Brown et al., 2012; Zhao et al., 2019). Se ha informado que los microbios del intestino delgado pueden usar varios nutrientes en la dieta, proteínas secretadas endógenas y urea circulante en el intestino para sintetizar aminoácidos (Bergen y Wu, 2009). Un estudio mostró que los microbios en el intestino delgado pueden metabolizar ampliamente varios aminoácidos, con una tasa metabólica de más del 50% (Yang et al., 2014). El método de etiquetado de isótopos puede confirmar mejor esta conclusión. En un informe sobre la lisina, la lisina fue sintetizada por microorganismos intestinales y utilizada por el cuerpo (Metges, 2000). Aunque la tasa de contribución puede ser pequeña, todavía no es despreciable en el caso de una ingesta limitada (Matthews, 2020). Además, varios aminoácidos sintetizados por microorganismos intestinales pueden promover el metabolismo muscular como factores reguladores (Lin et al., 2017). Como el triptófano estimula la vía mTOR en las células musculares, promoviendo la expresión génica involucrada en la síntesis de miofibrillas (Dukes et al., 2015).
Se ha informado que los microorganismos intestinales utilizan proteínas para producir una gran cantidad de metabolitos microbianos, algunos de los cuales son productos intermedios y otros son productos finales (Bishu, 2016). Algunos de estos metabolitos bacterianos son absorbidos por las células epiteliales intestinales y tienen un efecto positivo. Los metabolitos bacterianos no absorbidos se transportan a la vena porta y ejercen efectos fisiológicos sobre el hígado y los órganos periféricos (Dávila et al., 2013). Por ejemplo, el triptófano es metabolizado por microorganismos para producir indol y sus derivados (Roager y Licht, 2018), que pueden inducir a las células endocrinas intestinales a secretar péptido similar al glucagón 1, inhibir la secreción gástrica y el peristaltismo, y promover la saciedad (Jardon et al., 2022). La microbiota intestinal metaboliza y produce muchas de las vitaminas que el cuerpo necesita, especialmente ácido fólico y vitamina B12 (LeBlanc et al., 2013), que puede mejorar el anabolismo muscular (Kuo et al., 2007). Por lo tanto, existe una estrecha relación entre los microbios intestinales y el metabolismo del nitrógeno.
El estrés al destete interrumpe el equilibrio microbiano intestinal y reduce la absorción de mamíferos para la síntesis de aminoácidos y proteínas (Wang et al., 2019; Yi et al., 2019). Se ha demostrado que los probióticos equilibran los microbios intestinales y afectan la absorción de aminoácidos del cuerpo (Utzschneider et al., 2016; Geng et al., 2021). Además, los probióticos tienen efectos beneficiosos sobre el rendimiento del crecimiento y el metabolismo del nitrógeno durante el destete en mamíferos (Khanian et al., 2019; Wang y Kim, 2021). Nuestro estudio anterior mostró que L. rhamnosus GG ATCC53103 y L. plantarum JL01 mejoraron el rendimiento de crecimiento y la función inmune intestinal de cerdos destetados (Geng et al., 2021). Sin embargo, los efectos de L. rhamnosus GG ATCC53103 y L. plantarum JL01 sobre el metabolismo del nitrógeno de cerdos destetados aún no estaban claros. Por lo tanto, el presente estudio exploró los efectos de la alimentación de L. rhamnosus GG ATCC53103 y L. plantarum JL01 sobre el metabolismo del nitrógeno y los microbios intestinales en lechones destetados y utilizó la metagenómica para analizar el papel de la flora dominante. Finalmente, la metagenómica y la metabolómica están vinculadas para explorar el mecanismo de su impacto en el metabolismo del nitrógeno.
2. Materiales y métodos
2.1. Declaración ética
Todos los procedimientos experimentales con animales fueron aprobados por el Comité de Ética de la Universidad Agrícola de Jilin (Changchun, Jilin, China).
2.2. Animales y diseño experimental
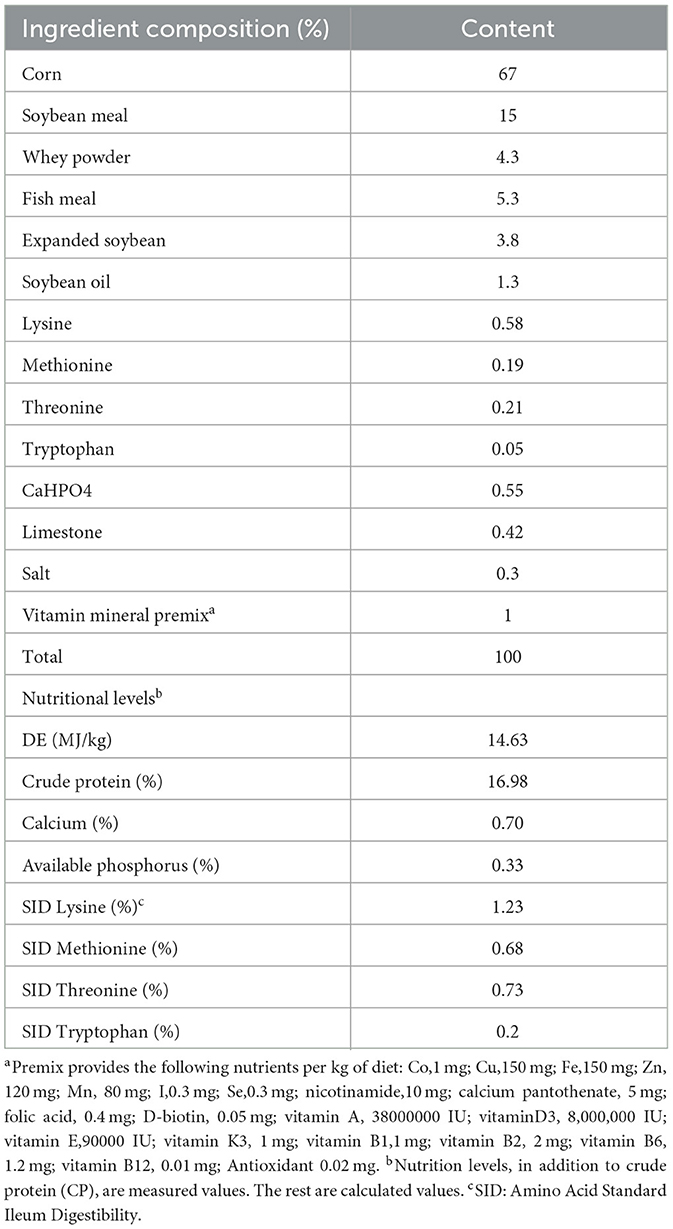
Cuarenta y ocho lechones destetados «Yorkshire-Landrace-Duroc» de 35 días de edad (promedio 11.59 kg) se dividieron aleatoriamente en cuatro grupos de dieta (n = 4 cada uno, i = 3 cada uno). Cada tres lechones se mantienen en una jaula. Después de un período de aclimatación de 7 días, se inició un experimento animal de 28 días. Los probióticos se cultivaron en medio MRS a 37°C durante 18h. El grupo de control fue alimentado con la dieta basal del Consejo Nacional de Investigación (2012) requerimientos nutricionales, y cada lechón fue alimentado con 10 mL de MRS sin bacterias por día. El grupo experimental fue alimentado con la dieta basal con MRS que contenía probióticos, y la dosis de probióticos por cerdo por día se incluyó 10 mL que contenían L. plantarum JL01 (1 × 109 Grupo CFU/mL Lac), L. rhamnosus GG ATCC53103 (1 × 109 UFC/ml, grupo Rha) o ambos probióticos (0,5 × 109 UFC/ml L. plantarum JL01 y 0,5 × 109 UFC/ml L. rhamnosus GG ATCC53103, grupo mixto). Los probióticos o MRS se mezclaron en una pequeña cantidad de la dieta básica y luego se alimentaron a los lechones por separado con una cuchara para garantizar que todos los lechones destetados comieran la dosis completa. Todos los lechones pueden obtener agua y alimento gratis. La composición de la dieta se muestra en la Tabla 1.
2.3. Recogida de muestras
El día 23 del período de cría. Los experimentos metabólicos se realizaron durante 5 días. Utilizando el método de recolección de heces y orina, seleccionamos al azar tres lechones de cada grupo para la prueba de metabolismo del nitrógeno, el peso fecal y el volumen de orina se midieron utilizando un dispositivo de recolección separado para heces y orina en jaulas metabólicas, y las heces y la orina se recolectaron a las 8:00 y 16:00 todos los días (Feyera et al., 2019). Las muestras de orina se agregaron con ácido sulfúrico al 10% para la fijación de nitrógeno y se almacenaron a -80 ° C para la prueba. Mezclamos las muestras fecales recolectadas todos los días, las guardamos en bolsas con cierre hermético, registramos el peso, sacamos 200 g de muestras fecales, agregamos rápidamente ácido sulfúrico al 10% para fijar el nitrógeno, las horneamos a 60 ° C a peso constante, luego pulverizamos y almacenamos en un refrigerador de -80 ° C para su análisis. En la mañana del día 28, tres lechones destetados fueron seleccionados al azar de cada grupo para recolectar muestras intestinales, y se utilizó una solución de barbital sódico al 3% como anestesia para lechones destetados. El yeyuno y el íleon se diseccionaron, y el contenido del yeyuno y el íleon se exprimieron cuidadosamente, y luego, el quimo se recolectó en viales criogénicos estériles. Posteriormente, se recogieron 10 ml de sangre de la vena porta hepática y se colocaron en un tubo de recolección de sangre que contenía heparina sódica. Después de la centrifugación, las capas de plasma se extrajeron y distribuyeron en tubos de criopreservación de 1,5 ml. Después, el músculo esquelético (glúteo mayor) de los lechones se recolectó rápidamente con un tubo EP estéril y se congeló rápidamente en nitrógeno líquido. Las muestras se almacenaron en un refrigerador de -80 ° C.
2.4. Experimento sobre el metabolismo del nitrógeno
El contenido de nitrógeno en piensos y heces se midió utilizando un aparato de determinación de nitrógeno (FOSS Kjeltec 8420 Kjeldahl + aparato de determinación de nitrógeno, FOSS [Technology and Trade Co. Ltd., Beijing, China]). Las fórmulas utilizadas fueron las siguientes: emisión de nitrógeno (g/d) = nitrógeno fecal (FN) (g/d) + nitrógeno en orina (UN) (g/d), digestibilidad aparente del nitrógeno (%) = [(ingesta de nitrógeno (NI) – FN)/NI] × 100%, tasa de deposición de nitrógeno (%) = [(NI-FN-UN)/NI] × 100%, y tasa de utilización de nitrógeno (%) = [(NI-FN-UN) / (NI-FN)] × 100%.
2.5. PCR cuantitativa en tiempo real
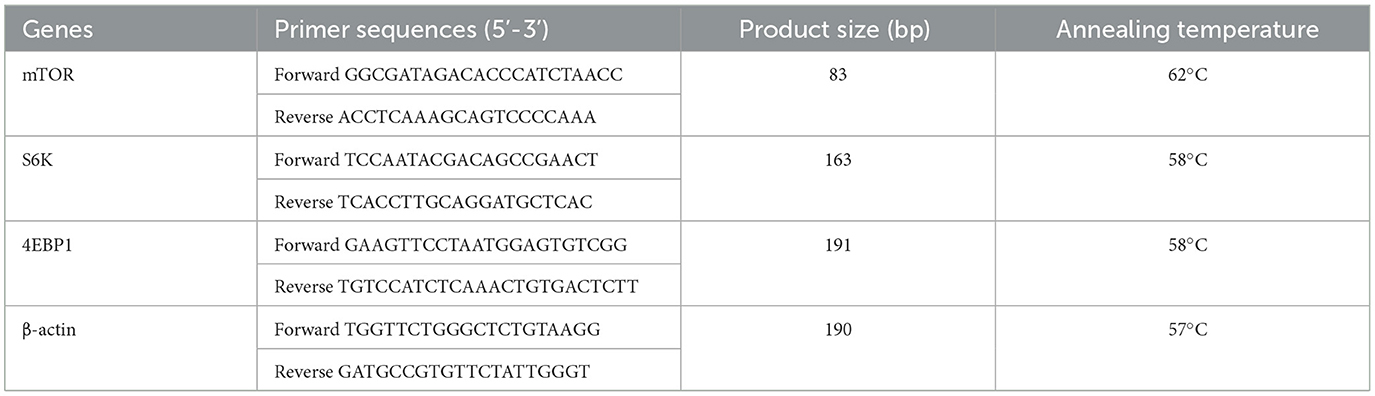
El ARN total del tejido muscular se extrajo utilizando TRIzol. Los pasos para la transcripción inversa de ARN y RT-qPCR fueron los mismos que antes (Ding et al., 2020). Se utilizó el kit de reactivos PrimeScript™ RT y SYBR Premix Ex T aq™ II para los procesos de transcripción inversa y RT-qPCR, respectivamente. El análisis de RT-qPCR se realizó utilizando el sistema qPCR Agilent Stratagene Mx3005P (Agilent Technologies, Inc.). Estos ensayos de RT-qPCR se realizaron por triplicado para cada muestra, y los resultados se normalizaron a la expresión de β-actina. La expresión se analizó mediante el método de comparación 2-ΔΔCt. Las secuencias de cebadores se proporcionan en la Tabla 2.®
2.6. Extracción de ADN y secuenciación del ARNr 16S
El ADN microbiano del quimo intestinal se extrajo utilizando el kit de ADN en heces TIANamp (Tiangen, Beijing, China).
Las secuencias de genes 16S rDNA se analizaron utilizando la plataforma de secuenciación Illumina HiSeq™ 2500. Los datos sin procesar se filtraron utilizando el software FASTP. Todas las muestras se agruparon y la similitud efectiva de la secuencia ≥ del 97% se agrupó en unidades de clasificación de operaciones (OTU). La curva de dispersión de OTU y la curva de abundancia de rango se trazaron en QIIME. Se realizó un análisis de Venn intergrupal en R (versión 3.4.1) para identificar otus únicos y comunes. Usamos QIIME para calcular Chao1, Simpson, etc. α Índice de Diversidad.
2.7. Preparación de la muestra para el análisis LC-MS
Las muestras de plasma de la vena porta que pesaban 50 mg se midieron cuidadosamente y se transfirieron a un tubo de EP. Posteriormente, el tubo EP se llenó con el disolvente (acetileno-metano-agua, 2: 2: 1, incluidos los estándares internos). Después de girar durante 30 segundos a una velocidad promedio de 45 Hz, la muestra se sometió a un baño de agua helada de 4 minutos. Luego, fue ultrasonido durante 5 minutos. El ultrasonido promedio se repite tres veces y luego se almacena a -20 ° C durante 1 h y durante 15 minutos durante 12, 000 RPM y 4 ° C. El Shangqing obtenido se transfirió a la botella LC-MC, se analizó en UHPLC-QE o orBITRAP MS, y se mezcló con el equivalente de todas las muestras para eliminar la muestra de control de calidad (QC).
2.8. Análisis funcional
Se utilizó la prueba T como análisis variable para cribar los diferentes metabolitos, y el umbral VIP se estableció en 1, y se seleccionaron las diferencias entre los dos grupos de metabolitos de un valor de p de < 0,05 y un VIP de ≥ 1. Los metabolitos se enriquecieron en la vía metabólica KEGG para el análisis de enriquecimiento. En comparación con todo el fondo, el análisis de enriquecimiento determinó el enriquecimiento significativo de metabolitos o canales de transición de señal en la diferencia.
2.9. Secuenciación
Utilizamos la biblioteca de ADN Nextera XT para preparar la biblioteca para el grupo macrobase y secuenciar la plataforma Illumina Hiseq. También utilizamos Metaphlan (V.2.2) (Truong et al., 2015) y Humann2 para analizar datos de grupos macroscópicos. Luego, usamos el Unigene para consultar la base de datos del comité de enzimas (EC) a través del software Diamond. La base de datos RefSeq del NCBI se utilizó para construir un índice microbiano con diferencias significativas en cerdos anotados por Metaothello.
2.10. Análisis estadístico
Los resultados se expresaron como la media ± DE. El análisis estadístico se realizó utilizando el software Prism (GraphPad Software, San Diego, CA) para realizar un ANOVA unidireccional seguido de un análisis post-hoc de Bonferroni. Los valores de p < 0,05 fueron considerados significativos. Se dibujaron diferentes funciones microbianas, genéticas, metabolitos y canales metabólicos a través del software Adobe Illustrator (EE.
3. Resultados
3.1. Metabolismo del nitrógeno
Los efectos de L. plantarum JL01 y L. rhamnosus GG ATCC53103 sobre el metabolismo nitrogenado de lechones destetados se muestran en la Tabla 3. En comparación con el grupo Con, el nitrógeno ingerido de los lechones en el grupo Rha, el grupo Lac y el grupo mix aumentó significativamente (P < 0,05), el nitrógeno fecal, el nitrógeno urinario y las emisiones totales de nitrógeno mostraron una tendencia decreciente, la digestibilidad aparente del nitrógeno de los grupos Rha y Lac tendió a aumentar; la digestibilidad aparente del nitrógeno del grupo de mezcla aumentó en un 1,88% (P < 0,05), y la tasa de deposición de nitrógeno aumentó respectivamente en 4,17, 4,49 y 5,65% (P < 0,05), y la tasa de utilización de nitrógeno aumentó en 2,82, 3,23 y 3,70%, respectivamente (P < 0,05).
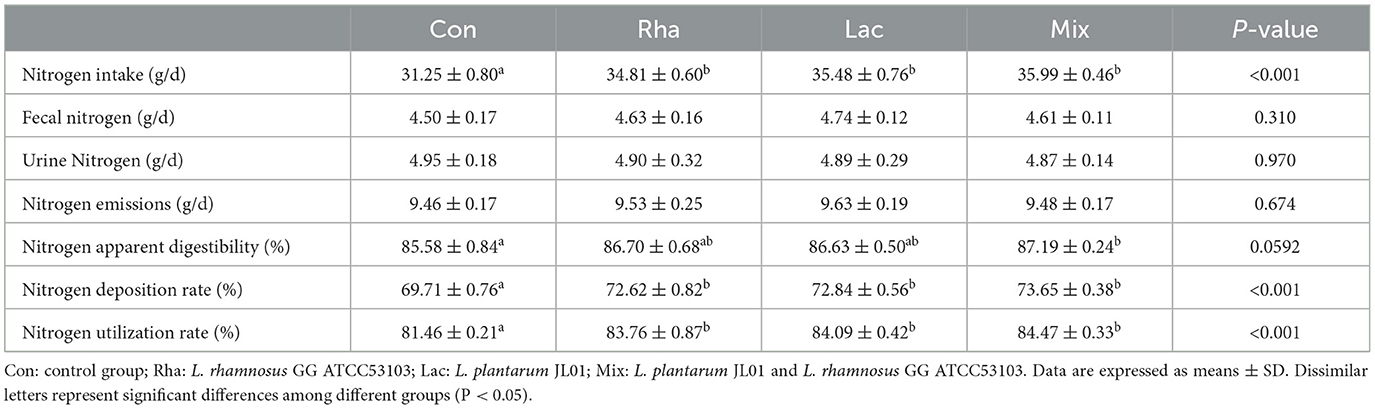 Tabla 3. Efectos de L. plantarum JL01, L. rhamnosus GG ATCC53103 sobre el metabolismo del nitrógeno de lechones destetados.
Tabla 3. Efectos de L. plantarum JL01, L. rhamnosus GG ATCC53103 sobre el metabolismo del nitrógeno de lechones destetados.
3.2. Expresión génica del depósito de proteínas del músculo esquelético
Como se muestra en la Figura 1, la expresión relativa de mTOR y ARNm S6K en el músculo esquelético de lechones en los grupos Rha, Lac y mix aumentó significativamente en comparación con el grupo Con (P < 0,05).
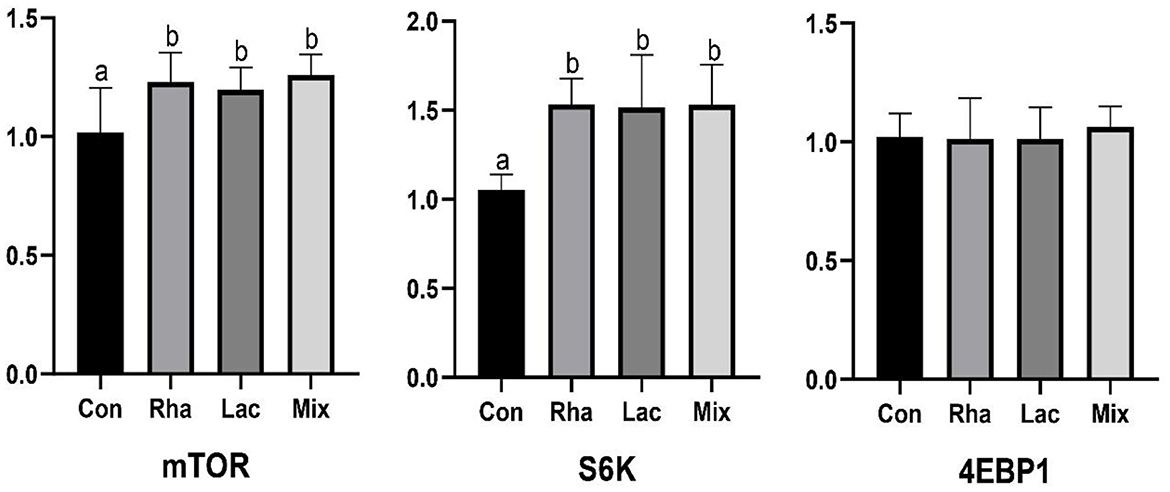 Figura 1. La expresión relativa de mTOR, S6K, 4EBP1 mRNA del músculo dorsal más largo de lechones destetados. Contra: grupo de control; Rha: L.rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (P < 0,05).
Figura 1. La expresión relativa de mTOR, S6K, 4EBP1 mRNA del músculo dorsal más largo de lechones destetados. Contra: grupo de control; Rha: L.rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (P < 0,05).
3.3. Datos adquiridos a partir de secuenciación de alto rendimiento y mediciones de diversidad alfa
En este experimento se obtuvieron un total de 2, 800, 880 secuencias, y se filtraron las secuencias originales, y finalmente, se obtuvieron 2, 591, 938 secuencias válidas, con un promedio de 128, 160 secuencias válidas por muestra para su posterior análisis. La diversidad microbiana es la diversidad en un hábitat o ecosistema particular y utiliza dos parámetros, riqueza de especies y uniformidad, para reflejar la diversidad y abundancia de microorganismos (Grice et al., 2009). Se utilizaron los índices Chao1 y ACE para estimar la información de riqueza de especies de las muestras, y Simpson y Shannon se utilizaron para estimar la información sobre la diversidad de especies (Segata et al., 2011). Los efectos de L. plantarum JL01 y L. rhamnosus GG ATCC53103 sobre la diversidad microbiana del intestino delgado de lechones destetados se muestran en la Figura 2. En comparación con el grupo Con, los índices de Shannon y Simpson de los grupos Rha, Lac y mixtos mostraron una tendencia creciente en el yeyuno, pero fueron insignificantes. En el íleon, los índices ACE, Chao1, Shannon y Simpson de lechones en el grupo mixto aumentaron significativamente en comparación con el grupo Con (P < 0,05), mientras que la uniformidad y abundancia de otros contenidos intestinales no fueron significativas.
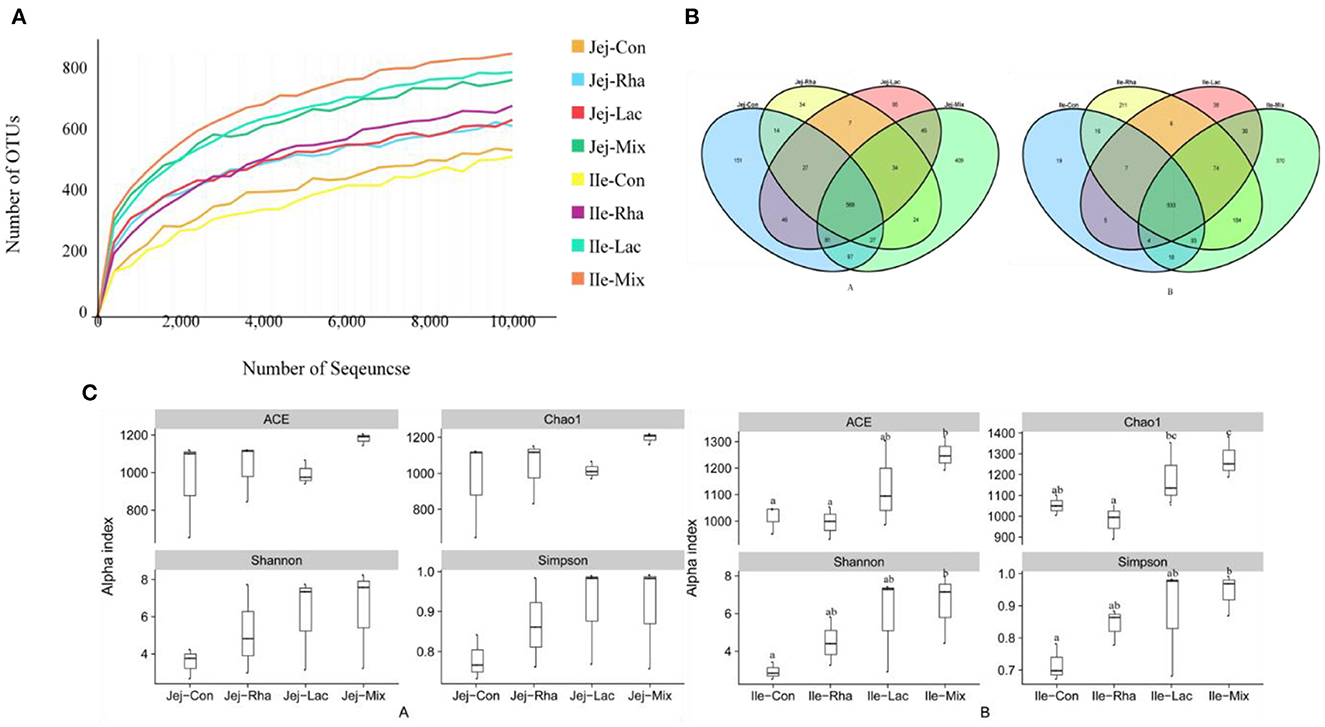 Figura 2. (A) Curvas de rarefacción de OTU de microbiota en yeyuno e íleon de lechones destetados. (A) Análisis comparativo OTU de la microbiota intestinal de lechones destetados (A) yeyuno (B) íleon. (C) Patrón de caja del índice de diversidad microbiana alfa de lechones destetados (A) yeyuno (B) íleon; Contra: grupo de control; Rha: L. rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (P < 0,05).
Figura 2. (A) Curvas de rarefacción de OTU de microbiota en yeyuno e íleon de lechones destetados. (A) Análisis comparativo OTU de la microbiota intestinal de lechones destetados (A) yeyuno (B) íleon. (C) Patrón de caja del índice de diversidad microbiana alfa de lechones destetados (A) yeyuno (B) íleon; Contra: grupo de control; Rha: L. rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (P < 0,05).
3.4. Cambios en la microbiota intestinal
Se analizó la composición microbiana del contenido intestinal de los lechones destetados. La Figura 3A muestra los 10 filos más abundantes entre todos los filos. Firmicutes, Proteobacteria y Bacteroidetes son los filos dominantes comunes en cada grupo de tratamiento, y sus abundancias relativas juntas explican las abundancias bacterianas en cada grupo de tratamiento. Representaron más del 93% del total de bacterias. Como se muestra en la Figura 3B, en comparación con el grupo Con, en el yeyuno, los Firmicutes en el grupo mixto disminuyeron significativamente (P < 0,05). La abundancia relativa de Bacteroidetes aumentó significativamente (P < 0,05), y la abundancia relativa de Proteus disminuyó significativamente (P < 0,05) en los grupos Lac y mixto. Firmicutes/Bacteroidetes aumentaron significativamente en lechones en los grupos Lac y mixtos (P < 0,05). En el íleon, la abundancia relativa de Firmicutes en los grupos Rha y mix fue significativamente menor que en el grupo Con (P < 0,05). La abundancia relativa de Bacteroidetes aumentó significativamente en Rha, Lac y grupos mixtos (P < 0,05). Firmicutes/Bacteroidetes aumentaron significativamente en Rha, Lac y grupos mixtos (P < 0,05).
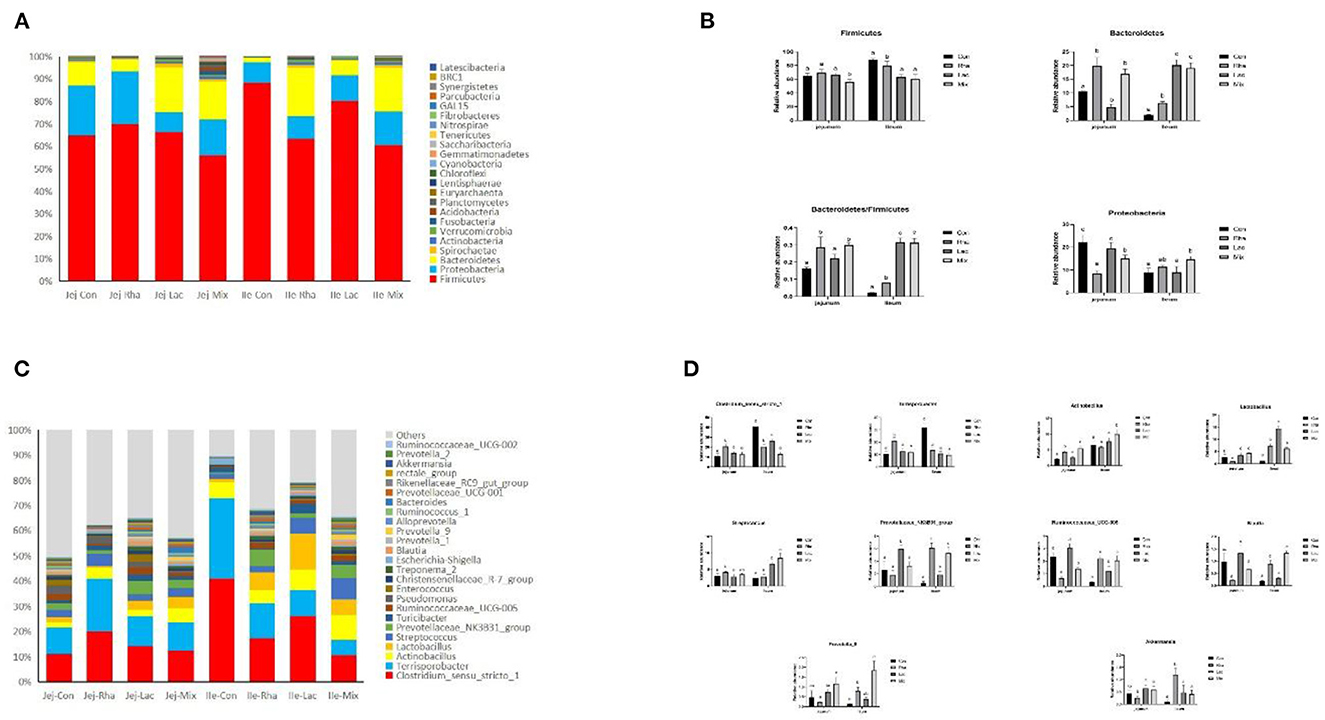 Figura 3. (A) Se presentan los 10 filos más abundantes. (B) Diferencias de composición bacteriana en el intestino delgado a nivel del filo. Contra: grupo de control; Rha: L. rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (p < 0,05). (C) Abundancias relativas de los diez géneros principales. (D) Diferencias de composición bacteriana en el intestino delgado a nivel de género. Contra: grupo de control; Rha: L. rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (P < 0,05).
Figura 3. (A) Se presentan los 10 filos más abundantes. (B) Diferencias de composición bacteriana en el intestino delgado a nivel del filo. Contra: grupo de control; Rha: L. rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (p < 0,05). (C) Abundancias relativas de los diez géneros principales. (D) Diferencias de composición bacteriana en el intestino delgado a nivel de género. Contra: grupo de control; Rha: L. rhamnosus GG ATCC53103; Lac: L. plantarum JL01; Mezcla: L. plantarum JL01 y L. rhamnosus GG ATCC53103. Los datos se expresan como medias ± DE. Las letras disímiles representan diferencias significativas entre los diferentes grupos (P < 0,05).
A nivel de género, se identificaron 181 géneros de todas las muestras. Las abundancias relativas de los 25 géneros principales se muestran en la Figura 3C. Como se muestra en la Figura 2D, en comparación con el grupo control, la abundancia relativa de Lactobacillus, Actinobacillus y Prevotella_9 en el grupo mixto aumentó significativamente (P < 0,05), y la abundancia relativa de Ruminococcaceae_UCG-005 disminuyó significativamente (P < 0,05). Las abundancias relativas de Ruminococcaceae_UCG-005, Actinobacillus, Streptococcus, Blautia y Prevotella_9 aumentaron significativamente (P < 0,05), mientras que las abundancias relativas de Clostridium_sensu_stricto_1 y Terrisporobacter disminuyeron significativamente (P < 0,05). Se encontró que el grupo Rha en el yeyuno, incluidos Clostridium_sensu_stricto_1, Terrisporobacter y Actinobacillus, era significativamente diferente. La abundancia relativa de Lactobacillus, Ruminococcaceae_UCG-005 y Streptococcus disminuyó significativamente (P < 0,05). Por el contrario, en el íleon, la abundancia relativa del grupo Rha, Prevotellaceae_NK3B31_group, Lactobacillus, Ruminococcaceae_UCG-005 y Akkermansia, aumentó significativamente (P < 0,05). Además, la abundancia relativa de Clostridium_sensu_stricto_1 y Terrisporobacter disminuyó significativamente (P < 0,05). La abundancia relativa del grupo Lac, incluyendo Prevotellaceae_NK3B31_group, Lactobacillus, Ruminococcaceae_UCG-005, Prevotella_9, mostró un aumento significativo (P < 0,05). Del mismo modo, en el íleon, la abundancia relativa del grupo Lac, que comprende Prevotellaceae_NK3B31_group, Lactobacillus, Ruminococcaceae_UCG-005 y Streptococcus, también aumentó significativamente (P < 0,05). En contraste, la abundancia relativa de Clostridium_sensu_stricto_1 y Terrisporobacter exhibió una disminución significativa (P < 0,05).
3.5. Mapa de redes de genes microbianos y funcionales diferenciados del intestino delgado
Este experimento solo se centra en los microorganismos diferenciados en el intestino delgado y los genes funcionales que codifican el metabolismo de aminoácidos y la síntesis de proteínas. Como se muestra en la Figura 4, Clostridium_ sensu_ stricto_ 1 y ruminocaceae_ Ucg-005 tienen un gen que codifica la d-3-fosfoglicerato deshidrogenasa; Actinobacillus, Clostridium, Lactobacillus y Streptococcus tienen genes que codifican carbamato quinasa y carbamoil fosfato sintasa; Akkermansia y Ruminococcus tienen genes que codifican carbamoil fosfato sintasa; Actinomycetes, Clostridium, Prevotellaceae_ NK3B31_ Group, Prevotella_ 9, Rumencocci y Streptococcus tienen genes que codifican la aconitato hidratasa; Actinomycetes, Klebsiella, Streptococcus, Rumencocci, Prevotellaceae_ NK3B31_ Group, Prevotella_ 9 y Clostridium tiene un gen que codifica la triptófano sintasa.
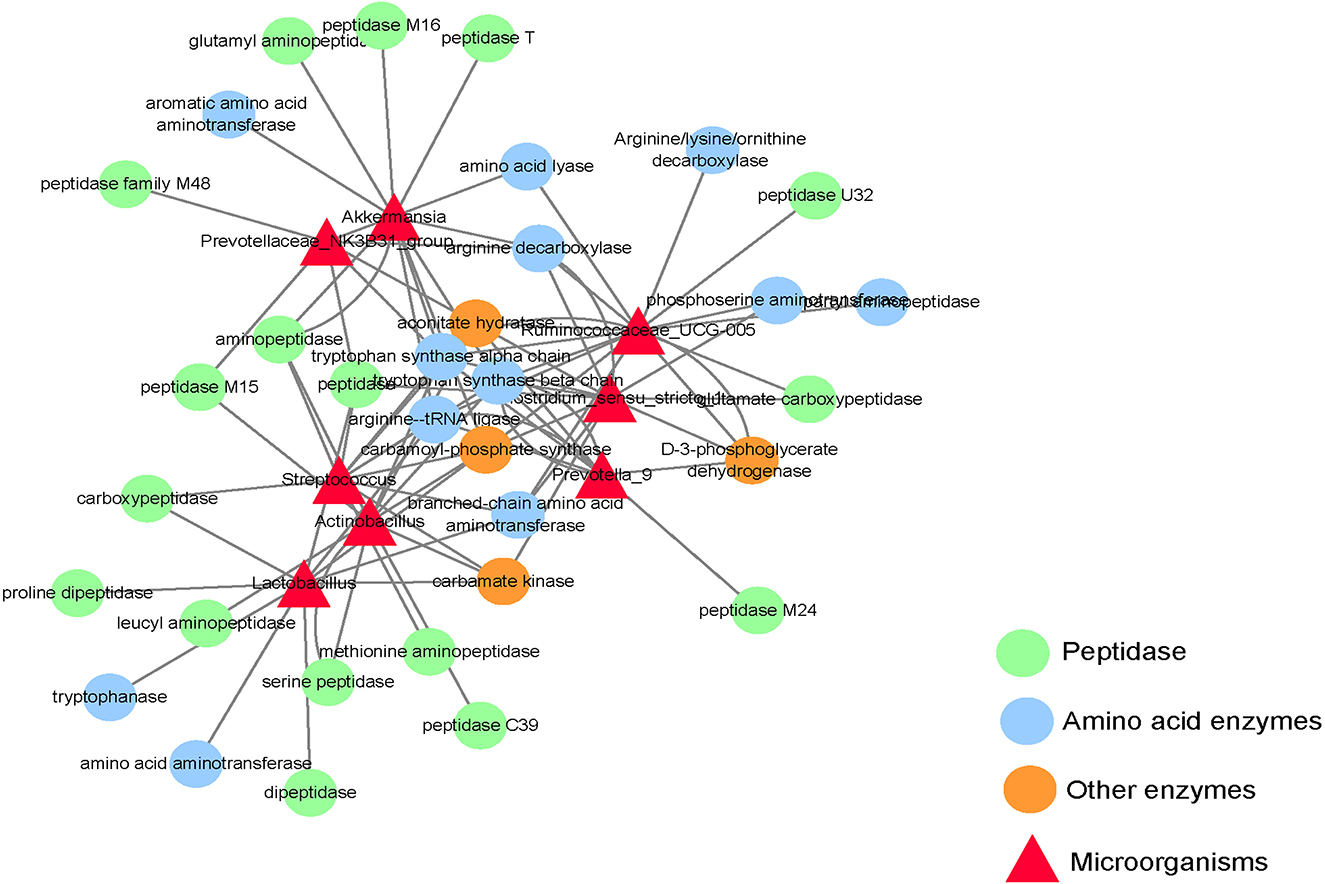 Figura 4. Diagrama de red de microbios diferenciales y genes funcionales en el intestino delgado de los lechones destetados.
Figura 4. Diagrama de red de microbios diferenciales y genes funcionales en el intestino delgado de los lechones destetados.
3.6. Perfil metabolómico de la vena porta
En esta prueba, no se detectaron picos significativos en la muestra en blanco, lo que indica que no hubo contaminación cruzada entre las muestras en esta prueba. La identificación de sustancias se llevó a cabo utilizando un paquete de programa R escrito por ellos mismos y una base de datos de espectrometría de masas secundaria autoconstruida. La espectrometría de masas de segundo orden coincide con el grado de corte = 0,2. Un total de 11.875 metabolitos se obtuvieron de los resultados de detección de este estudio, y 617 sustancias fueron identificadas por el espectro secundario.
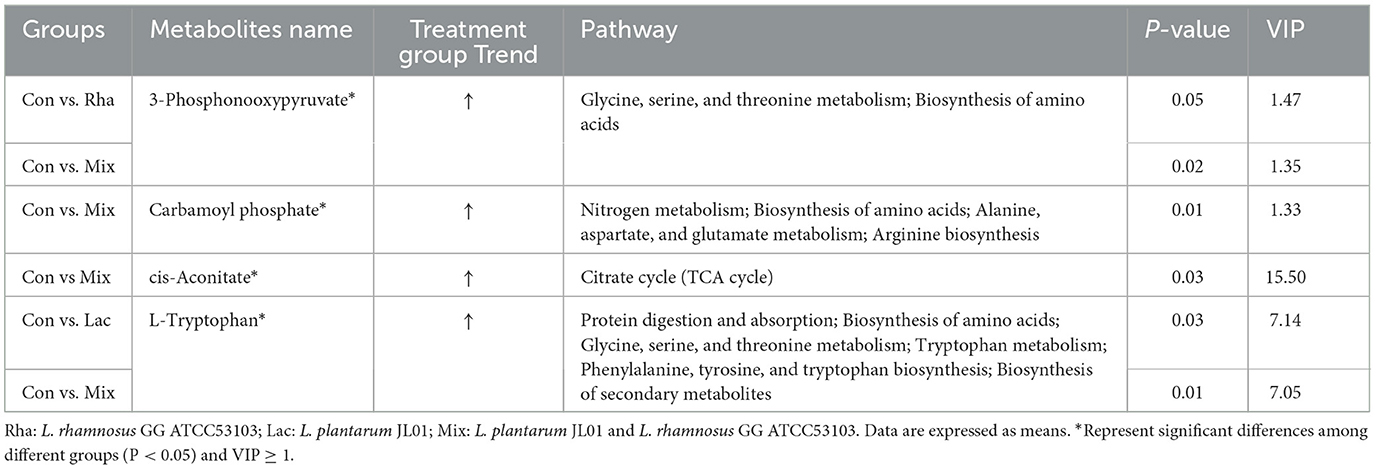
Como se muestra en la Figura 5A, cada grupo de tratamiento tuvo una separación aparente en comparación con el grupo control, lo que indica que la alimentación de L. rhamnosus y L. plantarum tuvo un efecto significativo sobre los metabolitos plasmáticos de la vena porta hepática de los lechones destetados. En este estudio, los metabolitos diferenciales relacionados con el metabolismo del nitrógeno fueron seleccionados para el análisis. Como se muestra en la Tabla 4, en los metabolitos de la vena porta, en comparación con Con, los metabolitos diferenciales producidos por el grupo Rha fueron 3-Fosfonooxipiruvato (P < 0,05), los metabolitos diferenciales producidos por el grupo Lac fueron L-Triptófano (P < 0,05), y los metabolitos diferenciales producidos por el grupo de mezcla fueron L-triptófano, 3-Fosfonooxipiruvato, cis-Aconitato y Carbamoil fosfato (P < 0,05). Como se muestra en las Figuras 4A-D, después de enriquecer los metabolitos, se seleccionaron las primeras 20 rutas con el valor q de enriquecimiento más bajo para dibujar el mapa. Este experimento solo se centró en la vía metabólica de los nutrientes del nitrógeno. Los resultados mostraron que el metabolito diferencial 3-fosfodesoxipiruvato enriquecido en la vena porta de lechones destetados producido por la alimentación de las bacterias mixtas de L. rhamnosus y L. plantarum fue la vía del metabolismo de la glicina, la serina y la treonina; la vía metabólica enriquecida por el metabolito diferencial L-triptófano es la vía metabólica del triptófano; la vía metabólica enriquecida por el metabolito diferencial cis-aconitato es la vía del ciclo del citrato (ciclo TCA); La vía metabólica para el metabolito diferencial fosfato carbamoil es la vía de biosíntesis de arginina.
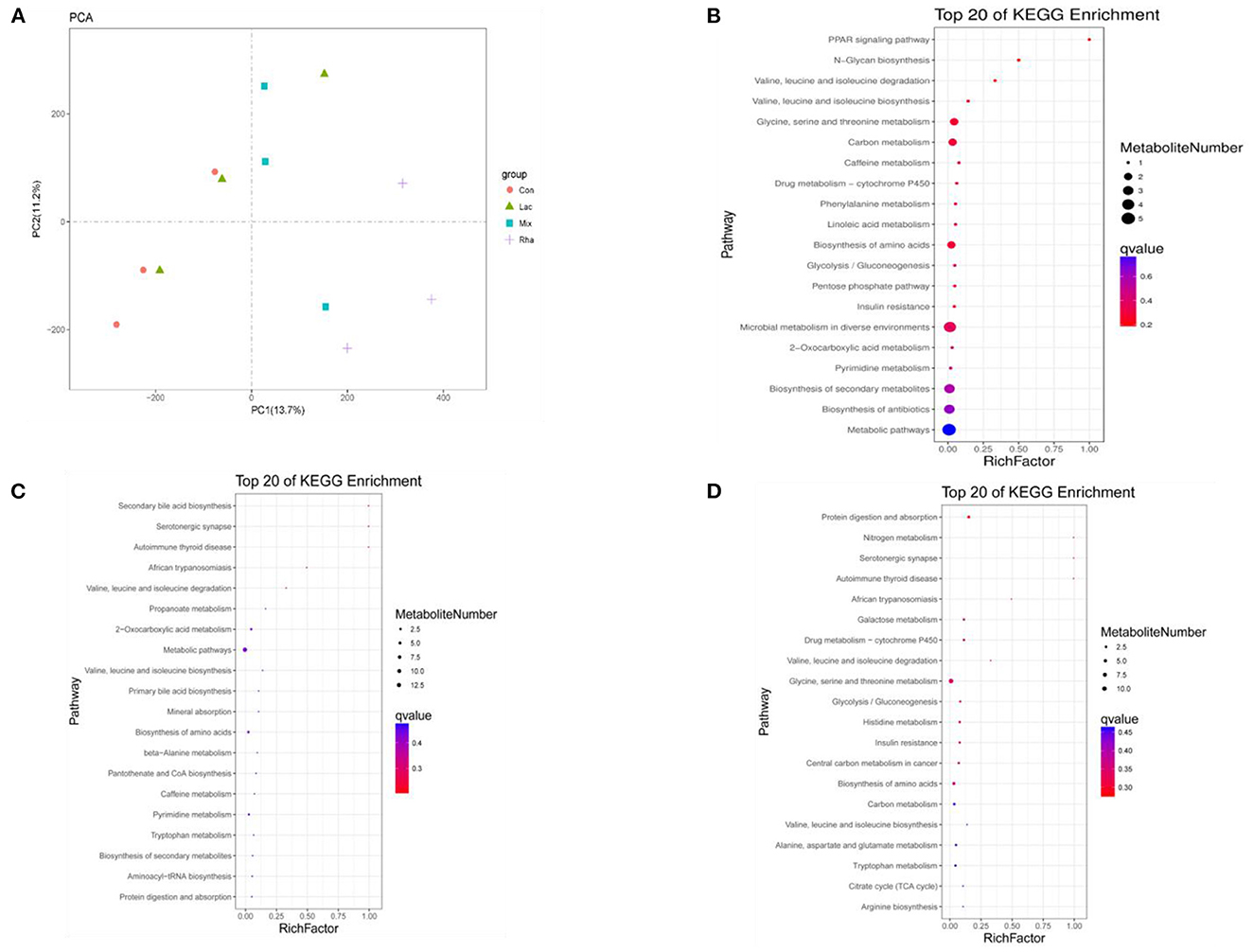 Figura 5. (A) Mapa de puntuación PCA de metabolitos en plasma de la vena porta hepática. Distribución de la vía KEGG enriquecida de metabolitos diferenciales en plasma de la vena porta hepática (B) Con vs. Rha (C) Con vs. Lac (D) Con vs. Mix.
Figura 5. (A) Mapa de puntuación PCA de metabolitos en plasma de la vena porta hepática. Distribución de la vía KEGG enriquecida de metabolitos diferenciales en plasma de la vena porta hepática (B) Con vs. Rha (C) Con vs. Lac (D) Con vs. Mix.
3.7. Relación entre el microbioma intestinal y la metabolómica no dirigida
Para estudiar más a fondo la relación entre la microflora intestinal y los metabolitos, visualizamos la correlación entre la microflora y los metabolitos del yeyuno y el íleon y llevamos a cabo un análisis de correlación de Spearman sobre la abundancia relativa de diferentes especies microbianas y metabolitos. Como se muestra en la Figura 6A, en el yeyuno, L-triptófano y cis-aconitato se correlacionaron positivamente con Streptococcus, Lactobacillus y Actinobacillus y se correlacionaron negativamente con Akkermansia. El fosfato de carbamoil se correlacionó positivamente con Akkermansia, Lactobacillus y Actinobacillus y se correlacionó negativamente con Streptococcus. El 3-fosfonoxipiruvato se correlacionó positivamente con Lactobacillus y Actinobacillus y se correlacionó negativamente con Akkermansia y Streptococcus. Como se muestra en la Figura 6B, en el íleon, encontramos 3-Phosphonooxypyruvato, Carbamoyl phosphate, cis-Aconitate, L-Tryptophan y Actinobacillus, Blautia, Lactobacillus, Streptococcus, Ruminococcaceae_UCG-005, Prevotella_9, Prevotellaceae_NK3B31_group y Akkermansia se correlacionaron positivamente y negativamente con Clostridium_sensu_stricto_1.
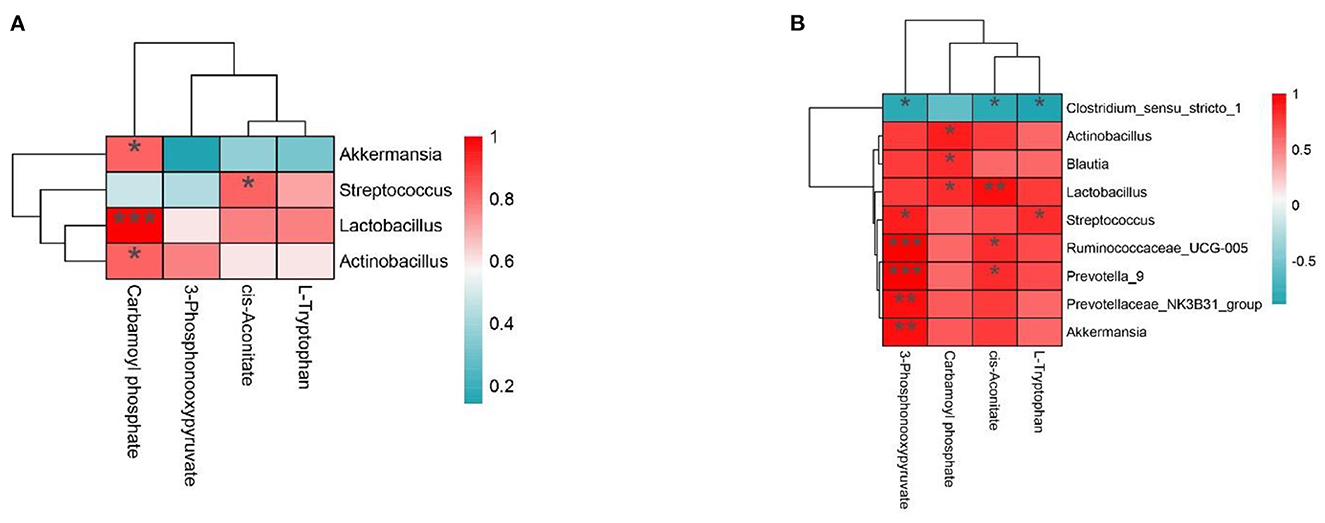 Figura 6. (A, B) La interacción entre la flora intestinal y el metaboloma de la vena porta en el grupo Con vs. grupo Mix. P < 0,05, P < 0,01 y P < 0,001 indican diferencias significativas entre la microbiota y los metabolitos. El azul representa correlaciones positivas; El rojo representa correlaciones negativas.******
Figura 6. (A, B) La interacción entre la flora intestinal y el metaboloma de la vena porta en el grupo Con vs. grupo Mix. P < 0,05, P < 0,01 y P < 0,001 indican diferencias significativas entre la microbiota y los metabolitos. El azul representa correlaciones positivas; El rojo representa correlaciones negativas.******
4. Discusión
Durante la producción porcina, aproximadamente el 46% del nitrógeno se retuvo en el cuerpo del animal, y el 54% restante se excretó principalmente en las heces y la orina. Por lo tanto, mejorar la eficiencia de convertir el alimento de proteína vegetal en proteína animal podría reducir la contaminación causada durante la producción de carne de cerdo y posiblemente aumentar la eficiencia económica (Millet et al., 2018). El intestino delgado es el sitio principal de absorción de proteínas dietéticas. La mayoría de los nutrientes nitrogenados en la dieta se digieren en el intestino delgado y se convierten en aminoácidos o péptidos pequeños; la parte no absorbida se excreta directamente con las heces, y parte de la parte absorbida es utilizada por el cuerpo animal para convertirse en nitrógeno depositado, y la otra parte se pasa a través del ciclo de ornitina para generar nitrógeno ureico y se excreta en la orina (Zhao et al., 2020). Al mismo tiempo, los microbios intestinales también juegan un papel importante. Utilizan fuentes de nitrógeno en el intestino para sintetizar aminoácidos y participar en el metabolismo del nitrógeno (van der Wielen et al., 2017). A través del análisis del metabolismo del nitrógeno, se refleja la situación preliminar de la utilización de nitrógeno en el cuerpo. En el presente estudio, encontramos que la alimentación combinada de L. plantarum JL01 y L. rhamnosus GG ATCC53103 tuvo el mejor efecto, aumentando la digestibilidad aparente del nitrógeno en un 1,31%, la tasa de deposición de nitrógeno en un 5,65% y la tasa de utilización de nitrógeno en un 3,70% en lechones destetados y fue consistente con informes anteriores (Zhu et al., 2017).
Los microorganismos intestinales juegan un papel importante en el sistema digestivo de los animales (Sun et al., 2019). Se ha informado que cuanto mayor es la diversidad de microorganismos intestinales, mayor es la estabilidad del microecosistema intestinal del cuerpo, promoviendo así el crecimiento de los animales y la absorción y utilización de nutrientes (Cani y Delzenne, 2009; McCann et al., 2017). Estudios anteriores han demostrado que la suplementación dietética con probióticos mejora la diversidad de la flora intestinal, lo que es consistente con nuestros resultados (Sun et al., 2023). Además, una gran cantidad de aminoácidos en el intestino delgado son transformados, metabolizados y utilizados por microorganismos intestinales, lo que no solo mantiene la composición y cantidad de los propios microorganismos, sino que también produce metabolitos que regulan las funciones fisiológicas del huésped (Nicholson et al., 2012), y también podría utilizar varios sustratos para sintetizar nuevos aminoácidos microbianos (Torrallardona et al., 2003; Metges y Petzke, 2005). En nuestro estudio, encontramos que la administración conjunta de los dos probióticos mejoró significativamente la abundancia de Prevotella, Streptococcus, Actinomyces y Klebsiella en el yeyuno y el íleon quimo aumentó significativamente en lechones. En la actualidad, hay pocos artículos sobre la relación entre los microorganismos del intestino delgado y los aminoácidos, por lo que detectamos los genes funcionales de los microorganismos del intestino delgado a través del macrogenoma para predecir la relación entre los microorganismos del intestino delgado y los aminoácidos. En nuestro experimento, los datos macrogenómicos mostraron que Prevotella, Streptococcus, Actinomyces y Klebsiella secretaron proteinasas y peptidasas alcalinas que hidrolizan proteínas y aminoácidos. Esto juega un papel importante en la digestión de proteínas de mamíferos y la absorción de aminoácidos. Actinomyces, Klebsiella, Streptococcus, Ruminococcus, Prevotella, Lactobacillus y Clostridium tienen genes que codifican varias peptidasas. Clostridium, Lactobacillus, Ruminococcus y Streptococcus tienen genes que codifican aminotransferasas de aminoácidos de cadena ramificada. Además, Actinomyces, Klebsiella, Streptococcus, Ruminococcus, Prevotella, Lactobacillus y Clostridium tienen genes que codifican arginasa. Estudios previos han demostrado que Klebsiella puede sintetizar ácido aspártico, ácido glutámico y triptófano, Streptococcus puede sintetizar leucina, lisina y serina, y Clostridium puede sintetizar serina, prolina y valina, lo que se alinea con nuestros resultados (Hooper et al., 2002; Dai et al., 2011). En resumen, cuando se combinan L. rhamnosus GG ATCC53103 y L. plantarum JL01, puede aumentar el número de bacterias beneficiosas en el intestino del cerdo y ajustar los microorganismos intestinales.
La vena porta hepática es una vena grande que ingresa al hígado desde el portal hepático después de que convergen las venas mesentéricas del tracto digestivo. Está estrechamente relacionado con el metabolismo de proteínas, lípidos, carbohidratos, vitaminas y hormonas (Johnson et al., 2016). En este estudio, el análisis de plasma de la vena porta se realizó en los grupos Con, Rha, Lac y mixto. En comparación con el grupo Con, se detectaron cuatro metabolitos importantes, 3-fosfonooxipiruvato, fosfato de carbamoil, cis-aconitato y L-triptófano, en el grupo mixto. Para detectar la relación entre metabolitos y microflora intestinal, analizamos los genes funcionales de la microflora intestinal a través del macrogenoma para analizar las razones del aumento de metabolitos (Figura 7). Nuestros datos muestran que la D-3-fosfoglicerato deshidrogenasa fue codificada por Clostridium, Prevotella y Ruminococcus, que es una enzima que convierte el 3-fosfoglicerato en fosfopiruvato. Los genes de la carbamato quinasa y carbamoil fosfato sintasa están codificados por Actinomycetes, Clostridium, Lactobacillus y Streptococcus, que pueden convertir el amoníaco en carbamato. La hidratasa del ácido aconítico está codificada por Actinomycetes, Clostridium, Prevotella, Ruminococcus y Streptococcus, que pueden convertir el ácido cítrico y el ácido isocítrico en ácido cis-aconítico. El L-triptófano está codificado por Actinomycetes, Klebsiella, Streptococcus, Ruminococcus, Prevotella y Clostridium, que tienen genes para la triptófano sintasa. En comparación con el grupo Con, estos microorganismos aumentaron significativamente en el grupo mixto. Además, nuestros resultados mostraron que la mayoría de los microorganismos se correlacionaron positivamente con los metabolitos. A través del análisis de enriquecimiento de KEGG, se encontró que el ácido cis-aconítico se enriqueció en el ciclo de TCA y fue un producto intermedio importante del ciclo de TCA. Su aumento puede promover el ciclo de TCA y conducir a un aumento de α-cetoglutarato (AKG). Es bien sabido que AKG juega un papel importante en el metabolismo de proteínas y el transporte transmembrana de aminoácidos (Wernerman y Hammarqvist, 1999; Zhao et al., 2020) encontró que agregar AKG a una dieta baja en proteínas puede mejorar la eficiencia del metabolismo del nitrógeno de los lechones destetados. Además, los niveles de ARNm de S6K, mTOR y 4EBP1 y proteína fosforilada en el músculo fueron más altos. En un estudio in vitro realizado por Cai et al. (2020), se demostró que AKG promueve la síntesis de proteínas en miotubos C2C12 a través de la vía de señalización Akt / mTOR. Nuestros datos mostraron que la expresión relativa de mTOR y S6K mRNA en el músculo esquelético aumentó significativamente. Por lo tanto, el ácido cis-aconítico puede promover la síntesis de proteínas en el músculo esquelético al aumentar el contenido de AKG, mejorando así la eficiencia del metabolismo del nitrógeno. Además, los metabolitos significativamente elevados 3-fosfonooxipiruvato y fosfato de carbamilo en la vena porta también pueden promover la síntesis de proteínas al promover el aumento de AKG causado por el ciclo de TCA. El 3-fosfonooxipiruvato es una sustancia intermedia en la vía metabólica de la glicina, la serina y la treonina y tiene la función de promover el metabolismo de la glicina, la serina y la treonina para producir metabolitos como el acetil CoA, el ácido propiónico y el ácido α-aminobutírico (Cogo et al., 2018). El acetil-CoA producido por el metabolismo del 3-fosfonooxipiruvato puede estar involucrado en el ciclo del TCA mediante la síntesis de ácido cítrico. CarbEl fosfato de amoil podría participar en el ciclo de la urea para generar ácido fumárico y participar en el ciclo de TCA. El triptófano es un aminoácido especial que puede participar en la síntesis de proteínas del huésped, aumentar la ingesta de alimento del huésped y garantizar el rendimiento del crecimiento del huésped (Sterndale et al., 2020). Estudios previos han demostrado que la suplementación dietética de triptófano moderado reduce significativamente la urea sérica y mejora el rendimiento del crecimiento (Ruan et al., 2014; Rao et al., 2021), lo cual es consistente con nuestros resultados. Además de producir acetil-CoA para participar en el ciclo de TCA, el triptófano también se puede utilizar como materia prima para la síntesis de proteínas para promover directamente la síntesis de proteínas.
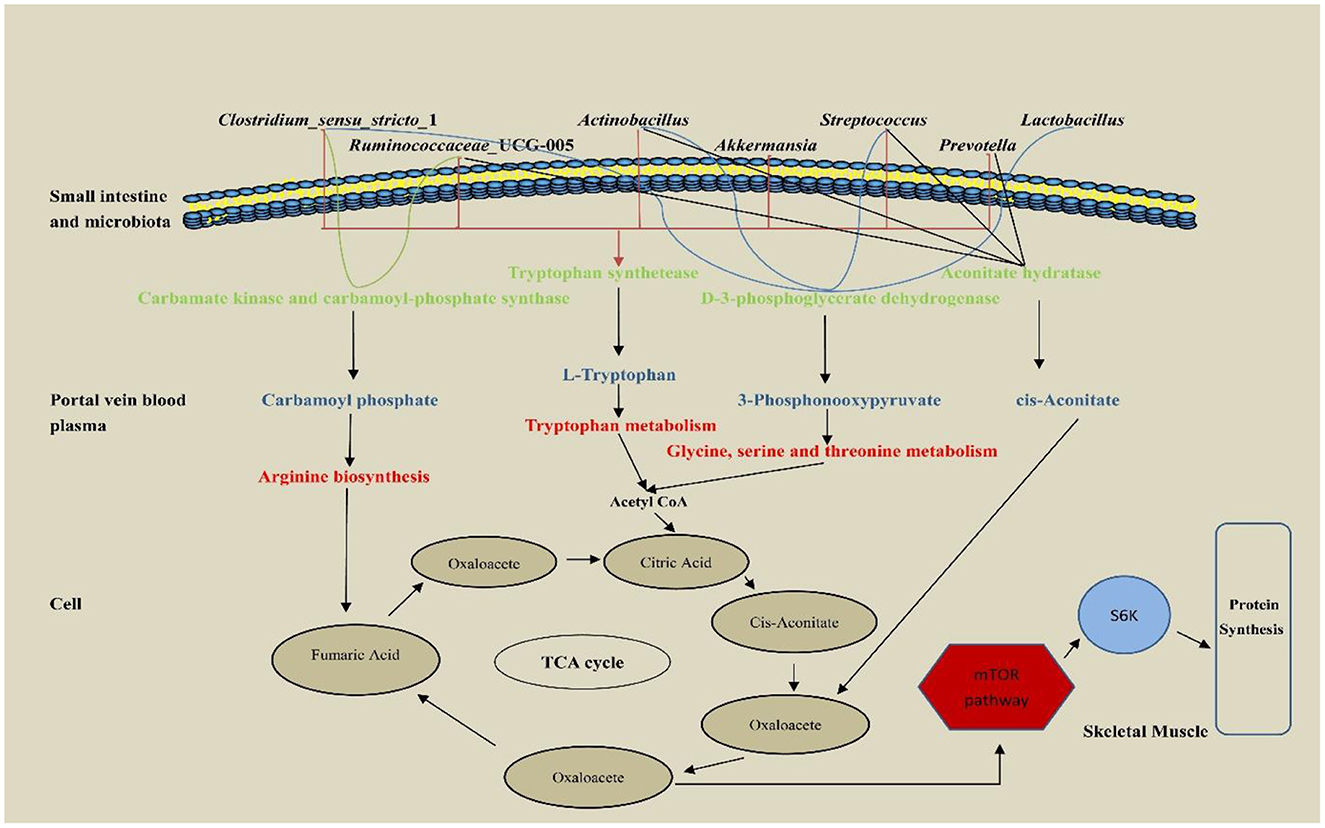 Figura 7. La vía de la vena porta hepática de metabolitos diferenciales reguladores de la síntesis de proteínas de lechones destetados.
Figura 7. La vía de la vena porta hepática de metabolitos diferenciales reguladores de la síntesis de proteínas de lechones destetados.
Curiosamente, el fosfato de carbamoil también puede metabolizar la arginina para promover la síntesis de proteínas. Un estudio previo ha demostrado que la arginina es el portador de nitrógeno más abundante para la síntesis de proteínas en el cuerpo (Wu et al., 1999), y en los mamíferos, la arginina tiene la capacidad de estimular la secreción de factores de crecimiento en la glándula pituitaria anterior (Flynn et al., 2002), por lo que los animales jóvenes son más sensibles a los requerimientos de arginina que son particularmente altos (Watanabe et al., 2009). Además, la suplementación dietética con arginina puede mejorar la eficiencia del uso de nitrógeno y mejorar la síntesis de proteínas del huésped (Guoyao et al., 2007), lo cual es consistente con nuestros resultados. Por lo tanto, el aumento del metabolismo del nitrógeno en la coadministración de L. rhamnosus GG ATCC53103 y L. plantarum JL01 en lechones destetados puede estar mediado por L-triptófano, 3-fosfonooxipiruvato, cis-aconitato y fosfato de carbamoil. El aumento de estos metabolitos puede estar relacionado con los genes funcionales contenidos en la flora significativamente aumentada.
5. Conclusión
En resumen, la administración oral de L. plantarum JL01 y L. rhamnosus GG ATCC53103 a lechones destetados aumentó la digestión aparente de nitrógeno. Se encontró que la suplementación dietética de L. plantarum JL01 y L. rhamnosus GG ATCC53103 mejoró la diversidad microbiana intestinal de los lechones destetados. Con la ayuda de genes funcionales codificados por bacterias intestinales dominantes, se promueve la producción de metabolitos dominantes L-triptófano 3-fosfonooxipiruvato, cis-aconitato y fosfato de carbamoil, y estos metabolitos promueven el ciclo de TCA de diferentes maneras, lo que puede conducir al aumento de AKG, y luego desempeñar un papel regulador positivo en la deposición de proteínas del músculo esquelético. Además, el L-triptófano tiene un efecto regulador positivo sobre el metabolismo del nitrógeno. Este hallazgo destaca aún más la importancia de la microbiota intestinal para el metabolismo del nitrógeno y demuestra un efecto de L. plantarum JL01 y L. rhamnosus GG ATCC53103 para promover el metabolismo del nitrógeno en lechones destetados.
Declaración de disponibilidad de datos
Los datos presentados en el estudio están depositados en el repositorio figshare, número de acceso 10.6084/m9.figshare.23537733.
Declaración ética
El estudio en animales fue revisado y aprobado por el Comité de Ética de la Universidad Agrícola de Jilin.
Contribuciones del autor
Diseño FH, XJ, TG, SS y HS. FH escribió este manuscrito. CW, JH, LZ, YZ, NB y LP participaron en la modificación del manuscrito. HS supervisó la redacción de este manuscrito. Todos los autores leyeron y aprobaron la versión final del manuscrito.
Financiación
Este estudio fue apoyado por el Plan Nacional Clave de Investigación y Desarrollo de China (2017YFD0500506) y la Fundación de Ciencias Naturales de la Provincia de Jilin (20210101019JC).
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
Bergen, W. G., y Wu, G. (2009). Reciclaje y utilización de nitrógeno intestinal en la salud y la enfermedad. J. Nutr. 139, 821–825. DOI: 10.3945/JN.109.104497
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Bishu, S. (2016). Detección de nutrientes y microbios en el intestino. Curr. Opinar. Gastroenterol. 32, 86–95. doi: 10.1097/MOG.0000000000000246
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cai, X., Zhu, C., Xu, Y., Jing, Y., Yuan, Y., Wang, L., et al. (2020). Nota de retracción: el alfa-cetoglutarato promueve la hipertrofia del músculo esquelético y la síntesis de proteínas a través de las vías de señalización Akt / mtor. Sci Rep. 10, 18721. DOI: 10.1038/S41598-020-72330-X
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cani, P. D., y Delzenne, N. M. (2009). El papel de la microbiota intestinal en el metabolismo energético y la enfermedad metabólica. Curr. Pharm. Diseño. 15, 1546–1558. DOI: 10.2174/138161209788168164
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Cogo, C., Pérez-Giménez, J., Rajeswari, C. B., Luna, M. F. y Lodeiro, A. R. (2018). Inducción por bradyrhizobium diazoefficiens de diferentes vías de crecimiento en d-manitol o l-arabinosa que conduce a diferencias pronunciadas en co2 fijación, o2 consumo, y producción de flagelos laterales. Frente. Microbiol. 9, 1189. DOI: 10.3389/fmicb.2018.01189
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dai, Z. L., Wu, G. y Zhu, W. Y. (2011). Metabolismo de aminoácidos en bacterias intestinales: vínculos entre la ecología intestinal y la salud del huésped. Frente. Biosci. 16, 1768–1786. DOI: 10.2741/3820
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dávila, A. M., Blachier, F., Gotteland, M., Andriamihaja, M., Benetti, P. H., Sanz, Y., et al. (2013). Metabolismo luminal intestinal del nitrógeno: papel de la microbiota intestinal y consecuencias para el huésped. Pharmacol. 68, 95–107. doi: 10.1016/j.phrs.2012.11.005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ding, H., Zhao, J., Liu, H., Wang, J. y Lu, W. (2020). Bmal1 knockdown promovió la apoptosis y redujo la secreción de testosterona en la línea celular tm3 leydig. Gen. 747, 144672. doi: 10.1016/j.gene.2020.144672
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Dukes, A., Davis, C., El, R. M., Upadhyay, S., Mork, S., Arounleut, P., et al. (2015). El aminoácido aromático triptófano estimula la señalización del músculo esquelético igf1/p70s6k/mtor in vivo y la expresión de genes miogénicos in vitro. Nutrición. 31, 1018–1024. doi: 10.1016/j.nut.2015.02.011
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Feyera, T., Zhou, P., Nuntapaitoon, M., Sorensen, K. U., Krogh, U., Bruun, T. S., et al. (2019). Metabolismo mamario y colostrogénesis en cerdas durante la gestación tardía y el período colostral. J. Anim. 97, 231–245. doi: 10.1093/jas/sky395
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Flynn, N. E., Meininger, C. J., Haynes, T. E. y Wu, G. (2002). Las bases metabólicas de la nutrición y farmacoterapia de arginina. Biomed. Farmacología. 56, 427–438. doi: 10.1016/S0753-3322(02)00273-1
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Geng, T., He, F., Su, S., Sun, K., Zhao, L., Zhao, Y., et al. (2021). Los probióticos lactobacillus rhamnosus gg atcc53103 y lactobacillus plantarum jl01 inducen alteraciones de citoquinas por la producción de tcda, dha, y ácidos succínico y palmítico, y mejoran la inmunidad de los lechones destetados. Res. Vet. 137, 56–67. doi: 10.1016/j.rvsc.2021.04.011
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Grice, E. A., Kong, H. H., Conlan, S., Deming, C. B., Davis, J., Young, A. C., et al. (2009). Diversidad topográfica y temporal del microbioma de la piel humana. Ciencia. 324, 1190–1192. doi: 10.1126/science.1171700
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Guoyao, W., Fuller, W. B., Teresa, A. D., Laurie, A. J., Greg, A. J., Sung, W. K., et al. (2007). Funciones importantes para la familia de aminoácidos arginina en la nutrición y producción porcina. Livest. Sci. 112, 3. doi: 10.1016/j.livsci.2007.07.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Hooper, L. V., Midtvedt, T. y Gordon, J. I. (2002). Cómo las interacciones huésped-microbiano dan forma al entorno nutricional del intestino de los mamíferos. Nutr. 22, 283–307. doi: 10.1146/annurev.nutr.22.011602.092259
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Jardon, K. M., Canfora, E. E., Goossens, G. H. y Blaak, E. E. (2022). Macronutrientes dietéticos y el microbioma intestinal: un enfoque de nutrición de precisión para mejorar la salud cardiometabólica. Intestino. 71, 1214–1226. DOI: 10.1136/GuTJNL-2020-323715
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Johnson, C. H., Ivanisevic, J. y Siuzdak, G. (2016). Metabolómica: más allá de los biomarcadores y hacia los mecanismos. Nat. Rev. 17, 25. doi: 10.1038/nrm.2016.25
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Khanian, M., Karimi-Torshizi, M. A. y Allameh, A. (2019). Alivio del daño oxidativo relacionado con la aflatoxinas en el hígado y mejora del rendimiento de crecimiento en pollos de engorde consumidos por Lactobacillus plantarum 299v durante todo el período de crecimiento. Toxicon. 158, 57–62. doi: 10.1016/j.toxicon.2018.11.431
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Krajmalnik-Brown, R., Ilhan, Z. E., Kang, D. W. y DiBaise, J. K. (2012). Efectos de los microbios intestinales en la absorción de nutrientes y la regulación energética. Nutr. Clin. Práctica. 27, 201–214. DOI: 10.1177/0884533611436116
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Kuo, H. K., Liao, K. C., Leveille, S. G., Bean, J. F., Yen, C. J., Chen, J. H., et al. (2007). Relación de los niveles de homocisteína con la fuerza del cuádriceps, la velocidad de la marcha y la discapacidad tardía en adultos mayores. J. Gerontol. Ser. A-Biol. Sci. Med. 62, 434–439. doi: 10.1093/gerona/62.4.434
Resumen de PubMed | Texto completo de CrossRef | Google Académico
LeBlanc, J. G., Milani, C., de Giori, G. S., Sesma, F., van Sinderen, D. y Ventura, M. (2013). Las bacterias como proveedores de vitaminas para su huésped: una perspectiva de la microbiota intestinal. Curr. Opinar. Biotecnología. 24, 160–168. doi: 10.1016/j.copbio.2012.08.005
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Lin, R., Liu, W., Piao, M. y Zhu, H. (2017). Una revisión de la relación entre la microbiota intestinal y el metabolismo de los aminoácidos. Aminoácidos. 49, 2083–2090. DOI: 10.1007/S00726-017-2493-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Matthews, D. E. (2020). Revisión del metabolismo de la lisina con un enfoque en humanos. J. Nutr. 150, 2548S−2555S. doi: 10.1093/jn/nxaa224
Resumen de PubMed | Texto completo de CrossRef | Google Académico
McCann, J. C., Elolimy, A. A. y Loor, J. J. (2017). Microbioma ruminal, probióticos y aditivos de fermentación. Veterinario Clin. N. Am.-Food Anim. 33, 539–553. doi: 10.1016/j.cvfa.2017.06.009
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Metges, C. C. (2000). Aporte de aminoácidos microbianos a la homeostasis de aminoácidos del huésped. J. Nutr. 130, 1857S−1864S. doi: 10.1093/jn/130.7.1857S
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Metges, C. C. y Petzke, K. J. (2005). Utilización de aminoácidos esenciales sintetizados en la microbiota intestinal de mamíferos monogástricos. Fr. J. Nutr. 94, 621–622. doi: 10.1079/BJN20051509
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Millet, S., Aluwe, M., Van den Broeke, A., Leen, F., Boever, D. E., De Campeneere, J., et al. (2018). Revisión: producción de carne de cerdo con máxima eficiencia nitrogenada. Animal. 12, 1060–1067. doi: 10.1017/S1751731117002610
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Consejo Nacional de Investigación. (2012). Requerimientos de nutrientes de los cerdos: Undécima edición revisada. Washington, DC: The National Academies Press.
Nicholson, J. K., Holmes, E., Kinross, J., Burcelin, R., Gibson, G., Jia, W., et al. (2012). Interacciones metabólicas de la microbiota huésped-intestino. Ciencia. 336, 1262–1267. doi: 10.1126/science.1223813
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Rao, Z., Li, J., Shi, B., Zeng, Y., Liu, Y., Sun, Z., et al. (2021). Los niveles dietéticos de triptófano afectan el rendimiento del crecimiento y la ecología microbiana intestinal en lechones destetados a través de metabolitos de triptófano y péptidos antimicrobianos intestinales. Animales. 11, 817. DOI: 10.3390/ANI11030817
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Roager, H. M. y Licht, T. R. (2018). Catabolitos de triptófano microbiano en la salud y la enfermedad. Nat. Commun. 9, 3294. DOI: 10.1038/S41467-018-05470-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Ruan, Z., Yang, Y., Wen, Y., Zhou, Y., Fu, X., Ding, S., et al. (2014). Análisis metabolómico del metabolismo de aminoácidos y grasas en ratas con suplementos de l-triptófano. Aminoácidos. 46, 2681–2691. DOI: 10.1007/S00726-014-1823-Y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Segata, N., Izard, J., Waldron, L., Gevers, D., Miropolsky, L., Garrett, W. S., et al. (2011). Descubrimiento y explicación de biomarcadores metagenómicos. Genoma Biol. 12, R60. DOI: 10.1186/GB-2011-12-6-R60
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Sterndale, S. O., Miller, D. W., Mansfield, J. P., Kim, J. C. y Pluske, J. R. (2020). El aumento del triptófano en la dieta junto con la disminución de otros aminoácidos neutros grandes aumenta el aumento de peso y la ingesta de alimento en cerdos destetados, independientemente de la infección experimental con escherichia coli enterotoxigénica. J. Anim. 98, 190. doi: 10.1093/jas/skaa190
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Sun, W., Chen, W., Meng, K., Cai, L., Li, G., Li, X., et al. (2023). La suplementación dietética con bacilo probiótico licheniformis s6 mejora la integridad intestinal mediante la modulación de la función de barrera intestinal y la diversidad microbiana en lechones destetados. Biología-Basilea. 12, 238. doi: 10.3390/biología12020238
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Sun, Y., Sun, Y., Shi, Z., Liu, Z., Zhao, C., Lu, T., et al. (2019). Microbiota intestinal del ciervo almizclero alpino salvaje y cautivo (moschus chrysogaster). Frente. Microbiol. 10, 3156. DOI: 10.3389/fmicb.2019.03156
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Torrallardona, D., Harris, C. I. y Fuller, M. F. (2003). La lisina sintetizada por la microflora gastrointestinal de los cerdos se absorbe, principalmente en el intestino delgado. Am. J. Physiol.-Endocrinol. Metab. 284, E1177–E1180. doi: 10.1152/ajpendo.00465.2002
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Truong, D. T., Franzosa, E. A., Tickle, T. L., Scholz, M., Weingart, G. y Pasolli, E., et al. (2015). Metaphlan2 para mejorar el perfil taxonómico metagenómico. Métodos nat. 12, 902–903. DOI: 10.1038/NMETH.3589
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Utzschneider, K. M., Kratz, M., Damman, C. J. y Hullar, M. (2016). Mecanismos que vinculan el microbioma intestinal y el metabolismo de la glucosa. J. Clin. Endocrino. Metab. 101, 1445–1454. DOI: 10.1210/JC.2015-4251
Resumen de PubMed | Texto completo de CrossRef | Google Académico
van der Wielen, N., Moughan, P. J. y Mensink, M. (2017). Absorción de aminoácidos en el intestino grueso de humanos y modelos porcinos. J. Nutr. 147, 1493–1498. DOI: 10.3945/JN.117.248187
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Wang, H., y Kim, I. H. (2021). Evaluación de la suplementación con probióticos dietéticos (lactobacillus plantarum bg0001) sobre el rendimiento de crecimiento, digestibilidad de nutrientes, perfil sanguíneo, emisión de gases fecales y microbiota fecal en cerdos destetados. Animales. 11, 1–14. doi: 10.3390/ani11082232
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Wang, Q., Sun, Q., Qi, R., Wang, J., Qiu, X., Liu, Z., et al. (2019). Efectos de lactobacillus plantarum sobre la morfología intestinal, la función de barrera intestinal y la composición de la microbiota de lechones lactantes. J. Anim. Physiol. Anim. Nutr. 103, 1908–1918. doi: 10.1111/jpn.13198
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Watanabe, K., Arozal, W., Tanaka, H., Ma, M., Satoh, S., Veeraveedu, P. T., et al. (2009). Efecto beneficioso del sustituto alimenticio que contiene l-arginina, ácido graso poliinsaturado omega-3 y ácido ribonucleico en la prevención o mejora del síndrome metabólico: un estudio en 15 pacientes con sobrepeso y un estudio del metabolismo de los ácidos grasos en animales. J. Clin. Bioquímica. Nutr. 44, 266–274. DOI: 10.3164/JCBN.08-241
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Wernerman, J., y Hammarqvist, F. (1999). Glutamina: nutriente necesario para el paciente de cuidados intensivos. Int. J. Colorrectal Dis. 14, 137–142. doi: 10.1007/s003840050199
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Wu, G., Ott, T. L., Knabe, D. A. y Bazer, F. W. (1999). Composición de aminoácidos del cerdo fetal. El J. Nutr. 129, 1031 doi: 10.1093/jn/129.5.1031
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Yang, Y. X., Dai, Z. L. y Zhu, W. Y. (2014). Impactos importantes de las bacterias intestinales en la utilización de aminoácidos dietéticos en cerdos. Aminoácidos. 46, 2489–2501. DOI: 10.1007/S00726-014-1807-Y
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Yi, H., Yang, G., Xiong, Y., Wu, Q., Xiao, H., Wen, X., et al. (2019). El perfil metabolómico y proteómico integrado revela la promoción de Lactobacillus reuteri LR1 en el metabolismo de aminoácidos en el eje intestino-hígado de cerdos destetados. Función de alimentos. 10, 7387–7396. doi: 10.1039/C9FO01781J
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhao, J., Zhang, X., Liu, H., Brown, M. A. y Qiao, S. (2019). Composición y función de las proteínas dietéticas y la microbiota intestinal. Curr. Proteína Pept. Sci. 20, 145–154. doi: 10.2174/1389203719666180514145437
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhao, L., Guo, H. y Sun, H. (2020). Efectos de la suplementación dietética baja en proteínas con alfa-cetoglutarato sobre el rendimiento del crecimiento, el metabolismo del nitrógeno y la vía de señalización mtor del músculo esquelético en lechones. J. Anim. Physiol. Anim. Nutr. 104, 300–309. DOI: 10.1111/JPN.13230
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Zhu, J., Gao, M., Zhang, R., Sun, Z., Wang, C., Yang, F., et al. (2017). Efectos de la harina de soja fermentada por l. Plantarum, b. Subtilis y s. Cerevisieae sobre crecimiento, función inmune y morfología intestinal en lechones destetados. Microbio. Celda. Hecho. 16, 191. DOI: 10.1186/S12934-017-0809-3
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: metabolitos, metabolismo del nitrógeno, lechones destetados, Lactobacillus rhamnosus GG ATCC53103, Lactobacillus plantarum JL01
Cita: He F, Jin X, Wang C, Hu J, Su S, Zhao L, Geng T, Zhao Y, Pan L, Bao N y Sun H (2023) Lactobacillus rhamnosus GG ATCC53103 y Lactobacillus plantarum JL01 mejoraron el metabolismo del nitrógeno en lechones destetados mediante la regulación de la estructura de la flora intestinal y los metabolitos de la vena porta. Frente. Microbiol. 14:1200594. doi: 10.3389/fmicb.2023.1200594
Recibido: 05 Abril 2023; Aprobado: 30 de mayo de 2023;
Publicado: 29 junio 2023.
Editado por:
Hao Zhong, Universidad Tecnológica de Zhejiang, China
Revisado por:
Yu Pi, Instituto de Investigación de Piensos (CAAS), China Miaomiao Bai, Instituto de Agricultura Subtropical (CAS), China Liping Gan, Universidad Tecnológica de Henan, China
Derechos de autor © 2023 He, Jin, Wang, Hu, Su, Zhao, Geng, Zhao, Pan, Bao y Sun. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Hui Sun, hui1688@163.com
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía