Monitoreo de la resistencia antimicrobiana a los aminoglucósidos y macrólidos en Campylobacter coli y Campylobacter jejuni de ganado sano en España (2002-2018)

Monitoreo de la resistencia antimicrobiana a los aminoglucósidos y macrólidos en Campylobacter coli y Campylobacter jejuni de ganado sano en España (2002-2018)
 Vicente López-Chavarrias1,
Vicente López-Chavarrias1,  María Ugarte-Ruiz1, Carmen Barcena1, Adolfo Olarra2, María García1, José Luis Saez3, Cristina de Frutos4, Tania Serrano5, Iratxe Pérez4,
María Ugarte-Ruiz1, Carmen Barcena1, Adolfo Olarra2, María García1, José Luis Saez3, Cristina de Frutos4, Tania Serrano5, Iratxe Pérez4,  Miguel Ángel Moreno6, Lucas Domínguez1,6 y
Miguel Ángel Moreno6, Lucas Domínguez1,6 y  Julio Álvarez1,6*
Julio Álvarez1,6*- 1Centro de Vigilancia Sanitaria de 1VISAVET, Universidad Complutense de Madrid, Madrid, España
- 2Facultad de Medicina Veterinaria y Zootecnia, Universidad Nacional Autónoma de México, Ciudad de México, México
- 3Subdirección General de Sanidad e Higiene Animal y Trazabilidad, Dirección General de la Producción Agraria, Ministerio de Agricultura, Pesca y Alimentación, Madrid, España
- 4Laboratorio Central de Veterinaria (LCV Algete), Ministerio de Agricultura, Pesca y Alimentación, Madrid, España
- 5TRAGSATEC, Tecnologías y Servicios Agrarios S.A., Madrid, España
- 6Departamento de Sanidad Animal, Facultad de Veterinaria, Universidad Complutense de Madrid, Madrid, España
Resistencia a los antimicrobianos (AMR) en Campylobacter spp. (Campylobacter coli y Campylobacter jejuni) es una preocupación debido a su importancia en la salud pública, particularmente cuando se trata de aminoglucósidos y macrólidos, medicamentos de elección para el tratamiento de casos humanos. La co-resistencia a estas dos clases de antimicrobianos implica la transferencia de elementos genéticos y/o la adquisición de mutaciones en diferentes loci genéticos, que a su vez pueden propagarse a través de fenómenos de transferencia de genes verticales u horizontal (HGT), con cada ruta teniendo diferentes implicaciones potenciales. Este estudio tuvo como objetivo evaluar la asociación entre la presencia de resistencia fenotípica a estas dos clases de antimicrobianos en C. coli y C. jejuni recuperados del ganado en mataderos en España (como parte del programa de vigilancia de la resistencia a los antimicrobianos), y a evaluar la heterogeneidad genética entre aislados resistentes y susceptibles mediante el análisis de la Durante el período 2002-2018, se recogieron los resultados de la prueba de susceptibilidad antimicrobiana de 10.965 aislados de Campylobacter recuperados de muestras fecales de pollos de engorde, pavos, cerdos y ganado para comparar la proporción de aislados resistentes y las concentraciones mínimas inhibidoras (MIC) con seis antimicrobianos, incluida la gentamicina Se determinaron los genes asociados a la AMR para un grupo de 51 aislados sometidos a la secuenciación del genoma completo, y el SVR flaA de un subconjunto de 168 aislados de todos los huéspedes con diferentes resistotipos se utilizó para construir un árbol filogenético basado en Neighbor-Joining y evaluar la existencia de grupos mediante el análisis de «uso de co La proporción de aislados resistentes a los antimicrobianos, tanto aminoglucósidos como de macrólidos, varió ampliamente para C. coli (7-91%) y menos para C. jejuni (todos huéspedes entre 0 a 1 %). En todos los huéspedes, estas proporciones fueron del 7-56% en aves de corral, del 12-82% en el ganado y del 22-91% en cerdos para C. coli y del 0-8% en aves de corral y del 1-1% en el ganado para C. jejuni. La comparación de las distribuciones del MIC reveló diferencias significativas específicas del huésped solo para ERY en C. jejuni (p = 0,023). Se observó una asociación significativa en la presentación simultánea de la RAM a ambas clases de antimicrobianos entre los huéspedes/especies bacterianas. El análisis del gen flaA mostró un agrupamiento de aislados que comparten resistotipo y, en menor grado, especies bacterianas y hospedantes. Se encontraron varios marcadores de resistencia asociados con la resistencia a los aminoglucósidos y macrólidos entre los aislados secuenciados. La asociación consistente entre la presentación simultánea de AMR a aminoglucósidos y macrólidos en todos los huéspedes podría deberse a la persistencia de cepas y/o mecanismos de resistencia en las poblaciones de Campylobacter en el ganado a lo largo del tiempo. Se necesitan más estudios basados en la secuenciación del genoma completo para evaluar los vínculos epidemiológicos entre los huéspedes y las cepas bacterianas.
Introducción
Campylobacter coli y Campylobacter jejuni, bacterias termófilas del género Campylobacter spp., son los patógenos zoonóticos gastrointestinales humanos notificados con más frecuencia en la Unión Europea (UE) desde 2005 (EFSA, 2005, 2006, 2007). Tradicionalmente, C. jejuni se aisló con más frecuencia de casos humanos que C. coli, pero en 2017, el 24,1 % de las infecciones humanas confirmadas por Campylobacter en la UE fueron causadas por C. coli frente al 22,2 % debido a C. jejuni, lo que sugiere que este patrón puede variar (EFSA-ECDC, 2019b). Los productos avícolas y avícolas se consideran la principal fuente de campilobacteriosis humana, seguidos de los rumiantes (carne de res, ganado lechero y su estiércol) y las fuentes ambientales (Ravel et al., 2017; Rosner et al., 2017; An et al., 2018; Thepault et al., 2018). El consumo de carne de cerdo se ha relacionado con casos humanos causados por C. coli (Rosner et al., 2017). Aunque la prevalencia de Campylobacter spp. en la carne de cerdo es alta, estudios anteriores encontraron que solo se asoció con el 2 % de todos los casos humanos (Kittl et al., 2013), mientras que la carne de vacuno se ha relacionado con el 19 % de los casos humanos (Boysen et al., 2014).
El tratamiento de los casos humanos, cuando sea necesario, puede verse obstaculizado por la creciente amenaza de resistencia a los antimicrobianos (RAM) observada en los últimos años (Friedrich, 2019). Los macrólidos [eritromicina (ERY) y azitromicina] y las fluoroquinolonas [ciprofloxacina (CIP)] son los medicamentos de elección en pacientes humanos que requieren tratamiento antibiótico, pero esta última clase no se recomienda para los niños. Cuando estos medicamentos son ineficaces, la administración sistémica de aminoglucósidos es la única opción que queda (Bolinger y Kathariou, 2017; Organización Mundial de la Salud (OMS), 2017). Los niveles de RAM a los aminoglucósidos y macrólidos en Campylobacter termofílico clínico de los seres humanos están aumentando (Aarestrup y Wegener, 1999; Bolinger y Kathariou, 2017; EFSA-ECDC, 2019b), y se ha observado una tendencia similar para los macrólidos en aislados de cerdos y pollo En Europa, la proporción de aislados resistentes de Campylobacter spp. de animales productores de alimentos y seres humanos varía según el país. Los aislados originarios de España mostraron niveles más altos de resistencia a los aminoglucósidos [gentamicina (GEN) y estreptomicina (STR)], macrólidos (ERY), quinolonas [CIP y ácido nalidísidico (NAL)] y tetraciclina (TET) en C. coli de pollos de engorde, pavo C. coli ha mostrado tradicionalmente niveles más altos de RAM para la mayoría de los antimicrobianos en comparación con C. jejuni tanto en humanos como en animales (EFSA-ECDC, 2017, 2018a, 2019b). Por lo tanto, el problema de la resistencia a los antimicrobianos puede intensificarse si la importancia de la C. coli como patógeno humano sigue aumentando. C. coli es ahora tan frecuente como C. jejuni en los pollos de engorde en algunos países (Wieczorek y Osek, 2013), por lo tanto, el monitoreo de los niveles de C. coli y C. jejuni AMR, particularmente de macrólidos y aminoglucósidos, es igualmente importante (EFSA-ECDC, 2018a
La resistencia a los aminoglucósidos y macrólidos puede estar mediada por múltiples mecanismos, incluidas las mutaciones cromosómicas y los elementos transferibles de genes horizontales (Davies y Wright, 1997; Saenz et al., 2000; Moore et al., 2006; Luangtongkum et al., 2009; Wieczorek y Os Las enzimas modificadoras de antibióticos (AME) están comúnmente involucradas en la resistencia a los aminoglucósidos (Saenz et al., 2000; Wieczorek y Osek, 2013; Garneau-Tsodikova y Labby, 2016), mientras que las metiltransferasas de ribosomas (RMT) están frecuentemente relacionadas con la resistencia a los mac La co-resistencia tanto a los aminoglucósidos como a los macrólidos, así como a la resistencia multifarmactección (MDR) a clases de antimicrobianos adicionales, se puede adquirir a través de varios mecanismos como RMT de ARNr 16S (RmtB, ArmA) codificados en islas genómicas de resistencia a múltiples fármaco Los mecanismos adicionales pueden incluir islas genómicas transferibles que llevan múltiples genes de resistencia a los aminoglucósidos que codifican para AME en C. coli (Davies y Wright, 1997; Luangtongkum et al., 2009; Qin et al., 2012; Wieczorek y Osek, 2013) y bombas de eflujo de macró fármacos Aunque varios de estos mecanismos se descubrieron por primera vez en C. coli, a partir de entonces se mostró evidencia de transferencia a C. jejuni (EFSA-ECDC, 2018a).
Los estudios epidemiológicos complementados con análisis genéticos son esenciales para ayudar a comprender los mecanismos por los cuales la co-resistencia y la MDR a los aminoglucósidos y macrólidos pueden estar surgiendo en el Campylobacter termofílico de animales y humanos (Luangtongkum et al., 2009; Wieczorek y Osek, 2013). Aquí, se analizaron los datos del programa nacional de vigilancia de la resistencia a los antimicrobianos en Campylobacter spp. de pollos, pavos, cerdos y ganado en España para evaluar la prevalencia de las especies de Campylobacter en diferentes huéspedes animales y los patrones de co-resistencia fenotípica de la resistencia a los macrolíidos y aminoglucósidos Los resultados de esta investigación pueden contribuir a explicar mejor los fenómenos de coelección de AMR/MDR entre estas dos clases de antimicrobianos en Campylobacter del ganado.
Materiales y métodos
Población del estudio
Los datos analizados aquí se basan en el trabajo de recolección de muestras, cultivo y pruebas de susceptibilidad antimicrobiana (AST) llevados a cabo durante 2002-2018 en aislados recuperados a través del programa nacional de monitoreo veterinario español de AMR para Campylobacter spp. en aves de corral (brolecinos y pavos), cerdos y ganado, de acuerdo con la legislación Se recogieron muestras para cada especie animal, procedentes de múltiples granjas, en mataderos que cubrían el 60 % del rendimiento nacional (Figuras Suplementarias 1, 2). Las muestras de pollos de engorde se recuperaban cada año de 2002 a 2014 y cada 2 años a partir de entonces, muestras de pavo cada 2 años de 2014 a 2018, muestras de cerdo cada año de 2002 a 2013 y cada 2 años a partir de entonces, y muestras de ganado cada año de 2007 a 2013 y cada 2 años a partir de entonces. Las muestras en común recogidas cada año para cada especie huésped oscilaron entre 76 y 500 (media = 228) para pollos de engorde, 467 y 500 (media = 485) para pavos, 171 y 384 (media = 268) para cerdos, y 163 y 384 (media = 261) para el ganado (Tabla complementaria 1).
En relación con el muestreo y el cultivo, se recogieron piscinas hechas de muestras de animales pertenecientes a la misma granja (de 10 caeca en aves de corral o del contenido de ciego de 2 animales en cerdos y ganado) se recogieron en el matadero y se transportaron refrigerados al laboratorio, donde se procesaron dentro de las 24 horas posteriores a la recolección de acuerdo con la norma Las colonias individuales con morfología compatible con Campylobacter spp. se identificaron como «C. coli«, «C. jejuni» o «C. spp». utilizando tiras API (hasta 2010) y una PCR múltiple (2010 en adelante) (Ugarte-Ruiz et al., 2012).
Con respecto a AST, el fenotipo AMR de los aislados de Campylobacter spp. se determinó utilizando el método de referencia de microdilución de caldo de dos veces (calculando las concentraciones mínimas inhibidoras – MIC, de acuerdo con la norma ISO 20776-1:2006) o la técnica de difusión (calculando los diámetros de las zonas de inhibición – IZD)
Los resultados de las pruebas de susceptibilidad a los antimicrobianos para los seis antimicrobianos enumerados en los programas de vigilancia de AMR para Campylobacter spp. en la UE (EC, 2013) estaban disponibles para aislados de todas las especies y años: CIP, TET, NAL, STR, ERY y GEN. Para ERY en pollos de engorde, los IZD se usaron hasta 2004 (incluidos) y los MIC se usaron a partir de entonces. Para STR en pollos de engorde y cerdos, los IZD se utilizaron hasta 2005 (incluidos) y los MIC se utilizaron a partir de entonces.
Se excluyeron del análisis las muestras en las que faltaba información sobre el resultado del cultivo, la identificación molecular y/o la tipificación de la resistencia almicrobiano. La siguiente información estaba disponible para todas las muestras del estudio: especies huésped, resultado de crecimiento de Campylobacter, especie de Campylobacter, año y resultado de AST. Los aislados se clasificaron como «susceptibles» (cresiones de tipo salvaje) o «no susceptibles» (crema resistentes) según los puntos de corte epidemiológicos (ECOFF) proporcionados por el «Comité Europeo de Pruebas de Susceptibilidad Antimicrobiana» (EUCAST)1 (Tabla 1). Las proporciones de aislados resistentes para cada especie y período bacteriano y huésped se definieron como muy bajos (<1%), bajos (1,1%), moderados (10,1–20%), altos (20,10–50%), muy altos (50,10,70 %) y extremadamente altos (71,100 %), según lo recomendado por la EFSA (EFSA-ECDC, 2019b).
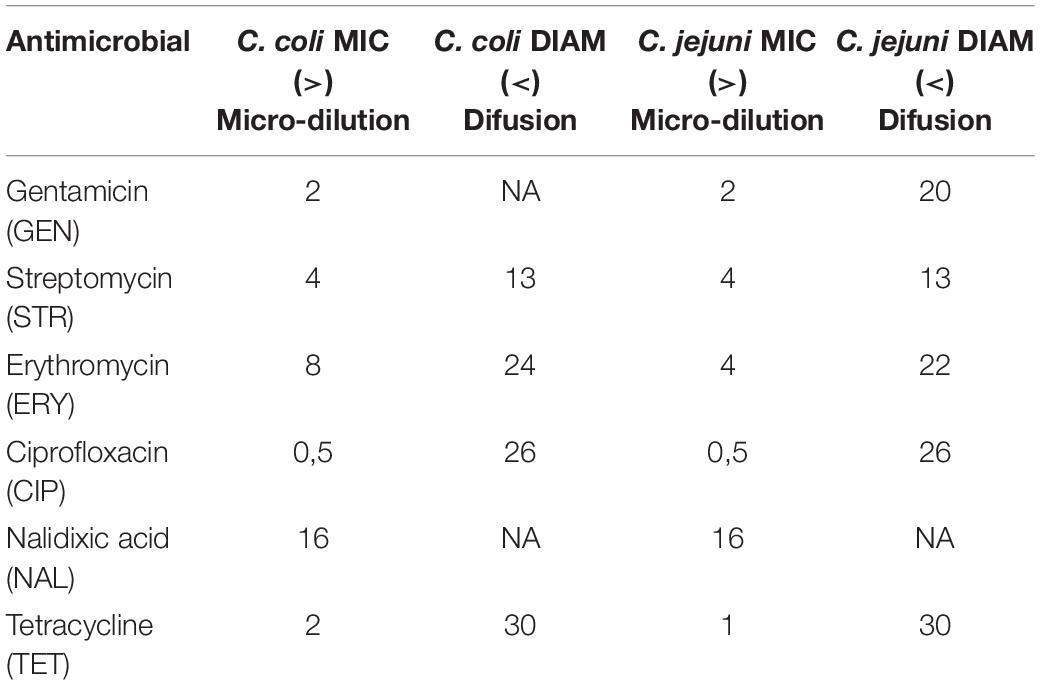 TABLA 1. Cortes epidemiológicos (ECOFF) utilizados para la interpretación de los CMI en Campylobacter spp. (Fuente EUCAST).
TABLA 1. Cortes epidemiológicos (ECOFF) utilizados para la interpretación de los CMI en Campylobacter spp. (Fuente EUCAST).
Análisis estadísticos
Las proporciones de aislados resistentes de C. coli y C. jejuni a cada antimicrobiano de los diferentes huéspedes se compararon utilizando pruebas Z, ajustadas para comparaciones múltiples por el método Holm. Se utilizaron regresiones logísticas de Cochrane-Armitage para probar las tendencias de la resistencia fenotípica a la resistencia a la resistencia a la resistencia a la resistencia a la resistencia a la resistencia a la C. y a la especie anfitriona y a la especie anfitriona, y el cambio relativo en la proporción de aislados resistentes al año, y a su intervalo La asociación en la presentación simultánea de la resistencia fenotípica a STR/ERY, GEN/ERY y GEN/STR durante todo el período de estudio y en diferentes períodos de tiempo (2002-2006, 2007-2012 y 2013-2014) se evaluó aún más para cada especie bacteriana y huésped utilizando riesgos relativos y pruebas exactas de chi cuadrado y Fisher.
Con el fin de evaluar las diferencias en la distribución de los valores de los CMI en C. coli y C. jejuni de las cuatro especies huésped, los datos disponibles se representaron como «squashtograms». La existencia de diferencias estadísticas en las distribuciones de las CMI en aislados susceptibles y no sensibles (aquí denominados «resistentes») dependiendo de la especie bacteriana (para un huésped determinado) o en el huésped (para una especie bacteriana determinada) se evaluó utilizando las pruebas de Mann-Whitney U o Kruskal-Wallis seguidas de las pruebas post hoc de Dunn, corrigiendo las
Caracterización molecular basada en la secuenciación de flaA
Se utilizó un subconjunto de 125 aislados que incluye todas las combinaciones de huéspedes, especies bacterianas, año de recuperación y fenotipo de RAM, elegidos al azar dentro de cada categoría, para evaluar su relación genética comparando la secuencia de genes de la región de variable corta (SVRC) de flagelelina flaA según lo descrito por otros autores (Ugarte-Ruiz et Los aislados seleccionados se clasificaron en dos categorías: aislados con resistencia fenotípica simultánea a los aminoglucósidos y ERY (n = 53, «casos») y aislados que no presentan esta resistencia simultánea (n = 72, «controles»). La amplificación de la secuencia del gen flaA SVR por PCR se realizó como se describió anteriormente (Ugarte-Ruiz et al., 2013), y los amplicones obtenidos se secuenciaron. Además, se extrajo la secuencia flaA de cincuenta y un aislados (8 «casos» y 43 «controles») previamente sometida a la secuenciación del genoma completo (códigos de acceso de GenBank SRX5575129 a SRX5587545) (junto con información sobre la presencia de genes de resistencia) utilizando un script Python casero. Las 176 secuencias resultantes se alinearon utilizando MUSCLE (Edgar, 2004) y se construyó un árbol filogenético de vecindad (NJ) con 1.000 bootstraps para evaluar la relación filogenética entre los aislados. El gen flaA de la cepa NCTC 1168 C. jejuni (1719 posiciones cromosómicas bacterianas de largo y nucleótidos 1269232 a 1270950) se utilizó como referencia externa. Se realizó un análisis de correspondencia múltiple (MCA) de los valores relativos sinónimos de uso de codones (RSCU) categorizados como > 1 ( sesgo positivo) o < 1 ( sesgo negativo) como se describió anteriormente (Meinersmann et al., 2005). El MCA incluía, junto con la RSCU de codones variables, otras covariables disponibles (especies bacterianas, especies hospedantes, resistencia a GEN, ERY y STR, y clado según lo determinado en el árbol filogenético de NJ).
Microsoft Access se utilizó para el manejo de datos y el análisis inicial de bases de datos. Los datos se manejaron con Microsoft Excel y se importaron a la versión 3.6.3 de «R» (R Core Team, 2020). Los paquetes R «FSA» (Ogle et al., 2020), «plyr» (Wickham, 2011) y «ggplot2», «dplyr», «reshape2» y «tidyr» (Wickham et al., 2019) se utilizaron para el análisis y la representación visual de los datos. La información sobre los marcadores asociados a la resistencia de las cepas secuenciadas se extrajo con ResFinder (Bortolaia et al., 2020). MEGA-X (Kumar et al., 2018) y DnaSP6 (Rozas et al., 2017) se utilizaron en secuencias de ADN importadas para la preparación y el análisis de alineaciones de secuencias. Los paquetes R «BiocManager» («coRdon») (Morgan y Ramos, 2019) y «seqinr» (Charif y Lobry, 2007) se utilizaron para el cálculo de RSCU. «Corrplot» (Wei et al., 2017), «FactoMineR» (Lê et al., 2008), «factoextra» (Kassambara y Mundt, 2020) y «ggtheme» se utilizaron para el análisis de MCA. Todas las figuras se generaron usando R, excepto la Figura 1 (Excel) y la Figura 6 (MEGA-X).
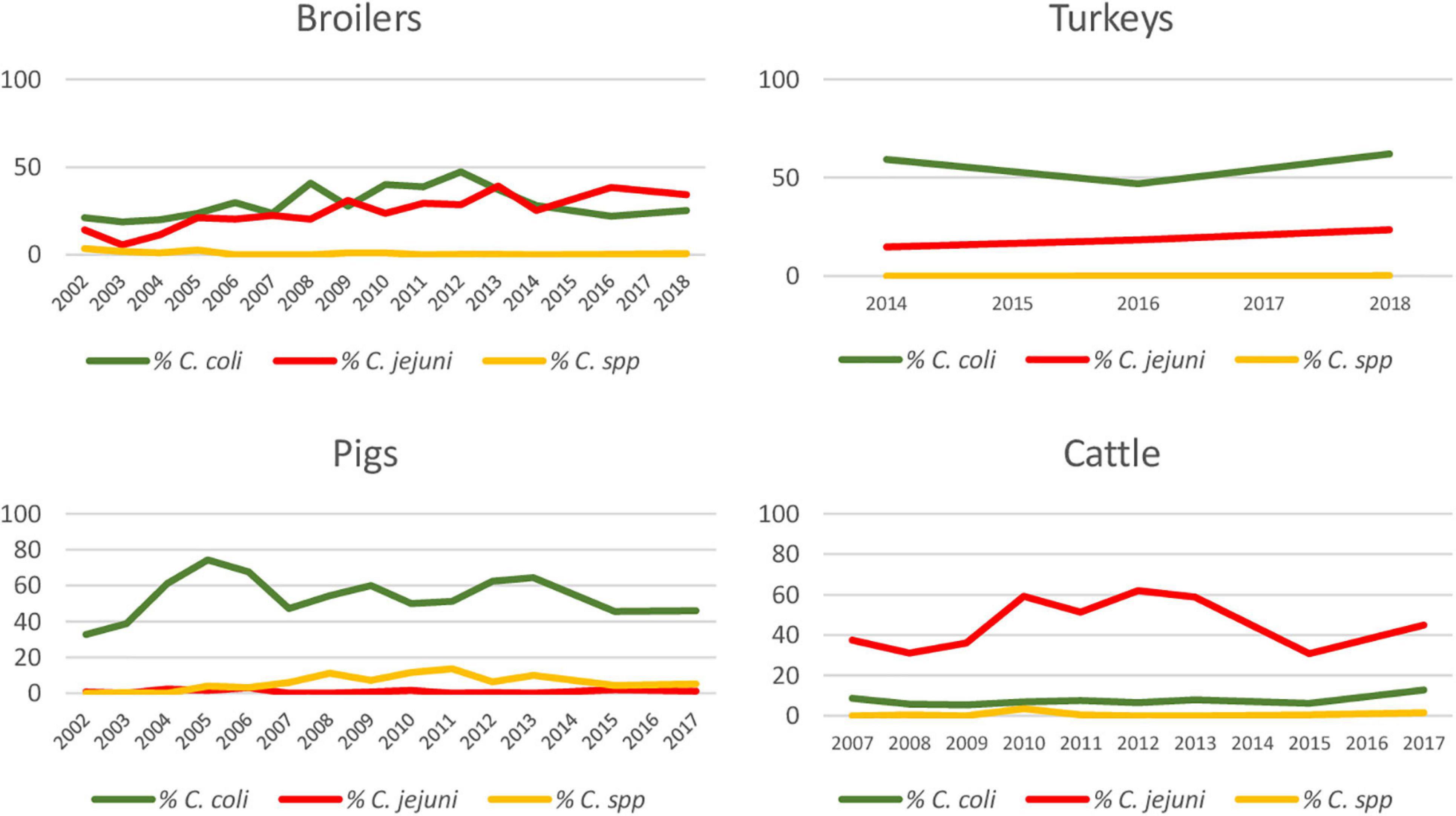 FIGURA 1. Porcentaje de aislados de Campylobacter sobre el total muestreado, identificado como C. coli, C. jejuni y C. spp. para cada especie huésped durante el período de estudio.
FIGURA 1. Porcentaje de aislados de Campylobacter sobre el total muestreado, identificado como C. coli, C. jejuni y C. spp. para cada especie huésped durante el período de estudio.
Resultados
En total, se incluyeron en el análisis 3.413 muestras independientes de pollos de engorde, 1.455 de pavos, 3.750 de cerdos y 2.347 de ganado, de las cuales 2.000 (58,6%), 1.090 (74,9%), 2.218 (59,2%) y 1.273 (54,3%) dieron lugar al aislamiento de Campylobacter spp., respectivamente El número de muestras analizadas, aislados recuperados y aislados sometidos a AST varió según el año y la especie huésped (Tabla complementaria 2).
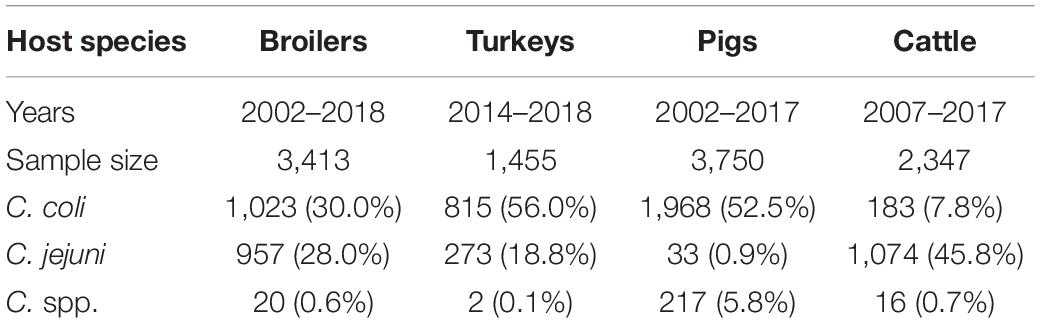 TABLA 2. Período de recolección, número de muestras fecales y muestras positivas para el aislamiento de Campylobacter de cada especie huésped incluida en el estudio.
TABLA 2. Período de recolección, número de muestras fecales y muestras positivas para el aislamiento de Campylobacter de cada especie huésped incluida en el estudio.
Durante todo el período de estudio, C. coli fue la especie más frecuentemente aislada en muestras de cerdo (88,7%; 1.968/2,218) y pavo (74,8%; 815/1,090), mientras que C. jejuni fue la especie más frecuente en el ganado (84,4%; 1.074/1,273). En los pollos de engorde, la proporción de C. coli y C. jejuni fue muy similar (51,2%; 1.023/2.000 y 47,8%; 957/2.000, respectivamente) (Tabla 2).
Aunque hubo diferencias dependiendo del año, la proporción de muestras positivas a C. coli y C. jejuni se mantuvo relativamente constante durante todo el período de estudio en cerdos y ganado, siendo una especie bacteriana más frecuente que la otra, mientras que la situación fue más variable en los pollos de engorde (Figura 1). Se observaron tendencias de aumento significativas en la proporción de muestras positivas de C. jejuni en pollos de engorde y pavos, con tasas anuales-anuales de aumento del 9,7 % (IC del 95%: 6,16-13,3%) y del 26,2% (IC del 95%: 15,81–37,43%), respectivamente (Figura 1).
La proporción general de aislados resistentes a CIP, NAL y TET fue extremadamente alta (>80%) tanto en C. coli como en C. jejuni de todas las especies hospedantes (Tabla 3 y Figuras 2-5), con valores anuales superiores al 70% durante todo el período de estudio, excepto en C. jejuni de ganado (Figura 5). Aún así, hubo diferencias significativas en la proporción de aislados resistentes a estos tres antimicrobianos dependiendo del huésped (Tabla 3). La proporción de aislados de C. coli resistentes a CIP y NAL fue significativamente menor en el ganado en comparación con los pollos de engorde y los pavos (p < 0,05) (y en los cerdos en comparación con los pavos para el CIP, p < 0,001). En el caso de TET, los aislados de C. coli de cerdos eran significativamente más resistentes que C. coli de ganado y pollos de engorde (p < 0,001), aunque la resistencia seguía siendo superior al 95 % en todas las especies (Tabla 3). En el caso de C. jejuni, los aislados de ganado eran significativamente menos resistentes a los tres antimicrobianos en comparación con los aislados de aves de corral (p < 0,05).
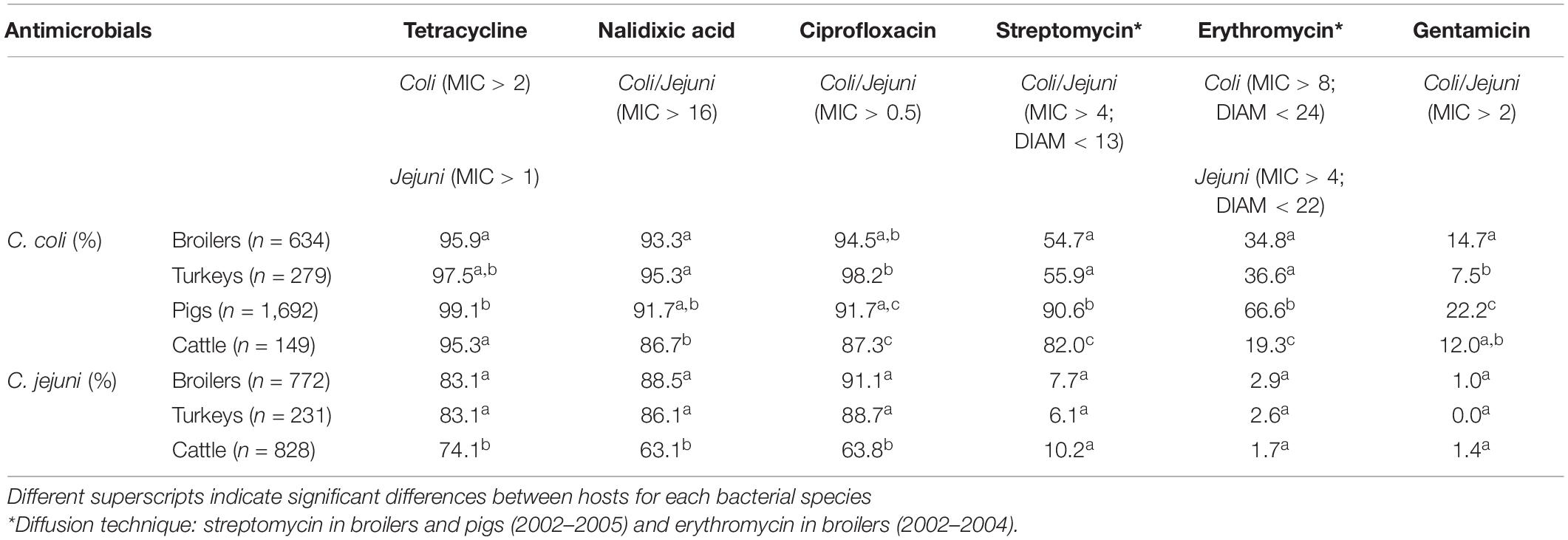 TABLA 3. Porcentaje de aislados de Campylobacter no susceptibles (resistentes) a cada antimicrobiano en las cuatro especies huésped durante el período estudiado.
TABLA 3. Porcentaje de aislados de Campylobacter no susceptibles (resistentes) a cada antimicrobiano en las cuatro especies huésped durante el período estudiado.
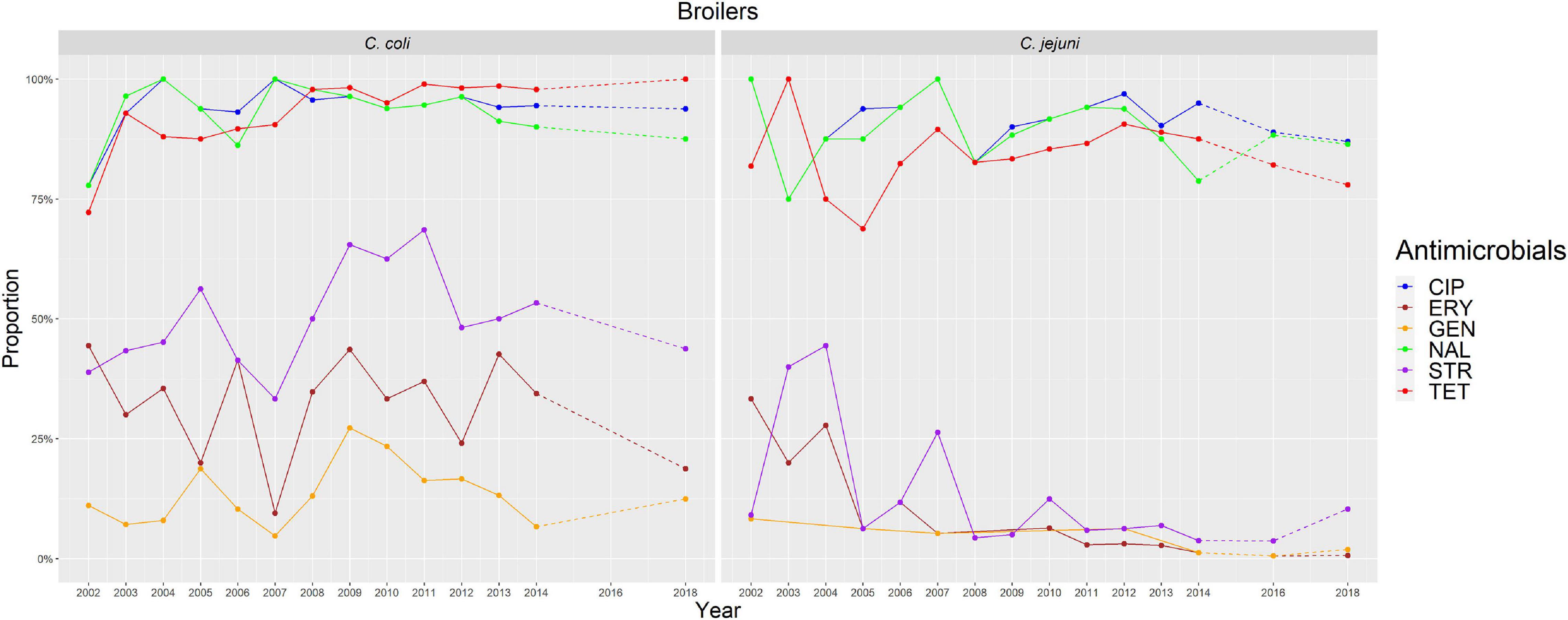 FIGURA 2. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli y C. jejuni de pollos de engorde para 2002-2003. Los años en los que se realizó AST se indican en el eje X; las líneas discontinuas indican períodos en los que no se realizó AST todos los años (no se realizó AST en C. coli en 2016).
FIGURA 2. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli y C. jejuni de pollos de engorde para 2002-2003. Los años en los que se realizó AST se indican en el eje X; las líneas discontinuas indican períodos en los que no se realizó AST todos los años (no se realizó AST en C. coli en 2016).
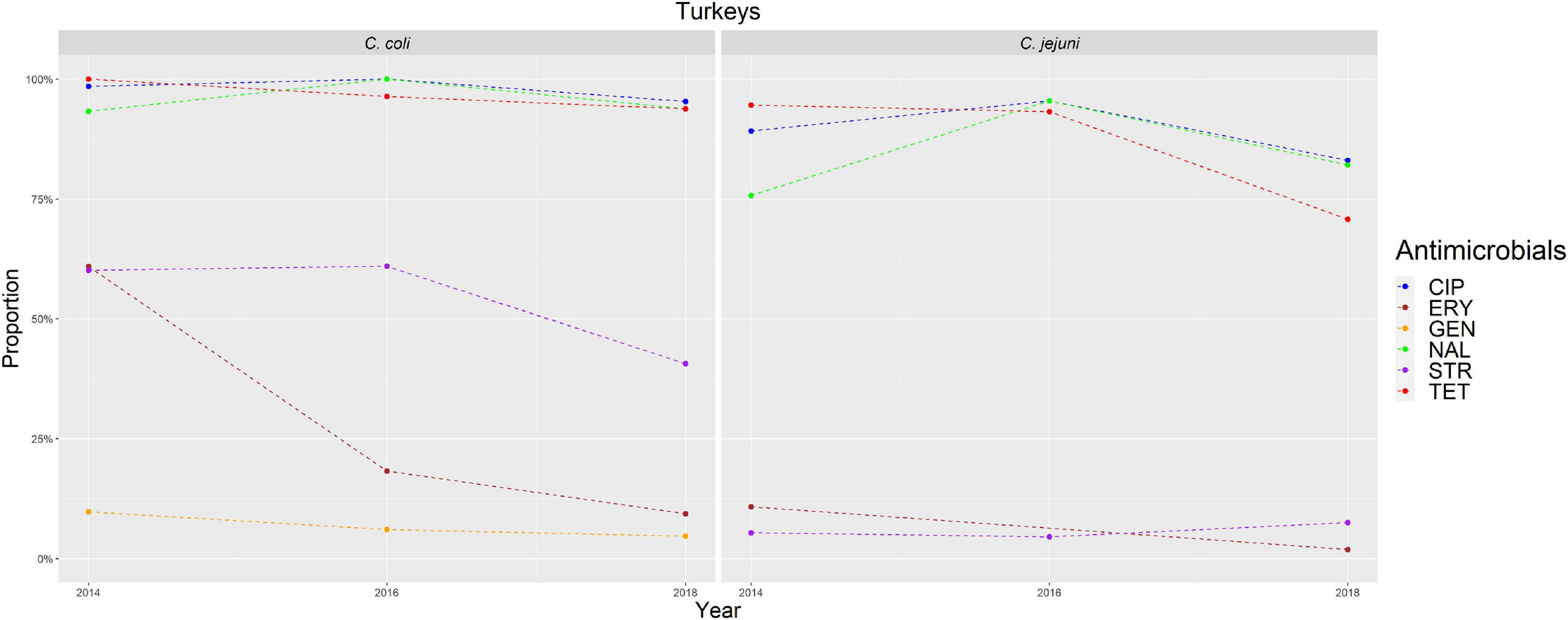 FIGURA 3. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli y C. jejuni de pavos para 2014-2018. Los años en los que se realizó el AST se indican en el eje X; las líneas discontinuas indican los períodos en los que no se realizó el AST todos los años.
FIGURA 3. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli y C. jejuni de pavos para 2014-2018. Los años en los que se realizó el AST se indican en el eje X; las líneas discontinuas indican los períodos en los que no se realizó el AST todos los años.
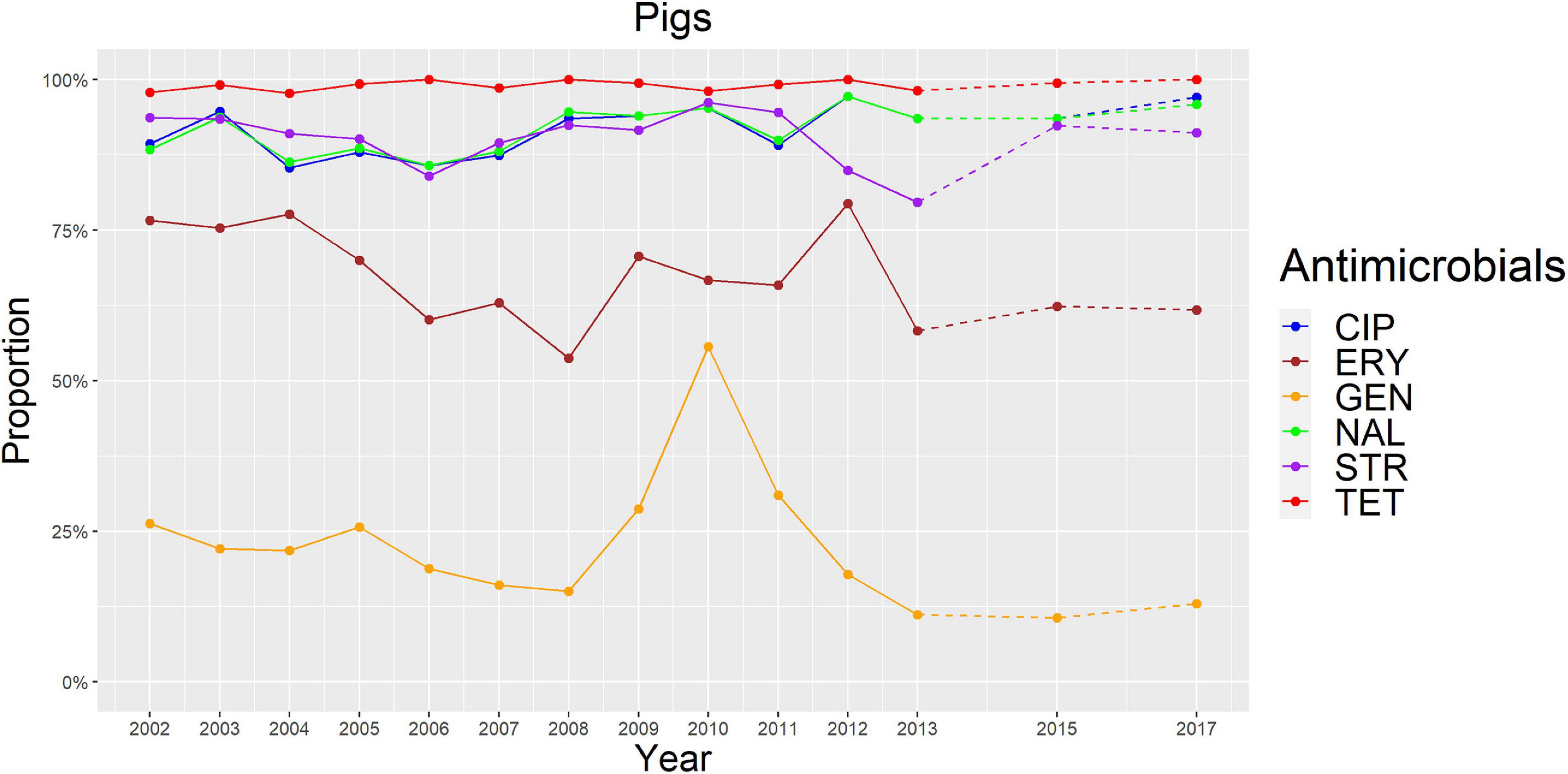 FIGURA 4. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli de cerdos para 2002-2003. Los años en los que se realizó el AST se indican en el eje X; las líneas discontinuas indican los períodos en los que no se realizó el AST todos los años.
FIGURA 4. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli de cerdos para 2002-2003. Los años en los que se realizó el AST se indican en el eje X; las líneas discontinuas indican los períodos en los que no se realizó el AST todos los años.
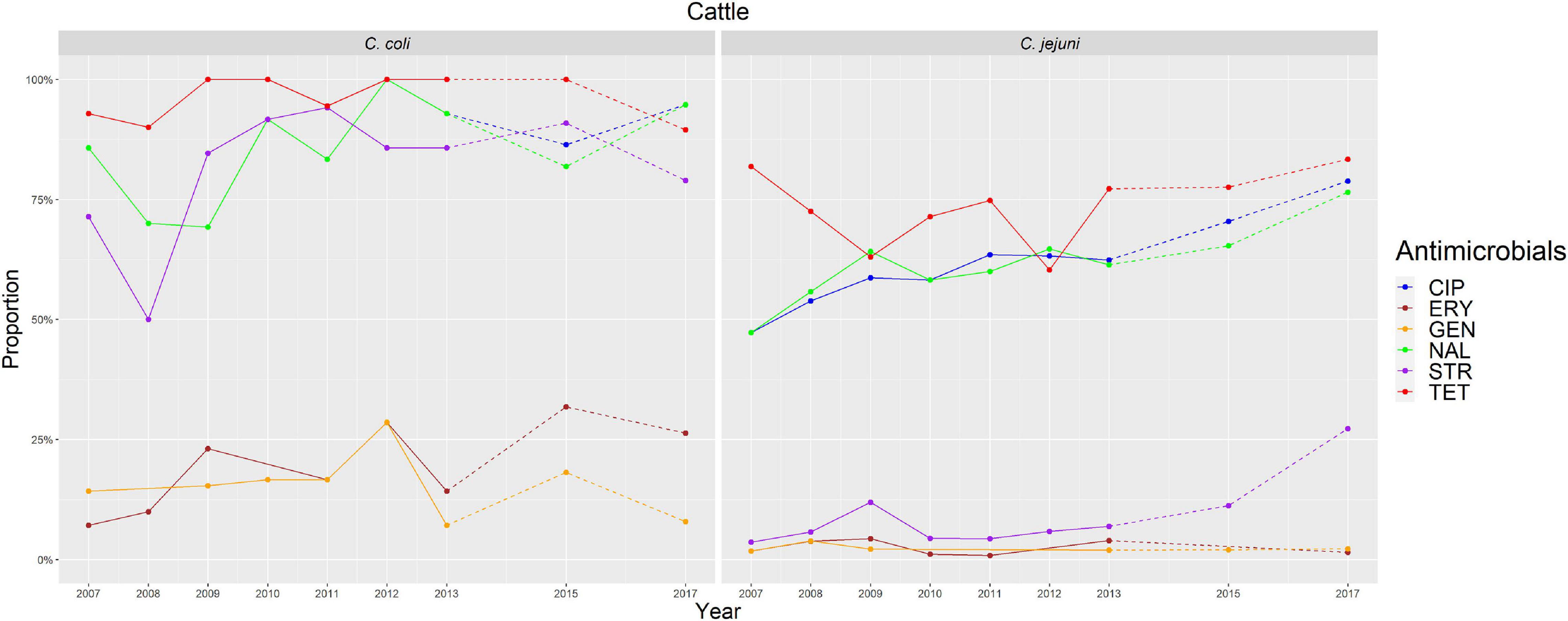 FIGURA 5. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli y C. jejuni del ganado para 2007-2017. Los años en los que se realizó el AST se indican en el eje X; las líneas discontinuas indican los períodos en los que no se realizó el AST todos los años.
FIGURA 5. Representación gráfica de las proporciones de RAM para cada antimicrobiano en C. coli y C. jejuni del ganado para 2007-2017. Los años en los que se realizó el AST se indican en el eje X; las líneas discontinuas indican los períodos en los que no se realizó el AST todos los años.
La proporción de resistencia a los tres antimicrobianos restantes (STR, ERY y GEN) fue mucho más variable (Tabla 3 y Figuras 2-5). En el caso de STR, se encontraron niveles extremadamente altos (80-91%) o de alto a muy alto (∼55%) en C. coli de ganado y cerdos y de pollos de engorde y pavos, respectivamente, con diferencias significativas entre todas las especies de huéspedes, excepto entre pollos de engorde y pavos (cerdos > ganado > aves de corral Por el contrario, se observaron valores <11% en C. jejuni de las tres especies hospedantes de las que se recuperó esta especie bacteriana (bról, pavos y ganado). Aunque los niveles de resistencia en C. jejuni siempre fueron significativamente más bajos que en C. coli para cualquier especie huésped dada, no se observaron diferencias significativas entre las especies huésped.
La proporción de resistencia a ERY en C. coli fue muy alta (67%) para los cerdos, alta (35%) para pollos de engorde y pavos, y moderada (19%) para el ganado (cerdos > aves de corral > ganado) (Tabla 3 y Figuras 2-5). Los valores generales en C. jejuni de todas las especies huésped fueron < 3% y significativamente más bajos que los de C. coli, de nuevo sin diferencias significativas entre los huéspedes.
Finalmente, la proporción de aislados resistentes a GEN fue baja (<25%) en C. coli de todas las especies (Tabla 3 y Figuras 2-5), aunque la proporción en cerdos volvió a ser significativamente más alta que la observada en otras especies huésped (p < 0,001). Los niveles de resistencia en C. jejuni fueron más bajos (<2%) y significativamente diferentes de los observados en C. coli del mismo huésped y, una vez más, no se observaron diferencias significativas entre los huéspedes.
Se observaron tendencias significativas (p < 0,05) en la proporción de aislados resistentes al ganado asociado al aumento de las tasas anuales de ERY y C. coli (29,9%, aunque con un amplio IC95%: −31,36 a 145,77) y STR y C. jejuni (16,7%, IC95%: 2.89-32,28) (Figura 5). También se observaron tendencias significativas (p < 0,001) en la proporción de aislados resistentes de C. jejuni recuperados anualmente de pollos de engorde para dos antimicrobianos, en ambos casos asociados a la disminución de las tasas anuales: STR (−9,9%, IC95%: −16,13 a −3,14) y
Se encontraron otras tendencias significativas (p < 0,001) asociadas con modestas tasas anuales de aumento en C. jejuni en el ganado para el CIP (5,22 %, IC95%: 3,91–6,54) y NAL (4,11 %, IC del 95%: 2,20–6,06), en C. coli en los pollos de engorde para TET (1, Para el resto de los antimicrobianos, las especies huésped y bacterianas no se detectaron tendencias significativas (Figuras 2-5).
Para cualquier especie bacteriana dada, un análisis de los resultados cuantitativos de AST entre las especies de huéspedes reveló diferencias significativas (p = 0,032) en la distribución de los valores de CMI entre los aislados «susceptibles» y «no susceptibles» solo para las cepas de C. jejuni de pavos «no susceptibles» a la ERY (MIC �� Por el contrario, para cualquier especie huésped dada, no se observaron diferencias significativas entre las distribuciones de C. coli «susceptible» y «no susceptible» vs. C. aislados de jejuni.
Perfiles fenotípicos de co-resistencia y MDR
Los principales perfiles de resistencia observados en cada especie bacteriana y huésped se muestran en la Tabla Suplementaria 3. De todos los aislados de C. coli de todas las especies anfitrionas, >85 % eran resistentes a tres (CIP-TET-NAL, TET-ERY-STR) o más antimicrobianos y >60 % eran resistentes a tres o más clases de antimicrobianos (MDR). Los perfiles de resistencia más comunes para C. coli de cada huésped fueron CIP-TET-NAL y CIP-TET-NAL-STR en pollos de engorde y pavos (del 18 % del 28 % de todos los aislados en cada especie huésped para cada perfil), CIP-TET-NAL-STR en ganado (∼ 50 % de todos los aislados de C. La proporción de aislados pan-susceptibles para C. coli en todas las especies huésped fue baja (0-4%), y menor que la proporción de aislados resistentes a los seis antimicrobianos (0–16%).
En comparación, la proporción de aislados de C. jejuni de todos los huéspedes resistentes a tres o más antimicrobianos fue del 54-76%, mientras que la proporción de MDR fue del 6 al9%. El perfil de resistencia más común para C. jejuni de todos los huéspedes fue CIP-TET-NAL, que ascendió a entre el ∼45 % de todo el ganado y el 60-70% de todos los aislados de pollos de engorde y pavos. En este caso, la proporción de aislados sensibles a los pan (5-15 %) fue mayor que la de aislados resistentes a los seis antimicrobianos en todas las especies (<1%).
Asociación entre la resistencia a GEN, STR y ERY
En general, se observó una asociación significativa entre la aparición de resistencia fenotípica a los aminoglucósidos y macrólidos, por lo que C. coli y C. jejuni de todas las especies hospedantes (excepto C. coli en cerdos y pavos y C. jejuni en pavos) resistentes a uno de los dos aminoglucósidos ( Esta asociación fue más fuerte en los aislados de C. jejuni y/o ganado.
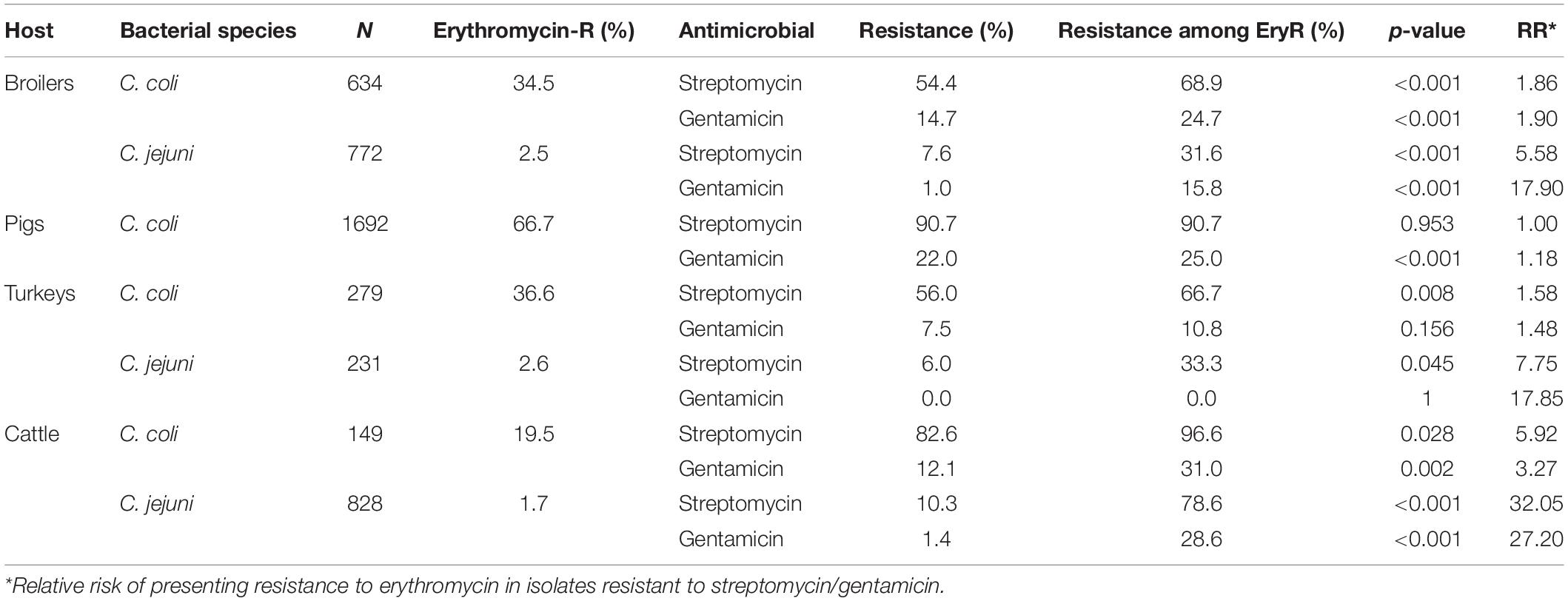 TABLA 4. Asociación entre la resistencia fenotípica a la gentamicina, la estreptomicina y la eritromicina en aislados de C. coli y C. jejuni del ganado.
TABLA 4. Asociación entre la resistencia fenotípica a la gentamicina, la estreptomicina y la eritromicina en aislados de C. coli y C. jejuni del ganado.
Análisis de los genes flaA y AMR
Se determinaron correctamente más de 300 pb (incluido el SVR de 267 pb de largo utilizado en el análisis) de la secuencia del gen flaA en 168 de los 176 aislados elegidos (todos excepto 2 «casos» y 6 «controles»).La figura 6 muestra el árbol filogenético construido utilizando los 168 aislados finales seleccionados (59 «casos» y 109 «controles») más la cepa de referencia. En general, se encontraron un total de 127 polimorfismos de un solo nucleótido (SNP) ubicados en 100 sitios polimórficos, lo que llevó a 73 secuencias únicas de genes flaA SVR. La diversidad de haplotipos (Hd – probabilidad de que dos secuencias seleccionadas al azar sean diferentes) fue de 0,975, y cada dos secuencias diferían en promedio en 27 SNP con una distancia evolutiva media general (d) entre las dos secuencias de 0,09.
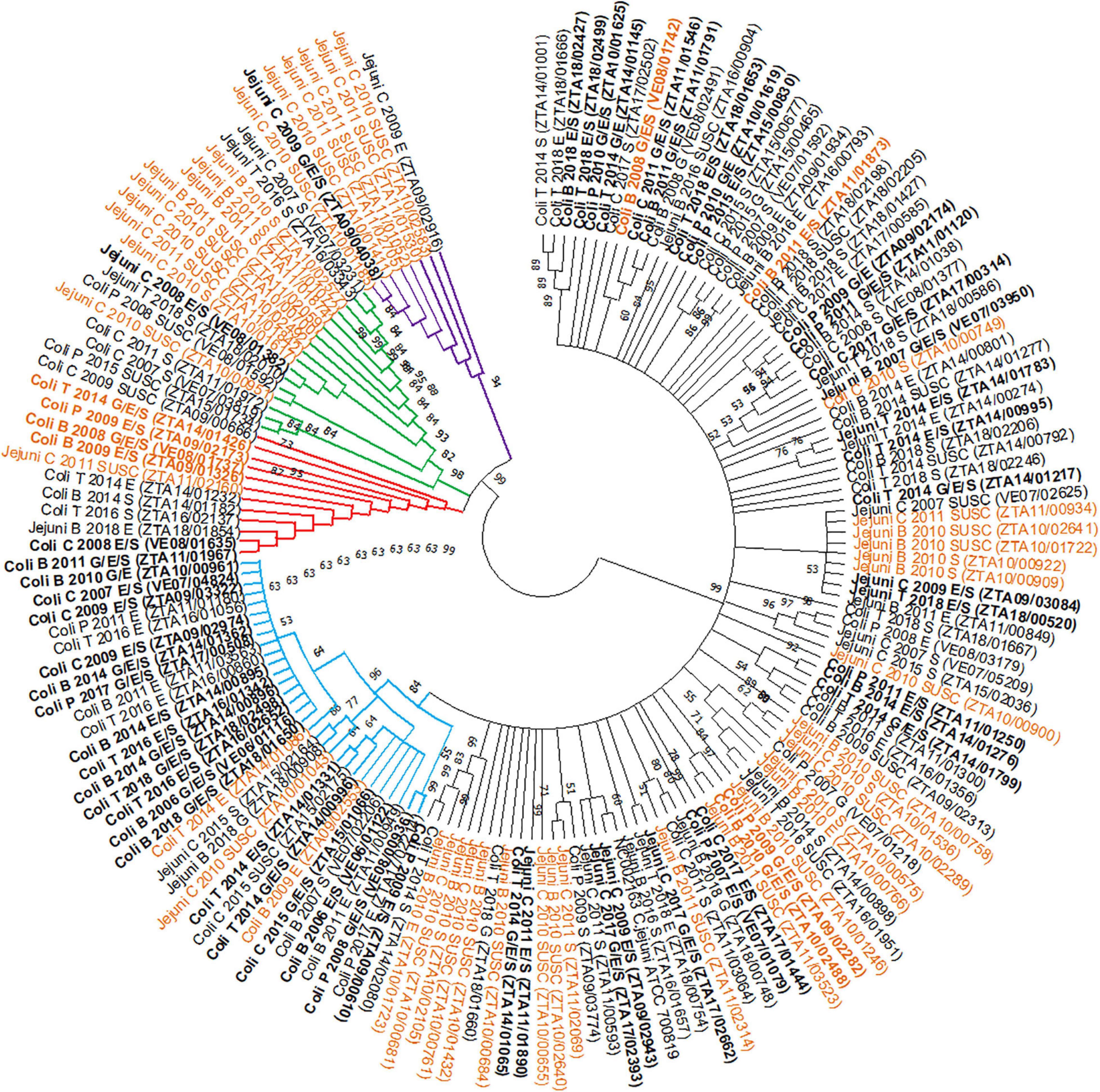 FIGURA 6. Árbol filogenético de la región variable corta (SVR) de genes flaA de 168 aislados seleccionados de Campylobacter, que combinan especies bacterianas, huésped, año de recuperación y fenotipo AMR, obtenido por el método de unión de vecinos [Grupo 1 = negro; Grupo 2 = azul; Grupo 3 = rojo; Grupo 4 = verde; Grupo 5 = violeta; NC002163
FIGURA 6. Árbol filogenético de la región variable corta (SVR) de genes flaA de 168 aislados seleccionados de Campylobacter, que combinan especies bacterianas, huésped, año de recuperación y fenotipo AMR, obtenido por el método de unión de vecinos [Grupo 1 = negro; Grupo 2 = azul; Grupo 3 = rojo; Grupo 4 = verde; Grupo 5 = violeta; NC002163
Los 168 aislados se clasificaron en cinco grupos basados en la topología del árbol: grupo 1, incluyendo la mayoría de los aislados secuenciados (n = 100 aislados), grupo 2 (n = 31 aislados), grupo 3 (n = 12 aislados), grupo 4 (n = 17 aislados) y grupo 5 (n = 8 aislados) (Figura 6). Los grupos 2 a 5 formaron clados separados del grupo 1 (alfal de arranque > 60). Los grupos 2 y 3, que consisten principalmente en cepas de C. coli (28/31 y 10/12, respectivamente) predominantemente de aves de corral (>60%), mostraron proporciones similares de aislados resistentes a aminoglucósidos y macrólidos («casos») y de «controles» (18/31 y 6/12, respectivamente) (Tabla 5). Los grupos 4 y 5 mostraron una mayor proporción de aislados de C. jejuni (13/17 en el grupo 4 y 8/8 en el grupo 5) de ganado (9/17 y 8/8), y la frecuencia de aislados con resistencia simultánea a ambas clases de antimicrobianos («casos») en estos grupos fue mucho menor (1/17 y 1/8, respectivamente) (Tabla 5).
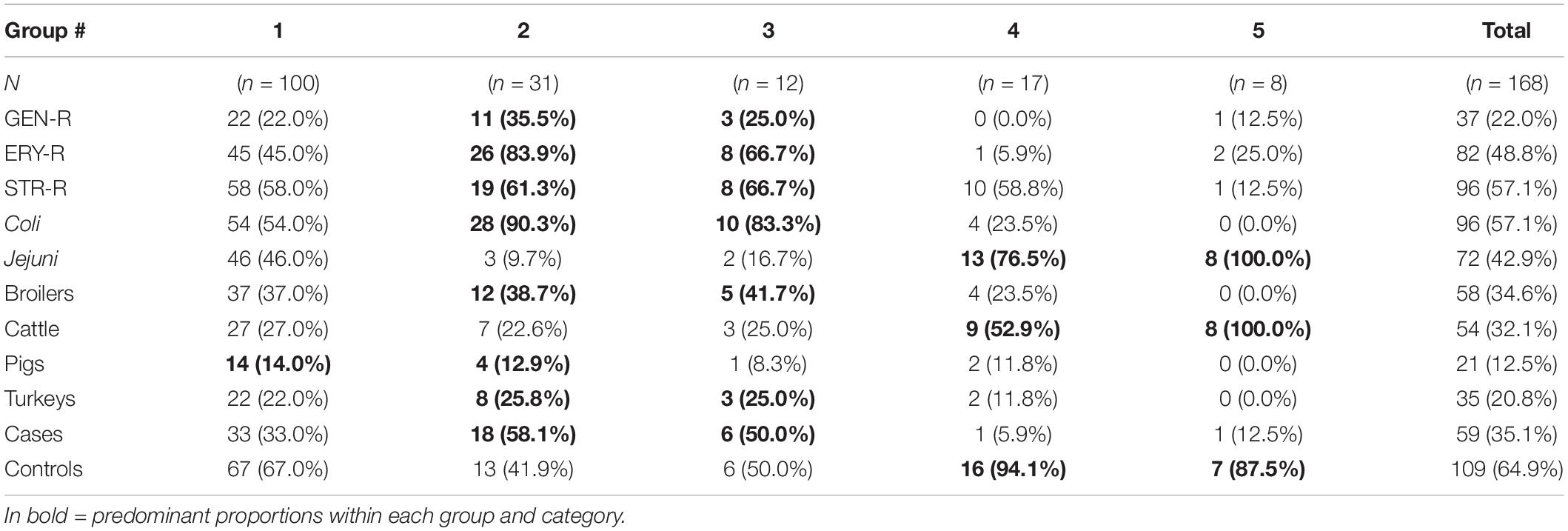 TABLA 5. Números y proporciones de aislados incluidos en cada uno de los grupos formados a partir del análisis filogenético basado en la susceptibilidad fenotípica de la resistencia a la resistencia a los antimicrobianos, las especies bacterianas, las especies huésped y las categorías de casos/controles.
TABLA 5. Números y proporciones de aislados incluidos en cada uno de los grupos formados a partir del análisis filogenético basado en la susceptibilidad fenotípica de la resistencia a la resistencia a los antimicrobianos, las especies bacterianas, las especies huésped y las categorías de casos/controles.
Solo se incluyeron en el análisis de MCA los valores completos de RSCU de los 20 codones variables entre los 168 aislados. Las dos primeras dimensiones identificadas en el MCA explicaron el 38 % de la variabilidad total observada. Los aislados incluidos en cada uno de los cinco grupos identificados en el árbol filogenético también se agruparon de acuerdo con las dos primeras dimensiones del MCA (Figura 7).
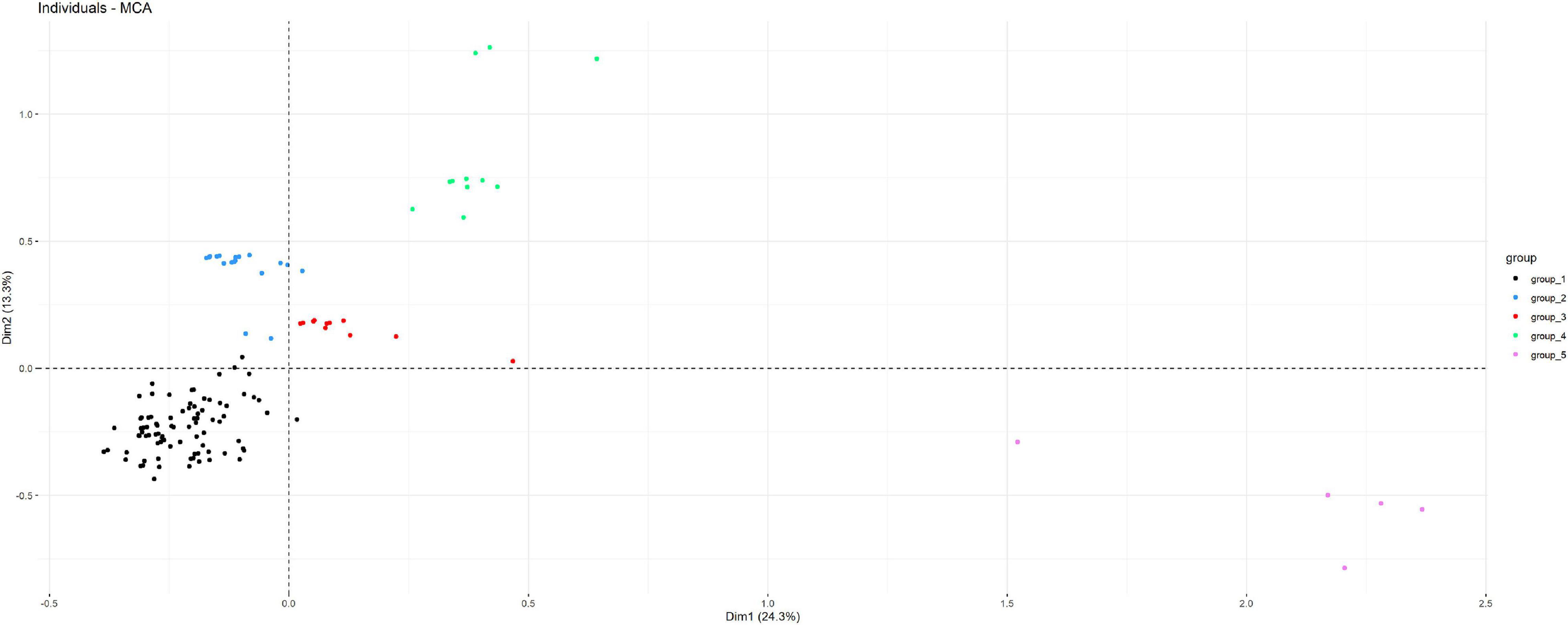 FIGURA 7. Distribución de los 168 aislados sometidos a la secuenciación de flaA gen de acuerdo con las dos primeras dimensiones de un análisis de correspondencia múltiple (MCA) realizado teniendo en cuenta la información sobre la RSCU de codones variables, especies bacterianas y huésped, resistencia a la gentamicina, eritromicina y estreptomicina, y clado según lo determinado en el árbol filogenético de
FIGURA 7. Distribución de los 168 aislados sometidos a la secuenciación de flaA gen de acuerdo con las dos primeras dimensiones de un análisis de correspondencia múltiple (MCA) realizado teniendo en cuenta la información sobre la RSCU de codones variables, especies bacterianas y huésped, resistencia a la gentamicina, eritromicina y estreptomicina, y clado según lo determinado en el árbol filogenético de
Se encontraron diferentes marcadores de resistencia involucrados en la AMR contra macrólidos y aminoglucósidos en los aislados sometidos a WGS. Varios genes involucrados en la bomba de eflujo CmeABC (cmeB, cmeC y cmeR) estaban presentes en los aislados secuenciados de C. jejuni 39-40/40 y faltaban en las cepas de C. coli 10-11/11, mientras que el gen cmeA estaba presente en 34/40 y 4/11 de las cepas de C. je Entre los 12 aislados resistentes a ERY, tres presentaron mutaciones asociadas con la resistencia a los macrólidos en el gen de codificación de ARNr 23S y otros tres llevaban el gen erm(B), mientras que no se encontró ningún marcador de resistencia en los seis aislados restantes. Con respecto a los genes asociados a la resistencia a los aminoglucósidos, se encontraron siete genes diferentes en una o más cepas (entre 0 y 3 por cepa), y su presencia se asoció con resistencia a STR y/o GEN, excepto en una cepa susceptible de C. jejuni de ganado (Archivo Excel complementario 2). No se observó ninguna asociación aparente entre un grupo de genes flaA específico y la presencia de ninguno de los marcadores de resistencia.
Discusión
La resistencia a los antimicrobianos se está convirtiendo en un problema importante para el tratamiento de enfermedades causadas por bacterias zoonóticas como el Campylobacter termófilo. Los mecanismos por los que la RAM puede propagarse en una población bacteriana (vertical o horizontalmente) tienen enormes implicaciones, ya que puede determinar la velocidad a la que se difunden los fenotipos de la RAM. De particular preocupación son los rasgos genéticos que confieren MDR (Magiorakos et al., 2012), particularmente cuando se transmiten juntos. Por lo tanto, es de suma importancia explorar los mecanismos genéticos implicados en la RAM en C. coli y C. jejuni de los diferentes huéspedes involucrados en la epidemiología de la infección en humanos (EFSA-ECDC, 2020). Como se describe en otra parte, este estudio de datos fenotípicos basados en aislados frente a datos agregados también ha demostrado ser un medio confiable para obtener información sobre dichos mecanismos (Alvarez et al., 2020), y la evaluación de los patrones de susceptibilidad fenotípica que se encuentran aquí puede guiar el análisis genético en un enfoque «de arriba hacia abajo» (Shepp
La proporción anual de muestras positivas de Campylobacter encontradas en nuestras muestras de pollos de engorde recogidas durante un período de 17 años (que va del 26,2 al 76,7%) fue mayor que los valores notificados por la EFSA de los estados miembros de la UE (26 %) (EFSA-ECDC, 2019a) y en su mayoría más altas que los valores notificados en La proporción de muestras positivas de Campylobacter en ganado de 2007 a 2017 (37-69,5%) también fue mucho más alta que los valores notificados por la EFSA para 10 países de la UE (1,5-3,5%) (EFSA-ECDC, 2018b, 2019a), aunque los estudios específicos de Finlandia (Hakkinen et al., 2007) y Lituania (Ramonaite et Del mismo modo, el porcentaje de muestras de cerdo de las que se recuperaron los aislados de Campylobacter en nuestro estudio (3,3,80%) estaba en el rango de resultados reportados para Grecia (49,1%) (Papadopoulos et al., 2020), mucho más alto que el reportado anteriormente por la EFSA para 8 países de la UE (2-7%) (EFSA-E Sin embargo, los porcentajes que encontramos en los pavos (65,4–85,9%) fueron similares a un informe de la UE que comprendía 5 países (71,6%) (EFSA-ECDC, 2019a), pero inferiores a los encontrados en un estudio alemán (90-100%) (Ahmed et al., 2016).
Como era de esperar, las especies huésped estaban fuertemente asociadas con las especies de Campylobacter recuperadas en muestras positivas, aunque las proporciones encontradas para cada especie bacteriana pueden variar dependiendo de los protocolos de aislamiento utilizados. El predominio de C. coli en las muestras de cerdos encontradas en nuestro estudio está de acuerdo con estudios anteriores de Dinamarca (Boes et al., 2005). Sin embargo, y a pesar de que esta especie bacteriana se ha asociado tradicionalmente con cerdos, se está volviendo más común en las aves de corral (Miller et al., 2006). En nuestra colección, C. coli fue de hecho la especie predominante en el pavo, mientras que se informó de una distribución más equilibrada entre C. coli y C. jejuni en muestras de pavo de Alemania (Ahmed et al., 2016). En los pollos de engorde, también se observó una distribución cercana al 50/50 para C. coli/C. jejuni, como la que se encuentra aquí, también se observó en muestras de China (Tang et al., 2020). Sin embargo, la EFSA informó de un predominio de C. jejuni con 2.452 muestras positivas de Campylobacter de 16 países (EFSA-ECDC, 2019a). En el ganado, un estudio de Dinamarca (Nielsen et al., 1997) encontró proporciones similares para cada especie bacteriana (6,8% C. coli, 90,9% C. jejuni y 2,3% C. spp.) que nuestro estudio (14,4% C. coli, 84,4% C. jejuni y 1,2% C. spp.).
Como se supone, el nivel de resistencia a los antimicrobianos utilizados en nuestro estudio estaba estrechamente relacionado con las especies bacterianas de Campylobacter encontradas, con niveles más altos de resistencia en C. coli que en C. jejuni de acuerdo con la investigación anterior (Pergola et al., 2017; Alvarez et al., 2020). La falta de barreras para la transferencia horizontal de genes (HGT) en C. coli puede explicar los niveles más altos de MDR observados en esta especie bacteriana en comparación con C. jejuni (Pearson et al., 2015).
De los seis antimicrobianos evaluados aquí, se encontraron niveles de resistencia de alto a extremadamente altos para tres de ellos (CIP, NAL y TET) en C. coli, mientras que en C. jejuni oscilaron entre medio y muy alto. Para CIP y NAL, estos niveles fueron consistentemente más altos que los descritos para los aislados de animales de comida en otros países europeos, con la excepción de C. jejuni en pavos (niveles iguales a Italia, Polonia y Portugal al 70 %) y al ganado (niveles iguales a Italia al 80 %) (EFSA-ECDC, 2020). Para TET en C. jejuni en el ganado, los niveles en España (85 %) se mantuvieron entre los niveles notificados en Austria, Dinamarca y los Países Bajos (60 %) y los niveles notificados en Italia (95 %) (EFSA-ECDC, 2020).
Los niveles de resistencia a los tres antimicrobianos restantes analizados en este estudio fueron mucho más variables, una vez más consistentemente más altos que en otros países europeos entre los huéspedes y las especies bacterianas. Las excepciones fueron C. jejuni de pavos (15% de ERY en Portugal vs. 2,6 % en España; 20 % STR en Polonia frente a 6,1% en España) y ganado (10 % ERY en Italia vs. 1,7 % en España) (EFSA-ECDC, 2020).
En general, se encontró consistentemente una asociación significativa entre la presentación de resistencia fenotípica a ERY (macrolida) y STR y GEN (aminoglucósidos) tanto para C. coli como para C. jejuni de la mayoría de las especies huésped (Tabla 3). Cuando se analizó la asociación entre pares de antimicrobianos estratificando por períodos de tiempo (2002-2006, 2007-2012 y 2013-2018), ciertas categorías no se asociaron significativamente, probablemente debido a que los tamaños de muestra más pequeños (los datos no se muestran). Como era de esperar, los aislados resistentes a STR tenían una probabilidad significativamente mayor de ser también resistentes a GEN, lo que se esperaba dado que pertenecen a la misma clase de antimicrobianos (aminoglucósidos) y, por lo tanto, comparten mecanismos de resistencia, principalmente basados en la transformación natural, la recombinación homóloga y el intercambio de MGE (Davi
Campylobacter se considera un patógeno de alto riesgo en términos de AMR debido a los altos niveles de HGT y la asociación de los genes AMR en los MDRGI. Algunos autores argumentan que es probable que la transferencia de MDRGI conduzca a fenómenos de coelección después de su movilización genética. Esto podría explicar por qué Campylobacter se adapta tan rápidamente en su interacción con el huésped, obteniendo constantemente fenotipos mejorados (Sheppard y Maiden, 2015).
El gen erm(B), descrito anteriormente solo en Asia (Qin et al., 2014) y posiblemente originario de bacterias Gram-positivas, se encontró en España en un C. coli de pollo de engorde en 2015 (Florez-Cuadrado et al., 2016) y dos C. coli de pavos en 2017 (Florez-Cuadrado et al., Este fue el primer informe europeo de este gen, asociado con otros genes en MDRGI que tienen resistencia a ERY, CIP, TET y NAL, e involucrados en AMR a STR (y presente en aislados que pueden ser susceptibles a GEN). Las tres cepas portadoras de erm(B), incluidas en este estudio, se encontraron en aislados que mostraban resistencia simultánea a los aminoglucósidos y se agruparon en diferentes clados (1, 2 y 3). Sin embargo, dado el número muy limitado de cepas secuenciadas resistentes a ERY, no se pueden sacar conclusiones con respecto a su asociación con poblaciones genéticas específicas. La inclusión de genes erm(B) en plásmidos que codifican genes de resistencia adicionales a otros antibióticos en C. coli de animales de comida podría allanar el camino para una rápida diseminación de la resistencia a los macrólidos (EFSA-ECDC, 2018a, 2019b). Además, los niveles de resistencia a ERY notificados en humanos han sido consistentemente más altos para C. coli que para C. jejuni (EFSA-ECDC, 2018a), y se han hecho informes similares en aves de corral (Pergola et al., 2017) de acuerdo con nuestros hallazgos. Dado que los macrólidos son una de las tres clases de «microbianos de Importancia Crítica» utilizadas para el tratamiento de la campilobacteriosis humana (junto con fluoroquinolonas y aminoglucósidos) (Organización Mundial de la Salud (OMS), 2017), se justifica un conocimiento más profundo de sus mecanismos de resistencia.
Las crecientes tasas de resistencia a la ERY en C. coli y a la STR en C. jejuni de origen bovino descritas aquí sugieren que esta especie huésped podría desempeñar un papel cada vez más importante en la epidemiología de la AMR inCampylobacter. Un estudio de casos y controles a nivel nacional realizado en Luxemburgo identificó el consumo de carne de vacuno como una fuente importante de infección por C. coli (Mossong et al., 2016), lo que sugiere que el ganado puede ser un reservorio relevante para este patógeno transmitido por los alimentos.
Los valores de las CMI en los aislados clasificados como «susceptibles» o «no susceptibles» pueden indicar la presencia o ausencia de diferentes determinantes de la RAM en el genoma bacteriano. Los valores de MIC significativamente más altos observados en este estudio para ERY en aislados resistentes al pavo (Archivo Excel complementario 1) podrían indicar la presencia del gen erm(B) transferible. Sin embargo, de los 12 aislados resistentes a ERY sometidos a WGS, solo tres llevaban el gen erm(B) (y tenían MICs que oscilaban entre 32 y 256 ug/ml), y, además, se encontraron mutaciones en el gen de codificación 23S rRNA en solo tres aislados (Archivo complementario Excel 2). Esto sugiere que otros mecanismos pueden estar involucrados en el aumento observado de los CMI en ciertos aislados, como la resistencia a la mutacional que afecta a la expresión de la bomba de eflujo CmeABC en C. jejuni (Zhang et al., 2017). Esto, relacionado con la alta proporción de aislados resistentes a ERY encontrados en C. coli de pavos en otros países europeos (EFSA-ECDC, 2017, 2020), pone de relieve aún más la necesidad de aclarar los mecanismos de resistencia presentes en los aislados resistentes de este huésped. De hecho, la EFSA recomienda investigar los mecanismos moleculares de la resistencia a los macrólidos, especialmente en aislados resistentes a altas concentraciones de ERY, con el fin de detectar mutaciones cromosómicas o la presencia del gen erm(B) transferible (EFSA-ECDC, 2019b). Además, estas mismas pautas recomiendan buscar genes resistentes a ERY, no solo en cepas resistentes que presenten resistencia concomitante a los aminoglucósidos o a un fenotipo MDR, sino también en aislados susceptibles. Por lo tanto, se necesitaría una caracterización profunda de los aislados resistentes para confirmar esta hipótesis. La integración de análisis fenotípicos y genómicos puede permitir predecir diferencias en los niveles de resistencia más allá de los umbrales de resistencia (Bolinger y Kathariou, 2017; EFSA-ECDC, 2019b).
Los estudios filogenéticos basados en la secuenciación del gen flaA SVR se han utilizado en el pasado para estudiar la epidemiología de Campylobacter spp. de diferentes fuentes (Zhang et al., 2018). Anteriormente, los estudios basados en la secuencia génica flaA no habían encontrado una relación entre la RAM y los genotipos específicos (Corcoran et al., 2006). Sin embargo, en nuestra colección de cepas se identificaron cinco grupos distintos, dos de los cuales se asociaron con una mayor proporción de resistencia simultánea a los aminoglucósidos y macrólidos (grupos 2 y 3), formados predominantemente por aislados de C. coli de pollos de engorde y pavos (Tabla 5). Por el contrario, los aislados de los grupos 4 y 5 eran principalmente C. jejuni de origen bovino. Se ha especulado anteriormente sobre la existencia de «lineados C. jejuni especialistas en ganado», lo que implica que la adaptación de C. jejuni al ganado podría estar asociada con la presencia de elementos genéticos que favorezcan su supervivencia en el intestino del ganado (Sheppard y Maiden, 2015) y con una ganancia y pérdida genética significativa (Mourkas et al., 2020) Curiosamente, el ganado C. jejuni mostró la asociación más fuerte entre la resistencia a los aminoglucósidos y los macrólidos (con RR > 25), pero solo 2 aislados co-resistentes (de los 7 aislados de ganado C. jejuni coresistente secuenciados) se clasificaron en los grupos 4 y 5.
Entre sus limitaciones, el tamaño de la muestra utilizado en la primera parte de este largo período de estudio (2002-2008) fue relativamente pequeño. Además, AST en los primeros años (2002-2005) se basó en la determinación de los IZD y los MIC en función del antimicrobiano considerado. Por lo tanto, las conclusiones basadas en datos de ese período deben interpretarse cuidadosamente. Además, solo el 1,5 % (168) del número total de aislados se sometió al análisis del gen flaA, y solo 51 de ellos fueron analizados por WGS.
Sin embargo, nuestros hallazgos confirman que se observaron constantemente altos niveles de resistencia en Campylobacter spp. de animales productores de alimentos, y que la resistencia a los macrólidos y aminoglucósidos estaba fuertemente asociada entre los huéspedes y las especies bacterianas. Se necesitarían más estudios basados en WGS para determinar los determinantes genéticos detrás de esta resistencia y la posible existencia de linajes más prevalentes.
En este estudio, los aislados de C. coli, más frecuentes en cerdos y aves de corral (especialmente pavos), mostraron niveles de resistencia significativamente más altos que las cepas de C. jejuni. La asociación significativa en la presentación simultánea de la resistencia fenotípica a los aminoglucósidos y macrólidos, confirmada en los aislados de C. coli independientemente de las especies hospedantes de origen, sugiere la posible circulación de genes de resistencia contra ambas clases de antimicrobianos. Tales mecanismos de resistencia podrían haberse transmitido juntos o, de lo contrario, se habían difundido a través de clones resistentes en las especies ganaderas estudiadas. El análisis genético reveló la presencia de algunos aislados más genéticamente relacionados con fenotipos resistentes en aves de corral y otros con fenotipos susceptibles en el ganado. Para probar estas hipótesis, sería necesario caracterizar los mecanismos de resistencia presentes en los aislados de las diferentes especies mediante un análisis molecular exhaustivo de todo su genoma.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio se incluyen en el artículo/Material complementario, se pueden dirigir más consultas al autor correspondiente.
Declaración ética
No se requirió una revisión y aprobación ética porque los animales incluidos en este estudio fueron muestreados en el matadero durante el procesamiento rutinario del ganado y no fueron sometidos a ningún manejo adicional de ningún tipo. Las muestras se recogieron en el marco de los programas oficiales de supervisión de acuerdo con la legislación nacional y de la UE.
Contribuciones del autor
VL-C, LD y JA: conceptualización. VL-C y JA: investigación, redacción: preparación del borrador original, metodología, software, análisis formal y validación. JA: Adquisición de fondos y administración y supervisión de proyectos. JS, CF, TS, IP, MU-R, CB y MG: suministro de datos. MU-R, CB y MG: curación de datos. VL-C y AO: trabajo de laboratorio. VL-C, MU-R, CB, AO, MG, JS, IP, MM, LD y JA: escritura: revisión y edición. Todos los autores contribuyeron al artículo y aprobaron la versión presentada.
Financiación
Este trabajo fue apoyado por la financiación del programa de Investigación e Innovación Horizonte 2020 de la Unión Europea en virtud del acuerdo de subvención No 773830: Programa Conjunto Europeo de Salud y es una contribución al Proyecto de Investigación Conjunta DiSCoVeR.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Agradecimientos
Nos gustaría dar las gracias a la ayuda de los técnicos de laboratorio Estefanía Martínez, Nisrin Maasoumi y Estefanía Rivero de la «Unidad de Zoonosis y Resistencia a los Antibióticos de Transmisión Alimentaria» (ZTA), y Bernabé Diéguez y Pilar Pozo del Centro de Vigilancia de Salud Veterinaria VISAVET por su ayuda.
Material complementario
El material complementario de este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fmicb.2021.689262/full#supplementary-material
Nota a pie de página
- ^ EUCAST-Sociedad Europea de Microbiología Clínica y Enfermedades Infecciosas, MIC y distribuciones de zonas y ECOFF http://www.eucast.org/mic_distributions_and_ecoffs/ (consultado el 14/02/2020).
Referencias
Aarestrup, F. M. (2005). Resistencia a los antimicrobianos en bacterias de origen animal. Washington, DC: Sociedad Americana de Microbiología. doi: 10.1128/9781555817534
Aarestrup, F. M. y Wegener, H. C. (1999). Los efectos del uso de antibióticos en animales destinados a la alimentación en el desarrollo de resistencia a los antimicrobianos de importancia para los seres humanos en Campylobacter y Escherichia coli. Infectar microbios. 1, 639-644. doi: 10.1016/s1286-4579(99)80064-1
Ahmed, M. F., El-Adawy, H., Hotzel, H., Tomaso, H., Neubauer, H., Kemper, N., et al. (2016). Prevalencia, genotipado y factores de riesgo de propagación de Campylobacter termófila en granjas de pavos orgánicos en Alemania. Gut Pathog.8:28. doi: 10.1186/s13099-016-0108-2
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Alvarez, J., Lopez, G., Muellner, P., De Frutos, C., Ahlstrom, C., Serrano, T., et al. (2020). Identificar las tendencias emergentes en la resistencia a los antimicrobianos utilizando datos de vigilancia de Salmonella en aves de corral en España. Transbound. Emerg. Dis. 67, 250-262. doi: 10.1111/tbed.13346
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
An, J. U., Ho, H., Kim, J., Kim, W. H., Kim, J., Lee, S., et al. (2018). Ganado lechero, un posible reservorio de campilobacteriosis humana: caracterización epidemiológica y molecular de Campylobacter jejuni de granjas ganaderas.Frente. Microbiol. 9:3136. doi: 10.3389/fmicb.2018.03136
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Boes, J., Nersting, L., Nielsen, E. M., Kranker, S., Enoe, C., Wachmann, H. C., et al. (2005). Prevalencia y diversidad de Campylobacter jejuni en rebaños de cerdos en granjas con y sin ganado o aves de corral. J. Food Prot. 68, 722-727. doi: 10.4315/0362-028x-68.4.722
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Bolinger, H., y Kathariou, S. (2017). El estado actual de la resistencia a los macrólidos en Campylobacter spp.: tendencias e impactos de los mecanismos de resistencia. Appl. Environ. Microbiol. 83, e416-e417. doi: 10.1128/AEM.00416-17
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Bortolaia, V., Kaas, R. S., Ruppe, E., Roberts, M. C., Schwarz, S., Cattoir, V., et al. (2020). ResFinder 4.0 para predicciones de fenotipos a partir de genotipos. J. Antimicrobiano. Madre. 75, 3491–3500. doi: 10.1093/jac/dkaa345
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Boysen, L., Rosenquist, H., Larsson, J. T., Nielsen, E. M., Sorensen, G., Nordentoft, S., et al. (2014). Atribución de la fuente de campilobacteriosis humana en Dinamarca. Epidemiol. Infectar. 142, 1599–1608. doi: 10.1017/s0950268813002719
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Charif, D., y Lobry, J. R. (2007). «SeqinR 1.0-2: un paquete aportado al proyecto r para la computación estadística dedicado a la recuperación y el análisis de secuencias biológicas», en Structural Approaches to Sequence Evolution, eds M. Oporto, EE. UU. Bastolla, H. E. Roman y M. Vendruscolo (Berlín: Springer). doi: 10.1007/978-3-540-35306-5_10
Corcoran, D., Quinn, T., Cotter, L., Whyte, P. y Fanning, S. (2006). Perfilado de resistencia antimicrobiana y tipificación de fla de Campylobacter spp. termofilílico irlandés de origen humano y avícola. Lett. Appl. Microbiol. 43, 560-565. doi: 10.1111/j.1472-765x.2006.01987.x
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Davies, J. y Wright, G. D. (1997). Resistencia bacteriana a los antibióticos aminoglucósidos. Tendencias Microbiol. 5, 234–240. doi: 10.1016/s0966-842x(97)01033-0
CE (2013). Decisión de ejecución de la Comisión de 12 de noviembre de 2013 sobre el seguimiento y la notificación de la resistencia a los antimicrobianos en bacterias zoonóticas y comensal (notificada en el documento C (2013) 7145). Oficial J. Eur. Unión L 303, 26-39.
Edgar, R. C. (2004). MÚSCULO: alineación de secuencias múltiples con alta precisión y alto rendimiento. Ácidos nucleicos Res. 32, 1792–1797. doi: 10.1093/nar/gkh340
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
EFSA (2005). El informe resumido de la Comunidad sobre las tendencias y fuentes de zoonosis, agentes zoonóticos y resistencia a los antimicrobianos en la Unión Europea en 2004. EFSA J. 3:310ar. doi: 10.2903/j.efsa.2005.310ar
EFSA (2006). El informe resumido de la Comunidad sobre las tendencias y fuentes de zoonosis, agentes zoonóticos, resistencia a los antimicrobianos y brotes transmitidos por los alimentos en la Unión Europea en 2005. EFSA J. 4:94r. doi: 10.2903/j.efsa.2006.94r
EFSA (2007). El informe resumido de la Comunidad sobre las tendencias y fuentes de zoonosis, agentes zoonóticos, resistencia a los antimicrobianos y brotes transmitidos por los alimentos en la Unión Europea en 2006. EFSA J. 5:130r. doi: 10.2903/j.efsa.2007.130r
EFSA-ECDC (2016). El informe resumido de la Unión Europea sobre la resistencia a los antimicrobianos en bacterias zoonóticas e indicadoras de seres humanos, animales y alimentos en 2014. EFSA J. 14:4380. doi: 10.2903/j.efsa.2016.4380
EFSA-ECDC (2017). El informe resumido de la Unión Europea sobre la resistencia a los antimicrobianos en bacterias zoonóticas e indicadoras de seres humanos, animales y alimentos en 2015. EFSA J. 15:e04694.
EFSA-ECDC (2018a). El informe resumido de la Unión Europea sobre la resistencia a los antimicrobianos en bacterias zoonóticas e indicadoras de seres humanos, animales y alimentos en 2016. EFSA J. 16:e05182. doi: 10.2903/j.efsa.2018.5182
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
EFSA-ECDC (2018b). El informe resumido de la Unión Europea sobre las tendencias y fuentes de zoonosis, agentes zoonóticos y brotes transmitidos por los alimentos en 2017. EFSA J. 16:e05500. doi: 10.2903/j.efsa.2018.5500
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
EFSA-ECDC (2019a). El Informe de Zoonosis One Health 2018 de la Unión Europea. EFSA J. 17:e05926. doi: 10.2903/j.efsa.2019.5926
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
EFSA-ECDC (2019b). El informe resumido de la Unión Europea sobre la resistencia a los antimicrobianos en bacterias zoonóticas e indicadoras de seres humanos, animales y alimentos en 2017. EFSA J. 17:e05598. doi: 10.2903/j.efsa.2019.5598
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
EFSA-ECDC (2020). El informe resumido de la Unión Europea sobre la resistencia a los antimicrobianos en bacterias zoonóticas e indicadoras de seres humanos, animales y alimentos en 2017/2018. EFSA J. 18:e06007. doi: 10.2903/j.efsa.2020.6007
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Florez-Cuadrado, D., Ugarte-Ruiz, M., Meric, G., Quesada, A., Porrero, M. C., Pascoe, B., et al. (2017). La comparación del genoma de campylobacter resistente a la eritromicina de Turquía identifica los huéspedes y las vías para la propagación horizontal de los genes erm(B). Frente. Microbiol. 8:2240. doi: 10.3389/fmicb.2017.02240
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Florez-Cuadrado, D., Ugarte-Ruiz, M., Quesada, A., Palomo, G., Domínguez, L. y Porrero, M. C. (2016). Descripción de un aislado de Campylobacter coli con erm(B) en Europa. J. Antimicrobiano. Madre. 71, 841–843. doi: 10.1093/jac/dkv383
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Friedrich, M. J. (2019). Resistencia a los antimicrobianos en aumento de las bacterias zoonóticas en Europa. JAMA 321:1448. doi: 10.1001/jama.2019.3829
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Garneau-Tsodikova, S., y Labby, K. J. (2016). Mecanismos de resistencia a los antibióticos aminoglucósidos: visión general y perspectivas. Medchemcomm 7, 11-27. doi: 10.1039/c5md00344j
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Hakkinen, M., Heiska, H. y Hanninen, M. L. (2007). Prevalencia de Campylobacter spp. en ganado en Finlandia y susceptibilidades antimicrobianas de las cepas de Campylobacter jejuni bovinas. Appl. Environ. Microbiol. 73, 3232-3238. doi: 10.1128/aem.02579-06
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Kassambara, A., y Mundt, F. (2020). «Extraer y visualizar los resultados de los análisis de datos multivariables». 1.0.7 ed.
Kittl, S., Heckel, G., Korczak, B. M. y Kuhnert, P. (2013). Atribución de la fuente de aislados de Campylobacter humano por MLST y la tipificación de fla y asociación de genotipos con resistencia a las quinolonas. PLoS One 8:e81796. doi: 10.1371/journal.pone.0081796
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Kumar, S., Stecher, G., Li, M., Knyaz, C. y Tamura, K. (2018). MEGA X: análisis de genética evolutiva molecular en todas las plataformas informáticas. Mol. Biol. Evol. 35, 1547–1549. doi: 10.1093/molbev/msy096
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Lê, S., Josse, J. y Husson, F. (2008). FactoMineR: AnRPackage para análisis multivariable. J. Estadísticas. Suave. 25, 1-18. doi: 10.18637/jss.v025.i01
Luangtongkum, T., Jeon, B., Han, J., Plummer, P., Logue, C. M. y Zhang, Q. (2009). Resistencia a los antibióticos en Campylobacter: emergencia, transmisión y persistencia. Futuro Microbiol. 4, 189-200. doi: 10.2217/17460913.4.2.189
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Magiorakos, A. P., Srinivasan, A., Carey, R. B., Carmeli, Y., Falagas, M. E., Giske, C. G., et al. (2012). Bacterias multirresistentes, ampliamente resistentes a los medicamentos y panresistentes: una propuesta de expertos internacionales para definiciones provisionales de normas para la resistencia adquirida. Clin. Microbiol. Infectar. 18, 268-281. doi: 10.1111/j.1469-0691.2011.03570.x
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Meinersmann, R. J., Phillips, R. W., Hiett, K. L. y Fedorka-Cray, P. (2005). Diferenciación de las poblaciones de campilobacter como lo demuestran las secuencias de regiones variables cortas de flagelina. Appl. Environ. Microbiol. 71, 6368–6374. doi: 10.1128/aem.71.10.6368-6374.2005
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Miller, W. G., Englen, M. D., Kathariou, S., Wesley, I. V., Wang, G., Pittenger-Alley, L., et al. (2006). Identificación de alelos asociados al huésped mediante la tipificación de secuencia multilocus de cepas de Campylobacter coli de animales de origen alimentarios. Microbiología (lectura) 152, 245-255. doi: 10.1099/mic.0.28348-0
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Moore, J. E., Barton, M. D., Blair, I. S., Corcoran, D., Dooley, J. S., Fanning, S., et al. (2006). La epidemiología de la resistencia a los antibióticos en Campylobacter. Microbes Infect 8, 1955–1966. doi: 10.1016/j.micinf.2005.12.030
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Morgan, M. y Ramos, M. (2019). «BiocManager: Accede al repositorio de paquetes de proyectos de Bioconductor». R paquete versión 1.30.10. ed.
Mossong, J., Mughini-Gras, L., Penny, C., Devaux, A., Olinger, C., Losch, S., et al. (2016). Campilobacteriosis humana en Luxemburgo, 2010-2013: un estudio de casos y controles combinado con la tipificación de secuencias multilocus para la atribución de la fuente y el análisis de factores de riesgo. Sci. Rep. 6:20939. doi: 10.1038/srep20939
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Mourkas, E., Taylor, A. J., Meric, G., Bayliss, S. C., Pascoe, B., Mageiros, L., et al. (2020). La intensificación agrícola y la evolución de la especialidad del huésped en el patógeno entéricos Campylobacter jejuni. Proc. Natl. Acad. Sci. EE. UU. 117, 11018–11028. doi: 10.1073/pnas.1917168117
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Nielsen, E. M., Engberg, J. y Madsen, M. (1997). Distribución de los serotipos de Campylobacter jejuni y C. coli de pacientes daneses, aves de corral, ganado y cerdos. FEMS Immunol. Med. Microbiol. 19, 47-56. doi: 10.1016/s0928-8244(97)00049-7
Ogle, D. h., Wheeler, P. y Dinno, a (2020). «FSA: Métodos simples de evaluación de las poblaciones de pesca». versión 0.8.27.9000 ed.
Papadopoulos, D., Petridou, E., Filioussis, G., Papadopoulos, T., Papageorgiou, K., Chatzistilianou, M., et al. (2020). Prevalencia y resistencia a los antibióticos de Campylobacter coli y Campylobacter jejuni en granjas porcinas griegas. Am. J. Microbiol. Immunol. 5:6. doi: 10.28933/ajmi-2020-02-2605
Pearson, B. M., Louwen, R., Van Baarlen, P., y Van Vliet, A. H. (2015). La distribución diferencial de los sistemas CRISPR-Cas de tipo II en los aislados agrícolas y no agrícolas de Campylobacter coli y Campylobacter jejuni se correlaciona con la falta de entornos compartidos. Genome Biol. Evol. 7, 2663–2679. doi: 10.1093/gbe/evv174
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Pérgola, S., Franciosini, M. P., Comitini, F., Ciani, M., De Luca, S., Bellucci, S., et al. (2017). Diversidad genética y perfiles de resistencia a los antimicrobianos de Campylobacter coli y Campylobacter jejuni aislados de pollo de engorde en granjas y en el momento del sacrificio en el centro de Italia. J. Appl. Microbiol. 122, 1348–1356. doi: 10.1111/jam.13419
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Qin, S., Wang, Y., Zhang, Q., Chen, X., Shen, Z., Deng, F., et al. (2012). Identificación de una nueva isla genómica que confiere resistencia a múltiples antibióticos aminoglucósidos en Campylobacter coli. Antimicrobiano. Agentes Chemother.56, 5332–5339. doi: 10.1128/aac.00809-12
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Qin, S., Wang, Y., Zhang, Q., Zhang, M., Deng, F., Shen, Z., et al. (2014). Informe del gen de la metilasa de ARN ribosómica erm(B) en Campylobacter coli multirresistente. J. Antimicrobiano. Madre. 69, 964-968. doi: 10.1093/jac/dkt492
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Equipo principal de R (2020). R: un lenguaje y un entorno para la computación estadística. Viena: Fundación R para la Computación Estadística.
Ramonaite, S., Rokaityte, A., Tamuleviciene, E., Malakauskas, A., Alter, T. y Malakauskas, M. (2013). Prevalencia, carga cuantitativa y diversidad genética de Campylobacter spp. en rebaños de ganado lechero en Lituania. Acta Vet. Escaneado.55:87. doi: 10.1186/1751-0147-55-87
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Ravel, A., Hurst, M., Petrica, N., David, J., Mutschall, S. K., Pintar, K., et al. (2017). Atribución de la fuente de campilobacteriosis humana en el punto de exposición combinando la evaluación comparativa de la exposición y la comparación de subtipos basada en la huella dactilar genómica comparativa. PLoS One 12:e0183790. doi: 10.1371/journal.pone.0183790
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Rosner, B. M., Schielke, A., Didelot, X., Kops, F., Breidenbach, J., Willrich, N., et al. (2017). Un estudio combinado de casos y control y atribución de fuentes moleculares de infecciones humanas por Campylobacter en Alemania, 2011-2014. Sci. Rep. 7:5139. doi: 10.1038/s41598-017-05227-x
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Rozas, J., Ferrer-Mata, A., Sánchez-Delbarrio, J. C., Guirao-Rico, S., Librado, P., Ramos-Onsins, S. E., et al. (2017). DnaSP 6: Análisis de polimorfismo de secuencia de ADN de grandes conjuntos de datos. Mol. Biol. Evol. 34, 3299–3302. doi: 10.1093/molbev/msx248
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Saenz, Y., Zarazaga, M., Lantero, M., Gastanares, M. J., Baquero, F. y Torres, C. (2000). Resistencia a los antibióticos en cepas deCampylobacter aisladas de animales, alimentos y seres humanos en España en 1997-1998. Antimicrobiano. Agentes Chemother. 44, 267-271. doi: 10.1128/aac.44.2.267-271.2000
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Sheppard, S. K., y Maiden, M. C. (2015). La evolución de Campylobacter jejuni y Campylobacter coli. Harb de primavera fría. Perspectiva. Biol. 7:a018119. doi: 10.1101/cshperspect.a018119
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Tang, Y., Jiang, Q., Tang, H., Wang, Z., Yin, Y., Ren, F., et al. (2020). Caracterización y prevalencia de Campylobacter spp. Desde el período de cría de pollos de engorde hasta el proceso de sacrificio en el este de China. Frente. Veterinario. Sci.7:227. doi: 10.3389/fvets.2020.00227
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Thepault, A., Rose, V., Quesne, S., Poezevara, T., Beven, V., Hirchaud, E., et al. (2018). Rumiante y pollo: fuentes importantes de campilobacteriosis en Francia a pesar de una variación en la atribución de la fuente en 2009 y 2015. Sci. Rep. 8:9305. doi: 10.1038/s41598-018-27558-z
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Ugarte-Ruiz, M., Florez-Cuadrado, D., Wassenaar, T. M., Porrero, M. C. y Domínguez, L. (2015). Comparación de métodos para mejorar la recuperación, el aislamiento y la detección cualitativa de C. jejuni y C. coli a partir de muestras de efluentes de aguas residuales. Int. J. Environ. Res. Salud Pública 12, 2749-2764. doi: 10.3390/ijerph120302749
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Ugarte-Ruiz, M., Gómez-Barrero, S., Porrero, M. C., Álvarez, J., García, M., Comeron, M. C., et al. (2012). Evaluación de cuatro protocolos para la detección y el aislamiento de Campylobacter termófilo de diferentes matrices. J. Appl. Microbiol.113, 200-208. doi: 10.1111/j.1365-2672.2012.05323.x
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Ugarte-Ruiz, M., Wassenaar, T. M., Gómez-Barrero, S., Porrero, M. C., Navarro-Gonzalez, N., y Domínguez, L. (2013). El efecto de los diferentes protocolos de aislamiento en la detección y caracterización molecular de Campylobacter de aves de corral. Lett. Appl. Microbiol. 57, 427-435. doi: 10.1111/lam.12130
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Wang, Y., Dong, Y., Deng, F., Liu, D., Yao, H., Zhang, Q., et al. (2016). Cambio de especies y resistencia múltiple a las drogas de Campylobacter de pollos y cerdos, China, 2008-14. J. Antimicrobiano. Madre. 71, 666-669. doi: 10.1093/jac/dkv382
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Wei, T., Simko, V., Levy, M., Xie, Y., Jin, Y., y Zemla, J. (2017). «Paquete R «corrplot»: Visualización de una matriz de correlación». 0,84 ed.
Wickham, H. (2011). La estrategia de división de aplicaciones y combinar para el análisis de datos. J. Estadísticas. Suave. 40, 1-29. doi: 10.18637/jss.v040.i01
Wickham, H., Averick, M., Bryan, J., Chang, W., Mcgowan, L., François, R., et al. (2019). Bienvenido al Tidyverse. J. Softw de código abierto. 4:1686. doi: 10.21105/joss.01686
Wieczorek, K. y Osek, J. (2013). Mecanismos de resistencia a los antimicrobianos entre Campylobacter. Biomed. Res. Int.2013:340605. doi: 10.1155/2013/340605
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Organización Mundial de la Salud (OMS) (2017). Antimicrobianos de Importancia Crítica para la Medicina Humana – 5a Revisión. Ginebra: OMS.
Zhang, Q., Al-Ghalith, G. A., Kobayashi, M., Segawa, T., Maeda, M., Okabe, S., et al. (2018). Secuenciación de región variable corta de flaA de alto rendimiento para evaluar la diversidad de Campylobacter en muestras fecales de aves. Frente. Microbiol. 9:2201. doi: 10.3389/fmicb.2018.02201
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Zhang, T., Cheng, Y., Luo, Q., Lu, Q., Dong, J., Zhang, R., et al. (2017). Correlación entre el polimorfismo de la caja gyrA y CmeR y la resistencia a las fluoroquinolonas en los aislados de Campylobacter jejuni en China. Antimicrobiano. Agentes Chemother. 61:e00422-17. doi: 10.1128/AAC.00422-17
Resumen de PubMed | Texto completo de CrossRef | Google Scholar
Palabras clave: Campylobacter, antibióticos, resistencia a los antimicrobianos, aminoglucósidos, macrólidos, flagelina, genes
Cita: Lopez-Chavarrias V, Ugarte-Ruiz M, Barcena C, Olarra A, Garcia M, Saez JL, de Frutos C, Serrano T, Pérez I, Moreno MA, Dominguez L y Alvarez J (2021) Monitoreo de la resistencia antimicrobiana a los aminoglucósidos y macrólidos en Campylobacter coli Frente. Microbiol.12:689262. doi: 10.3389/fmicb.2021.689262
Editado por:
Byeonghwa Jeon, Universidad de Minnesota Twin Cities, Estados Unidos
Revisado por:
Niwat Chansiripornchai, Universidad de Chulalongkorn, Tailandia
Mostafa Y. Abdel-Glil, Friedrich Loeffler Institut, Alemania
Copyright © 2021 López-Chavarrias, Ugarte-Ruiz, Barcena, Olarra, García, Saez, de Frutos, Serrano, Pérez, Moreno, Domínguez y Álvarez. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución de Creative Commons (CC BY). *Correspondencia: Julio Álvarez, jalvarez@visavet.ucm.es
Descargo de responsabilidad: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamación que pueda ser fabricado por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía

