Multirresistencia en aislados patógenos de Escherichia coli de infecciones del tracto urinario en perros, España
Multirresistencia en aislados patógenos de Escherichia coli de infecciones del tracto urinario en perros, España
 Ana Abad-Fau1
Ana Abad-Fau1  Eloísa Sevilla1,2*
Eloísa Sevilla1,2*  Ainara Oro1
Ainara Oro1 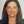 Inmaculada Martín-Burriel3
Inmaculada Martín-Burriel3  Bernardino Moreno1,2
Bernardino Moreno1,2  Mariano Morales1,4
Mariano Morales1,4  Rosa Bolea1,2
Rosa Bolea1,2- 1Departamento de Patología Animal, Facultad de Veterinaria, Instituto Agroalimentario de Aragón-IA2, Universidad de Zaragoza, Zaragoza, España
- 2Centro de Encefalopatías y Enfermedades Transmisibles Emergentes, Facultad de Veterinaria, Universidad de Zaragoza, Zaragoza, España
- 3Laboratorio de Genética Bioquímica, Facultad de Veterinaria, Instituto Agroalimentario de Aragón, Universidad de Zaragoza, Zaragoza, España
- 4Laboratorios Albéitar, Zaragoza, España
Escherichia coli (E. coli) es un patógeno frecuentemente aislado en casos de infecciones del tracto urinario (ITU) tanto en humanos como en perros, y existe evidencia de que los perros son reservorios de infecciones humanas. Además, E. coli se asocia con el aumento de las tasas de resistencia a los antimicrobianos. Este estudio se centra en el análisis de la resistencia a los antimicrobianos y la presencia de genes de virulencia seleccionados en aislamientos de E. coli de una población canina española con infección urinaria. Esta colección de aislados mostró un nivel extremadamente alto de resistencia fenotípica a las cefalosporinas de 1ª a 3ª generación, seguidas de las penicilinas, las fluoroquinolonas y los anfenicoles. Además, el 13,46% de ellos fueron considerados productores de betalactamasas de espectro extendido. También se detectó un porcentaje alarmantemente alto (71,15%) de aislamientos multirresistentes. Hubo una buena correlación entre los genes de resistencia a los antimicrobianos encontrados y la resistencia fenotípica expresada. La mayoría de los aislados fueron clasificados como E. coli patógeno extraintestinal, y otros dos albergaban factores de virulencia relacionados con patotipos diarreicogénicos. Se encontró una relación significativa entre la baja resistencia a los antibióticos y el alto portador del factor de virulencia, pero los mecanismos detrás de ella aún no se conocen bien. La detección de altas tasas de resistencia a los antimicrobianos a los tratamientos de primera elección pone de manifiesto la necesidad de una vigilancia constante de la resistencia a los antimicrobianos, así como de una revisión continua de las guías terapéuticas para las infecciones urinarias caninas para adaptarlas a los cambios en los patrones de resistencia a los antimicrobianos.
1 Introducción
Las infecciones del tracto urinario son una de las causas más comunes de supervisión veterinaria de atención primaria en perros y un desafío de tratamiento debido a su alta recurrencia e implicaciones terapéuticas. Escherichia coli es la bacteria más común aislada en infecciones urinarias en perros y humanos (1-3). Además, la bacteriemia por E. coli en humanos (la causa más común de bacteriemia en los países de ingresos altos) es causada por infecciones del tracto urinario en más del 50% de los casos (4). Escherichia coli también se ha asociado con un aumento de la resistencia a los antimicrobianos (2, 5). La evidencia sugiere que los perros actúan como reservorio de infecciones humanas por E. coli uropatógena (UPEC) y son una fuente de propagación de la resistencia a los antimicrobianos (6).
Escherichia coli se clasifica en varios patotipos en función de la presencia de factores de virulencia. Las E. coli uropatógenas se incluyen dentro del grupo de E. coli patógenas extraintestinales (ExPEC) y se caracterizan por un factor de virulencia específico. Algunos de estos factores de virulencia son las p-fimbrias (papC), la α-hemolisina (hlyA) y el factor necrotizante citotóxico tipo 1 (cnf1) (7). La Eae, el gen que codifica la intimina y está asociada con las cepas diarreicas, también se puede encontrar en cepas uropatógenas (8). Otros factores virulentos relevantes de E. coli incluyen las toxinas Shiga (Stx), también conocidas como verotoxinas y características de la E. coli productora de toxina Shiga (STEC), que están relacionadas con la diarrea hemorrágica y el síndrome urémico hemolítico, y se han descrito en varias especies, aunque las infecciones naturales rara vez se describen en perros (9, 10 ). Existe un riesgo zoonótico potencial asociado a la presencia de estos elementos genéticos en animales de compañía y otras especies (11, 12). Las cepas híbridas han ganado atención recientemente, especialmente aquellas que albergan varios factores de virulencia tradicionalmente asociados con diferentes patotipos. Estos nuevos tipos de cepas se consideran «heteropatógenas» o híbridas, como las cepas STEC/UPEC (2), y se consideran capaces de producir ambos resultados, diarrea o ITU (13).
Los genes de virulencia están codificados por plásmidos, bacteriófagos o islas de patogenicidad (PAI). Las islas de patogenicidad son fragmentos móviles e inestables de ADN presentes en las cepas patógenas, pero ausentes en las cepas no patógenas relacionadas, que pueden ser compartidas por transmisión horizontal. PapC, hlyA y cnf1, entre otros genes de virulencia, suelen estar codificados simultáneamente dentro de los PAI en UPEC (14, 15). Las p-fimbrias, codificadas por el gen papC, juegan un papel importante en la adherencia renal y en la respuesta inflamatoria (16). α-Hemolisina es una toxina conocida por producir lesiones renales, y aunque el mecanismo aún no está claro, cnf1 no juega un papel importante en la gravedad de la enfermedad, pero generalmente se asocia con otros genes de virulencia (15).
Se ha demostrado previamente que algunos aislados caninos de UPEC son clonales con los aislados de humanos, lo que sugiere su potencial zoonótico. También se ha propuesto que los perros podrían actuar como reservorio de este patotipo de E. coli, de ahí la importancia del estudio de las posibles implicaciones de las ITU en esta especie animal (17, 18).
En las últimas décadas ha habido una creciente preocupación por el aumento en el número de aislados de E. coli que presentan un perfil multirresistente (MDR) (19, 20). Se ha descrito que la tenencia de animales de compañía podría ser un factor de riesgo en la propagación de cepas patógenas de E. coli entre humanos y mascotas, favoreciendo también la diseminación de resistencia a los antimicrobianos en la comunidad (21-23).
Es común encontrar resistencia a antibióticos en aislados de E. coli de casos de ITU, lo que resalta la importancia de monitorear la susceptibilidad de la cepa al tratamiento antibiótico, incluso si ya se ha implementado un tratamiento experimental. De hecho, las cepas de UPEC aisladas de perros han sido descritas como reservorios de MDR en varios países (24, 25) y como portadoras de genes betalactamasas de espectro extendido (BLEE) (5). La E. coli productora de BLEE se ha encontrado previamente en gatos y perros, y también se ha demostrado la portación conjunta de humanos y perros en el mismo hogar en muestras fecales (26-28). En general, la BLEE y la presencia de otros mecanismos de resistencia a los antibióticos pueden dificultar el tratamiento de enfermedades infecciosas y, por lo tanto, dar lugar a infecciones crónicas complicadas.
Aunque se recomienda el cultivo microbiológico y las pruebas de susceptibilidad antes de establecer cualquier tratamiento antimicrobiano, con frecuencia se establece un tratamiento empírico y las recomendaciones más comunes para tratar estas infecciones en animales de compañía incluyen amoxicilina (sin ácido clavulánico) y trimetoprim-sulfonamidas como primer abordaje (29). Por lo tanto, se necesita información actualizada sobre los patrones de susceptibilidad a los antimicrobianos.
El objetivo de este estudio fue determinar la presencia de E. coli en muestras de orina de una población canina española que presentaba signos clínicos de infecciones del tracto urinario y caracterizar los aislamientos según los factores de virulencia seleccionados y su patrón de resistencia a los antimicrobianos, un campo de investigación escasamente investigado en España.
2 Materiales y métodos
2.1 Colección de aislados de Escherichia coli
Este estudio se realizó en un total de 52 aislamientos de E. coli. Esta colección de aislados provino de muestras de orina de perros diagnosticados con ITU. Las muestras se recogieron asépticamente mediante cistocentesis como parte de la actividad diaria de los veterinarios privados en Zaragoza, España. Los criterios seguidos para diagnosticar la ITU fueron los utilizados en la clínica diaria, que incluyen micción frecuente, dolor al orinar, fiebre o vómitos, entre otros. El período de muestreo osciló entre 2017 y 2019, y todas las muestras de orina se tomaron antes de establecer cualquier tratamiento. La edad media de los individuos fue de 8,97 años (IC 95%: 4,11-13,83%). En cuanto al sexo de estos individuos, 21 de ellos eran hombres y 31 mujeres.
Los aislados se identificaron utilizando VITEK® (bioMérieux, Francia) y los confirmados como E. coli se almacenaron a -20 °C para su posterior análisis.
2.2 Detección del gen de la virulencia
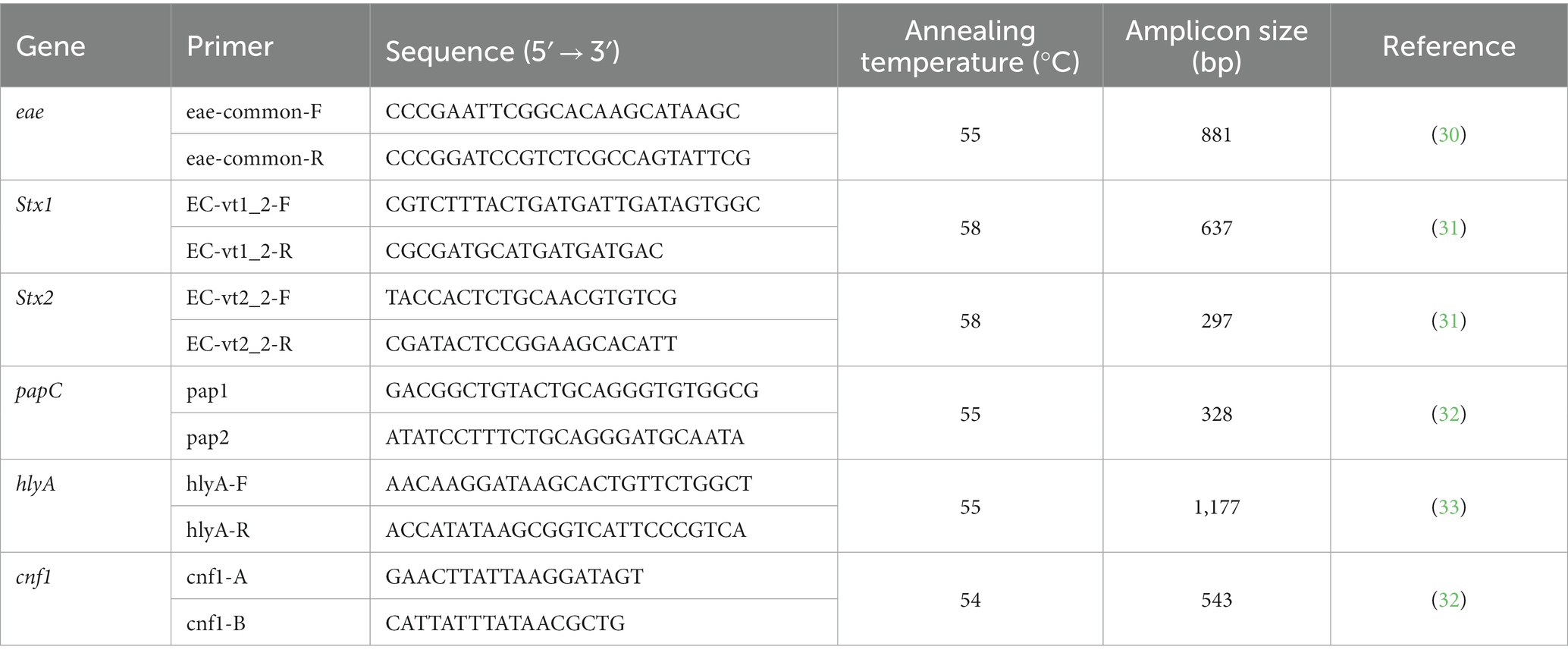
El ADN se extrajo hirviendo de 3 a 5 colonias de cultivos puros y luego se realizó una PCR convencional para la detección de genes relacionados con la virulencia. Estos genes incluían eae (intimina), Stx1 y Stx2 (toxinas Shiga 1 y 2), papC (P-fimbrias), hlyA (α-hemolisina) y cnf1 (factor necrotizante citotóxico tipo 1). Los cebadores utilizados en este estudio fueron los descritos en la Tabla 1. La PCR se realizó en un termociclador BiometraTRIO 48 (Analytik Jena, Alemania), y los productos de PCR se analizaron bajo luz UV en geles de agarosa al 1,5% teñidos con GelGreen® (Biotium, Estados Unidos).
La cepa CECT 4783 se utilizó como control positivo para los genes eae, Stx1 y Stx2; La cepa C136b fue el control positivo para los genes hlyA y cnf1, amablemente proporcionado por el Dr. J. A. Orden, Universidad Complutense de Madrid, España. Se utilizó una cepa canina previamente aislada por nuestro grupo de investigación (cepa Pe8, número de acceso GenBank MK034302) como control positivo para el gen papC.
Los aislamientos de Escherichia coli se clasificaron en patotipos según la presencia de los genes del factor de virulencia analizados. Las E. coli enterohemorrágicas (EHEC) se describen como aquellas cepas de E. coli que albergan tanto la intimina como las toxinas Shiga (12). Cuando solo uno de estos factores de virulencia estaba presente, los aislados se clasificaron como E. coli enteropatógena (EPEC) o STEC, respectivamente. Si se encontró alguno de los otros genes del factor de virulencia analizado, es decir, hlyA, PapC y/o cnf1, los aislados se clasificaron como E. coli patógena extraintestinal (ExPEC) (34).
2.3 Pruebas de sensibilidad a los antimicrobianos
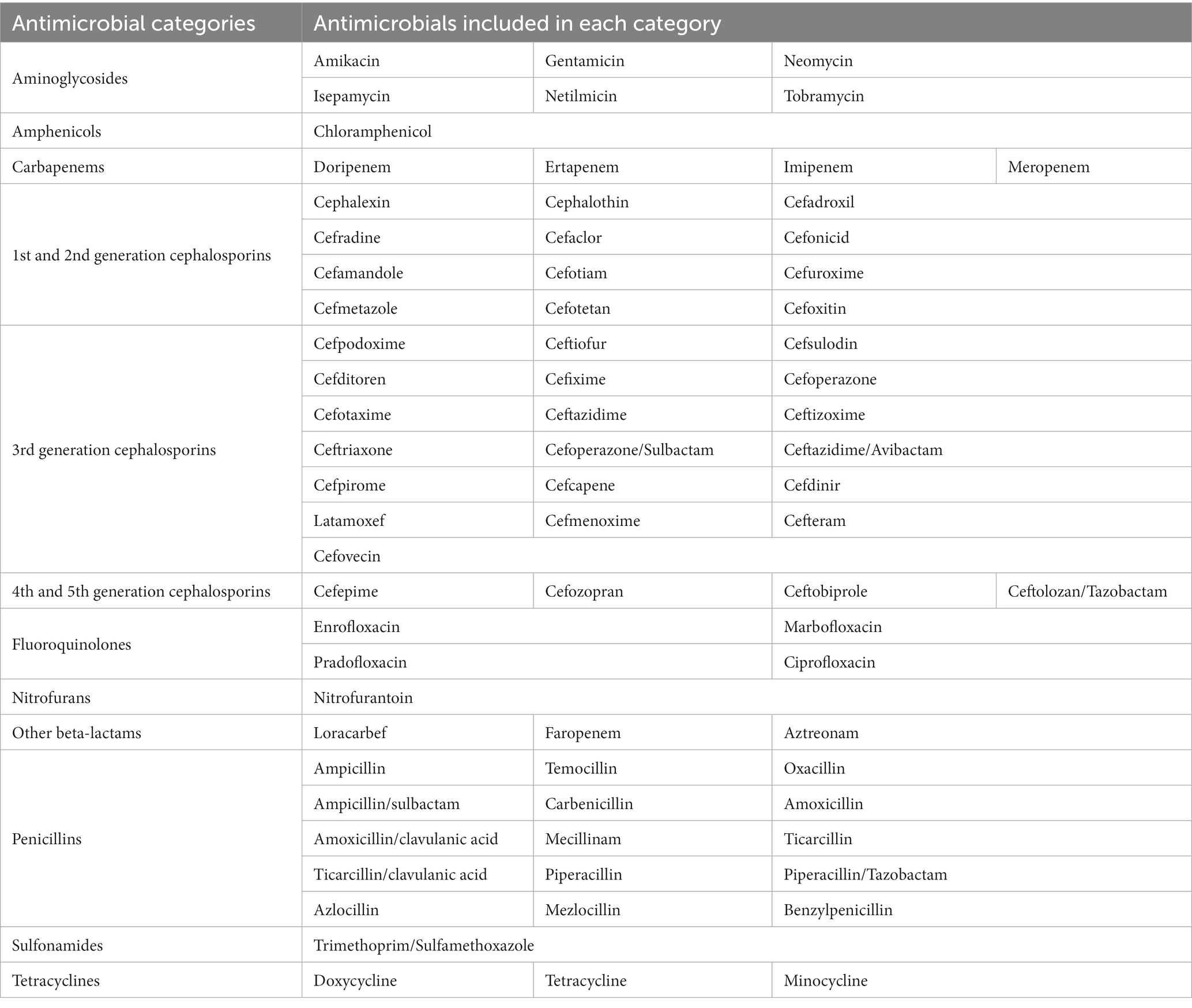
La susceptibilidad a 74 antimicrobianos diferentes se determinó utilizando VITEK® (bioMérieux, Francia). Los agentes antimicrobianos seleccionados para probar la susceptibilidad de cada cepa aislada dependieron de las directrices de VITEK, ya que los antimicrobianos clínicamente relevantes recomendados por VITEK®® variaron durante el período en el que se realizó el estudio. Los antimicrobianos se clasificaron en 12 categorías: aminoglucósidos, anfenicoles, carbapenémicos, fluoroquinolonas, nitrofuranos, otros β-lactámicos, penicilinas, tetraciclinas, sulfonamidas, cefalosporinas de 1ª-2ª generación, cefalosporinas de 3ª generación y cefalosporinas de 4ª-5ª generación, como se muestra en la Tabla 2. Todos los aislados se probaron con al menos un antibiótico de cada categoría, excepto las cefalosporinas de 4ª y 5ª generación, que se añadieron a mitad del estudio. En el caso de los aislados que no tenían información sobre las cefalosporinas de 4ª y 5ª generación, no se incluyeron ni la susceptibilidad ni la resistencia, por lo que se excluyeron del análisis de prevalencia para ese grupo. La resistencia a una categoría de antimicrobianos se definió como la resistencia a al menos uno de los agentes de esa categoría. Los aislados de MDR se definieron como aquellos aislados con no susceptibilidad a tres o más categorías de antimicrobianos (35).
Además, se utilizó la prueba VITEK 2 ESBL (bioMérieux) en estos aislados para la detección rápida de la producción de β-lactamasas de espectro extendido (BLEE), que se basa en la evaluación simultánea de los efectos inhibidores de cefepima, cefotaxima y ceftazidima, solos y en presencia de clavulanato.
Los puntos de corte para la interpretación de los resultados de la concentración inhibitoria mínima (CMI) se aplicaron de acuerdo con los criterios establecidos por bioMérieux para animales pequeños (AST-GN97, bioMérieux, Francia), que incluyen la resistencia natural y los puntos de corte del Instituto de Estándares Clínicos y de Laboratorio (36).
La categoría de resistencia intermedia proporciona una información flexible en la práctica clínica. Sin embargo, se ha encontrado previamente que los aislados de E. coli albergan genes de resistencia (37) Por esta razón, cuando tuvieron que ser categorizados en variantes dicotómicas fueron evaluados como resistentes.
2.4 Secuenciación del genoma completo
Los aislados que mostraban la tasa más alta de resistencia fenotípica, es decir, resistencia a seis o más categorías de antimicrobianos, se seleccionaron para su posterior caracterización mediante secuenciación del genoma completo (WGS). Un total de diez aislados de E. coli se cultivaron durante 24 h en agar nutriente (Oxoid, Reino Unido) y luego se extrajo el ADN utilizando el kit de purificación de ADN genómico Wizard (Promega, Madison, WI, Estados Unidos). Los parámetros de calidad del ADN se comprobaron tanto en el Qubit 4 (Invitrogen) como en la electroforesis en gel. La secuenciación del genoma se realizó en una plataforma Illumina Miseq con una longitud de lectura de extremo pareado de 150 pb. Las secuencias se recortaron en Galaxy (versión 0.3.8.1) y se ensamblaron con Unicycler (versión Galaxy 0.5.0 + Galaxy 1). Todos los datos de secuenciación se han enviado a la base de datos genómica del NCBI en el marco del PRJNA1031085 BioProject, y los números de acceso individuales son los siguientes: SAMN37924970 (aislado 258.883), SAMN37926527 (aislado 262.947), SAMN37926528 (aislado 263.715), SAMN37926529 (aislado 266.493), SAMN37926530 (aislado 267.252), SAMN37926531 (aislado 269.901), SAMN37926532 (aislado 271.550), SAMN37926533 (aislado 271.758), SAMN37926534 (aislado 271.811) y SAMN37926535 (aislado 271.960).
Los genes de resistencia a los antibióticos, los factores de virulencia, los serotipos y los tipos de secuencia (ST) se asignaron a estos genomas secuenciados utilizando herramientas que incluían ResFinder 4.1 (38-40), PathogenFinder 1.1 (41), VirulenceFinder 2.0 (39, 42), MLST 2.0 (E. coli #1 y #2) (39, 43-48), cgMLSTFinder 1.2 (42, 49), MGE v1.0.3 (39, 50-52) y SeroTypeFinder 2.0 (53). La visualización de los datos genómicos se llevó a cabo con Prok (ver 54). Se creó un árbol filogenético con la tubería Roary (55) basado en la anotación Prokka (56), y seguido por el uso del software IQ-TREE (57).
2.5 Análisis estadístico
La prevalencia se calculó con intervalos de confianza (IC) del 95%. Para probar la relación simple entre los factores de virulencia y los antibióticos, se utilizó la Prueba Exacta de Fisher y se determinaron los valores de p, considerándolos estadísticamente significativos cuando el valor de p ≤0,05. Los valores numéricos se calcularon mediante el coeficiente de Pearson. Los aislados que mostraron resistencia intermedia a los antibióticos se consideraron resistentes para las comparaciones estadísticas. Todos los análisis y cálculos se realizaron utilizando R versión 4.1.1 y RCommander 2.7-1.
3 Resultados
3.1 Análisis factorial de virulencia
Según el análisis del factor de virulencia realizado, la prevalencia de los genes relacionados con la virulencia fue la siguiente: 1,92% para eae (IC 95%: 0-5,66%), 1,92% para Stx2 (IC 95%: 0-5,66%), 59,62% para papC (IC 95%: 46,28-72,95%), 53,85% para hlyA (IC 95%: 40,30-67,4%) y 32,69% para cnf1 (IC 95%: 19,97-45,44%). Sin embargo, no se encontró el gen Stx1 en este estudio.
En cuanto a la clasificación del patotipo de E. coli, el 82,69% (IC 95%: 79-87%) de los aislamientos se clasificaron como ExPEC, y alrededor del 20% (9/43) de ellos albergaban simultáneamente los tres factores de virulencia extraintestinal analizados. Además, el 1,92% (IC 95%: 0-5,66%) de los aislamientos se definieron como EPEC, y el mismo valor se encontró para STEC. Sin embargo, no se detectaron aislamientos de EHEC. Ninguno de los factores de virulencia analizados en este estudio se encontró en el 13,46% (IC95%: 3-23%) de los aislamientos.
3.2 Prevalencia de resistencia fenotípica a los antimicrobianos
De acuerdo con los perfiles de resistencia a los antimicrobianos observados, solo uno de los 52 aislamientos de E. coli (IC 95%: 0-5,66%) fue susceptible a todos los antimicrobianos probados. Además, todas las categorías antimicrobianas presentaron aislamientos resistentes, aunque en un porcentaje variable.
De acuerdo con los niveles de resistencia a los antimicrobianos definidos por la Autoridad Europea de Seguridad Alimentaria (58), se encontró un nivel de resistencia extremadamente alto para las categorías de cefalosporinas de 1ª, 2ª y 3ª generación, seguidas de una resistencia muy alta a las penicilinas y fluoroquinolonas. Estos aislados también mostraron un alto nivel de resistencia a los anfenicoles (Figura 1). Se encontró un bajo nivel de resistencia en 5 de las 12 categorías: carbapenémicos, nitrofuranos, otros β-lactámicos, cefalosporinas de 4ª-5ª generación y aminoglucósidos.
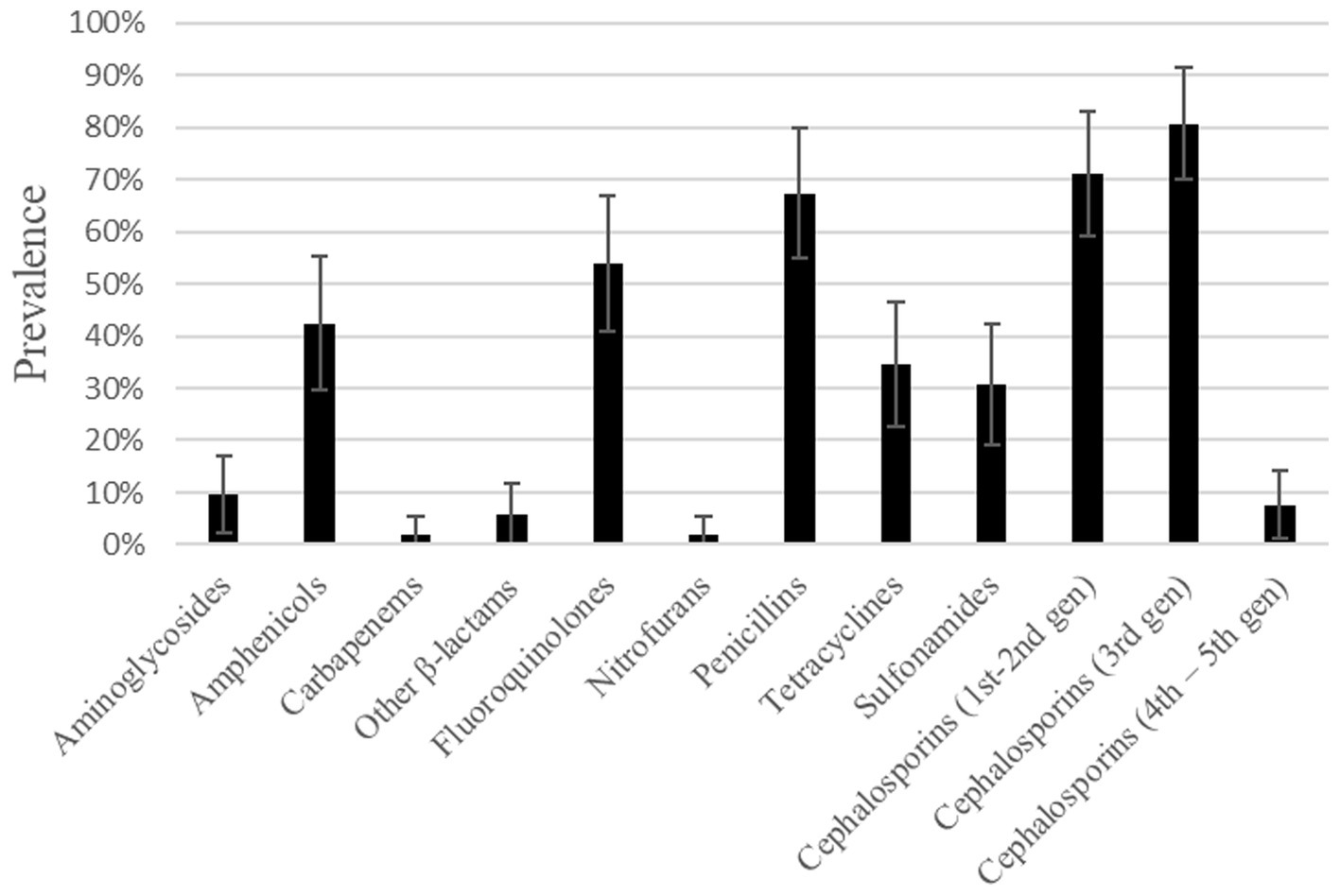 Figura 1. Prevalencia de resistencia a diferentes categorías antimicrobianas encontradas en aislados de E. coli de orina de perro.
Figura 1. Prevalencia de resistencia a diferentes categorías antimicrobianas encontradas en aislados de E. coli de orina de perro.
Varios aislamientos mostraron resistencia a los antibióticos, que se consideran antimicrobianos de importancia crítica y están clasificados en la categoría A (59). Por ejemplo, tres (de 30) aislados fueron resistentes al β-lactámico aztreonam, y hubo varios otros que se encontraron resistentes a los antibióticos de categoría A del grupo de penicilina: tres (de 17) aislados fueron resistentes a la carbenicilina, 18 (de 31) a la ticarcilina, uno (de 33) a la piperacilina y tres (de tres) a la mezlocilina. También hubo un aislado que mostró resistencia intermedia, y por lo tanto clasificado como resistente, a un agente de la categoría de carbapenémicos (imipenem).
Aparte de eso, el 13,46% (IC 95%: 4,17-22,73%) de los aislados se consideraron productores de BLEE, y casi el 60% (4/7) de ellos mostraron resistencia a 9 o más de las 12 categorías de antibióticos probadas.
3.3 Perfiles multirresistentes
Un total del 71,15% (IC 95%: 58,84-83,46%) de los aislados estudiados se describieron como MDR.
Se observaron dos perfiles principales de MDR, con una prevalencia del 7,69% (IC 95%: 0,45-14,93%) cada uno de ellos. Los aislamientos incluidos en uno de estos perfiles mostraron resistencia a las siguientes categorías antimicrobianas: penicilinas, cefalosporinas de 1ª-2ª y 3ª generación; mientras que el otro perfil comprendió aquellos aislados resistentes a anfenicoles, fluoroquinolonas, penicilinas y cefalosporinas de 1ª, 2ª y 3ª generación.
3.4 Análisis genómico de aislados seleccionados
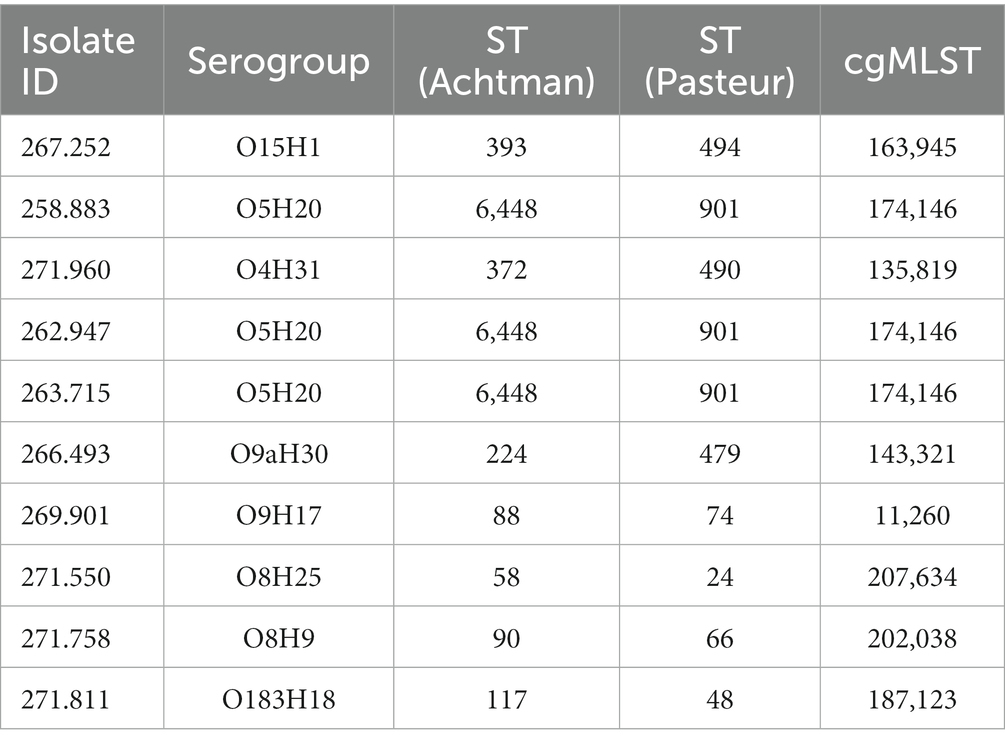
La tipificación molecular in silico se realizó en los genomas secuenciados de los aislados fenotípicamente resistentes seleccionados (Tabla 3). Se asignaron tres nomenclaturas diferentes para la tipificación de secuencias a cada aislado de acuerdo con el esquema MLST de Achtman, el esquema MLST de Pasteur y el genoma central (cg)-MLST.
Tres de estos aislamientos, es decir, 258.883, 262.947 y 263.715, compartían el mismo serotipo (O5H20), y el tipo de secuencia correspondiente (Pasteur ST 901 / Achtman ST 6448) y el tipo de secuencia del genoma central (cg-ST 174146), lo que convierte a este tipo de E. coli en el más prevalente entre los aislados estudiados. El resto de los aislados presentaron tipos moleculares únicos, aunque los aislados 271.758 y 269.901 pertenecían al mismo complejo clonal (CC ST23) y estaban emparejados en el árbol filogenético (Figura 2). La comparación anotada de los aislados (Figura 3) no mostró ninguna región faltante importante.
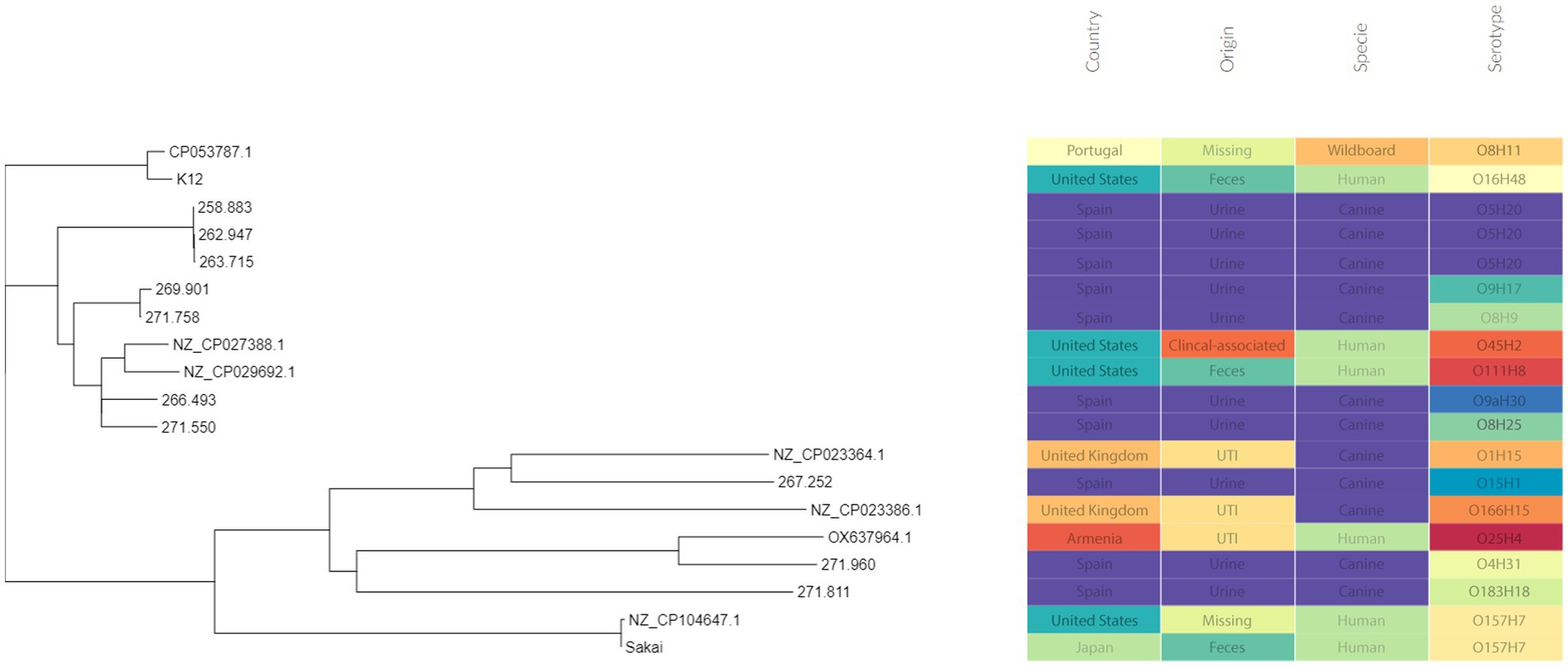 Figura 2. Árbol filogenético que incluye los aislados secuenciados de E. coli. Los metadatos se añadieron mediante la aplicación web Phandango (60).
Figura 2. Árbol filogenético que incluye los aislados secuenciados de E. coli. Los metadatos se añadieron mediante la aplicación web Phandango (60).
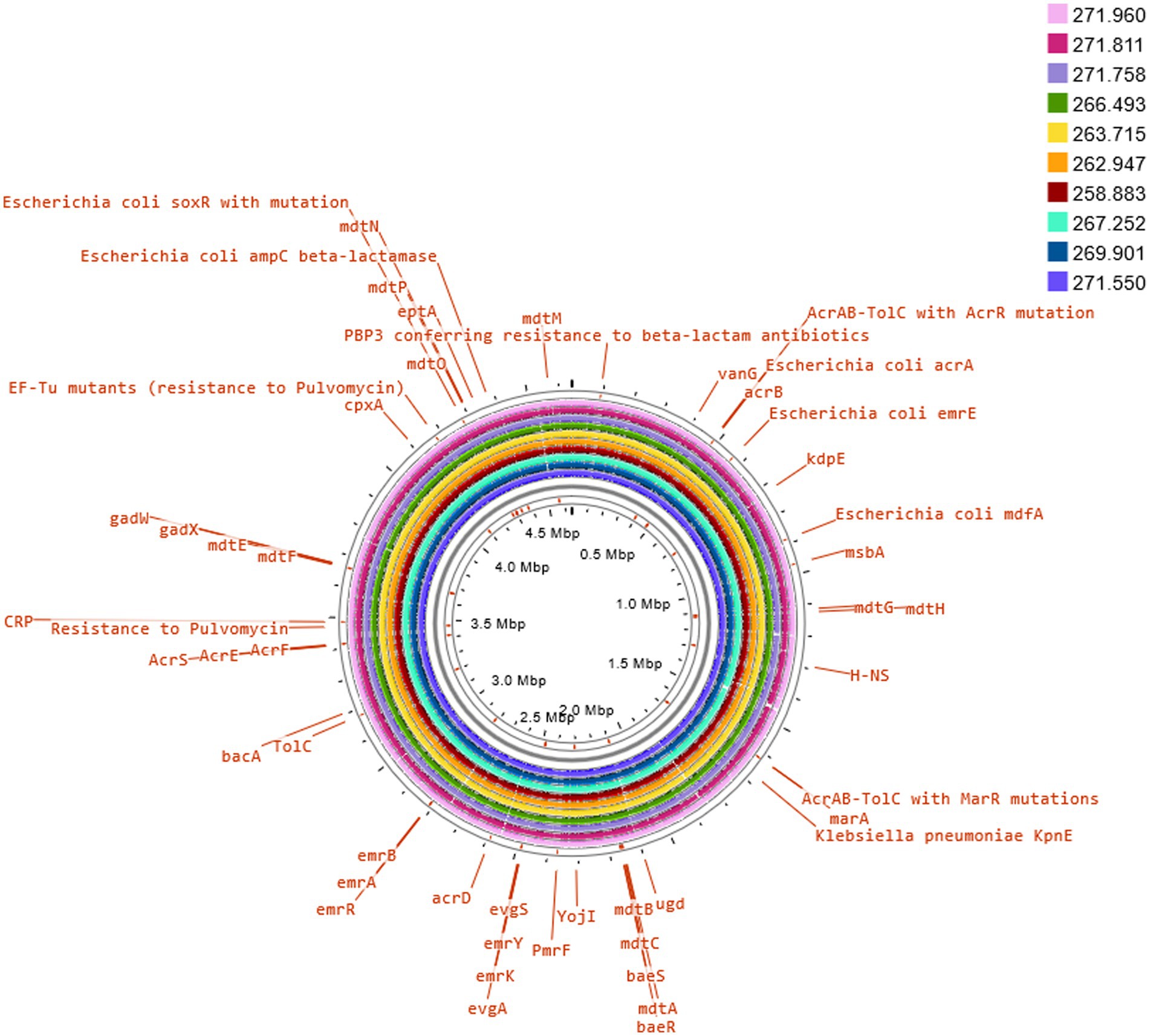 Figura 3. Comparación del genoma de referencia de E. coli K12 con aislados secuenciados. La anotación de genes seleccionados de resistencia a los antimicrobianos se llevó a cabo en Proksee Server del Grupo de Investigación Stothard (Universidad de Alberta, Canadá) que utiliza el análisis BLAST para ilustrar secuencias genómicas conservadas y faltantes (disponible en línea: https://proksee.ca/).
Figura 3. Comparación del genoma de referencia de E. coli K12 con aislados secuenciados. La anotación de genes seleccionados de resistencia a los antimicrobianos se llevó a cabo en Proksee Server del Grupo de Investigación Stothard (Universidad de Alberta, Canadá) que utiliza el análisis BLAST para ilustrar secuencias genómicas conservadas y faltantes (disponible en línea: https://proksee.ca/).
Se detectaron varios genes y mutaciones asociados con resistencia a diferentes categorías de antimicrobianos en los aislados secuenciados y se detallan en la Tabla 4. Además, algunos de estos genes se asociaron a elementos genéticos móviles (MGE), que se describen en la Tabla 5.
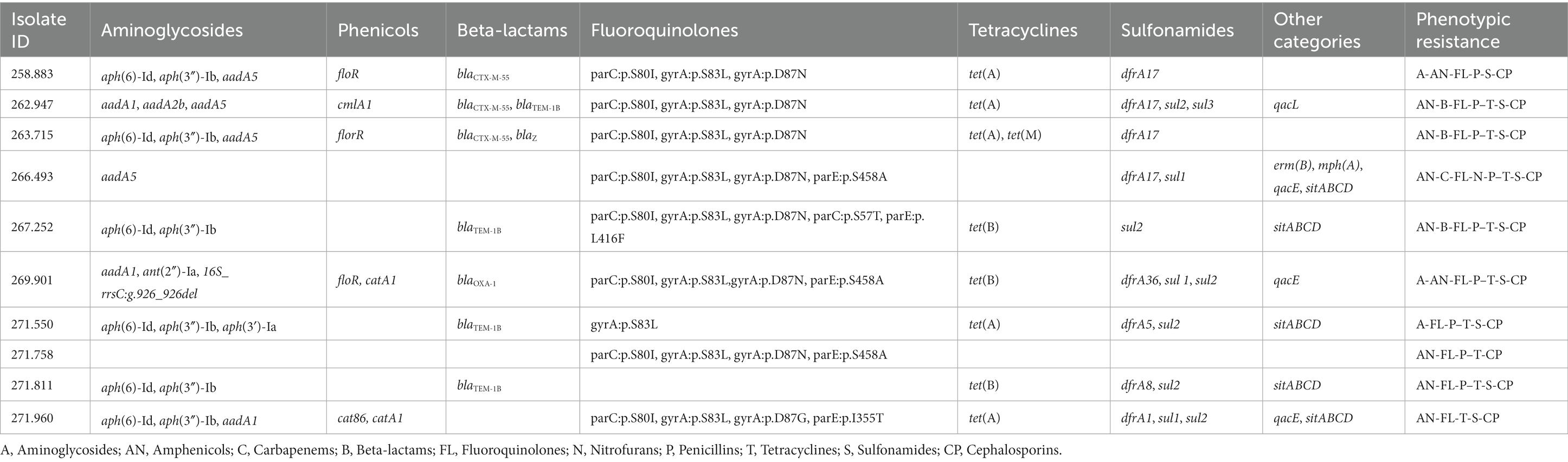 Tabla 4. Genes de resistencia a los antimicrobianos y mutaciones encontradas en los genomas secuenciados de los aislados de E. coli seleccionados, así como el patrón de resistencia fenotípica correspondiente mostrado en el ensayo de prueba de susceptibilidad.
Tabla 4. Genes de resistencia a los antimicrobianos y mutaciones encontradas en los genomas secuenciados de los aislados de E. coli seleccionados, así como el patrón de resistencia fenotípica correspondiente mostrado en el ensayo de prueba de susceptibilidad.
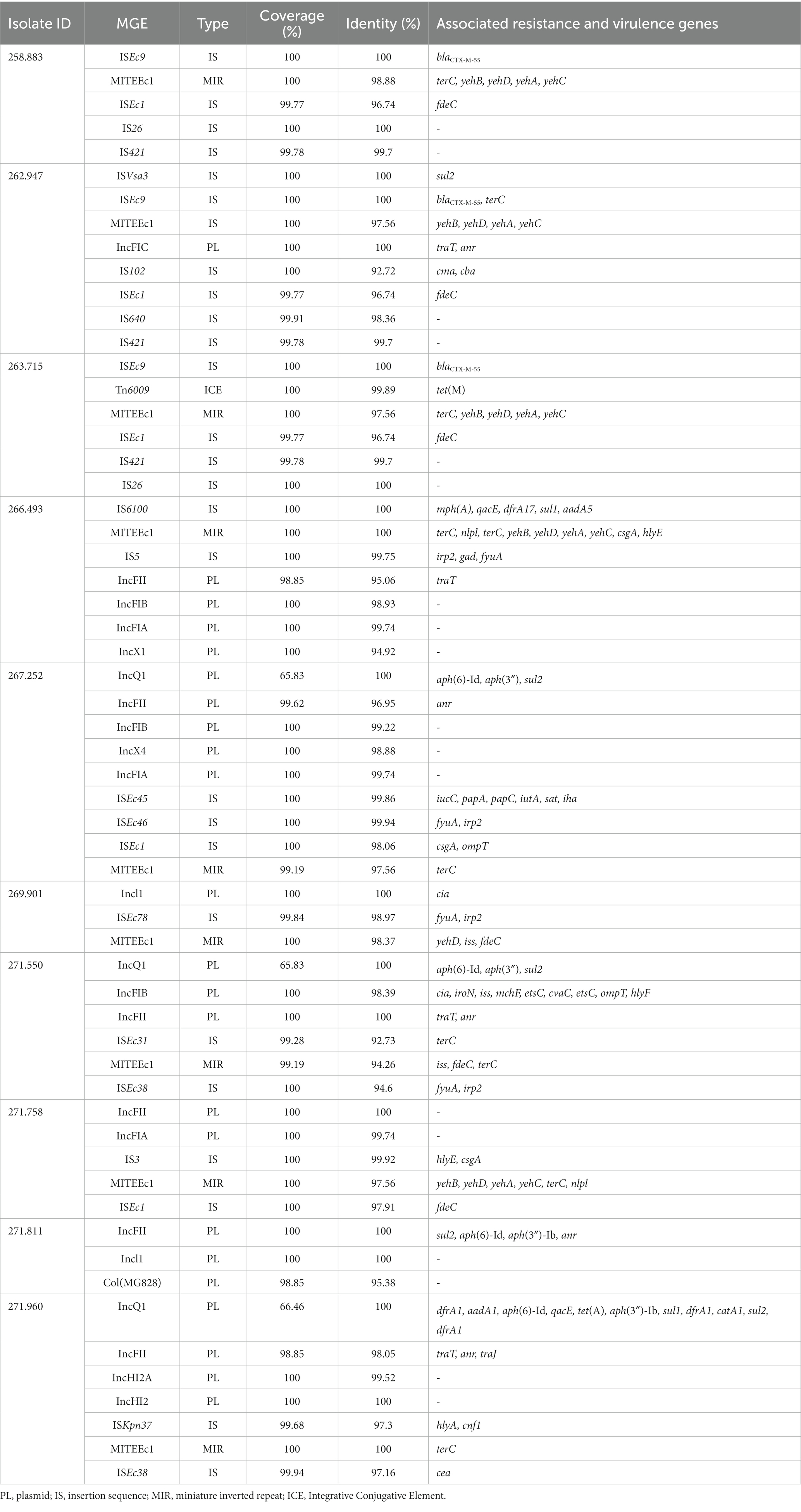 Tabla 5. Presencia de elementos genéticos móviles (MGE) en aislados secuenciados, y resistencia a antibióticos y genes de virulencia asociados.
Tabla 5. Presencia de elementos genéticos móviles (MGE) en aislados secuenciados, y resistencia a antibióticos y genes de virulencia asociados.
Curiosamente, hubo dos MGE de particular interés debido a su asociación con genes de resistencia importantes o un alto número de ellos, es decir, IS6100 e ISEc9, que se pueden ver en la Figura 4.
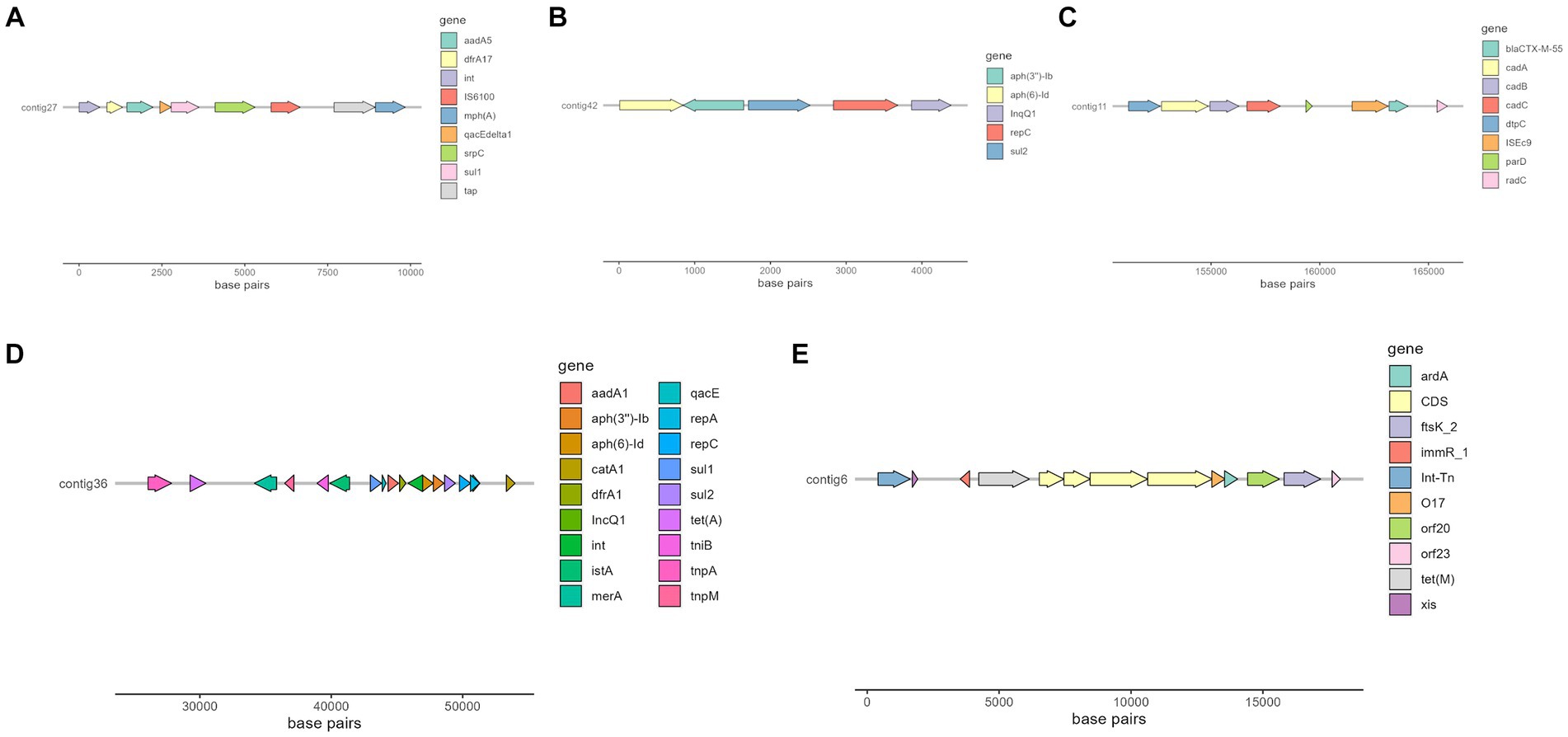 Figura 4. Se seleccionaron contigs de diferentes aislados que contenían genes de resistencia a los antimicrobianos, así como MGE anotados con Prokka (56). (A) Organización de un fragmento de contig 27 en el aislado 266.493, que contiene los genes aadA5, dfrA17, qacE, sul1 y mphA. (B) Organización del contig 42 en el aislado 269.901, que contiene los genes aph(6)-Id, aph(3″)-Ib y sul2. (C) Organización de un fragmento de contig 11 en el aislado 258.883, que contiene blaCTX-M-55 gen. (D) Organización de un fragmento de contig 36 en el aislado 271.960, que contiene varios genes de resistencia, como aadA1, sul2 y tet(A). (E) Organización de un fragmento de aislado 263.715, que contiene el gen de resistencia tet(M).
Figura 4. Se seleccionaron contigs de diferentes aislados que contenían genes de resistencia a los antimicrobianos, así como MGE anotados con Prokka (56). (A) Organización de un fragmento de contig 27 en el aislado 266.493, que contiene los genes aadA5, dfrA17, qacE, sul1 y mphA. (B) Organización del contig 42 en el aislado 269.901, que contiene los genes aph(6)-Id, aph(3″)-Ib y sul2. (C) Organización de un fragmento de contig 11 en el aislado 258.883, que contiene blaCTX-M-55 gen. (D) Organización de un fragmento de contig 36 en el aislado 271.960, que contiene varios genes de resistencia, como aadA1, sul2 y tet(A). (E) Organización de un fragmento de aislado 263.715, que contiene el gen de resistencia tet(M).
3.5 Asociación entre la resistencia a los antimicrobianos y los factores de virulencia
Al probar las relaciones simples entre la resistencia fenotípica a los antimicrobianos y la presencia de factores de virulencia en estos aislados, el porte del gen cnf1 mostró una asociación significativa con la resistencia a varias penicilinas, así como con la propia categoría de penicilina (Valor de p = 0,01). El gen Cnf1 también mostró una asociación significativa (Valor de p = 0,019) con la categoría MDR. Aparte de eso, la edad de los individuos se asoció significativamente con los aislados de E. coli que mostraron resistencia a varias cefalosporinas y a la categoría de cefalosporinas de 4ª-5ª generación (valor de p = 0,034). El sexo se asoció con los aislados resistentes a aminoglucósidos (Valor de p = 0,007).
La producción de BLEE se asoció con la resistencia a cinco de las 12 categorías antimicrobianas analizadas (anfenicoles, otros β lactámicos, cefalosporinas de 4ª-5ª generación, sulfonamidas y fluoroquinolonas). No se observó una relación significativa entre el número de genes relacionados con la virulencia y la producción de BLEE, sin embargo, se encontró una relación negativa entre el número de categorías antimicrobianas a las que los aislados mostraron resistencia y el número de genes relacionados con la virulencia (coeficiente de Pearson = 0,33, valor de – = 0,014).
4 Discusión
Este estudio ha evaluado la resistencia antimicrobiana tanto fenotípica como genotípica, así como la presencia de genes de virulencia seleccionados en aislamientos de E. coli de perros españoles con ITU y ha demostrado que los perros pueden ser reservorios de cepas uropatógenas resistentes de E. coli.
Comparando con los resultados obtenidos en un estudio previo (61) en el que se recogieron muestras en un tiempo y ubicación geográfica similares, aunque tenían un origen diferente (heces), encontramos que los aislamientos de E. coli en orina eran más susceptibles a los aminoglucósidos que los obtenidos en heces de perro (resistencia fenotípica encontrada en el 9,62% frente al 40% de los aislados). En general, la prevalencia de resistencias antimicrobianas encontradas es similar a la encontrada en E. coli aislada de pacientes con ITU (62, 63), excepto en el grupo de penicilina, que fue ligeramente mayor en este estudio (67,31% vs. ~ 45-50%). Esta alta prevalencia también es contraria a la tendencia decreciente de la resistencia a la penicilina en aislados de E. coli en Europa (20). Algunas hipótesis para este fenómeno podrían ser la tendencia al aumento de la resistencia a los antibióticos año tras año o el hecho de que en este trabajo se estudiaron más antibióticos del grupo de la penicilina.
La resistencia a los aminoglucósidos de Escherichia coli fue el único tipo de resistencia ligada al sexo del animal, encontrándose exclusivamente en individuos machos. De hecho, el sexo masculino se ha asociado previamente con la resistencia a los aminoglucósidos en bacterias gramnegativas (64).
Al tener en cuenta algunos de los antibióticos considerados clínicamente importantes para la salud humana y animal por la Agencia Europea del Medicamento (59), vale la pena mencionar que en este estudio se detectaron aislamientos de E. coli que mostraban resistencia a varios de estos antibióticos, incluso a antibióticos de categoría A («Evitar», incluye antibióticos no autorizados en medicina veterinaria en la Unión Europea). como ciertas penicilinas o carbapenémicos. Otra categoría relevante de antibióticos es la categoría B («Restringida»), que incluye aquellos enumerados como antimicrobianos de importancia crítica de mayor prioridad (HP-CIA) según la categorización de la Organización Mundial de la Salud, por ejemplo, cefalosporinas de 3ª generación o fluoroquinolonas. De hecho, hasta el 80,77 y el 53,85% de estos aislados se consideraron resistentes a las cefalosporinas y fluoroquinolonas de 3ª generación, respectivamente. La alta cantidad de resistencia general encontrada entre todas las categorías podría estar sesgada por el hecho de que las ITU complicadas se solicitan con más frecuencia para las pruebas de cultivo y antibiograma que los casos más simples de ITU.
Actualmente se recomienda el tratamiento con amoxicilina o trimetoprim-sulfonamidas como agentes de primera línea para el manejo de las infecciones urinarias bacterianas en perros (29). Sin embargo, teniendo en cuenta estos resultados, parece que el uso de estos antimicrobianos puede ser ineficaz en un alto porcentaje de casos, ya que el 58,33% de los aislados se encontraron resistentes a la amoxicilina y el 30,77% a la trimetoprima-sulfonamidas. Antes de sugerir cualquier cambio en las guías actuales para el tratamiento antibiótico en las infecciones urinarias caninas, hay que tener en cuenta que E. coli no es el único patógeno responsable de las infecciones urinarias y que los datos analizados en este estudio podrían estar sobreestimando la resistencia basal, principalmente debido a la selección de los pacientes. En cualquier caso, el uso de pruebas de resistencia a antibióticos como rutina permite no solo el seguimiento de la epidemiología de los perfiles de resistencia a los antibióticos, sino también la implementación más rápida de un tratamiento en caso de fracaso del empírico.
Cabe destacar el alto porcentaje de aislamientos de MDR encontrados (71,15%). Entre los aislamientos de MDR encontrados, más del 80% (30/37) fueron clasificados como ExPEC, y uno de ellos correspondió al patotipo EPEC. Este tipo de cepas poseen un riesgo zoonótico potencial y también pueden servir como reservorio de genes de resistencia (18), contribuyendo aún más a la diseminación de la resistencia a los antibióticos y limitando las opciones para el tratamiento de enfermedades infecciosas tanto en humanos como en animales.
La alta resistencia a los antibióticos se ha asociado con MGE en Enterobacteriaceae. Las bacterias que albergan estos elementos móviles pueden convertirse en un reservorio de resistencias a los antibióticos y transmitirse de las mascotas a sus dueños o al medio ambiente. Este fenómeno plantea un grave problema de salud debido a la propagación de la resistencia y al fracaso de los tratamientos antibióticos actuales (65).
De especial interés es la presencia del transposón Tn6009 portador del gen tet(M) en el aislado 263.715. Este elemento ha sido descrito previamente en otras bacterias Gram-negativas, como Enterococcus fecalis (66), y se asocia con resistencia a tetraciclina debido a la presencia de tet(M) (Figura 4E). Este transposón conjugativo no compuesto es de importancia clínica en bacterias Gram-positivas y tiene un papel potencial en la diseminación de la resistencia (67, 68). La resistencia a los betalactámicos fue codificada por blaCTX–M–55 gen en tres aislados, que se localizó en una secuencia de inserción ISEc9 (similar a IS 1380). La región ISEc9 se ha asociado previamente con genes BLEE (69) y se ha descrito en otras bacterias como Vibrio vulnificus (70). En todos los casos, el gen y la secuencia de inserción estaban a 46 pb de distancia. Uno de ellos (aislado 263.715) también albergaba un blaZ y otro (aislado 263.715) tenía una copia de BLATEM-1B. Genes que codifican para betalactamasas como el blaTEM-1B y BLAOXA-1 también se identificaron en otros aislamientos. Por ejemplo, blaTEM-1B estuvo presente en dos aislamientos que no mostraron resistencia fenotípica a betalactámicos (aislados 271.550 y 271.811), y en uno que sí lo hizo (aislado 267.252). Solo había un aislado (269.901) que contenía un blaOXA-1 gen. Como era de esperar, este aislado fue resistente a varios antibióticos en el grupo de penicilina (ampicilina, amoxicilina + ácido clavulánico, amoxicilina), y fue negativo en la prueba de producción de BLEE BlaOXA-1 se ha encontrado en ST131 o se ha asociado con otros genes en plásmidos (71). A pesar de que este gen se describió originalmente en MGE (72), solo identificamos plásmido IncI1 en este aislado, y no se asoció con ningún gen de resistencia a antibióticos. En la mayoría de los genomas secuenciados (7/10) se encontraron genes de resistencia a sulfonamida (sul 1 o 2), y en cinco de ellos estos genes se localizaron en un MGE (IS6100 e IncQ1 para sul1, e ISVsa3, IncQ1 o IncFII para sul2). Todos estos MGE también albergaban otros genes de resistencia, incluidos los relacionados con la estreptomicina (aadA5, aph(6)-Id, aph(3″)-Ib y aadA1 en IS 6100, IncFII e IncQ1) (73-76), trimetoprima (dfrA17 y dfrA1 en IS 6100 e IncQ1) (77, 78), antisépticos (qacL y qacE en IS 6100 e IncQ1, aunque todos los genes de resistencia a los antisépticos estaban incompletos) (79), eritromicina (mph(A) en IS6100) (80), doxiciclina y tetraciclina (tet(A) en IncQ1) (81) y cloranfenicol (catA1 en IncQ1) (82).
La transposasa ISVsa3 se encontró en uno de los aislados secuenciados (262.947) y contenía el gen sul2, que también se ha identificado en otros enteropatógenos (83-85). El aislado 266.493 albergaba la estructura de integrón dfrA17-aadA5-qacEdelta1-IS6100-mph(A)-sul1, que se identifica comúnmente en el patotipo ExPEC (86).
En cuanto a los plásmidos identificados, se ha documentado previamente en España que el plásmido IncFII está frecuentemente relacionado con la producción de BLEE (87). En este estudio, el plásmido solo se identificó en un aislado, aunque se clasificó como no productor de BLEE.
Otro plásmido identificado fue IncQ1, comúnmente encontrado en E. coli y con capacidad de transferencia entre diferentes especies bacterianas y cepas, lo que facilita la diseminación de resistencia a antibióticos en poblaciones bacterianas (88). Este plásmido se detectó en los genomas de tres aislados (267.252, 271.550 y 271.960) y se encontró cerca de genes de resistencia relacionados con la resistencia a aminoglucósidos (aph(6)-Id y aph(3″)-Ib). Estos tres aislados estaban distantes en el árbol filogenético, lo que sugiere que el plásmido probablemente se adquirió de forma independiente. Uno de estos aislados albergaba nueve genes de resistencia más cerca del plásmido detectado (Figura 4D), lo que indica un punto caliente potencial para la diseminación de resistencia a los antibióticos.
Al estudiar los genes de resistencia a antibióticos en aislados secuenciados, hubo en general una correlación consistente entre la resistencia fenotípica y genética. Sin embargo, hubo dos excepciones significativas a este patrón. Al examinar los aminoglucósidos, varios aislados mostraron susceptibilidad a esta categoría a pesar de ser portadores de genes de resistencia relacionados con la estreptomicina y la espectinomicina, que se incluyen en esta categoría de antimicrobianos. La segunda excepción se observó con la tetraciclina, donde la relación entre los genes de resistencia y la resistencia fenotípica no se alineó de manera consistente. Esta discrepancia en el aislado 258.883 puede atribuirse a una sustitución de nucleótidos en la posición 924 dentro del gen tet(A) (posición 3.323, GenBank: AF534183.1), específicamente en la transición de citosina (C) a timina (T). Debido a esta alteración, hay un cambio en la composición de la proteína de la alanina (Ala) en la posición 118 a la treonina (Thr). Aunque estos no son los primeros aislados de E. coli que albergan esta mutación genética (89), hasta donde saben los autores, nuestro estudio es el primero que asocia esta mutación en tet(A) a una falla en la respuesta fenotípica. Además, en más del 40% (4/9) de los aislamientos considerados fenotípicamente resistentes a los anfenicoles, no se encontró ningún gen de resistencia asociado a esta categoría antimicrobiana.
El árbol filogenético (Figura 3) mostró que los aislados caninos se agruparon, excepto el aislado 271.960 (ST 372), que se agrupó con un aislado humano (referencia de ensamblaje OX637964.1) que pertenece a ST131. ST 131 es uno de los tipos de secuencia predominantes dentro del patotipo ExPEC en todo el mundo (71, 90). De hecho, E. coli O25b:H4/ST131 fue descrita como un clon prevalente en la población humana española. De acuerdo con la bibliografía, este aislado canino no se asoció con resistencia a BLEE y tuvo un perfil de resistencia similar al de las cepas humanas (91, 92).
De acuerdo con los factores de virulencia analizados, la mayoría de los aislamientos de E. coli encontrados en las muestras de orina se categorizaron en el patotipo ExPEC, como se esperaba. Los factores de virulencia detectados con mayor frecuencia fueron papC y hlyA, seguidos de cnf1. La prevalencia de Cnf1 en estos aislamientos fue similar a la encontrada en aislamientos tanto de perros como de humanos, mientras que la prevalencia de papC y hlyA fue mayor en este estudio (18, 93-97). Sin embargo, la mayoría de las poblaciones muestreadas en estos estudios incluyen animales sanos, lo que podría reducir la prevalencia de los factores de virulencia de E. coli. La prevalencia de estos tres factores de virulencia fue mayor en los aislados de perros que en los encontrados en humanos (95, 98-100). Algunos de estos factores fueron encontrados en la MGE (Tabla 5), lo que pone de manifiesto su potencial de propagación a otras cepas.
Casi el 20% de estos aislamientos de ExPEC se mostraron en combinación con los tres genes relacionados con la virulencia extraintestinal analizados (papC, hlyA y cnf1), probablemente debido a la presencia de un PAI (101). Este tipo de factores de virulencia se encuentran con frecuencia en cepas de E. coli causantes de enfermedad extraintestinal tanto en humanos como en perros, siendo así esta especie animal un posible reservorio del patotipo ExPEC (102).
Aparte de eso, se estudió un aislado Stx2 positivo, que no albergaba ningún otro factor de virulencia. Se cree que la toxina Shiga 2 está asociada con el desarrollo del síndrome urémico hemolítico (11) y se produce mejor cuando se encuentra en combinación con otras cepas o bacterias (103). Sin embargo, también hay algunas descripciones de aislados de E. coli productores de toxina Shiga asociados con casos de infecciones urinarias, y se ha propuesto que las toxinas Shiga pueden unirse a los receptores de las células epiteliales de la vejiga urinaria y dañarlas (104, 105). Además, también se detectó un aislado de EPEC. No es la primera vez que se encuentra un aislado eae positivo entre las cepas asociadas a ITU, aunque su frecuencia también parece ser bastante baja (8, 106, 107). El papel de este producto génico (es decir, la intimina) en la patogénesis de las infecciones urinarias no se comprende completamente y su importancia aún no se ha estudiado (8).
Teniendo en cuenta que la población fecal de E. coli podría tener una relación con la patogénesis de las infecciones urinarias (108), se puede sugerir que ciertos patotipos diarreicos también tienen el potencial de causar infecciones urinarias, aunque son poco frecuentes. Sin embargo, el papel de estas cepas en el desarrollo de ITU y las causas moleculares y patogénicas detrás de ella aún no se conocen bien, y se necesita más investigación en este campo para comprender los mecanismos y las causas epidemiológicas. Sin embargo, la capacidad de estas cepas para causar una infección extraintestinal en el huésped no solo depende de sus genes relacionados con la virulencia, sino también de factores de riesgo como la edad o la inmunosupresión (106).
También es importante tener en cuenta que en la literatura se ha descrito una amplia variedad de rasgos de virulencia asociados al sector extraintestinal. Por lo tanto, además de estos genes de virulencia típicos de las cepas diarreicas, estos dos aislados también podrían albergar otros factores de virulencia extraintestinal diferentes a los analizados en este estudio. En este sentido, algunas cepas de E. coli han sido clasificadas recientemente como híbridas por albergar factores de virulencia generalmente asociados con diversos patotipos, por ejemplo, cepas STEC/UPEC (109). La plasticidad del genoma de este microorganismo promueve el intercambio y la combinación de determinantes de virulencia intestinal y extraintestinal, lo que resulta en un potencial heteropatogénico (106, 107). Por lo tanto, debe vigilarse la posible aparición de patotipos híbridos no solo en humanos sino también en animales.
El hallazgo de ocho aislamientos (15,38%) considerados aislados patógenos intestinales ni extraintestinales pudo explicarse por el hecho de que solo se probó una selección de factores de virulencia. Por lo tanto, estas E. coli podrían albergar otros genes diferentes relacionados con la virulencia no analizados en este estudio. Sin embargo, otra explicación podría ser que el agente causal de la infección urinaria en estos perros era diferente de E. coli, o incluso una causa no infecciosa. También cabe destacar que la detección de genes del factor de virulencia no significa que se expresen fenotípicamente, por lo que la gravedad de la enfermedad no podría evaluarse únicamente con esta información. Sin embargo, se sabe que la gravedad de la enfermedad no es causada por un solo factor de virulencia, sino por una combinación de ellos (110, 111).
El serotipo aislado con mayor frecuencia en este estudio fue O5H20. En este sentido, O5:H(−) se ha asociado con cepas STEC, y las toxinas Shiga también se han descrito en cepas de E. coli que causan ITU (112, 113). Sin embargo, estos aislados no albergaban ningún gen de la toxina Shiga. El resto de los serotipos de E. coli se distribuyen a lo largo de diferentes ST y patrones de resistencia a antibióticos, mostrando una distribución heterogénica.
Curiosamente, los patrones de baja resistencia a los antibióticos se relacionaron con un mayor número de factores de virulencia. Existe cierta literatura (114, 115) que sugiere una relación positiva entre los factores de virulencia y el MDR. Sin embargo, en los aislamientos de este estudio, solo el portador de cnf1 mostró una asociación significativa con MDR, mientras que un portador de factor de virulencia alto se asoció con perfiles de baja resistencia. La razón de este mecanismo aún no está clara, pero se plantea la hipótesis de que la adquisición de MDR se «sacrifica» a cambio de factores de virulencia, o que la baja presencia de factores de virulencia facilita la adquisición de resistencia a los antibióticos (116, 117). Al analizar la correlación entre todos los factores de virulencia encontrados y la presencia de genes de resistencia a antibióticos en aislados secuenciados del genoma completo, la relación no fue significativa (p = 0,14). Sin embargo, hubo un sesgo en la selección de los aislados, ya que solo se eligieron los más resistentes.
5 Conclusión
Con base en estos datos, un porcentaje muy alto de aislamientos de E. coli encontrados en muestras de orina de perros que padecían ITU se consideraron MDR, la mayoría de ellos clasificados como ExPEC. También se observó con frecuencia la resistencia fenotípica a los antimicrobianos de primera línea recomendada en el tratamiento de las infecciones urinarias, lo que podría asociarse a un fracaso del tratamiento. Además, se identificaron varios genes de resistencia a los antimicrobianos, algunos de ellos contenidos en MGE, en el genoma de aislados resistentes seleccionados. El uso de la secuenciación del genoma completo pudo identificar algunos de los mecanismos genéticos que subyacen a la resistencia a los antimicrobianos, aunque se encontraron algunas discordancias entre la resistencia fenotípica y los genes. La combinación de datos fenotípicos y genéticos mejora nuestra comprensión de la resistencia a los antibióticos y mejora la eficiencia de la selección de tratamientos.
En general, estos hallazgos son preocupantes tanto para la salud animal como para la salud pública, ya que los perros podrían actuar como reservorios de E. coli patógena MDR y contribuir a la propagación de la resistencia a los antimicrobianos. Por lo tanto, la vigilancia de la resistencia a los antimicrobianos y la revisión de las directrices terapéuticas deben abordarse continuamente en los entornos veterinarios clínicos.
Declaración de disponibilidad de datos
Las contribuciones originales presentadas en el estudio están a disposición del público. Estos datos se pueden encontrar en: https://www.ncbi.nlm.nih.gov/bioproject; PRJNA1031085.
Declaración ética
No se requirió aprobación ética para los estudios con animales de acuerdo con la legislación local y los requisitos institucionales, ya que las muestras se recogieron como parte de la actividad diaria de los veterinarios privados. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones de los autores
AA-F: Curación de datos, Análisis formal, Investigación, Redacción – borrador original, Redacción – revisión y edición. ES: Curación de datos, Análisis formal, Supervisión, Redacción – borrador original, Redacción – revisión y edición. AO: Investigación, Escritura – borrador original. IM-B: Análisis formal, Supervisión, Redacción – revisión y edición. BM: Recursos, Supervisión, Redacción – Revisión y Edición. MM: Conceptualización, Recursos, Supervisión. RB: Conceptualización, Obtención de Fondos, Recursos, Supervisión, Redacción, Revisión y Edición.
Financiación
El/los autor/es declara(n) haber recibido apoyo financiero para la investigación, autoría y/o publicación de este artículo. Este trabajo fue apoyado por el proyecto Microbiologia Veterinaria (Nº 2019/1180, OTRI UNIZAR).
Reconocimientos
Los autores agradecen al personal de Laboratorios Albéitar S. C. (Zaragoza, España) su excelente asistencia técnica y el suministro de algunas muestras para el estudio. Además, queremos hacer un reconocimiento al Departamento de Microbiología de la Facultad de Veterinaria de Zaragoza, en el que se realizaron algunas técnicas.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o afirmación que pueda ser hecha por su fabricante, no está garantizado ni respaldado por el editor.
Referencias
1. Flores-Mireles, AL, Walker, JN, Caparon, M, y Hultgren, SJ. Infecciones del tracto urinario: epidemiología, mecanismos de infección y opciones de tratamiento. Nat Rev Microbiol. (2015) 13:269–84. doi: 10.1038/nrmicro3432
Resumen de PubMed | Texto completo de Crossref | Google Académico
2. Hall, JL, Holmes, MA, y Baines, SJ. Prevalencia y resistencia antimicrobiana de patógenos del tracto urinario canino. Rec. Veterinario (2013) 173:549. doi: 10.1136/vr.101482
3. Teh, H. Una revisión de los conceptos actuales en las infecciones del tracto urinario canino. Aust Vet J. (2022) 100:56–62. doi: 10.1111/avj.13127
Resumen de PubMed | Texto completo de Crossref | Google Académico
4. Bonten, M, Johnson, JR, van den Biggelaar, AHJ, Georgalis, L, Geurtsen, J, de Palacios, PI, et al. Epidemiología de la bacteriemia por Escherichia coli: una revisión sistemática de la literatura. Clin Infect Dis. (2021) 72:1211–9. doi: 10.1093/cid/ciaa210
Resumen de PubMed | Texto completo de Crossref | Google Académico
5. Yousefi, A, y Torkan, S. Escherichia coli uropatógena en las muestras de orina de perros iraníes: patrón de resistencia a los antimicrobianos y distribución de genes de resistencia a los antibióticos. Biomed Res Int. (2017) 2017:1–10. doi: 10.1155/2017/4180490
6. Jacob, J, y Lorber, B. Enfermedades transmitidas por el mejor amigo del hombre: el perro. Microbiol Spectr. (2015) 3:15. DOI: 10.1128/microbiolspec. IOL5-0002-2015
Resumen de PubMed | Texto completo de Crossref | Google Académico
7. Landraud, L, Gibert, M, Popoff, MR, Boquet, P y Gauthier, M. La expresión de cnf1 por Escherichia coli J96 involucra una gran región de ADN aguas arriba que incluye el operón hlyCABD y está regulada por la proteína RfaH. Mol Microbiol. (2003) 47:1653–67. doi: 10.1046/j.1365-2958.2003.03391.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
8. Abe, CM, Salvador, FA, Falsetti, IN, Vieira, MA, Blanco, J, Blanco, JE, et al. Las cepas de Escherichia coli uropatógenas (UPEC) pueden tener propiedades de virulencia de E. coli diarreicogénica. FEMS Immunol Med Microbiol. (2008) 52:397–406. doi: 10.1111/j.1574-695X.2008.00388.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
9. de Brito, BG, Leite, DS, Linhares, RE, y Vidotto, MC. Factores asociados a la virulencia de cepas uropatógenas de Escherichia coli aisladas de cerdos. Microbiol veterinario. (1999) 65:123–32. doi: 10.1016/S0378-1135(98)00277-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
10. Dell’Orco, M, Bertazzolo, W, Pagliaro, L, Roccabianca, P, y Comazzi, S. Síndrome urémico hemolítico en un perro. Veterinario Clin Pathol. (2005) 34:264–9. doi: 10.1111/j.1939-165X.2005.tb00052.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
11. Persson, S, Olsen, KE, Ethelberg, S, y Scheutz, F. Método de subtipificación de las variantes de la toxina Shiga de Escherichia coli (verocitotoxina) 2 y correlaciones con las manifestaciones clínicas. J Clin Microbiol. (2007) 45:2020–4. doi: 10.1128/JCM.02591-06
Resumen de PubMed | Texto completo de Crossref | Google Académico
12. García, A, y Fox, JG. Una perspectiva de una sola salud para definir y descifrar el potencial patogénico de Escherichia coli en múltiples huéspedes. Comp Med. (2021) 71:3–45. doi: 10.30802/AALAS-CM-20-000054
Resumen de PubMed | Texto completo de Crossref | Google Académico
13. Gati, NS, Temme, IJ, Middendorf-Bauchart, B, Kehl, A, Dobrindt, U y Mellmann, A. Caracterización fenotípica comparativa de Escherichia coli híbrida productora de toxina Shiga / uropatógena, Escherichia coli uropatógena canónica y productora de toxina Shiga. Int J Med Microbiol. (2021) 311:151533. doi: 10.1016/j.ijmm.2021.151533
Resumen de PubMed | Texto completo de Crossref | Google Académico
14. Lloyd, AL, Rasko, DA, y Mobley, HL. Definición de islas genómicas y genes específicos de uropatógenos en Escherichia coli uropatógena. J Bacteriol. (2007) 189:3532–46. doi: 10.1128/JB.01744-06
Resumen de PubMed | Texto completo de Crossref | Google Académico
15. Martin, HR, Taylor, NS, Buckley, EM, Marini, RP, Patterson, MM y Fox, JG. Caracterización de cepas de Escherichia coli productoras de factor necrotizante citotóxico 1 a partir de heces de macacos sanos. J Med Microbiol. (2009) 58:1354–8. doi: 10.1099/jmm.0.012088-0
16. Lane, MC, y Mobley, HL. Papel de la adherencia mediada por P-fimbrial en la pielonefritis y persistencia de Escherichia coli uropatógena (UPEC) en el riñón de mamíferos. Riñón Int. (2007) 72:19–25. doi: 10.1038/sj.ki.5002230
Resumen de PubMed | Texto completo de Crossref | Google Académico
17. Johnson, JR, O’Bryan, TT, Low, DA, Ling, G, Delavari, P, Fasching, C, et al. Evidencia de similitud entre cepas de Escherichia coli patógenas extraintestinales caninas y humanas que expresan el alelo III papG. Infectar a Immun. (2000) 68:3327–36. doi: 10.1128/IAI.68.6.3327-3336.2000
Resumen de PubMed | Texto completo de Crossref | Google Académico
18. Johnson, JR, Stell, AL, y Delavari, P. Heces caninas como reservorio de Escherichia coli patógena extraintestinal. Infectar a Immun. (2001) 69:1306–14. doi: 10.1128/IAI.69.3.1306-1314.2001
Resumen de PubMed | Texto completo de Crossref | Google Académico
19. Silwedel, C, Vogel, U, Claus, H, Glaser, K, Speer, CP y Wirbelauer, J. Brote de secuencia de Escherichia coli multirresistente tipo 131 en una unidad de cuidados intensivos neonatales: la vigilancia activa eficiente evitó el desenlace fatal. J Hosp Infectar. (2016) 93:181–6. doi: 10.1016/j.jhin.2016.02.014
Resumen de PubMed | Texto completo de Crossref | Google Académico
20. ECDC. Resistencia a los antimicrobianos en la UE/EEE (EARS-net) – Informe epidemiológico anual 2019: Estocolmo: ECDC. (2020). Disponible en: https://www.ecdc.europa.eu/en/publications-data/surveillance-antimicrobial-resistance-europe-2019.
21. Ukah, UV, Glass, M, Avery, B, Daignault, D, Mulvey, MR, Reid-Smith, RJ, et al. Factores de riesgo para la adquisición de Escherichia coli multirresistente y el desarrollo de infecciones del tracto urinario adquiridas en la comunidad. Epidemiol Infect. (2018) 146:46–57. doi: 10.1017/S0950268817002680
Resumen de PubMed | Texto completo de Crossref | Google Académico
22. Köck, R, Daniels-Haardt, I, Becker, K, Mellmann, A, Friedrich, AW, Mevius, D, et al. Enterobacteriaceae resistentes a carbapenémicos en animales silvestres, productores de alimentos y de compañía: una revisión sistemática. Clin Microbiol Infectar. (2018) 24:1241–50. doi: 10.1016/j.cmi.2018.04.004
Resumen de PubMed | Texto completo de Crossref | Google Académico
23. Zechner, V, Sofka, D, Paulsen, P y Hilbert, F. Resistencia a los antimicrobianos en Escherichia coli y genes de resistencia en colifagos de una clínica de animales pequeños y en un perro paciente con infección crónica del tracto urinario. Antibióticos (Basilea). (2020) 9:652. doi: 10.3390/antibióticos9100652
24. Rzewuska, M, Czopowicz, M, Kizerwetter-Świda, M, Chrobak, D, Błaszczak, B, y Binek, M. Resistencia a múltiples fármacos en cepas de Escherichia coli aisladas de infecciones en perros y gatos en Polonia (2007-2013). ScientificWorldJournal. (2015) 2015:408205:1–8. doi: 10.1155/2015/408205
25. Yudhanto, S, Hung, CC, Maddox, CW y Varga, C. Resistencia antimicrobiana en bacterias aisladas de muestras de orina canina enviadas a un laboratorio de diagnóstico veterinario, Illinois, Estados Unidos. Front Vet Sci. (2022) 9:867784. doi: 10.3389/fvets.2022.867784
Resumen de PubMed | Texto completo de Crossref | Google Académico
26. Costa, D, Poeta, P, Briñas, L, Sáenz, Y, Rodrigues, J, y Torres, C. Detección de betalactamasas CTX-M-1 y TEM-52 en cepas de Escherichia coli de mascotas sanas en Portugal. J Quimiotera antimicrobiana. (2004) 54:960–1. doi: 10.1093/jac/dkh444
Resumen de PubMed | Texto completo de Crossref | Google Académico
27. O’Keefe, A, Hutton, TA, Schifferli, DM y Rankin, SC. Primera detección de betalactamasas de espectro extendido CTX-M y SHV en aislados del tracto urinario de Escherichia coli de perros y gatos en los Estados Unidos. Agentes antimicrobianos quimiotera. (2010) 54:3489–92. doi: 10.1128/AAC.01701-09
Resumen de PubMed | Texto completo de Crossref | Google Académico
28. van den Bunt, G, Fluit, AC, Spaninks, MP, Timmerman, AJ, Geurts, Y, Kant, A, et al. Transporte fecal, factores de riesgo, adquisición y persistencia de enterobacterias productoras de BLEE en perros y gatos y coportador con humanos pertenecientes al mismo hogar. J Quimiotera antimicrobiana. (2020) 75:342–50. doi: 10.1093/jac/dkz462
Resumen de PubMed | Texto completo de Crossref | Google Académico
29. Weese, JS, Blondeau, J, Boothe, D, Guardabassi, LG, Gumley, N, Papich, M, et al. Directrices de la Sociedad Internacional de Enfermedades Infecciosas de los Animales de Compañía (ISCAID) para el diagnóstico y tratamiento de las infecciones bacterianas del tracto urinario en perros y gatos. Veterinario J. (2019) 247:8–25. doi: 10.1016/j.tvjl.2019.02.008
Resumen de PubMed | Texto completo de Crossref | Google Académico
30. Oswald, E, Schmidt, H, Morabito, S, Karch, H, Marchès, O y Caprioli, A. Tipificación de genes de intimina en Escherichia coli enterohemorrágica y enteropatógena humana y animal: caracterización de una nueva variante de intimina. Infectar a Immun. (2000) 68:64–71. doi: 10.1128/IAI.68.1.64-71.2000
Resumen de PubMed | Texto completo de Crossref | Google Académico
31. Oh, KH, Kim, SB, Park, MS y Cho, SH. Desarrollo de un ensayo de PCR de un solo paso con nueve pares de cebadores para la detección de cinco tipos de Escherichia coli diarreica. J Microbiol Biotechnol. (2014) 24:862–8. doi: 10.4014/jmb.1312.12031
Resumen de PubMed | Texto completo de Crossref | Google Académico
32. Blanco, M, Blanco, JE, Rodríguez, E, Abalia, I, Alonso, MP, y Blanco, J. Detección de genes de virulencia en Escherichia coli uropatógena mediante reacción en cadena de la polimerasa (PCR): comparación con los resultados obtenidos mediante métodos fenotípicos. J Métodos de Microbiol. (1997) 31:37–43. doi: 10.1016/S0167-7012(97)00087-0
33. Yamamoto, S, Terai, A, Yuri, K, Kurazono, H, Takeda, Y y Yoshida, O. Detección de factores de urovirulencia en Escherichia coli mediante reacción en cadena de la polimerasa múltiple. FEMS Immunol Med Microbiol. (1995) 12:85–90. doi: 10.1111/j.1574-695X.1995.tb00179.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
34. Osugui, L, de Castro, AF, Iovine, R, Irino, K, y Carvalho, VM. Genotipos de virulencia, resistencia a antibióticos y antecedentes filogenéticos de Escherichia coli patógena extraintestinal aislada de infecciones del tracto urinario de perros y gatos en Brasil. Microbiol veterinario. (2014) 171:242–7. doi: 10.1016/j.vetmic.2014.03.027
Resumen de PubMed | Texto completo de Crossref | Google Académico
35. Magiorakos, AP, Srinivasan, A, Carey, RB, Carmeli, Y, Falagas, ME, Giske, CG, et al. Bacterias multirresistentes, extensivamente resistentes a los fármacos y panrresistentes: una propuesta internacional de expertos para definiciones estándar provisionales de la resistencia adquirida. Clin Microbiol Infectar. (2012) 18:268–81. doi: 10.1111/j.1469-0691.2011.03570.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
36. CLSI. Estándares de rendimiento M100 para pruebas de susceptibilidad a los antimicrobianos, un suplemento de CLSI para aplicación global. Berwyn, PA: CLSI (2018).
37. Zhang, X, Zhang, Y, Wang, F, Wang, C, Chen, L, Liu, H, et al. Desentrañar los mecanismos de resistencia a la nitrofurantoína y las características epidemiológicas entre los aislados clínicos de Escherichia coli. Int J Agentes antimicrobianos. (2018) 52:226–32. doi: 10.1016/j.ijantimicag.2018.04.021
Resumen de PubMed | Texto completo de Crossref | Google Académico
38. Bortolaia, V, Kaas, RS, Ruppe, E, Roberts, MC, Schwarz, S, Cattoir, V, et al. ResFinder 4.0 para predicciones de fenotipos a partir de genotipos. J Quimiotera antimicrobiana. (2020) 75:3491–500. doi: 10.1093/jac/dkaa345
Resumen de PubMed | Texto completo de Crossref | Google Académico
39. Camacho, C, Coulouris, G, Avagyan, V, Ma, N, Papadopoulos, J, Bealer, K, et al. BLAST+: arquitectura y aplicaciones. BMC Bioinformática. (2009) 10:421. doi: 10.1186/1471-2105-10-421
40. Zankari, E, Allesøe, R, Joensen, KG, Cavaco, LM, Lund, O y Aarestrup, FM. PointFinder: una novedosa herramienta web para la detección basada en WGS de la resistencia a los antimicrobianos asociada con mutaciones puntuales cromosómicas en patógenos bacterianos. J Quimiotera antimicrobiana. (2017) 72:2764–8. doi: 10.1093/jac/dkx217
Resumen de PubMed | Texto completo de Crossref | Google Académico
41. Cosentino, S, Voldby Larsen, M, Møller Aarestrup, F y Lund, O. PathogenFinder: distinguir al amigo del enemigo utilizando datos de secuencia del genoma completo bacteriano. PLoS Uno. (2013) 8:e77302. doi: 10.1371/journal.pone.0077302
Resumen de PubMed | Texto completo de Crossref | Google Académico
42. Clausen, PTLC, Aarestrup, FM y Lund, O. Alineación rápida y precisa de lecturas sin procesar contra bases de datos redundantes con KMA. BMC Bioinformática. (2018) 19:307. doi: 10.1186/s12859-018-2336-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
43. Bartual, SG, Seifert, H, Hippler, C, Luzón, MA, Wisplinghoff, H, y Rodríguez-Valera, F. Desarrollo de un esquema de tipificación de secuencias multilocus para la caracterización de aislados clínicos de Acinetobacter baumannii. J Clin Microbiol. (2005) 43:4382–90. doi: 10.1128/JCM.43.9.4382-4390.2005
Resumen de PubMed | Texto completo de Crossref | Google Académico
44. Griffiths, D, Fawley, W, Kachrimanidou, M, Bowden, R, Crook, DW, Fung, R, et al. Tipificación de secuencias multilocus de Clostridium difficile. J Clin Microbiol. (2010) 48:770–8. doi: 10.1128/JCM.01796-09
45. Jaureguy, F, Landraud, L, Passet, V, Diancourt, L, Frapy, E, Guigon, G, et al. Diversidad filogenética y genómica de cepas bacteriémicas humanas de Escherichia coli. BMC Genómica. (2008) 9:560. doi: 10.1186/1471-2164-9-560
Resumen de PubMed | Texto completo de Crossref | Google Académico
46. Larsen, MV, Cosentino, S, Rasmussen, S, Friis, C, Hasman, H, Marvig, RL, et al. Tipificación de secuencias multilocus de bacterias secuenciadas en el genoma total. J Clin Microbiol. (2012) 50:1355–61. doi: 10.1128/JCM.06094-11
Resumen de PubMed | Texto completo de Crossref | Google Académico
47. Lemee, L, Dhalluin, A, Pestel-Caron, M, Lemeland, JF, y Pons, JL. Análisis de tipificación de secuencias multilocus de aislados humanos y animales de Clostridium difficile de varios tipos toxigénicos. J Clin Microbiol. (2004) 42:2609–17. doi: 10.1128/JCM.42.6.2609-2617.2004
Resumen de PubMed | Texto completo de Crossref | Google Académico
48. Wirth, T, Falush, D, Lan, R, Colles, F, Mensa, P, Wieler, LH, et al. Sexo y virulencia en Escherichia coli: una perspectiva evolutiva. Mol Microbiol. (2006) 60:1136–51. doi: 10.1111/j.1365-2958.2006.05172.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
49. Alikhan, NF, Zhou, Z, Sargento, MJ, y Achtman, M. Una visión genómica de la estructura poblacional de Salmonella. PLoS Genet. (2018) 14:e1007261. doi: 10.1371/journal.pgen.1007261
Resumen de PubMed | Texto completo de Crossref | Google Académico
50. Carattoli, A, Zankari, E, García-Fernández, A, Voldby Larsen, M, Lund, O, Villa, L, et al. Detección in silico y tipificación de plásmidos mediante PlasmidFinder y tipificación de secuencias multilocus de plásmidos. Agentes antimicrobianos quimiotera. (2014) 58:3895–903. doi: 10.1128/AAC.02412-14
Resumen de PubMed | Texto completo de Crossref | Google Académico
51. Joensen, KG, Scheutz, F, Lund, O, Hasman, H, Kaas, RS, Nielsen, EM, et al. Secuenciación del genoma completo en tiempo real para la tipificación rutinaria, la vigilancia y la detección de brotes de Escherichia coli verotoxigénica. J Clin Microbiol. (2014) 52:1501–10. doi: 10.1128/JCM.03617-13
Resumen de PubMed | Texto completo de Crossref | Google Académico
52. Zankari, E, Hasman, H, Cosentino, S, Vestergaard, M, Rasmussen, S, Lund, O, et al. Identificación de genes adquiridos de resistencia a antimicrobianos. J Quimiotera antimicrobiana. (2012) 67:2640–4. doi: 10.1093/jac/dks261
Resumen de PubMed | Texto completo de Crossref | Google Académico
53. Joensen, KG, Tetzschner, AM, Iguchi, A, Aarestrup, FM y Scheutz, F. Serotipado in silico rápido y fácil de aislados de Escherichia coli mediante el uso de datos de secuenciación del genoma completo. J Clin Microbiol. (2015) 53:2410–26. doi: 10.1128/JCM.00008-15
Resumen de PubMed | Texto completo de Crossref | Google Académico
54. Grant, JR, Enns, E, Marinier, E, Mandal, A, Herman, EK, Chen, CY, et al. Proksee: caracterización y visualización en profundidad de genomas bacterianos. Ácidos nucleicos Res. (2023) 51:W484–92. doi: 10.1093/nar/gkad326
Resumen de PubMed | Texto completo de Crossref | Google Académico
55. Page, AJ, Cummins, CA, Hunt, M, Wong, VK, Reuter, S, Holden, MT, et al. Roary: análisis rápido del genoma de la panola procariota a gran escala. Bioinformática. (2015) 31:3691–3. doi: 10.1093/bioinformatics/btv421
Resumen de PubMed | Texto completo de Crossref | Google Académico
56. Seemann, T. Prokka: anotación rápida del genoma procariota. Bioinformática. (2014) 30:2068–9. doi: 10.1093/bioinformatics/btu153
Resumen de PubMed | Texto completo de Crossref | Google Académico
57. Nguyen, LT, Schmidt, HA, von Haeseler, A, y Minh, BQ. IQ-TREE: un algoritmo estocástico rápido y eficaz para estimar filogenias de máxima verosimilitud. Mol Biol Evol. (2015) 32:268–74. DOI: 10.1093/Molbev/MSU300
Resumen de PubMed | Texto completo de Crossref | Google Académico
58. EFSA, ECDC. Informe resumido de la Unión Europea sobre la resistencia a los antimicrobianos en bacterias zoonóticas e indicadoras de seres humanos, animales y alimentos en 2017/2018. EFSA J. (2020) 18:E06007. doi: 10.2903/j.efsa.2020.6007
59. EMA, CVMP, CHMP. Categorización de los antibióticos en la Unión Europea. Nº de informe: Supplement_1. (2019). Disponible en: https://www.ema.europa.eu/en/documents/report/categorisation-antibiotics-european-union-answer-request-european-commission-updating-scientific_en.pdf.
60. Hadfield, J, Croucher, NJ, Goater, RJ, Abudahab, K, Aanensen, DM, Harris, SR, et al. Phandango: un visor interactivo para la genómica de poblaciones bacterianas. Bioinfor. (2018) 34:292–293. doi: 10.1093/bioinformatics/btx610
61. Sevilla, E, Mainar-Jaime, RC, Moreno, B, Martín-Burriel, I, Morales, M, Andrés-Lasheras, S, et al. Resistencia antimicrobiana en aislamientos de Escherichia coli entérica canina y prevalencia de factores de adhesión-borrado y virulencia patógena extraintestinal en España. Acta Vet Hung. (2020) 68:1–7. doi: 10.1556/004.2020.00013
62. Chang, SK, Lo, DY, Wei, HW y Kuo, HC. Resistencia antimicrobiana de aislados de Escherichia coli de infecciones del tracto urinario canino. J Vet Med Sci. (2015) 77:59–65. doi: 10.1292/jvms.13-0281
Resumen de PubMed | Texto completo de Crossref | Google Académico
63. Yu, Z, Wang, Y, Chen, Y, Huang, M, Wang, Y, Shen, Z, et al. Resistencia antimicrobiana de patógenos bacterianos aislados de infecciones del tracto urinario canino. Microbiol veterinario. (2020) 241:108540. doi: 10.1016/j.vetmic.2019.108540
Resumen de PubMed | Texto completo de Crossref | Google Académico
64. Richter, SE, Miller, L, Needleman, J, Uslan, DZ, Bell, D, Watson, K, et al. Factores de riesgo para el desarrollo de resistencia a aminoglucósidos en bacilos gramnegativos. Am J Health Syst Pharm. (2019) 76:1838–47. doi: 10.1093/ajhp/zxz201
Resumen de PubMed | Texto completo de Crossref | Google Académico
65. Perdiz, SR, Kwong, SM, Firth, N, y Jensen, SO. Elementos genéticos móviles asociados a la resistencia a los antimicrobianos. Clin Microbiol Rev. (2018) 31:17. doi: 10.1128/CMR.00088-17
Resumen de PubMed | Texto completo de Crossref | Google Académico
66. Tóth, AG, Csabai, I, Judge, MF, Maróti, G, Becsei, Á, Spisák, S, et al. Genes móviles de resistencia a antimicrobianos en probióticos. Antibióticos (Basilea). (2021) 10:287. doi: 10.3390/antibióticos10111287
67. Rice, LB. Transposones conjugativos de la familia Tn916 y diseminación de determinantes de resistencia a los antimicrobianos. Agentes antimicrobianos quimiotera. (1998) 42:1871–7. doi: 10.1128/AAC.42.8.1871
Resumen de PubMed | Texto completo de Crossref | Google Académico
68. Soge, OO, Beck, NK, White, TM, No, DB y Roberts, MC. Un nuevo transposón, Tn6009, compuesto por un elemento Tn916 unido a un operón Staphylococcus aureus mer. J Quimiotera antimicrobiana. (2008) 62:674–80. doi: 10.1093/jac/dkn255
Resumen de PubMed | Texto completo de Crossref | Google Académico
69. Medugu, N, Tickler, IA, Duru, C, Egah, R, James, AO, Odili, V, et al. Caracterización fenotípica y molecular de Enterobacterales multirresistentes a betalactámicos aislados de pacientes que acuden a seis hospitales del norte de Nigeria. Sci Rep. (2023) 13:10306. doi: 10.1038/s41598-023-37621-z
Resumen de PubMed | Texto completo de Crossref | Google Académico
70. Nakayama, T, Yamaguchi, T, Jinnai, M, Kumeda, Y, y Hase, A. Vibrio vulnificus y V. alginolyticus productores de BLEE albergan un plásmido que codifica ISEc9 aguas arriba de blaCTX-M-55 y qnrS2 aislado de mariscos importados. Arch Microbiol. (2023) 205:569. doi: 10.1007/s00203-023-03569-x
71. Nicolas-Chanoine, MH, Bertrand, X, y Madec, JY. Escherichia coli ST131, un intrigante grupo clonal. Clin Microbiol Rev. (2014) 27:543–74. doi: 10.1128/CMR.00125-13
Resumen de PubMed | Texto completo de Crossref | Google Académico
72. Evans, BA, y Amyes, SG. OXA β-lactamasas. Clin Microbiol Rev. (2014) 27:241–63. doi: 10.1128/CMR.00117-13
Resumen de PubMed | Texto completo de Crossref | Google Académico
73. Sandvang, D. Nuevo gen de resistencia a la estreptomicina y a la espectinomicina como un casete de genes dentro de un integrón de clase 1 aislado de Escherichia coli. Agentes antimicrobianos quimiotera. (1999) 43:3036–8. doi: 10.1128/AAC.43.12.3036
Resumen de PubMed | Texto completo de Crossref | Google Académico
74. Meyer, R. Replicación y movilización conjugativa de plásmidos IncQ de amplio rango de huéspedes. plásmido. (2009) 62:57–70. doi: 10.1016/j.plasmid.2009.05.001
Resumen de PubMed | Texto completo de Crossref | Google Académico
75. Scholz, P, Haring, V, Wittmann-Liebold, B, Ashman, K, Bagdasarian, M y Scherzinger, E. Secuencia completa de nucleótidos y organización génica del plásmido de amplio rango de huéspedes RSF1010. Gen. (1989) 75:271–88. doi: 10.1016/0378-1119(89)90273-4
Resumen de PubMed | Texto completo de Crossref | Google Académico
76. Hollingshead, S y Vapnek, D. Análisis de secuencia de nucleótidos de un gen que codifica una estreptomicina/espectinomicina adenililtransferasa. plásmido. (1985) 13:17–30. doi: 10.1016/0147-619X(85)90052-6
Resumen de PubMed | Texto completo de Crossref | Google Académico
77. Sandalli, C, Buruk, CK, Sancaktar, M, y Ozgumus, OB. Prevalencia de integrones y una nueva variante dfrA17 en bacilos gramnegativos que causan infecciones adquiridas en la comunidad. Microbiol Immunol. (2010) 54:164–9. doi: 10.1111/j.1348-0421.2010.00197.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
78. Sáenz, Y, Briñas, L, Domínguez, E, Ruiz, J, Zarazaga, M, Vila, J, et al. Mecanismos de resistencia en cepas de Escherichia coli multirresistentes a antibióticos de origen humano, animal y alimentario. Agentes antimicrobianos quimiotera. (2004) 48:3996–4001. doi: 10.1128/AAC.48.10.3996-4001.2004
Resumen de PubMed | Texto completo de Crossref | Google Académico
79. Kazama, H, Hamashima, H, Sasatsu, M y Arai, T. Caracterización del gen de resistencia antiséptica qacE delta 1 aislado de aislados clínicos y ambientales de Vibrio parahaemolyticus y Vibrio cholerae non-O1. FEMS Microbiol Lett. (1999) 174:379–84.
80. Pawlowski, AC, Stogios, PJ, Koteva, K, Skarina, T, Evdokimova, E, Savchenko, A, et al. Evolución de la discriminación de sustratos en enzimas de resistencia a antibióticos macrólidos. Nat Commun. (2018) 9:112. doi: 10.1038/s41467-017-02680-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
81. Roberts, MC. Actualización sobre los genes adquiridos de resistencia a la tetraciclina. FEMS Microbiol Lett. (2005) 245:195–203. doi: 10.1016/j.femsle.2005.02.034
Resumen de PubMed | Texto completo de Crossref | Google Académico
82. Alton, NK y Vapnek, D. Análisis de la secuencia de nucleótidos del transposón de resistencia al cloranfenicol Tn9. Naturaleza. (1979) 282:864–9. doi: 10.1038/282864a0
Resumen de PubMed | Texto completo de Crossref | Google Académico
83. Lewis, GL, Fenton, RJ, Moriyama, EN, Loy, JD, y Moxley, RA. Asociación de IS Vsa3 con multirresistencia en aislados de Salmonella enterica de bovinos (Bos taurus). Microorganismos. (2023) 11:631. DOI: 10.3390/microorganismos11030631
Resumen de PubMed | Texto completo de Crossref | Google Académico
84. García, P, Guerra, B, Bances, M, Mendoza, MC y Rodicio, MR. Los plásmidos IncA/C median la resistencia antimicrobiana ligada a genes de virulencia en el clon español del serotipo emergente de Salmonella enterica. J Quimiotera antimicrobiana. (2011) 66:543–9. doi: 10.1093/jac/dkq481
Resumen de PubMed | Texto completo de Crossref | Google Académico
85. Zhou, S, Zhu, Y, Yan, Y, Wang, W y Wang, Y. Desciframiento de genes extracelulares de resistencia a antibióticos (eARG) en lodos activados por metagenoma. Res. de Agua. (2019) 161:610–20. doi: 10.1016/j.watres.2019.06.048
Resumen de PubMed | Texto completo de Crossref | Google Académico
86. Jarocki, VM, Heß, S, Anantanawat, K, Berendonk, TU y Djordjevic, SP. Linaje multirresistente de Escherichia coli enterotoxigénica ST182 con serotipo O169:H41 en desechos de aerolíneas. Microbiol frontal. (2021) 12:731050. doi: 10.3389/fmicb.2021.731050
Resumen de PubMed | Texto completo de Crossref | Google Académico
87. Novais, A, Cantón, R, Moreira, R, Peixe, L, Baquero, F, y Coque, TM. La aparición y diseminación de aislados de Enterobacteriaceae productores de enzimas similares a CTX-M-1 en España se asocian a plásmidos IncFII (CTX-M-15) y de amplio rango de hospedadores (CTX-M-1, −3 y −32). Agentes antimicrobianos quimiotera. (2007) 51:796–9. doi: 10.1128/AAC.01070-06
Resumen de PubMed | Texto completo de Crossref | Google Académico
88. Rawlings, DE, y Tietze, E. Biología comparativa de IncQ y plásmidos similares a IncQ. Microbiol Mol Biol Rev. (2001) 65:481–96. doi: 10.1128/MMBR.65.4.481-496.2001
Resumen de PubMed | Texto completo de Crossref | Google Académico
89. Vereecke, N, Van Hoorde, S, Sperling, D, Theuns, S, Devriendt, B y Cox, E. Virotipado y pruebas genéticas de susceptibilidad antimicrobiana de cepas porcinas ETEC/STEC y tipos de plásmidos asociados. Microbiol frontal. (2023) 14:312. doi: 10.3389/fmicb.2023.1139312
90. Bogema, DR, McKinnon, J, Liu, M, Hitchick, N, Miller, N, Venturini, C, et al. El análisis del genoma completo de la secuencia extraintestinal de Escherichia coli tipo 73 de un solo hospital durante un período de 2 años identificó diferentes grupos clonales circulantes. Genoma Microb. (2020) 6:255. doi: 10.1099/mgen.0.000255
91. López-Cerero, L, MeM, B, Serrano, L, Liró, J, Cisneros, JM, Rodríguez-Baño, J, et al. Escherichia coli O25b:H4/ST131 son prevalentes en España y a menudo no se asocian con BLEE o resistencia a quinolonas. Enferm Infecc Microbiol Clin. (2013) 31:385–8. doi: 10.1016/j.eimc.2012.09.005
92. Dautzenberg, MJ, Haverkate, MR, Bonten, MJ y Bootsma, MC. Potencial epidémico de Escherichia coli ST131 y Klebsiella pneumoniae ST258: una revisión sistemática y metaanálisis. BMJ Open. (2016) 6:E009971. doi: 10.1136/bmjopen-2015-009971
Resumen de PubMed | Texto completo de Crossref | Google Académico
93. LeCuyer, TE, Byrne, BA, Daniels, JB, Díaz-Campos, DV, Hammac, GK, Miller, CB, et al. Estructura poblacional y resistencia antimicrobiana de Escherichia coli uropatógena canina. J Clin Microbiol. (2018) 56:18. doi: 10.1128/JCM.00788-18
Resumen de PubMed | Texto completo de Crossref | Google Académico
94. Torkan, S, Bahadoranian, M, Khamesipour, F y Anyanwu, M. Detección de genes de virulencia y resistencia a los antimicrobianos en aislados de Escherichia coli de perros diarrhoiec en Irán. Arch med vet. (2016) 48:181–90. doi: 10.4067/S0301-732X2016000200008
95. Tramuta, C, Robino, P, Nucera, D, Salvarani, S, Banche, G, Malabaila, A, et al. Caracterización molecular y resistencia antimicrobiana de Escherichia coli fecal y urinaria aislada de perros y humanos en Italia. Vet Ital. (2014) 50:23–30. doi: 10.12834/VetIt.1304.09
Resumen de PubMed | Texto completo de Crossref | Google Académico
96. Siqueira, AK, Ribeiro, MG, DaS, L, Tiba, MR, Moura, C, Lopes, MD, et al. Factores de virulencia en cepas de Escherichia coli aisladas de infecciones del tracto urinario y casos de piometra y de heces de perros sanos. Res Vet Sci. (2009) 86:206–10. doi: 10.1016/j.rvsc.2008.07.018
Resumen de PubMed | Texto completo de Crossref | Google Académico
97. Yuri, K, Nakata, K, Katae, H, Tsukamoto, T y Hasegawa, A. Serotipos y factores de virulencia de cepas de Escherichia coli aisladas de perros y gatos. J Vet Med Sci. (1999) 61:37–40. doi: 10.1292/jvms.61.37
Resumen de PubMed | Texto completo de Crossref | Google Académico
98. Blum, G, Falbo, V, Caprioli, A, y Hacker, J. Grupos de genes que codifican el factor necrotizante citotóxico tipo 1, Prs-fimbrias y alfa-hemolisina forman la isla de patogenicidad II de la cepa uropatógena de Escherichia coli J96. FEMS Microbiol Lett. (1995) 126:189–95.
99. Landraud, L, Gauthier, M, Fosse, T y Boquet, P. Frecuencia de cepas de Escherichia coli que producen el factor necrotizante citotóxico (CNF1) en infecciones del tracto urinario nosocomial. Lett Appl Microbiol. (2000) 30:213–6. doi: 10.1046/j.1472-765x.2000.00698.x
Resumen de PubMed | Texto completo de Crossref | Google Académico
100. Johnson, JR, Delavari, P, Kuskowski, M y Stell, AL. Distribución filogenética de los rasgos asociados a la virulencia extraintestinal en Escherichia coli. J Infectar Dis. (2001) 183:78–88. doi: 10.1086/317656
Resumen de PubMed | Texto completo de Crossref | Google Académico
101. Diard, M, Garry, L, Selva, M, Mosser, T, Denamur, E y Matic, I. Las islas asociadas a la patogenicidad en Escherichia coli patógena extraintestinal son elementos de aptitud involucrados en la colonización intestinal. J Bacteriol. (2010) 192:4885–93. doi: 10.1128/JB.00804-10
Resumen de PubMed | Texto completo de Crossref | Google Académico
102. Johnson, JR, Stell, AL, Delavari, P, Murray, AC, Kuskowski, M y Gaastra, W. Similitudes filogenéticas y patotípicas entre los aislados de Escherichia coli de infecciones del tracto urinario en perros e infecciones extraintestinales en humanos. J Infectar Dis. (2001) 183:897–906. doi: 10.1086/319263
Resumen de PubMed | Texto completo de Crossref | Google Académico
103. Xiaoli, L, Figler, HM, Goswami Banerjee, K, Hayes, CS y Dudley, EG. La Escherichia coli no patógena mejora la producción de Stx2a de E. coli O157:H7 a través de mecanismos dependientes e independientes de bamA. Microbiol frontal. (2018) 9:1325. doi: 10.3389/fmicb.2018.01325
Resumen de PubMed | Texto completo de Crossref | Google Académico
104. Toval, F, Schiller, R, Meisen, I, Putze, J, Kouzel, IU, Zhang, W, et al. Caracterización de Escherichia coli productora de toxina Shiga asociada a infección del tracto urinario. Infectar a Immun. (2014) 82:4631–42. doi: 10.1128/IAI.01701-14
Resumen de PubMed | Texto completo de Crossref | Google Académico
105. Beutin, L, Karch, H, Aleksić, S, Spencker, FB y Rosenbaum, U. Aparición de Escherichia coli productora de verotoxina (toxina similar a Shiga) en la infección del tracto urinario humano. Infección. (1994) 22:425. doi: 10.1007/BF01715504
Resumen de PubMed | Texto completo de Crossref | Google Académico
106. Toval, F, Köhler, CD, Vogel, U, Wagenlehner, F, Mellmann, A, Fruth, A, et al. Caracterización de aislamientos de Escherichia coli de pacientes hospitalizados o ambulatorios con infección del tracto urinario. J Clin Microbiol. (2014) 52:407–18. doi: 10.1128/JCM.02069-13
Resumen de PubMed | Texto completo de Crossref | Google Académico
107. Valiatti, TB, Santos, FF, Santos, ACM, Nascimento, JAS, Silva, RM, Carvalho, E, et al. Características genéticas y de virulencia de una cepa híbrida atípica de Escherichia coli enteropatógena y uropatógena (aEPEC/UPEC). Las células frontales infectan el microbiol. (2020) 10:492. doi: 10.3389/fcimb.2020.00492
Resumen de PubMed | Texto completo de Crossref | Google Académico
108. Moreno, E, Andreu, A, Pigrau, C, Kuskowski, MA, Johnson, JR, y Prats, G. Relación entre las cepas de Escherichia coli causantes de cistitis aguda en mujeres y la población fecal de E. coli del huésped. J Clin Microbiol. (2008) 46:2529–34. doi: 10.1128/JCM.00813-08
Resumen de PubMed | Texto completo de Crossref | Google Académico
109. Gati, NS, Middendorf-Bauchart, B, Bletz, S, Dobrindt, U y Mellmann, A. Origen y evolución de cepas híbridas de Escherichia coli productoras de toxina Shiga y uropatógenas del tipo de secuencia 141. J Clin Microbiol. (2019) 58:19. doi: 10.1128/JCM.01309-19
Resumen de PubMed | Texto completo de Crossref | Google Académico
110. Brzuszkiewicz, E, Brüggemann, H, Liesegang, H, Emmerth, M, Olschläger, T, Nagy, G, et al. Cómo convertirse en uropatógeno: análisis genómico comparativo de cepas de Escherichia coli patógenas extraintestinales. Proc Natl Acad Sci USA. (2006) 103:12879–84. doi: 10.1073/pnas.0603038103
Resumen de PubMed | Texto completo de Crossref | Google Académico
111. Dale, AP, y Woodford, N. Escherichia coli patógena extraintestinal (ExPEC): enfermedad, portador y clones. J Infectar. (2015) 71:615–26. doi: 10.1016/j.jinf.2015.09.009
Resumen de PubMed | Texto completo de Crossref | Google Académico
112. McLean, C, Bettelheim, KA, Kuzevski, A, Falconer, L, y Djordjevic, SP. Aislamiento de Escherichia coli O5: H −, que posee genes para la toxina Shiga 1, la intitina-β y la enterohemolisina, a partir de una biopsia intestinal de un caso adulto de diarrea sanguinolenta: evidencia de dos patotipos distintos O5: H −. J Med Microbiol. (2005) 54:605–7. doi: 10.1099/jmm.0.45938-0
Resumen de PubMed | Texto completo de Crossref | Google Académico
113. Misra, RN. Serotipificación y enterotoxigenicidad de Escherichia coli aislada de infecciones del tracto urinario. Med J Fuerzas Armadas de la India. (1997) 53:83–6. doi: 10.1016/S0377-1237(17)30671-8
Resumen de PubMed | Texto completo de Crossref | Google Académico
114. Karam, MRA, Habibi, M y Bouzari, S. Relaciones entre los factores de virulencia y la resistencia a los antimicrobianos entre Escherichia coli aislada de infecciones del tracto urinario y aislados comensales en Teherán, Irán. Salud Pública de Osong Res Perspect. (2018) 9:217–24. doi: 10.24171/j.phrp.2018.9.5.02
Resumen de PubMed | Texto completo de Crossref | Google Académico
115. Shah, C, Baral, R, Bartaula, B, y Shrestha, LB. Factores de virulencia de Escherichia coli uropatógena (UPEC) y correlación con la resistencia a los antimicrobianos. BMC Microbiol. (2019) 19:204. doi: 10.1186/s12866-019-1587-3
Resumen de PubMed | Texto completo de Crossref | Google Académico
116. Wagner, S., Gally, DL, y Argyle, S.A. Las Escherichia coli multirresistentes de las infecciones del tracto urinario canino tienden a tener filotipos comensales, menor prevalencia de determinantes de virulencia y ampC-replicones. Microbiol veterinario. (2014) 169:171–8. doi: 10.1016/j.vetmic.2014.01.003
Resumen de PubMed | Texto completo de Crossref | Google Académico
Palabras clave: perro, Escherichia coli, multirresistencia, infección del tracto urinario, factores de virulencia
Cita: Abad-Fau A, Sevilla E, Oro A, Martín-Burriel I, Moreno B, Morales M y Bolea R (2024) Multirresistencia en aislados patógenos de Escherichia coli de infecciones del tracto urinario en perros, España. Frente. Vet. Sci. 11:1325072. doi: 10.3389/fvets.2024.1325072
Recibido: 20 de octubre de 2023; Aceptado: 12 de febrero de 2024;
Publicado: 23 de febrero de 2024.
Editado por:
Barbara Moroni, Instituto Zooprofiláctico Experimental para Piamonte, Liguria y Valle de Aosta (IZSPLVA), Italia
Revisado por:
Yasmine Hasanine Tartor, Universidad de Zagazig, Egipto
Nirajkumar Makadiya, Clínica Veterinaria Westbrook, Canadá
Copyright © 2024 Abad-Fau, Sevilla, Oro, Martín-Burriel, Moreno, Morales y Bolea. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia Creative Commons Attribution License (CC BY).
*Correspondencia: Eloísa Sevilla, esevillr@unizar.es
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, ni las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o afirmación que pueda hacer su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía